[0001] Настоящая заявка испрашивает приоритет и преимущество на основании совместно рассматриваемой предварительной заявки на патент США № 62/720015, поданной 20 августа 2018 г., которая полностью включена в данный документ посредством ссылки.
ОБЛАСТЬ ТЕХНИКИ
[0002] Настоящее изобретение относится к антигенсвязывающим полипептидам, которые связывают PD-L1 человека, фармацевтическим композициям и их применениям. Аспекты изобретения также относятся к экспрессионной системе, продуцирующей такие антигенсвязывающие полипептиды или антитела. Описанные в изобретении антигенсвязывающие полипептиды или фармацевтические композиции подходят для лечения субъекта, нуждающегося в этом, от патологического состояния, такого как рак млекопитающих, инфекция и так далее.
УРОВЕНЬ ТЕХНИКИ
[0003] Иммунные клетки имеют на поверхности костимулирующие и ингибирующие рецепторы, которые взаимодействуют с мембраносвязанными и растворимыми лигандами. Эти рецепторы служат для регуляции активности, продолжительности и типа иммунного ответа, путем изменения пороговых значений и продолжительности активации или ингибирования иммунной клетки. Их часто собирательно именуют иммунными контрольными точками. Многие молекулы таких контрольных точек являются членами суперсемейства молекул B7 или суперсемейства молекул фактора некроза опухолей (ФНО).
[0004] Семейство В7 включает как ингибирующие, так и стимулирующие корецепторы. Например, с одной стороны, лигирование белка запрограммированной (клеточной) гибели 1 (PD-1) и цитотоксического Т-лимфоцит-ассоциированного белка 4 (CTLA-4) с соответствующими им лигандами (PD-L1, PD-L2 и B7-1, B7-2, соответственно) приводит к подавлению активации или генерации регуляторных Т-клеток, анергии, истощению и апоптозу. С другой стороны, лигирование рецепторов кластера дифференцировки (CD28) и индуцируемого костимулятора T-лимфоцитов (ICOS) с соответствующими им лигандами ведет к повышенной пролиферации и продуцированию цитокина. И напротив, семейство костимулирующих рецепторов ФНО включает только стимулирующие молекулы, такие как OX40, 4-1BB, CD40, CD27 и их лиганды, способствующие пролиферации и дифференцировке эффекторной функции. Кроме того, существуют другие корецепторы, которые также принадлежат к любому из указанных семейств, например, Tim-3, LAG-3, Ceacam-1 и т.п.
[0005] За последние пару десятилетий стало ясно, что многие типы рака создают иммуносупрессивную среду внутри опухоли посредством множества механизмов. Ключевым моментом является эктопическая экспрессия ингибирующего лиганда иммунной контрольной точки (а именно PDL1), который подавляет внутриопухолевые Т-клетки. Также появляется все больше доказательств того, что блокирование указанной опосредованной опухолью супрессии иммунитета может дерепрессировать внутриопухолевые Т-клетки и позволить им убить опухоль. (Adachi K, Tamada K. Cancer Sci. 2015;106(8):945-50; Rafiq S, et al., Nat Biotechnol. 2018 Aug 13; Hargadon KM, et al., Int Immunopharmacol. 2018;62:29-39). Блокирование можно осуществить с помощью антитела или множеством других способов. Это отличается от традиционного лечения противораковыми антителами, когда антитело связывается с раковой клеткой и задействует комплементзависимую цитотоксичность (КЗЦ), а также антителозависимую клеточную цитотоксичность (АЗКЦ), чтобы напрямую убивать опухолевые клетки.
[0006] Антитела CTLA-4 были первыми из класса иммунотерапевтических средств, основанных на блокаде иммунных контрольных точек, получившими одобрение FDA. Другие мишени блокады, например, PD1 и связанные с ним молекулы, предлагают бóльшие и отличающиеся возможности для усиления противоопухолевого иммунитета в клинических условиях.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[0007] В настоящем изобретении предложены антигенсвязывающие полипептиды, которые связывают PD-L1 (или, взаимозаменяемо, «анти-PD-L1 полипептид(ы)», «PD-L1-связывающие полипептиды»), предпочтительно PD-L1 человека; полипептид имеет одну или обе из следующих характеристик: (a) связывается с PD-L1 и ингибирует его способность взаимодействовать с PD1; и (b) имеет изотип или константную область, который/которая может запускать АЗКЦ и/или КЗЦ. Итоговое антитело может убивать опухолевые клетки двумя синергетическими путями - за счет дерепрессии Т-клеток и прямой цитотоксичности. Полипептиды по настоящему изобретению могут применяться для лечения опухолей отдельно либо в комбинации с: (a) антителами, нацеленными на другие иммуносупрессивные пути; (b) химиотерапией или радиационной терапией; (c) другими механизмами блокирования иммуносупрессивных путей, например, аптамерами или РНК-интерференцией; или (d) другими иммунотерапевтическими агентами, например, цитокинами, нацеленной терапией, и т.д.
[0008] Согласно одному аспекту настоящего изобретения предложен антигенсвязывающий полипептид, например, антитело, его фрагмент, производное или аналог, который относится к изотипу IgG1 и связывается с эпитопом PD-L1, предпочтительно с аффинностью связывания, равной по меньшей мере 10-6M, и имеющий последовательность вариабельного домена тяжелой цепи «состоящую по существу из» последовательностей аминокислот, выбранным из группы, состоящей из SEQ ID NO:2, SEQ ID NO:6, SEQ ID NO:10, SEQ ID NO:14, SEQ ID NO:18, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:30, SEQ ID NO:34, SEQ ID NO:38, SEQ ID NO:42, SEQ ID NO:46, SEQ ID NO:50, SEQ ID NO:54, SEQ ID NO:58, SEQ ID NO:62, SEQ ID NO:66, SEQ ID NO:70, SEQ ID NO:74, SEQ ID NO:78, SEQ ID NO:82, SEQ ID NO:86, SEQ ID NO:90, SEQ ID NO:94, SEQ ID NO:98, SEQ ID NO:102, SEQ ID NO:106 и их комбинаций, что означает, в настоящем документе, последовательность по меньшей мере на 80%, или, более предпочтительно, на 85%, 90%, 95% или даже 100% идентичную указанным последовательностям аминокислот; и последовательность вариабельного домена легкой цепи, состоящую по существу из последовательностей, то есть по меньшей мере на 80%, или, более предпочтительно, 85%, 90%, 95% или даже 100% идентичную последовательностям аминокислот, выбранным из группы, состоящей из SEQ ID NO:4, SEQ ID NO:8, SEQ ID NO:12, SEQ ID NO:16, SEQ ID NO:20, SEQ ID NO:24, SEQ ID NO:28, SEQ ID NO:32, SEQ ID NO:36, SEQ ID NO:40, SEQ ID NO:44, SEQ ID NO:48, SEQ ID NO:52, SEQ ID NO:56, SEQ ID NO:60, SEQ ID NO:64, SEQ ID NO:68, SEQ ID NO:72, SEQ ID NO: 76, SEQ ID NO:80, SEQ ID NO:84, SEQ ID NO:88, SEQ ID NO:92, SEQ ID NO:96, SEQ ID NO:100, SEQ ID NO:104, SEQ ID NO:108, и их комбинаций.
[0009] В предпочтительных вариантах осуществления антигенсвязывающие полипептид или антигенсвязывающее антитело по настоящему изобретению включает пару вариабельной области тяжелой цепи и вариабельной области легкой цепи, причем их соответствующие последовательности по существу состоят из следующих пар: (a) SEQ ID NO:18 и SEQ ID NO:20; (b) SEQ ID NO:42 и SEQ ID NO:44; или (c) SEQ ID NO:34 и SEQ ID NO:36.
[00010] В других предпочтительных вариантах осуществления антигенсвязывающий полипептид или антигенсвязывающее антитело по настоящему изобретению включает пару вариабельной области тяжелой цепи и вариабельной области легкой цепи, причем их соответствующие последовательности по существу состоят из следующих пар: (a) SEQ ID NO:22 и SEQ ID NO:24; (b) SEQ ID NO:2 и SEQ ID NO:4; (c) SEQ ID NO:62 и SEQ ID NO:64; или (d) SEQ ID NO:82 и SEQ ID NO:84.
[00011] В других предпочтительных вариантах осуществления антигенсвязывающий полипептид или антигенсвязывающее антитело по настоящему изобретению включает пару вариабельной области тяжелой цепи и вариабельной области легкой цепи, причем их соответствующие последовательности по существу состоят из следующих пар: (a) SEQ ID NO:70 и SEQ ID NO:72; (b) SEQ ID NO:50 и SEQ ID NO:52; (c) SEQ ID NO:102 и SEQ ID NO:104; или (d) SEQ ID NO:30 и SEQ ID NO:32.
[00012] В других предпочтительных вариантах осуществления антигенсвязывающий полипептид или антигенсвязывающее антитело по настоящему изобретению включает пару вариабельной области тяжелой цепи и вариабельной области легкой цепи, причем их соответствующие последовательности по существу состоят из следующих пар: (a) SEQ ID NO:6 и SEQ ID NO:8; (b) SEQ ID NO:10 и SEQ ID NO:12; (c) SEQ ID NO:14 и SEQ ID NO:16; (d) SEQ ID NO:26 и SEQ ID NO:28; (e) SEQ ID NO:38 и SEQ ID NO:40; (f) SEQ ID NO:46 и SEQ ID NO:48; (g) SEQ ID NO:54 и SEQ ID NO:56; или (h) SEQ ID NO:58 и SEQ ID NO:60.
[00013] В других предпочтительных вариантах осуществления антигенсвязывающий полипептид или антигенсвязывающее антитело по настоящему изобретению включает пару вариабельной области тяжелой цепи и вариабельной области легкой цепи, причем их соответствующие последовательности по существу состоят из следующих пар: (a) SEQ ID NO:66 и SEQ ID NO:68; (b) SEQ ID NO:74 и SEQ ID NO:76; (c) SEQ ID NO:78 и SEQ ID NO:80; (d) SEQ ID NO:86 и SEQ ID NO:88; (e) SEQ ID NO:90 и SEQ ID NO:92; (f) SEQ ID NO:94 и SEQ ID NO:96; (g) SEQ ID NO:98 и SEQ ID NO:100; или (h) SEQ ID NO:106 и SEQ ID NO:108.
[00014] Предпочтительно указанный антигенсвязывающий полипептид является полностью человеческим или иным образом гуманизированным. В предпочтительном варианте осуществления указанный антигенсвязывающий полипептид дополнительно содержит константную область человека. Согласно одному признаку указанной константной областью человека является IgG1. В некотором варианте осуществления антитело по настоящему изобретению дополнительно включает вторую пару вариабельных областей тяжелой и легкой цепей, которая, например, по существу идентична первой паре.
[00015] В предпочтительном варианте связывание анти-PD-L1 полипептида с PD-L1 блокирует взаимодействие PD-L1 с PD1. Это может быть связано либо с тем, что эпитоп для связывания с PD-L1 расположен в интерфейсе или рядом с интерфейсом взаимодействия PD1, либо с аллостерическим изменением конформации интерфейса взаимодействия PD1.
[00016] Согласно другому аспекту настоящего изобретения предложены молекулы нуклеиновой кислоты, которые кодируют вышеупомянутые полипептиды. Молекула нуклеиновой кислоты может быть молекулой ДНК или РНК. В предпочтительных вариантах осуществления молекула нуклеиновой кислоты является молекулой ДНК, кодирующей вариабельную область тяжелой цепи и вариабельную область легкой цепи антигенсвязывающих полипептида или антитела по настоящему изобретению, где последовательности ДНК состоят по существу, соответственно, из следующих пар: (a) SEQ ID NO:17 и SEQ ID NO:19; (b) SEQ ID NO:33 и SEQ ID NO:35; (c) SEQ ID NO:41 и SEQ ID NO:43.
[00017] В других предпочтительных вариантах осуществления молекула нуклеиновой кислоты является молекулой ДНК, кодирующей вариабельную область тяжелой цепи и вариабельную область легкой цепи антигенсвязывающих полипептида или антитела по настоящему изобретению, где последовательности ДНК состоят по существу, соответственно, из следующих пар (a) SEQ ID NO:21 и SEQ ID NO:23; (b) SEQ ID NO:1 и SEQ ID NO:3; (c) SEQ ID NO:61 и SEQ ID NO:63; или (d) SEQ ID NO:81 и SEQ ID NO:83.
[00018] В других предпочтительных вариантах осуществления молекула нуклеиновой кислоты является молекулой ДНК, кодирующей вариабельную область тяжелой цепи и вариабельную область легкой цепи антигенсвязывающих полипептида или антитела по настоящему изобретению, где последовательности ДНК состоят по существу, соответственно, из следующих пар: (a) SEQ ID NO:69 и SEQ ID NO:71; (b) SEQ ID NO:49 и SEQ ID NO:51; (c) SEQ ID NO:101 и SEQ ID NO:103; или (d) SEQ ID NO:29 и SEQ ID NO:31.
[00019] В других предпочтительных вариантах осуществления молекула нуклеиновой кислоты является молекулой ДНК, кодирующей вариабельную область тяжелой цепи и вариабельную область легкой цепи антигенсвязывающих полипептида или антитела по настоящему изобретению, где последовательности ДНК состоят по существу, соответственно, из следующих пар: (a) SEQ ID NO:5 и SEQ ID NO:7; (b) SEQ ID NO:9 и SEQ ID NO:11; (c) SEQ ID NO:13 и SEQ ID NO:15; (d) SEQ ID NO:25 и SEQ ID NO:27; (e) SEQ ID NO:37 и SEQ ID NO:39; (f) SEQ ID NO:45 и SEQ ID NO:47; (g) SEQ ID NO:53 и SEQ ID NO:55; или (h) SEQ ID NO:57 и SEQ ID NO:59.
[00020] В других предпочтительных вариантах осуществления молекула нуклеиновой кислоты является молекулой ДНК, кодирующей вариабельную область тяжелой цепи и вариабельную область легкой цепи антигенсвязывающих полипептида или антитела по настоящему изобретению, где последовательности ДНК состоят по существу, соответственно, из следующих пар: (a) SEQ ID NO:65 и SEQ ID NO:67; (b) SEQ ID NO:73 и SEQ ID NO:75; (c) SEQ ID NO:77 и SEQ ID NO:79; (d) SEQ ID NO:85 и SEQ ID NO:87; (e) SEQ ID NO:89 и SEQ ID NO:91; (f) SEQ ID NO:93 и SEQ ID NO:95; (g) SEQ ID NO:97 и SEQ ID NO:99; или (h) SEQ ID NO:105 и SEQ ID NO:107.
[00021] Согласно другому аспекту настоящего изобретения предложена фармацевтическая композиция, включающая антигенсвязывающий полипептид, например, анти-PD-L1 антитело, его фрагмент, производное или аналог согласно описанию в настоящем документе. Фармацевтическая композиция дополнительно включает фармацевтически приемлемые вспомогательное вещество, носитель или разбавитель.
[00022] Согласно сходному аспекту настоящего изобретения предложен способ терапевтического лечения субъекта, нуждающегося в этом, от патологического состояния, включающий введение указанному субъекту терапевтически эффективного количества анти-PD-L1 полипептида или антитела, раскрытых в данном документе. Указанный способ может дополнительно включать этап введения второго, другого терапевтического антитела против по меньшей мере одного антигена клеточной поверхности, свидетельствующего об указанном состоянии. Состояние, которое лечат, может быть раком млекопитающих, инфекцией и т. п. В различных вариантах осуществления указанный анти-PD-L1 полипептид может быть антителом, фрагментом антитела, производным антитела или аналогом антитела.
[00023] Предпочтительно, спектр подлежащих лечению видов рака млекопитающих выбран из группы, состоящей из: рака яичников, рака толстого кишечника, рака молочной железы, рака легких, миелом, опухолей ЦНС нейробластного происхождения, моноцитарных лейкозов, B-клеточных лейкозов, Т-клеточных лейкозов, B-клеточных лимфом, Т-клеточных лимфом, опухолей, происходящих из тучных клеток, меланомы, рака мочевого пузыря, рака желудка, рака печени, уротелиальной карциномы, кожной карциномы, рака почек, рака головы и шеи, рака поджелудочной железы и их комбинаций. В более широком смысле предусмотрено, что любые виды рака, при которых по меньшей мере значительная часть опухолевых клеток экспрессирует детектируемое количество PD-L1, являются мишенью для лечения композицией по настоящему изобретению.
[00024] Согласно еще одному аспекту настоящего изобретения предложен способ профилактического лечения нуждающегося в этом субъекта при аналогичных состояниях, включающий введение указанному субъекту профилактически эффективного количества фармацевтической композиции по настоящему изобретению. Указанный способ может дополнительно включать этап введения вакцины против указанного состояния. В одном из вариантов осуществления указанное состояние представляет собой рак.
[00025] Согласно дополнительному аспекту настоящего изобретения предложена экспрессионная система млекопитающих, продуцирующая антигенсвязывающий полипептид, например, антитело, его фрагмент, производное или аналог, который связывается с эпитопом PD-L1 согласно описанию в настоящем документе.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[00026] На фиг. 1 схематически изображен скрининг на антигенсвязывающие полипептиды с использованием технологий твердофазного фагового пэннинга, в частности, с применением непрямого покрытия тестируемыми белками пробирок для иммуносорбции, согласно варианту осуществления настоящего изобретения.
[00027] На фиг. 2 схематически изображен скрининг антигенсвязывающих полипептидов с использованием технологий твердофазного фагового пэннинга, в частности, с применением прямого покрытия тестируемыми белками пробирок для иммуносорбции, согласно варианту осуществления настоящего изобретения.
[00028] Фиг. 3 представляет собой таблицу данных, которые характеризуют способность связывать hPDL1 репрезентативных одноцепочечных вариабельных фрагментов (scfv), полученных во варианте осуществления настоящего изобретения в непрямом анализе связывания ИФА ELISA. «ОК» означает отрицательный контроль.
[00029] Фиг. 4 представляет собой таблицу данных, которые характеризуют способность связывать hPDL1 репрезентативных одноцепочечных вариабельных фрагментов (scfv), полученных во варианте осуществления настоящего изобретения в FACS-анализе связывания. «ПК» означает положительный контроль с применением окрашенных APC-конъюгированным антителом против hPDL1 клеток hPDL1/293T (10 мкг/мл). «ОК» означает отрицательный контроль с неокрашенными клетками hPDL1/293T.
[00030] Фиг. 5 представляет собой таблицу данных, которые характеризуют способность блокировать взаимодействие между hPD1 и hPDL1 различных одноцепочечных вариабельных фрагментов (scfv), полученных во варианте осуществления настоящего изобретения в анализе блокирования рецепторов (планшеты, покрытые hPDL1). «ПК» означает положительный контроль с добавленным биотин-hPD1-Fc. «ОК» означает отрицательный контроль, в который добавляли только буфер.
[00031] Фиг. 6 представляет собой таблицу данных, которые характеризуют способность блокировать взаимодействие между hPD1 и hPDL1различных одноцепочечных вариабельных фрагментов (scfv), полученных во варианте осуществления настоящего изобретения в анализе блокирования рецепторов (планшеты, покрытые hPD1). «ПК» означает положительный контроль с добавленным биотин-hPDL1-Fc. «ОК» означает отрицательный контроль, в который добавляли только буфер.
[00032] Фиг. 7 иллюстрирует способность одноцепочечных вариабельных фрагментов (scfv), полученных во варианте осуществления настоящего изобретения в прямых ИФА ELISA, связывать hPDL1-Fc, mPDL1-Fc (PDL1 мыши) и hIgG1.
[00033] На фиг. 8A и 8B показано полноразмерное антитело 4-1E8, охарактеризованное с помощью ДСН-ПААГ (фиг. 8A) и эксклюзионной хроматографии (фиг. 8B).
[00034] На фиг. 9A и 9B показано полноразмерное антитело 3-1B11, охарактеризованное с помощью ДСН-ПААГ (фиг. 9A) и эксклюзионной хроматографии (фиг. 9B).
[00035] На фиг. 10A и 10B показано полноразмерное антитело 3-1E4, охарактеризованное с помощью ДСН-ПААГ (фиг. 10A) и эксклюзионной хроматографии (фиг. 10B).
[00036] На фиг. 11B и 11C показаны результаты количественного анализа связывания некоторых вариантов осуществления полноразмерного антитела по настоящему изобретению с hPDL1 в формате ИФА ELISA согласно фиг. 11A.
[00037] На фиг. 12A и 12B показаны результаты количественного FACS для некоторых вариантов осуществления полноразмерного антитела по настоящему изобретению, где происходит связывание с hPDL1-экспрессирующими клетками 293T (верхний график), и hPDL1-отрицательными клетками 293T (нижний график).
[00038] На фиг. 13B показаны результаты анализа блокирования рецепторов для наилучших кандидатных антител по настоящему изобретению в формате RBA 1 (фиг. 13A): с покрытием hPDL1-Fc и с добавлением Биотин-hPD1-Fc.
[00039] На фиг. 14B показаны результаты анализа блокирования рецепторов для наилучших кандидатных антител по настоящему изобретению в формате RBA 2 (фиг. 14A): с покрытием hPD1-Fc и с добавлением Биотин-hPDL1-Fc.
[00040] Фиг. 15 представляет собой таблицу данных, которые характеризуют различные полноразмерные антитела, полученные во варианте осуществления настоящего изобретения.
[00041] На фиг. 16A-16D показана аффинность в отношении PD-L1 лучших кандидатных антител при использовании BIAcore: на фиг. 16A схематично представлен формат BIAcore, используемый согласно примеру настоящего изобретения; на фиг. 16B перечислены результаты тестирования аффинности в отношении PD-L1 лучших кандидатных антител с использованием BIAcore; на фиг. 16C приведена кривая отклика антитела с кодовым названием 4-1E8 при тестировании аффинности BIAcore; и на фиг. 16D изображена кривая отклика антитела с кодовым названием 3-1B11 при тестировании аффинности BIAcore.
[00042] На фиг. 17A схематически изображен формат биннинга эпитопов, используемого в соответствии с примером настоящего изобретения. На фиг. 17B схематически изображены группы («бины») эпитопов для лучших кандидатных антител согласно варианту осуществления настоящего изобретения. На фиг. 17C перечислена матрица биннинга эпитопов для лучших кандидатных антител с применением формата, представленного на фиг. 17A.
[00043] На фиг. 18A-18D показана способность к связыванию: контролей (фиг. 18A), антител по настоящему изобретению с кодовыми названиями «4-1E8» (фиг. 18B), «3-1E4» (фиг. 18C) и «3-1B11» (фиг. 18D) с клетками 293T, трансфицированными экспрессирующей PDL1-GFP резуса конструкцией (наверху), и исходными клетками 293T (внизу) в FACS-анализах.
[00044] На фиг. 19A-19D показана способность к связыванию: контролей (фиг. 19A), антител по настоящему изобретению с кодовыми названиями «4-1E8» (фиг. 19B), «3-1E4» (фиг. 19C) и «3-1B11» (фиг. 19D) с клетками 293T, трансфицированными экспрессирующей PDL1 резуса конструкцией (наверху), и исходными клетками 293T (внизу) в FACS-анализах.
[00045] На фиг. 20 приведены репрезентативные данные EC50 для эксперимента с продуцированием ИЛ-2 согласно вариантам осуществления изобретения.
[00046] На фиг. 21 показана АЗКЦ-активность варианта осуществления полипептида с кодовым названием «4-1E8» в сравнении с коммерчески доступным антителом против PDL1 атезолизумабом.
[00047] На фиг. 22A-22C показана АЗКЦ-активность варианта осуществления полипептида с кодовым названием «4-1E8» в сравнении с вариантами осуществления с кодовыми названиями «3-1B11» (фиг. 22A) и «3-1E4» (фиг. 22B); в таблице обобщены основные точки данных (фиг. 22C).
[00048] На фиг. 23A, 23B и 23C представлены три группы экспериментальных данных по способности продуцировать ИЛ-2 для мононуклеарных клеток периферической крови (МКПК), совместно культивируемых с опухолевыми PDL1+ клетками MDA-MB-231, в присутствии наилучших антител по настоящему изобретению, в сравнении с коммерчески доступными антителами против PDL1.
[00049] На фиг. 24 представлены результаты анализа способности продуцировать ИФН-γ у CD8 Т-клеток, совместно культивируемых с опухолевыми клетками PDL1+ MDA-MB-231, в присутствии наилучших антител по настоящему изобретению, в сравнении с коммерчески доступными антителами против PDL1.
[00050] На фиг. 25A и 25B показаны результаты реакции смешанной культуры лимфоцитов для наилучших антител в соответствии с вариантами осуществления изобретения.
[00051] На фиг. 26A и 26B показана специфичность связывания антителами по настоящему изобретению с кодовыми названиями «4-1E8» (фиг. 26A) и «3-1B11» (фиг. 26B).
[00052] На фиг. 27A и 27B показана способность антител по настоящему изобретению E8 (фиг. 27A) и B11 (фиг. 27B) блокировать связывание CD80 с клетками, экспрессирующими PD-L1 (кривые, область под которыми закрашена серым цветом), в сравнении с CD80 по отдельности (сплошная линия) и вторичным антителом по отдельности (пунктирная линия).
[00053] На фиг. 28 показано измерение времени полужизни варианта осуществления антитела с применением мышей Tg32.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[00054] Если не указано иное, технические термины используются в соответствии с общепринятыми значениями.
[00055] В настоящем описании термины в единственном числе могут означать один или более. При использовании в настоящем описании термины в единственном числе вместе со словом «содержащий» могут означать один или более. Используемый в настоящем описании термин «другой» может означать второй или более. Более того, если иное не требуется по контексту, термины в единственном числе включают термины во множественном числе, а термины во множественном числе включают термины в единственном числе.
[00056] В настоящем описании «приблизительно» относится к числовому значению, включая, например, целые числа, дроби и проценты, независимо от того, указаны они явным образом или нет. Термин «приблизительно», в целом, относится к диапазону числовых значений (например, +/-5-10% относительно приведенного значения), которые рядовой специалист в данной области техники счел бы эквивалентными приведенному значению (например, имеющим ту же функцию или результат). В некоторых случаях термин «приблизительно» может включать числовые значения, округленные до ближайшего значащего числа. Если не указано иное, «приблизительно» представляет собой +/-10% от приведенного значения (значений).
[00057] «Антигенсвязывающий полипептид» представляет собой полипептид, содержащий часть, которая связывается с антигеном. Примеры антигенсвязывающих полипептидов включают антитела, фрагменты (например, антигенсвязывающий участок антитела), производные и аналоги антител.
[00058] Антигенсвязывающий полипептид или белок может иметь, например, структуру встречающегося в природе антитела (также известного как «иммуноглобулин»). Каждое встречающееся в природе антитело состоит из двух идентичных пар полипептидных цепей, каждая пара имеет одну «легкую» (приблизительно 25 кДа) и одну «тяжелую» цепь (приблизительно 50-70 кДа). Вариабельные области каждой пары легкой/тяжелой цепи образуют сайт связывания антитела таким образом, что интактное антитело имеет два сайта связывания.
[00059] Вариабельные области встречающихся в природе цепей антител демонстрируют одинаковую общую структуру относительно консервативных каркасных областей (FR), соединенных тремя гипервариабельными областями, также называемыми областями, определяющими комплементарность, или CDR. От N-конца до C-конца как легкая, так и тяжелая цепи содержат домены FR1, CDR1, FR2, CDR2, FR3, CDR3 и FR4. Назначение аминокислот каждому домену соответствует определениям Kabat et al. в источнике: Sequences of Proteins of Immunological Interest, 5th Ed., US Dept. of Health and Human Services, PHS, NIH, NIH Publication no. 91-3242, 1991. Другие системы нумерации аминокислот в цепях иммуноглобулинов включают IMGT (международная информационная система ImMunoGeneTics; Lefranc et al., Dev. Comp. Immunol. 29:185-203; 2005) и AHo (Honegger and Pluckthun, J. Mol. Biol. 309(3):657-670; 2001).
[00060] Антитела могут быть получены из таких источников, как сыворотка или плазма, которые содержат иммуноглобулины с различной антигенной специфичностью. Если такие антитела подвергают аффинной очистке, они могут быть обогащены определенной антигенной специфичностью. Такие обогащенные составы с антителами обычно состоят из менее чем приблизительно 10% антител, обладающих специфической связывающей активностью в отношении конкретного антигена. Подвергая эти составы нескольким этапам аффинной очистки, можно увеличить долю антитела, обладающего специфической связывающей активностью в отношении антигена. Антитела, полученные таким образом, часто называют «моноспецифическими». Препараты моноспецифических антител могут состоять из приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 99% или 99,9% антитела, обладающего специфической связывающей активностью в отношении конкретного антигена.
[00061] Термин «антитело» или «Аb» (и их формы во множественном числе) в настоящем описании в широком смысле относится к любой молекуле иммуноглобулина (Ig), состоящей из четырех полипептидных цепей, двух тяжелых (H) цепей и двух легких (L), или к любым функциональным фрагменту (фрагментам), мутанту (мутантам), варианту (вариантам), производному (производным) или аналогу (аналогам), которые сохраняют существенные и специфические признаки связывания эпитопа молекулой Ig. Такие форматы фрагментов, мутантов, вариантов, производных или аналогов антител известны в данной области техники и включают, среди прочего, Fab, F(ab'), F(ab')2, Fv, одноцепочечные антитела (scFv), однодоменные антитела (sdAb), фрагменты определяющей комплементарность области (CDR), химерные антитела, диатела, триатела, тетратела, и полипептиды, которые содержат по меньшей мере часть иммуноглобулина, достаточную для обеспечения специфического связывания антигена с полипептидом. Фрагменты, производные и аналоги антител могут быть получены методами рекомбинантной ДНК, или путем ферментативного или химического расщепления интактных антител.
[00062] Фрагмент Fab представляет собой моновалентный фрагмент, имеющий домены VL, VH, CL и CH1; фрагмент F(ab')2 представляет собой бивалентный фрагмент, содержащий два фрагмента Fab, связанных дисульфидным мостиком в шарнирной области; фрагмент Fd имеет домены VH и CH1; фрагмент Fv имеет домены VL и VH одного плеча антитела; и фрагмент dAb имеет домены VH, домен VL, или антигенсвязывающий фрагмент домена VH или VL (см., например, патенты США № 6846634; 6696245, опубликованные заявки на патент США 20/0202512; 2004/0202995; 2004/0038291; 2004/0009507; 2003/0039958, и Ward et al., Nature 341:544-546, 1989).
[00063] Одноцепочечное антитело (scFv) представляет собой антитело, в котором области VL и VH соединены через линкер (например, синтетическую последовательность аминокислотных остатков) с образованием непрерывной белковой цепи, при этом линкер достаточно длинный, чтобы позволять белковой цепи сворачиваться и формировать моновалентный антигенсвязывающий сайт (см., например, Bird et al., 1988, Science 242:423-26 и Huston et al., 1988, Proc. Natl. Acad. Sci. USA 85:5879-83). Диатела представляют собой бивалентные антитела, содержащие две полипептидные цепи, где каждая полипептидная цепь включает домены VH и VL, соединенные линкером, слишком коротким, чтобы позволять спаривание между двумя доменами одной цепи, что позволяет каждому домену спариваться с комплементарным доменом другой полипептидной цепи (см., например, Holliger et al., 1993, Proc. Natl. Acad. Sci. USA 90:6444-48, и Poljak et al., 1994, Structure 2:1121-23). Если две полипептидные цепи диатела идентичны, то диатело, полученное в результате их спаривания, будет иметь два идентичных антигенсвязывающих сайта. Полипептидные цепи, имеющие разные последовательности, можно применять для получения диатела с двумя разными антигенсвязывающими сайтами. Аналогичным образом, триатела и тетратела представляют собой антитела, содержащие три и четыре полипептидных цепи, соответственно, и образующие, соответственно, три и четыре антигенсвязывающих сайта, которые могут быть одинаковыми или разными.
[00064] Определяющие комплементарность области (CDRи) и каркасные участки (FR) данного антитела могут быть идентифицированы с использованием системы, описанной в источниках: Kabat et al. выше; Lefranc et al., выше, и/или Honegger and Pluckthun, выше. Одна или несколько CDR могут быть включены в молекулу ковалентным или нековалентным образом, превращая ее в антигенсвязывающий белок. Антигенсвязывающий полипептид может включать область или области CDR в качестве части большей полипептидной цепи, может ковалентно связывать область или области CDR с другой полипептидной цепью или может нековалентным образом включать область или области CDR. CDR позволяют антигенсвязывающему белку специфически связываться с конкретным представляющим интерес антигеном.
[00065] Антигенсвязывающий полипептид может иметь один или более сайтов связывания. Если существует более одного сайта связывания, то сайты связывания могут быть идентичны друг другу или могут быть разными. Например, встречающийся в природе иммуноглобулин человека обычно имеет два идентичных сайта связывания, тогда как «биспецифическое» или «бифункциональное» антитело имеет два разных сайта связывания.
[00066] В настоящем документе термин «антитело человека» или «гуманизированное антитело» включает все антитела, которые имеют одну или несколько вариабельных и константных областей, происходящих из последовательностей иммуноглобулина человека. Согласно одному варианту осуществления все вариабельные и константные домены происходят из последовательностей иммуноглобулина человека (полностью человеческое или гуманизированное антитело). Эти антитела могут быть получены различными способами, в том числе путем иммунизации представляющим интерес антигеном мыши, которая была генетически модифицирована для экспрессии антител, происходящих из генов, кодирующих тяжелую и/или легкую цепь человека. Гуманизированное антитело имеет последовательность, которая отличается от последовательности антитела, происходящего от не являющегося человеком вида, одной или несколькими заменами, делециями и/или добавлениями аминокислот, такими, чтобы гуманизированное антитело с меньшей вероятностью вызывало иммунный ответ и/или вызывало менее тяжелый иммунный ответ по сравнению с антителом не являющегося человеком вида при введении субъекту-человеку. Согласно одному варианту осуществления определенные аминокислоты в каркасных и константных доменах тяжелой и/или легкой цепей антитела не являющегося человеком вида мутированы с образованием гуманизированного антитела. Согласно другому варианту осуществления константный домен (домены) из человеческого антитела сливают с вариабельным(и) доменом (доменами) от не являющегося человеком вида. Согласно другому варианту осуществления один или несколько аминокислотных остатков в одной или нескольких последовательностях CDR антитела не являющегося человеком вида изменяют для снижения вероятной иммуногенности указанного антитела не являющегося человеком вида при его введении субъекту-человеку, причем измененные аминокислотные остатки либо не являются критическими для иммуноспецифического связывания антитела с его антигеном, либо внесенные в аминокислотную последовательность изменения являются консервативными изменениями, так что связывание гуманизированного антитела с антигеном не является значимо худшим, чем связывание антитела не являющегося человеком вида с антигеном. Примеры получения гуманизированных антител можно найти в патентах США № 6054297, 5886152 и 5877293.
[00067] В настоящем документе термин «химерное антитело» относится к антителу, которое содержит одну или более областей одного антитела и одну или более областей по меньшей мере другого антитела. Согласно одному варианту осуществления области CDR от более чем одного антитела против PD-L1 человека свободно комбинируют в различных сочетаниях в химерном антителе.
[00068] Активированные Т-клетки экспрессируют PD1 на клеточной поверхности. Связывание PD-L1 с PD1 активирует PD1 и подавляет PD1+ Т-клетки. Термин «нейтрализующее антитело» или «ингибирующее антитело» в настоящем документе относится к антителу, блокирующему активацию PD1, когда избыток антитела против PD-L1 снижает уровень указанной активации по меньшей мере приблизительно на 20% в анализе, описанном здесь в примерах. Согласно различным вариантам осуществления антигенсвязывающий белок снижает уровень активации PD1 по меньшей мере на 30%, 40%, 50%, 60%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 99% и 99,9%.
[00069] Фрагменты или аналоги антител могут быть легко получены специалистами в данной области техники при следовании принципам, описанным в настоящем документе и использовании методов, известных в данной области техники. Предпочтительные аминоконцы и карбоксильные концы фрагментов или аналогов расположены вблизи границ функциональных доменов. Структурные и функциональные домены могут быть идентифицированы путем сравнения данных нуклеотидных и/или аминокислотных последовательностей с общедоступными или проприетарными базами данных последовательностей. Компьютеризированные методы сравнения могут применяться для идентификации мотивов последовательностей или доменов предсказанной конформации белка, которые встречаются в других белках известной структуры и/или функции. Известны методы идентификации белковых последовательностей, которые укладываются в известную трехмерную структуру. См. Bowie et al., 1991, Science 253:164.
[00070] В настоящем документе антигенсвязывающие полипептид «специфически связывается» с антигеном (например, PD-L1 человека), если он связывается с антигеном со 100-наномолярной или меньшей константой диссоциации.
[00071] В настоящем документе «антигенсвязывающий домен», «антигенсвязывающая область» или «антигенсвязывающий сайт» представляет собой участок антигенсвязывающего белка, который содержит аминокислотные остатки (или другие фрагменты), которые взаимодействуют с антигеном и вносят вклад в специфичность антигенсвязывающего белка и аффинность к антигену. Чтобы антитело специфически связывалось со своим антигеном, оно должно включать по меньшей мере часть по меньшей мере одного из его доменов CDR.
[00072] В настоящем документе «эпитоп» представляет собой часть молекулы, которую связывает антигенсвязывающий белок (например, антитело). Эпитоп может содержать несмежные части молекулы (например, аминокислотные остатки в полипептиде, которые не являются смежными в первичной последовательности полипептида, но в условиях третичной и четвертичной структуры полипептида находятся достаточно близко друг к другу, чтобы быть связанными антигенсвязывающим белком).
[00073] В настоящем документе термины «полинуклеотид», «олигонуклеотид» и «нуклеиновая кислота» применяются взаимозаменяемо и включают молекулы ДНК (например, кДНК или геномной ДНК), молекулы РНК (например, мРНК), аналоги ДНК или РНК, созданные с применением аналогов нуклеотидов (например, пептидных нуклеиновых кислот и не встречающихся в природе аналогов нуклеотидов), и их гибриды. Молекула нуклеиновой кислоты может быть одноцепочечной или двуцепочечной. Согласно одному варианту осуществления молекулы нуклеиновой кислоты по настоящему изобретению содержат непрерывную открытую рамку считывания, кодирующую антитело или его фрагмент, производное, мутант или вариант.
[00074] «Вектор» в контексте настоящего документа означает нуклеиновую кислоту, которую можно применять для введения другой, связанной с ней нуклеиновой кислоты в клетку. Одним из типов вектора является «плазмида», представляющая собой линейную или кольцевую двуцепочечную молекулу ДНК, в которую могут быть лигированы дополнительные сегменты нуклеиновой кислоты. Другой тип вектора представляет собой вирусный вектор (например, дефектные по репликации ретровирусы, аденовирусы и аденоассоциированные вирусы), при этом дополнительные сегменты ДНК могут быть введены в вирусный геном. Определенные векторы способны к автономной репликации в клетке-хозяине, в которую они введены (например, бактериальные векторы, содержащие бактериальную точку начала репликации, и эписомные векторы млекопитающих). Другие векторы (например, неэписомные векторы млекопитающих) интегрируются в геном клетки-хозяина после введения в клетку-хозяина и, таким образом, реплицируются вместе с геномом хозяина. «Экспрессионный вектор» - это тип вектора, который может управлять экспрессией выбранного полинуклеотида.
[00075] В настоящем документе нуклеотидная последовательность является «функционально связанной» с регуляторной последовательностью, если регуляторная последовательность влияет на экспрессию (например, уровень, временную динамику или локализацию экспрессии) указанной нуклеотидной последовательности. «Регуляторная последовательность» представляет собой нуклеиновую кислоту, влияющую на экспрессию (например, уровень, временную динамику или локализацию экспрессии) нуклеиновой кислоты, с которой она функционально связана. Регуляторная последовательность может, например, оказывать на регулируемую нуклеиновую кислоту действие непосредственно или через действие одной или нескольких других молекул (например, полипептидов, которые связываются с регуляторной последовательностью и/или нуклеиновой кислотой). Примеры регуляторных последовательностей включают промоторы, энхансеры и другие элементы контроля экспрессии (например, сигналы полиаденилирования). Дополнительные примеры регуляторных последовательностей описаны, например, в источниках: Goeddel, 1990, Gene Expression Technology: Methods in Enzymology 185, Academic Press, San Diego, Calif. и Baron et al., 1995, Nucleic Acids Res. 23:3605-06.
[00076] Предпочтительно, широкий спектр видов рака млекопитающих, подлежащих лечению композициями по настоящему изобретению, выбран из группы, состоящей из: рака яичников, рака толстого кишечника, рака молочной железы, рака легких, миелом, опухолей ЦНС нейробластного происхождения, моноцитарных лейкозов, B-клеточных лейкозов, Т-клеточных лейкозов, B-клеточных лимфом, Т-клеточных лимфом, опухолей, происходящих из тучных клеток, меланомы, рака мочевого пузыря, рака желудка, рака печени, уротелиальной карциномы, кожной карциномы, рака почек, рака головы и шеи, рака поджелудочной железы и их комбинации. В более широком смысле, любой рак, при котором по меньшей мере часть опухолевых клеток экспрессирует заметное количество PD-L1, потенциально можно лечить композицией по настоящему изобретению.
[00077] Полипептиды согласно настоящему описанию могут быть получены с применением любых стандартных методов, известных в данной области техники. В одном примере указанные полипептиды получают методами рекомбинантной ДНК путем инсерции последовательности нуклеиновой кислоты (например, кДНК), кодирующей полипептид, в рекомбинантный экспрессионный вектор, и экспрессирования указанной последовательности ДНК в условиях, способствующих экспрессии.
[00078] Нуклеиновые кислоты, кодирующие любой из множества полипептидов, раскрытых в настоящем документе, могут быть синтезированы химическим путем. Использование кодонов может быть подобрано так, чтобы улучшать экспрессию в клетке. Такое применение кодонов будет зависеть от выбранного типа клетки. Специализированные схемы использования кодонов были разработаны для E. coli и других бактерий, а также для клеток млекопитающих, клеток растений, дрожжевых клеток и клеток насекомых. См., например: Mayfield et al., Proc. Natl. Acad. Sci. USA. 2003 100(2):438-42; Sinclair et al. Protein Expr. Purif. 2002 (1):96-105; Connell N D. Curr. Opin. Biotechnol. 2001 12(5):446-9; Makrides et al. Microbiol. Rev. 1996 60(3):512-38; и Sharp et al. Yeast. 1991 7(7):657-78.
[00079] Общие методы манипуляции с нуклеиновыми кислотами описаны, например, в источниках: Sambrook et al., Molecular Cloning: A Laboratory Manual, Vols. 1-3, Cold Spring Harbor Laboratory Press, 2 ed., 1989, или F. Ausubel et al., Current Protocols in Molecular Biology (Green Publishing и Wiley-Interscience: New York, 1987), с периодическими обновлениями, включенных в настоящий документ посредством ссылки. ДНК, кодирующая полипептид, функционально связана с подходящими регуляторными элементами транскрипции или трансляции, полученными из генов млекопитающих, вирусов или насекомых. Такие регуляторные элементы включают транскрипционный промотор, необязательную последовательность оператора для контроля транскрипции, последовательность, кодирующую подходящие сайты связывания рибосомы с мРНК, и последовательности, которые контролируют терминацию транскрипции и трансляции. Дополнительно включают способность к репликации в хозяине, обычно обеспечиваемую точкой начала репликации, и селективный ген для облегчения распознавания трансформантов.
[00080] Рекомбинантная ДНК по настоящему изобретению может также включать любой тип последовательности белковой метки, которая может быть полезна для очистки белка. Примеры белковых меток включают, не ограничиваясь перечисленными, гистидиновую метку, метку FLAG, метку myc, HA-метку или GST-метку. Описание подходящих клонирующих и экспрессионных векторов для применения с бактериальными, грибными, дрожжевыми клетками-хозяевами и клетками-хозяевами млекопитающих можно найти в источнике: Cloning Vectors: A Laboratory Manual, (Elsevier, N. Y., 1985).
[00081] Экспрессионную конструкцию по настоящему изобретению вводят в клетку-хозяина с применением способа, подходящего указанной для клетки-хозяина. В данной области техники известны различные способы введения нуклеиновых кислот в клетки-хозяева, включающие, не ограничиваясь перечисленными: электропорацию; трансфекцию с использованием хлорида кальция, хлорида рубидия, фосфата кальция, DEAE-декстрана или других веществ; бомбардировку микрочастицами; липофекцию; и инфекцию (где вектор является инфекционным агентом). Подходящие клетки-хозяева включают клетки прокариот, дрожжей, млекопитающих, или бактериальные клетки.
[00082] Описанные в настоящем документе белки также могут быть получены с применением систем клеточной трансляции. Для таких целей нуклеиновые кислоты, кодирующие полипептид, должны быть модифицированы, чтобы позволять транскрипцию in vitro для продуцирования мРНК, и позволять бесклеточную трансляцию мРНК в конкретной используемой бесклеточной системе, эукариотической, такой как бесклеточная система трансляции млекопитающих или дрожжей, или прокариотической, такой как бесклеточная система трансляции бактерий.
[00083] PD-L1-связывающие полипептиды также могут быть получены химическим синтезом (например, способами, описанными в источнике: Solid Phase Peptide Synthesis, 2nd ed., 1984, The Pierce Chemical Co., Rockford, Ill.). Модификации в белке также могут быть получены путем химического синтеза.
[00084] Полипептиды по настоящему изобретению могут быть очищены методами выделения/очистки белков, широко известными в области химии белков. Неограничивающие примеры включают экстракцию, перекристаллизацию, высаливание (например, с сульфатом аммония или сульфатом натрия), центрифугирование, диализ, ультрафильтрацию, адсорбционную хроматографию, ионообменную хроматографию, гидрофобную хроматографию, нормально-фазовую хроматографию, обращенно-фазовую хроматографию, гель-фильтрацию, эксклюзионную хроматографию, аффинную хроматографию, электрофорез, противоточное распределение или любые их комбинации. После очистки буфер полипептидов может быть заменен на другие буферы и/или полипептиды могут быть сконцентрированы любым из множества способов, известных в данной области техники, включая, но не ограничиваясь указанными, фильтрацию и диализ.
[00085] Очищенный полипептид предпочтительно имеет чистоту по меньшей мере 85%, более предпочтительно по меньшей мере 90% или 95%, и наиболее предпочтительно по меньшей мере 98%. Независимо от точного числового значения чистоты, полипептид очищен достаточно для применения в качестве фармацевтического продукта.
Посттрансляционные модификации полипептидов
[00086] Согласно определенному варианту осуществления связывающие полипептиды изобретения могут дополнительно содержать посттрансляционные модификации. Примеры посттрансляционных модификаций белков включают фосфорилирование, ацетилирование, метилирование, АДФ-рибозилирование, убиквитинирование, гликозилирование, карбонилирование, сумоилирование, биотинилирование, или добавление боковой цепи полипептида, или гидрофобной группы. В результате модифицированные растворимые полипептиды могут содержать неаминокислотные элементы, такие как липиды, поли- или моносахариды и фосфаты. Предпочтительной формой гликозилирования является сиалирование, при котором один или более фрагментов сиаловой кислоты конъюгируют с полипептидом. Фрагменты сиаловой кислоты улучшают растворимость и увеличивают период полужизни в сыворотке, а также снижают возможную иммуногенность белка. См. Raju et al. Biochemistry. 2001 31; 40(30):8868-76. Действие таких неаминокислотных элементов на функциональность полипептида может быть протестировано применительно к его антагонизирующей роли в отношении функции PD-L1 или PD-1, например, его ингибирующего действия на ангиогенез или рост опухоли.
[00087] Согласно одному варианту осуществления модифицированные формы рассматриваемых полипептидов включают связывание рассматриваемых растворимых полипептидов с небелковыми полимерами. В одном конкретном варианте осуществления указанный полимер представляет собой полиэтиленгликоль («ПЭГ»), полипропиленгликоль или полиоксиалкилены, как изложено в патентах США №4640835; №4496689; №4301144; №4670417; №4791192 или №4179337.
[00088] Согласно одному признаку пегилированные варианты связывающих полипептидов по настоящему изобретению предпочтительно сохраняют по меньшей мере 25%, 50%, 60%, 70%, 80%, 90%, 95% или 100% биологической активности, ассоциированной с немодифицированным белком. Согласно одному варианту осуществления биологическая активность относится к способности связываться с PD-L1, которую оценивают по скоростям KD, kon или koff. Согласно одному конкретному варианту осуществления пегилированный связывающий полипептид белок демонстрирует увеличение связывания с PD-L1 человека по сравнению с непегилированным аналогом. Согласно другому варианту осуществления биологическая активность относится к блокаде взаимодействия PD-L1/PD1.
Терапевтические средства, вакцины и введение
[00089] Настоящее изобретение также относится к способам лечения состояний или предотвращения предварительных состояний, которые откликаются на ингибирование биологической активности PD-L1. Предпочтительными примерами являются состояния, которые характеризуются клеточной гиперпролиферацией и длительной инфекцией. Методы и дозировки для введения варьируют в зависимости от типа конкретного полипептида и конкретного состояния, которое лечат. Поскольку регулирующие органы требуют, чтобы белковый реагент, используемый в качестве терапевтического средства, был представлен в составе с приемлемо низкими уровнями пирогенов, терапевтические составы по настоящему изобретению можно отличить от других составов по тому, что они по существу не содержат пирогенов или по меньшей мере содержат не более чем приемлемые уровни пирогенов, определенные соответствующим регулирующим органом (например, FDA в США).
[00090] Фармацевтические композиции по настоящему изобретению могут включать по меньшей мере один фармацевтически приемлемый разбавитель, носитель или вспомогательное вещество. Вспомогательные вещества, включенные в композиции, имеют разное назначение в зависимости, например, от типа используемой генной конструкции или эффекторных клеток, и от способа введения. Примеры широко используемых носителей включают, без ограничения: физиологический раствор, забуференный физиологический раствор, декстрозу, воду для инъекций, глицерин, этанол и их комбинации, стабилизирующие агенты, солюбилизирующие агенты и поверхностно-активные вещества, буферы и консерванты, агенты для регуляции тоничности, объемообразующие агенты и смазывающие агенты.
[00091] Согласно другому варианту осуществления изобретения фармацевтическую композицию по изобретению вводят пациенту. Примеры способов введения включают, не ограничиваясь указанным, внутривенную инъекцию. Другие способы включают, без ограничения, внутриопухолевый, внутрикожный, подкожный (п/к, п.к., sub-Q, Hypo), внутримышечный (в/м), внутрибрюшинный (в/б), внутриартериальный, внутрикостный, внутрисердечный, внутрисуставный (в сустав), внутрисуставной (в область синовиальной жидкости), внутричерепной, внутрипозвоночный и интратекальный (в спинномозговую жидкость). Любое известное устройство, пригодное для парентеральной инъекции или инфузии составов, может быть использовано для осуществления такого введения. В настоящем документе термины «лечить» и «лечение» имеют обычные и общепринятые значения, и включают одно или более из: блокирования, улучшения или уменьшения тяжести и/или частоты симптома заболевания (например, рака) у субъекта, и/или ингибирования роста, деления, распространения или пролиферации раковых клеток, или прогрессирования рака (например, появления новых опухолей) у субъекта. Лечение означает блокирование, улучшение, уменьшение или ингибирование приблизительно на 5-100% по сравнению с субъектом, у которого не применяли на практике способы по настоящему изобретению. Предпочтительно, блокирование, улучшение, уменьшение или ингибирование составляет приблизительно 100%, 99%, 95%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10% или 5% по сравнению с субъектом, у которого не применяли на практике способы по настоящему изобретению.
[00092] Согласно настоящему изобретению предложен набор, включающий один или более контейнеров, заполненных некоторым количеством генных конструкций, кодирующих полипептиды по настоящему изобретению, с фармацевтически приемлемыми вспомогательными веществами. В набор также может быть включена инструкция по применению. Кроме того, с набором может быть предоставлено уведомление в форме, предписанной государственным органом, регулирующим производство, применение или продажу фармацевтических или биологических продуктов, отражающее одобрение указанным органом производства, применения или продажи для введения человеку.
ПРИМЕРЫ
[00093] Скрининг антигенсвязывающего полипептида с использованием методов фагового дисплея:
[00094] Непрямое покрытие: Фиг. 1 - PDL1-связывающие одноцепочечные вариабельные фрагменты (scFv) идентифицировали стандартным методом фагового дисплея. Наивные библиотеки scFv человека получали путем реконструкции на основе ПЦР из В-клеток от 50 здоровых доноров. Твердофазный пэннинг в пробирках для иммуносорбции проводили с применением слитого белка hPDL1-Fc и нерелевантного слитого с Fc белка, непрямо иммобилизованных в пробирке для иммуносорбции, покрытой антителом IgG к Fc человека. Чтобы отсортировать белки с сильным связыванием, сначала истощали Fc-связывающие scFv с использованием нерелевантных слитых с Fc белков, а затем несвязанные фаги отбирали для связывания со слитым белком hPDL1-Fc. Элюированные фаги амплифицировали в бактериях. Эти этапы повторяли 3-4 раза; титры фагов и вариабельность определяли после второго этапа и далее. После того, как была замечена конвергенция в последовательности (этапы 3 и 4), отдельные клоны фага были протестированы на способность связывать hPDL1 в ИФА ELISA.
[00095] Прямое покрытие: Проводили путем прямого нанесения Fc белков на пробирку для иммуносорбции без применения антитела против Fc человека (фиг. 2).
[00096] ИФА ELISA со связыванием фага:
[00097] ИФА ELISA выполняли с применением той же стратегии, что и для пэннинга. Для клонов, полученных при непрямом пэннинге, планшеты сначала покрывали антителом против Fc человека, а затем белком Fc. Для клонов, полученных в результате прямого пэннинга, планшеты непосредственно покрывали белком Fc. В непрямых ИФА ELISA фаги тестировали на способность связывать hPDL1-Fc и нерелевантный белок Fc (или hIgG1) в параллельных анализах. Фаги, которые показали незначительное связывание с нерелевантным белком Fc и высокое связывание с hPDL1, отбирали для дальнейшего секвенирования и вторичного скрининга. Данные представлены на фиг. 3. Неспецифическое связывание большинства клонов незначительно (величина сигнала для белка Fc (разведение 1:10) составляет менее 0,2). В прямых ИФА ELISA фаги тестировали на способность связывать hPDL1-Fc, mPDL1-Fc (PDL1 мыши) и hIgG1 в параллельных анализах. Фаги продемонстрировали отсутствие значимого связывания с PDL1 мыши ни одной из наилучших молекул согласно настоящему изобретению, а именно, ни одна из наилучших молекул не демонстрировала значимой перекрестной реактивности с PDL1 мыши. Данные представлены на фиг. 7.
[00098] Секвенирование:
[00099] Уникальные клоны идентифицировали, секвенируя сначала область CDR3 тяжелой цепи. Позже эти результаты подтверждали при секвенировании полной последовательности. Небольшая подгруппа клонов содержала одинаковые CDR3, но имела значимые расхождения в других частях их последовательностей.
[000100] Вторичный скрининг с помощью FACS:
[000101] Фаги, лизаты фагов или лизаты бактерий, экспрессирующих scFv, тестировали на способность преимущественно связываться с клетками 293T, которые экспрессируют hPDL1, но не с исходными клетками 293T. Отношение средней интенсивности флуоресценции (MFI) использовали в качестве основы для идентификации положительных клонов. Данные представлены на фиг. 4. Большинство клонов показало высокое отношение, что можно идентифицировать как положительные клоны.
[000102] Идентификация блокаторов:
[000103] Фаги, лизаты фагов или лизаты бактерий, экспрессирующих scFv, тестировали на способность блокировать взаимодействие между hPD1 и hPDL1. Анализы связывания проводили путем нанесения на планшеты hPD1-Fc или hPDL1-Fc. Связывание биотинилированного лиганда (hPDL1 или hPD1) детектировали с применением стрептавидина-HRP стандартными методами. Потерю связывания в присутствии scFv использовали для идентификации потенциальных блокаторов. Результаты представлены на фиг. 5 и 6.
[000104] Получение и характеризация слитых с Fc белков:
[000105] Поскольку scFv относительно нестабильны, некоторые scFv преобразовали в слитые Fc и экспрессировали в клетках млекопитающих. Их очищали на колонках с белком A и тестировали на способность блокировать взаимодействие PD1-PDL1, а также на способность связывать клетки 293T, экспрессирующие PDL1.
[000106] Получение полноразмерных антител:
[000107] Гены полноразмерного антитела конструировали с помощью ПЦР-амплификации областей VH и VL из индивидуальных клонов scFv и клонировали в соответствующие экспрессионные векторы с применением стандартных методов, известных специалистам в данной области техники. Белки полноразмерных антител получали путем временной трансфекции клеток 293T, выращенных в суспензии, и очищали на колонке с белком A стандартными методами, известными специалистам в данной области техники.
[000108] Характеризация полноразмерных антител:
[000109] Примеры полноразмерных антител были охарактеризованы с помощью ДСН-ПААГ и эксклюзионной хроматографии (результаты представлены на фиг. 8A, 8B, 9A, 9B, 10A и 10B), а также количественной оценки их активности при (a) специфическом связывании hPDL1 в ИФА ELISA (результаты показаны на фиг. 11B и 11C); (b) специфическом связывании экспрессирующих hPDL1 клеток 293T и неочищенных клеток 293T (результаты показаны на фиг. 12A и 12B); и (в) блокировании взаимодействия PD1-PDL1 в обеих версиях анализа блокирования. Полученные данные для примеров наилучших кандидатных антител в Формате 1 и Формате 2 показаны на фиг. 13B и 14B. Данные результатов для 27 вариантов осуществления антител по настоящему изобретению показаны на фиг. 15.
[000110] Аффинность взаимодействия PD-L1 в анализе BIAcore:
[000111] Наилучшие кандидатные антитела тестировали на аффинность к PD-L1, используя BIAcore (фиг. 16B-16D). Вкратце, биотинилированный hPDL1 захватывали за счет стрептавидина на поверхности сенсорного чипа. Антитело прогоняли по чипу, и рассчитывали параметры реакции с использованием одноциклового кинетического метода, основанного на стабильности взаимодействия. Значения KD оценивали с применением программного обеспечения для оценки BIAcore X100 2.0 в модели связывания бивалентного аналита.
[000112] Связывание PD-L1 резуса при FACS
[000113] (A) Клетки 293T временно трансфицировали конструкцией для экспрессии PDL1-GFP резуса. Варианты осуществления 4-1E8, 3-1E4 и 3-1B11 тестировали и сравнивали с контролем. Результаты показаны на фиг. 18A-18D: все три антитела связывали PDL1 резуса.
[000114] (b) Клетки 293T временно трансфицировали конструкцией для экспрессии PDL1 резуса экспрессирующей конструкцией PDL1. Варианты осуществления 4-1E8, 3-1E4 и 3-1B11 тестировали и сравнивали с контролем. Результаты показаны на фиг. 19A-19D: все три антитела связывали PDL1 резуса.
[000115] Индукция ИЛ-2 и определение EC50
[000116] Мононуклеарные клетки периферической крови (МКПК) выделяли из крови человека в градиенте фиколла с последующим лизисом эритроцитов, используя стандартные протоколы. Для анализа готовили среду RPMI+ следующим образом: 10% ФСБ, 1% антибиотик-антимикотик (Gibco) и 1% заменимых аминокислот (Gibco) добавляли в среду RPMI в модификации ATCC (Gibco). После выделения из крови МКПК ресуспендировали в 10-20 мл RPMI+ и культивировали в течение ночи при 37ºC с 5% CO2. Затем МКПК высевали в 96-луночный планшет для тканевых культур (Corning) в концентрации 100 000 МКПК/96 лунок; конечный объем в каждой лунке составлял 200 мкл. Стафилококковый энтеротоксин B (SEB) добавляли в концентрации 1 нг/мл, а наилучшие антитела добавляли в концентрации 20 мкг/мл (для скрининга) или в диапазоне концентраций от 50 мкг/мл до 0,003 мкг/мл. В качестве контроля использовали клетки без SEB (например, без стимуляции); с SEB по отдельности или с SEB и изотипическим контролем (например, исходный уровень).
[000117] После 76-часовой инкубации при 37°C с 5% CO2 МКПК центрифугировали при 1200 об/мин в течение 15 минут при комнатной температуре, супернатанты собирали и хранили при -20°C. ИФА ELISA на ИЛ-2 проводили с использованием коммерческого набора для ИФА ELISA на ИЛ-2 (Biolegend или Thermofisher), следуя инструкциям производителя. Для ИФА ELISA супернатанты разбавляли до 1/20 - 1/80. Поглощение измеряли с применением считывающего устройства для микропланшетов Spectramax3 M3 (Molecular Devices), и анализировали данные с использованием программного обеспечения Graphpad. Наилучшие кандидатные антитела сравнивали с коммерчески доступными антителами против PD1. Результаты показаны на фиг. 20. В экспериментах по совместному культивированию опухолей с клетками MDA-MB-231 (см. фиг. 23A-23C) 4-1E8 стабильно демонстрировал лучшие результаты, чем 3-1B11 и 3-1E4, в отношении инактивации ИЛ-2 (см. фиг. 23A-23C) и ИФН-γ (см. фиг. 24). Однако все три антитела были настолько же или более эффективны, чем коммерческие антитела против PDL1 - атезолизумаб («Atezo») и дурвалумаб («Durva»), в аналогичных экспериментах по совместному культивированию с Т-клетками и MDA-MB-231.
[000118] АЗКЦ-активность
[000119] Как показано на фиг. 21 и 22, все три наилучших антитела продемонстрировали выраженную АЗКЦ-активность, в то время как атезолизумаб (сконструированный так, чтобы быть отрицательным по АЗКЦ) продемонстрировал отсутствие активности. Из трех вариантов осуществления изобретения 4-1E8 продемонстрировал наибольшую АЗКЦ-активность.
[000120] Реакция смешанной культуры лимфоцитов
[000121] Мононуклеарные клетки периферической крови (МКПК) выделяли из крови человека в градиенте фиколла с последующим лизисом эритроцитов, используя стандартные протоколы. Клетки культивировали в бессывороточной среде RPMI 1640 в течение 1 часа при 37°C. Неадгезивные клетки удаляли и оставшиеся моноциты культивировали в RPMI 1640 с добавлением 5% AB сыворотки человека, 2 нг/мл ГМ-КСФ и 10 нг/мл ИЛ-4 (BD Biosciences). Свежую среду с добавлением цитокинов добавляли каждые 2-3 суток. Зрелые дендритные клетки индуцировали добавлением 20 нг/мл ФНО-альфа (BD Biosciences) на 6 день и культивировали в течение 24 часов.
[000122] Дендритные клетки собирали, определяли фенотип и замораживали для последующего применения. CD4 Т-клетки выделяли из МКПК с применением магнитных микроносителей (Dynal) в соответствии с инструкциями производителя. CD4 Т-клетки культивировали в 96-луночных планшетах с плоским дном (Costar) вместе с аллогенными дендритными клетками в соотношении 1:2,5, используя RPMI 1640 с добавлением 10% AB сыворотки человека. Перед добавлением дендритные клетки обрабатывали 100 мг/мл митомицина C (Sigma). Пролиферацию Т-клеток измеряли методом разведений CFSE (или аналогичного красителя). Высвобождение ИФН-g измеряли с использованием коммерческого набора для ИФА ELISA на ИФН-g, следуя инструкциям производителя. Поглощение измеряли с применением считывающего устройства для микропланшетов Spectramax3 M3 (Molecular Devices) и анализировали данные с использованием программного обеспечения Graphpad. В указанных исследованиях наилучшие кандидатные антитела согласно вариантам осуществления настоящего изобретения демонстрировали результаты, сопоставимые с другими коммерчески доступными антителами против PD1 и против PDL1. Примеры данных представлены на фиг. 25A и 25B.
[000123] Специфичность связывания
[000124] Получали линии клеток Expi293, которые стабильно экспрессировали множество членов семейства B7 и их рецепторов. Потенциал антител против PDL1 тестировали с помощью FACS с использованием флуоресцентных антител против IgG человека. Полученные данные для примеров наилучших кандидатных антител приведены на фиг. 26A и 26B.
[000125] Блокирование связывания CD80-PDL1
[000126] Клетки DLD1, сконструированные так, чтобы экспрессировать PDL1, применяли для детекции связывания биотинилированного CD80-Fc в присутствии или в отсутствие антител против PDL1 с последующей обработкой флуоресцентным стрептавидином. Полученные данные для примеров наилучших кандидатных антител по настоящему изобретению приведены на фиг. 27A и 27B.
[000127] Измерение времени полужизни
[000128] Время полужизни сыворотки измеряли с применением гомозиготных самцов мышей Tg32 (B6.Cg-Fcgrttm1Dcr Tg(FCGRT)32Dcr/DcrJ, Jackson Labs). 2 мг/кг антитела инъецировали внутривенно на 0 день; кровь брали на 1 день и в различные последующие моменты времени. Готовили плазму и измеряли титры антител с применением сэндвич-анализа ELISA. Титры нормировали по титрам 1 дня. Также измеряли ответ против антител, и образцы с высокими титрами исключали из анализа, поскольку в ИФА ELISA они часто демонстрировали неожиданные изменения. Полученные данные для примеров наилучших кандидатных антител по настоящему изобретению приведены на фиг. 28. Время полужизни разных антител варьировало от 6,9 дней (3-1E4, см. подробное описание последовательности в примере 9 ниже) до 10,5 дней (3-1B11, см. подробное описание последовательности в примере 11 ниже) и 12,3 дней (4-1E8, см. подробное описание последовательности в примере 5 ниже).
[000129] Последовательности полипептидов
[000130] Примеры последовательностей PD-L1-связывающих полипептидов по настоящему изобретению приведены ниже:
Пример 1: Код антитела: 4-1A2
VH
[000131] ДНК (SEQ ID NO:1)
CAGGTTCAGCTGGTGCAGTCTGGGACTGAGGTGAAGAAGCCTGGGGCCTCAGTGAAGGTC
TCCTGCAAGGCTTCTGGATACACCTTCACCAGTTATGATATCAACTGGGTGCGACAGGCC
ACTGGACAAGGGCTTGAGTGGATGGGATGGATCAACCCTAACAGTGGTGGCACAAACTAT
GCACAGAAGTTTCAGGGCAGGGTCACCATGACCACAGACACTTCTACGGGCACAGCCTAC
ATGGAGCTGAGGAGCCTGAGATCTGACGACACGGCCGTGTATTACTGTGCGAGATTTTTA
TGGGGTTCGGGGAGTTATGACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000132] АМИНОКИСЛОТА (SEQ ID NO:2)
QVQLVQSGTEVKKPGASVKVSCKASGYTFTSYDINWVRQATGQGLEWMGWINPNSGGTNY
AQKFQGRVTMTTDTSTGTAYMELRSLRSDDTAVYYCARFLWGSGSYDYWGQGTLVTVSS
VL
[000133] ДНК (SEQ ID NO:3)
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACC
ATCACTTGCCGGGCAAGTCAGAGCATTAGCAGCTATTTAAATTGGTATCAGCAGAAACCA
GGGAAAGCCCCTAAGCTCCTGATCTATGCTGCATCCAGTTTGCAAAGTGGGGTCCCATCA
AGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGTCTGCAACCT
GAAGATTTCGCAACTTACTACTGTCAACAGACTTACACATTCCCGCACACTTTTGCCCAG
GGGACCAACCTGGAGATCAAA
[000134] АМИНОКИСЛОТА (SEQ ID NO:4)
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQTYTFPHTFAQGTNLEIK
Пример 2: Код антитела: 4-1A12
VH
[000135] ДНК (SEQ ID NO:5)
CAAGTCCAGCTGGTACAATCTGGAGCTGAGGTGAAGAAGCCTGGGGCCTCAGTGAAGGTC
TCCTGCAAGGCTTCTGGTTACACCTTTACCAGCTATGGTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGATGGATCAGCGCTTACAATGGTAACACAAACTAT
GCACAGAAGCTCCAGGGCAGAGTCACCATGACCACAGACACATCCACGAGCACAGCCTAC
ATGGAGCTGAGGAGCCTGAGATCTGACGACACGGCCGTGTATTACTGTGCGAGAGATTGG
ATACAGCTATGGTTACCCCTTGACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000136] АМИНОКИСЛОТА (SEQ ID NO:6)
QVQLVQSGAEVKKPGASVKVSCKASGYTFTSYGISWVRQAPGQGLEWMGWISAYNGNTNY
AQKLQGRVTMTTDTSTSTAYMELRSLRSDDTAVYYCARDWIQLWLPLDYWGQGTLVTVSS
VL
[000137] ДНК (SEQ ID NO:7)
GACATCCAGTTGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACC
ATCACTTGCCGGGCAAGTCAGAGCATTAGCAGCTATTTAAATTGGTATCAACAGAAACCA
GGGAAAGCCCCTAAGCTCCTGATCTATGGTGCATCCAGTTTGGAAAGTGGGGTCCCATCA
AGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGTCTGCAACCT
GAAGATTTTGCAACTTACTACTGTCAACAGAGTCACAGTTCCCCCCTCACTTTCGGCGGA
GGGACCAAGGTGGACATCAAA
[000138] АМИНОКИСЛОТА (SEQ ID NO:8)
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYGASSLESGVPS
RFSGSGSGTDFTLTISSLQPEDFATYYCQQSHSSPLTFGGGTKVDIK
Пример 3: Код антитела: 4-1B9
VH
[00139] ДНК (SEQ ID NO:9)
GAAGTGCAGCTGGTGCAGTCTGGGGGAGGCTTGGTCCAGCCTGGGAGGTCCCTGAGACTC
TCCTGTGCAGCCTCTGGATTCACCTTCAGTAGCTATGGCATGCACTGGGTCCGCCAGGCT
CCAGGCAAGGGGCTGGAGTGGGTGGCAGTTATATCATATGATGGAAGTAATAAATACTAT
GCAGACTCCGTGAAGGGCCGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTAT
CTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCGAAAGATTTG
ATCCCGTTGCGAGATAGTAGGGGGGGGTACTACTACGGTATGGACGTCTGGGGCCAAGGG
ACCACGGTCACCGTCTCCTCAGGGAGT
[000140] АМИНОКИСЛОТА (SEQ ID NO:10)
EVQLVQSGGGLVQPGRSLRLSCAASGFTFSSYGMHWVRQAPGKGLEWVAVISYDGSNKYY
ADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCAKDLIPLRDSRGGYYYGMDVWGQG
TTVTVSS
VL
[000141] ДНК (SEQ ID NO:11)
TCTTCTGAGCTGACTCAGGACCCTGCTGTGTCTGTGGCCTTGGGACAGACAGTCAGGATC
ACATGCCAAGGAGACAGCCTCAGAGACTATTATGCAAGCTGGTACCAGCAGAAGCCAGGA
CAGGCCCCTGTACTTGTCATCTATGGTAAAAACAACCGGCCCTCAGGAATCCCAGACCGA
TTCTCTGGCTCCAGCTCAGGAAACACAGCTTCCTTGACCATCACTGGGACTCAGGCGGAA
GATGAGGCTGACTATTACTGTAACTCCCGTGACAGCGGTGCTTACCATTATGTCTTCGGA
ACTGGGACCAAGGTCACCGTCCTA
[000142] АМИНОКИСЛОТА
SSELTQDPAVSVALGQTVRITCQGDSLRDYYASWYQQKPGQAPVLVIYGKNNRPSGIPDR
FSGSSSGNTASLTITGTQAEDEADYYCNSRDSGAYHYVFGTGTKVTVL
Пример 4: Код антитела: 4-1B12
VH
[000143] ДНК (SEQ ID NO:13)
CAAATCCAGCTGGTGCAGTCTGGGGGAGGCGTGGTCCAGCCTGGGAGGTCCCTGAGACTC
TCCTGTGCAGCCTCTGGATTCACCTTCAGTAGCTATGGCATGCACTGGGTCCGCCAGGCT
CCAGGCAAGGGGCTGGAGTGGGTGGCAGTTATATCATATGATGGAAGTAATAAATACTAT
GCAGACTCCGTGAAGGGCCGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTAT
CTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCGAAAGGAAGT
ATTATAGGGGATGGTGCTTTTGATATCTGGGGCCAAGGGACAATGGTCACCGTCTCTTCA
[000144] АМИНОКИСЛОТА (SEQ ID NO:14)
QIQLVQSGGGVVQPGRSLRLSCAASGFTFSSYGMHWVRQAPGKGLEWVAVISYDGSNKYY
ADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCAKGSIIGDGAFDIWGQGTMVTVSS
VL
[000145] ДНК (SEQ ID NO:15)
GATATTGTGATGACCCAGTCTCCACTCTCCCTGCCCGTCACCCTTGGAGAGCCGGCCTCC
ATCTCCTGCAGGTCTAGTCAGACCCTCCTGCATAATGGATTCAACTTTTTGGATTGGTAC
CTGCAGAAGCCAGGGCAGTCTCCACAACTCCTGATGTATTTGGCCTCTAGCCGGGCCTCC
GGGGTCCCTGACAGGTTCAGTGGCAGTGGATCGGGCACAGATTTCACACTGAAAATCAGC
AGAGTGGAGGCTGAGGATGTTGGGGTTTATTACTGCATGCAAGGTACACACTGGCCGTAC
ACTTTTGGCCAGGGGACCAAGCTGGATATCAAA
[000146] АМИНОКИСЛОТА (SEQ ID NO:16)
DIVMTQSPLSLPVTLGEPASISCRSSQTLLHNGFNFLDWYLQKPGQSPQLLMYLASSRAS
GVPDRFSGSGSGTDFTLKISRVEAEDVGVYYCMQGTHWPYTFGQGTKLDIK
Пример 5: Код антитела: 4-1E8
VH
[000147] ДНК (SEQ ID NO:17)
CAAATCCAGCTGGTACAATCTGGGGCTGAGGTGAAGATGCCTGGGGCCTCAGTGACGATT
TCCTGCGAGGCGTCTGGATACAACTTCATCAGCTACTATATACACTGGGTGCGACAGGCC
CCTGGACAAGGCCTTGAGTGGATGGGATTCGTCGTCCCTAGTGGTGGTGCCGCAGGCTAC
ACACAGAAGTTCCAGGGCAGACTCACCGTGACCAGGGACACGTCCACGAGCACAGTCTAC
ATGGACCTGAACAGCCTGACATCTGACGACACGGCCGTGTATTACTGTGTGCGAGAAATG
AGTGGTGGCTGGTTTGATTTCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCG
[000148] АМИНОКИСЛОТА (SEQ ID NO:18)
QIQLVQSGAEVKMPGASVTISCEASGYNFISYYIHWVRQAPGQGLEWMGFVVPSGGAAGY
TQKFQGRLTVTRDTSTSTVYMDLNSLTSDDTAVYYCVREMSGGWFDFWGQGTLVTVSS
VL
[000149] ДНК (SEQ ID NO:19)
GACATCGTGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACC
ATCACTTGCCGGGCAAGTCAGGGCATTAGAAATGATTTAGGCTGGTATCAGCAAAAACCA
GGGAAAGCCCCTAAGCTCCTGATCTATGCTGCATCCACTTTGCAAAGTGGGGTCCCATCA
AGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGGCT
GAAGATGTGGCAGTTTATTACTGTCAGCAATATTATAGTACTCCTCTCACTTTCGGCCCT
GGGACCAAAGTGGATATCAAA
[000150] АМИНОКИСЛОТА (SEQ ID NO:20)
DIVMTQSPSSLSASVGDRVTITCRASQGIRNDLGWYQQKPGKAPKLLIYAASTLQSGVPS
RFSGSGSGTDFTLTISSLQAEDVAVYYCQQYYSTPLTFGPGTKVDIK
Пример 6: Код антитела: 4-1G7
VH
[000151] ДНК (SEQ ID NO:21)
GAGGTGCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGTTACACCTTTACCAGCTATGGTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGATGGATCAGCGCTTACAATGGTAACACAAACTAT
GCACAGAAGCTCCAGGGCAGAGTCACCATGACCACAGACACATCCACGAGCACAGCCTAC
ATGGAGCTGAGGAGCCTGAGATCTGACGACACGGCCGTGTATTACTGTGCGAGAGCCTCA
CCGGTACAGCAGCCCATATGGTGGGCGGAGTACTGGGGCCAGGGAACCCTGGTCACCGTC
TCCTCA
[000152] АМИНОКИСЛОТА (SEQ ID NO:22)
EVQLVQSGAEVKKPGSSVKVSCKASGYTFTSYGISWVRQAPGQGLEWMGWISAYNGNTNY
AQKLQGRVTMTTDTSTSTAYMELRSLRSDDTAVYYCARASPVQQPIWWAEYWGQGTLVTV
SS
VL
[000153] ДНК (SEQ ID NO:23)
CAGTCTGCCCTGACTCAGCCTGCCTCCGTGTCTGGGTCTCCTGGACAGTCGATCACCATC
TCCTGCACTGGAACCAGCAGTGACGTTGGTGGTTATAACTATGTCTCCTGGTACCAACAG
CACCCAGGCAAAGCCCCCAAACTCATGATTTCTGATGTCAGTAAGCGGCCCTCAGGGGTT
TCTAATCGCTTCTCTGGCTCCAAGTCTGGCAACACGGCCTCCCTGACCATCTCTGGGCTC
CAGGCTGAGGACGAGGCTGATTATTACTGCAGCTCATATACAAGCAACTACACTTTGGTA
TTCGGCGGAGGGACCAAGCTGACCGTCCTA
[000154] АМИНОКИСЛОТА (SEQ ID NO:24)
QSALTQPASVSGSPGQSITISCTGTSSDVGGYNYVSWYQQHPGKAPKLMISDVSKRPSGV
SNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSNYTLVFGGGTKLTVL
Пример 7: Код антитела: 4-1H10
VH
[000155] ДНК (SEQ ID NO:25)
CAGCTGCAGCTACAGCAGTCCGGAGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTCCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAAGGATCATCCCTATCCTTGGTATAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACAAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAAGACACGGCCGTGTATTACTGTGCGAGTCATGGT
CGGGCAGCAGCTGGTAGGTACGCTATGGACGTCTGGGGCCAAGGGACCACGGTCACCGTC
TCCTCA
[000156] АМИНОКИСЛОТА (SEQ ID NO:26)
QLQLQQSGAEVKKPGSSVKVSCKAPGGTFSSYAISWVRQAPGQGLEWMGRIIPILGIANY
AQKFQGRVTITADKSTSTAYMELSSLRSEDTAVYYCASHGRAAAGRYAMDVWGQGTTVTV
SS
VL
[000157] ДНК (SEQ ID NO:27)
AATTTTATGCTGACTCAGCCCCACTCTGTGTCGGATTCTCCGGGGAAGACGGTAACCATC
TCCTGCACCCGCAGCAGTGGCAGCATTGCCAGCAACTATGTGCAGTGGTACCAGCAGCGC
CCGGGCAGTGCCCCCACCACTGTGATCTATGACGATAAGCAAAGACCCTCTGGGGTCCCT
GATCGGTTCTCGGGCTCCATCGACAGCTCCTCCAACTCTGCCTCCCTCACCATCTCTGGA
CTGACGACTGAGGACGAGGCTGACTACTACTGTCAGTCCTTTGATGGCAGCAGTGTCATC
TTCGGCGGAGGGACCAAGCTGACCGTCCTG
[000158] АМИНОКИСЛОТА (SEQ ID NO:28)
NFMLTQPHSVSDSPGKTVTISCTRSSGSIASNYVQWYQQRPGSAPTTVIYDDKQRPSGVP
DRFSGSIDSSSNSASLTISGLTTEDEADYYCQSFDGSSVIFGGGTKLTVL
Пример 8: Код антитела: 3-1H2
VH
[000159] ДНК (SEQ ID NO:29)
CAGGTTCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAAAGGAG
CGTTTCTATGATAGTAGTGGTTATTACGATGCTTTTGATATCTGGGGCCAAGGGACAATG
GTCACCGTCTCTTCA
[000160] АМИНОКИСЛОТА (SEQ ID NO:30)
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARKERFYDSSGYYDAFDIWGQGTM
VTVSS
VL
[000161] ДНК (SEQ ID NO:31)
CAGTCTGCCCTGACTCAGCCTCGCTCAGTGTCCGGGTCTCCTGGGCAGTCAGTCACCATC
TCCTGCACTGGAACCAGCAATGATGTTGGTGGTTATAACTATGTCTCCTGGTACCAACAG
CACCCAGGCAAAGCCCCCAAACTCATGATTTATGATGTCACTAAGCGGCCCTCAGGGGTC
CCTGATCGCTTCTCTGGCTCCAAGTCTGGCAACACGGCCTCCCTGACCGTCTCTGGCCTC
CAGCCTGAGGATGAGGCTGACTATTATTGCGCCTCTTATGGAGGCAGGAACAATTTGCTT
TTTGGCGGAGGGACTCAACTGACCGTCTTA
[000162] АМИНОКИСЛОТА (SEQ ID NO:32)
QSALTQPRSVSGSPGQSVTISCTGTSNDVGGYNYVSWYQQHPGKAPKLMIYDVTKRPSGV
PDRFSGSKSGNTASLTVSGLQPEDEADYYCASYGGRNNLLFGGGTQLTVL
Пример 9: Код антитела: 3-1E4
VH
[000163] ДНК (SEQ ID NO:33)
CAAATCCAGCTGGTACAATCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACAAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCCGGAGGGGGA
GCAGTGGCGGACAATAGTTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000164] АМИНОКИСЛОТА (SEQ ID NO:34)
QIQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADKSTSTAYMELSSLRSEDTAVYYCAGGGAVADNSYWGQGTLVTVSS
VL
[000165] ДНК (SEQ ID NO:35)
GACATCCGGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACC
ATCACTTGCCGGGCAAGTCAGGGCATTAGAAATGATTTAGGCTGGTATCAGCAGAAACCA
GGGAAAGCCCCTAAGCTCCTGATCTATGCTGCATCCAGTTTACAAAGTGGGGTCCCATCA
AGGTTCAGCGGCAGTGGATCTGGCACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCT
GAAGATTTTGCAACTTATTACTGTCTACAAGATTACAATTACCCTCGAACGTTCGGCCAA
GGGACCAAGGTGGAAATCAAA
[000166] АМИНОКИСЛОТА (SEQ ID NO:36)
DIRMTQSPSSLSASVGDRVTITCRASQGIRNDLGWYQQKPGKAPKLLIYAASSLQSGVPS
RFSGSGSGTDFTLTISSLQPEDFATYYCLQDYNYPRTFGQGTKVEIK
Пример 10: Код антитела: 3-1A8
VH
[000167] ДНК (SEQ ID NO:37)
CAAATCCAGCTGGTACAATCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACGGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGACGGT
TCGTATAGCAGCAGCTGGTACTCGTTTGACTACTGGGGCCAGGGAACCCTGGTCACCGTC
TCCTCA
[000168] АМИНОКИСЛОТА (SEQ ID NO:38)
QIQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARDGSYSSSWYSFDYWGQGTLVTV
SS
VL
[000169] ДНК (SEQ ID NO:39)
CAGTCTGCCCTGACTCAGCCTGCCTCCGTGTCTGGGTCTCCTGGACAGTCGATCACCATC
TCCTGCACTGGAACCAGCAGTGACGTCGGTGGTTATAACTATGTCTCCTGGTACCAACAG
CACCCAGGCAAAGCCCCCAAACTCATGATTTATGATGTCAGTAATCGGCCCTCAGGGGTT
TCTAATCGCTTCTCTGGCTCCAAGTCTGGCAACACGGCCTCCCTGACCATCTCTGGGCTC
CAGGCTGAGGACGAGGCTGATTATTACTGCTCCTCATATGCAGGTGATATTAGTTATGTA
CTGTTCGGCGGCGGGACCAAGCTGACCGTCCTA
[000170] АМИНОКИСЛОТА (SEQ ID NO:40)
QSALTQPASVSGSPGQSITISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGV
SNRFSGSKSGNTASLTISGLQAEDEADYYCSSYAGDISYVLFGGGTKLTVL
Пример 11: Код антитела: 3-1B11
VH
[000171] ДНК (SEQ ID NO:41)
GAAGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGAGGGTCCCTGAGACTC
TCCTGTGCAGCCTCTGGATTCACTTTTAGTGACTATGACATGATCTGGGTCCGCCAGGCT
CCAGGCAAGGGGCTGGAGTGGGTGGCAGTTATATCATATGATGGAAGTAATAAATACTAT
GCAGACTCCGTGAAGGGCCGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTAT
CTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCGAAAGAGTTC
TTTGGTGCTTTTGATATCTGGGGCCAAGGGACAATGGTCACCGTCTCTTCA
[000172] АМИНОКИСЛОТА (SEQ ID NO:42)
EVQLVESGGGLVQPGGSLRLSCAASGFTFSDYDMIWVRQAPGKGLEWVAVISYDGSNKYY
ADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCAKEFFGAFDIWGQGTMVTVSS
VL
[000173] ДНК (SEQ ID NO:43)
TCTTCTGAGCTGACTCAGGACCCTGCTGTGTCGGTGGCCTTGGGACAGACAGTCACGATC
ACATGCCAAGGAGACAGCCTCAATTACTATTATGCAAACTGGTTCCAGCTGAAGCCAGGG
CAGGCCCCTGTACTTGTCCTCTTTGGTAAAAACAACCGGCCCTCAGGGATCCCAGACCGA
TTCTCTGGCTCCTACTCGGGAAGCACAGCTTCCTTGACCATCACTGGGGCTCAGGCGGAA
GATGACGCTGACTATTACTGTAATTCGCGGGACAGCGGTGGTAATCCTTGGGTGTTCGGC
GGAGGGACCAAGCTGACCGTCCTA
[000174] АМИНОКИСЛОТА (SEQ ID NO:44)
SSELTQDPAVSVALGQTVTITCQGDSLNYYYANWFQLKPGQAPVLVLFGKNNRPSGIPDR
FSGSYSGSTASLTITGAQAEDDADYYCNSRDSGGNPWVFGGGTKLTVL
Пример 12: Код антитела: 4-1F3
VH
[000175] ДНК (SEQ ID NO:45)
CAAATCCAGCTGGTACAATCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAAGGATCATCCCTATCCTTGGTATAGCAGACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACAAATCCACGAGCACAGCCTAC
ATGGAACTGAGTAGCCTGGGATCTGAGGACACGGCCGTGTATTTTTGTGCGAGAGAGGGG
GGATCCTTTAGGCACTTTGACTTCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000176] АМИНОКИСЛОТА (SEQ ID NO:46)
QIQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGRIIPILGIADY
AQKFQGRVTITADKSTSTAYMELSSLGSEDTAVYFCAREGGSFRHFDFWGQGTLVTVSS
VL
[000177] ДНК (SEQ ID NO:47)
CAGCCTGTGCTGACTCAGCCACCCTCAGTCTCTGGGGCCCCAGGGCAGAGGGTCACCATC
TCCTGCGCTGGGAGCGACCCCAACATCGGGACAGGTCATGATGTGCACTGGTACCAGCAA
CTTCCAGGAACAGCCCCCAAACTCGTCATCTATGGTAACACCAATCGGCCCTCAGGGGTC
CCTGAGCGATTCACTGCCTCCAAGTCTGGCACCTCAGCCTCCCTGGCCATCACTGGGCTC
CAGGCTGAGGATGAGGCTGATTATTACTGCCAGGCCTACGACAGGAGCCTGCGTGGTTAT
GTCTTCGGGACTGGGACCAAGGTCACCGTCCTG
[000178] АМИНОКИСЛОТА (SEQ ID NO:48)
QPVLTQPPSVSGAPGQRVTISCAGSDPNIGTGHDVHWYQQLPGTAPKLVIYGNTNRPSGV
PERFTASKSGTSASLAITGLQAEDEADYYCQAYDRSLRGYVFGTGTKVTVL
Пример 13: Код антитела: 4-1G5
VH
[000179] ДНК (SEQ ID NO:49)
CAAATCCAGCTGGTACAGTCTGGTGCTGAAGTGAAGAAGCCTGGGGCCTCAGTGAAGGTC
TCCTGCAAGACTTCTGGTTACACCTTTACCAGCTATGGTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGATGGATCAGCGCTTACAATGGTAACACAAACTAT
GCACAGAAGCTCCAGGGCAGAGTCACCATGACCACAGACACATCCACGAGCACAGCCTAC
ATGGAGCTGAGGAGCCTGAGATCTGACGACACGGCCGTGTATTACTGTGCGAGAACTACA
GGTGACGAGTGGCTACGATTGGCTATAAATGACTACTGGGGCCAGGGAACCCTGGTCACC
GTCTCCTCA
[000180] АМИНОКИСЛОТА (SEQ ID NO:50)
QIQLVQSGAEVKKPGASVKVSCKTSGYTFTSYGISWVRQAPGQGLEWMGWISAYNGNTNY
AQKLQGRVTMTTDTSTSTAYMELRSLRSDDTAVYYCARTTGDEWLRLAINDYWGQGTLVT
VSS
VL
[000181] ДНК (SEQ ID NO:51)
GATATTGTGATGACACAGTCTCCCCTCTCCCTGCCCGTCACCCCTGGAGAGCCGGCCTCC
ATCTCCTGCAGGTCTAGTCTGCGCCTCATGCATCCTAATGGACTCAACTATTTGGATTGG
TACCTGCAGAAGCCAGGGCAGTCTCCACAGCTCCTAATCTTTTTGGGTTCTCAGCGGGCC
TCCGGGGTCCCTGACAGGTTCAGTGGCAGTGGATCAGGCACAGATTTTACACTGAAAATC
AGCAGAGTGGAGGCTGAGGATGTTGGCATTTATTACTGCATGCAAGCTCTAGAACCTCCG
TACACTTTTGGCCAGGGGACCAAGCTGGAGATCAAA
[000182] АМИНОКИСЛОТА (SEQ ID NO:52)
DIVMTQSPLSLPVTPGEPASISCRSSLRLMHPNGLNYLDWYLQKPGQSPQLLIFLGSQRA
SGVPDRFSGSGSGTDFTLKISRVEAEDVGIYYCMQALEPPYTFGQGTKLEIK
Пример 14: Код антитела: 4-1C9
VH
[000183] ДНК (SEQ ID NO:53)
CAGGTCCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGATCCC
GGGTATAGCAGTGGCTGGAAAGATGATGCTTTTGATATCTGGGGCCAAGGGACAATGGTC
ACCGTCTCTTCA
[000184] АМИНОКИСЛОТНАЯ (SEQ ID NO:54)
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARDPGYSSGWKDDAFDIWGQGTMV
TVSS
VL
[000185] ДНК (SEQ ID NO:55)
GAAATTGTGATGACACAGTCTCCAGGCACCCTGTCTTTGTCTCCAGGGGATACAGCCTCC
CTCTCCTGCAGGGCCAGTCAGACTGTTAGCAGCAACTACTTAGCCTGGTACCAACAGAAA
CCTGGCCAGGCTCCCAGGCTCCTCATCTATGATACATCCAACAGGGCCGCTGGCATCCCG
GCCAGGTTCAGTGGCAGTGGGTCTGGGACAGACTTCACTCTCACCATCAGTAGCCTAGAG
CCTGAAGATTTTGCAGTGTATTACTGTCAGCAGTACGGTAGCTCACTCTGGACGTTCGGC
CAAGGGACCAAGGTGGAAATCAAA
[000186] АМИНОКИСЛОТНАЯ (SEQ ID NO:56)
EIVMTQSPGTLSLSPGDTASLSCRASQTVSSNYLAWYQQKPGQAPRLLIYDTSNRAAGIP
ARFSGSGSGTDFTLTISSLEPEDFAVYYCQQYGSSLWTFGQGTKVEIK
Пример 15: Код антитела 11-A4
VH
[000187] ДНК (SEQ ID NO:57)
CAGGTGCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGCGGGG
CAGCAGCTGGTAGCCCTTTGGTACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000188] АМИНОКИСЛОТНАЯ (SEQ ID NO:58)
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARAGQQLVALWYYWGQGTLVTVSS
VL
[000189] ДНК (SEQ ID NO:59)
CAGTCTGCCCTGACTCAGCCTCCCTCCGCGTCCGGGTCTCGTGGACAGTCAGTCTCCATC
TCCTGCAGTGGAAGTCGCAGTGACATTGGATATTATAACTATGTCTCCTGGTATCAACAA
CACCCAGGCAAAGCCCCCAAACTCATCATTTTTGACGTCAATAAGCGGCCCTCAGGGGTC
CCTGATCGCTTCTCTGGCTCCAAGTCTGGCAACACGGCCTCCCTGACCGTCTCTGGCCTC
CAGCCTGAGGATGAGGCTGACTATTATTGCGCCTCTTATGGAGGCAGGAACAATTTGCTT
TTTGGCGGAGGGACTCAACTGACCGTCTTA
[000190] АМИНОКИСЛОТНАЯ (SEQ ID NO:60)
QSALTQPPSASGSRGQSVSISCSGSRSDIGYYNYVSWYQQHPGKAPKLIIFDVNKRPSGV
PDRFSGSKSGNTASLTVSGLQPEDEADYYCASYGGRNNLLFGGGTQLTVL
Пример 16: Код антитела: 21-A1
VH
[000191] ДНК (SEQ ID NO:61)
CAGGTGCAACTGCAGGAGTCGGGCCCAGGACTGGTGGAGCCTTCGGAGACCCTGTCCCTC
ACCTGCACTGTCTCTGGTGGCTCCATCAGTAGTTTCTACTGGAGCTGGATCCGGCAGCCC
CCAGGGAAGGGACTGGAGTGGATTGGCTATATCAATTACAGTGGGAGCACCAACTACAAC
CCCTCCCTCAAGAGTCGAGTCACCATATCAGTAGACACGTCCAAGAACCAGTTCTCCCTG
AAGCTGAGCTCTGTGACCGCCGCAGACACGGCTGTGTATTACTGTGCGAGACAGATATTA
TGGTTCGGGGAGTTAAGGTGGTTCGACCCCTGGGGCCAGGGAACCCTGGTCACCGTCTCC
TCA
[000192] АМИНОКИСЛОТНАЯ (SEQ ID NO:62)
QVQLQESGPGLVEPSETLSLTCTVSGGSISSFYWSWIRQPPGKGLEWIGYINYSGSTNYN
PSLKSRVTISVDTSKNQFSLKLSSVTAADTAVYYCARQILWFGELRWFDPWGQGTLVTVS
S
VL
[000193] ДНК (SEQ ID NO:63)
CAGTCTGCCCTGACTCAGCCTCCCTCCGCGTCCGGGTCTCCTGGACAGTCAGTCACCATC
TCCTGCACTGGAACCAGCAGTGACATTGGTGGTTATAACTATGTCTCCTGGTACCAACTG
CGCCCAGGCAAAGCCCCCAAACTCATGATTTATGACGTCACCAAGCGGCCCTCAGGGGTC
CCTGATCGCTTCTCTGGCTCCAAGTCTGGCAACACGGCCTCCCTGACCGTCTCTGGGCTC
CAGGCTGAGGATGAGGCTGATTATTACTGCAGCTCATATGCAGGCAGCAACAATGTGGTA
TTCGGCGGAGGGACCAAGCTGACCGTCCTA
[000194] АМИНОКИСЛОТНАЯ (SEQ ID NO:64)
QSALTQPPSASGSPGQSVTISCTGTSSDIGGYNYVSWYQLRPGKAPKLMIYDVTKRPSGV
PDRFSGSKSGNTASLTVSGLQAEDEADYYCSSYAGSNNVVFGGGTKLTVL
Пример 17: Код антитела: 21-H12
VH
[000195] ДНК (SEQ ID NO:65)
CAAGTCCAGCTGGTACAGTCTGGGGCTGAGGTGAAGAAGCCTGGGGCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAAATCCC
TACGGTTTCAACTGGTTCGACCCCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000196] АМИНОКИСЛОТНАЯ (SEQ ID NO:66)
QVQLVQSGAEVKKPGASVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARNPYGFNWFDPWGQGTLVTVSS
VL
[000197] ДНК (SEQ ID NO:67)
AATTTTATGCTGACTCAGCCCCACTCTGTGTCGGAGTCTCCGGGGAAGACGGTAACCATC
TCCTGCACCCGCAGCAGTGGCAGCATTGCCAGCAACTATGTGCAGTGGTACCAGCAGCGC
CCGGGCAGTGCCCCCACCACTGTGATCTATGAGGATAACCAAAGACCCTCTGGGGTCCCT
GATCGGTTCTCTGGCTCCATCGACAGCTCCTCCAACTCTGCCTCCCTCACCATCTCCGGA
CTGAAGACTGAGGACGAGGCTGACTACTACTGTCAGTCTTATGATGGCTTCAATCAGGTG
TTCGGCGGAGGGACCAAGCTGACCGTCCTA
[000198] АМИНОКИСЛОТНАЯ (SEQ ID NO:68)
NFMLTQPHSVSESPGKTVTISCTRSSGSIASNYVQWYQQRPGSAPTTVIYEDNQRPSGVP
DRFSGSIDSSSNSASLTISGLKTEDEADYYCQSYDGFNQVFGGGTKLTVL
Пример 18: Код антитела: 7-D12
VH
[000199] ДНК (SEQ ID NO:69)
CAAATGCAGCTGGTACAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACAAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAACCGGT
AGTAGTGGTTATGTACGTTGGAGCAACTGGTTCGACCCCTGGGGCCAGGGAACCCTGGTC
ACCGTCTCCTCA
[000200] АМИНОКИСЛОТНАЯ (SEQ ID NO:70)
QMQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADKSTSTAYMELSSLRSEDTAVYYCARTGSSGYVRWSNWFDPWGQGTLV
TVSS
VL
[000201] ДНК (SEQ ID NO:71)
GACATCCAGATGACCCAGTCTCCCTCCACCCTGTCTGCATTTGTAGGAGACAGAGTCACC
ATCACTTGCCGGGCCAGTGAGAGTATTAGTAGGTGGTTGGCCTGGTATCAGCAGAAACCA
GGGAAAGCCCCTAAACTCCTAATCTCTAAGACGTCTAATTTAGAAAGCGGGGTCCCGTCA
AGGTTCAGTGGCGCTGGATCTGGGACAGATTTCACTCTCACCATTAGCAGTCTGCAACCT
GAGGATTTTGCAACTTACTTCTGTCAACAGGGTTCCAAAATGCCTCCGACTTTCGGCGGA
GGGACCAAGGTGGAGATCAAG
[000202] АМИНОКИСЛОТНАЯ (SEQ ID NO:72)
DIQMTQSPSTLSAFVGDRVTITCRASESISRWLAWYQQKPGKAPKLLISKTSNLESGVPS
RFSGAGSGTDFTLTISSLQPEDFATYFCQQGSKMPPTFGGGTKVEIK
Пример 19: Код антитела: 9-E3
VH
[000203] ДНК (SEQ ID NO:73)
CAGGTGCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGGGGCC
TACGGTGGTAACTCCGCTTTTGACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000204] АМИНОКИСЛОТНАЯ (SEQ ID NO:74)
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAYGGNSAFDYWGQGTLVTVSS
VL
[000205] ДНК (SEQ ID NO:75)
CAGTCTGTGCTGACGCAGCCGCCCTCAGTGTCTGGGGCCCCAGGGCAGAGGGTCACCATC
TCCTGCACTGGGAGCAGCTCCAACATCGGGGCAGGTTATGATGTACACTGGTACCAGCAG
CTTCCAGGAACAGCCCCCAAACTCCTCATGTACAGTAATGATCAGCGGCCCTCAGGGGTC
ACTGAGCGATTCTCTGGCTCCAAGTCTGGCACCTCAGCCTCCCTGGCCATCAGTGGGCTC
CAGTCTGAAGATGAGGGTGATTACTACTGCCAGTCCTATGACAGAAGCCTGAGAGGTTCG
GTCTTCGGCGGAGGGACCAAGCTGACCGTCCTC
[000206] АМИНОКИСЛОТНАЯ (SEQ ID NO:76)
QSVLTQPPSVSGAPGQRVTISCTGSSSNIGAGYDVHWYQQLPGTAPKLLMYSNDQRPSGV
TERFSGSKSGTSASLAISGLQSEDEGDYYCQSYDRSLRGSVFGGGTKLTVL
Пример 20: Код антитела: 10-A6
VH
[000207] ДНК (SEQ ID NO:77)
GAGGTGCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGGCCTCAGTGAAGGTT
TCCTGCAAGGCTTCTGGTTACACCTTTACCAGCTATGGTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGATGGATCAGCGCTTACAATGGTAACACAAACTAT
GCACAGAAGCTCCAGGGCAGAGTCACCATGACCACAGACACATCCACGAGCACAGCCTAC
ATGGAGCTGAGGAGCCTGAGATCTGACGACACGGCCGTGTATTACTGTGCGAGAGATTCC
ATAGCAGCAGCTGGTACTCCGTTCGACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCC
TCA
[000208] АМИНОКИСЛОТНАЯ (SEQ ID NO:78)
EVQLVQSGAEVKKPGASVKVSCKASGYTFTSYGISWVRQAPGQGLEWMGWISAYNGNTNY
AQKLQGRVTMTTDTSTSTAYMELRSLRSDDTAVYYCARDSIAAAGTPFDYWGQGTLVTVS
S
VL
[000209] ДНК (SEQ ID NO:79)
AATTTTATGCTGACTCAGCCCCACTCTGTGTCGGAGTCTCCGGGGAAGACGGTCACCATC
TCCTGCACCCGCAGCAGTGGCATCATTGCCAGCAAATATGTGCACTGGTACCAGCAGCGC
CCGGGCAGTGCCCCCACCACTGTGATCTATGAGGATAACCAAAGACCGTCTGGGGTCCCT
GATCGATTCTCTGGCTCCATCGACAACTCCTCCAACTCTGCCTCCCTCACCATCTCTGGA
CTGCAGACTGAGGACGAGGCTGACTACTACTGTCAGTCTCATGACGGCATCAATCAGGTT
TTCGGCGGAGGGACCAAGGTCACCGTCCTA
[000210] АМИНОКИСЛОТНАЯ (SEQ ID NO:80)
NFMLTQPHSVSESPGKTVTISCTRSSGIIASKYVHWYQQRPGSAPTTVIYEDNQRPSGVP
DRFSGSIDNSSNSASLTISGLQTEDEADYYCQSHDGINQVFGGGTKVTVL
Пример 21: Код антитела: 12-A4
VH
[000211] ДНК (SEQ ID NO:81)
GAGGTGCAGCTGGTGGAGTCCCGGGGAGGCTTGGTACAGCCGGGGGGGTCCCTGAGACTC
TCCTGTGTAACTTCTGGATTCAGCTTTAACAACTATGCCATGAACTGGGTCCGCCAGGCT
CCGGGGAAGGGGCTGGAGTGGGTCTCAGCTGTTAGTGGTAGTGGTGGTACCACATACTAC
GCAGACTCCGTGAAGGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTTT
GTGCAGATGGACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCGAAAGGACTT
TTCCCTACGATTTTTGGAGTAGGAGCAATGTTTGACTACTGGGGCCAGGGAACCCTGGTC
ACCGTCTCCTCA
[000212] АМИНОКИСЛОТНАЯ (SEQ ID NO:82)
EVQLVESRGGLVQPGGSLRLSCVTSGFSFNNYAMNWVRQAPGKGLEWVSAVSGSGGTTYY
ADSVKGRFTISRDNSKNTLFVQMDSLRAEDTAVYYCAKGLFPTIFGVGAMFDYWGQGTLV
TVSS
VL
[000213] ДНК (SEQ ID NO:83)
TCTTCTGAGCTGACTCAGCCACCCTCAGCGTCTGGGACCCCCGGGCAGAGGGTCACCATC
TCTTGTTCTGGAAGCAGCTCCAACATCGGAAGTAATGCTGTTAACTGGTATCAGCAGCTC
CCAGGAACGGCCCCCAAACTCCTCATCTATGATAATAATCACCGGCCCTCAGGGGTCCCT
GACCGATTCTCTGGCTCCAAGTCTGGCACCTCAGCCTCCCTGGCCATCAGTGGGCTCCAG
TCTGAGGATGAGGCTGATTATTATTGTGCAGCATGGGATGACACCATTCCTGGTGTGCTA
TTCGCCGGAGGGACCAAGCTGACCGTCCTA
[000214] АМИНОКИСЛОТНАЯ (SEQ ID NO:84)
SSELTQPPSASGTPGQRVTISCSGSSSNIGSNAVNWYQQLPGTAPKLLIYDNNHRPSGVP
DRFSGSKSGTSASLAISGLQSEDEADYYCAAWDDTIPGVLFAGGTKLTVL
Пример 22: Код антитела: 14-G10
VH
[000215] ДНК (SEQ ID NO:85)
GAAGTGCAGCTGGTGGAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGGTGTT
TCTTACTACTACGGTATGGACGTCTGGGGCCAAGGGACCACGGTCACCGTCTCCTCA
[000216] АМИНОКИСЛОТНАЯ (SEQ ID NO:86)
EVQLVESGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGVSYYYGMDVWGQGTTVTVSS
VL
[000217] ДНК (SEQ ID NO:87)
CAGGCTGTGCTGACTCAGCCACCCTCGGTGTCCGTGTCCCCAGGACAGACAGCCATCATC
TCCTGTTCTGGACATAAATTGGGTGATAAGTATGTTTCCTGGTATCAACAGCAGCCAGGC
CAGTCCCCTGTGCTGGTCCTCTTTCAGGATACCAAGCGGCCCTCAGGGATCCCTGAGCGA
TTCTCTGGCTCCAACTCTGGGAACACAGCCACTCTGACCATCAGCGCGACCCAGGCTGCG
GATGAGGCTGACTATTACTGTCAGGCGGGGGACACCAAGTCTGTGATCTTCGGCGGCGGG
ACCAAGCTGACCGTCCTA
[000218] АМИНОКИСЛОТНАЯ (SEQ ID NO:88)
QAVLTQPPSVSVSPGQTAIISCSGHKLGDKYVSWYQQQPGQSPVLVLFQDTKRPSGIPER
FSGSNSGNTATLTISATQAADEADYYCQAGDTKSVIFGGGTKLTVL
Пример 23: Код антитела: 22-A6
VH
[000219] ДНК (SEQ ID NO:89)
CAGGTTCAGGTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGGCAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGGATAC
AGCTATGGTTCAGGACACCTTGACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000220] АМИНОКИСЛОТНАЯ (SEQ ID NO:90)
QVQVVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGYSYGSGHLDYWGQGTLVTVSS
VL
[000221] ДНК (SEQ ID NO:91)
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACC
ATCACTTGCCAGGCGAGTCAGGACATTAGCAACTATTTAAATTGGTATCAGCAGAAACCA
GGGAAAGCCCCTAAGCTCCTGATCTACGATGCATCCAATTTGGAAACAGGGGTCCCATCA
AGGTTCAGTGGCAGTGGATCTGGGACAGATTTCGCTCTCACCATCAGCAGTCTCCAACCT
GAAGATTTTGCAACTTATTACTGTCTACAGCATAATAGTTACCCTCGGACTTTTGGCCAG
GGGACCAAGCTGGAGATCAAA
[000222] АМИНОКИСЛОТНАЯ (SEQ ID NO:92)
DIQMTQSPSSLSASVGDRVTITCQASQDISNYLNWYQQKPGKAPKLLIYDASNLETGVPS
RFSGSGSGTDFALTISSLQPEDFATYYCLQHNSYPRTFGQGTKLEIK
Пример 24: Код антитела: 35-B1
VH
[000223] ДНК (SEQ ID NO:93)
GAGGTGCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGGCCTCAGTGAAGGTC
TCCTGCAAGGCTTCTGGATACACCTTCACCGGCTACTATATGCACTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGATGGATGAACCCTAACAGTGGTGACACAGCCTAT
ACACAGAACTTCCAGGGCAGAGTCACCATGACCAGGAACCCCTCCATAAGCACAGCCTAC
ATGGAGCTGAGCAACCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGAGGCCGG
GGGTTCGCGGAGAAGCCCCTTGGGTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000224] АМИНОКИСЛОТНАЯ (SEQ ID NO:94)
EVQLVQSGAEVKKPGASVKVSCKASGYTFTGYYMHWVRQAPGQGLEWMGWMNPNSGDTAY
TQNFQGRVTMTRNPSISTAYMELSNLRSEDTAVYYCARGRGFAEKPLGYWGQGTLVTVSS
VL
[000225] ДНК (SEQ ID NO:95)
GATATTGTGATGACTCAGTCTCCAGACTCCCTGGCTGTGTCTCTGGGCGGGAGGGCCACC
ATCAACTGCAAGTCCAGCCAGAGTATTTTATCCAGCTCCAATAATAAGAACTATTTAGCT
TGGTACCAGCAGAAACCAGGTCAGCCTCCTAAGCTGCTCATTTACTGGGCATCTACCCGG
GAATCCGGGGTCCCTGACCGGTTCAGCGGCAGCGGGTCTGGGACAGATTTCACTCTCACC
ATCAGCAGCCTGCAGGCTGAAGATGTGGCAGTTTATTACTGTCAGCAATATTATAGTACT
CCTCCGACATTCGGCCAAGGGACCAAGGTGGAAATCAAA
[000226] АМИНОКИСЛОТНАЯ (SEQ ID NO:96)
DIVMTQSPDSLAVSLGGRATINCKSSQSILSSSNNKNYLAWYQQKPGQPPKLLIYWASTR
ESGVPDRFSGSGSGTDFTLTISSLQAEDVAVYYCQQYYSTPPTFGQGTKVEIK
Пример 25: Код антитела: 3-1F4
VH
[000227] ДНК (SEQ ID NO:97)
GAGGTGCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGTCCTCGGTGAAGGTC
TCCTGCAAGGCTTCTGGAGGCACCTTCAGCAGCTATGCTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGAGGGATCATCCCTATCTTTGGTACAGCAAACTAC
GCACAGAAGTTCCAGGGCAGAGTCACGATTACCGCGGACGAATCCACGAGCACAGCCTAC
ATGGAGCTGAGCAGCCTGAGATCTGAGGACACGGCCGTGTATTACTGTGCGAGGGCCCCT
CGAGGGCAGTGGCTGGTTCACTACTTTGACTACTGGGGCCAGGGAACCCTGGTCACCGTC
TCCTCA
[000228] АМИНОКИСЛОТНАЯ (SEQ ID NO:98)
EVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTANY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARAPRGQWLVHYFDYWGQGTLVTV
SS
VL
[000229] ДНК (SEQ ID NO:99)
GAAATTGTGTTGACGCAGTCTCCAGCCACCCTCTCTCTGTCTCCAGGGGAAAGAGCCACC
CTCTCCTGCTGGGCCAGTCAGGATGTTAGCAACTACTTAGCCTGGTACCAACAGAAGCCT
GGCCAGGCTCCCAGGCTCCTCATCTATGATGCATCCAACAGGGCCACTGGCATCCCAGCC
AGGTTCAGCGGCAGTGGGTCTGGGACAGACTTCACTCTCACCATCAGCAGCCTAGAGCCT
GAAGATTTTGCAGTGTATTACTGTCAGCAACGTAGCAACTGGCCTCTCACTTTCGGCGGC
GGGACCAAGGTGGAGCTCAAA
[000230] АМИНОКИСЛОТНАЯ (SEQ ID NO:100)
EIVLTQSPATLSLSPGERATLSCWASQDVSNYLAWYQQKPGQAPRLLIYDASNRATGIPA
RFSGSGSGTDFTLTISSLEPEDFAVYYCQQRSNWPLTFGGGTKVELK
Пример 26: Код антитела: 4-1B3
VH
[000232] ДНК (SEQ ID NO:101)
CAGGTTCAGCTGGTGCAGTCTGGAGCTGAGGTGAAGAAGCCTGGGGCCTCAGTGAAGGTC
TCCTGCAAGGCTTCTGGTTACACCTTTACCAGCTATGGTATCAGCTGGGTGCGACAGGCC
CCTGGACAAGGGCTTGAGTGGATGGGATGGATCAGCGCTTACAATGGTAACACAAACTAT
GCACAGAAGCTCCAGGGCAGAGTCACCATGACCACAGACACATCCACGAGCACAGCCTAC
ATGGAGCTGAGGAGCCTGAGATCTGACGACACGGCCGTGTATTACTGTGCGAGAGAGTCC
TACTCGTCCGCAGGTATTGACTACTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
[000232] АМИНОКИСЛОТНАЯ (SEQ ID NO:102)
QVQLVQSGAEVKKPGASVKVSCKASGYTFTSYGISWVRQAPGQGLEWMGWISAYNGNTNY
AQKLQGRVTMTTDTSTSTAYMELRSLRSDDTAVYYCARESYSSAGIDYWGQGTLVTVSS
VL
[000233] ДНК (SEQ ID NO:103)
GATATTGTGATGACTCAGTCTCCACTCTCCCTGCCCGTCACCCCTGGAGAGCCGGCCTCC
ATCTCCTGCAGGTCTAGTCAGACCCTCCTGCATAGTAATGGATTCAACTATTTGGATTGG
TACCTGCAGAAGCCAGGGCAGTCTCCACAACTCCTGATGTATTTGGGCTCTAGCCGGGCC
TCCGGGGTCCCTGACAGGTTCAGTGGCAGTGGATCGGGCACAGATTTCACACTGAAAATC
AGCAGAGTGGAGGCTGAGGATGTTGGGGTTTATTACTGCATGCAAACTCTACAAACTCCT
CCGGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
[000234] АМИНОКИСЛОТНАЯ (SEQ ID NO:104)
DIVMTQSPLSLPVTPGEPASISCRSSQTLLHSNGFNYLDWYLQKPGQSPQLLMYLGSSRA
SGVPDRFSGSGSGTDFTLKISRVEAEDVGVYYCMQTLQTPPAFGGGTKVEIK
Пример 27: Код антитела: 21-G1
VH
[000235] ДНК (SEQ ID NO:105)
CAGGTCCAGCTGGTGCAGTCTGGGGCTGAGGTGAAGAAGCCTGGGGCCTCAGTGACGATT
TCCTGCGAGGCGTCTGGATACAACTTCATCAGCTACTATATACACTGGGTGCGACAGGCC
CCTGGACAAGGCCTTGAGTGGATGGGATTCGTCGTCCCTAGTGGTGGTGCCGCAGGCTAC
ACACAGAAGTTCCAGGGCAGACTCACCGTGACCAGGGACACGTCCACGAGCACAGTCTAC
ATGGACCTGAACAGCCTGACATCTGACGACACGGCCGTGTATTACTGTGTGCGAGAAATG
AGTGGTGGCTGGTTTGATTTCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCG
[000236] АМИНОКИСЛОТНАЯ (SEQ ID NO:106)
QVQLVQSGAEVKKPGASVTISCEASGYNFISYYIHWVRQAPGQGLEWMGFVVPSGGAAGY
TQKFQGRLTVTRDTSTSTVYMDLNSLTSDDTAVYYCVREMSGGWFDFWGQGTLVTVSS
VL
[000237] ДНК (SEQ ID NO:107)
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACC
ATCACTTGCCGGGCAAGTCAGAGCATTAGCAGCTATTTAAATTGGTATCAGCAGAAACCA
GGGAAAGCCCCTAAGCTCCTGATCTATGCTGCATCCAGTTTGCAAAGTGGGGTCCCATCA
AGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGTCTGCAACCT
GAAGATTTTGCAACTTACTACTGTCAACAGAGTTACAGTACCCCGATCACCTTCGGCCAA
GGGACACGACTGGAGATTAAA
[000238] АМИНОКИСЛОТНАЯ (SEQ ID NO:108)
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSYSTPITFGQGTRLEIK
[000239] Для специалистов в данной области техники будет очевидно, что в настоящее изобретение могут быть внесены различные модификации и изменения, не выходящие за рамки объема или сущности изобретения. Другие варианты реализации изобретения будут очевидны для специалистов в данной области техники после изучения описания и практической реализации изобретения, раскрытого в настоящем документе. Предполагается, что настоящее описание и примеры являются исключительно иллюстративными, а истинные объем и сущность изобретения определены приведенной ниже формулой изобретения.
[000240] В тексте настоящей заявки приведены ссылки на различные публикации, патенты и/или патентные заявки для более полного описания уровня техники, к которой относится настоящее изобретение. Содержание этих публикаций, патентов и/или патентных заявок включено в настоящий документ посредством ссылок полностью в той же степени, как если бы каждая независимая публикация, патент и/или патентная заявка были конкретно и индивидуально указаны как включенные посредством ссылки.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> 1GLOBE BIOMEDICAL CO., LTD./ 1ГЛОБ БИОМЕДИКАЛ КО., ЛТД.
1GLOBE HEALTH INSTITUTE LLC/ 1ГЛОБ ХЕЛС ИНСТИТЬЮТ ЛЛС
LI, Chiang J./ ЛИ, Чан Дж.
UNNIRAMAN, Shyam/ УННИРАМАН, Шьям
BADER, Hannah/ БЕЙДЕР, Ханна
JHA, Mithilesh/ ДЖА, Митилеш
<120> НОВЫЕ КОМПОЗИЦИИ АНТИТЕЛ ДЛЯ ИММУНОТЕРАПИИ РАКА
<130> PI19033299
<140> PCT/CN 2019/101659
<141> 2019-08-20
<150> US 62/720015
<151> 2018-08-20
<160> 108
<170> PatentIn, версия 3.5
<210> 1
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1A2 VH
<400> 1
caggttcagc tggtgcagtc tgggactgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc agttatgata tcaactgggt gcgacaggcc 120
actggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accacagaca cttctacggg cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagattttta 300
tggggttcgg ggagttatga ctactggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 2
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1A2 VH
<400> 2
Gln Val Gln Leu Val Gln Ser Gly Thr Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asp Ile Asn Trp Val Arg Gln Ala Thr Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Asn Ser Gly Gly Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Gly Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Leu Trp Gly Ser Gly Ser Tyr Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 3
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1A2 VL
<400> 3
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagatttcg caacttacta ctgtcaacag acttacacat tcccgcacac ttttgcccag 300
gggaccaacc tggagatcaa a 321
<210> 4
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1A2 VL
<400> 4
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Thr Phe Pro His
85 90 95
Thr Phe Ala Gln Gly Thr Asn Leu Glu Ile Lys
100 105
<210> 5
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1A12 VH
<400> 5
caagtccagc tggtacaatc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagattgg 300
atacagctat ggttacccct tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 6
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1A12 VH
<400> 6
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Trp Ile Gln Leu Trp Leu Pro Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 7
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1A12 VL
<400> 7
gacatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca acagaaacca 120
gggaaagccc ctaagctcct gatctatggt gcatccagtt tggaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agtcacagtt cccccctcac tttcggcgga 300
gggaccaagg tggacatcaa a 321
<210> 8
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1A12 VL
<400> 8
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser His Ser Ser Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Asp Ile Lys
100 105
<210> 9
<211> 387
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1B9 VH
<400> 9
gaagtgcagc tggtgcagtc tgggggaggc ttggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaaagatttg 300
atcccgttgc gagatagtag gggggggtac tactacggta tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc agggagt 387
<210> 10
<211> 127
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1B9 VH
<400> 10
Glu Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Leu Ile Pro Leu Arg Asp Ser Arg Gly Gly Tyr Tyr Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 11
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1B9 VL
<400> 11
tcttctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagagactat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcaggaat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactgggac tcaggcggaa 240
gatgaggctg actattactg taactcccgt gacagcggtg cttaccatta tgtcttcgga 300
actgggacca aggtcaccgt ccta 324
<210> 12
<211> 108
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1B9 VL
<400> 12
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Asp Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Thr Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp Ser Gly Ala Tyr His
85 90 95
Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105
<210> 13
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1B12 VH
<400> 13
caaatccagc tggtgcagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaaaggaagt 300
attatagggg atggtgcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 360
<210> 14
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1B12 VH
<400> 14
Gln Ile Gln Leu Val Gln Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Ser Ile Ile Gly Asp Gly Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 15
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1B12 VL
<400> 15
gatattgtga tgacccagtc tccactctcc ctgcccgtca cccttggaga gccggcctcc 60
atctcctgca ggtctagtca gaccctcctg cataatggat tcaacttttt ggattggtac 120
ctgcagaagc cagggcagtc tccacaactc ctgatgtatt tggcctctag ccgggcctcc 180
ggggtccctg acaggttcag tggcagtgga tcgggcacag atttcacact gaaaatcagc 240
agagtggagg ctgaggatgt tggggtttat tactgcatgc aaggtacaca ctggccgtac 300
acttttggcc aggggaccaa gctggatatc aaa 333
<210> 16
<211> 111
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1B12 VL
<400> 16
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Thr Leu Leu His Asn
20 25 30
Gly Phe Asn Phe Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro
35 40 45
Gln Leu Leu Met Tyr Leu Ala Ser Ser Arg Ala Ser Gly Val Pro Asp
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser
65 70 75 80
Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly Thr
85 90 95
His Trp Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Asp Ile Lys
100 105 110
<210> 17
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1E8 VH
<400> 17
caaatccagc tggtacaatc tggggctgag gtgaagatgc ctggggcctc agtgacgatt 60
tcctgcgagg cgtctggata caacttcatc agctactata tacactgggt gcgacaggcc 120
cctggacaag gccttgagtg gatgggattc gtcgtcccta gtggtggtgc cgcaggctac 180
acacagaagt tccagggcag actcaccgtg accagggaca cgtccacgag cacagtctac 240
atggacctga acagcctgac atctgacgac acggccgtgt attactgtgt gcgagaaatg 300
agtggtggct ggtttgattt ctggggccag ggaaccctgg tcaccgtctc ctcg 354
<210> 18
<211> 118
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1E8 VH
<400> 18
Gln Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Met Pro Gly Ala
1 5 10 15
Ser Val Thr Ile Ser Cys Glu Ala Ser Gly Tyr Asn Phe Ile Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Val Val Pro Ser Gly Gly Ala Ala Gly Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Leu Thr Val Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Asp Leu Asn Ser Leu Thr Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Arg Glu Met Ser Gly Gly Trp Phe Asp Phe Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 19
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1E8 VL
<400> 19
gacatcgtga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcaaaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccactt tgcaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcaggct 240
gaagatgtgg cagtttatta ctgtcagcaa tattatagta ctcctctcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 20
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1E8 VL
<400> 20
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Ala
65 70 75 80
Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Thr Pro Leu
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 21
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1G7 VH
<400> 21
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagcctca 300
ccggtacagc agcccatatg gtgggcggag tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 22
<211> 122
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1G7 VH
<400> 22
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Ser Pro Val Gln Gln Pro Ile Trp Trp Ala Glu Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 23
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1G7 VL
<400> 23
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tctgatgtca gtaagcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg acgaggctga ttattactgc agctcatata caagcaacta cactttggta 300
ttcggcggag ggaccaagct gaccgtccta 330
<210> 24
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1G7 VL
<400> 24
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Ser Asp Val Ser Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Asn
85 90 95
Tyr Thr Leu Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 25
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1H10 VH
<400> 25
cagctgcagc tacagcagtc cggagctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg ctcctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaagg atcatcccta tccttggtat agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaagac acggccgtgt attactgtgc gagtcatggt 300
cgggcagcag ctggtaggta cgctatggac gtctggggcc aagggaccac ggtcaccgtc 360
tcctca 366
<210> 26
<211> 122
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1H10 VH
<400> 26
Gln Leu Gln Leu Gln Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Pro Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser His Gly Arg Ala Ala Ala Gly Arg Tyr Ala Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 27
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1H10 VL
<400> 27
aattttatgc tgactcagcc ccactctgtg tcggattctc cggggaagac ggtaaccatc 60
tcctgcaccc gcagcagtgg cagcattgcc agcaactatg tgcagtggta ccagcagcgc 120
ccgggcagtg cccccaccac tgtgatctat gacgataagc aaagaccctc tggggtccct 180
gatcggttct cgggctccat cgacagctcc tccaactctg cctccctcac catctctgga 240
ctgacgactg aggacgaggc tgactactac tgtcagtcct ttgatggcag cagtgtcatc 300
ttcggcggag ggaccaagct gaccgtcctg 330
<210> 28
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1H10 VL
<400> 28
Asn Phe Met Leu Thr Gln Pro His Ser Val Ser Asp Ser Pro Gly Lys
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Arg Ser Ser Gly Ser Ile Ala Ser Asn
20 25 30
Tyr Val Gln Trp Tyr Gln Gln Arg Pro Gly Ser Ala Pro Thr Thr Val
35 40 45
Ile Tyr Asp Asp Lys Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Ile Asp Ser Ser Ser Asn Ser Ala Ser Leu Thr Ile Ser Gly
65 70 75 80
Leu Thr Thr Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Phe Asp Gly
85 90 95
Ser Ser Val Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 29
<211> 375
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1H2 VH
<400> 29
caggttcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaaaggag 300
cgtttctatg atagtagtgg ttattacgat gcttttgata tctggggcca agggacaatg 360
gtcaccgtct cttca 375
<210> 30
<211> 125
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1H2 VH
<400> 30
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Lys Glu Arg Phe Tyr Asp Ser Ser Gly Tyr Tyr Asp Ala Phe
100 105 110
Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120 125
<210> 31
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1H2 VL
<400> 31
cagtctgccc tgactcagcc tcgctcagtg tccgggtctc ctgggcagtc agtcaccatc 60
tcctgcactg gaaccagcaa tgatgttggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgatgtca ctaagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccgt ctctggcctc 240
cagcctgagg atgaggctga ctattattgc gcctcttatg gaggcaggaa caatttgctt 300
tttggcggag ggactcaact gaccgtctta 330
<210> 32
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1H2 VL
<400> 32
Gln Ser Ala Leu Thr Gln Pro Arg Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Asn Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Thr Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Tyr Gly Gly Arg
85 90 95
Asn Asn Leu Leu Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
<210> 33
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1E4 VH
<400> 33
caaatccagc tggtacaatc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc cggaggggga 300
gcagtggcgg acaatagtta ctggggccag ggaaccctgg tcaccgtctc ctca 354
<210> 34
<211> 118
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1E4 VH
<400> 34
Gln Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gly Gly Ala Val Ala Asp Asn Ser Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 35
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1E4 VL
<400> 35
gacatccgga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tacaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tggcacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtctacaa gattacaatt accctcgaac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
<210> 36
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1E4 VL
<400> 36
Asp Ile Arg Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Asp Tyr Asn Tyr Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 37
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1A8 VH
<400> 37
caaatccagc tggtacaatc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacggcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagagacggt 300
tcgtatagca gcagctggta ctcgtttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 38
<211> 122
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1A8 VH
<400> 38
Gln Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Ser Tyr Ser Ser Ser Trp Tyr Ser Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 39
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1A8 VL
<400> 39
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacgtcggt ggttataact atgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgatgtca gtaatcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg acgaggctga ttattactgc tcctcatatg caggtgatat tagttatgta 300
ctgttcggcg gcgggaccaa gctgaccgtc cta 333
<210> 40
<211> 111
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1A8 VL
<400> 40
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Ala Gly Asp
85 90 95
Ile Ser Tyr Val Leu Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 41
<211> 351
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1B11 VH
<400> 41
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt cacttttagt gactatgaca tgatctgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagtaa taaatactat 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaaagagttc 300
tttggtgctt ttgatatctg gggccaaggg acaatggtca ccgtctcttc a 351
<210> 42
<211> 117
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1B11 VH
<400> 42
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Asp Met Ile Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Phe Phe Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
100 105 110
Val Thr Val Ser Ser
115
<210> 43
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1B11 VL
<400> 43
tcttctgagc tgactcagga ccctgctgtg tcggtggcct tgggacagac agtcacgatc 60
acatgccaag gagacagcct caattactat tatgcaaact ggttccagct gaagccaggg 120
caggcccctg tacttgtcct ctttggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct cctactcggg aagcacagct tccttgacca tcactggggc tcaggcggaa 240
gatgacgctg actattactg taattcgcgg gacagcggtg gtaatccttg ggtgttcggc 300
ggagggacca agctgaccgt ccta 324
<210> 44
<211> 108
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1B11 VL
<400> 44
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Thr Ile Thr Cys Gln Gly Asp Ser Leu Asn Tyr Tyr Tyr Ala
20 25 30
Asn Trp Phe Gln Leu Lys Pro Gly Gln Ala Pro Val Leu Val Leu Phe
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Tyr Ser Gly Ser Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Asp Ala Asp Tyr Tyr Cys Asn Ser Arg Asp Ser Gly Gly Asn Pro
85 90 95
Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 45
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1F3 VH
<400> 45
caaatccagc tggtacaatc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaagg atcatcccta tccttggtat agcagactac 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggaactga gtagcctggg atctgaggac acggccgtgt atttttgtgc gagagagggg 300
ggatccttta ggcactttga cttctggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 46
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1F3 VH
<400> 46
Gln Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Gly Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Glu Gly Gly Ser Phe Arg His Phe Asp Phe Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 47
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1F3 VL
<400> 47
cagcctgtgc tgactcagcc accctcagtc tctggggccc cagggcagag ggtcaccatc 60
tcctgcgctg ggagcgaccc caacatcggg acaggtcatg atgtgcactg gtaccagcaa 120
cttccaggaa cagcccccaa actcgtcatc tatggtaaca ccaatcggcc ctcaggggtc 180
cctgagcgat tcactgcctc caagtctggc acctcagcct ccctggccat cactgggctc 240
caggctgagg atgaggctga ttattactgc caggcctacg acaggagcct gcgtggttat 300
gtcttcggga ctgggaccaa ggtcaccgtc ctg 333
<210> 48
<211> 111
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1F3 VL
<400> 48
Gln Pro Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ala Gly Ser Asp Pro Asn Ile Gly Thr Gly
20 25 30
His Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Val Ile Tyr Gly Asn Thr Asn Arg Pro Ser Gly Val Pro Glu Arg Phe
50 55 60
Thr Ala Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Tyr Asp Arg Ser
85 90 95
Leu Arg Gly Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 49
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1G5 VH
<400> 49
caaatccagc tggtacagtc tggtgctgaa gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaaga cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagaactaca 300
ggtgacgagt ggctacgatt ggctataaat gactactggg gccagggaac cctggtcacc 360
gtctcctca 369
<210> 50
<211> 123
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1G5 VH
<400> 50
Gln Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Thr Gly Asp Glu Trp Leu Arg Leu Ala Ile Asn Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 51
<211> 336
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1G5 VL
<400> 51
gatattgtga tgacacagtc tcccctctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtct gcgcctcatg catcctaatg gactcaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctaatct ttttgggttc tcagcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggcatt tattactgca tgcaagctct agaacctccg 300
tacacttttg gccaggggac caagctggag atcaaa 336
<210> 52
<211> 112
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1G5 VL
<400> 52
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Leu Arg Leu Met His Pro
20 25 30
Asn Gly Leu Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Gly Ser Gln Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Glu Pro Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 53
<211> 372
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1C9 VH
<400> 53
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagagatccc 300
gggtatagca gtggctggaa agatgatgct tttgatatct ggggccaagg gacaatggtc 360
accgtctctt ca 372
<210> 54
<211> 124
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1C9 VH
<400> 54
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Pro Gly Tyr Ser Ser Gly Trp Lys Asp Asp Ala Phe Asp
100 105 110
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 55
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1C9 VL
<400> 55
gaaattgtga tgacacagtc tccaggcacc ctgtctttgt ctccagggga tacagcctcc 60
ctctcctgca gggccagtca gactgttagc agcaactact tagcctggta ccaacagaaa 120
cctggccagg ctcccaggct cctcatctat gatacatcca acagggccgc tggcatcccg 180
gccaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag tagcctagag 240
cctgaagatt ttgcagtgta ttactgtcag cagtacggta gctcactctg gacgttcggc 300
caagggacca aggtggaaat caaa 324
<210> 56
<211> 108
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1C9 VL
<400> 56
Glu Ile Val Met Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Asp Thr Ala Ser Leu Ser Cys Arg Ala Ser Gln Thr Val Ser Ser Asn
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Thr Ser Asn Arg Ala Ala Gly Ile Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Leu
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 57
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 11-A4 VH
<400> 57
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagagcgggg 300
cagcagctgg tagccctttg gtactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 58
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 11-A4 VH
<400> 58
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Gly Gln Gln Leu Val Ala Leu Trp Tyr Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 59
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 11-A4 VL
<400> 59
cagtctgccc tgactcagcc tccctccgcg tccgggtctc gtggacagtc agtctccatc 60
tcctgcagtg gaagtcgcag tgacattgga tattataact atgtctcctg gtatcaacaa 120
cacccaggca aagcccccaa actcatcatt tttgacgtca ataagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccgt ctctggcctc 240
cagcctgagg atgaggctga ctattattgc gcctcttatg gaggcaggaa caatttgctt 300
tttggcggag ggactcaact gaccgtctta 330
<210> 60
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 11-A4 VL
<400> 60
Gln Ser Ala Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Arg Gly Gln
1 5 10 15
Ser Val Ser Ile Ser Cys Ser Gly Ser Arg Ser Asp Ile Gly Tyr Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Phe Asp Val Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Tyr Gly Gly Arg
85 90 95
Asn Asn Leu Leu Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
<210> 61
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 21-A1 VH
<400> 61
caggtgcaac tgcaggagtc gggcccagga ctggtggagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagt agtttctact ggagctggat ccggcagccc 120
ccagggaagg gactggagtg gattggctat atcaattaca gtgggagcac caactacaac 180
ccctccctca agagtcgagt caccatatca gtagacacgt ccaagaacca gttctccctg 240
aagctgagct ctgtgaccgc cgcagacacg gctgtgtatt actgtgcgag acagatatta 300
tggttcgggg agttaaggtg gttcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 62
<211> 121
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 21-A1 VH
<400> 62
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Glu Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Phe
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Asn Tyr Ser Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gln Ile Leu Trp Phe Gly Glu Leu Arg Trp Phe Asp Pro Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 63
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 21-A1 VL
<400> 63
cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc agtcaccatc 60
tcctgcactg gaaccagcag tgacattggt ggttataact atgtctcctg gtaccaactg 120
cgcccaggca aagcccccaa actcatgatt tatgacgtca ccaagcggcc ctcaggggtc 180
cctgatcgct tctctggctc caagtctggc aacacggcct ccctgaccgt ctctgggctc 240
caggctgagg atgaggctga ttattactgc agctcatatg caggcagcaa caatgtggta 300
ttcggcggag ggaccaagct gaccgtccta 330
<210> 64
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 21-A1 VL
<400> 64
Gln Ser Ala Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Ile Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Leu Arg Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Thr Lys Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Val Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Ala Gly Ser
85 90 95
Asn Asn Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 65
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 21-H12 VH
<400> 65
caagtccagc tggtacagtc tggggctgag gtgaagaagc ctggggcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaaatccc 300
tacggtttca actggttcga cccctggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 66
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 21-H12 VH
<400> 66
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asn Pro Tyr Gly Phe Asn Trp Phe Asp Pro Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 67
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 21-H12 VL
<400> 67
aattttatgc tgactcagcc ccactctgtg tcggagtctc cggggaagac ggtaaccatc 60
tcctgcaccc gcagcagtgg cagcattgcc agcaactatg tgcagtggta ccagcagcgc 120
ccgggcagtg cccccaccac tgtgatctat gaggataacc aaagaccctc tggggtccct 180
gatcggttct ctggctccat cgacagctcc tccaactctg cctccctcac catctccgga 240
ctgaagactg aggacgaggc tgactactac tgtcagtctt atgatggctt caatcaggtg 300
ttcggcggag ggaccaagct gaccgtccta 330
<210> 68
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 21-H12 VL
<400> 68
Asn Phe Met Leu Thr Gln Pro His Ser Val Ser Glu Ser Pro Gly Lys
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Arg Ser Ser Gly Ser Ile Ala Ser Asn
20 25 30
Tyr Val Gln Trp Tyr Gln Gln Arg Pro Gly Ser Ala Pro Thr Thr Val
35 40 45
Ile Tyr Glu Asp Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Ile Asp Ser Ser Ser Asn Ser Ala Ser Leu Thr Ile Ser Gly
65 70 75 80
Leu Lys Thr Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Gly
85 90 95
Phe Asn Gln Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 69
<211> 372
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 7-D12 VH
<400> 69
caaatgcagc tggtacagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaaccggt 300
agtagtggtt atgtacgttg gagcaactgg ttcgacccct ggggccaggg aaccctggtc 360
accgtctcct ca 372
<210> 70
<211> 124
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 7-D12 VH
<400> 70
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Gly Ser Ser Gly Tyr Val Arg Trp Ser Asn Trp Phe Asp
100 105 110
Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 71
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 7-D12 VL
<400> 71
gacatccaga tgacccagtc tccctccacc ctgtctgcat ttgtaggaga cagagtcacc 60
atcacttgcc gggccagtga gagtattagt aggtggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaactcct aatctctaag acgtctaatt tagaaagcgg ggtcccgtca 180
aggttcagtg gcgctggatc tgggacagat ttcactctca ccattagcag tctgcaacct 240
gaggattttg caacttactt ctgtcaacag ggttccaaaa tgcctccgac tttcggcgga 300
gggaccaagg tggagatcaa g 321
<210> 72
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 7-D12 VL
<400> 72
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Phe Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Ser Ile Ser Arg Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Lys Thr Ser Asn Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ala Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Gly Ser Lys Met Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 73
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 9-E3 VH
<400> 73
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaggggcc 300
tacggtggta actccgcttt tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 74
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 9-E3 VH
<400> 74
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Tyr Gly Gly Asn Ser Ala Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 75
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 9-E3 VL
<400> 75
cagtctgtgc tgacgcagcc gccctcagtg tctggggccc cagggcagag ggtcaccatc 60
tcctgcactg ggagcagctc caacatcggg gcaggttatg atgtacactg gtaccagcag 120
cttccaggaa cagcccccaa actcctcatg tacagtaatg atcagcggcc ctcaggggtc 180
actgagcgat tctctggctc caagtctggc acctcagcct ccctggccat cagtgggctc 240
cagtctgaag atgagggtga ttactactgc cagtcctatg acagaagcct gagaggttcg 300
gtcttcggcg gagggaccaa gctgaccgtc ctc 333
<210> 76
<211> 111
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 9-E3 VL
<400> 76
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Met Tyr Ser Asn Asp Gln Arg Pro Ser Gly Val Thr Glu Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu
65 70 75 80
Gln Ser Glu Asp Glu Gly Asp Tyr Tyr Cys Gln Ser Tyr Asp Arg Ser
85 90 95
Leu Arg Gly Ser Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 77
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 10-A6 VH
<400> 77
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagattcc 300
atagcagcag ctggtactcc gttcgactac tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 78
<211> 121
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 10-A6 VH
<400> 78
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ser Ile Ala Ala Ala Gly Thr Pro Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 79
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 10-A6 VL
<400> 79
aattttatgc tgactcagcc ccactctgtg tcggagtctc cggggaagac ggtcaccatc 60
tcctgcaccc gcagcagtgg catcattgcc agcaaatatg tgcactggta ccagcagcgc 120
ccgggcagtg cccccaccac tgtgatctat gaggataacc aaagaccgtc tggggtccct 180
gatcgattct ctggctccat cgacaactcc tccaactctg cctccctcac catctctgga 240
ctgcagactg aggacgaggc tgactactac tgtcagtctc atgacggcat caatcaggtt 300
ttcggcggag ggaccaaggt caccgtccta 330
<210> 80
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 10-A6 VL
<400> 80
Asn Phe Met Leu Thr Gln Pro His Ser Val Ser Glu Ser Pro Gly Lys
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Arg Ser Ser Gly Ile Ile Ala Ser Lys
20 25 30
Tyr Val His Trp Tyr Gln Gln Arg Pro Gly Ser Ala Pro Thr Thr Val
35 40 45
Ile Tyr Glu Asp Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Ile Asp Asn Ser Ser Asn Ser Ala Ser Leu Thr Ile Ser Gly
65 70 75 80
Leu Gln Thr Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser His Asp Gly
85 90 95
Ile Asn Gln Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 81
<211> 372
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 12-A4 VH
<400> 81
gaggtgcagc tggtggagtc ccggggaggc ttggtacagc cgggggggtc cctgagactc 60
tcctgtgtaa cttctggatt cagctttaac aactatgcca tgaactgggt ccgccaggct 120
ccggggaagg ggctggagtg ggtctcagct gttagtggta gtggtggtac cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgttt 240
gtgcagatgg acagcctgag agctgaggac acggctgtgt attactgtgc gaaaggactt 300
ttccctacga tttttggagt aggagcaatg tttgactact ggggccaggg aaccctggtc 360
accgtctcct ca 372
<210> 82
<211> 124
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 12-A4 VH
<400> 82
Glu Val Gln Leu Val Glu Ser Arg Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Val Thr Ser Gly Phe Ser Phe Asn Asn Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Val Ser Gly Ser Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Val Gln Met Asp Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Leu Phe Pro Thr Ile Phe Gly Val Gly Ala Met Phe Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 83
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 12-A4 VL
<400> 83
tcttctgagc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagcagctc caacatcgga agtaatgctg ttaactggta tcagcagctc 120
ccaggaacgg cccccaaact cctcatctat gataataatc accggccctc aggggtccct 180
gaccgattct ctggctccaa gtctggcacc tcagcctccc tggccatcag tgggctccag 240
tctgaggatg aggctgatta ttattgtgca gcatgggatg acaccattcc tggtgtgcta 300
ttcgccggag ggaccaagct gaccgtccta 330
<210> 84
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 12-A4 VL
<400> 84
Ser Ser Glu Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Ala Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn His Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp Asp Thr Ile
85 90 95
Pro Gly Val Leu Phe Ala Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 85
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 14-G10 VH
<400> 85
gaagtgcagc tggtggagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaggtgtt 300
tcttactact acggtatgga cgtctggggc caagggacca cggtcaccgt ctcctca 357
<210> 86
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 14-G10 VH
<400> 86
Glu Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Val Ser Tyr Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 87
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 14-G10 VL
<400> 87
caggctgtgc tgactcagcc accctcggtg tccgtgtccc caggacagac agccatcatc 60
tcctgttctg gacataaatt gggtgataag tatgtttcct ggtatcaaca gcagccaggc 120
cagtcccctg tgctggtcct ctttcaggat accaagcggc cctcagggat ccctgagcga 180
ttctctggct ccaactctgg gaacacagcc actctgacca tcagcgcgac ccaggctgcg 240
gatgaggctg actattactg tcaggcgggg gacaccaagt ctgtgatctt cggcggcggg 300
accaagctga ccgtccta 318
<210> 88
<211> 106
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 14-G10 VL
<400> 88
Gln Ala Val Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ile Ile Ser Cys Ser Gly His Lys Leu Gly Asp Lys Tyr Val
20 25 30
Ser Trp Tyr Gln Gln Gln Pro Gly Gln Ser Pro Val Leu Val Leu Phe
35 40 45
Gln Asp Thr Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Ala Thr Gln Ala Ala
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Gly Asp Thr Lys Ser Val Ile
85 90 95
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105
<210> 89
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 22-A6 VH
<400> 89
caggttcagg tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcggcaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaggatac 300
agctatggtt caggacacct tgactactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 90
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 22-A6 VH
<400> 90
Gln Val Gln Val Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Tyr Ser Tyr Gly Ser Gly His Leu Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 91
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 22-A6 VL
<400> 91
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcgctctca ccatcagcag tctccaacct 240
gaagattttg caacttatta ctgtctacag cataatagtt accctcggac ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 92
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 22-A6 VL
<400> 92
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Ala Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 93
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 35-B1 VH
<400> 93
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atgaacccta acagtggtga cacagcctat 180
acacagaact tccagggcag agtcaccatg accaggaacc cctccataag cacagcctac 240
atggagctga gcaacctgag atctgaggac acggccgtgt attactgtgc gagaggccgg 300
gggttcgcgg agaagcccct tgggtactgg ggccagggaa ccctggtcac cgtctcctca 360
<210> 94
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 35-B1 VH
<400> 94
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Met Asn Pro Asn Ser Gly Asp Thr Ala Tyr Thr Gln Asn Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asn Pro Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Asn Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Arg Gly Phe Ala Glu Lys Pro Leu Gly Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 95
<211> 339
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 35-B1 VL
<400> 95
gatattgtga tgactcagtc tccagactcc ctggctgtgt ctctgggcgg gagggccacc 60
atcaactgca agtccagcca gagtatttta tccagctcca ataataagaa ctatttagct 120
tggtaccagc agaaaccagg tcagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg gttcagcggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtact 300
cctccgacat tcggccaagg gaccaaggtg gaaatcaaa 339
<210> 96
<211> 113
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 35-B1 VL
<400> 96
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gly Arg Ala Thr Ile Asn Cys Lys Ser Ser Gln Ser Ile Leu Ser Ser
20 25 30
Ser Asn Asn Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Leu Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 97
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1F4 VH
<400> 97
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagggcccct 300
cgagggcagt ggctggttca ctactttgac tactggggcc agggaaccct ggtcaccgtc 360
tcctca 366
<210> 98
<211> 122
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1F4 VH
<400> 98
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Pro Arg Gly Gln Trp Leu Val His Tyr Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 99
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 3-1F4 VL
<400> 99
gaaattgtgt tgacgcagtc tccagccacc ctctctctgt ctccagggga aagagccacc 60
ctctcctgct gggccagtca ggatgttagc aactacttag cctggtacca acagaagcct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagcg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtgtatta ctgtcagcaa cgtagcaact ggcctctcac tttcggcggc 300
gggaccaagg tggagctcaa a 321
<210> 100
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 3-1F4 VL
<400> 100
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Trp Ala Ser Gln Asp Val Ser Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Leu Lys
100 105
<210> 101
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1B3 VH
<400> 101
caggttcagc tggtgcagtc tggagctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggtta cacctttacc agctatggta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcagcgctt acaatggtaa cacaaactat 180
gcacagaagc tccagggcag agtcaccatg accacagaca catccacgag cacagcctac 240
atggagctga ggagcctgag atctgacgac acggccgtgt attactgtgc gagagagtcc 300
tactcgtccg caggtattga ctactggggc cagggaaccc tggtcaccgt ctcctca 357
<210> 102
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1B3 VH
<400> 102
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Ser Ala Tyr Asn Gly Asn Thr Asn Tyr Ala Gln Lys Leu
50 55 60
Gln Gly Arg Val Thr Met Thr Thr Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Ser Tyr Ser Ser Ala Gly Ile Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 103
<211> 336
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 4-1B3 VL
<400> 103
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gaccctcctg catagtaatg gattcaacta tttggattgg 120
tacctgcaga agccagggca gtctccacaa ctcctgatgt atttgggctc tagccgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcgggca cagatttcac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaaactct acaaactcct 300
ccggctttcg gcggagggac caaggtggag atcaaa 336
<210> 104
<211> 112
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 4-1B3 VL
<400> 104
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Thr Leu Leu His Ser
20 25 30
Asn Gly Phe Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Met Tyr Leu Gly Ser Ser Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Thr
85 90 95
Leu Gln Thr Pro Pro Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 105
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 21-G1 VH
<400> 105
caggtccagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgacgatt 60
tcctgcgagg cgtctggata caacttcatc agctactata tacactgggt gcgacaggcc 120
cctggacaag gccttgagtg gatgggattc gtcgtcccta gtggtggtgc cgcaggctac 180
acacagaagt tccagggcag actcaccgtg accagggaca cgtccacgag cacagtctac 240
atggacctga acagcctgac atctgacgac acggccgtgt attactgtgt gcgagaaatg 300
agtggtggct ggtttgattt ctggggccag ggaaccctgg tcaccgtctc ctcg 354
<210> 106
<211> 118
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 21-G1 VH
<400> 106
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Thr Ile Ser Cys Glu Ala Ser Gly Tyr Asn Phe Ile Ser Tyr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Phe Val Val Pro Ser Gly Gly Ala Ala Gly Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Leu Thr Val Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Asp Leu Asn Ser Leu Thr Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Arg Glu Met Ser Gly Gly Trp Phe Asp Phe Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 107
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 21-G1 VL
<400> 107
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagta ccccgatcac cttcggccaa 300
gggacacgac tggagattaa a 321
<210> 108
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 21-G1 VL
<400> 108
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105
<---
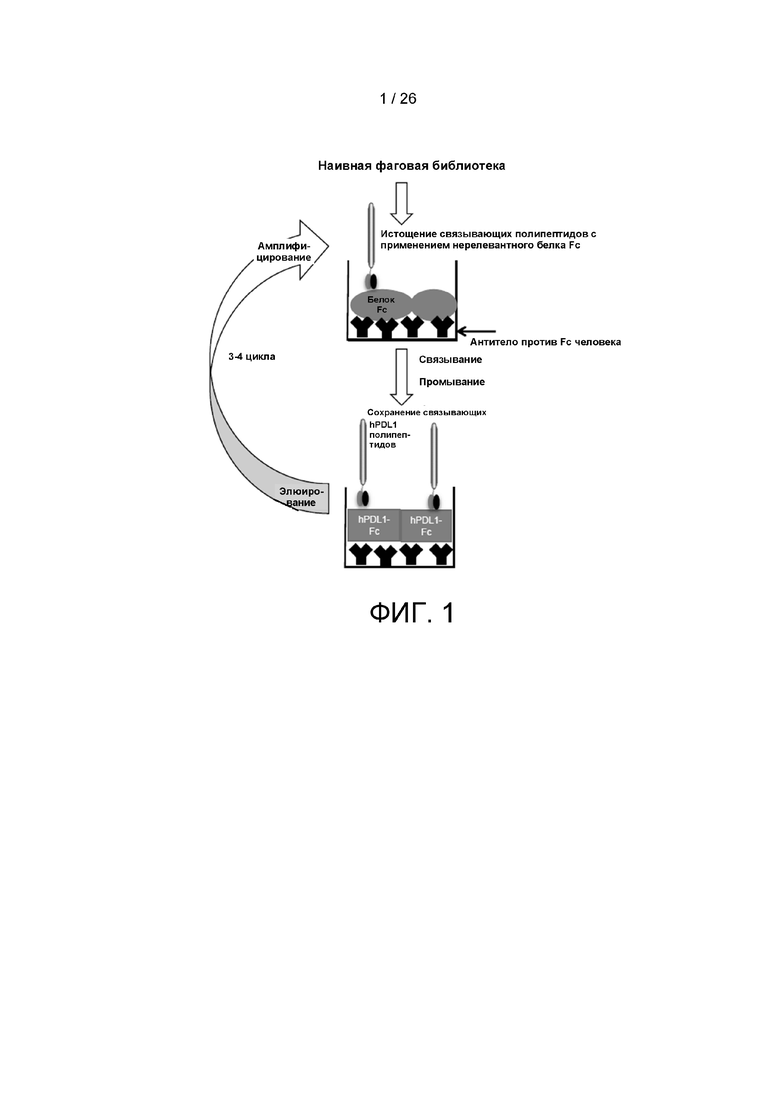
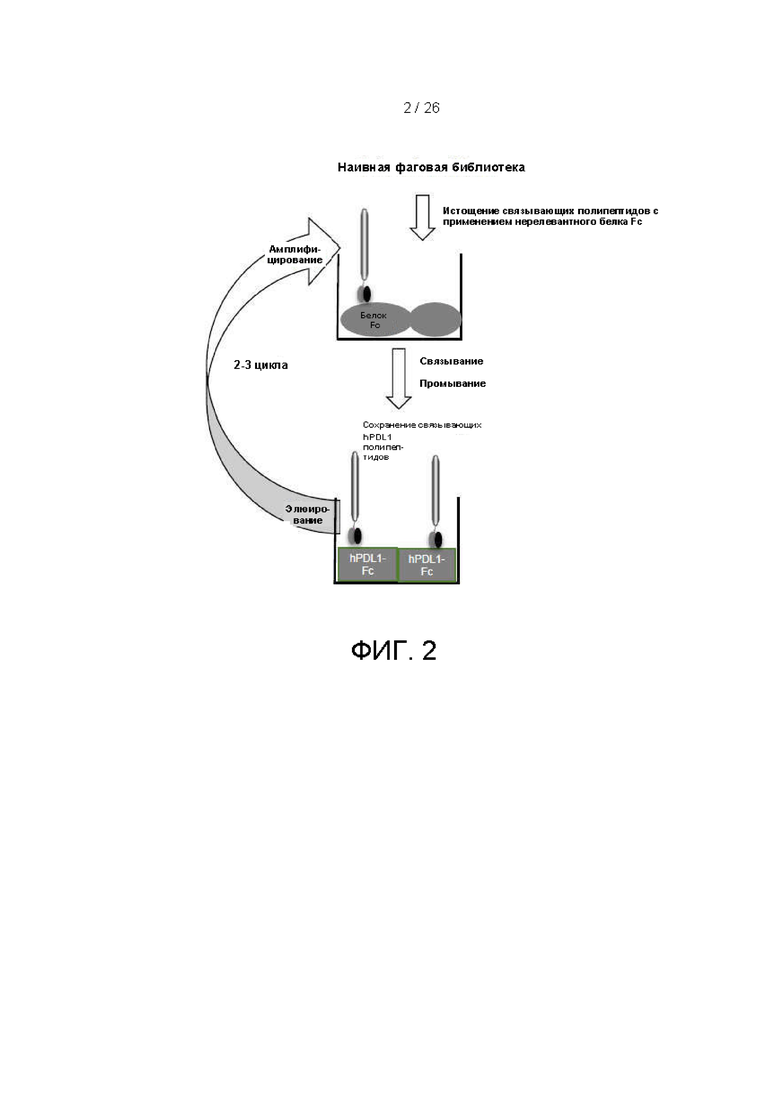
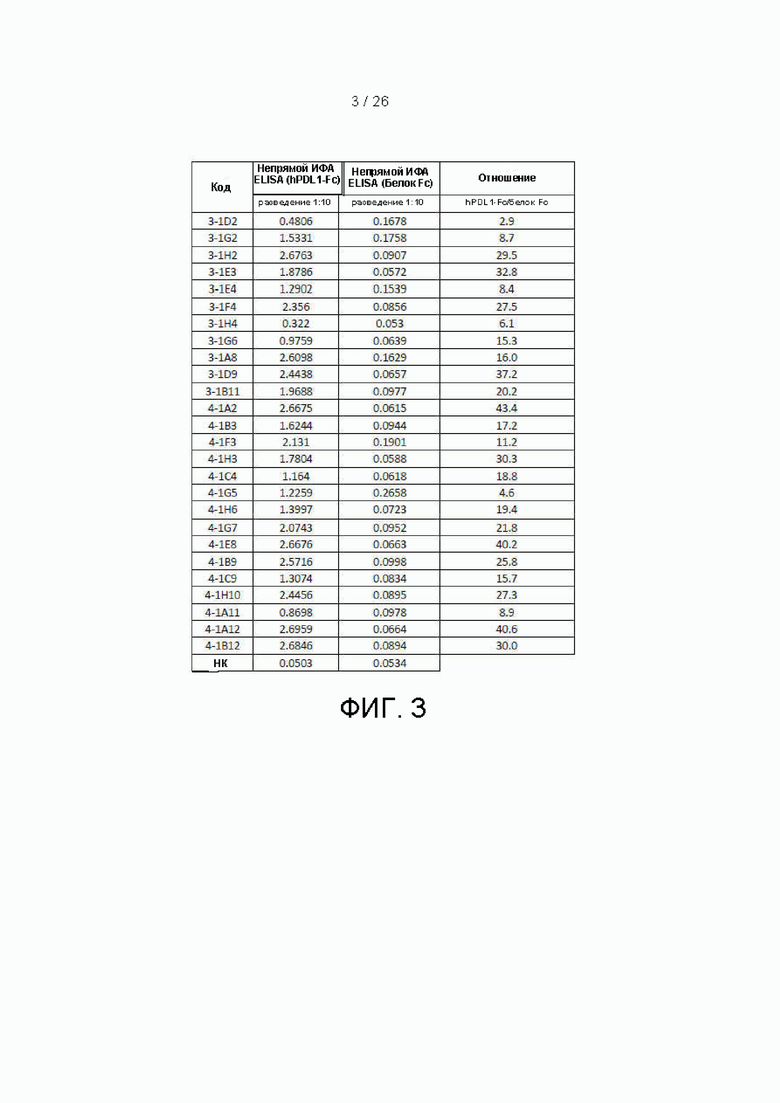
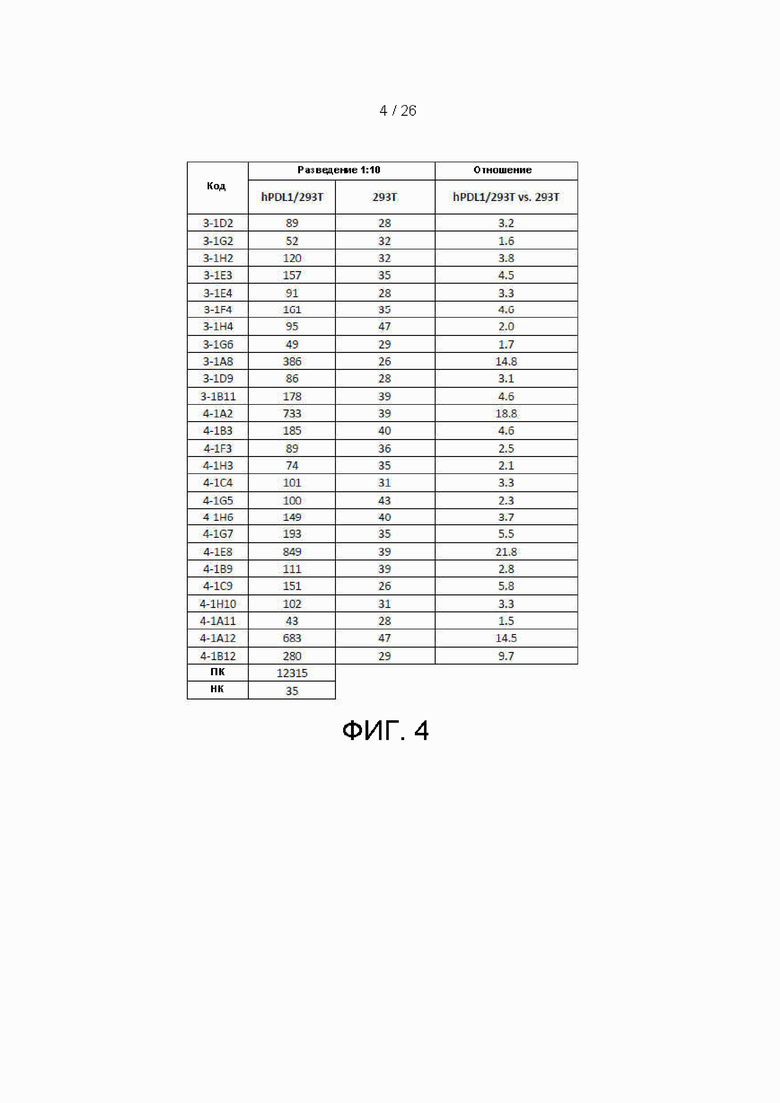
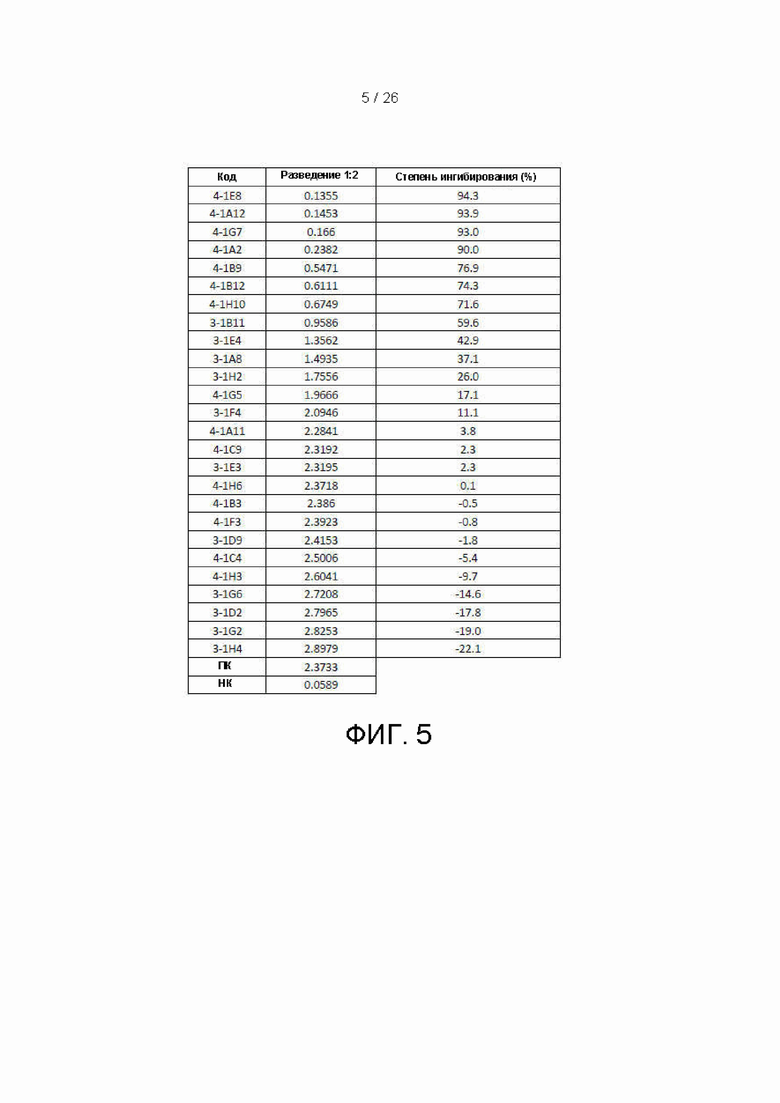
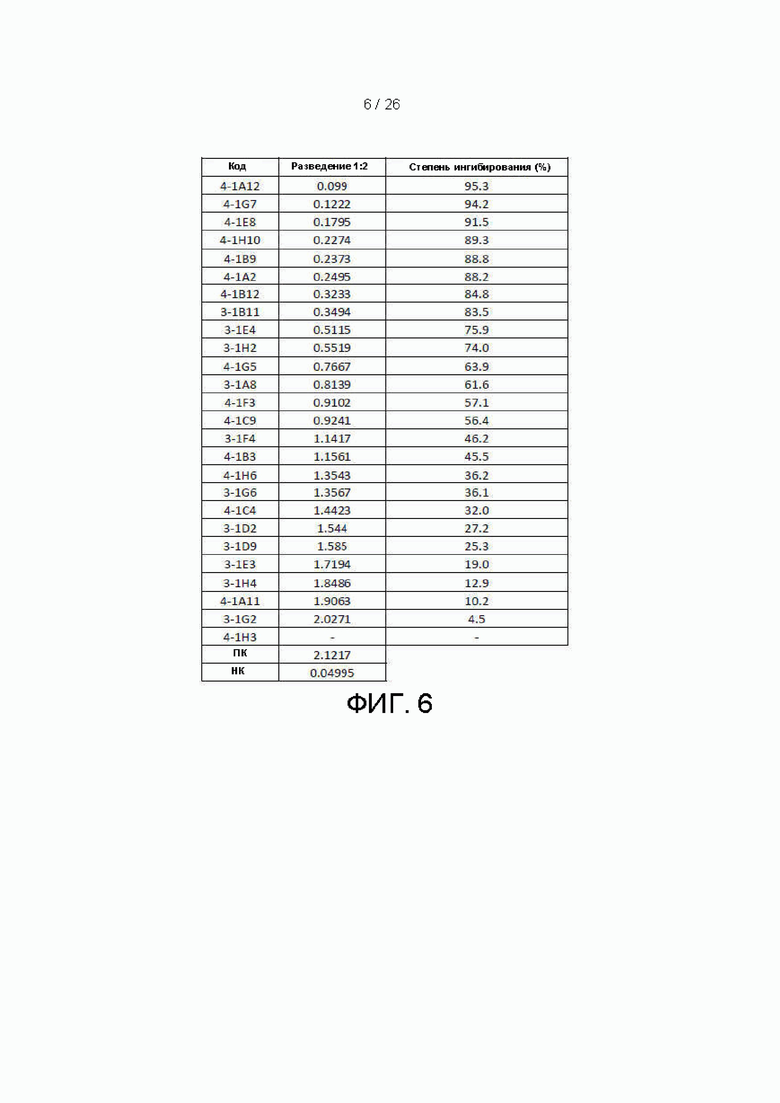
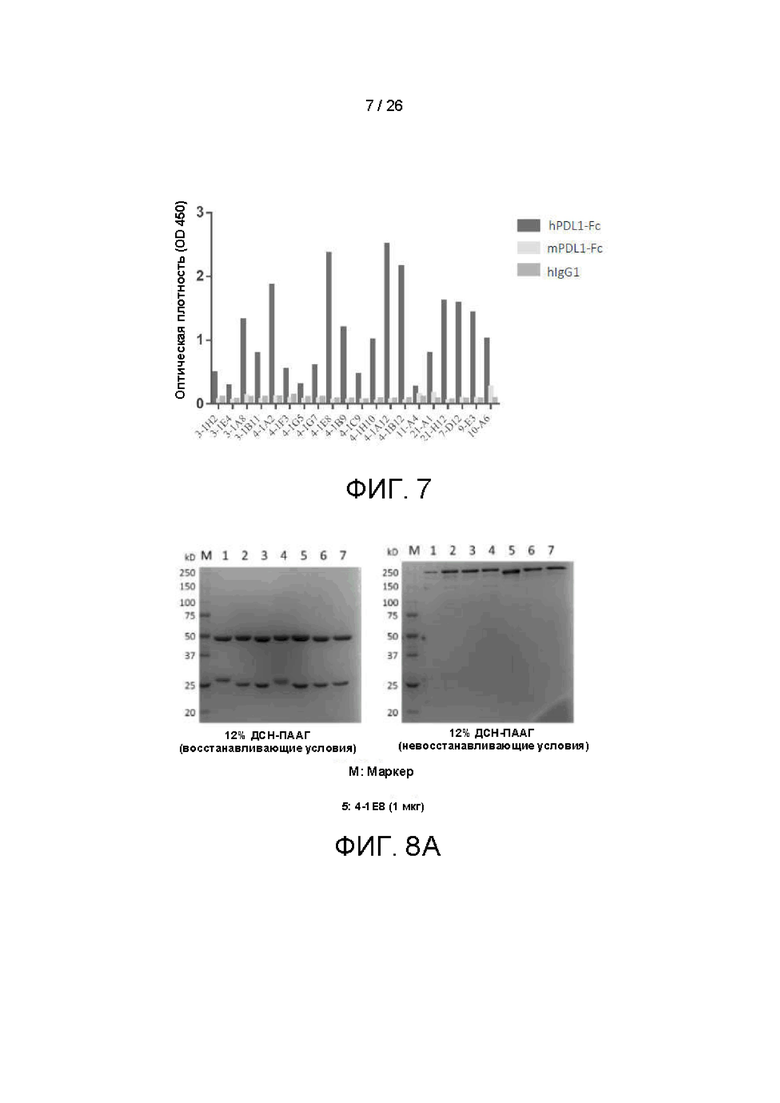
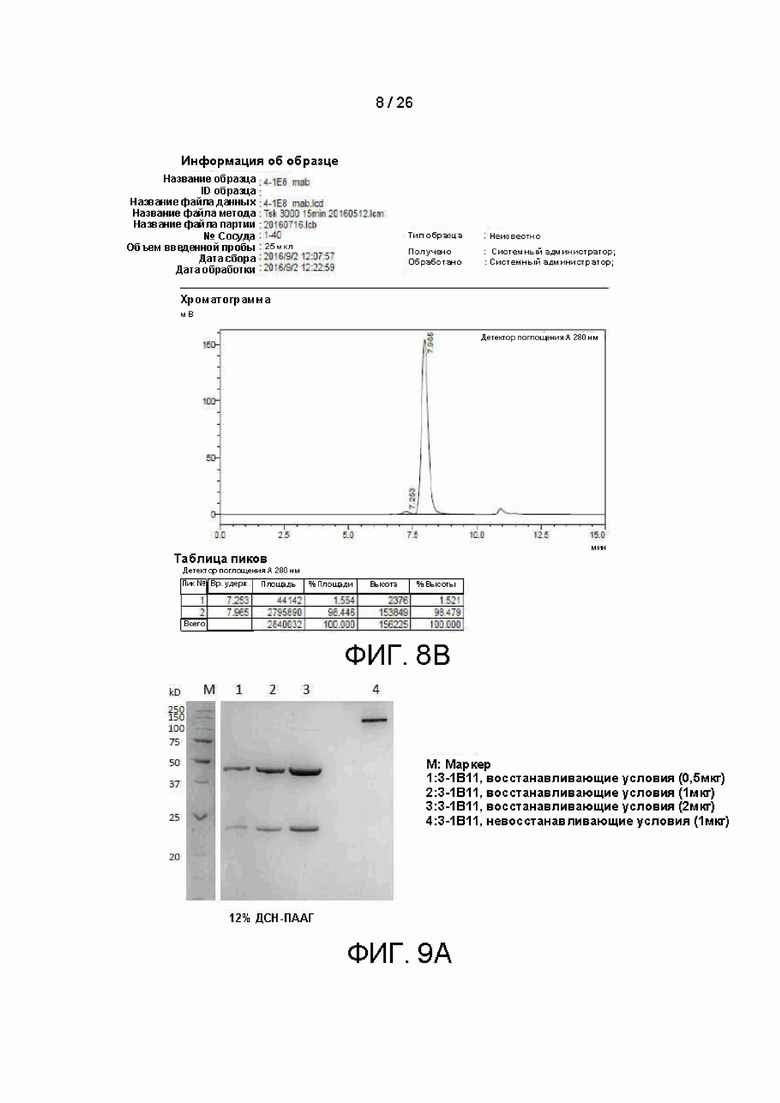
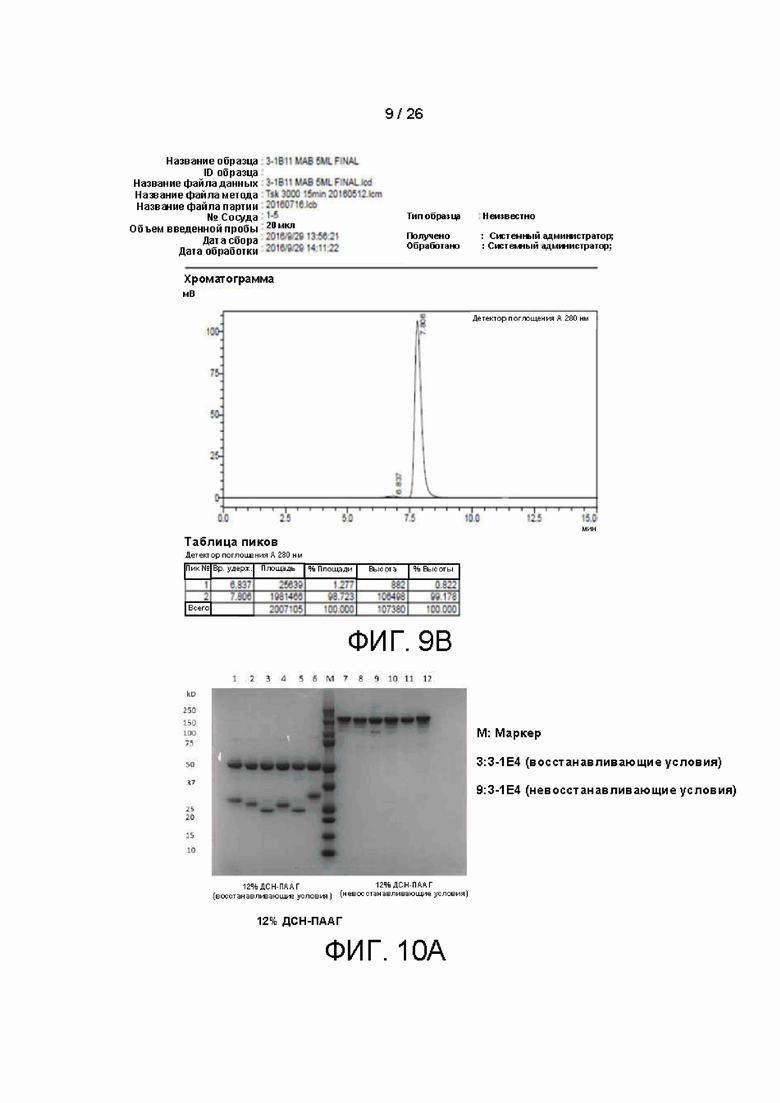
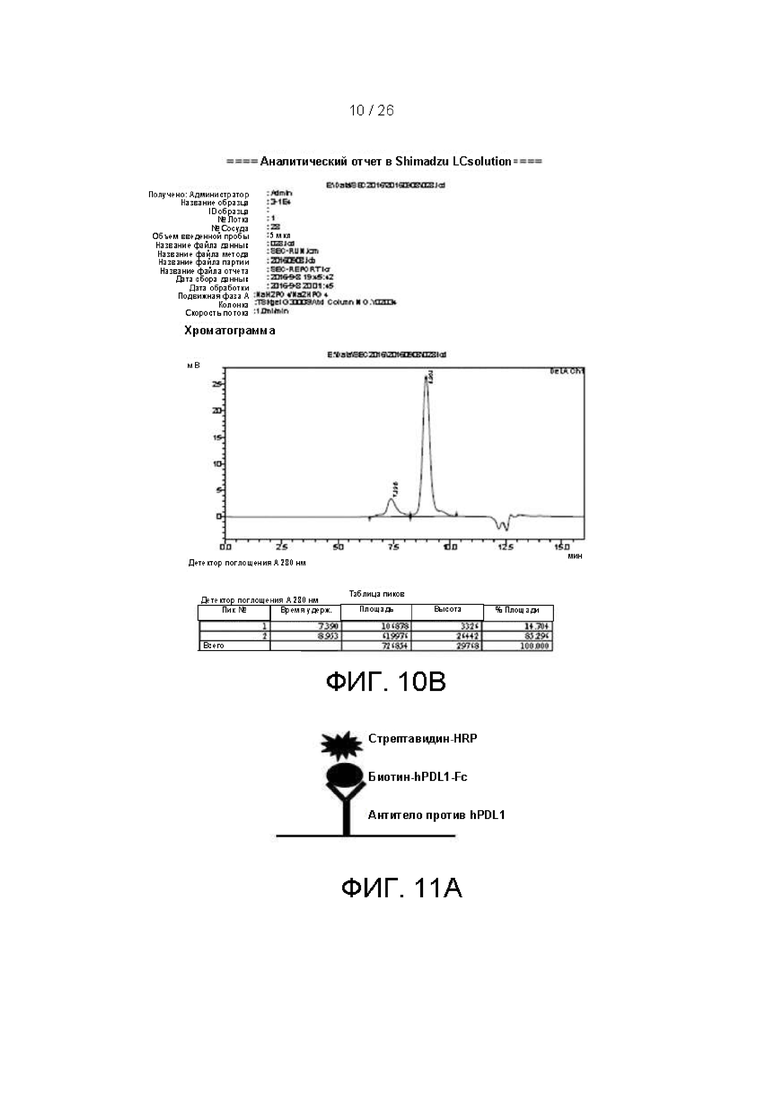
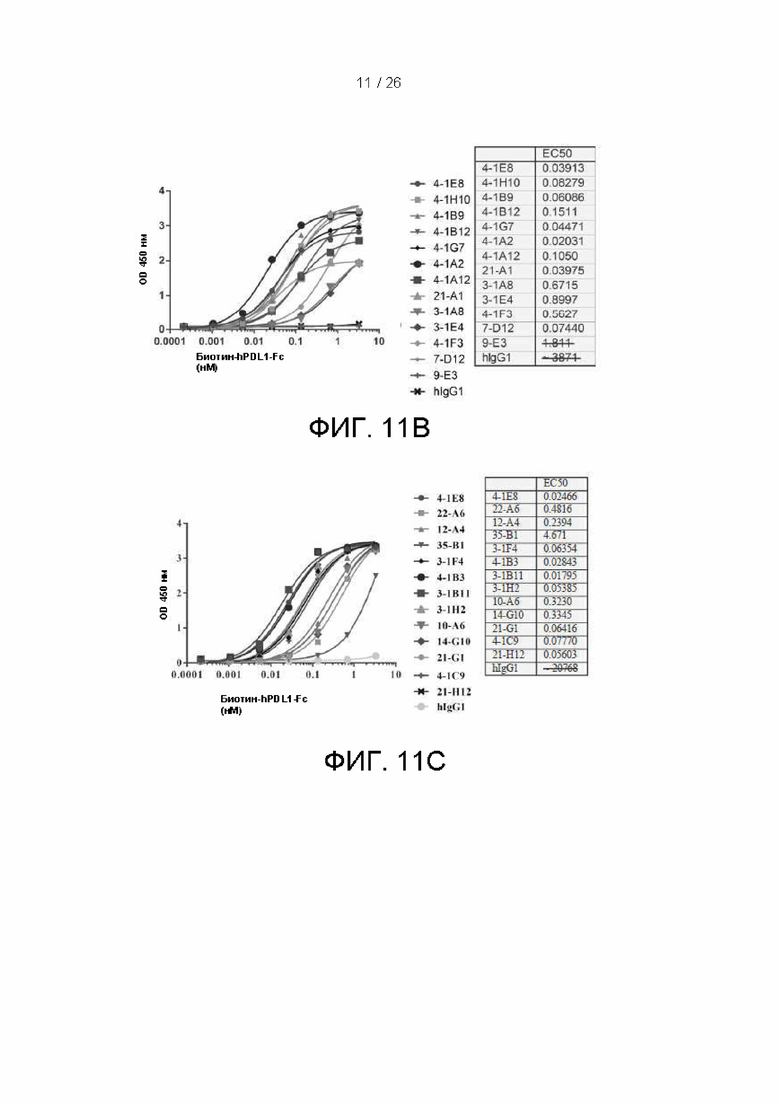
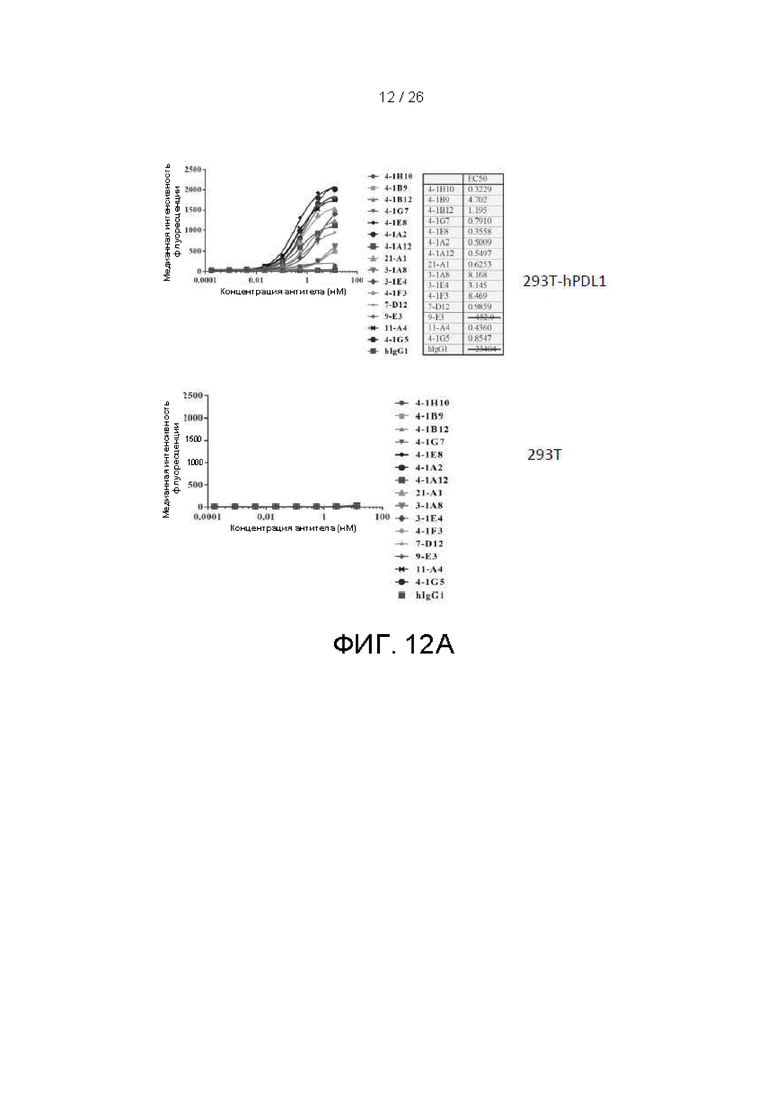
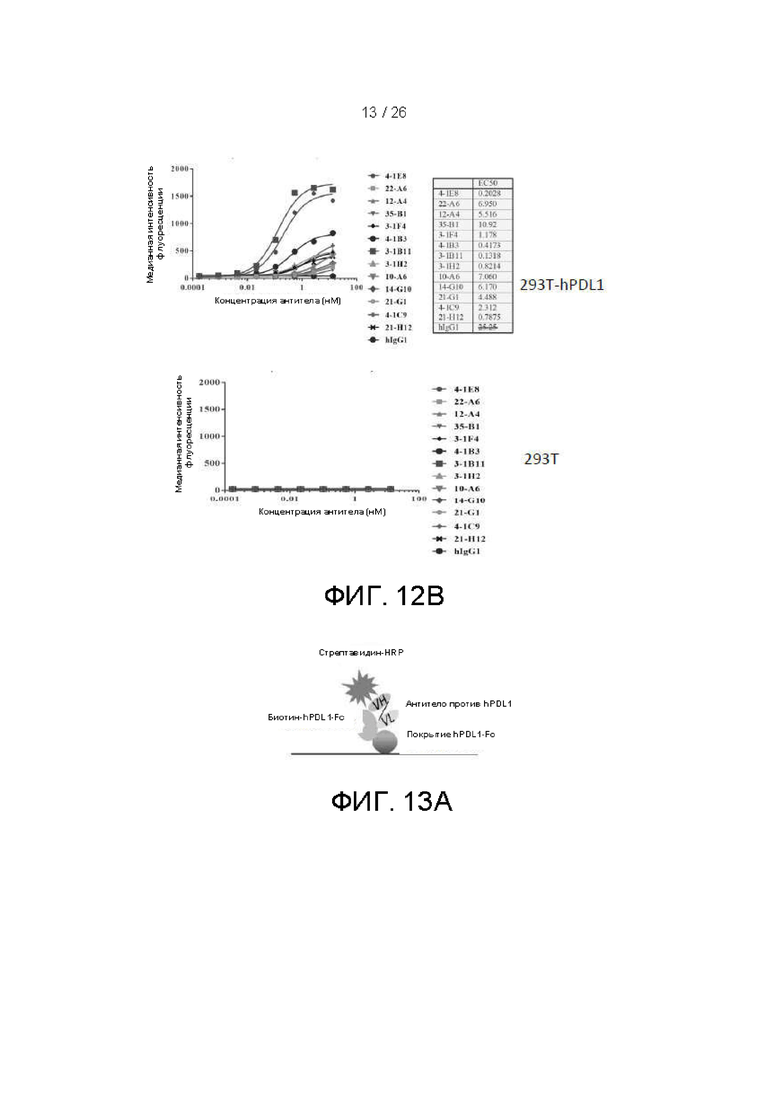
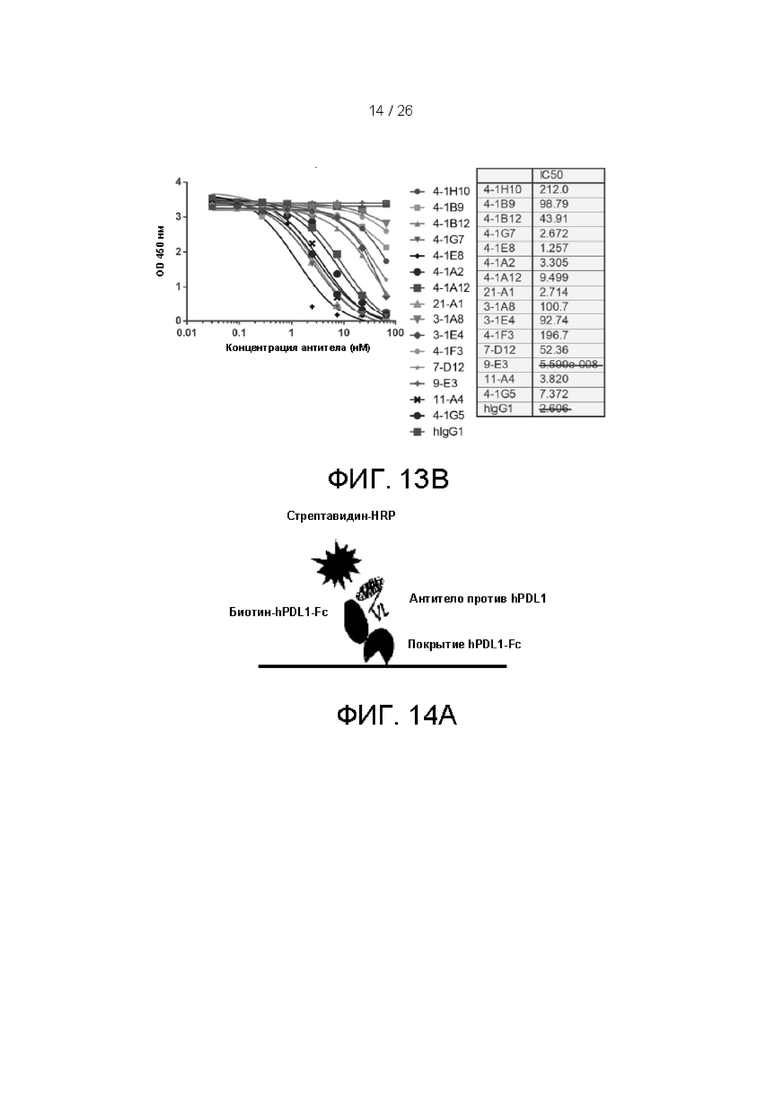
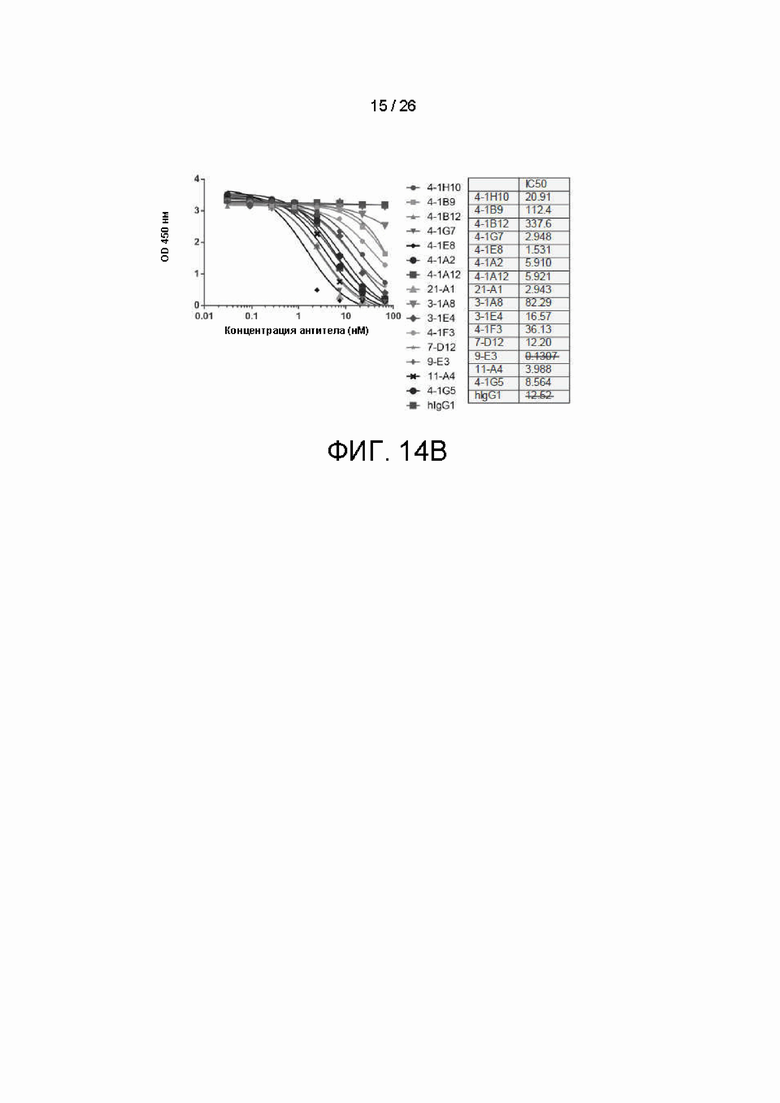

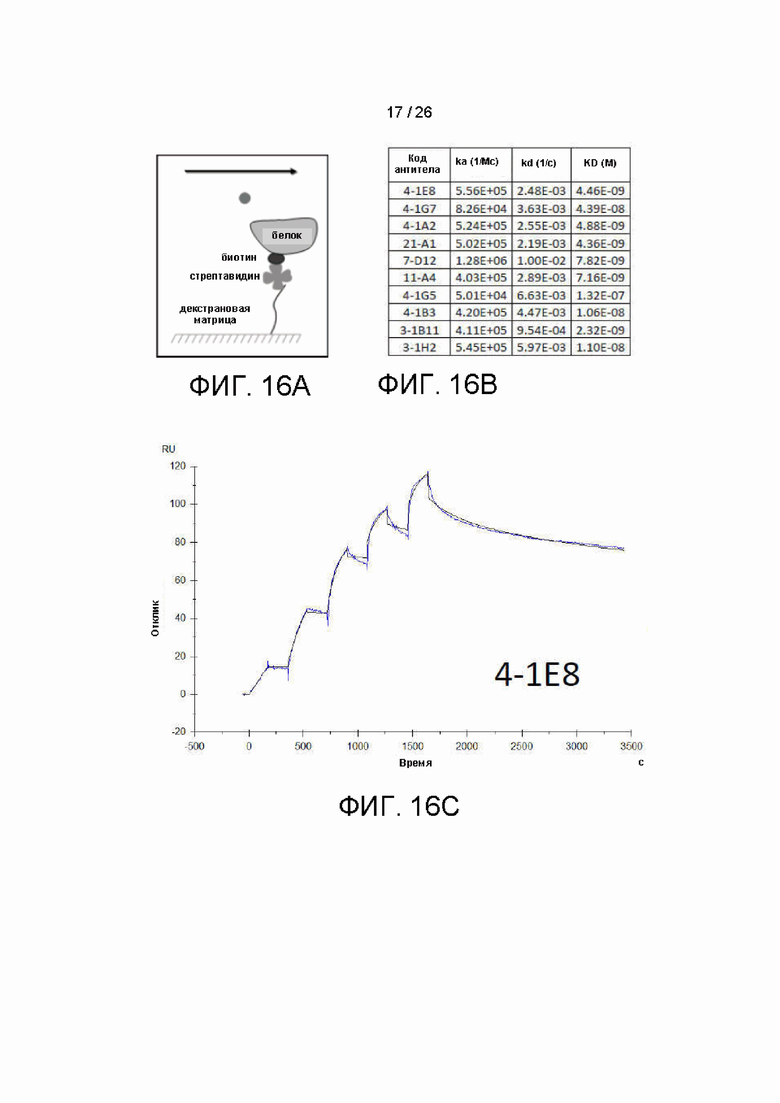
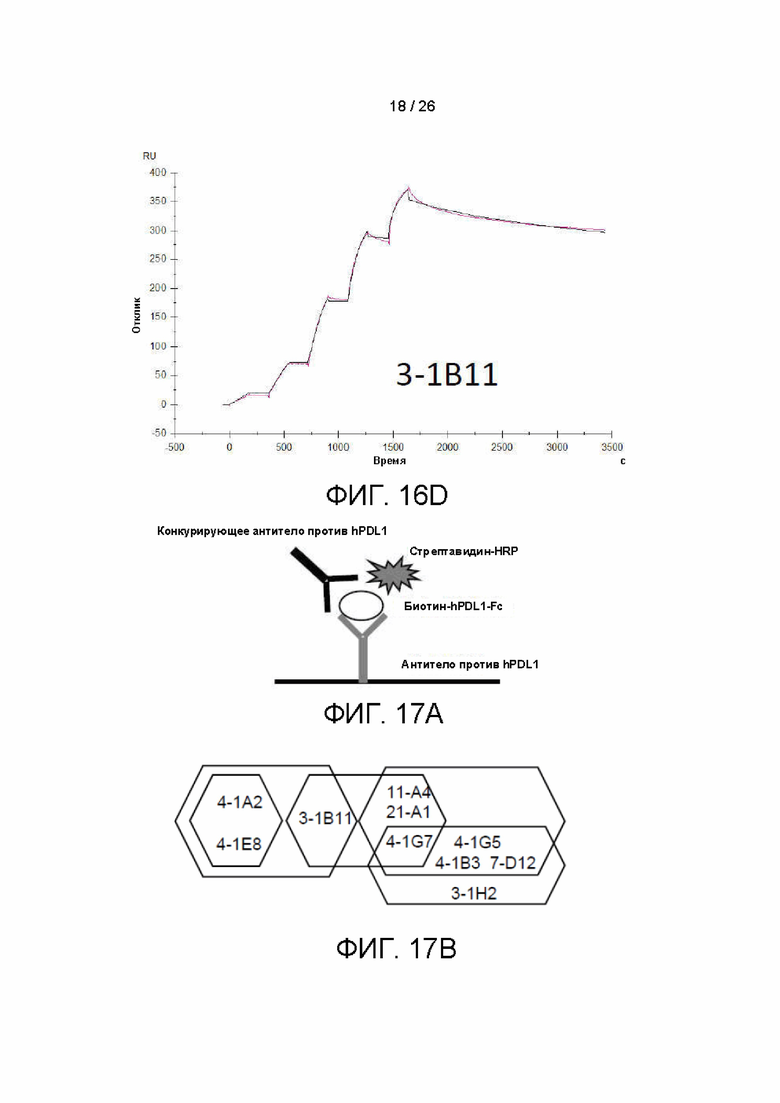
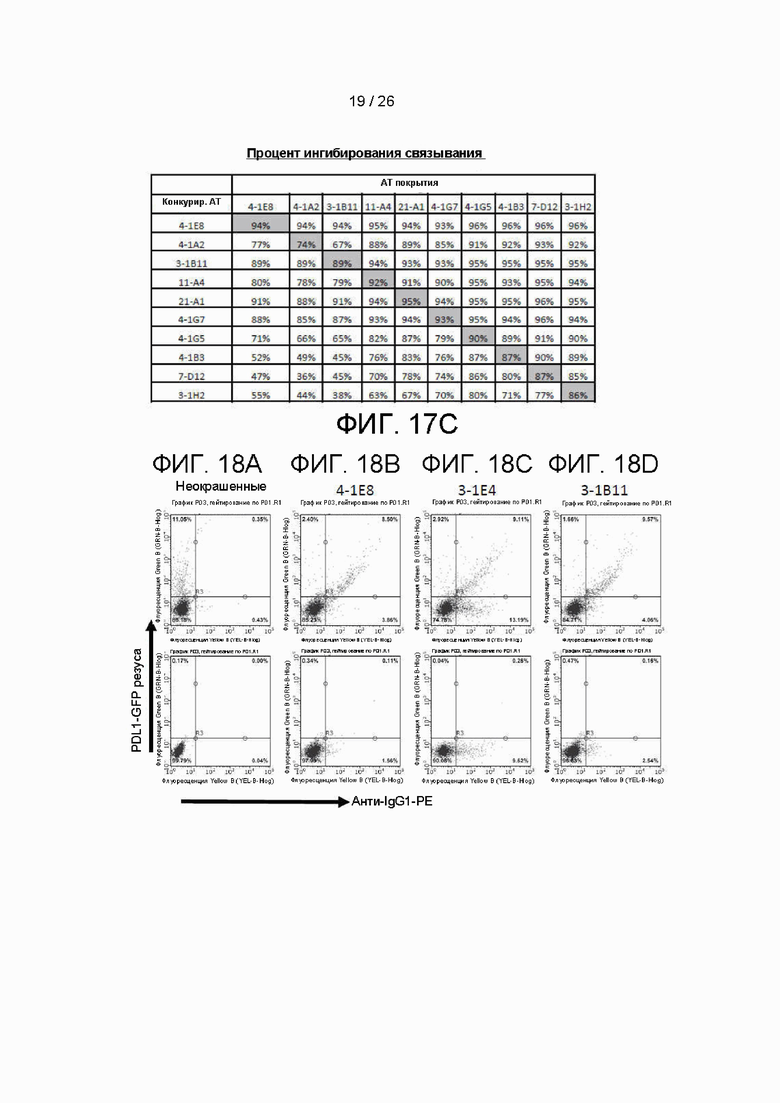
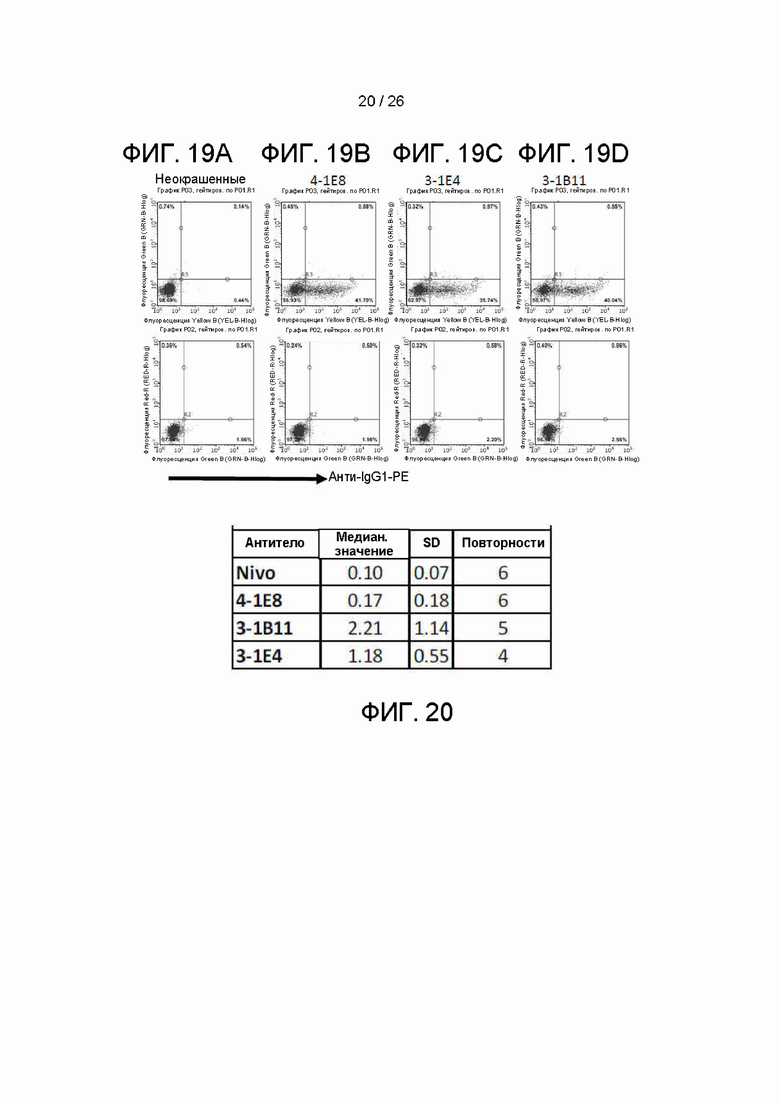

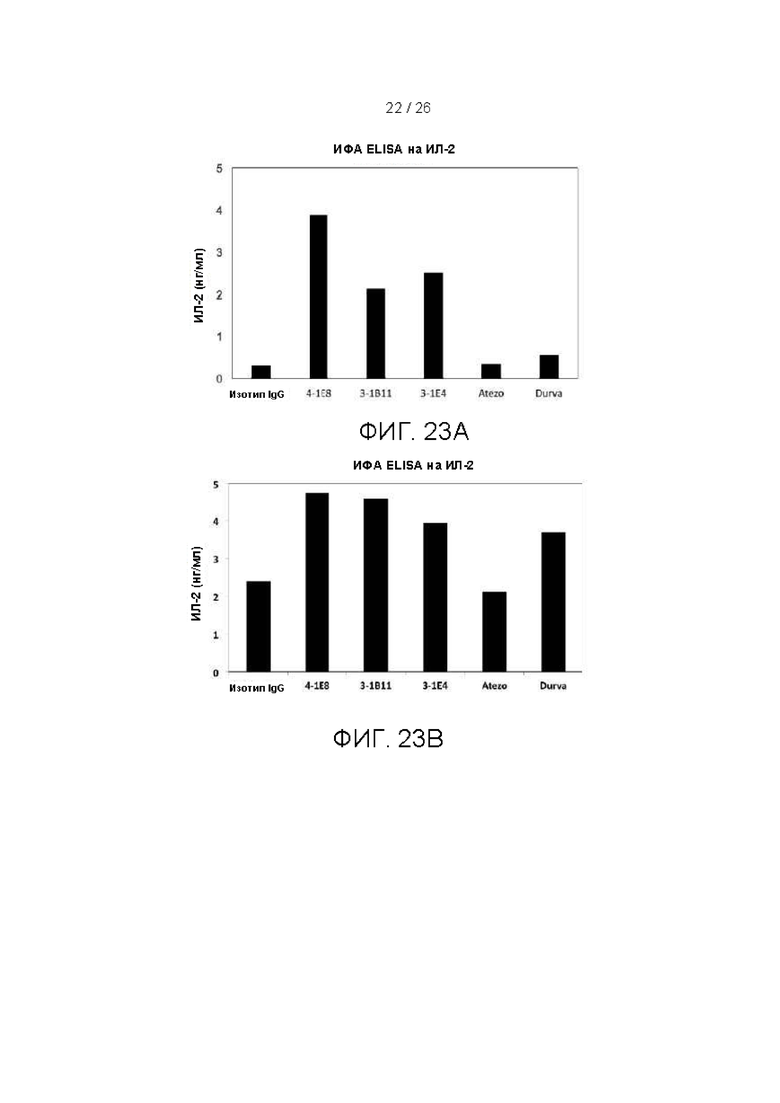

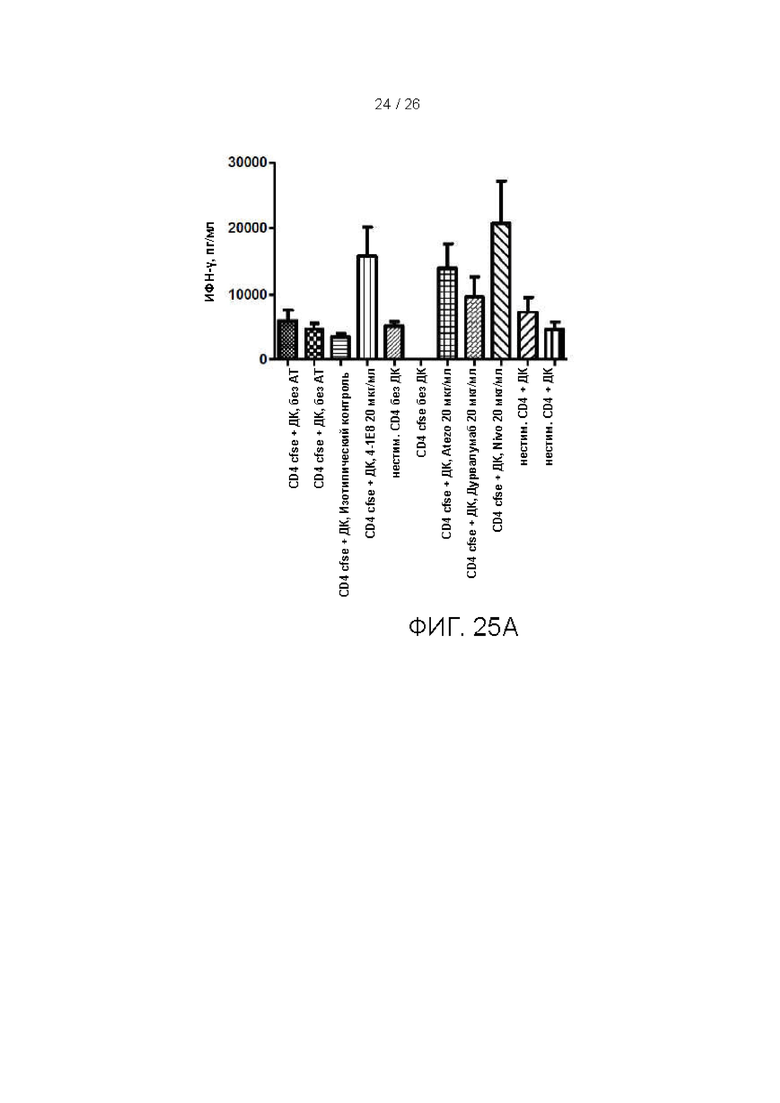
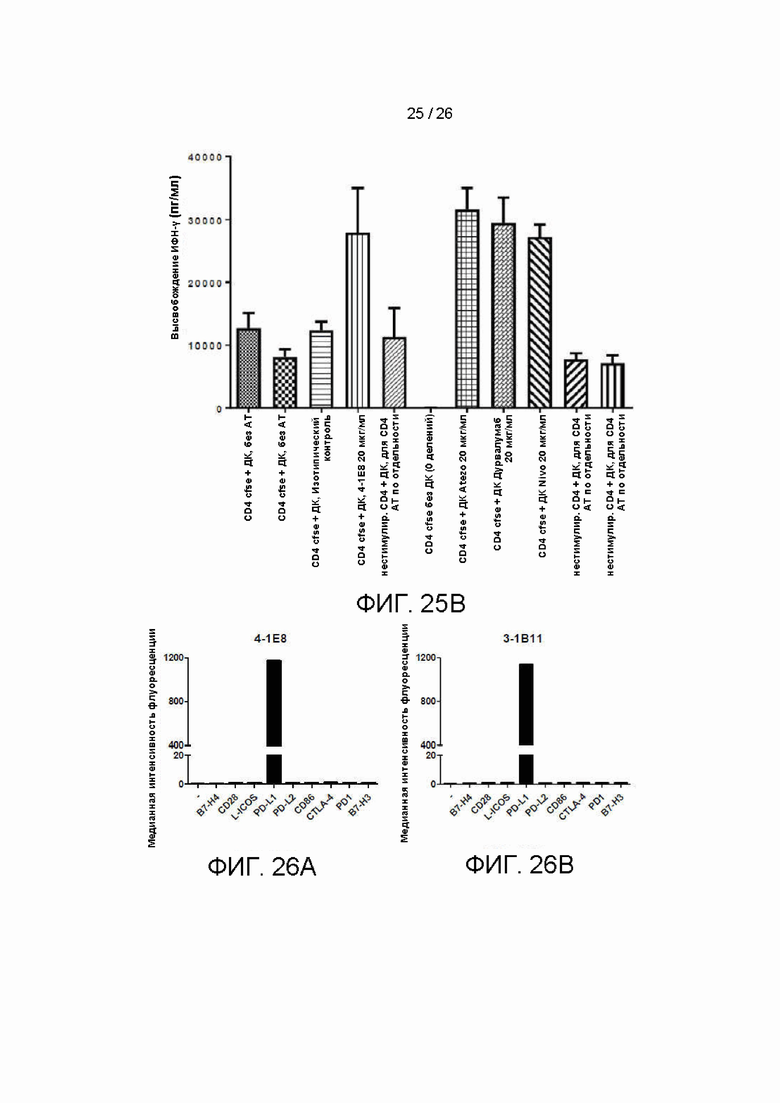
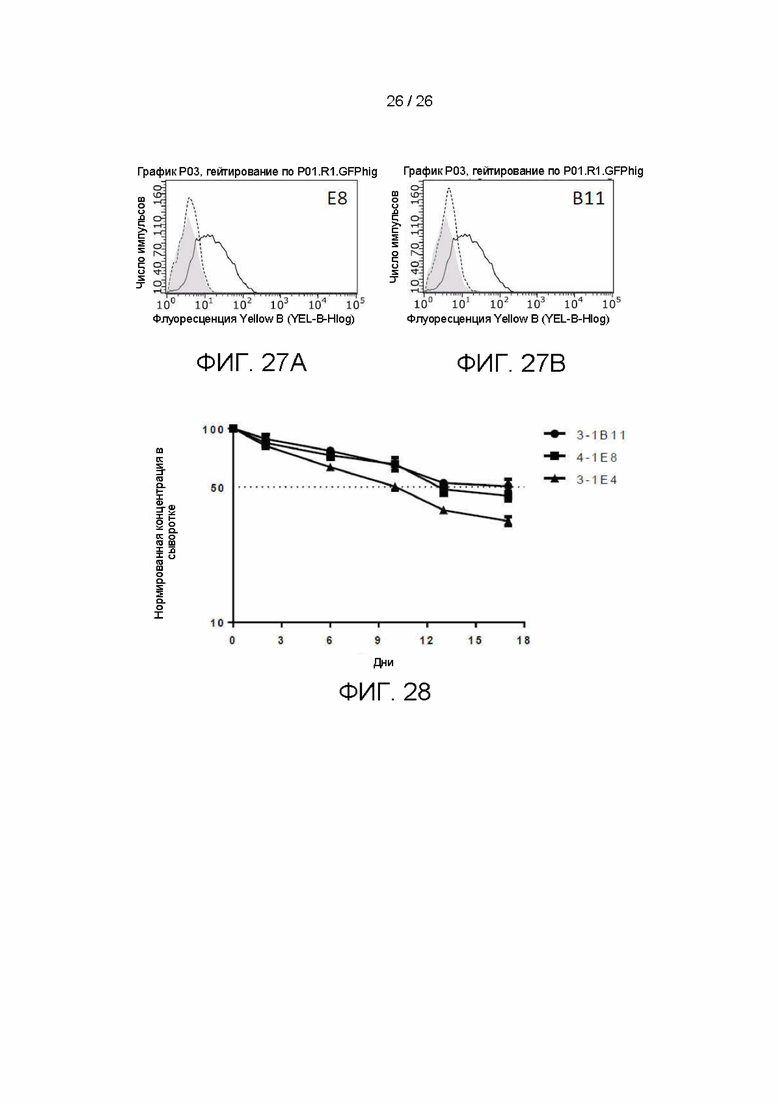
Группа изобретений относится к биотехнологии. Предложены: антигенсвязывающий полипептид, который связывается с эпитопом PD-L1 человека, содержащий вариабельный домен тяжелой цепи и вариабельный домен легкой цепи, причем их соответствующие последовательности по существу состоят из пар последовательностей, выбранных из определенной группы, молекула нуклеиновой кислоты, кодирующая антигенсвязывающий полипептид, фармацевтическая композиция для применения в ингибировании биологической активности PD-L1, а также способ терапевтического лечения субъекта. Изобретение применяется для лечения рака, содержащего часть опухолевых клеток, экспрессирующих детектируемое количество PD-L1. 4 н. и 8 з.п. ф-лы, 28 ил., 27 пр.
1. Антигенсвязывающий полипептид, который связывается с эпитопом PD-L1 человека, содержащий вариабельный домен тяжелой цепи и вариабельный домен легкой цепи, причем их соответствующие последовательности по существу состоят из пар последовательностей, выбранных из группы, состоящей из:
(a) SEQ ID NO: 18 и SEQ ID NO: 20;
(b) SEQ ID NO: 42 и SEQ ID NO: 44;
(с) SEQ ID NO: 34 и SEQ ID NO: 36;
(d) SEQ ID NO: 22 и SEQ ID NO: 24;
(e) SEQ ID NO: 2 и SEQ ID NO: 4;
(f) SEQ ID NO: 62 и SEQ ID NO: 64;
(g) SEQ ID NO: 82 или SEQ ID NO: 84;
(h) SEQ ID NO: 70 и SEQ ID NO: 72;
(i) SEQ ID NO: 50 и SEQ ID NO: 52;
(j) SEQ ID NO: 102, и SEQ ID NO: 104;
(k) SEQ ID NO: 30 и SEQ ID NO: 32;
(l) SEQ ID NO: 6 и SEQ ID NO: 8;
(m) SEQ ID NO: 10 и SEQ ID NO: 12;
(n) SEQ ID NO: 14 и SEQ ID NO: 16;
(o) SEQ ID NO: 26 и SEQ ID NO: 28;
(p) SEQ ID NO: 38 и SEQ ID NO: 40;
(q) SEQ ID NO: 46 и SEQ ID NO: 48;
(r) SEQ ID NO: 54 и SEQ ID NO: 56;
(s) SEQ ID NO: 58 и SEQ ID NO: 60;
(t) SEQ ID NO: 66 и SEQ ID NO: 68;
(u) SEQ ID NO: 74 и SEQ ID NO: 76;
(v) SEQ ID NO: 78 и SEQ ID NO: 80;
(w) SEQ ID NO: 86 и SEQ ID NO: 88;
(x) SEQ ID NO: 90 и SEQ ID NO: 92;
(y) SEQ ID NO: 94 и SEQ ID NO: 96;
(z) SEQ ID NO: 98 и SEQ ID NO: 100;
и
(aa) SEQ ID NO: 106 и SEQ ID NO: 108.
2. Антигенсвязывающий полипептид по п. 1, отличающийся тем, что вариабельный домен тяжелой цепи и вариабельный домен легкой цепи указанного полипептида имеют аминокислотные последовательности, состоящие из SEQ ID NO: 18 и SEQ ID NO: 20.
3. Антигенсвязывающий полипептид по п. 1, отличающийся тем, что вариабельный домен тяжелой цепи и вариабельный домен легкой цепи имеют аминокислотные последовательности, состоящие из SEQ ID NO: 42 и SEQ ID NO: 44.
4. Антигенсвязывающий полипептид по п. 1, отличающийся тем, что вариабельный домен тяжелой цепи и вариабельный домен легкой цепи имеют аминокислотные последовательности SEQ ID NO: 34 и SEQ ID NO: 36.
5. Антигенсвязывающий полипептид по пп. 1-4, отличающийся тем, что указанный полипептид является полностью человеческим антителом или гуманизированным антителом.
6. Антигенсвязывающий полипептид по п. 5, отличающийся тем, что указанное антитело содержит константную область человека, имеющую АЗКЦ-активность и/или КЗЦ-активность.
7. Молекула нуклеиновой кислоты, кодирующая антигенсвязывающий полипептид по пп. 1-4, причем указанная молекула нуклеиновой кислоты представляет собой молекулу ДНК или молекулу РНК.
8. Молекула нуклеиновой кислоты по п. 7, отличающаяся тем, что указанная молекула нуклеиновой кислоты содержит последовательности ДНК, кодирующие вариабельный домен тяжелой цепи и вариабельный домен легкой цепи, причем указанные последовательности ДНК состоят по существу из пар последовательностей, выбранных из группы, состоящей из: (a) SEQ ID NO: 17 и SEQ ID NO:19; (b) SEQ ID NO:33 и SEQ ID NO:35; и (c) SEQ ID NO:41 и SEQ ID NO:43.
9. Фармацевтическая композиция для применения в ингибировании биологической активности PD-L1, содержащая эффективное количество антигенсвязывающего полипептида по любому из пп. 1-6 и фармацевтически приемлемое вспомогательное вещество, фармацевтически приемлемый носитель или разбавитель.
10. Способ терапевтического лечения субъекта, нуждающегося в этом, от состояния, причем указанный способ включает введение указанному субъекту терапевтически эффективного количества антигенсвязывающего полипептида по любому из пп. 1-6, отличающийся тем, что состояние представляет собой рак, содержащий по меньшей мере часть опухолевых клеток, экспрессирующих детектируемое количество PD-L1.
11. Способ по п. 10, дополнительно включающий введение в комбинации с (a) антителами, нацеленными на другие иммуносупрессивные пути; (b) химиотерапией или радиационной терапией; (c) другими механизмами блокирования иммуносупрессивных путей; или (d) другими агентами для иммунотерапии.
12. Способ по любому из пп. 10, 11, отличающийся тем, что указанное состояние представляет собой рак млекопитающих, выбранный из группы, состоящей из: рака яичников, рака толстого кишечника, рака молочной железы, рака легких, миелом, опухолей ЦНС нейробластного происхождения, моноцитарных лейкозов, B-клеточных лейкозов, Т-клеточных лейкозов, B-клеточных лимфом, Т-клеточных лимфом, опухолей, происходящих из тучных клеток, меланомы, рака мочевого пузыря, рака желудка, рака печени, уротелиальной карциномы, кожной карциномы, рака почек, рака головы и шеи, рака поджелудочной железы и комбинаций перечисленного.
| CN 106432501 A, 22.02.2017 | |||
| CN 104736168 A, 24.06.2015 | |||
| CN 102264762 A, 30.11.2011 | |||
| CN 105777906 A, 20.07.2016 | |||
| CN 106496327 A, 15.03.2017 | |||
| CN 107973854 A, 01.05.2018 | |||
| АНТИТЕЛА К PD-L1 И ИХ ПРИМЕНЕНИЕ ДЛЯ УСИЛЕНИЯ ФУНКЦИИ Т-КЛЕТОК | 2009 |
|
RU2636023C2 |
Авторы
Даты
2023-10-02—Публикация
2019-08-20—Подача