Область техники настоящего изобретения
Настоящее изобретение относится к способу получения биомолекулы, включающему превращение белка в полимер в мультимерной форме. В частности, настоящее изобретение относится к способу получения биомолекулы, рекомбинантно экспрессируемой из клетки-хозяина, в многофункциональный мультиспецифический биомолекулярный полимер, имеющий повышенную продолжительность эффективности in vivo, с использованием системы убиквитинирования.
Предшествующий уровень техники настоящего изобретения
Получение биомолекул и/или низкомолекулярных химических соединений, включая белки, пептиды, полипептиды, антитела, ДНК и РНК в мультимерной форме, имеет различные преимущества. Например, физико-химические свойства белка, такие как растворимость, гелеобразование, термическая стабильность и рН-стабильность, могут быть улучшены путем связывания двух или более гомогенных или гетерологичных белков с использованием слияния или сшивающего линкера (или сшивающего агента). Например, CLEA (сшитый агрегат ферментов), лакказа, образованный путем множественного связывания через сшивающий агент, показал более повышенную стабильность и эффективность в ходе окисления крахмала, а CLEA другого фермента, нитрилгидратазы, показал превосходное увеличение активности при превращении акрилонитрила в акриламид и не терял активности в течение 36 рециклов.
Кроме того, многие белки образуют в клетках комплексы для выполнения сложных функций, что, как известно, связано с эффектом близости белков. Например, целлюлаза (Novozymes Cellic® CTec3), полученная путем получения ферментов, необходимых для расщепления лигноцеллюлозы, как например, целлюлаза, бета-глюкозидаза (β-глюкозидаза), гемицеллюлаза и т.п., в виде сложной смеси с использованием каркаса, как известно, проявляет 3,5-кратное и более повышение эффекта при расщеплении лигноцеллюлозы. Кроме того, такой белок в мультимерной форме проявляет эффект каналирования. То есть, если ферменты, участвующие в сопряженной реакции, находятся рядом друг с другом, перенос промежуточного соединения является эффективным, и эффективность всей реакции значительно увеличивается. Кроме того, предполагается желательным для повышения его эффективности использование гомогенного или гетерологичного белка в мультимерной форме при анализе любого вещества с использованием белка, иммобилизованного на шарике или подложке, или при разделении и/или очистке вещества, подлежащего обнаружению.
Как описано выше, хотя белок в мультимерной форме обеспечивает различные преимущества в промышленных и медицинских применениях, известно, что сложно получить белок, имеющий такую структуру. Например, существует способ разработки и получения мультимерного белка в качестве нового фермента слияния путем конструирования каркаса на генетической стадии. Однако, поскольку новый белок должен быть разработан и произведен, его разработка занимает много времени, и в реальности сложно слить два или более ферментов. Кроме того, в случае способа получения белковой мультимерной конструкции (CLEA) с использованием химического сшивающего агента активность может подавляться, поскольку химическая связь не возникает в определенном месте, а может возникать в любом месте на поверхности белка. Белки, образующие мультимерную конструкцию, должны быть способны к получению посредством синтеза или микробной экспрессии, и активные сайты этих белков не должны нарушаться.
В качестве способа выделения и/или очистки представляющего интерес белка был предложен способ использования убиквитина. Это способ, при котором сначала ген, кодирующий белок, связанный с убиквитином, экспрессируется в прокариотических клетках с получением слитого белка, связанного с убиквитином, а затем обрабатывается ферментом, расщепляющим убиквитин, для эффективного отделения и очистки только представляющего интерес белка от белка слияния с убиквитином. Заявка на патент США № 10/504785 относится к экспрессии рекомбинантного гена и очистке экспрессированного белка, и в ней описывается получение слитого белка, в котором нуклеотид, кодирующий С-концевой домен убиквитин-подобного белка (Ubl), функционально связан с нуклеотидом, кодирующим представляющий интерес белок, и экспрессируется в клетке-хозяине. Корейская патентная заявка № 10-2005-0050824 описывает использование убиквитина в качестве партнера по слиянию при экспрессии рекомбинантного белка. Кроме того, заявка на патент Кореи № 10-2015-0120852 относится к использованию колонки с убиквитином для очистки белка и описывает, что полиубиквитиновая цепь загружается в колонку, и белок очищается с использованием убиквитинирования in vitro, включая E2. Кроме того, заявка на патент США № 12/249334 предназначена для решения проблемы растворимости в воде и укладки, которая является проблемой при получении путем экспрессии рекомбинантного белка, и описывает использование SUMO, имеющего сайт расщепления, распознаваемый протеазой Ulp1 (Ubl -специфическая протеаза 1) для облегчения экспрессии, выделения и очистки рекомбинантного белка и для повышения активности белка. Однако эти способы описывают только использование убиквитина для экспрессии белка и не описывают и не предполагают получение белка в мультимерной форме, а поскольку белок, который нужно отделить и очистить, случайным образом связывается с убиквитином, эти способы все же имеют ограничения эффективности разделения или анализа.
С другой стороны, биомолекулы, такие как белки или пептиды или рекомбинантно полученные белки или пептиды, являются нестабильными молекулами, которые демонстрируют короткий период полувыведения из сыворотки. В частности, эти белки или пептиды очень нестабильны при получении в водных растворах для диагностических или терапевтических целей. Кроме того, такие белковые или пептидные лекарственные средства невыгодны из-за их короткого периода полувыведения из сыворотки in vivo и должны вводиться с высокой частотой или в более высокой дозе. Однако частый прием препарата вызывает различные побочные эффекты и причиняет больному дискомфорт. Например, известно много проблем, возникающих у пациентов, которым требуется частое введение лекарственных средств, например, у пациентов с диабетом или у пациентов, страдающих рассеянным склерозом. Были изучены различные способы повышения стабильности или периода полувыведения таких биомолекул in vivo. Например, компонент, способный увеличить время полувыведения, ковалентно присоединен к биомолекуле, такой как белок или пептид. Например, хорошо известно, что присоединение полимеров, таких как полиэтиленгликоль или ПЭГ, к полипептидам может увеличить время полувыведения этих пептидов в сыворотке.
Соответственно, авторы настоящего изобретения прилагали неустанные усилия для разработки способа получения мультифункционального мультиспецифического биомолекулярного полимера, имеющего высокую степень интеграции и повышенную продолжительность эффективности или период полувыведения in vivo без ингибирования активности белка. В результате биомолекула, связанная с убиквитином, рекомбинантно экспрессировалась из клетки-хозяина и реагировала in vitro с ферментом, связанным с убиквитинированием, с образованием мультифункционального мультимерного биомолекулярного полимера, связанного с полиубиквитиновым каркасом. На основании вышеизложенного авторы настоящего изобретения создали настоящее изобретение.
Документы предшествующего уровня техники
Патентный документ
(Патентный документ 1) Заявка на патент Кореи № 10-2005-0050824
(Патентный документ 2) Заявка на патент Кореи № 10-2015-0120852
(Патентный документ 3) Заявка на патент США № 12/249,334
Подробное раскрытие настоящего изобретения
Техническая задача
Как описано выше, задачей настоящего изобретения является создание мультифункциональной мультиспецифической биомолекулы, имеющей повышенную продолжительность эффективности in vivo, путем связывания биомолекулы-мишени с полиубиквитиновым каркасом.
Другой задачей настоящего изобретения является предоставление способа получения мультифункциональной мультиспецифической биомолекулы, имеющей повышенную продолжительность эффективности in vivo, путем связывания биомолекулы-мишени с полиубиквитиновым каркасом.
Другой задачей настоящего изобретения является предоставление фармацевтической композиции, содержащей мультифункциональную мультиспецифическую биомолекулу.
Другой задачей настоящего изобретения является предоставление способа получения фармацевтической композиции, содержащей мультифункциональную мультиспецифическую биомолекулу.
Решение задача
Для достижения вышеуказанных целей настоящее изобретение обеспечивает мультифункциональный мультиспецифический мультимерный биомолекулярный полимер, который состоит из полиубиквитинового каркаса, образованного ковалентным связыванием двух или более убиквитинов, и от 2 до 10 биомолекул, содержащих связывающие фрагменты, каждый из которых специфичен для разных сайтов связывания, где биомолекула содержит активные сайты, которые специфически связываются с другими биомолекулами, низкомолекулярными химическими соединениями или наночастицами и непосредственно связана с N-концом, С-концом или как с N-концом, так и с С-концом убиквитина, или связана линкером, и носитель, который продлевает стабильность и/или продолжительность эффективности биомолекулы in vivo, непосредственно связанный с N- концом, С-концом или как с N-концом, так и с С-концом убиквитина или связанный линкером.
В одном варианте осуществления, относящемся к этому, линкер может представлять собой комбинацию от 1 до 30 повторов GGGGS или EAAAK, но без ограничения. В другом варианте осуществления, относящемся к этому, биомолекула, связанная с N-концом убиквитина, может быть дистальным концом мультимерного биомолекулярного полимера, и биомолекула, связанная с С-концом, N-концом или как с С-концом, так и с N-концом убиквитина может быть проксимальным концом мультимерного биомолекулярного полимера.
В контексте настоящего изобретения термин «полимер» относится к группе мономеров ряда биомолекул, связанных вместе. Полимер может быть линейным или разветвленным (разветвленная форма). Когда полимер является разветвленным, каждая полимерная цепь может называться «полимерным плечом». Конец полимерного плеча, связанный с фрагментом инициатора, является проксимальным концом, а конец полимерного плеча с растущей цепью является дистальным концом.
В контексте настоящего изобретения термин «линкер» относится к химическому фрагменту, который соединяет вместе две группы. Линкер может быть расщепляемым или нерасщепляемым. Расщепляемый линкер может быть гидролизуемым, ферментативно расщепляемым, рН-чувствительным, фотолабильным или дисульфидным линкером среди прочих. Другие линкеры включают гомобифункциональные и гетеробифункциональные линкеры.
В одном варианте осуществления настоящего изобретения носитель выполняет функцию увеличения продолжительности эффективности биомолекулы in vivo. Носитель может быть одним или несколькими, выбранными из группы, состоящей из альбумина, фрагмента антитела, Fc-домена, трансферрина, XTEN (генетическое слияние неточных повторяющихся пептидных последовательностей), CTP (карбоксиконцевой пептид), PAS (пролин-аланин-сериновый полимер), ELK (эластиноподобный пептид), HAP (гомоаминокислотный полимер), GLK (желатиноподобный белок), PEG (полиэтиленгликоль) и жирной кислоты, но без ограничения.
В другом варианте осуществления настоящего изобретения полиубиквитиновый каркас может быть образован путем ковалентного связывания донорного убиквитина, в котором один или несколько лизинов убиквитина замещены другими аминокислотами, включая аргинин или аланин, и акцепторного убиквитина, в котором 6-й, 11-й, 27-й, 29-й, 33-й, 48-й или 63-й лизин от N-конца замещен другими аминокислотами, включая аргинин или аланин. Кроме того, в другом варианте осуществления настоящего изобретения 73-й лейцин от N-конца убиквитина может быть заменен другими аминокислотами, включая пролин.
В контексте настоящего изобретения термин «биомолекула» относится к молекулам, обладающим биологической активностью в живом организме. В одном варианте осуществления настоящего изобретения биомолекула может быть выбрана из группы, состоящей из инсулина, аналога инсулина, глюкагона, глюкагоноподобных пептидов, GLP -1 и двойного агониста глюкагона, GLP -1 и двойного агониста GLP, GLP -1 и глюкагона и тройного агониста GLP, экзендин-4, аналога экзендина-4, пептида, секретирующего инсулин, и его аналога, гормона роста человека, гормона, высвобождающий гормон роста (GHRH), пептида, высвобождающего гормон роста, гранулоцитарного колониестимулирующего фактора (G-CSF), пептида против ожирения, рецептора, связанного с G-белком, лептина, GIP (желудочный ингибирующий полипептид), интерлейкинов, рецепторов интерлейкина, белков, связывающих интерлейкин, интерферонов, рецепторов интерферона, белков, связывающих цитокины, активатора макрофагов, пептида макрофагов, В-клеточного фактора, Т-клеточного фактора, супрессивного фактора аллергии, гликопротеина некроза клеток, иммунотоксина, лимфотоксина, фактора некроза опухоли (TNF), фактора ингибирования опухоли, фактора роста метастазов, альфа-1-антитрипсина, альбумина, α-лактальбумина, аполипопротеина-Е, эритропоэтина (EPO), высокогликозилированного эритропоэтина, ангиопоэтинов, гемоглобина, тромбина, пептида, активирующего рецептор тромбина, тромбомодулина, факторов крови VII, VIIa , VIII, IX и XIII, активаторов плазминогена, фибрин-связывающего пептида, урокиназы, стрептокиназы, гирудин, белка С, С-реактивного белка, ингибитора ренина, ингибитора коллагеназы, супероксиддисмутазы, тромбоцитарного фактора роста, эпителиального фактора роста, эпидермального фактора роста, ангиостатина, ангиотензина, костного морфогенетического фактора роста, костного морфогенетического белка, кальцитонина, атриопептина, фактора, индуцирующего хрящ, элкатонина, активатора соединительной ткани, ингибитора пути тканевого фактора, фолликулостимулирующего гормона (FSH), лютеинизирующего гормона (LH), гормона высвобождения лютеинизирующего гормона (LHRH), факторов роста нервов, паратиреоидного гормона (PTH), релаксина, секретина, соматомедина, гормона коры надпочечников, холецистокинина, панкреатического полипептида, гастрин-высвобождающего пептида, кортикотропин-высвобождающего фактора, тироид-стимулирующего гормона (TSH), аутотаксина, лактоферрина, миостатина, рецептора, антагониста рецептора, фактора роста фибробластов, адипонектина, антагониста рецептора интерлейкина, антигена клеточной поверхности, вирусного вакцинного антигена, моноклонального антитела, поликлонального антитела и фрагментов антител.
В контексте настоящего изобретения термин «сайт связывания» относится к сайту, который связан с другим веществом или компонентом, а «фрагмент связывания» относится к компоненту, включающему часть, способную связываться с другим веществом или компонентом. Кроме того, в контексте настоящего изобретения термин «активный сайт» относится к сайту, который индуцирует активность путем взаимодействия с лигандом или рецептором.
Кроме того, UCT (С-концевая метка убиквитина) относится к специфической последовательности С-концевого участка убиквитина, и UCT конъюгирован со специфическим лизином другого убиквитина посредством ковалентной связи.
Кроме того, настоящее изобретение предоставляет способ получения мультифункционального мультиспецифического мультимерного биомолекулярного полимера, в котором полиубиквитиновый каркас, две или более биомолекул, содержащих связывающие фрагменты, каждый из которых специфичен для разных сайтов связывания, и носитель, который продлевает продолжительность эффективности in vivo, непосредственно связаны с N-концом или С-концом убиквитина или связанны линкером, где способ включает (i) рекомбинантную экспрессию биомолекулы, с которой С-концевая метка убиквитина слита или связана линкером, из клетки-хозяина, включая прокариотические клетки или эукариотические клетки, и (ii) добавление ферментов E1, E2 и E3 или ферментов E1 и E2 для убиквитинирования в клеточные лизаты или очищенные продукты клетки-хозяина и взаимодействие с ними, где полиубиквитиновый каркас образован путем ковалентного связывания двух или более убиквитинов, и биомолекула состоит из от 2 до 10 биомолекул, имеет активные сайты, которые специфически связываются с другими биомолекулами, низкомолекулярными химическими соединениями или наночастицами и связаны с N-концом, C-концом или как с N-концом, так и с C-концом убиквитина линкером.
В одном варианте осуществления, относящимся к этому, фермент Е2 может связываться с 6-м, 11-м, 27-м, 29-м, 33-м, 48-м или 63-м лизином от N-конца убиквитина или может представлять собой фермент конъюгации убиквитина Е2-25К или ферментный комплекс Ucb13-MMS2 конъюгации убиквитина, но без ограничения.
В другом варианте осуществления, относящемся к этому, С-концевая метка убиквитина может представлять собой метку, в которой два или более убиквитина повторно связаны в форме «голова к хвосту» или в разветвленной форме (форма разветвленного типа или изопептидная форма типа разветвления), где убиквитин, связанный в форме «голова к хвосту» или в разветвленной форме, может представлять собой убиквитин, в котором 75-й и 76-й глицины от N-конца заменены другими аминокислотами, включая валин.
Эффекты настоящего изобретения
В соответствии с настоящим изобретением, поскольку связь между полимерами или комплексами биомолекул обеспечивается полиубиквитиновым каркасом, полиубиквитин может действовать как жесткий каркас или линкер, который поддерживает расстояние и ориентацию между биомолекулами, связанными с полиубиквитином. Таким образом, мультифункциональный мультиспецифический мультимерный биомолекулярный полимер может быть получен без вмешательства активного сайта.
Кроме того, согласно настоящему изобретению мультифункциональный мультиспецифический мультимерный биомолекулярный полимер предоставляется в форме, которая связана с молекулой, способной повышать продолжительность эффективности in vivo, и, таким образом, может использоваться для получения лекарственных средств, требующих повышенной стабильности in vivo и продолжительности эффективности.
В настоящем изобретении биомолекула может быть одной или несколькими, выбранными из группы, состоящей из белка, пептида, полипептида, антитела, фрагмента антитела, ДНК и РНК, и, например, с помощью гетерологичных белков модульная функциональность может быть придана линейному мультифункциональному мультимерному полимеру. Кроме того, согласно настоящему изобретению мультифункциональный мультиспецифический мультимерный биомолекулярный полимер предоставляется в форме, которая связана с молекулой, способной увеличивать продолжительность эффективности in vivo и, таким образом, может обеспечивать повышенную стабильность и продолжительность эффективности биомолекулы in vivo.
Краткое описание чертежей
На фиг. 1 показан способ получения линейного мультифункционального мультимерного слитого белка (UniStac) согласно настоящему изобретению.
На фиг. 2 и 3 показаны результаты подтверждения слитого белка UCT в мультимерной форме, образованного реакцией UniStac согласно настоящему изобретению.
На фиг. 4 показаны различные формы применения линейного мультифункционального мультимерного слитого белка согласно настоящему изобретению.
На фиг. 5 показаны результаты получения UniStac с использованием только E1-E2.
На фиг. 6 схематически показано получение линейного мультифункционального мультимерного слитого белка согласно настоящему изобретению и его применение путем иммобилизации.
На фиг. 7 схематически показаны способ UCT «голова к хвосту» и UniStac.
На фиг. 8 показаны результаты подтверждения с помощью SDS-PAGE после очистки ксилозоредуктазы (XR), полученной в соответствии с настоящим изобретением, с помощью GPC.
На фиг. 9 показаны результаты подтверждения с помощью SDS-PAGE после очистки оксалоацетатдекарбоксилазы (OAC), полученной в соответствии с настоящим изобретением, с помощью GPC.
На фиг. 10 показаны результаты подтверждения с помощью SDS-PAGE после очистки ксилитдегидрогеназы (XDH), полученной в соответствии с настоящим изобретением, с помощью GPC.
На фиг. 11 показаны результаты подтверждения с помощью SDS-PAGE после очистки триозофосфатизомеразы (TIM), полученной согласно настоящему изобретению, с помощью GPC.
На фиг. 12 показаны результаты подтверждения с помощью SDS-PAGE после очистки альдолазы (ALD), полученной согласно настоящему изобретению, с помощью GPC.
На фиг. 13 показаны результаты подтверждения с помощью SDS-PAGE после очистки фруктозо-1,6-бисфосфатазы (FBP), полученной согласно настоящему изобретению, с помощью GPC.
На фиг. 14 показаны результаты подтверждения с помощью SDS-PAGE после очистки пируватоксидазы (POPG), полученной в соответствии с настоящим изобретением, с помощью GPC.
На фиг. 15 показаны результаты анализа активности ксилозоредуктазы.
На фиг. 16 показаны результаты анализа стабильности ксилозоредуктазы.
На фиг. 17 показаны результаты анализа активности оксалоацетатдекарбоксилазы.
На фиг. 18 показаны результаты анализа стабильности оксалоацетатдекарбоксилазы.
На фиг. 19 показаны результаты анализа активности ксилитдегидрогеназы.
На фиг. 20 показаны результаты анализа стабильности ксилитдегидрогеназы.
На фиг. 21 показаны результаты анализа активности пируватоксидазы.
На фиг. 22 показан полимер UniStac структуры, с которой связаны три фермента, TIM, ALD и FBP.
На фиг. 23 показан синергетический эффект ферментов TIM, ALD и FBP.
На фиг. 24 показаны результаты получения и подтверждения линейных мультифункциональных мультимерных комплексов белков A и белков G.
На фиг. 25 показаны результаты получения и подтверждения hGH, в котором аспартат удлинен на С-концевой части 76-го глицина С-концевой метки убиквитина.
На фиг. 26 показаны результаты получения и подтверждения происхождения полимера из E3.
На фиг. 27 показаны результаты получения и подтверждения полимера hGH в зависимости от наличия или отсутствия DUB.
На фиг. 28 показана активность связывания IgG человеческого происхождения с шариками, на которых иммобилизован мономер белка А, и шариками, на которых иммобилизован полимер белка А.
На фиг. 29 показана структура линейного многофункционального мультимерного биомолекулярного полимера, связанного с N-концом, С-концом или как с N-концом, так и с С-концом убиквитина, соответственно, и результат его получения.
На фиг. 30 показаны результаты фармакокинетического профиля, показывающие, что время полужизни в крови находится на том же уровне, а скорость биоабсорбции (AUC) является более высокой при сравнении биомолекулярного полимера, в котором носитель связан с двойным убиквитином, и сывороточного альбумина человека.
На фиг. 31 показан вектор pcDNA3.1 (+), с которым связан ген, экспрессирующий акцепторный белок на основе Fc.
На фиг. 32 показан результат подтверждения экспрессии акцепторного белка на основе Fc.
На фиг. 33 показан результат подтверждения экспрессии акцепторного белка на основе Fc, который специфически связывается с антителом Fc IgG.
На фиг. 34 и 35 показаны результаты очистки акцепторного белка на основе Fc.
На фиг. 36 показан результат подтверждения убиквитин-IL-1RA с помощью метода анализа SDS-PAGE.
На фиг. 37 показан процесс очистки убиквитин- IL-1РА.
На фиг. 38 и 39 показаны результаты очистки меченого His-SUMO белка убиквитин-IL-1RA.
На фиг. 40 показан результат подтверждения меченого His-SUMO убиквитина-IL-1RA с помощью метода анализа SDS-PAGE.
На фиг. 41 и 42 показаны результаты подтверждения убиквитин-IL-1RA без метки His-SUMO.
На фиг. 43 и 44 показаны результаты очистки белка убиквитин-IL-1RA.
На фиг. 45 показан результат подтверждения степени конъюгации между акцептором и донором с помощью метода анализа SDS-PAGE.
На фиг. 46 показан результат анализа выхода конъюгата с использованием метода анализа μCE- SDS.
На фиг. 47 показан процесс очистки конъюгата.
На фиг. 48 и 49 показаны результаты подтверждения конъюгата, очищенной с помощью Ni-сефарозы.
На фиг. 50 и 51 показан результаты подтверждения очищенного конъюгата.
На фиг. 52 и 53 показаны результаты подтверждения конечного полимера UniStac в SDS-PAGE (восстановительные и нативные условия).
На фиг. 54 показан результат подтверждения мономера с использованием метода анализа μCE- SDS.
На фиг. 55 показан результат подтверждения мономера с помощью SEC-HPLC.
На фиг. 56 показаны структуры донорного белка (D-192), белка UniStac (C-193) и слитого белка (C-192; сравнительная группа) с использованием акцепторного белка и сывороточного альбумина человека в качестве носителя.
На фиг. 57 показан график зависимости концентрации лекарственного средства в крови от времени после подкожного введения слитых белков (С-192 и D-192).
На фиг. 58 показан график зависимости концентрации лекарственного средства в крови от времени после подкожного введения слитых белков (С-193 и D-192).
Наилучший способ осуществления настоящего изобретения
В одном варианте осуществления настоящее изобретение предоставляет способ получения мультифункционального мультиспецифического мультимерного биомолекулярного полимера, в котором полиубиквитиновый каркас, две или более биомолекул, содержащих связывающие фрагменты, каждый из которых специфичен для разных сайтов связывания, и носитель, который продлевает продолжительность эффективности in vivo, непосредственно связаны с N-концом или С-концом убиквитина или связанны линкером, где способ включает (i) рекомбинантную экспрессию биомолекулы, с которой С-концевая метка убиквитина слита или связана линкером, из клетки-хозяина, включая прокариотические клетки или эукариотические клетки, и (ii) добавление ферментов E1, E2 и E3 или ферментов E1 и E2 для убиквитинирования в клеточные лизаты или очищенные продукты клетки-хозяина и взаимодействие с ними, где полиубиквитиновый каркас образован путем ковалентного связывания двух или более убиквитинов, и биомолекула состоит из от 2 до 10 биомолекул, имеет активные сайты, которые специфически связываются с другими биомолекулами, низкомолекулярными химическими соединениями или наночастицами и связаны с N-концом, C-концом или как с N-концом, так и с C-концом убиквитина линкером.
В настоящем изобретении инициатор, который инициирует образование мультифункционального мультиспецифического мультимерного биомолекулярного полимера или комплекса могут представлять собой Е3, Е2, Е1, свободный убиквитин или субстрат-мишень Е3. В настоящем документе фермент Е2 может связываться с 48-м или 63-м лизином убиквитина, а фермент Е2 может представлять собой фермент конъюгации убиквитина Е2-25К, или ферментный комплекс, конъюгации убиквитина, Ucb13-MMS2.
В настоящем изобретении каждая из биомолекул предпочтительно связывается с N-концом убиквитина. Кроме того, мультимерный биомолекулярный полимер может состоять из 2-30 биомолекул.
Реакция UniStac согласно настоящему изобретению схематически показана на фиг. 1.
Кроме того, результаты подтверждения слитого белка UCT в мультимерной форме, образованного реакцией UniStac, показаны на фиг. 2 и 3.
Кроме того, мультифункциональный мультиспецифический мультимерный биомолекулярный полимер согласно настоящему изобретению может быть получен в различных формах. Конкретные примеры показаны на фиг. 4, 6 и 7. То есть на первом чертеже схематически показан способ получения линейного полимера фермента UniStac путем взаимодействия смеси UniStac с ферментом, меченым на С-конце убиквитином, как показано на фиг. 1, с последующей фильтрацией. На втором чертеже показан способ получения ферментного агрегата UniStac путем взаимодействия смеси UniStac с ферментом, меченым на С-конце убиквитином, с последующим осаждением с сшивающим линкером. На третьем чертеже схематически показан способ иммобилизации белка, меченого убиквитином на С-конце, на подложке или шарике.
Далее настоящее изобретение должно быть описано более подробно посредством следующих примеров. Эти примеры предназначены только для более подробного описания настоящего изобретения, и специалистам в данной области очевидно, что объем настоящего изобретения не ограничивается этими примерами в соответствии с сущностью настоящего изобретения.
Примеры получения
Пример получения 1. Клонирование, экспрессия и очистка С-концевого слитого белка
Ген, кодирующий слитый белок UCT (убиквитиновая С-концевая метка) (SEQ ID NO: 1), используемый в примерах настоящего изобретения, был получен у Genscript Inc.
Для получения генной конструкции Ub out, не содержащей убиквитиновой метки на С-конце, была использована система быстрого клонирования (Li C, Wen A, Shen B, Lu J, Huang Y, Chang Y (2011). Fast cloning: a highly simplified, purification-free, sequence- and ligation-independent PCR cloning method. BMC Biotechnol 11, 92.). Этот способ представляет собой технологию, позволяющую связывать гены (вставка, удаление или замена), в которой, если продукт ПЦР обрабатывают непосредственно только Dpn1 в отсутствие рестриктазы и лигазы, Dpn1 играет роль рестриктазы и лигазы через механизм, который еще не идентифицирован вместе с полимеразой. В этом способе используется праймер, предназначенный для перекрывания обоих концов полимеразы Phusion (Thermo Fisher Scientific), ПЦР (95°C в течение 3 минут, 95°C в течение 15 секунд, 55°C в течение 1 минуты, 72°C в течение 1 минуты/kb повтор 18 раз, 72°C в течение 5 минут, 12°C в течение 20 минут) проводили на всех векторах, за исключением удаляемой области. Затем полученный продукт ПЦР подвергали обработке Dpn1 в течение 1 часа при 37°C и трансформировали в E. coli DH5α (Novagen), а затем получали представляющую интерес плазмиду. Все генные конструкции были идентифицированы коммерческим секвенированием ДНК.
Для сверхэкспрессии слитого белка UCT каждую генную конструкцию трансформировали в штаммы E. coli BL21 DE3 (Novagen) (XR, TIM, ALD), Rosetta pLysS DE3 (Novagen) (XDH, OAC, POPG), Origami2 DE3 (Novagen) (FBP). Клетки, содержащие плазмиду для экспрессии белка (pET21a, Genscript), инкубировали в среде LB (Miller) при 37°C. Когда значение OD600 достигало около 0,6, экспрессию белка индуцировали 250 мкМ изопропил-β-D-1-тиогалактопиранозида (изопропил-бета-D-тиогалактопиранозида) (IPTG) при 16°C в течение 20 часов. Затем после центрифугирования (при 3500 об/мин при 4°C в течение 15 минут) клеточный осадок ресуспендировали в лизирующем буфере (20 мМ трис-HCl, pH 8,0, 500 мМ NaCl2, 20 мМ имидазол) и лизировали ультразвуком (50 % амплитуды, плюс включение 3 секунды-выключение 5 секунд, последние 15 минут). Затем лизат дополнительно центрифугировали при 14000 об/мин при 4°С в течение 30 минут. Водорастворимую фракцию белка, содержащую N-концевую His-метку, очищали гель-фильтрационной хроматографией с использованием колонки для гель-фильтрации Superdex 75 пг 16/600 (GE Healthcare), предварительно уравновешенной аффинным никелем и буфером FPLC (Ni-NTA агароза, QIAGEN, 20 мМ трис-HCl, pH 8,0, 150 мМ NaCl2). Все белки UCT концентрировали до 100 мкМ для анализа ферментативной активности. Все целевые белки оценивали с помощью SDS-PAGE. На фиг. 8-14 показаны результаты подтверждения целевых белков. Слитые белки UCT, используемые в настоящем изобретении, показаны в таблице 1 ниже.
Пример получения 2. Получение линейной конструкции UniStac
В настоящем изобретении реакция получения слитого белка в линейной мультифункциональной мультимерной форме была обозначена как реакция UniStac. Реакцию UniStac (общий объем 50 мкл) проводили в буфере UniStac (25 мМ HEPES (Sigma- aldrich), pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), а смесь UniStac для реакции UniStac (0,5 мкМ Е1, 5 мкМ Е2, 1 мкМ Е3, 4 мМ АТФ) добавляли к слитому белку UCT согласно настоящему изобретению для инициации реакции. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа.
Соотношение белков, используемых в реакции, составляло от 10 мкМ до 20 мкМ слитого белка UCT на 1 мкМ фермента Е3 (соотношение от 1:10 до 1:20), что было условием, установленным для этой цели для того, чтобы по меньшей мере 10 слитых мономеров образовывали линейный многофункциональный мультимер в течение 1 часа посредством реакции UniStac. E1, E2 и E3, используемые в настоящем изобретении, являются соответственно следующими:
Пример получения 3. Получение UniStac с использованием только E1-E2 (E2 платформа)
E2-UniStac получали с использованием E2-25K (GenBank ID-U58522.1) (E2 человека), Ucb13 (дрожжевой E2)-MMS2 (GenBank ID-U66724.1) (вариант дрожжевого убиквитин-конъюгирующего фермента) (GenBank ID -U66724.1). Использовали плазмиду рекомбинантной ДНК, синтезированную Genscript. Реакцию E2-UniStac проводили в условиях буфера (50 мМ Трис, pH 8,0, 5 мМ MgCl2), и смесь E2-UniStac (1 мкМ E1, 10 мкМ E2, 4 мМ АТФ) добавляли в раствор свободного убиквитина (20 мкМ), чтобы инициировать реакцию. Реакцию E2-UniStac проводили при встряхивании при комнатной температуре в течение 1 часа. Результаты представлены на фиг. 5.
Примеры
Пример 1. Анализ активности и стабильности ксилозоредуктазы (XR)
Анализ активности ксилозоредуктазы
Реакцию UniStac проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2) и добавляли смесь UniStac (0,5 мкМ E1, 5 мкМ E2, 1 мкМ E3, 4 мМ АТФ) в раствор белка XR, чтобы инициировать реакцию. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа, а затем анализировали каталитическую активность. Каталитическую активность XR анализировали путем измерения изменения поглощения при 340 нм, вызванного окислением NADH.
Реакцию для анализа каталитической активности инициировали добавлением NADH (2 мМ) к смеси XR (10 мкМ) и ксилозы (200 мМ) в 100 мМ буфере NaCl (pH 7,0), содержащем 1 мМ MgCl2 и 0,02% Твин-20. XR представлял собой образец в форме мономера, который не содержал убиквитиновой метки на С-конце XR и не образовывал полимер при тех же условиях смешивания UniStac. Статистический анализ проводили с помощью программы Prism 6 (GraphPad Software, Inc). Результаты показаны на фиг. 15.
Как показано на фиг. 15, XR в соответствии с настоящим изобретением способствует восстановлению D-ксилозы до ксилита за счет использования NADH в качестве ко-субстрата. Поглощение представляет собой количество NADH в растворе. Полимер UniStac XR (нижняя кривая) показал более быстрое потребление NADH по сравнению с мономерной формой (верхняя кривая). Обе реакции содержали одинаковое количество мономеров. Следовательно, повышенная скорость реакции зависит исключительно от ковалентных связей между мономерами. В итоге было подтверждено, что активность полимера XR UniStac увеличилась в 10 раз по сравнению с мономером XR без убиквитиновой метки.
Анализ рН-стабильности ксилозоредуктазы
Как мономер XR, так и полимер UniStac обрабатывали в течение 30 минут при указанном значении рН перед инициированием реакции добавлением NADH и ксилозы. Как показано на фиг. 16, при pH 5,5 и 6,5 полимер XR UniStac показал значительно повышенную стабильность по сравнению с мономером XR без убиквитиновой метки. Результаты представляют собой среднее значение трех экспериментов.
Пример 2. Анализ активности и стабильности оксалоацетатдекарбоксилазы (ОАС)
Анализ активности OAC
OAC, участвующую в глюконеогенезе, использовали для исследования повреждения печени в сочетании с AST-ALT. Реакцию UniStac проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2) и добавляли смесь UniStac (0,5 мкМ E1, 5 мкМ E2, 1 мкМ E3, 4 мМ АТФ) к раствору белка OAC, чтобы инициировать реакцию. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа, а затем анализировали каталитическую активность. Анализ активности OAC был основан на снижении поглощения (340 нм) по мере потребления NADH при следующих условиях: 45 мМ буфер TEA, рН 8,0, 0,45 мМ MnCl2, 2 мМ NADH, 11 ЕД LDH, 5 мкМ OAC, 2,5 мМ.
OAC представлял собой образец в форме мономера, который не содержал убиквитиновой метки на С-конце OAC и не образовывал полимер при тех же условиях смешивания UniStac. Статистический анализ проводили с помощью программы Prism 6 (GraphPad Software, Inc). Результаты показаны на фиг. 17.
Как показано на фиг. 17, в результате сравнения активности мономера без Ub (OAC) на С-конце и активности полимера (UniStaced OAC) активность полимера увеличилась в 9 раз. Поглощение представляет собой количество NADH в растворе. Полимер UniStac OAC (нижняя кривая) показал более быстрое потребление NADH по сравнению с мономерной формой (верхняя кривая). Обе реакции содержали одинаковое количество мономеров. Следовательно, повышенная скорость реакции зависит исключительно от ковалентных связей между мономерами. В итоге было подтверждено, что активность полимера OAC UniStac была увеличена в 9 раз по сравнению с мономером OAC без убиквитиновой метки (OAC).
Анализ стабильности OAC
Как мономер OAC, так и полимер UniStac обрабатывали в течение 30 минут при указанном значении pH перед инициированием реакции добавлением NADH и оксалоацетата. Как показано на фиг. 18, при низком значении рН от 4,5 до 6,5 полимер OAC UniStac показал значительно повышенную стабильность pH по сравнению с мономером OAC без убиквитиновой метки (OAC). Результаты представляют собой среднее значение трех экспериментов.
Пример 3. Анализ активности и стабильности ксилитдегидрогеназы (XDH)
Анализ активности XDH
XDH представляет собой фермент, принадлежащий к пути катаболизма D-ксилозы, и известно, что он превращает ксилит, продукт XR, в ксилулозу с использованием NAD+. Для анализа активности XDH реакцию UniStac сначала проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), и смесь UniStac (0,5 мкМ E1, 5 мкМ E2, 1 мкМ E3, 4 мМ АТФ) добавляли к раствору белка XDH для инициации реакции. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа, а затем анализировали каталитическую активность. Активность XDH измеряли, наблюдая за восстановлением NAD+ при 340 нм. Реакцию инициировали добавлением NADH (2 мМ) к смеси XDH (20 мкМ) и ксилозы (200 мМ) в 100 мМ буфере NaCl (pH 7,0), содержащем 1 мМ MgCl2 и 0,02% Tween-20. XDH представлял собой образец в форме мономера, который не содержал убиквитиновой метки на С-конце XDH и не образовывал полимер при тех же условиях смешивания UniStac. Статистический анализ проводили с помощью программы Prism 6 (GraphPad Software, Inc). Результаты показаны на фиг. 19.
Как показано на фиг. 19, при pH 5,5 полимер UniStac из XDH (верхняя кривая) показал более высокую скорость потребления NADH+ по сравнению с его мономерной формой (нижняя кривая). Обе реакции содержали одинаковое количество мономеров. Следовательно, разница в активности зависит исключительно от ковалентных связей между мономерами. В итоге было подтверждено, что активность полимера XDH UniStac была увеличена в 10 раз по сравнению с мономером XDH без убиквитиновой метки (XDH).
Анализ стабильности XDH
Как мономер XDH, так и полимер UniStac обрабатывали в течение 30 минут при указанном значении pH перед инициированием реакции добавлением NAD+ и ксилита. Как показано на фиг. 20, при всех измеренных значениях pH полимер XR UniStac показал значительно более высокую стабильность pH по сравнению с XDH. Результаты представляют собой среднее значение трех экспериментов.
Пример 4. Анализ активности пируватоксидазы (POPG)
Известно, что POPG используется для исследования повреждения печени путем обнаружения таких ферментов, как AST-ALT, фермент, участвующий в процессе глюконеогенеза. Для анализа активности POPG сначала проводили реакцию UniStac в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), и смесь UniStac (0,5 мкМ E1, 5 мкМ E2, 1 мкМ E3, 4 мМ АТФ) добавляли к раствору белка POPG для инициации реакции. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа, а затем анализировали каталитическую активность. Для анализа каталитической активности измеряли количество H2 O2, полученного в процессе окисления пирувата POPG с помощью ABTS. Реакцию инициировали добавлением POPG (5 мкМ) к смеси пирувата (100 мМ), пирофосфата (6 мМ), ABTS (2,2'-азино-бис(3-этилбензотиазолин-6-сульфокислота) (10 мМ) и HRP (пероксидаза хрена) (0,2 ед/мл) в натрий-фосфатном буфере.
Мономер POPG (POPG) представлял собой образец в форме мономера, который не содержал убиквитиновой метки на С-конце POPG и не образовывал полимер при тех же условиях смешивания UniStac. Статистический анализ проводили с помощью программы Prism 6 (GraphPad Software, Inc). Как показано на фиг. 21, при рН 5,5 POPG (верхняя кривая) проявлял более высокую активность по сравнению с его мономерной формой (нижняя кривая). Обе реакции содержали одинаковое количество мономеров. Следовательно, разница в активности зависит исключительно от ковалентных связей между мономерами. В итоге было подтверждено, что активность полимера POPG UniStac увеличилась в 2 раза по сравнению с мономером POPG без убиквитиновой метки (POPG).
Пример 5. Анализ синергетического эффекта убиквитинового фермента
Известно, что триозофосфатизомераза (TIM), фруктозобисфосфатальдолаза (ALD) и фруктозобисфосфатаза (FBP) образуют каскадную реакцию для получения F6P в качестве конечного продукта из DHAP (дигидроксиацетонфосфата). Анализ синергетического эффекта фермента UniStac проводили путем измерения фруктозо-6-фосфата (F6P), продукта TIM, ферментного комплекса ALD и FBP. F6P изомеризуется в глюкозо-6-фосфат (G6P) фосфоглюкозоизомеразой (PGI), а равное количество NAD+ в качестве субстрата модифицируется глюкозо-6-фосфатдегидрогеназой (G6PDH). Авторы настоящего изобретения определяли ферментативную активность путем измерения количества новообразованного NADH при 340 нм путем добавления 2,5 мМ ферментного комплекса (дигидроксиацетонфосфат, DHAP), 20 ед./мл фермента для анализа (PGI и G6PDH) и 2,5 мМ комплекса фермента NAD+ к смесь 4 мкМ ферментного комплекса TIM, ALD и FBP в буферных условиях HEPES (200 мМ HEPES, pH 7,5, 10 мМ MgCl2, 0,5 мМ MnCl2, 1 мМ CaCl2).
Сложная смесь ферментов представляла собой образец в виде мономера, который не содержал убиквитиновой метки на С-конце фермента и не образовывал полимер в тех же условиях смеси UniStac. Статистический анализ проводили с помощью программы Prism 6 (GraphPad Software, Inc). В указанный момент времени реакцию останавливали и измеряли количество F6P с помощью фосфоглюкозоизомеразы (PGI), которая использует NAD+ для превращения F6P в глюкозо-6-фосфат (G6P). Поглощение представляет собой количество F6P.
Результаты эксперимента представлены на фиг. 23. Как показано на фиг. 23, полимер UniStac из трех различных ферментов (верхняя кривая) показал более высокую активность в пять раз, чем мономерная смесь ферментов (нижняя кривая), подтверждая синергетический эффект ферментов UniStac. На фиг. 22 показан конечный продукт структуры (UniStac Polymer), в которой связаны три фермента, TIM, ALD и FBP.
Пример 6. Способ многостадийного мечения (протезирования) убиквитином
Биомолекулу с С-концевой меткой убиквитина синтезировали в соответствии с примерами получения согласно настоящему изобретению. Затем полимер (полиэтиленгликоль), содержащий гидроксиламин, реагировал с указанной выше биомолекулой. В результате было подтверждено, что полимер был помечен убиквитином по оксимной связи. Оксимная связь может быть использована в качестве средства, позволяющего создать полимерную систему доставки лекарственного средства.
Пример 7. Получение линейного мультимерного полимера белка А и белка G
Реакцию UniStac (общий объем 50 мкл) проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), и смесь UniStac (0,5 мкМ Е1, 5 мкМ Е2, 1 мкМ E3, 4 мМ АТФ) добавляли к раствору белка A или белка G, чтобы инициировать реакцию. Использовали плазмиды рекомбинантной ДНК, содержащие последовательности, соответствующие белку A (GenBank ID-AAB05743.1) и белку G (CAA27638.1), которые были синтезированы Genscript. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа, а затем проводили SDS-PAGE.
Как показано на фиг. 24, по сравнению с образцом без смеси UniStac было подтверждено, что мономерная полоса белка A или белка G уменьшилась в образце, к которому была добавлена смесь UniStac, а полоса с высокой молекулярной массой (линейный мультимерный полимер) вновь появлялась. Кроме того, было подтверждено, что некоторые линейные мультимерные полимеры не проходят через стэкинг-гель из-за увеличения молекулярной массы до нескольких сотен кДа.
Пример 8. hGH, в котором С-конец 76-го глицина С-концевой метки убиквитина удлиняется аспартатом
Для сверхэкспрессии слитого белка UCT каждую генную конструкцию трансформировали в штамм E. coli BL21 DE3 (Novagen). В этом примере в качестве белка использовали hGH (SEQ ID NO: 18). Клетки, содержащие плазмиду для экспрессии белка (pET21a, Genscript), инкубировали в среде LB (Miller) при 37°C. Когда значение OD600 достигало около 0,6, экспрессию белка индуцировали 250 мкМ изопропил-β-D-1-тиогалактопиранозида (изопропил-бета-D-тиогалактопиранозида) (IPTG) при 16°C в течение 20 часов. Затем после центрифугирования (при 3500 об/мин при 4°C в течение 15 минут) клеточный осадок ресуспендировали в лизирующем буфере (20 мМ трис-HCl, pH 8,0, 500 мМ NaCl2, 20 мМ имидазол) и лизировали ультразвуком (50 % амплитуды, плюс включение 3 секунды-выключение 5 секунд, последние 15 минут). Затем лизат дополнительно центрифугировали при 14000 об/мин при 4°С в течение 30 минут. Водорастворимую фракцию белка, содержащую N-концевую His-метку, очищали гель-фильтрационной хроматографией с использованием колонки для гель-фильтрации Superdex 75 пг 16/600 (GE Healthcare), предварительно уравновешенной аффинным никелем и буфером FPLC (Ni-NTA агароза - QIAGEN, 20 мМ трис-HCl, pH 8,0, 150 мМ NaCl2). Очищенный hGH концентрировали до 100 мкМ и оценивали с помощью SDS-PAGE. Результаты показаны на фиг. 25.
Пример 9. Получение полиубиквитинового каркаса, происходящего из Е3 (Rsp5)
Реакцию UniStac (общий объем 50 мкл) проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), и смесь UniStac (0,5 мкМ Е1, 5 мкМ Е2 (Ubch5a или Ubch7), 1 мкМ E3, 4 мМ АТФ) добавляли к раствору белка для инициации реакции. Реакцию UniStac проводили при встряхивании при комнатной температуре в течение 1 часа, а затем проводили SDS-PAGE.
Как показано на фиг. 26, было подтверждено, что количество Е3 было снижено в образце, к которому была добавлена смесь UniStac, по сравнению с образцом без смеси UniStac. Это связано с тем, что полоса Е3, молекулярная масса которой была увеличена за счет образования полиубиквитинового каркаса, происходящего от Е3, была смещена вверх. Кроме того, по сравнению со смесью UniStac, содержащей Ubch5a E2 (фиг. 26а), в результате добавления смеси UniStac, содержащей слабореакционноспособный Ubch7 E2 (фиг. 26b), было подтверждено, что молекулярная масса постепенно увеличивалась за счет связывая убиквитин один за другим с E3 (Rsp5) с течением времени.
Пример 10. Получение полимера hGH в зависимости от присутствия или отсутствия DUB
Реакцию UniStac hGH (SEQ ID NO: 18) сравнивали в условиях, когда DUB присутствовал вместе, и в условиях, когда DUB был исключен. В данном случае используется hGH, в котором две убиквитиновые метки неоднократно соединяются на С-конце в форме голова к хвосту, а С-конец убиквитиновой метки удлиняется с помощью аспартата. Следовательно, если аспартат на С-конце убиквитиновой метки не расщепляется с помощью DUB, реакция UniStac не происходит.
Реакцию UniStac для подтверждения образования полимера hGH, биомолекулы, проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), и смесь UniStac (1 мкМ E1, 5 мкМ E2 (ubch5a), 1 мкМ E3, 4 мМ АТФ) добавляли в 20 мкМ раствор белка hGH для инициации реакции.
Кроме того, реакцию E2-UniStac, в которой был исключен E3, проводили в буфере E2-UniStac (50 мМ Tris pH 8,0, 5 мМ MgCl2), и смесь E2-UniStac (1 мкМ E1, 10 мкМ E2 (Ucb13-MMS2 комплекс), 4 мМ АТФ) добавляли в 20 мкМ раствор белка hGH для инициации реакции.
Для совместного подтверждения активности DUB реакцию проводили одновременно в условиях, когда DUB (YUH1) отсутствовал, и в условиях, когда DUB (YUH1) присутствовал при концентрации 2 мкМ соответственно. Все реакции проводили при встряхивании при комнатной температуре в течение от 1 до 4 часов, а затем подтверждали с помощью SDS-PAGE.
Как показано на фиг. 27, было подтверждено, что полимер hGH образовывался только в условиях, когда присутствовал DUB, и было подтверждено, что полимер не образовывался, поскольку аспартат на С-конце UCT hGH не был расщеплен в условиях отсутствия DUB.
Пример 11. Связывающая активность полимера белка А, иммобилизованного на шарике
Реакцию UniStac для получения полимера белка А проводили в буфере UniStac (25 мМ HEPES, pH 7,5, 50 мМ NaCl, 4 мМ MgCl2). Смесь UniStac (0,5 мкМ Е1, 5 мкМ Е2 (Ubch5a или Ubch7), 1 мкМ Е3, 4 мМ АТФ) добавляли к раствору белка А для инициирования реакции. Реакцию UniStac проводили путем встряхивания при комнатной температуре в течение 1 часа, а затем смешивания в соотношении 1:1 вместе с латексными шариками при концентрации 50%, а затем встряхивания при температуре окружающей среды в течение 4 часов и иммобилизации полимера белка А на шарике. После реакции иммобилизации для удаления неиммобилизованного белка проводили трехкратную промывку буфером PBS (10 мМ Na2HPO4 pH 7,4, 1,8 мМ KH2PO4, 137 мМ NaCl, 2,7 мМ КСl). После промывания к шарикам добавляли иммуноглобулин G (IgG), полученный из сыворотки человека, при концентрации 2 мг/мл для анализа связывающей активности полимера белка А, иммобилизованного на шариках. Реакцию связывания проводили путем встряхивания при температуре окружающей среды в течение 1 часа, а затем трижды промывали буфером PBS таким же образом, как в описанном выше способе промывки, и затем подтверждали с помощью SDS-PAGE.
Как показано на фиг. 28, было подтверждено, что активность связывания IgG человеческого происхождения с шариками, на которых был иммобилизован полимер белка А, повышалась на 15% или более по сравнению с шариками, на которых мономер белка А был иммобилизован таким же образом, за исключением того, что смесь UniStac не добавляли.
Пример 12. Получение линейного мультивалентного биомолекулярного полимера, связанного с N-концом, С-концом или как с N-концом, так и С-концом убиквитина, соответственно
Образование димера UniStac подтверждали получением димера донорного убиквитина, в котором hGH был связан с N-концом (SEQ ID NO: 18), и акцепторного убиквитина, в котором hGH был связан с N-концом (SEQ ID №: 19) (фиг. 29, а)), димера донорного убиквитина, в котором hGH был связан с N-концом (SEQ ID NO: 18), и акцепторного убиквитина, в котором hGH был связан с C-концом (SEQ ID NO: 20) (фиг. 29 (b). )), и димера донорного убиквитина, в котором hGH был связан с N-концом (SEQ ID NO: 18), и акцепторного убиквитина, в котором SUMO и hGH были связаны с N-концом и C-концом, соответственно, (SEQ ID №: 21) (фиг. 29 (c)), соответственно.
Акцепторным убиквитином является форма, в которой 73-й лейцин замещен пролином, форма, в которой другие лизины, кроме 48-го лизина (фиг. 29 (c)) или 63-го лизина (фиг. 29 (а) и (b)) акцепторного убиквитина замещены аргинином, и форма, в которой С-конец удлинен аспартатом или биомолекулой (hGH).
Реакцию UniStac (рис. 29 (а) и (б b проводили в буфере UniStac (25 мМ HEPES pH 7,5, 50 мМ NaCl, 4 мМ MgCl2), и смесь UniStac (1 мкМ E1, 5 мкМ E2 (комплекс Ubc13-MMS2), 4 мМ АТФ) добавляли к раствору (общая концентрация убиквитина 20 мкМ), в котором были смешаны 10 мкМ акцепторного убиквитинового белка и донорного убиквитинового белка, чтобы инициировать реакцию.
Кроме того, реакцию UniStac (фиг. 29 (c)) инициировали заменой Е2 на Е2-25К, отличный от комплекса Ubc13-MMS2, и акцепторного убиквитина на белок, имеющий только 48-й Lys, в отличие от 63-го Lys, при тех же условиях, как описанная выше реакция. Реакцию UniStac проводили путем встряхивания при 27°C в течение 4 часов, а затем подтверждали с помощью SDS-PAGE. В реакции UniStac (фиг. 29 (b)) использовали акцепторный убиквитин в форме Ub-hGH, в которой His-sumo отщепляли с помощью фермента SENP1 от белка в форме His-sumo-Ub-hGH, и было подтверждено, что в это время оставшийся SENP1 был включен в реакцию UniStac, и, таким образом, донор hGH, Ubc13 и His-sumo MMS2 также были расщеплены вместе, а полоса димера и E2 (Ubc13, MMS2) после реакции была смещена.
Как показано на фиг. 29, было подтверждено, что димер UniStac образовывался во всех формах, в которых биомолекула была связана с N-концом, С-концом или как с N-концом, так и с С-концом, соответственно. SEQ ID NO белков и т.п., используемых в этом примере, являются следующими: донорный убиквитин, в котором hGH был связан с N-концом (SEQ ID NO: 18), акцепторный убиквитин, в котором hGH был связан с N-концом (SEQ ID NO: 19), акцепторный убиквитин, в котором hGH был связан с С-концом (SEQ ID NO: 20), акцепторный убиквитин, в котором SUMO и hGH были связаны с N-концом и С-концом, соответственно (SEQ ID NO: 21).
Пример 13. Подтверждение фармакокинетики
Диубиквитин-альбумин (OGB1) и альбумин (OGB3) однократно вводили подкожно 9-недельным самцам крыс Spraque-Dawley, затем собирали кровь для каждого периода времени и анализировали концентрацию лекарственного средства в сыворотке. Диубиквитин-альбумин вводили в дозе 0,833 мг/кг, а альбумин вводили в дозе 1 мг/кг, и каждая группа состояла из 12 самцов крыс. Забор крови для анализа концентрации лекарственного средства в крови осуществляли у 3 крыс на группу до введения (контроль) и через 0,5, 1, 2, 4, 6, 8, 12, 24, 36, 48 и 72 часа после введения (всего 12 моментов времени).
Контрольная группа состояла из5 крыс-самцов, забор крови проводили через 1 и 24 часа после введения. Сыворотку отделяли от собранной крови и хранили в криогенно-замороженном состоянии при температуре -70 ± 10°С. Анализ проводили путем измерения концентрации лекарственного средства в образцах крови, собранных для каждого периода времени с помощью набора для ELISA сывороточного альбумина человека. Сыворотку для разведения готовили путем смешивания буфера для разведения (1xPBS, 1% BSA) и контрольной крысиной сыворотки в соотношении 1:1 и использовали для анализа ELISA. Диубиквитин-альбумин разводили в сыворотке от 800 нг/мл до 15,625 нг/мл и вносили в каждую лунку. Каждый образец разбавляли с использованием контрольной крысиной сыворотки и буфера для разведения с получением сыворотки для разведения в конечном соотношении 1:1 и распределяли в каждую лунку. Раствор смеси антител готовили путем разбавления антител Capture и Detector из набора сывороточного альбумина человека в растворе СР для разведения антител. В каждую лунку вносили по 100 мкл раствора смеси антител и инкубировали при 400 об/мин при комнатной температуре в течение 1 часа. По окончании инкубации в каждую лунку вносили по 300 мкл промывочного раствора и трижды повторяли процесс встряхивания. В каждую лунку вносили по 100 мкл раствора субстрата TMB и инкубировали при 400 об/мин при комнатной температуре в течение 10 минут, затем в каждую лунку вносили по 100 мкл стоп-раствора, помещали в прибор и измеряли оптическую плотность (OD450). Альбумин разбавляли от 100 нг/мл до 2,5 нг/мл и вносили в каждую лунку, а остальные разведения образца и экспериментальную процедуру выполняли таким же образом для измерения поглощения. Калибровочную кривую рассчитывали с 4 параметрами на основе значений поглощения, измеренных для каждой концентрации, и, наконец, концентрацию лекарственного средства в сыворотке рассчитывали на основе значений поглощения, измеренных в образце, по сравнению с калибровочной кривой. Фармакокинетические параметры рассчитывали с помощью программы Phoenix WinNonlin (Ver. 8.1, компания Pharsight-A Certara, США) по результатам измерения концентрации испытуемого вещества в сыворотке крови для оценки фармакокинетики.
Как показано на фиг. 30, было подтверждено, что диубиквитин-альбумин (OGB1) проявлял более высокую концентрацию в плазме. Кроме того, было подтверждено, что AUC увеличилась в 1,8 раза, а Cмакс увеличилась более чем в 2 раза в группе, получавшей диубиквитин-альбумин (OGB1), по сравнению с группой, получавшей альбумин (OGB3). Таким образом, можно видеть, что полимер диубиквитин-альбумин согласно настоящему изобретению имеет более превосходный фармакокинетический эффект при более низкой концентрации.
Наконец, биомолекулярный полимер согласно настоящему изобретению предоставляется в форме, которая связана с молекулой, способной увеличивать продолжительность эффективности in vivo, и, таким образом, может использоваться для получения фармацевтической композиции, требующей увеличения продолжительности эффективности in vivo.
Пример 14. Получение рекомбинантной экспрессионной плазмидной ДНК, способной экспрессировать акцепторный белок на основе Fc (акцепторный белок)
Слитый белок, в котором носитель был непосредственно связан с С-концом акцепторного убиквитина, получали следующим способом. Фрагмент антитела (Fc-домен IgG) использовали в качестве белка-носителя слитого белка, и этот слитый белок называют «акцепторным белком на основе Fc».
Сигнальный пептид, который вызывает секрецию белка из клеток для проявления растворимости в клетках, акцепторный убиквитин, шарнир и ген Fc были сконструированы для вставки в вектор pcDNA3.1(+). Вектор pcDNA3.1(+) представляет собой вектор экспрессии для клеток животных, имеющих промотор CMV, ген устойчивости к ампициллину и т.п.
На фиг. 31 показан вектор pcDNA3.1 (+), с которым связан ген, экспрессирующий акцепторный белок на основе Fc. Нуклеотидная последовательность и аминокислотная последовательность, экспрессирующие акцепторный белок на основе Fc, показаны в таблицах 3 и 4 ниже.
(SEQ ID NO: 22)
(SEQ ID NO: 23)
(SEQ ID NO: 26)
(SEQ ID NO: 27)
Был получен указанный выше рекомбинантный вектор экспрессии акцепторного белка на основе Fc.
Плазмидную ДНК акцепторного белка на основе Fc помещали в компетентные клетки DH5α, трансформированные путем обработки тепловым шоком при 42°C в течение 1 минуты и высевали на твердую среду LB, содержащую 100 мкг/мл ампициллина. Посеянный планшет с твердой средой LB стационарно инкубировали при 37°C в течение по меньшей мере 16 часов с получением колоний. Брали одну колонию, инокулировали в 5 мл среды LB, а затем инкубировали при 37°C и 220 об/мин в течение 16 часов. Часть культурального раствора инокулировали в среду LB, содержащую ампициллин, и затем инкубировали при 37°С и 220 об/мин в течение 16 часов. Культуральный раствор центрифугировали при 3500 об/мин в течение 30 минут с получением осадка E. coli, а затем добавляли растворы P1, P2 и P3 из набора для выделения ДНК (QIAGEN) для разрушения клеточной стенки и разделяли белки с получением суспензии ДНК. Осадок плазмидной ДНК получали из полученной суспензии ДНК с помощью колонки для очистки набора для выделения ДНК (QIAGEN), и сушили естественным путем. К высушенному осадку ДНК добавляли воду для культивирования клеток (Sigma Aldrich), растворяли и затем фильтровали через фильтр 0,22 мкм. Конечную экстрагированную плазмидную ДНК использовали для экспрессии белка после измерения концентрации и чистоты ДНК с использованием прибора с нанокаплями (IMPLEN).
Пример 15. Экспрессия и подтверждение идентификации акцепторного белка на основе Fc (акцепторного белка)
Экспрессия акцепторного белка на основе Fc
Клетки человека Expi293F получены из линии клеток эмбриональной почки человека 293 и обладают высокой эффективностью трансфекции и экспрессии белка.
За 24 часа до процесса трансфекции клетки Expi293F (Gibco) инокулировали в количестве 3x106 жизнеспособных клеток/мл, помещали на орбитальный шейкер в инкубатор с 8 % CO2 и инкубировали в течение 24 часов при 37°C, 80 % влажность или выше, 95 об/мин (диаметр встряхивания 50 мм). Клетки подсчитывали для определения жизнеспособности и количества клеток, разбавляли экспрессионной средой Expi293 (Gibco) до конечной концентрации 3×106 жизнеспособных клеток/мл и общего объема 200 мл и инокулировали в колбе Эрленмейера, объемом 1 л.
200 мкг рекомбинантного экспрессионного вектора ДНК акцепторного белка на основе Fc разводили в 12 мл среды Opti-MEM I с уменьшенной сывороткой (Gibco), а 640 мкл реагента ExpiFectamineTM 293 (Gibco) разводили в 11,2 мл среды Opti- -MEM I с уменьшенной сывороткой (Gibco). Затем реакцию проводили при температуре окружающей среды в течение 5 минут. Раствор, содержащий реагент ExpiFectamineTM 293, помещали в раствор, содержащий рекомбинантного вектора экспрессии ДНК акцепторного белка на основе Fc, перемешивали и подвергали реакции при температуре окружающей среды в течение 12 минут. Трансфекцию осуществляли путем медленного дозирования в 1-литровую колбу, инокулированную до 3×106 жизнеспособных клеток/ мл. Помещали на орбитальный шейкер в инкубатор с 8% СО2 и инкубировали в течение 18 часов при 37°С, влажности 80% или выше, 95 об/мин (диаметр встряхивания 50 мм). Через 18 часов добавляли 1,2 мл Enhancer 1 (Gibco) и 12 мл Enhancer 2 (Gibco), соответственно, помещали на орбитальный шейкер в инкубатор с 8 % CO2 и инкубировали в течение 7 дней при 37°C, 80 % влажности или выше, 95 об/мин (диаметр встряхивания 50 мм).
Идентификация экспрессии акцепторного белка на основе Fc
Культуральный раствор, полученный выше, центрифугировали в течение по меньшей мере 30 минут при 3500 об/мин с получением только культурального раствора экспрессии акцепторного белка на основе Fc, за исключением клеточного осадка. Полученный культуральный раствор фильтровали через фильтр для удаления примесей. Для того, чтобы определить уровень экспрессии акцепторного белка на основе Fc из культурального раствора брали 80 мкл и добавляли к нему 20 мкл 5X невосстанавливающего красителя загрузки образца, перемешивали и оставляли стоять при 95°C в течение 10 минут.
Для качественного анализа уровня экспрессии брали 80 мкл бычьего сывороточного альбумина, разведенного до 31,25, 62,5, 125, 250, 500, 750 и 1000 мг/мл, и 20 мкл 5Х невосстанавливающего красителя загрузки образца добавляли, перемешивали и оставляли стоять при 95°C в течение 10 минут. Каждый образец и маркерный белок для проверки размера загружали в 10% трис-глициновый гель и белки разделяли при 80 вольтах (В) в течение около 20 минут и при 120 вольтах (В) в течение 90 минут. После завершения обработки геля проводили окрашивание кумасси бриллиантовым синим R при осторожном встряхивании, а окрашивающий реагент удаляли из окрашенного геля с помощью буфера, содержащего 10 % уксусной кислоты, при осторожном встряхивании. Обесцвеченный гель получали в виде файла изображения, и концентрацию акцепторного белка на основе Fc по сравнению с количеством полосы белка бычьего сывороточного альбумина анализировали качественно и количественно с помощью программы Image J, и результаты показаны на фиг. 32.
Как показано на фиг. 32, было подтверждено, что образовался акцепторный белок на основе Fc, имеющий размер 100 кДа, и было подтверждено, что образовалось 264,1 мг/мл белка.
Подтверждение целевой специфичности акцепторного белка на основе Fc
Вестерн-блоттинг выполняли для подтверждения специфической для мишени экспрессии акцепторного белка на основе Fc. Брали 80 мкл экспрессированного культурального раствора, добавляли 20 мкл 5X невосстанавливающего красителя загрузки образца, перемешивали и оставляли стоять при 95°C в течение 10 минут. Подготовленный образец и маркерный белок для проверки размера загружали в 10% трис-глициновый гель и белки разделяли при 80 вольтах (В) в течение около 20 минут и при 120 вольтах (В) в течение 90 минут. Гель переносил белок на поливинилиденфторидную мембрану (мембрану PVDF) в течение около 2 часов при 0,3 ампер (А) для электрофореза. Мембрану блокировали для устранения неспецифических реакций при осторожном встряхивании в течение 1 часа в 1X PBST (фосфатно-солевом буфере с Tween 20), содержащем 5% обезжиренного молока. Козий антикроличий IgG (H+L), который специфически связывается с антителом Fc IgG человека, и HRP использовали для подтверждения специфической экспрессии акцепторного белка на основе Fc. Результаты представлены на фиг. 3. Отрицательный контроль представляет собой образец, не содержащий плазмидной ДНК.
Как показано на фиг. 33, была подтверждена экспрессия акцепторного белка на основе Fc, который специфически связывается с антителом IgG Fc и имеет размер 100 кДа.
Очистка акцепторного белка на основе Fc
Культуральный раствор акцепторного белка на основе Fc, экспрессированного выше, наносили на колонку MabSelect Prism A (Cytiva), уравновешенную уравновешивающим буфером (20 мМ фосфат натрия, 150 мМ NaCl, pH 7,4). Уравновешивающий буфер, используемый для удаления примесей, которые не были связаны с колонкой, и элюирующий буфер 1 (50 мМ ацетата натрия при рН 4,5) и элюирующий буфер 2 (50 мМ ацетата натрия при рН 4,0) использовали в качестве ступенчатой элюции для извлечения акцепторного белка. К выделенному акцепторному белку добавляли 1 М Трис, чтобы обеспечить pH выделенного белка 7,5. Акцепторный белок, полученный после титрования рН, подвергали диализу с 25 мМ трис-буфером, рН 7,5, с последующей ультрафильтрацией. Результаты очистки акцепторного белка с помощью SDS-PAGE были подтверждены с использованием 10% собственного геля. Результаты показаны на фиг. 34 и 35.
Пример 16. Экспрессия и очистка белка убиквитин-IL-1RA (донорный белок)
Экспрессия белка убиквитин-IL-1RA
Убиквитин-биомолекулярный белок, в котором биомолекула непосредственно связана с С-концом донорного убиквитина, получали следующим образом.
Последовательность гена, кодирующего белок убиквитин-биомолекула, трансформировали в вектор pET21a, меченный His-SUMO, а затем 0,5 мкл плазмиды, в которую был вставлен ген, помещали в Е-пробирку, содержащую 50 мкл компетентной клетки E. coli BL21 (DE3). После этого перемешивали постукиванием и оставляли на льду на 20 минут. Для применения теплового шока E-пробирку оставляли на водяной бане при 42°C на 50 секунд, а затем оставляли на льду на 5 минут. После этого в E-пробирку добавляли 300 мкл свежей среды LB и инкубировали во встряхивающем инкубаторе при 37°C в течение 1 часа до завершения трансформации. Трансформированные клетки наносили на LB-планшет, содержащий ампициллин в концентрации 100 мг/мл в BSC при соотношении 1/1000, и инкубировали в течение ночи в стационарном инкубаторе при 37°С. После этого полученную одиночную колонию брали и инокулировали в 100 мл среды ТБ, содержащей ампициллин в концентрации 100 мг/мл при соотношении 1/1000, а затем посевной материал культивировали при 37°С и 220 об/мин в течение 6 часов.
В случае белка убиквитин-IL-1RA (донор, D-192), содержащего IL-1RA в качестве биомолекулы, посевной раствор инокулировали в 3 л собственной среды TB, содержащей ампициллин при соотношении 1/1000, при концентрации 100 мг/мл при соотношении 1:100 и проводили основное культивирование. Инкубацию проводили при 37°С, в растворенном кислороде 40% в течение 4 часов с использованием ферментера Biocanvas (Centrion), и проводили индукцию 1 М сток-раствора IPTG при конечной концентрации 200 мкМ. Для того, чтобы довести количество растворенного кислорода в культивируемом растворе до 40%, число оборотов лопасти автоматически регулировали от 300 до 700 об/мин в течение времени инкубации. После индукции инкубацию продолжали еще 14 часов и инкубацию прекращали. Инкубированный раствор центрифугировали при 7000 g в течение 30 минут с получением влажных клеток E. coli.
Оставшиеся питательные вещества и оптическую плотность измеряли после завершения инкубации с использованием анализатора Cedex BIO Analyzer (Roche). Результаты показаны в Таблице 5 ниже.
метр
(мг/л)
(мг/л)
(мг/л)
(мг/л)
(мг/л)
Убиквитин-IL-1RA идентифицировали с помощью способа анализа SDS-PAGE. Результаты показаны на фиг. 36. Кроме того, была измерена концентрация белка убиквитин-IL-1RA, и результат анализа составил 963,32 мг/л.
Очистка белка убиквитин-IL-1RA
Белок убиквитин-IL-1RA очищали следующим способом (фиг. 37).
(A) Лизис/обработка ультразвуком
Ресуспендирование влажных клеток, полученных при инкубации, проводили с использованием буфера для лизиса (20 мМ фосфата натрия, рН 7,0). Его осуществляли добавлением 9 мл лизирующего буфера на 1 г влажных клеток. Образцы лизиса помещали на лед и обрабатывали ультразвуком в течение 20 минут в условиях включения/выключения Pules = 3 с/5 с, амплитуда 45 %. Лизат центрифугировали при 14000 об/мин в течение 30 минут для того, чтобы получить только супернатант.
(B) Очистка с захватом
Лизат загружали на Ni-сефарозную смолу (Cytiva). После того, как загрузка образца была завершена, его достаточно промыли с использованием промывочного буфера (20 мМ фосфата натрия, рН 7,0, 0,02 М имидазола) для удаления неспецифического белка. После этого меченный Hig-SUMO белок убиквитин-IL-1RA выделяли с использованием буфера для элюции (20 мМ фосфата натрия, рН 7,0, 0,2 М имидазол). Восстановленный белок убиквитин-IL-1RA, меченный His-SUMO, подвергали диализу с 20 мМ буфером фосфата натрия, pH 7,0, для удаления имидазола. Очистку донорного белка через колонку с никелем подтверждали с помощью SDS-PAGE. Результаты показаны на фиг. 38 и 39.
(C) Расщепление ферментом SENP1
Меченный His-SUMO белок убиквитин-IL-1RA и SENP1 подвергали расщеплению ферментом SENP1 при соотношении 100 мг белка убиквитин-IL-1RA: 1 мг SENP1. Концентрацию выделенного белка определяли количественно с помощью Ni-очистки, и соответствующий рекомбинантный SNEP1 смешивали на основе количества меченого His-SUMO белка убиквитин-IL-1RA. Реакционную смесь оставляли при температуре окружающей среды (от 15 до 25°С) на 1 час. Расщепление ферментом SENP1 подтверждали с помощью SDS-PAGE. Результаты показаны на фиг. 40.
(D) Удаление His-SUMO
Реакционную смесь наносили на Ni-сефарозную смолу (Cytiva), уравновешенную уравновешивающим буфером (20 мМ фосфата натрия, рН 7,0, 0,02 М имидазол). Выполняли загрузку образца и позволяли протекать белку убиквитин-IL-1RA, в котором была расщеплена метка His-SUMO. После завершения загрузки образца оставшийся белок убиквитин-IL-1RA извлекали с использованием уравновешивающего буфера (20 мМ фосфата натрия, pH 7,0, 0,02 М имидазола), а восстановленный белок убиквитин-IL-1RA подвергали диализу с 20 мМ фосфата натрия, буфер pH 7,0 для удаления имидазола. Процесс удаления His-SUMO был подтвержден с помощью SDS-PAGE. Результаты показаны на фиг. 41 и 42.
(E) Очистка
Белок убиквитин-IL-1RA, извлеченный на предыдущей стадии, загружали на колонку с анионообменной смолой, уравновешенную уравновешивающим буфером (20 мМ фосфата натрия, буфер pH 7,0). Пропускали белок убиквитин-IL-1RA. Выделенный белок убиквитин-IL-1RA подвергали ультрафильтрации до конечной концентрации 10 мг/мл. Результаты процесса с анионообменной смолой показаны на фиг. 43 и 44.
Пример 17. Конъюгирование
Производительность и выход конъюгирования
Конъюгирование выполняли с использованием акцепторного белка, полученного в примере 18, и донорного белка, полученного в примере 19. Молярное соотношение акцепторного и донорного белков составляло 1:3. В этом случае акцепторный белок можно установить в диапазоне от 10 мкМ до 50 мкМ. Кроме того, для инициирования реакции к смеси UniStac добавляли Е1, Е2, Е3 и АТФ. Реакцию проводили при 25°С в течение 16 часов в стационарном состоянии.
Для полученного продукта реакции конъюгации (C-193) степень конъюгации качественно анализировали путем загрузки 1,12 мкг акцептора на SDS-PAGE с градиентом от 4 до 12%, и результаты показаны на фиг. 45.
Кроме того, для полученного продукта реакции количественно анализировали степень конъюгации с использованием метода анализа μCE-SDS. Предварительную обработку образцов проводили с использованием набора реагентов HT Protein Express Reagent Kit, а затем проводили анализ с использованием лабораторного чипа Protein Express Assay Labchip, и результаты анализа выхода показаны в таблице 6 ниже и на фиг. 46.
Уравнение 1

Выход конъюгации в приведенной выше таблице 6 был рассчитан с использованием приведенного выше уравнения 1, и выход конъюгации UniStac составил 97,21 %, что указывает на очень высокое специфическое значение акцептора.
Очистка конъюгата UniStac
Для того, чтобы восстановить только образец конъюгации (С-193), очистку проводили с использованием следующего процесса (фиг. 47).
Для полученного продукта реакции готовили Ni-сефарозную смолу, уравновешенную уравновешивающим буфером (25 мМ Трис, рН 8,0, 0,5 М NaCl). Небольшое количество 5 М NaCl было добавлено к образцу загрузки, чтобы довести проводимость до 50 мСм/см. После завершения загрузки подготовленного образца с соответствующей проводимостью в подготовленную колонку для удаления примесей использовали уравновешивающий буфер (25 мМ Трис, рН 8,0, 0,5 М NaCl). После этого элюирующий буфер (25 мМ Трис, рН 8,0) использовали для извлечения конъюгата (С-193). Извлеченный конъюгат (С-193) подвергали диализу с натрий-фосфатным буфером без соли с рН 7,0 для удаления соли и имидазола. Ni-очистка была подтверждена хроматографией, и результаты показаны на фиг. 48 и 49.
Конъюгат наносили на колонку с анионообменной смолой (анионообменная хроматография), уравновешенную уравновешивающим буфером (25 мМ фосфата натрия, буфер pH 7,0). Для извлечения конъюгата использовали элюирующий буфер (25 мМ фосфата натрия, рН 7,0, 250 мМ NaCl), и результаты показаны на фиг. 50 и 51.
Конечный UniStac
Конъюгат (С-193) подвергали диализу с буфером для состава (4,6 мМ гистидина, 5,7 мМ Трис, pH 7,5, 10 мМ аргинина, 0,1 г/мл трегалозы) для получения состава. Конечный продукт UniStac готовили путем разбавления до 1,1 мг/мл и 0,5 мг/ мл. Полученные образцы хранили в морозильной камере при температуре -70°С.
Пример 18. Физико-химический анализ конечного полимера UniStac
Для измерения чистоты конечного полимера UniStac (лекарственный субстрат, C-193) были проведены SDS-PAGE (восстановительные и нативные условия), μCE-SDS и анализ SEC-HPLC.
Анализ SDS-PAGE
Для анализа конечного продукта конъюгата (DS, C-193) в восстанавливающих условиях проводили восстанавливающий анализ SDS-PAGE с использованием 4-12% геля Bis-Tris Plus и рабочего буфера MES SDS. 3 мкг образца загружали в каждый полученный PAGE, и результаты показаны на фиг. 52.
Для анализа конечного продукта конъюгата (C-193 DS) в нативных условиях был проведен анализ нативного PAGE с использованием 4-15% T/G-PAG-BC без SDS и нативного прогонного буфера с трис-глицином. 4,5 мкг образца загружали в каждый подготовленный PAGE, и результаты показаны на фиг. 53.
Анализ μCE-SDS
Степень включения фрагментов в образец C-193 DS анализировали с использованием метода анализа μCE-SDS (Perkin Elmer Labchip GX II Touch.). Предварительную обработку образцов проводили с использованием набора реагентов HT Protein Express Reagent Kit (Perkin Elmer), а затем проводили анализ с использованием лабораторного чипа Protein Express Assay Labchip (Perkin Elmer). Результаты показаны в таблице 7 ниже и на фиг. 54.
Уравнение 2
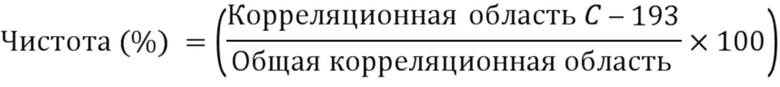
Чистоту мономера рассчитывали по уравнению 2, и результаты представлены в таблице 7, подтверждая, что чистота мономера составляет 98% или более.
Анализ SEC-HPLC
Степень высокомолекулярного включения в образце C-193 DS анализировали с использованием колонки SEC-HPLC и прибора Alliance e2695 XC HPLC. Около 30 мкг образца C-193 DS вводили в каждую из подготовленных колонок для проведения анализа, и результаты показаны в таблице 8 ниже и на фиг. 55.
Чистота мономера была рассчитана с использованием приведенного выше уравнения 2, и результаты показаны в таблице 8 выше, подтверждая, что чистота мономера была очень высокой и составляла 100%.
Пример 19. Сравнение фармакокинетики слитых белков (C-192, C-193 и D-192)
Получали донорный белок (D-192), белок UniStac (C-193) и слитый белок (C-192) с использованием акцепторного белка и сывороточного альбумина человека в качестве носителя. Структуры трех образцов показаны на фиг. 56 ниже.
9-недельным самцам мышей ICR однократно подкожно вводили три образца, а затем собирали кровь для каждого периода времени и проводили тест измерения фармакокинетики для анализа концентрации лекарственного средства в крови и расчета фармакокинетических параметров.
8-недельных самцов мышей ICR, приобретенных у Orient BIO, Корея, помещали на карантин и акклиматизировали в течение 7 дней. После карантина и акклиматизации всех животных ранжировали по массе тела и проводили разделение на группы (n=2 на момент времени забора крови) таким образом, чтобы средняя масса тела каждой группы была равномерно распределена. После этого тестируемое вещество С-192 в дозе 10 мг/кг, тестируемое вещество С-193 в дозе 1 мг/кг и тестируемое вещество D-192 в дозе 1 мг/кг однократно вводили каждой мыши подкожно. Точки сбора крови: до введения, 2, 6, 8, 10, 16, 32, 24, 48, 72 и 96 часов после введения С-192, 0,5, 1, 2, 4, 6, 8, 10, 16, 24, 32, 40, 48, 56 и 72 часа после введения С-193 и 1, 2, 3, 4, 5 и 6 часов после введения D-192, соответственно. Сыворотку отделяли от собранной крови центрифугированием и хранили при -70 ± 10°С для анализа концентрации лекарственного средства в крови.
Для измерения концентрации тестируемого вещества в крови использовали набор для ELISA человеческого IL-1RA (Abcam, UK), обладающий специфической реактивностью к IL-1RA. Сначала в 96-луночный планшет вносили по 50 мкл стандартного вещества и сыворотки для каждого момента времени, а затем в каждую лунку вносили по 50 мкл коктейля антител из набора IL-1RA ELISA для человека и проводили реакцию в смесительном устройстве при 25°C (термомикромиксер) при 400 об/мин в течение 1 часа. Раствор в лунке планшета сливали и встряхивали, чтобы не осталось никаких остатков. В каждую лунку вносили по 300 мкл промывочного раствора, затем трижды повторяли процедуру отбрасывания и встряхивания. В каждую лунку вносили по 100 мкл цветообразователя, а затем проводили реакцию в смесителе при 25°С при 400 об/мин в течение 10 минут.
Наконец, в каждую лунку вносили 100 мкл стоп-раствора, а затем измеряли оптическую плотность при 450 нм с использованием прибора для измерения абсорбции (многорежимного считывателя микропланшетов). Концентрацию лекарственного средства в сыворотке крови для каждого момента времени, рассчитанную относительно стандартного вещества, использовали для расчета фармакокинетических параметров. На фиг. 57 и 58 показаны графики зависимости концентрации лекарственного средства в крови от времени после подкожного введения слитых белков (С-192, С-193 и D-192) мышам.
Кроме того, фармакокинетические параметры были рассчитаны на основе экспериментальных результатов, и результаты показаны в Таблице 9 ниже.
Как показано в Таблице 9 выше, когда три слитых белка сравнивали и оценивали на основе периода полувыведения, указывающего на стабильность лекарственного средства in vivo, было подтверждено, что период полувыведения слитого белка (С-192) с использованием сывороточный альбумин человека в качестве носителя был увеличен около в 23 раза от 0,4 часа до 9,3 часа по сравнению с акцепторным белком (D-192).
Кроме того, было подтверждено, что время полувыведения слитого белка (С-193) с использованием Fc в качестве носителя увеличилось около в 33 раза от 0,4 часа до 13,7 часа по сравнению с акцепторным белком (D-192).
Наконец, слитый белок, в котором альбумин и носитель Fc слиты, обладает превосходными фармакокинеическими свойствами за счет увеличения времени полувыведения, AUC, Tмакс и Cмакс, и может использоваться в качестве превосходного лекарственного средства, которое можно наносить в желаемый сайт при небольшой дозе.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> УАНДЖИН БАЙОТЕКНОЛОДЖИ ИНК.
<120> МУЛЬТИФУНКЦИОНАЛЬНЫЙ МУЛЬТИСПЕЦИФИЧЕСКИЙ МУЛЬТИМЕРНЫЙ
БИОМОЛЕКУЛЯРНЫЙ ПОЛИМЕР, ИМЕЮЩИЙ ПРОЛОНГИРОВАННУЮ ЭФФЕКТИВНОСТЬ
IN-VIVO
<130> OGB20P-0008-WO
<150> KR 10-2019-0154945
<151> 2019-11-27
<160> 29
<170> KoPatentIn 3.0
<210> 1
<211> 76
<212> PRT
<213> Искусственная последовательность
<220>
<223> C-концевая метка убиквитина
<400> 1
Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp
20 25 30
Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
65 70 75
<210> 2
<211> 506
<212> PRT
<213> Искусственная последовательность
<220>
<223> ксилозоредуктаза
<400> 2
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Pro Ser Ile Lys Leu Asn Ser
100 105 110
Gly Tyr Asp Met Pro Ala Val Gly Phe Gly Cys Trp Lys Val Asp Val
115 120 125
Asp Thr Cys Ser Glu Gln Ile Tyr Arg Ala Ile Lys Thr Gly Tyr Arg
130 135 140
Leu Phe Asp Gly Ala Glu Asp Tyr Ala Asn Glu Lys Leu Val Gly Ala
145 150 155 160
Gly Val Lys Lys Ala Ile Asp Glu Gly Ile Val Lys Arg Glu Asp Leu
165 170 175
Phe Leu Thr Ser Lys Leu Trp Asn Asn Tyr His His Pro Asp Asn Val
180 185 190
Glu Lys Ala Leu Asn Arg Thr Leu Ser Asp Leu Gln Val Asp Tyr Val
195 200 205
Asp Leu Phe Leu Ile His Phe Pro Val Thr Phe Lys Phe Val Pro Leu
210 215 220
Glu Glu Lys Tyr Pro Pro Gly Phe Tyr Cys Gly Lys Gly Asp Asn Phe
225 230 235 240
Asp Tyr Glu Asp Val Pro Ile Leu Glu Thr Trp Lys Ala Leu Glu Lys
245 250 255
Leu Val Lys Ala Gly Lys Ile Arg Ser Ile Gly Val Ser Asn Phe Pro
260 265 270
Gly Ala Leu Leu Leu Asp Leu Leu Arg Gly Ala Thr Ile Lys Pro Ser
275 280 285
Val Leu Gln Val Glu His His Pro Tyr Leu Gln Gln Pro Arg Leu Ile
290 295 300
Glu Phe Ala Gln Ser Arg Gly Ile Ala Val Thr Ala Tyr Ser Ser Phe
305 310 315 320
Gly Pro Gln Ser Phe Val Glu Leu Asn Gln Gly Arg Ala Leu Asn Thr
325 330 335
Ser Pro Leu Phe Glu Asn Glu Thr Ile Lys Ala Ile Ala Ala Lys His
340 345 350
Gly Lys Ser Pro Ala Gln Val Leu Leu Arg Trp Ser Ser Gln Arg Gly
355 360 365
Ile Ala Ile Ile Pro Lys Ser Asn Thr Val Pro His Leu Leu Glu Asn
370 375 380
Lys Asp Val Asn Ser Phe Asp Leu Asp Glu Gln Asp Phe Ala Asp Ile
385 390 395 400
Ala Lys Leu Asp Ile Asn Leu Arg Phe Asn Asp Pro Trp Asp Trp Asp
405 410 415
Lys Ile Pro Ile Phe Val Gly Gly His His His His His His Met Gln
420 425 430
Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu
435 440 445
Pro Ser Asp Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu
450 455 460
Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu
465 470 475 480
Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr
485 490 495
Leu His Leu Val Leu Arg Leu Arg Gly Gly
500 505
<210> 3
<211> 542
<212> PRT
<213> Искусственная последовательность
<220>
<223> ксилитдегидрогеназа
<400> 3
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Thr Ala Asn Pro Ser Leu Val
100 105 110
Leu Asn Lys Ile Asp Asp Ile Ser Phe Glu Thr Tyr Asp Ala Pro Glu
115 120 125
Ile Ser Glu Pro Thr Asp Val Leu Val Gln Val Lys Lys Thr Gly Ile
130 135 140
Cys Gly Ser Asp Ile His Phe Tyr Ala His Gly Arg Ile Gly Asn Phe
145 150 155 160
Val Leu Thr Lys Pro Met Val Leu Gly His Glu Ser Ala Gly Thr Val
165 170 175
Val Gln Val Gly Lys Gly Val Thr Ser Leu Lys Val Gly Asp Asn Val
180 185 190
Ala Ile Glu Pro Gly Ile Pro Ser Arg Phe Ser Asp Glu Tyr Lys Ser
195 200 205
Gly His Tyr Asn Leu Cys Pro His Met Ala Phe Ala Ala Thr Pro Asn
210 215 220
Ser Lys Glu Gly Glu Pro Asn Pro Pro Gly Thr Leu Cys Lys Tyr Phe
225 230 235 240
Lys Ser Pro Glu Asp Phe Leu Val Lys Leu Pro Asp His Val Ser Leu
245 250 255
Glu Leu Gly Ala Leu Val Glu Pro Leu Ser Val Gly Val His Ala Ser
260 265 270
Lys Leu Gly Ser Val Ala Phe Gly Asp Tyr Val Ala Val Phe Gly Ala
275 280 285
Gly Pro Val Gly Leu Leu Ala Ala Ala Val Ala Lys Thr Phe Gly Ala
290 295 300
Lys Gly Val Ile Val Val Asp Ile Phe Asp Asn Lys Leu Lys Met Ala
305 310 315 320
Lys Asp Ile Gly Ala Ala Thr His Thr Phe Asn Ser Lys Thr Gly Gly
325 330 335
Ser Glu Glu Leu Ile Lys Ala Phe Gly Gly Asn Val Pro Asn Val Val
340 345 350
Leu Glu Cys Thr Gly Ala Glu Pro Cys Ile Lys Leu Gly Val Asp Ala
355 360 365
Ile Ala Pro Gly Gly Arg Phe Val Gln Val Gly Asn Ala Ala Gly Pro
370 375 380
Val Ser Phe Pro Ile Thr Val Phe Ala Met Lys Glu Leu Thr Leu Phe
385 390 395 400
Gly Ser Phe Arg Tyr Gly Phe Asn Asp Tyr Lys Thr Ala Val Gly Ile
405 410 415
Phe Asp Thr Asn Tyr Gln Asn Gly Arg Glu Asn Ala Pro Ile Asp Phe
420 425 430
Glu Gln Leu Ile Thr His Arg Tyr Lys Phe Lys Asp Ala Ile Glu Ala
435 440 445
Tyr Asp Leu Val Arg Ala Gly Lys Gly Ala Val Lys Cys Leu Ile Asp
450 455 460
Gly Pro Glu Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile
465 470 475 480
Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala Lys
485 490 495
Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe
500 505 510
Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile
515 520 525
Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly
530 535 540
<210> 4
<211> 407
<212> PRT
<213> Искусственная последовательность
<220>
<223> оксалоацетатдекарбоксилаза
<400> 4
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Tyr Glu Leu Gly Val Val Tyr
100 105 110
Arg Asn Ile Gln Arg Ala Asp Arg Ala Ala Ala Asp Gly Leu Ala Ala
115 120 125
Leu Gly Ser Ala Thr Val His Glu Ala Met Gly Arg Val Gly Leu Leu
130 135 140
Lys Pro Tyr Met Arg Pro Ile Tyr Ala Gly Lys Gln Val Ser Gly Thr
145 150 155 160
Ala Val Thr Val Leu Leu Gln Pro Gly Asp Asn Trp Met Met His Val
165 170 175
Ala Ala Glu Gln Ile Gln Pro Gly Asp Ile Val Val Ala Ala Val Thr
180 185 190
Ala Glu Cys Thr Asp Gly Tyr Phe Gly Asp Leu Leu Ala Thr Ser Phe
195 200 205
Gln Ala Arg Gly Ala Arg Ala Leu Ile Ile Asp Ala Gly Val Arg Asp
210 215 220
Val Lys Thr Leu Gln Glu Met Asp Phe Pro Val Trp Ser Lys Ala Ile
225 230 235 240
Ser Ser Lys Gly Thr Ile Lys Ala Thr Leu Gly Ser Val Asn Ile Pro
245 250 255
Ile Val Cys Ala Gly Met Leu Val Thr Pro Gly Asp Val Ile Val Ala
260 265 270
Asp Asp Asp Gly Val Val Cys Val Pro Ala Ala Arg Ala Val Glu Val
275 280 285
Leu Ala Ala Ala Gln Lys Arg Glu Ser Phe Glu Gly Glu Lys Arg Ala
290 295 300
Lys Leu Ala Ser Gly Val Leu Gly Leu Asp Met Tyr Lys Met Arg Glu
305 310 315 320
Pro Leu Glu Lys Ala Gly Leu Lys Tyr Ile Asp Met Gln Ile Phe Val
325 330 335
Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp
340 345 350
Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro
355 360 365
Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly
370 375 380
Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu
385 390 395 400
Val Leu Arg Leu Arg Gly Gly
405
<210> 5
<211> 430
<212> PRT
<213> Искусственная последовательность
<220>
<223> триозофосфатизомераза
<400> 5
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Arg Arg Val Leu Val Ala Gly
100 105 110
Asn Trp Lys Met His Lys Thr Pro Ser Glu Ala Arg Val Trp Phe Ala
115 120 125
Glu Leu Lys Arg Leu Leu Pro Pro Leu Gln Ser Glu Ala Ala Val Leu
130 135 140
Pro Ala Phe Pro Ile Leu Pro Val Ala Lys Glu Val Leu Ala Glu Thr
145 150 155 160
Gln Val Gly Tyr Gly Ala Gln Asp Val Ser Ala His Lys Glu Gly Ala
165 170 175
Tyr Thr Gly Glu Val Ser Ala Arg Met Leu Ser Asp Leu Gly Cys Arg
180 185 190
Tyr Ala Ile Val Gly His Ser Glu Arg Arg Arg Tyr His Gly Glu Thr
195 200 205
Asp Ala Leu Val Ala Glu Lys Ala Lys Arg Leu Leu Glu Glu Gly Ile
210 215 220
Thr Pro Ile Leu Cys Val Gly Glu Pro Leu Glu Val Arg Glu Lys Gly
225 230 235 240
Glu Ala Val Pro Tyr Thr Leu Arg Gln Leu Arg Gly Ser Leu Glu Gly
245 250 255
Val Glu Pro Pro Gly Pro Glu Ala Leu Val Ile Ala Tyr Glu Pro Val
260 265 270
Trp Ala Ile Gly Thr Gly Lys Asn Ala Thr Pro Glu Asp Ala Glu Ala
275 280 285
Met His Gln Glu Ile Arg Lys Ala Leu Ser Glu Arg Tyr Gly Glu Ala
290 295 300
Phe Ala Ser Arg Val Arg Ile Leu Tyr Gly Gly Ser Val Asn Pro Lys
305 310 315 320
Asn Phe Ala Asp Leu Leu Ser Met Pro Asn Val Asp Gly Gly Leu Val
325 330 335
Gly Gly Ala Ser Leu Glu Leu Glu Ser Phe Leu Ala Leu Leu Arg Ile
340 345 350
Ala Gly Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr
355 360 365
Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala Lys Ile
370 375 380
Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala
385 390 395 400
Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln
405 410 415
Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
420 425 430
<210> 6
<211> 496
<212> PRT
<213> Искусственная последовательность
<220>
<223> альдолаза
<400> 6
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Val Thr Met Pro Tyr Val Lys
100 105 110
Asn Thr Lys Glu Ile Leu Glu Lys Ala Ser Lys Glu Arg Tyr Ala Ile
115 120 125
Gly Ala Phe Asn Phe Asn Asn Met Glu Phe Leu Gln Ala Ile Leu Glu
130 135 140
Ala Ala Glu Glu Glu Lys Ala Pro Val Ile Val Ala Thr Ser Glu Gly
145 150 155 160
Ala Ile Lys Tyr Ile Gly Lys Gly Asp Ile Glu Thr Gly Ala Lys Leu
165 170 175
Ala Val Glu Met Val Arg Thr Tyr Ala Glu Lys Leu Ser Val Pro Val
180 185 190
Ala Leu His Leu Asp His Gly Arg Asp Phe Lys Val Ile Met Ala Ala
195 200 205
Ile Lys Ala Gly Tyr Ser Ser Val Met Ile Asp Ala Ser His Leu Pro
210 215 220
Phe Glu Glu Asn Leu Arg Glu Thr Lys Arg Ile Val Glu Ile Ala His
225 230 235 240
Ala Val Gly Ile Ser Val Glu Ala Glu Leu Gly Lys Leu Lys Gly Ile
245 250 255
Glu Asp Asn Val Val Glu Lys Glu Ser Val Leu Val Asp Pro Glu Glu
260 265 270
Ala Lys Val Phe Val Lys Glu Thr Glu Val Asp Phe Leu Ala Pro Ala
275 280 285
Ile Gly Thr Ser His Gly Ala Phe Lys Phe Lys Gly Glu Ala Gln Leu
290 295 300
Asp Phe Glu Arg Leu Lys Lys Val Lys Glu Tyr Thr Gln Ile Pro Leu
305 310 315 320
Val Leu His Gly Ala Ser Met Val Pro Gln Asp Ile Val Lys Leu Ala
325 330 335
Asn Glu Tyr Gly Ala Glu Leu Ser Gly Ala Lys Gly Val Pro Glu Asp
340 345 350
Met Leu Lys Lys Ala Ile Glu Leu Gly Ile Asn Lys Ile Asn Thr Asp
355 360 365
Thr Asp Leu Arg Ile Thr Phe Val Ala Tyr Leu Arg Lys Val Leu Ser
370 375 380
Glu Asp Lys Ser Gln Ile Asp Pro Arg Lys Ile Phe Lys Pro Val Phe
385 390 395 400
Glu Gln Val Lys Glu Ile Val Lys Glu Arg Ile Arg Ile Phe Gly Ser
405 410 415
Ser Gly Lys Ala Met Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr
420 425 430
Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu Asn Val Lys Ala
435 440 445
Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile
450 455 460
Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn
465 470 475 480
Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly
485 490 495
<210> 7
<211> 437
<212> PRT
<213> Искусственная последовательность
<220>
<223> фруктозо-1,6-бисфосфатаза
<400> 7
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Leu Asp Arg Leu Asp Phe Ser
100 105 110
Ile Lys Leu Leu Arg Lys Val Gly His Leu Leu Met Ile His Trp Gly
115 120 125
Arg Val Asp Asn Val Glu Lys Lys Thr Gly Phe Lys Asp Ile Val Thr
130 135 140
Glu Ile Asp Arg Glu Ala Gln Arg Met Ile Val Asp Glu Ile Arg Lys
145 150 155 160
Phe Phe Pro Asp Glu Asn Ile Met Ala Glu Glu Gly Ile Phe Glu Lys
165 170 175
Gly Asp Arg Leu Trp Ile Ile Asp Pro Ile Asp Gly Thr Ile Asn Phe
180 185 190
Val His Gly Leu Pro Asn Phe Ser Ile Ser Leu Ala Tyr Val Glu Asn
195 200 205
Gly Glu Val Lys Leu Gly Val Val His Ala Pro Ala Leu Asn Glu Thr
210 215 220
Leu Tyr Ala Glu Glu Gly Ser Gly Ala Phe Phe Asn Gly Glu Arg Ile
225 230 235 240
Arg Val Ser Glu Asn Ala Ser Leu Glu Glu Cys Val Gly Ser Thr Gly
245 250 255
Ser Tyr Val Asp Phe Thr Gly Lys Phe Ile Glu Arg Met Glu Lys Arg
260 265 270
Thr Arg Arg Ile Arg Ile Leu Gly Ser Ala Ala Leu Asn Ala Ala Tyr
275 280 285
Val Gly Ala Gly Arg Val Asp Phe Phe Val Thr Trp Arg Ile Asn Pro
290 295 300
Trp Asp Ile Ala Ala Gly Leu Ile Ile Val Lys Glu Ala Gly Gly Met
305 310 315 320
Val Thr Asp Phe Ser Gly Lys Glu Ala Asn Ala Phe Ser Lys Asn Phe
325 330 335
Ile Phe Ser Asn Gly Leu Ile His Asp Glu Val Val Lys Val Val Asn
340 345 350
Glu Val Val Glu Glu Ile Gly Gly Lys Met Gln Ile Phe Val Lys Thr
355 360 365
Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile
370 375 380
Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp
385 390 395 400
Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr
405 410 415
Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu
420 425 430
Arg Leu Arg Gly Gly
435
<210> 8
<211> 772
<212> PRT
<213> Искусственная последовательность
<220>
<223> пируватоксидаза
<400> 8
Met Gly His His His His His His Ser Asp Gln Glu Ala Lys Pro Ser
1 5 10 15
Thr Glu Asp Leu Gly Asp Lys Lys Glu Gly Glu Tyr Ile Lys Leu Lys
20 25 30
Val Ile Gly Gln Asp Ser Ser Glu Ile His Phe Lys Val Lys Met Thr
35 40 45
Thr His Leu Lys Lys Leu Lys Glu Ser Tyr Cys Gln Arg Gln Gly Val
50 55 60
Pro Met Asn Ser Leu Arg Phe Leu Phe Glu Gly Gln Arg Ile Ala Asp
65 70 75 80
Asn His Thr Pro Lys Glu Leu Gly Met Glu Glu Glu Asp Val Ile Glu
85 90 95
Val Tyr Gln Glu Gln Thr Gly Gly Met Ser Asp Asn Lys Ile Asn Ile
100 105 110
Gly Leu Ala Val Met Lys Ile Leu Glu Ser Trp Gly Ala Asp Thr Ile
115 120 125
Tyr Gly Ile Pro Ser Gly Thr Leu Ser Ser Leu Met Asp Ala Met Gly
130 135 140
Glu Glu Glu Asn Asn Val Lys Phe Leu Gln Val Lys His Glu Glu Val
145 150 155 160
Gly Ala Met Ala Ala Val Met Gln Ser Lys Phe Gly Gly Asn Leu Gly
165 170 175
Val Thr Val Gly Ser Gly Gly Pro Gly Ala Ser His Leu Ile Asn Gly
180 185 190
Leu Tyr Asp Ala Ala Met Asp Asn Ile Pro Val Val Ala Ile Leu Gly
195 200 205
Ser Arg Pro Gln Arg Glu Leu Asn Met Asp Ala Phe Gln Glu Leu Asn
210 215 220
Gln Asn Pro Met Tyr Asp His Ile Ala Val Tyr Asn Arg Arg Val Ala
225 230 235 240
Tyr Ala Glu Gln Leu Pro Lys Leu Val Asp Glu Ala Ala Arg Met Ala
245 250 255
Ile Ala Lys Arg Gly Val Ala Val Leu Glu Val Pro Gly Asp Phe Ala
260 265 270
Lys Val Glu Ile Asp Asn Asp Gln Trp Tyr Ser Ser Ala Asn Ser Leu
275 280 285
Arg Lys Tyr Ala Pro Ile Ala Pro Ala Ala Gln Asp Ile Asp Ala Ala
290 295 300
Val Glu Leu Leu Asn Asn Ser Lys Arg Pro Val Ile Tyr Ala Gly Ile
305 310 315 320
Gly Thr Met Gly His Gly Pro Ala Val Gln Glu Leu Ala Arg Lys Ile
325 330 335
Lys Ala Pro Val Ile Thr Thr Gly Lys Asn Phe Glu Thr Phe Glu Trp
340 345 350
Asp Phe Glu Ala Leu Thr Gly Ser Thr Tyr Arg Val Gly Trp Lys Pro
355 360 365
Ala Asn Glu Thr Ile Leu Glu Ala Asp Thr Val Leu Phe Ala Gly Ser
370 375 380
Asn Phe Pro Phe Ser Glu Val Glu Gly Thr Phe Arg Asn Val Asp Asn
385 390 395 400
Phe Ile Gln Ile Asp Ile Asp Pro Ala Met Leu Gly Lys Arg His His
405 410 415
Ala Asp Val Ala Ile Leu Gly Asp Ala Gly Leu Ala Ile Asp Glu Ile
420 425 430
Leu Asn Lys Val Asp Ala Val Glu Glu Ser Ala Trp Trp Thr Ala Asn
435 440 445
Leu Lys Asn Ile Ala Asn Trp Arg Glu Tyr Ile Asn Met Leu Glu Thr
450 455 460
Lys Glu Glu Gly Asp Leu Gln Phe Tyr Gln Val Tyr Asn Ala Ile Asn
465 470 475 480
Asn His Ala Asp Glu Asp Ala Ile Tyr Ser Ile Asp Val Gly Asn Ser
485 490 495
Thr Gln Thr Ser Ile Arg His Leu His Met Thr Pro Lys Asn Met Trp
500 505 510
Arg Thr Ser Pro Leu Phe Ala Thr Met Gly Ile Ala Ile Pro Gly Gly
515 520 525
Leu Gly Ala Lys Asn Thr Tyr Pro Asp Arg Gln Val Trp Asn Ile Ile
530 535 540
Gly Asp Gly Ala Phe Ser Met Thr Tyr Pro Asp Val Val Thr Asn Val
545 550 555 560
Arg Tyr Asn Met Pro Val Ile Asn Val Val Phe Ser Asn Thr Glu Tyr
565 570 575
Ala Phe Ile Lys Asn Lys Tyr Glu Asp Thr Asn Lys Asn Leu Phe Gly
580 585 590
Val Asp Phe Thr Asp Val Asp Tyr Ala Lys Ile Ala Glu Ala Gln Gly
595 600 605
Ala Lys Gly Phe Thr Val Ser Arg Ile Glu Asp Met Asp Arg Val Met
610 615 620
Ala Glu Ala Val Ala Ala Asn Lys Ala Gly His Thr Val Val Ile Asp
625 630 635 640
Cys Lys Ile Thr Gln Asp Arg Pro Ile Pro Val Glu Thr Leu Lys Leu
645 650 655
Asp Ser Lys Leu Tyr Ser Glu Asp Glu Ile Lys Ala Tyr Lys Glu Arg
660 665 670
Tyr Glu Ala Ala Asn Leu Val Pro Phe Arg Glu Tyr Leu Glu Ala Glu
675 680 685
Gly Leu Glu Ser Lys Tyr Ile Lys Met Gln Ile Phe Val Lys Thr Leu
690 695 700
Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser Asp Thr Ile Glu
705 710 715 720
Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln
725 730 735
Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu
740 745 750
Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg
755 760 765
Leu Arg Gly Gly
770
<210> 9
<211> 1024
<212> PRT
<213> Искусственная последовательность
<220>
<223> Дрожжевой UBE1
<400> 9
Met Ser Ser Asn Asn Ser Gly Leu Ser Ala Ala Gly Glu Ile Asp Glu
1 5 10 15
Ser Leu Tyr Ser Arg Gln Leu Tyr Val Leu Gly Lys Glu Ala Met Leu
20 25 30
Lys Met Gln Thr Ser Asn Val Leu Ile Leu Gly Leu Lys Gly Leu Gly
35 40 45
Val Glu Ile Ala Lys Asn Val Val Leu Ala Gly Val Lys Ser Met Thr
50 55 60
Val Phe Asp Pro Glu Pro Val Gln Leu Ala Asp Leu Ser Thr Gln Phe
65 70 75 80
Phe Leu Thr Glu Lys Asp Ile Gly Gln Lys Arg Gly Asp Val Thr Arg
85 90 95
Ala Lys Leu Ala Glu Leu Asn Ala Tyr Val Pro Val Asn Val Leu Asp
100 105 110
Ser Leu Asp Asp Val Thr Gln Leu Ser Gln Phe Gln Val Val Val Ala
115 120 125
Thr Asp Thr Val Ser Leu Glu Asp Lys Val Lys Ile Asn Glu Phe Cys
130 135 140
His Ser Ser Gly Ile Arg Phe Ile Ser Ser Glu Thr Arg Gly Leu Phe
145 150 155 160
Gly Asn Thr Phe Val Asp Leu Gly Asp Glu Phe Thr Val Leu Asp Pro
165 170 175
Thr Gly Glu Glu Pro Arg Thr Gly Met Val Ser Asp Ile Glu Pro Asp
180 185 190
Gly Thr Val Thr Met Leu Asp Asp Asn Arg His Gly Leu Glu Asp Gly
195 200 205
Asn Phe Val Arg Phe Ser Glu Val Glu Gly Leu Asp Lys Leu Asn Asp
210 215 220
Gly Thr Leu Phe Lys Val Glu Val Leu Gly Pro Phe Ala Phe Arg Ile
225 230 235 240
Gly Ser Val Lys Glu Tyr Gly Glu Tyr Lys Lys Gly Gly Ile Phe Thr
245 250 255
Glu Val Lys Val Pro Arg Lys Ile Ser Phe Lys Ser Leu Lys Gln Gln
260 265 270
Leu Ser Asn Pro Glu Phe Val Phe Ser Asp Phe Ala Lys Phe Asp Arg
275 280 285
Ala Ala Gln Leu His Leu Gly Phe Gln Ala Leu His Gln Phe Ala Val
290 295 300
Arg His Asn Gly Glu Leu Pro Arg Thr Met Asn Asp Glu Asp Ala Asn
305 310 315 320
Glu Leu Ile Lys Leu Val Thr Asp Leu Ser Val Gln Gln Pro Glu Val
325 330 335
Leu Gly Glu Gly Val Asp Val Asn Glu Asp Leu Ile Lys Glu Leu Ser
340 345 350
Tyr Gln Ala Arg Gly Asp Ile Pro Gly Val Val Ala Phe Phe Gly Gly
355 360 365
Leu Val Ala Gln Glu Val Leu Lys Ala Cys Ser Gly Lys Phe Thr Pro
370 375 380
Leu Lys Gln Phe Met Tyr Phe Asp Ser Leu Glu Ser Leu Pro Asp Pro
385 390 395 400
Lys Asn Phe Pro Arg Asn Glu Lys Thr Thr Gln Pro Val Asn Ser Arg
405 410 415
Tyr Asp Asn Gln Ile Ala Val Phe Gly Leu Asp Phe Gln Lys Lys Ile
420 425 430
Ala Asn Ser Lys Val Phe Leu Val Gly Ser Gly Ala Ile Gly Cys Glu
435 440 445
Met Leu Lys Asn Trp Ala Leu Leu Gly Leu Gly Ser Gly Ser Asp Gly
450 455 460
Tyr Ile Val Val Thr Asp Asn Asp Ser Ile Glu Lys Ser Asn Leu Asn
465 470 475 480
Arg Gln Phe Leu Phe Arg Pro Lys Asp Val Gly Lys Asn Lys Ser Glu
485 490 495
Val Ala Ala Glu Ala Val Cys Ala Met Asn Pro Asp Leu Lys Gly Lys
500 505 510
Ile Asn Ala Lys Ile Asp Lys Val Gly Pro Glu Thr Glu Glu Ile Phe
515 520 525
Asn Asp Ser Phe Trp Glu Ser Leu Asp Phe Val Thr Asn Ala Leu Asp
530 535 540
Asn Val Asp Ala Arg Thr Tyr Val Asp Arg Arg Cys Val Phe Tyr Arg
545 550 555 560
Lys Pro Leu Leu Glu Ser Gly Thr Leu Gly Thr Lys Gly Asn Thr Gln
565 570 575
Val Ile Ile Pro Arg Leu Thr Glu Ser Tyr Ser Ser Ser Arg Asp Pro
580 585 590
Pro Glu Lys Ser Ile Pro Leu Cys Thr Leu Arg Ser Phe Pro Asn Lys
595 600 605
Ile Asp His Thr Ile Ala Trp Ala Lys Ser Leu Phe Gln Gly Tyr Phe
610 615 620
Thr Asp Ser Ala Glu Asn Val Asn Met Tyr Leu Thr Gln Pro Asn Phe
625 630 635 640
Val Glu Gln Thr Leu Lys Gln Ser Gly Asp Val Lys Gly Val Leu Glu
645 650 655
Ser Ile Ser Asp Ser Leu Ser Ser Lys Pro His Asn Phe Glu Asp Cys
660 665 670
Ile Lys Trp Ala Arg Leu Glu Phe Glu Lys Lys Phe Asn His Asp Ile
675 680 685
Lys Gln Leu Leu Phe Asn Phe Pro Lys Asp Ala Lys Thr Ser Asn Gly
690 695 700
Glu Pro Phe Trp Ser Gly Ala Lys Arg Ala Pro Thr Pro Leu Glu Phe
705 710 715 720
Asp Ile Tyr Asn Asn Asp His Phe His Phe Val Val Ala Gly Ala Ser
725 730 735
Leu Arg Ala Tyr Asn Tyr Gly Ile Lys Ser Asp Asp Ser Asn Ser Lys
740 745 750
Pro Asn Val Asp Glu Tyr Lys Ser Val Ile Asp His Met Ile Ile Pro
755 760 765
Glu Phe Thr Pro Asn Ala Asn Leu Lys Ile Gln Val Asn Asp Asp Asp
770 775 780
Pro Asp Pro Asn Ala Asn Ala Ala Asn Gly Ser Asp Glu Ile Asp Gln
785 790 795 800
Leu Val Ser Ser Leu Pro Asp Pro Ser Thr Leu Ala Gly Phe Lys Leu
805 810 815
Glu Pro Val Asp Phe Glu Lys Asp Asp Asp Thr Asn His His Ile Glu
820 825 830
Phe Ile Thr Ala Cys Ser Asn Cys Arg Ala Gln Asn Tyr Phe Ile Glu
835 840 845
Thr Ala Asp Arg Gln Lys Thr Lys Phe Ile Ala Gly Arg Ile Ile Pro
850 855 860
Ala Ile Ala Thr Thr Thr Ser Leu Val Thr Gly Leu Val Asn Leu Glu
865 870 875 880
Leu Tyr Lys Leu Ile Asp Asn Lys Thr Asp Ile Glu Gln Tyr Lys Asn
885 890 895
Gly Phe Val Asn Leu Ala Leu Pro Phe Phe Gly Phe Ser Glu Pro Ile
900 905 910
Ala Ser Pro Lys Gly Glu Tyr Asn Asn Lys Lys Tyr Asp Lys Ile Trp
915 920 925
Asp Arg Phe Asp Ile Lys Gly Asp Ile Lys Leu Ser Asp Leu Ile Glu
930 935 940
His Phe Glu Lys Asp Glu Gly Leu Glu Ile Thr Met Leu Ser Tyr Gly
945 950 955 960
Val Ser Leu Leu Tyr Ala Ser Phe Phe Pro Pro Lys Lys Leu Lys Glu
965 970 975
Arg Leu Asn Leu Pro Ile Thr Gln Leu Val Lys Leu Val Thr Lys Lys
980 985 990
Asp Ile Pro Ala His Val Ser Thr Met Ile Leu Glu Ile Cys Ala Asp
995 1000 1005
Asp Lys Glu Gly Glu Asp Val Glu Val Pro Phe Ile Thr Ile His Leu
1010 1015 1020
<210> 10
<211> 147
<212> PRT
<213> Искусственная последовательность
<220>
<223> Ubch5a (Homo sapiens)
<400> 10
Met Ala Leu Lys Arg Ile Gln Lys Glu Leu Ser Asp Leu Gln Arg Asp
1 5 10 15
Pro Pro Ala His Cys Ser Ala Gly Pro Val Gly Asp Asp Leu Phe His
20 25 30
Trp Gln Ala Thr Ile Met Gly Pro Pro Asp Ser Ala Tyr Gln Gly Gly
35 40 45
Val Phe Phe Leu Thr Val His Phe Pro Thr Asp Tyr Pro Phe Lys Pro
50 55 60
Pro Lys Ile Ala Phe Thr Thr Lys Ile Tyr His Pro Asn Ile Asn Ser
65 70 75 80
Asn Gly Ser Ile Cys Leu Asp Ile Leu Arg Ser Gln Trp Ser Pro Ala
85 90 95
Leu Thr Val Ser Lys Val Leu Leu Ser Ile Cys Ser Leu Leu Cys Asp
100 105 110
Pro Asn Pro Asp Asp Pro Leu Val Pro Asp Ile Ala Gln Ile Tyr Lys
115 120 125
Ser Asp Lys Glu Lys Tyr Asn Arg His Ala Arg Glu Trp Thr Gln Lys
130 135 140
Tyr Ala Met
145
<210> 11
<211> 154
<212> PRT
<213> Искусственная последовательность
<220>
<223> Ubch7 (Homo sapiens)
<400> 11
Met Ala Ala Ser Arg Arg Leu Met Lys Glu Leu Glu Glu Ile Arg Lys
1 5 10 15
Cys Gly Met Lys Asn Phe Arg Asn Ile Gln Val Asp Glu Ala Asn Leu
20 25 30
Leu Thr Trp Gln Gly Leu Ile Val Pro Asp Asn Pro Pro Tyr Asp Lys
35 40 45
Gly Ala Phe Arg Ile Glu Ile Asn Phe Pro Ala Glu Tyr Pro Phe Lys
50 55 60
Pro Pro Lys Ile Thr Phe Lys Thr Lys Ile Tyr His Pro Asn Ile Asp
65 70 75 80
Glu Lys Gly Gln Val Cys Leu Pro Val Ile Ser Ala Glu Asn Trp Lys
85 90 95
Pro Ala Thr Lys Thr Asp Gln Val Ile Gln Ser Leu Ile Ala Leu Val
100 105 110
Asn Asp Pro Gln Pro Glu His Pro Leu Arg Ala Asp Leu Ala Glu Glu
115 120 125
Tyr Ser Lys Asp Arg Lys Lys Phe Cys Lys Asn Ala Glu Glu Phe Thr
130 135 140
Lys Lys Tyr Gly Glu Lys Arg Pro Val Asp
145 150
<210> 12
<211> 200
<212> PRT
<213> Искусственная последовательность
<220>
<223> E2-25K (Homo sapiens)
<400> 12
Met Ala Asn Ile Ala Val Gln Arg Ile Lys Arg Glu Phe Lys Glu Val
1 5 10 15
Leu Lys Ser Glu Glu Thr Ser Lys Asn Gln Ile Lys Val Asp Leu Val
20 25 30
Asp Glu Asn Phe Thr Glu Leu Arg Gly Glu Ile Ala Gly Pro Pro Asp
35 40 45
Thr Pro Tyr Glu Gly Gly Arg Tyr Gln Leu Glu Ile Lys Ile Pro Glu
50 55 60
Thr Tyr Pro Phe Asn Pro Pro Lys Val Arg Phe Ile Thr Lys Ile Trp
65 70 75 80
His Pro Asn Ile Ser Ser Val Thr Gly Ala Ile Cys Leu Asp Ile Leu
85 90 95
Lys Asp Gln Trp Ala Ala Ala Met Thr Leu Arg Thr Val Leu Leu Ser
100 105 110
Leu Gln Ala Leu Leu Ala Ala Ala Glu Pro Asp Asp Pro Gln Asp Ala
115 120 125
Val Val Ala Asn Gln Tyr Lys Gln Asn Pro Glu Met Phe Lys Gln Thr
130 135 140
Ala Arg Leu Trp Ala His Val Tyr Ala Gly Ala Pro Val Ser Ser Pro
145 150 155 160
Glu Tyr Thr Lys Lys Ile Glu Asn Leu Cys Ala Met Gly Phe Asp Arg
165 170 175
Asn Ala Val Ile Val Ala Leu Ser Ser Lys Ser Trp Asp Val Glu Thr
180 185 190
Ala Thr Glu Leu Leu Leu Ser Asn
195 200
<210> 13
<211> 153
<212> PRT
<213> Искусственная последовательность
<220>
<223> Ubc13 (Saccharomyces cerevisiae)
<400> 13
Met Ala Ser Leu Pro Lys Arg Ile Ile Lys Glu Thr Glu Lys Leu Val
1 5 10 15
Ser Asp Pro Val Pro Gly Ile Thr Ala Glu Pro His Asp Asp Asn Leu
20 25 30
Arg Tyr Phe Gln Val Thr Ile Glu Gly Pro Glu Gln Ser Pro Tyr Glu
35 40 45
Asp Gly Ile Phe Glu Leu Glu Leu Tyr Leu Pro Asp Asp Tyr Pro Met
50 55 60
Glu Ala Pro Lys Val Arg Phe Leu Thr Lys Ile Tyr His Pro Asn Ile
65 70 75 80
Asp Arg Leu Gly Arg Ile Cys Leu Asp Val Leu Lys Thr Asn Trp Ser
85 90 95
Pro Ala Leu Gln Ile Arg Thr Val Leu Leu Ser Ile Gln Ala Leu Leu
100 105 110
Ala Ser Pro Asn Pro Asn Asp Pro Leu Ala Asn Asp Val Ala Glu Asp
115 120 125
Trp Ile Lys Asn Glu Gln Gly Ala Lys Ala Lys Ala Arg Glu Trp Thr
130 135 140
Lys Leu Tyr Ala Lys Lys Lys Pro Glu
145 150
<210> 14
<211> 137
<212> PRT
<213> Искусственная последовательность
<220>
<223> MMS2 (UEV - вариант фермента конъюгации убиквитина)
(Saccharomyces
cerevisiae)
<400> 14
Met Ser Lys Val Pro Arg Asn Phe Arg Leu Leu Glu Glu Leu Glu Lys
1 5 10 15
Gly Glu Lys Gly Phe Gly Pro Glu Ser Cys Ser Tyr Gly Leu Ala Asp
20 25 30
Ser Asp Asp Ile Thr Met Thr Lys Trp Asn Gly Thr Ile Leu Gly Pro
35 40 45
Pro His Ser Asn His Glu Asn Arg Ile Tyr Ser Leu Ser Ile Asp Cys
50 55 60
Gly Pro Asn Tyr Pro Asp Ser Pro Pro Lys Val Thr Phe Ile Ser Lys
65 70 75 80
Ile Asn Leu Pro Cys Val Asn Pro Thr Thr Gly Glu Val Gln Thr Asp
85 90 95
Phe His Thr Leu Arg Asp Trp Lys Arg Ala Tyr Thr Met Glu Thr Leu
100 105 110
Leu Leu Asp Leu Arg Lys Glu Met Ala Thr Pro Ala Asn Lys Lys Leu
115 120 125
Arg Gln Pro Lys Glu Gly Glu Thr Phe
130 135
<210> 15
<211> 807
<212> PRT
<213> Искусственная последовательность
<220>
<223> E3 RSP5
<400> 15
Met Pro Ser Ser Ile Ser Val Lys Leu Val Ala Ala Glu Ser Leu Tyr
1 5 10 15
Lys Arg Asp Val Phe Arg Ser Pro Asp Pro Phe Ala Val Leu Thr Ile
20 25 30
Asp Gly Tyr Gln Thr Lys Ser Thr Ser Ala Ala Lys Lys Thr Leu Asn
35 40 45
Pro Tyr Trp Asn Glu Thr Phe Lys Phe Asp Asp Ile Asn Glu Asn Ser
50 55 60
Ile Leu Thr Ile Gln Val Phe Asp Gln Lys Lys Phe Lys Lys Lys Asp
65 70 75 80
Gln Gly Phe Leu Gly Val Val Asn Val Arg Val Gly Asp Val Leu Gly
85 90 95
His Leu Asp Glu Asp Thr Ala Thr Ser Ser Gly Arg Pro Arg Glu Glu
100 105 110
Thr Ile Thr Arg Asp Leu Lys Lys Ser Asn Asp Gly Met Ala Val Ser
115 120 125
Gly Arg Leu Ile Val Val Leu Ser Lys Leu Pro Ser Ser Ser Pro His
130 135 140
Ser Gln Ala Pro Ser Gly His Thr Ala Ser Ser Ser Thr Asn Thr Ser
145 150 155 160
Ser Thr Thr Arg Thr Asn Gly His Ser Thr Ser Ser Thr Arg Asn His
165 170 175
Ser Thr Ser His Pro Ser Arg Gly Thr Ala Gln Ala Val Glu Ser Thr
180 185 190
Leu Gln Ser Gly Thr Thr Ala Ala Thr Asn Thr Ala Thr Thr Ser His
195 200 205
Arg Ser Thr Asn Ser Thr Ser Ser Ala Thr Arg Gln Tyr Ser Ser Phe
210 215 220
Glu Asp Gln Tyr Gly Arg Leu Pro Pro Gly Trp Glu Arg Arg Thr Asp
225 230 235 240
Asn Phe Gly Arg Thr Tyr Tyr Val Asp His Asn Thr Arg Thr Thr Thr
245 250 255
Trp Lys Arg Pro Thr Leu Asp Gln Thr Glu Ala Glu Arg Gly Gln Leu
260 265 270
Asn Ala Asn Thr Glu Leu Glu Arg Arg Gln His Arg Gly Arg Thr Leu
275 280 285
Pro Gly Gly Ser Ser Asp Asn Ser Ser Val Thr Val Gln Val Gly Gly
290 295 300
Gly Ser Asn Ile Pro Pro Val Asn Gly Ala Ala Ala Ala Ala Phe Ala
305 310 315 320
Ala Thr Gly Gly Thr Thr Ser Gly Leu Gly Glu Leu Pro Ser Gly Trp
325 330 335
Glu Gln Arg Phe Thr Pro Glu Gly Arg Ala Tyr Phe Val Asp His Asn
340 345 350
Thr Arg Thr Thr Thr Trp Val Asp Pro Arg Arg Gln Gln Tyr Ile Arg
355 360 365
Thr Tyr Gly Pro Thr Asn Thr Thr Ile Gln Gln Gln Pro Val Ser Gln
370 375 380
Leu Gly Pro Leu Pro Ser Gly Trp Glu Met Arg Leu Thr Asn Thr Ala
385 390 395 400
Arg Val Tyr Phe Val Asp His Asn Thr Lys Thr Thr Thr Trp Asp Asp
405 410 415
Pro Arg Leu Pro Ser Ser Leu Asp Gln Asn Val Pro Gln Tyr Lys Arg
420 425 430
Asp Phe Arg Arg Lys Val Ile Tyr Phe Arg Ser Gln Pro Ala Leu Arg
435 440 445
Ile Leu Pro Gly Gln Cys His Ile Lys Val Arg Arg Lys Asn Ile Phe
450 455 460
Glu Asp Ala Tyr Gln Glu Ile Met Arg Gln Thr Pro Glu Asp Leu Lys
465 470 475 480
Lys Arg Leu Met Ile Lys Phe Asp Gly Glu Glu Gly Leu Asp Tyr Gly
485 490 495
Gly Val Ser Arg Glu Phe Phe Phe Leu Leu Ser His Glu Met Phe Asn
500 505 510
Pro Phe Tyr Cys Leu Phe Glu Tyr Ser Ala Tyr Asp Asn Tyr Thr Ile
515 520 525
Gln Ile Asn Pro Asn Ser Gly Ile Asn Pro Glu His Leu Asn Tyr Phe
530 535 540
Lys Phe Ile Gly Arg Val Val Gly Leu Gly Val Phe His Arg Arg Phe
545 550 555 560
Leu Asp Ala Phe Phe Val Gly Ala Leu Tyr Lys Met Met Leu Arg Lys
565 570 575
Lys Val Val Leu Gln Asp Met Glu Gly Val Asp Ala Glu Val Tyr Asn
580 585 590
Ser Leu Asn Trp Met Leu Glu Asn Ser Ile Asp Gly Val Leu Asp Leu
595 600 605
Thr Phe Ser Ala Asp Asp Glu Arg Phe Gly Glu Val Val Thr Val Asp
610 615 620
Leu Lys Pro Asp Gly Arg Asn Ile Glu Val Thr Asp Gly Asn Lys Lys
625 630 635 640
Glu Tyr Val Glu Leu Tyr Thr Gln Trp Arg Ile Val Asp Arg Val Gln
645 650 655
Glu Gln Phe Lys Ala Phe Met Asp Gly Phe Asn Glu Ile Pro Glu Asp
660 665 670
Leu Val Thr Val Phe Asp Glu Arg Glu Leu Glu Leu Leu Ile Gly Gly
675 680 685
Ile Ala Glu Ile Asp Ile Glu Asp Trp Lys Lys His Thr Asp Tyr Arg
690 695 700
Gly Tyr Gln Glu Ser Asp Glu Val Ile Gln Trp Phe Trp Lys Cys Val
705 710 715 720
Ser Glu Trp Asp Asn Glu Gln Arg Ala Arg Leu Leu Gln Phe Thr Thr
725 730 735
Gly Thr Ser Arg Ile Pro Val Asn Gly Phe Lys Asp Leu Gln Gly Ser
740 745 750
Asp Gly Pro Arg Arg Phe Thr Ile Glu Lys Ala Gly Glu Val Gln Gln
755 760 765
Leu Pro Lys Ser His Thr Cys Phe Asn Arg Val Asp Leu Pro Gln Tyr
770 775 780
Val Asp Tyr Asp Ser Met Lys Gln Lys Leu Thr Leu Ala Val Glu Glu
785 790 795 800
Thr Ile Gly Phe Gly Gln Glu
805
<210> 16
<211> 1319
<212> PRT
<213> Искусственная последовательность
<220>
<223> E3 DOA10
<400> 16
Met Asp Val Asp Ser Asp Val Asn Val Ser Arg Leu Arg Asp Glu Leu
1 5 10 15
His Lys Val Ala Asn Glu Glu Thr Asp Thr Ala Thr Phe Asn Asp Asp
20 25 30
Ala Pro Ser Gly Ala Thr Cys Arg Ile Cys Arg Gly Glu Ala Thr Glu
35 40 45
Asp Asn Pro Leu Phe His Pro Cys Lys Cys Arg Gly Ser Ile Lys Tyr
50 55 60
Met His Glu Ser Cys Leu Leu Glu Trp Val Ala Ser Lys Asn Ile Asp
65 70 75 80
Ile Ser Lys Pro Gly Ala Asp Val Lys Cys Asp Ile Cys His Tyr Pro
85 90 95
Ile Gln Phe Lys Thr Ile Tyr Ala Glu Asn Met Pro Glu Lys Ile Pro
100 105 110
Phe Ser Leu Leu Leu Ser Lys Ser Ile Leu Thr Phe Phe Glu Lys Ala
115 120 125
Arg Leu Ala Leu Thr Ile Gly Leu Ala Ala Val Leu Tyr Ile Ile Gly
130 135 140
Val Pro Leu Val Trp Asn Met Phe Gly Lys Leu Tyr Thr Met Met Leu
145 150 155 160
Asp Gly Ser Ser Pro Tyr Pro Gly Asp Phe Leu Lys Ser Leu Ile Tyr
165 170 175
Gly Tyr Asp Gln Ser Ala Thr Pro Glu Leu Thr Thr Arg Ala Ile Phe
180 185 190
Tyr Gln Leu Leu Gln Asn His Ser Phe Thr Ser Leu Gln Phe Ile Met
195 200 205
Ile Val Ile Leu His Ile Ala Leu Tyr Phe Gln Tyr Asp Met Ile Val
210 215 220
Arg Glu Asp Val Phe Ser Lys Met Val Phe His Lys Ile Gly Pro Arg
225 230 235 240
Leu Ser Pro Lys Asp Leu Lys Ser Arg Leu Lys Glu Arg Phe Pro Met
245 250 255
Met Asp Asp Arg Met Val Glu Tyr Leu Ala Arg Glu Met Arg Ala His
260 265 270
Asp Glu Asn Arg Gln Glu Gln Gly His Asp Arg Leu Asn Met Pro Ala
275 280 285
Ala Ala Ala Asp Asn Asn Asn Asn Val Ile Asn Pro Arg Asn Asp Asn
290 295 300
Val Pro Pro Gln Asp Pro Asn Asp His Arg Asn Phe Glu Asn Leu Arg
305 310 315 320
His Val Asp Glu Leu Asp His Asp Glu Ala Thr Glu Glu His Glu Asn
325 330 335
Asn Asp Ser Asp Asn Ser Leu Pro Ser Gly Asp Asp Ser Ser Arg Ile
340 345 350
Leu Pro Gly Ser Ser Ser Asp Asn Glu Glu Asp Glu Glu Ala Glu Gly
355 360 365
Gln Gln Gln Gln Gln Gln Pro Glu Glu Glu Ala Asp Tyr Arg Asp His
370 375 380
Ile Glu Pro Asn Pro Ile Asp Met Trp Ala Asn Arg Arg Ala Gln Asn
385 390 395 400
Glu Phe Asp Asp Leu Ile Ala Ala Gln Gln Asn Ala Ile Asn Arg Pro
405 410 415
Asn Ala Pro Val Phe Ile Pro Pro Pro Ala Gln Asn Arg Ala Gly Asn
420 425 430
Val Asp Gln Asp Glu Gln Asp Phe Gly Ala Ala Val Gly Val Pro Pro
435 440 445
Ala Gln Ala Asn Pro Asp Asp Gln Gly Gln Gly Pro Leu Val Ile Asn
450 455 460
Leu Lys Leu Lys Leu Leu Asn Val Ile Ala Tyr Phe Ile Ile Ala Val
465 470 475 480
Val Phe Thr Ala Ile Tyr Leu Ala Ile Ser Tyr Leu Phe Pro Thr Phe
485 490 495
Ile Gly Phe Gly Leu Leu Lys Ile Tyr Phe Gly Ile Phe Lys Val Ile
500 505 510
Leu Arg Gly Leu Cys His Leu Tyr Tyr Leu Ser Gly Ala His Ile Ala
515 520 525
Tyr Asn Gly Leu Thr Lys Leu Val Pro Lys Val Asp Val Ala Met Ser
530 535 540
Trp Ile Ser Asp His Leu Ile His Asp Ile Ile Tyr Leu Tyr Asn Gly
545 550 555 560
Tyr Thr Glu Asn Thr Met Lys His Ser Ile Phe Ile Arg Ala Leu Pro
565 570 575
Ala Leu Thr Thr Tyr Leu Thr Ser Val Ser Ile Val Cys Ala Ser Ser
580 585 590
Asn Leu Val Ser Arg Gly Tyr Gly Arg Glu Asn Gly Met Ser Asn Pro
595 600 605
Thr Arg Arg Leu Ile Phe Gln Ile Leu Phe Ala Leu Lys Cys Thr Phe
610 615 620
Lys Val Phe Thr Leu Phe Phe Ile Glu Leu Ala Gly Phe Pro Ile Leu
625 630 635 640
Ala Gly Val Met Leu Asp Phe Ser Leu Phe Cys Pro Ile Leu Ala Ser
645 650 655
Asn Ser Arg Met Leu Trp Val Pro Ser Ile Cys Ala Ile Trp Pro Pro
660 665 670
Phe Ser Leu Phe Val Tyr Trp Thr Ile Gly Thr Leu Tyr Met Tyr Trp
675 680 685
Phe Ala Lys Tyr Ile Gly Met Ile Arg Lys Asn Ile Ile Arg Pro Gly
690 695 700
Val Leu Phe Phe Ile Arg Ser Pro Glu Asp Pro Asn Ile Lys Ile Leu
705 710 715 720
His Asp Ser Leu Ile His Pro Met Ser Ile Gln Leu Ser Arg Leu Cys
725 730 735
Leu Ser Met Phe Ile Tyr Ala Ile Phe Ile Val Leu Gly Phe Gly Phe
740 745 750
His Thr Arg Ile Phe Phe Pro Phe Met Leu Lys Ser Asn Leu Leu Ser
755 760 765
Val Pro Glu Ala Tyr Lys Pro Thr Ser Ile Ile Ser Trp Lys Phe Asn
770 775 780
Thr Ile Leu Leu Thr Leu Tyr Phe Thr Lys Arg Ile Leu Glu Ser Ser
785 790 795 800
Ser Tyr Val Lys Pro Leu Leu Glu Arg Tyr Trp Lys Thr Ile Phe Lys
805 810 815
Leu Cys Ser Arg Lys Leu Arg Leu Ser Ser Phe Ile Leu Gly Lys Asp
820 825 830
Thr Pro Thr Glu Arg Gly His Ile Val Tyr Arg Asn Leu Phe Tyr Lys
835 840 845
Tyr Ile Ala Ala Lys Asn Ala Glu Trp Ser Asn Gln Glu Leu Phe Thr
850 855 860
Lys Pro Lys Thr Leu Glu Gln Ala Glu Glu Leu Phe Gly Gln Val Arg
865 870 875 880
Asp Val His Ala Tyr Phe Val Pro Asp Gly Val Leu Met Arg Val Pro
885 890 895
Ser Ser Asp Ile Val Ser Arg Asn Tyr Val Gln Thr Met Phe Val Pro
900 905 910
Val Thr Lys Asp Asp Lys Leu Leu Lys Pro Leu Asp Leu Glu Arg Ile
915 920 925
Lys Glu Arg Asn Lys Arg Ala Ala Gly Glu Phe Gly Tyr Leu Asp Glu
930 935 940
Gln Asn Thr Glu Tyr Asp Gln Tyr Tyr Ile Val Tyr Val Pro Pro Asp
945 950 955 960
Phe Arg Leu Arg Tyr Met Thr Leu Leu Gly Leu Val Trp Leu Phe Ala
965 970 975
Ser Ile Leu Met Leu Gly Val Thr Phe Ile Ser Gln Ala Leu Ile Asn
980 985 990
Phe Val Cys Ser Phe Gly Phe Leu Pro Val Val Lys Leu Leu Leu Gly
995 1000 1005
Glu Arg Asn Lys Val Tyr Val Ala Trp Lys Glu Leu Ser Asp Ile Ser
1010 1015 1020
Tyr Ser Tyr Leu Asn Ile Tyr Tyr Val Cys Val Gly Ser Val Cys Leu
1025 1030 1035 1040
Ser Lys Ile Ala Lys Asp Ile Leu His Phe Thr Glu Gly Gln Asn Thr
1045 1050 1055
Leu Asp Glu His Ala Val Asp Glu Asn Glu Val Glu Glu Val Glu His
1060 1065 1070
Asp Ile Pro Glu Arg Asp Ile Asn Asn Ala Pro Val Asn Asn Ile Asn
1075 1080 1085
Asn Val Glu Glu Gly Gln Gly Ile Phe Met Ala Ile Phe Asn Ser Ile
1090 1095 1100
Phe Asp Ser Met Leu Val Lys Tyr Asn Leu Met Val Phe Ile Ala Ile
1105 1110 1115 1120
Met Ile Ala Val Ile Arg Thr Met Val Ser Trp Val Val Leu Thr Asp
1125 1130 1135
Gly Ile Leu Ala Cys Tyr Asn Tyr Leu Thr Ile Arg Val Phe Gly Asn
1140 1145 1150
Ser Ser Tyr Thr Ile Gly Asn Ser Lys Trp Phe Lys Tyr Asp Glu Ser
1155 1160 1165
Leu Leu Phe Val Val Trp Ile Ile Ser Ser Met Val Asn Phe Gly Thr
1170 1175 1180
Gly Tyr Lys Ser Leu Lys Leu Phe Phe Arg Asn Arg Asn Thr Ser Lys
1185 1190 1195 1200
Leu Asn Phe Leu Lys Thr Met Ala Leu Glu Leu Phe Lys Gln Gly Phe
1205 1210 1215
Leu His Met Val Ile Tyr Val Leu Pro Ile Ile Ile Leu Ser Leu Val
1220 1225 1230
Phe Leu Arg Asp Val Ser Thr Lys Gln Ile Ile Asp Ile Ser His Gly
1235 1240 1245
Ser Arg Ser Phe Thr Leu Ser Leu Asn Glu Ser Phe Pro Thr Trp Thr
1250 1255 1260
Arg Met Gln Asp Ile Tyr Phe Gly Leu Leu Ile Ala Leu Glu Ser Phe
1265 1270 1275 1280
Thr Phe Phe Phe Gln Ala Thr Val Leu Phe Ile Gln Trp Phe Lys Ser
1285 1290 1295
Thr Val Gln Asn Val Lys Asp Glu Val Tyr Thr Lys Gly Arg Ala Leu
1300 1305 1310
Glu Asn Leu Pro Asp Glu Ser
1315
<210> 17
<211> 278
<212> PRT
<213> Искусственная последовательность
<220>
<223> E3 MARCH5
<400> 17
Met Pro Asp Gln Ala Leu Gln Gln Met Leu Asp Arg Ser Cys Trp Val
1 5 10 15
Cys Phe Ala Thr Asp Glu Asp Asp Arg Thr Ala Glu Trp Val Arg Pro
20 25 30
Cys Arg Cys Arg Gly Ser Thr Lys Trp Val His Gln Ala Cys Leu Gln
35 40 45
Arg Trp Val Asp Glu Lys Gln Arg Gly Asn Ser Thr Ala Arg Val Ala
50 55 60
Cys Pro Gln Cys Asn Ala Glu Tyr Leu Ile Val Phe Pro Lys Leu Gly
65 70 75 80
Pro Val Val Tyr Val Leu Asp Leu Ala Asp Arg Leu Ile Ser Lys Ala
85 90 95
Cys Pro Phe Ala Ala Ala Gly Ile Met Val Gly Ser Ile Tyr Trp Thr
100 105 110
Ala Val Thr Tyr Gly Ala Val Thr Val Met Gln Val Val Gly His Lys
115 120 125
Glu Gly Leu Asp Val Met Glu Arg Ala Asp Pro Leu Phe Leu Leu Ile
130 135 140
Gly Leu Pro Thr Ile Pro Val Met Leu Ile Leu Gly Lys Met Ile Arg
145 150 155 160
Trp Glu Asp Tyr Val Leu Arg Leu Trp Arg Lys Tyr Ser Asn Lys Leu
165 170 175
Gln Ile Leu Asn Ser Ile Phe Pro Gly Ile Gly Cys Pro Val Pro Arg
180 185 190
Ile Pro Ala Glu Ala Asn Pro Leu Ala Asp His Val Ser Ala Thr Arg
195 200 205
Ile Leu Cys Gly Ala Leu Val Phe Pro Thr Ile Ala Thr Ile Val Gly
210 215 220
Lys Leu Met Phe Ser Ser Val Asn Ser Asn Leu Gln Arg Thr Ile Leu
225 230 235 240
Gly Gly Ile Ala Phe Val Ala Ile Lys Gly Ala Phe Lys Val Tyr Phe
245 250 255
Lys Gln Gln Gln Tyr Leu Arg Gln Ala His Arg Lys Ile Leu Asn Tyr
260 265 270
Pro Glu Gln Glu Glu Ala
275
<210> 18
<211> 1116
<212> PRT
<213> Искусственная последовательность
<220>
<223> His-SUMO-hGH-Ub(D)
<400> 18
Met Glu Thr Gly Leu Tyr His Ile Ser His Ile Ser His Ile Ser His
1 5 10 15
Ile Ser His Ile Ser His Ile Ser Ser Glu Arg Ala Ser Pro Gly Leu
20 25 30
Asn Gly Leu Ala Ala Leu Ala Leu Tyr Ser Pro Arg Ala Ser Glu Arg
35 40 45
Thr His Arg Gly Leu Ala Ala Ser Pro Leu Glu Ala Gly Leu Tyr Ala
50 55 60
Ser Pro Leu Tyr Ser Leu Tyr Ser Gly Leu Ala Gly Leu Tyr Gly Leu
65 70 75 80
Ala Thr Tyr Arg Ile Leu Glu Leu Tyr Ser Leu Glu Ala Leu Tyr Ser
85 90 95
Val Ala Leu Ile Leu Glu Gly Leu Tyr Gly Leu Asn Ala Ser Pro Ser
100 105 110
Glu Arg Ser Glu Arg Gly Leu Ala Ile Leu Glu His Ile Ser Pro His
115 120 125
Glu Leu Tyr Ser Val Ala Leu Leu Tyr Ser Met Glu Thr Thr His Arg
130 135 140
Thr His Arg His Ile Ser Leu Glu Ala Leu Tyr Ser Leu Tyr Ser Leu
145 150 155 160
Glu Ala Leu Tyr Ser Gly Leu Ala Ser Glu Arg Thr Tyr Arg Cys Tyr
165 170 175
Ser Gly Leu Asn Ala Arg Gly Gly Leu Asn Gly Leu Tyr Val Ala Leu
180 185 190
Pro Arg Ala Met Glu Thr Ala Ser Asn Ser Glu Arg Leu Glu Ala Ala
195 200 205
Arg Gly Pro His Glu Leu Glu Ala Pro His Glu Gly Leu Ala Gly Leu
210 215 220
Tyr Gly Leu Asn Ala Arg Gly Ile Leu Glu Ala Leu Ala Ala Ser Pro
225 230 235 240
Ala Ser Asn His Ile Ser Thr His Arg Pro Arg Ala Leu Tyr Ser Gly
245 250 255
Leu Ala Leu Glu Ala Gly Leu Tyr Met Glu Thr Gly Leu Ala Gly Leu
260 265 270
Ala Gly Leu Ala Ala Ser Pro Val Ala Leu Ile Leu Glu Gly Leu Ala
275 280 285
Val Ala Leu Thr Tyr Arg Gly Leu Asn Gly Leu Ala Gly Leu Asn Thr
290 295 300
His Arg Gly Leu Tyr Gly Leu Tyr Pro His Glu Pro Arg Ala Thr His
305 310 315 320
Arg Ile Leu Glu Pro Arg Ala Leu Glu Ala Ser Glu Arg Ala Arg Gly
325 330 335
Leu Glu Ala Pro His Glu Ala Ser Pro Ala Ser Asn Ala Leu Ala Met
340 345 350
Glu Thr Leu Glu Ala Ala Arg Gly Ala Leu Ala His Ile Ser Ala Arg
355 360 365
Gly Leu Glu Ala His Ile Ser Gly Leu Asn Leu Glu Ala Ala Leu Ala
370 375 380
Pro His Glu Ala Ser Pro Thr His Arg Thr Tyr Arg Gly Leu Asn Gly
385 390 395 400
Leu Ala Pro His Glu Gly Leu Ala Gly Leu Ala Ala Leu Ala Thr Tyr
405 410 415
Arg Ile Leu Glu Pro Arg Ala Leu Tyr Ser Gly Leu Ala Gly Leu Asn
420 425 430
Leu Tyr Ser Thr Tyr Arg Ser Glu Arg Pro His Glu Leu Glu Ala Gly
435 440 445
Leu Asn Ala Ser Asn Pro Arg Ala Gly Leu Asn Thr His Arg Ser Glu
450 455 460
Arg Leu Glu Ala Cys Tyr Ser Pro His Glu Ser Glu Arg Gly Leu Ala
465 470 475 480
Ser Glu Arg Ile Leu Glu Pro Arg Ala Thr His Arg Pro Arg Ala Ser
485 490 495
Glu Arg Ala Ser Asn Ala Arg Gly Gly Leu Ala Gly Leu Ala Thr His
500 505 510
Arg Gly Leu Asn Gly Leu Asn Leu Tyr Ser Ser Glu Arg Ala Ser Asn
515 520 525
Leu Glu Ala Gly Leu Ala Leu Glu Ala Leu Glu Ala Ala Arg Gly Ile
530 535 540
Leu Glu Ser Glu Arg Leu Glu Ala Leu Glu Ala Leu Glu Ala Ile Leu
545 550 555 560
Glu Gly Leu Asn Ser Glu Arg Thr Arg Pro Leu Glu Ala Gly Leu Ala
565 570 575
Pro Arg Ala Val Ala Leu Gly Leu Asn Pro His Glu Leu Glu Ala Ala
580 585 590
Arg Gly Ser Glu Arg Val Ala Leu Pro His Glu Ala Leu Ala Ala Ser
595 600 605
Asn Ser Glu Arg Leu Glu Ala Val Ala Leu Thr Tyr Arg Gly Leu Tyr
610 615 620
Ala Leu Ala Ser Glu Arg Ala Ser Pro Ser Glu Arg Ala Ser Asn Val
625 630 635 640
Ala Leu Thr Tyr Arg Ala Ser Pro Leu Glu Ala Leu Glu Ala Leu Tyr
645 650 655
Ser Ala Ser Pro Leu Glu Ala Gly Leu Ala Gly Leu Ala Gly Leu Tyr
660 665 670
Ile Leu Glu Gly Leu Asn Thr His Arg Leu Glu Ala Met Glu Thr Gly
675 680 685
Leu Tyr Ala Arg Gly Leu Glu Ala Gly Leu Ala Ala Ser Pro Gly Leu
690 695 700
Tyr Ser Glu Arg Pro Arg Ala Ala Arg Gly Thr His Arg Gly Leu Tyr
705 710 715 720
Gly Leu Asn Ile Leu Glu Pro His Glu Leu Tyr Ser Gly Leu Asn Thr
725 730 735
His Arg Thr Tyr Arg Ser Glu Arg Leu Tyr Ser Pro His Glu Ala Ser
740 745 750
Pro Thr His Arg Ala Ser Asn Ser Glu Arg His Ile Ser Ala Ser Asn
755 760 765
Ala Ser Pro Ala Ser Pro Ala Leu Ala Leu Glu Ala Leu Glu Ala Leu
770 775 780
Tyr Ser Ala Ser Asn Thr Tyr Arg Gly Leu Tyr Leu Glu Ala Leu Glu
785 790 795 800
Ala Thr Tyr Arg Cys Tyr Ser Pro His Glu Ala Arg Gly Leu Tyr Ser
805 810 815
Ala Ser Pro Met Glu Thr Ala Ser Pro Leu Tyr Ser Val Ala Leu Gly
820 825 830
Leu Ala Thr His Arg Pro His Glu Leu Glu Ala Ala Arg Gly Ile Leu
835 840 845
Glu Val Ala Leu Gly Leu Asn Cys Tyr Ser Ala Arg Gly Ser Glu Arg
850 855 860
Val Ala Leu Gly Leu Ala Gly Leu Tyr Ser Glu Arg Cys Tyr Ser Gly
865 870 875 880
Leu Tyr Pro His Glu Met Glu Thr Gly Leu Asn Ile Leu Glu Pro His
885 890 895
Glu Val Ala Leu Ala Arg Gly Thr His Arg Leu Glu Ala Thr His Arg
900 905 910
Gly Leu Tyr Ala Arg Gly Thr His Arg Ile Leu Glu Thr His Arg Leu
915 920 925
Glu Ala Gly Leu Ala Val Ala Leu Gly Leu Ala Pro Arg Ala Ser Glu
930 935 940
Arg Ala Ser Pro Thr His Arg Ile Leu Glu Gly Leu Ala Ala Ser Asn
945 950 955 960
Val Ala Leu Ala Arg Gly Ala Leu Ala Ala Arg Gly Ile Leu Glu Gly
965 970 975
Leu Asn Ala Ser Pro Ala Arg Gly Gly Leu Ala Gly Leu Tyr Ile Leu
980 985 990
Glu Pro Arg Ala Pro Arg Ala Ala Ser Pro Gly Leu Asn Gly Leu Asn
995 1000 1005
Ala Arg Gly Leu Glu Ala Ile Leu Glu Pro His Glu Ala Leu Ala Gly
1010 1015 1020
Leu Tyr Ala Arg Gly Gly Leu Asn Leu Glu Ala Gly Leu Ala Ala Ser
1025 1030 1035 1040
Pro Gly Leu Tyr Ala Arg Gly Thr His Arg Leu Glu Ala Ser Glu Arg
1045 1050 1055
Ala Ser Pro Thr Tyr Arg Ala Ser Asn Ile Leu Glu Gly Leu Asn Ala
1060 1065 1070
Arg Gly Gly Leu Ala Ser Glu Arg Thr His Arg Leu Glu Ala His Ile
1075 1080 1085
Ser Leu Glu Ala Val Ala Leu Leu Glu Ala Ala Arg Gly Leu Glu Ala
1090 1095 1100
Ala Arg Gly Gly Leu Tyr Gly Leu Tyr Ala Ser Pro
1105 1110 1115
<210> 19
<211> 1116
<212> PRT
<213> Искусственная последовательность
<220>
<223> His-SUMO- hGH-Ub(A)
<400> 19
Met Glu Thr Gly Leu Tyr His Ile Ser His Ile Ser His Ile Ser His
1 5 10 15
Ile Ser His Ile Ser His Ile Ser Ser Glu Arg Ala Ser Pro Gly Leu
20 25 30
Asn Gly Leu Ala Ala Leu Ala Leu Tyr Ser Pro Arg Ala Ser Glu Arg
35 40 45
Thr His Arg Gly Leu Ala Ala Ser Pro Leu Glu Ala Gly Leu Tyr Ala
50 55 60
Ser Pro Leu Tyr Ser Leu Tyr Ser Gly Leu Ala Gly Leu Tyr Gly Leu
65 70 75 80
Ala Thr Tyr Arg Ile Leu Glu Leu Tyr Ser Leu Glu Ala Leu Tyr Ser
85 90 95
Val Ala Leu Ile Leu Glu Gly Leu Tyr Gly Leu Asn Ala Ser Pro Ser
100 105 110
Glu Arg Ser Glu Arg Gly Leu Ala Ile Leu Glu His Ile Ser Pro His
115 120 125
Glu Leu Tyr Ser Val Ala Leu Leu Tyr Ser Met Glu Thr Thr His Arg
130 135 140
Thr His Arg His Ile Ser Leu Glu Ala Leu Tyr Ser Leu Tyr Ser Leu
145 150 155 160
Glu Ala Leu Tyr Ser Gly Leu Ala Ser Glu Arg Thr Tyr Arg Cys Tyr
165 170 175
Ser Gly Leu Asn Ala Arg Gly Gly Leu Asn Gly Leu Tyr Val Ala Leu
180 185 190
Pro Arg Ala Met Glu Thr Ala Ser Asn Ser Glu Arg Leu Glu Ala Ala
195 200 205
Arg Gly Pro His Glu Leu Glu Ala Pro His Glu Gly Leu Ala Gly Leu
210 215 220
Tyr Gly Leu Asn Ala Arg Gly Ile Leu Glu Ala Leu Ala Ala Ser Pro
225 230 235 240
Ala Ser Asn His Ile Ser Thr His Arg Pro Arg Ala Leu Tyr Ser Gly
245 250 255
Leu Ala Leu Glu Ala Gly Leu Tyr Met Glu Thr Gly Leu Ala Gly Leu
260 265 270
Ala Gly Leu Ala Ala Ser Pro Val Ala Leu Ile Leu Glu Gly Leu Ala
275 280 285
Val Ala Leu Thr Tyr Arg Gly Leu Asn Gly Leu Ala Gly Leu Asn Thr
290 295 300
His Arg Gly Leu Tyr Gly Leu Tyr Pro His Glu Pro Arg Ala Thr His
305 310 315 320
Arg Ile Leu Glu Pro Arg Ala Leu Glu Ala Ser Glu Arg Ala Arg Gly
325 330 335
Leu Glu Ala Pro His Glu Ala Ser Pro Ala Ser Asn Ala Leu Ala Met
340 345 350
Glu Thr Leu Glu Ala Ala Arg Gly Ala Leu Ala His Ile Ser Ala Arg
355 360 365
Gly Leu Glu Ala His Ile Ser Gly Leu Asn Leu Glu Ala Ala Leu Ala
370 375 380
Pro His Glu Ala Ser Pro Thr His Arg Thr Tyr Arg Gly Leu Asn Gly
385 390 395 400
Leu Ala Pro His Glu Gly Leu Ala Gly Leu Ala Ala Leu Ala Thr Tyr
405 410 415
Arg Ile Leu Glu Pro Arg Ala Leu Tyr Ser Gly Leu Ala Gly Leu Asn
420 425 430
Leu Tyr Ser Thr Tyr Arg Ser Glu Arg Pro His Glu Leu Glu Ala Gly
435 440 445
Leu Asn Ala Ser Asn Pro Arg Ala Gly Leu Asn Thr His Arg Ser Glu
450 455 460
Arg Leu Glu Ala Cys Tyr Ser Pro His Glu Ser Glu Arg Gly Leu Ala
465 470 475 480
Ser Glu Arg Ile Leu Glu Pro Arg Ala Thr His Arg Pro Arg Ala Ser
485 490 495
Glu Arg Ala Ser Asn Ala Arg Gly Gly Leu Ala Gly Leu Ala Thr His
500 505 510
Arg Gly Leu Asn Gly Leu Asn Leu Tyr Ser Ser Glu Arg Ala Ser Asn
515 520 525
Leu Glu Ala Gly Leu Ala Leu Glu Ala Leu Glu Ala Ala Arg Gly Ile
530 535 540
Leu Glu Ser Glu Arg Leu Glu Ala Leu Glu Ala Leu Glu Ala Ile Leu
545 550 555 560
Glu Gly Leu Asn Ser Glu Arg Thr Arg Pro Leu Glu Ala Gly Leu Ala
565 570 575
Pro Arg Ala Val Ala Leu Gly Leu Asn Pro His Glu Leu Glu Ala Ala
580 585 590
Arg Gly Ser Glu Arg Val Ala Leu Pro His Glu Ala Leu Ala Ala Ser
595 600 605
Asn Ser Glu Arg Leu Glu Ala Val Ala Leu Thr Tyr Arg Gly Leu Tyr
610 615 620
Ala Leu Ala Ser Glu Arg Ala Ser Pro Ser Glu Arg Ala Ser Asn Val
625 630 635 640
Ala Leu Thr Tyr Arg Ala Ser Pro Leu Glu Ala Leu Glu Ala Leu Tyr
645 650 655
Ser Ala Ser Pro Leu Glu Ala Gly Leu Ala Gly Leu Ala Gly Leu Tyr
660 665 670
Ile Leu Glu Gly Leu Asn Thr His Arg Leu Glu Ala Met Glu Thr Gly
675 680 685
Leu Tyr Ala Arg Gly Leu Glu Ala Gly Leu Ala Ala Ser Pro Gly Leu
690 695 700
Tyr Ser Glu Arg Pro Arg Ala Ala Arg Gly Thr His Arg Gly Leu Tyr
705 710 715 720
Gly Leu Asn Ile Leu Glu Pro His Glu Leu Tyr Ser Gly Leu Asn Thr
725 730 735
His Arg Thr Tyr Arg Ser Glu Arg Leu Tyr Ser Pro His Glu Ala Ser
740 745 750
Pro Thr His Arg Ala Ser Asn Ser Glu Arg His Ile Ser Ala Ser Asn
755 760 765
Ala Ser Pro Ala Ser Pro Ala Leu Ala Leu Glu Ala Leu Glu Ala Leu
770 775 780
Tyr Ser Ala Ser Asn Thr Tyr Arg Gly Leu Tyr Leu Glu Ala Leu Glu
785 790 795 800
Ala Thr Tyr Arg Cys Tyr Ser Pro His Glu Ala Arg Gly Leu Tyr Ser
805 810 815
Ala Ser Pro Met Glu Thr Ala Ser Pro Leu Tyr Ser Val Ala Leu Gly
820 825 830
Leu Ala Thr His Arg Pro His Glu Leu Glu Ala Ala Arg Gly Ile Leu
835 840 845
Glu Val Ala Leu Gly Leu Asn Cys Tyr Ser Ala Arg Gly Ser Glu Arg
850 855 860
Val Ala Leu Gly Leu Ala Gly Leu Tyr Ser Glu Arg Cys Tyr Ser Gly
865 870 875 880
Leu Tyr Pro His Glu Met Glu Thr Gly Leu Asn Ile Leu Glu Pro His
885 890 895
Glu Val Ala Leu Ala Arg Gly Thr His Arg Leu Glu Ala Thr His Arg
900 905 910
Gly Leu Tyr Ala Arg Gly Thr His Arg Ile Leu Glu Thr His Arg Leu
915 920 925
Glu Ala Gly Leu Ala Val Ala Leu Gly Leu Ala Pro Arg Ala Ser Glu
930 935 940
Arg Ala Ser Pro Thr His Arg Ile Leu Glu Gly Leu Ala Ala Ser Asn
945 950 955 960
Val Ala Leu Ala Arg Gly Ala Leu Ala Ala Arg Gly Ile Leu Glu Gly
965 970 975
Leu Asn Ala Ser Pro Ala Arg Gly Gly Leu Ala Gly Leu Tyr Ile Leu
980 985 990
Glu Pro Arg Ala Pro Arg Ala Ala Ser Pro Gly Leu Asn Gly Leu Asn
995 1000 1005
Ala Arg Gly Leu Glu Ala Ile Leu Glu Pro His Glu Ala Leu Ala Gly
1010 1015 1020
Leu Tyr Ala Arg Gly Gly Leu Asn Leu Glu Ala Gly Leu Ala Ala Ser
1025 1030 1035 1040
Pro Gly Leu Tyr Ala Arg Gly Thr His Arg Leu Glu Ala Ser Glu Arg
1045 1050 1055
Ala Ser Pro Thr Tyr Arg Ala Ser Asn Ile Leu Glu Gly Leu Asn Leu
1060 1065 1070
Tyr Ser Gly Leu Ala Ser Glu Arg Thr His Arg Leu Glu Ala His Ile
1075 1080 1085
Ser Leu Glu Ala Val Ala Leu Leu Glu Ala Ala Arg Gly Pro Arg Ala
1090 1095 1100
Ala Arg Gly Gly Leu Tyr Gly Leu Tyr Ala Ser Pro
1105 1110 1115
<210> 20
<211> 804
<212> PRT
<213> Искусственная последовательность
<220>
<223> Ub(A)-hGH
<400> 20
Met Glu Thr Gly Leu Asn Ile Leu Glu Pro His Glu Val Ala Leu Ala
1 5 10 15
Arg Gly Thr His Arg Leu Glu Ala Thr His Arg Gly Leu Tyr Ala Arg
20 25 30
Gly Thr His Arg Ile Leu Glu Thr His Arg Leu Glu Ala Gly Leu Ala
35 40 45
Val Ala Leu Gly Leu Ala Pro Arg Ala Ser Glu Arg Ala Ser Pro Thr
50 55 60
His Arg Ile Leu Glu Gly Leu Ala Ala Ser Asn Val Ala Leu Ala Arg
65 70 75 80
Gly Ala Leu Ala Ala Arg Gly Ile Leu Glu Gly Leu Asn Ala Ser Pro
85 90 95
Ala Arg Gly Gly Leu Ala Gly Leu Tyr Ile Leu Glu Pro Arg Ala Pro
100 105 110
Arg Ala Ala Ser Pro Gly Leu Asn Gly Leu Asn Ala Arg Gly Leu Glu
115 120 125
Ala Ile Leu Glu Pro His Glu Ala Leu Ala Gly Leu Tyr Ala Arg Gly
130 135 140
Gly Leu Asn Leu Glu Ala Gly Leu Ala Ala Ser Pro Gly Leu Tyr Ala
145 150 155 160
Arg Gly Thr His Arg Leu Glu Ala Ser Glu Arg Ala Ser Pro Thr Tyr
165 170 175
Arg Ala Ser Asn Ile Leu Glu Gly Leu Asn Leu Tyr Ser Gly Leu Ala
180 185 190
Ser Glu Arg Thr His Arg Leu Glu Ala His Ile Ser Leu Glu Ala Val
195 200 205
Ala Leu Leu Glu Ala Ala Arg Gly Pro Arg Ala Ala Arg Gly Gly Leu
210 215 220
Tyr Gly Leu Tyr Ala Ser Pro Pro His Glu Pro Arg Ala Thr His Arg
225 230 235 240
Ile Leu Glu Pro Arg Ala Leu Glu Ala Ser Glu Arg Ala Arg Gly Leu
245 250 255
Glu Ala Pro His Glu Ala Ser Pro Ala Ser Asn Ala Leu Ala Met Glu
260 265 270
Thr Leu Glu Ala Ala Arg Gly Ala Leu Ala His Ile Ser Ala Arg Gly
275 280 285
Leu Glu Ala His Ile Ser Gly Leu Asn Leu Glu Ala Ala Leu Ala Pro
290 295 300
His Glu Ala Ser Pro Thr His Arg Thr Tyr Arg Gly Leu Asn Gly Leu
305 310 315 320
Ala Pro His Glu Gly Leu Ala Gly Leu Ala Ala Leu Ala Thr Tyr Arg
325 330 335
Ile Leu Glu Pro Arg Ala Leu Tyr Ser Gly Leu Ala Gly Leu Asn Leu
340 345 350
Tyr Ser Thr Tyr Arg Ser Glu Arg Pro His Glu Leu Glu Ala Gly Leu
355 360 365
Asn Ala Ser Asn Pro Arg Ala Gly Leu Asn Thr His Arg Ser Glu Arg
370 375 380
Leu Glu Ala Cys Tyr Ser Pro His Glu Ser Glu Arg Gly Leu Ala Ser
385 390 395 400
Glu Arg Ile Leu Glu Pro Arg Ala Thr His Arg Pro Arg Ala Ser Glu
405 410 415
Arg Ala Ser Asn Ala Arg Gly Gly Leu Ala Gly Leu Ala Thr His Arg
420 425 430
Gly Leu Asn Gly Leu Asn Leu Tyr Ser Ser Glu Arg Ala Ser Asn Leu
435 440 445
Glu Ala Gly Leu Ala Leu Glu Ala Leu Glu Ala Ala Arg Gly Ile Leu
450 455 460
Glu Ser Glu Arg Leu Glu Ala Leu Glu Ala Leu Glu Ala Ile Leu Glu
465 470 475 480
Gly Leu Asn Ser Glu Arg Thr Arg Pro Leu Glu Ala Gly Leu Ala Pro
485 490 495
Arg Ala Val Ala Leu Gly Leu Asn Pro His Glu Leu Glu Ala Ala Arg
500 505 510
Gly Ser Glu Arg Val Ala Leu Pro His Glu Ala Leu Ala Ala Ser Asn
515 520 525
Ser Glu Arg Leu Glu Ala Val Ala Leu Thr Tyr Arg Gly Leu Tyr Ala
530 535 540
Leu Ala Ser Glu Arg Ala Ser Pro Ser Glu Arg Ala Ser Asn Val Ala
545 550 555 560
Leu Thr Tyr Arg Ala Ser Pro Leu Glu Ala Leu Glu Ala Leu Tyr Ser
565 570 575
Ala Ser Pro Leu Glu Ala Gly Leu Ala Gly Leu Ala Gly Leu Tyr Ile
580 585 590
Leu Glu Gly Leu Asn Thr His Arg Leu Glu Ala Met Glu Thr Gly Leu
595 600 605
Tyr Ala Arg Gly Leu Glu Ala Gly Leu Ala Ala Ser Pro Gly Leu Tyr
610 615 620
Ser Glu Arg Pro Arg Ala Ala Arg Gly Thr His Arg Gly Leu Tyr Gly
625 630 635 640
Leu Asn Ile Leu Glu Pro His Glu Leu Tyr Ser Gly Leu Asn Thr His
645 650 655
Arg Thr Tyr Arg Ser Glu Arg Leu Tyr Ser Pro His Glu Ala Ser Pro
660 665 670
Thr His Arg Ala Ser Asn Ser Glu Arg His Ile Ser Ala Ser Asn Ala
675 680 685
Ser Pro Ala Ser Pro Ala Leu Ala Leu Glu Ala Leu Glu Ala Leu Tyr
690 695 700
Ser Ala Ser Asn Thr Tyr Arg Gly Leu Tyr Leu Glu Ala Leu Glu Ala
705 710 715 720
Thr Tyr Arg Cys Tyr Ser Pro His Glu Ala Arg Gly Leu Tyr Ser Ala
725 730 735
Ser Pro Met Glu Thr Ala Ser Pro Leu Tyr Ser Val Ala Leu Gly Leu
740 745 750
Ala Thr His Arg Pro His Glu Leu Glu Ala Ala Arg Gly Ile Leu Glu
755 760 765
Val Ala Leu Gly Leu Asn Cys Tyr Ser Ala Arg Gly Ser Glu Arg Val
770 775 780
Ala Leu Gly Leu Ala Gly Leu Tyr Ser Glu Arg Cys Tyr Ser Gly Leu
785 790 795 800
Tyr Pro His Glu
<210> 21
<211> 1116
<212> PRT
<213> Искусственная последовательность
<220>
<223> SUMO-Ub(A)-hGH
<400> 21
Met Glu Thr Gly Leu Tyr His Ile Ser His Ile Ser His Ile Ser His
1 5 10 15
Ile Ser His Ile Ser His Ile Ser Ser Glu Arg Ala Ser Pro Gly Leu
20 25 30
Asn Gly Leu Ala Ala Leu Ala Leu Tyr Ser Pro Arg Ala Ser Glu Arg
35 40 45
Thr His Arg Gly Leu Ala Ala Ser Pro Leu Glu Ala Gly Leu Tyr Ala
50 55 60
Ser Pro Leu Tyr Ser Leu Tyr Ser Gly Leu Ala Gly Leu Tyr Gly Leu
65 70 75 80
Ala Thr Tyr Arg Ile Leu Glu Leu Tyr Ser Leu Glu Ala Leu Tyr Ser
85 90 95
Val Ala Leu Ile Leu Glu Gly Leu Tyr Gly Leu Asn Ala Ser Pro Ser
100 105 110
Glu Arg Ser Glu Arg Gly Leu Ala Ile Leu Glu His Ile Ser Pro His
115 120 125
Glu Leu Tyr Ser Val Ala Leu Leu Tyr Ser Met Glu Thr Thr His Arg
130 135 140
Thr His Arg His Ile Ser Leu Glu Ala Leu Tyr Ser Leu Tyr Ser Leu
145 150 155 160
Glu Ala Leu Tyr Ser Gly Leu Ala Ser Glu Arg Thr Tyr Arg Cys Tyr
165 170 175
Ser Gly Leu Asn Ala Arg Gly Gly Leu Asn Gly Leu Tyr Val Ala Leu
180 185 190
Pro Arg Ala Met Glu Thr Ala Ser Asn Ser Glu Arg Leu Glu Ala Ala
195 200 205
Arg Gly Pro His Glu Leu Glu Ala Pro His Glu Gly Leu Ala Gly Leu
210 215 220
Tyr Gly Leu Asn Ala Arg Gly Ile Leu Glu Ala Leu Ala Ala Ser Pro
225 230 235 240
Ala Ser Asn His Ile Ser Thr His Arg Pro Arg Ala Leu Tyr Ser Gly
245 250 255
Leu Ala Leu Glu Ala Gly Leu Tyr Met Glu Thr Gly Leu Ala Gly Leu
260 265 270
Ala Gly Leu Ala Ala Ser Pro Val Ala Leu Ile Leu Glu Gly Leu Ala
275 280 285
Val Ala Leu Thr Tyr Arg Gly Leu Asn Gly Leu Ala Gly Leu Asn Thr
290 295 300
His Arg Gly Leu Tyr Gly Leu Tyr Met Glu Thr Gly Leu Asn Ile Leu
305 310 315 320
Glu Pro His Glu Val Ala Leu Ala Arg Gly Thr His Arg Leu Glu Ala
325 330 335
Thr His Arg Gly Leu Tyr Ala Arg Gly Thr His Arg Ile Leu Glu Thr
340 345 350
His Arg Leu Glu Ala Gly Leu Ala Val Ala Leu Gly Leu Ala Pro Arg
355 360 365
Ala Ser Glu Arg Ala Ser Pro Thr His Arg Ile Leu Glu Gly Leu Ala
370 375 380
Ala Ser Asn Val Ala Leu Ala Arg Gly Ala Leu Ala Ala Arg Gly Ile
385 390 395 400
Leu Glu Gly Leu Asn Ala Ser Pro Ala Arg Gly Gly Leu Ala Gly Leu
405 410 415
Tyr Ile Leu Glu Pro Arg Ala Pro Arg Ala Ala Ser Pro Gly Leu Asn
420 425 430
Gly Leu Asn Ala Arg Gly Leu Glu Ala Ile Leu Glu Pro His Glu Ala
435 440 445
Leu Ala Gly Leu Tyr Leu Tyr Ser Gly Leu Asn Leu Glu Ala Gly Leu
450 455 460
Ala Ala Ser Pro Gly Leu Tyr Ala Arg Gly Thr His Arg Leu Glu Ala
465 470 475 480
Ser Glu Arg Ala Ser Pro Thr Tyr Arg Ala Ser Asn Ile Leu Glu Gly
485 490 495
Leu Asn Ala Arg Gly Gly Leu Ala Ser Glu Arg Thr His Arg Leu Glu
500 505 510
Ala His Ile Ser Leu Glu Ala Val Ala Leu Leu Glu Ala Ala Arg Gly
515 520 525
Pro Arg Ala Ala Arg Gly Gly Leu Tyr Gly Leu Tyr Ala Ser Pro Pro
530 535 540
His Glu Pro Arg Ala Thr His Arg Ile Leu Glu Pro Arg Ala Leu Glu
545 550 555 560
Ala Ser Glu Arg Ala Arg Gly Leu Glu Ala Pro His Glu Ala Ser Pro
565 570 575
Ala Ser Asn Ala Leu Ala Met Glu Thr Leu Glu Ala Ala Arg Gly Ala
580 585 590
Leu Ala His Ile Ser Ala Arg Gly Leu Glu Ala His Ile Ser Gly Leu
595 600 605
Asn Leu Glu Ala Ala Leu Ala Pro His Glu Ala Ser Pro Thr His Arg
610 615 620
Thr Tyr Arg Gly Leu Asn Gly Leu Ala Pro His Glu Gly Leu Ala Gly
625 630 635 640
Leu Ala Ala Leu Ala Thr Tyr Arg Ile Leu Glu Pro Arg Ala Leu Tyr
645 650 655
Ser Gly Leu Ala Gly Leu Asn Leu Tyr Ser Thr Tyr Arg Ser Glu Arg
660 665 670
Pro His Glu Leu Glu Ala Gly Leu Asn Ala Ser Asn Pro Arg Ala Gly
675 680 685
Leu Asn Thr His Arg Ser Glu Arg Leu Glu Ala Cys Tyr Ser Pro His
690 695 700
Glu Ser Glu Arg Gly Leu Ala Ser Glu Arg Ile Leu Glu Pro Arg Ala
705 710 715 720
Thr His Arg Pro Arg Ala Ser Glu Arg Ala Ser Asn Ala Arg Gly Gly
725 730 735
Leu Ala Gly Leu Ala Thr His Arg Gly Leu Asn Gly Leu Asn Leu Tyr
740 745 750
Ser Ser Glu Arg Ala Ser Asn Leu Glu Ala Gly Leu Ala Leu Glu Ala
755 760 765
Leu Glu Ala Ala Arg Gly Ile Leu Glu Ser Glu Arg Leu Glu Ala Leu
770 775 780
Glu Ala Leu Glu Ala Ile Leu Glu Gly Leu Asn Ser Glu Arg Thr Arg
785 790 795 800
Pro Leu Glu Ala Gly Leu Ala Pro Arg Ala Val Ala Leu Gly Leu Asn
805 810 815
Pro His Glu Leu Glu Ala Ala Arg Gly Ser Glu Arg Val Ala Leu Pro
820 825 830
His Glu Ala Leu Ala Ala Ser Asn Ser Glu Arg Leu Glu Ala Val Ala
835 840 845
Leu Thr Tyr Arg Gly Leu Tyr Ala Leu Ala Ser Glu Arg Ala Ser Pro
850 855 860
Ser Glu Arg Ala Ser Asn Val Ala Leu Thr Tyr Arg Ala Ser Pro Leu
865 870 875 880
Glu Ala Leu Glu Ala Leu Tyr Ser Ala Ser Pro Leu Glu Ala Gly Leu
885 890 895
Ala Gly Leu Ala Gly Leu Tyr Ile Leu Glu Gly Leu Asn Thr His Arg
900 905 910
Leu Glu Ala Met Glu Thr Gly Leu Tyr Ala Arg Gly Leu Glu Ala Gly
915 920 925
Leu Ala Ala Ser Pro Gly Leu Tyr Ser Glu Arg Pro Arg Ala Ala Arg
930 935 940
Gly Thr His Arg Gly Leu Tyr Gly Leu Asn Ile Leu Glu Pro His Glu
945 950 955 960
Leu Tyr Ser Gly Leu Asn Thr His Arg Thr Tyr Arg Ser Glu Arg Leu
965 970 975
Tyr Ser Pro His Glu Ala Ser Pro Thr His Arg Ala Ser Asn Ser Glu
980 985 990
Arg His Ile Ser Ala Ser Asn Ala Ser Pro Ala Ser Pro Ala Leu Ala
995 1000 1005
Leu Glu Ala Leu Glu Ala Leu Tyr Ser Ala Ser Asn Thr Tyr Arg Gly
1010 1015 1020
Leu Tyr Leu Glu Ala Leu Glu Ala Thr Tyr Arg Cys Tyr Ser Pro His
1025 1030 1035 1040
Glu Ala Arg Gly Leu Tyr Ser Ala Ser Pro Met Glu Thr Ala Ser Pro
1045 1050 1055
Leu Tyr Ser Val Ala Leu Gly Leu Ala Thr His Arg Pro His Glu Leu
1060 1065 1070
Glu Ala Ala Arg Gly Ile Leu Glu Val Ala Leu Gly Leu Asn Cys Tyr
1075 1080 1085
Ser Ala Arg Gly Ser Glu Arg Val Ala Leu Gly Leu Ala Gly Leu Tyr
1090 1095 1100
Ser Glu Arg Cys Tyr Ser Gly Leu Tyr Pro His Glu
1105 1110 1115
<210> 22
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Сигнальный пептид (IgGk)
<400> 22
atggaaactg atactctgct gctgtgggtg ctgctgctgt gggtgcccgg ctcaactggt 60
60
<210> 23
<211> 231
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Ub (A)
<400> 23
atgcagatct tcgtgaggac cctgacagat cggaccatca cactggaggt ggagccaagc 60
gacaccatcg agaacgtgag ggccagaatc caggaccggg agggcatccc ccctgatcag 120
cagagactga tcttcgctgg ccgccagctg gaggacggaa ggaccctgag cgattacaat 180
atccagaaag agtctacact gcacctggtg ctgagaccgc gcgtcgtgga t 231
<210> 24
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Hinge (IgG1)
<400> 24
gagccaaaat cttgtgacaa aactcataca tgtccc 36
<210> 25
<211> 660
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Fc (IgG1)
<400> 25
ccatgtcccg cacctgaact gctgggcgga cctagcgtgt ttctgttccc acctaagcca 60
aaggacaccc tgatgatctc caggaccccc gaggtgacat gcgtggtggt ggacgtgagc 120
cacgaggacc ccgaggtgaa gttcaactgg tacgtggatg gcgtggaggt gcataatgcc 180
aagacaaagc caagggagga gcagtacaac agcacctatc gggtggtgtc tgtgctgaca 240
gtgctgcacc aggactggct gaacggcaag gagtataagt gcaaggtgtc taataaggcc 300
ctgcccgctc ctatcgagaa gaccatctcc aaggccaagg gccagccaag agagccccag 360
gtgtacacac tgccccctag ccgcgacgag ctgaccaaga accaggtgtc tctgacatgt 420
ctggtgaagg gcttctatcc ttctgatatc gctgtggagt gggagtccaa tggccagcca 480
gagaacaatt acaagaccac accacccgtg ctggactctg atggctcctt ctttctgtat 540
tccaagctga ccgtggataa gagcagatgg cagcagggca acgtgttctc ctgtagcgtg 600
atgcatgaag cactgcataa tcactatacc cagaagtcac tgtcactgag tcccggtaaa 660
660
<210> 26
<211> 20
<212> PRT
<213> Искусственная последовательность
<220>
<223> Сигнальный пептид (IgGk)
<400> 26
Met Glu Thr Asp Thr Leu Leu Leu Trp Val Leu Leu Leu Trp Val Pro
1 5 10 15
Gly Ser Thr Gly
20
<210> 27
<211> 77
<212> PRT
<213> Искусственная последовательность
<220>
<223> Ub(A)
<400> 27
Met Gln Ile Phe Val Arg Thr Leu Thr Asp Arg Thr Ile Thr Leu Glu
1 5 10 15
Val Glu Pro Ser Asp Thr Ile Glu Asn Val Arg Ala Arg Ile Gln Asp
20 25 30
Arg Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Arg
35 40 45
Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu
50 55 60
Ser Thr Leu His Leu Val Leu Arg Pro Arg Val Val Asp
65 70 75
<210> 28
<211> 12
<212> PRT
<213> Искусственная последовательность
<220>
<223> Hinge (IgG1)
<400> 28
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
1 5 10
<210> 29
<211> 660
<212> PRT
<213> Искусственная последовательность
<220>
<223> Fc (IgG1)
<400> 29
Pro Arg Ala Cys Tyr Ser Pro Arg Ala Ala Leu Ala Pro Arg Ala Gly
1 5 10 15
Leu Ala Leu Glu Ala Leu Glu Ala Gly Leu Tyr Gly Leu Tyr Pro Arg
20 25 30
Ala Ser Glu Arg Val Ala Leu Pro His Glu Leu Glu Ala Pro His Glu
35 40 45
Pro Arg Ala Pro Arg Ala Leu Tyr Ser Pro Arg Ala Leu Tyr Ser Ala
50 55 60
Ser Pro Thr His Arg Leu Glu Ala Met Glu Thr Ile Leu Glu Ser Glu
65 70 75 80
Arg Ala Arg Gly Thr His Arg Pro Arg Ala Gly Leu Ala Val Ala Leu
85 90 95
Thr His Arg Cys Tyr Ser Val Ala Leu Val Ala Leu Val Ala Leu Ala
100 105 110
Ser Pro Val Ala Leu Ser Glu Arg His Ile Ser Gly Leu Ala Ala Ser
115 120 125
Pro Pro Arg Ala Gly Leu Ala Val Ala Leu Leu Tyr Ser Pro His Glu
130 135 140
Ala Ser Asn Thr Arg Pro Thr Tyr Arg Val Ala Leu Ala Ser Pro Gly
145 150 155 160
Leu Tyr Val Ala Leu Gly Leu Ala Val Ala Leu His Ile Ser Ala Ser
165 170 175
Asn Ala Leu Ala Leu Tyr Ser Thr His Arg Leu Tyr Ser Pro Arg Ala
180 185 190
Ala Arg Gly Gly Leu Ala Gly Leu Ala Gly Leu Asn Thr Tyr Arg Ala
195 200 205
Ser Asn Ser Glu Arg Thr His Arg Thr Tyr Arg Ala Arg Gly Val Ala
210 215 220
Leu Val Ala Leu Ser Glu Arg Val Ala Leu Leu Glu Ala Thr His Arg
225 230 235 240
Val Ala Leu Leu Glu Ala His Ile Ser Gly Leu Asn Ala Ser Pro Thr
245 250 255
Arg Pro Leu Glu Ala Ala Ser Asn Gly Leu Tyr Leu Tyr Ser Gly Leu
260 265 270
Ala Thr Tyr Arg Leu Tyr Ser Cys Tyr Ser Leu Tyr Ser Val Ala Leu
275 280 285
Ser Glu Arg Ala Ser Asn Leu Tyr Ser Ala Leu Ala Leu Glu Ala Pro
290 295 300
Arg Ala Ala Leu Ala Pro Arg Ala Ile Leu Glu Gly Leu Ala Leu Tyr
305 310 315 320
Ser Thr His Arg Ile Leu Glu Ser Glu Arg Leu Tyr Ser Ala Leu Ala
325 330 335
Leu Tyr Ser Gly Leu Tyr Gly Leu Asn Pro Arg Ala Ala Arg Gly Gly
340 345 350
Leu Ala Pro Arg Ala Gly Leu Asn Val Ala Leu Thr Tyr Arg Thr His
355 360 365
Arg Leu Glu Ala Pro Arg Ala Pro Arg Ala Ser Glu Arg Ala Arg Gly
370 375 380
Ala Ser Pro Gly Leu Ala Leu Glu Ala Thr His Arg Leu Tyr Ser Ala
385 390 395 400
Ser Asn Gly Leu Asn Val Ala Leu Ser Glu Arg Leu Glu Ala Thr His
405 410 415
Arg Cys Tyr Ser Leu Glu Ala Val Ala Leu Leu Tyr Ser Gly Leu Tyr
420 425 430
Pro His Glu Thr Tyr Arg Pro Arg Ala Ser Glu Arg Ala Ser Pro Ile
435 440 445
Leu Glu Ala Leu Ala Val Ala Leu Gly Leu Ala Thr Arg Pro Gly Leu
450 455 460
Ala Ser Glu Arg Ala Ser Asn Gly Leu Tyr Gly Leu Asn Pro Arg Ala
465 470 475 480
Gly Leu Ala Ala Ser Asn Ala Ser Asn Thr Tyr Arg Leu Tyr Ser Thr
485 490 495
His Arg Thr His Arg Pro Arg Ala Pro Arg Ala Val Ala Leu Leu Glu
500 505 510
Ala Ala Ser Pro Ser Glu Arg Ala Ser Pro Gly Leu Tyr Ser Glu Arg
515 520 525
Pro His Glu Pro His Glu Leu Glu Ala Thr Tyr Arg Ser Glu Arg Leu
530 535 540
Tyr Ser Leu Glu Ala Thr His Arg Val Ala Leu Ala Ser Pro Leu Tyr
545 550 555 560
Ser Ser Glu Arg Ala Arg Gly Thr Arg Pro Gly Leu Asn Gly Leu Asn
565 570 575
Gly Leu Tyr Ala Ser Asn Val Ala Leu Pro His Glu Ser Glu Arg Cys
580 585 590
Tyr Ser Ser Glu Arg Val Ala Leu Met Glu Thr His Ile Ser Gly Leu
595 600 605
Ala Ala Leu Ala Leu Glu Ala His Ile Ser Ala Ser Asn His Ile Ser
610 615 620
Thr Tyr Arg Thr His Arg Gly Leu Asn Leu Tyr Ser Ser Glu Arg Leu
625 630 635 640
Glu Ala Ser Glu Arg Leu Glu Ala Ser Glu Arg Pro Arg Ala Gly Leu
645 650 655
Tyr Leu Tyr Ser
660
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НОВЫЙ БЕЛКОВЫЙ КОНЪЮГАТ И ЕГО ПРИМЕНЕНИЕ ДЛЯ ПРОФИЛАКТИКИ ИЛИ ЛЕЧЕНИЯ НЕАЛКОГОЛЬНОГО СТЕАТОГЕПАТИТА, ОЖИРЕНИЯ И ДИАБЕТА | 2021 |
|
RU2822001C1 |
| МОДИФИЦИРОВАННЫЕ МИТОХОНДРИИ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2811467C2 |
| БЕЛКИ С ДВОЙНОЙ ФУНКЦИЕЙ И СОДЕРЖАЩАЯ ИХ ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ | 2016 |
|
RU2741345C2 |
| ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ ДЛЯ ПРЕДОТВРАЩЕНИЯ ИЛИ ЛЕЧЕНИЯ ГЕПАТИТА, ФИБРОЗА ПЕЧЕНИ И ЦИРРОЗА ПЕЧЕНИ, ВКЛЮЧАЮЩАЯ СЛИТЫЕ БЕЛКИ | 2017 |
|
RU2795548C2 |
| МУТИРОВАННЫЙ БЕЛОК SPOT И СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ С ЕГО ПРИМЕНЕНИЕМ | 2022 |
|
RU2837657C2 |
| СЛИТЫЙ БЕЛОК GDF15 ДЛИТЕЛЬНОГО ДЕЙСТВИЯ И СОДЕРЖАЩАЯ ЕГО ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ | 2020 |
|
RU2836280C1 |
| СПОСОБ ПОЛУЧЕНИЯ БИФУНКЦИОНАЛЬНЫХ БЕЛКОВ И ИХ ПРОИЗВОДНЫХ | 2018 |
|
RU2795970C2 |
| СЛИТЫЙ БЕЛОК, ВКЛЮЧАЮЩИЙ IL-12 И АНТИТЕЛО ПРОТИВ FAP, И ЕГО ПРИМЕНЕНИЕ | 2021 |
|
RU2831612C1 |
| КОМПОЗИЦИЯ ДЛЯ КУЛЬТИВИРОВАНИЯ РЕГУЛЯТОРНЫХ Т-КЛЕТОК И ЕЕ ПРИМЕНЕНИЕ | 2020 |
|
RU2807802C1 |
| Новый вариант О-сукцинилгомосеринтрансферазы и способ получения О-сукцинилгомосерина с использованием этого варианта | 2018 |
|
RU2747493C1 |
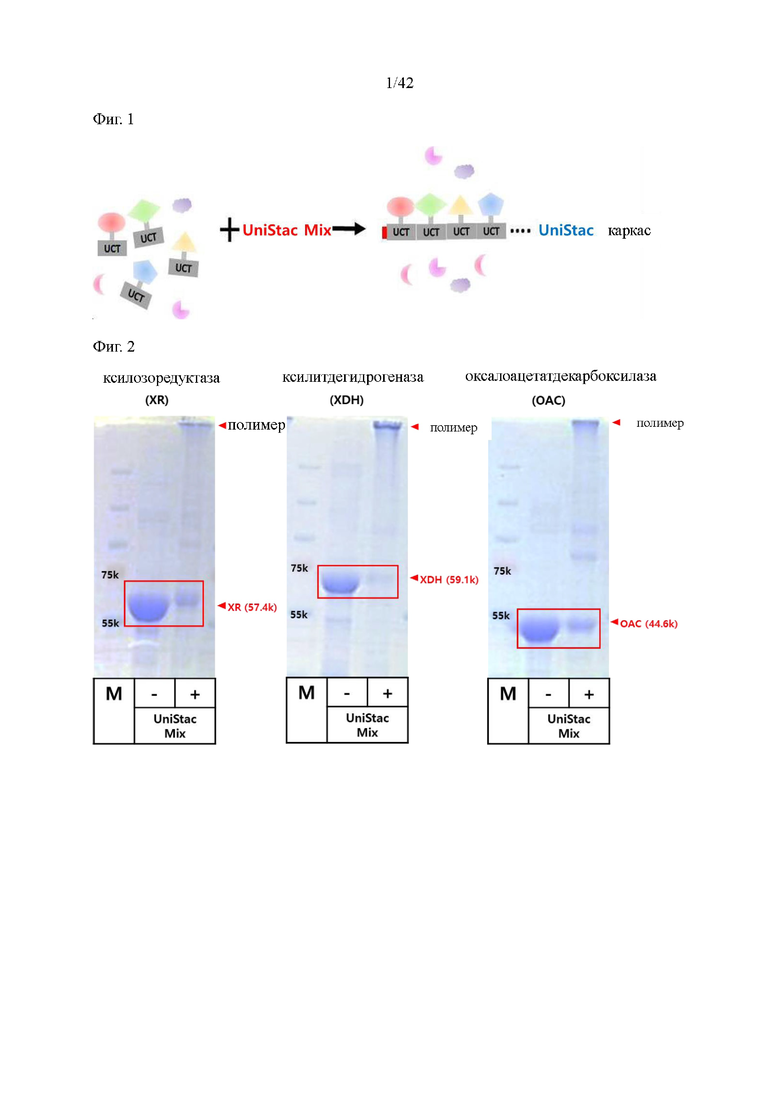




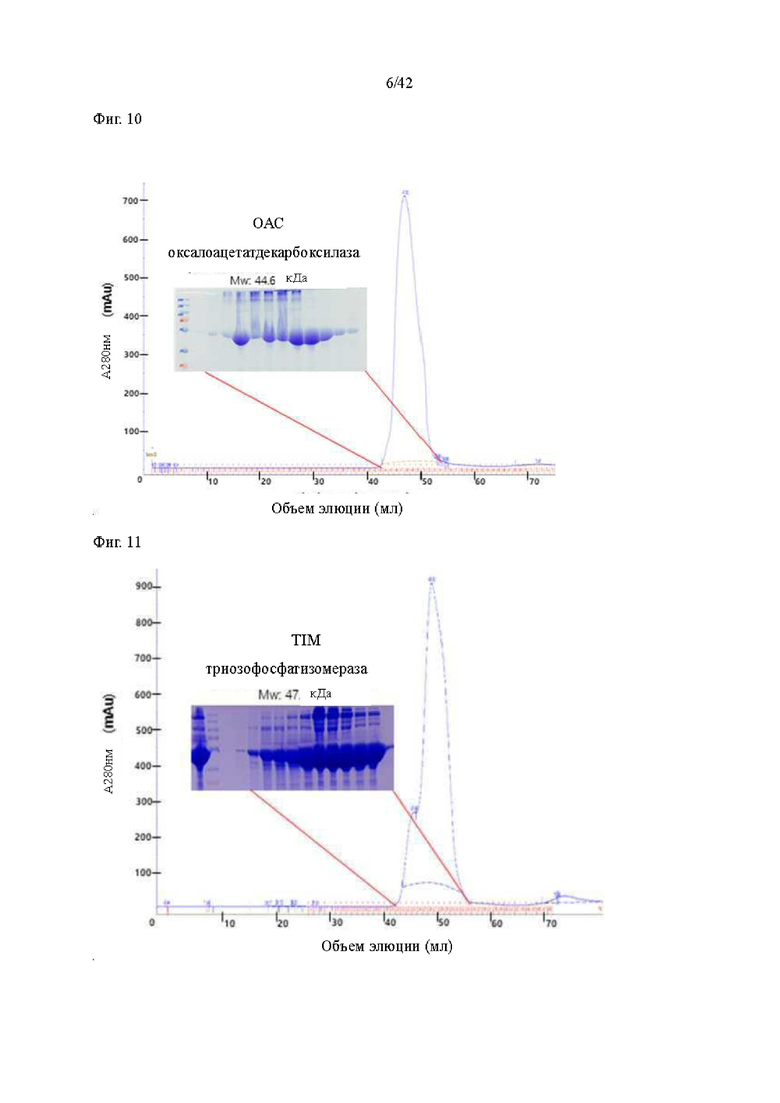




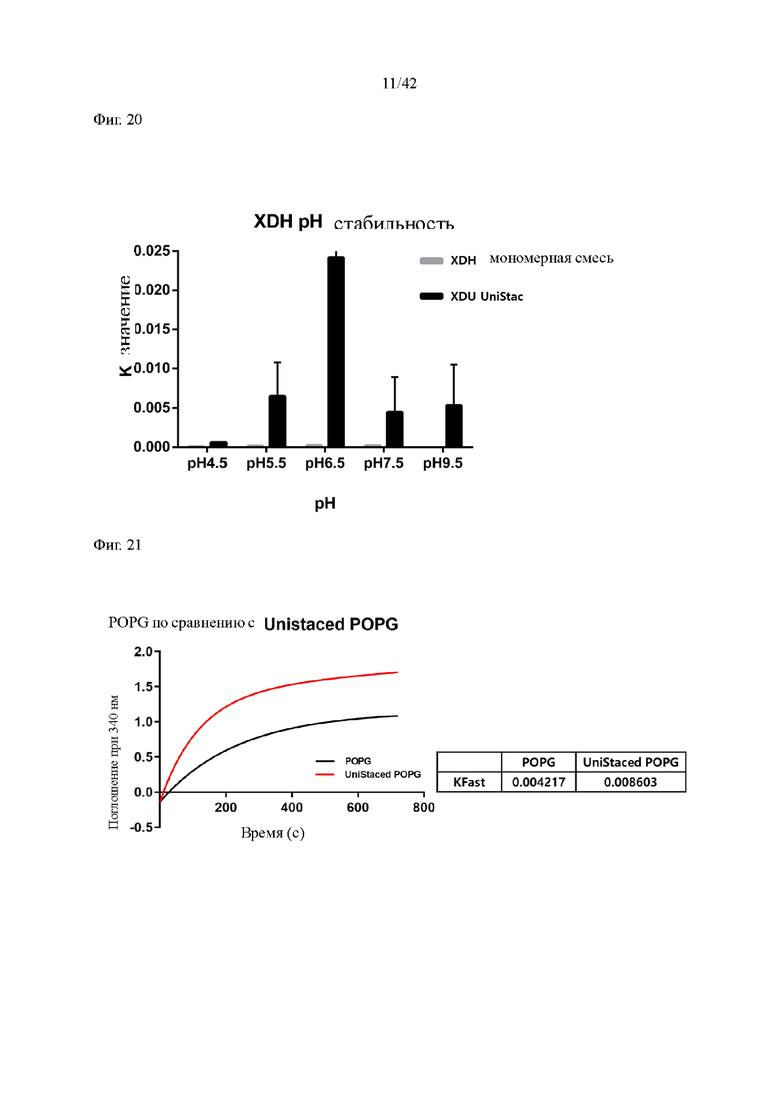
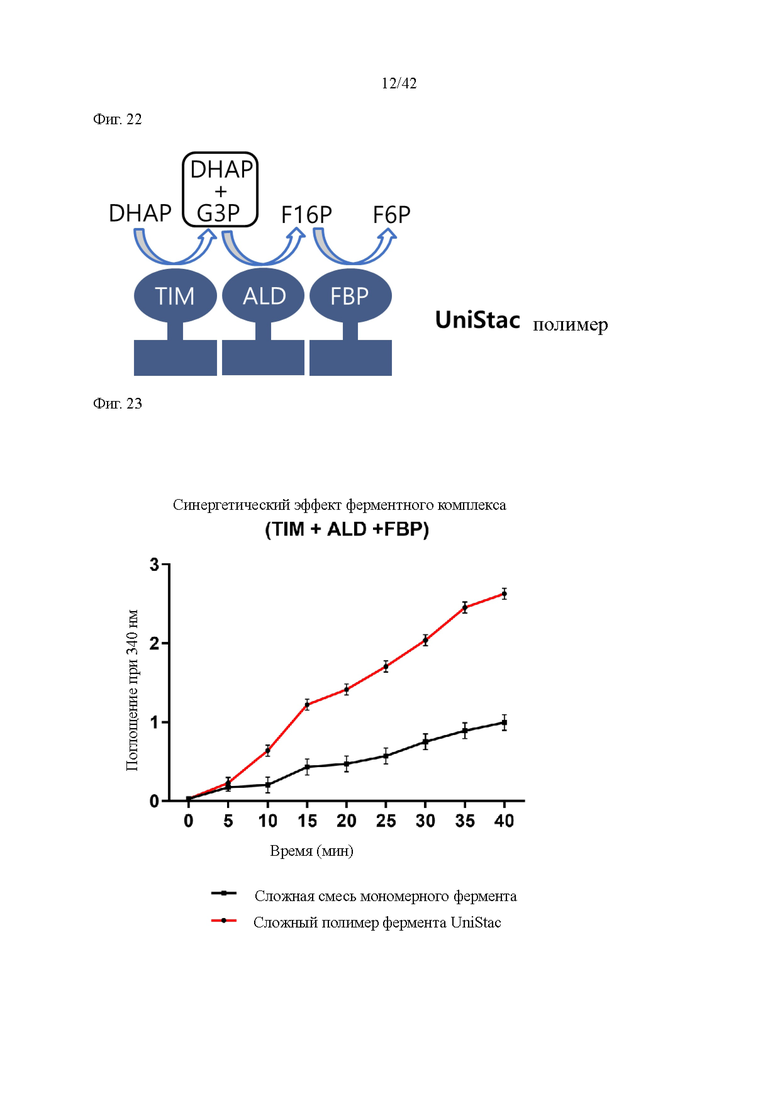










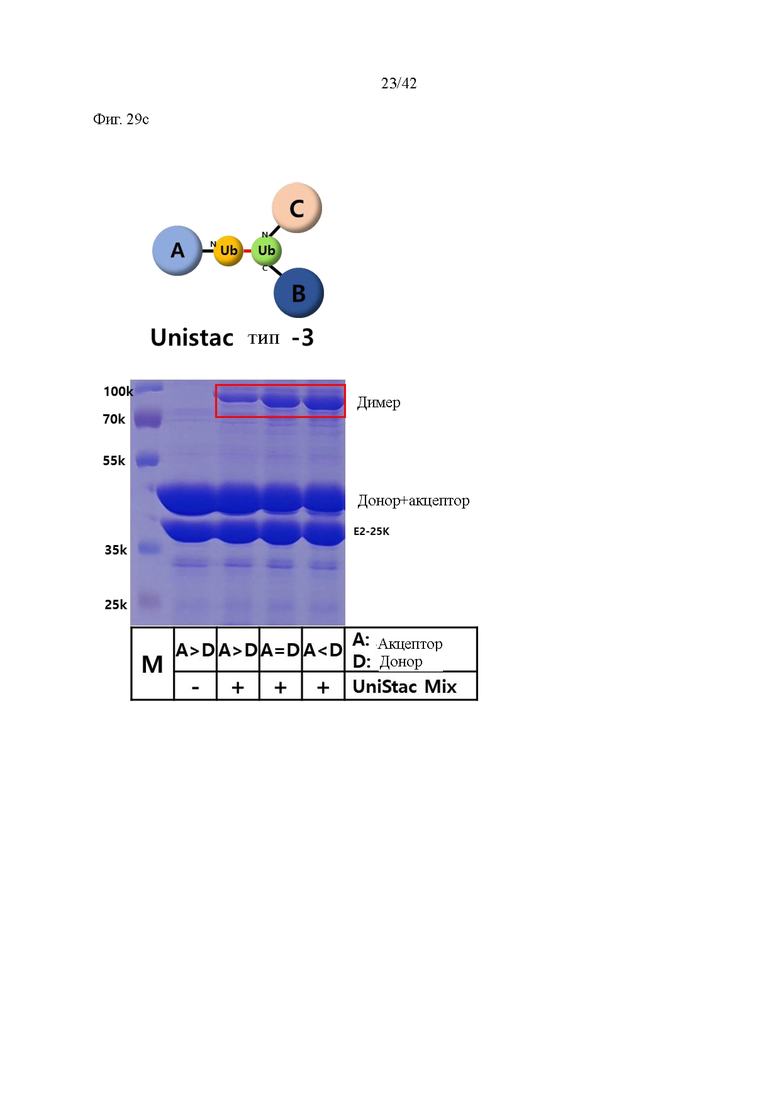



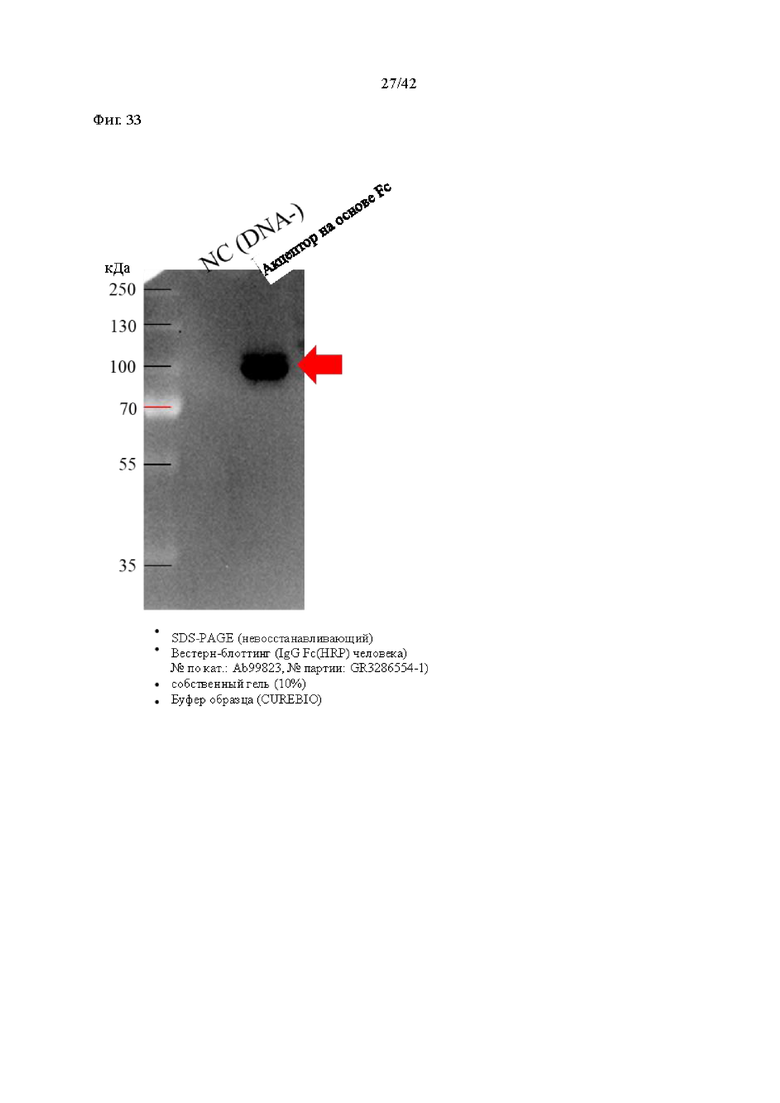



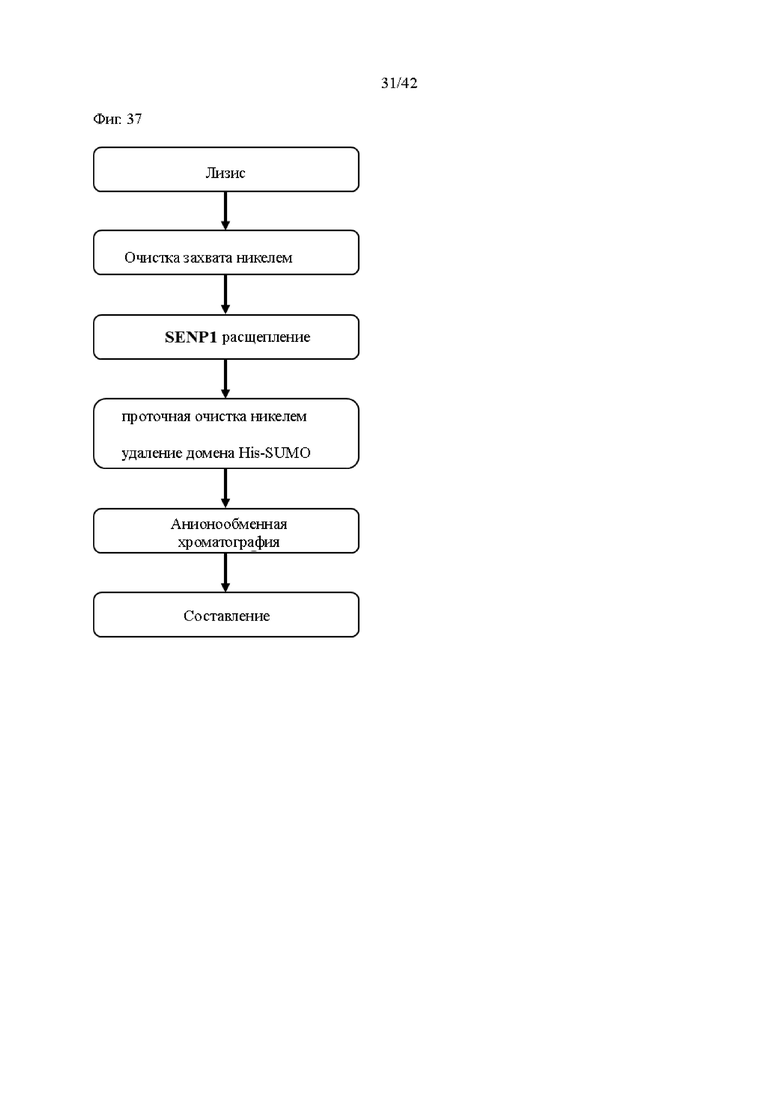





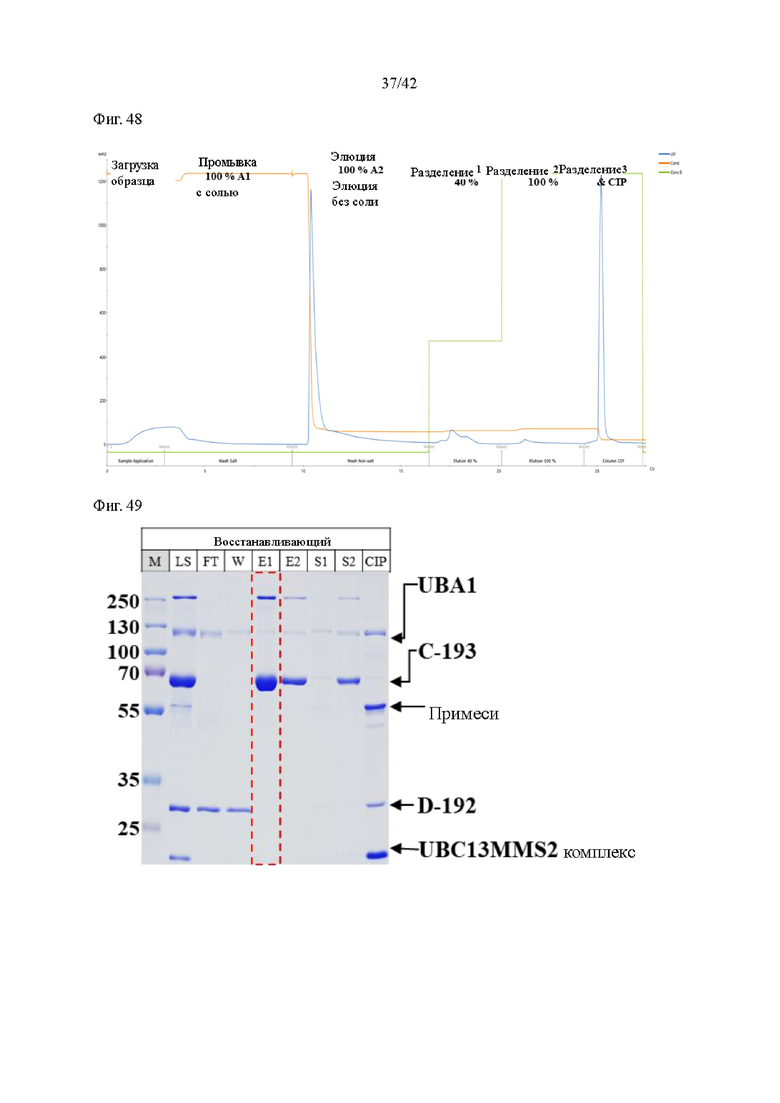





Настоящее изобретение относится к области биотехнологии. Изобретение раскрывает новый полимер на основе убиквитина, причем биологически активные молекулы, связанные с таким полимерным каркасом, обладают увеличенным временем полужизни in vivo, что может быть использовано при получении лекарственных средств и терапевтически эффективных форм из указанных биологически активных веществ. 2 н. и 11 з.п. ф-лы, 19 пр., 9 табл., 58 ил.
1. Применение полимера, содержащего полиубиквитиновый каркас, который образован ковалентным связыванием двух или более убиквитинов, биомолекулу и носитель, для продления стабильности и продолжительности эффективности биомолекулы in vivo, где
биомолекула состоит из от 2 до 10 биомолекул и содержит активные сайты, которые специфически связываются с другими биомолекулами, низкомолекулярными химическими соединениями или наночастицами, и непосредственно связана с N-концом, С-концом или как с N-концом, так и с С-концом убиквитина, или связана линкером, и где
носитель непосредственно связан с N-концом, С-концом или как с N-концом, так и с С-концом убиквитина или связан линкером, где
носитель является одним или несколькими, выбранными из группы, состоящей из альбумина, фрагмента антитела, Fc-домена, трансферрина, XTEN (генетическое слияние неточных повторяющихся пептидных последовательностей), CTP (карбоксиконцевой пептид), PAS (пролин-аланин-сериновый полимер), ELK (эластиноподобный пептид), HAP (гомоаминокислотный полимер), GLK (желатиноподобный белок), PEG (полиэтиленгликоль) и жирной кислоты.
2. Применение по п. 1, где линкер представляет собой комбинацию от 1 до 30 повторов GGGGS или EAAAK.
3. Применение по п. 1 или 2, где биомолекула, связанная с N-концом убиквитина, является дистальным концом мультимерного биомолекулярного полимера.
4. Применение по п. 1 или 2, где биомолекула, связанная с С-концом, N-концом или как с С-концом, так и с N-концом убиквитина, является проксимальным концом мультимерного биомолекулярного полимера.
5. Применение по п. 1 или 2, где полиубиквитиновый каркас образован путем ковалентного связывания донорного убиквитина, в котором один или несколько лизинов убиквитина замещены другими аминокислотами, включая аргинин или аланин, и акцепторного убиквитина, в котором 6-й, 11-й, 27-й, 29-й, 33-й, 48-й или 63-й лизин от N-конца замещен другими аминокислотами, включая аргинин или аланин.
6. Применение по п. 1 или 2, где 73-й лейцин от N-конца убиквитина заменен другими аминокислотами, включая пролин.
7. Применение по п. 1 или 2, где биомолекула выбрана из группы, состоящей из инсулина, аналога инсулина, глюкагона, глюкагоноподобных пептидов, GLP-1 и двойного агониста глюкагона, GLP-1 и двойного агониста GLP, GLP-1 и глюкагона и тройного агониста GLP, экзендин-4, аналога экзендина-4, пептида, секретирующего инсулин, и его аналога, гормона роста человека, гормона, высвобождающий гормон роста (GHRH), пептида, высвобождающего гормон роста, гранулоцитарного колониестимулирующего фактора (G-CSF), пептида против ожирения, рецептора, связанного с G-белком, лептина, GIP (желудочный ингибирующий полипептид), интерлейкинов, рецепторов интерлейкина, белков, связывающих интерлейкин, интерферонов, рецепторов интерферона, белков, связывающих цитокины, активатора макрофагов, пептида макрофагов, В-клеточного фактора, Т-клеточного фактора, супрессивного фактора аллергии, гликопротеина некроза клеток, иммунотоксина, лимфотоксина, фактора некроза опухоли (TNF), фактора ингибирования опухоли, фактора роста метастазов, альфа-1-антитрипсина, альбумина, альфа -лактальбумина, аполипопротеина-Е, эритропоэтина (EPO), высокогликозилированного эритропоэтина, ангиопоэтинов, гемоглобина, тромбина, пептида, активирующего рецептор тромбина, тромбомодулина, факторов крови VII, VIIa , VIII, IX и XIII, активаторов плазминогена, фибрин-связывающего пептида, урокиназы, стрептокиназы, гирудин, белка С, С-реактивного белка, ингибитора ренина, ингибитора коллагеназы, супероксиддисмутазы, тромбоцитарного фактора роста, эпителиального фактора роста, эпидермального фактора роста, ангиостатина, ангиотензина, костного морфогенетического фактора роста, костного морфогенетического белка, кальцитонина, атриопептина, фактора, индуцирующего хрящ, элкатонина, активатора соединительной ткани, ингибитора пути тканевого фактора, фолликулостимулирующего гормона (FSH), лютеинизирующего гормона (LH), гормона высвобождения лютеинизирующего гормона (LHRH), факторов роста нервов, паратиреоидного гормона (PTH), релаксина, секретина, соматомедина, гормона коры надпочечников, холецистокинина, панкреатического полипептида, гастрин-высвобождающего пептида, кортикотропин-высвобождающего фактора, тироид-стимулирующего гормона (TSH), аутотаксина, лактоферрина, миостатина, рецептора, антагониста рецептора, фактора роста фибробластов, адипонектина, антагониста рецептора интерлейкина, антигена клеточной поверхности, вирусного вакцинного антигена, моноклонального антитела, поликлонального антитела и фрагментов антител.
8. Способ получения полимера, в котором полиубиквитиновый каркас, две или более биомолекул, содержащих связывающие фрагменты, каждый из которых специфичен для разных сайтов связывания, и носитель, который продлевает продолжительность эффективности in vivo, непосредственно связаны с N-концом или С-концом убиквитина или связанны линкером, где способ включает
(i) рекомбинантную экспрессию биомолекулы, с которой С-концевая метка убиквитина слита или связана линкером, из клетки-хозяина, включая прокариотические клетки или эукариотические клетки, и
(ii) добавление ферментов E1, E2 и E3 или ферментов E1 и E2 для убиквитинирования в клеточные лизаты или очищенные продукты клетки-хозяина и взаимодействие с ними,
где полиубиквитиновый каркас образован путем ковалентного связывания двух или более убиквитинов, и
биомолекула состоит из от 2 до 10 биомолекул, имеет активные сайты, которые специфически связываются с другими биомолекулами, низкомолекулярными химическими соединениями или наночастицами и связаны с N-концом, C-концом или как с N-концом, так и с C-концом убиквитина линкером.
9. Способ по п. 8, где фермент Е2 связывается с 6-м, 11-м, 27-м, 29-м, 33-м, 48-м или 63-м лизином от N-конца убиквитина.
10. Способ по п. 8 или 9, где фермент E2 представляет собой фермент конъюгации убиквитина E2-25K.
11. Способ по п. 8 или 9, где фермент E2 представляет собой ферментный комплекс Ucb13-MMS2 конъюгации убиквитина.
12. Способ по п. 8 или 9, где С-концевая метка убиквитина представляет собой метку, в которой два или более убиквитина повторно связаны в форме «голова к хвосту» или в разветвленной форме (форма разветвленного типа или изопептидная форма типа разветвления).
13. Способ по п. 12, где убиквитин, связанный в форме «голова к хвосту» или в разветвленной форме, представляет собой убиквитин, в котором 75-й и 76-й глицины от N-конца заменены другими аминокислотами, включая валин.
| RU 2014141617 A1, 10.05.2016 | |||
| WO 2019007869 A1, 10.01.2019 | |||
| WO 2009009773 A1, 15.01.2009 | |||
| US 20130267680 A1, 10.10.2013. |
Авторы
Даты
2023-10-13—Публикация
2020-11-27—Подача