Изобретение относится к плазмидной ДНК-матрице C1M-BSI и к молекуле мРНК mRNA-BSI, обеспечивающим синтез искусственного полиэпитопного белка-иммуногена BSI-COV, содержащего консервативные эпитопы антигенов вируса SARS-CoV-2, и может быть использовано в области биотехнологии, медицины и иммунологии. Данная мРНК индуцирует SARS-CoV-2-специфический Т-клеточный и гуморальный иммунитет у лабораторных животных.
Во время пандемии COVID-19 для массового использования были одобрены первые мРНК-вакцины и полученные результаты стали свидетельством больших перспектив данного вида вакцинных разработок. Важно, что мРНК-вакцины способны очень эффективно индуцировать Т-клеточный иммунный ответ, необходимый для формирования активного и пролонгированного противовирусного иммунитета. Существуют технологии синтеза мРНК in vitro, что позволяет избежать необходимости трудоемкой очистки получаемого препарата от белков линии-продуцента.
Известны мРНК-вакцины против COVID-19, запатентованные исследователями Калифорнийского университета США (WO 2023069551, МПК А61К 39/215, опубл. 27.04.2023 г.). Данная заявка включает описание трех конструкций, которые могут быть использованы как дополнительные к основным зарегистрированным вакцинам, так и самостоятельно. Одна вакцина содержит только CD8+ эпитопы в различных комбинациях. Другая - эпитопы CD4+ фолликулярных Т-хелперов и консервативные линейные В-клеточные эпитопы. Третья - CD4+, CD8+ и В-клеточные эпитопы. Во всех конструкциях эпитопы представлены короткими последовательностями, перемежающимися со спейсерами из двух или четырех аминокислот, а также включают различные регуляторные последовательности, такие как сайт расщепления фурином или катепсином, сайты для повышения секреции, мембранные сигнальные пептиды и т.д. В качестве возможных средств доставки указанных мРНК-вакцин в патенте указаны многочисленные липосомальные частицы и/или любые биосовместимые полимеры. Однако, ни для одной из указанных конструкций ни в патенте, ни в публикациях нет данных о биологической и иммунологической активности.
Наиболее близким аналогом (прототипом) является два варианта универсальной мРНК-вакцины, разработанной китайскими исследователями из Сучжоу (CN 116808190, МПК А61К 39/215, опубл. 29.09.2023 г.). Каждая из вариантов вакцин кодирует последовательность, обеспечивающую индукцию нейтрализующих антител: белки S1, RBD и HR1/HR2, а также одну из последовательностей, отвечающую за индукцию Т-клеточного ответа. Между этими двумя частями расположен сайт протеолиза. Один из вариантов, нацеленных на индукцию клеточного ответа - последовательность, состоящая из 100 Т-клеточных эпитопов, находящихся в консервативных областях вирусных белков, как белка-шипа, так и других. Другой вариант -последовательность, кодирующая белки Е, М, N и RdRp. Доставка обеих мРНК-вакцин осуществлялась с помощью липидной системы и было показано, что введение данных вакцин вызывает иммунный ответ, способный обеспечить защиту животных от заражения вирусом. Однако нет никаких данных о том, были ли отобранные эпитопы проанализированы с помощью известных инструментов на иммуногенность. Исходя из текста патента, можно заключить, что в состав вакцины вошли консервативные регионы вируса SARS-CoV-2, имеющие высокую гомологию среди разных штаммов, а также в сравнении с родственными вирусами SARS-CoV и MERS-CoV. К тому же конструкция представляется довольно сложной и громоздкой, когда включает сразу последовательности, кодирующие Т-клеточные и В-клеточные эпитопы, представленные целыми белками. Также есть данные, демонстрирующие более низкую эффективность экспрессии генов, находящихся не на первой позиции в полицистронных конструкциях.
Данную проблему можно решить, располагая В-клеточные и Т-клеточные иммуногены в разных конструкциях, к тому же это позволит менять В-клеточную компоненту в соответствии с актуальным штаммом, ведь зачастую именно наиболее богатые эпитопами нейтрализующих антител фрагменты вирусных белков являются быстро мутирующими под давлением отбора, и вакцины, содержащие в качестве мишеней такие белки, со временем становятся не эффективными.
Техническим результатом заявляемого изобретения является создание более простой по конструкции полиэпитопной Т-клеточной мРНК вакцины, кодирующей только Т-клеточные консервативные эпитопы и содержащей высоко иммуногенные эпитопы из различных белков вируса SARS-CoV-2, консервативных для разных штаммов этого вируса и рестриктируемых широким спектром аллелей МНС, которая может быть как самостоятельным иммунобиологическим препаратом, так и основной универсальной частью вакцинного комплекса, способного индуцировать специфический цитотоксический и Т-хелперный иммунитет против различных штаммов вируса SARS-CoV-2.
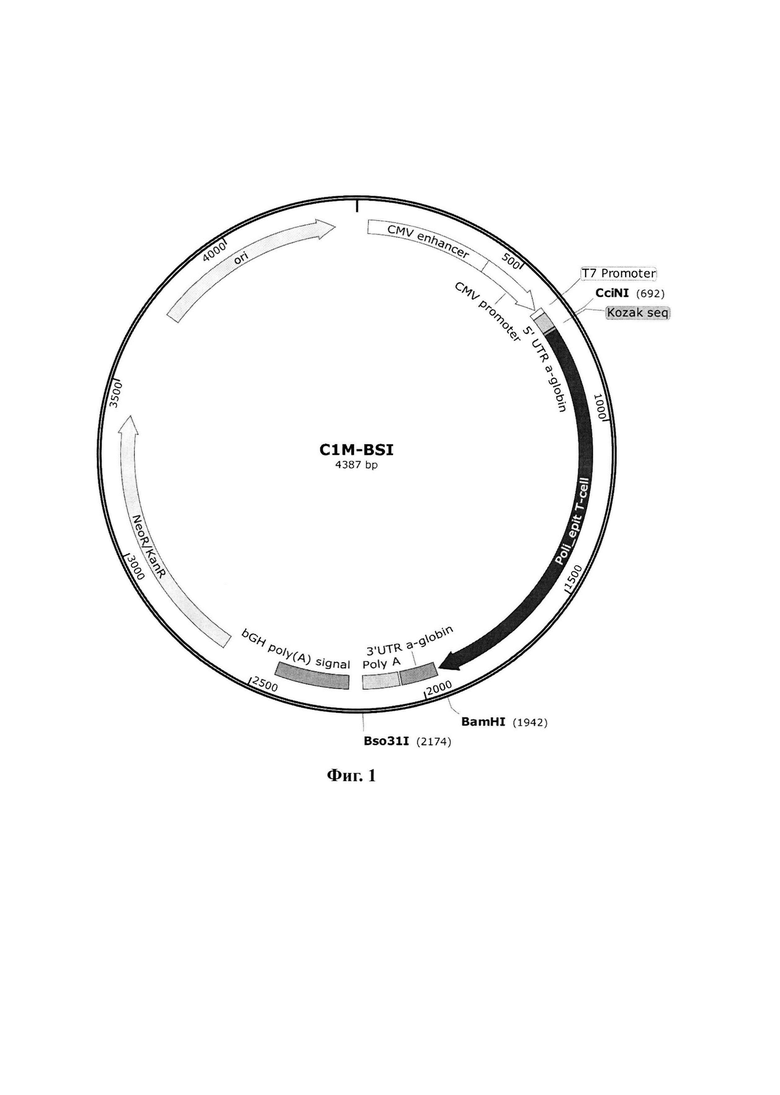
Указанный технический результат достигается созданием плазмидной ДНК-матрицы C1M-BSI, имеющей нуклеотидную последовательность SEQ ID NO: 1 размером 4387 п.о., молекулярную массу 2,8 МДа, содержащей целевой ген bsi-cov, кодирующий искусственный Т-клеточный иммуноген, перед которым находится промотор ДНК-зависимой РНК-полимеразы фага Т7, обеспечивающий синтез in vitro mRNA-BSI с помощью рекомбинантной РНК-полимеразы фага Т7, и состоящей в соответствии с физической и генетической карты представленной на фиг. 1 из следующих фрагментов:
- векторный фрагмент плазмиды pVAX с координатами 2175-641 и размером 2855 п.о., содержащий промотор РНК-полимеразы фага Т7 с координатами 625-641, размером 17 п.о., ген устойчивости к неомицину/канамицину (NeoR/KanR), имеющий координаты 2614-3408, и размер 795 п.о., точку начала репликации Co1E1 ori, имеющий координаты 3734-4322 и размер 589 п.о., а также энхансер и промотор цитомегаловируса, имеющий координаты 36-619 и размер 584 п.о.;
- фрагмент, имеющий координаты 643-2174 и размер 1532 п.о., содержащий целевой искусственный ген bsi-cov, имеющий координаты 703-1938 и размер 1236 п.о., а также последовательность Козак, имеющая координаты 695-702 и размер 8 п.о. и последовательности, кодирующие 5'-нетранслируемую область, имеющую координаты 643-692 и размер 50 п.о. и 3'-нетранслируемую область, имеющую координаты 1948-2057 и размер 110 п.о., полиаденозиновый (полиА) хвост, имеющий координаты 2064-2174 и размер 111 п.о.
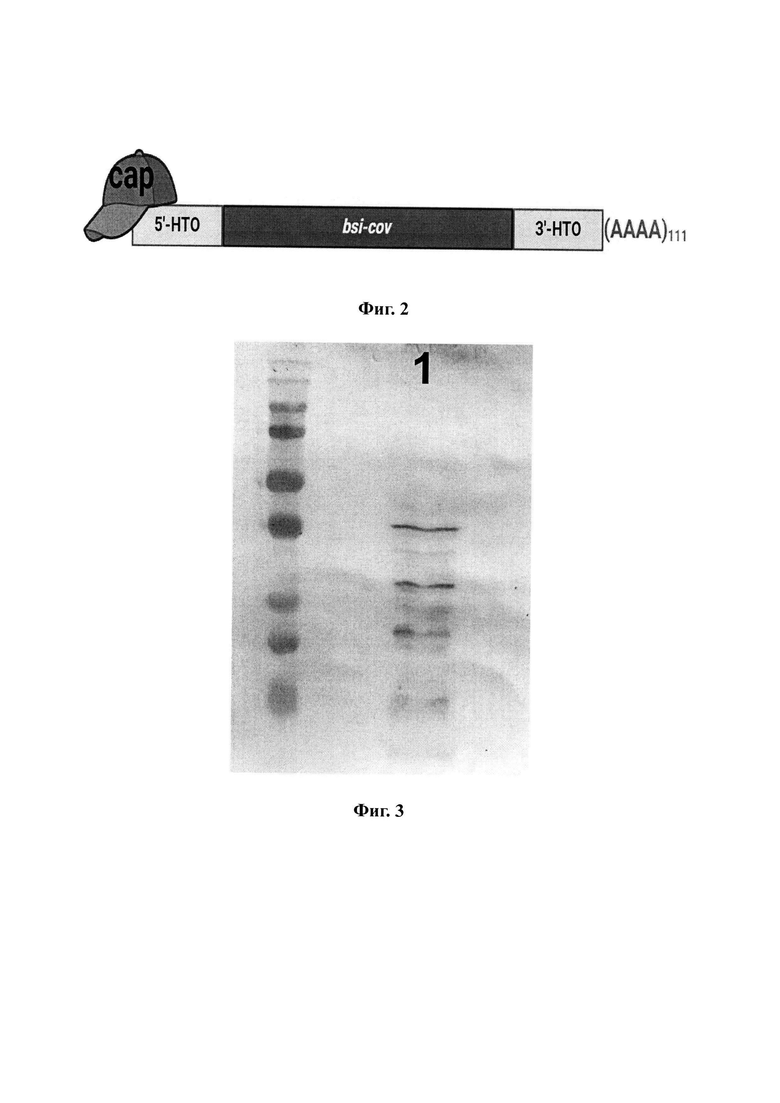
Указанный технический результат достигается также созданием молекулы mRNA-BSI, имеющей нуклеотидную последовательность SEQ ID NO: 2, длиной 1532 п.о., обеспечивающую синтез искусственного иммуногена в организме млекопитающих и содержащую, кроме целевого гена, консенсусную последовательность Козак, 7-метилгуанозиновый кэп на 5'-конце мРНК, полиаденозиновый (полиА) хвост на 3'-конце мРНК (длиной 111 нуклеотидов), 5'- и 3'-нетранслируемые области (НТО) по обе стороны от рамки считывания (фиг. 2).
В качестве средства доставки мРНК в данной заявке использовался липосомальный комплекс, однако указанный метод не входит в состав заявки, и мРНК-вакцина mRNA-BSI может быть введена любым доступным способом.
Изобретение иллюстрируется следующими графическими материалами.
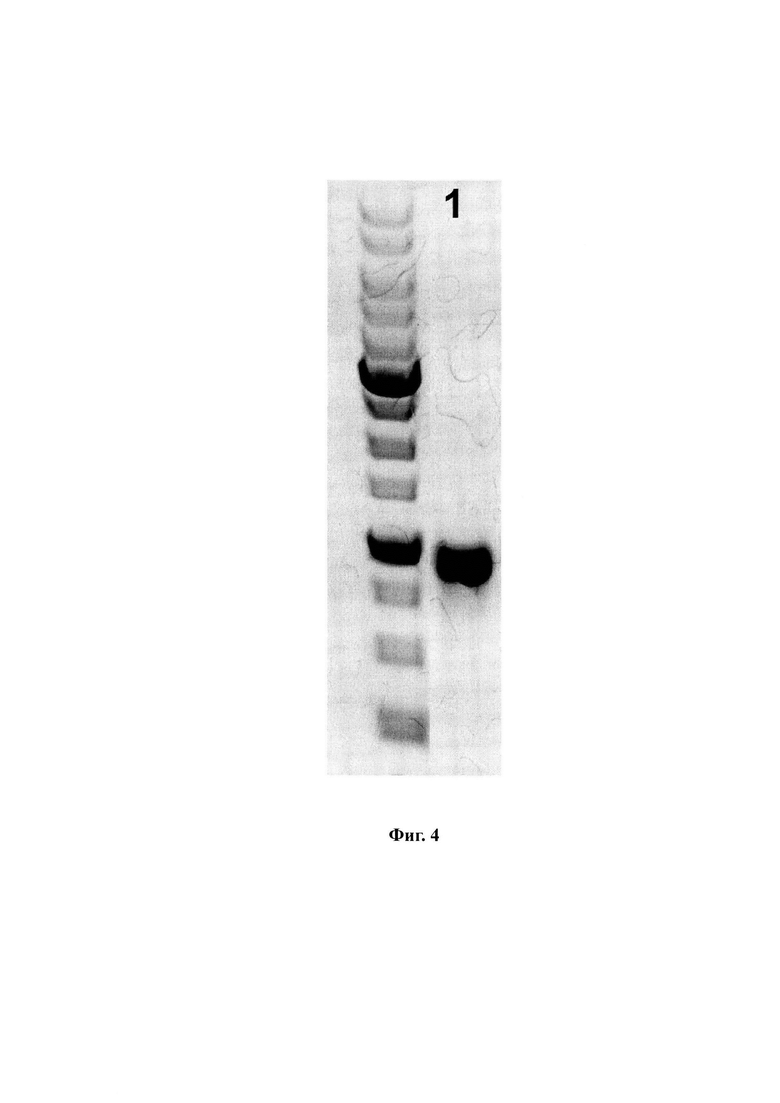
На фиг. 1 приведена физическая и генетическая карта плазмидной матрицы C1M-BSI. На фиг. 2 представлена схема структуры mRNA-BSI. На фиг. 3 представлен анализ продукции белка BSI-COV в клетках HEK293T, трансфицированных плазмидной ДНК C1M-BSI, с помощью МКА 29F2, специфичных к маркерному эпитопу в составе иммуногена, и вестерн-блоттинга:
Дорожка 1 - лизат HEK293T, трансфицированных C1M-BSI;
Дорожка 2 - лизат нетрансфицированных HEK293T;
Слева - маркер молекулярных масс Precision Plus Protein Dual Color Standards (Bio-rad, США).
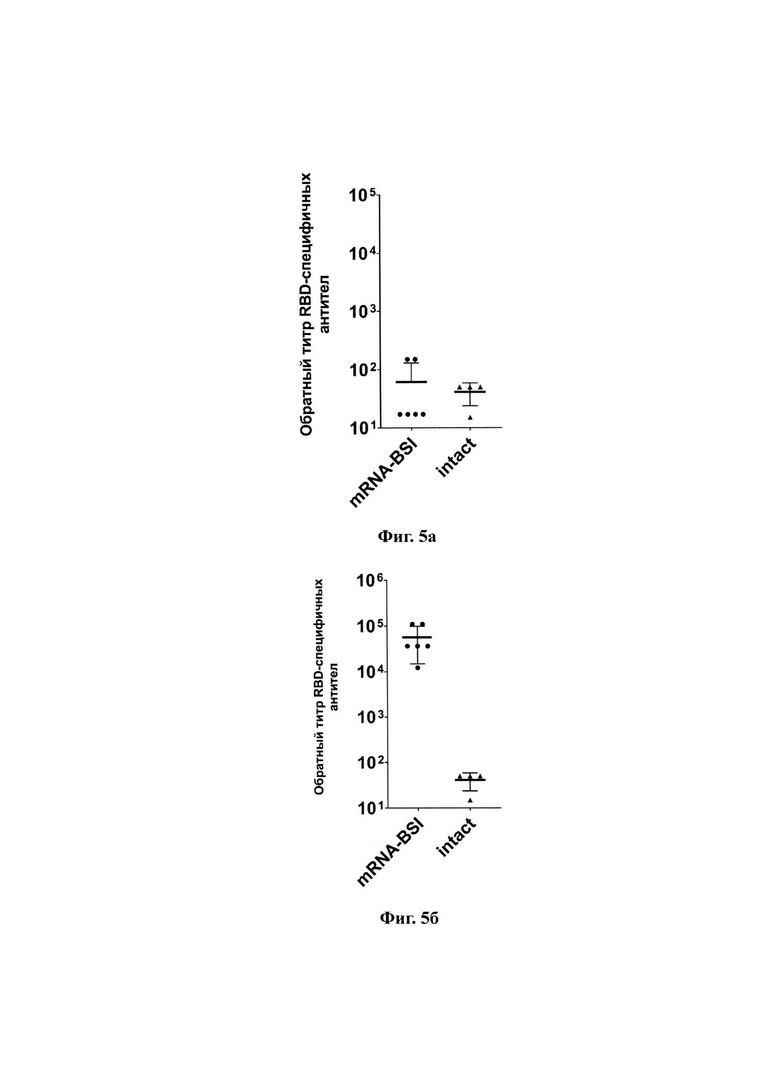
На фиг. 4 приведен электрофоретический анализ мРНК, синтезированной in vitro, после обработки ДНКазой, в 2% агарозном геле.
Слева - маркер длин ДНК для ориентировочной оценки длин мРНК;
Дорожка 1 - мРНК mRNA-BSI, синтезированная in vitro.
На фиг. 5а представлены диаграммы оценки RBD-специфических антител в сыворотках мышей через три недели после первой иммунизации.
Группа mRNA-BSI - иммунизирована мРНК mRNA-BSI;
Группа intact - не иммунизированные мыши.
На фиг. 5б приведены диаграммы оценки RBD-специфических антител в сыворотках мышей через две недели после второй иммунизации.
Группа mRNA-BSI - иммунизирована мРНК mRNA-BSI;
Группа intact - не иммунизированные мыши.
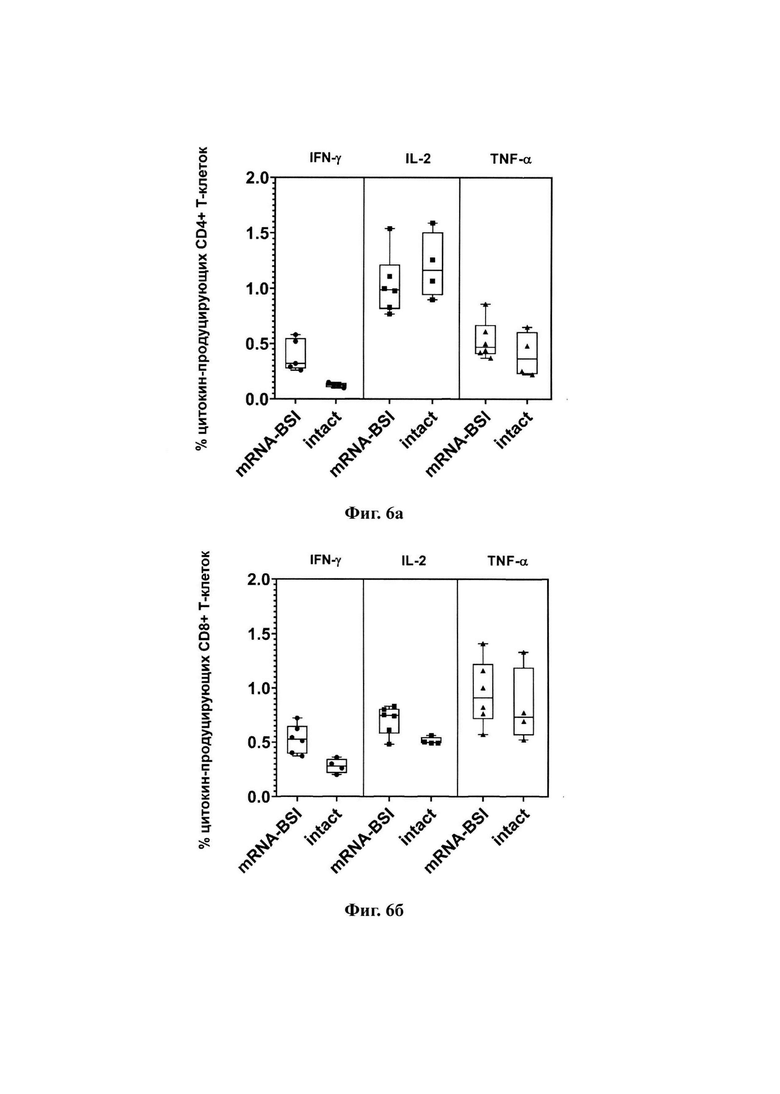
На фиг. 6а представлена доля SARS-CoV-2-специфических цитокин-продуцирующих CD4+ Т-клеток (по данным ICS и проточной цитометрии).
Группа mRNA-BSI - иммунизирована мРНК mRNA-BSI;
Группа intact - не иммунизированные мыши.
На фиг. 6б представлена доля SARS-CoV-2-специфических цитокин-продуцирующих CD8+ Т-клеток (по данным ICS и проточной цитометрии).
Группа mRNA-BSI - иммунизирована мРНК mRNA-BSI;
Группа intact - не иммунизированные мыши.
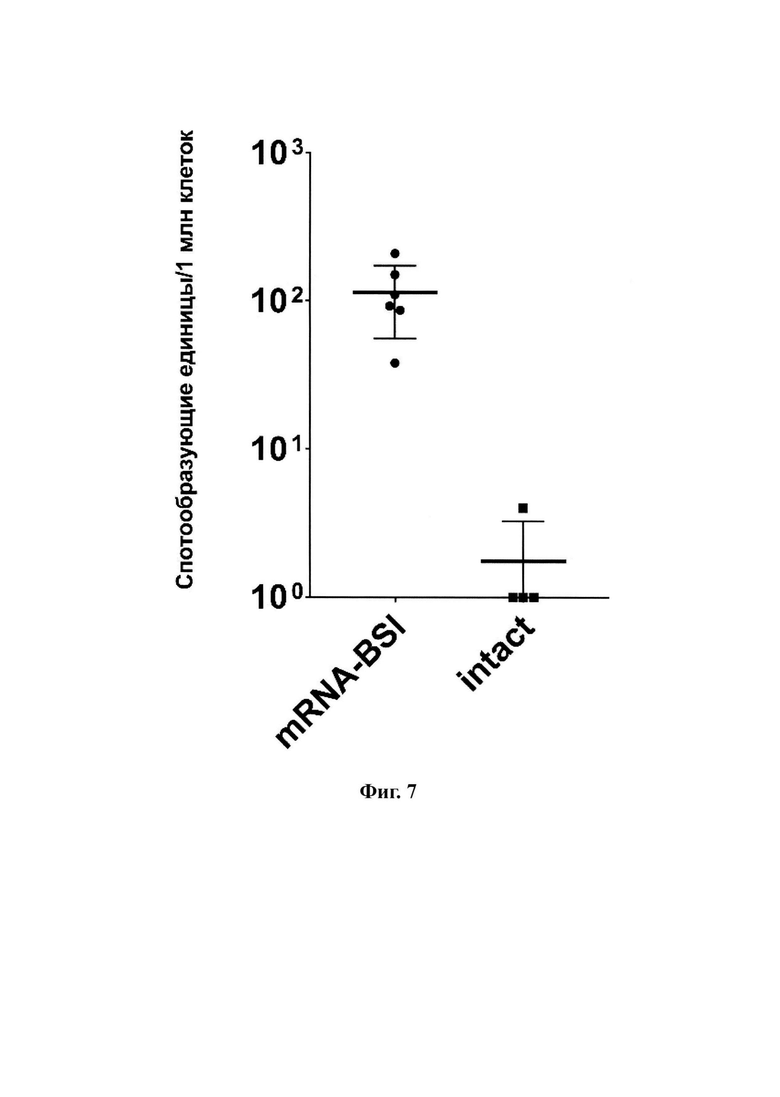
На фиг. 7 приведены результаты оценки вирус-специфических Т-лимфоцитов у иммунизированных животных на 35 сутки после начала иммунизации с помощью метода ELISpot.
Группа mRNA-BSI - иммунизирована мРНК mRNA-BSI;
Группа intact - не иммунизированные мыши.
На фиг. 8 приведена нуклеотидная последовательность SEQ ID NO: 1 плазмидной ДНК-матрицы C1M-BSI, имеющей размер 4387 п.о.
На фиг. 9 представлена нуклеотидная последовательность SEQ ID NO: 2 молекулы mRNA-BSI, имеющей размер 1532 п.о.
Ниже приведены примеры конкретного выполнения изобретения.
Пример 1. Дизайн плазмиды C1M-BSI.
Ген bsi-cov амплифицировали с конструкции pBSI-COV (патент РФ №2806556, опубл. 01.11.2023 г.) с помощью специфических праймеров (Borg BSI to C1 - F: 5'-tttttgcggccgccaccatggccaag-3', Borg BSI to C1 - R: 5' tttttGGATCCttagcgcagggtcttgtagaagc-3'), затем обрабатывали эндонуклеазами рестрикции CciNI и BamHI и встраивали в вектор С1М в окружение 5'- и 3'- НТО. Структуру полученной конструкции подтверждали секвенированием по методу Сэнгера в ЦКП "Геномика" (Россия). В результате была получена ДНК-конструкция, кодирующая искусственный иммуноген BSI-COV, которая была обозначена как C1M-BSI (фиг. 1).
Пример 2. Проверка эффективности экспрессии целевого гена в условиях in vitro
2.1 Трансфекция клеток HEK293T
Перевиваемые клетки почечного эмбрионального эпителия человека HEK-293Т (Human embryonic kidney), получены из коллекции клеточных культур ГНЦ ВБ «Вектор». Культуру клеток HEK-293Т культивировали при температуре 37°С, 80% относительной влажности воздуха и 5% содержании СО2. В качестве ростовой среды использовали среду DMEM с добавлением 10% термоинактивированной (30 мин при температуре 56°С) фетальной бычьей сыворотки (FBS), 2 мМ L-глутамина и антибиотика (гентамицин (50 мкг/мл)). Трансфекцию производили в 12-луночных планшетах с использованием реагента Matra-A, содержащего магнитные частицы, в соответствии с инструкцией производителя. По истечении 48 часов после трансформации клетки снимали с использованием раствора Версена и центрифугировали 5000 об/мин, осадок растворяли в 30 мкл PBS для дальнейшей работы.
2.2 Исследование продукции целевых белков
Лизат трансфицированных клеток исследовали на наличие целевого белка с помощью иммуноблотинга с использованием МКА 29F2 к маркерному эпитопу. Полосы, соответствующие белку с наибольшим весом, соответствуют теоретически рассчитанному продукту гена bsi-cov (примерно 46 кДа). Наличие лесенки из дискретных белков свидетельствует об эффективном процессинге Т-клеточных иммуногенов в клетке (фиг. 3).
Пример 3. Описание мРНК mRNA-BSI
Синтез мРНК, кодирующей искусственный иммуноген BSI-COV вируса SARS-COV-2, проводили с соответствующей линеаризованной и очищенной ДНК-матрицы (C1M-BSI-Ub). ДНК-матрица C1M-BSI, имеющая нуклеотидную последовательность SEQ ID NO: 1 и карту, представленную на фиг. 1, содержит промотор ДНК-зависимой РНК-полимеразы бактериофага Т7 и целевой ген bsi-cov.
Линеаризацию матрицы проводили с использованием эндонуклеазы рестрикции Bso31I, после чего матрицу очищали от компонентов реакционной смеси и использовали для синтеза мРНК из расчета 1 мкг матрицы на 150 мкг синтезируемой мРНК с помощью набора фирмы «Биолабмикс» (Россия) по инструкции производителя. Смесь рибонуклеотидов содержала псевдоуридин вместо уридина, а также в реакцию был добавлен аналог Сар. После синтеза мРНК была обработана ДНКазой, очищена переосаждением AcNH4 и проанализирована с помощью электрофореза в 2% агарозном геле (фиг. 4).
В результате, была получена мРНК mRNA-BSI, имеющая нуклеотидную последовательность SEQ ID NO: 2.
Пример 4. Иммуногенность mRNA-BSI
4.1 Иммунизация лабораторных животных, подготовка образцов для исследования
Работа с животными проводилась в соответствии с «Руководством по уходу и использованию лабораторных животных». Протоколы были одобрены Комитетом по уходу и использованию животных (IACUC) при Государственном научном центре вирусологии и биотехнологии «Вектор» (ГНЦ ВБ «Вектор»/08-02.2024, Протокол №3 от 29.02.2024).
Для оценки иммуногенности созданной конструкции использовали самок мышей BALB/c массой 16-18 г. Мышей разделили на две группы по 6 животных в каждой и иммунизировали группу 1 (mRNA-BSI) - 10 мкг мРНК, кодирующей искусственный иммуноген BSI-COV, а группу intact не иммунизировали. Иммунизацию проводили дважды внутримышечно (в верхнюю часть бедра задней конечности) в дни 0 и 21 с помощью липосомальных частиц. Спустя 14 дней после второй иммунизации у животных были взяты кровь и селезенки для исследования иммунного ответа.
Кровь собирали из орбитального синуса через три недели после начала эксперимента и в конечной точке, инкубировали 1 ч при 37°С, сыворотки отделяли от клеточных элементов центрифугированием (10000 об/мин, 5 мин), затем инактивировали в течение 30 мин при 56°С и хранили при -20°С до момента анализа.
Селезенки последовательно измельчали на нейлоновых фильтрах для клеток с диаметром пор 70 и 40 мкм (BD Falcon, США). После лизиса эритроцитов лизирующим буфером (Sigma, США) спленоциты дважды промывали полной средой RPMI и помещали в 1 мл среды RPMI с 2 мМ L-глутамина, 50 мкг/мл гентамицина и 10% FBS (Thermo Fisher Scientific, США). Клетки подсчитывали с использованием автоматического счетчика клеток ТС20 (Bio-Rad, Hercules, СА, USA).
4.2 Иммуноферментный анализ сывороток
В качестве иммуносорбента в ИФА использовали рекомбинантный белок RBD, который сорбировали на 96-луночные планшеты (Nunc, Дания) в концентрации 1 мкг/мл в течение ночи при 4°С в 2М растворе мочевины (рН 8.0). На следующий день лунки промывали, блокировали 1% раствором казеина и добавляли серийные разведения сывороток, начиная с 1:50, заканчивая 1:109350. Сыворотки инкубировали 1 ч, затем отмывали и добавляли в лунки раствор антител против IgG мыши, конъюгированных с пероксидазой хрена (Sigma, США), инкубировали в течение 1 ч, после чего в отмытые лунки добавляли хромоген ТМВ (Имтек, Россия). После остановки реакции стоп-раствором оптическую плотность измеряли при длине волны 450 нм с помощью ИФА-ридера Varioskan LUX (Thermo Fisher Scientific, США).
Данные ИФА представлены на фиг. 5а и 5б. Уровень антител в группе mRNA-BSI составил в среднем 1:61 через три недели после первой иммунизации и 1:56700 - в конечной точке (две недели после второй иммунизации) (фиг. 5б). Эти данные свидетельствуют об эффективности созданной мРНК-вакцины, кодирующей полиэпитопный иммуноген BSI-COV, несмотря на то, что лишь небольшая часть иммуногена представляет из себя последовательность, взятую из RBD белка S вируса SARS-CoV-2.
4.3 Исследование поствакцинального Т-клеточного ответа с помощью ICS и проточной цитофлуориметрии
Для анализа CD4+ и CD8+ Т-лимфоцитов, реагирующих выбросом IFN-γ при стимуляции вирус-специфическими пептидами, 7*105 клеток пассировали в лунки 96-луночных культуральных планшетов с круглым дном(Jet-BIOFIL, Китай) и стимулировали смесью пептидов, входящих в состав иммуногена BSI-COV. Каждый пептид добавляли в концентрации 20 мкг/мл на лунку и клетки инкубировали в течение 4 ч при 37°С в присутствии 5% СО2 и еще 16 ч с брефельдином А (5 мкг/мл, GolgiPlug BD Biosciences). На следующий день клетки окрашивали анти-CD3, конъюгированным с Alexa Fluor 700 (Biolegend, США), анти-CD4, конъюгированным с BV785 (Biolegend, США), и анти-CD8, конъюгированным с FITC (Biolegend, США); фиксировали 1% параформальдегидом в PBS; и пермеабилизировали 0,5% Tween 20 в PBS. Затем клетки окрашивали для выявления внутриклеточных цитокинов анти-IFNγ-APC, анти-IL2-BV421 и анти-TNFα-PE (Biolegend, США). Образцы анализировали с использованием проточного цитометра ZE5 (Bio-Rad) и программного обеспечения Everest.
Анализ Т-клеточного ответа с помощью метода ICS показал, что при иммунизации мышей мРНК-вакциной mRNA-BSI среди CD3+/CD8+ Т-клеток формируются клетки, продуцирующие IFN-γ, IL-2 и TNF-α в ответ на стимуляцию вирусными пептидами (фиг. 6а). Это говорит об индукции эффекторных цитотоксических Т-лимфоцитов, важных для распознавания и уничтожения клеток, инфицированных SARS-CoV-2. Для CD3+/CD4+ лимфоцитов было показано поствакцинальное повышение доли клеток, продуцирующих IFN-γ и TNF-α, что говорит о формировании пула специфических Т-хелперов преимущественно 1-го типа, участвующих в поддержке цитотоксических лимфоцитов и дополнительно активирующих дендритные клетки и макрофаги (фиг. 6б). Этот факт свидетельствует о формировании выраженного вирус-специфического Т-клеточного ответа. Значения в опытной группе достоверно отличались от контрольной группы.
4.4 Исследование поствакцинального Т-клеточного ответа с помощью метода ELISpot
Анализ Т-клеточного иммунного ответа проводили с использованием набора Mouse IFN-γ ELISpot HRP (Mabtech, Швеция) в соответствии с инструкциями производителя. Спленоциты пассировали в количестве 2,5*105 клеток/лунку и добавляли среду RPMI с 10% FBS (отрицательный контроль), или смесь пептидов (каждого в концентрации 20 мкг/мл), или конканавалин А (положительный контроль). Клетки инкубировали в течение 20 ч при 37°С в присутствии 5% СО2. Затем планшеты промывали и инкубировали с первичными антителами против IFN-γ. Планшеты снова промывали и затем инкубировали со вторичным антителом, конъюгированным со пероксидазой хрена. После промывки планшеты инкубировали с ТМВ. Количество клеток, продуцирующих IFN-γ, подсчитывали с использованием ELISpot ридера (Carl Zeiss, Оберкохен, Германия). Спленоциты, выделенные от иммунизированных мышей BALB/c, стимулировали пулом пептидов, входящих в состав иммуногена BSI-COV, рестриктированных главным комплексом гистосовместимости (МНС) класса I (H2-Dd, H-2-Kd и H-2-Ld) и МНС класса II (H2-IAd и H2-IEd) мышей BALB/c. Пептиды были подобраны в соответствии с эпитопами, входящими в состав иммуногена и предсказанными с использованием инструментов IEDB Analysis Resource и синтезированы AtaGenix Laboratories (Ухань, Китай); чистота пептидов >80%.
Данные IFN-γ-ELISpot показали, что через две недели после второй иммунизации наблюдалось формирование Т-клеточного иммунитета у мышей, которым вводили мРНК-вакцинную конструкцию. В группе, иммунизированной mRNA-BSI, среднее количество Т-лимфоцитов, секретирующих IFN-γ, в расчете на 106 спленоцитов составляло 114 (фиг. 7). Значения в опытной группе достоверно отличались от контрольной группы.
Таким образом, преимущество заявляемой вакцинной платформы по сравнению с другими аналогами заключается в том, что более простая по конструкции мРНК-вакцина mRNA-BSI кодирует только Т-клеточные эпитопы, которые являются консервативными, и может быть как самостоятельным иммунобиологическим препаратом, так и универсальной составной частью вакцин, другие компоненты которых могут меняться в соответствии с эпидемиологической актуальностью. Данная вакцина эффективно индуцирует гуморальный и клеточный ответ при дозе введения всего 10 мкг.
Список источников патентной и научно-технической информации
1. Ng O.W., Chia A., Tan А.Т., Jadi R.S., Leong H.N., Bertoletti A., Tan Y.J. Memory T cell responses targeting the SARS coronavirus persist up to 11 years post-infection. Vaccine. 2016 Apr 12;34(17):2008-14. doi: 10.1016/j.vaccine.2016.02.063.
2. Walsh E.E., Frenck R.W. Jr, Falsey A.R., Kitchin N., Absalon J., Gurtman A., Lockhart S., Neuzil K., Mulligan M.J., Bailey R., Swanson K.A., Li R, Koury K., Kalina W., Cooper D., Fontes-Garfias C, Shi P.Y., Tiireci O., Tompkins K.R., Lyke K.E., Raabe V., Dormitzer P.R., Jansen K.U., §ahin U., Gruber W.C. Safety and Immunogenicity of Two RNA-Based Covid-19 Vaccine Candidates. N Engl J Med. 2020 Dec 17;383(25):2439-2450. doi: 10.1056/NEJMoa2027906.
3. Nathan A., Rossin E.J., Kaseke C, Park R.J., Khatri A., Koundakjian D., Urbach J.M., Singh N.K., Bashirova A., Tano-Menka R., Senjobe R, Waring M.T., Piechocka-Trocha A., Garcia-Beltran W.F., Iafrate A.J., Naranbhai V, Carrington M., Walker B.D., Gaiha G.D. Structure-guided T cell vaccine design for SARS-CoV-2 variants and sarbecoviruses. Cell. 2021 Aug 19;184(17):4401-4413.e10. doi: 10.1016/j.cell.2021.06.029.
4. Wang Q., Lei C, Wan H., Liu Q. Improved cellular immune response elicited by a ubiquitin-fused DNA vaccine against Mycobacterium tuberculosis. DNA Cell Biol. 2012 Apr;31(4):489-95. doi: 10.1089/dna.2011.1309.
5. Safavi A., Kefayat A., Mahdevar E., Abiri A., Ghahremani F. Exploring the out of sight antigens of SARS-CoV-2 to design a candidate multi-epitope vaccine by utilizing immunoinformatics approaches. Vaccine. 2020 Nov 10;38(48):7612-7628. doi: 10.1016/j.vaccine.2020.10.016.
6. Borgoyakova M.B., Karpenko L.I., Rudometov A.P., Starostina E.V., Zadorozhny A.M., Kisakova L.A., Kisakov D.N., Sharabrin S.V., Ilyichev A.A., Bazhan S.I. Artificial COVID-19 T-Cell Immunogen. Bull Exp Biol Med. 2023 Oct;175(6):804-809. doi: 10.1007/s10517-023-05951-7.
7. Международная заявка № WO 2023069551, МПК A61K 39/215, A61K 31/7088, A61K 31/7105, A61K 31/711, A61K 31/713 Multi-epitope mRNA SARS-CoV-2 vaccine for boosting immunity through the activation of CD4 and CD8 T cells as well as В lymphocytes, опубл. 27.04.2023 г.
8. Патент Китая № CN 116808190, МПК A61K 39/215, C12N 15/10, A61K 9/127, A61K 47/18, A61K 47/24, A61K 47/28. Preparation of SARS-Cov-2 mRNA universal vaccine, опубл. 29.09.2023.
9. Патент РФ №2806556, МПК A61K 39/215, C12N 15/62, C07K 14/165, А61Р 31/14, заявка №2023114102: заявл. 29.05.2023: опубл. 01.11.2023 / Бажан С.И., Боргоякова М.Б., Карпенко Л.И., Рудометов А.П., Старостина Е.В., Ильичев А.А.
10. Патент РФ №2806590, МПК A61K 39/215, C12N 15/62, C07K 14/165, А61Р 31/14, заявка №2023114101: заявл. 29.05.2023: опубл. 01.11.2023 / Бажан С.И., Боргоякова М.Б., Карпенко Л.И., Рудометов А.П., Старостина Е.В., Ильичев А.А.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="mRNA-BSI.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-10-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-10-27</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>State Research Center of Virology and
Biotechnology "Vector"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Плазмидная ДНК-матрица и молекула
мРНК, полученная с использованием данной ДНК-матрицы и обеспечивающая
экспрессию гена искусственного полиэпитопного белка-иммуногена,
содержащего консервативные эпитопы антигенов вируса SARS-CoV-2 и
индуцирующего SARS-CoV-2-специфический иммунитет</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>4387</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4387</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>36..415</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>CMV enhanser</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>416..619</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>CMV promoter</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>625..641</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>T7 promoter</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>2217..2441</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>bGH poly(A) signal</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>2614..3408</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>aminoglycoside phosphotransferase from
Tn5 (Neomycin/kanamycin resistance)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>oriT</INSDFeature_key>
<INSDFeature_location>3734..4322</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>high-copy-number ColE1/pMB1/pBR322/pUC
origin of replication</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>703..1938</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gene of artificial multi-epitope
immunogen</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>695..702</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>Kozak sequence</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location>643..692</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>1948..2057</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>polyA_site</INSDFeature_key>
<INSDFeature_location>2064..2174</INSDFeature_location>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactcttcgcgatgtacgggccagatatacgcgttgacattgattattg
actagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacat
aacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgta
tgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggactatttacggtaaactgcc
cacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggc
ccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagt
catcgctattaccatggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgg
ggatttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacgggactttc
caaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctatat
aagcagagctcaaattaatacgactcactataagagaataaactagtattcttctggtccccacagactc
agagagaacccgcggccgccaccatggccaagttcgtggccgcctggaccctgaaggccgccgccaagaa
gaacatcgccgactacaactacaagctgcccgacgacttcaccggctgcgtgatcgcctggaacagcaac
aacctggacagcaaggtgggcggcaactacaactacctgtaccgcctgttccgcaagagcaacctgaagc
ccttcgagagcatcatcgcctacaccatgagcctgggcgccgagaacagcgtggcctacagcaacaacag
catcgccatccccaccaacttcaccatcggcctgaccgtgctgccccccctgctgaccgacgagatgatc
gcccagtacaccagcgccctgctggccggcaccatcaccagcggctggaccttcggcgccggcgccgccc
acctgatgagcttcccccagagcgccccccacggcgtggtgttcctgcacgtgacctacgtgcccgccca
ggagaagaacctgaacgagagcctgatcgacctgcaggagctgggcaagtacgagcagtacatcaagtgg
ccctggtacatctggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgagcctgg
tgaagcccagcttctacgtgtacagccgcgtgaagaacctgaacagcagccgcgtgcccgacctgctggt
gaccctggcctgcttcgtgctggccgccgtgtaccgcatcaactggatcaccggcggcatcgccatcgcc
atggcctgcctggtgggcctgaaggagatcaccgtggccaccagccgcaccctgagctactacaagctgg
gcgccagccagcgcgtggccggcgacagcggcttcgccgcctacttcccccgcggccagggcgtgcccat
caacaccaacagcagccccgacgaccagatcggctactaccgccgcgccacccgccgcatccgcggcggc
gacggcaagtggccccagatcgcccagttcgcccccagcgccagcgccttcttcggcatgagccgcatcg
gcatggaggtgacccccagcggcacctggctgacctacaccggcgccatcaagctggacgacaaggaccc
caacttcaaggaccaggtgatcctgctgaacaagcacatcgacgcctacaagaccttcccccccaccgag
cccaagaaggacaagaagaagaaggagcccttccgcgactacgtggaccgcttctacaagaccctgcgct
aaggatccgctggagcctcggtggcctagcttcttgccccttgggcctccccccagcccctcctcccctt
cctgcacccgtacccccgtggtctttgaataaagtctgagtgggcggcgaattcaaaaaaaaaaaaaaaa
aaaaaaaaaaaaaaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaagagaccgggcccgtttaaacccgctgatcagcctcgactg
tgccttctagttgccagccatctgttgtttgcccctcccccgtgccttccttgaccctggaaggtgccac
tcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtcattctattctg
gggggtggggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgctggggatgcgg
tgggctctatggcttctactgggcggttttatggacagcaagcgaaccggaattgccagctggggcgccc
tctggtaaggttgggaagccctgcaaagtaaactggatggctttctcgccgccaaggatctgatggcgca
ggggatcaagctctgatcaagagacaggatgaggatcgtttcgcatgattgaacaagatggattgcacgc
aggttctccggccgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggctgctct
gatgccgccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccggtg
ccctgaatgaactgcaagacgaggcagcgcggctatcgtggctggccacgacgggcgttccttgcgcagc
tgtgctcgacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccggggcaggatctc
ctgtcatctcaccttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgcatacgc
ttgatccggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactcggatgga
agccggtcttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgcc
aggctcaaggcgagcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccgaata
tcatggtggaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccgctatca
ggacatagcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgaccgcttcctcgtg
ctttacggtatcgccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttcttctgaa
ttattaacgcttacaatttcctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcat
acaggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaata
tgtatccgctcatgagacaataaccctgataaatgcttcaataatagcacgtgctaaaacttcattttta
atttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcg
ttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaa
tctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaac
tctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtag
ttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtgg
ctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgca
gcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgaga
tacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaa
gcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcc
tgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatgg
aaaaacgccagcaacgcggcctttttacggttcctgggcttttgctggccttttgctcacatgttctt</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1532</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1532</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>unsure</INSDFeature_key>
<INSDFeature_location>53..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>map</INSDQualifier_name>
<INSDQualifier_value>Kozak sequence</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>gene</INSDFeature_key>
<INSDFeature_location>61..1296</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>gene</INSDQualifier_name>
<INSDQualifier_value>gene of artificial multi-epitope
immunogen</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>1306..1415</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>polyA_site</INSDFeature_key>
<INSDFeature_location>1422..1532</INSDFeature_location>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagaataaactagtattcttctggtccccacagactcagagagaacccg
cggccgccaccatggccaagttcgtggccgcctggaccctgaaggccgccgccaagaagaacatcgccga
ctacaactacaagctgcccgacgacttcaccggctgcgtgatcgcctggaacagcaacaacctggacagc
aaggtgggcggcaactacaactacctgtaccgcctgttccgcaagagcaacctgaagcccttcgagagca
tcatcgcctacaccatgagcctgggcgccgagaacagcgtggcctacagcaacaacagcatcgccatccc
caccaacttcaccatcggcctgaccgtgctgccccccctgctgaccgacgagatgatcgcccagtacacc
agcgccctgctggccggcaccatcaccagcggctggaccttcggcgccggcgccgcccacctgatgagct
tcccccagagcgccccccacggcgtggtgttcctgcacgtgacctacgtgcccgcccaggagaagaacct
gaacgagagcctgatcgacctgcaggagctgggcaagtacgagcagtacatcaagtggccctggtacatc
tggctgggcttcatcgccggcctgatcgccatcgtgatggtgaccatcatgagcctggtgaagcccagct
tctacgtgtacagccgcgtgaagaacctgaacagcagccgcgtgcccgacctgctggtgaccctggcctg
cttcgtgctggccgccgtgtaccgcatcaactggatcaccggcggcatcgccatcgccatggcctgcctg
gtgggcctgaaggagatcaccgtggccaccagccgcaccctgagctactacaagctgggcgccagccagc
gcgtggccggcgacagcggcttcgccgcctacttcccccgcggccagggcgtgcccatcaacaccaacag
cagccccgacgaccagatcggctactaccgccgcgccacccgccgcatccgcggcggcgacggcaagtgg
ccccagatcgcccagttcgcccccagcgccagcgccttcttcggcatgagccgcatcggcatggaggtga
cccccagcggcacctggctgacctacaccggcgccatcaagctggacgacaaggaccccaacttcaagga
ccaggtgatcctgctgaacaagcacatcgacgcctacaagaccttcccccccaccgagcccaagaaggac
aagaagaagaaggagcccttccgcgactacgtggaccgcttctacaagaccctgcgctaaggatccgctg
gagcctcggtggcctagcttcttgccccttgggcctccccccagcccctcctccccttcctgcacccgta
cccccgtggtctttgaataaagtctgagtgggcggcgaattcaaaaaaaaaaaaaaaaaaaaaaaaaaaa
aaagcatatgactaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa
aaaaaaaaaaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Искусственный ген, кодирующий белок-иммуноген BSI-COV-Ub, рекомбинантная плазмидная ДНК pBSI-COV-Ub, обеспечивающая экспрессию целевого гена, и искусственный полиэпитопный белок-иммуноген BSI-COV-Ub, содержащий убиквитин и эпитопы антигенов вируса SARS-CoV-2 и индуцирующий SARS-CoV-2-специфический Т-клеточный иммунитет | 2023 |
|
RU2806590C1 |
| Искусственный ген, кодирующий белок-иммуноген BSI-COV, рекомбинантная плазмидная ДНК pBSI-COV, обеспечивающая экспрессию целевого гена, и искусственный полиэпитопный белок-иммуноген BSI-COV, содержащий эпитопы антигенов вируса SARS-CoV-2 и индуцирующий SARS-CoV-2-специфический T-клеточный иммунитет | 2023 |
|
RU2806556C1 |
| Плазмидная ДНК-матрица pVAX-RBD, молекул мРНК-RBD, полученная с использованием ДНК-матрицы pVAX-RBD, обеспечивающая синтез и секрецию белка RBD SARS-CoV 2 в клетках организма млекопитающих и комплекс в виде наночастиц, содержащих молекулы мРНК-RBD, индуцирующих SARS-CoV-специфические антитела, обладающие вируснейтрализующей активностью | 2021 |
|
RU2754230C1 |
| Конъюгат белка рецепторсвязывающего домена (RBD) поверхностного гликопротеина S вируса SARS-CoV-2 с полимером полиглюкин-спермидин (PGS) и вакцинный комплекс против коронавирусной инфекции COVID-19 на основе указанного конъюгата и плазмидной ДНК pVAX-RBD | 2022 |
|
RU2781294C1 |
| Плазмидная ДНК-кассета pVAX-C3-PolyA, плазмидная ДНК-матрица pVAX-C3-RBD-PolyA для синтеза мРНК и полинуклеотидная вакцина против SARS-CoV-2, представляющая собой молекулы мРНК-C3-RBD-PolyA, индуцирующие SARS-CoV-2-специфические антитела | 2024 |
|
RU2839841C1 |
| Интегративный плазмидный вектор pVEAL3-RBDdel, обеспечивающий синтез и секрецию рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, рекомбинантный штамм клеточной линии CHO-K1- RBDdelta и рекомбинантный белок RBDdelta SARS-CoV-2, продуцируемый штаммом клеточной линии | 2023 |
|
RU2816175C1 |
| Искусственный ген, кодирующий бицистронную структуру, образованную последовательностями рецептор-связующего домена гликопротеина S коронавируса SARS-CoV-2, P2A-пептида и гликопротеина G VSV, рекомбинантная плазмида pStem-rVSV-Stbl_RBD_SC2, обеспечивающая экспрессию искусственного гена и рекомбинантный штамм вируса везикулярного стоматита rVSV-Stbl_RBD_SC2, используемого для создания вакцины против коронавируса SARS-CoV-2 | 2020 |
|
RU2733831C1 |
| СПОСОБ ПОЛУЧЕНИЯ БАКТЕРИАЛЬНОГО ПРОДУЦЕНТА РЕКОМБИНАНТНОГО БЕЛКА НУКЛЕОКАПСИДА NC ВИРУСА SARS-COV-2 | 2023 |
|
RU2812347C1 |
| Рекомбинантный штамм Sen-Sdelta(M) вируса Сендай, продуцирующий S белок SARS-CoV-2 и используемый для получения интраназальной вакцины против COVID-19 | 2024 |
|
RU2837536C1 |
| Искусственный ген EctoS_SC2, кодирующий эктодомен гликопротеина S коронавируса SARS-CoV-2 с C-концевым тримеризующим доменом, рекомбинантная плазмида pStem-rVSV-EctoS_SC2, обеспечивающая экспрессию искусственного гена, и рекомбинантный штамм вируса везикулярного стоматита rVSV-EctoS_SC2, используемый для создания вакцины против коронавируса SARS-CoV-2 | 2020 |
|
RU2733834C1 |



Изобретение относится к биотехнологии. Создана плазмидная матрица C1M-BSI, имеющая нуклеотидную последовательность SEQ ID NO: 1, размером 4387 п.о., молекулярную массу 2,8 МДа, содержащая целевой ген, кодирующий искусственный полиэпитопный белок BSI-COV, обеспечивающая синтез мРНК mRNA-BSI in vitro. Получена мРНК mRNA-BSI, имеющая нуклеотидную последовательность SEQ ID NO: 2, длиной 1532 п.о., кодирующая ген bsi-cov и имеющая 5'- и 3'-НТО, консенсусную последовательность Козак, 7-метилгуанозиновый кэп на 5'-конце и полиаденозиновый (полиА) хвост на 3'-конце. При введении лабораторным животным данная мРНК индуцирует формирование гуморального и клеточного иммунного ответа. Техническим результатом заявляемого изобретения является создание универсальной, безопасной и эффективной полиэпитопной мРНК-вакцины, способной индуцировать специфический цитотоксический и Т-хелперный ответ против различных штаммов вируса SARS-CoV-2. 2 н.п. ф-лы, 9 ил., 4 пр.
1. Плазмидная ДНК-матрица C1M-BSI, имеющая нуклеотидную последовательность SEQ ID NO: 1, размер 4387 п.о., молекулярную массу 2,8 МДа, содержащая целевой ген bsi-cov, кодирующий искусственный Т-клеточный иммуноген, перед которым находится промотор ДНК-зависимой РНК-полимеразы фага Т7, обеспечивающий синтез in vitro mRNA-BSI с помощью рекомбинантной РНК-полимеразы фага Т7, и состоящая из следующих фрагментов:
- векторный фрагмент плазмиды pVAX, имеющий координаты 2175-641 и размер 2855 п.о., содержащий промотор РНК-полимеразы фага Т7 с координатами 625-641 и размером 17 п.о., ген устойчивости к неомицину/канамицину (NeoR/KanR) с координатами 2614-3408 и размером 795 п.о., точку начала репликации Co1E1 ori с координатами 3734-4322 и размером 589 п.о., а также энхансер и промотор цитомегаловируса с координатами 36-619 и размером 584 п.о.;
- фрагмент, имеющий координаты 643-2174 и размер 1532 п.о., содержащий целевой искусственный ген bsi-cov с координатами 703-1938 и размером 1236 п.о., а также последовательность Козак, имеющая координаты 695-702 и размер 8 п.о., последовательности, кодирующие 5'-нетранслируемую область с координатами 643-692 и размером 50 п.о. и 3'-нетранслируемую область с координатами 1948-2057 и размером 110 п.о., полиаденозиновый (полиА) хвост, имеющий координаты 2064-2174 и размер 111 п.о.
2. Молекула mRNA-BSI, используемая для создания вакцины против коронавируса, имеющая нуклеотидную последовательность SEQ ID NO: 2 и длину 1532 п.о., полученная с использованием плазмидной ДНК-матрицы по п. 1 и обеспечивающая экспрессию искусственного гена bsi-cov и синтез в организме млекопитающих полиэпитопного белка-иммуногена, включающего консервативные эпитопы антигенов вируса SARS-CoV-2, индуцирующих SARS-CoV-2-специфический иммунитет, и содержащая, кроме целевого гена bsi-cov, консенсусную последовательность Козак, 7-метилгуанозиновый кэп на 5'-конце мРНК, полиаденозиновый (полиА) хвост на 3'-конце мРНК длиной 111 нуклеотидов, 5'- и 3'-нетранслируемые области (НТО), расположенные по обе стороны от рамки считывания.
| СПОСОБ ПОЛУЧЕНИЯ РЕКОМБИНАНТНОГО ИММУНОГЛОБУЛИНА IGA2m1-ИЗОТИПА В КЛЕТКАХ МЛЕКОПИТАЮЩИХ | 2022 |
|
RU2801178C1 |
| Искусственный ген N1new, кодирующий нуклеокапсидный белок коронавируса SARS-CoV-2, и рекомбинантная плазмида pET-28a-N1new, обеспечивающая экспрессию искусственного гена | 2021 |
|
RU2762962C1 |
| WO 2022036337 A1, 17.02.2022. | |||
Авторы
Даты
2025-04-30—Публикация
2024-11-14—Подача