Область техники
Настоящее изобретение относится к биотехнологии и молекулярной биологии, в частности к способу выделения тотальной ДНК бактерий из образцов почвы, к способу оценки содержания микроорганизмов в образцах, в частности, в образцах почвы, и изучения их разнообразия методом массового параллельного секвенирования библиотек ампликонов V3 и V4 вариабельного региона гена, кодирующего 16S субъединицу рибосомальной РНК, а также к наборам для осуществления указанных способов.
Уровень техники
Содержание и идентификация бактерий в образцах окружающей среды часто осуществляется с помощью методов, зависящих от культивирования бактерий. Однако далеко не все бактерии возможно культивировать в лабораторных условиях. Ввиду этого, метагеномный анализ является мощным инструментом среди современных молекулярных технологий нового поколения. Метагеномика применяет набор геномных технологий и инструментов биоинформатики для прямого доступа к генетическому содержанию целых сообществ организмов посредством так называемого «массового секвенирования» без необходимости выделения и лабораторного культивирования отдельных видов. Эта технология обычно используется для описания профилей микробных сообществ различных экосистем, таких как моря и почвы, для установления профилей микробных сообществ различных биологических образцов и для изучения микробных сообществ пищевых продуктов.
Задача определения видового состава сообщества решается с помощью секвенирования определённых генов, которые должны быть у всех организмов в сообществе. Некоторые участки таких последовательностей геномной ДНК, как например ген, кодирующий 16S рРНК, состоят из высококонсервативных последовательностей и гипервариабельных участков (McCabe K. M.и др. Bacterial species identification after DNA amplification with a universal primer pair. Molecular genetics and metabolism, 1999, т. 66, № 3, стр. 205-211, doi:10.1006/mgme.1998.2795). Эта особенность позволяет использовать праймеры для секвенирования, которые комплементарны консервативным участкам для получения последовательностей гипервариабельных участков. Полученные последовательности позволяют отнести организм к тому или иному виду (Tringe S.G., Hugenholtz P. A renaissance for the pioneering 16S rRNA gene. Current Opinion in Microbiology, 2008, т. 11, №. 5, стр. 442-446. doi:10.1016/j.mib.2008.09.011; Fadrosh D. W. И др. An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumina MiSeq platform. Microbiome, 2014, т. 2, статья 6, doi:10.1186/2049-2618-2-6).
Анализ бактериального состава почв представляет интерес, например, для определения биологического разнообразия бактериальных сообществ почв, для оценки загрязнённости почв по изменению состава таких сообществ, для обнаружения залежей углеводородного сырья по наличию метанотрофных бактерий и т.п.
Одним из важнейших этапов в проведении подобных исследований является выделение ДНК бактерий из образцов почв, поскольку от цельности, чистоты и количества выделенной ДНК зависит успешность проведения исследования.
Основной проблемой выделения ДНК бактерий из образцов почв является наличие почти во всех типах почв гуминовых кислот и фульвокислот, наличие даже незначительных количеств которых приводит к частичному или полному ингибированию ДНК-полимеразы и невозможности проведения реакции ПЦР и последующего массового параллельного секвенирования. В случае неполной очистки образца ДНК от гуминовых кислот и фульвокислот образец имеет окраску от светло- до тёмно-коричневого цвета и для достижения необходимой эффективности ПЦР требуется предварительно разводить образец выделенной ДНК. Это приводит не только к уменьшению концентрации ингибиторов, но также и к уменьшению концентрации ДНК исследуемого образца, что, в свою очередь, может приводить к невозможности амплификации целевых фрагментов исследуемой ДНК, необходимых для их последующего секвенирования.
Для решения этой проблемы обычно используют сложные, трудоёмкие и дорогие способы выделения и очистки ДНК.
Известны коммерческие наборы реактивов для выделения метагеномной почвенной ДНК. Выделение ДНК проводят в соответствии с протоколом производителей. Так, например, набор для выделения геномной ДНК из почв FastDNA Spin Kit For Soil (MP Biomedicals) (FastDNA Spin Kit For Soil, MP Biomedicals. https://www.dia-m.ru/lab/gomogenizatory-fastprep/acs/17086/) используют с гомогенизатором FastPrep или с альтернативным ему прибором. До 500 мг почвенной пробы помещают в 2 мл пробирки, содержащие лизирующую матрицу Е - смесь стеклянных (4 мм в диаметре), кварцевых (0,1 мм в диаметре) и керамических (1,4 мм в диаметре) шариков, способных разрушать все почвенные организмы. Гомогенизацию проводят в присутствии МТ буфера и фосфата натрия. По завершению лизиса образцы центрифугируют. Полученную ДНК очищают с использованием технологии Geneclean, включающей применение силикагелевых фильтров SPIN для устранения гуминовых кислот и полифенолов. Однако этот набор имеет высокую стоимость, примерно 400 долларов на 50 выделений, выход ДНК низкий и составляет порядка 1-5 мкг на 500 мг почвы. При этом, в документе не указана степень чистоты выделенной ДНК.
Также известен набор NucleoSpin® Soil (MACHEREY-NAGEL) (Genomic DNA from soil. User manual. NucleoSpin® Soil. November 2017 / Rev. 07. http://www.mnnet.com/Portals/8/attachments/Redakteure_Bio/Protocols/Genomic%20DNA/UM_gDNASoil.pdf), который предназначен для выделения тотальной ДНК из почвы. Набор включает два альтернативных лизирующих буфера и добавку Enhancer SX, которую можно комбинировать с обоими лизирующими буферами. Для количественного связывания ДНК используют оптимизированные колонки с диоксидом кремния - NucleoSpin® Soil Columns. NucleoSpin® Bead Tubes, содержащие керамические бусины, которые позволяют наиболее эффективно лизировать микроорганизмы в образце. Для удобного удаления загрязняющих веществ в набор включены колонки для удаления ингибиторов NucleoSpin® Inhibitor Removal Columns. Вначале материал образца ресуспендируют в буфере для лизиса SL1 или SL2, дополненном Enhancer SX и механически разрушают с использованием керамических бусин. Белки и ингибиторы ПЦР осаждают буфером для лизиса SL3, а затем осаждают путем центрифугирования вместе с керамическими шариками и образцом материала. Супернатант (надосадочную жидкость) отбирают и очищают, пропуская его через NucleoSpin® Inhibitor Removal Column колонку для удаления ингибиторов. Связывание ДНК регулируется добавлением буфера SB и лизат загружают в колонку NucleoSpin® Soil Column. Остаточные гуминовые вещества, особенно гуминовые кислоты и другие ингибиторы ПЦР, удаляют путем промывки буфером SB и промывочными буферами SW1, а затем SW2. После стадии высушивания готовая к использованию ДНК может быть элюирована буфером SE SEPLUS - 5 мМ Трис/HCl, рН 8,5. Данный набор имеет высокую стоимость, примерно 370 долларов на 50 выделений, и невысокий выход ДНК порядка 2-10 мкг из 0,5 г почвы. При этом, в документе также не указана степень чистоты выделенной ДНК.
Известен набор для выделения ДНК (RU 2650865, опубл. 17.04.2018), включающий лизирующий, промывочные и элюирующий буферные растворы. Лизирующий буферный раствор содержит гуанидин тиоцианат, Трис-гидрохлорид, тритон и сорбент в виде суспензии из магнитных микросфер в солевом растворе. Промывочный буфер №1 содержит гуанидин тиоцианат, Трис-гидрохлорид и этиловый спирт. Промывочный буфер №2 содержит Трис-гидрохлорид, хлорид натрия и этиловый спирт. Элюирующий раствор представляет собой деионизированную воду. Известный набор реактивов для выделения ДНК имеет небольшой выход ДНК - от 200 до 500-1000 нг из 5 г исходного материала.
Для облегчения выхода ДНК из клеток, кроме определенных реагентов, используют способы с применением физических факторов воздействия, например, ультразвука.
Например, в способе выделения ДНК из почвы (Bintrim S.B., Donohue T.J., Handelsman J., Roberts G.P., Goodman R.M. Molecular phylogeny of Archaea from soil // Proc. Natl. Acad. Sci. USA. - 1997. - V. 94 (1). - P. 277-282. https://doi.org/10.1073/pnas.94.L277) для облегчения выхода ДНК из клеток используют ультразвук. 500 миллиграммов почвы ресуспендируют в 500 мкл раствора А, содержащего 250 мМ NaCl и 100 мМ Na2ЭДТА (двунатриевая соль этилендиаминтетрауксусной кислоты), и обрабатывают ультразвуком в ультразвуковой ванночке Branson 2200 в течение 3 минут. Затем добавляют лизоцим до концентрации 0,5 мг/мл и смесь инкубируют при 37°C в течение 30 мин с периодическим перемешиванием. Далее добавляют протеиназу K до конечной концентрации 2,0 мг/мл и смесь инкубируют еще 30 мин. После инкубации добавляют 500 мкл раствора В, который содержит 250 мМ NaCl, 100 мМ Na2ЭДТА, 4% (вес/об.) додецилсульфата натрия (SDS), и 75 мкл 5 М гуанидин изотиоцианата и смесь осторожно перемешивают. Смесь инкубируют при 68°C в течение 1 часа при периодическом перемешивании. После инкубации образец смешивают в соотношении 1:1 с гранулами диоксида циркония/кремнезема диаметром 0,1 мм и гомогенизируют в Mini-Beadbeater (тип ВХ-4, Biospec Products, США) при 3000 об/мин в течение 45 секунд. Образцы центрифугируют, чтобы удалить гранулы, и отбирают надосадочную жидкость, в которую добавляют 150 микролитров раствора цетилтриметиламмоний бромида (СТАВ), концентрации 2% (вес./об.), 100 мМ ТрисHCl рН 8.0, 20 мМ Na2ЭДТА, 1.4 М NaCl, и перемешивают. Смесь инкубируют при 65°C в течение 15 мин с периодическим перемешиванием и затем последовательно экстрагируют равными объемами смеси хлороформ : изоамиловый спирт (24:1), фенол : хлороформ : изоамиловый спирт (24:24:1) и хлороформ : изоамиловый спирт (24:1). Равный объем изопропанола добавляют в супернатант, и тотальную ДНК извлекают центрифугированием. ДНК ресуспендируют в 500 мкл 10 мМ ТрисHCl (рН 8.0) и для амплификации ПЦР очищают 4-кратной ультрафильтрацией с использованием микроконцентраторов Microcon-100 (Amicon). Недостатком известного способа является трудоемкость, а использование ультразвука может вызвать повреждение структуры ДНК (Grokhovsky S.L. Specificity of DNA cleavage by ultrasound // Molecular Biology. - 2006. - V. 40 (2). - P. 276-283; Нечипуренко Ю.Д, Головкин M.B., Нечипуренко Д.Ю., Ильичева И.А., Панченко Л.А., Полозов Р.В., Гроховский С.Л. Характерные особенности расщепления ДНК ультразвуком // Журнал структурной химии. - 2009. - Т. 50, №5. - С. 1045-1052). При этом, в документе также не указана степень чистоты выделенной ДНК и её целостности.
Известен способ выделения ДНК, в котором образец почвы после смешивания с лизирующим раствором, содержащим NaCl, SDS, инкубируют при температуре 72°C в течение 45 мин для лизиса бактериальных клеток (Sagar K., Singh S., Goutam K.K., Konwar В.K. Assessment of five soil DNA extraction methods and a rapid laboratory-developed method for quality soil DNA extraction for 16S rDNA-based amplification and library construction. // J Microbiol. Methods. - 2014. - V. 97. - P. 68-73. doi: 10.1016/j.mimet.2013.11.008). Далее образец центрифугируют при ускорении 13000 g в течение 5 мин при 4°C и супернатант переносят в 2-мл центрифужную пробирку. 100 мкл 6 М ацетата калия и 400 мкл 50% полиэтиленгликоля (ПЭГ) вносят в надосадочную жидкость и смесь оставляют для осаждения на 20 мин при -20°C и затем центрифугируют при 4°C в течение 5 мин. Супернатант удаляют и осадок высушивают на воздухе. Далее его растворяют в 500 мкл ТЕ-буфера (Трис-ЭДТА буфер) рН 8.0, добавляют 500 мкл хлороформа и центрифугируют с ускорением 13000 g при 4°C в течение 5 мин. Экстракцию хлороформом повторяют дважды и в супернатант добавляют 500 мкл изопропанола. Затем оставляют для осаждения водной фракции ДНК на 5 мин при 4°C и снова центрифугируют при ускорении 13000 g в течение 5 мин. Осадок ДНК суспендируют в 100 мкл 1×ТЕ (10 мM Трис-HCl, 1 мМ ЭДТА (этилендиаминтетрауксусная кислота)). Недостатком метода является невысокий выход ДНК, который составляет 3.8 мкг/г почвы, что может быть обусловлено неэффективной процедурой клеточного лизиса. Также в документе не указана степень чистоты выделенной ДНК.
В другом способе выделения ДНК из почвы для клеточного лизиса используют порошок из стекла (Devi S.G., Fathima A.A., Radha S., Arunraj R., Curtis W.R., Ramya M. Rapid and economical method for efficient DNA extraction from diversesoils suitable for metagenomic applications // PLoS One. - 2015. - V. 10 (7):e0132441. doi: 10.1371/journal.pone.0132441). Для выделения ДНК 1 г образца почвы и 1 г стерильного порошка из измельченного лабораторного стекла помещают в стерильную ступку и растирают примерно в течение 5 мин. Для экстракции ДНК добавляют 1 мл буфера 100 мМ Трис, 100 мМ ЭДТА, 1.5 М NaCl (рН 8.0) и 10 мг порошкообразного активированного угля и перемешивают несколько раз посредством пипетирования. Переносят смесь в 2-миллилитровую пробирку. Инкубируют пробирку при 65°C в течение 10 минут на водяной бане, а затем центрифугируют при ускорении 12000 g в течение 5 минут при 4°C. Переносят 500 мкл супернатанта в новую микроцентрифужную пробирку объемом 2 мл. В полученную надосадочную жидкость добавляют 100 мкл ацетата натрия, рН 5.2, и 400 мкл 30% ПЭГ (MW-8000). Дают смеси остыть при -20°C в течение 20 минут в морозильной камере. Медленно оттаивают пробирки, а затем центрифугируют при 12000 g, в течение 5 минут при 4°C. Удаляют надосадочную жидкость и снова суспендируют осадок с 500 мкл ТЕ-буфера 10 мМ Трис, 1 мМ ЭДТА рН 8.0. Добавляют равный объем (500 мкл) смеси хлороформ : изоамиловый спирт, приготовленной в соотношении 24:1. Центрифугируют при 12000 g в течение 5 минут при 4°C. Переносят водную фазу в новую пробирку и добавляют 500 мкл ледяного изопропанола. Проводят осаждение в течение 5 мин при 4°C и центрифугируют при ускорении 12000 g в течение 10 мин при 4°C. Удаляют супернатант и промывают осадок 70% этанолом. Центрифугируют при 12000 g в течение 2 минут при 4°C. Удаляют супернатант, высушивают на воздухе осадок и растворяют в 100 мкл буфера ТЕ (рН 8.0). Недостатком метода также является невысокий выход ДНК, равный 5.48 мкг/г почвы. Также в документе нет указаний на степень чистоты выделенной ДНК.
Известен способ выделения ДНК из образцов почвы (Выделение ДНК из образцов почвы: Методические указания. Российская академия сельскохозяйственных наук. Всероссийский научно исследовательский институт сельскохозяйственной микробиологии. - СПб., 2011. - 27 с.), в котором в качестве основного детергента для выделения ДНК используют додецилсульфат натрия. К навеске 0.2 г замороженной почвы, помещенной в пробирку, добавляют количество стеклянных бусин, по объему примерно равное почве, и вносят раствор гуанидина - гуанидина изотиоцианат 240 мM, натрий-фосфатный буфер 200 мM, рН 7,0, и 350 мкл 1% раствора SDS - Трис HCl 500 мM, SDS 1% (вес/об.), рН 7,9, а также 400 мкл смеси фенол-хлороформ. Пробирку помещают во встряхиватель и гомогенизируют образец в течение 1-15 мин, в зависимости от мощности прибора (FastPrep 24-1 мин при максимальной мощности, Vortex Genie® 2-15 мин при максимальной скорости). Затем центрифугируют при ускорении 10-15×103 g при максимальной скорости в течение 5 мин. Водную фазу отбирают, добавляют 400 мкл хлороформа, центрифугируют также, как и на предыдущей стадии, и отбирают водную фазу. К неочищенному экстракту ДНК добавляют равный объем изопропилового спирта, центрифугируют на максимальной скорости в течение 5 мин, промывают 70% (об./об.) этанолом, слегка подсушивают на воздухе и растворяют осадок при 65°C в течение 5-10 мин в 100 мкл воды. Выход ДНК составляет 1750 нг из 0,2 г образца чернозема, что недостаточно для метагеномных исследований, например, для массового секвенирования. Также в документе нет указаний на степень чистоты выделенной ДНК.
Известен способ выделения ДНК из образцов почвы включающий гомогенизацию образца почвы путем вибрации в присутствии мелющих тел в растворе, содержащем гуанидин НCl, натрий-фосфатный буфер, Трис-HCl, додецилсульфат натрия (SDS), смесь фенол-хлороформ, с последующим центрифугированием и осаждением ДНК изопропанолом, в котором раствор для гомогенизации почвы дополнительно содержит детергент лаурилсаркозинат натрия при следующем соотношении исходных компонентов, вес.%: лаурилсаркозинат натрия - 2-6, додецилсульфат натрия - 1-4 (патент РФ №2696052, 30.07.2019). Гомогенизацию образца почвы осуществляют при помощи вибромельницы в диапазоне частот 25-35 Гц. В качестве мелющих тел используют смесь стеклянных бусин диаметром 0,5 мм и 1,0 мм и керамических бусин диаметром 2.0 мм в весовом соотношении 3:1:1 соответственно. Выход ДНК составляет порядка 10000 нг на 0,2 г чернозема. При этом, в документе нет указаний на степень чистоты выделенной ДНК и её целостности.
Описание настоящего изобретения
Задачей настоящего изобретения является создание простого, дешёвого и высокоэффективного способа выделения метагеномной ДНК бактерий почвы с высокой степенью очистки.
Указанный технический результат достигается тем, что способ выделения ДНК бактерий из почвы включает одновременный лизис клеток и гомогенизацию с использованием керамических шариков в режиме перемешивания со скоростью 1400 об./мин и термостатирования при 65°С в течение часа; центрифугирование полученной смеси и отделение надосадочной жидкости; выделение ДНК бактерий из надосадочной жидкости связыванием на кремниевом сорбенте, диоксиде кремния; последовательную промывку сорбента с ДНК, раствором №1, содержащим 100 мМ Трис-HCl, 240 мМ NaCl, 2 мМ ЭДТА, 0,2% Твин-20, 63% этанол в dd H2O, затем раствором №2, содержащим 50 мМ NaCl, 73,7% этанол в dd H2O; промывку 100% ацетоном; высушивание промытого сорбента при температуре 65°С до полного испарения ацетона и элюцию ДНК с кремниевого сорбента буфером, содержащим 1х TE.
Сочетание указанных выше стадий и компонентов в заявляемых концентрациях приводит к повышению эффективности выделения ДНК бактерий из почвы с высокой степенью чистоты. При этом степень чистоты выделенной ДНК является высокой и достаточной для осуществления способов оценки бактериального состава почв по существу стандартными методами без необходимости какой-либо существенной адаптации этих методов для решения конкретной задачи по оценке содержания микроорганизмов в образцах, в частности, в образцах почвы и изучения разнообразия микроорганизмов методом массового параллельного секвенирования библиотек. Осуществление способа оценки бактериального состава почв, также раскрытого в настоящем документе, посредством метагеномного секвенирования фрагмента V3-V4 гена, кодирующего субъединицу 16 рибосомальной РНК (16S рРНК), с использованием ДНК, выделенной указанным выше способом, служит подтверждением этого утверждения.
Способ выделения ДНК бактерий из почвы согласно осуществляют следующим образом.
Образцы почв, которые хранят и транспортируют в растворе 95% этанола с целью сохранения метагеномного состава при температуре -20°С, центрифугируют для отделения фракции почвы, и осуществляют одновременный лизис клеток и гомогенизацию с использованием керамических шариков в режиме перемешивания со скоростью 1400 об./мин и термостатирования при 65°С в течение часа. Затем полученную смесь центрифугируют и отделяют надосадочную жидкость. ДНК бактерий выделяют из полученной надосадочной жидкости осаждением на сорбенте, представляющем собой диоксид кремния. Затем осуществляют последовательную промывку сорбента со связанной ДНК, раствором №1, содержащим 100 мМ Трис-HCl, 240 мМ NaCl, 2 мМ ЭДТА, 0,2% Твин-20, 63% этанол в dd H2O, затем раствором №2, содержащим 50 мМ NaCl, 73,7% этанол в dd H2O, и, наконец, 100% ацетоном. Промытый сорбент высушивают при температуре 65°С до полного испарения ацетона. ДНК бактерий элюируют с кремниевого сорбента буфером, содержащим 10 мМ Трис-HCl, 1 мМ ЭДТА, рН 8,0. Полученный раствор содержит очищенную тотальную ДНК бактерий, пригодную для подготовки метагеномных библиотек.
Настоящее изобретение также предоставляет набор для выделения тотальной метагеномной ДНК бактерий из образцов почвы описанным выше способом. Указанный набор содержит керамические шарики для гомогенизации; лизирующий раствор, содержащий 500 мМ NaCl, 50 мМ ЭДТА, 2% поливинилпирролидон, 1,4% додецилсульфат натрия, 100 мМ ацетат натрия, рH=5,5; диоксид кремния; ДНК-осаждающий раствор, содержащий 2,5 М GuSCN, 20 мМ ЭДТА, 80 мМ Трис-HCl, 0,432 мМ ПЭГ (мол вес 6 кДа), 0,09% Тритон X-100, 18% этанол, 2,86 г/л трикарбоксиэтилфосфин в dd H2O, pH=7,2-7,3 (доведённый KOH); промывочный раствор №1, содержащий 100 мМ Трис-HCl, 240 мМ NaCl, 2 мМ ЭДТА, 0,2% Твин-20, 63% этанол в dd H2O; промывочный раствор №2, содержащий 50 мМ NaCl, 73,7% этанол в dd H2O; 100% ацетон; элюирующий буфер, содержащий 10 мМ Трис-HCl, 1 мМ ЭДТА, рН 8,0. В одном из вариантов настоящего изобретения, указанный набор также может дополнительно содержать водный раствор 95% этанола для хранения и транспортировки образцов почвы.
Также настоящее изобретение раскрывает способ оценки бактериального состава почв посредством метагеномного секвенирования фрагмента гена, кодирующего субъединицу 16 рибосомальной РНК (16S рРНК), включающий следующие стадии:
а) сбор образцов почвы;
б) выделение тотальной ДНК бактерий из образцов почвы описанным выше способом;
в) оценка качества и количества полученной бактериальной ДНК с помощью ПЦР в реальном времени;
г) проведение первой ПЦР для каждого из образцов тотальной ДНК, выделенных на стадии б), для введения адаптеров во фрагменты V3-V4 гена, кодирующего 16S рРНК;
д) очистка продукта ПЦР, полученного на стадии г), на магнитных частицах;
е) проведение второй ПЦР для каждого продукта ПЦР, выделенного на стадии д), с использованием пары праймеров, содержащих уникальные индексы для каждого конкретного образца;
ж) очистка продуктов ПЦР, полученных на стадии е), на магнитных частицах;
з) оценка длин фрагментов амплификации, полученных на стадии ж), с помощью электрофореза в агарозном геле;
и) измерение концентрации очищенных продуктов ПЦР, полученных на стадии ж), и смешивание всех указанных продуктов ПЦР в равной концентрации с получением геномной библиотеки;
к) полногеномное секвенирование геномной библиотеки, полученной на стадии и), с получением набора последовательностей нуклеотидов фрагмента V3-V4 гена, кодирующего 16S рРНК, для каждого из образцов тотальной ДНК бактерий;
л) классификация ДНК бактерий по известным таксонам и определение качественного и количественного состава бактерий почвы.
Настоящее изобретение также предоставляет комплект наборов для оценки бактериального состава почв посредством метагеномного секвенирования указанным выше способом. Комплект наборов содержит:
а) описанный выше набор для выделения бактерий из образцов почвы;
б) набор для оценки концентрации бактериальной ДНК методом ПЦР в реальном времени, содержащий:
- раствор праймеров SEQ ID NO: 1 и 2, 10 мкМ,
- буфер, содержащий 20% маннитол, 3 мМ МgCl2, 2% трегалоза, 1х краситель EVA Green, 1% NaN3, 500 мМ Трис HCl, 500 мМ KCl, 0,5% глицерин, 0,1% Твин-20, 20 мг/мл бычьего сывороточного альбумина (БСА) в dd H2O, рН=9,0;
- раствор термостабильной Taq ДНК-полимеразы, 5 ед/мкл,
- раствор дНТФ, 2,5 мМ;
- dd H2O.
в) набор для первой ПЦР, содержащий:
- раствор праймеров с адаптерами SEQ ID NO: 3 и 4, 5 мкМ;
- буфер, содержащий 60 мМ Tрис-HCl, 25 мМ KCl, 2 мМ MgCl2, 0,1% Тритон Х-100, рН 8,8;
- раствор ДНК-полимеразы высокоточной, 2 ед/мкл;
- раствор БСА, 20 мг/мл;
- раствор дНТФ, 2,5 мМ;
- dd H2O.
г) набор для второй ПЦР, содержащий:
- растворы праймеров с индексами SEQ ID NO: 5-48, 10 мкМ;
- буфер, содержащий 60 мМ Tрис-HCl, 25 мМ KCl, 2 мМ MgCl2, 0,1% Тритон Х-100, рН 8,8;
- раствор ДНК-полимеразы высокоточной, 2 ед/мкл;
- раствор БСА, 20 мг/мл;
- раствор дНТФ, 2,5 мМ;
- dd H2O.
д) набор для очистки продуктов амплификации на магнитных частицах, содержащий:
- магнитные частицы;
- промывочный буфер, представляющий собой 80% водный этиловый спирт;
-буфер для элюции ДНК, содержащий 10 мМ Tрис-HCl, рН 8,0.
Описание рисунков
На Фиг. 1 показан результат анализа 62 образцов тотальной ДНК, выделенной из почвы, методом ПЦР в режиме реального времени по каналу специфичной реакции карбоксифлуоресцеина (FAM). Интерфейс Bio-Rad CFX Manager; ось абсцисс - пороговый цикл (Цикл), ось ординат - относительные единицы флуоресценции (ОЕФ).
На Фиг. 2 показаны данные электрофоретической детекции метагеномных библиотек в агарозном геле. 1 и 17 - маркеры длин ДНК на 1 т.п.о.; 2 - очищенные на магнитных частицах ампликоны 2A после второй ПЦР с индексами; 3 и 12 - очищенные на магнитных частицах ампликоны 3A после второй ПЦР с индексами; 4 - очищенные на магнитных частицах ампликоны 5A1 после второй ПЦР с индексами; 5 - очищенные на магнитных частицах ампликоны 5AB после второй ПЦР с индексами; 6 - очищенные на магнитных частицах ампликоны 15А1 после второй ПЦР с индексами; 7 - очищенные на магнитных частицах ампликоны 6A1 после второй ПЦР с индексами; 8 - очищенные на магнитных частицах ампликоны 9A1 после второй ПЦР с индексами; 9 - очищенные на магнитных частицах ампликоны 2A2/AB после второй ПЦР с индексами; 10 - очищенные на магнитных частицах ампликоны 7A2 после второй ПЦР с индексами; 11 - очищенные на магнитных частицах ампликоны 16A1 после второй ПЦР с индексами; 12 - очищенные на магнитных частицах ампликоны 6В после второй ПЦР с индексами; 13 - очищенные на магнитных частицах ампликоны 13A1 после второй ПЦР с индексами; 14 - очищенные на магнитных частицах ампликоны 6A1 после первой ПЦР c адаптерами; 15 - очищенные на магнитных частицах ампликоны 5A1 после первой ПЦР c адаптерами; 16 - очищенные на магнитных частицах ампликоны 2A2/AB после первой ПЦР c адаптерами.
На Фиг. 3 показано количество прочтений 10 библиотек.
На Фиг. 4 показаны средние показатели качества запуска.
На Фиг. 5 показано распределение длин последовательностей.
На Фиг. 6 показан процент содержания адаптеров по FastQC.
На Фиг. 7 показана классификация прочтений по доменам.
На Фиг. 8 представлено распределение бактерий по филумам в каждом из исследованных образцов.
На Фиг. 9 представлено распределение прочтений между бактериальными филумами в образце 2А2/АВ (А), в образце 3А (Б), в образце 5А1 (В), в образце 5АВ (Г), в образце 7А2 (Д), в образце 9А1 (Е), в образце 13А1 (Ж), в образце 15А1 (З).
Далее приведены примеры вариантов осуществления настоящего изобретения. Указанные иллюстративные примеры приведены исключительно для целей разъяснения сути настоящего изобретения и не ограничивают каким-либо образом рамки настоящего изобретения, определяемые формулой настоящего изобретения.
Примеры
Пример 1. Выделение тотальной ДНК из образцов почвы
1) Транспортировка и хранение.
Образцы почв транспортировали под раствором 95% этанола с целью сохранения метагеномного состава в неизменном состоянии. Дальнейшее хранение образцов осуществляли при температуре -20°С.
2) Пробоподготовка.
Перед выделением осуществляли отбор фракции почвы из спиртового раствора в 1,5-мл пробирки, центрифугирование отобранных проб при 5000 об./мин в течение трех минут с дальнейшим удалением надосадочной жидкости. Осадок прогревали в термостате при 50°С в пробирках с открытыми крышками до полного высыхания образцов почвы и исчезновению запаха спирта.
3) Гомогенизация и лизис.
Для автоматической гомогенизации использовали ротационный гомогенизатор Precellys Evolution («BertinTechnologies», Франция). Навеску почвы массой 250-300 миллиграмм переносили в специализированные 2-мл пробирки для гомогенизации с предварительно внесенными керамическими шариками и в каждую пробирку добавляли по 1 мл лизирующего раствора следующего состава: 500 мМ NaCl, 50 мМ ЭДТА, 2% поливинилпирролидон, 1,4% додецилсульфат натрия, 100 мМ ацетат натрия, рH=5,5. Пробирки помещали в гомогенизатор и проводили запуск прибора по следующей программе: 8 000 об/мин в течение 15 сек, пауза 10 сек, 4 повтора. Полученный раствор термостатировать в режиме перемешивания со скоростью 1400 об/мин при 65°С в течение часа с последующим центрифугированием со скоростью 13000 об/мин в течение трех минут.
Для ручной гомогенизации использовали керамические ступки, в которые вносили навеску почвы, массой 400-500 мг с последующим перетиранием керамическим пестиком до получения однородной массы. Из полученной однородной массы почвы отбирали 250-300 мг, переносили в пробирки объемом 1,5 мл и в каждую пробирку добавляли 1 лизирующего раствора указанного выше состава. Полученный раствор термостатировали в режиме перемешивания со скоростью 1400 об/мин при 65°С в течение часа с последующим центрифугированием со скоростью 13000 об/мин в течение трех минут.
4) Осаждение ДНК на кремниевом сорбенте
В новые пробирки объемом 1,5 мл вносили по 500 мкл осаждающего раствора следующего состава: 2,5 М GuSCN, 20 мМ ЭДТА, 80 мМ Трис-HCl, 0,432 мМ ПЭГ (мол вес 6 кДа), 0,09% Тритон X-100, 18% этанол, 2,86 г/л трикарбоксиэтилфосфин в dd H2O, pH=7,2-7,3 (доведённый KOH). Туда же вносили 50 мкл раствора со взвесью сорбента диоксида кремния (Sigma-Aldrich, США, кат № S5631, 0,5-10 мкм (примерно 80% в диапазоне 1-5 мкм)), а также вносили 600 мкл надосадочной жидкости, полученной на предыдущем этапе после центрифугирования. Смесь перемешивали на вортексе в течение нескольких секунд и термостатировали с перемешиванием со скоростью 1400 об/мин в течение 5 минут при 65°С с последующим центрифугированием со скоростью 5000 об/мин в течение двух минут.
5) Промывка.
Надосадочную жидкость полностью удаляли, не затрагивая сорбент, осевший на дне пробирки. В каждую пробирку вносили по 500 мкл промывочного раствора №1, содержащего 100 мМ Трис-HCl, 240 мМ NaCl, 2 мМ ЭДТА, 0,2% Твин-20, 63% этанол в dd H2O. Пробирки с образцами тщательно перемешивали и центрифугировали на микроцентрифуге вортексного типа в течение минуты на максимальной скорости (7000 об/мин). Далее надосадочную жидкость полностью удаляли, не затрагивая сорбент. Аналогичные промывки производили раствором № 2, содержащим 50 мМ NaCl, 73,7% этанол в dd H2O, и 100% ацетоном. После промывки ацетоном пробирки помещали в термостат с открытыми крышками и высушивали осадок в течение 5-10 минут при температуре 65°С до полного испарения промывочного раствора.
6) Элюция.
К высушенному осадку добавляли 200 мкл элюирующего буфера следующего состава: 10 мМ Трис-HCl, 1 мМ ЭДТА, рН 8,0. Далее пробирки с образцами тщательно перемешивали и термостатировали при перемешивании со скоростью 1400 об/мин при 65°С в течение 10 минут. Затем центрифугировали пробирки при 13000 об/мин в течение трех минут. После чего 180 мкл надосадочной жидкости переносили в новые пробирки.
Полученная надосадочная жидкость содержит раствор очищенной тотальной ДНК, пригодной для подготовки метагеномных библиотек.
Пример 2. Оценка качества и количества полученной ДНК с помощью ПЦР в реальном времени (ПЦР-РВ)
Оценку качества и количества полученной ДНК осуществляли с помощью ПЦР в реальном времени (ПЦР-РВ). 2,5х буфер для ПЦР в реальном времени содержал 20% маннитол, 3 мМ МgCl2, 2% трегалоза, 1х краситель EVA Green (ООО «НПФ Синтол», РФ), 1% NaN3, 500 мМ Трис HCl, 500 мМ KCl, 0,5% глицерин, 0,1% Твин-20, 20 мг/мл бычьего сывороточного альбумина (БСА) в деионизированной воде (dd H2O), рН=9,0. В реакционную смесь добавляли праймеры F (SEQ ID NO: 1) и R (SEQ ID NO: 2) к региону V3-V4 гена 16S рибосомальной РНК (рРНК), 2,5 ед. термостабильной Taq ДНК-полимеразы (ООО «НПФ Синтол», РФ) и 5 мкл раствора выделенной ДНК. Состав реакционной смеси для ПЦР-РВ показан в Таблице 1.
Таблица 1. Реакционная смесь для ПЦР-РВ.
ПЦР-РВ осуществляли на амплификаторе «ДТпрайм» (ООО «ДНК-Технология», РФ). Амплификацию осуществляли по следующему протоколу: температура 95°С, 5 минут, число циклов - 1; затем температура 95°С, 15 секунд, температура 60°С, 20 секунд, температура 72°С, 30 секунд, число циклов - 50. Детекцию сигнала флуоресценции осуществляли при температуре 60°С.
Образцы, для которых значение порогового цикла (Сq) составило 30 или менее, могут быть использованы для дальнейшего анализа. Образцы со значением Сq более 30 требуют повторного выделения. Результат ПЦР-РВ анализа ДНК, выделенной из 62 образцов почвы, представлен на Фиг. 1 и в Таблице 2.
Таблица 2. Результаты ПЦР-РВ с 62 образцами тотальной ДНК, выделенной из почвы, с праймерами к региону V3-V4 ПЦР-РВ гена 16S рРНК.
Как видно из результатов ПЦР-РВ, все образцы тотальной ДНК, выделенной из почвы, содержали количество ДНК, достаточное для проведения реакции ПЦР, при этом чистота ДНК также была достаточно высокой для проведения по существу стандартной реакции ПЦР с высокоточной ДНК-полимеразой. Настоящие данные подтверждают высокую эффективность заявленного в настоящем изобретении способа выделения тотальной метагеномной ДНК бактерий из образцов почвы и высокую чистоту выделенной этим способом ДНК.
Пример 3. Введение адаптеров в ампликоны участка V3-V4 16S рРНК путем постановки первой ПЦР
Адаптеры в ампликоны участка V3-V4 16S рРНК вводили путем постановки первой ПЦР. 2,5х буфер для первой ПЦР содержал 60 мМ Tрис-HCl, 25 мМ KCl, 2 мМ MgCl2, 0,1% Тритон Х-100 в деионизированной воде (dd H2O), рН 8,8. В реакционную смесь добавляли праймеры FА (SEQ ID NO: 3) и RА (SEQ ID NO: 4) с адаптерами, 5 пмоль. Структура праймеров показана в Таблице 3. Также в реакционную смесь добавляли 0,22 мкл бычьего сывороточного альбумина (БСА), 20 мг/мл, 1 ед. высокоточной ДНК-полимеразы (HF-Fuzz ДНК полимераза, Диалат Лтд., РФ) и 2,5 мкл раствора выделенной ДНК. Состав реакционной смеси для первой ПЦР показан в Таблице 4.
Таблица 3. Прямой (FА) и обратный (RА) праймеры с адаптерами к региону V3-V4 гена 16S рРНК.
Таблица 4. Состав ПЦР смеси (25 мкл) для получения ампликонов с адаптерами.
Первую ПЦР осуществляли на амплификаторе «ДТклассик» (ООО «ДНК-Технология», РФ). Амплификацию осуществляли по следующему протоколу: температура 98°С, 1 минута, число циклов - 1; затем температура 98°С, 7 секунд, температура 55°С, 30 секунд, температура 72°С, 30 секунд, число циклов - 25; финальная стадия при температуре 72°С, 5 минут.
Пример 4. Очистка полученных ампликонов с адаптерами
Очистку полученных ампликонов с адаптерами осуществляли с использованием магнитных частиц “SynMag” (ООО «НПФ Синтол», РФ).
К ампликонам в стрипах на 0,2 мл добавляли 20 мкл предварительно тщательно перемешанных магнитных частиц (соотношение раствор ампликона : раствор магнитных частиц 1:0,8). Перемешивали, сбрасывали капли кратким центрифугированием. Инкубировали на столе 5 минут. Перемещали стрипы на магнитный планшет и инкубировали на нем 5 минут. Не задевая магнитные частицы, отбирали жидкость. Добавляли 200 мкл 80% водного этанола. Инкубировали 30 секунд. Не задевая магнитные частицы, полностью удаляли этанол. Повторяли промывку 80% водным этанолом еще один раз. Не задевая магнитные частицы, полностью удаляли этанол. Сушили пробирки с открытыми крышками в течение 10 минут. К магнитным частицам добавляли 52,5 мкл буфера для элюции ДНК, содержащего 10 мМ Tрис-HCl, рН 8,0. Перемешивали, сбрасывали капли кратким центрифугированием. Инкубировали на столе 2 минуты. Перемещали стрипы на магнитный планшет и инкубировали на нем 2 минуты. Не задевая магнитные частицы, отбирали 50 мкл очищенного ПЦР продукта и переносили в чистые стрипы.
Пример 5. Постановка второй ПЦР с использованием праймеров, содержащих индексы
Индексирование метагеномных библиотек осуществляли с помощью постановки второй ПЦР с полученными на предыдущем этапе очищенными ампликонами с адаптерами. На данном этапе использовали праймеры с включенными в них индексными последовательностями. Рекомендованные последовательности индексов представлены в Таблице 5.
Таблица 5.
Индексные последовательности 701-729 расположены между адапторным участком и локус-специфическим участком прямого праймера FА (SEQ ID NO: 3), а индексные последовательности 501-522 расположены между адапторным участком и локус-специфическим участком обратного праймера RА (SEQ ID NO: 4). При этом, для ДНК, выделенной из каждого образца, берётся уникальная пара прямого обратного праймера с индексными последовательностями.
2,5х буфер для второй ПЦР содержал 60 мМ Tрис-HCl, 25 мМ KCl, 2 мМ MgCl2, 0,1% Тритон Х-100 в деионизированной воде (dd H2O), рН 8,8. В реакционную смесь для каждого образца добавляли уникальную пару праймеров с индексами i7 и i5, 5 пмоль. Также в реакционную смесь добавляли 0,44 мкл бычьего сывороточного альбумина (БСА), 20 мг/мл, 2 ед. высокоточной ДНК-полимеразы (HF-Fuzz ДНК полимераза, Диалат Лтд., РФ) и 5 мкл раствора выделенной ДНК. Состав реакционной смеси для второй ПЦР показан в Таблице 6.
Таблица 6. Состав ПЦР смеси (50 мкл) для получения ампликонов с индексами.
Вторую ПЦР осуществляли на амплификаторе «ДТклассик» (ООО «ДНК-Технология», РФ). Амплификацию осуществляли по следующему протоколу: температура 98°С, 1 минута, число циклов - 1; затем температура 98°С, 7 секунд, температура 55°С, 30 секунд, температура 72°С, 30 секунд, число циклов - 8; финальная стадия при температуре 72°С, 5 минут. Ожидаемая длина полученного фрагмента с учетом длин индексов 550 п.н.
Очистку полученных ампликонов с адаптерами осуществляли как описано в Примере 4 за исключением того, что вначале к ампликонам в стрипах на 0,2 мл добавляли 56 мкл предварительно тщательно перемешанных магнитных частиц (соотношение раствор ампликона : раствор магнитных частиц 1:1,1), а для элюции ДНК после промывки добавляли 27,5 мкл буфера для элюции и отбирали 25 мкл очищенного ПЦР продукта.
Пример 6. Проведение оценки длин фрагментов амплификации с помощью электрофореза в агарозном геле.
Оценку длин фрагментов полученных библиотек проводить методом электрофореза в 1% агарозном геле с добавлением этидиума бромида. После первой ПЦР на электрофорез брали 7 мкл образца, смешанного с 5 мкл красителя бромфенолового синего, после второй ПЦР 2 мкл библиотек смешивали с 5 мкл красителя.
Результаты анализа показаны на Фиг. 2. Как видно из приведённой картины геля, длины полученных фрагментов с учетом длин индексов соответствовали ожидаемой длине продуктов второй ПЦР.
Пример 7. Измерение концентрации готовых очищенных продуктов второй ПЦР и расчет молярных концентраций.
Концентрации готовых очищенных продуктов второй ПЦР измеряли следующим образом. Готовили смесь для измерения на N образцов (где N-количество образцов) из расчета 200 мкл буфера для измерения из набора СинКвант HS ДНК (СинКвант ДНК HS буфер 1-кратный, ООО «НПФ Синтол», РФ) и 1 мкл красителя (PicoGreen488) на один образец. Смесь готовили на N+2 количеств измерений. В пробирки на 0,5 мл вносили по 199 мкл смеси для измерения и по 1 мкл образца. Также допустимо соотношение 198 мкл смеси и 2 мкл образца, 195 мкл смеси и 5 мкл образца. Для калибровки в 2 дополнительные пробирки на 0,5 мл вносили 190 мкл смеси, а также 10 мкл стандарта 1 (СинКвант ДНК HS стандарт ДНК в TE в буфере 0 нг/мкл, ООО «НПФ Синтол», РФ) в первую пробирку и 10 мкл стандарта 2 (СинКвант ДНК HS стандарт ДНК в TE в буфере 10 нг/мкл, ООО «НПФ Синтол», РФ) во вторую пробирку. Смеси перемешивали и убирали в темное место на 5 минут. Калибровали флуориметр Qubix (Айвок, РФ) по указанным стандартам в соответствии с рекомендациями производителя (ООО «НПФ Синтол», Россия), измеряли концентрации образцов. Полученные концентрации в нг/мкл переводили в моль/л. Полученные результаты по оценке концентрации продуктов второй ПЦР с их переводом в молярные концентрации для нескольких образцов представлены в Таблице 7.
Таблица 7. Итоговые концентрации (C) ряда продуктов второй ПЦР с переводом в молярные концентрации.
Пример 8. Сборка итоговых метагеномных библиотек и их массовое параллельное секвенирование
Готовили пул библиотек с концентрацией 4 нМ, соблюдая равное соотношение между концентрациями всех библиотек в пуле. Расчеты для разведения каждой библиотеки до загрузочной концентрации и смешивания пула удобно производить на специализированных сайтах (https://support.illumina.com/help/pooling-calculator/pooling-calculator.htm).
Готовили пул библиотек с концентрацией 2 нМ. Для этого 5 мкл пула с концентрацией 4 нМ смешивали с 5 мкл NaOH (0,2 М), перемешивали и инкубировали 5 минут.
Готовили контрольную библиотеку PhiX с концентрацией 2 нМ. Для этого смешивали 2 мкл библиотеки PhiX (10 нМ, Illumina, Inc., США) с 3 мкл ddH2O и 5 мкл NaOH (0,2 М).
Готовили пул библиотек с концентрацией 20 пМ. Для этого к 10 мкл пула библиотек с концентрацией 2 нМ добавляли 990 мкл гибридизационного буфера HT1 (Illumina, Inc., США).
Готовили контрольную библиотеку PhiX с концентрацией 20 пМ. Для этого к 10 мкл библиотеки PhiX с концентрацией 2 нМ добавляли 990 мкл гибридизационного буфера HT1.
Готовили пул библиотек с концентрацией 8 пМ. Для этого в пробирке смешивали 360 мкл гибридизационного буфера HT1 и 240 мкл пула библиотек с концентрацией 20 пМ.
Готовили контрольную библиотеку PhiX с концентрацией 8 пМ. Для этого в пробирке смешивали 360 мкл гибридизационного буфера HT1 и 240 мкл библиотеки PhiX с концентрацией 20 пМ.
В чистую пробирку вносили 480 мкл пула библиотек с концентрацией 8 пМ и 120 мкл библиотеки PhiX с концентрацией 8 пМ (в соотношении пул исследуемых библиотек: контрольная библиотека 4:1).
Метагеномную библиотеку анализировали на полногеномном секвенаторе (MiSeq, Illumina, Inc., США). В ходе работы была произведена серия запусков, в процессе которых использовали картридж MiSeq Reagent Nano Kit v2 (500 циклов) (Illumina, Inc., США) с реактивами, рассчитанными на прочтения 2х250. В один запуск оптимально брать 10-12 образцов для получения в среднем около 70000-90000 прочтений на образец.
Пример 8. Результаты массового параллельного секвенирования и их интерпретация
В анализ вошел пул из 10 метагеномных библиотек, приготовленных согласно описанной выше технологии. По оценке Q30 прочтения стабильно хорошие. Не замечено N-последовательностей. Статистика по длине прочтений и оценке качества представлена на Фиг. 3 - 6. В ходе метагеномного анализа 98-99% прочтений в каждом образце были классифицированы как бактериальные (Фиг. 7). На Фиг. 8 представлены распределения бактерий по филумам в каждом из исследованных образцов. На Фиг. 9 (А)-(З) показаны распределения прочтений между бактериальными филумами в различных образцах.
Хотя настоящее изобретение описано в деталях выше, для специалиста в данной области техники очевидно, что могут быть сделаны изменения и произведены эквивалентные замены, и такие изменения и замены не выходят за рамки настоящего изобретения, которые определяются приложенной формулой изобретения.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Soil bacteria
DNAs.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-07-04">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>Soil bacterial DNA</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Федеральный исследовательский центр
«Институт биологии южных морей имени А.О. Ковалевского РАН» (ФИЦ
ИнБЮМ) </ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Scientific Institution
Federal Research Center "A.O. Kovalevsky Institute of Biology of
the Southern Seas of the Russian Academy of Sciences" (FRC
InBYuM)</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ВЫДЕЛЕНИЯ ТОТАЛЬНОЙ ДНК
БАКТЕРИЙ ИЗ ОБРАЗЦОВ ПОЧВЫ, СПОСОБ ОЦЕНКИ БАКТЕРИАЛЬНОГО СОСТАВА ПОЧВ
ПОСРЕДСТВОМ МЕТАГЕНОМНОГО СЕКВЕНИРОВАНИЯ И НАБОРЫ ДЛЯ ОСУЩЕСТВЛЕНИЯ
СПОСОБОВ</InventionTitle>
<SequenceTotalQuantity>48</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctacgggnggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactachvgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagcctacgggnggcwgca
g</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>55</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..55</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacaggactachvgggtatc
taatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtcgccttacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagctagtacgcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagttctgcctcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggctcaggacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagaggagtcccctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagcatgcctacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggtagagagcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagcctctctgcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagagcgtagccctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagcagcctcgcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtgcctcttcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtcctctaccctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtcatgagccctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagcctgagatcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtagcgagtcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggtagctcccctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtactacgccctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q49">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagaggctccgcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggcagcgtacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagctgcgcatcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q55">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggagcgctacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q57">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagcgctcagtcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q59">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggtcttaggcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q61">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagactgatcgcctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q63">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacagtagctgcacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q65">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcggcagcgtcagatgtgtataagagacaggacgtcgacctacggg
nggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q67">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagtagatcgcgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q69">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagctctctatgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q71">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagtatcctctgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q73">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagagagtagagactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q75">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacaggtaaggaggactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q77">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagactgcatagactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q79">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagaaggagtagactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q81">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagctaagcctgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q83">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagcgtctaatgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q85">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagtctctccggactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q87">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagtcgactaggactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q89">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagttctagctgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q91">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagcctagagtgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q93">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacaggcgtaagagactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q95">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagctattaaggactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q97">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagaaggctatgactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q99">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacaggagccttagactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q101">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctcgtgggctcggagatgtgtataagagacagttatgcgagactach
vgggtatctaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799413C1 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |
| Наборы олигонуклеотидов для выявления ДНК бактерии Helicobacter pylori в клиническом материале методом полимеразной цепной реакции в режиме реального времени | 2024 |
|
RU2839157C1 |
| Панель праймеров для полногеномного секвенирования вирусов парагриппа человека 1 и 2 типа | 2024 |
|
RU2833687C1 |
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ГЕНОТИПИРОВАНИЯ ГРИБОВ ВИДОВОГО КОМПЛЕКСА COLLETOTRICHUM ACUTATUM | 2022 |
|
RU2807352C1 |
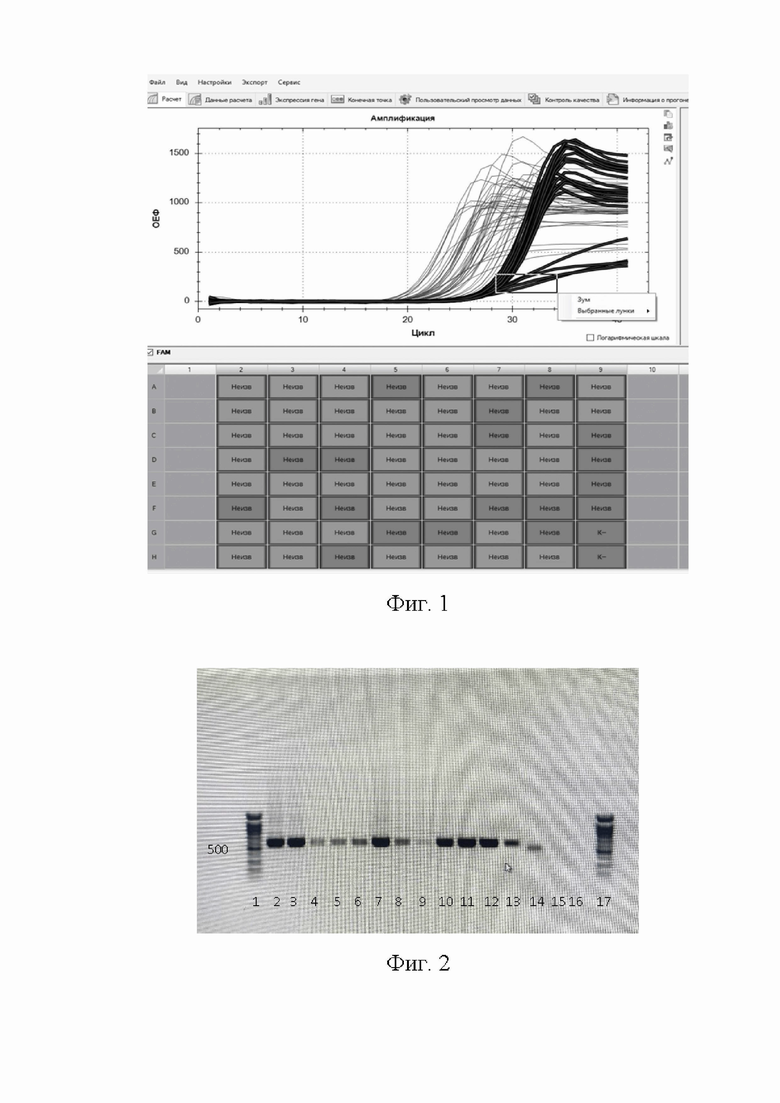
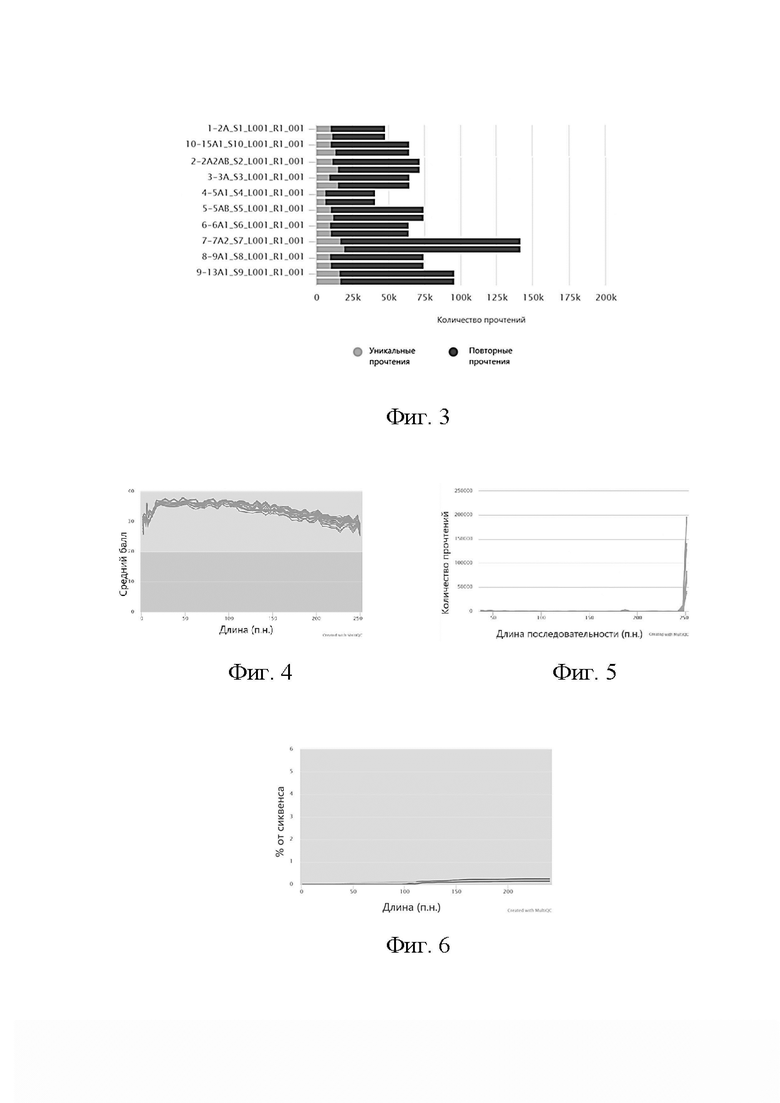
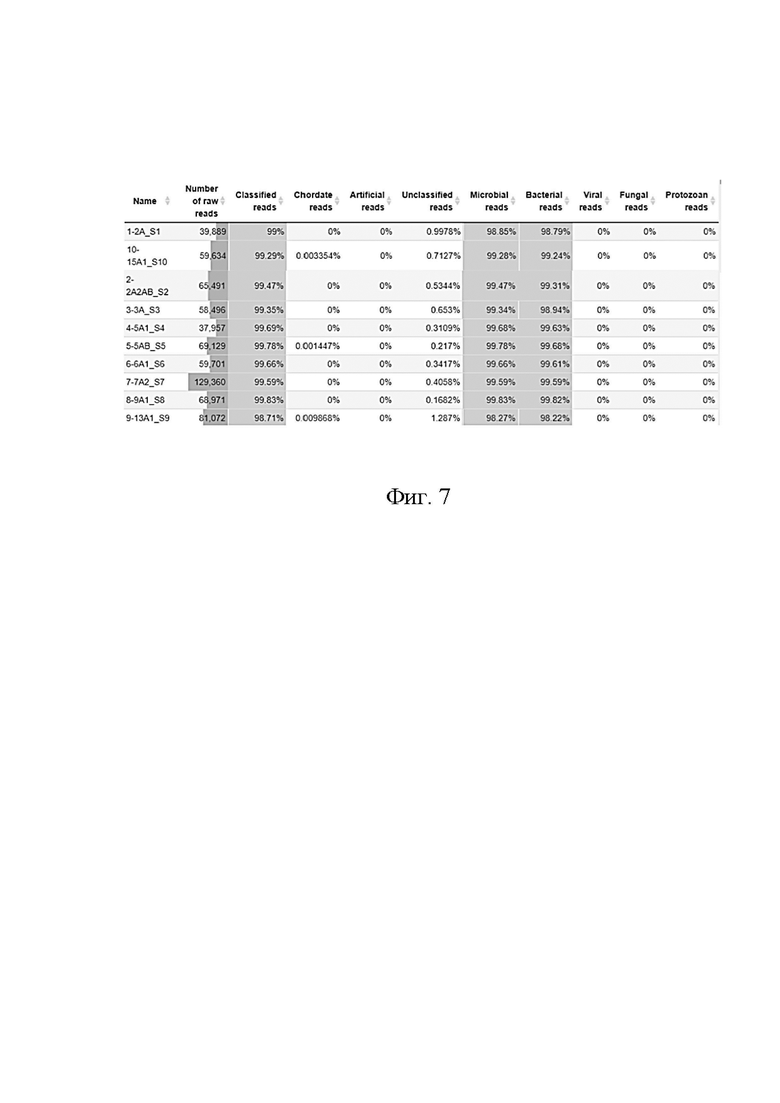
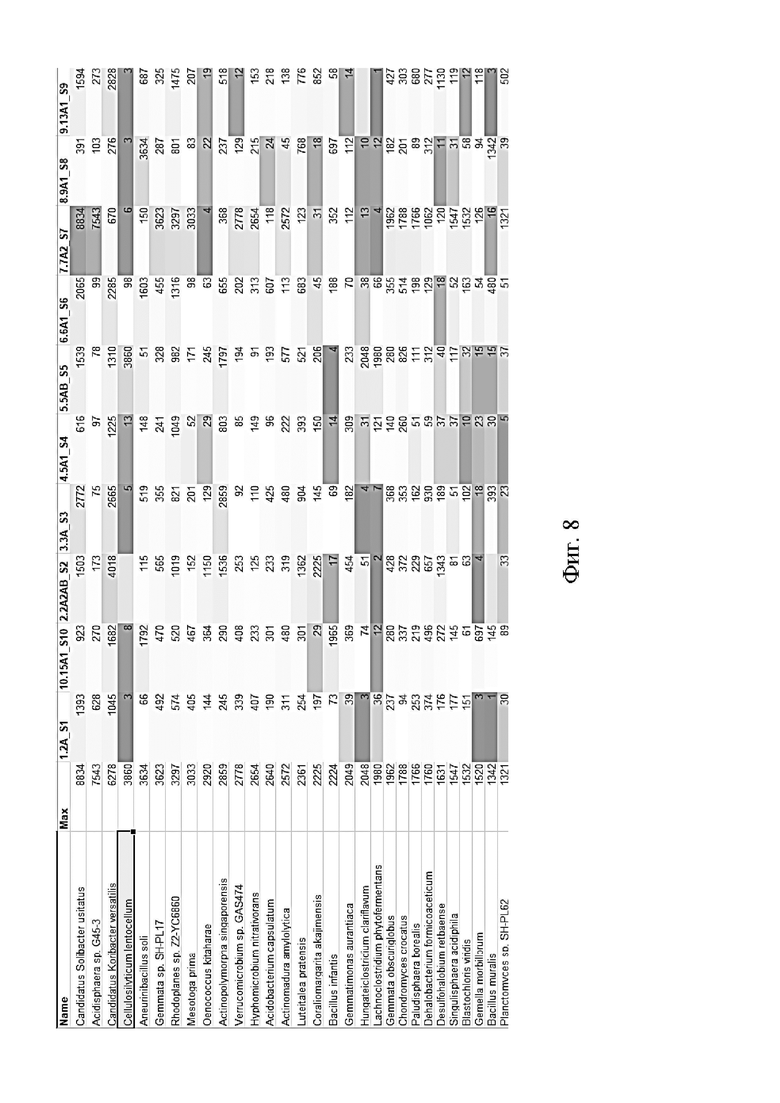
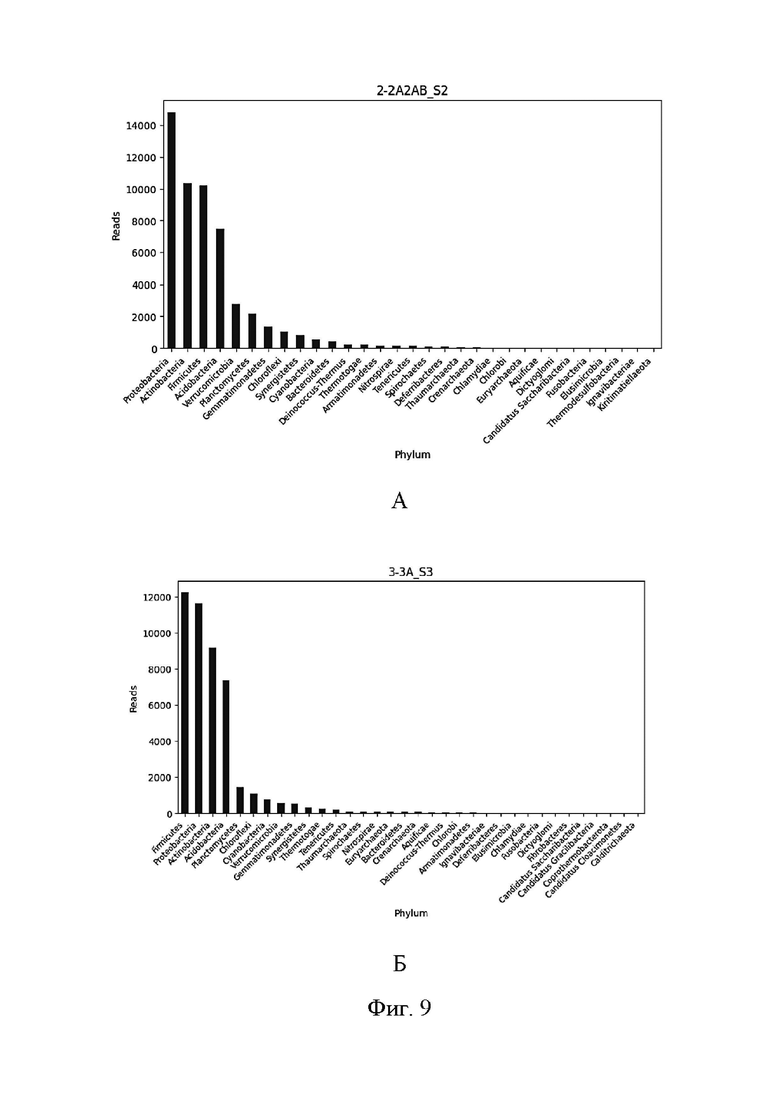
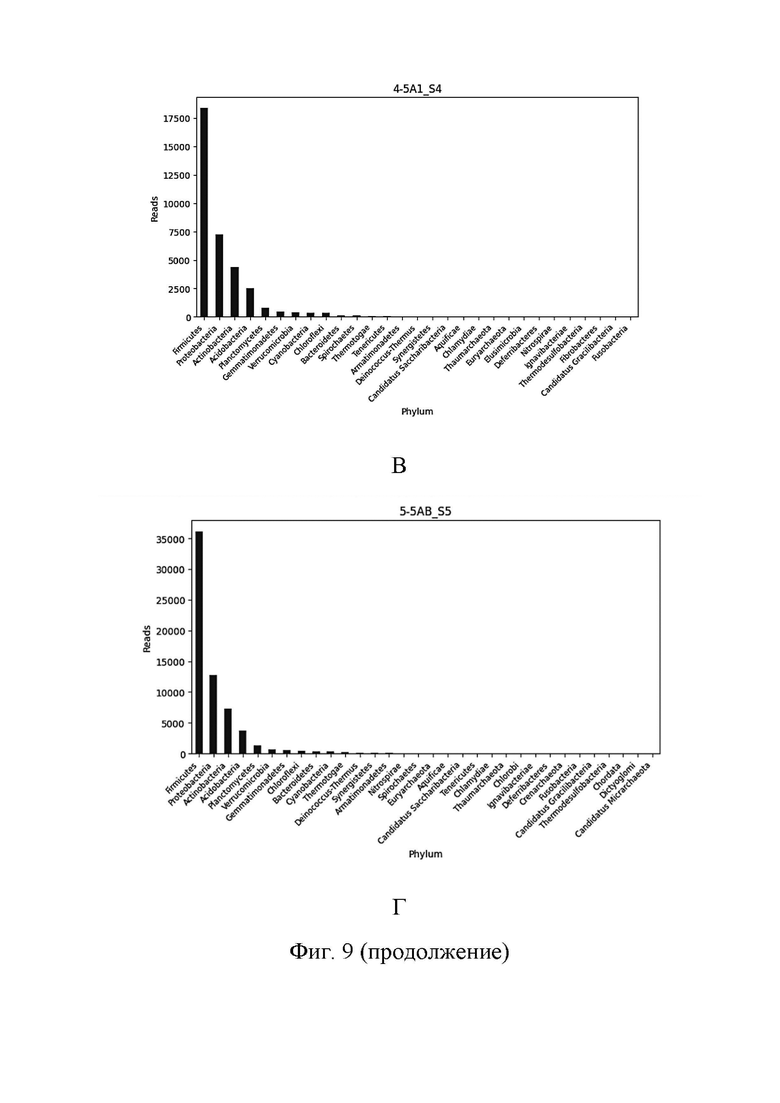
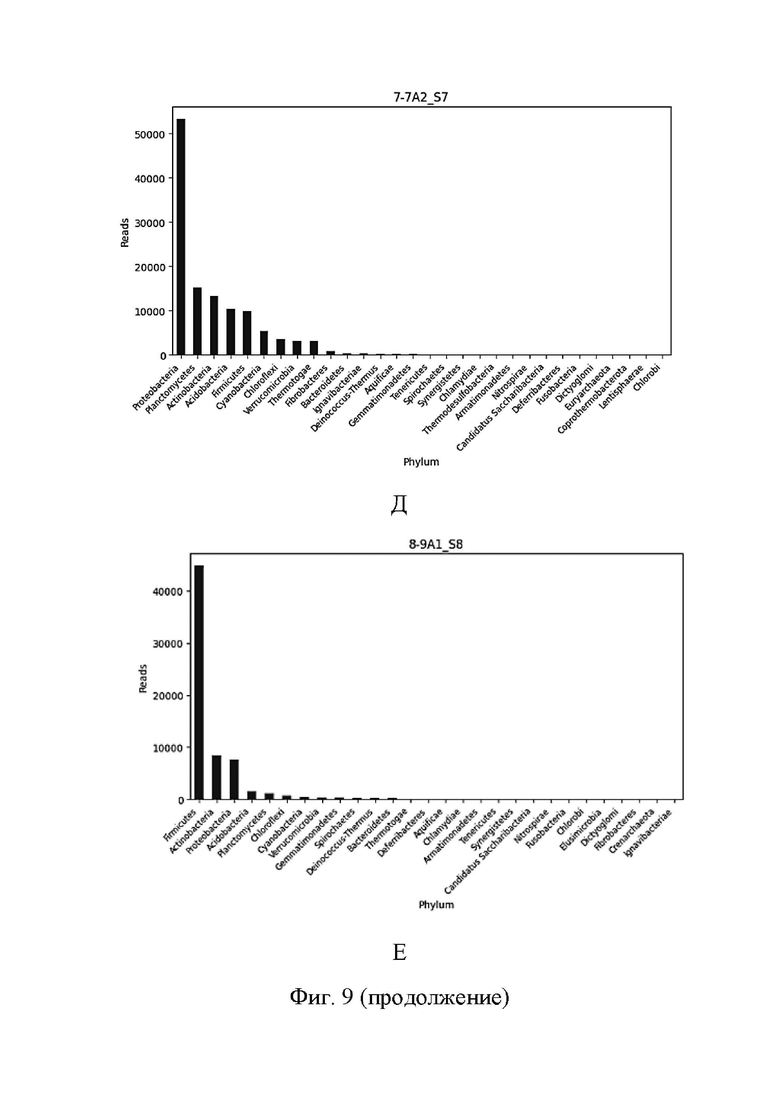
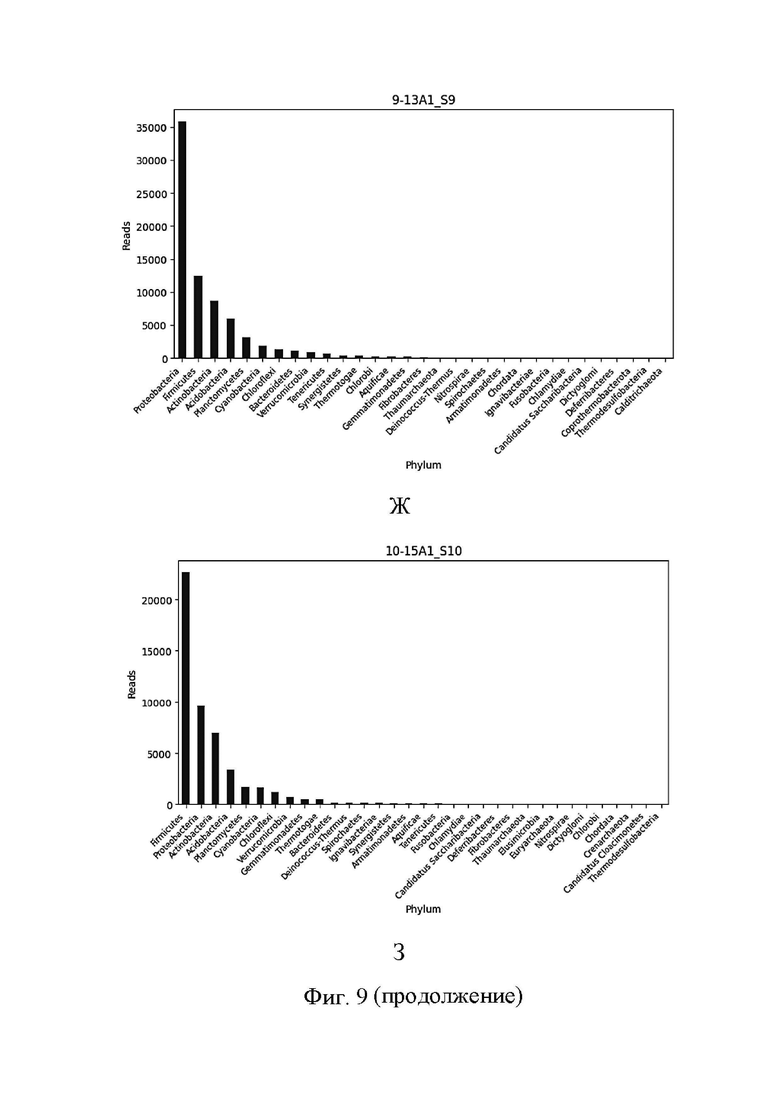
Изобретение относится к области биотехнологии, в частности к способу выделения тотальной метагеномной ДНК бактерий из образцов почвы. Указанный способ включает: сбор образцов почвы, одновременный лизис бактериальных клеток и гомогенизацию с использованием лизирующего раствора, центрифугирование указанной смеси с последующим осаждением ДНК бактерий из надосадочной жидкости, последовательную промывку сорбента с ДНК, высушивание промытого сорбента и элюцию ДНК. Изобретение также относится к набору для осуществления указанного способа, а также способу оценки бактериального состава почв и комплекту наборов, позволяющему осуществлять такую оценку. Настоящее изобретение обеспечивает высокоэффективный способ выделения метагеномной ДНК бактерий почвы с высокой степенью очистки. 4 н. и 1 з.п. ф-лы, 9 ил., 7 табл., 8 пр.
1. Способ выделения тотальной метагеномной ДНК бактерий из образцов почвы, включающий:
а) сбор образцов почвы и их обработку 95% водным раствором этанола;
б) одновременный лизис клеток бактерий из образцов почвы и гомогенизацию с использованием лизирующего раствора, содержащего 500 мМ NaCl, 50 мМ ЭДТА, 2% поливинилпирролидон, 1,4% додецилсульфат натрия, 100 мМ ацетат натрия, рH=5,5, и керамических шариков в режиме перемешивания со скоростью 1400 об/мин и термостатирования при 65°С в течение часа;
в) центрифугирование смеси, полученной на стадии б), и отделение надосадочной жидкости;
г) осаждение ДНК бактерий из надосадочной жидкости, полученной на стадии в), на сорбенте, представляющем собой диоксид кремния с использованием осаждающего раствора, содержащего 2,5 М GuSCN, 20 мМ ЭДТА, 80 мМ Трис-HCl, 0,432 мМ ПЭГ с мол. вес. 6 кДа, 0,09% Тритон X-100, 18% этанол, 2,86 г/л трикарбоксиэтилфосфин в dd H2O, pH=7,2-7,3, доведённый KOH;
д) последовательную промывку сорбента с ДНК раствором № 1, содержащим 100 мМ Трис-HCl, 240 мМ NaCl, 2 мМ ЭДТА, 0,2% Твин-20, 63% этанол в dd H2O, затем раствором № 2, содержащим 50 мМ NaCl, 73,7% этанол в dd H2O; промывку 100% ацетоном;
е) высушивание промытого сорбента при температуре 65°С до полного испарения ацетона;
ж) элюцию ДНК с кремниевого сорбента буфером, содержащим 10 мМ Трис-HCl, 1 мМ ЭДТА, рН 8,0.
2. Набор для выделения тотальной метагеномной ДНК бактерий из образцов почвы способом по п. 1, содержащий:
- керамические шарики для гомогенизации;
- лизирующий раствор, содержащий 500 мМ NaCl, 50 мМ ЭДТА, 2% поливинилпирролидон, 1,4% додецилсульфат натрия, 100 мМ ацетат натрия, рH=5,5;
- диоксид кремния;
- осаждающий раствор, содержащий 2,5 М GuSCN, 20 мМ ЭДТА, 80 мМ Трис-HCl, 0,432 мМ ПЭГ с мол. вес. 6 кДа, 0,09% Тритон X-100, 18% этанол, 2,86 г/л трикарбоксиэтилфосфин в dd H2O, pH=7,2-7,3 доведённый KOH;
- промывочный раствор № 1, содержащий 100 мМ Трис-HCl, 240 мМ NaCl, 2 мМ ЭДТА, 0,2% Твин-20, 63% этанол в dd H2O;
- промывочный раствор № 2, содержащий 50 мМ NaCl, 73,7% этанол в dd H2O;
- 100% ацетон;
- элюирующий буфер, содержащий 10 мМ Трис-HCl, 1 мМ ЭДТА, рН 8,0.
3. Набор по п. 2, дополнительно содержащий водный раствор 95% этанола.
4. Способ оценки бактериального состава почв посредством метагеномного секвенирования фрагмента гена, кодирующего субъединицу 16 рибосомальной РНК (16S рРНК), включающий следующие стадии:
а) выделение тотальной ДНК бактерий из образцов почвы способом по п. 1;
б) оценка качества и количества полученной бактериальной ДНК с помощью ПЦР в реальном времени с использованием праймеров SEQ ID NO: 1 и 2;
в) проведение первой ПЦР для каждого из образцов тотальной ДНК, выделенных на стадии а), для введения адаптеров во фрагменты V3-V4 гена, кодирующего 16S рРНК, с использованием праймеров SEQ ID NO: 3 и 4;
г) очистка продукта ПЦР, полученного на стадии в), на магнитных частицах;
д) проведение второй ПЦР для каждого продукта ПЦР, выделенного на стадии г), с использованием для каждого конкретного продукта ПЦР, выделенного на стадии г), уникальной пары праймеров, содержащих индексы, где один праймер из такой пары выбран из праймеров SEQ ID NO: 5-30, а второй праймер выбран из праймеров SEQ ID NO: 31-48;
е) очистка продуктов ПЦР, полученных на стадии д), на магнитных частицах;
ж) оценка длин фрагментов амплификации, полученных на стадии е), с помощью электрофореза в агарозном геле;
з) измерение концентрации очищенных продуктов ПЦР, полученных на стадии е), и смешивание всех указанных продуктов ПЦР в равной концентрации с получением геномной библиотеки;
и) полногеномное секвенирование геномной библиотеки, полученной на стадии з), с получением набора последовательностей нуклеотидов фрагмента V3-V4 гена, кодирующего 16S рРНК, для каждого из образцов тотальной ДНК бактерий;
к) классификация ДНК бактерий по известным таксонам и определение качественного и количественного состава бактерий почвы.
5. Комплект наборов для оценки бактериального состава почв посредством метагеномного секвенирования способом по п. 4, включающий в себя:
а) набор по п. 2 для выделения бактерий из образцов почвы;
б) набор для оценки концентрации бактериальной ДНК методом ПЦР в реальном времени, содержащий:
- раствор праймеров SEQ ID NO: 1 и 2, 10 мкМ,
- буфер, содержащий 20% маннитол, 3 мМ МgCl2, 2% трегалоза, 1х краситель EVA Green, 1% NaN3, 500 мМ Трис HCl, 500 мМ KCl, 0,5% глицерин, 0,1% Твин-20, 20 мг/мл бычьего сывороточного альбумина (БСА) в dd H2O, рН=9,0;
- раствор Taq ДНК-полимеразы, 5 ед./мкл;
- раствор дНТФ, 2,5 мМ;
- dd H2O,
в) набор для первой ПЦР, содержащий:
- раствор праймеров с адаптерами SEQ ID NO: 3 и 4, 5 мкМ;
- буфер, содержащий 60 мМ Tрис-HCl, 25 мМ KCl, 2 мМ MgCl2, 0,1% Тритон Х-100, рН 8,8;
- раствор ДНК-полимеразы высокоточной, 2 ед./мкл;
- раствор БСА, 20 мг/мл;
- раствор дНТФ, 2,5 мМ;
- dd H2O,
г) набор для второй ПЦР, содержащий:
- растворы праймеров с индексами SEQ ID NO: 5-48, каждый 10 мкМ;
- буфер, содержащий 60 мМ Tрис-HCl, 25 мМ KCl, 2 мМ MgCl2, 0,1% Тритон Х-100, рН 8,8;
- раствор ДНК-полимеразы высокоточной, 2 ед./мкл;
- раствор БСА, 20 мг/мл;
- раствор дНТФ, 2,5 мМ;
- dd H2O,
д) набор для очистки продуктов амплификации на магнитных частицах, содержащий:
- магнитные частицы;
- промывочный буфер, представляющий собой 80% водный этиловый спирт;
- буфер для элюции ДНК, содержащий 10 мМ Tрис-HCl, рН 8,0.
| Способ выделения ДНК из почвы | 2018 |
|
RU2696052C1 |
| Универсальный способ выделения ДНК и лизирующая смесь для его осуществления | 2022 |
|
RU2807254C1 |
| ZIELIŃSKA, S | |||
| et al., The choice of the DNA extraction method may influence the outcome of the soil microbial community structure analysis, MicrobiologyOpen, 2017, 6 (4), e00453 | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| ПРОБИРКА ДЛЯ УДАРНОЙ ОБРАБОТКИ ШАРИКАМИ И СПОСОБ ЭКСТРАКЦИИ ДЕЗОРИБОНУКЛЕИНОВОЙ КИСЛОТЫ И/ИЛИ РИБОНУКЛЕИНОВОЙ КИСЛОТЫ ИЗ МИКРООРГАНИЗМОВ | 2017 |
|
RU2743140C2 |
| VERMA, S | |||
| K | |||
| et al., An improved method suitable for | |||
Авторы
Даты
2024-11-05—Публикация
2024-07-05—Подача