Область техники, к которой относится изобретение
Изобретение, помимо прочего, относится к новому вирусу рыб, указанному как коронавирус, который является причиной гибели рыбы, а также к способам обнаружения указанного вируса у рыбы и защиты рыбы от заражения указанным вирусом, и к связанным с этим средствам (реагентам) и применениям.
Уровень техники
Рыба является основным источником пищи, и рыбоводство стало важной отраслью, в частности потому, что показатели вылова дикой рыбы невысоки или снижаются из-за чрезмерного вылова рыбы и сокращения естественной среды обитания. Примеры выращиваемой в рыбоводческих хозяйствах рыбы включают атлантического лосося (Salmo salar) и пинагора (Cyclopterus lumpus).
Однако производству рыбы в аквакультуре угрожают инфекционные заболевания, которые также могут влиять на природные популяции рыбы. Например, известно, что воспаление сердца и скелетных мышц (HSMI) часто является смертельным заболеванием у выращиваемого в рыбоводческих хозяйствах атлантического лосося. У пораженной рыбы часто наблюдают пониженный аппетит, и впоследствии аномальное поведение при плавании и, в некоторых случаях, внезапную смерть. Внешние поражения обычно не наблюдаются. При вскрытии сердце часто выглядит бледным и несколько рыхлым. В некоторых случаях перикард наполнен кровью. Гистологические исследования показывают, что у большинства рыб в пораженных сетных садках обнаруживают серьезные повреждения, хотя рыбы выглядят здоровыми. Впервые обнаруженное в Норвегии в 1999 году (Kongtorp et al., J Fish Dis 27, 2004), HSMI впоследствии было связано с несколькими вспышками на других фермах Норвегии и Великобритании. Считается, что реовирус рыб (PRV) принадлежит к семейству Reoviridae, подсемейству Spinareovirinae, и является вероятным возбудителем HSMI (Kibenge et al., Virol J. 10, 2013). С 1999 года наблюдается рост числа вспышек заболевания, при этом считается, что это заболевание оказывает негативное экономическое воздействие на лососеводство.
Синдром кардиомиопатии (CMS) является тяжелым заболеванием сердца, поражающим главным образом крупного атлантического лосося на второй год в морской воде перед сбором. Пораженные рыбы могут внезапно гибнуть без проявления внешних признаков заболевания, или могут демонстрировать такие симптомы, как нарушенное поведение плавания и анорексию. Заболевание впервые было выявлено у выращиваемого атлантического лосося в Норвегии в 1985 году, а затем у выращиваемого лосося на Фарерских островах, в Великобритании и Ирландии. CMS также был описан у атлантического лосося в естественной среде в Норвегии. В 2010 году двухцепочечный РНК-вирус семейства Totiviridae, названный вирусом миокардита рыб (PMCV), был описан как возбудитель CMS (Haugland et al, J. Virol, 85, 2011). PMCV считается одной из самых крупных проблем в производстве атлантического лосося, приводящей к крупным финансовым потерям компаний-производителей лосося.
Проблемы, связанные с болезнями при производстве пинагора (Cyclopterus lumpus), в некоторой степени обусловлены бактериальными инфекциями. Из них наиболее значимыми видами являлись подвиды Aeromonas salmonicida (атипичный фурункулез), виды Pasteurella, Vibrio anguillarum и виды Tenacibaculum. Применение программ целевой вакцинации, систематический мониторинг заболеваний и улучшение процесса производства привели к постепенному снижению числа случаев атипичного фурунколоза, вибриоза и пастереллеза. Однако несколько видов вирусов были обнаружены у диких пинагоров, включая вирусную геморрагическую септицемию (VHSV) (Guðmundsdóttir et al, J. Fish Dis, 42, 2019), вирусный некроз нервов (VNN) и новый ранавирус. Недавно был обнаружен вирус, поражающий пинагора в аквакультуре: флавивирус пинагора (LFV/CLuV) (Skoge et al, Arch Virol, 163, 2018). Вирус LFV/CLuV демонстрирует низкое, но четкое сходство с неклассифцированным вирусом летучих мышей Тамана (TABV). Было обнаружено, что LFV/CLuV присутствует во всех видах тканей пораженных пинагоров, однако патология в основном наблюдалась в печени и почках. Вирус связывают с серьезным заболеванием пинагора. После изучения свойств LFV/CLuV, определение мирового распространения вируса и его связи с заболеванием показало, что он широко распространен с относительно высокой ассоциированной распространенностью.
Некоторые рыбоводческие хозяйства в настоящее время сталкиваются с высокой смертностью, например, до 80% в некоторых популяциях пинагора, хотя ОТ-ПЦР в реальном времени и гистологическое исследование не подтверждали присутствие каких-либо известных патогенов у рыбы.
Таким образом, сохраняется потребность в идентификации других патогенов, которые инфицируют и вызывают гибель рыбы, особенно выращиваемого в аквакультуре пинагора. Кроме того, сохраняется потребность в способах контроля при производстве рыбы в аквакультуре на присутствие инфицирования патогенами, чтобы предотвратить вспышки инфекции и потенциально лечить зараженную рыбу.
Сущность изобретения
В настоящем изобретении был неожиданно обнаружен новый вирус пинагора, именуемый в настоящем изобретении коронавирус Cyclopterus lumpus (CLuCV). Длина и организация генома вместе с анализом последовательности показывают, что CLuCV является торовирусом из семейства Coronaviridae, ближайшим родственником которого является вирус Берн. Вирусы рода Torovirus обычно поражают кишечник и могут вызывать тяжелую диарею у пораженных животных. Вирус, раскрытый в настоящем изобретении, является первым торовирусом, обнаруженным у рыб.
Таким образом, в одном аспекте изобретение относится к нуклеиновая кислота, включающая по меньшей мере одну последовательность открытой рамки считывания (ORF), выбранной из группы, состоящей из ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5; где
ORF-1 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В другом аспекте изобретение относится к нуклеиновой кислоте, где: (a) последовательность указанной нуклеиновой кислоты комплементарна любой из SEQ ID NO: 1 - SEQ ID NO: 5; и/или (b) последовательность указанной нуклеиновой кислоты комплементарна SEQ ID NO: 6.
В другом аспекте изобретение относится к вирусному полипептиду, включающему аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену.
В другом аспекте изобретение относится к вирусу, который инфицирует и способен вызывать гибель пинагора (Cyclopterus lumpus), где вирусный геном включает последовательность нуклеиновой кислоты, раскрытую в настоящем изобретении, где указанная последовательность нуклеиновой кислоты содержит основание урацил (U) вместо основания тимина (T), и/или где вирус включает вирусный полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену.
В другом аспекте изобретение относится к олигонуклеотидному праймеру, включающему последовательность по меньшей мере из 9 нуклеотидов, где указанная последовательность комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении.
В другом аспекте изобретение относится к олигонуклеотидному праймеру, который включает: (a) последовательность по меньшей мере из 9 последовательных нуклеотидов, где указанная последовательность комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, (b) включает по меньшей мере 9 последовательных нуклеотидов последовательности, которая представляет собой или комплементарна части референсной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 или SEQ ID NO: 6, или (c) включает по меньшей мере 9 последовательных нуклеотидов последовательности, которая по меньшей мере на 80% идентична последовательности, которая представляет собой или комплементарна последовательности, выбранной из группы, состоящей из SEQ ID NO: 12 - SEQ ID NO: 80; предпочтительно при условии, что олигонуклеотидный праймер не включает последовательность, выбранную из группы, состоящей из SEQ ID NO: 21 - SEQ ID NO: 23.
В другом аспекте изобретение относится к способу обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающему следующие стадии:
(a) контакт нуклеиновой кислоты, выделенной из биологического образца рыбы, по меньшей мере с одним олигонуклеотидным праймером, с образованием смеси, где по меньшей мере один олигонуклеотидный праймер комплементарен последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, и
(b) определение, присутствует ли после амплификации смеси из a) продукт амплификации, где присутствие продукта амплификации указывает на присутствие РНК, ассоциированной с вирусом, и следовательно, присутствие вируса в биологическом образце.
В другом аспекте изобретение относится к способу обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии:
(a) секвенирование нуклеиновой кислоты, выделенной из биологического образца рыбы, и
(b) сравнение полученной последовательности нуклеиновой кислоты с последовательностью нуклеиновой кислоты, которая представляет собой или комплементарна референсной последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6, где по меньшей мере 80% идентичность последовательности двух последовательностей указывает на присутствие вируса в биологическом образце.
В другом аспекте изобретение относится к способу обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии:
(a) секвенирование нуклеиновой кислоты, выделенной из биологического образца рыбы, и
(b) трансляцию полученной последовательности нуклеиновой кислоты в аминокислотную последовательность или трансляцию последовательности нуклеиновой кислоты, комплементарной указанной полученной последовательности нуклеиновой кислоты, в аминокислотную последовательность, и
(c) сравнение полученной аминокислотной последовательности с референсной последовательностью, выбранной из группы, состоящей из SEQ ID NO 7-11, где по меньшей мере 80% идентичность последовательности двух последовательностей указывает на присутствие вируса в биологическом образце.
В другом аспекте изобретение относится ко антителу, которое связывает полипептид, где полипептид кодируется последовательностью нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, и/или где полипептид включает аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену.
В другом аспекте изобретение относится к набору для обнаружения вируса в биологическом образце рыбы, где набор включает олигонуклеотидный праймер, раскрытый в настоящем изобретении, и/или антитело, раскрытое в настоящем изобретении.
В другом аспекте изобретение относится к антителу для применения в лечении рыбы, инфицированной вирусом, раскрытым в настоящем изобретении.
В другом аспекте изобретение относится к применению вируса, раскрытого в настоящем изобретении, для получения вакцины.
В другом аспекте изобретение относится к вакцине, включающей:
(i) последовательность нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении;
(ii) последовательность нуклеиновой кислоты, раскрытую в настоящем изобретении;
(iii) вирусный полипептид, кодируемый последовательностью нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении;
(iv) вирусный полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену; или
(v) вирус, раскрытый в настоящем изобретении.
В еще одном аспекте изобретение относится к молекуле интерферирующей РНК (иРНК) для применения в лечении рыбы, инфицированной вирусом, где молекула иРНК включает по меньшей мере 12 последовательных нуклеотидов или комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении.
Краткое описание фигур
Фигура 1: Схема из последовательности CluCV, идентифицированной в настоящем изобретении, которая имеет длину 24613 нуклеотидов и содержит пять возможных открытых рамок считывания (ORF).
Фигура 2: Геномная нуклеотидная последовательность CLuCV.
Фигура 3: Нуклеотидная последовательность ORF-1 CLuCV.
Фигура 4: Нуклеотидная последовательность ORF-2 CLuCV.
Фигура 5: Нуклеотидная последовательность ORF-3, ORF-4 и ORF-5 CLuCV.
Фигура 6: Аминокислотная последовательность ORF-1 CLuCV.
Фигура 7: Аминокислотная последовательность ORF-2 CLuCV.
Фигура 8: Аминокислотная последовательность ORF-3, ORF-4 и ORF-5 CLuCV.
Фигура 9: Окрашивание гематоксилином и эозином целого среза пораженного пинагора, на котором показано накопление жидкости в желудке (стрелка).
Фигура 10: Окрашивание гематоксилином и эозином среза кишечника пинагора, на котором показано накопление слизи и выброс клеточного содержимого (стрелки).
Фигура 11: Окрашивание гематоксилином и эозином среза кишечника пинагора, на котором показано накопление слизи (стрелка) и выброс клеточного содержимого.
Фигура 12: Окрашивание гематоксилином и эозином среза кишечника пинагора, на котором показано накопление слизи (стрелка).
Подробное описание изобретения
Определения
Для облегчения понимания настоящего изобретения ниже представлены несколько определений терминов, используемых при описании изобретения.
При использовании в настоящем изобретении термин "пинагор" предназначен для обозначения любых видов, выбранных из всего семейства Пинагоровые (Cyclopteridae). Наиболее предпочтительным видом согласно изобретению является Cyclopterus lumpus.
Термин "нуклеиновая кислота" включает молекулы ДНК (например, кДНК или геномную ДНК), молекулы РНК (например, мРНК), аналоги ДНК или РНК, созданные с использованием аналогов нуклеотидов (например, пептидонуклеиновые кислоты и неприродные аналоги нуклеотидов), а также их гибриды. Таким образом, хотя в последовательностях нуклеиновых кислот, представленных на Фигурах 2-5 и SEQ ID NO: 1-6, используются основания гуанин, цитозин, аденин и тимин, варианты осуществления изобретения относятся к соответствующим последовательностям РНК, в которых используются основания гуанин, цитозин, аденин и урацил (т.е. урацил вместо тимина), поэтому такие последовательности РНК также представлены в настоящем изобретении. Молекула нуклеиновой кислоты может быть одноцепочечной или двухцепочечной. Если не определено иное, левый конец любой одноцепочечной последовательности нуклеиновой кислоты, обсуждаемой в настоящем изобретении, является 5'-концом. Направление 5'→3' присоединения растущих РНК-транскриптов является направлением транскрипции.
Термин "олигонуклеотид" означает нуклеиновую кислоту, включающую 200 или меньше нуклеотидов. Олигонуклеотиды могут быть одноцепочечными, например, при использовании в качестве праймеров, клонирующих праймеров или гибридизационных зондов, или они могут быть двухцепочечными, например, при использовании в конструировании мутантного гена. Олигонуклеотиды могут быть смысловыми или антисмысловыми олигонуклеотидами. Олигонуклеотид может включать метку, в том числе радиоактивную метку, флуоресцентную метку, гаптен или антигенную метку, для анализов обнаружения.
При использовании в настоящем изобретении следует понимать, что термины "олигонуклеотидный праймер" или "праймер" относятся к нуклеиновой кислоте (например, длиной по меньшей мере 9 нуклеотидов и меньше 60 нуклеотидов), подходящей для направления активности в области нуклеиновой кислоты, например, для амплификации последовательности нуклеиновой кислоты-мишени с помощью полимеразной цепной реакции (ПЦР), или для гибридизации in situ.
При использовании в настоящем изобретении термин "комплементарный" в отношении последовательностей нуклеиновой кислоты означает последовательности нуклеиновых кислот, которые формируют двухцепочечную структуру при совпадении пар оснований (A с T (или U); и G с C). Например, последовательностью нуклеиновой кислоты, комплементарной G-T-A-C, является C-A-T-G. Другие примеры комплементарных последовательностей нуклеиновых кислот являются следующими:
Комплементарная последовательность нуклеиновой кислоты (например, в случае, если нуклеиновой кислотой является ДНК):
5'-ATTCGCTTAACGCAA-3'
3'-TAAGCGAATTGCGTT-5'
Соответствующие комплементарные последовательности, в которых тимин заменен урацилом (например, в случае, если нуклеиновой кислотой является РНК):
5'-AUUCGCUUAACGCAA-3'
3'-UAAGCGAAUUGCGUU-5'
При использовании в настоящем изобретении термин "аминокислота" относится к одной из 20 природных аминокислот или любым неприродным аналогам. Предпочтительно термин "аминокислота" относится к одной из 20 природных аминокислот.
Термины "полипептид" или "белок" означают макромолекулу, состоящую из последовательности аминокислот. Белок может быть нативным белком, то есть белком, продуцированным природной и нерекомбинантной клеткой; или он может быть продуцирован генетически модифицированной или рекомбинантной клеткой, и включает молекулы, имеющие аминокислотную последовательность нативного белка, или молекулы, имеющие делеции, вставки и/или замены одной или более аминокислот в сравнении с нативной последовательностью. Термины также включают полимеры аминокислот, в которых одна или больше аминокислот являются химическими аналогами соответствующего природного полимера аминокислот.
Термин "идентичность последовательностей" указывает количественный показатель степени гомологии между двумя последовательностями, которые могут быть последовательностями нуклеиновых кислот (также называемыми нуклеотидными последовательностями) или аминокислотными последовательностями. Если две сравниваемые последовательности имеют разную длину, требуется произвести их выравнивание с получением наилучшего соответствия, допуская вставку пропусков или, в альтернативе, усечение на концах последовательностей нуклеиновых кислот или аминокислотных последовательностей.
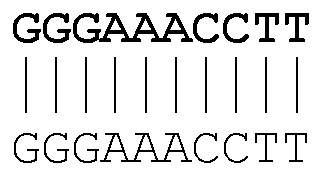
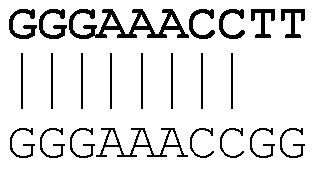
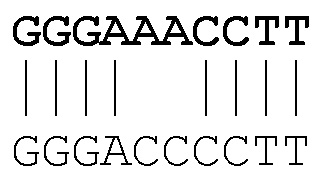
В случае нуклеотидной последовательности, например, термин "по меньшей мере на 80% идентичный" таким образом означает, что по меньшей мере 80% нуклеотидов во всей последовательности могут быть выровнены с идентичными нуклеотидами из другой последовательности. Указанный процент нуклеотидов может упоминаться как, например, идентичный на 80%, идентичный на 85%, идентичный на 90%, идентичный на 95%, идентичный на 99% или больше на протяжении указанной области, при сравнении и выравнивании с максимальным соответствием. Например, последовательность, которая имеет длину 10 нуклеотидов, например GGGAAACCTT, может быть на 80% идентична непрерывной последовательности (например, GGGAAACCGG) или ненепрерывной последовательности (например, GGGACCCCTT):
пример 100% идентичности:
пример 80% идентичности:
пример 80% идентичности:
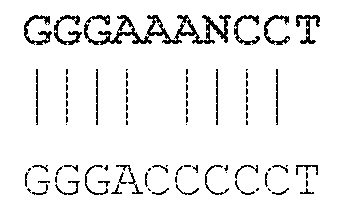
В случае если основание, обозначенное "N", найдено в конкретном положении в референсной нуклеотидной последовательности, идентичность последовательностей дается для любого из оснований аденина (A), цитозина (C), гуанина (G) и тимина (T) или урацила (U) в соответствующем положении на сравниваемой последовательности. См. следующий пример идентичности. То же относится к сравнениям аминокислотных последовательностей, т.е. в случае, если аминокислота, обозначенная "X", найдена в конкретном положении в референсной аминокислотной последовательности, идентичность последовательностей дается для любой аминокислоты в соответствующем положении в сравниваемой последовательности.
пример 80% идентичности:
Специалисту известно, что доступны разные средства для сравнения последовательностей (см. ниже).
При использовании в настоящем изобретении термин "содержащий консервативную замену" в отношении аминокислоты означает, что аминокислота может быть заменена другой аминокислотой в ее соответствующей группе, согласно следующим шести группам: [1] Аланин (A), Серин (S), Треонин (T); [2] Аспарагиновая кислота (D), Глутаминовая кислота (E); [3] Аспарагин (N), Глутамин (Q); [4] Аргинин (R), Лизин (K); [5] Изолейцин (I), Лейцин (L), Метионин (M), Валин (V); и [6] Фенилаланин (F), Тирозин (Y), Триптофан (W). "Вариант, содержащий консервативную замену", в отношении полипептида или белка означает, что любая из аминокислот в указанном полипептиде или белке может быть консервативно заменена, как определено выше.
При использовании в настоящем изобретении термин "антитело" относится к гликопротеину, включающему по меньшей мере две тяжелых (H) цепи и две легких (L) цепи, соединенные дисульфидными связями, или соответствующему фрагменту антитела (антигенсвязывающей части). Каждая тяжелая цепь состоит из вариабельной области тяжелой цепи (VH) и константной области тяжелой цепи (CH). Константная область тяжелой цепи состоит из трех доменов, CH1, CH2 и CH3. Каждая легкая цепь состоит из вариабельной области легкой цепи (VL) и константной области легкой цепи (CL). Константная область легкой цепи состоит из одного домена, CL. Области VH и VL можно далее подразделить на области гипервариабельности, называемые определяющими комплементарность областями (CDR), которые чередуются с областями, которые являются более консервативными, называемыми каркасными областями (FR). Каждая VH и VL состоит из трех CDR-областей и четырех FR-областей, расположенных от N-конца к C-концу в следующем порядке: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4. Вариабельные области тяжелой и легкой цепи содержат связывающий домен, который взаимодействует с антигеном. Константные области антител могут опосредовать связывание иммуноглобулина с клетками-хозяевам или факторами, включая различные клетки иммунной системы (например, эффекторные клетки) и первый компонент (C1q) классической системы комплемента. Антитела согласно изобретению включают моноклональные антитела (включая полноразмерные моноклональные антитела) и поликлональные антитела, целые антитела, химерные антитела, гуманизированные антитела, человеческие антитела или гибридные антитела с двойной или множественной антигенной или эпитопной специфичностью, фрагменты антител и субфрагменты антител, например, Fab, Fab', F(ab')2, фрагменты и т.п., включая гибридные фрагменты любого иммуноглобулина или любого природного, синтетического или генетически модифицированного белка, действующего как антитело путем связывания со специфическим антигеном с образованием комплекса. Антитела согласно изобретению могут быть также Fc-слитыми белками.
"Вектор" представляет собой нуклеиновую кислоту, которая может использоваться для введения другой нуклеиновой кислоты (или "конструкции"), связанной с ней, в клетку. Одним из типов вектора является "плазмида", которая относится к линейной или кольцевой двухцепочечной молекуле ДНК, в которой могут быть лигированы дополнительные сегменты нуклеиновой кислоты. Другим типом вектора является вирусный вектор (например, репликационно-дефектные ретровирусы, аденовирусы и аденоассоциированные вирусы), где в вирусный геном могут быть введены дополнительные сегменты ДНК. Некоторые векторы способны к автономной репликации в клетке-хозяине, в которую они введены (например, бактериальные векторы, содержащие бактериальную точку начала репликации и эписомные векторы млекопитающих). Другие векторы (например, неэписомные векторы млекопитающих) интегрируются в геном клетки-хозяина после введения в клетку-хозяина и культивирования под селективным давлением и, таким образом, реплицируются вместе с геномом хозяина. Вектор может использоваться для направления экспрессии выбранной нуклеиновой кислоты в клетке.
"Клетка-хозяин" является клеткой, которая может использоваться для экспрессии нуклеиновой кислоты, например, нуклеиновой кислоты, раскрытой в настоящем изобретении. Клетка-хозяин может быть прокариотом, например E. coli, или она может быть эукариотом, например одноклеточным эукариотом (например, дрожжами или другим грибом), клеткой растения (например, клеткой растения табака или томата), клеткой животного (например, клеткой человека, клеткой обезьяны, клеткой хомяка, клеткой крысы, клеткой мыши или клеткой насекомого) или гибридомой. Примеры клеток-хозяев включают линии клеток яичников китайского хомячка (CHO) или их производные. Как правило, клетка-хозяин представляет собой культивируемую клетку, которая может быть трансформирована или трансфицирована кодирующей полипептид нуклеиновой кислотой, которая может затем экспрессироваться в клетке-хозяине. Следует понимать, что термин клетка-хозяин относится не только к клетке конкретного субъекта, но и к потомству или потенциальному потомству такой клетки. Поскольку некоторые модификации могут произойти в последующих поколениях, например, в результате мутации или воздействия факторов внешней среды, такое потомство может не быть, по существу, идентичным родительской клетке, но при этом все еще будет включено в рамки данного термина, используемого в настоящем изобретении.
Термины "лечить" и "лечение" включают терапевтическое лечение, профилактическое лечение и применения, которые уменьшают симптомы нарушения или снижают риск развития у субъекта (например, рыбы) нарушения (например, симптомов вирусной инфекции).
Термин "вакцина" при использовании в настоящем изобретении относится к материалу, который может вызывать иммунный ответ, который блокирует, частично или полностью, инфекционность возбудителя инфекции, который в отношении настоящего изобретения является вирусом, воздействующим на рыбу, например пинагоров. Таким образом, при введении рыбе, вакцина согласно изобретению иммунизирует рыбу против заболевания, вызванного вирусом. Иммунизирующим компонентом вакцины может быть, например, ДНК, как в вакцине ДНК, РНК, как в вакцине РНК, рекомбинантный белок или его фрагмент согласно настоящему изобретению, или живой или ослабленный рекомбинантный вирус.
"Средство иРНК" (сокращение от "средства интерферирующей РНК") при использовании в настоящем изобретении представляет собой средство на основе РНК, которое может даунрегулировать (снижать) экспрессию целевого гена, например, белка, кодируемого ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5. Средство иРНК может действовать по одному или более механизмам, включая посттранскрипционное расщепление мРНК-мишени, иногда именуемое в уровне техники как "РНКи", или предтранскрипционные или предтрансляционные механизмы. Средство иРНК может быть двухцепочечным (дц) средством иРНК. Средство иРНК также может быть "малой интерферирующей РНК" (миРНК).
Термины "изобретения" или "согласно изобретению" при использовании в настоящем изобретении обозначают все аспекты и варианты осуществления изобретения, раскрытого и/или заявленного в настоящем изобретении. С другой стороны любые аспекты, объекты или варианты осуществления, указанные в настоящем изобретении как "раскрытые в настоящем изобретении" или "описанные в настоящем изобретении", следует понимать как аспекты, объекты или варианты осуществления "изобретения" или "согласно изобретению".
При использовании в настоящем изобретении термин "содержащий" следует рассматривать как охватывающий "включающий" и "состоящий из", причем оба этих значения являются специально предусмотренными и, следовательно, индивидуально раскрытыми вариантами осуществления согласно настоящему изобретению.
При использовании в настоящем изобретении формы единственного числа не должны ограничивать число случаев (т.е. случаев появления) элемента или компонента. Таким образом, формы единственного числа следует читать как включающие один или по меньшей мере один, при этом форма единственного числа элемента или компонента также включает множественное число, если в явной форме не подразумевается, что такое число должно быть единственным.
При использовании в настоящем изобретении термин "приблизительно", дополняющий количество используемого вещества, ингредиента, компонента или параметра, относится к вариации числового количества, которая может возникать, например, в результате стандартных процедур измерения и обработки, например, процедуры обработки жидкости, используемых при изготовлении концентратов или растворов. Кроме того, вариация может возникать вследствие непреднамеренной погрешности в процедурах измерения, различий в производстве, источника или чистоты ингредиентов, используемых для выполнения способов, и т.п. В одном варианте осуществления термин "приблизительно" означает в пределах 10% от приведенного числового значения. В более конкретном варианте осуществления термин "приблизительно" означает в пределах 5% от приведенного числового значения.
Вирусные последовательности нуклеиновых кислот и вирусные полипептиды
В одном аспекте изобретение относится к нуклеиновой кислоте, включающей по меньшей мере одну последовательность открытой рамки считывания (ORF), выбранную из группы, состоящей из ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5; где
ORF-1 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В некоторых вариантах осуществления:
ORF-1 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В предпочтительных вариантах осуществления:
ORF-1 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В более предпочтительных вариантах осуществления:
ORF-1 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В еще более предпочтительных вариантах осуществления:
ORF-1 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В еще более предпочтительных вариантах осуществления:
ORF-1 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
В особенно предпочтительных вариантах осуществления:
ORF-1 представляет собой последовательность нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 представляет собой последовательность нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 представляет собой последовательность нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 представляет собой последовательность нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 представляет собой последовательность нуклеиновой кислоты SEQ ID NO: 5.
В конкретных вариантах осуществления нуклеиновая кислота, раскрытая в настоящем изобретении, включает по меньшей мере ORF-1, ORF-2, ORF-3 и/или ORF-4, согласно любому из их вариантов осуществления, раскрытых в настоящем изобретении.
Последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, может быть по меньшей мере на 80% идентична вирусному геному согласно SEQ ID NO: 6. В некоторых вариантах осуществления последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, по меньшей мере на 85% идентична вирусному геному согласно SEQ ID NO: 6. В предпочтительных вариантах осуществления последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, по меньшей мере на 90% идентична вирусному геному согласно SEQ ID NO: 6. В более предпочтительных вариантах осуществления последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, по меньшей мере на 95% идентична вирусному геному согласно SEQ ID NO: 6. В еще более предпочтительных вариантах осуществления последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, по меньшей мере на 98% идентична вирусному геному согласно SEQ ID NO: 6. В еще более предпочтительных вариантах осуществления последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, по меньшей мере на 99% идентична вирусному геному согласно SEQ ID NO: 6. В наиболее предпочтительных вариантах осуществления последовательность нуклеиновой кислоты, раскрытой в настоящем изобретении, обладает 100% идентичностью с последовательностью вирусного генома согласно SEQ ID NO: 6 (CLuCV).
Также изобретение относится к нуклеиновой кислоте, последовательность которой комплементарна последовательности любой из нуклеиновых кислот, раскрытых в настоящем изобретении.
Последовательность указанной нуклеиновой кислоты может быть комплементарна ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5 согласно любому из соответствующих вариантов осуществления, раскрытых в настоящем изобретении. Последовательность нуклеиновой кислоты также может быть комплементарна последовательности нуклеиновой кислоты, которая по меньшей мере на 80% идентична, в некоторых вариантах осуществления по меньшей мере на 85% идентична, в предпочтительных вариантах осуществления по меньшей мере на 90% идентична, в более предпочтительных вариантах осуществления по меньшей мере на 95% идентична, в еще более предпочтительных вариантах осуществления по меньшей мере на 98% идентична, в еще более предпочтительных вариантах осуществления по меньшей мере на 99% идентична, и в наиболее предпочтительных вариантах осуществления на 100% идентична последовательности вирусного генома согласно SEQ ID NO: 6 (CLuCV).
Таким образом, изобретение также относится к нуклеиновой кислоте, где: (a) последовательность указанной нуклеиновой кислоты комплементарна любой из SEQ ID NO: 1 - SEQ ID NO: 5; и/или (b) последовательность указанной нуклеиновой кислоты комплементарна SEQ ID NO: 6.
В предпочтительных вариантах осуществления последовательности нуклеиновых кислот, раскрытые в настоящем изобретении, представляют собой РНК-последовательности нуклеиновых кислот, т.е. они содержат основание урацил (U) вместо основания тимина (T). Таким образом, вирусы, раскрытые в настоящем изобретении, содержат свою генетическую информацию в форме таких РНК-последовательностей нуклеиновых кислот.
Специалисту будет известно, что изменения последовательности нуклеиновой кислоты, приводящие к модификациям кодируемой ею аминокислотной последовательности белка, могут оказывать малое, если таковое вообще будет присутствовать, воздействие на конечную трехмерную структуру белка. Например, кодон аминокислоты аланина, гидрофобной аминокислоты, может быть заменен кодоном, кодирующим другой менее гидрофобный остаток, такой как глицин, или более гидрофобный остаток, такой как валин, лейцин или изолейцин. Аналогичным образом, можно также ожидать, что изменения, которые приводят к замене одного отрицательно заряженного остатка другим остатком, таким как аспарагиновая кислота вместо глутаминовой кислоты, или одного положительно заряженного остатка другим остатком, таким как лизин вместо аргинина, будут давать белок, обладающий по существу такой же функциональной активностью.
Каждая из следующих шести групп содержит аминокислоты, которые являются типичными консервативными заменами друг для друга: [1] Аланин (A), Серин (S), Треонин (T); [2] Аспарагиновая кислота (D), Глутаминовая кислота (E); [3] Аспарагин (N), Глутамин (Q); [4] Аргинин(R), Лизин (K); [5] Изолейцин (I), Лейцин (L), Метионин (M), Валин (V); и [6] Фенилаланин (F), Тирозин (Y), Триптофан (W) (см., например, патентную публикацию США 20100291549).
Предпочтительно ORF 1-5 в настоящем изобретении кодируют вирусные полипептиды, включающие аминокислотные последовательности SEQ ID NO: 7-11, соответственно, или аминокислотные последовательности, которые по меньшей мере на 80% идентичны (например, по меньшей мере на 85% идентичны или по меньшей мере на 90% идентичны, или по меньшей мере на 91% идентичны, по меньшей мере на 92% идентичны, по меньшей мере на 93% идентичны, по меньшей мере на 94% идентичны, по меньшей мере на 95% идентичны, по меньшей мере на 96% идентичны, по меньшей мере на 97% идентичны, по меньшей мере на 98% идентичны или по меньшей мере на 99% идентичны) аминокислотным последовательностям SEQ ID NO: 7-11, соответственно. В некоторых вариантах осуществления ORF 1-5 кодируют вирусные полипептиды, которые являются содержащими консервативную замену вариантами SEQ ID NO 7-11, соответственно, как описано выше.
Таким образом, в другом аспекте изобретение относится к вирусным полипептидам, включающим аминокислотные последовательности SEQ ID NO: 7-11, соответственно, или аминокислотные последовательности, которые по меньшей мере на 80% идентичны (например, по меньшей мере на 85% идентичны или по меньшей мере на 90% идентичны, или по меньшей мере на 91% идентичны, по меньшей мере на 92% идентичны, по меньшей мере на 93% идентичны, по меньшей мере на 94% идентичны, по меньшей мере на 95% идентичны, по меньшей мере на 96% идентичны, по меньшей мере на 97% идентичны, по меньшей мере на 98% идентичны или по меньшей мере на 99% идентичны) аминокислотным последовательностям SEQ ID NO: 7-11, соответственно. В некоторых вариантах осуществления вирусные полипептиды являются содержащими консервативную замену вариантами SEQ ID NO 7-11, соответственно, как описано выше. Также предложены векторы, например, плазмидные векторы или вирусные векторы, включающие последовательности нуклеиновых кислот, кодирующие вирусные полипептиды согласно изобретению, как описано выше.
Идентичность (гомологию) последовательностей белков и/или нуклеиновых кислот можно оценивать при использовании любых из множества известных алгоритмов и программ для сравнения последовательностей. Для сравнения последовательностей обычно одна последовательность выступает в качестве референсной последовательности (например, последовательности, раскрытой в настоящем изобретении), с которой сравнивают тестируемые последовательности. Затем алгоритм сравнения последовательностей вычисляет процент идентичности последовательностей для тестируемых последовательностей по отношению к референсной последовательности на основе параметров программы.
Процент идентичности двух аминокислотных последовательностей или двух последовательностей нуклеиновых кислот можно определить, например, путем сравнения информации о последовательностях при использовании компьютерной программы GAP, т.е. Genetics Computer Group (GCG; Мэдисон, Висконсин), версии 10.0 в пакете Wisconsin, GAP (Devereux et al. (1984), Nucleic Acids Res. 12:387-95). При вычислении процента идентичности сравниваемые последовательности обычно выравнивают таким образом, чтобы получить наибольшее совпадение между последовательностями. Предпочтительные параметры по умолчанию для программы GAP включают следующее: (1) Реализация GCG унарной матрицы сравнения (содержащей значение 1 для идентичностей и 0 для неидентичностей) для нуклеотидов и матрица сравнения аминокислот Грибскова и Берджесса (Gribskov and Burgess (1986) Nucleic Acids Res. 14:6745), как описано в Atlas of Polypeptide Sequence and Structure, Schwartz and Dayhoff, eds., National Biomedical Research Foundation, pp. 353-358 (1979), или другие сопоставимые матрицы сравнения; (2) штраф 8 баллов за каждый пропуск и дополнительный штраф 2 балла за каждый символ в каждом пропуске для аминокислотных последовательностей или штраф 50 баллов за каждый пропуск и дополнительный штраф 3 балла за каждый символ в каждом пропуске для нуклеотидных последовательностей; (3) отсутствие штрафа за пропуски на концах; и (4) отсутствие максимального штрафа за длинные пропуски.
Идентичность и/или подобие последовательностей также можно определить при использовании алгоритма локальной идентичности последовательностей Смита и Уотермана (Smith and Waterman, 1981, Adv. Appl. Math. 2:482), алгоритма выравнивания идентичности последовательностей Нидлмана и Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48:443), метода поиска подобия Пирсона и Липмана (Pearson and Lipman, 1988, Proc. Nat. Acad. Sci. U.S.A. 85:2444), компьютерных реализаций этих алгоритмов (BESTFIT, FASTA и TFASTA в пакете программ Wisconsin Genetics, Genetics Computer Group, 575 Science Drive, Madison, Wis.).
Другим примером полезного алгоритма является PILEUP. PILEUP создает множественное выравнивание последовательностей из группы родственных последовательностей при использовании прогрессивных парных выравниваний. Он также позволяет построить дерево, показывающее отношения кластеризации, используемые при создании выравнивания. В PILEUP используется упрощение метода прогрессивного выравнивания Фенга и Дулитла (Feng & Doolittle, 1987, J. Mol. Evol. 35:351-360); метод подобен описанному в публикации Higgins and Sharp, 1989, CABIOS 5:151-153. Полезные параметры PILEUP включают вес пропуска по умолчанию 3,00, вес длины пропуска по умолчанию 0,10 и взвешенные концевые пропуски.
Другим примером полезного алгоритма является алгоритм BLAST, описанный в публикации Altschul et al., 1990, J. Mol. Biol. 215:403-410; Altschul et al., 1997, Nucleic Acids Res. 25:3389-3402; и Karin et al., 1993, Proc. Natl. Acad. Sci. U.S.A. 90:5873-5787. Особенно полезной программой BLAST является программа WU-BLAST-2, созданная на основе публикации Altschul et al., 1996, Methods in Enzymology 266:460-480. В WU-BLAST-2 используется несколько параметров поиска, значения большинства из которых установлены по умолчанию. Изменяемые параметры устанавливают со следующими значениями: размах перекрывания=1, доля перекрывания=0,125, порог слова (T) = II. Параметры HSP S и HSP S2 являются динамическими значениями и устанавливаются самой программой в зависимости от состава конкретной последовательности и состава конкретной базы данных, в которой производят поиск интересующей последовательности; однако значения можно изменять для повышения чувствительности.
Дополнительным полезным алгоритмом является gapped BLAST, описанный в публикации Altschul et al., 1993, Nucl. Acids Res. 25:3389-3402. В gapped BLAST используется матрица замен BLOSUM-62; пороговый параметр T, равный 9; метод с двумя совпадениями, способствующий выбору продолжаемых последовательностей без пропусков, штрафы за длину пропуска k в размере 10+k; Xu, равный 16, и Xg, равный 40 для стадии поиска по базе данных и 67 для стадии вычисления результата алгоритмов. Выравнивания с пропусками инициируются баллом, соответствующим приблизительно 22 битам.
Способы получения вирусных полипептидов, описанных в настоящем изобретении, хорошо известны специалистам в данной области. В качестве примера и без ограничения, последовательности нуклеиновых кислот, кодирующие вирусные полипептиды, включающие SEQ ID NO 7-11, или последовательности, идентичные им по меньшей мере на 80%, включая содержащие консервативную замену варианты SEQ ID NO 7-11, можно клонировать в вектор, такой как, например, плазмидный или вирусный вектор, и экспрессировать в подходящем хозяине, таком как клетки рыб, клетки млекопитающих, клетки бактерий, клетки растений и клетки насекомых, а затем выделять из них экспрессированные вирусные полипептиды.
Вирусы
В другом аспекте изобретение относится к вирусу, который инфицирует и способен вызывать гибель пинагора (Cyclopterus lumpus), где вирусный геном включает последовательность нуклеиновой кислоты, раскрытую в настоящем изобретении, где указанная последовательность нуклеиновой кислоты содержит основание урацил (U) вместо основания тимина (T).
В другом аспекте изобретение относится к вирусу, который инфицирует и способен вызывать гибель пинагора (Cyclopterus lumpus), где вирус включает вирусный полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену.
В другом аспекте изобретение относится к вирусу, который инфицирует и способен вызывать гибель пинагора (Cyclopterus lumpus), где вирусный геном включает последовательность нуклеиновой кислоты, раскрытую в настоящем изобретении, где указанная последовательность нуклеиновой кислоты содержит основание урацил (U) вместо основания тимина (T), и где вирус включает вирусный полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену.
В предпочтительных вариантах осуществления нуклеиновая кислота, содержащаяся в вирусе, находится в форме одноцепочечной РНК (оцРНК).
В некоторых вариантах осуществления вирусный геном включает последовательность нуклеиновой кислоты, включающую по меньшей мере одну из ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5 согласно любому из их вариантов осуществления, раскрытых в настоящем изобретении. В предпочтительных вариантах осуществления последовательность вирусного генома включает по меньшей мере ORF-1, ORF-2, ORF-3 и ORF-4 согласно любому из их вариантов осуществления, раскрытых в настоящем изобретении. В некоторых вариантах осуществления вирусный геном включает последовательность нуклеиновой кислоты, которая представляет собой или комплементарна последовательности нуклеиновой кислоты, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно на 98%, наиболее предпочтительно на 99% или даже на 100% идентична последовательности вирусного генома согласно SEQ ID NO: 6 (CLuCV).
В некоторых вариантах осуществления вирус включает ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5; где
ORF-1 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5;
и где ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5 кодируют вирусные полипептиды, включающие SEQ ID NO 7-11, соответственно, или последовательности, которые по меньшей мере на 80% идентичны SEQ ID NO 7-11, соответственно.
В некоторых вариантах осуществления ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5 кодируют вирусные полипептиды, включающие соответствующие аминокислотные последовательности, включающие SEQ ID NO 7-11, или последовательности, которые по меньшей мере на 80% идентичны им (например, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны SEQ ID NO 7-11). Предпочтительно вирус включает ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5, как определено в настоящем изобретении, где указанные ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5 кодируют вирусные полипептиды, которые по меньшей мере на 95% идентичны SEQ ID NO 7-11, соответственно. Более предпочтительно вирус включает ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5, как определено в настоящем изобретении, где указанные ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5 кодируют вирусные полипептиды, которые являются содержащими консервативные замены вариантами SEQ ID NO 7-11, соответственно, или вирусные полипептиды, включающие аминокислотные последовательности SEQ ID NO 7-11, соответственно.
В некоторых вариантах осуществления инфицирование пинагора вирусом, раскрытым в настоящем изобретении, вызывает следующие симптомы у рыбы:
(i) повреждение ткани в кишечнике, и/или
(ii) диарею.
В других вариантах осуществления инфицирование пинагора вирусом, раскрытым в настоящем изобретении, дополнительно вызывает анорексию.
Повреждение ткани в кишечнике можно диагностировать с помощью гистологического исследования или электронной микроскопии, которые позволяют наблюдать разрушение ткани кишечника, например, разрушение структуры ворсинок, увеличенную толщину других слоев. Предпочтительно срезы ткани окрашивают с использованием гистологической окраски гематоксилином и эозином (ГЭ). При использовании такой окраски у инфицированных особей можно наблюдать накопление жидкостей и непереваренных частиц корма, как в желудке, так и в кишечнике (см., например, Фигуры 9-12), что приводит к диареяподобному состоянию у этих рыб. Кроме того, может наблюдаться повреждение стенки кишечника с выбросом клеточного содержимого и увеличением продукции слизи (см., например, Фигуры 10-12). Диарея рыб может наблюдаться в резервуарах с водой у разводимой рыбы.
Изменения биомакромолекулярных компонентов (белков в целом, сидерофильных белков, нейтральных мукополисахаридов, гликогена и кислых мукополисахаридов) также можно наблюдать в образцах ткани кишечника с помощью стандартных методов, например, вестерн-блоттинга, ИФА, окрашивания срезов тканей и т.д.
Анорексию у рыб можно наблюдать, когда рыба отказывается от корма.
В некоторых вариантах осуществления вирус представляет собой оболочечный вирус. Оболоченные вирусы имеют защитный слой (оболочку), покрывающий их белковые капсиды. Оболочки обычно образуются из частей мембран клетки-хозяина (фосфолипидов и белков), но также включают некоторые вирусные гликопротеины. Они могут помогать вирусам уклоняться от иммунной системы хозяина.
В некоторых вариантах осуществления вирус является коронавирусом. В предпочтительных вариантах осуществления вирусом является торовирусом. Такие вирусы имеют оболочку круглой формы, но плеоморфную, диаметром приблизительно 100-150 нм. Вирусная частица, как правило, имеет поверхностные спайковые белки, которые являются булавовидными и равномерно распределены по поверхности.
В некоторых вариантах осуществления вирус, раскрытый в настоящем изобретении, включает 5'-нетранслируемую область (5'-UTR), которая функционирует в качестве участка внутренней посадки рибосомы (IRES).
В предпочтительных вариантах осуществления ORF-1 кодирует полипротеин, ORF-2 кодирует спайковый гликопротеин, ORF-3 кодирует мембранный белок, и ORF-4 кодирует белок нуклеокапсида.
Также изобретение относится к вектору, включающему нуклеиновую кислоту, кодирующую по меньшей мере одну ORF, как раскрыто в настоящем изобретении, со всеми соответствующими вариантами осуществления. В некоторых вариантах осуществления вектор содержит нуклеиновую кислоту, которая кодирует полный вирус, раскрытый в данном документе. Вектор может использоваться для введения указанной нуклеиновой кислоты (кислот) в клетку, такую как клетка-хозяин.
Также изобретение относится к клетке-хозяину, включающей вирус, раскрытый в настоящем изобретении. Клетка-хозяин может быть клеткой бактерии, клеткой рыбы или клеткой млекопитающего.
Олигонуклеотидные праймеры
В другом аспекте изобретение относится к олигонуклеотидному праймеру, включающему последовательность по меньшей мере из 9 нуклеотидов, где указанная последовательность комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении.
В другом аспекте изобретение относится к олигонуклеотидному праймеру, который включает: (a) последовательность по меньшей мере из 9 последовательных нуклеотидов, где указанная последовательность комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, (b) включает по меньшей мере 9 последовательных нуклеотидов последовательности, которая представляет собой часть или комплементарна части референсной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 или SEQ ID NO: 6, или (c) включает по меньшей мере 9 последовательных нуклеотидов последовательности, которая по меньшей мере на 80% идентична последовательности, которая представляет собой или комплементарна последовательности, выбранной из группы, состоящей из SEQ ID NO: 12 - SEQ ID NO: 80; предпочтительно при условии, что указанный олигонуклеотидный праймер не включает последовательность, выбранную из группы, состоящей из SEQ ID NO: 21 - SEQ ID NO: 23.
В некоторых вариантах осуществления олигонуклеотидный праймер имеет длину 9-60 нуклеотидов. В предпочтительных вариантах осуществления олигонуклеотидный праймер имеет длину 12-40 нуклеотидов. В более предпочтительных вариантах осуществления олигонуклеотидный праймер имеет длину 15-30 нуклеотидов. В еще более предпочтительных вариантах осуществления олигонуклеотидный праймер имеет длину 18-25 нуклеотидов.
Для специалиста будет очевидно, что олигонуклеотидный праймер, который комплементарен последовательности нуклеиновой кислоты, будет гибридизоваться с этой последовательностью в строгих условиях. "Строгие условия" относятся к условиям температуры, ионной силы и присутствия других соединений, таких как органические растворители, при которых проводят гибридизацию нуклеиновых кислот. В строгих условиях спаривание оснований нуклеиновых кислот будет происходить только между последовательностями нуклеиновых кислот, имеющими высокую частоту комплементарных оснований. Строгие условия гибридизации известны специалисту (см., например, Green M. R., Sambrook, J., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press; 4th edition, 2012). Точные условия строгой гибридизации обычно зависят от последовательности и будут отличаться в разных обстоятельствах, что сумеет легко понять специалист. Более длинные последовательности гибридизуются при более высоких температурах по сравнению с более короткими последовательностями. Обычно строгие условия выбирают примерно на 5°C ниже температуры плавления (Тп) конкретной последовательности. Тп определяют как температуру, при которой 50% дуплексных молекул диссоциировали на составляющие их одиночные цепи. Поскольку последовательности-мишени обычно присутствуют в избытке, при Тп 50% нуклеотидных праймеров обычно будут заняты при равновесии. Как правило, строгие условия будут такими, при которых концентрация соли составляет меньше чем приблизительно 1,0 М ионов натрия, обычно приблизительно от 0,01 до 1,0 М ионов натрия (или других солей), при рН 6,8-8,3, и температура составляет по меньшей мере приблизительно 30°C для коротких праймеров (например, от 10 нуклеотидов до 50 нуклеотидов) и по меньшей мере приблизительно 60°C для более длинных праймеров. Строгие условия также могут быть достигнуты путем добавления дестабилизирующих агентов, таких как формамид. Для специалиста будет очевидно, что из-за комплементарности последовательностей олигонуклеотидный праймер согласно изобретению, таким образом, гибридизуется с последовательностью нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении.
Олигонуклеотидный праймер согласно настоящему изобретению может быть помечен молекулярным маркером, чтобы обеспечить возможность визуализации гибридизации с последовательностью-мишенью или количественного определения амплификации последовательности-мишени. Различные молекулярные маркеры или метки известны специалисту.
В конкретном варианте осуществления изобретение относится к олигонуклеотидному праймеру, который включает по меньшей мере 9 последовательных нуклеотидов последовательности, которая представляет собой или комплементарна части (например, длиной 9-60 нуклеотидов, предпочтительно длиной 12-40 нуклеотидов, более предпочтительно длиной 15-30 нуклеотидов, еще более предпочтительно длиной 18-25 нуклеотидов) референсной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 или SEQ ID NO: 6; предпочтительно при условии, что указанный олигонуклеотидный праймер не включает последовательность, выбранную из группы, состоящей из SEQ ID NO: 21 - SEQ ID NO: 23. В наиболее предпочтительном варианте осуществления последовательность указанного олигонуклеотидного праймера состоит из указанной последовательности последовательных нуклеотидов.
В другом конкретном варианте осуществления изобретение относится к олигонуклеотидному праймеру, который включает по меньшей мере 9 (предпочтительно по меньшей мере 12, более предпочтительно по меньшей мере 15, еще более предпочтительно по меньшей мере 18) последовательных нуклеотидов последовательности, которая по меньшей мере на 80% идентична последовательности, которая представляет собой или комплементарна последовательности, выбранной из группы, состоящей из SEQ ID NO: 12 - SEQ ID NO: 80; предпочтительно при условии, что указанный олигонуклеотидный праймер не включает последовательность, выбранную из группы, состоящей из SEQ ID NO: 21 - SEQ ID NO: 23.
В другом аспекте изобретение относится к применению по меньшей мере одного олигонуклеотидного праймера в способе обнаружения вируса, раскрытого в настоящем изобретении, где по меньшей мере один праймер включает последовательность по меньшей мере из 9 нуклеотидов, например из 9 последовательных нуклеотидов, (предпочтительно по меньшей мере 12, более предпочтительно по меньшей мере 15, еще более предпочтительно по меньшей мере 18), и где указанная последовательность комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме указанного вируса.
В некоторых вариантах осуществления по меньшей мере один олигонуклеотидный праймер представляет собой пару праймеров, т.е. два праймера, один прямой праймер и один обратный праймер, которые комплементарны двум областям в последовательности нуклеиновой кислоты, и которые могут использоваться для амплификации последовательности между двумя указанными областями. Специалисту хорошо известно и находится в рамках его/ее компетенции, как проводить поиск олигонуклеотидных праймеров, подходящих для составления пары. Согласно применению изобретения, "пара праймеров" может использоваться для амплификации последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении.
В некоторых вариантах осуществления применение включает синтез кДНК в способе обнаружения вируса, раскрытого в настоящем изобретении. Например, случайные олигонуклеотидные праймеры (например, гексануклеотиды) используют для синтеза кДНК с последовательности нуклеиновой кислоты, которая содержится в геноме указанного вируса.
В некоторых вариантах осуществления применение включает ПЦР в способе обнаружения вируса, раскрытого в настоящем изобретении. В некоторых вариантах осуществления применение включает ОТ-ПЦР в способе обнаружения вируса, раскрытого в настоящем изобретении. В некоторых вариантах осуществления применение включает ОТ-кПЦР в способе обнаружения вируса, раскрытого в настоящем изобретении. В некоторых вариантах осуществления применение включает случайную мультиплексную ОТ-ПЦР в способе обнаружения вируса, раскрытого в настоящем изобретении; в данном способе используют смесь праймеров, подобранных так, что они устойчивы к образованию димеров праймеров при амплификации (см. Clem et al, Virol J, 4, 2007). В некоторых вариантах осуществления применение включает транскрипционно-опосредованную амплификацию (TMA) в способе обнаружения вируса, раскрытого в настоящем изобретении. В некоторых вариантах осуществления применение включает амплификацию с замещением цепей (SDA) в способе обнаружения вируса, раскрытого в настоящем изобретении.
В некоторых вариантах осуществления применение включает обнаружение in situ, которое также называют гибридизацией in situ (ISH), в способе обнаружения вируса, раскрытого в настоящем изобретении, например, флуоресцентную гибридизацию in situ (FISH). В ISH используется меченая комплементарная ДНК, РНК или модифицированная последовательность олигонуклеотидного праймера (зонд), которые позволяют проводить визуализацию спецфических нуклеиновых кислот в морфологически сохраненных клетках и срезах ткани. Зонд может быть помечен радиоизотопными, флуоресцентными или антигенными метками (например, дигоксигенином), локализацию которых затем можно установить и количественно определить в ткани с помощью ауторадиографии, флуоресцентной микроскопии или иммуногистохимического исследования, соответственно.
Диагностические методы
В другом аспекте изобретение относится к способу обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии:
(a) контакт нуклеиновой кислоты, выделенной из биологического образца рыбы, по меньшей мере с одним олигонуклеотидным праймером, с образованием смеси, где по меньшей мере один олигонуклеотидный праймер комплементарен последовательности нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, и
(b) определение, присутствует ли после амплификации смеси из a) продукт амплификации, где присутствие продукта амплификации указывает на присутствие РНК, ассоциированной с вирусом, и, следовательно, на присутствие вируса в биологическом образце.
В некоторых вариантах осуществления нуклеиновую кислоту на стадии (a) способа, например РНК, выделяют из биологических образцов при помощи твердофазной экстракции, например, очистки на колонке с использованием твердой фазы мембраны с силикагелем. В некоторых вариантах осуществления нуклеиновую кислоту на стадии (a) способа, например РНК, выделяют из биологических образцов при помощи экстракции фенолом/хлороформом.
В способе может использоваться любой подходящий олигонуклеотидный праймер, раскрытый в настоящем изобретении. Как равило, по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b), имеющего длину от 45 нуклеотидов до 3000 нуклеотидов. Однако продукты амплификации даже меньшей или большей длины могут быть соответственно получены с применением способов и олигонуклеотидного праймера, раскрытых в настоящем изобретении.
В некоторых вариантах осуществления по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b) для анализа ПЦР или ОТ-ПЦР, который имеет длину от 100 нуклеотидов до 2500 нуклеотидов. В предпочтительных вариантах осуществления по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b) для анализа ПЦР или ОТ-ПЦР, который имеет длину от 200 нуклеотидов до 1500 нуклеотидов. В более предпочтительных вариантах осуществления по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b) для анализа ПЦР или ОТ-ПЦР, который имеет длину от 300 нуклеотидов до 1000 нуклеотидов.
В некоторых вариантах осуществления по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b) для анализа ОТ-ПЦР в реальном времени, который имеет длину от 45 нуклеотидов до 500 нуклеотидов. В предпочтительных вариантах осуществления по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b) для анализа ОТ-ПЦР в реальном времени, который имеет длину от 50 нуклеотидов до 350 нуклеотидов. В более предпочтительных вариантах осуществления по меньшей мере один олигонуклеотидный праймер стадии (a) способа выбирают для получения продукта амплификации согласно стадии (b) для анализа ОТ-ПЦР в реальном времени, который имеет длину от 55 нуклеотидов до 250 нуклеотидов.
В некоторых вариантах осуществления для амплификации на стадии (b) способа используют ПЦР. В некоторых вариантах осуществления для амплификации на стадии (b) способа используют ОТ-ПЦР. В некоторых вариантах осуществления для амплификации на стадии (b) способа используют ОТ-кПЦР.
В некоторых вариантах осуществления продукт амплификации стадии (b) способа определяют с помощью Саузерн-блоттинга. В некоторых вариантах осуществления продукт амплификации стадии (b) способа определяют с помощью Нозерн-блоттинга. В некоторых вариантах осуществления продукт амплификации стадии (b) способа определяют с помощью спектрофотометрии. В некоторых вариантах осуществления продукт амплификации стадии (b) способа определяют с использованием красителя для ДНК. В некоторых вариантах осуществления продукт амплификации стадии (b) способа определяют путем количественного определения присутствия меченого олигонуклеотидного праймера, например, количественного определения присутствия флуоресцентно-меченного олигонуклеотидного праймера.
В другом аспекте изобретение относится к способу обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии:
(a) секвенирование нуклеиновой кислоты, выделенной из биологического образца рыбы, и
(b) сравнение полученной последовательности нуклеиновой кислоты с последовательностью нуклеиновой кислоты, которая представляет собой или комплементарна референсной последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6, где по меньшей мере 80% идентичность последовательности двух указанных последовательностей указывает на присутствие вируса в биологическом образце.
В некоторых вариантах осуществления нуклеиновую кислоту на стадии (a) способа, например РНК, выделяют из биологических образцов при помощи твердофазной экстракции, например, очистки на колонке с использованием твердой фазы мембраны с силикагелем. В некоторых вариантах осуществления нуклеиновую кислоту на стадии (a) метода, например РНК, выделяют из биологических образцов с помощью экстракции фенолом/хлороформом.
В предпочтительных вариантах осуществления секвенирование на стадии (a) способа дает последовательность ДНК, которую можно непосредственно сравнивать с референсными последовательностями ДНК, выбранными из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6.
В некоторых вариантах осуществления секвенирование на стадии (a) способа выполняют методом секвенирования по Сэнгеру (метод обрыва цепи). В предпочтительных вариантах осуществления секвенирование на стадии (a) способа выполняют методом секвенирования следующего поколения (NGS), предпочтительно секвенирования на платформе Illumina (Solexa), секвенирования Roche 454, Ion Torrent или секвенирования SOLiD (Goodwin S, et al., (2016) Coming of age: Ten years of next-generation sequencing technologies. Nature reviews, Genetics, 17, 333-351).
В некоторых вариантах осуществления по меньшей мере 85% идентичность последовательности двух указанных последовательностей на стадии (b) способа указывает на присутствие вируса в биологическом образце.
В предпочтительных вариантах осуществления по меньшей мере 90% идентичность последовательности двух указанных последовательностей на стадии (b) способа указывает на присутствие вируса в биологическом образце.
В более предпочтительных вариантах осуществления по меньшей мере 95% идентичность последовательности двух указанных последовательностей на стадии (b) способа указывает на присутствие вируса в биологическом образце.
В еще более предпочтительных вариантах осуществления по меньшей мере 98% идентичность последовательности двух указанных последовательностей на стадии (b) способа указывает на присутствие вируса в биологическом образце.
В еще более предпочтительных вариантах осуществления по меньшей мере 99% идентичность последовательности двух указанных последовательностей на стадии (b) способа указывает на присутствие вируса в биологическом образце.
В особенно предпочтительном варианте осуществления 100% идентичность последовательности двух указанных последовательностей на стадии (b) способа указывает на присутствие вируса в биологическом образце.
В другом аспекте изобретение относится к способу обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии:
(a) секвенирование нуклеиновой кислоты, выделенной из биологического образца рыбы, и
(b) трансляцию полученной последовательности нуклеиновой кислоты в аминокислотную последовательность или трансляцию последовательности нуклеиновой кислоты, комплементарной указанной полученной последовательности нуклеиновой кислоты, с получением аминокислотной последовательности, и
(c) сравнение полученной аминокислотной последовательности с референсной последовательностью, выбранной из группы, состоящей из SEQ ID NO 7-11, где по меньшей мере 80% идентичность последовательности двух указанных последовательностей указывает на присутствие вируса в биологическом образце.
Антитела
В другом аспекте изобретение относится к антителу, которое связывает полипептид, где полипептид кодируется последовательностью нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, и/или где полипептид включает аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену.
В некоторых вариантах осуществления полипептид выбран из группы, состоящей из:
(i) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 7;
(ii) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 8;
(iii) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 9;
(iv) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 10; и
(v) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 11.
В некоторых вариантах осуществления антитело является поликлональным антителом. В некоторых вариантах осуществления антитело является моноклональным антителом.
Антитела, раскрытые в настоящем изобретении, могут быть получены методами генетической иммунизации, в которых нативные белки экспрессируются in vivo с обычными посттранскрипционными модификациями, избегая выделения или синтеза антигена. Например, гидродинамическая доставка в хвостовую вену или вену конечности голых плазмидных ДНК векторов экспрессии может использоваться для получения представляющего интерес антигена in vivo у мышей, крыс и кроликов, и, таким образом, индуцирует антигенспецифичные антитела (Tang et al, Nature 356(6365): 152-4 (1992); Tighe et al, Immunol. Today 19(2) 89-97 (1998); Bates et al, Biotechniques, 40(2) 199-208 (2006)). Это обеспечивает эффективное получение высокого титра антигенспецифичных антител. Антитела также могут быть получены методами in vitro. Подходящие примеры включают, без ограничения этим, технологии гибридом, фаговый дисплей, дрожжевой дисплей и т.п.
Наборы
В другом аспекте изобретение относится к набору для обнаружения вируса в биологическом образце рыбы, где набор включает олигонуклеотидный праймер, раскрытый в настоящем изобретении, и/или антитело, раскрытое в настоящем изобретении.
В некоторых вариантах осуществления набор представляет собой тест ОТ-ПЦР в реальном времени, например, набор является тестом ОТ-кПЦР в реальном времени.
В некоторых вариантах осуществления набор предназначен для обнаружения коронавируса в биологическом образце рыбы. В предпочтительных вариантах осуществления набор предназначен для обнаружения торовируса в биологическом образце рыбы. В более предпочтительных вариантах осуществления набор предназначен для обнаружения торовируса в биологическом образце пинагора.
Медицинские применения и вакцины
В другом аспекте изобретение относится к антителу для применения в лечении рыбы, в частности пинагора, от заболевания, вызванного коронавирусной, в частности торовирусной, инфекцией. В предпочтительном варианте осуществления антитело предназначено для применения в лечении рыбы, в частности пинагора, от заболевания, вызванного вирусом, раскрытым в настоящем изобретении (CLuCV). Антитело связывает полипептид, кодируемый последовательностью нуклеиновой кислоты, содержащейся в геноме вируса, раскрытого в настоящем изобретении.
В предпочтительных вариантах осуществления рыбой является пинагор.
В некоторых вариантах осуществления у рыбы наблюдаются следующие симптомы:
(i) повреждение ткани в кишечнике, и/или
(ii) диарея.
В других вариантах осуществления у рыбы дополнительно наблюдаются симптомы анорексии.
Симптомы могут быть определены, как описано выше.
В другом аспекте изобретение относится к применению вируса, раскрытого в настоящем изобретении, для получения вакцины против заболевания, вызванного указанным вирусом.
В другом аспекте изобретение относится к вакцине, включающей:
(i) последовательность нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении;
(ii) последовательность нуклеиновой кислоты, раскрытую в настоящем изобретении;
(iii) вирусный полипептид, кодируемый последовательностью нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении;
(iv) вирусный полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80%, по меньшей мере на 90% или по меньшей мере на 95% идентична любой из SEQ ID NO 7-11, или которая является любой из SEQ ID NO 7-11, или их вариантом, содержащим консервативную замену; или
(v) вирус, раскрытый в настоящем изобретении.
В некоторых вариантах осуществления вакцина содержит несколько вирусных полипептидов, например, первый полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80% идентична SEQ ID NO: 7, и второй полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80% идентична SEQ ID NO: 8. Вакцина может содержать один, два, три, четыре или пять вирусных полипептидов.
Вакцина для защиты рыбы, в частности пинагора, от заболевания, вызванного коронавирусной, в частности торовирусной, инфекцией. В предпочтительном варианте осуществления вакцина предназначена для защиты рыбы, в частности пинагора, от заболевания, вызванного заражением вирусом, раскрытым в настоящем изобретении (CLuCV).
В некоторых вариантах осуществления, где вакцина включает последовательность нуклеиновой кислоты, содержащуюся в геноме вируса, раскрытого в настоящем изобретении, указанная последовательность нуклеиновой кислоты включает по меньшей мере одну из ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5 согласно любому из их вариантов осуществления, раскрытых в настоящем изобретении. В предпочтительных вариантах осуществления, где вакцина включает последовательность нуклеиновой кислоты, содержащуюся в геноме вируса, раскрытого в настоящем изобретении, указанная последовательность нуклеиновой кислоты включает по меньшей мере ORF-1, ORF-2, ORF-3 и ORF-4 согласно любому из их вариантов осуществления, раскрытых в настоящем изобретении. В некоторых вариантах осуществления, где вакцина включает последовательность нуклеиновой кислоты, содержащуюся в геноме вируса, раскрытого в настоящем изобретении, указанная последовательность нуклеиновой кислоты является или комплементарна последовательности нуклеиновой кислоты, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична последовательности вирусного генома согласно SEQ ID NO: 6 (CLuCV).
Нуклеиновой кислотой может быть ДНК или РНК.
В некоторых вариантах осуществления, где вакцина включает вирусный полипептид, кодируемый последовательностью нуклеиновой кислоты, которая содержится в геноме вируса, раскрытого в настоящем изобретении, указанный полипептид выбран из группы, состоящей из:
(i) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 7;
(ii) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 8;
(iii) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 9;
(iv) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 10; и
(v) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична, или представляет собой, SEQ ID NO: 11.
В некоторых вариантах осуществления, где вакцина включает вирус, раскрытый в настоящем изобретении, геном указанного вируса включает последовательность нуклеиновой кислоты, которая является РНК последовательностью нуклеиновой кислоты, включающей по меньшей мере одну из ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5 согласно любому из их вариантов осуществления, раскрытых в настоящем изобретении. В некоторых вариантах осуществления, где вакцина включает вирус, раскрытый в настоящем изобретении, геном указанного вируса включает последовательность нуклеиновой кислоты, которая является РНК последовательностью нуклеиновой кислоты, которая представляет собой или комплементарна последовательности нуклеиновой кислоты, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно на 98%, особенно предпочтительно на 99% или даже на 100% идентична последовательности вирусного генома согласно SEQ ID NO: 6 (CLuCV).
В некоторых вариантах осуществления вакцина включает количество антигена, которое составляет в пределах 0,05-1,0 мг/мл, такое как от 0,1 до 0,5 мг/мл, от 0,15 до 0,4 мг/мл или от 0,2 до 0,3 мг/мл. Вакцина может быть предназначена для введения в дозах 0,005-0,5 мг/индивид, предпочтительно 0,01-0,05 мг/индивид, более предпочтительно 0,01-0,02 мг/индивид.
В некоторых вариантах осуществления вакцина включает количество антигена, соответствующее TCID50 105-1010 на дозу, предпочтительно TCID50 106-109 на дозу.
Вакцина может находиться в форме суспензии вируса или может быть лиофилизированной. В лиофилизированную вакцину может потребоваться добавить один или больше стабилизаторов. Подходящие стабилизаторы представляют собой, например, углеводы, такие как сорбит, маннит, крахмал, сахарозу, декстран; белоксодержащие вещества, такие как бычья сыворотка или обезжиренное молоко; и буферные вещества, такие как фосфаты щелочных металлов.
Вакцина согласно изобретению также может быть в составе, включающем адъювант. Примерами адъювантов, часто применяемых при разведении рыбы и моллюсков, является мурамилдипептиды, липолисахариды, некоторые глюканы и гликаны, минеральное масло, Montanide™ и Carbopol®. Обзор вспомогательных веществ, подходящих для применения в вакцинах для рыб, приведен в обзоре Sommerset (Expert Rev. Vaccines 4(1), 89-101 (2005)).
Вакцина согласно изобретению может дополнительно содержать подходящий фармацевтический носитель. В некоторых вариантах осуществления вакцина изготовлена в форме эмульсии типа вода в масле. Вакцина также может содержать так называемый "носитель". Носителем является такое средство, к которому прикреплен антиген, но не связан с ним ковалентно. Такие носители, помимо прочего, представляют собой биоразлагаемые нано/микрочастицы или капсулы PLGA (сополимер лактида и гликолевой кислоты), альгината или хитозана, липосомы, ниосомы, мицеллы, множественные эмульсии и макрозоли, известные в уровне техники. Особой формой такого носителя, в котором антиген частично заключен в носитель, является так называемый ISCOM.
Кроме того, вакцина может включать одно или больше подходящих поверхностно-активных соединений или эмульгаторов, например, Cremophore®, Tween® и Span®. Кроме того, могут использоваться адъюванты, такие как интерлейкин, CpG и гликопротеины.
В некоторых вариантах осуществления вакцина представлена в виде корма для рыбы, при этом указанный корм может быть, например, гранулированным или экструдированным кормом.
В другом аспекте изобретение относится к молекуле интерферирующей РНК (иРНК) для применения в лечении рыбы, в частности пинагора, от заболевания, вызванного коронавирусной, в частности торовирусной, инфекцией. В предпочтительном варианте осуществления иРНК предназначена для применения в лечении рыбы, в частности пинагора, от заболевания, вызванного вирусом, раскрытым в настоящем изобретении (CLuCV). В конкретном варианте молекула иРНК включает по меньшей мере 12 (предпочтительно смежных) нуклеотидов из, или комплементарных, последовательности нуклеиновой кислоты, содержащейся в геноме вируса, раскрытого в настоящем изобретении.
В предпочтительных вариантах осуществления рыбой является пинагор.
В некоторых вариантах осуществления у рыбы наблюдаются следующие симптомы:
(i) повреждение ткани в кишечнике, и/или
(ii) диарея.
В других вариантах осуществления у рыбы дополнительно наблюдаются симптомы анорексии.
Симптомы могут быть определены, как описано выше.
В некоторых вариантах осуществления молекула иРНК включает по меньшей мере 12 (предпочтительно смежных) нуклеотидов из, или комплементарных, последовательности нуклеиновой кислоты, включающей ORF-1, ORF-2, ORF-3, ORF-4 или ORF-5 согласно любому из соответствующих вариантов осуществления, раскрытых в настоящем изобретении. В некоторых вариантах осуществления молекула иРНК включает по меньшей мере 12 (предпочтительно смежных) нуклеотидов из, или комплементарных, последовательности нуклеиновой кислоты, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно на 98%, особенно предпочтительно на 99% или даже на 100% идентична последовательности вирусного генома согласно SEQ ID NO: 6 (CLuCV).
Последовательности
Последовательности, указанные в настоящем описании, являются следующими (см. также Фигуры и список последовательностей):
Таблица 1
Дополнительные полезные праймеры, указанные в настоящем изобретении, представлены в Таблице 2 ниже:
Другие олигонуклеотидные праймеры, указанные в настоящем изобретении, представлены в Таблице 3:
Примеры
Следующие примеры иллюстрируют настоящее изобретение. Они предназначены для помощи в понимании изобретения и не должны рассматриваться в качестве какого-либо ограничения объема изобретения.
Пример 1: Идентификация CLuCV
Образцы рыбы получали на предприятии по разведению пинагора (Cyclopterus lumpus), которое столкнулось с высокой смертностью (60-80%). Образцы исследовали с помощью стандартного ОТ-ПЦР в реальном времени и гистологического исследования. С помощью ОТ-ПЦР в реальном времени не обнаружили никаких известных патогенов. Гистологическое исследование показало симптомы повреждения ткани в кишечнике у пораженных особей, однако при этом не наблюдали никаких потенциально патогенных бактерий и микропаразитов.
Суммарную РНК выделяли из погибающей рыбы с использованием методики экстракции фенолом/хлороформом (набор Qiagen RNeasy® 96 Universal Tissue Kit) и использовали эту РНК в качестве матрицы в анализе методом секвенирования следующего поколения (NGS). Анализ NGS, который проводили в компании BaseClear (https://www.baseclear.com/), дал приблизительно 98 миллионов чтений последовательностей, большинство из которых происходили из транскриптома хозяина. Приблизительно 28600 собранных чтений, полученных в анализе NGS, как обнаружили, не показали никакого значимого совпадения нуклеотидов в базе GenBank Национального центра биотехнологической информации США (NCBI). На основе этих чтений авторами изобретения была идентифицирована и исследована последовательность нового вируса, называемого в настоящем изобретении коронавирусом Cyclopterus lumpus или CLuCV. Последовательности ДНК, полученные в результате анализа NGS, предоставлены на Фигурах 2-5 (см. также SEQ ID NO: 1-6). Однако следует понимать, что в вирусе CLuCV присутствуют соответствующие последовательности РНК.
Было установлено, что последовательность CLuCV имеет длину 24613 нуклеотидов и содержит пять возможных открытых рамок считывания (ORF). Схема показана на Фигуре 1. Родство между CLuCV и другими известными вирусами может быть определено только при сравнении транслированных аминокислотных последовательностей. Длина и организация генома в сочетании с последующим анализом последовательности указывают, что это - новый торовирус из семейства Coronaviridae, к которому наиболее близок вирус Берн (выделенный у лошадей).
Предварительные результаты указывают, что, по меньшей мере в Норвегии, CLuCV демонстрирует значимое присутствие в аквакультурах пинагора.
Патология, связанная с CLuTV у пинагора, показана на Фигурах 9-12. Секционный материал пинагора окрашивали с использованием гистологической окраски гематоксилином и эозином (Г-Э). Фигура 9 представляет собой цельный срез пораженного пинагора, на котором показано накопление жидкости в желудке (стрелка). Фигура 10 представляет собой срез кишечника пинагора, на котором показано накопление слизи и выброс клеточного содержимого (стрелки). Фигура 11 представляет собой срез кишечника пинагора, на котором показано накопление слизи (стрелка) и выброс клеточного содержимого. Фигура 12 представляет собой срез кишечника пинагора, на котором показано накопление слизи (стрелка).
Пример 2: Анализ нуклеотидой последовательности CLuCV
При выполнении стандартного поиска нуклеотидных последовательностей в BLAST с использованием последовательности CLuCV, в базе GeneBank NCBI не было обнаружено никаких совпадений. При изменении параметров поиска с включением в поиск более отличающихся последовательностей, были обнаружены некоторые области последовательности из других известных вирусов. Процент идентичности нуклеотидной последовательности ("Seq. Id.") между этими областями CLuCV и областями из других известных вирусов показан в Таблице 2.
Таблица 2. Наилучшее совпадение нуклеотидной последовательности, обнаруженное между CLuCV и известными вирусами в GeneBank NCBI
(NCBI GeneBank)
Id. (%)
Пример 3: Анализ аминокислотной последовательности CLuCV
Трансляция последовательностей ORF CLuCV дает в общей сложности пять потенциальных белков. Идентичность аминокислотных последовательностей ORF CLuCV с другими известными белками других вирусов показана в Таблице 3. Представлены только наилучшие совпадения для каждой ORF.
Таблица 3. Наилучшие совпадения аминокислотной последовательности, обнаруженные между CLuCV и известными вирусами в NCBI GeneBank
ORF-1 соответствует полипротеину. ORF-2 соответствует спайковому гликопротеину. ORF-3 соответствует мембранному гликопротеину. ORF-4 соответствует белку нуклеокапсида. ORF-5 соответствует неизвестному белку.
Пример 4: Оценка присутствия CLuCV в культурных популяциях пинагора
Были разработаны три отдельных анализа ОТ-ПЦР в реальном времени (TCL-ORF-1, TCL-M и TCL-S) с использованием праймеров согласно SEQ ID NO: 12-14, 15-17 и 18-20, соответственно. TCL-ORF-1 направлен на ORF-1, TCL-M направлен на ORF-3 (мембранный белок), и TCL-S направлен на ORF-2 (спайковый гликопротеин). С помощью всех трех анализов было подтверждено, что исходный материал образцов рыбы был положительным на CLuCV.
Для более широкого исследования анализ TCL-ORF-1 выбрали для оценки присутствия CLuCV в существующих культурных популяциях пинагора. Результаты скрининга различных популяций пинагора в Норвегии показаны в Таблице 4.
Таблица 4. Результаты тестирования материала NGS и полевого материала
Как показано в таблице, в некоторых популяциях пинагора, в особенности в округах Тренделаг и Рогаланд, наблюдается высокая распространенность CLuCV.
Ввиду представленного в настоящем изобретении описания нужно понимать, что настоящее изобретение также охватывает следующие объекты:
1. Нуклеиновая кислота, где последовательность указанной нуклеиновой кислоты включает:
(a) по меньшей мере одну последовательность открытой рамки считывания (ORF), выбранной из группы, состоящей из ORF-1, ORF-2, ORF-3, ORF-4 и ORF-5, или
(b) последовательность, комплементарную ей;
где
ORF-1 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 80% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
2. Нуклеиновая кислота по пункту 1, где:
ORF-1 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 85% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
3. Нуклеиновая кислота по пункту 1 или 2, где:
ORF-1 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 90% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
4. Нуклеиновая кислота по любому из пунктов 1-3, где:
ORF-1 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 95% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
5. Нуклеиновая кислота по любому из пунктов 1-4, где:
ORF-1 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 98% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
6. Нуклеиновая кислота по любому из пунктов 1-5, где:
ORF-1 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 по меньшей мере на 99% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
7. Нуклеиновая кислота по любому из пунктов 1-6, где:
ORF-1 на 100% идентична последовательности нуклеиновой кислоты SEQ ID NO: 1,
ORF-2 на 100% идентична последовательности нуклеиновой кислоты SEQ ID NO: 2,
ORF-3 на 100% идентичен последовательности нуклеиновой кислоты SEQ ID NO: 3,
ORF-4 на 100% идентична последовательности нуклеиновой кислоты SEQ ID NO: 4, и
ORF-5 на 100% идентична последовательности нуклеиновой кислоты SEQ ID NO: 5.
8. Нуклеиновая кислота, где последовательность указанной нуклеиновой кислоты по меньшей мере на 80% идентична соответствующей последовательности, присутствующей в SEQ ID NO: 6, предпочтительно по меньшей мере на 85% идентична, более предпочтительно по меньшей мере на 90% идентична, еще более предпочтительно по меньшей мере на 95% идентична, еще более предпочтительно по меньшей мере на 98% идентична, еще более предпочтительно по меньшей мере на 99% идентична, и особенно предпочтительно на 100% идентична.
9. Нуклеиновая кислота, где последовательность указанной нуклеиновой кислоты по меньшей мере на 80% идентична соответствующей последовательности, присутствующей в последовательности, которая комплементарна SEQ ID NO: 6, предпочтительно по меньшей мере на 85% идентична, более предпочтительно по меньшей мере на 90% идентична, еще более предпочтительно по меньшей мере на 95% идентична, еще более предпочтительно по меньшей мере на 98% идентична, еще более предпочтительно по меньшей мере на 99% идентична, и особенно предпочтительно на 100% идентична.
10. Нуклеиновая кислота по пунктам 8 или 9, где последовательность указанной нуклеиновой кислоты включает 200 нуклеотидов или меньше.
11. Нуклеиновая кислота по любому из пунктов 8-10, где последовательность указанной нуклеиновой кислоты включает по меньшей мере 60 нуклеотидов.
12. Нуклеиновая кислота по пункту 11, где последовательность указанной нуклеиновой кислоты включает по меньшей мере 100 нуклеотидов, предпочтительно по меньшей мере 150 нуклеотидов.
13. Нуклеиновая кислота по пункту 8 или 9, где последовательность указанной нуклеиновой кислоты включает по меньшей мере 200 нуклеотидов.
14. Применение нуклеиновой кислоты по любому из пунктов 1-13:
(a) в качестве гибридизационного зонда;
(b) для обнаружения вируса, раскрытого в настоящем изобретении, в биологическом образце рыбы, в частности пинагора;
(c) в способе обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, в частности пинагора;
(d) в способе обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, в частности пинагора, согласно любому из соответствующих способов, раскрытых в настоящем изобретении;
(e) для изготовления вакцины для защиты рыбы, в частности пинагора, от заболевания, вызванного коронавирусной, в частности торовирусной, инфекцией; или
(f) для изготовления вакцины для защиты рыбы, в частности пинагора, от заболевания, вызванного вирусом, раскрытым в настоящем изобретении, таким как вирус по пунктам 18-25 ниже.
15. Нуклеиновая кислота по пункту 1, где последовательность указанной нуклеиновой кислоты включает по меньшей мере ORF-1, ORF-2, ORF-3 и ORF-4, как определено в любом из пунктов 1-7, или последовательность, которая комплементарна ей.
16. Нуклеиновая кислота по пункту 1, где последовательность указанной нуклеиновой кислоты по меньшей мере на 80% идентична вирусному геному согласно SEQ ID NO: 6, предпочтительно по меньшей мере на 85% идентична, более предпочтительно по меньшей мере на 90% идентична, еще более предпочтительно по меньшей мере на 95% идентична, еще более предпочтительно по меньшей мере на 98% идентична, еще более предпочтительно по меньшей мере на 99% идентична, и особенно предпочтительно на 100% идентична.
17. Нуклеиновая кислота по пункту 1, где последовательность указанной нуклеиновой кислоты по меньшей мере на 80% идентична последовательности, которая комплементарна SEQ ID NO: 6, предпочтительно по меньшей мере на 85% идентична, более предпочтительно по меньшей мере на 90% идентична, еще более предпочтительно по меньшей мере на 95% идентична, еще более предпочтительно по меньшей мере на 98% идентична, еще более предпочтительно по меньшей мере на 99% идентична, и особенно предпочтительно на 100% идентична.
18. Вирус, в частности вирус, который инфицирует и способен вызывать гибель пинагора (такого как Cyclopterus lumpus), где вирусный геном включает последовательность нуклеиновой кислоты по любому из пунктов 1-13 и 15-17, где указанная последовательность нуклеиновой кислоты, содержащаяся в вирусном геноме, содержит основание урацил (U) вместо основания тимина (T).
19. Вирус по пункту 18, где заражение пинагора вирусом вызывает у рыбы следующие симптомы:
(i) повреждение ткани в кишечнике, и/или
(ii) диарею.
20. Вирус, включающий одну или больше, предпочтительно две или больше, более предпочтительно три или больше, более предпочтительно четыре или больше, еще более предпочтительно пять из ORF 1-5, где указанные ORF 1-5 кодируют вирусные полипептиды SEQ ID NO 7-11, соответственно, или вирусные полипептиды, которые по меньшей мере на 80% идентичны SEQ ID NO 7-11, соответственно.
21. Вирус по пункту 20, где указанные ORF 1-5 кодируют вирусные полипептиды SEQ ID NO 7-11, соответственно, или вирусные полипептиды, которые по меньшей мере на 90% идентичны SEQ ID NO 7-11, соответственно.
22. Вирус по пункту 20, где указанные ORF 1-5 кодируют вирусные полипептиды SEQ ID NO 7-11, соответственно, или вирусные полипептиды, которые по меньшей мере на 95% идентичны SEQ ID NO 7-11, соответственно.
23. Вирус по любому из пунктов 20-22, где указанные ORF 1-5 кодируют вирусные полипептиды, которые представляют собой содержащие консервативные замены варианты SEQ ID NO 7-11, соответственно.
24. Вирус по любому из пунктов 18-23, где вирус является оболочечным вирусом.
25. Вирус по любому из пунктов 18-24, где вирус является коронавирусом, предпочтительно торовирусом.
26. Олигонуклеотидный праймер, включающий последовательность по меньшей мере из 9 последовательных нуклеотидов, предпочтительно по меньшей мере из 12 последовательных нуклеотидов, более предпочтительно по меньшей мере из 15 последовательных нуклеотидов, и особенно предпочтительно по меньшей мере из 18 последовательных нуклеотидов, где указанная последовательность содержится в геноме вируса по любому из пунктов 18-25, предпочтительно при условии, что указанный праймер не включает SEQ ID NO 21-23.
27. Олигонуклеотидный праймер, включающий последовательность по меньшей мере из 9 последовательных нуклеотидов, предпочтительно по меньшей мере из 12 последовательных нуклеотидов, более предпочтительно по меньшей мере из 15 последовательных нуклеотидов, и особенно предпочтительно по меньшей мере из 18 последовательных нуклеотидов, где указанная последовательность комплементарна последовательности нуклеиновой кислоты, содержащейся в геноме вируса по любому из пунктов 18-25, предпочтительно при условии, что указанный праймер не включает SEQ ID NO 21-23.
28. Олигонуклеотидный праймер, включающий последовательность по меньшей мере из 9 последовательных нуклеотидов, предпочтительно по меньшей мере из 12 последовательных нуклеотидов, более предпочтительно по меньшей мере из 15 последовательных нуклеотидов, и особенно предпочтительно по меньшей мере из 18 последовательных нуклеотидов, где указанная последовательность по меньшей мере на 80% идентична последовательности, содержащейся в SEQ ID NO: 6, предпочтительно при условии, что указанный праймер не включает SEQ ID NO 21-23.
29. Олигонуклеотидный праймер, включающий последовательность по меньшей мере из 9 последовательных нуклеотидов, предпочтительно по меньшей мере из 12 последовательных нуклеотидов, более предпочтительно по меньшей мере из 15 последовательных нуклеотидов, и особенно предпочтительно по меньшей мере из 18 последовательных нуклеотидов, где указанная последовательность по меньшей мере на 80% идентична последовательности, содержащейся в последовательности, комплементарной SEQ ID NO: 6, предпочтительно при условии, что указанный праймер не включает SEQ ID NO 21-23.
30. Олигонуклеотидный праймер по пункту 28 или 29, где указанный процент идентичности последовательности составляет по меньшей мере 85%, предпочтительно по меньшей мере 90%, более предпочтительно по меньшей мере 95%, еще более предпочтительно по меньшей мере 98%, еще более предпочтительно по меньшей мере 99% и особенно предпочтительно 100%.
31. Олигонуклеотидный праймер по любому из пунктов 26-30, где указанный олигонуклеотидный праймер имеет длину 9-60 нуклеотидов, предпочтительно длину 12-40 нуклеотидов, более предпочтительно длину 15-30 нуклеотидов, особенно предпочтительно длину 18-25 нуклеотидов.
32. Олигонуклеотидный праймер по любому из пунктов 26-28, где указанная последовательность, содержащаяся в нем, представляет собой часть или комплементарна части референсной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 или SEQ ID NO: 6.
33. Олигонуклеотидный праймер, который включает по меньшей мере 9 последовательных нуклеотидов, предпочтительно по меньшей мере 12 последовательных нуклеотидов, более предпочтительно по меньшей мере 15 последовательных нуклеотидов и особенно предпочтительно по меньшей мере 18 последовательных нуклеотидов последовательности, которая по меньшей мере на 80% идентична последовательности, которая представляет собой или комплементарна последовательности, выбранной из группы, состоящей из SEQ ID NO: 12 - SEQ ID NO: 80, предпочтительно при условии, что указанный праймер не включает SEQ ID NO 21-23.
34. Применение по меньшей мере одного олигонуклеотидного праймера в способе обнаружения вируса по любому из пунктов 18-25, где по меньшей мере один праймер включает последовательность по меньшей мере из 9 последовательных нуклеотидов, предпочтительно по меньшей мере из 12 последовательных нуклеотидов, более предпочтительно по меньшей мере из 15 последовательных нуклеотидов, и особенно предпочтительно по меньшей мере из 18 последовательных нуклеотидов, и где указанная последовательность комплементарна последовательности нуклеиновой кислоты, которая содержится в геноме указанного вируса, предпочтительно при условии, что указанный праймер не включает SEQ ID NO 21-23.
35. Применение по меньшей мере одной нуклеиновой кислоты по любому из пунктов 1-13 или 15-17, или по меньшей мере одного олигонуклеотидного праймера по любому из пунктов 26-34, в способе обнаружения вируса рыб, необязательно где вирус инфицирует и способен вызывать гибель пинагора.
36. Применение по пункту 35, где указанная нуклеиновая кислота или указанный олигонуклеотидный праймер применяется в способе обнаружения вируса по любому из пунктов 18-24.
37. Способ обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, в частности пинагора, включающий следующие стадии:
(a) контакт нуклеиновой кислоты, выделенной из биологического образца рыбы, по меньшей мере с одним олигонуклеотидным праймером, с получением смеси, где по меньшей мере один олигонуклеотидный праймер комплементарен последовательности нуклеиновой кислоты, которая содержится в геноме вируса по любому из пунктов 18-24,
(b) определение, присутствует ли при амплификации смеси a) продукт амплификации, где присутствие продукта амплификации указывает на присутствие РНК, ассоциированной с вирусом, и, следовательно, на присутствие вируса в биологическом образце.
38. Способ по пункту 37, где олигонуклеотидный праймер является олигонуклеотидным праймером по любому из пунктов 26-34.
39. Способ по пунктам 37 или 38, где нуклеиновую кислоту на стадии (a) способа, например РНК, выделяют из биологических образцов при помощи твердофазной экстракции, например, очистки на колонке с помощью твердой фазы мембраны с силикагелем.
40. Способ по любому из пунктов 37-38, где нуклеиновую кислоту на стадии (a) способа, например РНК, выделяют из биологических образцов с помощью экстракции фенолом/хлороформом.
41. Способ обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, в частности пинагора, включающий следующие стадии:
(a) секвенирование нуклеиновой кислоты, выделенной из биологического образца рыбы, и
(b) сравнение полученной последовательности нуклеиновой кислоты с последовательностью нуклеиновой кислоты, которая представляет собой или комплементарна референсной последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6, где по меньшей мере 80% идентичность последовательности между двумя последовательностями указывает на присутствие вируса в биологическом образце.
42. Способ по пункту 41, где процент идентичности последовательности между двумя последовательностями, который указывает на присутствие вируса в биологическом образце, составляет по меньшей мере 85%, предпочтительно по меньшей мере 90%, более предпочтительно по меньшей мере 95%, еще более предпочтительно по меньшей мере 98%, еще более предпочтительно по меньшей мере 99% и наиболее предпочтительно 100%.
43. Вирусный полипептид, включающий аминокислотную последовательность, которая по меньшей мере на 80% идентична любой из SEQ ID NO 7-11.
44. Вирусный полипептид по пункту 43, включающий аминокислотную последовательность, которая по меньшей мере на 90% идентична любой из SEQ ID NO 7-11.
45. Вирусный полипептид по пункту 43 или 44, включающий аминокислотную последовательность, которая по меньшей мере на 95% идентична любой из SEQ ID NO 7-11.
46. Вирусный полипептид по любому из пунктов 43-45, включающий аминокислотную последовательность, включающую любую из SEQ ID NO: 7-11, или ее вариант, содержащий консервативную замену.
47. Вирусный полипептид по любому из пунктов 43-45, включающий аминокислотную последовательность, включающую SEQ ID NO 7-11.
48. Антитело, которое связывает полипептид, где полипептид кодируется последовательностью нуклеиновой кислоты, которая содержится в геноме вируса по любому из пунктов 18-25, или является вирусным полипептидом, кодируемым нуклеиновой кислотой по любому из пунктов 1-13 и 15-17.
49. Антитело, которое связывает полипептид, выбранный из группы, состоящей из:
(i) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична SEQ ID NO: 7;
(ii) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична SEQ ID NO: 8;
(iii) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична SEQ ID NO: 9;
(iv) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична SEQ ID NO: 10; и
(v) полипептида, включающего аминокислотную последовательность, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно по меньшей мере на 98%, особенно предпочтительно по меньшей мере на 99% или даже на 100% идентична SEQ ID NO: 11.
50. Набор для обнаружения вируса в биологическом образце рыбы, где набор включает нуклеиновую кислоту по любому из пунктов 1-13 или 15-17, олигонуклеотидный праймер по любому из пунктов 26-34 и/или антитело по пункту 48 или по пункту 49.
51. Набор по пункту 50, где набор подходит для поведения или предназначен для применения при проведении анализа ОТ-ПЦР в реальном времени.
52. Антитело по пункту 49 или по пункту 48 для применения в лечении рыбы, инфицированной вирусом, в частности пинагора.
53. Антитело по пункту 452, где вирус является вирусом по любому из пунктов 18-25.
54. Применение вируса по любому из пунктов 18-25 для изготовления вакцины.
55. Вакцина, включающая:
(i) последовательность нуклеиновой кислоты, которая содержится в геноме вируса по любому из пунктов 18-25;
(ii) последовательность нуклеиновой кислоты по любому из пунктов 1-13 или 15-17;
(iii) вирусный полипептид, кодируемый последовательностью нуклеиновой кислоты, содержащейся в геноме вируса по любому из пунктов 18-25;
(iv) вирусный полипептид, кодируемый последовательностью нуклеиновой кислоты по любому из пунктов 1-13 или 15-17;
(v) вирусный полипептид по любому из пунктов 43-47, или
(vi) вирус по любому из пунктов 18-25.
56. Вакцина по пункту 55, где последовательность нуклеиновой кислоты является последовательностью нуклеиновой кислоты, указанной в любом из пунктов 1-13 или 17, где указанная последовательность нуклеиновой кислоты содержит основание урацил (U) вместо основания тимина (T).
57. Молекула интерферирующей РНК (иРНК) для применения в лечении рыбы, инфицированной вирусом, в частности пинагора, где молекула иРНК включает по меньшей мере 12 (предпочтительно смежных) нуклеотидов, или комплементарна, последовательности нуклеиновой кислоты, содержащейся в геноме вируса по любому из пунктов 18-25.
58. Молекула интерферирующей РНК (иРНК) для применения в лечении рыбы, инфицированной вирусом, в частности пинагора, где молекула иРНК включает по меньшей мере 12 (предпочтительно смежных) нуклеотидов, или комплементарна, последовательности нуклеиновой кислоты, которая по меньшей мере на 80%, предпочтительно по меньшей мере на 85%, более предпочтительно по меньшей мере на 90%, еще более предпочтительно по меньшей мере на 95%, еще более предпочтительно на 98%, особенно предпочтительно на 99% или даже на 100% идентична последовательности вирусного генома согласно SEQ ID NO: 6 (CLuCV).
59. Интерферирующая РНК по пункту 58 или по пункту 57, где указанная молекула иРНК включает по меньшей мере 15 и более предпочтительно по меньшей мере 18 из указанных (предпочтительно смежных) нуклеотидов.
Специалисты в данной области поймут или смогут определить, при использовании не более чем рутинных экспериментов и/или общедоступных знаний, многочисленные эквиваленты конкретных аспектов, объектов и вариантов осуществления, раскрытых в настоящем изобретении как в примерах, так и в основной части всего патентного описания. Предполагается, что такие эквиваленты входят в объем данного изобретения и должны быть охвачены следующей формулой изобретения, или любой формулой изобретения, которой могут придерживаться на основании настоящего описания.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Pharmaq Analytiq
<120> Новый коронавирус рыб
<130> ZOE20235PCT
<160> 80
<170> BiSSAP 1.3.6
<210> 1
<211> 17601
<212> ДНК
<213> Вирусы
<220>
<223> нуклеотидная последовательность ORF-1 ("n" в положениях 2490-2503, 2523,
2524 и 9771 обозначает любое основание)
<400> 1
atgaaaaaaa ttgaagatgc tttaggaaca cttaaacctg cctttaaggc acacatagac 60
tctctacctc ctttcatggg aaaactagcc acaatgctag ctgagacaag acgaggaaaa 120
actccccctc ttttgatcta tgtcatatca acaattttgg aaacgaatat tacagttcat 180
tatgtcagcc acacaataac ttcataccat tcttcaaacg cacacactca cactcatgaa 240
ttcgactcag aagattacac tcctgacaat cagattttaa ctaaagtcaa ccgacatggt 300
aatgacctta atcattcata tatccatggc gccaccaata tgtacaaccc tgtctaccag 360
agacacccac caaacatgtg ctacatgctt acgggactgt acatgttgtc aggcttacag 420
gaattgtatg ccatggctga agacaacttg acaacatgcc aaatcaacct tctcagatgt 480
ctgtttgatt tgaatcaaga tgaatttgat gtagattaca catttgtaat atacactcct 540
agcaaatctc aggagtgtgc cttcaaatat cttcaggaga tagtccacca ctgtgaactc 600
accattttta gacatacaac aacaagtgtc ttcagttgca acaaatgcaa ccatgtggaa 660
actgtcattt ctagttgttc tttgaatctt gtatatataa ctgattctat tgaaaaagca 720
tttcaaccca ctgtagaagc taataccgac tacatgtgtg aaaactgcgg tctacgcgac 780
cataaactta aaacaacagt aacaaatcca gacctccgtt tggcacaact aaactatcca 840
acggattcaa aatacactat ctttcttgat gaacaagctc cttttgtctt ccattccatt 900
gcaaaacacg ttggaactgc taattccgga cattggagtg cattgaatgt aaattcagat 960
atgttgtcag actctaacga gcgacaacat tattatacaa cacctagtat tgttttgctt 1020
gcatttcttc ctgaggaaga gctacagaac attagaaatt catcacctct tcaggaccat 1080
caaccagatg atgttgaaga cgttgaatct cccctaccag gttctatatt ttatactaca 1140
gatgacattt tttctactaa gagtctatcc atagctcact gtgtagcccg cgactttcac 1200
atgtccggcg gcatagcaaa aattttctca gataaattcg gctctaaaac tttcttgaaa 1260
tcacaaaacc ccgttatagg cggtttttcc attttactta gagagtgtcg tgacatgtac 1320
tacctcgtca caaaagagaa aacttcagat aagccgacat accaagacct taaaaactcc 1380
ttgggttcta tgacagagaa tttggttcgc aagaatcata atactctttc aattccatat 1440
ataggctgtg ggattgatgg tttacaatgg gcaaccgttg aaaaacaagt caaagaaatt 1500
gtctgcgctc gaggtattga tgtaacagtc caccacctcg aaaatgaagt taaacacact 1560
ccagaacaac aaacagcttc tgacaattca gtcaaattag ttcaaaaact ctttacagaa 1620
acacctcaag ctatacctat agttgttcct tcagatgact cagacagcga catagatgag 1680
tcagccgatg tctttttacc aggacctgaa tctgaatctg attcaaaatc agaatctggg 1740
tcagattacg actttaaatc ggcttctgaa ccagaagatg aattggagcc cactccaatc 1800
tctgaacttg aactaacacc agcgtcaagt ttaactgtag agtccgatga caatccagac 1860
acaagccagg aaacattacc agaatcaaac tctgaggaaa ccaaacctga gcaaacacct 1920
gacacaacat caaaggtgtc ctccgattcg aagcttgatc cacaatcaga attagaagaa 1980
gaactggcca acaaaccaga atcggcttct gaaccacaat ctgagactga atcgagttct 2040
gaatctgaag aggagcttga gccacaatca gaatcagagg aagaaccggc caacaaaccg 2100
gaatctcctt ctgaatcaca atctgaaaat ggatcgagtt ctgaacccga agaggagtct 2160
gaaaaaccat cggagtctgc tgaaacagca acagaggata gcccggaaac aacaccggaa 2220
acaaccttag agttaaccac acaactcaaa cctgcttcag aatctgacga caaaccggac 2280
acaccagcac catcaccttc accaattcaa ccagagaaaa acttggacac cacccctgaa 2340
caaacttcac aaccaaccac acaactggaa ttgacgttag aaacacagga acaaccagac 2400
actacaccag aagtgccatc tgtctcagaa gataaaccag acacacttga agaatcttct 2460
gaatcaacac cagaactctc agaattggan nnnnnnnnnn nnnccaccag taccaagacc 2520
tanngaacca gacctccaac acctagacca agatctgcac gtggagctag gactagatcc 2580
tgtgctggca cacctatacc agttattttt gatattatag ataatacaag ccagcctcaa 2640
gttccccttg acttcccaga agcactgcaa gaactgaaca aacctagtga agtaatccca 2700
gcggctagtg aaaaacctgt ggaaaaacaa ataatccata gttttgtaag tgtcgaaaca 2760
ccttgcaaac ccaaagccac taaagttacg aattatgtag ctgcacaatc taatgcaatt 2820
ctaaattgta ttaaggcttt cattcctagc aacccgcttt cactattcaa tagaaaacca 2880
gcttttagaa aaataatatt cactgaagac acttcagaac cagatagcga tgatgatgat 2940
tgtgaataca ctccaccaac atcaccattc cctgaacttc tggcattggt agatgaagac 3000
attgaagtag aacaaactca atctgtaatt ccaaaaacag actctgcttc aattgtggag 3060
gatcttaaaa aacaagaatc ctctactttg tcattggaca ccaacacatc gaaacctaca 3120
agctctccgc gaagacagcc tagggaagta gaaagtgttg atgaatccag tgatgactca 3180
tctaaaccaa aaacaatttc aacattagac aaacctgcta tgaatagtga cacgaaacct 3240
acagactctt cgcgaaaaga gcctctggaa gtaccagtta ctacatcttt aagcacccct 3300
gctaaaaacc aagataaaaa atcttcaaaa tctgcaaaag taataaaaga ctattctttg 3360
acccctaaca cagtcaaaca gcaagtctat tcactctacg gtgaatcagt agatgcagtt 3420
aaataccttg ttcaaacata cccagacagg gctaaacaaa cagctggtat tgcttatttc 3480
cttataacta cttatttaat atggaccatc ggtctcatag gagtaccaat ggcctttaaa 3540
ataccaatgt ttttatgtct tttataccaa gttaatggat taaatatagc accatttgtt 3600
actaaccaaa agttacaata tgttgcattt ccactttggt ataagctcta tgaagtaata 3660
tcagtccgtt ttgtggcgaa tatagcacaa tttattgtta aaacaccacc tatagatgtt 3720
ttaaacaagc taattcgttc taataaagac aagccagtca aattaacacc aaataaacat 3780
actttgatgt taattcatga cttagcttta gagtctgtcg acggaaaaga aaaccgctat 3840
tataatactg atgttacaac tttcacaaaa aggcatagca cttctaatat ttcatatgtt 3900
ctaaagtcta ctttaatcaa atatgtcatg gaccattgtt atgtaaatat tgcagttttt 3960
actttagtta gatacttaac tttattagtg tttattcaac atttctctaa tccttatgtt 4020
cttgaagcaa atagccaatc acataccgtt ttacagtatc tgttttcaca cttgagacca 4080
tttggaaggc ctttgtgccc aaccctcaat gactacatga cgacagcaac accacgcgat 4140
gcacatgtac aagcaggttc tcacttcagt gaattttgtg ttcctattca ttatacaaca 4200
ccaatcatta aaagcacaat ggcagaacca tcactttttc tactttttaa cccagtctta 4260
tggcctttgg ttatggttgt atatttttat cctccaatga tgtttatagc aaatgcagtc 4320
gcttattcat gccttccttt agtggtcttg ttacaatggc tttatgccat gtggttttct 4380
tgtacatgct atggcaccaa aagatgtgcc aagcatttgc ataaaaatga agtggttaaa 4440
ccaatggaat ccacttcaac taagaaccgc atgacattta ctccatcaac gaccttttgt 4500
agtaaacata acttcttctg tccagatgca ccacatataa tgactcttgc aatggctagg 4560
caacttacaa actactacaa tttgacagat acagtaatac ctgacatcca ggaatactcc 4620
cacgagaacc ctactgtaca atttattcac tttgatccac ttaaacacgg tgccgacaca 4680
attttggaac caattacaag cgctagcgcc agttcaattg ttgcatggta ctctctcctc 4740
tttaatcaaa agtttgtcct ttcacattat agctacagaa ccccagtagc cgtagttgac 4800
aaaccagagg aaacagatgg tgatgataca aaatcattag catctgacac ttctgataac 4860
tttgagtcta ttagaaagac caaccataag aatcagagca aacaacagtt taggccaaac 4920
ggtcaccaaa gaccaagtaa gactttcaaa cgccattcaa gaataatgac atctgaacag 4980
aagaacagct taattgaaac ttttaagggt ttaacaaatg gcacagcagc catcccacag 5040
cctttaatca tttttatttg ggttatcctt atggtaatac caacactctt tttagtcgcc 5100
agttccagca gaacagctgc aacaatgcct ttaaaccgct actcaggcgt caaccccact 5160
ggaattatgt ttcaccaagc acctccttac atccattcgg aaccaccaaa ggaaacttac 5220
tacaaactca gttatcctta tccgtcagca acagttgtga gaaccttgaa aggccatctc 5280
tattaccata gcgatgatac cgttcaacaa aattgtacca tgcaatattc acttatagct 5340
gcttctacaa agcacgtgtg tggcaaggta gtttacacta taccagccca tgtctcaatt 5400
ggctcactta aactgttgct tgtccacccg gatcaaacaa atttaccatt tgaactacca 5460
gtttcagatg aagtccgtct ttgctacctc acaaccttga acgcaccaag atgcatgccc 5520
tctcaactag ccatgtcaaa taaccaattt gccgctgtaa gccttgtttt gttaataaca 5580
ttagtttctt taattaaagt ttatataatg ttttttactg tttttaaaca ctacacaaca 5640
actgttttta tacttgtagc tgtgactact atcacaatgt tggtgtcctt cttagctcct 5700
ccacttctca tagtcgttct tctttcacta gcatggctat ggtacggcaa tacaattgta 5760
ttgtgccata tcatgctttt gatagtctta gtcgtctcat ggaaagtggc tgctgtctgt 5820
ttcatcttcg ccttattgta ctttggaaaa tgtgctatgc ttagcaagaa cattaaatac 5880
gtacagggtg gagttaaatt ttcaggaacc tttgaagaaa tagctcagtc aaccttcttc 5940
attaactacg gagtagcttg tcagcttctg gaacatactg gacagacaat tgaggatata 6000
atgcaactta gaaccgcggg tggagcccca gcaaggcttg cgcgctcaat atacgattgc 6060
ttttccacaa atgcctctgt cttgtacagt cccaggtcat tttcaccaca gtcacttata 6120
acaaaatatt tatacccagg ttcgatccct gtcggcagag cccctgtctt attaggcaaa 6180
atctccggca tgacttgctt agggcgtgaa cagtccacct gtttccaatc atcagctaca 6240
accattacta cgtgtaccca tgctgtaaat actgctggaa cattcatgtc tcaaattaaa 6300
tgtgttatag ataataaaat atatacagtt caacccgaga atataaccat aaccggaatg 6360
aaagctacat ttgaagttga aggactacct ccattcacca acgatgtaac agtggcccca 6420
aagccgctga agcattacat ggatggaaag agacaccttg ttctctacac taaaagtgag 6480
agcatagtct actcttcaat aatgtggccg actgaaaacg gtttattctc gtcatcagtt 6540
tctgacccag gagattcagg tgcaccctac ttttcagaca atgtcatagt aggaatacac 6600
caaggtcgca acgaagcaac caacaatcct gccattttag caagtggtat ggatggtgag 6660
tctccctgtg taggttacga tgaccaatca tatggccttc cacttcaaga atatttcact 6720
cacattgtct tatcaaataa gccaagtgac tttggtgctc catctaacgt ggcgccaaat 6780
aaatactaca acaaaaaatc atttgaacaa ttagctgacg aagataagac ttatttaaat 6840
agtttatcat atcccctgtc ctcatctaat tattgttact ttaatagctt caaaacccaa 6900
tcaagcacaa caatgctcga caacgctgaa gttattaaat atgtagtttt gcttctcatg 6960
atcttggatt atttcttttc aatcatttgc gaagatgctt taaacccagc atcttacgct 7020
atgttagtta tcgttttggt tcaggctttt attacaaaaa ttacagtttt cagaacaggt 7080
atctatatcc aggcagccgt ttttcaagca tttattgtac ctatagtcag tcaaattaca 7140
ttgatactgg ctgcagatac tgcaagaagt tttttaacgt tccacttttt tgtacttgct 7200
gttttgacat atttcgttct ttgccgtatt gctgtagatt tttggcgttc catgtttttg 7260
ctatttttga caagcgtctt tgcaaccatc atatggacta caaagaatga cttcaatatt 7320
ttacatgaaa ctggcgtggt tctaacaccc acggcagaat tagctcttat agtagctttt 7380
acttatataa tttatgcttc atgtatgtta acacctgtac cactgtatac tatttgtgtt 7440
ttcttttcat ttttatcaaa tgctccactc taccttgccg tcctctcatt cggcattcta 7500
gtttctttca aaacaaacca agattttgga cgtctagtgg ataaagtgtt ttctttaaat 7560
atgctctatg aataccatgc ttaccaaaac tatgttattc aaaactcagg tcaacaccca 7620
ggattttaca ggtcactctt tgcttttttt atcaatttga ccacccaacc aaaaacaaca 7680
tacaaatgtt tcaaacccca gacagcaagt ggttacagag taatatatca aactcccact 7740
acagagttca ataaatctct gcaacatgcc agtatcacaa aagatgacaa ctccaaccat 7800
ataattatgt ttgctgacgg ctcatctgat aatctcaatt gggcaaaaga aatggtcgca 7860
accattcatc taaccaaccc aaatttgcag ccactcatca ttggatacta ccacaactcc 7920
atggacgtca taaccaaggg aacttacatg caacatgaat tcataaaaat gccagctgtt 7980
atcttaactc aagatcctct aactgaacca atcagtcatt tagcagcagc agcatttact 8040
tcaatttctg gaaaacctca ggcacagaaa aacaacgttg tttcaaactc caaagcgcgc 8100
ataaacacag ccgttcacga cgctgtcgaa agcgtttatt caggagaaac atacgttgcc 8160
cccaaaccta tagtctcagg aaaaactgtt gtagagaaac cattctctac aaccgaaacc 8220
accatgtaca taatgcgtgg tttacccggt tctggaaaat ctttcaaagt tagtcaatta 8280
gttgctaaag atccaaattt agtcgtagct tccgcagacc actttagata ttcaaatgac 8340
aaaactggaa aagccgtata cacctacatt ccagaagcaa ctagttctgt acatttacaa 8400
tgtcagaata gagcccgcaa agctctagaa aacggccaat ctgtgtgcat tgataataca 8460
aatctaacac tcttagaaat gagaccttac gtcttattag cccgttcttt taactataac 8520
attgaattca tacactcaga ctctccctgg gccttaaacc ttgacctgtt acatgctaaa 8580
ggtgtacata atgttcctag agcaaagctc gtaatcatgt atgatagatt ctttgaccgt 8640
gataatcaaa tcgatgcaga cagtcttata cagtatgtta ttgaagcaat tgatccaaaa 8700
cttgttgctc caatcatgaa ccgtttccct gccgactgtg atcttatcct tcaatctgcc 8760
ctaacaccag accttgaagt attaaagcaa aactacgaca gagcaaacgc aacataccaa 8820
gatgtttctt tagatgatcc tccggcttta aaggcagcac gtcgtgctat gaatatagct 8880
aaatctgaat atgaggcagg cgaagcaggc cagcgtcgca ttgagaaatt tttagaaaga 8940
caggatgtag cagcactcaa ccaaacgctc acaactgtca atcaatctaa attcatagca 9000
gcgatccgtt ccatctacct aagcaccatt agcaatttga gactaaaaac ccgtcatatg 9060
ggtgaaggat catatgcagt tacatcaggt actaatacta ccgataaagt tttagttaac 9120
acgccacaac gtatgactag aattgaagat ggcatttata agcttgttgc aaacggtttt 9180
gaaatcacaa tgtgcgacgg cagcaactta gccggtgtta cttttgaaca ggatataaat 9240
cctagcatgt acccttttgt ttttacatta atgtcaaata tagctgtacc tgttttaacc 9300
cgccaagcaa atgttggcta tcttgatatg tcaaataaat tcatctgtaa agatggcact 9360
gttcaatttc aaggtgtcat ctatgcctac cacactccat caaatgagag tgctgacttc 9420
aaagtaggca ataccagttg gaccctccag aaaaacatca atttgactgc ttttattcct 9480
gcaattcata aaactgcaac cttcgcagca caatcagtgt tcttaggagg actacccatg 9540
gaagagcacc aagccttttc cgacacaccc acagcctcaa acaaatttaa agtttttgtt 9600
tcatccacag tctgcgcctc aaccgtgtgc aaagtaaatc ataaaactta tgtacagata 9660
ccagatgaca ttcaagatcc ttttacatat atgcatcaca gcgtttgttc acacaacaaa 9720
tttttatcaa accatgaaac cagatgtcaa atctgtcctt taaactgtta nagcgcaaat 9780
ccgtgtgtgt ctacggcttg cgctctattt gataatggca cattacctcg gtcaacacat 9840
tatattaatg ttagcaccac ttcaaatgtt ggcttgttta aggcagtaaa gaagtctact 9900
cgtcaactaa acattgacgg ttttccttac atgctaaagc aggttaaaga cgactcagaa 9960
cttgtaagtt ctcttaaaat aggtctacct aatatcctcc cacatcacat ggtggaaact 10020
aagtcaaaaa cataccttct taggggcccc acaacggctt actcacttgg cgatttatgc 10080
tacgcactct ttaatggcga ctttgattat attcgcgaaa atataaactc tgatttcgtt 10140
ttggaccgtg aagccggaat gcctgataca gaaacacgta cgtggctgtt cagcatttta 10200
aactttgcag tacctagagt gtgtgctata attgaccaga tgatttctga gaacgtcttc 10260
tataaactga ctttggataa cttagatcta tacggatcac tctatgattt tgacgactat 10320
cctactgaag gctttaacag gcctgatgat gtgatacgta tgttaaagga gatatggtcc 10380
ttctgtagac gtccactacc tgccgacctt cttaaatacc atgaagacat cggtgcagca 10440
gccactcaag aaatattgct gcatgcaccc ttcattgata aagtttgtgc tctaaatgac 10500
agattagctg ttgttgataa tagagcaagt caatactttt tctgtgaaga agaaggtgtc 10560
tttacccata tttacaatcc agtctacgga actttagcat tcgataacaa gttgatccaa 10620
tcaaaggatc cttcatgtac attacagcgc ctcattacta tacaaggccc tttgtctacg 10680
aatgctagtc ccgtgatctc tatttctgat tccactcata ttgccaacaa tattaatcca 10740
tctaaccaaa agacaacacc gttgtactac gatttggaac ttgcgcaaga attcattgac 10800
gcaggtttaa atattgatgg cgtttccaac tacttcttct atggaccgtc tagagcgggt 10860
gtagtgtctg atttcttact atatgaattc caaggaactc aatggtttga caataacatg 10920
ctgcgctctc tttattcttt catattgaag aattcagagt gttacagaac aacagatcaa 10980
ctggacttta gaggtggaaa accccgtaaa tcctcaatgg gacatggtgt tactggcttt 11040
aagcaagacg tcgtgtacgc tgctttaggc cctgatatga ttgaaacctt gtatgaaacg 11100
gcaaaacaaa caccattgcc gttttgtaca aaaataactg ccaagtatgc attaacagca 11160
aagcctagag ctcgtacagt tgcagcatgc tcctttgtag cctcaactat ttttaggtac 11220
gctcacaagc ctctaactaa taatatggtc tcaaaagcac agcagggttt gggttattgt 11280
ttaattggaa tttctaaatt ccacggtcga tttaataaat ttgttaagtc tagggtaggc 11340
actgtcgaag actttaatgt tttcggtagt gactacacta aatgtgaccg tacatttccc 11400
ttagctttgc gtgctctttc agctgccctt attttcgatc ttggcggcca tgacccagac 11460
aactgtcttt ttattaacga gcttaatgca tacatgctag acattgtttc agtcgaagac 11520
tcctttgcaa ataaaccagg aggtacttca tcaggagatg ccactacagc atactccaac 11580
actctgtata actttgcagt ccactatatt atcatgtgga aaacattctt gacagtcaat 11640
gacccttcta ccaaggtcat acgcagtgca gctcatcacg ccctaacaag tggtgacttc 11700
tctatgtaca atgacatgat acaagacatg ttggatgtag actatacact caacttcctc 11760
tctgacgatt catacatctg ttcaaaacca agcgcttttc cgatctttac gctcgagaac 11820
tatccttcta aactgcagtc tatactccac acagcagtag atagcaaaaa atcctgggaa 11880
gcaaagggtg agattaaaga attctgttcc tctcacatag tcaacgttga cggcgactac 11940
cactttaaac cagaaaagga tagaatattg gcttcattgc tgatattatc gaaaatcgct 12000
gacatggaca tcttctttat gaggttcgtt gcgttattgg ctgaatccgc cgtatatata 12060
cgcatcgatc ctacattttg gctggccctg tttggtgttt tcgaaaaccg cgtaacagcg 12120
tttaaatctg aaacattgct ctcacctgtt cctgaacaac tcatgaaggt ggctttttat 12180
gaatcgcttg tctttgccga cgtggatgct acagccttat atggtttcct tgatggtttt 12240
aaaatgcaaa gtcaaactct ccacccagac ggtgttgagg gttttgacaa gcaaagtgac 12300
cgagtaaaac actgttttgc ttgtgacaat atatcagttg gacactgttc gatttgtccc 12360
gttccccttc ctttgtgctc tttttgcttc tatgagcatg ctctgctcaa tgaacattat 12420
gaagcttctg gaattgcgtg tgaatgcgga gacgctgaca ttagacaact tcacttaaaa 12480
ataaccaatc aaccatcctc gcacaatttt atctgtgctg aatgtcccac tgtagctatg 12540
aagctgccaa tcttcaactc tttccaagga aaagtactgc ttccaatgtt ccgtatgaat 12600
acgccattgc cttcctcagt ctctgtaatt gttgatgtac gttccaaccc aaaagcacct 12660
aagatgctgt gggacgacgt ccagaatttc agagaaaatt gtactaggat agcatacgaa 12720
tccgtttcgt gtgctgaact agctagggag gtggtttact atccatatga agtgattgaa 12780
tccaaagcag gtcaagcacg acttagaata cagaacttta aatgttcacc aactacttat 12840
gttcagttct acaaagtccg tcaaaatgga aagtattgtc tagtagccaa agcaactcta 12900
acgccggctt ttgaaaacca aacagacatt ttctccgttt ttcaaccaaa caacttttca 12960
ccttggaata catcatcagt gtttgcagta gaacaatacg ctgcaatata ccctcccata 13020
ccaaaggaac cagtcaatgc tacgttcgtc ttaggacctc caggctgtgg taaaacatac 13080
tacatagcca aaacgtactt ttcacaggct tctgagacat gtccggtcgt atactgcgca 13140
cctactcaca gattagtttt agatatggac gcagaatata gtggtgtagt ttcaaaatct 13200
ctctacaata atagagtgta caaaaatcca gcctacaaaa caggcgaacc attcaaatta 13260
tgtttcacca cacacaacac gatgccagtt caaaagaaag cgatcctcat tatagatgaa 13320
gtgtctttaa ttacacccca ctctctattt tcgatcattg gtaaagggtt ctatgagata 13380
gtactcgtag gagacccttt tcagctctcg gctgtttttc caggttttgt tgtcaatcac 13440
acatatgacg ggttttacat ccgccggcta gtaaataagg tcaaacacct aacagtttgt 13500
taccgttgtc cacaagaaat cttggacata ttttctaagc cctatcatga tgttgggatt 13560
gacctcacaa ccggaaatac caatccagga aaggcatcca tttatacact aaattggctt 13620
caagcagatg taggtactaa aaatccggac aaactcagac aactctttgc gcaatatcca 13680
ggctttaaaa ttatcaccaa ctacagatgt gttgttgatg cagctaaaag ttacggtatt 13740
aacgtcgaaa ccatcgactc atcccaagga accaccggag ataggcatct ggtggtaatt 13800
tgcggcagta ccaacttttc taaactttta aacaggttta tagtagcagc ctctcgttca 13860
acaactgaac tagttatagt catgttgcca gagctttaca actatttaac agagacgttt 13920
aacttcaaac cgttacaatt gcaaaatgtg catgtaccga tcgcagtatc ttctacagca 13980
ttctgcgata tagaatttta tcactttcaa aagaagttct atgttggtga aataagcgta 14040
agcacaagta ccactatgac atgtcagttg ggttgttata ttaatggctc ctacatgctc 14100
ccacctgtgc ttgaaaactc tgaagaccgt ctctacgttc cttctagatg gagacgtatg 14160
ataagaaaat accctactga atctatgcac atttccttac tggacagact tctgaggcac 14220
attttattaa caactactgg agaaattcat ttcgtaatgt tctctgcaga caatgatctc 14280
attgcactgg atccgtactt tataccaccc actctatgtg agtgtggtag tgcgggtcta 14340
gtggaagtag acatcactgt tttctgccgc aattgtttgc ctaaagatgg taaagccact 14400
cgtttggtaa aaccgtctac actagatgtc cagactgaaa aactcagact tgcaaaagtt 14460
catgctaagg tttgtaaaat caagcatggc agtgctcaca acgctgatgt tgatgctatt 14520
atgactcaat gtatatatgc taatagctta acattcacac caacaaccca actagttgtt 14580
aacactgatg agttcacctt ttacatgcta cctaggccgt caaaccgtca tttgagaatc 14640
attcataaga acgacaaacg tttctatgct atcactcatg aagaagaaga tctcttcttt 14700
actaacatct cagcagtggt agacccaatt cctgcaaaat tcaacattgc acactctaca 14760
agcttcctca ccatcaaaag tggttgcgca ggtaataaga cttgtaccag atgctattat 14820
ttacacttag catacacgga atttgtttct caacacaagt atgaaccatt cacttgtgtg 14880
tcttttaaga tacggtttga cttttcacaa ttcactgact cagtagatac tttcctccga 14940
caaggcttaa taacctttca tccggagatg aattcactgc aaaaatcact tttattagca 15000
gtggataagg tctattgtga taacttcacc tcaaacggta gaaggtttag actttacgac 15060
aacaatttgg ttaaatccat aatcaaaggt tcagtggctc aaaactccat catcatgcca 15120
ctcgactcag ttttacacgg gttgaacatt gatttcacag tcggatgtgc cgtagataac 15180
ttttcctgca aagaagcagc gagtgttagg tactcagaag tagtactttc catcaccaag 15240
ttgcccccag gcacttgcca gttatactac gtcatatctt acggcctgaa ctctcccaag 15300
accacttatg ctggtcacca attgttcgac ggctttgaga ctgttattgt tgttaatcgt 15360
aaagataaac ccccttacgt cctcacacag tatattaatg atgttgtagt tgcaatgcca 15420
gagtccctct tttcaacagg tcgattctac agagaaaaac catatcccgt ccttatgaac 15480
gaggatttaa gtggcttaaa ccatcacatt ttctctggtg actatacaga cgaatctctt 15540
acattaggag gtgtccatca tatagtaact ttaaacacct atgaccacaa gctcaactat 15600
atccaaacga aagctacatg tgccgcctca gtttcaactg gcggacgtgg tcataaaatt 15660
actacactgt ttgacgttca tgcaaatcaa cttgctgatg aaattaccag agttacatct 15720
gttgttacaa cacagtctaa agttattaat ttgacaatag attatcagca agttccttgt 15780
atgtactggt cttcaccgac cggcataaga accttctacc ctcaggctgt tagactggac 15840
gcaaagttta taccatacta cgtagaatat cccaatattc taccggcagt tgttgaagac 15900
caggtgtacg atttgtctaa ttacaatcaa ccacctttag gccaaaactg ccctgtaaac 15960
tttcacaagt acgtccagct aactcacttt attttagatc atgtgaaaat ccccgaaaag 16020
ggtttgatat atcatatcgg tgcagcaggt actaagcaat gttcacctgg agacttaata 16080
ttggaacaat ttttcaataa atccatcata tactcaagtg accttcttcc ttaccaatca 16140
cctgctgtgc aggttgcatt ggatgtaagg ttttcggctt cactcatcat ttcagactgc 16200
tattcgaaag aaccgcagcc tgatttgttg agtaagttga ttaacaaact agtgtatggt 16260
ggaactctca tttttaagac caccgagact ttcacatgtg acccagcctt ttatgttgct 16320
cattttaact gtataaagtt ttttactgcc gctgttaatc actcatcatc agaagtttat 16380
attgcgttca tcggaaaact ccctaaacca aacaacaact ttttagcctc agactatttc 16440
cagagattaa ctcaacatag aaataaagta gttaaacagc cttacgctca cacatgggac 16500
acatctttta cgtacccata cccctcaaat gttcttcaag ttagtcgtaa aaacctttta 16560
tatctatttg aaaccagagg agctgcagta ggtactttga tttttgaaga accatcaaaa 16620
cctgctgtaa agatacctac aaagtgtcaa accacacaac cctcgtgtgt cattgaggtt 16680
ggtaaccaat acgattgttg cattcaagac atcattaccc tcctcaatgg aaaatccttc 16740
acagtgaagg tgcccaactc agaatcctta ctgcgcgata tctgcacact tgcgcttagc 16800
cagagttatt ccatcaatat tcgcggaaaa acactctaca cccttagttc cctacttaga 16860
attaggcaac aatccttact gttttacgga gagaaggtca aaaaccctcg accccgtaat 16920
gtcttgaaca aatataccaa ctacctcaag gcaaaagtga ttaggcatta caccaagcct 16980
caatcaacag ttttggacat tggtacagga aaaggacaag atttgagaaa atactcgtta 17040
gcaggggtta aatccctcac ttgtgtcgag cctagtcccg agtctgtgac tgaactttca 17100
ataatagcta gtccccttga tatggagaca cacacagtta tgagttctgc ccagaaattc 17160
gagacctcgc tgacgtttga cttggctttc tctttctttg ccttgcacta tgcattggat 17220
gacgtttgta tgtctgaaac actcaacaat gttttttgca aacttaacag taattcacag 17280
ttgatcttag tagttccaaa tgctggcagg atgcaatcca taccttccct tggtttaaca 17340
gtcactcatc tagatgatga taaagtttgg tttaaatact cagactatat agactgcgaa 17400
gaaccgttag tagacaaaga aaaactactt acgtgtttag ctacatatgg aacaattgtt 17460
actgactcac cattctatga cggtgcaaac aaaatcctag accaaaaatg ctcatccatg 17520
tatagagcat cgacagccca tctaaatccc gatgaaattc aatatattaa tatgtatgat 17580
ttaattgttg tcattaagaa t 17601
<210> 2
<211> 4173
<212> ДНК
<213> Вирусы
<220>
<223> нуклеотидная последовательность ORF-2
<400> 2
atgttcgcgc tcgttctaac cctcacaata gcttcggcta ttgcccaaga tttccccgca 60
tatgacccgt gtcctacttg ctcaaccccc ggtaataaaa taccggctcc gagcacagtt 120
gcccagtatt caacaaacta cggtgcgaac ttctttaccg tagtctttga tggtattatc 180
ttcaaccaat ttagggagag ttattaccac caatgtagac caacacctga atactgccca 240
gatgcaatca attgcgcctt aaacagaaca ggcgcatcct gcaaaccttt cgcaactggc 300
ccgaattcac aatgtcagaa cagtttcgag ggcaacatcg acatatgtgc aacatgtagc 360
cctctaaaac aagaaactcc attcatctgc tacaatagat acgggataat tatatacccg 420
acagcagata tcgttctctc cgctaggttt aagataggct ctttttcacc caaggcttgt 480
gataactacc taaacgactt aaattgtgat tcaaaaacgg caaggtcata tgtcatttcc 540
cgaccgcagt ctttttcact gcaatatcct aactcattag gcccctatca gctgaaacga 600
ttttctcttg caaaggagat cgttgactta cgtgctggcg ttttaacctc actcccaaac 660
cgaggttata agggtagaac aacatactct tatcccgtca ctgcactctc actcttggct 720
cgttccaaag tggctgaagc cgacaaattc ttttatatcg aggctaaaat tctactgtac 780
gcttggtcac agaaacctca aatccgcttt ctaggtgcat actgtcccac agacgtgtca 840
tgccctgatt caactgccct cggctgttgt ttttccggaa gtggatctga gttttactac 900
gcctttcgcc agtggtacta cgcaagcctg ggtatggaag acctagtcga ctttgataat 960
tcaacagtct taagtctctc gcctgatact cctcaaatta cacccgttgt gtcttatttt 1020
ctagaaaaag ttttaccttt gtttaaatca catgtacccg gacgtgtttt ttactgccat 1080
tcacttatgt ctaacggtgt atgtactttt gaccatgttg ttgtaaatat taatgccgag 1140
gccgtctttt ttgacctcga agtagacata ggcagcataa ttgctgacgc atatcgcgtt 1200
gaaaggccta atactttatg ttatgataca aactgtactc ttgccacaag caggaccact 1260
gagtataatt acgctgctta tgttgtttat atactcttca atttgtattc tagtaatcgc 1320
attgcgatag atttcaacac acactcaatc ttgcaaggat tactacaaca caatagcaac 1380
taccagactg ctaatttaga ctatctgttt gttggagcac tttttacagg tacttttaaa 1440
catattacaa gcaatcaagc ttacccagta cctttaactt atccaattgt taagacatat 1500
gtagggccgt caaaccaata ctcaatgtca aataaactgt tttcatatac tcacaatttg 1560
acggctcaag cccattcagg catatgtaac tctttttact gttataaacc acgttttgta 1620
ccaattgatg tttttattca tagtgcttta acccctgaca gcttgatgga aacagaatct 1680
tttgtttgtg tctctttgcg ttcaccatct gcaggatcaa catccgcagg tagtttttat 1740
ttgcaatgtc tcaattcttc catcgatttg catccaggtt catttgtacc cgtttcctca 1800
agtccagagt cttccagccg cgtaacagct gagctggctt ttaatactag aaatggtata 1860
ttttctcctt gtcttaacgg tacatgtgta ctcgcaccta ctgacccaat tgtttttatg 1920
cgtcagggtg cctggtttac aaaatcttta cactttgatg tttcaccatg caaacctatg 1980
cattttccag acatagatat acagccccca acatacaatg tctcctctat caagatggac 2040
gacaatgctg tattggttca agaccttact tcgggtttag taattgacca caatttaggc 2100
tccatactca gaccgaaagg tagagctttg gaagtttcgt attatgctca ctccatttta 2160
cgttaccttg aaccggattc ttgtctacct gacaactttc ttaactttgt cacttgttta 2220
gactatatct gttcagactc gtcaccttgc cgtgctgccg caagccagta ctgtcaggca 2280
ggcatttatt ttgagtctgc atttaataag tctaggtatt ctttgcttaa cgcttacacg 2340
ctttttaaca caagtcttca aaccttattg cctgagactt ttcttgagat agaagatgat 2400
gaaccccata gcagatcaaa gagatcaatt gatactacaa gcaatattcg ccctagtcaa 2460
ttgcttgtta atggacgtat tccgtctaca agttcagctt ttgctgttaa cgtcgctcgt 2520
ggtcgaggaa cgattatgcc tcgtcctgga actggtggca tgggttcgtc cttttctgct 2580
gtttctaggt cgggtagtat ttcttcctta tcctcggttg gctcctcaac acctttgatc 2640
tctaattgga gaacatcttc atctcaactc aaaactctca acctcaacat taacactaaa 2700
attcctaaga tttcaacaaa gtcaggtttt gccagtatta catctttgtt tgcttcaggt 2760
ttaggagtcg tcgatctagg tctatctatt ttcaacatga tagaacagcg tagagttgct 2820
gagatcactc agatgcaaat tagccaactg gctgactcta tagtgtatct tgctgatgtg 2880
acatttgaag ctatcaagaa tttggaactc tcggttaact ccttgggtac gttcttatcg 2940
gaattttcca ctcagatgtc gatcaccata agccaaatac aatcatcatt tgaagagcag 3000
caagatgcta caaatgatgc gttgtactac actaacgctg ctgcgtcata ccaagcctcc 3060
atggcgtatg tcatttcaga gttaaacgca atatctctgt ctgtcactag atcctacgac 3120
tcttacacca gttgcatcac ttctggcatt aatgggctca ttacaccatc atgcttgcca 3180
gcccaccagt tgttacagtt actcgacacc gttatcaatt ccacagcagg aacaggatgc 3240
cgtcccatct acggcagaga agaagtggtg aaatactaca ctttacctct aatcaatcaa 3300
ggttattcct ttaacgggtc gattttcttc gtctttaaca ttcccatcac ttgccaggga 3360
attgccggag atgtatatga agtagaacca cctatacttg tagatgtacc atcaaagact 3420
gctttacgca tgattacacc atcaaacgta gtcgcaacac aagcaggatt agctgaatta 3480
gatttgcgtc attgcgaaag gtaccataac gagttcctat gcgattcttc agcattcctt 3540
tctacacctt caaaatacat agactgttta acaaacgcaa ctgactgttc tttgcaattc 3600
atcacacaac acgttccaga tccttgcgtt tacacatcgc cagcttcttt atattgttat 3660
tattcaccca tatgtgatca atgtcacata gtagccggtt gtaatgaatc tcagcagtac 3720
aacttcactt ctgctgatgg cggcgtagtc ttttattcca tacaagacag agactgtggc 3780
cacttccccc acatcactgt tactacgcct gcagccatac aagaagactt cactgtcgga 3840
ccgtatttac catcgctgcc aattcacacc gcctacgtca atgttacctg gaatgtaaca 3900
ctaccaggaa attggacctg ggaaaatatc accctaacag ccaattggac ccaacacttc 3960
attgagatga aaaaaaacat cacaatgatg gctgaagaaa tagataacct taccaacttc 4020
ggtaaggttt tagttggcca gctaaatagc tttttatcat ctttgtttaa cataccatta 4080
ggtttgatga cgttttgctt ttctgtagcc gctttaggcc tgtccattat tgctttactt 4140
gtgttatgtt ttccacagaa gccacataaa tta 4173
<210> 3
<211> 780
<212> ДНК
<213> Вирусы
<220>
<223> нуклеотидная последовательность ORF-3
<400> 3
atgatgttta ccctagtagt gctttttacc ctcctcggcc tttccatggc ctccacagag 60
ctgaatttcg atcctactct acccctcccc tctcctataa atgccctcgt cgacattttc 120
ggaaacaaca gcttgtttct caaagagtcc ctgctcggca aatccaccgg agccgtctac 180
gcatacttgt acagcagtgc catctctctc ctgctgctac tttgggtaac tgtatggagt 240
attgctactt cacactttaa cgtaactcgc attccaacca tcgcggttct cactaatgcg 300
agtatgtttt tgctgttggc atcggctact gttacaacct ggtttctccc aactgtgacg 360
aacgtcttct tttatacact cactgcgctg ttcaccttct tttcctttgt gttcttactg 420
tggttggttt actatatgtt tactaccatt agggcatatc gaagggtcgg ttcatggcgc 480
gttgtgttta acggaaaata ttctctactt gctggaactc aggctgtttg cctttgcaga 540
cccgccatac atctggttct aaccaaaacg aacacagata catactggtg tctagatgga 600
acccccatct acaatgttga cttactacaa ttagttggcc ccaaaggatt atatccttac 660
aaaagaatga ctacaatcac tgcaccaaaa ggcacaaaaa catctgctgc cgtttacacc 720
cttcaaaaag aagaagtttg tgctctctca gaaatcacag tacataatga tactgatttt 780
<210> 4
<211> 603
<212> ДНК
<213> Вирусы
<220>
<223> нуклеотидная последовательность ORF-4
<400> 4
atgtcttacc cggtttacta cgaacagcgt cgttattccc cccgccaatt caacaatggc 60
ggagggtata atcctacacc tcaacctaga gtagttcgta ctaatcctgg taaccaagct 120
tacaaccccc ggcgtaaccg aaacgccact ccgaaccaac aacaaatggt tccttaccag 180
cctcagtatc aagcacctcc tcagccaagg gtggtctatg tagatcgccc tcaagaacct 240
gtagtaattt acagagctcc tccacaagga aaaaaacaat caggcaaacg ccacacagca 300
gaagaacgct ggtatcaagg cgaaaaacct gtgcagaaga aacaggcacc caaaggaaaa 360
tcaaagaaag cagcaacacc tgctaatcct aaaaagcagc ctacacaatc tgacaaagtt 420
cccatcgcct acccagacaa tcatcccttc catgacctcg caccagctga catccgcgct 480
ttcaaaaagc agctgatcca aaatctggac cttggacatg gtgaaatgaa tcaactgcgg 540
ctttcaatcg atctgttgcc catcaagaaa ccagcaccaa caccagcggt gccagctcct 600
ctg 603
<210> 5
<211> 339
<212> ДНК
<213> Вирусы
<220>
<223> нуклеотидная последовательность ORF-5
<400> 5
atgtttaccc ttgtgcttat tatcctgctt agtttttcta tggcttttaa tgcttttaca 60
tttctgctgt tattattttt tacttttaag tgcattataa cccgcacttt agtcgtagtt 120
cccattgact acccagaaaa tcatcctttc aatggcctct caccagagga aatcatcagc 180
tacaaatcac agctgatcca aaatctcgat cttggacatg gtgaagtaat taaacatcga 240
ttctcaattg atttacttcc cctcaaaaca acaagcactc ctaccaccag tgctatttta 300
tggaaaaggt tcaaaacctc ccataaagaa aacaaccac 339
<210> 6
<211> 24613
<212> ДНК
<213> Вирусы
<220>
<223> Геномная нуклеотидная последовательность ("n" в положениях 2748-2761, 2781, 2782 и 10029 обозначает любое основание)
<400> 6
gcagtccacc aacacaacgt ggctctctgc ttacctgtaa gggcacgccc ttatgctgaa 60
ataatcattc aggaacacct ttttgtttca agaaatagta ggagttttaa accatccttc 120
tatcttgttc caacaaagga gccgcaatca aacactctaa cccttcatgg gggtgatttt 180
gatgcaaagc gacacttggg taagaaacca gtaaagtgca atattcttga cattgttgac 240
aaaaaccaca caatagcaat gaaaaaaatt gaagatgctt taggaacact taaacctgcc 300
tttaaggcac acatagactc tctacctcct ttcatgggaa aactagccac aatgctagct 360
gagacaagac gaggaaaaac tccccctctt ttgatctatg tcatatcaac aattttggaa 420
acgaatatta cagttcatta tgtcagccac acaataactt cataccattc ttcaaacgca 480
cacactcaca ctcatgaatt cgactcagaa gattacactc ctgacaatca gattttaact 540
aaagtcaacc gacatggtaa tgaccttaat cattcatata tccatggcgc caccaatatg 600
tacaaccctg tctaccagag acacccacca aacatgtgct acatgcttac gggactgtac 660
atgttgtcag gcttacagga attgtatgcc atggctgaag acaacttgac aacatgccaa 720
atcaaccttc tcagatgtct gtttgatttg aatcaagatg aatttgatgt agattacaca 780
tttgtaatat acactcctag caaatctcag gagtgtgcct tcaaatatct tcaggagata 840
gtccaccact gtgaactcac catttttaga catacaacaa caagtgtctt cagttgcaac 900
aaatgcaacc atgtggaaac tgtcatttct agttgttctt tgaatcttgt atatataact 960
gattctattg aaaaagcatt tcaacccact gtagaagcta ataccgacta catgtgtgaa 1020
aactgcggtc tacgcgacca taaacttaaa acaacagtaa caaatccaga cctccgtttg 1080
gcacaactaa actatccaac ggattcaaaa tacactatct ttcttgatga acaagctcct 1140
tttgtcttcc attccattgc aaaacacgtt ggaactgcta attccggaca ttggagtgca 1200
ttgaatgtaa attcagatat gttgtcagac tctaacgagc gacaacatta ttatacaaca 1260
cctagtattg ttttgcttgc atttcttcct gaggaagagc tacagaacat tagaaattca 1320
tcacctcttc aggaccatca accagatgat gttgaagacg ttgaatctcc cctaccaggt 1380
tctatatttt atactacaga tgacattttt tctactaaga gtctatccat agctcactgt 1440
gtagcccgcg actttcacat gtccggcggc atagcaaaaa ttttctcaga taaattcggc 1500
tctaaaactt tcttgaaatc acaaaacccc gttataggcg gtttttccat tttacttaga 1560
gagtgtcgtg acatgtacta cctcgtcaca aaagagaaaa cttcagataa gccgacatac 1620
caagacctta aaaactcctt gggttctatg acagagaatt tggttcgcaa gaatcataat 1680
actctttcaa ttccatatat aggctgtggg attgatggtt tacaatgggc aaccgttgaa 1740
aaacaagtca aagaaattgt ctgcgctcga ggtattgatg taacagtcca ccacctcgaa 1800
aatgaagtta aacacactcc agaacaacaa acagcttctg acaattcagt caaattagtt 1860
caaaaactct ttacagaaac acctcaagct atacctatag ttgttccttc agatgactca 1920
gacagcgaca tagatgagtc agccgatgtc tttttaccag gacctgaatc tgaatctgat 1980
tcaaaatcag aatctgggtc agattacgac tttaaatcgg cttctgaacc agaagatgaa 2040
ttggagccca ctccaatctc tgaacttgaa ctaacaccag cgtcaagttt aactgtagag 2100
tccgatgaca atccagacac aagccaggaa acattaccag aatcaaactc tgaggaaacc 2160
aaacctgagc aaacacctga cacaacatca aaggtgtcct ccgattcgaa gcttgatcca 2220
caatcagaat tagaagaaga actggccaac aaaccagaat cggcttctga accacaatct 2280
gagactgaat cgagttctga atctgaagag gagcttgagc cacaatcaga atcagaggaa 2340
gaaccggcca acaaaccgga atctccttct gaatcacaat ctgaaaatgg atcgagttct 2400
gaacccgaag aggagtctga aaaaccatcg gagtctgctg aaacagcaac agaggatagc 2460
ccggaaacaa caccggaaac aaccttagag ttaaccacac aactcaaacc tgcttcagaa 2520
tctgacgaca aaccggacac accagcacca tcaccttcac caattcaacc agagaaaaac 2580
ttggacacca cccctgaaca aacttcacaa ccaaccacac aactggaatt gacgttagaa 2640
acacaggaac aaccagacac tacaccagaa gtgccatctg tctcagaaga taaaccagac 2700
acacttgaag aatcttctga atcaacacca gaactctcag aattggannn nnnnnnnnnn 2760
nccaccagta ccaagaccta nngaaccaga cctccaacac ctagaccaag atctgcacgt 2820
ggagctagga ctagatcctg tgctggcaca cctataccag ttatttttga tattatagat 2880
aatacaagcc agcctcaagt tccccttgac ttcccagaag cactgcaaga actgaacaaa 2940
cctagtgaag taatcccagc ggctagtgaa aaacctgtgg aaaaacaaat aatccatagt 3000
tttgtaagtg tcgaaacacc ttgcaaaccc aaagccacta aagttacgaa ttatgtagct 3060
gcacaatcta atgcaattct aaattgtatt aaggctttca ttcctagcaa cccgctttca 3120
ctattcaata gaaaaccagc ttttagaaaa ataatattca ctgaagacac ttcagaacca 3180
gatagcgatg atgatgattg tgaatacact ccaccaacat caccattccc tgaacttctg 3240
gcattggtag atgaagacat tgaagtagaa caaactcaat ctgtaattcc aaaaacagac 3300
tctgcttcaa ttgtggagga tcttaaaaaa caagaatcct ctactttgtc attggacacc 3360
aacacatcga aacctacaag ctctccgcga agacagccta gggaagtaga aagtgttgat 3420
gaatccagtg atgactcatc taaaccaaaa acaatttcaa cattagacaa acctgctatg 3480
aatagtgaca cgaaacctac agactcttcg cgaaaagagc ctctggaagt accagttact 3540
acatctttaa gcacccctgc taaaaaccaa gataaaaaat cttcaaaatc tgcaaaagta 3600
ataaaagact attctttgac ccctaacaca gtcaaacagc aagtctattc actctacggt 3660
gaatcagtag atgcagttaa ataccttgtt caaacatacc cagacagggc taaacaaaca 3720
gctggtattg cttatttcct tataactact tatttaatat ggaccatcgg tctcatagga 3780
gtaccaatgg cctttaaaat accaatgttt ttatgtcttt tataccaagt taatggatta 3840
aatatagcac catttgttac taaccaaaag ttacaatatg ttgcatttcc actttggtat 3900
aagctctatg aagtaatatc agtccgtttt gtggcgaata tagcacaatt tattgttaaa 3960
acaccaccta tagatgtttt aaacaagcta attcgttcta ataaagacaa gccagtcaaa 4020
ttaacaccaa ataaacatac tttgatgtta attcatgact tagctttaga gtctgtcgac 4080
ggaaaagaaa accgctatta taatactgat gttacaactt tcacaaaaag gcatagcact 4140
tctaatattt catatgttct aaagtctact ttaatcaaat atgtcatgga ccattgttat 4200
gtaaatattg cagtttttac tttagttaga tacttaactt tattagtgtt tattcaacat 4260
ttctctaatc cttatgttct tgaagcaaat agccaatcac ataccgtttt acagtatctg 4320
ttttcacact tgagaccatt tggaaggcct ttgtgcccaa ccctcaatga ctacatgacg 4380
acagcaacac cacgcgatgc acatgtacaa gcaggttctc acttcagtga attttgtgtt 4440
cctattcatt atacaacacc aatcattaaa agcacaatgg cagaaccatc actttttcta 4500
ctttttaacc cagtcttatg gcctttggtt atggttgtat atttttatcc tccaatgatg 4560
tttatagcaa atgcagtcgc ttattcatgc cttcctttag tggtcttgtt acaatggctt 4620
tatgccatgt ggttttcttg tacatgctat ggcaccaaaa gatgtgccaa gcatttgcat 4680
aaaaatgaag tggttaaacc aatggaatcc acttcaacta agaaccgcat gacatttact 4740
ccatcaacga ccttttgtag taaacataac ttcttctgtc cagatgcacc acatataatg 4800
actcttgcaa tggctaggca acttacaaac tactacaatt tgacagatac agtaatacct 4860
gacatccagg aatactccca cgagaaccct actgtacaat ttattcactt tgatccactt 4920
aaacacggtg ccgacacaat tttggaacca attacaagcg ctagcgccag ttcaattgtt 4980
gcatggtact ctctcctctt taatcaaaag tttgtccttt cacattatag ctacagaacc 5040
ccagtagccg tagttgacaa accagaggaa acagatggtg atgatacaaa atcattagca 5100
tctgacactt ctgataactt tgagtctatt agaaagacca accataagaa tcagagcaaa 5160
caacagttta ggccaaacgg tcaccaaaga ccaagtaaga ctttcaaacg ccattcaaga 5220
ataatgacat ctgaacagaa gaacagctta attgaaactt ttaagggttt aacaaatggc 5280
acagcagcca tcccacagcc tttaatcatt tttatttggg ttatccttat ggtaatacca 5340
acactctttt tagtcgccag ttccagcaga acagctgcaa caatgccttt aaaccgctac 5400
tcaggcgtca accccactgg aattatgttt caccaagcac ctccttacat ccattcggaa 5460
ccaccaaagg aaacttacta caaactcagt tatccttatc cgtcagcaac agttgtgaga 5520
accttgaaag gccatctcta ttaccatagc gatgataccg ttcaacaaaa ttgtaccatg 5580
caatattcac ttatagctgc ttctacaaag cacgtgtgtg gcaaggtagt ttacactata 5640
ccagcccatg tctcaattgg ctcacttaaa ctgttgcttg tccacccgga tcaaacaaat 5700
ttaccatttg aactaccagt ttcagatgaa gtccgtcttt gctacctcac aaccttgaac 5760
gcaccaagat gcatgccctc tcaactagcc atgtcaaata accaatttgc cgctgtaagc 5820
cttgttttgt taataacatt agtttcttta attaaagttt atataatgtt ttttactgtt 5880
tttaaacact acacaacaac tgtttttata cttgtagctg tgactactat cacaatgttg 5940
gtgtccttct tagctcctcc acttctcata gtcgttcttc tttcactagc atggctatgg 6000
tacggcaata caattgtatt gtgccatatc atgcttttga tagtcttagt cgtctcatgg 6060
aaagtggctg ctgtctgttt catcttcgcc ttattgtact ttggaaaatg tgctatgctt 6120
agcaagaaca ttaaatacgt acagggtgga gttaaatttt caggaacctt tgaagaaata 6180
gctcagtcaa ccttcttcat taactacgga gtagcttgtc agcttctgga acatactgga 6240
cagacaattg aggatataat gcaacttaga accgcgggtg gagccccagc aaggcttgcg 6300
cgctcaatat acgattgctt ttccacaaat gcctctgtct tgtacagtcc caggtcattt 6360
tcaccacagt cacttataac aaaatattta tacccaggtt cgatccctgt cggcagagcc 6420
cctgtcttat taggcaaaat ctccggcatg acttgcttag ggcgtgaaca gtccacctgt 6480
ttccaatcat cagctacaac cattactacg tgtacccatg ctgtaaatac tgctggaaca 6540
ttcatgtctc aaattaaatg tgttatagat aataaaatat atacagttca acccgagaat 6600
ataaccataa ccggaatgaa agctacattt gaagttgaag gactacctcc attcaccaac 6660
gatgtaacag tggccccaaa gccgctgaag cattacatgg atggaaagag acaccttgtt 6720
ctctacacta aaagtgagag catagtctac tcttcaataa tgtggccgac tgaaaacggt 6780
ttattctcgt catcagtttc tgacccagga gattcaggtg caccctactt ttcagacaat 6840
gtcatagtag gaatacacca aggtcgcaac gaagcaacca acaatcctgc cattttagca 6900
agtggtatgg atggtgagtc tccctgtgta ggttacgatg accaatcata tggccttcca 6960
cttcaagaat atttcactca cattgtctta tcaaataagc caagtgactt tggtgctcca 7020
tctaacgtgg cgccaaataa atactacaac aaaaaatcat ttgaacaatt agctgacgaa 7080
gataagactt atttaaatag tttatcatat cccctgtcct catctaatta ttgttacttt 7140
aatagcttca aaacccaatc aagcacaaca atgctcgaca acgctgaagt tattaaatat 7200
gtagttttgc ttctcatgat cttggattat ttcttttcaa tcatttgcga agatgcttta 7260
aacccagcat cttacgctat gttagttatc gttttggttc aggcttttat tacaaaaatt 7320
acagttttca gaacaggtat ctatatccag gcagccgttt ttcaagcatt tattgtacct 7380
atagtcagtc aaattacatt gatactggct gcagatactg caagaagttt tttaacgttc 7440
cacttttttg tacttgctgt tttgacatat ttcgttcttt gccgtattgc tgtagatttt 7500
tggcgttcca tgtttttgct atttttgaca agcgtctttg caaccatcat atggactaca 7560
aagaatgact tcaatatttt acatgaaact ggcgtggttc taacacccac ggcagaatta 7620
gctcttatag tagcttttac ttatataatt tatgcttcat gtatgttaac acctgtacca 7680
ctgtatacta tttgtgtttt cttttcattt ttatcaaatg ctccactcta ccttgccgtc 7740
ctctcattcg gcattctagt ttctttcaaa acaaaccaag attttggacg tctagtggat 7800
aaagtgtttt ctttaaatat gctctatgaa taccatgctt accaaaacta tgttattcaa 7860
aactcaggtc aacacccagg attttacagg tcactctttg ctttttttat caatttgacc 7920
acccaaccaa aaacaacata caaatgtttc aaaccccaga cagcaagtgg ttacagagta 7980
atatatcaaa ctcccactac agagttcaat aaatctctgc aacatgccag tatcacaaaa 8040
gatgacaact ccaaccatat aattatgttt gctgacggct catctgataa tctcaattgg 8100
gcaaaagaaa tggtcgcaac cattcatcta accaacccaa atttgcagcc actcatcatt 8160
ggatactacc acaactccat ggacgtcata accaagggaa cttacatgca acatgaattc 8220
ataaaaatgc cagctgttat cttaactcaa gatcctctaa ctgaaccaat cagtcattta 8280
gcagcagcag catttacttc aatttctgga aaacctcagg cacagaaaaa caacgttgtt 8340
tcaaactcca aagcgcgcat aaacacagcc gttcacgacg ctgtcgaaag cgtttattca 8400
ggagaaacat acgttgcccc caaacctata gtctcaggaa aaactgttgt agagaaacca 8460
ttctctacaa ccgaaaccac catgtacata atgcgtggtt tacccggttc tggaaaatct 8520
ttcaaagtta gtcaattagt tgctaaagat ccaaatttag tcgtagcttc cgcagaccac 8580
tttagatatt caaatgacaa aactggaaaa gccgtataca cctacattcc agaagcaact 8640
agttctgtac atttacaatg tcagaataga gcccgcaaag ctctagaaaa cggccaatct 8700
gtgtgcattg ataatacaaa tctaacactc ttagaaatga gaccttacgt cttattagcc 8760
cgttctttta actataacat tgaattcata cactcagact ctccctgggc cttaaacctt 8820
gacctgttac atgctaaagg tgtacataat gttcctagag caaagctcgt aatcatgtat 8880
gatagattct ttgaccgtga taatcaaatc gatgcagaca gtcttataca gtatgttatt 8940
gaagcaattg atccaaaact tgttgctcca atcatgaacc gtttccctgc cgactgtgat 9000
cttatccttc aatctgccct aacaccagac cttgaagtat taaagcaaaa ctacgacaga 9060
gcaaacgcaa cataccaaga tgtttcttta gatgatcctc cggctttaaa ggcagcacgt 9120
cgtgctatga atatagctaa atctgaatat gaggcaggcg aagcaggcca gcgtcgcatt 9180
gagaaatttt tagaaagaca ggatgtagca gcactcaacc aaacgctcac aactgtcaat 9240
caatctaaat tcatagcagc gatccgttcc atctacctaa gcaccattag caatttgaga 9300
ctaaaaaccc gtcatatggg tgaaggatca tatgcagtta catcaggtac taatactacc 9360
gataaagttt tagttaacac gccacaacgt atgactagaa ttgaagatgg catttataag 9420
cttgttgcaa acggttttga aatcacaatg tgcgacggca gcaacttagc cggtgttact 9480
tttgaacagg atataaatcc tagcatgtac ccttttgttt ttacattaat gtcaaatata 9540
gctgtacctg ttttaacccg ccaagcaaat gttggctatc ttgatatgtc aaataaattc 9600
atctgtaaag atggcactgt tcaatttcaa ggtgtcatct atgcctacca cactccatca 9660
aatgagagtg ctgacttcaa agtaggcaat accagttgga ccctccagaa aaacatcaat 9720
ttgactgctt ttattcctgc aattcataaa actgcaacct tcgcagcaca atcagtgttc 9780
ttaggaggac tacccatgga agagcaccaa gccttttccg acacacccac agcctcaaac 9840
aaatttaaag tttttgtttc atccacagtc tgcgcctcaa ccgtgtgcaa agtaaatcat 9900
aaaacttatg tacagatacc agatgacatt caagatcctt ttacatatat gcatcacagc 9960
gtttgttcac acaacaaatt tttatcaaac catgaaacca gatgtcaaat ctgtccttta 10020
aactgttana gcgcaaatcc gtgtgtgtct acggcttgcg ctctatttga taatggcaca 10080
ttacctcggt caacacatta tattaatgtt agcaccactt caaatgttgg cttgtttaag 10140
gcagtaaaga agtctactcg tcaactaaac attgacggtt ttccttacat gctaaagcag 10200
gttaaagacg actcagaact tgtaagttct cttaaaatag gtctacctaa tatcctccca 10260
catcacatgg tggaaactaa gtcaaaaaca taccttctta ggggccccac aacggcttac 10320
tcacttggcg atttatgcta cgcactcttt aatggcgact ttgattatat tcgcgaaaat 10380
ataaactctg atttcgtttt ggaccgtgaa gccggaatgc ctgatacaga aacacgtacg 10440
tggctgttca gcattttaaa ctttgcagta cctagagtgt gtgctataat tgaccagatg 10500
atttctgaga acgtcttcta taaactgact ttggataact tagatctata cggatcactc 10560
tatgattttg acgactatcc tactgaaggc tttaacaggc ctgatgatgt gatacgtatg 10620
ttaaaggaga tatggtcctt ctgtagacgt ccactacctg ccgaccttct taaataccat 10680
gaagacatcg gtgcagcagc cactcaagaa atattgctgc atgcaccctt cattgataaa 10740
gtttgtgctc taaatgacag attagctgtt gttgataata gagcaagtca atactttttc 10800
tgtgaagaag aaggtgtctt tacccatatt tacaatccag tctacggaac tttagcattc 10860
gataacaagt tgatccaatc aaaggatcct tcatgtacat tacagcgcct cattactata 10920
caaggccctt tgtctacgaa tgctagtccc gtgatctcta tttctgattc cactcatatt 10980
gccaacaata ttaatccatc taaccaaaag acaacaccgt tgtactacga tttggaactt 11040
gcgcaagaat tcattgacgc aggtttaaat attgatggcg tttccaacta cttcttctat 11100
ggaccgtcta gagcgggtgt agtgtctgat ttcttactat atgaattcca aggaactcaa 11160
tggtttgaca ataacatgct gcgctctctt tattctttca tattgaagaa ttcagagtgt 11220
tacagaacaa cagatcaact ggactttaga ggtggaaaac cccgtaaatc ctcaatggga 11280
catggtgtta ctggctttaa gcaagacgtc gtgtacgctg ctttaggccc tgatatgatt 11340
gaaaccttgt atgaaacggc aaaacaaaca ccattgccgt tttgtacaaa aataactgcc 11400
aagtatgcat taacagcaaa gcctagagct cgtacagttg cagcatgctc ctttgtagcc 11460
tcaactattt ttaggtacgc tcacaagcct ctaactaata atatggtctc aaaagcacag 11520
cagggtttgg gttattgttt aattggaatt tctaaattcc acggtcgatt taataaattt 11580
gttaagtcta gggtaggcac tgtcgaagac tttaatgttt tcggtagtga ctacactaaa 11640
tgtgaccgta catttccctt agctttgcgt gctctttcag ctgcccttat tttcgatctt 11700
ggcggccatg acccagacaa ctgtcttttt attaacgagc ttaatgcata catgctagac 11760
attgtttcag tcgaagactc ctttgcaaat aaaccaggag gtacttcatc aggagatgcc 11820
actacagcat actccaacac tctgtataac tttgcagtcc actatattat catgtggaaa 11880
acattcttga cagtcaatga cccttctacc aaggtcatac gcagtgcagc tcatcacgcc 11940
ctaacaagtg gtgacttctc tatgtacaat gacatgatac aagacatgtt ggatgtagac 12000
tatacactca acttcctctc tgacgattca tacatctgtt caaaaccaag cgcttttccg 12060
atctttacgc tcgagaacta tccttctaaa ctgcagtcta tactccacac agcagtagat 12120
agcaaaaaat cctgggaagc aaagggtgag attaaagaat tctgttcctc tcacatagtc 12180
aacgttgacg gcgactacca ctttaaacca gaaaaggata gaatattggc ttcattgctg 12240
atattatcga aaatcgctga catggacatc ttctttatga ggttcgttgc gttattggct 12300
gaatccgccg tatatatacg catcgatcct acattttggc tggccctgtt tggtgttttc 12360
gaaaaccgcg taacagcgtt taaatctgaa acattgctct cacctgttcc tgaacaactc 12420
atgaaggtgg ctttttatga atcgcttgtc tttgccgacg tggatgctac agccttatat 12480
ggtttccttg atggttttaa aatgcaaagt caaactctcc acccagacgg tgttgagggt 12540
tttgacaagc aaagtgaccg agtaaaacac tgttttgctt gtgacaatat atcagttgga 12600
cactgttcga tttgtcccgt tccccttcct ttgtgctctt tttgcttcta tgagcatgct 12660
ctgctcaatg aacattatga agcttctgga attgcgtgtg aatgcggaga cgctgacatt 12720
agacaacttc acttaaaaat aaccaatcaa ccatcctcgc acaattttat ctgtgctgaa 12780
tgtcccactg tagctatgaa gctgccaatc ttcaactctt tccaaggaaa agtactgctt 12840
ccaatgttcc gtatgaatac gccattgcct tcctcagtct ctgtaattgt tgatgtacgt 12900
tccaacccaa aagcacctaa gatgctgtgg gacgacgtcc agaatttcag agaaaattgt 12960
actaggatag catacgaatc cgtttcgtgt gctgaactag ctagggaggt ggtttactat 13020
ccatatgaag tgattgaatc caaagcaggt caagcacgac ttagaataca gaactttaaa 13080
tgttcaccaa ctacttatgt tcagttctac aaagtccgtc aaaatggaaa gtattgtcta 13140
gtagccaaag caactctaac gccggctttt gaaaaccaaa cagacatttt ctccgttttt 13200
caaccaaaca acttttcacc ttggaataca tcatcagtgt ttgcagtaga acaatacgct 13260
gcaatatacc ctcccatacc aaaggaacca gtcaatgcta cgttcgtctt aggacctcca 13320
ggctgtggta aaacatacta catagccaaa acgtactttt cacaggcttc tgagacatgt 13380
ccggtcgtat actgcgcacc tactcacaga ttagttttag atatggacgc agaatatagt 13440
ggtgtagttt caaaatctct ctacaataat agagtgtaca aaaatccagc ctacaaaaca 13500
ggcgaaccat tcaaattatg tttcaccaca cacaacacga tgccagttca aaagaaagcg 13560
atcctcatta tagatgaagt gtctttaatt acaccccact ctctattttc gatcattggt 13620
aaagggttct atgagatagt actcgtagga gacccttttc agctctcggc tgtttttcca 13680
ggttttgttg tcaatcacac atatgacggg ttttacatcc gccggctagt aaataaggtc 13740
aaacacctaa cagtttgtta ccgttgtcca caagaaatct tggacatatt ttctaagccc 13800
tatcatgatg ttgggattga cctcacaacc ggaaatacca atccaggaaa ggcatccatt 13860
tatacactaa attggcttca agcagatgta ggtactaaaa atccggacaa actcagacaa 13920
ctctttgcgc aatatccagg ctttaaaatt atcaccaact acagatgtgt tgttgatgca 13980
gctaaaagtt acggtattaa cgtcgaaacc atcgactcat cccaaggaac caccggagat 14040
aggcatctgg tggtaatttg cggcagtacc aacttttcta aacttttaaa caggtttata 14100
gtagcagcct ctcgttcaac aactgaacta gttatagtca tgttgccaga gctttacaac 14160
tatttaacag agacgtttaa cttcaaaccg ttacaattgc aaaatgtgca tgtaccgatc 14220
gcagtatctt ctacagcatt ctgcgatata gaattttatc actttcaaaa gaagttctat 14280
gttggtgaaa taagcgtaag cacaagtacc actatgacat gtcagttggg ttgttatatt 14340
aatggctcct acatgctccc acctgtgctt gaaaactctg aagaccgtct ctacgttcct 14400
tctagatgga gacgtatgat aagaaaatac cctactgaat ctatgcacat ttccttactg 14460
gacagacttc tgaggcacat tttattaaca actactggag aaattcattt cgtaatgttc 14520
tctgcagaca atgatctcat tgcactggat ccgtacttta taccacccac tctatgtgag 14580
tgtggtagtg cgggtctagt ggaagtagac atcactgttt tctgccgcaa ttgtttgcct 14640
aaagatggta aagccactcg tttggtaaaa ccgtctacac tagatgtcca gactgaaaaa 14700
ctcagacttg caaaagttca tgctaaggtt tgtaaaatca agcatggcag tgctcacaac 14760
gctgatgttg atgctattat gactcaatgt atatatgcta atagcttaac attcacacca 14820
acaacccaac tagttgttaa cactgatgag ttcacctttt acatgctacc taggccgtca 14880
aaccgtcatt tgagaatcat tcataagaac gacaaacgtt tctatgctat cactcatgaa 14940
gaagaagatc tcttctttac taacatctca gcagtggtag acccaattcc tgcaaaattc 15000
aacattgcac actctacaag cttcctcacc atcaaaagtg gttgcgcagg taataagact 15060
tgtaccagat gctattattt acacttagca tacacggaat ttgtttctca acacaagtat 15120
gaaccattca cttgtgtgtc ttttaagata cggtttgact tttcacaatt cactgactca 15180
gtagatactt tcctccgaca aggcttaata acctttcatc cggagatgaa ttcactgcaa 15240
aaatcacttt tattagcagt ggataaggtc tattgtgata acttcacctc aaacggtaga 15300
aggtttagac tttacgacaa caatttggtt aaatccataa tcaaaggttc agtggctcaa 15360
aactccatca tcatgccact cgactcagtt ttacacgggt tgaacattga tttcacagtc 15420
ggatgtgccg tagataactt ttcctgcaaa gaagcagcga gtgttaggta ctcagaagta 15480
gtactttcca tcaccaagtt gcccccaggc acttgccagt tatactacgt catatcttac 15540
ggcctgaact ctcccaagac cacttatgct ggtcaccaat tgttcgacgg ctttgagact 15600
gttattgttg ttaatcgtaa agataaaccc ccttacgtcc tcacacagta tattaatgat 15660
gttgtagttg caatgccaga gtccctcttt tcaacaggtc gattctacag agaaaaacca 15720
tatcccgtcc ttatgaacga ggatttaagt ggcttaaacc atcacatttt ctctggtgac 15780
tatacagacg aatctcttac attaggaggt gtccatcata tagtaacttt aaacacctat 15840
gaccacaagc tcaactatat ccaaacgaaa gctacatgtg ccgcctcagt ttcaactggc 15900
ggacgtggtc ataaaattac tacactgttt gacgttcatg caaatcaact tgctgatgaa 15960
attaccagag ttacatctgt tgttacaaca cagtctaaag ttattaattt gacaatagat 16020
tatcagcaag ttccttgtat gtactggtct tcaccgaccg gcataagaac cttctaccct 16080
caggctgtta gactggacgc aaagtttata ccatactacg tagaatatcc caatattcta 16140
ccggcagttg ttgaagacca ggtgtacgat ttgtctaatt acaatcaacc acctttaggc 16200
caaaactgcc ctgtaaactt tcacaagtac gtccagctaa ctcactttat tttagatcat 16260
gtgaaaatcc ccgaaaaggg tttgatatat catatcggtg cagcaggtac taagcaatgt 16320
tcacctggag acttaatatt ggaacaattt ttcaataaat ccatcatata ctcaagtgac 16380
cttcttcctt accaatcacc tgctgtgcag gttgcattgg atgtaaggtt ttcggcttca 16440
ctcatcattt cagactgcta ttcgaaagaa ccgcagcctg atttgttgag taagttgatt 16500
aacaaactag tgtatggtgg aactctcatt tttaagacca ccgagacttt cacatgtgac 16560
ccagcctttt atgttgctca ttttaactgt ataaagtttt ttactgccgc tgttaatcac 16620
tcatcatcag aagtttatat tgcgttcatc ggaaaactcc ctaaaccaaa caacaacttt 16680
ttagcctcag actatttcca gagattaact caacatagaa ataaagtagt taaacagcct 16740
tacgctcaca catgggacac atcttttacg tacccatacc cctcaaatgt tcttcaagtt 16800
agtcgtaaaa accttttata tctatttgaa accagaggag ctgcagtagg tactttgatt 16860
tttgaagaac catcaaaacc tgctgtaaag atacctacaa agtgtcaaac cacacaaccc 16920
tcgtgtgtca ttgaggttgg taaccaatac gattgttgca ttcaagacat cattaccctc 16980
ctcaatggaa aatccttcac agtgaaggtg cccaactcag aatccttact gcgcgatatc 17040
tgcacacttg cgcttagcca gagttattcc atcaatattc gcggaaaaac actctacacc 17100
cttagttccc tacttagaat taggcaacaa tccttactgt tttacggaga gaaggtcaaa 17160
aaccctcgac cccgtaatgt cttgaacaaa tataccaact acctcaaggc aaaagtgatt 17220
aggcattaca ccaagcctca atcaacagtt ttggacattg gtacaggaaa aggacaagat 17280
ttgagaaaat actcgttagc aggggttaaa tccctcactt gtgtcgagcc tagtcccgag 17340
tctgtgactg aactttcaat aatagctagt ccccttgata tggagacaca cacagttatg 17400
agttctgccc agaaattcga gacctcgctg acgtttgact tggctttctc tttctttgcc 17460
ttgcactatg cattggatga cgtttgtatg tctgaaacac tcaacaatgt tttttgcaaa 17520
cttaacagta attcacagtt gatcttagta gttccaaatg ctggcaggat gcaatccata 17580
ccttcccttg gtttaacagt cactcatcta gatgatgata aagtttggtt taaatactca 17640
gactatatag actgcgaaga accgttagta gacaaagaaa aactacttac gtgtttagct 17700
acatatggaa caattgttac tgactcacca ttctatgacg gtgcaaacaa aatcctagac 17760
caaaaatgct catccatgta tagagcatcg acagcccatc taaatcccga tgaaattcaa 17820
tatattaata tgtatgattt aattgttgtc attaagaatt agcacaacaa aatgttcgcg 17880
ctcgttctaa ccctcacaat agcttcggct attgcccaag atttccccgc atatgacccg 17940
tgtcctactt gctcaacccc cggtaataaa ataccggctc cgagcacagt tgcccagtat 18000
tcaacaaact acggtgcgaa cttctttacc gtagtctttg atggtattat cttcaaccaa 18060
tttagggaga gttattacca ccaatgtaga ccaacacctg aatactgccc agatgcaatc 18120
aattgcgcct taaacagaac aggcgcatcc tgcaaacctt tcgcaactgg cccgaattca 18180
caatgtcaga acagtttcga gggcaacatc gacatatgtg caacatgtag ccctctaaaa 18240
caagaaactc cattcatctg ctacaataga tacgggataa ttatataccc gacagcagat 18300
atcgttctct ccgctaggtt taagataggc tctttttcac ccaaggcttg tgataactac 18360
ctaaacgact taaattgtga ttcaaaaacg gcaaggtcat atgtcatttc ccgaccgcag 18420
tctttttcac tgcaatatcc taactcatta ggcccctatc agctgaaacg attttctctt 18480
gcaaaggaga tcgttgactt acgtgctggc gttttaacct cactcccaaa ccgaggttat 18540
aagggtagaa caacatactc ttatcccgtc actgcactct cactcttggc tcgttccaaa 18600
gtggctgaag ccgacaaatt cttttatatc gaggctaaaa ttctactgta cgcttggtca 18660
cagaaacctc aaatccgctt tctaggtgca tactgtccca cagacgtgtc atgccctgat 18720
tcaactgccc tcggctgttg tttttccgga agtggatctg agttttacta cgcctttcgc 18780
cagtggtact acgcaagcct gggtatggaa gacctagtcg actttgataa ttcaacagtc 18840
ttaagtctct cgcctgatac tcctcaaatt acacccgttg tgtcttattt tctagaaaaa 18900
gttttacctt tgtttaaatc acatgtaccc ggacgtgttt tttactgcca ttcacttatg 18960
tctaacggtg tatgtacttt tgaccatgtt gttgtaaata ttaatgccga ggccgtcttt 19020
tttgacctcg aagtagacat aggcagcata attgctgacg catatcgcgt tgaaaggcct 19080
aatactttat gttatgatac aaactgtact cttgccacaa gcaggaccac tgagtataat 19140
tacgctgctt atgttgttta tatactcttc aatttgtatt ctagtaatcg cattgcgata 19200
gatttcaaca cacactcaat cttgcaagga ttactacaac acaatagcaa ctaccagact 19260
gctaatttag actatctgtt tgttggagca ctttttacag gtacttttaa acatattaca 19320
agcaatcaag cttacccagt acctttaact tatccaattg ttaagacata tgtagggccg 19380
tcaaaccaat actcaatgtc aaataaactg ttttcatata ctcacaattt gacggctcaa 19440
gcccattcag gcatatgtaa ctctttttac tgttataaac cacgttttgt accaattgat 19500
gtttttattc atagtgcttt aacccctgac agcttgatgg aaacagaatc ttttgtttgt 19560
gtctctttgc gttcaccatc tgcaggatca acatccgcag gtagttttta tttgcaatgt 19620
ctcaattctt ccatcgattt gcatccaggt tcatttgtac ccgtttcctc aagtccagag 19680
tcttccagcc gcgtaacagc tgagctggct tttaatacta gaaatggtat attttctcct 19740
tgtcttaacg gtacatgtgt actcgcacct actgacccaa ttgtttttat gcgtcagggt 19800
gcctggttta caaaatcttt acactttgat gtttcaccat gcaaacctat gcattttcca 19860
gacatagata tacagccccc aacatacaat gtctcctcta tcaagatgga cgacaatgct 19920
gtattggttc aagaccttac ttcgggttta gtaattgacc acaatttagg ctccatactc 19980
agaccgaaag gtagagcttt ggaagtttcg tattatgctc actccatttt acgttacctt 20040
gaaccggatt cttgtctacc tgacaacttt cttaactttg tcacttgttt agactatatc 20100
tgttcagact cgtcaccttg ccgtgctgcc gcaagccagt actgtcaggc aggcatttat 20160
tttgagtctg catttaataa gtctaggtat tctttgctta acgcttacac gctttttaac 20220
acaagtcttc aaaccttatt gcctgagact tttcttgaga tagaagatga tgaaccccat 20280
agcagatcaa agagatcaat tgatactaca agcaatattc gccctagtca attgcttgtt 20340
aatggacgta ttccgtctac aagttcagct tttgctgtta acgtcgctcg tggtcgagga 20400
acgattatgc ctcgtcctgg aactggtggc atgggttcgt ccttttctgc tgtttctagg 20460
tcgggtagta tttcttcctt atcctcggtt ggctcctcaa cacctttgat ctctaattgg 20520
agaacatctt catctcaact caaaactctc aacctcaaca ttaacactaa aattcctaag 20580
atttcaacaa agtcaggttt tgccagtatt acatctttgt ttgcttcagg tttaggagtc 20640
gtcgatctag gtctatctat tttcaacatg atagaacagc gtagagttgc tgagatcact 20700
cagatgcaaa ttagccaact ggctgactct atagtgtatc ttgctgatgt gacatttgaa 20760
gctatcaaga atttggaact ctcggttaac tccttgggta cgttcttatc ggaattttcc 20820
actcagatgt cgatcaccat aagccaaata caatcatcat ttgaagagca gcaagatgct 20880
acaaatgatg cgttgtacta cactaacgct gctgcgtcat accaagcctc catggcgtat 20940
gtcatttcag agttaaacgc aatatctctg tctgtcacta gatcctacga ctcttacacc 21000
agttgcatca cttctggcat taatgggctc attacaccat catgcttgcc agcccaccag 21060
ttgttacagt tactcgacac cgttatcaat tccacagcag gaacaggatg ccgtcccatc 21120
tacggcagag aagaagtggt gaaatactac actttacctc taatcaatca aggttattcc 21180
tttaacgggt cgattttctt cgtctttaac attcccatca cttgccaggg aattgccgga 21240
gatgtatatg aagtagaacc acctatactt gtagatgtac catcaaagac tgctttacgc 21300
atgattacac catcaaacgt agtcgcaaca caagcaggat tagctgaatt agatttgcgt 21360
cattgcgaaa ggtaccataa cgagttccta tgcgattctt cagcattcct ttctacacct 21420
tcaaaataca tagactgttt aacaaacgca actgactgtt ctttgcaatt catcacacaa 21480
cacgttccag atccttgcgt ttacacatcg ccagcttctt tatattgtta ttattcaccc 21540
atatgtgatc aatgtcacat agtagccggt tgtaatgaat ctcagcagta caacttcact 21600
tctgctgatg gcggcgtagt cttttattcc atacaagaca gagactgtgg ccacttcccc 21660
cacatcactg ttactacgcc tgcagccata caagaagact tcactgtcgg accgtattta 21720
ccatcgctgc caattcacac cgcctacgtc aatgttacct ggaatgtaac actaccagga 21780
aattggacct gggaaaatat caccctaaca gccaattgga cccaacactt cattgagatg 21840
aaaaaaaaca tcacaatgat ggctgaagaa atagataacc ttaccaactt cggtaaggtt 21900
ttagttggcc agctaaatag ctttttatca tctttgttta acataccatt aggtttgatg 21960
acgttttgct tttctgtagc cgctttaggc ctgtccatta ttgctttact tgtgttatgt 22020
tttccacaga agccacataa attataatcg tggtttcgct tgtaaatatt gatcaattga 22080
ggttttttac actttagtgt ttttcctcaa ccaattagac cagaggtttt tttacaccaa 22140
agtgtttttc ctctacaaag aattgaggtt ttttacactc tagtgttttt cctcaaactt 22200
atatatataa aatttcatta gtttgacatt tcattataaa tagcacaaca aatacattca 22260
ggcgacttgc atgatgttta ccctagtagt gctttttacc ctcctcggcc tttccatggc 22320
ctccacagag ctgaatttcg atcctactct acccctcccc tctcctataa atgccctcgt 22380
cgacattttc ggaaacaaca gcttgtttct caaagagtcc ctgctcggca aatccaccgg 22440
agccgtctac gcatacttgt acagcagtgc catctctctc ctgctgctac tttgggtaac 22500
tgtatggagt attgctactt cacactttaa cgtaactcgc attccaacca tcgcggttct 22560
cactaatgcg agtatgtttt tgctgttggc atcggctact gttacaacct ggtttctccc 22620
aactgtgacg aacgtcttct tttatacact cactgcgctg ttcaccttct tttcctttgt 22680
gttcttactg tggttggttt actatatgtt tactaccatt agggcatatc gaagggtcgg 22740
ttcatggcgc gttgtgttta acggaaaata ttctctactt gctggaactc aggctgtttg 22800
cctttgcaga cccgccatac atctggttct aaccaaaacg aacacagata catactggtg 22860
tctagatgga acccccatct acaatgttga cttactacaa ttagttggcc ccaaaggatt 22920
atatccttac aaaagaatga ctacaatcac tgcaccaaaa ggcacaaaaa catctgctgc 22980
cgtttacacc cttcaaaaag aagaagtttg tgctctctca gaaatcacag tacataatga 23040
tactgatttt taggtcatat aaaaaagcta acacatctaa aaaatgtctt acccggttta 23100
ctacgaacag cgtcgttatt ccccccgcca attcaacaat ggcggagggt ataatcctac 23160
acctcaacct agagtagttc gtactaatcc tggtaaccaa gcttacaacc cccggcgtaa 23220
ccgaaacgcc actccgaacc aacaacaaat ggttccttac cagcctcagt atcaagcacc 23280
tcctcagcca agggtggtct atgtagatcg ccctcaagaa cctgtagtaa tttacagagc 23340
tcctccacaa ggaaaaaaac aatcaggcaa acgccacaca gcagaagaac gctggtatca 23400
aggcgaaaaa cctgtgcaga agaaacaggc acccaaagga aaatcaaaga aagcagcaac 23460
acctgctaat cctaaaaagc agcctacaca atctgacaaa gttcccatcg cctacccaga 23520
caatcatccc ttccatgacc tcgcaccagc tgacatccgc gctttcaaaa agcagctgat 23580
ccaaaatctg gaccttggac atggtgaaat gaatcaactg cggctttcaa tcgatctgtt 23640
gcccatcaag aaaccagcac caacaccagc ggtgccagct cctctgtaat ttatggaaaa 23700
ggtgcaagac ctcccataaa taagtagata tgttcattac cctcattatg atcttcgcca 23760
tcttggcttt cccttcaaca tctgaaggag cagcccaaga actactcaaa gctgtaaaat 23820
ctgctgctat catggaaaag gtgcaagacc tcccatgaaa atagtagccg gtttcaaaag 23880
ttgataatta ttgcattatg tttacccttg tgcttattat cctgcttagt ttttctatgg 23940
cttttaatgc ttttacattt ctgctgttat tattttttac ttttaagtgc attataaccc 24000
gcactttagt cgtagttccc attgactacc cagaaaatca tcctttcaat ggcctctcac 24060
cagaggaaat catcagctac aaatcacagc tgatccaaaa tctcgatctt ggacatggtg 24120
aagtaattaa acatcgattc tcaattgatt tacttcccct caaaacaaca agcactccta 24180
ccaccagtgc tattttatgg aaaaggttca aaacctccca taaagaaaac aaccactaaa 24240
caaccatgga aaaggtgcaa gacctcccat gaaattagtg gttgctttca aaaattaata 24300
aatattgtga taaatgtcta tttctaacca cccttaaaaa taggtacccc cactatatct 24360
agccgacgtt aactcctgga tatgttatag tgttctctcc ccaatcgttc atctgctcgc 24420
ttttagaact gctaggctgt atctgttaaa tgtttaatct ttagactcaa tttacgttct 24480
tttttacata aaatcctcct attttgctat cccttatttt aattaaaccc ctttagtatc 24540
accagtatcc ctaatcactc ccctagcctc ccttgttctg acctgtatga aatgtcaaaa 24600
aactaaatga aaa 24613
<210> 7
<211> 5867
<212> БЕЛОК
<213> Вирусы
<220>
<223> аминокислотная последовательность ORF-1 ("X" в положениях 830-835, 841, 842 и 3257 обозначает любую аминокислоту)
<400> 7
Met Lys Lys Ile Glu Asp Ala Leu Gly Thr Leu Lys Pro Ala Phe Lys
1 5 10 15
Ala His Ile Asp Ser Leu Pro Pro Phe Met Gly Lys Leu Ala Thr Met
20 25 30
Leu Ala Glu Thr Arg Arg Gly Lys Thr Pro Pro Leu Leu Ile Tyr Val
35 40 45
Ile Ser Thr Ile Leu Glu Thr Asn Ile Thr Val His Tyr Val Ser His
50 55 60
Thr Ile Thr Ser Tyr His Ser Ser Asn Ala His Thr His Thr His Glu
65 70 75 80
Phe Asp Ser Glu Asp Tyr Thr Pro Asp Asn Gln Ile Leu Thr Lys Val
85 90 95
Asn Arg His Gly Asn Asp Leu Asn His Ser Tyr Ile His Gly Ala Thr
100 105 110
Asn Met Tyr Asn Pro Val Tyr Gln Arg His Pro Pro Asn Met Cys Tyr
115 120 125
Met Leu Thr Gly Leu Tyr Met Leu Ser Gly Leu Gln Glu Leu Tyr Ala
130 135 140
Met Ala Glu Asp Asn Leu Thr Thr Cys Gln Ile Asn Leu Leu Arg Cys
145 150 155 160
Leu Phe Asp Leu Asn Gln Asp Glu Phe Asp Val Asp Tyr Thr Phe Val
165 170 175
Ile Tyr Thr Pro Ser Lys Ser Gln Glu Cys Ala Phe Lys Tyr Leu Gln
180 185 190
Glu Ile Val His His Cys Glu Leu Thr Ile Phe Arg His Thr Thr Thr
195 200 205
Ser Val Phe Ser Cys Asn Lys Cys Asn His Val Glu Thr Val Ile Ser
210 215 220
Ser Cys Ser Leu Asn Leu Val Tyr Ile Thr Asp Ser Ile Glu Lys Ala
225 230 235 240
Phe Gln Pro Thr Val Glu Ala Asn Thr Asp Tyr Met Cys Glu Asn Cys
245 250 255
Gly Leu Arg Asp His Lys Leu Lys Thr Thr Val Thr Asn Pro Asp Leu
260 265 270
Arg Leu Ala Gln Leu Asn Tyr Pro Thr Asp Ser Lys Tyr Thr Ile Phe
275 280 285
Leu Asp Glu Gln Ala Pro Phe Val Phe His Ser Ile Ala Lys His Val
290 295 300
Gly Thr Ala Asn Ser Gly His Trp Ser Ala Leu Asn Val Asn Ser Asp
305 310 315 320
Met Leu Ser Asp Ser Asn Glu Arg Gln His Tyr Tyr Thr Thr Pro Ser
325 330 335
Ile Val Leu Leu Ala Phe Leu Pro Glu Glu Glu Leu Gln Asn Ile Arg
340 345 350
Asn Ser Ser Pro Leu Gln Asp His Gln Pro Asp Asp Val Glu Asp Val
355 360 365
Glu Ser Pro Leu Pro Gly Ser Ile Phe Tyr Thr Thr Asp Asp Ile Phe
370 375 380
Ser Thr Lys Ser Leu Ser Ile Ala His Cys Val Ala Arg Asp Phe His
385 390 395 400
Met Ser Gly Gly Ile Ala Lys Ile Phe Ser Asp Lys Phe Gly Ser Lys
405 410 415
Thr Phe Leu Lys Ser Gln Asn Pro Val Ile Gly Gly Phe Ser Ile Leu
420 425 430
Leu Arg Glu Cys Arg Asp Met Tyr Tyr Leu Val Thr Lys Glu Lys Thr
435 440 445
Ser Asp Lys Pro Thr Tyr Gln Asp Leu Lys Asn Ser Leu Gly Ser Met
450 455 460
Thr Glu Asn Leu Val Arg Lys Asn His Asn Thr Leu Ser Ile Pro Tyr
465 470 475 480
Ile Gly Cys Gly Ile Asp Gly Leu Gln Trp Ala Thr Val Glu Lys Gln
485 490 495
Val Lys Glu Ile Val Cys Ala Arg Gly Ile Asp Val Thr Val His His
500 505 510
Leu Glu Asn Glu Val Lys His Thr Pro Glu Gln Gln Thr Ala Ser Asp
515 520 525
Asn Ser Val Lys Leu Val Gln Lys Leu Phe Thr Glu Thr Pro Gln Ala
530 535 540
Ile Pro Ile Val Val Pro Ser Asp Asp Ser Asp Ser Asp Ile Asp Glu
545 550 555 560
Ser Ala Asp Val Phe Leu Pro Gly Pro Glu Ser Glu Ser Asp Ser Lys
565 570 575
Ser Glu Ser Gly Ser Asp Tyr Asp Phe Lys Ser Ala Ser Glu Pro Glu
580 585 590
Asp Glu Leu Glu Pro Thr Pro Ile Ser Glu Leu Glu Leu Thr Pro Ala
595 600 605
Ser Ser Leu Thr Val Glu Ser Asp Asp Asn Pro Asp Thr Ser Gln Glu
610 615 620
Thr Leu Pro Glu Ser Asn Ser Glu Glu Thr Lys Pro Glu Gln Thr Pro
625 630 635 640
Asp Thr Thr Ser Lys Val Ser Ser Asp Ser Lys Leu Asp Pro Gln Ser
645 650 655
Glu Leu Glu Glu Glu Leu Ala Asn Lys Pro Glu Ser Ala Ser Glu Pro
660 665 670
Gln Ser Glu Thr Glu Ser Ser Ser Glu Ser Glu Glu Glu Leu Glu Pro
675 680 685
Gln Ser Glu Ser Glu Glu Glu Pro Ala Asn Lys Pro Glu Ser Pro Ser
690 695 700
Glu Ser Gln Ser Glu Asn Gly Ser Ser Ser Glu Pro Glu Glu Glu Ser
705 710 715 720
Glu Lys Pro Ser Glu Ser Ala Glu Thr Ala Thr Glu Asp Ser Pro Glu
725 730 735
Thr Thr Pro Glu Thr Thr Leu Glu Leu Thr Thr Gln Leu Lys Pro Ala
740 745 750
Ser Glu Ser Asp Asp Lys Pro Asp Thr Pro Ala Pro Ser Pro Ser Pro
755 760 765
Ile Gln Pro Glu Lys Asn Leu Asp Thr Thr Pro Glu Gln Thr Ser Gln
770 775 780
Pro Thr Thr Gln Leu Glu Leu Thr Leu Glu Thr Gln Glu Gln Pro Asp
785 790 795 800
Thr Thr Pro Glu Val Pro Ser Val Ser Glu Asp Lys Pro Asp Thr Leu
805 810 815
Glu Glu Ser Ser Glu Ser Thr Pro Glu Leu Ser Glu Leu Xaa Xaa Xaa
820 825 830
Xaa Xaa Xaa Thr Ser Thr Lys Thr Xaa Xaa Thr Arg Pro Pro Thr Pro
835 840 845
Arg Pro Arg Ser Ala Arg Gly Ala Arg Thr Arg Ser Cys Ala Gly Thr
850 855 860
Pro Ile Pro Val Ile Phe Asp Ile Ile Asp Asn Thr Ser Gln Pro Gln
865 870 875 880
Val Pro Leu Asp Phe Pro Glu Ala Leu Gln Glu Leu Asn Lys Pro Ser
885 890 895
Glu Val Ile Pro Ala Ala Ser Glu Lys Pro Val Glu Lys Gln Ile Ile
900 905 910
His Ser Phe Val Ser Val Glu Thr Pro Cys Lys Pro Lys Ala Thr Lys
915 920 925
Val Thr Asn Tyr Val Ala Ala Gln Ser Asn Ala Ile Leu Asn Cys Ile
930 935 940
Lys Ala Phe Ile Pro Ser Asn Pro Leu Ser Leu Phe Asn Arg Lys Pro
945 950 955 960
Ala Phe Arg Lys Ile Ile Phe Thr Glu Asp Thr Ser Glu Pro Asp Ser
965 970 975
Asp Asp Asp Asp Cys Glu Tyr Thr Pro Pro Thr Ser Pro Phe Pro Glu
980 985 990
Leu Leu Ala Leu Val Asp Glu Asp Ile Glu Val Glu Gln Thr Gln Ser
995 1000 1005
Val Ile Pro Lys Thr Asp Ser Ala Ser Ile Val Glu Asp Leu Lys Lys
1010 1015 1020
Gln Glu Ser Ser Thr Leu Ser Leu Asp Thr Asn Thr Ser Lys Pro Thr
1025 1030 1035 1040
Ser Ser Pro Arg Arg Gln Pro Arg Glu Val Glu Ser Val Asp Glu Ser
1045 1050 1055
Ser Asp Asp Ser Ser Lys Pro Lys Thr Ile Ser Thr Leu Asp Lys Pro
1060 1065 1070
Ala Met Asn Ser Asp Thr Lys Pro Thr Asp Ser Ser Arg Lys Glu Pro
1075 1080 1085
Leu Glu Val Pro Val Thr Thr Ser Leu Ser Thr Pro Ala Lys Asn Gln
1090 1095 1100
Asp Lys Lys Ser Ser Lys Ser Ala Lys Val Ile Lys Asp Tyr Ser Leu
1105 1110 1115 1120
Thr Pro Asn Thr Val Lys Gln Gln Val Tyr Ser Leu Tyr Gly Glu Ser
1125 1130 1135
Val Asp Ala Val Lys Tyr Leu Val Gln Thr Tyr Pro Asp Arg Ala Lys
1140 1145 1150
Gln Thr Ala Gly Ile Ala Tyr Phe Leu Ile Thr Thr Tyr Leu Ile Trp
1155 1160 1165
Thr Ile Gly Leu Ile Gly Val Pro Met Ala Phe Lys Ile Pro Met Phe
1170 1175 1180
Leu Cys Leu Leu Tyr Gln Val Asn Gly Leu Asn Ile Ala Pro Phe Val
1185 1190 1195 1200
Thr Asn Gln Lys Leu Gln Tyr Val Ala Phe Pro Leu Trp Tyr Lys Leu
1205 1210 1215
Tyr Glu Val Ile Ser Val Arg Phe Val Ala Asn Ile Ala Gln Phe Ile
1220 1225 1230
Val Lys Thr Pro Pro Ile Asp Val Leu Asn Lys Leu Ile Arg Ser Asn
1235 1240 1245
Lys Asp Lys Pro Val Lys Leu Thr Pro Asn Lys His Thr Leu Met Leu
1250 1255 1260
Ile His Asp Leu Ala Leu Glu Ser Val Asp Gly Lys Glu Asn Arg Tyr
1265 1270 1275 1280
Tyr Asn Thr Asp Val Thr Thr Phe Thr Lys Arg His Ser Thr Ser Asn
1285 1290 1295
Ile Ser Tyr Val Leu Lys Ser Thr Leu Ile Lys Tyr Val Met Asp His
1300 1305 1310
Cys Tyr Val Asn Ile Ala Val Phe Thr Leu Val Arg Tyr Leu Thr Leu
1315 1320 1325
Leu Val Phe Ile Gln His Phe Ser Asn Pro Tyr Val Leu Glu Ala Asn
1330 1335 1340
Ser Gln Ser His Thr Val Leu Gln Tyr Leu Phe Ser His Leu Arg Pro
1345 1350 1355 1360
Phe Gly Arg Pro Leu Cys Pro Thr Leu Asn Asp Tyr Met Thr Thr Ala
1365 1370 1375
Thr Pro Arg Asp Ala His Val Gln Ala Gly Ser His Phe Ser Glu Phe
1380 1385 1390
Cys Val Pro Ile His Tyr Thr Thr Pro Ile Ile Lys Ser Thr Met Ala
1395 1400 1405
Glu Pro Ser Leu Phe Leu Leu Phe Asn Pro Val Leu Trp Pro Leu Val
1410 1415 1420
Met Val Val Tyr Phe Tyr Pro Pro Met Met Phe Ile Ala Asn Ala Val
1425 1430 1435 1440
Ala Tyr Ser Cys Leu Pro Leu Val Val Leu Leu Gln Trp Leu Tyr Ala
1445 1450 1455
Met Trp Phe Ser Cys Thr Cys Tyr Gly Thr Lys Arg Cys Ala Lys His
1460 1465 1470
Leu His Lys Asn Glu Val Val Lys Pro Met Glu Ser Thr Ser Thr Lys
1475 1480 1485
Asn Arg Met Thr Phe Thr Pro Ser Thr Thr Phe Cys Ser Lys His Asn
1490 1495 1500
Phe Phe Cys Pro Asp Ala Pro His Ile Met Thr Leu Ala Met Ala Arg
1505 1510 1515 1520
Gln Leu Thr Asn Tyr Tyr Asn Leu Thr Asp Thr Val Ile Pro Asp Ile
1525 1530 1535
Gln Glu Tyr Ser His Glu Asn Pro Thr Val Gln Phe Ile His Phe Asp
1540 1545 1550
Pro Leu Lys His Gly Ala Asp Thr Ile Leu Glu Pro Ile Thr Ser Ala
1555 1560 1565
Ser Ala Ser Ser Ile Val Ala Trp Tyr Ser Leu Leu Phe Asn Gln Lys
1570 1575 1580
Phe Val Leu Ser His Tyr Ser Tyr Arg Thr Pro Val Ala Val Val Asp
1585 1590 1595 1600
Lys Pro Glu Glu Thr Asp Gly Asp Asp Thr Lys Ser Leu Ala Ser Asp
1605 1610 1615
Thr Ser Asp Asn Phe Glu Ser Ile Arg Lys Thr Asn His Lys Asn Gln
1620 1625 1630
Ser Lys Gln Gln Phe Arg Pro Asn Gly His Gln Arg Pro Ser Lys Thr
1635 1640 1645
Phe Lys Arg His Ser Arg Ile Met Thr Ser Glu Gln Lys Asn Ser Leu
1650 1655 1660
Ile Glu Thr Phe Lys Gly Leu Thr Asn Gly Thr Ala Ala Ile Pro Gln
1665 1670 1675 1680
Pro Leu Ile Ile Phe Ile Trp Val Ile Leu Met Val Ile Pro Thr Leu
1685 1690 1695
Phe Leu Val Ala Ser Ser Ser Arg Thr Ala Ala Thr Met Pro Leu Asn
1700 1705 1710
Arg Tyr Ser Gly Val Asn Pro Thr Gly Ile Met Phe His Gln Ala Pro
1715 1720 1725
Pro Tyr Ile His Ser Glu Pro Pro Lys Glu Thr Tyr Tyr Lys Leu Ser
1730 1735 1740
Tyr Pro Tyr Pro Ser Ala Thr Val Val Arg Thr Leu Lys Gly His Leu
1745 1750 1755 1760
Tyr Tyr His Ser Asp Asp Thr Val Gln Gln Asn Cys Thr Met Gln Tyr
1765 1770 1775
Ser Leu Ile Ala Ala Ser Thr Lys His Val Cys Gly Lys Val Val Tyr
1780 1785 1790
Thr Ile Pro Ala His Val Ser Ile Gly Ser Leu Lys Leu Leu Leu Val
1795 1800 1805
His Pro Asp Gln Thr Asn Leu Pro Phe Glu Leu Pro Val Ser Asp Glu
1810 1815 1820
Val Arg Leu Cys Tyr Leu Thr Thr Leu Asn Ala Pro Arg Cys Met Pro
1825 1830 1835 1840
Ser Gln Leu Ala Met Ser Asn Asn Gln Phe Ala Ala Val Ser Leu Val
1845 1850 1855
Leu Leu Ile Thr Leu Val Ser Leu Ile Lys Val Tyr Ile Met Phe Phe
1860 1865 1870
Thr Val Phe Lys His Tyr Thr Thr Thr Val Phe Ile Leu Val Ala Val
1875 1880 1885
Thr Thr Ile Thr Met Leu Val Ser Phe Leu Ala Pro Pro Leu Leu Ile
1890 1895 1900
Val Val Leu Leu Ser Leu Ala Trp Leu Trp Tyr Gly Asn Thr Ile Val
1905 1910 1915 1920
Leu Cys His Ile Met Leu Leu Ile Val Leu Val Val Ser Trp Lys Val
1925 1930 1935
Ala Ala Val Cys Phe Ile Phe Ala Leu Leu Tyr Phe Gly Lys Cys Ala
1940 1945 1950
Met Leu Ser Lys Asn Ile Lys Tyr Val Gln Gly Gly Val Lys Phe Ser
1955 1960 1965
Gly Thr Phe Glu Glu Ile Ala Gln Ser Thr Phe Phe Ile Asn Tyr Gly
1970 1975 1980
Val Ala Cys Gln Leu Leu Glu His Thr Gly Gln Thr Ile Glu Asp Ile
1985 1990 1995 2000
Met Gln Leu Arg Thr Ala Gly Gly Ala Pro Ala Arg Leu Ala Arg Ser
2005 2010 2015
Ile Tyr Asp Cys Phe Ser Thr Asn Ala Ser Val Leu Tyr Ser Pro Arg
2020 2025 2030
Ser Phe Ser Pro Gln Ser Leu Ile Thr Lys Tyr Leu Tyr Pro Gly Ser
2035 2040 2045
Ile Pro Val Gly Arg Ala Pro Val Leu Leu Gly Lys Ile Ser Gly Met
2050 2055 2060
Thr Cys Leu Gly Arg Glu Gln Ser Thr Cys Phe Gln Ser Ser Ala Thr
2065 2070 2075 2080
Thr Ile Thr Thr Cys Thr His Ala Val Asn Thr Ala Gly Thr Phe Met
2085 2090 2095
Ser Gln Ile Lys Cys Val Ile Asp Asn Lys Ile Tyr Thr Val Gln Pro
2100 2105 2110
Glu Asn Ile Thr Ile Thr Gly Met Lys Ala Thr Phe Glu Val Glu Gly
2115 2120 2125
Leu Pro Pro Phe Thr Asn Asp Val Thr Val Ala Pro Lys Pro Leu Lys
2130 2135 2140
His Tyr Met Asp Gly Lys Arg His Leu Val Leu Tyr Thr Lys Ser Glu
2145 2150 2155 2160
Ser Ile Val Tyr Ser Ser Ile Met Trp Pro Thr Glu Asn Gly Leu Phe
2165 2170 2175
Ser Ser Ser Val Ser Asp Pro Gly Asp Ser Gly Ala Pro Tyr Phe Ser
2180 2185 2190
Asp Asn Val Ile Val Gly Ile His Gln Gly Arg Asn Glu Ala Thr Asn
2195 2200 2205
Asn Pro Ala Ile Leu Ala Ser Gly Met Asp Gly Glu Ser Pro Cys Val
2210 2215 2220
Gly Tyr Asp Asp Gln Ser Tyr Gly Leu Pro Leu Gln Glu Tyr Phe Thr
2225 2230 2235 2240
His Ile Val Leu Ser Asn Lys Pro Ser Asp Phe Gly Ala Pro Ser Asn
2245 2250 2255
Val Ala Pro Asn Lys Tyr Tyr Asn Lys Lys Ser Phe Glu Gln Leu Ala
2260 2265 2270
Asp Glu Asp Lys Thr Tyr Leu Asn Ser Leu Ser Tyr Pro Leu Ser Ser
2275 2280 2285
Ser Asn Tyr Cys Tyr Phe Asn Ser Phe Lys Thr Gln Ser Ser Thr Thr
2290 2295 2300
Met Leu Asp Asn Ala Glu Val Ile Lys Tyr Val Val Leu Leu Leu Met
2305 2310 2315 2320
Ile Leu Asp Tyr Phe Phe Ser Ile Ile Cys Glu Asp Ala Leu Asn Pro
2325 2330 2335
Ala Ser Tyr Ala Met Leu Val Ile Val Leu Val Gln Ala Phe Ile Thr
2340 2345 2350
Lys Ile Thr Val Phe Arg Thr Gly Ile Tyr Ile Gln Ala Ala Val Phe
2355 2360 2365
Gln Ala Phe Ile Val Pro Ile Val Ser Gln Ile Thr Leu Ile Leu Ala
2370 2375 2380
Ala Asp Thr Ala Arg Ser Phe Leu Thr Phe His Phe Phe Val Leu Ala
2385 2390 2395 2400
Val Leu Thr Tyr Phe Val Leu Cys Arg Ile Ala Val Asp Phe Trp Arg
2405 2410 2415
Ser Met Phe Leu Leu Phe Leu Thr Ser Val Phe Ala Thr Ile Ile Trp
2420 2425 2430
Thr Thr Lys Asn Asp Phe Asn Ile Leu His Glu Thr Gly Val Val Leu
2435 2440 2445
Thr Pro Thr Ala Glu Leu Ala Leu Ile Val Ala Phe Thr Tyr Ile Ile
2450 2455 2460
Tyr Ala Ser Cys Met Leu Thr Pro Val Pro Leu Tyr Thr Ile Cys Val
2465 2470 2475 2480
Phe Phe Ser Phe Leu Ser Asn Ala Pro Leu Tyr Leu Ala Val Leu Ser
2485 2490 2495
Phe Gly Ile Leu Val Ser Phe Lys Thr Asn Gln Asp Phe Gly Arg Leu
2500 2505 2510
Val Asp Lys Val Phe Ser Leu Asn Met Leu Tyr Glu Tyr His Ala Tyr
2515 2520 2525
Gln Asn Tyr Val Ile Gln Asn Ser Gly Gln His Pro Gly Phe Tyr Arg
2530 2535 2540
Ser Leu Phe Ala Phe Phe Ile Asn Leu Thr Thr Gln Pro Lys Thr Thr
2545 2550 2555 2560
Tyr Lys Cys Phe Lys Pro Gln Thr Ala Ser Gly Tyr Arg Val Ile Tyr
2565 2570 2575
Gln Thr Pro Thr Thr Glu Phe Asn Lys Ser Leu Gln His Ala Ser Ile
2580 2585 2590
Thr Lys Asp Asp Asn Ser Asn His Ile Ile Met Phe Ala Asp Gly Ser
2595 2600 2605
Ser Asp Asn Leu Asn Trp Ala Lys Glu Met Val Ala Thr Ile His Leu
2610 2615 2620
Thr Asn Pro Asn Leu Gln Pro Leu Ile Ile Gly Tyr Tyr His Asn Ser
2625 2630 2635 2640
Met Asp Val Ile Thr Lys Gly Thr Tyr Met Gln His Glu Phe Ile Lys
2645 2650 2655
Met Pro Ala Val Ile Leu Thr Gln Asp Pro Leu Thr Glu Pro Ile Ser
2660 2665 2670
His Leu Ala Ala Ala Ala Phe Thr Ser Ile Ser Gly Lys Pro Gln Ala
2675 2680 2685
Gln Lys Asn Asn Val Val Ser Asn Ser Lys Ala Arg Ile Asn Thr Ala
2690 2695 2700
Val His Asp Ala Val Glu Ser Val Tyr Ser Gly Glu Thr Tyr Val Ala
2705 2710 2715 2720
Pro Lys Pro Ile Val Ser Gly Lys Thr Val Val Glu Lys Pro Phe Ser
2725 2730 2735
Thr Thr Glu Thr Thr Met Tyr Ile Met Arg Gly Leu Pro Gly Ser Gly
2740 2745 2750
Lys Ser Phe Lys Val Ser Gln Leu Val Ala Lys Asp Pro Asn Leu Val
2755 2760 2765
Val Ala Ser Ala Asp His Phe Arg Tyr Ser Asn Asp Lys Thr Gly Lys
2770 2775 2780
Ala Val Tyr Thr Tyr Ile Pro Glu Ala Thr Ser Ser Val His Leu Gln
2785 2790 2795 2800
Cys Gln Asn Arg Ala Arg Lys Ala Leu Glu Asn Gly Gln Ser Val Cys
2805 2810 2815
Ile Asp Asn Thr Asn Leu Thr Leu Leu Glu Met Arg Pro Tyr Val Leu
2820 2825 2830
Leu Ala Arg Ser Phe Asn Tyr Asn Ile Glu Phe Ile His Ser Asp Ser
2835 2840 2845
Pro Trp Ala Leu Asn Leu Asp Leu Leu His Ala Lys Gly Val His Asn
2850 2855 2860
Val Pro Arg Ala Lys Leu Val Ile Met Tyr Asp Arg Phe Phe Asp Arg
2865 2870 2875 2880
Asp Asn Gln Ile Asp Ala Asp Ser Leu Ile Gln Tyr Val Ile Glu Ala
2885 2890 2895
Ile Asp Pro Lys Leu Val Ala Pro Ile Met Asn Arg Phe Pro Ala Asp
2900 2905 2910
Cys Asp Leu Ile Leu Gln Ser Ala Leu Thr Pro Asp Leu Glu Val Leu
2915 2920 2925
Lys Gln Asn Tyr Asp Arg Ala Asn Ala Thr Tyr Gln Asp Val Ser Leu
2930 2935 2940
Asp Asp Pro Pro Ala Leu Lys Ala Ala Arg Arg Ala Met Asn Ile Ala
2945 2950 2955 2960
Lys Ser Glu Tyr Glu Ala Gly Glu Ala Gly Gln Arg Arg Ile Glu Lys
2965 2970 2975
Phe Leu Glu Arg Gln Asp Val Ala Ala Leu Asn Gln Thr Leu Thr Thr
2980 2985 2990
Val Asn Gln Ser Lys Phe Ile Ala Ala Ile Arg Ser Ile Tyr Leu Ser
2995 3000 3005
Thr Ile Ser Asn Leu Arg Leu Lys Thr Arg His Met Gly Glu Gly Ser
3010 3015 3020
Tyr Ala Val Thr Ser Gly Thr Asn Thr Thr Asp Lys Val Leu Val Asn
3025 3030 3035 3040
Thr Pro Gln Arg Met Thr Arg Ile Glu Asp Gly Ile Tyr Lys Leu Val
3045 3050 3055
Ala Asn Gly Phe Glu Ile Thr Met Cys Asp Gly Ser Asn Leu Ala Gly
3060 3065 3070
Val Thr Phe Glu Gln Asp Ile Asn Pro Ser Met Tyr Pro Phe Val Phe
3075 3080 3085
Thr Leu Met Ser Asn Ile Ala Val Pro Val Leu Thr Arg Gln Ala Asn
3090 3095 3100
Val Gly Tyr Leu Asp Met Ser Asn Lys Phe Ile Cys Lys Asp Gly Thr
3105 3110 3115 3120
Val Gln Phe Gln Gly Val Ile Tyr Ala Tyr His Thr Pro Ser Asn Glu
3125 3130 3135
Ser Ala Asp Phe Lys Val Gly Asn Thr Ser Trp Thr Leu Gln Lys Asn
3140 3145 3150
Ile Asn Leu Thr Ala Phe Ile Pro Ala Ile His Lys Thr Ala Thr Phe
3155 3160 3165
Ala Ala Gln Ser Val Phe Leu Gly Gly Leu Pro Met Glu Glu His Gln
3170 3175 3180
Ala Phe Ser Asp Thr Pro Thr Ala Ser Asn Lys Phe Lys Val Phe Val
3185 3190 3195 3200
Ser Ser Thr Val Cys Ala Ser Thr Val Cys Lys Val Asn His Lys Thr
3205 3210 3215
Tyr Val Gln Ile Pro Asp Asp Ile Gln Asp Pro Phe Thr Tyr Met His
3220 3225 3230
His Ser Val Cys Ser His Asn Lys Phe Leu Ser Asn His Glu Thr Arg
3235 3240 3245
Cys Gln Ile Cys Pro Leu Asn Cys Xaa Ser Ala Asn Pro Cys Val Ser
3250 3255 3260
Thr Ala Cys Ala Leu Phe Asp Asn Gly Thr Leu Pro Arg Ser Thr His
3265 3270 3275 3280
Tyr Ile Asn Val Ser Thr Thr Ser Asn Val Gly Leu Phe Lys Ala Val
3285 3290 3295
Lys Lys Ser Thr Arg Gln Leu Asn Ile Asp Gly Phe Pro Tyr Met Leu
3300 3305 3310
Lys Gln Val Lys Asp Asp Ser Glu Leu Val Ser Ser Leu Lys Ile Gly
3315 3320 3325
Leu Pro Asn Ile Leu Pro His His Met Val Glu Thr Lys Ser Lys Thr
3330 3335 3340
Tyr Leu Leu Arg Gly Pro Thr Thr Ala Tyr Ser Leu Gly Asp Leu Cys
3345 3350 3355 3360
Tyr Ala Leu Phe Asn Gly Asp Phe Asp Tyr Ile Arg Glu Asn Ile Asn
3365 3370 3375
Ser Asp Phe Val Leu Asp Arg Glu Ala Gly Met Pro Asp Thr Glu Thr
3380 3385 3390
Arg Thr Trp Leu Phe Ser Ile Leu Asn Phe Ala Val Pro Arg Val Cys
3395 3400 3405
Ala Ile Ile Asp Gln Met Ile Ser Glu Asn Val Phe Tyr Lys Leu Thr
3410 3415 3420
Leu Asp Asn Leu Asp Leu Tyr Gly Ser Leu Tyr Asp Phe Asp Asp Tyr
3425 3430 3435 3440
Pro Thr Glu Gly Phe Asn Arg Pro Asp Asp Val Ile Arg Met Leu Lys
3445 3450 3455
Glu Ile Trp Ser Phe Cys Arg Arg Pro Leu Pro Ala Asp Leu Leu Lys
3460 3465 3470
Tyr His Glu Asp Ile Gly Ala Ala Ala Thr Gln Glu Ile Leu Leu His
3475 3480 3485
Ala Pro Phe Ile Asp Lys Val Cys Ala Leu Asn Asp Arg Leu Ala Val
3490 3495 3500
Val Asp Asn Arg Ala Ser Gln Tyr Phe Phe Cys Glu Glu Glu Gly Val
3505 3510 3515 3520
Phe Thr His Ile Tyr Asn Pro Val Tyr Gly Thr Leu Ala Phe Asp Asn
3525 3530 3535
Lys Leu Ile Gln Ser Lys Asp Pro Ser Cys Thr Leu Gln Arg Leu Ile
3540 3545 3550
Thr Ile Gln Gly Pro Leu Ser Thr Asn Ala Ser Pro Val Ile Ser Ile
3555 3560 3565
Ser Asp Ser Thr His Ile Ala Asn Asn Ile Asn Pro Ser Asn Gln Lys
3570 3575 3580
Thr Thr Pro Leu Tyr Tyr Asp Leu Glu Leu Ala Gln Glu Phe Ile Asp
3585 3590 3595 3600
Ala Gly Leu Asn Ile Asp Gly Val Ser Asn Tyr Phe Phe Tyr Gly Pro
3605 3610 3615
Ser Arg Ala Gly Val Val Ser Asp Phe Leu Leu Tyr Glu Phe Gln Gly
3620 3625 3630
Thr Gln Trp Phe Asp Asn Asn Met Leu Arg Ser Leu Tyr Ser Phe Ile
3635 3640 3645
Leu Lys Asn Ser Glu Cys Tyr Arg Thr Thr Asp Gln Leu Asp Phe Arg
3650 3655 3660
Gly Gly Lys Pro Arg Lys Ser Ser Met Gly His Gly Val Thr Gly Phe
3665 3670 3675 3680
Lys Gln Asp Val Val Tyr Ala Ala Leu Gly Pro Asp Met Ile Glu Thr
3685 3690 3695
Leu Tyr Glu Thr Ala Lys Gln Thr Pro Leu Pro Phe Cys Thr Lys Ile
3700 3705 3710
Thr Ala Lys Tyr Ala Leu Thr Ala Lys Pro Arg Ala Arg Thr Val Ala
3715 3720 3725
Ala Cys Ser Phe Val Ala Ser Thr Ile Phe Arg Tyr Ala His Lys Pro
3730 3735 3740
Leu Thr Asn Asn Met Val Ser Lys Ala Gln Gln Gly Leu Gly Tyr Cys
3745 3750 3755 3760
Leu Ile Gly Ile Ser Lys Phe His Gly Arg Phe Asn Lys Phe Val Lys
3765 3770 3775
Ser Arg Val Gly Thr Val Glu Asp Phe Asn Val Phe Gly Ser Asp Tyr
3780 3785 3790
Thr Lys Cys Asp Arg Thr Phe Pro Leu Ala Leu Arg Ala Leu Ser Ala
3795 3800 3805
Ala Leu Ile Phe Asp Leu Gly Gly His Asp Pro Asp Asn Cys Leu Phe
3810 3815 3820
Ile Asn Glu Leu Asn Ala Tyr Met Leu Asp Ile Val Ser Val Glu Asp
3825 3830 3835 3840
Ser Phe Ala Asn Lys Pro Gly Gly Thr Ser Ser Gly Asp Ala Thr Thr
3845 3850 3855
Ala Tyr Ser Asn Thr Leu Tyr Asn Phe Ala Val His Tyr Ile Ile Met
3860 3865 3870
Trp Lys Thr Phe Leu Thr Val Asn Asp Pro Ser Thr Lys Val Ile Arg
3875 3880 3885
Ser Ala Ala His His Ala Leu Thr Ser Gly Asp Phe Ser Met Tyr Asn
3890 3895 3900
Asp Met Ile Gln Asp Met Leu Asp Val Asp Tyr Thr Leu Asn Phe Leu
3905 3910 3915 3920
Ser Asp Asp Ser Tyr Ile Cys Ser Lys Pro Ser Ala Phe Pro Ile Phe
3925 3930 3935
Thr Leu Glu Asn Tyr Pro Ser Lys Leu Gln Ser Ile Leu His Thr Ala
3940 3945 3950
Val Asp Ser Lys Lys Ser Trp Glu Ala Lys Gly Glu Ile Lys Glu Phe
3955 3960 3965
Cys Ser Ser His Ile Val Asn Val Asp Gly Asp Tyr His Phe Lys Pro
3970 3975 3980
Glu Lys Asp Arg Ile Leu Ala Ser Leu Leu Ile Leu Ser Lys Ile Ala
3985 3990 3995 4000
Asp Met Asp Ile Phe Phe Met Arg Phe Val Ala Leu Leu Ala Glu Ser
4005 4010 4015
Ala Val Tyr Ile Arg Ile Asp Pro Thr Phe Trp Leu Ala Leu Phe Gly
4020 4025 4030
Val Phe Glu Asn Arg Val Thr Ala Phe Lys Ser Glu Thr Leu Leu Ser
4035 4040 4045
Pro Val Pro Glu Gln Leu Met Lys Val Ala Phe Tyr Glu Ser Leu Val
4050 4055 4060
Phe Ala Asp Val Asp Ala Thr Ala Leu Tyr Gly Phe Leu Asp Gly Phe
4065 4070 4075 4080
Lys Met Gln Ser Gln Thr Leu His Pro Asp Gly Val Glu Gly Phe Asp
4085 4090 4095
Lys Gln Ser Asp Arg Val Lys His Cys Phe Ala Cys Asp Asn Ile Ser
4100 4105 4110
Val Gly His Cys Ser Ile Cys Pro Val Pro Leu Pro Leu Cys Ser Phe
4115 4120 4125
Cys Phe Tyr Glu His Ala Leu Leu Asn Glu His Tyr Glu Ala Ser Gly
4130 4135 4140
Ile Ala Cys Glu Cys Gly Asp Ala Asp Ile Arg Gln Leu His Leu Lys
4145 4150 4155 4160
Ile Thr Asn Gln Pro Ser Ser His Asn Phe Ile Cys Ala Glu Cys Pro
4165 4170 4175
Thr Val Ala Met Lys Leu Pro Ile Phe Asn Ser Phe Gln Gly Lys Val
4180 4185 4190
Leu Leu Pro Met Phe Arg Met Asn Thr Pro Leu Pro Ser Ser Val Ser
4195 4200 4205
Val Ile Val Asp Val Arg Ser Asn Pro Lys Ala Pro Lys Met Leu Trp
4210 4215 4220
Asp Asp Val Gln Asn Phe Arg Glu Asn Cys Thr Arg Ile Ala Tyr Glu
4225 4230 4235 4240
Ser Val Ser Cys Ala Glu Leu Ala Arg Glu Val Val Tyr Tyr Pro Tyr
4245 4250 4255
Glu Val Ile Glu Ser Lys Ala Gly Gln Ala Arg Leu Arg Ile Gln Asn
4260 4265 4270
Phe Lys Cys Ser Pro Thr Thr Tyr Val Gln Phe Tyr Lys Val Arg Gln
4275 4280 4285
Asn Gly Lys Tyr Cys Leu Val Ala Lys Ala Thr Leu Thr Pro Ala Phe
4290 4295 4300
Glu Asn Gln Thr Asp Ile Phe Ser Val Phe Gln Pro Asn Asn Phe Ser
4305 4310 4315 4320
Pro Trp Asn Thr Ser Ser Val Phe Ala Val Glu Gln Tyr Ala Ala Ile
4325 4330 4335
Tyr Pro Pro Ile Pro Lys Glu Pro Val Asn Ala Thr Phe Val Leu Gly
4340 4345 4350
Pro Pro Gly Cys Gly Lys Thr Tyr Tyr Ile Ala Lys Thr Tyr Phe Ser
4355 4360 4365
Gln Ala Ser Glu Thr Cys Pro Val Val Tyr Cys Ala Pro Thr His Arg
4370 4375 4380
Leu Val Leu Asp Met Asp Ala Glu Tyr Ser Gly Val Val Ser Lys Ser
4385 4390 4395 4400
Leu Tyr Asn Asn Arg Val Tyr Lys Asn Pro Ala Tyr Lys Thr Gly Glu
4405 4410 4415
Pro Phe Lys Leu Cys Phe Thr Thr His Asn Thr Met Pro Val Gln Lys
4420 4425 4430
Lys Ala Ile Leu Ile Ile Asp Glu Val Ser Leu Ile Thr Pro His Ser
4435 4440 4445
Leu Phe Ser Ile Ile Gly Lys Gly Phe Tyr Glu Ile Val Leu Val Gly
4450 4455 4460
Asp Pro Phe Gln Leu Ser Ala Val Phe Pro Gly Phe Val Val Asn His
4465 4470 4475 4480
Thr Tyr Asp Gly Phe Tyr Ile Arg Arg Leu Val Asn Lys Val Lys His
4485 4490 4495
Leu Thr Val Cys Tyr Arg Cys Pro Gln Glu Ile Leu Asp Ile Phe Ser
4500 4505 4510
Lys Pro Tyr His Asp Val Gly Ile Asp Leu Thr Thr Gly Asn Thr Asn
4515 4520 4525
Pro Gly Lys Ala Ser Ile Tyr Thr Leu Asn Trp Leu Gln Ala Asp Val
4530 4535 4540
Gly Thr Lys Asn Pro Asp Lys Leu Arg Gln Leu Phe Ala Gln Tyr Pro
4545 4550 4555 4560
Gly Phe Lys Ile Ile Thr Asn Tyr Arg Cys Val Val Asp Ala Ala Lys
4565 4570 4575
Ser Tyr Gly Ile Asn Val Glu Thr Ile Asp Ser Ser Gln Gly Thr Thr
4580 4585 4590
Gly Asp Arg His Leu Val Val Ile Cys Gly Ser Thr Asn Phe Ser Lys
4595 4600 4605
Leu Leu Asn Arg Phe Ile Val Ala Ala Ser Arg Ser Thr Thr Glu Leu
4610 4615 4620
Val Ile Val Met Leu Pro Glu Leu Tyr Asn Tyr Leu Thr Glu Thr Phe
4625 4630 4635 4640
Asn Phe Lys Pro Leu Gln Leu Gln Asn Val His Val Pro Ile Ala Val
4645 4650 4655
Ser Ser Thr Ala Phe Cys Asp Ile Glu Phe Tyr His Phe Gln Lys Lys
4660 4665 4670
Phe Tyr Val Gly Glu Ile Ser Val Ser Thr Ser Thr Thr Met Thr Cys
4675 4680 4685
Gln Leu Gly Cys Tyr Ile Asn Gly Ser Tyr Met Leu Pro Pro Val Leu
4690 4695 4700
Glu Asn Ser Glu Asp Arg Leu Tyr Val Pro Ser Arg Trp Arg Arg Met
4705 4710 4715 4720
Ile Arg Lys Tyr Pro Thr Glu Ser Met His Ile Ser Leu Leu Asp Arg
4725 4730 4735
Leu Leu Arg His Ile Leu Leu Thr Thr Thr Gly Glu Ile His Phe Val
4740 4745 4750
Met Phe Ser Ala Asp Asn Asp Leu Ile Ala Leu Asp Pro Tyr Phe Ile
4755 4760 4765
Pro Pro Thr Leu Cys Glu Cys Gly Ser Ala Gly Leu Val Glu Val Asp
4770 4775 4780
Ile Thr Val Phe Cys Arg Asn Cys Leu Pro Lys Asp Gly Lys Ala Thr
4785 4790 4795 4800
Arg Leu Val Lys Pro Ser Thr Leu Asp Val Gln Thr Glu Lys Leu Arg
4805 4810 4815
Leu Ala Lys Val His Ala Lys Val Cys Lys Ile Lys His Gly Ser Ala
4820 4825 4830
His Asn Ala Asp Val Asp Ala Ile Met Thr Gln Cys Ile Tyr Ala Asn
4835 4840 4845
Ser Leu Thr Phe Thr Pro Thr Thr Gln Leu Val Val Asn Thr Asp Glu
4850 4855 4860
Phe Thr Phe Tyr Met Leu Pro Arg Pro Ser Asn Arg His Leu Arg Ile
4865 4870 4875 4880
Ile His Lys Asn Asp Lys Arg Phe Tyr Ala Ile Thr His Glu Glu Glu
4885 4890 4895
Asp Leu Phe Phe Thr Asn Ile Ser Ala Val Val Asp Pro Ile Pro Ala
4900 4905 4910
Lys Phe Asn Ile Ala His Ser Thr Ser Phe Leu Thr Ile Lys Ser Gly
4915 4920 4925
Cys Ala Gly Asn Lys Thr Cys Thr Arg Cys Tyr Tyr Leu His Leu Ala
4930 4935 4940
Tyr Thr Glu Phe Val Ser Gln His Lys Tyr Glu Pro Phe Thr Cys Val
4945 4950 4955 4960
Ser Phe Lys Ile Arg Phe Asp Phe Ser Gln Phe Thr Asp Ser Val Asp
4965 4970 4975
Thr Phe Leu Arg Gln Gly Leu Ile Thr Phe His Pro Glu Met Asn Ser
4980 4985 4990
Leu Gln Lys Ser Leu Leu Leu Ala Val Asp Lys Val Tyr Cys Asp Asn
4995 5000 5005
Phe Thr Ser Asn Gly Arg Arg Phe Arg Leu Tyr Asp Asn Asn Leu Val
5010 5015 5020
Lys Ser Ile Ile Lys Gly Ser Val Ala Gln Asn Ser Ile Ile Met Pro
5025 5030 5035 5040
Leu Asp Ser Val Leu His Gly Leu Asn Ile Asp Phe Thr Val Gly Cys
5045 5050 5055
Ala Val Asp Asn Phe Ser Cys Lys Glu Ala Ala Ser Val Arg Tyr Ser
5060 5065 5070
Glu Val Val Leu Ser Ile Thr Lys Leu Pro Pro Gly Thr Cys Gln Leu
5075 5080 5085
Tyr Tyr Val Ile Ser Tyr Gly Leu Asn Ser Pro Lys Thr Thr Tyr Ala
5090 5095 5100
Gly His Gln Leu Phe Asp Gly Phe Glu Thr Val Ile Val Val Asn Arg
5105 5110 5115 5120
Lys Asp Lys Pro Pro Tyr Val Leu Thr Gln Tyr Ile Asn Asp Val Val
5125 5130 5135
Val Ala Met Pro Glu Ser Leu Phe Ser Thr Gly Arg Phe Tyr Arg Glu
5140 5145 5150
Lys Pro Tyr Pro Val Leu Met Asn Glu Asp Leu Ser Gly Leu Asn His
5155 5160 5165
His Ile Phe Ser Gly Asp Tyr Thr Asp Glu Ser Leu Thr Leu Gly Gly
5170 5175 5180
Val His His Ile Val Thr Leu Asn Thr Tyr Asp His Lys Leu Asn Tyr
5185 5190 5195 5200
Ile Gln Thr Lys Ala Thr Cys Ala Ala Ser Val Ser Thr Gly Gly Arg
5205 5210 5215
Gly His Lys Ile Thr Thr Leu Phe Asp Val His Ala Asn Gln Leu Ala
5220 5225 5230
Asp Glu Ile Thr Arg Val Thr Ser Val Val Thr Thr Gln Ser Lys Val
5235 5240 5245
Ile Asn Leu Thr Ile Asp Tyr Gln Gln Val Pro Cys Met Tyr Trp Ser
5250 5255 5260
Ser Pro Thr Gly Ile Arg Thr Phe Tyr Pro Gln Ala Val Arg Leu Asp
5265 5270 5275 5280
Ala Lys Phe Ile Pro Tyr Tyr Val Glu Tyr Pro Asn Ile Leu Pro Ala
5285 5290 5295
Val Val Glu Asp Gln Val Tyr Asp Leu Ser Asn Tyr Asn Gln Pro Pro
5300 5305 5310
Leu Gly Gln Asn Cys Pro Val Asn Phe His Lys Tyr Val Gln Leu Thr
5315 5320 5325
His Phe Ile Leu Asp His Val Lys Ile Pro Glu Lys Gly Leu Ile Tyr
5330 5335 5340
His Ile Gly Ala Ala Gly Thr Lys Gln Cys Ser Pro Gly Asp Leu Ile
5345 5350 5355 5360
Leu Glu Gln Phe Phe Asn Lys Ser Ile Ile Tyr Ser Ser Asp Leu Leu
5365 5370 5375
Pro Tyr Gln Ser Pro Ala Val Gln Val Ala Leu Asp Val Arg Phe Ser
5380 5385 5390
Ala Ser Leu Ile Ile Ser Asp Cys Tyr Ser Lys Glu Pro Gln Pro Asp
5395 5400 5405
Leu Leu Ser Lys Leu Ile Asn Lys Leu Val Tyr Gly Gly Thr Leu Ile
5410 5415 5420
Phe Lys Thr Thr Glu Thr Phe Thr Cys Asp Pro Ala Phe Tyr Val Ala
5425 5430 5435 5440
His Phe Asn Cys Ile Lys Phe Phe Thr Ala Ala Val Asn His Ser Ser
5445 5450 5455
Ser Glu Val Tyr Ile Ala Phe Ile Gly Lys Leu Pro Lys Pro Asn Asn
5460 5465 5470
Asn Phe Leu Ala Ser Asp Tyr Phe Gln Arg Leu Thr Gln His Arg Asn
5475 5480 5485
Lys Val Val Lys Gln Pro Tyr Ala His Thr Trp Asp Thr Ser Phe Thr
5490 5495 5500
Tyr Pro Tyr Pro Ser Asn Val Leu Gln Val Ser Arg Lys Asn Leu Leu
5505 5510 5515 5520
Tyr Leu Phe Glu Thr Arg Gly Ala Ala Val Gly Thr Leu Ile Phe Glu
5525 5530 5535
Glu Pro Ser Lys Pro Ala Val Lys Ile Pro Thr Lys Cys Gln Thr Thr
5540 5545 5550
Gln Pro Ser Cys Val Ile Glu Val Gly Asn Gln Tyr Asp Cys Cys Ile
5555 5560 5565
Gln Asp Ile Ile Thr Leu Leu Asn Gly Lys Ser Phe Thr Val Lys Val
5570 5575 5580
Pro Asn Ser Glu Ser Leu Leu Arg Asp Ile Cys Thr Leu Ala Leu Ser
5585 5590 5595 5600
Gln Ser Tyr Ser Ile Asn Ile Arg Gly Lys Thr Leu Tyr Thr Leu Ser
5605 5610 5615
Ser Leu Leu Arg Ile Arg Gln Gln Ser Leu Leu Phe Tyr Gly Glu Lys
5620 5625 5630
Val Lys Asn Pro Arg Pro Arg Asn Val Leu Asn Lys Tyr Thr Asn Tyr
5635 5640 5645
Leu Lys Ala Lys Val Ile Arg His Tyr Thr Lys Pro Gln Ser Thr Val
5650 5655 5660
Leu Asp Ile Gly Thr Gly Lys Gly Gln Asp Leu Arg Lys Tyr Ser Leu
5665 5670 5675 5680
Ala Gly Val Lys Ser Leu Thr Cys Val Glu Pro Ser Pro Glu Ser Val
5685 5690 5695
Thr Glu Leu Ser Ile Ile Ala Ser Pro Leu Asp Met Glu Thr His Thr
5700 5705 5710
Val Met Ser Ser Ala Gln Lys Phe Glu Thr Ser Leu Thr Phe Asp Leu
5715 5720 5725
Ala Phe Ser Phe Phe Ala Leu His Tyr Ala Leu Asp Asp Val Cys Met
5730 5735 5740
Ser Glu Thr Leu Asn Asn Val Phe Cys Lys Leu Asn Ser Asn Ser Gln
5745 5750 5755 5760
Leu Ile Leu Val Val Pro Asn Ala Gly Arg Met Gln Ser Ile Pro Ser
5765 5770 5775
Leu Gly Leu Thr Val Thr His Leu Asp Asp Asp Lys Val Trp Phe Lys
5780 5785 5790
Tyr Ser Asp Tyr Ile Asp Cys Glu Glu Pro Leu Val Asp Lys Glu Lys
5795 5800 5805
Leu Leu Thr Cys Leu Ala Thr Tyr Gly Thr Ile Val Thr Asp Ser Pro
5810 5815 5820
Phe Tyr Asp Gly Ala Asn Lys Ile Leu Asp Gln Lys Cys Ser Ser Met
5825 5830 5835 5840
Tyr Arg Ala Ser Thr Ala His Leu Asn Pro Asp Glu Ile Gln Tyr Ile
5845 5850 5855
Asn Met Tyr Asp Leu Ile Val Val Ile Lys Asn
5860 5865
<210> 8
<211> 1391
<212> БЕЛОК
<213> Вирусы
<220>
<223> аминокислотная последовательность ORF-2
<400> 8
Met Phe Ala Leu Val Leu Thr Leu Thr Ile Ala Ser Ala Ile Ala Gln
1 5 10 15
Asp Phe Pro Ala Tyr Asp Pro Cys Pro Thr Cys Ser Thr Pro Gly Asn
20 25 30
Lys Ile Pro Ala Pro Ser Thr Val Ala Gln Tyr Ser Thr Asn Tyr Gly
35 40 45
Ala Asn Phe Phe Thr Val Val Phe Asp Gly Ile Ile Phe Asn Gln Phe
50 55 60
Arg Glu Ser Tyr Tyr His Gln Cys Arg Pro Thr Pro Glu Tyr Cys Pro
65 70 75 80
Asp Ala Ile Asn Cys Ala Leu Asn Arg Thr Gly Ala Ser Cys Lys Pro
85 90 95
Phe Ala Thr Gly Pro Asn Ser Gln Cys Gln Asn Ser Phe Glu Gly Asn
100 105 110
Ile Asp Ile Cys Ala Thr Cys Ser Pro Leu Lys Gln Glu Thr Pro Phe
115 120 125
Ile Cys Tyr Asn Arg Tyr Gly Ile Ile Ile Tyr Pro Thr Ala Asp Ile
130 135 140
Val Leu Ser Ala Arg Phe Lys Ile Gly Ser Phe Ser Pro Lys Ala Cys
145 150 155 160
Asp Asn Tyr Leu Asn Asp Leu Asn Cys Asp Ser Lys Thr Ala Arg Ser
165 170 175
Tyr Val Ile Ser Arg Pro Gln Ser Phe Ser Leu Gln Tyr Pro Asn Ser
180 185 190
Leu Gly Pro Tyr Gln Leu Lys Arg Phe Ser Leu Ala Lys Glu Ile Val
195 200 205
Asp Leu Arg Ala Gly Val Leu Thr Ser Leu Pro Asn Arg Gly Tyr Lys
210 215 220
Gly Arg Thr Thr Tyr Ser Tyr Pro Val Thr Ala Leu Ser Leu Leu Ala
225 230 235 240
Arg Ser Lys Val Ala Glu Ala Asp Lys Phe Phe Tyr Ile Glu Ala Lys
245 250 255
Ile Leu Leu Tyr Ala Trp Ser Gln Lys Pro Gln Ile Arg Phe Leu Gly
260 265 270
Ala Tyr Cys Pro Thr Asp Val Ser Cys Pro Asp Ser Thr Ala Leu Gly
275 280 285
Cys Cys Phe Ser Gly Ser Gly Ser Glu Phe Tyr Tyr Ala Phe Arg Gln
290 295 300
Trp Tyr Tyr Ala Ser Leu Gly Met Glu Asp Leu Val Asp Phe Asp Asn
305 310 315 320
Ser Thr Val Leu Ser Leu Ser Pro Asp Thr Pro Gln Ile Thr Pro Val
325 330 335
Val Ser Tyr Phe Leu Glu Lys Val Leu Pro Leu Phe Lys Ser His Val
340 345 350
Pro Gly Arg Val Phe Tyr Cys His Ser Leu Met Ser Asn Gly Val Cys
355 360 365
Thr Phe Asp His Val Val Val Asn Ile Asn Ala Glu Ala Val Phe Phe
370 375 380
Asp Leu Glu Val Asp Ile Gly Ser Ile Ile Ala Asp Ala Tyr Arg Val
385 390 395 400
Glu Arg Pro Asn Thr Leu Cys Tyr Asp Thr Asn Cys Thr Leu Ala Thr
405 410 415
Ser Arg Thr Thr Glu Tyr Asn Tyr Ala Ala Tyr Val Val Tyr Ile Leu
420 425 430
Phe Asn Leu Tyr Ser Ser Asn Arg Ile Ala Ile Asp Phe Asn Thr His
435 440 445
Ser Ile Leu Gln Gly Leu Leu Gln His Asn Ser Asn Tyr Gln Thr Ala
450 455 460
Asn Leu Asp Tyr Leu Phe Val Gly Ala Leu Phe Thr Gly Thr Phe Lys
465 470 475 480
His Ile Thr Ser Asn Gln Ala Tyr Pro Val Pro Leu Thr Tyr Pro Ile
485 490 495
Val Lys Thr Tyr Val Gly Pro Ser Asn Gln Tyr Ser Met Ser Asn Lys
500 505 510
Leu Phe Ser Tyr Thr His Asn Leu Thr Ala Gln Ala His Ser Gly Ile
515 520 525
Cys Asn Ser Phe Tyr Cys Tyr Lys Pro Arg Phe Val Pro Ile Asp Val
530 535 540
Phe Ile His Ser Ala Leu Thr Pro Asp Ser Leu Met Glu Thr Glu Ser
545 550 555 560
Phe Val Cys Val Ser Leu Arg Ser Pro Ser Ala Gly Ser Thr Ser Ala
565 570 575
Gly Ser Phe Tyr Leu Gln Cys Leu Asn Ser Ser Ile Asp Leu His Pro
580 585 590
Gly Ser Phe Val Pro Val Ser Ser Ser Pro Glu Ser Ser Ser Arg Val
595 600 605
Thr Ala Glu Leu Ala Phe Asn Thr Arg Asn Gly Ile Phe Ser Pro Cys
610 615 620
Leu Asn Gly Thr Cys Val Leu Ala Pro Thr Asp Pro Ile Val Phe Met
625 630 635 640
Arg Gln Gly Ala Trp Phe Thr Lys Ser Leu His Phe Asp Val Ser Pro
645 650 655
Cys Lys Pro Met His Phe Pro Asp Ile Asp Ile Gln Pro Pro Thr Tyr
660 665 670
Asn Val Ser Ser Ile Lys Met Asp Asp Asn Ala Val Leu Val Gln Asp
675 680 685
Leu Thr Ser Gly Leu Val Ile Asp His Asn Leu Gly Ser Ile Leu Arg
690 695 700
Pro Lys Gly Arg Ala Leu Glu Val Ser Tyr Tyr Ala His Ser Ile Leu
705 710 715 720
Arg Tyr Leu Glu Pro Asp Ser Cys Leu Pro Asp Asn Phe Leu Asn Phe
725 730 735
Val Thr Cys Leu Asp Tyr Ile Cys Ser Asp Ser Ser Pro Cys Arg Ala
740 745 750
Ala Ala Ser Gln Tyr Cys Gln Ala Gly Ile Tyr Phe Glu Ser Ala Phe
755 760 765
Asn Lys Ser Arg Tyr Ser Leu Leu Asn Ala Tyr Thr Leu Phe Asn Thr
770 775 780
Ser Leu Gln Thr Leu Leu Pro Glu Thr Phe Leu Glu Ile Glu Asp Asp
785 790 795 800
Glu Pro His Ser Arg Ser Lys Arg Ser Ile Asp Thr Thr Ser Asn Ile
805 810 815
Arg Pro Ser Gln Leu Leu Val Asn Gly Arg Ile Pro Ser Thr Ser Ser
820 825 830
Ala Phe Ala Val Asn Val Ala Arg Gly Arg Gly Thr Ile Met Pro Arg
835 840 845
Pro Gly Thr Gly Gly Met Gly Ser Ser Phe Ser Ala Val Ser Arg Ser
850 855 860
Gly Ser Ile Ser Ser Leu Ser Ser Val Gly Ser Ser Thr Pro Leu Ile
865 870 875 880
Ser Asn Trp Arg Thr Ser Ser Ser Gln Leu Lys Thr Leu Asn Leu Asn
885 890 895
Ile Asn Thr Lys Ile Pro Lys Ile Ser Thr Lys Ser Gly Phe Ala Ser
900 905 910
Ile Thr Ser Leu Phe Ala Ser Gly Leu Gly Val Val Asp Leu Gly Leu
915 920 925
Ser Ile Phe Asn Met Ile Glu Gln Arg Arg Val Ala Glu Ile Thr Gln
930 935 940
Met Gln Ile Ser Gln Leu Ala Asp Ser Ile Val Tyr Leu Ala Asp Val
945 950 955 960
Thr Phe Glu Ala Ile Lys Asn Leu Glu Leu Ser Val Asn Ser Leu Gly
965 970 975
Thr Phe Leu Ser Glu Phe Ser Thr Gln Met Ser Ile Thr Ile Ser Gln
980 985 990
Ile Gln Ser Ser Phe Glu Glu Gln Gln Asp Ala Thr Asn Asp Ala Leu
995 1000 1005
Tyr Tyr Thr Asn Ala Ala Ala Ser Tyr Gln Ala Ser Met Ala Tyr Val
1010 1015 1020
Ile Ser Glu Leu Asn Ala Ile Ser Leu Ser Val Thr Arg Ser Tyr Asp
1025 1030 1035 1040
Ser Tyr Thr Ser Cys Ile Thr Ser Gly Ile Asn Gly Leu Ile Thr Pro
1045 1050 1055
Ser Cys Leu Pro Ala His Gln Leu Leu Gln Leu Leu Asp Thr Val Ile
1060 1065 1070
Asn Ser Thr Ala Gly Thr Gly Cys Arg Pro Ile Tyr Gly Arg Glu Glu
1075 1080 1085
Val Val Lys Tyr Tyr Thr Leu Pro Leu Ile Asn Gln Gly Tyr Ser Phe
1090 1095 1100
Asn Gly Ser Ile Phe Phe Val Phe Asn Ile Pro Ile Thr Cys Gln Gly
1105 1110 1115 1120
Ile Ala Gly Asp Val Tyr Glu Val Glu Pro Pro Ile Leu Val Asp Val
1125 1130 1135
Pro Ser Lys Thr Ala Leu Arg Met Ile Thr Pro Ser Asn Val Val Ala
1140 1145 1150
Thr Gln Ala Gly Leu Ala Glu Leu Asp Leu Arg His Cys Glu Arg Tyr
1155 1160 1165
His Asn Glu Phe Leu Cys Asp Ser Ser Ala Phe Leu Ser Thr Pro Ser
1170 1175 1180
Lys Tyr Ile Asp Cys Leu Thr Asn Ala Thr Asp Cys Ser Leu Gln Phe
1185 1190 1195 1200
Ile Thr Gln His Val Pro Asp Pro Cys Val Tyr Thr Ser Pro Ala Ser
1205 1210 1215
Leu Tyr Cys Tyr Tyr Ser Pro Ile Cys Asp Gln Cys His Ile Val Ala
1220 1225 1230
Gly Cys Asn Glu Ser Gln Gln Tyr Asn Phe Thr Ser Ala Asp Gly Gly
1235 1240 1245
Val Val Phe Tyr Ser Ile Gln Asp Arg Asp Cys Gly His Phe Pro His
1250 1255 1260
Ile Thr Val Thr Thr Pro Ala Ala Ile Gln Glu Asp Phe Thr Val Gly
1265 1270 1275 1280
Pro Tyr Leu Pro Ser Leu Pro Ile His Thr Ala Tyr Val Asn Val Thr
1285 1290 1295
Trp Asn Val Thr Leu Pro Gly Asn Trp Thr Trp Glu Asn Ile Thr Leu
1300 1305 1310
Thr Ala Asn Trp Thr Gln His Phe Ile Glu Met Lys Lys Asn Ile Thr
1315 1320 1325
Met Met Ala Glu Glu Ile Asp Asn Leu Thr Asn Phe Gly Lys Val Leu
1330 1335 1340
Val Gly Gln Leu Asn Ser Phe Leu Ser Ser Leu Phe Asn Ile Pro Leu
1345 1350 1355 1360
Gly Leu Met Thr Phe Cys Phe Ser Val Ala Ala Leu Gly Leu Ser Ile
1365 1370 1375
Ile Ala Leu Leu Val Leu Cys Phe Pro Gln Lys Pro His Lys Leu
1380 1385 1390
<210> 9
<211> 260
<212> БЕЛОК
<213> Вирусы
<220>
<223> аминокислотная последовательность ORF-3
<400> 9
Met Met Phe Thr Leu Val Val Leu Phe Thr Leu Leu Gly Leu Ser Met
1 5 10 15
Ala Ser Thr Glu Leu Asn Phe Asp Pro Thr Leu Pro Leu Pro Ser Pro
20 25 30
Ile Asn Ala Leu Val Asp Ile Phe Gly Asn Asn Ser Leu Phe Leu Lys
35 40 45
Glu Ser Leu Leu Gly Lys Ser Thr Gly Ala Val Tyr Ala Tyr Leu Tyr
50 55 60
Ser Ser Ala Ile Ser Leu Leu Leu Leu Leu Trp Val Thr Val Trp Ser
65 70 75 80
Ile Ala Thr Ser His Phe Asn Val Thr Arg Ile Pro Thr Ile Ala Val
85 90 95
Leu Thr Asn Ala Ser Met Phe Leu Leu Leu Ala Ser Ala Thr Val Thr
100 105 110
Thr Trp Phe Leu Pro Thr Val Thr Asn Val Phe Phe Tyr Thr Leu Thr
115 120 125
Ala Leu Phe Thr Phe Phe Ser Phe Val Phe Leu Leu Trp Leu Val Tyr
130 135 140
Tyr Met Phe Thr Thr Ile Arg Ala Tyr Arg Arg Val Gly Ser Trp Arg
145 150 155 160
Val Val Phe Asn Gly Lys Tyr Ser Leu Leu Ala Gly Thr Gln Ala Val
165 170 175
Cys Leu Cys Arg Pro Ala Ile His Leu Val Leu Thr Lys Thr Asn Thr
180 185 190
Asp Thr Tyr Trp Cys Leu Asp Gly Thr Pro Ile Tyr Asn Val Asp Leu
195 200 205
Leu Gln Leu Val Gly Pro Lys Gly Leu Tyr Pro Tyr Lys Arg Met Thr
210 215 220
Thr Ile Thr Ala Pro Lys Gly Thr Lys Thr Ser Ala Ala Val Tyr Thr
225 230 235 240
Leu Gln Lys Glu Glu Val Cys Ala Leu Ser Glu Ile Thr Val His Asn
245 250 255
Asp Thr Asp Phe
260
<210> 10
<211> 201
<212> БЕЛОК
<213> Вирусы
<220>
<223> аминокислотная последовательность ORF-4
<400> 10
Met Ser Tyr Pro Val Tyr Tyr Glu Gln Arg Arg Tyr Ser Pro Arg Gln
1 5 10 15
Phe Asn Asn Gly Gly Gly Tyr Asn Pro Thr Pro Gln Pro Arg Val Val
20 25 30
Arg Thr Asn Pro Gly Asn Gln Ala Tyr Asn Pro Arg Arg Asn Arg Asn
35 40 45
Ala Thr Pro Asn Gln Gln Gln Met Val Pro Tyr Gln Pro Gln Tyr Gln
50 55 60
Ala Pro Pro Gln Pro Arg Val Val Tyr Val Asp Arg Pro Gln Glu Pro
65 70 75 80
Val Val Ile Tyr Arg Ala Pro Pro Gln Gly Lys Lys Gln Ser Gly Lys
85 90 95
Arg His Thr Ala Glu Glu Arg Trp Tyr Gln Gly Glu Lys Pro Val Gln
100 105 110
Lys Lys Gln Ala Pro Lys Gly Lys Ser Lys Lys Ala Ala Thr Pro Ala
115 120 125
Asn Pro Lys Lys Gln Pro Thr Gln Ser Asp Lys Val Pro Ile Ala Tyr
130 135 140
Pro Asp Asn His Pro Phe His Asp Leu Ala Pro Ala Asp Ile Arg Ala
145 150 155 160
Phe Lys Lys Gln Leu Ile Gln Asn Leu Asp Leu Gly His Gly Glu Met
165 170 175
Asn Gln Leu Arg Leu Ser Ile Asp Leu Leu Pro Ile Lys Lys Pro Ala
180 185 190
Pro Thr Pro Ala Val Pro Ala Pro Leu
195 200
<210> 11
<211> 113
<212> БЕЛОК
<213> Вирусы
<220>
<223> аминокислотная последовательность ORF-5
<400> 11
Met Phe Thr Leu Val Leu Ile Ile Leu Leu Ser Phe Ser Met Ala Phe
1 5 10 15
Asn Ala Phe Thr Phe Leu Leu Leu Leu Phe Phe Thr Phe Lys Cys Ile
20 25 30
Ile Thr Arg Thr Leu Val Val Val Pro Ile Asp Tyr Pro Glu Asn His
35 40 45
Pro Phe Asn Gly Leu Ser Pro Glu Glu Ile Ile Ser Tyr Lys Ser Gln
50 55 60
Leu Ile Gln Asn Leu Asp Leu Gly His Gly Glu Val Ile Lys His Arg
65 70 75 80
Phe Ser Ile Asp Leu Leu Pro Leu Lys Thr Thr Ser Thr Pro Thr Thr
85 90 95
Ser Ala Ile Leu Trp Lys Arg Phe Lys Thr Ser His Lys Glu Asn Asn
100 105 110
His
<210> 12
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер TCL-ORF-1
<400> 12
acactatacc agcccatg 18
<210> 13
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Taqman зонд TCL-ORF-1
<400> 13
caagcaacag tttaagtgag cc 22
<210> 14
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер TCL-ORF-1
<400> 14
tgtgaggtag caaagacg 18
<210> 15
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер TCL-M
<400> 15
gtgttcttac tgtggttgg 19
<210> 16
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Taqman зонд TCL-M
<400> 16
cattagggca tatcgaaggg tc 22
<210> 17
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер TCL-M
<400> 17
tgagttccag caagtagag 19
<210> 18
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер TCL-S
<400> 18
tcaaaactct caacctcaac 20
<210> 19
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Taqman зонд TCL-S
<400> 19
aagatgtaat actggcaaaa cctg 24
<210> 20
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер TCL-S
<400> 20
acgactccta aacctgaag 19
<210> 21
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> SEQ ID NO:21
<400> 21
taatttgact gactatag 18
<210> 22
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> SEQ ID NO:22
<400> 22
taagaaacta taccagtcca tgtcg 25
<210> 23
<211> 12
<212> ДНК
<213> Искусственная последовательность
<220>
<223> SEQ ID NO:23
<400> 23
agtttaagtg ag 12
<210> 24
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F1
<400> 24
cagtccacca acacaacgtg 20
<210> 25
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R1
<400> 25
cccaagtgtc gctttgcatc 20
<210> 26
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F2
<400> 26
ccaacaaagg agccgcaatc 20
<210> 27
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R2
<400> 27
gcacatgttt ggtgggtgtc 20
<210> 28
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F3
<400> 28
gacacccacc aaacatgtgc 20
<210> 29
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R3
<400> 29
gccaaacgga ggtctggatt 20
<210> 30
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F4
<400> 30
aatccagacc tccgtttggc 20
<210> 31
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R4
<400> 31
tcaatacctc gagcgcagac 20
<210> 32
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F5
<400> 32
tgtctgcgct cgaggtattg 20
<210> 33
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R5
<400> 33
tttgttcagg ggtggtgtcc 20
<210> 34
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F6
<400> 34
tctgacgaca aaccggacac 20
<210> 35
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R6
<400> 35
agccgctggg attacttcac 20
<210> 36
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F7
<400> 36
ttcctagcaa cccgctttca 20
<210> 37
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R7
<400> 37
agcggttttc ttttccgtcg 20
<210> 38
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F8
<400> 38
cgacggaaaa gaaaaccgct 20
<210> 39
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R8
<400> 39
actacggcta ctggggttc 19
<210> 40
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F9
<400> 40
ccccagtagc cgtagttgac 20
<210> 41
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R9
<400> 41
tcatgccgga gattttgcct 20
<210> 42
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F10
<400> 42
aggcaaaatc tccggcatga 20
<210> 43
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R10
<400> 43
agatgagccg tcagcaaaca 20
<210> 44
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F11
<400> 44
tgtttgctga cggctcatct 20
<210> 45
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R11
<400> 45
tgcgtttgct ctgtcgtagt 20
<210> 46
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F12
<400> 46
actacgacag agcaaacgca 20
<210> 47
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R12
<400> 47
atagagcgca agccgtagac 20
<210> 48
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F13
<400> 48
gtctacggct tgcgctctat 20
<210> 49
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R13
<400> 49
gccagtaaca ccatgtccca 20
<210> 50
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F14
<400> 50
tgggacatgg tgttactggc 20
<210> 51
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R14
<400> 51
aacgctgtta cgcggttttc 20
<210> 52
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F15
<400> 52
gaaaaccgcg taacagcgtt 20
<210> 53
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R15
<400> 53
ctggcatcgt gttgtgtgtg 20
<210> 54
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F16
<400> 54
cacacacaac acgatgccag 20
<210> 55
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R16
<400> 55
tagacccgca ctaccacact 20
<210> 56
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F17
<400> 56
agtgtggtag tgcgggtcta 20
<210> 57
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R17
<400> 57
ttttatgacc acgtccgcca 20
<210> 58
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F18
<400> 58
tggcggacgt ggtcataaaa 20
<210> 59
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R18
<400> 59
ctgcagctcc tctggtttca 20
<210> 60
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F19
<400> 60
tgaaaccaga ggagctgcag 20
<210> 61
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R19
<400> 61
gccgaagcta ttgtgagggt 20
<210> 62
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F20
<400> 62
accctcacaa tagcttcggc 20
<210> 63
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R20
<400> 63
cggaaaaaca acagccgagg 20
<210> 64
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F21
<400> 64
cctcggctgt tgtttttccg 20
<210> 65
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R21
<400> 65
tgggtcagta ggtgcgagta 20
<210> 66
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F22
<400> 66
tactcgcacc tactgaccca 20
<210> 67
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R22
<400> 67
ggagccaacc gaggataagg 20
<210> 68
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F23
<400> 68
tggcatgggt tcgtcctttt 20
<210> 69
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R23
<400> 69
ggtacctttc gcaatgacgc 20
<210> 70
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F24
<400> 70
gcgtcattgc gaaaggtacc 20
<210> 71
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R24
<400> 71
accctccgcc attgttgaat 20
<210> 72
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F25
<400> 72
attccaacca tcgcggttct 20
<210> 73
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R25
<400> 73
ttcgccttga taccagcgtt 20
<210> 74
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F26
<400> 74
acgctggtat caaggcgaaa 20
<210> 75
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R26
<400> 75
atccaggagt taacgtcggc 20
<210> 76
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_F27
<400> 76
gccgacgtta actcctggat 20
<210> 77
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> TCL_R27
<400> 77
aggtcagaac aagggaggct a 21
<210> 78
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Анализ 1 мембраны праймер 1
<400> 78
catctacctc tcccatactc 20
<210> 79
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Анализ 1 мембраны праймер 2
<400> 79
actgcttcca aaactgatta cct 23
<210> 80
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Анализ 1 мембраны праймер 3
<400> 80
gggcgtaaag agaatgtaag 20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МУТАНТНАЯ П-ГИДРОКСИФЕНИЛПИРУВАТДИОКСИГЕНАЗА, НУКЛЕИНОВАЯ КИСЛОТА, КОДИРУЮЩАЯ ЕЕ, И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2781830C2 |
| ГОМОЛОГ ГЛИЦЕРОЛ-3-ФОСФАТ-АЦИЛТРАНСФЕРАЗЫ И ЕГО ИСПОЛЬЗОВАНИЕ | 2011 |
|
RU2545375C2 |
| КОМПОЗИЦИИ И СПОСОБЫ РЕДАКТИРОВАНИЯ ГЕНОВ | 2019 |
|
RU2804665C2 |
| Т-КЛЕТКИ С ДВУМЯ ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ CAR | 2021 |
|
RU2839662C1 |
| НЕВИРУСНЫЙ ИММУНОТАРГЕТИНГ | 2020 |
|
RU2833379C2 |
| СПОСОБ ПРОДУЦИРОВАНИЯ АРЕНАВИРУСА, А ТАКЖЕ МУТАНТОВ АРЕНАВИРУСА С ПРОТИВООПУХОЛЕВЫМИ СВОЙСТВАМИ | 2019 |
|
RU2812046C2 |
| ПРОТИВОРАКОВЫЕ ВАКЦИНЫ | 2017 |
|
RU2718663C2 |
| Выделенная нуклеиновая кислота, которая кодирует слитый белок на основе FVIII-BDD и гетерологичного сигнального пептида, и ее применение | 2022 |
|
RU2818229C2 |
| ДИСПЛЕЙ ИНТЕГРАЛЬНОГО МЕМБРАННОГО БЕЛКА НА ВНЕКЛЕТОЧНЫХ ОБОЛОЧЕЧНЫХ ВИРИОНАХ ПОКСВИРУСА | 2017 |
|
RU2759846C2 |
| АBC-ТРАНСПОРТЕРЫ ДЛЯ ВЫСОКОЭФФЕКТИВНОГО ПРОИЗВОДСТВА РЕБАУДИОЗИДОВ | 2020 |
|
RU2795855C2 |
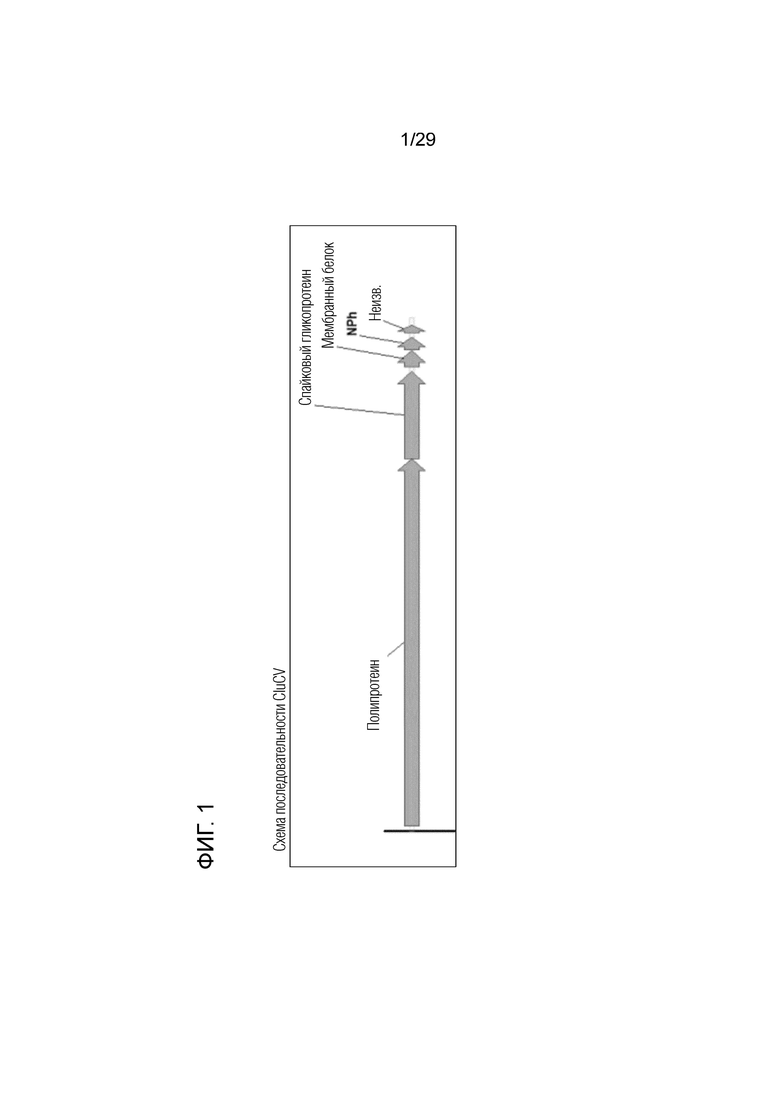
























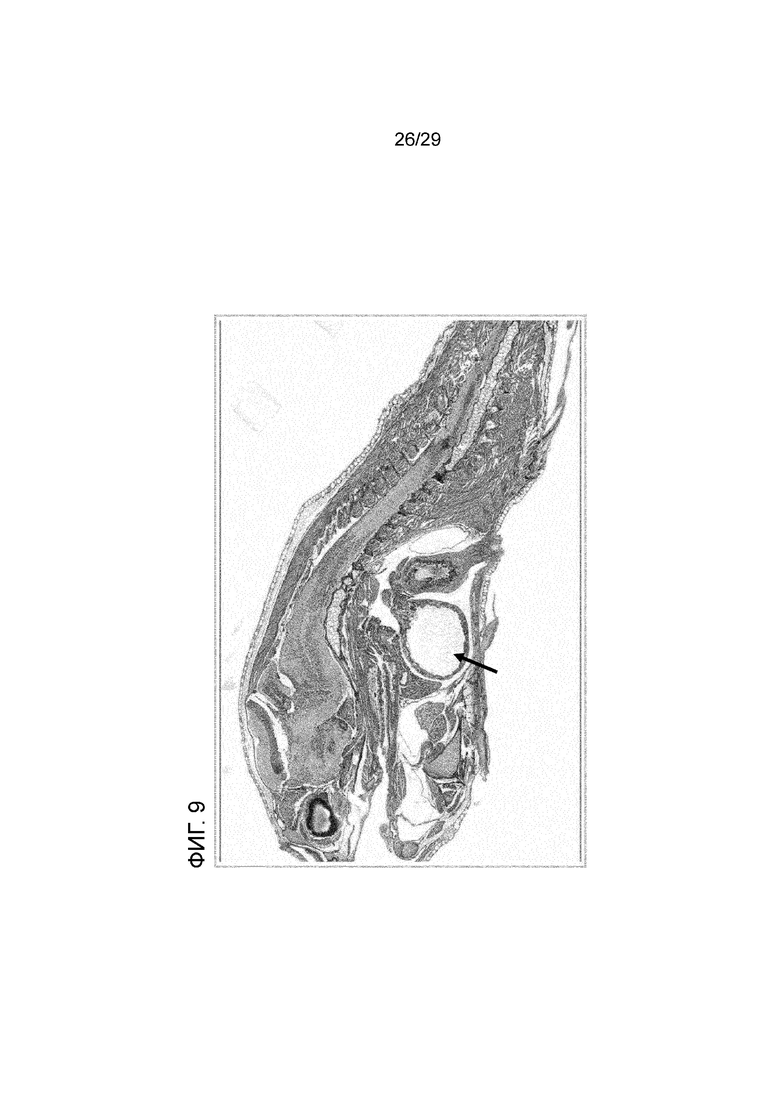
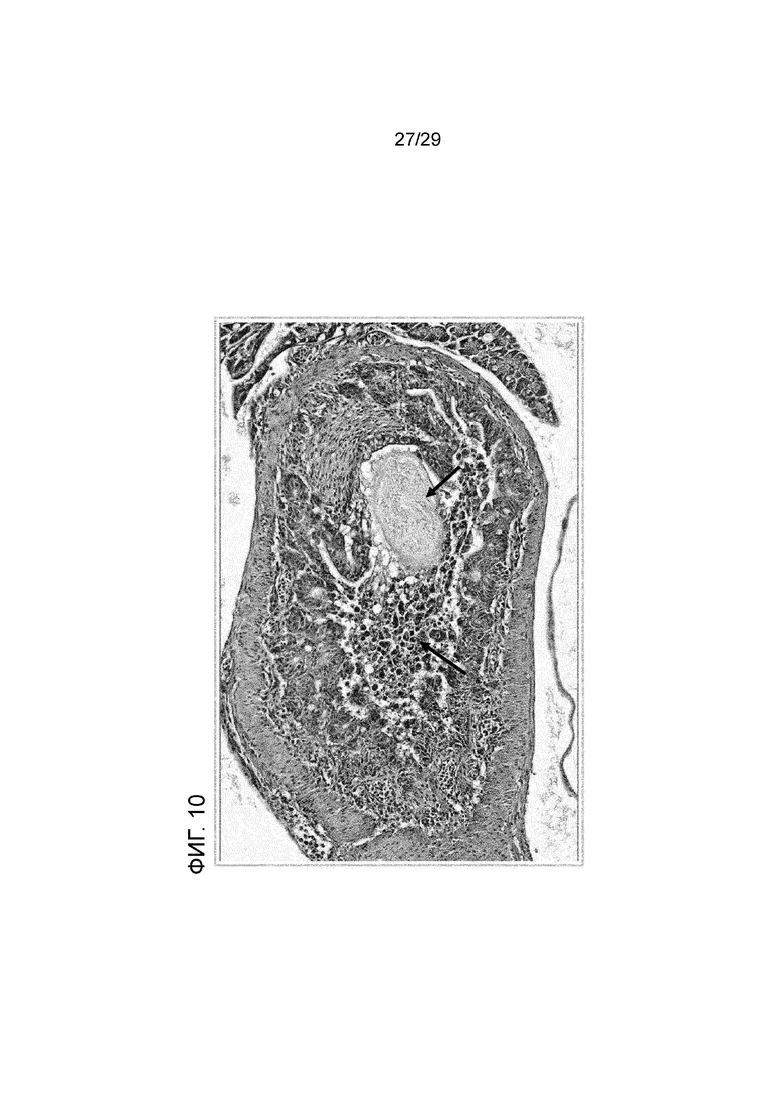
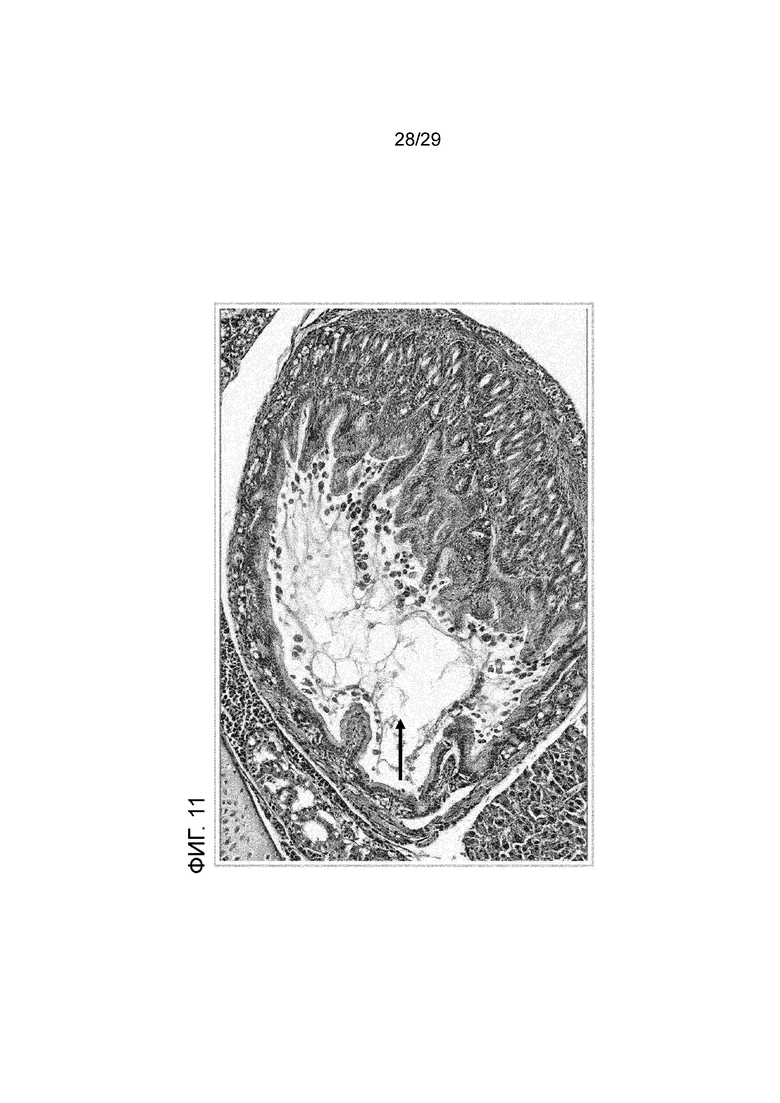
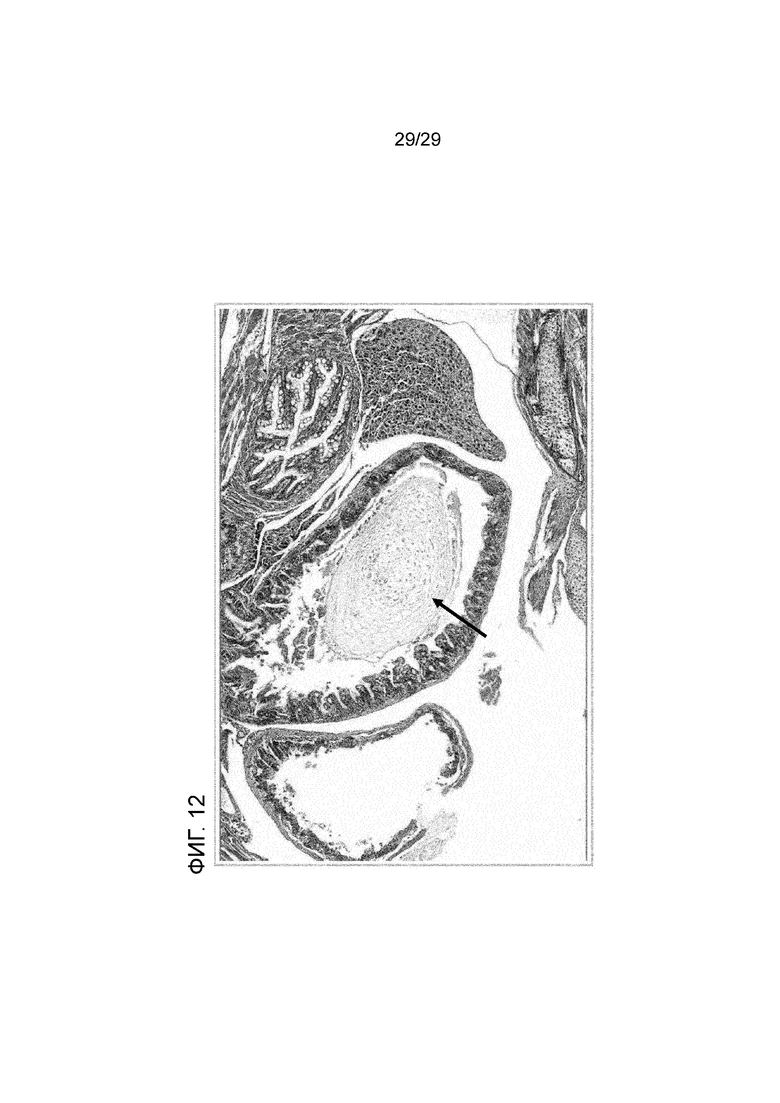
Изобретение относится к биотехнологии. Описан олигонуклеотидный праймер, который: (a) включает по меньшей мере 18 последовательных нуклеотидов последовательности, которая представляет собой часть или комплементарна части, референсной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 или SEQ ID NO: 6, или (b) включает по меньшей мере 18 последовательных нуклеотидов последовательности, которая по меньшей мере на 80% идентична последовательности, которая представляет собой или комплементарна последовательности, выбранной из группы, состоящей из SEQ ID NO: 12 - SEQ ID NO: 80; при условии, что указанный олигонуклеотидный праймер не включает последовательность, выбранную из группы, состоящей из SEQ ID NO: 21 - SEQ ID NO: 23. Раскрыто применение по меньшей мере одного указанного олигонуклеотидного праймера в способе обнаружения вируса, который инфицирует и способен вызывать гибель пинагора (Cyclopterus lumpus), где вирусный геном включает последовательность нуклеиновой кислоты, которая по меньшей мере на 90% идентична любой из SEQ ID NO: 1, 2, 3, 4, 5, 6, и где указанная последовательность нуклеиновой кислоты содержит основание урацил (U) вместо основания тимин (T). Представляен способ обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии: (a) контакт нуклеиновой кислоты, выделенной из биологического образца рыбы, по меньшей мере с одним олигонуклеотидным праймером по п.1 с получением смеси, (b) определение, присутствует ли после амплификации смеси a) продукт амплификации, где присутствие продукта амплификации указывает на присутствие РНК, ассоциированной с вирусом, и, следовательно, на присутствие вируса в биологическом образце. Раскрыт набор для обнаружения вируса в биологическом образце рыбы, где набор включает олигонуклеотидный праймер. Изобретение расширяет арсенал средств для выявления болезней рыб. 4 н. и 1 з.п. ф-лы, 12 ил., 6 табл., 4 пр.
1. Олигонуклеотидный праймер, который:
(a) включает по меньшей мере 18 последовательных нуклеотидов последовательности, которая представляет собой часть или комплементарна части, референсной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5 или SEQ ID NO: 6, или
(b) включает по меньшей мере 18 последовательных нуклеотидов последовательности, которая по меньшей мере на 80% идентична последовательности, которая представляет собой или комплементарна последовательности, выбранной из группы, состоящей из SEQ ID NO: 12 - SEQ ID NO: 80;
при условии, что указанный олигонуклеотидный праймер не включает последовательность, выбранную из группы, состоящей из SEQ ID NO: 21 - SEQ ID NO: 23.
2. Применение по меньшей мере одного олигонуклеотидного праймера по п.1 в способе обнаружения вируса, который инфицирует и способен вызывать гибель пинагора (Cyclopterus lumpus), где вирусный геном включает последовательность нуклеиновой кислоты, которая по меньшей мере на 90% идентична любой из SEQ ID NO: 1, 2, 3, 4, 5, 6, и где указанная последовательность нуклеиновой кислоты содержит основание урацил (U) вместо основания тимин (T).
3. Способ обнаружения вируса, который инфицирует и способен вызывать гибель рыбы, включающий следующие стадии:
(a) контакт нуклеиновой кислоты, выделенной из биологического образца рыбы, по меньшей мере с одним олигонуклеотидным праймером по п.1 с получением смеси,
(b) определение, присутствует ли после амплификации смеси a) продукт амплификации, где присутствие продукта амплификации указывает на присутствие РНК, ассоциированной с вирусом, и, следовательно, на присутствие вируса в биологическом образце.
4. Набор для обнаружения вируса в биологическом образце рыбы, где набор включает олигонуклеотидный праймер по п.1.
5. Набор по п.4, где указанный набор является тестом ОТ-ПЦР в реальном времени.
| WO 2016075277 A1, 19.05.2016 | |||
| Р | |||
| РАХКОНЕН, П | |||
| ВЕННЕЗСТРЕМ и др | |||
| Здоровая рыба | |||
| Профилактика, диагностикa и лечение болезней, Nykypaino, Helsinki 2013, стр.52-66 | |||
| Е.Л | |||
| МИКУЛИЧ УЧЕБНО-МЕТОДИЧЕСКИЙ КОМПЛЕКС ПО ДИСЦИПЛИНЕ "ИХТИОПАТОЛОГИЯ", Учреждение образования "Белорусская государственная сельскохозяйственная академия", 2011, Горки 2011, стр | |||
| Прибор для массовой выработки лекал | 1921 |
|
SU118A1 |
Авторы
Даты
2024-02-16—Публикация
2020-02-04—Подача