ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к новому гену ацилтрансферазы и его использованию. Ген ацилтрансферазы по настоящему изобретению может являться геном глицерол-3-фосфат-ацилтрансферазы (GPAT) и/или геном глицеронфосфат-O-ацилтрансферазы (GNPAT).
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Жирные кислоты являются важными компонентами, образующими липиды, такие как фосфолипиды и триацилглицерол. Жирные кислоты, имеющие два или более ненасыщенных участка связывания, носят общее название полиненасыщенные жирные кислоты (PUFA). В частности, например, известны арахидоновая кислота, дигомо-γ-линоленовая кислота, эйкозапентаеновая кислота, и докозагексаеновая кислота, и сообщают об их различных биологических активностях (непатентный документ 1). Некоторые из полиненасыщенных жирных кислот не могут синтезироваться в организмах животных, и такие полиненасыщенные жирные кислоты должны поступать в организм из пищевых продуктов как незаменимые жирные кислоты.
В организмах животных полиненасыщенные жирные кислоты содержаться в различных органах и тканях. Например, арахидоновую кислоту выделяют из липидов, выделяемых из надпочечника и печени животных. Однако количества этих полиненасыщенных жирных кислот, содержащихся в органах животных, невелики, и полиненасыщенные жирные кислоты, экстрагируемые и выделяемые только из органов животных, недостаточны для получения их большого количества. Таким образом, разрабатывают микробиологические способы получения полиненасыщенных жирных кислот посредством культивирования различных микроорганизмов. В частности, известно, что микроорганизмы рода Mortierella эффективно продуцируют липиды, содержащие полиненасыщенные жирные кислоты, такие как арахидоновая кислота. Также предпринимают другие попытки продуцировать полиненасыщенные жирные кислоты в растениях. Как известно, полиненасыщенные жирные кислоты представляют запасные липиды, такие как триацилглицерол, и накапливаются в клетках микроорганизмов или семенах растений.
Триацилглицерол в качестве запасного липида генерируется в живых организмах следующим образом: глицерол-3-фосфат-ацилтрансфераза переносит ацильную группу на глицерол-3-фосфат для получения лизофосфатидной кислоты. Ацилтрансфераза лизофосфатидной кислоты переносит другую ацильную группу на лизофосфатидную кислоту для получения фосфатидной кислоты. Фосфатаза фосфатидной кислоты дефосфорилирует фосфатидную кислоту для получения диацилглицерола. Диацилглицерол-ацилтрансфераза переносит дополнительную ацильную группу на диацилглицерол для получения, в конечном итоге, триацилглицерола.
Известно, что в указанном выше пути биосинтеза триацилглицерола или пути биосинтеза фосфолипидов глицерол-3-фосфат-ацилтрансфераза (далее в настоящем описании, также обозначаемая как "GPAT": EC 2.3.1.15) задействует реакцию генерации лизофосфатидной кислоты через ацилирование глицерол-3-фосфата.
Сообщали о существовании гена GPAT в некоторых организмах. В качестве генов GPAT, полученных из млекопитающих, клонируют два типа генов GPAT, т.е. микросомальный тип (мембраносвязанная форма) и митохондриальный тип (мембраносвязанная форма) (непатентный документ 2). В качестве генов GPAT, полученных из растений, клонируют три типа генов GPAT, т.е. микросомальный тип (мембраносвязанная форма), митохондриальный тип (мембраносвязанная форма) и хлоропластный тип (свободная форма), (непатентный документ 3).
В качестве генов GPAT, полученных из грибков Saccharomyces cerevisiae, клонируют два типа генов GPAT, т.е. микросомальный тип (мембраносвязанная форма) GPT2/GAT1 (YKR067w) и SCT1/GAT2 (YBL011w), и известно, что одновременная делеция этих типов генов GPAT приводит к гибели (непатентный документ 4). Показано, что GPT2 обладает активностью, проявляя широкую субстратную специфичность к жирным кислотам от пальмитиновой кислоты (16:0) до олеиновой кислоты (18:1), где SCT1 демонстрирует высокую субстратную селективность к жирным кислотам, имеющим 16 атомов углерода, таким как пальмитиновая кислота (16:0) и пальмитолеиновая кислота (16:1) (непатентный документ 4).
Кроме того, ген GPAT клонируют из различных биологических видов. В частности, сообщают, что получают GPAT из продуцирующего липиды грибка рода Mortierella.
Что касается GPAT, полученной из Mortierella ramanniana, выделяют микросомальный тип GPAT, и показано, что эта GPAT в качестве донора ацильной группы, предпочтительно, использует олеиновую кислоту (18:1) с селективностью в 5,4 раз выше, чем для пальмитиновой кислоты (16:0) (непатентный документ 5). Что касается GPAT, полученной из Mortierella alpina (далее в настоящем описании, также обозначаемого как "M. alpina"), сообщают, что активность глицерол-3-фосфат-ацилтрансферазы свойственна микросомальной фракции (непатентный документ 6).
Показано, что, если присутствующая в микросоме M. alpina GPAT (мембраносвязанная форма) реагирует с различными ацил-CoA in vitro, GPAT использует в качестве субстратов широкий диапазон полиненасыщенных жирных кислот, таких как олеиновая кислота (18:1), линолевая кислота (18:2), дигомо-γ-линоленовая кислота (DGLA) (20:3) и арахидоновая кислота (20:4), с сохранением своей высокой активности (патентный документ 1).
Показано, что клонированную из M. alpina GPAT (ATCC № 16266) (далее в настоящем описании, обозначаемую как MaGPAT1 (ATCC № 16266)) экспрессировали в Yarrowia lipolytica, которую трансформировали таким образом, что эйкозапентаеновая кислота (EPA) может биологически синтезироваться, и в результате среди всех жирных кислот возрастает доля дигомо-γ-линоленовой кислоты (DGLA) (20:3), в то время как доля олеиновой кислоты (18:1) снижается. Это показывает, что избирательно включается полиненасыщенная жирная кислота с более длинной цепью и более высокой степенью ненасыщенности (патентный документ 2).
В недавних исследованиях гомолог GPAT MaGPAT2 выделяли из M. alpina (штамм 1S-4), и сообщают, что гомолог обладает субстратной специфичностью, отличающейся от MaGPAT1 (патентный документ 3). Т.е. при экспрессии в дрожжах MaGPAT1 повышает содержание пальмитиновой кислоты в продуцируемых дрожжами липидах, в то время как MaGPAT2 повышает содержание олеиновой кислоты в продуцируемых дрожжами липидах.
СПИСОК ЛИТЕРАТУРЫ
ПАТЕНТНЫЕ ДОКУМЕНТЫ
Патентный документ 1: международная публикация № WO2004/087902.
Патентный документ 2: патентная публикация США № 2006/0094091.
Патентный документ 3: международная публикация № WO2008/156026.
НЕПАТЕНТНЫЕ ДОКУМЕНТЫ
Непатентный документ 1: Lipids, 39, 1147-1161, 2004
Непатентный документ 2: Biochimica et Biophysica Acta, 1348, 17-26, 1997.
Непатентный документ 3: Biochimica et Biophysica Acta, 1348, 10-16, 1997.
Непатентный документ 4: The Journal of Biological Chemistry, 276 (45), 41710-41716, 2001.
Непатентный документ 5: The Biochemical Journal, 355, 315-322, 2001.
Непатентный документ 6: Biochemical Society Transactions, 28, 707-709, 2000.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
ЗАДАЧИ, ПОДЛЕЖАЩИЕ РЕШЕНИЮ ПОСРЕДСТВОМ ИЗОБРЕТЕНИЯ
Когда гены GPAT, о которых сообщали ранее, встраивают в клетки-хозяева и экспрессируют в них, композиция продуцируемых хозяином жирных кислот ограничена их субстратной специфичностью. Необходима идентификация нового гена, продукт которого может продуцировать заданную композицию жирных кислот при встраивании или экспрессии в клетку-хозяина.
Объект настоящего изобретения относится к белку и нуклеиновой кислоте, посредством экспрессии или встраивания в клетки-хозяева которых можно достигать продукции жира с заданным композиционным отношением жирных кислот, можно повышать содержание заданной жирной кислоты или можно повышать количество запасного липида триацилглицерола (TG).
СРЕДСТВА РЕШЕНИЯ ЗАДАЧ
Автор изобретения проводил тщательное исследование для решения указанных выше проблем. Во-первых, автор изобретения исследовал геном продуцирующего липиды гриба Mortierella alpina и выделял из генома последовательности с высокой степенью гомологии с известными генами глицерол-3-фосфат-ацилтрансферазы (GPAT). Кроме того, для получения полноразмерной открытой рамки считывания (ORF), кодирующей GPAT, клонировали полноразмерную кДНК скринингом библиотеки или ПЦР кДНК библиотеки. Автор изобретения исследовал продукцию композиции жирных кислот, встраивая ген в клетки-хозяева с высокой пролиферативной способностью, такие как дрожжи, и, в результате, автор изобретения успешно клонировал ген, относящийся к новой GPAT с отличающейся субстратной специфичностью и способной генерировать композицию жирных кислот, отличающуюся от композиции жирных кислот, продуцируемой клетками-хозяевами, экспрессирующими общепринятую GPAT, и настоящее изобретение было завершено. Т.е. настоящее изобретение состоит в следующем.
(1) Нуклеиновая кислота по любому из пунктов, выбранных из пунктов (a)-(g) ниже:
(a) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотную последовательность, приведенную в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(b) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(c) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(d) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(e) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующей белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(f) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающей экзоны, кодирующие белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью; и
(g) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающей экзоны, кодирующие белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью.
(2) Нуклеиновая кислота по п.(1), где нуклеиновая кислота является любой, выбранной из пунктов (a)-(g) ниже:
(a) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотную последовательность, приведенную в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(b) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующей белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(c) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 90% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующей белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(d) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, обладающей идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(e) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующую белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(f) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающую экзоны, кодирующие белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью; и
(g) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 90% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающую экзоны, кодирующие белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью.
(3) Нуклеиновая кислота по любому из пунктов, выбранных из пунктов (a)-(d) ниже:
(a) нуклеиновая кислота, содержащая нуклеотидную последовательность, приведенную в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, или ее фрагмент;
(b) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, или ее фрагмент;
(c) нуклеиновая кислота, содержащая нуклеотидную последовательность, приведенную в SEQ ID NO:3, SEQ ID NO:6 или SEQ ID NO:11, или ее фрагмент; и
(d) нуклеиновая кислота, содержащая нуклеотидную последовательность приведенную в SEQ ID NO:7 или SEQ ID NO:12, или ее фрагмент.
(4) Нуклеиновая кислота по любому из пунктов, выбранных из пунктов (a)-(g) ниже:
(a) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотную последовательность, приведенную в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности глицерол-3-фосфат-ацилтрансферазы (далее в настоящем описании также обозначаемая как "недостаточность GPAT") дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим нуклеиновую кислоту, кодирующую белок, по сравнению с продукцией в нетрансформированном вектором хозяине;
(b) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(c) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(d) нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количства триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(e) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующую белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(f) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине; и
(g) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине.
(5) нуклеиновая кислота по п.(4), где нуклеиновая кислота является любой, выбранной из пунктов (a)-(g) ниже:
(a) нуклеиновая кислота, содержащая нуклеотидную последовательность, включающую экзоны, кодирующие белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(b) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, в условиях 2×SSC при 50°C, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(c) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 90% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(d) нуклеиновая кислота, содержащая нуклеотидную последовательность, включающую экзоны, кодирующие белок, состоящий из аминокислотной последовательности, обладающей идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(e) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, в условиях 2×SSC при 50°C, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине;
(f) нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую с нуклеиновой кислотой, содержащей нуклеотидную последовательность, комплементарную нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12, в условиях 2×SSC при 50°C, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине; и
(g) нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 90% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающую экзоны, кодирующие белок, обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине.
(6) Белок, выбранный из пунктов (a) и (b) ниже:
(a) белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или a глицеронфосфат-ацилтрансферазной активностью; и
(b) белок, состоящий из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью.
(7) Белок, выбранный из пунктов (a) и (b) ниже:
(a) белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью; и
(b) белок, состоящий из аминокислотной последовательности, обладающей идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью.
(8) Белок, выбранный из пунктов (a) и (b) ниже:
(a) белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине; и
(b) белок, состоящий из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине.
(9) Белок, выбранный из пунктов (a) и (b) ниже:
(a) белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине; и
(b) белок, состоящий из аминокислотной последовательности, обладающей идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий одной из следующих активностей по пунктам i)-v):
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности GPAT дрожжей (S. cerevisiae); и
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине.
(10) Белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9.
(11) Рекомбинантный вектор, содержащий нуклеиновую кислоту по любому из пунктов (1)-(5).
(12) Трансформант, трансформированный рекомбинантным вектором по п.(11).
(13) Композиция жирных кислот, содержащая жирную кислоту или липид, получаемый культивированием трансформанта по п.(12).
(14) Способ получения композиции жирных кислот, включающий сбор жирной кислоты или липида из культуры, получаемой культивированием трансформанта по п.(12).
(15) Пища, содержащая композицию жирных кислот по п.(13).
ПРЕИМУЩЕСТВА ИЗОБРЕТЕНИЯ
GPAT по настоящему изобретению обладает субстратной специфичностью, отличающейся от таковой общепринятой GPAT, и может позволять хозяину продуцировать жирные кислоты, обладающие композицией, отличающейся от композиции жирных кислот, получаемой экспрессией общепринятой GPAT хозяином. Это может обеспечивать получение липидов с заданными характеристиками и эффектами и, таким образом, применимо в пище, косметике, лекарственных средствах, мыле и т.д.
GPAT по настоящему изобретению может повышать продукцию жирных кислот и запасных липидов, и, таким образом, может повышать продукцию полиненасыщенных жирных кислот в микроорганизмах и растениях и является предпочтительной.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
На фиг.1-1 показано сравнение геномной последовательности (SEQ ID NO:7) и последовательности CDS (SEQ ID NO:3) MaGPAT4, полученной из M. alpina штамма 1S-4.
Фиг.1-2 является продолжением фигуры 1-1.
Фиг.1-3 является продолжением фигуры 1-2.
На фиг.2-1 показана последовательность CDS (SEQ ID NO:3) MaGPAT4, полученной из M. alpina штамма 1S-4, и предсказанная из нее аминокислотная последовательность (SEQ ID NO:2), где двойным подчеркиванием указывают, что является совпадением в качестве области, обладающей высокой гомологией с мотивом ацилтрансферазы (инвентарный номер PF01553) из Pfam.
Фиг.2-2 является продолжением фиг.2-1.
На фиг.3-1 показано сравнение геномной последовательности (SEQ ID NO:12) и последовательности CDS (SEQ ID NO:10) MaGPAT5, полученной из M. alpina штамма 1S-4.
Фиг.3-2 является продолжением фиг.3-1.
На фиг.4-1 показана последовательность кДНК (SEQ ID NO:11) MaGPAT5, полученной из M. alpina штамма 1S-4, и предсказанная из нее аминокислотная последовательность (SEQ ID NO:9).
Фиг.4-2 является продолжением фиг.4-1.
На фиг.5 показано сравнение предсказанной аминокислотной последовательности (SEQ ID NO:2) MaGPAT4, полученной M. alpina штамма 1S-4, аминокислотной последовательности (SEQ ID NO:21; инвентарный номер GenBank XP_001224211) предполагаемого белка, полученного из аскомицета Chaetomium globosum CBS 148.51, и аминокислотной последовательности (SEQ ID NO:22; инвентарный номер GenBank BAE78043) GPAT, белка plsB, полученного из E. coli, где одинарным подчеркиванием указывают области, обладающие высокой гомологией с мотивом ацилтрансферазы (инвентарный номер PF01553) из Pfam. В частности, область консервативного гомолога GPAT указана двойным подчеркиванием, символом "∗" указывают аминокислотный остаток, важный для ацилтрансферазной активности, и символом "+" указывают аминокислотный остаток, необходимый для связывания с G3P.
На фиг.6-1 показано сравнение предсказанной аминокислотной последовательности (SEQ ID NO:9) MaGPAT5, полученной из M. alpina штамма 1S-4, аминокислотной последовательности (SEQ ID NO:23; инвентарный номер GenBank XP_759516) предполагаемого белка UM03369, полученного из базидиомицета Ustilago maydis 521, и аминокислотных последовательностей SCT1 (YBL011W) (SEQ ID NO:24) и GPT2 (YKR067W) (SEQ ID NO:25) GPAT, полученной из S. cerevisiae, где двойным подчеркиванием указывают область консервативного гомолога GPAT, символом "∗" указывают аминокислотный остаток, важный для ацилтрансферазной активности, и символом "+" указывают аминокислотный остаток, необходимый для связывания с G3P.
Фиг.6-2 является продолжением фигуры 6-1.
Фиг.7 представляет собой график композиционного отношения жирных кислот в липидных фракциях, когда экспрессию индуцировали галактозой посредством культивирования дрожжей, трансформированных плазмидой, содержащей MaGPAT4-long или MaGPAT4, связанную с галактоза-индуцибельным промотором, в среде SG-Trp.
Фиг.8 представляет собой график, демонстрирующий композицию всех жирных кислот, когда дрожжи, трансформированные плазмидой, содержащей MaGPAT4-long или MaGPAT4, связанную с галактоза-индуцибельным промотором, культивировали в среде SC-Trp, не содержащей галактозу.
Фиг.9 представляет собой график, демонстрирующий зависящее от времени изменение продукции арахидоновой кислоты на сухой вес клеток, когда GPAT4 гиперэкспрессировали в M. alpina.
ОПИСАНИЕ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ
Настоящее изобретение относится к новому гену ацилтрансферазы, полученному из Mortierella, и его использованию. Ацилтрансфераза по настоящему изобретению может являться ацилтрансферазой, ацилирующей глицерол-3-фосфат для получения лизофосфатидной кислоты и/или переносящей ацильную группу на гидроксильную группу глицеронфосфата.
Ацилтрансфераза по настоящему изобретению является ферментом, катализирующим реакцию переноса ацильной группы на глицерол-3-фосфат и/или глицеронфосфат. Акцептором ацильной группы для фермента по настоящему изобретению, как правило, является глицерол-3-фосфат и/или глицеронфосфат, но не ограничивается ими.
Таким образом, ацилтрансфераза по настоящему изобретению может обладать активностью глицерол-3-фосфат-ацилтрансферазы (GPAT) и/или глицеронфосфат-O-ацилтрансферазы (GNPAT). Однако в настоящем описании фермент по настоящему изобретению также можно условно обозначать как "глицерол-3-фосфат-ацилтрансфераза" или "GPAT", независимо от его фактической активности.
Нуклеиновая кислота, кодирующая глицерол-3-фосфат-ацилтрансферазу по настоящему изобретению
Примеры глицерол-3-фосфат-ацилтрансферазы (GPAT) по настоящему изобретению включают MaGPAT4, MaGPAT4-long и MaGPAT5. Соответствие кДНК, CDS и ORF, кодирующих MaGPAT4, MaGPAT4-long или MaGPAT5, и их предсказанной аминокислотной последовательностью представлено в таблице 1.
Последовательности, относящиеся к MaGPAT4 по настоящему изобретению, включают SEQ ID NO:2, представляющую аминокислотную последовательность MaGPAT4; SEQ ID NO:1, представляющую последовательность области ORF MaGPAT4; и SEQ ID NO:3, представляющую последовательность CDS или кДНК MaGPAT4. Среди этих последовательностей SEQ ID NO:1 соответствует нуклеотидам с 1 по 2475 в последовательности, приведенной в SEQ ID NO:3. Последовательности, относящиеся к MaGPAT4-long по настоящему изобретению, включают SEQ ID NO:5, представляющую аминокислотную последовательность MaGPAT4-long; SEQ ID NO:4, представляющую последовательность области ORF MaGPAT4-long; и SEQ ID NO:6, представляющую последовательность области CDS или кДНК MaGPAT4-long. Среди них SEQ ID NO:1 соответствует нуклеотидам с 1 по 2475 в последовательности, приведенной в SEQ ID NO:3. Среди этих последовательностей SEQ ID NO:4 соответствует нуклеотидам с 1 по 2643 в последовательности, приведенной в SEQ ID NO:6. Как показано в таблице, аминокислотная последовательность и нуклеотидная последовательность MaGPAT4 являются частями аминокислотной последовательности и нуклеотидной последовательности MaGPAT4-long, соответственно. SEQ ID NO:7 представляет геномную нуклеотидную последовательность, кодирующую MaGPAT4 и MaGPAT4-long по настоящему изобретению. В случае кодирования MaGPAT4 геномная последовательность, приведенная в SEQ ID NO:7, состоит из десяти экзонов и девяти интронов, и экзонные области соответствуют нуклеотидам с 596 по 744, с 850 по 924, с 1302 по 1396, с 1480 по 1726, с 1854 по 2279, с 2370 по 2632, с 2724 по 3299, с 3390 по 3471, с 3575 по 4024 и с 4133 по 4248 в SEQ ID NO:7. В случае кодирования MaGPAT4-long геномная последовательность, приведенная в SEQ ID NO:7, состоит из десяти экзонов и девяти интронов, и экзонные области соответствуют нуклеотидам с 428 по 744, с 850 по 924, с 1302 по 1396, с 1480 по 1726, с 1854 по 2279, с 2370 по 2632, с 2724 по 3299, с 3390 по 3471, с 3575 по 402 и с 4133 по 4248 в SEQ ID NO:7.
Последовательности, относящиеся к MaGPAT5 по настоящему изобретению, включают SEQ ID NO:9, представляющую аминокислотную последовательность MaGPAT5; SEQ ID NO:8, представляющую последовательность области ORF MaGPAT5; SEQ ID NO:10, представляющую последовательность области CDS MaGPAT5; и SEQ ID NO:11, представляющую последовательность кДНК MaGPAT5. Среди этих последовательностей SEQ ID NO:10 соответствует нуклеотидам с 225 по 2591 в последовательности, приведенной в SEQ ID NO:11; и SEQ ID NO:8 соответствует нуклеотидам с 225 по 2588 в последовательности, приведенной в SEQ ID NO:11, и нуклеотидам с 1 по 2364 в последовательности, приведенной в SEQ ID NO:10. SEQ ID NO:12 представляет геномную нуклеотидную последовательность, кодирующую MaGPAT5 по настоящему изобретению. Геномная последовательность, приведенная в SEQ ID NO:12, состоит из трех экзонов и двух интронов, и экзонные области соответствуют нуклеотидам с 1 по 302, с 457 по 1676 и с 1754 по 2598 в SEQ ID NO:12.
Нуклеиновые кислоты по настоящему изобретению включают одноцепочечные и двухцепочечные ДНК, и а также комплементарную им РНК, которые могут являться природными или искусственно полученными. Неограничивающие примеры ДНК включают геномную ДНК, кДНК, соответствующую геномной ДНК, химически синтезированную ДНК, амплифицированную посредством ПЦР ДНК, их сочетания и гибриды ДНК/РНК.
Предпочтительные варианты осуществления нуклеиновых кислот по настоящему изобретению включают (a) нуклеиновые кислоты, содержащие нуклеотидную последовательность, приведенную в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, (b) нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, (c) нуклеиновые кислоты, содержащие нуклеотидную последовательность, приведенную в SEQ ID NO:3, SEQ ID NO:6 или SEQ ID NO:11, и (d) нуклеиновые кислоты, содержащие нуклеотидную последовательность, приведенную в SEQ ID NO:7 или SEQ ID NO:12.
Для получения этих нуклеотидных последовательностей, для поиска нуклеотидной последовательности, кодирующей белок, обладающий высокой идентичностью с известными белками, обладающими GPAT-активностью, можно использовать данные о нуклеотидной последовательности EST или геномной ДНК организмов с GPAT-активностью. Предпочтительные организмы с GPAT-активностью являются продуцирующими липиды грибками, включая, в качестве неограничивающих примеров, M. alpina.
Для анализа EST сначала получали библиотеку кДНК. Библиотеку кДНК можно получать, обращаясь к "Molecular Cloning, A Laboratory Manual 3rd ed." (Cold Spring Harbor Press (2001)). Альтернативно, можно использовать коммерчески доступный набор для получения библиотеки кДНК. Примеры способа получения библиотеки кДНК, подходящей для настоящего изобретения, являются следующими. А именно, подходящий штамм продуцирующего липиды грибка M. alpina инокулируют в подходящую среду и предварительно культивируют в течение подходящего периода времени. Подходящими для этого предварительного культивирования условиями культивирования являются, например, композиция среды из 1,8% глюкозы и 1% дрожжевого экстракта, pH 6,0, время культивирования от 3 до 4 дней, и температура культивирования 28°C. Затем предварительно культивируемый продукт подвергают основному культивированию в подходящих условиях. Подходящей для основного культивирования композицией среды является, например, 1,8% глюкозы, 1% соевого порошка, 0,1% оливкового масла, 0,01% адеканола, 0,3% KH2PO4, 0,1% Na2SO4, 0,05% CaCl2·2H2O, и 0,05% MgCl2·6H2O и pH 6,0. Подходящими для основного культивирования условиями культивирования являются, например, аэрация и перемешивание культуры при 300 об./мин., 1 vvm и 26°C в течение 8 дней. При культивировании можно добавлять подходящее количество глюкозы. В течение основного культивирования в подходящие моменты времени отбирают культивируемый продукт, клетки из которого собирают для получения тотальной РНК. Тотальную РНК можно получать любым известным способом, таким как способ с использованием гидрохлорида гуанидина/CsCl. Из получаемой тотальной РНК можно очищать поли(A)+РНК с использованием коммерчески доступного набора и можно получать библиотеку кДНК с использованием коммерчески доступного набора. Нуклеотидную последовательность любого клона из полученной библиотеки кДНК определяют с использованием праймеров, сконструированных по вектору для определения нуклеотидной последовательности вставки. В результате можно получать EST. Например, при использовании набора ZAP-cDNA GigapackIII Gold Cloning Kit (Stratagene) для получения библиотеки кДНК, возможно направленное клонирование.
При анализе геномной ДНК культивируют клетки организма с GPAT-активностью и из клеток получают геномную ДНК. Определяют нуклеотидную последовательность получаемой геномной ДНК и составляют определенную нуклеотидную последовательность. В полученной в конечном итоге последовательности суперконтига ищут последовательность, кодирующую аминокислотную последовательность, обладающую высокой гомологией с аминокислотной последовательностью известного белка, обладающего GPA-активностью. Праймеры получают из последовательности суперконтига, давшей совпадение с последовательностью, кодирующей такую аминокислотную последовательность. ПЦР осуществляют с использованием библиотеки кДНК в качестве матрицы, и получаемый фрагмент ДНК встраивают в плазмиду для клонирования. ПЦР осуществляют с использованием клонированной плазмиды в качестве матрицы и указанных выше праймеров для получения зонда. Проводят скрининг библиотеки кДНК с использованием получаемого зонда.
Поиск гомологии предсказанных аминокислотных последовательностей MaGPAT4 и MaGPAT5 по настоящему изобретению осуществляли с использованием программного обеспечения BLASTp среди аминокислотных последовательностей, зарегистрированных в GenBank. Аминокислотной последовательностью, обладающей высокой идентичностью с последовательностью MaGPAT4, является аминокислотная последовательность (инвентарный номер GenBank XP_001224211) предполагаемого белка, полученного из аскомицета Chaetomium globosum CBS 148.51, и идентичность составляет 39,3%. Аминокислотная последовательность MaGPAT4 также обладает гомологией с глицеронфосфат-O-ацилтрансферазой (GNPAT; инвентарный номер GenBank AAH00450), полученной из человека, и идентичность аминокислот составляет 22,6%. Кроме того, идентичность аминокислот между MaGPAT4 и белком plsB (инвентарный номер GenBank BAE78043), являющимся GPAT, полученной из Escherichia coli (E. coli), составляет 17,6%. Аминокислотной последовательностью, обладающей высокой идентичностью с последовательностью MaGPAT5, является аминокислотная последовательность (инвентарный номер GenBank XP_759516) предполагаемого белка, полученного из базидиомицета Ustilago maydis 521, и идентичность составляет 15,4%.
Настоящее изобретение также включает нуклеиновые кислоты, функционально эквивалентные нуклеиновой кислоте, содержащей нуклеотидную последовательность, приведенную в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8 (далее в настоящем описании также обозначаемую как "нуклеотидная последовательность по настоящему изобретению"), или нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9 (далее в настоящем описании также обозначаемой как "аминокислотная последовательность по настоящему изобретению"). Термин "функционально эквивалентный" относится к тому, что белок, кодируемый нуклеотидной последовательностью по настоящему изобретению, и белок, состоящий из аминокислотной последовательности по настоящему изобретению, обладают глицерол-3-фосфат-ацилтрансферазной (GPAT) активностью и/или a глицеронфосфат-O-ацилтрансферазной (GNPAT) активностью. Кроме того, термин "функционально эквивалентный" может относиться к наличию любой из следующих активностей относительно композиционного отношения жирных кислот в хозяине, экспрессирующем белок, кодируемый нуклеотидной последовательностью по настоящему изобретению, или белок, состоящий из аминокислотной последовательности по настоящему изобретению:
i) активность, относящаяся к выработке композиции жирных кислот, содержащей большую долю пальмитиновой кислоты и меньшую долю пальмитолеиновой кислоты, в экспрессирующих белок дрожжах по сравнению с их долями в композиции жирных кислот в неэкспрессирующем белок хозяине;
ii) активность, относящаяся к получению больших долей жирных кислот в экспрессирующих белок дрожжах по сравнению с таковым в неэкспрессирующем белок хозяине;
iii) активность, относящаяся к получению большего количества триацилглицерола (TG) в экспрессирующих белок дрожжах по сравнению с TG в неэкспрессирующем белок хозяине;
iv) активность, относящаяся к восполнению недостаточности глицерол-3-фосфат-ацилтрансферазы (GPAT недостаточность) дрожжей (S. cerevisiae), где недостаточность GPAT связана с недостаточностями, предпочтительно, обоих генов SCT1 и GPT2; и/или
v) активность, относящаяся к повышению продукции арахидоновой кислоты в хозяине, трансформированном рекомбинантным вектором, содержащим кодирующую белок нуклеиновую кислоту, по сравнению с продукцией в нетрансформированном вектором хозяине.
Такие нуклеиновые кислоты, функционально эквивалентные нуклеиновым кислотам по настоящему изобретению, включают нуклеиновые кислоты, содержащие нуклеотидные последовательности, представленные в любом пункте, выбранном из пунктов (a)-(g) ниже. Следует отметить, что в приведенных ниже описаниях нуклеотидных последовательностей термин "активность по настоящему изобретению" относится к "GPAT-активности", "GNPAT-активности", или, по меньшей мере, одной активности, выбранной из представленных выше активностей по пунктам i)-v).
(a) Нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий активностью по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, кодирующие белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий активностью по настоящему изобретению.
В частности, нуклеотидная последовательность, содержащаяся в нуклеиновой кислоте по настоящему изобретению, является нуклеотидной последовательностью, кодирующей белок, обладающий описываемой выше активностью по настоящему изобретению и состоящий из:
(i) аминокислотной последовательности с делецией одной или нескольких (предпочтительно, от одной до нескольких (например, от 1 до 250, от 1 до 200, от 1 до 150, от 1 до 100, от 1 до 80, от 1 до 75, от 1 до 50, от 1 до 30, от 1 до 25, от 1 до 20 или от 1 до 15, более предпочтительно 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1)) аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(ii) аминокислотной последовательности с заменой одной или нескольких (предпочтительно, от одной до нескольких (например, от 1 до 250, от 1 до 200, от 1 до 150, от 1 до 100, от 1 до 80, от 1 до 75, от 1 до 50, от 1 до 30, от 1 до 25, от 1 до 20 или от 1 до 15, более предпочтительно 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1)) аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(iii) аминокислотной последовательности с добавлением одной или нескольких (предпочтительно, от одной до нескольких (например, от 1 до 250, от 1 до 200, от 1 до 150, от 1 до 100, от 1 до 80, от 1 до 75, от 1 до 50, от 1 до 30, от 1 до 25, от 1 до 20 или от 1 до 15, более предпочтительно 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1)) аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9; или
(iv) аминокислотной последовательности в любой комбинации указанных выше (i)-(iii).
Среди указанных выше, замена является предпочтительно консервативной, что означает замену конкретного аминокислотного остатка другим остатком со схожими физическими и химическими свойствами. Она может являться любой заменой, по-существу, не изменяющей структурные свойства исходной последовательности. Например, возможна любая замена при условии, что замещенные аминокислоты не разрушают спираль исходной последовательности или не разрушают любой другой тип вторичной структуры, характеризующий исходную последовательность.
Как правило, консервативную замену встраивают синтезом с использованием биологической системы или химическим пептидным синтезом, предпочтительно, химическим пептидным синтезом. В таком случае заместители могут включать неприродный аминокислотный остаток, пептидомиметик или обратную или инвертированную форму, где незамещенная область является обратной или инвертированной в аминокислотной последовательности.
Неограничивающие примеры взаимозаменяемых аминокислотных остатков классифицируют и приводят ниже:
Группа A: лейцин, изолейцин, норлейцин, валин, норвалин, аланин, 2-аминобутановая кислота, метионин, O-метилсерин, трет-бутилглицин, трет-бутилаланин и циклогексилаланин;
Группа B: аспарагиновая кислота, глутаминовая кислота, изоаспарагиновая кислота, изоглутаминовая кислота, 2-аминоадипиновая кислота и 2-аминосубериновая кислота;
Группа C: аспарагин и глутамин;
Группа D: лизин, аргинин, орнитин, 2,4-диаминобутановая кислота и 2,3-диаминопропионовая кислота;
Группа E: пролин, 3-гидроксипролин и 4-гидроксипролин;
Группа F: серин, треонин и гомосерин; и
Группа G: фенилаланин и тирозин.
При неконсервативной замене члена одной из указанных выше групп можно заменять членом другой группы. В таком случае для сохранения биологической функции белка по настоящему изобретению предпочтительно учитывать индексы гидрофобности аминокислот (Kyte, et al., J. Mol. Biol., 157: 105-131 (1982)).
В случае неконсервативной замены можно выполнять замены аминокислот на основе их гидрофильности.
Необходимо отметить, что при консервативной замене или неконсервативной замене аминокислотные остатки, соответствующие 316-ой, 319-ой и 351-ой аминокислоте в SEQ ID NO:2, желательно, представляют собой глицин, серин и пролин, соответственно. В SEQ ID NO:9 аминокислотные остатки, соответствующие 430-ой, 432-ой, и 465-ой аминокислоте, желательно, представляют собой глицин, серин и пролин, соответственно.
На всем протяжении описания и чертежей нуклеотиды, аминокислоты и их аббревиатуры указаны в соответствии с рекомендациями Комиссии IUPAC-IUB по биохимической номенклатуре или как общепринято используют в этой области, например, как описано в Immunology-A Synthesis (2-е издание, издательство E.S. Golub и D.R. Gren, Sinauer Associates, Sunderland, Massachusetts (1991)). Кроме того, аминокислоты, которые могут иметь оптические изомеры, предназначены для обозначения своих L-изомеров, если не указано иначе.
Стереоизомеры, такие как D-аминокислоты указанных выше аминокислот, неприродные аминокислоты, такие как α,α-незамещенные аминокислоты, N-алкиламинокислоты, молочная кислота и другие нестандартные аминокислоты также могут входить в состав белков по настоящему изобретению.
Необходимо отметить, что в обозначении белков, используемом на всем протяжении описания, направление влево переставляет собой направление в сторону аминоконца, и направление вправо представляет собой направление в сторону карбоксиконца в соответствии с общепринятым использованием и условным обозначением в данной области.
Аналогично, в основном, если не указано иначе, левый конец одноцепочечных полинуклеотидных последовательностей представляет собой 5'-конец, и направление влево в двухцепочечных полинуклеотидных последовательностях обозначают как 5'-направление.
Специалисты в данной области могут конструировать и получать подходящих мутантов представленных в описании белков с использованием известных в данной области способов. Например, они могут идентифицировать область молекулы белка, подходящую для изменения структуры белка по настоящему изобретению без нарушения биологической активности белка посредством воздействия на область, по-видимому, менее важную для биологической активности белка. Специалисты в данной области также могут идентифицировать остаток или область, консервативную для схожих белков. Специалисты в данной области также могут встраивать консервативную аминокислотную замену в область, по-видимому, важную для биологической активности или структуры белка по настоящему изобретению, без нарушения биологической активности и без нарушения полипептидной структуры белка.
Специалисты в данной области могут проводить, так называемое, структурно-функциональное исследование, при котором идентифицируют остатки в пептиде, схожем с пептидной последовательностью белка по настоящему изобретению, важные для биологической активности или структуры белка, сравнивают аминокислотные остатки этих двух пептидов и, таким образом, прогнозируют, какой остаток в белке, схожем с белком по настоящему изобретению, является аминокислотным остатком, соответствующим аминокислотному остатку, важному для биологической активности или структуры. Они также могут выбирать мутанта, сохраняющего биологическую активность белка по настоящему изобретению, посредством селекции аминокислотного заместителя, химически схожего с соответствующим предсказанным аминокислотным остатком. Кроме того, специалисты в данной области могут анализировать трехмерную структуру и аминокислотную последовательность этого мутантного белка. Кроме того, специалисты в данной области могут прогнозировать выравнивание аминокислотных остатков, включенных в трехмерную структуру белка на основе полученных, таким образом, аналитических результатов. Хотя, аминокислотные остатки, находящиеся, как предсказано, на поверхности белка, могут участвовать в важных взаимодействиях с другими молекулами, специалисты в данной области будут способны получать мутанта, не вызывающего изменение в этих аминокислотных остатках, находящихся, как предсказано, на поверхности белка, на основе аналитических результатов, как указано выше. Специалисты в данной области также могут получать мутанта с одной заменой аминокислоты для любых аминокислотных остатков в составе белка по настоящему изобретению. Эти мутанты можно подвергать скринингу посредством любого известного анализа для получения информации об отдельных мутантах, что, в свою очередь, делает возможной оценку применимости отдельных аминокислотных остатков в составе белка по настоящему изобретению посредством сравнения случая, когда с заменой конкретного аминокислотного остатка демонстрирует более низкую биологическую активность, чем белок по настоящему изобретению, случая, когда мутант не демонстрирует биологическую активность, или случая, когда мутант демонстрирует неподходящую активность, ингибирующую биологическую активность белка по настоящему изобретению. Кроме того, специалисты в данной области легко могут анализировать замены аминокислот, нежелательные для мутантов белка по настоящему изобретению, на основе информации, собранной при таких общепринятых экспериментах по отдельности или в комбинации с другими мутациями.
Как описано выше, белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, можно получать способами, такими как сайт-специфический мутагенез, как описано, например, в "Molecular Cloning, A Laboratory Manual 3rd ed." (Cold Spring Harbor Press (2001)); "Current Protocols in Molecular Biology" (John Wiley & Sons (1987-1997); Kunkel, (1985), Proc. Natl. Acad. Sci. USA, 82: 488-92; или Kunkel, (1988), Method Enzymol, 85: 2763-6). Получение мутанта с такой мутацией, включающей делецию, замену или добавление аминокислоты, можно осуществлять, например, известными способами, такими как мутагенез по Кюнкелю или мутагенез с дуплексными разрывами с использованием набора для встраивания мутаций на основе сайт-специфического мутагенеза, такого как набор для сайт-специфического мутагенеза QuikChangeTM (производства Stratagene), система для сайт-специфического мутагенеза GeneTailorTM (производства Invitrogen) или система для сайт-специфического мутагенеза TaKaRa (например, Mutan-K, Mutan-Super Express Km; производства Takara Bio Inc.).
В дополнение к указанному выше сайт-специфическому мутагенезу способы встраивания делеции, замены или добавления одной или нескольких аминокислот в аминокислотной последовательности белка при сохранении его активности включают способ обработки гена мутагеном и способ избирательного расщепления гена и делеции, замены или добавления выбранного нуклеотида, и затем лигирования гена.
Нуклеотидная последовательность, содержащаяся в нуклеиновой кислоте по настоящему изобретению предпочтительно является нуклеотидной последовательностью, кодирующей белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающей GPAT-активностью и/или GNPAT-активностью.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению также предпочтительно включают нуклеотидные последовательности, кодирующие белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий активностью по настоящему изобретению.
Количество и участки мутаций или модификаций аминокислот в белке по настоящему изобретению не ограничены при условии сохранения активности по настоящему изобретению.
Активность по настоящему изобретению, представляющую собой GPAT-активность белка, можно измерять известным способом, например, смотри Biochem. J., 355, 315-322, 2001.
Например, "GPAT-активность" по настоящему изобретению можно измерять следующим образом: Из дрожжей, экспрессирующих GPAT по настоящему изобретению, микросомальную фракцию получают, например, способом, описываемым в J. Bacteriology, 173, 2026-2034 (1991) или подобным. Микросомальную фракцию добавляю в реакционный раствор, содержащий 0,44 мМ глицерол-3-фосфата, 0,36 мМ ацил-CoA, 0,5 мМ DTT, 1 мг/мл BSA, 2 мМ MgCl2 и 50 мМ трис-HCl (pH 7,5), с последующей реакцией при 28°C в течение подходящего периода времени. Реакцию останавливают добавлением смеси хлороформа и метанола и экстрагируют липиды. Для измерения количества полученной лизофосфатидной кислоты полученные липиды фракционируют тонкослойной хроматографией или подобным способом.
Активность по настоящему изобретению, представленную в указанных выше пунктах i), ii) или v), можно измерять, например, определением долей жирных кислот в клетке-хозяине, экспрессирующей белок по настоящему изобретению (например, дрожжи, M. alpina). Смесь хлороформа и метанола, доведенную до подходящего соотношения, добавляют к лиофилизированным клеткам, полученным способом получения композиции жирных кислот по настоящему изобретению, и получаемую смесь перемешивают и затем нагревают в течение подходящего периода времени. Клетки разделяют посредством центрифугирования для удаления растворителя. Эту процедуру повторяют несколько раз. Затем липиды высушивают подходящим способом, а затем для получения образца снова растворяют в растворителе, таком как хлороформ. Из подходящего количества этого образца посредством способа с использованием соляной кислоты и метанола жирные кислоты клеток превращают в сложный метиловый эфир и экстрагируют гексаном. Гексан отгоняют с последующим анализом посредством газовой хроматографии.
Активность по настоящему изобретению, представленную в указанном выше п. iii), можно измерять, например, определением количества триацилглицерола (TG) дрожжей, экспрессирующих белок по настоящему изобретению. Липиды экстрагируют и собирают из клеток, как описано выше, и фракцию TG собирают посредством фракционирования, например, посредством тонкослойной хроматографии (TLC). Жирные кислоты, составляющие TG, в собранной фракции TG превращают в сложный метиловый эфир посредством способа с использованием соляной кислоты и метанола и экстрагируют гексаном. Гексан отгоняют с последующим количественным анализом посредством газовой хроматографии.
Активность по настоящему изобретению, представленную в указанном выше п. iv), можно измерять, например, подтверждая, может ли встроенный белок по настоящему изобретению восполнять недостаточность GPAT дрожжей (S. cerevisiae). У дрожжей SCT1 и GPT2 известны как гены, участвующие в GPAT-активности, и известно, что одновременная недостаточность этих генов приводит к гибели. Т.е. дрожжи с недостаточностью и гена SCT1, и гена GPT2, как правило, не могут расти, но могут расти в случае восполнения, когда экспрессируется ген с функцией, схожей с этими генами, т.е. белок с GPAT-активностью. Рассматривая GPAT по настоящему изобретению, способ подтверждения восполнения недостаточности GPAT дрожжей может являться любым способом, с помощью которого подтверждают восстановление GPAT-активности штамма дрожжей с недостаточностью гена SCT1 и гена GPT2 посредством экспрессии гена GPAT по настоящему изобретению. Например, как в частности описывают ниже в примере 8, в гомозиготных по Δgpt2 диплоидных дрожжах получают гетерозиготный штамм с недостаточностью только одного аллеля гена SCT1. Затем, получают штамм, где одну экспрессирующую кассету гена GPAT по настоящему изобретению встраивают в гетерозиготный штамм на хромосоме, отличающейся от хромосомы, на которой присутствует SCT1, или штамм, где плазмидный вектор с экспрессирующей кассетой гена GPAT по настоящему изобретению встраивают в гетерозиготный штамм. Получаемый штамм переносят на среду для спорообразования для образования аскоспор. Получаемые клетки подвергают анализу случайной выборки спор или тетрадному анализу для получения гаплоидного штамма, получаемого из спор. Исследуют генотип получаемых таким образом гаплоидных дрожжей. Если подтверждают, что штамм Δgpt2Δsct1, который, по существу, не может расти, может расти только при наличии экспрессирующей кассеты гена GPAT по настоящему изобретению, GPAT по настоящему изобретению можно определять, как способную восполнять GPAT-активность у дрожжей.
(b) Нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий активностью по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, гибридизуемые в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующей белок, обладающий активностью по настоящему изобретению.
Такую нуклеотидную последовательность можно получать, например, из библиотеки кДНК или геномной библиотеки известным способом гибридизации, таким как гибридизация колоний, клонирование способом бляшкообразования или саузерн-блоттинг, с использованием зонда, получаемого из подходящего фрагмента способом, известным специалистам в данной области.
Подробное описание способа гибридизации можно найти в "Molecular Cloning, A Laboratory Manual 3rd ed." (Cold Spring Harbor Press (2001), в частности, Разделы 6 и 7), "Current Protocols in Molecular Biology" (John Wiley & Sons (1987-1997), в частности, Разделы 6.3 и 6.4), и "DNA Cloning 1: Core Techniques, A Practical Approach 2nd ed." (Oxford University (1995), в частности, раздел 2.10 по условиям гибридизации).
Строгость условий гибридизации определяют, главным образом, на основе условий гибридизации, более предпочтительно - на основе условий гибридизации и условий промывания. Термин "строгие условия", используемый на всем протяжении описания, предназначен для включения умеренно или очень строгих условий.
В частности, примеры умеренно строгих условий включают условия гибридизации от 1×SSC до 6×SSC при температуре от 42°C до 55°C, более предпочтительно - от 1×SSC до 3×SSC при температуре от 45°C до 50°C, и наиболее предпочтительно - 2×SSC при 50°C. В случае гибридизации применяют раствор, содержащий, например, приблизительно 50% формамида, температуру гибридизации ниже указанной выше температуры на 5°C-15°C. Условиями промывания являются, например, от 0,5×SSC до 6×SSC при температуре 40°C до 60°C. В гибридизационной раствор и промывочный раствор, как правило, можно добавлять от 0,05% до 0,2% SDS, предпочтительно - приблизительно 0,1% SDS.
Очень строгие условия включают гибридизацию и/или промывание при более высокой температуре и/или более низкой концентрации соли по сравнению с умеренно строгими условиями. Примеры условий гибридизации включают от 0,1×SSC до 2×SSC при температуре 55°C до 65°C, более предпочтительно - от 0,1×SSC до 1×SSC при температуре 60°C до 65°C, и наиболее предпочтительно 0,2×SSC при 63°C. Условиями промывания являются, например, от 0,2×SSC до 2×SSC при температуре от 50°C до 68°C и, более предпочтительно, 0,2×SSC при температуре от 60°C до 65°C.
Примеры условий гибридизации, в частности, используемых в настоящем изобретении, включают, в качестве неограничивающих примеров, прегибридизацию в 5×SSC, 1% SDS, 50 мМ Трис-HCl (pH 7,5) и 50% формамида при 42°C, инкубацию в течение ночи при 42°C в присутствии зонда для образования гибридов и трехкратное промывание в 0,2×SSC, 0,1% SDS при 65°C в течение 20 минут.
Также можно использовать коммерчески доступный набор для гибридизации без использования радиоактивного вещества в качестве зонда. В частности, например, для гибридизации используют набор для определения нуклеиновых кислот с использованием DIG (Roche Diagnostics) или систему прямого мечения и определения ECL (производства Amersham).
Предпочтительные примеры нуклеотидной последовательности по настоящему изобретению включают нуклеотидные последовательности, гибридизуемые с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, в условиях 2×SSC при 50°C и кодирующие белок, обладающий GPAT-активностью и/или GNPAT активностью.
(c) Нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий активностью по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, обладающие идентичностью по меньшей мере 70% с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующие белок, обладающий активностью по настоящему изобретению.
Предпочтительно, например, нуклеиновая кислота содержит нуклеотидную последовательность, обладающую идентичностью по меньшей мере 75%, более предпочтительно - 80% или более (например, 85% или более, более предпочтительно - 90% или более, и наиболее предпочтительно - 95%, 98%, или 99% или более) с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующую белок, обладающий активностью по настоящему изобретению.
Процентную долю идентичности между двумя нуклеотидными последовательностями можно определять визуальным осмотром и математическим вычислением, но предпочтительно определять сравнением информации о последовательности двух нуклеиновых кислот с использованием компьютерной программы. В качестве компьютерных программ можно использовать компьютерные программы для сравнения последовательностей, например, программное обеспечение BLASTN (Altschul et al., (1990), J. Mol. Biol., 215: 403-10) версии 2.2.7, доступное через веб-сайт National Library of Medicine: http://www.ncbi.nlm.nih.gov/blast/bl2seq/bls.html, или алгоритм WU-BLAST 2.0. Стандартные параметры настроек по умолчанию для WU-BLAST 2.0 описывают на следующем веб-сайте: http://blast.wustl.edu.
(d) Нуклеиновая кислота, содержащая нуклеотидную последовательность, кодирующую аминокислотную последовательность, обладающую идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующую белок, обладающий активностью по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, кодирующие аминокислотную последовательность, обладающую идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующие белок, обладающий активностью по настоящему изобретению. Белок, кодируемый нуклеиновой кислотой по настоящему изобретению, может являться любым белком, обладающим идентичностью с аминокислотной последовательностью MaGPAT4, MaGPAT4-long или MaGPAT5, при условии, что белок функционально эквивалентен белку, обладающему активностью по настоящему изобретению.
Конкретные примеры белка включают аминокислотные последовательности, обладающие идентичностью 75% или более, предпочтительно - 80% или более, более предпочтительно - 85% или более, и наиболее предпочтительно - 90% или более (например, 95% или более, кроме того, 98% или более) с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9.
Нуклеотидная последовательность, содержащаяся в нуклеиновой кислоте по настоящему изобретению предпочтительно является нуклеотидной последовательностью, кодирующей аминокислотную последовательность, обладающую идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующей белок, обладающий активностью по настоящему изобретению. Более предпочтительно, нуклеотидная последовательность, кодирующая аминокислотную последовательность, обладающую идентичностью 95% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующая белок, обладающий активностью по настоящему изобретению.
Процентную долю идентичности между двумя аминокислотными последовательностями можно определять визуальным осмотром и математическим вычислением или можно определять с использованием компьютерной программы. Примеры такой компьютерной программы включают BLAST, FASTA (Altschul et al., J. Mol. Biol., 215: 403-410, (1990)) и ClustalW. В частности, различные условия (параметры) поиска идентичности с использованием программного обеспечения BLAST описывают Altschul et al. (Nucl. Acids. Res., 25, pp. 3389-3402, 1997), и они общедоступны через веб-сайт National Center for Biotechnology Information (NCBI) США или DNA Data Bank of Japan (DDBJ) (BLAST Manual, Altschul et al., NCB/NLM/NIH Bethesda, MD 20894; Altschul et al.). Для определения процентной доли идентичности также можно использовать программное обеспечение, такое как программное обеспечение для обработки генетической информации GENETYX Ver. 7 (Genetyx Corporation), DINASIS Pro (Hitachisoft) или Vector NTI (Infomax).
Конкретная схема выравнивания для выравнивания множества аминокислотных последовательностей может демонстрировать совпадение последовательностей также в конкретной короткой области, и, таким образом, можно определять область, обладающую очень высокой идентичностью последовательности в такой короткой области, даже если полноразмерные последовательности не имеют значительного родства между собой. Кроме того, в алгоритме BLAST можно использовать матрицу аминокислотных замен BLOSUM62 и следующие параметры выбора: (A) включение фильтров для скрытия части запрашиваемой последовательности с низким композиционным уровнем сложности (как определяют посредством программного обеспечения SEG, Wootton и Federhen (Computers и Chemistry, 1993); также смотри Wootton и Federhen, 1996, "Analysis of compositionally biased regions in sequence databases", Methods Enzymol., 266: 554-71) или для скрытия сегментов с короткой периодичность внутренних повторов (как определяют посредством программного обеспечения XNU, Claverie и States (Computers и Chemistry, 1993), и (B) порог статистического значения для сообщаемых совпадений с последовательностями в базе данных, или ожидаемой вероятности совпадений, обнаруженных лишь случайно, по статистической модели E-score (Karlin и Altschul, 1990); если статистическая значимость, приписываемая совпадению, выше этого порога E-score, о совпадении не сообщают.
(e) Нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующей белок, обладающий активностью по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, гибридизуемые в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующей белок, обладающий активностью по настоящему изобретению.
Белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и условия гибридизации описывают выше. Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, гибридизуемые в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующей белок, обладающий активностью по настоящему изобретению.
(f) Нуклеиновая кислота, содержащая нуклеотидную последовательность, гибридизуемую в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающей экзоны, кодирующие белок, обладающий активностью по настоящему изобретению.
Нуклеотидные последовательности, приведенные в SEQ ID NO:7 и SEQ ID NO:12, являются последовательностями геномной ДНК, кодирующей MaGPAT4 (и MaGPAT4-long) и MaGPAT5, соответственно, по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, гибридизуемые в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включают экзоны, кодирующие белок, обладающий активностью по настоящему изобретению.
Такую нуклеотидную последовательность можно получать способом, известным специалистам в данной области, например, из геномной библиотеки известным способом гибридизации, таким как гибридизация колоний, клонирование способом бляшкообразования, или саузерн-блоттинг, с использованием зонда, получаемого с использованием подходящего фрагмента. Условия гибридизации описывают выше.
(g) Нуклеиновая кислота, содержащая нуклеотидную последовательность, состоящую из нуклеотидной последовательности, обладающей идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающую экзоны, кодирующие белок, обладающий активностью по настоящему изобретению.
Примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, обладающие идентичностью по меньшей мере 70% с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и кодирующие белок, обладающий активностью по настоящему изобретению. Предпочтительные примеры нуклеотидной последовательности включают последовательности, обладающие идентичностью по меньшей мере 75%, более предпочтительно - 80% или более (например, 85% или более, более предпочтительно - 90% или более, и наиболее предпочтительно - 95%, 98% или 99% или более) с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и включающие экзоны, кодирующие белок, обладающий активностью по настоящему изобретению. Процентная доля идентичности между двумя нуклеотидными последовательностями можно определять, как описано выше.
Последовательность геномной ДНК, приведенная в SEQ ID NO:7, состоит из десяти экзонов и девяти интронов. В SEQ ID NO:7 экзонные области соответствуют нуклеотидам с 428 по 744, или с 596 по 744, с 850 по 924, с 1302 по 1396, с 1480 по 1726, с 1854 по 2279, с 2370 по 2632, с 2724 по 3299, с 3390 по 3471, с 3575 по 4024 и с 4133 по 4248. Последовательность геномной ДНК, приведенная в SEQ ID NO:12, состоит из трех экзонов и двух интронов. В SEQ ID NO:12 экзонные области соответствуют нуклеотидам с 1 по 302, с 457 по 1676 и с 1754 по 2598.
В другом варианте осуществления примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, включающие интронные области, обладающие идентичностью нуклеотидной последовательности 100% с последовательностью геномной ДНК, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и экзонные области, обладающие идентичностью нуклеотидной последовательности по меньшей мере 70% или более, более предпочтительно, 75% или более и, более предпочтительно, 80% или более (например, 85% или более, более предпочтительно - 90% или более, и наиболее предпочтительно - 95%, 98% или 99% или более) с последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, где экзоны кодируют белок, обладающий активностью по настоящему изобретению.
В другом варианте осуществления примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, включающие экзонные области, обладающие идентичностью нуклеотидной последовательности 100% с последовательностью геномной ДНК, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и интронные области, обладающие идентичностью нуклеотидной последовательности по меньшей мере 70% или более, более предпочтительно, 75% или более и, более предпочтительно, 80% или более (например, 85% или более, более предпочтительно - 90% или более, и наиболее предпочтительно - 95%, 98% или 99% или более) с последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, где интронные области могут удаляться в процессе сплайсинга, и, таким образом, экзонные области лигируются для кодирования белка, обладающего активностью по настоящему изобретению.
В другом варианте осуществления примеры нуклеотидной последовательности, содержащейся в нуклеиновой кислоте по настоящему изобретению, включают нуклеотидные последовательности, включающие интронные области, обладающие идентичностью нуклеотидной последовательности по меньшей мере 70% или более, более предпочтительно, 75% или более и, более предпочтительно, 80% или более (например, 85% или более, более предпочтительно - 90% или более, и наиболее предпочтительно - 95%, 98% или 99% или более) с последовательностью геномной ДНК, приведенной в SEQ ID NO:7 или SEQ ID NO:12, и экзонные области, обладающие идентичностью нуклеотидной последовательности по меньшей мере 70% или более, более предпочтительно, 75% или более и, более предпочтительно, 80% или более (например, 85% или более, более предпочтительно, 90% или более и, наиболее предпочтительно, 95% или более, 98% или более, или 99% или более) с последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12, где интронные области могут удаляться в процессе сплайсинга, и, таким образом, экзонные области лигируются для кодирования белка, обладающего активностью по настоящему изобретению.
Процентную долю идентичности между двумя нуклеотидными последовательностями можно определять описываемым выше способом.
Примеры нуклеиновой кислоты по настоящему изобретению включают нуклеиновые кислоты, каждая из которых состоит из нуклеотидной последовательности с делецией, заменой или добавлением одной или нескольких нуклеотидов в нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирует белок, обладающий активностью по настоящему изобретению. Более конкретно, применимая нуклеиновая кислота включает любую из следующих нуклеотидных последовательностей:
(i) нуклеотидная последовательность с делецией одной или нескольких (предпочтительно, от одной до нескольких (например, от 1 до 720, от 1 до 600, от 1 до 500, от 1 до 400, от 1 до 300, от 1 до 250, от 1 до 200, от 1 до 150, от 1 до 100, от 1 до 50, от 1 до 30, от 1 до 25, от 1 до 20 или от 1 до 15, более предпочтительно - 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1)) нуклеотидов в нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(ii) нуклеотидная последовательность с заменой одной или нескольких (предпочтительно, от одной до нескольких (например, от 1 до 720, от 1 до 600, от 1 до 500, от 1 до 400, от 1 до 300, от 1 до 250, от 1 до 200, от 1 до 150, от 1 до 100, от 1 до 50, от 1 до 30, от 1 до 25, от 1 до 20 или от 1 до 15, более предпочтительно - 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1)) нуклеотидов в нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(iii) нуклеотидная последовательность с добавлением одной или нескольких (предпочтительно, от одной до нескольких (например, от 1 до 720, от 1 до 600, от 1 до 500, от 1 до 400, от 1 до 300, от 1 до 250, от 1 до 200, от 1 до 150, от 1 до 100, от 1 до 50, от 1 до 30, от 1 до 25, от 1 до 20 или от 1 до 15, более предпочтительно - 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1)) нуклеотидов в нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8; и
(iv) нуклеотидная последовательность с любой комбинацией из указанных выше пунктов(i)-(iii),
где нуклеотидная последовательность кодирует белок, обладающий активностью по настоящему изобретению.
Предпочтительный вариант осуществления нуклеиновой кислоты по настоящему изобретению также включает нуклеиновые кислоты, содержащие фрагмент нуклеотидной последовательности, приведенной в любом из пунктов (a)-(d) ниже:
(a) нуклеотидная последовательность, приведенная в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(b) нуклеотидная последовательность, кодирующая белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(c) нуклеотидная последовательность, приведенная в SEQ ID NO:3, SEQ ID NO:6, или SEQ ID NO:11; и
(d) нуклеотидная последовательность, приведенная в SEQ ID NO:7 или SEQ ID NO:12.
(A) Нуклеотидная последовательность, приведенная в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, (b) нуклеотидная последовательность, кодирующая белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и (c) нуклеотидная последовательность, приведенная в SEQ ID NO:3, SEQ ID NO:6 или SEQ ID NO:11, представлены в таблице 1. Нуклеотидную последовательность, приведенную в SEQ ID NO:7 или SEQ ID NO:12, также описывают выше. Фрагменты этих последовательностей включают ORF, CDS, биологически активную область, область, используемую в качестве праймера, как описано ниже, или область, которая может служит в качестве зонда, содержащиеся в этих нуклеотидных последовательностях, и могут являться природными или искусственно полученными.
Примеры нуклеиновой кислоты по настоящему изобретению включают следующие нуклеиновые кислоты.
(1) Нуклеиновые кислоты, представленные в любом из пунктов (a)-(g) ниже:
(a) нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(b) нуклеиновые кислоты, гибридизуемые в строгих условиях из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(c) нуклеиновые кислоты, содержащие нуклеотидную последовательность, обладающую идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(d) нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(e) нуклеиновые кислоты, гибридизуемые в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(f) нуклеиновые кислоты, гибридизуемые в строгих условиях с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12; и
(g) нуклеиновые кислоты, содержащие нуклеотидную последовательность, обладающую идентичностью 70% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12.
(2) Нуклеиновые кислоты, описываемые выше в п.(1), представленные ниже в любом из пунктов (a)-(g):
(a) нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(b) нуклеиновые кислоты, гибридизуемые в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(c) нуклеиновые кислоты, содержащие нуклеотидную последовательность, обладающую идентичностью 90% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(d) нуклеиновые кислоты, содержащие нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, обладающей идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(e) нуклеиновые кислоты, гибридизуемые в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(f) нуклеиновые кислоты, гибридизуемые в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, приведенной в SEQ ID NO:7 или SEQ ID NO:12; и
(g) нуклеиновые кислоты, содержащие нуклеотидную последовательность, обладающую идентичностью 90% или более с нуклеотидной последовательностью, приведенной в SEQ ID NO:7 или SEQ ID NO:12.
Глицерол-3-фосфат-ацилтрансфераза по настоящему изобретению
Примеры белка по настоящему изобретению включают белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и белки, функционально эквивалентные такому белку. Эти белки могут являться природными или искусственно полученными. Белок, состоящий из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, описывают выше. Выражение "функционально эквивалентные белки" относится к белкам, обладающим "активностью по настоящему изобретению", описываемой выше в разделе "Нуклеиновая кислота, кодирующая глицерол-3-фосфат-ацилтрансферазу по настоящему изобретению".
В настоящем изобретении, примеры белков, функционально эквивалентных белку, состоящему из аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, включают белки по любому из пунктов (a) и (b) ниже:
(a) белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий активностью по настоящему изобретению; и
(b) белок, состоящий из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий активностью по настоящему изобретению.
В приведенном выше аминокислотная последовательность с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, или аминокислотная последовательность, обладающая идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, является такой, как описано выше в разделе "Нуклеиновая кислота, кодирующая глицерол-3-фосфат-ацилтрансферазу по настоящему изобретению". "Белок, обладающий активностью по настоящему изобретению" включает мутантов белков, кодируемых нуклеиновой кислотой, содержащей нуклеотидную последовательность, приведенную в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8; мутантные белки со многими типами модификаций, такими как делеция, замена и добавление одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9; модифицированные белки, например, с модифицированными боковыми цепями аминокислот; и белки, слитные с другими белками, где эти белки обладают GPAT-активностью, GNPAT-активностью и/или активностью по п. i) или активностью по п. ii), описываемыми выше в разделе "Нуклеиновая кислота, кодирующая глицерол-3-фосфат-ацилтрансферазу по настоящему изобретению".
Белок по настоящему изобретению можно получать искусственно. В таком случае белок можно получать химическим синтезом, таким как Fmoc-способ (флуоренилметилоксикарбонильный способ) или tBoc-способ (трет-бутилоксикарбонильный способ). Кроме того, для химического синтеза можно использовать синтезатор пептидов, поставляемый Advanced ChemTech, Perkin Elmer, Pharmacia, Protein Technology Instrument, Synthecell-Vega, PerSeptive, Shimadzu Corporation или другим производителем.
Примеры белка по настоящему изобретению дополнительно включают следующие белки.
(1) Белки по любому из пунктов (a) и (b) ниже:
(a) белки, состоящие из аминокислотной последовательности с делецией, заменой или добавлением одной или нескольких аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9; и
(b) белки, состоящие из аминокислотной последовательности, обладающей идентичностью 70% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9.
(2) Белки, описываемые выше в п.(1), представленные ниже в любом из пунктов (a) и (b):
(a) белки, состоящие из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 80 аминокислот в аминокислотной последовательности, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9; и
(b) белки, состоящие из аминокислотной последовательности, обладающей идентичностью 90% или более с аминокислотной последовательностью, приведенной в SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9.
Клонирование нуклеиновой кислоты по настоящему изобретению
Нуклеиновую кислоту GPAT по настоящему изобретению можно клонировать, например, скринингом библиотеки кДНК с использованием подходящего зонда. Клонирование можно осуществлять посредством ПЦР-амплификации с использованием подходящих праймеров и последующего лигирования с подходящим вектором. Клонированную нуклеиновую кислоту можно дополнительно субклонировать в другой вектор.
Можно использовать коммерчески доступные плазмидные векторы, такие как pBlue-ScriptTM SK(+) (Stratagene), pGEM-T (Promega), pAmp (TM: Gibco-BRL), p-Direct (Clontech) или pCR2.1-TOPO (Invitrogen). При ПЦР-амплификации праймер может представлять собой любую область, например, нуклеотидной последовательности, приведенной в SEQ ID NO:3, 6 или 11. Например, можно использовать, праймеры описываемые ниже в примерах. Затем, осуществляют ПЦР с использованием кДНК, полученной из клеток M. alpina, с использованием указанных выше праймеров, ДНК-полимеразы и любого другого вещества. Хотя специалисты в данной области легко могут осуществлять этот способ, например, в соответствие с "Molecular Cloning, A Laboratory Manual 3rd ed." (Cold Spring Harbor Press (2001)), условия ПЦР в настоящем изобретении могут являться, например, следующими:
Температура денатурации: 90°C до 95°C,
Температура отжига: 40°C до 60°C,
Температура элонгации: 60°C до 75°C, и
Количество циклов: 10 или более циклов.
Получаемый продукт ПЦР можно осуществлять известным способом, например, с использованием набора, такого как GENECLEAN kit (Funakoshi Co., Ltd.), QIAquick PCR purification (QIAGEN) или ExoSAP-IT (GE Healthcare Bio-Sciences); DEAE-целлюлозного фильтра или диализной трубки. В случае использования агарозного геля продукт ПЦР подвергают электрофорезу в агарозном геле, и фрагменты нуклеотидной последовательности вырезают из агарозного геля и очищают, например, набором GENECLEAN (Funakoshi Co., Ltd.) или QIAquick Gel extraction kit (QIAGEN) или способом замораживания-сдавливания.
Нуклеотидную последовательность клонированной нуклеиновой кислоты можно определять с использованием секвенатора.
Конструирование вектора для экспрессии GPAT и получения трансформанта
Настоящее изобретение также относится к рекомбинантному вектору, содержащему нуклеиновую кислоту, кодирующую GPAT по настоящему изобретению. Настоящее изобретение дополнительно относится к трансформанту, трансформированному таким рекомбинантным вектором.
Рекомбинантный вектор и трансформант можно получать следующим образом: плазмиду, содержащую нуклеиновую кислоту, кодирующую GPAT по настоящему изобретению, расщепляют ферментом рестрикции. Примеры ферментов рестрикции включают, в качестве неограничивающих примеров, EcoRI, KpnI, BamHI и SalI. Конец можно затуплять T4-полимеразой. Расщепленный фрагмент ДНК очищают посредством электрофореза в агарозном геле. Этот фрагмент ДНК встраивают в экспрессирующий вектор известным способом для получения вектора экспрессии GPAT. Этот экспрессирующий вектор встраивают в хозяина для получения трансформанта, предназначенного для экспрессии желаемого белка.
В этом случае экспрессирующий вектор и хозяин могут принадлежать любому типу, позволяющему экспрессировать желаемый белок. Примеры хозяина включают грибы, бактерии, растения, животных и их клетки. Примеры грибов включают нитевидные грибы, такие как продуцирующий липиды M. alpina, и штаммы дрожжей, такие как Saccharomyces cerevisiae. Примеры бактерий включают Escherichia coli и Bacillus subtilis. Примеры растений включают масличные растения, такие как рапс, соя, хлопок, сафлор и лен.
В качестве продуцирующих липиды можно использовать микроорганизмы, например, штаммов, описываемых в MYCOTAXON, Vol. XLIV, NO. 2, pp. 257-265 (1992), и их конкретные примеры включают микроорганизмы, принадлежащие роду Mortierella, такие как микроорганизмы, принадлежащие подроду Mortierella, например, Mortierella elongata IFO8570, Mortierella exigua IFO8571, Mortierella hygrophila IFO5941, Mortierella alpina IFO8568, ATCC16266, ATCC32221, ATCC42430, CBS219.35, CBS224.37, CBS250.53, CBS343.66, CBS527.72, CBS528.72, CBS529.72, CBS608.70, и CBS754.68; и микроорганизмы, принадлежащие подроду Micromucor, например, Mortierella isabellina CBS194.28, IFO6336, IFO7824, IFO7873, IFO7874, IFO8286, IFO8308, IFO7884, Mortierella nana IFO8190, Mortierella ramanniana IFO5426, IFO8186, CBS112.08, CBS212.72, IFO7825, IFO8184, IFO8185, IFO8287 и Mortierella vinacea CBS236.82. В частности, Mortierella alpina является предпочтительным.
При использовании гриба в качестве хозяина нуклеиновая кислота по настоящему изобретению предпочтительно является самореплицируемой в хозяине или предпочтительно обладает структурой, встраиваемой в хромосому гриба. Предпочтительно, нуклеиновая кислота также включает промотор и терминатор. При использовании M. alpina в качестве хозяина в качестве экспрессирующего вектора можно использовать, например, pD4, pDuraSC или pDura5. Можно использовать любой промотор, делающий возможной экспрессию в хозяине, и его примеры включают промоторы, полученные из M. alpina, такие как промотор гена гистона H4.1, промотор гена GAPDH (глицеральдегид-3-фосфат-дегидрогеназы) и промотор гена TEF (фактор элонгации трансляции).
Примеры способа встраивания рекомбинантного вектора в нитевидные грибы, такие как M. alpina, включают электропорацию, сферопластный способ, способ доставки частиц и прямую микроинъекцию ДНК в ядра. В случае использования ауксотрофного штамма хозяина трансформированный штамм можно получать селекцией штамма, вырастающего на селективной среде без конкретного питательного вещества(веществ). Альтернативно, при трансформации с использованием маркерного гена устойчивости к лекарственному средству колонию устойчивых к лекарственному средству клеток можно получать культивированием клеток-хозяев на селективной среде, содержащей лекарственное средство.
При использовании дрожжей в качестве хозяина в качестве экспрессирующего вектора можно использовать, например, pYE22m. Альтернативно, можно использовать коммерчески доступные дрожжи, экспрессирующие векторы, такие как pYES (Invitrogen) или pESC (Stratagene). Примеры хозяина, подходящего для настоящего изобретения, включают, в качестве неограничивающих примеров, Saccharomyces cerevisiae штамм EH13-15 (trp1, MATα). Промотором, который можно использовать, является, например, полученный из дрожжей промотор, такой как промотор GAPDH, промотор gal1 или промотор gal10.
Примеры способа встраивания рекомбинантного вектора в дрожжи включают способ с использованием ацетата лития, электропорацию, сферопластный способ, декстран-опосредованную трансфекцию, осаждение фосфатом кальция, полибрен-опосредованную трансфекцию, слияние протопластов, инкапсуляцию полинуклеотида(полинуклеотидов) в липосомах и прямую микроинъекцию ДНК в ядра.
При применении бактерии, такой как E. coli, в качестве хозяина в качестве экспрессирующего вектора можно использовать, например, pGEX или pUC18, поставляемые Pharmacia. Промотор, который можно использовать, включает промотор, полученный из, например, E. coli или фага, такой как промотор trp, промотор lac, промотор PL и промотор PR. Примеры способа встраивания рекомбинантного вектора в бактерии включают электропорацию и способ с использованием хлорида кальция.
Способ получения композиции жирных кислот по настоящему изобретению
Настоящее изобретение относится к способу получения композиции жирных кислот из описываемого выше трансформанта, т.е. способу получения композиции жирных кислот из культивируемого продукта, получаемого культивированием трансформанта. Композиция жирных кислот содержит смесь одной или нескольких жирных кислот. Жирные кислоты могут являться свободными жирными кислотами или могут присутствовать в форме липидов, содержащих жирные кислоты, такие как триглицерид или фосфолипид. В частности, композицию жирных кислот по настоящему изобретению можно получать следующим способом. Альтернативно, композицию жирных кислот также можно получать любым другим известным способом.
Средой, используемой для культивирования организма, экспрессирующего GPAT, может являться любой культуральный раствор (среда) с подходящим pH и осмотическим давлением, и она может содержать любые биологические материалы, такие как питательные вещества, необходимые для роста каждого хозяина, микроэлементы, сыворотку и антибиотики. Например, в случае экспрессии GPAT трансформированными дрожжами неограничивающие примеры среды включают среду SC-Trp, среду YPD и среду YPD5. Композиция конкретной среды, например, среды SC-Trp, является следующей: один литр среды включает 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г глюкозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденина, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 1,8 г лейцина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина и 0,6 г урацила).
Можно использовать любые условия культивирования, подходящие для роста хозяина и для стабильного сохранения получаемого фермента. В частности, можно регулировать отдельные условия, включая степень аэробности, время культивирования, температуру, влажность, и можно получать статическую культуру или культивирование с использованием перемешивания. Культивирование можно осуществлять в одних и тех же условиях (одноступенчатое культивирование) или посредством, так называемого, двухступенчатого или трехступенчатого культивирования с использованием двух или более различных условий культивирования. Для крупномасштабного культивирования предпочтительным является культивирование в два или более этапов по причине высокой эффективности такого культивирования.
При двухступенчатом культивировании с использованием дрожжей в качестве хозяина композицию жирных кислот по настоящему изобретению можно получать следующим образом: в качестве прекультуры колонию трансформанта инокулируют, например, в среде SC-Trp и культивируют при перемешивании при 30°C в течение 2 дней. Затем, в качестве основной культуры 500 мкл раствора прекультуры добавляют к 10 мл среды YPD5 (2% дрожжевого экстракта, 1% полипептона и 5% глюкозы) с последующим культивированием при перемешивании при 30°C в течение 2 дней.
Композиция жирных кислот по настоящему изобретению
Настоящее изобретение также относится к композиции жирных кислот в качестве смеси одной или нескольких жирных кислот в клетках, экспрессирующих GPAT по настоящему изобретению, предпочтительно, композиции жирных кислот, получаемой культивированием трансформанта, экспрессирующего GPAT по настоящему изобретению. Жирные кислоты могут являться свободными жирными кислотами или могут присутствовать в форме липидов, содержащих жирные кислоты, таких как триглицерид или фосфолипид.
Жирные кислоты, содержащиеся в композиции жирных кислот по настоящему изобретению, являются линейными или разветвленными монокарбоновыми кислотами длинноцепочечных углеводов, и их примеры включают, в качестве неограничивающих примеров, миристиновую кислоту (тетрадекановую кислоту) (14:0), миристолеиновую кислоту (тетрадеценовую кислоту) (14:1), пальмитиновую кислоту (гексадекановую кислоту) (16:0), пальмитолеиновую кислоту (9-гексадеценовую кислоту) (16:1), стеариновую кислоту (октадекановую кислоту) (18:0), олеиновую кислоту (цис-9-октадеценовую кислоту) (18:1(9)), вакценовую кислоту (11-октадеценовую кислоту) (18:1(11)), линолевую кислоту (цис,цис-9,12-октадекадиеновую кислоту) (18:2(9,12)), α-линоленовую кислоту (9,12,15-октадекатриеновую кислоту) (18:3(9,12,15)), γ-линоленовую кислоту (6,9,12- октадекатриеновую кислоту) (18:3(6,9,12)), стеаридоновую кислоту (6,9,12,15-октадекатетраеновую кислоту) (18:4(6,9,12,15)), арахидовую кислоту (эйкозановую кислоту) (20:0), (8,11-эйкозадиеновую кислоту) (20:2(8,11)), mead acid (5,8,11-эйкозатриеновую кислоту) (20:3(5,8,11)), дигомо-γ-линоленовую кислоту (8,11,14-эйкозатриеновую кислоту) (20:3(8,11,14)), арахидоновую кислоту (5,8,11,14-эйкозатетраеновую кислоту) (20:4(5,8,11,14)), эйкозатетраеновую кислоту (8,11,14,17-эйкозатетраеновую кислоту) (20:4(8,11,14,17)), эйкозапентаеновую кислоту (5,8,11,14,17-эйкозапентаеновую кислоту) (20:5(5,8,11,14,17)), бегеновую кислоту (докозановую кислоту) (22:0), (7,10,13,16-докозатетраеновую кислоту) (22:4(7,10,13,16)), (7,10,13,16,19-докозапентаеновую кислоту) (22:5(7,10,13,16,19)), (4,7,10,13,16-докозапентаеновую кислоту) (22:5(4,7,10,13,16)), (4,7,10,13,16,19-докозагексаеновую кислоту) (22:6(4,7,10,13,16,19)), лигноцериновую кислоту (тетрадокозановую кислоту) (24:0), ацетэруковую кислоту (цис-15-тетрадокозановую кислоту) (24:1) и церотовую кислоту (гексадокозановую кислоту) (26:0). Необходимо отметить, что названия веществ являются общепринятыми названиями, определенными IUPAC Biochemical Nomenclature, и их систематические названия даны в скобках вместе с числами, обозначающими номера атомов углерода и положения двойных связей.
Композиция жирных кислот по настоящему изобретению может состоять из любого количества и любого типа жирных кислот при условии, что она является комбинацией одной или нескольких жирных кислот, выбранных из указанных выше жирных кислот.
Доли жирных кислоты в композиции жирных кислот по настоящему изобретению можно определять способом определения композиционного отношения или долей жирных кислот, описываемым в разделе "Нуклеиновая кислота, кодирующая глицерол-3-фосфат-ацилтрансферазу по настоящему изобретению" описания.
Пища или другие продукты, содержащие композицию жирных кислот по настоящему изобретению
Настоящее изобретение также относится к пищевому продукту, содержащему описываемую выше композицию жирных кислот. Композицию жирных кислот по настоящему изобретению можно использовать, например, для получения пищевых продуктов, содержащих жиры и масла, и получения промышленного сырья (например, сырья для косметики, лекарственных средств (например, для наружного применения на коже) и мыла) в общепринятых способах. Косметические средства (косметические композиции) или лекарственные средства (фармацевтические композиции) можно составлять в любой лекарственной форме, включая, в качестве неограничивающих примеров, растворы, пасты, гели, твердые вещества и порошки. Примеры форм пищевых продуктов включают фармацевтические составы, такие как капсулы; натуральную жидкую пищу, полуперевариваемую пищу, богатую питательными веществами, и элементарную пищу, богатую питательными веществами, где композицию жирных кислот по настоящему изобретению смешивают с белками, сахарами, жирами, микроэлементами, витаминами, эмульгаторами и ароматизаторами; и обработанные формы, такие как питьевые продукты и энтеральное питание.
Кроме того, примеры пищевого продукта по настоящему изобретению включают, в качестве неограничивающих примеров, питательные добавки, диетическую пищу, функциональную пищу, пищу для детей, модифицированное молоко для новорожденных, модифицированное молоко для недоношенных детей и пищу для пожилых людей. На всем протяжении описания термин "пища" используют как обобщенный термин для съедобных материалов в твердой, жидкой форме или их смеси.
Термин "питательные добавки" относится к пищевым продуктам, обогащенным конкретными питательными ингредиентами. Термин "диетическая пища" относится к пищевым продуктам, оздоровляющим или полезным для здоровья, и включает питательные добавки, натуральную пищу и диетическую пищу. Термин "функциональная пища" относится к пищевым продуктам для обеспечения питательными ингредиентами, помогающими организму контролировать его функции, и является синонимом пищи для специального лечебного использования. Термин "пища для детей" относится к пищевым продуктам, которые дают детям возрастом приблизительно до 6 лет. Термин "пища для пожилых" относится к пищевым продуктам, обработанным для облегчения их переваривания и абсобции по сравнению с необработанной пищей. Термин "модифицированное молоко для новорожденных" относится к модифицированному молоку, которое дают детям возрастом приблизительно до одного года. Термин "модифицированное молоко для недоношенных детей" относится к модифицированному молоку, которое дают недоношенным детям приблизительно до 6 месяцев после рождения.
Примеры этих пищевых продуктов включают натуральную пищу (обработанную жирами и маслами), такую как мясо, рыба и орехи; пищу, дополненную жирами и маслами при приготовлении, такую как китайская пища, китайская лапша и супы; пищевые продукты, приготовленные с использованием жиров и масел в качестве теплоносителя, такие как темпура (жареные во фритюре рыба и овощи), жареная во фритюре пища, жареный тофу, китайский жареный рис, пончики и японские жареные печенья (каринто)); пищу на основе жира и масла или обработанную пищу, дополненную жирами или маслами при приготовлении, такую как сливочное масло, маргарин, майонез, соусы, шоколад, лапша быстрого приготовления, карамель, печенье, пироги и мороженое; и пищу, опрыснутую или покрытую жирами или маслами после приготовления, такую как рисовое печенье, твердое печенье и пирожки с пастой из красных бобов. Однако, пищевые продукты по настоящему изобретению не ограничены пищей, содержащей жиры и масла, и их другие примеры включают сельскохозяйственные пищевые продукты, такие как хлебобулочные изделия, лапша, вареный рис, сладости (например, конфеты, жевательную резинку, таблетки, японские сладости), тофу и продукты их обработки; ферментированные пищевые продукты, такие как очищенное саке, медицинский ликер, выдержанный ликер (мирин), уксус, соевый соус и мисо; пищевые продукты животноводства, такие как йогурт, ветчина, бекон и сосиски; морепродукты, такие как рыбная паста (камабоко), жареная во фритюре рыбная паста (агетэн) и рыбный пирог (хантен); и фруктовые напитки, безалкогольные напитки, спортивные напитки, алкогольные напитки и чай.
Способ оценки и выбора штамма с использованием кодирующей GPAT нуклеиновой кислоты или белка GPAT по настоящему изобретению
Настоящее изобретение также относится к способу оценки и выбора продуцирующего липиды гриба с использованием кодирующей GPAT нуклеиновой кислоты или белка GPAT по настоящему изобретению. Подробности приведены ниже.
(1) Способ оценки
Одним из вариантов осуществления настоящего изобретения является способ оценки продуцирующего липиды гриба с использованием кодирующей GPAT нуклеиновой кислоты или белка GPAT по настоящему изобретению. В способе оценки по настоящему изобретению, например, штамм продуцирующего липиды гриба в качестве тестового штамма оценивают по активности по настоящему изобретению с использованием праймеров или зондов, сконструированных на основе нуклеотидной последовательности по настоящему изобретению. Такую оценку можно осуществлять известными способами, например, описываемыми в международной публикации № WO01/040514 и JP-A-8-205900. Способ оценки в кратком изложении описывают ниже.
Первым этапом является получение генома тестового штамма. Геном можно получать любым известным способом, таким как способ Херефорда или способ с использованием ацетата калия (смотри, например, Methods in Yeast Genetics, Cold Spring Harbor Laboratory Press, p. 130 (1990)).
Праймеры или зонды конструируют на основе нуклеотидной последовательности по настоящему изобретению, предпочтительно, последовательности, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8. Эти праймеры или зонды могут представлять собой любые области нуклеотидной последовательности по настоящему изобретению, и их можно конструировать известным способом. Количество нуклеотидов в полинуклеотиде, используемом в качестве праймера, как правило, составляет 10 или более, предпочтительно - от 15 до 25. Количество нуклеотидов, подходящих для фланкируемой праймерами области, как правило, составляет от 300 до 2000.
Полученные выше праймеры или зонды используют для исследования того, содержит ли геном тестового штамма последовательность, специфичную для нуклеотидной последовательности по настоящему изобретению или нет. Последовательность, специфичную для нуклеотидной последовательности по настоящему изобретению, можно определять известным способом. Например, полинуклеотид, содержащий часть или всю последовательность, специфичную для нуклеотидной последовательности по настоящему изобретению, или полинуклеотид, содержащий нуклеотидную последовательность, комплементарную нуклеотидной последовательности, используют в качестве одного праймера, и полинуклеотид, содержащий часть или всю последовательность, локализованную выше или ниже этой последовательности, или полинуклеотид, содержащий нуклеотидную последовательность, комплементарную нуклеотидной последовательности, используют в качестве другого праймера, и нуклеиновую кислоту из тестового штамма амплифицируют посредством ПЦР или других способов. Кроме того, например, можно определять наличие или отсутствие продукта амплификации и молекулярную массу продукта амплификации.
Условия ПЦР, подходящие для способа по настоящему изобретению, в частности, не ограничены и могут являться, например, следующими:
Температура денатурации: от 90°C до 95°C
Температура отжига: от 40°C до 60°C
Температура элонгации: от 60°C до 75°C
Количество циклов: 10 или более циклов.
Для определения молекулярной массы продукта амплификации получаемые продукты реакции можно разделять электрофорезом в агарозном геле или любым другим способом. Можно прогнозировать или оценивать активность по настоящему изобретению у тестового штамма, подтверждая, является ли молекулярная масса продукта амплификации достаточной для перекрывания молекулы нуклеиновой кислоты, соответствующей области, специфичной для нуклеотидной последовательности по настоящему изобретению. Кроме того, активность по настоящему изобретению можно прогнозировать или оценивать с высокой точностью, анализируя нуклеотидную последовательность продукта амплификации описываемым выше способом. Способ оценки активности по настоящему изобретению описан выше.
Альтернативно, при оценке по настоящему изобретению активность по настоящему изобретению у тестового штамма можно оценивать культивированием тестового штамма и измерением уровня экспрессии GPAT, кодируемой нуклеотидной последовательностью по настоящему изобретению, например, последовательностью, приведенной в SEQ ID NO:1 или SEQ ID NO:6. Уровень экспрессии GPAT можно измерять культивированием тестового штамма в подходящих условиях и количественным анализом мРНК или белка GPAT. мРНК или белок можно определять количественно известным способом. Например, мРНК можно определять количественно нозерн-гибридизацией или количественной ПЦР в реальном времени и белок можно определять количественно вестерн-блоттингом (Current Protocols in Molecular biology, John Wiley & Sons, 1994-2003).
(2) Способ выбора
Другим вариантом осуществления настоящего изобретения является способ выбора продуцирующего липиды гриба с использованием кодирующей GPAT нуклеиновой кислоты или белка GPAT по настоящему изобретению. При выборе по настоящему изобретению штамм с желаемой активностью можно выбирать культивированием тестового штамма, измерением уровня экспрессии GPAT, кодируемой нуклеотидной последовательностью по настоящему изобретению, например, последовательностью, приведенной в SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и выбора штамма с желаемым уровнем экспрессии. Альтернативно, желаемый штамм можно выбирать, получая стандартный штамм, культивируя стандартный штамм и тестовый штамм раздельно, измеряя уровень экспрессии каждого штамма и сравнивая уровень экспрессии стандартного штамма с уровнем экспрессии тестового штамма. В частности, например, стандартный штамм и тестовые штаммы культивируют в подходящих условиях и измеряют уровень экспрессии каждого штамма. Можно выбирать штамм, проявляющий желаемую активность, выбирая тестовый штамм, демонстрирующий более высокую или более низкую экспрессию, чем стандартный штамм. Желаемую активность можно определять, например, измерением уровня экспрессии GPAT и композиции жирных кислот, продуцируемой GPAT, как описано выше.
При выборе по настоящему изобретению тестовый штамм с желаемой активностью также можно выбирать культивированием тестовых штаммов и выбором штамма с высокой или низкой активностью по настоящему изобретению. Желаемую активность можно определять, например, измерением уровня экспрессии GPAT и композиции жирных кислот, продуцируемой GPAT, как описано выше.
Примеры тестового штамма и стандартного штамма включают, в качестве неограничивающих примеров, штаммы, трансформированные с использованием вектора по настоящему изобретению, штаммы, модифицированные для супрессии экспрессии нуклеиновых кислот по настоящему изобретению, подвергнутые мутагенезу штаммы и мутировавшие естественным образом штаммы. Активность по настоящему изобретению можно измерять, например, способом, описываемым в разделе "Нуклеиновая кислота, кодирующая глицерол-3-фосфат-ацилтрансферазу по настоящему изобретению" описания. Примеры способов мутагенеза включают, в качестве неограничивающих примеров, физические способы, такие как облучение ультрафиолетом или радиацией; и химические способы обработки химическими веществами, такими как EMS (этилметан сульфонат) или N-метил-N-нитрозогуанидин (смотри, например, Yasuji Oshima ed., Biochemistry Experiments vol. 39, Experimental Protocols for Yeast Molecular Genetics, pp. 67-75, Japan Scientific Societies Press).
Примеры штамма, используемого в качестве стандартного штамма по настоящему изобретению или тестового штамма, включают, в качестве неограничивающих примеров, описываемые выше продуцирующий липиды гриб и дрожжи. В частности, стандартный штамм и тестовый штамм могут являться любой комбинацией штаммов, принадлежащих различным родам или видам, и можно одновременно использовать один или несколько тестовых штаммов.
Настоящее изобретение более подробно описано ниже в следующих примерах, непредназначенных для ограничения объема изобретения.
ПРИМЕРЫ
Пример 1: Анализ генома Mortierella alpina
M. alpina штамма 1S-4 инокулировали в 100 мл среды GY2:1 (2% глюкозы, 1% дрожжевого экстракта, pH 6,0) и культивировали при перемешивании при 28°C в течение 2 дней. Клетки собирали фильтрацией и получали геномную ДНК с использованием DNeasy (QIAGEN).
Нуклеотидную последовательность геномной ДНК определяли с использованием Roche 454 GS FLX Standard. В этом случае нуклеотид из библиотеки фрагментов секвенировали в два этапа, и нуклеотид из библиотеки "mate pair" секвенировали в три этапа. Получаемые нуклеотидные последовательности собирали для получения 300 суперконтигов.
Пример 2: Синтез кДНК и конструирование библиотеки кДНК
M. alpina штамма 1S-4 инокулировали в 4 мл среды (2% глюкозы, 1% дрожжевого экстракта, pH 6,0) и культивировали при 28°C в течение 4 дней. Клетки собирали фильтрацией и выделяли РНК набором RNeasy для растений (QIAGEN). Комплементарную ДНК синтезировали с использованием системы SuperScript First-Strand для ПЦР с обратной транскрипцией (Invitrogen). Кроме того, из тотальной РНК выделяли поли(A)+ РНК с использованием набора для очистки Oligotex-dT30<Super>mRNA (Takara Bio Inc.). Библиотеку кДНК конструировали с использованием набора для клонирования ZAP-cDNA Gigapack III Gold (Stratagene).
Пример 3: Поиск гомолога GPAT
Аминокислотную последовательность (инвентарный номер GenBank BAE78043) plsB, GPAT, полученной из Escherichia coli (E. coli), подвергали поиску с использованием tblastn на наличие геномных нуклеотидных последовательностей M. alpina штамма 1S-4. В результате суперконтиг, включающий последовательность, приведенную в SEQ ID NO:7, демонстрировал совпадение. Аминокислотную последовательность GPAT, полученной из дрожжей (S. cerevisiae), SCT1 (YBL011W) или GPT2 (YKR067W), подвергали поиску с использованием tblastn на наличие геномных нуклеотидных последовательностей M. alpina штамма 1S-4. В результате суперконтиги, включающие последовательность, приведенную в SEQ ID NO:12, SEQ ID NO:13, SEQ ID NO:14 или SEQ ID NO:15, демонстрировали совпадение.
SEQ ID NO:13 являлась геномной последовательностью MaGPAT1, и SEQ ID NO:15 являлась геномной последовательностью MaGPAT2 (WO2008/156026). SEQ ID NO:14 являлась геномной последовательностью MaGPAT3, отдельно идентифицированной авторами настоящего изобретения ранее (данные не опубликованы на момент подачи настоящей заявки).
Гены, относящиеся к SEQ ID NO:7 и SEQ ID NO:12, по-видимому являются новыми. Ген, относящийся к SEQ ID NO:7, называли MaGPAT4, и ген, относящийся к SEQ ID NO:12, называли MaGPAT5.
Пример 4: Клонирование MaGPAT4 и MaGPAT5
(1) Клонирование кДНК MaGPAT4
Для клонирования кДНК MaGPAT4 получали следующий праймер.
Нуклеотидную последовательность суперконтига, содержащего последовательность, приведенную в SEQ ID NO:4, подвергали анализу с использованием BLAST и сравнивали с известным гомологом GPAT. Результат позволил предполагать, что TAA в положениях 4246-4248 в последовательности, приведенной в SEQ ID NO:4, являлся стоп-кодоном. Положение инициаторного кодона было тяжело прогнозировать из сравнения известных гомологов. Таким образом, предпринимали попытку клонирования 5'-конца кДНК способом 5'-RACE. Т.е. кДНК на 5'-стороне выше следующего праймера:
Праймер GPAT4-S: 5'-CAAGGATGTTGTTGATGAGGAAGGCGAAG-3' (SEQ ID NO:16) клонировали с использованием праймера в качестве 5'-геноспецифичного праймера и набора Gene Racer (Invitrogen). В результате получали последовательность, содержащую нуклеотидную последовательность в положениях 93-595 SEQ ID NO:3. Однако, эта последовательность не содержала последовательность, прогнозируемую в качестве инициаторного кодона, и сравнение с другими гомологами GPAT также позволяло предполагать, что последовательность не содержит инициаторный кодон MaGPAT4. Таким образом, геномные последовательности, последовательность, приведенную в SEQ ID NO:7, и последовательность в положениях 93-595 SEQ ID NO:3, сравнивали друг с другом и подробно исследовали область выше 5', где обе последовательности совпадали друг с другом. В результате обнаруживали ATG, который может служить инициаторным кодоном, в двух положениях, 596-598 и 428-430, ниже стоп-кодона, впервые появившегося в геномной последовательности в рамке, вероятно, кодирующей MaGPAT4. Для клонирования CDS MaGPAT4 следующие праймеры получали с использованием ATG в положениях 596-598 в качестве стартового кодона:
Праймер SacI-GPAT4-1: 5'-GAGCTCATGCCCATCGTTCCAGCTCAGC-3' (SEQ ID NO:17), и
Праймер Sal-GPAT4-2: 5'-GTCGACTTATAATTTCGGGGCGCCATCGC-3' (SEQ ID NO:18).
ПЦР осуществляли с ExTaq (Takara Bio Inc.) с использованием кДНК в качестве матрицы и комбинации праймера SacI-GPAT4-1 и праймера Sal-GPAT4-2 при 94°C в течение 2 мин и затем 30 циклов (94°C в течение 1 мин, 55°C в течение 1 мин и 72°C в течение 1 мин). Амплифицированный фрагмент ДНК приблизительно 2,5 т.п.н. клонировали с использованием набора для клонирования TOPO-TA (Invitrogen). Определяли нуклеотидную последовательность вставленной области и плазмиду с нуклеотидной последовательностью, приведенной в SEQ ID NO:3, называли pCR-MaGPAT4. Нуклеотидная последовательность CDS, кодирующего MaGPAT4, представлена в SEQ ID NO:3, нуклеотидная последовательность ORF представлена в SEQ ID NO:1, и аминокислотная последовательность MaGPAT4, предсказанная из этих нуклеотидных последовательностей, представлена в SEQ ID NO:2.
В случае использования ATG в положениях 428-430 в качестве инициаторного кодона последовательность определяли как MaGPAT4-long. Нуклеотидная последовательность CDS, кодирующего MaGPAT4-long, представлена в SEQ ID NO:6, нуклеотидная последовательность ORF представлена в SEQ ID NO:4, и аминокислотная последовательность MaGPAT4-long, предсказанная из этих нуклеотидных последовательностей, представлена в SEQ ID NO:5.
(2) Клонирование кДНК MaGPAT5
Для клонирования кДНК MaGPAT5 получали следующие праймеры:
Праймер GPAT5-1F: 5'-TTCCCTGAAGGCGTATCGAGCGACGATT-3' (SEQ ID NO:19), и
Праймер GPAT5-3R: 5'-CAAATGTTGACAGCAGCCAGAACG-3' (SEQ ID NO:20).
ПЦР осуществляли с ExTaq (Takara Bio Inc.) с использованием сконструированной таким образом библиотеки в качестве матрицы и комбинации праймера GPAT5-1F и праймера GPAT5-3R при 94°C в течение 2 мин и затем 30 циклов (94°C в течение 1 мин, 55°C в течение 1 мин и 72°C в течение 1 мин). Получаемый фрагмент ДНК приблизительно 0,9 т.п.н. клонировали с использованием набора для клонирования TOPO-TA (Invitrogen). Определяли нуклеотидную последовательность вставленной области и плазмиду с нуклеотидной последовательностью положений 1503-2385 SEQ ID NO:8 называли pCR-MaGPAT5-P.
Затем посредством ПЦР с использованием этих плазмид в качестве матриц и указанных выше праймеров получали зонды. В реакции использовали ExTaq (Takara Bio Inc., Japan), за исключением того, что для получения зондов MaGPAT5 вместо смеси меченых дигоксигенином (DIG) dNTP для мечения ДНК, подлежащей амплификации, использовали смесь для мечения при ПЦР (Roche Diagnostics). Библиотеку кДНК подвергали скринингу с этими зондами.
Условия гибридизации являлись следующими:
Буфер: 5×SSC, 1% SDS, 50 мМ Трис-HCl (pH 7,5), 50% формамида,
Температура: 42°C (в течение ночи), и
Условия промывания: 0,2×SSC, в 0,1% растворе SDS (65°C) в течение 20 мин (три раза).
Для определения использовали набор для определения нуклеиновых кислот с DIG (Roche Diagnostics). Для получения каждой плазмидной ДНК плазмиды вырезали эксцизией in vivo из клонов фага, получаемых при скрининге. Плазмида с наибольшей вставкой среди плазмид, получаемых при скрининге с использованием зонда MaGPAT5, включала нуклеотидную последовательность, приведенную в SEQ ID NO:11, и ее называли плазмида pB-MaGPAT5. Проводили поиск ORF в нуклеотидной последовательности, приведенной в SEQ ID NO:11. В результате обнаруживали CDS с иницаторным кодом в положениях 225-227 SEQ ID NO:11 и стоп-кодоном в положениях 2589-2591 SEQ ID NO:11. Результат поиска аминокислотной последовательности, предсказанной по этой последовательности, с использованием blastp и другая информация позволяли предполагать, что этот CDS кодирует MaGPAT5. CDS гена, кодирующего MaGPAT5, представлен в SEQ ID NO:10, ORF представлена в SEQ ID NO:8, и аминокислотная последовательность MaGPAT5, предсказанная из этих нуклеотидных последовательностей, представлена в SEQ ID NO:9.
(3) Анализ последовательности
Геномную последовательность (SEQ ID NO:7) и последовательность CDS (SEQ ID NO:3) гена MaGPAT4 сравнивали друг с другом. Результат позволял предполагать, что геномная последовательность этого гена состоит из десяти экзонов и девяти интронов и кодирует белок, состоящий из 825 аминокислотных остатков (фиг.1 и 2). Сравнение геномной последовательности (SEQ ID NO:12) и последовательности CDS (SEQ ID NO:10) гена MaGPAT5 позволяла предполагать, что геномная последовательность этого гена состоит из трех экзонов и двух интронов и кодирует белок, состоящий из 788 аминокислотных остатков (фиг.3 и 4).
MaGPAT4 и MaGPAT5 сравнивали с известным гомологом GPAT, полученным из Mortierella alpina. В таблицах 2 и 3 показаны последовательности CDS и идентичность аминокислотных последовательностей, предсказанных из последовательностей CDS.
Предсказанную аминокислотную последовательность (SEQ ID NO:2) MaGPAT4 подвергали анализу гомологии с аминокислотными последовательностями, зарегистрированными в GenBank nr, с использованием BLASTp. Аминокислотной последовательностью, показавшей наименьшую величину E при сравнении с этой последовательностью, т.е. обладающей наибольшей идентичностью, являлась аминокислотная последовательность (инвентарный номер GenBank XP_001224211) предсказанного белка, полученного из аскомицета Chaetomium globosum CBS 148.51, и его идентичность составляла 39,3%. Аминокислотная последовательность также обладала гомологией с глицеронфосфат-O-ацилтрансферазой (GNPAT), полученной из животного, и идентичность с GNPAT человека (инвентарный номер GenBank AAH00450) составляла 22,6%. Идентичность аминокислотной последовательности (инвентарный номер GenBank BAE78043) с GPAT, полученной из Escherichia coli (E. coli), белком plsB, составляла 17,6%. На фиг.5 показано выравнивание MaGPAT4 с этими аминокислотными последовательностями.
Аналогично, предсказанную аминокислотную последовательность (SEQ ID NO:9) MaGPAT5 подвергали анализу гомологии с аминокислотными последовательностями, зарегистрированными в GenBank nr, с использованием BLASTp. Аминокислотной последовательностью, показавшей наименьшую величину E при сравнении с этой последовательностью, т.е. обладающей наибольшей идентичностью, являлась аминокислотная последовательность (инвентарный номер GenBank XP_759516) предсказанного белка UM03369, полученного из аскомицета Ustilago maydis 521, и его идентичность составляла 15,4%. На фиг.6 показано выравнивание MaGPAT5 с этими аминокислотными последовательностями.
И MaGPAT4, и MaGPAT5 сохраняют область, консервативную в ацилтрансферазе, в частности, консервативный остаток глицина (G) и остаток пролина (P), указанные символом "∗", считается важным для ацилтрансферазной активности. Кроме того, также сохраняют остаток серина (S), указанный символом "+", считающийся важным для связывания с G3P (фиг.5 и 6).
Пример 5: Функциональный анализ MaGPAT4
(1) Конструирование вектора, экспрессирующего MaGPAT4 в дрожжах
Фрагмент ДНК, полученный расщеплением экспрессирующего вектора pYE22m дрожжей (Biosci. Biotech. Biochem., 59, 1221-1228, 1995) ферментами рестрикции EcoRI и SalI, и фрагмент ДНК приблизительно 2,1 т.п.н., полученный расщеплением плазмиды pCR-MaGPAT4 ферментами рестрикции EcoRI и SalI, соединяли друг с другом с использованием Ligation High (TOYOBO) для конструирования плазмиды pYE-MaGPAT4.
Отдельно для сравнения конструировали экспрессирующие векторы, экспрессирующие MaGPAT1, MaGPAT2 или MaGPAT3 в дрожжах. Векторы, экспрессирующие MaGPAT1 и MaGPAT2 в дрожжах, конструировали, как описано в WO2008/156026, и называли pYE-MaGPAT1 и pYE-MaGPAT2, соответственно. Вектор, экспрессирующий MaGPAT3 в дрожжах, получали следующим образом. Осуществляли поиск с использованием tblastn с геномными нуклеотидными последовательностями M. alpina штамма 1S-4, полученными как в примере 1, с использованием аминокислотной последовательности MaGPAT1 (ATCC № 16266) в качестве поискового запроса. В результате идентифицировали ген с гомологией с MaGPAT1 и называли MaGPAT3 (данные не опубликованы на момент подачи настоящей заявки). Получали следующие праймеры:
Eco-MaGPAT3-F: 5'-GAATTCATGGGTCTCCAGATCTATGACTTCGTCTC-3' (SEQ ID NO:26), и
Sal-MaGPAT3-R: 5'-GTCGACTTATGCCTCCTTAGACTTGACTGCATCC-3' (SEQ ID NO:27).
ПЦР осуществляли с KOD-Plus- (TOYOBO) с использованием кДНК, описываемой в примере 2, в качестве матрицы и этих праймеров. В результате амплифицировали фрагмент ДНК приблизительно 2,2 т.п.н. Фрагмент клонировали с использованием набора для клонирования Zero Blunt TOPO PCR (Invitrogen), и получаемую плазмиду называли pCR-MaGPAT3. Для получения фрагмента ДНК приблизительно 2,3 т.п.н. плазмиду pCR-MaGPAT3 расщепляли ферментами рестрикции EcoRI и SalI и для получения плазмиды pYE-MaGPAT3 этот фрагмент ДНК встраивали в участки EcoRI и SalI экспрессирующего вектора pYE22m для дрожжей.
(2) Получение трансформанта
Дрожжи S. cerevisiae штамма EH13-15 (trp1, MATα) (Appl. Microbiol. Biotechnol., 30, 515-520, 1989) трансформировали способом с использованием ацетата лития с использованием плазмид pYE22m, pYE-MaGPAT4, pYE-MaGPAT1, pYE-MaGPAT2 или pYE-MaGPAT3. Выбирали трансформанты, выращенные на агарной среде (2% агара) SC-Trp (содержащей 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г глюкозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденин, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 1,8 г лейцина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина и 0,6 г урацила) на литр).
(3) Культура дрожжей
Два произвольно выбранных штамма из штаммов, полученных трансформацией плазмидой pYE22m, называли штамм C-1 и штамм C-2, и два произвольно выбранных штамма из штаммов, полученных трансформацией плазмидой pYE-MaGPAT4, называли штамм MaGPAT4-1 и штамм MaGPAT4-2. Эти штаммы подвергали следующему эксперименту по культивированию. Для сравнения два штамма, произвольно выбранных из штаммов, получаемых трансформацией плазмидой pYE-MaGPAT1, называли штамм MaGPAT1-1 и штамм MaGPAT1-2, два штамма, произвольно выбранных из штаммов, получаемых трансформацией плазмидой pYE-MaGPAT2, называли штамм MaGPAT2-1 и штамм MaGPAT2-2, и два штамма, произвольно выбранных из штаммов, получаемых трансформацией плазмидой pYE-MaGPAT3, называли штамм MaGPAT3-1 и штамм MaGPAT3-2, и эти штаммы также подвергали следующему эксперименту по культивированию в качестве штамма MaGPAT4-1 и штамма MaGPAT4-2.
В качестве прекультуры одну платиновую петлю дрожжевых клеток из планшета инокулировали в 10 мл среды SC-Trp и культивировали при перемешивании при 30°C в течение 1 дня. В основной культуре 500 мкл раствора прекультуры добавляли к 10 мл среды SC-Trp или среды YPD (2% дрожжевого экстракта, 1% полипептона и 2% глюкозы) с последующим культивирование при перемешивании при 30°C в течение 2 дней.
(4) Анализ клеточных жирных кислот
Дрожжевые клетки собирали посредством центрифугирования культурального раствора. Клетки промывали 10 мл стерилизованной воды, опять собирали посредством центрифугирования и лиофилизировали. К лиофилизированным дрожжевым клеткам добавляли 4 мл смеси, содержащей хлороформ:метанол (2:1), и энергично перемешивали, и затем позволяли отстаиваться при 70°C в течение 1 часа. Дрожжевые клетки разделяли посредством центрифугирования и собирали растворитель. К оставшимся клеткам снова добавляли 4 мл смеси хлороформ:метанол (2:1) и раствор собирали подобным образом. Липиды высушивали с использованием Speedback и добавляли в него 2 мл хлороформа для растворения липидом. Отбирали двести микролитров этого раствора, превращали жирные кислоты клеток в метиловый сложный эфир посредством способа с использованием соляной кислоты и метанола и экстрагировали гексаном. Гексан отгоняли с последующим анализом газовой хроматографией. Результаты представлены в таблицах 4-7.
Как показано в таблицах 4 и 5, композиционное отношение жирных кислот в составе липида в клетках штамма, высокоэкспрессирующего MaGPAT4, обладает высокой долей пальмитиновой кислоты (16:0) и низкой долей пальмитолеиновой кислоты (16:1) по сравнению с таковыми контрольного штамма, в который встраивали только вектор. Эта тенденция отличается от тенденций, когда использовали другую GPAT, полученную из Mortierella alpina, т.е. MaGPAT1, MaGPAT2 или MaGPAT3. Таким образом, MaGPAT4 по настоящему изобретению обладает субстратной специфичностью, отличающейся от таковой других GPAT, полученных из Mortierella alpina.
Кроме того, как показано в таблицах 6 и 7, содержание жирных кислот в клетках штамма, высокоэкспрессирующих MaGPAT4, повышалось приблизительно в 2 раза по сравнению в контрольном штамме.
Пример 6: Функциональный анализ MaGPAT4-long и MaGPAT4
(1) Конструирование галактоза-индуцибельного экспрессирующего вектора
Для стимуляции экспрессии MaGPAT4-long или MaGPAT4 галактозой плазмиду, содержащую галактоза-индуцибельный промотор, конструировали следующим образом.
ПЦР осуществляли с ExTaq (Takara Bio Inc.) с использованием кДНК, полученной в примере 2, в качестве матрицы и комбинации праймера Not-MaGPAT4-F1 и праймера Bam-MaGPAT4-R или комбинации праймера Not-MaGPAT4-F2 и праймера Bam-MaGPAT4-R при 94°C в течение 2 мин и затем 30 циклов (94°C в течение 1 мин, 55°C в течение 1 мин и 72°C в течение 1 мин). Амплифицированные фрагменты ДНК приблизительно 2,7 т.п.н. и приблизительно 2,5 т.п.н. клонировали с использованием набора для клонирования TOPO-TA (Invitrogen) и подтверждали наличие последовательности CDS GPAT4-long, приведенной в SEQ ID NO:6, и последовательности CDS GPAT4, приведенной в SEQ ID NO:3, соответственно, и называли плазмида pCR-long MaGPAT4-1 и плазмида pCR-MaGPAT4-1, соответственно. Фрагмент ДНК приблизительно 6,5 т.п.н., полученный расщеплением вектора pESC-TRP (Stratagene) ферментами рестрикции NotI и BglII, соединяли с фрагментами ДНК приблизительно 2,7 т.п.н. или 2,5 т.п.н., получаемыми расщеплением плазмиды pCR-long MaGPAT4-1 или плазмиды pCR-MaGPAT4-1 ферментами рестрикции NotI и BamHI с использованием Ligation High (TOYOBO) для получения плазмиды pESC-T-MaGPAT4-long или плазмиды pESC-T-MaGPAT4.
Праймер Not-MaGPAT4-F1: 5'-GCGGCCGCATGACAACCGGCGACAGTACCG-3' (SEQ ID NO:28)
Праймер Not-MaGPAT4-F2: 5'-GCGGCCGCATGCCCATCGTTCCAGCTCAG-3' (SEQ ID NO:29)
Праймер Bam-MaGPAT4-R: 5'-GGATCCTTATAATTTCGGGGCGCCATCG-3' (SEQ ID NO:30)
(2) Получение трансформанта
Дрожжи S. cerevisiae штамма EH13-15 трансформировали способом с использованием ацетата лития с использованием pESC-TRP, pESC-T-MaGPAT4-long или pESC-T-MaGPAT4. Выбирали трансформанты, выраставшие на агарной среде SC-Trp.
(3) Культура дрожжей
В качестве прекультуры каждый из четырех произвольно выбранных штаммов из трансформантов, получаемых трансформацией с каждой плазмидой, инокулировали в 10 мл жидкой среды SD-Trp и культивировали при перемешивании при 30°C в течение 1 дня. В основной культуре каждый 1 мл раствора прекультуры инокулировали в 10 мл жидкой среды SG-Trp (содержащей, 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г глюкозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденина, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 1,8 г лейцина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина и 0,6 г урацила) на литр) в параллели с последующим культивированием при перемешивании при 30°C в течение 2 дней для стимуляции экспрессии MaGPAT4-long или MaGPAT4.
(4) Анализ жирных кислот
Дрожжевые клетки собирали посредством центрифугирования культурального раствора. Клетки промывали 10 мл стерилизованной воды, снова собирали посредством центрифугирования и лиофилизировали. Жирные кислоты в клетках одной линии каждого штамма превращали в метиловый сложный эфир способом с использованием соляной кислоты и метанола и подвергали анализу газовой хроматографии тотальных жирных кислот, содержащихся в клетках (Таблица 8).
Липиды в клетках другой линии каждого штамма экстрагировали следующим образом. Т.е. 1 мл смеси, содержащей хлороформ:метанол (2:1) и стеклянные бусы добавляли в клетки, и клетки разрушали на гомогенизаторе типа "bead beater". Затем проводили центрифугирование и супернатант собирали. Затем один миллилитр смеси хлороформ:метанол (2:1) добавляли к оставшимся клеткам и аналогично собирали супернатант. Эту процедуру повторяли и липиды экстрагировали в общей сложности 4 мл смеси хлороформ:метанол (2:1). Растворитель отгоняли с использованием Speedback и остаток растворяли в небольшом количестве хлороформа. Липиды фракционировали на пластине силикагеля 60 (Merck) посредством тонкослойной хроматографией с использованием смеси гексан:диэтиловый простой эфир:уксусная кислота в соотношении 70:30:1 в качестве элюента. Липиды визуализировали распылением раствора примулина и последующим облучением УФ. Фракцию триацилглицерола (TG), фракцию свободных жирных кислот (FFA), фракцию диацилглицерола (DG) и фракцию фосфолипида (PL) соскребали с пластины и соответственно помещали в пробирки. Жирные кислоты превращали в метиловые сложные эфиры способом с использованием соляной кислоты и метанола и подвергали анализу газовой хроматографией жирных кислот (таблица 9, Фиг.7).
Результаты анализа каждого штамма со встроенным геном представлены ниже. Штамм со встроенным вектором pESC-TRP использовали в качестве контроля.
TG
DG
PL
Общие количества жирных кислот в штамме, экспрессирующем MaGPAT4-long, и в штамме, экспрессирующем MaGPAT4, повышались в 1,7 и 2,0 раза, соответственно, по сравнению с таковым в контроле (таблица 8).
Количества PL в штамме, экспрессирующем MaGPAT4-long, и в штамме, экспрессирующем MaGPAT4, составляли, соответственно, в 1,1 и 1,2 раза больше, чем таковые в контроле. Таким образом, эти штаммы почти не демонстрировали различий относительно контроля. В отличие от этого, количества TG в штамме, экспрессирующем MaGPAT4-long, и в штамме, экспрессирующем MaGPAT4, значительно повышались, в 2,8 и 3,5 раза, соответственно, относительно контроля. Т.е. экспрессия MaGPAT4-long или MaGPAT4 может активировать биосинтез жирных кислот и повышает продукцию запасного липида TG. Кроме того, количества DG и FFA в штамме, экспрессирующем MaGPAT4-long, и в штамме, экспрессирующем MaGPAT4, повышались по сравнению с таковыми в контроле (таблица 9).
Доли жирных кислот в каждой фракции липидов представлены на фиг.7. Во фракции FFA доли жирных кислот в штамме, экспрессирующем MaGPAT4-long, и в штамме, экспрессирующем MaGPAT4, почти не отличаются от таковых в контроле, в то время как в других фракциях наблюдали тенденцию повышения насыщенных жирных кислот и снижения ненасыщенных жирных кислот в штамме, экспрессирующем MaGPAT4-long, и в штамме, экспрессирующем MaGPAT4. В частности, повышалось содержание 16:0 (пальмитиновой кислоты), и снижалось содержание 16:1 (пальмитолеиновая кислота).
Кроме того, основное культивирование осуществляли с использованием жидкой среды SC-Trp вместо жидкой среды SG-Trp. Т.к. жидкая среда SC-Trp не содержит галактозу, экспрессия MaGPAT4-long или MaGPAT4, встраиваемой при трансформации, не стимулирует при основном культивировании с использованием этой среды. В эксперименте с использованием жидкой среды SC-Trp не наблюдали различий в количестве и долях тотальных жирных кислот в клетках между контролем и штаммом со встроенной MaGPAT4-long или MaGPAT4 (таблица 10, фиг.8). Это также позволяло предполагать, что экспрессию MaGPAT4-long или MaGPAT4 стимулировала галактоза, приводя к повышению жирных кислот в клетках в среде SG-Trp.
Пример 7: Дополнительный эксперимент с дрожжами S. cerevisiae Δsct1 и Δgpt2
У дрожжей S. cerevisiae SCT1 и GPT2 известны как гены, вовлеченные в GPAT-активность, и известно, что одновременная недостаточность в этих генах приводит к гибели. Для подтверждения того, обладают ли продукты MaGPAT4 и MaGPAT4-long, полученные из Mortierella alpina, GPAT-активностью, осуществляли дополнительный эксперимент с Δsct1 и Δgpt2. В таблице 11 приведены генотипы штаммов, полученных как описано ниже.
(1) Получение штамма GP-1
Ген SCT1 гомозиготных по Δgpt2 диплоидных дрожжей (кат. № YSC1021-663938) из коллекции нокаутных дрожжей (Open Biosystems) разрушали следующим образом. ДНК выделяли S. cerevisiae штамма S288C клетки с использованием Dr. GenTLE (из дрожжей) (TaKaRa Bio Inc.). Фрагмент гена SCT1 амплифицировали посредством ПЦР с KOD-Plus-(TOYOBO) с использованием получаемой ДНК в качестве матрицы, праймера Xba1-Des-SCT1-F: 5'-TCTAGAATGCCTGCACCAAAACTCAC-3' (SEQ ID NO:31) и праймера Xba1-Des-SCT1-R: 5'-TCTAGACCACAAGGTGATCAGGAAGA-3' (SEQ ID NO:32). Амплифицированный фрагмент ДНК приблизительно 1,3 т.п.н. клонировали с использованием набора для клонирования Zero Blunt TOPO PCR (Invitrogen) и получаемую плазмиду называли pCR-SCT1P. Затем фрагмент ДНК приблизительно 2,2 т.п.н., содержащий CDS гена LEU2, получаемый расщеплением плазмиды YEp13 ферментами рестрикции SalI и XhoI, соединяли с фрагментом ДНК приблизительно 4,4 т.п.н., получаемым расщеплением плазмиды pCR-SCT1P ферментом рестрикции SalI с использованием Ligation High (TOYOBO) для получения плазмиды pCR-Δsct1:LEU2, где ген LEU2 встраивают в обратной ориентации в отношении гена SCT1. Ее расщепляли ферментом рестрикции XbaI и гомозиготные по Δgpt2 диплоидные дрожжи трансформировали способом с использованием ацетата лития. Выбирали трансформант, вырастающий на агарной (2% агара) среде SD-Leu (содержащей 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г глюкозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденина, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина, 1,2 г триптофана и 0,6 г урацила) на литр). ДНК выделяли из получаемых клеток трансформанта описываемым выше способом. Для подтверждения генотипа, т.е. подтверждения того, что генотип представляет собой SCT1/Δsct1:LEU2, осуществляли ПЦР с использованием комбинации (a) праймера SCT1outORF-F: 5'-AGTGTAGGAAGCCCGGAATT-3' (SEQ ID NO:33) и праймера SCT1inORF-R: 5'-GCGTAGATCCAACAGACTAC-3' (SEQ ID NO:34) (0,5 т.п.н.) или комбинации (b) праймера SCT1outORF-F и праймера LEU2inORF-F: 5'-TTGCCTCTTCCAAGAGCACA-3' (SEQ ID NO:35) (1,2 т.п.н.) и штамм называли штамм GP-1.
(2) Конструирование галактоза-индуцибельного экспрессирующего вектора с использованием URA3 в качестве маркера
Для получения плазмиды pESC-U-MaGPAT4-long и плазмиды pESC-U-MaGPAT4 фрагмент ДНК приблизительно 6,6 т.п.н., получаемый расщеплением вектора pESC-URA (Stratagene) ферментами рестрикции NotI и BglII, соединяли с фрагментом ДНК приблизительно 2,7 т.п.н. или 2,5 т.п.н., получаемым расщеплением плазмиды pCR-MaGPAT4-long-1 или плазмиды pCR-MaGPAT4-1 ферментами рестрикции NotI и BamHI с использованием Ligation High (TOYOBO).
(3) Получение трансформанта
Дрожжи штамма GP-1 трансформировали способом с использованием ацетата лития с использованием pESC-URA, pESC-U-MaGPAT4-long или pESC-U-MaGPAT4. Выбирали трансформанты, вырастающие на агарной среде (2% агара) SC-Ura (содержащей 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г глюкозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденина, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина, 1,2 г триптофана и 1,8 г лейцина) на литр). Штамм, получаемый трансформацией с использованием pESC-URA называли штамм C-D, и штаммы, получаемые трансформацией с использованием pESC-U-MaGPAT4-long и pESC-U-MaGPAT4, называли штамм MaGPAT4-long-D и штамм MaGPAT4-D, соответственно.
(4) Спорообразование и тетрадный анализ
Каждый из штамма C-D, штамма MaGPAT4-long-D и штамма MaGPAT4-D наносили на агарную среду YPD и культивировали при 30°C в течение 1 дня. Выращенные клетки наносили на агарную среду для спорообразования (0,5% ацетата калия, 2% агара) и культивировали при 20°C в течение 7 дней. Соскребали подходящее количество получаемых клеток и суспендировали в 100 мкл раствора зимолиазы (0,125 мг/мл зимолиазы 100T, 1M сорбита, 40 мМ калий-фосфатный буфер (pH 6,8)). Суспензию инкубировали при комнатной температуре в течение 30 мин, и содержащую суспензию пробирку помещали на лед. После подтверждения образования аскоспор при микроскопии посредством микроманипулирования выделяли четыре аскоспоры на агарной среде YPDGal (2% дрожжевого экстракта, 1% пептона и 2% галактозы) с последующей инкубацией при 30°C в течение 2 дней для получения колоний, получаемых из каждой споры.
Для исследования урациловой ауксотрофии и лейциновой ауксотрофии получаемые клоны спор реплицировали инкубацией на агарной среде (2% агара) SG-Ura (содержащей 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г галактозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденина, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина, 1,2 г триптофана и 1,8 г лейцина) на литр) и агарной среде (2% агара) SG-Leu (содержащей 6,7 г дрожжевых азотных оснований без аминокислот (DIFCO), 20 г галактозы и 1,3 г порошка аминокислот (смесь 1,25 г сульфата аденина, 0,6 г аргинина, 3 г аспарагиновой кислоты, 3 г глутаминовой кислоты, 0,6 г гистидина, 0,9 г лизина, 0,6 г метионина, 1,5 г фенилаланина, 11,25 г серина, 0,9 г тирозина, 4,5 г валина, 6 г треонина, 1,2 г триптофана и 0,6 г урацила) на литр) при 30°C в течение 3 дней. Считали, что клон, демонстрирующий урациловую ауксотрофию, утрачивал встроенную плазмиду. Таким образом, такой клон не подвергали последующему анализу.
Среди четырех аскоспор, выделенных из штамма C-D, две аскоспоры могли расти на агарной среда YPGal, и все штаммы демонстрировали лейциновую ауксотрофию. Все четыре аскоспоры, выделенные из штамма MaGPAT4-long-D или штамма MaGPAT4-D, могли расти на агарной среде YPGal. По результатам исследования лейциновой ауксотрофии две из четырех аскоспор демонстрировали лейциновую ауксотрофию и другие две не демонстрировали лейциновую ауксотрофию.
Генотипы получаемых штаммов исследовали посредством выделения ДНК из клеток, как описано выше, и осуществления ПЦР с использованием комбинации (a) праймера SCT1outORF-F и праймер SCT1inORF-R и комбинации (b) праймера SCT1outORF-F и праймера LEU2inORF-F. В результате неауксотрофные по лейцину штаммы не амплифицировали посредством ПЦР с использованием комбинации (a), но амплифицировали посредством ПЦР с использованием комбинации (b). Таким образом, подтверждали, что эти штаммы имеют аллели Δsct1:LEU2. Ауксотрофные по лейцину штаммы амплифицировали посредством ПЦР с использованием комбинации (a), но не амплифицировали посредством ПЦР с использованием комбинации (b). Таким образом, подтверждали, что эти штаммы имеют аллели SCT1.
Кроме того, каждый из штаммов, получаемых из четырех аскоспор, выделенных из штамма MaGPAT4-long-D или штамма MaGPAT4-D, наносили на агарную среду YPD. Два штамма росли хорошо, но другие два штамма росли достаточно плохо. Штаммы, демонстрировавшие плохой рост, соответствовали неауксотрофным по лейцину штаммам с аллелями Δsct1:LEU2.
Эти результаты подтверждали, что, хотя наличие в штамме Δsct1 Δgpt2 недостаточности двух генов, вовлеченных в GPAT-активность дрожжей, приводит к гибели, штамм может расти, экспрессируя MaGPAT4-long или MaGPAT4. Т.е., это позволяло с уверенностью предполагать, что белок MaGPAT4-long и белок MaGPAT4 обладают GPAT-активностью.
Пример 8: Гиперэкспрессия GPAT4 в Mortierella alpina
(1) Конструирование экспрессирующего вектора для M. alpina
Для экспрессии GPAT4 в M. alpina векторы конструировали следующим образом.
Вектор pUC18 расщепляли ферментами рестрикции EcoRI и HindIII, и адаптер, полученный отжигом олигонуклеотидов, MCS-for-pUC18-F2 и MCS-for-pUC18-R2 встраивали для конструирования плазмиды pUC18-RF2.
MCS-for-pUC18-F2: 5'-AATTCATAAGAATGCGGCCGCTAAACTATTCTAGACTAGGTCGACGGCGCGCCA-3' (SEQ ID NO:36)
MCS-for-pUC18-R2: 5'-AGCTTGGCGCGCCGTCGACCTAGTCTAGAATAGTTTAGCGGCCGCATTCTTATG-3' (SEQ ID NO:37)
Фрагмент ДНК приблизительно 0,5 т.п.н., амплифицированный посредством ПЦР с KOD-plus- (TOYOBO) с использованием генома M. alpina в качестве матрицы, праймера Not1-GAPDHt-F и праймера EcoR1-Asc1-GAPDHt-R, клонировали с использованием набора для клонирования Zero Blunt TOPO PCR (Invitrogen). После подтверждения нуклеотидной последовательности встроенной области для конструирования плазмиды pDG-1 фрагмент ДНК приблизительно 0,9 т.п.н., получаемый расщеплением ферментами рестрикции NotI и EcoRI, встраивали в участки NotI и EcoRI плазмиды pUC18-RF2.
Праймер Not1-GAPDHt-F: 5'-AGCGGCCGCATAGGGGAGATCGAACC-3' (SEQ ID NO:38)
Праймер EcoR1-Asc1-GAPDHt-R: 5'-AGAATTCGGCGCGCCATGCACGGGTCCTTCTCA-3' (SEQ ID NO:39)
Фрагмент ДНК, амплифицированный посредством ПЦР с KOD-plus- (TOYOBO) с использованием генома M. alpina в качестве матрицы, праймера URA5g-F1 и праймера URA5g-R1, клонировали с использованием набора для клонирования Zero Blunt TOPO PCR (Invitrogen). После подтверждения нуклеотидной последовательности встроенной области для конструирования плазмиды pDuraG фрагмент ДНК приблизительно 2 т.п.н., получаемый расщеплением ферментом рестрикции SalI, встраивали в участок SalI плазмиды pDG-1 таким образом, что 5'-конец гена URA5 находился в участке EcoRI вектора.
Праймер URA5g-F1: 5'-GTCGACCATGACAAGTTTGC-3' (SEQ ID NO:40)
Праймер URA5g-R1: 5'-GTCGACTGGAAGACGAGCACG-3' (SEQ ID NO:41)
Затем фрагмент ДНК приблизительно 1,0 т.п.н., амплифицированный посредством ПЦР с KOD-plus- (TOYOBO) с использованием генома M. alpina в качестве матрицы, праймера hisHp+URA5-F и праймера hisHp+MGt-F соединяли с фрагментом ДНК приблизительно 5,3 т.п.н., амплифицированным посредством ПЦР с KOD-plus- (TOYOBO) с использованием pDuraG в качестве матрицы, праймера pDuraSC-GAPt-F и праймера URA5gDNA-F с использованием набора для клонирования In-Fusion (зарегистрированная торговая марка) Advantage PCR (TaKaRa Bio Inc.) для получения плазмид pDUra-RhG.
Праймер hisHp+URA5-F: 5'-GGCAAACTTGTCATGAAGCGAAAGAGAGATTATGAAAACAAGC-3' (SEQ ID NO:42)
Праймер hisHp+MGt-F: 5'-CACTCCCTTTTCTTAATTGTTGAGAGAGTGTTGGGTGAGAGT-3' (SEQ ID NO:43)
Праймер pDuraSC-GAPt-F: 5'-TAAGAAAAGGGAGTGAATCGCATAGGG-3' (SEQ ID NO:44)
Праймер URA5gDNA-F: 5'-CATGACAAGTTTGCCAAGATGCG-3' (SEQ ID NO:45)
Фрагмент ДНК приблизительно 6,3 т.п.н. амплифицировали посредством ПЦР с KOD-plus- (TOYOBO) с использованием плазмиды pDUra-RhG в качестве матрицы, праймера pDuraSC-GAPt-F и праймера pDurahG-hisp-R.
Праймер pDurahG-hisp-R: 5'-ATTGTTGAGAGAGTGTTGGGTGAGAGTG-3' (SEQ ID NO:46)
Фрагмент ДНК приблизительно 2,5 т.п.н. амплифицировали посредством ПЦР с KOD-plus- (TOYOBO) с использованием плазмиды pCR-MaGPAT4-1 в качестве матрицы, праймера MaGPAT4+hisp-F и праймер MaGPAT4+MGt-R.
Праймер MaGPAT4+hisp-F: 5' CACTCTCTCAACAATATGACAACCGGCGACAGTACCGC-3' (SEQ ID NO:47)
Праймер MaGPAT4+MGt-R: 5' CACTCCCTTTTCTTATTATAATTTCGGGGCGCCATCGC-3' (SEQ ID NO:48)
Для получения плазмиды pDUraRhG-GPAT4 получаемый фрагмент 2,5 т.п.н. соединяли с указанным выше фрагментом ДНК 6,3 т.п.н. с использованием набора для клонирования In-Fusion (зарегистрированная торговая марка) Advantage PCR (TaKaRa Bio Inc.).
(2) Получение трансформанта M. alpina
Трансформацию осуществляли с использованием в качестве хозяина ауксотрофного по урацилу штамма Δura-3, полученного из M. alpina штамма 1S-4 по способу, описываемому в патентном документе (WO2005/019437, название изобретения: "Method of breeding lipid-producing fungus"), способом доставки частиц с использованием плазмиды pDUraRhG-GPAT4. Трансформанты выбирали с использованием агарной среды SC (0,5% дрожжевых азотных оснований без аминокислот и сульфата аммония (Difco), 0,17% сульфата аммония, 2% глюкозы, 0,002% аденина, 0,003% тирозина, 0,0001% метионина, 0,0002% аргинина, 0,0002% гистидина, 0,0004% лизина, 0,0004% триптофана, 0,0005% треонина, 0,0006% изолейцина, 0,0006% лейцина, 0,0006% фенилаланина, 2% агара).
(3) Оценка трансформированного M. alpina
Получаемый трансформант инокулировали в 4 мл среды GY и культивировали при перемешивании при 28°C в течение 2 дней. Клетки собирали фильтрацией. РНК выделяли с использованием набора для растений RNeasy (QIAGEN) и синтезировали кДНК с использованием системы SuperScript First-Strand для ПЦР с обратной транскрипцией (Invitrogen). Для подтверждения экспрессии каждого гена из встроенной конструкции осуществляли ПЦР в реальном времени с использованием комбинации следующих праймеров:
Праймер GPAT4-RT1: 5'-GAGTGCTTCATCGAGGGCACC-3' (SEQ ID NO:49), и
Праймер GPAT4-RT2: 5'-TCCTTCACTGTCAACCTCGATCAC-3' (SEQ ID NO:50).
Один из штаммов с подтвержденной гиперэкспрессией инокулировали в 10 мл среды GY (2% глюкозы, 1% дрожжевого экстракта) и культивировали при перемешивании при 28°C и 300 об./мин. в течение 3 дней. Весь культуральный раствор добавляли к 500 мл среды GY (в 2-литровых флаконах Сакагучи) и культивировали при перемешивании при 28°C и 120 об./мин. Пять миллилитров и десять миллилитров культурального раствора отбирали на третий, седьмой, десятый и двенадцатый дни и фильтровали. Клетки высушивали при 120°°C. Жирные кислоты превращали в метиловые сложные эфиры способом с использованием соляной кислоты и метанола и подвергали анализу газовой хроматографией жирных кислот. Исследовали изменение с течением времени количества продуцируемой арахидоновой кислоты на высушенные клетки. В качестве контроля использовали хозяина для трансформации штамм Δura-3. Результаты представлены на фиг.9.
Количество арахидоновой кислоты (AA) на клетки повышалось в M. alpina, гиперэкспрессирующем GPAT4, по сравнению с таковым в контроле.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ В ПРОИЗВОЛЬНОМ ФОРМАТЕ
SEQ ID NO:16: праймер GPAT4-S
SEQ ID NO:17: праймер SacI-GPAT4-1
SEQ ID NO:18: праймер Sal-GPAT4-2
SEQ ID NO:19: праймер GPAT5-1F
SEQ ID NO:20: праймер GPAT5-3R
SEQ ID NO:26: праймер Eco-MaGPAT3-F
SEQ ID NO:27: праймер Sal-MaGPAT3-R
SEQ ID NO:28: праймер Not-MaGPAT4-F1
SEQ ID NO:29: праймер Not-MaGPAT4-F2
SEQ ID NO:30: праймер Bam-MaGPAT4-R
SEQ ID NO:31: праймер Xba1-Des-SCT1-F
SEQ ID NO:32: праймер Xba1-Des-SCT1-R
SEQ ID NO:33: праймер SCT1outORF-F
SEQ ID NO:34: праймер SCT1inORF-R
SEQ ID NO:35: праймер LEU2inORF-F
SEQ ID NO:36: олигонуклеотид MCS-for-pUC18-F2
SEQ ID NO:37: олигонуклеотид MCS-for-pUC18-R2
SEQ ID NO:38: праймер Not1-GAPDHt-F
SEQ ID NO:39: праймер EcoR1-Asc1-GAPDHt-R
SEQ ID NO:40: праймер URA5g-F1
SEQ ID NO:41: праймер URA5g-R1
SEQ ID NO:42: праймер hisHp+URA5-F
SEQ ID NO:43: праймер hisHp+MGt-F
SEQ ID NO:44: праймер pDuraSC-GAPt-F
SEQ ID NO:45: праймер URA5gDNA-F
SEQ ID NO:46: праймер pDurahG-hisp-R
SEQ ID NO:47: праймер MaGPAT4+hisp-F
SEQ ID NO:48: праймер MaGPAT4+MGt-R
SEQ ID NO:49: праймер GPAT4-RT1
SEQ ID NO:50: праймер GPAT4-RT2
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Suntory Holdings, Ltd.
<120> Гомолог глицерол-3-фосфат-ацилтрансферазы и его использование
<130> FA0008-11002
<140> PCT/JP2011/052258
<141> 2011-02-03
<150> JP 2010-22125
<151> 2010-02-03
<160> 50
<170> PatentIn version 3.1
<210> 1
<211> 2475
<212> ДНК
<213> Mortierella alpina
<220>
<221> CDS
<222> (1)..(2475)
<223>
<400> 1
atg ccc atc gtt cca gct cag caa gtc gac tcc tcc tcg tgc cct ccc 48
Met Pro Ile Val Pro Ala Gln Gln Val Asp Ser Ser Ser Cys Pro Pro
1 5 10 15
tct ggt gaa tct agc ccg ttg att ccc agt ttg ggc gaa gga gtg cat 96
Ser Gly Glu Ser Ser Pro Leu Ile Pro Ser Leu Gly Glu Gly Val His
20 25 30
tcg ggt cat gga cac gtt gtc gac aat gac gag tcc ggc gtg gag aac 144
Ser Gly His Gly His Val Val Asp Asn Asp Glu Ser Gly Val Glu Asn
35 40 45
att acc aaa aag cac gca gga cga att aga gaa gat ccg gtc ggc ttc 192
Ile Thr Lys Lys His Ala Gly Arg Ile Arg Glu Asp Pro Val Gly Phe
50 55 60
gtg gtg cag act gca gct ttc tat cag ggc acg ggt tgg aga agc tac 240
Val Val Gln Thr Ala Ala Phe Tyr Gln Gly Thr Gly Trp Arg Ser Tyr
65 70 75 80
agc aac tat gtt gga acg cgc att ctt tac gaa ggc ttc tct gca acc 288
Ser Asn Tyr Val Gly Thr Arg Ile Leu Tyr Glu Gly Phe Ser Ala Thr
85 90 95
ttc aag gag cga att ctc gcc agt tca aaa gtg aat gat ctc atc aag 336
Phe Lys Glu Arg Ile Leu Ala Ser Ser Lys Val Asn Asp Leu Ile Lys
100 105 110
gac atg gcc aac aag cag ttg gat gtc ctg atc aag caa aga caa gat 384
Asp Met Ala Asn Lys Gln Leu Asp Val Leu Ile Lys Gln Arg Gln Asp
115 120 125
gcg tat gac gca gag agg act gca aat gca gga aaa aag aac ttc aag 432
Ala Tyr Asp Ala Glu Arg Thr Ala Asn Ala Gly Lys Lys Asn Phe Lys
130 135 140
ccc aaa gtt cga ctc ctt cgc cca gaa gat atc gag gct cgt cgc aaa 480
Pro Lys Val Arg Leu Leu Arg Pro Glu Asp Ile Glu Ala Arg Arg Lys
145 150 155 160
aca tta gaa gcc gag ctt gtt gcg gtg gca aag tca aat atc gac aaa 528
Thr Leu Glu Ala Glu Leu Val Ala Val Ala Lys Ser Asn Ile Asp Lys
165 170 175
ctt gtt tgt gat atg aac agt atg aaa ttc atc agg ttc ttc gcc ttc 576
Leu Val Cys Asp Met Asn Ser Met Lys Phe Ile Arg Phe Phe Ala Phe
180 185 190
ctc atc aac aac atc ctt gtg aga atg tac cat caa gga att cac atc 624
Leu Ile Asn Asn Ile Leu Val Arg Met Tyr His Gln Gly Ile His Ile
195 200 205
aag gag tcc gag ttc ttg gag ctg cgg agg ata gct gag tac tgc gca 672
Lys Glu Ser Glu Phe Leu Glu Leu Arg Arg Ile Ala Glu Tyr Cys Ala
210 215 220
gag aaa aag tat tcg atg gtg gtg ttg cca tgc cac aag tca cac atc 720
Glu Lys Lys Tyr Ser Met Val Val Leu Pro Cys His Lys Ser His Ile
225 230 235 240
gac tac ctc gtc gtc tcg tac att ttc ttc cgc atg gga tta gct tta 768
Asp Tyr Leu Val Val Ser Tyr Ile Phe Phe Arg Met Gly Leu Ala Leu
245 250 255
cct cac att gct gct ggc gat aac ctg gac atg ccc att gtc gga aag 816
Pro His Ile Ala Ala Gly Asp Asn Leu Asp Met Pro Ile Val Gly Lys
260 265 270
gca ctc aaa gga gca ggc gcg ttc ttc att cgc cgt tct tgg gct gac 864
Ala Leu Lys Gly Ala Gly Ala Phe Phe Ile Arg Arg Ser Trp Ala Asp
275 280 285
gat caa ctt tac acc agc att gtt cag gaa tat gtt cag gag ctt ttg 912
Asp Gln Leu Tyr Thr Ser Ile Val Gln Glu Tyr Val Gln Glu Leu Leu
290 295 300
gag gga gga tac aat atc gag tgc ttc atc gag ggc acc cga agc aga 960
Glu Gly Gly Tyr Asn Ile Glu Cys Phe Ile Glu Gly Thr Arg Ser Arg
305 310 315 320
aca gga aaa ctt ttg cca cca aag ctg gga gtc cta aag att atc atg 1008
Thr Gly Lys Leu Leu Pro Pro Lys Leu Gly Val Leu Lys Ile Ile Met
325 330 335
gat gct atg ctt tcg aac cgc atc caa gac tgc tac atc gtg ccc atc 1056
Asp Ala Met Leu Ser Asn Arg Ile Gln Asp Cys Tyr Ile Val Pro Ile
340 345 350
tct atc ggt tat gac aag gtc atc gaa acc gag act tat atc aat gag 1104
Ser Ile Gly Tyr Asp Lys Val Ile Glu Thr Glu Thr Tyr Ile Asn Glu
355 360 365
ctt ctc gga atc ccc aag gaa aag gag agt ttg tgg ggt gtt att acg 1152
Leu Leu Gly Ile Pro Lys Glu Lys Glu Ser Leu Trp Gly Val Ile Thr
370 375 380
aat tcg agg ctg ctc cag ctc aag atg ggc cgc att gat gtc cga ttt 1200
Asn Ser Arg Leu Leu Gln Leu Lys Met Gly Arg Ile Asp Val Arg Phe
385 390 395 400
gca aag ccg tac agt ttg cga aac ttt atg aat cat gag atc gag cgc 1248
Ala Lys Pro Tyr Ser Leu Arg Asn Phe Met Asn His Glu Ile Glu Arg
405 410 415
aga gag atc atc aat aag cgg gaa gac acc gat agt gtg gcg aaa tct 1296
Arg Glu Ile Ile Asn Lys Arg Glu Asp Thr Asp Ser Val Ala Lys Ser
420 425 430
cag ctg cta aag gca ttg ggc tac aag gtc ttg gca gac atc aac tcg 1344
Gln Leu Leu Lys Ala Leu Gly Tyr Lys Val Leu Ala Asp Ile Asn Ser
435 440 445
gtc tct gta gta atg ccc acg gcc ctc gtg ggt act gtc atc ctt aca 1392
Val Ser Val Val Met Pro Thr Ala Leu Val Gly Thr Val Ile Leu Thr
450 455 460
ctc cga gga cga ggt gtt ggc cgt aat gag ctg atc cgt cgt gtt gag 1440
Leu Arg Gly Arg Gly Val Gly Arg Asn Glu Leu Ile Arg Arg Val Glu
465 470 475 480
tgg ctg aag cgc gag att ctt tcc aag ggt ggt cgc gtt gcc aac ttt 1488
Trp Leu Lys Arg Glu Ile Leu Ser Lys Gly Gly Arg Val Ala Asn Phe
485 490 495
agc ggg atg gaa act ggc gag gtt gta gat cga gca ttg ggc gtt ctt 1536
Ser Gly Met Glu Thr Gly Glu Val Val Asp Arg Ala Leu Gly Val Leu
500 505 510
aag gac ctt gtg gcg ctg cag aag aat ttg ctc gag ccc gtc ttc tat 1584
Lys Asp Leu Val Ala Leu Gln Lys Asn Leu Leu Glu Pro Val Phe Tyr
515 520 525
gcg gtc aag cgc ttc gag ctt tcg ttc tac agg aat cag ctc atc cac 1632
Ala Val Lys Arg Phe Glu Leu Ser Phe Tyr Arg Asn Gln Leu Ile His
530 535 540
ctc ttt gtc cat gag gcc atc atc gcc gtg acg atg tac acc cgc atc 1680
Leu Phe Val His Glu Ala Ile Ile Ala Val Thr Met Tyr Thr Arg Ile
545 550 555 560
aag att ggt ggc gcc aag tct aca caa cac att agt cag aat gag ctg 1728
Lys Ile Gly Gly Ala Lys Ser Thr Gln His Ile Ser Gln Asn Glu Leu
565 570 575
ctg aac gag gtc acc ttc ctg agc cgc ctg ctc aag acc gac ttt atc 1776
Leu Asn Glu Val Thr Phe Leu Ser Arg Leu Leu Lys Thr Asp Phe Ile
580 585 590
tac aac cct ggc gat att gag agt aac ttg gag cat aca ttg gat tac 1824
Tyr Asn Pro Gly Asp Ile Glu Ser Asn Leu Glu His Thr Leu Asp Tyr
595 600 605
ctc aag aaa tcc aat gtg atc gag gtt gac agt gaa gga tat gtc gga 1872
Leu Lys Lys Ser Asn Val Ile Glu Val Asp Ser Glu Gly Tyr Val Gly
610 615 620
ctc tct gat gct gaa cgc agc aag ggc cga gag aac tat gac ttt tat 1920
Leu Ser Asp Ala Glu Arg Ser Lys Gly Arg Glu Asn Tyr Asp Phe Tyr
625 630 635 640
tgt ttc ctg ctc tgg ccc ttc gtg gag aca tac tgg ctc gca gcc gtg 1968
Cys Phe Leu Leu Trp Pro Phe Val Glu Thr Tyr Trp Leu Ala Ala Val
645 650 655
tcc ctg tat acc ctg att ccc acc gcc aaa gag ttg act cag cag ttg 2016
Ser Leu Tyr Thr Leu Ile Pro Thr Ala Lys Glu Leu Thr Gln Gln Leu
660 665 670
gac agc aac gga gag cct cag gtt cac tgg gtt gag gag cgc gtg ttc 2064
Asp Ser Asn Gly Glu Pro Gln Val His Trp Val Glu Glu Arg Val Phe
675 680 685
atg gag aag acg caa atg ttc gga aag acg ctt tac tac cag gga gac 2112
Met Glu Lys Thr Gln Met Phe Gly Lys Thr Leu Tyr Tyr Gln Gly Asp
690 695 700
ctc tcc tac ttt gag tct gtc aac atg gag acg ctc aag aat ggt ttt 2160
Leu Ser Tyr Phe Glu Ser Val Asn Met Glu Thr Leu Lys Asn Gly Phe
705 710 715 720
aat cgt ctg tgc gat tat ggc atc ctt atg atg aag cga ccc acc aat 2208
Asn Arg Leu Cys Asp Tyr Gly Ile Leu Met Met Lys Arg Pro Thr Asn
725 730 735
gcc aag gag aag aca aag gtt gct ctc cac cct gat ttt atg cca agc 2256
Ala Lys Glu Lys Thr Lys Val Ala Leu His Pro Asp Phe Met Pro Ser
740 745 750
cga ggt gct gac ggt cat gtc att gcc agc ggc gca ctt tgg gat atg 2304
Arg Gly Ala Asp Gly His Val Ile Ala Ser Gly Ala Leu Trp Asp Met
755 760 765
gtc gaa cat atc ggc acg ttc aga cgt gaa ggc aag aat cgt cgt gat 2352
Val Glu His Ile Gly Thr Phe Arg Arg Glu Gly Lys Asn Arg Arg Asp
770 775 780
aac gcc aca gtt tcc tcc cgt gtc ctg cgg ttt gca gag gtc gtc gcg 2400
Asn Ala Thr Val Ser Ser Arg Val Leu Arg Phe Ala Glu Val Val Ala
785 790 795 800
aac gct cca gct ccg gtt aag gta ccc ttg ccc aat ccg gca ccc aaa 2448
Asn Ala Pro Ala Pro Val Lys Val Pro Leu Pro Asn Pro Ala Pro Lys
805 810 815
agg aca ggc gat ggc gcc ccg aaa tta 2475
Arg Thr Gly Asp Gly Ala Pro Lys Leu
820 825
<210> 2
<211> 825
<212> PRT
<213> Mortierella alpina
<400> 2
Met Pro Ile Val Pro Ala Gln Gln Val Asp Ser Ser Ser Cys Pro Pro
1 5 10 15
Ser Gly Glu Ser Ser Pro Leu Ile Pro Ser Leu Gly Glu Gly Val His
20 25 30
Ser Gly His Gly His Val Val Asp Asn Asp Glu Ser Gly Val Glu Asn
35 40 45
Ile Thr Lys Lys His Ala Gly Arg Ile Arg Glu Asp Pro Val Gly Phe
50 55 60
Val Val Gln Thr Ala Ala Phe Tyr Gln Gly Thr Gly Trp Arg Ser Tyr
65 70 75 80
Ser Asn Tyr Val Gly Thr Arg Ile Leu Tyr Glu Gly Phe Ser Ala Thr
85 90 95
Phe Lys Glu Arg Ile Leu Ala Ser Ser Lys Val Asn Asp Leu Ile Lys
100 105 110
Asp Met Ala Asn Lys Gln Leu Asp Val Leu Ile Lys Gln Arg Gln Asp
115 120 125
Ala Tyr Asp Ala Glu Arg Thr Ala Asn Ala Gly Lys Lys Asn Phe Lys
130 135 140
Pro Lys Val Arg Leu Leu Arg Pro Glu Asp Ile Glu Ala Arg Arg Lys
145 150 155 160
Thr Leu Glu Ala Glu Leu Val Ala Val Ala Lys Ser Asn Ile Asp Lys
165 170 175
Leu Val Cys Asp Met Asn Ser Met Lys Phe Ile Arg Phe Phe Ala Phe
180 185 190
Leu Ile Asn Asn Ile Leu Val Arg Met Tyr His Gln Gly Ile His Ile
195 200 205
Lys Glu Ser Glu Phe Leu Glu Leu Arg Arg Ile Ala Glu Tyr Cys Ala
210 215 220
Glu Lys Lys Tyr Ser Met Val Val Leu Pro Cys His Lys Ser His Ile
225 230 235 240
Asp Tyr Leu Val Val Ser Tyr Ile Phe Phe Arg Met Gly Leu Ala Leu
245 250 255
Pro His Ile Ala Ala Gly Asp Asn Leu Asp Met Pro Ile Val Gly Lys
260 265 270
Ala Leu Lys Gly Ala Gly Ala Phe Phe Ile Arg Arg Ser Trp Ala Asp
275 280 285
Asp Gln Leu Tyr Thr Ser Ile Val Gln Glu Tyr Val Gln Glu Leu Leu
290 295 300
Glu Gly Gly Tyr Asn Ile Glu Cys Phe Ile Glu Gly Thr Arg Ser Arg
305 310 315 320
Thr Gly Lys Leu Leu Pro Pro Lys Leu Gly Val Leu Lys Ile Ile Met
325 330 335
Asp Ala Met Leu Ser Asn Arg Ile Gln Asp Cys Tyr Ile Val Pro Ile
340 345 350
Ser Ile Gly Tyr Asp Lys Val Ile Glu Thr Glu Thr Tyr Ile Asn Glu
355 360 365
Leu Leu Gly Ile Pro Lys Glu Lys Glu Ser Leu Trp Gly Val Ile Thr
370 375 380
Asn Ser Arg Leu Leu Gln Leu Lys Met Gly Arg Ile Asp Val Arg Phe
385 390 395 400
Ala Lys Pro Tyr Ser Leu Arg Asn Phe Met Asn His Glu Ile Glu Arg
405 410 415
Arg Glu Ile Ile Asn Lys Arg Glu Asp Thr Asp Ser Val Ala Lys Ser
420 425 430
Gln Leu Leu Lys Ala Leu Gly Tyr Lys Val Leu Ala Asp Ile Asn Ser
435 440 445
Val Ser Val Val Met Pro Thr Ala Leu Val Gly Thr Val Ile Leu Thr
450 455 460
Leu Arg Gly Arg Gly Val Gly Arg Asn Glu Leu Ile Arg Arg Val Glu
465 470 475 480
Trp Leu Lys Arg Glu Ile Leu Ser Lys Gly Gly Arg Val Ala Asn Phe
485 490 495
Ser Gly Met Glu Thr Gly Glu Val Val Asp Arg Ala Leu Gly Val Leu
500 505 510
Lys Asp Leu Val Ala Leu Gln Lys Asn Leu Leu Glu Pro Val Phe Tyr
515 520 525
Ala Val Lys Arg Phe Glu Leu Ser Phe Tyr Arg Asn Gln Leu Ile His
530 535 540
Leu Phe Val His Glu Ala Ile Ile Ala Val Thr Met Tyr Thr Arg Ile
545 550 555 560
Lys Ile Gly Gly Ala Lys Ser Thr Gln His Ile Ser Gln Asn Glu Leu
565 570 575
Leu Asn Glu Val Thr Phe Leu Ser Arg Leu Leu Lys Thr Asp Phe Ile
580 585 590
Tyr Asn Pro Gly Asp Ile Glu Ser Asn Leu Glu His Thr Leu Asp Tyr
595 600 605
Leu Lys Lys Ser Asn Val Ile Glu Val Asp Ser Glu Gly Tyr Val Gly
610 615 620
Leu Ser Asp Ala Glu Arg Ser Lys Gly Arg Glu Asn Tyr Asp Phe Tyr
625 630 635 640
Cys Phe Leu Leu Trp Pro Phe Val Glu Thr Tyr Trp Leu Ala Ala Val
645 650 655
Ser Leu Tyr Thr Leu Ile Pro Thr Ala Lys Glu Leu Thr Gln Gln Leu
660 665 670
Asp Ser Asn Gly Glu Pro Gln Val His Trp Val Glu Glu Arg Val Phe
675 680 685
Met Glu Lys Thr Gln Met Phe Gly Lys Thr Leu Tyr Tyr Gln Gly Asp
690 695 700
Leu Ser Tyr Phe Glu Ser Val Asn Met Glu Thr Leu Lys Asn Gly Phe
705 710 715 720
Asn Arg Leu Cys Asp Tyr Gly Ile Leu Met Met Lys Arg Pro Thr Asn
725 730 735
Ala Lys Glu Lys Thr Lys Val Ala Leu His Pro Asp Phe Met Pro Ser
740 745 750
Arg Gly Ala Asp Gly His Val Ile Ala Ser Gly Ala Leu Trp Asp Met
755 760 765
Val Glu His Ile Gly Thr Phe Arg Arg Glu Gly Lys Asn Arg Arg Asp
770 775 780
Asn Ala Thr Val Ser Ser Arg Val Leu Arg Phe Ala Glu Val Val Ala
785 790 795 800
Asn Ala Pro Ala Pro Val Lys Val Pro Leu Pro Asn Pro Ala Pro Lys
805 810 815
Arg Thr Gly Asp Gly Ala Pro Lys Leu
820 825
<210> 3
<211> 2478
<212> ДНК
<213> Mortierella alpina
<400> 3
atgcccatcg ttccagctca gcaagtcgac tcctcctcgt gccctccctc tggtgaatct 60
agcccgttga ttcccagttt gggcgaagga gtgcattcgg gtcatggaca cgttgtcgac 120
aatgacgagt ccggcgtgga gaacattacc aaaaagcacg caggacgaat tagagaagat 180
ccggtcggct tcgtggtgca gactgcagct ttctatcagg gcacgggttg gagaagctac 240
agcaactatg ttggaacgcg cattctttac gaaggcttct ctgcaacctt caaggagcga 300
attctcgcca gttcaaaagt gaatgatctc atcaaggaca tggccaacaa gcagttggat 360
gtcctgatca agcaaagaca agatgcgtat gacgcagaga ggactgcaaa tgcaggaaaa 420
aagaacttca agcccaaagt tcgactcctt cgcccagaag atatcgaggc tcgtcgcaaa 480
acattagaag ccgagcttgt tgcggtggca aagtcaaata tcgacaaact tgtttgtgat 540
atgaacagta tgaaattcat caggttcttc gccttcctca tcaacaacat ccttgtgaga 600
atgtaccatc aaggaattca catcaaggag tccgagttct tggagctgcg gaggatagct 660
gagtactgcg cagagaaaaa gtattcgatg gtggtgttgc catgccacaa gtcacacatc 720
gactacctcg tcgtctcgta cattttcttc cgcatgggat tagctttacc tcacattgct 780
gctggcgata acctggacat gcccattgtc ggaaaggcac tcaaaggagc aggcgcgttc 840
ttcattcgcc gttcttgggc tgacgatcaa ctttacacca gcattgttca ggaatatgtt 900
caggagcttt tggagggagg atacaatatc gagtgcttca tcgagggcac ccgaagcaga 960
acaggaaaac ttttgccacc aaagctggga gtcctaaaga ttatcatgga tgctatgctt 1020
tcgaaccgca tccaagactg ctacatcgtg cccatctcta tcggttatga caaggtcatc 1080
gaaaccgaga cttatatcaa tgagcttctc ggaatcccca aggaaaagga gagtttgtgg 1140
ggtgttatta cgaattcgag gctgctccag ctcaagatgg gccgcattga tgtccgattt 1200
gcaaagccgt acagtttgcg aaactttatg aatcatgaga tcgagcgcag agagatcatc 1260
aataagcggg aagacaccga tagtgtggcg aaatctcagc tgctaaaggc attgggctac 1320
aaggtcttgg cagacatcaa ctcggtctct gtagtaatgc ccacggccct cgtgggtact 1380
gtcatcctta cactccgagg acgaggtgtt ggccgtaatg agctgatccg tcgtgttgag 1440
tggctgaagc gcgagattct ttccaagggt ggtcgcgttg ccaactttag cgggatggaa 1500
actggcgagg ttgtagatcg agcattgggc gttcttaagg accttgtggc gctgcagaag 1560
aatttgctcg agcccgtctt ctatgcggtc aagcgcttcg agctttcgtt ctacaggaat 1620
cagctcatcc acctctttgt ccatgaggcc atcatcgccg tgacgatgta cacccgcatc 1680
aagattggtg gcgccaagtc tacacaacac attagtcaga atgagctgct gaacgaggtc 1740
accttcctga gccgcctgct caagaccgac tttatctaca accctggcga tattgagagt 1800
aacttggagc atacattgga ttacctcaag aaatccaatg tgatcgaggt tgacagtgaa 1860
ggatatgtcg gactctctga tgctgaacgc agcaagggcc gagagaacta tgacttttat 1920
tgtttcctgc tctggccctt cgtggagaca tactggctcg cagccgtgtc cctgtatacc 1980
ctgattccca ccgccaaaga gttgactcag cagttggaca gcaacggaga gcctcaggtt 2040
cactgggttg aggagcgcgt gttcatggag aagacgcaaa tgttcggaaa gacgctttac 2100
taccagggag acctctccta ctttgagtct gtcaacatgg agacgctcaa gaatggtttt 2160
aatcgtctgt gcgattatgg catccttatg atgaagcgac ccaccaatgc caaggagaag 2220
acaaaggttg ctctccaccc tgattttatg ccaagccgag gtgctgacgg tcatgtcatt 2280
gccagcggcg cactttggga tatggtcgaa catatcggca cgttcagacg tgaaggcaag 2340
aatcgtcgtg ataacgccac agtttcctcc cgtgtcctgc ggtttgcaga ggtcgtcgcg 2400
aacgctccag ctccggttaa ggtacccttg cccaatccgg cacccaaaag gacaggcgat 2460
ggcgccccga aattataa 2478
<210> 4
<211> 2643
<212> ДНК
<213> Mortierella alpina
<220>
<221> CDS
<222> (1)..(2643)
<223>
<400> 4
atg aca acc ggc gac agt acc gct gct gac ggt agc agc agc agt agc 48
Met Thr Thr Gly Asp Ser Thr Ala Ala Asp Gly Ser Ser Ser Ser Ser
1 5 10 15
agc aac aac agc acc aac att gcc agt act agt aac ggc aag gct gct 96
Ser Asn Asn Ser Thr Asn Ile Ala Ser Thr Ser Asn Gly Lys Ala Ala
20 25 30
ccc cac cca ctc caa ggg gga tca ccc gct cct gta gct cca gtt ttg 144
Pro His Pro Leu Gln Gly Gly Ser Pro Ala Pro Val Ala Pro Val Leu
35 40 45
gaa ttg aag cct ctc aag aat gtt atg ccc atc gtt cca gct cag caa 192
Glu Leu Lys Pro Leu Lys Asn Val Met Pro Ile Val Pro Ala Gln Gln
50 55 60
gtc gac tcc tcc tcg tgc cct ccc tct ggt gaa tct agc ccg ttg att 240
Val Asp Ser Ser Ser Cys Pro Pro Ser Gly Glu Ser Ser Pro Leu Ile
65 70 75 80
ccc agt ttg ggc gaa gga gtg cat tcg ggt cat gga cac gtt gtc gac 288
Pro Ser Leu Gly Glu Gly Val His Ser Gly His Gly His Val Val Asp
85 90 95
aat gac gag tcc ggc gtg gag aac att acc aaa aag cac gca gga cga 336
Asn Asp Glu Ser Gly Val Glu Asn Ile Thr Lys Lys His Ala Gly Arg
100 105 110
att aga gaa gat ccg gtc ggc ttc gtg gtg cag act gca gct ttc tat 384
Ile Arg Glu Asp Pro Val Gly Phe Val Val Gln Thr Ala Ala Phe Tyr
115 120 125
cag ggc acg ggt tgg aga agc tac agc aac tat gtt gga acg cgc att 432
Gln Gly Thr Gly Trp Arg Ser Tyr Ser Asn Tyr Val Gly Thr Arg Ile
130 135 140
ctt tac gaa ggc ttc tct gca acc ttc aag gag cga att ctc gcc agt 480
Leu Tyr Glu Gly Phe Ser Ala Thr Phe Lys Glu Arg Ile Leu Ala Ser
145 150 155 160
tca aaa gtg aat gat ctc atc aag gac atg gcc aac aag cag ttg gat 528
Ser Lys Val Asn Asp Leu Ile Lys Asp Met Ala Asn Lys Gln Leu Asp
165 170 175
gtc ctg atc aag caa aga caa gat gcg tat gac gca gag agg act gca 576
Val Leu Ile Lys Gln Arg Gln Asp Ala Tyr Asp Ala Glu Arg Thr Ala
180 185 190
aat gca gga aaa aag aac ttc aag ccc aaa gtt cga ctc ctt cgc cca 624
Asn Ala Gly Lys Lys Asn Phe Lys Pro Lys Val Arg Leu Leu Arg Pro
195 200 205
gaa gat atc gag gct cgt cgc aaa aca tta gaa gcc gag ctt gtt gcg 672
Glu Asp Ile Glu Ala Arg Arg Lys Thr Leu Glu Ala Glu Leu Val Ala
210 215 220
gtg gca aag tca aat atc gac aaa ctt gtt tgt gat atg aac agt atg 720
Val Ala Lys Ser Asn Ile Asp Lys Leu Val Cys Asp Met Asn Ser Met
225 230 235 240
aaa ttc atc agg ttc ttc gcc ttc ctc atc aac aac atc ctt gtg aga 768
Lys Phe Ile Arg Phe Phe Ala Phe Leu Ile Asn Asn Ile Leu Val Arg
245 250 255
atg tac cat caa gga att cac atc aag gag tcc gag ttc ttg gag ctg 816
Met Tyr His Gln Gly Ile His Ile Lys Glu Ser Glu Phe Leu Glu Leu
260 265 270
cgg agg ata gct gag tac tgc gca gag aaa aag tat tcg atg gtg gtg 864
Arg Arg Ile Ala Glu Tyr Cys Ala Glu Lys Lys Tyr Ser Met Val Val
275 280 285
ttg cca tgc cac aag tca cac atc gac tac ctc gtc gtc tcg tac att 912
Leu Pro Cys His Lys Ser His Ile Asp Tyr Leu Val Val Ser Tyr Ile
290 295 300
ttc ttc cgc atg gga tta gct tta cct cac att gct gct ggc gat aac 960
Phe Phe Arg Met Gly Leu Ala Leu Pro His Ile Ala Ala Gly Asp Asn
305 310 315 320
ctg gac atg ccc att gtc gga aag gca ctc aaa gga gca ggc gcg ttc 1008
Leu Asp Met Pro Ile Val Gly Lys Ala Leu Lys Gly Ala Gly Ala Phe
325 330 335
ttc att cgc cgt tct tgg gct gac gat caa ctt tac acc agc att gtt 1056
Phe Ile Arg Arg Ser Trp Ala Asp Asp Gln Leu Tyr Thr Ser Ile Val
340 345 350
cag gaa tat gtt cag gag ctt ttg gag gga gga tac aat atc gag tgc 1104
Gln Glu Tyr Val Gln Glu Leu Leu Glu Gly Gly Tyr Asn Ile Glu Cys
355 360 365
ttc atc gag ggc acc cga agc aga aca gga aaa ctt ttg cca cca aag 1152
Phe Ile Glu Gly Thr Arg Ser Arg Thr Gly Lys Leu Leu Pro Pro Lys
370 375 380
ctg gga gtc cta aag att atc atg gat gct atg ctt tcg aac cgc atc 1200
Leu Gly Val Leu Lys Ile Ile Met Asp Ala Met Leu Ser Asn Arg Ile
385 390 395 400
caa gac tgc tac atc gtg ccc atc tct atc ggt tat gac aag gtc atc 1248
Gln Asp Cys Tyr Ile Val Pro Ile Ser Ile Gly Tyr Asp Lys Val Ile
405 410 415
gaa acc gag act tat atc aat gag ctt ctc gga atc ccc aag gaa aag 1296
Glu Thr Glu Thr Tyr Ile Asn Glu Leu Leu Gly Ile Pro Lys Glu Lys
420 425 430
gag agt ttg tgg ggt gtt att acg aat tcg agg ctg ctc cag ctc aag 1344
Glu Ser Leu Trp Gly Val Ile Thr Asn Ser Arg Leu Leu Gln Leu Lys
435 440 445
atg ggc cgc att gat gtc cga ttt gca aag ccg tac agt ttg cga aac 1392
Met Gly Arg Ile Asp Val Arg Phe Ala Lys Pro Tyr Ser Leu Arg Asn
450 455 460
ttt atg aat cat gag atc gag cgc aga gag atc atc aat aag cgg gaa 1440
Phe Met Asn His Glu Ile Glu Arg Arg Glu Ile Ile Asn Lys Arg Glu
465 470 475 480
gac acc gat agt gtg gcg aaa tct cag ctg cta aag gca ttg ggc tac 1488
Asp Thr Asp Ser Val Ala Lys Ser Gln Leu Leu Lys Ala Leu Gly Tyr
485 490 495
aag gtc ttg gca gac atc aac tcg gtc tct gta gta atg ccc acg gcc 1536
Lys Val Leu Ala Asp Ile Asn Ser Val Ser Val Val Met Pro Thr Ala
500 505 510
ctc gtg ggt act gtc atc ctt aca ctc cga gga cga ggt gtt ggc cgt 1584
Leu Val Gly Thr Val Ile Leu Thr Leu Arg Gly Arg Gly Val Gly Arg
515 520 525
aat gag ctg atc cgt cgt gtt gag tgg ctg aag cgc gag att ctt tcc 1632
Asn Glu Leu Ile Arg Arg Val Glu Trp Leu Lys Arg Glu Ile Leu Ser
530 535 540
aag ggt ggt cgc gtt gcc aac ttt agc ggg atg gaa act ggc gag gtt 1680
Lys Gly Gly Arg Val Ala Asn Phe Ser Gly Met Glu Thr Gly Glu Val
545 550 555 560
gta gat cga gca ttg ggc gtt ctt aag gac ctt gtg gcg ctg cag aag 1728
Val Asp Arg Ala Leu Gly Val Leu Lys Asp Leu Val Ala Leu Gln Lys
565 570 575
aat ttg ctc gag ccc gtc ttc tat gcg gtc aag cgc ttc gag ctt tcg 1776
Asn Leu Leu Glu Pro Val Phe Tyr Ala Val Lys Arg Phe Glu Leu Ser
580 585 590
ttc tac agg aat cag ctc atc cac ctc ttt gtc cat gag gcc atc atc 1824
Phe Tyr Arg Asn Gln Leu Ile His Leu Phe Val His Glu Ala Ile Ile
595 600 605
gcc gtg acg atg tac acc cgc atc aag att ggt ggc gcc aag tct aca 1872
Ala Val Thr Met Tyr Thr Arg Ile Lys Ile Gly Gly Ala Lys Ser Thr
610 615 620
caa cac att agt cag aat gag ctg ctg aac gag gtc acc ttc ctg agc 1920
Gln His Ile Ser Gln Asn Glu Leu Leu Asn Glu Val Thr Phe Leu Ser
625 630 635 640
cgc ctg ctc aag acc gac ttt atc tac aac cct ggc gat att gag agt 1968
Arg Leu Leu Lys Thr Asp Phe Ile Tyr Asn Pro Gly Asp Ile Glu Ser
645 650 655
aac ttg gag cat aca ttg gat tac ctc aag aaa tcc aat gtg atc gag 2016
Asn Leu Glu His Thr Leu Asp Tyr Leu Lys Lys Ser Asn Val Ile Glu
660 665 670
gtt gac agt gaa gga tat gtc gga ctc tct gat gct gaa cgc agc aag 2064
Val Asp Ser Glu Gly Tyr Val Gly Leu Ser Asp Ala Glu Arg Ser Lys
675 680 685
ggc cga gag aac tat gac ttt tat tgt ttc ctg ctc tgg ccc ttc gtg 2112
Gly Arg Glu Asn Tyr Asp Phe Tyr Cys Phe Leu Leu Trp Pro Phe Val
690 695 700
gag aca tac tgg ctc gca gcc gtg tcc ctg tat acc ctg att ccc acc 2160
Glu Thr Tyr Trp Leu Ala Ala Val Ser Leu Tyr Thr Leu Ile Pro Thr
705 710 715 720
gcc aaa gag ttg act cag cag ttg gac agc aac gga gag cct cag gtt 2208
Ala Lys Glu Leu Thr Gln Gln Leu Asp Ser Asn Gly Glu Pro Gln Val
725 730 735
cac tgg gtt gag gag cgc gtg ttc atg gag aag acg caa atg ttc gga 2256
His Trp Val Glu Glu Arg Val Phe Met Glu Lys Thr Gln Met Phe Gly
740 745 750
aag acg ctt tac tac cag gga gac ctc tcc tac ttt gag tct gtc aac 2304
Lys Thr Leu Tyr Tyr Gln Gly Asp Leu Ser Tyr Phe Glu Ser Val Asn
755 760 765
atg gag acg ctc aag aat ggt ttt aat cgt ctg tgc gat tat ggc atc 2352
Met Glu Thr Leu Lys Asn Gly Phe Asn Arg Leu Cys Asp Tyr Gly Ile
770 775 780
ctt atg atg aag cga ccc acc aat gcc aag gag aag aca aag gtt gct 2400
Leu Met Met Lys Arg Pro Thr Asn Ala Lys Glu Lys Thr Lys Val Ala
785 790 795 800
ctc cac cct gat ttt atg cca agc cga ggt gct gac ggt cat gtc att 2448
Leu His Pro Asp Phe Met Pro Ser Arg Gly Ala Asp Gly His Val Ile
805 810 815
gcc agc ggc gca ctt tgg gat atg gtc gaa cat atc ggc acg ttc aga 2496
Ala Ser Gly Ala Leu Trp Asp Met Val Glu His Ile Gly Thr Phe Arg
820 825 830
cgt gaa ggc aag aat cgt cgt gat aac gcc aca gtt tcc tcc cgt gtc 2544
Arg Glu Gly Lys Asn Arg Arg Asp Asn Ala Thr Val Ser Ser Arg Val
835 840 845
ctg cgg ttt gca gag gtc gtc gcg aac gct cca gct ccg gtt aag gta 2592
Leu Arg Phe Ala Glu Val Val Ala Asn Ala Pro Ala Pro Val Lys Val
850 855 860
ccc ttg ccc aat ccg gca ccc aaa agg aca ggc gat ggc gcc ccg aaa 2640
Pro Leu Pro Asn Pro Ala Pro Lys Arg Thr Gly Asp Gly Ala Pro Lys
865 870 875 880
tta 2643
Leu
<210> 5
<211> 881
<212> PRT
<213> Mortierella alpina
<400> 5
Met Thr Thr Gly Asp Ser Thr Ala Ala Asp Gly Ser Ser Ser Ser Ser
1 5 10 15
Ser Asn Asn Ser Thr Asn Ile Ala Ser Thr Ser Asn Gly Lys Ala Ala
20 25 30
Pro His Pro Leu Gln Gly Gly Ser Pro Ala Pro Val Ala Pro Val Leu
35 40 45
Glu Leu Lys Pro Leu Lys Asn Val Met Pro Ile Val Pro Ala Gln Gln
50 55 60
Val Asp Ser Ser Ser Cys Pro Pro Ser Gly Glu Ser Ser Pro Leu Ile
65 70 75 80
Pro Ser Leu Gly Glu Gly Val His Ser Gly His Gly His Val Val Asp
85 90 95
Asn Asp Glu Ser Gly Val Glu Asn Ile Thr Lys Lys His Ala Gly Arg
100 105 110
Ile Arg Glu Asp Pro Val Gly Phe Val Val Gln Thr Ala Ala Phe Tyr
115 120 125
Gln Gly Thr Gly Trp Arg Ser Tyr Ser Asn Tyr Val Gly Thr Arg Ile
130 135 140
Leu Tyr Glu Gly Phe Ser Ala Thr Phe Lys Glu Arg Ile Leu Ala Ser
145 150 155 160
Ser Lys Val Asn Asp Leu Ile Lys Asp Met Ala Asn Lys Gln Leu Asp
165 170 175
Val Leu Ile Lys Gln Arg Gln Asp Ala Tyr Asp Ala Glu Arg Thr Ala
180 185 190
Asn Ala Gly Lys Lys Asn Phe Lys Pro Lys Val Arg Leu Leu Arg Pro
195 200 205
Glu Asp Ile Glu Ala Arg Arg Lys Thr Leu Glu Ala Glu Leu Val Ala
210 215 220
Val Ala Lys Ser Asn Ile Asp Lys Leu Val Cys Asp Met Asn Ser Met
225 230 235 240
Lys Phe Ile Arg Phe Phe Ala Phe Leu Ile Asn Asn Ile Leu Val Arg
245 250 255
Met Tyr His Gln Gly Ile His Ile Lys Glu Ser Glu Phe Leu Glu Leu
260 265 270
Arg Arg Ile Ala Glu Tyr Cys Ala Glu Lys Lys Tyr Ser Met Val Val
275 280 285
Leu Pro Cys His Lys Ser His Ile Asp Tyr Leu Val Val Ser Tyr Ile
290 295 300
Phe Phe Arg Met Gly Leu Ala Leu Pro His Ile Ala Ala Gly Asp Asn
305 310 315 320
Leu Asp Met Pro Ile Val Gly Lys Ala Leu Lys Gly Ala Gly Ala Phe
325 330 335
Phe Ile Arg Arg Ser Trp Ala Asp Asp Gln Leu Tyr Thr Ser Ile Val
340 345 350
Gln Glu Tyr Val Gln Glu Leu Leu Glu Gly Gly Tyr Asn Ile Glu Cys
355 360 365
Phe Ile Glu Gly Thr Arg Ser Arg Thr Gly Lys Leu Leu Pro Pro Lys
370 375 380
Leu Gly Val Leu Lys Ile Ile Met Asp Ala Met Leu Ser Asn Arg Ile
385 390 395 400
Gln Asp Cys Tyr Ile Val Pro Ile Ser Ile Gly Tyr Asp Lys Val Ile
405 410 415
Glu Thr Glu Thr Tyr Ile Asn Glu Leu Leu Gly Ile Pro Lys Glu Lys
420 425 430
Glu Ser Leu Trp Gly Val Ile Thr Asn Ser Arg Leu Leu Gln Leu Lys
435 440 445
Met Gly Arg Ile Asp Val Arg Phe Ala Lys Pro Tyr Ser Leu Arg Asn
450 455 460
Phe Met Asn His Glu Ile Glu Arg Arg Glu Ile Ile Asn Lys Arg Glu
465 470 475 480
Asp Thr Asp Ser Val Ala Lys Ser Gln Leu Leu Lys Ala Leu Gly Tyr
485 490 495
Lys Val Leu Ala Asp Ile Asn Ser Val Ser Val Val Met Pro Thr Ala
500 505 510
Leu Val Gly Thr Val Ile Leu Thr Leu Arg Gly Arg Gly Val Gly Arg
515 520 525
Asn Glu Leu Ile Arg Arg Val Glu Trp Leu Lys Arg Glu Ile Leu Ser
530 535 540
Lys Gly Gly Arg Val Ala Asn Phe Ser Gly Met Glu Thr Gly Glu Val
545 550 555 560
Val Asp Arg Ala Leu Gly Val Leu Lys Asp Leu Val Ala Leu Gln Lys
565 570 575
Asn Leu Leu Glu Pro Val Phe Tyr Ala Val Lys Arg Phe Glu Leu Ser
580 585 590
Phe Tyr Arg Asn Gln Leu Ile His Leu Phe Val His Glu Ala Ile Ile
595 600 605
Ala Val Thr Met Tyr Thr Arg Ile Lys Ile Gly Gly Ala Lys Ser Thr
610 615 620
Gln His Ile Ser Gln Asn Glu Leu Leu Asn Glu Val Thr Phe Leu Ser
625 630 635 640
Arg Leu Leu Lys Thr Asp Phe Ile Tyr Asn Pro Gly Asp Ile Glu Ser
645 650 655
Asn Leu Glu His Thr Leu Asp Tyr Leu Lys Lys Ser Asn Val Ile Glu
660 665 670
Val Asp Ser Glu Gly Tyr Val Gly Leu Ser Asp Ala Glu Arg Ser Lys
675 680 685
Gly Arg Glu Asn Tyr Asp Phe Tyr Cys Phe Leu Leu Trp Pro Phe Val
690 695 700
Glu Thr Tyr Trp Leu Ala Ala Val Ser Leu Tyr Thr Leu Ile Pro Thr
705 710 715 720
Ala Lys Glu Leu Thr Gln Gln Leu Asp Ser Asn Gly Glu Pro Gln Val
725 730 735
His Trp Val Glu Glu Arg Val Phe Met Glu Lys Thr Gln Met Phe Gly
740 745 750
Lys Thr Leu Tyr Tyr Gln Gly Asp Leu Ser Tyr Phe Glu Ser Val Asn
755 760 765
Met Glu Thr Leu Lys Asn Gly Phe Asn Arg Leu Cys Asp Tyr Gly Ile
770 775 780
Leu Met Met Lys Arg Pro Thr Asn Ala Lys Glu Lys Thr Lys Val Ala
785 790 795 800
Leu His Pro Asp Phe Met Pro Ser Arg Gly Ala Asp Gly His Val Ile
805 810 815
Ala Ser Gly Ala Leu Trp Asp Met Val Glu His Ile Gly Thr Phe Arg
820 825 830
Arg Glu Gly Lys Asn Arg Arg Asp Asn Ala Thr Val Ser Ser Arg Val
835 840 845
Leu Arg Phe Ala Glu Val Val Ala Asn Ala Pro Ala Pro Val Lys Val
850 855 860
Pro Leu Pro Asn Pro Ala Pro Lys Arg Thr Gly Asp Gly Ala Pro Lys
865 870 875 880
Leu
<210> 6
<211> 2646
<212> ДНК
<213> Mortierella alpina
<400> 6
atgacaaccg gcgacagtac cgctgctgac ggtagcagca gcagtagcag caacaacagc 60
accaacattg ccagtactag taacggcaag gctgctcccc acccactcca agggggatca 120
cccgctcctg tagctccagt tttggaattg aagcctctca agaatgttat gcccatcgtt 180
ccagctcagc aagtcgactc ctcctcgtgc cctccctctg gtgaatctag cccgttgatt 240
cccagtttgg gcgaaggagt gcattcgggt catggacacg ttgtcgacaa tgacgagtcc 300
ggcgtggaga acattaccaa aaagcacgca ggacgaatta gagaagatcc ggtcggcttc 360
gtggtgcaga ctgcagcttt ctatcagggc acgggttgga gaagctacag caactatgtt 420
ggaacgcgca ttctttacga aggcttctct gcaaccttca aggagcgaat tctcgccagt 480
tcaaaagtga atgatctcat caaggacatg gccaacaagc agttggatgt cctgatcaag 540
caaagacaag atgcgtatga cgcagagagg actgcaaatg caggaaaaaa gaacttcaag 600
cccaaagttc gactccttcg cccagaagat atcgaggctc gtcgcaaaac attagaagcc 660
gagcttgttg cggtggcaaa gtcaaatatc gacaaacttg tttgtgatat gaacagtatg 720
aaattcatca ggttcttcgc cttcctcatc aacaacatcc ttgtgagaat gtaccatcaa 780
ggaattcaca tcaaggagtc cgagttcttg gagctgcgga ggatagctga gtactgcgca 840
gagaaaaagt attcgatggt ggtgttgcca tgccacaagt cacacatcga ctacctcgtc 900
gtctcgtaca ttttcttccg catgggatta gctttacctc acattgctgc tggcgataac 960
ctggacatgc ccattgtcgg aaaggcactc aaaggagcag gcgcgttctt cattcgccgt 1020
tcttgggctg acgatcaact ttacaccagc attgttcagg aatatgttca ggagcttttg 1080
gagggaggat acaatatcga gtgcttcatc gagggcaccc gaagcagaac aggaaaactt 1140
ttgccaccaa agctgggagt cctaaagatt atcatggatg ctatgctttc gaaccgcatc 1200
caagactgct acatcgtgcc catctctatc ggttatgaca aggtcatcga aaccgagact 1260
tatatcaatg agcttctcgg aatccccaag gaaaaggaga gtttgtgggg tgttattacg 1320
aattcgaggc tgctccagct caagatgggc cgcattgatg tccgatttgc aaagccgtac 1380
agtttgcgaa actttatgaa tcatgagatc gagcgcagag agatcatcaa taagcgggaa 1440
gacaccgata gtgtggcgaa atctcagctg ctaaaggcat tgggctacaa ggtcttggca 1500
gacatcaact cggtctctgt agtaatgccc acggccctcg tgggtactgt catccttaca 1560
ctccgaggac gaggtgttgg ccgtaatgag ctgatccgtc gtgttgagtg gctgaagcgc 1620
gagattcttt ccaagggtgg tcgcgttgcc aactttagcg ggatggaaac tggcgaggtt 1680
gtagatcgag cattgggcgt tcttaaggac cttgtggcgc tgcagaagaa tttgctcgag 1740
cccgtcttct atgcggtcaa gcgcttcgag ctttcgttct acaggaatca gctcatccac 1800
ctctttgtcc atgaggccat catcgccgtg acgatgtaca cccgcatcaa gattggtggc 1860
gccaagtcta cacaacacat tagtcagaat gagctgctga acgaggtcac cttcctgagc 1920
cgcctgctca agaccgactt tatctacaac cctggcgata ttgagagtaa cttggagcat 1980
acattggatt acctcaagaa atccaatgtg atcgaggttg acagtgaagg atatgtcgga 2040
ctctctgatg ctgaacgcag caagggccga gagaactatg acttttattg tttcctgctc 2100
tggcccttcg tggagacata ctggctcgca gccgtgtccc tgtataccct gattcccacc 2160
gccaaagagt tgactcagca gttggacagc aacggagagc ctcaggttca ctgggttgag 2220
gagcgcgtgt tcatggagaa gacgcaaatg ttcggaaaga cgctttacta ccagggagac 2280
ctctcctact ttgagtctgt caacatggag acgctcaaga atggttttaa tcgtctgtgc 2340
gattatggca tccttatgat gaagcgaccc accaatgcca aggagaagac aaaggttgct 2400
ctccaccctg attttatgcc aagccgaggt gctgacggtc atgtcattgc cagcggcgca 2460
ctttgggata tggtcgaaca tatcggcacg ttcagacgtg aaggcaagaa tcgtcgtgat 2520
aacgccacag tttcctcccg tgtcctgcgg tttgcagagg tcgtcgcgaa cgctccagct 2580
ccggttaagg tacccttgcc caatccggca cccaaaagga caggcgatgg cgccccgaaa 2640
ttataa 2646
<210> 7
<211> 4248
<212> ДНК
<213> Mortierella alpina
<400> 7
gttgacatag tctttttggt agcagtagca ttgttgtagt ttctcttact caacggacag 60
cagcagcacg accgccgagg taaagcggtg gatcccatag agactctttt gccctcgtct 120
ccactgtcgc tctccccaca gaaaaacatt tttatttcct ttcccgcatt tttttcttct 180
ttttttcctg cctctcttgt ctctcctccc ctaagcacca cctaacccgc tctatcgccc 240
aaccacttca actcagcacc ctcacggagc ctgccaacca acgttgcaag aaggaaaaaa 300
agtacagcgc atcttgtagt ccagcgcgga gcagtggaaa aacctctcca ttccgaccta 360
acagatcacg cagtctgatc agctagtttg ccccacagtt attgcatcac cctttcaacc 420
agtccacatg acaaccggcg acagtaccgc tgctgacggt agcagcagca gtagcagcaa 480
caacagcacc aacattgcca gtactagtaa cggcaaggct gctccccacc cactccaagg 540
gggatcaccc gctcctgtag ctccagtttt ggaattgaag cctctcaaga atgttatgcc 600
catcgttcca gctcagcaag tcgactcctc ctcgtgccct ccctctggtg aatctagccc 660
gttgattccc agtttgggcg aaggagtgca ttcgggtcat ggacacgttg tcgacaatga 720
cgagtccggc gtggagaaca ttacgcaagt tcaaattgaa tattccagcg gctatgaacg 780
agtttggatg cttgtttagt tttgtattaa cacgaacttt cgctcttgtt attgttgtta 840
tcaatgcagc aaaaagcacg caggacgaat tagagaagat ccggtcggct tcgtggtgca 900
gactgcagct ttctatcagg gcacggtacg ttcaatatct gacatcactg ctttagtgga 960
gccgcagagg aaagcacttc ttaacgacaa ctgcagctgg atcggtcggt acctcctgct 1020
caacgagcta tcagcccttc tccgccgcca ttgtgagggt gatttgtctc ataccaggaa 1080
cagcagaaga aaaagaggaa tcgtgttcac aaagcatcgc ttcggggttg cgctcacgct 1140
cggtcctctt gattgctccc tggaagtctc ccttctgcaa actgtcactc tcccacgttc 1200
cctttttttt ttttttttta ctatcctccc atcccctgcc tctcgtcatg cttgagctga 1260
ataatgctaa attcttatcc gcatatcgtc tttgcttttt agggttggag aagctacagc 1320
aactatgttg gaacgcgcat tctttacgaa ggcttctctg caaccttcaa ggagcgaatt 1380
ctcgccagtt caaaaggtaa cacaagatag ggtgctgtct gtacttcaac acgattcgtc 1440
aaaatggcat gatctaacga accctacctt gacctcctag tgaatgatct catcaaggac 1500
atggccaaca agcagttgga tgtcctgatc aagcaaagac aagatgcgta tgacgcagag 1560
aggactgcaa atgcaggaaa aaagaacttc aagcccaaag ttcgactcct tcgcccagaa 1620
gatatcgagg ctcgtcgcaa aacattagaa gccgagcttg ttgcggtggc aaagtcaaat 1680
atcgacaaac ttgtttgtga tatgaacagt atgaaattca tcaggtatgg accaacacga 1740
gaataacgca gcgtggaact gaacagtgtg gcagaagagg gatggcgaga tatatttgtt 1800
gtttgctaga caccagatgt taacaacttt cctccttggc tgatgtgtta ggttcttcgc 1860
cttcctcatc aacaacatcc ttgtgagaat gtaccatcaa ggaattcaca tcaaggagtc 1920
cgagttcttg gagctgcgga ggatagctga gtactgcgca gagaaaaagt attcgatggt 1980
ggtgttgcca tgccacaagt cacacatcga ctacctcgtc gtctcgtaca ttttcttccg 2040
catgggatta gctttacctc acattgctgc tggcgataac ctggacatgc ccattgtcgg 2100
aaaggcactc aaaggagcag gcgcgttctt cattcgccgt tcttgggctg acgatcaact 2160
ttacaccagc attgttcagg aatatgttca ggagcttttg gagggaggat acaatatcga 2220
gtgcttcatc gagggcaccc gaagcagaac aggaaaactt ttgccaccaa agctgggagg 2280
ttcgttcaca gctttggtct tgtttttgct actgggcacg ctggcgatct ctttgggtat 2340
taaccttcac accaatccac ctttactagt cctaaagatt atcatggatg ctatgctttc 2400
gaaccgcatc caagactgct acatcgtgcc catctctatc ggttatgaca aggtcatcga 2460
aaccgagact tatatcaatg agcttctcgg aatccccaag gaaaaggaga gtttgtgggg 2520
tgttattacg aattcgaggc tgctccagct caagatgggc cgcattgatg tccgatttgc 2580
aaagccgtac agtttgcgaa actttatgaa tcatgagatc gagcgcagag agtaagcaga 2640
acctgtgttt tgttgtgcaa gacgttttca aaactggaga ggaattatgt tgacccaggg 2700
ctatttgttt ttctgcattt aggatcatca ataagcggga agacaccgat agtgtggcga 2760
aatctcagct gctaaaggca ttgggctaca aggtcttggc agacatcaac tcggtctctg 2820
tagtaatgcc cacggccctc gtgggtactg tcatccttac actccgagga cgaggtgttg 2880
gccgtaatga gctgatccgt cgtgttgagt ggctgaagcg cgagattctt tccaagggtg 2940
gtcgcgttgc caactttagc gggatggaaa ctggcgaggt tgtagatcga gcattgggcg 3000
ttcttaagga ccttgtggcg ctgcagaaga atttgctcga gcccgtcttc tatgcggtca 3060
agcgcttcga gctttcgttc tacaggaatc agctcatcca cctctttgtc catgaggcca 3120
tcatcgccgt gacgatgtac acccgcatca agattggtgg cgccaagtct acacaacaca 3180
ttagtcagaa tgagctgctg aacgaggtca ccttcctgag ccgcctgctc aagaccgact 3240
ttatctacaa ccctggcgat attgagagta acttggagca tacattggat tacctcaagg 3300
tgagttatct cgcacaggaa taagggacag ctgcaattcg ctgaaagtag acctgagcgc 3360
aacggtctaa cattatcgtt cttttctaga aatccaatgt gatcgaggtt gacagtgaag 3420
gatatgtcgg actctctgat gctgaacgca gcaagggccg agagaactat ggtaatgggc 3480
tattctattt gtactcacta cagacgtgat gtgcatgttg tatcggccga gaaagtcgtt 3540
tctgactgaa cctctcattt tatcattact ctagactttt attgtttcct gctctggccc 3600
ttcgtggaga catactggct cgcagccgtg tccctgtata ccctgattcc caccgccaaa 3660
gagttgactc agcagttgga cagcaacgga gagcctcagg ttcactgggt tgaggagcgc 3720
gtgttcatgg agaagacgca aatgttcgga aagacgcttt actaccaggg agacctctcc 3780
tactttgagt ctgtcaacat ggagacgctc aagaatggtt ttaatcgtct gtgcgattat 3840
ggcatcctta tgatgaagcg acccaccaat gccaaggaga agacaaaggt tgctctccac 3900
cctgatttta tgccaagccg aggtgctgac ggtcatgtca ttgccagcgg cgcactttgg 3960
gatatggtcg aacatatcgg cacgttcaga cgtgaaggca agaatcgtcg tgataacgcc 4020
acaggtaagg aacatgtgtc ttgacattgc tcgaaacgaa atttgttgct ctgtatgctt 4080
tgtcaccagg ggtactaatg gcttgtgctc ttgcttacac tttccaacct agtttcctcc 4140
cgtgtcctgc ggtttgcaga ggtcgtcgcg aacgctccag ctccggttaa ggtacccttg 4200
cccaatccgg cacccaaaag gacaggcgat ggcgccccga aattataa 4248
<210> 8
<211> 2364
<212> ДНК
<213> Mortierella alpina
<220>
<221> CDS
<222> (1)..(2364)
<223>
<400> 8
atg gaa gga gac gca gta cgg cct gct ttg gcc aga aag atc cct ggt 48
Met Glu Gly Asp Ala Val Arg Pro Ala Leu Ala Arg Lys Ile Pro Gly
1 5 10 15
ctc tac agc ttc atc aaa ctc ctt tgc agg acg ctt ttt cac atc ttc 96
Leu Tyr Ser Phe Ile Lys Leu Leu Cys Arg Thr Leu Phe His Ile Phe
20 25 30
ttc agg gat tac gac gcc ttt cat acc cag ttt gtt cca cag gac gaa 144
Phe Arg Asp Tyr Asp Ala Phe His Thr Gln Phe Val Pro Gln Asp Glu
35 40 45
cca ttg cta gtt atc tcc aac cat ggc aac tac ctt ctg gat ggc ctc 192
Pro Leu Leu Val Ile Ser Asn His Gly Asn Tyr Leu Leu Asp Gly Leu
50 55 60
gcc ttg ttg gcc acc ttt cca ggc cag atc tcc ttt ttg atg gca cag 240
Ala Leu Leu Ala Thr Phe Pro Gly Gln Ile Ser Phe Leu Met Ala Gln
65 70 75 80
ccc aat ttc aag act gca att ggt ggc atc gcc agg aag att ggt gcc 288
Pro Asn Phe Lys Thr Ala Ile Gly Gly Ile Ala Arg Lys Ile Gly Ala
85 90 95
att cca gta ctg aga cca cag gac gcg gcc aga tat gac ggt gcg agt 336
Ile Pro Val Leu Arg Pro Gln Asp Ala Ala Arg Tyr Asp Gly Ala Ser
100 105 110
atg gtc aca atc gct cag gat ggc aac tca gtc ctc ggt cag ggg att 384
Met Val Thr Ile Ala Gln Asp Gly Asn Ser Val Leu Gly Gln Gly Ile
115 120 125
ggc aag cag ctg act ttg ggc gat act gtc tat atc gag tgt ggg acg 432
Gly Lys Gln Leu Thr Leu Gly Asp Thr Val Tyr Ile Glu Cys Gly Thr
130 135 140
ttc cag gac gct ggc agg gac aat cgc gtc acg caa tgt tat ggc gtg 480
Phe Gln Asp Ala Gly Arg Asp Asn Arg Val Thr Gln Cys Tyr Gly Val
145 150 155 160
gtc agt gcg atc gtc agt gac aac gag gtg ttg ttc aag gct ccc ggt 528
Val Ser Ala Ile Val Ser Asp Asn Glu Val Leu Phe Lys Ala Pro Gly
165 170 175
ttg aaa tgg att ccc gca tcc ttg aca tcg gaa cgc gac att gcc tat 576
Leu Lys Trp Ile Pro Ala Ser Leu Thr Ser Glu Arg Asp Ile Ala Tyr
180 185 190
atc aaa tcg cga aag att gtt cgg cat ggg tca ctc aag atc aga gtg 624
Ile Lys Ser Arg Lys Ile Val Arg His Gly Ser Leu Lys Ile Arg Val
195 200 205
gaa cgt ggc aac acc tgg gtc gga atc aat gag gcg ctt aaa gca cag 672
Glu Arg Gly Asn Thr Trp Val Gly Ile Asn Glu Ala Leu Lys Ala Gln
210 215 220
gag cag cag aac aat ggc tcg ttg gca agc agc gca acg ggg acg atc 720
Glu Gln Gln Asn Asn Gly Ser Leu Ala Ser Ser Ala Thr Gly Thr Ile
225 230 235 240
ggc aag ttt gtt cac aag ata ttt tca aag tcg ccg gat gca gac gca 768
Gly Lys Phe Val His Lys Ile Phe Ser Lys Ser Pro Asp Ala Asp Ala
245 250 255
aga tca gat gat gtg cat ttg gcc gag aat ggg tat tcc gga gca gat 816
Arg Ser Asp Asp Val His Leu Ala Glu Asn Gly Tyr Ser Gly Ala Asp
260 265 270
atc ccc ggg tcc ttg acc gct cca gcc aac ttt cac aca act gag act 864
Ile Pro Gly Ser Leu Thr Ala Pro Ala Asn Phe His Thr Thr Glu Thr
275 280 285
aca cca cta ctc aaa aag gcg cgc tcg tca aac agc tcc agt cat cct 912
Thr Pro Leu Leu Lys Lys Ala Arg Ser Ser Asn Ser Ser Ser His Pro
290 295 300
ata tac aca gta cca aag cgc gca gac tcg aac gcg agg ctt tca tca 960
Ile Tyr Thr Val Pro Lys Arg Ala Asp Ser Asn Ala Arg Leu Ser Ser
305 310 315 320
tac tct acc act cac agc aca aat gca gtg gcc gat gcc gac aac gac 1008
Tyr Ser Thr Thr His Ser Thr Asn Ala Val Ala Asp Ala Asp Asn Asp
325 330 335
gac gag acc acg cgc cct gca aat gga ctt agg aac gcg caa ggc gga 1056
Asp Glu Thr Thr Arg Pro Ala Asn Gly Leu Arg Asn Ala Gln Gly Gly
340 345 350
cac aac ccc gct gga acc aat ggc gtt gtc aat gga ggc gca tcc aca 1104
His Asn Pro Ala Gly Thr Asn Gly Val Val Asn Gly Gly Ala Ser Thr
355 360 365
tcc atg agc cca cga agc act cca ttg acc tcc cct aca ctt cac agc 1152
Ser Met Ser Pro Arg Ser Thr Pro Leu Thr Ser Pro Thr Leu His Ser
370 375 380
tcc aca tcg gcc gtg tca cac ttc ccc tcg cga ccc tgc ccc ttc cag 1200
Ser Thr Ser Ala Val Ser His Phe Pro Ser Arg Pro Cys Pro Phe Gln
385 390 395 400
ttc tca cac cca atc gac cat tct gtg atc tac gag agc gtc tgg aag 1248
Phe Ser His Pro Ile Asp His Ser Val Ile Tyr Glu Ser Val Trp Lys
405 410 415
aac ttt gag gat ggt cgc acc gtt gct gta ttc cct gaa ggc gta tcg 1296
Asn Phe Glu Asp Gly Arg Thr Val Ala Val Phe Pro Glu Gly Val Ser
420 425 430
agc gac gat tat cac ttg ctc gac ttc aaa tat ggc tgc acc atc atg 1344
Ser Asp Asp Tyr His Leu Leu Asp Phe Lys Tyr Gly Cys Thr Ile Met
435 440 445
gtt ctt gga tac ctg gct cag cat cgc tct aag act cta agg att ata 1392
Val Leu Gly Tyr Leu Ala Gln His Arg Ser Lys Thr Leu Arg Ile Ile
450 455 460
cca tgc gga ctg aac ttc ttt aat cgc cat cga ttt cga tcc cgg ttc 1440
Pro Cys Gly Leu Asn Phe Phe Asn Arg His Arg Phe Arg Ser Arg Phe
465 470 475 480
tac gcc gac tac tcc cat ccg ctc acc gtc ccc gac cac ctt gta gag 1488
Tyr Ala Asp Tyr Ser His Pro Leu Thr Val Pro Asp His Leu Val Glu
485 490 495
atg tat cgc gaa gga gga gaa gcc aag aag caa gcc tgt act gag ctt 1536
Met Tyr Arg Glu Gly Gly Glu Ala Lys Lys Gln Ala Cys Thr Glu Leu
500 505 510
ctg cag atg att cac tcg gct gtg gag ggg ctg act ctt aac gca cca 1584
Leu Gln Met Ile His Ser Ala Val Glu Gly Leu Thr Leu Asn Ala Pro
515 520 525
aac tac gac gag ctg cga ctt tac aag gca acg cga cga ctc tac agc 1632
Asn Tyr Asp Glu Leu Arg Leu Tyr Lys Ala Thr Arg Arg Leu Tyr Ser
530 535 540
act ggg aag aag ctt acc gtg cca cag aaa ttg gag cta act cgc cgt 1680
Thr Gly Lys Lys Leu Thr Val Pro Gln Lys Leu Glu Leu Thr Arg Arg
545 550 555 560
ttt gcg aaa ggt tat caa aat ctg gtc atg acg ccg agc atg gct gca 1728
Phe Ala Lys Gly Tyr Gln Asn Leu Val Met Thr Pro Ser Met Ala Ala
565 570 575
ttg aaa cgc gac att gat gct tat gac aag cat ctc tcc agc agc ggc 1776
Leu Lys Arg Asp Ile Asp Ala Tyr Asp Lys His Leu Ser Ser Ser Gly
580 585 590
gtc cga gac gca caa ctg acc gca aac cca agc att ctg gct gcc ctt 1824
Val Arg Asp Ala Gln Leu Thr Ala Asn Pro Ser Ile Leu Ala Ala Leu
595 600 605
gta ttc ata ttg ccc gcg tta ttc ctc ttg cca gtg ctg ttc ctg ctt 1872
Val Phe Ile Leu Pro Ala Leu Phe Leu Leu Pro Val Leu Phe Leu Leu
610 615 620
tcg tta ccc ggg acg ttg ctc ttc gga cct gtt gga ctt ttg gcg tcg 1920
Ser Leu Pro Gly Thr Leu Leu Phe Gly Pro Val Gly Leu Leu Ala Ser
625 630 635 640
tgg gcg gca aag cag aaa gga cag cag gcc atg ctt gcc ttt cag tct 1968
Trp Ala Ala Lys Gln Lys Gly Gln Gln Ala Met Leu Ala Phe Gln Ser
645 650 655
tat ctc cca gtt tca cgt tgg cct ggc cga gat gtg atc gcc acc tgg 2016
Tyr Leu Pro Val Ser Arg Trp Pro Gly Arg Asp Val Ile Ala Thr Trp
660 665 670
aag atc gtg gtg tcc ttg gcg ttg atg ccc gtc tgc ttt att ctg gac 2064
Lys Ile Val Val Ser Leu Ala Leu Met Pro Val Cys Phe Ile Leu Asp
675 680 685
gca aca ctg ctc acg atc cta gca cac cat tgg gaa gca ctg cag gag 2112
Ala Thr Leu Leu Thr Ile Leu Ala His His Trp Glu Ala Leu Gln Glu
690 695 700
tac tgg act atg ggt cgt cta gtt gcg ttc tgg ctg ctg tca aca ttt 2160
Tyr Trp Thr Met Gly Arg Leu Val Ala Phe Trp Leu Leu Ser Thr Phe
705 710 715 720
gtg att ttt cca acg atg gcg tat ggt acg gtc tgg ctt tgg gag tgg 2208
Val Ile Phe Pro Thr Met Ala Tyr Gly Thr Val Trp Leu Trp Glu Trp
725 730 735
cag att gat ttg aag atg cag atc tat gta tgg tgg tgg aag ctc tgc 2256
Gln Ile Asp Leu Lys Met Gln Ile Tyr Val Trp Trp Trp Lys Leu Cys
740 745 750
gga ggc aat gca gag atg aag cgc tgg aga cag gat ctt gtc gag aga 2304
Gly Gly Asn Ala Glu Met Lys Arg Trp Arg Gln Asp Leu Val Glu Arg
755 760 765
atg gat gca ctt gtc gaa agg atg ggc ggt aga agg gtg ttt gat gta 2352
Met Asp Ala Leu Val Glu Arg Met Gly Gly Arg Arg Val Phe Asp Val
770 775 780
tcg ggc tat tat 2364
Ser Gly Tyr Tyr
785
<210> 9
<211> 788
<212> PRT
<213> Mortierella alpina
<400> 9
Met Glu Gly Asp Ala Val Arg Pro Ala Leu Ala Arg Lys Ile Pro Gly
1 5 10 15
Leu Tyr Ser Phe Ile Lys Leu Leu Cys Arg Thr Leu Phe His Ile Phe
20 25 30
Phe Arg Asp Tyr Asp Ala Phe His Thr Gln Phe Val Pro Gln Asp Glu
35 40 45
Pro Leu Leu Val Ile Ser Asn His Gly Asn Tyr Leu Leu Asp Gly Leu
50 55 60
Ala Leu Leu Ala Thr Phe Pro Gly Gln Ile Ser Phe Leu Met Ala Gln
65 70 75 80
Pro Asn Phe Lys Thr Ala Ile Gly Gly Ile Ala Arg Lys Ile Gly Ala
85 90 95
Ile Pro Val Leu Arg Pro Gln Asp Ala Ala Arg Tyr Asp Gly Ala Ser
100 105 110
Met Val Thr Ile Ala Gln Asp Gly Asn Ser Val Leu Gly Gln Gly Ile
115 120 125
Gly Lys Gln Leu Thr Leu Gly Asp Thr Val Tyr Ile Glu Cys Gly Thr
130 135 140
Phe Gln Asp Ala Gly Arg Asp Asn Arg Val Thr Gln Cys Tyr Gly Val
145 150 155 160
Val Ser Ala Ile Val Ser Asp Asn Glu Val Leu Phe Lys Ala Pro Gly
165 170 175
Leu Lys Trp Ile Pro Ala Ser Leu Thr Ser Glu Arg Asp Ile Ala Tyr
180 185 190
Ile Lys Ser Arg Lys Ile Val Arg His Gly Ser Leu Lys Ile Arg Val
195 200 205
Glu Arg Gly Asn Thr Trp Val Gly Ile Asn Glu Ala Leu Lys Ala Gln
210 215 220
Glu Gln Gln Asn Asn Gly Ser Leu Ala Ser Ser Ala Thr Gly Thr Ile
225 230 235 240
Gly Lys Phe Val His Lys Ile Phe Ser Lys Ser Pro Asp Ala Asp Ala
245 250 255
Arg Ser Asp Asp Val His Leu Ala Glu Asn Gly Tyr Ser Gly Ala Asp
260 265 270
Ile Pro Gly Ser Leu Thr Ala Pro Ala Asn Phe His Thr Thr Glu Thr
275 280 285
Thr Pro Leu Leu Lys Lys Ala Arg Ser Ser Asn Ser Ser Ser His Pro
290 295 300
Ile Tyr Thr Val Pro Lys Arg Ala Asp Ser Asn Ala Arg Leu Ser Ser
305 310 315 320
Tyr Ser Thr Thr His Ser Thr Asn Ala Val Ala Asp Ala Asp Asn Asp
325 330 335
Asp Glu Thr Thr Arg Pro Ala Asn Gly Leu Arg Asn Ala Gln Gly Gly
340 345 350
His Asn Pro Ala Gly Thr Asn Gly Val Val Asn Gly Gly Ala Ser Thr
355 360 365
Ser Met Ser Pro Arg Ser Thr Pro Leu Thr Ser Pro Thr Leu His Ser
370 375 380
Ser Thr Ser Ala Val Ser His Phe Pro Ser Arg Pro Cys Pro Phe Gln
385 390 395 400
Phe Ser His Pro Ile Asp His Ser Val Ile Tyr Glu Ser Val Trp Lys
405 410 415
Asn Phe Glu Asp Gly Arg Thr Val Ala Val Phe Pro Glu Gly Val Ser
420 425 430
Ser Asp Asp Tyr His Leu Leu Asp Phe Lys Tyr Gly Cys Thr Ile Met
435 440 445
Val Leu Gly Tyr Leu Ala Gln His Arg Ser Lys Thr Leu Arg Ile Ile
450 455 460
Pro Cys Gly Leu Asn Phe Phe Asn Arg His Arg Phe Arg Ser Arg Phe
465 470 475 480
Tyr Ala Asp Tyr Ser His Pro Leu Thr Val Pro Asp His Leu Val Glu
485 490 495
Met Tyr Arg Glu Gly Gly Glu Ala Lys Lys Gln Ala Cys Thr Glu Leu
500 505 510
Leu Gln Met Ile His Ser Ala Val Glu Gly Leu Thr Leu Asn Ala Pro
515 520 525
Asn Tyr Asp Glu Leu Arg Leu Tyr Lys Ala Thr Arg Arg Leu Tyr Ser
530 535 540
Thr Gly Lys Lys Leu Thr Val Pro Gln Lys Leu Glu Leu Thr Arg Arg
545 550 555 560
Phe Ala Lys Gly Tyr Gln Asn Leu Val Met Thr Pro Ser Met Ala Ala
565 570 575
Leu Lys Arg Asp Ile Asp Ala Tyr Asp Lys His Leu Ser Ser Ser Gly
580 585 590
Val Arg Asp Ala Gln Leu Thr Ala Asn Pro Ser Ile Leu Ala Ala Leu
595 600 605
Val Phe Ile Leu Pro Ala Leu Phe Leu Leu Pro Val Leu Phe Leu Leu
610 615 620
Ser Leu Pro Gly Thr Leu Leu Phe Gly Pro Val Gly Leu Leu Ala Ser
625 630 635 640
Trp Ala Ala Lys Gln Lys Gly Gln Gln Ala Met Leu Ala Phe Gln Ser
645 650 655
Tyr Leu Pro Val Ser Arg Trp Pro Gly Arg Asp Val Ile Ala Thr Trp
660 665 670
Lys Ile Val Val Ser Leu Ala Leu Met Pro Val Cys Phe Ile Leu Asp
675 680 685
Ala Thr Leu Leu Thr Ile Leu Ala His His Trp Glu Ala Leu Gln Glu
690 695 700
Tyr Trp Thr Met Gly Arg Leu Val Ala Phe Trp Leu Leu Ser Thr Phe
705 710 715 720
Val Ile Phe Pro Thr Met Ala Tyr Gly Thr Val Trp Leu Trp Glu Trp
725 730 735
Gln Ile Asp Leu Lys Met Gln Ile Tyr Val Trp Trp Trp Lys Leu Cys
740 745 750
Gly Gly Asn Ala Glu Met Lys Arg Trp Arg Gln Asp Leu Val Glu Arg
755 760 765
Met Asp Ala Leu Val Glu Arg Met Gly Gly Arg Arg Val Phe Asp Val
770 775 780
Ser Gly Tyr Tyr
785
<210> 10
<211> 2367
<212> ДНК
<213> Mortierella alpina
<400> 10
atggaaggag acgcagtacg gcctgctttg gccagaaaga tccctggtct ctacagcttc 60
atcaaactcc tttgcaggac gctttttcac atcttcttca gggattacga cgcctttcat 120
acccagtttg ttccacagga cgaaccattg ctagttatct ccaaccatgg caactacctt 180
ctggatggcc tcgccttgtt ggccaccttt ccaggccaga tctccttttt gatggcacag 240
cccaatttca agactgcaat tggtggcatc gccaggaaga ttggtgccat tccagtactg 300
agaccacagg acgcggccag atatgacggt gcgagtatgg tcacaatcgc tcaggatggc 360
aactcagtcc tcggtcaggg gattggcaag cagctgactt tgggcgatac tgtctatatc 420
gagtgtggga cgttccagga cgctggcagg gacaatcgcg tcacgcaatg ttatggcgtg 480
gtcagtgcga tcgtcagtga caacgaggtg ttgttcaagg ctcccggttt gaaatggatt 540
cccgcatcct tgacatcgga acgcgacatt gcctatatca aatcgcgaaa gattgttcgg 600
catgggtcac tcaagatcag agtggaacgt ggcaacacct gggtcggaat caatgaggcg 660
cttaaagcac aggagcagca gaacaatggc tcgttggcaa gcagcgcaac ggggacgatc 720
ggcaagtttg ttcacaagat attttcaaag tcgccggatg cagacgcaag atcagatgat 780
gtgcatttgg ccgagaatgg gtattccgga gcagatatcc ccgggtcctt gaccgctcca 840
gccaactttc acacaactga gactacacca ctactcaaaa aggcgcgctc gtcaaacagc 900
tccagtcatc ctatatacac agtaccaaag cgcgcagact cgaacgcgag gctttcatca 960
tactctacca ctcacagcac aaatgcagtg gccgatgccg acaacgacga cgagaccacg 1020
cgccctgcaa atggacttag gaacgcgcaa ggcggacaca accccgctgg aaccaatggc 1080
gttgtcaatg gaggcgcatc cacatccatg agcccacgaa gcactccatt gacctcccct 1140
acacttcaca gctccacatc ggccgtgtca cacttcccct cgcgaccctg ccccttccag 1200
ttctcacacc caatcgacca ttctgtgatc tacgagagcg tctggaagaa ctttgaggat 1260
ggtcgcaccg ttgctgtatt ccctgaaggc gtatcgagcg acgattatca cttgctcgac 1320
ttcaaatatg gctgcaccat catggttctt ggatacctgg ctcagcatcg ctctaagact 1380
ctaaggatta taccatgcgg actgaacttc tttaatcgcc atcgatttcg atcccggttc 1440
tacgccgact actcccatcc gctcaccgtc cccgaccacc ttgtagagat gtatcgcgaa 1500
ggaggagaag ccaagaagca agcctgtact gagcttctgc agatgattca ctcggctgtg 1560
gaggggctga ctcttaacgc accaaactac gacgagctgc gactttacaa ggcaacgcga 1620
cgactctaca gcactgggaa gaagcttacc gtgccacaga aattggagct aactcgccgt 1680
tttgcgaaag gttatcaaaa tctggtcatg acgccgagca tggctgcatt gaaacgcgac 1740
attgatgctt atgacaagca tctctccagc agcggcgtcc gagacgcaca actgaccgca 1800
aacccaagca ttctggctgc ccttgtattc atattgcccg cgttattcct cttgccagtg 1860
ctgttcctgc tttcgttacc cgggacgttg ctcttcggac ctgttggact tttggcgtcg 1920
tgggcggcaa agcagaaagg acagcaggcc atgcttgcct ttcagtctta tctcccagtt 1980
tcacgttggc ctggccgaga tgtgatcgcc acctggaaga tcgtggtgtc cttggcgttg 2040
atgcccgtct gctttattct ggacgcaaca ctgctcacga tcctagcaca ccattgggaa 2100
gcactgcagg agtactggac tatgggtcgt ctagttgcgt tctggctgct gtcaacattt 2160
gtgatttttc caacgatggc gtatggtacg gtctggcttt gggagtggca gattgatttg 2220
aagatgcaga tctatgtatg gtggtggaag ctctgcggag gcaatgcaga gatgaagcgc 2280
tggagacagg atcttgtcga gagaatggat gcacttgtcg aaaggatggg cggtagaagg 2340
gtgtttgatg tatcgggcta ttattga 2367
<210> 11
<211> 2632
<212> ДНК
<213> Mortierella alpina
<400> 11
gtttgtctct cgacctttcg tcatcactct ctcgtcgtca tcccagccag ccttttgctt 60
ctttttcatt tctttgccgc atggactaac ggctgacact ctcacgccct ctcctccatc 120
gcaaagtatt tcttgcactg cttgccctgt ttttaatcga ctcctcgtca agcaatcacc 180
tccagcggat cccaggtggc cgcagcccta taccccaact gccaatggaa ggagacgcag 240
tacggcctgc tttggccaga aagatccctg gtctctacag cttcatcaaa ctcctttgca 300
ggacgctttt tcacatcttc ttcagggatt acgacgcctt tcatacccag tttgttccac 360
aggacgaacc attgctagtt atctccaacc atggcaacta ccttctggat ggcctcgcct 420
tgttggccac ctttccaggc cagatctcct ttttgatggc acagcccaat ttcaagactg 480
caattggtgg catcgccagg aagattggtg ccattccagt actgagacca caggacgcgg 540
ccagatatga cggtgcgagt atggtcacaa tcgctcagga tggcaactca gtcctcggtc 600
aggggattgg caagcagctg actttgggcg atactgtcta tatcgagtgt gggacgttcc 660
aggacgctgg cagggacaat cgcgtcacgc aatgttatgg cgtggtcagt gcgatcgtca 720
gtgacaacga ggtgttgttc aaggctcccg gtttgaaatg gattcccgca tccttgacat 780
cggaacgcga cattgcctat atcaaatcgc gaaagattgt tcggcatggg tcactcaaga 840
tcagagtgga acgtggcaac acctgggtcg gaatcaatga ggcgcttaaa gcacaggagc 900
agcagaacaa tggctcgttg gcaagcagcg caacggggac gatcggcaag tttgttcaca 960
agatattttc aaagtcgccg gatgcagacg caagatcaga tgatgtgcat ttggccgaga 1020
atgggtattc cggagcagat atccccgggt ccttgaccgc tccagccaac tttcacacaa 1080
ctgagactac accactactc aaaaaggcgc gctcgtcaaa cagctccagt catcctatat 1140
acacagtacc aaagcgcgca gactcgaacg cgaggctttc atcatactct accactcaca 1200
gcacaaatgc agtggccgat gccgacaacg acgacgagac cacgcgccct gcaaatggac 1260
ttaggaacgc gcaaggcgga cacaaccccg ctggaaccaa tggcgttgtc aatggaggcg 1320
catccacatc catgagccca cgaagcactc cattgacctc ccctacactt cacagctcca 1380
catcggccgt gtcacacttc ccctcgcgac cctgcccctt ccagttctca cacccaatcg 1440
accattctgt gatctacgag agcgtctgga agaactttga ggatggtcgc accgttgctg 1500
tattccctga aggcgtatcg agcgacgatt atcacttgct cgacttcaaa tatggctgca 1560
ccatcatggt tcttggatac ctggctcagc atcgctctaa gactctaagg attataccat 1620
gcggactgaa cttctttaat cgccatcgat ttcgatcccg gttctacgcc gactactccc 1680
atccgctcac cgtccccgac caccttgtag agatgtatcg cgaaggagga gaagccaaga 1740
agcaagcctg tactgagctt ctgcagatga ttcactcggc tgtggagggg ctgactctta 1800
acgcaccaaa ctacgacgag ctgcgacttt acaaggcaac gcgacgactc tacagcactg 1860
ggaagaagct taccgtgcca cagaaattgg agctaactcg ccgttttgcg aaaggttatc 1920
aaaatctggt catgacgccg agcatggctg cattgaaacg cgacattgat gcttatgaca 1980
agcatctctc cagcagcggc gtccgagacg cacaactgac cgcaaaccca agcattctgg 2040
ctgcccttgt attcatattg cccgcgttat tcctcttgcc agtgctgttc ctgctttcgt 2100
tacccgggac gttgctcttc ggacctgttg gacttttggc gtcgtgggcg gcaaagcaga 2160
aaggacagca ggccatgctt gcctttcagt cttatctccc agtttcacgt tggcctggcc 2220
gagatgtgat cgccacctgg aagatcgtgg tgtccttggc gttgatgccc gtctgcttta 2280
ttctggacgc aacactgctc acgatcctag cacaccattg ggaagcactg caggagtact 2340
ggactatggg tcgtctagtt gcgttctggc tgctgtcaac atttgtgatt tttccaacga 2400
tggcgtatgg tacggtctgg ctttgggagt ggcagattga tttgaagatg cagatctatg 2460
tatggtggtg gaagctctgc ggaggcaatg cagagatgaa gcgctggaga caggatcttg 2520
tcgagagaat ggatgcactt gtcgaaagga tgggcggtag aagggtgttt gatgtatcgg 2580
gctattattg agcaacgttc atgtataaag tcatttggcc caattcttct cc 2632
<210> 12
<211> 2598
<212> ДНК
<213> Mortierella alpina
<400> 12
atggaaggag acgcagtacg gcctgctttg gccagaaaga tccctggtct ctacagcttc 60
atcaaactcc tttgcaggac gctttttcac atcttcttca gggattacga cgcctttcat 120
acccagtttg ttccacagga cgaaccattg ctagttatct ccaaccatgg caactacctt 180
ctggatggcc tcgccttgtt ggccaccttt ccaggccaga tctccttttt gatggcacag 240
cccaatttca agactgcaat tggtggcatc gccaggaaga ttggtgccat tccagtactg 300
aggcaagtct ttgaaaaacc acaaaataca gagcgaaata gtgaccatag gctcgacata 360
tggggtggcg tgatctgcgc atcctgtctt ttcgtcaacc ctttttgctt tgcccacttg 420
ctgaccctgt gattatgctc tttgtccaat ctatagacca caggacgcgg ccagatatga 480
cggtgcgagt atggtcacaa tcgctcagga tggcaactca gtcctcggtc aggggattgg 540
caagcagctg actttgggcg atactgtcta tatcgagtgt gggacgttcc aggacgctgg 600
cagggacaat cgcgtcacgc aatgttatgg cgtggtcagt gcgatcgtca gtgacaacga 660
ggtgttgttc aaggctcccg gtttgaaatg gattcccgca tccttgacat cggaacgcga 720
cattgcctat atcaaatcgc gaaagattgt tcggcatggg tcactcaaga tcagagtgga 780
acgtggcaac acctgggtcg gaatcaatga ggcgcttaaa gcacaggagc agcagaacaa 840
tggctcgttg gcaagcagcg caacggggac gatcggcaag tttgttcaca agatattttc 900
aaagtcgccg gatgcagacg caagatcaga tgatgtgcat ttggccgaga atgggtattc 960
cggagcagat atccccgggt ccttgaccgc tccagccaac tttcacacaa ctgagactac 1020
accactactc aaaaaggcgc gctcgtcaaa cagctccagt catcctatat acacagtacc 1080
aaagcgcgca gactcgaacg cgaggctttc atcatactct accactcaca gcacaaatgc 1140
agtggccgat gccgacaacg acgacgagac cacgcgccct gcaaatggac ttaggaacgc 1200
gcaaggcgga cacaaccccg ctggaaccaa tggcgttgtc aatggaggcg catccacatc 1260
catgagccca cgaagcactc cattgacctc ccctacactt cacagctcca catcggccgt 1320
gtcacacttc ccctcgcgac cctgcccctt ccagttctca cacccaatcg accattctgt 1380
gatctacgag agcgtctgga agaactttga ggatggtcgc accgttgctg tattccctga 1440
aggcgtatcg agcgacgatt atcacttgct cgacttcaaa tatggctgca ccatcatggt 1500
tcttggatac ctggctcagc atcgctctaa gactctaagg attataccat gcggactgaa 1560
cttctttaat cgccatcgat ttcgatcccg gttctacgcc gactactccc atccgctcac 1620
cgtccccgac caccttgtag agatgtatcg cgaaggagga gaagccaaga agcaaggtaa 1680
ggaagaccaa aactgcctcc ctcacatatc gcagtctgtc gaagtttttc tcacatatct 1740
ctttttctct tagcctgtac tgagcttctg cagatgattc actcggctgt ggaggggctg 1800
actcttaacg caccaaacta cgacgagctg cgactttaca aggcaacgcg acgactctac 1860
agcactggga agaagcttac cgtgccacag aaattggagc taactcgccg ttttgcgaaa 1920
ggttatcaaa atctggtcat gacgccgagc atggctgcat tgaaacgcga cattgatgct 1980
tatgacaagc atctctccag cagcggcgtc cgagacgcac aactgaccgc aaacccaagc 2040
attctggctg cccttgtatt catattgccc gcgttattcc tcttgccagt gctgttcctg 2100
ctttcgttac ccgggacgtt gctcttcgga cctgttggac ttttggcgtc gtgggcggca 2160
aagcagaaag gacagcaggc catgcttgcc tttcagtctt atctcccagt ttcacgttgg 2220
cctggccgag atgtgatcgc cacctggaag atcgtggtgt ccttggcgtt gatgcccgtc 2280
tgctttattc tggacgcaac actgctcacg atcctagcac accattggga agcactgcag 2340
gagtactgga ctatgggtcg tctagttgcg ttctggctgc tgtcaacatt tgtgattttt 2400
ccaacgatgg cgtatggtac ggtctggctt tgggagtggc agattgattt gaagatgcag 2460
atctatgtat ggtggtggaa gctctgcgga ggcaatgcag agatgaagcg ctggagacag 2520
gatcttgtcg agagaatgga tgcacttgtc gaaaggatgg gcggtagaag ggtgtttgat 2580
gtatcgggct attattga 2598
<210> 13
<211> 3087
<212> ДНК
<213> Mortierella alpina
<400> 13
atggcccttc agatctacga cttcgtgtcg ttcttcttca ctatcctgct cgacatcttc 60
ttcagggaga ttcgtcccag aggcgcacac aaaattccac aaaaaggacc cgtgatcttt 120
gttgccgctc ctcatgccaa tcaggtacgt gcacatgagg gctcgctttt ctcggagcgg 180
gcttcgtcca aaaagctgca caggctgaaa tgagagcgca tattcacctg gacacgggca 240
cagagttcat gcatgataag ccggatagca ggagccgaca ctgcagaaga gaccctctgg 300
accaacaaag aaaaacatca tgacggaact agccttctcc ggaagagcca atggacgtca 360
ttggtagacg caaccgttcc aactaaccgg cgctcttgtc ctcatagttt gtcgatcctc 420
tcgtcttgat gcgtgagtgt ggccgtagag tctcattcct tgcggccaaa aagtctatgg 480
accgccggtg gattggtgct atggcacgct cgatgaatgc gagtaagttg cttggacttt 540
gacacgaaac tgttgccacg tcaagaatat tcccaccctc ccgactcgac cacctccagc 600
tctcacctac acacacaaaa caggaaattt cagtgttctt caaggctttg gtggtgcttc 660
gaatggcgct acaaaaggcc tgctctaggg tgatagagtg ttggctttga tccttccgac 720
attctcggct cccttaccaa gacatttgtc ctgaatgctg attgatccga cactgctgaa 780
ccattcctac tcatagttcc tgttgaacgc ccgcaggatc ttgctaaagc gggttcggga 840
gtcatcaaac ttttggatcg ctatggtgat cctcttcgag tgacaggtgt cggcactaaa 900
ttcacaaagg agctacttgt gggagatcag atatctctcc caaaggacgt tggctcctca 960
gccgtggtcg agatcatatc tgataccgag ctgattgtca agaaggaatt caaggagctc 1020
aaggccctcg aattattgac cagccctgat ggaaccaagt ataaatgcct acctcacatg 1080
gaccagacga atgtatacaa aactgtcttt gagcgcctca acgctggaca ttgcgttggc 1140
attttccccg aaggtggatc ccacgatcgc gctgagatgc tgccattgaa aggtacgtgt 1200
gctcgtgctt ctgcacagag cagagtagtt gatatggaac agaagaaaaa agacacgcga 1260
ccagctttga ttaacagcca cgtgtttcct ttacctttgc gaaacattat agctggagtc 1320
accatcatgg ctctgggcgc gttggccgcc aacccttcgt tggacctcaa gattgtcacc 1380
tgcggcctca actactttca tcctcatcgc ttccgctcgc gtgcagtggt cgagtttggc 1440
gagccactga cggtccctcc tgagctggtc gaaatgtaca agcgaggcgg ggctgagaag 1500
cgtgaagcgt gcggaaagtt gctggataca atctatgagg ctcttcgcgg tgtcactctc 1560
aatgcacctg attacgaaac gttgatggta tggagcaaag gaccatagcg tggatgaagg 1620
aggacgtgga aaggacaagc accgctcaca gatttctcac tcttgtattt gtgattatct 1680
ctaggtcatt caagcggccc gtcgccttta caagcccact catcgcaagc tgcagatctc 1740
acaagtcgtg gagttgaacc gcaggttcgt cgcaggatac atgcacttca aggacaaccc 1800
taaagtcatt gaagccaagg acaaggtcat gcattacaac actcaacttc gataccatgg 1860
actgcgcgat catcaggtga acattcgcac aaccaggaaa cacgctatcg gcatgctcat 1920
ctcacggctc attcagatga tctttttgag ttgtctggct ctacctgggt aagcacagct 1980
tgaatctcga ccaggtcccg caatgatccc attgcggaga agtcactgac gcttgctctt 2040
cccgtgcttt tttgaataga accctgatga atcttccggt cgctattgtc gctcgtgtca 2100
tcagcaacaa gaaggccaaa ggtacgcctt gcggtttgtt atcttttcgt gtttgctttt 2160
gtgctcgcca ctggaaacta atatttctac atcactctgc aactggtaga ggcgctggct 2220
gcctcgacag tcaagattgc tggaagggat gtcctggcta catggaagct gctggtcgct 2280
ctaggattga tgcctgtcct ctacttcaca tattccgtca tggtctttat ctattgtggc 2340
cgcttcgaca tatcgttcaa gtcgcgtctc ttgatcgctt gggcagcatg ggcgctaatt 2400
cctttcgtaa cgtatgcaag catacgcttc ggtgaagttg gtatcgatat tttcaaatct 2460
atccgcccat tgttcttgtc catcatccca ggtgaagaga gcacgatcaa cgacttgcgc 2520
aaagcccgag cggaactcca gaagactatc accaatctta tcaatgagct ggcgccgcag 2580
atttatcccg actttgattc gaagcgcatc ctcgatccgt ctcctgcaga tcgccccagc 2640
cgctcggcat caggtaccaa ccttgcacag acaatcttca acacggccgc tcagcctttg 2700
aaccaatggc taggcaagga cggccgcttt gaatgggagc gcaccgagga ttcggatgca 2760
gatgatgtgt tcttcttttt ggacccagca agaggaattc ttggacggtc gagggcgtcg 2820
tcctggggag gaggggcatt tacacctgcc gccgatgggt cgcgatcccg gaatcggagc 2880
aggacaagca gcttcacgtc gggacagatc cagcttggcg agggcttcaa actcgaggca 2940
ttgacggaac tgccgaggga caagcctttt gcagaggtga cgaggcggct gagtgtgagc 3000
cgcatgcaga gatacgggtt ggagggtatg acgcgctcgg acacggacga aaacgaaggc 3060
tctacagcca agtcaaaaga tatctag 3087
<210> 14
<211> 3280
<212> ДНК
<213> Mortierella alpina
<400> 14
atgggtctcc agatctatga cttcgtctca ttcttcttca ccctcctgct cgacatcttc 60
ttcagggaaa tccgtcctcg gggggcccac aagatccccc gacaaggacc cgtcattttt 120
gtcgccgccc ctcatgccaa tcaggtaggc cattgtcttt aatttggaag cttaaagtca 180
ccactttatg cgcaagagcc tcatggtccg taaggtacta atcgctgctt gcatgaaact 240
agtttgttga tccactcgtg ttgatgcgcg aatgtggccg acgagtctcg ttcctggctg 300
ccaaaaagtc aatggaccgt cgctggatag gcgccatggc gcgttccatg aatgccagta 360
agtacaaaaa aaaaaaaaaa aaaaaaaaaa aacgacctat tcggcgtgag agtggcaacg 420
aaggggagag agatacacaa acgggccttt tgagtgcgtg tgtgcgtggg aaaagaatac 480
gagaacattc tcttgccagc aaaacgcgct tcctttctct tttctccgac gttgttgatg 540
gcgccttttg tatacacttc ctatccatcg tccctatccg agagatcaag gttttctgcg 600
tcgtgttatt ttaggccact ccgccgagat aacagacacg agctactgac cgattcaacg 660
agggttcaca caccgtccac aaccacctga gaaatgtgca ggccatgacg agtgtcgtgt 720
gccgtttctt ctttgatacc ataccgctcc tgcagtcatg ggccatccgt tatgccctga 780
cctcagcatt ggaatctgac gtttttttct gctcgctctc ttgtcatctg cgttagttcc 840
tgtggagcgt cctcaggact tggctaaggc tggttcggga acgatcaaac tggtggatcg 900
ctatggcgac cctctccgca tcactgggct cggtaccaaa ttcacaaagg aactctttgt 960
cggcgaccag atttcgctcc caaaggacgt tggaacctcg gctgtggtcg agatcatctc 1020
ggacactgaa ctgattgtca agaaagagtt caaggagctc aaggcactgg agcttttaac 1080
cagtgctgag ggatccaagt acaagtgcat gcctcacatg gaccagagca aggtctacaa 1140
gactgttttt gaacgcttaa atgctggcca ttgtgttggc atctttcctg aaggaggttc 1200
acacgatcgt gcagagatgc tgcccctgaa aggtaagcgc ccctcgtgag catcccaaca 1260
taacgggaat taccccacac ttgccttgtc cttgcgcatc tctgtatgaa cgtacactga 1320
ttgcattgca tatctccgtt tggattggat agctggtgtc accatcatgg cgcttggcgc 1380
actggccgct aaccctgacc tggaccttaa aattgtgacc tgcggcctga actatttcca 1440
cccccatcga ttccgttctc gcgccgttgt tgagtttggt gagcctttga ccgtgcctcc 1500
agaacttgtc gagatgtaca agaggggtgg agctgagaag cgcgaggcct gcggaaagct 1560
cctcgacacc atctacgatg cactcaagaa tgtcacactg aacgcccccg attacgaaac 1620
actcatggtg aggattggcg gtgtttttgc atgcggctta tgtcatttgg agcaattgag 1680
accaacgtta acttaaaggg ctttaatatg gctggactgg atgtaggtta ttcaagctgc 1740
ccgccgtctc tacaagccaa cacatcgcaa gctgcagatc tctcaagttg tggagttgaa 1800
ccgtcgcttc gtcgctggtt atctgcactt tcaggacaac ccaaaggtga ttgatacaaa 1860
ggacaaagtt atgcactaca acactcagct acgctatcat ggacttcgcg accaccaggt 1920
caacatccgc acaactcggc gacatgccat tgaactattg atttggcgag ttgtgcagat 1980
ggtctttttg agtctactag cgcttccagg gtaagaacga atgcagcagt ggtgtcatgt 2040
cacagacttt tgtgtgggcg gttagattga ggctagcact acttcacgcg attggatatt 2100
agactaacgc tttctctact tttagtacca tgatgaatct tccagttgcc atcgttgctc 2160
gcatcatcag caacaagaag gccaagggta tgtaatcgtc gcaatgacag cgacaaatct 2220
tttgattatc gggagaatgg cgtcagagga aaaaggccaa ggctaacgct ataatcattt 2280
tcacaattta acagaggctt tggctgcatc gaccgtgaaa attgcaggaa gggacgttct 2340
agccacatgg aagttgcttg tggccctggg attgatgccc gtcctttact tttcgtactc 2400
gtttgttatc tttttgctgt gtggacggtt cgacattacg ctcaagaccc gcctcctgat 2460
cgcttgggcg gcttgggcct gcattccgtt tgtgacctac gccagtatcc gtttcggtga 2520
ggtgggtatc gatatcttca agtcgatccg tcctctcttc ttgtcaatca ttcccggcga 2580
ggaaaacacg atcaatgagc tccgcaagtc gcgtgcagag cttcaaaaga ccatcaacga 2640
gctcatcaat gagctggcgc cggaaatata ccccgacttt gattccaaac gaatcttgga 2700
cccttcgcca aacgatcgac ccagccgcgg cgcgtcgcgc tccgcctcgg gcaccaacct 2760
tgcgcagacc attttcaaca ccatgaacac ggccacacag ccgctaaacc aatggctcgg 2820
tatggatggg cgcttcgagt gggagcgtgt ggacgactcg gatgcggacg acgtgttctt 2880
tttcctcaac cctgcaggag ccatccaagg gcgatcgagg acgtcttctt ggggtgctgg 2940
agcatggacg ccttcgtctg ctggcgatgg ttctcggtct cggtcaagga gtcgcagtcg 3000
gacgagctcg tttgcgtcag ggcagattca gctgggcgaa gggttcaagc tggaggcatt 3060
gacagagctg ccaaaggata agccttttgg cgaggtgaca cgacgactca gcttgagtcg 3120
caagcagaag catggactgg tgggcgacat gaccaaggat cccgaggaga ttgagaagca 3180
gggcggattt atgcatgagg gacactttgt cagcacaccg cccatcacgg ttcagaacat 3240
ggatgatcct atggatgcag tcaagtctaa ggaggcataa 3280
<210> 15
<211> 2896
<212> ДНК
<213> Mortierella alpina
<400> 15
atggccaaac ttcggaagcg gacctcccag tcaaaggagg gcgccgctga caccaacggc 60
acaaggcgag acagcgtcga tgacacgaac agcgtcggca gctacgatcc acgcgccctc 120
tccaacgatc caccaaagat gtacagggcg atccggttct tcttcaagat gtgcctgcac 180
tccttctatg gccatgtgga ggtcgagggc accgagaata ttgcaccaaa caactaccct 240
gctatccttg gtaacgttct taggacaaat attcctttgc aggatgatca cacattatgt 300
catggccttt ggtcttgaaa tgttagtcta accgttcgga tgcacactcg tcgatagttg 360
cgaaccacag caacagtttg acggatgcga ttgccattat gtcgactgtt cctcccaaga 420
gcaggagcat gattaggatg accgcaaagg acacgttttg gcataagcca ggcgtcttca 480
agtatgcact gccagcctag cctgaacaca cctcgtccga tgcctccttt cactgaccgg 540
atatcttttt tgtctttgaa tggcgcgccc tcctcgtgca ctcttcgtct ccttctccgt 600
tattgatagt tatgtcatca aaaacgctgg cactgtcccg atcaaaagac gcaaggatta 660
tgagaaccaa aaggtcgaca acactgacgc gatgggtgca ttgatcgata cccttggagc 720
aggaagttgt gtatggtaag tggagggtcc ttgtctgcat accgcgctgc acttgaaggc 780
tttctacact tccaatgaca gggatagctg cactgcatgt ttgattgtaa agcgtaacat 840
agagagaaga gggggaagag ggggtgggga aaactgaatc aatatcaagg accatatgaa 900
tatgattcaa agcactggtt gcaatgctgt gttgttttgc atgaaaaacg cagctccttg 960
cctcgactca cccctgcatc ttcatatacc accttccaac cgcaaaaaaa aatcaaagca 1020
tgttcccgga gggcatctcg cgctatcacc cacaacttgc tccgttcaag gccggtgtcg 1080
ccatgattgc cagcgatgta agtgttcgct tgatgaactt ttattatttt tgacgttccg 1140
accgtgctgg ctcctcgcga ctgcaactgt gatttgtcat ggcaaatacc acagcgctct 1200
cactgacatg aattttcatg ctctctttca cgctcctctg tctctcatgc ttgccctaat 1260
attcttttgt acacgaccga tgatacacga ccgatgatac acgacggggg ggtttctcgg 1320
tttgcgacat tgtgccatgg atgtgtttgt ctcaattaga cgctctcccg gtttcaagac 1380
acgcccgatt tttctctcac gctcatgaca gcgtcgatca actatcttca ccgtgaaaag 1440
ttccgatctg atgttctcgt cacgttccat gcacccattg tgctaacccc gcaacaagac 1500
tcgaagctgt tttcgactga cctggaagtc aagaaggaag cgatccgaaa actgacagag 1560
ttgctcgagg gcaccgtccg atcaactcta ctggatgccg aggactggca aacagtccga 1620
gtgggtcatg ttgccaggaa gctctatgct ggcgatctgg gaactcggat ttcgctggga 1680
cagtacgtgc gtttgaccag gaagtttgtc acggcgttca gtcagcacaa gcaggaggag 1740
gaggcagcgg tcgatgacga gcgttatggt caggagaagc acgggggcgg tgccgagagg 1800
aatggtgatt ctttggagat gaggcatcct gagcgcatgg ataaggcgac tagaaagaaa 1860
atcgacgagc tcgccaggga tttggctgta agcatgagta attcattcag tcgtctcaca 1920
cacggcatgc tctttcaaaa gtggacgcta acggatacat tattcttatt tttattttct 1980
tgacggtgat cataacagga ctaccaaaac cagctggact tctatcacct caaggactat 2040
cgtatcaagc aaggcaagcc aagtgcaaag attcttatcg gacgtctttt ccaaagattc 2100
ttgcttgctt gccttttgtc gaccatttgc attcctggac tgttcctttg ggcacctgtg 2160
tttatcgccg tgaagtacgc aagacattgg gttgacactg ataccttgcc aacaattaag 2220
cacgaactct ccttctcgat acctgctatt tttaacactg atgtaatcgt ctcttttttt 2280
ttctctctag gtacaaagag agtcagctta ggcgcaaggg acccttggag gacaacttgg 2340
atgaaattgc ccagtacaag ttgatgatct cgactttctt cttgccgatc atctgggggt 2400
tctggatcgt aatgaccttg ccaattgcgc tctttagcgc gccgggcatc gttgttctga 2460
tgtggctgta agtcaagaga ggaagaagat gcatatcaag accttttcaa gttatcaaga 2520
cactatgact attgctgcga cgaagactaa cttatatttc cattgtgcgt gtgatagtac 2580
gatccgctgg cttgaggact tgatccacaa cgcgaaatcg atgttgtccc ttttgcgatt 2640
gctgtttatg acggaggata ccatgtactc gttgagagac taccgtcagg ggctggcgca 2700
tcgtgtgcac gattttgcgg tcgatcatct gaagttgcct gaggaccctg aggttctggt 2760
caaggagaac aagaccaaga aggtcgacag tggctggatg ggcaagttgt cgggcagcta 2820
cttctcgatc aagaggagaa gaagaaagga ctggaacgag gttatgcgat tgcacgatgt 2880
ttctcactat gactga 2896
<210> 16
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер GPAT4-S
<400> 16
caaggatgtt gttgatgagg aaggcgaag 29
<210> 17
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер SacI-GPAT41
<400> 17
gagctcatgc ccatcgttcc agctcagc 28
<210> 18
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Sal-GPAT4-2
<400> 18
gtcgacttat aatttcgggg cgccatcgc 29
<210> 19
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер GPAT5-1F
<400> 19
ttccctgaag gcgtatcgag cgacgatt 28
<210> 20
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер GPAT5-3R
<400> 20
caaatgttga cagcagccag aacg 24
<210> 21
<211> 835
<212> PRT
<213> Chaetomium globosum
<400> 21
Met Ala Pro Asp Asp Arg His Asp Ser Ala Pro Asp Leu Arg Ile Leu
1 5 10 15
Gly Asp Arg Ile Thr Leu Gln Pro Ser Gly Phe Val Glu Pro Glu His
20 25 30
Thr Gly Glu Gly Lys Glu Glu Ala Leu Met Lys Asn Met Ala Arg Phe
35 40 45
Arg Ser Glu Pro Leu Arg Phe Leu Arg Glu Val Ser Leu Tyr Val Ser
50 55 60
Gly Thr Gly Trp Arg Ala Tyr Asp Asn Val Ile Gly Gln Pro Ile Phe
65 70 75 80
Tyr Ser Gly Phe Ser Glu His Ile Lys Thr Glu Met Met Ser Ala Thr
85 90 95
Leu Leu Gln Thr Lys Ile Ala Gln Leu Ala Asp Met Arg Val Ala Val
100 105 110
Glu Glu Lys Glu Gly Leu Leu Asn Gly Ser Asp Ser Asp Leu Ala Ala
115 120 125
Lys Lys Ala Arg Arg Arg Ala Ala Leu Val Gln Asn Leu Gln Glu Val
130 135 140
Ala Glu Lys Leu Ala Asp Asn Met Ile Cys Lys Phe Asp Ser Lys Pro
145 150 155 160
Phe Ile Arg Gly Ala Tyr Tyr Leu Val Thr Gln Leu Leu Leu Arg Ala
165 170 175
Tyr His Gln Gly Ile His Val Ser Ser Glu Glu Val Leu Arg Leu Arg
180 185 190
Ser Val Ala Glu Glu Ala Ala Arg Lys Lys Gln Ser Ile Ile Phe Leu
195 200 205
Pro Cys His Arg Ser His Val Asp Tyr Val Ser Leu Gln Leu Leu Cys
210 215 220
Tyr Arg Leu Gly Leu Ala Leu Pro Val Val Val Ala Gly Asp Asn Leu
225 230 235 240
Asn Phe Pro Val Val Gly Ser Phe Leu Gln His Ala Gly Ala Met Tyr
245 250 255
Ile Arg Arg Ser Phe Gly Asp Asp Gln Leu Tyr Ser Thr Leu Val Gln
260 265 270
Thr Tyr Ile Asp Val Met Leu Gln Gly Gly Tyr Asn Leu Glu Cys Phe
275 280 285
Ile Glu Gly Gly Arg Ser Arg Thr Gly Lys Leu Leu Pro Pro Lys Phe
290 295 300
Gly Ile Leu Asn Phe Val Leu Asp Ser Leu Leu Ser Gly Arg Val Glu
305 310 315 320
Asp Ala Ile Ile Cys Pro Val Ser Thr Gln Tyr Asp Lys Val Ile Glu
325 330 335
Thr Glu Gly Tyr Val Thr Glu Leu Leu Gly Val Pro Lys Lys Lys Glu
340 345 350
Asn Leu Ala Asp Phe Leu Thr Gly Gly Ser Ser Ile Leu Ser Leu Arg
355 360 365
Leu Gly Arg Val Asp Val Arg Phe His Glu Pro Trp Ser Leu Arg Gly
370 375 380
Phe Ile Asp Glu Gln Leu Ser Arg Leu Ser Lys Met Pro Ser Ala Ile
385 390 395 400
Asn Val Asp Trp Arg Asp Met Lys Asn Gln Met Leu Arg Gln Lys Leu
405 410 415
Leu Arg Thr Leu Gly Tyr Lys Val Leu Ala Asp Ile Asn Ala Val Ser
420 425 430
Val Val Met Pro Thr Ala Leu Ile Gly Thr Val Leu Leu Thr Leu Arg
435 440 445
Gly Arg Gly Val Gly Arg Arg Glu Leu Ile Leu Arg Val Glu Trp Leu
450 455 460
Thr Asn Arg Val Arg Ala Lys Gly Gly Arg Val Ala His Phe Gly Asn
465 470 475 480
Ala Pro Leu Ser Asp Val Val Gln Arg Gly Leu Asp Val Leu Gly Lys
485 490 495
Asp Leu Val Gly Val Val Glu Gly Leu Pro Glu Pro Thr Tyr Tyr Ala
500 505 510
Val Asp Arg Phe Gln Leu Ser Phe Tyr Arg Asn Met Thr Ile His Leu
515 520 525
Phe Ile Ser Glu Thr Leu Val Ala Thr Ala Leu Tyr Thr Lys Val Lys
530 535 540
Gln Gly Gly Gly Pro Ser Ile Gln Asp Ile Pro Tyr Arg Asp Leu Ser
545 550 555 560
Asp Gln Val Leu Phe Leu Ser Ser Leu Phe Arg Gly Glu Phe Ile Tyr
565 570 575
Ser Gly Glu Gly Leu Ala Thr Asn Leu Glu Arg Thr Leu Leu Gly Leu
580 585 590
Glu Ala Asp Asn Val Ile Leu Leu Glu Arg Asp Glu Glu Gly Asn Ile
595 600 605
Thr Lys Val Gly Leu Ser Gly Ala Glu Arg Ala Ala Gly Arg Glu Asn
610 615 620
Phe Asp Phe Tyr Cys Phe Leu Ile Trp Pro Phe Ile Glu Ala Ser Trp
625 630 635 640
Leu Ala Ala Val Ser Leu Met Gly Leu Val Pro Pro Ala Gly Gln Lys
645 650 655
Asp Asp Ile Trp Val Gln Val Ser Lys Ala Gln Asp Ser Ala Gln Leu
660 665 670
Leu Gly Lys Thr Leu Tyr His Gln Gly Asp Leu Ser Tyr Phe Glu Ala
675 680 685
Val Asn Lys Glu Thr Leu Lys Asn Ser Tyr Gln Arg Phe Ala Glu Glu
690 695 700
Gly Ile Ile Gln Val Val Lys Ser Lys Asp Thr Arg Ile Pro Pro Arg
705 710 715 720
Leu Arg Ile Ala Pro Glu Trp Arg Pro Gly Arg Asp Lys Glu Thr Gly
725 730 735
Glu Leu Leu Pro Ala Gly Arg Leu Trp Asp Phe Thr Glu Lys Ile Ala
740 745 750
Ser Ser Arg Arg Glu Gly Lys Asn Arg Arg Asp Gly Ala Thr Val Ser
755 760 765
Ser Arg Val Leu Arg Leu Thr Asn Glu Leu Gly Arg Lys Leu Phe Glu
770 775 780
Glu Ala Val Glu Gly Glu Thr Gly Lys Gly Lys Gly Lys Gly Lys Gly
785 790 795 800
Lys Ala Ala Val Pro Ala Arg Leu Ser Ser Glu Asp Glu Arg Ser Leu
805 810 815
Ser Arg Thr Val Arg Glu Gln Glu Arg Lys Arg Lys Leu Glu Arg Arg
820 825 830
Ala His Leu
835
<210> 22
<211> 807
<212> PRT
<213> Escherichia coli
<400> 22
Met Ser Gly Trp Pro Arg Ile Tyr Tyr Lys Leu Leu Asn Leu Pro Leu
1 5 10 15
Ser Ile Leu Val Lys Ser Lys Ser Ile Pro Ala Asp Pro Ala Pro Glu
20 25 30
Leu Gly Leu Asp Thr Ser Arg Pro Ile Met Tyr Val Leu Pro Tyr Asn
35 40 45
Ser Lys Ala Asp Leu Leu Thr Leu Arg Ala Gln Cys Leu Ala His Asp
50 55 60
Leu Pro Asp Pro Leu Glu Pro Leu Glu Ile Asp Gly Thr Leu Leu Pro
65 70 75 80
Arg Tyr Val Phe Ile His Gly Gly Pro Arg Val Phe Thr Tyr Tyr Thr
85 90 95
Pro Lys Glu Glu Ser Ile Lys Leu Phe His Asp Tyr Leu Asp Leu His
100 105 110
Arg Ser Asn Pro Asn Leu Asp Val Gln Met Val Pro Val Ser Val Met
115 120 125
Phe Gly Arg Ala Pro Gly Arg Glu Lys Gly Glu Val Asn Pro Pro Leu
130 135 140
Arg Met Leu Asn Gly Val Gln Lys Phe Phe Ala Val Leu Trp Leu Gly
145 150 155 160
Arg Asp Ser Phe Val Arg Phe Ser Pro Ser Val Ser Leu Arg Arg Met
165 170 175
Ala Asp Glu His Gly Thr Asp Lys Thr Ile Ala Gln Lys Leu Ala Arg
180 185 190
Val Ala Arg Met His Phe Ala Arg Gln Arg Leu Ala Ala Val Gly Pro
195 200 205
Arg Leu Pro Ala Arg Gln Asp Leu Phe Asn Lys Leu Leu Ala Ser Arg
210 215 220
Ala Ile Ala Lys Ala Val Glu Asp Glu Ala Arg Ser Lys Lys Ile Ser
225 230 235 240
His Glu Lys Ala Gln Gln Asn Ala Ile Ala Leu Met Glu Glu Ile Ala
245 250 255
Ala Asn Phe Ser Tyr Glu Met Ile Arg Leu Thr Asp Arg Ile Leu Gly
260 265 270
Phe Thr Trp Asn Arg Leu Tyr Gln Gly Ile Asn Val His Asn Ala Glu
275 280 285
Arg Val Arg Gln Leu Ala His Asp Gly His Glu Leu Val Tyr Val Pro
290 295 300
Cys His Arg Ser His Met Asp Tyr Leu Leu Leu Ser Tyr Val Leu Tyr
305 310 315 320
His Gln Gly Leu Val Pro Pro His Ile Ala Ala Gly Ile Asn Leu Asn
325 330 335
Phe Trp Pro Ala Gly Pro Ile Phe Arg Arg Leu Gly Ala Phe Phe Ile
340 345 350
Arg Arg Thr Phe Lys Gly Asn Lys Leu Tyr Ser Thr Val Phe Arg Glu
355 360 365
Tyr Leu Gly Glu Leu Phe Ser Arg Gly Tyr Ser Val Glu Tyr Phe Val
370 375 380
Glu Gly Gly Arg Ser Arg Thr Gly Arg Leu Leu Asp Pro Lys Thr Gly
385 390 395 400
Thr Leu Ser Met Thr Ile Gln Ala Met Leu Arg Gly Gly Thr Arg Pro
405 410 415
Ile Thr Leu Ile Pro Ile Tyr Ile Gly Tyr Glu His Val Met Glu Val
420 425 430
Gly Thr Tyr Ala Lys Glu Leu Arg Gly Ala Thr Lys Glu Lys Glu Ser
435 440 445
Leu Pro Gln Met Leu Arg Gly Leu Ser Lys Leu Arg Asn Leu Gly Gln
450 455 460
Gly Tyr Val Asn Phe Gly Glu Pro Met Pro Leu Met Thr Tyr Leu Asn
465 470 475 480
Gln His Val Pro Asp Trp Arg Glu Ser Ile Asp Pro Ile Glu Ala Val
485 490 495
Arg Pro Ala Trp Leu Thr Pro Thr Val Asn Asn Ile Ala Ala Asp Leu
500 505 510
Met Val Arg Ile Asn Asn Ala Gly Ala Ala Asn Ala Met Asn Leu Cys
515 520 525
Cys Thr Ala Leu Leu Ala Ser Arg Gln Arg Ser Leu Thr Arg Glu Gln
530 535 540
Leu Thr Glu Gln Leu Asn Cys Tyr Leu Asp Leu Met Arg Asn Val Pro
545 550 555 560
Tyr Ser Thr Asp Ser Thr Val Pro Ser Ala Ser Ala Ser Glu Leu Ile
565 570 575
Asp His Ala Leu Gln Met Asn Lys Phe Glu Val Glu Lys Asp Thr Ile
580 585 590
Gly Asp Ile Ile Ile Leu Pro Arg Glu Gln Ala Val Leu Met Thr Tyr
595 600 605
Tyr Arg Asn Asn Ile Ala His Met Leu Val Leu Pro Ser Leu Met Ala
610 615 620
Ala Ile Val Thr Gln His Arg His Ile Ser Arg Asp Val Leu Met Glu
625 630 635 640
His Val Asn Val Leu Tyr Pro Met Leu Lys Ala Glu Leu Phe Leu Arg
645 650 655
Trp Asp Arg Asp Glu Leu Pro Asp Val Ile Asp Ala Leu Ala Asn Glu
660 665 670
Met Gln Arg Gln Gly Leu Ile Thr Leu Gln Asp Asp Glu Leu His Ile
675 680 685
Asn Pro Ala His Ser Arg Thr Leu Gln Leu Leu Ala Ala Gly Ala Arg
690 695 700
Glu Thr Leu Gln Arg Tyr Ala Ile Thr Phe Trp Leu Leu Ser Ala Asn
705 710 715 720
Pro Ser Ile Asn Arg Gly Thr Leu Glu Lys Glu Ser Arg Thr Val Ala
725 730 735
Gln Arg Leu Ser Val Leu His Gly Ile Asn Ala Pro Glu Phe Phe Asp
740 745 750
Lys Ala Val Phe Ser Ser Leu Val Leu Thr Leu Arg Asp Glu Gly Tyr
755 760 765
Ile Ser Asp Ser Gly Asp Ala Glu Pro Ala Glu Thr Met Lys Val Tyr
770 775 780
Gln Leu Leu Ala Glu Leu Ile Thr Ser Asp Val Arg Leu Thr Ile Glu
785 790 795 800
Ser Ala Thr Gln Gly Glu Gly
805
<210> 23
<211> 1092
<212> PRT
<213> Ustilago maydis
<400> 23
Met Thr Gly Thr Ser Asn Leu Pro Ala Ala Ser Ser Gln Asp Ser Ala
1 5 10 15
Val Ile Thr Thr Leu Ser Ser Pro Ser Glu Ala His Leu Ser Thr Ala
20 25 30
Ser His Pro Ser Ala Ala Ala Ser Ser Thr Ala Ser Ala Gln His Pro
35 40 45
Pro Thr Ile Asp Thr His Pro Thr Arg Leu Ser Ser Asn Asp Pro Leu
50 55 60
Lys Lys His Pro Asn Thr Ala Ile Ala Lys Gly Thr Ala Ala Glu Val
65 70 75 80
Gly Ser Lys Gln Lys Glu Phe Gln Arg Arg Ala Gln His Leu Ser Leu
85 90 95
Thr Ser Lys Pro Ile Pro Tyr Ala Arg Gln Gly Asp Ser Pro Arg Leu
100 105 110
Leu Val Val Leu Lys Asp Phe Asp Arg Leu Leu Arg Ser Leu Pro Ile
115 120 125
Pro Leu His His Ile Val Pro Thr Ile Val Ala Arg Phe Ile Cys Arg
130 135 140
Val Phe Arg Ser Gln Asn Ile Met Ala Ser Asn Ile Ala Phe Asp Ile
145 150 155 160
Ala Leu Phe Phe Trp Arg Ile Ile Ile Asn Leu Phe Phe Arg Glu Ile
165 170 175
Arg Pro Arg Ser Ser Trp Arg Ile Pro Arg Glu Gly Pro Val Ile Phe
180 185 190
Val Ala Ala Pro His His Asn Gln Phe Leu Asp Pro Leu Leu Leu Ala
195 200 205
Ser Glu Val Arg Arg Ala Ser Gly Arg Arg Val Ala Phe Leu Ile Ala
210 215 220
Glu Lys Ser Ile Lys Arg Arg Phe Val Gly Ala Ala Ala Arg Ile Met
225 230 235 240
Gln Ser Ile Pro Val Ala Arg Ala Ala Asp Ser Ala Lys Ala Gly Lys
245 250 255
Gly Tyr Ile Ser Leu His Pro Ser Gly Asp Pro Leu Leu Ile Gln Gly
260 265 270
His Gly Thr Ala Phe Lys Ser Gln Leu Gln Leu Lys Gly Gln Ile Met
275 280 285
Leu Pro Lys Ala Cys Gly His Ala Thr Val Glu Val Val Glu Val Ile
290 295 300
Ser Asp Thr Glu Leu Lys Ile Lys Lys Glu Phe Lys Asp Pro Arg Ala
305 310 315 320
Leu Asp Met Leu Arg Gly Lys Val Pro Gln Pro Glu Pro Thr Lys Ser
325 330 335
Asp Lys Lys Pro Ser Lys Ser Ser Ser Ser Lys Ala Leu Glu Lys Val
340 345 350
Ala Ala Asp Leu Phe Glu Asn Gln Gly Cys Arg Tyr Ser Cys Leu Pro
355 360 365
Phe Val Asp Gln Thr Gln Met Tyr Ala Lys Val Tyr Asp Lys Leu Ala
370 375 380
Glu Gly Gly Cys Leu Gly Ile Phe Pro Glu Gly Gly Ser His Asp Arg
385 390 395 400
Thr Asp Leu Leu Pro Leu Lys Ala Gly Val Val Ile Met Ala Leu Gly
405 410 415
Ala Met Ser Ala Asn Arg Asp Leu Asn Val Arg Ile Val Pro Val Gly
420 425 430
Leu Ser Tyr Phe His Pro His Lys Phe Arg Ser Arg Ala Val Val Glu
435 440 445
Phe Gly Ala Pro Ile Asp Val Pro Arg Gln Leu Val Gly Gln Phe Asp
450 455 460
Glu Gly Gly Glu Gly Lys Arg Lys Ala Val Gly Gln Met Met Asp Ile
465 470 475 480
Val Tyr Asp Gly Leu Lys Gly Val Thr Leu Arg Ala Pro Asp Tyr Glu
485 490 495
Thr Leu Met Val Val Gln Ala Gly Arg Arg Leu Tyr Arg Ala Pro Gly
500 505 510
Gln Ser Leu Ser Leu Gly Gln Thr Val Ala Leu Asn Arg Lys Phe Ile
515 520 525
Met Gly Tyr Leu Gln Phe Lys Asp Glu Pro Arg Val Val Lys Leu Arg
530 535 540
Asp Glu Val Leu Arg Tyr Asn Lys Lys Leu Arg Tyr Ala Gly Leu Arg
545 550 555 560
Asp His Gln Val Glu Arg Ala Thr Arg Ala Gly Trp Arg Ser Leu Gly
565 570 575
Leu Leu Ala Tyr Arg Leu Gly Leu Leu Gly Leu Trp Gly Gly Leu Ala
580 585 590
Leu Pro Gly Ala Val Leu Asn Ser Pro Ile Ile Ile Leu Ala Lys Ile
595 600 605
Ile Ser His Lys Lys Ala Lys Glu Ala Leu Ala Ala Ser Gln Val Lys
610 615 620
Val Ala Gly Arg Asp Val Leu Ala Thr Trp Lys Val Leu Val Ser Leu
625 630 635 640
Gly Val Ala Pro Ile Leu Tyr Ser Phe Tyr Ala Ala Leu Ala Thr Tyr
645 650 655
Leu Ala His Arg Leu Glu Leu Ser Pro Arg Thr Arg Ala Leu Met Pro
660 665 670
Leu Tyr Thr Leu Ile Val Leu Pro Thr Met Ser Tyr Ser Ala Leu Lys
675 680 685
Phe Ala Glu Val Gly Ile Asp Ile Tyr Lys Ser Leu Pro Pro Leu Phe
690 695 700
Ile Ser Leu Ile Pro Gly Asn His Lys Val Ile Leu Asp Leu Gln Gln
705 710 715 720
Thr Arg Thr Lys Ile Ser Ala Asp Met His Ala Leu Ile Asp Glu Leu
725 730 735
Ala Pro Gln Val Trp Glu Asp Phe Ala Glu Asn Arg Met Leu Pro Ser
740 745 750
Ala Ser Ala Pro Pro Thr Pro Ser Arg Glu Ala Leu Val Trp Lys Asp
755 760 765
Lys Lys Gln Ser Ser Ser Ala Ala Ser Asp Ala Leu Ser His Pro Leu
770 775 780
Gln Trp Met Asp Glu Arg Leu Phe Gly Trp Gly Arg Arg Arg His Ser
785 790 795 800
Ser Thr Arg Arg Ser Leu Thr Ala Glu Glu Ile Lys His Leu Arg Ser
805 810 815
Pro Ser Leu Thr Arg Gly Ser Ala Lys Asp Glu Asn Ser Val Leu Asp
820 825 830
Asp Glu Asp Gly Ala Arg Phe Glu Gly Glu Gly Asp Gly Ser Leu Asp
835 840 845
Asp Val Ser Glu Gly Ser Ser Ser Phe Ile Glu Ser Gly Glu Glu Asp
850 855 860
Glu Gly Asp Tyr Glu Ala Val Phe Ser Met Leu Asn Pro Gln Asn Leu
865 870 875 880
Leu Asn Gly Leu Arg Asn Gly Gly Leu Ser Pro Gly Thr Pro Gly Ser
885 890 895
Gly Gly Arg Arg Ser Arg Thr His Ser Arg Ser Arg Ser Gly Ser Arg
900 905 910
Gly Ser Ile Gly Gly Val Ala Ser Gly Glu Thr Phe Ala Glu Lys Arg
915 920 925
Asn Arg Ser Ser Gln Asp Leu Arg Ala Leu Met Arg Glu Gly Gly Ala
930 935 940
Met Ser Pro Thr Thr Thr Arg Ser Ser Gly Ile Leu Glu Gly Ala Gly
945 950 955 960
Ser Lys Gln Ser Ser Arg Thr Ser Ala Leu Pro Ala Ser Leu Ile Thr
965 970 975
Thr Asn Ile Ser Gly Asn Asp Gly Glu Asn Gly Ala Ser Lys Ala Ser
980 985 990
Asn Thr Ser Arg Arg Asn Arg Thr His Ser Leu Ser Glu Asp Val His
995 1000 1005
Val Ser Glu Leu Lys Asn Ala Gly Pro Ala Ala Lys Lys Leu Pro
1010 1015 1020
Phe Ser Ala Ala Ser Gln Ala Phe Glu Met Gln His Glu Ala Ser
1025 1030 1035
Gly Gly Gln Ala Gly Tyr Arg Pro Lys Leu Glu Asn Met Gln Ser
1040 1045 1050
Ala Glu Asn Thr Pro Pro Ala Thr Pro Lys His Glu Asp Lys Lys
1055 1060 1065
Val Leu Thr Ser Ala Ser Ser Val Pro Thr Asp Ser Asn Asp Leu
1070 1075 1080
Asp Gly Lys Asp Asp Gly Lys Ala Gln
1085 1090
<210> 24
<211> 759
<212> PRT
<213> Saccharomyces cerevisiae
<400> 24
Met Pro Ala Pro Lys Leu Thr Glu Lys Phe Ala Ser Ser Lys Ser Thr
1 5 10 15
Gln Lys Thr Thr Asn Tyr Ser Ser Ile Glu Ala Lys Ser Val Lys Thr
20 25 30
Ser Ala Asp Gln Ala Tyr Ile Tyr Gln Glu Pro Ser Ala Thr Lys Lys
35 40 45
Ile Leu Tyr Ser Ile Ala Thr Trp Leu Leu Tyr Asn Ile Phe His Cys
50 55 60
Phe Phe Arg Glu Ile Arg Gly Arg Gly Ser Phe Lys Val Pro Gln Gln
65 70 75 80
Gly Pro Val Ile Phe Val Ala Ala Pro His Ala Asn Gln Phe Val Asp
85 90 95
Pro Val Ile Leu Met Gly Glu Val Lys Lys Ser Val Asn Arg Arg Val
100 105 110
Ser Phe Leu Ile Ala Glu Ser Ser Leu Lys Gln Pro Pro Ile Gly Phe
115 120 125
Leu Ala Ser Phe Phe Met Ala Ile Gly Val Val Arg Pro Gln Asp Asn
130 135 140
Leu Lys Pro Ala Glu Gly Thr Ile Arg Val Asp Pro Thr Asp Tyr Lys
145 150 155 160
Arg Val Ile Gly His Asp Thr His Phe Leu Thr Asp Cys Met Pro Lys
165 170 175
Gly Leu Ile Gly Leu Pro Lys Ser Met Gly Phe Gly Glu Ile Gln Ser
180 185 190
Ile Glu Ser Asp Thr Ser Leu Thr Leu Arg Lys Glu Phe Lys Met Ala
195 200 205
Lys Pro Glu Ile Lys Thr Ala Leu Leu Thr Gly Thr Thr Tyr Lys Tyr
210 215 220
Ala Ala Lys Val Asp Gln Ser Cys Val Tyr His Arg Val Phe Glu His
225 230 235 240
Leu Ala His Asn Asn Cys Ile Gly Ile Phe Pro Glu Gly Gly Ser His
245 250 255
Asp Arg Thr Asn Leu Leu Pro Leu Lys Ala Gly Val Ala Ile Met Ala
260 265 270
Leu Gly Cys Met Asp Lys His Pro Asp Val Asn Val Lys Ile Val Pro
275 280 285
Cys Gly Met Asn Tyr Phe His Pro His Lys Phe Arg Ser Arg Ala Val
290 295 300
Val Glu Phe Gly Asp Pro Ile Glu Ile Pro Lys Glu Leu Val Ala Lys
305 310 315 320
Tyr His Asn Pro Glu Thr Asn Arg Asp Ala Val Lys Glu Leu Leu Asp
325 330 335
Thr Ile Ser Lys Gly Leu Gln Ser Val Thr Val Thr Cys Ser Asp Tyr
340 345 350
Glu Thr Leu Met Val Val Gln Thr Ile Arg Arg Leu Tyr Met Thr Gln
355 360 365
Phe Ser Thr Lys Leu Pro Leu Pro Leu Ile Val Glu Met Asn Arg Arg
370 375 380
Met Val Lys Gly Tyr Glu Phe Tyr Arg Asn Asp Pro Lys Ile Ala Asp
385 390 395 400
Leu Thr Lys Asp Ile Met Ala Tyr Asn Ala Ala Leu Arg His Tyr Asn
405 410 415
Leu Pro Asp His Leu Val Glu Glu Ala Lys Val Asn Phe Ala Lys Asn
420 425 430
Leu Gly Leu Val Phe Phe Arg Ser Ile Gly Leu Cys Ile Leu Phe Ser
435 440 445
Leu Ala Met Pro Gly Ile Ile Met Phe Ser Pro Val Phe Ile Leu Ala
450 455 460
Lys Arg Ile Ser Gln Glu Lys Ala Arg Thr Ala Leu Ser Lys Ser Thr
465 470 475 480
Val Lys Ile Lys Ala Asn Asp Val Ile Ala Thr Trp Lys Ile Leu Ile
485 490 495
Gly Met Gly Phe Ala Pro Leu Leu Tyr Ile Phe Trp Ser Val Leu Ile
500 505 510
Thr Tyr Tyr Leu Arg His Lys Pro Trp Asn Lys Ile Tyr Val Phe Ser
515 520 525
Gly Ser Tyr Ile Ser Cys Val Ile Val Thr Tyr Ser Ala Leu Ile Val
530 535 540
Gly Asp Ile Gly Met Asp Gly Phe Lys Ser Leu Arg Pro Leu Val Leu
545 550 555 560
Ser Leu Thr Ser Pro Lys Gly Leu Gln Lys Leu Gln Lys Asp Arg Arg
565 570 575
Asn Leu Ala Glu Arg Ile Ile Glu Val Val Asn Asn Phe Gly Ser Glu
580 585 590
Leu Phe Pro Asp Phe Asp Ser Ala Ala Leu Arg Glu Glu Phe Asp Val
595 600 605
Ile Asp Glu Glu Glu Glu Asp Arg Lys Thr Ser Glu Leu Asn Arg Arg
610 615 620
Lys Met Leu Arg Lys Gln Lys Ile Lys Arg Gln Glu Lys Asp Ser Ser
625 630 635 640
Ser Pro Ile Ile Ser Gln Arg Asp Asn His Asp Ala Tyr Glu His His
645 650 655
Asn Gln Asp Ser Asp Gly Val Ser Leu Val Asn Ser Asp Asn Ser Leu
660 665 670
Ser Asn Ile Pro Leu Phe Ser Ser Thr Phe His Arg Lys Ser Glu Ser
675 680 685
Ser Leu Ala Ser Thr Ser Val Ala Pro Ser Ser Ser Ser Glu Phe Glu
690 695 700
Val Glu Asn Glu Ile Leu Glu Glu Lys Asn Gly Leu Ala Ser Lys Ile
705 710 715 720
Ala Gln Ala Val Leu Asn Lys Arg Ile Gly Glu Asn Thr Ala Arg Glu
725 730 735
Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu
740 745 750
Glu Gly Lys Glu Gly Asp Ala
755
<210> 25
<211> 743
<212> PRT
<213> Saccharomyces cerevisiae
<400> 25
Met Ser Ala Pro Ala Ala Asp His Asn Ala Ala Lys Pro Ile Pro His
1 5 10 15
Val Pro Gln Ala Ser Arg Arg Tyr Lys Asn Ser Tyr Asn Gly Phe Val
20 25 30
Tyr Asn Ile His Thr Trp Leu Tyr Asp Val Ser Val Phe Leu Phe Asn
35 40 45
Ile Leu Phe Thr Ile Phe Phe Arg Glu Ile Lys Val Arg Gly Ala Tyr
50 55 60
Asn Val Pro Glu Val Gly Val Pro Thr Ile Leu Val Cys Ala Pro His
65 70 75 80
Ala Asn Gln Phe Ile Asp Pro Ala Leu Val Met Ser Gln Thr Arg Leu
85 90 95
Leu Lys Thr Ser Ala Gly Lys Ser Arg Ser Arg Met Pro Cys Phe Val
100 105 110
Thr Ala Glu Ser Ser Phe Lys Lys Arg Phe Ile Ser Phe Phe Gly His
115 120 125
Ala Met Gly Gly Ile Pro Val Pro Arg Ile Gln Asp Asn Leu Lys Pro
130 135 140
Val Asp Glu Asn Leu Glu Ile Tyr Ala Pro Asp Leu Lys Asn His Pro
145 150 155 160
Glu Ile Ile Lys Gly Arg Ser Lys Asn Pro Gln Thr Thr Pro Val Asn
165 170 175
Phe Thr Lys Arg Phe Ser Ala Lys Ser Leu Leu Gly Leu Pro Asp Tyr
180 185 190
Leu Ser Asn Ala Gln Ile Lys Glu Ile Pro Asp Asp Glu Thr Ile Ile
195 200 205
Leu Ser Ser Pro Phe Arg Thr Ser Lys Ser Lys Val Val Glu Leu Leu
210 215 220
Thr Asn Gly Thr Asn Phe Lys Tyr Ala Glu Lys Ile Asp Asn Thr Glu
225 230 235 240
Thr Phe Gln Ser Val Phe Asp His Leu His Thr Lys Gly Cys Val Gly
245 250 255
Ile Phe Pro Glu Gly Gly Ser His Asp Arg Pro Ser Leu Leu Pro Ile
260 265 270
Lys Ala Gly Val Ala Ile Met Ala Leu Gly Ala Val Ala Ala Asp Pro
275 280 285
Thr Met Lys Val Ala Val Val Pro Cys Gly Leu His Tyr Phe His Arg
290 295 300
Asn Lys Phe Arg Ser Arg Ala Val Leu Glu Tyr Gly Glu Pro Ile Val
305 310 315 320
Val Asp Gly Lys Tyr Gly Glu Met Tyr Lys Asp Ser Pro Arg Glu Thr
325 330 335
Val Ser Lys Leu Leu Lys Lys Ile Thr Asn Ser Leu Phe Ser Val Thr
340 345 350
Glu Asn Ala Pro Asp Tyr Asp Thr Leu Met Val Ile Gln Ala Ala Arg
355 360 365
Arg Leu Tyr Gln Pro Val Lys Val Arg Leu Pro Leu Pro Ala Ile Val
370 375 380
Glu Ile Asn Arg Arg Leu Leu Phe Gly Tyr Ser Lys Phe Lys Asp Asp
385 390 395 400
Pro Arg Ile Ile His Leu Lys Lys Leu Val Tyr Asp Tyr Asn Arg Lys
405 410 415
Leu Asp Ser Val Gly Leu Lys Asp His Gln Val Met Gln Leu Lys Thr
420 425 430
Thr Lys Leu Glu Ala Leu Arg Cys Phe Val Thr Leu Ile Val Arg Leu
435 440 445
Ile Lys Phe Ser Val Phe Ala Ile Leu Ser Leu Pro Gly Ser Ile Leu
450 455 460
Phe Thr Pro Ile Phe Ile Ile Cys Arg Val Tyr Ser Glu Lys Lys Ala
465 470 475 480
Lys Glu Gly Leu Lys Lys Ser Leu Val Lys Ile Lys Gly Thr Asp Leu
485 490 495
Leu Ala Thr Trp Lys Leu Ile Val Ala Leu Ile Leu Ala Pro Ile Leu
500 505 510
Tyr Val Thr Tyr Ser Ile Leu Leu Ile Ile Leu Ala Arg Lys Gln His
515 520 525
Tyr Cys Arg Ile Trp Val Pro Ser Asn Asn Ala Phe Ile Gln Phe Val
530 535 540
Tyr Phe Tyr Ala Leu Leu Val Phe Thr Thr Tyr Ser Ser Leu Lys Thr
545 550 555 560
Gly Glu Ile Gly Val Asp Leu Phe Lys Ser Leu Arg Pro Leu Phe Val
565 570 575
Ser Ile Val Tyr Pro Gly Lys Lys Ile Glu Glu Ile Gln Thr Thr Arg
580 585 590
Lys Asn Leu Ser Leu Glu Leu Thr Ala Val Cys Asn Asp Leu Gly Pro
595 600 605
Leu Val Phe Pro Asp Tyr Asp Lys Leu Ala Thr Glu Ile Phe Ser Lys
610 615 620
Arg Asp Gly Tyr Asp Val Ser Ser Asp Ala Glu Ser Ser Ile Ser Arg
625 630 635 640
Met Ser Val Gln Ser Arg Ser Arg Ser Ser Ser Ile His Ser Ile Gly
645 650 655
Ser Leu Ala Ser Asn Ala Leu Ser Arg Val Asn Ser Arg Gly Ser Leu
660 665 670
Thr Asp Ile Pro Ile Phe Ser Asp Ala Lys Gln Gly Gln Trp Lys Ser
675 680 685
Glu Gly Glu Thr Ser Glu Asp Glu Asp Glu Phe Asp Glu Lys Asn Pro
690 695 700
Ala Ile Val Gln Thr Ala Arg Ser Ser Asp Leu Asn Lys Glu Asn Ser
705 710 715 720
Arg Asn Thr Asn Ile Ser Ser Lys Ile Ala Ser Leu Val Arg Gln Lys
725 730 735
Arg Glu His Glu Lys Lys Glu
740
<210> 26
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Eco-MaGPAT3-F
<400> 26
gaattcatgg gtctccagat ctatgacttc gtctc 35
<210> 27
<211> 34
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Sal-MaGPAT3-R
<400> 27
gtcgacttat gcctccttag acttgactgc atcc 34
<210> 28
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Not-MaGPAT4-F1
<400> 28
gcggccgcat gacaaccggc gacagtaccg 30
<210> 29
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Not-MaGPAT4-F2
<400> 29
gcggccgcat gcccatcgtt ccagctcag 29
<210> 30
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Bam-MaGPAT4-R
<400> 30
ggatccttat aatttcgggg cgccatcg 28
<210> 31
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Xba1-Des-SCT1-F
<400> 31
tctagaatgc ctgcaccaaa actcac 26
<210> 32
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Xba1-Des-SCT1-R
<400> 32
tctagaccac aaggtgatca ggaaga 26
<210> 33
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер SCT1outORF-F
<400> 33
agtgtaggaa gcccggaatt 20
<210> 34
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер SCT1inORF-R
<400> 34
gcgtagatcc aacagactac 20
<210> 35
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер LEU2inORF-F
<400> 35
ttgcctcttc caagagcaca 20
<210> 36
<211> 54
<212> ДНК
<213> Искусственная последовательность
<220>
<223> олиго ДНК MCS-for-pUC18-F2
<400> 36
aattcataag aatgcggccg ctaaactatt ctagactagg tcgacggcgc gcca 54
<210> 37
<211> 54
<212> ДНК
<213> Искусственная последовательность
<220>
<223> олиго ДНК MCS-for-pUC18-R2
<400> 37
agcttggcgc gccgtcgacc tagtctagaa tagtttagcg gccgcattct tatg 54
<210> 38
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер Not1-GAPDHt-F
<400> 38
agcggccgca taggggagat cgaacc 26
<210> 39
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер EcoR1-Asc1-GAPDHt-R
<400> 39
agaattcggc gcgccatgca cgggtccttc tca 33
<210> 40
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер URA5g-F1
<400> 40
gtcgaccatg acaagtttgc 20
<210> 41
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер URA5g-R1
<400> 41
gtcgactgga agacgagcac g 21
<210> 42
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер hisHp+URA5-F
<400> 42
ggcaaacttg tcatgaagcg aaagagagat tatgaaaaca agc 43
<210> 43
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер hisHp+MGt-F
<400> 43
cactcccttt tcttaattgt tgagagagtg ttgggtgaga gt 42
<210> 44
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер pDuraSC-GAPt-F
<400> 44
taagaaaagg gagtgaatcg cataggg 27
<210> 45
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер URA5gДНК-F
<400> 45
catgacaagt ttgccaagat gcg 23
<210> 46
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер pDurahG-hisp-R
<400> 46
attgttgaga gagtgttggg tgagagtg 28
<210> 47
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер MaGPAT4+hisp-F
<400> 47
cactctctca acaatatgac aaccggcgac agtaccgc 38
<210> 48
<211> 38
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер MaGPAT4+MGt-R
<400> 48
cactcccttt tcttattata atttcggggc gccatcgc 38
<210> 49
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер GPAT4-RT1
<400> 49
gagtgcttca tcgagggcac c 21
<210> 50
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер GPAT4-RT2
<400> 50
tccttcactg tcaacctcga tcac 24
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБЫ ПОЛУЧЕНИЯ ПРОМЫШЛЕННЫХ ПРОДУКТОВ ИЗ РАСТИТЕЛЬНЫХ ЛИПИДОВ | 2015 |
|
RU2743384C2 |
| РАСТЕНИЯ С МОДИФИЦИРОВАННЫМИ ПРИЗНАКАМИ | 2017 |
|
RU2809117C2 |
| Выделенная нуклеиновая кислота, которая кодирует слитый белок на основе FVIII-BDD и гетерологичного сигнального пептида, и ее применение | 2022 |
|
RU2818229C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| МУТАНТНЫЙ ПОЛИПЕПТИД ГИДРОКСИФЕНИЛПИРУВАТДИОКСИГЕНАЗА, КОДИРУЮЩИЙ ЕГО ГЕН И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2822892C1 |
| Мутантная нитрилгидратаза, нуклеиновая кислота, кодирующая указанную мутантную нитрилгидратазу, вектор экспрессии и трансформант, содержащие указанную нуклеиновую кислоту, способ получения указанной мутантной нитрилгидратазы и способ получения амидного соединения | 2017 |
|
RU2736086C1 |
| РЕКОМБИНАНТНЫЕ КЛЕТКИ, ПРОДУЦИРУЮЩИЕ ГИАЛУРОНОВУЮ КИСЛОТУ | 2022 |
|
RU2839983C2 |
| Вакцина на основе AAV5 для индукции специфического иммунитета к вирусу SARS-CoV-2 и/или профилактики коронавирусной инфекции, вызванной SARS-CoV-2 | 2020 |
|
RU2760301C1 |
| НЕ ТОКСИЧЕСКИЕ ПОЛИПЕПТИДЫ КЛОСТРИДИАЛЬНОГО НЕЙРОТОКСИНА ДЛЯ ПРИМЕНЕНИЯ В ЛЕЧЕНИИ НЕВРОЛОГИЧЕСКИХ НАРУШЕНИЙ | 2020 |
|
RU2839368C1 |
| ПРОТИВОРАКОВЫЕ ВАКЦИНЫ | 2017 |
|
RU2718663C2 |
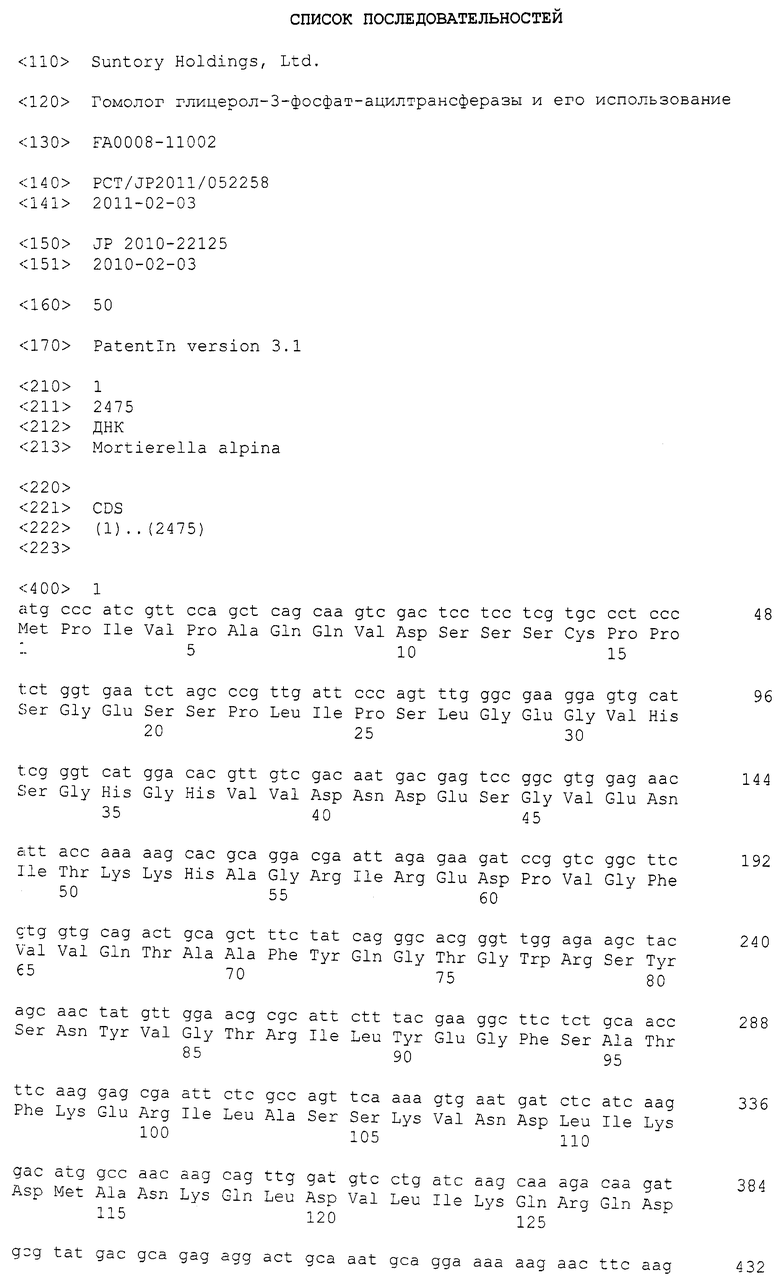



























































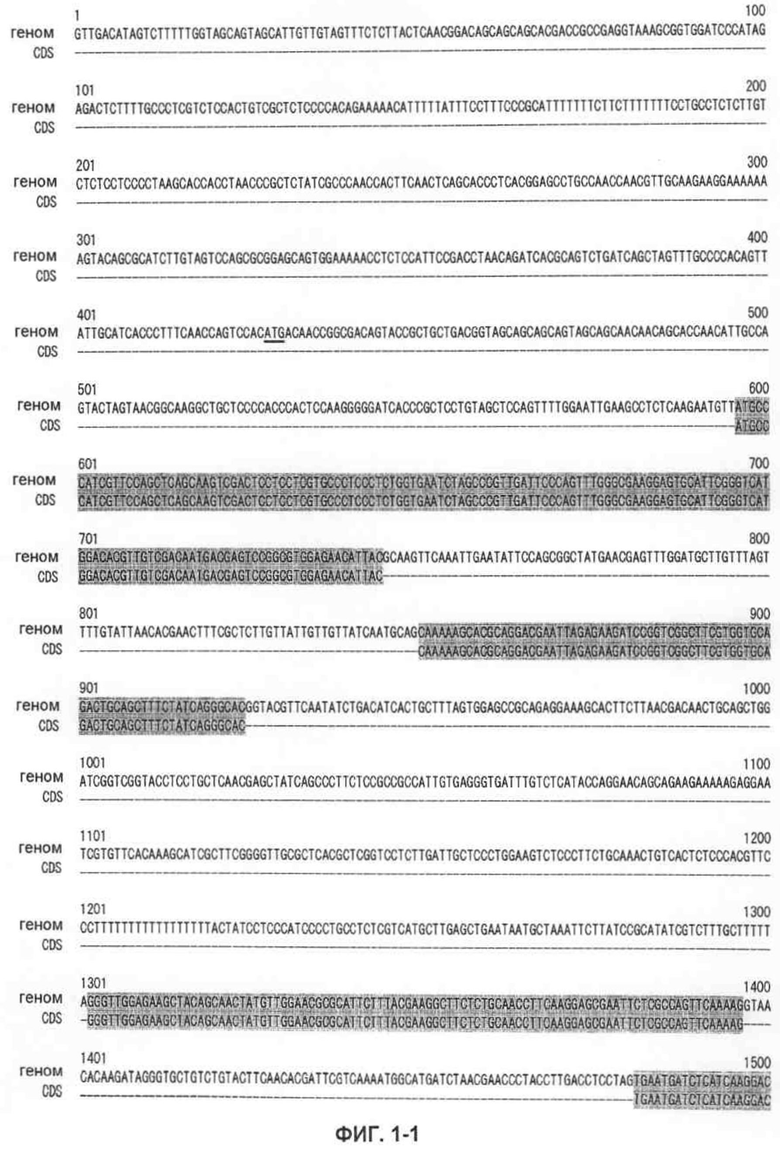
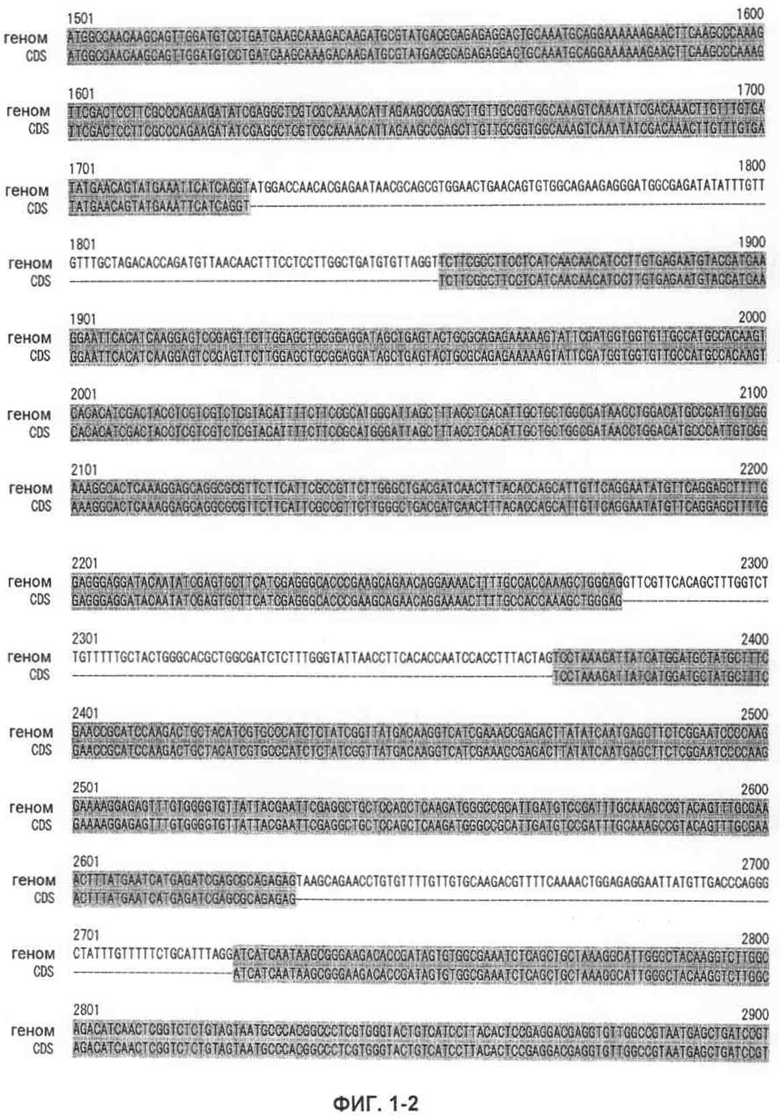






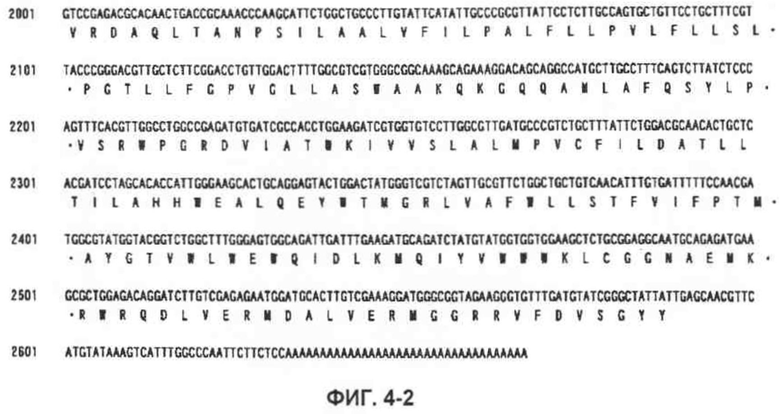

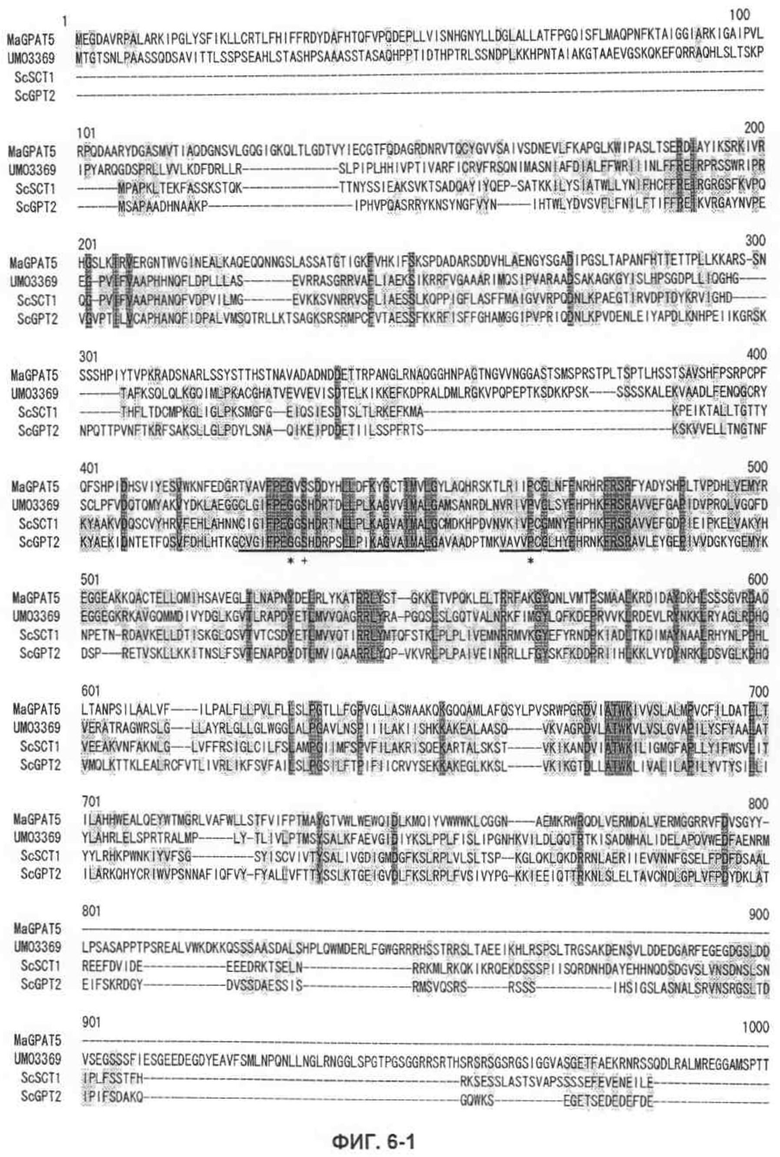

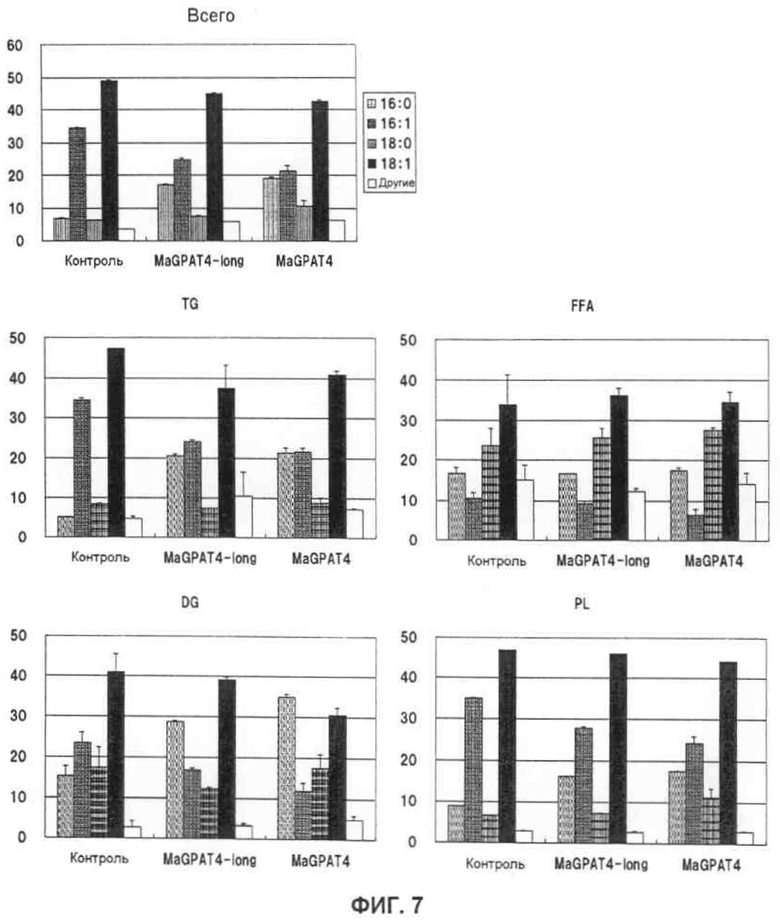
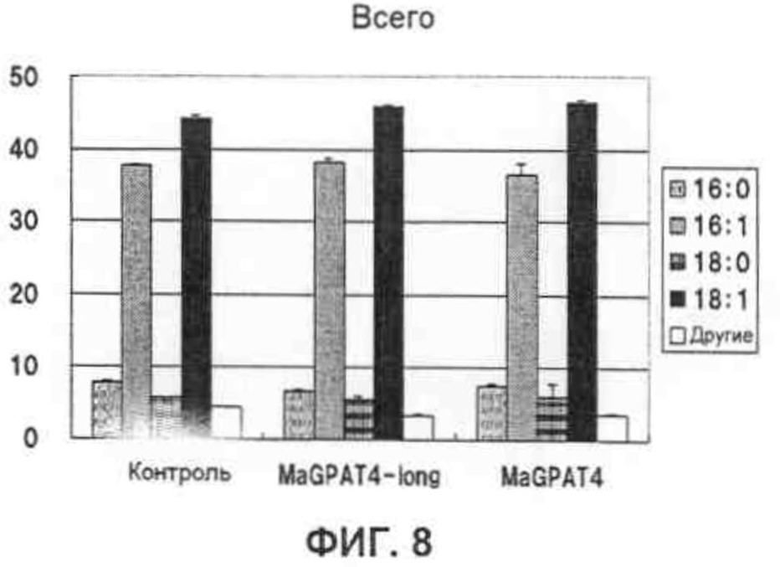
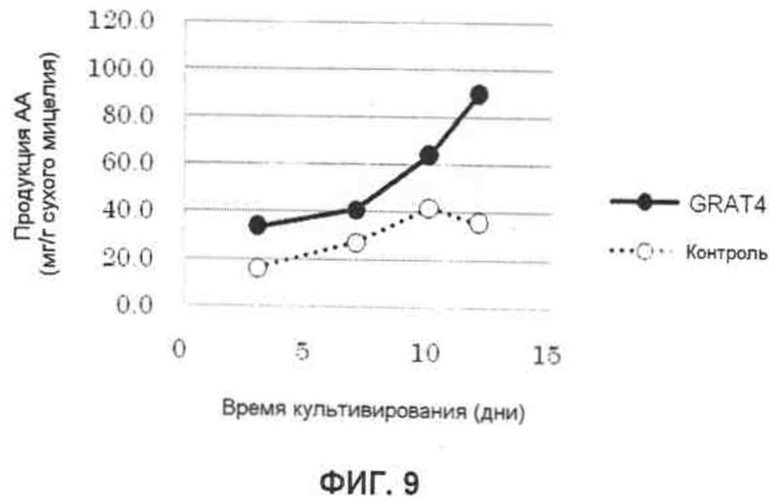
Группа изобретений относится к биологии, в частности к новому гену глицерол-3-фосфат-ацилтрансферазы и/или глицеролфосфат - ацилтрансферазы. Изобретение относится также к белку, кодируемому данным геном, вектору экспрессии, содержащему заявленную нуклеиновую кислоту, и трансформанту, трансформированному указанным вектором. Изобретение относится к композиции жирных кислот, продуцируемых заявленным трансформантом, и к способу получения указанной композиции. Группа изобретений обеспечивает возможность получения продукции жира с заданным композиционным отношением жирных кислот с обеспечением повышения содержания заданной жирной кислоты или повышением количества запасного липида триацилглицерола (TG). 9 н. п. ф-лы, 11 табл., 8 пр.
1. Нуклеиновая кислота, выбранная из (a)-(g) ниже:
(a) нуклеиновой кислоты, содержащей нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 15 аминокислот в аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9 и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(b) нуклеиновой кислоты, содержащей нуклеотидную последовательность, гибридизуемую в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующей белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(c) нуклеиновой кислоты, содержащей нуклеотидную последовательность, состоящую из нуклеотидной последовательности, которая на 95% или более идентична нуклеотидной последовательности SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8, и кодирующей белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(d) нуклеиновой кислоты, содержащей нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности, которая на 95% или более идентична аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(e) нуклеиновой кислоты, содержащей нуклеотидную последовательность, гибридизуемую в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей белок, состоящий из аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и кодирующей белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью;
(f) нуклеиновой кислоты, содержащей нуклеотидную последовательность, гибридизуемую в условиях 2×SSC при 50°C с нуклеиновой кислотой, состоящей из нуклеотидной последовательности, комплементарной нуклеотидной последовательности SEQ ID NO:7 или SEQ ID NO:12, и включающей экзоны, кодирующие белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью; и
(g) нуклеиновой кислоты, содержащей нуклеотидную последовательность, состоящую из нуклеотидной последовательности, которая на 95% или более идентична нуклеотидной последовательности SEQ ID NO:7 или SEQ ID NO:12, и включающей экзоны, кодирующие белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью.
2. Нуклеиновая кислота, кодирующая белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью по любому из подпунктов, выбранных из (a)-(d) ниже:
(a) нуклеиновой кислоты, содержащей нуклеотидную последовательность SEQ ID NO:1, SEQ ID NO:4 или SEQ ID NO:8;
(b) нуклеиновой кислоты, содержащей нуклеотидную последовательность, кодирующую белок, состоящий из аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9;
(c) нуклеиновой кислоты, содержащей нуклеотидную последовательность SEQ ID NO:3, SEQ ID NO:6 или SEQ ID NO:11; и
(d) нуклеиновой кислоты, содержащей нуклеотидную последовательность SEQ ID NO:7 или SEQ ID NO:12.
3. Белок, выбранный из (a) и (b) ниже:
(a) белка, состоящего из аминокислотной последовательности с делецией, заменой или добавлением от 1 до 15 аминокислот в аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и с глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью; и
(b) белок, состоящий из аминокислотной последовательности, которая на 90% или более идентична аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9, и обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью.
4. Белок, обладающий глицерол-3-фосфат-ацилтрансферазной активностью и/или глицеронфосфат-ацилтрансферазной активностью, состоящий из аминокислотной последовательности SEQ ID NO:2, SEQ ID NO:5 или SEQ ID NO:9.
5. Рекомбинантный вектор экспрессии, содержащий нуклеиновую кислоту по любому из пп. 1 или 2.
6. Трансформант, предназначенный для продукции жирных кислот, трансформированный рекомбинантным вектором по п. 5.
7. Композиция жирных кислот, состоящая из жирных кислот, продуцируемых культивируемым трансформантом по п. 6, для получения пищевого продукта, косметической композиции или фармацевтической композиции, или липида, содержащего жирные кислоты, продуцируемые культивируемым трансформантом по п. 6, для продукции пищевого продукта, косметической композиции или фармацевтической композиции.
8. Способ получения композиции, состоящей из жирных кислот, включающий сбор композиции жирных кислот или липидов, содержащих жирные кислоты, из культуры, получаемой культивированием трансформанта по п. 6.
9. Пищевой продукт, содержащий композицию жирных кислот по п. 7.
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| ТЕПЛООБМЕННИК | 2007 |
|
RU2341751C1 |
| WO 2008156026 A1, 24.12.2008 | |||
| WO 2006052824 A2, 18.05.2006 | |||
| НОВЫЙ КЛАСС ФЕРМЕНТОВ В БИОСИНТЕТИЧЕСКОМ ПУТИ ПОЛУЧЕНИЯ ТРИАЦИЛГЛИЦЕРИНА И РЕКОМБИНАНТНЫЕ МОЛЕКУЛЫ ДНК, КОДИРУЮЩИЕ ЭТИ ФЕРМЕНТЫ | 2000 |
|
RU2272073C2 |
Авторы
Даты
2015-03-27—Публикация
2011-02-03—Подача