Область техники, к которой относится изобретение
Настоящее изобретение относится к полипептиду, содержащему антигенсвязывающий домен и транспортирующий фрагмент, который имеет ингибирующий домен, ингибирующий антигенсвязывающую активность антигенсвязывающего домена, и обладающему более длительным временем полужизни, чем время полужизни антигенсвязывающего домена, в том случае, если он присутствует индивидуально, к способам получения и скрининга полипептидов, фармацевтическим композициям, которые содержат полипептид, способам получения и скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL-, VH- или VHH-доменом, и к библиотекам слитых полипептидов, каждый из которых содержит однодоменное антитело, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL, VH или VHH.
Предпосылки создания изобретения
Антитела привлекли внимание в качестве лекарственных средств, поскольку они обладают высокой стабильностью в плазме и вызывают небольшое количество побочных действий. В частности, большое количество лекарственных средств на основе антител IgG-типа находится в продаже, и большое количество лекарственных средств на основе антител находится настоящее время в разработке (непатентные документы 1 и 2).
Антитела привлекают внимание в качестве лекарственных средств благодаря их высокой стабильности в плазме и небольшому количеству побочных действий. Среди них большое количество лекарственных средства на основе антител IgG-типа поступают в продажу, и в настоящее время много лекарственных средств на основе антител находится в разработке (NPL 1 и 2).
Ритуксан против CD20, цетуксимаб против EGFR, герцептин против HER2, и т.п. к настоящему времени одобрены в качестве терапевтических лекарственных средств в виде лекарственных средств на основе антител против рака (NPL 3). Указанные молекулы антител связываются с их антигенами, которые экспрессируются на раковых клетках, и в результате проявляют цитотоксическую активность в отношении раковых клеток посредством ADCC, ингибирования сигнала и т.д. Известно, что указанная цитотоксическая активность, основанная на ADCC-активности и т.д., зависит от количества антигенов, экспрессируемых на клетках, которые являются мишенями терапевтических антител (NPL 4). Кроме того, высокие уровни экспрессии таргетных антигенов являются предпочтительными с точки зрения воздействий терапевтических антител. Однако, если антиген, хотя и имеющий высокий уровень экспрессии, экспрессируется в здоровых тканях, то цитотоксическая активность, основанная на ADCC-активности и т.д., оказывает воздействие на здоровые клетки. В результате побочные действия становятся серьезной проблемой. В связи с этим, предпочтительно, чтобы антигены, таргетируемые терапевтическими антителами, которые применяют в качестве терапевтических лекарственных средств при раке, экспрессировались специфически на раковых клетках. Например, молекула антитела против ЕрСАМ (молекула клеточной адгезии эпителия), которая известна в качестве ракового антигена, рассматривалась в качестве перспективной в качестве терапевтического лекарственного средства при раке. Однако известно, что ЕрСАМ экспрессируется также в поджелудочной железе. Так, описано, что по результатам клинических испытаний введение антитела к ЕрСАМ вызывает панкреатит в качестве нежелательной реакции из-за цитотоксической активности в отношении поджелудочной железы (NPL 5).
После успехов, достигнутых в отношении лекарственных средств на основе антител, обладающих цитотоксической активностью, основанной на ADCC-активности, опубликованы данные о втором поколении улучшенных молекул антител, обладающих сильной цитотоксической активностью, обусловленной, например, повышением ADCC-активности путем удаления фукозы из N-связанного олигосахарида в нативной Fc-области человеческого IgG1 (NPL 6) или повышением ADCC-активности путем усиления связывания с FcγRIIIa посредством аминокислотной замены в нативной Fc-области человеческого IgG1 (непатентный документ 7). Улучшенные молекулы антител, обладающие более сильной цитотоксической активностью, такие как конъюгат антитела и лекарственного средства (ADC), содержащий антитело, конъюгированное с лекарственным средством, которое обладает сильной цитотоксической активностью (NPL 8), и низкомолекулярное антитело, обладающее цитотоксической активностью в отношении раковых клеток в результате рекрутинга Т-клеток к раковым клеткам (NPL 9), описаны также в качестве лекарственных средств на основе антител, обладающих цитотоксической активностью в отношении раковых клеток, с использованием механизма, отличного от упомянутой выше опосредуемой NK-клетками ADCC-активности.
Указанные молекулы антител, обладающие более сильной цитотоксической активностью, могут проявлять цитотоксическую активность даже в отношении раковых клеток, которые экспрессирует невысокие уровни антигена, но также проявлять цитотоксическую активность в отношении здоровых тканей, в которых экспрессируются низкие уровни антигена, аналогичного антигену раковых клеток. Так, EGFR-BiTE, биспецифическое антитело против CD3 и EGFR, может проявлять сильную цитотоксическую активность в отношении раковых клеток и обладать противоопухолевым действием путем рекрутинга Т-клеток к раковым клеткам, сравнимым с действием цетуксимаба, нативного человеческого IgG1 против EGFR. С другой стороны, установлено также, что серьезные побочные действия проявляются при введении EGFR-BiTE обезьянам циномолгус, поскольку EGFR экспрессируется также в здоровых тканях (NPL 10). Кроме того, в клинических условиях обнаружено, что ADC биватузумаба с мертансином, который содержит мертансин, конъюгированный с антителом против CD44v6, отличающегося высоким уровнем экспрессии на раковых клетках, вызывает серьезную кожную токсичность и гепатотоксичность, поскольку CD44v6 экспрессируется также в здоровых тканях (NPL 11).
Как отмечалось выше, применение антитела, которое может обладать сильной цитотоксической активностью даже в отношении раковых клеток, экспрессирующих низкие уровни антигена, требует, чтобы антиген-мишень экспрессировался исключительно специфическим для рака образом. Однако с учетом того, что антиген-мишень HER2 герцептина или антиген-мишень EGFR цетуксимаба экспрессируется также в здоровых тканях, лишь ограниченное количество раковых антигенов может экспрессироваться исключительно специфическим для рака образом. Поэтому побочные действия, опосредуемые цитотоксическим действием на здоровые ткани, могут становиться проблемой, хотя цитотоксическая активность в отношении раковых клеток может повышаться.
В настоящее время установлено, что ипилимумаб, который повышает опухолевый иммунитет путем ингибирования CTLA4, участвующего в иммуносупрессии при раке, удлиняет общую выживаемость при метастатической меланоме (NPL 12). Однако ипилимумаб системно ингибирует CTLA4 и поэтому вызывает напоминающие аутоиммунитет серьезные побочные действия из-за системной активации иммунитета, хотя повышает опухолевый иммунитет (NPL 13).
При этом лекарственные средства на основе антител, обладающие терапевтическим действием путем ингибирования воспалительных цитокинов при воспалительных и аутоиммунных заболеваниях, известны в качестве лекарственных средств на основе антител, предназначенных для лечения болезней, отличных от рака (NPL 14). Известно, что, например, ремикад или хумира, таргетирующие TNF, и актемра, таргетирующая IL-6R, обладают высоким терапевтическим действием в отношении ревматоидного артрита, при этом инфекционное заболевание проявляется в качестве побочного действия, связанного с системной нейтрализацией этих цитокинов (NPL15).
В качестве методов, пригодных для второго поколения лекарственных средств на основе антител, разработаны различные методы. Например, опубликованы данные о методах, повышающих эффекторные функции, способность связывать антиген, фармакокинетические параметры или стабильность, или снижающих риск иммуногенности (NPL 16). Однако к настоящему времени известно лишь небольшое количество сведений о методах, которые позволяют лекарственным средствам на основе антител действовать специфически на ткань-мишень для разрешения проблемы, обусловленной побочными действиями, указанными выше. Описанные методы включают метод, в котором соединяют антитело с маскирующим пептидом через линкер, который расщепляется протеазой, которая экспрессируется в пораженной области, такой как раковая ткань или воспалительная ткань, маскируя тем самым антигенсвязывающий сайт антитела с помощью маскирующего пептида и ингибируя антигенсвязывающую активность антитела; и отделяют от него в результате диссоциации маскирующий пептид с помощью расщепления протеазой этого линкера так, что антитело сохраняет свою антигенсвязывающую активность и приобретает способность связываться с антигеном в патологической ткани-мишени (NPL 17 и 18 и PTL 1).
Перечень процитированных документов
[Патентные документы]
[PTL 1] WO 2010/081173.
[Непатентные документы]
[NPL 1] Janice М. Reichert, Clark J. Rosensweig, Laura В. Faden и Matthew С. Dewitz, Monoclonal antibody successes in the clinic. Nat. Biotechnol. 23, 2005, cc. 1073-1078.
[NPL 2] Pavlou A.K., Belsey M.J., The therapeutic antibodies market to 2008. Eur. J. Pharm. Biopharm. 59 (3), 2005, cc. 389-396.
[NPL 3] Weiner L.M., Surana R., Wang S., Monoclonal antibodies: versatile platforms for cancer immunotherapy. Nat. Rev. Immunol. 10 (5), 2010, cc. 317-327.
[NPL 4] Lewis G.D, Figari I., Fendly В., Wong W.L., Carter P., Gorman C., Shepard H.M., Differential responses of human tumor cell lines to anti-p185HER2 monoclonal antibodies. Cancer Immunol. Immunotherapy 37, 1993, cc. 255-263.
[NPL 5] de Bono J.S., Tolcher A.W., Forero A., Vanhove G.F., Takimoto C., Bauer R.J., Hammond L. A., Patnaik A., White M.L., Shen S., Khazaeli M.B., Rowinsky E.K., LoBuglio A.F., ING-1, a monoclonal antibody targeting Ep-CAM in patients with advanced adenocarcinomas. Clin. Cancer Res. 10 (22), 2004, cc. 7555-7565.
[NPL 6] Satoh M., Iida S., Shitara K., Non-fucosylated therapeutic antibodies as next-generation therapeutic antibodies. Expert Opin. Biol. Ther. 6 (11), 2006, cc. 1161-1173.
[NPL 7] Desjarlais J.R., Lazar G.A., Zhukovsky E.A., Chu S.Y., Optimizing engagement of the immune system by anti-tumor antibodies: an engineer's perspective. Drug Discov. Today 12 (21-22), 2007, cc. 898-910.
[NPL 8] Alley S.C., Okeley N.M., Senter P.D., Antibody-drug conjugates: targeted drug delivery for cancer. Curr. Opin. Chem. Biol. 14 (4), 2010, cc. 529-537.
[NPL 9] BiTE: Teaching antibodies to engage T-cells for cancer therapy. Baeuerle P.A., Kufer P., Bargou R., Curr. Opin. Mol. Ther. 11 (1), 2009, cc. 22-30.
[NPL 10] Lutterbuese R., Raum T., Kischel R., Hoffmann P., Mangold S., Rattel B., Friedrich M., Thomas O., Lorenczewski G., Rau D., Schaller E., Herrmann I., Wolf A., Urbig T., Baeuerle P.A., Kufer P., T cell-engaging BiTE antibodies specific for EGFR potently eliminate KRAS- and BRAF-mutated colorectal cancer cells. Proc. Natl. Acad. Sci. U.S.A. 107 (28), 2010, cc. 12605-12610.
[NPL 11] Riechelmann H., Sauter A., Golze W., Hanft G., Schroen C., Hoermann K., Erhardt T., Gronau S., Phase I trial with the CD44v6-targeting immunoconjugate bivatuzumab mertansine in head and neck squamous cell carcinoma. Oral Oncol. 44 (9), 2008, cc. 823-829.
[NPL 12] Trinh V.A., Hwu W. J., Ipilimumab in the treatment of melanoma. Expert Opin. Biol. Ther., 14 апреля 2012 г. (doi: 10.1517/14712598.2012.675325).
[NPL 13] Juszczak A., Gupta A., Karavitaki N., Middleton M.R., Grossman A., IPILIMUMAB - A NOVEL IMMUNOMODULATING THERAPY CAUSING AUTOIMMUNE HYPOPHYSITIS: A CASE REPORT AND REVIEW. Eur. J. Endocrinol. 10 апреля 2012 г. (doi: 10.1530/EJE-12-0167).
[NPL 14] Takeuchi T., Kameda H., The Japanese experience with biologic therapies for rheumatoid arthritis. Nat. Rev. Rheumatol. 6 (11), 2010, cc. 644-652.
[NPL 15] Nam J.L., Winthrop K.L., van Vollenhoven R.F., Pavelka K., Valesini G., Hensor E.M., Worthy G., Landewe R., Smolen J.S., Emery P., Buch M.H., Current evidence for the management of rheumatoid arthritis with biological disease-modifying antirheumatic drugs: a systematic literature review informing the EULAR recommendations for the management of RA. Ann. Rheum. Dis. 69 (6), 2010, cc. 976-986.
[NPL 16] Kim S.J., Park Y., Hong H.J., Antibody engineering for the development of therapeutic antibodies. Mol. Cells. 20 (1), 2005, cc. 17-29.
[NPL 17] Desnoyers L.R., Vasiljeva O., Richardson J.H., Yang A., Menendez E.E., Liang T.W., Wong C, Bessette P.H., Kamath K., Moore S. J., Sagert J.G., Hostetter D.R., Han F., Gee J., Flandez J., Markham K., Nguyen M., Krimm M., Wong K.R., Liu S., Daugherty P.S., West J.W., Lowman H.B., Tumor-specific activation of an EGFR-targeting probody enhances therapeutic index. Sci Transl Med. 5(207), 16 октября 2013 г„ 207ra144.
[NPL 18] Polu K.R., Lowman H.B., Probody therapeutics for targeting antibodies to diseased tissue. Expert Opin Biol Ther. 14(8), август 2014 г., cc. 1049-1053.
Краткое изложение сущности изобретения
Задача, положенная в основу изобретения
При создании настоящего изобретения высказано предположение о том, что описанные выше методики диссоциации путем расщепления протеазой маскирующего пептида, который ингибирует антигенсвязывающую активность антитела так, что антитело сохраняет свою антигенсвязывающую активность, могут вызывать побочные действия, поскольку антитело, расщепленное в пораженной области, может попадать в здоровые ткани с кровотоком, поскольку расщепление протеазой является необратимым.
Настоящее изобретение создано на основе указанной идеи. Объектом настоящего изобретения является фармацевтическая композиция, которую можно применять для лечения заболевания, снижая при этом побочные действия, и ее действующее вещество. Другим объектом настоящего изобретения являются способы скрининга и получения фармацевтической композиции и действующего вещества.
Решение указанной задачи
При создании настоящего изобретения проведены широкие исследования и в результате были разработаны полипептиды, содержащие антигенсвязывающий домен и транспортирующий фрагмент, который имеет ингибирующий домен, ингибирующий связывающую активность антигенсвязывающего домена, и обладающий более длительными временем полужизни, чем время полужизни антигенсвязывающего домена в том случае, когда он присутствует индивидуально. Предполагается, что применение полипептида может обеспечивать восстановление у антигенсвязывающего домена его антигенсвязывающей активности в пораженной(ых) болезнью ткани(ях) и проявление антигенсвязывающей активности в пораженной(ых) болезнью ткани(ях). Кроме того, системное распределение активированной формы антигенсвязывающего домена может подавляться благодаря различию во временах полужизни между полипептидом, содержащим антигенсвязывающий домен, антигенсвязывающая активность которого ингибируется, и полипептидом, содержащим антигенсвязывающий домен, антигенсвязывающая активность которого восстанавливается. Кроме того, при создании настоящего изобретения установлено, что полипептиды или фармацевтические композиции, содержащие полипептид, можно применять для лечения заболевания, и установлено также, что полипептиды или фармацевтические композиции можно применять для лечения заболевания, где применение включает введение полипептида; и что полипептиды можно применять для приготовления лекарственного средства, предназначенного для лечения заболевания. И, наконец, при создании настоящего изобретения разработаны также способы скрининга и получения полипептида, способы получения и скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL, VH или VHH, и библиотека, включающая однодоменное антитело, антигенсвязывающую активность которого можно ингибировать путем ассоциации с конкретным VL, VH или VHH.
Настоящее изобретение базируется на указанных данных, и оно конкретно включает представленные ниже варианты осуществления изобретения.
(1) Полипептид, содержащий антигенсвязывающий домен и транспортирующий фрагмент, где транспортирующий фрагмент имеет ингибирующий домен, который ингибирует антигенсвязывающую активность антигенсвязывающего домена, и антигенсвязывающий домен, который имеет более короткое время полужизни в крови, чем транспортирующий фрагмент.
(2) Полипептид по п. (1), в котором молекулярная масса антигенсвязывающего домена меньше, чем молекулярная масса транспортирующего фрагмента.
(3) Полипептид по п. (1) или п. (2), в котором молекулярная масса антигенсвязывающего домена составляет 60 кДа или менее.
(4) Полипептид по одному из п.п. (1)-(3), в котором транспортирующий фрагмент обладает FcRn-связывающей активностью, а антигенсвязывающий домен не обладает FcRn-связывающей активностью или имеет более слабую FcRn-связывающую активность, чем активность транспортирующего фрагмента.
(5) Полипептид по одному из п.п. (1)-(4), в котором антигенсвязывающий домен обладает способностью высвобождаться из полипептида, и антигенсвязывающий домен, высвободившийся из полипептида, имеет более высокую антигенсвязывающую активность, чем его активность до высвобождения.
(6) Полипептид по одному из п.п. (1)-(5), в котором ингибирующий домен транспортирующего фрагмента находится в ассоциации с антигенсвязывающим доменом и тем самым ингибирует антигенсвязывающую активность антигенсвязывающего домена.
(7) Полипептид по п. (5), где полипептид содержит сайт расщепления, где сайт расщепления расщепляется таким образом, что антигенсвязывающий домен приобретает способность высвобождаться из полипептида.
(8) Полипептид по п. (6), где полипептид содержит сайт расщепления, где сайт расщепления расщепляется таким образом, что аннулируется ассоциация ингибирующего домена транспортирующего фрагмента с антигенсвязывающим доменом.
(9) Полипептид по п. (7) или п. (8), в котором сайт расщепления содержит расщепляемую протеазой последовательность.
(10) Полипептид по п. (9), где протеаза представляет собой протеазу, специфическую для ткани-мишени.
(11) Полипептид по п. (10), где ткань-мишень представляет собой раковую ткань или воспалительную ткань.
(12) Полипептид по п. (9), где протеаза представляет собой по меньшей мере одну протеазу, выбранную из матриптазы, урокиназы (uPA) и металлопротеиназы.
(13) Полипептид по п. (12), где протеаза представляет собой по меньшей мере одну протеазу, выбранную из MT-SP1, uPA, ММР-2, ММР-9, ADAMTS5, ММР-7 и ММР-13.
(14) Полипептид по п. (9), в котором расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 12, 25, 26, 78-81, 83, 84, 91, 168-178, 193-195, 833-852 и 1062-1081, и последовательностей, представленных в таблице 1.
(15) Полипептид по одному из п.п. (9)-(14), в котором к одному концу расщепляемой протеазой последовательности дополнительно присоединен первый гибкий линкер.
(16) Полипептид по п. (15), в котором к другому концу расщепляемой протеазой последовательности дополнительно присоединен второй гибкий линкер.
(17) Полипептид по п. (15), в котором первый гибкий линкер представляет собой гибкий линкер, состоящий из глицин-серинового полимера.
(18) Полипептид по п. (16), в котором второй гибкий линкер представляет собой гибкий линкер, состоящий из глицин-серинового полимера.
(19) Полипептид по одному из п.п. (1)-(18), в котором антигенсвязывающий домен содержит однодоменное антитело или представляет собой однодоменное антитело, в котором ингибирующий домен транспортирующего фрагмента ингибирует антигенсвязывающую активность однодоменного антитела.
(20) Полипептид по п. (19), в котором однодоменное антитело представляет собой VHH, VH, который сам обладает антигенсвязывающей активностью, или VL, который сам обладает антигенсвязывающей активностью.
(21) Полипептид по одному из п.п. (1)-(20), в котором антигенсвязывающий домен содержит однодоменное антитело, и ингибирующий домен транспортирующего фрагмента представляет собой VHH, VH антитела или VL антитела, в котором антигенсвязывающая активность однодоменного антитела ингибируется VHH, VH антитела или VL антитела.
(22) Полипептид по одному из п.п. (1)-(21), в котором антигенсвязывающий домен содержит однодоменное антитело, и ингибирующий домен транспортирующего фрагмента представляет собой VHH, VH антитела или VL антитела, в котором антигенсвязывающая активность однодоменного антитела ингибируется путем его ассоциации с VHH, VH антитела или VL антитела.
(23) Полипептид по одному из п.п. (19)-(22), в котором однодоменное антитело представляет собой VHH или VH, который сам обладает антигенсвязывающей активностью, и ингибирующий домен транспортирующего фрагмента представляет собой VL антитела, в котором антигенсвязывающая активность VHH или VH, который сам обладает антигенсвязывающей активностью, ингибирует путем его ассоциации с VL антитела.
(24) Полипептид по одному из п.п. (19)-(23), в котором однодоменное антитело представляет собой VHH, где VHH имеет аминокислотную замену по меньшей мере в одном положении, выбранном из аминокислотных положений 37, 44, 45 и 47 (все пронумерованы согласно нумерации Кэбота).
(25) Полипептид по одному из п.п. (19)-(23), в котором однодоменное антитело представляет собой VHH, где VHH содержит по меньшей мере одну аминокислоту, выбранную из аминокислот 37V, 44G, 45L и 47W (все пронумерованы согласно нумерации Кэбота).
(26) Полипептид по одному из п.п. (19)-(23), в котором однодоменное антитело представляет собой VHH, где VHH содержит по меньшей мере одну аминокислотную замену, выбранную из аминокислотных замен F37V, Y37V, E44G, Q44G, R45L, H45L, G47W, F47W, L47W, T47W и S47W (все пронумерованы согласно нумерации Кэбота).
(27) Полипептид по одному из п.п. (19)-(23), в котором однодоменное антитело представляет собой VHH, где VHH имеет аминокислотные замены по меньшей мере в одном наборе положений, выбранном из положений 37/44, положений 37/45, положений 37/47, положений 44/45, положений 44/47, положений 45/47, положений 37/44/45, положений 37/44/47, положений 37/45/47, положений 44/45/47 и положений 37/44/45/47 (все пронумерованы согласно нумерации Кэбота).
(28) Полипептид по одному из п.п. (19)-(23), в котором однодоменное антитело представляет собой VHH, где VHH содержит по меньшей мере один набор аминокислот, выбранный из 37V/44G, 37V/45L, 37V/47W, 44G/45L, 44G/47W, 45L/47W, 37V/44G/45L, 37V/44G/47W, 37V/45L/47W, 44G/45L/47W и 37V/44G/45L/47W (все пронумерованы согласно нумерации Кэбота).
(29) Полипептид по одному из п.п. (19)-(23), в котором однодоменное антитело представляет собой VHH, где VHH содержит по меньшей мере один набор аминокислотных замен, выбранный из F37V/R45L, F37V/G47W, R45L/G47W и F37V/R45L/G47W (все пронумерованы согласно нумерации Кэбота).
(30) Полипептид по одному из п.п. (19)-(22), в котором однодоменное антитело представляет собой VL, который сам обладает антигенсвязывающей активностью, а ингибирующий домен транспортирующего фрагмента представляет собой VH антитела, в котором антигенсвязывающая активность VL, который сам обладает антигенсвязывающей активностью, ингибируется путем его ассоциации с VH антитела.
(31) Полипептид по одному из п.п. (1)-(30), в котором транспортирующий фрагмент имеет FcRn-связывающую область.
(32) Полипептид по одному из п.п. (1)-(31), в котором транспортирующий фрагмент содержит константную область антитела.
(33) Полипептид по п. (32), в котором константная область антитела транспортирующего фрагмента и антигенсвязывающий домен слиты через линкер или без линкера.
(34) Полипептид по п. (32), в котором транспортирующий фрагмент содержит константную область тяжелой цепи антитела, в котором константная область тяжелой цепи антитела и антигенсвязывающий домен слиты через линкер или без линкера.
(35) Полипептид по п. (32), в котором транспортирующий фрагмент содержит константную область легкой цепи антитела, в котором константная область легкой цепи антитела и антигенсвязывающий домен слиты через линкер или без линкера.
(36) Полипептид по п. (34), где в полипептиде N-конец константной области тяжелой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, и полипептид дополнительно имеет расщепляемую протеазой последовательность, в котором расщепляемая протеазой последовательность локализована в последовательности антигенсвязывающего домена или в константной области тяжелой цепи антитела на стороне, примыкающей к антигенсвязывающему домену за аминокислотой в положении 122 (EU-нумерация).
(37) Полипептид по п. (35), где в полипептиде N-конец константной области легкой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, и полипептид дополнительно имеет расщепляемую протеазой последовательность, в котором расщепляемая протеазой последовательность локализована в последовательности антигенсвязывающего домена или в константной области легкой цепи антитела на стороне, примыкающей к антигенсвязывающему домену за аминокислотой в положении 113 (EU-нумерация) (положение 113 согласно нумерации Кэбота).
(38) Полипептид по одному из п.п. (33)-(35), где в полипептиде N-конец константной области антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH или VHH, и полипептид дополнительно имеет расщепляемую протеазой последовательность, в котором расщепляемая протеазой последовательность локализована в последовательности константной области антитела или в однодоменном антителе антигенсвязывающего домена на стороне, примыкающей к константной области антитела за аминокислотным положением 109 (нумерация Кэбота).
(39) Полипептид по п. (33), где в полипептиде N-конец константной области антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, и полипептид дополнительно имеет расщепляемую протеазой последовательность, в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью антитела.
(40) Полипептид по (34), где в полипептиде N-конец константной области тяжелой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, и полипептид дополнительно имеет расщепляемую протеазой последовательность, в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью тяжелой цепи антитела.
(41) Полипептид по (35), где в полипептиде N-конец константной области легкой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, и полипептид дополнительно имеет расщепляемую протеазой последовательность, в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью легкой цепи антитела.
(42) Полипептид по п. (40), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH, или VHH, и расщепляемая протеазой последовательность локализована в любом положении между аминокислотным положением 109 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 122 (EU-нумерация) константной области тяжелой цепи антитела.
(43) Полипептид по п. (41), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH, или VHH, и расщепляемая протеазой последовательность локализована в любом положении между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 113 (EU-нумерация) (положение 113 согласно нумерации Кэбота) константной области легкой цепи антитела.
(44) Полипептид по п. (40), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VL, и расщепляемая протеазой последовательность локализована в любом положении между аминокислотой в положении 104 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 122 (EU-нумерация) константной области тяжелой цепи антитела.
(45) Полипептид по п. (41), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VL, и расщепляемая протеазой последовательность локализована в любом положении между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 113 (EU-нумерация) (положение 113 согласно нумерации Кэбота) константной области легкой цепи антитела.
(46) Полипептид по одному из п.п. (32)-(45), в котором константная область антитела полипептида представляет собой константную область антитела IgG-типа.
(47) Полипептид по одному из п.п. (1)-(46), где полипептид представляет собой молекулу, подобную антителу IgG-типа.
(48) Полипептид по одному из п.п. (1)-(47), в котором при анализе антигенсвязывающего домена в нерасщепленном состоянии с использованием BLI (интерферометрия биослоя) (система Octet) связывание антигенсвязывающего домена с антигеном не выявляется.
(49) Полипептид по одному из п.п. (1)-(48), в котором второй антигенсвязывающий домен дополнительно связан с антигенсвязывающим доменом.
(50) Полипептид по п. (49), в котором второй антигенсвязывающий домен имеет антигенсвязывающую специфичность, отличную от специфичности антигенсвязывающего домена.
(51) Полипептид по п. (49) или п. (50), в котором второй антигенсвязывающий домен содержит второе однодоменное антитело.
(52) Полипептид по п. (51), в котором антигенсвязывающий домен представляет собой однодоменное антитело, второй антигенсвязывающий домен представляет собой второе однодоменное антитело, и антигенсвязывающий домен и второй антигенсвязывающий домен обладают способностью высвобождаться из полипептида, в котором однодоменное антитело и второе однодоменное антитело образуют биспецифическую антигенсвязывающую молекулу, когда антигенсвязывающий домен и второй антигенсвязывающий домен находятся в высвободившихся состояниях.
(53) Полипептид по одному из п.п. (49)-(52), в котором второй антигенсвязывающий домен направлен против HER2 или GPC3 в качестве антигена-мишени.
(54) Полипептид по одному из п.п. (1)-(53), где полипептид имеет также дополнительный антигенсвязывающий домен, отличный от антигенсвязывающего домена, при этом антигенсвязывающая активность дополнительного антигенсвязывающего домена также ингибируется при его связывании с транспортирующим фрагментом полипептида.
(55) Полипептид по п. (54), в котором дополнительный антигенсвязывающий домен и антигенсвязывающий домен отличаются по антигенсвязывающей специфичности.
(56)Полипептид по одному из п.п. (1)-(55), в котором антигенсвязывающий домен представляет собой антигенсвязывающий домен, направленный против плексина A1, IL-6R или CD3 в качестве антигена-мишени.
(57) Фармацевтическая композиция, содержащая полипептид по одному из п.п. (1)-(56).
(58) Способ получения полипептида по одному из п.п. (1)-(56).
(59) Способ получения по п. (58), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом таким образом, что антигенсвязывающая активность однодоменного антитела ингибируется ингибирующим доменом транспортирующего фрагмента, с образованием полипептида-предшественника; и
(в) интродукция расщепляемой протеазой последовательности в полипептид-предшественник.
(60) Способ получения по п. (58), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом таким образом, что антигенсвязывающая активность однодоменного антитела ингибируется ингибирующим доменом транспортирующего фрагмента, с образованием полипептида-предшественника; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между однодоменным антителом и транспортирующим фрагментом.
(61) Способ получения по п. (58), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью; и
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом через расщепляемую протеазой последовательность таким образом, что антигенсвязывающая активность однодоменного антитела ингибируется ингибирующим доменом транспортирующего фрагмента, с образованием полипептида.
(62) Способ получения по одному из п.п. (59)-(61), дополнительно включающий следующую стадию:
(г) подтверждение того, что связывающая активность однодоменного антитела, включенного в полипептид или полипептид-предшественник, в отношении антигена-мишени ослабляется или утрачивается.
(63) Способ получение по одному из п.п. (59)-(62), дополнительно включающий следующую стадию:
(д) высвобождение однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившееся однодоменное антитело связывается с антигеном.
(64) Способ получения по п. (58), в котором полипептид представляет собой молекулу, подобную антителу IgG-типа.
(65) Способ получения по п. (64), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с VL в качестве замены VH антитела IgG-типа, или обеспечение осуществления ассоциации однодоменного антитела с VH в качестве замены VL антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело; и
(в) интродукция расщепляемой протеазой последовательности в подобную антителу IgG-типа молекулу-предшественника, которая несет однодоменное антитело.
(66) Способ получения по п. (64), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с VL в качестве замены VH антитела IgG-типа, или обеспечение осуществления ассоциации однодоменного антитела с VH в качестве замены VL антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между однодоменным антителом и константной областью антитела в подобную антителу IgG-типа молекулу-предшественника.
(67) Способ получения по п. (64), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), в качестве замены VH или VL антитела IgG-типа с константной областью тяжелой цепи или константной областью легкой цепи антитела IgG-типа через расщепляемую протеазой последовательность, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы, которая несет однодоменное антитело.
(68) Способ получения по одному из п.п. (65)-(67), дополнительно включающий следующую стадию:
(г) подтверждение того, что связывающая активность однодоменного антитела, находящегося в подобной антителу IgG-типа молекуле или подобной антителу IgG-типа молекуле-предшественнике, в отношении антигена-мишени ослабляется или утрачивается.
(69) Способ получения по одному из п.п. (65)-(68), дополнительно включающий следующую стадию:
(д) высвобождение однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившееся однодоменное антитело связывается с антигеном-мишенью.
(70) Способ получения по п. (64), включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации однодоменного антитела с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации однодоменного антитела с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH антитела или обеспечение осуществления ассоциации варианта однодоменного антитела с VL антитела, в результате чего антигенсвязывающая активность варианта однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела; и
(в) интродукция расщепляемой протеазой последовательности в подобную антителу IgG-типа молекулу-предшественника, которая несет вариант однодоменного антитела.
(71) Способ получения по п. (64), включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH антитела или обеспечение осуществления ассоциации варианта однодоменного антитела с VL антитела, в результате чего антигенсвязывающая активность варианта однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между вариантом однодоменного антитела и константной областью в подобную антителу IgG-типа молекулу-предшественника.
(72) Способ получения по п. (64), включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени; и
(б) сцепление варианта однодоменного антитела, полученного на стадии (а), с константной областью тяжелой цепи антитела IgG-типа через расщепляемую протеазой последовательность или сцепление варианта однодоменного антитела с константной областью легкой цепи антитела IgG-типа через расщепляемую протеазой последовательность, в результате чего антигенсвязывающая активность варианта однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела.
(73) Способ получения по одному из п.п. (70)-(72), дополнительно включающий следующую стадию:
(г) подтверждение того, что связывающая активность варианта однодоменного антитела, находящегося в подобной антителу IgG-типа молекуле, или связывающая активность варианта однодоменного антитела, находящегося в подобной антителу IgG-типа молекуле-предшественнике, в отношении антигена-мишени ослабляется или утрачивается.
(74) Способ получения по одному из п.п. (70)-(73), дополнительно включающий следующую стадию:
(д) высвобождение варианта однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившийся вариант однодоменного антитела связывается с антигеном-мишенью.
(75) Полинуклеотид, кодирующий полипептид по одному из п.п. (1)-(56).
(76) Вектор, содержащий полинуклеотид по п. (75).
(77) Клетка-хозяин, содержащая полинуклеотид по п. (75) или вектор по п. (76).
(78) Способ получения полипептида по одному из п.п. (1)-(56), включающий стадию культивирования клетки-хозяина по п. (77).
(79) Способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL, путем его ассоциации с конкретным VH или путем его ассоциации с конкретным VHH.
(80) Способ скрининга по п. (79), где способ представляет способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL.
(81) Способ скрининга по п. (80), включающий следующие стадии:
(а) получение однодоменного антитела, имеющего целевую антигенсвязывающую активность (активность в отношении антигена-мишени);
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с конкретным VL; и
(в) подтверждение того, что связывающая активность однодоменного антитела, ассоциированного с конкретным VL на стадии (б), в отношении антигена ослабляется по сравнению с активностью до ассоциации или утрачивается.
(82) Способ скрининга по п. (80), включающий следующие стадии:
(а) обеспечение осуществления ассоциации однодоменного антитела с конкретным VL;
(б) выбор продукта(ов) ассоциации, образованного(ых) VL и однодоменным антителом, на основе того, что однодоменное антитело, ассоциированное с конкретным VL на стадии (а), не обладает связывающей активностью в отношении антигена или его связывающая активность соответствует предварительно установленному уровню или ниже его; и
(в) подтверждение того, что однодоменное антитело в продукте(ах) ассоциации, выбранном(ых) на стадии (б), имеет более сильную связывающую активность в отношении антигена в неассоциированном с конкретным VL состоянии, чем в ассоциированном с ним состоянии.
(83) Способ скрининга по п. (79), где способ представляет собой способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VH.
(84) Способ скрининга по п. (83), включающий следующие стадии:
(а) получение однодоменного антитела, имеющего целевую антигенсвязывающую активность;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с конкретным VH; и
(в) подтверждение того, что связывающая активность однодоменного антитела, ассоциированного с конкретным VH на стадии (б), в отношении антигена ослабляется по сравнению с активностью до ассоциации или утрачивается.
(85) Способ скрининга по п. (83), включающий следующие стадии:
(а) обеспечение осуществления ассоциации однодоменного антитела с конкретным VH;
(б) выбор продукта(ов) ассоциации, образованного(ых) VH и однодоменным антителом, на основе того, что однодоменное антитело, ассоциированное с конкретным VH на стадии (а), не обладает связывающей активностью в отношении антигена или его связывающая активность соответствует предварительно установленному уровню или ниже его; и
(в) подтверждение того, что однодоменное антитело в продукте(ах) ассоциации, выбранном(ых) на стадии (б), имеет более сильную связывающую активность в отношении антигена в неассоциированном с конкретным VH состоянии, чем в ассоциированном с ним состоянии.
(86) Способ скрининга по п. (79), где способ представляет собой способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VHH.
(87) Способ скрининга по п. (86), включающий следующие стадии:
(а) получение однодоменного антитела, имеющего целевую антигенсвязывающую активность;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с конкретным VHH; и
(в) подтверждение того, что связывающая активность однодоменного антитела, ассоциированного с конкретным VHH на стадии (б), в отношении антигена ослабляется по сравнению с активностью до ассоциации или утрачивается.
(88) Способ скрининга по п. (86), включающий следующие стадии:
(а) обеспечение осуществления ассоциации однодоменного антитела с конкретным VHH;
(б) выбор продукта(ов) ассоциации, образованного(ых) VHH и однодоменным антителом, на основе того, что однодоменное антитело, ассоциированное с конкретным VHH на стадии (а), не обладает связывающей активностью в отношении антигена или его связывающая активность соответствует предварительно установленному уровню или ниже его; и
(в) подтверждение того, что однодоменное антитело в продукте(ах) ассоциации, выбранном(ых) на стадии (б), имеет более сильную связывающую активность в отношении антигена в неассоциированном с конкретным VHH состоянии, чем в ассоциированном с ним состоянии.
(89) Способ получения однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL, путем его ассоциации с конкретным VH или путем его ассоциации с конкретным VHH.
(90) Способ получения по п. (89), где способ представляет собой способ получения однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL.
(91) Способ получения по п. (90), включающий следующую стадию:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени.
(92) Способ получения по п. (91), дополнительно включающий следующие стадии:
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VL; и
(в) подтверждение того, что антигенсвязывающая активность варианта однодоменного антитела, ассоциированного с VL, ослабляется по сравнению с активностью до ассоциации или утрачивается.
(93) Способ получения по п. (89), где способ представляет собой способ получения однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VH.
(94) Способ получения по п. (93), включающий следующую стадию:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени.
(95) Способ получения по п. (94), дополнительно включающий следующие стадии:
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH; и
(в) подтверждение того, что антигенсвязывающая активность варианта однодоменного антитела, ассоциированного с VH, ослабляется по сравнению с активностью до ассоциации или утрачивается.
(96) Способ получения по п. (89), где способ представляет собой способ получения однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VHH.
(97) Способ получения по п. (96), включающий следующую стадию:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VHH, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени.
(98) Способ получения по п. (97), дополнительно включающий следующие стадии:
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VHH; и
(в) подтверждение того, что антигенсвязывающая активность варианта однодоменного антитела, ассоциированного с VHH, ослабляется по сравнению с активностью до ассоциации или утрачивается.
(99) Библиотека, содержащая множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом, где однодоменные антитела включают однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VL, однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VH, или однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VHH.
(100) Библиотека по п. (99), где фрагмент, представляющий собой однодоменное антитело, в каждом слитом полипептиде в библиотеке включает однодоменное антитело, полученное из животного семейства верблюдовых (Camelidae) или трансгенного животного, несущего ген, который может кодировать однодоменное антитело или гуманизированное антитело, однодоменное антитело, полученное иммунизацией животного семейства Camelidae или трансгенного животного, несущего ген, который может кодировать однодоменное антитело или гуманизированное антитело, или искусственно созданное однодоменное антитело, полученное из VH или VL человеческого антитела.
(101) Библиотека по п. (99) или п. (100), представляющая собой библиотеку, которая содержит множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом, где однодоменные антитела включают однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VL.
(102) Библиотека по п. (99) или п. (100), представляющая собой библиотеку, которая содержит множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом, где однодоменное антитело включает однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VH.
(103) Библиотека по п. (99) или п. (100), представляющая собой библиотеку, которая содержит множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом, где однодоменное антитело включает однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VHH.
(104) Способ скрининга библиотеки по п. (99) или п. (100) в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться при его ассоциации с конкретным VL, однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VH, или однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VHH.
(105) Способ скрининга библиотеки по п. (101) в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться при его ассоциации с конкретным VL.
(106) Способ скрининга по п. (105), включающий следующие стадии
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов библиотеки;
(б) получение партнера по ассоциации второго поддерживающего ассоциацию домена, слитого с конкретным VL;
(в) обеспечение осуществления ассоциации каждого из слитых полипептидов, экспонированных на стадии (а), с партнером по ассоциации, полученным на стадии (б), и отбор слитого(ых) полипептида(ов), который(ые) не связывается(ются) с антигеном или имеет(ют) антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в состоянии, в котором однодоменное антитело находится в ассоциации с VL; и
(г) выбор из слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, соответствующую предварительно установленному уровню или выше его в состоянии, в котором однодоменное антитело, входящее в него, не находится в ассоциации с VL.
(107) Способ скрининга по п. (106), в котором партнер по ассоциации, полученный на стадии (б), дополнительно содержит расщепляемую протеазой последовательность, и стадия (г) включает расщепление партнера по ассоциации обработкой протеазой таким образом, что ассоциация однодоменного антитела с VL аннулируется.
(108) Способ скрининга по п. (107), в котором расщепляемая протеазой последовательность партнера по ассоциации, полученного на стадии (б), локализована вблизи границы между конкретным VL и вторым поддерживающим ассоциацию доменом.
(109) Способ скрининга по п. (106), в котором слитые полипептиды библиотеки дополнительно содержат расщепляемую протеазой последовательность, и стадия (г) включает расщепление слитого(ых) полипептида(ов) обработкой протеазой таким образом, что ассоциация однодоменного антитела с VL аннулируется.
(110) Способ скрининга по п. (109), в котором расщепляемая протеазой последовательность, входящая в каждый слитый полипептид, локализована вблизи границы между однодоменным антителом и первым поддерживающим ассоциацию доменом.
(111) Способ скрининга по п. (106), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro полноразмерого(ых) слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), или их фрагментов, содержащих однодоменные антитела.
(112) Способ скрининга по п. (106), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro полноразмерого(ых) слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), и отбор слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его в состоянии ассоциации только со вторым поддерживающим ассоциацию доменом.
(113) Способ скрининга библиотеки по п. (102) в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться при его ассоциации с конкретным VH.
(114) Способ скрининга по п. (113), включающий следующие стадии
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов библиотеки;
(б) получение партнера по ассоциации второго поддерживающего ассоциацию домена, слитого с конкретным VH;
(в) обеспечение осуществления ассоциации каждого из слитых полипептидов, экспонированных на стадии (а), с партнером по ассоциации, полученным на стадии (б), и отбор слитого(ых) полипептида(ов), который(ые) не связывается(ются) с антигеном или имеет(ют) антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело находится в ассоциации с VH; и (г) выбор из слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, соответствующую предварительно установленному уровню или выше его в состоянии, в котором однодоменное антитело, входящее в него, не находится в ассоциации с VH.
(115) Способ скрининга по п. (114), в котором партнер по ассоциации, полученный на стадии (б), дополнительно содержит расщепляемую протеазой последовательность, и стадия (г) включает расщепление партнера по ассоциации обработкой протеазой таким образом, что ассоциация однодоменного антитела с VH аннулируется.
(116) Способ скрининга по п. (115), в котором расщепляемая протеазой последовательность партнера по ассоциации, полученного на стадии (б), локализована вблизи границы между конкретным VH и вторым поддерживающим ассоциацию доменом.
(117) Способ скрининга по п. (114), в котором слитые полипептиды библиотеки дополнительно содержат расщепляемую протеазой последовательность, и стадия (г) включает расщепление слитого(ых) полипептида(ов) обработкой протеазой таким образом, что ассоциация однодоменного антитела с VH аннулируется.
(118) Способ скрининга по п. (117), в котором расщепляемая протеазой последовательность, входящая в каждый слитый полипептид, локализована вблизи границы между однодоменным антителом и первым поддерживающим ассоциацию доменом.
(119) Способ скрининга по п. (114), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro полноразмерого(ых) слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), или их фрагментов, содержащих однодоменные антитела.
(120) Способ скрининга по п. (114), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro полноразмерого(ых) слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), и отбор слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его в состоянии ассоциации только со вторым поддерживающим ассоциацию доменом.
(121) Способ скрининга библиотеки по п. (103) в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться при его ассоциации с конкретным VHH.
(122) Способ скрининга по п. (121), включающий следующие стадии
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов библиотеки;
(б) получение партнера по ассоциации второго поддерживающего ассоциацию домена, слитого с конкретным VHH;
(в) обеспечение осуществления ассоциации слитых полипептидов, экспонированных на стадии (а), с партнером по ассоциации, полученным на стадии (б), и отбор слитого(ых) полипептида(ов), который(ые) не связывается(ются) с антигеном или имеет(ют) антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело находится в ассоциации с конкретным VHH; и
(г) выбор из слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, соответствующую предварительно установленному уровню или выше его в состоянии, в котором однодоменное антитело, входящее в него, не находится в ассоциации с VHH.
(123) Способ скрининга по п. (122), в котором партнер по ассоциации, полученный на стадии (б), дополнительно содержит расщепляемую протеазой последовательность, и стадия (г) включает расщепление партнера по ассоциации посредством обработки протеазой таким образом, что ассоциация однодоменного антитела с VHH аннулируется.
(124) Способ скрининга по п. (123), в котором расщепляемая протеазой последовательность партнера по ассоциации, полученного на стадии (б), локализована вблизи границы между конкретным VHH и вторым поддерживающим ассоциацию доменом.
(125) Способ скрининга по п. (122), в котором слитые полипептиды библиотеки дополнительно содержат расщепляемую протеазой последовательность, и стадия (г) включает расщепление слитого(ых) полипептида(ов) обработкой протеазой таким образом, что ассоциация однодоменного антитела с VHH аннулируется.
(126) Способ скрининга по п. (125), в котором расщепляемая протеазой последовательность, входящая в каждый слитый полипептид, локализована вблизи границы между однодоменным антителом и первым поддерживающим ассоциацию доменом.
(127) Способ скрининга по п. (122), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro полноразмерого(ых) слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), или их фрагментов, содержащих однодоменные антитела.
(128) Способ скрининга по п. (122), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro полноразмерого(ых) слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), и отбор слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его в состоянии ассоциации только со вторым поддерживающим ассоциацию доменом.
(129) Способ скрининга по одному из п.п. (106)-(112), (114)-(120) и (122)-(128), в котором стадия получения партнера по ассоциации, указанная на стадии (б), представляет собой стадию обеспечения осуществления совместного экспонирования партнера по ассоциации и слитых полипептидов.
(130) Библиотека по одному из п.п. (99)-(103), в которой первый поддерживающий ассоциацию домен содержит СН1-домен антитела IgG-типа или константную область легкой цепи антитела.
(131) Способ скрининга по одному из п.п. (106)-(112), (114)-(120) и (122)-(128), в котором первый поддерживающий ассоциацию домен содержит CHI-домен антитела IgG-типа, а второй поддерживающий ассоциацию домен содержит константную область легкой цепи антитела.
(132) Способ скрининга по одному из п.п. (106)-(112), (114)-(120) и (122)-(128), в котором первый поддерживающий ассоциацию домен содержит константную область легкой цепи антитела, а второй поддерживающий ассоциацию домен содержит CH1-домен антитела IgG-типа.
(133) Способ скрининга по п. (105), включающий следующие стадии:
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов библиотеки;
(б) получение партнера по ассоциации второго поддерживающего ассоциацию домена, слитого с конкретным VL;
(в) отбор слитого(ых) полипептида(ов), содержащего(их) однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его; и
(г) обеспечение осуществления ассоциации слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), с партнером по ассоциации, полученным на стадии (б), и отбор слитого полипептида, который не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело находится в ассоциации с VL.
(134) Способ скрининга по п. (129), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro слитых полипептидов, отобранных на стадии (в).
(135) Способ скрининга по п. (133), в котором стадия (в) включает обеспечение осуществления ассоциации слитого(ых) полипептида(ов) только со вторым поддерживающим ассоциацию доменом или подтверждение связывания антигена однодоменным антителом, входящим в слитый полипептид, которое находится в ассоциации только со вторым поддерживающим ассоциацию доменом.
(136) Способ скрининга по п. (113), включающий следующие стадии:
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов библиотеки;
(б) получение партнера по ассоциации второго поддерживающего ассоциацию домена, слитого с конкретным VH;
(в) отбор слитого(ых) полипептида(ов), содержащего(их) однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его; и
(г) обеспечение осуществления ассоциации слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), с партнером по ассоциации, полученным на стадии (б), и отбор слитого полипептида, который не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело находится в ассоциации с VH.
(137) Способ скрининга по п. (136), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro слитых полипептидов, отобранных на стадии (в).
(138) Способ скрининга по п. (136), в котором стадия (в) включает обеспечение осуществления ассоциации слитого полипептида только со вторым поддерживающим ассоциацию доменом или подтверждение связывания антигена однодоменным антителом, входящим в слитый полипептид, которое находится в ассоциации только со вторым поддерживающим ассоциацию доменом.
(139) Способ скрининга по п. (121), включающий следующие стадии:
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов библиотеки;
(б) получение партнера по ассоциации второго поддерживающего ассоциацию домена, слитого с конкретным VHH;
(в) отбор слитого(ых) полипептида(ов), содержащего(их) однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его; и
(г) обеспечение осуществления ассоциации слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), с партнером по ассоциации, полученным на стадии (б), и отбор слитого полипептида, который не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело находится в ассоциации с VHH.
(140) Способ скрининга по п. (139), в котором стадия (г) включает обеспечение осуществления повторного экспонирования in vitro слитых полипептидов, отобранных на стадии (в).
(141) Способ скрининга по п. (139), в котором стадия (в) включает обеспечение осуществления ассоциации слитого(ых) полипептида(ов) только со вторым поддерживающим ассоциацию доменом или подтверждение связывания антигена однодоменным антителом, входящим в слитый полипептид, которое находится в ассоциации только со вторым поддерживающим ассоциацию доменом.
(142) Способ скрининга по одному из п.п. (133)-(141), в котором стадия обеспечения осуществления ассоциации слитого(ых) полипептида(ов) с партнером по ассоциации, указанная на стадии (г), представляет собой стадию обеспечения осуществления совместного экспонирования партнера по ассоциации и слитых полипептидов.
(143) Способ скрининга по одному из п.п. (133)-(142), в котором первый поддерживающий ассоциацию домен содержит СН1-домен антитела IgG-типа, а второй поддерживающий ассоциацию домен содержит константную область легкой цепи антитела.
(144) Способ скрининга по одному из п.п. (133)-(142), в котором первый поддерживающий ассоциацию домен содержит константную область легкой цепи антитела, а второй поддерживающий ассоциацию домен содержит СН1-домен антитела IgG-типа.
В частности, настоящее изобретение может включать также конкретные варианты осуществления изобретения, примеры которых представлены ниже.
(Б1) Полипептид, содержащий антигенсвязывающий домен и транспортирующий фрагмент, где транспортирующий фрагмент имеет ингибирующий домен, который ингибирует антигенсвязывающую активность антигенсвязывающего домена, и где полипептид имеет расщепляемую протеазой последовательность, содержащую одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б2) Полипептид по п. (Б1), в котором ингибирование антигенсвязывающей активности антигенсвязывающего домена с помощью ингибирующего домена в состоянии, в котором расщепляемая протеазой последовательность является расщепленной протеазой, слабее, чем ингибирование антигенсвязывающей активности антигенсвязывающего домена с помощью ингибирующего домена в условиях, в которых расщепляемая протеазой последовательность является нерасщепленной.
(Б3) Полипептид по п. (Б1) или п. (Б2), в котором антигенсвязывающий домен имеет более короткое время полужизни в крови, чем транспортирующий фрагмент.
(Б4) Полипептид по одному из п.п. (Б1)-(Б3), в котором молекулярная масса антигенсвязывающего домена меньше, чем молекулярная масса транспортирующего фрагмента.
(Б5) Полипептид по одному из п.п. (Б1)-(Б4), в котором молекулярная масса антигенсвязывающего домена составляет 60 кДа или менее.
(Б6) Полипептид по одному из п.п. (Б1)-(Б5), в котором транспортирующий фрагмент обладает FcRn-связывающей активностью, а антигенсвязывающий домен не обладает FcRn-связывающей активностью или имеет более слабую FcRn-связывающую активность, чем транспортирующий фрагмент.
(Б7) Полипептид по одному из п.п. (Б1)-(Б6), в котором антигенсвязывающий домен обладает способностью высвобождаться из полипептида, и антигенсвязывающий домен имеет более высокую антигенсвязывающую активность в высвободившемся из полипептида состоянии, чем антигенсвязывающая активность в невысвободившемся из полипептида состоянии.
(Б8) Полипептид по одному из п.п. (Б1)-(Б7), в котором антигенсвязывающая активность антигенсвязывающего домена ингибируется в результате ассоциации ингибирующего домена транспортирующего фрагмента с антигенсвязывающим доменом.
(Б9) Полипептид по п. (Б7), в котором расщепляемая протеазой последовательность расщепляется протеазой таким образом, что антигенсвязывающий домен приобретает способность высвобождаться из полипептида.
(Б10) Полипептид по п. (Б8), в котором расщепляемая протеазой последовательность расщепляется протеазой таким образом, что ассоциация ингибирующего домена транспортирующего фрагмента с антигенсвязывающим доменом аннулируется.
(Б11) Полипептид по одному из п.п. (Б1)-(Б 10), где протеаза представляет собой протеазу, специфическую для ткани-мишени.
(Б12) Полипептид по п. (Б 11), где ткань-мишень представляет собой раковую ткань или воспалительную ткань, и протеаза представляет собой специфическую для раковой ткани протеазу или специфическую для воспалительной ткани протеазу.
(Б13) Полипептид по одному из п.п. (Б1)-(Б12), где протеаза представляет собой по меньшей мере одну протеазу, выбранную из матриптазы, урокиназы (иРА) и металлопротеиназы.
(Б14) Полипептид по одному из п.п. (Б1)-(Б12), где протеаза представляет собой по меньшей мере одну протеазу, выбранную из MT-SP1, uPA, ММР-2, ММР-9, ADAMTS5, ММР-7 и ММР-13.
(Б15) Полипептид по одному из п.п. (Б1)-(Б14), в котором первый гибкий линкер дополнительно присоединен к одному концу расщепляемой протеазой последовательности.
(Б16) Полипептид по п. (Б15), в котором первый гибкий линкер представляет собой гибкий линкер, состоящий из глицин-серинового полимера.
(Б17) Полипептид по п. (Б 15) или п. (Б16), в котором второй гибкий линкер дополнительно присоединен к другому концу расщепляемой протеазой последовательности.
(Б18) Полипептид по п. (Б17), в котором второй гибкий линкер представляет собой гибкий линкер, состоящий из глицин-серинового полимера.
(Б19) Полипептид по одному из п.п. (Б1)-(Б 18), в котором антигенсвязывающий домен содержит однодоменное антитело или представляет собой однодоменное антитело, в котором ингибирующий домен транспортирующего фрагмента ингибирует антигенсвязывающую активность однодоменного антитела.
(Б20) Полипептид по п. (Б 19), в котором однодоменное антитело представляет собой VHH, VH, который сам обладает антигенсвязывающей активностью, или VL, который сам обладает антигенсвязывающей активностью.
(Б21) Полипептид по одному из п.п. (Б1)-(Б20), в котором антигенсвязывающий домен содержит однодоменное антитело, в котором ингибирующий домен транспортирующего фрагмента представляет собой VHH, VH антитела или VL антитела и в котором антигенсвязывающая активность однодоменного антитела ингибируется VHH, VH антитела или VL антитела.
(Б22) Полипептид по одному из п.п. (Б1)-(Б21), в котором антигенсвязывающий домен содержит однодоменное антитело, в котором ингибирующий домен транспортирующего фрагмента представляет собой VHH, VH антитела или VL антитела и в котором антигенсвязывающая активность однодоменного антитела ингибируется путем его ассоциации с VHH, VH антитела или VL антитела.
(Б23) Полипептид по одному из п.п. (Б19)-(Б22), в котором однодоменное антитело представляет собой VHH или VH, который сам обладает антигенсвязывающей активностью, в котором ингибирующий домен транспортирующего фрагмента представляет собой VL антитела и в котором антигенсвязывающая активность VHH или VH, который сам обладает антигенсвязывающей активностью, ингибирует путем его ассоциации с VL антитела.
(Б24) Полипептид по одному из п.п. (Б19)-(Б23), в котором однодоменное антитело представляет собой VHH, где VHH имеет аминокислотную замену по меньшей мере в одном положении, выбранном из аминокислот в положениях 37, 44, 45 и 47 (все пронумерованы согласно нумерации Кэбота).
(Б25) Полипептид по одному из п.п. (Б19)-(Б23), в котором однодоменное антитело представляет собой VHH и в котором VHH содержит по меньшей мере одну аминокислоту, выбранную из аминокислот 37V, 44G, 45L и 47W (все пронумерованы согласно нумерации Кэбота).
(Б26) Полипептид по одному из п.п. (Б19)-(Б23), в котором однодоменное антитело представляет собой VHH и в котором VHH содержит по меньшей мере одну аминокислотную замену, выбранную из аминокислотных замен F37V, Y37V, E44G, Q44G, R45L, H45L, G47W, F47W, L47W, T47W и S47W (все пронумерованы согласно нумерации Кэбота).
(Б27) Полипептид по одному из п.п. (Б19)-(Б23), в котором однодоменное антитело представляет собой VHH и в котором VHH имеет аминокислотные замены по меньшей мере в одном наборе положений, выбранном из положений 37/44, положений 37/45, положений 37/47, положений 44/45, положений 44/47, положений 45/47, положений 37/44/45, положений 37/44/47, положений 37/45/47, положений 44/45/47 и положений 37/44/45/47 (все пронумерованы согласно нумерации Кэбота).
(Б28) Полипептид по одному из п.п. (Б19)-(Б23), в котором однодоменное антитело представляет собой VHH и в котором VHH содержит по меньшей мере один набор аминокислот, выбранный из 37V/44G, 37V/45L, 37V/47W, 44G/45L, 44G/47W, 45L/47W, 37V/44G/45L, 37V/44G/47W, 37V/45L/47W, 44G/45L/47W и 37V/44G/45L/47W (все пронумерованы согласно нумерации Кэбота).
(Б29) Полипептид по одному из п.п. (Б19)-(Б23), в котором однодоменное антитело представляет собой VHH и в котором VHH содержит по меньшей мере один набор аминокислотных замен, выбранный из F37V/R45L, F37V/G47W, R45L/G47W и F37V/R45L/G47W (все пронумерованы согласно нумерации Кэбота).
(Б30) Полипептид по одному из п.п. (Б19)-(Б22), в котором однодоменное антитело представляет собой VL, который сам обладает антигенсвязывающей активностью, в котором ингибирующий домен транспортирующего фрагмента представляет собой VH антитела и в котором антигенсвязывающая активность VL, который сам обладает антигенсвязывающей активностью, ингибируется путем его ассоциации с VH антитела.
(Б31) Полипептид по одному из п.п. (Б1)-(Б30), в котором транспортирующий фрагмент имеет FcRn-связывающую область.
(Б32) Полипептид по одному из п.п. (Б1)-(Б31), в котором транспортирующий фрагмент содержит константную область антитела.
(Б33) Полипептид по п. (Б32), в котором константная область антитела транспортирующего фрагмента и антигенсвязывающий домен слиты через линкер или без линкера.
(Б34) Полипептид по п. (Б32), в котором транспортирующий фрагмент содержит константную область тяжелой цепи антитела и в котором константная область тяжелой цепи антитела и антигенсвязывающий домен слиты через линкер или без линкера.
(Б35) Полипептид по п. (Б32), в котором транспортирующий фрагмент содержит константную область легкой цепи антитела и в котором константная область легкой цепи антитела и антигенсвязывающий домен слиты через линкер или без линкера.
(Б36) Полипептид по п. (Б34), в котором N-конец константной области тяжелой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера и в котором расщепляемая протеазой последовательность локализована в последовательности антигенсвязывающего домена или в константной области тяжелой цепи антитела на стороне, примыкающей к антигенсвязывающему домену за аминокислотой в положении 122 (EU-нумерация).
(Б37) Полипептид по п. (Б35), в котором N-конец константной области легкой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера и в котором расщепляемая протеазой последовательность локализована в последовательности антигенсвязывающего домена или в константной области легкой цепи антитела на стороне, примыкающей к антигенсвязывающему домену за аминокислотой в положении 113 (EU-нумерация) (положение 113 согласно нумерации Кэбота).
(Б38) Полипептид по одному из п.п. (Б33)-(Б36), в котором N-конец константной области антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера, в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH, или представляет собой VHH и в котором расщепляемая протеазой последовательность локализована в последовательности константной области антитела или в однодоменном антителе антигенсвязывающего домена на стороне, примыкающей к константной области антитела за аминокислотой в положении 109 (нумерация Кэбота).
(Б39) Полипептид по п. (БЗЗ), в котором N-конец константной области антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера и в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью антитела.
(Б40) Полипептид по п. (Б34), в котором N-конец константной области тяжелой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера и в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью тяжелой цепи антитела.
(Б41) Полипептид по п. (Б35), в котором N-конец константной области легкой цепи антитела транспортирующего фрагмента и С-конец антигенсвязывающего домена слиты через линкер или без линкера и в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью легкой цепи антитела.
(Б42) Полипептид по п. (Б40), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH, или представляет собой VHH и в котором расщепляемая протеазой последовательность локализована между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 122 (EU-нумерация) константной области тяжелой цепи антитела.
(Б43) Полипептид по п. (Б41), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH, или представляет собой VHH и в котором расщепляемая протеазой последовательность локализована между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 113 (EU-нумерация) (положение 113 согласно нумерации Кэбота) константной области легкой цепи антитела.
(Б44) Полипептид по п. (Б40), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VL, и в котором расщепляемая протеазой последовательность локализована между аминокислотой в положении 104 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 122 (EU-нумерация) константной области тяжелой цепи антитела.
(Б45) Полипептид по п. (Б41), в котором антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VL, и в котором расщепляемая протеазой последовательность локализована между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела антигенсвязывающего домена и аминокислотой в положении 113 (EU-нумерация) (положение 113 согласно нумерации Кэбота) константной области легкой цепи антитела.
(Б46) Полипептид по одному из п.п. (Б32)-(Б45), в котором константная область антитела полипептида представляет собой константную область антитела IgG-типа.
(Б47) Полипептид по одному из п.п. (Б1)-(Б46), где полипептид представляет собой молекулу, подобную антителу IgG-типа.
(Б48) Полипептид по одному из п.п. (Б1)-(Б47), в котором при анализе антигенсвязывающего домена в нерасщепленном состоянии с использованием интерферометрии биослоя (ВЫ) (система Octet) связывание антигенсвязывающего домена с антигеном не выявляется.
(Б49) Полипептид по одному из п.п. (Б1)-(4Б8), в котором второй антигенсвязывающий домен дополнительно сцеплен с антигенсвязывающим доменом.
(Б50) Полипептид по п. (Б49), в котором второй антигенсвязывающий домен имеет антигенсвязывающую специфичность, отличную от специфичности антигенсвязывающего домена.
(51) Полипептид по п. (49) или п. (50), в котором второй антигенсвязывающий домен содержит второе однодоменное антитело.
(Б52) Полипептид по п. (Б51), в котором антигенсвязывающий домен представляет собой однодоменное антитело, в котором второй антигенсвязывающий домен представляет собой второе однодоменное антитело, в котором антигенсвязывающий домен и второй антигенсвязывающий домен обладают способностью высвобождаться из полипептида и в котором однодоменное антитело и второе однодоменное антитело образуют биспецифическую антигенсвязывающую молекулу, когда антигенсвязывающий домен и второй антигенсвязывающий домен находятся в высвободившемся состоянии.
(Б53) Полипептид по одному из п.п. (Б49)-(Б52), в котором второй антигенсвязывающий домен направлен против HER2 или GPC3 в качестве антигена-мишени.
(Б54) Полипептид по одному из п.п. (Б1)-(Б53), где полипептид имеет также дополнительный антигенсвязывающий домен, отличный от антигенсвязывающего домена, и где антигенсвязывающая активность дополнительного антигенсвязывающего домена также ингибируется при его сцеплении с транспортирующим фрагментом полипептида.
(Б55) Полипептид по п. (Б54), в котором дополнительный антигенсвязывающий домен и антигенсвязывающий домен имеют различные антигенсвязывающие специфичности.
(Б56) Полипептид по одному из п.п. (Б1)-(Б55), в котором антигенсвязывающий домен представляет собой антигенсвязывающий домен, направленный против плексина A1, IL-6R или CD3 в качестве антигена-мишени.
(Б57) Фармацевтическая композиция, содержащая полипептид по одному из п.п. (Б1)-(Б56).
(Б58) Способ получения полипептида по одному из п.п. (Б1)-(Б56).
(Б59) Способ получения по п. (Б58), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом таким образом, что антигенсвязывающая активность однодоменного антитела ингибируется ингибирующим доменом транспортирующего фрагмента, с образованием полипептида-предшественника; и
(в) интродукция расщепляемой протеазой последовательности в полипептид-предшественник.
(Б60) Способ получения по п. (Б58), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом таким образом, что антигенсвязывающая активность однодоменного антитела ингибируется ингибирующим доменом транспортирующего фрагмента, с образованием полипептида-предшественника; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между однодоменным антителом и транспортирующим фрагментом,
где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б61) Способ получения по п. (Б58), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью; и
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом через расщепляемую протеазой последовательность таким образом, что антигенсвязывающая активность однодоменного антитела ингибируется ингибирующим доменом транспортирующего фрагмента, с образованием полипептида,
где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б62) Способ получения по одному из п.п. (Б59)-(Б61), дополнительно включающий следующую стадию:
(г) подтверждение того, что связывающая активность однодоменного антитела, включенного в полипептид или полипептид-предшественник, в отношении антигена-мишени ослабляется или утрачивается.
(Б63) Способ получение по одному из п.п. (Б59)-(Б62), дополнительно включающий следующую стадию:
(д) высвобождение однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившееся однодоменное антитело связывается с антигеном.
(Б64) Способ получения по п. (Б58), в котором полипептид представляет собой молекулу, подобную антителу IgG-типа.
(Б65) Способ получения по п. (Б64), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с VL в качестве замены VH антитела IgG-типа или обеспечение осуществления ассоциации однодоменного антитела с VH в качестве замены VL антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело;и
(в) интродукция расщепляемой протеазой последовательности в подобную антителу IgG-типа молекулу-предшественника, которая несет однодоменное антитело, где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б66) Способ получения по п. (Б64), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с VL в качестве замены VH антитела IgG-типа или обеспечение осуществления ассоциации однодоменного антитела с VH в качестве замены VL антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело;и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между однодоменным антителом и константной областью антитела в подобную антителу IgG-типа молекулу-предшественника, где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б67) Способ получения по п. (Б64), включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), в качестве замены VH или VL антитела IgG-типа с константной областью тяжелой цепи или константной областью легкой цепи антитела IgG-типа через расщепляемую протеазой последовательность, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело, где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б68) Способ получения по одному из п.п. (Б65)-(Б67), дополнительно включающий следующую стадию:
(г) подтверждение того, что связывающая активность однодоменного антитела, интродуцированного в подобную антителу IgG-типа молекулу или подобную антителу IgG-типа молекулу-предшественника, в отношении антигена-мишени ослабляется или утрачивается.
(Б69) Способ получения по одному из п.п. (Б65)-(Б68), дополнительно включающий следующую стадию:
(д) высвобождение однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившееся однодоменное антитело связывается с антигеном-мишенью.
(Б70) Способ получения по п. (Б64), включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH антитела или обеспечение осуществления ассоциации варианта однодоменного антитела с VL антитела, в результате чего антигенсвязывающая активность варианта однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела; и
(в) интродукция расщепляемой протеазой последовательности в подобную антителу IgG-типа молекулу-предшественника, которая несет вариант однодоменного антитела, где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б71) Способ получения по п. (Б64), включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации однодоменного антитела с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH антитела или обеспечение осуществления ассоциации варианта однодоменного антитела с VL антитела, в результате чего антигенсвязывающая активность варианта однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между вариантом однодоменного антитела и константной областью в подобной антителу IgG-типа молекуле-предшественнике, где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б72) Способ получения по п. (Б64), включающий следующие стадии: (а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени; и
(б) сцепление варианта однодоменного антитела, полученного на стадии (а), с константной областью тяжелой цепи антитела IgG-типа через расщепляемую протеазой последовательность или сцепление варианта однодоменного антитела с константной областью легкой цепи антитела IgG-типа через расщепляемую протеазой последовательность, в результате чего антигенсвязывающая активность варианта однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела, где расщепляемая протеазой последовательность содержит одну или несколько последовательностей, выбранных из последовательностей, представленных в SEQ ID NO: 833-852 и SEQ ID NO: 1062-1081, и последовательностей, представленных в таблице 1.
(Б73) Способ получения по одному из п.п. (Б70)-(Б72), дополнительно включающий следующую стадию:
(г) подтверждение того, что связывающая активность варианта однодоменного антитела, интродуцированного в подобную антителу IgG-типа молекулу или интродуцированного в подобную антителу IgG-типа молекулу-предшественника, в отношении антигена-мишени ослабляется или утрачивается.
(Б74) Способ получения по одному из п.п. (Б70)-(Б73), дополнительно включающий следующую стадию:
(д) высвобождение варианта однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившийся вариант однодоменного антитела связывается с антигеном-мишенью.
(Б75) Полинуклеотид, кодирующий полипептид по одному из п.п. (Б1)-(Б56).
(Б76) Вектор, содержащий полинуклеотид по п. (Б75).
(Б77) Клетка-хозяин, содержащая полинуклеотид по п. (Б75) или вектор по п. (Б76).
(Б78) Способ получения полипептида по одному из п.п. (Б1)-(Б56), включающий стадию культивирования клетки-хозяина по п. (Б77).
Краткое описание чертежей
На чертежах показано:
на фиг. 1 диаграмма, демонстрирующая концепцию технологии Probody™. Probody представляет собой молекулу антитела, антигенсвязывающая активность которого ингибируется путем сцепления антитела с пептидом, маскирующим антигенсвязывающий сайт антитела, через линкер, который расщепляется протеазой, экспрессируемой в области повреждения;
на фиг. 2 диаграмма, демонстрирующая причину побочных действий, которые могут обусловливаться Probody. Активированное Probody при накоплении в крови может вызывать побочные действия при связывании с антигеном, который экспрессируется в здоровой ткани;
на фиг. 3 - диаграмма, демонстрирующая причину побочных действий, которые могут обусловливаться Probody. Probody находится в равновесии между состоянием, в котором маскирующий пептид, сцепленный через линкер, связан с антигенсвязывающим сайтом, и состоянием, в котором маскирующий пептид диссоциирован. Молекула в диссоциированном состоянии может связываться с антигеном;
на фиг. 4 - диаграмма, демонстрирующая причину побочных действий, которые могут обусловливаться Probody. Антитело к лекарственному средству против маскирующего пептида (антитело к маскирующему пептиду) может связываться с маскирующим пептидом Probody до активации и тем самым активировать Probody без расщепления протеазой;
на фиг. 5 - диаграмма, демонстрирующая концепцию, касающуюся полипептида, который содержит антигенсвязывающий домен и транспортирующий фрагмент.(А) Полипептид с антигенсвязывающим доменом, сцепленным с транспортирующим фрагментом, имеет продолжительное время полужизни и не связывается с антигеном. (Б) Антигенсвязывающий домен высвобождает, например, в результате расщепления в сайте расщепления, для связывания с антигеном, и высвободившийся таким образом домен имеет короткое время полужизни;
на фиг. 6 - диаграмма, демонстрирующая один из вариантов способа получения полипептида, предлагаемого в настоящем изобретении. В настоящем варианте осуществления изобретения представляющий интерес полипептид представляет собой подобную антителу IgG-типа молекулу. (А) Получают однодоменное антитело, связывающееся с антигеном-мишенью. (Б) Давали осуществляться ассоциации однодоменного антитела с VL в качестве заместителя VH антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется. (В) Расщепляемую протеазой последовательность интродуцируют в подобную антителу IgG-типа молекулу-предшественника, которая несет однодоменное антитело;
на фиг. 7 - диаграмма, демонстрирующая один из вариантов полипептида, предлагаемого в настоящем изобретении. В настоящем варианте осуществления изобретения полипептид представляет собой подобную антителу IgG-типа молекулу, а антигенсвязывающие домены создают соответственно на фрагментах, которые соответствуют двум вариабельным областям антитела IgG-типа. Два антигенсвязывающих домена могут иметь одинаковую антигенсвязывающую специфичность или могут отличаться их антигенсвязывающей специфичностью;
на фиг. 8 - диаграмма, демонстрирующая вариант осуществления изобретения, в котором второй антигенсвязывающий домен дополнительно сцеплен с антигенсвязывающий доменом, предлагаемым в настоящем изобретении. В этом варианте осуществления изобретения антигенсвязывающий домен и второй антигенсвязывающий домен образуют после высвобождения биспецифическую антигенсвязывающую молекулу. На фиг. 8(A) показана диаграмма, демонстрирующая полипептид в невысвободившемся состоянии. Антигенсвязывающая активность антигенсвязывающего домена ингибируется. На фиг. 8(Б) показана диаграмма высвобождения биспецифической антигенсвязывающей молекулы, образованной антигенсвязывающий доменом и вторым антигенсвязывающий доменом. На фиг. 8(B) показана диаграмма, демонстрирующая биспецифическую антигенсвязывающую молекулу, направленную, например, против поверхностного антигена Т-клеток и поверхностного антигена раковых клеток, в качестве примера биспецифической антигенсвязывающей молекулы после высвобождения;
на фиг. 9А - диаграмма, демонстрирующая один пример способа скрининга слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться в результате его ассоциации с конкретным ингибирующим доменом, из библиотеки, которая содержит множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. На фиг. 9А(1) показана диаграмма, на которой продемонстрирована библиотека, содержащая множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. На фиг. 9А(2) показана диаграмма, демонстрирующая, что антигенсвязывающая активность каждого однодоменного антитела подтверждена в состоянии, в котором слитый полипептид находится в ассоциации с партером по ассоциации. Отобран слитый полипептид, содержащий однодоменное антитело, которое не связывается с антигеном-мишенью или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в указанном состоянии ассоциации. На фиг. 9А(3) показана диаграмма, демонстрирующая, что ассоциация однодоменного антитела в слитом полипептиде, отобранном на фиг. 9А(2), с ингибирующим доменом в партнере по ассоциации аннулирована, и антигенсвязывающая активность однодоменного антитела подтверждена. Отобран слитый полипептид, содержащий однодоменное антитело, которое связывается с антигеном-мишенью или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, в указанном состоянии отсутствия ассоциации. На фиг. 9А(2') показана диаграмма, на которой продемонстрировано подтверждение антигенсвязывающей активности однодоменного антитела в каждом слитом полипептиде. Отобран слитый полипептид, содержащий однодоменное антитело, которое связывается с антигеном-мишенью или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, в указанном состоянии его индивидуального существования. На фиг. 9А(3') показана диаграмма, демонстрирующая, что антигенсвязывающая активность однодоменного антитела подтверждена в состоянии, в котором слитый полипептид, отобранный на фиг. 9А(2'), находится в ассоциации с партнером по ассоциации. Отобран слитый полипептид, содержащий однодоменное антитело, которое не связывается с антигеном-мишенью или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в указанном состоянии ассоциации;
на фиг. 9Б - диаграмма, демонстрирующая один более конкретный пример способа скрининга слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться в результате его ассоциации с конкретным ингибирующим доменом, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. (1) Слитые полипептиды, каждый из которых содержит однодоменное антитело и первый поддерживающий ассоциацию домен, и партнера по ассоциации, который несет расщепляемую протеазой последовательность между ингибирующим доменом и вторым поддерживающим ассоциацию доменом, экспонировали совместно в форме Fab-подобной структуры; (2) отбирали из экспонированных таким образом Fab-подобных структур структуру, которая не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его; и (3) расщепляли партнера по ассоциации протеазой и отбирали фрагмент, содержащий однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его;
на фиг. 9В - диаграмма, демонстрирующая другой более конкретный пример способа скрининга слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться в результате его ассоциации с конкретным ингибирующим доменом, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. (1) Слитые полипептиды, каждый из которых содержит расщепляемую протеазой последовательность между однодоменным антителом и первым поддерживающим ассоциацию доменом, и партнера по ассоциации ингибирующего домена, сцепленного со вторым поддерживающим ассоциацию доменом, экспонировали совместно в форме Fab-подобной структуры; (2) отбирали из экспонированных таким образом Fab-подобных структур структуру, которая не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его; и (3) расщепляли слитый полипептид протеазой и отбирали фрагмент, содержащий однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его;
на фиг. 9Г - диаграмма, демонстрирующая альтернативный пример способа скрининга слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться в результате его ассоциации с конкретным ингибирующим доменом, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. (1) Слитые полипептиды, каждый из которых содержит однодоменное антитело и первый поддерживающий ассоциацию домен, и партнера по ассоциации ингибирующего домена, сцепленного со вторым поддерживающим ассоциацию доменом, экспонировали совместно в форме Fab-подобной структуры, и отбирали из экспонированных таким образом Fab-подобных структур структуру, которая не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его; и (2) отобранные таким образом, указанные на фиг. 9Г(1) фрагменты, содержащие однодоменные антитела в виде Fab-подобный структур, экспонировали вновь так, чтобы в это же время не происходила экспрессия ингибирующего домена, и отбирали фрагмент, который связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его. На каждой из фиг. 9Г(2') и 9Г(2") показана диаграмма, демонстрирующая альтернативный вариант, в котором фрагменты, содержащие однодоменные антитела, которые указаны на фиг. 9Г(2), экспонировали вновь так, чтобы вместе с ними не происходила экспрессия ингибирующего домена. Порядок осуществления (1) и (2), (2') или (2'') может представлять собой осуществление (2), (2') или (2'') до осуществления (1). В частности, фрагменты, содержащие однодоменные антитела, экспонируют таким образом, чтобы вместе с ними не происходила экспрессия ингибирующего домена, и отбирают фрагмент, имеющий антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его. Далее слитые полипептиды, каждый из которых содержит однодоменное антитело, содержащее фрагмент, имеющий связывающую активность, которая соответствует предварительно установленному уровню или выше его, и первый поддерживающий ассоциацию домен, и партнера по ассоциации ингибирующего домена, сцепленного со вторым поддерживающим ассоциацию доменом, экспонируют совместно с получением Fab-подобной структуры, и отбирают из экспонированных таким образом Fab-подобных структур структуру, которая не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его;
на фиг. 10 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим IL-6R подобных антителу молекул, которые получали, давая осуществляться ассоциации различных легких цепей с конструкцией IL6R90-G1m, содержащей VHH к человеческому IL-6R (IL6R90), слитый с константной областью человеческого IgG1 (СН1-шарнир-СН2-СН3). Время начала действия подобных антителу молекул на сенсоры с иммобилизованным антигеном обозначено как исходная точка на оси абсцисс;
на фиг. 11 (А) - диаграмма, на которой продемонстрирована модель подобной антителу молекулы, полученная путем встраивания расщепляемой протеазой последовательности вблизи границы между VHH и константной областью в IL6R90-G1m. На фиг. 11(Б) показана диаграмма, на которой представлены обозначения каждой полученной тяжелой цепи антитела, сайт инсерции аминокислотной последовательности и встроенная аминокислотная последовательность. Сайт инсерции обозначен как [вставка];
на фиг. 12-1 - диаграмма, на которой продемонстрированы результаты оценки степени расщепления, полученные с помощью ДСН-ПААГ в восстанавливающих условиях после обработки протеазой (MT-SP1) IL6R90-G1m или подобной антителу молекулы, полученной путем инсерции расщепляемой протеазой последовательности вблизи границы между VHH и константной областью в IL6R90-G1m. Из двух новых полос, образовавшихся в результате обработки протеазой, полоса, соответствующая 25 кДа или менее, представляет собой полосу, полученную из VHH, а полоса, соответствующая 25-50 кДа, представляет собой полосу, полученную из константной области;
на фиг. 12-2 - диаграмма, на которой представлено продолжение фиг. 12-1;
на фиг. 13 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим IL-6R конструкции IL-6R90-G1m или подобных антителу молекул, полученных путем инсерции расщепляемой протеазой последовательности вблизи границы между VHH и константной областью в IL-6R90-G1m, или результаты оценки этих образцов после обработки протеазой (MT-SP1). На сенсограммах, обозначенных как протеаза-, представлены результаты оценки связывания с антигеном не обработанных протеазой подобных антителу молекул, а на сенсограммах, обозначенных как протеаза+, представлены результаты оценки связывания с антигеном обработанных протеазой подобных антителу молекул. Момент времени, соответствующий 30 с до начала действия подобных антителу молекул на сенсоры с иммобилизованным антигеном, обозначен как исходная точка на оси абсцисс;
на фиг. 14 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим IL-6R подобных антителу молекул, полученных путем обеспечения осуществления ассоциации различных легких цепей с конструкцией 20A11-G1m, содержащей VHH к человеческому IL-6R (20А11), слитый с константной областью человеческого IgG1 (СН1-шарнир-СН2-СН3). Момент времени, соответствующий 30 с до начала действия подобных антителу молекул на сенсоры с иммобилизованным антигеном, обозначен как исходная точка на оси абсцисс;
на фиг. 15 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим IL-6R конструкции 20A11-G1m или подобных антителу молекул, полученных путем интродукции мутаций в аминокислоты, присутствующие на поверхности раздела между 20А11 и VL, и давая осуществляться ассоциации различных легких цепей с конструкцией 20Allhu-G1m, содержащей полученную таким образом конструкцию 20A11hu, слитую с константной областью человеческого IgG1 (СН1-шарнир-СН2-СН3). Момент времени, соответствующий 60 с до начала действия подобных антителу молекул на сенсоры с иммобилизованным антигеном, обозначен как исходная точка на оси абсцисс;
на фиг. 16 - диаграмма, на которой продемонстрированы результаты оценки степени расщепления, полученные с помощью ДСН-ПААГ в восстанавливающих условиях после обработки протеазой (MT-SP1) 20A11-G1m или 4 типов подобных антителу молекул, полученных путем инсерции расщепляемой протеазой последовательности вблизи границы между 20A11hu и константной областью в 20A11hu-G1m. Из двух новых полос, образовавшихся в результате обработки протеазой, полоса, соответствующая 25 кДа или менее, представляет собой полосу, полученную из VHH, а полоса, соответствующая 25-50 кДа, представляет собой полосу, полученную из константной области;
на фиг. 17 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим IL-6R конструкции 20A11-G1m или подобных антителу молекул, полученных путем инсерции расщепляемой протеазой последовательности вблизи границы между VHH и константной областью в 20A11hu-G1m, или результаты оценки этих образцов после обработки протеазой (MT-SP1). На сенсограммах, обозначенных как протеаза-, представлены результаты оценки связывания с антигеном не обработанных протеазой подобных антителу молекул, а на сенсограммах, обозначенных как протеаза+, представлены результаты оценки связывания с антигеном обработанных протеазой подобных антителу молекул. Момент времени, соответствующий 60 с до начала действия подобных антителу молекул на сенсоры с иммобилизованным антигеном, обозначен как исходная точка на оси абсцисс.Образец, обозначенный как «не изучен», соответствует образцу, который не анализировали;
на фиг. 18 - диаграмма, на которой продемонстрированы результаты оценки степени расщепления, полученные с помощью электрофореза ДСН-ПААГ в восстанавливающих условиях и детекции с помощью СВВ (кумасси бриллиантовый голубой), после обработки протеазой (MT-SP1) подобных антителу молекул, которые имели VHH к человеческому CD3 в вариабельных областях их тяжелых цепей и которые получали путем инсерции расщепляемой протеазой последовательности вблизи границы между VHH и константной областью тяжелой цепи. Из двух новых полос, образовавшихся в результате обработки протеазой, полоса, соответствующая 10-15 кДа, представляет собой полосу, полученную из VHH, а полоса, соответствующая примерно 37 кДа, представляет собой полосу, полученную из константной области тяжелой цепи;
на фиг. 19 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим CD3ed-Fc образцов после обработки протеазой (МТ-SP1) подобных антителу молекул, которые имели VHH к человеческому CD3 в вариабельных областях их тяжелых цепей и которые получали путем инсерции расщепляемой протеазой последовательности вблизи границы между VHH и константной областью тяжелой цепи. На сенсограммах, обозначенных как протеаза-, представлены результаты оценки связывания с антигеном не обработанных протеазой подобных антителу молекул, а на сенсограммах, обозначенных как протеаза+, представлены результаты оценки связывания с антигеном обработанных протеазой подобных антителу молекул. Момент времени, соответствующий 30 с до начала действия подобных антителу молекул на сенсоры с иммобилизованным антигеном, обозначен как исходная точка на оси абсцисс. При оценке связывания ответ до начала связывания антигена принимали за 0, а ответ перед началом действия антител принимали за 100. Время указано от момента, соответствующего 30 с до начала действия антител;
на фиг. 20 - диаграмма, на которой продемонстрированы результаты оценки степени расщепления, полученные с помощью электрофореза ДСН-ПААГ в восстанавливающих условиях и детекции с помощью СВВ после обработки протеазой (MT-SP1) молекулы, которая имеет IL-6R90-G1m в качестве тяжелой цепи и Vk1-39-k0MT в качестве легкой цепи, или подобных антителу молекул, полученных путем инсерции расщепляемой протеазой последовательности вблизи границы между вариабельной областью легкой цепи и константной областью легкой цепи молекулы, которая имеет IL-6R90-G1m в качестве тяжелой цепи и Vk1-39-k0MT в качестве легкой цепи. Две полосы, происходящие из легкой цепи, образовались в результате обработки протеазой, и легкая цепь расщеплялась протеазой;
на фиг. 21 - диаграмма, на которой продемонстрированы результаты оценки связывания с человеческим IL-6R образцов после обработки протеазой (MT-SP1) молекулы, имеющей IL-6R90-G1m в качестве тяжелой цепи и Vk1-39-k0MT в качестве легкой цепи, или подобных антителу молекул, полученных путем инсерции расщепляемой протеазой последовательности вблизи границы между вариабельной областью легкой цепи и константной областью легкой цепи молекулы, которая имеет IL-6R90-G1m в качестве тяжелой цепи и Vk1-39-k0MT в качестве легкой цепи. На сенсограммах, обозначенных как протеаза-, представлены результаты оценки связывания с антигеном не обработанных протеазой подобных антителу молекул, а на сенсограммах, обозначенных как протеаза+, представлены результаты оценки связывания с антигеном обработанных протеазой подобных антителу молекул. Антитело (MRA), для которого подтверждено связывание с IL-6R, применяли в качестве положительного контроля. Момент времени, соответствующий началу действия подобных антителу молекул на сенсоры с иммобилизованным антигеном, представляет собой исходную точку на оси абсцисс;
на фиг. 22 - диаграмма, на которой продемонстрированы полученные с помощью ДСН-ПААГ результаты оценки расщепления протеазой подобных антителу IgG-типа молекул с включенным VHH, который связывается с человеческим плексином А1. Дорожка, обозначенная как протеаза (+), соответствует обработанным протеазой образцам, а дорожка, обозначенная как протеаза (-), соответствует не обработанным протеазой образцам, которые используют в качестве отрицательного контроля;
на фиг. 23 - диаграмма, на которой продемонстрированы полученные с помощью системы Octet сенсограммы оценки связывания с человеческим плексином A1 VHH, который высвободился в результате расщепления протеазой подобных антителу IgG-типа молекул с включенным VHH, который связывается с человеческим плексином А1. Сенсограмма, обозначенная как протеаза (+), соответствует обработанным протеазой образцам, а сенсограмма, обозначенная как протеаза (-), соответствует не обработанным протеазой образцам. Концентрации, в которых применяли подобные антителу IgG-типа молекулы, указаны на левой стороне диаграммы;
на фиг. 24 - диаграмма, на которой продемонстрированы полученные с помощью ДСН-ПААГ результаты оценки расщепления протеазой полипептидов, содержащих биспецифические VHH-VHH;
на фиг. 25 - диаграмма, на которой продемонстрированы данные о люциферазной активности до и после расщепления протеазой. Пунктирной линией обозначены образцы, не обработанные протеазой, а сплошной линией обозначены образцы, обработанные протеазой;
на фиг. 26 диаграмма, на которой продемонстрированы данные о люциферазной активности до и после расщепления протеазой. Пунктирной линией обозначены образцы, не обработанные протеазой, а сплошной линией обозначены образцы, обработанные протеазой;
на фиг. 27 - диаграмма, на которой продемонстрированы полученные с помощью ДСН-ПААГ результаты оценки расщепления протеазой подобной антителу IgG-типа молекулы, содержащей VHH к человеческому IL-6R;
на фиг. 28 - диаграмма, на которой продемонстрированы результаты оценки расщепления протеазой подобных антителу IgG-типа молекул, несущих расщепляемую протеазой последовательность в их легких цепях;
на фиг. 29 - диаграмма, на которой продемонстрированы результаты оценки степени активации в зависимости от присутствия или отсутствия обработки протеазой подобных антителу IgG-типа молекул, несущих расщепляемую протеазой последовательность в их легких цепях;
на фиг. 30А - диаграмма, на которой продемонстрированы результаты оценки расщепления протеазой подобных антителу IgG-типа молекул, несущих расщепляемую протеазой последовательность в их тяжелых цепях;
на фиг. 30Б - диаграмма, на которой продемонстрированы результаты оценки расщепления протеазой подобных антителу IgG-типа молекул, несущих расщепляемую протеазой последовательность в их тяжелых цепях. Расщепление протеазой осуществляли, используя буфер для анализа (набор для анализа активности ММР (флуорометрический - зеленый) (ab112146), компонент С: буфер для анализа);
на фиг. 31 - диаграмма, на которой продемонстрированы результаты оценки эффективности расщепления in vivo, полученные при введении мышам молекулы антитела со встроенной расщепляемой протеазой последовательностью.
Описание вариантов осуществления изобретения
Согласно настоящему изобретению полипептид, как правило, представляет собой пептид, имеющий длину порядка 4 аминокислот или более, и белок. Кроме того, согласно настоящему изобретению полипептид, как правило, состоит из искусственно созданной последовательности (но он конкретно не ограничен только ею). Например, можно применять полученный из организма полипептид. Альтернативному этому, полипептид согласно настоящему изобретению может представлять собой любой встречающийся в естественных условиях полипептид, синтетический полипептид, рекомбинантный полипептид и т.п.Кроме того, согласно настоящему изобретению фрагменты указанных полипептидов также подпадают под понятие полипептид.
В настоящем описании каждую аминокислоту обозначают однобуквенным или трехбуквенным кодом или ими обоими, например, следующим образом: Ala/A, Leu/L, Arg/R, Lys/K, Asn/N, Met/M, Asp/D, Phe/F, Cys/C, Pro/P, Gln/Q, Ser/S, Glu/E, Thr/T, Gly/G, Trp/W, His/H, Tyr/Y, Ile/I или Val/V. Для обозначения аминокислоты, локализованной в конкретном положении, можно применять соответственно обозначение с использованием номера, соответствующего конкретному положению, в сочетании с однобуквенным кодом или трехбуквенным кодом аминокислоты. Например, аминокислота 37V, которая представляет собой аминокислоту, содержащуюся в однодоменном антителе, представляет собой Val, локализованный в положении 37 согласно нумерации Кэбота.
Для изменения аминокислоты в аминокислотной последовательности полипептида можно соответствующим образом адаптировать известный в данной области метод, такой как сайтнаправленный мутагенез (Kunkel и др., Proc. Natl. Acad. Sci. USA 82, 1985, cc. 488-492) или ПЦР с удлинением перекрытий. Множество методов, известных в данной области, можно также адаптировать в качестве альтернативных методов замены аминокислот на аминокислоты, отличные от встречающихся в естественных условиях аминокислот (Annu. Rev. Biophys. Biomol. Struct. 35, 2006, cc. 225-249; и Proc. Natl. Acad. Sci. USA 100(11), 2003, cc. 6353-6357). Можно применять, например, бесклеточную систему трансляции, содержащую тРНК, в которой не встречающаяся в естественных условиях аминокислота слита с супрессорной тРНК, подавляющей амбер-мутацию, комплементарной UAG-кодону (амбер-кодон), который представляет собой стоп-кодон (фирма Clover Direct (Protein Express)). В настоящем описании примеры изменения включают (но, не ограничиваясь только ими) замену.
В контексте настоящего описания подразумевается, что понятие «и/или», применяемое для обозначения сайтов аминокислотных изменений, включает каждую комбинацию, к которой применимо «и» и «или». В частности, например, фраза «аминокислоты в положениях 37, 45 и/или 47 заменены» включает следующие вариации аминокислотных изменений:
(а) положение 37, (б) положение 45, (в) положение 47, (г) положения 37 и 45, (д) положения 37 и 47, (е) положения 45 и 47 и (ж) положения 37, 45 и 47.
В настоящем описании выражение, в котором однобуквенные коды или трехбуквенные коды аминокислот до и после изменения указаны перед или после номера, обозначающего конкретное положение, можно применять соответственно для обозначения аминокислотного изменения. Например, изменение F37V или Phe37Val, применяемое для обозначения замены аминокислоты, содержащейся в вариабельной области антитела или однодоменном антителе, обозначает замену Phe в положении 37 согласно нумерации Кэбота на Val. В частности, номер обозначает аминокислотное положение согласно нумерации Кэбота; однобуквенный или трехбуквенный код аминокислоты, предшествующий номеру, означает аминокислоту до замены; а однобуквенный код или трехбуквенный код аминокислоты после номера означает аминокислоту после замены. Аналогично этому, изменение Р238А или Pro238Ala, применяемое для обозначения замены аминокислоты в Fc-области, содержащейся в константной области антитела, означает замену Pro в положении 238 согласно EU-нумерации на Ala. В частности, номер обозначает аминокислотное положение согласно EU-нумерации; однобуквенный или трехбуквенный код аминокислоты, предшествующий номеру, означает аминокислоту до замены; а однобуквенный код или трехбуквенный код аминокислоты после номера означает аминокислоту после замены.
В настоящем описании понятие «антитело» используется в его наиболее широком смысле и относится к различным структурам антител, включая (но, не ограничиваясь только ими) моноклональное антитело, поликлональное антитело, мультиспецифическое антитело (например, биспецифическое антитело), однодоменное антитело и фрагменты антител, при условии, что они обладают требуемой антигенсвязывающей активностью.
Понятие «фрагмент антитела» относится к молекуле, отличной от интактного антитела, включающей часть интактного антитела, связывающуюся с антигеном, с которым связывается интактное антитело. Примеры фрагментов антитела включают (но, не ограничиваясь только ими) Fv, Fab, Fab', Fab'-SH, F(ab')2, димерные антитела (диабоди), линейные антитела, молекулы одноцепочечных антител (например, scFv) и мультиспецифические антитела, образованные из фрагментов антител.
Понятия «полноразмерное антитело», «интактное антитело» и «цельное антитело» в контексте настоящего описания используют взаимозаменяемо для обозначения антитела, имеющего структуру, практически сходную со структурой нативного антитела, или имеющего тяжелые цепи, которые содержат представленную в настоящем описании Fc-область.
Понятие «вариабельная область» или «вариабельный домен» относится к домену тяжелой или легкой цепи антитела, который участвует в связывании антитела с его антигеном. Вариабельные домены тяжелой цепи и легкой цепи (VH и VL соответственно) нативного антитела, как правило, имеют сходные структуры, при этом каждый домен содержит четыре консервативных каркасных участка (FR) и три гипервариабельных участка (CDR) (см., например, Kindt и др., Kuby Immunology, 6-ое изд., изд-во W.H. Freeman and Co., 2007, с. 91). Одного VH- или VL-домена может быть достаточно для обеспечения специфичности связывания антигена.
Понятие «гипервариабельный участок» или «CDR» в контексте настоящего описания относится к участку, последовательность которого является гипервариабельной и/или формирует петли определенной структуры («гипервариабельные петли»), и/или относится к контактирующим с антигеном остаткам («контакты с антигеном») или к каждому из участков вариабельного домена антитела. Как правило, антитела содержат шесть CDR: три в VH (H1, Н2, Н3) и три в VL (L1, L2, L3). Согласно настоящему описанию примеры CDR включают:
(а) гипервариабельные петли, включающие аминокислотные остатки 26-32 (L1), 50-52 (L2), 91-96 (L3), 26-32 (H1), 53-55 (Н2) и 96-101 (Н3) (Chothia и Lesk, J. Mol. Biol. 196, 1987, cc. 901-917);
(б) CDR, включающие аминокислотные остатки 24-34 (L1), 50-56 (L2), 89-97 (L3), 31-35b (H1), 50-65 (Н2) и 95-102 (Н3) (Kabat и др., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991, публикация NIH 91-3242);
(в) области контактов с антигеном, включающие аминокислотные остатки 27 с-36 (L1), 46-55 (L2), 89-96 (L3), 30-35b (H1), 47-58 (Н2) и 93-101 (Н3) (MacCallum и др., J. Mol. Biol. 262, 1996, сс. 732-745); и
(г) комбинации остатков, указанных в подпунктах (а), (б) и/или (в), включающие аминокислотные остатки HVR 46-56 (L2), 47-56 (L2), 48-56 (L2), 49-56 (L2), 26-35 (H1), 26-35b (H1), 49-65 (Н2), 93-102 (Н3) и 94-102 (Н3).
Как правило, однодоменное антитело содержит три CDR: CDR1, CDR2 и CDR3. Когда однодоменное антитело представляет собой однодоменное антитело, полученное из VHH или VH антитела, то приведенные в качестве примеров CDR однодоменного антитела включают:
(а) гипервариабельные петли, включающие аминокислотные остатки 26-32 (CDR1), 53-55 (CDR2) и 96-101 (CDR3) (Chothia и Lesk, J. Mol. Biol. 196, 1987, сс. 901-917);
(б) CDR, включающие аминокислотные остатки 31-35b (CDR1), 50-65 (CDR2) и 95-102 (CDR3) (Kabat и др., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991);
(в) области контактов с антигеном, включающие аминокислотные остатки 30-35b (CDR1), 47-58 (CDR2) и 93-101 (CDR3) (MacCallum и др., J. Mol. Biol. 262, 1996, сс. 732-745); и
(г) комбинации остатков, указанных в подпунктах (а), (б) и/или (в), включающие аминокислотные остатки CDR 26-35 (CDR1), 26-35b (CDR1), 49-65 (CDR2), 93-102 (CDR3) или 94-102 (CDR3).
Когда однодоменное антитело представляет собой однодоменное антитело, полученное из VL антитела, то приведенные в качестве примеров CDR однодоменного антитела включают:
(а) гипервариабельные петли, включающие аминокислотные остатки 26-32 (CDR1), 50-52 (CDR2) и 91-96 (CDR3) (Chothia и Lesk, J. Mol. Biol. 196, 1987, сс. 901-917);
(б) CDR, включающие аминокислотные остатки 24-34 (CDR1), 50-56 (CDR2) и 89-97 (CDR3) (Kabat и др., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991);
(в) области контактов с антигеном, включающие аминокислотные остатки 27 с-36 (CDR1), 46-55 (CDR2) и 89-96 (CDR3) (MacCallum и др., J. Mol. Biol. 262, 1996, сс.732-745); и
(г) комбинации остатков, указанных в подпунктах (а), (б) и/или (в), включающие аминокислотные остатки CDR 46-56 (CDR2), 47-56 (CDR2), 48-56 (CDR2) или 49-56 (CDR2).
Если специально не указано иное, то в настоящем описании остатки в CDR и другие остатки в вариабельном домене (например, остатки в FR) нумеруют согласно Kabat и др., выше.
Понятие «каркасный участок» или «FR» относится к остаткам вариабельного домена, отличным от остатков гипервариабельного участка (CDR). FR вариабельного домена, как правило, состоит из четырех FR-доменов: FR1, FR2, FR3 и FR4. Таким образом, последовательности CDR и FR, как правило, имеют следующее расположение в VH (или VL): FR1-H1(L1)-FR2-H2(L2)-FR3-H3(L3)-FR4. В однодоменном антителе последовательности CDR и FR, как правило, расположены в следующем порядке: FR1-CDR1-FR2-CDR2-FR3-CDR3-FR4.
Как правило, однодоменное антитело, предлагаемое в настоящем изобретении, может представлять собой полипептид, который содержит:
а) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 11 согласно нумерации Кэбота выбирают из группы, состоящей из L, М, S, V и W, и предпочтительно он представляет собой L); и/или
б) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 37 согласно нумерации Кэбота выбирают из группы, состоящей из F, Y, Н, I, L и V, и предпочтительно он представляет собой F или Y); и/или в) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 44 согласно нумерации Кэбота выбирают из группы, состоящей из G, Е, A, D, Q, R, S и L, предпочтительно он представляет собой G, Е или Q и более предпочтительно G или Е); и/или
г) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 45 согласно нумерации Кэбота выбирают из группы, состоящей из L, R, С, I, L, Р, Q и V, и предпочтительно он представляет собой L или R); и/или
д) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 47 согласно нумерации Кэбота выбирают из группы, состоящей из W, L, F, A, G, I, М, R, S, V и Y, и предпочтительно он представляет собой W, L, F или R); и/или
е) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 83 согласно нумерации Кэбота выбирают из группы, состоящей R, K, N, Е, G, I, М, Q и Т, предпочтительно он представляет собой K или R и более предпочтительно K); и/или
ж) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 84 согласно нумерации Кэбота выбирают из группы, состоящей из Р, A, L, R, S, Т, D и V, и предпочтительно он представляет собой Р); и/или
з) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 103 согласно нумерации Кэбота выбирают из группы, состоящей из W, Р, R и S, и предпочтительно он представляет собой W); и/или
и) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 104 согласно нумерации Кэбота представляет собой G или D, и предпочтительно он представляет собой G); и/или
к) аминокислотную последовательность, состоящую из четырех каркасных участков/последовательностей, которая имеет три гипервариабельных участка/последовательности, встроенные между ними (аминокислотный остаток в положении 108 согласно нумерации Кэбота выбирают из группы, состоящей из Q, L и R, и предпочтительно он представляет собой Q или L).
Более конкретно (но, не ограничиваясь только указанным) однодоменное антитело, предлагаемое в настоящем изобретении, может представлять собой полипептид, содержащий одну из аминокислотных последовательностей, состоящих из четырех каркасных участков/последовательностей, которые имеют три гипервариабельных участка/последовательности, встроенные между ними:
л) аминокислотная последовательность, в которой аминокислотные остатки в положениях 43-46 согласно нумерации Кэбота представляют собой KERE или KQRE;
м) аминокислотная последовательность, в которой аминокислотные остатки в положениях 44-47 согласно нумерации Кэбота представляют собой GLEW; и
н) аминокислотная последовательность, в которой аминокислотные остатки в положениях 83-84 согласно нумерации Кэбота представляют собой KP или ЕР.
В настоящем описании понятие «константная область» или «константный домен» относится к области или домену, отличному от вариабельных областей антитела. Например, антитело IgG-типа представляет собой гетеротетрамерный гликопротеин с молекулярной массой примерно 150000 Да, состоящий из двух идентичных легких цепей и двух идентичных тяжелых цепей, связанных дисульфидными мостиками. В направлении от N-конца к С-концу, каждая тяжелая цепь имеет вариабельную область (VH), которую называют также вариабельным тяжелым доменом или вариабельным доменом тяжелой цепи, за которой следует три константных домена (CH1, СН2 и СН3). Аналогично этому, в направлении от N-конца к С-концу, каждая легкая цепь имеет вариабельную область (VL), которую называют также вариабельным легким доменом или вариабельным доменом легкой цепи, за которой следует константный домен легкой цепи (CL). Легкие цепи нативных антител в зависимости от аминокислотной последовательности их константных доменов могут принадлежать к одному из двух типов, которые обозначают каппа (κ) и лямбда (λ).
В настоящем описании понятие «Fc-область» применяют для обозначения С-концевой области тяжелой цепи иммуноглобулина, которая содержит по меньшей мере часть константной области. Это понятие включает Fc-области, имеющие нативные последовательности, и вариант Fc-областей. В одном из объектов изобретения Fc-область человеческого IgG простирается от Cys226 или от Pro230 до карбоксильного конца тяжелой цепи. Однако С-концевой лизин (Lys447) или глицин-лизин (Gly446-Lys447) Fc-области может присутствовать или может отсутствовать. Если специально не указано иное, то в настоящем описании нумерация аминокислотных остатков в Fc-области или константной области соответствует системе нумерации EU, которую обозначают также как EU-индекс, описанной у Kabat и др., ., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991.
Понятие «класс» антитела относится к типу константного домена или константной области, который/которая входит в его тяжелую цепь. Известно пять основных классов антител: IgA, IgD, IgE, IgG и IgM, и некоторые из них можно подразделять дополнительно на подклассы (изотипы), например, IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2. Константные домены тяжелой цепи, которые соответствуют различным классам иммуноглобулинов, обозначают как α, δ, ε, y и μ соответственно.
В настоящем описании «антигенсвязывающий домен» ограничен только способностью связываться с представляющим интерес антигеном. Антигенсвязывающий домен может представлять собой домен, имеющий любую структуру, если применяемый домен связывается с представляющим интерес антигеном. Примеры указанного домена включают (но, не ограничиваясь только ими), вариабельную область тяжелой цепи антитела (VH), вариабельную область легкой цепи антитела (VL), однодоменное антитело (scIAt), модуль, состоящий примерно из 35 аминокислот, обозначенный как А-домен, который входит в авимер (Avimer), белок клеточной мембраны, присутствующий in vivo (WO 2004/044011 и WO 2005/040229), аднектин (Adnectin), содержащий 10Fn3-домен, служащий в качестве связывающего белок домена, полученного из гликопротеина фибронектина, экспрессируемого на клеточных мембранах (WO 2002/032925), аффибоди (Affibody), имеющий каркас IgG-связывающего домена, состоящий из трехспирального пучка, включающего 58 аминокислот белка А (WO 1995/001937), DARPin (сконструированные белки с анкириновыми повторами), которые представляют собой области анкириновых повторов (AR), экспонируемые на молекулярной поверхности, каждый из которых имеет структуру, в которой повторно уложена субъединица с изгибом, содержащая 33 аминокислотных остатка, две антипараллельные спирали и петлю (WO 2002/020565), антикалин (Anticalin), который имеет состоящие из четырех петель области, поддерживающие одну сторону центрально закрученной бочкообразной структуры, состоящей из восьми антипараллельных цепей, которые являются высоко консервативными среди молекул липокаинов, таких как липокалин, ассоциированный с желатиназой нейтрофилов (NGAL) (WO 2003/029462), и вогнутую область, образованную структурой в виде параллельных тяжей внутри подковообразной структуры, образованной уложенными повторами, которые состоят из богатых лейцином повторяющихся (LRR) модулей вариабельного рецептора лимфоцитов (VLR), который не имеет иммуноглобулиновой структуры и применяется в системе приобретенной устойчивости у бесчелюстных позвоночных, таких как миноги или миксины (WO 2008/016854).
Предпочтительные примеры антигенсвязывающего домена, предлагаемого в настоящем изобретении, включают антигенсвязывающий домен, который может проявлять антигенсвязывающую функцию посредством молекулы, состоящей только из антигенсвязывающего домена, и антигенсвязывающий домен, который сам может проявлять антигенсвязывающую функцию после освобождения от дополнительного пептида, сцепленного с ним. Примеры такого антигенсвязывающего домена включают (но, не ограничиваясь только ими) однодоменные антитела, scFv, Fv, Fab, Fab' и F(ab')2.
Одним из предпочтительных примеров антигенсвязывающего домена, предлагаемого в настоящем изобретении, является антигенсвязывающий домен с молекулярной массой 60 кДа или менее. Примеры такого антигенсвязывающего домена включают (но, не ограничиваясь только ими) однодоменные антитела, scFv, Fab и Fab'. По-видимому, антигенсвязывающий домен с молекулярной массой 60 кДа или менее, как правило, должен подвергаться почечному клиренсу, когда присутствует в виде мономера в крови (см. J Biol Chem. 263 (29), 15 октября 1988 г., сс.15064-15070).
С другой точки зрения, один из предпочтительных примеров антигенсвязывающего домена, предлагаемого в настоящем изобретении, включает антигенсвязывающий домен, который имеет время полужизни в крови 12 ч или менее. Примеры такого антигенсвязывающего домена включают (но, не ограничиваясь только ими) однодоменные антитела, scFv, Fab и Fab'.
Один из предпочтительных примеров антигенсвязывающего домена, предлагаемого в настоящем изобретении, включает однодоменное антитело (sdAт).
В настоящем описании понятие «однодоменное антитело» не ограничено его структурой, если домен может сам обладать антигенсвязывающей активностью. Известно, что обычное (каноническое) антитело, например, антитело IgG-типа, обладает антигенсвязывающей активностью в состоянии, в котором вариабельная область образуется путем спаривания VH и VL, в то время как собственная структура домена однодоменного антитела сама может обладать антигенсвязывающей активностью без спаривания с другим доменом. Как правило, однодоменное антитело имеет относительно низкую молекулярную массу и присутствует в форме мономера.
Примеры однодоменного антитела включают (но, не ограничиваясь только ими) антигенсвязывающие молекулы, в которых исходно отсутствует легкая цепь, такие как VHH животного семейства Camelidae и акульи VNAR, и фрагменты антител, которые содержат полный VH-домен антитела или его часть или полный VL-домен антитела или его часть. Примеры однодоменного антитела, которое представляет собой фрагмент антитела, содержащий полный VH-домен или VL-домен антитела или его часть, включают (но, не ограничиваясь только ими) искусственно созданные однодоменные антитела, происходящие из VH человеческого антитела или VL человеческого антитела, которые описании в US №6248516 В1 и др. В некоторых вариантах осуществления настоящего изобретения одно однодоменное антитело имеет три CDR (CDR1, CDR2 и CDR3).
Однодоменное антитело можно получать из животного, в организме которого может продуцироваться однодоменное антитело, или путем иммунизации животного, в организме которого может продуцироваться однодоменное антитело. Примеры животного, в организме которого может продуцироваться однодоменное антитело, включают (но, не ограничиваясь только ими) животных семейства Camelidae и трансгенных животных, несущих ген, который может кодировать однодоменное антитело. Животные семейства Camelidae включают верблюдов, лам, альпак, одногорбых верблюдов и гуанако и др. Примеры трансгенных животных, несущих ген, который может кодировать однодоменное антитело, включают (но, не ограничиваясь только ими) трансгенных животных, которые описаны в WO 2015/143414 и публикации патента США US2011/0123527 А1. Каркасные последовательности однодоменного антитела, полученного из животного, можно превращать в последовательности человеческих зародышевых линий или в сходные с ними последовательности с получением гуманизированного однодоменного антитела. Гуманизированное однодоменное антитело (например, гуманизированный VHH) также является одним из вариантов однодоменного антитела, предлагаемого в настоящем изобретении. Понятие «гуманизированное однодоменное антитело» относится к химерному однодоменному антителу, которое содержит аминокислотные остатки из нечеловеческих CDR и аминокислотные остатки из человеческих FR. В некоторых вариантах осуществления изобретения в гуманизированном однодоменном антителе все или практически все CDR соответствуют участкам нечеловеческого антитела, а все или практически все FR соответствуют участкам человеческого антитела. В гуманизированном антителе, даже, если часть остатков в FR не соответствует остаткам человеческого антитела, практически все FR рассматриваются в качестве примера, соответствующего остаткам человеческого антитела. Например, при гуманизации VHH, который является вариантом однодоменного антитела, необходимо, чтобы часть остатков FR представляла собой остатки, которые не соответствуют остаткам человеческого антитела (С.Vincke и др., The Journal of Biological Chemistry 284, cc. 3273-3284).
Альтернативно этому, однодоменное антитело можно получать с помощью ELISA, пэннинга или т.п. из полипептидной библиотеки, содержащей однодоменные антитела. Примеры полипептидных библиотек, содержащих однодоменные антитела, включают (но, не ограничиваясь только ими) библиотеки наивных антител, полученных из различных животных или людей (см., например, Methods in Molecular Biology 911, 2012, cc. 65-78; и Biochimica et Biophysica Acta - Proteins and Proteomics 1764: 8, 2006, 1307-1319), библиотеки антител, полученных путем иммунизации различных животных (см., например, Journal of Applied Microbiology 117: 2, 2014, cc. 528-536), и библиотеки синтетических антител, полученных из генов антител различных животных или людей (см., например, Journal of Biomolecular Screening 21: 1, 2016, cc. 35-43; Journal of Biological Chemistry 291:24, 2016, cc. 12641-12657; и AIDS 30: 11, 2016, cc. 1691-1701).
В настоящем описании «антиген» ограничен только наличием эпитопа, с которым связывается антигенсвязывающий домен. Предпочтительные примеры антигена включают (но, не ограничиваясь только ими) пептиды, полипептиды и белки животного или человеческого происхождения. Предпочтительные примеры антигена, предназначенного для применения для лечения заболевания, вызываемого тканью-мишенью, включают (но, не ограничиваясь только ими) молекулы, экспрессируемые на поверхности клеток-мишеней (например, раковых клеток и воспалительных клеток), молекулы, экспрессируемые на поверхности других клеток в тканях, содержащих клетки-мишени, молекулы, экспрессируемые на поверхности клеток, которые имеют иммунологическую роль в отношении клеток-мишеней и тканей, содержащих клетки-мишени, и макромолекулы, присутствующие в строме тканей, содержащих клетки-мишени.
Примеры антигена могут включать следующие молекулы: 17-IA, 4-1ВВ, 4Dc, 6-кето-PGF1a, 8-изо-PGF2a, 8-оксо-dG, А1 аденозиновый рецептор, А33, АСЕ, АСЕ-2, активин, активин А, активин АВ, активин В, активин С, активин RIA, активин RIA ALK-2, активин RIB ALK-4, активин RIIA, активин RIIB, ADAM, ADAM 10, ADAM12, ADAM15, ADAM17/TACE, ADAM8, ADAM9, ADAMTS, ADAMTS4, ADAMTS5, адрессин, aFGF, ALCAM, ALK, ALK-1, ALK-7, альфа-1-антитрипсин, антагонист альфа-V/бета-1, ANG, Ang, APAF-1, APE, APJ, APP, APRIL, AR, ARC, ART, артемин, анти-Id, ASPARTIC, предсердный натрийуретический фактор, интегрин av/b3, Axl, b2M, В7-1, В7-2, В7-Н, стимулятор В-лимфоцитов (BlyS), ВАСЕ, ВАСЕ-1, Bad, BAFF, BAFF-R, Bag-1, BAK, Вах, ВСА-1, ВСАМ, Bel, ВСМА, BDNF, b-ECGF, bFGF, BID, Bik, BIM, BLC, BL-CAM, BLK, BMP, BMP-2 BMP-2a, BMP-3 остеогенин, BMP-4 BMP-2b, BMP-5, BMP-6 Vgr-1, BMP-7 (OP-1), BMP-8 (BMP-8a, OP-2), BMPR, BMPR-IA (ALK-3), BMPR-IB (ALK-6), BRK-2, RPK-1, BMPR-II (BRK-3), BMP, b-NGF, BOK, бомбезин, нейтротрофический фактор костного мозга, BPDE, BPDE-DNA, ВТС, фактор системы комплемента 3 (С3), С3а, С4, С5, С5а, С10, СА125, CAD-8, кальцитонин, цАМФ, карциноэмбриональный антиген (СЕА), ассоциированные с раком антигены, катепсин А, катепсин В, катепсин C/DPPI, катепсин D, катепсин Е, катепсин Н, катепсин L, катепсин О, катепсин S, катепсин V, катепсин X/Z/P, CBL, CC1, ССК2, CCL, CCL1, CCL11, CCL12, CCL13, CCL14, CCL15, CCL16, CCL17, CCL18, CCL19, CCL2, CCL20, CCL21, CCL22, CCL23, CCL24, CCL25, CCL26, CCL27, CCL28, CCL3, CCL4, CCL5, CCL6, CCL7, CCL8, CCL9/10, CCR, CCR1, CCR10, CCR10, CCR2, CCR3, CCR4, CCR5, CCR6, CCR7, CCR8, CCR9, CD1, CD2, CD3, CD3E, CD4, CD5, CD6, CD7, CD8, CD10, CD11a, CD11b, CD11c, CD13, CD14, CD15, CD16, CD18, CD19, CD20, CD21, CD22, CD23, CD25, CD27L, CD28, CD29, CD30, CD30L, CD32, CD33 (белок р67), CD34, CD38, CD40, CD40L, CD44, CD45, CD46, CD49a, CD52, CD54, CD55, CD56, CD61, CD64, CD66e, CD74, CD80 (В7-1), CD89, CD95, CD123, CD137, CD138, CD140a, CD146, CD147, CD148, CD152, CD164, CEACAM5, CFTR, цГМФ, CINC, ботулинический токсин, токсин Clostridium perfringens, CKb8-1, CLC, CMV, CMV UL, CNTF, CNTN-1, COX, C-Ret, CRG-2, CT-1, CTACK, CTGF, CTLA-4, PD-1, PD-L1, LAG3, TIM3, галектин-9, CX3CL1, CX3CR1, CXCL, CXCL1, CXCL2, CXCL3, CXCL4, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL11, CXCL12, CXCL13, CXCL14, CXCL15, CXCL16, CXCR, CXCR1, CXCR2, CXCR3, CXCR4, CXCR5, CXCR6, антигены, ассоциированные с цитокератином опухоли, DAN, DCC, DcR3, DC-SIGN, регулирующий комплемент фактор (фактор ускорения диссоциации), des(1-3)-IGF-I (IGF-1 головного мозга), Dhh, дигоксин, DNAM-1, ДНКаза, Dpp, DPPIV/CD26, Dtk, ECAD, EDA, EDA-A1, EDA-A2, EDAR, EGF, EGFR (ErbB-1), EMA, EMMPRIN, ENA, эндотелиновый рецептор, энкефалиназа, eNOS, Eot, эотаксин 1, EpCAM, эфрин B2/EphB4, EPO, ERCC, Е-селектин, ET-1, фактор IIa, фактор VII, фактор VIIIc, фактор IX, фибробласт-активирующий белок (FAP), Fas, FcR1, FEN-1, ферритин, FGF, FGF-19, FGF-2, FGF3, FGF-8, FGFR, FGFR-3, фибрин, FL, FLIP, Flt-3, Flt-4, фолликулостимулирующий гормон, фракталкин, FZD1, FZD2, FZD3, FZD4, FZD5, FZD6, FZD7, FZD8, FZD9, FZD10, G250, Gas6, GCP-2, GCSF, GD2, GD3, GDF, GDF-1, GDF-3 (Vgr-2), GDF-5 (BMP-14, CDMP-1), GDF-6 (BMP-13, CDMP-2), GDF-7 (BMP-12, CDMP-3), GDF-8 (миостатин), GDF-9, GDF-15 (MIC-1), GDNF, GDNF, GFAP, GFRa-1, GFR-альфа 1, GFR-альфа 2, GFR-альфа 3, GITR, глюкагон, Glut4, гликопротеин IIb/IIIa (GPIIb/IIIa), GM-CSF, gp130, gp72, GRO, релизин-фактор ростового гормона, гаптен (NP-кэп или NIP-кэп), HB-EGF, НСС, оболочечный гликопротеин HCMV gB, оболочечный гликопротеин HCMV gH, HCMV UL, гематопоэтический фактор роста (HGF), Hep В gp120, гепараназа, Her2, Her2/neu (ErbB-2), Her3 (ErbB-3), Her4 (ErbB-4), гликопротеин вируса герпеса простого (HSV) gB, гликопротеин HSV gDn, HGF А, высокомолекулярный ассоциированный с меланомой антиген (HMW-MAA), HIV gp120, HIV IIIB gp 120 V3 loop, HLA, HLA-DR, HM1.24, HMFG РЕМ, HRG, Hrk, человеческий сердечный миозин, человеческий цитомегаловирус (HCMV), человеческий гормон роста (HGH), HVEM, 1-309, IAP, ICAM, ICAM-1, ICAM-3, ICE, ICOS, IFNg, Ig, IgA-рецептор, IgE, IGF, IGF-связывающий белок, IGF-1R, IGFBP, IGF-I, IGF-II, IL, IL-1, IL-1R, IL-2, IL-2R, IL-4, IL-4R, IL-5, IL-5R, IL-6, IL-6R, IL-8, IL-9, IL-10, IL-12, IL-13, IL-15, IL-18, IL-18R, IL-21, IL-23, IL-27, интерферон (INF)-альфа, INF-бета, INF-гамма, ингибин, iNOS, А-цепь инсулина, В-цепь инсулина, инсулиноподобный фактор роста 1, интегрин альфа 2, интегрин альфа 3, интегрин альфа 4, интегрин альфа 4/бета 1, интегрин альфа 4/бета 7, интегрин альфа 5 (альфа V), интегрин альфа 5/бета 1, интегрин альфа 5/бета 3, интегрин альфа 6, интегрин бета 1, интегрин бета 2, интерферон гамма, IP-10, I-TAC, JE, калликреин 2, калликреин 5, калликреин 6, калликреин 11, калликреин 12, калликреин 14, калликреин 15, калликреин L1, калликреин L2, калликреин L3, калликреин L4, KC, KDR, фактор роста кератиноцитов (KGF), ламинин 5, LAMP, LAP, LAP (TGF-1), латентный TGF-1, латентный TGF-1 bp1, LBP, LDGF, LECT2, фактор Lefty, антиген Льюис-Y, антиген, ассоциированный с системой Льюис, LFA-1, LFA-3, Lfo, LIF, LIGHT, липопротеин, LIX, LKN, Lptn, L-селектин, LT-a, LT-b, LTB4, LTBP-1, антиген поверхности легкого, лютеинизирующий гормон, рецептор лимфотоксина бета, Мас-1, MAdCAM, MAG, МАР2, MARC, МСАМ, МСК-2, МСР, M-CSF, MDC, Mer, металлопротеазы, MGDF-рецептор, MGMT, МНС (HLA-DR), MIF, MIG, MIP, MIP-1-альфа, MK, ММАС1, ММР, ММР-1, ММР-10, ММР-11, ММР-12, ММР-13, ММР-14, ММР-15, ММР-2, ММР-24, ММР-3, ММР-7, ММР-8, ММР-9, MPIF, Мро, MSK, MSP, муцин (Muc1), MUC18, ингибирующее вещество Мюллера, Mug, MuSK, NAIP, NAP, NCAD, N-кадхерин, NCA 90, NCAM, неприлизин, нейротрофин-3, -4 или -6, нейротурин, фактор роста нерва (NGF), NGFR, NGF-бета, nNOS, NO, NOS, Npn, NRG-3, NT, NTN, OB, OGG1, OPG, OPN, OSM, OX40L, OX40R, pi50, p95, PADPr, паратиреоидный гормон, PARC, PARP, PBR, PBSF, PCAD, Р-кадхерин, PCNA, PDGF, PDK-1, PECAM, РЕМ, PF4, PGE, PGF, PGI2, PGJ2, PIN, PLA2, плацентарная щелочная фосфатаза (PLAP), PIGF, PLP, PP14, проинсулин, прорелаксин, белок С, PS, PSA, PSCA, простатспецифический мембранный антиген (PSMA), PTEN, PTHrp, Ptk, PTN, R51, RANK, RANKL, RANTES, цепь А релаксина, цепь В релаксина, ренин, респираторно-синцитиальный вирус (RSV) F, RSV Fgp, Ret, ревматоидный фактор, RLIP76, RPA2, RSK, S100, SCF/KL, SDF-1, SERINE, сывороточный альбумин, sFRP-3, Shh, SIGIRR, SK-1, SLAM, SLPI, SMAC, SMDF, SMOH, SOD, SPARC, Stat, STEAP, STEAP-II, TACE, TACI, TAG-72 (ассоциированный с опухолью гликопротеин-72), TARC, ТСА-3, Т-клеточный рецептор (например, Т-клеточный рецептор альфа/бета), TdT, ТЕСК, ТЕМ1, ТЕМ5, ТЕМ7, ТЕМ8, TERT, PLAP-подобная щелочная фосфатаза яичек, TfR, TGF, TGF-альфа, TGF-бета, TGF-бета Pan-специфический, TGF-бeтaRI (ALK-5), TGF-бетаRII, TGF-бетаRIIb, TGF-бетаRIII, TGF-бета1, TGF-бета2, TGF-бета3, TGF-бета4, TGF-бета5, тромбин, Ck-1 тимуса, тиреотропный гормон, Tie, TIMP, TIQ, тканевый фактор, TMEFF2, Tmpo, TMPRSS2, TNF, TNF-альфа, TNF-альфа,бета, TNF-бета2, TNFc, TNF-RI, TNF-RII, TNFRSF10A (TRAIL R1 Apo-2, DR4), TNFRSF10B (TRAIL R2 DR5, KILLER, TRICK-2A, TRICK-B), TNFRSF10C (TRAIL R3 DcR1, LIT, TRID), TNFRSF10D (TRAIL R4 DcR2, TRUNDD), TNFRSF11A (RANK ODF R, TRANCE R), TNFRSF11B (OPG OCIF, TR1), TNFRSF12 (TWEAK R FN14), TNFRSF13B (TACI), TNFRSF13C (BAFF R), TNFRSF14 (HVEM ATAR, HveA, LIGHT R, TR2), TNFRSF16 (NGFR p75NTR), TNFRSF17 (BCMA), TNFRSF18 (GITR AITR), TNFRSF19 (TROY TAJ, TRADE), TNFRSF19L (RELT), TNFRSF1A (TNF RI CD120a, p55-60), TNFRSF1B (TNF RII CD120b, p75-80), TNFRSF26 (TNFRH3), TNFRSF3 (LTbR TNF RIII, TNFC R), TNFRSF4 (OX40 ACT35, TXGP1 R), TNFRSF5 (CD40 p50), TNFRSF6 (Fas Аро-1, АРТ1, CD95), TNFRSF6B (DcR3 M68, TR6), TNFRSF7 (CD27), TNFRSF8 (CD30), TNFRSF9 (4-1BB CD 137, ILA), TNFRSF21 (DR6), TNFRSF22 (DcTRAIL R2 TNFRH2), TNFRST23 (DcTRAIL R1 TNFRH1), TNFRSF25 (DR3 Apo-3, LARD, TR-3, TRAMP, WSL-1), TNFSF10 (TRAIL Аро-2-лиганд, TL2), TNFSF11 (TRANCE/RANK-лиганд ODF, OPG-лиганд), TNFSF12 (TWEAK Аро-3-лиганд, DR3-лиганд), TNFSF13 (APRIL TALL2), TNFSF13B (BAFF BLYS, TALL1, THANK, TNFSF20), TNFSF14 (LIGHT HVEM-лиганд, LTg), TNFSF15 (TL1A/VEGI), TNFSF18 (GITR-лиганд AITR-лиганд, TL6), TNFSF1A (TNF-a конектин, DIF, TNFSF2), TNFSF1B (TNF-b LTa, TNFSF1), TNFSF3 (LTb TNFC, p33), TNFSF4 (ОХ40-лиганд gp34, TXGP1), TNFSF5 (CD40-лиганд CD154, gp39, HIGM1, IMD3, TRAP), TNFSF6 (Fas-лиганд Аро-1-лиганд, APT1-лиганд), TNFSF7 (CD27-лиганд CD70), TNFSF8 (CD30-лиганд CD153), TNFSF9 (4-1ВВ-лиганд CD137-лиганд), TP-1, t-PA, Тро, TRAIL, TRAIL R, TRAIL-R1, TRAIL-R2, TRANCE, рецептор трансферрина, TRF, Trk, TROP-2, TLR (Toll-подобный рецептор) 1, TLR2, TLR3, TLR4, TLR5, TLR6, TLR7, TLR8, TLR9, TLR10, TSG, TSLP, ассоциированный с опухолью антиген СА125, ассоциированный с опухолью антиген, экспрессирующий ассоциированные с антигеном Льюис-Y углеводы, TWEAK, ТХВ2, Ung, uPAR, uPAR-1, урокиназа, VCAM, VCAM-1, VECAD, VE-кадхерин, УЕ-кадхерин-2, VEFGR-1 (flt-1), VEGF, VEGFR, VEGFR-3 (flt-4), VEGI, VIM, вирусный антиген, VLA, VLA-1, VLA-4, VNR интегрин, фактор фон Виллебранда, WIF-1, WNT1, WNT2, WNT2B/13, WNT3, WNT3A, WNT4, WNT5A, WNT5B, WNT6, WNT7A, WNT7B, WNT8A, WNT8B, WNT9A, WNT9B, WNT10A, WNT10B, WNT11, WNT16, XCL1, XCL2, XCR1, XCR1, XEDAR, XIAP, XPD, HMGB1, IgA, А бета, CD81, CD97, CD98, DDR1, DKK1, EREG, Hsp90, IL-17/IL-17R, IL-20/IL-20R, окисленный LDL, PCSK9, прекалликреин, RON, TMEM16F, SOD1, хромогранин А, хромогранин В, tau, VAP1, высокомолекулярный кининоген, IL-31, IL-31R, Nav1.1, Nav1.2, Nav1.3, Nav1.4, Nav1.5, Nav1.6, Nav1.7, Nav1.8, Nav1.9, EPCR, C1, C1q, C1r, C1s, C2, C2a, C2b, С3, С3а, C3b, C4, C4a, C4b, C5, C5a, C5b, C6, C7, C8, C9, фактор В, фактор D, фактор H, пропердин, склеростин, фибриноген, фибрин, протромбин, тромбин, тканевый фактор, фактор V, фактор Va, фактор VII, фактор VIIa, фактор VIII, фактор VIIIa, фактор IX, фактор ТХа, фактор X, фактор Ха, фактор XI, фактор XIa, фактор XII, фактор XIIa, фактор XIII, фактор XIIIa, TFPI, антитромбин III, EPCR, тромбомодулин, TAPI, tPA, плазминоген, плазмин, PAI-1, PAI-2, GPC3, синдекан-1, синдекан-2, синдекан-3, синдекан-4, LPA и S1P; и рецепторы для гормонов и факторов роста.
Хотя перечисленные выше примеры антигенов включают также рецепторы, указанные рецепторы, даже присутствующие в растворимой форме в общей воде организма, можно применять в качестве антигена, с которым связывается антигенсвязывающий домен, предлагаемый в настоящем изобретении. Один из примеров (но, не ограничиваясь только им) растворимой формы такого рецептора может являться белок, представленный в SEQ ID NO: 35, который представляет собой растворимый IL-6R, описанный у Mullberg и др., J. Immunol. 152 (10), 1994, сс.4958-4968.
Перечисленные выше примеры антигенов включают мембранные молекулы, которые экспрессируются на клеточных мембранах, и растворимые молекулы, которые секретируются из клеток наружу от клеток. Когда антигенсвязывающий домен, предлагаемый в настоящем изобретении, связывается с растворимой молекулой, секретируемой из клеток, то антигенсвязывающий домен предпочтительно обладает нейтрализующей активностью.
Раствор, в котором содержится растворимая молекула, не ограничен, и указанная растворимая молекула может присутствовать в общей воде организма, т.е. в любой жидкости сосудов или любой жидкости, заполняющей пространство между тканями и клетками в живых организмах. Растворимая молекула, с которой антигенсвязывающий домен, предлагаемый в настоящем изобретении, связывается, может присутствовать во внеклеточной жидкости (но, не ограничиваясь только указанным). Внеклеточная жидкость является общим названием плазмы, межклеточной жидкости, лимфы, плотных соединительных тканей, спинномозговой жидкости, спинальной жидкости, аспиратов, синовиальной жидкости или таких компонентов в кости и хряще, альвеолярной жидкости (жидкость бронхоальвеолярного лаважа), асцитной жидкости, плеврального выпота, сердечного выпота, цистной жидкости, внутриглазной жидкости (гидатоид) или указанных трансклеточных жидкостей (различные жидкости в гранулярных полостях, образовавшиеся в результате активного транспорта или секреторной активности клеток, и жидкости в полости кишечника и других полостей в организме) у позвоночных животных.
Эпитоп, который означает антигенную детерминанту, присутствующую в антигене, представляет собой область с которой связывается антигенсвязывающий домен, указанный в настоящем описании. Так, например, эпитоп можно характеризовать на основе его структуры. Альтернативно этому, эпитоп можно характеризовать на основе антигенсвязывающей активности антигенсвязывающего домена, который распознает эпитоп. Когда антиген представляет собой пептид или полипептид, то эпитоп можно определять по аминокислотным остаткам, образующим эпитоп. Когда эпитоп представляет собой сахарную цепь, то эпитоп можно определять по специфической для него структуре сахарной цепи.
Линейный эпитоп представляет собой эпитоп, который содержит эпитоп, который распознается по его первичной последовательности аминокислот. Указанный линейный эпитоп, как правило, содержит по меньшей мере 3 и наиболее часто по меньшей мере 5, например, примерно от 8 до 10 или от 6 до 20 аминокислотных остатков в его уникальной последовательности.
В отличие от линейного эпитопа конформационный эпитоп представляет собой эпитоп, в котором первичная последовательность аминокислот содержит компонент, который не является единственной детерминантой распознаваемого эпитопа (например, эпитоп, первичная последовательность аминокислот которого может не распознаваться специфическим в отношении эпитопа антителом). Конформационный эпитоп может содержать увеличенное по сравнению с линейными эпитопами количество аминокислот. Для распознавания конформационного эпитопа антигенсвязывающий домен распознает трехмерную структуру пептида или белка. Например, когда белковая молекула уложена и образует трехмерную структуру, то некоторые аминокислоты и/или основная цепь полипептида, которые образуют конформационный эпитоп, располагаются параллельно, что обеспечивает распознавание эпитопа. Примеры методов определения конформации эпитопа включают (но, не ограничиваясь только ими) рентгеновскую кристаллографию, двухмерный ядерный магнитный резонанс и сайтспецифическое спиновое мечение, и электронный парамагнитный резонанс (см., например, Epitope Mapping Protocols in Methods in Molecular Biology, под ред. Morris, т.66, 1996).
Структуру антигенсвязывающего домена, связывающегося с эпитопом, называют паратопом. Паратоп стабильно связывается с эпитопом посредством водородной связи, электростатической силы, Ван-дер-ваальсовой силы, гидрофобной связи или т.п., действующей между эпитопом и паратопом. Указанную силу связывания между эпитопом и паратопом называют аффинностью. Общую силу связывания, когда множество антигенсвязывающих доменов связывается с множеством антигенов, называют авидностью. Аффинность действует синергетически, когда, например, антитело, содержащее множество антигенсвязывающих доменов (т.е. многовалентное антитело), связывается с множеством эпитопов. Таким образом, авидность превышает аффинность.
В конкретном варианте осуществления изобретения антигенсвязывающий домен, представленный в настоящем описании, характеризуется константой диссоциации (Kd)≤1 мкМ, ≤100нМ, ≤10нМ, ≤1нМ, ≤0,1нМ, ≤0,01нМ или ≤0,001нМ (например, 10-8 М или менее, например, от 10-8 М до 10-13 М, например, от 10-9М до 10-13М).
Ниже в настоящем описании представлены примеры методов подтверждения связывания с эпитопом антигенсвязывающего домена против IL-6R, или полипептида, содержащего антигенсвязывающий домен. Однако метод подтверждения связывания с эпитопом антигенсвязывающего домена против антигена, отличного от IL-6R, или полипептида, содержащего антигенсвязывающий домен, можно осуществлять также соответственно согласно описанному ниже примеру.
Например, для подтверждения того, что антигенсвязывающий домен против IL-6R распознает линейный эпитоп в молекуле IL-6R, можно применять описанный ниже метод: синтезируют для указанной выше цели линейный пептид, содержащий аминокислотную последовательность, которая содержит внеклеточный домен IL-6R. Пептид можно синтезировать химически. Альтернативно этому, пептид получают с помощью методов генной инженерии, используя область в кДНК IL-6R, которая кодирует аминокислотную последовательность, соответствующую внеклеточному домену. Затем оценивают антигенсвязывающий домен против IL-6R в отношении связывающей активности с линейным пептидом, который содержит аминокислотную последовательность, образующую внеклеточный домен. Например, иммобилизованный линейный пептид можно применять в качестве антигена при осуществлении ELISA для оценки связывающей активности антигенсвязывающей домена в отношении пептида. Альтернативно этому, связывающую активность в отношении линейного пептида можно оценивать по уровню ингибирования линейным пептидом связывания антигенсвязывающего домена с экспрессирующими IL-6R клетками. С помощью этих анализов можно определять связывающую активность антигенсвязывающего домена в отношении линейного пептида.
Кроме того, способность антигенсвязывающего домена против IL-6R распознавать конформационный эпитоп можно подтверждать следующим методом: для описанной выше цели получали экспрессирующие IL-6R клетки. Считается, например, что антигенсвязывающий домен против IL-6R распознает конформационный эпитоп, если он при контакте отличается сильным связыванием с экспрессирующими IL-6R клетками, но при этом антигенсвязывающий домен в незначительной степени связывается с иммобилизованным линейным пептидом, который содержит аминокислотную последовательность, образующую внеклеточный домен IL-6R, или с денатурированным (с помощью обычного денатурирующего агента, такого как гуанидин) линейным пептидом, который содержит аминокислотную последовательность, образующую внеклеточный домен IL-6R. В этом контексте понятие «связывается в незначительной степени» означает, что активность связывания составляет 80% или менее, как правило, 50% или менее, предпочтительно 30% или менее и наиболее предпочтительно 15% или менее по сравнению с активностью связывания с клетками, экспрессирующими человеческий IL-6R.
Методы подтверждения антигенсвязывающей активности антигенсвязывающего домена включают также методы измерения величины Kd, например, с помощью анализа связывания несущего радиоактивную метку антигена (РИА). В одном из вариантов осуществления изобретения РИА осуществляют с использованием представляющего интерес антигенсвязывающего домена и его антигена. Например, измеряют в растворе аффинность связывания антигенсвязывающего домена и его антигена путем уравновешивания антигенсвязывающего домена минимальной концентрацией меченного 125I антигена в присутствии полученных титрованием серийных разведений немеченого антигена, и осуществляя затем «захват» связанного антигена с помощью планшета, сенсибилизированного антигенсвязывающим доменом (см., например, Chen и др., J. Mol Biol 293, 1999, сс.865-881).
В альтернативном варианте осуществления изобретения Kd измеряют методом поверхностного плазмонного резонанса с помощью устройства BIACORE®. Например, анализ осуществляют с помощью устройства BIACORE®-2000 или BIACORE®-3000 (фирма BIAcore Inc., Пискатавей, шт. Нью-Джерси) при 25°С с использованием антигена, иммобилизованного на СМ5-чипах с уровнем иммобилизации, соответствующим примерно 10 единицам ответа (RU). В одном из вариантов осуществления изобретения биосенсорные чипы из карбоксиметилированного декстрана (СМ5, фирма BIAcore Inc.) активируют с помощью гидрохлорида N-этил-N'-(3-диметиламинопропил)карбодиимида (EDC) и N-гидроксисукцинимида (NHS) согласно инструкциям поставщика. Антиген разводят 10мМ ацетатом натрия, рН 4,8 до концентрации 5 мкг/мл (примерно 0,2 мкМ) перед инъекцией со скоростью потока 5 мкл/мин для достижения связывания белка, соответствующего примерно 10 единицам ответа (RU). После инъекции антигена инъецируют 1М этаноламин для блокады непрореагировавших групп. Для кинетических измерений инъецируют двукратные серийные разведения (от 0,78 до 500нМ) антигенсвязывающего домена в ЗФР, дополненном 0,05% полисорбата 20 (TWEEN-20™) в качестве сурфактанта (ЗФР-Т), при 25°С со скоростью потока примерно 25 мкл/мин. Скорость реакции ассоциации (kon) и реакции диссоциации (koff) рассчитывают с использованием простой модели связывания Ленгмюра 1:1 (программное обеспечение Evaluation, версия 3.2, BIACORE®) путем одновременной аппроксимации сенсограмм ассоциации и диссоциации. Константу равновесия реакции диссоциации (Kd) рассчитывают как соотношение koff/kon. Кроме того, кажущуюся константу диссоциации (Kd) можно определять с помощью анализа равновесия. Для осуществления этой процедуры можно применять протокол, прилагаемый к BIACORE® (см., например, Chen и др., J Mol Biol 293, 1999, сс.865-881 и Methods Enzymol. 323, 2000, сс.325-340). При осуществлении анализа методом поверхностного плазмонного резонанса специалисты в данной области могут в значительной степени изменять количество иммобилизованного белка, количество применяемого в реакции белка, температуру и состав раствора.
Если скорость реакции ассоциации превышает 106 М-1 с-1 при оценке с помощью описанного выше метода поверхностного плазмонного резонанса, то скорость реакции ассоциации можно определять с помощью метода гашения флуоресценции, который позволяет измерять повышение или снижение интенсивности излучения флуоресценции (длина волны возбуждения 295 нм; длина волны испускания 340 нм, полоса пропускания 16 нм) при 25°С, применяя в концентрации 20нМ антигенсвязывающий домен в ЗФР, рН 7,2, в присутствии возрастающих концентраций антигена, осуществляя измерения с помощью спектрометра (например, спектрофотометра, снабженного блоком для остановки потока (фирма Aviv Instruments), или спектрофотометра SLM-AMINCO™ серии 8000 (фирма ThermoSpectronic/ Thermo Fisher Scientific Inc.) при перемешивании содержимого кюветы).
Кроме того, антигенсвязывающую активность антигенсвязывающего домена можно измерять с помощью известного метода измерения взаимодействия типа молекула-молекула, такого как электрогенерированная хемилюминесценция.
Примеры метода измерения связывающей активности антигенсвязывающего домена с IL-6R в отношении экспрессирующих IL-6R клеток включают методы, описанные в: Antibodies: A Laboratory Manual, под ред. Harlow, David Lane, изд-во Cold Spring Harbor Laboratory, 1988, cc. 359-420. В частности, оценку связывающей активности можно осуществлять на основе принципа ELIS А или метода FACS (разделение клеток на основе возбуждения флуоресценции), используя экспрессирующие IL-6R клетки в качестве антигена.
В формате ELISA связывающую активность антигенсвязывающего домена с IL-6R в отношении экспрессирующих IL-6R клеток, можно оценивать количественно путем сравнения уровней сигналов, возникающих в процессе ферментативной реакции. В частности, тестируемый антигенсвязывающий домен добавляют в планшет для ELIS А, на котором иммобилизованы экспрессирующие IL-6R клетки. Затем тестируемый антигенсвязывающий домен, связанный с клетками, выявляют с помощью меченного ферментом антитела, которое распознает тестируемый антигенсвязывающий домен. Альтернативно этому, когда применяют FACS, приготавливают серийные разведения тестируемого антигенсвязывающего домена, и титр антитела, связывающегося с экспрессирующими IL-6R клетками, можно определять путем сравнения активности тестируемого антигенсвязывающего домена с экспрессирующими IL-6R клетками.
Связывание тестируемого антигенсвязывающего домена с антигеном, который экспрессируется на поверхности клеток, суспендированных в буферном растворе или аналогичной среде, можно выявлять с помощью проточного цитометра. Известными проточными цитометрами являются, например, следующие устройства:
FACSCanto™ II,
FACSAria™,
FACS Array™,
FACS Vantage™ SE
FACSCalibur™ (все товарные знаки фирмы BD Biosciences),
EPICS ALTRA HyPerSort,
Cytomics FC 500,
EPICS XL-MCL ADC EPICS XL ADC,
Cell Lab Quanta/Cell Lab Quanta SC (все товарные знаки фирмы Beckman Coulter).
Одним из предпочтительных примеров метода измерения антигенсвязывающей активности антигенсвязывающего домена в отношении IL-6R является следующий метод: сначала экспрессирующие IL-6R клетки, подвергнутые взаимодействию с тестируемым антигенсвязывающий доменом, окрашивают меченным с помощью ФИТЦ вторичным антителом, которое распознает тестируемый антигенсвязывающий домен. Тестируемый антигенсвязывающий домен соответствующим образом разводят приемлемым буферным раствором с получением антигенсвязывающего домена в требуемой для применения концентрации. Например, антигенсвязывающий домен можно применять в концентрации, составляющей от 10 мкг/мл до 10 нг/мл. Затем определяют интенсивность флуоресценции и количество клеток с помощью FACSCalibur (фирма Becton, Dickinson and Company). Интенсивность флуоресценции, определенная с помощью анализов на основе программы CELL QUEST (фирма Becton, Dickinson and Company), а именно, выраженная в виде средних геометрических значений, отражает количество связанного тестируемого антигенсвязывающего домена. В целом, активность связывания антигенсвязывающего домена, выраженную в количестве связанного тестируемого антигенсвязывающего домена, можно оценивать, определяя среднее геометрическое значение.
Имеет ли антигенсвязывающий домен против IL-6R общий эпитоп с каким-либо антигенсвязывающим доменом, можно оценивать по конкуренции между этими антигенсвязывающими доменами за один и тот же эпитоп.Конкуренцию между антигенсвязывающими доменами определяют путем анализа перекрестной блокады или с помощью аналогичного анализа. Например, предпочтительным анализом перекрестной блокады является конкурентный ELISA-анализ.
В частности, при осуществлении анализа перекрестной блокады белок IL-6R, иммобилизованный в лунках титрационного микропланшета, предварительно инкубируют в присутствии антигенсвязывающего домена, рассматриваемого в качестве конкурента-кандидата, или без него. Затем вносят тестируемый антигенсвязывающий домен. Количество связанного с белком IL-6R тестируемого антигенсвязывающего домена в лунках находится в обратной корреляции с активностью связывания антигенсвязывающего домена, рассматриваемого в качестве конкурента-кандидата, который конкурирует за связывание с этим же эпитопом. В целом, это означает, что чем выше аффинность антигенсвязывающего домена, рассматриваемого в качестве конкурента-кандидата, к одному и тому же эпитопу, тем ниже активность связывания тестируемого антигенсвязывающего домена с сенсибилизированными белком IL-6R лунками.
Количество тестируемого антигенсвязывающего домена, связанного через белок IL-6R с лунками, легко определять путем предварительного мечения антигенсвязывающего домена. Например, меченный биотином антигенсвязывающий домен оценивают с использованием конъюгата авидин/пероксидаза и соответствующего субстрата. В частности, анализ перекрестной блокады, в котором применяют в качестве меток ферменты, такие как пероксидаза, называют «ELISA-анализом в конкурентных условиях (конкурентный анализ)». Антигенсвязывающий домен можно метить также с помощью альтернативных предназначенных для мечения субстанций, которые можно выявлять или оценивать. В частности, в данной области известны радиоактивные метки, флуоресцентные метки и т.п.
Когда антигенсвязывающий домен, рассматриваемый в качестве конкурента-кандидата, может блокировать связывание антигенсвязывающего домена против IL-6R по меньшей мере на 20%, предпочтительно по меньшей мере на 20-50% и предпочтительно по меньшей мере на 50% по сравнению со связывающей активностью, установленной в контрольном эксперименте, который проводят в отсутствии антигенсвязывающего домена, рассматриваемого в качестве конкурента-кандидата, то считается, что для тестируемого антигенсвязывающего домена характерна выраженная способность к связыванию с тем же эпитопом, с которым связывается антигенсвязывающий домен, рассматриваемый в качестве конкурента, или он конкурирует за связывание с тем же самым эпитопом.
Если структура эпитопа, с которым связывается антигенсвязывающий домен против IL-6R, уже идентифицирована, то для решения вопроса о том, имеют ли тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен один и тот же эпитоп, можно сравнивать связывающие активности двух указанных антигенсвязывающих доменов с пептидом или полипептидом, полученным путем интродукции аминокислотной мутации в пептид, образующий эпитоп.
При осуществлении указанного метода измерения связывающих активностей, например, можно сравнивать связывающие активности тестируемого антигенсвязывающего домена и контрольного антигенсвязывающего домена с линейным пептидом, в который интродуцирована мутация, с помощью описанного выше формата ELISA. Помимо метода ELISA связывающую активность в отношении мутантного пептида, связанного с колонкой, можно определять, пропуская через колонку тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен и затем оценивая количество антигенсвязывающего домена, элюированное в раствор для элюции. Известны методы адсорбции мутантного пептида на колонке, например, в форме слитого с GST пептида.
Когда идентифицированный эпитоп представляет собой конформационный эпитоп, то для решения вопроса о том, имеют ли тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен общий эпитоп, можно применять следующий метод: сначала получают экспрессирующие IL-6R клетки и клетки, экспрессирующие IL-6R с мутацией, интродуцированной в эпитоп. Тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен добавляют в клеточные суспензии, полученные путем суспендирования этих клеток в приемлемом буферном растворе, таком как ЗФР. Затем клеточные суспензии соответствующим образом промывают буферным раствором и добавляют меченное с помощью ФИТЦ антитело, которое распознает тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен. Определяют интенсивность флуоресценции и количество клеток, окрашенных меченым антителом, используя FACSCalibur (фирма Becton, Dickinson and Company). Тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен соответствующим образом разводят приемлемым буферным раствором и применяют в требуемой концентрации. Указанные антигенсвязывающие домены применяют, например, в любой из концентраций от 10 мкг/мл до 10 нг/мл. Интенсивность флуоресценции определяют с помощью анализа, для оценки результатов которого применяют программу CELL QUEST (фирма Becton, Dickinson and Company), а именно, определяют среднее геометрическое значение, которое отражает количество меченого антитела, связанного с клетками. В целом, это означает, что связывающую активность тестируемого антигенсвязывающего домена и контрольного антигенсвязывающего домена, которые характеризуются количеством связанного меченого антитела, можно определять, оценивая среднее геометрическое значение.
Конкуренцию антигенсвязывающего домена с другими антигенсвязывающим доменом за один и тот же эпитоп помимо описанных выше анализов ELISA или FACS можно определять также с помощью анализа связывания несущего радиоактивную метку антигена (РИА), анализа поверхностного плазмонного резонанса BIACORE®, электрогенерированной хемилюминесции или т.п.
При осуществлении описанного выше метода для решения вопроса о том, имеет ли место «незначительное связывание с клетками, которые экспрессируют мутантный IL-6R», можно применять, например, следующий метод: сначала тестируемый антигенсвязывающий домен и контрольный антигенсвязывающий домен, связанные с клетками, которые экспрессируют мутантный IL-6R, окрашивают меченым антителом. Затем определяют интенсивность флуоресценции в клетках. Когда для оценки флуоресценции с помощью проточной цитометрии используют FACSCalibur, то полученную интенсивность флуоресценции можно анализировать с помощью программы CELL QUEST. На основе средних геометрических значений в присутствии полипептида, являющегося продуктом ассоциации, и без него можно рассчитывать согласно приведенной ниже формуле I используемое для сравнения значение (ΔGeo-Mean), характеризующее степень увеличения интенсивности флуоресценции в результате связывания с антигенсвязывающим доменом.
(Формула I)
ΔGeo-Mean=Geo-Mean (в присутствии полипептида, являющегося продуктом ассоциации)/Geo-Mean (в отсутствии полипептида, являющегося продуктом ассоциации)
Используемое для сравнения среднее геометрическое значение (значение AGeo-Mean для мутантной молекулы IL-6R), определенное с помощью описанного выше анализа, которое отражает количество тестируемого антигенсвязывающего домена, связанного с клетками, которые экспрессируют мутантный IL-6R, сравнивают с используемым для сравнения значением ΔGeo-Mean, которое отражает количество тестируемого антигенсвязывающего домена, связанного с экспрессирующими IL-6R клетками. В этом случае концентрации тестируемого антигенсвязывающего домена, применяемые для определения используемых для сравнения (относительных) значений ΔGeo-Mean для экспрессирующих IL-6R клеток и клеток, экспрессирующих мутантный IL-6R, наиболее предпочтительно регулируют таким образом, чтобы концентрации были одинаковыми или практически одинаковыми. Антигенсвязывающий домен, для которого уже подтверждена способность распознавать эпитоп в IL-6R, применяют в качестве контрольного антигенсвязывающего домена.
Если используемое для сравнения значение ΔGeo-Mean тестируемого антигенсвязывающего домена в отношении клеток, экспрессирующих мутантный IL-6R, ниже чем используемое для сравнения значение ΔGeo-Mean тестируемого антигенсвязывающего домена в отношении клеток, экспрессирующих IL-6R, по меньшей мере на 80%, предпочтительно на 50%, более предпочтительно на 30% и наиболее предпочтительно на 15%, то считают, что тестируемый антигенсвязывающий домен «незначительно связывается с клетками, которые экспрессируют мутантный IL-6R». Формула для определения значения Geo-Mean (среднее геометрическое значение) описана в руководстве по применению программы CELL QUEST (фирма BD biosciences). Эпитоп для тестируемого антигенсвязывающего домена и эпитоп для контрольного антигенсвязывающего домена можно рассматривать как одинаковые, когда путем сравнения установлено, что используемые для сравнения значения являются практически эквивалентными.
В настоящем описании понятие «транспортирующий фрагмент» относится к фрагменту, отличному от антигенсвязывающего домена в полипептиде. Транспортирующий фрагмент, предлагаемый в настоящем изобретении, как правило, представляет собой пептид или полипептид, состоящий из аминокислот. В конкретном варианте осуществления изобретения транспортирующий фрагмент представляет собой полипептид, сцепленный с антигенсвязывающим доменом через сайт расщепления. Транспортирующий фрагмент, предлагаемый в настоящем изобретении, может представлять собой серию пептидов или полипептидов, соединенных через амидную(ые) связь(и), или может представлять собой комплекс, образованный из множества пептидов или полипептидов с помощью ковалентной(ых) связи(ей), такой(их) как дисульфидная связь, или нековалентной(ых) связи(ей), такой(их) как водородная связь или гидрофобное взаимодействие.
Транспортирующий фрагмент, предлагаемый в настоящем изобретении, имеет ингибирующий домен, который ингибирует антигенсвязывающую активность антигенсвязывающего домена. В настоящем описании понятие «ингибирующий домен» ограничено только ингибированием антигенсвязывающей активности антигенсвязывающего домена. Ингибирующий домен может представлять собой домен, имеющий любую структуру, если применяемый домен может ингибировать антигенсвязывающую активность антигенсвязывающего домена. Примеры указанного ингибирующего домена включают (но, не ограничиваясь только ими) вариабельные области тяжелой цепи антитела (VH), вариабельные области легкой цепи антитела (VL), рецепторы пре-В-клеток и однодоменные антитела. Ингибирующий домен может полностью образовывать транспортирующий фрагмент или может образовывать часть транспортирующего фрагмента.
В некоторых вариантах осуществления настоящего изобретения высвободившийся из полипептида антигенсвязывающий домен имеет более высокую антигенсвязывающую активность по сравнению с активностью до высвобождения. Другими словами, антигенсвязывающая активность антигенсвязывающего домена ингибируется с помощью ингибирующего домена в невысвободившемся из полипептида состоянии антигенсвязывающего домена. Решение вопроса о том, ингибируется ли антигенсвязывающая активность антигенсвязывающего домена с помощью ингибирующего домена, осуществляют с помощью такого метода как FACS ((разделение клеток на основе возбуждения флуоресценции), ELISA (твердофазный иммуноферментный анализ), ECL (электрогенерированная хемилюминесция), метод SPR (поверхностный плазмонный резонанс) (система Biacore), BLI (интерферометрия биослоя) (система Octet). В некоторых вариантах осуществления настоящего изобретения антигенсвязывающая активность антигенсвязывающего домена, высвободившегося из полипептида, имеет величину, равную или превышающую в 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 2000 или 3000 раз связывающую активность антигенсвязывающего домена, невысвободившегося из полипептида. В некоторых более конкретных вариантах осуществления настоящего изобретения связывание антигенсвязывающего домена с антигеном до высвобождения не поддается обнаружению, когда антигенсвязывающую активность антигенсвязывающего домена измеряют с помощью одного из методов, выбранного из перечисленных выше методов.
В некоторых объектах настоящего изобретения сайт расщепления расщепляется так, что антигенсвязывающий домен приобретает способность высвобождаться из полипептида. Таким образом, в указанных объектах изобретения антигенсвязывающую активность можно сравнивать до и после расщепления полипептида. В частности, антигенсвязывающая активность, измеренная при использовании расщепленного полипептида, имеет величину равную или превышающую в 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 2000 или 3000 раз антигенсвязывающую активность, измеренную с использованием нерасщепленного полипептида. В некоторых более конкретных вариантах осуществления изобретения связывание с антигеном антигенсвязывающего домена нерасщепленного полипептида не поддается обнаружению, когда антигенсвязывающую активность измеряют с помощью одного из методов, выбранного из перечисленных выше методов.
В некоторых объектах настоящего изобретения сайт расщепления расщепляется протеазой. Таким образом, в указанных объектах изобретения антигенсвязывающую активность можно сравнивать до и после обработки полипептида протеазой. В частности, антигенсвязывающая активность, измеренная с использованием полипептида после обработки протеазой, имеет величину равную или превышающую в 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 2000 или 3000 раз антигенсвязывающую активность, измеренную с использованием полипептида, не обработанного протеазой. В некоторых более конкретных вариантах осуществления изобретения связывание с антигеном антигенсвязывающего домена необработанного протеазой полипептида не поддается обнаружению, когда антигенсвязывающую активность измеряют с помощью одного из методов, выбранного из перечисленных выше методов.
В настоящем изобретении полипептид, содержащий антигенсвязывающий домен и транспортирующий фрагмент, имеет более продолжительное время полужизни в крови, чем антигенсвязывающий домен, присутствующий в индивидуальном виде. В некоторых вариантах осуществления настоящего изобретения для удлинения времени полужизни полипептида транспортирующий фрагмент создают так, чтобы он имел более продолжительное время полужизни в крови. В указанных вариантах осуществления изобретения примеры подхода, удлиняющего время полужизни в крови транспортирующего фрагмента, включают (но, не ограничиваясь только ими) большую молекулярную массу транспортирующего фрагмента, FcRn-связывающую активность, которой наделен транспортирующий фрагмент, альбуминсвязывающую активность, которой наделен транспортирующий фрагмент, и ПЭГилирование транспортирующего фрагмента. В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент имеет более продолжительное время полужизни в крови, чем антигенсвязывающий домен (другими словами, антигенсвязывающий домен имеет более короткое время полужизни в крови, чем транспортирующий фрагмент).
В настоящем изобретения время полужизни антигенсвязывающего домена индивидуально и полипептида или время полужизни в крови
антигенсвязывающего домена и транспортирующего фрагмента предпочтительно сравнивают, как время их полужизни в крови людей. Если время полужизни в крови трудно измерять в организме человека, то его можно прогнозировать на основе времени их полужизни в крови мышей (например, обычных мышей, трансгенных мышей, экспрессирующих человеческий антиген, и трансгенных мышей, экспрессирующий человеческий FcRn) или обезьян (например, обезьян циномолгус).
В одном из вариантов осуществления изобретения подход, обеспечивающий удлинение времени полужизни в крови транспортирующего фрагмента, включает применение транспортирующего фрагмента, имеющего большую молекулярную массу. В одном из вариантов осуществления изобретения подход, обеспечивающий более длительное время полужизни транспортирующего фрагмента по сравнению с антигенсвязывающим доменом, включает создание транспортирующего фрагмента, имеющего большую молекулярную массу по сравнению с антигенсвязывающим доменом.
В одном из вариантов осуществления изобретения подход, обеспечивающий удлинение времени полужизни в крови транспортирующего фрагмента, включает придание транспортирующему фрагменту FcRn-связывающей активности. Транспортирующий фрагмент можно, как правило, наделять FcRn-связывающей активностью с помощью метода создания FcRn-связывающей области в транспортирующем фрагменте. FcRn-связывающая область означает область, которая обладает связывающей активностью в отношении FcRn, и она может иметь любую структуру, если применяемая область обладает связывающей активностью в отношении FcRn.
Транспортирующий фрагмент, содержащий FcRn-связывающую область, обладает способностью проникать в клетки и затем расщепляться в плазме через путь спасения FcRn. Например, молекула IgG имеет относительно продолжительное время циркуляции в плазме (медленно исчезновение), благодаря функции FcRn, известного в качестве рецептора спасения молекулы IgG. Молекула IgG, поглощенная эндосомой посредством пиноцитоза, связывается с FcRn, который экспрессируется в эндосоме в условиях кислой среды внутри эндосомы. Молекула IgG, которая не может связываться с FcRn, передвигается в лизосому и расщепляется в ней, в то время как молекула IgG, связанная с FcRn, переносится на клеточную поверхность, затем отщепляется в результате диссоциации от FcRn в нейтральных условиях в плазме и затем возвращается в плазму.
FcRn-связывающая область предпочтительно представляет собой область, которая непосредственно связывается с FcRn. Предпочтительные примеры FcRn-связывающей области могут включать Fc-области антител. Однако область, обладающая способностью связываться с полипептидом, таким как альбумин, или IgG, который обладает способностью связываться с FcRn, может непосредственно связываться с FcRn через альбумин, IgG или т.п. Таким образом, FcRn-связывающая область, предлагаемая в настоящем изобретении, может представлять собой область, которая связывается с указанным полипептидом, обладающим FcRn-связывающей способностью.
Связывающую активность FcRn-связывающей области, предлагаемой в настоящем изобретении, в отношении FcRn, прежде всего человеческого FcRn, можно измерять с помощью метода, известного специалистам в данной области, который упомянут выше в разделе, в котором описана связывающая активность. Пригодные для этой цели условия могут определять соответственно специалисты в данной области. Связывающую активность в отношении человеческого FcRn можно оценивать в виде KD (константа диссоциации), кажущаяся KD (кажущаяся константа диссоциации), kd (скорость реакции диссоциации) или кажущаяся kd (кажущаяся скорость реакции диссоциации) и т.д. Указанные величины можно измерять методами, известными специалистам в данной области. Например, можно использовать Biacore (фирма GE Healthcare Japan Corp.), метод Скэтчарда, проточные цитометры и т.п.
Условия измерения связывающей активности FcRn-связывающей области в отношении FcRn не ограничены конкретно, и их могут выбирать соответственно специалисты в данной области. Связывающую активность можно измерять в условиях, включающих, например, MES-буфер при 37°С, как описано, например, в WO 2009/125825. Кроме того, связывающую активность FcRn-связывающей области, предлагаемой в настоящем изобретении, в отношении FcRn можно измерять методом, известным специалистам в данной области, и можно оценивать с использованием, например, Biacore (фирма GE Healthcare Japan Corp.). При измерении связывающей активности FcRn-связывающей области в отношении FcRn можно инъецировать в качестве аналита FcRn и FcRn-связывающую область или транспортирующий фрагмент, содержащий FcRn-связывающую область, на чипы, на которых иммобилизованы FcRn-связывающая область или транспортирующий фрагмент, содержащий FcRn-связывающую область, и FcRn соответственно, с последующей их оценкой.
Что касается значения рН, применяемого в условиях измерения, то связывающую активность FcRn-связывающей области с FcRn можно оценивать при любом рН от 4,0 до 6,5. Предпочтительно применяют значение рН от 5,8 до 6,0, близкое к рН в ранней эндосоме in vivo, для определения аффинности связывания FcRn-связывающей области с человеческим FcRn. Что касается температуры, применяемой в условиях измерения, то связывающую активность FcRn-связывающей области с FcRn можно оценивать при любой температуре от 10°С до 50°С. Для определения аффинности связывания FcRn-связывающей области с человеческим FcRn применяют предпочтительно температуру от 15°С до 40°С. Более предпочтительно для определения аффинности связывания FcRn-связывающей области с FcRn применяют также любую температуру от 20°С до 35°С, например, любую температуру 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 и 35°С. Согласно настоящему изобретению температура 25°С является одним из не ограничивающих объем изобретения примеров температуры.
Один из примеров FcRn-связывающей области включает (но, не ограничиваясь только ею) Fc-область антитела IgG-типа. В случае применения Fc-области антитела IgG-типа, ее тип не ограничен только указанным и можно применять, например, Fc-область IgG1, IgG2, IgG3 или IgG4. Например, можно применять Fc-область, содержащую одну из последовательностей, выбранную из аминокислотных последовательностей, представленных в SEQ ID NO: 21, 22, 23, и 24.
Можно применять нативную Fc-область антитела IgG-типа, а также вариант Fc-области, имеющий одну или несколько аминокислотных замен, если Fc-область обладает FcRn-связывающей активностью.
Например, можно применять вариант Fc-области, который содержит аминокислотную последовательность, полученную из Fc-области антитела IgG-типа путем замены по меньшей мере одной аминокислоты, выбранной из аминокислоты, находящейся в следующих положениях согласно EU-нумерации: 237, 238, 239, 248, 250, 252, 254, 255, 256, 257, 258, 265, 270, 286, 289, 297, 298, 303, 305, 307, 308, 309, 311, 312, 314, 315, 317, 325, 332, 334, 360, 376, 380, 382, 384, 385, 386, 387, 389, 424, 428, 433, 434 и 436, на другую аминокислоту.
Более конкретно, вариант Fc-области содержит по меньшей мере одну аминокислотную замену, выбранную из следующих замен:
аминокислотная замена для замены Gly в положении 237 на Met,
аминокислотная замена для замены Pro в положении 238 на Ala,
аминокислотная замена для замены Ser в положении 239 на Lys,
аминокислотная замена для замены Lys в положении 248 на Ile,
аминокислотная замена для замены Thr в положении 250 на Ala, Phe, Ile, Met, Gln, Ser, Val, Trp или Tyr,
аминокислотная замена для замены Met в положении 252 на Phe, Trp или Tyr,
аминокислотная замена для замены Ser в положении 254 на Thr,
аминокислотная замена для замены Arg в положении 255 на Glu,
аминокислотная замена для замены Thr в положении 256 на Asp, Glu или Gln,
аминокислотная замена для замены Pro в положении 257 на Ala, Gly, Ile, Leu, Met, Asn, Ser, Thr или Val,
аминокислотная замена для замены Glu в положении 258 на His,
аминокислотная замена для замены Asp в положении 265 на Ala,
аминокислотная замена для замены Asp в положении 270 на Phe,
аминокислотная замена для замены Asn в положении 286 на Ala или Glu,
аминокислотная замена для замены Thr в положении 289 на His,
аминокислотная замена для замены Asn в положении 297 на Ala,
аминокислотная замена для замены Ser в положении 298 на Gly,
аминокислотная замена для замены Val в положении 303 на Ala,
аминокислотная замена для замены Val в положении 305 на Ala,
аминокислотная замена для замены Thr в положении 307 на Ala, Asp, Phe,
Gly, His, Ile, Lys, Leu, Met, Asn, Pro, Gln, Arg, Ser, Val, Trp или Tyr,
аминокислотная замена для замены Val в положении 308 на Ala, Phe, Ile, Leu, Met, Pro, Gln или Thr,
аминокислотная замена для замены Leu или Val в положении 309 на Ala, Asp, Glu, Pro или Arg,
аминокислотная замена для замены Gln в положении 311 на Ala, His или Ile,
аминокислотная замена для замены Asp в положении 312 на Ala или His,
аминокислотная замена для замены Leu в положении 314 на Lys или Arg,
аминокислотная замена для замены Asn в положении 315 на Ala или His,
аминокислотная замена для замены Lys в положении 317 на Ala,
аминокислотная замена для замены Asn в положении 325 на Gly,
аминокислотная замена для замены Не в положении 332 на Val,
аминокислотная замена для замены Lys в положении 334 на Leu,
аминокислотная замена для замены Lys в положении 360 на His,
аминокислотная замена для замены Asp в положении 376 на Ala,
аминокислотная замена для замены Glu в положении 380 на Ala,
аминокислотная замена для замены Glu в положении 382 на Ala,
аминокислотная замена для замены Asn или Ser в положении 384 на Ala,
аминокислотная замена для замены Gly в положении 385 на Asp или His,
аминокислотная замена для замены Gln в положении 386 на Pro,
аминокислотная замена для замены Pro в положении 387 на Glu,
аминокислотная замена для замены Asn в положении 389 на Ala или Ser,
аминокислотная замена для замены Ser в положении 424 на Ala,
аминокислотная замена для замены Met в положении 428 на Ala, Asp, Phe, Gly, His, Ile, Lys, Leu, Asn, Pro, Gln, Ser, Thr, Val, Trp или Tyr,
аминокислотная замена для замены His в положении 433 на Lys,
аминокислотная замена для замены Asn в положении 434 на Ala, Phe, His, Ser, Trp или Tyr и
аминокислотная замена для замены Tyr или Phe в положении 436 на His (все согласно EU-нумерации) в Fc-области антитела IgG-типа.
С другой точки зрения, можно применять Fc-область, содержащую по меньшей мере одну аминокислоту, выбранную из
Met в качестве аминокислоты в положении 237,
Ala в качестве аминокислоты в положении 238,
Lys в качестве аминокислоты в положении 239,
Ile в качестве аминокислоты в положении 248,
Ala, Phe, Ile, Met, Gln, Ser, Val, Trp или Tyr в качестве аминокислоты в положении 250,
Phe, Trp или Tyr в качестве аминокислоты в положении 252,
Thr в качестве аминокислоты в положении 254,
Glu в качестве аминокислоты в положении 255,
Asp, Glu или Gln в качестве аминокислоты в положении 256,
Ala, Gly, Ile, Leu, Met, Asn, Ser, Thr или Val в качестве аминокислоты в положении 257,
His в качестве аминокислоты в положении 258,
Ala в качестве аминокислоты в положении 265,
Phe в качестве аминокислоты в положении 270,
Ala или Glu в качестве аминокислоты в положении 286,
His в качестве аминокислоты в положении 289,
Ala в качестве аминокислоты в положении 297,
Gly в качестве аминокислоты в положении 298,
Ala в качестве аминокислоты в положении 303,
Ala в качестве аминокислоты в положении 305,
Ala, Asp, Phe, Gly, His, Ile, Lys, Leu, Met, Asn, Pro, Gln, Arg, Ser, Val, Trp или Tyr в качестве аминокислоты в положении 307,
Ala, Phe, Ile, Leu, Met, Pro, Gln или Thr в качестве аминокислоты в положении 308,
Ala, Asp, Glu, Pro или Arg в качестве аминокислоты в положении 309,
Ala, His или Ile в качестве аминокислоты в положении 311,
Ala или His в качестве аминокислоты в положении 312,
Lys или Arg в качестве аминокислоты в положении 314,
Ala или His в качестве аминокислоты в положении 315,
Ala в качестве аминокислоты в положении 317,
Gly в качестве аминокислоты в положении 325,
Val в качестве аминокислоты в положении 332,
Leu в качестве аминокислоты в положении 334,
His в качестве аминокислоты в положении 360,
Ala в качестве аминокислоты в положении 376,
Ala в качестве аминокислоты в положении 380,
Ala в качестве аминокислоты в положении 382,
Ala в качестве аминокислоты в положении 384,
Asp или His в качестве аминокислоты в положении 385,
Pro в качестве аминокислоты в положении 386,
Glu в качестве аминокислоты в положении 387,
Ala или Ser в качестве аминокислоты в положении 389,
Ala в качестве аминокислоты в положении 424,
Ala, Asp, Phe, Gly, His, Ile, Lys, Leu, Asn, Pro, Gin, Ser, Thr, Val, Trp или Tyr в качестве аминокислоты в положении 428,
Lys в качестве аминокислоты в положении 433,
Ala, Phe, His, Ser, Trp или Tyr в качестве аминокислоты в положении 434 и His в качестве аминокислоты в положении 436 (все согласно EU-нумерации),
в Fc-области антитела IgG-типа.
Тот факт, что транспортирующий фрагмент обладает FcRn-связывающей активностью, не означает, что антигенсвязывающий домен не обладает FcRn-связывающей активностью. В вариантах осуществления изобретения, в которых транспортирующий фрагмент имеет более продолжительное время полужизни в крови, чем антигенсвязывающий домен, антигенсвязывающий домен, как должно быть очевидно, может не обладать FcRn-связывающей активностью, или антигенсвязывающий домен может обладать FcRn-связывающей активностью, если FcRn-связывающая активность слабее, чем активность транспортирующего фрагмента.
В одном из вариантов осуществления изобретения метод удлинения времени полужизни в крови транспортирующего фрагмента включает связывание транспортирующего фрагмента с альбумином. Поскольку альбумин не поддается экскреции почками и обладает FcRn-связывающей способностью, его время полужизни в крови составляет от 17 дней до 19 дней (J Clin Invest. 32 (8), август 1953 г., сс. 746-768). Так, установлено, что белок, связанный с альбумином, становится более объемным и обладает способностью опосредованно связываться с FcRn и поэтому имеет удлиненное время полужизни в крови (Antibodies 4 (3), 2015, сс. 141-156.
В одном из вариантов осуществления изобретения альтернативный метод удлинения времени полужизни в крови транспортирующего фрагмента включает ПЭГилирование транспортирующего фрагмента. Считается, что ПЭГилирование белка придает белку объем и подавляет также его расщепление протеазой в крови, тем самым удлиняя время полужизни белка в крови (J Pharm Sci. 97 (10), октябрь 2008 г., сс. 4167-4183).
В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент содержит Fc-область антитела. В конкретном варианте осуществления изобретения транспортирующий фрагмент содержит СН2-домен и СН3-домен человеческого антитела IgG-типа. В конкретном варианте осуществления изобретения транспортирующий фрагмент содержит фрагмент, простирающийся от Cys226 или Pro230 тяжелой цепи человеческого антитела IgG1-изотипа до карбоксильного конца тяжелой цепи. Однако C-концевой лизин (Lys447) или глицин-лизин (Gly446-Lys447) Fc-области может присутствовать или может отсутствовать.
В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент содержит константную область антитела. В более предпочтительном варианте осуществления изобретения транспортирующий фрагмент содержит константную область антитела IgG-типа. В другом предпочтительном варианте осуществления изобретения транспортирующий фрагмент содержит константную область человеческого антитела IgG-типа.
В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент содержит: область, практически сходную по структуре с константной областью тяжелой цепи антитела; и область, практически сходную по структуре с легкой цепью антитела, соединенную с областью через ковалентную связь, такую как дисульфидная связь, или нековалентную связь, такую как водородная связь или гидрофобное взаимодействие.
В настоящем описании «полипептид, содержащий антигенсвязывающий домен и транспортирующий фрагмент», как правило, представляет собой серию полипептидов, соединенных через амидную(ые) связь(и), или белок, содержащий множество полипептидов, соединенных через амидную(ые) связи.
В некоторых вариантах осуществления настоящего изобретения антигенсвязывающий домен обладает способностью высвобождаться из полипептида, и антигенсвязывающий домен, высвободившийся из полипептида, имеет более высокую антигенсвязывающую активность. В настоящем описании понятие «высвобождается» относится к взаимному разделению двух фрагментов полипептида. Высвобождение антигенсвязывающего домена из полипептида может быть связано с упразднением взаимодействия между антигенсвязывающий доменом и транспортирующим фрагментом. Антигенсвязывающая активность антигенсвязывающего домена, включенного в полипептид, ингибируется. Следовательно, тот факт, что антигенсвязывающий домен высвободился из полипептида, можно подтверждать путем измерения антигенсвязывающей активности в организме индивидуума и сравнения ее с антигенсвязывающей активностью антигенсвязывающего домена, включенного в полипептид.
В некоторых вариантах осуществления изобретения полипептид содержит сайт расщепления, и сайт расщепления расщепляется так, что
антигенсвязывающий домен высвобождается из полипептида. Сайт расщепления можно расщеплять, например, ферментом, можно восстанавливать восстановителем или можно подвергать фоторазложению. Сайт расщепления можно помещать в любое положение в полипептиде, если антигенсвязывающий домен может высвобождаться и не утрачивает свою антигенсвязывающую активность после высвобождения. Полипептид может содержать также дополнительный сайт расщепления, отличный от сайт расщепления, предназначенного для высвобождения антигенсвязывающего домена. В одном из вариантов осуществления настоящего изобретения сайт расщепления содержит расщепляемую протеазой последовательность и может расщепляться протеазой(амии).
В настоящем описании понятие «расщепленные» относится к состоянию, в котором антигенсвязывающий домен и транспортирующий фрагмент отделены друг от друга после изменения сайта расщепления протеазой, восстановления цистеин-цистеиновой дисульфидной связи в сайте расщепления и/или фотоактивации. В настоящем описании понятие «нерасщепленный» относится к состоянию антигенсвязывающего домена, в котором он сцеплен с транспортирующим фрагментом в отсутствии расщепления протеазой сайта расщепления, в отсутствии восстановления цистеин-цистеиновой дисульфидной связи в сайте расщепления и/или в отсутствии света.
Расщепление сайта расщепления можно определять, подвергая раствор, содержащий полипептид, имеющий сайт расщепления, ДСН-ПААГ (электрофорез в полиакриламидном геле) и измеряя молекулярные массы фрагментов или определяя изменения в молекулярной массе до и после расщепления.
Сайт расщепления может быть специфически модифицирован (расщеплен, восстановлен или расщеплен в результате воздействия света) агентом (т.е. протеазой, восстановителем или светом) со скоростью, составляющей примерно от 0,001 до 1500×104 M-1c-1, или по меньшей мере 0,001, 0,005, 0,01, 0,05, 0,1, 0,5, 1, 2,5, 5, 7,5, 10, 15, 20, 25, 50, 75, 100, 125, 150, 200, 250, 500, 750, 1000, 1250 или 1500×104 M-1c-1.
Специфическое расщепление протеазой осуществляют путем контакта между протеазой и сайтом расщепления или молекулой, содержащей сайт расщепления. Сайт расщепления может расщепляться в присутствии достаточной ферментативной активности. Достаточная ферментативная активность может означать способность фермента осуществлять расщепление при контакте с сайтом расщепления.
В настоящем описании понятие «протеаза» относится к ферменту, такому как эндопептидаза или экзопептидаза, который гидролизует пептидную связь, как правило, эндопептидаза. Протеаза, применяемая в настоящем изобретении, ограничена только способностью расщеплять последовательность, расщепляемую протеазой, и практически не ограничена ее типом. В некоторых вариантах осуществления изобретения применяют специфическую для ткани-мишени протеазу. Специфическая для ткани-мишени протеаза может представлять собой, например, любую из следующих протеаз:
(1) протеазу, которая отличается более высоким уровнем экспрессии в ткани-мишени, чем в здоровой ткани,
(2) протеазу, которая обладает более высокой активностью в ткани-мишени, чем в здоровой ткани,
(3) протеазу, которая отличается более высоким уровнем экспрессии в клетках-мишенях, чем в здоровой клетке, и
(4) протеазу, которая обладает более высокой активностью в клетках-мишенях, чем в здоровой клетке.
В более конкретном варианте осуществления изобретения применяют специфическую для раковой ткани протеазу или специфическую для воспалительной ткани протеазу.
В настоящем описании понятие «ткань-мишень» относится к ткани, содержащей по меньшей мере одну клетку-мишень. В некоторых вариантах осуществления настоящего изобретения ткань-мишень представляет собой раковую ткань. В некоторых вариантах осуществления настоящего изобретения ткань-мишень представляет собой воспалительную ткань.
Понятие «раковая ткань» относится к ткани, содержащей по меньшей мере одну раковую клетку. Таким образом, с учетом того, что, например, раковая ткань содержит раковые клетки и кровеносные сосуды, каждый тип клеток, который принимает участие в формировании опухолевой массы, содержащей раковые клетки и эндотелиальные клетки, подпадает под объем настоящего изобретения. В настоящем описании опухолевая масса относится к очагу опухолевой ткани. Понятие «опухоль», как правило, применяют для обозначения доброкачественной неоплазмы или злокачественной неоплазмы.
В настоящем описании примеры «воспалительной ткани» включают:
суставную ткань при ревматоидном артрите или остеоартрите,
легкое (альвеолы) при бронхиальной астме или COPD,
ткань органа пищеварительной системы при воспалительном заболевании кишечника, болезни Крона или неспецифическом язвенном колите,
фиброзную ткань при фиброзе печени, почки или легкого,
ткань, образующуюся при отторжении трансплантата органа,
кровеносный сосуд или сердце (сердечная мышца) при артериосклерозе или сердечной недостаточности,
висцеральную жировую ткань при метаболическом синдроме,
кожную ткань при атопическом дерматите и других дерматидах, и
спинномозговой нерв при грыже диска или хроническом люмбаго.
В частности, специфически экспрессируемая или специфически активируемая протеаза или протеаза, которая рассматривается в качестве связанной с болезненным состоянием ткани-мишени (специфическая для ткани-мишени протеаза), известна для некоторых типов тканей-мишеней. Например, в WO 2013/128194, WO 2010/081173 и WO 2009/025846 описана протеаза, специфически экспрессируемая в раковой ткани. Кроме того, в J Inflamm (Lond). 7, 2010, с. 45, Nat Rev Immunol. 6 (7), июль 2006 г., cc. 541-550, Nat Rev Drug Discov. 13 (12), декабрь 2014 г., cc. 904-927, Respir Res. 17, 4 марта 2016 г., с. 23, Dis Model Mech. 7 (2), февраль 2014 г., cc. 193-203 и Biochim Biophys Acta. 1824 (1), январь 2012 г., cc. 133-145 описана протеаза, которая, как считается, связана с воспалением.
Помимо протеазы, специфически экспрессируемой в ткани-мишени, существует также протеаза, специфически активируемая в ткани-мишени. Например, протеаза может экспрессироваться в неактивной форме и затем превращаться в активную форму. Многие ткани содержат субстанцию, ингибирующую активную протеазу и контролирующую активность в результате процесса активации и присутствия ингибитора (Nat Rev Cancer. 3 (7), июль 2003 г., cc. 489-501). В ткани-мишени активная протеаза может специфически активироваться, избегая ингибирования.
Активную протеазу можно оценивать путем применения метода с использованием антитела, распознающего активную протеазу (PNAS 110 (1), 2 января 2013 г., сс. 93-98), или метода с использованием флуоресцентно меченного пептида, распознаваемого протеазой, флуоресцентность которого гасится перед расщеплением, но испускается после расщепления (Nat Rev Drug Discov. 9 (9), сентябрь 2010, сс. 690-701. doi: 10.1038/nrd3053).
Согласно одной из точек зрения понятие «специфическая для ткани-мишени протеаза» может относиться к любой из следующих протеаз:
(I) протеаза, которая отличается более высоким уровнем экспрессии в ткани-мишени, чем в здоровой ткани,
(II) протеаза, которая обладает более высокой активностью в ткани-мишени, чем в здоровой ткани,
(III) протеаза, которая отличается более высоким уровнем экспрессии в клетке-мишени, чем в здоровой клетке, и
(IV) протеаза, которая обладает более высокой активностью в клетке-мишени, чем в здоровой клетке.
Конкретные примеры протеазы включают (но, не ограничиваясь только ими) цистеиновую протеазу (включая семейства катепсина В, L, S и т.д.), аспартилпротеазу (катепсины D, Е, K, О и т.д.), сериновую протеазу (включая матриптазу (в том числе MT-SP1), катепсины А и G, тромбин, плазмин, урокиназу (uPA), тканевый активатор плазминогена (tPA), эластазу, протеиназу 3, тромбин, калликреин, триптазу и химазу), металлопротеиназы (металлопротеиназы (ММР 1-28), включая как связанные с мембраной формы (ММР 14-17 и ММР 24-25), так и секретируемые формы (ММР 1-13, ММР 18-23 и ММР 26-28), дизинтегрин А и металлопротеиназы (ADAM), дизинтегрин А и металлопротеиназу с мотивами тромбоспондина (ADAMTS), меприн (меприн альфа и меприн бета), CD10 (CALLA), простатический антиген (PSA), легумаин, TMPRSS3, TMPRSS4, эластазу нейтрофилов (HNE), бета секретазу (ВАСЕ), фибробласт-активирующий белок альфа (FAP), гранзим В, гуанидинобензоатазу (GB), гепсин, неприлизин, NS3/4A, HCV-NS3/4, калпаин, ADAMDEC1, ренин, катепсин С, катепсин V/L2, катепсин X/Z/P, крузипаин, отубаин 2, родственные калликреину пептидазы (KLK) (KLK3, KLK4, KLK5, KLK6, KLK7, KLK8, KLK10, KLK11, KLK13 и KLK14)), белок, участвующий в остеогенезе 1 (ВМР-1), активированный белок С, связанные с коагуляцией крови протеазы (фактор VIIa, фактор IXa, фактор Ха, фактор XIa и фактор XIIa), HtrA1, лактоферрин, марапсин, РАСЕ4, DESC1, дипептидилпептидазу 4 (DPP-4), TMPRSS2, катепсин F, катепсин H, катепсин L2, катепсин О, катепсин S, гранзим А, гепсин, калпаин 2, глутаматкарбоксипептидазу 2, AMSH-подобные протеазы, AMSH, гамма секретазу, антиплазминрасщепляющий фермент (АРСЕ), децисин 1, фермент, подобный N-ацетилированной альфа-связанной кислой дипептидазе 1 (NAALADL1), и фурин.
Согласно другой точке зрения понятие «специфическая для ткани-мишени протеаза» может относиться к специфической для раковой ткани протеазе или специфической для воспалительной ткани протеазе.
Примеры специфической для раковой ткани протеазы включают протеазу, которая специфически экспрессируется в раковой ткани, описанную в WO 2013/128194, WO 2010/081173 и WO 2009/025846.
Касательно типа специфической для раковой ткани протеазы обработка протеазой, которая обладает более высокой специфичностью экспрессии в раковой ткани, является более эффективной для снижения побочных действий. Предпочтительная специфическая для раковой ткани протеаза имеет концентрацию в раковой ткани, превышающую в 5 раз или более, более предпочтительно в 10 раз или более, еще более предпочтительно в 500 раз или более, наиболее предпочтительно в 1000 раз или более, ее концентрацию в здоровой ткани. Кроме того, предпочтительная специфическая для раковой ткани протеаза имеет активность в раковой ткани, превышающую в 2 раза или более, более предпочтительно в 3 раза или более, в 4 раза или более, в 5 раз или более или в 10 раз или более, еще более предпочтительно в 100 раз или более, особенно предпочтительно в 500 раз или более, наиболее предпочтительно в 1000 раз или более ее активность в здоровых тканях.
Специфическая для раковой ткани протеаза может находиться в форме, связанной с мембраной раковой клетки, или может находиться в секретируемой внеклеточной форме, не связанной с клеточной мембраной. Когда специфическая для раковой ткани протеаза не связана с мембраной раковой клетки, то предпочтительной для опосредуемой иммуноцитами цитотоксичности является специфичность в отношении раковых клеток, поскольку специфическая для раковой ткани протеаза должна присутствовать в раковой ткани или в непосредственной близости от нее. В настоящем описании понятие «в непосредственной близости от раковой ткани» означает местоположение, в котором расщепляемая протеазой последовательность, специфическая для раковой ткани, расщепляется таким образом, чтобы антигенсвязывающий домен проявлял антигенсвязывающую активность. Однако предпочтительно, чтобы повреждение здоровых клеток в данном местоположении было минимизировано.
Согласно альтернативной точки зрения специфическая для раковой ткани протеаза представляет собой любую из следующих протеаз:
(I) протеазу, которая отличается более высоким уровнем экспрессии в раковой ткани, чем в здоровой ткани,
(II) протеазу, которая обладает более высокой активностью в раковой ткани, чем в здоровых ткани,
(III) протеазу, которая отличается более высоким уровнем экспрессии в раковой клетке, чем в здоровой клетке, и
(IV) протеазу, которая обладает более высокой активностью в раковой клетке, чем в здоровой клетке.
Один тип специфической для раковой ткани протеазы можно применять индивидуально или можно объединять два или большее количество типов специфических для раковой ткани протеаз. Количество типов специфической для раковой ткани протеазы могут определять соответственно специалисты в данной области с учетом типа рака, подлежащего лечению.
Исходя из указанных точек зрения, специфическая для раковой ткани протеаза предпочтительно представляет собой сериновые протеазы или металлопротеиназы, более предпочтительно матриптазы (включая MT-SP1), урокиназу (uPA) или металлопротеиназы, предпочтительно также MT-SP1, uPA, ММР-2 или ММР-9, из перечисленных выше протеаз.
Касательно типа специфической для воспалительной ткани протеазы обработка протеазой, которая обладает более высокой специфичностью экспрессии в воспалительной ткани, является более эффективной для снижения побочных действий. Предпочтительная специфическая для воспалительной ткани протеаза имеет концентрацию в воспалительной ткани, превышающую в 5 раз или более, более предпочтительно в 10 раз или более, еще более предпочтительно в 500 раз или более, наиболее предпочтительно в 1000 раз или более, ее концентрацию в здоровой ткани. Кроме того, предпочтительная специфическая для воспалительной ткани протеаза имеет активность в воспалительной ткани, превышающую в 2 раза или более, более предпочтительно в 3 раза или более, в 4 раза или более, в 5 раз или более или в 10 раз или более, еще более предпочтительно в 100 раз или более, особенно предпочтительно в 500 раз или более, наиболее предпочтительно в 1000 раз или более ее активность в здоровых тканях.
Специфическая для воспалительной ткани протеаза может находиться в форме, связанной с мембраной воспалительной клетки, или может находиться в секретируемой внеклеточной форме, не связанной с клеточной мембраной. Когда специфическая для воспалительной ткани протеаза не связана с мембраной воспалительной клетки, то предпочтительной для опосредуемой иммуноцитами цитотоксичности является специфичность в отношении воспалительных клеток, поскольку специфическая для воспалительной ткани протеаза должна присутствовать в воспалительной ткани или в непосредственной близости от нее. В настоящем описании понятие «в непосредственной близости от воспалительной ткани» означает местоположение, в котором расщепляемая протеазой последовательность, специфическая для воспалительной ткани, расщепляется таким образом, чтобы антигенсвязывающий домен проявлял антигенсвязывающую активность. Однако предпочтительно, чтобы повреждение здоровых клеток в данном местоположении было минимизировано.
Согласно альтернативной точки зрения специфическая для воспалительной ткани протеаза представляет собой любую из следующих протеаз:
(I) протеазу, которая отличается более высоким уровнем экспрессии в воспалительной ткани, чем в здоровой ткани,
(II) протеазу, которая обладает более высокой активностью в воспалительной ткани, чем в здоровых ткани,
(III) протеазу, которая отличается более высоким уровнем экспрессии в воспалительной клетке, чем в здоровой клетке, и
(IV) протеазу, которая обладает более высокой активностью в воспалительной клетке, чем в здоровой клетке.
Один тип специфической для воспалительной ткани протеазы можно применять индивидуально или можно объединять два или большее количество типов специфических для воспалительной ткани протеаз. Количество типов специфической для воспалительной ткани протеазы могут определять соответственно специалисты в данной области с учетом патологического состояния, подлежащего лечению.
Исходя из указанных точек зрения, специфическая для воспалительной ткани протеаза предпочтительно из перечисленных выше протеаз представляет собой металлопротеиназу. Металлопротеиназа более предпочтительно представляет собой ADAMTS5, ММР-2, ММР-7, ММР-9 или ММР-13.
Расщепляемая протеазой последовательность представляет собой конкретную аминокислотную последовательность, которая специфически распознается специфической для ткани-мишени протеазой, когда полипептид гидролизуется специфической для ткани-мишени протеазой в водном растворе.
Расщепляемая протеазой последовательность предпочтительно представляет собой аминокислотную последовательность, которая гидролизуется с высокой специфичностью протеазой, специфической для ткани-мишени, более специфически экспресируемой в ткани-мишени или клетках-мишенях, подлежащих обработке, или более специфически активируемой в ткани/клетках-мишенях, подлежащих обработке, с точки зрения снижения побочных действий.
Конкретные примеры расщепляемой протеазой последовательности включают последовательности-мишени, которые специфически гидролизуются с помощью указанной выше протеазы, специфически экспрессируемой в раковой ткани, которые описаны в WO 2013/128194, WO 2010/081173 и WO 2009/025846, специфической для воспалительной ткани протеазой и т.п. Можно применять также последовательность, искусственно измененную, например, путем соответствующей интродукции аминокислотной мутации в последовательность-мишень, которая специфически гидролизуется известной протеазой. Альтернативно этому, можно применять расщепляемую протеазой последовательность, идентифицированную с помощью метода, известного специалистам в данной области, который описан в Nature Biotechnology 19, 2001, сс. 661-667.
Кроме того, можно применять встречающуюся в естественных условиях расщепляемую протеазой последовательность. Например, TGFβ превращают в латентную форму путем расщепления протеазой. Аналогично этому, можно применять расщепляемую протеазой последовательность в белке, который изменяет молекулярную форму при расщеплении протеазой.
Примеры расщепляемой протеазой последовательности, которую можно применять, включают (но, не ограничиваясь только ими) последовательности, описанные в WO 2015/116933, WO 2015/048329, WO 2016/118629, WO 2016/179257, WO 2016/179285, WO 2016/179335, WO 2016/179003, WO 2016/046778, WO 2016/014974, в публикациях патентов США US 2016/0289324, US 2016/0311903, в PNAS 97, 2000, сс. 7754-7759, Biochemical Journal 426, 2010, сс. 219-228 и Beilstein J Nanotechnol. 7, 2016, сс. 364-373.
Расщепляемая протеазой последовательность более предпочтительно представляет собой аминокислотную последовательность, которая специфически гидролизуется с помощью приемлемой для ткани-мишени специфической протеазы, упомянутой выше. Аминокислотная последовательность, которая специфически гидролизуется с помощью специфической для ткани-мишени протеазы, предпочтительно представляет собой любую из следующих аминокислотных последовательностей:
LSGRSDNH (SEQ ID NO: 12, расщепляемая MT-SP1 или uPA),
PLGLAG (SEQ ID NO: 25, расщепляемая ММР-2 или ММР-9) и
VPLSLTMG (SEQ ID NO: 26, расщепляемая ММР-7).
Любую из приведенных ниже последовательностей можно применять также в качестве расщепляемой протеазой последовательности:
TSTSGRSANPRG (SEQ ID NO: 74, расщепляемая MT-SP1 или uPA),
ISSGLLSGRSDNH (SEQ ID NO: 75, расщепляемая MT-SP1 или uPA),
AVGLLAPPGGLSGRSDNH (SEQ ID NO: 76, расщепляемая MT-SP1 или uPA),
GAGVPMSMRGGAG (SEQ ID NO: 77, расщепляемая MMP-1),
GAGIPVSLRSGAG (SEQ ID NO: 78, расщепляемая MMP-2),
GPLGIAGQ (SEQ ID NO: 79, расщепляемая MMP-2),
GGPLGMLSQS (SEQ ID NO: 80, расщепляемая MMP-2),
PLGLWA (SEQ ID NO: 81, расщепляемая MMP-2),
GAGRPFSMIMGAG (SEQ ID NO: 82, расщепляемая MMP-3),
GAGVPLSLTMGAG (SEQ ID NO: 83, расщепляемая MMP-7),
GAGVPLSLYSGAG (SEQ ID NO: 84, расщепляемая MMP-9),
AANLRN (SEQ ID NO: 85, расщепляемая MMP-11),
AQAYVK (SEQ ID NO: 86, расщепляемая MMP-11),
AANYMR (SEQ ID NO: 87, расщепляемая MMP-11),
AAALTR (SEQ ID NO: 88, расщепляемая MMP-11),
AQNLMR (SEQ ID NO: 89, расщепляемая MMP-11),
AANYTK (SEQ ID NO: 90, расщепляемая MMP-11),
GAGPQGLAGQRGIVAG (SEQ ID NO: 91, расщепляемая MMP-13),
PRFKIIGG (SEQ ID NO: 92, расщепляемая проурокиназой),
PRFRIIGG (SEQ ID NO: 93, расщепляемая проурокиназой),
GAGSGRSAG (SEQ ID NO: 94, расщепляемая uPA),
SGRSA (SEQ ID NO: 95, расщепляемая uPA),
GSGRSA (SEQ ID NO: 96, расщепляемая uPA),
SGKSA (SEQ ID NO: 97, расщепляемая uPA),
SGRSS (SEQ ID NO: 98, расщепляемая uPA),
SGRRA (SEQ ID NO: 99, расщепляемая uPA),
SGRNA (SEQ ID NO: 100, расщепляемая uPA),
SGRKA (SEQ ID NO: 101, расщепляемая uPA),
QRGRSA (SEQ ID NO: 102, расщепляемая tPA),
GAGSLLKSRMVPNFNAG (SEQ ID NO: 103, расщепляемая катепсином В)
TQGAAA (SEQ ID NO: 104, расщепляемая катепсином В),
GAAAAA (SEQ ID NO: 105, расщепляемая катепсином В),
GAGAAG (SEQ ID NO: 106, расщепляемая катепсином В),
AAAAAG (SEQ ID NO: 107, расщепляемая катепсином В),
LCGAAI (SEQ ID NO: 108, расщепляемая катепсином В),
FAQALG (SEQ ID NO: 109, расщепляемая катепсином В),
LLQANP (SEQ ID NO: 110, расщепляемая катепсином В),
LAAANP (SEQ ID NO: 111, расщепляемая катепсином В),
LYGAQF (SEQ ID NO: 112, расщепляемая катепсином В),
LSQAQG (SEQ ID NO: 113, расщепляемая катепсином В),
ASAASG (SEQ ID NO: 114, расщепляемая катепсином В),
FLGASL (SEQ ID NO: 115, расщепляемая катепсином В),
AYGATG (SEQ ID NO: 116, расщепляемая катепсином В),
LAQATG (SEQ ID NO: 117, расщепляемая катепсином В),
GAGSGVVIATVIVITAG (SEQ ID NO: 118, расщепляемая катепсином L),
APMAEGGG (SEQ ID NO: 119, расщепляемая меприном альфа или меприном бета),
EAQGDKII (SEQ ID NO: 120, расщепляемая меприном альфа или меприном бета),
LAFSDAGP (SEQ ID NO: 121, расщепляемая меприном альфа или меприном бета),
YVADAPK (SEQ ID NO: 122, расщепляемая меприном альфа или меприном бета),
RRRRR (SEQ ID NO: 123, расщепляемая фурином),
RRRRRR (SEQ ID NO: 124, расщепляемая фурином),
GQSSRHRRAL (SEQ ID NO: 125, расщепляемая фурином),
SSRHRRALD (SEQ ID NO: 126),
RKSSIIIRMRDVVL (SEQ ID NO: 127, расщепляемая плазминогеном),
SSSFDKGKYKKGDDA (SEQ ID NO: 128, расщепляемая стафилокиназой),
SSSFDKGKYKRGDDA (SEQ ID NO: 129, расщепляемая стафилокиназой),
IEGR (SEQ ID NO: 130, расщепляемая фактором Xa),
IDGR (SEQ ID NO: 131, расщепляемая фактором Xa),
GGSIDGR (SEQ ID NO: 132, расщепляемая фактором Xa),
GPQGIAGQ (SEQ ID NO: 133, расщепляемая коллагеназой),
GPQGLLGA (SEQ ID NO: 134, расщепляемая коллагеназой),
GIAGQ (SEQ ID NO: 135, расщепляемая коллагеназой),
GPLGIAG (SEQ ID NO: 136, расщепляемая коллагеназой),
GPEGLRVG (SEQ ID NO: 137, расщепляемая коллагеназой),
YGAGLGVV (SEQ ID NO: 138, расщепляемая коллагеназой),
AGLGVVER (SEQ ID NO: 139, расщепляемая коллагеназой),
AGLGISST (SEQ ID NO: 140, расщепляемая коллагеназой),
EPQALAMS (SEQ ID NO: 141, расщепляемая коллагеназой),
QALAMSAI (SEQ ID NO: 142, расщепляемая коллагеназой),
AAYHLVSQ (SEQ ID NO: 143, расщепляемая коллагеназой),
MDAFLESS (SEQ ID NO: 144, расщепляемая коллагеназой),
ESLPVVAV (SEQ ID NO: 145, расщепляемая коллагеназой),
SAPAVESE (SEQ ID NO: 146, расщепляемая коллагеназой),
DVAQFVLT (SEQ ID NO: 147, расщепляемая коллагеназой),
VAQFVLTE (SEQ ID NO: 148, расщепляемая коллагеназой),
AQFVLTEG (SEQ ID NO: 149, расщепляемая коллагеназой),
PVQPIGPQ (SEQ ID NO: 150, расщепляемая коллагеназой),
LVPRGS (SEQ ID NO: 151, расщепляемая тромбином),
TSGSGRSANARG (SEQ ID NO: 168, расщепляемая uPA и MT-SP1),
TSQSGRSANQRG (SEQ ID NO: 169, расщепляемая uPA и MT-SP1),
TSPSGRSAYPRG (SEQ ID NO: 170, расщепляемая uPA и MT-SP1),
TSGSGRSATPRG (SEQ ID NO: 171, расщепляемая uPA и MT-SP1),
TSQSGRSATPRG (SEQ ID NO: 172, расщепляемая uPA и MT-SP1),
TSASGRSATPRG (SEQ ID NO: 173, расщепляемая uPA и MT-SP1),
TSYSGRSAVPRG (SEQ ID NO: 174, расщепляемая uPA и MT-SP1),
TSYSGRSANFRG (SEQ ID NO: 175, расщепляемая uPA и MT-SP1),
TSSSGRSATPRG (SEQ ID NO: 176, расщепляемая uPA и MT-SP1),
TSTTGRSASPRG (SEQ ID NO: 177, расщепляемая uPA и MT-SP1) и
TSTSGRSANPRG (SEQ ID NO: 178, расщепляемая uPA и MT-SP1).
В качестве расщепляемых протеазой последовательностей можно применять также расщепляемые протеазой последовательности, представленные в таблице 1.
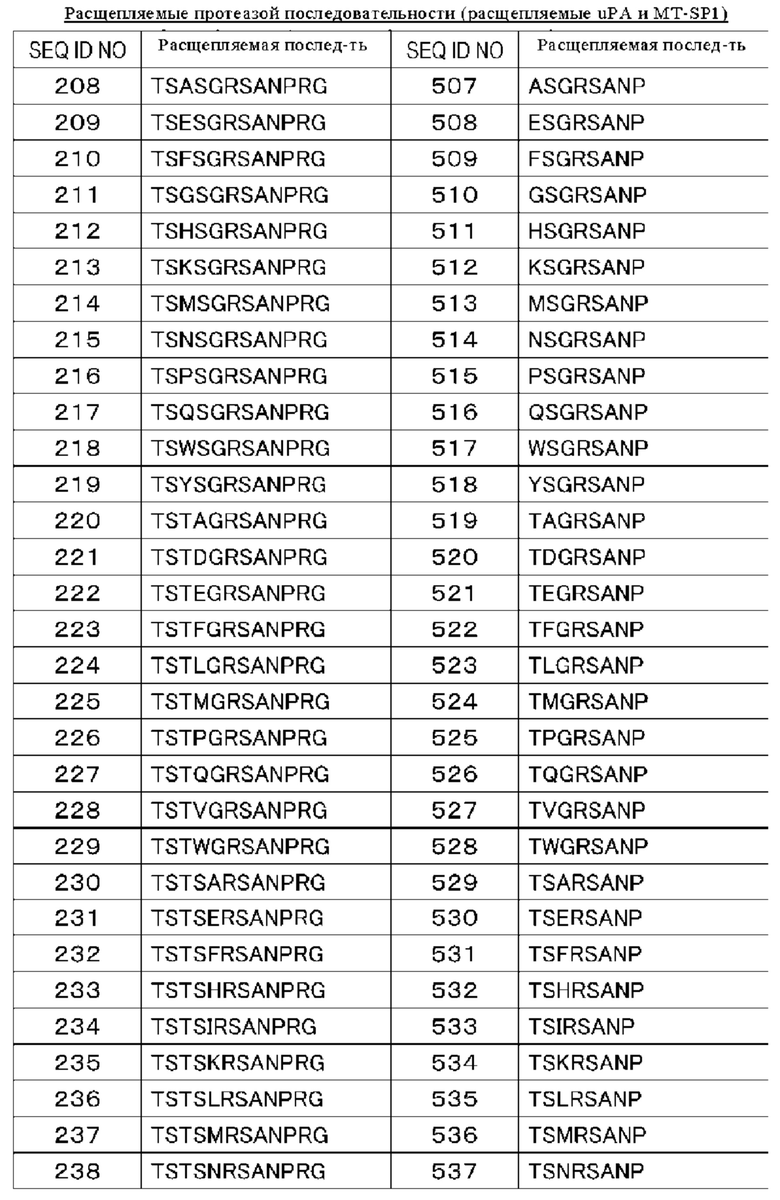
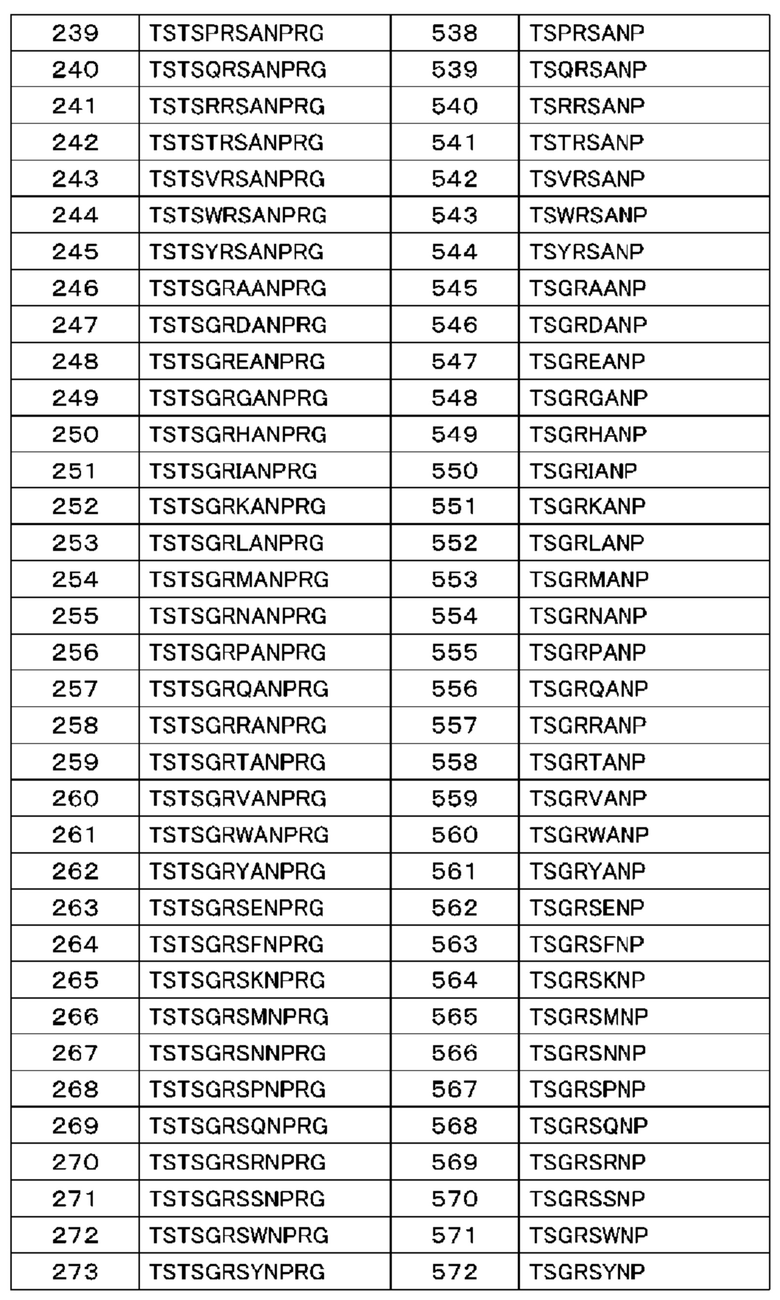
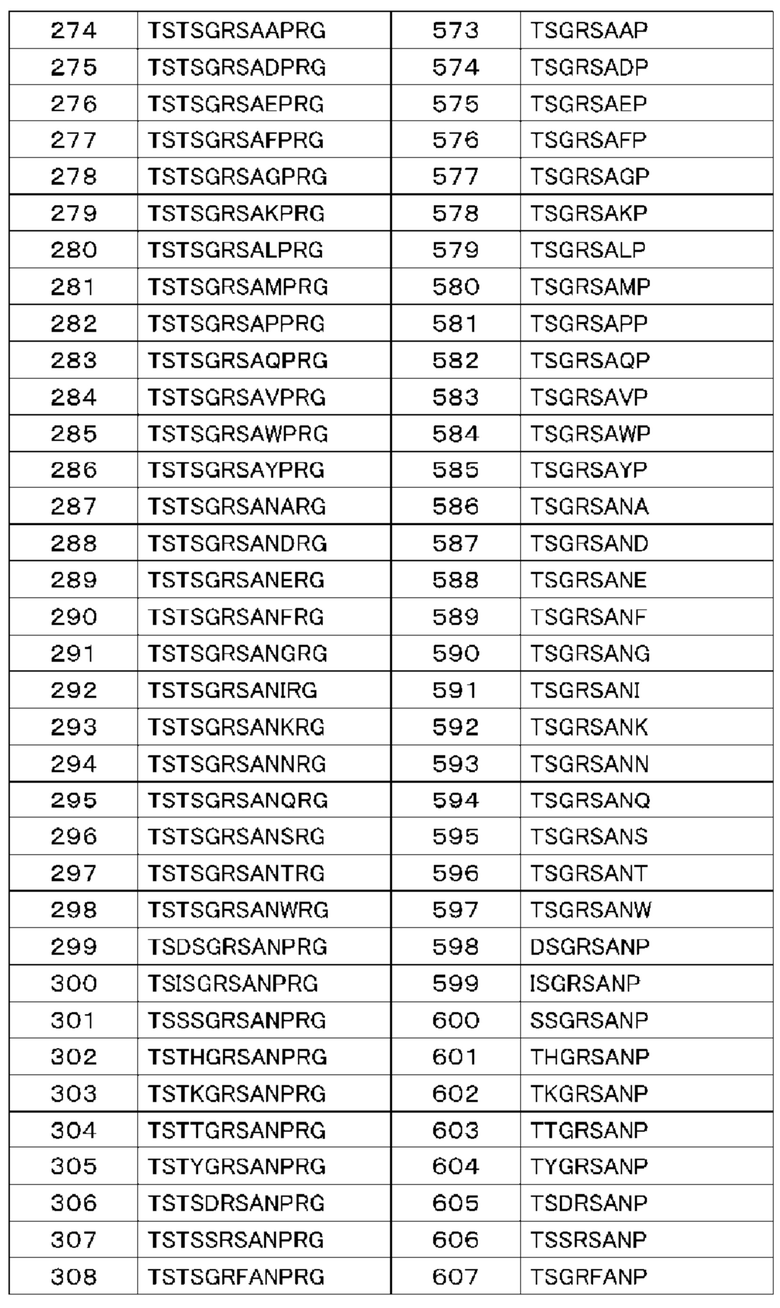
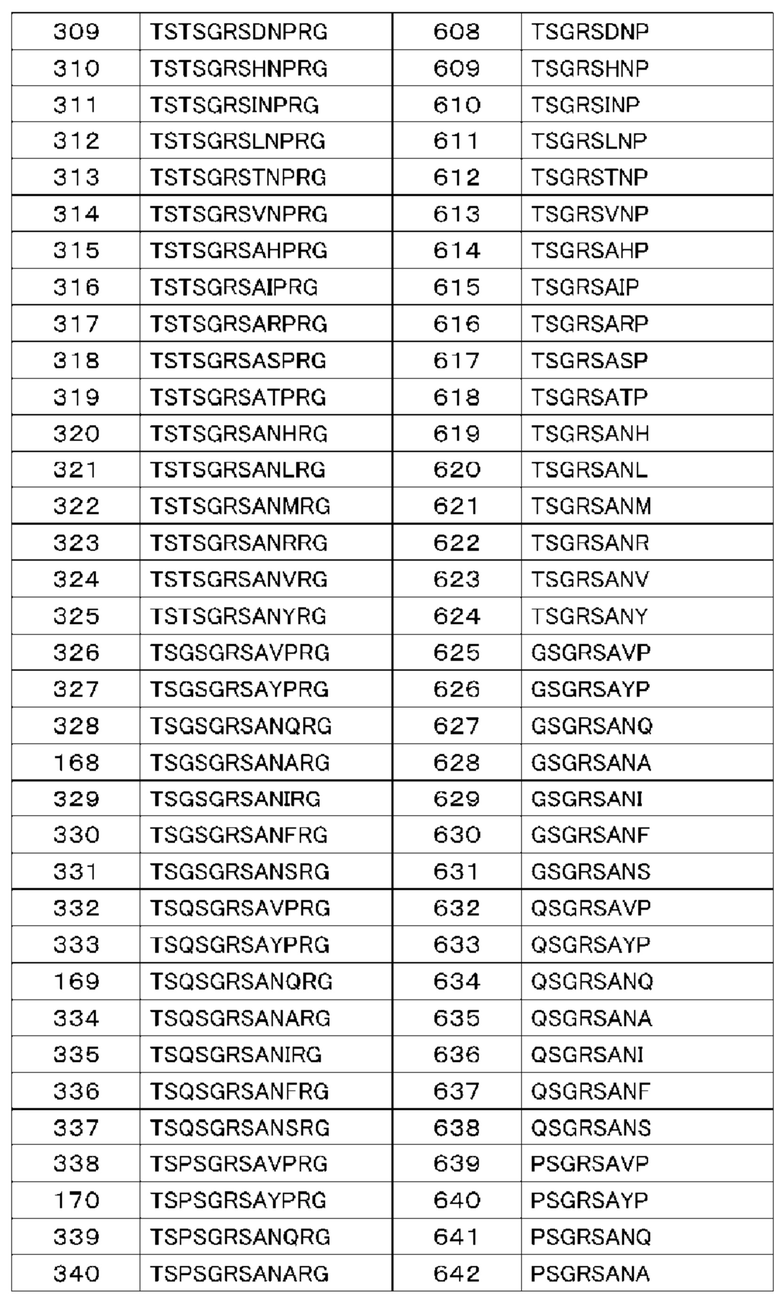

Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 833),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, К, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 834),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из А, Е, F, G, Н, K, М, N, Р, Q, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, К, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 835),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, F, L, М, Р, Q, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, T, V, W и Y; X4 обозначает R; X5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 836),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из А, Е, F, Н, I, K, L, М, N, Р, Q, R, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 837),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, G, Н, I, K, L, М, N, Q, R, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 838),
в которой Х1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из Е, F, K, М, N, Р, Q, R, S и W; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 839),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, F, G, L, М, Р, Q, V и W; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 840),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, I, K, N, Т и W.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 841),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, G, I, Р, Q, S и Y; Х2 обозначает аминокислоту, выбранную из K или Т; Х3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает А; Х7 обозначает аминокислоту, выбранную из Н, I и V; и Х8 обозначает аминокислоту, выбранную из Н, V и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 842),
в которой X1-Х8 каждый обозначает индивидуальную аминокислоту, X1 обозначает Y; Х2 обозначает аминокислоту, выбранную из S и Т; Х3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает аминокислоту, выбранную из А и Е; Х7 обозначает аминокислоту, выбранную из N и V; и Х8 обозначает аминокислоту, выбранную из Н, Р, V и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 843),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает
аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, T, V, W и Y; X8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 844),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из А, Е, F, G, Н, K, М, N, Р, Q, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из А, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 845),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, F, L, М, Р, Q, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 846),
в которой X1-X9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из А, Е, F, Н, I, K, L, М, N, Р, Q, R, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из А, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 847),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, G, Н, I, K, L, М, N, Q, R, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из А, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 848),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из Е, F, K, М, N, Р, Q, R, S и W; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 849),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; Х3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, F, G, L, М, Р, Q, V и W; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 850),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, I, K, N, T и W; и X9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 851),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает аминокислоту, выбранную из A, G, I, Р, Q, S и Y; Х2 обозначает аминокислоту, выбранную из K или Т; X3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает А; Х7 обозначает аминокислоту, выбранную из Н, I и V; Х8 обозначает аминокислоту, выбранную из Н, V и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 852),
в которой X1-Х9 каждый обозначает индивидуальную аминокислоту, X1 обозначает Y; Х2 обозначает аминокислоту, выбранную из S и Т; X3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает аминокислоту, выбранную из А и Е; Х8 обозначает аминокислоту, выбранную из Н, Р, V и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1062),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1063),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из А, Е, F, G, Н, K, М, N, Р, Q, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1064),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, F, L, М, Р, Q, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1065),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из А, Е, F, Н, I, K, L, М, N, Р, Q, R, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1066),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, G, Н, I, K, L, М, N, Q, R, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1067),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; X6 обозначает аминокислоту, выбранную из Е, F, K, М, N, Р, Q, R, S и W; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1068),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, F, G, L, М, Р, Q, V и W; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1069),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, I, K, N, Т и W.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1070),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, G, I, Р, Q, S и Y; Х2 обозначает аминокислоту, выбранную из K или Т; X3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает А; Х7 обозначает аминокислоту, выбранную из Н, I и V; и Х8 обозначает аминокислоту, выбранную из Н, V и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8 (SEQ ID NO: 1071),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает Y; Х2 обозначает аминокислоту, выбранную из S и Т; X3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает аминокислоту, выбранную из А и Е; Х7 обозначает аминокислоту, выбранную из N и V; и Х8 обозначает аминокислоту, выбранную из Н, Р, V и Y.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1072),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1073),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из А, Е, F, G, Н, K, М, N, Р, Q, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из А, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1074),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, F, L, М, Р, Q, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1075),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из А, Е, F, Н, I, K, L, М, N, Р, Q, R, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из А, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1076),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, G, Н, I, K, L, М, N, Q, R, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из А, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1077),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; X2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из Е, F, K, М, N, Р, Q, R, S и W; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1078),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, F, G, L, М, Р, Q, V и W; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1079),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, М, N, Р, Q, S, Т, W и Y; Х2 обозначает аминокислоту, выбранную из A, D, Е, F, Н, K, L, М, Р, Q, S, Т, V, W и Y; X3 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х4 обозначает R; Х5 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; X6 обозначает аминокислоту, выбранную из A, D, Е, F, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х7 обозначает аминокислоту, выбранную из A, D, Е, F, G, Н, I, K, L, М, N, Р, Q, R, S, Т, V, W и Y; Х8 обозначает аминокислоту, выбранную из A, D, Е, F, G, I, K, N, Т и W; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1080),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает аминокислоту, выбранную из A, G, I, Р, Q, S и Y; Х2 обозначает аминокислоту, выбранную из K или Т; X3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает А; Х7 обозначает аминокислоту, выбранную из Н, I и V; Х8 обозначает аминокислоту, выбранную из Н, V и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
Следующую последовательность можно применять также в качестве расщепляемой протеазой последовательности:
Х10-Х11-Х1-Х2-Х3-Х4-Х5-Х6-Х7-Х8-Х9 (SEQ ID NO: 1081),
в которой X1-X11 каждый обозначает индивидуальную аминокислоту, Х10 обозначает аминокислоту, выбранную из I, Т и Y; X11 обозначает S; X1 обозначает Y; Х2 обозначает аминокислоту, выбранную из S и Т; X3 обозначает G; Х4 обозначает R; Х5 обозначает S; Х6 обозначает аминокислоту, выбранную из А и Е; Х7 обозначает аминокислоту, выбранную из N и V; Х8 обозначает аминокислоту, выбранную из Н, Р, V и Y; и Х9 обозначает аминокислоту, выбранную из A, G, Н, I, L и R.
В дополнение к упомянутым выше последовательностям, расщепляемым протеазой, можно получать также новые расщепляемые протеазой последовательности путем скрининга. Например, основываясь на результате анализа кристаллической структуры известной расщепляемой протеазой последовательности, новые расщепляемые протеазой последовательности можно изучать путем изменения взаимодействия активных остатков/распознаваемых остатков расщепляемой последовательности и фермента. Новые расщепляемые протеазой последовательности можно изучать также на основе изменения аминокислот в известной расщепляемой протеазой последовательности и оценки взаимодействия между измененной последовательностью и протеазой. В качестве другого примера расщепляемые протеазой последовательности можно изучать путем оценки взаимодействия протеазы с библиотекой пептидов, экспонированной с использованием метода дисплея in vitro, такого как фаговый дисплей и рибосомный дисплей, или с помощью массива пептидов, иммобилизованных на чипе или гранулах.
Взаимодействие между расщепляемой протеазой последовательностью и протеазой можно оценивать путем тестирования расщепления последовательности с помощью протеазы in vitro или in vivo.
Расщепленные фрагменты после обработки протеазой можно разделять с помощью электрофореза, такого как ДСН-ПААГ, и количественно оценивать для исследования расщепляемой протеазой последовательности, активности протеазы и степени расщепления молекулы, в которую интродуцирована расщепляемая протеазой последовательность. Пример метода (но, не ограничиваясь только им) оценки степени расщепления молекулы, в которую интродуцирована расщепляемая протеазой последовательность, включает следующий метод: например, когда степень расщепления варианта антитела, в который интродуцирована расщепляемая протеазой последовательность, оценивают с использованием рекомбинантного человеческого активатора и-плазминогена/урокиназы (человеческая uPA, huPA) (фирма R&D Systems; 1310-SE-010) или рекомбинантной человеческой матриптазы / каталитический домен ST14 (человеческая MT-SP1, hMT-SP1) (фирма R&D Systems; 3946-SE-010), то 100 мкг/мл варианта антитела подвергают взаимодействию с 40 нМ huPA или 3 нМ hMT-SP1 в ЗФР при 37°С в течение 1 ч, а затем подвергают иммуноанализу на основе капиллярного электрофореза. Иммуноанализ на основе капиллярного электрофореза можно осуществлять с использованием системы Wes (фирма Protein Simple), но применяемый в настоящем описании метод не ограничен указанным. В качестве альтернативы иммуноанализу на основе капиллярного электрофореза для разделения можно применять ДСН-ПААГ и т.д. с последующей детекцией Вестерн-блоттингом. Применяемый в настоящем описании метод не ограничен указанными методами. До и после расщепления легкую цепь можно выявлять с использованием антитела к человеческой лямбда-цепи, меченное HRP (фирма abeam; ab9007), но можно применять любое антитело, с помощью которого можно выявлять расщепленные фрагменты. Площадь каждого пика, полученного после обработки протеазой, определяют с помощью программного обеспечения для системы Wes (программное обеспечение Compass для систем SW; фирма Protein Simple), и степень расщепления (%) варианта антитела можно определять с использованием следующей формулы:
(площадь пика расщепленной легкой цепи) × 100 / (площадь пика расщепленной легкой цепи + площадь пика нерасщепленной легкой цепи).
Степени расщепления можно определять, если белковые фрагменты поддаются обнаружению до и после обработки протеазой. Степени расщепления можно определять не только для вариантов антител, но также и для различных белковых молекул, в которые интродуцирована расщепляемая протеазой последовательность.
Степень расщепления in vivo молекулы, в которую интродуцирована расщепляемая протеазой последовательность, можно определять путем введения молекулы животным и детекции введенной молекулы в образцах крови. Например, вариант антитела, в который интродуцирована расщепляемая протеазой последовательность, вводят мышам и из образцов их крови получают плазму. Антитело очищают из плазмы с помощью метода, известного специалистам в данной области, с использованием Dynabeads с белком А (фирма Thermo; 10001D), и затем подвергают иммуноанализу на основе капиллярного электрофореза для оценки степени расщепления протеазой варианта антитела. Иммуноанализ на основе капиллярного электрофореза можно осуществлять с использованием системы Wes (фирма Protein Simple), но применяемый в настоящем описании метод не ограничен указанным. В качестве альтернативы иммуноанализу на основе капиллярного электрофореза для разделения можно применять ДСН-ПААГ и т.д. с последующей детекцией Вестерн-блоттингом. Применяемый в настоящем описании метод не ограничен указанными методами. До и после расщепления легкую цепь можно выявлять с использованием антитела к человеческой лямбда-цепи, меченное HRP (фирма abeam; ab9007), но можно применять любое антитело, с помощью которого можно выявлять расщепленные фрагменты. Когда площадь каждого пика, полученного с помощью иммуноанализа на основе капиллярного электрофореза, определяют с помощью программного обеспечения для системы Wes (программное обеспечение Compass для систем SW; фирма Protein Simple), то долю оставшейся легкой цепи можно рассчитывать как [площадь пика легкой цепи] / [площадь пика тяжелой цепи], для определения доли полноразмерной легкой цепи, оставшейся нерасщепленной в организме мыши. Эффективность расщепления in vivo можно определять, если белковые фрагменты, полученные из живого организма, поддаются обнаружению. Степени расщепления можно определять не только для вариантов антител, но также и для различных белковых молекул, в которые интродуцирована расщепляемая протеазой последовательность. Расчет степеней расщепления с помощью указанных выше методов позволяет, например, сравнивать in vivo степени расщепления вариантов антител, в которые интродуцированы различные расщепляемые последовательности, и сравнивать степень расщепления одного варианта антитела на различных животных моделях, таких как модель, созданная с использованием здоровых мышей, и модель, созданная с использованием мышей с трансплантированными опухолями.
Например, все расщепляемые протеазой последовательности, представленные в таблице 1, впервые открыты при создании настоящего изобретения. Полипептиды, содержащие указанные расщепляемые протеазой последовательности, все можно применять в качестве субстратов протеаз, которые гидролизуются под действием протеаз. Таким образом, в настоящем изобретении предложены субстраты протеаз, которые содержат последовательность, выбранную из SEQ ID NO: 833-852 и 1062-1081, и последовательностей, перечисленных в таблице 1. Субстраты протеаз, предлагаемые в настоящем изобретении, можно применять в качестве, например, библиотеки, из которой субстрат, обладающий свойствами, пригодными для данной цели, можно выбирать для включения в полипептид, предлагаемый в настоящем изобретении. В частности, для избирательного расщепления полипептида, предлагаемого в настоящем изобретении, протеазой, локализованной в повреждении, субстраты можно оценивать в отношении их чувствительности к указанной протеазе. Когда полипептид, предлагаемый в настоящем изобретении, вводят in vivo, то молекула может приходить в контакт с различными протеазами до достижения повреждения. Поэтому молекула предпочтительно должна обладать чувствительностью к протеазе, локализованной в повреждении, а также максимально высокой устойчивостью к другим протеазам. Для отбора требуемой расщепляемой протеазой последовательности в зависимости от поставленной цели каждый субстрат протеазы можно заранее всесторонне анализировать в отношении чувствительности к различным протеазам для определения его устойчивости к протеазам. На основе установленного спектра устойчивости к протеазам можно выявлять расщепляемую протеазой последовательность с необходимой чувствительностью и устойчивостью.
Альтернативно этому, полипептид, в который интродуцирована расщепляемая протеазой последовательность, до достижения повреждения подвергается не только ферментативным действиям, связанным с протеазами, но также и различным связанным с окружающей средой стрессам, таким как изменения рН, температурный и окислительно/восстановительный стресс.На основе сравнения информации об устойчивости субстратов протеаз к указанным внешним факторам можно отбирать расщепляемую протеазой последовательность с требуемыми свойствами.
В одном из вариантов осуществления настоящего изобретения к одному из концов или к обоим концам расщепляемой протеазой последовательности дополнительно присоединяют гибкий линкер. Гибкий линкер на одном конце расщепляемой протеазой последовательности можно обозначать как первый гибкий линкер, а гибкий линкер на другом конце можно обозначать как второй гибкий линкер. В конкретном варианте осуществления применяют одну из следующих комбинаций расщепляемой протеазой последовательности и гибкого линкера:
(расщепляемая протеазой последовательность),
(первый гибкий линкер)-(расщепляемая протеазой последовательность),
(расщепляемая протеазой последовательность)-(второй гибкий линкер), и
(первый гибкий линкер)-(расщепляемая протеазой последовательность)-(второй гибкий линкер).
Гибкий линкер, указанный в данном варианте осуществления изобретения, предпочтительно представляет собой пептидный линкер. Первый гибкий линкер и второй гибкий линкер каждый присутствует независимо и произвольно, и они представляют собой идентичные или различные гибкие линкеры, каждый из которых содержит по меньшей мере одну гибкую аминокислоту (Gly и т.д.). Гибкий линкер содержит, например, достаточное количество остатков (аминокислоты, произвольно выбранные из Arg, Ile, Gln, Glu, Cys, Tyr, Trp, Thr, Val, His, Phe, Pro, Met, Lys, Gly, Ser, Asp, Asn, Ala и т.д., прежде всего Gly, Ser, Asp, Asn и Ala, в частности, Gly и Ser, особенно предпочтительно Gly и т.д.) для расщепляемой протеазой последовательности для получения требуемой доступности протеазы.
Гибкий линкер, пригодный для применения на обоих концах расщепляемой протеазой последовательности, как правило, представляет собой гибкий линкер, который облегчает доступ протеазы к расщепляемой протеазой последовательности и повышает расщепляющую эффективность протеазы. Приемлемый гибкий линкер можно легко выбирать и его можно предпочтительно выбирать из линкеров различной длины, таких как имеющие длину от 1 аминокислоты (Gly и т.д.) до 20 аминокислот, от 2 аминокислот до 15 аминокислот или от 3 аминокислот до 12 аминокислот, в том числе от 4 аминокислот до 10 аминокислот, от 5 аминокислот до 9 аминокислот, от 6 аминокислот до 8 аминокислот или от 7 аминокислот до 8 аминокислот.В некоторых вариантах осуществления настоящего изобретения гибкий линкер представляет собой пептидный линкер, состоящий из 1-7 аминокислот.
Примеры гибкого линкера включают (но, не ограничиваясь только ими) глициновые полимеры (G)n, глицин-сериновые полимеры (включая, например, (GS)n, (GSGGS: SEQ ID NO: 27)n и (GGGS: SEQ ID NO: 28)n, в которых n обозначает целое число, равное по меньшей мере 1), глицин-аланиновые полимеры, аланин-сериновые полимеры и другие гибкие линкеры, широко применяемые в общепринятых методиках.
Среди них глициновые полимеры и глицин-сериновые полимеры заслуживают внимания в связи с тем, что эти аминокислоты являются относительно неструктурированными и способны функционировать в виде нейтральных связок между компонентами.
Примеры гибкого линкера, состоящего из глицин-серинового полимера, могут включать (но, не ограничиваясь только ими)

В настоящем описании понятие «ассоциация» может относиться, например, к состоянию, в котором две или большие количество полипептидных областей взаимодействуют друг с другом. Как правило, гидрофобная связь, водородная связь, ионная связь или т.п. образуется между требуемыми пептидными областям с образованием продукта ассоциации. В качестве одного из примеров обычной ассоциации, известно, что антитело, представляющее собой нативное антитело, сохраняет спаренную структуру вариабельной области тяжелой цепи (VH) и вариабельной области легкой цепи (VL) благодаря нековалентной связи или т.п. между ними.
В некоторых вариантах осуществления настоящего изобретения ингибирующий домен транспортирующего фрагмента находится в ассоциации с антигенсвязывающим доменом. Ингибирующий домен может представлять собой часть транспортирующего фрагмента или может состоять полностью из транспортирующего фрагмента. С другой точки зрения, ингибирующий домен можно определять также как фрагмент, вступающий в ассоциацию с антигенсвязывающим доменом в транспортирующем фрагменте.
В более конкретном варианте осуществления изобретения антигенсвязывающий домен, который представляет собой однодоменное антитело, и ингибирующий домен, который представляет собой VL, VH или VHH, образуют такую же ассоциацию, как и ассоциация, присутствующая между VH антитела и VL антитела. В другом конкретном варианте осуществления изобретения антигенсвязывающий домен, который представляет собой однодоменное антитело, и ингибирующий домен, который представляет собой VL, VH или VHH, образуют такую же ассоциацию, как и ассоциация, присутствующая между VH антитела и VL антитела, и в состоянии созданной таким образом ассоциации ингибирующий домен конформационно ингибирует связывание антигенсвязывающего домена с антигеном или конформационно изменяет антигенсвязывающий сайт антигенсвязывающего домена так, что антигенсвязывающая активность однодоменного антитела ингибируется с помощью VL, VH или VHH. В варианте осуществления изобретения, в котором VHH применяют в качестве однодоменного антитела, считается, что связывание VHH с антигеном конформационно ингибируется с помощью ингибирующего домена, когда CDR3, основной антигенсвязывающий сайт VHH, или соседний к нему сайт присутствует на поверхности раздела ассоциации с ингибирующим доменом.
Ассоциацию антигенсвязывающего домена с ингибирующим доменом можно аннулировать, например, путем расщепления сайта расщепления. Аннулирование (упразднение) ассоциации можно применять взаимозаменяемо, например, с аннулированием состояния, в котором два или большее количество полипептидных областей взаимодействуют друг с другом. Взаимодействие между двумя или большим количеством полипептидных областей можно полностью аннулировать или взаимодействие между двумя или большим количеством полипептидных областей можно частично аннулировать.
В настоящем описании понятие «поверхность раздела», как правило, относится к границе, на которой происходит ассоциация или взаимодействие двух областей друг с другом. Аминокислотные остатки, формирующие поверхность раздела, представляют собой, как правило, один или множество аминокислотных остатков, которые содержатся в каждой полипептидной области, подвергаемой ассоциации, и более предпочтительно представляют собой аминокислотные остатки, которые соприкасаются друг с другом при ассоциации и участвуют в образовании взаимодействия. В частности, взаимодействие включает нековалентные связи, такие как водородная связь, электростатическое взаимодействие или образование соляного мостика между аминокислотными остатками, соприкасающимися друг с другом при ассоциации.
В настоящем описании понятие «аминокислотные остатки, формирующие поверхность раздела» относится, в частности, к аминокислотным остаткам, содержащимся в полипептидных областях, образующих поверхность раздела. Например, полипептидные области, образующие поверхность раздела, означают полипептидные области, ответственные за внутримолекулярное или межмолекулярное избирательное связывание антител, лигандов, рецепторов, субстратов и т.д. Конкретные примеры таких полипептидных областей в антителах могут включать вариабельные области тяжелых цепей и вариабельные области легких цепей. В некоторых вариантах осуществления настоящего изобретения примеры указанных полипептидных областей могут включать антигенсвязывающие домены и ингибирующие домены.
Примеры аминокислотных остатков, формирующих поверхность раздела, включают (но, не ограничиваясь только ими) аминокислотные остатки, сближающиеся друг с другом при ассоциации. Аминокислотные остатки, сближающиеся друг с другом при ассоциации, можно выявлять, например, анализируя конформации полипептидов и изучая аминокислотные последовательности полипептидных областей, формирующих поверхность раздела при ассоциации полипептидов.
В некоторых вариантах осуществления настоящего изобретения аминокислотный(ые) остаток(ки), участвующий(ие) в ассоциации в антигенсвязывающем домене, или аминокислотный(ые) остаток(ки), участвующий(ие) в ассоциации в ингибирующем домене, можно изменять для усиления ассоциации антигенсвязывающего домена с ингибирующим доменом. В другом конкретном варианте осуществления изобретения можно изменять аминокислотный(ые) остаток(ки), формирующий(ие) поверхность раздела с ингибирующим доменом в антигенсвязывающем домене, или аминокислотный(ые) остаток(ки), формирующий(ие) поверхность раздела с антигенсвязывающим доменом в ингибирующем домене. В предпочтительном варианте осуществления изобретения аминокислотный(ые) остаток(ки), формирующий(ие) поверхность раздела, можно изменять с помощью метода интродукции мутации(ий) в аминокислотный(ые) остаток(ки) на поверхности раздела, таким образом, чтобы два или большее количество аминокислотных остатков, формирующих поверхность раздела, приобретали различные заряды. Изменение аминокислотного(ых) остатка(ов), приводящее к различным зарядам, включает изменение положительно заряженного аминокислотного остатка на отрицательно заряженный(ые) аминокислотный(ые) остаток(ки) или незаряженный(ые) аминокислотный(ие) остаток(ки), изменение отрицательно заряженного аминокислотного остатка на положительно заряженный(ые) аминокислотный(ые) остаток(ки) или незаряженный(ые) аминокислотный(ые) остаток(ки), и изменение незаряженного(ых) аминокислотного(ых) остатка(ов) на положительно или отрицательно заряженный(ые) аминокислотный(ые) остаток(ки). Указанное аминокислотное изменение осуществляют с целью усиления ассоциации, и оно не ограничено положением аминокислотного изменения или типом аминокислоты, если при этом можно достигать цели, состоящей в усилении ассоциации. Примеры изменения включают (но, не ограничиваясь только указанным) замену.
В некоторых вариантах осуществления настоящего изобретения имеет место ассоциация между VHH, служащим в качестве антигенсвязывающего домена, и VL, служащим в качестве ингибирующего домена. Аминокислотный остаток в VHH, участвующий в ассоциации с VL, можно обозначать, например, как аминокислотный остаток, формирующий поверхность раздела между VHH и VL. Примеры аминокислотного остатка в VHH, участвующего в ассоциации с VL, включают (но, не ограничиваясь только ими) аминокислотные остатки в положениях 37, 44, 45 и 47 (J. Mol. Biol. 350, 2005, сс. 112-125). Активность VHH ингибируется в результате усиления ассоциации между VHH и VL. Аналогично этому, аминокислотный остаток в VL, участвующий в ассоциации с VHH, можно обозначать, например, как аминокислотный остаток, формирующий поверхность раздела между VHH и VL.
Аминокислотный остаток в VHH, участвующий в ассоциации с VL, можно изменять для усиления ассоциации между VHH и VL. Примеры указанной аминокислотной замены включают (но, не ограничиваясь только ими) F37V, Y37V, E44G, Q44G, R45L, H45L, G47W, F47W, L47W, T47W или/и S47W. Вместо изменения указанного остатка в VHH, можно применять также VHH, в котором исходно присутствует аминокислотный остаток 37V, 44G, 45 L или/и 47W.
Вместо аминокислоты в VHH можно изменять аминокислотный остаток в VL, участвующий в ассоциации с VHH, и аминокислотные изменения можно интродуцировать также и в VHH, и в VL, если при этом можно достигать цели, состоящей в усилении ассоциации между VHH и VL.
В некоторых альтернативных вариантах осуществления настоящего изобретения можно осуществлять ассоциацию антигенсвязывающего домена и ингибирующего домена друг с другом, используя VHH в качестве антигенсвязывающего домена и используя VH или VHH в качестве ингибирующего домена. Аминокислотный остаток, участвующий в ассоциации с VH или VHH, служащим в качестве ингибирующего домена, в VHH, служащим в качестве антигенсвязывающего домена, можно идентифицировать и изменять для усиления ассоциации антигенсвязывающего домена VHH с ингибирующим доменом VH или VHH. Кроме того, можно идентифицировать и изменять аминокислотные остатки, участвующие в ассоциации с VHH, служащим в качестве антигенсвязывающего домена, в VH или VHH, служащих в качестве ингибирующего домена.
В случае применения однодоменного антитела, отличного от VHH, в качестве антигенсвязывающего домена, аминокислотные остатки, участвующие в ассоциации, в антигенсвязывающем домене или в ингибирующем домене, можно также идентифицировать и изменять аналогично описанному выше.
В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент и антигенсвязывающий домен сливают через линкер. В более конкретном варианте осуществления изобретения транспортирующий фрагмент и антигенсвязывающий домен сливают через линкер, содержащий сайт расщепления. В альтернативном конкретном варианте осуществления изобретения транспортирующий фрагмент и антигенсвязывающий домен сливают через линкер, полученный таким образом слитый белок содержит сайт расщепления.
В другом варианте осуществления настоящего изобретения транспортирующий фрагмент и антигенсвязывающий домен сливают без линкера. В более конкретном варианте осуществления изобретения формируют аминную связь между N-концевой аминокислотой транспортирующего фрагмента и С-концевой аминокислотой антигенсвязывающего домена, получая слитый белок. Образовавшийся слитый белок содержит сайт расщепления. В конкретном варианте осуществления изобретения от одной до нескольких N-концевых аминокислот транспортирующего фрагмента или/и от одной до нескольких С-концевых аминокислот антигенсвязывающего домена изменяют, и N-конец транспортирующего фрагмента и С-конец антигенсвязывающего домена сливают с получением сайта расщепления вблизи положения слияния. Более конкретно, сайт расщепления можно формировать, например, путем превращения четырех С-концевых аминокислот антигенсвязывающего домена в последовательность LSGR и превращения четырех N-концевых аминокислот транспортирующего фрагмента в последовательность SDNH.
В некоторых вариантах осуществления настоящего изобретения сайт расщепления полипептида, содержащего транспортирующий фрагмент и антигенсвязывающий домен, содержит расщепляемую протеазой последовательность. Расщепляемую протеазой последовательность можно помещать в любое положение в полипептиде, если антигенсвязывающий домен высвобождается расщеплением протеазой и не утрачивает свою антигенсвязывающую активность после высвобождения.
В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент содержит константную область антитела, и N-конец константной области антитела и С-конец антигенсвязывающего домена слиты через линкер или без линкера.
В конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в константной области антитела, входящей в транспортирующий фрагмент. В этом случае расщепляемая протеазой последовательность может быть локализована в константной области антитела так, чтобы антигенсвязывающий домен высвобождался расщеплением протеазой. В конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в константной области тяжелой цепи антитела, входящей в транспортирующий фрагмент, и более конкретно локализована в константной области тяжелой цепи антитела на стороне, примыкающей к антигенсвязывающему домену, за аминокислотой в положении 140 (EU-нумерация), предпочтительно в константной области тяжелой цепи антитела на стороне, примыкающей к антигенсвязывающему домену, за аминокислотой в положении 122 (EU-нумерация). В альтернативном конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в константной области легкой цепи антитела, входящей в транспортирующий фрагмент, и более конкретно локализована в константной области легкой цепи антитела на стороне, примыкающей к антигенсвязывающему домену, за аминокислотой в положении 130 (EU-нумерация) (нумерация положения 130 по Кэботу), предпочтительно в константной области легкой цепи антитела на стороне, примыкающей к антигенсвязывающему домену, за аминокислотой в положении 113 (EU-нумерация) (нумерация положения 113 по Кэботу).
В некоторых вариантах осуществления настоящего изобретения антигенсвязывающий домен представляет собой однодоменное антитело, и С-конец однодоменного антитела и N-конец транспортирующего фрагмента слиты через линкер или без линкера.
В конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в однодоменном антителе. В более конкретном варианте осуществления изобретения однодоменное антитело представляет собой однодоменное антитело, полученное из VH или VHH, и расщепляемая протеазой последовательность локализована в однодоменном антителе на стороне, примыкающей к транспортирующему фрагменту, за аминокислотой в положении 35b (нумерация Кэбота), предпочтительно в однодоменном антителе на стороне, примыкающей к транспортирующему фрагменту, за аминокислотой в положении 95 (нумерация Кэбота), более предпочтительно в однодоменном антителе на стороне, примыкающей к транспортирующему фрагменту, за аминокислотой в положении 109 (нумерация Кэбота). В альтернативном конкретном варианте осуществления изобретения однодоменное антитело представляет собой однодоменное антитело, полученное из VL, и расщепляемая протеазой последовательность локализована в однодоменном антителе на стороне, примыкающей к транспортирующему фрагменту, за аминокислотой в положении 32 (нумерация Кэбота), предпочтительно в однодоменном антителе на стороне, примыкающей к транспортирующему фрагменту, за аминокислотой в положении 91 (нумерация Кэбота), более предпочтительно в однодоменном антителе на стороне, примыкающей к транспортирующему фрагменту, за аминокислотой в положении 104 (нумерация Кэбота).
В некоторых вариантах осуществления настоящего изобретения транспортирующий фрагмент содержит константную область антитела, антигенсвязывающий домен представляет собой однодоменное антитело, и константная область антитела и однодоменное антитело слиты через линкер или без линкера. В более конкретном варианте осуществления изобретения N-конец константной области антитела и С-конец однодоменного антитела слиты через линкер или без линкера. В альтернативном конкретном варианте осуществления изобретения С-конец константной области антитела и N-конец однодоменного антитела слиты через линкер или без линкера.
В конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в константной области антитела, входящей в транспортирующий фрагмент. В более конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в константной области тяжелой цепи антитела на стороне, примыкающей к однодоменному антителу, за аминокислотой в положении 140 (EU-нумерация), предпочтительно в константной области тяжелой цепи антитела на стороне, примыкающей к однодоменному антителу, за аминокислотой в положении 122 (EU-нумерация). В альтернативном конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в константной области легкой цепи антитела на стороне, примыкающей к однодоменному антителу, за аминокислотой в положении 130 (EU-нумерация) (нумерация положения 130 по Кэботу), предпочтительно в константной области легкой цепи антитела на стороне, примыкающей к антигенсвязывающему домену, за аминокислотой в положении 113 (EU-нумерация) (нумерация положения 113 по Кэботу).
В конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована в однодоменном антителе. В более конкретном варианте осуществления изобретения однодоменное антитело представляет собой однодоменное антитело, полученное из VH или VHH, и расщепляемая протеазой последовательность локализована в однодоменном антителе на стороне, примыкающей к константной области антитела, за аминокислотой в положении 35b (нумерация Кэбота), предпочтительно в однодоменном антителе на стороне, примыкающей к константной области антитела, за аминокислотой в положении 95 (нумерация Кэбота), более предпочтительно в однодоменном антителе на стороне, примыкающей к константной области антитела, за аминокислотой в положении 109 (нумерация Кэбота). В альтернативном конкретном варианте осуществления изобретения однодоменное антитело представляет собой однодоменное антитело, полученное из VL, и расщепляемая протеазой последовательность локализована в однодоменном антителе на стороне, примыкающей к константной области антитела, за аминокислотой в положении 32 (нумерация Кэбота), предпочтительно в однодоменном антителе на стороне, примыкающей к константной области антитела, за аминокислотой в положении 91 (нумерация Кэбота), более предпочтительно в однодоменном антителе на стороне, примыкающей к константной области антитела, за аминокислотой в положении 104 (нумерация Кэбота) однодоменного антитела.
В конкретном варианте осуществления изобретения расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и транспортирующим фрагментом. Фраза «вблизи границы между антигенсвязывающим доменом и транспортирующим фрагментом» относится к фрагменту, расположенному в прямом направлении или в обратном направлении от сайта сцепления между антигенсвязывающим доменом и транспортирующим фрагментом, и который не влияет существенно на вторичную структуру антигенсвязывающего домена.
В более конкретном варианте осуществления изобретения антигенсвязывающий домен сцеплен с константной областью антитела, входящей в транспортирующий фрагмент, и расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью антитела. Фраза «вблизи границы между антигенсвязывающим доменом и константной областью антитела» может относиться к положению вблизи границы между антигенсвязывающим доменом и константной областью тяжелой цепи антитела или вблизи границы между антигенсвязывающим доменом и константной областью легкой цепи антитела. Когда антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH или VHH, и он соединен с константной областью тяжелой цепи антитела, то фраза «вблизи границы между антигенсвязывающим доменом и константной областью антитела» может относиться к положению между аминокислотой в положении 101 (нумерация Кэбота) однодоменного антитела и аминокислотой в положении 140 (EU-нумерация) константной области тяжелой цепи антитела и может предпочтительно относиться к положению между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела и аминокислотой в положении 122 (EU-нумерация) константной области тяжелой цепи антитела. Когда антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VH или VHH, и оно соединено с константной областью легкой цепи антитела, то фраза «вблизи границы между антигенсвязывающим доменом и константной областью легкой цепи антитела» может относиться к положению между аминокислотой в положении 101 (нумерация Кэбота) однодоменного антитела и аминокислотой в положении 130 (EU-нумерация) (нумерация положения 130 по Кэботу) константной области легкой цепи антитела и может предпочтительно относиться к положению между аминокислотой в положении 109 (нумерация Кэбота) однодоменного антитела и аминокислотой в положении 113 (EU-нумерация) (нумерация положения 113 по Кэботу) константной области легкой цепи антитела. Когда антигенсвязывающий домен представляет собой однодоменное антитело, полученное из VL, то фраза «вблизи границы между антигенсвязывающим доменом и константной областью антитела» относится к положению между аминокислотой в положении 96 (нумерация Кэбота) однодоменного антитела и предписанным положением в константной области антитела, предпочтительно между аминокислотой в положении 104 (нумерация Кэбота) однодоменного антитела и предписанным положением в константной области антитела.
В некоторых вариантах осуществления настоящего изобретения полипептид представляет собой молекулу, подобную антителу IgG-типа. Примеры таких вариантов осуществления изобретения включают (но, не ограничиваясь только ими): вариант осуществления изобретения, в котором транспортирующий фрагмент содержит константную область антитела IgG-типа, однодоменное антитело, служащее в качестве антигенсвязывающего домена, занимает место VH антитела IgG-типа, и антигенсвязывающая активность ингибируется VL; вариант осуществления изобретения, в котором транспортирующий фрагмент содержит константную область антитела IgG-типа, однодоменное антитело, служащее в качестве антигенсвязывающего домена, занимает место VL антитела IgG-типа, и антигенсвязывающая активность ингибируется VH; и вариант осуществления изобретения, в котором транспортирующий фрагмент содержит константную область антитела IgG-типа, однодоменное антитело, служащее в качестве антигенсвязывающего домена, занимает место одного из VH и VL антитела IgG-типа, и дополнительное однодоменное антитело, ингибирующее антигенсвязывающую активность антигенсвязывающего домена, занимает место другого домена антитела IgG-типа.
Понятие «молекула, подобная антителу IgG-типа», применяемое в настоящем описании, используют для обозначения молекулы, которая имеет фрагменты, практически сходные по структуре с константными доменами или константными областями в антителе IgG-типа, и фрагменты, практически сходные по структуре с вариабельными доменами или вариабельными областями в антителе IgG-типа, и которая конформационно имеет практическое сходство с антителом IgG-типа. В молекуле, подобной антителу IgG-типа, домен, сходный с СН1 антитела, и домен, сходный с CL антитела, можно применять взаимозаменяемо; это означает, что, если взаимодействие является сходным с взаимодействием между СН1 и CL антитела IgG-типа, имеющим место между доменами, то домены, сцепленные с участком, сходным с шарнирной область антитела, могут представлять собой СН1-домен антитела или CL-домен антитела. Однако в настоящем описании «молекула, подобная антителу IgG-типа» может проявлять или может не проявлять антигенсвязывающую активность, сохраняя при этом структуры, сходные со структурами антитела IgG-типа.
Полипептид может содержать один или множество антигенсвязывающих доменов. Один или множество ингибирующих доменов может(гут) ингибировать антигенсвязывающую активность множества антигенсвязывающих доменов. Каждый из множества антигенсвязывающих доменов может быть ассоциирован с ингибирующим доменом. Каждый из множества антигенсвязывающих доменов может быть слит с транспортирующим фрагментом. Каждый из множества антигенсвязывающих доменов может обладать способностью высвобождаться из полипептидов. Сайт(ы) расщепления для высвобождения множества антигенсвязывающих доменов может(ут) представлять собой множество сайтов расщепления, соответствующих количеству антигенсвязывающих доменов.
Когда полипептид представляет собой молекулу, подобную антителу IgG-типа, антигенсвязывающие домены можно создавать соответственно на фрагментах, которые соответствуют двум вариабельным областям антитела IgG-типа, как продемонстрировано на фиг. 7. Указанный вариант осуществления изобретения должен быть очевиден специалистам в данной области после ознакомления с настоящим изобретением. Антигенсвязывающие домены, включенные в оба плеча, могут иметь одинаковую антигенсвязывающую специфичность или могут отличаться по антигенсвязывающей специфичности. Указанный вариант осуществления изобретения должен быть очевиден специалистам в данной области после ознакомления с настоящим изобретением. Очевидно, что указанные варианты осуществления изобретения включены в объем настоящего изобретения.
В некоторых вариантах осуществления настоящего изобретения антигенсвязывающий домен дополнительно сцеплен со вторым антигенсвязывающим доменом. Примеры второго антигенсвязывающего домена включают (но, не ограничиваясь только ими) однодоменные антитела, фрагменты антител, модуль, состоящий примерно из 35 аминокислот, обозначенный как А-домен, который входит в авимер (Avimer), белок клеточной мембраны, присутствующий in vivo (WO 2004/044011 и WO 2005/040229), аднектин (Adnectin), содержащий 10Fn3-домен, служащий в качестве связывающего белок домена, полученного из гликопротеина фибронектина, экспрессируемого на клеточных мембранах (WO 2002/032925), аффибоди (Affibody), имеющий каркас IgG-связывающего домена, состоящий из трехспирального пучка, включающего 58 аминокислот белка A (WO 1995/001937), DARPin (сконструированные белки с анкириновыми повторами), которые представляют собой области, экспонируемые на молекулярной поверхности анкириновых повторов (AR), каждый из которых имеет структуру, в которой повторно уложена субъединица с изгибом, содержащая 33 аминокислотных остатка, две антипараллельной спирали и петлю (WO 2002/020565), антикалины (Anticalin), которые имеют состоящие из четырех петель области, поддерживающие одну сторону центрально закрученной бочкообразной структуры, состоящей из восьми антипараллельных цепей, которые являются высоко консервативными среди молекул липокалинов, таких как липокалин, ассоциированный с желатиназой нейтрофилов (NGAL) (WO 2003/029462), и вогнутую область, образованную структурой в виде параллельных тяжей внутри подковообразной структуры, образованной уложенными повторами, которые состоят из богатых лейцином повторяющихся (LRR) модулей вариабельного рецептора лимфоцитов (VLR), который не имеет иммуноглобулиновой структуры и применяется в системе приобретенной устойчивости у бесчелюстных позвоночных, таких как миноги или миксины (WO 2008/016854). В предпочтительном варианте осуществления изобретения второй антигенсвязывающий домен имеет антигенсвязывающую специфичность, отличную от специфичности антигенсвязывающего домена. В предпочтительном варианте осуществления изобретения молекулярная масса антигенсвязывающего домена и второго сцепленного антигенсвязывающего домена составляет 60 кДа или менее.
В некоторых более конкретных вариантах осуществления изобретения антигенсвязывающий домен и второй антигенсвязывающий домен представляют собой однодоменные антитела, отличающиеся по антигенсвязывающим специфичностям, антигенсвязывающий домен и второй сцепленный антигенсвязывающий домен обладают способностью высвобождаться из полипептида, и антигенсвязывающий домен и второй антигенсвязывающий домен образуют биспецифическую антигенсвязывающую молекулу после высвобождения. Примеры такой биспецифической антигенсвязывающей молекулы включают (но не ограничиваясь только ими) биспецифическую антигенсвязывающую молекулу, которая имеет антигенсвязывающий домен, специфически связывающийся с поверхностным антигеном клетки-мишени, и второй антигенсвязывающий домен, специфически связывающийся с поверхностным антигеном иммуноцита, биспецифическую антигенсвязывающую молекулу, которая имеет антигенсвязывающий домен и второй антигенсвязывающий домен, связывающийся с другими субъединицами одного и того же антигена, и биспецифическую антигенсвязывающую молекулу, которая имеет антигенсвязывающий домен и второй антигенсвязывающий домен, которые связываются с различными эпитопами одного и того же антигена. Указанная биспецифическая антигенсвязывающая молекула может обеспечивать рекрутинг иммуноцитов в окрестность клеток-мишеней, и поэтому считается, что является ценным для лечения заболевания, вызываемого клетками-мишенями.
Антигенсвязывающая активность второго антигенсвязывающего домена может ингибироваться или может не ингибироваться транспортирующим фрагментом. Второй антигенсвязывающий домен может находиться в ассоциации или может не находиться в ассоциации со структурой, являющейся частью транспортирующего фрагмента. В частности, когда антигенсвязывающий домен и второй антигенсвязывающий домен отличаются по антигенсвязывающим специфичностям, то антигенсвязывающий домен в невысвободившемся состоянии не может проявлять антигенсвязывающую активность, что продемонстрировано, например, на фиг. 8, даже, если антигенсвязывающая активность второго антигенсвязывающего домена не ингибируется, и даже, если второй антигенсвязывающий домен не находится в ассоциации со структурой, являющейся частью транспортирующего фрагмента. Указанная биспецифическая антигенсвязывающая молекула, содержащая антигенсвязывающий домен, сцепленный со вторым антигенсвязывающим доменом, не может проявлять функцию, состоящую в биспецифическом связывании с двумя типами антигенов.
На фиг. 8 продемонстрирована одна приведенная в качестве примера форма, в которой антигенсвязывающий домен дополнительно сцеплен со вторым антигенсвязывающим доменом.
В настоящем описании понятие «специфичность» относится к способности, с помощью которой одна из специфически связывающихся молекул не может связываться в значительной степени с молекулой, отличной от одной или нескольких молекул, которые являются ее партнерами по связыванию. Это понятие используют также, когда антигенсвязывающий домен обладает специфичностью в отношении эпитопа, входящего в конкретный антиген. Это понятие используют также, когда антигенсвязывающий домен обладает специфичностью в отношении конкретного эпитопа среди множества эпитопов, входящих в антиген. В этом контексте понятие «не связывается специфически» при определении с помощью метода, описанного в разделе, касающемся активности связывания, означает, что связывающая активность специфически связывающейся молекулы в отношении молекулы, отличной от ее партнера(ов) по связыванию, составляет 80% или менее, как правило, 50% или менее, предпочтительно 30% или менее, особенно предпочтительно 15% или менее, от ее связывающей активности в отношении молекул, которые являются ее партнером(ами) по связыванию.
Настоящее изобретение относится также к фармацевтическим композициям (лекарственным средствам), которые содержат полипептид, предлагаемый в настоящем изобретении, и фармацевтически приемлемый носитель.
Применяемое в настоящем описании понятие «лечение» (и его грамматические вариации, такие как «лечить» или «процесс лечения») относится к клиническому вмешательству с целью изменения естественного течения болезни у индивидуума, подлежащего лечению, и его можно осуществлять либо для профилактики, либо в процессе развития клинического патологического состояния. Требуемыми действиями лечения являются (но, не ограничиваясь только ими) предупреждение возникновения или рецидива болезни, облегчение симптомов, уменьшение любых прямых или косвенных патологических последствий болезни, предупреждение метастазов, снижение скорости развития болезни, облегчение или временное ослабление болезненного состояния и ремиссия или улучшение прогноза. В некоторых вариантах осуществления изобретения полипептид, предлагаемый в настоящем изобретении, применяют для замедления возникновения заболевания(ий) или замедления развития заболевай ия(ий).
В настоящем изобретении понятие «фармацевтическая композиция» относится, как правило, к лекарственному средству, предназначенному для лечения или предупреждения заболевания или для обследования или постановки диагноза. В настоящем изобретении понятие «фармацевтическая композиция, содержащая полипептид» можно применять взаимозаменяемо с понятием «способ лечения заболевания, включающий введение полипептида индивидууму, подлежащему лечению» и можно применять взаимозаменяемо с понятием «применение полипептида для производства лекарственного средства, предназначенного для лечения заболевания». Кроме того, понятие «фармацевтическая композиция, содержащая полипептид» можно применять взаимозаменяемо с понятием «применение полипептида для лечения заболевания».
Фармацевтические композиции, предлагаемые в настоящем изобретении, можно приготавливать для применения методом, известным специалистам в данной области. Например, фармацевтические композиции можно применять парентерально, например, в форме стерильного раствора или суспензии для инъекции в воде или любых других фармацевтически приемлемых жидкостях. Фармацевтические композиции можно приготавливать, например, путем объединения соответственно полипептида с фармакологически приемлемым носителем или средой, в частности, стерильной водой или физиологическим соляным раствором, растительным маслом, эмульгатором, суспендирующим агентом, поверхностно-активным веществом, стабилизатором, корригентом, эксципиентом, наполнителем, антисептиком, связывающим агентом и т.д., и смешивать их с получением стандартной дозы лекарственного средства, требуемой для общепринятой фармацевтической практики. Количество действующего вещества в указанных композициях регулируют так, чтобы получать приемлемый объем в предварительно определенном диапазоне.
Стерильную композицию для инъекции можно приготавливать согласно общепринятой фармацевтической практике с использованием такого наполнителя, как дистиллированная вода, пригодная для инъекций. Примеры пригодного для инъекций водного раствора включают изотонические растворы, которые содержат физиологический соляной раствор, глюкозу или другие адъюванты (например, D-сорбит, D-маннозу, D-маннит и хлорид натрия). Водный раствор можно применять в комбинации с соответствующим солюбилизатором, например, спиртом (этанол и т.д.), многоатомным спиртом (пропиленгликоль, полиэтиленгликоль и т.д.) или неионогенным поверхностно-активным веществом (Polysorbate 80™, НСО-50 и т.д.).
Примеры масляного раствора включают кунжутное масло и соевое масло. Масляный раствор можно применять также в комбинации с бензилбензоатом и/или бензиловым спиртом в качестве солюбилизатора. Масляный раствор можно дополнять буфером (например, раствором фосфатного буфера и раствором натрий-ацетатного буфера), смягчающим агентом (например, гидрохлоридом прокаина), стабилизатором (например, бензиловым спиртом и фенолом) и антиоксидантом. Полученным раствором для инъекции, как правило, заполняют соответствующую ампулу.
Фармацевтическую композицию, предлагаемую в настоящем изобретении, предпочтительно вводят парентеральным путем. Например, вводят композицию в лекарственной форме, пригодной для инъекции, трансназального, транспульмонального или чрескожного введения. Фармацевтическую композицию можно вводить системно или применять место, например, путем внутривенной инъекции, внутримышечной инъекции, внутрибрюшинной инъекции или подкожной инъекции.
Метод введения можно выбирать соответственно в зависимости от возраста и симптомов у пациента. Дозу фармацевтической композиции, содержащей полипептид, можно выбирать из диапазона, составляющего, например, от 0,0001 до 1000 мг на кг веса тела на дозу. Альтернативно этому, дозу фармацевтической композиции, содержащей полипептид, можно выбирать из дозы, составляющей, например, от 0,001 до 100000 мг на пациента. Однако настоящее изобретение не обязательно ограничено указанными численными величинам. Хотя доза и метод введения варьируются в зависимости от веса тела, возраста, симптомов и т.д. пациента, специалисты в данной области могут определять соответствующую дозу и метод введения с учетом указанных обстоятельств.
Настоящее изобретение относится также к способам получения полипептида, содержащего транспортирующий фрагмент, который имеет ингибирующий домен, и антигенсвязывающий домен.
Один из способов получения полипептида, предлагаемого в настоящем изобретении, является способ, который включает: получение антигенсвязывающего домена, обладающего антигенсвязывающей активностью; сцепление антигенсвязывающего домена с транспортирующим фрагментом так, чтобы антигенсвязывающая активность антигенсвязывающего домена ингибировалась ингибирующим доменом, с получением полипептида-предшественника; и дополнительная инсерция сайта расщепления в полипептид-предшественник или замена части полипептида-предшественника на сайт расщепления. Метод интродукции сайта расщепления может представлять собой любую инсерцию сайта расщепления и изменение части полипептида-предшественника так, чтобы сайт расщепление мог быть интродуцирован в полипептид-предшественник. Альтернативно этому, сайт изменения можно интродуцировать в полипептид-предшественник путем объединения обоих подходов. Указанный вариант осуществления изобретения должен быть очевиден специалистам в данной области после ознакомления с настоящим описанием и включен в объем настоящего изобретения.
Другой способ получения полипептида, предлагаемого в настоящем изобретении, представляет собой способ, который включает: получение антигенсвязывающего домена, обладающего антигенсвязывающей активность; и сцепление антигенсвязывающего домена с транспортирующим фрагментом через сайт расщепления так, чтобы антигенсвязывающая активность антигенсвязывающего домена ингибировалась ингибирующим доменом, с получением полипептида. Когда антигенсвязывающий домен сцеплен с транспортирующим фрагментом через сайт расщепления, то сайт расщепления может представлять собой «прослойку» между антигенсвязывающим доменом и транспортирующим фрагментом, или часть антигенсвязывающего домена или/и часть транспортирующего фрагмента может быть изменена и может применяться в качестве части сайта расщепления.
«Встраивание, инсерция» аминокислотной последовательности А в аминокислотную последовательность Б означает расщепление аминокислотной последовательности Б на две части без делеции и сцепление двух частей с аминокислотной последовательностью А (т.е. получение такой аминокислотной последовательности, как «первая половина аминокислотной последовательности Б - аминокислотная последовательность А вторая половина аминокислотной последовательности Б»). «Интродукция» аминокислотной последовательности А в аминокислотную последовательность Б означает расщепление аминокислотной последовательности Б на две части и связывание двух частей с аминокислотной последовательностью А. Это включает не только указанное выше «встраивание» аминокислотной последовательности А в аминокислотную последовательность Б, но также сцепление двух частей с аминокислотной последовательностью А после делеции одного или нескольких аминокислотных остатков аминокислотной последовательности Б, включая остатки, примыкающие к аминокислотной последовательности А (т.е. замену части аминокислотной последовательности Б на аминокислотную последовательность А).
Одним из вариантов осуществления изобретения являются способы получения полипептида, в которых используют однодоменное антитело в качестве антигенсвязывающего домена и используют расщепляемую протеазой последовательность в качестве сайта расщепления, которые будут описаны ниже.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, который содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом так, чтобы антигенсвязывающая активность однодоменного антитела ингибировалась с помощью ингибирующего домена транспортирующего фрагмента, с образованием полипептида-предшественника; и
(в) интродукция расщепляемой протеазой последовательности в полипептид-предщественник.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, который содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом так, чтобы антигенсвязывающая активность однодоменного антитела ингибировалась с помощью ингибирующего домена транспортирующего фрагмента, с образованием полипептида-предшественника; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между однодоменным антителом и транспортирующим фрагментом.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, который содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью; и
(б) сцепление однодоменного антитела, полученного на стадии (а), с транспортирующим фрагментом через расщепляемую протеазой последовательность так, чтобы антигенсвязывающая активность однодоменного антитела ингибировалась с помощью ингибирующего домена транспортирующего фрагмента, с образованием полипептида.
В конкретном варианте осуществления изобретения способ получения полипептида, который содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий дополнительно следующую стадию:
(г) подтверждение того, что связывающая активность однодоменного антитела, включенного в полипептид или полипептид-предшественник, в отношении антигена-мишени ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до сцепления, и уровень указанного снижения не ограничен.
В конкретном варианте осуществления изобретения способ получения полипептида, который содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий дополнительно следующую стадию:
(д) высвобождение однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившееся однодоменное антитело связывается с антигеном- мишенью.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, который представляет собой молекулу, подобную антителу IgG-типа, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с VL в качестве замены VH антитела IgG-типа или обеспечение осуществления ассоциации однодоменного антитела с VH в качестве замены VL антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело; и
(в) интродукция расщепляемой протеазой последовательности в подобную антителу IgG-типа молекулу-предшественника, которая несет однодоменное антитело.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, который представляет собой молекулу, подобную антителу IgG-типа, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с VL в качестве замены VH антитела IgG-типа или обеспечение осуществления ассоциации однодоменного антитела с VH в качестве замены VL антитела IgG-типа, в результате чего антигенсвязывающая активность однодоменного антитела ингибируется, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет однодоменное антитело; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между однодоменным антителом и константной областью антитела в подобную антителу IgG-типа молекулу-предшественника.
В одном из вариантов осуществления изобретения способ получения полипептида, который представляет собой молекулу, подобную антителу IgG-типа, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) получение однодоменного антитела, связывающегося с антигеном-мишенью; и
(б) сцепление однодоменного антитела, полученного на стадии (а), в качестве замены VH или VL антитела IgG-типа с константной областью тяжелой цепи или с константной областью легкой цепи антитела IgG-типа через расщепляемую протеазой последовательность так, чтобы антигенсвязывающая активность однодоменного антитела ингибировалась, с образованием подобной антителу IgG-типа молекулы, которая несет однодоменное антитело.
В конкретном варианте осуществления изобретения способ получения полипептида, который представляет собой молекулу, подобную антителу IgG-типа, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий дополнительно следующую стадию:
(г) подтверждение того, что связывающая активность однодоменного антитела, интродуцированного в подобную антителу IgG-типа молекулу или в подобную антителу IgG-типа молекулу-предшественника, в отношении антигена-мишени ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации или сцепления, и уровень указанного снижения не ограничен.
В конкретном варианте осуществления настоящего изобретения способ получения полипептида, представляющего собой подобную антителу IgG-типа молекулу, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий дополнительно следующую стадию:
(д) высвобождение однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившееся однодоменное антитело связывается с антигеном-мишенью.
В случае применения VH, VL или VHH в качестве ингибирующего домена способ ингибирования антигенсвязывающей активности однодоменного антитела с помощью ингибирующего домена транспортирующего фрагмента включает способ обеспечения осуществления ассоциации однодоменного антитела с VH, VL или VHH. VH, VL или VHH, который ингибирует антигенсвязывающую активность полученного однодоменного антитела, можно отбирать с помощью скрининга, обеспечивая осуществление ассоциации известного VH, VL или VHH с однодоменным антителом и сравнивая антигенсвязывающую активность однодоменного антитела до и после ассоциации.
В другом способе для ингибирования антигенсвязывающей активности однодоменного антитела с помощью конкретного VH, VL или VHH аминокислотный остаток, участвующий в ассоциации с VH, VL или VHH в однодоменном антителе, можно заменять для усиления ассоциации, или пару однодоменное антитело/ингибирующий домен, обладающую требуемым уровнем различия в антигенсвязывающей активности до и после ассоциации, можно получать также, используя однодоменное антитело, которое в исходном состоянии имеет в качестве аминокислотного остатка остаток, который может усиливать ассоциацию.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, представляющего собой подобную антителу IgG-типа молекулу, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена- мишени;
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH антитела или обеспечение осуществления ассоциации варианта однодоменного антитела с VL антитела так, чтобы антигенсвязывающая активность варианта однодоменного антитела ингибировалась, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела; и
(в) интродукция расщепляемой протеазой последовательности в подобную антителу IgG-типа молекулу-предшественника, которая несет вариант однодоменного антитела.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, представляющего собой подобную антителу IgG-типа молекулу, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с VH антитела или обеспечение осуществления ассоциации варианта однодоменного антитела с VL антитела так, чтобы антигенсвязывающая активность варианта однодоменного антитела ингибировалась, с образованием подобной антителу IgG-типа молекулы-предшественника, которая несет вариант однодоменного антитела; и
(в) интродукция расщепляемой протеазой последовательности вблизи границы между вариантом однодоменного антитела и константной областью в подобную антителу IgG-типа молекулу-предшественника.
В одном из вариантов осуществления настоящего изобретения способ получения полипептида, представляющего собой подобную антителу IgG-типа молекулу, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий следующие стадии:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, или замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени;
(б) сцепление варианта однодоменного антитела, полученного на стадии (а), с константной областью тяжелой цепи антитела IgG-типа через расщепляемую протеазой последовательность или сцепление варианта однодоменного антитела с константной областью легкой цепи антитела IgG-типа через расщепляемую протеазой последовательность так, чтобы антигенсвязывающая активность варианта однодоменного антитела ингибировалась, с образованием подобной антителу IgG-типа молекулы, которая несет вариант однодоменного антитела.
В конкретном варианте осуществления изобретения способ получения полипептида, представляющего собой подобную антителу IgG-типа молекулу, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий дополнительно следующую стадию:
(г) подтверждение того, что связывающая активность варианта однодоменного антитела, находящегося в подобной антителу IgG-типа молекуле или подобной антителу IgG-типа молекуле-предшественнике, в отношении антигена-мишени ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации или сцепления, и уровень указанного снижения не ограничен.
В конкретном варианте осуществления изобретения способ получения полипептида, представляющего собой подобную антителу IgG-типа молекулу, которая содержит транспортирующий фрагмент, имеющий ингибирующий домен, и антигенсвязывающий домен, представляет собой способ получения, включающий дополнительно следующую стадию:
(д) высвобождение варианта однодоменного антитела посредством расщепления протеазой расщепляемой протеазой последовательности и подтверждение того, что высвободившийся вариант однодоменного антитела связывается с антигеном-мишенью.
Настоящее изобретение относится также к полинуклеотидам, каждый из которых кодирует полипептид, содержащий транспортирующий фрагмент, который имеет ингибирующий домен, и антигенсвязывающий домен.
Полинуклеотид, предлагаемый в настоящем изобретении, как правило, присутствует в (или встроен в) соответствующем векторе и им трансфектируют клетки-хозяева. Вектор не ограничен конкретным вектором, если он стабильно сохраняет встроенную нуклеиновую кислоту. Например, когда в качестве хозяина применяют Е. coli, то вектор pBluescript (производства фирмы Stratagene Corp.) или подобные векторы являются предпочтительными в качестве вектора для клонирования. Можно применять различные поступающие в продажу векторы. В случае применения вектора для получения полипептида, предлагаемого в настоящем изобретении, наиболее ценным является экспрессионный вектор. Экспрессионный вектор не ограничен конкретным вектором, если он сохраняет способность обеспечивать экспрессию полипептида in vitro, в Е. coli, в культивируемых клетках или организме индивидуумов. Например, предпочтительно можно применять экспрессионный вектор, такой как вектор pBEST (производства фирмы Promega Corp.) для экспрессии in vitro; вектор рЕТ (производства фирмы Invitrogen Corp.) для экспрессии в Е. coli; вектор pME18S-FL3 (GenBank, код доступа № АВ009864) для экспрессии в культуре клеток и вектор pME18S (Takebe и др., Mol. Cell Biol. 8, 1988, сс. 466-472) для экспрессии в организме индивидуумов. Встраивание ДНК, предлагаемой в настоящем изобретении, в вектор можно осуществлять общепринятым методом, например, с помощью реакции в присутствии лигазы с использованием сайтов, распознаваемых рестриктазами (Current protocols in Molecular Biology, под ред. Ausubel и др., изд-во Publish. John Wiley & Sons, раздел 11.4-11.11, 1987).
Клетки-хозяева не ограничены конкретными клетками и различные клетки-хозяева можно применять в зависимости от поставленной цели. Примеры клеток для экспрессии полипептида могут включать бактериальные клетки (например, Streptococcus, Staphylococcus, Е. coli, Streptomyces и Bacillus subtilis), грибные клетки (например, дрожжей и Aspergillus), клетки насекомых (например, Drosophila S2 и Spodoptera SF9), клетки животных (например,СНО, COS, HeLa, С127, 3Т3, ВНК, НЕК293 и клетки бычьей меланомы) и клетки растений. Трансфекцию вектором клеток-хозяев можно осуществлять с помощью метода, известного в данной области, например, метода осаждения фосфатом кальция, метода электропорации (Current protocols in Molecular Biology под ред. Ausubel и др., изд-во Publish. John Wiley & Sons, раздел 9.1-9.9, 1987), метода с использованием липофектамина (производства фирмы GIBCO-BRL) или метода микроинъекции.
Соответствующий секреторный сигнал можно включать в представляющий интерес полипептид для того, чтобы секретировать полипептид, экспрессируемый в клетках-хозяевах, в полость эндоплазматического ретикулума, периплазматическое пространство или внеклеточное окружение. Сигнал может быть эндогенным для представляющего интерес полипептида или может представлять собой чужеродный сигнал.
Когда полипептид, предлагаемый в настоящем изобретении, секретируется в среду, то выделение полипептида в процессе производства осуществляют путем выделения среды. Когда полипептид, предлагаемый в настоящем изобретении, получают в клетках, то клетки сначала лизируют, а затем выделяют полипептид.
Методы, известные в данный области, включающие осаждение сульфатом аммония или этанолом, экстракцию кислотой, анионо- или катионообменную хроматографию, хроматографию на фосфоцеллюлозе, хроматографию гидрофобных взаимодействий, аффинную хроматографию, хроматографию на гидроксиаппатите и хроматографию лектинов можно применять для выделения и очистки полипептида, предлагаемого в настоящем изобретении, из культуры рекомбинантных клеток.
Примеры антигенсвязывающего домена, применяемого в некоторых вариантах осуществления настоящего изобретения, включают однодоменное антитело. В указанных вариантах осуществления изобретения антигенсвязывающую активность однодоменного антитела можно ингибировать путем его ассоциации с конкретным VL, путем его ассоциации с конкретным VH или путем его ассоциации с конкретным VHH. Настоящее изобретение относится также к способам скрининга указанных однодоменных антител. VL, VH или VHH, имеющий известную последовательность, например, VL, VH или VHH, имеющий последовательность, которая зарегистрирована в IMGT или базе данных Кэбота, можно применять в качестве VL, VH или VHH, который ингибирует антигенсвязывающую активность однодоменного антитела. Кроме того, можно примерять последовательность VL, VH или VHH, впервые идентифицированную в библиотеке человеческих антител или т.п. VL, VH или VHH, который ингибирует связывающую активность однодоменного антитела, можно отбирать, получая белок путем комбинации этих последовательностей и измеряя связывающую активность с помощью описанного выше метода.
В некоторых вариантах осуществления настоящего изобретения можно применять VL, VH или VHH, имеющий последовательность человеческой зародышевой линии, в качестве VL, VH или VHH, который ингибирует антигенсвязывающую активность однодоменного антитела. Например, в случае применения VL в качестве ингибирующего домена, можно применять VL, имеющий каркасные последовательности каппа-цепи, или VL, имеющий каркасные последовательности лямбда-цепи. Кроме того, можно применять VL, имеющий модифицированные каркасные последовательности, такие как объединенные каркасные последовательности каппа-цепи и лямбда-цепи.
В одном из вариантов осуществления настоящего изобретения предложен способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VL, включающий следующие стадии:
(а) получение однодоменного антитела, обладающего связывающей активностью в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с конкретным VL; и
(в) подтверждение того, что связывающая активность однодоменного антитела, ассоциированного с конкретным VL на стадии (б), в отношении антигена ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации, и уровень указанного снижения не ограничен.
В одном из вариантов осуществления настоящего изобретения предложен способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VH, включающий следующие стадии:
(а) получение однодоменного антитела, обладающего связывающей активностью в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с конкретным VH; и
(в) подтверждение того, что связывающая активность однодоменного антитела, ассоциированного с конкретным VH на стадии (б), в отношении антигена ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации, и уровень указанного снижения не ограничен.
В одном из вариантов осуществления настоящего изобретения предложен способ скрининга однодоменного антитела, антигенсвязывающую активность которого можно ингибировать путем его ассоциации с конкретным VHH, включающий следующие стадии:
(а) получение однодоменного антитела, обладающего связывающей активностью в отношении антигена-мишени;
(б) обеспечение осуществления ассоциации однодоменного антитела, полученного на стадии (а), с конкретным VHH; и
(в) подтверждение того, что связывающая активность однодоменного антитела, ассоциированного с конкретным VHH на стадии (б), в отношении антигена ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации, и уровень указанного снижения не ограничен.
Примеры способа (обеспечения) осуществления ассоциации однодоменного антитела с конкретным VL, VH или VHH включают способ создания молекулы, которая имеет последовательность однодоменного антитела в качестве замены последовательности одного из VH и VL в антителе или фрагменте антитела, который содержит и VH, и VL, таком как интактное антитело, Fab, Fab' или (Fab)2, и экспрессирует полипептид, имеющий указанную последовательность.
Настоящее изобретение относится также к способу получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем усиления ассоциации однодоменного антитела с конкретным VL, путем усиления ассоциации однодоменного антитела с конкретным VH или путем усиления ассоциации однодоменного антитела с конкретным VHH, в дополнение к скринингу однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VL, путем его ассоциации с конкретным VH или путем его ассоциации с конкретным VHH.
В одном из вариантов осуществления настоящего изобретения предложен способ получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VL, включающий следующую стадию:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VL антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени.
В конкретном варианте осуществления настоящего изобретения предложен способ получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VL, дополнительно включающий следующие стадии:
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с конкретным VL; и
(в) подтверждение того, что антигенсвязывающая активность варианта однодоменного антитела, ассоциированного с VL, ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации, и уровень указанного снижения не ограничен.
В одном из вариантов осуществления настоящего изобретения предложен способ получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VH, включающий следующую стадию:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VH антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени.
В конкретном варианте осуществления настоящего изобретения предложен способ получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VH, дополнительно включающий следующие стадии:
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с конкретным VH; и
(в) подтверждение того, что антигенсвязывающая активность варианта однодоменного антитела, ассоциированного с VH, ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации, и уровень указанного снижения не ограничен.
В одном из вариантов осуществления настоящего изобретения предложен способ получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VHH, включающий следующую стадию:
(а) замена аминокислотного остатка в однодоменном антителе, который участвует в ассоциации с VHH антитела, с получением варианта однодоменного антитела, который сохраняет связывающую активность однодоменного антитела в отношении антигена-мишени.
В конкретном варианте осуществления настоящего изобретения предложен способ получения однодоменного антитела, антигенсвязывающая активность которого ингибируется путем его ассоциации с конкретным VHH, дополнительно включающий следующие стадии:
(б) обеспечение осуществления ассоциации варианта однодоменного антитела, полученного на стадии (а), с конкретным VHH; и
(в) подтверждение того, что антигенсвязывающая активность варианта однодоменного антитела, ассоциированного с VHH, ослабляется или утрачивается.
В настоящем изобретении фраза «связывающая активность ослабляется» означает, что связывающая активность в отношении антигена-мишени снижается по сравнению с активностью до ассоциации, и уровень указанного снижения не ограничен.
Стадию обеспечения осуществления ассоциации однодоменного антитела с конкретным VL, VH или VHH осуществляют с помощью способа создания молекулы, которая имеет последовательность однодоменного антитела в качестве замены последовательности одного из VH и VL в антителе или фрагменте антитела, который содержит и VH, и VL, таком как интактное антитело, Fab, Fab' или (Fab)2, и экспрессирует полипептид, имеющий указанную последовательность.
Согласно конкретному варианту осуществления настоящего изобретения однодоменное антитело, предлагаемое в настоящем изобретении, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным VL, VH или VHH, можно получать из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом.
Согласно настоящему описанию вариант «библиотеки» может представлять собой библиотеку, обеспечивающую эффективное получение однодоменного антитела, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным VL, VH или VHH.
В настоящем описании понятие «библиотека» относится к набору множества слитых полипептидов, имеющих различные последовательности, или набору нуклеиновых кислот или полинуклеотидов, кодирующих эти слитые полипептиды. Множество слитых полипептидов, находящихся в библиотеке, представляют собой слитые полипептиды, отличающиеся по последовательности друг от друга, не имеющие сигнальной последовательности.
В настоящем описании понятие «отличающиеся по последовательности друг от друга» касательно слитых полипептидов, отличающихся по последовательности друг от друга, означает, что индивидуальные слитые полипептиды в библиотеке имеют различные последовательности. Более предпочтительно, понятие означает, что фрагменты однодоменных антител в индивидуальных слитых полипептидах в библиотеке имеют различные последовательности. В частности, количество различных последовательностей в библиотеке отражает количество независимых клонов, отличающихся по последовательности, в библиотеке, и это обозначают также как «размер библиотеки». Размер библиотеки обычной фаговой дисплейной библиотеки составляет от 106 до 1012 и может быть увеличен до 1014 с помощью методики, известной в данной области, такой как метод рибосомного дисплея. Однако фактическое количество фаговых частиц для применения в селекции с помощью пэннинга фаговой библиотеки, как правило, в 10-10000 раз превышает размер библиотеки. Указанная чрезмерная кратность, которую обозначают также как «количество эквивалентов библиотеки», означает, что от 10 до 10000 индивидуальных клонов могут иметь одинаковую аминокислотную последовательность. Таким образом, понятие «отличающиеся по последовательности друг от друга» согласно настоящему изобретению означает, что индивидуальные полипептиды в библиотеке, исключая количество эквивалентов библиотеки, имеют различные последовательности, и более конкретно означает, что библиотека имеет от 106 до 1014 молекул, предпочтительно от 107 до 1012 молекул полипептидов, отличающихся по последовательности друг от друга.
Понятие «множество» применительно к библиотеке, состоящей в основном из множества слитых полипептидов, предлагаемых в настоящем изобретении, относится к набору из двух или большего типов субстанций, таких, например, как полипептид, полинуклеотидная молекула, вектор или вирус, предлагаемые в настоящем изобретении. При условии, что, например, две или большее количество субстанций отличаются друг от друга конкретным признаком, это означает, что субстанции относятся к двум или большему количеству типов. Их примеры могут включать мутантную аминокислоту, обнаруженную в конкретном аминокислотном положении в аминокислотной последовательности. Например, два или большее количество полипептидов, предлагаемых в настоящем изобретении, которые имеют практически одинаковые, предпочтительно идентичные последовательности, за исключением конкретных мутантных аминокислот, экспонируемых на поверхности, в совершенно различных аминокислотных положениях, рассматриваются в качестве множества полипептидов, предлагаемых в настоящем изобретении. В другом примере две или большее количество молекул полинуклеотидов, предлагаемых в настоящем изобретении, которые имеют практически одинаковые, предпочтительно идентичные последовательности, за исключением оснований, кодирующих конкретные мутантные аминокислоты, экспонируемые на поверхности, в совершенно различных аминокислотных положениях, рассматриваются в качестве множества молекул полинуклеотидов, предлагаемых в настоящем изобретении.
Методы пэннинга, которые применяют для фаговых векторов, предпочтительно применяют также в качестве метода скрининга слитых полипептидов с применением с качестве показателя связывающей активности. Ген, кодирующий каждое однодоменное антитело, и ген, кодирующий CH1-домен или константную область легкой цепи антитела IgG-типа, можно соединять в соответствующей форме с получением слитого полипептида. Гены, кодирующие полученные таким образом слитые полипептиды, можно встраивать в фаговые векторы с получением фагов, которые экспрессируют на поверхности слитые полипептиды. После контакта фагов с требуемым антигеном фаги, связанные с антигеном, можно выделять, выделяя ДНК, кодирующие слитые полипептиды, которые имеют представляющую интерес связывающую активность. Эту операцию можно при необходимости повторять для обогащения слитыми полипептидами, которые имеют требуемую связывающую активность.
В дополнение к методу фагового дисплея методика, основанная на применении бесклеточной системы трансляции, методика презентации слитых полипептидов на поверхности клетки или вируса, методика, основанная на применении эмульсии, и т.п., известны в качестве методик получения слитых полипептидов путем пэннинга с применением библиотеки. Например, метод рибосомного дисплея, включающий формирование комплекса мРНК и транслируемого белка с участием рибосомы путем удаления стоп-кодона и т.д., метод дисплея кДНК или мРНК, включающий ковалентное связывание генной последовательности с транслируемым белком с использованием такого соединения, как пуромицин, или метод CIS-дисплея, включающий формирование комплекса гена и транслируемого белка с использованием связывающей белок нуклеиновой кислоты, можно применять в качестве методики, основанной на применении бесклеточной системы трансляции. Например, можно применять метод фагового дисплея, а также метод дисплея на Е. coli, метод дисплея на грамположительных бактериях, метод дрожжевого дисплея, метод дисплея на клетках млекопитающих или метод вирусного дисплея, в качестве методики презентации слитых полипептидов на поверхности клетки или вируса. Например, метод вирусного дисплея in vitro с использованием эмульсии, содержащей ген и связанную с трансляцией молекулу, можно применять в качестве методики, основанной на применении эмульсии. Эти методы уже известные в данной области (Nat Biotechnol. 18(12), декабрь 2000 г., сс. 1287-1292, Nucleic Acids Res. 34(19), 2006, е127, Proc Natl Acad Sci USA. 101(9), 2 марта 2004 г., сс. 2806-2810, Proc Natl Acad Sci USA. 101(25), 22 июня 2004 г., сс. 9193-9198, Protein Eng Des Sel. 21(4), апреля 2008 г., сс. 247-255, Proc Natl Acad Sci USA. 97(20), 26 сентября 2000 г., сс. 10701-10705, MAbs. 2(5), сентябрь-октябрь 2010 г., сс. 508-518, Methods Mol Biol. 911, 2012, сс. 183-198).
Партнера по ассоциации ингибирующего домена, сцепленного со вторым поддерживающим ассоциацию доменом, можно применять в способе получения представляющего интерес однодоменного антитела из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом.
В настоящем описании понятие «первый поддерживающий ассоциацию домен» и «второй поддерживающий ассоциацию домен» относится к доменам, которые могут взаимодействовать друг с другом через связь, такую как гидрофобная связь, водородная связь или ионная связь, с образованием продукта ассоциации. Предпочтительные примеры первого поддерживающего ассоциацию домена и второго поддерживающего ассоциацию домена включают (но, не ограничиваясь только ими) константные области легкой цепи (CL) и СН1-домены константных областей тяжелой цепи антитела.
Первый поддерживающий ассоциацию домен и второй поддерживающий ассоциацию домен могут взаимодействовать друг с другом и образовывать ассоциацию слитого полипептида с партнером по ассоциации, вне зависимости от степени способности к ассоциации (степени сочетательности) между однодоменным антителом и ингибирующий доменом.
В альтернативном варианте осуществления настоящего изобретения предложена библиотека, содержащая множество слитых полипептидов однодоменных антител, сцепленных с константной областью легкой цепи антитела IgG-типа, в которой однодоменные антитела включают однодоменное антитело, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным VL, VH или VHH, и способ скрининга библиотеки в отношении однодоменного антитела, антигенсвязывающую активность которого можно ингибировать, или она может утрачиваться при ассоциации с конкретным VL, VH или VHH.
В конкретном варианте осуществления изобретения, который представлен на фиг. 9А(1), 9А(2), 9А(3), 9Б и 9В,
(1) слитые полипептиды однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом, экспонируют на поверхности фагов или т.п. с использованием метода дисплея, такого как фаговый дисплей.
(2) Получают партнера по ассоциации ингибирующего домена, сцепленного со вторым поддерживающим ассоциацию доменом, и осуществляют ассоциацию слитых полипептидов с партнером по ассоциации. Отбирают слитый полипептид, который не связывается с антигеном-мишенью или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в указанном состоянии слитого полипептида, ассоциированного с партнером по ассоциации.
(3) Ассоциацию однодоменного антитела в слитом полипептиде, отобранном в подпункте (2), с ингибирующий доменом в партнере по ассоциации аннулируют. Отбирают слитый полипептид, который связывается с антигеном-мишенью или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, в состоянии, в котором однодоменное антитело не находится в ассоциации с ингибирующим доменом.
В этом контексте, например, способ расщепления партнера по ассоциации вблизи границы между ингибирующим доменом и вторым поддерживающим ассоциацию доменом, который проиллюстрирован на фиг. 9Б, или способ расщепления слитого полипептида вблизи границы между однодоменным антителом и первым поддерживающим ассоциацию доменом, который проиллюстрирован на фиг. 9В, можно применять в качестве способа аннулирования ассоциации однодоменного антитела с ингибирующим доменом.
Другим вариантом осуществления настоящего изобретения является способ, включающий, как продемонстрировано на фиг. 9Г, сравнение различия в связывающей активности однодоменного антитела в случае, когда однодоменное антитело и ингибирующий домен экспрессируют совместно, и в случае, когда однодоменное антитело экспрессируют таким образом, чтобы не имела место его совместная экспрессия с ингибирующий доменом, вместо сравнения различия в связывающей активности однодоменного антитела в случае аннулированной ассоциации и неаннулированной ассоциации однодоменного антитела с ингибирующим доменом, как продемонстрировано на фиг. 9А-9В.
Как продемонстрировано на фиг. 9Г(1), однодоменное антитело и ингибирующий домен экспрессируют совместно таким образом, чтобы происходила ассоциация. Отбирают слитый полипептид, содержащий однодоменное антитело, которое не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в указанном состоянии. Как продемонстрировано на фиг. 9Г(2), 9Г(2') и 9Г(2''), однодоменное антитело экспрессируют так, чтобы вместе с ним не экспрессировался ингибирующий домен. Отбирают слитый полипептид, содержащий однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, в указанном состоянии. Таким образом, в результате скрининга можно отбирать однодоменное антитело, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным ингибирующим доменом, например, VH, VL или VHH, из библиотеки, которая содержит множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. Альтернативно этому, однодоменное антитело экспрессируют так, чтобы вместе с ним не экспрессировался ингибирующий домен. Отбирают полипептид, содержащий однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, в указанном состоянии. Затем однодоменное антитело и ингибирующий домен экспрессируют совместно таким образом, чтобы происходила ассоциации. Отбирают полипептид, содержащий однодоменное антитело, которое не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в указанном состоянии. С помощью этого способа можно также отбирать в результате скрининга однодоменное антитело, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным ингибирующим доменом, например, VH, VL или VHH, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом. Альтернативно этому, как продемонстрировано на фиг. 9Г(2), 9Г(2') и 9Г(2''), однодоменное антитело экспрессируют так, чтобы вместе с ним не экспрессировался ингибирующий домен (экспрессируют только однодоменное антитело; экспрессируют только слитый полипептид, содержащий однодоменное антитело и первый поддерживающий ассоциацию домен; или слитый полипептид, содержащий однодоменное антитело и первый поддерживающий ассоциацию домен, объединяют путем ассоциации только со вторым поддерживающим ассоциацию доменом), и отбирают слитый полипептид, содержащий однодоменное антитело, которое связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, в указанном состоянии. Затем, как продемонстрировано на фиг. 9Г(1), экспрессируют совместно однодоменное антитело в отобранном слитом полипептиде и ингибирующий домен с образованием ассоциации. Отбирают слитый полипептид, содержащий однодоменное антитело, которое не связывается с антигеном или имеет антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его, в указанном состоянии. В результате можно также отбирать с помощью скрининга однодоменное антитело, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным ингибирующим доменом, например, VH, VL или VHH, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с первым поддерживающим ассоциацию доменом.
«Антигенсвязывающий активность, которая соответствует предварительно установленному уровню или ниже его» может означать, например, антигенсвязывающую активность, которая снижается ниже предварительно установленного референс-уровня, когда антигенсвязывающую активность измеряют с помощью метода, указанного в настоящем описании. Аналогично этому, «антигенсвязывающий активность, которая соответствует предварительно установленному уровню или выше его» может означать, например, антигенсвязывающую активность, которая превышает предварительно установленный референс-уровень, когда антигенсвязывающую активность измеряют с помощью метода, указанного в настоящем описании. Слитый полипептид, имеющий антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его, связывается сильнее с антигеном, чем слитый полипептид, имеющий антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его.
Слитый полипептид, отобранный в описанном выше подпункте (3), содержит однодоменное антитело, которое не обладает или обладает слабой антигенсвязывающей активностью в состоянии ассоциации с ингибирующим доменом и имеет (или имеет сильную) антигенсвязывающую активность в состоянии отсутствия ассоциации с ингибирующим доменом. Последовательность слитого полипептида, отобранного указанным способом, можно анализировать для дополнительного получения данных о последовательности однодоменного антитела, содержащегося в нем. Таким образом можно получать однодоменное антитело.
Для способов скрининга в отношении слитого полипептида, содержащего представляющее интерес однодоменное антитело, путем использования слитых полипептидов и партнера по ассоциации, важно сравнивать антигенсвязывающую активность однодоменного антитела в состоянии ассоциации и в состоянии отсутствия ассоциации с ингибирующим доменом. Как продемонстрировано на фиг. 9А(2') и 9А(3'), сначала подтверждают антигенсвязывающую активность экспонированных слитых полипептидов и отбирают слитые полипептиды, которые связываются с антигеном или имеют антигенсвязывающую активность, которая соответствует предварительно установленному уровню или выше его. Затем дают осуществляться ассоциации отобранных таким образом слитых полипептидов с партнером по ассоциации. Отбирают слитые полипептиды, которые не связываются с антигеном или имеют антигенсвязывающую активность, которая соответствует предварительно установленному уровню или ниже его в указанном состоянии. С помощью указанного способа можно получать также слитый полипептид, содержащий представляющее интерес однодоменное антитело.
Ниже в настоящем описании представлено несколько вариантов осуществления изобретения, в которых использовали СН1-домен антитела IgG-типа в качестве первого поддерживающего ассоциацию домена и использовали CL антитела IgG-типа в качестве второго поддерживающего ассоциацию домена.
Слитый полипептид, содержащий представляющее интерес однодоменное антитело, можно подвергать скринингу, используя библиотеку, содержащую множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с СН1-доменом антитела IgG-типа.
Некоторыми вариантами осуществления настоящего изобретения является библиотека, содержащая множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с СН1-доменом антитела IgG-типа, где однодоменные антитела включают однодоменное антитело, антигенсвязывающая активность которого ингибируется или утрачивается при его ассоциации с конкретным VL, VH или VHH, и способ скрининга библиотеки в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или утрачиваться при его ассоциации с конкретным VL, VH или VHH.
Конкретным вариантом осуществления настоящего изобретения является способ скрининга в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться при его ассоциации с конкретным VL, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с СН1-доменом антитела IgG-типа. В частности, в настоящем изобретении предложен способ скрининга в отношении однодоменного антитела, включающий следующие стадии:
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов из библиотеки, предлагаемой в настоящем изобретении;
(б) получение партнера по ассоциации, включающего константную область легкой цепи антитела IgG-типа, слитую с конкретным VL;
(в) обеспечение осуществления ассоциации каждого из слитых полипептидов, экспонированных на стадии (а), с партнером по ассоциации, полученным на стадии (б), и отбор слитого(ых) полипептида(ов), который(ые) не связывается(ются) с антигеном или имеет(ют) антигенсвязывающую активность, соответствующую предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело ассоциировано с VL; и
(г) выбор из слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, соответствующую предварительно установленному уровню или выше его в состоянии, в котором содержащееся в нем однодоменное антитело не находится в ассоциации с VL.
Партнер по ассоциации, полученный на стадии (б), дополнительно содержит расщепляемую протеазой последовательность. В этом случае на стадии (г) ассоциация однодоменного антитела с VL аннулируется обработкой протеазой, и антигенсвязывающую активность однодоменного антитела можно подтверждать в состоянии, в котором однодоменное антитело не находится в ассоциации с VL. Расщепляемая протеазой последовательность в партнере по ассоциации не ограничена ее положением, если ассоциация однодоменного антитела с VL аннулируется расщеплением. В качестве примера положения расщепляемая протеазой последовательность может быть локализована, например, вблизи границы между VL и константной областью легкой цепи антитела IgG-типа в партнере по ассоциации, предпочтительно в любом положении между аминокислотой в положении 96 (нумерация Кэбота) VL и аминокислотой в положении 130 (EU-нумерация) (нумерация положения 130 по Кэботу) константной области легкой цепи антитела, более предпочтительно в любом положении между аминокислотой в положении 104 (нумерация Кэбота) VL и аминокислотой в положении 113 (EU-нумерация) (нумерация положения 113 по Кэботу) константной области легкой цепи антитела.
Вместо применения партнера по ассоциации, содержащего расщепляемую протеазой последовательность, расщепляемую протеазой последовательность можно интродуцировать в слитые полипептиды в библиотеке, и слитые полипептиды можно расщеплять протеазой так, чтобы аннулировать ассоциацию однодоменного антитела с VL. Положение расщепляемой протеазой последовательности в каждом слитом полипептиде не ограничено ее положением, если ассоциация однодоменного антитела с VL аннулируется расщеплением, и однодоменное антитело сохраняет свою антигенсвязывающую активность после расщепления. В качестве примера положения расщепляемая протеазой последовательность может быть локализована, например, вблизи границы между однодоменным антителом и СН1-доменом антитела IgG-типа в слитом полипептиде.
На стадии (г) полноразмерный(ые) слитый(ые) полипептид(ы), отобранный(ые) на стадии (в), или их фрагменты, содержащие однодоменные антитела, можно вновь экспонировать и антигенсвязывающую активность однодоменного антитела можно подтверждать в состоянии, в котором однодоменное антитело не находится в ассоциации с VL.
Конкретным вариантом осуществления изобретения является способ скрининга в отношении слитого полипептида, содержащего однодоменное антитело, антигенсвязывающая активность которого может ингибироваться или может утрачиваться при его ассоциации с конкретным VH, из библиотеки, содержащей множество слитых полипептидов однодоменных антител, каждое из которых сцеплено с константной областью легкой цепи антитела IgG-типа. В частности, в настоящем изобретении предложен способ скрининга в отношении однодоменного антитела, включающий следующие стадии:
(а) обеспечение осуществления экспонирования in vitro слитых полипептидов из библиотеки, предлагаемой в настоящем изобретении;
(б) получение партнера по ассоциации, включающего CHI-домен антитела IgG-типа, слитый с конкретным VH;
(в) обеспечение осуществления ассоциации слитых полипептидов, экспонированных на стадии (а), с партнером по ассоциации, полученным на стадии (б), и отбор слитого(ых) полипептида(ов), который(ые) не связывается(ются) с антигеном или имеет(ют) антигенсвязывающую активность, соответствующую предварительно установленному уровню или ниже его в состоянии, в котором однодоменное антитело ассоциировано с VH; и
(г) выбор из слитого(ых) полипептида(ов), отобранного(ых) на стадии (в), слитого полипептида, который связывается с антигеном или имеет антигенсвязывающую активность, соответствующую предварительно установленному уровню или выше его в состоянии, в котором содержащееся в нем однодоменное антитело не находится в ассоциации с VH.
Партнер по ассоциации, полученный на стадии (б), дополнительно содержит расщепляемую протеазой последовательность. В этом случае на стадии (г) ассоциация однодоменного антитела с VH аннулируется обработкой протеазой, и антигенсвязывающую активность однодоменного антитела можно подтверждать в состоянии, в котором однодоменное антитело не находится в ассоциации с VH. Расщепляемая протеазой последовательность в партнере по ассоциации не ограничена ее положением, если ассоциация однодоменного антитела с VH аннулируется расщеплением. В качестве примера положения расщепляемая протеазой последовательность может быть локализована, например, вблизи границы между VH и СН1-доменом антитела IgG-типа в партнере по ассоциации, предпочтительно в любом положении между аминокислотой в положении 101 (нумерация Кэбота) VH и аминокислотой в положении 140 (EU-нумерация) константной области тяжелой цепи антитела, более предпочтительно в любом положении между аминокислотой в положении 109 (нумерация Кэбота) VH и аминокислотой в положении 122 (EU-нумерация) константной области тяжелой цепи антитела.
Вместо применения партнера по ассоциации, содержащего расщепляемую протеазой последовательность, расщепляемую протеазой последовательность можно интродуцировать в слитые полипептиды в библиотеке, и слитые полипептиды можно расщеплять протеазой так, чтобы аннулировать ассоциацию однодоменного антитела с VH. Положение расщепляемой протеазой последовательности в каждом слитом полипептиде не ограничено ее положением, если ассоциация однодоменного антитела с VH аннулируется расщеплением, и однодоменное антитело сохраняет свою антигенсвязывающую активность после расщепления. В качестве примера положения расщепляемая протеазой последовательность может быть локализована, например, вблизи границы между однодоменным антителом и константной областью легкой цепи антитела IgG-типа в слитом полипептиде.
На стадии (г) полноразмерный(ые) слитый(ые) полипептид(ы), отобранный(ые) на стадии (в), или их фрагменты, содержащие однодоменные антитела, можно вновь экспонировать и антигенсвязывающую активность однодоменного антитела можно подтверждать в состоянии, в котором однодоменное антитело не находится в ассоциации с VH.
Аминокислоты, содержащиеся в каждой аминокислотной последовательности, указанной в настоящем описании, можно подвергать посттрансляционной модификации (например, модификация N-концевого глутамина в пироглутаминовую кислоту путем пироглутамилирования представляет собой модификацию, хорошо известную специалистам в данной области). Как должно быть очевидно, указанная аминокислотная последовательность, содержащая подвергнутую пост-трансляционной модификации аминокислоту, также относится к аминокислотной последовательности, предлагаемой в настоящем изобретении.
Специалистам в данной области должно быть очевидно, что произвольные комбинации одного или нескольких вариантов осуществления изобретения, представленных в настоящем описании, можно включать также в настоящее изобретение, если это технически не противоречит основам, известным специалистам в данной области техники.
Примеры
Ниже представлены примеры способа и композиций, предлагаемых в настоящем изобретении. Должно быть очевидно, что можно осуществлять различные другие варианты в свете упомянутого выше общего описания изобретения.
Пример 1. Проблема, связанная с известными активируемыми протеазой антителами
Описан способ получения антитела, которое проявляет антигенсвязывающую активность только после расщепления протеазой, которая экспрессируется в области повреждения, такой как раковая ткань или воспалительная ткань. Указанное антитело, называемое Probody, представляет собой молекулу антитела, представленную на фиг. 1, антигенсвязывающая активность которого ингибируется путем соединения антитела с пептидом, маскирующим антигенсвязывающий сайт антитела, через линкер, который расщепляется протеазой, экспрессируемой в области повреждения (NPL 18). Маскирующий пептид отделяется от Probody в результате расщепления компонента линкера с помощью протеазы, которая экспрессируется в патологически измененной области-мишени, так, что полученная в результате молекула антитела восстанавливает свою антигенсвязывающую активность и приобретает способность связываться с антигеном в патологически измененной ткани-мишени.
Предполагается, что Probody может связываться с антигеном избирательно в патологически измененной области-мишени с помощью описанного выше механизма и поэтому имеет расширенное терапевтическое окно. Однако, поскольку расщепление антитела протеазой является необратимым в случае Probody, то существует вероятность того, что антитело, расщепленное в патологически измененной области, может возвращаться в кровь из патологически измененной области и связываться с антигеном, который экспрессируется в здоровой ткани, в результате попадания антитела в здоровые ткани через кровоток. Probody, активированное протеазой, сохраняет Fc-область, такую же как у Probody до активации, и поэтому обладает длительным временем удержания в крови. Поэтому антитело, активированное протеазой, которая экспрессируется в патологически измененной области, может длительное время циркулировать в крови. Протеаза, даже если она характеризуется повышенным уровнем экспрессии в патологически измененной области, экспрессируется также и в здоровых тканях, и свободная протеаза, продуцируемая в патологически измененной области, может проникать в кровь (The Chinese-German Journal of Clinical Oncology т. 3, №. 2, июнь 2004 г., cc. 78-80). Таким образом, Probody может активироваться указанной свободной протеазой. Следовательно, существует вероятность того, что Probody будет активироваться в области, отличенной от патологически измененной области. Активированное таким образом Probody также циркулирует в крови в течение длительного времени. Таким образом, существует возможность того, что происходит постоянная активация Probody в патологически измененной области, в здоровых тканях и в крови, и активированное Probody, если имеет продолжительное время удержания в крови, накапливается в крови. Активированное Probody, накопленное в крови, может вызывать побочные действия путем связывания с антигеном который экспрессируется в здоровых тканях (фиг. 2).
Антигенсвязывающая активность Probody ингибируется маскирующим пептидом, сцепленным с антителом через линкер, однако антигенсвязывающая активность не полностью ингибируется. Probody находится в равновесии между состоянием, в котором маскирующий пептид, сцепленный через линкер, связан с антигенсвязывающим сайтом, и состоянием, в котором маскирующий пептид отделен от него в результате диссоциации. Молекула в диссоциированном состоянии может связываться с антигеном (фиг. 3). Фактически, Probody, представляющее собой антитело к EGFR, описанное в NPL 17, обладает связывающей активностью в отношении EGFR даже до расщепления протеазой линкера. Хотя антигенсвязывающая активность повышается в 30-100 раз в результате расщепления линкера протеазой, Probody, которое присутствует в высокой концентрации до активации, может вызывать побочные действия при связывании с антигеном, который экспрессируется в здоровых тканях, поскольку связывающая активность Probody до активации составляет от 1/30 до 1/100 от связывающей активности активированного Probody.
В Probody применяют искусственный пептид для маскировки антигенсвязывающего сайта антитела. Искусственный пептид имеет последовательность, которая отсутствует в нативных человеческих белках, и поэтому может обладать иммуногенностью для людей. Известно, что указанная иммуногенность снижает воздействия лекарственных средств на основе антител путем индукции антител к лекарственным средствам (Blood. 127 (13), 31 марта 2016 г., сс. 1633-1641).
Возможные антитела к Probody в виде антител к лекарственным средствам представляют собой антитело к лекарственному средству против комплекса антитела и маскирующего пептида (Probody до активации), антитело к лекарственному средству против антитела, отделившегося в результате диссоциации от маскирующего пептида (активированное Probody), антитело к лекарственному средству против маскирующего пептида (маскирующий пептид, отделившийся в результате диссоциации от активированного Probody) и т.п. Среди них антитело к лекарственному средству против маскирующего пептида (антитело к маскирующему пептиду) может связываться с маскирующим пептидом Probody до активации и тем самым активировать Probody даже без расщепления протеазой (фиг. 4). Probody, активированное антителом к маскирующему пептиду, может вызывать побочные действия при связывании с антигеном, который экспрессируется в здоровых тканях.
Пример 2. Концепция, касающаяся активируемого протеазой полипептида, содержащего однодоменное антитело
Как продемонстрировано в примере 1, с технологией Probody связаны следующие проблемы:
1. Probody, активированное расщеплением протеазой, имеет длительное время удержания в крови
2. Даже до расщепления протеазой Probody имеет связывающую активность в отношении антигена.
3. Маскирующий пептид представляет собой искусственную нечеловеческую последовательность и может индуцировать антитела против маскирующего пептида.
При создании изобретения высказано предположение о том, что путь, который можно применять для решения указанных проблем и создания лекарственного средства на основе антитела, обладающего активностью в патологически измененных областей, должен удовлетворять следующим условиям:
1. Антигенсвязывающий домен, активированный расщеплением протеазой, имеет короткое время полужизни в крови.
2. Антигенсвязывающая активность молекулы до расщепления протеазой является минимизированной.
3. Маскирующий пептид, имеющий искусственную нечеловеческую последовательность, не применяется.
При создании настоящего изобретения создана молекула, представленная на фиг. 5, в качестве одного примера полипептида, который удовлетворяет описанным выше условиям. Полипептид с антигенсвязывающим доменом, сцепленным с транспортирующим фрагментом, имеет длительное время полужизни и не связывается с антигеном, поскольку антигенсвязывающая активность антигенсвязывающего домена заингибирована (А). Антигенсвязывающий домен высвобождается и высвободившийся таким образом антигенсвязывающий домен восстанавливает свою антигенсвязывающую активность и кроме того, имеет короткое время полужизни (Б). Полипептид, представленный на фиг. 5, имеет различные варианты. В случае применения подобной антителу IgG-типа молекулы, полипептид можно получать с помощью способа получения, проиллюстрированного на фиг. 6. Сначала получают однодоменное антитело (например, VH или VHH), связывающееся с антигеном-мишенью (А). Дают осуществляться ассоциации однодоменного антитела в качестве замены одного из доменов VH и VL в антителе IgG-типа, которое имеет последовательность зародышевой линии, с одним другим доменом (VL или VH) с получением подобной антителу IgG-типа молекулы (Б). Расщепляемую протеазой последовательность интродуцируют в подобную антителу IgG-типа молекулу (В). Примеры положения, в которое осуществляют интродукцию, включают положение вблизи границы между включенным однодоменным антителом (VH или VHH) и константной областью (СН1 или CL).
Однодоменное антитело имеет антигенсвязывающую активность в индивидуальном состоянии, но утрачивает свою антигенсвязывающую активность после формирования вариабельной области с использованием VL, VH, VHH или т.п. VL или VH имеет последовательность нативного антитела, имеющего последовательность зародышевой линии, и поэтому имеет низкий риск иммуногенности и с низкой долей вероятности может индуцировать антитело к лекарственному средству, распознающее указанный домен VL или VH. В случае формирования вариабельной области однодоменного антитела с использованием VHH, гуманизация VHH снижает риск иммуногенности и снижает вероятность индукции антитела к лекарственному средству, распознающего гуманизированный VHH. Расщепляемая протеазой последовательность, встроенная в подобную антителу IgG-типа молекулу, расщепляется протеазой таким образом, чтобы высвобождалось однодоменное антитело. Высвободившееся однодоменное антитело обладает антигенсвязывающей активностью. Подобная антителу IgG-типа молекула до расщепления структурно схожа с каноническими молекулами IgG и поэтому имеет продолжительное время удержания в крови, в то время как однодоменное антитело, высвободившееся в результате расщепления протеазой, имеет молекулярную массу примерно 13 кДа, у него не сохраняется Fc-область и поэтому оно быстро выводится посредством почечной экскреции. Фактически время полужизни полноразмерного IgG составляет порядка 2-3 недель (Blood. 127 (13), 31 марта 2016 г., 1633-1641), в то время как время полужизни однодоменного антитела составляет примерно 2 ч (Antibodies 4 (3), 2015, сс. 141-156). Поэтому антигенсвязывающая молекула, активированная протеазой, имеет короткое время полужизни в крови и с низкой долей вероятности может связываться с антигенами в здоровых тканях.
Когда однодоменное антитело представляет собой VL, то можно применять описанную выше концепцию, например, интродуцируя расщепляемую протеазой последовательность вблизи границы между VL и CL.
Пример 3. Получение активируемого протеазой полипептида с использованием VHH, связывающегося с IL-6R
3-1 Получение полипептида с включенным VHH, который связывается с IL-6R.
Экспрессионный вектор, кодирующий конструкцию IL6R90-G1m (SEQ ID NO: 2), которая содержит IL6R90 (SEQ ID NO: 1), VHH, который обладает связывающей и нейтрализующей активностью в отношении человеческого IL-6R, описанный в WO 2010/115998, слитый с константной областью человеческого IgGI (СН1-шарнир-СН2-СН3), получали с помощью метода, известного специалистам в данной области.
Экспрессионные векторы, кодирующие VK1-39-k0MT (SEQ ID NO: 3), VK2-28-k0MT (SEQ ID NO: 4), VK3-20-k0MT (SEQ ID NO: 5), VL1-40-lamL (SEQ ID NO: 6), VL1-44-lamL (SEQ ID NO: 7), VL2-14-lamL (SEQ ID NO: 8), VL3-21-lamL (SEQ ID NO: 9), k0 (SEQ ID NO: 10) и lamL (SEQ ID NO: 11) в качестве легких цепей (вариабельная область - константная область) различных подклассов, имеющих последовательность человеческой зародышевой линии, получали с помощью метода, известного специалистам в данной области.
Подобные антителу IgG-типа молекулы IL6R90-G1m/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 3), IL6R90-G1m/VK2-28-k0MT (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 4), IL6R90-G1m/VK3-20-k0MT (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 5), IL6R90-G1m/VL1-40-lamL (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 6), IL6R90-G1m/VL1-44-lamL (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 7), IL6R90-G1m/VL2-14-lamL (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 8), IL6R90-G1m/VL3-21-lamL (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 9), IL6R90-G1m/k0 (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 10) и IL6R90-G1m/lamL (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 11) экспрессировали путем кратковременной экспрессии с использованием клеток FreeStyle 293 (фирма Invitrogen Corp.) с помощью метода, известного специалистам в данной области, и очищали с помощью метода, известного специалистам в данной области, используя белок А.
3-2 Оценка связывания с IL-6R полипептида, в который включен VHH, связывающийся с человеческим IL-6R
Связывающую активность IL6R90-G1m/VK1-39-k0MT, IL6R90-G1m/VK2-28-k0MT, IL6R90-G1m/VK3-20-k0MT, IL6R90-G1m/VL1-40-lamL, IL6R90-G1m/VL1-44-lamL, IL6R90-G1m/VL2-14-lamL, IL6R90-G1m/VL3-21-lamL, IL6R90-G1m/k0 и IL6R90-G1m/lamL в отношении IL-6R оценивали следующим методом.
Применяемый в качестве антигена рекомбинантный человеческий IL-6R получали следующим образом: линию СНО, стабильно экспрессирующую растворимый человеческий IL-6R (ниже в настоящем описании обозначен также как hsIL-6R, IL-6R или IL-6R), состоящий из аминокислотной последовательности, простирающейся от положения 1 до 357, начиная с N-конца, который описан в J. Immunol. 152, 1994, сс. 4958-4968, конструировали с помощью метода, известного специалистам в данной области, культивировали и давали осуществляться экспрессии hsIL-6R. hsIL-6R очищали из полученного супернатанта культуры с помощью 2 стадий, таких как хроматография на колонке Blue Сефароза 6 FF и гель-фильтрация. Фракцию, элюированную в качестве основного пика на финальной стадии, применяли в качестве конечного очищенного продукта.
Оценку связывания с hsIL-6R каждой молекулы осуществляли с помощью системы Octet HTX (фирма Pall ForteBio Corp.). В частности, каждой молекуле давали связываться с биосенсором/белком А (ProA) (фирма Pall ForteBio Corp., 18-5013) и давали осуществляться взаимодействию hsIL-6R с ней, после чего оценивали связывание при 30°С. Сенсограммы, на которых продемонстрированы ответы, характеризующие связывание в реальном времени, полученные с помощью Octet HTX, представлены на фиг. 10. Установлено, что IL6R90-G1m/k0 и IL6R90-G1m/lamL, у которых отсутствовал VL, связывались с hsIL-6R, в то время как IL6R90-G1m/VK1-39-k0MT, IL6R90-G1m/VK2-28-k0MT, IL6R90-G1m/VK3-20-k0MT, IL6R90-G1m/VL1-40-lamL, IL6R90-G1m/VL1-44-lamL и IL6R90-G1m/VL2-14-lamL содержащие вариабельную область, сформированную с использованием VL, не обладали способностью связываться с hsIL-6R. Исходя из этого, установлено, что VHH, обладающий связывающей активностью в отношении человеческого IL-6R, может утрачивать свою IL-6R-связывающую активность при формировании вариабельной области посредством ассоциации с VL.
3-3 Интродукция расщепляемой протеазой последовательности в полипептид с включенным VHH, связывающимся с IL-6R
Проводили исследование, в котором осуществляли инсерцию расщепляемой протеазой последовательности вблизи границы между антителом к человеческому IL-6R VHH IL6R90 и СН1. Создавали шесть типов тяжелых цепей, которые продемонстрированы на фиг. 11, при этом последовательность пептида A (SEQ ID NO: 12), описанную последовательность, расщепляемую специфически экспрессируемой при раке урокиназой (uPA) и MT-SP1, встраивали в 3 сайта вблизи границы между IL6R90 и СН1 с помощью глицин-серинового линкера или без него. Экспрессионные векторы, кодирующие IL6R90H1001 (SEQ ID NO: 13), IL6R90H1002 (SEQ ID NO: 14), IL6R90H1003 (SEQ ID NO: 15), IL6R90H1004 (SEQ ID NO: 16), IL6R90H1005 (SEQ ID NO: 17) и IL6R90H1006 (SEQ ID NO: 18), получали с помощью метода, известного специалистам в данной области.
Подобные антителу IgG-типа молекулы IL6R90H1001/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 13, легкая цепь: SEQ ID NO: 3), IL6R90H1002/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 14, легкая цепь: SEQ ID NO: 3), IL6R90H1003/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 15, легкая цепь: SEQ ID NO: 3), IL6R90H1004/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 16, легкая цепь: SEQ ID NO: 3), IL6R90H1005/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 17, легкая цепь: SEQ ID NO: 3) и IL6R90H1006/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 18, легкая цепь: SEQ ID NO: 3) экспрессировали путем кратковременной экспрессии с использованием указанных тяжелых цепей и VK1-39-k0MT (SEQ ID NO: 3) в качестве легкой цепи, применяя клетки FreeStyle 293 (фирма Invitrogen Corp.), с помощью метода, известного специалистам в данной области, и очищали с помощью метода, известного специалистам в данной области, используя белок А.
3-4 Активация полипептида. несущего расщепляемую протеазой последовательность, с помощью расщепления протеазой
Проверяли, будет ли происходить высвобождение VHH из конструкций IL6R90H1001/VK1-39-k0MT, IL6R90H1002/VK1-39-k0MT, IL6R90H1003/VK1-39-k0MT, IL6R90H1004/VK1-39-k0MT, IL6R90H1005/VK1-39-k0MT и IL6R90H1006/VK1-39-k0MT, обладающих связывающей активностью в отношении IL-6R, при расщеплении протеазой.
Растворимый человеческий IL-6R получали с помощью метода, известного специалистам в данной области. Полученный растворимый человеческий IL-6R биотинилировали с помощью метода, известного специалистам в данной области.
Для присоединения биотина к С-концу растворимого человеческого IL-6R (который обозначают также как hsIL-6R или растворимый человеческий IL-6R; SEQ ID NO: 35) генный фрагмент, кодирующий специфическую последовательность (последовательность AviTag (Avi-метка); SEQ ID NO: 36), подлежащую биотинилированию с помощью биотинлигазы, связывали через генной фрагмент, кодирующий линкер, в прямом направлении относительно генного фрагмента, кодирующего hsIL-6R. Генный фрагмент, кодирующий белок, который содержит hsIL-6R, сцепленный с последовательностью Avi-метки (hsIL-6R-Avitag; SEQ ID NO: 37), встраивали в вектор для экспрессии в клетках животных. Сконструированным плазмидным вектором трансфектировали клетки FreeStyle 293 (фирма Invitrogen Corp.), используя 293Fectin (фирма Invitrogen Corp.). При осуществлении этой операции клетки совместно трансфектировали геном для экспрессии EBNA1 (SEQ ID NO: 57) и геном для экспрессии биотинлигазы (BirA; SEQ ID NO: 58), и к ним дополнительно добавляли биотин для мечения с помощью биотина hsIL-6R-Avitag. Клетки, трансфектированные с использованием описанных выше процедур, культивировали при 37°С в атмосфере с 8% СО2; и давали представляющему интерес белку (hsIL-6R-ВАР1) секретироваться в супернатант культуры. Раствор указанной клеточной культуры фильтровали, используя емкость с 0,22-микрометровым верхним фильтром, для получения супернатанта культуры.
Антитело к человеческому IL-6R иммобилизовывали на HiTrap NHS-активированной HP (фирма GE Healthcare Japan Corp.) согласно протоколу производителя для получения колонки (колонка с антителом к человеческому IL-6R). Супернатант культуры вносили на колонку с антителом к человеческому IL-6R, уравновешенную TBS, с последующей элюцией связанного hsIL-6R с помощью 2М аргинина (pH 4,0). Затем элюат из колонки с антителом к человеческому IL-6R разводили TBS и затем вносили на авидиновую колонку SoftLink (фирма Promega Corp.), уравновешенную TBS, с последующей элюцией hsIL-6R-BAP1 с помощью 5 мМ биотина, 50 мМ Трис-НС1 (рН 8,0) и 2М аргинина (рН 4,0). Из этого элюата удаляли агрегаты hsIL-6R-BAP1 с помощью гель-фильтрации, используя Супердекс 200 (фирма GE Healthcare Japan Corp.) для получения очищенного hsIL-6R-BAP1, осуществляя замену буфера на D-ЗФР и 0,05% CHAPS.
В качестве протеазы использовали рекомбинантную человеческую матриптазу/каталитический домен ST14 (фирма R&D Systems, Inc., 3946-SE-010). 12,5нМ протеазу и 100 мкг/мл каждой подобной антителу IgG-типа молекулы инкубировали в ЗФР при 37°С в течение 20 ч. Затем расщепление протеазой оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 12. В результате для конструкций IL6R90H1002/VK1-39-k0MT, IL6R90H1004/VK1-39-k0MT, IL6R90H1005/VK1-39-k0MT и IL6R90H1006/VK1-39-k0MT подтверждено расщепление протеазой расщепляемой протеазой последовательности вблизи границы между VHH и константной областью тяжелой цепи.
Затем осуществляли оценку связывания с IL-6R VHH, который высвобождался после обработки протеазой, с использованием Octet HTX (фирма Pall ForteBio Corp.). В частности, hsIL-6R-BAPl давали связываться со стрептавидиновым сенсором (фирма Pall ForteBio Corp., 18-5021) и каждой расщепленной подобной антителу IgG-типа молекуле давали взаимодействовать с ним, после чего оценивали связывание при 30°С. Сенсограммы, на которых продемонстрированы ответы, характеризующие связывание в реальном времени, полученные с помощью Octet HTX, представлены на фиг. 13. В результате связывание было подтверждено для IL6R90H1002/VK1-39-k0MT, IL6R90H1004/VK1-39-k0MT, IL6R90H1005/VK1-39-k0MT и IL6R90H1006/VK1-39-k0MT. Для IL6R90-G1m/k0 и IL6R90-G1m/lamL установлено двухвалентное связывание на основе авидности, в то время как высвободившийся VHH связывается на основе аффинности. Таким образом, для обработанных протеазой IL6R90H1002/VK1-39-k0MT, IL6R90H1004/VK1-39-k0MT, IL6R90H1005/VK1-39-k0MT и IL6R90H1006/VK1-39-k0MT характерна более высокая скорость диссоциации от IL-6R, чем для IL6R90-G1m/k0 и IL6R90-G1m/lamL. Кроме того, VHH имеет меньшую молекулярную массу по сравнению с IL6R90-G1m/k0 и IL6R90-G1m/lamL. Поэтому его ответ в виде уровня связывания был более низким.
Эти результаты продемонстрировали, что IL6R90H1002/VK1-39-k0MT, IL6R90H1004/VK1-39-k0MT, IL6R90H1005/VK1-39-k0MT или IL6R90H1006/VK1-39-k0MT не обладают связывающей активностью в отношении IL-6R, в отличие от варианта, в котором последовательность пептида А, встроенная вблизи границы между VHH и константной областью тяжелой цепи, расщеплялась при обработке протеазой, в результате чего VHH-домен высвобождается, и высвободившийся VHH может связываться с IL-6R. Исходя из указанного, можно заключить, что фактически удалось получить молекулу, подтверждающую концепцию, описанную в примере 2.
Пример 4. Получение активируемого протеазой полипептида путем изменения с использованием VHH, связывающегося с IL-6R
4-1 Оценка связывания с IL-6R полипептида с включенным VHH. который связывается с IL-6R
Получали экспрессионный вектор, кодирующий конструкцию 20A11-G1m (SEQ ID NO: 38), которая содержит 20А11 (SEQ ID NO: 19), VHH, который обладает связывающей и нейтрализующей активностью в отношении IL-6R, описанный в WO 2010/115998, слитый с константной областью человеческого IgGI (СН1-шарнир-СН2-СН3), аналогично методу, описанному в примере 3, с использованием методики, известной специалистам в данной области.
Полипептиды 20A11-G1m/VK1-39-k0MT, 20A11-G1m/VK2-28-k0MT, 20А11-G1m/VK3-20-k0MT, 20A11-G1m/VL1-40-lamL, 20A11-G1m/VL1-44-lamL, 20A11-G1m/VL2-14-lamL и 20 A11-G1m/VL3-21-lamL экспрессировали и очищали аналогично методу, описанному в примере 3, используя указанную тяжелую цепь и конструкции VK1-39-k0MT (SEQ ID NO: 3), VK2-28-k0MT (SEQ ID NO: 4), VK3-20-k0MT (SEQ ID NO: 5), VL1-40-lamL (SEQ ID NO: 6), VL1-44-lamL (SEQ ID NO: 7), VL2-14-lamL (SEQ ID NO: 8) и VL3-21-lamL (SEQ ID NO: 9) в качестве легких цепей.
Оценивали полученные конструкции 20A11-G1m/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 3), 20A11-G1m/VK2-28-k0MT (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 4), 20A11-G1m/VK3-20-k0MT (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 5), 20A11-G1m/VL1-40-lamL (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 6), 20A11-G1m/VL1-44-lamL (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 7), 20A11-G1m/VL2-14-lamL (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 8) и 20A11-G1m/VL3-21-lamL (тяжелая цепь: SEQ ID NO: 38, легкая цепь: SEQ ID NO: 9) в отношении их связывания с IL-6R аналогично методу, описанному в примере 3. Результаты представлены на фиг. 14. В результате установлено, что ни одна из легких цепей, примененных в этом примере, не ингибировала IL-6R-связывающую активность 20А11 при ассоциации с тяжелой цепью, содержащей 20А11, слитый с константной областью IgGI человеческой зародышевой линии (СН1-шарнир-СН2-СН3).
Это, вероятно, является следствием того, что 20А11 не образовывал стабильную вариабельную область с VL, применяемыми в этом примере.
4-2 Интродукция аминокислотного изменения в сайт поверхности раздела между VHH и VL в полипептиде с включенным VHH без утраты антигенсвязывающей способности
Для формирования стабильной вариабельной области между 20А11 и VL интродуцировали мутации в аминокислоты, присутствующие на поверхности раздела между 20А11 и VL. Получали экспрессионный вектор, кодирующий конструкцию 20A11hu-G1m (SEQ ID NO: 39), содержащую 20A11hu (получен из 20А11 путем интродукции мутаций, приводящих к замене F в положении 37 на V (F37V), R в положении 45 на L и G в положении 47 на W (вся нумерация Кэбота)) (SEQ ID NO: 20), слитый с константной областью человеческого IgGI (СН1-шарнир-СН2-СН3), аналогично методу, описанному в примере 3, с помощью методики, известной специалистам в данной области.
Полипептиды 20A11hu-G1m/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 3), 20A11hu-G1m/VK2-28-k0MT (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 4), 20A11hu-G1m/VK3-20-k0MT (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 5), 20A11hu-G1m/VL1-40-lamL (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 6), 20A11hu-G1m/VL1-44-lamL (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 7), 20A11hu-G1m/VL2-14-lamL (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 8) и 20A11hu-G1m/VL3-21-lamL (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 9) экспрессировали и очищали аналогично методу, описанному в примере 3, используя указанную тяжелую цепь и VK1-39-k0MT (SEQ ID NO: 3), VK2-28-k0MT (SEQ ID NO: 4), VK3-20-k0MT (SEQ ID NO: 5), VL1-40-lamL (SEQ ID NO: 6), VL1-44-lamL (SEQ ID NO: 7), VL2-14-lamL (SEQ ID NO: 8) и VL3-21-lamL (SEQ ID NO: 9) в качестве легких цепей.
4-3 Оценка связывания с IL-6R полипептида с включенным VHH, который содержит аминокислотное изменение в сайте поверхности раздела между VHH и VL
Полученные конструкции 20A11hu-G1m/VK1-39-k0MT, 20A11hu-G1m/VK2-28-k0MT, 20A11hu-G1m/VK3-20-k0MT, 20A11hu-G1m/VL1-40-lamL, 20A11hu-G1m/VL1-44-lamL, 20A11hu-G1m/VL2-14-lamL и 20A11hu-G1m/VL3-21-lamL оценивали в отношении их связывания с IL-6R при 30°С или 25°С, аналогично методу, описанному в примере 3. Результаты представлены на фиг. 15.
В результате установлено, что 20A11hu-G1m/VK1-39-k0MT, 20A11hu-G1m/VK2-28-k0MT, 20A11hu-G1m/VK3-20-k0MT, 20A11hu-G1m/VL1-40-lamL, 20A11hu-G1m/VL1-44-lamL и 20A11hu-G1m/VL2-14-lamL не обладали способностью связываться с IL-6R.
Указанные результаты продемонстрировали, что VHH 20A11, который не утрачивал свою IL-6R-связывающую активность при ассоциации с VL, применяемым в примере 3, может формировать стабильную вариабельную область с VL и может утрачивать свою IL-6R-связывающую активность при изменении аминокислот, присутствующих в сайте поверхности раздела между VHH и VL, на 37V, 45L и 47W (нумерация Кэбота) и, тем самым, изменяясь с 20A11 на 20A11hu.
4-4 Интродукция расщепляемой протеазой последовательности в полипептид с включенным VHH, который содержит аминокислотное изменение в сайте поверхности раздела между VHH и VL
Тяжелые цепи 20A11huH1001 (SEQ ID NO: 40), 20A11huH1002 (SEQ ID NO: 41), 20A11huH1004 (SEQ ID NO: 42) и 20A11huH1006 (SEQ ID NO: 43) получали аналогично методу, описанному в примере 3, встраивая расщепляемую протеазой последовательность (SEQ ID NO: 12) или расщепляемую протеазой последовательность, сцепленную с гибким линкером (SEQ ID NO: 44), вблизи границы между 20A11hu и СН1.
Полипептиды 20A11huH1001/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 40, легкая цепь: SEQ ID NO: 3), 20A11huH1002/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 41, легкая цепь: SEQ ID NO: 3), 20A11huH1004/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 42, легкая цепь: SEQ ID NO: 3) и 20A11huH1006/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 43, легкая цепь: SEQ ID NO: 3) экспрессировали и очищали аналогично методу, описанному в примере 3, применяя эти тяжелые цепи и VK1-39-k0MT (SEQ ID NO: 3) в качестве легкой цепи.
4-5 Активация полипептида. несущего расщепляемую протеазой последовательность, путем расщепления протеазой
20A11huH1001/VK1-39-k0MT, 20A11huH1002/VK1-39-k0MT, 20A11huH1004/VK1-39-k0MT и 20A11huH1006/VK1-39-k0MT расщепляли протеазой аналогично методу, описанному в примере 3, и степень расщепления оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 16.
В результате подтверждено, что 20A11huH1002/VK1-39-k0MT, 20A11huH1004/VK1-39-k0MT и 20A11huH1006/VK1-39-k0MT подвергались расщеплению протеазой вблизи границы между VHH и СН1.
Затем оценку связывания с IL-6R VHH, высвободившегося в результате обработки протеазой, проводили при 30°С или 25°С аналогично методу, описанному в примере 3. Полученные с помощью Octet сенсограммы представлены на фиг. 17.
В результате связывание с IL-6R продемонстрировано для 20A11huH1002/VK1-39-k0MT, 20A11huH1004/VK1-39-k0MT и 20A11huH1006/VK1-39-k0MT, для которых подтверждено расщепление вблизи границы между VHH и СН1 при обработке протеазой.
Эти результаты продемонстрировали, что, даже, если VHH, включенный в полипептид, не утрачивает свою антигенсвязывающую активность немедленно после ассоциации с конкретным VL, антигенсвязывающая активность может утрачиваться путем интродукции усиливающей ассоциацию мутации в аминокислоту, присутствующую на поверхности раздела между VHH и VL.
Исходя из указанных результатов, можно заключить, что молекулу, удовлетворяющую концепции, которая описана в примере 2, можно получать также методом объединения легкой цепи с VHH, который содержит замененную аминокислоту, участвующую в ассоциации с легкой цепью, в дополнение к методу объединения легкой цепи с VHH, получение которого описано выше в примере 3.
Пример 5. Создание активируемого протеазой полипептида с использованием VHH, полученного из иммунизированных альпак
5-1 Получение VHH из иммунизированных альпак
Альпак иммунизировали IL-6R, CD3 или плексином А1 с помощью метода, известного специалистам в данной области. Через 4 и 8 недель собирали РВМС. Из собранных РВМС ген VHH амплифицировали аналогично методу, описанному в J. Immunol. Methods 324, 2007, с. 13. Амплифицированный фрагмент гена VHH соединяли с геном 3 и встраивали в фагмидный вектор. Фагмидным вектором, имеющим вставку в виде VHH-фрагмента, трансфектировали Е. coli с помощью метода электропорации, и фаги, презентирующие VHH, получали методом, уже известным специалистам в данной области. Полученные фаги оценивали в отношении их связывания с IL-6R, CD3 или плексином А1 с помощью ELISA. Последовательность связанного клона анализировали с помощью метода, известного специалистам в данной области для идентификации VHH, связывающегося с антигеном.
5-2 Увеличение количества (обогащение) VHH, связывающегося с CD3
VHH, связывающийся с человеческим CD3, идентифицировали из библиотеки VHH, конструирование которой описано в примере 5-1. Клоны VHH, обладающие способностью связываться с человеческим CD3, обогащали, используя меченный биотином белок, содержащий ε-цепь человеческого CD3 (CD3ε) и δ-цепь человеческого CD3 (CD3δ), сцепленные с константной областью человеческого антитела (человеческий CD3ed-Fc), в качестве антигена. Человеческий CD3ed-Fc получали следующими образом: экспрессионным вектором для клеток животных, имеющим ген, который кодирует аминокислотную последовательность, представленную в SEQ ID NO: 59, ген, который кодирует аминокислотную последовательность, представленную в SEQ ID NO: 60, и ген, который кодирует BirA (SEQ ID NO: 58), трансфектировали клетки FreeStyle 293 (фирма Invitrogen Corp.). После трансфекции к ним добавляли L-биотин и биотинилирование осуществляли в культуральном растворе. Клетки культивировали в виде встряхиваемой культуры при 37°С согласно протоколу. Через 4-5 дней собирали супернатант. Из супернатанта получали белок, слитый с константной областью антитела, с помощью колонки с белком A (Eshmuno А (фирма Merck KGaA)). Для дополнительного получения только гетеродимера CD3εδ фракцию гетеродимера CD3εδ, слитого с константной областью антитела (обозначенную как CD3ed-Fc), отделяли с помощью колонки с антителом к FLAG M2. Затем осуществляли гель-фильтрацию (Супердекс 200, фирма GE Healthcare Japan Corp.) с получением представляющей интерес фракции гетеродимера CD3εδ (обозначен как человеческий CD3ed-Fc).
Производство фага осуществляли из Е. coli, сохраняя сконструированные фагмиды для фагового дисплея. Популяцию фага осаждали, добавляя 2,5М NaCl/10% ПЭГ к культуральному раствору Е. coli после производства фага, а затем разводили TBS с получением раствора, содержащего фагововую библиотеку. Затем добавляли БСА в раствор, содержащий фаговую библиотеку, с получением конечной концентрация БСА 4%. Пэннинг осуществляли согласно принятому методу пэннинга, используя антиген, иммобилизованный на магнитных гранулах (J. Immunol. Methods. 332 (1-2), 2008, сс. 2-9; J. Immunol. Methods. 247 (1-2), 2001, сс. 191-203; Biotechnol. Prog. 18 (2), 2002, сс. 212-220; и Mol. Cell Proteomics 2 (2), 2003, сс. 61-69). Применяемые магнитные гранулы представляли собой покрытые нейтравидином (NeutrAvidin) гранулы (FG bead NeutrAvidin) или покрытые стрептавидином гранулы (Dynabeads MyOne Streptavidin T1).
В частности, 100 пмолей меченного биотином антигена добавляли в полученный раствор фаговой библиотеки, и раствор фаговой библиотеки приводили в контакт с антигеном при комнатной температуре в течение 60 мин. К ним добавляли магнитные гранулы, блокированные БСА, и комплексам антигена и фагов давали связываться с магнитными гранулами при комнатной температуре в течение 15 мин. Гранулы промывали дважды 0,5 мл TBST (TBS, содержащий 0,1% Твин 20; TBS производства фирмы Takara Bio Inc.) и затем дополнительно промывали однократно 0,5 мл TBS. Затем к ним добавляли 0,5 мл трипсина в концентрации 1 мг/мл и гранулы суспендировали при комнатной температуре в течение 15 мин и после этого немедленно разделяли, используя магнитный стенд для выделения раствора фагов. Восстановленный раствора фагов добавляли к 20 мл штамма E. coli ER2738 на экспоненциальной стадии роста (ОП600: 0,4-0,5). Е. coli культивировали при слабом перемешивании при 37°С в течение 1 ч, осуществляя тем самым заражение фагами. Зараженными E. coli инокулировали планшет размером 225 мм × 225 мм. Затем фаги выделяли из культурального раствора инокулированных Е. coli с получением раствора фаговой библиотеки. Указанный цикл, называемый пэннингом, повторяли в целом дважды. Во втором цикле пэннинга гранулы промывали три раза TBST и затем дважды TBS. Кроме того, в случае пэннинга в отношении человеческого CD3ed-Fc добавляли 4 нмоля человеческой Fc-области.
5-3 Получение активируемой протеазой подобной антителу IgG-типа молекулы с включенным VHH. который связывается CD3
Нуклеотидную последовательность, кодирующую последовательность VHH (таблица 2) каждого связывающегося с человеческим CD3 клона, полученного согласно методу, описанному в примере 5-1 или 5-2, соединяли с нуклеотидной последовательностью, кодирующей сайт расщепления протеазой и константную область, с помощью метода, описанного в примере 3, и встраивали в экспрессионный вектор для клеток животных. Полученный продукт применяли в качестве тяжелой цепи подобной антителу IgG-типа молекуле.

Активируемые протеазой подобные антителу IgG-типа молекулы, которые представлены ниже в таблице 3, экспрессировали путем кратковременной экспрессии, используя клетки Free Style 293 (фирма Invitrogen Corp.), с помощью метода, известного специалистам в данной области, и очищали с помощью метода, известного специалистам в данной области, используя белок А.

5-4 Активация активируемых протеазой подобных антителу IgG-типа молекул путем расщепления протеазой
Подобные антителу IgG-типа молекулы, полученные согласно методу, описанному в примере 5-3, расщепляли протеазой аналогично методу, описанному в примере 3, и степень расщепления оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 18. Концентрацию протеазы доводили до 25нМ, и в этом анализе применяли Octet RED (фирма Pall ForteBio Corp.).
В результате подтверждено, что подобные антителу IgG-типа молекулы подвергались расщеплению протеазой в расщепляемой протеазой последовательности.
Затем осуществляли оценку CD3-связываю щей способности VHH, высвободившегося в результате обработки протеазой, аналогично методу, описанному в примере 3. Полученные с помощью Octet сенсограммы представлены на фиг. 19.
В результате установлено, что подобные антителу IgG-типа молекулы bC3edL1R1N160H01-G1mISHI01/VK1-39-k0MT, bC3edL1R1N161H01-G1mISHI01/VK1-39-k0MT и bC3edL1R1N164H01-G1mISHI01/VK1-39-k0MT не обладали способностью связывать антиген до обработки протеазой, при этом их способность связывать антиген подтверждена после обработки протеазой. Множество молекул VHH, связывающихся с CD3, полученных таким же образом, что и VHH, представленные в таблице 2, применяли также для получения подобной антителу IgG-типа молекулы, содержащей такой же сайт расщепления протеазой, что и подобные антителу IgG-типа молекулы, представленные в таблице 3. В результате способность связывать антиген была подтверждена после обработки протеазой. Эти результаты продемонстрировали, что помимо полипептидов, описанных в примерах 3 и 4, подобная антителу IgG-типа молекула, несущая расщепляемую протеазой последовательность, может подвергаться расщеплению в расщепляемой протеазой последовательности при обработке протеазой и тем самым высвобождать антигенсвязывающий домен, а высвободившийся антигенсвязывающий домен может связываться с антигеном.
Пример 6. Полипептид, несущий расщепляемую протеазой последовательность в легкой цепи
Легкие цепи VK1-39P-2-Pk0MT (SEQ ID NO: 67), VK1-39P-1-Pk0MT (SEQ ID NO: 68), VK1-39P-Pk0MT (SEQ ID NO: 69), VK1-39P+2-Pk0MT (SEQ ID NO: 70), VK1-39P+3-Pk0MT (SEQ ID NO: 71), VK1-39P+4-Pk0MT (SEQ ID NO: 72) и VK1-39P+5-Pk0MT (SEQ ID NO: 73), несущие расщепляемую протеазой последовательность в любом положении, получали аналогично методу, описанному в примере 3.
Подобные антителу IgG-типа молекулы экспрессировали и очищали аналогично методу, описанному в примере 3, используя указанные легкие цепи и IL6R90-G1m (SEQ ID NO: 2) в качестве тяжелой цепи. Концентрацию протеазы доводили до 25нМ. IL6R90-G1m/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 3) применяли в качестве подобной антителу IgG-типа молекулы, не имеющей расщепляемой последовательности.
Затем полученные подобные антителу IgG-типа молекулы расщепляли протеазой аналогично методу, описанному в примере 3, и степень расщепления оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 20. В результате подтверждено, что VK1-39P+2-Pk0MT (SEQ ID NO: 70), VK1-39P+3-Pk0MT (SEQ ID NO: 71), VK1-39P+4-Pk0MT (SEQ ID NO: 72) и VK1-39P+5-Pk0MT (SEQ ID NO: 73) подвергались расщеплению протеазой в расщепляемой протеазой последовательности. Дополнительно проводили оценку способности связывать IL-6R для VHH, обработанного протеазой, аналогично методу, описанному в примере 3. Полученные с помощью Octet сенсограммы представлены на фиг. 21. В результате связывание подтверждено также при обработке протеазой, когда расщепляемую последовательность интродуцировали в легкую цепь, демонстрируя, что активируемый протеазой полипептид, несущий расщепляемую протеазой последовательность в его легкой цепи, можно получать так, чтобы антигенсвязывающий домен экспонировался, проявляя антигенсвязывающую способность при расщеплении протеазой легкой цепи.
Пример 7. Библиотека, содержащая тяжелую цепь. которая имеет антигенсвязывающий домен, и легкую цепь, несущую расщепляемую протеазой последовательность, и получение активируемого протеазой полипептида методом фагового дисплея с использованием библиотеки
Как установлено в примере 6, даже, когда расщепляемую протеазой последовательность интродуцировали в легкую цепь активируемого протеазой полипептида, антигенсвязывающий домен экспонировался после расщепления легкой цепи, связываясь с антигеном.
Соответственно тяжелую цепь, содержащую антигенсвязывающий домен, такой как однодоменное антитело, легкую цепь, несущую расщепляемую протеазой последовательность, включали в фагмиду и давали презентоваться с помощью фага. Конструировали множество фагмид для фагового дисплея, содержащих различные типы антигенсвязывающих доменов, с последующим получением фагов из Е. coli, в которых сохранялись эти фагмиды. Популяцию фага осаждали, добавляя 2,5М NaCl/10% ПЭГ к культуральному раствору E. coli после производства фага, а затем разводили TBS с получением раствора фаговой библиотеки. Затем добавляли БСА в раствор фаговой библиотеки с получением конечной концентрации БСА 4%.
Таким образом получали активируемый протеазой полипептид путем пэннинга из созданной фаговой библиотеки. Пэннинг осуществляли согласно принятому метода пэннинга, используя антиген, иммобилизованный на магнитных гранулах (J. Immunol. Methods. 332 (1-2), 2008, cc. 2-9; J. Immunol. Methods. 247 (1-2), 2001, cc. 191-203; Biotechnol. Prog. 18 (2), 2002, cc. 212-220; и Mol. Cell Proteomics 2 (2), 2003, cc. 61-69). Фаги, несвязанные с иммобилизованными антигеном магнитными гранулами, извлекали до добавления протеазы, а фаги, связанные с иммобилизованными антигеном магнитными гранулами, извлекали после добавления протеазы. Применяемые магнитные гранулы представляли собой покрытые нейтравидином гранулы (Sera-Mag SpeedBead NeutrAvidin-coated FG bead NeutrAvidin) или покрытые стрептавидином гранулы (Dynabeads M-280 Streptavidin). Антигенсвязывающий клон можно отбирать из извлеченных фагов с помощью ELISA для фагов, который описан в предыдущем разделе, или можно ген антитела субклонировать в векторе для экспрессии в животных и экспрессировать с использованием клеток животных и связывающую активность сравнивать до и после обработки протеазой для селекции связывающихся клонов.
Пример 8. Библиотека, содержащая тяжелую цепь, которая имеет антигенсвязывающий домен, и легкую цепь, и получение тяжелой цепи, антигенсвязывающая способность которой контролируется легкой цепью, методом фагового дисплея из библиотеки
Как установлено в примере 3, антигенсвязывающая способность тяжелой цепи, содержащей антигенсвязывающий домен, контролируется путем ассоциации с легкой цепью. Таким образом, методом фагового дисплея получали тяжелую цепь, которая утрачивала свою антигенсвязывающую способность при ассоциации с легкой цепью и проявляла антигенсвязывающую способность при презентации индивидуально или в комбинации с константной областью легкой цепи.
Тяжелую цепь, содержащую антигенсвязывающий домен, такой как однодоменное антитело, включали в фагмиду и презентовали с помощью фага. Конструировали множество фагмид для фагового дисплея, содержащих различные типы антигенсвязывающих доменов, с последующим получением фагов из Е. coli, в которых сохранялись эти фагмиды. Популяцию фага осаждали, добавляя 2,5М NaCl/10% ПЭГ к культуральному раствору Е. coli после производства фага, а затем разводили TBS с получением раствора фаговой библиотеки. Затем добавляли БСА в раствор, содержащий фаговую библиотеку, с получением конечной концентрации БСА 4%.
Тяжелую цепь, которая проявляет антигенсвязывающую способность, когда презентируется индивидуально или в комбинации с константной областью легкой цепи и утрачивает свою антигенсвязывающую способность при ассоциации с вариабельной областью легкой цепи, получали с помощью пэннинга из созданной таким образом фаговой библиотеки. Пэннинг осуществляли в соответствии с методом пэннинга, используя антиген, иммобилизованный на магнитных гранулах, согласно методу, описанному в примере 5. Фаги, связанные с иммобилизованными антигеном магнитными гранулами, извлекали из фаговой библиотеки, презентирующей тяжелые цепи или тяжелые цепи с константными областями легких цепей. Указанным извлеченным фагам давали инфицировать Е. coli, и фаги, презентирующие тяжелые и легкие цепи, получали с использованием хелперного фага, экспрессирующего легкую цепь. Фаги, презентирующие тяжелую цепь, которая содержит антигенсвязывающий домен, и легкую цепь, получали описанным выше методом из культурального раствора Е. coli после производства фага. Фаги, несвязанные с иммобилизованными антигеном магнитными гранулами, извлекали из популяции фагов, презентирующих тяжелые и легкие цепи.
Как продемонстрировано на фиг. 9Г, пэннинг можно осуществлять путем изменения порядка извлечения популяции фага, презентирующего тяжелую цепь, либо индивидуально, либо в комбинации с константной областью легкой цепи, связывающегося с иммобилизованными антигеном магнитными гранулами, и извлечения популяции фага, презентирующего тяжелые и легкие цепи, несвязывающегося с иммобилизованными антигеном магнитными гранулами. В дополнение в методу экспрессии легкой цепи с использованием хелперного фага, область, кодирующую легкую цепь, и область, кодирующую тяжелую цепь, можно включать, как обычно, в одну и ту же фагмиду, и можно включать ген, кодирующий только константную область легкой цепи или полноразмерную легкую цепь, и применять в каждом цикле пэннинга.
Антигенсвязывающий клон можно отбирать из извлеченных фагов с помощью ELISA для фагов, который описан в предыдущем разделе, или можно ген антитела субклонировать в векторе для экспрессии в животных и экспрессировать с использованием клеток животных и связывающую активность сравнивать до и после обработки протеазой для селекции связывающихся клонов.
Пример 9. Получение VHH, антигенсвязывающая способность которого контролируется легкой цепью, путем применения метода фагового дисплея, и получение подобной антителу IgG-типа молекулы, содержащей VHH
В примере 3 установлено, что антигенсвязывающая способность VHH, который входит в качестве заместителя VH в тяжелую цепь, контролируется путем ассоциации с легкой цепью. Соответственно VHH, который утрачивал свою антигенсвязывающую способность при ассоциации с конкретной легкой цепью, и проявлял антигенсвязывающую способность при презентации тяжелой цепи индивидуально или в комбинации с константной областью легкой цепи, т.е. без ассоциации с вариабельной областью легкой цепи, получали из фаговой библиотеки, презентирующей СН1, сцепленный с VHH, полученным из РВМС иммунизированных альпак. Получали подобную антителу IgG-типа молекулу, содержащую VHH.
9-1 Конструирование экспрессирующих легкую цепь хелперных фагов с интегрированной экспрессионной единицей легкой цепи
На основе метода, описанного в WO 2015/046554, промотор, сигнальную последовательность, гены вариабельной области легкой цепи и константной области легкой цепи антитела или ген константной области легкой цепи и т.д. интегрировали в геном хелперного фага, создавая экспрессирующий легкую цепь хелперный фаг. Е. coli, инфицированные указанным хелперным фагом, могли экспрессировать вариабельную область легкой цепи и константную область легкой цепи антитела или только константную область легкой цепи.
В частности, геном получали из хелперного фага M13KO7TC, сконструированного согласно методу, описанному в WO 2015/046554, и интродуцировали в геном экспрессионную единицу легкой цепи. Ген, кодирующий вариабельную область легкой цепи и константную область легкой цепи (VK1-39-k0MTdC; SEQ ID NO: 152), или ген. кодирующий константную область легкой цепи (k0MTdC; SEQ ID NO: 153), применяли в качестве гена легкой цепи, подлежащего интродукции. Конструкцию lac промотор-pelB сигнальная последовательность-ген легкой цепи встраивали в M13KO7TC/SacI с помощью описанного выше метода и трансфектировали штамм Е. coli ER2738 методом электропорации.
Полученные Е. coli культивировали и в супернатант культуры добавляли 2,5М NaCl/10% ПЭГ для очистки хелперных фагов методом осаждения с помощью ПЭГ. Титры полученных хелперных фагов M13KO7TC-Vk1-39-k0MTdC и M13KO7TC-k0MTdC определяли с помощью общепринятого метода бляшкообразования.
9-2 Получение библиотеки, содержащей множество молекул VHH-CH1
Альпак иммунизировали методом, известным специалистам в данной области, используя 4 типа иммуногенов: внеклеточный домен человеческого IL-6R, гетеродимер человеческого CD3εγ, гетеродимер обезьяньего CD3εγ и клеточный домен человеческого плексина А1. Через 4 недели собирали РВМС. Гетеродимеры CD3εγ получали согласно методу, описанному в Journal of Molecular Biology 302, 2000, сс. 899-916. Из собранных РВМС ген VHH амплифицировали согласно методу, описанному в J. Immunol. Methods 324, 2007, с. 13. Амплифицированный фрагмент гена VHH соединяли с геном 3 CH1-гена и встраивали с фагмидые векторы с получением библиотеки, содержащей множество молекул VHH-CH1, содержащих VHH, сцепленный с СН1.
9-3 Метод получения популяции фага, презентирующего конструкцию VHH-CH1/полноразмерная легкая цепь или VHH-CH1/константная область легкой цепи
Фагмидным вектором, имеющим вставку в виде гена, кодирующего VHH-CH1, трансфектировали Е. coli методом электропорации. Полученных Е. coli можно культивировать и инфицировать хелперным фагом M13KO7TC-Vk1-39-k0MTdC, полученным согласно методу, описанному в примере 9-1, в результате чего конструкция VHH-CH1, экспрессированная фагмидным вектором, и полноразмерная легкая цепь, экспрессированная хелперным фагом, формировали Fab-структуру, для получения популяции фага, презентирующего VHH-CH1/полноразмерная легкая цепь (VHH-CH1/Vk1-39-k0MTdC) на поверхности фагмид, содержащих ген, кодирующий VHH-CH1. Кроме того, Е. coli, несущих фагмидный вектор, который имеет вставку в виде гена, кодирующего VHH-CH1, можно культивировать и инфицировать хелперным фагом M13KO7TC-k0MTdC, полученным согласно методу, описанному в примере 9-1, в результате чего конструкция VHH-CH1, экспрессированная фагмидным вектором, и константная область легкой цепь, экспрессированная хелперным фагом, формировали структуру в виде VHH-CH1, ассоциированных с CL, для получения популяции фага, презентирующего VHH-CH1/константная область легкой цепи (VHH-CH1/k0MTdC). Можно добавлять в супернатант культуры 2,5М NaCl/10% ПЭГ для очистки фагов методом осаждения ПЭГ. Титры полученных фагов можно определять с помощью общепринятого метода бляшкообразования.
9-4 Получение конструкции VHH-CH1. содержащей связывающийся с плексином A1 VHH, антигенсвязывающая способность которого ингибируется при ассоциации с вариабельной областью легкой цепи, и который обладает антигенсвязывающей способностью в отсутствии вариабельной области легкой цепи. из фаговой библиотеки VHH-CH1
Получали конструкцию VHH-CH1, содержащую VHH, антигенсвязывающая способность которого ингибируется при ассоциации с вариабельной областью легкой цепи, и который обладает антигенсвязывающей способностью в отсутствии вариабельной области легкой цепи, путем пэннинга из библиотеки VHH-CH1, полученной согласно методу, описанному в примере 9-2.
Применяемый антиген представлял собой меченный биотином человеческий плексин A1, получение которого описано в приведенном для справки примере (референс-пример).
Метод пэннинга осуществляли с использованием следующих стадий:
(1) Популяцию фага, презентирующего VHH-CH1/константная область легкой цепи (VHH-CH1/k0MTdC), получали согласно методу, описанному в примере 9-3, из фаговой библиотеки VHH-CH1, полученной согласно методу, описанному в примере 9-2, и из популяции извлекали фаги, связанные с магнитными гранулами с иммобилизованным антигеном.
(2) Популяцию фага, презентирующего VHH-CH1/полноразмерная легкая цепь (VHH-CH1/Vk1-39-k0MTdC), получали согласно методу, описанному в примере 9-3, из извлеченных фагов, и из популяции извлекали фаги, несвязанные с магнитными гранулами с иммобилизованным антигеном.
(3) Извлеченные фаги повторно подвергали стадиям (1) и (2) для извлечения требуемого фага.
С помощью пэннинга можно отбирать множество молекул VHH-CH1, связывание которых с плексином А1 ингибировалось в результате ассоциации с легкой цепью Vk1-39-k0MTdC, и которые обладали связывающей способностью в отношении плексина А1 в отсутствии вариабельной области легкой цепи.
Другой метод пэннинга осуществляли с использованием следующих стадий:
(1) Популяцию фага, презентирующего VHH-CH1/константная область легкой цепи (VHH-CH1/k0MTdC), получали согласно методу, описанному в примере 9-3, из фаговой библиотеки VHH-CH1, полученной согласно методу, описанному в примере 9-2, и из популяции извлекали фаги, связанные с магнитными гранулами с иммобилизованным антигеном.
(2) Популяцию фага, презентирующего VHH-CH1/полноразмерная легкая цепь (VHH-CH1/Vk1-39-k0MTdC), получали согласно методу, описанному в примере 9-3, из извлеченных фагов, и из популяции извлекали фаги, несвязанные с иммобилизованными антигеном магнитными гранулами. Дополнительно из извлеченных фагов выделяли фаги, связывающиеся с магнитными гранулами с иммобилизованным антителом к легкой цепи (фирма EY Laboratories, Inc., Cat. BAT-2107-2).
(3) Извлеченные фаги повторно подвергали стадиям (1) и (2) для извлечения требуемого фага.
С помощью пэннинга можно отбирать множество молекул VHH-CH1, связывание которых с плексином А1 ингибировалось в результате ассоциации с легкой цепью Vk1-39-k0MTdC, и которые обладали связывающей способностью в отношении плексина А1 в отсутствии вариабельной области легкой цепи.
VHH в VHH-CH1, отобранный с помощью пэннинга, можно применять для получения подобных антителу IgG-типа молекул.
9-5 Получение активируемых протеазой подобных антителу IgG-типа молекул с включенным VHH. который связывается с плексином А1
Нуклеотидную последовательность, кодирующую VHH, который содержится в каждой молекуле VHH-CH1, отобранной согласно методу, описанному в примере 9-4, соединяли с нуклеотидной последовательностью, кодирующей сайт расщепления протеазой и константную область тяжелой цепи, согласно методу, описанному в примере 3. Образовавшуюся конструкцию применяли в качестве тяжелой цепи подобной антителу IgG-типа молекулы и объединяли с полноразмерной легкой цепью VK1-39-k0MT (SEQ ID NO: 3). Подобные антителу IgG-типа молекулы экспрессировали путем кратковременной экспрессии, используя клетки FreeStyle 293 (фирма Invitrog en Corp.), с помощью метода, известного специалистам в данной области, и очищали с помощью метода, известного специалистам в данной области, используя белок А.
Полученные подобные антителу IgG-типа молекулы представлены в таблице 4.

9-6 Активация активируемой протеазой подобной антителу IgG-типа молекулы путем расщепления протеазой
Подобные антителу IgG-типа молекулы, полученные согласно методу, описанному в примере 9-4, расщепляли протеазой, аналогично методу, описанному в примере 3, и степень расщепления оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 22. Концентрацию протеазы доводили до 25нМ.
В результате установлено, что каждая из полученных подобных антителу IgG-типа молекул подвергалась расщеплению протеазой в расщепляемой протеазой последовательности.
Далее оценивали связывание с человеческим плексином A1 VHH, высвободившегося в результате обработки протеазой, аналогично методу, описанному в примере 3. Полученные с помощью Octet сенсограммы представлены на фиг. 23.
В результате установлено, что каждая из полученных подобных антителу IgG-типа молекул не проявляла антигенсвязывающую способность до обработки протеазой, однако подтверждена антигенсвязывающая способность высвободившегося VHH после обработки протеазой.
Пример 10. Полипептиды, содержащие биспецифическую молекулу VHH-VHH
10-1 Биспецифическая конструкция VHH-VHH, связывающаяся с раковым антигеном и CD3, и получение полипептида, содержащего биспецифическую молекулу VHH-VHH
Как продемонстрировано на фиг. 8, активируемый протеазой антигенсвязывающий домен может образовывать биспецифическую антигенсвязывающую молекулу со вторым антигенсвязывающим доменом.
VHH HN3 (SEQ ID NO: 159), распознающий человеческий глипикан 3, и VHH G03 (SEQ ID NO: 160), распознающий CD3, соединяли через линкер, состоящий из глицина и серина, с получением биспецифической конструкции VHH-VHH HN3G03. Константную область тяжелой цепи антитела, представленную в SEQ ID NO: 161, дополнительно соединяли с ней через расщепляемую протеазой последовательность, и образовавшуюся тяжелую цепь HN3G03-cF760mnHIF (SEQ ID NO: 162), содержащую биспецифическую конструкцию VHH-VHH, встраивали в вектор для экспрессии в животных.
VHH HerF07 (SEQ ID NO: 163), распознающий Her2, и VHH G03 (SEQ ID NO: 160), распознающий CD3, соединяли через линкер, состоящий из глицина и серина, с получением биспецифической конструкции VHH-VHH HerF07G03. Константную область тяжелой цепи антитела, представленную в SEQ ID NO: 161, дополнительно соединяли с ней через расщепляемую протеазой последовательность, и образовавшуюся тяжелую цепь HerF07G03-cF760mnHIF (SEQ ID NO: 164), содержащую биспецифическую конструкцию VHH-VHH, встраивали в вектор для экспрессии в животных.
Клетки Expi293 (фирма Life Technologies Corp.) совместно трансфектировали каждой тяжелой цепью, содержащей биспецифическую конструкцию VHH-VHH, и векторами для экспрессии в животных соответственно, которые имели вставки в виде легкой цепи VK1.39-k0MT (SEQ ID NO: 3), человеческой последовательностью константной области VHn-Kn010dGK (SEQ ID NO: 166), простирающейся от шарнирной области до С-конца, для экспрессии полипептида, содержащего биспецифическую конструкцию VHH-VHH. Затем, полипептид, содержащий биспецифическую конструкцию VHH-VHH, очищали с помощью метода, известного специалистам в данной области, используя 96-луночный планшет типа MonoSpin ProA (фирма GL Sciences Inc., каталожный №7510-11312). Полипептид, содержащий биспецифическую конструкцию VHH-VHH HN3G03, представлял собой HN3G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, а полипептид, содержащий биспецифическую конструкцию VHH-VHH HerF07G03, представлял собой HerF07G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT.
Для обработки протеазой uPA (рекомбинантный активатор человеческого и-плазминогена, фирма R&D Systems, Inc.) (конечная концентрация: 25нМ) добавляли к 40 мкг каждого очищенного полипептида, содержащего биспецифическую конструкцию VHH-VHH, и инкубировали при 37°С в течение 20 ч или более. Необработанные протеазой образцы инкубировали после добавления ЗФР вместо протеазы в таком же количестве, что и протеаза. Подвергается ли расщепляемый протеазой полипептид, содержащий биспецифическую конструкцию VHH-VHH, требуемому расщеплению, определяли с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 24. Как продемонстрировано на фиг. 24, можно предположить, что биспецифическая конструкция VHH-VHH отделялась от цельной молекулы путем расщепления протеазой.
10-2 Оценка активации CD3 полипептидом, содержащим биспецифическую конструкцию VHH-VHH, направленную против GPC3 и CD3, путем расщепления протеазой
Агонистическую активность в отношении CD3 оценивали с использованием репортерных клеток Jurkat-NFAT (клетки линии NFAT luc2_jurkat). Репортерные клетки Jurkat-NFAT представляют собой клеточную линию экспрессирующих CD3 полученных из человеческого острого Т-клеточного лейкоза клеток, слитую с респондерным элементом NFAT и люциферазой (luc2P), и экспрессирующую люциферазу при активации сигнала, действующего в прямом направлении относительно CD3. Клетки-мишени, применяемые для антител к GPC3, представляли собой клетки линии SK-рса60, созданные путем усиления способности экспрессировать человеческий GPC3 в полученной из клеток человеческого рака печени линии SK-HEP-1. Клетки-мишени и эффекторные клетки добавляли из расчета 1,25Е+04 клеток/лунку и 7.50Е+04 клеток/лунку соответственно в каждую лунку 96-луночного планшета для анализа с белым дном (фирма Costar, 3917). Конструкцию HN3G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, обработанную или необработанную протеазой, добавляли в лунки в конечной концентрации 1нМ, 10нМ или 100нМ. После 24-часовой инкубации при 37°С в присутствии 5% СО2; активность фермента люциферазы измеряли по интенсивности люминисценции, используя систему для анализа люциферазы Bio-Glo (фирма Promega Corp., G7940) согласно прилагаемому протоколу. Для детекции использовали устройство 2104 En Vision. Результаты представлены на фиг. 25. Никакого повышения люциферазной активности не обнаружено в образце, необработанном протеазой, в то время как повышение люциферазной активности выявлено при использовании конструкции HN3G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, обработанной протеазой. В частности, для обработанной протеазой конструкции HN3G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, удалось подтвердить наличие агонистической активности в отношении CD3, в то время как биспецифическая конструкция VHH-VHH, направленная против GPC3 и CD3, высвобождалась из HN3G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT при расщеплении протеазой и проявляла CD3-связывающую активность, заингибированную в отсутствии расщепления.
10-3 Оценка активации CD3 полипептидом, содержащим биспецифическую конструкцию VHH-VHH, направленную против Her2 и CD3, путем расщепления протеазой
Агонистическую активность в отношении CD3 оценивали с использованием репортерных клеток Jurkat-NFAT (клетки линии NFAT luc2_jurkat). Репортерные клетки Jurkat-NFAT (эффекторные клетки) представляют собой клеточную линию экспрессирующих CD3 полученных из человеческого острого Т-клеточного лейкоза клеток, слитую с респондерным элементом NFAT и люциферазой (luc2P), и экспрессирующую люциферазу при активации сигнала, действующего в прямом направлении относительно CD3. Применяемые клетки-мишени представляли собой клеточную линию LS1034. Клетки-мишени и эффекторные клетки добавляли из расчета 2.50Е+04 клеток/лунку и 7,50Е+04 клеток/лунку соответственно в каждую лунку 96-луночного планшета для анализа с белым дном (фирма Costar, 3917). Конструкцию HerF07G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, обработанную или необработанную протеазой, добавляли в лунки в конечной концентрации 0,01нМ, 0,1нМ и 1нМ. После 24-часовой инкубации при 37°С в присутствии 5% СО2; активность фермента люциферазы измеряли по интенсивности люминисценции, используя систему для анализа люциферазы Bio-Glo (фирма Promega Corp., G7940) согласно прилагаемому протоколу. Для детекции использовали устройство 2104 EnVision. Результаты представлены на фиг. 26. Никакого повышения люциферазной активности не обнаружено в образце, необработанном протеазой, в то время как повышение люциферазной активности выявлено при использовании конструкции HerF07G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, обработанной протеазой. В частности, для обработанной протеазой конструкции HerF07G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT, удалось подтвердить наличие агонистической активности в отношении CD3, в то время как биспецифическая конструкция VHH-VHH, направленная против Her2 и CD3, высвобождалась из HerF07G03-cF760mnHIF/VHn-Kn010dGK/VK1.39-k0MT при расщеплении протеазой и проявляла CD3-связывающую активность, заингибированную в отсутствии расщепления.
Пример 11. Интродукция различных сайтов расщепления протеазой в полипептид с включенным VHH
11-1 Интродукция различных расщепляемых протеазой последовательностей в полипептид с включенным VHH, который связывается с IL-6R
Получали экспрессионный вектор, кодирующий конструкцию IL6R90-G1T4 (SEQ ID NO: 167), которая содержит IL6R90 (SEQ ID NO: 1), т.е. VHH, который обладает связывающей и нейтрализующей активностью в отношении человеческого IL-6R, описанный в WO 2010/115998, слитый с константной областью человеческого IgGI (СН1-шарнир-СН2-СН3), с использованием метода, известного специалистам в данной области. Подобную антителу IgG-типа молекулу IL6R90-G1T4/VK1-39-k0MT (тяжелая цепь: SEQ ID NO: 167, легкая цепь: SEQ ID NO: 3) экспрессировали путем кратковременной экспрессии с использованием клеток FreeStyle 293 (фирма Invitrogen Corp.) или клеток Expi293 (фирма Life Technologies Corp.) с помощью метода, известного специалистам в данной области, и очищали с помощью метода, известного специалистам в данной области, используя белок А.
Различные расщепляемые протеазой последовательности встраивали вблизи границы между VHH и СН1 в тяжелую цепь IL6R90-G1T4/VK1-39-k0MT. Экспрессионные векторы, в которые расщепляемую протеазой последовательность, представленную в таблице 5, встраивали вблизи границы между VHH и СН1, получали с помощью методов, известных специалистам в данной области. Последовательности содержащих VHH тяжелых цепей, несущих расщепляемую протеазой последовательность, представлены в таблице 6.
Эти тяжелые цепи объединяли с легкой цепью. Подобные антителу IgG1-изотипа молекулы, представленные в таблице 7, которые несут расщепляемую протеазой последовательность вблизи границы между VHH и СН1, кратковременно экспрессировали с использованием клеток FreeStyle 293 (фирма Invitrogen Corp.) или клеток Expi293 cells (фирма Life Technologies Corp.) согласно методу, известному специалистам в данной области, и очищали согласно методу, известному специалистам в данной области, используя белок А.


11-2 Оценка расщепления протеазой множества подобных антителу IgG-типа молекул, содержащих VHH к человеческому IL-6R. которые несут расщепляемую протеазой последовательность в области тяжелой цепи
Изучали, могут ли подобные антителу IgG-типа молекулы, полученные согласно методу, описанному в примере 11-1, расщепляться протеазой. В качестве протеазы применяли рекомбинантную человеческую матриптазу/каталитический домен ST14 (MT-SP1) (фирма R&D Systems, Inc., 3946-SE-010). Подвергали взаимодействию 10нМ протеазу и 50 мкг/мл антитела в ЗФР при 37°С в течение 20 ч, после чего осуществляли ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 27. Установлено, что обработка протеазой подобных антителу IgG-типа молекул, несущих любую из расщепляемых протеазой последовательностей, приводила к созданию новой полосы, соответствующей молекулярной массе порядка 37 кДа. Таким образом, подтверждено, что подобные антителу IgG-типа молекулы подвергались расщеплению протеазой в расщепляемых протеазой последовательностях, которые представлены в таблице 5, встроенных вблизи границы между VHH и СН1. Кроме того, с использованием аналогичного метода подтверждено, что расщепляемые протеазой последовательности, представленные в таблице 5, могли расщепляться человеческой uPA и мышиной uPA, при их встраивании в антитело IgG-типа.
Пример 12. Оценка уровня активации в результате расщепления протеазой подобной антителу IgG-типа молекулы, несущей расщепляемую протеазой последовательность в ее легкой цепи
Получали экспрессионный вектор, кодирующий конструкцию IL6R75-G1m (SEQ ID NO: 191), которая содержит IL6R75 (SEQ ID NO: 190), т.е. VHH, который обладает связывающей и нейтрализующей активностью в отношении человеческого IL-6R, описанный в WO 2010/115998, слитый с константной областью человеческого IgG1 (СН1-шарнир-СН2-СН3), с использованием метода, известного специалистам в данной области. IL6R75hu-G1m (SEQ ID NO: 192) получали путем интродукции аминокислотных изменений в сайте на поверхности раздела между VHH и VL, аналогично методу, описанному в примере 4-2. Подобные антителу IgG-типа молекулы IL6R90-G1m/VK1-39P+4-Pk0MT (тяжелая цепь: SEQ ID NO: 2, легкая цепь: SEQ ID NO: 72), 20A11hu-G1m/VK1-39P+4-Pk0MT (тяжелая цепь: SEQ ID NO: 39, легкая цепь: SEQ ID NO: 72) и IL6R75hu-G1m/VK1-39P+4-Pk0MT (тяжелая цепь: SEQ ID NO: 192, легкая цепь: SEQ ID NO: 72) экспрессировали и очищали, аналогично методу, описанному в примере 3, используя легкую цепь с включенной расщепляемой протеазой последовательностью VK1-39P+4-Pk0MT (SEQ ID NO: 72), и IL6R90-G1m (SEQ ID NO: 2), 20A11hu-G1m (SEQ ID NO: 39) и IL6R75hu-G1m (SEQ ID NO: 192) в качестве тяжелых цепей.
IL6R90-G1m/VK1-39P+4-Pk0MT, 20A11hu-G1m/VK1-39P+4-Pk0MT и IL-6R75hu-G1m/VK1-39P+4-Pk0MT расщепляли протеазой, аналогично методу, описанному в примере 3, и оценивали степень расщепления. Результаты представлены на фиг. 28. В частности, в качестве протеазы применяли рекомбинантную человеческую матриптазу/каталитический домен ST14 (МТ-SP1) (фирма R&D Systems, Inc., 3946-SE-010). Подвергали взаимодействию 50 нМ протеазу и 50 мкг/мл каждой из подобных антителу IgG-типа молекул в ЗФР при 37°С в течение 20 ч. Затем расщепление протеазой оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. В результате подтверждено, что IL6R90-G1m/VK1-39P+4-Pk0MT, 20A11hu-G1m/VK1-39P+4-Pk0MT и IL-6R75hu-G1m/VK1-39P+4-Pk0MT подвергались расщеплению протеазой вблизи границы между VL и CL.
Затем способность VHH, высвободившегося в результате обработки протеазой, связываться с IL-6R оценивали с помощью ELISA. В частности, конструкцию hsIL-6R-BAP1, применяемую в примере 3, иммобилизовывали на покрытом стрептавидином 384-луночном планшете (фирма Greiner Bio-One GmbH, 781990) и каждой расщепленной подобной антителу IgG-типа молекуле давали связываться с ним при комнатной температуре. После осуществления реакции в течение 30 мин конструкции давали взаимодействовать с меченным HRP антителом к человеческому IgG (фирма Sigma-Aldrich Co. LLC, SAB3701362-2MG) при комнатной температуре в течение 10 мин и добавляли для взаимодействия хромогенный раствор ТМВ (фирма Life Technologies Corp., 002023). После осуществления реакции при комнатной температуре в течение 30 мин реакцию прекращали с помощью серной кислоты, а затем измеряли абсорбцию при 450 нм с помощью мультимодального ридера Synergy НТХ (фирма BioTek Instruments, Inc.). Рассчитывали соотношение для иммобилизованных антителом лунок и неиммобилизованных лунок и применяли в качестве отношения S/N. Строили график зависимости отношения S/N (среднее), установленного с помощью ELISA, отложенного на оси ординат, от концентрации каждой подобной антителу IgG-типа молекулы, отложенной на оси абсцисс. Результаты представлены на фиг. 29. Эти результаты продемонстрировали, что обработанная протеазой подобная антителу IgG-типа молекула 20A11hu-G1m/VK1-39P+4-Pk0MT, несущая расщепляемую последовательность в ее легкой цепи, в 10 или более раз превышала по IL-6R-связывающей активности необработанную протеазой подобную антителу IgG-типа молекулу, а обработанная протеазой подобная антителу IgG-типа молекула IL6R90-G1m/VK1-39P+4-Pk0MT в 1000 или более раз превышала по IL-6R-связывающей активности необработанную протеазой молекулу.
Пример 13. Получение и оценка подобных антителу IgG-типа молекул, несущих различные расщепляемые протеазой последовательности
13-1 Получение полипептидов, несущих различные расщепляемые протеазой последовательности
Подобные антителу IgG-типа молекулы получали аналогично методу, описанному в примере 3, используя последовательности, распознаваемые протеазами, отличными отурокиназы или матриптазы. Каждую из различных пептидных последовательностей, для которой известно, что она расщепляется ММР-2, ММР-7, ММР-9 или ММР-13, встраивали вблизи границы между вариабельной и константной областями IL6R90-G1m, и пептидную последовательность, которая содержит гибкий линкер, состоящий из глицин-серинового полимера, встраивали в непосредственной близости от указанных расщепляемых последовательностей. Встроенные последовательности представлены в таблице 8.
Тяжелые цепи создавали так, чтобы эти последовательности были встроены вблизи границы между вариабельными и константными областями IL6R90-G1m. Экспрессионные векторы, кодирующие варианты тяжелых цепей 6R90EIVHEMP2.1-6R90EICHEMP2.1G1m (SEQ ID NO: 165), 6R90EIVHEMP2.2-6R90EICHEMP2.2G1m (SEQ ID NO: 202), 6R90EIVHEMP2.3-6R90EICHEMP2.3G1m (SEQ ID NO: 203), 6R90EIVHEMP2.4-6R90EICHEMP2.4G1m (SEQ ID NO: 204), 6R90EIVHEMP7.1-6R90EICHEMP7.1G1m (SEQ ID NO: 205), 6R90EIVHEMP7.2-6R90EICHEMP7.2G1m (SEQ ID NO: 206), 6R90EIVHEMP13-6R90EICHEMP13G1m (SEQ ID NO: 207), 6R90EIVHEG4SMP2MP9G4S-6R90EICHEG4SMP2MP9G4SG1m (SEQ ID NO: 196), 6R90EIVHEG4SMP2.2G4S-6R90EIVHEG4SMP2.2G4SG1m (SEQ ID NO: 197) и 6R90EIVHEG4SMP9G4S-6R90EIVHEG4SMP9G4SG1m (SEQ ID NO: 198), получали с помощью метода, известного специалистам в данной области.
В таблице 9 представлены подобные антителу IgG-типа молекулы, в которых объединены указанные варианты тяжелых цепей с легкой цепью и которые несут расщепляемую протеазой последовательность вблизи границы между вариабельной и константной областями тяжелой цепи. Указанные подобные антителу IgG-типа молекулы экспрессировали путем кратковременной экспрессии с использованием клеток FreeStyle 293 (фирма Invitrogen Corp.) или клеток Expi293 (фирма Life Technologies Corp.) с помощью метода, известного специалистам в данной области, и очищали с помощью метода, известного специалистам в данной области, используя белок А.
13-2 Оценка расщепления протеазой подобных антителу IgG-типа молекул, несущих различные расщепляемые протеазой последовательности
Изучали, могут ли подобные антителу IgG-типа молекулы, полученные согласно методу, описанному в примере 13-1, расщепляться протеазой. В качестве протеазы применяли рекомбинантную человеческую ММР-2 (фирма R&D Systems, Inc., 902-МР-010), рекомбинантную человеческую ММР-7 (фирма R&D Systems, Inc., 907-МР-010), рекомбинантную человеческую ММР-9 (фирма R&D Systems, Inc., 911-МР-010) или рекомбинантную человеческую ММР-13 (фирма R&D Systems, Inc., 511-MM-010). Каждую из MMP-2, MMP-7, ММР-9 и ММР-13 применяли после смешения с 1 мМ ацетатом пара-аминофенилртути (АРМА; фирма Abeam PLC, ab112146) и активировали при 37°С в течение 1 ч или 24 ч. Повергали взаимодействию протеазу в концентрации 50, 100 или 500 нМ и каждую подобную антителу IgG-типа молекулу в концентрации 50 мкг/мл или 100 мкг/мл в ЗФР или 20 мМ Трис-HCl, 150 мМ NaCl и 5 мМ CaCl2 (рН 7,2) (ниже в контексте настоящего описания обозначен как Трис), при 37°С в течение 20 ч. Затем расщепление протеазой оценивали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты представлены на фиг. 30А и 30Б. На 30Б представлены результаты, полученные при осуществлении расщепления протеазой в буфере для анализа (набор для оценки активности ММР-(флуорометрический - зеленый) (ab112146), компонент С: буфер для анализа).
В результате установлено, что 6R90EIVHEMP2.1-6R90EICHEMP2.1G1m/VK1-39-k0MT, 6R90EIVHEMP2.2-6R90EICHEMP2.2G1m/VK1-39-k0MT, 6R90EIVHEMP2.3-6R90EICHEMP2.3G1m/VK1-39-k0MT, 6R90EIVHEMP2.4-6R90EICHEMP2.4G1m/VK1-39-k0MT, 6R90EIVHEG4SMP2MP9G4S-6R90EICHEG4SMP2MP9G4SG1m/VK1-39-k0MT и 6R90EIVHEG4SMP2.2G4S-6R90EICHEG4SMP2.2G4SG1m/VK1-39-k0MT расщеплялись MMP-2. Установлено, что 6R90EIVHEMP7.1-6R90EICHEMP7.1G1m/VK1-39-k0MT и 6R90EIVHEMP7.2-6R90EICHEMP7.2G1m/VK1-39-k0MT расщеплялись ММР-7. Установлено, что 6R90EIVHEG4SMP2MP9G4S-6R90EICHEG4SMP2MP9G4SGlm/VK1-39-k0MT и 6R90EIVHEG4SMP9G4S-6R90EICHEG4SMP9G4SG1m/VK1-39-k0MT расщеплялись ММР-9. Установлено, что конструкция 6R90EIVHEMP13-6R90EICHEMP13G1m/VK1-39-k0MT расщеплялась ММР-13.
Пример 14. Оценка различных расщепляемых протеазой последовательностей
14-1 Получение вариантов антител, несущих различные расщепляемые протеазой последовательности
Расщепляемые протеазой последовательности, представленные в таблице 10, встраивали вблизи границы между вариабельной областью легкой цепи и константной областью антител, которые имели тяжелую цепь, представленную в SEQ ID NO: 831, и легкую цепь, представленную в SEQ ID NO: 832, с получением вариантов легкой цепи, несущих различные расщепляемые протеазой последовательности (таблица 11).
Варианты легкой цепи, несущие расщепляемую протеазой последовательности, которые получали согласно описанному выше методу, объединяли с тяжелой цепью, имеющей SEQ ID NO: 831, и варианты антител, которые представлены в таблице 12, кратковременно экспрессировали с использованием клеток Expi293 (фирма Life technologies) согласно методу, известному специалисту в данной области, и очищали с использованием белка А согласно методу, известному специалисту в данной области.

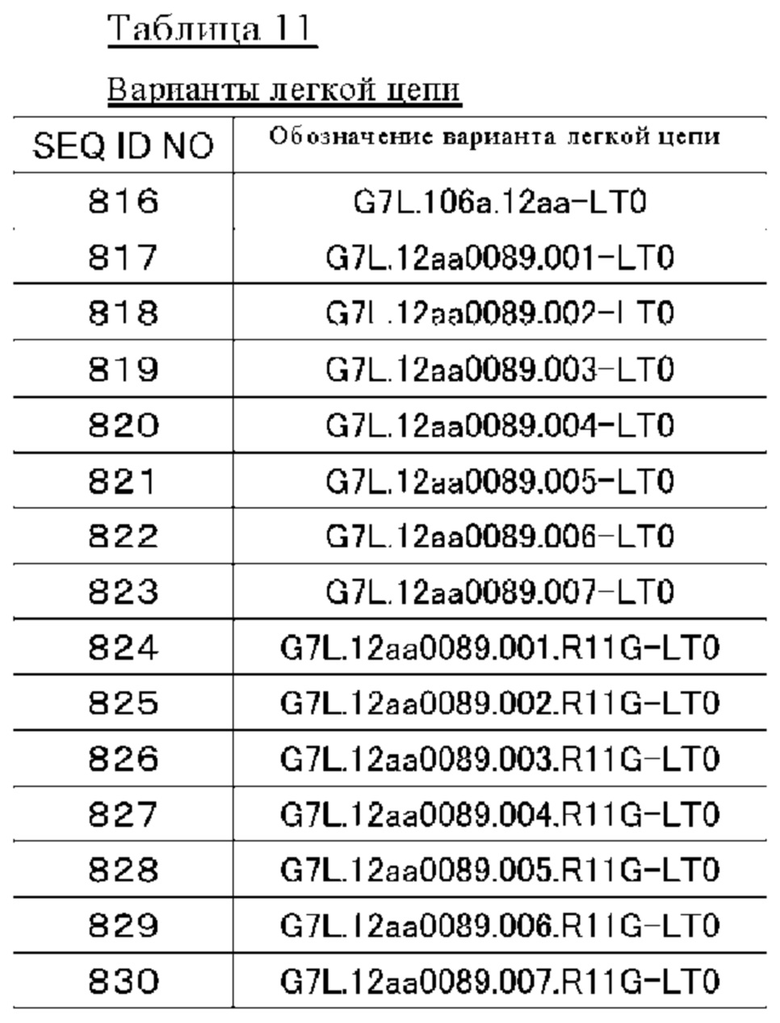
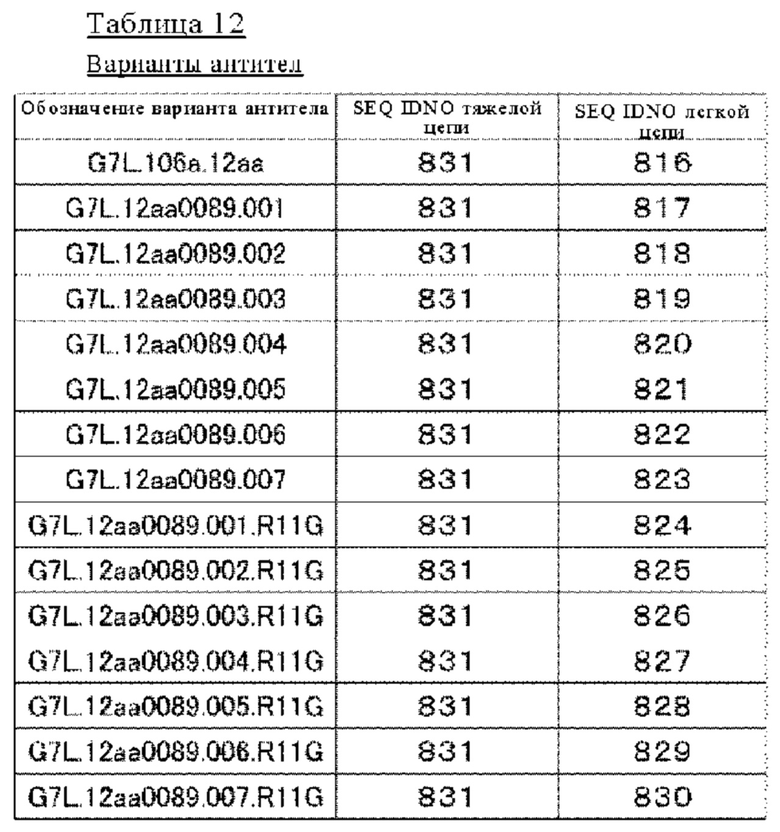
14-2 Оценка расщепляемых протеазой вариантов антител, несущих расщепляемую протеазой последовательность
Варианты антител, полученные согласно методу, описанному в примере 14-1, тестировали для решения вопроса о том, будут ли они расщепляться обработкой протеазой. Применяемая протеаза представляла собой рекомбинантный человеческий активатора u-плазминогена/урокиназу (человеческая uPA, huPA) (фирма R&D Systems; 1310-SE-010). Антителам давали взаимодействовать в течение 1 ч в следующих условиях: 40 нМ протеаза, 100 мкг/мл варианта антитела, ЗФР и 37°С, и затем анализировали с помощью ДСН-ПААГ в восстанавливающих условиях. Результаты подтвердили, что все варианты антител, несущие расщепляемую протеазой последовательность, расщеплялись обработкой протеазой. Другими словами, установлено, что расщепляемые протеазой последовательности, представленные в таблице 10, могут расщепляться протеазой. Кроме того, варианты антител, отличные от G7L.106а.12аа, все расщеплялись более эффективно, чем G7L.106а.12аа.
Пример 15. Оценка различных расщепляемых протеазой последовательностей
15-1 Получение вариантов антител, несущих расщепляемые протеазой последовательности
Помимо расщепляемых протеазой последовательностей, описанных в примере 14, исследовали дополнительно расщепляемые протеазой последовательности для повышения эффективности расщепления и селективности протеаз. Расщепляемые протеазой последовательности, представленные в таблице 13, встраивали вблизи границы между вариабельной областью легкой цепи и константной областью антител, которые имели тяжелую цепь, представленную в SEQ ID NO: 831, и легкую цепь, представленную в SEQ ID NO: 832, с получением вариантов легкой цепи, несущих различные расщепляемые протеазой последовательности (таблицы 14 и 15).
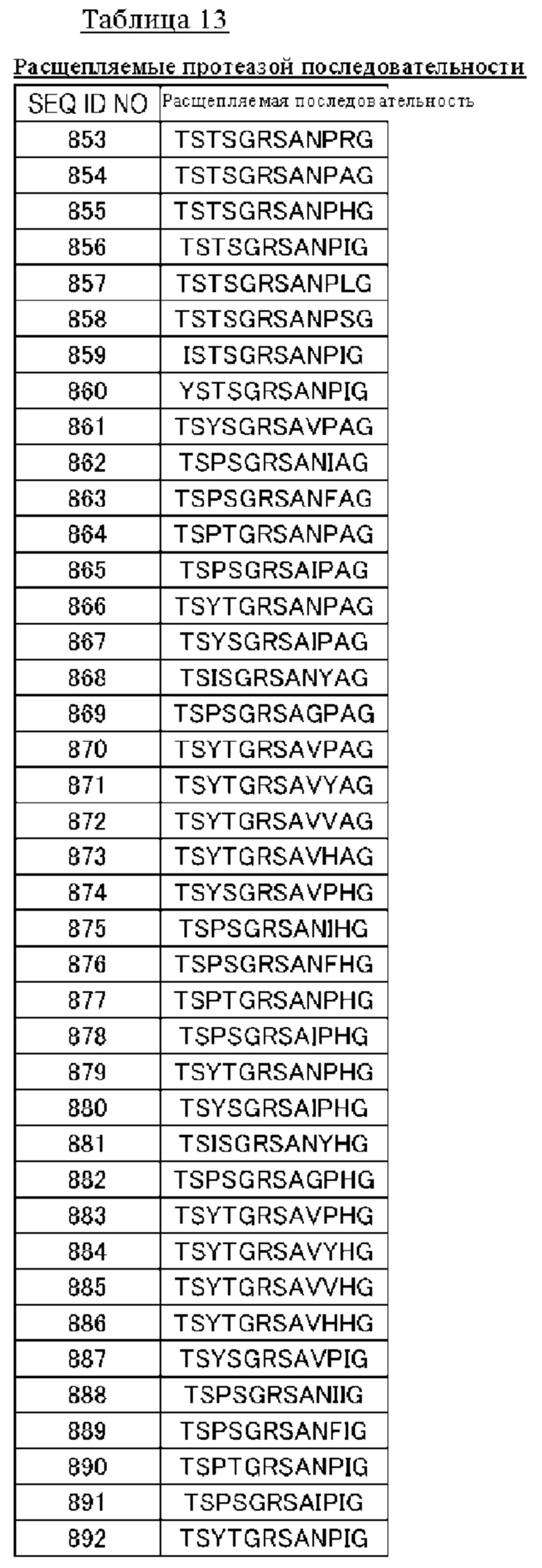
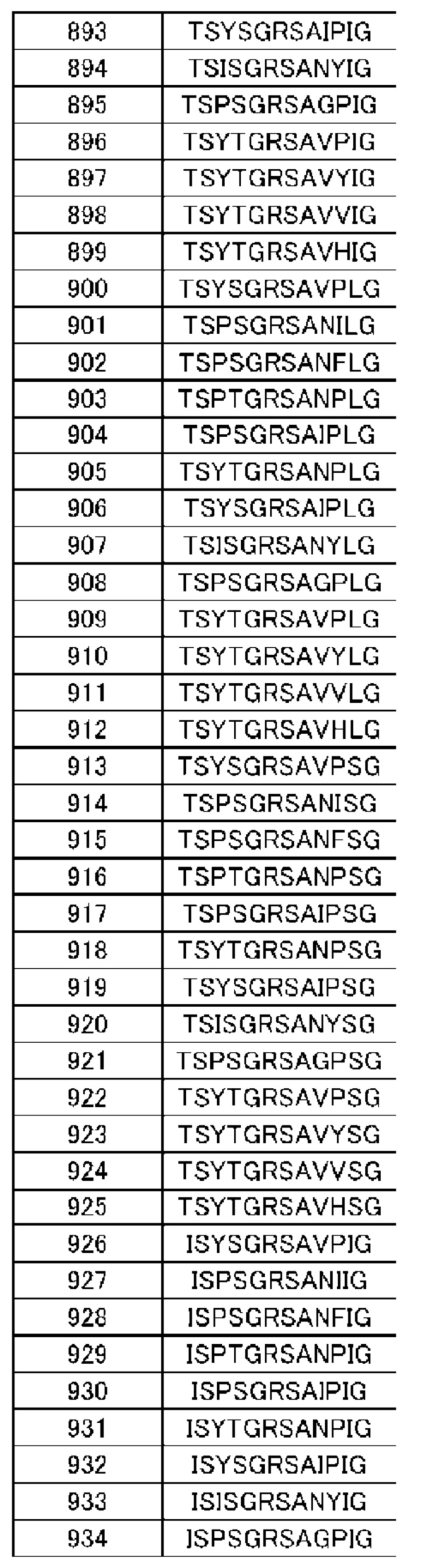
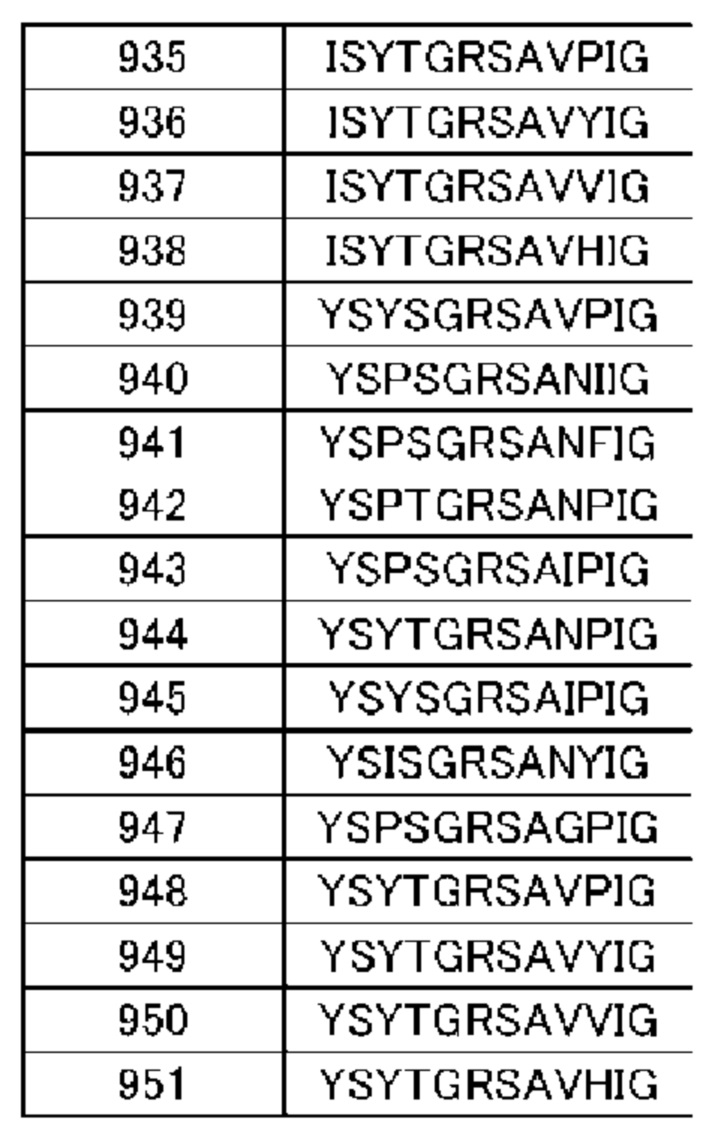
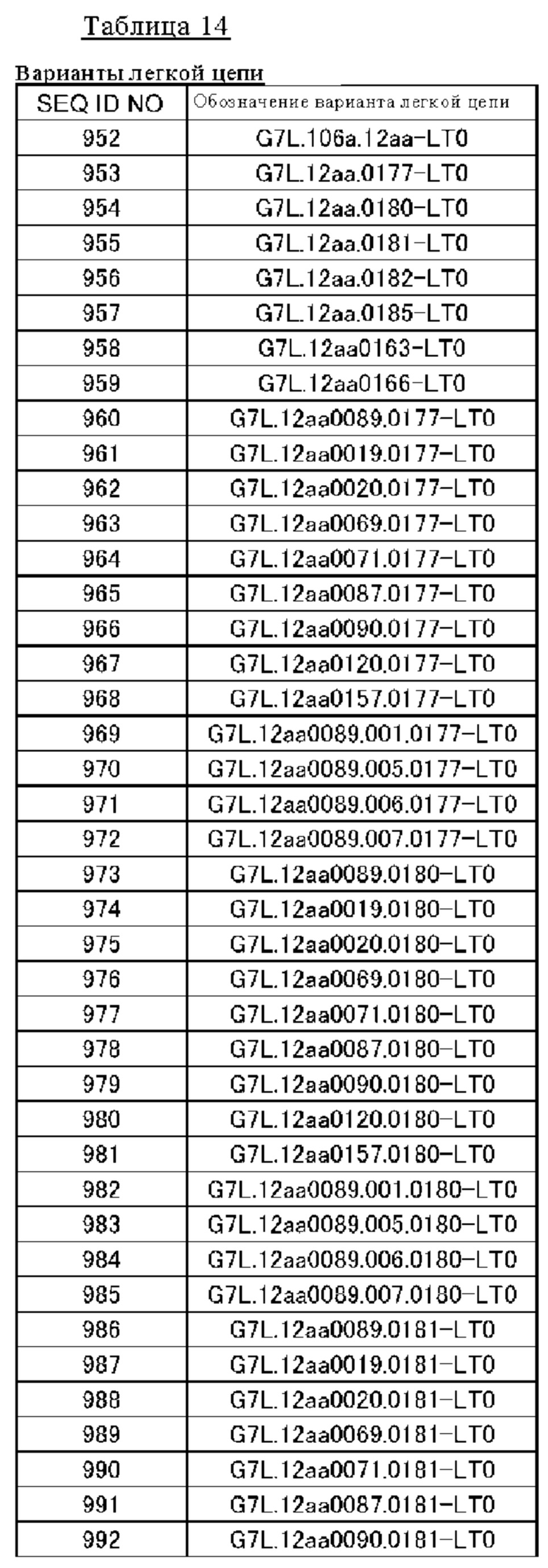
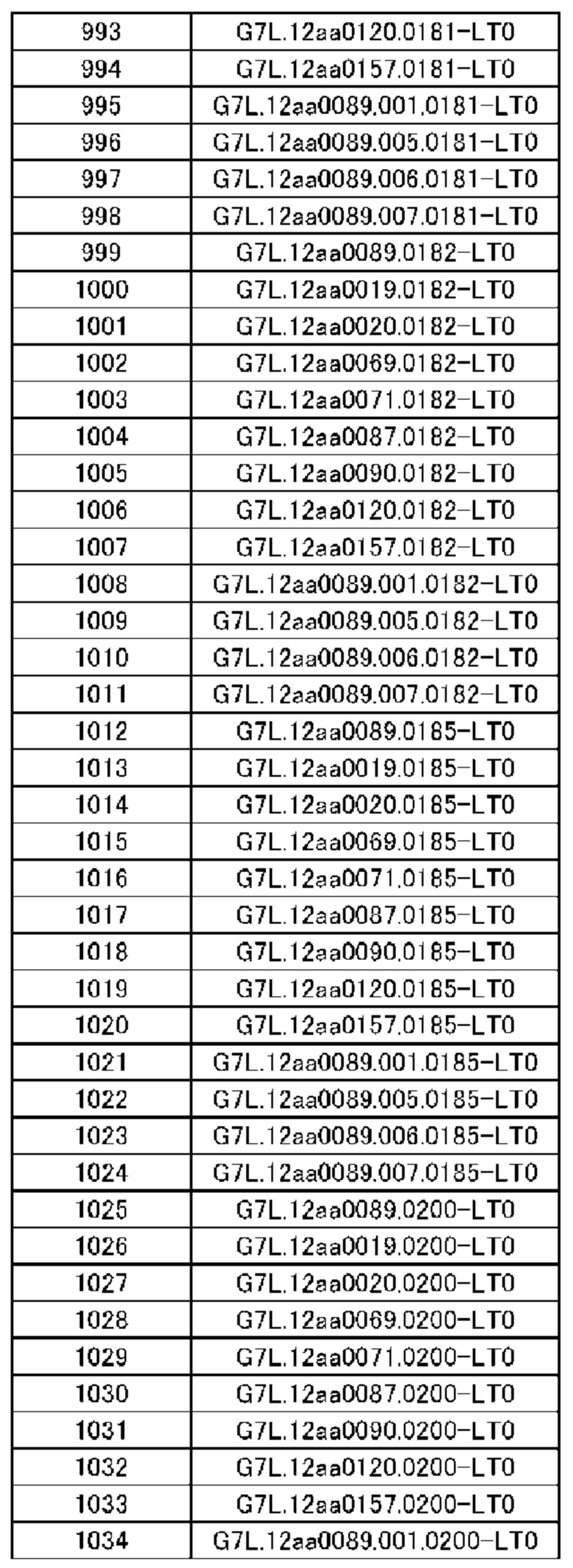
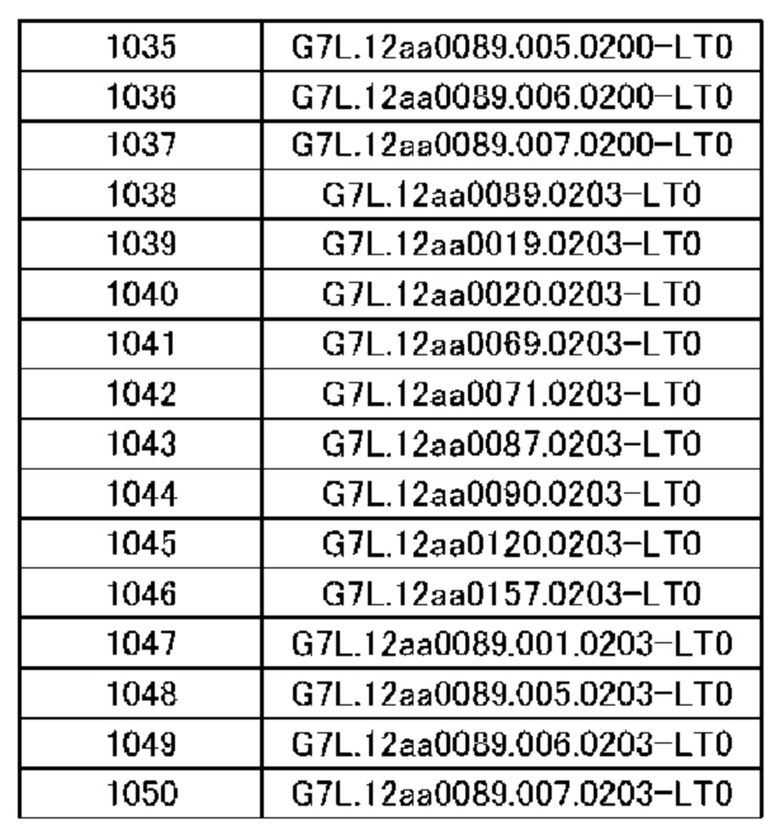
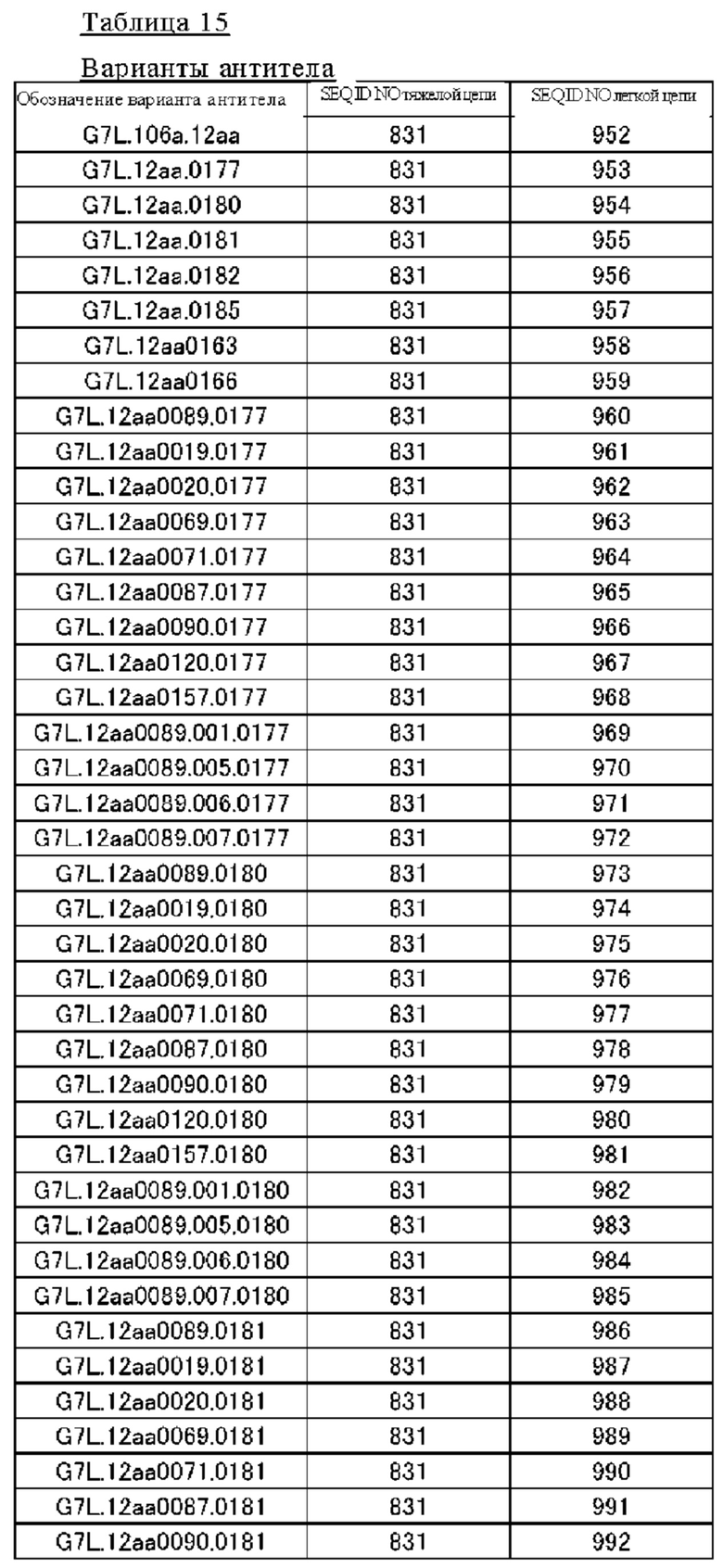
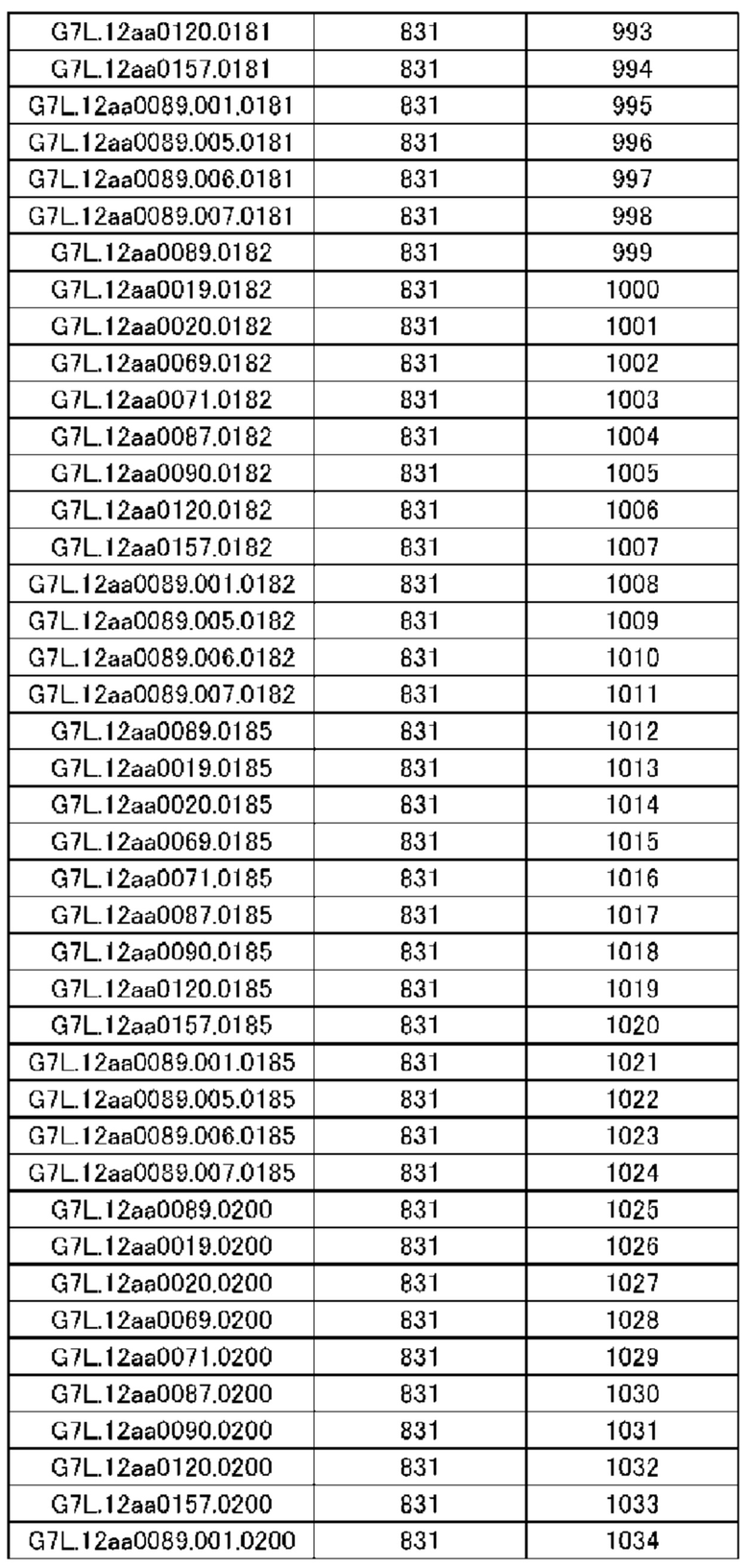
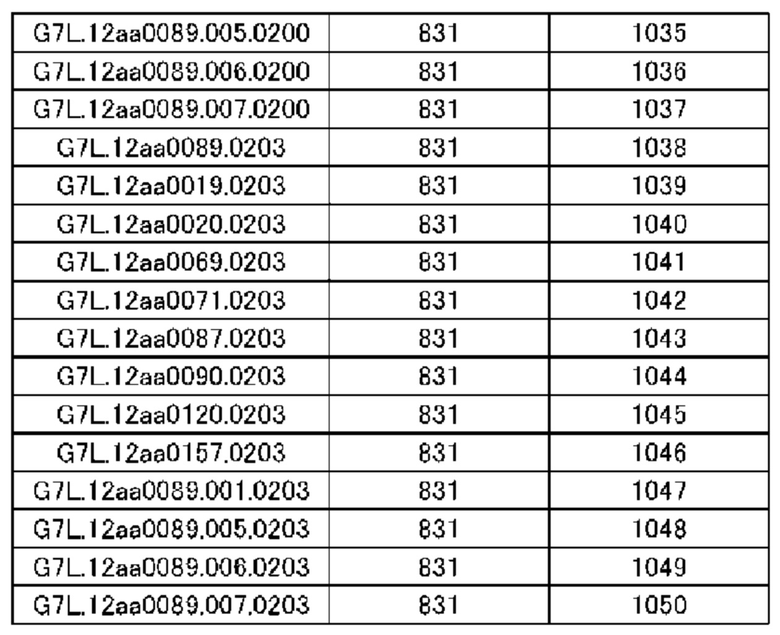
15-2 Оценка расщепляемых протеазой вариантов антител, несущих расщепляемую протеазой последовательность
Варианты антител, полученные согласно методу, описанному в примере 15-1, тестировали для решения вопроса о том, будут ли они расщепляться обработкой протеазой. Применяемые протеазы представляли собой рекомбинантный человеческий активатора u-плазминогена/урокиназу (человеческая uPA, huPA) (фирма R&D Systems; 1310-SE-010) или рекомбинантную человеческую матриптазу/каталитический домен ST14 (человеческая MT-SP1, hMT-SP1) (фирма R&D Systems, Inc., 3946-SE-010). Вариантам антител давали взаимодействовать в течение 1 ч в следующих условиях: 40 нМ huPA или 3 нМ hMT-SP1, 100 мкг/мл варианта антитела, ЗФР и 37°С, и затем подвергали иммуноанализу на основе капиллярного электрофореза. Иммуноанализ на основе капиллярного электрофореза осуществляли с использованием системы Wes (фирма Protein Simple), и антитело к человеческой лямбда-цепи, меченное HRP (фирма abcam; ab9007), применяли для детекции легких цепей до и после расщепления. В результате установлено, что пик, соответствующий примерно 36 кДа, обнаруженный до обработки протеазой, исчезал и появлялся новый пик, соответствующий примерно 20 кДа. Это позволяет предположить, что пик с молекулярной массой примерно 36 кДа соответствовал нерасщепленной легкой цепи варианта антитела, а пик с молекулярной массой примерно 20 кДа соответствовал расщепленной легкой цепи. Площадь каждого пика, полученного после обработки протеазой, определяли с помощью программного обеспечения для системы Wes (программное обеспечение Compass для систем SW; фирма Protein Simple), и степень расщепления (%) варианта антитела определяли с использованием следующей формулы:
(площадь пика расщепленной легкой цепи) × 100 / (площадь пика расщепленной легкой цепи + площадь пика нерасщепленной легкой цепи).
Степени расщепления (%) вариантов антител, обработанных huPA, представлены в таблице 16, а степени расщепления вариантов антител, обработанных hMT-SP1, представлены в таблице 17. Из тех вариантов антител, которые представлены в указанных выше таблицах 16 и 17, варианты с более высокой степенью расщепления huPA, но с более низкой степенью расщепления hMT-SP1, иными словами, варианты с более высокой селективностью в отношении huPA, чем G7L.106а.12аа (тяжелая цепь: G7H-G1T4 (SEQ ID NO: 831), легкая цепь: G7L.106а.12aa-LT0 (SEQ ID NO: 952)), представлены в таблице 18.
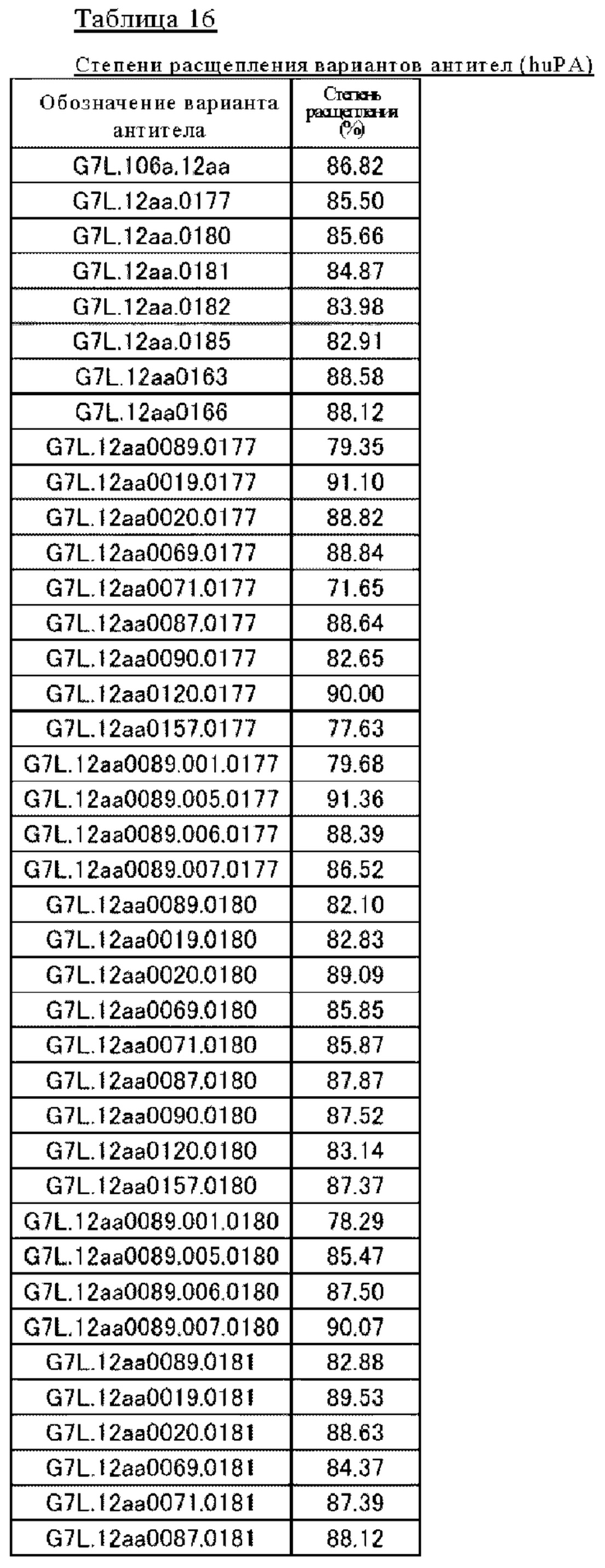
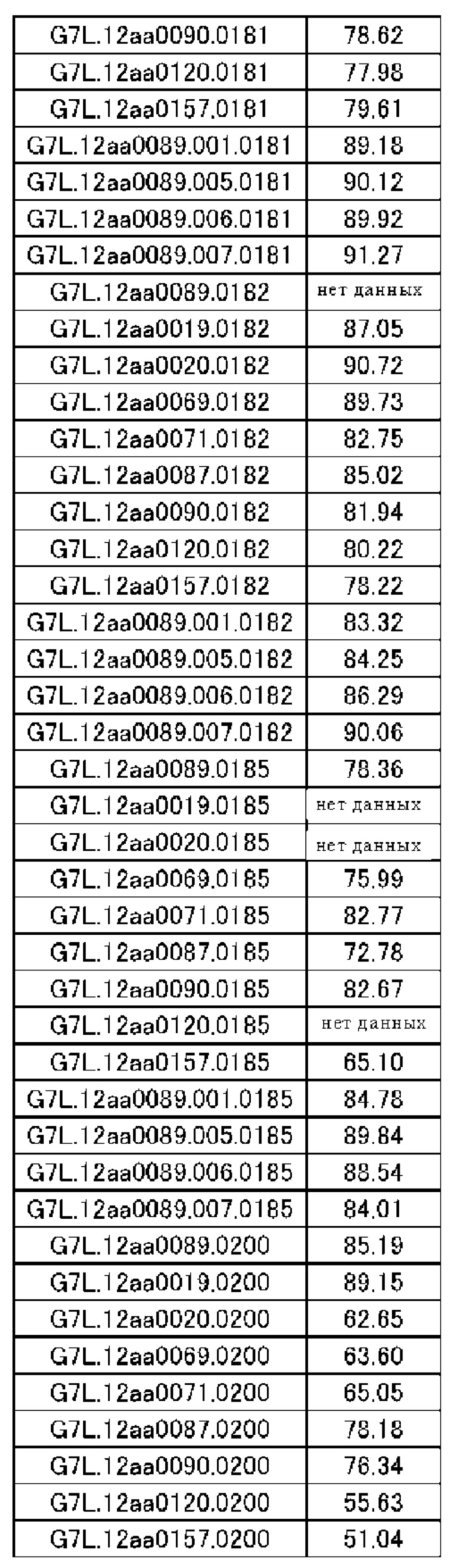
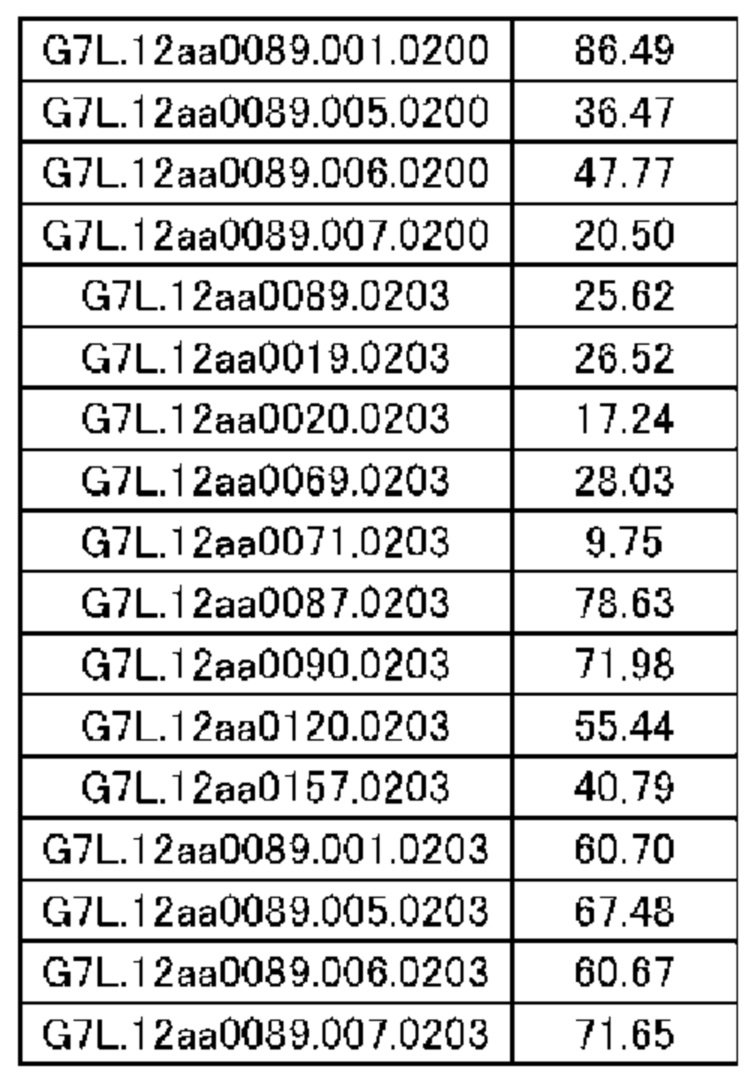
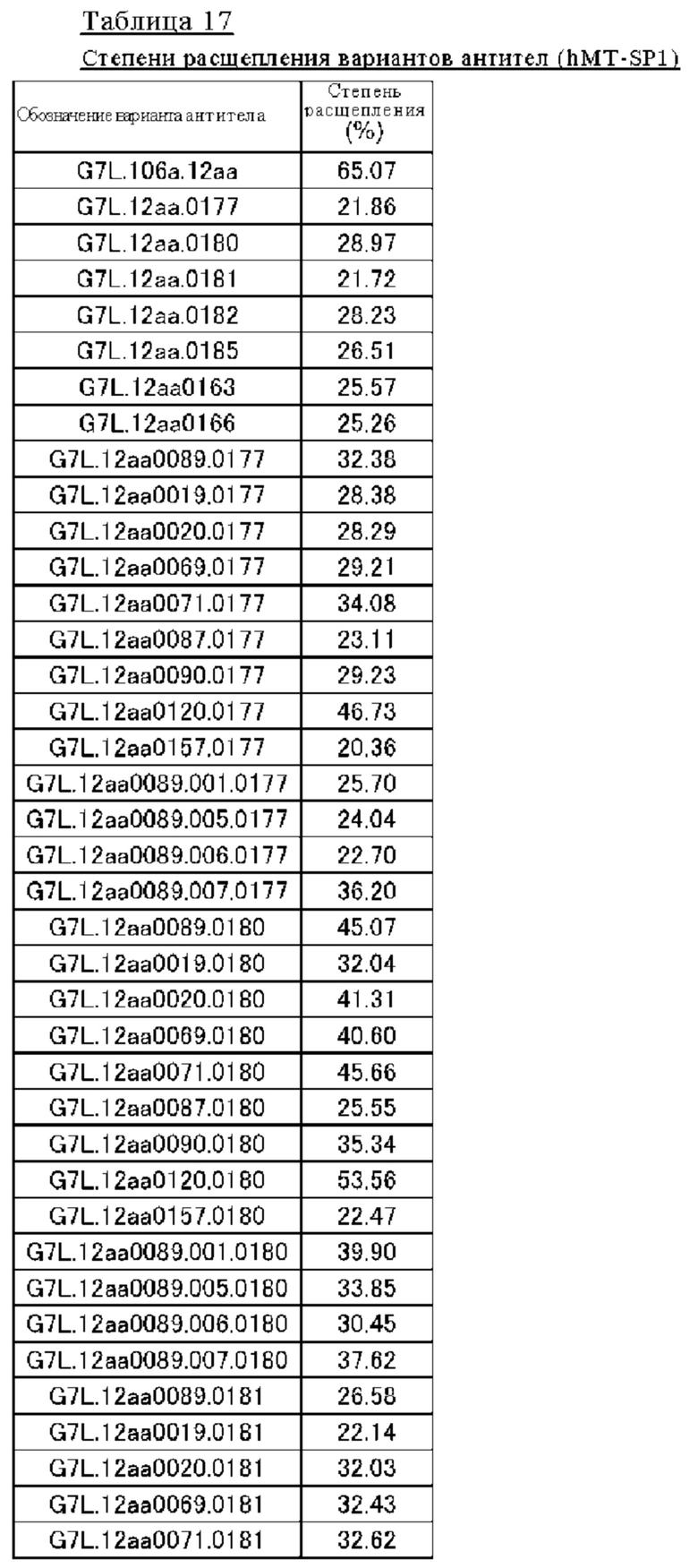
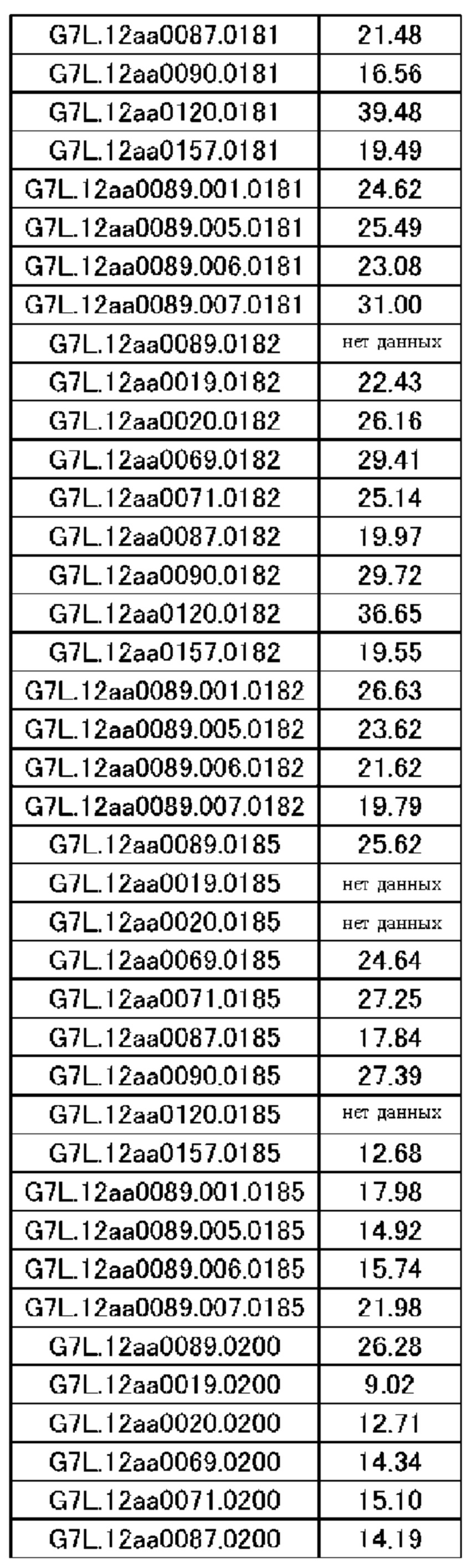
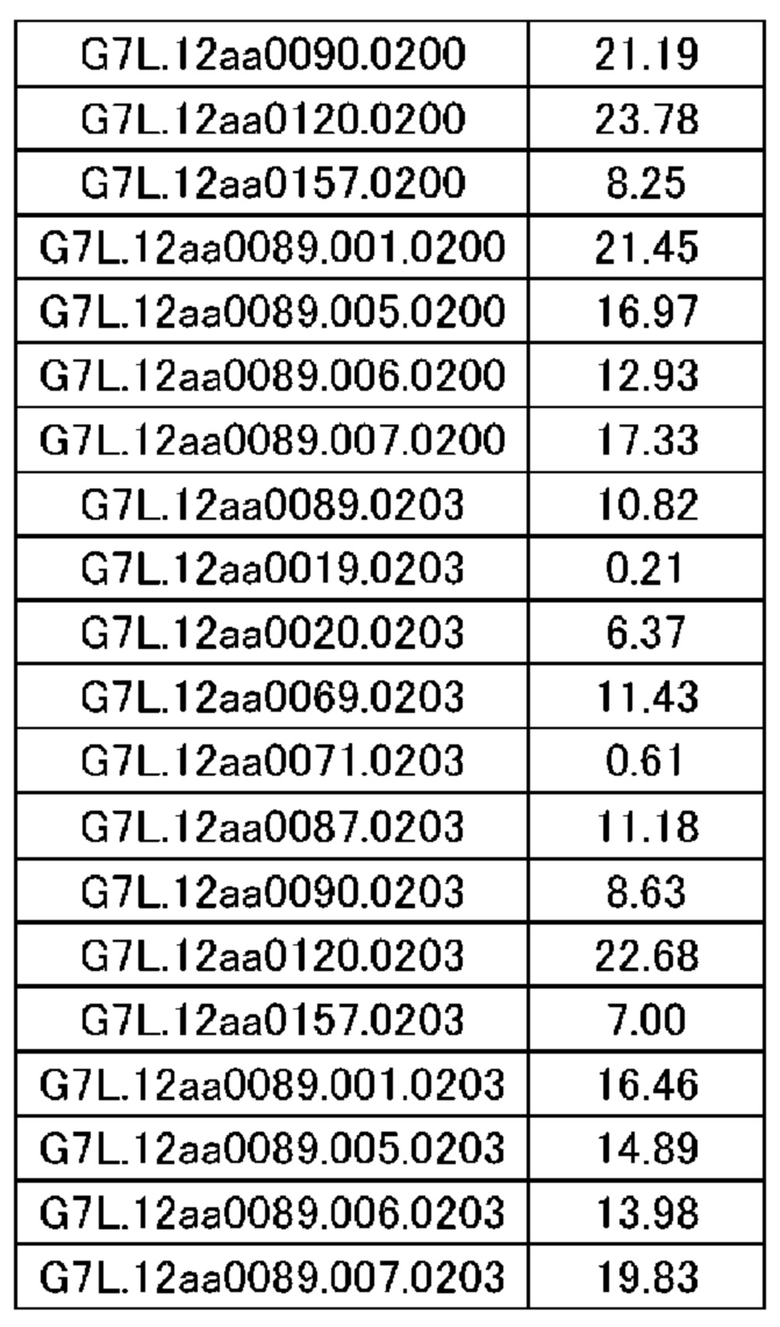
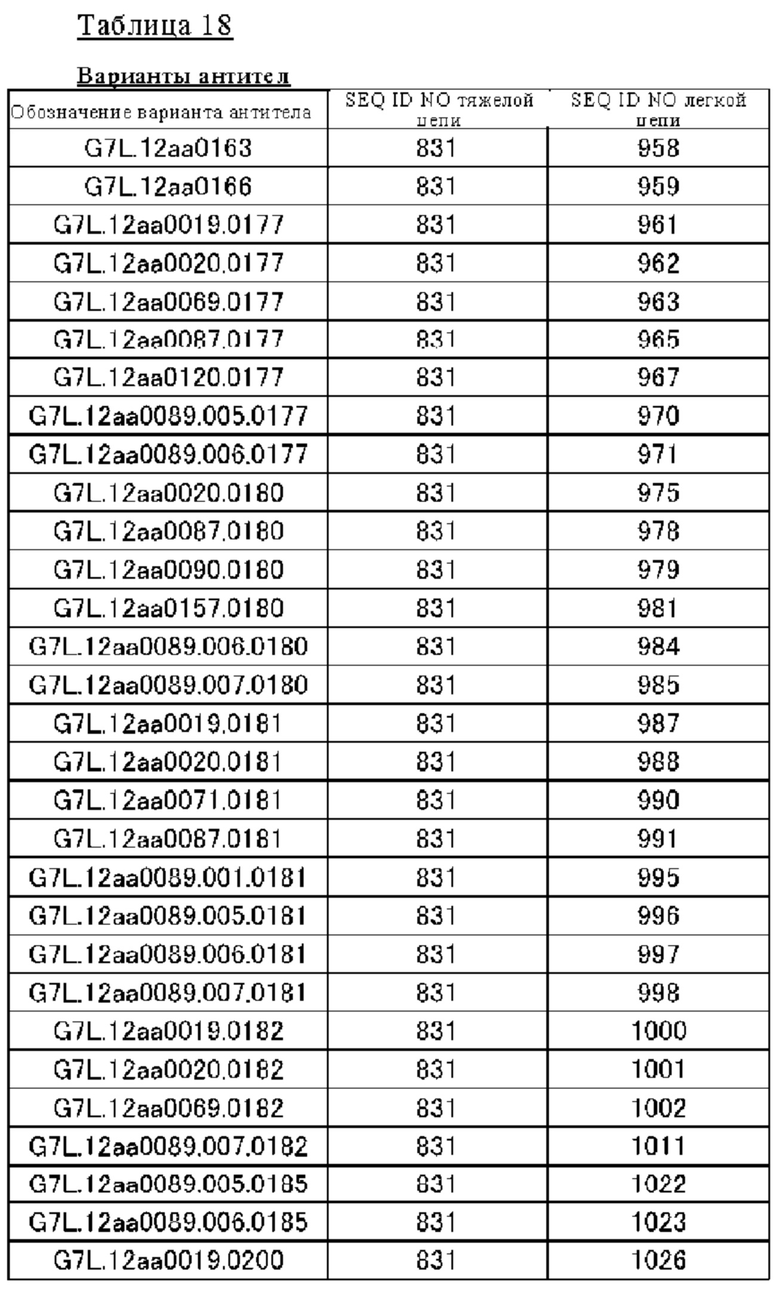
Пример 16. Оценка in vivo расщепляемых протеазой вариантов антител, несущих различные расщепляемые протеазой последовательности
16.1 Получение биспеиифических антител, несущих расщепляемую протеазой последовательность
Расщепляемые протеазой последовательности, представленные в таблице 19, встраивали вблизи границы между вариабельной областью легкой цепи и константной областью антител (родительские антитела), содержащих тяжелую цепь, имеющую SEQ ID NO: 1051, и легкую цепь, имеющую SEQ ID NO: 832, с получением вариантов легкой цепи, несущих различные расщепляемые протеазой последовательности (таблица 20).
Варианты легкой цепи, несущие расщепляемые протеазой последовательности, полученные согласно описанному выше методу, и легкую цепь, имеющую SEQ ID NO: 832, объединяли с тяжелой цепью, имеющей SEQ ID NO: 1051, и варианты антител, представленные в таблице 21, кратковременно экспрессировали с использованием клеток Expi293 (фирма Life technologies) согласно методу, известному специалистам в данной области, и очищали с использованием белка А согласно методу, известному специалистам в данной области.
Кроме того, антитело против гемоцианина лимфы улитки, MabKLHn (тяжелая цепь: IC17HdK-F760mnN17 (SEQ ID NO: 1390), легкая цепь: IC17L-k0 (SEQ ID NO: 1391)), также кратковременно экспрессировали с использованием клеток Expi293 (фирма Life technologies) согласно методу, известному специалистам в данной области, и очищали с использованием белка А согласно методу, известному специалистам в данной области.
MabKLHn смешивали с вариантами антител, представленными в таблице 21, или с родительским антителом, содержащем тяжелую цепь, имеющую SEQ ID NO: 1051, и легкую цепь, имеющую SEQ ID NO: 832, с получением биспецифических антител, которые представлены в таблице 22, с использованием метода, описанного в WO 2015046467.


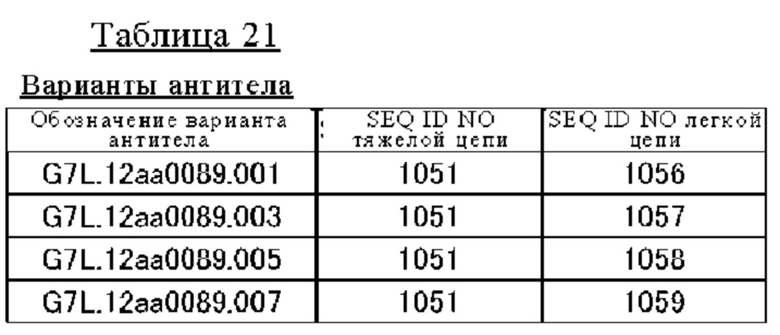
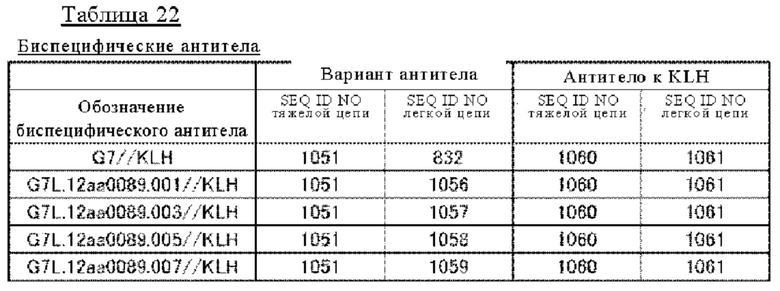
16-2 Получение клеточной линии, стабильно экспрессирующей протеазу B16F10/chGPC3/muPA применяли в качестве стабильно экспрессирующей протеазу клеточной линии для трансплантации мышам. Эту клеточную линию получали путем интродукции гена модифицированного мышиного химерного глипикана 3 (chGPC3) и гена мышиной uPA (muPA: NM_008873) в клеточную линию мышиной меланомы B16F10, и создания и последующего клонирования стабильно экспрессирующей клеточной линии. Клетки B16F10/chGPC3/muPA культивировали в среде RPMI1640 (фирма Nacalai Tesque), содержащей 10% FBS (фирма SIGMA), 0,5 мг/мл генетицина (фирма Gibco) и 1,5 мкг/мл пуромицина (фирма Gibco).
16-3 Получение сингенной мышиной модели с трансплантированной опухолевой линией
Предназначенные для трансплантации мыши представляли собой мышей линии C57BL/6NCrl (возрастом 6 недель, самки), которых покупали у фирмы Charles River Laboratories. Клетки B16F10/chGPC3/muPA трансплантировали подкожно мышам линии C57BL/6NCrl (1Е6 клеток на животное). Когда средний объем трансплантированной опухоли достигал объема примерно от 200 до 300 мм3, мышам, которых использовали в качестве мышиной модели, вводили вариант антитела.
Объем трансплантата опухоли рассчитывали с помощью следующей формулы:
Объем опухоли = длинный диаметр × короткий диаметр × короткий диаметр/2.
16-4 Получение агентов, предназначенных для введения Варианты антител, несущие расщепляемые протеазой последовательности, которые представлены в таблице 22, полученные согласно методу, описанному в примере 16-1, применяли в качестве агентов для введения применяемым в качестве моделей мышам, которым трансплантировали клетки B16F10/chGPC3/muPA. Предназначенные для введения агенты получали с использованием ЗФРТ-буфера (буфер ЗФР+0,05% Твин20) так, чтобы концентрация варианта антитела составляла 0,1 мг/мл.
16-5 Тест по введению вариантов антител для оценки расщепления пр отеазой
Через 11 дней после трансплантации мышам, трансплантированным клетками B16F10/chGPC3/muPA, вводили пять образцов вариантов антител, несущих различные расщепляемые протеазой последовательности, через хвостовую вену в дозе 1 мг/кг (мг вводимого антитела на кг веса тела мыши). Обозначения вариантов антител, дозы, методы введения и другие детали опыта по введению представлены в таблице 23.
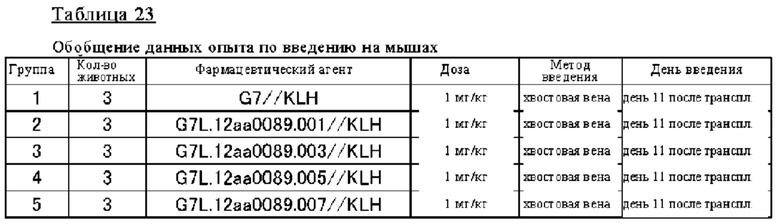
16-6 Сбор крови из орбитального синуса у применяемых в качестве моделей мышей, транслантированных клетками B16F10/chGPC3/muPA
В дни 1 и 3 после введения варианта антитела кровь получали из глазницы применяемых в качестве моделей мышей, трансплантированных клетками
B16F10/chGPC3/muPA. Сбор крови осуществляли под анестезией изофлурананом. Собранную кровь центрифугировали при 1900×g, 4°С в течение 10 мин. После центрифугирования из компонентов плазмы получали супернатант и хранили при -30°С.
16-7 Оценка расщепления введенных антител, полученных из организма мышей
Антитела очищали из плазмы, собранной согласно методу, описанному в примере 16-6, используя Dynabeads с белком А (фирма Thermo; 10001D) согласно методу, известному специалистам в данной области, и затем подвергали иммуноанализу на основе капиллярного электрофореза для оценки эффективности расщепления протеазой вариантов антител. Систему Wes (фирма Protein Simple) применяли для осуществления иммуноанализа на основе капиллярного электрофореза. Для детекции легкой цепи антитела применяли антитело к человеческой лямбда-цепи, меченное HRP (фирма abeam; ab9007). Для детекции тяжелой цепи антитела применяли антитело к человеческой тяжелой цепи, меченное HRP (фирма Protein Simple; 043-491). В результате установлено, что пик, соответствующий нерасщепленной полноразмерной легкой цепи, обнаруженный с помощью антитела к человеческой лямбда-цепи, соответствовал примерно 36 кДа, а пик полноразмерной тяжелой цепи, обнаруженный с помощью антитела к человеческой тяжелой цепи, соответствовал примерно 56 кДа. Легкая цепь MabKLHn представляет собой каппа-цепь, которая не поддавалась обнаружению при использовании антитела к человеческой лямбда-цепи. Таким образом, антитело к человеческой лямбда-цепи можно применять для оценки эффективности расщепления легкой цепи, несущей расщепляемую протеазой последовательность. Площадь каждого пика, полученного с помощью иммуноанализа на основе капиллярного электрофореза, определяли с использованием программного обеспечения для системы Wes (программное обеспечение Compass для систем SW; фирма Protein Simple), и долю оставшейся легкой цепи рассчитывали как [площадь пика легкой цепи]/[площадь пика тяжелой цепи], для определения доли полноразмерной легкой цепи, оставшейся нерасщепленной в организме мышей.
Данные о долях оставшихся легких цепей антител, полученных из организма мышей через один день и три дня после введения мышам, представлены на фиг. 31. В результате установлено, что варианты антител, несущие расщепляемую протеазой последовательность, которые представлены в таблице 21, имели меньшую долю оставшихся легких цепей по сравнению с родительским антителом, содержащим тяжелую цепь, имеющую SEQ ID NO: 1051, и легкую цепь, имеющую SEQ ID NO: 832, в организме мышей с трансплантированными опухолями. Таким образом, установлено, что легкие цепи, несущие расщепляемую протеазой последовательность, эффективно расщеплялись in vivo в организме мышей с трансплантированными опухолями. Референс-пример 1. Получение биотинилированного плексина А1 Биотинилированный плексин А1 (обозначенный также как меченный биотином человеческий плексин А1) получали с помощью метода, известного специалистам в данной области. В частности, генный фрагмент, кодирующий конкретную последовательность (последовательность Avi-метки последовательность; SEQ ID NO: 36), подлежащую биотинилированию с помощью биотинлигазы, и генный фрагмент, кодирующий последовательность FLAG-метки (SEQ ID NO: 199; DYKDDDDK), соединяли через генный фрагмент, кодирующий линкер, состоящий из глицина и серина, в прямом направлении относительно генного фрагмента, кодирующего внеклеточную область плексина А1. Генный фрагмент, кодирующий белок, который содержит плексин А1, сцепленный с последовательностью Avi-метки и последовательностью FLAG-метки (SEQ ID NO: 200), встраивали в вектор для экспрессии в клетках животных. Сконструированным плазмидным вектором трансфектировали клетки FreeStyle 293 (фирма Invitrogen Corp.), используя 293Fectin (фирма Invitrogen Corp.). При осуществлении указанной операции клетки совместно трансфектировали геном для экспрессии EBNA1 (SEQ ID NO: 57), геном для экспрессии биотинлигазы (BirA; SEQ ID NO: 58) и к ним дополнительно добавляли биотин для мечения с помощью биотина плексина А1. Клетки, трансфектированные с помощью упомянутых выше процедур, культивировали при 37°С в атмосфере с 8% СО2 и в результате происходила секреция представляющего интерес белка (биотинилированный плексин А1) в супернатант культуры. Этот раствор клеточной культуры фильтровали, используя емкость с 0,22-микрометровым верхним фильтром, для получения супернатанта культуры.
Колонку упаковывали агарозой с антителом к FLAG М2 (Анти FLAG М2) (фирма Sigma-Aldrich Co. LLC, №A2220) с получением колонки для FLAG. Колонку для FLAG уравновешивали предварительно D-ЗФР(-). В нее вносили супернатант культуры для связывания биотинилированного плексина А1 с колонкой. Затем биотинилированный плексин А1 элюировали, используя пептид FLAG, растворенный в D-ЗФР(-). Агрегаты удаляли из этого элюата с помощью гель-фильтрации, используя HiLoad 26/600 Супердекс 200 пг, 320 мл (фирма GE Healthcare Japan Corp., 28-9893-36), с получением очищенного биотинилированного плексина А1.
С целью лучшего понимания упомянутые выше варианты осуществления изобретения описаны подробно со ссылкой на фактические примеры и иллюстративные примеры. Однако описание и иллюстрации, представленные в настоящем описании, не следует интерпретировать как ограничивающие объем настоящего изобретения. Содержание всей патентной литературы и научной литературы, процитированной в настоящем описании, специально полностью включено в него в качестве ссылки.
Промышленная применимость
Полипептиды, предлагаемые в настоящем изобретении, которые содержат антигенсвязывающий домен и транспортирующий фрагмент, обладающий более длительным временем полужини в крови по сравнению с антигенсвязывающим доменом, и имеющие ингибирующий домен, который ингибирует связывающую активность антигенсвязывающего домена, и фармацевтические композиции, содержащие полипептид, могут транспортировать антигенсвязывающий домен в кровь, ингибируя при этом антигенсвязывающую активность антигенсвязывающего домена. Кроме того, применение полипептида, предлагаемого в настоящем изобретении, может позволять антигенсвязывающему домену проявлять свою антигенсвязывающую активность конкретно в пораженных болезнью областях. Кроме того, поскольку антигенсвязывающий домен имеет более короткое время полужизни в момент проявления свой антигенсвязывающей активности по сравнению со временем транспортировки, то снижается риск системного действия. Таким образом, полипептиды и фармацевтические композиции, предлагаемые в настоящем изобретении, являются очень ценными для лечения заболеваний.
Однодоменное антитело, антигеневязывающая активность которого ингибируется при ассоциации с конкретным VL, VH или VHH, можно подвергать скринингу или получать в качестве одного из примеров антигенсвязывающего домена, предназначенного для эффективного получения полипептида, предлагаемого в настоящем изобретении. Кроме того, необходимые антигенсвязывающие домены можно эффективно создавать, когда полипептид, предлагаемый в настоящем изобретении, получают с использованием библиотеки, включающей однодоменное антитело, антигенсвязывающая активность которого ингибируется при ассоциации с конкретным VL, VH или VHH, в качестве одного из примеров антигенсвязывающего домена, который можно применять в полипептидах, предлагаемых в настоящем изобретении.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ЧУГАИ СЕЙЯКУ КАБУСИКИ КАЙСЯ
<120> ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И
ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ
<130> C1-A1724P
<150> JP 2017-227650
<151> 2017-11-28
<150> JP 2018-103682
<151> 2018-05-30
<160> 1061
<170> PatentIn version 3.5
<210> 1
<211> 127
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 2
<211> 455
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 2
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro
450 455
<210> 3
<211> 214
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 3
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 4
<211> 219
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 4
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Arg
85 90 95
Leu His Trp Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 5
<211> 215
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 5
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Arg Ser Pro
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 6
<211> 217
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 6
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Asn Ser
85 90 95
Leu Asn Ala Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu
115 120 125
Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe
130 135 140
Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val
145 150 155 160
Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys
165 170 175
Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser
180 185 190
His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu
195 200 205
Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 7
<211> 216
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 7
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Gly Gly Val
85 90 95
Thr Gly Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 8
<211> 217
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 8
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Arg Ser Gly
85 90 95
Tyr Thr Leu Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu
115 120 125
Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe
130 135 140
Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val
145 150 155 160
Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys
165 170 175
Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser
180 185 190
His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu
195 200 205
Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 9
<211> 213
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 9
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Lys
1 5 10 15
Thr Ala Arg Ile Thr Cys Gly Gly Asn Asn Ile Gly Ser Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Tyr Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Ser Ser Thr Asp Val
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala
100 105 110
Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala
115 120 125
Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala
130 135 140
Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val
145 150 155 160
Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser
165 170 175
Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr
180 185 190
Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala
195 200 205
Pro Thr Glu Cys Ser
210
<210> 10
<211> 107
<212> PRT
<213> Homo sapiens
<400> 10
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 11
<211> 106
<212> PRT
<213> Homo sapiens
<400> 11
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
1 5 10 15
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
20 25 30
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
35 40 45
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
50 55 60
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
65 70 75 80
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
85 90 95
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
100 105
<210> 12
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 12
Leu Ser Gly Arg Ser Asp Asn His
1 5
<210> 13
<211> 463
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 13
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Leu Ser Gly Arg Ser Asp Asn His Thr Lys Gly Pro Ser Val Phe
130 135 140
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
145 150 155 160
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
165 170 175
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
195 200 205
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
210 215 220
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
225 230 235 240
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
245 250 255
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
260 265 270
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
275 280 285
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
290 295 300
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
305 310 315 320
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
325 330 335
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
340 345 350
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
355 360 365
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
370 375 380
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
385 390 395 400
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
405 410 415
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
420 425 430
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
435 440 445
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 14
<211> 473
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 14
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn His Gly Ser
130 135 140
Ser Gly Gly Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
145 150 155 160
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
165 170 175
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
180 185 190
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
195 200 205
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
210 215 220
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
225 230 235 240
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
245 250 255
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
260 265 270
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
275 280 285
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
290 295 300
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
305 310 315 320
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
325 330 335
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
340 345 350
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
355 360 365
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
370 375 380
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
385 390 395 400
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
405 410 415
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
420 425 430
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
435 440 445
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
450 455 460
Thr Gln Lys Ser Leu Ser Leu Ser Pro
465 470
<210> 15
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 15
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Leu Ser Gly
115 120 125
Arg Ser Asp Asn His Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 16
<211> 473
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 16
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Gly Gly
115 120 125
Ser Gly Leu Ser Gly Arg Ser Asp Asn His Gly Ser Ser Gly Gly Ser
130 135 140
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
145 150 155 160
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
165 170 175
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
180 185 190
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
195 200 205
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
210 215 220
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
225 230 235 240
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
245 250 255
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
260 265 270
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
275 280 285
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
290 295 300
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
305 310 315 320
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
325 330 335
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
340 345 350
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
355 360 365
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
370 375 380
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
385 390 395 400
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
405 410 415
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
420 425 430
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
435 440 445
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
450 455 460
Thr Gln Lys Ser Leu Ser Leu Ser Pro
465 470
<210> 17
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 17
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Leu Ser Gly Arg Ser Asp Asn His Gly Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 18
<211> 473
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 18
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn His
130 135 140
Gly Ser Ser Gly Gly Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
145 150 155 160
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
165 170 175
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
180 185 190
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
195 200 205
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
210 215 220
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
225 230 235 240
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
245 250 255
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
260 265 270
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
275 280 285
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
290 295 300
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
305 310 315 320
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
325 330 335
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
340 345 350
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
355 360 365
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
370 375 380
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
385 390 395 400
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
405 410 415
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
420 425 430
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
435 440 445
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
450 455 460
Thr Gln Lys Ser Leu Ser Leu Ser Pro
465 470
<210> 19
<211> 121
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 19
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Tyr Arg Gln Ala Pro Gly Lys Gly Arg Glu Leu Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 20
<211> 121
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 20
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 21
<211> 330
<212> PRT
<213> Homo sapiens
<400> 21
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 22
<211> 326
<212> PRT
<213> Homo sapiens
<400> 22
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 23
<211> 377
<212> PRT
<213> Homo sapiens
<400> 23
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Leu Lys Thr Pro Leu Gly Asp Thr Thr His Thr Cys Pro
100 105 110
Arg Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg
115 120 125
Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys
130 135 140
Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys Pro
145 150 155 160
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
165 170 175
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
180 185 190
Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Lys Trp Tyr
195 200 205
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
210 215 220
Gln Tyr Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Leu His
225 230 235 240
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
245 250 255
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln
260 265 270
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
275 280 285
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
290 295 300
Ser Asp Ile Ala Val Glu Trp Glu Ser Ser Gly Gln Pro Glu Asn Asn
305 310 315 320
Tyr Asn Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu
325 330 335
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Ile
340 345 350
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn Arg Phe Thr Gln
355 360 365
Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375
<210> 24
<211> 327
<212> PRT
<213> Homo sapiens
<400> 24
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 25
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 25
Pro Leu Gly Leu Ala Gly
1 5
<210> 26
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 26
Val Pro Leu Ser Leu Thr Met Gly
1 5
<210> 27
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 27
Gly Ser Gly Gly Ser
1 5
<210> 28
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 28
Gly Gly Gly Ser
1
<210> 29
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 29
Gly Gly Ser Gly
1
<210> 30
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 30
Gly Gly Ser Gly Gly
1 5
<210> 31
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 31
Gly Ser Gly Ser Gly
1 5
<210> 32
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 32
Gly Ser Gly Gly Gly
1 5
<210> 33
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 33
Gly Gly Gly Ser Gly
1 5
<210> 34
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 34
Gly Ser Ser Ser Gly
1 5
<210> 35
<211> 338
<212> PRT
<213> Homo sapiens
<400> 35
Leu Ala Pro Arg Arg Cys Pro Ala Gln Glu Val Ala Arg Gly Val Leu
1 5 10 15
Thr Ser Leu Pro Gly Asp Ser Val Thr Leu Thr Cys Pro Gly Val Glu
20 25 30
Pro Glu Asp Asn Ala Thr Val His Trp Val Leu Arg Lys Pro Ala Ala
35 40 45
Gly Ser His Pro Ser Arg Trp Ala Gly Met Gly Arg Arg Leu Leu Leu
50 55 60
Arg Ser Val Gln Leu His Asp Ser Gly Asn Tyr Ser Cys Tyr Arg Ala
65 70 75 80
Gly Arg Pro Ala Gly Thr Val His Leu Leu Val Asp Val Pro Pro Glu
85 90 95
Glu Pro Gln Leu Ser Cys Phe Arg Lys Ser Pro Leu Ser Asn Val Val
100 105 110
Cys Glu Trp Gly Pro Arg Ser Thr Pro Ser Leu Thr Thr Lys Ala Val
115 120 125
Leu Leu Val Arg Lys Phe Gln Asn Ser Pro Ala Glu Asp Phe Gln Glu
130 135 140
Pro Cys Gln Tyr Ser Gln Glu Ser Gln Lys Phe Ser Cys Gln Leu Ala
145 150 155 160
Val Pro Glu Gly Asp Ser Ser Phe Tyr Ile Val Ser Met Cys Val Ala
165 170 175
Ser Ser Val Gly Ser Lys Phe Ser Lys Thr Gln Thr Phe Gln Gly Cys
180 185 190
Gly Ile Leu Gln Pro Asp Pro Pro Ala Asn Ile Thr Val Thr Ala Val
195 200 205
Ala Arg Asn Pro Arg Trp Leu Ser Val Thr Trp Gln Asp Pro His Ser
210 215 220
Trp Asn Ser Ser Phe Tyr Arg Leu Arg Phe Glu Leu Arg Tyr Arg Ala
225 230 235 240
Glu Arg Ser Lys Thr Phe Thr Thr Trp Met Val Lys Asp Leu Gln His
245 250 255
His Cys Val Ile His Asp Ala Trp Ser Gly Leu Arg His Val Val Gln
260 265 270
Leu Arg Ala Gln Glu Glu Phe Gly Gln Gly Glu Trp Ser Glu Trp Ser
275 280 285
Pro Glu Ala Met Gly Thr Pro Trp Thr Glu Ser Arg Ser Pro Pro Ala
290 295 300
Glu Asn Glu Val Ser Thr Pro Met Gln Ala Leu Thr Thr Asn Lys Asp
305 310 315 320
Asp Asp Asn Ile Leu Phe Arg Asp Ser Ala Asn Ala Thr Ser Leu Pro
325 330 335
Val Gln
<210> 36
<211> 15
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 36
Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu
1 5 10 15
<210> 37
<211> 358
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 37
Leu Ala Pro Arg Arg Cys Pro Ala Gln Glu Val Ala Arg Gly Val Leu
1 5 10 15
Thr Ser Leu Pro Gly Asp Ser Val Thr Leu Thr Cys Pro Gly Val Glu
20 25 30
Pro Glu Asp Asn Ala Thr Val His Trp Val Leu Arg Lys Pro Ala Ala
35 40 45
Gly Ser His Pro Ser Arg Trp Ala Gly Met Gly Arg Arg Leu Leu Leu
50 55 60
Arg Ser Val Gln Leu His Asp Ser Gly Asn Tyr Ser Cys Tyr Arg Ala
65 70 75 80
Gly Arg Pro Ala Gly Thr Val His Leu Leu Val Asp Val Pro Pro Glu
85 90 95
Glu Pro Gln Leu Ser Cys Phe Arg Lys Ser Pro Leu Ser Asn Val Val
100 105 110
Cys Glu Trp Gly Pro Arg Ser Thr Pro Ser Leu Thr Thr Lys Ala Val
115 120 125
Leu Leu Val Arg Lys Phe Gln Asn Ser Pro Ala Glu Asp Phe Gln Glu
130 135 140
Pro Cys Gln Tyr Ser Gln Glu Ser Gln Lys Phe Ser Cys Gln Leu Ala
145 150 155 160
Val Pro Glu Gly Asp Ser Ser Phe Tyr Ile Val Ser Met Cys Val Ala
165 170 175
Ser Ser Val Gly Ser Lys Phe Ser Lys Thr Gln Thr Phe Gln Gly Cys
180 185 190
Gly Ile Leu Gln Pro Asp Pro Pro Ala Asn Ile Thr Val Thr Ala Val
195 200 205
Ala Arg Asn Pro Arg Trp Leu Ser Val Thr Trp Gln Asp Pro His Ser
210 215 220
Trp Asn Ser Ser Phe Tyr Arg Leu Arg Phe Glu Leu Arg Tyr Arg Ala
225 230 235 240
Glu Arg Ser Lys Thr Phe Thr Thr Trp Met Val Lys Asp Leu Gln His
245 250 255
His Cys Val Ile His Asp Ala Trp Ser Gly Leu Arg His Val Val Gln
260 265 270
Leu Arg Ala Gln Glu Glu Phe Gly Gln Gly Glu Trp Ser Glu Trp Ser
275 280 285
Pro Glu Ala Met Gly Thr Pro Trp Thr Glu Ser Arg Ser Pro Pro Ala
290 295 300
Glu Asn Glu Val Ser Thr Pro Met Gln Ala Leu Thr Thr Asn Lys Asp
305 310 315 320
Asp Asp Asn Ile Leu Phe Arg Asp Ser Ala Asn Ala Thr Ser Leu Pro
325 330 335
Val Gln Gly Gly Gly Gly Ser Gly Leu Asn Asp Ile Phe Glu Ala Gln
340 345 350
Lys Ile Glu Trp His Glu
355
<210> 38
<211> 449
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 38
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Tyr Arg Gln Ala Pro Gly Lys Gly Arg Glu Leu Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 39
<211> 449
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 39
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro
<210> 40
<211> 457
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 40
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Tyr Arg Gln Ala Pro Gly Lys Gly Arg Glu Leu Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Leu Ser Gly Arg Ser
115 120 125
Asp Asn His Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455
<210> 41
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 41
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Tyr Arg Gln Ala Pro Gly Lys Gly Arg Glu Leu Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Ser Gly Gly Ser Gly
115 120 125
Leu Ser Gly Arg Ser Asp Asn His Gly Ser Ser Gly Gly Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 42
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 42
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Tyr Arg Gln Ala Pro Gly Lys Gly Arg Glu Leu Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg
115 120 125
Ser Asp Asn His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 43
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 43
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Val Phe Lys Ile Asn
20 25 30
Val Met Ala Trp Tyr Arg Gln Ala Pro Gly Lys Gly Arg Glu Leu Val
35 40 45
Ala Gly Ile Ile Ser Gly Gly Ser Thr Ser Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Phe Ile Thr Thr Glu Ser Asp Tyr Asp Leu Gly Arg Arg Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Ser Gly Gly
115 120 125
Ser Gly Leu Ser Gly Arg Ser Asp Asn His Gly Ser Ser Gly Gly Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 44
<211> 18
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 44
Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn His Gly Ser Ser
1 5 10 15
Gly Gly
<210> 45
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 45
Leu Ser Gly Arg Ser Asp Asn His Gly
1 5
<210> 46
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 46
Gly Ser Gly Gly
1
<210> 47
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 47
Ser Gly Gly Gly
1
<210> 48
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 48
Gly Ser Ser Gly
1
<210> 49
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 49
Gly Gly Gly Gly Ser
1 5
<210> 50
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 50
Gly Gly Gly Gly Gly Ser
1 5
<210> 51
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 51
Ser Gly Gly Gly Gly
1 5
<210> 52
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 52
Gly Ser Ser Gly Gly
1 5
<210> 53
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 53
Ser Gly Gly Ser Gly
1 5
<210> 54
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 54
Ser Gly Gly Gly Gly Gly
1 5
<210> 55
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 55
Gly Gly Gly Gly Gly Gly Ser
1 5
<210> 56
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 56
Ser Gly Gly Gly Gly Gly Gly
1 5
<210> 57
<211> 641
<212> PRT
<213> Epstein-Barr virus (EBV)
<400> 57
Met Ser Asp Glu Gly Pro Gly Thr Gly Pro Gly Asn Gly Leu Gly Glu
1 5 10 15
Lys Gly Asp Thr Ser Gly Pro Glu Gly Ser Gly Gly Ser Gly Pro Gln
20 25 30
Arg Arg Gly Gly Asp Asn His Gly Arg Gly Arg Gly Arg Gly Arg Gly
35 40 45
Arg Gly Gly Gly Arg Pro Gly Ala Pro Gly Gly Ser Gly Ser Gly Pro
50 55 60
Arg His Arg Asp Gly Val Arg Arg Pro Gln Lys Arg Pro Ser Cys Ile
65 70 75 80
Gly Cys Lys Gly Thr His Gly Gly Thr Gly Ala Gly Ala Gly Ala Gly
85 90 95
Gly Ala Gly Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly Ala Gly
100 105 110
Gly Gly Ala Gly Gly Ala Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly
115 120 125
Gly Ala Gly Ala Gly Gly Gly Ala Gly Gly Ala Gly Gly Ala Gly Ala
130 135 140
Gly Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly Gly Ala Gly Ala Gly
145 150 155 160
Gly Gly Ala Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly
165 170 175
Ala Gly Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly Gly
180 185 190
Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly Ala Gly Gly Ala Gly
195 200 205
Gly Ala Gly Gly Ala Gly Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala
210 215 220
Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly Ala Gly Ala Gly Gly Ala
225 230 235 240
Gly Ala Gly Gly Ala Gly Ala Gly Gly Ala Gly Gly Ala Gly Ala Gly
245 250 255
Gly Ala Gly Gly Ala Gly Ala Gly Gly Ala Gly Gly Ala Gly Ala Gly
260 265 270
Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly Gly Ala Gly
275 280 285
Ala Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly Ala Gly Gly Ala Gly
290 295 300
Ala Gly Gly Ala Gly Gly Ala Gly Ala Gly Gly Gly Ala Gly Ala Gly
305 310 315 320
Gly Ala Gly Ala Gly Gly Gly Gly Arg Gly Arg Gly Gly Ser Gly Gly
325 330 335
Arg Gly Arg Gly Gly Ser Gly Gly Arg Gly Arg Gly Gly Ser Gly Gly
340 345 350
Arg Arg Gly Arg Gly Arg Glu Arg Ala Arg Gly Gly Ser Arg Glu Arg
355 360 365
Ala Arg Gly Arg Gly Arg Gly Arg Gly Glu Lys Arg Pro Arg Ser Pro
370 375 380
Ser Ser Gln Ser Ser Ser Ser Gly Ser Pro Pro Arg Arg Pro Pro Pro
385 390 395 400
Gly Arg Arg Pro Phe Phe His Pro Val Gly Glu Ala Asp Tyr Phe Glu
405 410 415
Tyr His Gln Glu Gly Gly Pro Asp Gly Glu Pro Asp Val Pro Pro Gly
420 425 430
Ala Ile Glu Gln Gly Pro Ala Asp Asp Pro Gly Glu Gly Pro Ser Thr
435 440 445
Gly Pro Arg Gly Gln Gly Asp Gly Gly Arg Arg Lys Lys Gly Gly Trp
450 455 460
Phe Gly Lys His Arg Gly Gln Gly Gly Ser Asn Pro Lys Phe Glu Asn
465 470 475 480
Ile Ala Glu Gly Leu Arg Ala Leu Leu Ala Arg Ser His Val Glu Arg
485 490 495
Thr Thr Asp Glu Gly Thr Trp Val Ala Gly Val Phe Val Tyr Gly Gly
500 505 510
Ser Lys Thr Ser Leu Tyr Asn Leu Arg Arg Gly Thr Ala Leu Ala Ile
515 520 525
Pro Gln Cys Arg Leu Thr Pro Leu Ser Arg Leu Pro Phe Gly Met Ala
530 535 540
Pro Gly Pro Gly Pro Gln Pro Gly Pro Leu Arg Glu Ser Ile Val Cys
545 550 555 560
Tyr Phe Met Val Phe Leu Gln Thr His Ile Phe Ala Glu Val Leu Lys
565 570 575
Asp Ala Ile Lys Asp Leu Val Met Thr Lys Pro Ala Pro Thr Cys Asn
580 585 590
Ile Arg Val Thr Val Cys Ser Phe Asp Asp Gly Val Asp Leu Pro Pro
595 600 605
Trp Phe Pro Pro Met Val Glu Gly Ala Ala Ala Glu Gly Asp Asp Gly
610 615 620
Asp Asp Gly Asp Glu Gly Gly Asp Gly Asp Glu Gly Glu Glu Gly Gln
625 630 635 640
Glu
<210> 58
<211> 321
<212> PRT
<213> Escherichia coli
<400> 58
Met Lys Asp Asn Thr Val Pro Leu Lys Leu Ile Ala Leu Leu Ala Asn
1 5 10 15
Gly Glu Phe His Ser Gly Glu Gln Leu Gly Glu Thr Leu Gly Met Ser
20 25 30
Arg Ala Ala Ile Asn Lys His Ile Gln Thr Leu Arg Asp Trp Gly Val
35 40 45
Asp Val Phe Thr Val Pro Gly Lys Gly Tyr Ser Leu Pro Glu Pro Ile
50 55 60
Gln Leu Leu Asn Ala Lys Gln Ile Leu Gly Gln Leu Asp Gly Gly Ser
65 70 75 80
Val Ala Val Leu Pro Val Ile Asp Ser Thr Asn Gln Tyr Leu Leu Asp
85 90 95
Arg Ile Gly Glu Leu Lys Ser Gly Asp Ala Cys Ile Ala Glu Tyr Gln
100 105 110
Gln Ala Gly Arg Gly Arg Arg Gly Arg Lys Trp Phe Ser Pro Phe Gly
115 120 125
Ala Asn Leu Tyr Leu Ser Met Phe Trp Arg Leu Glu Gln Gly Pro Ala
130 135 140
Ala Ala Ile Gly Leu Ser Leu Val Ile Gly Ile Val Met Ala Glu Val
145 150 155 160
Leu Arg Lys Leu Gly Ala Asp Lys Val Arg Val Lys Trp Pro Asn Asp
165 170 175
Leu Tyr Leu Gln Asp Arg Lys Leu Ala Gly Ile Leu Val Glu Leu Thr
180 185 190
Gly Lys Thr Gly Asp Ala Ala Gln Ile Val Ile Gly Ala Gly Ile Asn
195 200 205
Met Ala Met Arg Arg Val Glu Glu Ser Val Val Asn Gln Gly Trp Ile
210 215 220
Thr Leu Gln Glu Ala Gly Ile Asn Leu Asp Arg Asn Thr Leu Ala Ala
225 230 235 240
Met Leu Ile Arg Glu Leu Arg Ala Ala Leu Glu Leu Phe Glu Gln Glu
245 250 255
Gly Leu Ala Pro Tyr Leu Ser Arg Trp Glu Lys Leu Asp Asn Phe Ile
260 265 270
Asn Arg Pro Val Lys Leu Ile Ile Gly Asp Lys Glu Ile Phe Gly Ile
275 280 285
Ser Arg Gly Ile Asp Lys Gln Gly Ala Leu Leu Leu Glu Gln Asp Gly
290 295 300
Ile Ile Lys Pro Trp Met Gly Gly Glu Ile Ser Leu Arg Ser Ala Glu
305 310 315 320
Lys
<210> 59
<211> 373
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 59
Gln Asp Gly Asn Glu Glu Met Gly Gly Ile Thr Gln Thr Pro Tyr Lys
1 5 10 15
Val Ser Ile Ser Gly Thr Thr Val Ile Leu Thr Cys Pro Gln Tyr Pro
20 25 30
Gly Ser Glu Ile Leu Trp Gln His Asn Asp Lys Asn Ile Gly Gly Asp
35 40 45
Glu Asp Asp Lys Asn Ile Gly Ser Asp Glu Asp His Leu Ser Leu Lys
50 55 60
Glu Phe Ser Glu Leu Glu Gln Ser Gly Tyr Tyr Val Cys Tyr Pro Arg
65 70 75 80
Gly Ser Lys Pro Glu Asp Ala Asn Phe Tyr Leu Tyr Leu Arg Ala Arg
85 90 95
Val Cys Glu Asn Cys Met Glu Met Asp Asp Ile Glu Gly Arg Met Asp
100 105 110
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
115 120 125
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
130 135 140
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
145 150 155 160
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
165 170 175
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
180 185 190
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
195 200 205
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
210 215 220
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
225 230 235 240
Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
245 250 255
Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
260 265 270
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
275 280 285
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
290 295 300
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
305 310 315 320
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
325 330 335
Leu Ser Leu Ser Pro Gly Gly Ser Gly Gly Ser Gly Glu Asn Leu Tyr
340 345 350
Phe Gln Gly Ser Gly Gly Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys
355 360 365
Ile Glu Trp His Glu
370
<210> 60
<211> 323
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 60
Phe Lys Ile Pro Ile Glu Glu Leu Glu Asp Arg Val Phe Val Asn Cys
1 5 10 15
Asn Thr Ser Ile Thr Trp Val Glu Gly Thr Val Gly Thr Leu Leu Ser
20 25 30
Asp Ile Thr Arg Leu Asp Leu Gly Lys Arg Ile Leu Asp Pro Arg Gly
35 40 45
Ile Tyr Arg Cys Asn Gly Thr Asp Ile Tyr Lys Asp Lys Glu Ser Thr
50 55 60
Val Gln Val His Tyr Arg Met Cys Gln Ser Cys Val Glu Leu Asp Asp
65 70 75 80
Ile Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys
85 90 95
Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu
100 105 110
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
115 120 125
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
130 135 140
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
145 150 155 160
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
165 170 175
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
180 185 190
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
195 200 205
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
210 215 220
Arg Cys Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys
225 230 235 240
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
245 250 255
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
260 265 270
Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
275 280 285
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
290 295 300
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp
305 310 315 320
Asp Asp Lys
<210> 61
<211> 123
<212> PRT
<213> Vicugna pacos
<400> 61
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn His Cys
20 25 30
Trp Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asn Thr Asp Gly Gly Ser Ala Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Trp Ala Pro Leu Val Asp Tyr Tyr Tyr Gly Met Asp Tyr
100 105 110
Trp Gly Lys Gly Thr Leu Val Thr Val Ser Gly
115 120
<210> 62
<211> 121
<212> PRT
<213> Vicugna pacos
<400> 62
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Phe Asp Asn Tyr
20 25 30
Val Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Ser Ala Ile Ser Trp Asn Gly Gly Ser Thr Tyr Tyr Ala Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Pro Arg Val Tyr Tyr Ser Gly Ser Pro Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Gln Val Thr Val Ser Gly
115 120
<210> 63
<211> 118
<212> PRT
<213> Vicugna pacos
<400> 63
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Val Tyr
20 25 30
Gly Met Ser Trp Val Arg His Ser Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Asn Trp Asn Gly Gly Ser Thr Tyr Tyr Ala Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Phe Ala Pro Trp Asp Gly Asp Met Glu Trp Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly
115
<210> 64
<211> 469
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 64
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn His Cys
20 25 30
Trp Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Val Ile Asn Thr Asp Gly Gly Ser Ala Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Trp Ala Pro Leu Val Asp Tyr Tyr Tyr Gly Met Asp Tyr
100 105 110
Trp Gly Lys Gly Thr Leu Val Thr Val Ser Gly Gly Ser Gly Leu Ser
115 120 125
Gly Arg Ser Asp Asn His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr
130 135 140
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
145 150 155 160
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
165 170 175
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
180 185 190
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
195 200 205
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
210 215 220
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
225 230 235 240
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
245 250 255
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
260 265 270
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
275 280 285
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
290 295 300
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
305 310 315 320
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
325 330 335
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
340 345 350
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
355 360 365
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
370 375 380
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
385 390 395 400
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
405 410 415
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
420 425 430
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
435 440 445
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
450 455 460
Leu Ser Leu Ser Pro
465
<210> 65
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 65
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Phe Asp Asn Tyr
20 25 30
Val Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Ser Ala Ile Ser Trp Asn Gly Gly Ser Thr Tyr Tyr Ala Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Ser Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Pro Arg Val Tyr Tyr Ser Gly Ser Pro Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg
115 120 125
Ser Asp Asn His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 66
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 66
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Val Tyr
20 25 30
Gly Met Ser Trp Val Arg His Ser Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Asn Trp Asn Gly Gly Ser Thr Tyr Tyr Ala Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Phe Ala Pro Trp Asp Gly Asp Met Glu Trp Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn
115 120 125
His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 67
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 67
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Leu Ser Gly Arg Ser Asp Asn
100 105 110
His Ile Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 68
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 68
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Leu Ser Gly Arg Ser Asp
100 105 110
Asn His Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 69
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 69
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Leu Ser Gly Arg Ser
100 105 110
Asp Asn His Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 70
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 70
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Leu Ser Gly
100 105 110
Arg Ser Asp Asn His Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 71
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 71
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Leu Ser
100 105 110
Gly Arg Ser Asp Asn His Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 72
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 72
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Leu
100 105 110
Ser Gly Arg Ser Asp Asn His Ala Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 73
<211> 222
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 73
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Leu Ser Gly Arg Ser Asp Asn His Pro Ser Val Phe Ile Phe Pro Pro
115 120 125
Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu
130 135 140
Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn
145 150 155 160
Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser
165 170 175
Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala
180 185 190
Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly
195 200 205
Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 74
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 74
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 75
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 75
Ile Ser Ser Gly Leu Leu Ser Gly Arg Ser Asp Asn His
1 5 10
<210> 76
<211> 18
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 76
Ala Val Gly Leu Leu Ala Pro Pro Gly Gly Leu Ser Gly Arg Ser Asp
1 5 10 15
Asn His
<210> 77
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 77
Gly Ala Gly Val Pro Met Ser Met Arg Gly Gly Ala Gly
1 5 10
<210> 78
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 78
Gly Ala Gly Ile Pro Val Ser Leu Arg Ser Gly Ala Gly
1 5 10
<210> 79
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 79
Gly Pro Leu Gly Ile Ala Gly Gln
1 5
<210> 80
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 80
Gly Gly Pro Leu Gly Met Leu Ser Gln Ser
1 5 10
<210> 81
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 81
Pro Leu Gly Leu Trp Ala
1 5
<210> 82
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 82
Gly Ala Gly Arg Pro Phe Ser Met Ile Met Gly Ala Gly
1 5 10
<210> 83
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 83
Gly Ala Gly Val Pro Leu Ser Leu Thr Met Gly Ala Gly
1 5 10
<210> 84
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 84
Gly Ala Gly Val Pro Leu Ser Leu Tyr Ser Gly Ala Gly
1 5 10
<210> 85
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 85
Ala Ala Asn Leu Arg Asn
1 5
<210> 86
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 86
Ala Gln Ala Tyr Val Lys
1 5
<210> 87
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 87
Ala Ala Asn Tyr Met Arg
1 5
<210> 88
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 88
Ala Ala Ala Leu Thr Arg
1 5
<210> 89
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 89
Ala Gln Asn Leu Met Arg
1 5
<210> 90
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 90
Ala Ala Asn Tyr Thr Lys
1 5
<210> 91
<211> 16
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 91
Gly Ala Gly Pro Gln Gly Leu Ala Gly Gln Arg Gly Ile Val Ala Gly
1 5 10 15
<210> 92
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 92
Pro Arg Phe Lys Ile Ile Gly Gly
1 5
<210> 93
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 93
Pro Arg Phe Arg Ile Ile Gly Gly
1 5
<210> 94
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 94
Gly Ala Gly Ser Gly Arg Ser Ala Gly
1 5
<210> 95
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 95
Ser Gly Arg Ser Ala
1 5
<210> 96
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 96
Gly Ser Gly Arg Ser Ala
1 5
<210> 97
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 97
Ser Gly Lys Ser Ala
1 5
<210> 98
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 98
Ser Gly Arg Ser Ser
1 5
<210> 99
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 99
Ser Gly Arg Arg Ala
1 5
<210> 100
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 100
Ser Gly Arg Asn Ala
1 5
<210> 101
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 101
Ser Gly Arg Lys Ala
1 5
<210> 102
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 102
Gln Arg Gly Arg Ser Ala
1 5
<210> 103
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 103
Gly Ala Gly Ser Leu Leu Lys Ser Arg Met Val Pro Asn Phe Asn Ala
1 5 10 15
Gly
<210> 104
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 104
Thr Gln Gly Ala Ala Ala
1 5
<210> 105
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 105
Gly Ala Ala Ala Ala Ala
1 5
<210> 106
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 106
Gly Ala Gly Ala Ala Gly
1 5
<210> 107
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 107
Ala Ala Ala Ala Ala Gly
1 5
<210> 108
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 108
Leu Cys Gly Ala Ala Ile
1 5
<210> 109
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 109
Phe Ala Gln Ala Leu Gly
1 5
<210> 110
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 110
Leu Leu Gln Ala Asn Pro
1 5
<210> 111
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 111
Leu Ala Ala Ala Asn Pro
1 5
<210> 112
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 112
Leu Tyr Gly Ala Gln Phe
1 5
<210> 113
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 113
Leu Ser Gln Ala Gln Gly
1 5
<210> 114
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 114
Ala Ser Ala Ala Ser Gly
1 5
<210> 115
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 115
Phe Leu Gly Ala Ser Leu
1 5
<210> 116
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 116
Ala Tyr Gly Ala Thr Gly
1 5
<210> 117
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 117
Leu Ala Gln Ala Thr Gly
1 5
<210> 118
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 118
Gly Ala Gly Ser Gly Val Val Ile Ala Thr Val Ile Val Ile Thr Ala
1 5 10 15
Gly
<210> 119
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 119
Ala Pro Met Ala Glu Gly Gly Gly
1 5
<210> 120
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 120
Glu Ala Gln Gly Asp Lys Ile Ile
1 5
<210> 121
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 121
Leu Ala Phe Ser Asp Ala Gly Pro
1 5
<210> 122
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 122
Tyr Val Ala Asp Ala Pro Lys
1 5
<210> 123
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 123
Arg Arg Arg Arg Arg
1 5
<210> 124
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 124
Arg Arg Arg Arg Arg Arg
1 5
<210> 125
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 125
Gly Gln Ser Ser Arg His Arg Arg Ala Leu
1 5 10
<210> 126
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 126
Ser Ser Arg His Arg Arg Ala Leu Asp
1 5
<210> 127
<211> 14
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 127
Arg Lys Ser Ser Ile Ile Ile Arg Met Arg Asp Val Val Leu
1 5 10
<210> 128
<211> 15
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 128
Ser Ser Ser Phe Asp Lys Gly Lys Tyr Lys Lys Gly Asp Asp Ala
1 5 10 15
<210> 129
<211> 15
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 129
Ser Ser Ser Phe Asp Lys Gly Lys Tyr Lys Arg Gly Asp Asp Ala
1 5 10 15
<210> 130
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 130
Ile Glu Gly Arg
1
<210> 131
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 131
Ile Asp Gly Arg
1
<210> 132
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 132
Gly Gly Ser Ile Asp Gly Arg
1 5
<210> 133
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 133
Gly Pro Gln Gly Ile Ala Gly Gln
1 5
<210> 134
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 134
Gly Pro Gln Gly Leu Leu Gly Ala
1 5
<210> 135
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 135
Gly Ile Ala Gly Gln
1 5
<210> 136
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 136
Gly Pro Leu Gly Ile Ala Gly
1 5
<210> 137
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 137
Gly Pro Glu Gly Leu Arg Val Gly
1 5
<210> 138
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 138
Tyr Gly Ala Gly Leu Gly Val Val
1 5
<210> 139
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 139
Ala Gly Leu Gly Val Val Glu Arg
1 5
<210> 140
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 140
Ala Gly Leu Gly Ile Ser Ser Thr
1 5
<210> 141
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 141
Glu Pro Gln Ala Leu Ala Met Ser
1 5
<210> 142
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 142
Gln Ala Leu Ala Met Ser Ala Ile
1 5
<210> 143
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 143
Ala Ala Tyr His Leu Val Ser Gln
1 5
<210> 144
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 144
Met Asp Ala Phe Leu Glu Ser Ser
1 5
<210> 145
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 145
Glu Ser Leu Pro Val Val Ala Val
1 5
<210> 146
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 146
Ser Ala Pro Ala Val Glu Ser Glu
1 5
<210> 147
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 147
Asp Val Ala Gln Phe Val Leu Thr
1 5
<210> 148
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 148
Val Ala Gln Phe Val Leu Thr Glu
1 5
<210> 149
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 149
Ala Gln Phe Val Leu Thr Glu Gly
1 5
<210> 150
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 150
Pro Val Gln Pro Ile Gly Pro Gln
1 5
<210> 151
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 151
Leu Val Pro Arg Gly Ser
1 5
<210> 152
<211> 214
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 152
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Phe Asp Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Ala
210
<210> 153
<211> 107
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 153
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Ala
100 105
<210> 154
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 154
Gln Leu Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Ser Phe Ser Ile Ser
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Thr Ile Thr Ser Gly Gly Glu Ala Tyr Tyr Ala Asn Ser Met Lys
50 55 60
Asp Arg Phe Thr Ile Ser Arg Asp Asn Ala Glu Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Arg Leu Lys Pro Glu Asp Ser Ala Val Tyr Tyr Cys Asn
85 90 95
Thr Lys Phe Pro Trp Ser Thr Asp Trp Asp Ser Arg Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn
115 120 125
His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 155
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 155
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Ser Phe Ser Ile Ser
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Thr Ile Thr Ser Gly Gly Glu Ala Tyr Tyr Ala Asn Ser Met Lys
50 55 60
Asp Arg Phe Thr Ile Ser Arg Asp Asn Ala Glu Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Arg Leu Lys Pro Glu Asp Ser Ala Val Tyr Tyr Cys Asn
85 90 95
Thr Lys Phe Pro Trp Ser Thr Asp Trp Asp Ser Arg Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn
115 120 125
His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 156
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 156
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Ser Phe Ser Ile Ser
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Thr Ile Thr Ser Gly Gly Glu Ala Tyr Tyr Ala Asn Ser Met Lys
50 55 60
Asp Arg Phe Thr Ile Ser Arg Asp Asn Ala Glu Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Arg Leu Lys Pro Glu Asp Ser Ala Val Tyr Tyr Cys Asn
85 90 95
Thr Lys Phe Pro Trp Ser Thr Asp Trp Asn Ala Arg Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn
115 120 125
His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 157
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 157
Gln Leu Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Ser Phe Ser Ile Ser
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Thr Ile Ala Ser Gly Gly Glu Ala Tyr Tyr Ala Asn Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Glu Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Arg Leu Lys Pro Glu Asp Ser Ala Val Tyr Tyr Cys Asn
85 90 95
Thr Lys Phe Pro Trp Ser Thr Asp Trp Asn Ala Arg Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn
115 120 125
His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 158
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 158
Gln Leu Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Ser Phe Ser Ile Ser
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Thr Ile Ala Ser Gly Gly Glu Ala Tyr Tyr Ala Asn Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Glu Tyr Ala Glu Asn Thr Val Phe Leu
65 70 75 80
Gln Met Asn Arg Leu Lys Pro Glu Asp Ser Ala Val Tyr Tyr Cys Asn
85 90 95
Thr Lys Phe Pro Trp Ser Thr Asp Trp Asn Ala Arg Gly Gln Gly Thr
100 105 110
Gln Val Thr Val Ser Gly Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn
115 120 125
His Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
130 135 140
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
145 150 155 160
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
165 170 175
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
180 185 190
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
195 200 205
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
210 215 220
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
225 230 235 240
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
275 280 285
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 159
<211> 116
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 159
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Tyr Phe Asp Phe Asp Ser Tyr
20 25 30
Glu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr His Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Thr Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Val Asn Met Asp Arg Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 160
<211> 124
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 160
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Pro Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Tyr Arg Gly Tyr
20 25 30
Ser Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ala Ala Ile Val Trp Ser Gly Gly Asn Thr Tyr Tyr Glu Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Thr Ser Leu Lys Pro Glu Asp Ser Ala Thr Tyr Tyr Cys
85 90 95
Ala Ala Lys Ile Arg Pro Tyr Ile Phe Lys Ile Ala Gly Gln Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 161
<211> 354
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 161
Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn His Gly Ser Ser Gly Gly
1 5 10 15
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
20 25 30
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
35 40 45
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
50 55 60
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
65 70 75 80
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
85 90 95
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
100 105 110
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
115 120 125
Pro Cys Pro Ala Pro Glu Leu Arg Gly Gly Pro Lys Val Phe Leu Phe
130 135 140
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
145 150 155 160
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
165 170 175
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
180 185 190
Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr
195 200 205
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
210 215 220
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
225 230 235 240
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
245 250 255
Cys Glu Met Thr Lys Asn Gln Val Ser Leu Ser Cys Ala Val Lys Gly
260 265 270
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
275 280 285
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
290 295 300
Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
305 310 315 320
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
325 330 335
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys Asp Asp Asp
340 345 350
Asp Lys
<210> 162
<211> 629
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 162
Gln Val Gln Leu Val Gln Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Tyr Phe Asp Phe Asp Ser Tyr
20 25 30
Glu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Ser Ile Tyr His Ser Gly Ser Thr Tyr Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Thr Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Val Asn Met Asp Arg Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
145 150 155 160
Gly Pro Val Gln Ala Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
165 170 175
Gly Arg Thr Tyr Arg Gly Tyr Ser Met Gly Trp Phe Arg Gln Ala Pro
180 185 190
Gly Lys Glu Arg Glu Phe Val Ala Ala Ile Val Trp Ser Gly Gly Asn
195 200 205
Thr Tyr Tyr Glu Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
210 215 220
Asn Ala Lys Asn Thr Met Tyr Leu Gln Met Thr Ser Leu Lys Pro Glu
225 230 235 240
Asp Ser Ala Thr Tyr Tyr Cys Ala Ala Lys Ile Arg Pro Tyr Ile Phe
245 250 255
Lys Ile Ala Gly Gln Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr
260 265 270
Val Ser Ser Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn His Gly Ser
275 280 285
Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
290 295 300
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
305 310 315 320
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
325 330 335
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
340 345 350
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
355 360 365
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
370 375 380
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
385 390 395 400
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Arg Gly Gly Pro Lys Val
405 410 415
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
420 425 430
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
435 440 445
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
450 455 460
Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser
465 470 475 480
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
485 490 495
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
500 505 510
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
515 520 525
Pro Ser Arg Cys Glu Met Thr Lys Asn Gln Val Ser Leu Ser Cys Ala
530 535 540
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
545 550 555 560
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
565 570 575
Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
580 585 590
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
595 600 605
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp Tyr Lys
610 615 620
Asp Asp Asp Asp Lys
625
<210> 163
<211> 118
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 163
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Ser Ile Asn
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Leu Ile Ser Ser Ile Gly Asp Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Lys
85 90 95
Arg Phe Arg Thr Ala Ala Gln Gly Thr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 164
<211> 631
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 164
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Ser Ile Asn
20 25 30
Thr Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Leu Val
35 40 45
Ala Leu Ile Ser Ser Ile Gly Asp Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Lys
85 90 95
Arg Phe Arg Thr Ala Ala Gln Gly Thr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
145 150 155 160
Gly Gly Gly Pro Val Gln Ala Gly Gly Ser Leu Arg Leu Ser Cys Ala
165 170 175
Ala Ser Gly Arg Thr Tyr Arg Gly Tyr Ser Met Gly Trp Phe Arg Gln
180 185 190
Ala Pro Gly Lys Glu Arg Glu Phe Val Ala Ala Ile Val Trp Ser Gly
195 200 205
Gly Asn Thr Tyr Tyr Glu Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
210 215 220
Arg Asp Asn Ala Lys Asn Thr Met Tyr Leu Gln Met Thr Ser Leu Lys
225 230 235 240
Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Ala Ala Lys Ile Arg Pro Tyr
245 250 255
Ile Phe Lys Ile Ala Gly Gln Tyr Asp Tyr Trp Gly Gln Gly Thr Leu
260 265 270
Val Thr Val Ser Ser Gly Ser Gly Leu Ser Gly Arg Ser Asp Asn His
275 280 285
Gly Ser Ser Gly Gly Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
290 295 300
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
305 310 315 320
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
325 330 335
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
340 345 350
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
355 360 365
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
370 375 380
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
385 390 395 400
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Arg Gly Gly Pro
405 410 415
Lys Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
420 425 430
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
435 440 445
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
450 455 460
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val
465 470 475 480
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
485 490 495
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
500 505 510
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
515 520 525
Leu Pro Pro Ser Arg Cys Glu Met Thr Lys Asn Gln Val Ser Leu Ser
530 535 540
Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
545 550 555 560
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
565 570 575
Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys
580 585 590
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
595 600 605
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Asp
610 615 620
Tyr Lys Asp Asp Asp Asp Lys
625 630
<210> 165
<211> 468
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 165
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Ala Gly Ile Pro Val Ser Leu Arg Ser Gly Ala Gly Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro
465
<210> 166
<211> 230
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 166
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro
225 230
<210> 167
<211> 455
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 167
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro
450 455
<210> 168
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 168
Thr Ser Gly Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 169
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 169
Thr Ser Gln Ser Gly Arg Ser Ala Asn Gln Arg Gly
1 5 10
<210> 170
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 170
Thr Ser Pro Ser Gly Arg Ser Ala Tyr Pro Arg Gly
1 5 10
<210> 171
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 171
Thr Ser Gly Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 172
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 172
Thr Ser Gln Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 173
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 173
Thr Ser Ala Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 174
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 174
Thr Ser Tyr Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 175
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 175
Thr Ser Tyr Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 176
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 176
Thr Ser Ser Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 177
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 177
Thr Ser Thr Thr Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 178
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 178
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 179
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 179
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Gly Ser Gly Arg Ser Ala Asn Ala Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 180
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 180
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Gln Ser Gly Arg Ser Ala Asn Gln Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 181
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 181
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Pro Ser Gly Arg Ser Ala Tyr Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 182
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 182
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Gly Ser Gly Arg Ser Ala Thr Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 183
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 183
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Gln Ser Gly Arg Ser Ala Thr Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 184
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 184
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Ala Ser Gly Arg Ser Ala Thr Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 185
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 185
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Tyr Ser Gly Arg Ser Ala Val Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 186
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 186
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Tyr Ser Gly Arg Ser Ala Asn Phe Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 187
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 187
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Ser Ser Gly Arg Ser Ala Thr Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 188
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 188
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Thr Thr Gly Arg Ser Ala Ser Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 189
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 189
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr
115 120 125
Ser Thr Ser Gly Arg Ser Ala Asn Pro Arg Gly Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
245 250 255
Arg Arg Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
340 345 350
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
370 375 380
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro
465
<210> 190
<211> 127
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 190
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Asp Ile Gly Trp Phe Arg Gln Ala Pro Gly Lys Gly Arg Glu Gly Val
35 40 45
Ser Gly Ile Ser Ser Ser Asp Gly Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Glu Pro Pro Asp Ser Ser Trp Tyr Leu Asp Gly Ser Pro Glu
100 105 110
Phe Phe Lys Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 191
<211> 455
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 191
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Asp Ile Gly Trp Phe Arg Gln Ala Pro Gly Lys Gly Arg Glu Gly Val
35 40 45
Ser Gly Ile Ser Ser Ser Asp Gly Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Glu Pro Pro Asp Ser Ser Trp Tyr Leu Asp Gly Ser Pro Glu
100 105 110
Phe Phe Lys Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro
450 455
<210> 192
<211> 455
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 192
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Asp Ile Gly Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Ser Ser Asp Gly Asn Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Glu Pro Pro Asp Ser Ser Trp Tyr Leu Asp Gly Ser Pro Glu
100 105 110
Phe Phe Lys Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro
450 455
<210> 193
<211> 16
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 193
Gly Gly Gly Gly Ser Pro Leu Gly Leu Ala Gly Gly Gly Gly Gly Ser
1 5 10 15
<210> 194
<211> 18
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 194
Gly Gly Gly Gly Ser Gly Pro Leu Gly Ile Ala Gly Gln Gly Gly Gly
1 5 10 15
Gly Ser
<210> 195
<211> 23
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 195
Gly Gly Gly Gly Ser Gly Ala Gly Val Pro Leu Ser Leu Tyr Ser Gly
1 5 10 15
Ala Gly Gly Gly Gly Gly Ser
20
<210> 196
<211> 471
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 196
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Gly Gly Gly Ser Pro Leu Gly Leu Ala Gly Gly Gly Gly Gly Ser
130 135 140
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
145 150 155 160
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
165 170 175
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
180 185 190
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
195 200 205
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
210 215 220
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
225 230 235 240
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Pro
465 470
<210> 197
<211> 473
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 197
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Gly Gly Gly Ser Gly Pro Leu Gly Ile Ala Gly Gln Gly Gly Gly
130 135 140
Gly Ser Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
145 150 155 160
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
165 170 175
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
180 185 190
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
195 200 205
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
210 215 220
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
225 230 235 240
Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
245 250 255
Cys Pro Ala Pro Glu Leu Arg Arg Gly Pro Ser Val Phe Leu Phe Pro
260 265 270
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
275 280 285
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
290 295 300
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
305 310 315 320
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
325 330 335
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
340 345 350
Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys
355 360 365
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
370 375 380
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
385 390 395 400
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
405 410 415
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
420 425 430
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
435 440 445
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
450 455 460
Thr Gln Lys Ser Leu Ser Leu Ser Pro
465 470
<210> 198
<211> 478
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 198
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Gly Gly Gly Ser Gly Ala Gly Val Pro Leu Ser Leu Tyr Ser Gly
130 135 140
Ala Gly Gly Gly Gly Gly Ser Ser Thr Lys Gly Pro Ser Val Phe Pro
145 150 155 160
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
165 170 175
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
180 185 190
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
195 200 205
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
210 215 220
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
225 230 235 240
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
245 250 255
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
260 265 270
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
275 280 285
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
290 295 300
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
305 310 315 320
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
325 330 335
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
340 345 350
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
355 360 365
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
370 375 380
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
385 390 395 400
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
405 410 415
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
420 425 430
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
435 440 445
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
450 455 460
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
465 470 475
<210> 199
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 199
Asp Tyr Lys Asp Asp Asp Asp Lys
1 5
<210> 200
<211> 1267
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 200
Met Trp Trp Arg Leu Trp Trp Leu Leu Leu Leu Leu Leu Leu Leu Trp
1 5 10 15
Pro Met Val Trp Ala Glu Ala Gly Leu Pro Arg Ala Gly Gly Gly Ser
20 25 30
Gln Pro Pro Phe Arg Thr Phe Ser Ala Ser Asp Trp Gly Leu Thr His
35 40 45
Leu Val Val His Glu Gln Thr Gly Glu Val Tyr Val Gly Ala Val Asn
50 55 60
Arg Ile Tyr Lys Leu Ser Gly Asn Leu Thr Leu Leu Arg Ala His Val
65 70 75 80
Thr Gly Pro Val Glu Asp Asn Glu Lys Cys Tyr Pro Pro Pro Ser Val
85 90 95
Gln Ser Cys Pro His Gly Leu Gly Ser Thr Asp Asn Val Asn Lys Leu
100 105 110
Leu Leu Leu Asp Tyr Ala Ala Asn Arg Leu Leu Ala Cys Gly Ser Ala
115 120 125
Ser Gln Gly Ile Cys Gln Phe Leu Arg Leu Asp Asp Leu Phe Lys Leu
130 135 140
Gly Glu Pro His His Arg Lys Glu His Tyr Leu Ser Ser Val Gln Glu
145 150 155 160
Ala Gly Ser Met Ala Gly Val Leu Ile Ala Gly Pro Pro Gly Gln Gly
165 170 175
Gln Ala Lys Leu Phe Val Gly Thr Pro Ile Asp Gly Lys Ser Glu Tyr
180 185 190
Phe Pro Thr Leu Ser Ser Arg Arg Leu Met Ala Asn Glu Glu Asp Ala
195 200 205
Asp Met Phe Gly Phe Val Tyr Gln Asp Glu Phe Val Ser Ser Gln Leu
210 215 220
Lys Ile Pro Ser Asp Thr Leu Ser Lys Phe Pro Ala Phe Asp Ile Tyr
225 230 235 240
Tyr Val Tyr Ser Phe Arg Ser Glu Gln Phe Val Tyr Tyr Leu Thr Leu
245 250 255
Gln Leu Asp Thr Gln Leu Thr Ser Pro Asp Ala Ala Gly Glu His Phe
260 265 270
Phe Thr Ser Lys Ile Val Arg Leu Cys Val Asp Asp Pro Lys Phe Tyr
275 280 285
Ser Tyr Val Glu Phe Pro Ile Gly Cys Glu Gln Ala Gly Val Glu Tyr
290 295 300
Arg Leu Val Gln Asp Ala Tyr Leu Ser Arg Pro Gly Arg Ala Leu Ala
305 310 315 320
His Gln Leu Gly Leu Ala Glu Asp Glu Asp Val Leu Phe Thr Val Phe
325 330 335
Ala Gln Gly Gln Lys Asn Arg Val Lys Pro Pro Lys Glu Ser Ala Leu
340 345 350
Cys Leu Phe Thr Leu Arg Ala Ile Lys Glu Lys Ile Lys Glu Arg Ile
355 360 365
Gln Ser Cys Tyr Arg Gly Glu Gly Lys Leu Ser Leu Pro Trp Leu Leu
370 375 380
Asn Lys Glu Leu Gly Cys Ile Asn Ser Pro Leu Gln Ile Asp Asp Asp
385 390 395 400
Phe Cys Gly Gln Asp Phe Asn Gln Pro Leu Gly Gly Thr Val Thr Ile
405 410 415
Glu Gly Thr Pro Leu Phe Val Asp Lys Asp Asp Gly Leu Thr Ala Val
420 425 430
Ala Ala Tyr Asp Tyr Arg Gly Arg Thr Val Val Phe Ala Gly Thr Arg
435 440 445
Ser Gly Arg Ile Arg Lys Ile Leu Val Asp Leu Ser Asn Pro Gly Gly
450 455 460
Arg Pro Ala Leu Ala Tyr Glu Ser Val Val Ala Gln Glu Gly Ser Pro
465 470 475 480
Ile Leu Arg Asp Leu Val Leu Ser Pro Asn His Gln Tyr Leu Tyr Ala
485 490 495
Met Thr Glu Lys Gln Val Thr Arg Val Pro Val Glu Ser Cys Val Gln
500 505 510
Tyr Thr Ser Cys Glu Leu Cys Leu Gly Ser Arg Asp Pro His Cys Gly
515 520 525
Trp Cys Val Leu His Ser Ile Cys Ser Arg Arg Asp Ala Cys Glu Arg
530 535 540
Ala Asp Glu Pro Gln Arg Phe Ala Ala Asp Leu Leu Gln Cys Val Gln
545 550 555 560
Leu Thr Val Gln Pro Arg Asn Val Ser Val Thr Met Ser Gln Val Pro
565 570 575
Leu Val Leu Gln Ala Trp Asn Val Pro Asp Leu Ser Ala Gly Val Asn
580 585 590
Cys Ser Phe Glu Asp Phe Thr Glu Ser Glu Ser Val Leu Glu Asp Gly
595 600 605
Arg Ile His Cys Arg Ser Pro Ser Ala Arg Glu Val Ala Pro Ile Thr
610 615 620
Arg Gly Gln Gly Asp Gln Arg Val Val Lys Leu Tyr Leu Lys Ser Lys
625 630 635 640
Glu Thr Gly Lys Lys Phe Ala Ser Val Asp Phe Val Phe Tyr Asn Cys
645 650 655
Ser Val His Gln Ser Cys Leu Ser Cys Val Asn Gly Ser Phe Pro Cys
660 665 670
His Trp Cys Lys Tyr Arg His Val Cys Thr His Asn Val Ala Asp Cys
675 680 685
Ala Phe Leu Glu Gly Arg Val Asn Val Ser Glu Asp Cys Pro Gln Ile
690 695 700
Leu Pro Ser Thr Gln Ile Tyr Val Pro Val Gly Val Val Lys Pro Ile
705 710 715 720
Thr Leu Ala Ala Arg Asn Leu Pro Gln Pro Gln Ser Gly Gln Arg Gly
725 730 735
Tyr Glu Cys Leu Phe His Ile Pro Gly Ser Pro Ala Arg Val Thr Ala
740 745 750
Leu Arg Phe Asn Ser Ser Ser Leu Gln Cys Gln Asn Ser Ser Tyr Ser
755 760 765
Tyr Glu Gly Asn Asp Val Ser Asp Leu Pro Val Asn Leu Ser Val Val
770 775 780
Trp Asn Gly Asn Phe Val Ile Asp Asn Pro Gln Asn Ile Gln Ala His
785 790 795 800
Leu Tyr Lys Cys Pro Ala Leu Arg Glu Ser Cys Gly Leu Cys Leu Lys
805 810 815
Ala Asp Pro Arg Phe Glu Cys Gly Trp Cys Val Ala Glu Arg Arg Cys
820 825 830
Ser Leu Arg His His Cys Ala Ala Asp Thr Pro Ala Ser Trp Met His
835 840 845
Ala Arg His Gly Ser Ser Arg Cys Thr Asp Pro Lys Ile Leu Lys Leu
850 855 860
Ser Pro Glu Thr Gly Pro Arg Gln Gly Gly Thr Arg Leu Thr Ile Thr
865 870 875 880
Gly Glu Asn Leu Gly Leu Arg Phe Glu Asp Val Arg Leu Gly Val Arg
885 890 895
Val Gly Lys Val Leu Cys Ser Pro Val Glu Ser Glu Tyr Ile Ser Ala
900 905 910
Glu Gln Ile Val Cys Glu Ile Gly Asp Ala Ser Ser Val Arg Ala His
915 920 925
Asp Ala Leu Val Glu Val Cys Val Arg Asp Cys Ser Pro His Tyr Arg
930 935 940
Ala Leu Ser Pro Lys Arg Phe Thr Phe Val Thr Pro Thr Phe Tyr Arg
945 950 955 960
Val Ser Pro Ser Arg Gly Pro Leu Ser Gly Gly Thr Trp Ile Gly Ile
965 970 975
Glu Gly Ser His Leu Asn Ala Gly Ser Asp Val Ala Val Ser Val Gly
980 985 990
Gly Arg Pro Cys Ser Phe Ser Trp Arg Asn Ser Arg Glu Ile Arg Cys
995 1000 1005
Leu Thr Pro Pro Gly Gln Ser Pro Gly Ser Ala Pro Ile Ile Ile
1010 1015 1020
Asn Ile Asn Arg Ala Gln Leu Thr Asn Pro Glu Val Lys Tyr Asn
1025 1030 1035
Tyr Thr Glu Asp Pro Thr Ile Leu Arg Ile Asp Pro Glu Trp Ser
1040 1045 1050
Ile Asn Ser Gly Gly Thr Leu Leu Thr Val Thr Gly Thr Asn Leu
1055 1060 1065
Ala Thr Val Arg Glu Pro Arg Ile Arg Ala Lys Tyr Gly Gly Ile
1070 1075 1080
Glu Arg Glu Asn Gly Cys Leu Val Tyr Asn Asp Thr Thr Met Val
1085 1090 1095
Cys Arg Ala Pro Ser Val Ala Asn Pro Val Arg Ser Pro Pro Glu
1100 1105 1110
Leu Gly Glu Arg Pro Asp Glu Leu Gly Phe Val Met Asp Asn Val
1115 1120 1125
Arg Ser Leu Leu Val Leu Asn Ser Thr Ser Phe Leu Tyr Tyr Pro
1130 1135 1140
Asp Pro Val Leu Glu Pro Leu Ser Pro Thr Gly Leu Leu Glu Leu
1145 1150 1155
Lys Pro Ser Ser Pro Leu Ile Leu Lys Gly Arg Asn Leu Leu Pro
1160 1165 1170
Pro Ala Pro Gly Asn Ser Arg Leu Asn Tyr Thr Val Leu Ile Gly
1175 1180 1185
Ser Thr Pro Cys Thr Leu Thr Val Ser Glu Thr Gln Leu Leu Cys
1190 1195 1200
Glu Ala Pro Asn Leu Thr Gly Gln His Lys Val Thr Val Arg Ala
1205 1210 1215
Gly Gly Phe Glu Phe Ser Pro Gly Thr Leu Gln Val Tyr Ser Asp
1220 1225 1230
Ser Leu Leu Thr Leu Pro Asp Tyr Lys Asp Asp Asp Asp Lys Gly
1235 1240 1245
Gly Gly Gly Ser Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile
1250 1255 1260
Glu Trp His Glu
1265
<210> 201
<211> 347
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 201
Met Val Leu Ala Ser Ser Thr Thr Ser Ile His Thr Met Leu Leu Leu
1 5 10 15
Leu Leu Met Leu Ala Gln Pro Ala Met Ala Met Lys Asp Asn Thr Val
20 25 30
Pro Leu Lys Leu Ile Ala Leu Leu Ala Asn Gly Glu Phe His Ser Gly
35 40 45
Glu Gln Leu Gly Glu Thr Leu Gly Met Ser Arg Ala Ala Ile Asn Lys
50 55 60
His Ile Gln Thr Leu Arg Asp Trp Gly Val Asp Val Phe Thr Val Pro
65 70 75 80
Gly Lys Gly Tyr Ser Leu Pro Glu Pro Ile Gln Leu Leu Asn Ala Lys
85 90 95
Gln Ile Leu Gly Gln Leu Asp Gly Gly Ser Val Ala Val Leu Pro Val
100 105 110
Ile Asp Ser Thr Asn Gln Tyr Leu Leu Asp Arg Ile Gly Glu Leu Lys
115 120 125
Ser Gly Asp Ala Cys Ile Ala Glu Tyr Gln Gln Ala Gly Arg Gly Arg
130 135 140
Arg Gly Arg Lys Trp Phe Ser Pro Phe Gly Ala Asn Leu Tyr Leu Ser
145 150 155 160
Met Phe Trp Arg Leu Glu Gln Gly Pro Ala Ala Ala Ile Gly Leu Ser
165 170 175
Leu Val Ile Gly Ile Val Met Ala Glu Val Leu Arg Lys Leu Gly Ala
180 185 190
Asp Lys Val Arg Val Lys Trp Pro Asn Asp Leu Tyr Leu Gln Asp Arg
195 200 205
Lys Leu Ala Gly Ile Leu Val Glu Leu Thr Gly Lys Thr Gly Asp Ala
210 215 220
Ala Gln Ile Val Ile Gly Ala Gly Ile Asn Met Ala Met Arg Arg Val
225 230 235 240
Glu Glu Ser Val Val Asn Gln Gly Trp Ile Thr Leu Gln Glu Ala Gly
245 250 255
Ile Asn Leu Asp Arg Asn Thr Leu Ala Ala Met Leu Ile Arg Glu Leu
260 265 270
Arg Ala Ala Leu Glu Leu Phe Glu Gln Glu Gly Leu Ala Pro Tyr Leu
275 280 285
Ser Arg Trp Glu Lys Leu Asp Asn Phe Ile Asn Arg Pro Val Lys Leu
290 295 300
Ile Ile Gly Asp Lys Glu Ile Phe Gly Ile Ser Arg Gly Ile Asp Lys
305 310 315 320
Gln Gly Ala Leu Leu Leu Glu Gln Asp Gly Ile Ile Lys Pro Trp Met
325 330 335
Gly Gly Glu Ile Ser Leu Arg Ser Ala Glu Lys
340 345
<210> 202
<211> 463
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 202
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Pro Leu Gly Ile Ala Gly Gln Ser Thr Lys Gly Pro Ser Val Phe
130 135 140
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
145 150 155 160
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
165 170 175
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
195 200 205
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
210 215 220
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
225 230 235 240
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
245 250 255
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
260 265 270
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
275 280 285
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
290 295 300
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
305 310 315 320
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
325 330 335
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
340 345 350
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
355 360 365
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
370 375 380
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
385 390 395 400
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
405 410 415
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
420 425 430
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
435 440 445
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 203
<211> 465
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 203
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Gly Pro Leu Gly Met Leu Ser Gln Ser Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
245 250 255
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
260 265 270
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
275 280 285
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
290 295 300
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
305 310 315 320
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
325 330 335
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
340 345 350
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
355 360 365
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
370 375 380
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
385 390 395 400
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
405 410 415
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
420 425 430
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
435 440 445
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
450 455 460
Pro
465
<210> 204
<211> 461
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 204
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Pro Leu Gly Leu Trp Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
130 135 140
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
145 150 155 160
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
165 170 175
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
180 185 190
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
195 200 205
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
210 215 220
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
225 230 235 240
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
245 250 255
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
260 265 270
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
275 280 285
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
290 295 300
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
305 310 315 320
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
325 330 335
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
340 345 350
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
355 360 365
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
370 375 380
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
385 390 395 400
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
405 410 415
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
420 425 430
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
435 440 445
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 205
<211> 463
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 205
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Val Pro Leu Ser Leu Thr Met Gly Ser Thr Lys Gly Pro Ser Val Phe
130 135 140
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
145 150 155 160
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
165 170 175
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
180 185 190
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
195 200 205
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
210 215 220
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
225 230 235 240
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
245 250 255
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
260 265 270
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
275 280 285
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
290 295 300
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
305 310 315 320
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
325 330 335
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
340 345 350
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
355 360 365
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
370 375 380
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
385 390 395 400
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
405 410 415
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
420 425 430
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
435 440 445
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 206
<211> 468
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 206
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Ala Gly Val Pro Leu Ser Leu Thr Met Gly Ala Gly Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro
465
<210> 207
<211> 471
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 207
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Ala Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Trp Asn Gly Asn Asn Thr Tyr Tyr Thr Glu Ser Met
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Ser Thr Ala Ile Val Gly Val Pro Pro Thr Tyr Pro Asp
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
115 120 125
Gly Ala Gly Pro Gln Gly Leu Ala Gly Gln Arg Gly Ile Val Ala Gly
130 135 140
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
145 150 155 160
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
165 170 175
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
180 185 190
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
195 200 205
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
210 215 220
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
225 230 235 240
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Pro
465 470
<210> 208
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 208
Thr Ser Ala Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 209
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 209
Thr Ser Glu Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 210
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 210
Thr Ser Phe Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 211
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 211
Thr Ser Gly Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 212
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 212
Thr Ser His Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 213
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 213
Thr Ser Lys Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 214
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 214
Thr Ser Met Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 215
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 215
Thr Ser Asn Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 216
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 216
Thr Ser Pro Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 217
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 217
Thr Ser Gln Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 218
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 218
Thr Ser Trp Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 219
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 219
Thr Ser Tyr Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 220
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 220
Thr Ser Thr Ala Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 221
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 221
Thr Ser Thr Asp Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 222
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 222
Thr Ser Thr Glu Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 223
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 223
Thr Ser Thr Phe Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 224
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 224
Thr Ser Thr Leu Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 225
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 225
Thr Ser Thr Met Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 226
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 226
Thr Ser Thr Pro Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 227
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 227
Thr Ser Thr Gln Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 228
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 228
Thr Ser Thr Val Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 229
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 229
Thr Ser Thr Trp Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 230
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 230
Thr Ser Thr Ser Ala Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 231
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 231
Thr Ser Thr Ser Glu Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 232
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 232
Thr Ser Thr Ser Phe Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 233
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 233
Thr Ser Thr Ser His Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 234
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 234
Thr Ser Thr Ser Ile Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 235
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 235
Thr Ser Thr Ser Lys Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 236
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 236
Thr Ser Thr Ser Leu Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 237
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 237
Thr Ser Thr Ser Met Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 238
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 238
Thr Ser Thr Ser Asn Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 239
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 239
Thr Ser Thr Ser Pro Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 240
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 240
Thr Ser Thr Ser Gln Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 241
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 241
Thr Ser Thr Ser Arg Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 242
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 242
Thr Ser Thr Ser Thr Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 243
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 243
Thr Ser Thr Ser Val Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 244
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 244
Thr Ser Thr Ser Trp Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 245
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 245
Thr Ser Thr Ser Tyr Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 246
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 246
Thr Ser Thr Ser Gly Arg Ala Ala Asn Pro Arg Gly
1 5 10
<210> 247
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 247
Thr Ser Thr Ser Gly Arg Asp Ala Asn Pro Arg Gly
1 5 10
<210> 248
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 248
Thr Ser Thr Ser Gly Arg Glu Ala Asn Pro Arg Gly
1 5 10
<210> 249
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 249
Thr Ser Thr Ser Gly Arg Gly Ala Asn Pro Arg Gly
1 5 10
<210> 250
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 250
Thr Ser Thr Ser Gly Arg His Ala Asn Pro Arg Gly
1 5 10
<210> 251
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 251
Thr Ser Thr Ser Gly Arg Ile Ala Asn Pro Arg Gly
1 5 10
<210> 252
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 252
Thr Ser Thr Ser Gly Arg Lys Ala Asn Pro Arg Gly
1 5 10
<210> 253
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 253
Thr Ser Thr Ser Gly Arg Leu Ala Asn Pro Arg Gly
1 5 10
<210> 254
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 254
Thr Ser Thr Ser Gly Arg Met Ala Asn Pro Arg Gly
1 5 10
<210> 255
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 255
Thr Ser Thr Ser Gly Arg Asn Ala Asn Pro Arg Gly
1 5 10
<210> 256
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 256
Thr Ser Thr Ser Gly Arg Pro Ala Asn Pro Arg Gly
1 5 10
<210> 257
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 257
Thr Ser Thr Ser Gly Arg Gln Ala Asn Pro Arg Gly
1 5 10
<210> 258
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 258
Thr Ser Thr Ser Gly Arg Arg Ala Asn Pro Arg Gly
1 5 10
<210> 259
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 259
Thr Ser Thr Ser Gly Arg Thr Ala Asn Pro Arg Gly
1 5 10
<210> 260
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 260
Thr Ser Thr Ser Gly Arg Val Ala Asn Pro Arg Gly
1 5 10
<210> 261
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 261
Thr Ser Thr Ser Gly Arg Trp Ala Asn Pro Arg Gly
1 5 10
<210> 262
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 262
Thr Ser Thr Ser Gly Arg Tyr Ala Asn Pro Arg Gly
1 5 10
<210> 263
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 263
Thr Ser Thr Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 264
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 264
Thr Ser Thr Ser Gly Arg Ser Phe Asn Pro Arg Gly
1 5 10
<210> 265
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 265
Thr Ser Thr Ser Gly Arg Ser Lys Asn Pro Arg Gly
1 5 10
<210> 266
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 266
Thr Ser Thr Ser Gly Arg Ser Met Asn Pro Arg Gly
1 5 10
<210> 267
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 267
Thr Ser Thr Ser Gly Arg Ser Asn Asn Pro Arg Gly
1 5 10
<210> 268
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 268
Thr Ser Thr Ser Gly Arg Ser Pro Asn Pro Arg Gly
1 5 10
<210> 269
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 269
Thr Ser Thr Ser Gly Arg Ser Gln Asn Pro Arg Gly
1 5 10
<210> 270
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 270
Thr Ser Thr Ser Gly Arg Ser Arg Asn Pro Arg Gly
1 5 10
<210> 271
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 271
Thr Ser Thr Ser Gly Arg Ser Ser Asn Pro Arg Gly
1 5 10
<210> 272
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 272
Thr Ser Thr Ser Gly Arg Ser Trp Asn Pro Arg Gly
1 5 10
<210> 273
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 273
Thr Ser Thr Ser Gly Arg Ser Tyr Asn Pro Arg Gly
1 5 10
<210> 274
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 274
Thr Ser Thr Ser Gly Arg Ser Ala Ala Pro Arg Gly
1 5 10
<210> 275
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 275
Thr Ser Thr Ser Gly Arg Ser Ala Asp Pro Arg Gly
1 5 10
<210> 276
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 276
Thr Ser Thr Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 277
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 277
Thr Ser Thr Ser Gly Arg Ser Ala Phe Pro Arg Gly
1 5 10
<210> 278
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 278
Thr Ser Thr Ser Gly Arg Ser Ala Gly Pro Arg Gly
1 5 10
<210> 279
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 279
Thr Ser Thr Ser Gly Arg Ser Ala Lys Pro Arg Gly
1 5 10
<210> 280
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 280
Thr Ser Thr Ser Gly Arg Ser Ala Leu Pro Arg Gly
1 5 10
<210> 281
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 281
Thr Ser Thr Ser Gly Arg Ser Ala Met Pro Arg Gly
1 5 10
<210> 282
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 282
Thr Ser Thr Ser Gly Arg Ser Ala Pro Pro Arg Gly
1 5 10
<210> 283
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 283
Thr Ser Thr Ser Gly Arg Ser Ala Gln Pro Arg Gly
1 5 10
<210> 284
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 284
Thr Ser Thr Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 285
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 285
Thr Ser Thr Ser Gly Arg Ser Ala Trp Pro Arg Gly
1 5 10
<210> 286
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 286
Thr Ser Thr Ser Gly Arg Ser Ala Tyr Pro Arg Gly
1 5 10
<210> 287
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 287
Thr Ser Thr Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 288
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 288
Thr Ser Thr Ser Gly Arg Ser Ala Asn Asp Arg Gly
1 5 10
<210> 289
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 289
Thr Ser Thr Ser Gly Arg Ser Ala Asn Glu Arg Gly
1 5 10
<210> 290
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 290
Thr Ser Thr Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 291
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 291
Thr Ser Thr Ser Gly Arg Ser Ala Asn Gly Arg Gly
1 5 10
<210> 292
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 292
Thr Ser Thr Ser Gly Arg Ser Ala Asn Ile Arg Gly
1 5 10
<210> 293
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 293
Thr Ser Thr Ser Gly Arg Ser Ala Asn Lys Arg Gly
1 5 10
<210> 294
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 294
Thr Ser Thr Ser Gly Arg Ser Ala Asn Asn Arg Gly
1 5 10
<210> 295
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 295
Thr Ser Thr Ser Gly Arg Ser Ala Asn Gln Arg Gly
1 5 10
<210> 296
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 296
Thr Ser Thr Ser Gly Arg Ser Ala Asn Ser Arg Gly
1 5 10
<210> 297
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 297
Thr Ser Thr Ser Gly Arg Ser Ala Asn Thr Arg Gly
1 5 10
<210> 298
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 298
Thr Ser Thr Ser Gly Arg Ser Ala Asn Trp Arg Gly
1 5 10
<210> 299
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 299
Thr Ser Asp Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 300
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 300
Thr Ser Ile Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 301
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 301
Thr Ser Ser Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 302
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 302
Thr Ser Thr His Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 303
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 303
Thr Ser Thr Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 304
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 304
Thr Ser Thr Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 305
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 305
Thr Ser Thr Tyr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 306
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 306
Thr Ser Thr Ser Asp Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 307
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 307
Thr Ser Thr Ser Ser Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 308
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 308
Thr Ser Thr Ser Gly Arg Phe Ala Asn Pro Arg Gly
1 5 10
<210> 309
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 309
Thr Ser Thr Ser Gly Arg Ser Asp Asn Pro Arg Gly
1 5 10
<210> 310
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 310
Thr Ser Thr Ser Gly Arg Ser His Asn Pro Arg Gly
1 5 10
<210> 311
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 311
Thr Ser Thr Ser Gly Arg Ser Ile Asn Pro Arg Gly
1 5 10
<210> 312
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 312
Thr Ser Thr Ser Gly Arg Ser Leu Asn Pro Arg Gly
1 5 10
<210> 313
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 313
Thr Ser Thr Ser Gly Arg Ser Thr Asn Pro Arg Gly
1 5 10
<210> 314
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 314
Thr Ser Thr Ser Gly Arg Ser Val Asn Pro Arg Gly
1 5 10
<210> 315
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 315
Thr Ser Thr Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 316
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 316
Thr Ser Thr Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 317
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 317
Thr Ser Thr Ser Gly Arg Ser Ala Arg Pro Arg Gly
1 5 10
<210> 318
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 318
Thr Ser Thr Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 319
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 319
Thr Ser Thr Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 320
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 320
Thr Ser Thr Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 321
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 321
Thr Ser Thr Ser Gly Arg Ser Ala Asn Leu Arg Gly
1 5 10
<210> 322
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 322
Thr Ser Thr Ser Gly Arg Ser Ala Asn Met Arg Gly
1 5 10
<210> 323
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 323
Thr Ser Thr Ser Gly Arg Ser Ala Asn Arg Arg Gly
1 5 10
<210> 324
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 324
Thr Ser Thr Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 325
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 325
Thr Ser Thr Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 326
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 326
Thr Ser Gly Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 327
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 327
Thr Ser Gly Ser Gly Arg Ser Ala Tyr Pro Arg Gly
1 5 10
<210> 328
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 328
Thr Ser Gly Ser Gly Arg Ser Ala Asn Gln Arg Gly
1 5 10
<210> 329
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 329
Thr Ser Gly Ser Gly Arg Ser Ala Asn Ile Arg Gly
1 5 10
<210> 330
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 330
Thr Ser Gly Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 331
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 331
Thr Ser Gly Ser Gly Arg Ser Ala Asn Ser Arg Gly
1 5 10
<210> 332
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 332
Thr Ser Gln Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 333
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 333
Thr Ser Gln Ser Gly Arg Ser Ala Tyr Pro Arg Gly
1 5 10
<210> 334
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 334
Thr Ser Gln Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 335
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 335
Thr Ser Gln Ser Gly Arg Ser Ala Asn Ile Arg Gly
1 5 10
<210> 336
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 336
Thr Ser Gln Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 337
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 337
Thr Ser Gln Ser Gly Arg Ser Ala Asn Ser Arg Gly
1 5 10
<210> 338
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 338
Thr Ser Pro Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 339
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 339
Thr Ser Pro Ser Gly Arg Ser Ala Asn Gln Arg Gly
1 5 10
<210> 340
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 340
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 341
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 341
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ile Arg Gly
1 5 10
<210> 342
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 342
Thr Ser Pro Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 343
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 343
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ser Arg Gly
1 5 10
<210> 344
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 344
Thr Ser Ala Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 345
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 345
Thr Ser Ala Ser Gly Arg Ser Ala Tyr Pro Arg Gly
1 5 10
<210> 346
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 346
Thr Ser Ala Ser Gly Arg Ser Ala Asn Gln Arg Gly
1 5 10
<210> 347
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 347
Thr Ser Ala Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 348
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 348
Thr Ser Ala Ser Gly Arg Ser Ala Asn Ile Arg Gly
1 5 10
<210> 349
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 349
Thr Ser Ala Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 350
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 350
Thr Ser Ala Ser Gly Arg Ser Ala Asn Ser Arg Gly
1 5 10
<210> 351
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 351
Thr Ser Tyr Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 352
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 352
Thr Ser Gly Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 353
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 353
Thr Ser Gln Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 354
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 354
Thr Ser Pro Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 355
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 355
Thr Ser Ala Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 356
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 356
Thr Ser His Ser Gly Arg Ser Glu Asn Pro Arg Gly
1 5 10
<210> 357
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 357
Thr Ser Thr Ser Gly Arg Ser Glu Asn Gln Arg Gly
1 5 10
<210> 358
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 358
Thr Ser Thr Ser Gly Arg Ser Glu Asn Ala Arg Gly
1 5 10
<210> 359
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 359
Thr Ser Thr Ser Gly Arg Ser Glu Asn Ile Arg Gly
1 5 10
<210> 360
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 360
Thr Ser Thr Ser Gly Arg Ser Glu Asn Phe Arg Gly
1 5 10
<210> 361
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 361
Thr Ser Thr Ser Gly Arg Ser Glu Asn Ser Arg Gly
1 5 10
<210> 362
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 362
Thr Ser Tyr Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 363
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 363
Thr Ser Gly Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 364
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 364
Thr Ser Gln Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 365
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 365
Thr Ser Pro Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 366
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 366
Thr Ser Ala Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 367
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 367
Thr Ser His Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 368
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 368
Thr Ser Thr Ser Gly Arg Ser Ala Glu Gln Arg Gly
1 5 10
<210> 369
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 369
Thr Ser Thr Ser Gly Arg Ser Ala Glu Ala Arg Gly
1 5 10
<210> 370
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 370
Thr Ser Thr Ser Gly Arg Ser Ala Glu Ile Arg Gly
1 5 10
<210> 371
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 371
Thr Ser Thr Ser Gly Arg Ser Ala Glu Phe Arg Gly
1 5 10
<210> 372
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 372
Thr Ser Thr Ser Gly Arg Ser Ala Glu Ser Arg Gly
1 5 10
<210> 373
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 373
Thr Ser Gly Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 374
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 374
Thr Ser Gly Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 375
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 375
Thr Ser Gly Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 376
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 376
Thr Ser Gly Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 377
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 377
Thr Ser Gly Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 378
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 378
Thr Ser Gly Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 379
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 379
Thr Ser Gly Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 380
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 380
Thr Ser Gly Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 381
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 381
Thr Ser Gln Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 382
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 382
Thr Ser Gln Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 383
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 383
Thr Ser Gln Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 384
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 384
Thr Ser Gln Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 385
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 385
Thr Ser Gln Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 386
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 386
Thr Ser Gln Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 387
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 387
Thr Ser Gln Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 388
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 388
Thr Ser Gln Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 389
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 389
Thr Ser Pro Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 390
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 390
Thr Ser Pro Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 391
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 391
Thr Ser Pro Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 392
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 392
Thr Ser Pro Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 393
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 393
Thr Ser Pro Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 394
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 394
Thr Ser Pro Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 395
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 395
Thr Ser Pro Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 396
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 396
Thr Ser Pro Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 397
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 397
Thr Ser Pro Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 398
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 398
Thr Ser Ala Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 399
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 399
Thr Ser Ala Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 400
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 400
Thr Ser Ala Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 401
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 401
Thr Ser Ala Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 402
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 402
Thr Ser Ala Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 403
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 403
Thr Ser Ala Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 404
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 404
Thr Ser Ala Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 405
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 405
Thr Ser Ala Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 406
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 406
Thr Ser Tyr Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 407
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 407
Thr Ser Tyr Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 408
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 408
Thr Ser Tyr Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 409
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 409
Thr Ser Tyr Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 410
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 410
Thr Ser Tyr Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 411
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 411
Thr Ser Tyr Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 412
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 412
Thr Ser Tyr Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 413
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 413
Thr Ser Tyr Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 414
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 414
Thr Ser Tyr Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 415
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 415
Thr Ser Tyr Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 416
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 416
Thr Ser Ser Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 417
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 417
Thr Ser Ser Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 418
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 418
Thr Ser Ser Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 419
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 419
Thr Ser Ser Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 420
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 420
Thr Ser Ser Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 421
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 421
Thr Ser Ser Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 422
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 422
Thr Ser Ser Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 423
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 423
Thr Ser Ser Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 424
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 424
Thr Ser Ser Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 425
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 425
Thr Ser Ser Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 426
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 426
Thr Ser Ser Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 427
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 427
Thr Ser Ile Thr Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 428
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 428
Thr Ser Ile Lys Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 429
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 429
Thr Ser Ile Ser Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 430
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 430
Thr Ser Ile Ser Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 431
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 431
Thr Ser Ile Ser Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 432
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 432
Thr Ser Ile Ser Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 433
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 433
Thr Ser Ile Ser Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 434
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 434
Thr Ser Ile Ser Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 435
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 435
Thr Ser Ile Ser Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 436
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 436
Thr Ser Ile Ser Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 437
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 437
Thr Ser Ile Ser Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 438
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 438
Thr Ser Ile Ser Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 439
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 439
Thr Ser Thr Thr Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 440
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 440
Thr Ser Thr Thr Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 441
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 441
Thr Ser Thr Thr Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 442
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 442
Thr Ser Thr Thr Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 443
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 443
Thr Ser Thr Thr Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 444
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 444
Thr Ser Thr Thr Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 445
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 445
Thr Ser Thr Thr Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 446
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 446
Thr Ser Thr Thr Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 447
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 447
Thr Ser Thr Thr Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 448
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 448
Thr Ser Thr Lys Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 449
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 449
Thr Ser Thr Lys Gly Arg Ser Ala Ile Pro Arg Gly
1 5 10
<210> 450
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 450
Thr Ser Thr Lys Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 451
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 451
Thr Ser Thr Lys Gly Arg Ser Ala Ser Pro Arg Gly
1 5 10
<210> 452
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 452
Thr Ser Thr Lys Gly Arg Ser Ala His Pro Arg Gly
1 5 10
<210> 453
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 453
Thr Ser Thr Lys Gly Arg Ser Ala Asn Ala Arg Gly
1 5 10
<210> 454
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 454
Thr Ser Thr Lys Gly Arg Ser Ala Asn Phe Arg Gly
1 5 10
<210> 455
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 455
Thr Ser Thr Lys Gly Arg Ser Ala Asn Tyr Arg Gly
1 5 10
<210> 456
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 456
Thr Ser Thr Lys Gly Arg Ser Ala Asn Val Arg Gly
1 5 10
<210> 457
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 457
Thr Ser Thr Lys Gly Arg Ser Ala Asn His Arg Gly
1 5 10
<210> 458
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 458
Thr Ser Thr Ser Gly Arg Ser Ala Val Tyr Arg Gly
1 5 10
<210> 459
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 459
Thr Ser Thr Ser Gly Arg Ser Ala Val Val Arg Gly
1 5 10
<210> 460
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 460
Thr Ser Thr Ser Gly Arg Ser Ala Val His Arg Gly
1 5 10
<210> 461
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 461
Thr Ser Thr Ser Gly Arg Ser Ala Ile Tyr Arg Gly
1 5 10
<210> 462
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 462
Thr Ser Thr Ser Gly Arg Ser Ala Ile Val Arg Gly
1 5 10
<210> 463
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 463
Thr Ser Thr Ser Gly Arg Ser Ala Ile His Arg Gly
1 5 10
<210> 464
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 464
Thr Ser Thr Ser Gly Arg Ser Ala Ser Tyr Arg Gly
1 5 10
<210> 465
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 465
Thr Ser Thr Ser Gly Arg Ser Ala Ser Val Arg Gly
1 5 10
<210> 466
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 466
Thr Ser Thr Ser Gly Arg Ser Ala Ser His Arg Gly
1 5 10
<210> 467
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 467
Thr Ser Thr Ser Gly Arg Ser Ala His Tyr Arg Gly
1 5 10
<210> 468
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 468
Thr Ser Thr Ser Gly Arg Ser Ala His Val Arg Gly
1 5 10
<210> 469
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 469
Thr Ser Thr Ser Gly Arg Ser Ala His His Arg Gly
1 5 10
<210> 470
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 470
Thr Ser Pro Ser Gly Arg Ser Glu Val Pro Arg Gly
1 5 10
<210> 471
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 471
Thr Ser Pro Ser Gly Arg Ser Ala Glu Pro Arg Gly
1 5 10
<210> 472
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 472
Thr Ser Pro Ser Gly Arg Ser Ala Gly Pro Arg Gly
1 5 10
<210> 473
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 473
Thr Ser Ala Ser Gly Arg Ser Glu Asn Ala Arg Gly
1 5 10
<210> 474
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 474
Thr Ser Ala Ser Gly Arg Ser Ala Glu Ala Arg Gly
1 5 10
<210> 475
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 475
Thr Ser Ala Ser Gly Arg Ser Ala Gly Ala Arg Gly
1 5 10
<210> 476
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 476
Thr Ser Gly Thr Gly Arg Ser Ala Thr Pro Arg Gly
1 5 10
<210> 477
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 477
Thr Ser Gly Ser Gly Arg Ser Ala Thr Tyr Arg Gly
1 5 10
<210> 478
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 478
Thr Ser Gly Ser Gly Arg Ser Ala Thr Val Arg Gly
1 5 10
<210> 479
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 479
Thr Ser Gly Ser Gly Arg Ser Ala Thr His Arg Gly
1 5 10
<210> 480
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 480
Thr Ser Gly Thr Gly Arg Ser Ala Thr Tyr Arg Gly
1 5 10
<210> 481
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 481
Thr Ser Gly Thr Gly Arg Ser Ala Thr Val Arg Gly
1 5 10
<210> 482
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 482
Thr Ser Gly Thr Gly Arg Ser Ala Thr His Arg Gly
1 5 10
<210> 483
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 483
Thr Ser Gly Ser Gly Arg Ser Glu Thr Pro Arg Gly
1 5 10
<210> 484
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 484
Thr Ser Gly Thr Gly Arg Ser Glu Thr Pro Arg Gly
1 5 10
<210> 485
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 485
Thr Ser Gly Ser Gly Arg Ser Glu Thr Tyr Arg Gly
1 5 10
<210> 486
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 486
Thr Ser Gly Ser Gly Arg Ser Glu Thr Val Arg Gly
1 5 10
<210> 487
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 487
Thr Ser Gly Ser Gly Arg Ser Glu Thr His Arg Gly
1 5 10
<210> 488
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 488
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 489
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 489
Thr Ser Tyr Ser Gly Arg Ser Ala Val Tyr Arg Gly
1 5 10
<210> 490
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 490
Thr Ser Tyr Ser Gly Arg Ser Ala Val Val Arg Gly
1 5 10
<210> 491
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 491
Thr Ser Tyr Ser Gly Arg Ser Ala Val His Arg Gly
1 5 10
<210> 492
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 492
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Arg Gly
1 5 10
<210> 493
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 493
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val Arg Gly
1 5 10
<210> 494
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 494
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Arg Gly
1 5 10
<210> 495
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 495
Thr Ser Tyr Ser Gly Arg Ser Glu Val Pro Arg Gly
1 5 10
<210> 496
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 496
Thr Ser Tyr Thr Gly Arg Ser Glu Val Pro Arg Gly
1 5 10
<210> 497
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 497
Thr Ser Tyr Ser Gly Arg Ser Glu Val Tyr Arg Gly
1 5 10
<210> 498
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 498
Thr Ser Tyr Ser Gly Arg Ser Glu Val Val Arg Gly
1 5 10
<210> 499
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 499
Thr Ser Tyr Ser Gly Arg Ser Glu Val His Arg Gly
1 5 10
<210> 500
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 500
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Gly Gly
1 5 10
<210> 501
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 501
Thr Ser Tyr Ser Gly Arg Ser Ala Val Tyr Gly Gly
1 5 10
<210> 502
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 502
Thr Ser Tyr Ser Gly Arg Ser Ala Val Val Gly Gly
1 5 10
<210> 503
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 503
Thr Ser Tyr Ser Gly Arg Ser Ala Val His Gly Gly
1 5 10
<210> 504
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 504
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Gly Gly
1 5 10
<210> 505
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 505
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val Gly Gly
1 5 10
<210> 506
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 506
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Gly Gly
1 5 10
<210> 507
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 507
Ala Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 508
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 508
Glu Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 509
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 509
Phe Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 510
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 510
Gly Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 511
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 511
His Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 512
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 512
Lys Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 513
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 513
Met Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 514
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 514
Asn Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 515
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 515
Pro Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 516
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 516
Gln Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 517
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 517
Trp Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 518
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 518
Tyr Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 519
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 519
Thr Ala Gly Arg Ser Ala Asn Pro
1 5
<210> 520
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 520
Thr Asp Gly Arg Ser Ala Asn Pro
1 5
<210> 521
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 521
Thr Glu Gly Arg Ser Ala Asn Pro
1 5
<210> 522
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 522
Thr Phe Gly Arg Ser Ala Asn Pro
1 5
<210> 523
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 523
Thr Leu Gly Arg Ser Ala Asn Pro
1 5
<210> 524
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 524
Thr Met Gly Arg Ser Ala Asn Pro
1 5
<210> 525
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 525
Thr Pro Gly Arg Ser Ala Asn Pro
1 5
<210> 526
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 526
Thr Gln Gly Arg Ser Ala Asn Pro
1 5
<210> 527
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 527
Thr Val Gly Arg Ser Ala Asn Pro
1 5
<210> 528
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 528
Thr Trp Gly Arg Ser Ala Asn Pro
1 5
<210> 529
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 529
Thr Ser Ala Arg Ser Ala Asn Pro
1 5
<210> 530
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 530
Thr Ser Glu Arg Ser Ala Asn Pro
1 5
<210> 531
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 531
Thr Ser Phe Arg Ser Ala Asn Pro
1 5
<210> 532
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 532
Thr Ser His Arg Ser Ala Asn Pro
1 5
<210> 533
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 533
Thr Ser Ile Arg Ser Ala Asn Pro
1 5
<210> 534
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 534
Thr Ser Lys Arg Ser Ala Asn Pro
1 5
<210> 535
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 535
Thr Ser Leu Arg Ser Ala Asn Pro
1 5
<210> 536
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 536
Thr Ser Met Arg Ser Ala Asn Pro
1 5
<210> 537
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 537
Thr Ser Asn Arg Ser Ala Asn Pro
1 5
<210> 538
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 538
Thr Ser Pro Arg Ser Ala Asn Pro
1 5
<210> 539
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 539
Thr Ser Gln Arg Ser Ala Asn Pro
1 5
<210> 540
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 540
Thr Ser Arg Arg Ser Ala Asn Pro
1 5
<210> 541
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 541
Thr Ser Thr Arg Ser Ala Asn Pro
1 5
<210> 542
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 542
Thr Ser Val Arg Ser Ala Asn Pro
1 5
<210> 543
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 543
Thr Ser Trp Arg Ser Ala Asn Pro
1 5
<210> 544
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 544
Thr Ser Tyr Arg Ser Ala Asn Pro
1 5
<210> 545
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 545
Thr Ser Gly Arg Ala Ala Asn Pro
1 5
<210> 546
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 546
Thr Ser Gly Arg Asp Ala Asn Pro
1 5
<210> 547
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 547
Thr Ser Gly Arg Glu Ala Asn Pro
1 5
<210> 548
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 548
Thr Ser Gly Arg Gly Ala Asn Pro
1 5
<210> 549
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 549
Thr Ser Gly Arg His Ala Asn Pro
1 5
<210> 550
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 550
Thr Ser Gly Arg Ile Ala Asn Pro
1 5
<210> 551
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 551
Thr Ser Gly Arg Lys Ala Asn Pro
1 5
<210> 552
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 552
Thr Ser Gly Arg Leu Ala Asn Pro
1 5
<210> 553
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 553
Thr Ser Gly Arg Met Ala Asn Pro
1 5
<210> 554
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 554
Thr Ser Gly Arg Asn Ala Asn Pro
1 5
<210> 555
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 555
Thr Ser Gly Arg Pro Ala Asn Pro
1 5
<210> 556
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 556
Thr Ser Gly Arg Gln Ala Asn Pro
1 5
<210> 557
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 557
Thr Ser Gly Arg Arg Ala Asn Pro
1 5
<210> 558
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 558
Thr Ser Gly Arg Thr Ala Asn Pro
1 5
<210> 559
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 559
Thr Ser Gly Arg Val Ala Asn Pro
1 5
<210> 560
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 560
Thr Ser Gly Arg Trp Ala Asn Pro
1 5
<210> 561
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 561
Thr Ser Gly Arg Tyr Ala Asn Pro
1 5
<210> 562
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 562
Thr Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 563
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 563
Thr Ser Gly Arg Ser Phe Asn Pro
1 5
<210> 564
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 564
Thr Ser Gly Arg Ser Lys Asn Pro
1 5
<210> 565
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 565
Thr Ser Gly Arg Ser Met Asn Pro
1 5
<210> 566
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 566
Thr Ser Gly Arg Ser Asn Asn Pro
1 5
<210> 567
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 567
Thr Ser Gly Arg Ser Pro Asn Pro
1 5
<210> 568
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 568
Thr Ser Gly Arg Ser Gln Asn Pro
1 5
<210> 569
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 569
Thr Ser Gly Arg Ser Arg Asn Pro
1 5
<210> 570
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 570
Thr Ser Gly Arg Ser Ser Asn Pro
1 5
<210> 571
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 571
Thr Ser Gly Arg Ser Trp Asn Pro
1 5
<210> 572
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 572
Thr Ser Gly Arg Ser Tyr Asn Pro
1 5
<210> 573
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 573
Thr Ser Gly Arg Ser Ala Ala Pro
1 5
<210> 574
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 574
Thr Ser Gly Arg Ser Ala Asp Pro
1 5
<210> 575
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 575
Thr Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 576
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 576
Thr Ser Gly Arg Ser Ala Phe Pro
1 5
<210> 577
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 577
Thr Ser Gly Arg Ser Ala Gly Pro
1 5
<210> 578
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 578
Thr Ser Gly Arg Ser Ala Lys Pro
1 5
<210> 579
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 579
Thr Ser Gly Arg Ser Ala Leu Pro
1 5
<210> 580
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 580
Thr Ser Gly Arg Ser Ala Met Pro
1 5
<210> 581
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 581
Thr Ser Gly Arg Ser Ala Pro Pro
1 5
<210> 582
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 582
Thr Ser Gly Arg Ser Ala Gln Pro
1 5
<210> 583
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 583
Thr Ser Gly Arg Ser Ala Val Pro
1 5
<210> 584
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 584
Thr Ser Gly Arg Ser Ala Trp Pro
1 5
<210> 585
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 585
Thr Ser Gly Arg Ser Ala Tyr Pro
1 5
<210> 586
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 586
Thr Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 587
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 587
Thr Ser Gly Arg Ser Ala Asn Asp
1 5
<210> 588
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 588
Thr Ser Gly Arg Ser Ala Asn Glu
1 5
<210> 589
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 589
Thr Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 590
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 590
Thr Ser Gly Arg Ser Ala Asn Gly
1 5
<210> 591
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 591
Thr Ser Gly Arg Ser Ala Asn Ile
1 5
<210> 592
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 592
Thr Ser Gly Arg Ser Ala Asn Lys
1 5
<210> 593
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 593
Thr Ser Gly Arg Ser Ala Asn Asn
1 5
<210> 594
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 594
Thr Ser Gly Arg Ser Ala Asn Gln
1 5
<210> 595
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 595
Thr Ser Gly Arg Ser Ala Asn Ser
1 5
<210> 596
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 596
Thr Ser Gly Arg Ser Ala Asn Thr
1 5
<210> 597
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 597
Thr Ser Gly Arg Ser Ala Asn Trp
1 5
<210> 598
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 598
Asp Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 599
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 599
Ile Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 600
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 600
Ser Ser Gly Arg Ser Ala Asn Pro
1 5
<210> 601
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 601
Thr His Gly Arg Ser Ala Asn Pro
1 5
<210> 602
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 602
Thr Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 603
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 603
Thr Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 604
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 604
Thr Tyr Gly Arg Ser Ala Asn Pro
1 5
<210> 605
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 605
Thr Ser Asp Arg Ser Ala Asn Pro
1 5
<210> 606
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 606
Thr Ser Ser Arg Ser Ala Asn Pro
1 5
<210> 607
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 607
Thr Ser Gly Arg Phe Ala Asn Pro
1 5
<210> 608
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 608
Thr Ser Gly Arg Ser Asp Asn Pro
1 5
<210> 609
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 609
Thr Ser Gly Arg Ser His Asn Pro
1 5
<210> 610
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 610
Thr Ser Gly Arg Ser Ile Asn Pro
1 5
<210> 611
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 611
Thr Ser Gly Arg Ser Leu Asn Pro
1 5
<210> 612
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 612
Thr Ser Gly Arg Ser Thr Asn Pro
1 5
<210> 613
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 613
Thr Ser Gly Arg Ser Val Asn Pro
1 5
<210> 614
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 614
Thr Ser Gly Arg Ser Ala His Pro
1 5
<210> 615
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 615
Thr Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 616
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 616
Thr Ser Gly Arg Ser Ala Arg Pro
1 5
<210> 617
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 617
Thr Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 618
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 618
Thr Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 619
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 619
Thr Ser Gly Arg Ser Ala Asn His
1 5
<210> 620
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 620
Thr Ser Gly Arg Ser Ala Asn Leu
1 5
<210> 621
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 621
Thr Ser Gly Arg Ser Ala Asn Met
1 5
<210> 622
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 622
Thr Ser Gly Arg Ser Ala Asn Arg
1 5
<210> 623
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 623
Thr Ser Gly Arg Ser Ala Asn Val
1 5
<210> 624
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 624
Thr Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 625
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 625
Gly Ser Gly Arg Ser Ala Val Pro
1 5
<210> 626
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 626
Gly Ser Gly Arg Ser Ala Tyr Pro
1 5
<210> 627
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 627
Gly Ser Gly Arg Ser Ala Asn Gln
1 5
<210> 628
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 628
Gly Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 629
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 629
Gly Ser Gly Arg Ser Ala Asn Ile
1 5
<210> 630
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 630
Gly Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 631
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 631
Gly Ser Gly Arg Ser Ala Asn Ser
1 5
<210> 632
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 632
Gln Ser Gly Arg Ser Ala Val Pro
1 5
<210> 633
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 633
Gln Ser Gly Arg Ser Ala Tyr Pro
1 5
<210> 634
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 634
Gln Ser Gly Arg Ser Ala Asn Gln
1 5
<210> 635
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 635
Gln Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 636
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 636
Gln Ser Gly Arg Ser Ala Asn Ile
1 5
<210> 637
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 637
Gln Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 638
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 638
Gln Ser Gly Arg Ser Ala Asn Ser
1 5
<210> 639
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 639
Pro Ser Gly Arg Ser Ala Val Pro
1 5
<210> 640
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 640
Pro Ser Gly Arg Ser Ala Tyr Pro
1 5
<210> 641
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 641
Pro Ser Gly Arg Ser Ala Asn Gln
1 5
<210> 642
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 642
Pro Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 643
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 643
Pro Ser Gly Arg Ser Ala Asn Ile
1 5
<210> 644
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 644
Pro Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 645
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 645
Pro Ser Gly Arg Ser Ala Asn Ser
1 5
<210> 646
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 646
Ala Ser Gly Arg Ser Ala Val Pro
1 5
<210> 647
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 647
Ala Ser Gly Arg Ser Ala Tyr Pro
1 5
<210> 648
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 648
Ala Ser Gly Arg Ser Ala Asn Gln
1 5
<210> 649
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 649
Ala Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 650
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 650
Ala Ser Gly Arg Ser Ala Asn Ile
1 5
<210> 651
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 651
Ala Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 652
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 652
Ala Ser Gly Arg Ser Ala Asn Ser
1 5
<210> 653
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 653
Tyr Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 654
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 654
Gly Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 655
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 655
Gln Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 656
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 656
Pro Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 657
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 657
Ala Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 658
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 658
His Ser Gly Arg Ser Glu Asn Pro
1 5
<210> 659
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 659
Thr Ser Gly Arg Ser Glu Asn Gln
1 5
<210> 660
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 660
Thr Ser Gly Arg Ser Glu Asn Ala
1 5
<210> 661
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 661
Thr Ser Gly Arg Ser Glu Asn Ile
1 5
<210> 662
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 662
Thr Ser Gly Arg Ser Glu Asn Phe
1 5
<210> 663
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 663
Thr Ser Gly Arg Ser Glu Asn Ser
1 5
<210> 664
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 664
Tyr Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 665
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 665
Gly Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 666
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 666
Gln Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 667
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 667
Pro Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 668
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 668
Ala Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 669
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 669
His Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 670
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 670
Thr Ser Gly Arg Ser Ala Glu Gln
1 5
<210> 671
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 671
Thr Ser Gly Arg Ser Ala Glu Ala
1 5
<210> 672
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 672
Thr Ser Gly Arg Ser Ala Glu Ile
1 5
<210> 673
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 673
Thr Ser Gly Arg Ser Ala Glu Phe
1 5
<210> 674
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 674
Thr Ser Gly Arg Ser Ala Glu Ser
1 5
<210> 675
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 675
Gly Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 676
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 676
Gly Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 677
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 677
Gly Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 678
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 678
Gly Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 679
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 679
Gly Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 680
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 680
Gly Ser Gly Arg Ser Ala His Pro
1 5
<210> 681
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 681
Gly Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 682
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 682
Gly Ser Gly Arg Ser Ala Asn Val
1 5
<210> 683
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 683
Gly Ser Gly Arg Ser Ala Asn His
1 5
<210> 684
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 684
Gln Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 685
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 685
Gln Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 686
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 686
Gln Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 687
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 687
Gln Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 688
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 688
Gln Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 689
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 689
Gln Ser Gly Arg Ser Ala His Pro
1 5
<210> 690
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 690
Gln Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 691
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 691
Gln Ser Gly Arg Ser Ala Asn Val
1 5
<210> 692
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 692
Gln Ser Gly Arg Ser Ala Asn His
1 5
<210> 693
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 693
Pro Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 694
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 694
Pro Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 695
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 695
Pro Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 696
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 696
Pro Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 697
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 697
Pro Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 698
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 698
Pro Ser Gly Arg Ser Ala His Pro
1 5
<210> 699
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 699
Pro Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 700
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 700
Pro Ser Gly Arg Ser Ala Asn Val
1 5
<210> 701
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 701
Pro Ser Gly Arg Ser Ala Asn His
1 5
<210> 702
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 702
Ala Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 703
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 703
Ala Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 704
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 704
Ala Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 705
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 705
Ala Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 706
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 706
Ala Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 707
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 707
Ala Ser Gly Arg Ser Ala His Pro
1 5
<210> 708
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 708
Ala Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 709
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 709
Ala Ser Gly Arg Ser Ala Asn Val
1 5
<210> 710
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 710
Ala Ser Gly Arg Ser Ala Asn His
1 5
<210> 711
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 711
Tyr Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 712
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 712
Tyr Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 713
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 713
Tyr Ser Gly Arg Ser Ala Val Pro
1 5
<210> 714
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 714
Tyr Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 715
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 715
Tyr Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 716
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 716
Tyr Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 717
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 717
Tyr Ser Gly Arg Ser Ala His Pro
1 5
<210> 718
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 718
Tyr Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 719
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 719
Tyr Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 720
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 720
Tyr Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 721
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 721
Tyr Ser Gly Arg Ser Ala Asn Val
1 5
<210> 722
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 722
Tyr Ser Gly Arg Ser Ala Asn His
1 5
<210> 723
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 723
Ser Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 724
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 724
Ser Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 725
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 725
Ser Ser Gly Arg Ser Ala Val Pro
1 5
<210> 726
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 726
Ser Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 727
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 727
Ser Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 728
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 728
Ser Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 729
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 729
Ser Ser Gly Arg Ser Ala His Pro
1 5
<210> 730
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 730
Ser Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 731
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 731
Ser Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 732
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 732
Ser Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 733
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 733
Ser Ser Gly Arg Ser Ala Asn Val
1 5
<210> 734
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 734
Ser Ser Gly Arg Ser Ala Asn His
1 5
<210> 735
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 735
Ile Thr Gly Arg Ser Ala Asn Pro
1 5
<210> 736
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 736
Ile Lys Gly Arg Ser Ala Asn Pro
1 5
<210> 737
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 737
Ile Ser Gly Arg Ser Ala Val Pro
1 5
<210> 738
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 738
Ile Ser Gly Arg Ser Ala Ile Pro
1 5
<210> 739
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 739
Ile Ser Gly Arg Ser Ala Thr Pro
1 5
<210> 740
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 740
Ile Ser Gly Arg Ser Ala Ser Pro
1 5
<210> 741
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 741
Ile Ser Gly Arg Ser Ala His Pro
1 5
<210> 742
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 742
Ile Ser Gly Arg Ser Ala Asn Ala
1 5
<210> 743
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 743
Ile Ser Gly Arg Ser Ala Asn Phe
1 5
<210> 744
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 744
Ile Ser Gly Arg Ser Ala Asn Tyr
1 5
<210> 745
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 745
Ile Ser Gly Arg Ser Ala Asn Val
1 5
<210> 746
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 746
Ile Ser Gly Arg Ser Ala Asn His
1 5
<210> 747
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 747
Thr Thr Gly Arg Ser Ala Val Pro
1 5
<210> 748
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 748
Thr Thr Gly Arg Ser Ala Ile Pro
1 5
<210> 749
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 749
Thr Thr Gly Arg Ser Ala Thr Pro
1 5
<210> 750
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 750
Thr Thr Gly Arg Ser Ala Ser Pro
1 5
<210> 751
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 751
Thr Thr Gly Arg Ser Ala His Pro
1 5
<210> 752
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 752
Thr Thr Gly Arg Ser Ala Asn Ala
1 5
<210> 753
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 753
Thr Thr Gly Arg Ser Ala Asn Phe
1 5
<210> 754
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 754
Thr Thr Gly Arg Ser Ala Asn Tyr
1 5
<210> 755
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 755
Thr Thr Gly Arg Ser Ala Asn Val
1 5
<210> 756
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 756
Thr Thr Gly Arg Ser Ala Asn His
1 5
<210> 757
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 757
Thr Lys Gly Arg Ser Ala Val Pro
1 5
<210> 758
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 758
Thr Lys Gly Arg Ser Ala Ile Pro
1 5
<210> 759
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 759
Thr Lys Gly Arg Ser Ala Thr Pro
1 5
<210> 760
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 760
Thr Lys Gly Arg Ser Ala Ser Pro
1 5
<210> 761
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 761
Thr Lys Gly Arg Ser Ala His Pro
1 5
<210> 762
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 762
Thr Lys Gly Arg Ser Ala Asn Ala
1 5
<210> 763
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 763
Thr Lys Gly Arg Ser Ala Asn Phe
1 5
<210> 764
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 764
Thr Lys Gly Arg Ser Ala Asn Tyr
1 5
<210> 765
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 765
Thr Lys Gly Arg Ser Ala Asn Val
1 5
<210> 766
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 766
Thr Lys Gly Arg Ser Ala Asn His
1 5
<210> 767
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 767
Thr Ser Gly Arg Ser Ala Val Tyr
1 5
<210> 768
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 768
Thr Ser Gly Arg Ser Ala Val Val
1 5
<210> 769
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 769
Thr Ser Gly Arg Ser Ala Val His
1 5
<210> 770
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 770
Thr Ser Gly Arg Ser Ala Ile Tyr
1 5
<210> 771
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 771
Thr Ser Gly Arg Ser Ala Ile Val
1 5
<210> 772
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 772
Thr Ser Gly Arg Ser Ala Ile His
1 5
<210> 773
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 773
Thr Ser Gly Arg Ser Ala Ser Tyr
1 5
<210> 774
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 774
Thr Ser Gly Arg Ser Ala Ser Val
1 5
<210> 775
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 775
Thr Ser Gly Arg Ser Ala Ser His
1 5
<210> 776
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 776
Thr Ser Gly Arg Ser Ala His Tyr
1 5
<210> 777
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 777
Thr Ser Gly Arg Ser Ala His Val
1 5
<210> 778
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 778
Thr Ser Gly Arg Ser Ala His His
1 5
<210> 779
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 779
Pro Ser Gly Arg Ser Glu Val Pro
1 5
<210> 780
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 780
Pro Ser Gly Arg Ser Ala Glu Pro
1 5
<210> 781
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 781
Pro Ser Gly Arg Ser Ala Gly Pro
1 5
<210> 782
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 782
Ala Ser Gly Arg Ser Glu Asn Ala
1 5
<210> 783
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 783
Ala Ser Gly Arg Ser Ala Glu Ala
1 5
<210> 784
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 784
Ala Ser Gly Arg Ser Ala Gly Ala
1 5
<210> 785
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 785
Gly Thr Gly Arg Ser Ala Thr Pro
1 5
<210> 786
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 786
Gly Ser Gly Arg Ser Ala Thr Tyr
1 5
<210> 787
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 787
Gly Ser Gly Arg Ser Ala Thr Val
1 5
<210> 788
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 788
Gly Ser Gly Arg Ser Ala Thr His
1 5
<210> 789
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 789
Gly Thr Gly Arg Ser Ala Thr Tyr
1 5
<210> 790
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 790
Gly Thr Gly Arg Ser Ala Thr Val
1 5
<210> 791
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 791
Gly Thr Gly Arg Ser Ala Thr His
1 5
<210> 792
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 792
Gly Ser Gly Arg Ser Glu Thr Pro
1 5
<210> 793
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 793
Gly Thr Gly Arg Ser Glu Thr Pro
1 5
<210> 794
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 794
Gly Ser Gly Arg Ser Glu Thr Tyr
1 5
<210> 795
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 795
Gly Ser Gly Arg Ser Glu Thr Val
1 5
<210> 796
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 796
Gly Ser Gly Arg Ser Glu Thr His
1 5
<210> 797
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 797
Tyr Thr Gly Arg Ser Ala Val Pro
1 5
<210> 798
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 798
Tyr Ser Gly Arg Ser Ala Val Tyr
1 5
<210> 799
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 799
Tyr Ser Gly Arg Ser Ala Val Val
1 5
<210> 800
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 800
Tyr Ser Gly Arg Ser Ala Val His
1 5
<210> 801
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 801
Tyr Thr Gly Arg Ser Ala Val Tyr
1 5
<210> 802
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 802
Tyr Thr Gly Arg Ser Ala Val Val
1 5
<210> 803
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 803
Tyr Thr Gly Arg Ser Ala Val His
1 5
<210> 804
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 804
Tyr Ser Gly Arg Ser Glu Val Pro
1 5
<210> 805
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 805
Tyr Thr Gly Arg Ser Glu Val Pro
1 5
<210> 806
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 806
Tyr Ser Gly Arg Ser Glu Val Tyr
1 5
<210> 807
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 807
Tyr Ser Gly Arg Ser Glu Val Val
1 5
<210> 808
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 808
Tyr Ser Gly Arg Ser Glu Val His
1 5
<210> 809
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 809
Tyr Thr Gly Arg Ser Ala Val Pro
1 5
<210> 810
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 810
Tyr Ser Gly Arg Ser Ala Val Tyr
1 5
<210> 811
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 811
Tyr Ser Gly Arg Ser Ala Val Val
1 5
<210> 812
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 812
Tyr Ser Gly Arg Ser Ala Val His
1 5
<210> 813
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 813
Tyr Thr Gly Arg Ser Ala Val Tyr
1 5
<210> 814
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 814
Tyr Thr Gly Arg Ser Ala Val Val
1 5
<210> 815
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 815
Tyr Thr Gly Arg Ser Ala Val His
1 5
<210> 816
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 816
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 817
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 817
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 818
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 818
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Tyr Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 819
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 819
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Val Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 820
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 820
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val His Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 821
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 821
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 822
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 822
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 823
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 823
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 824
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 824
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 825
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 825
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Tyr Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 826
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 826
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Val Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 827
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 827
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val His Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 828
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 828
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 829
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 829
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 830
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 830
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Gly Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 831
<211> 462
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 831
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Thr Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Phe
20 25 30
Ser Ile Thr Trp Leu Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Glu Ile Thr Pro Met Phe Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ser Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Gly Leu Thr Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Arg Phe Asp Val Ser Asp Leu Leu Thr Asp Lys Pro
100 105 110
Lys Val Thr Ile Asn Tyr Asn Gly Met Asp Val Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Arg Arg Gly Pro Ser
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 832
<211> 216
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 832
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 833
<400> 833
000
<210> 834
<400> 834
000
<210> 835
<400> 835
000
<210> 836
<400> 836
000
<210> 837
<400> 837
000
<210> 838
<400> 838
000
<210> 839
<400> 839
000
<210> 840
<400> 840
000
<210> 841
<400> 841
000
<210> 842
<400> 842
000
<210> 843
<400> 843
000
<210> 844
<400> 844
000
<210> 845
<400> 845
000
<210> 846
<400> 846
000
<210> 847
<400> 847
000
<210> 848
<400> 848
000
<210> 849
<400> 849
000
<210> 850
<400> 850
000
<210> 851
<400> 851
000
<210> 852
<400> 852
000
<210> 853
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 853
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Arg Gly
1 5 10
<210> 854
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 854
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Ala Gly
1 5 10
<210> 855
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 855
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro His Gly
1 5 10
<210> 856
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 856
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 857
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 857
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Leu Gly
1 5 10
<210> 858
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 858
Thr Ser Thr Ser Gly Arg Ser Ala Asn Pro Ser Gly
1 5 10
<210> 859
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 859
Ile Ser Thr Ser Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 860
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 860
Tyr Ser Thr Ser Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 861
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 861
Thr Ser Tyr Ser Gly Arg Ser Ala Val Pro Ala Gly
1 5 10
<210> 862
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 862
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ile Ala Gly
1 5 10
<210> 863
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 863
Thr Ser Pro Ser Gly Arg Ser Ala Asn Phe Ala Gly
1 5 10
<210> 864
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 864
Thr Ser Pro Thr Gly Arg Ser Ala Asn Pro Ala Gly
1 5 10
<210> 865
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 865
Thr Ser Pro Ser Gly Arg Ser Ala Ile Pro Ala Gly
1 5 10
<210> 866
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 866
Thr Ser Tyr Thr Gly Arg Ser Ala Asn Pro Ala Gly
1 5 10
<210> 867
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 867
Thr Ser Tyr Ser Gly Arg Ser Ala Ile Pro Ala Gly
1 5 10
<210> 868
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 868
Thr Ser Ile Ser Gly Arg Ser Ala Asn Tyr Ala Gly
1 5 10
<210> 869
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 869
Thr Ser Pro Ser Gly Arg Ser Ala Gly Pro Ala Gly
1 5 10
<210> 870
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 870
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Ala Gly
1 5 10
<210> 871
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 871
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Ala Gly
1 5 10
<210> 872
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 872
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val Ala Gly
1 5 10
<210> 873
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 873
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Ala Gly
1 5 10
<210> 874
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 874
Thr Ser Tyr Ser Gly Arg Ser Ala Val Pro His Gly
1 5 10
<210> 875
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 875
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ile His Gly
1 5 10
<210> 876
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 876
Thr Ser Pro Ser Gly Arg Ser Ala Asn Phe His Gly
1 5 10
<210> 877
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 877
Thr Ser Pro Thr Gly Arg Ser Ala Asn Pro His Gly
1 5 10
<210> 878
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 878
Thr Ser Pro Ser Gly Arg Ser Ala Ile Pro His Gly
1 5 10
<210> 879
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 879
Thr Ser Tyr Thr Gly Arg Ser Ala Asn Pro His Gly
1 5 10
<210> 880
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 880
Thr Ser Tyr Ser Gly Arg Ser Ala Ile Pro His Gly
1 5 10
<210> 881
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 881
Thr Ser Ile Ser Gly Arg Ser Ala Asn Tyr His Gly
1 5 10
<210> 882
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 882
Thr Ser Pro Ser Gly Arg Ser Ala Gly Pro His Gly
1 5 10
<210> 883
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 883
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro His Gly
1 5 10
<210> 884
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 884
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr His Gly
1 5 10
<210> 885
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 885
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val His Gly
1 5 10
<210> 886
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 886
Thr Ser Tyr Thr Gly Arg Ser Ala Val His His Gly
1 5 10
<210> 887
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 887
Thr Ser Tyr Ser Gly Arg Ser Ala Val Pro Ile Gly
1 5 10
<210> 888
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 888
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ile Ile Gly
1 5 10
<210> 889
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 889
Thr Ser Pro Ser Gly Arg Ser Ala Asn Phe Ile Gly
1 5 10
<210> 890
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 890
Thr Ser Pro Thr Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 891
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 891
Thr Ser Pro Ser Gly Arg Ser Ala Ile Pro Ile Gly
1 5 10
<210> 892
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 892
Thr Ser Tyr Thr Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 893
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 893
Thr Ser Tyr Ser Gly Arg Ser Ala Ile Pro Ile Gly
1 5 10
<210> 894
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 894
Thr Ser Ile Ser Gly Arg Ser Ala Asn Tyr Ile Gly
1 5 10
<210> 895
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 895
Thr Ser Pro Ser Gly Arg Ser Ala Gly Pro Ile Gly
1 5 10
<210> 896
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 896
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Ile Gly
1 5 10
<210> 897
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 897
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Ile Gly
1 5 10
<210> 898
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 898
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val Ile Gly
1 5 10
<210> 899
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 899
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Ile Gly
1 5 10
<210> 900
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 900
Thr Ser Tyr Ser Gly Arg Ser Ala Val Pro Leu Gly
1 5 10
<210> 901
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 901
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ile Leu Gly
1 5 10
<210> 902
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 902
Thr Ser Pro Ser Gly Arg Ser Ala Asn Phe Leu Gly
1 5 10
<210> 903
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 903
Thr Ser Pro Thr Gly Arg Ser Ala Asn Pro Leu Gly
1 5 10
<210> 904
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 904
Thr Ser Pro Ser Gly Arg Ser Ala Ile Pro Leu Gly
1 5 10
<210> 905
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 905
Thr Ser Tyr Thr Gly Arg Ser Ala Asn Pro Leu Gly
1 5 10
<210> 906
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 906
Thr Ser Tyr Ser Gly Arg Ser Ala Ile Pro Leu Gly
1 5 10
<210> 907
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 907
Thr Ser Ile Ser Gly Arg Ser Ala Asn Tyr Leu Gly
1 5 10
<210> 908
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 908
Thr Ser Pro Ser Gly Arg Ser Ala Gly Pro Leu Gly
1 5 10
<210> 909
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 909
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Leu Gly
1 5 10
<210> 910
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 910
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Leu Gly
1 5 10
<210> 911
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 911
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val Leu Gly
1 5 10
<210> 912
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 912
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Leu Gly
1 5 10
<210> 913
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 913
Thr Ser Tyr Ser Gly Arg Ser Ala Val Pro Ser Gly
1 5 10
<210> 914
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 914
Thr Ser Pro Ser Gly Arg Ser Ala Asn Ile Ser Gly
1 5 10
<210> 915
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 915
Thr Ser Pro Ser Gly Arg Ser Ala Asn Phe Ser Gly
1 5 10
<210> 916
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 916
Thr Ser Pro Thr Gly Arg Ser Ala Asn Pro Ser Gly
1 5 10
<210> 917
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 917
Thr Ser Pro Ser Gly Arg Ser Ala Ile Pro Ser Gly
1 5 10
<210> 918
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 918
Thr Ser Tyr Thr Gly Arg Ser Ala Asn Pro Ser Gly
1 5 10
<210> 919
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 919
Thr Ser Tyr Ser Gly Arg Ser Ala Ile Pro Ser Gly
1 5 10
<210> 920
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 920
Thr Ser Ile Ser Gly Arg Ser Ala Asn Tyr Ser Gly
1 5 10
<210> 921
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 921
Thr Ser Pro Ser Gly Arg Ser Ala Gly Pro Ser Gly
1 5 10
<210> 922
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 922
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Ser Gly
1 5 10
<210> 923
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 923
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Ser Gly
1 5 10
<210> 924
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 924
Thr Ser Tyr Thr Gly Arg Ser Ala Val Val Ser Gly
1 5 10
<210> 925
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 925
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Ser Gly
1 5 10
<210> 926
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 926
Ile Ser Tyr Ser Gly Arg Ser Ala Val Pro Ile Gly
1 5 10
<210> 927
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 927
Ile Ser Pro Ser Gly Arg Ser Ala Asn Ile Ile Gly
1 5 10
<210> 928
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 928
Ile Ser Pro Ser Gly Arg Ser Ala Asn Phe Ile Gly
1 5 10
<210> 929
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 929
Ile Ser Pro Thr Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 930
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 930
Ile Ser Pro Ser Gly Arg Ser Ala Ile Pro Ile Gly
1 5 10
<210> 931
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 931
Ile Ser Tyr Thr Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 932
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 932
Ile Ser Tyr Ser Gly Arg Ser Ala Ile Pro Ile Gly
1 5 10
<210> 933
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 933
Ile Ser Ile Ser Gly Arg Ser Ala Asn Tyr Ile Gly
1 5 10
<210> 934
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 934
Ile Ser Pro Ser Gly Arg Ser Ala Gly Pro Ile Gly
1 5 10
<210> 935
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 935
Ile Ser Tyr Thr Gly Arg Ser Ala Val Pro Ile Gly
1 5 10
<210> 936
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 936
Ile Ser Tyr Thr Gly Arg Ser Ala Val Tyr Ile Gly
1 5 10
<210> 937
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 937
Ile Ser Tyr Thr Gly Arg Ser Ala Val Val Ile Gly
1 5 10
<210> 938
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 938
Ile Ser Tyr Thr Gly Arg Ser Ala Val His Ile Gly
1 5 10
<210> 939
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 939
Tyr Ser Tyr Ser Gly Arg Ser Ala Val Pro Ile Gly
1 5 10
<210> 940
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 940
Tyr Ser Pro Ser Gly Arg Ser Ala Asn Ile Ile Gly
1 5 10
<210> 941
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 941
Tyr Ser Pro Ser Gly Arg Ser Ala Asn Phe Ile Gly
1 5 10
<210> 942
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 942
Tyr Ser Pro Thr Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 943
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 943
Tyr Ser Pro Ser Gly Arg Ser Ala Ile Pro Ile Gly
1 5 10
<210> 944
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 944
Tyr Ser Tyr Thr Gly Arg Ser Ala Asn Pro Ile Gly
1 5 10
<210> 945
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 945
Tyr Ser Tyr Ser Gly Arg Ser Ala Ile Pro Ile Gly
1 5 10
<210> 946
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 946
Tyr Ser Ile Ser Gly Arg Ser Ala Asn Tyr Ile Gly
1 5 10
<210> 947
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 947
Tyr Ser Pro Ser Gly Arg Ser Ala Gly Pro Ile Gly
1 5 10
<210> 948
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 948
Tyr Ser Tyr Thr Gly Arg Ser Ala Val Pro Ile Gly
1 5 10
<210> 949
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 949
Tyr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Ile Gly
1 5 10
<210> 950
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 950
Tyr Ser Tyr Thr Gly Arg Ser Ala Val Val Ile Gly
1 5 10
<210> 951
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 951
Tyr Ser Tyr Thr Gly Arg Ser Ala Val His Ile Gly
1 5 10
<210> 952
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 952
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 953
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 953
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 954
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 954
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 955
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 955
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 956
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 956
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 957
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 957
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 958
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 958
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 959
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 959
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Thr Ser Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 960
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 960
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 961
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 961
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 962
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 962
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 963
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 963
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 964
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 964
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 965
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 965
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 966
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 966
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 967
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 967
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 968
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 968
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 969
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 969
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 970
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 970
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 971
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 971
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 972
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 972
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Ala Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 973
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 973
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 974
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 974
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 975
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 975
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 976
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 976
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 977
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 977
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 978
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 978
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 979
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 979
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 980
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 980
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 981
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 981
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 982
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 982
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 983
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 983
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 984
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 984
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 985
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 985
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His His Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 986
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 986
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 987
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 987
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 988
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 988
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 989
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 989
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 990
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 990
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 991
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 991
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 992
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 992
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 993
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 993
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 994
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 994
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 995
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 995
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 996
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 996
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 997
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 997
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 998
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 998
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 999
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 999
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1000
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1000
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1001
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1001
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1002
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1002
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1003
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1003
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1004
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1004
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1005
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1005
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1006
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1006
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1007
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1007
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1008
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1008
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1009
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1009
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1010
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1010
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1011
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1011
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Leu Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1012
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1012
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1013
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1013
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1014
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1014
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1015
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1015
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1016
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1016
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1017
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1017
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1018
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1018
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1019
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1019
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1020
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1020
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1021
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1021
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1022
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1022
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1023
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1023
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1024
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1024
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Ser Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1025
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1025
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1026
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1026
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1027
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1027
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1028
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1028
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1029
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1029
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1030
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1030
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1031
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1031
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1032
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1032
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1033
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1033
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1034
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1034
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1035
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1035
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1036
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1036
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1037
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1037
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ile Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1038
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1038
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1039
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1039
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Ile Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1040
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1040
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Asn Phe Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1041
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1041
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Pro Thr Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1042
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1042
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Ile Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1043
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1043
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Asn Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1044
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1044
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Ile Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1045
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1045
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Ile Ser Gly Arg Ser Ala Asn Tyr Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1046
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1046
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Pro Ser Gly Arg Ser Ala Gly Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1047
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1047
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1048
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1048
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1049
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1049
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Val Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1050
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1050
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Tyr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Ile Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1051
<211> 462
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1051
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Thr Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Phe
20 25 30
Ser Ile Thr Trp Leu Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Glu Ile Thr Pro Met Phe Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ser Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Gly Leu Thr Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Arg Phe Asp Val Ser Asp Leu Leu Thr Asp Lys Pro
100 105 110
Lys Val Thr Ile Asn Tyr Asn Gly Met Asp Val Trp Gly Gln Gly Thr
115 120 125
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
130 135 140
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
145 150 155 160
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
165 170 175
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
180 185 190
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
195 200 205
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
210 215 220
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
225 230 235 240
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Arg Gly Gly Pro Lys
245 250 255
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
260 265 270
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
275 280 285
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
290 295 300
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
305 310 315 320
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
325 330 335
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
340 345 350
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
355 360 365
Pro Pro Ser Arg Lys Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
370 375 380
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
385 390 395 400
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Tyr Leu Asp
405 410 415
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
420 425 430
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
435 440 445
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
450 455 460
<210> 1052
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1052
Thr Ser Tyr Thr Gly Arg Ser Ala Val Pro Arg Gly
1 5 10
<210> 1053
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1053
Thr Ser Tyr Ser Gly Arg Ser Ala Val Val Arg Gly
1 5 10
<210> 1054
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1054
Thr Ser Tyr Thr Gly Arg Ser Ala Val Tyr Arg Gly
1 5 10
<210> 1055
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1055
Thr Ser Tyr Thr Gly Arg Ser Ala Val His Arg Gly
1 5 10
<210> 1056
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1056
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Pro Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1057
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1057
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Ser Gly Arg Ser Ala Val Val Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1058
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1058
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val Tyr Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1059
<211> 228
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1059
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn
20 25 30
Thr Val Asn Trp Tyr Gln Arg Leu Pro Gly Ala Ala Pro Gln Leu Leu
35 40 45
Ile Tyr Asn Asn Asp Gln Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Gly Ser Leu Val Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ser Trp Asp Asp Ser Leu
85 90 95
Asn Gly Arg Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Thr Ser
100 105 110
Tyr Thr Gly Arg Ser Ala Val His Arg Gly Gly Gln Pro Lys Ala Ala
115 120 125
Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala Asn
130 135 140
Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala Val
145 150 155 160
Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val Glu
165 170 175
Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser Ser
180 185 190
Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr Ser
195 200 205
Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala Pro
210 215 220
Thr Glu Cys Ser
225
<210> 1060
<211> 444
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1060
Gln Val Gln Leu Gln Gln Ser Gly Pro Gln Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Ser Tyr
20 25 30
Trp Met His Trp Val Asn Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Met Ile Asp Pro Ser Tyr Ser Glu Thr Arg Leu Asn Gln Lys Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Pro Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Leu Tyr Gly Asn Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Leu Arg Gly Gly Pro Lys Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Tyr Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Glu Ser Leu Ser Leu Ser Pro
435 440
<210> 1061
<211> 214
<212> PRT
<213> Artificial sequence
<220>
<223> an artificially synthesized sequence
<400> 1061
Asp Ile Gln Met Thr Gln Ser Ser Ser Ser Phe Ser Val Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Glu Asp Ile Tyr Asn Arg
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Asn Ala Pro Arg Leu Leu Ile
35 40 45
Ser Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Lys Asp Tyr Thr Leu Ser Ile Thr Ser Leu Gln Thr
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Val Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<---
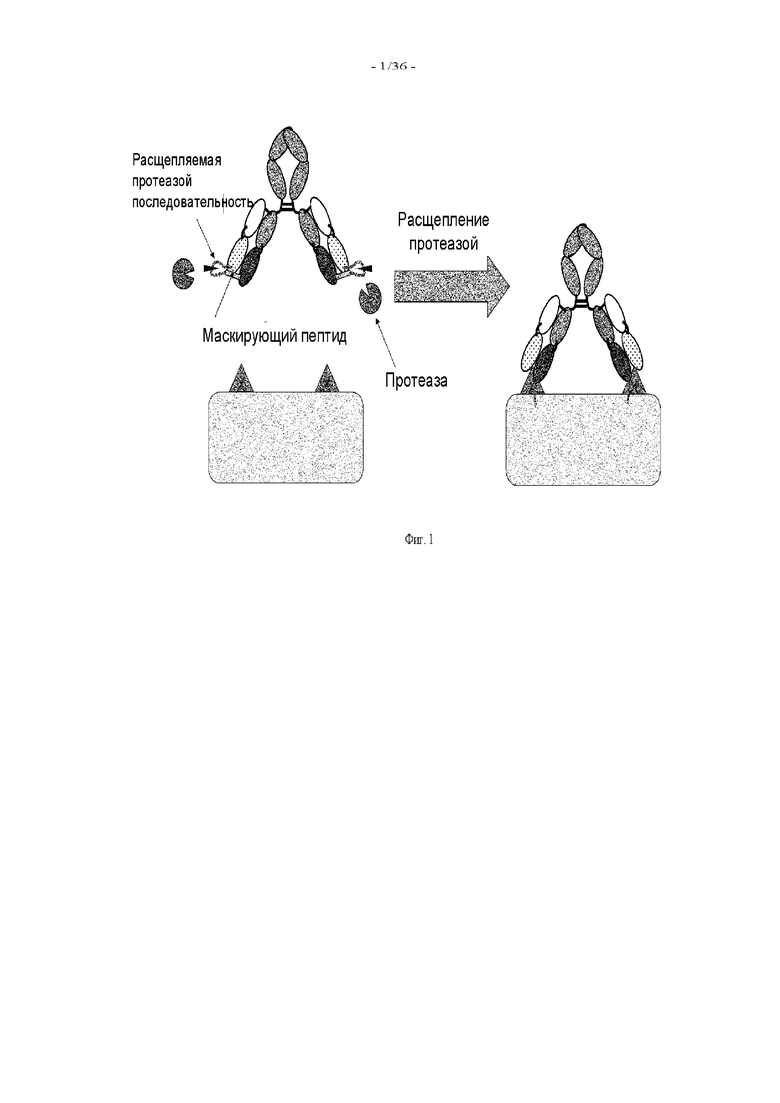
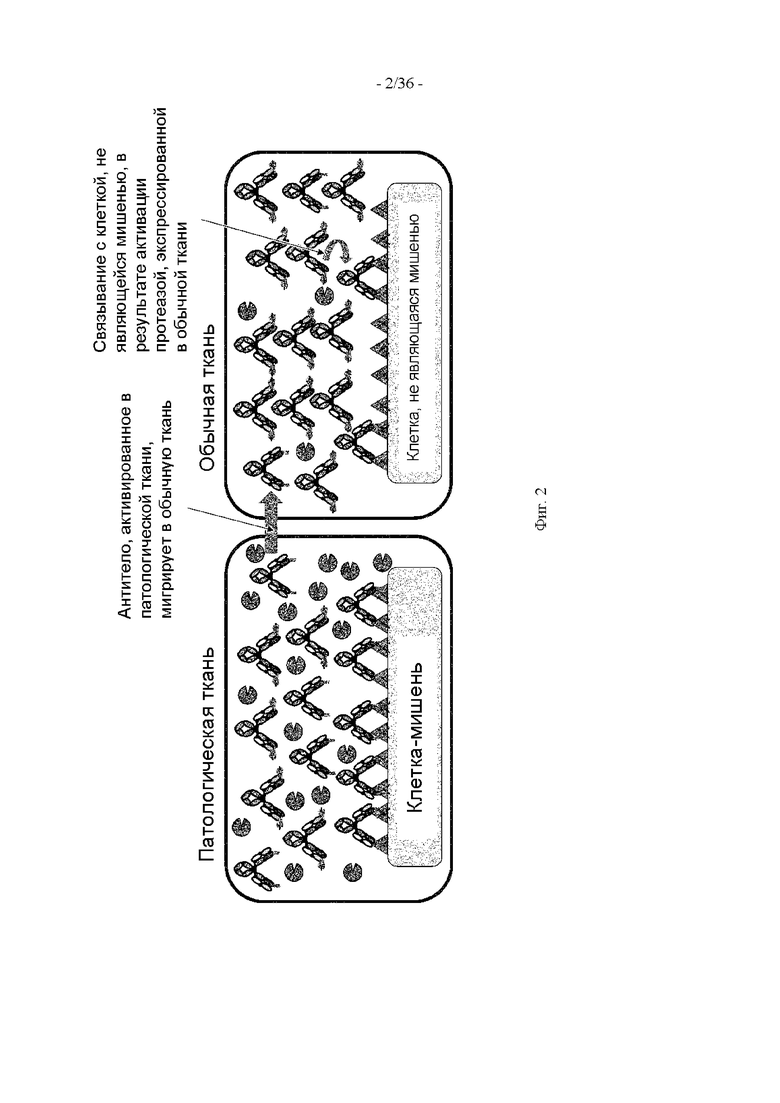
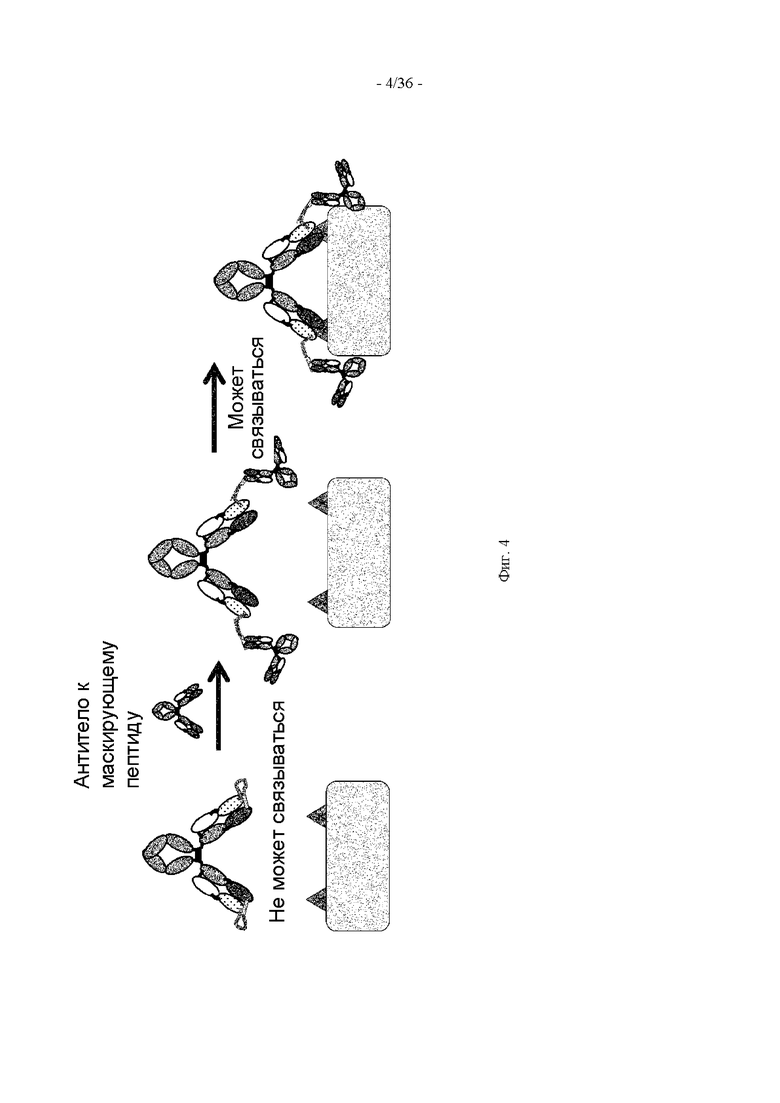
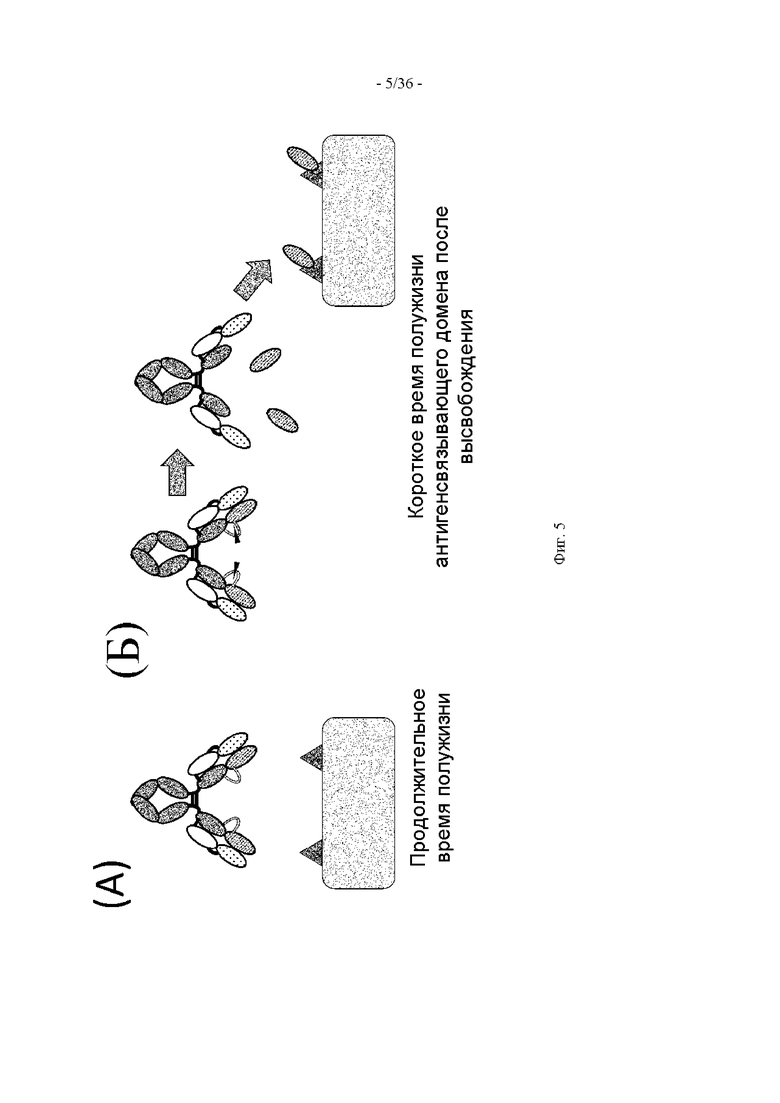
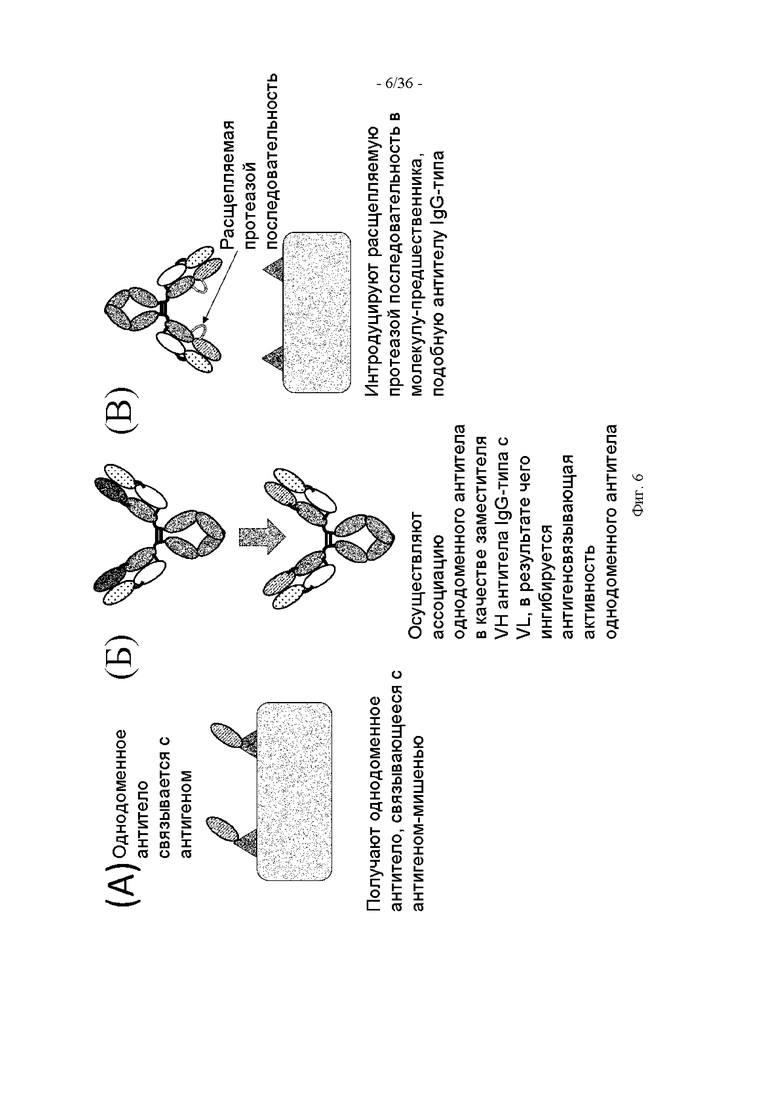
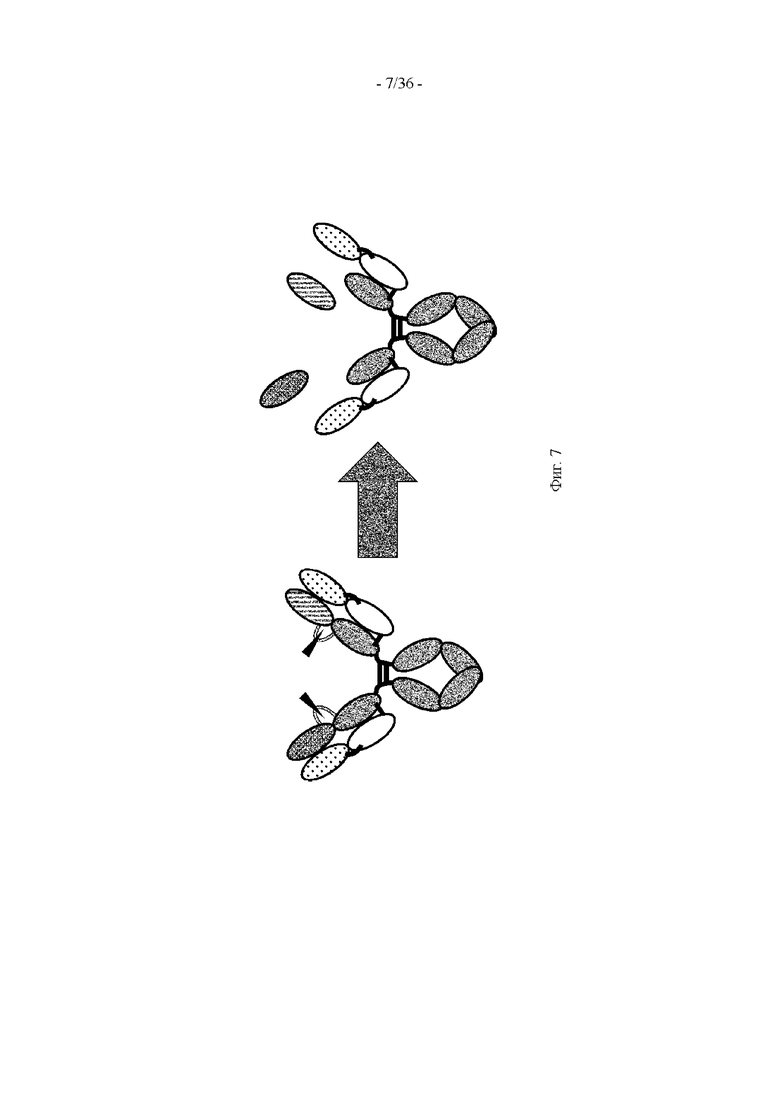
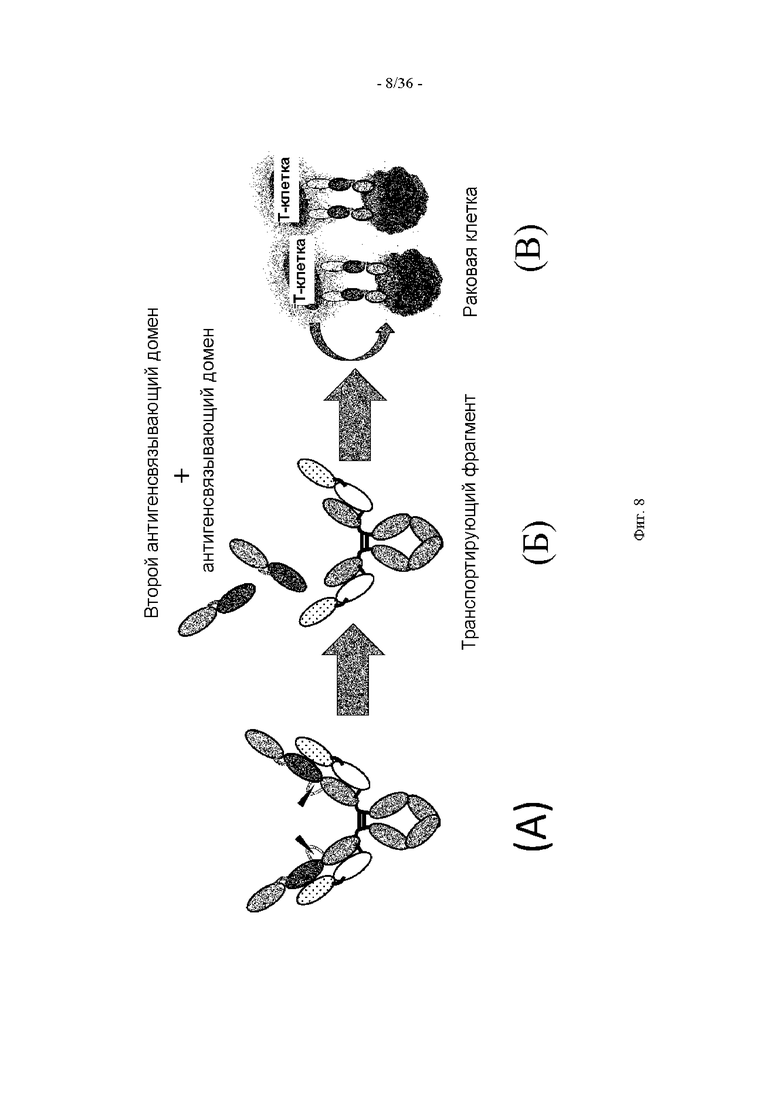
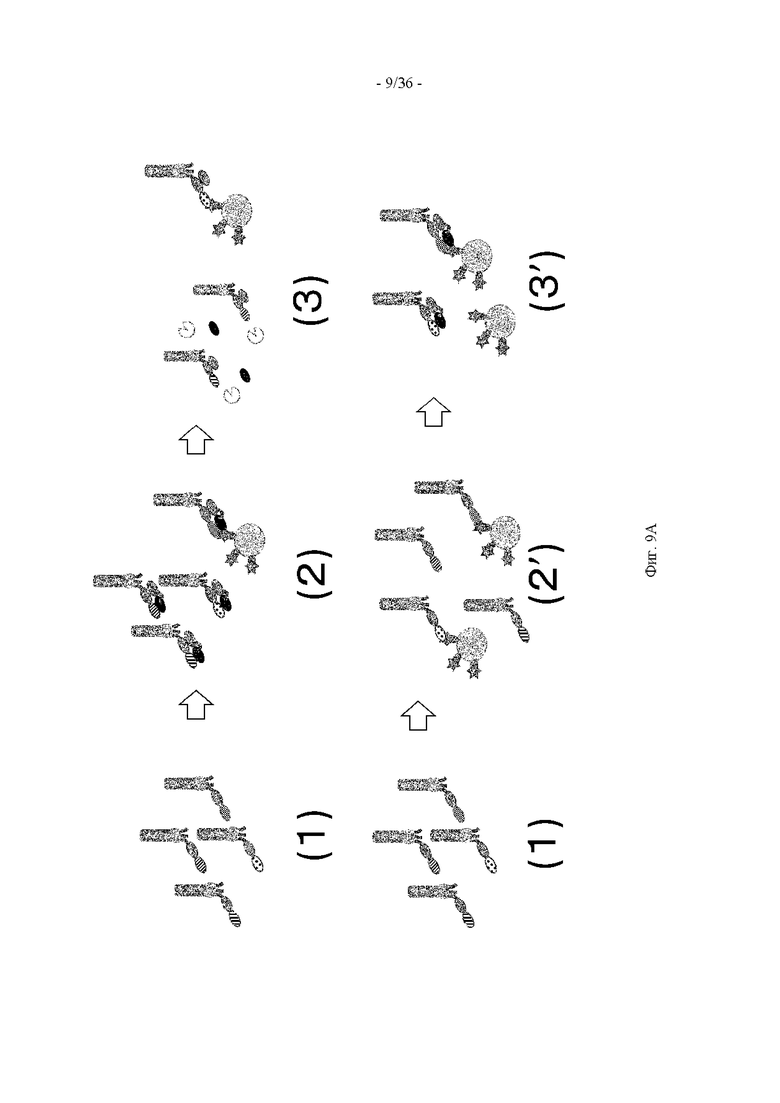
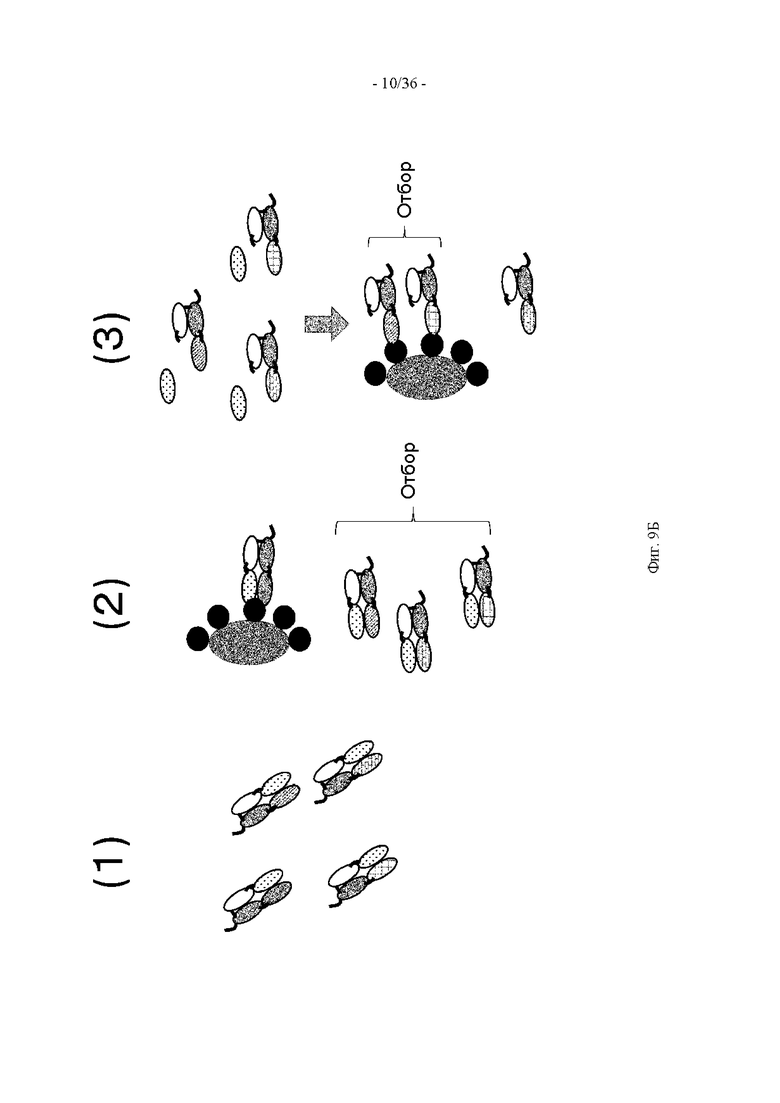
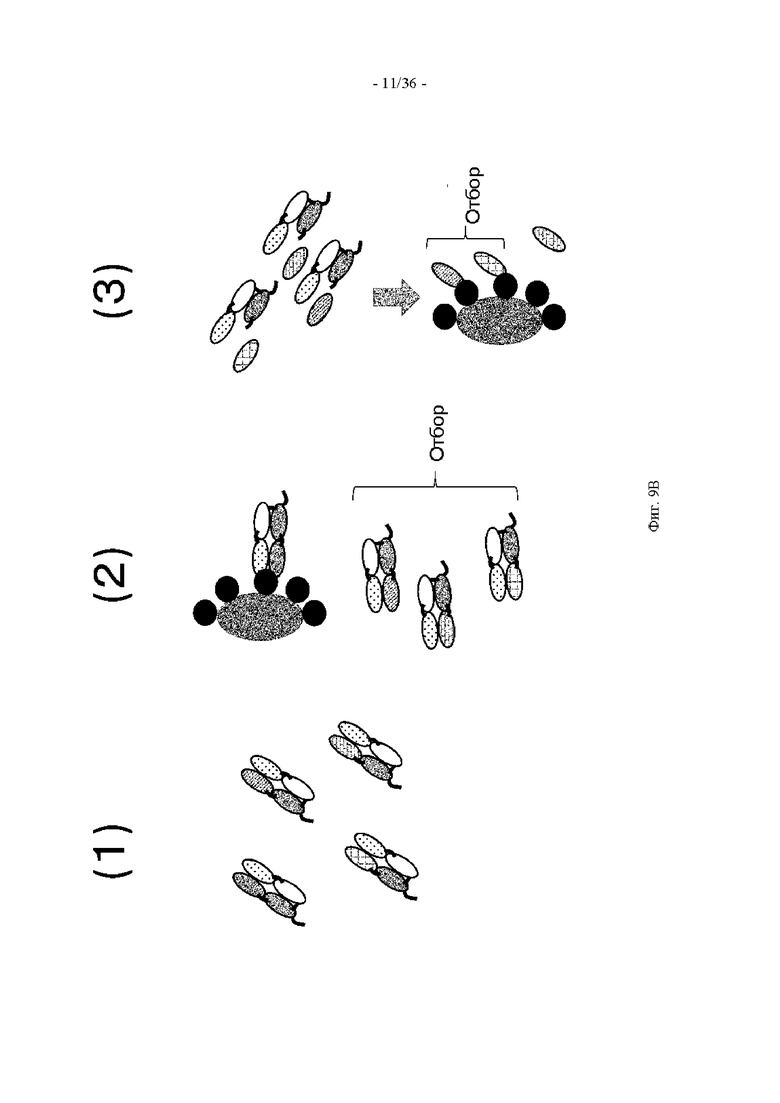
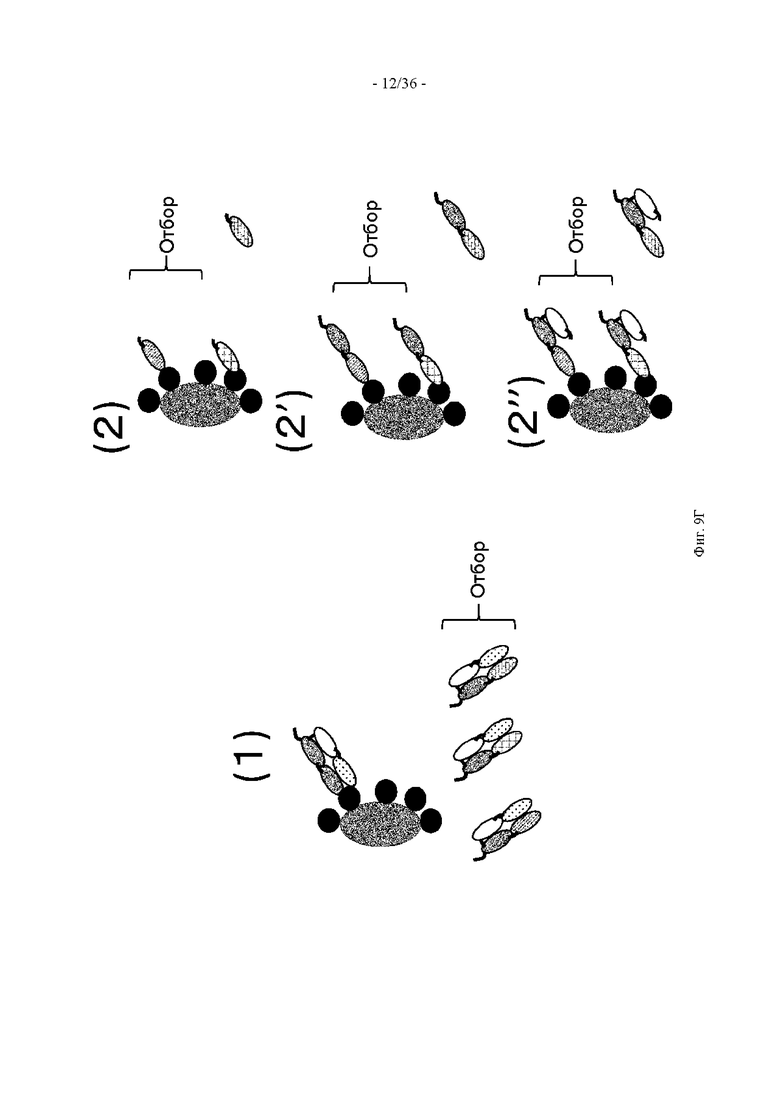
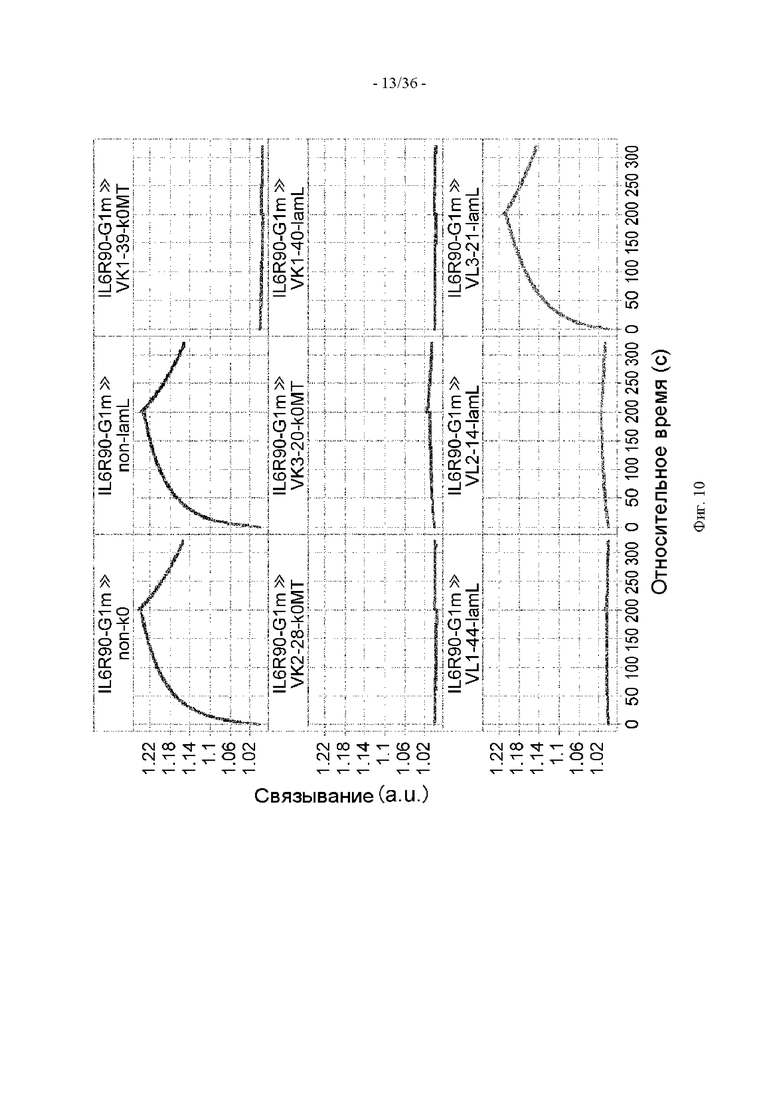

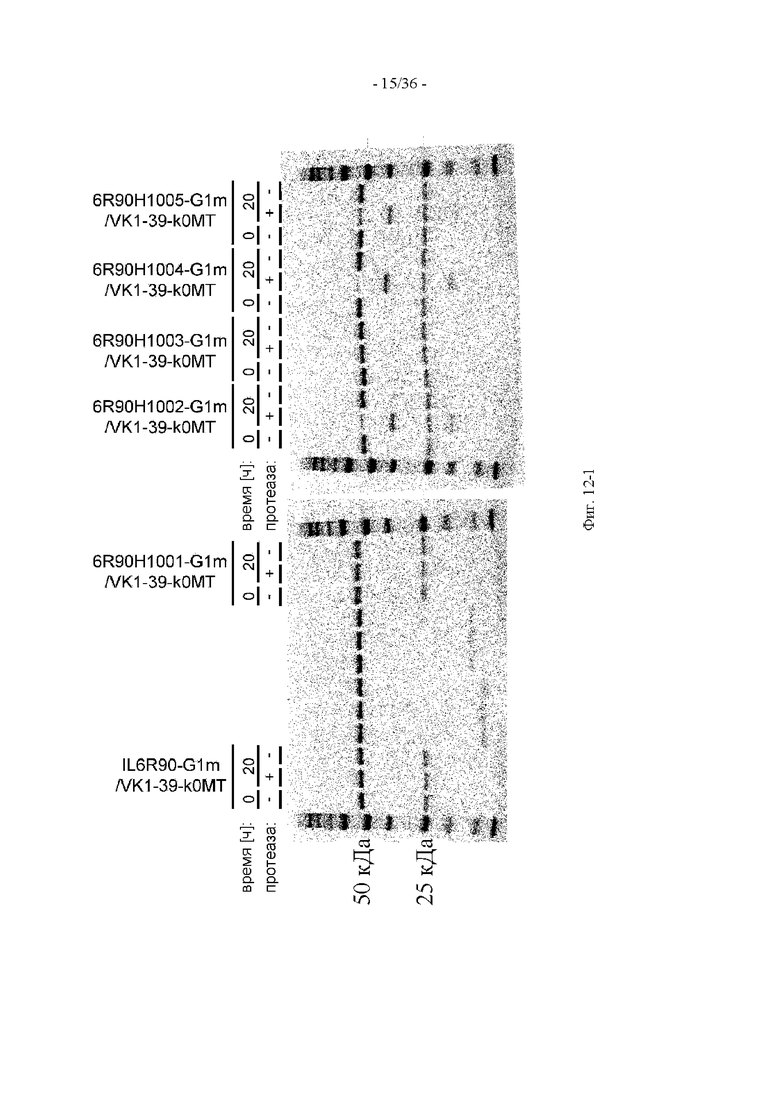
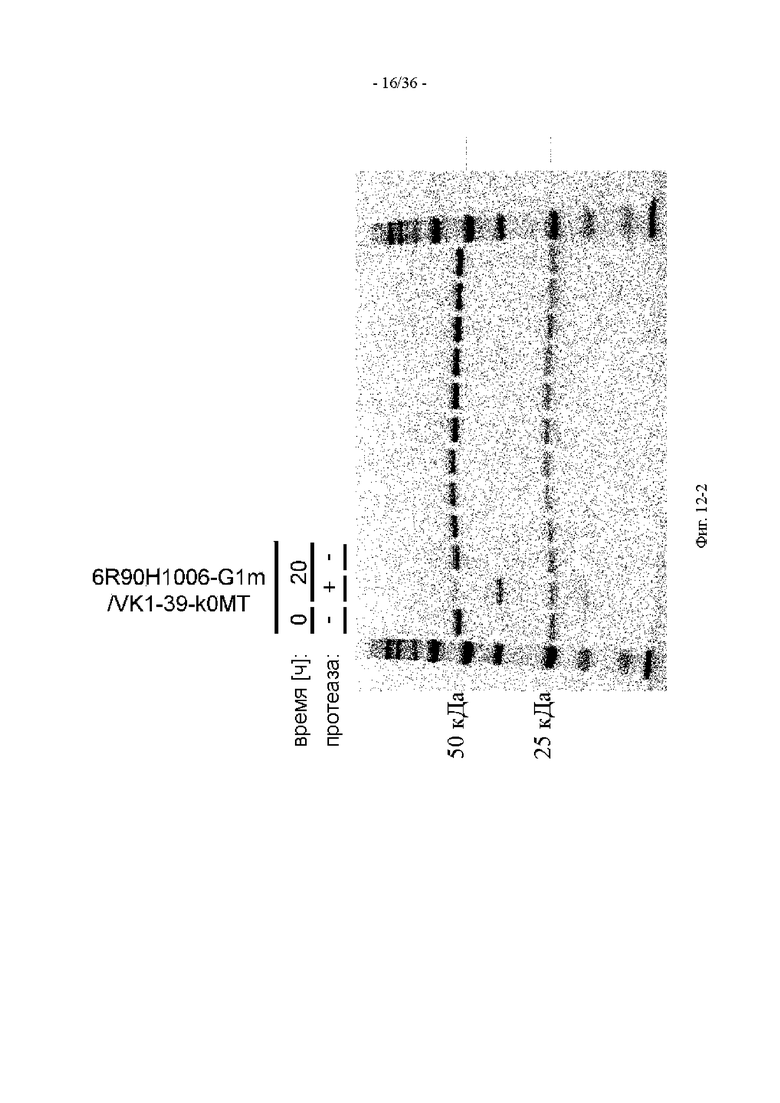
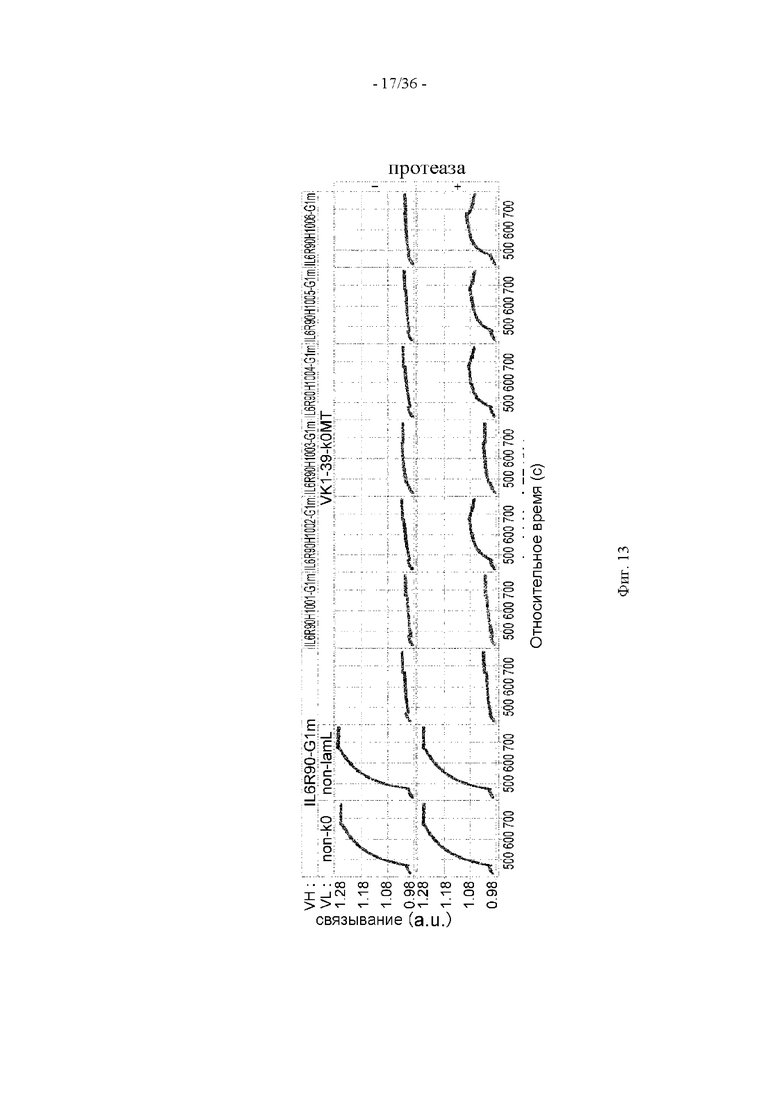
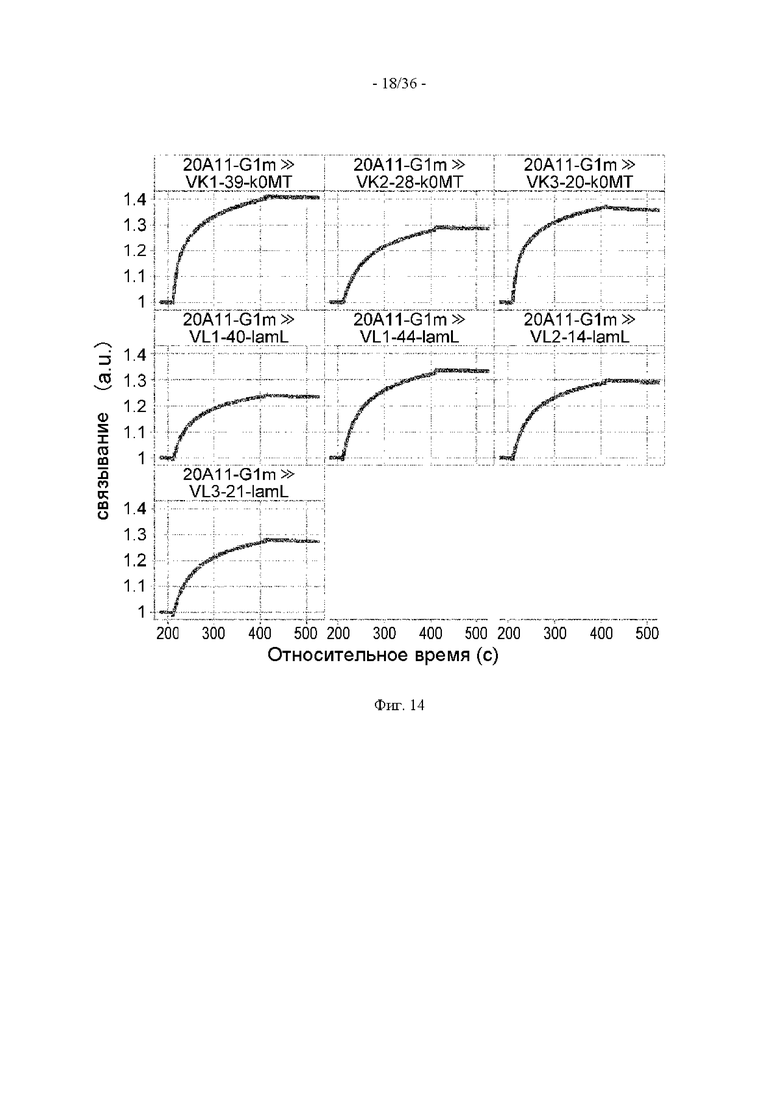
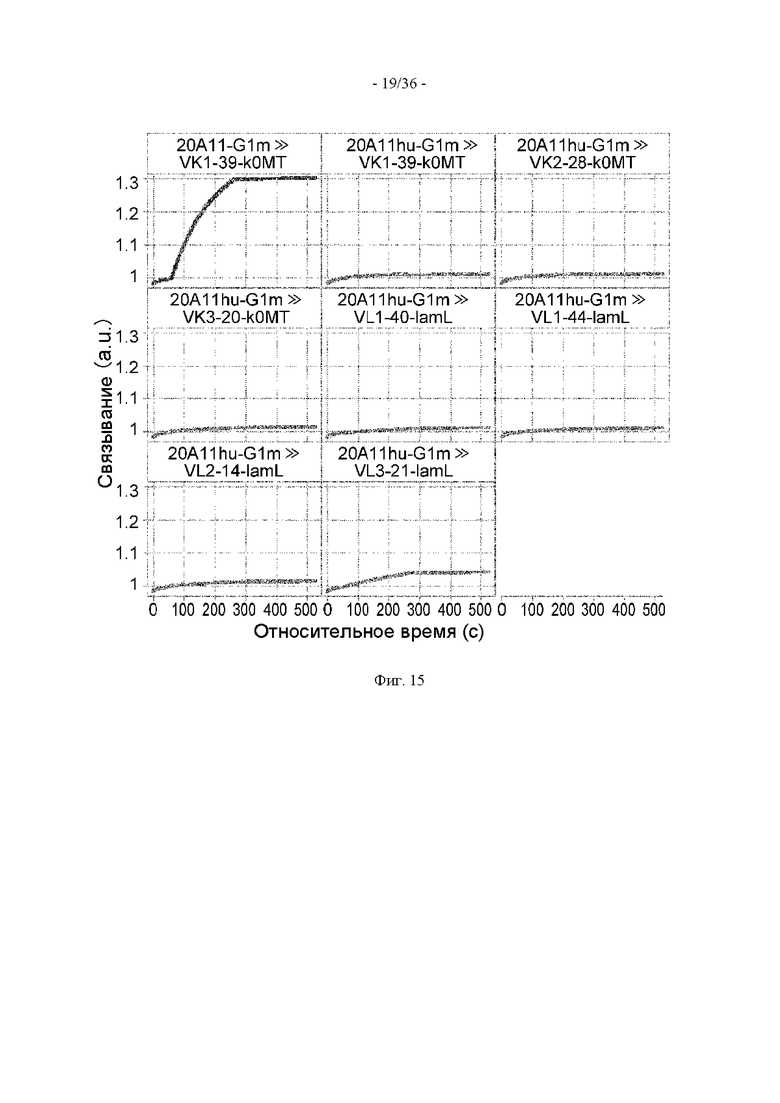
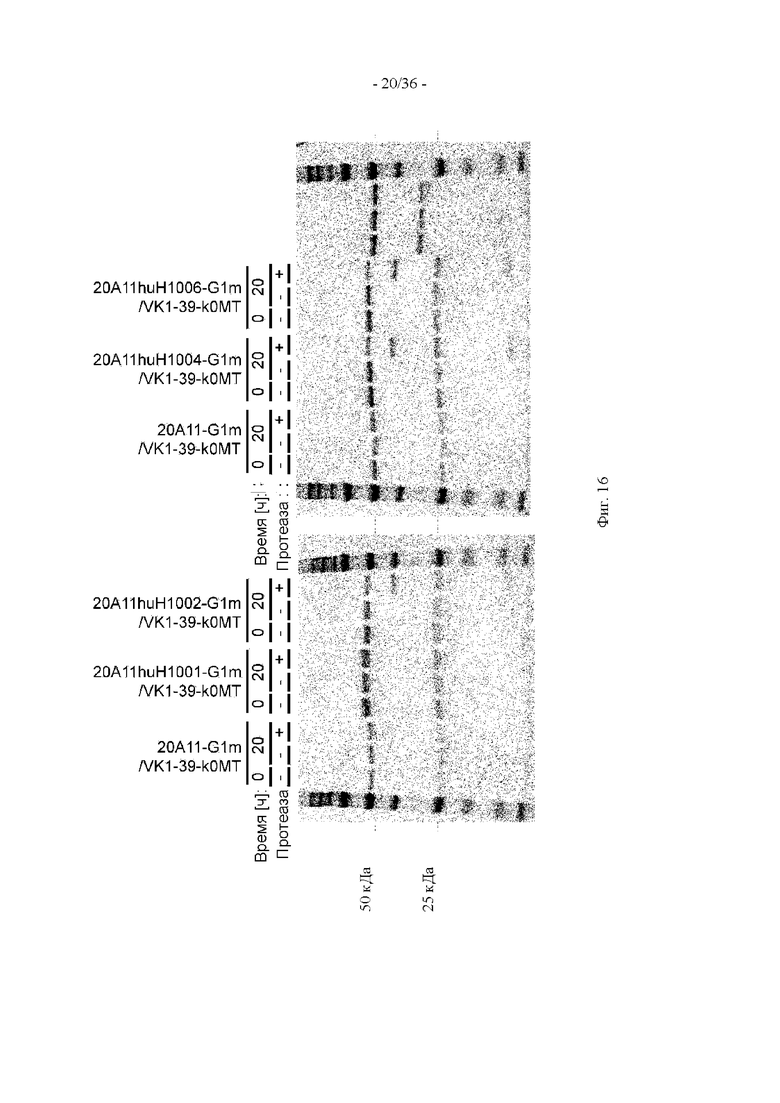
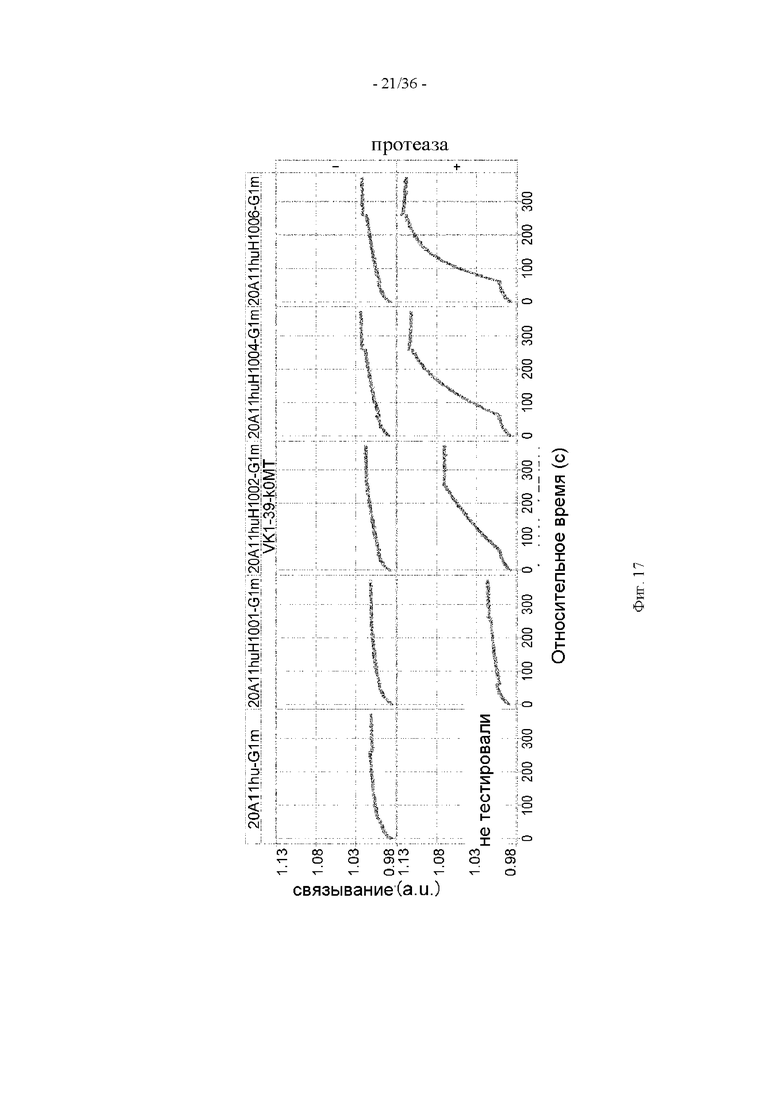
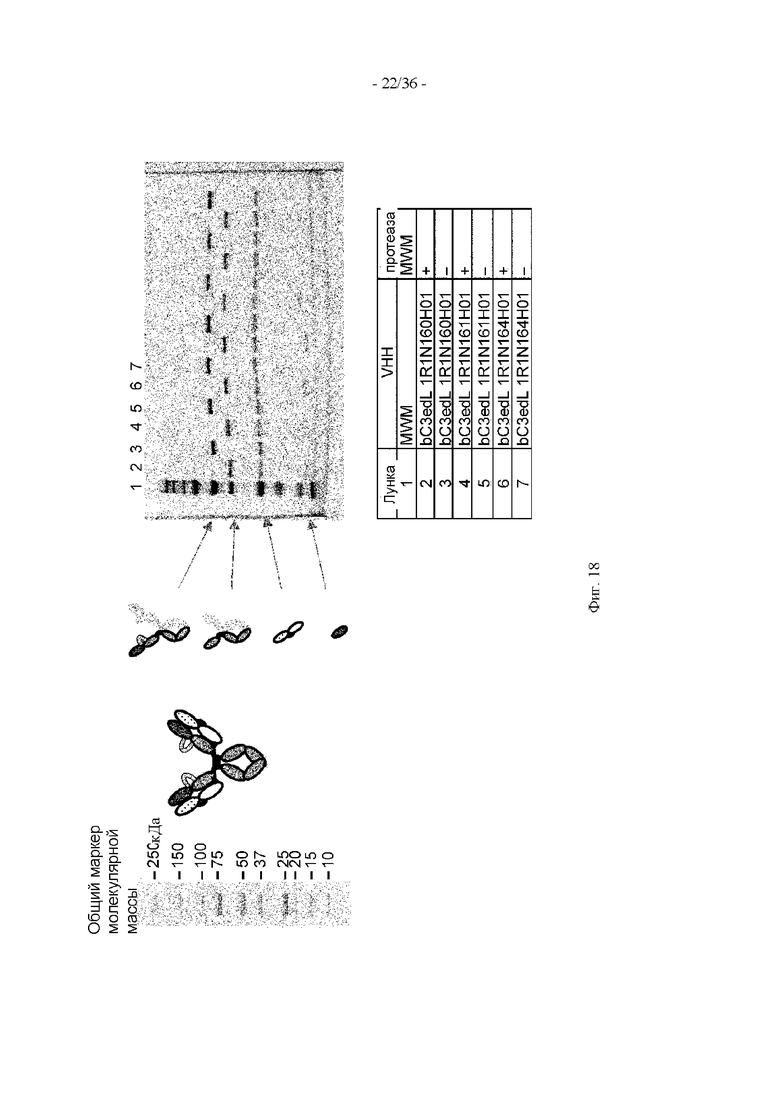
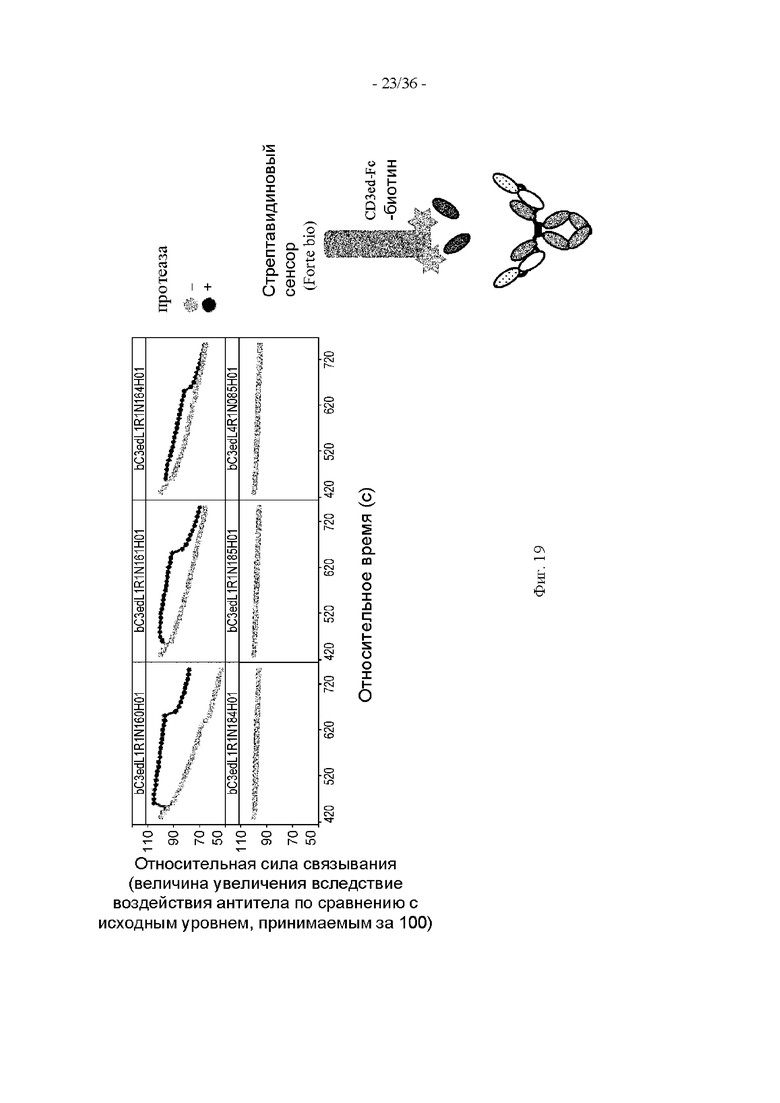
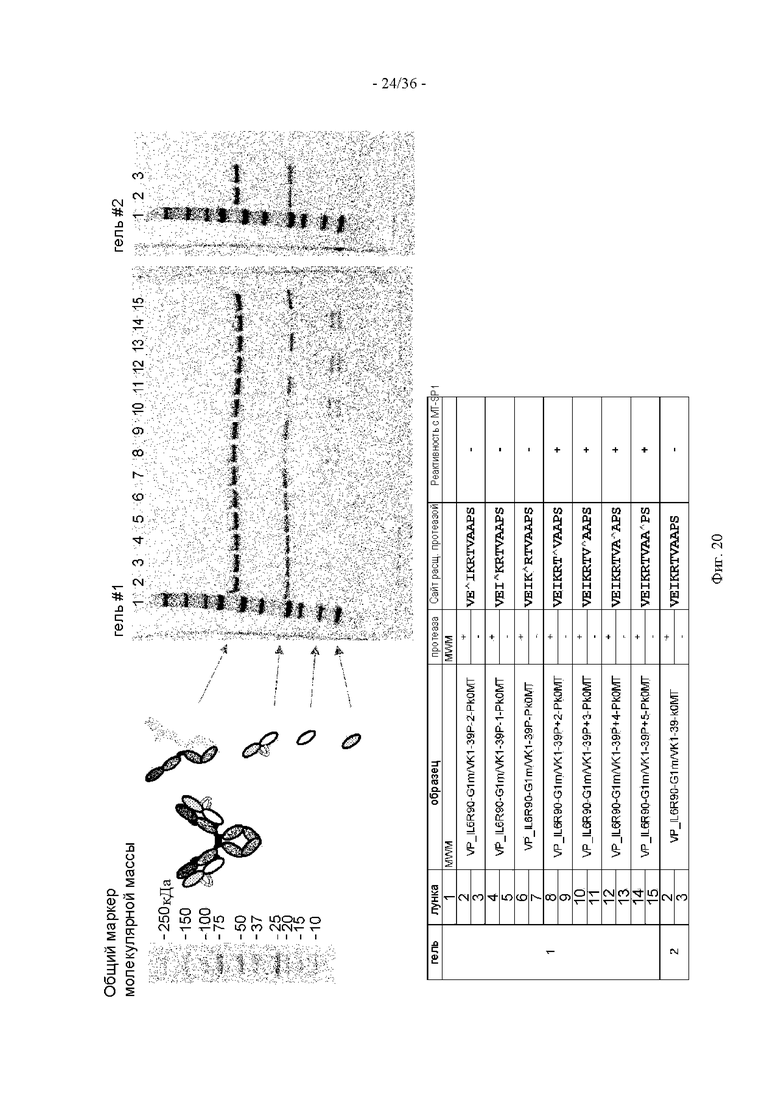
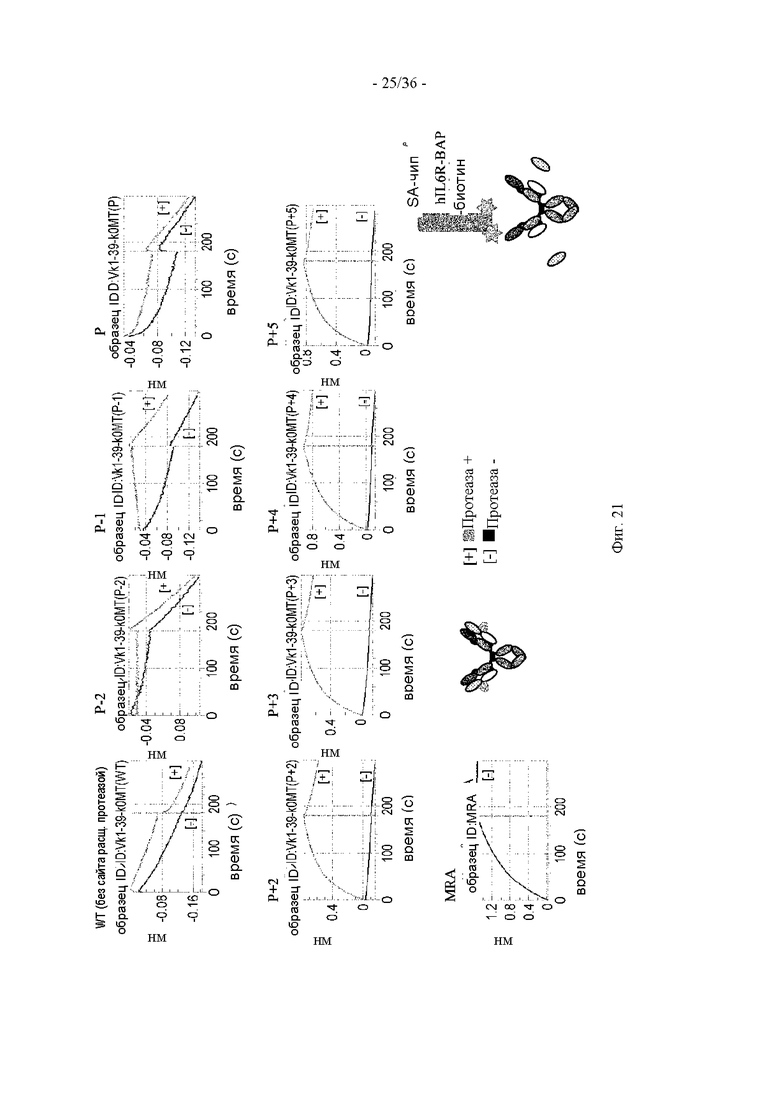
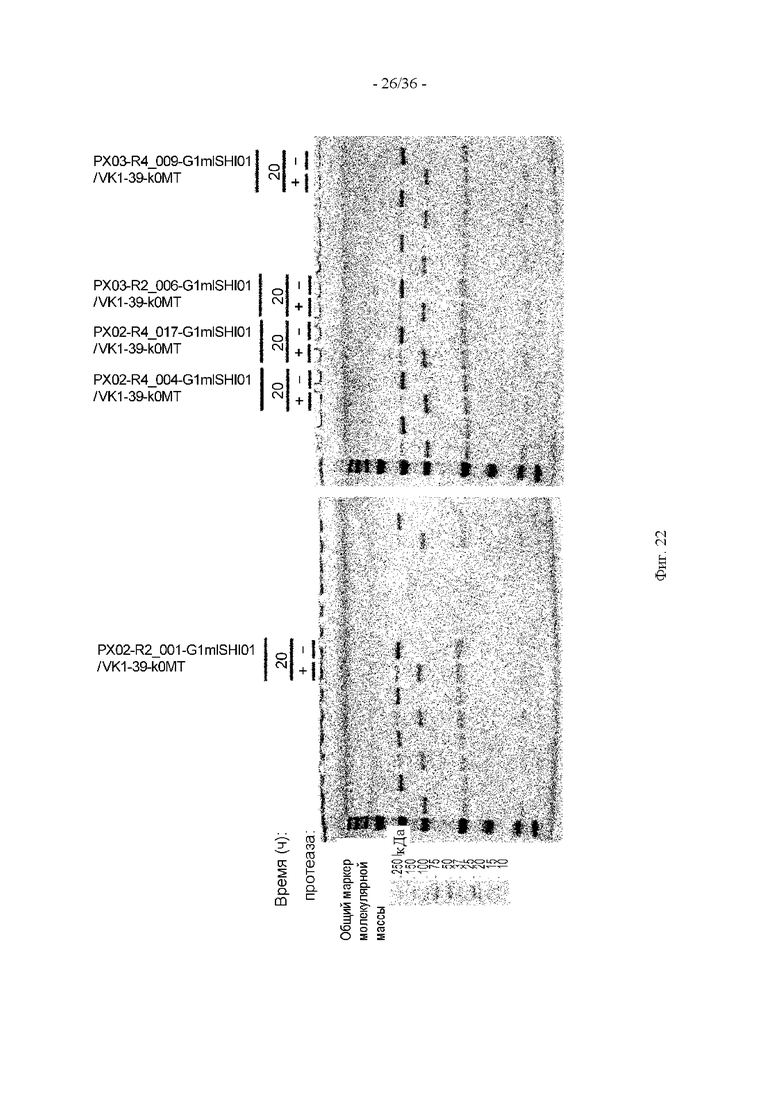
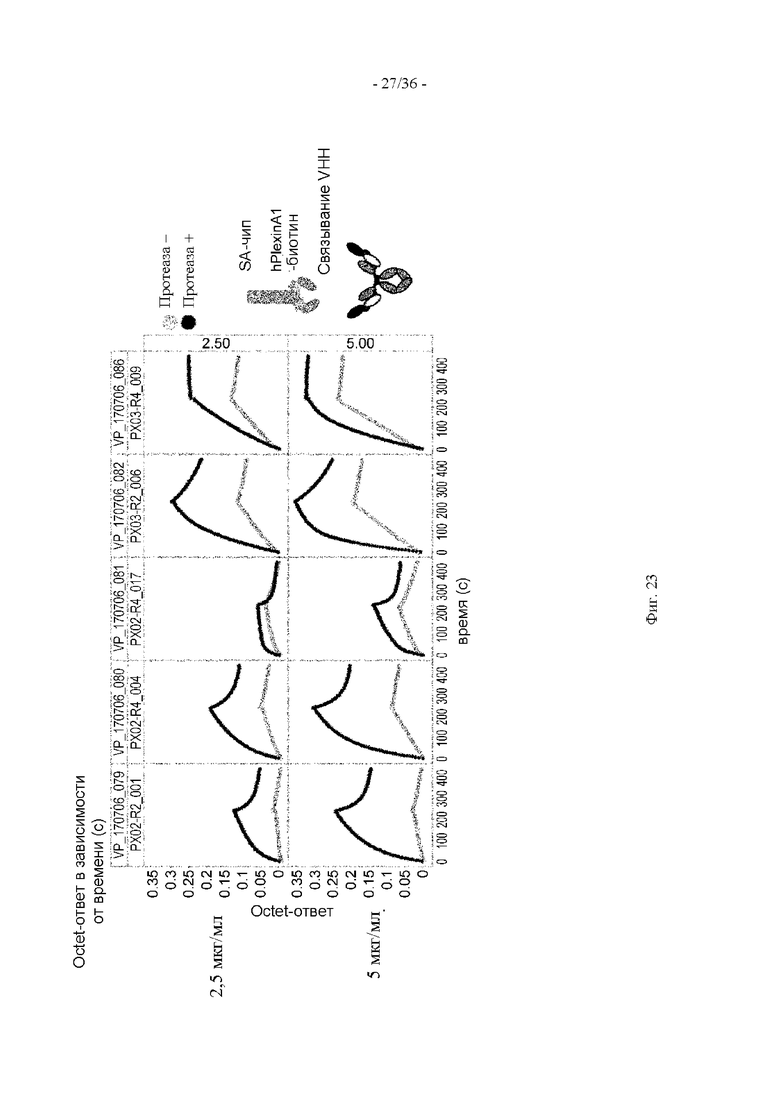
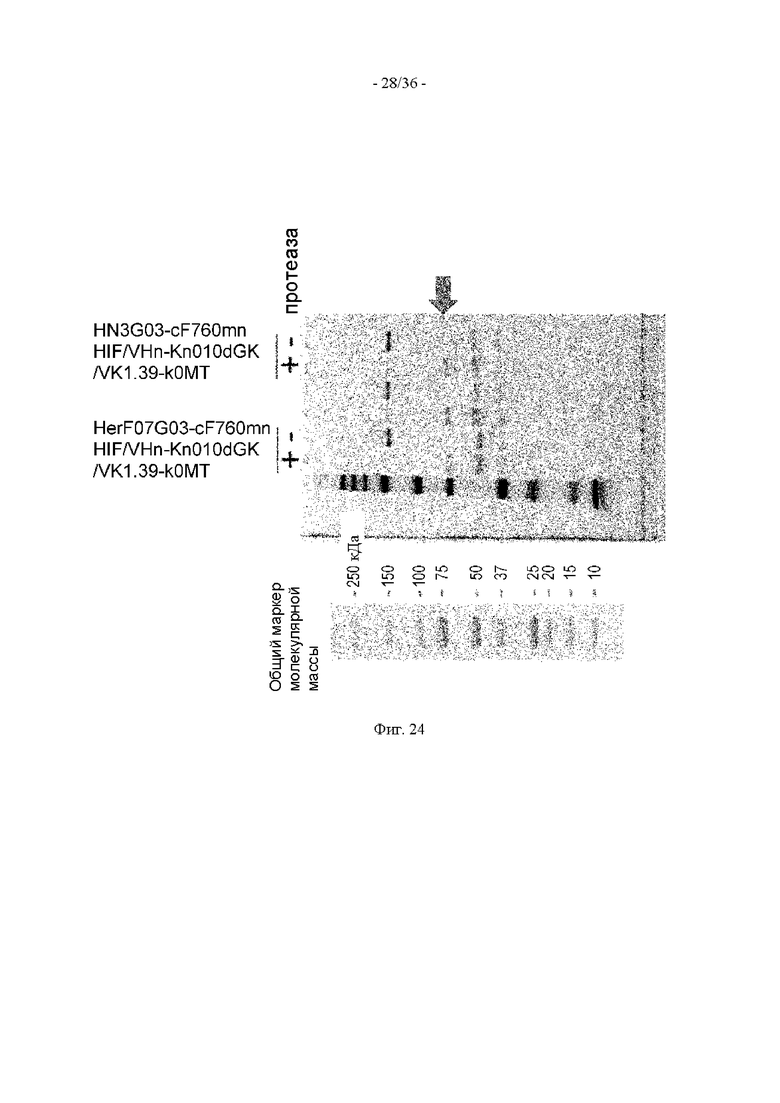
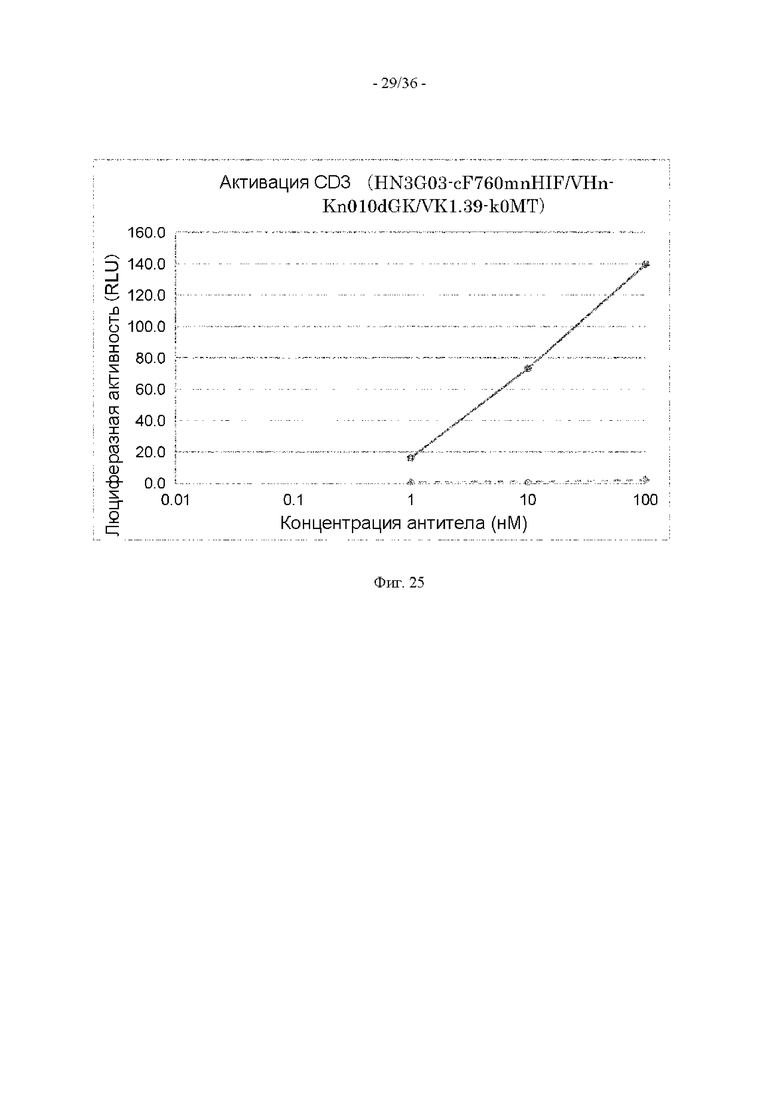
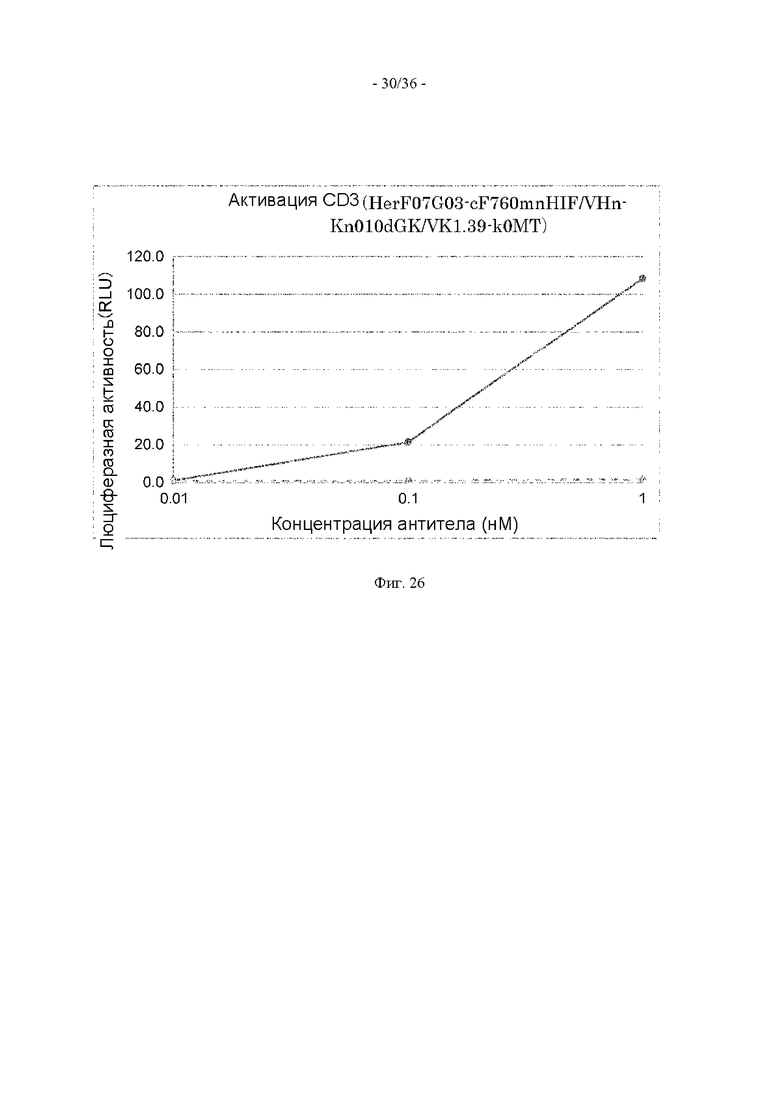

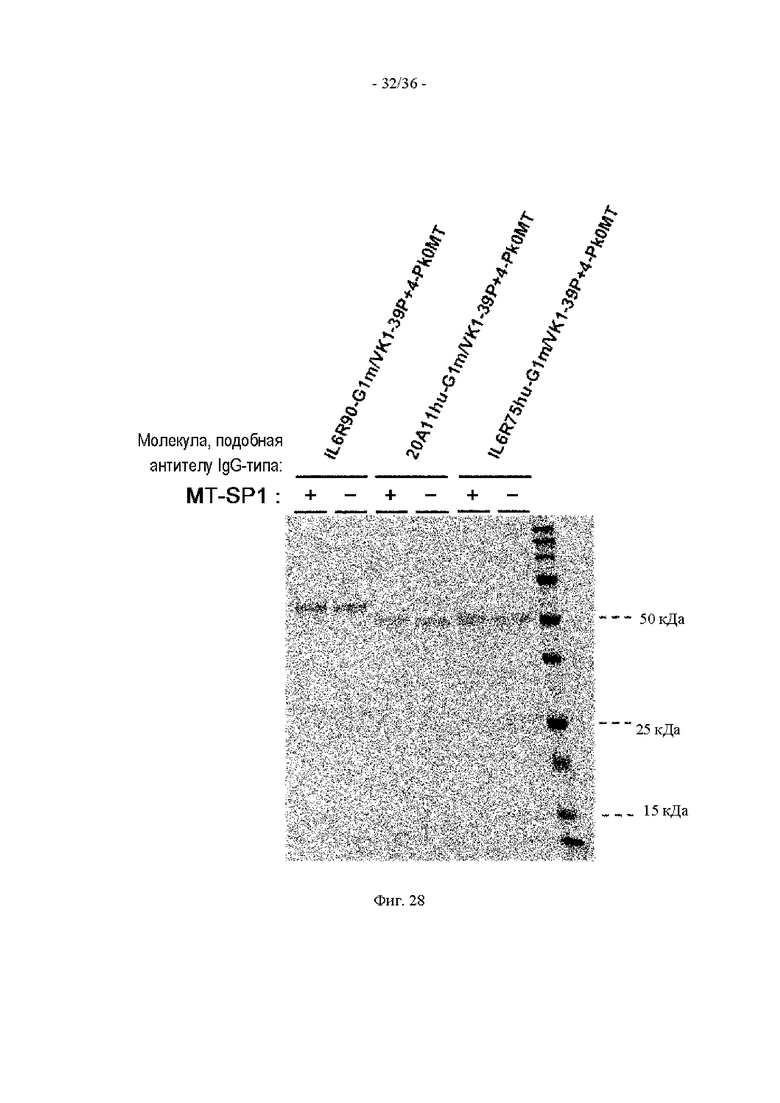
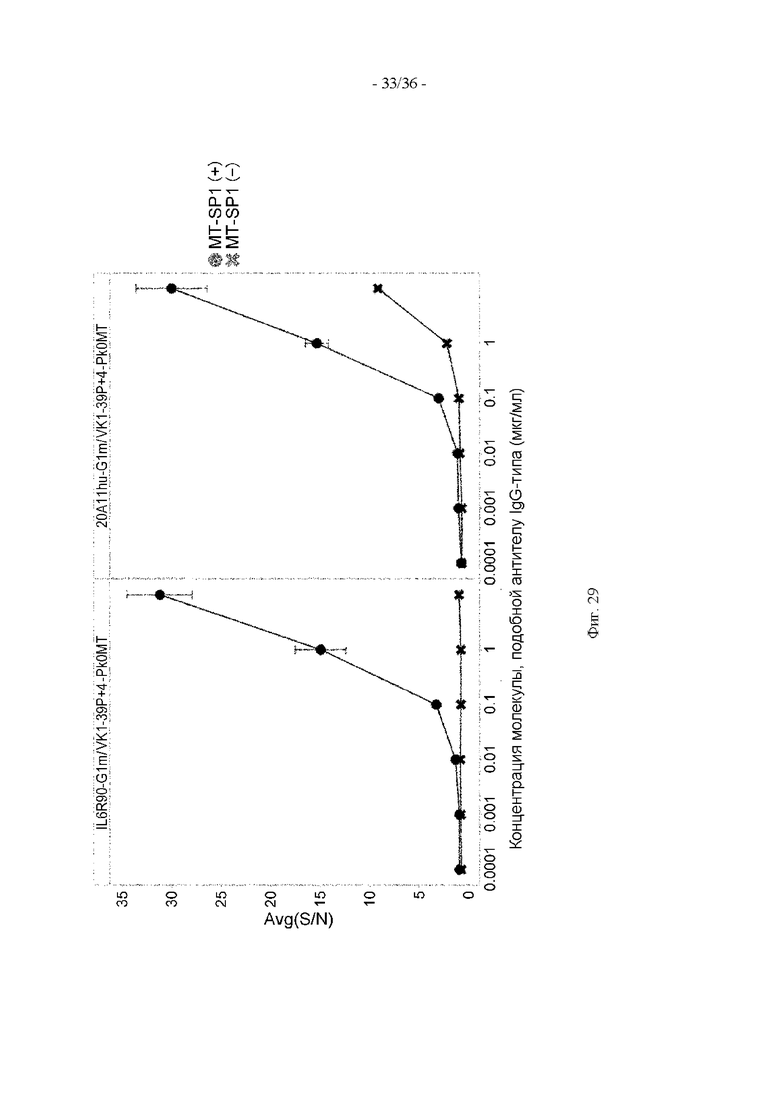

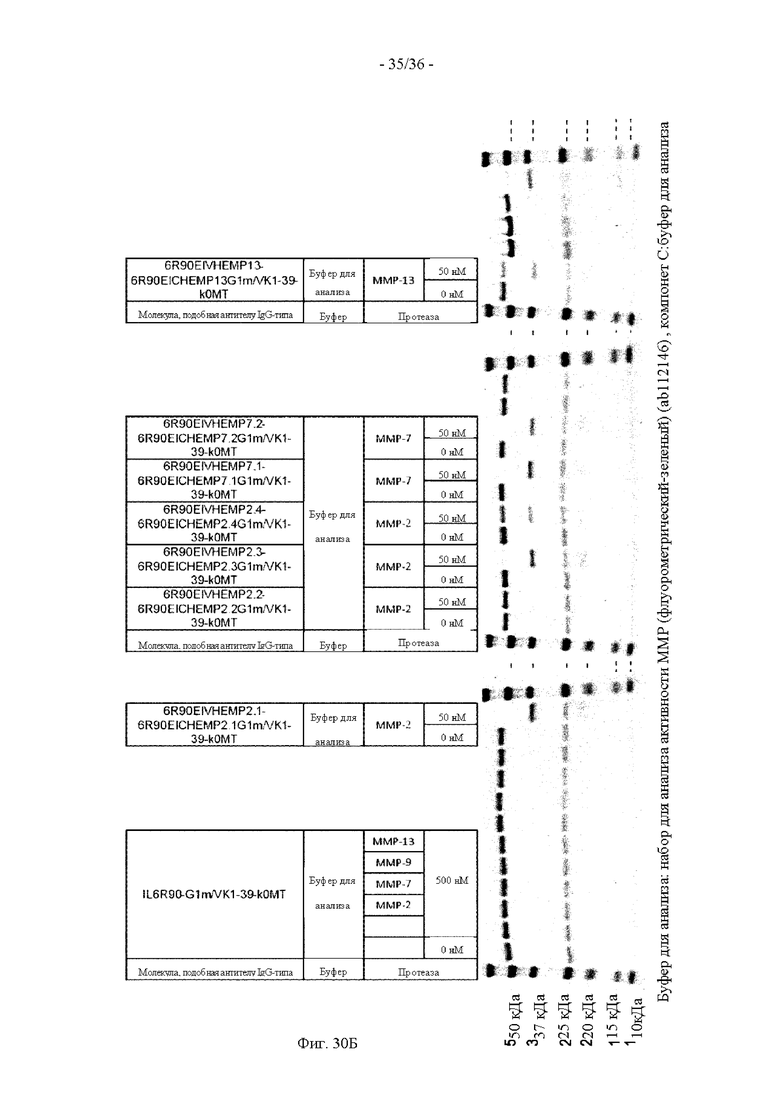
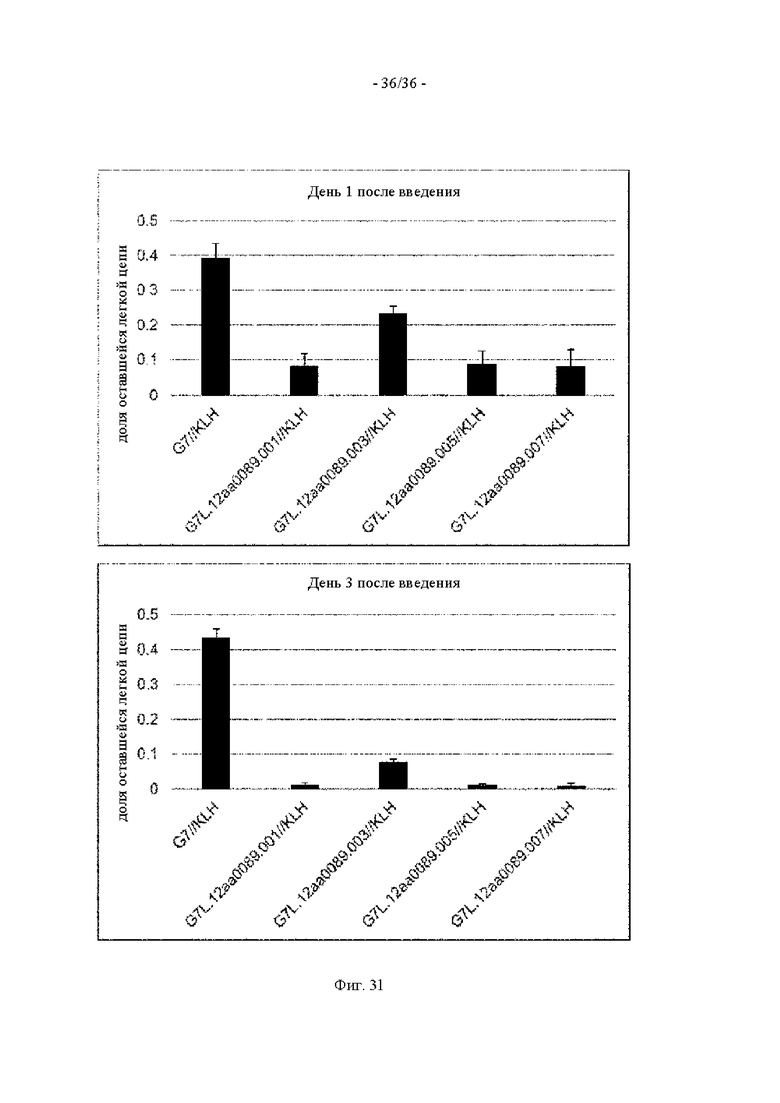
Изобретение относится к области биохимии, в частности к полипептиду, специфичному для ткани-мишени и содержащему антигенсвязывающий домен и транспортирующий фрагмент, в котором транспортирующий фрагмент имеет ингибирующий домен, ингибирующий антигенсвязывающую активность антигенсвязывающего домена, в котором антигенсвязывающий домен имеет более короткое время полужизни в крови, чем транспортирующий фрагмент. Раскрыта фармацевтическая композиция, содержащая указанный полипептид. Также раскрыт способ получения указанного полипептида. Изобретение позволяет эффективно получать указанные полипептиды. 3 н. и 11 з.п. ф-лы, 23 табл., 31 ил., 16 пр.
1. Полипептид, специфичный для ткани-мишени, содержащий антигенсвязывающий домен и транспортирующий фрагмент, в котором транспортирующий фрагмент имеет ингибирующий домен, ингибирующий антигенсвязывающую активность антигенсвязывающего домена, в котором антигенсвязывающий домен имеет более короткое время полужизни в крови, чем транспортирующий фрагмент, и где полипептид имеет расщепляемую протеазой последовательность, которая содержит одну или множество последовательностей, выбранную(ых) из последовательностей SEQ ID NO: 171, 168-170, 172-177 и 208-815.
2. Полипептид по п. 1, в котором ингибирование антигенсвязывающей активности антигенсвязывающего домена ингибирующим доменом в состоянии, в котором расщепляемая протеазой последовательность расщеплена протеазой, является более слабым, чем ингибирование антигенсвязывающей активности антигенсвязывающего домена ингибирующим доменом в состоянии, в котором расщепляемая протеазой последовательность нерасщеплена.
3. Полипептид по п. 1 или 2, в котором антигенсвязывающий домен обладает способностью высвобождаться из полипептида, и в котором антигенсвязывающий домен имеет более высокую антигенсвязывающую активность в высвободившемся из полипептида состоянии по сравнению с антигенсвязывающей активностью в невысвободившемся из полипептида состоянии.
4. Полипептид по одному из пп. 1-3, в котором антигенсвязывающая активность антигенсвязывающего домена ингибируется путем ассоциации ингибирующего домена транспортирующего фрагмента с антигенсвязывающим доменом.
5. Полипептид по п. 3 или 4, в котором расщепляемая протеазой последовательность расщепляется протеазой таким образом, что антигенсвязывающий домен приобретает способность высвобождаться из полипептида и/или таким образом, что ассоциация ингибирующего домена транспортирующего фрагмента с антигенсвязывающим доменом аннулируется.
6. Полипептид по одному из пп. 1-5, где протеаза представляет собой специфическую для раковой ткани протеазу или специфическую для воспалительной ткани протеазу.
7. Полипептид по одному из пп. 1-6, в котором антигенсвязывающий домен содержит однодоменное антитело или представляет собой однодоменное антитело и в котором ингибирующий домен транспортирующего фрагмента ингибирует антигенсвязывающую активность однодоменного антитела.
8. Полипептид по одному из пп. 1-7, в котором антигенсвязывающий домен содержит однодоменное антитело, в котором ингибирующий домен транспортирующего фрагмента представляет собой VHH, VH антитела или VL антитела и в котором антигенсвязывающая активность однодоменного антитела ингибируется посредством VHH, VH антитела или VL антитела.
9. Полипептид по одному из пп. 1-8, в котором транспортирующий фрагмент содержит константную область антитела.
10. Полипептид по п. 9, в котором N-конец константной области антитела транспортирующего фрагмента и C-конец антигенсвязывающего домена слиты через линкер или без линкера и в котором расщепляемая протеазой последовательность локализована вблизи границы между антигенсвязывающим доменом и константной областью антитела.
11. Полипептид по п. 9 или 10, в котором константная область антитела полипептида представляет собой константную область антитела IgG-типа.
12. Полипептид по одному из пп. 1-11, в котором полипептид представляет собой молекулу, подобную антителу IgG-типа.
13. Фармацевтическая композиция для лечения заболевания, вызванного тканью-мишенью, содержащая эффективное количество полипептида по одному из пп. 1-12 и фармацевтически приемлемый носитель, где заболевание представляет собой рак или воспалительное заболевание, или расстройство.
14. Способ получения полипептида по одному из пп. 1-12, включающий стадии:
а) трансформации клетки-хозяина вектором экспрессии, в который был инсертицирован полинуклеотид, кодирующий полипептид по одному из пп. 1-12.
б) культивирование трансформированной клетки, и
в) выделение целевого полипептида.
| WO 2012025525 A1 (ROCHE G | |||
| [CH]), 01.03.2012 | |||
| TURK B.E | |||
| and et al., Determination of protease cleavage site motifs using mixture-based oriented peptide libraries, 2001 Nature Publishing Group | |||
| RU 2015101803 A, 20.08.2016. |
Авторы
Даты
2024-03-15—Публикация
2018-11-28—Подача