Область техники, к которой относится изобретение
Изобретение относится к области конструирования библиотеки из ДНК плазмы, более конкретно, относится к композиции двухсторонних библиотечных меток и ее применению в платформе секвенирования MGI.
Предшествующий уровень техники
Для того, чтобы реализовать секвенирование большего количества образцов в процессе секвенирования на высокопроизводительном секвенаторе MGI, каждый образец необходимо пометить отличающимся индексом и отсеквенировать, а затем данные разделяются посредством биоинформатического анализа в зависимости от информации с индексов. Однако в настоящее время в платформе секвенирования MGI в основном используются односторонние библиотечные метки. Поскольку односторонние библиотечные метки (индексы) имеют естественные дефекты, это легко приводит к перекрестным помехам в данных от разных образцов. Из-за загрязнения адаптеров или праймеров при синтезе, ходе эксперимента или секвенирования проблемы перекрестных помех неизбежны. Следовательно, необходимо решить проблему низкочастотных взаимных перекрестных помех между разными образцами. Наилучший способ - использовать двухсторонние библиотечные метки, которые могут эффективно устранить проблемы взаимных перекрестных помех между разными образцами.
Однако по сравнению с односторонними библиотечными метками применение двухсторонних библиотечных меток, независимо от того, может ли секвенатор точно считывать библиотечные двухсторонние метки или нет, серьезно повлияет на эффективное разделение данных секвенирования посредством биоинформатического анализа. Если возникнет проблема со считыванием последовательностей двухсторонних библиотечных меток, скорость разделения данных секвенирования будет снижена, что приведет к увеличению стоимости секвенирования.
Следовательно, проблемой, требующей решения, является каким образом применить двухсторонние метки для маркировки объединенных библиотек, чтобы не только уменьшить проблемы перекрестных помех образцов, но и улучшить скорость разделения данных секвенирования.
Сущность
Основная цель изобретения заключается в том, чтобы обеспечить композицию двухсторонних библиотечных меток и ее применение в платформе секвенирования MGI, чтобы решить проблему с перекрестными помехами образцов при применении односторонних библиотечных меток в платформе секвенирования MGI.
Для достижения этой цели в соответствии с аспектом изобретения предлагается композиция двухсторонних библиотечных меток, где данная композиция двухсторонних библиотечных меток включает множество 5'-концевых библиотечных меток и множество 3'-концевых библиотечных меток, длины 5'-концевых библиотечных меток одинаковы, длины 3'-концевых библиотечных меток одинаковы, и частоты каждого основания в одном и том же положении также одинаковы.
Кроме того, длины 5'-концевых библиотечных меток одинаковы с длинами 3'-концевых библиотечных меток, предпочтительно любые фиксированные длины составляют от 6 до 10 п.о.; желательно, чтобы в составе двухсторонних библиотечных меток было не менее 3 различий в основаниях между любыми двумя библиотечными метками, количество непрерывных одинаковых оснований в любой библиотечной метке не превышало 3, а содержание GC во всех библиотечных метках было равно 40-60%. Предпочтительно, композиция двухсторонних библиотечных меток содержит комбинацию из сбалансированных по 4 двухсторонних библиотечных меток или комбинацию из сбалансированных по 8 двухсторонних библиотечных меток, где комбинация из сбалансированных по 4 двухсторонних библиотечных меток содержит 4n 5'-концевых библиотечных меток и 4n 3'-концевых библиотечных меток, а комбинация сбалансированных по 8 двухсторонних библиотечных меток включает 8n 5'-концевых библиотечных меток и 8n 3'-концевых библиотечных меток, где n представляет собой целое число, большее или равное 1.
Кроме того, в комбинации из сбалансированных по 4 двухсторонних библиотечных меток, 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 96 групп, показанных в таблице 1, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких групп из 96 групп, показанных в таблице 1, которые отличаются от 5'-концевых библиотечных меток.
Кроме того, в комбинации из сбалансированных по 8 двухсторонних библиотечных меток, 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких групп из 48 групп, показанных в Таблице 2, которые отличаются от 5'-концевых библиотечных меток.
Согласно второму аспекту изобретения, в изобретении предлагается композиция праймеров для амплификации с двусторонними библиотечными метками для платформы секвенирования MGI, и композиция праймеров для амплификации включает множество пар праймеров для амплификации с двусторонними библиотечными метками, каждая пара праймеров для амплификации содержит 5’ концевую библиотечную метку и 3’ концевую библиотечную метку, и длины множества 5'-концевых библиотечных меток в парах праймеров для амплификации одинаковы, и длины множества 3'-концевых библиотечных меток в парах праймеров для амплификации одинаковы, и частоты каждого основания в одном и том же положении также одинаковы.
Кроме того, длины множества 5'-конца библиотечных меток в парах праймеров для амплификации одинаковы с длинами множества 3'-концевых библиотечных меток в парах праймеров для амплификации; предпочтительно, чтобы длины множества 5'-концевых библиотечных меток и длины множества 3'-концевых библиотечных меток были любой фиксированной длины между 6~10 п.о.; желательно, чтобы в составе было не менее 3 отличий оснований между любыми двумя библиотечными метками, а количество непрерывных одинаковых оснований в любой библиотечной метке не превышало 3; желательно, чтобы содержание GC во всех библиотечных метках составляло 40-60%; предпочтительно композиция содержит комбинацию 4n пар праймеров для амплификации сбалансированных по 4 или комбинацию 8n пар праймеров для амплификации сбалансированных по 8, где n представляет собой целое число, большее или равное 1.
Кроме того, в комбинации 4n пар праймеров для амплификации сбалансированных по 4 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 96 групп, показанных в таблице 1, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких из 96 групп, показанных в таблице 1, которые отличаются от 5'-концевых библиотечных меток; предпочтительно, в комбинации 8n пар праймеров для амплификации сбалансированных по 8, 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, которые отличаются от 5'-концевых библиотечных меток.
Кроме того, каждая пара праймеров для амплификации дополнительно содержит 5'-концевую универсальную последовательность для амплификации и 3'-концевую универсальную последовательность для амплификации, причем 5'-концевая универсальная последовательность для амплификации содержит универсальную вышележащую последовательность 5'-концевой библиотечной метки и универсальную нижележащую последовательность 5'-концевой библиотечной метки, а 3'-концевая универсальная последовательность для амплификации содержит универсальную вышележащую последовательность 3'-концевой библиотечной метки и универсальную нижележащую последовательность 3'-концевой библиотечной метки; предпочтительно, универсальная вышележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 793, а универсальная нижележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 794; универсальная вышележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 795, а универсальная нижележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 796; или
универсальная вышележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 793, а универсальная нижележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 797; универсальная вышележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 795, а универсальная нижележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 798.
В соответствии с третьим аспектом изобретения в изобретении также предлагается набор для создания библиотеки для секвенирования, который включает любую из указанных выше композиций праймеров для амплификации.
Кроме того, набор дополнительно содержит «пузырьковые» адаптеры, при этом «пузырьковые» адаптеры содержат первую последовательность адаптера и вторую последовательность адаптера, первая последовательность адаптера представляет собой SEQ ID NO: 769, а вторая последовательность адаптера представляет собой SEQ ID NO: 770, или первая последовательность адаптера представляет собой SEQ ID NO: 773, а вторая последовательность адаптера представляет собой SEQ ID NO: 774.
В соответствии с четвертым аспектом изобретения в изобретении предлагается способ конструирования библиотеки для секвенирования на основе платформы секвенирования MGI, включающий применение любого из указанных наборов для конструирования.
В соответствии с пятым аспектом изобретения в изобретении предлагается библиотека для секвенирования, включающая указанную выше комбинацию двухсторонних библиотечных меток или любую из указанных выше комбинаций праймеров для амплификации.
Путем введения двухсторонних библиотечных меток и оптимизации комбинации двухсторонних библиотечных меток, при применении этих двухсторонних библиотечных меток для разделения данных секвенирования можно решить проблемы перекрестных помех, вызванные синтезом, экспериментальным процессом и машинным секвенированием, и результаты будут точнее. Кроме того, контролируя одинаковые длины 5'-концевых библиотечных меток и длины 3'-концевых библиотечных меток, и ограничивая частоты каждого основания в одном и том же положении, основания двухсторонних меток в композиции имеют одинаковую частоту, поэтому при синтезе адаптеров или праймеров для амплификации двухсторонних меток композиции можно получить несколько библиотек с хорошо сбалансированными по основаниям метками с двух концов. Когда эти несколько библиотек объединены и секвенированы машинным образом, последовательности двусторонних меток могут быть точно прочитаны, а данные секвенирования могут быть эффективно разделены.
Краткое описание чертежей
Сопроводительные чертежи, которые составляют часть этой заявки, предназначены для дальнейшего понимания настоящего изобретения. Иллюстративные воплощения настоящего изобретения и их описание предназначены для объяснения настоящего изобретения, а не для его ограничения. На чертежах:
На Фигуре 1A, Фигуре 1B и Фигуре 1C показаны преимущества платформы секвенирования MGI с использованием двухсторонних меток по сравнению с односторонними метками для устранения проблем с перекрестными помехами;
На фигурах 2А и 2В показаны две формы адаптера для MGI с односторонней меткой;
На фигурах 3А и 3В показаны две формы адаптера для MGI с двухсторонними метками;
На Фигуре 4 показан процесс построения библиотеки с использованием двух двухсторонних меток на основе платформы MGI;
На Фигуре 5 показано, что решения, в которых используются двухсторонние метки по настоящему изобретению, совместимы с решениями, в которых используются односторонние метки;
На Фигуре 6 показан адаптер, в котором праймеры для амплификации двухсторонних меток и праймеры для амплификации односторонних меток совместимы;
Фигуры 7А и 7В показывают сбалансированные по основаниям последовательности, относящиеся к типу сбалансированных по 4 и сбалансированных по 8;
На Фигуре 8 показано сравнение баланса оснований между метками, сбалансированными по 4 и сбалансированными по 8;
На Фигуре 9 показано сравнение выходных данных двух способов построения библиотек;
На фигуре 10 показана разница в разделении данных секвенирования между метками, сбалансированными по 4 и сбалансированными по 8, в процессах секвенирования 12 объединенных образцов.
Подробное описание воплощений
Следует отметить, что воплощения в настоящей заявке и признаки в воплощениях могут быть объединены друг с другом без конфликта. Настоящее изобретение будет подробно описано ниже со ссылкой на воплощения.
Толкование конкретных терминов:
Адаптеры двусторонних меток: для высокопроизводительного секвенирования требуется универсальный адаптер секвенирования для присоединения к концам каждого фрагмента. Каждая некомплементарная область адаптера имеет вариабельную последовательность, которая представляет собой последовательность метки, которая используется для разделения данных во время секвенирования.
Баланс оснований в последовательностях меток: последовательность ДНК состоит из четырех оснований, а именно A, T, G и C. Для эффективного считывания во время секвенирования набор последовательностей меток объединяется, с тем чтобы гарантировать, что соотношение оснований в каждом положении в последовательности меток одинаково.
Как упоминалось в «предшествующем уровне техники», при использовании односторонних меток для создания библиотек для высокопроизводительного секвенирования MGI возникают некоторые проблемы с перекрестными помехами между образцами (Это явление также существует на платформе секвенирования Illumina. Хотя платформа MGI сильно отличается от платформы Illumina, процесс синтеза последовательности адаптера, создания библиотеки и гибридизации неизбежно вызывает проблемы с перекрестными помехами между образцами). Как показано на Фигуре 1А, если в экспериментальном процессе возникает 1 % взаимных перекрестных помех, будь то синтез адаптера, создание библиотеки, гибридизационный захват или машинное секвенирование, будут те же проблемы с перекрестными помехами. Лучший способ решить проблемы с перекрестными помехами между образцами - это ввести двухсторонние метки в процессе конструирования библиотеки. Как показано на Фигуре 1B, проблемы с перекрестными помехами можно решить только путем введения двухсторонних меток при максимально возможном контроле экспериментальных процессов. Как показано на Фигуре 1C, двухсторонние метки уменьшат проблемы перекрестных помех в 100 раз (от 1% до 0,01%) относительно односторонних меток.
Чтобы решить проблемы с перекрестными помехами в образцах для платформы секвенирования MGI, в этом изобретении также делается попытка заменить односторонние метки двусторонними метками. Идея и процесс исследований и разработок заключался в следующем.
Пузырьковые адапторы используются при конструировании библиотеки MGI. В отличие от адаптеров Y-типа у Illumina, односторонние метки MGI можно включить в адаптеры (как показано на Фигуре 2B) или использовать отдельно (Фигура 2A); в то время как последовательности двухсторонней метки не могут быть объединены с последовательностью переднего конца (как показано на Фигуре 3B, поскольку область переднего конца составляет всего 7 п.о. если последовательность метки включена на переднем конце, структура «пузырька» будет длиннее, и стабильность этой структуры крайне плоха, а эффективность очень низка. И эффективность при осуществлении будет ниже чем у усеченной структуры, где праймеры последовательности метки и универсальный адаптер разделены). И универсальный адаптер и праймеры для амплификации двухсторонних меток можно использовать отдельно (как показано на Фигуре 3A). Двусторонние метки были соединены в соответствии со структурой, показанной на Фигуре 3А, и авторы изобретения обнаружили, что большая «везикула» в середине «пузырькового» адаптера будет влиять на стабильность вторичной структуры стадии отжига и влиять на лигирование адаптеров (средняя эффективность составляет 20%-40%). «Пузырьковый» адаптер MGI отличается от Y-образного адаптера Illumina, в котором двухсторонние метки могут быть объединены вместе.
Дальнейшие исследования показали, что, когда неспаренные основания в средней области «пузырьковых» адаптеров MGI могут составлять 30 ± 5 п.о., а спаренные основания - 20 ± 2 п.о., легче сформировать стабильное лигирование при отжиге, повышая эффективность лигирования, как показано в Решении 1 на Фигуре 4. Когда неспаренные основания в средней области также могут иметь размер 45 ± 5 п.о., а спаренные основания - 25 ± 2 п.о., легче сформировать стабильное лигирование отжига, повышая эффективность лигирования, как показано в Решении 2 на Фигуре 4. Кроме того, авторы изобретения обнаружили, что по сравнению с Решением 2 Решение 1 имеет следующие преимущества: во-первых, когда «пузырьковая» область составляет 30±5 п.о., адаптеры стабильно отжигаются, комплементарная область является короткой и стабильность полезна для лигирования. Во-вторых, при совместимости с ампликонами с односторонними метками, ампликоны можно переключать между односторонними и двухсторонними метками, как показано на Фигуре 5. Оно также совместимо с адаптерами с односторонними метками, как показано на Фигуре 6.
Изобретатели также обнаружили, что, хотя Решение 2 имеет много преимуществ по сравнению с Решением 1, оба решения могут работать, если нужно получить библиотеку секвенирования на платформе секвенирования MGI с двусторонними метками. Если созданная библиотека с двусторонними метками используется для машинного секвенирования, а данные секвенирования разделяются после секвенирования, авторы обнаружили, что требования к балансу оснований в адаптерах с двусторонними метками для MGI во время секвенирования являются более строгими, чем у адаптеров с односторонними метками, и данные секвенирования могут быть разделены только в том случае, если последовательности меток на двух концах являются правильными, как показано на Фигуре 1B. То есть, хотя двухсторонние метки решают проблемы перекрестных помех между образцами, требования к балансу оснований для машинного секвенирования чрезвычайно строгие, а плохой баланс оснований серьезно повлияет на точность считывания данных секвенирования, что, в свою очередь, повлияет на эффективное разделение данных секвенирования.
Для более точного разделения данных секвенирования, взяв в качестве примера количество оснований в двухсторонних метках, равное 10, авторы изобретения оптимизировали баланс оснований в двухсторонних метках в соответствии со следующими правилами: 1) между каждой последовательностью меток есть различия в 3 основаниях; 2) содержание GC каждой последовательности составляет 0,4-0,6; 3) количество непрерывных одинаковых оснований не может превышать 3. В соответствии с этими правилами была оценена вторичная структура каждой последовательности выбранной метки, чтобы увидеть, образуется ли вторичная структура, например, шпилечные складки, между последовательностью метки и универсальным праймером на 3'-конце праймера для амплификации, что уменьшит эффективность амплификации, есть ли влияние на баланс каждого основания метки в объединенных библиотеках образцов, есть ли дополнительное влияние на точность считывания последовательности метки и, следовательно, есть ли снижение точности разделения данных секвенирования.
В соответствии с приведенными выше оптимизированными правилами скрининга настоящее изобретение оптимизирует 384 типа последовательностей меток, сбалансированных по 4, и 384 типа последовательностей меток, сбалансированных по 8. Метки, сбалансированные по 4, относятся к группе последовательностей меток, показанных на Фигуре 7А (первые 1-4 метки показаны в таблице 4). Группа из 4 последовательностей меток относится к основаниям A, T, G или C, встречающимся один раз в каждом положении с 1-й по 10-е положение каждой метки. Аналогично, метки, сбалансированные по 8, относятся к группе из 8 последовательностей меток, как показано на Фигуре 7В (первые 1-8 меток показаны в таблице 5). Группа из 8 последовательностей меток относится к основанию A, T, G или C, встречающемуся дважды в каждом положении с 1-й по 10-е положение каждой метки.
Согласно многочисленным тестам изобретения, группа из меток, сбалансированных по 4, является наименьшей единицей баланса и лучшей комбинацией. Комбинации меток, сбалансированных по 4, могут быть объединены в 4, 8, 12 и 16 комбинаций, которые являются 4-кратно сбалансированными, а комбинации меток, сбалансированных по 8, должны быть объединены в 8 и 16 комбинаций, которые являются 8-кратно сбалансированными. Как показано на Фигуре 8 (последовательность метки в комбинации меток, сбалансированных по 4, слева соответствует комбинации меток библиотеки, содержащейся в первых 4 наборах из числа наборов праймеров для амплификации в таблице 1, и библиотечная метка комбинации меток, сбалансированных по 8, справа соответствует комбинации библиотечных меток, содержащейся в первых двух наборах наборов праймеров для амплификации в таблице 2), когда библиотеки из меток, сбалансированных по 4, объединены и секвенированы на устройстве, основания сбалансированы, и пропорция каждого основания составляет 25%. А при использовании комбинаций меток, сбалансированных по 8, доля каждого основания составляет 0-50%. При объединении 8 кратных, например, 8 или 16 образцов, пропорция каждого основания после комбинации библиотечных меток может быть сбалансирована, и каждая составляет 25%. Когда на устройстве объединены и секвенированы 12 образцов, доля каждого основания в комбинации из меток, сбалансированных по 8, составляет от 16,7% до 33,3%.
Кроме того, баланс нецелой кратности 4 меток также лучше, чем комбинация меток, сбалансированных по 8, и применение меток, сбалансированных по 4, более удобно. По мере того, как производительность секвенирования секвенатора MGI становится все выше и выше, оптимизированные 384 типа комбинаций меток, сбалансированных по 4, в настоящей заявке обеспечивают эффективное секвенирование четырех закрытых библиотек с метками, сбалансированными по 4 (см. Таблицу 1 для комбинаций меток, сбалансированных по 4). Оптимизированные комбинации 384 меток, сбалансированных по 8, также обеспечивают эффективное секвенирование восьми закрытых библиотек с метками, сбалансированными по 8 (см. Таблицу 2 для комбинации меток, сбалансированных по 8).
Предпочтительно, когда две сбалансированные метки используются для формирования двухсторонних праймеров для амплификации, последовательность праймера 1 представляет собой прямое расположение 384 номеров, а праймер 2 представляет собой обратное расположение 384 номеров, что является рекомендуемым расположением настоящего изобретения. В практических приложениях также можно комбинировать и размещать в соответствии с фактическими потребностями. Например, как показано в таблице 1, когда праймер 1 выбран в любой из 96 групп, праймер 2 может быть выбран в любой из оставшихся 95 групп. Конечно, если количество объединяемых образцов больше 4, например 8 или 12, количество групп меток праймера 1 просто должно отличаться от числа групп меток праймера 2. Например, праймер 1 выбирают из первых 3 групп, а праймер 2 можно выбирать из любых 3 групп из оставшихся 93 групп. Пока на устройстве объединены и секвенированы 4 кратные образцы, двусторонние библиотечные метки можно выбирать в соответствии с этим правилом.
Когда количество объединенных образцов не является числом кратным 4, 4 образца с большим объемом данных секвенирования должны быть расположены в одном наборе комбинаций сбалансированных меток, а образцы с небольшим объемом данных секвенирования должны быть расположены в другом наборе других комбинаций сбалансированных меток. В этой ситуации комбинации из меток, сбалансированных по 4, имеют очевидные преимущества перед комбинациями из меток, сбалансированных по 8. Комбинации меток, сбалансированных по 4, имеют преимущество перед комбинациями меток, сбалансированных по 8, при целочисленной кратности 4 (4, 12, 20), а комбинация нецелочисленной кратности 4 также лучше, чем комбинация меток, сбалансированных по 8, а баланс лучше, чем у комбинации меток, сбалансированных по 8, когда число объединяемых образцов равно 4n+1 и 4n+2. Таким образом, комбинация из меток, сбалансированных по 4, имеет следующие преимущества: 1) Комбинаций из меток, сбалансированных по 4, в два раза больше, чем из меток, сбалансированных по 8; 2) для трех групп несбалансированных расположений баланс в комбинациях групп 4n+1 и 4n+2 также лучше, чем в комбинации меток, сбалансированных по 8; 3) Когда есть разница в количестве данных секвенирования между образцами, комбинации меток, сбалансированных по 4, лучше располагать близко к балансу, и образцы для большого количества данных секвенирования имеют приоритет в сбалансированной комбинации, и это может быть несбалансированным для образцов для небольшого количества данных секвенирования.
Таблица 1. Группа, сбалансированная по 4
группы
ID
NO:
группы
ID
NO:
Таблица 2. 384 типа последовательностей меток, сбалансированных по 8
груп-пы
ID
NO:
группы
ID
NO:
группы
ID NO:
Скорость разделения данных секвенирования в группах из меток сбалансированных по 4, будет выше, потому что секвенатор более точно считывает основания со сбалансированной композицией, а несбалансированные основания вызовут ошибки чтения и уменьшат скорость разделения данных секвенирования. Когда 12 образцов объединены в равных пропорциях, метки, сбалансированные по 4, и метки, сбалансированные по 8, использовались для создания библиотек для секвенирования. Из результатов, показанных на Фигуре 10, для меток, сбалансированных по 4, 12 образцов имеют почти одинаковое разделение данных секвенирования. Для меток, сбалансированных по 8, некоторые образцы из 12 образцов значительно снизили разделение данных.
На основании результатов вышеуказанных исследований авторы изобретения предложили технические решения изобретения.
В типичном воплощении изобретения предлагается композиция двухсторонних библиотечных меток. Композиция двухсторонних библиотечных меток включает множество 5'-концевых библиотечных меток и множество 3'-концевых библиотечных меток. Все длины 5'-концевых библиотечных одинаковы, длины 3'-концевых библиотечных меток одинаковы, и частоты каждого основания в одном и том же положении в композиции двухсторонних библиотечных меток также одинаковы.
В композиции двухсторонних библиотечных меток, предлагаемых изобретением, за счет контроля того, что длины библиотечных меток для 5'-конца одинаковы, длины библиотечных меток для 3'-конца одинаковы, и частоты каждого основания в одном и том же положении одинаковы, можно получить несколько библиотек с хорошо сбалансированными двусторонними метками. Когда несколько библиотек объединены для секвенирования, последовательность двухсторонних меток может быть прочитана более точно, а данные секвенирования могут быть разделены более эффективно.
Для дальнейшего улучшения баланса оснований и точности считывания библиотечных меток в предпочтительном воплощении длина 5'-концевых библиотечных меток совпадает с длиной 3'-концевых библиотечных меток для, предпочтительно равна любой фиксированной длине между 6-10 п.о. Длина библиотечных меток на обоих концах одинакова, так что при разделении образцов одинаковое количество оснований в библиотечных метках на обоих концах участвует в определении источника образца, поэтому вероятность поддержки, обеспечиваемой библиотеками, с обоих концов одинаковы. Это позволяет избежать того, что один конец библиотечной метки будет длиннее и референсная вероятность поддержки будет выше, а другой конец библиотечной метки будет короче и референсная вероятность поддержки будет ниже, что приведет к результату, который больше полагается на метки для одного конца.
Предпочтительно, чтобы в композиции двухсторонних библиотечных меток было не менее 3 отличий оснований между любыми двумя библиотечными метками, а количество непрерывных одинаковых оснований в любой библиотечной метке не превышало 3, и, желательно, чтобы содержимое GC во всех библиотечных метках составляло 40-60%. Когда библиотечные метки соответствуют вышеуказанным принципам оптимизации оснований и используются вместе, баланс оснований лучше, результаты считывания точнее, и скорость разделения данных также выше.
Предпочтительно, композиция двухсторонних библиотечных меток включает композицию из двухсторонних библиотечных меток, сбалансированных по 4, или композицию из двухсторонних библиотечных меток, сбалансированных по 8, комбинация из двухсторонних библиотечных меток, сбалансированных по 4, включает 4n 5'-концевых библиотечных меток и 4n 3'-концевых библиотечных меток. Комбинация из двухсторонних библиотечных меток, сбалансированных по 8, включает 8n 5'-концевых библиотечных меток и 8n 3'-концевых библиотечных меток для, n - целое число, большее или равное 1.
В предпочтительном воплощении в композиции двухсторонних библиотечных меток, сбалансированных по 4, 5'-концевые библиотечные метки выбирают из любой из 96 групп, показанных в таблице 1, а 3'-концевые библиотечные метки выбирают из любой из 96 групп, показанных в Таблице 1, отличающихся от группы 5'-концевых библиотечных меток.
В предпочтительном воплощении в составе сбалансированных по 8 двухсторонних библиотечных меток 5'-концевые библиотечные метки выбирают из любой из 48 групп, показанных в таблице 2, а 3'-концевые библиотечные метки выбирают из любой из 48 групп, показанных в таблице 2, отличающихся от группы 5'-концевых библиотечных меток.
Во втором типичном воплощении изобретения предлагается композиция праймеров для амплификации с двухсторонними библиотечными метками для платформы секвенирования MGI, где композиция праймеров для амплификации включает множество пар праймеров для амплификации с двухсторонними библиотечными метками, при этом каждая пара праймеров для амплификации включает 5'-концевую библиотечную метку и 3'-концевую библиотечную метку, длины 5'-концевых библиотечных меток одинаковы, и длины 3'-концевых библиотечных меток в парах праймеров для амплификации одинаковы, и частоты каждого основания в одном и том же положении также одинаковы.
За счет контроля одинаковых длин 5'-концевых библиотечных меток и одинаковых длин 3'-концевых библиотечных меток множества пар праймеров для амплификации, а также чтобы частоты каждого основания в одном и том же положении также были одинаковыми, когда двухсторонние метки в композиции праймеров для амплификации используются для маркировки нескольких объединенных образцов для секвенирования, считывание оснований меток сбалансировано, результаты более точны, и данные образцов, разделенных по меткам, также более точны, что улучшает скорость разделения данных секвенирования.
На основании одинаковых длин 5'-концевых библиотечных меток и одинаковых длин 3'-концевых библиотечных меток вышеуказанных объединенных образцов, чтобы дополнительно улучшить баланс оснований и точность считывания библиотечных меток, в предпочтительном воплощении, длины 5'-концевых библиотечных меток и длины 3'-концевых библиотечных меток множества пар праймеров для амплификации одинаковы. Длины библиотечных меток для обоих концов каждой пары праймеров для амплификации одинаковы, так что при разделении образцов одинаковое количество оснований в библиотечных метках на обоих концах участвует в определении источника образца, и вероятность поддержки, предоставляемой библиотеками на обоих концах, одинакова. Это позволяет избежать того, что один конец библиотечной метки будет длиннее и референсная вероятность поддержки выше, а другой конец библиотечной метки будет короче, а референсная вероятность поддержки ниже, что приведет к результату, который больше полагается на метки для одного конца.
Более предпочтительно, длины 5'-концевых библиотечных меток и 3'-концевых библиотечных меток фиксирована и имеет любую длину между 6-10 п.о., кроме того, предпочтительная длина составляет 10 п.о., что обеспечивает большую дискриминацию и большие преимущества, чем другие длины, такие как 6 п.о. или 8 п.о.
Чтобы обеспечить более сбалансированные библиотечные метки, в предпочтительном воплощении в составе праймеров для амплификации имеется по меньшей мере 3 различия в основаниях между любыми двумя библиотечными метками, и количество непрерывных одинаковых оснований в любой из библиотечных меток не превышает 3, а содержание GC в библиотечных метках составляет 40-60%. Когда библиотечные метки соответствуют описанному выше принципу оптимизации оснований и используются совместно, баланс чтения оснований становится лучше, результат более точен, а скорость разделения данных секвенирования также повышается.
В предпочтительном воплощении указанная композиция праймеров для амплификации включает комбинацию 4n пар праймеров для амплификации с метками, сбалансированными по 4, или комбинацию 8n пар праймеров для амплификации с метками, сбалансированными по 8, где n представляет собой целое число, большее или равное 1. Более предпочтительно, в 4n парах праймеров для амплификации с метками, сбалансированными по 4, 5' концевые библиотечные метки выбирают из любой одной или нескольких из 96 групп, показанных в таблице 1, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких из 96 групп, отличных от 5' концевых библиотечных меток, показанных в таблице 1. Количество групп в данном документе определяется в соответствии с фактическими потребностями. Комбинации 96 групп последовательностей меток в таблице 1 обеспечивают более высокую точность считывания, поэтому разделение данных секвенирования является более точным, а скорость разделения также повышается.
В другом предпочтительном воплощении в 8n парах праймеров для амплификации с метками, сбалансированными по 8, 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, которые отличаются от групп 5'-концевых библиотечных меток.
В приведенной выше композиции праймеров для амплификации каждая пара праймеров для амплификации дополнительно включает 5'-концевую универсальную последовательность для амплификации и 3'-концевую универсальную последовательность для амплификации, и 5'-концевая универсальная последовательность для амплификации включает универсальную нижележащую последовательность 5'-концевых библиотечных меток и универсальную вышележащую последовательность 5'-концевых библиотечных меток, 3'-концевая универсальная последовательность для амплификации включает универсальную нижележащую последовательность 3'-концевых библиотечных меток и универсальную вышележащую последовательность для 3'-концевых библиотечных меток. Специфическую последовательность универсальной последовательности для амплификации в каждой паре праймеров для амплификации определяют в соответствии с существующими универсальными последовательностями платформы секвенирования MGI. Комбинация праймеров для амплификации, образованная парами праймеров для амплификации, содержащими вышеуказанные библиотечные метки, может повысить точность считывания библиотечных меток, когда образцы объединены и секвенированы на машине, тем самым повышая точность данных секвенирования каждого образца.
Как упоминалось выше, в конструкции библиотеки может использоваться относительно короткий «пузырьковый» адаптер (то есть количество неспаренных оснований в средней области составляет 30 ± 5 п.о.) или относительно длинный «пузырьковый» адаптер (количество неспаренных оснований в средней области составляет 45 ± 5 п.о.). Соответственно, универсальная последовательность в паре праймеров для амплификации в данном документе также может быть адаптирована к более длинной или более короткой универсальной последовательности для амплификации в зависимости от длины «пузырькового» адаптера.
В предпочтительном воплощении, соответствующем применению более короткого «пузырькового» адаптера, универсальная вышележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 793, а универсальная нижележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ. ID №: 794; универсальная вышележащая последовательность 3' концевой библиотечной метки представляет собой SEQ ID NO: 795, а универсальная нижележащая последовательность 3' концевой библиотечной метки представляет собой SEQ ID NO: 796.
В другом предпочтительном воплощении, соответствующем применению более длинного «пузырькового» адаптера, универсальная вышележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 793, а универсальная нижележащая последовательность 5'-концевой библиотечной метки для представляет собой SEQ. ID №: 797; универсальная вышележащая последовательность 3' концевой библиотечной метки представляет собой SEQ ID NO: 795, а универсальная нижележащая последовательность 3' концевой библиотечной метки представляет собой SEQ ID NO: 798.
В третьем воплощении изобретения также предлагается набор библиотечных конструктов на основе платформы секвенирования MGI, набор включает любую композицию праймеров для амплификации, упомянутую выше. В двухсторонних библиотечных метках в праймерах для амплификации соблюден баланс оснований, поэтому последовательности меток каждого образца после секвенирования могут быть точно прочитаны, а точность разделения данных объединенных образцов повышается.
Для дальнейшего повышения удобства создания библиотеки набор может дополнительно включать «пузырьковый» адаптер платформы секвенирования MGI, «пузырьковый» адаптер включает первую последовательность адаптера и вторую последовательность адаптера, и первая последовательность адаптера представляет собой SEQ ID NO: 769, вторая последовательность адаптера представляет собой SEQ ID NO: 770, или первая последовательность адаптера представляет собой SEQ ID NO: 773, вторая последовательность адаптера представляет собой SEQ ID NO: 774. По сравнению с относительно более длинным «пузырьковым» адаптером, более короткий «пузырьковый» адаптер может не только улучшить стабильность лигирования и повысить эффективность лигирования, но также более совместим с последующими процедурами ПЦР-амплификации после лигирования адаптера.
В четвертом воплощении изобретения предлагается способ создания библиотеки для секвенирования с применением любого из вышеуказанных наборов на основе платформы секвенирования MGI. Когда библиотеки, созданные с вышеуказанными наборами, секвенируются на устройстве, баланс библиотечных меток становится лучше, и скорость разделения данных точности считывания становится выше.
В пятом воплощении изобретения также предлагается библиотека секвенирования. Библиотека секвенирования включает любую композицию праймеров для амплификации или конструируется любым из вышеуказанных способов. Баланс библиотечных меток в библиотеке секвенирования лучше, и точность чтения библиотечных меток после секвенирования выше, и скорость разделения данных выше.
Преимущества изобретения будут дополнительно описаны ниже в воплощении. Следует отметить, что в примерах ниже для создания библиотек используется набор для подготовки библиотек NadPrep™ DNA library prep kit.
Артикул №: 1002212 NadPrep® Plasma Free DNA double-end tag library prep kit (для MGI).
Артикул №: 1003811 User's Guide V1. 0 (Nanodigmbio, Нанкин).
Кратко способ описывается следующим образом.
Фрагмент образца ДНК --- репарация концов и присоединение аденозина к тупым концам фрагмента ДНК --- Лигирование --- Отбор фрагментов --- ПЦР-амплификация --- Очистка библиотеки, количественный анализ и контроль качества --- Секвенирование или целевое секвенирование на платформе MGI.
Следует также отметить, что следующие примеры являются просто иллюстративными и не ограничивают способ изобретения следующими способами.
Пример 1. Решение 1 и Решение 2 для подготовки библиотеки.
Стадии: см. набор для подготовки библиотеки ДНК DNA library prep kit (для MGI) (201909Version2.0)
Различия заключаются в последовательности «пузырькового» адаптера и последовательности праймера для амплификации.
(1) Решение 1:
Последовательность «пузырькового» адаптера:
SEQ ID NO:769 (адаптер 1) и SEQ ID NO:770 (адаптер 2):
SEQ ID NO: 769: (31 п.о.) /phos/agtcggaggccaagcggtcttaggaagacaa;
SEQ ID NO: 770 (40 п.о.): ttgtcttcctaacaggaacgacatggctacgatccgact*t.
SEQ ID NO:771 (праймер для амплификации 1) - SEQ ID NO:772 (праймер для амплификации 2):
SEQ ID NO: 771: (64 п.о.)
/phos/ctctcagtacgtcagcagttnnnnnnnnnncaactccttggctcacagaacgacatggctacga, где последовательность перед nnnnnnnnnn (/phos/ctctcagtacgtcagcagtt) представляет собой SEQ ID NO: 793, последовательность после nnnnnnnn (caactccttggctcacagaacgacatggctacga) представляет собой SEQ ID NO: 794, (gacatggctacga представляет собой удлиненную часть по сравнению с решением 2).
SEQ ID NO: 772: (52 п.о.)
gcatggcgaccttatcagnnnnnnnnttgtcttcctaagaccgcttggcc, где последовательность перед nnnnnnnn (gcatggcgaccttatcag) представляет собой SEQ ID NO:795, последовательность после nnnnnnnnnn (ttgtcttcctaagaccgcttggcc) представляет собой SEQ ID NO:796 (cc представляет собой удлиненную часть по сравнению с Решением 2).
Характеристики Решения 1:
1. Комплементарная часть адаптера 7 + 13 п.о. (принадлежит области 20 ± 2 п.о.), область везикулярной структуры 20 + 12 п.о. (принадлежит области 30 ± 5 п.о.);
2. Праймер для амплификации немного длиннее.
Преимущества:
1. Структура «пузырьков» короче, поэтому отожженная структура стабильна.
2. Праймер для амплификации совместим с односторонними праймерами для амплификации и односторонними метками (см. 201910229527.4).
(2) Решение 2:
Последовательность адаптера
SEQ ID NO: 773 (адаптер 1) - SEQ ID NO: 774 (адаптер 2).
SEQ ID NO: 773 (35 п.о.):/phos/agtcggaggccaagcggtcttaggaagacaatcag.
SEQ ID NO: 774 (59 п.о.):
ctgattgtcttcctaagcaactccttggctcacagaacgacatggctacgatccgactt.
SEQ ID NO:775 (праймер для амплификации 1) и SEQ ID NO:776 (праймер для амплификации 2).
SEQ ID NO: 775: (51 п.о.)
/фос/ctctcagtacgtcagcagttnnnnnnnnnncaactccttggctcacagaac; где последовательность перед nnnnnnnnnn (/phos/CTCtcagtacgtcagcagtt) представляет собой SEQ ID NO:793, последовательность после nnnnnnnnnn (caactccttggctcacagaac) представляет собой SEQ ID NO:797.
SEQ ID NO: 776: (50 п.о.)
gcatggcgaccttatcagnnnnnnnnttgtcttcctaagaccgcttgg, где последовательность перед nnnnnnnnnn (gcatggcgaccttatcag) представляет собой SEQ ID NO:795, последовательность после nnnnnnnnnn (ttgtcttcctaagaccgcttgg) представляет собой SEQ ID NO:798.
Характеристики Решения 2:
1. Комплементарная часть адаптера 7+17 п.о. (относится к области 25±2 п.о.), «пузырьковая» структура 34+12 п.о. (относится к области 45±5 п.о.);
2. Праймер для амплификации короче.
Недостатки по сравнению с решением 1:
1. «Пузырьковая» структура длиннее, поэтому загибридизованная структура относительно нестабильна.
2. Праймер для амплификации не совместим с другими решениями (праймер для амплификации короче, и нет повторяющейся последовательности с «пузырьковой» структурой).
Результаты адаптерных структур и праймеров для амплификации решения 1 и решения 2 показаны на Фигуре 4. Могут быть получены обе библиотеки с двусторонними метками для секвенирования MGI. Для конструирования библиотеки в процессе эксперимента вводили 25 нг и 100 нг ДНК. Информация представлена в таблице ниже.
Таблица 3. Выходы библиотеки Решения 1 и Решения 2
количество ДНК
Библиотеки с двусторонними метками для MGI могут быть получены как из Решения 1, так и из Решения 2, и выход библиотек аналогичен, как показано на Фигуре 9. Но Решение 2 не совместимо с односторонними праймерами для амплификации и адаптерами с односторонними метками.
Пример 2. Сравнение разделения данных в 12 объединенных образцах между метками, сбалансированными по 4, и метками, сбалансированными по 8.
Решение с использованием двусторонних меток может эффективно решить проблемы перекрестных помех между образцами (также называемые перескоком меток). Но только когда оба конца меток правильные, данные секвенирования могут быть эффективно разделены. Таким образом, требования к балансу двусторонних меток более строгие, чем для односторонних меток. Настоящее изобретение оптимизирует два набора решений с метками, сбалансированными по 4, и метками, сбалансированными по 8. В этом примере использовались как метки, сбалансированные по 4, так и метки, сбалансированные по 8, а также объединены 12 библиотек для секвенирования для определения скорости разделения каждого образца для двух наборах решений. Экспериментальные стадии и информация представлены ниже:
Стадии: См. инструкции к набору для подготовки библиотеки ДНК NadPrep™ (для MGI) (201909Version2.0). Различие лишь в том, что адаптер с односторонними метками был заменен на адаптер с двусторонними метками.
Последовательность меток, сбалансированных по 4, использованная в эксперименте, показана в таблице 4, соседние 4 метки представляют собой группу баланса, и каждая группа выделяется жирным или неутолщенным шрифтом. Метка 1 представляет собой прямое распределение 384 последовательностей меток, а метка 2 представляет собой обратное распределение 384 последовательностей меток. Праймер 1 с меткой 1 и праймер 2 с меткой 384 составляют комбинацию первой группы праймеров с двусторонней меткой. Праймер 1 с меткой 2 и праймер 2 с меткой 383 составляют комбинацию второй группы праймеров с двусторонней меткой. Всего будет 384 комбинации.
Расположения меток, сбалансированных по 8, и расположения меток, сбалансированных по 4, одинаковы. Единственным отличием является 8 меток в группе, как показано в таблице 5. При объединении 12 библиотечных меток первые 8 являются сбалансированными, а последние 4 - несбалансированными. Для комбинации из меток, сбалансированных по 4, 12 библиотечных меток точно сбалансированы.
Таблица 4. 12 комбинаций меток, сбалансированных по 4
Таблица 5. 12 комбинаций меток, сбалансированных по 4
Для стандарта - мгеномной ДНК человека создаются библиотеки с 12 комбинациями двусторонних меток, сбалансированных по 4, и меток, сбалансированных по 8. Последовательности двусторонних меток, сбалансированных по 4, показаны в таблице 4, а последовательности двусторонних меток, сбалансированных по 8, показаны в таблице 5. Библиотеки, сбалансированные по 4, и библиотеки, сбалансированные по 8, секвенируют и анализируют на платформе секвенирования MGI.
Две группы библиотек были разделены на два раунда, в первом раунде для разделения использовалась максимальная отказоустойчивость (разделит ошибки секвенирования), а во втором раунде для разделения была разрешена только одна отказоустойчивость на метку. Результаты разделения данных показаны на Фигуре 10, скорость разделения данных 12 библиотек с метками, сбалансированными по 4, более стабильна, а скорость разделения данных 12 библиотек с метками, сбалансированными по 8, нестабильна. Результаты показывают, что сбалансированные двухсторонние метки в большей степени способствуют эффективному разделению данных секвенатора MGI, в данном документе дизайн меток, сбалансированных по 8, позволяет повысить эффективную скорость разделения данных лишь в некоторой степени, но дизайн меток, сбалансированных по 4, повышает лучше.
Пример 3
Чтобы обеспечить разницу в эффективности между 48 группами комбинаций сбалансированных по 8 меток, предусмотренных настоящим изобретением, и 12 группами комбинаций сбалансированных по 8 меток, обеспечиваемых производством MGI, при их дизайне учитывалась совместимость. Существует разница в 3 основания в любых двух последовательностях между 48 группами комбинаций меток, сбалансированных по 8, и 12 группами комбинаций меток, сбалансированных по 8, предоставленных производством MGI.
Кроме того, есть и другие важные отличия:
1. Композиция оснований последовательности меток в настоящем изобретении более сбалансирована и содержание GC составляет 40%-60%, при этом содержание GC в метках производства MGI составляет от 20% до 80%.
2. Последовательность метки по настоящему изобретению обладает свойством соответствия адаптерной последовательности Решения 1, чтобы гарантировать равномерное получение амплифицированных библиотек. Но некоторые последовательности производства MGI не удовлетворяют сбалансированному требованию по эффективности амплификации библиотеки.
Чтобы дополнительно проверить разницу эффективности в балансе амплификации, была выбрана группа из меток, сбалансированных по 8, комбинаций MDI001-MDI008 по изобретению и группа из меток, сбалансированных по 8, комбинаций MGI001-MGI008 производства MGI (показана в таблице 6) для конструирования библиотек: 100 нг ДНК в качестве исходного материала, ПЦР-амплификация в течение 5 циклов для определения выхода библиотеки, результаты представлены в таблице 7.
Как показано в Таблице 7, выходы библиотек по изобретению одинаковы, в то время как выход одной библиотеки производства MGI составляет менее половины нормального значения, что указывает на то, что оптимизированные последовательности меток по настоящему изобретению имеют лучший баланс. Кроме того, эффективность амплификации более стабильна. В то же время, благодаря высокой пропускной способности секвенатора MGI, две группы из 384 меток в настоящем изобретении оказались лучше, чем 120 меток производства MGI, чтобы удовлетворить потребность в высокой производительности для объединенного секвенирования.
Таблица 6. 8 комбинаций меток, сбалансированных по 8, производства MGI
Таблица 7
Согласно вышеприведенным воплощениям, двухсторонние библиотечные метки настоящего изобретении вводили в платформе секвенирования MGI для решения проблем перекрестных помех образцов, вызванных синтезом, экспериментальным процессом и процессом секвенирования, что сделает результаты обнаружения более точными. Кроме того, авторы изобретения обнаружили, путем тестирования и оптимизации, что когда средняя структура «пузырькового» адаптера имеет длину 30 ± 5 п.о., а количество спаренных оснований равно 20 ± 2 п.о., отжиг «пузырьковых» адаптеров является наиболее стабильным. Между тем, праймер для амплификации представляет собой удлиненный праймер для амплификации, который может быть совместим с ампликонами с односторонними метками и адаптерами с молекулярными односторонними метками. «Пузырьковые» адаптеры с такой композиционной структурой используются вместе с удлиненными праймерами для амплификации при конструировании библиотеки, которые могут быть совместимы с существующим решением с односторонними метками для платформы MGI и удобны для приложения секвенирования MGI.
На основании вышеизложенного, чтобы получить лучшее разделение данных, настоящее изобретение оптимизировало 384 комбинации последовательностей меток, сбалансированных по 4, и меток, сбалансированных по 8, соответственно, что обеспечивает оптимальное решение для высокопроизводительного секвенирования и разделения данных секвенирования для платформы MGI.
Приведенное выше описание представляет собой только предпочтительное воплощение настоящего изобретения и не предназначено для ограничения настоящего изобретения, и специалисты в данной области техники могут внести в настоящее изобретение различные модификации и изменения. Любые модификации, эквивалентные замены, усовершенствования и т.д., сделанные в рамках сущности и объема настоящего изобретения, предназначены для включения в объем охраны настоящего изобретения.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> НАНОДИГМБАЙО (НАНЬЦЗИН) БАЙОТЕКНОЛОДЖИ КО., ЛТД.
<120> КОМПОЗИЦИЯ ДВУХСТОРОННИХ БИБЛИОТЕЧНЫХ МЕТОК И ИХ ПРИМЕНЕНИЕ
В ПЛАТФОРМЕ СЕКВЕНИРОВАНИЯ MGI
<130> PN204453NAGD
<150> PCT/CN2020/139919
<151> 2020-12-28
<150> CN 202010838955.X
<151> 2020-08-19
<160> 798
<170> PatentIn version 3.5
<210> 1
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 1
tcacattgct 10
<210> 2
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 2
aatggcgctc 10
<210> 3
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 3
gtctcaatga 10
<210> 4
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 4
cggatgcaag 10
<210> 5
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 5
tcgcttaagc 10
<210> 6
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 6
cgaggcttag 10
<210> 7
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 7
gtctaaggct 10
<210> 8
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 8
aatacgccta 10
<210> 9
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 9
aagcctattg 10
<210> 10
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 10
cgctactgca 10
<210> 11
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 11
tcaagagcat 10
<210> 12
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 12
gttgtgcagc 10
<210> 13
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 13
agacaggaat 10
<210> 14
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 14
ccttgccgta 10
<210> 15
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 15
gtcattacgg 10
<210> 16
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 16
taggcattcc 10
<210> 17
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 17
catatcatcg 10
<210> 18
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 18
gcacaacaat 10
<210> 19
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 19
ttgtcgtggc 10
<210> 20
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 20
agcggtgcta 10
<210> 21
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 21
agccagtagg 10
<210> 22
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 22
gtaagtgtac 10
<210> 23
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 23
tagtcacgtt 10
<210> 24
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 24
cctgtcacca 10
<210> 25
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 25
atcgtggatg 10
<210> 26
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 26
tggagatcga 10
<210> 27
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 27
cctcacagat 10
<210> 28
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 28
gaatctctcc 10
<210> 29
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 29
gcagactgac 10
<210> 30
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 30
ctcattaacg 10
<210> 31
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 31
tggtgagctt 10
<210> 32
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 32
aatccgctga 10
<210> 33
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 33
tcgcatcaac 10
<210> 34
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 34
agaacagtga 10
<210> 35
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 35
catgtctcct 10
<210> 36
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 36
gtctggagtg 10
<210> 37
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 37
gaggtctgtg 10
<210> 38
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 38
ctatagacgt 10
<210> 39
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 39
tgcagtgacc 10
<210> 40
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 40
actccactaa 10
<210> 41
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 41
gcgaagtagg 10
<210> 42
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 42
tgcctaacct 10
<210> 43
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 43
aatggtctac 10
<210> 44
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 44
ctatccggta 10
<210> 45
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 45
tacgcttcag 10
<210> 46
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 46
cggagcatct 10
<210> 47
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 47
gtactagatc 10
<210> 48
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 48
acttagcgga 10
<210> 49
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 49
atcactccat 10
<210> 50
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 50
gatcgcagtg 10
<210> 51
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 51
ccggaattcc 10
<210> 52
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 52
tgattggaga 10
<210> 53
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 53
cacaaggtcg 10
<210> 54
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 54
tctcgcagga 10
<210> 55
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 55
gtggtatcat 10
<210> 56
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 56
agatctcatc 10
<210> 57
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 57
gatggagatt 10
<210> 58
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 58
ctcattctgc 10
<210> 59
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 59
tggcagacaa 10
<210> 60
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 60
acatcctgcg 10
<210> 61
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 61
acgtcgcaga 10
<210> 62
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 62
tgtataggct 10
<210> 63
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 63
caacacattg 10
<210> 64
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 64
gtcggttcac 10
<210> 65
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 65
agccataagc 10
<210> 66
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 66
gtattccgag 10
<210> 67
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 67
cataggttca 10
<210> 68
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 68
tcggcagctt 10
<210> 69
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 69
gttcggtcct 10
<210> 70
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 70
tggattgtag 10
<210> 71
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 71
ccagaacgtc 10
<210> 72
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 72
aactccaaga 10
<210> 73
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 73
cgtacactgg 10
<210> 74
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 74
gcacacagca 10
<210> 75
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 75
atggtgtatc 10
<210> 76
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 76
tactgtgcat 10
<210> 77
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 77
cggcaatcag 10
<210> 78
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 78
gtagttcgga 10
<210> 79
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 79
tacaggaact 10
<210> 80
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 80
acttccgttc 10
<210> 81
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 81
tgctccacga 10
<210> 82
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 82
aatcaaggtc 10
<210> 83
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 83
gtaggtcaat 10
<210> 84
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 84
ccgatgttcg 10
<210> 85
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 85
gacgtgtgca 10
<210> 86
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 86
tcgccacttc 10
<210> 87
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 87
ataagcacgt 10
<210> 88
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 88
cgttatgaag 10
<210> 89
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 89
aatagagcca 10
<210> 90
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 90
gtacctcgac 10
<210> 91
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 91
ccggtgattg 10
<210> 92
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 92
tgctactagt 10
<210> 93
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 93
gatccggact 10
<210> 94
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 94
ttaggcacaa 10
<210> 95
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 95
agcattcttg 10
<210> 96
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 96
ccgtaatggc 10
<210> 97
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 97
gtatagctgc 10
<210> 98
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 98
cagacatctg 10
<210> 99
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 99
agcggtggat 10
<210> 100
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 100
tctctcaaca 10
<210> 101
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 101
catccactgt 10
<210> 102
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 102
gccgtgaaca 10
<210> 103
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 103
ataagcggag 10
<210> 104
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 104
tggtattctc 10
<210> 105
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 105
acagttctca 10
<210> 106
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 106
cattgagagc 10
<210> 107
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 107
gtcacgactg 10
<210> 108
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 108
tggcactgat 10
<210> 109
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 109
aatgattcgc 10
<210> 110
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 110
cgcttaagta 10
<210> 111
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 111
ttgagcgacg 10
<210> 112
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 112
gcaccgctat 10
<210> 113
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 113
acgaacggat 10
<210> 114
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 114
gaacggacta 10
<210> 115
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 115
tgctctctgg 10
<210> 116
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 116
cttgtatacc 10
<210> 117
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 117
caccagcaca 10
<210> 118
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 118
ttgtctgtag 10
<210> 119
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 119
agtatcactc 10
<210> 120
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 120
gcaggatggt 10
<210> 121
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 121
ttatccacgt 10
<210> 122
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 122
acggttcgtc 10
<210> 123
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 123
gaccagtaag 10
<210> 124
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 124
cgtagagtca 10
<210> 125
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 125
acgttaaggt 10
<210> 126
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 126
ctcagcttag 10
<210> 127
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 127
tgtgctccta 10
<210> 128
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 128
gaacaggacc 10
<210> 129
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 129
gcgaccttga 10
<210> 130
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 130
atcgtgagtt 10
<210> 131
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 131
cgtcgtcaac 10
<210> 132
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 132
taataagccg 10
<210> 133
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 133
ccacttagta 10
<210> 134
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 134
agtagctagt 10
<210> 135
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 135
gacgaactcg 10
<210> 136
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 136
ttgtcggcac 10
<210> 137
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 137
agcatatcgt 10
<210> 138
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 138
gtagacggag 10
<210> 139
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 139
cattctcatc 10
<210> 140
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 140
tcgcggatca 10
<210> 141
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 141
ctggaggcaa 10
<210> 142
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 142
gatacttgtg 10
<210> 143
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 143
tgcctccact 10
<210> 144
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 144
acatgaatgc 10
<210> 145
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 145
tcagcagagg 10
<210> 146
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 146
ctgtgcatta 10
<210> 147
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 147
agtaatcgac 10
<210> 148
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 148
agtaatcgac 10
<210> 149
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 149
aagcggtgaa 10
<210> 150
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 150
ctatcacact 10
<210> 151
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 151
gccgttatgc 10
<210> 152
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 152
tgtaacgctg 10
<210> 153
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 153
gcgtgtaact 10
<210> 154
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 154
catgtaccac 10
<210> 155
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 155
tgccactgta 10
<210> 156
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 156
ataacggtgg 10
<210> 157
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 157
cttgaaggtt 10
<210> 158
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 158
gcctctatgg 10
<210> 159
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 159
tagatccacc 10
<210> 160
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 160
agacggtcaa 10
<210> 161
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 161
gctggattaa 10
<210> 162
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 162
taacaccggc 10
<210> 163
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 163
atgactgccg 10
<210> 164
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 164
cgcttgaatt 10
<210> 165
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 165
ctatagcgag 10
<210> 166
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 166
aacgttgttc 10
<210> 167
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 167
gctaccacgt 10
<210> 168
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 168
tggcgataca 10
<210> 169
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 169
tccgccaatc 10
<210> 170
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 170
caactagtgt 10
<210> 171
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 171
agtaagccaa 10
<210> 172
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 172
gtgtgttgcg 10
<210> 173
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 173
gatcagatgg 10
<210> 174
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 174
ctcgtaggtc 10
<210> 175
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 175
tcgtgtccat 10
<210> 176
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 176
agaacctaca 10
<210> 177
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 177
ccgcattcct 10
<210> 178
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 178
gttggacata 10
<210> 179
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 179
tgatcgaggc 10
<210> 180
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 180
aacatcgtag 10
<210> 181
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 181
gttgcgcgaa 10
<210> 182
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 182
acaagtaagc 10
<210> 183
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 183
cgcttctcct 10
<210> 184
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 184
tagcaagttg 10
<210> 185
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 185
aggcctcttc 10
<210> 186
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 186
gtaatgtcgt 10
<210> 187
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 187
cactgcagag 10
<210> 188
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 188
tctgaagaca 10
<210> 189
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 189
gctaaggata 10
<210> 190
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 190
cacggttggt 10
<210> 191
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 191
tgaccaccag 10
<210> 192
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 192
atgttcatcc 10
<210> 193
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 193
agctactctg 10
<210> 194
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 194
caacgtgagt 10
<210> 195
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 195
tcgatgctaa 10
<210> 196
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 196
gttgcaagcc 10
<210> 197
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 197
cgacatgtgt 10
<210> 198
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 198
gatgcgcata 10
<210> 199
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 199
tcgtgatcag 10
<210> 200
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 200
atcatcagcc 10
<210> 201
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 201
tcaatggcgg 10
<210> 202
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 202
cagtaactct 10
<210> 203
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 203
gttgcctgac 10
<210> 204
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 204
agccgtaata 10
<210> 205
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 205
ctaataggct 10
<210> 206
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 206
tctcctccac 10
<210> 207
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 207
agctgcatga 10
<210> 208
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 208
gaggagtatg 10
<210> 209
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 209
accacgtagc 10
<210> 210
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 210
gaatgcagta 10
<210> 211
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 211
ttggtagcct 10
<210> 212
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 212
cgtcatctag 10
<210> 213
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 213
tcaatgaggt 10
<210> 214
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 214
agcgaagctg 10
<210> 215
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 215
ctgtcttaac 10
<210> 216
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 216
gatcgcctca 10
<210> 217
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 217
gttccgaatg 10
<210> 218
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 218
tacgtacgca 10
<210> 219
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 219
cggagttcac 10
<210> 220
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 220
acatacgtgt 10
<210> 221
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 221
ctgaagagat 10
<210> 222
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 222
gaagcctcca 10
<210> 223
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 223
tgtcttcatc 10
<210> 224
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 224
acctgagtgg 10
<210> 225
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 225
gtacgtcctt 10
<210> 226
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 226
aactaggtca 10
<210> 227
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 227
tgtgtcaggc 10
<210> 228
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 228
ccgacataag 10
<210> 229
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 229
tccacacgtc 10
<210> 230
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 230
cggcacatga 10
<210> 231
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 231
gtatgttcct 10
<210> 232
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 232
aatgtggaag 10
<210> 233
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 233
agacagacgt 10
<210> 234
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 234
ttctgtggag 10
<210> 235
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 235
caggtcttcc 10
<210> 236
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 236
gctacacata 10
<210> 237
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 237
ctagcgacac 10
<210> 238
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 238
aggcattact 10
<210> 239
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 239
gacatccgga 10
<210> 240
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 240
tcttgagttg 10
<210> 241
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 241
acaacagaag 10
<210> 242
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 242
cgcttgtgga 10
<210> 243
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 243
gttggtctcc 10
<210> 244
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 244
tagcacactt 10
<210> 245
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 245
gcttgcaata 10
<210> 246
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 246
tggacgtgct 10
<210> 247
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 247
aacgaaccgg 10
<210> 248
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 248
ctacttgtac 10
<210> 249
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 249
cggtgagtga 10
<210> 250
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 250
gcaatgcatt 10
<210> 251
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 251
ttccattcac 10
<210> 252
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 252
aatgccagcg 10
<210> 253
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 253
tactcttctc 10
<210> 254
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 254
aggaaggtaa 10
<210> 255
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 255
gttggacggt 10
<210> 256
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 256
ccactcaacg 10
<210> 257
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 257
aatgactggt 10
<210> 258
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 258
gtgccacaac 10
<210> 259
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 259
cgaatgatcg 10
<210> 260
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 260
tcctgtgcta 10
<210> 261
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 261
tgtgaattgg 10
<210> 262
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 262
aaccggcctt 10
<210> 263
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 263
ctatccgacc 10
<210> 264
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 264
gcgattagaa 10
<210> 265
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 265
cgtaaccgca 10
<210> 266
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 266
gactgataac 10
<210> 267
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 267
atgcttactg 10
<210> 268
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 268
tcagcggtgt 10
<210> 269
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 269
acataacacc 10
<210> 270
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 270
cacctgaggt 10
<210> 271
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 271
gtgactgtaa 10
<210> 272
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 272
tgtggctctg 10
<210> 273
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 273
ctcagactct 10
<210> 274
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 274
acacatgcta 10
<210> 275
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 275
tgtgcctaag 10
<210> 276
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 276
gagttgaggc 10
<210> 277
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 277
agccgttctc 10
<210> 278
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 278
ttgttggtct 10
<210> 279
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 279
caagacaaga 10
<210> 280
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 280
gctacacgag 10
<210> 281
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 281
gtgacgcgat 10
<210> 282
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 282
aacctctctg 10
<210> 283
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 283
tcttgagaga 10
<210> 284
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 284
cgagatatcc 10
<210> 285
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 285
gaaggattca 10
<210> 286
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 286
tcgcctggtt 10
<210> 287
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 287
agtaacacgg 10
<210> 288
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 288
ctcttgcaac 10
<210> 289
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 289
agagacttac 10
<210> 290
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 290
cttatggccg 10
<210> 291
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 291
gcctgacgtt 10
<210> 292
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 292
tagcctaaga 10
<210> 293
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 293
aagcatatcc 10
<210> 294
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 294
ctaatccgtt 10
<210> 295
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 295
gcttggtcga 10
<210> 296
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 296
tgcgcagaag 10
<210> 297
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 297
gatcctgata 10
<210> 298
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 298
tcaagcacgg 10
<210> 299
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 299
ctgtagcgct 10
<210> 300
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 300
agcgtattac 10
<210> 301
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 301
tgtctgattg 10
<210> 302
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 302
accagacggt 10
<210> 303
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 303
gtgtctgacc 10
<210> 304
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 304
caagactcaa 10
<210> 305
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 305
caagactcaa 10
<210> 306
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 306
agacgaggtc 10
<210> 307
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 307
ctggattact 10
<210> 308
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 308
gatatgctga 10
<210> 309
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 309
ctggtcaagg 10
<210> 310
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 310
gctccattcc 10
<210> 311
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 311
tgatgtcgaa 10
<210> 312
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 312
aacaaggctt 10
<210> 313
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 313
catctagaca 10
<210> 314
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 314
gcggatacag 10
<210> 315
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 315
ttctgcttgt 10
<210> 316
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 316
agaacgcgtc 10
<210> 317
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 317
aagacgaact 10
<210> 318
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 318
ccagtactgc 10
<210> 319
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 319
gttcgctgta 10
<210> 320
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 320
tgctatgcag 10
<210> 321
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 321
tacacgcgca 10
<210> 322
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 322
aggtacgcag 10
<210> 323
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 323
gttcgattgt 10
<210> 324
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 324
ccagttaatc 10
<210> 325
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 325
taagatcgga 10
<210> 326
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 326
agctcggctt 10
<210> 327
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 327
gttcgataag 10
<210> 328
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 328
ccgatcatcc 10
<210> 329
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 329
actggactca 10
<210> 330
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 330
caatagaggc 10
<210> 331
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 331
gtcacttaag 10
<210> 332
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 332
tggctcgctt 10
<210> 333
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 333
cgcactatgg 10
<210> 334
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 334
gaggtacatt 10
<210> 335
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 335
tcttagtgac 10
<210> 336
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 336
atacgcgcca 10
<210> 337
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 337
tgaggcatat 10
<210> 338
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 338
attctatggc 10
<210> 339
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 339
ccgtagcatg 10
<210> 340
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 340
gacactgcca 10
<210> 341
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 341
acggcattaa 10
<210> 342
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 342
gtaagcgagg 10
<210> 343
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 343
cattatcgct 10
<210> 344
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 344
tgcctgactc 10
<210> 345
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 345
gactcatcca 10
<210> 346
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 346
acgaacatac 10
<210> 347
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 347
ttacgtcggt 10
<210> 348
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 348
cgtgtggatg 10
<210> 349
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 349
cctaacattc 10
<210> 350
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 350
atcgcacgca 10
<210> 351
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 351
gaacgttcgg 10
<210> 352
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 352
tggttggaat 10
<210> 353
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 353
aacaagtgag 10
<210> 354
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 354
gttgttgctc 10
<210> 355
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 355
tgacgcaact 10
<210> 356
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 356
ccgtcactga 10
<210> 357
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 357
tgttattccg 10
<210> 358
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 358
ctactcaaga 10
<210> 359
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 359
acgagacgtc 10
<210> 360
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 360
gacgcggtat 10
<210> 361
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 361
gatgacgtta 10
<210> 362
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 362
agccgatacc 10
<210> 363
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 363
ttatctcgag 10
<210> 364
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 364
ccgatgacgt 10
<210> 365
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 365
gtgagttcgc 10
<210> 366
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 366
acacaacatg 10
<210> 367
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 367
cgtgcgatca 10
<210> 368
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 368
tacttcggat 10
<210> 369
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 369
ctatcggtgt 10
<210> 370
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 370
gacattcaag 10
<210> 371
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 371
agtgacacca 10
<210> 372
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 372
tcgcgatgtc 10
<210> 373
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 373
cgagtcagtc 10
<210> 374
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 374
ttgccagtga 10
<210> 375
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 375
aacaagcact 10
<210> 376
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 376
gcttgttcag 10
<210> 377
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 377
tagtgatgtg 10
<210> 378
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 378
cgtgtgacat 10
<210> 379
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 379
atccaccacc 10
<210> 380
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 380
gcaactgtga 10
<210> 381
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 381
atccaccggt 10
<210> 382
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 382
ccgtcattac 10
<210> 383
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 383
tgagtggcta 10
<210> 384
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 384
gatagtaacg 10
<210> 385
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 385
cgtcgatgac 10
<210> 386
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 386
atataaggcg 10
<210> 387
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 387
gatcgtgctc 10
<210> 388
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 388
cagtcttcgg 10
<210> 389
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 389
agaacgatct 10
<210> 390
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 390
ttggtgcatt 10
<210> 391
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 391
gccgtcataa 10
<210> 392
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 392
tccaaccaga 10
<210> 393
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 393
gatagcaaga 10
<210> 394
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 394
accgtgcttc 10
<210> 395
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 395
gcagatgtaa 10
<210> 396
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 396
tgttggagcg 10
<210> 397
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 397
ttgtatccac 10
<210> 398
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 398
cgcacagatg 10
<210> 399
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 399
caactctcgt 10
<210> 400
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 400
atgccatgct 10
<210> 401
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 401
aggcagctta 10
<210> 402
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 402
tagcctagcg 10
<210> 403
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 403
atcacgtgcg 10
<210> 404
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 404
cgttatgcgc 10
<210> 405
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 405
caaggatcga 10
<210> 406
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 406
gttgtcgtat 10
<210> 407
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 407
gcaatcaatc 10
<210> 408
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 408
tcctgacaat 10
<210> 409
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 409
aggtgcctta 10
<210> 410
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 410
aagaaccaag 10
<210> 411
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 411
catacatgac 10
<210> 412
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 412
tgcctggtga 10
<210> 413
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 413
tctcggagtt 10
<210> 414
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 414
gtcgtagact 10
<210> 415
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 415
gcatataccg 10
<210> 416
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 416
ctagcttcgc 10
<210> 417
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 417
gacgtatcaa 10
<210> 418
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 418
cctgctagga 10
<210> 419
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 419
caacttggcg 10
<210> 420
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 420
atgcgacctc 10
<210> 421
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 421
gttaaggacg 10
<210> 422
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 422
tccaagttgt 10
<210> 423
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 423
aggtccattc 10
<210> 424
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 424
tgatgccaat 10
<210> 425
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 425
cctaagagtt 10
<210> 426
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 426
gatactagct 10
<210> 427
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 427
aaggcatctc 10
<210> 428
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 428
tggcatctgg 10
<210> 429
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 429
acactggagc 10
<210> 430
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 430
gtatgccaca 10
<210> 431
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 431
ctcttagtag 10
<210> 432
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 432
tgcggctcaa 10
<210> 433
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 433
taaggctaga 10
<210> 434
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 434
gaggagataa 10
<210> 435
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 435
cgttagcact 10
<210> 436
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 436
aggctaggat 10
<210> 437
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 437
atctcactgg 10
<210> 438
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 438
tccagttctc 10
<210> 439
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 439
gtaaccgctc 10
<210> 440
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 440
cctcttagcg 10
<210> 441
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 441
actaaggctg 10
<210> 442
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 442
gaggagtatg 10
<210> 443
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 443
tagtcaacgc 10
<210> 444
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 444
ccatcaagga 10
<210> 445
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 445
agcctctgct 10
<210> 446
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 446
ttcgttcaca 10
<210> 447
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 447
gtaagtgtac 10
<210> 448
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 448
cgtcgcctat 10
<210> 449
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 449
tgtgaaggag 10
<210> 450
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 450
agaccggttc 10
<210> 451
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 451
ctcgcctaac 10
<210> 452
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 452
ccgctgtcta 10
<210> 453
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 453
gacataacct 10
<210> 454
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 454
gagaatagca 10
<210> 455
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 455
ttatgtcagg 10
<210> 456
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 456
acttgcctgt 10
<210> 457
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 457
tgctggatct 10
<210> 458
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 458
agtagtgttg 10
<210> 459
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 459
ctccaatcct 10
<210> 460
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 460
cctatgtgta 10
<210> 461
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 461
taatatccgc 10
<210> 462
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 462
gtgccacaac 10
<210> 463
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 463
gcagtcagga 10
<210> 464
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 464
aaggccgaag 10
<210> 465
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 465
cgtgttagag 10
<210> 466
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 466
acaggacgat 10
<210> 467
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 467
cggataacgg 10
<210> 468
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 468
gttagtgcct 10
<210> 469
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 469
aagcacgaca 10
<210> 470
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 470
gtctccttgc 10
<210> 471
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 471
tcaccgtata 10
<210> 472
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 472
tactagcttc 10
<210> 473
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 473
accattcacg 10
<210> 474
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 474
agtagctagt 10
<210> 475
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 475
ttccgaaggt 10
<210> 476
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 476
gtaccaacca 10
<210> 477
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 477
tatgtgtgtc 10
<210> 478
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 478
cgatcggtac 10
<210> 479
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 479
gaggatctag 10
<210> 480
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 480
ccgtacgcta 10
<210> 481
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 481
gtcaactcgg 10
<210> 482
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 482
tggaagcaca 10
<210> 483
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 483
tcgttgtagc 10
<210> 484
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 484
agccgaagtt 10
<210> 485
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 485
gatgcaacct 10
<210> 486
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 486
acttccgttc 10
<210> 487
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 487
caagttggaa 10
<210> 488
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 488
ctacgtctag 10
<210> 489
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 489
tgtcgttaag 10
<210> 490
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 490
gtctcaatga 10
<210> 491
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 491
gatcaagcca 10
<210> 492
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 492
aactccgatc 10
<210> 493
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 493
ctaagtcttc 10
<210> 494
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 494
tgagagcgct 10
<210> 495
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 495
acgatctcgg 10
<210> 496
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 496
ccggtgagat 10
<210> 497
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 497
catctcatga 10
<210> 498
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 498
ctgtgactcg 10
<210> 499
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 499
agaccttgga 10
<210> 500
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 500
actgtcgacg 10
<210> 501
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 501
gtgaagacac 10
<210> 502
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 502
tactgtgcat 10
<210> 503
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 503
gcagagcgtt 10
<210> 504
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 504
tgcacatatc 10
<210> 505
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 505
cctgtgtaac 10
<210> 506
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 506
aacgatgcca 10
<210> 507
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 507
tcgagcatag 10
<210> 508
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 508
gaattcctgt 10
<210> 509
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 509
agaacgtccg 10
<210> 510
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 510
cgccaaggta 10
<210> 511
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 511
ttgtgaaggc 10
<210> 512
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 512
gttcctcatt 10
<210> 513
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 513
cgtcactatt 10
<210> 514
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 514
gcgatgcaga 10
<210> 515
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 515
cgtgtcctag 10
<210> 516
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 516
gaagaatgga 10
<210> 517
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 517
atgtgtggct 10
<210> 518
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 518
tcctgtacac 10
<210> 519
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 519
ttaccgattc 10
<210> 520
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 520
aacacagccg 10
<210> 521
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 521
tcataccaag 10
<210> 522
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 522
gaagcttact 10
<210> 523
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 523
gtcagtaggt 10
<210> 524
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 524
ctgtgcgtag 10
<210> 525
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 525
agtgaatgga 10
<210> 526
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 526
tccacggttc 10
<210> 527
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 527
aatctgcctc 10
<210> 528
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 528
cggctaacca 10
<210> 529
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 529
gacatacagt 10
<210> 530
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 530
agaggcctca 10
<210> 531
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 531
cctgatattg 10
<210> 532
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 532
gtgctgaact 10
<210> 533
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 533
cgatggtcag 10
<210> 534
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 534
aatacagcgc 10
<210> 535
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 535
tcgtcctgac 10
<210> 536
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 536
ttccatggta 10
<210> 537
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 537
cgttcgactg 10
<210> 538
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 538
cgctacactc 10
<210> 539
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 539
atagatcggt 10
<210> 540
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 540
accagattca 10
<210> 541
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 541
gatacggagg 10
<210> 542
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 542
ttaggaggaa 10
<210> 543
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 543
gcgcttctac 10
<210> 544
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 544
tagctctact 10
<210> 545
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 545
ccacctgctt 10
<210> 546
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 546
cgtaggtctg 10
<210> 547
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 547
tcttagtgac 10
<210> 548
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 548
atcctagaac 10
<210> 549
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 549
aggtgtaggt 10
<210> 550
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 550
gtaaccatca 10
<210> 551
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 551
taggtccaga 10
<210> 552
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 552
gacgaactcg 10
<210> 553
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 553
ccaacagatt 10
<210> 554
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 554
cctctatctc 10
<210> 555
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 555
gagtgtctca 10
<210> 556
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 556
ttggcttaag 10
<210> 557
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 557
agtcgcatgg 10
<210> 558
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 558
atcttgcggc 10
<210> 559
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 559
tgcaaggcct 10
<210> 560
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 560
gaagacagaa 10
<210> 561
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 561
acagactcat 10
<210> 562
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 562
gagttgaggc 10
<210> 563
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 563
gttctagacg 10
<210> 564
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 564
agttcaggcg 10
<210> 565
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 565
cagacgatga 10
<210> 566
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 566
tccaacctat 10
<210> 567
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 567
ttacgttctc 10
<210> 568
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 568
cgcggtcata 10
<210> 569
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 569
aatccttccg 10
<210> 570
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 570
gaggattgaa 10
<210> 571
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 571
atcggagtgc 10
<210> 572
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 572
gcgttaagtg 10
<210> 573
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 573
cctcggcaat 10
<210> 574
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 574
ttaacggctt 10
<210> 575
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 575
tgattccaca 10
<210> 576
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 576
cgcaacatgc 10
<210> 577
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 577
tcaatgaggt 10
<210> 578
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 578
gtccaagctg 10
<210> 579
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 579
gcgcactaag 10
<210> 580
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 580
cggtgagtga 10
<210> 581
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 581
tgaatccgac 10
<210> 582
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 582
aatgctcact 10
<210> 583
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 583
atcggttcca 10
<210> 584
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 584
cattcgattc 10
<210> 585
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 585
ttatgcctac 10
<210> 586
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 586
atcctccgac 10
<210> 587
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 587
cgcagatata 10
<210> 588
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 588
acaacatgtg 10
<210> 589
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 589
tcggatgagg 10
<210> 590
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 590
cgtgcgatca 10
<210> 591
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 591
gatcttacgt 10
<210> 592
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 592
gagtaggcct 10
<210> 593
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 593
aagagagaag 10
<210> 594
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 594
gttctcacgg 10
<210> 595
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 595
aagcggtgaa 10
<210> 596
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 596
cgctcagtta 10
<210> 597
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 597
ctagacactc 10
<210> 598
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 598
gcttattgct 10
<210> 599
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 599
tgcgttctgc 10
<210> 600
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 600
tcaacgcact 10
<210> 601
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 601
cttgcttaca 10
<210> 602
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 602
ctcctcgcaa 10
<210> 603
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 603
tctcagcctc 10
<210> 604
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 604
gacggagtac 10
<210> 605
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 605
acgtctatct 10
<210> 606
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 606
gaatagagtg 10
<210> 607
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 607
aggatacggt 10
<210> 608
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 608
tgaagctagg 10
<210> 609
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 609
aacctcgcac 10
<210> 610
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 610
gatccggact 10
<210> 611
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 611
gtgtctacag 10
<210> 612
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 612
acggagaatc 10
<210> 613
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 613
tgttacctca 10
<210> 614
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 614
tcaatacgtg 10
<210> 615
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 615
ctaagttggt 10
<210> 616
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 616
cgcggattga 10
<210> 617
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 617
acctcacata 10
<210> 618
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 618
cggactgtct 10
<210> 619
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 619
cgtggcagaa 10
<210> 620
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 620
attcgaatgc 10
<210> 621
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 621
taggttcaag 10
<210> 622
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 622
gtccactctc 10
<210> 623
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 623
gaatagtgct 10
<210> 624
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 624
tcaatggcgg 10
<210> 625
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 625
aagcatcctg 10
<210> 626
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 626
gtggtgttca 10
<210> 627
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 627
gtacaacgtt 10
<210> 628
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 628
cctgcagcat 10
<210> 629
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 629
tctatcgtac 10
<210> 630
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 630
cactcgtagc 10
<210> 631
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 631
tgcagcagca 10
<210> 632
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 632
agatgtaagg 10
<210> 633
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 633
gtgtaaccgc 10
<210> 634
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 634
catcggaaga 10
<210> 635
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 635
gaggaattac 10
<210> 636
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 636
tgttcgctct 10
<210> 637
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 637
accgtctata 10
<210> 638
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 638
tgcctcagag 10
<210> 639
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 639
acaagtgccg 10
<210> 640
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 640
ctaactggtt 10
<210> 641
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 641
cagaacgtgg 10
<210> 642
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 642
gttcttctgt 10
<210> 643
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 643
cggtgaagtc 10
<210> 644
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 644
gtaagatgag 10
<210> 645
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 645
tgccatcaca 10
<210> 646
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 646
tcctcggata 10
<210> 647
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 647
acagtgtcct 10
<210> 648
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 648
aatgccacac 10
<210> 649
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 649
gaccactcga 10
<210> 650
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 650
atggacaaca 10
<210> 651
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 651
aacctacggt 10
<210> 652
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 652
tctaggattg 10
<210> 653
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 653
cggattctcg 10
<210> 654
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 654
cgatcttctc 10
<210> 655
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 655
tcatcggaat 10
<210> 656
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 656
gttggaggac 10
<210> 657
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 657
acacatgcta 10
<210> 658
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 658
ccttaggacg 10
<210> 659
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 659
ctaaccaatc 10
<210> 660
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 660
aacgcacgag 10
<210> 661
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 661
gtcggttcac 10
<210> 662
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 662
tggctcatgt 10
<210> 663
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 663
tggtggttgt 10
<210> 664
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 664
gatatacgca 10
<210> 665
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 665
cacgaacact 10
<210> 666
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 666
tgcgtgagca 10
<210> 667
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 667
gtactgacga 10
<210> 668
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 668
ccgtgagttc 10
<210> 669
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 669
acttacttgg 10
<210> 670
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 670
agaagctcat 10
<210> 671
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 671
gttactggac 10
<210> 672
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 672
tagcctcatg 10
<210> 673
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 673
tgacaatgac 10
<210> 674
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 674
aatcacctct 10
<210> 675
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 675
aacagacctg 10
<210> 676
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 676
gctagtgtgt 10
<210> 677
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 677
gcggttgata 10
<210> 678
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 678
ctagcgacac 10
<210> 679
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 679
cggttgaacg 10
<210> 680
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 680
ttctcctgga 10
<210> 681
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 681
agttagctgg 10
<210> 682
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 682
agtcctgtaa 10
<210> 683
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 683
taaggccggt 10
<210> 684
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 684
gccttatcct 10
<210> 685
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 685
cagcattcaa 10
<210> 686
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 686
tcgagcaatc 10
<210> 687
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 687
gtaacgaacg 10
<210> 688
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 688
ctcgtaggtc 10
<210> 689
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 689
ctggattact 10
<210> 690
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 690
gtatgttcct 10
<210> 691
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 691
tgttcgcgac 10
<210> 692
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 692
cacggaattg 10
<210> 693
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 693
agcacaatgc 10
<210> 694
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 694
acacaccgga 10
<210> 695
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 695
tatctcgcta 10
<210> 696
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 696
gcgatggaag 10
<210> 697
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 697
taactcgtgg 10
<210> 698
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 698
acaaccagta 10
<210> 699
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 699
agcgtacgtc 10
<210> 700
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 700
tcgactctgt 10
<210> 701
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 701
ctgtatgcca 10
<210> 702
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 702
gacgagacct 10
<210> 703
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 703
cgttggtaac 10
<210> 704
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 704
gttcgataag 10
<210> 705
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 705
caagcgcgat 10
<210> 706
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 706
gtgtgagtgc 10
<210> 707
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 707
gcagcataat 10
<210> 708
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 708
ttcttgtgca 10
<210> 709
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 709
tggcacattg 10
<210> 710
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 710
aacaatacgc 10
<210> 711
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 711
cgtattgctg 10
<210> 712
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 712
actcgccaca 10
<210> 713
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 713
gctgcttggt 10
<210> 714
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 714
ttgtggatac 10
<210> 715
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 715
accaaggcga 10
<210> 716
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 716
agaagaccta 10
<210> 717
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 717
tagctctgtc 10
<210> 718
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 718
cgacttctat 10
<210> 719
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 719
ctctccaacg 10
<210> 720
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 720
gatgaagacg 10
<210> 721
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 721
tccagctcat 10
<210> 722
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 722
accaagactc 10
<210> 723
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 723
caactgcgca 10
<210> 724
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 724
agtggatatc 10
<210> 725
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 725
gtactaagag 10
<210> 726
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 726
gtgtacgtcg 10
<210> 727
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 727
cggtctctgt 10
<210> 728
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 728
tatgctgaga 10
<210> 729
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 729
aataggtagg 10
<210> 730
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 730
agcttgcgct 10
<210> 731
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 731
tggagccgat 10
<210> 732
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 732
ccgcatacta 10
<210> 733
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 733
ttcgatgacg 10
<210> 734
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 734
gatccaatac 10
<210> 735
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 735
gcattctcgc 10
<210> 736
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 736
ctagcagtta 10
<210> 737
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 737
cgttgacgct 10
<210> 738
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 738
agtatatgcg 10
<210> 739
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 739
acctccgcta 10
<210> 740
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 740
caacacgaat 10
<210> 741
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 741
ttgagtaagg 10
<210> 742
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 742
gaagctctta 10
<210> 743
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 743
tccgagatgc 10
<210> 744
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 744
gtgctgtcac 10
<210> 745
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 745
agcttccagc 10
<210> 746
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 746
tgcctatcgc 10
<210> 747
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 747
caatctcgcg 10
<210> 748
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 748
cttcctacaa 10
<210> 749
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 749
acgaggtact 10
<210> 750
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 750
gatgaaggat 10
<210> 751
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 751
tcgagcgtta 10
<210> 752
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 752
gtagagattg 10
<210> 753
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 753
acggctagag 10
<210> 754
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 754
gctatagctt 10
<210> 755
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 755
tgcgtcatgg 10
<210> 756
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 756
ttccaacatc 10
<210> 757
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 757
cggtgttgga 10
<210> 758
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 758
gattgcgcct 10
<210> 759
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 759
ataacgctca 10
<210> 760
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 760
caacagtaac 10
<210> 761
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 761
actggaggag 10
<210> 762
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 762
gccggtaagt 10
<210> 763
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 763
ctcacactta 10
<210> 764
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 764
agacagtggc 10
<210> 765
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 765
ctgatgtcct 10
<210> 766
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 766
gagttcacaa 10
<210> 767
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 767
tgttctcttc 10
<210> 768
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<400> 768
taacacgacg 10
<210> 769
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(31)
<223> solution 1 adapter 1
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5phosphorylation
<400> 769
agtcggaggc caagcggtct taggaagaca a 31
<210> 770
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(40)
<223> solution 1 adapter 2
<220>
<221> misc_feature
<222> (40)..(40)
<223> 3'-OH phosphorothioate
<400> 770
ttgtcttcct aacaggaacg acatggctac gatccgactt 40
<210> 771
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(64)
<223> solution 1 primer1
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5'phosphorylation
<220>
<221> misc_feature
<222> (21)..(30)
<223> library tag sequence
<400> 771
ctctcagtac gtcagcagtt nnnnnnnnnn caactccttg gctcacagaa cgacatggct 60
acga 64
<210> 772
<211> 52
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(52)
<223> solution 1 primer2
<220>
<221> misc_feature
<222> (19)..(28)
<223> library tag sequence
<400> 772
gcatggcgac cttatcagnn nnnnnnnntt gtcttcctaa gaccgcttgg cc 52
<210> 773
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(35)
<223> solution 2 adapter 1
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5'phosphorylation
<400> 773
agtcggaggc caagcggtct taggaagaca atcag 35
<210> 774
<211> 59
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(59)
<223> solution 2 adapter 2
<400> 774
ctgattgtct tcctaagcaa ctccttggct cacagaacga catggctacg atccgactt 59
<210> 775
<211> 51
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(51)
<223> solution 2 primer1
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5'phosphorylation
<220>
<221> misc_feature
<222> (21)..(30)
<223> library tag sequence
<400> 775
ctctcagtac gtcagcagtt nnnnnnnnnn caactccttg gctcacagaa c 51
<210> 776
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(50)
<223> solution 2 primer2
<220>
<221> misc_feature
<222> (19)..(28)
<223> library tag sequence
<400> 776
gcatggcgac cttatcagnn nnnnnnnntt gtcttcctaa gaccgcttgg 50
<210> 777
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI001tag1
<400> 777
atgcatctaa 10
<210> 778
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI002tag1
<400> 778
agctctggac 10
<210> 779
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI003tag1
<400> 779
ctatcacgtg 10
<210> 780
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI004tag1
<400> 780
ggactagtgg 10
<210> 781
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI005tag1
<400> 781
gccaagtcca 10
<210> 782
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI006tag1
<400> 782
cctgtcaagc 10
<210> 783
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI007tag1
<400> 783
tagaggtctt 10
<210> 784
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI008tag1
<400> 784
tatggcaact 10
<210> 785
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI001tag2
<400> 785
tagaggacaa 10
<210> 786
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI002tag2
<400> 786
cctagcgaat 10
<210> 787
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI003tag2
<400> 787
gtagtcatcg 10
<210> 788
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI004tag2
<400> 788
gctgagctgt 10
<210> 789
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI005tag2
<400> 789
aacctagata 10
<210> 790
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI006tag2
<400> 790
ttgccatctc 10
<210> 791
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI007tag2
<400> 791
agatcttgcg 10
<210> 792
<211> 10
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(10)
<223> MGI008tag2
<400> 792
cgctatcggc 10
<210> 793
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(20)
<223> universial amplification sequence
<220>
<221> misc_feature
<222> (1)..(1)
<223> 5'
<400> 793
ctctcagtac gtcagcagtt 20
<210> 794
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(34)
<223> universial primer
<400> 794
caactccttg gctcacagaa cgacatggct acga 34
<210> 795
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(18)
<223> universial primer
<400> 795
gcatggcgac cttatcag 18
<210> 796
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(24)
<223> universial primer
<400> 796
ttgtcttcct aagaccgctt ggcc 24
<210> 797
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(21)
<223> universial amplification sequence
<400> 797
caactccttg gctcacagaa c 21
<210> 798
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> tag sequence for base balance
<220>
<221> misc_feature
<222> (1)..(22)
<223> universial amplification sequence
<400> 798
ttgtcttcct aagaccgctt gg 22
<---
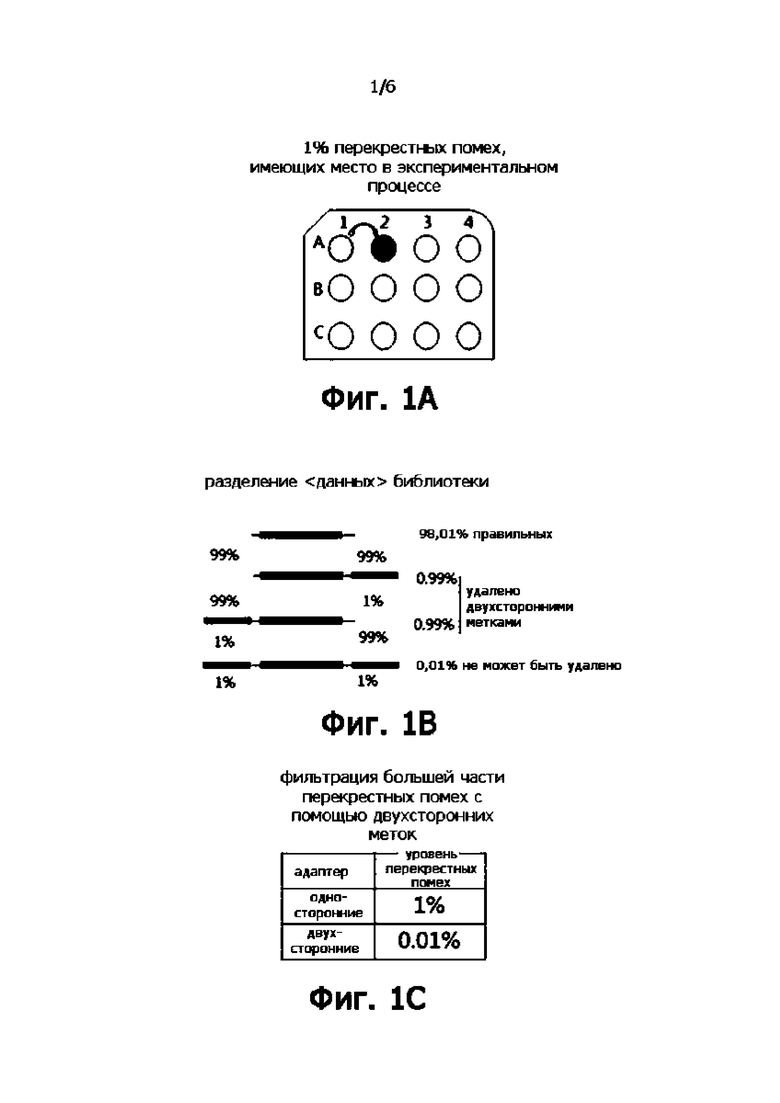
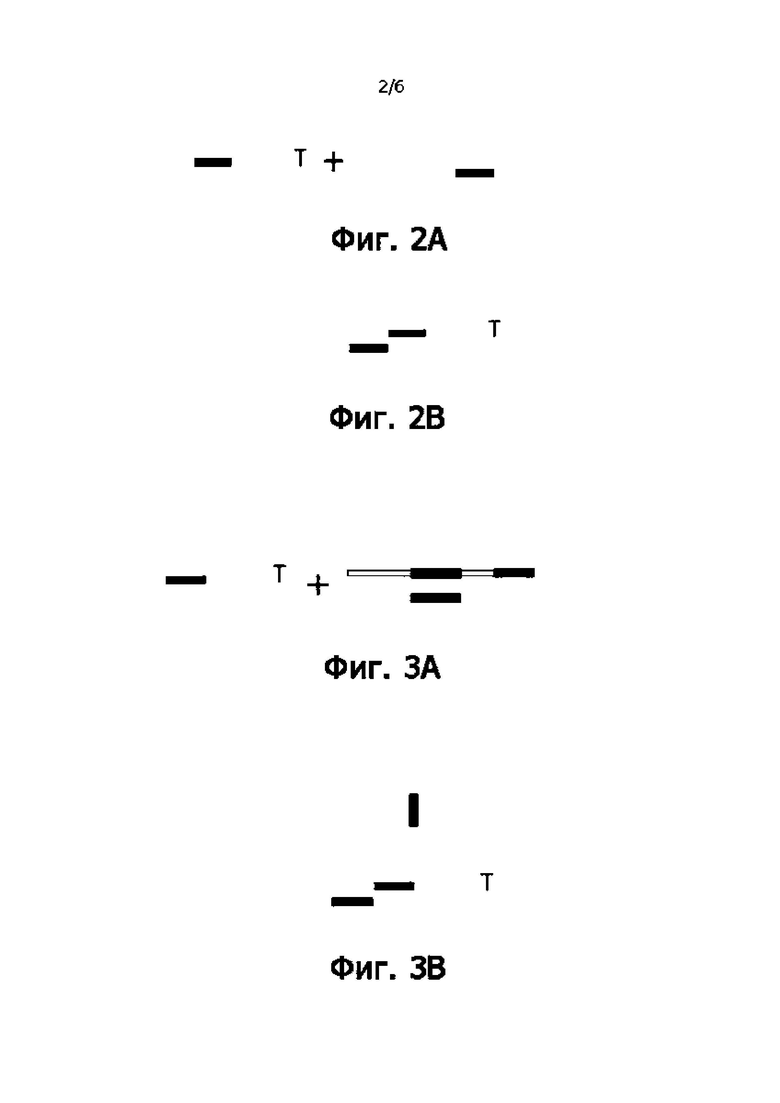
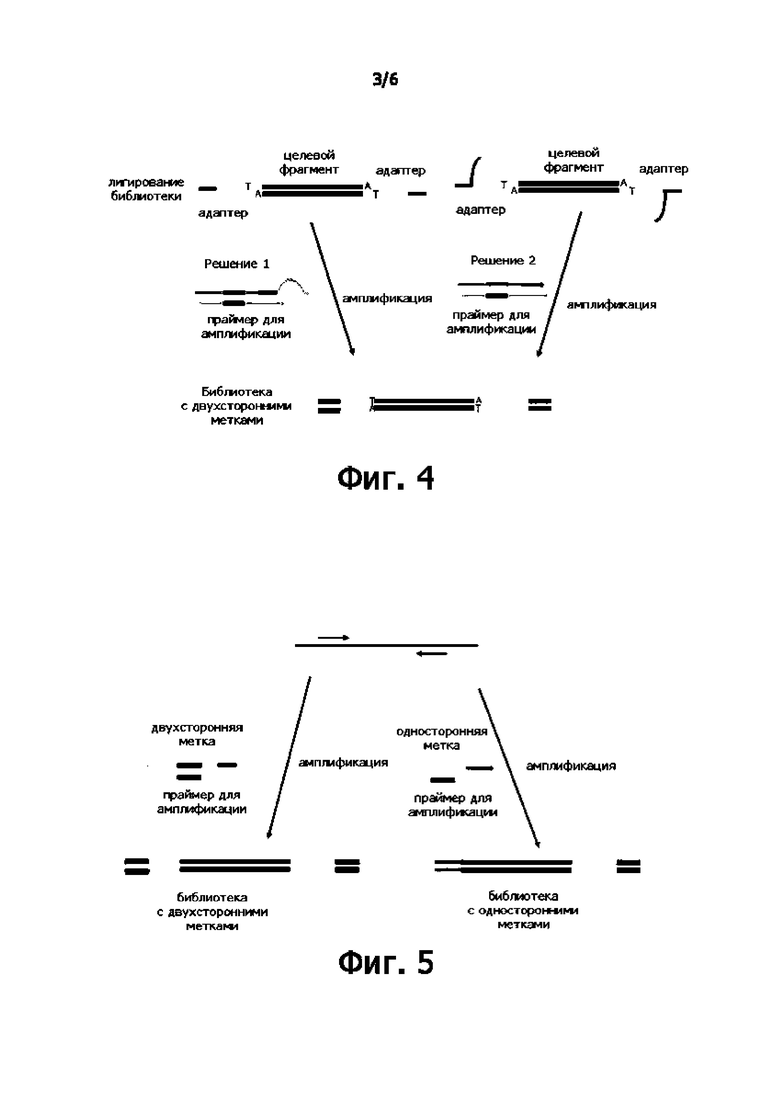
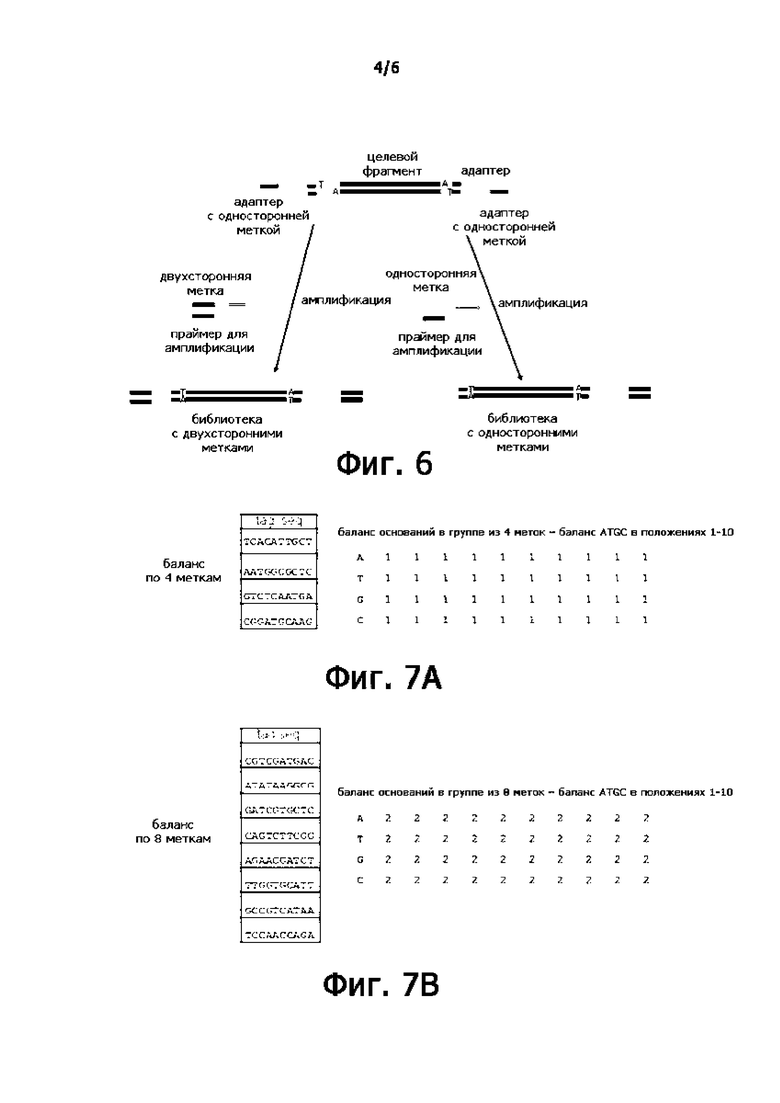


Изобретение относится к биотехнологии. Описана композиция двухсторонних библиотечных меток и ее применение на платформе секвенирования MGI. Композиция двухсторонних библиотечных меток включает множество 5'-концевых библиотечных меток и множество 3'-концевых библиотечных меток, длины множества 5'-концевых библиотечных меток одинаковы, длины множества 3'-концевых библиотечных меток одинаковы, и в композиции двухсторонних библиотечных меток частоты для каждого основания в одном и том же положении одинаковы. Использование оптимизированных двухсторонних библиотечных меток для разделения данных секвенирования может решить проблемы перекрестных помех, возникающих во время синтеза, экспериментального процесса и машинного секвенирования. Так, контролируя одинаковые длины 5'-концевых библиотечных меток, одинаковые длины для 3'-концевых библиотечных меток, и одинаковые частоты каждого основания в одном и том же положении, получают множество библиотек с хорошо сбалансированными по основаниям двусторонними метками. При объединении нескольких библиотек для секвенирования двухсторонние метки считываются более точно, эффективно повышая скорость разделения данных секвенирования библиотеки. 5 н. и 2 з.п. ф-лы, 10 ил., 7 табл., 3 пр.
1. Композиция двухсторонних библиотечных меток на основе платформы секвенирования MGI, включающая: множество 5'-концевых библиотечных меток и множество 3'-концевых библиотечных меток, длины множества 5'-концевых библиотечных меток одинаковы, длины множества 3'-концевых библиотечных меток одинаковы, и частоты для каждого основания в одном и том же положении одинаковы;
где длины 5'-концевых библиотечных меток одинаковы с длинами 3'-концевых библиотечных меток;
в композиции двухсторонних библиотечных меток есть не менее 3 отличий оснований между любыми двумя библиотечными метками, а количество непрерывных одинаковых оснований в любой библиотечной метке не превышало 3;
содержание GC во всех библиотечных метках составляет 40-60%;
где композиция двухсторонних библиотечных меток содержит комбинацию из двухсторонних библиотечных меток, сбалансированных по 4, или комбинацию из двухсторонних библиотечных меток, сбалансированных по 8, где комбинация из двухсторонних библиотечных меток, сбалансированных по 4, содержит 4n 5'-концевых библиотечных меток и 4n 3'-концевых библиотечных меток, а комбинация двухсторонних библиотечных меток, сбалансированных по 8, включает 8n 5'-концевых библиотечных меток и 8n 3'-концевых библиотечных меток, где n представляет собой целое число, большее или равное 1;
где в комбинации двухсторонних библиотечных меток, сбалансированных по 4, 5'-концевые библиотечные метки выбраны из любой одной или нескольких из 96 групп, показанных в таблице 1, и 3'-концевые библиотечные метки выбраны из любой одной или нескольких из 96 групп, показанных в таблице 1, которые отличаются от 5'-концевых библиотечных меток;
где в комбинации из двухсторонних библиотечных меток, сбалансированных по 8, 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, а 3'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, которые отличаются от 5'-концевых библиотечных меток.
2. Композиция праймеров для амплификации с двусторонними библиотечными метками на основе платформы секвенирования MGI, содержащая:
множество пар праймеров для амплификации с двусторонними библиотечными метками, каждая пара праймеров для амплификации содержит 5'-концевую библиотечную метку и 3'-концевую библиотечную метку,
при этом все длины множества 5'-концевых библиотечных меток в парах праймеров для амплификации одинаковы, и все длины множества 3'-концевых библиотечных меток в парах праймеров для амплификации одинаковы, и частоты каждого основания в одном и том же положении тоже все одинаковы;
где все длины множества 5'-концевых библиотечных меток в парах праймеров для амплификации совпадают с длинами множества 3'-концевых библиотечных меток в парах праймеров для амплификации;
в составе есть не менее 3 отличий оснований между любыми двумя библиотечными метками, а количество непрерывных одинаковых оснований в любой библиотечной метке не превышало 3;
содержание GC во всех библиотечных метках составляет 40-60%;
композиция содержит комбинацию 4n сбалансированных по 4 пар праймеров для амплификации или комбинацию 8n сбалансированных по 8 пар праймеров для амплификации, где n представляет собой целое число, большее или равное 1;
в комбинации 4n пар праймеров для амплификации, сбалансированных по 4, 5'-концевые библиотечные метки выбраны из любой одной или нескольких из 96 групп, показанных в таблице 1, а 3'-концевые библиотечные метки выбраны из любой одной или более из 96 групп, показанных в таблице 1, которые отличаются от 5'-концевых библиотечных меток;
в комбинации 8n сбалансированных по 8 пар праймеров для амплификации 5'-концевые библиотечные метки выбирают из любой одной или нескольких из 48 групп, показанных в таблице 2, а 3'-концевые библиотечные метки выбирают из любой одной или больше из 48 групп, показанных в Таблице 2, которые отличаются от 5'-концевых библиотечных меток.
3. Композиция по п.2, отличающаяся тем, что каждая пара праймеров для амплификации дополнительно содержит 5'-концевую универсальную последовательность для амплификации и 3'-концевую универсальную последовательность для амплификации, при этом 5'-концевая универсальная последовательность для амплификации содержит универсальную вышележащую последовательность 5'-концевой библиотечной метки и универсальную нижележащую последовательность 5'-концевой библиотечной метки, а 3'-концевая универсальная последовательность для амплификации содержит универсальную вышележащую последовательность 3'-концевой библиотечной метки и универсальную нижележащую последовательность 3'-концевой библиотечной метки;
где универсальная вышележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 793, а универсальная нижележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 794; универсальная вышележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 795, а универсальная нижележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 796; или
универсальная вышележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 793, а универсальная нижележащая последовательность 5'-концевой библиотечной метки представляет собой SEQ ID NO: 797; универсальная вышележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 795, а универсальная нижележащая последовательность 3'-концевой библиотечной метки представляет собой SEQ ID NO: 798.
4. Набор для создания библиотеки для секвенирования, содержащий композицию праймеров для амплификации по любому из пп. 2, 3.
5. Набор по п. 4, отличающийся тем, что набор дополнительно содержит «пузырьковые» адаптеры, при этом «пузырьковые» адаптеры содержат первую последовательность адаптера и вторую последовательность адаптера, первая последовательность адаптера представляет собой SEQ ID NO: 769, а вторая последовательность адаптера представляет собой SEQ ID NO: 770 или
первая последовательность адаптера представляет собой SEQ ID NO: 773, а вторая последовательность адаптера представляет собой SEQ ID NO: 774.
6. Способ создания библиотеки, предназначенной для секвенирования на основе платформы секвенирования MGI, включающий применение набора по п. 4 или 5 для конструирования указанной библиотеки.
7. Библиотека для секвенирования, содержащая композицию двусторонних библиотечных меток по п. 1.
| CN 111534518 A, 14.08.2020 | |||
| Способ вулканизации галош | 1932 |
|
SU34141A1 |
| АРТЕМ НЕДОЛУЖКО, ОЛЬГА ПТАШНИК, 12 методов в картинках: секвенирование нуклеиновых кислот, Биомолекула, 11 АВГУСТА 2017, найдено в интернет 06.09.2023 https://biomolecula.ru/articles/metody-v-kartinkakh-sekvenirovanie-nukleinovykh-kislot. | |||
Авторы
Даты
2024-03-28—Публикация
2020-12-28—Подача