ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННУЮ ЗАЯВКУ
[0001] Данная заявка заявляет приоритет по предварительной заявке США № 62/701065, поданной 20 июля 2018 г., и предварительной заявке США № 62/844958, поданной 8 мая 2019 г., содержание которых включено в данный документ в полном объеме посредством ссылки.
ОБЛАСТЬ ТЕХНИКИ ИЗОБРЕТЕНИЯ
[0002] Предложены анти-CD112R антитела, а также их применение для усиления, повышения и поддержания противоопухолевого иммунного ответа, лечения рака и усиления взаимодействий CD226 с CD112.
УРОВЕНЬ ТЕХНИКИ ИЗОБРЕТЕНИЯ
[0003] Звенья и врожденной, и адаптивного иммунной системы используют узкоспециализированные иммунные клетки для инспекции организма в поисках признаков злокачественного новообразования. Врожденный иммунитет обеспечивает первую линию защиты и быстрый ответ с использованием таких механизмов, как барьеры и деструктивные пептиды, которые являются неспецифическими и существуют в природе. Природные клетки-киллеры (NK) представляют собой тип лимфоцитов, которые являются частью врожденной иммунной системы и могут распознавать и уничтожать инфицированные вирусом и опухолевые клетки, используя гранзимы, хранящиеся в их цитоплазме.
[0004] Адаптивный иммунитет развивается со временем в ответ на антиген и обеспечивает длительный иммунитет. Цитотоксические лимфоциты (ЦТЛ), также известные как CD8+ T-клетки, являются частью адаптивного иммунного ответа, поскольку они распознают антигены вируса и опухоли, представленные антигенпрезентирующими клетками (АПК). ЦТЛ активируются при взаимодействии с АПК, такой как дендритная клетка или макрофаг. АПК представляет опухолевые антигены в контексте молекул ГКГС к T-клеточному рецептору (ТКР) на поверхности T-клеток. Во время этого когнатного взаимодействия АПК обеспечивает костимулирующий сигнал, который приводит к активации Т-клеток, пролиферации Т-клеток и уменьшению или устранению клеток, экспрессирующих антиген, посредством цитотоксических механизмов.
[0005] Применение анти-CD112R иммунотерапии дает возможность повышать, усиливать и поддерживать иммунные ответы. CD112R представляет собой ингибирующий рецептор, в первую очередь экспрессируемый Т-клетками и NK-клетками, и который конкурирует за связывание CD112 с активирующим рецептором CD226. Взаимодействие CD112 с CD112R имеет более высокую аффинность, чем с CD226, и тем самым эффективно регулирует активацию клеток, опосредованную CD226. Анти-CD112R антитела, которые блокируют взаимодействие с CD112, ограничивают подавляющую передачу сигналов непосредственно нижестоящему CD112R, одновременно способствуя большей активации иммунных клеток за счет увеличения взаимодействий CD226 с CD112. В исследованиях in vitro было показано, что анти-CD112R антитела увеличивают пролиферацию, активацию и цитотоксичность иммунных эффекторных клеток.
[0006] Экспрессия мРНК CD112R обнаруживается в ряде раковых тканей и основана на прогнозном анализе с использованием набора данных TCGA (Атлас ракового генома). Его экспрессия наиболее сильна в опухолях, обогащенных Т- и NK-клетками. Помимо экспрессии на миелоидных клетках, экспрессия лиганда CD112R, CD112, обычно повышается на опухолевых клетках различного происхождения. Учитывая эти обстоятельства, взаимодействие CD112R с инфильтрирующими опухоль иммунными клетками имеет сильный потенциал для негативной регуляции местных иммунных ответов в микроокружении опухоли.
[0007] Таким образом, терапевтическое лечение анти-CD112R антителами дает возможность снизить подавляющую передачу сигналов, которая предположительно возникает, когда иммунные клетки, экспрессирующие CD112R, взаимодействуют с CD112 на опухолевых клетках и/или миелоидных клетках в микроокружении опухоли и могут усиливать, повышать и поддерживать противоопухолевые иммунные ответы.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[0008] В некоторых вариантах осуществления предложено выделенное анти-CD112R антитело. Такое выделенное анти-CD112R антитело связывается с CD112R человека, при этом указанное антитело блокирует связывающее взаимодействие между CD112 человека и CD112R человека и не блокирует связывающее взаимодействие между мышиным CD112 и мышиным CD112R, причем указанное антитело необязательно полностью человеческое или гуманизированное.
[0009] В некоторых вариантах осуществления в настоящем изобретении предложено выделенное антитело, содержащее:
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 5; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 6; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 101; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 102; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 103; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 104; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 105; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 106; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 201; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 202; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 203; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 204; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 205; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 206; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 301; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 302; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 303; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 304; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 305; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 306; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 401; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 402; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 403; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 404; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 405; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 406; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 501; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 502; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 503; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 504; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 505; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 506; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 601; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 602; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 603; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 604; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 605; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 606; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 701; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 702; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 703; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 704; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 705; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 706; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 801; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 802; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 803; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 804; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 805; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 806; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 901; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 902; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 903; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 904; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 905; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 906; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 2003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 2006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 3001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 3002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 3004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 3005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 4003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 4006.
[0010] В некоторых вариантах осуществления антитело содержит вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL), при этом:
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 12 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 18; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 112 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 118; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 212 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 218; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 312 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 318; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 412 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 418; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 512 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 518; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 612 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 618; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 712 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 718; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 812 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 818; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 912 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 918; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 1012 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 1018; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 2012 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 2018; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 3012 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 3018; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 4012 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 4018.
[0011] В некоторых вариантах осуществления антитело содержит шесть CDR (HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3), как описано в данном документе, и последовательности VH и/или VL, которые по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичны аминокислотной последовательности VH и/или VL, описанной в данном документе. В некоторых вариантах осуществления последовательности VH и/или VL не на 100% идентичны аминокислотной последовательности, описанной в данном документе. В некоторых вариантах осуществления антитело содержит аминокислотные последовательности HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, описанные в настоящем документе, и вариацию последовательности в последовательностях VH и/или VL, которая находится за пределами последовательностей CDR. В таких вариантах осуществления вариация последовательностей VH и/или VL находится в пределах одной или более каркасных областей VH и/или VL.
[0012] В некоторых вариантах осуществления антитело содержит последовательности VH и/или VL, которые по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100%. идентичны аминокислотной последовательности, описанной в данном документе. В некоторых вариантах осуществления последовательности VH и/или VL не на 100% идентичны аминокислоте, описанной в данном документе. В таких вариантах осуществления вариация последовательностей VH и/или VL находится в и/или за пределами последовательностей CDR, если не указано иное.
[0013] В некоторых вариантах осуществления антитело содержит вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL), при этом:
VH содержит аминокислотную последовательность SEQ ID NO: 12, а VL содержит аминокислотную последовательность SEQ ID NO: 18; или
VH содержит аминокислотную последовательность SEQ ID NO: 112, а VL содержит аминокислотную последовательность SEQ ID NO: 118; или
VH содержит аминокислотную последовательность SEQ ID NO: 212, а VL содержит аминокислотную последовательность SEQ ID NO: 218; или
VH содержит аминокислотную последовательность SEQ ID NO: 312, а VL содержит аминокислотную последовательность SEQ ID NO: 318; или
VH содержит аминокислотную последовательность SEQ ID NO: 412, а VL содержит аминокислотную последовательность SEQ ID NO: 418; или
VH содержит аминокислотную последовательность SEQ ID NO: 512, а VL содержит аминокислотную последовательность SEQ ID NO: 518; или
VH содержит аминокислотную последовательность SEQ ID NO: 612, а VL содержит аминокислотную последовательность SEQ ID NO: 618; или
VH содержит аминокислотную последовательность SEQ ID NO: 712, а VL содержит аминокислотную последовательность SEQ ID NO: 718; или
VH содержит аминокислотную последовательность SEQ ID NO: 812, а VL содержит аминокислотную последовательность SEQ ID NO: 818; или
VH содержит аминокислотную последовательность SEQ ID NO: 912, а VL содержит аминокислотную последовательность SEQ ID NO: 918; или
VH содержит аминокислотную последовательность SEQ ID NO: 1012, а VL содержит аминокислотную последовательность SEQ ID NO: 1018; или
VH содержит аминокислотную последовательность SEQ ID NO: 2012, а VL содержит аминокислотную последовательность SEQ ID NO: 2018; или
VH содержит аминокислотную последовательность SEQ ID NO: 3012, а VL содержит аминокислотную последовательность SEQ ID NO: 3018; или
VH содержит аминокислотную последовательность SEQ ID NO: 4012, а VL содержит аминокислотную последовательность SEQ ID NO: 4018.
[0014] В некоторых вариантах осуществления предложены анти-CD112R антитела, которые активируют NK-клетки. В некоторых вариантах осуществления предложены анти-CD112R антитела, которые повышают регуляцию CD137 на NK-клетках. В некоторых вариантах осуществления анти-CD112R антитела, которые активируют NK-клетки и/или повышают регуляцию CD137 на NK-клетках, представляют собой любые антитела, описанные в Таблице последовательностей, такие как, например, антитела 32, 33, 34, 35 и 36. В некоторых вариантах осуществления анти-CD112R антитела, которые активируют NK-клетки и/или повышают регуляцию CD137 на NK-клетках, содержат шесть CDR антител 32, 33, 34, 35 и 36, соответственно (см. Таблицу последовательностей).
[0015] В некоторых вариантах осуществления антитело представляет собой моноклональное антитело. В некоторых вариантах осуществления антитело представляет собой гуманизированное антитело. В некоторых вариантах осуществления антитело представляет собой полностью человеческое антитело. В некоторых вариантах осуществления антитело представляет собой фрагмент антитела, выбранный из Fab, Fab', Fv, scFv или (Fab')2. В некоторых вариантах осуществления антитело представляет собой полноразмерное антитело. В некоторых вариантах осуществления выделенное антитело содержит Fc-область IgG1, IgG2, IgG3 или IgG4. В некоторых вариантах осуществления в описании предложена композиция, содержащая раскрытое в данном документе антитело и фармацевтически приемлемый носитель.
[0016] В некоторых вариантах осуществления антитело усиливает дегрануляцию NK-клеток, увеличивает активацию NK-клеток, увеличивает активацию внутриопухолевых NK-клеток при их представлении в комбинации с анти-TIGIT антителом, ингибирует рост опухоли in vivo и/или предотвращает приживление опухоли при повторном заражении опухолью. В некоторых таких вариантах осуществления антитело содержит константную область тяжелой цепи IgG1 человека, и антитело увеличивает дегрануляцию NK-клеток, увеличивает активацию NK-клеток, увеличивает активацию внутриопухолевых NK-клеток при их представлении в комбинации с анти-TIGIT антителом, ингибирует рост опухоли in vivo и/или предотвращает приживление опухоли при повторном заражении опухолью по сравнению с идентичным в остальном антителом, содержащим константную область тяжелой цепи IgG человека другого изотипа.
[0017] В некоторых вариантах осуществления в описании предложен способ усиления, повышения и/или поддержания противоопухолевого иммунного ответа у субъекта, включающий введение антитела или композиции, описанной в данном документе, субъекту, имеющему опухоль.
[0018] В некоторых вариантах осуществления в описании предложен способ лечения рака у субъекта, включающий введение антитела или композиции, описанной в данном документе, субъекту, имеющему рак. В некоторых вариантах осуществления рак представляет собой карциному, лимфому, бластому, саркому или лейкоз. В некоторых вариантах осуществления рак представляет собой плоскоклеточный рак, мелкоклеточный рак легкого, рак гипофиза, рак пищевода, астроцитому, саркому мягких тканей, немелкоклеточный рак легкого (в том числе плоскоклеточный немелкоклеточный рак легкого), аденокарциному легкого, плоскоклеточную карциному легкого, рак брюшины, гепатоцеллюлярный рак, рак желудочно-кишечного тракта, рак поджелудочной железы, глиобластому, рак шейки матки, рак яичников, рак печени, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки, рак толстой и прямой кишок, карциному эндометрия или матки, карциному слюнной железы, рак почки, почечно-клеточную карциному, рак печени, рак предстательной железы, рак влагалища, рак щитовидной железы, карциному печени, рак головного мозга, рак эндометрия, рак яичка, холангиокарциному, рак желчного пузыря, рак желудка, меланому или различные типы рака головы и шеи (в том числе плоскоклеточную карциному головы и шеи).
[0019] В некоторых вариантах осуществления в описании предложен способ усиления взаимодействий CD226 с CD112 у субъекта, включающий введение субъекту антитела или композиции, описанной в данном документе.
[0020] В некоторых вариантах осуществления в описании предложен способ усиления активации CD8 Т-клеток у субъекта, включающий введение антитела или композиции, описанной в данном документе, субъекту, нуждающемуся в активации CD8 Т-клеток.
[0021] В некоторых вариантах осуществления в описании предложен способ усиления продукции гамма-интерферона CD8 Т-клетками у субъекта, включающий введение антитела или композиции, описанной в настоящем документе, субъекту, нуждающемуся в продукции гамма-интерферона CD8 Т-клетками.
[0022] В некоторых вариантах осуществления в описании предложен способ усиления активации NK-клеток у субъекта, включающий введение антитела или композиции, описанной в данном документе, субъекту, нуждающемуся в активации NK-клеток.
[0023] В некоторых вариантах осуществления в описании предложен способ усиления цитотоксичности, опосредованной NK-клетками, у субъекта, включающий введение антитела или композиции, описанной в данном документе, субъекту, нуждающемуся в повышении цитотоксичности, опосредованной NK-клетками.
[0024] В некоторых вариантах осуществления способы, описанные в настоящем документе, дополнительно включают введение второй терапии. В некоторых вариантах осуществления вторая терапия представляет собой лучевую терапию, хирургическое вмешательство или введение второго агента. В некоторых вариантах осуществления вторая терапия представляет собой второй агент. В некоторых таких вариантах осуществления второй агент представляет собой антагонист PD-1, PD-L1, CTLA-4, Lag-3 или TIM-3. В некоторых вариантах осуществления второй агент представляет собой антагонист TIGIT или CD96. В некоторых вариантах осуществления второй агент представляет собой антагонист PVRL1, PVRL2, PVRL3, PVRL4 или CD155. В некоторых вариантах осуществления второй агент представляет собой антагонист CD47, CD39 или IL-27. В некоторых вариантах осуществления второй агент представляет собой агонист STING.
[0025] В некоторых вариантах осуществления в описании предложено применение антитела или композиции, описанной в настоящем документе, для усиления и/или увеличения и/или поддержания противоопухолевого иммунитета и/или лечения рака и/или усиления взаимодействий CD226 с CD112.
[0026] В некоторых вариантах осуществления в описании предложено применение антитела или композиции, описанной в настоящем документе, для приготовления лекарственного средства для усиления и/или увеличения и/или поддержания противоопухолевого иммунного ответа и/или лечения рака и/или усиления взаимодействий CD226 с CD112.
[0027] В некоторых вариантах осуществления в описании предложена нуклеиновая кислота, кодирующая антитело, описанное в данном документе.
[0028] В некоторых вариантах осуществления в описании предложена клетка-хозяин, содержащая нуклеиновую кислоту, кодирующую антитело, описанное в данном документе.
[0029] В некоторых вариантах осуществления в описании предложен способ получения описанного в данном документе антитела, включающий культивирование клетки-хозяина, содержащей нуклеиновую кислоту, кодирующую антитело, описанное в данном документе, при этом клетка-хозяин культивируется в условиях, при которых экспрессируется антитело. В некоторых вариантах осуществления способ дополнительно включает очищение антитела.
[0030] Иллюстративные варианты осуществления изобретения включают следующее:
Варианты осуществления 1. Выделенное анти-CD112R антитело, которое связывается с CD112R человека, при этом указанное антитело блокирует связывающее взаимодействие между CD112 человека и CD112R человека и не блокирует связывающее взаимодействие между мышиным CD112 и мышиным CD112R, причем указанное антитело необязательно полностью человеческое или гуманизированное.
Варианты осуществления 2. Выделенное антитело по варианту осуществления 1, в котором выделенное антитело содержит:
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 701; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 702; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 703; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 704; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 705; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 706; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 2003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 2006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 3001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 3002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 3004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 3005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 4003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 4006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1001 с 1, 2 или 3 аминокислотными заменами в положениях 4, 5 и/или 6 SEQ ID NO: 1001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1002 с 1, 2, 3, 4 или 5 аминокислотными заменами в положениях 1, 3, 5, 6 и/или 8 SEQ ID NO: 1002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1006.
Варианты осуществления 3. Выделенное антитело по варианту осуществления 1 или 2, в котором указанное антитело содержит вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL), при этом:
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 712, и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 718; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 1012, и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 1018; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 2012, и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 2018; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 3012, и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 3018; или
VH по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 4012 и VL по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 4018, необязательно при условии, что если какая-либо вариация последовательности присутствует в CDR, такая вариация последовательности находится в пределах HCDR1 или HCDR2 с не более чем 3-мя аминокислотными заменами, например, не более чем 2-мя аминокислотными заменами в положениях 4, 5 и/или 6 HCDR1 и с не более чем 5-ю аминокислотными заменами, например, не более чем 2-мя аминокислотными заменами в положениях 1, 3, 5, 6 и/или 8 HCDR2, необязательно, при этом вариация не находится в пределах HCDR3, LCDR1, LCDR2 и LCDR3.
Варианты осуществления 4. Выделенное антитело по любому из вариантов осуществления 1-3, в котором антитело содержит вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL), при этом:
VH содержит аминокислотную последовательность SEQ ID NO: 712 и VL содержит аминокислотную последовательность SEQ ID NO: 718; или
VH содержит аминокислотную последовательность SEQ ID NO: 1012 и VL содержит аминокислотную последовательность SEQ ID NO: 1018; или
VH содержит аминокислотную последовательность SEQ ID NO: 2012 и VL содержит аминокислотную последовательность SEQ ID NO: 2018; или
VH содержит аминокислотную последовательность SEQ ID NO: 3012 и VL содержит аминокислотную последовательность SEQ ID NO: 3018; или
VH содержит аминокислотную последовательность SEQ ID NO: 4012 и VL содержит аминокислотную последовательность SEQ ID NO: 4018.
Варианты осуществления 5. Выделенное антитело по любому из предыдущих вариантов осуществления, в котором указанное антитело представляет собой моноклональное антитело.
Варианты осуществления 6. Выделенное антитело по любому из предыдущих вариантов осуществления, в котором антитело представляет собой полноразмерное антитело или фрагмент антитела, необязательно Fab, Fab', Fv, scFv или (Fab')2.
Варианты осуществления 7. Выделенное антитело по любому из вариантов осуществления 1-5, в котором антитело содержит Fc-область IgG1, IgG2, IgG3 или IgG4, при этом антитело необязательно содержит константную область тяжелой цепи IgG1 человека, константную область тяжелой цепи IgG4 человека или мутантную константную область тяжелой цепи IgG4 человека, при этом мутантная константная область тяжелой цепи IgG4 человека необязательно содержит мутацию, выбранную из замены в Ser228, замены в Leu235, замены в Asn297 или их комбинации, нумерация в соответствии с нумерацией EU, или замена S228P и замена L235E, нумерация в соответствии с нумерацией EU.
Варианты осуществления 8. Выделенное антитело по любому из вариантов осуществления 1-7, в котором антитело
увеличивает дегрануляцию NK-клеток; и/или
увеличивает активацию NK-клеток; и/или
увеличивает активацию внутриопухолевых NK-клеток при представлении в комбинации с анти-TIGIT антителом; и/или
подавляет рост опухоли in vivo; и/или
предотвращает приживление опухоли при повторном заражении опухолью, необязательно, при этом антитело представляет собой IgG1 или IgG4.
Варианты осуществления 9. Фармацевтическая композиция, содержащая антитело по любому из вариантов осуществления 1-8 и фармацевтически приемлемый носитель, при этом композиция необязательно содержит опсонизирующий агент, агент, истощающий популяцию регуляторных Т-клеток, химиотерапию и/или антагонист PD-1, PD- L1, CTLA-4, Lag-3 или TIM-3.
Варианты осуществления 10. Выделенное антитело по любому из вариантов осуществления 1-8 или фармацевтическая композиция по варианту осуществления 9 для применения в усилении, повышении и/или поддержании противоопухолевого иммунного ответа у субъекта, необязательно, при этом у субъекта усиливается активация CD8 Т-клеток или увеличивается продукция гамма-интерферона CD8 Т-клетками, или, необязательно, при этом у субъекта усиливается активация NK-клеток, или у субъекта усиливается цитотоксичность, опосредованная NK-клетками, или, необязательно, при этом у субъекта усиливается взаимодействия CD226 с CD112.
Варианты осуществления 11. Выделенное антитело по любому из вариантов осуществления 1-8 или фармацевтическая композиция по варианту осуществления 9 для применения при лечении рака у субъекта, при этом рак, необязательно, представляет собой карциному, лимфому, бластому, саркому или лейкемию, или при этом рак, необязательно, представляет собой плоскоклеточный рак, мелкоклеточный рак легкого, рак гипофиза, рак пищевода, астроцитому, саркому мягких тканей, немелкоклеточный рак легкого (в том числе плоскоклеточный немелкоклеточный рак легкого), аденокарциному легкого, плоскоклеточную карциному легкого, рак брюшины, гепатоцеллюлярный рак, рак желудочно-кишечного тракта, рак поджелудочной железы, глиобластому, рак шейки матки, рак яичников, рак печени, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки, рак толстой и прямой кишок, карциному эндометрия или матки, карциному слюнной железы, рак почки, почечно-клеточную карциному, рак печени, рак предстательной железы, рак влагалища, рак щитовидной железы, карциному печени, рак головного мозга, рак эндометрия, рак яичка, холангиокарциному, рак желчного пузыря, рак желудка, меланому или различные типы рака головы и шеи (в том числе плоскоклеточную карциному головы и шеи).
Варианты осуществления 12. Выделенное антитело или фармацевтическая композиция для применения в соответствии с вариантом осуществления 10 или 11, при этом применение дополнительно включает введение второй терапии, причем вторая терапия необязательно представляет собой лучевую терапию, хирургическое вмешательство или введение второго агента, где второй агент необязательно является антагонистом PD-1, PD-L1, CTLA-4, Lag-3 или TIM-3, или антагонистом TIGIT или CD96, или антагонистом PVRL1, PVRL2, PVRL3, PVRL4 и CD155, или антагонистом CD47, или является антагонистом CD39, или является антагонистом IL-27, или является агонистом STING, при этом второй агент необязательно представляет собой антагонистическое антитело.
Варианты осуществления 13. Нуклеиновая кислота, кодирующая антитело по любому из вариантов осуществления 1-8.
Варианты осуществления 14. Клетка-хозяин, содержащая нуклеиновую кислоту по варианту осуществления 13.
Варианты осуществления 15. Способ получения антитела по любому из вариантов осуществления 1-8, включающий культивирование клетки-хозяина согласно варианту осуществления 14 в условиях, при которых экспрессируется антитело, необязательно дополнительно включающий очищение антитела.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
[0031] На Фиг. 1 показана способность анти-CD112R антител по сравнению с контрольным антителом изотипа IgG1 связываться с клетками Jurkat, сконструированными для сверхэкспрессии CD112R человека. Интенсивность связывания оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) метки антитела Alexa Fluor® 647.
[0032] На Фиг. 2A-2B показана способность анти-CD112R антител по сравнению с контрольным антителом изотипа IgG1 блокировать взаимодействие CD112R с CD112 на клетках Jurkat, сконструированных для сверхэкспрессии CD112R человека. На Фиг. 2А, клетки предварительно инкубировали либо с изотипическим контролем IgG1, либо с анти-CD112R антителом. После промывки клетки одновременно окрашивали биотинилированным his-меченным CD112 человека и стрептавидином-PE. Способность анти-CD112R антител блокировать связывание CD112 с CD112R оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) PE-метки и отображали как процент ингибирования. Процент ингибирования рассчитывали как [100 - ((MFI тестового образца/Макс. MFI) * 100%)]. На Фиг. 2B показан процент ингибирования, измеренный с помощью ИФА, взаимодействия между CD112R человека и CD112 с помощью описанных в данном документе анти-CD112R антител.
[0033] На Фиг. 3 показана усиленная цитотоксичность, опосредованная Nk-клетками человека, против клеток REH (линия клеток лейкемии человека) в присутствии анти-CD112R антител по сравнению с контрольным антителом изотипа IgG1. Активированные NK-клетки и клетки REH, меченные CellTrace violet, культивировали совместно в течение четырех часов. После совместного культивирования жизнеспособность клеток REH оценивали путем окрашивания 7-AAD. Цитотоксичность (процент относительно изотипического контроля) рассчитывали как ((процент мертвых клеток с тестовым антителом минус процент мертвых клеток с антителом изотипического контроля), деленный на процент мертвых клеток с антителом изотипического контроля) x 100.
[0034] На Фиг. 4 показана усиленная антиген-управляемая активация CD8+ Т-клеток, измеренная посредством секреции ИФН-γ, в присутствии анти-CD112R антител по сравнению с контрольным антителом изотипа IgG1. Клетки Colo205 обрабатывали пептидом pp65 и совместно культивировали с CMV-специфическими Т-клетками человека в присутствии анти-CD112R антитела или контрольного антитела изотипа IgG1. Уровни ИФН-γ в супернатантах культивируемых клеток измеряли с помощью Luminex. CD8+ Т-клетки, обработанные анти-CD112R антителами 2 и 5, приводили к большей секреции ИФН-γ, чем наблюдаемая с изотипическим контролем.
[0035] На Фиг. 5 показан график, показывающий, что комбинация мышиных анти-CD112R антител и мышиных анти-TIGIT антител оказывает терапевтический эффект на мышиной сингенной модели опухоли CT-26. Мышей с опухолью рандомизировали на четыре группы и дважды в неделю в течение двух недель им вводили внутрибрюшинно инъекцию 1) антитела изотипического контроля; 2) анти-TIGIT антитела; 3) анти-CD112R антитела; или 4) анти-TIGIT антитела в сочетании с анти-CD112R антителом. Средние объемы опухолей для каждой группы лечения показаны в зависимости от времени. Результаты демонстрируют, что комбинация анти-CD112R антитела с анти-TIGIT антителом была эффективной для снижения роста опухоли по сравнению с животными, получавшими изотипический контроль, в то время как монотерапия анти-CD112R антителом или анти-TIGIT антителом показала либо отсутствие активности, либо лишь умеренный эффект на снижение роста опухоли.
[0036] На Фиг. 6 показан график, изображающий повышенную экспрессию CD112R в PBMC после активации анти-CD3 антителом. РВМС человека стимулировали in vitro анти-CD3 антителом, а экспрессию CD112R оценивали с помощью проточной цитометрии. Количественное определение связывания антитела к CD112R оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) метки антитела Alexa Fluor® 647 для указанного типа клеток. CD112R показан как кратное изменение по сравнению с отрицательным контролем (FON, (gMFI CD112R, деленный на gMFI изотипического контроля)).
[0037] На Фиг. 7 показана усиленная дегрануляция, опосредованная NK-клетками, в ответ на опухолевые клетки в присутствии анти-CD112R антител с изотипом IgG1. NK-клетки человека и клетки Raji.CD112 совместно культивировали в течение четырех часов с антителом к CD107a PE в присутствии антител к CD112R. После совместного культивирования дегрануляцию NK-клеток определяли по частоте встречаемости NK-клеток, которые были CD107a-положительными.
[0038] На Фиг. 8A-8D показана усиленная активация NK-клеток в присутствии антитела к CD112R с изотипом IgG1. PBMC человека от 2 разных доноров и клетки K562 совместно культивировали в течение 16 часов в присутствии антитела к CD112R. После совместного культивирования активацию NK-клеток определяли по частоте встречаемости NK-клеток, которые были CD137-положительными. Результаты для донора 1 и донора 2 в двух независимых анализах показаны на Фиг. 8A-8D, соответственно.
[0039] На Фиг. 9 показано ингибирование роста опухоли у мышей, которых лечили анти-CD112R антителом. На Фигуре показаны сводные данные 3-х экспериментов, N=44-45 на группу. Статистический анализ был выполнен с помощью теста Манна-Уитни на 24 день после имплантации.
[0040] На Фиг. 10A-10B показано, что введение анти-CD112R антитела увеличивает общую выживаемость мышей, инокулированных опухолями CT-26, и защищает мышей от повторного заражения опухолью. На Фиг.10А показана частота выживания мышей после заражения первичной опухолью с введением анти-CD112R антитела. У выживших мышей не было пальпируемых опухолей после 50-го дня инокуляции, и считалось, что они являются мышами с полным клиническим ответом. На Фиг. 10B показано ингибирование роста опухоли у выживших мышей после повторного заражения опухолью по сравнению с наивными контрольными мышами. Статистический анализ выполняли с помощью теста Мантела-Кокса на 50-й день после имплантации (Фиг. 10A) и теста Манна-Уитни на 15-й день после имплантации (Фиг. 10B).
[0041] На Фиг. 11 показано, что эффективность блокады CD112R in vivo на мышиной модели опухоли CT26 зависит от NK-клеток и CD8 T-клеток. На Фигуре показано ингибирование роста опухоли у мышей, которых лечили анти-CD112R антителом, с одновременным истощением популяции либо NK-клеток, либо CD8 T-клеток.
[0042] На Фиг. 12A-12B показана экспрессия CD69 (Фиг. 12A) и гранзима B (Фиг. 12B) на внутриопухолевых NK-клетках в модели опухоли CT-26 после введения анти-CD112R антитела.
[0043] На Фиг. 13A-13F показаны средние (Фиг. 13A) и индивидуальные (Фиг. 13 B-E) значения измерений объема опухоли в зависимости от времени на модели опухоли CT-26 после введения только анти-CD112R антител и в комбинации с анти-PD1 антителами. На Фиг. 13F показана общая выживаемость без опухолей на 50-й день после имплантации как доля выживших без опухолей на группу.
[0044] На Фиг. 14A-14F показано выравнивание последовательностей CDR описанных в данном документе антител. На Фиг. 14A-14C показаны последовательности CDR вариабельной области тяжелой цепи, а на Фиг. 14D-14F показаны последовательности CDR вариабельной области легкой цепи. На каждой из Фиг. 14A - 14F в первом столбце показаны HCDR1 (Фиг. 14A), HCDR2 (Фиг. 14B) и HCDR3 (Фиг. 14C), LCDR1 (Фиг. 14D), LCDR2 (Фиг. 14E) и LCDR3 (Фиг. 14F), при этом номер клона антитела указан перед H1 (для HCDR1), H2 (для HCDR2), H3 (для HCDR3), L1 (для LCDR1), L2 (для LCDR2) и L3 (для LCDR3). Во втором столбце показана последовательность, в третьем столбце показано количество аминокислот в последовательности, а в последнем столбце показан процент идентичности каждой последовательности относительно последовательности клона 32 исходного антитела. Клоны 32, 33, 34, 35 и 36 членов семейства выделены жирным шрифтом.
[0045] На Фиг. 15A-15H показано выравнивание последовательностей каркасной области описанных в данном документе антител. На Фиг. 15A-15D показаны последовательности каркасной области вариабельной области тяжелой цепи, а на Фиг. 15E-15H показаны последовательности каркасной области вариабельной области легкой цепи. На каждой из Фиг. 15A - 15F в первом столбце показаны FR1 тяжелой цепи (Фиг. 15A), FR2 (Фиг. 15B), FR3 (Фиг. 15C), FR4 (Фиг. 15D) и FR1 легкой цепи (Фиг. 15E), FR2 (Фиг. 15F), а также FR3 (Фиг. 15G) и FR4 (Фиг. 15H), при этом номер клона антитела указан перед VH (для HFR1, HFR3, HFR3 и HFR4) и VL (для LFR1, LFR2, LFR3 и LFR4). Во втором столбце показана последовательность, а в последнем столбце показан процент идентичности каждой последовательности относительно последовательности из клона 32 исходного антитела. Клоны 32, 33, 34, 35 и 36 членов семейства выделены жирным шрифтом.
[0046] На Фиг. 16A-16B показано выравнивание последовательностей вариабельной области описанных в данном документе антител. На Фиг. 16A показаны последовательности вариабельной области тяжелой цепи, а на Фиг. 16B показаны последовательности вариабельной области легкой цепи. Каждая последовательность помечена соответствующим номером клона. Показан процент идентичности каждой последовательности относительно последовательности из клона 32 антитела. Клоны 32, 33, 34, 35 и 36 членов семейства выделены жирным шрифтом.
[0047] На Фиг. 17 показана степень связывания описанных в данном документе анти-CD112R антител и дополнительных анти-CD112R антител (антител A, B и C, которые связывают CD112R человека) с клетками, экспрессирующими CD112R мыши.
[0048] На Фиг. 18 показана степень связывания описанных в данном документе анти-CD112R антител и дополнительных анти-CD112R антител (антител A, B и C, которые связывают CD112R человека) с растворимым CD112R мыши.
[0049] На Фиг. 19 показано процентное значение ингибирования взаимодействия между CD11R мыши и CD112 мыши с помощью описанных в данном документе анти-CD112R антител и дополнительных анти-CD112R антител (антител A, B и C, которые связывают CD112R человека).
ПОДРОБНОЕ ОПИСАНИЕ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
ОПРЕДЕЛЕНИЯ
[0050] В данной заявке использование «или» означает «и/или», если не указано иное. В контексте пункта формулы со множественной зависимостью использование «или» относится к более чем одному предшествующему независимому или зависимому пункту формулы только в альтернативе. Термины «содержащий», «включающий» и «имеющий» могут использоваться взаимозаменяемо в данном документе.
[0051] Термины «CD112R», «PVR-связанный домен иммуноглобулина, содержащий», «рецептор CD112», «белок, содержащий домен иммуноглобулина, связанный с рецептором полиовируса», «домен иммуноглобулина, связанный с рецептором полиовируса, содержащий», «рецептор нектина-2», «C7orf15» и «трансмембранный белок PVRIG» используются взаимозаменяемо и относятся к нативному CD112R человека, если специально не указано иное (например, CD112R мыши, CD112R яванского макака и т.д.). Этот термин включает полноразмерный непроцессированный CD112R, а также любую форму CD112R, которая возникает в результате процесинга в клетке. Термин охватывает встречающиеся в природе варианты CD112R человека, например, варианты сплайсинга или аллельные варианты. Внешние идентификаторы гена CD112R включают Entrez Gene: 79037, Ensembl: ENSG00000213413, OMIM: 617012 и UniProtKB: Q6DKI7.
[0052] «Аффинность» относится к силе суммарного количества нековалентных взаимодействий между одним сайтом связывания молекулы (например, антитела) и ее партнером по связыванию (например, антигеном). Если не указано иное, в контексте данного документа «аффинность связывания» относится к действительной аффинности связывания, которая отражает взаимодействие в соотношении 1:1 между членами связывающей пары (например, антителом и антигеном). Аффинность молекулы X к ее партнеру Y обычно может быть представлена константой диссоциации (KD). Аффинность можно измерить обычными методами, известными в данной области, включая описанные в данном документе. Конкретные иллюстративные и примерные варианты осуществления для измерения аффинности связывания описаны ниже.
[0053] Антитело с «созревшей аффинностью» относится к антителу с одним или более изменениями в одной или более гипервариабельных областях (HVR) по сравнению с исходным антителом, которое не имеет таких изменений, при этом такие изменения необязательно приводят к повышению аффинности антитела к антигену.
[0054] Термин «антитело» в данном документе используется в самом широком смысле и охватывает различные структуры антител, включая, помимо прочего, моноклональные антитела, поликлональные антитела, мультиспецифические антитела (например, биспецифические антитела) и фрагменты антител, если они проявляют желаемую антигенсвязывающую активность.
[0055] «Фрагмент антитела» относится к молекуле, отличной от интактного антитела, которая включает часть интактного антитела, которая связывает антиген, с которым связывается интактное антитело. Примеры фрагментов антител включают Fv, Fab, Fab', Fab'-SH, F(ab')2; диатела; линейные антитела; молекулы одноцепочечных антител (например, scFv); и мультиспецифические антитела, образованные из фрагментов антител.
[0056] Термин «блокировать» в контексте взаимодействия между двумя или более молекулами используется в данном документе для обозначения ингибирования или предотвращения указанного взаимодействия между двумя или более молекулами, при этом ингибирование или предотвращение указанного взаимодействия между двумя или более молекулами является полным или почти полным по меньшей мере при одном условии. «Почти полное» ингибирование - это процентное ингибирование примерно на 70-99,9%, а «полное» ингибирование составляет 100%. Например, говорят, что молекула «блокирует» взаимодействие между двумя или более другими молекулами, если она полностью или почти полностью ингибирует такое взаимодействие при определенных концентрациях дозозависимым образом.
[0057] Термин «рак» применяют в данном документе для обозначения группы клеток, которые демонстрируют аномально высокие уровни пролиферации и роста. Рак может быть доброкачественным (также называемым доброкачественной опухолью), предзлокачественным или злокачественным. Раковые клетки могут представлять собой раковые клетки солидных опухолей или лейкемические раковые клетки. Термин «опухоль» используется в данном документе для обозначения клетки или клеток, которые содержат рак. Термин «рост опухоли» используется в данном документе для обозначения пролиферации или роста клетки или клеток, которые содержат рак, что приводит к соответствующему увеличению размера или степени рака.
[0058] Термин «химерное» антитело относится к антителу, в котором фрагмент тяжелой и/или легкой цепи получен из конкретного источника или вида, в то время как часть тяжелой и/или легкой цепи получены из другого источника или вида.
[0059] «Класс» антитела относится к типу константного домена или константной области, которой обладает его тяжелая цепь. Есть пять основных классов антител: IgA, IgD, IgE, IgG и IgM, и некоторые из них могут быть дополнительно разделены на подклассы (изотипы), например,IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2. Константные домены тяжелой цепи, которые соответствуют различным классам иммуноглобулинов, называются α, δ, ε, γ и μ, соответственно.
[0060] Введение «в комбинации с» одним или более дополнительными терапевтическими средствами включает одновременное (параллельное) и последовательное (непрерывное) введение в любом порядке.
[0061] Используемый в данном документе термин «цитотоксический агент» относится к веществу, которое ингибирует или предотвращает клеточную функцию и/или вызывает гибель или разрушение клеток. Цитотоксические агенты включают радиоактивные изотопы (например, At211, I131, I125, Y90, Re186, Re188, Sm153, Bi212, P32, Pb212и радиоактивные изотопы Lu); химиотерапевтические агенты или лекарственные препараты (например, метотрексат, адриамицин, алкалоиды барвинка (винкристин, винбластин, этопозид), доксорубицин, мелфалан, митомицин С, хлорамбуцил, даунорубицин или другие интеркалирующие агенты); агенты, ингибирующие рост; ферменты и их фрагменты, такие как нуклеолитические ферменты; антибиотики; токсины, такие как низкомолекулярные токсины или ферментативно активные токсины бактериального, грибкового, растительного или животного происхождения, включая их фрагменты и/или варианты; и различные противоопухолевые или противораковые средства, описанные ниже, но не ограничиваются ими.
[0062] «Эффекторные функции» относятся к биологической активности, присущей Fc-области антитела, которая варьируется в зависимости от изотипа антитела. Примеры эффекторных функций антитела включают: связывание C1q и комплемент-зависимую цитотоксичность (КЗЦ); связывание с Fc-рецептором; антителозависимую клеточно-опосредованную цитотоксичность (АЗКЦ); фагоцитоз; подавление рецепторов клеточной поверхности (например рецептора В-клеток); и активацию В-клеток.
[0063] «Эффективное количество» агента, например фармацевтического состава, относится к количеству, эффективному в дозах и в течение периодов времени, необходимых для достижения желаемого терапевтического или профилактического результата.
[0064] Термин «Fc-область» в данном документе используется для определения С-концевой области тяжелой цепи иммуноглобулина, которая содержит по меньшей мере часть константной области. Этот термин включает нативную последовательность Fc-областей и варианты Fc-областей.В некоторых вариантах осуществления Fc-область тяжелой цепи IgG человека простирается от Cys226 или от Pro230 до карбоксильного конца тяжелой цепи. Однако C-концевой лизин (Lys447) Fc-области может присутствовать, а может и не присутствовать (нумерация в этом абзаце соответствует системе нумерации EU, также называемой индексом EU, как описано в Kabat et al., Sequences of Proteins of Immunological Interest, 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD, 1991).
[0065] «Каркас», «каркасная область» или «FR» относится к остаткам вариабельного домена, отличным от остатков гипервариабельной области (HVR). FR вариабельного домена обычно состоит из четырех доменов FR: FR1, FR2, FR3 и FR4. Соответственно, последовательности HVR и FR обычно появляются в следующей последовательности в VH (или VL): FR1-H1(L1)-FR2-H2(L2)-FR3-H3(L3)-FR4.
[0066] Термины «полноразмерное антитело», «интактное антитело» и «целое антитело» используются в данном документе взаимозаменяемо для обозначения антитела, имеющего структуру, по существу аналогичную структуре нативного антитела, или имеющего тяжелые цепи, которые содержат Fc-область, как определено в данном документе.
[0067] Термины «клетка-хозяин», «линия клеток-хозяев» и «культура клеток-хозяев» используются взаимозаменяемо и относятся к клеткам, в которые была введена экзогенная нуклеиновая кислота, включая потомство таких клеток. Клетки-хозяева включают «трансформанты» и «трансформированные клетки», которые включают первичную трансформированную клетку и потомство, полученное от нее, независимо от количества пассажей. Потомство может не быть полностью идентичным по содержанию нуклеиновой кислоты родительской клетке и может содержать мутации. В данный документ включены мутантные потомки, которые обладают той же функцией или биологической активностью, что и проверенная или отобранная первоначально трансформированная клетка.
[0068] «Антитело человека» представляет собой антитело, которое имеет аминокислотную последовательность, которая соответствует таковой антитела, продуцируемого у человека или клеткой человека, или полученного из источника, отличного от человека, который использует репертуары человеческих антител или другие последовательности, кодирующие антитела человека. Это определение человеческого антитела специально исключает гуманизированное антитело, содержащее нечеловеческие антигенсвязывающие остатки.
[0069] Термин «вариабельная область» или «вариабельный домен» относится к домену тяжелой или легкой цепи антитела, который участвует в связывании антитела с антигеном. Вариабельные домены тяжелой цепи и легкой цепи (VH и VL, соответственно) нативного антитела обычно имеют сходные структуры, причем каждый домен включает четыре консервативные каркасные области (FR) и три гипервариабельные области (HVR). (См., например, Kindt et al. Kuby Immunology, 6th ed., W.H. Freeman and Co., page 91 (2007).) Одного домена VH или VL может быть достаточно для придания антигенсвязывающей специфичности. Кроме того, антитела, которые связывают конкретный антиген, могут быть выделены с использованием домена VH или VL из антитела, связывающего антиген, для скрининга библиотеки комплементарных доменов VL или VH, соответственно. См., например,, Portolano et al., J. Immunol. 150:880-887 (1993); Clarkson et al., Nature 352:624-628 (1991).
[0070] «Человеческий консенсусный каркас» представляет собой каркас, который представляет собой наиболее часто встречающиеся аминокислотные остатки при выборе последовательностей каркасных участков VL или VH иммуноглобулина человека. Как правило, выбор последовательностей VL или VH иммуноглобулина человека осуществляется из подгруппы последовательностей вариабельных доменов. Как правило, подгруппа последовательностей является подгруппой, как описано в Kabat et al., Sequences of Proteins of Immunological Interest, Fifth Edition, NIH Publication 91-3242, Bethesda MD (1991), vols. 1-3. В некоторых вариантах осуществления для VL подгруппа представляет собой подгруппу каппа I, как описано в Kabat et al., выше. В некоторых вариантах осуществления для VH подгруппа представляет собой подгруппу III, как описано в Kabat et al., выше.
[0071] Термин «гипервариабельная область» или «HVR» в контексте настоящего описания относится к каждой из областей вариабельного домена антитела, которые являются гипервариабельными по последовательности («определяющие комплементарность области» или «CDR») и/или образуют структурно определенные петли («гипервариабельные области») и/или содержат остатки, контактирующие с антигеном («контакты антигена»). Обычно антитела содержат шесть HVR: три в VH (H1, H2, H3) и три в VL (L1, L2, L3).
[0072] «Иммуноконъюгат» представляет собой антитело, конъюгированное с одной или более гетерологичными молекулами, включая цитотоксический агент, но не ограничиваясь им.
[0073] «Человек» или «субъект» - это млекопитающее. Млекопитающие включают домашних животных (например, коров, овец, кошек, собак и лошадей), приматов (например, людей и приматов, не являющихся людьми, таких как обезьяны), кроликов и грызунов (например, мышей и крыс), но не ограничиваются ими. В некоторых вариантах осуществления индивидуум или субъект является человеком.
[0074] «Выделенное» антитело - это антитело, которое было отделено от компонента его естественного окружения. В некоторых вариантах осуществления антитело очищают до чистоты более 95% или 99%, что определяется, например, электрофоретически (например, ДСН-ПААГ, изоэлектрическим фокусированием (IEF), капиллярным электрофорезом) или хроматографией (например, ионным обменом или ВЭЖХ с обратной фазой). Для обзора методов оценки чистоты антител см., например, Flatman et al., J. Chromatogr. B 848:79-87 (2007).
[0075] Термин «моноклональное антитело» в контексте настоящего описания относится к антителу, полученному из популяции по существу гомогенных антител, т.е. отдельные антитела, составляющие популяцию, идентичны и/или связываются с одним и тем же эпитопом, за исключением возможных вариантных антител, например, содержащих встречающиеся в природе мутации или возникающих во время получения препарата моноклонального антитела, такие варианты обычно присутствуют в незначительных количествах. В отличие от препаратов поликлональных антител, которые обычно включают разные антитела, направленные против разных детерминант (эпитопов), каждое моноклональное антитело в препарате моноклонального антитела направлено против одной детерминанты на антигене. Таким образом, определитель «моноклональное» указывает на характер антитела как полученного из по существу гомогенной популяции антител и не должен толковаться как требующий получения антитела каким-либо конкретным методом.Например, моноклональные антитела, которые будут использоваться в соответствии с настоящим изобретением, могут быть получены различными методами, включая, помимо прочего, гибридомный метод, методы рекомбинантной ДНК, методы фагового дисплея и методы с использованием трансгенных животных, содержащих все или часть локусов иммуноглобулина человека, такие способы и другие иллюстративные способы получения моноклональных антител, описанные в данном документе.
[0076] «Чистое антитело» относится к антителу, которое не конъюгировано с гетерологичным фрагментом (например, цитотоксическим фрагментом) или радиоактивной меткой. Чистое антитело может присутствовать в фармацевтическом составе.
[0077] «Нативные антитела» относятся к природным молекулам иммуноглобулинов с различными структурами. Например, нативные IgG-антитела представляют собой гетеротетрамерные гликопротеины примерно 150000 дальтон, состоящие из двух идентичных легких цепей и двух идентичных тяжелых цепей, связанных дисульфидной связью. От N- до C-конца каждая тяжелая цепь имеет вариабельную область (VH), также называемую вариабельным доменом тяжелой цепи, за которой следуют три константных домена (CH1, CH2 и CH3). Аналогично, от N- до С-конца каждая легкая цепь имеет вариабельную область (VL), также называемую вариабельным доменом легкой цепи, за которой следует константный домен легкой цепи (CL). Легкая цепь антитела может быть отнесена к одному из двух типов, называемых каппа (κ) и лямбда (λ), на основе аминокислотной последовательности ее константного домена.
[0078] «Процент (%) идентичности аминокислотной последовательности» по отношению к эталонной полипептидной последовательности определяется как процент аминокислотных остатков в последовательности-кандидате, которые идентичны аминокислотным остаткам в эталонной полипептидной последовательности, после выравнивания последовательностей и введения пробелов при необходимости для достижения максимального процента идентичности последовательностей, но без учета каких-либо консервативных замен как части идентичности последовательностей. Выравнивание для определения процента идентичности аминокислотной последовательности может быть достигнуто различными способами, которые известны специалистам в данной области, например, с использованием общедоступного компьютерного программного обеспечения, такого как программное обеспечение BLAST, BLAST-2, ALIGN или Megalign (DNASTAR). Специалисты в данной области техники могут определить соответствующие параметры для выравнивания последовательностей, включая любые алгоритмы, необходимые для достижения максимального выравнивания по всей длине сравниваемых последовательностей. Однако для целей настоящего описания % значения идентичности аминокислотных последовательностей получают с использованием компьютерной программы сравнения последовательностей ALIGN-2. Компьютерная программа для сравнения последовательностей ALIGN-2 была разработана Genentech, Inc., а исходный код был подан вместе с пользовательской документацией в Бюро регистрации авторских прав США, Вашингтон, округ Колумбия, 20559, где он зарегистрирован под номером регистрации авторских прав США TXU510087. Программа ALIGN-2 находится в открытом доступе от Genentech, Inc., Южный Сан-Франциско, Калифорния, или может быть скомпилирована из исходного кода. Программа ALIGN-2 должна быть скомпилирована для использования в операционной системе UNIX, включая цифровой UNIX V4.0D. Все параметры сравнения последовательностей задаются программой ALIGN-2 и не меняются.
[0079] В ситуациях, когда ALIGN-2 используется для сравнения аминокислотных последовательностей,% идентичности аминокислотной последовательности данной аминокислотной последовательности A относительно, с или по сравнению с данной аминокислотной последовательности B (которая альтернативно может быть сформулирована как данная аминокислота последовательность A, которая имеет или содержит определенный % идентичности аминокислотной последовательности относительно, с или по сравнению с данной аминокислотной последовательности B) рассчитывается следующим образом:
100 умножить на дробь X/Y
где X - количество аминокислотных остатков, оцененных как идентичные совпадения программой выравнивания последовательностей ALIGN-2 при выравнивании этой программой A и B, и где Y - общее количество аминокислотных остатков в B. Понятно, что если длина аминокислотной последовательности A не равна длине аминокислотной последовательности B,% идентичности аминокислотной последовательности A к B не будет равняться % идентичности аминокислотной последовательности B к A. Если специально не указано иное, все значения % идентичности аминокислотной последовательности, используемые в данном документе, получены как описано в непосредственно предшествующем абзаце с использованием компьютерной программы ALIGN-2.
[0080] Термин «фармацевтический состав» или «фармацевтическая композиция» относится к препарату, который находится в такой форме, чтобы обеспечить эффективность биологической активности содержащегося в нем активного ингредиента, и который не содержит дополнительных компонентов, которые являются неприемлемо токсичными для субъекта, которому будет вводиться состав.
[0081] «Фармацевтически приемлемый носитель» относится к ингредиенту в фармацевтическом составе или композиции, отличному от активного ингредиента, который нетоксичен для субъекта. Фармацевтически приемлемый носитель включает, без ограничения, буфер, эксципиент, стабилизатор или консервант.
[0082] Используемый в данном документе термин «лечение» (и его грамматические вариации, такие как «лечить» или «лечение») относится к клиническому вмешательству в попытке изменить естественное течение болезни у индивидуума, проходящего лечение, и может проводиться либо для профилактики, либо во время курса клинической патологии. Желаемые эффекты лечения включают предотвращение возникновения или рецидива заболевания, облегчение симптомов, уменьшение любых прямых или косвенных патологических последствий заболевания, предотвращение метастазирования, снижение скорости прогрессирования заболевания, улучшение или временное облегчение состояния болезни, и ремиссию или улучшенный прогноз, но не ограничиваются ими. В некоторых вариантах осуществления антитела по изобретению используются для задержки развития заболевания или замедления прогрессирования заболевания.
[0083] Используемый в данном документе термин «вектор» относится к молекуле нуклеиновой кислоты, способной размножать другую нуклеиновую кислоту, с которой она связана. Термин включает вектор как самореплицирующуюся структуру нуклеиновой кислоты, а также вектор, включенный в геном клетки-хозяина, в которую он был введен. Некоторые векторы способны управлять экспрессией нуклеиновых кислот, с которыми они функционально связаны. Такие векторы называются в данном документе «векторами экспрессии».
КОМПОЗИЦИИ И СПОСОБЫ
[0084] Предложены анти-CD112R антитела, композиции, содержащие описанные антитела, и способы их применения.
Типичные анти-CD112R антитела
[0085] В таблице последовательностей ниже представлены последовательности определенных вариантов осуществления антител, описанных и заявленных в данном документе.
[0086] В некоторых вариантах осуществления предложены антитела, которые связываются с CD112R и/или блокируют связывание CD112R с CD112 и/или усиливают активацию Т-клеток и NK-клеток. В некоторых вариантах осуществления предложены антитела, которые связываются с CD112R. В некоторых вариантах осуществления предложены антитела, которые блокируют связывание CD112R с CD112. В некоторых вариантах осуществления предложены антитела, которые усиливают активацию CD226, Т-клеток и/или NK-клеток.
[0087] Ингибирование связывания между CD112R и CD112, например, с Т- и NK-клетками, можно определить путем измерения ингибирования связывания клеток, с которыми связывается CD112R, в присутствии и в отсутствие антитела.
[0088] В данном документе предложены антитела, которые специфически связываются с CD112R.
[0089] В некоторых вариантах осуществления антитела связываются с CD112R человека.
[0090] В некоторых вариантах осуществления антитела связываются с CD112R человека и блокируют взаимодействие CD112R человека с CD112 человека. В некоторых вариантах осуществления антитела связываются с CD112R человека, блокируют взаимодействие CD112R человека с CD112 человека, но не блокируют взаимодействие CD112R мыши с CD112 мыши. В некоторых вариантах осуществления антитела, которые связываются с CD112R человека, блокируют взаимодействие CD112R человека с CD112 человека, но не блокируют взаимодействие CD112R мыши с CD112 мыши, включают антитела 32, 33, 34, 35 и 36.
[0091] В некоторых вариантах осуществления антитело к CD112R содержит вариабельную область тяжелой цепи («VH»), содержащую CDR1, CDR2 и/или CDR3 VH любого из антител к CD112R, представленных в настоящем документе (т.е. клоны антител с номерами 2, 5, 44, 58, 10 , 38, 15, 35, 46, 47, 32, 33, 34 или 36).
[0092] В некоторых вариантах осуществления антитело к CD112R содержит VH, содержащую CDR1, CDR2 и/или CDR3 VH любого из антител к CD112R, представленных в настоящем документе, и VL, содержащую CDR1, CDR2 и/или CDR3 любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VH, содержащую CDR1, CDR2 и/или CDR3 VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, и VL, содержащую CDR1, CDR2 и/или CDR3 VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, при этом CDR VH и VL необязательно происходят из одного и того же клона антитела.
[0093] В некоторых вариантах осуществления предложены антитела, содержащие следующее:
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 5; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 6; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 101; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 102; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 103; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 104; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 105; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 106; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 201; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 202; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 203; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 204; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 205; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 206; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 301; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 302; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 303; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 304; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 305; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 306; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 401; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 402; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 403; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 404; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 405; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 406; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 501; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 502; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 503; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 504; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 505; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 506; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 601; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 602; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 603; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 604; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 605; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 606; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 701; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 702; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 703; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 704; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 705; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 706; или
HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 801; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 802; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 803; с или без (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 804; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 805; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 806; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 901; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 902; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 903; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 904; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 905; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 906; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 2003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 2006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 3001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 3002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 3004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 3005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 3006; или
(a) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4001; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4002; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 4003; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 4004; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4005; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 4006.
[0094] В некоторых вариантах осуществления антитело к CD112R содержит VL, содержащую CDR1, CDR2 и CDR3 VL любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VL, содержащую CDR1, CDR2 и CDR3 VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36.
[0095] В некоторых вариантах осуществления антитело к CD112R может содержать:
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 2, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 2; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 5, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 5; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 44, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 44; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 58, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 58; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 10, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 10; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 38, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 38; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 15, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 15; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 35, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 35; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 47, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 47; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 46, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 46; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 32, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 32; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 33, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 33; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 34, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 34; или
VH, содержащую аминокислотную последовательность CDR1, CDR2 и CDR3 VH клона антитела номер 36, и VL, содержащую CDR1, CDR2 и CDR3 VL клона антитела номер 36.
[0096] В приведенной ниже таблице последовательностей представлены последовательности вариабельной области тяжелой и легкой цепей определенных описанных антител.
[0097] В некоторых вариантах осуществления антитело к CD112R содержит VH, содержащую аминокислотную последовательность VH любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VH, содержащую аминокислотную последовательность VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36.
[0098] В некоторых вариантах осуществления антитело к CD112R содержит VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, но с 1, 2, 3, 4 или 5 аминокислотными заменами вне определяющих комплементарность областей (CDR), например 1, 2, 3, 4 или 5 консервативными заменами вне CDR. В некоторых вариантах осуществления антитело к CD112R содержит VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, но с 1, 2, 3, 4 или 5 реверсивными заменами вне определяющих комплементарность областей (CDR).
[0099] В некоторых вариантах осуществления антитело к CD112R содержит VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, но с 1, 2, 3, 4 или 5 аминокислотными заменами в каркасных областях последовательности VH, например, 1, 2, 3, 4 или 5 консервативными заменами. В некоторых вариантах осуществления антитело к CD112R содержит VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, но с 1, 2, 3, 4 или 5 реверсивными заменами в каркасных областях последовательности VH.
[00100] В некоторых вариантах осуществления антитело к CD112R содержит CDR VH и VL любого из антител к CD112R, описанных в данном документе, при этом каждая CDR содержит 0, 1, 2 или 3 аминокислотных добавления, замены (например, консервативные замены) или делеции.
[00101] В некоторых вариантах осуществления антитело к CD112R содержит CDR1, CDR2 и CDR3 VH, содержащие аминокислотные последовательности CDR VH любого из антител к CD112R, представленных в настоящем документе, и содержит VH, которая по меньшей мере на 90%, по меньшей мере на 91%, по крайней мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентична VH любого из представленных в данном документе антител к CD112R. В некоторых вариантах осуществления антитело к CD112R содержит VH, содержащую аминокислотную последовательность, которая по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентична аминокислотной последовательности VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46 , 47, 32, 33, 34 или 36. В некоторых вариантах осуществления VH антитела отличается от VH последовательностей, показанных в Таблице последовательностей, из-за 1, 2, 3, 4 или 5 аминокислотных замен в каркасных областях последовательности VH, например, 1, 2, 3, 4 или 5 консервативных замен. В некоторых вариантах осуществления VH антитела отличается от VH последовательностей, показанных в Таблице последовательностей, из-за 1, 2, 3, 4 или 5 реверсивных замен в каркасных областях последовательности VH.
[00102] В некоторых вариантах осуществления антитело к CD112R содержит VH, состоящую из аминокислотной последовательности VH любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VH, которая состоит из аминокислотной последовательности VH любого из клонов антитела с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36.
[00103] В некоторых вариантах осуществления антитело к CD112R содержит VL, содержащую аминокислотную последовательность VL любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VL, содержащую аминокислотную последовательность VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36. В некоторых вариантах осуществления антитело к CD112R содержит CDR1, CDR2 и CDR3 VL, содержащие аминокислотные последовательности CDR VL любого из антител к CD112R, представленных в настоящем документе, и содержит VL, которая по меньшей мере на 90%, по меньшей мере на 91%, по крайней мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентична VL любого из представленных в данном документе антител к CD112R. В некоторых вариантах осуществления антитело к CD112R содержит VL, содержащую аминокислотную последовательность, которая по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентична аминокислотной последовательности VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46 , 47, 32, 33, 34 или 36. В некоторых вариантах осуществления VL антитела отличается от VL последовательностей, показанных в Таблице последовательностей, из-за 1, 2, 3, 4 или 5 аминокислотных замен в каркасных областях последовательности VL, например, 1, 2, 3, 4 или 5 консервативных замен. В некоторых вариантах осуществления VL антитела отличается от VL последовательностей, показанных в Таблице последовательностей, из-за 1, 2, 3, 4 или 5 реверсивных замен.
[00104] В некоторых вариантах осуществления антитело к CD112R содержит VL, состоящую из аминокислотной последовательности VL любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VL, которая состоит из аминокислотной последовательности VL любого из клонов антитела с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36.
[00105] В некоторых вариантах осуществления антитело к CD112R содержит VH, содержащую аминокислотную последовательность VH любого из представленных в данном документе антител к CD112R, и содержит VL, содержащую аминокислотную последовательность VL любого из тех же антител к CD112R, представленных в настоящем документе. В некоторых из этих вариантов осуществления антитело к CD112R содержит VH, содержащую аминокислотную последовательность VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, и VL, содержащую аминокислотную последовательность VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, необязательно где VH и VL относятся к одному и тому же номеру клона антитела.
[00106] В некоторых вариантах осуществления VH антитела представляет собой VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, но с 1, 2, 3, 4 или 5 аминокислотными заменами в каркасных областях последовательности VH, таких как 1, 2, 3, 4 или 5 консервативных замены, а VL представляет собой VL любого из тех же антител из списка выше. Однако в некоторых вариантах осуществления VH антитела представляет собой VH любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36, но с 1, 2, 3, 4 или 5 заменами в каркасных областях последовательности VH.
[00107] В некоторых вариантах осуществления антитело к CD112R содержит VH и VL, содержащие аминокислотные последовательности VH и VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36.
[00108] В некоторых вариантах осуществления антитело к CD112R содержит CDR1, CDR2 и CDR3 VH, содержащие аминокислотные последовательности CDR VH любого из антител к CD112R, представленных в настоящем документе, а также CDR1, CDR2 и CDR3 VL, содержащие аминокислотные последовательности CDR VL любого из представленных в данном документе антител к CD112R, а также содержит VH и VL, каждая из которых по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны соответствующим VH и VL любого из представленных в данном документе антител к CD112R. В некоторых вариантах осуществления VH и VL антитела отличаются от последовательностей VH и VL, показанных в Таблице последовательностей, из-за 1, 2, 3, 4 или 5 аминокислотных замен в каркасных областях последовательностей, таких как 1, 2, 3, 4 или 5 консервативных замен или таких как 1, 2, 3, 4 или 5 реверсивных замен.
[00109] В некоторых вариантах осуществления антитело к CD112R содержит VH и VL, состоящие из аминокислотной последовательности VH и VL любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит VH и VL, каждая из которых состоит из аминокислотных последовательностей VH и VL любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 и 36.
[00110] Антитело к CD112R может содержать:
VH, содержащую аминокислотную последовательность VH клона антитела номер 2, и VL, содержащую аминокислотную последовательность VL клона антитела номер 2; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 5, и VL, содержащую аминокислотную последовательность VL клона антитела номер 5; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 44, и VL, содержащую аминокислотную последовательность VL клона антитела номер 44; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 58, и VL, содержащую аминокислотную последовательность VL клона антитела номер 58; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 10, и VL, содержащую аминокислотную последовательность VL клона антитела номер 10; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 38, и VL, содержащую аминокислотную последовательность VL клона антитела номер 38; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 15, и VL, содержащую аминокислотную последовательность VL клона антитела номер 15; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 35, и VL, содержащую аминокислотную последовательность VL клона антитела номер 35; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 47, и VL, содержащую аминокислотную последовательность VL клона антитела номер 47;
VH, содержащую аминокислотную последовательность VH клона антитела номер 46, и VL, содержащую аминокислотную последовательность VL клона антитела номер 46;
VH, содержащую аминокислотную последовательность VH клона антитела номер 32, и VL, содержащую аминокислотную последовательность VL клона антитела номер 32; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 33, и VL, содержащую аминокислотную последовательность VL клона антитела номер 33; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 34, и VL, содержащую аминокислотную последовательность VL клона антитела номер 34; или
VH, содержащую аминокислотную последовательность VH клона антитела номер 36, и VL, содержащую аминокислотную последовательность VL клона антитела номер 36; или.
[00111] Антитело к CD112R может содержать:
VH, содержащую CDR VH клона антитела номер 2, и VL, содержащую CDR VL клона антитела номер 2, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 2; или
VH, содержащую CDR VH клона антитела номер 5, и VL, содержащую CDR VL клона антитела номер 5, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 5; или
VH, содержащую CDR VH клона антитела номер 44, и VL, содержащую CDR VL клона антитела номер 44, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 44; или
VH, содержащую CDR VH клона антитела номер 58, и VL, содержащую CDR VL клона антитела номер 58, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 58; или
VH, содержащую CDR VH клона антитела номер 10, и VL, содержащую CDR VL клона антитела номер 10, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 10; или
VH, содержащую CDR VH клона антитела номер 38, и VL, содержащую CDR VL клона антитела номер 38, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 38; или
VH, содержащую CDR VH клона антитела номер 15, и VL, содержащую CDR VL клона антитела номер 15, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 15; или
VH, содержащую CDR VH клона антитела номер 35, и VL, содержащую CDR VL клона антитела номер 35, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 35; или
VH, содержащую CDR VH клона антитела номер 47, и VL, содержащую CDR VL клона антитела номер 47, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 47; или
VH, содержащую CDR VH клона антитела номер 46, и VL, содержащую CDR VL клона антитела номер 46, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 46; или
VH, содержащую CDR VH клона антитела номер 32, и VL, содержащую CDR VL клона антитела номер 32, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 32; или
VH, содержащую CDR VH клона антитела номер 33, и VL, содержащую CDR VL клона антитела номер 33, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 33; или
VH, содержащую CDR VH клона антитела номер 34, и VL, содержащую CDR VL клона антитела номер 34, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 34; или
VH, содержащую CDR VH клона антитела номер 36, и VL, содержащую CDR VL клона антитела номер 36, и аминокислотные последовательности VH и VL, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны VH и VL клона антитела номер 36.
[00112] В некоторых из вышеупомянутых вариантов осуществления VH и/или VL могут отличаться от последовательности каждого из видов наличием 1, 2, 3, 4 или 5 аминокислотных замен, таких как 1, 2, 3, 4 или 5 консервативных замен. В некоторых вариантах осуществления VH может содержать 1, 2, 3, 4 или 5 реверсивных замен.
[00113] Антитело к CD112R может содержать:
VH, состоящую из аминокислотной последовательности VH клона антитела номер 2, и VL, состоящую из VL клона антитела номер 2; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 5, и VL, состоящую из VL клона антитела номер 5; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 44, и VL, состоящую из VL клона антитела номер 44; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 58, и VL, состоящую из VL клона антитела номер 58; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 10, и VL, состоящую из VL клона антитела номер 10; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 38, и VL, состоящую из VL клона антитела номер 38; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 15, и VL, состоящую из VL клона антитела номер 15; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 35, и VL, состоящую из VL клона антитела номер 35; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 47, и VL, состоящую из VL клона антитела номер 47; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 46, и VL, состоящую из VL клона антитела номер 46; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 32, и VL, состоящую из VL клона антитела номер 32; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 33, и VL, состоящую из VL клона антитела номер 33; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 34, и VL, состоящую из VL клона антитела номер 34; или
VH, состоящую из аминокислотной последовательности VH клона антитела номер 36, и VL, состоящую из VL клона антитела номер 36.
[00114] В некоторых вариантах осуществления антитело к CD112R содержит любую из вариабельных областей и/или CDR 1-3 вариабельной области антител, описанных выше и в других местах в настоящем документе, например, в Таблице последовательностей.
[00115] В некоторых вариантах осуществления антитело к CD112R представляет собой антитело IgG, такое как антитело IgG1, IgG2, IgG3 или IgG4, или его модифицированную форму, как описано в разделе ниже. В некоторых вариантах осуществления константная область выполняет эффекторную функцию, а в некоторых вариантах осуществления константная область не является эффекторной.
[00116] В некоторых вариантах осуществления антитело к CD112R содержит тяжелую цепь (НС), содержащую аминокислотную последовательность тяжелой цепи любого из антител к CD112R, представленных в настоящем документе. В некоторых вариантах осуществления антитело к CD112R содержит тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи любого из клонов антител с номерами 2, 5, 44, 58, 10, 38, 15, 35, 46, 47, 32, 33, 34 или 36.
[00117] В некоторых вариантах осуществления антитело к CD112R может содержать:
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 2, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 2; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 5, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 5; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 44, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 44; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 58, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 58; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 10, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 10; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 38, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 38; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 15, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 15; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 35, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 35; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 47, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 47; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 46, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 46; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 32, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 32; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 33, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 33; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 34, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 34; или
тяжелую цепь, содержащую аминокислотную последовательность тяжелой цепи клона антитела номер 36, и легкую цепь, содержащую аминокислотную последовательность легкой цепи клона антитела номер 36.
[00118] Антитело к CD112R может содержать:
тяжелую цепь (HC), содержащую CDR HC клона антитела номер 2, и легкую цепь (LC), содержащую CDR LC клона антитела номер 2, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 2, соответственно; или
HC, содержащую CDR HC клона антитела номер 5, и легкую цепь (LC), содержащую CDR LC клона антитела номер 5, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 5, соответственно; или
HC, содержащую CDR HC клона антитела номер 44, и легкую цепь (LC), содержащую CDR LC клона антитела номер 44, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 44, соответственно; или
HC, содержащую CDR HC клона антитела номер 58, и легкую цепь (LC), содержащую CDR LC клона антитела номер 58, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 58, соответственно; или
HC, содержащую CDR HC клона антитела номер 10, и легкую цепь (LC), содержащую CDR LC клона антитела номер 10, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 10, соответственно; или
HC, содержащую CDR HC клона антитела номер 38, и легкую цепь (LC), содержащую CDR LC клона антитела номер 38, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 38, соответственно; или
HC, содержащую CDR HC клона антитела номер 15, и легкую цепь (LC), содержащую CDR LC клона антитела номер 15, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 15, соответственно; или
HC, содержащую CDR HC клона антитела номер 35, и легкую цепь (LC), содержащую CDR LC клона антитела номер 35, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 35, соответственно; или
HC, содержащую CDR HC клона антитела номер 47, и легкую цепь (LC), содержащую CDR LC клона антитела номер 47, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 47, соответственно; или
HC, содержащую CDR HC клона антитела номер 46, и легкую цепь (LC), содержащую CDR LC клона антитела номер 46, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 46, соответственно; или
HC, содержащую CDR HC клона антитела номер 32, и LC, содержащую CDR LC клона антитела номер 32, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 32, соответственно; или
HC, содержащую CDR HC клона антитела номер 33, и легкую цепь (LC), содержащую CDR LC клона антитела номер 33, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 33, соответственно; или
HC, содержащую CDR HC клона антитела номер 34, и легкую цепь (LC), содержащую CDR LC клона антитела номер 34, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 34, соответственно; или
HC, содержащую CDR HC клона антитела номер 36, и легкую цепь (LC), содержащую CDR LC клона антитела номер 36, и аминокислотные последовательности HC и LC, которые по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны HC и LC клона антитела номер 36, соответственно.
[00119] В некоторых из вышеупомянутых вариантов осуществления HC и/или LC могут отличаться от последовательности каждого из видов наличием 1, 2, 3, 4 или 5 аминокислотных замен, таких как 1, 2, 3, 4 или 5 консервативных замен. В некоторых из вышеупомянутых вариантов осуществления HC и/или LC могут отличаться от последовательности каждого из видов наличием 1, 2, 3, 4 или 5 аминокислотных замен, таких как 1, 2, 3, 4 или 5 реверсивных замен.
1. Примерный класс антител с общими структурными и функциональными признаки
[00120] В некоторых вариантах осуществления предложены анти-CD112R антитела. В некоторых вариантах осуществления антитела к CD112 имеют общие структурные и/или функциональные признаки. В некоторых вариантах осуществления класс антител включает исходное антитело и его варианты с созревшей аффинностью. Один типичный класс антител включает исходный клон 32 и его варианты с созревшей аффинностью, но не ограничивается ими. В некоторых вариантах осуществления варианты с созревшей аффинностью включают антитела 33, 34, 35 и 36. В некоторых вариантах осуществления варианты с созревшей аффинностью включают антитела с консервативными заменами по сравнению с антителами 32, 33, 34, 35 и 36.
a. Структурные признаки типичного класса антител
[00121] В некоторых вариантах осуществления анти-CD112R антитела имеют общие структурные признаки, такие как, например, показанные на Фигурах 14 и 15. Когда в данном документе описывается «класс» или «члены класса» антител, следует понимать, что охватываются/предполагаются варианты осуществления, описывающие одно анти-CD112R антитело или множество анти-CD112R антител. В некоторых вариантах осуществления анти-CD112R антитела содержат идентичные HCDR3. В некоторых вариантах осуществления анти-CD112R антитела содержат идентичные LCDR1. В некоторых вариантах осуществления анти-CD112R антитела содержат идентичные LCDR2. В некоторых вариантах осуществления анти-CD112R антитела содержат идентичные LCDR3. В некоторых вариантах осуществления анти-CD112R антитела содержат идентичные HCDR3 и идентичные LCDR1, LCDR2 и/или LCDR3. В некоторых вариантах осуществления анти-CD112R антитела содержат HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1003, и/или LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1004, и/или LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1005, и/или LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 1006.
[00122] В некоторых вариантах осуществления каждый член класса антител содержит каркасные области тяжелой цепи, содержащие аминокислотные последовательности SEQ ID NO: 1007, 1008, 1009 и 1010. В некоторых вариантах осуществления каждый член класса антител содержит аминокислотную последовательность вариабельной области тяжелой цепи, которая по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92% или по меньшей мере на 93% идентична аминокислотной последовательности SEQ ID NO: 1012, при этом любая и каждая вариация последовательности относительно SEQ ID NO: 1012 находится в HCDR1 и/или HCDR2. В некоторых вариантах осуществления каждый член класса антител содержит каркасные области легкой цепи, содержащие аминокислотные последовательности SEQ ID NO: 1013, 1014, 1015 и 1016. В некоторых вариантах осуществления каждый член класса антител содержит аминокислотную последовательность вариабельной области легкой цепи SEQ ID NO: 1018.
[00123] Члены этого типичного класса антител могут включать некоторые варианты аминокислотных последовательностей HCDR1 и HCDR2. В некоторых вариантах осуществления каждый член класса антител содержит HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1001 или аминокислотную последовательность SEQ ID NO: 1001 с 1, 2 или 3 аминокислотными заменами в положениях 4, 5 и/или 6 SEQ ID NO: 1001. В некоторых вариантах осуществления класс антител содержит HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 1001 с 1, 2 или 3 аминокислотными заменами в положениях, которые варьируются между аминокислотами SEQ ID NO: 1001, 2001, 3001, 4001 и 701, как показано на Фигуре 14. В некоторых вариантах осуществления одна или более из 1, 2 или 3 аминокислотных замен не являются консервативными заменами. В некоторых вариантах осуществления одна или более из 1, 2 или 3 аминокислотных замен являются консервативными заменами. В некоторых вариантах осуществления член класса антител содержит HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 2001, 3001, 701 или 4001.
[00124] В некоторых вариантах осуществления каждый член класса антител содержит HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1002 или аминокислотную последовательность SEQ ID NO: 1002 с 1, 2, 3, 4 или 5 аминокислотными заменами в положениях 1, 3, 5, 6 и/или 8 SEQ ID NO: 1002. В некоторых вариантах осуществления класс антител содержит HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 1002 с 1, 2, 3, 4 или 5 аминокислотными заменами в положениях, которые варьируются между аминокислотами SEQ ID NO: 1002, 2002, 3002, 4002 и 702, как показано на Фигуре 14. В некоторых вариантах осуществления одна или более из 1, 2, 3, 4 или 5 аминокислотных замен не являются консервативными заменами. В некоторых вариантах осуществления одна или более из 1, 2, 3, 4 или 5 аминокислотных замен являются консервативными заменами. В некоторых вариантах осуществления член класса антител содержит HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 2002, 3002, 702 или 4002.
[00125] В некоторых вариантах осуществления каждый член класса антител содержит идентичные каркасные области тяжелой цепи и легкой цепи.
б. Функциональные признаки типичного класса антител
[00126] В некоторых вариантах осуществления предложены антитела к CD112R, при этом антитела обладают особым техническим эффектом связывания с CD112R человека, блокируя взаимодействие CD112R человека с CD112 и не блокируя взаимодействие CD112R мыши с CD112. В некоторых вариантах осуществления каждый член класса антител связывается с CD112R человека и блокирует связывающее взаимодействие между CD112 человека и CD112R человека. В некоторых вариантах осуществления каждый член класса антител не блокирует связывающее взаимодействие между CD112 мыши и CD112R мыши. Хотя члены класса антител не блокируют взаимодействие между CD112 мыши и CD112R мыши, члены класса антител либо частично ингибируют связывающее взаимодействие между CD112 мыши и CD112R мыши, либо не ингибируют связывающее взаимодействие между CD112 мыши и CD112R мыши. В некоторых таких вариантах осуществления ни один член класса антител не ингибирует взаимодействие между CD112R мыши и CD112 мыши более чем на 50%. В некоторых вариантах осуществления каждый член класса антител проявляет по меньшей мере некоторое связывание с растворимым CD112R мыши. В некоторых вариантах осуществления антитело является полностью человеческим или гуманизированным. В некоторых вариантах осуществления каждый член класса антител связывает один и тот же эпитоп на CD112R человека.
2. Фрагменты антител
[00127] В некоторых вариантах осуществления антитело, представленное в настоящем документе, представляет собой фрагмент антитела. Фрагменты антител включают фрагменты Fab, Fab', Fab'-SH, F(ab')2, Fv и scFv, и другие фрагменты, описанные ниже. Для обзора некоторых фрагментов антител см. Hudson et al. Nat. Med. 9:129-134 (2003). Для обзора фрагментов scFv см. например, Pluckthün, in The Pharmacology of Monoclonal Antibodies, vol. 113, Rosenburg and Moore eds., (Springer-Verlag, New York), pp. 269-315 (1994); см. также WO 93/16185; и патенты США № 5571894 и 5587458. Обсуждения фрагментов Fab и F(ab')2, содержащих остатки эпитопа, связывающего рецептор реутилизации, и имеющих увеличенный период полужизни in vivo, приведены в Патенте США № 5869046.
[00128] Диатела представляют собой фрагменты антител с двумя антигенсвязывающими сайтами, которые могут быть бивалентными или биспецифическими. См., например, EP 404097; WO 1993/01161; Hudson et al., Nat. Med. 9:129-134 (2003); и Hollinger et al., Proc. Natl. Acad. Sci. USA 90: 6444-6448 (1993). Триатела и тетратела также описаны в Hudson et al., Nat. Med. 9:129-134 (2003).
[00129] Однодоменные антитела представляют собой фрагменты антител, содержащие весь или часть вариабельного домена тяжелой цепи или весь или часть вариабельного домена легкой цепи антитела. В некоторых вариантах осуществления однодоменное антитело представляет собой однодоменное антитело человека (Domantis, Inc., Waltham, MA; см., например, Патент США № 6248516 B1).
[00130] Фрагменты антител могут быть получены различными методами, включая, помимо прочего, протеолитическое расщепление интактного антитела, а также продукцию рекомбинантными клетками-хозяевами (например, E. сoli или фагом), как описано в данном документе.
3. Мультиспецифические антитела
[00131] В некоторых вариантах осуществления антитело, представленное в настоящем документе, представляет собой мультиспецифическое антитело, например биспецифическое антитело. Мультиспецифические антитела - это моноклональные антитела, обладающие специфичностью связывания по крайней мере с двумя разными сайтами. В некоторых вариантах осуществления одна из специфичностей связывания относится к CD112R, а другая - к любому другому антигену. В некоторых вариантах осуществления одна из специфичностей связывания относится к CD112R, а другая независимо выбрана из одного (в случае биспецифического) или более (в случае мультиспецифического) из PD-1, PD-L1, CTLA-4, Lag-3, TIM-3, TIGIT, CD96, PVRL1, PVRL2, PVRL3, PVRL4, CD155, STING, CD47, CD39 и IL-27. В некоторых вариантах осуществления биспецифические антитела могут связываться с двумя разными эпитопами CD112R. Биспецифические антитела также можно использовать для локализации цитотоксических агентов в клетках, экспрессирующих CD112R. Биспецифические антитела можно получить в виде полноразмерных антител или фрагментов антител.
[00132] Методы получения мультиспецифических антител включают рекомбинантную коэкспрессию двух пар тяжелая цепь-легкая цепь иммуноглобулина, имеющих разные специфичности (см. Milstein and Cuello, Nature 305: 537 (1983)), WO 93/08829, и Traunecker et al., EMBO J. 10: 3655 (1991)), и конструирование «выступ-во-впадину» (см., например, Патент США № 5731168), но не ограничиваются ими. Мультиспецифические антитела также могут быть получены путем создания электростатических управляющих эффектов для создания Fc-гетеродимерных молекул антитела (WO 2009/089004 A1); перекрестного сшивания двух или более антител или фрагментов (см., например, Патент США № 4676980 и Brennan et al., Science, 229: 81 (1985)); использование лейциновых застежек для выработки биспецифических антител (см., например, Kostelny et al., J. Immunol., 148(5):1547-1553 (1992)); использования технологии «диател» для получения фрагментов биспецифических антител (см., например, Hollinger et al., Proc. Natl. Acad. Sci. USA, 90:6444-6448 (1993)); и использования одноцепочечных димеров Fv (sFv) (см., например Gruber et al., J. Immunol., 152:5368 (1994)); и получение триспецифических антител, как описано, например, в Tutt et al. J. Immunol. 147: 60 (1991).
[00133] Сконструированные антитела с тремя или более функциональными антигенсвязывающими сайтами, включая «антитела-осьминоги», также включены в данный документ (см., например US 2006/0025576 A1).
[00134] Антитело или фрагмент в данном документе также включает «Fab двойного действия» или «DAF», содержащий антигенсвязывающий сайт, который связывается с CD112R, а также с другим, отличным антигеном (см., например, US 2008/0069820).
4. Варианты антител
[00135] В некоторых вариантах осуществления предложены варианты аминокислотной последовательности антител, представленных в настоящем документе. Например, может быть желательно улучшить аффинность связывания и/или другие биологические свойства антитела. Варианты аминокислотной последовательности антитела могут быть получены путем внесения соответствующих модификаций в нуклеотидную последовательность, кодирующую антитело, или путем пептидного синтеза. Такие модификации включают, например, делеции и/или вставки, и/или замены остатков в аминокислотных последовательностях антитела. Любая комбинация делеции, вставки и замены может быть использована для получения конечной конструкции при условии, что конечная конструкция обладает желаемыми характеристиками, например, антигенсвязывающими характеристиками.
5. Варианты замены, вставки и делеции
[00136] В некоторых вариантах осуществления предложены варианты антител, содержащие одну или более аминокислотных замен. Сайты, представляющие интерес для замещающего мутагенеза, включают HVR и FR. Консервативные замены показаны в Таблице 1 как «примерные замены». Аминокислотные замены могут быть введены в представляющее интерес антитело, и продукты могут быть проверены на желаемую активность, например, сохранение/улучшение связывания антигена, снижение иммуногенности или улучшение АЗКЦ или КЗЦ.
[00137] ТАБЛИЦА 1
остаток
замены
замены
Аминокислоты могут быть сгруппированы в соответствии с общими свойствами боковой цепи:
(1) гидрофобные: норлейцин, Met, Ala, Val, Leu, Ile;
(2) нейтральные гидрофильные: Cys, Ser, Thr, Asn, Gln;
(3) кислые: Asp, Glu;
(4) основные: His, Lys, Arg;
(5) остатки, влияющие на ориентацию цепи: Gly, Pro;
(6) ароматные: Trp, Tyr, Phe.
[00138] Неконсервативные замены повлекут за собой замену члена одного из этих классов на другой класс.
[00139] В HVR могут быть внесены изменения (например, замены), например, для повышения аффинности антитела. Такие изменения могут быть сделаны в «горячих точках» HVR, то есть в остатках, кодируемых кодонами, которые подвергаются мутации с высокой частотой в процессе соматического созревания (см., например, Chowdhury, Methods Mol. Biol. 207:179-196 (2008)), и/или остатках, которые контактируют с антигеном, при этом полученный вариант VH или VL тестируется на аффинность связывания. Созревание аффинности путем конструирования и повторного выбора из вторичных библиотек описано, например, в Hoogenboom et al. in Methods in Molecular Biology 178:1-37 (O'Brien et al., ed., Human Press, Totowa, NJ, (2001).) В некоторых вариантах осуществления созревание аффинности, разнообразие вводится в вариабельные гены, выбранные для созревания, любым из множества способов (например, подверженной ошибкам ПЦР, перетасовкой цепей или олигонуклеотид-направленным мутагенезом). Затем создается вторичная библиотека. Затем библиотека проверяется для выявления любых вариантов антител с желаемой аффинностью. Другой метод введения разнообразия включает подходы, направленные на HVR, в которых несколько остатков HVR (например, 4-6 остатков за раз) рандомизируются. Остатки HVR, участвующие в связывании антигена, могут быть специфически идентифицированы, например, с использованием аланин-сканирующего мутагенеза или моделирования. CDR-H3 и CDR-L3, в частности, часто становятся мишенью.
[00140] В некоторых вариантах осуществления замены, вставки или делеции могут происходить в одной или более HVR, если такие изменения существенно не снижают способность антитела связывать антиген. Например, консервативные изменения (например, консервативные замены, как предусмотрено в данном документе), которые существенно не снижают аффинность связывания, могут быть внесены в HVR. Такие изменения могут, например, находиться за пределами контактирующих с антигеном остатков HVR. В некоторых вариантах осуществления вариантов последовательностей VH и VL, представленных выше, каждая HVR либо не изменена, либо содержит не более одной, двух или трех аминокислотных замен.
[00141] Применимый способ идентификации остатков или областей антитела, которые могут быть мишенями для мутагенеза, называется «аланин-сканирующий мутагенез», как описано в Cunningham and Wells (1989) Science, 244:1081-1085. В этом способе остаток или группа целевых остатков (например, заряженные остатки, такие как arg, asp, his, lys и glu) идентифицируются и заменяются нейтральной или отрицательно заряженной аминокислотой (например, аланином или полиаланином) для определения влияет ли это на взаимодействие антитела с антигеном. Дальнейшие замены могут быть введены в аминокислотные положения, демонстрирующие функциональную чувствительность к начальным заменам. Альтернативно или дополнительно, кристаллическая структура комплекса антиген-антитело может быть использована для определения точек контакта между антителом и антигеном. Такие контактные остатки и соседние остатки могут быть нацелены или исключены как кандидаты для замены. Варианты могут быть проверены, чтобы определить, содержат ли они желаемые свойства.
[00142] Вставки аминокислотной последовательности включают амино- и/или карбокси-концевые слияния, имеющие длину в диапазоне от одного остатка до полипептидов, содержащих сто или более остатков, а также вставки внутри последовательности одного или нескольких аминокислотных остатков. Примеры концевых вставок включают антитело с N-концевым остатком метионила. Другие инсерционные варианты молекулы антитела включают слияние N- или C-конца антитела с ферментом (например, ADEPT) или полипептидом, который увеличивает время полужизни антитела в сыворотке.
6. Варианты гликозилирования
[00143] В определенных вариантах осуществления антитело, представленное в настоящем документе, изменено для увеличения или уменьшения степени, в которой антитело является гликозилированным. Добавление или удаление сайтов гликозилирования к антителу может быть удобно выполнено путем изменения аминокислотной последовательности таким образом, что создается или удаляется один или более сайтов гликозилирования.
[00144] Когда антитело содержит Fc-область, углевод, присоединенный к нему, может быть изменен. Нативные антитела, продуцируемые клетками млекопитающих, обычно содержат разветвленный двухантенный олигосахарид, который обычно присоединяется посредством N-связи к Asn297 домена CH2 Fc-области. См., например, Wright et al. TIBTECH 15:26-32 (1997). Олигосахарид может включать в себя различные углеводы, например, маннозу, N-ацетилглюкозамин (GlcNAc), галактозу и сиаловую кислоту, а также фукозу, присоединенную к GlcNAc в «стволе» двухантенной структуры олигосахаридов. В некоторых вариантах осуществления могут быть сделаны модификации олигосахарида в антителе по изобретению для создания вариантов антител с определенными улучшенными свойствами.
[00145] В некоторых вариантах осуществления представлены варианты антител, имеющие углеводную структуру, в которой отсутствует фукоза, присоединенная (прямо или косвенно) к Fc-области. Например, количество фукозы в таком антителе может составлять от 1% до 80%, от 1% до 65%, от 5% до 65% или от 20% до 40%. Количество фукозы определяется путем расчета среднего количества фукозы в сахарной цепи в Asn297 относительно суммы всех гликоструктур, присоединенных к Asn 297 (например, сложных, гибридных и структур с высоким содержанием маннозы), как измерено с помощью масс-спектрометрии MALDI-TOF, как описано, например, в WO 2008/077546. Asn297 относится к остатку аспарагина, расположенному приблизительно в положении 297 в Fc-области (нумерация Eu остатков Fc-области); однако Asn297 также может быть расположен приблизительно на ± 3 аминокислоты выше или ниже положения 297, то есть между положениями 294 и 300, из-за незначительных вариаций последовательности в антителах. Такие варианты фукозилирования могут иметь улучшенную функцию АЗКЦ. См., например, патентные публикации США № US 2003/0157108 (Presta, L.); US 2004/0093621 (Kyowa Hakko Kogyo Co., Ltd). Примеры публикаций, относящихся к «дефукозилированным» или «дефицитным по фукозе» вариантам антител, включают: US 2003/0157108; WO 2000/61739; WO 2001/29246; US 2003/0115614; US 2002/0164328; US 2004/0093621; US 2004/0132140; US 2004/0110704; US 2004/0110282; US 2004/0109865; WO 2003/085119; WO 2003/084570; WO 2005/035586; WO 2005/035778; WO 2005/053742; WO 2002/031140; Okazaki et al. J. Mol. Biol. 336:1239-1249 (2004); Yamane-Ohnuki et al. Biotech. Bioeng. 87: 614 (2004). Примеры клеточных линий, способных продуцировать дефукозилированные антитела, включают клетки Lec13 CHO, дефицитные по фукозилированию белка (Ripka et al. Arch. Biochem. Biophys. 249:533-545 (1986); заявка на патент США № US 2003/0157108 A1, Presta, L; и WO 2004/056312 A1, Adams et al., особенно в примере 11), и линии клеток с нокаутом, таких как ген альфа-1,6-фукозилтрансферазы, FUT8, клетки с нокаутом СНО (см., например, Yamane-Ohnuki et al. Biotech. Bioeng. 87: 614 (2004); Kanda, Y. et al., Biotechnol. Bioeng., 94(4):680-688 (2006); и WO 2003/085107).
[00146] Варианты антител дополнительно содержат олигосахариды, разделенные пополам, например, в которых двухантенный олигосахарид, присоединенный к Fc-области антитела, разделен пополам GlcNAc. Такие варианты антител могут иметь пониженное фукозилирование и/или улучшенную функцию АЗКЦ. Примеры таких вариантов антител описаны, например, в WO 2003/011878 (Jean-Mairet et al.); патенте США № 6 602 684 (Umana et al.); и US 2005/0123546 (Umana et al.). Также предложены варианты антител с по меньшей мере одним остатком галактозы в олигосахариде, присоединенном к Fc-области. Такие варианты антител могут иметь улучшенную функцию КЗЦ. Такие варианты антител описаны, например, в WO 1997/30087 (Patel et al.); WO 1998/58964 (Raju, S.); и WO 1999/22764 (Raju, S.).
7. Варианты Fc-области
[00147] В некоторых вариантах осуществления одна или более аминокислотных модификаций могут быть введены в Fc-область антитела, представленного в настоящем документе, тем самым генерируя вариант Fc-области. Вариант Fc-области может содержать последовательность Fc-области человека (например, Fc-область человеческого IgG1, IgG2, IgG3 или IgG4), содержащую аминокислотную модификацию (например, замену) в одном или более аминокислотных положениях.
[00148] В некоторых вариантах осуществления в изобретении рассмотрен вариант антитела, который обладает некоторыми, но не всеми эффекторными функциями, что делает его подходящим кандидатом для применений, в которых время полужизни антитела in vivo является важным, но некоторые эффекторные функции (такие как комплемент и АЗКЦ) ненужны или вредны. Анализы цитотоксичности in vitro и/или in vivo могут быть проведены для подтверждения снижения/истощения активности КЗЦ и/или АЗКЦ. Например, могут быть проведены анализы связывания Fc-рецептора (FcR), чтобы убедиться, что у антитела отсутствует связывание с FcγR (следовательно, вероятно, не обладает активностью АЗКЦ), но сохраняется способность связывания с FcRn. Первичные клетки для опосредования АЗКЦ, NK-клетки, экспрессируют только FcγRIII, тогда как моноциты экспрессируют FcγRI, FcγRII и FcγRIII. Данные по экспрессии FcR на гемопоэтических клетках обобщены в таблице 3 на странице 464 публикации Ravetch and Kinet, Annu. Rev. Immunol. 9:457-492 (1991). Неограничивающие примеры анализов in vitro для оценки активности АЗКЦ интересующей молекулы описаны в патенте США № 5500362 (см., например, Hellstrom, I. et al. Proc. Nat'l Acad. Sci. USA 83:7059-7063 (1986)) и Hellstrom, I et al., Proc. Nat'l Acad. Sci. USA 82:1499-1502 (1985); 5821337 (см. Bruggemann, M. et al., J. Exp. Med. 166:1351-1361 (1987)). В качестве альтернативы можно использовать методы нерадиоактивных анализов (см., например, анализ нерадиоактивной цитотоксичности ACTI™ для проточной цитометрии (CellTechnology, Inc. Mountain View, CA; и анализ нерадиоактивной цитотоксичности CytoTox 96® (Promega, Madison, WI). Эффекторные клетки, пригодные для такого анализа, включают мононуклеарные клетки периферической крови МКПК (PBMC) и естественные клетки-киллеры (NK). В качестве альтернативы или дополнения, АЗКЦ-активность молекулы, представляющей интерес, можно оценить in vivo, например, на животной модели, такой как описанная в публикации Clynes et al. Proc. Nat'l Acad. Sci. USA 95:652-656 (1998). Анализы связывания C1q также могут быть выполнены для подтверждения того, что антитело неспособно связывать C1q и, следовательно, не имеет активности КЗЦ. См., например, ИФА связывания C1q и C3c в WO 2006/029879 и WO 2005/100402. Для оценки активации комплемента может быть проведен анализ КЗЦ (см., например, Gazzano-Santoro et al., J. Immunol. Methods 202:163 (1996); Cragg, M.S. et al., Blood 101:1045-1052 (2003); and Cragg, M.S. И M.J. Glennie, Blood 103:2738-2743 (2004)). Связывание FcRn и определение клиренса/периода полужизни in vivo также можно проводить с использованием методов, известных в данной области (см., например, Petkova, S.B. et al., Int'l. Immunol. 18(12):1759-1769 (2006)).
[00149] Антитела с пониженной эффекторной функцией включают антитела с заменой одного или более остатков 238, 265, 269, 270, 297, 327 и 329 Fc-области (патент США № 6737056). Такие Fc-мутанты включают Fc-мутантов с заменами в двух или более аминокислотных положениях 265, 269, 270, 297 и 327, включая так называемого Fc-мутанта «DANA» с заменой остатков 265 и 297 на аланин (патент США № 7332581).
[00150] Описаны некоторые варианты антител с улучшенным или ослабленным связыванием с FcR. (См., например, Патент США № 6737056; WO 2004/056312, и Shields et al., J. Biol. Chem. 9(2): 6591-6604 (2001).)
[00151] В некоторых вариантах осуществления вариант антитела содержит Fc-область с одной или более аминокислотными заменами, которые улучшают АЗКЦ, например, заменами в положениях 298, 333 и/или 334 Fc-области (нумерация остатков по EU).
[00152] В некоторых вариантах осуществления в Fc-область вносятся изменения, которые приводят к измененному (т.е. улучшенному или ослабленному) связыванию C1q и/или комплемент-зависимой цитотоксичности (КЗЦ), например, как описано в патенте США № 6194551, WO 99/51642 и Idusogie et al. J. Immunol. 164: 4178-4184 (2000).
[00153] Антитела с увеличенным периодом полужизни и улучшенным связыванием с неонатальным Fc-рецептором (FcRn), который отвечает за перенос материнских IgG к плоду (Guyer et al., J. Immunol. 117:587 (1976) и Kim et al., J. Immunol. 24:249 (1994)), описаны в US 2005/0014934 A1 (Hinton et al.).Эти антитела содержат Fc-область с одной или более заменами в ней, которые улучшают связывание Fc-области с FcRn. Такие Fc-варианты включают варианты с заменами в одном или более остатках Fc-области: 238, 252, 254, 256, 265, 272, 286, 303, 305, 307, 311, 312, 317, 340, 356, 360, 362, 376, 378, 380, 382, 413, 424 или 434, например, заменой остатка 434 Fc-области (например, Патент США № 7371826).
[00154] См. также Duncan & Winter, Nature 322:738-40 (1988); патент США № 5648260; патент США № 5624821; и WO 94/29351, касающиеся других примеров вариантов Fc-области.
[00155] В некоторых вариантах осуществления предложено антитело в соответствии с Таблицей последовательностей, при этом его изотип представляет собой IgG1 человека. В некоторых вариантах осуществления предложено антитело в соответствии с Таблицей последовательностей, при этом его изотип представляет собой IgG4 человека. В некоторых вариантах осуществления предложено антитело в соответствии с Таблицей последовательностей, при этом его изотип представляет собой IgG4 человека, и при этом имеется единственная мутация серина 228 на пролин (S228P).
8. Варианты сконструированных антител с цистеином
[00156] В некоторых вариантах осуществления может быть желательно создать сконструированные антитела с цистеином, например, «THIOM-антитела», в которых один или более остатков антитела замещены остатками цистеина. В конкретных вариантах осуществления замещенные остатки находятся в доступных сайтах антитела. За счет замещения этих остатков цистеином реактивные тиоловые группы тем самым размещаются в доступных сайтах антитела и могут использоваться для конъюгирования антитела с другими фрагментами, такими как фрагменты лекарственного средства или линкер-лекарственные фрагменты, для создания иммуноконъюгата, как описано далее в настоящем документе. В некоторых вариантах осуществления любой один или более из следующих остатков могут быть замещены цистеином: V205 (нумерация по Кабат) легкой цепи; A118 (нумерация EU) тяжелой цепи; и S400 (нумерация EU) Fc-области тяжелой цепи. Сконструированные антитела с цистеином могут быть получены, как описано, например, в патенте США № 7521541.
9. Производные антител
[00157] В некоторых вариантах осуществления антитело, представленное в настоящем документе, может быть дополнительно модифицировано, чтобы содержать дополнительные небелковые фрагменты, которые известны в данной области и легко доступны. Фрагменты, подходящие для дериватизации антитела, включают водорастворимые полимеры, но не ограничиваются ими. Неограничивающие примеры водорастворимых полимеров включают полиэтиленгликоль (ПЭГ), сополимеры этиленгликоля/пропиленгликоля, карбоксиметилцеллюлозу, декстран, поливиниловый спирт, поливинилпирролидон, поли-1,3-диоксолан, поли-1,3,6-триоксан, сополимер этилена и малеинового ангидрида, полиаминокислоты (либо гомополимеры, либо статистические сополимеры) и декстран или поли(н-винилпирролидон)полиэтиленгликоль, гомополимеры пропиленгликоля, сополимеры пролипропиленоксида/этиленоксида, полиоксиэтилированные полиолы (например, глицерин), поливиниловый спирт и их смеси, но не ограничиваются ими. Пропиональдегид полиэтиленгликоля может иметь преимущества при производстве из-за его стабильности в воде. Полимер может меть любую молекулярную массу и может быть разветвленным или неразветвленным. Количество полимеров, присоединенных к антителу, может варьироваться, и если присоединено более одного полимера, они могут быть одинаковыми или разными молекулами. В общем, количество и/или тип полимеров, используемых для дериватизации, может быть определено на основании соображений, включая конкретные свойства или функции антитела, подлежащего улучшению, будет ли производное антитело использоваться в терапии при определенных условиях и т. д., но не ограничиваясь ими.
[00158] В другом варианте осуществления предложены конъюгаты антитела и небелкового фрагмента, которые можно избирательно нагревать под воздействием излучения. В некоторых вариантах осуществления небелковый фрагмент представляет собой углеродную нанотрубку (Kam et al., Proc. Natl. Acad. Sci. USA 102: 11600-11605 (2005)). Излучение может иметь любую длину волны и включает, помимо прочего, длины волн, которые не повреждают обычные клетки, но нагревают небелковый фрагмент до температуры, при которой гибнут клетки, проксимальные к антитело-небелковому фрагменту.
Рекомбинантные методы
[00159] Антитела могут быть получены с использованием рекомбинантных методов и композиций, например, как описано в патенте США № 4816567. В некоторых вариантах осуществления предусмотрена выделенная нуклеиновая кислота, кодирующая описанное в данном документе анти-CD112R антитело. Такая нуклеиновая кислота может кодировать аминокислотную последовательность, содержащую VL, и/или аминокислотную последовательность, содержащую VH антитела (например, легкую и/или тяжелую цепи антитела). В дополнительном варианте осуществления предложен один или более векторов (например, векторов экспрессии), содержащих такую нуклеиновую кислоту. В дополнительном варианте осуществления предложена клетка-хозяин, содержащая такую нуклеиновую кислоту. В одном таком варианте осуществления клетка-хозяин содержит (например, была трансформирована им): (1) вектор, содержащий нуклеиновую кислоту, кодирующую аминокислотную последовательность, содержащую VL антитела, и аминокислотную последовательность, содержащую VH антитела, или (2) первый вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую VL антитела, и второй вектор, содержащий нуклеиновую кислоту, которая кодирует аминокислотную последовательность, содержащую VH антитела. В некоторых вариантах осуществления клетка-хозяин является эукариотической, например клеткой яичника китайского хомячка (СНО) или лимфоидной клеткой (например, клеткой Y0, NS0, Sp20). В некоторых вариантах осуществления предложен способ получения анти-CD112R антитела, где способ включает культивирование клетки-хозяина, содержащей нуклеиновую кислоту, кодирующую антитело, как указано выше, в условиях, подходящих для экспрессии антитела, и необязательно выделение антитела из клетки-хозяина (или среды для культивирования клеток-хозяев).
[00160] Для получения рекомбинантного анти-CD112R антитела нуклеиновую кислоту, кодирующую антитело, например, как описано выше, выделяют и вставляют в один или более векторов для дальнейшего клонирования и/или экспрессии в клетке-хозяине. Такую нуклеиновую кислоту можно легко выделять и секвенировать с использованием общепринятых процедур (например, с использованием олигонуклеотидных зондов, способных специфически связываться с генами, кодирующими тяжелые и легкие цепи антитела).
[00161] Клетки-хозяева, подходящие для клонирования или экспрессии векторов, кодирующих антитело, включают прокариотические или эукариотические клетки, описанные в данном документе. Например, антитела могут продуцироваться в бактериях, в частности, когда гликозилирование и эффекторная функция Fc не нужны. Для экспрессии фрагментов антител и полипептидов в бактериях см., например, патенты США № 5648237, 5789199 и 5840523. (См. также Charlton, Methods in Molecular Biology, Vol. 248 (BKC Lo, ed., Humana Press, Totowa, NJ, 2003), pp. 245-254, описывающую экспрессию фрагментов антител в E. coli). После экспрессии антитело может быть выделено из биомассы бактериальных клеток в растворимой фракции и может быть дополнительно очищено.
[00162] Помимо прокариот, подходящими хозяевами для клонирования или экспрессии векторов, кодирующих антитела, являются эукариотические микроорганизмы, такие как мицелиальные грибы или дрожжи, включая штаммы грибов и дрожжей, чьи метаболические пути гликозилирования были «гуманизированы», что приводит к продукции антител с частично или полностью человеческим профилем гликозилирования. См. Gerngross, Nat. Biotech. 22:1409-1414 (2004) и Li et al., Nat. Biotech. 24:210-215 (2006).
[00163] Клетки-хозяева, подходящие для экспрессии гликозилированного антитела, также можно получать из многоклеточных организмов (беспозвоночных и позвоночных). Примеры клеток беспозвоночных включают клетки растений и насекомых. Идентифицированы многочисленные бакуловирусные штаммы, которые можно использовать в сочетании с клетками насекомых, в частности для трансфекции клеток Spodoptera frugiperda.
[00164] В качестве хозяев также можно использовать культуры растительных клеток. См., например, патенты США № 5959177, 6040498, 6420548, 7125978 и 6417429 (описывающие технологию PLANTIBODIES™ для получения антител в трансгенных растениях).
[00165] В качестве хозяев также можно использовать клетки позвоночных. Например, можно использовать линии клеток млекопитающих, адаптированные для роста в суспензии. Другими примерами применимых линий клеток-хозяев млекопитающих являются линия CV1 почек обезьяны, трансформированная SV40 (COS-7); линия почек эмбриона человека (293 или клетки 293, как описано, например, у Graham et al., J. Gen Virol. 36:59 (1977); клетки почек детенышей хомячка (BHK); клетки Сертоли мыши (клетки TM4, как описано, например, у Mather, Biol. Reprod. 23:243-251 (1980)); клетки почки обезьяны (CV1); клетки почки африканской зеленой мартышки (VERO-76); клетки карциномы шейки матки человека (HELA); клетки почки собаки (MDCK; клетки печени крысы линии buffalo (BRL 3A); клетки легкого человека (W138); клетки печени человека (Hep G2); клетки опухоли молочной железы мыши (MMT 060562); клетки TRI, как описано, например, у Mather et al., Annals N.Y. Acad. Sci. 383:44-68 (1982); клетки MRC 5 и клетки FS4. Другие применимые линии клеток-хозяев млекопитающих включают клетки яичника китайского хомячка (СНО), включая клетки DHFR-CHO (Urlaub et al., Proc. Natl. Acad. Sci. USA 77:4216 (1980)); и линии миеломных клеток, такие как Y0, NS0 и Sp2/0. Для обзора некоторых линий клеток-хозяев млекопитающих, подходящих для продукции антител, см., например, Yazaki and Wu, Methods in Molecular Biology, Vol. 248 (B.K.C. Lo, ed., Humana Press, Totowa, NJ), pp. 255-268 (2003).
Иммуноконъюгаты
[00166] В изобретении также предложены иммуноконъюгаты, содержащие анти-CD112R антитело, конъюгированное в данном документе с одним или более другими терапевтическими агентами или радиоактивными изотопами.
[00167] В другом варианте осуществления иммуноконъюгат содержит антитело, как описано в данном документе, конъюгированное с радиоактивным атомом с образованием радиоконъюгата. Для производства радиоконъюгатов доступны различные радиоактивные изотопы. Примеры включают At211, I131, I125, Y90, Re186, Re188, Sm153, Bi212, P32, Pb212 и радиоактивные изотопы Lu. Когда радиоконъюгат используется для обнаружения, он может содержать радиоактивный атом для сцинтиграфических исследований, например tc99m или I123, или спиновую метку для изображений ядерного магнитного резонанса (ЯМР) (также известную как магнитно-резонансная томография, МРТ), например снова йод-123, йод-131, индий-111, фтор-19, углерод-13, азот-15, кислород-17, гадолиний, марганец или железо.
[00168] Конъюгаты антитела могут быть получены с использованием различных бифункциональных агентов, связывающих болок, таких как N-сукцинимидил-3-(2-пиридилдитио) пропионат (SPDP), сукцинимидил-4-(N-малеимидометил) циклогексан-1-карбоксилат (SMCC), иминотиолан (IT), бифункциональные производные имидоэфиров (такие как диметиладипимидат HCl), активные сложные эфиры (такие как дисукцинимидил суберат), альдегиды (такие как глутаральдегид), бис-азидосоединения (такие как бис (п-азидобензоил) гександиамин), производные бис-диазония (такие как бис-(п-диазонийбензоил)-этилендиамин), диизоцианаты (такие как 2,6-диизоцианат толуола) и бис-активные соединения фтора (такие как 1,5-дифтор-2,4-динитробензол). Например, иммунотоксин рицин может быть получен, как описано в Vitetta et al., Science 238:1098 (1987). Меченная углеродом-14 1-изотиоцианатобензил-3-метилдиэтилентриаминпентауксусная кислота (MX-DTPA) является типичным хелатирующим агентом для конъюгации радионуклеотида с антителом. См. WO 94/11026. Линкер может быть «расщепляемым линкером», способствующим высвобождению цитотоксического лекарственного средства в клетке. Могут быть использованы, например, кислотолабильный линкер, чувствительный к пептидазе линкер, фотолабильный линкер, диметиловый линкер или дисульфидсодержащий линкер (Chari et al., Cancer Res. 52:127-131 (1992); патент США № 5208020).
[00169] Иммуноконъюгаты или ADC в настоящем документе явно предполагают конъюгаты, полученные с помощью перекрестно-линкерных реагентов, включая, помимо прочего, BMPS, EMCS, GMBS, HBVS, LC-SMCC, MBS, MPBH, SBAP, SIA, SIAB, SMCC, SMPB, SMPH, сульфо-EMCS, сульфо-GMBS, сульфо-KMUS, сульфо-MBS, сульфо-SIAB, сульфо-SMCC и сульфо-SMPB, а также SVSB (сукцинимидил-(4-винилсульфон) бензоат), которые являются коммерчески доступными (например, от Pierce Biotechnology, Inc., Rockford, IL., U.S.A), но не ограничиваются ими.
Фармацевтические составы и композиции
[00170] Фармацевтические препараты или композиции анти-CD112R-антитела, как описано в данном документе, получают путем смешивания такого антитела, имеющего желаемую степень чистоты, с одним или более необязательными фармацевтически приемлемыми носителями, разбавителями и/или наполнителями (Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed. (1980)), в виде лиофилизированных составов или водных растворов. Фармацевтически приемлемые носители, разбавители и эксципиенты обычно нетоксичны для реципиентов в используемых дозировках и концентрациях и включают: стерильную воду, буферы, такие как фосфат, цитрат и другие органические кислоты; антиоксиданты, включая аскорбиновую кислоту и метионин; консерванты (такие как хлорид октадецилдиметилбензиламмония; хлорид гексаметония; хлорид бензалкония, хлорид бензетония; фенол, бутиловый или бензиловый спирт; алкилпарабены, такие как метил или пропилпарабен; катехол; резорцинол; циклогексанол; 3-пентанол; и м-крезол); низкомолекулярные (менее чем около 10 остатков) полипептиды; белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, включая глюкозу, маннозу или декстрины; хелатообразующие агенты, такие как ЭДТК; сахара, такие как сахароза, маннит, трегалоза или сорбит; солеобразующие противоионы, такие как натрий; комплексы металлов (например, комплексы Zn-белок); и/или неионные поверхностно-активные вещества, такие как полиэтиленгликоль (ПЭГ), но не ограничиваются ими. Примеры фармацевтически приемлемых носителей в данном документе дополнительно включают агенты, диспергирующие лекарственные средства, такие как растворимые нейтрально-активные гликопротеины гиалуронидазы (sHASEGP), например растворимые гликопротеины гиалуронидазы PH-20 человека, такие как rHuPH20 (HYLENEX®, Baxter International, Inc.).Некоторые иллюстративные sHASEGP и способы их использования, включая rHuPH20, описаны в публикациях патентов США № 2005/0260186 и 2006/0104968.В одном аспекте sHASEGP комбинируется с одной или более дополнительными гликозаминогликаназами, такими как хондроитиназы.
[00171] Типичные составы лиофилизированных антител описаны в патенте США № 6267958. Водные составы антител включают те, которые описаны в патенте США № 6171586 и WO2006/044908, причем последние составы включают гистидин-ацетатный буфер.
[00172] Состав или композиция, описанная в данном документе, также может содержать более одного активного ингредиента, если это необходимо для конкретного показания, подлежащего лечению, предпочтительно ингредиенты с дополнительной активностью, которые не оказывают неблагоприятного воздействия друг на друга. Такие активные ингредиенты присутствуют в надлежащей комбинации в количествах, которые эффективны для предполагаемого применения.
[00173] Активные ингредиенты могут быть заключены в микрокапсулы, полученные, например, методами коацервации или межфазной полимеризации, например, микрокапсулы из гидроксиметилцеллюлозы или желатиновые микрокапсулы и полиметилметацилатные микрокапсулы, соответственно, в коллоидных системах доставки лекарств (например, липосомы, альбумин микросферы, микроэмульсии, наночастицы и нанокапсулы) или в макроэмульсиях. Такие методы раскрыты в Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed. (1980).
[00174] Можно изготовить препараты с замедленным высвобождением. Подходящие примеры препаратов с замедленным высвобождением включают полупроницаемые матрицы из твердых гидрофобных полимеров, содержащие антитело, причем матрицы представлены в форме, например пленок или микрокапсул.
[00175] Составы или композиции, используемые для введения in vivo, обычно стерильны. Стерильность может быть легко достигнута, например, путем фильтрации через стерильные фильтрующие мембраны.
Терапевтические способы
[00176] Любые из представленных в данном документе анти-CD112R антител могут использоваться в терапевтических методах. Везде, где обсуждается «антитело», также следует понимать, что композиция, содержащая антитело, также включена.
[00177] В одном аспекте предусмотрено анти-CD112R антитело для использования в качестве лекарственного средства. В некоторых вариантах осуществления предложено анти-CD112R антитело для применения для усиления, повышения и/или поддержания противоопухолевого иммунного ответа у субъекта, имеющего опухоль. В некоторых вариантах осуществления опухоль является раковой. В некоторых вариантах осуществления предложено анти-CD112R антитело для использования при лечении рака. В некоторых вариантах осуществления предложено анти-CD112R антитело для использования для усиления взаимодействий CD226 с CD112.
[00178] В дополнительном аспекте в изобретении предложено применение анти-CD112R антитела при производстве или приготовлении лекарственного средства. В некоторых вариантах осуществления лекарственное средство предназначено для использования для усиления, повышения и/или поддержания противоопухолевого иммунного ответа у субъекта, имеющего опухоль. В некоторых вариантах осуществления опухоль является раковой. В некоторых вариантах осуществления лекарственное средство предназначено для лечения рака. В некоторых вариантах осуществления лекарственное средство предназначено для усиления взаимодействий CD226 с CD112.
[00179] В дополнительных аспектах в изобретении предложены способы лечения заболеваний и/или расстройств, при которых желательно блокирование CD112R. В некоторых вариантах осуществления предусмотрены способы усиления, повышения и/или поддержания противоопухолевого иммунного ответа у субъекта, имеющего опухоль, включающие введение анти-CD112R антитела, как описано в данном документе. В некоторых вариантах осуществления опухоль является раковой. В некоторых вариантах осуществления предложены способы лечения рака у субъекта, имеющего рак, включающие введение анти-CD112R антитела, как описано в данном документе. В некоторых вариантах осуществления предусмотрены способы усиления взаимодействий CD226 с CD112 у субъекта, необязательно имеющего рак, включающие введение анти-CD112R антитела, как описано в данном документе.
[00180] В некоторых аспектах в изобретении предложен способ облегчения одного или более симптомов заболевания или расстройства, связанного с белком CD112R; или анти-CD112R антитело, или лекарственное средство, содержащее анти-CD112R антитело, для облегчения одного или более симптомов заболевания или расстройства, связанного с белком CD112R (такого как любое из заболеваний или расстройств, описанных в данном документе, например, рака). В некоторых аспектах в изобретении предложен способ уменьшения количества симптомов или тяжести одного или более симптомов заболевания или расстройства, связанных с белком CD112R; или анти-CD112R антитело, или лекарственное средство, содержащее анти-CD112R антитело, для уменьшения количества симптомов или тяжести одного или более симптомов заболевания или нарушения, связанного с белком CD112R (например, любого из заболеваний или расстройств, описанных в данном документе, например, рака). В конкретном варианте осуществления симптомом заболевания или расстройства, связанного с белком CD112R, является опухоль, а уменьшение представляет собой уменьшение размера опухоли, неспособность опухоли к росту или устранение опухоли.
[00181] Описанные в данном документе антитела можно использовать, например, для лечения рака. В некоторых вариантах осуществления предложены способы лечения рака, включающие введение субъекту эффективного количества описанного в данном документе антитела. В некоторых вариантах осуществления антитела могут запускать или усиливать иммунный ответ у субъекта, такой как антигенспецифический иммунный ответ. В некоторых вариантах осуществления антитела могут стимулировать активность Т-клеток. В некоторых вариантах осуществления антитела могут ингибировать рост по меньшей мере одной опухоли у субъекта.
[00182] В настоящем документе предложены способы лечения субъекта, имеющего рак, включающие введение субъекту терапевтически эффективного количества анти-CD112R антитела, описанного в данном документе, таким образом, чтобы субъект проходил лечение. Анти-CD112R антитело можно использовать отдельно. Альтернативно, анти-CD112R антитело можно использовать в сочетании с другим агентом, как описано ниже.
[00183] Виды рака могут представлять собой рак с солидными опухолями или злокачественные новообразования крови (например, опухоли жидких тканей).
[00184] Неограничивающие примеры рака для лечения включают плоскоклеточный рак, мелкоклеточный рак легкого, немелкоклеточный рак легкого, плоскоклеточный немелкоклеточный рак легкого (NSCLC), неплоскоклеточный NSCLC, глиому, рак желудочно-кишечного тракта, рак почек (например, светлоклеточная карцинома), рак яичников, рак печени, колоректальный рак, рак эндометрия, рак почки (например, почечно-клеточная карцинома (RCC)), рак предстательной железы (например, гормонорезистентная аденокарцинома предстательной железы), рак щитовидной железы, нейробластома, рак поджелудочной железы, глиобластому (мультиформнуюглиобластому), рак шейки матки, рак желудка, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки и рак головы и шеи (или карциному), рак желудка, герминогенную опухоль, саркому у детей, синоназальная опухоль из клеток-натуральных киллеров, меланому (например, метастатическая злокачественная меланома, такая как кожная или внутриглазная злокачественная меланома), рак костей, рак кожи, рак матки, рак анальной области, рак яичек, карциному фаллопиевых труб, карциному эндометрия, карциному шейки матки, карциному влагалища, карциному вульвы, рак пищевода, рак тонкой кишки, рак эндокринной системы, рак паращитовидной железы, рак надпочечника, саркома мягких тканей, рак уретры, рак полового члена, солидные опухоли у детей, рак мочеточника, карциному почечной лоханки, новообразование центральной нервной системы (ЦНС), первичную лимфому ЦНС, опухолевый ангиогенез, опухоль оси позвоночника, рак головного мозга, глиому ствола головного мозга, аденому гипофиза, саркому Капоши, эпидермоидный рак, плоскоклеточный рак, Т-клеточную лимфому, раковые образования, связанные с воздействием окружающей среды, в том числе индуцированные асбестом, связанные с вирусом раковые опухоли или раковые опухоли вирусного происхождения (например, вирус папилломы человека (опухоли, связанные с HPV-, или опухоли с HPV-происхождением)), а также гематологические злокачественные новообразования, происходящие от одного из двух основных линий клеток крови то есть линии миелоидных клеток (которая продуцирует гранулоциты, эритроциты, тромбоциты, макрофаги и тучные клетки) или линии лимфоидных клеток (которая продуцирует B, T, NK и плазматические клетки), например, все типы лейкозов, лимфом и миелом, например, острые, хронические, лимфоцитарные и/или миелогенные лейкозы, такие как острый лейкоз (ALL), острый миелогенный лейкоз (AML), хронический лимфоцитарный лейкоз (CLL) и хронический миелолейкоз (CML), недифференцированный AML (MO), миелобластный лейкоз (M1), миелобластный лейкоз (M2; с созреванием клеток), промиелоцитарный лейкоз (вариант M3 или M3 [M3V]), миеломоноцитарный лейкоз (M4 или вариант M4 с эозинофилией [M4E]), моноцитарный лейкоз (M5), эритролейкоз (M6), мегакариобластный лейкоз (M7), изолированная гранулоцитарная саркома и хлорома; лимфомы, такие как лимфома Ходжкина (HL), неходжкинская лимфома (NHL), В-клеточные гематологические злокачественные новообразования, например, B-клеточные лимфомы, T-клеточные лимфомы, лимфоплазмацитоидные лимфомы, моноцитоидные B-клеточные лимфомы, лимфома лимфоидной ткани, ассоциированная со слизистой оболочкой (MALT), анапластическая (например, Ki 1+) крупноклеточная лимфома, Т-клеточная лимфома/лейкоз взрослых, лимфома из мантийных клеток, ангио-иммунобластная Т-клеточная лимфома, ангиоцентрическая лимфома, интестинальная Т-клеточная лимфома, первичная медиастинальная B-клеточная лимфома, предшественница Т-лимфобластной лимфомы, Т-лимфобластная лимфома; и лимфома/лейкоз (T-Lbly/T-ALL), периферическая Т-клеточная лимфома, лимфобластная лимфома, посттрансплантационное лимфопролиферативное заболевание, истинная гистиоцитарная лимфома, первичная лимфома центральной нервной системы, первичная эффузионная лимфома, B-клеточная лимфома, лимфобластная лимфома (LBL), гемопоэтические опухоли лимфоидной линии, острый лимфобластный лейкоз, диффузная В-крупноклеточная лимфома, лимфома Беркитта, фолликулярная лимфома, диффузная гистиоцитарная лимфома (DHL), иммунобластная крупноклеточная лимфома, предшественница B-лимфобластной лимфомы (CTLC-лимфома), также называемая грибовидным микозом или синдромом Сезари) и лимфоплазмацитоидная лимфома (LPL) с макроглобулинемией Вальденстрема; миеломы, такие как миелома IgG, миелома легких цепей, несекреторная миелома, вялотекущая миелома (также называемая индолентной миеломой), одиночная плазмоцитома и множественные миеломы, хронический лимфолейкоз (CLL), волосисто-клеточная лимфома; гемопоэтические опухоли миелоидного происхождения, опухоли мезенхимального происхождения, включая фибросаркому и рабдомиоскаркому; семинома, тератокарцинома, опухоли центральной и периферической нервной системы, включая астроцитому, шванномы; опухоли мезенхимального происхождения, включая фибросаркому, рабдомиоскарому и остеосаркому; и другие опухоли, в том числе меланома, пигментная ксеродермия, кератоакантома, семинома, фолликулярный рак щитовидной железы и тератокарцинома, гемопоэтические опухоли лимфоидной линии, например Т-клеточные и В-клеточные опухоли, включая, помимо прочего, Т-клеточные нарушения, такие как Т- пролимфоцитарный лейкоз (T-PLL), включая типы с мелкоклеточными и церебриформными клетками; лейкоз из крупных гранулярных лимфоцитов (LGL) Т-клеточного типа; и T-NHL гепатолиенальная лимфома; периферическая/посттимическая Т-клеточная лимфома (плеоморфные и иммунобластные подтипы); ангиоцентрическая (назальная) Т-клеточная лимфома; рак головы или шеи, рак почек, рак прямой кишки, рак щитовидной железы; острая миелоидная лимфома, а также любые комбинации указанных видов рака. Описанные в данном документе способы также могут быть использованы для лечения метастатических злокачественных опухолей, неоперабельных, рефрактерных злокачественных опухолей (например, злокачественных опухолей, рефрактерных к предыдущей иммунотерапии, например, блокирующему антителу к CTLA-4 или PD-1) и/или рецидивирующих злокачественных опухолей.
[00185] В некоторых вариантах осуществления описанное в данном документе антитело вводят субъектам, имеющим рак, который проявил неадекватный ответ на предшествующее лечение или прогрессировал, например, на предшествующее лечение иммуноонкологическим или иммунотерапевтическим препаратом. В некоторых вариантах осуществления рак является рефрактерным или резистентным к предшествующему лечению, либо по своей природе рефрактерным, либо резистентным (например, рефрактерным к антагонисту пути PD-1), или при котором приобретается резистентное или рефрактерное состояние. Например, описанное в данном документе антитело можно вводить субъектам, которые не реагируют или недостаточно реагируют на первую терапию, или у которых наблюдается прогрессирование заболевания после лечения, например, лечения антагонистом пути анти-PD-1, либо отдельно, либо в комбинации с другой терапией (например, с терапией антагонистами пути анти-PD-1). В других вариантах осуществления описанное в данном документе антитело вводят субъектам, которые ранее не получали (т.е. не лечились) иммуноонкологический агент, например антагонист пути PD-1.
Комбинации
[00186] Антитела по изобретению можно использовать в терапии либо отдельно, либо в комбинации с другими агентами. Например, антитело по изобретению можно совместно вводить по меньшей мере с одним дополнительным терапевтическим агентом (например, дополнительно включающим введение второй терапии).
[00187] В некоторых вариантах осуществления нацеливание на дополнительный независимый путь ингибирования или их комбинации могут привести к дальнейшему усилению активации иммунных клеток по сравнению с монотерапией.
[00188] В некоторых вариантах осуществления дополнительный терапевтический агент или второй агент представляет собой химиотерапевтический агент, опсонизирующий агент, агент, истощающий популяцию регуляторных Т-клеток («Treg»), антагонист мишени, отличной от CD112R, или агонист мишени, отличной от CD112R. В некоторых вариантах осуществления второй агент представляет собой химиотерапевтический агент, описанный в данном документе, или любой известный химиотерапевтический агент. В некоторых вариантах осуществления второй агент представляет собой опсонизирующий агент, где опсонизирующий агент представляет собой антитело, отличное от анти-CD112R антитела, которое нацелено на раковые или опухолевые клетки. В некоторых вариантах осуществления второй агент представляет собой описанный в данном документе агент, истощающий Treg, или любой известный агент, истощающий Treg. В некоторых вариантах осуществления второй агент является антагонистом мишени, отличной от CD112R. В некоторых вариантах осуществления второй агент является агонистом мишени, отличной от CD112R.
[00189] В некоторых случаях второй агент нацелен на независимый путь ингибирования, такой как, например, путь с участием PD-1, PD-L1, CTLA-4, Lag-3 или TIM-3. В некоторых вариантах осуществления второй агент противодействует одному или более из PD-1, PD-L1, CTLA-4, Lag-3 и TIM-3. Подходящие антагонисты для использования в описанной в данном документе комбинированной терапии включают, без ограничения, лиганды, антитела (например, моноклональные антитела и биспецифические антитела) и поливалентные агенты. В одном варианте осуществления антагонист представляет собой слитый белок, например слитый белок Fc, такой как AMP-244. В некоторых вариантах осуществления антагонист PD-1 представляет собой анти-PD-1 или анти-PD-L1 антитело.
[00190] Типичным анти-PD-1 антителом является ниволумаб (BMS-936558) или антитело, которое содержит CDR или вариабельные области одного из антител 17D8, 2D3, 4H1, 5C4, 7D3, 5F4 и 4A11, описанные в WO 2006/121168. В некоторых вариантах осуществления анти-PD-1 антитело представляет собой MK-3475 (ламбролизумаб), описанное в WO 2012/145493; AMP-514, описанное в WO 2012/145493; или PDR001. Другие известные антитела к PD-1 и другие ингибиторы PD-1 включают антитела, описанные в WO 2009/014708, WO 03/099196, WO 2009/114335, WO 2011/066389, WO 2011/161699, WO 2012/145493, патентах США №№ 7635757 и 8217149, и публикации патента США № 2009/0317368. Также можно использовать любое из анти-PD-1 антител, описанных в WO2013/173223. Анти-PD-1 антитело, которое конкурирует за связывание и/или связывается с тем же эпитопом на PD-1, что и одно из этих антител, также можно использовать в комбинированном лечении.
[00191] В некоторых вариантах осуществления анти-PD-L1 антитело, используемое в комбинированной терапии, представляет собой BMS-936559 (обозначается как 12A4 в WO 2007/005874 и патенте США № 7943743) или антитело, которое содержит CDR или вариабельные области 3G10, 12A4, 10A5, 5F8, 10H10, 1B12, 7H1, 11E6, 12B7 и 13G4, которое описано в публикации PCT WO 07/005874 и патенте США № 7943743. В определенном варианте осуществления анти-PD-L1 антителом является MEDI4736 (также известный как дурвалумаб и анти-B7-H1), MPDL3280A (также известный как атезолизумаб и RG7446), MSB0010718C (также известный как авелумаб; WO2013/79174) или rHigM12B7. Также можно использовать любое из анти-PD-L1 антител, раскрытых в WO 2013/173223, WO 2011/066389, WO 2012/145493, патентах США № 7635757 и 8217149 и публикации США № 2009/145493. Анти-PD-L1 антитела, которые конкурируют и/или связываются с тем же эпитопом, что и эпитоп любого из этих антител, также можно использовать в комбинированном лечении.
[00192] В некоторых вариантах осуществления антитело к CD112R по данному описанию можно использовать с антагонистом CTLA-4, например, анти-CTLA-4 антителом. В одном из вариантов осуществления анти-CTLA-4 антитело представляет собой антитело, выбранное из группы: Yervoy® (ипилимумаб или антитело к 10D1, описанное в публикации РСТ WO 01/14424), тремелимумаб (ранее тицилимумаб, CP-675,206), моноклональное антитело или анти-CTLA-4 антитело, описанное в любой из следующих публикаций: WO 98/42752; WO 00/37504; патенте США № 6207156; Hurwitz et al. (1998) Pro. Natl. Acad. Sci. USA 95(17): 10067-10071; Camacho et al. (2004) J. Clin. Oncology 22(145): antibodiestract No. 2505 (антитело к CP-675206); и Mokyr et al. (1998) Cancer Res. 58:5301-5304. Также можно использовать любое из анти-CTLA-4 антител, описанных в WO2013/173223.
[00193] В некоторых вариантах осуществления анти-CD112R антитело по настоящему описанию используется в комбинации с антагонистом LAG-3 (также называемым в данном и других документах как LAG3). Примеры анти-LAG3 антител включают антитела, содержащие CDR или вариабельные области антител 25F7, 26H10, 25E3, 8B7, 11F2 или 17E5, которые описаны в публикации патента США № US 2011/0150892, WO 10/19570 и WO 2014/008218. В одном варианте осуществления анти-LAG-3 антитело представляет собой BMS-986016. Другие признанные в данной области анти-LAG-3 антитела, которые можно использовать, включают IMP731 и IMP-321, описанные в US 2011/007023, WO 08/132601 и WO 09/44273. Анти-LAG-3 антитела, которые конкурируют и/или связываются с тем же эпитопом, что и эпитоп любого из этих антител, также можно использовать в комбинированном лечении.
[00194] В некоторых вариантах осуществления нацеливание на два или более рецепторов TIGIT, CD96 и CD112R одновременно увеличивает опосредованную CD226 передачу сигналов по сравнению с монотерапией анти-CD112R. Следовательно, в некоторых вариантах осуществления второй агент является антагонистом TIGIT и/или CD96. Подходящие антагонисты для использования в описанной в данном документе комбинированной терапии включают, без ограничения, лиганды, антитела (например, моноклональные антитела и биспецифические антитела) и поливалентные агенты.
[00195] В некоторых вариантах осуществления члены семейства генов PVR активируются в опухолевых клетках и могут проявлять присущие опухолевые свойства. Нацеливание на дополнительных членов семейства генов PVR в сочетании с анти-CD112R антителами приводит к повышенной чувствительности опухолей по сравнению с монотерапией. Следовательно, в некоторых вариантах осуществления второй агент выбирается из одного или более антагонистов PVRL1, PVRL2, PVRL3, PVRL4 и CD155. Подходящие антагонисты для применения в описанной в данном документе комбинированной терапии включают, без ограничения, лиганды, антитела (например, моноклональные антитела и биспецифические антитела) и поливалентные агенты.
[00196] Агонисты STING индуцируют активацию клеток врожденного иммунитета, что приводит к усилению примирования Т-клеток и привлечению иммунных клеток в микроокружение опухоли. Нацеливание на агонисты STING в комбинации с CD112R может привести к еще большему увеличению набора и активации Т-клеток и NK-клеток.
[00197] Повышенный фагоцитоз, опосредованный анти-CD47 антителами, может привести к увеличению презентирования антигенов рака макрофагами Т-клеткам. Комбинированное лечение анти-CD47 антителом и анти-CD112R антителом, таким как анти-CD112R антитело, предложенное в настоящем документе, дает возможность усилить ответ Т-клеток, специфичных к раку, и полностью охвачено данным документом.
[00198] Аденозин через аденозиновые рецепторы, экспрессируемые на иммунных клетках, ингибирует активацию Т-клеток и NK-клеток. Анти-CD39 антитела подавляют образование аденозина, предотвращая гидролиз аденозинтрифосфата (АТФ). Комбинированное лечение анти-CD39 антителом и анти-CD112R антителом, таким как анти-CD112R антитело, представленное в настоящем документе, дает возможность дополнительно усилить терапию CD112R путем ингибирования опосредованной аденозином передачи клеточного сигнала в иммунных клетках.
[00199] Цитокины могут эффективно модулировать активацию Т-клеток и NK-клеток. IL-27 представляет собой иммуносупрессивный цитокин, который ингибирует ответы, опосредованные Т-клетками и NK-клетками. Анти-IL-27 антитела дают возможность усилить терапию CD112R за счет ограничения передачи сигналов иммуносупрессивных цитокинов в иммунных клетках. Таким образом, обеспечивается комбинированное лечение анти-IL-27 антителом и анти-CD112R антителом, таким как анти-CD112R антитело, предложенное в настоящем документе.
[00200] Антитела в данном документе также могут быть предоставлены до, по существу одновременно с или после других способов лечения, например хирургического вмешательства, химиотерапии, лучевой терапии или введения биологического препарата, такого как другое терапевтическое антитело. В некоторых вариантах осуществления рак рецидивировал или прогрессировал после терапии, выбранной из хирургического вмешательства, химиотерапии и лучевой терапии, или их комбинации. Например, анти-CD112R антитело, описанное в данном документе, можно вводить в качестве дополнительной терапии, когда существует риск наличия микрометастазов и/или для снижения риска рецидива.
[00201] Для лечения рака комбинации можно вводить в сочетании с одним или более дополнительными противораковыми агентами, такими как химиотерапевтический агент, агент, ингибирующий рост, противораковая вакцина, такая как вакцина для генной терапии, агент против ангиогенеза и/или противоопухолевый состав.
[00202] В некоторых вариантах осуществления с комбинацией можно вводить противовоспалительное лекарственное средство, такое как стероид или нестероидное противовоспалительное лекарственное средство (NSAID). В случаях, когда желательно привести в состояние покоя аберрантно пролиферативные клетки, субъекту также можно вводить гормоны и стероиды (включая синтетические аналоги), такие как 17a-этинилэстрадиол, диэтилстильбестрол, тестостерон, преднизон, флуоксиместерон, дроматостанолон, тестолактон, мегестролацетат, метилпреднизолон, метил-тестостерон, преднизолон, триамцинолон, хлортрианизен, гидроксипрогестерон, аминоглутетимид, эстрамустин, медроксипрогестеронацетат, леупролид, флутамид, ZOLADEX®, Торемид, в сочетании с или до лечения антителами к CD112R, описанными в данном документе. При использовании способов или композиций, описанных в данном документе, при желании также можно вводить другие агенты, используемые для модуляции роста или метастазирования опухоли в клинических условиях, такие как антимиметики.
[00203] Такие комбинированные терапии, указанные выше, включают комбинированное введение (когда два или более терапевтических агентов включены в одни и те же или отдельные составы или композиции) и раздельное введение, и в этом случае введение антитела по изобретению может происходить до, одновременно и/или после введения дополнительного терапевтического агента или агентов. В некоторых вариантах осуществления введение анти-CD112R антитела и введение дополнительного терапевтического агента происходит в течение примерно одного месяца, или в течение примерно одной, двух или трех недель, или в течение примерно одного, двух, трех, четырех, пяти или шести дней друг от друга.
[00204] Антитело по изобретению (и любое дополнительное терапевтическое средство) можно вводить любыми подходящими способами, включая парентеральное, внутрилегочное и интраназальное, и при желании местного лечения, интралезиальное введение. Парентеральные инфузии включают внутримышечное, внутривенное, внутриартериальное, внутрибрюшинное или подкожное введение. Введение дозы может осуществляться любым подходящим путем, например инъекциями, такими как внутривенные или подкожные инъекции, частично в зависимости от того, является ли введение краткосрочным или хроническим. В данном документе предусмотрены различные схемы введения дозы, включая, помимо прочего, однократное или многократное введение в различные моменты времени, болюсное введение и пульсовую инфузию.
[00205] Антитела по изобретению могут быть приготовлены, дозированы и введены способом, совместимым с надлежащей медицинской практикой. Факторы, которые следует учитывать в этом контексте, включают конкретное заболевание, которое лечат, конкретное млекопитающее, которое лечат, клиническое состояние отдельного субъекта, причину расстройства, место доставки агента, способ введения, схема введения и другие факторы, известные практикующим врачам. Используемый в данном документе термин «разделенная доза» означает разделение единичной стандартной дозы или общей суточной дозы на две или более доз, например, два или более введений единичной стандартной дозы. Антитело можно вводить в виде «разделенной дозы».
[00206] Антитело не обязательно, но опционально приготовлено с одним или более агентами, используемыми в настоящее время для предотвращения или лечения рассматриваемого нарушения. Эффективное количество таких других агентов зависит от количества антитела, присутствующего в составе или композиции, типа расстройства или лечения и других факторов, обсуждаемых выше. Их обычно используют в тех же дозах и с путями введения, как описано в данном документе, или примерно от 1 до 99% доз, описанных в данном документе, или в любой дозе и любым путем, которые эмпирически/клинически определены как подходящие. В некоторых вариантах осуществления антитело представлено в составе для немедленного высвобождения, а другой агент приготовлен для пролонгированного высвобождения или наоборот.
Промышленные изделия
[00207] В другом аспекте изобретения предложено промышленное изделие, содержащее материалы, пригодные для лечения, профилактики и/или диагностики расстройств, описанных выше. Изделие содержит контейнер и размещенную с контейнером или находящуюся на нем этикетку или вкладыш в упаковку. Подходящие контейнеры включают, например, бутыли, флаконы, шприцы, пакеты с растворами для в/в введения и т. д. Контейнеры можно формировать из различных материалов, например из стекла или пластика. Контейнер содержит композицию, которая сама по себе или в комбинации с другой композицией, эффективна для лечения, профилактики и/или диагностики состояния, и может иметь стерильный отверстие доступа (например, контейнер может представлять собой пакет или флакон с раствором для внутривенного введения, имеющий пробку, к имеющий пробку, выполненную с возможностью прокалывания иглой для подкожных инъекций). По крайней мере один активный агент в композиции представляет собой антитело по изобретению. На этикетке или на листке-вкладыше в упаковку указано, что композицию применяют для лечения предпочтительного патологического состояния. Кроме того, изделие может содержать (а) первый контейнер с содержащейся в нем композицией, причем композиция содержит антитело по изобретению; и (b) второй контейнер с содержащейся в нем композицией, причем композиция содержит дополнительный цитотоксический или иной терапевтический агент. Промышленное изделие в этом варианте осуществления изобретения может дополнительно содержать вкладыш в упаковку, указывающий, что композиции могут использоваться для лечения конкретного состояния. Альтернативно или дополнительно изделие может дополнительно содержать второй (или третий) контейнер, содержащий фармацевтически приемлемый буфер, такой как бактериостатическая воду для инъекций (BWFI), фосфатно-солевой буферный раствор, раствор Рингера и раствор декстрозы. Оно может дополнительно включать другие материалы, желательные с коммерческой точки зрения и с точки зрения пользователя, включая другие буферы, разбавители, фильтры, иглы и шприцы.
[00208] Понятно, что любое из вышеуказанных промышленных изделий может включать иммуноконъюгат по изобретению вместо или в дополнение к анти-CD112R антителу.
ПРИМЕРЫ
Пример 1. Получение анти-CD112R антител.
[00209] Антигены биотинилировали с использованием набора для сульфо-NHS-биотинилирования EZ-Link от Pierce. Козий F(ab')2, конъюгированный FITC, к каппа-цепи человека (LC-FITC), ExtrAvidin-PE (EA-PE) и стрептавидин-AF633 (SA-633) были получены от Southern Biotech, Sigma и Molecular Probes, соответственно. Разделительные колонки со стрептавидиновыми микрогранулами и MACS LC были приобретены у Miltenyi Biotec. Козье антитело, конъюгированное с РЕ, к IgG человека (Human-PE) было получено от Southern Biotech.
[00210] Первичное обнаружение.
[00211] Восемь наивных синтетических дрожжевых библиотек человека, каждая из ~ 109 вариантов, размножали, как описано ранее (см., например, Y. Xu et al, Addressing polyspecificity of antibodies selected from an in vitro yeast presentation system: a FACS-based, high-throughput selection and analytical tool. PEDS 26.10, 663-70 (2013); WO 2009036379; WO 2010105256; и WO 2012009568). Для первых двух раундов отбора была выполнена методика магнитной сортировки гранул с использованием системы Miltenyi MACS, как описано ранее (см., например, Siegel et al, High efficiency recovery and epitope-specific sorting of an scFv yeast display library". J Immunol Methods 286(1-2), 141-153 (2004). Вкратце, дрожжевые клетки (~ 1010 клеток/библиотеку) инкубировали с 1,5 мл 10 нМ биотинилированного Fc-слитого антигена в течение 15 минут при 30°C в промывочном буфере (фосфатно-солевой буфер (ФСБ)/0,1% бычий сывороточный альбумин (БСА)). После однократного промывания 40 мл ледяного промывочного буфера осадок клеток ресуспендировали в 20 мл промывочного буфера, к дрожжам добавляли стрептавидиновые микрогранулы (500 мкл) и инкубировали в течение 15 мин при 4°C. Затем дрожжи осаждали, ресуспендировали в 20 мл промывочного буфера и наносили на колонку Miltenyi LS. После загрузки 20 мл на колонку, ее промывали 3 раза 3 мл промывочного буфера. Колонку затем удаляли из магнитного поля и дрожжи элюировали 5 мл питательной среды и затем выращивали в течение ночи. Следующие раунды селекции были выполнены с использованием проточной цитометрии. Приблизительно 2×107 дрожжей осаждали, трижды промывали промывочным буфером и инкубировали при 30°C либо с уменьшающимися концентрациями биотинилированного антигена (от 100 до 1 нМ) в условиях равновесия, либо с реагентом истощения полиспецифичности (PSR), чтобы удалить неспецифические антитела из выборки. Для истощения PSR библиотеки инкубировали разведением 1:10 биотинилированного реагента PSR, как описано ранее (см., например, Y. Xu et al, Addressing polyspecificity of antibodies selected from an in vitro yeast presentation system: a FACS-based, high-throughput selection and analytical tool. PEDS 26.10, 663-70 (2013). Затем дрожжи дважды промывали промывочным буфером и окрашивали вторичными реагентами LC-FITC (разбавленным 1:100) и либо SA-633 (разбавленным 1:500), либо EAPE (разбавленным 1:50) в течение 15 минут при 4°C. После двукратной промывки промывочным буфером осадки клеток ресуспендировали в 0,3 мл промывочного буфера и переносили в сортирующие пробирки с сетчатыми крышками. Отбор с использованием давления аффинности для отбора и выделения антител с более высокой аффинностью осуществляли путем конкуренции с холодным (т.е. немеченым) антигеном.
[00212] Сортировку проводили с использованием сортировщика FACS ARIA (BD Biosciences) и определяли сортировочные интервалы для отбора антител с желаемыми характеристиками. Отборочные раунды повторяли до тех пор, пока не была получена популяция со всеми желаемыми характеристиками. После последнего раунда сортировки дрожжи высевали и отбирали отдельные колонии для характеристики.
[00213] Перетасовывание партии легкой цепи.
[00214] Протокол диверсификации легкой цепи использовался на этапе первичного обнаружения для дальнейшего обнаружения и улучшения антител.
[00215] Протокол диверсификации партии легких цепей: тяжелые цепи из наивной селекции экстрагировали из дрожжей с помощью ПЦР и трансформировали в библиотеку легких цепей с количеством вариантов 5×106. Селекции были выполнены с одним раундом MACS и тремя раундами FACS, как описано в наивном обнаружении. В различных раундах FACS библиотеки проверяли на связывание PSR и давление аффинности путем титрования антигена до 0,5 нМ. Сортировка проводилась для получения популяции с желаемыми характеристиками.
[00216] Оптимизация антител
[00217] Оптимизацию антител проводили путем получения вариантов вариабельных областей тяжелой цепи, как описано ниже.
[00218] Селекция CDRH1 и CDRH2: CDRH3 одного антитела рекомбинировали в предварительно созданную библиотеку с вариантами CDRH1 и CDRH2 с количеством вариантов 1 × 108, и отбор выполнялся с помощью одного раунда MACS и трех раундов FACS, как описано в наивном обнаружении. В различных раундах FACS библиотеки исследовали на предмет связывания PSR, перекрестной реактивности у мыши, и давления аффинности путем титрования или давления аффинности путем предварительного образования комплекса антигена с исходным Fab или исходным IgG для обогащения связывающими веществами с более высокой аффинностью, чем исходный IgG. Сортировка проводилась для получения популяции с желаемыми характеристиками.
[00219] Производство и очистка антител
[00220] Клоны дрожжей выращивали до насыщения и затем индуцировали в течение 48 часов при 30°С при встряхивании. После индукции дрожжевые клетки осаждали и супернатанты собирали для очистки. IgG очищали с использованием колонки с белком А и элюировали уксусной кислотой, рН 2,0. Фрагменты Fab были получены путем расщепления папаином и очищены с помощью KappaSelect (GE Healthcare LifeSciences).
[00221] Измерения KD с использованием ForteBio
[00222] Измерения аффинности с использованием ForteBio выполняли на Octet RED384 в целом, как описано ранее (см., например, Estep et al., High throughput solution-based measurement of antibody-antigen affinity and epitope binning. Mabs 5(2), 270-278 (2013)). Вкратце, измерения аффинности с использованием ForteBio выполняли путем загрузки IgG в оперативном режиме на сенсоры AHQ. Сенсоры уравновешивали в автономном режиме в буфере для анализа в течение 30 минут, а затем контролировали в оперативном режиме в течение 60 секунд для установления исходного уровня. Сенсоры с нагруженными IgG подвергались воздействию 100 нМ антигена в течение 3 минут, а затем переносились в буфер для анализа на 3 минуты для измерения скорости диссоциации. Все кинетики анализировали с использованием модели связывания 1:1.
[00223] Биннинг эпитопа/блокирование лиганда с использованием ForteBio
[00224] Биннинг эпитопа/блокирование лигандов проводили с использованием стандартного сэндвич-анализа перекрестного блокирования. Контрольный IgG к мишени или рецептор нагружали на сенсоры AHQ, а незанятые Fc-связывающие сайты на сенсоре блокировали нерелевантным (нецелевым) антителом IgG1 человека. Затем сенсоры подвергали воздействию 100 нМ целевого антигена, а затем второго антитела к мишени. Дополнительное связывание вторым антителом после ассоциации антигена указывает на незанятый эпитоп (не конкурентный), в то время как отсутствие связывания указывает на блокирование эпитопа (блокирование конкурента или лиганда).
[00225] Кинтетический анализ Biacore
[00226] Для измерений на основе Biacore антиген был ковалентно связан с чипом C1 захвата Fc к антителу мыши с использованием набора иммобилизации по аминогруппе (GE Healthcare Bio-Sciences). Связь между антигеном и пятиточечным титрованием антитела трехкратным разведением, начиная с 27 нМ, измеряли в течение 300 секунд. Затем в течение 3600 секунд измеряли диссоциацию между антигеном и антителом. Кинетические данные были проанализированы и адаптированы в целом с использованием модели связывания 1:1.
Пример 2. Анти-CD112R антитела связываются с CD112R.
[00227] Анализ связывания на клетках
[00228] Оценивали способность анти-CD112R антител связываться с CD112R, экспрессируемым на клетках. 1×105 клеток Jurkat (линия клеток острой Т-клеточной лейкемии, ATCC № TIB-152), которые были либо дикого типа, либо сконструированы для сверхэкспрессии CD112R человека (Jurkat-CD112R OE), добавляли в каждую лунку 96-луночного планшета с V-образным дном и окрашивали либо анти-CD112R антителами, либо изотипическим контролем IgG1 (0,63 мкг/мл) в течение 1 часа при 4°C. Клетки дважды промывали ФСБ+ 2% ФБС и ресуспендировали с антителом к Fc IgG человека, конъюгированным с Alexa Fluor® 647 (Biolegend, № по каталогу 409320), разведенным 1:100 в ФБС+2% ФБС, и инкубировали при 4°C в течение 30 минут. Затем клетки дважды промывали и ресуспендировали в ФСБ+2% ФБС. Клеточные данные были получены с использованием LSRFortessa X-20 (BD Biosciences) и проанализированы с помощью программного обеспечения FlowJo (Tree Star).
[00229] Результаты представлены на Фиг. 1. Количественное связывание антител с клетками Jurkat-CD112R OE оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) сигнала Alexa Fluor® 647. Эти результаты демонстрируют, что анти-CD112R антитела связываются с клетками, экспрессирующими CD112R. Сводная информация о связывании антител представлена в Таблице 2.
Пример 3. Анти-CD112R антитела блокируют связывание CD112 с клетками, экспрессирующими CD112R.
[00230] Анализ блокировки на клетках
[00231] Оценивали способность анти-CD112R антител блокировать связывание CD112 с клетками, экспрессирующими CD112R. 1×105 клеток Jurkat (линия клеток острой Т-клеточной лейкемии, ATCC № TIB-152), которые были сконструированы для сверхэкспрессии CD112R человека (Jurkat-CD112R OE), добавляли в каждую лунку 96-луночного планшета с V-образным дном и окрашивали серийными разведениями либо анти-CD112R антител, либо изотипического контроля IgG1 (максимальная концентрация, 10 мкг/мл) в течение 1 часа при 4°C. Клетки дважды промывали ФСБ+2% ФБС и ресуспендировали с биотинилированным his-меченным CD112 человека (3 мкг/мл) (BPS Bioscience, № 71234) и PE-стрептавидином (5 мкг/мл) (Biolegend, № 405204) в ФСБ+2% ФБС, и инкубировали 2 часа при 4°C. Затем клетки дважды промывали и ресуспендировали в ФСБ+2% ФБС. Клеточные данные были получены с использованием LSRFortessa X-20 (BD Biosciences) и проанализированы с помощью программного обеспечения FlowJo (Tree Star).
[00232] Результаты представлены на Фиг. 2А и в Таблице 2. Количественное определение связывания CD112 с клетками оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) сигнала PE и отображали как процент ингибирования. Процент ингибирования рассчитывали как [100 - ((MFI тестируемого образца/Макс. MFI)*100%)]. Эти результаты демонстрируют, что анти-CD112R антитела ингибируют способность CD112 связываться с экспрессирующими CD112R клетками дозозависимым образом. Сводная информация о связывании и блокировании антител представлена в Таблице 2.
[00233] Способность анти-CD112R антител блокировать CD112R человека также оценивали с помощью ИФА. Вкратце, 96-луночные планшеты Nunc Maxisorp покрывали 1 мкг/мл слитого белка CD112R-hIgG4 в ФСБ в течение ночи при 4°C. Затем планшеты промывали 6 раз ФСБ+0,01% Tween-20 (ФБСТ) и затем блокировали 200 мкл ФСБ+1% БСА в течение 1,5 часов при комнатной температуре. После блокирования планшеты промывали 6 раз ФСБТ. Затем добавляли 100 мкл анти-CD112R антител в ФСБ+1% БСА до конечной исходной концентрации 10 мкг/мл с 4-кратными серийными разведениями. Планшеты инкубировали при комнатной температуре 1,5 часа. Следующие планшеты промывали 6 раз ФСБТ и затем инкубировали с 1 мкг/мл белка Fc CD112 (R&D Systems, кат № 9317-N2-050), который был биотинилирован с помощью набора для сульфо-NHS-биотинилирования (Thermo Fisher, кат. № 21925) в 100 мкл ФСБ+1% БСА в течение 1 часа при комнатной температуре. Затем планшеты промывали 6 раз ФСБТ и затем инкубировали со стрептавидин-HRP (Biolegend, № по каталогу 405210), разведенным в ФСБ+1% БСА, в соответствии с рекомендациями производителя в течение 1 часа при комнатной температуре. После этой инкубации планшеты промывали 6 раз ФСБТ и проявляли субстратом TMB (Life Technologies, № по каталогу 002023). Реакцию останавливали равным объемом стоп-реагента (Life Technologies, № по каталогу SS04). Поглощение при 450 нм (ОП 450) измеряли на планшет-ридере SpectraMax.
[00234] Результаты представлены на Фиг. 2В и в Таблице 2. Эти результаты демонстрируют, что анти-CD112R антитела блокируют связывание CD112R человека с CD112 человека. Процент ингибирования рассчитывали как [100 - ((ОП 450 тестового образца/Макс. ОП 450)*100%)] Макс. ОП 450 определяли как поглощение при 450 нм в отсутствие антитела.
[00235] Таблица 2: Сводная информация о связывании и блокировании антител
Пример 4. Анти-CD112R антитела усиливают опосредованное NК-клетками уничтожение.
[00236] Анализ цитотоксичности NK
[00237] Чтобы определить влияние анти-CD112R антител человека на цитотоксичность, опосредованную NK-клетками, NK-клетки человека совместно культивировали с клетками-мишенями REH (линия клеток острого лимфоцитарного лейкоза, отличная от T/B, ATCC № CRL-8286) в присутствии анти-CD112R антитела или изотипического контроля.
[00238] Вкратце, NK-клетки были выделены из PBMC трех здоровых доноров посредством отрицательной селекции (набор для выделения NK-клеток Easysep™, Stemcell № 17955) и активированы в течение 16 часов с помощью IL-2 (10 единиц/мл) (Peprotech № 200-02) и IL-12 (20 нг/мл (Peprotech № 200-12) в RPMI+10% ФБС+1% пенициллин-стрептомицин (R10) (ThermoFisher). Клетки REH промывали, ресуспендировали в ФСБ (ThermoFisher) и метили с помощью CellTrace ™violet (CTV) (ThermoFisher № C34557) в течение 12 минут при 37°C. Затем клетки REH промывали ФСБ+10% ФБС и затем ресуспендировали в R10. После активации NK-клетки промывали и ресуспендировали в R10. 2,5 × 105 NK-клеток и 5 × 104 клеток REH добавляли в каждую лунку 96-луночного планшета с плоским дном для соотношения эффекторных клеток-мишеней 5:1. Анти-CD112R антитело и антитело изотипа IgG1 разводили в R10 и также добавляли в каждую лунку до конечной концентрации 10 мкг/мл. Каждое условие выполнялось в двух экземплярах. Затем планшеты инкубировали в течение 4 часов при 37°C. Затем клетки промывали и инкубировали при комнатной температуре в течение 30 минут в темноте с красителем для определения жизнеспособности 7-AAD (1 мкг/мл) (Biolegend № 420404) для специфической маркировки мертвых клеток. Данные жизнеспособности клеток были получены с использованием LSRFortessa X-20 (BD Biosciences) и проанализированы с помощью программного обеспечения FlowJo (Tree Star). Мертвые клетки REH были определены как двойные положительные по CTV и 7-AAD клетки.
[00239] Результаты представлены на Фиг. 3. Цитотоксичность (процент относительно изотипического контроля) рассчитывали как ((процент мертвых клеток с тестовым антителом минус процент мертвых клеток с антителом изотипического контроля), деленный на процент мертвых клеток с антителом изотипического контроля) x 100. Обработка NK-клеток каждым из описанных в данном документе анти-CD112R антител приводила к усилению опосредованной клетками цитотоксичности против клеток REH по сравнению с изотипическим контролем. Эти результаты демонстрируют, что анти-CD112R антитела усиливают опосредованное уничтожение NK-клеток.
Пример 5. Анти-CD112R антитела усиливают активацию CD8+ Т-клеток, управляемую антигеном.
[00240] Антигенспецифический анализ CD8+ Т-клеток.
[00241] Оценивали влияние анти-CD112R антител на активацию CD8+ Т-клеток, управляемую антигеном. Первичная ограниченная по HLA-A*0201 цитомегаловирусная (CMV) линия CD8+ Т-клеток (Astarte Biologics № 1049, партия № 3782DE17) инкубировали с активированными пептидами клеток Colo205 (клеточная линия аденокарциномы толстой кишки, ATCC № CCL-222) в присутствии анти-CD112R антитела или изотипического контроля.
[00242] Вкратце, CMV-специфические Т-клетки размораживали, промывали и ресуспендировали в X-VIVO 10 (ThermoFisher № BW04380Q). 2 × 104 CMV Т-клеток добавляли в каждую лунку 96-луночного круглодонного планшета и оставляли в покое в течение 4 часов при 37°C. После начального периода покоя в каждую лунку добавляли 5 × 104 клеток Colo205 и пептид CMV pp65 (1 нг/мл) (Anaspec № AS-63937). Затем анти-CD112R антитело и изотипическое антитело разводили в X-VIVO 10 и добавляли в каждую лунку до конечной концентрации 10 мкг/мл. Каждое условие выполнялось в двух экземплярах. Затем планшеты инкубировали в течение 16 часов при 37°C. Супернатанты собирали из каждой лунки и подвергали одному циклу замораживания/оттаивания перед оценкой цитокинов. После оттаивания аналитические супернатанты разбавляли 1:5 в X-VIVO 10, а затем измеряли гамма-интерферон (ИФН-g) в аналитическом супернатанте с помощью мультиплексного анализа с использованием магнитных гранул Luminex для CD8+ T-клеток человека (Millipore Sigma № HCD8MAG-15K) и проводили на Millipore FlexMap 3D.
[00243] Результаты представлены на Фиг. 4. Условия испытаний. Уровни ИФН-g определяли количественно на основе стандартной кривой, построенной для определенных концентраций IFNg. CD8+ Т-клетки, обработанные анти-CD112R антителами 2 и 5, приводили к большей секреции ИФН-g, чем наблюдаемая с изотипическим контролем. Эти результаты демонстрируют, что анти-CD112R антитела усиливают активацию CD8+ Т-клеток, управляемую антигеном.
Пример 6. Комбинация антитела к CD112R мыши и антитела к TIGIT мыши оказывает терапевтический эффект на модели опухоли CT-26 мыши.
[00244] Эффективность комбинированной блокады CD112R и Tigit in vivo
[00245] Эффективность блокады CD112R и TIGIT в качестве отдельных и комбинированных агентов была протестирована на мышиной сингенной модели опухоли аденокарциномы толстой кишки CT26. Самкам мышей Balb/c в возрасте 7 недель (Charles River Laboratories, № 028) имплантировали подкожно 0,1 × 106 клеток CT26.WT (ATCC № CRL-2638) в 0,1 мл инокуляционной матрицы 50% матригеля. Мышей рандомизировали на группы по 10 мышей в каждой (всего 40 мышей) стратифицированным способом с объемом опухоли в диапазоне 80-120 мм3 и обрабатывали дважды в неделю в течение двух недель путем внутрибрюшинной инъекции, как показано в Таблице 3.
[00246] Таблица 3: Детали группы лечения
[00247] Объемы опухолей измеряли каждые 2-3 дня, пока опухоли не достигли предельного размера IACUC (<2000 мм3).
[00248] Таблица 4: Сведения об антителах
[00249] Результаты представлены на Фиг 5. На графике показаны средние объемы опухолей для каждой группы лечения в зависимости от времени. Результаты демонстрируют, что комбинация анти-CD112R антитела с анти-TIGIT антителом была эффективной для снижения роста опухоли по сравнению с животными, получавшими изотипический контроль, в то время как монотерапия анти-CD112R антителом или анти-TIGIT антителом показала либо отсутствие активности, либо лишь умеренный эффект на снижение роста опухоли. Хотя одно анти-CD112R антитело не показало активности в этом анализе, другие эксперименты, представленные в настоящем документе, демонстрируют пользу монотерапии анти-CD112R антителом. См., например, Фиг. 9.
Пример 7. Повышенная экспрессия CD112R в PBMC после активации анти-CD3 антителом.
[00250] CD112R активируется в активированных РВМС in vitro
[00251] Чтобы определить влияние клеточной активации на экспрессию CD112R, PBMC человека стимулировали in vitro анти-CD3 антителом. Мононуклеарные клетки периферической крови (PBMC) от здоровых доноров выделяли из лейкоцитов (Research Blood Components). Отдельные лейкоцитарные пленки обрабатывали отдельно. В каждую коническую пробирку объемом 50 мл (Corning № 430290) добавляли 15 мл лейкоцитарной пленки и разбавляли 15 мл ФСБ (Thermofisher № 14190144) + 2 мМ ЭДТА (Fisher Scientific № BP2482-500) до общего объема 30 мл в каждой пробирке. Разбавленные лейкоцитарные пленки покрывали 14 мл Ficolpaque (GE Healthcare Life Science № 17-544203) и центрифугировали при 2000 об/мин в течение 20 минут при комнатной температуре с выключенным тормозом. Градиентную интерфазу собирали и дважды промывали ФСБ+2 мМ ЭДТА. Выделенные клетки подсчитывали и ресуспендировали в концентрации 2,5-5 × 107 клеток/мл в 10% ДМСО (Sigma-Aldrich № 472301) + 90% инактивированной нагреванием ФБС (ThermoFisher № 16140-071).
[00252] Замороженные РВМС быстро оттаивали и ресуспендировали в среде с добавлением RPMI, которая содержала RPMI+GlutaMax (1x) (ThermoFisher № 61870-036), 10% инактивированный нагреванием ФБС, 1x раствор незаменимых аминокислот MEM (ThermoFisher № 15140-122), 1 мМ пирувата натрия (ThermoFisher № 11360070), 100 Ед/мл Пеницилина/Стрептомицина (ThermoFisher № 15140-122), 1x 2-меркаптоэтанола (ThermoFisher № 21985023), 10 мМ Hepes (ThermoFisher № 15630-080). Выделенные PBMC промывали, подсчитывали и ресуспендировали в концентрации 0,5 × 106 клеток/мл. 1 × 106 клеток на лунку помещали в 24-луночный планшет с плоским дном (Corning № 3526) и стимулировали 0,25 мкг/мл анти-CD3 антитела (клон UCHT1, Biolegend № 300414). Клетки собирали в указанные моменты времени, промывали буфером для FACS, содержащим 1x ФСБ, 2% ФБС и 2 мМ ЭДТА, и переносили в 96-луночный планшет с V-образным дном (Costar № 3894) для окрашивания антител.
[00253] Клетки центрифугировали при 1500 об/мин в течение 3 минут, и супернатант сливали. Клетки ресуспендировали в буфере FACS и инкубировали в течение 1 часа при 4°C с первичными антителами, указанными в Таблице 5, следующим образом:
[00254] Таблица 5
[00255] Клетки дважды промывали и инкубировали в течение 30 минут при 4°C с разведенным 1:100 Alexa 647-конъюгированным антителом к IgG человека (клон HP6017, Biolegend № 409320). Клетки промывали один раз буфером FACS и окрашивали красителем Live/Dead Aqua для определения жизнеспособности, разведенным 1:500 в 1x ФСБ (Thermofisher № L34966) в течение 10 минут при 4°C. Клетки промывали один раз и собирали непосредственно на проточном цитометре X-20 Fortessa (BD Biosciences). Данные анализировали с помощью Flowjo (TreeStar) и Graphpad prism (программное обеспечение Graphpad).
[00256] Результаты представлены на Фиг. 6. Количественное определение связывания антитела к CD112R оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) сигнала Alexa Fluor® 647 для указанного типа клеток. Связывание анти-CD112R антитела показано как кратное по сравнению с отрицательным контролем (FON, (gMFI CD112R, деленное на gMFI изотипического контроля)). Эти результаты демонстрируют, что экспрессия CD112R увеличивается в NK-клетках и Т-клетках после активации анти-CD3 антителом.
Пример 8. Анти-CD112R антитела усиливают дегрануляцию NK-клеток в совместных культурах опухолевых клеток.
[00257] Чтобы определить влияние анти-CD112R антитела на дегрануляцию, опосредованную NK-клетками, NK-клетки человека культивировали совместно с клетками-мишенями Raji (линия клеток лимфомы Беркитта, ATCC № CCL-86), которые ранее были трансдуцированы лентивирусом для экспрессии CD112 (Origene, № RC213693L2), в присутствии антител 35, 38, 44 и изотипического контроля.
[00258] Вкратце, NK-клетки выделяли и объединяли из PBMC трех здоровых доноров посредством отрицательной селекции (набор для выделения NK-клеток Easysep™, Stemcell № 17955) и культивировали в течение 16 часов в среде DMEM+10% ФБС+1% пенициллин-стрептомицин (D10) (ThermoFisher). После инкубации в течение ночи при 37°C NK-клетки промывали и ресуспендировали в D10. Клетки Raji.CD112 собирали, промывали и затем ресуспендировали в D10. 1 × 105 NK-клеток и 5 × 104 клеток Raji.CD112 добавляли в каждую лунку 96-луночного планшета с плоским дном для соотношения эффекторных клеток-мишеней 2:1. Анти-CD112R антитела и контрольное антитело изотипа IgG1 разводили в D10 и добавляли в каждую лунку при начальной концентрации 10 мкг/мл при 10-кратных серийных разведениях. Каждое условие выполнялось в двух экземплярах. Анти-CD107a антитело, конъюгированное с PE (Biolegend, № 328608), и монензин (Biolegend, № 420701) также добавляли в каждую лунку в концентрациях, указанных производителем. Конечный объем каждой лунки составлял 200 мкл. Затем планшеты инкубировали в течение 4 часов при 37°C. Через 4 часа анти-CD3 антитело, конъюгированное с FITC (Biolegend, № 300306), и анти-NKp46 антитело, конъюгированное с APC (Biolegend № 331914), разводили в D10 и добавляли по 50 мкл в каждую лунку. Затем планшеты инкубировали еще 30 минут при 4°C для окрашивания. Затем клетки переносили в планшеты с V-образным дном, дважды промывали и ресуспендировали в ФСБ+2% ФБС. Данные были получены с использованием проточного цитометра LSRFortessa X-20 (BD Biosciences) и проанализированы с помощью программного обеспечения FlowJo (Tree Star). Дегрануляцию NK определяли как частоту встречаемости клеток Cd107a+ в лимфоцитарных воротах CD3- NKp46+.
[00259] Результаты представлены на Фиг. 7. Обработка NK-клеток анти-CD112R антителами, описанная в данном документе, привела к усилению дегрануляции NK, как измерено с помощью окрашивания CD107a, по сравнению с изотипическим контролем.
Пример 9. Анти-CD112R антитела повышают активацию NK-клеток в совместных культурах опухолевых клеток PBMC.
[00260] Чтобы оценить влияние антител к CD112R на активацию NK-клеток, несколько антител оценивали в совместных культурах опухолевых клеток РВМС. Повышенная регуляция CD137 (4-1BB), который ранее был установлен как маркер активации NK-клеток (Baessler et al. (2010) Blood 115 (15); André et al. (2018) Cell 175, 1731-1743), измеряли на NK-клетках из PBMC, совместно культивируемых с клетками-мишенями K562 (линия клеток хронического миелогенного лейкоза, ATCC № CCL-243) с анти-CD112R антителами или изотипическими контрольными антителами.
[00261] Вкратце, замороженные PBMC, выделенные из лейкоцитов здоровых доноров, размораживали, промывали, ресуспендировали в DMEM+10% ФБС+1% пенициллин-стрептомицин (D10) и высевали в 96-луночные планшеты с плоским дном в концентрации 5×105 клеток на лунку и выдерживали 4 часа при 37°C перед добавлением клеток-мишеней и антител. Затем в первом эксперименте (Фиг. 8A-8B) антитела к CD112R и контрольное антитело изотипа IgG1 разводили в D10 и добавляли в каждую лунку в начальной концентрации 10 мкг/мл при 10-кратных серийных разведениях. В следующем эксперименте (Фиг. 8C-8D) в каждую лунку добавляли одну концентрацию (1 мкг/мл) анти-CD112R антитела или контрольного антитела изотипа IgG1. Для обоих экспериментов каждое условие выполнялось в двух повторностях. Затем клетки K562 собирали, промывали и ресуспендировали в D10 и добавляли в каждую лунку в концентрации 5 × 104 клеток на лунку. Конечный объем каждой лунки составлял 200 мкл. Затем планшеты инкубировали в течение 16 часов при 37°C. Через 16 часов клетки переносили на планшеты с V-образным дном и дважды промывали ФСБ+2% ФБС. Клетки окрашивали анти-CD3 антителом, конъюгированным с FITC (Biolegend, № 300306), анти-NKp46 антителом, конъюгированным с BV421 (Biolegend № 331914) и анти-CD137 антителом, конъюгированным с APC (Biolegend, № 309810) в ФСБ+2% ФБС в течение 30 минут при 4°C. Затем клетки дважды промывали и ресуспендировали в ФСБ+2% FBS. Данные были получены с использованием проточного цитометра LSRFortessa X-20 (BD Biosciences) и проанализированы с помощью программного обеспечения FlowJo (Tree Star). Активация NK-клеток определялась как частота встречаемости CD137+ клеток в лимфоцитарных воротах CD3-NKp46+.
[00262] Результаты от двух отдельных доноров из двух независимых экспериментов представлены на Фиг. 8A-8D. Добавление анти-CD112R антитела к совместным культурам клеток PBMC-K562 привело к значительной активации NK-клеток по сравнению с изотипическим контролем, как измерено с помощью активации CD137 на NK-клетках.
Пример 10. Анти-CD112R антитело снижает рост опухоли в модели CT-26.
[00263] Эффективность блокады CD112R in vivo оценивали на мышиной сингенной модели опухоли аденокарциномы толстой кишки CT26.WT. Самкам мышей BALB/cAnNTac в возрасте 7 недель (Taconic Biosciences, кат. № BALB-F) подкожно имплантировали в правый бок 0,2×106 CT26.WT (ATCC, кат. № CRL-2638) в 0,1 мл бессывороточной среды с 50% Geltrex (GIBCO, кат. № A1432-02) и 50% RPMI-1640 (GIBCO, кат. № A10491-01). Мышей с пальпируемыми опухолями рандомизировали на 4-й день после имплантации и лечили внутрибрюшинно два раза в неделю в течение трех недель, начиная со дня рандомизации, как показано в Таблице 6.
[00264] Таблица 6:
[00265] Объемы опухолей измеряли штангенциркулем каждые 2-3 дня, пока опухоли не достигли предельного размера в соответствии с IACUC (<2000 мм 3). Объем опухоли (мм3) рассчитывали следующим образом: ширина (мм) x [длина (мм)] 2×0,5.
[00266] Результаты представлены на Фиг. 9. На графике представлены объединенные данные трех независимых экспериментов, показывающие средние объемы опухолей для каждой группы лечения в зависимости от времени. Эти результаты демонстрируют, что лечение мышей с опухолью антителом к CD112R приводило к значительному ингибированию роста опухоли, измеренному на 24 день после инокуляции.
Пример 11. Блокада CD112R приводит к противоопухолевому иммунитету у мышей с полным отторжением опухоли в модели CT26.
[00267] Противоопухолевый иммунитет оценивали у мышей, которых лечили анти-CD112R антителом, и которые демонстрировали полные ответы на первичные заражения опухолью CT26.WT. Для первичного заражения самкам мышей BALB/cAnNTac в возрасте 7 недель (Taconic Biosciences, кат. № BALB-F) подкожно имплантировали в правый бок 0,2×106 CT26.WT (ATCC, кат. № CRL-2638) в 0,1 мл бессывороточной среды с 50% Geltrex (GIBCO, кат. № A1432-02) и 50% RPMI-1640 (GIBCO, кат. № A10491-01). Мышей с пальпируемыми опухолями рандомизировали на 4-й день после имплантации и лечили внутрибрюшинно два раза в неделю в течение трех недель, начиная со дня рандомизации, как показано в Таблице 7.
[00268] Таблица 7:
[00269] Объемы опухолей измеряли штангенциркулем каждые 2-3 дня, пока опухоли не достигли предельного размера в соответствии с IACUC (<2000 мм 3). Объем опухоли (мм3) рассчитывали следующим образом: ширина (мм) x [длина (мм)] 2×0,5.
[00270] Все выжившие мыши на 50-й день после имплантации, у которых отсутствовали какие-либо заметные опухоли, считались выжившими/с полным клиническим ответом. Мышей с полным ответом (n=8) из группы, получавшей анти-CD112R антитело, повторно заражали путем инокуляции в левый бок 1 × 106 клеток CT26.WT (ATCC, кат. № CRL-2638) в 0,1 мл бессывороточной среды с 50% Geltrex (GIBCO, кат. № A1432-02) и 50% RPMI-1640 (GIBCO, кат. № A10491-01), что в пять раз превышает дозу первичной инокуляции. В качестве контроля равным по возрасту наивным самкам мышей Balb/c (n=5) также аналогичным образом инокулировали в левый бок 1×106 клеток CT26.WT в 0,1 мл бессывороточной среды с 50% Geltrex и 50% RPMI-1640. Мыши не получали дальнейшего лечения. Объемы опухолей измеряли каждые 2-3 дня, пока опухоли не достигли предельного размера в соответствии с IACUC (<2000 мм 3). Объем опухоли (мм3) рассчитывали следующим образом: ширина (мм) x [длина (мм)] 2×0,5.
[00271] Результаты представлены на Фиг. 10A-10B. На Фиг. 10А представлены объединенные данные 3 независимых экспериментов, показывающие частоту выживания мышей, которым имплантированы первичные опухоли CT26, которых лечили анти-CD112R антителом. На Фиг. 11В показаны средние объемы опухолей у группы лечения анти-CD112R антителом с полным клиническим ответом после повторного заражения опухолью и у наивных мышей, подвергнутых стимуляции контролем, в зависимости от времени. Статистический анализ выполняли с помощью теста Мантела-Кокса на 50-й день после имплантации (Фиг. 10A) и теста Манна-Уитни на 15-й день после имплантации (Фиг. 10B). Эти результаты демонстрируют, что мыши, которых лечили анти-CD112R антителом, проявляли полные клинические ответы после заражения первичной опухолью и последующее быстрое отторжение при повторном заражении опухолью у этих мышей также демонстрирует, что лечение анти-CD112R антителом приводит к развитию иммунологической памяти и защитного иммунитета.
Пример 12. И NK-клетки, и CD8-Т-клетки вносят вклад в терапевтическую активность анти-CD112R антитела при заражении опухолью CT26.
[00272] Эффективность блокады CD112R in vivo оценивали на мышиной сингенной модели опухоли CT26 после истощения популяции NK-клеток или CD8 T-клеток. Для истощения популяции NК и CD8 Т-клетки мышей обрабатывали дважды в неделю в течение трех недель, начиная с рандомизации, антителом Asialo-GM1 («asGM1» на Фиг. 11; Biolegend; кат. № 146002; доза 100 мкл/мышь; внутрибрюшинно) и анти-CD8a антителом (Bioxcell; кат. № BE0085; 200 мкг/мышь; внутрибрюшинно), соответственно.
[00273] Самкам мышей BALB/cAnNTac в возрасте 7 недель (Taconic Biosciences, кат. № BALB-F) подкожно имплантировали в правый бок 0,2 x106 CT26.WT (ATCC, кат. № CRL-2638) в 0,1 мл бессывороточной среды с 50% Geltrex (GIBCO, кат. № A1432-02) и 50% RPMI-1640 (GIBCO, кат. № A10491-01). Мышей с пальпируемыми опухолями рандомизировали на 4-й день после имплантации и лечили внутрибрюшинно два раза в неделю в течение трех недель, начиная со дня рандомизации, с помощью антитела 46 (IgG2a мыши к CD112R; 12,5 мг/кг; внутрибрюшинно).
[00274] Объемы опухолей измеряли штангенциркулем каждые 2-3 дня, пока опухоли не достигли предельного размера в соответствии с IACUC (<2000 мм3). Объем опухоли (мм3) рассчитывали следующим образом: ширина (мм) x [длина (мм)] 2×0,5.
[00275] Результаты представлены на Фиг. 11. На графике показаны средние объемы опухолей для каждой группы лечения в зависимости от времени. Эти результаты показывают, что терапевтический эффект анти-CD112R антитела значительно снижается после истощения популяции NK-клеток или CD8 T-клеток. Эти результаты показывают, что как CD8 Т-клетки, так и NK-клетки необходимы для эффективного ингибирования роста опухоли, опосредованного анти-CD112R антителом.
Пример 13. Блокада CD112R активирует опухолевые NK-клетки in vivo.
[00276] Оценка ex vivo маркеров активации NK после введения дозы монотерапии анти-CD112R антитела.
[00277] Чтобы определить влияние анти-CD112R антитела на активацию NK-клеток in vivo, самкам мышей BALB/cAnNTac в возрасте 7 недель (Taconic Biosciences, кат. № BALB-F) имплантировали подкожно в правый бок 0,2×106 CT26.WT (ATCC, кат. № CRL-2638) в 0,1 мл 50% Geltrex (GIBCO, кат. № A1432-02). Мышей с пальпируемыми опухолями рандомизировали на 4-й день после имплантации и им вводили 500 мкг либо антитела изотипического контроля (клон C1.18.4, BioXcell,кат. № 0085), либо антитела 46 (IgG2a мыши к CD112R). Обоим группам также совместно вводили 500 мкг изотипического контроля (клон MOPC-21, Bioxcell, кат № 0083). Терапии готовили в стерильном 1x ФСБ (GIBCO, кат. № 14190-136), и общий объем 100 мкл вводили внутрибрюшинно.
[00278] Обработка опухолей: мышей умерщвляли и опухоли резецировали через 24 часа после обработки. Опухоль была переработана в суспензии единичных клеток путем разрушения ткани через фильтр с размером ячеек 440 микрон (Costar № 3480), помещенный в центрифужную пробирку объемом 50 мл (Falcon № 352350), с использованием грубого конца поршня шприца объемом 3 мл (BD 301077) в буфере FACS (1x ФСБ, 2% термоинактивированный ФБС, GIBCO, кат. № 16140-071; 2 мМ ЭДТА, Fisher Bioreagents, кат. № BP2482-500). Вытесненные опухоли еще раз фильтровали через сетчатый фильтр размером 70 микрон (Falcon, кат. № 352350), а оставшуюся ткань дополнительно разрушали с помощью поршня шприца объемом 3 мл. Клетки центрифугировали при 800 g в течение 10 минут. Осадки клеток ресуспендировали в буфере FACS.
[00279] Повторная стимуляция ex vivo: Примерно половину суспензии единичных клеток переносили в 96-луночные планшеты из полипропилена с U-образным дном глубиной 2 мл (Thermofisher № AB-0932) и центрифугировали при 1000 g в течение 5 минут при 4°C. Клетки ресуспендировали в предварительно нагретой среде 1x RPMI+Glutamax (GIBCO № 61870-035) с 10% инактивированной нагреванием ФБС, содержащей 20 нг/мл PMA (Abcam № ab120297), 500 нг/мл соли иономицина Ca2+ (Abcam № ab120116 ), 5 мкг/мл брефельдина А (Biolegend № 420601) и 2 мкМ монензина (Biolegend № 420701). Клетки инкубировали течение 3,5 часов при 37°C, 5% CO2. Клетки снова центрифугировали, как описано выше.
[00280] Окрашивание поверхностного антигена антителом для FACS: Осадки клеток один раз промывали 500 мкл холодного буфера FACS. Клетки ресуспендировали в 100 мкл буфера FACS с TruStain fcX™ (антитело к CD16/32 мыши, при разведении, указанном в Таблице 8. Клетки инкубировали на льду в течение 15 минут. Готовили коктейль из антител к поверхностным антигенам (подробности см. в Таблице 8) и добавляли непосредственно к предварительно заблокированным клеткам. Клетки инкубировали 1 час на льду. Затем клетки дважды промывали 500 мкл буфера FACS.
[00281] Окрашивание красителями в отношении жизнеспособности: клетки окрашивали красителем для оценки жизнеспособности Live/Dead Aqua, разведенным 1:500 в 1x ФСБ (Thermofisher № L34966) в течение 10 минут при 4°C.
[00282] Фиксация: клетки промывали один раз и фиксировали 200 мкл буфера для фиксации/пермеабилизации eBioscience Foxp3 (Thermofisher № 00-5523-00, используя протокол производителя для рекомендаций по разведению) в течение ночи при 4°C.
[00283] Окрашивание внутриклеточного антигена антителом для FACS: Клетки были пермеабилизированы путем добавления непосредственно 200 мкл 1х буфера для пермеабилизации eBioscience (см. протокол производителя для рекомендаций по разведению). Клетки центрифугировали при 1000 g в течение 5 минут и окрашивали внутриклеточной панелью антител при конечном разведении в 1x буфере для пермеабилизации eBioscience. Клетки инкубировали в течение 1 часа при комнатной температуре. Клетки дважды промывали 500 мкл буфера для пермеабилизации, ресуспендировали в 150 мкл буфера FACS.
[00284] Клетки собирали на проточном цитометре X-20 Fortessa (BD Biosciences). Данные анализировали с помощью Flowjo (FlowJo, LLC) и Graphpad prism (программное обеспечение Graphpad).
[00285] Таблица 8: Антитела, используемые на Фиг. 12 A-B.
Fire™
Dead
[00286] Результаты представлены на Фиг. 12A-B. На Фиг. 12А показана частота инфильтрирующих опухоль NK-клеток, экспрессирующих CD69. На Фиг. 12В показана частота инфильтрующих опухоль NK-клеток, экспрессирующих гранзим В, после рестимуляции ex vivo. NK-клетки гейтировали следующим образом: популяции CD45+, CD45+ SSC-ALow, живых, синглетных, NKp46+ TCRb-. Положительный гейт был установлен на основе отрицательного контроля флуоресценции минус один (FMO). P-значение было получено с использованием непарного t-критерия (*, p <0,05; **, p <0,01, ***, p <0,001). Эти результаты демонстрируют, что блокада CD112R значительно увеличивает экспрессию маркера ранней активации CD69 и белка протеазосодержащей вакуоли, гранзима В, во внутриопухолевых NK-клетках в модели опухоли CT-26.
Пример 14. Комбинация анти-CD112R и анти-PD1 антител оказывает терапевтический эффект и увеличивает выживаемость без опухолей на модели опухоли CT-26 мыши.
[00287] Эффективность комбинированной блокады CD112R и PD-1 in vivo
[00288] Эффективность блокады CD112R и PD-1 в качестве монотерапии и в качестве комбинированной терапии была протестирована на сингенной модели опухоли аденокарциномы толстой кишки CT-26. Самкам мышей BALB/cAnNTac в возрасте 6 недель (Taconic Biosciences, Balb-F) подкожно имплантировали в правый бок 0,2×106 CT26.WT (ATCC, кат. № CRL-2638) в 0,1 мл бессывороточной среды с 50% Geltrex (GIBCO, кат. № A1432-02) и 50% RPMI-1640 (GIBCO, кат. № A10491-01). Мышей рандомизировали на группы по 10 мышей в каждой со средним объемом опухоли 90 мм3 с использованием метода рандомизации парного распределения. Мышей обрабатывали дважды в неделю в течение двух недель путем внутрибрюшинной инъекции, как показано в Таблице 9. Подробные сведения о средствах лечения включены в Таблицу 10.
[00289] Таблица 9: Подробности для группы лечения
[00290] Таблица 10: Сведения об антителах
[00291] Объемы опухолей измеряли дважды в неделю до тех пор, пока опухоли не достигли предельного размера в соответствии с IACUC (<2000 мм3). Объем опухоли (мм3) рассчитывали следующим образом: ширина (мм) x [длина (мм)] 2×0,52.
[00292] Результаты представлены на Фиг. 13A-F. На Фиг. 13A-E показаны измерения среднего и индивидуального объема опухоли, соответственно, для каждой группы лечения в зависимости от времени. Результаты, показанные на Фиг. 13A, демонстрируют, что комбинация анти-CD112R с анти-PD-1 антител была эффективной и статистически значимой (при измерении на 21 день с помощью непарного t-критерия) в отношении снижения роста опухоли по сравнению с животными, которым вводили изотипический контроль. Монотерапия анти-CD112R или анти-PD-1 антителами также продемонстрировала активность в снижении роста опухоли. На Фиг. 13F показана общая выживаемость без опухолей на 50-й день после имплантации, показанная как доля выживших без опухолей на группу после лечения, как описано выше. Эти результаты демонстрируют, что комбинация анти-CD112R антитела с анти-PD-1 антителом обеспечивает более высокую выживаемость без опухолей, чем изотипический контроль или монотерапевтический агент.
Пример 15. Связывание анти-CD112R антител с клетками, экспрессирующими мышиный CD112R
[00293] Способность анти-CD112R антител связываться с CD112R мыши оценивали на клетках, сверхэкспрессирующих CD112R мыши. 0,8 × 105 клеток 293T (ATCC CRL-3216), которые были сконструированы для сверхэкспрессии мышиного CD112R (293T.mCD112R), добавляли в каждую лунку 96-луночного планшета с V-образным дном и окрашивали либо анти-CD112R антителами, либо изотипическим контролем IgG1 при начальной концентрации 10 мкг/мл, в 3-кратных серийных разведениях в течение 30 минут при 4°C. Клетки дважды промывали ФСБ+ 2% ФБС и ресуспендировали с антителом к Fc IgG человека, конъюгированным с Alexa Fluor® 647 (Biolegend, № по каталогу 409320) , разведенным 1:100 в ФБС+2% ФБС, и инкубировали при 4°C в течение 20 минут. Затем клетки дважды промывали и ресуспендировали в ФСБ+2% ФБС. Клеточные данные были получены с использованием LSRFortessa X-20 (BD Biosciences) и проанализированы с помощью программного обеспечения FlowJo (Tree Star).
[00294] Результаты представлены на Фиг. 17 и в Таблице 11. Количественное связывание антител с клетками 293T.mCD112R оценивали по среднему геометрическому значению интенсивности флуоресценции (gMFI) сигнала Alexa Fluor® 647. Эти результаты демонстрируют, что несколько анти-CD112R антител связываются с клетками, экспрессирующими CD112R мыши.
Пример 16. Связывание анти-CD112R антител с растворимым мышиным CD112R
[00295] Способность анти-CD112R антител связываться с растворимым CD112R мыши оценивали с помощью ИФА. Вкратце, 96-луночные планшеты Nunc Maxisorp покрывали 1 мкг/мл анти-CD112R антител или изотипического контроля (Biolegend, кат. № 403502) в ФСБ в течение ночи при 4°C. Затем планшеты промывали 6 раз ФСБ+0,01% Tween-20 (ФБСТ) и затем блокировали 200 мкл ФСБ+1% БСА в течение 1,5 часов при комнатной температуре. После блокирования планшеты промывали 6 раз ФСБТ. Затем добавляли 100 мкл слитого белка CD112R-hIgG4 мыши в ФСБ+1% БСА до конечной исходной концентрации 10 мкг/мл с 4-кратными серийными разведениями. Планшеты инкубировали при комнатной температуре 1,5 часа. Следующие планшеты промывали 6 раз ФСБТ, а затем инкубировали с анти-IgG4 HRP антителом (Thermo Fisher, кат. № MA1-33437) в 100 мкл в течение 1 часа при комнатной температуре. Планшеты промывали 6 раз ФСБТ и проявляли субстратом TMB (Life Technologies, № по каталогу 002023). Реакцию останавливали равным объемом стоп-реагента (Life Technologies, № по каталогу SS04). Поглощение при 450 нм(ОП 450) измеряли на планшет-ридере SpectraMax.
[00296] Результаты представлены на Фиг. 18 и в Таблице 11. Эти результаты демонстрируют, что анти-CD112R антитела связываются с растворимым CD112R мыши.
Пример 17. Ингибирование или блокирование связывания мышиного CD112R с CD112
[00297] Способность видов анти-CD112R антител с перекрестной реактивностью блокировать связывание CD112R мыши с CD112 мыши оценивали с помощью ИФА. Вкратце, 96-луночные планшеты Nunc Maxisorp покрывали 1 мкг/мл CD112 мыши (Sino Biological, № по каталогу 50318-M08H) в ФСБ в течение ночи при 4°C. Затем планшеты промывали 6 раз ФСБ+0,01% Tween-20 (ФБСТ) и затем блокировали 200 мкл ФСБ+1% БСА в течение 1,5 часов при комнатной температуре. После блокирования планшеты промывали 6 раз ФСБТ. Затем добавляли 50 мкл анти-CD112R антител или изотипического контроля (Biolegend, № по каталогу 403502) в ФСБ+1% БСА до конечной начальной концентрации 40 мкг/мл с 2-кратными серийными разведениями. 50 мкл слитого белка CD112R-hIgG4 мыши также добавляли в каждую лунку до конечной концентрации 2 мкг/мл. Планшеты инкубировали при комнатной температуре 1,5 часа. Следующие планшеты промывали 6 раз ФСБТ, а затем инкубировали с анти-IgG4 HRP антителом (Thermo Fisher, кат. № MA1-33437) в 100 мкл в течение 1 часа при комнатной температуре. Планшеты промывали 6 раз ФСБТ и проявляли субстратом TMB (Life Technologies, № по каталогу 002023). Реакцию останавливали равным объемом стоп-реагента (Life Technologies, № по каталогу SS04). Поглощение при 450 нм(ОП 450) измеряли на планшет-ридере SpectraMax.
[00298] Результаты представлены на Фиг. 19 и в Таблице 11. Эти результаты демонстрируют, что анти-CD112R антитела ингибируют связывание CD112R мыши с CD112 мыши в различной степени. Процент ингибирования рассчитывали как [100 - ((ОП тестового образца 450/Макс. ОП 450)*100%)] Макс. ОП 450 определяли как поглощение при 450 нм в отсутствие антитела.
[00299] Как показано в Таблице 11 и на Фиг.17-19, антитела 32, 33, 34, 35 и 36 способны блокировать взаимодействие CD112 человека с CD112R человека, но согласно определению блокирования, описанному в данном документе, не способны блокировать связывающее взаимодействие между CD112R мыши и CD112 мыши. См., например, последние два столбца Таблицы 11 для антител 32, 33, 34, 35 и 36, показывающие % ингибирования взаимодействия у мыши на уровне 0, 28,3, 20,3, 24,2 и 41,6, соответственно, по сравнению с % ингибирования взаимодействия у человека на уровне 75,1, 78,8, 80, 81,7 и 86,2, соответственно. Антитела, не принадлежащие к типичному классу антител, родственных антителу 32, не проявляют такого дифференциального блокирования.
[00300] Таблица 11: Сводная информация о связывании и блокировании антител (мыши)
[00301] Хотя вышеизложенное изобретению было описано довольно подробно посредством иллюстрации и примера в целях ясности понимания, описания и примеры не следует истолковывать как ограничивающие объем изобретению. Содержание всей патентной и научной литературы, цитируемой в данном документе, явным образом и в полном объеме включено посредством ссылки.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> SURFACE ONCOLOGY, INC.
ADIMAB LLC
<120> КОМПОЗИЦИИ АНТИ-CD112R АНТИТЕЛ И СОПУТСТВУЮЩИЕ СПОСОБЫ
<130> 01219-0001-00PCT
<150> US 62/701 065
<151> 20.07.2018
<150> US 62/844 958
<151> 08.05.2019
<160> 4018
<170> PatentIn версия 3.5
<210> 1
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH CDR1
<400> 1
Phe Thr Phe Ser Glu Tyr Thr Met Asn
1 5
<210> 2
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH CDR2
<400> 2
Ala Ile Val Gly Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 3
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH CDR3
<400> 3
Ala Lys Asp Tyr Ser Ser Gly Asp Trp Ile Asp Tyr Gly Met Asp Val
1 5 10 15
<210> 4
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL CDR1
<400> 4
Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 5
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL CDR2
<400> 5
Asp Ala Ser Asn Leu Ala Thr
1 5
<210> 6
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL CDR3
<400> 6
Gln Gln Phe Asp Leu Leu Pro Pro Thr
1 5
<210> 7
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH FR1
<400> 7
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
20 25
<210> 8
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH FR2
<400> 8
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
1 5 10
<210> 9
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH FR3
<400> 9
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 10
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH FR4
<400> 10
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 11
<211> 369
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH ДНК
<400> 11
Gly Ala Gly Gly Thr Gly Cys Ala Gly Cys Thr Gly Gly Thr Gly Gly
1 5 10 15
Ala Gly Thr Cys Thr Gly Gly Gly Gly Gly Ala Gly Gly Cys Cys Thr
20 25 30
Gly Gly Thr Cys Ala Ala Gly Cys Cys Thr Gly Gly Gly Gly Gly Gly
35 40 45
Thr Cys Cys Cys Thr Gly Ala Gly Ala Cys Thr Cys Thr Cys Cys Thr
50 55 60
Gly Thr Gly Cys Ala Gly Cys Cys Thr Cys Thr Gly Gly Ala Thr Thr
65 70 75 80
Cys Ala Cys Cys Thr Thr Cys Ala Gly Thr Gly Ala Ala Thr Ala Thr
85 90 95
Ala Cys Cys Ala Thr Gly Ala Ala Cys Thr Gly Gly Gly Thr Cys Cys
100 105 110
Gly Cys Cys Ala Gly Gly Cys Thr Cys Cys Ala Gly Gly Gly Ala Ala
115 120 125
Gly Gly Gly Gly Cys Thr Gly Gly Ala Gly Thr Gly Gly Gly Thr Cys
130 135 140
Thr Cys Ala Gly Cys Thr Ala Thr Thr Gly Thr Ala Gly Gly Thr Ala
145 150 155 160
Gly Thr Gly Gly Thr Gly Ala Cys Ala Gly Cys Ala Cys Ala Thr Ala
165 170 175
Cys Thr Ala Cys Gly Cys Ala Gly Ala Cys Thr Cys Cys Gly Thr Gly
180 185 190
Ala Ala Gly Gly Gly Cys Cys Gly Gly Thr Thr Cys Ala Cys Cys Ala
195 200 205
Thr Cys Thr Cys Cys Ala Gly Ala Gly Ala Cys Ala Ala Thr Thr Cys
210 215 220
Cys Ala Ala Gly Ala Ala Cys Ala Cys Gly Cys Thr Gly Thr Ala Thr
225 230 235 240
Cys Thr Gly Cys Ala Ala Ala Thr Gly Ala Ala Cys Ala Gly Cys Cys
245 250 255
Thr Gly Ala Gly Ala Gly Cys Cys Gly Ala Gly Gly Ala Cys Ala Cys
260 265 270
Gly Gly Cys Gly Gly Thr Gly Thr Ala Cys Thr Ala Cys Thr Gly Cys
275 280 285
Gly Cys Cys Ala Ala Gly Gly Ala Cys Thr Ala Cys Ala Gly Cys Thr
290 295 300
Cys Cys Gly Gly Ala Gly Ala Cys Thr Gly Gly Ala Thr Cys Gly Ala
305 310 315 320
Thr Thr Ala Thr Gly Gly Ala Ala Thr Gly Gly Ala Cys Gly Thr Ala
325 330 335
Thr Gly Gly Gly Gly Cys Cys Ala Gly Gly Gly Ala Ala Cys Ala Ala
340 345 350
Cys Thr Gly Thr Cys Ala Cys Cys Gly Thr Cys Thr Cys Cys Thr Cys
355 360 365
Ala
<210> 12
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VH Белка
<400> 12
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Glu Tyr
20 25 30
Thr Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Val Gly Ser Gly Asp Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Tyr Ser Ser Gly Asp Trp Ile Asp Tyr Gly Met Asp Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 13
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL FR1
<400> 13
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 14
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL FR2
<400> 14
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 15
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL FR3
<400> 15
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys
20 25 30
<210> 16
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL FR4
<400> 16
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 17
<211> 321
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL DNA
<400> 17
Gly Ala Cys Ala Thr Cys Cys Ala Gly Ala Thr Gly Ala Cys Cys Cys
1 5 10 15
Ala Gly Thr Cys Thr Cys Cys Ala Thr Cys Cys Thr Cys Cys Cys Thr
20 25 30
Gly Thr Cys Thr Gly Cys Ala Thr Cys Thr Gly Thr Ala Gly Gly Ala
35 40 45
Gly Ala Cys Ala Gly Ala Gly Thr Cys Ala Cys Cys Ala Thr Cys Ala
50 55 60
Cys Thr Thr Gly Cys Cys Ala Gly Gly Cys Gly Ala Gly Thr Cys Ala
65 70 75 80
Gly Gly Ala Cys Ala Thr Thr Ala Gly Cys Ala Ala Cys Thr Ala Thr
85 90 95
Thr Thr Ala Ala Ala Thr Thr Gly Gly Thr Ala Thr Cys Ala Gly Cys
100 105 110
Ala Gly Ala Ala Ala Cys Cys Ala Gly Gly Gly Ala Ala Ala Gly Cys
115 120 125
Cys Cys Cys Thr Ala Ala Gly Cys Thr Cys Cys Thr Gly Ala Thr Cys
130 135 140
Thr Ala Cys Gly Ala Thr Gly Cys Ala Thr Cys Cys Ala Ala Thr Thr
145 150 155 160
Thr Gly Gly Cys Ala Ala Cys Ala Gly Gly Gly Gly Thr Cys Cys Cys
165 170 175
Ala Thr Cys Ala Ala Gly Gly Thr Thr Cys Ala Gly Thr Gly Gly Ala
180 185 190
Ala Gly Thr Gly Gly Ala Thr Cys Thr Gly Gly Gly Ala Cys Ala Gly
195 200 205
Ala Thr Thr Thr Thr Ala Cys Thr Thr Thr Cys Ala Cys Cys Ala Thr
210 215 220
Cys Ala Gly Cys Ala Gly Cys Cys Thr Gly Cys Ala Gly Cys Cys Thr
225 230 235 240
Gly Ala Ala Gly Ala Thr Ala Thr Thr Gly Cys Ala Ala Cys Ala Thr
245 250 255
Ala Thr Thr Ala Cys Thr Gly Thr Cys Ala Gly Cys Ala Gly Thr Thr
260 265 270
Cys Gly Ala Thr Cys Thr Cys Cys Thr Cys Cys Cys Thr Cys Cys Thr
275 280 285
Ala Cys Thr Thr Thr Thr Gly Gly Cys Gly Gly Ala Gly Gly Gly Ala
290 295 300
Cys Cys Ala Ala Gly Gly Thr Thr Gly Ala Gly Ala Thr Cys Ala Ala
305 310 315 320
Ala
<210> 18
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 2 - VL Белка
<400> 18
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Ala Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Phe Asp Leu Leu Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 19
<400> 19
000
<210> 20
<400> 20
000
<210> 21
<400> 21
000
<210> 22
<400> 22
000
<210> 23
<400> 23
000
<210> 24
<400> 24
000
<210> 25
<400> 25
000
<210> 26
<400> 26
000
<210> 27
<400> 27
000
<210> 28
<400> 28
000
<210> 29
<400> 29
000
<210> 30
<400> 30
000
<210> 31
<400> 31
000
<210> 32
<400> 32
000
<210> 33
<400> 33
000
<210> 34
<400> 34
000
<210> 35
<400> 35
000
<210> 36
<400> 36
000
<210> 37
<400> 37
000
<210> 38
<400> 38
000
<210> 39
<400> 39
000
<210> 40
<400> 40
000
<210> 41
<400> 41
000
<210> 42
<400> 42
000
<210> 43
<400> 43
000
<210> 44
<400> 44
000
<210> 45
<400> 45
000
<210> 46
<400> 46
000
<210> 47
<400> 47
000
<210> 48
<400> 48
000
<210> 49
<400> 49
000
<210> 50
<400> 50
000
<210> 51
<400> 51
000
<210> 52
<400> 52
000
<210> 53
<400> 53
000
<210> 54
<400> 54
000
<210> 55
<400> 55
000
<210> 56
<400> 56
000
<210> 57
<400> 57
000
<210> 58
<400> 58
000
<210> 59
<400> 59
000
<210> 60
<400> 60
000
<210> 61
<400> 61
000
<210> 62
<400> 62
000
<210> 63
<400> 63
000
<210> 64
<400> 64
000
<210> 65
<400> 65
000
<210> 66
<400> 66
000
<210> 67
<400> 67
000
<210> 68
<400> 68
000
<210> 69
<400> 69
000
<210> 70
<400> 70
000
<210> 71
<400> 71
000
<210> 72
<400> 72
000
<210> 73
<400> 73
000
<210> 74
<400> 74
000
<210> 75
<400> 75
000
<210> 76
<400> 76
000
<210> 77
<400> 77
000
<210> 78
<400> 78
000
<210> 79
<400> 79
000
<210> 80
<400> 80
000
<210> 81
<400> 81
000
<210> 82
<400> 82
000
<210> 83
<400> 83
000
<210> 84
<400> 84
000
<210> 85
<400> 85
000
<210> 86
<400> 86
000
<210> 87
<400> 87
000
<210> 88
<400> 88
000
<210> 89
<400> 89
000
<210> 90
<400> 90
000
<210> 91
<400> 91
000
<210> 92
<400> 92
000
<210> 93
<400> 93
000
<210> 94
<400> 94
000
<210> 95
<400> 95
000
<210> 96
<400> 96
000
<210> 97
<400> 97
000
<210> 98
<400> 98
000
<210> 99
<400> 99
000
<210> 100
<400> 100
000
<210> 101
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH CDR1
<400> 101
Phe Thr Phe Ser Asp Tyr Ala Met Ile
1 5
<210> 102
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH CDR2
<400> 102
Ala Ile Ser Gly Gly Gly Glu Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 103
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH CDR3
<400> 103
Ala Lys Asp Tyr Ser Ser Gly Asp Trp Ile Asp Tyr Gly Met Asp Val
1 5 10 15
<210> 104
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL CDR1
<400> 104
Gln Ala Ser Gln Asp Ile Ser Asn Tyr Leu Asn
1 5 10
<210> 105
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL CDR2
<400> 105
Asp Ala Ser Asn Leu Ala Thr
1 5
<210> 106
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL CDR3
<400> 106
Gln Gln Phe Asp Leu Leu Pro Pro Thr
1 5
<210> 107
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH FR1
<400> 107
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
20 25
<210> 108
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH FR2
<400> 108
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
1 5 10
<210> 109
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH FR3
<400> 109
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 110
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH FR4
<400> 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 111
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH ДНК
<400> 111
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc gactatgcca tgatatgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggtg gaggtgaaag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggcggtgt actactgcgc caaggactac 300
agctccggag actggatcga ttatggaatg gacgtatggg gccagggaac aactgtcacc 360
gtctcctca 369
<210> 112
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VH Белка
<400> 112
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Ala Met Ile Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Gly Gly Glu Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Tyr Ser Ser Gly Asp Trp Ile Asp Tyr Gly Met Asp Val
100 105 110
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 113
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL FR1
<400> 113
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 114
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL FR2
<400> 114
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 115
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL FR3
<400> 115
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys
20 25 30
<210> 116
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL FR4
<400> 116
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 117
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL ДНК
<400> 117
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggcaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcagcag ttcgatctcc tccctcctac ttttggcgga 300
gggaccaagg ttgagatcaa a 321
<210> 118
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 5 - VL Белка
<400> 118
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Ala Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Phe Asp Leu Leu Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 119
<400> 119
000
<210> 120
<400> 120
000
<210> 121
<400> 121
000
<210> 122
<400> 122
000
<210> 123
<400> 123
000
<210> 124
<400> 124
000
<210> 125
<400> 125
000
<210> 126
<400> 126
000
<210> 127
<400> 127
000
<210> 128
<400> 128
000
<210> 129
<400> 129
000
<210> 130
<400> 130
000
<210> 131
<400> 131
000
<210> 132
<400> 132
000
<210> 133
<400> 133
000
<210> 134
<400> 134
000
<210> 135
<400> 135
000
<210> 136
<400> 136
000
<210> 137
<400> 137
000
<210> 138
<400> 138
000
<210> 139
<400> 139
000
<210> 140
<400> 140
000
<210> 141
<400> 141
000
<210> 142
<400> 142
000
<210> 143
<400> 143
000
<210> 144
<400> 144
000
<210> 145
<400> 145
000
<210> 146
<400> 146
000
<210> 147
<400> 147
000
<210> 148
<400> 148
000
<210> 149
<400> 149
000
<210> 150
<400> 150
000
<210> 151
<400> 151
000
<210> 152
<400> 152
000
<210> 153
<400> 153
000
<210> 154
<400> 154
000
<210> 155
<400> 155
000
<210> 156
<400> 156
000
<210> 157
<400> 157
000
<210> 158
<400> 158
000
<210> 159
<400> 159
000
<210> 160
<400> 160
000
<210> 161
<400> 161
000
<210> 162
<400> 162
000
<210> 163
<400> 163
000
<210> 164
<400> 164
000
<210> 165
<400> 165
000
<210> 166
<400> 166
000
<210> 167
<400> 167
000
<210> 168
<400> 168
000
<210> 169
<400> 169
000
<210> 170
<400> 170
000
<210> 171
<400> 171
000
<210> 172
<400> 172
000
<210> 173
<400> 173
000
<210> 174
<400> 174
000
<210> 175
<400> 175
000
<210> 176
<400> 176
000
<210> 177
<400> 177
000
<210> 178
<400> 178
000
<210> 179
<400> 179
000
<210> 180
<400> 180
000
<210> 181
<400> 181
000
<210> 182
<400> 182
000
<210> 183
<400> 183
000
<210> 184
<400> 184
000
<210> 185
<400> 185
000
<210> 186
<400> 186
000
<210> 187
<400> 187
000
<210> 188
<400> 188
000
<210> 189
<400> 189
000
<210> 190
<400> 190
000
<210> 191
<400> 191
000
<210> 192
<400> 192
000
<210> 193
<400> 193
000
<210> 194
<400> 194
000
<210> 195
<400> 195
000
<210> 196
<400> 196
000
<210> 197
<400> 197
000
<210> 198
<400> 198
000
<210> 199
<400> 199
000
<210> 200
<400> 200
000
<210> 201
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH CDR1
<400> 201
Gly Thr Phe Asp Asn Tyr Tyr Ile Ser
1 5
<210> 202
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH CDR2
<400> 202
Gly Ile Phe Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 203
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH CDR3
<400> 203
Ala Arg Glu Val Gly His Tyr Ser Gly Ser Pro Tyr Tyr Met Asp Val
1 5 10 15
<210> 204
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL CDR1
<400> 204
Arg Ala Ser Gln Ser Ile Asn Ser Trp Leu Ala
1 5 10
<210> 205
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL CDR2
<400> 205
Asp Ala Ser Ser Leu Glu Ser
1 5
<210> 206
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL CDR3
<400> 206
Gln Gln Val Gly Pro Tyr Leu Thr
1 5
<210> 207
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH FR1
<400> 207
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 208
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH FR2
<400> 208
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 209
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH FR3
<400> 209
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 210
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH FR4
<400> 210
Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 211
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH ДНК
<400> 211
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcgac aactattaca tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcttcccta tcttcggtac cgcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagagaagtc 300
ggacactact ccggcagccc atactacatg gacgtatggg gcaagggtac aactgtcacc 360
gtctcctca 369
<210> 212
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VH Белка
<400> 212
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Asp Asn Tyr
20 25 30
Tyr Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Val Gly His Tyr Ser Gly Ser Pro Tyr Tyr Met Asp Val
100 105 110
Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 213
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL FR1
<400> 213
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 214
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL FR2
<400> 214
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Ser
1 5 10 15
<210> 215
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL FR3
<400> 215
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 216
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL FR4
<400> 216
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 217
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL ДНК
<400> 217
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattaat agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctccgat gcctccagtt tggaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccagcag gtcggcccct acctcacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 218
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 44 - VL Белка
<400> 218
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Asn Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Gly Pro Tyr Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 219
<400> 219
000
<210> 220
<400> 220
000
<210> 221
<400> 221
000
<210> 222
<400> 222
000
<210> 223
<400> 223
000
<210> 224
<400> 224
000
<210> 225
<400> 225
000
<210> 226
<400> 226
000
<210> 227
<400> 227
000
<210> 228
<400> 228
000
<210> 229
<400> 229
000
<210> 230
<400> 230
000
<210> 231
<400> 231
000
<210> 232
<400> 232
000
<210> 233
<400> 233
000
<210> 234
<400> 234
000
<210> 235
<400> 235
000
<210> 236
<400> 236
000
<210> 237
<400> 237
000
<210> 238
<400> 238
000
<210> 239
<400> 239
000
<210> 240
<400> 240
000
<210> 241
<400> 241
000
<210> 242
<400> 242
000
<210> 243
<400> 243
000
<210> 244
<400> 244
000
<210> 245
<400> 245
000
<210> 246
<400> 246
000
<210> 247
<400> 247
000
<210> 248
<400> 248
000
<210> 249
<400> 249
000
<210> 250
<400> 250
000
<210> 251
<400> 251
000
<210> 252
<400> 252
000
<210> 253
<400> 253
000
<210> 254
<400> 254
000
<210> 255
<400> 255
000
<210> 256
<400> 256
000
<210> 257
<400> 257
000
<210> 258
<400> 258
000
<210> 259
<400> 259
000
<210> 260
<400> 260
000
<210> 261
<400> 261
000
<210> 262
<400> 262
000
<210> 263
<400> 263
000
<210> 264
<400> 264
000
<210> 265
<400> 265
000
<210> 266
<400> 266
000
<210> 267
<400> 267
000
<210> 268
<400> 268
000
<210> 269
<400> 269
000
<210> 270
<400> 270
000
<210> 271
<400> 271
000
<210> 272
<400> 272
000
<210> 273
<400> 273
000
<210> 274
<400> 274
000
<210> 275
<400> 275
000
<210> 276
<400> 276
000
<210> 277
<400> 277
000
<210> 278
<400> 278
000
<210> 279
<400> 279
000
<210> 280
<400> 280
000
<210> 281
<400> 281
000
<210> 282
<400> 282
000
<210> 283
<400> 283
000
<210> 284
<400> 284
000
<210> 285
<400> 285
000
<210> 286
<400> 286
000
<210> 287
<400> 287
000
<210> 288
<400> 288
000
<210> 289
<400> 289
000
<210> 290
<400> 290
000
<210> 291
<400> 291
000
<210> 292
<400> 292
000
<210> 293
<400> 293
000
<210> 294
<400> 294
000
<210> 295
<400> 295
000
<210> 296
<400> 296
000
<210> 297
<400> 297
000
<210> 298
<400> 298
000
<210> 299
<400> 299
000
<210> 300
<400> 300
000
<210> 301
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH CDR1
<400> 301
Phe Thr Phe Gly Asp Tyr Ala Met Ser
1 5
<210> 302
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH CDR2
<400> 302
Phe Ile Gly Ser Lys Phe Tyr Gly Gly Glu Thr Glu Tyr Thr Ala Ser
1 5 10 15
Val Lys Gly
<210> 303
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH CDR3
<400> 303
Ala Arg Gly Pro Arg Arg Tyr Thr Tyr Gly Met Asp Val
1 5 10
<210> 304
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL CDR1
<400> 304
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 305
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL CDR2
<400> 305
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 306
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL CDR3
<400> 306
Gln Gln Ser Ser Thr Pro Leu Thr
1 5
<210> 307
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH FR1
<400> 307
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly
20 25
<210> 308
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH FR2
<400> 308
Trp Phe Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
1 5 10
<210> 309
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH FR3
<400> 309
Arg Phe Thr Ile Ser Arg Asp Gly Ser Lys Ser Ile Ala Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 310
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH FR4
<400> 310
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 311
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH ДНК
<400> 311
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc cagggcggtc cctgagactc 60
tcctgtacag cttctggatt cacctttggt gattatgcta tgagctggtt ccgccaggct 120
ccagggaagg ggctggagtg ggtaggtttc attggaagca aattctatgg tggggaaaca 180
gaatacaccg cgtctgtgaa aggcagattc accatctcaa gagatggttc caaaagcatc 240
gcctatctgc aaatgaacag cctgaaaacc gaggacacgg cggtgtacta ctgcgccaga 300
ggaccaagac gctacacata cggaatggac gtatggggcc agggaacaac tgtcaccgtc 360
tcctca 366
<210> 312
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VH Белка
<400> 312
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Gly Asp Tyr
20 25 30
Ala Met Ser Trp Phe Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Ser Lys Phe Tyr Gly Gly Glu Thr Glu Tyr Thr Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Gly Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Gly Pro Arg Arg Tyr Thr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 313
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL FR1
<400> 313
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 314
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL FR2
<400> 314
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 315
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL FR3
<400> 315
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 316
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL FR4
<400> 316
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 317
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL ДНК
<400> 317
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agctccaccc ccctcacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 318
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 58 - VL Белка
<400> 318
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Ser Thr Pro Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 319
<400> 319
000
<210> 320
<400> 320
000
<210> 321
<400> 321
000
<210> 322
<400> 322
000
<210> 323
<400> 323
000
<210> 324
<400> 324
000
<210> 325
<400> 325
000
<210> 326
<400> 326
000
<210> 327
<400> 327
000
<210> 328
<400> 328
000
<210> 329
<400> 329
000
<210> 330
<400> 330
000
<210> 331
<400> 331
000
<210> 332
<400> 332
000
<210> 333
<400> 333
000
<210> 334
<400> 334
000
<210> 335
<400> 335
000
<210> 336
<400> 336
000
<210> 337
<400> 337
000
<210> 338
<400> 338
000
<210> 339
<400> 339
000
<210> 340
<400> 340
000
<210> 341
<400> 341
000
<210> 342
<400> 342
000
<210> 343
<400> 343
000
<210> 344
<400> 344
000
<210> 345
<400> 345
000
<210> 346
<400> 346
000
<210> 347
<400> 347
000
<210> 348
<400> 348
000
<210> 349
<400> 349
000
<210> 350
<400> 350
000
<210> 351
<400> 351
000
<210> 352
<400> 352
000
<210> 353
<400> 353
000
<210> 354
<400> 354
000
<210> 355
<400> 355
000
<210> 356
<400> 356
000
<210> 357
<400> 357
000
<210> 358
<400> 358
000
<210> 359
<400> 359
000
<210> 360
<400> 360
000
<210> 361
<400> 361
000
<210> 362
<400> 362
000
<210> 363
<400> 363
000
<210> 364
<400> 364
000
<210> 365
<400> 365
000
<210> 366
<400> 366
000
<210> 367
<400> 367
000
<210> 368
<400> 368
000
<210> 369
<400> 369
000
<210> 370
<400> 370
000
<210> 371
<400> 371
000
<210> 372
<400> 372
000
<210> 373
<400> 373
000
<210> 374
<400> 374
000
<210> 375
<400> 375
000
<210> 376
<400> 376
000
<210> 377
<400> 377
000
<210> 378
<400> 378
000
<210> 379
<400> 379
000
<210> 380
<400> 380
000
<210> 381
<400> 381
000
<210> 382
<400> 382
000
<210> 383
<400> 383
000
<210> 384
<400> 384
000
<210> 385
<400> 385
000
<210> 386
<400> 386
000
<210> 387
<400> 387
000
<210> 388
<400> 388
000
<210> 389
<400> 389
000
<210> 390
<400> 390
000
<210> 391
<400> 391
000
<210> 392
<400> 392
000
<210> 393
<400> 393
000
<210> 394
<400> 394
000
<210> 395
<400> 395
000
<210> 396
<400> 396
000
<210> 397
<400> 397
000
<210> 398
<400> 398
000
<210> 399
<400> 399
000
<210> 400
<400> 400
000
<210> 401
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH CDR1
<400> 401
Phe Thr Phe Asp Asp Tyr Ala Val His
1 5
<210> 402
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH CDR2
<400> 402
Gly Ile Ser Trp Ser Ser Gly Leu Ile Gly Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 403
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH CDR3
<400> 403
Ala Lys Gly Pro Pro Thr Tyr Gln Asp Tyr Phe Asp Leu
1 5 10
<210> 404
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL CDR1
<400> 404
Arg Ala Ser Gln Ser Val Ser Arg Tyr Leu Ala
1 5 10
<210> 405
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL CDR2
<400> 405
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 406
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL CDR3
<400> 406
Gln Gln Val Ser Phe Phe Pro Pro Ile Thr
1 5 10
<210> 407
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH FR1
<400> 407
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
20 25
<210> 408
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH FR2
<400> 408
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
1 5 10
<210> 409
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH FR3
<400> 409
Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 410
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH FR4
<400> 410
Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 411
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH ДНК
<400> 411
gaagtgcagc tggtggagtc tgggggaggc ttggtacagc ctggcaggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttgat gattatgccg tgcactgggt ccggcaagct 120
ccagggaagg gcctggagtg ggtctcaggt attagttgga gtagtggact aataggctat 180
gcggactctg tgaagggccg attcaccatc tccagagaca acgccaagaa ctccctgtat 240
ctgcaaatga acagtctgag agctgaggac acggcggtgt actactgcgc caagggccct 300
cctacctacc aagactactt cgacctatgg gggagaggta ccttggtcac cgtctcctca 360
<210> 412
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VH Белка
<400> 412
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Val His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Ser Ser Gly Leu Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Pro Pro Thr Tyr Gln Asp Tyr Phe Asp Leu Trp Gly Arg
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 413
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL FR1
<400> 413
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys
20
<210> 414
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL FR2
<400> 414
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
1 5 10 15
<210> 415
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL FR3
<400> 415
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys
20 25 30
<210> 416
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL FR4
<400> 416
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 417
<211> 324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL ДНК
<400> 417
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc aggtacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcag gtcagtttct tccctcctat cacttttggc 300
ggagggacca aggttgagat caaa 324
<210> 418
<211> 108
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 10 - VL Белка
<400> 418
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Val Ser Phe Phe Pro Pro
85 90 95
Ile Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 419
<400> 419
000
<210> 420
<400> 420
000
<210> 421
<400> 421
000
<210> 422
<400> 422
000
<210> 423
<400> 423
000
<210> 424
<400> 424
000
<210> 425
<400> 425
000
<210> 426
<400> 426
000
<210> 427
<400> 427
000
<210> 428
<400> 428
000
<210> 429
<400> 429
000
<210> 430
<400> 430
000
<210> 431
<400> 431
000
<210> 432
<400> 432
000
<210> 433
<400> 433
000
<210> 434
<400> 434
000
<210> 435
<400> 435
000
<210> 436
<400> 436
000
<210> 437
<400> 437
000
<210> 438
<400> 438
000
<210> 439
<400> 439
000
<210> 440
<400> 440
000
<210> 441
<400> 441
000
<210> 442
<400> 442
000
<210> 443
<400> 443
000
<210> 444
<400> 444
000
<210> 445
<400> 445
000
<210> 446
<400> 446
000
<210> 447
<400> 447
000
<210> 448
<400> 448
000
<210> 449
<400> 449
000
<210> 450
<400> 450
000
<210> 451
<400> 451
000
<210> 452
<400> 452
000
<210> 453
<400> 453
000
<210> 454
<400> 454
000
<210> 455
<400> 455
000
<210> 456
<400> 456
000
<210> 457
<400> 457
000
<210> 458
<400> 458
000
<210> 459
<400> 459
000
<210> 460
<400> 460
000
<210> 461
<400> 461
000
<210> 462
<400> 462
000
<210> 463
<400> 463
000
<210> 464
<400> 464
000
<210> 465
<400> 465
000
<210> 466
<400> 466
000
<210> 467
<400> 467
000
<210> 468
<400> 468
000
<210> 469
<400> 469
000
<210> 470
<400> 470
000
<210> 471
<400> 471
000
<210> 472
<400> 472
000
<210> 473
<400> 473
000
<210> 474
<400> 474
000
<210> 475
<400> 475
000
<210> 476
<400> 476
000
<210> 477
<400> 477
000
<210> 478
<400> 478
000
<210> 479
<400> 479
000
<210> 480
<400> 480
000
<210> 481
<400> 481
000
<210> 482
<400> 482
000
<210> 483
<400> 483
000
<210> 484
<400> 484
000
<210> 485
<400> 485
000
<210> 486
<400> 486
000
<210> 487
<400> 487
000
<210> 488
<400> 488
000
<210> 489
<400> 489
000
<210> 490
<400> 490
000
<210> 491
<400> 491
000
<210> 492
<400> 492
000
<210> 493
<400> 493
000
<210> 494
<400> 494
000
<210> 495
<400> 495
000
<210> 496
<400> 496
000
<210> 497
<400> 497
000
<210> 498
<400> 498
000
<210> 499
<400> 499
000
<210> 500
<400> 500
000
<210> 501
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH CDR1
<400> 501
Phe Thr Phe Ser Gly His Leu Met Ser
1 5
<210> 502
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH CDR2
<400> 502
Ala Ile Ser Gly Ser Ala Gly Glu Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 503
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH CDR3
<400> 503
Ala Arg Asp Ala Tyr Tyr Asp Asp Trp Ser Gly Trp Ala Asp Trp Tyr
1 5 10 15
Phe Asp Leu
<210> 504
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL CDR1
<400> 504
Arg Ala Ser Gln Ser Val Ser Arg Tyr Leu Ala
1 5 10
<210> 505
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL CDR2
<400> 505
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 506
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL CDR3
<400> 506
Gln Gln Val Ser Leu Leu Pro Pro Thr
1 5
<210> 507
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH FR1
<400> 507
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
20 25
<210> 508
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH FR2
<400> 508
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
1 5 10
<210> 509
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH FR3
<400> 509
Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 510
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH FR4
<400> 510
Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 511
<211> 378
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH ДНК
<400> 511
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc ggacacctaa tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggat ccgcaggtga aacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggcggtgt actactgcgc cagagatgcg 300
tactacgacg actggagcgg atgggccgat tggtacttcg atttatgggg gagaggtacc 360
ttggtcaccg tctcctca 378
<210> 512
<211> 126
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VH Белка
<400> 512
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Ala Gly Glu Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ala Tyr Tyr Asp Asp Trp Ser Gly Trp Ala Asp Trp Tyr
100 105 110
Phe Asp Leu Trp Gly Arg Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 513
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL FR1
<400> 513
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys
20
<210> 514
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL FR2
<400> 514
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
1 5 10 15
<210> 515
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL FR3
<400> 515
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys
20 25 30
<210> 516
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL FR4
<400> 516
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 517
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL ДНК
<400> 517
gaaattgtgt tgacacagtc tccagccacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc aggtacttag cctggtacca acagaaacct 120
ggccaggctc ccaggctcct catctatgat gcatccaaca gggccactgg catcccagcc 180
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag cctagagcct 240
gaagattttg cagtttatta ctgtcagcag gtcagtctcc tccctcctac ttttggcgga 300
gggaccaagg ttgagatcaa a 321
<210> 518
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 38 - VL Белка
<400> 518
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Val Ser Leu Leu Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 519
<400> 519
000
<210> 520
<400> 520
000
<210> 521
<400> 521
000
<210> 522
<400> 522
000
<210> 523
<400> 523
000
<210> 524
<400> 524
000
<210> 525
<400> 525
000
<210> 526
<400> 526
000
<210> 527
<400> 527
000
<210> 528
<400> 528
000
<210> 529
<400> 529
000
<210> 530
<400> 530
000
<210> 531
<400> 531
000
<210> 532
<400> 532
000
<210> 533
<400> 533
000
<210> 534
<400> 534
000
<210> 535
<400> 535
000
<210> 536
<400> 536
000
<210> 537
<400> 537
000
<210> 538
<400> 538
000
<210> 539
<400> 539
000
<210> 540
<400> 540
000
<210> 541
<400> 541
000
<210> 542
<400> 542
000
<210> 543
<400> 543
000
<210> 544
<400> 544
000
<210> 545
<400> 545
000
<210> 546
<400> 546
000
<210> 547
<400> 547
000
<210> 548
<400> 548
000
<210> 549
<400> 549
000
<210> 550
<400> 550
000
<210> 551
<400> 551
000
<210> 552
<400> 552
000
<210> 553
<400> 553
000
<210> 554
<400> 554
000
<210> 555
<400> 555
000
<210> 556
<400> 556
000
<210> 557
<400> 557
000
<210> 558
<400> 558
000
<210> 559
<400> 559
000
<210> 560
<400> 560
000
<210> 561
<400> 561
000
<210> 562
<400> 562
000
<210> 563
<400> 563
000
<210> 564
<400> 564
000
<210> 565
<400> 565
000
<210> 566
<400> 566
000
<210> 567
<400> 567
000
<210> 568
<400> 568
000
<210> 569
<400> 569
000
<210> 570
<400> 570
000
<210> 571
<400> 571
000
<210> 572
<400> 572
000
<210> 573
<400> 573
000
<210> 574
<400> 574
000
<210> 575
<400> 575
000
<210> 576
<400> 576
000
<210> 577
<400> 577
000
<210> 578
<400> 578
000
<210> 579
<400> 579
000
<210> 580
<400> 580
000
<210> 581
<400> 581
000
<210> 582
<400> 582
000
<210> 583
<400> 583
000
<210> 584
<400> 584
000
<210> 585
<400> 585
000
<210> 586
<400> 586
000
<210> 587
<400> 587
000
<210> 588
<400> 588
000
<210> 589
<400> 589
000
<210> 590
<400> 590
000
<210> 591
<400> 591
000
<210> 592
<400> 592
000
<210> 593
<400> 593
000
<210> 594
<400> 594
000
<210> 595
<400> 595
000
<210> 596
<400> 596
000
<210> 597
<400> 597
000
<210> 598
<400> 598
000
<210> 599
<400> 599
000
<210> 600
<400> 600
000
<210> 601
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH CDR1
<400> 601
Phe Thr Phe Gly Asp Val Ala Met Ser
1 5
<210> 602
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH CDR2
<400> 602
Tyr Ile Gly Ser Lys Ala Tyr Gly Gly Glu Thr Glu Tyr Thr Ala Ser
1 5 10 15
Val Lys Gly
<210> 603
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH CDR3
<400> 603
Ala Arg Ala Gly His Ser Tyr Gly Ser Ile Ala Ser Asn Trp Phe Asp
1 5 10 15
Pro
<210> 604
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL CDR1
<400> 604
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 605
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL CDR2
<400> 605
Gly Ala Ser Ser Leu Gln Ser
1 5
<210> 606
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL CDR3
<400> 606
Gln Gln Gly Phe Tyr Thr Pro Trp Thr
1 5
<210> 607
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH FR1
<400> 607
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly
20 25
<210> 608
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH FR2
<400> 608
Trp Phe Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
1 5 10
<210> 609
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH FR3
<400> 609
Arg Phe Thr Ile Ser Arg Asp Gly Ser Lys Ser Ile Ala Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 610
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH FR4
<400> 610
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 611
<211> 378
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH ДНК
<400> 611
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc cagggcggtc cctgagactc 60
tcctgtacag cttctggatt cacctttggt gatgtcgcta tgtcctggtt ccgccaggct 120
ccagggaagg ggctggagtg ggtaggttac attggaagca aagcttatgg tggggaaaca 180
gaatacaccg cgtctgtgaa aggcagattc accatctcaa gagatggttc caaaagcatc 240
gcctatctgc aaatgaacag cctgaaaacc gaggacacgg cggtgtacta ctgcgccaga 300
gctggacaca gctacggatc catcgccagc aactggttcg acccatgggg acagggtaca 360
ttggtcaccg tctcctca 378
<210> 612
<211> 126
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VH Белка
<400> 612
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Gly Asp Val
20 25 30
Ala Met Ser Trp Phe Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Tyr Ile Gly Ser Lys Ala Tyr Gly Gly Glu Thr Glu Tyr Thr Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Gly Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ala Gly His Ser Tyr Gly Ser Ile Ala Ser Asn Trp
100 105 110
Phe Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 613
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL FR1
<400> 613
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 614
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL FR2
<400> 614
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 615
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL FR3
<400> 615
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 616
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL FR4
<400> 616
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 617
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL ДНК
<400> 617
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatggt gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa ggattctaca ctccttggac ttttggcgga 300
gggaccaagg ttgagatcaa a 321
<210> 618
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 15 - VL Белка
<400> 618
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Phe Tyr Thr Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 619
<400> 619
000
<210> 620
<400> 620
000
<210> 621
<400> 621
000
<210> 622
<400> 622
000
<210> 623
<400> 623
000
<210> 624
<400> 624
000
<210> 625
<400> 625
000
<210> 626
<400> 626
000
<210> 627
<400> 627
000
<210> 628
<400> 628
000
<210> 629
<400> 629
000
<210> 630
<400> 630
000
<210> 631
<400> 631
000
<210> 632
<400> 632
000
<210> 633
<400> 633
000
<210> 634
<400> 634
000
<210> 635
<400> 635
000
<210> 636
<400> 636
000
<210> 637
<400> 637
000
<210> 638
<400> 638
000
<210> 639
<400> 639
000
<210> 640
<400> 640
000
<210> 641
<400> 641
000
<210> 642
<400> 642
000
<210> 643
<400> 643
000
<210> 644
<400> 644
000
<210> 645
<400> 645
000
<210> 646
<400> 646
000
<210> 647
<400> 647
000
<210> 648
<400> 648
000
<210> 649
<400> 649
000
<210> 650
<400> 650
000
<210> 651
<400> 651
000
<210> 652
<400> 652
000
<210> 653
<400> 653
000
<210> 654
<400> 654
000
<210> 655
<400> 655
000
<210> 656
<400> 656
000
<210> 657
<400> 657
000
<210> 658
<400> 658
000
<210> 659
<400> 659
000
<210> 660
<400> 660
000
<210> 661
<400> 661
000
<210> 662
<400> 662
000
<210> 663
<400> 663
000
<210> 664
<400> 664
000
<210> 665
<400> 665
000
<210> 666
<400> 666
000
<210> 667
<400> 667
000
<210> 668
<400> 668
000
<210> 669
<400> 669
000
<210> 670
<400> 670
000
<210> 671
<400> 671
000
<210> 672
<400> 672
000
<210> 673
<400> 673
000
<210> 674
<400> 674
000
<210> 675
<400> 675
000
<210> 676
<400> 676
000
<210> 677
<400> 677
000
<210> 678
<400> 678
000
<210> 679
<400> 679
000
<210> 680
<400> 680
000
<210> 681
<400> 681
000
<210> 682
<400> 682
000
<210> 683
<400> 683
000
<210> 684
<400> 684
000
<210> 685
<400> 685
000
<210> 686
<400> 686
000
<210> 687
<400> 687
000
<210> 688
<400> 688
000
<210> 689
<400> 689
000
<210> 690
<400> 690
000
<210> 691
<400> 691
000
<210> 692
<400> 692
000
<210> 693
<400> 693
000
<210> 694
<400> 694
000
<210> 695
<400> 695
000
<210> 696
<400> 696
000
<210> 697
<400> 697
000
<210> 698
<400> 698
000
<210> 699
<400> 699
000
<210> 700
<400> 700
000
<210> 701
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH CDR1
<400> 701
Gly Thr Phe Ser Ser Ala Ala Ile Ser
1 5
<210> 702
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH CDR2
<400> 702
Asn Ile Ile Pro Ile Val Gly Ile Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 703
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH CDR3
<400> 703
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
1 5 10 15
<210> 704
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL CDR1
<400> 704
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 705
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL CDR2
<400> 705
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 706
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL CDR3
<400> 706
Gln Gln Ser Asp Ile Leu Tyr Thr
1 5
<210> 707
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH FR1
<400> 707
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 708
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH FR2
<400> 708
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 709
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH FR3
<400> 709
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 710
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH FR4
<400> 710
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 711
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH ДНК
<400> 711
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc tccgccgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaaac atcatcccta tcgtaggtat agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagagacacg 300
ggacggggat acaccagaca cttctggttt gacccctggg gacagggtac attggtcacc 360
gtctcctca 369
<210> 712
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VH Белка
<400> 712
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ala
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Ile Pro Ile Val Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 713
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL FR1
<400> 713
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 714
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL FR2
<400> 714
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 715
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL FR3
<400> 715
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 716
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL FR4
<400> 716
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 717
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL ДНК
<400> 717
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agcgacatcc tctacacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 718
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 35 - VL Белка
<400> 718
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asp Ile Leu Tyr Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 719
<400> 719
000
<210> 720
<400> 720
000
<210> 721
<400> 721
000
<210> 722
<400> 722
000
<210> 723
<400> 723
000
<210> 724
<400> 724
000
<210> 725
<400> 725
000
<210> 726
<400> 726
000
<210> 727
<400> 727
000
<210> 728
<400> 728
000
<210> 729
<400> 729
000
<210> 730
<400> 730
000
<210> 731
<400> 731
000
<210> 732
<400> 732
000
<210> 733
<400> 733
000
<210> 734
<400> 734
000
<210> 735
<400> 735
000
<210> 736
<400> 736
000
<210> 737
<400> 737
000
<210> 738
<400> 738
000
<210> 739
<400> 739
000
<210> 740
<400> 740
000
<210> 741
<400> 741
000
<210> 742
<400> 742
000
<210> 743
<400> 743
000
<210> 744
<400> 744
000
<210> 745
<400> 745
000
<210> 746
<400> 746
000
<210> 747
<400> 747
000
<210> 748
<400> 748
000
<210> 749
<400> 749
000
<210> 750
<400> 750
000
<210> 751
<400> 751
000
<210> 752
<400> 752
000
<210> 753
<400> 753
000
<210> 754
<400> 754
000
<210> 755
<400> 755
000
<210> 756
<400> 756
000
<210> 757
<400> 757
000
<210> 758
<400> 758
000
<210> 759
<400> 759
000
<210> 760
<400> 760
000
<210> 761
<400> 761
000
<210> 762
<400> 762
000
<210> 763
<400> 763
000
<210> 764
<400> 764
000
<210> 765
<400> 765
000
<210> 766
<400> 766
000
<210> 767
<400> 767
000
<210> 768
<400> 768
000
<210> 769
<400> 769
000
<210> 770
<400> 770
000
<210> 771
<400> 771
000
<210> 772
<400> 772
000
<210> 773
<400> 773
000
<210> 774
<400> 774
000
<210> 775
<400> 775
000
<210> 776
<400> 776
000
<210> 777
<400> 777
000
<210> 778
<400> 778
000
<210> 779
<400> 779
000
<210> 780
<400> 780
000
<210> 781
<400> 781
000
<210> 782
<400> 782
000
<210> 783
<400> 783
000
<210> 784
<400> 784
000
<210> 785
<400> 785
000
<210> 786
<400> 786
000
<210> 787
<400> 787
000
<210> 788
<400> 788
000
<210> 789
<400> 789
000
<210> 790
<400> 790
000
<210> 791
<400> 791
000
<210> 792
<400> 792
000
<210> 793
<400> 793
000
<210> 794
<400> 794
000
<210> 795
<400> 795
000
<210> 796
<400> 796
000
<210> 797
<400> 797
000
<210> 798
<400> 798
000
<210> 799
<400> 799
000
<210> 800
<400> 800
000
<210> 801
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH CDR1
<400> 801
Gly Thr Phe Ser Asn Tyr Ala Ile Ser
1 5
<210> 802
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH CDR2
<400> 802
Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 803
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH CDR3
<400> 803
Ala Arg Gly Arg Gly Ala Leu Ala Leu Val Gly Pro Tyr Tyr Gly Met
1 5 10 15
Asp Val
<210> 804
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL CDR1
<400> 804
Arg Ser Ser Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr Leu Asp
1 5 10 15
<210> 805
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL CDR2
<400> 805
Leu Gly Ser His Arg Ala Ser
1 5
<210> 806
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL CDR3
<400> 806
Met Gln Ala Leu Arg Ala Pro Thr
1 5
<210> 807
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH FR1
<400> 807
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 808
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH FR2
<400> 808
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 809
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH FR3
<400> 809
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 810
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH FR4
<400> 810
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 811
<211> 375
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH ДНК
<400> 811
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc aactatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagaggcaga 300
ggcgctctgg cactcgtcgg accatactac ggaatggacg tatggggcca gggaacaact 360
gtcaccgtct cctca 375
<210> 812
<211> 125
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VH Белка
<400> 812
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Asn Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Arg Gly Ala Leu Ala Leu Val Gly Pro Tyr Tyr Gly Met
100 105 110
Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 813
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL FR1
<400> 813
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys
20
<210> 814
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL FR2
<400> 814
Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile Tyr
1 5 10 15
<210> 815
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL FR3
<400> 815
Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys
20 25 30
<210> 816
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL FR4
<400> 816
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 817
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL ДНК
<400> 817
gatattgtga tgactcagtc tccactctcc ctgcccgtca cccctggaga gccggcctcc 60
atctcctgca ggtctagtca gagcctcctg catagtaatg gatacaacta tttggattgg 120
tacctgcaga agccagggca gtctccacag ctcctgatct atttgggttc tcatcgggcc 180
tccggggtcc ctgacaggtt cagtggcagt ggatcaggca cagattttac actgaaaatc 240
agcagagtgg aggctgagga tgttggggtt tattactgca tgcaggcact ccgagcccct 300
acttttggcg gagggaccaa ggttgagatc aaa 333
<210> 818
<211> 111
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 47 - VL Белка
<400> 818
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser His Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Arg Ala Pro Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 819
<400> 819
000
<210> 820
<400> 820
000
<210> 821
<400> 821
000
<210> 822
<400> 822
000
<210> 823
<400> 823
000
<210> 824
<400> 824
000
<210> 825
<400> 825
000
<210> 826
<400> 826
000
<210> 827
<400> 827
000
<210> 828
<400> 828
000
<210> 829
<400> 829
000
<210> 830
<400> 830
000
<210> 831
<400> 831
000
<210> 832
<400> 832
000
<210> 833
<400> 833
000
<210> 834
<400> 834
000
<210> 835
<400> 835
000
<210> 836
<400> 836
000
<210> 837
<400> 837
000
<210> 838
<400> 838
000
<210> 839
<400> 839
000
<210> 840
<400> 840
000
<210> 841
<400> 841
000
<210> 842
<400> 842
000
<210> 843
<400> 843
000
<210> 844
<400> 844
000
<210> 845
<400> 845
000
<210> 846
<400> 846
000
<210> 847
<400> 847
000
<210> 848
<400> 848
000
<210> 849
<400> 849
000
<210> 850
<400> 850
000
<210> 851
<400> 851
000
<210> 852
<400> 852
000
<210> 853
<400> 853
000
<210> 854
<400> 854
000
<210> 855
<400> 855
000
<210> 856
<400> 856
000
<210> 857
<400> 857
000
<210> 858
<400> 858
000
<210> 859
<400> 859
000
<210> 860
<400> 860
000
<210> 861
<400> 861
000
<210> 862
<400> 862
000
<210> 863
<400> 863
000
<210> 864
<400> 864
000
<210> 865
<400> 865
000
<210> 866
<400> 866
000
<210> 867
<400> 867
000
<210> 868
<400> 868
000
<210> 869
<400> 869
000
<210> 870
<400> 870
000
<210> 871
<400> 871
000
<210> 872
<400> 872
000
<210> 873
<400> 873
000
<210> 874
<400> 874
000
<210> 875
<400> 875
000
<210> 876
<400> 876
000
<210> 877
<400> 877
000
<210> 878
<400> 878
000
<210> 879
<400> 879
000
<210> 880
<400> 880
000
<210> 881
<400> 881
000
<210> 882
<400> 882
000
<210> 883
<400> 883
000
<210> 884
<400> 884
000
<210> 885
<400> 885
000
<210> 886
<400> 886
000
<210> 887
<400> 887
000
<210> 888
<400> 888
000
<210> 889
<400> 889
000
<210> 890
<400> 890
000
<210> 891
<400> 891
000
<210> 892
<400> 892
000
<210> 893
<400> 893
000
<210> 894
<400> 894
000
<210> 895
<400> 895
000
<210> 896
<400> 896
000
<210> 897
<400> 897
000
<210> 898
<400> 898
000
<210> 899
<400> 899
000
<210> 900
<400> 900
000
<210> 901
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH CDR1
<400> 901
Phe Thr Phe Gly Asp Tyr Ala Met Ser
1 5
<210> 902
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH CDR2
<400> 902
Phe Ile Gly Ser Lys Ala Tyr Gly Gly Thr Thr Glu Tyr Thr Ala Ser
1 5 10 15
Val Lys Gly
<210> 903
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH CDR3
<400> 903
Ala Arg Gly Pro Arg Arg Tyr Thr Tyr Gly Met Asp Val
1 5 10
<210> 904
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL CDR1
<400> 904
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 905
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL CDR2
<400> 905
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 906
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL CDR3
<400> 906
Gln Gln Ser Ser Thr Pro Leu Thr
1 5
<210> 907
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH FR1
<400> 907
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly
20 25
<210> 908
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH FR2
<400> 908
Trp Phe Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
1 5 10
<210> 909
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH FR3
<400> 909
Arg Phe Thr Ile Ser Arg Asp Gly Ser Lys Ser Ile Ala Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 910
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH FR4
<400> 910
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 911
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH ДНК
<400> 911
gaggtgcagc tggtggagtc tgggggaggc ttggtacagc cagggcggtc cctgagactc 60
tcctgtacag cttctggatt cacctttggt gattatgcta tgagctggtt ccgccaggct 120
ccagggaagg ggctggagtg ggtaggtttc attggaagca aagcttatgg tgggacaaca 180
gaatacaccg cgtctgtgaa aggcagattc accatctcaa gagatggttc caaaagcatc 240
gcctatctgc aaatgaacag cctgaaaacc gaggacacgg cggtgtacta ctgcgccaga 300
ggaccaagac gctacacata cggaatggac gtatggggcc agggaacaac tgtcaccgtc 360
tcctca 366
<210> 912
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VH Белка
<400> 912
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Gly Asp Tyr
20 25 30
Ala Met Ser Trp Phe Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Phe Ile Gly Ser Lys Ala Tyr Gly Gly Thr Thr Glu Tyr Thr Ala
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Gly Ser Lys Ser Ile
65 70 75 80
Ala Tyr Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Gly Pro Arg Arg Tyr Thr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 913
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL FR1
<400> 913
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 914
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL FR2
<400> 914
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 915
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL FR3
<400> 915
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 916
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL FR4
<400> 916
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 917
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL ДНК
<400> 917
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agctccaccc ccctcacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 918
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 46 - VL Белка
<400> 918
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Ser Thr Pro Leu Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 919
<400> 919
000
<210> 920
<400> 920
000
<210> 921
<400> 921
000
<210> 922
<400> 922
000
<210> 923
<400> 923
000
<210> 924
<400> 924
000
<210> 925
<400> 925
000
<210> 926
<400> 926
000
<210> 927
<400> 927
000
<210> 928
<400> 928
000
<210> 929
<400> 929
000
<210> 930
<400> 930
000
<210> 931
<400> 931
000
<210> 932
<400> 932
000
<210> 933
<400> 933
000
<210> 934
<400> 934
000
<210> 935
<400> 935
000
<210> 936
<400> 936
000
<210> 937
<400> 937
000
<210> 938
<400> 938
000
<210> 939
<400> 939
000
<210> 940
<400> 940
000
<210> 941
<400> 941
000
<210> 942
<400> 942
000
<210> 943
<400> 943
000
<210> 944
<400> 944
000
<210> 945
<400> 945
000
<210> 946
<400> 946
000
<210> 947
<400> 947
000
<210> 948
<400> 948
000
<210> 949
<400> 949
000
<210> 950
<400> 950
000
<210> 951
<400> 951
000
<210> 952
<400> 952
000
<210> 953
<400> 953
000
<210> 954
<400> 954
000
<210> 955
<400> 955
000
<210> 956
<400> 956
000
<210> 957
<400> 957
000
<210> 958
<400> 958
000
<210> 959
<400> 959
000
<210> 960
<400> 960
000
<210> 961
<400> 961
000
<210> 962
<400> 962
000
<210> 963
<400> 963
000
<210> 964
<400> 964
000
<210> 965
<400> 965
000
<210> 966
<400> 966
000
<210> 967
<400> 967
000
<210> 968
<400> 968
000
<210> 969
<400> 969
000
<210> 970
<400> 970
000
<210> 971
<400> 971
000
<210> 972
<400> 972
000
<210> 973
<400> 973
000
<210> 974
<400> 974
000
<210> 975
<400> 975
000
<210> 976
<400> 976
000
<210> 977
<400> 977
000
<210> 978
<400> 978
000
<210> 979
<400> 979
000
<210> 980
<400> 980
000
<210> 981
<400> 981
000
<210> 982
<400> 982
000
<210> 983
<400> 983
000
<210> 984
<400> 984
000
<210> 985
<400> 985
000
<210> 986
<400> 986
000
<210> 987
<400> 987
000
<210> 988
<400> 988
000
<210> 989
<400> 989
000
<210> 990
<400> 990
000
<210> 991
<400> 991
000
<210> 992
<400> 992
000
<210> 993
<400> 993
000
<210> 994
<400> 994
000
<210> 995
<400> 995
000
<210> 996
<400> 996
000
<210> 997
<400> 997
000
<210> 998
<400> 998
000
<210> 999
<400> 999
000
<210> 1000
<400> 1000
000
<210> 1001
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH CDR1
<400> 1001
Gly Thr Phe Ser Ser Tyr Ala Ile Ser
1 5
<210> 1002
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH CDR2
<400> 1002
Gly Ile Ile Pro Ile Ser Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 1003
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH CDR3
<400> 1003
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
1 5 10 15
<210> 1004
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL CDR1
<400> 1004
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 1005
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL CDR2
<400> 1005
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 1006
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL CDR3
<400> 1006
Gln Gln Ser Asp Ile Leu Tyr Thr
1 5
<210> 1007
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH FR1
<400> 1007
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 1008
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH FR2
<400> 1008
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 1009
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH FR3
<400> 1009
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 1010
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH FR4
<400> 1010
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 1011
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH ДНК
<400> 1011
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctctggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagagacacg 300
ggacggggat acaccagaca cttctggttt gacccctggg gacagggtac attggtcacc 360
gtctcctca 369
<210> 1012
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VH Белка
<400> 1012
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Ser Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1013
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL FR1
<400> 1013
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 1014
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL FR2
<400> 1014
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 1015
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL FR3
<400> 1015
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 1016
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL FR4
<400> 1016
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 1017
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL ДНК
<400> 1017
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agcgacatcc tctacacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 1018
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 32 - VL Белка
<400> 1018
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asp Ile Leu Tyr Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 1019
<400> 1019
000
<210> 1020
<400> 1020
000
<210> 1021
<400> 1021
000
<210> 1022
<400> 1022
000
<210> 1023
<400> 1023
000
<210> 1024
<400> 1024
000
<210> 1025
<400> 1025
000
<210> 1026
<400> 1026
000
<210> 1027
<400> 1027
000
<210> 1028
<400> 1028
000
<210> 1029
<400> 1029
000
<210> 1030
<400> 1030
000
<210> 1031
<400> 1031
000
<210> 1032
<400> 1032
000
<210> 1033
<400> 1033
000
<210> 1034
<400> 1034
000
<210> 1035
<400> 1035
000
<210> 1036
<400> 1036
000
<210> 1037
<400> 1037
000
<210> 1038
<400> 1038
000
<210> 1039
<400> 1039
000
<210> 1040
<400> 1040
000
<210> 1041
<400> 1041
000
<210> 1042
<400> 1042
000
<210> 1043
<400> 1043
000
<210> 1044
<400> 1044
000
<210> 1045
<400> 1045
000
<210> 1046
<400> 1046
000
<210> 1047
<400> 1047
000
<210> 1048
<400> 1048
000
<210> 1049
<400> 1049
000
<210> 1050
<400> 1050
000
<210> 1051
<400> 1051
000
<210> 1052
<400> 1052
000
<210> 1053
<400> 1053
000
<210> 1054
<400> 1054
000
<210> 1055
<400> 1055
000
<210> 1056
<400> 1056
000
<210> 1057
<400> 1057
000
<210> 1058
<400> 1058
000
<210> 1059
<400> 1059
000
<210> 1060
<400> 1060
000
<210> 1061
<400> 1061
000
<210> 1062
<400> 1062
000
<210> 1063
<400> 1063
000
<210> 1064
<400> 1064
000
<210> 1065
<400> 1065
000
<210> 1066
<400> 1066
000
<210> 1067
<400> 1067
000
<210> 1068
<400> 1068
000
<210> 1069
<400> 1069
000
<210> 1070
<400> 1070
000
<210> 1071
<400> 1071
000
<210> 1072
<400> 1072
000
<210> 1073
<400> 1073
000
<210> 1074
<400> 1074
000
<210> 1075
<400> 1075
000
<210> 1076
<400> 1076
000
<210> 1077
<400> 1077
000
<210> 1078
<400> 1078
000
<210> 1079
<400> 1079
000
<210> 1080
<400> 1080
000
<210> 1081
<400> 1081
000
<210> 1082
<400> 1082
000
<210> 1083
<400> 1083
000
<210> 1084
<400> 1084
000
<210> 1085
<400> 1085
000
<210> 1086
<400> 1086
000
<210> 1087
<400> 1087
000
<210> 1088
<400> 1088
000
<210> 1089
<400> 1089
000
<210> 1090
<400> 1090
000
<210> 1091
<400> 1091
000
<210> 1092
<400> 1092
000
<210> 1093
<400> 1093
000
<210> 1094
<400> 1094
000
<210> 1095
<400> 1095
000
<210> 1096
<400> 1096
000
<210> 1097
<400> 1097
000
<210> 1098
<400> 1098
000
<210> 1099
<400> 1099
000
<210> 1100
<400> 1100
000
<210> 1101
<400> 1101
000
<210> 1102
<400> 1102
000
<210> 1103
<400> 1103
000
<210> 1104
<400> 1104
000
<210> 1105
<400> 1105
000
<210> 1106
<400> 1106
000
<210> 1107
<400> 1107
000
<210> 1108
<400> 1108
000
<210> 1109
<400> 1109
000
<210> 1110
<400> 1110
000
<210> 1111
<400> 1111
000
<210> 1112
<400> 1112
000
<210> 1113
<400> 1113
000
<210> 1114
<400> 1114
000
<210> 1115
<400> 1115
000
<210> 1116
<400> 1116
000
<210> 1117
<400> 1117
000
<210> 1118
<400> 1118
000
<210> 1119
<400> 1119
000
<210> 1120
<400> 1120
000
<210> 1121
<400> 1121
000
<210> 1122
<400> 1122
000
<210> 1123
<400> 1123
000
<210> 1124
<400> 1124
000
<210> 1125
<400> 1125
000
<210> 1126
<400> 1126
000
<210> 1127
<400> 1127
000
<210> 1128
<400> 1128
000
<210> 1129
<400> 1129
000
<210> 1130
<400> 1130
000
<210> 1131
<400> 1131
000
<210> 1132
<400> 1132
000
<210> 1133
<400> 1133
000
<210> 1134
<400> 1134
000
<210> 1135
<400> 1135
000
<210> 1136
<400> 1136
000
<210> 1137
<400> 1137
000
<210> 1138
<400> 1138
000
<210> 1139
<400> 1139
000
<210> 1140
<400> 1140
000
<210> 1141
<400> 1141
000
<210> 1142
<400> 1142
000
<210> 1143
<400> 1143
000
<210> 1144
<400> 1144
000
<210> 1145
<400> 1145
000
<210> 1146
<400> 1146
000
<210> 1147
<400> 1147
000
<210> 1148
<400> 1148
000
<210> 1149
<400> 1149
000
<210> 1150
<400> 1150
000
<210> 1151
<400> 1151
000
<210> 1152
<400> 1152
000
<210> 1153
<400> 1153
000
<210> 1154
<400> 1154
000
<210> 1155
<400> 1155
000
<210> 1156
<400> 1156
000
<210> 1157
<400> 1157
000
<210> 1158
<400> 1158
000
<210> 1159
<400> 1159
000
<210> 1160
<400> 1160
000
<210> 1161
<400> 1161
000
<210> 1162
<400> 1162
000
<210> 1163
<400> 1163
000
<210> 1164
<400> 1164
000
<210> 1165
<400> 1165
000
<210> 1166
<400> 1166
000
<210> 1167
<400> 1167
000
<210> 1168
<400> 1168
000
<210> 1169
<400> 1169
000
<210> 1170
<400> 1170
000
<210> 1171
<400> 1171
000
<210> 1172
<400> 1172
000
<210> 1173
<400> 1173
000
<210> 1174
<400> 1174
000
<210> 1175
<400> 1175
000
<210> 1176
<400> 1176
000
<210> 1177
<400> 1177
000
<210> 1178
<400> 1178
000
<210> 1179
<400> 1179
000
<210> 1180
<400> 1180
000
<210> 1181
<400> 1181
000
<210> 1182
<400> 1182
000
<210> 1183
<400> 1183
000
<210> 1184
<400> 1184
000
<210> 1185
<400> 1185
000
<210> 1186
<400> 1186
000
<210> 1187
<400> 1187
000
<210> 1188
<400> 1188
000
<210> 1189
<400> 1189
000
<210> 1190
<400> 1190
000
<210> 1191
<400> 1191
000
<210> 1192
<400> 1192
000
<210> 1193
<400> 1193
000
<210> 1194
<400> 1194
000
<210> 1195
<400> 1195
000
<210> 1196
<400> 1196
000
<210> 1197
<400> 1197
000
<210> 1198
<400> 1198
000
<210> 1199
<400> 1199
000
<210> 1200
<400> 1200
000
<210> 1201
<400> 1201
000
<210> 1202
<400> 1202
000
<210> 1203
<400> 1203
000
<210> 1204
<400> 1204
000
<210> 1205
<400> 1205
000
<210> 1206
<400> 1206
000
<210> 1207
<400> 1207
000
<210> 1208
<400> 1208
000
<210> 1209
<400> 1209
000
<210> 1210
<400> 1210
000
<210> 1211
<400> 1211
000
<210> 1212
<400> 1212
000
<210> 1213
<400> 1213
000
<210> 1214
<400> 1214
000
<210> 1215
<400> 1215
000
<210> 1216
<400> 1216
000
<210> 1217
<400> 1217
000
<210> 1218
<400> 1218
000
<210> 1219
<400> 1219
000
<210> 1220
<400> 1220
000
<210> 1221
<400> 1221
000
<210> 1222
<400> 1222
000
<210> 1223
<400> 1223
000
<210> 1224
<400> 1224
000
<210> 1225
<400> 1225
000
<210> 1226
<400> 1226
000
<210> 1227
<400> 1227
000
<210> 1228
<400> 1228
000
<210> 1229
<400> 1229
000
<210> 1230
<400> 1230
000
<210> 1231
<400> 1231
000
<210> 1232
<400> 1232
000
<210> 1233
<400> 1233
000
<210> 1234
<400> 1234
000
<210> 1235
<400> 1235
000
<210> 1236
<400> 1236
000
<210> 1237
<400> 1237
000
<210> 1238
<400> 1238
000
<210> 1239
<400> 1239
000
<210> 1240
<400> 1240
000
<210> 1241
<400> 1241
000
<210> 1242
<400> 1242
000
<210> 1243
<400> 1243
000
<210> 1244
<400> 1244
000
<210> 1245
<400> 1245
000
<210> 1246
<400> 1246
000
<210> 1247
<400> 1247
000
<210> 1248
<400> 1248
000
<210> 1249
<400> 1249
000
<210> 1250
<400> 1250
000
<210> 1251
<400> 1251
000
<210> 1252
<400> 1252
000
<210> 1253
<400> 1253
000
<210> 1254
<400> 1254
000
<210> 1255
<400> 1255
000
<210> 1256
<400> 1256
000
<210> 1257
<400> 1257
000
<210> 1258
<400> 1258
000
<210> 1259
<400> 1259
000
<210> 1260
<400> 1260
000
<210> 1261
<400> 1261
000
<210> 1262
<400> 1262
000
<210> 1263
<400> 1263
000
<210> 1264
<400> 1264
000
<210> 1265
<400> 1265
000
<210> 1266
<400> 1266
000
<210> 1267
<400> 1267
000
<210> 1268
<400> 1268
000
<210> 1269
<400> 1269
000
<210> 1270
<400> 1270
000
<210> 1271
<400> 1271
000
<210> 1272
<400> 1272
000
<210> 1273
<400> 1273
000
<210> 1274
<400> 1274
000
<210> 1275
<400> 1275
000
<210> 1276
<400> 1276
000
<210> 1277
<400> 1277
000
<210> 1278
<400> 1278
000
<210> 1279
<400> 1279
000
<210> 1280
<400> 1280
000
<210> 1281
<400> 1281
000
<210> 1282
<400> 1282
000
<210> 1283
<400> 1283
000
<210> 1284
<400> 1284
000
<210> 1285
<400> 1285
000
<210> 1286
<400> 1286
000
<210> 1287
<400> 1287
000
<210> 1288
<400> 1288
000
<210> 1289
<400> 1289
000
<210> 1290
<400> 1290
000
<210> 1291
<400> 1291
000
<210> 1292
<400> 1292
000
<210> 1293
<400> 1293
000
<210> 1294
<400> 1294
000
<210> 1295
<400> 1295
000
<210> 1296
<400> 1296
000
<210> 1297
<400> 1297
000
<210> 1298
<400> 1298
000
<210> 1299
<400> 1299
000
<210> 1300
<400> 1300
000
<210> 1301
<400> 1301
000
<210> 1302
<400> 1302
000
<210> 1303
<400> 1303
000
<210> 1304
<400> 1304
000
<210> 1305
<400> 1305
000
<210> 1306
<400> 1306
000
<210> 1307
<400> 1307
000
<210> 1308
<400> 1308
000
<210> 1309
<400> 1309
000
<210> 1310
<400> 1310
000
<210> 1311
<400> 1311
000
<210> 1312
<400> 1312
000
<210> 1313
<400> 1313
000
<210> 1314
<400> 1314
000
<210> 1315
<400> 1315
000
<210> 1316
<400> 1316
000
<210> 1317
<400> 1317
000
<210> 1318
<400> 1318
000
<210> 1319
<400> 1319
000
<210> 1320
<400> 1320
000
<210> 1321
<400> 1321
000
<210> 1322
<400> 1322
000
<210> 1323
<400> 1323
000
<210> 1324
<400> 1324
000
<210> 1325
<400> 1325
000
<210> 1326
<400> 1326
000
<210> 1327
<400> 1327
000
<210> 1328
<400> 1328
000
<210> 1329
<400> 1329
000
<210> 1330
<400> 1330
000
<210> 1331
<400> 1331
000
<210> 1332
<400> 1332
000
<210> 1333
<400> 1333
000
<210> 1334
<400> 1334
000
<210> 1335
<400> 1335
000
<210> 1336
<400> 1336
000
<210> 1337
<400> 1337
000
<210> 1338
<400> 1338
000
<210> 1339
<400> 1339
000
<210> 1340
<400> 1340
000
<210> 1341
<400> 1341
000
<210> 1342
<400> 1342
000
<210> 1343
<400> 1343
000
<210> 1344
<400> 1344
000
<210> 1345
<400> 1345
000
<210> 1346
<400> 1346
000
<210> 1347
<400> 1347
000
<210> 1348
<400> 1348
000
<210> 1349
<400> 1349
000
<210> 1350
<400> 1350
000
<210> 1351
<400> 1351
000
<210> 1352
<400> 1352
000
<210> 1353
<400> 1353
000
<210> 1354
<400> 1354
000
<210> 1355
<400> 1355
000
<210> 1356
<400> 1356
000
<210> 1357
<400> 1357
000
<210> 1358
<400> 1358
000
<210> 1359
<400> 1359
000
<210> 1360
<400> 1360
000
<210> 1361
<400> 1361
000
<210> 1362
<400> 1362
000
<210> 1363
<400> 1363
000
<210> 1364
<400> 1364
000
<210> 1365
<400> 1365
000
<210> 1366
<400> 1366
000
<210> 1367
<400> 1367
000
<210> 1368
<400> 1368
000
<210> 1369
<400> 1369
000
<210> 1370
<400> 1370
000
<210> 1371
<400> 1371
000
<210> 1372
<400> 1372
000
<210> 1373
<400> 1373
000
<210> 1374
<400> 1374
000
<210> 1375
<400> 1375
000
<210> 1376
<400> 1376
000
<210> 1377
<400> 1377
000
<210> 1378
<400> 1378
000
<210> 1379
<400> 1379
000
<210> 1380
<400> 1380
000
<210> 1381
<400> 1381
000
<210> 1382
<400> 1382
000
<210> 1383
<400> 1383
000
<210> 1384
<400> 1384
000
<210> 1385
<400> 1385
000
<210> 1386
<400> 1386
000
<210> 1387
<400> 1387
000
<210> 1388
<400> 1388
000
<210> 1389
<400> 1389
000
<210> 1390
<400> 1390
000
<210> 1391
<400> 1391
000
<210> 1392
<400> 1392
000
<210> 1393
<400> 1393
000
<210> 1394
<400> 1394
000
<210> 1395
<400> 1395
000
<210> 1396
<400> 1396
000
<210> 1397
<400> 1397
000
<210> 1398
<400> 1398
000
<210> 1399
<400> 1399
000
<210> 1400
<400> 1400
000
<210> 1401
<400> 1401
000
<210> 1402
<400> 1402
000
<210> 1403
<400> 1403
000
<210> 1404
<400> 1404
000
<210> 1405
<400> 1405
000
<210> 1406
<400> 1406
000
<210> 1407
<400> 1407
000
<210> 1408
<400> 1408
000
<210> 1409
<400> 1409
000
<210> 1410
<400> 1410
000
<210> 1411
<400> 1411
000
<210> 1412
<400> 1412
000
<210> 1413
<400> 1413
000
<210> 1414
<400> 1414
000
<210> 1415
<400> 1415
000
<210> 1416
<400> 1416
000
<210> 1417
<400> 1417
000
<210> 1418
<400> 1418
000
<210> 1419
<400> 1419
000
<210> 1420
<400> 1420
000
<210> 1421
<400> 1421
000
<210> 1422
<400> 1422
000
<210> 1423
<400> 1423
000
<210> 1424
<400> 1424
000
<210> 1425
<400> 1425
000
<210> 1426
<400> 1426
000
<210> 1427
<400> 1427
000
<210> 1428
<400> 1428
000
<210> 1429
<400> 1429
000
<210> 1430
<400> 1430
000
<210> 1431
<400> 1431
000
<210> 1432
<400> 1432
000
<210> 1433
<400> 1433
000
<210> 1434
<400> 1434
000
<210> 1435
<400> 1435
000
<210> 1436
<400> 1436
000
<210> 1437
<400> 1437
000
<210> 1438
<400> 1438
000
<210> 1439
<400> 1439
000
<210> 1440
<400> 1440
000
<210> 1441
<400> 1441
000
<210> 1442
<400> 1442
000
<210> 1443
<400> 1443
000
<210> 1444
<400> 1444
000
<210> 1445
<400> 1445
000
<210> 1446
<400> 1446
000
<210> 1447
<400> 1447
000
<210> 1448
<400> 1448
000
<210> 1449
<400> 1449
000
<210> 1450
<400> 1450
000
<210> 1451
<400> 1451
000
<210> 1452
<400> 1452
000
<210> 1453
<400> 1453
000
<210> 1454
<400> 1454
000
<210> 1455
<400> 1455
000
<210> 1456
<400> 1456
000
<210> 1457
<400> 1457
000
<210> 1458
<400> 1458
000
<210> 1459
<400> 1459
000
<210> 1460
<400> 1460
000
<210> 1461
<400> 1461
000
<210> 1462
<400> 1462
000
<210> 1463
<400> 1463
000
<210> 1464
<400> 1464
000
<210> 1465
<400> 1465
000
<210> 1466
<400> 1466
000
<210> 1467
<400> 1467
000
<210> 1468
<400> 1468
000
<210> 1469
<400> 1469
000
<210> 1470
<400> 1470
000
<210> 1471
<400> 1471
000
<210> 1472
<400> 1472
000
<210> 1473
<400> 1473
000
<210> 1474
<400> 1474
000
<210> 1475
<400> 1475
000
<210> 1476
<400> 1476
000
<210> 1477
<400> 1477
000
<210> 1478
<400> 1478
000
<210> 1479
<400> 1479
000
<210> 1480
<400> 1480
000
<210> 1481
<400> 1481
000
<210> 1482
<400> 1482
000
<210> 1483
<400> 1483
000
<210> 1484
<400> 1484
000
<210> 1485
<400> 1485
000
<210> 1486
<400> 1486
000
<210> 1487
<400> 1487
000
<210> 1488
<400> 1488
000
<210> 1489
<400> 1489
000
<210> 1490
<400> 1490
000
<210> 1491
<400> 1491
000
<210> 1492
<400> 1492
000
<210> 1493
<400> 1493
000
<210> 1494
<400> 1494
000
<210> 1495
<400> 1495
000
<210> 1496
<400> 1496
000
<210> 1497
<400> 1497
000
<210> 1498
<400> 1498
000
<210> 1499
<400> 1499
000
<210> 1500
<400> 1500
000
<210> 1501
<400> 1501
000
<210> 1502
<400> 1502
000
<210> 1503
<400> 1503
000
<210> 1504
<400> 1504
000
<210> 1505
<400> 1505
000
<210> 1506
<400> 1506
000
<210> 1507
<400> 1507
000
<210> 1508
<400> 1508
000
<210> 1509
<400> 1509
000
<210> 1510
<400> 1510
000
<210> 1511
<400> 1511
000
<210> 1512
<400> 1512
000
<210> 1513
<400> 1513
000
<210> 1514
<400> 1514
000
<210> 1515
<400> 1515
000
<210> 1516
<400> 1516
000
<210> 1517
<400> 1517
000
<210> 1518
<400> 1518
000
<210> 1519
<400> 1519
000
<210> 1520
<400> 1520
000
<210> 1521
<400> 1521
000
<210> 1522
<400> 1522
000
<210> 1523
<400> 1523
000
<210> 1524
<400> 1524
000
<210> 1525
<400> 1525
000
<210> 1526
<400> 1526
000
<210> 1527
<400> 1527
000
<210> 1528
<400> 1528
000
<210> 1529
<400> 1529
000
<210> 1530
<400> 1530
000
<210> 1531
<400> 1531
000
<210> 1532
<400> 1532
000
<210> 1533
<400> 1533
000
<210> 1534
<400> 1534
000
<210> 1535
<400> 1535
000
<210> 1536
<400> 1536
000
<210> 1537
<400> 1537
000
<210> 1538
<400> 1538
000
<210> 1539
<400> 1539
000
<210> 1540
<400> 1540
000
<210> 1541
<400> 1541
000
<210> 1542
<400> 1542
000
<210> 1543
<400> 1543
000
<210> 1544
<400> 1544
000
<210> 1545
<400> 1545
000
<210> 1546
<400> 1546
000
<210> 1547
<400> 1547
000
<210> 1548
<400> 1548
000
<210> 1549
<400> 1549
000
<210> 1550
<400> 1550
000
<210> 1551
<400> 1551
000
<210> 1552
<400> 1552
000
<210> 1553
<400> 1553
000
<210> 1554
<400> 1554
000
<210> 1555
<400> 1555
000
<210> 1556
<400> 1556
000
<210> 1557
<400> 1557
000
<210> 1558
<400> 1558
000
<210> 1559
<400> 1559
000
<210> 1560
<400> 1560
000
<210> 1561
<400> 1561
000
<210> 1562
<400> 1562
000
<210> 1563
<400> 1563
000
<210> 1564
<400> 1564
000
<210> 1565
<400> 1565
000
<210> 1566
<400> 1566
000
<210> 1567
<400> 1567
000
<210> 1568
<400> 1568
000
<210> 1569
<400> 1569
000
<210> 1570
<400> 1570
000
<210> 1571
<400> 1571
000
<210> 1572
<400> 1572
000
<210> 1573
<400> 1573
000
<210> 1574
<400> 1574
000
<210> 1575
<400> 1575
000
<210> 1576
<400> 1576
000
<210> 1577
<400> 1577
000
<210> 1578
<400> 1578
000
<210> 1579
<400> 1579
000
<210> 1580
<400> 1580
000
<210> 1581
<400> 1581
000
<210> 1582
<400> 1582
000
<210> 1583
<400> 1583
000
<210> 1584
<400> 1584
000
<210> 1585
<400> 1585
000
<210> 1586
<400> 1586
000
<210> 1587
<400> 1587
000
<210> 1588
<400> 1588
000
<210> 1589
<400> 1589
000
<210> 1590
<400> 1590
000
<210> 1591
<400> 1591
000
<210> 1592
<400> 1592
000
<210> 1593
<400> 1593
000
<210> 1594
<400> 1594
000
<210> 1595
<400> 1595
000
<210> 1596
<400> 1596
000
<210> 1597
<400> 1597
000
<210> 1598
<400> 1598
000
<210> 1599
<400> 1599
000
<210> 1600
<400> 1600
000
<210> 1601
<400> 1601
000
<210> 1602
<400> 1602
000
<210> 1603
<400> 1603
000
<210> 1604
<400> 1604
000
<210> 1605
<400> 1605
000
<210> 1606
<400> 1606
000
<210> 1607
<400> 1607
000
<210> 1608
<400> 1608
000
<210> 1609
<400> 1609
000
<210> 1610
<400> 1610
000
<210> 1611
<400> 1611
000
<210> 1612
<400> 1612
000
<210> 1613
<400> 1613
000
<210> 1614
<400> 1614
000
<210> 1615
<400> 1615
000
<210> 1616
<400> 1616
000
<210> 1617
<400> 1617
000
<210> 1618
<400> 1618
000
<210> 1619
<400> 1619
000
<210> 1620
<400> 1620
000
<210> 1621
<400> 1621
000
<210> 1622
<400> 1622
000
<210> 1623
<400> 1623
000
<210> 1624
<400> 1624
000
<210> 1625
<400> 1625
000
<210> 1626
<400> 1626
000
<210> 1627
<400> 1627
000
<210> 1628
<400> 1628
000
<210> 1629
<400> 1629
000
<210> 1630
<400> 1630
000
<210> 1631
<400> 1631
000
<210> 1632
<400> 1632
000
<210> 1633
<400> 1633
000
<210> 1634
<400> 1634
000
<210> 1635
<400> 1635
000
<210> 1636
<400> 1636
000
<210> 1637
<400> 1637
000
<210> 1638
<400> 1638
000
<210> 1639
<400> 1639
000
<210> 1640
<400> 1640
000
<210> 1641
<400> 1641
000
<210> 1642
<400> 1642
000
<210> 1643
<400> 1643
000
<210> 1644
<400> 1644
000
<210> 1645
<400> 1645
000
<210> 1646
<400> 1646
000
<210> 1647
<400> 1647
000
<210> 1648
<400> 1648
000
<210> 1649
<400> 1649
000
<210> 1650
<400> 1650
000
<210> 1651
<400> 1651
000
<210> 1652
<400> 1652
000
<210> 1653
<400> 1653
000
<210> 1654
<400> 1654
000
<210> 1655
<400> 1655
000
<210> 1656
<400> 1656
000
<210> 1657
<400> 1657
000
<210> 1658
<400> 1658
000
<210> 1659
<400> 1659
000
<210> 1660
<400> 1660
000
<210> 1661
<400> 1661
000
<210> 1662
<400> 1662
000
<210> 1663
<400> 1663
000
<210> 1664
<400> 1664
000
<210> 1665
<400> 1665
000
<210> 1666
<400> 1666
000
<210> 1667
<400> 1667
000
<210> 1668
<400> 1668
000
<210> 1669
<400> 1669
000
<210> 1670
<400> 1670
000
<210> 1671
<400> 1671
000
<210> 1672
<400> 1672
000
<210> 1673
<400> 1673
000
<210> 1674
<400> 1674
000
<210> 1675
<400> 1675
000
<210> 1676
<400> 1676
000
<210> 1677
<400> 1677
000
<210> 1678
<400> 1678
000
<210> 1679
<400> 1679
000
<210> 1680
<400> 1680
000
<210> 1681
<400> 1681
000
<210> 1682
<400> 1682
000
<210> 1683
<400> 1683
000
<210> 1684
<400> 1684
000
<210> 1685
<400> 1685
000
<210> 1686
<400> 1686
000
<210> 1687
<400> 1687
000
<210> 1688
<400> 1688
000
<210> 1689
<400> 1689
000
<210> 1690
<400> 1690
000
<210> 1691
<400> 1691
000
<210> 1692
<400> 1692
000
<210> 1693
<400> 1693
000
<210> 1694
<400> 1694
000
<210> 1695
<400> 1695
000
<210> 1696
<400> 1696
000
<210> 1697
<400> 1697
000
<210> 1698
<400> 1698
000
<210> 1699
<400> 1699
000
<210> 1700
<400> 1700
000
<210> 1701
<400> 1701
000
<210> 1702
<400> 1702
000
<210> 1703
<400> 1703
000
<210> 1704
<400> 1704
000
<210> 1705
<400> 1705
000
<210> 1706
<400> 1706
000
<210> 1707
<400> 1707
000
<210> 1708
<400> 1708
000
<210> 1709
<400> 1709
000
<210> 1710
<400> 1710
000
<210> 1711
<400> 1711
000
<210> 1712
<400> 1712
000
<210> 1713
<400> 1713
000
<210> 1714
<400> 1714
000
<210> 1715
<400> 1715
000
<210> 1716
<400> 1716
000
<210> 1717
<400> 1717
000
<210> 1718
<400> 1718
000
<210> 1719
<400> 1719
000
<210> 1720
<400> 1720
000
<210> 1721
<400> 1721
000
<210> 1722
<400> 1722
000
<210> 1723
<400> 1723
000
<210> 1724
<400> 1724
000
<210> 1725
<400> 1725
000
<210> 1726
<400> 1726
000
<210> 1727
<400> 1727
000
<210> 1728
<400> 1728
000
<210> 1729
<400> 1729
000
<210> 1730
<400> 1730
000
<210> 1731
<400> 1731
000
<210> 1732
<400> 1732
000
<210> 1733
<400> 1733
000
<210> 1734
<400> 1734
000
<210> 1735
<400> 1735
000
<210> 1736
<400> 1736
000
<210> 1737
<400> 1737
000
<210> 1738
<400> 1738
000
<210> 1739
<400> 1739
000
<210> 1740
<400> 1740
000
<210> 1741
<400> 1741
000
<210> 1742
<400> 1742
000
<210> 1743
<400> 1743
000
<210> 1744
<400> 1744
000
<210> 1745
<400> 1745
000
<210> 1746
<400> 1746
000
<210> 1747
<400> 1747
000
<210> 1748
<400> 1748
000
<210> 1749
<400> 1749
000
<210> 1750
<400> 1750
000
<210> 1751
<400> 1751
000
<210> 1752
<400> 1752
000
<210> 1753
<400> 1753
000
<210> 1754
<400> 1754
000
<210> 1755
<400> 1755
000
<210> 1756
<400> 1756
000
<210> 1757
<400> 1757
000
<210> 1758
<400> 1758
000
<210> 1759
<400> 1759
000
<210> 1760
<400> 1760
000
<210> 1761
<400> 1761
000
<210> 1762
<400> 1762
000
<210> 1763
<400> 1763
000
<210> 1764
<400> 1764
000
<210> 1765
<400> 1765
000
<210> 1766
<400> 1766
000
<210> 1767
<400> 1767
000
<210> 1768
<400> 1768
000
<210> 1769
<400> 1769
000
<210> 1770
<400> 1770
000
<210> 1771
<400> 1771
000
<210> 1772
<400> 1772
000
<210> 1773
<400> 1773
000
<210> 1774
<400> 1774
000
<210> 1775
<400> 1775
000
<210> 1776
<400> 1776
000
<210> 1777
<400> 1777
000
<210> 1778
<400> 1778
000
<210> 1779
<400> 1779
000
<210> 1780
<400> 1780
000
<210> 1781
<400> 1781
000
<210> 1782
<400> 1782
000
<210> 1783
<400> 1783
000
<210> 1784
<400> 1784
000
<210> 1785
<400> 1785
000
<210> 1786
<400> 1786
000
<210> 1787
<400> 1787
000
<210> 1788
<400> 1788
000
<210> 1789
<400> 1789
000
<210> 1790
<400> 1790
000
<210> 1791
<400> 1791
000
<210> 1792
<400> 1792
000
<210> 1793
<400> 1793
000
<210> 1794
<400> 1794
000
<210> 1795
<400> 1795
000
<210> 1796
<400> 1796
000
<210> 1797
<400> 1797
000
<210> 1798
<400> 1798
000
<210> 1799
<400> 1799
000
<210> 1800
<400> 1800
000
<210> 1801
<400> 1801
000
<210> 1802
<400> 1802
000
<210> 1803
<400> 1803
000
<210> 1804
<400> 1804
000
<210> 1805
<400> 1805
000
<210> 1806
<400> 1806
000
<210> 1807
<400> 1807
000
<210> 1808
<400> 1808
000
<210> 1809
<400> 1809
000
<210> 1810
<400> 1810
000
<210> 1811
<400> 1811
000
<210> 1812
<400> 1812
000
<210> 1813
<400> 1813
000
<210> 1814
<400> 1814
000
<210> 1815
<400> 1815
000
<210> 1816
<400> 1816
000
<210> 1817
<400> 1817
000
<210> 1818
<400> 1818
000
<210> 1819
<400> 1819
000
<210> 1820
<400> 1820
000
<210> 1821
<400> 1821
000
<210> 1822
<400> 1822
000
<210> 1823
<400> 1823
000
<210> 1824
<400> 1824
000
<210> 1825
<400> 1825
000
<210> 1826
<400> 1826
000
<210> 1827
<400> 1827
000
<210> 1828
<400> 1828
000
<210> 1829
<400> 1829
000
<210> 1830
<400> 1830
000
<210> 1831
<400> 1831
000
<210> 1832
<400> 1832
000
<210> 1833
<400> 1833
000
<210> 1834
<400> 1834
000
<210> 1835
<400> 1835
000
<210> 1836
<400> 1836
000
<210> 1837
<400> 1837
000
<210> 1838
<400> 1838
000
<210> 1839
<400> 1839
000
<210> 1840
<400> 1840
000
<210> 1841
<400> 1841
000
<210> 1842
<400> 1842
000
<210> 1843
<400> 1843
000
<210> 1844
<400> 1844
000
<210> 1845
<400> 1845
000
<210> 1846
<400> 1846
000
<210> 1847
<400> 1847
000
<210> 1848
<400> 1848
000
<210> 1849
<400> 1849
000
<210> 1850
<400> 1850
000
<210> 1851
<400> 1851
000
<210> 1852
<400> 1852
000
<210> 1853
<400> 1853
000
<210> 1854
<400> 1854
000
<210> 1855
<400> 1855
000
<210> 1856
<400> 1856
000
<210> 1857
<400> 1857
000
<210> 1858
<400> 1858
000
<210> 1859
<400> 1859
000
<210> 1860
<400> 1860
000
<210> 1861
<400> 1861
000
<210> 1862
<400> 1862
000
<210> 1863
<400> 1863
000
<210> 1864
<400> 1864
000
<210> 1865
<400> 1865
000
<210> 1866
<400> 1866
000
<210> 1867
<400> 1867
000
<210> 1868
<400> 1868
000
<210> 1869
<400> 1869
000
<210> 1870
<400> 1870
000
<210> 1871
<400> 1871
000
<210> 1872
<400> 1872
000
<210> 1873
<400> 1873
000
<210> 1874
<400> 1874
000
<210> 1875
<400> 1875
000
<210> 1876
<400> 1876
000
<210> 1877
<400> 1877
000
<210> 1878
<400> 1878
000
<210> 1879
<400> 1879
000
<210> 1880
<400> 1880
000
<210> 1881
<400> 1881
000
<210> 1882
<400> 1882
000
<210> 1883
<400> 1883
000
<210> 1884
<400> 1884
000
<210> 1885
<400> 1885
000
<210> 1886
<400> 1886
000
<210> 1887
<400> 1887
000
<210> 1888
<400> 1888
000
<210> 1889
<400> 1889
000
<210> 1890
<400> 1890
000
<210> 1891
<400> 1891
000
<210> 1892
<400> 1892
000
<210> 1893
<400> 1893
000
<210> 1894
<400> 1894
000
<210> 1895
<400> 1895
000
<210> 1896
<400> 1896
000
<210> 1897
<400> 1897
000
<210> 1898
<400> 1898
000
<210> 1899
<400> 1899
000
<210> 1900
<400> 1900
000
<210> 1901
<400> 1901
000
<210> 1902
<400> 1902
000
<210> 1903
<400> 1903
000
<210> 1904
<400> 1904
000
<210> 1905
<400> 1905
000
<210> 1906
<400> 1906
000
<210> 1907
<400> 1907
000
<210> 1908
<400> 1908
000
<210> 1909
<400> 1909
000
<210> 1910
<400> 1910
000
<210> 1911
<400> 1911
000
<210> 1912
<400> 1912
000
<210> 1913
<400> 1913
000
<210> 1914
<400> 1914
000
<210> 1915
<400> 1915
000
<210> 1916
<400> 1916
000
<210> 1917
<400> 1917
000
<210> 1918
<400> 1918
000
<210> 1919
<400> 1919
000
<210> 1920
<400> 1920
000
<210> 1921
<400> 1921
000
<210> 1922
<400> 1922
000
<210> 1923
<400> 1923
000
<210> 1924
<400> 1924
000
<210> 1925
<400> 1925
000
<210> 1926
<400> 1926
000
<210> 1927
<400> 1927
000
<210> 1928
<400> 1928
000
<210> 1929
<400> 1929
000
<210> 1930
<400> 1930
000
<210> 1931
<400> 1931
000
<210> 1932
<400> 1932
000
<210> 1933
<400> 1933
000
<210> 1934
<400> 1934
000
<210> 1935
<400> 1935
000
<210> 1936
<400> 1936
000
<210> 1937
<400> 1937
000
<210> 1938
<400> 1938
000
<210> 1939
<400> 1939
000
<210> 1940
<400> 1940
000
<210> 1941
<400> 1941
000
<210> 1942
<400> 1942
000
<210> 1943
<400> 1943
000
<210> 1944
<400> 1944
000
<210> 1945
<400> 1945
000
<210> 1946
<400> 1946
000
<210> 1947
<400> 1947
000
<210> 1948
<400> 1948
000
<210> 1949
<400> 1949
000
<210> 1950
<400> 1950
000
<210> 1951
<400> 1951
000
<210> 1952
<400> 1952
000
<210> 1953
<400> 1953
000
<210> 1954
<400> 1954
000
<210> 1955
<400> 1955
000
<210> 1956
<400> 1956
000
<210> 1957
<400> 1957
000
<210> 1958
<400> 1958
000
<210> 1959
<400> 1959
000
<210> 1960
<400> 1960
000
<210> 1961
<400> 1961
000
<210> 1962
<400> 1962
000
<210> 1963
<400> 1963
000
<210> 1964
<400> 1964
000
<210> 1965
<400> 1965
000
<210> 1966
<400> 1966
000
<210> 1967
<400> 1967
000
<210> 1968
<400> 1968
000
<210> 1969
<400> 1969
000
<210> 1970
<400> 1970
000
<210> 1971
<400> 1971
000
<210> 1972
<400> 1972
000
<210> 1973
<400> 1973
000
<210> 1974
<400> 1974
000
<210> 1975
<400> 1975
000
<210> 1976
<400> 1976
000
<210> 1977
<400> 1977
000
<210> 1978
<400> 1978
000
<210> 1979
<400> 1979
000
<210> 1980
<400> 1980
000
<210> 1981
<400> 1981
000
<210> 1982
<400> 1982
000
<210> 1983
<400> 1983
000
<210> 1984
<400> 1984
000
<210> 1985
<400> 1985
000
<210> 1986
<400> 1986
000
<210> 1987
<400> 1987
000
<210> 1988
<400> 1988
000
<210> 1989
<400> 1989
000
<210> 1990
<400> 1990
000
<210> 1991
<400> 1991
000
<210> 1992
<400> 1992
000
<210> 1993
<400> 1993
000
<210> 1994
<400> 1994
000
<210> 1995
<400> 1995
000
<210> 1996
<400> 1996
000
<210> 1997
<400> 1997
000
<210> 1998
<400> 1998
000
<210> 1999
<400> 1999
000
<210> 2000
<400> 2000
000
<210> 2001
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH CDR1
<400> 2001
Gly Thr Phe Gly Asn Tyr Ala Ile Ser
1 5
<210> 2002
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH CDR2
<400> 2002
Gly Ile Ile Pro Ile Pro Gly Ile Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 2003
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH CDR3
<400> 2003
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
1 5 10 15
<210> 2004
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL CDR1
<400> 2004
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 2005
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL CDR2
<400> 2005
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 2006
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL CDR3
<400> 2006
Gln Gln Ser Asp Ile Leu Tyr Thr
1 5
<210> 2007
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH FR1
<400> 2007
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 2008
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH FR2
<400> 2008
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 2009
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH FR3
<400> 2009
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 2010
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH FR4
<400> 2010
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 2011
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH ДНК
<400> 2011
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcgga aactatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tcccaggtat cgcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagagacacg 300
ggacggggat acaccagaca cttctggttt gacccctggg gacagggtac attggtcacc 360
gtctcctca 369
<210> 2012
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VH Белка
<400> 2012
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Gly Asn Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Pro Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 2013
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL FR1
<400> 2013
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 2014
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL FR2
<400> 2014
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 2015
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL FR3
<400> 2015
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 2016
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL FR4
<400> 2016
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 2017
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL ДНК
<400> 2017
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agcgacatcc tctacacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 2018
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 33 - VL Белка
<400> 2018
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asp Ile Leu Tyr Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 2019
<400> 2019
000
<210> 2020
<400> 2020
000
<210> 2021
<400> 2021
000
<210> 2022
<400> 2022
000
<210> 2023
<400> 2023
000
<210> 2024
<400> 2024
000
<210> 2025
<400> 2025
000
<210> 2026
<400> 2026
000
<210> 2027
<400> 2027
000
<210> 2028
<400> 2028
000
<210> 2029
<400> 2029
000
<210> 2030
<400> 2030
000
<210> 2031
<400> 2031
000
<210> 2032
<400> 2032
000
<210> 2033
<400> 2033
000
<210> 2034
<400> 2034
000
<210> 2035
<400> 2035
000
<210> 2036
<400> 2036
000
<210> 2037
<400> 2037
000
<210> 2038
<400> 2038
000
<210> 2039
<400> 2039
000
<210> 2040
<400> 2040
000
<210> 2041
<400> 2041
000
<210> 2042
<400> 2042
000
<210> 2043
<400> 2043
000
<210> 2044
<400> 2044
000
<210> 2045
<400> 2045
000
<210> 2046
<400> 2046
000
<210> 2047
<400> 2047
000
<210> 2048
<400> 2048
000
<210> 2049
<400> 2049
000
<210> 2050
<400> 2050
000
<210> 2051
<400> 2051
000
<210> 2052
<400> 2052
000
<210> 2053
<400> 2053
000
<210> 2054
<400> 2054
000
<210> 2055
<400> 2055
000
<210> 2056
<400> 2056
000
<210> 2057
<400> 2057
000
<210> 2058
<400> 2058
000
<210> 2059
<400> 2059
000
<210> 2060
<400> 2060
000
<210> 2061
<400> 2061
000
<210> 2062
<400> 2062
000
<210> 2063
<400> 2063
000
<210> 2064
<400> 2064
000
<210> 2065
<400> 2065
000
<210> 2066
<400> 2066
000
<210> 2067
<400> 2067
000
<210> 2068
<400> 2068
000
<210> 2069
<400> 2069
000
<210> 2070
<400> 2070
000
<210> 2071
<400> 2071
000
<210> 2072
<400> 2072
000
<210> 2073
<400> 2073
000
<210> 2074
<400> 2074
000
<210> 2075
<400> 2075
000
<210> 2076
<400> 2076
000
<210> 2077
<400> 2077
000
<210> 2078
<400> 2078
000
<210> 2079
<400> 2079
000
<210> 2080
<400> 2080
000
<210> 2081
<400> 2081
000
<210> 2082
<400> 2082
000
<210> 2083
<400> 2083
000
<210> 2084
<400> 2084
000
<210> 2085
<400> 2085
000
<210> 2086
<400> 2086
000
<210> 2087
<400> 2087
000
<210> 2088
<400> 2088
000
<210> 2089
<400> 2089
000
<210> 2090
<400> 2090
000
<210> 2091
<400> 2091
000
<210> 2092
<400> 2092
000
<210> 2093
<400> 2093
000
<210> 2094
<400> 2094
000
<210> 2095
<400> 2095
000
<210> 2096
<400> 2096
000
<210> 2097
<400> 2097
000
<210> 2098
<400> 2098
000
<210> 2099
<400> 2099
000
<210> 2100
<400> 2100
000
<210> 2101
<400> 2101
000
<210> 2102
<400> 2102
000
<210> 2103
<400> 2103
000
<210> 2104
<400> 2104
000
<210> 2105
<400> 2105
000
<210> 2106
<400> 2106
000
<210> 2107
<400> 2107
000
<210> 2108
<400> 2108
000
<210> 2109
<400> 2109
000
<210> 2110
<400> 2110
000
<210> 2111
<400> 2111
000
<210> 2112
<400> 2112
000
<210> 2113
<400> 2113
000
<210> 2114
<400> 2114
000
<210> 2115
<400> 2115
000
<210> 2116
<400> 2116
000
<210> 2117
<400> 2117
000
<210> 2118
<400> 2118
000
<210> 2119
<400> 2119
000
<210> 2120
<400> 2120
000
<210> 2121
<400> 2121
000
<210> 2122
<400> 2122
000
<210> 2123
<400> 2123
000
<210> 2124
<400> 2124
000
<210> 2125
<400> 2125
000
<210> 2126
<400> 2126
000
<210> 2127
<400> 2127
000
<210> 2128
<400> 2128
000
<210> 2129
<400> 2129
000
<210> 2130
<400> 2130
000
<210> 2131
<400> 2131
000
<210> 2132
<400> 2132
000
<210> 2133
<400> 2133
000
<210> 2134
<400> 2134
000
<210> 2135
<400> 2135
000
<210> 2136
<400> 2136
000
<210> 2137
<400> 2137
000
<210> 2138
<400> 2138
000
<210> 2139
<400> 2139
000
<210> 2140
<400> 2140
000
<210> 2141
<400> 2141
000
<210> 2142
<400> 2142
000
<210> 2143
<400> 2143
000
<210> 2144
<400> 2144
000
<210> 2145
<400> 2145
000
<210> 2146
<400> 2146
000
<210> 2147
<400> 2147
000
<210> 2148
<400> 2148
000
<210> 2149
<400> 2149
000
<210> 2150
<400> 2150
000
<210> 2151
<400> 2151
000
<210> 2152
<400> 2152
000
<210> 2153
<400> 2153
000
<210> 2154
<400> 2154
000
<210> 2155
<400> 2155
000
<210> 2156
<400> 2156
000
<210> 2157
<400> 2157
000
<210> 2158
<400> 2158
000
<210> 2159
<400> 2159
000
<210> 2160
<400> 2160
000
<210> 2161
<400> 2161
000
<210> 2162
<400> 2162
000
<210> 2163
<400> 2163
000
<210> 2164
<400> 2164
000
<210> 2165
<400> 2165
000
<210> 2166
<400> 2166
000
<210> 2167
<400> 2167
000
<210> 2168
<400> 2168
000
<210> 2169
<400> 2169
000
<210> 2170
<400> 2170
000
<210> 2171
<400> 2171
000
<210> 2172
<400> 2172
000
<210> 2173
<400> 2173
000
<210> 2174
<400> 2174
000
<210> 2175
<400> 2175
000
<210> 2176
<400> 2176
000
<210> 2177
<400> 2177
000
<210> 2178
<400> 2178
000
<210> 2179
<400> 2179
000
<210> 2180
<400> 2180
000
<210> 2181
<400> 2181
000
<210> 2182
<400> 2182
000
<210> 2183
<400> 2183
000
<210> 2184
<400> 2184
000
<210> 2185
<400> 2185
000
<210> 2186
<400> 2186
000
<210> 2187
<400> 2187
000
<210> 2188
<400> 2188
000
<210> 2189
<400> 2189
000
<210> 2190
<400> 2190
000
<210> 2191
<400> 2191
000
<210> 2192
<400> 2192
000
<210> 2193
<400> 2193
000
<210> 2194
<400> 2194
000
<210> 2195
<400> 2195
000
<210> 2196
<400> 2196
000
<210> 2197
<400> 2197
000
<210> 2198
<400> 2198
000
<210> 2199
<400> 2199
000
<210> 2200
<400> 2200
000
<210> 2201
<400> 2201
000
<210> 2202
<400> 2202
000
<210> 2203
<400> 2203
000
<210> 2204
<400> 2204
000
<210> 2205
<400> 2205
000
<210> 2206
<400> 2206
000
<210> 2207
<400> 2207
000
<210> 2208
<400> 2208
000
<210> 2209
<400> 2209
000
<210> 2210
<400> 2210
000
<210> 2211
<400> 2211
000
<210> 2212
<400> 2212
000
<210> 2213
<400> 2213
000
<210> 2214
<400> 2214
000
<210> 2215
<400> 2215
000
<210> 2216
<400> 2216
000
<210> 2217
<400> 2217
000
<210> 2218
<400> 2218
000
<210> 2219
<400> 2219
000
<210> 2220
<400> 2220
000
<210> 2221
<400> 2221
000
<210> 2222
<400> 2222
000
<210> 2223
<400> 2223
000
<210> 2224
<400> 2224
000
<210> 2225
<400> 2225
000
<210> 2226
<400> 2226
000
<210> 2227
<400> 2227
000
<210> 2228
<400> 2228
000
<210> 2229
<400> 2229
000
<210> 2230
<400> 2230
000
<210> 2231
<400> 2231
000
<210> 2232
<400> 2232
000
<210> 2233
<400> 2233
000
<210> 2234
<400> 2234
000
<210> 2235
<400> 2235
000
<210> 2236
<400> 2236
000
<210> 2237
<400> 2237
000
<210> 2238
<400> 2238
000
<210> 2239
<400> 2239
000
<210> 2240
<400> 2240
000
<210> 2241
<400> 2241
000
<210> 2242
<400> 2242
000
<210> 2243
<400> 2243
000
<210> 2244
<400> 2244
000
<210> 2245
<400> 2245
000
<210> 2246
<400> 2246
000
<210> 2247
<400> 2247
000
<210> 2248
<400> 2248
000
<210> 2249
<400> 2249
000
<210> 2250
<400> 2250
000
<210> 2251
<400> 2251
000
<210> 2252
<400> 2252
000
<210> 2253
<400> 2253
000
<210> 2254
<400> 2254
000
<210> 2255
<400> 2255
000
<210> 2256
<400> 2256
000
<210> 2257
<400> 2257
000
<210> 2258
<400> 2258
000
<210> 2259
<400> 2259
000
<210> 2260
<400> 2260
000
<210> 2261
<400> 2261
000
<210> 2262
<400> 2262
000
<210> 2263
<400> 2263
000
<210> 2264
<400> 2264
000
<210> 2265
<400> 2265
000
<210> 2266
<400> 2266
000
<210> 2267
<400> 2267
000
<210> 2268
<400> 2268
000
<210> 2269
<400> 2269
000
<210> 2270
<400> 2270
000
<210> 2271
<400> 2271
000
<210> 2272
<400> 2272
000
<210> 2273
<400> 2273
000
<210> 2274
<400> 2274
000
<210> 2275
<400> 2275
000
<210> 2276
<400> 2276
000
<210> 2277
<400> 2277
000
<210> 2278
<400> 2278
000
<210> 2279
<400> 2279
000
<210> 2280
<400> 2280
000
<210> 2281
<400> 2281
000
<210> 2282
<400> 2282
000
<210> 2283
<400> 2283
000
<210> 2284
<400> 2284
000
<210> 2285
<400> 2285
000
<210> 2286
<400> 2286
000
<210> 2287
<400> 2287
000
<210> 2288
<400> 2288
000
<210> 2289
<400> 2289
000
<210> 2290
<400> 2290
000
<210> 2291
<400> 2291
000
<210> 2292
<400> 2292
000
<210> 2293
<400> 2293
000
<210> 2294
<400> 2294
000
<210> 2295
<400> 2295
000
<210> 2296
<400> 2296
000
<210> 2297
<400> 2297
000
<210> 2298
<400> 2298
000
<210> 2299
<400> 2299
000
<210> 2300
<400> 2300
000
<210> 2301
<400> 2301
000
<210> 2302
<400> 2302
000
<210> 2303
<400> 2303
000
<210> 2304
<400> 2304
000
<210> 2305
<400> 2305
000
<210> 2306
<400> 2306
000
<210> 2307
<400> 2307
000
<210> 2308
<400> 2308
000
<210> 2309
<400> 2309
000
<210> 2310
<400> 2310
000
<210> 2311
<400> 2311
000
<210> 2312
<400> 2312
000
<210> 2313
<400> 2313
000
<210> 2314
<400> 2314
000
<210> 2315
<400> 2315
000
<210> 2316
<400> 2316
000
<210> 2317
<400> 2317
000
<210> 2318
<400> 2318
000
<210> 2319
<400> 2319
000
<210> 2320
<400> 2320
000
<210> 2321
<400> 2321
000
<210> 2322
<400> 2322
000
<210> 2323
<400> 2323
000
<210> 2324
<400> 2324
000
<210> 2325
<400> 2325
000
<210> 2326
<400> 2326
000
<210> 2327
<400> 2327
000
<210> 2328
<400> 2328
000
<210> 2329
<400> 2329
000
<210> 2330
<400> 2330
000
<210> 2331
<400> 2331
000
<210> 2332
<400> 2332
000
<210> 2333
<400> 2333
000
<210> 2334
<400> 2334
000
<210> 2335
<400> 2335
000
<210> 2336
<400> 2336
000
<210> 2337
<400> 2337
000
<210> 2338
<400> 2338
000
<210> 2339
<400> 2339
000
<210> 2340
<400> 2340
000
<210> 2341
<400> 2341
000
<210> 2342
<400> 2342
000
<210> 2343
<400> 2343
000
<210> 2344
<400> 2344
000
<210> 2345
<400> 2345
000
<210> 2346
<400> 2346
000
<210> 2347
<400> 2347
000
<210> 2348
<400> 2348
000
<210> 2349
<400> 2349
000
<210> 2350
<400> 2350
000
<210> 2351
<400> 2351
000
<210> 2352
<400> 2352
000
<210> 2353
<400> 2353
000
<210> 2354
<400> 2354
000
<210> 2355
<400> 2355
000
<210> 2356
<400> 2356
000
<210> 2357
<400> 2357
000
<210> 2358
<400> 2358
000
<210> 2359
<400> 2359
000
<210> 2360
<400> 2360
000
<210> 2361
<400> 2361
000
<210> 2362
<400> 2362
000
<210> 2363
<400> 2363
000
<210> 2364
<400> 2364
000
<210> 2365
<400> 2365
000
<210> 2366
<400> 2366
000
<210> 2367
<400> 2367
000
<210> 2368
<400> 2368
000
<210> 2369
<400> 2369
000
<210> 2370
<400> 2370
000
<210> 2371
<400> 2371
000
<210> 2372
<400> 2372
000
<210> 2373
<400> 2373
000
<210> 2374
<400> 2374
000
<210> 2375
<400> 2375
000
<210> 2376
<400> 2376
000
<210> 2377
<400> 2377
000
<210> 2378
<400> 2378
000
<210> 2379
<400> 2379
000
<210> 2380
<400> 2380
000
<210> 2381
<400> 2381
000
<210> 2382
<400> 2382
000
<210> 2383
<400> 2383
000
<210> 2384
<400> 2384
000
<210> 2385
<400> 2385
000
<210> 2386
<400> 2386
000
<210> 2387
<400> 2387
000
<210> 2388
<400> 2388
000
<210> 2389
<400> 2389
000
<210> 2390
<400> 2390
000
<210> 2391
<400> 2391
000
<210> 2392
<400> 2392
000
<210> 2393
<400> 2393
000
<210> 2394
<400> 2394
000
<210> 2395
<400> 2395
000
<210> 2396
<400> 2396
000
<210> 2397
<400> 2397
000
<210> 2398
<400> 2398
000
<210> 2399
<400> 2399
000
<210> 2400
<400> 2400
000
<210> 2401
<400> 2401
000
<210> 2402
<400> 2402
000
<210> 2403
<400> 2403
000
<210> 2404
<400> 2404
000
<210> 2405
<400> 2405
000
<210> 2406
<400> 2406
000
<210> 2407
<400> 2407
000
<210> 2408
<400> 2408
000
<210> 2409
<400> 2409
000
<210> 2410
<400> 2410
000
<210> 2411
<400> 2411
000
<210> 2412
<400> 2412
000
<210> 2413
<400> 2413
000
<210> 2414
<400> 2414
000
<210> 2415
<400> 2415
000
<210> 2416
<400> 2416
000
<210> 2417
<400> 2417
000
<210> 2418
<400> 2418
000
<210> 2419
<400> 2419
000
<210> 2420
<400> 2420
000
<210> 2421
<400> 2421
000
<210> 2422
<400> 2422
000
<210> 2423
<400> 2423
000
<210> 2424
<400> 2424
000
<210> 2425
<400> 2425
000
<210> 2426
<400> 2426
000
<210> 2427
<400> 2427
000
<210> 2428
<400> 2428
000
<210> 2429
<400> 2429
000
<210> 2430
<400> 2430
000
<210> 2431
<400> 2431
000
<210> 2432
<400> 2432
000
<210> 2433
<400> 2433
000
<210> 2434
<400> 2434
000
<210> 2435
<400> 2435
000
<210> 2436
<400> 2436
000
<210> 2437
<400> 2437
000
<210> 2438
<400> 2438
000
<210> 2439
<400> 2439
000
<210> 2440
<400> 2440
000
<210> 2441
<400> 2441
000
<210> 2442
<400> 2442
000
<210> 2443
<400> 2443
000
<210> 2444
<400> 2444
000
<210> 2445
<400> 2445
000
<210> 2446
<400> 2446
000
<210> 2447
<400> 2447
000
<210> 2448
<400> 2448
000
<210> 2449
<400> 2449
000
<210> 2450
<400> 2450
000
<210> 2451
<400> 2451
000
<210> 2452
<400> 2452
000
<210> 2453
<400> 2453
000
<210> 2454
<400> 2454
000
<210> 2455
<400> 2455
000
<210> 2456
<400> 2456
000
<210> 2457
<400> 2457
000
<210> 2458
<400> 2458
000
<210> 2459
<400> 2459
000
<210> 2460
<400> 2460
000
<210> 2461
<400> 2461
000
<210> 2462
<400> 2462
000
<210> 2463
<400> 2463
000
<210> 2464
<400> 2464
000
<210> 2465
<400> 2465
000
<210> 2466
<400> 2466
000
<210> 2467
<400> 2467
000
<210> 2468
<400> 2468
000
<210> 2469
<400> 2469
000
<210> 2470
<400> 2470
000
<210> 2471
<400> 2471
000
<210> 2472
<400> 2472
000
<210> 2473
<400> 2473
000
<210> 2474
<400> 2474
000
<210> 2475
<400> 2475
000
<210> 2476
<400> 2476
000
<210> 2477
<400> 2477
000
<210> 2478
<400> 2478
000
<210> 2479
<400> 2479
000
<210> 2480
<400> 2480
000
<210> 2481
<400> 2481
000
<210> 2482
<400> 2482
000
<210> 2483
<400> 2483
000
<210> 2484
<400> 2484
000
<210> 2485
<400> 2485
000
<210> 2486
<400> 2486
000
<210> 2487
<400> 2487
000
<210> 2488
<400> 2488
000
<210> 2489
<400> 2489
000
<210> 2490
<400> 2490
000
<210> 2491
<400> 2491
000
<210> 2492
<400> 2492
000
<210> 2493
<400> 2493
000
<210> 2494
<400> 2494
000
<210> 2495
<400> 2495
000
<210> 2496
<400> 2496
000
<210> 2497
<400> 2497
000
<210> 2498
<400> 2498
000
<210> 2499
<400> 2499
000
<210> 2500
<400> 2500
000
<210> 2501
<400> 2501
000
<210> 2502
<400> 2502
000
<210> 2503
<400> 2503
000
<210> 2504
<400> 2504
000
<210> 2505
<400> 2505
000
<210> 2506
<400> 2506
000
<210> 2507
<400> 2507
000
<210> 2508
<400> 2508
000
<210> 2509
<400> 2509
000
<210> 2510
<400> 2510
000
<210> 2511
<400> 2511
000
<210> 2512
<400> 2512
000
<210> 2513
<400> 2513
000
<210> 2514
<400> 2514
000
<210> 2515
<400> 2515
000
<210> 2516
<400> 2516
000
<210> 2517
<400> 2517
000
<210> 2518
<400> 2518
000
<210> 2519
<400> 2519
000
<210> 2520
<400> 2520
000
<210> 2521
<400> 2521
000
<210> 2522
<400> 2522
000
<210> 2523
<400> 2523
000
<210> 2524
<400> 2524
000
<210> 2525
<400> 2525
000
<210> 2526
<400> 2526
000
<210> 2527
<400> 2527
000
<210> 2528
<400> 2528
000
<210> 2529
<400> 2529
000
<210> 2530
<400> 2530
000
<210> 2531
<400> 2531
000
<210> 2532
<400> 2532
000
<210> 2533
<400> 2533
000
<210> 2534
<400> 2534
000
<210> 2535
<400> 2535
000
<210> 2536
<400> 2536
000
<210> 2537
<400> 2537
000
<210> 2538
<400> 2538
000
<210> 2539
<400> 2539
000
<210> 2540
<400> 2540
000
<210> 2541
<400> 2541
000
<210> 2542
<400> 2542
000
<210> 2543
<400> 2543
000
<210> 2544
<400> 2544
000
<210> 2545
<400> 2545
000
<210> 2546
<400> 2546
000
<210> 2547
<400> 2547
000
<210> 2548
<400> 2548
000
<210> 2549
<400> 2549
000
<210> 2550
<400> 2550
000
<210> 2551
<400> 2551
000
<210> 2552
<400> 2552
000
<210> 2553
<400> 2553
000
<210> 2554
<400> 2554
000
<210> 2555
<400> 2555
000
<210> 2556
<400> 2556
000
<210> 2557
<400> 2557
000
<210> 2558
<400> 2558
000
<210> 2559
<400> 2559
000
<210> 2560
<400> 2560
000
<210> 2561
<400> 2561
000
<210> 2562
<400> 2562
000
<210> 2563
<400> 2563
000
<210> 2564
<400> 2564
000
<210> 2565
<400> 2565
000
<210> 2566
<400> 2566
000
<210> 2567
<400> 2567
000
<210> 2568
<400> 2568
000
<210> 2569
<400> 2569
000
<210> 2570
<400> 2570
000
<210> 2571
<400> 2571
000
<210> 2572
<400> 2572
000
<210> 2573
<400> 2573
000
<210> 2574
<400> 2574
000
<210> 2575
<400> 2575
000
<210> 2576
<400> 2576
000
<210> 2577
<400> 2577
000
<210> 2578
<400> 2578
000
<210> 2579
<400> 2579
000
<210> 2580
<400> 2580
000
<210> 2581
<400> 2581
000
<210> 2582
<400> 2582
000
<210> 2583
<400> 2583
000
<210> 2584
<400> 2584
000
<210> 2585
<400> 2585
000
<210> 2586
<400> 2586
000
<210> 2587
<400> 2587
000
<210> 2588
<400> 2588
000
<210> 2589
<400> 2589
000
<210> 2590
<400> 2590
000
<210> 2591
<400> 2591
000
<210> 2592
<400> 2592
000
<210> 2593
<400> 2593
000
<210> 2594
<400> 2594
000
<210> 2595
<400> 2595
000
<210> 2596
<400> 2596
000
<210> 2597
<400> 2597
000
<210> 2598
<400> 2598
000
<210> 2599
<400> 2599
000
<210> 2600
<400> 2600
000
<210> 2601
<400> 2601
000
<210> 2602
<400> 2602
000
<210> 2603
<400> 2603
000
<210> 2604
<400> 2604
000
<210> 2605
<400> 2605
000
<210> 2606
<400> 2606
000
<210> 2607
<400> 2607
000
<210> 2608
<400> 2608
000
<210> 2609
<400> 2609
000
<210> 2610
<400> 2610
000
<210> 2611
<400> 2611
000
<210> 2612
<400> 2612
000
<210> 2613
<400> 2613
000
<210> 2614
<400> 2614
000
<210> 2615
<400> 2615
000
<210> 2616
<400> 2616
000
<210> 2617
<400> 2617
000
<210> 2618
<400> 2618
000
<210> 2619
<400> 2619
000
<210> 2620
<400> 2620
000
<210> 2621
<400> 2621
000
<210> 2622
<400> 2622
000
<210> 2623
<400> 2623
000
<210> 2624
<400> 2624
000
<210> 2625
<400> 2625
000
<210> 2626
<400> 2626
000
<210> 2627
<400> 2627
000
<210> 2628
<400> 2628
000
<210> 2629
<400> 2629
000
<210> 2630
<400> 2630
000
<210> 2631
<400> 2631
000
<210> 2632
<400> 2632
000
<210> 2633
<400> 2633
000
<210> 2634
<400> 2634
000
<210> 2635
<400> 2635
000
<210> 2636
<400> 2636
000
<210> 2637
<400> 2637
000
<210> 2638
<400> 2638
000
<210> 2639
<400> 2639
000
<210> 2640
<400> 2640
000
<210> 2641
<400> 2641
000
<210> 2642
<400> 2642
000
<210> 2643
<400> 2643
000
<210> 2644
<400> 2644
000
<210> 2645
<400> 2645
000
<210> 2646
<400> 2646
000
<210> 2647
<400> 2647
000
<210> 2648
<400> 2648
000
<210> 2649
<400> 2649
000
<210> 2650
<400> 2650
000
<210> 2651
<400> 2651
000
<210> 2652
<400> 2652
000
<210> 2653
<400> 2653
000
<210> 2654
<400> 2654
000
<210> 2655
<400> 2655
000
<210> 2656
<400> 2656
000
<210> 2657
<400> 2657
000
<210> 2658
<400> 2658
000
<210> 2659
<400> 2659
000
<210> 2660
<400> 2660
000
<210> 2661
<400> 2661
000
<210> 2662
<400> 2662
000
<210> 2663
<400> 2663
000
<210> 2664
<400> 2664
000
<210> 2665
<400> 2665
000
<210> 2666
<400> 2666
000
<210> 2667
<400> 2667
000
<210> 2668
<400> 2668
000
<210> 2669
<400> 2669
000
<210> 2670
<400> 2670
000
<210> 2671
<400> 2671
000
<210> 2672
<400> 2672
000
<210> 2673
<400> 2673
000
<210> 2674
<400> 2674
000
<210> 2675
<400> 2675
000
<210> 2676
<400> 2676
000
<210> 2677
<400> 2677
000
<210> 2678
<400> 2678
000
<210> 2679
<400> 2679
000
<210> 2680
<400> 2680
000
<210> 2681
<400> 2681
000
<210> 2682
<400> 2682
000
<210> 2683
<400> 2683
000
<210> 2684
<400> 2684
000
<210> 2685
<400> 2685
000
<210> 2686
<400> 2686
000
<210> 2687
<400> 2687
000
<210> 2688
<400> 2688
000
<210> 2689
<400> 2689
000
<210> 2690
<400> 2690
000
<210> 2691
<400> 2691
000
<210> 2692
<400> 2692
000
<210> 2693
<400> 2693
000
<210> 2694
<400> 2694
000
<210> 2695
<400> 2695
000
<210> 2696
<400> 2696
000
<210> 2697
<400> 2697
000
<210> 2698
<400> 2698
000
<210> 2699
<400> 2699
000
<210> 2700
<400> 2700
000
<210> 2701
<400> 2701
000
<210> 2702
<400> 2702
000
<210> 2703
<400> 2703
000
<210> 2704
<400> 2704
000
<210> 2705
<400> 2705
000
<210> 2706
<400> 2706
000
<210> 2707
<400> 2707
000
<210> 2708
<400> 2708
000
<210> 2709
<400> 2709
000
<210> 2710
<400> 2710
000
<210> 2711
<400> 2711
000
<210> 2712
<400> 2712
000
<210> 2713
<400> 2713
000
<210> 2714
<400> 2714
000
<210> 2715
<400> 2715
000
<210> 2716
<400> 2716
000
<210> 2717
<400> 2717
000
<210> 2718
<400> 2718
000
<210> 2719
<400> 2719
000
<210> 2720
<400> 2720
000
<210> 2721
<400> 2721
000
<210> 2722
<400> 2722
000
<210> 2723
<400> 2723
000
<210> 2724
<400> 2724
000
<210> 2725
<400> 2725
000
<210> 2726
<400> 2726
000
<210> 2727
<400> 2727
000
<210> 2728
<400> 2728
000
<210> 2729
<400> 2729
000
<210> 2730
<400> 2730
000
<210> 2731
<400> 2731
000
<210> 2732
<400> 2732
000
<210> 2733
<400> 2733
000
<210> 2734
<400> 2734
000
<210> 2735
<400> 2735
000
<210> 2736
<400> 2736
000
<210> 2737
<400> 2737
000
<210> 2738
<400> 2738
000
<210> 2739
<400> 2739
000
<210> 2740
<400> 2740
000
<210> 2741
<400> 2741
000
<210> 2742
<400> 2742
000
<210> 2743
<400> 2743
000
<210> 2744
<400> 2744
000
<210> 2745
<400> 2745
000
<210> 2746
<400> 2746
000
<210> 2747
<400> 2747
000
<210> 2748
<400> 2748
000
<210> 2749
<400> 2749
000
<210> 2750
<400> 2750
000
<210> 2751
<400> 2751
000
<210> 2752
<400> 2752
000
<210> 2753
<400> 2753
000
<210> 2754
<400> 2754
000
<210> 2755
<400> 2755
000
<210> 2756
<400> 2756
000
<210> 2757
<400> 2757
000
<210> 2758
<400> 2758
000
<210> 2759
<400> 2759
000
<210> 2760
<400> 2760
000
<210> 2761
<400> 2761
000
<210> 2762
<400> 2762
000
<210> 2763
<400> 2763
000
<210> 2764
<400> 2764
000
<210> 2765
<400> 2765
000
<210> 2766
<400> 2766
000
<210> 2767
<400> 2767
000
<210> 2768
<400> 2768
000
<210> 2769
<400> 2769
000
<210> 2770
<400> 2770
000
<210> 2771
<400> 2771
000
<210> 2772
<400> 2772
000
<210> 2773
<400> 2773
000
<210> 2774
<400> 2774
000
<210> 2775
<400> 2775
000
<210> 2776
<400> 2776
000
<210> 2777
<400> 2777
000
<210> 2778
<400> 2778
000
<210> 2779
<400> 2779
000
<210> 2780
<400> 2780
000
<210> 2781
<400> 2781
000
<210> 2782
<400> 2782
000
<210> 2783
<400> 2783
000
<210> 2784
<400> 2784
000
<210> 2785
<400> 2785
000
<210> 2786
<400> 2786
000
<210> 2787
<400> 2787
000
<210> 2788
<400> 2788
000
<210> 2789
<400> 2789
000
<210> 2790
<400> 2790
000
<210> 2791
<400> 2791
000
<210> 2792
<400> 2792
000
<210> 2793
<400> 2793
000
<210> 2794
<400> 2794
000
<210> 2795
<400> 2795
000
<210> 2796
<400> 2796
000
<210> 2797
<400> 2797
000
<210> 2798
<400> 2798
000
<210> 2799
<400> 2799
000
<210> 2800
<400> 2800
000
<210> 2801
<400> 2801
000
<210> 2802
<400> 2802
000
<210> 2803
<400> 2803
000
<210> 2804
<400> 2804
000
<210> 2805
<400> 2805
000
<210> 2806
<400> 2806
000
<210> 2807
<400> 2807
000
<210> 2808
<400> 2808
000
<210> 2809
<400> 2809
000
<210> 2810
<400> 2810
000
<210> 2811
<400> 2811
000
<210> 2812
<400> 2812
000
<210> 2813
<400> 2813
000
<210> 2814
<400> 2814
000
<210> 2815
<400> 2815
000
<210> 2816
<400> 2816
000
<210> 2817
<400> 2817
000
<210> 2818
<400> 2818
000
<210> 2819
<400> 2819
000
<210> 2820
<400> 2820
000
<210> 2821
<400> 2821
000
<210> 2822
<400> 2822
000
<210> 2823
<400> 2823
000
<210> 2824
<400> 2824
000
<210> 2825
<400> 2825
000
<210> 2826
<400> 2826
000
<210> 2827
<400> 2827
000
<210> 2828
<400> 2828
000
<210> 2829
<400> 2829
000
<210> 2830
<400> 2830
000
<210> 2831
<400> 2831
000
<210> 2832
<400> 2832
000
<210> 2833
<400> 2833
000
<210> 2834
<400> 2834
000
<210> 2835
<400> 2835
000
<210> 2836
<400> 2836
000
<210> 2837
<400> 2837
000
<210> 2838
<400> 2838
000
<210> 2839
<400> 2839
000
<210> 2840
<400> 2840
000
<210> 2841
<400> 2841
000
<210> 2842
<400> 2842
000
<210> 2843
<400> 2843
000
<210> 2844
<400> 2844
000
<210> 2845
<400> 2845
000
<210> 2846
<400> 2846
000
<210> 2847
<400> 2847
000
<210> 2848
<400> 2848
000
<210> 2849
<400> 2849
000
<210> 2850
<400> 2850
000
<210> 2851
<400> 2851
000
<210> 2852
<400> 2852
000
<210> 2853
<400> 2853
000
<210> 2854
<400> 2854
000
<210> 2855
<400> 2855
000
<210> 2856
<400> 2856
000
<210> 2857
<400> 2857
000
<210> 2858
<400> 2858
000
<210> 2859
<400> 2859
000
<210> 2860
<400> 2860
000
<210> 2861
<400> 2861
000
<210> 2862
<400> 2862
000
<210> 2863
<400> 2863
000
<210> 2864
<400> 2864
000
<210> 2865
<400> 2865
000
<210> 2866
<400> 2866
000
<210> 2867
<400> 2867
000
<210> 2868
<400> 2868
000
<210> 2869
<400> 2869
000
<210> 2870
<400> 2870
000
<210> 2871
<400> 2871
000
<210> 2872
<400> 2872
000
<210> 2873
<400> 2873
000
<210> 2874
<400> 2874
000
<210> 2875
<400> 2875
000
<210> 2876
<400> 2876
000
<210> 2877
<400> 2877
000
<210> 2878
<400> 2878
000
<210> 2879
<400> 2879
000
<210> 2880
<400> 2880
000
<210> 2881
<400> 2881
000
<210> 2882
<400> 2882
000
<210> 2883
<400> 2883
000
<210> 2884
<400> 2884
000
<210> 2885
<400> 2885
000
<210> 2886
<400> 2886
000
<210> 2887
<400> 2887
000
<210> 2888
<400> 2888
000
<210> 2889
<400> 2889
000
<210> 2890
<400> 2890
000
<210> 2891
<400> 2891
000
<210> 2892
<400> 2892
000
<210> 2893
<400> 2893
000
<210> 2894
<400> 2894
000
<210> 2895
<400> 2895
000
<210> 2896
<400> 2896
000
<210> 2897
<400> 2897
000
<210> 2898
<400> 2898
000
<210> 2899
<400> 2899
000
<210> 2900
<400> 2900
000
<210> 2901
<400> 2901
000
<210> 2902
<400> 2902
000
<210> 2903
<400> 2903
000
<210> 2904
<400> 2904
000
<210> 2905
<400> 2905
000
<210> 2906
<400> 2906
000
<210> 2907
<400> 2907
000
<210> 2908
<400> 2908
000
<210> 2909
<400> 2909
000
<210> 2910
<400> 2910
000
<210> 2911
<400> 2911
000
<210> 2912
<400> 2912
000
<210> 2913
<400> 2913
000
<210> 2914
<400> 2914
000
<210> 2915
<400> 2915
000
<210> 2916
<400> 2916
000
<210> 2917
<400> 2917
000
<210> 2918
<400> 2918
000
<210> 2919
<400> 2919
000
<210> 2920
<400> 2920
000
<210> 2921
<400> 2921
000
<210> 2922
<400> 2922
000
<210> 2923
<400> 2923
000
<210> 2924
<400> 2924
000
<210> 2925
<400> 2925
000
<210> 2926
<400> 2926
000
<210> 2927
<400> 2927
000
<210> 2928
<400> 2928
000
<210> 2929
<400> 2929
000
<210> 2930
<400> 2930
000
<210> 2931
<400> 2931
000
<210> 2932
<400> 2932
000
<210> 2933
<400> 2933
000
<210> 2934
<400> 2934
000
<210> 2935
<400> 2935
000
<210> 2936
<400> 2936
000
<210> 2937
<400> 2937
000
<210> 2938
<400> 2938
000
<210> 2939
<400> 2939
000
<210> 2940
<400> 2940
000
<210> 2941
<400> 2941
000
<210> 2942
<400> 2942
000
<210> 2943
<400> 2943
000
<210> 2944
<400> 2944
000
<210> 2945
<400> 2945
000
<210> 2946
<400> 2946
000
<210> 2947
<400> 2947
000
<210> 2948
<400> 2948
000
<210> 2949
<400> 2949
000
<210> 2950
<400> 2950
000
<210> 2951
<400> 2951
000
<210> 2952
<400> 2952
000
<210> 2953
<400> 2953
000
<210> 2954
<400> 2954
000
<210> 2955
<400> 2955
000
<210> 2956
<400> 2956
000
<210> 2957
<400> 2957
000
<210> 2958
<400> 2958
000
<210> 2959
<400> 2959
000
<210> 2960
<400> 2960
000
<210> 2961
<400> 2961
000
<210> 2962
<400> 2962
000
<210> 2963
<400> 2963
000
<210> 2964
<400> 2964
000
<210> 2965
<400> 2965
000
<210> 2966
<400> 2966
000
<210> 2967
<400> 2967
000
<210> 2968
<400> 2968
000
<210> 2969
<400> 2969
000
<210> 2970
<400> 2970
000
<210> 2971
<400> 2971
000
<210> 2972
<400> 2972
000
<210> 2973
<400> 2973
000
<210> 2974
<400> 2974
000
<210> 2975
<400> 2975
000
<210> 2976
<400> 2976
000
<210> 2977
<400> 2977
000
<210> 2978
<400> 2978
000
<210> 2979
<400> 2979
000
<210> 2980
<400> 2980
000
<210> 2981
<400> 2981
000
<210> 2982
<400> 2982
000
<210> 2983
<400> 2983
000
<210> 2984
<400> 2984
000
<210> 2985
<400> 2985
000
<210> 2986
<400> 2986
000
<210> 2987
<400> 2987
000
<210> 2988
<400> 2988
000
<210> 2989
<400> 2989
000
<210> 2990
<400> 2990
000
<210> 2991
<400> 2991
000
<210> 2992
<400> 2992
000
<210> 2993
<400> 2993
000
<210> 2994
<400> 2994
000
<210> 2995
<400> 2995
000
<210> 2996
<400> 2996
000
<210> 2997
<400> 2997
000
<210> 2998
<400> 2998
000
<210> 2999
<400> 2999
000
<210> 3000
<400> 3000
000
<210> 3001
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH CDR1
<400> 3001
Gly Thr Phe Ser Ser Ala Ala Ile Ser
1 5
<210> 3002
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH CDR2
<400> 3002
Gly Ile Phe Pro Ile Ser Gly His Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 3003
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH CDR3
<400> 3003
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
1 5 10 15
<210> 3004
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL CDR1
<400> 3004
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 3005
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL CDR2
<400> 3005
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 3006
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL CDR3
<400> 3006
Gln Gln Ser Asp Ile Leu Tyr Thr
1 5
<210> 3007
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH FR1
<400> 3007
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 3008
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH FR2
<400> 3008
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 3009
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH FR3
<400> 3009
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 3010
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH FR4
<400> 3010
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 3011
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH ДНК
<400> 3011
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agcgccgcta tcagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcttcccta tctccggtca cgcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagagacacg 300
ggacggggat acaccagaca cttctggttt gacccctggg gacagggtac attggtcacc 360
gtctcctca 369
<210> 3012
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VH Белка
<400> 3012
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ala
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Pro Ile Ser Gly His Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 3013
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL FR1
<400> 3013
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 3014
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL FR2
<400> 3014
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 3015
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL FR3
<400> 3015
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 3016
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL FR4
<400> 3016
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 3017
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL ДНК
<400> 3017
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agcgacatcc tctacacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 3018
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 34 - VL Белка
<400> 3018
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asp Ile Leu Tyr Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 3019
<400> 3019
000
<210> 3020
<400> 3020
000
<210> 3021
<400> 3021
000
<210> 3022
<400> 3022
000
<210> 3023
<400> 3023
000
<210> 3024
<400> 3024
000
<210> 3025
<400> 3025
000
<210> 3026
<400> 3026
000
<210> 3027
<400> 3027
000
<210> 3028
<400> 3028
000
<210> 3029
<400> 3029
000
<210> 3030
<400> 3030
000
<210> 3031
<400> 3031
000
<210> 3032
<400> 3032
000
<210> 3033
<400> 3033
000
<210> 3034
<400> 3034
000
<210> 3035
<400> 3035
000
<210> 3036
<400> 3036
000
<210> 3037
<400> 3037
000
<210> 3038
<400> 3038
000
<210> 3039
<400> 3039
000
<210> 3040
<400> 3040
000
<210> 3041
<400> 3041
000
<210> 3042
<400> 3042
000
<210> 3043
<400> 3043
000
<210> 3044
<400> 3044
000
<210> 3045
<400> 3045
000
<210> 3046
<400> 3046
000
<210> 3047
<400> 3047
000
<210> 3048
<400> 3048
000
<210> 3049
<400> 3049
000
<210> 3050
<400> 3050
000
<210> 3051
<400> 3051
000
<210> 3052
<400> 3052
000
<210> 3053
<400> 3053
000
<210> 3054
<400> 3054
000
<210> 3055
<400> 3055
000
<210> 3056
<400> 3056
000
<210> 3057
<400> 3057
000
<210> 3058
<400> 3058
000
<210> 3059
<400> 3059
000
<210> 3060
<400> 3060
000
<210> 3061
<400> 3061
000
<210> 3062
<400> 3062
000
<210> 3063
<400> 3063
000
<210> 3064
<400> 3064
000
<210> 3065
<400> 3065
000
<210> 3066
<400> 3066
000
<210> 3067
<400> 3067
000
<210> 3068
<400> 3068
000
<210> 3069
<400> 3069
000
<210> 3070
<400> 3070
000
<210> 3071
<400> 3071
000
<210> 3072
<400> 3072
000
<210> 3073
<400> 3073
000
<210> 3074
<400> 3074
000
<210> 3075
<400> 3075
000
<210> 3076
<400> 3076
000
<210> 3077
<400> 3077
000
<210> 3078
<400> 3078
000
<210> 3079
<400> 3079
000
<210> 3080
<400> 3080
000
<210> 3081
<400> 3081
000
<210> 3082
<400> 3082
000
<210> 3083
<400> 3083
000
<210> 3084
<400> 3084
000
<210> 3085
<400> 3085
000
<210> 3086
<400> 3086
000
<210> 3087
<400> 3087
000
<210> 3088
<400> 3088
000
<210> 3089
<400> 3089
000
<210> 3090
<400> 3090
000
<210> 3091
<400> 3091
000
<210> 3092
<400> 3092
000
<210> 3093
<400> 3093
000
<210> 3094
<400> 3094
000
<210> 3095
<400> 3095
000
<210> 3096
<400> 3096
000
<210> 3097
<400> 3097
000
<210> 3098
<400> 3098
000
<210> 3099
<400> 3099
000
<210> 3100
<400> 3100
000
<210> 3101
<400> 3101
000
<210> 3102
<400> 3102
000
<210> 3103
<400> 3103
000
<210> 3104
<400> 3104
000
<210> 3105
<400> 3105
000
<210> 3106
<400> 3106
000
<210> 3107
<400> 3107
000
<210> 3108
<400> 3108
000
<210> 3109
<400> 3109
000
<210> 3110
<400> 3110
000
<210> 3111
<400> 3111
000
<210> 3112
<400> 3112
000
<210> 3113
<400> 3113
000
<210> 3114
<400> 3114
000
<210> 3115
<400> 3115
000
<210> 3116
<400> 3116
000
<210> 3117
<400> 3117
000
<210> 3118
<400> 3118
000
<210> 3119
<400> 3119
000
<210> 3120
<400> 3120
000
<210> 3121
<400> 3121
000
<210> 3122
<400> 3122
000
<210> 3123
<400> 3123
000
<210> 3124
<400> 3124
000
<210> 3125
<400> 3125
000
<210> 3126
<400> 3126
000
<210> 3127
<400> 3127
000
<210> 3128
<400> 3128
000
<210> 3129
<400> 3129
000
<210> 3130
<400> 3130
000
<210> 3131
<400> 3131
000
<210> 3132
<400> 3132
000
<210> 3133
<400> 3133
000
<210> 3134
<400> 3134
000
<210> 3135
<400> 3135
000
<210> 3136
<400> 3136
000
<210> 3137
<400> 3137
000
<210> 3138
<400> 3138
000
<210> 3139
<400> 3139
000
<210> 3140
<400> 3140
000
<210> 3141
<400> 3141
000
<210> 3142
<400> 3142
000
<210> 3143
<400> 3143
000
<210> 3144
<400> 3144
000
<210> 3145
<400> 3145
000
<210> 3146
<400> 3146
000
<210> 3147
<400> 3147
000
<210> 3148
<400> 3148
000
<210> 3149
<400> 3149
000
<210> 3150
<400> 3150
000
<210> 3151
<400> 3151
000
<210> 3152
<400> 3152
000
<210> 3153
<400> 3153
000
<210> 3154
<400> 3154
000
<210> 3155
<400> 3155
000
<210> 3156
<400> 3156
000
<210> 3157
<400> 3157
000
<210> 3158
<400> 3158
000
<210> 3159
<400> 3159
000
<210> 3160
<400> 3160
000
<210> 3161
<400> 3161
000
<210> 3162
<400> 3162
000
<210> 3163
<400> 3163
000
<210> 3164
<400> 3164
000
<210> 3165
<400> 3165
000
<210> 3166
<400> 3166
000
<210> 3167
<400> 3167
000
<210> 3168
<400> 3168
000
<210> 3169
<400> 3169
000
<210> 3170
<400> 3170
000
<210> 3171
<400> 3171
000
<210> 3172
<400> 3172
000
<210> 3173
<400> 3173
000
<210> 3174
<400> 3174
000
<210> 3175
<400> 3175
000
<210> 3176
<400> 3176
000
<210> 3177
<400> 3177
000
<210> 3178
<400> 3178
000
<210> 3179
<400> 3179
000
<210> 3180
<400> 3180
000
<210> 3181
<400> 3181
000
<210> 3182
<400> 3182
000
<210> 3183
<400> 3183
000
<210> 3184
<400> 3184
000
<210> 3185
<400> 3185
000
<210> 3186
<400> 3186
000
<210> 3187
<400> 3187
000
<210> 3188
<400> 3188
000
<210> 3189
<400> 3189
000
<210> 3190
<400> 3190
000
<210> 3191
<400> 3191
000
<210> 3192
<400> 3192
000
<210> 3193
<400> 3193
000
<210> 3194
<400> 3194
000
<210> 3195
<400> 3195
000
<210> 3196
<400> 3196
000
<210> 3197
<400> 3197
000
<210> 3198
<400> 3198
000
<210> 3199
<400> 3199
000
<210> 3200
<400> 3200
000
<210> 3201
<400> 3201
000
<210> 3202
<400> 3202
000
<210> 3203
<400> 3203
000
<210> 3204
<400> 3204
000
<210> 3205
<400> 3205
000
<210> 3206
<400> 3206
000
<210> 3207
<400> 3207
000
<210> 3208
<400> 3208
000
<210> 3209
<400> 3209
000
<210> 3210
<400> 3210
000
<210> 3211
<400> 3211
000
<210> 3212
<400> 3212
000
<210> 3213
<400> 3213
000
<210> 3214
<400> 3214
000
<210> 3215
<400> 3215
000
<210> 3216
<400> 3216
000
<210> 3217
<400> 3217
000
<210> 3218
<400> 3218
000
<210> 3219
<400> 3219
000
<210> 3220
<400> 3220
000
<210> 3221
<400> 3221
000
<210> 3222
<400> 3222
000
<210> 3223
<400> 3223
000
<210> 3224
<400> 3224
000
<210> 3225
<400> 3225
000
<210> 3226
<400> 3226
000
<210> 3227
<400> 3227
000
<210> 3228
<400> 3228
000
<210> 3229
<400> 3229
000
<210> 3230
<400> 3230
000
<210> 3231
<400> 3231
000
<210> 3232
<400> 3232
000
<210> 3233
<400> 3233
000
<210> 3234
<400> 3234
000
<210> 3235
<400> 3235
000
<210> 3236
<400> 3236
000
<210> 3237
<400> 3237
000
<210> 3238
<400> 3238
000
<210> 3239
<400> 3239
000
<210> 3240
<400> 3240
000
<210> 3241
<400> 3241
000
<210> 3242
<400> 3242
000
<210> 3243
<400> 3243
000
<210> 3244
<400> 3244
000
<210> 3245
<400> 3245
000
<210> 3246
<400> 3246
000
<210> 3247
<400> 3247
000
<210> 3248
<400> 3248
000
<210> 3249
<400> 3249
000
<210> 3250
<400> 3250
000
<210> 3251
<400> 3251
000
<210> 3252
<400> 3252
000
<210> 3253
<400> 3253
000
<210> 3254
<400> 3254
000
<210> 3255
<400> 3255
000
<210> 3256
<400> 3256
000
<210> 3257
<400> 3257
000
<210> 3258
<400> 3258
000
<210> 3259
<400> 3259
000
<210> 3260
<400> 3260
000
<210> 3261
<400> 3261
000
<210> 3262
<400> 3262
000
<210> 3263
<400> 3263
000
<210> 3264
<400> 3264
000
<210> 3265
<400> 3265
000
<210> 3266
<400> 3266
000
<210> 3267
<400> 3267
000
<210> 3268
<400> 3268
000
<210> 3269
<400> 3269
000
<210> 3270
<400> 3270
000
<210> 3271
<400> 3271
000
<210> 3272
<400> 3272
000
<210> 3273
<400> 3273
000
<210> 3274
<400> 3274
000
<210> 3275
<400> 3275
000
<210> 3276
<400> 3276
000
<210> 3277
<400> 3277
000
<210> 3278
<400> 3278
000
<210> 3279
<400> 3279
000
<210> 3280
<400> 3280
000
<210> 3281
<400> 3281
000
<210> 3282
<400> 3282
000
<210> 3283
<400> 3283
000
<210> 3284
<400> 3284
000
<210> 3285
<400> 3285
000
<210> 3286
<400> 3286
000
<210> 3287
<400> 3287
000
<210> 3288
<400> 3288
000
<210> 3289
<400> 3289
000
<210> 3290
<400> 3290
000
<210> 3291
<400> 3291
000
<210> 3292
<400> 3292
000
<210> 3293
<400> 3293
000
<210> 3294
<400> 3294
000
<210> 3295
<400> 3295
000
<210> 3296
<400> 3296
000
<210> 3297
<400> 3297
000
<210> 3298
<400> 3298
000
<210> 3299
<400> 3299
000
<210> 3300
<400> 3300
000
<210> 3301
<400> 3301
000
<210> 3302
<400> 3302
000
<210> 3303
<400> 3303
000
<210> 3304
<400> 3304
000
<210> 3305
<400> 3305
000
<210> 3306
<400> 3306
000
<210> 3307
<400> 3307
000
<210> 3308
<400> 3308
000
<210> 3309
<400> 3309
000
<210> 3310
<400> 3310
000
<210> 3311
<400> 3311
000
<210> 3312
<400> 3312
000
<210> 3313
<400> 3313
000
<210> 3314
<400> 3314
000
<210> 3315
<400> 3315
000
<210> 3316
<400> 3316
000
<210> 3317
<400> 3317
000
<210> 3318
<400> 3318
000
<210> 3319
<400> 3319
000
<210> 3320
<400> 3320
000
<210> 3321
<400> 3321
000
<210> 3322
<400> 3322
000
<210> 3323
<400> 3323
000
<210> 3324
<400> 3324
000
<210> 3325
<400> 3325
000
<210> 3326
<400> 3326
000
<210> 3327
<400> 3327
000
<210> 3328
<400> 3328
000
<210> 3329
<400> 3329
000
<210> 3330
<400> 3330
000
<210> 3331
<400> 3331
000
<210> 3332
<400> 3332
000
<210> 3333
<400> 3333
000
<210> 3334
<400> 3334
000
<210> 3335
<400> 3335
000
<210> 3336
<400> 3336
000
<210> 3337
<400> 3337
000
<210> 3338
<400> 3338
000
<210> 3339
<400> 3339
000
<210> 3340
<400> 3340
000
<210> 3341
<400> 3341
000
<210> 3342
<400> 3342
000
<210> 3343
<400> 3343
000
<210> 3344
<400> 3344
000
<210> 3345
<400> 3345
000
<210> 3346
<400> 3346
000
<210> 3347
<400> 3347
000
<210> 3348
<400> 3348
000
<210> 3349
<400> 3349
000
<210> 3350
<400> 3350
000
<210> 3351
<400> 3351
000
<210> 3352
<400> 3352
000
<210> 3353
<400> 3353
000
<210> 3354
<400> 3354
000
<210> 3355
<400> 3355
000
<210> 3356
<400> 3356
000
<210> 3357
<400> 3357
000
<210> 3358
<400> 3358
000
<210> 3359
<400> 3359
000
<210> 3360
<400> 3360
000
<210> 3361
<400> 3361
000
<210> 3362
<400> 3362
000
<210> 3363
<400> 3363
000
<210> 3364
<400> 3364
000
<210> 3365
<400> 3365
000
<210> 3366
<400> 3366
000
<210> 3367
<400> 3367
000
<210> 3368
<400> 3368
000
<210> 3369
<400> 3369
000
<210> 3370
<400> 3370
000
<210> 3371
<400> 3371
000
<210> 3372
<400> 3372
000
<210> 3373
<400> 3373
000
<210> 3374
<400> 3374
000
<210> 3375
<400> 3375
000
<210> 3376
<400> 3376
000
<210> 3377
<400> 3377
000
<210> 3378
<400> 3378
000
<210> 3379
<400> 3379
000
<210> 3380
<400> 3380
000
<210> 3381
<400> 3381
000
<210> 3382
<400> 3382
000
<210> 3383
<400> 3383
000
<210> 3384
<400> 3384
000
<210> 3385
<400> 3385
000
<210> 3386
<400> 3386
000
<210> 3387
<400> 3387
000
<210> 3388
<400> 3388
000
<210> 3389
<400> 3389
000
<210> 3390
<400> 3390
000
<210> 3391
<400> 3391
000
<210> 3392
<400> 3392
000
<210> 3393
<400> 3393
000
<210> 3394
<400> 3394
000
<210> 3395
<400> 3395
000
<210> 3396
<400> 3396
000
<210> 3397
<400> 3397
000
<210> 3398
<400> 3398
000
<210> 3399
<400> 3399
000
<210> 3400
<400> 3400
000
<210> 3401
<400> 3401
000
<210> 3402
<400> 3402
000
<210> 3403
<400> 3403
000
<210> 3404
<400> 3404
000
<210> 3405
<400> 3405
000
<210> 3406
<400> 3406
000
<210> 3407
<400> 3407
000
<210> 3408
<400> 3408
000
<210> 3409
<400> 3409
000
<210> 3410
<400> 3410
000
<210> 3411
<400> 3411
000
<210> 3412
<400> 3412
000
<210> 3413
<400> 3413
000
<210> 3414
<400> 3414
000
<210> 3415
<400> 3415
000
<210> 3416
<400> 3416
000
<210> 3417
<400> 3417
000
<210> 3418
<400> 3418
000
<210> 3419
<400> 3419
000
<210> 3420
<400> 3420
000
<210> 3421
<400> 3421
000
<210> 3422
<400> 3422
000
<210> 3423
<400> 3423
000
<210> 3424
<400> 3424
000
<210> 3425
<400> 3425
000
<210> 3426
<400> 3426
000
<210> 3427
<400> 3427
000
<210> 3428
<400> 3428
000
<210> 3429
<400> 3429
000
<210> 3430
<400> 3430
000
<210> 3431
<400> 3431
000
<210> 3432
<400> 3432
000
<210> 3433
<400> 3433
000
<210> 3434
<400> 3434
000
<210> 3435
<400> 3435
000
<210> 3436
<400> 3436
000
<210> 3437
<400> 3437
000
<210> 3438
<400> 3438
000
<210> 3439
<400> 3439
000
<210> 3440
<400> 3440
000
<210> 3441
<400> 3441
000
<210> 3442
<400> 3442
000
<210> 3443
<400> 3443
000
<210> 3444
<400> 3444
000
<210> 3445
<400> 3445
000
<210> 3446
<400> 3446
000
<210> 3447
<400> 3447
000
<210> 3448
<400> 3448
000
<210> 3449
<400> 3449
000
<210> 3450
<400> 3450
000
<210> 3451
<400> 3451
000
<210> 3452
<400> 3452
000
<210> 3453
<400> 3453
000
<210> 3454
<400> 3454
000
<210> 3455
<400> 3455
000
<210> 3456
<400> 3456
000
<210> 3457
<400> 3457
000
<210> 3458
<400> 3458
000
<210> 3459
<400> 3459
000
<210> 3460
<400> 3460
000
<210> 3461
<400> 3461
000
<210> 3462
<400> 3462
000
<210> 3463
<400> 3463
000
<210> 3464
<400> 3464
000
<210> 3465
<400> 3465
000
<210> 3466
<400> 3466
000
<210> 3467
<400> 3467
000
<210> 3468
<400> 3468
000
<210> 3469
<400> 3469
000
<210> 3470
<400> 3470
000
<210> 3471
<400> 3471
000
<210> 3472
<400> 3472
000
<210> 3473
<400> 3473
000
<210> 3474
<400> 3474
000
<210> 3475
<400> 3475
000
<210> 3476
<400> 3476
000
<210> 3477
<400> 3477
000
<210> 3478
<400> 3478
000
<210> 3479
<400> 3479
000
<210> 3480
<400> 3480
000
<210> 3481
<400> 3481
000
<210> 3482
<400> 3482
000
<210> 3483
<400> 3483
000
<210> 3484
<400> 3484
000
<210> 3485
<400> 3485
000
<210> 3486
<400> 3486
000
<210> 3487
<400> 3487
000
<210> 3488
<400> 3488
000
<210> 3489
<400> 3489
000
<210> 3490
<400> 3490
000
<210> 3491
<400> 3491
000
<210> 3492
<400> 3492
000
<210> 3493
<400> 3493
000
<210> 3494
<400> 3494
000
<210> 3495
<400> 3495
000
<210> 3496
<400> 3496
000
<210> 3497
<400> 3497
000
<210> 3498
<400> 3498
000
<210> 3499
<400> 3499
000
<210> 3500
<400> 3500
000
<210> 3501
<400> 3501
000
<210> 3502
<400> 3502
000
<210> 3503
<400> 3503
000
<210> 3504
<400> 3504
000
<210> 3505
<400> 3505
000
<210> 3506
<400> 3506
000
<210> 3507
<400> 3507
000
<210> 3508
<400> 3508
000
<210> 3509
<400> 3509
000
<210> 3510
<400> 3510
000
<210> 3511
<400> 3511
000
<210> 3512
<400> 3512
000
<210> 3513
<400> 3513
000
<210> 3514
<400> 3514
000
<210> 3515
<400> 3515
000
<210> 3516
<400> 3516
000
<210> 3517
<400> 3517
000
<210> 3518
<400> 3518
000
<210> 3519
<400> 3519
000
<210> 3520
<400> 3520
000
<210> 3521
<400> 3521
000
<210> 3522
<400> 3522
000
<210> 3523
<400> 3523
000
<210> 3524
<400> 3524
000
<210> 3525
<400> 3525
000
<210> 3526
<400> 3526
000
<210> 3527
<400> 3527
000
<210> 3528
<400> 3528
000
<210> 3529
<400> 3529
000
<210> 3530
<400> 3530
000
<210> 3531
<400> 3531
000
<210> 3532
<400> 3532
000
<210> 3533
<400> 3533
000
<210> 3534
<400> 3534
000
<210> 3535
<400> 3535
000
<210> 3536
<400> 3536
000
<210> 3537
<400> 3537
000
<210> 3538
<400> 3538
000
<210> 3539
<400> 3539
000
<210> 3540
<400> 3540
000
<210> 3541
<400> 3541
000
<210> 3542
<400> 3542
000
<210> 3543
<400> 3543
000
<210> 3544
<400> 3544
000
<210> 3545
<400> 3545
000
<210> 3546
<400> 3546
000
<210> 3547
<400> 3547
000
<210> 3548
<400> 3548
000
<210> 3549
<400> 3549
000
<210> 3550
<400> 3550
000
<210> 3551
<400> 3551
000
<210> 3552
<400> 3552
000
<210> 3553
<400> 3553
000
<210> 3554
<400> 3554
000
<210> 3555
<400> 3555
000
<210> 3556
<400> 3556
000
<210> 3557
<400> 3557
000
<210> 3558
<400> 3558
000
<210> 3559
<400> 3559
000
<210> 3560
<400> 3560
000
<210> 3561
<400> 3561
000
<210> 3562
<400> 3562
000
<210> 3563
<400> 3563
000
<210> 3564
<400> 3564
000
<210> 3565
<400> 3565
000
<210> 3566
<400> 3566
000
<210> 3567
<400> 3567
000
<210> 3568
<400> 3568
000
<210> 3569
<400> 3569
000
<210> 3570
<400> 3570
000
<210> 3571
<400> 3571
000
<210> 3572
<400> 3572
000
<210> 3573
<400> 3573
000
<210> 3574
<400> 3574
000
<210> 3575
<400> 3575
000
<210> 3576
<400> 3576
000
<210> 3577
<400> 3577
000
<210> 3578
<400> 3578
000
<210> 3579
<400> 3579
000
<210> 3580
<400> 3580
000
<210> 3581
<400> 3581
000
<210> 3582
<400> 3582
000
<210> 3583
<400> 3583
000
<210> 3584
<400> 3584
000
<210> 3585
<400> 3585
000
<210> 3586
<400> 3586
000
<210> 3587
<400> 3587
000
<210> 3588
<400> 3588
000
<210> 3589
<400> 3589
000
<210> 3590
<400> 3590
000
<210> 3591
<400> 3591
000
<210> 3592
<400> 3592
000
<210> 3593
<400> 3593
000
<210> 3594
<400> 3594
000
<210> 3595
<400> 3595
000
<210> 3596
<400> 3596
000
<210> 3597
<400> 3597
000
<210> 3598
<400> 3598
000
<210> 3599
<400> 3599
000
<210> 3600
<400> 3600
000
<210> 3601
<400> 3601
000
<210> 3602
<400> 3602
000
<210> 3603
<400> 3603
000
<210> 3604
<400> 3604
000
<210> 3605
<400> 3605
000
<210> 3606
<400> 3606
000
<210> 3607
<400> 3607
000
<210> 3608
<400> 3608
000
<210> 3609
<400> 3609
000
<210> 3610
<400> 3610
000
<210> 3611
<400> 3611
000
<210> 3612
<400> 3612
000
<210> 3613
<400> 3613
000
<210> 3614
<400> 3614
000
<210> 3615
<400> 3615
000
<210> 3616
<400> 3616
000
<210> 3617
<400> 3617
000
<210> 3618
<400> 3618
000
<210> 3619
<400> 3619
000
<210> 3620
<400> 3620
000
<210> 3621
<400> 3621
000
<210> 3622
<400> 3622
000
<210> 3623
<400> 3623
000
<210> 3624
<400> 3624
000
<210> 3625
<400> 3625
000
<210> 3626
<400> 3626
000
<210> 3627
<400> 3627
000
<210> 3628
<400> 3628
000
<210> 3629
<400> 3629
000
<210> 3630
<400> 3630
000
<210> 3631
<400> 3631
000
<210> 3632
<400> 3632
000
<210> 3633
<400> 3633
000
<210> 3634
<400> 3634
000
<210> 3635
<400> 3635
000
<210> 3636
<400> 3636
000
<210> 3637
<400> 3637
000
<210> 3638
<400> 3638
000
<210> 3639
<400> 3639
000
<210> 3640
<400> 3640
000
<210> 3641
<400> 3641
000
<210> 3642
<400> 3642
000
<210> 3643
<400> 3643
000
<210> 3644
<400> 3644
000
<210> 3645
<400> 3645
000
<210> 3646
<400> 3646
000
<210> 3647
<400> 3647
000
<210> 3648
<400> 3648
000
<210> 3649
<400> 3649
000
<210> 3650
<400> 3650
000
<210> 3651
<400> 3651
000
<210> 3652
<400> 3652
000
<210> 3653
<400> 3653
000
<210> 3654
<400> 3654
000
<210> 3655
<400> 3655
000
<210> 3656
<400> 3656
000
<210> 3657
<400> 3657
000
<210> 3658
<400> 3658
000
<210> 3659
<400> 3659
000
<210> 3660
<400> 3660
000
<210> 3661
<400> 3661
000
<210> 3662
<400> 3662
000
<210> 3663
<400> 3663
000
<210> 3664
<400> 3664
000
<210> 3665
<400> 3665
000
<210> 3666
<400> 3666
000
<210> 3667
<400> 3667
000
<210> 3668
<400> 3668
000
<210> 3669
<400> 3669
000
<210> 3670
<400> 3670
000
<210> 3671
<400> 3671
000
<210> 3672
<400> 3672
000
<210> 3673
<400> 3673
000
<210> 3674
<400> 3674
000
<210> 3675
<400> 3675
000
<210> 3676
<400> 3676
000
<210> 3677
<400> 3677
000
<210> 3678
<400> 3678
000
<210> 3679
<400> 3679
000
<210> 3680
<400> 3680
000
<210> 3681
<400> 3681
000
<210> 3682
<400> 3682
000
<210> 3683
<400> 3683
000
<210> 3684
<400> 3684
000
<210> 3685
<400> 3685
000
<210> 3686
<400> 3686
000
<210> 3687
<400> 3687
000
<210> 3688
<400> 3688
000
<210> 3689
<400> 3689
000
<210> 3690
<400> 3690
000
<210> 3691
<400> 3691
000
<210> 3692
<400> 3692
000
<210> 3693
<400> 3693
000
<210> 3694
<400> 3694
000
<210> 3695
<400> 3695
000
<210> 3696
<400> 3696
000
<210> 3697
<400> 3697
000
<210> 3698
<400> 3698
000
<210> 3699
<400> 3699
000
<210> 3700
<400> 3700
000
<210> 3701
<400> 3701
000
<210> 3702
<400> 3702
000
<210> 3703
<400> 3703
000
<210> 3704
<400> 3704
000
<210> 3705
<400> 3705
000
<210> 3706
<400> 3706
000
<210> 3707
<400> 3707
000
<210> 3708
<400> 3708
000
<210> 3709
<400> 3709
000
<210> 3710
<400> 3710
000
<210> 3711
<400> 3711
000
<210> 3712
<400> 3712
000
<210> 3713
<400> 3713
000
<210> 3714
<400> 3714
000
<210> 3715
<400> 3715
000
<210> 3716
<400> 3716
000
<210> 3717
<400> 3717
000
<210> 3718
<400> 3718
000
<210> 3719
<400> 3719
000
<210> 3720
<400> 3720
000
<210> 3721
<400> 3721
000
<210> 3722
<400> 3722
000
<210> 3723
<400> 3723
000
<210> 3724
<400> 3724
000
<210> 3725
<400> 3725
000
<210> 3726
<400> 3726
000
<210> 3727
<400> 3727
000
<210> 3728
<400> 3728
000
<210> 3729
<400> 3729
000
<210> 3730
<400> 3730
000
<210> 3731
<400> 3731
000
<210> 3732
<400> 3732
000
<210> 3733
<400> 3733
000
<210> 3734
<400> 3734
000
<210> 3735
<400> 3735
000
<210> 3736
<400> 3736
000
<210> 3737
<400> 3737
000
<210> 3738
<400> 3738
000
<210> 3739
<400> 3739
000
<210> 3740
<400> 3740
000
<210> 3741
<400> 3741
000
<210> 3742
<400> 3742
000
<210> 3743
<400> 3743
000
<210> 3744
<400> 3744
000
<210> 3745
<400> 3745
000
<210> 3746
<400> 3746
000
<210> 3747
<400> 3747
000
<210> 3748
<400> 3748
000
<210> 3749
<400> 3749
000
<210> 3750
<400> 3750
000
<210> 3751
<400> 3751
000
<210> 3752
<400> 3752
000
<210> 3753
<400> 3753
000
<210> 3754
<400> 3754
000
<210> 3755
<400> 3755
000
<210> 3756
<400> 3756
000
<210> 3757
<400> 3757
000
<210> 3758
<400> 3758
000
<210> 3759
<400> 3759
000
<210> 3760
<400> 3760
000
<210> 3761
<400> 3761
000
<210> 3762
<400> 3762
000
<210> 3763
<400> 3763
000
<210> 3764
<400> 3764
000
<210> 3765
<400> 3765
000
<210> 3766
<400> 3766
000
<210> 3767
<400> 3767
000
<210> 3768
<400> 3768
000
<210> 3769
<400> 3769
000
<210> 3770
<400> 3770
000
<210> 3771
<400> 3771
000
<210> 3772
<400> 3772
000
<210> 3773
<400> 3773
000
<210> 3774
<400> 3774
000
<210> 3775
<400> 3775
000
<210> 3776
<400> 3776
000
<210> 3777
<400> 3777
000
<210> 3778
<400> 3778
000
<210> 3779
<400> 3779
000
<210> 3780
<400> 3780
000
<210> 3781
<400> 3781
000
<210> 3782
<400> 3782
000
<210> 3783
<400> 3783
000
<210> 3784
<400> 3784
000
<210> 3785
<400> 3785
000
<210> 3786
<400> 3786
000
<210> 3787
<400> 3787
000
<210> 3788
<400> 3788
000
<210> 3789
<400> 3789
000
<210> 3790
<400> 3790
000
<210> 3791
<400> 3791
000
<210> 3792
<400> 3792
000
<210> 3793
<400> 3793
000
<210> 3794
<400> 3794
000
<210> 3795
<400> 3795
000
<210> 3796
<400> 3796
000
<210> 3797
<400> 3797
000
<210> 3798
<400> 3798
000
<210> 3799
<400> 3799
000
<210> 3800
<400> 3800
000
<210> 3801
<400> 3801
000
<210> 3802
<400> 3802
000
<210> 3803
<400> 3803
000
<210> 3804
<400> 3804
000
<210> 3805
<400> 3805
000
<210> 3806
<400> 3806
000
<210> 3807
<400> 3807
000
<210> 3808
<400> 3808
000
<210> 3809
<400> 3809
000
<210> 3810
<400> 3810
000
<210> 3811
<400> 3811
000
<210> 3812
<400> 3812
000
<210> 3813
<400> 3813
000
<210> 3814
<400> 3814
000
<210> 3815
<400> 3815
000
<210> 3816
<400> 3816
000
<210> 3817
<400> 3817
000
<210> 3818
<400> 3818
000
<210> 3819
<400> 3819
000
<210> 3820
<400> 3820
000
<210> 3821
<400> 3821
000
<210> 3822
<400> 3822
000
<210> 3823
<400> 3823
000
<210> 3824
<400> 3824
000
<210> 3825
<400> 3825
000
<210> 3826
<400> 3826
000
<210> 3827
<400> 3827
000
<210> 3828
<400> 3828
000
<210> 3829
<400> 3829
000
<210> 3830
<400> 3830
000
<210> 3831
<400> 3831
000
<210> 3832
<400> 3832
000
<210> 3833
<400> 3833
000
<210> 3834
<400> 3834
000
<210> 3835
<400> 3835
000
<210> 3836
<400> 3836
000
<210> 3837
<400> 3837
000
<210> 3838
<400> 3838
000
<210> 3839
<400> 3839
000
<210> 3840
<400> 3840
000
<210> 3841
<400> 3841
000
<210> 3842
<400> 3842
000
<210> 3843
<400> 3843
000
<210> 3844
<400> 3844
000
<210> 3845
<400> 3845
000
<210> 3846
<400> 3846
000
<210> 3847
<400> 3847
000
<210> 3848
<400> 3848
000
<210> 3849
<400> 3849
000
<210> 3850
<400> 3850
000
<210> 3851
<400> 3851
000
<210> 3852
<400> 3852
000
<210> 3853
<400> 3853
000
<210> 3854
<400> 3854
000
<210> 3855
<400> 3855
000
<210> 3856
<400> 3856
000
<210> 3857
<400> 3857
000
<210> 3858
<400> 3858
000
<210> 3859
<400> 3859
000
<210> 3860
<400> 3860
000
<210> 3861
<400> 3861
000
<210> 3862
<400> 3862
000
<210> 3863
<400> 3863
000
<210> 3864
<400> 3864
000
<210> 3865
<400> 3865
000
<210> 3866
<400> 3866
000
<210> 3867
<400> 3867
000
<210> 3868
<400> 3868
000
<210> 3869
<400> 3869
000
<210> 3870
<400> 3870
000
<210> 3871
<400> 3871
000
<210> 3872
<400> 3872
000
<210> 3873
<400> 3873
000
<210> 3874
<400> 3874
000
<210> 3875
<400> 3875
000
<210> 3876
<400> 3876
000
<210> 3877
<400> 3877
000
<210> 3878
<400> 3878
000
<210> 3879
<400> 3879
000
<210> 3880
<400> 3880
000
<210> 3881
<400> 3881
000
<210> 3882
<400> 3882
000
<210> 3883
<400> 3883
000
<210> 3884
<400> 3884
000
<210> 3885
<400> 3885
000
<210> 3886
<400> 3886
000
<210> 3887
<400> 3887
000
<210> 3888
<400> 3888
000
<210> 3889
<400> 3889
000
<210> 3890
<400> 3890
000
<210> 3891
<400> 3891
000
<210> 3892
<400> 3892
000
<210> 3893
<400> 3893
000
<210> 3894
<400> 3894
000
<210> 3895
<400> 3895
000
<210> 3896
<400> 3896
000
<210> 3897
<400> 3897
000
<210> 3898
<400> 3898
000
<210> 3899
<400> 3899
000
<210> 3900
<400> 3900
000
<210> 3901
<400> 3901
000
<210> 3902
<400> 3902
000
<210> 3903
<400> 3903
000
<210> 3904
<400> 3904
000
<210> 3905
<400> 3905
000
<210> 3906
<400> 3906
000
<210> 3907
<400> 3907
000
<210> 3908
<400> 3908
000
<210> 3909
<400> 3909
000
<210> 3910
<400> 3910
000
<210> 3911
<400> 3911
000
<210> 3912
<400> 3912
000
<210> 3913
<400> 3913
000
<210> 3914
<400> 3914
000
<210> 3915
<400> 3915
000
<210> 3916
<400> 3916
000
<210> 3917
<400> 3917
000
<210> 3918
<400> 3918
000
<210> 3919
<400> 3919
000
<210> 3920
<400> 3920
000
<210> 3921
<400> 3921
000
<210> 3922
<400> 3922
000
<210> 3923
<400> 3923
000
<210> 3924
<400> 3924
000
<210> 3925
<400> 3925
000
<210> 3926
<400> 3926
000
<210> 3927
<400> 3927
000
<210> 3928
<400> 3928
000
<210> 3929
<400> 3929
000
<210> 3930
<400> 3930
000
<210> 3931
<400> 3931
000
<210> 3932
<400> 3932
000
<210> 3933
<400> 3933
000
<210> 3934
<400> 3934
000
<210> 3935
<400> 3935
000
<210> 3936
<400> 3936
000
<210> 3937
<400> 3937
000
<210> 3938
<400> 3938
000
<210> 3939
<400> 3939
000
<210> 3940
<400> 3940
000
<210> 3941
<400> 3941
000
<210> 3942
<400> 3942
000
<210> 3943
<400> 3943
000
<210> 3944
<400> 3944
000
<210> 3945
<400> 3945
000
<210> 3946
<400> 3946
000
<210> 3947
<400> 3947
000
<210> 3948
<400> 3948
000
<210> 3949
<400> 3949
000
<210> 3950
<400> 3950
000
<210> 3951
<400> 3951
000
<210> 3952
<400> 3952
000
<210> 3953
<400> 3953
000
<210> 3954
<400> 3954
000
<210> 3955
<400> 3955
000
<210> 3956
<400> 3956
000
<210> 3957
<400> 3957
000
<210> 3958
<400> 3958
000
<210> 3959
<400> 3959
000
<210> 3960
<400> 3960
000
<210> 3961
<400> 3961
000
<210> 3962
<400> 3962
000
<210> 3963
<400> 3963
000
<210> 3964
<400> 3964
000
<210> 3965
<400> 3965
000
<210> 3966
<400> 3966
000
<210> 3967
<400> 3967
000
<210> 3968
<400> 3968
000
<210> 3969
<400> 3969
000
<210> 3970
<400> 3970
000
<210> 3971
<400> 3971
000
<210> 3972
<400> 3972
000
<210> 3973
<400> 3973
000
<210> 3974
<400> 3974
000
<210> 3975
<400> 3975
000
<210> 3976
<400> 3976
000
<210> 3977
<400> 3977
000
<210> 3978
<400> 3978
000
<210> 3979
<400> 3979
000
<210> 3980
<400> 3980
000
<210> 3981
<400> 3981
000
<210> 3982
<400> 3982
000
<210> 3983
<400> 3983
000
<210> 3984
<400> 3984
000
<210> 3985
<400> 3985
000
<210> 3986
<400> 3986
000
<210> 3987
<400> 3987
000
<210> 3988
<400> 3988
000
<210> 3989
<400> 3989
000
<210> 3990
<400> 3990
000
<210> 3991
<400> 3991
000
<210> 3992
<400> 3992
000
<210> 3993
<400> 3993
000
<210> 3994
<400> 3994
000
<210> 3995
<400> 3995
000
<210> 3996
<400> 3996
000
<210> 3997
<400> 3997
000
<210> 3998
<400> 3998
000
<210> 3999
<400> 3999
000
<210> 4000
<400> 4000
000
<210> 4001
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH CDR1
<400> 4001
Gly Thr Phe Ala Thr Tyr Ala Ile Ser
1 5
<210> 4002
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH CDR2
<400> 4002
Gly Ile Phe Pro Leu Ser Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 4003
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH CDR3
<400> 4003
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
1 5 10 15
<210> 4004
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL CDR1
<400> 4004
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 4005
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL CDR2
<400> 4005
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 4006
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL CDR3
<400> 4006
Gln Gln Ser Asp Ile Leu Tyr Thr
1 5
<210> 4007
<211> 26
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH FR1
<400> 4007
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly
20 25
<210> 4008
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH FR2
<400> 4008
Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10
<210> 4009
<211> 30
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH FR3
<400> 4009
Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 4010
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH FR4
<400> 4010
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 4011
<211> 369
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH ДНК
<400> 4011
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcgca acctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcttccctc tctccggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggcggtgt actactgcgc cagagacacg 300
ggacggggat acaccagaca cttctggttt gacccctggg gacagggtac attggtcacc 360
gtctcctca 369
<210> 4012
<211> 123
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VH Белка
<400> 4012
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ala Thr Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Phe Pro Leu Ser Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Thr Gly Arg Gly Tyr Thr Arg His Phe Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 4013
<211> 23
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL FR1
<400> 4013
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 4014
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL FR2
<400> 4014
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 4015
<211> 32
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL FR3
<400> 4015
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 4016
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL FR4
<400> 4016
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 4017
<211> 318
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL ДНК
<400> 4017
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcagcaa agcgacatcc tctacacttt tggcggaggg 300
accaaggttg agatcaaa 318
<210> 4018
<211> 106
<212> Белок
<213> Искусственная последовательность
<220>
<223> синтетический: 36 - VL Белка
<400> 4018
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Asp Ile Leu Tyr Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИТЕЛА К C10ORF54 И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2714232C2 |
| УСОВЕРШЕНСТВОВАННЫЕ ПРОКОАГУЛЯНТНЫЕ АНТИТЕЛА | 2019 |
|
RU2810748C2 |
| АНТИТЕЛА К C10ORF54 И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2819627C1 |
| АНТИТЕЛА ПРОТИВ СИГНАЛ-РЕГУЛЯТОРНОГО БЕЛКА АЛЬФА И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2771964C2 |
| Композиции и способы активации интегринов | 2020 |
|
RU2836262C1 |
| МОЛЕКУЛЫ АНТИТЕЛ ПРОТИВ ВИРУСА BK | 2021 |
|
RU2835801C1 |
| CD131-СВЯЗЫВАЮЩИЕ БЕЛКИ И ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2773927C2 |
| ТЕРАПЕВТИЧЕСКИЕ АНТИТЕЛА ПРОТИВ ЛИГАНДА CD40 | 2018 |
|
RU2770209C2 |
| АНТИТЕЛА К PD-1 И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2761640C2 |
| АНТИТЕЛА ПРОТИВ TNFRSF9 И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2812200C2 |

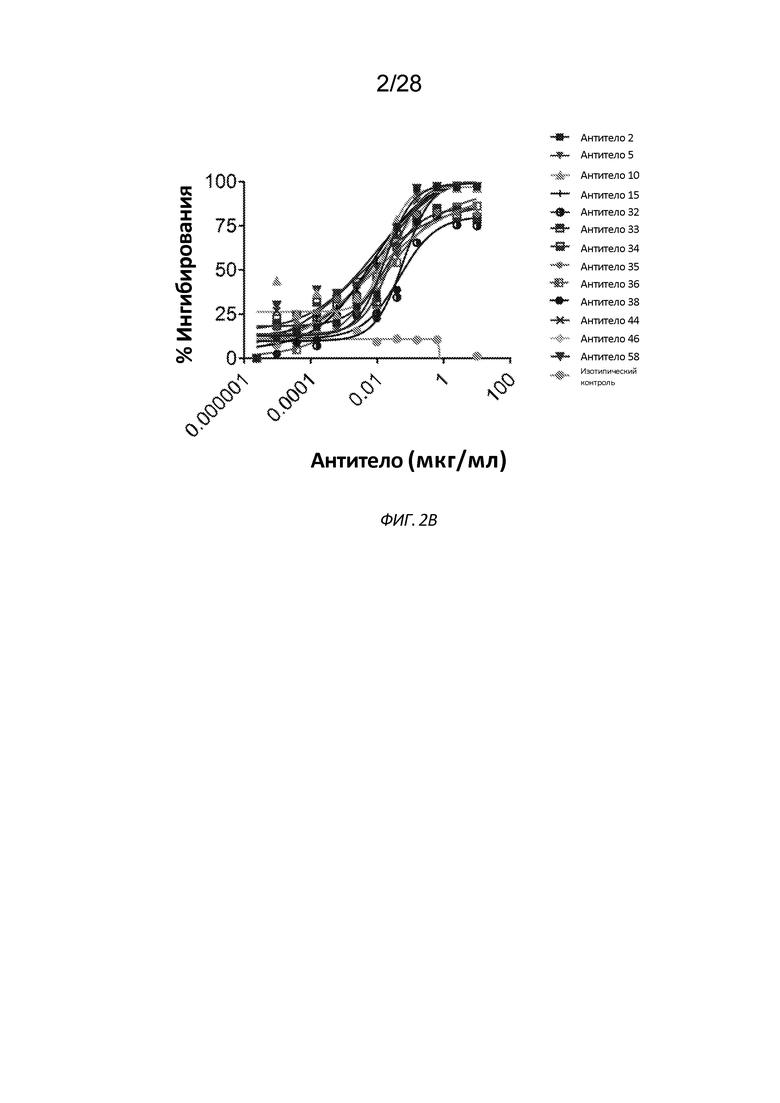





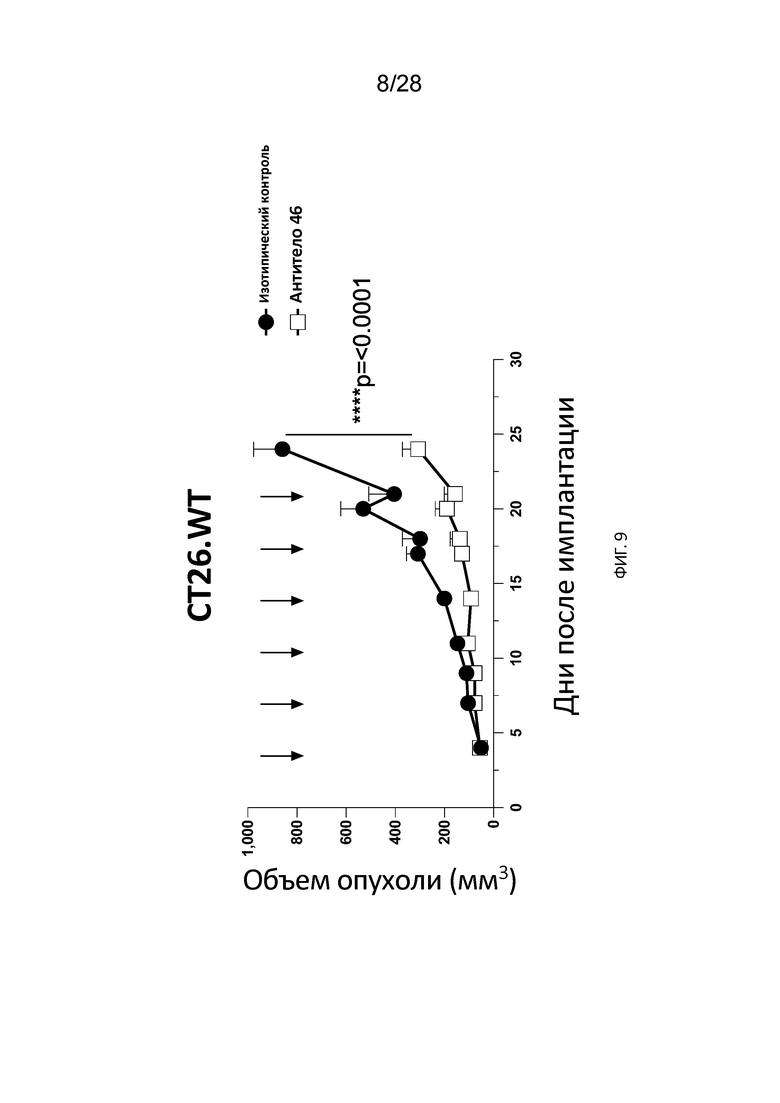


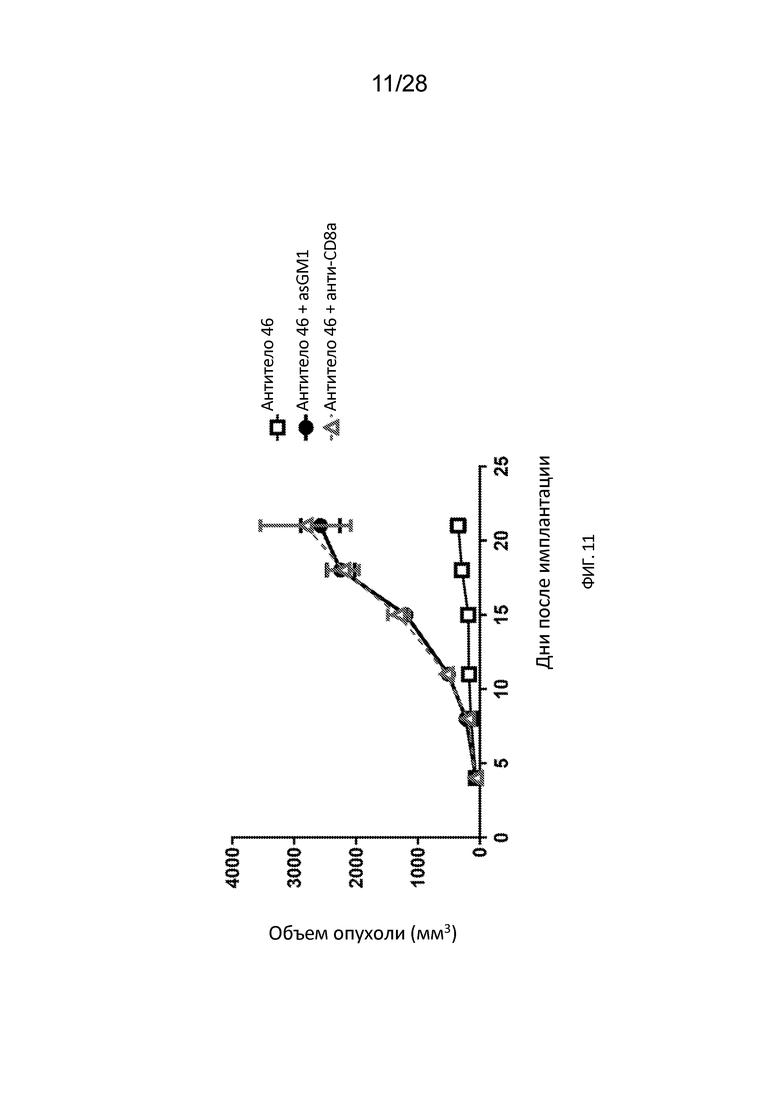
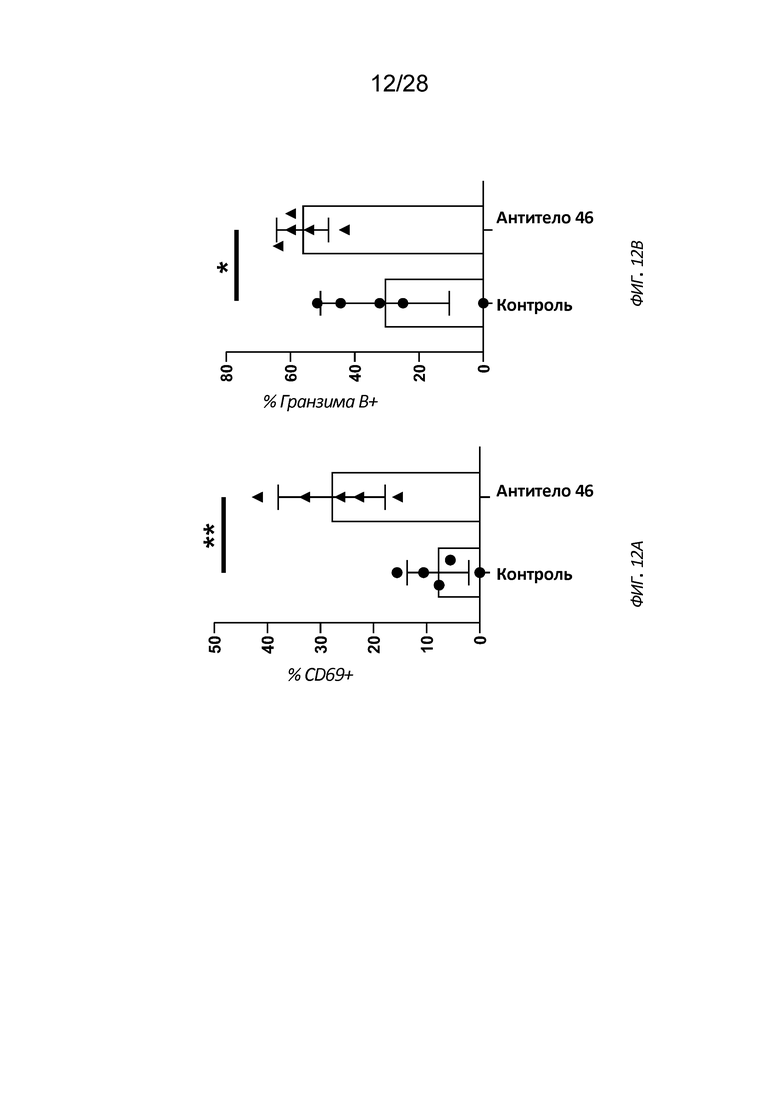

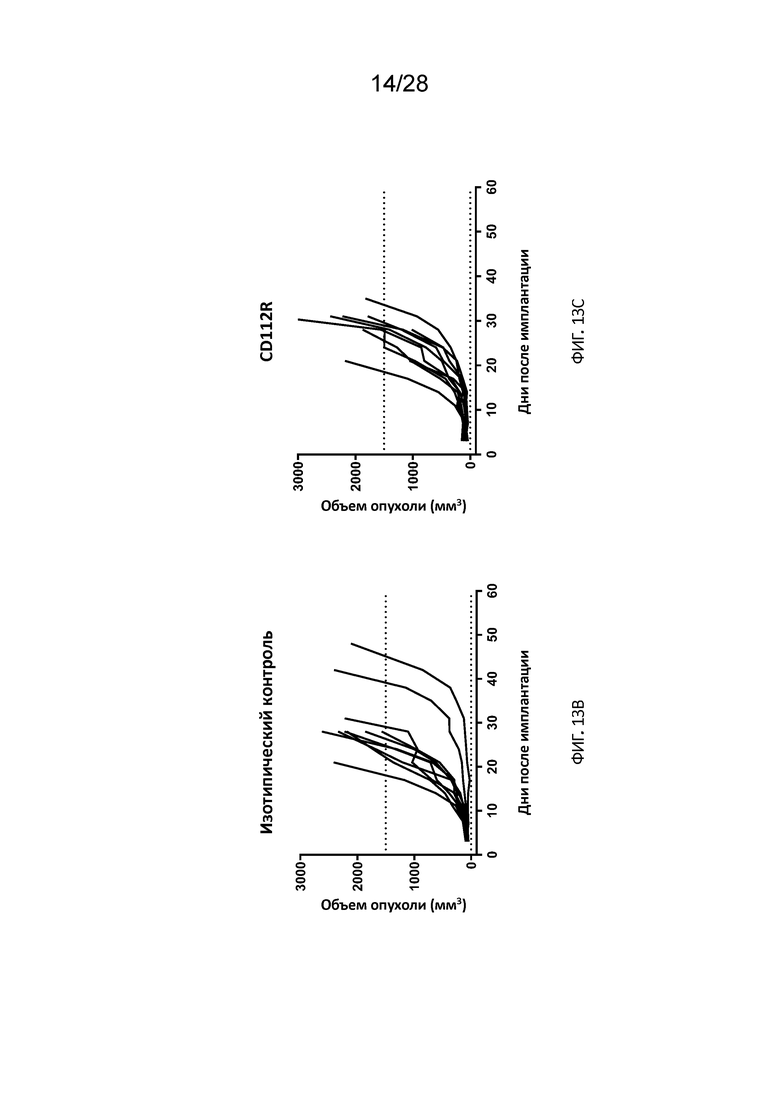
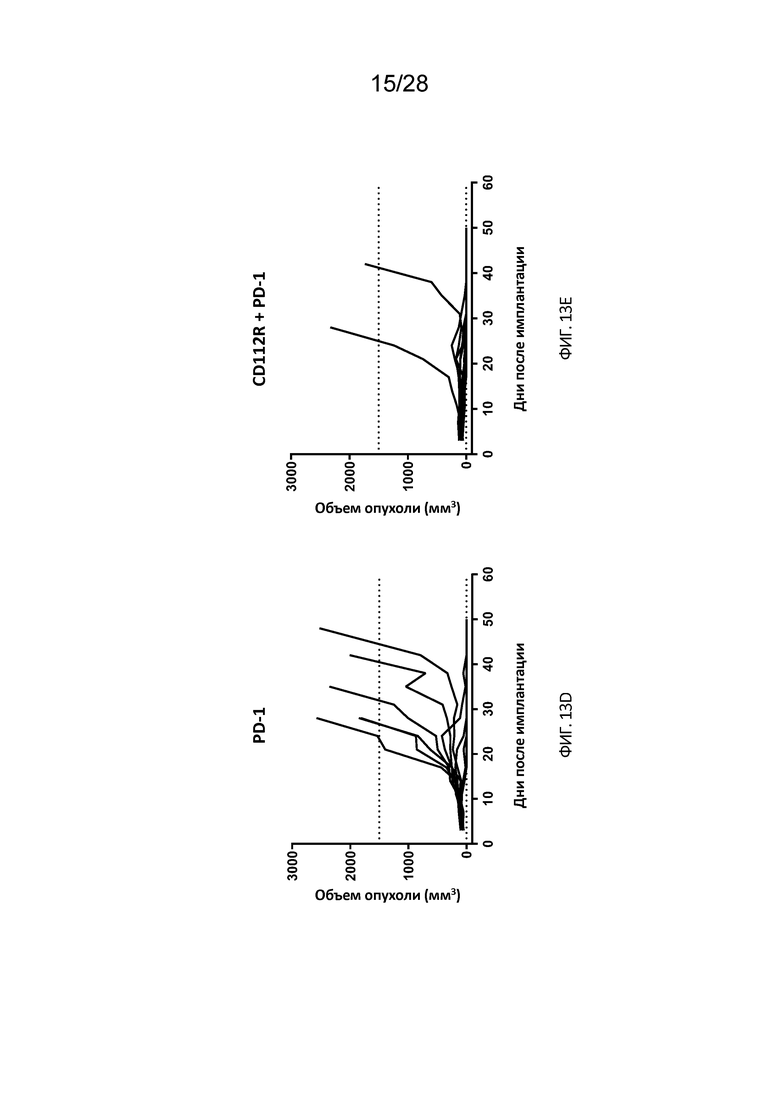













Изобретение относится к области биохимии, в частности к выделенному антителу анти-CD112R или его фрагменту. Также раскрыта композиция, содержащая указанное антитело; нуклеиновая кислота, кодирующая указанное антитело; клетка-хозяин, содержащая указанное антитело. Раскрыты способ усиления, повышения и/или поддержания противоопухолевого иммунного ответа у субъекта, с помощью указанного антитела, способ усиления активации CD8 Т-клеток или NK-клеток у субъекта, с помощью указанного антитела, способ усиления продукции гамма-интерферона CD8 Т-клетками, с помощью указанного антитела, способ усиления цитотоксичности, с помощью указанного антитела, способ усиления взаимодействий CD226 с CD112 у субъекта, с помощью указанного антитела, способ получения указанного антитела. Изобретение обладает способностью эффективно лечить заболевания, экспрессией CD112. 13 н. и 27 з.п. ф-лы, 19 ил., 12 табл., 17 пр.
1. Выделенное анти-CD112R антитело или его фрагмент, содержащие вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL), где VH содержит аминокислотную последовательность SEQ ID NO: 712 и VL содержит аминокислотную последовательность SEQ ID NO: 718.
2. Выделенное антитело или его фрагмент по п. 1, где указанное выделенное антитело представляет собой моноклональное антитело.
3. Выделенное антитело или его фрагмент по п. 1 или 2, где указанное выделенное антитело представляет собой фрагмент антитела.
4. Выделенное антитело или его фрагмент по п. 3, где указанный фрагмент представляет собой Fab, Fab’, Fv, scFv или (Fab’)2.
5. Выделенное антитело или его фрагмент по п. 1 или 2, где указанное выделенное антитело представляет собой полноразмерное антитело.
6. Выделенное антитело или его фрагмент по п. 5, где Fc-область указанного выделенного антитела содержит IgG1, IgG2, IgG3 или IgG4.
7. Выделенное антитело или его фрагмент по любому из пп. 1-6, где указанное выделенное антитело содержит константную область тяжелой цепи IgG1 человека.
8. Выделенное антитело или его фрагмент по любому из пп. 1-6, где указанное выделенное антитело содержит константную область тяжелой цепи IgG4 человека.
9. Выделенное антитело или его фрагмент по п. 8, где указанное выделенное антитело содержит мутантную константную область тяжелой цепи IgG4 человека.
10. Выделенное антитело или его фрагмент по п. 9, где указанная мутантная константная область тяжелой цепи IgG4 содержит мутацию, выбранную из замены в Ser228, замены в Leu235, замены в Asn297 или их комбинации, нумерация в соответствии с нумерацией EU.
11. Выделенное антитело или его фрагмент по п. 9, где указанная мутантная константная область тяжелой цепи IgG4 содержит замену S228P и замену L235E, нумерация в соответствии с нумерацией EU.
12. Выделенное антитело или его фрагмент по любому из пп. 1-7, где указанное выделенное антитело содержит константную область тяжелой цепи IgG1 человека, и при этом указанное выделенное антитело
i) увеличивает дегрануляцию NK-клеток; и/или
ii) увеличивает активацию NK-клеток; и/или
iii) увеличивает активацию внутриопухолевых NK-клеток при представлении в комбинации с анти-TIGIT антителом; и/или
iv) подавляет рост опухоли in vivo; и/или
v) предотвращает приживление опухоли при повторном заражении опухолью.
13. Выделенное антитело или его фрагмент по любому из предшествующих пунктов, где указанное антитело связывается с CD112R человека.
14. Полностью человеческое выделенное анти-CD112R антитело или его фрагмент по любому из предшествующих пунктов.
15. Композиция для лечения рака, при котором опухолевые клетки и/или миелоидные клетки в микроокружении опухоли экспрессируют CD112, содержащая выделенное антитело или его фрагмент по любому из пп. 1-14 и фармацевтически приемлемый носитель.
16. Композиция по п. 15, дополнительно содержащая антагонист PD-1, PD-L1, CTLA-4, Lag-3, TIGIT, CD96 или TIM-3.
17. Способ усиления, повышения и/или поддержания противоопухолевого иммунного ответа у субъекта, включающий введение субъекту, имеющему опухоль, антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16.
18. Способ усиления активации CD8 Т-клеток у субъекта, включающий введение антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16 субъекту, нуждающемуся в активации CD8 Т-клеток.
19. Способ усиления продукции гамма-интерферона CD8 Т-клетками у субъекта, включающий введение антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16 субъекту, нуждающемуся в продукции гамма-интерферона CD8 Т-клетками.
20. Способ усиления активации NK-клеток у субъекта, включающий введение антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16 субъекту, нуждающемуся в активации NK-клеток.
21. Способ усиления цитотоксичности, опосредованной NK-клетками, у субъекта, включающий введение антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16 субъекту, нуждающемуся в повышении цитотоксичности, опосредованной NK-клетками.
22. Способ лечения рака у субъекта, при котором опухолевые клетки и/или миелоидные клетки в микроокружении опухоли экспрессируют CD112, включающий введение антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16 субъекту, имеющему рак.
23. Способ по п. 22, где указанный рак представляет собой карциному, лимфому, бластому, саркому или лейкоз.
24. Способ по п. 22, где указанный рак представляет собой плоскоклеточный рак, мелкоклеточный рак легкого, рак гипофиза, рак пищевода, астроцитому, саркому мягких тканей, немелкоклеточный рак легкого (в том числе плоскоклеточный немелкоклеточный рак легкого), аденокарциному легкого, плоскоклеточную карциному легкого, рак брюшины, гепатоцеллюлярный рак, рак желудочно-кишечного тракта, рак поджелудочной железы, глиобластому, рак шейки матки, рак яичников, рак печени, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки, рак толстой и прямой кишок, карциному эндометрия или матки, карциному слюнной железы, рак почки, почечно-клеточную карциному, рак печени, рак предстательной железы, рак влагалища, рак щитовидной железы, карциному печени, рак головного мозга, рак эндометрия, рак яичка, холангиокарциному, рак желчного пузыря, рак желудка, меланому или различные типы рака головы и шеи (в том числе плоскоклеточную карциному головы и шеи).
25. Способ усиления взаимодействий CD226 с CD112 у субъекта, включающий введение субъекту антитела или его фрагмента по любому из пп. 1-14 или композиции по п. 15 или 16.
26. Способ по любому из пп. 17-25, где указанный способ дополнительно включает применение второй терапии.
27. Способ по п. 26, где указанная вторая терапия представляет собой лучевую терапию или хирургическое вмешательство.
28. Способ по п. 26, где указанная вторая терапия представляет собой применение химиотерапии, опсонизирующего агента или агента, истощающего популяцию регуляторных Т-клеток.
29. Способ по п. 26, где указанная вторая терапия представляет собой введение антагониста PD-1, PD-L1, CTLA-4, Lag-3 или TIM-3.
30. Способ по п. 26, где указанная вторая терапия представляет собой введение антагониста TIGIT или CD96.
31. Способ по п. 26, где указанная вторая терапия представляет собой введение антагониста PVRL1, PVRL2, PVRL3, PVRL4 и CD155.
32. Способ по п. 26, где указанная вторая терапия представляет собой введение антагониста CD47.
33. Способ по п. 26, где указанная вторая терапия представляет собой введение антагониста CD39.
34. Способ по п. 26, где указанная вторая терапия представляет собой введение антагониста IL-27.
35. Способ по п. 26, где указанная вторая терапия представляет собой введение агониста STING.
36. Способ по любому из пп. 29-34, где указанный антагонист представляет собой антитело.
37. Нуклеиновая кислота, кодирующая выделенное антитело или его фрагмент по любому из пп. 1-14.
38. Клетка-хозяин для получения анти-CD112R антитела или его фрагмента, содержащая нуклеиновую кислоту по п. 37.
39. Способ получения выделенного антитела или его фрагмента по любому из пп. 1-14, включающий культивирование клетки-хозяина по п. 38 в условиях, при которых экспрессируется указанное выделенное антитело или его фрагмент.
40. Способ по п. 39, дополнительно включающий очистку указанного выделенного антитела или его фрагмента.
| ОДНОЦЕПОЧЕЧНЫЕ МУЛЬТИВАЛЕНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ЭФФЕКТОРНОЙ ФУНКЦИЕЙ | 2007 |
|
RU2487888C2 |
| WO 2017221255 A1, 28.12.2017 | |||
| US 20170240613 A1, 24.08.2017. | |||
Авторы
Даты
2024-05-22—Публикация
2019-07-19—Подача