Изобретение относится к области медицины, вирусологии, микробиологии, молекулярной генетики и биотехнологии и может быть использовано в медицинской промышленности при производстве живых вакцин для профилактики и лечения инфекций, вызванных коронавирусом.
Коронавирусы (Coronaviridae)- семейство вирусов, включающее на январь 2020 года более 60 видов РНК-содержащих патогенных вирусов, объединенных в четыре подсемейства, которые поражают человека и животных. Название связано со строением вируса, который имеет шиловидные отростки. Они напоминают солнечную корону и содержат рецептор-связывающие белки, на которые реагируют трансмембранные рецепторы клеток. Коронавирусы имеют сходную организацию генома, представленного цепочкой из положительной цепи однонитевой РНК из 25-30 тысяч нуклеотидов.
Коронавирусы имеют глобальное распространение и поражают множество людей и животных-хозяев, вызывая заболевания, которые варьируются от инфекций верхних дыхательных путей у людей до инфекций желудочно-кишечного тракта, энцефалита и демиелинизации у животных; и могут быть смертельными. Международный комитет по таксономии вирусов (ICTV) сообщает о четырех родах коронавирусов, а именно альфакоронавирусах, бетакоронавирусах, гаммакоронавирусах и дельтакоронавирусах.
CoV представляют собой оболочечные одноцепочечные РНК-вирусы позитивного смысла с геномами от 26,2 до 31,7 кб, что является самым большим среди известных РНК-вирусов [Лай М.М., Кавана Д. Молекулярная биология коронавирусов. Adv. Virus Res. 1997;48:1-100]. Этот большой, закрытый и полиаденилированный геном содержит семь распространенных генов коронавируса в следующем консервативном порядке: 5'-ORF1a-ORF1b-S-ORF3-E-M-N-3' [Krzysztof Pyre, Ben Berkhout, and Lia van der Hoek. The Novel Human Coronaviruses NL63 andJ Virol., 2007 81(7): 3051-3057. doi: 10.1128/JVI.01466-06].
ORF1a/b охватывает две трети генома и продуцирует мРНК длиной в геном (mRNAl), которая кодирует два перекрывающихся вирусных репликазных белка в форме полипротеинов 1a (pp1a) и pp1ab [Fang S.G., Shen Н., Wang J., Tay F.P., Liu D.X. Proteolytic processing of polyproteins 1a and 1ab between non-structural proteins 10 and 11/12 of Coronavirus infectious bronchitis virus is dispensable for viral replication in cultured cells, Virology, 2008, 379(2): 175-180. doi: 10.1016/j.virol.2008.06.038].
Эти полипротеины образуются в результате сдвига рибосомального каркаса -1, который включает сложную структуру псевдокнотовой РНК [Брайерли И., Бурснелл М.Э., Биннс М.М., Билимория Б., Блок B.C., Браун Т.Д., Инглис С.С. Эффективный рибосомный сигнал сдвига кадров в кодирующей полимеразу области IBV коронавируса. EMBO J. 1987; 6:3779-3785], а затем протеолитически обрабатываются вирусно кодируемыми протеазами в зрелые неструктурные белки (от nsp1 до nsp16). Последняя треть генома продуцирует субгеномные (sg) мРНК, которые кодируют четыре структурных белка: спайк (S), оболочку (Е), мембрану (М) и нуклеокапсид (N), а также ряд вспомогательных белков.
Нуклеокапсидный (N) белок коронавируса с тяжелым острым респираторным синдромом (SARS-CoV) является наиболее распространенным белком в инфицированных вирусом клетках. Его основная функция заключается в упаковке молекулы РНК вирусного генома с положительной цепью длиной около 30 кб с 5'-шапкой в рибонуклеопротеиновый (RNP) комплекс, называемый капсидом. Упаковка рибонуклеокапсида является фундаментальной частью вирусной самосборки, а комплекс RNP представляет собой необходимую матрицу для репликации с помощью РНК-зависимого комплекса РНК-полимеразы. Кроме того, было показано, что N-белок SARS-CoV модулирует клеточный механизм хозяина и может выполнять регуляторные функции в течение жизненного цикла вируса [Ababou and Ladbury, 2007, Hsieh et al., 2005, Surjit et al., 2006].
До вспышки SARS структура N-белков коронавируса никогда не изучалась подробно. Самая ранняя структурная модель N-белка коронавируса была предложена Паркером и Мастерсом [Masters, (2006)]. В их модели белок N состоял из трех доменов, разделенных двумя спейсерами. Центральный домен действовал как РНК-связывающий домен, тогда как остальные два кислых домена, по-видимому, играли роль во взаимодействиях белок-белок. Хотя в то время модель давала общий обзор структуры белка N коронавируса, в ней отсутствовали необходимые детали, чтобы дать четкое представление о взаимосвязи структуры и функции белка. Пандемия SARS открыла новую эру структурных исследований структуры белка коронавируса. Белок N коронавируса представляет собой фосфопротеин массой 46 кДа из 422 аминокислот, идентичность последовательности которого составляет 20-30% с N-белками других коронавирусов.
Сравнение аминокислотных последовательностей показало, что N-белки коронавируса имеют три отдельных и высококонсервативных домена: две структурные и независимо свернутые структурные области, а именно N-концевой домен (NTD / домен 1) и С-концевой домен (CTD / домен 3), которые разделены внутренней неупорядоченной центральной областью (РНК-связывающий домен /домен 2). Было показано, что все три домена в разных CoV связываются с вирусной РНК [Хуан К., ЮЛ., Петрос A.M., Гунасекера А., Лю 3., Сюй Н., Хайдук П., Мак Дж., Фесик С.В., Олейничак Э.Т. Structure of the N-terminal RNA-binding domain of the SARS CoV nucleocapsid protein, Biochemistry, 2004; 43(20):6059-63, doi: 10.1021/bi036155b].
Из четырех основных структурных белков N-белок наиболее высоко экспрессируется в клетках-хозяевах. [Чанг, Чунг-ке; Хоу, Мин-Хон; Чанг, Чи-Фон; Сяо, Чван-Дэн; Хуан, Тай-хуан The SARS coronavirus nucleocapsid protein - Forms and functionsdoi: Antiviral Res. 2014 Mar; 103: 39-50. doi: 10.1016/j.antiviral. 2013.12.009]. Как и другие структурные белки, ген, кодирующий N-белок, расположен ближе к 3'-концу генома [Марджори; Филдинг, Бертрам, 2014. Вирусы. 6 (8): 2991-3018. doi: 10.3390/v608299].
N-белок локализуется преимущественно в цитоплазме. У многих коронавирусов популяция белка N локализована в ядрышке [Paul S. Masters, The molecular biology of coronaviruses Adv Virus Res. 2006; 66: 193-292. doi: 10.1016/S0065-3527(06)66005-3]. Считается, что это связано с его влиянием на клеточный цикл [Су, Минцзюнь; Чен, Япин; Ци, Шаньшань; Ши, Да; Фенг, Ли; Сунь, Дунбо, A Mini-Review on Cell Cycle Regulation of Coronavirus Infection, Front Vet Sci. 2020; 7: 586826, doi: 10.3389/fvets.2020.586826].
Свойства N-белка, заключающиеся в том, что он хорошо сохраняется, не часто рекомбинирует и вызывает сильный Т-клеточный ответ, привели к тому, что он был изучен в качестве потенциальной мишени для коронавирусных вакцин. [Alba Grifoni,1 Daniela Weiskopf,1 Sydney I. Ramirez, 1,2 Jose Mateus, «Targets of T Cell Responses to SARS-CoV-2 Coronavirus in Humans with COVID-19 Disease and Unexposed Individuals)), Cell. 181 (7): 1489-1501.el5. doi:10.1016/j.cell.2020.05.015; Renee L Hajnik, Jessica A Plante, Yuejin Liang Mohamad-Gabriel Alameh, Jinyi Tang, Srinivasa Reddy Bonam «Dual spike and nucleocapsid mRNA vaccination confer protection against SARS-CoV-2 Omicron and Delta variants in preclinical models)), Sci Transl Med., 2022 Sep, 114(662):eabq1945. dok10.1126/scitranslmed.abq1945; Хайник Р.Л., Планте Джа, Лян У, Аламех М.Г., Тан Дж., Бонам-старший, Чжун С., Адам А, Шартон Д., Рафаэль Г.Х., Лю У, Хейзелл Н.К., Сун Дж., Сун Л., Ши ПИ, Ван Т., Уокер Д.Х., Сан Дж., Вайсман Д., Уивер С.К., Планте К.С., Хьюстон 2022. "Dual spike and nucleocapsid mRNA vaccination confer protection against SARS-CoV-2 Omicron and Delta variants in preclinical models": Sci Transl Med, 2022 Sep 14; 14(662): eabql945, dok10.1126/scitranslmed.abq1945].
Кандидат на вакцину UB-612 является одной из таких экспериментальных вакцин, которая нацелена на белок N, наряду с другими вирусными белками, в попытке вызвать широкий иммунитет.[Daniel Martinez-Flores, Jesus Zepeda-Cervantes, Adolfo Cruz-Resendiz, Sergio Aguirre-Sampieri, Alicia Sampieri, and Luis Vaca " SARS-CoV-2 Vaccines Based on the Spike Glycoprotein and Implications of New Viral Variants". Front Immunol. 2021; 12: 701501, doi:10.3389/fimmu.2021.701501].
Одним из альтернативных подходов к созданию вакцин как против вирусов гриппа, так и против коронавирусов, является встраивание консервативных последовательностей вирусных белков в пробиотические векторы. То есть, альтернативой использованию химических адъювантов для вакцинации является применение так называемых живых вакцин на основе пробиотиков. Живые вакцины применяют, как правило, однократно, вводят подкожно, накожно или внутримышечно, а некоторые вакцины перорально и ингаляционно. Главным преимуществом живых вакцин является то, что они активируют все компоненты иммунной системы, вызывая сбалансированный прочный иммунный ответ.
Пробиотики - препараты, оказывающие общее благотворное влияние на организм человека (чаще всего молочнокислые бактерии). Установлено, что некоторые пробиотики являются эффективными неспецифическими стимуляторами выработки специфических иммуноглобулинов против различных инфекций [Vintini Е.О., Medina M.S., ВМС Immunol. 12: 46 (2011; Bermudez-Hurnarun L.G., Kharrat P., Chatel J.M., Microb. Cell. Fact. 10: 17-24 (2011)]. Пробиотики стали использоваться в качестве векторов, в часть из которых успешно внесены плазмидные конструкции, обеспечивающие экспрессию антигенов патогенных бактерий [ME Y., Luo Y., Huang X., Song F., Liu G. Microbiology, 158: 498-504 (2012), De Azevedo M., Karczzewski J., Lefeure F. Et al. BMC Microbiol. 12: 299 (2011)].
Пробиотические микроорганизмы рассматриваются в настоящее время как основа для получения рекомбинантных живых вакцин, экспрессирующих вакцинные антигены возбудителей актуальных инфекций Vintini Е.О., Medina M.S., ВМС Immunol, 12: 46 (2011); Bermudez-Hurnarun L.G., Kharrat P., Chatel J.M., Microb. Cell. Fact. 10: 17-24 (2011); MEY., Luo Y., Huang X., SongF., LiuG. Microbiology, 158: 498-504 (2012), De Azevedo M., Karczzewski J., Lefeure F. Et al. BMC Microbiol. 12: 299 (2011), [Lei H. et al. Virology. T. 476: 189-195 (2015)].
В 2015 г. Хан Л. со своими коллегами создали химерную конструкцию на основе бактерии Lactococcus lactis, содержащую на своей поверхности NA вируса гриппа А, и показали, что она способна защищать мышей от инфекции вирусами гриппа [Lei Н. et al. Virology. Т. 476: 189-195, (2015)]. Таким образом, они показали, что такая химерная конструкция может быть кандидатом для создания универсальной противогриппозной вакцины.
Известен пробиотический штамм Enterococcus faecium L3 ND-79 ВНИИСХМ, предназначенный для изготовления лечебно-профилактических средств и продуктов питания лечебно-профилактического назначения, обладающий устойчивостью к широкому спектру антибиотиков, более выраженным антагонизмом к патогенным бактериям, высокой жизнеспособностью и высоким уровнем продукции витаминов группы "В" и фолиевой кислоты; известный штамм положен в основу заявляемого изобретения [патент РФ №2220199 на штамм энтерококков Enterococcus faecium L3 для изготовления лечебно-профилактических средств].
Известен способ, основанный на введении bac гена патогенных СГВ в хромосомную ДНК в области кодирования белков пилей пробиотического штамма Enterococcus faecium L3 с последующей экспрессией белка Вас, кодируемого этим геном, в пилях. Пили энтерококков выступают за пределы бактериальных клеток и способны проникать сквозь капсулу, которая экранирует большинство бактериальных белков-антигенов. Пили состоят из белковых мономеров, способных к агрегации. За счет этого процесса может увеличиваться доза чужеродного антигена, встроенного в белок пилей. Последнее должно способствовать увеличению титров антител, специфичных к встраиваемому в структуру поверхностного белка энтерококка полипептидного фрагмента штамма патогенного СГВ. Способ введения гена патогенных стрептококков в пили пробиотического штамма Enterococcus faecium L3 интересен тем, что рекомбинантный белок экспрессируется не в цитоплазме, а на поверхности бактерии [патент РФ №2640250 на «Способ введения генов патогенных стрептококков в хромосомную ДНК пробиотического штамма Enterococcus faecium 13 для экспрессии в пилях», МПК: А61К 39/092; авторы - Суворов А.Н. и др.].
Известно, что штамм Enterococcus faecium L3 обладает выраженной антагонистической активностью в отношении грамположительных и грамотрицательных бактерий, способностью восстанавливать микробиоценоз кишечника на фоне дисбиотических состояний [Yermolenko Е., Suvorov А., Chernush A., et al. International Congress Series, 1289: 363-366 (2006)], а также оказывать иммуномодулирующее действие на организм хозяина [Tarasova Е., Yermolenko Е., Donets V., Sundukova Z., Bochkareva A., Borschev I., Suvorova M., Ilyasov. I., Simanenkov V., Suvorov A. The influence of probiotic enetrococci on the microbiota and cytokines expression in rats with dysbiosis induced by antibiotics // Beneficial Microbes. -2010 - Т. 1. -№3. - C. 265-270].
Осуществлено физическое картирование штамма Enterococcus faecium L3 [Suvorov A., Simanenkov V., Gromova L. et al. Prebiotics and probiotics potential for human health, 104-112, (2011)]. В экспериментах на здоровых самках мышей показано, что интравагинальное введение высоких доз штамма Enterococcus faecium L3 не только не оказывает токсического действия на организм, но не влияет на состояние слизистой оболочки влагалища [Суворов А.Н., Алехина Г.Г., Пигаревский П.В. и др. Гастробюллетень, 4: 29-31, (2001)] и способствует экспрессии IL-10 клетками слизистой оболочки влагалища крыс с экспериментальным вагинитом, вызванном S.agalactiae и Staphylococcus aureus [Tarasova Е., Yermolenko Е., Donets V., Sundukova Z., Bochkareva A., Borschev I., Suvorova M., Ilyasov I., Simanenkov V., Suvorov A. The influence of probiotic enetrococci on the microbiota and cytokines expression in rats with dysbiosis induced by antibiotics // Beneficial Microbes. -2010.- Т. 1. - №3. - C. 265-270].
В штамме Enterococcus faecium L3, как и у других грамположительных бактерий, имеются пили, которые представляют собой фимбрии длиной 0,3-3 щп и диаметром 2-1 Опт [9 - Telford JL, Barocchi MA, Margarit I, Rappuoli R, Grandi G. Pili in gram-positive pathogens. Nat Rev Microbiol. - 2006 - T.4, №7 - C. 509-519. doi: 10.1038/nrmicrol443. PMID: 16778837]. Это длинные белок-подобные полимеры, тянущиеся на поверхности бактерий, представляют собой субъединицы белка пилина, соединенные ковалентной связью. Они играют большую роль в адгезии колонизации хозяина. Пили являются высоко иммуногенными структурами, которые находятся под селективным давлением иммунных реакций хозяина [Danne С., Dramsi S. Pili of gram-positive bacteria: roles in host colonization. Res Microbiol. 2012 Nov-Dec; 163(9-10):645-58. doi: 10.1016/j.resmic.2012.10.012. Epub 2012 Oct 29. PMID: 23116627]. Принцип модификации пилей энтерококков вакцинными антигенами является приоритетным подходом при создании эффективных живых вакцин благодаря экспозиции целевого антигена на поверхности энтерококка.
Известен способ создания живой вакцины за счет введения генов патогенных стрептококков в хромосомную ДНК пробиотического штамма Enterococcus faecium L3 для экспрессии в пилях, и живая вакцина на основе модифицированного штамма пробиотиков Enterococcus faecium L3 для профилактики инфекции, вызванной Streptococcus pneumonie [Суворов А.Н., Гупалова Т.В., Кулешевич Е.В., Леонтьева Г.Ф., Крамская Т.А, Патент №2701733. (2019)], который принят в качестве прототипа.
Недостатком описанного в этом патенте способа создания живой вакцины является сложность конструирования такой вакцины за счет большого количества стадий, необходимых для создания суммарного фрагмента ДНК, состоящего из двух отдельных фрагментов гена пробиотика Enterococcus faecium L3 и химерного гена Streptococcus pneumonie - pspf
Указанный недостаток устраняется в способе создания кандидата живой вакцины за счет замены химерного гена Streptococcus pneumoniae - pspf на фрагмент гена sarsNl методом клонирования, что сокращает время получения вакцины в отличие от прототипа, а также позволяет создавать любые другие вакцинные кандидаты на этой платформе.
Существенно, что созданный вакцинный кандидат в отличие от вакцины-прототипа, предназначен для обеспечения специфической профилактики заболеваний, вызванных вирусом SARS-Cov-2, а не Streptococcus pneumoniae [Патент RU 2745626].
В ответ на попадание в организм возбудителя инфекции иммунная система начинает вырабатывать специфические белки - антитела. Наиболее иммуногенными белками вируса SARS-CoV-2 являются спайковый (S) и нуклеокапсидный (N) белки. Антитела к нуклеокапсидому белку SARS-CoV-2 могут обладать большей чувствительностью на ранних стадиях инфекции, в то время как антитела к спайковому белку SARS-CoV-2 позволяют судить о наличии протективного (защитного) иммунитета, который мог сформироваться после перенесенного заболевания или после вакцинации. Нуклеокапсидный белок (N) - это белок, который упаковывает геном позитивной РНК коронавирусов с образованием рибонуклеопротеиновых структур, заключенных в вирусный капсид. 14- белок является наиболее высоко экспрессируемым из четырех основных структурных белков коронавируса. В дополнение к своим взаимодействиям с РНК, N-белок образует белок-белковые взаимодействия с мембранным белком коронавируса (М) в процессе вирусной сборки.
Настоящее изобретение отличается тем, что был получен фрагмент гена, кодирующий белок N коронавируса SARS-Cov-2 в связи с тем, что N- белок коронавируса, хорошо сохраняется, не часто рекомбинирует и вызывает сильный Т-клеточный ответ. Поэтому может быть использован в качестве потенциальной мишени для коронавирусных вакцин.
Техническим результатом заявляемого изобретения является значительное упрощение способа получения живой вакцинного кандидата против коронавирусной инфекции.
Указанный технический результат достигается тем, что в способе создания живого вакцинного кандидата против коронавирусной инфекции на основе пробиотического штамма Enterococcus faecium L3 (по патенту РФ №2220199), модифицированного в результате электропорации культуры энтерококков штамма Enterococcus faecium L3 рекомбинантной плазмидной ДНК рentF-sarsNl, содержащей последовательность SEQ ID No:l в соответствии с заявленным изобретением, для создания SEQ ID No:l в плазмиде pentF-pspf (по патентуРФ №2701733), участок pspf был заменен на фрагмент гена нуклеокапсидного белка N коронавируса SARS-CoV-2, при
этом ДНК pentF-sarsNl кодирует аминокислотную последовательность SEQ ID No:2, которая стимулирует гуморальный и секреторный иммунитет в отношении нуклеокапсидного белка N коронавируса SARS-CoV-2.
При этом задача генетической части работы состояла в осуществлении интеграции участка ДНК вируса в структуру гена поверхностного белка пробиотика без нарушения открытой рамки считывания и повреждения участков кодирования компонентов, участвующих в процессинге поверхностного белка PilF.
Кроме того, указанный технический результат достигается тем, что вакцинный кандидат Enterococcus faecium L3-pentF-sarsNl, в которой штамм Enterococcus faecium экспрессирует ген слитого белка, индуцирующего образование специфических антител класса G и А в отношении инфекции.
В соответствии с заявленным изобретением, в плазмидную ДНК вводят фрагмент гена, кодирующего нуклеокапсидный белок N, осуществляющий стимуляцию гуморального и секреторного иммунитета в отношении N белка коронавируса SARS-CoV-2, т.е. созданием живого вакцинного кандидата, обладающего выраженным иммуногенным эффектом.
В известном способе, использованном в качестве прототипа на основе штамма пробиотика Enterococcus faecium L3 была сконструирована генно- инженерная живая вакцина со встроенным в хромосому, в области, кодирующей ген пилей, участком химерного гена pspf, сконструированного на основе генов патогенности Streptococcus pneumoniae. Штамм Enterococcus faecium L3-PSPF+ был получен в результате трансформации плазмидой pentF-pspf штамма Enterococcus faecium L3. Интраназальное введение вакцины Enterococcus faecium L3-PSPF+ стимулировало развитие специфического системного и местного иммунного ответа. На модели интраназальной летальной пневмококковой инфекции у мышей линии СВА было показано, что трехкратная интраназальная вакцинация созданной живой пробиотической вакциной повышала защиту от летальной пневмококковой инфекции.
При реализации заявленного способа создания вакцинного кандидата на основе пробиотического штамма Enterococcus faecium L3 был смоделирован и использован фрагмент ДНК нуклеокапсидного белка коронавируса, а затем получен методом обратной транскрипции (ДНК на матрице вирусной РНК была получена с помощью одношаговой обратно-транскриптазной полимеразно-цепной реакции (ПЦР) с праймерами CN1 и N2c помощью набора OneStep RT-PCR Kit (Qiagen, Германия). После электрофореза в 1.5% агарозном геле, полученный ПЦР продукт был вырезан из агарозного геля и гидролизован ферментами NdEI и EcoRI. При этом в плазмиде pentF-pspf участок гена pspf был заменен на фрагмент гена нуклеокапсидного белка.
В итоге реализации этого способа достигается решение конечной технической задачи заявленного изобретения, заключающееся в создании живого вакцинного кандидата против коронавируса, обладающей выраженными иммуногенными свойствам, на основе биологически активного штамма Enterococcus faecium L3 за счет замещения в структуре участка его гена пилей на участок гена нуклеокапсидного белка корановируса. Создание такой вакцинного кандидата против инфекции, вызываемой корановирусом, основано на введении участка ДНК, кодирующего антигенный фрагмент иммуногенных эпитопов коронавируса в структуру гена пилей пробиотика.
Реализация указанной технической задачи поясняется конкретными примерами введения гена sarsN1 коронавируса в хромосомную ДНК в области, кодирующей ген пилей, пробиотического штамма Enterococcus faecium L3 для экспрессии в пилях иммуногенного участка нуклеокапсидного белка N коронавируса:
а) получение слитого гена entF-sarsN1 и его клонирование;
б) выявление бактериальных клонов, содержащих ген слитого белка SARSN1 в хромосоме;
в) пероральная вакцинация мышей пробиотическим вакцинным кандидатом;
г) определение L3-SARSN1-специфических антител в крови и вагинальных смывах.
Технической сущностью предлагаемого изобретения является создание живого вакцинного кандидата против коронавируса, обладающей выраженными иммуногенными свойствами.
Пероральное введение вакцины Enterococcus faecium L3 с геном sarsN1 коронавируса стимулировало развитие специфического системного и местного иммунного ответа.
Для получения слитого гена entF-sarsN1 и его клонирования были сконструированы ДНК праймеры, представленные в таблице «Олигонуклеотидные праймеры», в которой в графе 1 приведены 4 названия праймеров; в графе 2 приведена их ориентация по отношению к положительной цепочке ДНК. а в графе 3 приведены нуклеотидные последовательности, которые были сконструированы в соответствии с направлением цепочки ДНК последовательность от 5'к 3'.

Нуклеотидные последовательности в графе 3 с участками, выделенными подчеркиванием, указывают на соответствующие им сайты гидролиза рестрикционными эндонуклеазами.
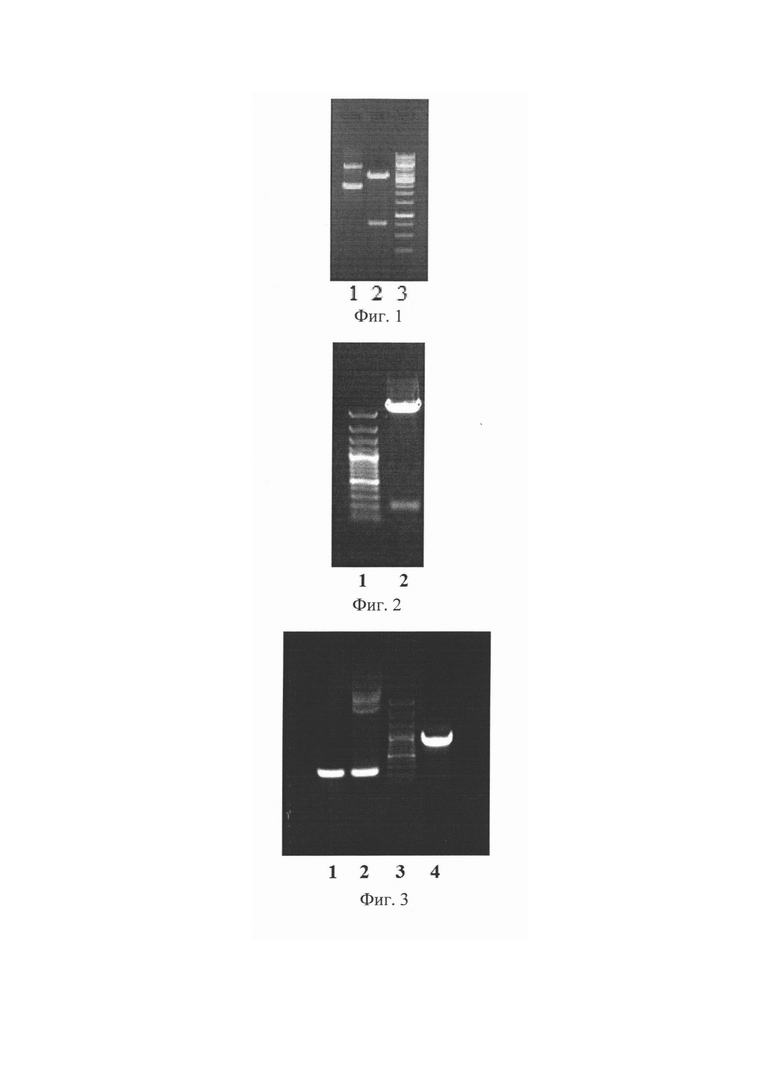
Рекомбинантная плазмидная ДНК pentF-pspf представляющая собой плазмидную ДНК, полученную в результате вставки суммарного фрагмента ДНК, состоящего из двух отдельных фрагментов гена пробиотика Enterococcus faecium L3 и фрагмента гена pspf описанная в предыдущей заявке на патент №2018144657 «Создание живой вакцины на основе штамма пробиотиков enterococcus faecium L3 для профилактики инфекции, вызванной streptococcus рпеитоте», (2018)] была использована для настоящей конструкции. Для этого из плазмидной ДНК pentF-pspf нужно было удалить вставку гена пневмококка pspf и заменить ее на вставку гена sarsN7. Для этого плазмидную ДНК pentF-pspf гидролизовали ферментами NdEI и EcoRI, сайты для которых ограничивают последовательность гена pspf (Фиг. 1). Полученный верхний фрагмент после гидролиза был использован для дальнейшего клонирования.
К-ДНК, полученная на матрице РНК коронавируса, была амплифицирована с праймерами CN1 и N2. Программа ПЦР состояла из: денатурации при 94°С - 30 сек, отжига праймеров - 55°С - 1 мин и синтеза -72°С - 2 мин. Этот цикл повторялся 30 раз, после чего смесь инкубировалась при 72°С 10 мин. Полученный ПЦР продукт был вырезан из геля и также гидролизован ферментами NdEI и EcoRI
Клонирование рестрицированного фрагмента после амплификации фрагмента ДНК, кодирующего фрагмент SARSN1, осуществляли, используя верхний фрагмент после гидролиза плазмиды pentF-pspf. ДНК рестрикты выделяли из агарозы с помощью набора «QIAquick Gel Extraction Kit» (Qiagen, USA), лигировали и трансформировали в гетерологичную систему E.coli DH5a. Среда для отбора содержала 500 мкг/мл эритромицина. В результате трансформации был получен один клон. Из этого клона была выделена плазмидная ДНК. Наличие вставки подтверждалось гидролизом плазмиды ферментами NdEI и EcoRI (Фиг. 2).
Продукты ПЦР плазмидной ДНК, полученные с праймерами CN1 и N2, и SeqF и N2 были просеквенированы. Результаты сиквенса показали правильность последовательности плазмидной ДНК, которая содержала нужные фрагменты ДНК энтерококка и ДНК коронавируса (перечень последовательностей SEQ ID No: 1).
В результате клонирования были получена плазмида pentF-sarsN 1 с прогнозируемой вставкой и геном устойчивости к эритромицину.
В результате электропорации энтерококков созданной интегративной плазмидой было получено 6 трансформантов. Они были проверены в реакции ПЦР с праймерами CN1 и N2. Для этого из трансформантов выделяли ДНК, используя набор ДНК-экспресс (Литех, Россия). Положительный ответ дали все трансформанты. Для определения интеграции плазмидной ДНК pentF-sarsN1 в хромосомную ДНК энтерококка была проведена амплификация ДНК, выделенных из 6 клонов с праймерами В1 и N2. Интеграция плазмидной ДНК pentF-sarsN1 в хромосомную ДНК энтерококка была обнаружена только у одного трансформанта (Фиг 3).
Секвенирование ДНК, выделенной из положительного клона, обозначенного, как L3 - SARS N1, было проведено с праймерами, соответствующими последовательности гена sarsN нуклеокапсидного белка (праймер N2) и последовательности хромосомной ДНК энтерококков (праймер В1) для подтверждения интеграции плазмидной ДНК pentF- sarsNB хромосомную ДНК энтерококка (перечень последовательностей SEQ ID No: 1).
Клон L3 - SARSN1, экспрессирующий фрагмент гена нуклеокапсидного белка N белка, был выбран в качестве вакцинного кандидата для дальнейшего исследования.
Мышам, линии СВА (n=10), самцы в возрасте 12-14 недель с помощью адаптера вводили перорально E.faecium L3 (n=10) и L3-SARSN1 (n=10). Курс вакцинации состоял из трех циклов иммунизации с интервалом в две недели. Каждый цикл включал трехкратное ежедневное введение 0,5 мл взвеси бактерий в дозе 10*8 КОЕ/мышь. Для вакцинации исходный и модифицированный варианты E.faecium культивировали в среде BL в течение 18 часов при 37°С в аэробных условиях. E.faecium, модифицированный участком гена коронавируса, культивировали в присутствии эритромицина в концентрации 5 мкг/мл. Суспензию клеток отмывали дважды в ЗФФР и вводили мышам.
Для оценки гуморального и местного специфического иммунного ответа через 4 дня после окончания второго и третьего курса вакцинации у контрольных и вакцинированных мышей забирали кровь. В сыворотке крови определяли уровень специфических L3-SARSN1 IgG и проводили анализ специфических IgA антител в смывах и помете мышей.
Пример 1. Получение слитого гена entF- sarsNl и его клонирование.
Рекомбинантная плазмидная ДНК pentF-pspf, представляющая собой плазмидную ДНК, полученную в результате вставки суммарного фрагмента ДНК, состоящего из двух отдельных фрагментов гена пробиотика Enterococcus faecium L3 и фрагмента гена pspf, описанная в предыдущей заявке на патент №2018144657 «Создание живой вакцины на основе штамма пробиотиков enterococcus faecium L3 для профилактики инфекции, вызванной streptococcus pneumonie», (2018)] была использована для создания слитого гена entF-sarsN1. Д ля этого из плазмидной ДНК pentF-pspf вырезали вставку гена пневмококка pspf проведя гидролиз плазмидной ДНК pentF-pspf ферментами NdEI и EcoRI Полученный верхний фрагмент после гидролиза был использован для дальнейшего клонирования.
На фиг. 1 представлена электрофореграмма рестрицированной плазмидной ДНК pentF-pspf ферментами NdEI и EcoRI, где обозначено:
1 – плазмида pentF-pspf - исходная;
2 - плазмида pentF-pspf рестрицированная;
3 - 100 п. н. ДНК - маркер (сверху вниз: 3000, 2000, 1000, 900, 800, 700, 600, 500, 400, 300, 200 и 100 нуклеотидных пар).
К-ДНК, полученная на матрице РНК вируса коронавируса, была амплифицирована с праймерами CN1 и N2. Программа ПЦР состояла из: денатурации при 94°С - 30 сек, отжига праймеров - 55°С - 1 мин и синтеза -72°С - 2 мин. Этот цикл повторялся 30 раз, после чего смесь инкубировали при 72°С 10 мин. Полученный ПЦР продукт был вырезан из геля и также гидролизован ферментами NdEI и EcoRI. Клонирование рестрицированного фрагмента после амплификации ДНК, кодирующего фрагмент N1 белка, осуществляли, используя верхний фрагмент после гидролиза плазмиды pentF-pspf. Продукты рестрикции разделяли с помощью электрофореза в 1% агарозном геле. ДНК рестрикты выделяли из агарозы с помощью набора «QIAquick Gel Extraction Kit» (Qiagen, USA), лигировали и трансформировали в гетерологичную систему E.coli DH5a. Среда для отбора содержала 500 мкг/мл эритромицина. Из полученного клона после трансформации была выделена ДНК, которая была проверена в реакции ПЦР с праймерами CN1 и N2 и дала положительный ответ. Из этого трансформанта была выделена плазмида, которая содержала ожидаемую вставку. На фиг. 2 представлена электрофореграмма рестрицированной плазмиды NdEI и EcoRI, где цифрами обозначено:
1-100 п. н. ДНК - маркер (сверху вниз: 3000, 2000, 1000, 900, 800, 700, 600, 500, 400, 300, 200 и 100 нуклеотидных пар);
2 - рестрицированная плазмида.
Для дальнейшей работы использовали эту плазмиду, обозначенную как pentF- sarsN1.
Пример 2. Электропорация энтерококков.
Для электропорации энтерококков культуру Enterococcus faecium L3 сеяли в 3 мл бульона Tood-Hewitt (ТНВ) («HiMedia», Индия) и выращивали в течение ночи при 37°С, затем пересевали в 50 мл бульона ТНВ 1 мл ночной культуры и выращивали ее до оптической плотности при длине волны 650 нм 0.3. После этого культуру помещали в лед и затем отмывали трижды в 20 мл 10% глицерола при температуре 4°С, полученный осадок суспендировали в 0.5 мл стерильного раствора глицерола, переосаждали и конечный осадок ресуспендировали в 0.3 мл того же раствора, разлили по 50 мкл в пробирки и проводили электропорацию в кювете с расстоянием между электродами 1 мм при напряжении 2100 В. Во льду к клеткам добавили ДНК (полученную интегративную плазмиду pentF-sarsN1 плазмиду, 300 нг). Оптимальная продолжительность импульса составила 4-5 миллисекунд. После проведения разряда тока в кювету добавляли 1 мл ТНВ, инкубировали 1 час и высевали на чашки с селективной средой, которая содержала 10 мкг/мл эритромицина. Затем ожидали появления трансформантов через 24 часа.
В результате электропорации энтерококков созданной интегративной плазмидой было получен 6 трансформантов. Они были проверены в реакции ПЦР с праймерами CN1 и N2. Для этого из трансформантов выделяли ДНК, используя набор ДНК-экспресс (Литех, Россия). Положительный ответ дали все трансформанты.
Для определения интеграции плазмидной ДНК pentF-sarsN1 в хромосомную ДНК энтерококка была проведена амплификация ДНК, выделенных из 6 клонов с праймерами В1 и N2. Интеграция плазмидной ДНК pentF-sarsN1 в хромосомную ДНК энтерококка была обнаружена только у одного трансформанта (фиг 3). Он был обозначен как L3-SARSN1. Секвенирование ДНК, выделенной из этого клона, было проведено с праймерами, соответствующими последовательности гена sarsN (праймер N2) и последовательности хромосомной ДНК энтерококков (праймер В1) для подтверждения интеграции плазмидной ДНК pentF-sarsN1 в хромосомную ДНК энтерококка (фиг. 4).
На фиг. 3 представлена электрофореграмма амплифицированных ДНК фрагментов с праймерами CN1 и N2, В1 и N2.
1 - продукт ПЦР ДНК из полученного клона после электропорации с праймерами CN1 и N2.
2 - продукт ПЦР плазмидной ДНК pentF-sarsN1,
3 - 100 п.н. ДНК - маркер (сверху вниз: 3000, 2000, 1000, 900, 800, 700, 600, 500, 400, 300, 200 и 100 нуклеотидных пар).
4 - продукт ПЦР ДНК из полученного клона после электропорации с праймерами В1 и N2.
На фиг. 4 представлена нуклеотидная последовательность, показывающая интеграцию плазмидной ДНК pentF-sarsN1 в хромосомную ДНК энтерококка.
Последовательности праймеров В1 и N2 выделены подчеркиванием, d2-1 представляет собой область гена энтерококка, кодирующую N-концевую часть белка энтерококка; d2-2 представляет собой область гена энтерококка, кодирующую центральную часть белка, которая была частично заменена фрагментом гена sarsN1.
Клон энтерококков L3-SARSN1, экспрессирующий ген слитого SARSN1 белка, выбран в качестве вакцинного кандидата для дальнейшего исследования.
Пример 3. Пероральная вакцинация мышей живой пробиотической вакциной.
Мышам линии СВА (10 самцов в возрасте 12-14 недель) с помощью адаптера вводили перорально E.faecium L3 (N=10) и L3-SARSN1 (N=10). Курс вакцинации состоял из трех циклов иммунизации с интервалом в две недели. Каждый цикл включал трехкратное ежедневное введение 0,5 мл взвеси бактерий в дозе 10*8 КОЕ/мышь. Для вакцинации исходный и модифицированный варианты E.faecium культивировали в среде BL в течение 18 часов при 37°С в аэробных условиях. E.faecium, модифицированный участком гена N белка коронавируса, культивировали в присутствии эритромицина в концентрации 5 мкг/мл. Суспензию клеток отмывали дважды в ЗФФР и вводили мышам.
Пример 4. Определение L3-SARSN1 - специфических IgG антител в крови мышей и анализ специфических IgA антител в смывах и помете мышей.
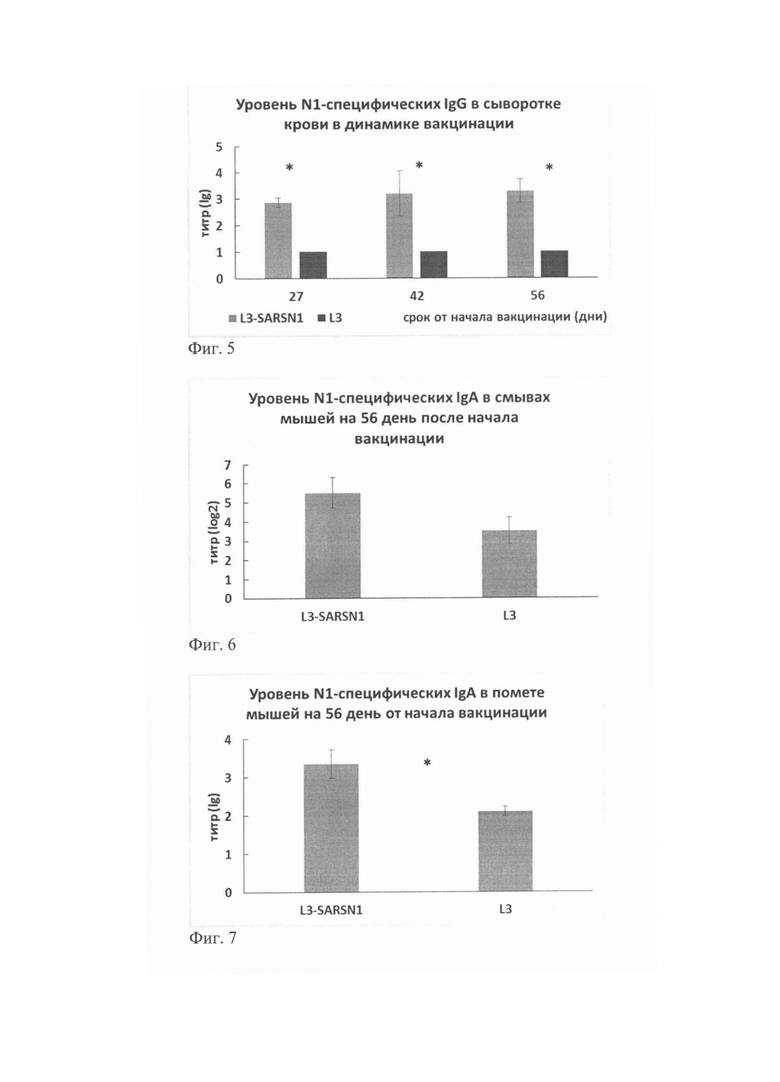
Через 27, 42 и 56 дней после окончания второго этапа иммунизации, у мышей забирали кровь для оценки уровня специфического гуморального иммунного ответа, а также на 56 день получали носовые смывы и помет для определения специфических IgA антител. Сыворотки крови и смывы использовали без дополнительной обработки, из помета готовили 10% (вес/объем) взвесь в ЗФФР, надосадок отделяли центрифугированием и далее использовали в анализе. Уровень специфических IgG и IgA определяли общепринятым методом ИФА. На дно 96-луночного планшета сорбировали рекомбинантный белок N (коммерческий аналог вирусной вставки в энтерококк) в концентрации 0,5 мкг/мл в течение ночи при 4°С. Последовательные 4-кратные разведения исследуемого биологического материала вносили в лунки в объеме 100 мкл и инкубировали 1 час при 37°С.Избыток антител отмывали ЗФФР с 0,5% Tween 20 и 2% сухого молока. Специфические антитела выявляли с помощью вторичных антител, конъюгированных с пероксидазой хрена. Для идентификации антител класса А использовали козий анти-мышь -IgA-HRP конъюгат (Sigma), антител класса G - козий анти-мышь-IgG-HRP конъюгат (Sigma). Раствор конъюгата вносили в объеме 100 мкл и инкубировали 1 час при 37°С. Планшеты отмывали четыре раза в ЗФФР с 0,5% Tween 20, связанный конъюгат выявляли в цветной реакции с ТМБ, интенсивность окраски оценивали при длине волны 450 нм. Анализ сывороток показал, что в крови вакцинированных животных через 27, 42 и 56 дней после последнего цикла вакцинации присутствовали антитела класса G, специфичные в отношении вирусного белка N, аналогичного вставке в энтерококк. Титры антител колебались в пределах 1:320-1:10240.
На фиг. 5 представлены результаты титрования иммуноглобулинов класса G в сыворотке мышей через 27, 42 и 56 дней после последнего цикла вакцинации. Показатели выражены в виде средних значений титров специфического IgG. Столбец 1 - титр специфического IgG из сыворотки мышей, получавших L3-SARSN1. Столбец 2 - титр специфического IgG из сыворотки мышей, получавших E.faecium L3 * - отличия между группами достоверны р≤0,05.
На фиг. 6 представлено содержание иммуноглобулинов класса А в носовых смывах мышей через 56 дней после окончания вакцинации, где в столбце 1 представлены средние титры специфического IgA из сыворотки мышей, получавших L3-SARSN1; в столбце 2 средний титр специфического IgA из сыворотки мышей, получавших E.faecium L3 * - отличия между группами достоверны р≤0,05.
На фиг. 7 представлено содержание иммуноглобулинов класса А в помете мышей через 56 дней после окончания вакцинации, где в столбце 1 представлены средние титры специфического IgA из сыворотки мышей, получавших L3-SARSN1; в столбце 2 средний титр специфического IgA из сыворотки мышей, получавших E.faecium L3 * - отличия между группами достоверны р≤0,05.
Определение уровня специфических антител класса А позволило установить, что вакцинация приводит к появлению в секретах слизистых оболочек мышей специфических IgA, значения колеблются в пределах 1:320-1:10240 для 10% взвеси помета, в пределах 1:32-1:128 в носовых смывах.
Как показывают результаты экспериментов, заявленное изобретение позволяет создать эффективный живой вакцинный кандидат с профилактическим эффектом в отношении коронавирусной инфекции.
Конструирование живого вакцинного кандидата на основе биологически активного пробиотического штамма Enterococcus faecium L3 за счет включения в его пили антигена SARS N коронавируса позволило объединить в одном препарате эффективность полезных свойств пробиотика и специфического антигенного стимула.
--->
<?xml version="1.0" encoding="UTF-8"?><!DOCTYPE ST26SequenceListing
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN"
"ST26SequenceListing_V1_3.dtd"><ST26SequenceListing
originalFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="/run/media/v20/USB DISK/0000 Патентовед кейс/600 Гупалова
ТВ 2022-11-28/02 Работа с авторами/К ответу 2023-02-21/ИЗ 2022133125
L3-SARSN1 (600).xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2023-02-27">
<ApplicationIdentification> <IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022133125</ApplicationNumberText>
<FilingDate>2022-12-15</FilingDate> </ApplicationIdentification>
<ApplicantFileReference>600</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022133125</ApplicationNumberText>
<FilingDate>2022-12-15</FilingDate>
</EarliestPriorityApplicationIdentification> <ApplicantName
languageCode="ru">Федеральное государственное бюджетное научное
учреждение "Институт экспериментальной
медицины"</ApplicantName> <ApplicantNameLatin>Federal State
Budgetary Scientific Institution "Institute of Experimental
Medicine"</ApplicantNameLatin> <InventionTitle
languageCode="ru">Способ создания рекомбинантного штамма энтерококка
L3-SARSN1 на основе биологически активного штамма Enterococcus
faecium L3</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity> <SequenceData
sequenceIDNumber="1"> <INSDSeq>
<INSDSeq_length>1317</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1317</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgagagcagctggtattgagttgaatgatacatttctatctatttacagtt
taaatggacagtatcagcaacgtgtgtcttggtataatgacaataatgaatctgtcggtgaacgtaatat
tgatatgagagaatttgttgggtatgaaaaaatgggtagcttaccttattttgtcacaacagatacagca
tgtgcagaatacaaagctcctgcgttatcaacaaacaatttaacttcaaaagtagtgggaggacgtgcag
aaaaggcttatagctcgaatgatcatttcaccgatgttgtaggagctgatacttatcacagaagtggtgt
aacgtatacgcttcaaggcgcttccccaacattcatgattggcgcaaatacgaatagtatgatgtttagc
tttgatactgcattgctatggacaccacaaccatcgaagcctacaaaagaagtgtttaacaaagctaata
ctgaagaggcagcacacaatattgacaaaaaagtgattccacaaggatcagatgtttactatcatattca
tcaaaagtttgatgcattaacagtcaacacaatgaacaaatacaaatcatttaaaatcactgataccttt
gacagcaaaaattttgatatggtatcggatgggaaaaactatgatggcgcattgcatatgactgagggag
ccttgaatacaccaaaagatcacattggcacccgcaatcctgctaacaatgctgcaatcgtgctacaact
tcctcaaggaacaacattgccaaaaggcttctacgcagaagggagcagaggcggcagtcaagcctcttct
cgttcctcatcacgtagtcgcaacagttcaagaaattcaactccaggcagcagtatggaattctacgaaa
aaaacctgaaagaatacaccgacaagctggataaactcgagaaaggttctgcgcgagtgatagatatgag
tacaggaaaagatattacttcagaaggtacactaacctatgatagcaatttaagaacgctgaaatgggaa
gcttccgctgatttcttaagtaaaaatcttttagatggacgagaaattcaactgatatttacagctaaaa
ctccactacagtcagaaaaaaatattgataaccaagccgtagcagcagtagagaatgtttctaataaaac
gaatgttgtaacaattggtgttgatcctaacttaccacaagtcattgttcctagaacaggttctacacat
ttagtaacaattttagtggttagcttagtattacttgtgttagctactcttagttatgtggctctgaagt
tcaat</INSDSeq_sequence> </INSDSeq> </SequenceData> <SequenceData
sequenceIDNumber="2"> <INSDSeq>
<INSDSeq_length>439</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..439</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRAAGIELNDTFLSIYSLNGQYQQRVSWYNDNNESVGERNIDMREFVGYEKM
GSLPYFVTTDTACAEYKAPALSTNNLTSKVVGGRAEKAYSSNDHFTDVVGADTYHRSGVTYTLQGASPTF
MIGANTNSMMFSFDTALLWTPQPSKPTKEVFNKANTEEAAHNIDKKVIPQGSDVYYHIHQKFDALTVNTM
NKYKSFKITDTFDSKNFDMVSDGKNYDGALHMTEGALNTPKDHIGTRNPANNAAIVLQLPQGTTLPKGFY
AEGSRGGSQASSRSSSRSRNSSRNSTPGSSMEFYEKNLKEYTDKLDKLEKGSARVIDMSTGKDITSEGTL
TYDSNLRTLKWEASADFLSKNLLDGREIQLIFTAKTPLQSEKNIDNQAVAAVENVSNKTNVVTIGVDPNL
PQVIVPRTGSTHLVTILVVSLVLLVLATLSYVALKFN</INSDSeq_sequence> </INSDSeq>
</SequenceData></ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ создания рекомбинантного штамма энтерококка L3-SARS"XBB.1.5" на основе биологически активного штамма Enterococcus faecium L3 | 2023 |
|
RU2840971C1 |
| Способ создания живого штамма энтерококка L3-SARS на основе биологически активного штамма Е. faecium L3 | 2021 |
|
RU2782529C1 |
| Способ создания живой вакцины против коронавирусной инфекции COVID-19 на основе пробиотического штамма Enterococcus faecium L3 и живая вакцина Enterococcus faecium L3-pentF-covid-19 | 2020 |
|
RU2745626C1 |
| Живая вакцина на основе штамма пробиотиков ENTEROCOCCUS FAECIUM L3 для профилактики инфекции, вызванной STREPTOCOCCUS PNEUMONIE | 2018 |
|
RU2701733C1 |
| СПОСОБ ВВЕДЕНИЯ ГЕНОВ ПАТОГЕННЫХ СТРЕПТОКОККОВ В ХРОМОСОМНУЮ ДНК ПРОБИОТИЧЕСКОГО ШТАММА ENTEROCOCCUS FAECIUM L3 ДЛЯ ЭКСПРЕССИИ В ПИЛЯХ | 2015 |
|
RU2640250C2 |
| Рекомбинантная ДНК, обеспечивающая получение рекомбинантного белка Cov1, обладающего иммуногенными свойствами в отношении вируса SARS-CoV-2 | 2021 |
|
RU2776484C1 |
| Интегративный плазмидный вектор pVEAL3-RBDdel, обеспечивающий синтез и секрецию рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, рекомбинантный штамм клеточной линии CHO-K1- RBDdelta и рекомбинантный белок RBDdelta SARS-CoV-2, продуцируемый штаммом клеточной линии | 2023 |
|
RU2816175C1 |
| СПОСОБ ПОЛУЧЕНИЯ БАКТЕРИАЛЬНОГО ПРОДУЦЕНТА РЕКОМБИНАНТНОГО БЕЛКА НУКЛЕОКАПСИДА NC ВИРУСА SARS-COV-2 | 2023 |
|
RU2812347C1 |
| Искусственная генетическая конструкция для гетерологической экспрессии рецептор-связывающего домена S-белка в слитной полипептидной цепи с нуклеокапсидным белком | 2022 |
|
RU2801597C1 |
| РЕКОМБИНАНТНЫЙ ВИРУС ГРИППА, ПРЕДНАЗНАЧЕННЫЙ ДЛЯ ПРОФИЛАКТИКИ COVID-19 И ГРИППА, СПОСОБ ЕГО ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2022 |
|
RU2802058C1 |

Изобретение относится к биотехнологии. Предложен способ создания рекомбинантного штамма энтерококка L3-SARSN1 на основе биологически активного штамма Е. faecium L3, отличающийся тем, что проводят электропорацию культуры энтерококков Enterococcus faecium L3 рекомбинантной плазмидной ДНК pentF-sarsN1, имеющей SEQ ID No: 1, и выбирают клон энтерококка L3-SARSN1, экспрессирующий белок, имеющий аминокислотную последовательность SEQ ID No: 2, стимулирующий гуморальный и секреторный иммунный ответ в отношении нуклеокапсидного белка N коронавируса SARS-Cov-2. Также предложена рекомбинантная плазмида pentF-sarsN1, имеющая нуклеотидную последовательность SEQ ID No: 1, предназначенная для получения клона энтерококка L3-SARSN1, экспрессирующего белок, имеющего аминокислотную последовательность SEQ ID No: 2, стимулирующий гуморальный и секреторный иммунный ответ в отношении нуклеокапсидного белка N коронавируса SARS-Cov-2. Изобретение обеспечивает эффективное получение живого вакцинного кандидата против коронавирусной инфекции. 2 н.п. ф-лы, 7 ил., 1 табл., 4 пр.
1. Способ создания рекомбинантного штамма энтерококка L3-SARSN1 на основе биологически активного штамма Е. faecium L3, отличающийся тем, что проводят электропорацию культуры энтерококков Enterococcus faecium L3 рекомбинантной плазмидной ДНК pentF-sarsN1, имеющей SEQ ID No: 1, и выбирают клон энтерококка L3-SARSN1, экспрессирующий белок, имеющий аминокислотную последовательность SEQ ID No: 2, стимулирующий гуморальный и секреторный иммунный ответ в отношении нуклеокапсидного белка N коронавируса SARS-Cov-2.
2. Рекомбинантная плазмида pentF-sarsN1, имеющая нуклеотидную последовательность SEQ ID No: 1, предназначенная для получения клона энтерококка L3-SARSN1, экспрессирующего белок, имеющего аминокислотную последовательность SEQ ID No: 2, стимулирующий гуморальный и секреторный иммунный ответ в отношении нуклеокапсидного белка N коронавируса SARS-Cov-2.
| Способ создания живой вакцины против коронавирусной инфекции COVID-19 на основе пробиотического штамма Enterococcus faecium L3 и живая вакцина Enterococcus faecium L3-pentF-covid-19 | 2020 |
|
RU2745626C1 |
| СПОСОБ ВВЕДЕНИЯ ГЕНОВ ПАТОГЕННЫХ СТРЕПТОКОККОВ В ХРОМОСОМНУЮ ДНК ПРОБИОТИЧЕСКОГО ШТАММА ENTEROCOCCUS FAECIUM L3 ДЛЯ ЭКСПРЕССИИ В ПИЛЯХ | 2015 |
|
RU2640250C2 |
| Живая вакцина на основе штамма пробиотиков ENTEROCOCCUS FAECIUM L3 для профилактики инфекции, вызванной STREPTOCOCCUS PNEUMONIE | 2018 |
|
RU2701733C1 |
| WO 2021202361 A1, 07.10.2021. | |||
Авторы
Даты
2024-05-28—Публикация
2022-12-15—Подача