Изобретение относится к биотехнологии и медицине, а именно к мониторингу и диагностике вируса Хендра (HENV) у здоровых, больных и лиц с подозрением на Хендра инфекцию.
Вирус Хендра - относительно новая зоонозная инфекция, впервые зафиксированная в 1994 в городе Брисбене, Австралия. Вирус вызвал вспышку острой респираторной инфекции среди лошадей, были заражены 2 человека, находившиеся в тесном контакте с больными животными. Один из них умер от тяжелой интерстициальной пневмонии.
Вирус Хендра принадлежит к семейству Paramyxoviridae подсемейства Paramyxovirinae, рода Henipavirus. Относится к плеоморфным и оболочечным вирусам с нуклеокапсидом в форме «елочки». Вирионы имеют размер от 40 до 600 нм в диаметре. Липидная оболочка вириона содержит гликопротеины и белки слияния, образующие шипы на ее поверхности. Геном представлен одноцепочечной нефрагментированной РНК отрицательной полярности и состоит из 6 генов, кодирующих 6 основных структурных белков: N (нуклеокапсидный белок), Р (фосфопротеин), М (матричный белок), F (белок слияния), G (гликопротеин) и L (большой белок). Прикрепление вируса опосредуется взаимодействием белка G с рецептором клетки-хозяина - эфрином В2/В3, в то время как слияние вируса и эндоцитоз опосредуются совместным действием как гликопротеина, так белка слияния (Lee В., Rota Р.А., 2012).
На сегодняшний день известно 62 вспышки данной инфекции с участием 108 лошадей (88 лошадей умерли, 20 лошадей умерли с неопределенным диагнозом, но имевшие схожие симптомы) и 7 человек (4 летальных случая). Все случаи заражения происходили на территории их распространения - Австралии. Уровень летальности у лошадей и людей составляет приблизительно 80% и 60% соответственно (Yuen K.Y., 2021).
Естественными хозяевами вируса Хендра в природе являются плодоядные крыланы, относящиеся к роду Pteropus, семейству Pteropodidae (Eaton В.Т., Wang L.-F., 2008). Основной механизм передачи возбудителя от животного к человеку - контакт с биологическими жидкостями инфицированной лошади, основная группа риска - ветеринары и дрессировщики. Лошади заражаются при употреблении пищи, загрязненной мочой, слюной или родовыми жидкостями крыланов. Вирус способен поражать домашних питомцев. Экспериментально были инфицированы свиньи, хорьки, африканские зеленые мартышки, кошки, хомяки и морские свинки. Предположительно они могут передавать вирус людям. Доказанных случаев заражения от человека к человеку зарегистрировано не было (Ruiz S.I. et. al., 2013).
Инкубационный период при Хендра инфекции у лошадей и людей колеблется от 3 до 16 дней. Клиническими особенностями у людей выступает острый фебрильный энцефалит, а общими клиническими - грипподобные симптомы, лихорадка, головная боль, сонливость. Летальный исход при тяжелых респираторных и/или неврологических симптомах. После выздоровления возможен рецидивирующий энцефалит, по-видимому, связанный с возобновлением размножения вируса в центральной нервной системе (Zaki S.R., Keating М.К., 2018).
Из-за небольшого количества зараженных, общую клиническую картину выявить не получается, тем не менее установлено, что основными органами-мишенями для данного патогена являются легкие, мозг, селезенка и почки, в результате поражения которых развивается полиорганная недостаточность. При этом доминирующим синдромом болезни в клинике заболевания человека является острый респираторный дистресс-синдром.
При работе с вирусом Хендра, в соответствии с анализом управления рисками в лаборатории, требуются обеспечения условий 4 уровня биобезопасности.
Существует вакцина против вируса Хендра, зарегистрированная в Австралии в 2012 году для лошадей. Для человека и других животных нет этиотропных средств лечения, а также вакцины. Основным принципом ведения пациентов является интенсивная поддерживающая терапия с устранением симптомов заболевания.
Существуют следующие методы для диагностики генипавирусов. Идентифицировать возбудителей можно методами ОТ-ПЦР и ГЩР в реальном времени, иммуногистохимическим методом, непрямым методом флуоресценции антител, твердофазным иммуноферментным анализом, реакцией нейтрализации вируса, анализом с микросферами (World Organisation for Animal Health, 2015).
Метод ОТ-ПЦР в реальном времени из-за высокой чувствительности является наиболее распространенными. Метод предполагает диагностирование вируса Хендра по наличию вирусной РНК в биологических образцах. В качестве анализируемых образцов для диагностики вируса Хендра могут выступать пробы сыворотки крови, образцы цельной крови, мазки со слизистой оболочки носо- и ротоглотки, мокроты, аутопаты легких, образцы мочи, образцы головного мозга, почек и селезенки (https://www.who.int/ru). Метод диагностики предусматривает экстракцию РНК из биологических образцов с последующим проведением полимеразной цепной реакции с обратной транскрипцией для целевых фрагментов РНК генома вируса Хендра с использованием набора праймеров и зондов.
Однако, не существует способов выявления РНК вируса Хендра методом ОТ-ПЦР в реальном времени, зарегистрированных на территории РФ. В настоящее время вирус Хендра вызвал небольшое количество вспышек в мире, тем не менее, из-за высокого уровня смертности патоген представляет проблему общественного здравоохранения. Для того чтобы сдержать распространение болезни, требуются инструменты для своевременной диагностики заболевших, а также для выявления носителей вируса Хендра еще до появления первых симптомов.
Целью создания настоящего изобретения является расширение арсенала средств эпиднадзора для мониторинга носительства вируса Хендра.
Технический результат, достигаемый при использовании изобретения - разработка высокочувствительного способа идентификации РНК Хендра вируса в биологических образцах.
Поставленная задача решалась путем:
- конструирования диагностических праймеров и флуоресцентно-меченного зонда на специфические для HENV кДНК фрагменты, полученные с РНК матрицы;
- конструирования рекомбинантной плазмидной ДНК и MS2-фага, несущих специфический участок РНК-матрицы;
- оптимизации концентраций компонентов реакционной смеси и условий проведения ПЦР.
Авторами предложен способ выявления вируса Хендра, согласно которому, выделенную из биологических образцов РНК анализируют в одной пробирке при помощи реакции обратной транскрипции и дальнейшей полимеразной цепной реакции полученных кДНК, при помощи олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участку гена, кодирующего нуклеокапсидный белок N вируса Хендра.
Анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию или отсутствию значения порогового цикла Ct, причем результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
На начальном этапе были подобраны и синтезированы пара специфических олигонуклеотидных праймеров и зондов для гибридизационно-флуоресцентной детекции продуктов ПЦР. Для этого в базе данных NCBI Mega BLAST (http://www.ncbi.nlm.nih. gov/) был выбран наиболее консервативный участок генома вируса Хендра. Были проанализированы все имеющиеся в базе данных последовательности. Также были подобраны праймеры и флуоресцентный зонд для ВКО в качестве внутреннего контроля ПЦР. Последовательности олигонуклеотидных праймеров и флуоресцентного зонда представлены в таблице 1 и таблице 2.
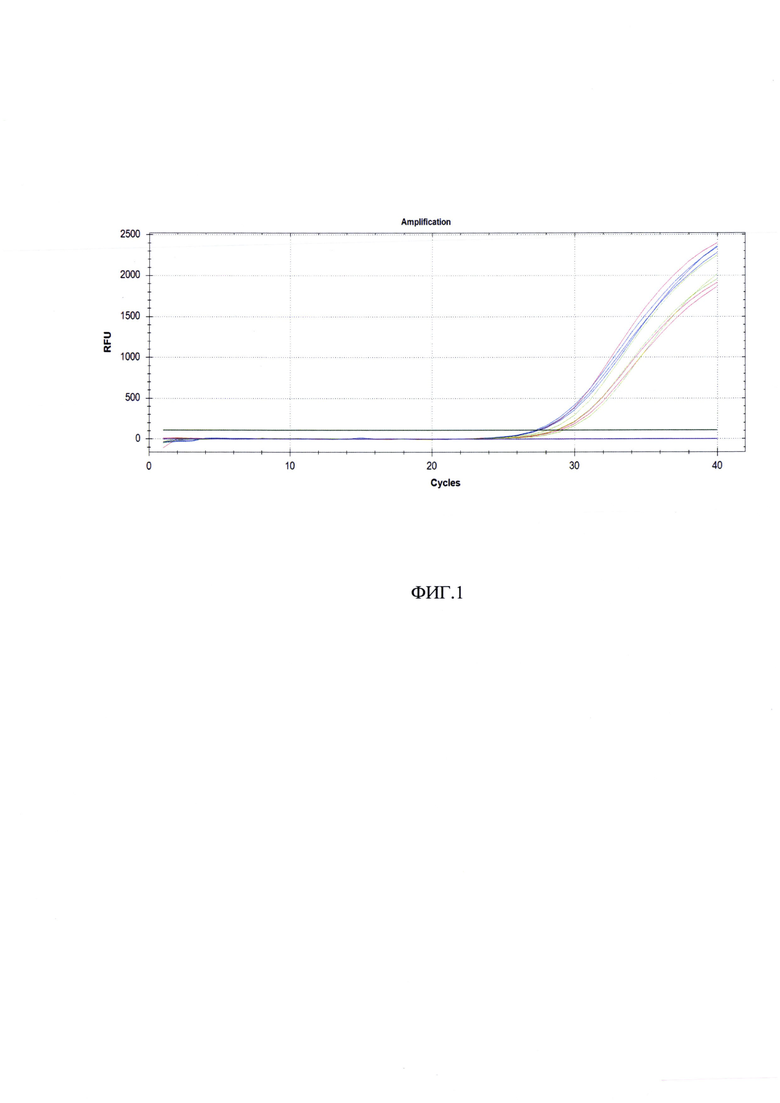
Сущность изобретения поясняется чертежом, где на Фиг. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции при анализе контрольных образцов. Каждая кривая выше пороговой линии отображает положительный результат на выявление в образце РНК Хендра вируса, каждая кривая ниже пороговой линии отображает отрицательные контроли, не содержащие РНК HENV.
Подбор и анализ свойств олигонуклеотидных праймеров и флуоресцентного зонда проводился с использованием программного обеспечения Oligonucleotide Properties Calculator и MFold.
Для контроля качества прохождения этапов обратной транскрипции и ПЦР в состав методики были введены рекомбинантные положительные контрольные образцы K+ и ПКО, а также внутренний контрольный образец -ВКО. Матрицу для создания рекомбинантных положительных контрольных образцов получали синтетическим методом на основе ампликона, включающего в себя диагностическую область-мишень и фланкирующие последовательности нуклеотидов. Ампликон получали методом ПЦР в один шаг.Конечный ПЦР-продукт лигировали в плазмидный вектор pGEM-T («Promega», USA) под контролем промотора Т7 РНК полимеразы и трансформировали им Escherichia coli (штамм XLl-Blue). Рекомбинантные плазмиды из индивидуальных клонов проверяли на правильность ориентации целевой последовательности и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом секвенирования по Сэнгеру с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500x1 («Applied Biosystems», США).
Соответствующие заданным критериям рекомбинантные плазмиды использовали для приготовления положительного контрольного образца этапа ПЦР (K+). Для этого определяли концентрацию ДНК в растворе рекомбинантной плазмиды и разводили стерильной водой до рабочей концентрации 1*107 копий/мл. Также плазмиды использовались для получения рекомбинантного РНК-содержащего положительного контрольного образца с защитной белковой оболочкой MS2-фага (ПКО). Для полученного продукта производили определение концентрации, затем разводили РНК-буфером (ФБУН ЦНИИ Эпидемиологии, Россия) до рабочей концентрации 1*106 копий/мл, которую использовали в качестве ПКО.
Оценку аналитической чувствительности метода производили на разведениях РНК-содержащего рекомбинантного положительного контрольного образца (ПКО), из которых экстрагировали РНК с помощью набора для выделения нуклеиновых кислот «Рибо-Преп» (ФБУН ЦНИИ Эпидемиологии, Россия), а затем с помощью специфических праймеров и флуоресцентных зондов определяли минимальное разведение, детектируемое как положительное в 100% случаев. Определенная таким образом аналитическая чувствительность метода составила 1*10 копий/мл.
Свойство изобретения специфически определять РНК вируса Хендра достигнуто путем подбора праймеров и зондов к высококонсервативным фрагментам РНК вируса, что минимизирует возможность перекрестных реакций с нуклеиновыми кислотами человека, близкородственных микроорганизмов, других инфекционных возбудителей.
Аналитическую специфичность оценивали при помощи тестирования следующих микроорганизмов: вирус парагриппа человека 3 типа, вирус гриппа А/ Puerto Rico/ 8/ 34/ (H1N1), вирус гриппа В/ Florida/ 04/ 06, аденовирус человека 5 типа, риновирус человека 1 типа, вирус клещевого энцефалита, лассавирус бешенства, вирус кори, респираторно-синцитиальный вирус человека, SARS-CoV-2, коронавирус человека ОС43. Неспецифические реакции отсутствуют.
Таким образом, в результате проведенных исследований был разработан и апробирован способ выявления РНК вируса Хендра в различных видах биологического материала. Технический результат достигается путем определения РНК HENV, включающим выделение РНК исследуемой пробы, проведение обратной транскрипции и ПЦР с учетом результатов в режиме реального времени, согласно изобретению.
Диагностика проводилась следующим образом: подготовку проб проводили согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Материалом для исследования могут служить клинические и биологические образцы.
Экстракция производится из 100 мкл полученной суспензии образца с лизирующим буфером на основе 6 моль гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения РНК, и последующим инкубированием 5 минут при температуре (65±1)°С. Выделение РНК осуществляют с помощью наборов «Рибо-Преп» и «Рибо-Сорб» (ФБУН ЦНИИ Эпидемиологии, Россия) в соответствии с инструкциями к наборам. Во все пробирки, включая контроль выделения, до этапа прогревания в лизирующем буфере добавляют 10 мкл ВКО. После выделения РНК приступают к постановке ОТ-ПЦР.
Для упрощения подготовки и стандартизации выполнения анализа в тест-систему входят 8 реактивов:
1) Реактив Amp 1RT - буферный раствор, содержащий фермент для обратной транскрипции. Представляет из себя буферный раствор, содержащий 50 мМ Трис-HCl, рН 8,0 (при 25°С), 100 мМ NaCl, 1 мМ ЭДТА, 5 мМ дитиотреитол, 50% (v/v) глицерин и 0,1% (v/v) NP-40, ингибитор РНК аз, M-MuLV -RH ревертазу и HS-Taq ДНК-полимеразу;
2) Реактив Amp 1В - буфер. Представляет собой буферный раствор, содержащий 100 мМ Трис-HCl, рН 8,3 (при 25°С), 150 мМ KCl, 0,6 мМ каждого дезоксинуклеозидтрифосфата, 10 мМ MgCl2, 8 мМ дитиотрейто л а, стабилизаторы и усилители ферментов;
3) Реактив Amp 2 "HENV" - содержит олигонуклеотидные праймеры и флуоресцентно-меченые олигонуклеотидные зонды к РНК вируса Хендра и к ВКО;
4) K+ "HENV" - положительный контроль этапа ПЦР. Представляет из себя смесь двух плазмидных ДНК: плазмиды, содержащей фрагмент вируса Хендра и плазмиды, содержащей фрагмент ВКО, с которых, при помощи реактива Атр2, амплифицируются специфические фрагменты;
5) К- - отрицательный контроль этапа ПЦР. Представляет из себя дистиллированную воду;
6) ПКО "HENV" - положительный контрольный образец этапа экстракции нуклеиновых кислот.Представляет собой псевдовирусные частицы на основе MS2-фага, содержащие РНК с фрагментом РНК вируса Хендра, которые после выделения и обратной транскрипции в кДНК, амплифицируются при помощи реактива Amp 2;
7) ОКО - отрицательный контрольный образец этапа экстракции нуклеиновых кислот.Представляет из себя дистиллированную воду;
8) ВКО - представляет из себя плазмиду, содержащую фрагмент ВКО, которая амплифицируются при помощи реактива Amp 2 HENV.
Реактивы ПКО, ВКО и ОКО хранятся при температуре +4°С, остальные компоненты хранятся при температуре -20°С.
Для постановки реакции ОТ-ПЦР в реальном времени нужно взять необходимое количество микропробирок объемом 0,2 мл, соответствующее числу исследуемых проб; плюс четыре пробирки для положительных и отрицательных контролей. Подготовить ПЦР-смесь из расчета на одну реакцию: 1 мкл реактив Amp1RT, 12,5 мкл реактив Amp1B, 1,5 мкл реактив Amp2. Смесь вортексируют и осаждают на настольной центрифуге. По 15 мкл полученной смеси вносят в пробирки, затем добавляют в каждую по 10 мкл РНК-пробы, экстрагированной из исследуемого материала. Готовят 4 контрольные реакции. Для этого в пробирку для положительного контроля ПЦР вносят 10 мкл К+, в пробирку для положительного контроля обратной транскрипции вносят 10 мкл образца, экстрагированного из ПКО. В пробирку для отрицательного контроля ПЦР вносят 10 мкл К-, в пробирку для отрицательного контроля экстракции вносят 10 мкл пробы, экстрагированной из ОКО. Конечный объем реакционной смеси составляет 25 мкл. Микропробирки переносят в программируемый амплификатор с функцией детекции флуоресценции в режиме «реального времени» по каналам (FAM/SybrGreen/Green и JOE/ HEX). Режим амплификации представлен в таблице 3.
Детекцию флуоресцентного сигнала производят при 60°С для приборов роторного типа и при 55°С для приборов планшетного типа по каналам Green (FAM) и Yellow (JOE/HEX). Учет и анализ результатов проводят с помощью программного обеспечения прибора на основании отсутствия или наличия сигнала флуоресценции в исследуемых пробах по детектируемым каналам. Результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию (или отсутствию) значения порогового цикла (Ct) в соответствующей графе в таблице результатов. Результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. Накопление флуоресцентного сигнала по каналу HEX свидетельствует о наличии в исследуемом материале РНК HENV. При этом результат считается валидным, если параллельно в пробе по каналу FAM регистрируется накопление флуоресцентного сигнала ВКО, а в образцах K+ и ПКО - по каналу HEX. Сигнал по каналу HEX в образцах К- и ОКО должен отсутствовать.
Таким образом, заявляемый способ выявления позволяет достоверно выявлять РНК вируса Хендра в клинических пробах.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="Перечень последовательностей Method for
detecting Hendra virus RNA of the species Hendra henipavirus by
real-time RT-PCR (HENV Amp PS).xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2023-07-14">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>000</ApplicationNumberText>
<FilingDate>2023-05-15</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>000</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>000</ApplicationNumberText>
<FilingDate>2023-05-15</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека»</ApplicantName>
<ApplicantNameLatin>Department of Epidemiology, Pasteur Institute,
Federal Service on Consumers' Rights Protection and Human
Well-Being Surveillance</ApplicantNameLatin>
<InventorName languageCode="ru">Дедков Владимир
Георгиевич</InventorName>
<InventorNameLatin>Dedkov Vladimir Georgievich</InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления РНК вируса Хендра
вида Hendra henipavirus методом ОТ-ПЦР в реальном времени (HENV Amp
PS)</InventionTitle>
<InventionTitle languageCode="en">Method for detecting Hendra virus
RNA of the species Hendra henipavirus by real-time RT-PCR (HENV Amp
PS)</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accagaagatgggcaaagtatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcagaatttccaccatgaattttctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl]-N-ethyl-6-(ethylamino)-2
7-dimethyl-3H-xanthen-3-iminium chloride) fluorophore at 5'
end</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Non-fluorescent Black Hole Quencher 1
(2-[N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4-methyl-2-nitrophen
yl)diazenyl]phenyl]diazenyl]anilino]ethanol) at 3'
end</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agggtcaatccattctttgccttaacccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления РНК вируса Henipavirus hendraense методом рекомбиназной полимеразной амплификации | 2024 |
|
RU2834950C1 |
| Способ выявления вируса Henipavirus hendraense методом DETECTR с изотермической амплификацией | 2024 |
|
RU2834907C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2834908C1 |
| Способ выявления вируса кори методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822430C1 |
| Способ выявления РНК модифицированного вакцинного полиовируса типа 2 (nOPV2) методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2795703C1 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
Изобретение относится к области биотехнологии. Описан способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени. Способ включает экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно меченного зонда, комплементарных участку гена, кодирующего нуклеокапсидный белок N вируса Хендра. Технический результат, достигаемый при использовании изобретения, состоит в разработке высокочувствительного способа идентификации РНК Хендра вируса в биологических образцах. 1 ил., 3 табл.
Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени, включающий экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно меченного зонда, комплементарных участку гена, кодирующего нуклеокапсидный белок N вируса Хендра, отличающийся тем, что в качестве прямого праймера используется последовательность acc aga aga tgg gca aag tat g, в качестве обратного праймера - tca gaa ttt cca cca tga att ttc tg, а в качестве зонда последовательность agg gtc aat cca ttc ttt gcc tta acc cag, у которой на 5'-конце расположена флуоресцирующая метка R6G, а на 3'-конце не флуоресцирующий гаситель BHQ1.
| WO 2013036993 A1, 21.03.2013 | |||
| US 20070150972 A1, 28.06.2007 | |||
| УНИВЕРСАЛЬНАЯ БИОСЕНСОРНАЯ СИСТЕМА ДЛЯ ДЕТЕКЦИИ АНАЛИТА | 2017 |
|
RU2737943C1 |
Авторы
Даты
2024-07-02—Публикация
2023-07-27—Подача