Изобретение относится к биотехнологии и медицине, решает проблему выявления генетических маркеров данного вируса. Предназначено для мониторинга и диагностики вируса Bandavirus dabieense у здоровых, больных и лиц с подозрением на лихорадку с тромбоцитопеническим синдромом и у людей, болеющих лихорадкой с тромбоцитопеническим синдромом.
Bandavirus dabieense (В. dabieense) представляет собой РНК-вирус, принадлежащий к роду Bandavirus семейства Phenuiviridae порядка Bunyavirales (https://ictv.global/taxonomy). Геном вируса В. dabieense состоит из трех сегментов - L, М и S. Бандавирус Dabieense, который ранее обозначался как SFTS virus (SFTSV), вирус Dabie или вирус Хуайяншань (Huaiyangshan virus) является возбудителем лихорадки с тромбоцитопеническим синдромом.
Тяжелая лихорадка с тромбоцитопеническим синдромом (severe fever with thrombocytopenia syndrome, SFTS) - вирусное заболевание, передаваемое клещами. Данное заболевание характеризуется острой лихорадкой, поражениями респираторного и желудочно-кишечного трактов, сопровождающееся прогрессирующей тромбоцитопенией, лейкопенией, с летальностью до 30% (Yu X.J. et al., 2011).
Вирус В. dabieense передается в основном через укусы клещей, таких как Haemaphysalis longicornis, Rhipicephalus microplus, Haemaphysalis flava, Haemaphysalis concinna, Amblyomma testudinarium и Ixodes nipponensis (Miao D. et al., 2020). Также зарегистрированы случаи передачи вируса В. dabieense при прямом контакте с кровью, биологическими жидкостями от заболевшего человека или животного (Hwang J. et al., 2016). Резервуаром вируса В. dabieense могут быть различные виды домашних (козы, свиньи, КРС, домашняя птица) и диких животных (грызуны, дикие олени) (Lina T.L. et al., 2020).
Заболевание чаще всего встречается в Китае, Южной Корее, Японии, Вьетнаме. На территории России природных очагов вируса В. dabieense на данный момент не обнаружено, не смотря на широкое распространение переносчика (Haemaphysalis longicorni) на территории Дальнего Востока РФ (Белов Ю.А. и др., 2019). Диагностика основана на выявлении РНК вируса методом ПЦР и/или специфических иммуноглобулинов класса М и G в зависимости от сроков начала болезни.
В качестве анализируемых образцов могут выступать клещи, сыворотка крови. Метод диагностики предусматривает экстракцию РНК из биологических образцов с последующим проведением полимеразной цепной реакции для целевых фрагментов РНК генома вируса В. dabieense с использованием набора праймеров и зонда, комплементарных участку L-сегменту вируса В. dabieense.
На данный момент времени на территории Российской Федерации не существует готовых наборов для выявления РНК вируса В. dabieense методом ОТ-ПЦР в реальном времени. Для решения таких задач, как проведения мониторинга, своевременной диагностики заболевших, а также для выявления носителей вируса В. dabieense еще до появления первых симптомов в короткие сроки необходимы современные инструменты. Создание такого инструмента и является целью нашего изобретения. Поставленная задача решалась путем:
- конструирования диагностических праймеров и флуоресцентно-меченного зонда на специфические для вируса В. dabieense кДНК фрагменты, полученные с РНК матрицы;
- конструирования рекомбинантной плазмидной ДНК и MS2-фага, несущих специфический участок РНК-матрицы;
- оптимизации концентраций компонентов реакционной смеси и условий проведения ПЦР.
- Технический результат - разработка высокочувствительного способа идентификации РНК вируса В. dabieense в биологических образцах и других вируссодержащих пробах (культуральная вируссодержащая жидкость, и т.д.).
Авторами предложен способ выявления вируса В. dabieense, согласно которому, выделенную из биологических образцов РНК анализируют в одной пробирке при помощи реакции обратной транскрипции и дальнейшей полимеразной цепной реакции полученных кДНК, при помощи олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участку гена L, кодирующего РНК-зависимую РНК-полимеразу B.dabiense
Анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию или отсутствию значения порогового цикла Ct, причем результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
На начальном этапе были подобраны и синтезированы пара специфических олигонуклеотидных праймеров и зонд для гибридизационно-флуоресцентной детекции продуктов ПЦР. Для этого в базе данных GenBank (https://www.ncbi.nlm.nih.gov/) был выбран наиболее консервативный участок генома вируса В. dabieense. Были проанализированы все имеющиеся в базе данных последовательности. Также, были подобраны праймеры и флуоресцентный зонд для внутреннего контрольного образца ПЦР. Последовательности олигонуклеотидных праймеров и флуоресцентного зонда представлены в Таблице 1 и Таблице 2.


Сущность изобретения поясняется чертежом, где на Фиг. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции при анализе контрольных образцов. Красные и зеленые кривые выше пороговой линии отображают положительный результат на выявление в образце РНК вируса В. dabieense, синие кривые ниже пороговой линии отображают отрицательные контроли, не содержащие РНК данного патогена.
Подбор и анализ свойств олигонуклеотидных праймеров и флуоресцентного зонда проводился с использованием программного обеспечения Oligonucleotide Properties Calculator и MFold.
Для контроля качества прохождения ПЦР в состав методики были введены рекомбинантные положительные контрольные образцы К+ и ПКО и внутренний контрольный образец (ВКО). Матрицу для создания рекомбинантных положительных контрольных образцов получали синтетическим методом на основе ампликона, включающего в себя диагностическую область-мишень и фланкирующие последовательности нуклеотидов. Ампликон получали методом ПЦР в два шага. Конечный ПЦР-продукт лигировали в плазмидный вектор pGEM-T («Promega», USA) и трансформировали им Escherichia coli (штамм Turbo). Рекомбинантные плазмиды из индивидуальных клонов проверяли на правильность ориентации целевой последовательности и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом секвенирования по Сэнгеру с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500x1 («Applied Biosystems», США).
Соответствующие заданным критериям рекомбинантные плазмиды использовали для приготовления положительного контрольного образца этапа ПЦР (К+). Для этого определяли концентрацию ДНК в растворе рекомбинантной плазмиды и разводили в 1xTE-буфере (рН 8.0) со стабилизаторами натрия азида 0,05% и ДНК спермы лосося 0,02 мг/мл до рабочей концентрации 1*107 копий/мл. Также, плазмиды использовались для получения рекомбинантного РНК-содержащего положительного контрольного образца (ПКО) с защитной белковой оболочкой MS2-фага. Для полученного продукта производили определение концентрации методом цифровой капельной ПЦР (ddPCR) («BioRad», USA). Рабочая концентрации составила 1*106 копий/мл, которую использовали в качестве ПКО в дальнейшем анализе.
Оценку предела обнаружения метода производили на десятикратных разведениях ПКО от начальной концентрации 1*106 копий/мл, из которых экстрагировали РНК с помощью набора для выделения нуклеиновых кислот «Рибо-Преп» (ФБУН ЦНИИ Эпидемиологии, Россия), а затем с помощью специфических праймеров и флуоресцентных зондов определяли минимальное разведение, детектируемое как положительное в 100% случаев. Определенная таким образом аналитическая чувствительность метода составила 1*103 копий/мл.
Свойство изобретения специфически определять РНК вируса В. dabieense достигнуто путем подбора праймеров и зондов к консервативным фрагментам РНК вируса, что минимизирует возможность перекрестных реакций с геномом человека, близкородственных микроорганизмов, других инфекционных возбудителей.
Аналитическую специфичность оценивали при помощи тестирования биологических образцов, содержащих следующие микроорганизмы: вируса денге, вируса Конго-крымской геморрагической лихорадки, вируса клещевого энцефалита, вируса геморрагической лихорадки с почечным синдромом, вируса краснухи, коронавируса SARS-CoV-2, вируса кори. Неспецифические реакции отсутствовали.
Таким образом, в результате проведенных исследований был разработан и апробирован способ выявления РНК вируса В. dabieense в различных видах биологического материала. Технический результат достигается путем определения РНК вируса В. dabieense, включающим выделение РНК исследуемой пробы, проведение обратной транскрипции и ПЦР с учетом результатов в режиме реального времени, согласно изобретению.
Подготовку проб проводили согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Материалом для исследования могут служить клинические и биологические образцы.
Экстракция производится из 100 мкл полученной суспензии образца с лизирующим буфером на основе 6 моль гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения РНК, и последующим инкубированием 5 минут при температуре (65±1)°С. Выделение РНК осуществляют с помощью наборов «Рибо-преп» и «Магно-Сорб» (ФБУН ЦНИИ Эпидемиологии, Россия) в соответствии с инструкциями к наборам. Во все пробирки, включая контроль выделения, до этапа прогревания в лизирующем буфере добавляют 10 мкл ВКО. После выделения РНК приступают к постановке ОТ-ПЦР.
Для упрощения подготовки и стандартизации выполнения анализа в тест-систему входят 8 реактивов:
1. Реактив Amp 1RT - буферный раствор, содержащий фермент для обратной транскрипции. Представляет из себя буферный раствор, содержащий 50 мМ Трис-HCl, рН 8,0 (при 25°С), 100 мМ NaCl, 1 мМ ЭДТА, 5 мМ дитиотреитол, 50% (v/v) глицерин и 0,1%) (v/v) NP-40, ингибитор РНКаз, M-MuLV -RH ревертазу и HS-Taq ДНК-полимеразу;
2. Реактив Amp 1 В - буферный раствор, содержащий 100 мМ Трис-HCl, рН 8,3 (при 25°С), 150 мМ KCl, 0,6 мМ каждого дезоксинуклеозидтрифосфата, 10 мМ MgCl2, 8 мМ дитиотрейтола, стабилизаторы и усилители ферментов;
3. Реактив Amp 2«SFTSV» - содержит олигонуклеотидные праймеры и флуоресцентно-меченые олигонуклеотидные зонды к РНК вируса В. dabieense и к ВКО;
4. К+ «SFTSV» - положительный контроль этапа ПЦР. Представляет из себя смесь двух плазмидных ДНК: плазмиды, содержащей фрагмент вируса В. dabieense и плазмиды, содержащей фрагмент ВКО, с которых, при помощи реактива Amp 2«SFTSV», амплифицируются специфические фрагменты;
5. К- - отрицательный контроль этапа ПЦР. Представляет из себя дистиллированную воду;
6. ПКО «SFTSV» - положительный контрольный образец этапа экстракции и обратной транскрипции нуклеиновых кислот. Представляет из себя псевдовирусные частицы на основе MS2-фага, содержащие РНК с фрагментом РНК вируса В. dabieense, которые после выделения и обратной транскрипции в кДНК, амплифицируются при помощи реактива Amp 2«SFTSV»;
7. ОКО - отрицательный контрольный образец этапа экстракции нуклеиновых кислот. Представляет из себя дистиллированную воду;
8. ВКО - внутренний контрольный образец этапа экстракции представляет из себя плазмиду, содержащую фрагмент ВКО, которая амплифицируются при помощи реактива Amp 2«SFTSV».
Реактивы ПКО, ВКО и ОКО хранятся при температуре +4°С, остальные хранятся при температуре от -22°С до -18°С.
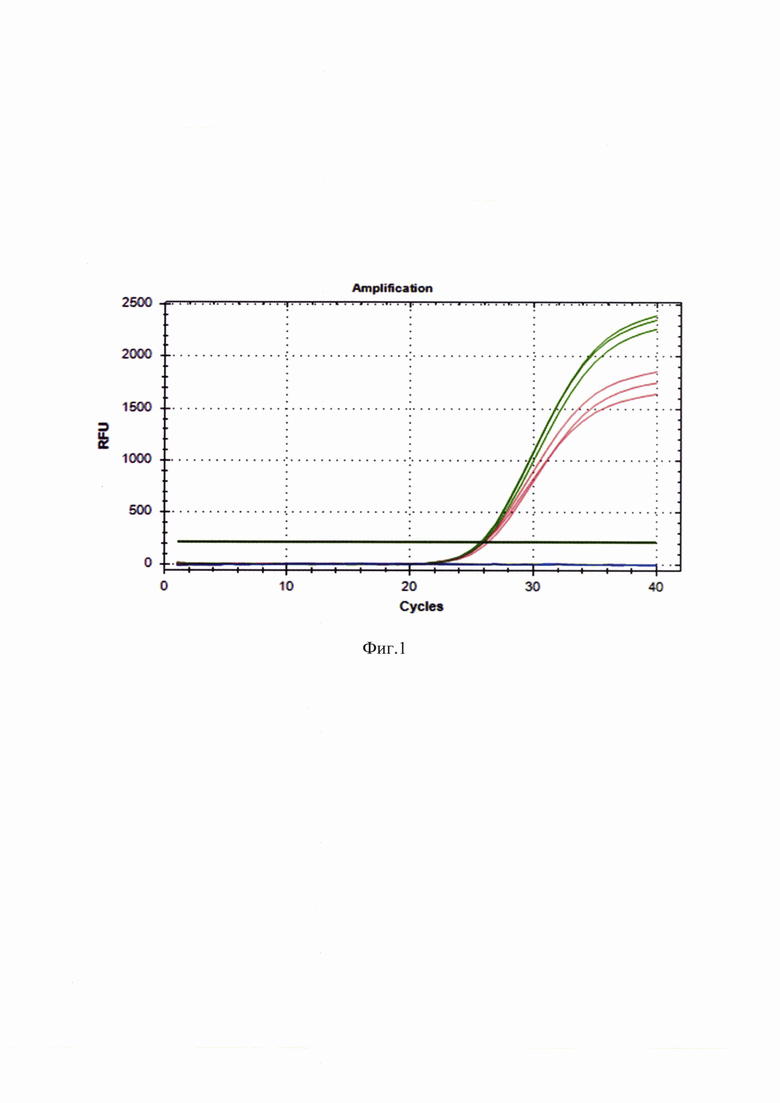
Для постановки реакции ОТ-ПЦР в реальном времени нужно взять необходимое количество микропробирок объемом 0,2 мл, соответствующее числу исследуемых проб; плюс четыре пробирки для положительных и отрицательных контролей. Подготовить ПЦР-смесь из расчета на одну реакцию: 1 мкл реактив Amp 1RT; 12,5 мкл реактив Amp 1 В; 1,5 мкл реактив Amp 2«SFTSV». Смесь вортексируют и осаждают на настольной центрифуге. По 15 мкл полученной смеси вносят в пробирки, затем добавляют в каждую по 10 мкл РНК-пробы, экстрагированной из исследуемого материала. Готовят 4 контрольные реакции. Для этого в пробирку для положительного контроля ПЦР вносят 10 мкл К+ «SFTSV», в пробирку для положительного контроля обратной транскрипции вносят 10 мкл образца, экстрагированного из ПКО «SFTSV». В пробирку для отрицательного контроля ПЦР вносят 10 мкл К-, в пробирку для отрицательного контроля экстракции вносят 10 мкл пробы, экстрагированной из ОКО. Конечный объем реакционной смеси составляет 25 мкл. Микропробирки переносят в программируемый амплификатор с функцией детекции флуоресценции в режиме «реального времени» по каналам (FAM /Green и JOE/HEX/Yellow). Режим амплификации представлен в таблице 3.
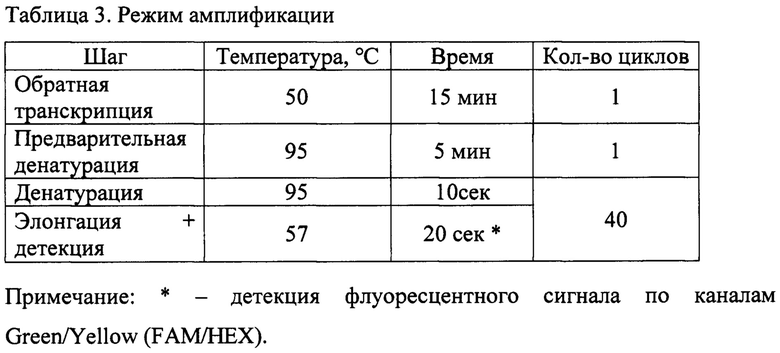
Детекцию флуоресцентного сигнала производят при 57°С по каналам Green (FAM) и Yellow (JOE/HEX). Учет и анализ результатов проводят с помощью программного обеспечения прибора на основании отсутствия или наличия сигнала флуоресценции в исследуемых пробах по детектируемым каналам. Результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию (или отсутствию) значения порогового цикла Ct в соответствующей графе в таблице результатов. Результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. Накопление флуоресцентного сигнала по каналу HEX свидетельствует о наличии в исследуемом материале РНК вируса В. dabieense. При этом результат считается валидным, если параллельно в пробе по каналу FAM регистрируется накопление флуоресцентного сигнала ВКО, а в образцах К+ «SFTSV» и ПКО «SFTSV» - по каналу HEX. Сигнал по каналу HEX в образцах К- и ОКО должен отсутствовать.
Таким образом, заявляемый способ выявления позволяет достоверно выявлять РНК вируса В. dabieense.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ выявления РНК вируса Bandavirus dabieense (SFTSV)
методом ОТ-ПЦР в реальном времени.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-05-03">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0000</ApplicationNumberText>
<FilingDate>2024-04-23</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>0000</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера» Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека</ApplicantName>
<ApplicantNameLatin>Department of Epidemiology, Pasteur Institute,
Federal Service on Consumers' Rights Protection and Human
Well-Being Surveillance</ApplicantNameLatin>
<InventorName languageCode="ru">Попова Маргарита Руслановна
</InventorName>
<InventorNameLatin>Popova Margarita Ruslanovna</InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления РНК вируса
Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном
времени</InventionTitle>
<InventionTitle languageCode="en">Method for detecting virus
Bandavirus dabieense e RNA by real-time RT-PCR </InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agatgracttggaagtgctttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaagtcatgattgatcttatatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Non-fluorescent Black Hole Quencher 1
(2-[N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4-methyl-2-nitrophen
yl)diazenyl]phenyl]diazenyl]anilino]ethanol) at 3'
end</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q8">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl]-N-ethyl-6-(ethylamino)-2
7-dimethyl-3H-xanthen-3-iminium chloride) fluorophore at 5'
end</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataaacgtggagaatgggctgtctcgttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2834908C1 |
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822161C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
| Способ выявления РНК модифицированного вакцинного полиовируса типа 2 (nOPV2) методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2795703C1 |
| Способ выявления вируса кори методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822430C1 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ВИРУСА ОБЕЗЬЯНЬЕЙ ОСПЫ ВИДА MONKEYPOX МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ (MPX AMP PS) | 2023 |
|
RU2803898C1 |
| Способ выявления вируса клещевого энцефалита методом ОТ-ПЦР в реальном времени | 2019 |
|
RU2744187C1 |
Изобретение относится к медицинской биотехнологии. Предложен способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени. Способ включает экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участку гена L, кодирующего РНК-зависимую РНК-полимеразу вируса В. dabieense. В качестве прямого и обратного праймеров используются последовательности SFTSV-F (SEQ ID NO.1) aga tgr act tgg aag tgc ttt g; SFTSV-R (SEQ ID NO.2) tga agt cat gat tga tct tat atg с, а в качестве зонда SFTSV-P (SEQ ID NO.3) R6G - ata aac gtg gag aat ggg ctg tct cgt tt - BHQ1. Способ решает проблему выявления генетических маркеров данного вируса и позволяет проводить мониторинг и диагностику вируса В. dabieense у здоровых, больных и лиц с подозрением на лихорадку с тромбоцитопеническим синдромом и у людей, болеющих лихорадкой с тромбоцитопеническим синдромом. 1 ил., 3 табл.
Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени, включающий экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участку гена L, кодирующего РНК-зависимую РНК-полимеразу вируса В. dabieense, отличающийся тем, что в качестве прямого и обратного праймеров используются последовательности
SFTSV-F (SEQ ID NO.1) aga tgr act tgg aag tgc ttt g
SFTSV-R (SEQ ID NO.2) tga agt cat gat tga tct tat atg с,
а в качестве зонда
SFTSV-P (SEQ ID NO.3) R6G - ata aac gtg gag aat ggg ctg tct cgt tt - BHQ1.
| KR 102510442 B1, 15.03.2023 | |||
| CN 102618669 B, 29.01.2014 | |||
| CN 103320524 A, 25.09.2013 | |||
| CN 103320524 B, 06.05.2015 | |||
| KR 102019804 B1, 04.11.2019 | |||
| CN 102618669 A, 01.08.2012 | |||
| Сизикова Т.Е | |||
| и др., Молекулярная эволюция вируса бандаби Даби (Phenuiviridae: Bandavirus: Dabie bandavirus) - возбудителя острой лихорадки с тромбоцитопеническим синдромом, |
Авторы
Даты
2024-12-05—Публикация
2024-05-28—Подача