Изобретение относится к биотехнологии и медицине, а именно к мониторингу и диагностике вируса геморрагической лихорадки Крым-Конго (CCHF) у здоровых, больных и лиц с подозрением на геморрагическую лихорадку, вызванную вирусом CCHF.
Вирус геморрагической лихорадки Крым-Конго (CCHFV=Crimean-Congo Hemorrhage Fever Virus) является возбудителем одноименной лихорадки, которая является широко распространенным заболеванием, охватывающим территории от восточного Китая до юга России и Черноморского региона, до южной оконечности Африки (ECDC Technical Report, 2023). По современной классификации вирус относится к порядку Bunyavirales, семейству Nairoviridae, к роду Orthonairovirus и имеет, по новой классификации, латинское название Orthonairovirus haemorrhagiae (https://ictv.global).
Заболевание было впервые описано в 1944 году на территории Крымского полуострова экспедицией советских врачей и получило название «Крымская Геморрагическая лихорадка». В 1967 году были выпущены статьи (Simpson et al. 1967; Woodall et al. 1967), которые описывали новый вирус с названием «вирус Конго» (Ergonul О., 2006). В 1968 году было предложено принятое в международном научном сообществе название «Вирус Крым-Конго Геморрагической Лихорадки» (Онищенко Г.Г., Куличенко А.Н., 2018).
По данным ВОЗ (WHO) и Европейского центра по контролю заболеваний (ECDC) в эндемичных регионах в Африке, на Балканах, на Ближнем Востоке и в азиатских странах к югу от 50-й параллели северной широты уровень инфицирования колеблется от 10,000 до 15,000 человек в год (в зависимости от статистики страны). У 88% заразившихся наблюдается субклиническая форма заболевания и для каждого 8-ого - развивается тяжелая форма болезни. Смертельный исход для тяжелобольных пациентов составляет от 10 до 40%, что по оценочным данным составляет от 50 до 750 смертельных исходов в год (WHO, 2023; ECDC, 2023). В 2023 году была зафиксирована крупная вспышка геморрагической лихорадки в Иране, из 511 подтвержденных случаев, 65 закончились летальным исходом (AtwanZ., Alhilfi R., et al. 2023).
CCHF вирус является зоонозом, с ежегодно увеличивающейся географией распространения, и циркулирующим в природе в энзоотическом состоянии. Природным резервуаром вируса являются клещи, преимущественно, семейства Ixodidae (16 видов) рода Hyalomma, которые считаются основными переносчиками инфекции человеку, также есть данные по виду Argas {Persicargas) persicus (семейство Argasidae) (Hoogstraal H., et al., 1979). Серологические данные обнаруживают наличие CCHF вируса у широкого круга видов домашних и диких животных: от кроликов до крупного рогатого скота, страусов и черепах (Spengler, Bergeron, et al. 2016 & Kar S., et al. 2020). Однако клинические симптомы развиваются только в людей и могут иметь различные исходы: от бессимптомных и легких инфекций до тяжелых и смертельных случаев (Ergonul О., 2006). Вирус может передаваться при укусе зараженного клеща, при прямом контакте с кровью или тканями зараженным животных, главным образом домашнего скота. Таким образом, риск заражения существует для людей, проводящих много времени на открытом воздухе (например, солдаты, фермеры, работники лесного хозяйства, охотники и туристы) и тех, кто находится в постоянном контакте с домашним скотом (например, пастухи, фермеры, мясники, работники скотобоен и ветеринары) (Aslam S., Latif MS., et al. 2016).
Вирус геморрагической лихорадки Крым-Конго представляет собой вирион сферической формы диаметром 80-120 нм с гетеродимерными шипами из гликопротеинов GN и GC на поверхности. Изолированный рибонуклеопротеиновые (RNP) комплексы состоят из отдельных сегментов геномной одноцепочечной (-) РНК. Комплексы RNP связаны с большим (L) белком (ICTV Virus Taxonomy Profile). Геном состоит из 3-х сегментов: S, М и L сегментов. S-сегмент, схожий по размерам с другими представителями Bunyaviruses, обладает размером ~1,6 kb и кодирует вирусный нуклеопротеиновый белок (NP - nucleoprotein) и малый неструктурный протеин (NSs - non-structural protein) (Zivcec M., Scholte F.E.M, et al. 2016). NP взаимодействует с РНК, формируя рибонуклеопротеиновый комплекс, а также обладает эндонуклеазной активностью, взаимодействует с белками теплового шока хозяина во время внутриклеточной репликации вируса и способствует транскрипции вирусной мРНК. М-сегмент (~5.4 kb) и L-сегмент (~12.1 kb) значительно больше, чем у других Bunyaviruses и содержат по одному гену. В М-сегменте зашифрован гликопротеиновый предшественник (GPC - glycoprotein precursor), влияющий на вирусную сборку, в L-сегменте - L-белок, содержащий вирусную РНК-зависимую РНК-полимеразу (Hoogstraal Н., et al. 1979).
Одной из особенностей вируса является высокая степень разнообразия последовательностей, с расхождением 20%, 31% и 22% для S, М и L сегментов среди данных GenBank. На основе филогенетического анализа вирусного S-сегмента было идентифицировано 7 линий (клад) (Deyde V., Khristova М., et al. 2006), которые коррелируют с географическим распространением вируса. Филогенетический анализ по М и L сегментам разделяется на 9 и 6 линий соответственно (Deyde V., Khristova М., et al. 2006; Hewson R., 2007). Реассортация и рекомбинация сегментов, происходящая при одновременном заражении переносчика разными вирусными штаммами, принадлежащими к разным линиям, приводит к формированию новых генетических вариантов. В 2022 году Ставропольским противочумным институтом Роспотребнадзора были опубликованы данные о новой линии вируса - Europe 3 на территории Ставропольского края (Volynkina A., et al. 2022).
Отсутствие других млекопитающих, кроме человека и новорожденных мышей, восприимчивых к инфицированию CCHFV, создает трудность в разработке подходящей животной модели для исследования вируса и создания вакцины (Büşra A., Bayram А., 2023). В настоящее время всемирно признанной вакцины против CCHFV не существует, однако есть ряд вакцин-кандидатов, находящиеся на разных этапах клинических испытаний (Büşra A., Bayram А., 2023).
Для диагностики CCHFV существуют разнообразные лабораторные тест-системы, однако анализы пациентов с подозрением на CCHF, в первую очередь тестируются с использованием RT-PCR, поскольку этот метод обеспечивает наибольшую чувствительность обнаружения вируса, особенно на ранних этапах. Выявление может быть осложнено высоким разнообразием штаммов CCHFV, поскольку тест-системы основаны на консервативных последовательностях для обнаружения. Для облегчения сбора и обработки биоматериала при диагностике CCHFV наиболее предпочтительны образцы мочи и слюны. Молекулярная диагностика подразумевает экстракцию РНК вируса из образцов с последующим проведением полимеразной цепной реакции с обратной транскриптазой для целевых фрагментов РНК с использованием набора праймеров и флуоресцентного зонда.
В настоящее время на территории РФ существует 1 зарегистрированный набор для выявления вируса Крым-Конго геморрагической лихорадки (CCHFV) - Ампли-Сенс® CCHFV-FL (Регистрационный номер РУ № ФСР 2012/12997; ФБУН ЦНИИ Эпидемиологии Роспотребнадзора). На основании новых данных генотипирования вируса были созданы индивидуальные праймеры для линии Europe 3.
Из-за высокого уровня смертности патоген представляет проблему общественного здравоохранения. Для того, чтобы сдержать распространение болезни, требуются инструменты для своевременной диагностики заболевших, а также для выявления носителей вируса Геморрагической лихорадки Крым-Конго еще до появления первых симптомов.
Целью создания данного изобретения является расширение арсенала средств эпидемиологического надзора для мониторинга носительства и распространения вируса геморрагической лихорадки Крым-Конго.
Технический результат - разработка высокочувствительного способа идентификации РНК вируса геморрагической лихорадки Крым-Конго в биологических образцах.
Поставленная задача решалась путем:
- конструирования диагностических праймеров и флуоресцентно-меченого зонда на специфические для CCHFV кДНК фрагменты, полученные с РНК матрицы;
- конструирования рекомбинантной плазмидной ДНК и MS2-фага, несущих специфический участок РНК-матрицы;
- оптимизации концентраций компонентов реакционной смеси и условий проведения ПНР.
Авторами предложен способ выявления вируса геморрагической лихорадки Крым-Конго, согласно которому выделенную из биологических образцов РНК анализируют в одной пробирке при помощи реакции обратной транскрипции и дальнейшей полимеразной цепной реакции, при помощи олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участкам S-сегмента, кодирующего нуклеопротеиновый белок вируса CCHFV.
Анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию или отсутствию значения порогового цикла Ct, причем результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
На начальном этапе были подобраны и синтезированы пара специфических олигонуклеотидных праймеров и зонд для гибридизационно-флуоресцентной детекции продуктов ПЦР. Для этого в базе данных GenBank (https://www.ncbi.nlm.nih.gov/) был выбран наиболее консервативный участок генома CCHFV. Были проанализированы все имеющиеся в базе данных последовательности. Также были подобраны праймеры и флуоресцентный зонд для внутреннего контрольного образца ПЦР. Последовательности олигонуклеотидных праймеров и флуоресцентного зонда представлены в Таблице 1 и Таблице 2.


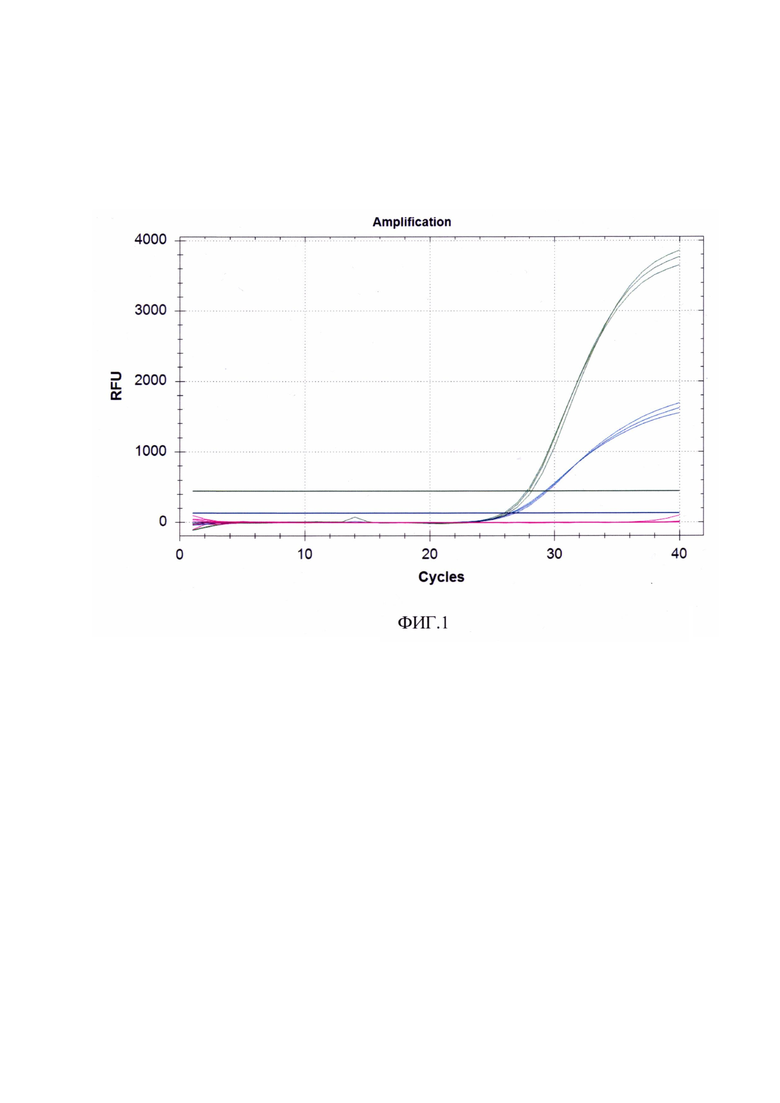
Сущность изобретения поясняется чертежом, где на Фиг. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции при анализе контрольных образцов. Кривые зеленого цвета (канал HEX) выше пороговой линии отображает положительный результат на выявление в образце РНК вируса Геморрагической лихорадки Крым-Конго, кривые розового цвета ниже пороговой линии отображает отрицательные контроли, не содержащие РНК данного патогена. Кривые синего цвета (канад FAM) выше пороговой линии отображают положительный результат для последовательности внутреннего контроля, подтверждающего качество прохождения полимеразной цепной реакции.
Подбор и анализ свойств олигонуклеотидных праймеров и флуоресцентного зонда проводился с использованием программного обеспечения Oligonucleotide Properties Calculator и MFold.
Для контроля качества прохождения этапов обратной транскрипции и ПЦР в состав методики были введены рекомбинантные положительные контрольные образцы K+ и ПКО, а также внутренний контрольный образец - ВКО. Матрицу для создания рекомбинантных положительных контрольных образцов получали синтетическим методом на основе ампликона, включающего в себя диагностическую область-мишень и фланкирующие последовательности нуклеотидов. Ампликон получали методом ПЦР в один шаг. Конечный ПЦР-продукт лигировали в плазмидный вектор pGEM-T («Promega», USA) под контролем промотора Т7 РНК полимеразы и трансформировали им Escherichia coli (штамм XL1-Blue). Рекомбинантные плазмиды из индивидуальных клонов проверяли на правильность ориентации целевой последовательности и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом секвенирования по Сэнгеру с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500x1 («Applied Biosystems», США).
Соответствующие заданным критериям рекомбинантные плазмиды использовали для приготовления положительного контрольного образца этапа ПЦР (К+). Для этого определяли концентрацию ДНК в растворе рекомбинантной плазмиды и разводили в 1х ТЕ-буфере (рН 8.0) со стабилизаторами натрия азида 0,05% и ДНК спермы лосося 0,02 мг/мл до рабочей концентрации 1*107 копий/мл. Также, плазмиды использовались для получения рекомбинантного РНК-содержащего положительного контрольного образца (ПКО) с защитной белковой оболочкой MS2-фага. Для полученного продукта производили определение концентрации методом цифровой капельной ПЦР (ddPCR) («BioRad», USA). Рабочая концентрации составила 1*107 копий/мл, которую использовали в качестве ПКО в дальнейшем анализе.
Оценку предела обнаружения метода производили на разведениях ПКО, из которых экстрагировали РНК с помощью набора для выделения нуклеиновых кислот «Рибо-Преп» (ФБУН ЦНИИ Эпидемиологии, Россия), а затем с помощью специфических праймеров и флуоресцентных зондов определяли минимальное разведение, детектируемое как положительное в 100% случаев. Определенная таким образом аналитическая чувствительность метода составила 1*103 копий/мл.
Свойство изобретения специфически определять РНК вируса геморрагической лихорадки Крым-Конго достигнуто путем подбора праймеров и зондов к высококонсервативным фрагментам РНК вируса, что минимизирует возможность перекрестных реакций с нуклеиновыми кислотами человека, близкородственных микроорганизмов, других инфекционных возбудителей.
Аналитическую специфичность оценивали при помощи тестирования образцов нуклеиновых кислот человека и следующих вирусов: вируса кори, вируса клещевого энцефалита, вируса парагриппа человека типа 4b, риновируса человека 17 типа, коронавируса SARS-CoV-2, аденовируса человека 6 типа, вируса Коксаки человека типа В1, эховируса человека 4 типа (вирус ECHO 4 типа), парэховируса человека 1 типа, вируса герпеса человека 5 типа, вируса эпидемического паротита, вируса краснухи. Неспецифические реакции отсутствуют.
Таким образом, в результате проведенных исследований был разработан и апробирован способ выявления РНК вируса Геморрагической лихорадки Крым-Конго в различных видах биологического материала. Технический результат достигается путем определения РНК Геморрагической лихорадки Крым-Конго, включающим выделение РНК исследуемой пробы, проведение обратной транскрипции и ПЦР с учетом результатов в режиме реального времени, согласно изобретению.
Подготовку проб проводили согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Материалом для исследования могут служить клинические и биологические образцы.
Экстракция производится из 100 мкл полученной суспензии образца с лизирующим буфером на основе 6 моль гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения РНК, и последующим инкубированием 5 минут при температуре (65±1)°С. Выделение РНК осуществляют с помощью наборов «Рибо-преп» и «Рибо-Сорб» (ФБУН ЦНИИ Эпидемиологии, Россия) в соответствии с инструкциями к наборам. Во все пробирки, включая контроль выделения, до этапа прогревания в лизирующем буфере добавляют 10 мкл ВКО. После выделения РНК приступают к постановке ОТ-ПЦР.
Для упрощения подготовки и стандартизации выполнения анализа в тест-систему входят 8 реактивов:
1) Реактив Amp 1RT - буферный раствор, содержащий фермент для обратной транскрипции. Представляет из себя буферный раствор, содержащий 50 мМ Трис-HCl, рН 8,0 (при 25°С), 100 мМ NaCl, 1 мМ ЭДТА, 5 мМ дитиотреитол, 50% (v/v) глицерин и 0,1% (v/v) NP-40, ингибитор РНКаз, M-MuLV -RH ревертазу и HS-Taq ДНК-полимеразу;
2) Реактив Amp 1В - буферный раствор, содержащий 100 мМ Трис-HCl, рН 8,3 (при 25°С), 150 мМ KCl, 0,6 мМ каждого дезоксинуклеозидтрифосфата, 10 мМ MgCl2, 8 мМ дитиотрейтола, стабилизаторы и усилители ферментов;
3) Реактив Amp 2 "CCHFV" - содержит олигонуклеотидные праймеры и флуоресцентно-меченые олигонуклеотидные зонды к РНК вируса Геморрагической лихорадки Крым-Конго и к ВКО;
4) К+ "CCHFV" - положительный контроль этапа ПЦР. Представляет из себя смесь двух плазмидных ДНК: плазмиды, содержащей фрагмент вируса Геморрагической лихорадки Крым-Конго и плазмиды, содержащей фрагмент ВКО, с которых, при помощи реактива Amp2, амплифицируются специфические фрагменты;
5) К- - отрицательный контроль этапа ПЦР. Представляет из себя дистиллированную воду;
6) ПКО "CCHFV" - положительный контрольный образец этапа экстракции и обратной транскрипции нуклеиновых кислот. Представляет из себя псевдовирусные частицы на основе MS2-фага, содержащие РНК с фрагментом РНК вируса Геморрагической лихорадки Крым-Конго, которые после выделения и обратной транскрипции в кДНК, амплифицируются при помощи реактива Amp 2;
7) ОКО - отрицательный контрольный образец этапа экстракции нуклеиновых кислот. Представляет из себя дистиллированную воду;
8) ВКО - внутренний контрольный образец этапа экстракции представляет из себя плазмиду, содержащую фрагмент ВКО, которая амплифицируются при помощи реактива Amp 2 "CCHFV".
Реактивы ПКО, ВКО и ОКО хранятся при температуре +4°С, остальные хранятся при температуре от -22°С до -18°С.
Для постановки реакции ОТ-ПЦР в реальном времени нужно взять необходимое количество микропробирок объемом 0,2 мл, соответствующее числу исследуемых проб; плюс четыре пробирки для положительных и отрицательных контролей. Подготовить ПЦР-смесь из расчета на одну реакцию: 1 мкл реактив Amp 1RT, 12,5 мкл реактив Amp 1В, 1,5 мкл реактив Amp2. Смесь вортексируют и осаждают на настольной центрифуге. По 15 мкл полученной смеси вносят в пробирки, затем добавляют в каждую по 10 мкл РНК-пробы, экстрагированной из исследуемого материала. Готовят 4 контрольные реакции. Для этого в пробирку для положительного контроля ПЦР вносят 10 мкл К+, в пробирку для положительного контроля обратной транскрипции вносят 10 мкл образца, экстрагированного из ПКО. В пробирку для отрицательного контроля ПЦР вносят 10 мкл К-, в пробирку для отрицательного контроля экстракции вносят 10 мкл пробы, экстрагированной из ОКО. Конечный объем реакционной смеси составляет 25 мкл. Микропробирки переносят в программируемый амплификатор с функцией детекции флуоресценции в режиме «реального времени» по каналам (FAM/SybrGreen/Green и JOE/HEX). Режим амплификации представлен в таблице 3.
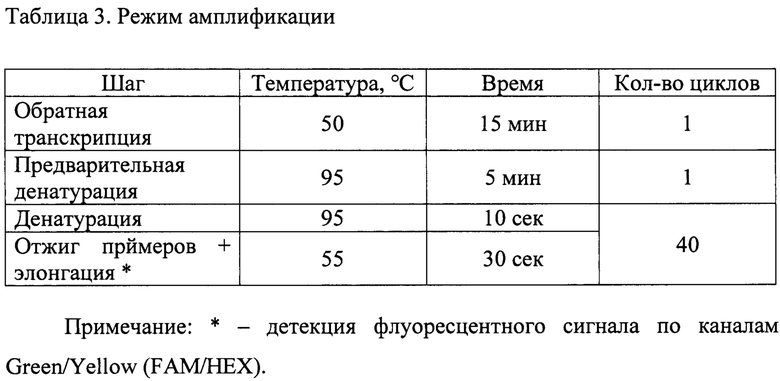
Детекцию флуоресцентного сигнала производят при 60°С по каналам Green (FAM) и Yellow (JOE/HEX). Учет и анализ результатов проводят с помощью программного обеспечения прибора на основании отсутствия или наличия сигнала флуоресценции в исследуемых пробах по детектируемым каналам. Результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию (или отсутствию) значения порогового цикла (Ct) в соответствующей графе в таблице результатов. Результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. Накопление флуоресцентного сигнала по каналу HEX свидетельствует о наличии в исследуемом материале РНК CCHFV. При этом результат считается валидным, если параллельно в пробе по каналу FAM регистрируется накопление флуоресцентного сигнала ВКО, а в образцах К+ и ПКО - по каналу HEX. Сигнал по каналу HEX в образцах К- и ОКО должен отсутствовать.
Таким образом, заявляемый способ выявления позволяет достоверно выявлять РНК вируса геморрагической лихорадки Крым-Конго в клинических пробах.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-11-27"
softwareVersion="2.3.0" softwareName="WIPO Sequence" fileName="Способ
выявления РНК вируса Геморрагической лихорадки Крым-Конго методом
ОТ-ПЦР в реальном времени.xml" dtdVersion="V1_3"
nonEnglishFreeTextLanguageCode="ru"
originalFreeTextLanguageCode="en">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText/>
<FilingDate/>
</ApplicationIdentification>
<ApplicantFileReference/>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека»</ApplicantName>
<ApplicantNameLatin>Department of Epidemiology, Pasteur Institute,
Federal Service on Consumers' Rights Protection and Human Well-Being
Surveillance</ApplicantNameLatin>
<InventorName languageCode="ru">Туренко Анастасия
Сергеевна</InventorName>
<InventorNameLatin>Anastasiia Turenko</InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления РНК вируса
Геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном
времени.</InventionTitle>
<InventionTitle languageCode="en">Detecting method Crimean-Congo
Hemorrhagic Fever Virus RNA using real-time PCR.</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcggcactagttgaagccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgtagctttcgtcccaagattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Non-fluorescent Black Hole Quencher 1
(2-[N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4- methyl-2-
nitrophenyl)diazenyl]phenyl]diazenyl]anilino]ethanol) at
3'-end</INSDQualifier_value>
<NonEnglishQualifier_value>Non-fluorescent Black Hole Quencher 1
(2-[N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4- methyl-2-
nitrophenyl)diazenyl]phenyl]diazenyl]anilino]ethanol) at
3'-end</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q8">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl]-N-ethyl-6-(ethylamino)-2
7-dimethyl-3H-xanthen-3- iminium chloride) fluorophore at 5'
end</INSDQualifier_value>
<NonEnglishQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl]-N-ethyl-6-(ethylamino)-2
7-dimethyl-3H-xanthen-3- iminium chloride) fluorophore at 5'
end</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcccatatatgagtgtgcctggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |
| Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822161C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления вируса кори методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822430C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
| СПОСОБ ВЫЯВЛЕНИЯ ВИРУСА ОБЕЗЬЯНЬЕЙ ОСПЫ ВИДА MONKEYPOX МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ (MPX AMP PS) | 2023 |
|
RU2803898C1 |
| Способ выявления вируса Mammarenavirus machupoense методом DETECTR с изотермической амплификацией | 2024 |
|
RU2832917C1 |
| Способ выявления РНК модифицированного вакцинного полиовируса типа 2 (nOPV2) методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2795703C1 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
Изобретение относится к биотехнологии и медицине. Заявлен способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени, включающий экстракцию РНК из биологических образцов с последующим проведением двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участку гена сегмента S, кодирующего белок нуклеокапсида вируса геморрагической лихорадки Крым-Конго, отличающийся тем, что в качестве прямого праймера используют последовательность - atc ggc act agt tga agc cac, в качестве обратного праймера - gtg tag ctt tcg tcc caa gat tt, в качестве зонда - cgc cca tat atg agt gtg cct ggg с флуоресцирующей меткой R6G на 5'-конце и с нефлуоресцирующим гасителем BHQ1 на 3'-конце. Изобретение позволяет осуществлять мониторинг и диагностику вируса геморрагической лихорадки Крым-Конго у здоровых, больных и лиц с подозрением на геморрагическую лихорадку, вызванную вирусом, и эпидемиологический мониторинг. 1 ил., 3 табл.
Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени, включающий экстракцию РНК из биологических образцов с последующим проведением двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно-меченого зонда, комплементарных участку гена сегмента S, кодирующего белок нуклеокапсида вируса геморрагической лихорадки Крым-Конго, отличающийся тем, что в качестве прямого праймера используют последовательность - atc ggc act agt tga agc cac, в качестве обратного праймера - gtg tag ctt tcg tcc caa gat tt, в качестве зонда - cgc cca tat atg agt gtg cct ggg с флуоресцирующей меткой R6G на 5'-конце и с нефлуоресцирующим гасителем BHQ1 на 3'-конце.
| Е.В.Найденова, И.Н.Шарова и др., ТЕСТ-СИСТЕМА ДЛЯ ВЫЯВЛЕНИЯ РНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ МЕТОДОМ ОТ-ПЦР, Проблемы особо опасных инфекций, вып | |||
| Автоматический огнетушитель | 0 |
|
SU92A1 |
| Mehmet Yapar, Hakan Aydogan et al., Rapid and quantitative detection of Crimean-Congo hemorrhagic fever virus by one-step real-time reverse transcriptase-PCR, | |||
Авторы
Даты
2025-02-17—Публикация
2024-04-27—Подача