Изобретение относится к биотехнологии и медицине, а именно к диагностике инфекционных заболеваний, в частности к проблеме выявления генетических маркеров вируса кори (Measles virus).
Обычное течение заболевания сопровождается температурой, покраснением глаз и сыпью. Часто (примерно в трети случаев) возникают осложнения, такие как конъюнктивит, воспаление среднего уха и диарея. К наиболее тяжелым осложнениям относятся пневмония, дегидратация, вызванная тяжелой диареей, слепота, энцефалит, дистрофическое поражение мозга. Наиболее подвержены риску осложнений дети до 5 лет, и взрослые старше 20 лет, а также лица, с ослабленной иммунной системой.
В случае инфицирования беременной женщины, возникает высокий риск перинатальной гибели или пороков развития плода. При заражении на поздних сроках беременности возможно рождение ребенка с симптомами инфекции. В таких случаях смертность достигает 30%.
Наиболее эффективным способом борьбы с корью является обязательная вакцинация населения. Взрослым рекомендуется приходить ревакцинацию каждые 10 лет. Тем не менее, из-за чрезвычайно высокой контагиозности заболевания, существует постоянная угроза вспышки. Это обуславливает необходимость разработки и улучшения методов для быстрого и точно обнаружения вируса.
Целью создания предлагаемого нами метода является расширение арсенала средств, используемых для обнаружения вируса кори. Технической задачей изобретения являлась разработка высокочувствительного способа обнаружения вируса кори, основанного на методе ОТ-ПЦР в режиме реального времени. Метод позволяет идентифицировать генетический материал Measles virus в биологических образцах и других вируссодержащих пробах (культуральная вируссодержащая жидкость, и т.д.).
Поставленная задача решалась путем: 1) конструирования диагностических праймеров и флуоресцентно-меченного зонда, соответствующих консервативным участкам гена белка L; 2) конструирования рекомбинантной плазмидной ДНК и М82-фага, несущих специфический участок ДНК- и РНК-матрицы; 3) оптимизации концентраций компонентов реакционной смеси и условий проведения ПНР.
Авторами предложен способ, согласно которому выделенную из биологических образцов (кровь и ее производные, мазок из носоглотки, моча) РНК анализируют с использованием метода двухстадийной реакции обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса кори с использованием смеси олигонуклеотидных праймеров и соответствующего флуоресцентно меченного зонда, комплементарных участку гена белка L вируса кори.
Анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию или отсутствию значения порогового цикла Ct, причем результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
На начальном этапе были подобрана и синтезирована 1 пара специфических олигонуклеотидных праймеров и зонд для гибридизационно-флуоресцентной детекции продуктов ПНР. Для этого в базе данных NCBI Mega BLAST (http://www.ncbi.nlm.nih.gov/) был выбран наиболее консервативный участок генома Measles virus. Были проанализированы все имеющиеся в базе данных последовательности. Также были подобраны праймеры и флуоресцентный зонд для ВКО в качестве внутреннего контроля ПЦР. Последовательности олигонуклеотидных праймеров и флуоресцентного зонда представлены в Таблице 1 и Таблице 2.

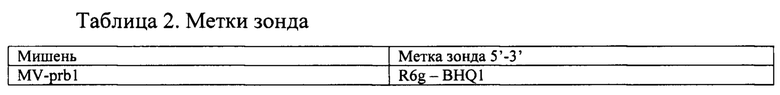
Подбор и анализ свойств олигонуклеотидных праймеров и флуоресцентного зонда проводился с использованием программного обеспечения Oligonucleotide Properties, Calculator и MFold.
Для контроля качества прохождения этапов обратной транскрипции и ПЦР в состав методики были введены рекомбинантные положительные контрольные образцы К+и ПКО и внутренний контрольный образец (ВКО). Матрицу для создания рекомбинантных положительных контрольных образцов получали синтетическим методом на основе ампликона, включающего в себя диагностическую область-мишень и фланкирующие последовательности нуклеотидов. Ампликон получали методом ПЦР в один шаг. Конечный ПЦР-продукт лигировали в плазмидный вектор pGEM-T («Promega», USA) под контролем промотора Т7 РНК полимеразы и трансформировали им Escherichia coli (штамм Turbo). Рекомбинантные плазмиды из индивидуальных клонов проверяли на правильность ориентации целевой последовательности и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом секвенирования по Сэнгеру с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500x1 («Applied Biosystems», США).
Соответствующие заданным критериям рекомбинантные плазмиды использовали для приготовления положительного контрольного образца этапа ПЦР (К+). Для этого определяли концентрацию ДНК в растворе рекомбинантной плазмиды и разводили стерильной водой до рабочей концентрации 1⋅107 копий/мл. Также данные рекомбинантные плазмиды использовались для получения рекомбинантного РНК-содержащего положительного контрольного образца с защитной белковой оболочкой MS2-фага (ПКО). Для полученного продукта также производили определение концентрации, затем разводили РНК-буфером (ФБУН ЦНИИ Эпидемиологии, Россия) до рабочей концентрации 1⋅107 копий/мл, которую использовали в качестве препарата ПКО.
Оценку аналитической чувствительности способа производили на разведениях РНК-содержащего рекомбинантного положительного контрольного образца (ПКО), из которых экстрагировали РНК с помощью набора для выделения нуклеиновых кислот «Рибо-Преп» (ФБУН ЦНИИ Эпидемиологии, Россия), а затем с помощью специфических праймеров и флуоресцентных зондов определяли минимальное разведение, детектируемое как положительное в 100% случаев. Определенная таким образом аналитическая чувствительность способа составила 2⋅102 копий/мл.
Аналитическую специфичность метода оценивали с помощью панели нуклеиновых кислот вирусов (таблица 3). В результате исследования перекрестных реакций не зафиксировано.
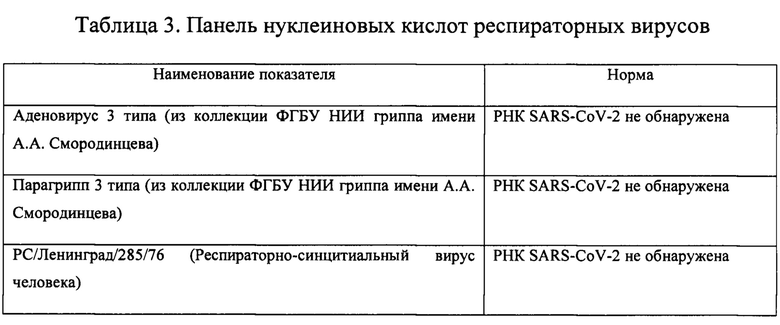

Таким образом, в результате проведенных исследований был разработан и апробирован способ выявления РНК вируса кори в различных видах биологического материала.
Технический результат, достигаемый при использовании изобретения - расширение арсенала средств, используемых для обнаружения вируса кори.
Технический результат достигается путем определения РНК вируса кори, включающим выделение РНК исследуемой пробы, проведение обратной транскрипции и ПЦР с учетом результатов в режиме реального времени, согласно изобретению.
Диагностика проводится следующим образом: Подготовку проб проводят согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Материалом для исследования могут служить: клинические и биологические образцы. Экстракция производится из 100 мкл полученной суспензии образца с лизирующим буфером на основе 6 моль гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения РНК, и последующим инкубированием 5 минут при температуре (65±1)°С. Выделение РНК осуществляют с помощью наборов «Рибо-Преп» и «Рибо-Сорб» (ФБУН ЦНИИ Эпидемиологии, Россия) в соответствии с инструкциями к наборам. Во все пробирки, включая контроль выделения, на этапе прогревания в лизирующем буфере добавляют 10 мкл внутреннего контрольного образца (ВКО).
После выделения РНК приступают к постановке ОТ-ПЦР. Для упрощения и стандартизации подготовки реактивов в тест-систему входят 8 смесей. Реактив AmplRT представляет из себя буферный раствор, содержащий 50 мМ Трис-HCl, рН 8.0 (при 25°С), 100 мМ NaCl, 1 мМ ЭДТА, 5 мМ дитиотреитол, 50% (v/v) глицерин и 0.1% (v/v) NP-40, ингибитор РНКаз, M-MuLV - RH ревертаза и HS-Taq ДНК-полимераза. Реактив AmplB представляет из себя буферный раствор, содержащий 100 мМ Трис-HCl, рН 8.3 (при 25°С), 150 мМ KСl, 0,6 мМ каждого дезоксинуклеозидтрифосфата, 10 мМ MgCl2, 8 мМ ДТТ, стабилизаторы и усилители ферментов. Реактив Атр2 содержит олигонуклеотидные праймеры и флуоресцентно-меченые олигонуклеотидные зонды к вирусу кори и к ВКО. K+ представляет собой смесь двух типов кДНК: к ДНК Measles virus и кДНК ВКО, содержащих специфическую последовательность нуклеотидов ДНК, амплифицируемую с участием реактива Аmр2. K - представляет собой дистиллированную стерильную воду. ПКО представляет собой псевдовирусные частицы на основе MS2 фага, содержащие фрагмент РНК вируса кори, амплифицируемый с участием Реактива Аmр2. ОКО - дистиллированная стерильная вода. ВКО представляет собой псевдовирусные частицы на основе MS2 фага, содержащие синтетическую последовательность РНК, амплифицируемую с участием праймеров к ВКО и детектируемую с участием зондов к ВКО в составе реактива Amp 2. Все реактивы, кроме ПКО и ВКО, хранятся при температуре -20°С. ПКО и ВКО хранятся при температуре +4°С.
Для постановки реакции ОТ-ПЦР в реальном времени отбирают необходимое количество пробирок объемом 0,2 мл, соответствующее числу исследуемых проб, а также четыре пробирки для положительных и отрицательных контролей. Далее по числу исследуемых проб смешивают реакционные смеси: 1 мкл реактив Amp1RT, 12,5 мкл реактив Amp1B, 1,5 мкл реактив Аmр2. По 15 мкл полученной смеси вносят в подготовленные пробирки, затем добавляют 10 мкл РНК-пробы, экстрагированной из исследуемого материала. Готовят 4 контрольные реакции. Для этого в пробирку для положительного контроля ПНР вносят 10 мкл K+, в пробирку для положительного контроля обратной транскрипции вносят 10 мкл образца, экстрагированного из ПКО. В пробирку для отрицательного контроля ПЦР вносят 10 мкл K-, в пробирку для отрицательного контроля экстракции вносят 10 мкл пробы, экстрагированной из ОКО. Конечный объем реакционной смеси составляет 25 мкл. Пробирки переносят в программируемый амплификатор с функцией амплификации в режиме «реального времени» с наличием 2 каналов детекции флуоресценции (FAM/Green и JOE/HEX/Yellow). Режим амплификации представлен в таблице 4.
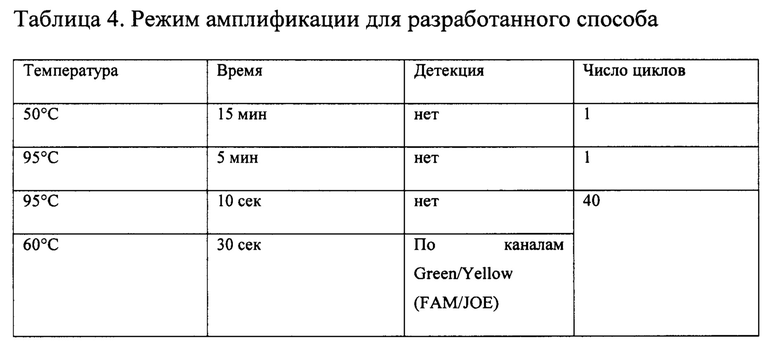
Детекцию флуоресцентного сигнала производят при 60°С по каналам Green (FAM) и Yellow (JOE/HEX). Учет и анализ результатов проводят с помощью программного обеспечения прибора на основании отсутствия или наличия сигнала флуоресценции в исследуемых пробах по детектируемым каналам. Результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию (или отсутствию) значения порогового цикла Ct в соответствующей графе в таблице результатов. Результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. Накопление флуоресцентного сигнала по каналу JOE свидетельствует о наличии в исследуемом материале РНК вируса кори. При этом результат считается валидным, если параллельно в пробе по каналу FAM регистрируется накопление флуоресцентного сигнала ВКО, а в образцах K+ и ПКО - по каналу JOE. Сигнал по каналу JOE в образцах K- и ОКО должен отсутствовать.
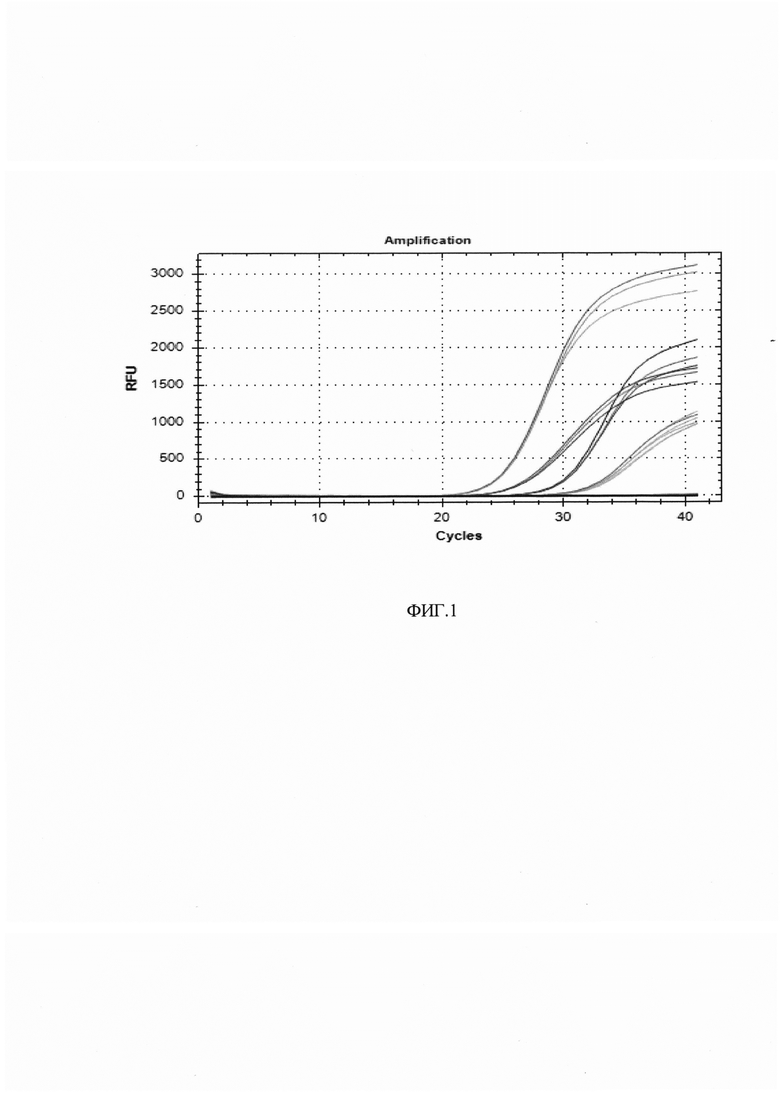
Сущность изобретения поясняется чертежом, где на ФИГ. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции при анализе образцов РНК вируса кори. Каждая кривая выше пороговой линии отображает положительный результат на выявление в образце РНК вируса кори, каждая кривая ниже пороговой линии отображает отрицательные контроли, не содержащие целевых участков РНК.
Таким образом, заявляемый метод позволяет достоверно выявлять РНК вируса кори в клинических пробах.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="21032023 MV.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2023-03-21">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>00000</ApplicationNumberText>
<FilingDate>2023-03-13</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>00000</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>20221218424</ApplicationNumberText>
<FilingDate>2022-10-31</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки "Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека"(ФБУН НИИ эпидемиологии и микробиологии имени
Пастера),ул.Мира,14,Санкт-Петербург,197101,Российская Федерация
</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur Institute
</ApplicantNameLatin>
<InventorName languageCode="ru">Дедков Владимир Георгиевич
</InventorName>
<InventorNameLatin>Dedkov Vladimir Gueorguievich</InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления вируса кори
методом ОТ-ПЦР в реальном времени .</InventionTitle>
<InventionTitle languageCode="en">A method for detecting Measles
virus by RT Real-Time PCR .</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctatagagcaggtgctgatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная последовательность
</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagcaatccatcttgccctgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl]-N-ethyl-6-(ethylamino)-2,7-dimethyl-3H-x
anthen-3-iminium chloride)</INSDQualifier_value>
<NonEnglishQualifier_value>Флуорофор Родамин 6Ж
(9-[2-(этоксикарбонил)фенил]-N-этил-6-(этиламино)-2,7-
диметил-3H-ксантен-3-иминия хлорид) на
5'-конце.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>>26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q6">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Non-fluorescent Black Hole Quencher 1
(2-[N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4-methyl-2-nitrophen
yl)diazenyl]phenyl]diazenyl]anilino]ethanol at 3' end
.)</INSDQualifier_value>
<NonEnglishQualifier_value>Не флуоресцентный тушитель Вlack
Hole Quеncher 1
(2-(N-(2-гидроксиэтил)-4-[[2-метокси-5-метил-4-[(4-метил-2-нитрофенил)
диазенил]фенил]диазенил]анилино]этанол) на 3'-конце
.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgcgggttggcaattaacggaccca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |
| Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822161C1 |
| Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2834908C1 |
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
| Способ выявления РНК модифицированного вакцинного полиовируса типа 2 (nOPV2) методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2795703C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ВИРУСА ОБЕЗЬЯНЬЕЙ ОСПЫ ВИДА MONKEYPOX МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ (MPX AMP PS) | 2023 |
|
RU2803898C1 |
| Способ выявления вируса клещевого энцефалита методом ОТ-ПЦР в реальном времени | 2019 |
|
RU2744187C1 |
Изобретение относится к области биотехнологии. Описан способ выявления РНК вируса кори методом ОТ-ПЦР в реальном времени. Он предусматривает экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции - обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса. Способ включает использование набора олигонуклеотидных праймеров и соответствующего флуоресцентно меченного зонда. Технический результат заключается в расширении арсенала средств, используемых для обнаружения вируса кори. 1 ил., 4 табл.
Способ выявления РНК вируса кори методом ОТ-ПЦР в реальном времени, предусматривающий экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции - обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома вируса, с использованием набора олигонуклеотидных праймеров и соответствующего флуоресцентно меченного зонда, включающего:
SEQ ID NO: 1 acctatagagcaggtgctgatc;
SEQ ID NO: 2 aagcaatccatcttgccctgag;
SEQ ID NO: 3 ttgcgggttggcaattaacggaccca,
комплементарных участку гена РНК-зависимой РНК полимеразы вируса кори, при этом зонд содержит метки R6G-BHQ1.
| НАБОР ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ ДЛЯ ИДЕНТИФИКАЦИИ ВИРУСА КРАСНУХИ В КЛИНИЧЕСКИХ ОБРАЗЦАХ | 2006 |
|
RU2317333C2 |
| US 20100119536 A1, 13.05.2010 | |||
| US 20060177849 A1, 10.08.2006. | |||
Авторы
Даты
2024-07-05—Публикация
2023-03-23—Подача