Изобретение относится к биотехнологии и медицине, а именно к диагностике инфекционных заболеваний, в частности к выявлению генетических маркеров вируса Mammarenavirus guanaritoense (GTOV).
GTOV - является одноцепочечным РНК-вирусом семейства Arenaviridae, вызывающим Венесуэльскую геморрагическую лихорадку (ВГЛ). Естественным резервуаром возбудителя в природе изначально считался камышовый короткохвостый хомячок Zygodontomys brevicauda - грызун, обитатель Центральной и Южной Америки. Однако, исследования показали, что данное животное является не единственным, восприимчивым к инфицированию GTOV. Вирус был также выделен у хлопковой крысы Олстона (S. alstoni) и хлопкового хомяка (S. hispidus). Обнаружена серопозитивность у рисовых хомяков (род. Oryzomys). Исследования распространенности ВГЛ в Венесуэле также продемонстрировали наличие вируса с серопозитивным течением инфекции у домашней крысы (R. rattus). В случае тесного контакта с человеком, R. Rattus становится важным источником инфекции путем контаминации почвы и сельскохозяйственных культур экскрементами, и другими биологическими жидкостями. С данным вирусом не связаны какие-либо значительные патологические изменения в популяции грызунов, нарушающих биоразнообразие.
GTOV демонстрирует специфический видовой тропизм, заражая естественным образом грызунов. Также было показано что GTOV экспериментально заражает морских свинок и зеленых мартышек. Данный спектр тропизма частично обусловлен тем фактом, что GTOV использует в качестве своих входных ворот, высоко консервативный среди млекопитающих рецептор трансферрина 1(TfR1).
Передача вируса происходит при контакте с биологическими жидкостями зараженного животного, через укусы или царапины инфицированных грызунов или путем вдыхания вируса и употребления загрязненных вирусом пищевых продуктов.
Факторы риска включают сельскохозяйственную деятельность в сезон дождей, с непосредственной близостью к инфицированным животным в эндемической местности. Мужчины входят в группу повышенного профессионального риска, в силу высокой степени занятости в вышеуказанной отрасли.
Первые случаи Венесуэльской геморрагической лихорадки (ВГЛ) были выявлены в конце 1980-х в венесуэльском штате Португеза и прилегающих к нему районах. У подтвержденных случаев, коэффициент летальности составил 60%. С сентября 1989 по декабрь 2006, было зарегистрировано 618 случаев ВГЛ. В 2021 г., министерством здравоохранения Боливарианской Республики Венесуэла было зафиксировано 36 случаев. Летальность составила 23.1%.
На сегодняшний день, коммерчески доступной вакцины не существует. Известен патент WO 2023175044, описывающий разработку потенциальной вакцины MOPEVACNEW. Однако, для подтверждения эффективности последней, требуются дополнительные клинические испытания.
Имеются очень редкие случаи передачи инфекции от человека к человеку. Чаще всего она происходит локально, в отдельных населенных пунктах, в основном в течение сезона дождей.
По прошествии инкубационного периода длительностью от 3 до 12 дней, на начальной стадии заболевания, наиболее распространенными симптомами ВГЛ являются резкий подъем температуры тела на фоне упадка сил, головная боль, упадок психической деятельности, миалгия, артралгия, кашель, боль в горле, тошнота, рвота, диарея, обезвоживание и сонливость. Отсутствие патогномонических признаков приводит к затруднению постановки диагноза на данном этапе.
На этапе геморрагической фазы проявления заболевания, наблюдаются носовое кровотечение, кровоточивость десен, гематемезис и мелена потенциально приводящие к тромбоцитопении и лейкопении. В тяжелых случаях, составляющих 25-30%, наблюдаются геморрагический шок, артериальная гипотензия и/или гиповолемический шок приводящие к геморрагическому застою, диффузному отеку легких, полиорганной недостаточности и летальному исходу.
Продолжительность заболевания в случаях, не связанных с летальным исходом, составляет около 10-14 дней. ВГЛ затрагивает все возрастные группы обоих полов. Самая высокая заболеваемость наблюдалась у лиц в возрасте от 15 до 44 лет.
В большинстве случаев, те кто выживает, обычно выздоравливают без осложнений. Однако, полное выздоровление требует длительной госпитализации.
ВГЛ поддается лечению лишь частично: Рибавирин является единственным противовирусным препаратом, доказавшим свою эффективность против аренавирусных инфекций на ранних стадиях. Применение препарата на поздних стадиях заболевания, приводит к незначительной положительной динамике.
Другой противовирусный препарат под названием «Фавипиравир», продемонстрировал широкий спектр активности in vitro, избирательно ингибируя РНК-зависимую РНК полимеразу GTOV, фермента участвующего в транскрипции и репликации вируса.
При симптоматическом лечении применяются; переливание цельной крови или ее компонентов (плазмы выздоровевших, концентратов тромбоцитов, фибриногена), прием витамина К, растворы для внутривенного введения, противовирусные препараты и оксигенотерапия.
Изначально, ВГЛ диагностировали методом выделения вируса GTOV из клеточных культур, который до сих пор остается золотым стандартом лабораторной диагностики геморрагических лихорадок (Silva-Ramos, С. R.,  С.,
С.,  A., & Rodas, J. D. (2022). An updated review and current challenges of Guanarito virus infection, Venezuelan hemorrhagic fever. Archives of virology, 167(9), 1727-1738. https://doi.org/10.1007/s00705-022-05453-3). Однако данный способ требует специальных условий проведения анализа в лаборатории с УББ 4 согласно СанПин 3.3686-21, в силу принадлежности вируса к I группе патогенности.
A., & Rodas, J. D. (2022). An updated review and current challenges of Guanarito virus infection, Venezuelan hemorrhagic fever. Archives of virology, 167(9), 1727-1738. https://doi.org/10.1007/s00705-022-05453-3). Однако данный способ требует специальных условий проведения анализа в лаборатории с УББ 4 согласно СанПин 3.3686-21, в силу принадлежности вируса к I группе патогенности.
В последние время, наибольшее распространение получила полимеразная цепная реакция (ПЦР) с обратной транскрипцией, позволяющая идентифицировать возбудитель путем выявления РНК в биологических образцах. В качестве анализируемых образцов могут выступать цельная кровь (с ЭДТА), плазма крови и мазки со слизистой оболочки ротоглотки. Метод диагностики предусматривает экстракцию РНК из биологических образцов с последующим проведением полимеразной цепной реакции для целевых фрагментов РНК генома вируса Mammarenavirus guanaritoense с использованием набора праймеров и зонда, комплементарных участку гена, белка L, РНК зависимой РНК полимеразы вируса.
Известна тест-система KR1020220159612 для обнаружения РНК вируса GTOV, основанная на использовании методов классической ПЦР и ОТ-ПЦР
(https://patentscope.wipo.int/search/en/detail.jsf?docId=KR382041077&_cid=P11-LUS6TG-84006-1). Недостаток данного метода заключается в его трудоемкости, связанной с применением электрофореза в агарозном геле, а также с высокой продолжительностью программ амплификации.
В настоящее время, учитывая масштабы изменения климата, и потенциальную возможность возникновения новых пандемий с ранее неизвестными человечеству возбудителями - актуальными становятся меры, предполагающие постоянную противоэпидемическую готовность которая включает в себя, кроме прочих мер, разработку новых инструментов для своевременной диагностики заболевших, с целью выявления носителей еще до появления первых симптомов.
Создание такого инструмента и является целью нашего изобретения. Технический результат - разработка высокочувствительного способа идентификации РНК вируса Mammarenavirus guanaritoense в биологических образцах и других вируссодержащих пробах (культуральная вируссодержащая жидкость, и т.д.).
Поставленная задача решалась путем:
- конструирования диагностических праймеров и флуоресцентно-меченного зонда на специфические для GTOV кДНК фрагменты, полученные с РНК матрицы;
- конструирования рекомбинантной плазмидной ДНК и MS2-фага, несущих специфический участок РНК-матрицы;
- оптимизации концентраций компонентов реакционной смеси и условий проведения ПЦР.
Авторами предложен способ выявления вируса Mammarenavirus guanaritoense, согласно которому, выделенную из биологических образцов РНК анализируют в одной пробирке при помощи реакции обратной транскрипции и дальнейшей полимеразной цепной реакции в реальном времени полученных кДНК, используя комплементарные участку гена белка L (кодирующего РНК-зависимую РНК-полимеразу вируса Гуанарито), олигонуклеотидные праймеры и соответствующий флуоресцентно меченый зонд.
Анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию или отсутствию значения порогового цикла Ct, причем результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
На начальном этапе были подобраны и синтезированы специфические олигонуклеотидные праймеры и зонд для гибридизационно-флуоресцентной детекции продуктов ПЦР. Для этого в базе данных Genbank (https://www.ncbi.nlm.nih.gov/genbanck/) был выбран наиболее консервативный участок генома вируса Mammarenavirus guanaritoense и проанализированы все имеющиеся в базе данных последовательности. Также были подобраны праймеры и флуоресцентный зонд для внутреннего контрольного образца ПЦР. Последовательности олигонуклеотидных праймеров и флуоресцентного зонда представлены в Таблице 1. Метки зонда представлены в Таблице 2. Наличие двух прямых праймеров f1 и f2 позволяет детектировать все имеющиеся на сей день последовательности гена белка L GTOV.

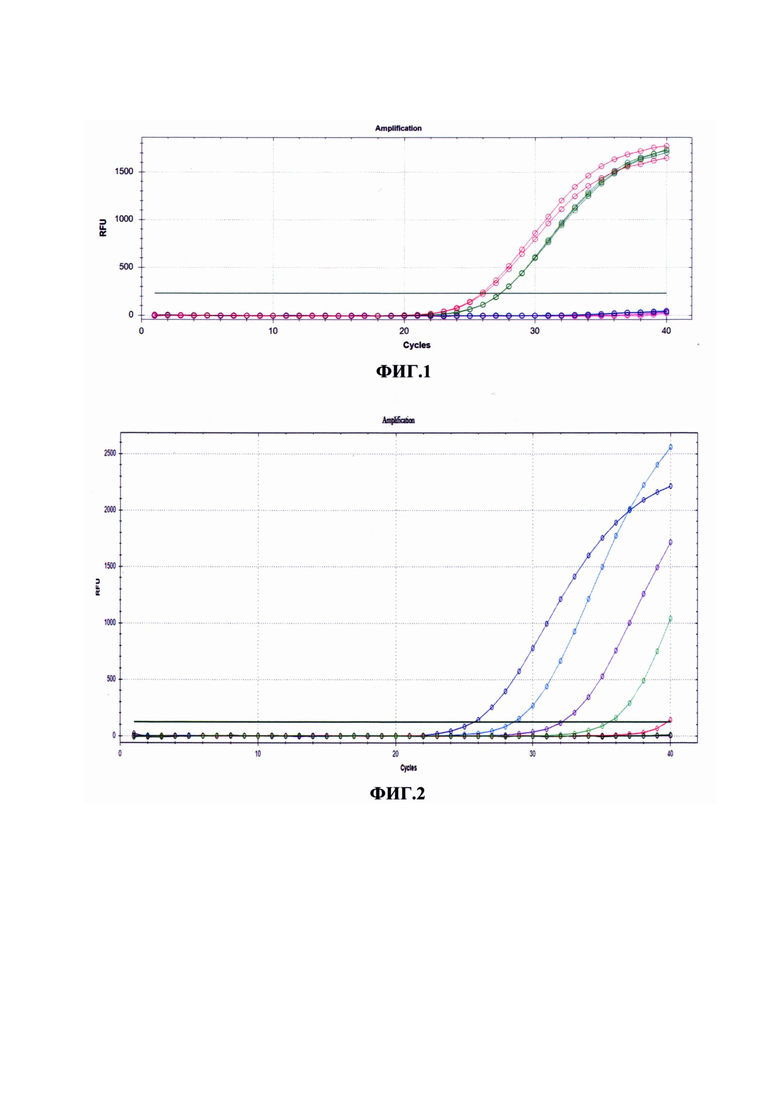
Сущность изобретения поясняется чертежом, где на ФИГ. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции при анализе контрольных образцов. Кривые красного и зеленого цветов выше пороговой линии, представляют собой контрольные образцы этапа ПЦР (К+) и этапа выделения РНК (ПКО) соответственно, и обозначают положительный результат на выявление в образце РНК вируса Mammarenavirus guanaritoense. Кривые синего и розового цветов ниже пороговой линии, являются отрицательными контролями этапа ПЦР (К-) и этапа выделения РНК (ОКО) соответственно, и не содержат РНК вируса. На ФИГ. 2 представлены данные подтверждающие аналитическую чувствительность метода. Кривые синего, голубого, фиолетового, зеленого и красного цветов представляют собой десятикратные разведения контрольного образца концентрацией от 107 до 103 копий / мл соответственно.
Подбор и анализ свойств олигонуклеотидных праймеров и флуоресцентного зонда проводился с использованием программного обеспечения Oligonucleotide Properties Calculator 3.27 и MFold.
Для контроля качества прохождения ПЦР в состав методики были введены рекомбинантные положительные контрольные образцы К+ и ПКО и внутренний контрольный образец (ВКО). Матрицу для создания рекомбинантных положительных контрольных образцов получали синтетическим методом на основе ампликона, включающего в себя диагностическую область-мишень и фланкирующие последовательности нуклеотидов. Ампликон получали методом ПЦР в один шаг. Конечный ПЦР-продукт лигировали в плазмидный вектор pGEM-T («Promega», USA) под контролем промотора Т7 РНК полимеразы и трансформировали им Escherichia coli (штамм XL1-Blue). Рекомбинантные плазмиды из индивидуальных клонов проверяли на правильность ориентации целевой последовательности и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом секвенирования по Сэнгеру с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500x1 («Applied Biosystems», США).
Соответствующие заданным критериям рекомбинантные плазмиды использовали для приготовления положительных контрольных образцов этапа ПЦР (К+). Для этого определяли концентрацию ДНК в растворе рекомбинантной плазмиды и разводили 1xTE-буфере (рН 8.0) со стабилизаторами натрия азида 0,05% и ДНК спермы лосося 0,02 мг/мл до рабочей концентрации 1*107 копий/мл.
Также плазмиды использовались для получения рекомбинантного РНК-содержащего положительного контрольного образца с защитной белковой оболочкой MS2-фага (ПКО). Для полученного продукта производили определение концентрации методом цифровой капельной ПЦР (ddPCR) («BioRad», USA), затем разводили РНК-буфером (ФБУН ЦНИИ Эпидемиологии, Россия) до рабочей концентрации 0,8*106 копий/мл, которую использовали в качестве ПКО в дальнейшем анализе.
Оценку аналитической чувствительности метода производили на десятикратных разведениях раствора ПКО от начальной концентрации 1*107 копий/мл, которые проводили через все стадии выделения с помощью набора для выделения нуклеиновых кислот «Рибо-преп» (ФБУН ЦНИИ Эпидемиологии, Россия), а затем с помощью специфических праймеров и флуоресцентных зондов определяли минимальное разведение, детектируемое как положительное в 100% случаев. Определенная таким образом аналитическая чувствительность метода составила 1*103 копий/мл (см. Фиг. 2).
Свойство изобретения специфически определять РНК вируса Mammarenavirus guanaritoense достигнуто путем подбора праймеров и зондов к высококонсервативным фрагментам РНК-генома вируса, что минимизирует возможность перекрестных реакций с геномом человека, близкородственных микроорганизмов и других инфекционных возбудителей.
Аналитическую специфичность оценивали при помощи тестирования образцов генома человека и следующих вирусов: Hendra, Machupo, Nipah, Junin, Sabia и SARS-CoV (тяжелый острый респираторный синдром). Неспецифические реакции отсутствовали.
Таким образом, в результате проведенных исследований был разработан и апробирован способ выявления РНК вируса Mammarenavirus guanaritoense в различных видах биологического материала. Технический результат достигается путем определения РНК вируса Mammarenavirus guanaritoense, включающим выделение РНК исследуемой пробы и проведение ОТ-ПЦР с учетом результатов в режиме реального времени, согласно изобретению.
Диагностика проводится следующим образом: Подготовку проб проводят согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Материалом для исследования могут служить клинические и биологические образцы.
Экстракция производится из 100 мкл. полученной суспензии образца с лизирующим буфером на основе 6 моль гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения нуклеиновых кислот, и последующим инкубированием 5 минут при температуре (65±1)°С. Выделение РНК осуществляют с помощью наборов «Рибо-преп» и «Рибо-Сорб» (ФБУН ЦНИИ Эпидемиологии, Россия) в соответствии с инструкциями к наборам. Во все пробирки, включая контроль выделения, до этапа прогревания в лизирующем буфере добавляют 10 мкл. внутреннего контрольного образца (ВКО). После выделения РНК приступают к постановке ОТ-ПЦР.
Для упрощения подготовки и стандартизации выполнения анализа в тест-систему входят 8 реактивов:
1) Реактив Amp 1RT, представляющий из себя буферный раствор, в состав которого входят 50 мМ Трис-HCl, рН 8.0 (при 25°С), 100 мМ NaCl, 1 мМ ЭДТА, 5 мМ дитиотреитол, 50% (v/v) глицерин и 0.1% (v/v) NP-40, ингибитор РНКаз, M-MuLV-RH-ревертаза и HS-Taq - полимераза;
2) Реактив Amp 1В который представляет из себя буферный раствор, содержащий 100 мМ Трис-HCl, рН 8.3 (при 25°С), 150 мМ KCl, 0,6 мМ каждого дезоксинуклеозидтрифосфата, 10 мМ MgCl2, 8 мМ ДТТ, стабилизаторы и усилители ферментов;
3) Реактив Amp 2, содержащий олигонуклеотидные праймеры и флуоресцентно-меченые олигонуклеотидные зонды к РНК вируса GTOV и к ВКО;
4) К+ “GTOV”, представляющий собой смесь двух плазмидных ДНК:
a) плазмида, содержащая фрагмент вируса GTOV;
b) плазмида, содержащая фрагмент ВКО;
с которых, при помощи реактива Amp 2, амплифицируются специфические фрагменты позволяющие контролировать корректность прохождения реакции ОТ-ПЦР;
5) К-, представляющий из себя стерильную, дистиллированную воду;
6) ПКО “GTOV” - положительный контрольный образец, в составе которого псевдовирусные частицы на основе MS2-фага, содержащие РНК с фрагментом РНК вируса GTOV, которые после выделения и обратной транскрипции в к, амплифицируются при помощи реактива Amp 2;
7) ОКО, представляющий собой дистиллированную стерильную воду;
8) ВКО, представляющий собой псевдовирусные частицы на основе MS2-фага, содержащие РНК с фрагментом РНК ВКО, которые после выделения и обратной транскрипции в к, амплифицируются при помощи реактива Amp 2 “GTOV”.
Все реактивы, кроме ПКО и ВКО, хранятся при температуре -20°С. ПКО и ВКО хранятся при температуре +4°С.
Для постановки реакции ПЦР в реальном времени нужно взять необходимое количество микропробирок объемом 0,2 мл, соответствующее числу исследуемых проб; плюс четыре пробирки для положительных и отрицательных контролей. Подготовить ПЦР-смесь из расчета на одну реакцию: 12,5 мкл реактива Amp1, 1,5 мкл реактива Amp2. По 15 мкл полученной смеси вносят в пробирки, затем добавляют в каждую по 10 мкл РНК-пробы, экстрагированной из исследуемого материала. Готовят 4 контрольные реакции. Для этого в пробирку для положительного контроля ПЦР вносят 10 мкл К+, в пробирку для положительного контроля экстракции вносят 10 мкл образца, экстрагированного из ПКО. В пробирку для отрицательного контроля ПЦР вносят 10 мкл К-, в пробирку для отрицательного контроля экстракции вносят 10 мкл пробы, экстрагированной из ОКО. Конечный объем реакционной смеси составляет 25 мкл. Микропробирки переносят в программируемый амплификатор с функцией детекции флуоресценции в режиме «реального времени» по каналам (FAM/Green и HEX/Yellow). Режим амплификации представлен в таблице 3.

Детекцию флуоресцентного сигнала производят при 55°С по каналам Green (FAM) и Yellow (JOE/HEX). Учет и анализ результатов проводят с помощью программного обеспечения прибора на основании отсутствия или наличия сигнала флуоресценции в исследуемых пробах по детектируемым каналам. Результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию (или отсутствию) значения порогового цикла Ct в соответствующей графе в таблице результатов. Результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. Накопление флуоресцентного сигнала по каналу HEX свидетельствует о наличии в исследуемом материале РНК вируса GTOV. При этом результат считается валидным, если параллельно в пробе по каналу FAM регистрируется накопление флуоресцентного сигнала ВКО, а в образцах К+ и ПКО - по каналу HEX. Сигнал по каналу HEX в образцах К- и ОКО должен отсутствовать.
Таким образом, заявляемый способ позволяет достоверно выявлять РНК вируса Mammarenavirus guanaritoense в биологических образцах.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ выявления РНК вируса Mammarenavirus guanaritoense
методом ОТ-ПЦР в реальном времени last.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2024-04-16">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>0000</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
"Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им.Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и брагополучия
человека" ФБУН НИИ эпидемиологии и микробиологии имени
Пастера.</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur Institute,Federal
Service for Consumer Rights Protection and Human Well-being,Saint
Petersburg,Russian Federation </ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ выявления РНК вируса
Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени
</InventionTitle>
<InventionTitle languageCode="en">A method for detecting the RNA of
the Mammarenavirus guanaritoense by the RT-Real Time PCR technique
</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaagcttgaagccctctgttaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggagcttgaagccctctgtta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcctaggcgaaatttgacaatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl]-N-ethyl-6(ethylamino)-2,7-dimethyl-3H-xa
nthen-3-iminium chloride) on 5' end.</INSDQualifier_value>
<NonEnglishQualifier_value>Флуорофор Родамин 6Ж
(9-[2-этоксикарбонил)фенил]-N-этил-6-(этиламино)-2,7-диметил-3Н-ксанте
н-3-иминия хлорид) на 5'-конце.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>>27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q10">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Non-fluorescent Black Hole Quencher 1
(2-(N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4-methyl-2-nitrophen
yl)diazenyl]phenyl]diazenyl]anilino]ethanol at 3`
end.</INSDQualifier_value>
<NonEnglishQualifier_value>Не флуоресцентный тушитель Black
Hole Quencher 1
(2-(N-(2-гидроксиэтил)-4-[2-метокси-5-метил-4-[[2-метокси-5-метил-2-ни
трофенил)диазенил]фенил]диазенил]анилино]этанол) на 3'- конце
.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctccttctgataagacagttcaggag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления вируса Mammarenavirus guanaritoense методом DETECTR с изотермической амплификацией | 2024 |
|
RU2835994C1 |
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |
| Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822161C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом рекомбиназной полимеразной амплификации | 2024 |
|
RU2834909C1 |
| Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2834908C1 |
| Способ выявления вируса кори методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822430C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
| Способ выявления вируса Guanarito методом, основанным на применении дезоксирибозима 10-23 | 2024 |
|
RU2827922C1 |
Изобретение относится к области биотехнологии. Описан способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени, включающий экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции - обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома Mammarenavirus guanaritoense, с использованием набора праймеров и зонда, комплементарных участку гена белка L, кодирующего РНК зависимую РНК полимеразу. Технический результат заключается в разработке высокочувствительного способа идентификации РНК вируса Mammarenavirus guanaritoense в биологических образцах и других вируссодержащих пробах. 2 ил., 3 табл.
Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени, включающий экстракцию РНК из биологических образцов, с последующим проведением двухстадийной реакции - обратной транскрипции и полимеразной цепной реакции для целевых фрагментов РНК генома Mammarenavirus guanaritoense, с использованием набора праймеров и зонда, комплементарных участку гена белка L, кодирующего РНК зависимую РНК полимеразу, отличающийся тем, что в качестве праймеров используются последовательности
а в качестве зонда
определяющие наличие вируса Mammarenavirus guanaritoense, в случае пересечения кривой флуоресценции пороговой линии.
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ ВИРУСА ЗАПАДНОГО НИЛА 1 ГЕНОТИПА | 2019 |
|
RU2715625C1 |
| WO 2022051667 A1 10.03.2022 | |||
| Sironi M., Forni D., de la Torre J | |||
| C | |||
| Mammarenavirus genetic diversity and its biological implications | |||
| Viral Fitness and Evolution: Population Dynamics and Adaptive Mechanisms, 2023, V.439, P | |||
| САННЫЙ ВЕЛОСИПЕД С ВЕДУЩИМ КОЛЕСОМ, СНАБЖЕННЫМ ШИПАМИ | 1921 |
|
SU265A1 |
Авторы
Даты
2025-05-15—Публикация
2024-04-19—Подача