Изобретение относится к области молекулярной и клеточной биологии и предназначено для оценки количества Т и В-лимфоцитов в исследуемых образцах ДНК человека с помощью цифровой капельной полимеразной реакции, основанной на методике абсолютной квантификации молекул ДНК, содержащих сигнальные фрагменты эксцизионных колец Т и В-лимфоцитов.
Иммунную систему можно условно разделить на две основные компоненты: врожденная иммунная система, которая обеспечивает немедленную, но неспецифическую защиту от чужеродных веществ, и адаптивная иммунная система, которая обеспечивает специфическую защиту от патогенов и обладает способностью запоминать предыдущие встречи с тем же возбудителем. Основными представителями адаптивной иммунной системы являются Т и В-лимфоциты (1, 2), которые распознают специфические антигенные пептиды при содействии главного комплекса гистосовместимости (ГКГС) (3, 4).
Распознавание специфических антигенных пептидов обеспечивается Т и В клеточными рецепторами, TCR и BCR, соответственно, находящимися на поверхности клетки. У Т-лимфоцитов TCR состоит из альфа- и бета-цепей (2). У В-лимфоцитов рецептор состоит из тяжелых и легких цепей иммуноглобулина. Объединяющим фактором лимфоцитов является их разнообразие по репертуарам за счет гипервариабельных областей их клеточных рецепторов.
Внутри цепи TCRβ определяющая комплементарность область 1 (CDR) 1 и CDR2 контактируют с альфа-спиралями ГКГС, в то время как гипервариабельные области CDR3 взаимодействуют в основном с антигенным пептидом (1, 2). CDR3 в альфа- и бета-цепях TCR имеют широкий диапазон разнообразных последовательней, обеспечивающих обнаружение антигенного пептида. Схожим механизмом распознавания обладают и иммуноглобулины, вырабатываемые В-лимфоцитами.
Несмотря на гипервариабельность локуса, обеспечивающего распознавание антигенного пептида, каждый лимфоцит обладает уникальной и неизменной последовательностью данного локуса. Подобная стабильность локуса обеспечивается за счёт механизмов перманентной перестройки собственного генома клетки.
В процессе рекомбинации TCR вырезанные фрагменты ДНК создают круги эксцизии Т-клеточных рецепторов (TREC), которые экспортируются в цитоплазму Т-клеток (5, 6). В частности, сигнальный совместный TREC δRec-ψJα (sjTREC) продуцируется во время делеции TCRD и обнаруживается примерно в 70% (альфа-бета) αβ Т-клеток. Они считаются наиболее оптимальной мишенью для оценки их продукции тимусом (7-9). Кольца вырезания рекомбинации с удалением каппа (KREC) образуются во время перестройки BCR в наивных В-клетках и аналогичны TREC (10). Сходным образом, sjKREC, образующийся во время реаранжировок intronRSS-Kde в локусе IGK, является надежной мишенью для оценки неогенеза В-клеток из костного мозга (11, 12).
Как TREC, так и KREC стабильны и нерепликативны во время пролиферации клеток (13, 14). Анализ TREC и KREC широко применяется в различных клинических условиях для оценки продукции T и B-клеток тимусом и костным мозгом. Измерение уровней TREC и KREC в периферической крови используется для скрининга и мониторинга иммунодефицитных заболеваний.
Существует ряд изобретений близких по заявляемой технической сущности (15-18). Кроме того, известны методы оценки количества TREC и KREC для диагностики первичных и вторичных иммунодефицитов у человека (19). Тем не менее, основной отличительной их особенностью является использование технологии полимеразной цепной реакции (ПЦР) в режиме реального времени, а также использования других разработанных наборов праймеров.
Предлагается способ с использованием цифровой ПЦР, а не ПЦР в режиме реального времени, что в значительной степени повышает точность измерения. Технология цифровой ПЦР (digital PCR, dPCR или digital droplet PCR, ddPCR) имеет преимущество в отношении меньшей чувствительности к ингибиторам ПЦР. Кроме того, она позволяет получить точные (абсолютные) измерения количества копий участков ДНК, путём проведения флуоресцентных анализов, протекающих в десятках тысяч независимых реакций (капель, лунок) (20, 21). Точность измерения повышается за счет измерения каждой введенной молекулы ДНК, содержащих целевой фрагмент, т.е. абсолютная квантификация ДНК. Подобная точность достигается за счет разобщения каждой молекулы ДНК на 20 тыс. независимых реакций.
Отличительной особенностью цифровой ПЦР от количественной ПЦР в режиме реального времени является измерение флуоресценции не во время ПЦР, а «по конечной точке», т.е. по завершению ПЦР. Тем самым достигается высокая точность анализа, который позволяет определить целочисленное количество копий в локусах, по сравнению с другими технологиями (20-22).
Новая техническая задача - подбор последовательностей (структуры) олигонуклеотидных проб, разработка протокола проведения цифровой ПЦР для абсолютной квантификации (точного определения количества) сигнальных фрагментов эксцизионных колец Т и В-клеточных рецепторов, содержащихся в биологическом образце человека.
Указанный результат достигается за счет разработки набора синтетических олигонуклеотидных последовательностей и проб, а также протокола проведения цифровой ПЦР для подсчета количества Т и В-лимфоцитов в биологическом образце человека.
Техническим результатом изобретения является повышение точности оценки количества Т и В-лимфоцитов в интересующих биологических образцах человека, по сравнению с существующими аналогами.
Описание изобретения
Разработан набор праймеров и флюоресцентных проб (по принципу TaqMan-проб) для генов TCRAC (Альфа-константа Т-клеточного рецептора) и эксцизионных колец TREC (эксцизионные кольца Т-клеточного рецептора) и KREC (каппа-делетированные рекомбинационные эксцизионные кольца). Учитывая особенность лимфоцитов, активно подверженных геномным рекомбинациям в период созревания, ген TCRAC был выбран как участок, который не участвует в рекомбинациях.
Сущность заявляемого технического решения иллюстрируется следующими фигурами:
фиг. 1. Структура набора синтетических олигонуклеотидных праймеров и проб;
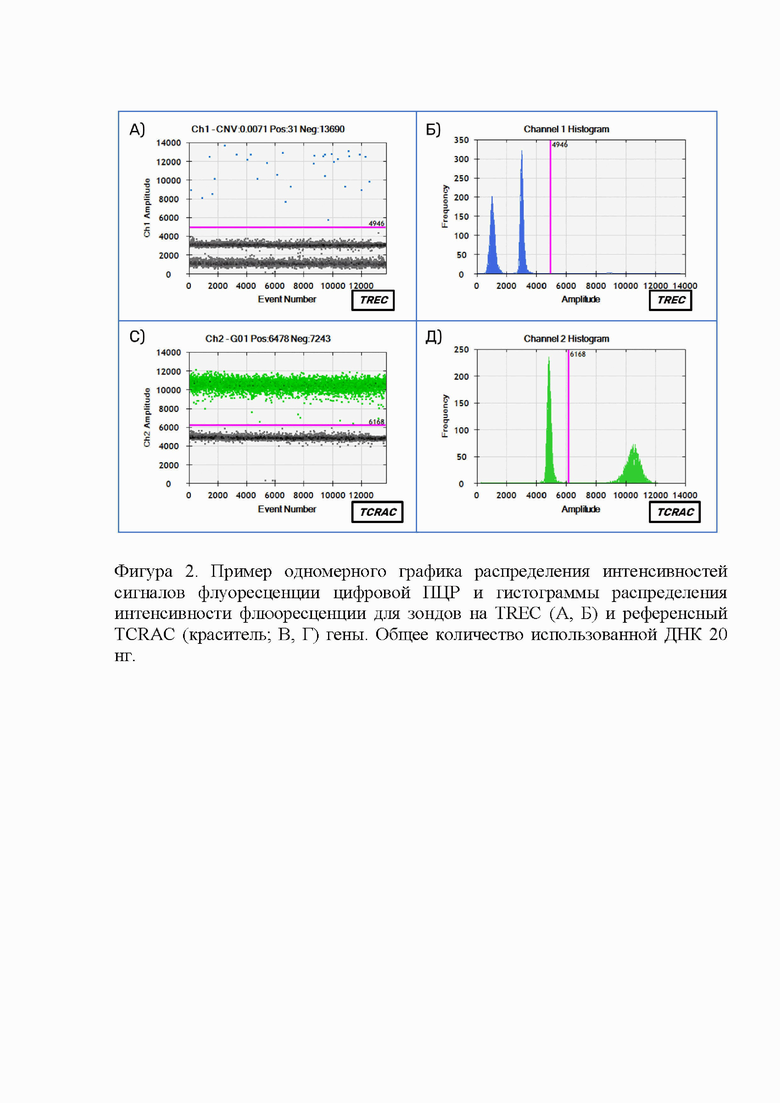
фиг 2. Пример одномерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР и гистограммы распределения интенсивности флюоресценции для зондов на TREC (А, Б) и референсный TCRAC гены. Общее количество использованной ДНК 20 нг;
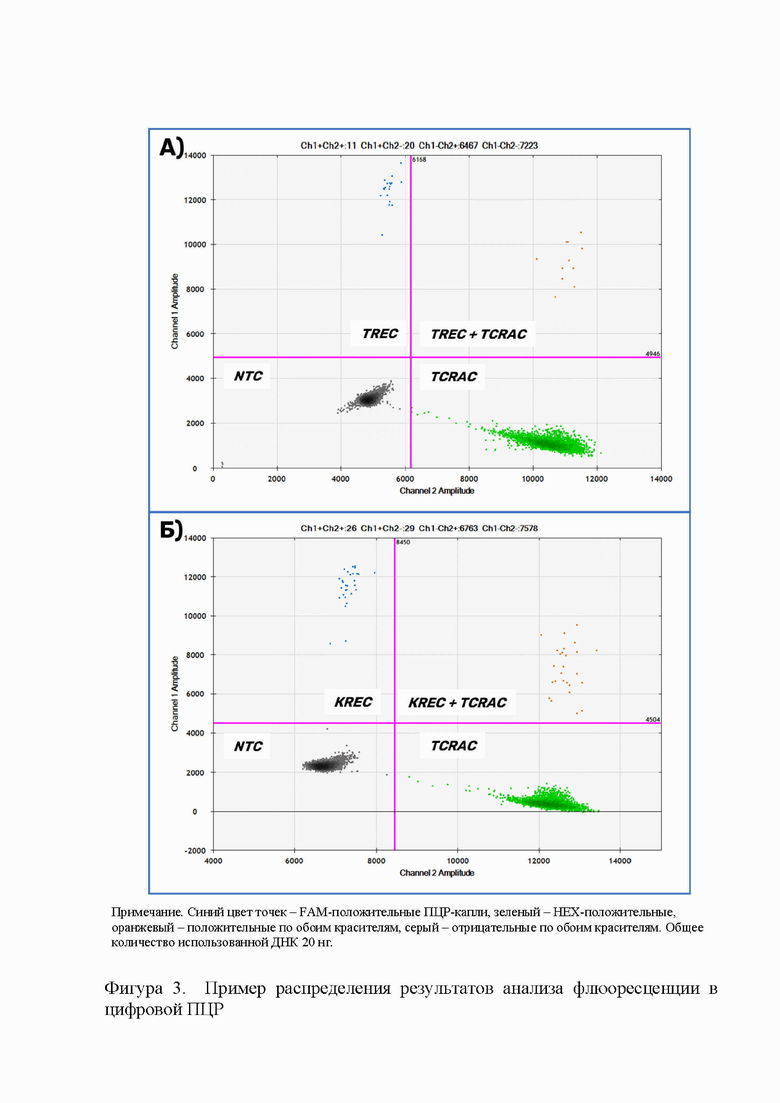
фиг. 3. Пример распределения результатов анализа флюоресценции в цифровой ПЦР.
Праймеры и пробы были протестированы на отсутствие сайтов отжига в ядерном геноме с помощью программы BLAST онлайн [23]. Оптимальной концентрацией является 450 нМ для каждого праймера и 250 нМ для зонда (фиг 1).
Генотипирование с помощью набора проб осуществляли по модифицированному протоколу генотипирования методом dPCR и ddPCR следующим образом:
1. В качестве материала для исследования можно использовать препарат ДНК индивида, выделенный из биообразца любым способом (включая сухие пятна крови), который позволяет использовать ДНК в качестве матрицы в полимеразной цепной реакции. Основным требованием к качеству используемой ДНК является минимальные признаки деградации (разрушения) ДНК. Рекомендуется провести оценку качества ДНК с помощью любого метода ДНК электрофореза, при котором должен присутствовать только высокомолекулярный бэнд тотальной ДНК, отсутствие бэндов примесей РНК и любых «шмеров» - признаков деградации ДНК. Выполнение данной рекомендации позволит избежать неточности в измерении.
2. Образец исследуемой ДНК необходимо развести до рабочей концентрации, оптимально 100 нг/мкл, для удобства последующих манипуляций. В реакционную смесь могут входить безнуклеазная вода, буферы для фермента рестрикции и иные компоненты, рекомендованные производителем.
3. Реакционная смесь для проведения мультиплексной цифровой ПЦР состоит из двух объемов мастер-микса производителя dPCR или ddPCR, мультиплекса из праймеров и зондов для исследуемых генов TCRAC, TREC и KREC, безнуклеазной воды и образца ДНК. Оптимальная концентрация ДНК выбирается согласно рекомендациям производителя прибора dPCR или ddPCR. Оптимальным соотношением является от 20 нг ДНК на 20 мкл реакционной смеси (фиг 2, 3).
4. Протокол программы ПЦР основан на стандартной 2х этапной ПЦР, включающей: этап денатурации при +94°С, этап одновременного отжига и элонгации при +57°С в течение 1 мин. Этап «горячего старта» и «горячего завершения» может быть добавлен согласно протоколу производителя. Хранение готовых продуктов ПЦР возможно при +10°С в течение 24 часов.
5. Общая методическая постановка dPCR (ddPCR) и получение сигналов флуоресценции проводится согласно протоколу производителя технологии ddPCR (капельная, микрофлюидная) или dPCR (на микрокристалле, нанопланшете, иное) [19, 20]. Основным требованием для оценки сигналов флуоресценции нанореакций является достаточное общее количество нанореакций, которое должно превышать более 10000 капель на реакцию для адекватной оценки результатов. Примеры получаемых результатов представлены на фиг. 3.
6. Определение доли Т и В-лимфоцитов (Rtarget) в биологическом образце на 10 млн. ядросодержащих клеток проводится по формуле,
где Ctarget - количество копий TREC (для определения Т-лимфоцитов) или KREC (для определения В-лимфоцитов), Cref - количество копий TCRAC.
7. Определение количества Т и В-лимфоцитов относительно объема выделенного из крови (Qtarget, клеток/мл крови) выполняется по следующей формуле,
где V - объем крови, использованной для выделения ДНК (мл), Efactor - фактор элюции, то есть в скольких микролитрах было получено ДНК (мкл), Ctarget - количество копий TREC (для определения Т-лимфоцитов) или KREC (для определения В-лимфоцитов).
Источники информации, принятые во внимание при составлении описания:
1. Davis M.M., Bjorkman P.J. T-Cell Antigen Receptor Genes and T-cell Recognition // Nature. 1988. Vol. 334. P. 395-402.
2. Krogsgaard M., Davis M.M. How T Cells ‘See’ Antigen // Nat. Immunol. 2005. Vol. 6. P. 239-245.
3. Rowen L. et al. The Complete 685-Kilobase DNA Sequence of the Human Beta T Cell Receptor Locus // Sci. 1999. Vol. 272 P. 1755-1762.
4. Glanville J. et al. Identifying Specificity Groups in the T Cell Receptor Repertoire // Nature. 2017. Vol. 547. P. 94-98.
5. Livak F., Schatz D.G. Identification of V(D)J recombination coding end intermediates in normal thymocytes // J. Mol. Biol. 1997. Vol. 267. P.1-9.
6. Takeshita S. et al. Excision products of the T cell receptor gene support a progressive rearrangement model of the alpha/delta locus // EMBO J. 1989. Vol. 8. P. 3261-3270.
7. Ye P., Kirschner D.E. Reevaluation of T cell receptor excision circles as a measure of human recent thymic emigrants // J. Immunol. 2002. Vol. 168. P. 4968-4979.
8. Hazenberg M.D. et al. T cell receptor excision circles as markers for recent thymic emigrants: basic aspects, technical approach, and guidelines for interpretation // J. Mol. Med. 2001. Vol. 79. P. 631-640.
9. Barbaro M. et al. Newborn screening for severe primary immunodeficiency diseases in Sweden-a 2-year pilot TREC and KREC screening study // J. Clin. Immunol. 2017. Vol. 37. P. 51-60.
10. Siminovitch K.A., et al. A uniform deleting element mediates the loss of kappa genes in human B cells // Nature. 1985. Vol. 316. P. 260-262.
11. van Zelm M.C. et al. PID comes full circle: applications of V(D)J recombination excision circles in research, diagnostics and newborn screening of primary immunodeficiency disorders // Front. Immunol. 2011. Vol. 2. P.12.
12. van Zelm M.C. et al. Replication history of B lymphocytes reveals homeostatic proliferation and extensive antigen-induced B cell expansion // J. Exp. Med. 2007. Vol. 204. P. 645-655.
13. Kong F.K. et al. T cell receptor gene deletion circles identify recent thymic emigrants in the peripheral T cell pool // Proc. Natl. Acad. Sci. 1999. Vol. 96. P. 1536-1540.
14. Livak F., Schatz D.G. T-cell receptor alpha locus V(D)J recombination by-products are abundant in thymocytes and mature T cells // Mol. Cell Biol. 1996. Vol. 16. P. 609-618.
15. RU 2756979 C2 (Семенов А.В и соавт.) 7 октября 2021, весь документ.
16. RU 2611393 C1 (Донецкова А.Д и соавт.) 21 февраля 2017, весь документ.
17. RU 2587540 C1 (Гордукова М.А. и соавт.) 20 июня 2015, весь документ.
18. EP 1849877 Al (van Dongen J.J.M. et al.) 24 апреля 2006, весь документ.
19. Borte S. et al. Neonatal screening for severe primary immunodeficiency diseases using high-throughput triplex real-time PCR // Blood. 2012. Vol. 119, № 11. P. 2552-2555.
20. Hindson B.J. et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number // Anal. Chem. 2011. Vol. 83, № 22. P. 8604-8610.
21. Pinheiro L.B. et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification // Anal. Chem. 2012. Vol. 84, № 2. P. 1003-1011.
22. Usher C.L. et al. Structural forms of the human amylase locus and their relationships to SNPs, haplotypes and obesity // Nat. Genet. Nature Publishing Group, 2015. Vol. 47, № 8. P. 921-925.
23. Ye J. et al. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. // BMC Bioinformatics. 2012. Vol. 13. P. 134.
24. Dong L. et al. Comparison of four digital PCR platforms for accurate quantification of DNA copy number of a certified plasmid DNA reference material // Sci. Rep. Nature Publishing Group, 2015. Vol. 5, № 1. P. 1-11.
25. Farr R.J. et al. Comparative analysis of diagnostic platforms for measurement of differentially methylated insulin DNA // J. Biol. Methods. Journal of Biological Methods, 2019. Vol. 6, № 2. P.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd" PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-04-05" softwareVersion="2.2.0" softwareName="WIPO Sequence" fileName="dPCR Tcell and Bcell counts.xml" dtdVersion="V1_3">
<ApplicantFileReference>2023-03</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное бюджетное научное учреждение Томский национальный исследовательский медицинский центр Российской академии наук</ApplicantName>
<ApplicantNameLatin>Federalnoe gosudarstvennoe byudjetnoe nauchnoe uchrejdenie Tomskii nacionalnii issledovatelskii medicinskii centr Rossiiskoi akademii nauk</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ количественной оценки Т и В-лимфоцитов по содержанию эксцизионных колец TREC и KREC в образцах ДНК с помощью цифровой полимеразной цепной реакции</InventionTitle>
<SequenceTotalQuantity>9</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgatgccacatccctttca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccagctgcagggtttag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actcctgtgcacggtgatgcatag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctctttcccttagtggcattat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agccagctcttaccctaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acagtgtgcgctgccaacct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagctctggtcaatgatgtctaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acaaggctcactgtttcttagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggtctcggccttatccatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Рекомбинантная плазмидная ДНК pGEM-TCRAC-sjTREC- sjKREC, содержащая нуклеотидные последовательности локуса гена Т-лимфоцитарного α-рецептора и сигнальные фрагменты эксцизионных колец Т- и В-клеточного рецепторов | 2022 |
|
RU2807806C1 |
| Способ определения делеции 7 экзона в гене SMN1 с определением количества копий гена SMN2 в образцах ДНК с помощью цифровой полимеразной цепной реакции | 2024 |
|
RU2838673C1 |
| Способ определения генотипа ротавирусов первой и второй геногрупп и реассортантов между ними методом мультиплексной ПЦР | 2024 |
|
RU2833073C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
| BCMA-СПЕЦИФИЧЕСКИЕ ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, СОДЕРЖАЩИЕ БЛОКАТОР ПЕРЕДАЧИ СИГНАЛА IL-6 | 2023 |
|
RU2828294C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |

Изобретение относится к области молекулярной биологии. Описан способ количественной оценки Т и В-лимфоцитов человека методом цифровой полимеразной цепной реакции, характеризующийся тем, что точная оценка количества Т и В-лимфоцитов человека в биологическом образце достигается путем использования подсчета копий сигнальных фрагментов эксцизионных колец генов ТREC и KREC, а также гена. Технический результат заключается в повышении точности количественной оценки Т и В-лимфоцитов в интересующих биологических образцах человека. 3 ил.
Способ количественной оценки Т и В-лимфоцитов человека методом цифровой полимеразной цепной реакции, характеризующийся тем, что точная оценка количества Т и В-лимфоцитов человека в биологическом образце достигается путем использования подсчета копий сигнальных фрагментов эксцизионных колец генов ТREC и KREC, а также гена TCRAC, выступающего в роли референсного гена для подсчета общего количества клеток в биологическом материале, с применением цифровой ПЦР, с использованием мультиплексного набора специфичных праймеров и зондов, представленных последовательностями SEQ ID NO:1-9.
| CN 110408713 A, 05.11.2019 | |||
| СПОСОБ ДИАГНОСТИКИ СОСТОЯНИЯ ИММУННОЙ СИСТЕМЫ ПАЦИЕНТА И НАБОР ПРАЙМЕРОВ, ЗОНДОВ И СТАНДАРТНЫХ ОБРАЗЦОВ ДЛЯ КОЛИЧЕСТВЕННОЙ ОЦЕНКИ ДНК МОЛЕКУЛ TREC, KREC И КОЛИЧЕСТВА ГЕНОМ ЭКВИВАЛЕНТОВ ДНК | 2015 |
|
RU2587540C1 |
| Sottini A et al | |||
| Simultaneous quantification of T-cell receptor excision circles (TRECs) and K-deleting recombination excision circles (KRECs) by real-time PCR | |||
| J Vis Exp | |||
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
Авторы
Даты
2024-07-03—Публикация
2023-11-16—Подача