Изобретение относится к области молекулярной и клеточной биологии и предназначено для оценки количества копий гена SMN1 и SMN2 с определением делеции 7 экзона гена SMN1 в исследуемых образцах ДНК человека с помощью цифровой капельной полимеразной реакции, основанной на методике аллель специфичной абсолютной квантификации локуса генов SMN1 и SMN2.
Белок выживаемости мотонейронов кодируется геном SMN1, который имеет высокую гомологию с геном SMN2, то есть данный ген является его паралогом (1). Ген SMN2 отличается от SMN1 пятью нуклеотидами, и среди них имеется одна критическая замена C на T в 7 экзоне, которая приводит к альтернативному сплайсингу во время транскрипции, в результате чего белок является функционально неполноценным (2). Дефицит SMN белка приводит к аутосомно-рецессивной спинальной мышечной атрофии (СМА), встречающейся с частотой 1:10000 и являющейся ведущей генетической причиной детской смертности (3).
Нарушение гена SMN1 может произойти как в результате делеции гена, так и в результате генной конверсии, то есть в результате замены функциональной части гена SMN1 на гомологичную, но нефункциональную часть гена SMN2. Однако до 95% случаев СМА делеция 7 экзона гена SMN1 является ведущей причиной (4). Несмотря на то, что наличие гена SMN2 не является критически важным для жизнедеятельности здоровых лиц, он является жизненно важным для пациентов со СМА, поскольку имеются наблюдения, что несмотря на низкую функциональную пригодность гена SMN2, его наличие у пациентов со СМА смягчает течение заболевания (4-6).
Поскольку определение дозы генов SMN1 и SMN2 имеет решающее значение в генетическом консультировании и прогностической оценке, разработка технологий, позволяющих точно идентифицировать количество копий генов является критической задачей. Как было показано, особенно важным является измерение не только количества копий гена SMN1, но и SMN2 (4, 6).
Технология цифровой ПЦР (digital PCR, dPCR или digital droplet PCR, ddPCR) имеет преимущество в отношении меньшей чувствительности к ингибиторам ПЦР. Кроме того, она позволяет получить точные (абсолютные) измерения количества копий участков ДНК, путём проведения флуоресцентных анализов, протекающих в десятках тысяч независимых реакций (капель, лунок) (7-8). Точность измерения повышается за счет измерения каждой введенной молекулы ДНК, содержащих целевой фрагмент, т.е. абсолютная квантификация ДНК. Подобная точность достигается за счет разобщения каждой молекулы ДНК на 20 тыс. независимых реакций.
Отличительной особенностью цифровой ПЦР от количественной ПЦР в режиме реального времени является измерение флуоресценции не во время ПЦР, а «по конечной точке», т.е. по завершению ПЦР. Тем самым достигается высокая точность анализа, который позволяет определить целочисленное количество копий в локусах, по сравнению с другими технологиями (7-9).
Существует ряд изобретений близких по заявляемой технической сущности (10-14). Однако результаты существующих разработок сопровождаются «эффектом дождя», т.е. слияние кластеров, что затрудняет оценку копий (15-18). Кроме того, используется разный набор зондов на разные однонуклеотидные замены, для дискриминации кластеров между генами SMN1 и SMN2. Также обычно выполняется несколько реакций, что увеличивает трудозатраты, либо работы фокусируются только на одном гене.
В данной работе авторы предлагают способ одновременной оценки количества копий 7 экзона генов SMN1 и SMN2 за одну реакцию. Зонды подбирались на один однонуклеотидный аллель в 7 экзоне, который характерен только конкретному гену. Так NC_000005.10:g.70951946C (GRCh38.p14) специфичен для гена SMN1, тогда как NC_000005.10:g.70076526T (GRCh38.p14) - для гена SMN2. Точность измерения повышается за счет использования абсолютной квантификации (т.е. применение цифровой ПЦР), оценки единого аллеля (аллельная дискриминация), универсальных праймеров на оба гена, и использование модифицированных зондов, а именно замкнутых нуклеиновых кислот.
Новая техническая задача - подбор последовательностей (структуры) олигонуклеотидных проб, разработка протокола проведения цифровой ПЦР для абсолютной квантификации (точного определения количества) генов SMN2 и SMN1 с определением делеции 7 экзона в биологическом образце человека.
Указанный результат достигается за счет разработки набора синтетических олигонуклеотидных последовательностей и проб, их модификации, а также модифицированного протокола проведения цифровой ПЦР для подсчета количества генов SMN2 и SMN1 с определением делеции 7 экзона в биологическом образце человека.
Техническим результатом изобретения является повышение точности оценки количества гена SMN2 и SMN1 с определением делеции 7 экзона в интересующих биологических образцах человека, по сравнению с существующими аналогами.
Разработан набор праймеров и флюоресцентных проб (по принципу TaqMan-проб) для генов SMN1 (белок выживаемости мотонейронов 1) и SMN2. Учитывая необходимость подсчета количества генов SMN1 и SMN2 относительно референсного региона, предполагается использовать данный набор с набором праймеров и флюоресцентных проб, специфичных на регионы генома, выступающих в качестве контрольных, такие как локусы гена HBB, TERT, RPP30.
Изобретение поясняется фигурами:
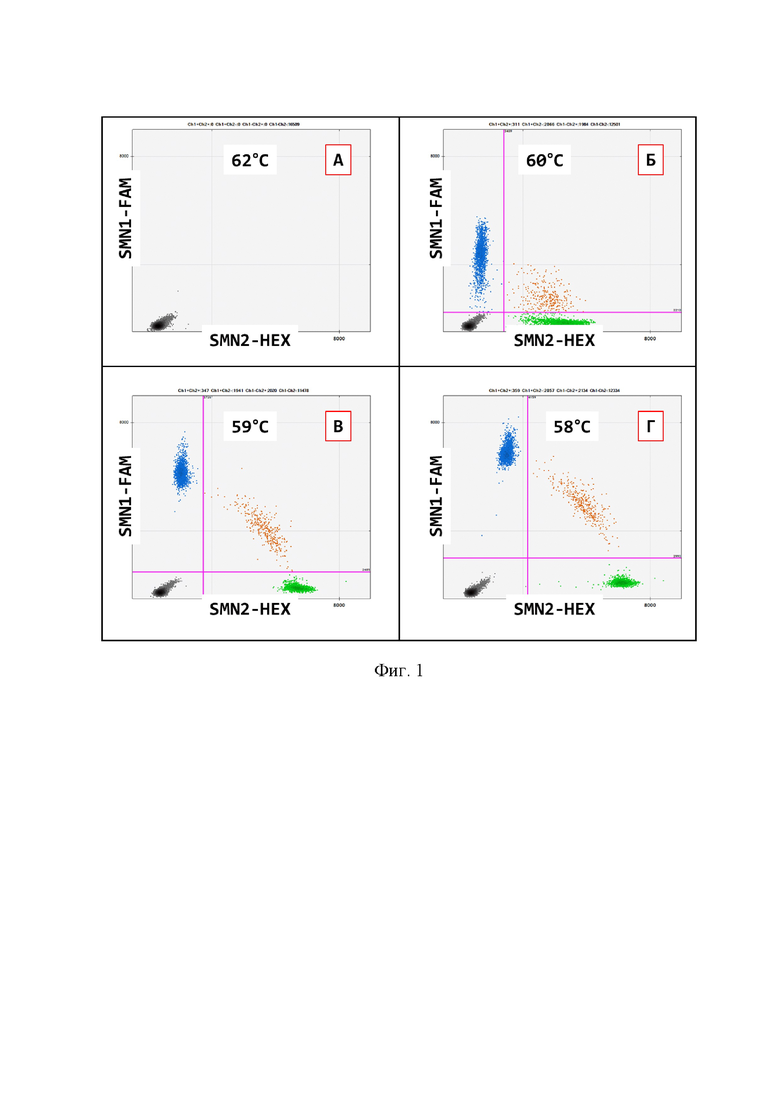
на фигуре 1 представлен пример двумерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР в зависимости от температуры элонгации;
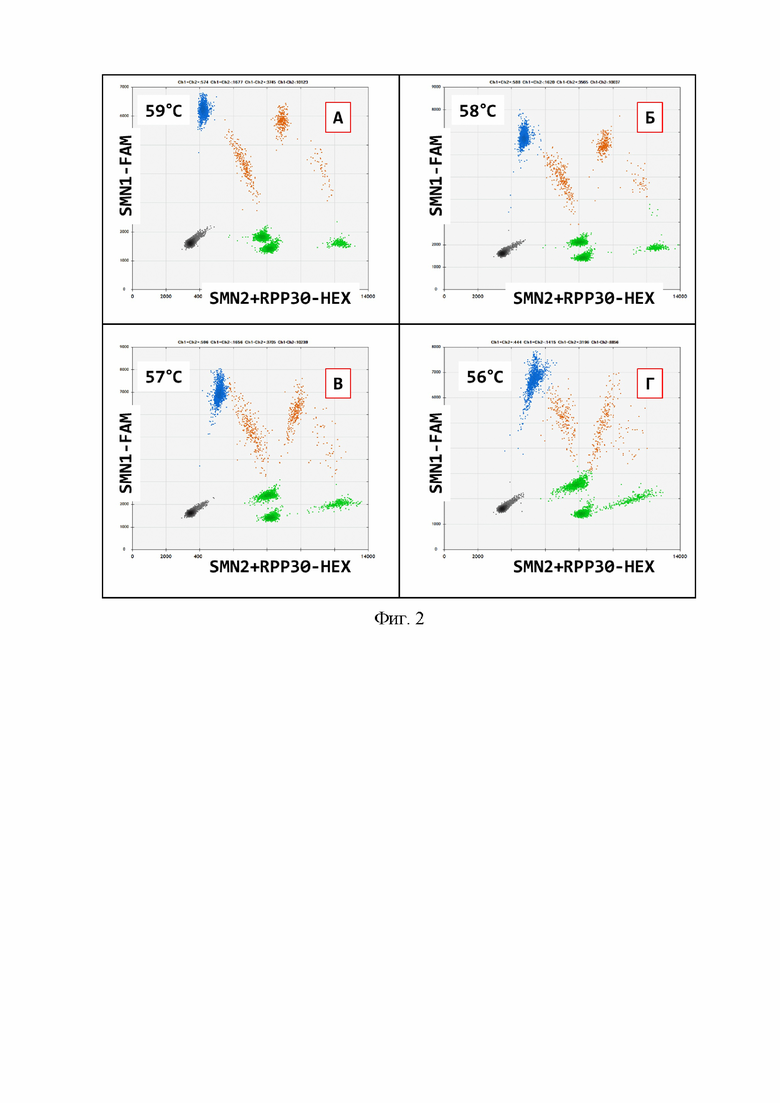
на фигуре 2 представлен пример двумерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР в зависимости от температуры элонгации при сочетании генов SMN1 и SMN2 с референсным геном RPP30 по каналу HEX с одинаковой концентрацией;
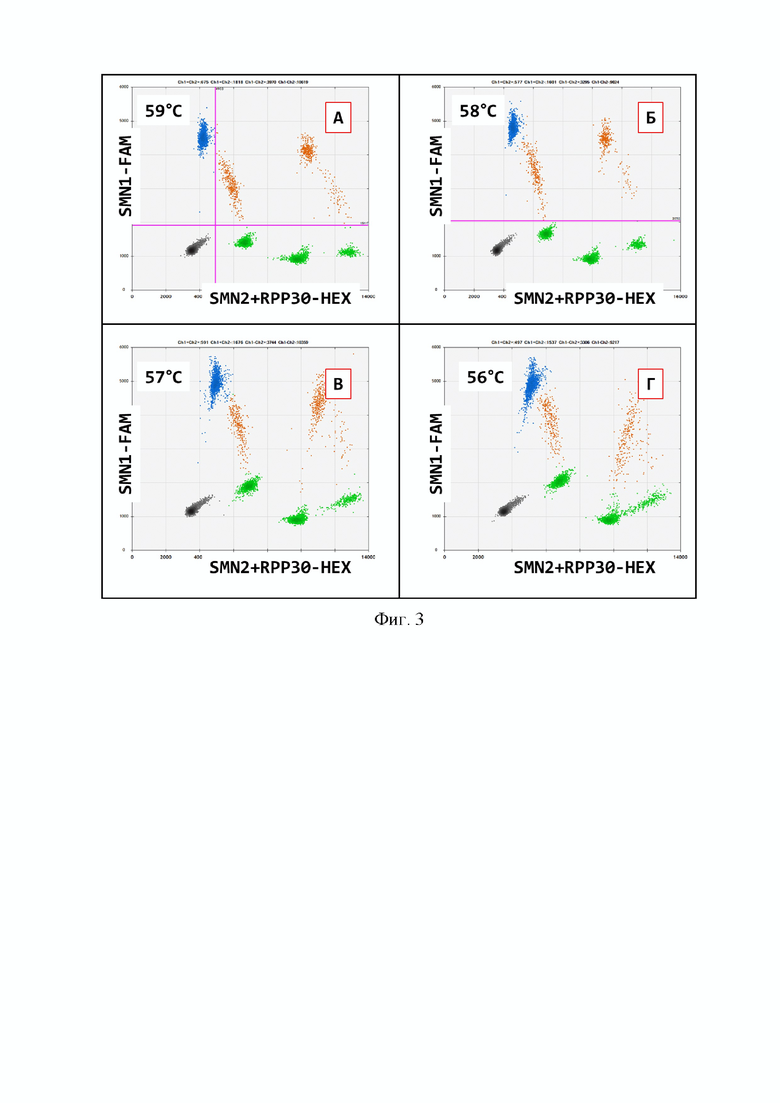
на фигуре 3 представлен пример двумерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР в зависимости от температуры элонгации при сочетании генов SMN1 и SMN2 с высокой концентрацией зонда для референсного гена RPP30 с каналом HEX;
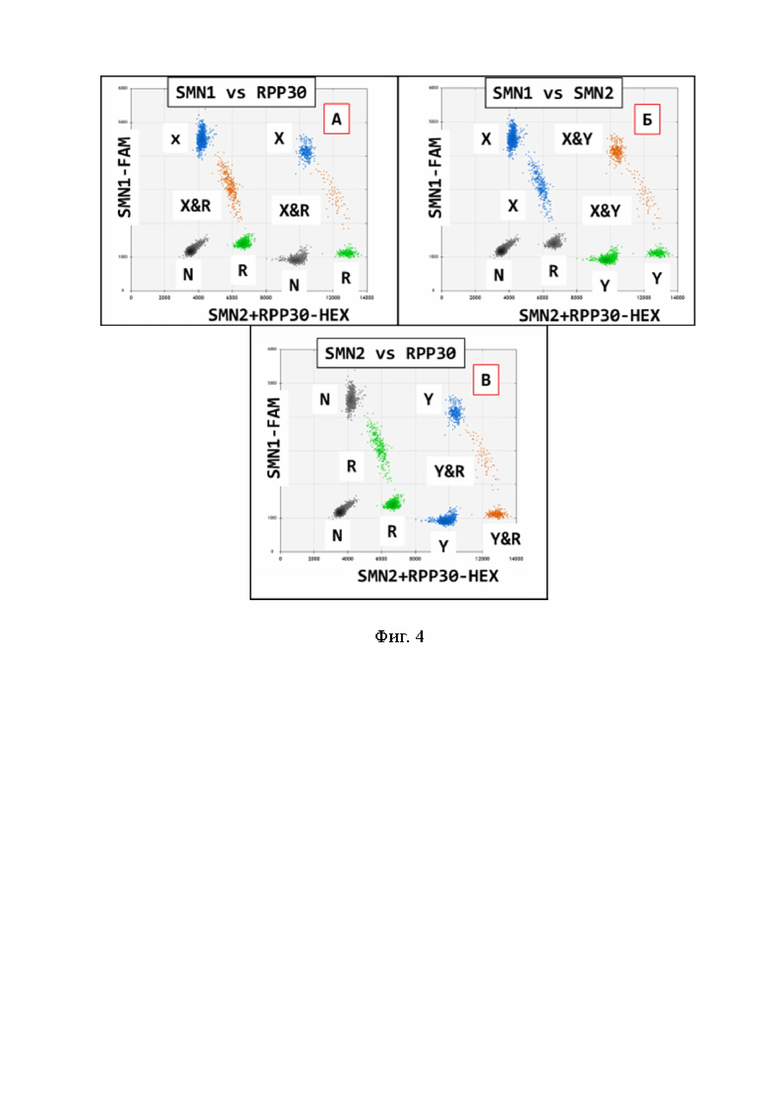
на фигуре 4 представлен пример результатов анализа и их принципа оценки соотношения SMN1 к SMN2, а также количества копий 7 экзона генов SMN1 и SMN2 при использовании референсного зонда на ген RPP30. SMN1 - X- положительные кластеры; SMN2 - Y - положительные кластеры;
R - кластеры эталонного региона; N - отрицательные кластеры.
Разработанные праймеры и пробы были протестированы на отсутствие сайтов отжига в ядерном геноме с помощью программы BLAST онлайн (19). Генотипирование с помощью набора проб осуществляли по модифицированному протоколу генотипирования методом dPCR и ddPCR следующим образом:
1. В качестве материала для исследования можно использовать препарат ДНК индивида, выделенный из биообразца любым способом (включая сухие пятна крови), который позволяет использовать ДНК в качестве матрицы в полимеразной цепной реакции. Основным требованием к качеству используемой ДНК является отсутствие минимальных признаков деградации (разрушения) ДНК. Рекомендуется провести оценку качества ДНК с помощью любого метода ДНК электрофореза, при котором должен присутствовать только высокомолекулярный бэнд тотальной ДНК, отсутствие бэндов примесей РНК и любых «шмеров» - признаков деградации ДНК. Выполнение данной рекомендации позволит избежать неточности в измерении.
2. Образец исследуемой ДНК необходимо развести до рабочей концентрации, оптимально 100 нг/мкл, для удобства последующих манипуляций. В реакционную смесь могут входить безнуклеазная вода, буферы для фермента рестрикции и иные компоненты, рекомендованные производителем.
3. Реакционная смесь для проведения мультиплексной цифровой ПЦР состоит из двух объемов мастер-микса производителя dPCR или ddPCR, мультиплекса из праймеров и зондов для исследуемых генов SMN1, SMN2 и референсного региона, безнуклеазной воды и образца ДНК.
4. Оптимальная концентрация ДНК выбирается согласно рекомендациям производителя прибора dPCR или ddPCR. Оптимальным соотношением является от 20 нг ДНК на 20 мкл реакционной смеси (фиг. 1-4).
5. Использовалась стандартная для TaqMan технологии концентрация праймеров, а именно 500 нM. Оптимальная концентрация проб, специфичных на гены SMN1 и SMN2, составляла 250 нМ (табл. 1), а для референсного региона - 500 нМ (фиг. 2-3).
Таблица 1. Структура набора синтетических олигонуклеотидных праймеров и проб.
Примечание: * «краситель» являются флуоресцентными метками, применяемых для мечения олигонуклеотидов. Подбор необходимого «красителя» осуществляется согласно спецификации производителя dPCR (ddPCR). Гаситель подбирается согласно выбранному красителю. +N - замкнутый нуклеозид (LNA, locked nucleiс acid).
6. Для одновременной оценки количества копий 7 экзона генов SMN1 и SMN2 за одну реакцию зонды подбирались на один однонуклеотидный аллель в 7 экзоне, который характерен только конкретному гену. Так NC_000005.10:g.70951946C (GRCh38.p14) специфичен для гена SMN1, тогда как NC_000005.10:g.70076526T (GRCh38.p14) для гена SMN2. Для проведения аллель-специфичной цифровой ПЦР зонды модифицировались «замкнутыми» нуклеиновыми кислотами (locked nucleiс acid), согласно схеме представленной в табл.1.
7. Протокол программы ПЦР основан на 2-этапной ПЦР, включающей: этап денатурации при +94°С, этап одновременного отжига и элонгации при +59°С в течение 1 мин. (фиг 1-3). Скорость снижения температуры 1,5°С в секунду, согласно рекомендациям технологии проведения цифровой ПЦР. Этап «горячего старта» и «горячего завершения» может быть добавлен согласно протоколу производителя. Хранение готовых продуктов ПЦР возможно при +10°С в течение 24 часов.
8. Общая методическая постановка dPCR и получение сигналов флуоресценции проводится согласно протоколу производителя технологии digital PCR (7). Основным требованием для оценки сигналов флуоресценции нанореакций является достаточное общее количество нанореакций, которое должно превышать более 10000 капель на реакцию для адекватной оценки результатов. Примеры получаемых результатов представлены на фигуре 4.
9. Определение количества копий 7 экзона гена SMN1 проводится согласно схеме, представленной на фигуре 4А, и по формуле,
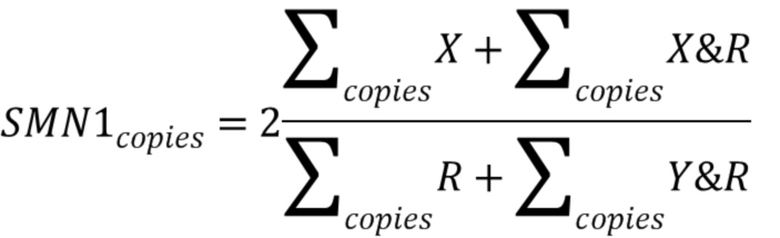 , [1]
, [1]
где 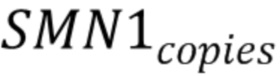 - количество копий 7 экзона гена SMN1 на клетку,
- количество копий 7 экзона гена SMN1 на клетку, 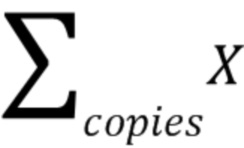 - сумма количества копий 7 экзона гена SMN1 в Х-кластере,
- сумма количества копий 7 экзона гена SMN1 в Х-кластере, 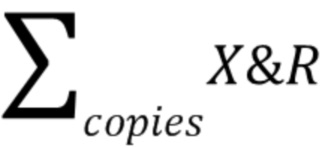 - сумма количества копий ДНК, содержащихся в кластере Х&R-кластере, в которой содержатся участки ДНК 7 экзона гена SMN1 и референсного региона гена RPP30,
- сумма количества копий ДНК, содержащихся в кластере Х&R-кластере, в которой содержатся участки ДНК 7 экзона гена SMN1 и референсного региона гена RPP30, 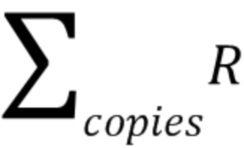 - количество копий гена RPP30 в R-кластере.
- количество копий гена RPP30 в R-кластере.
10. Определение количества копий 7 экзона гена SMN2 проводится согласно схеме, представленной на фигуре 4В, и по формуле
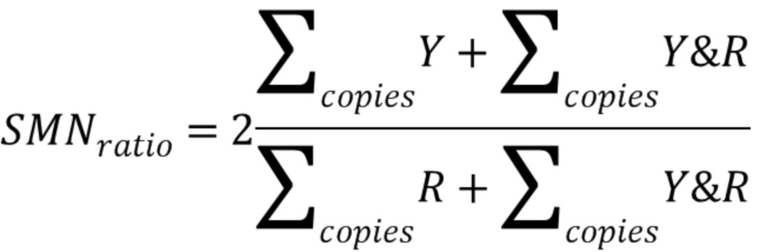 , [2]
, [2]
где 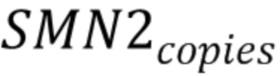 - количество копий 7 экзона гена SMN2 на клетку,
- количество копий 7 экзона гена SMN2 на клетку, 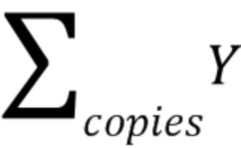 - сумма количества копий 7 экзона гена SMN2 в Y-кластере,
- сумма количества копий 7 экзона гена SMN2 в Y-кластере, 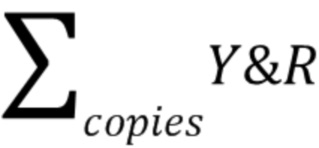 - сумма количества копий ДНК, содержащихся в кластере Y&R-кластере, в которой содержатся участки ДНК 7 экзона гена SMN2 и референсного региона гена RPP30,
- сумма количества копий ДНК, содержащихся в кластере Y&R-кластере, в которой содержатся участки ДНК 7 экзона гена SMN2 и референсного региона гена RPP30, 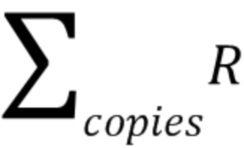 - количество копий гена RPP30 в R-кластере.
- количество копий гена RPP30 в R-кластере.
11. Определение соотношения генов SMN1 и SMN2 проводится согласно схеме, представленной на фигуре 4Б, и по формуле
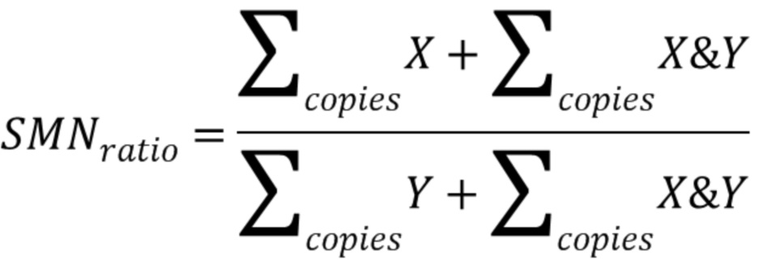 , [3]
, [3]
где 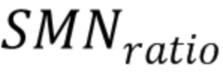 - соотношение количества генов SMN1 и SMN2,
- соотношение количества генов SMN1 и SMN2, 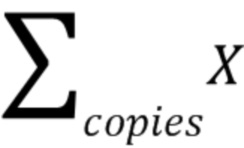 - сумма количества копий 7 экзона гена SMN2 в Х-кластере,
- сумма количества копий 7 экзона гена SMN2 в Х-кластере, 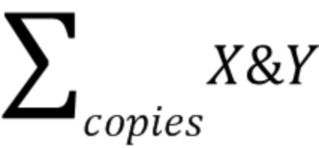 - сумма количества копий ДНК, содержащихся в кластере X&Y-кластере, в которой содержатся участки ДНК 7 экзона гена SMN1 и SMN2,
- сумма количества копий ДНК, содержащихся в кластере X&Y-кластере, в которой содержатся участки ДНК 7 экзона гена SMN1 и SMN2, 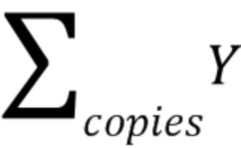 - сумма количества копий 7 экзона гена SMN2 в Y-кластере.
- сумма количества копий 7 экзона гена SMN2 в Y-кластере.
Фигура 1. Пример двумерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР в зависимости от температуры элонгации. Синий цвет точек - FAM-положительные ПЦР-капли по гену SMN1, зеленый - HEX-положительные по гену SMN2, оранжевый - положительные по обоим красителям, серый - отрицательные по обоим красителям. Общее количество использованной ДНК 20 нг.
Фигура 2. Пример двумерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР в зависимости от температуры элонгации при сочетании генов SMN1 и SMN2 с референсным геном RPP30 по каналу HEX с одинаковой концентрацией. Синий цвет точек - FAM-положительные ПЦР-капли, зеленый - HEX-положительные, оранжевый - положительные по обоим красителям, серый - отрицательные по обоим красителям. Общее количество использованной ДНК 20 нг.
Фигура 3. Пример двумерного графика распределения интенсивностей сигналов флуоресценции цифровой ПЦР в зависимости от температуры элонгации при сочетании генов SMN1 и SMN2 с высокой концентрацией зонда для референсного гена RPP30 с каналом HEX. Синий цвет точек - FAM-положительные ПЦР-капли, зеленый - HEX-положительные, оранжевый - положительные по обоим красителям, серый - отрицательные по обоим красителям. Общее количество использованной ДНК 20 нг.
Фигура 4. Пример результатов анализа и их принципа оценки соотношения SMN1 к SMN2, а также количества копий 7 экзона генов SMN1 и SMN2 при использовании референсного зонда на ген RPP30. Синий цвет точек - X-положительные кластеры по 7 экзону гена SMN1, Y-положительные кластеры по 7 экзону гена SMN2, R-положительный кластер референсного зонда по гену RPP30, N-кластер рассматривается как относительный негативный кластер, X&R-кластер содержит положительные капли по 7 экзону гена SMN1 и референсного региона по гену RPP30, Y&R-кластер содержит положительные капли по 7 экзону гена SMN2 и референсного региона по гену RPP30, X&Y-кластер содержит положительные капли по 7 экзону гена SMN1 и SMN2. Общее количество использованной ДНК 20 нг.
SMN1 - X- положительные кластеры; SMN2 - Y - положительные кластеры;
R - кластеры эталонного региона; N - отрицательные кластеры.
Источники информации, принятые во внимание при составлении описания:
1. Lefebvre S, Bürglen L, Reboullet S, et al. Identification and characterization of a spinal muscular atrophy-determining gene. Cell. 1995 Jan 13;80(1):155-65. doi: 10.1016/0092-8674(95)90460-3.
2. Munsat TL, Davies KE. International SMA consortium meeting. (26-28 June 1992, Bonn, Germany). Neuromuscul Disord. 1992;2(5-6):423-8. doi: 10.1016/s0960-8966(06)80015-5.
3. Ogino S, Wilson RB. Genetic testing and risk assessment for spinal muscular atrophy (SMA). Hum Genet. 2002 Dec;111(6):477-500. doi: 10.1007/s00439-002-0828-x.
4. Prior TW, Nagan N. Spinal Muscular Atrophy: Overview of Molecular Diagnostic Approaches. Curr Protoc Hum Genet. 2016 Jan 1;88:9.27.1-9.27.13. doi: 10.1002/0471142905.hg0927s88.
5. Ogino S, Wilson RB, Gold B. New insights on the evolution of the SMN1 and SMN2 region: simulation and meta-analysis for allele and haplotype frequency calculations. Eur J Hum Genet. 2004 Dec;12(12):1015-23. doi: 10.1038/sj.ejhg.5201288.
6. Cobb MS, Rose FF, Rindt H, et al. Development and characterization of an SMN2-based intermediate mouse model of Spinal Muscular Atrophy. Hum Mol Genet. 2013 May 1;22(9):1843-55. doi: 10.1093/hmg/ddt037.
7. Hindson BJ et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number // Anal. Chem. 2011. Vol. 83, № 22. P. 8604-8610.
8. Pinheiro LB et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification // Anal. Chem. 2012. Vol. 84, № 2. P. 1003-1011.
9. Usher CL et al. Structural forms of the human amylase locus and their relationships to SNPs, haplotypes and obesity // Nat. Genet. Nature Publishing Group, 2015. Vol. 47, № 8. P. 921-925.
10. Anhuf D et al. Determination of SMN1 and SMN2 copy number using TaqMan technology. Hum Mutat. 2003 Jul;22(1):74-8. doi: 10.1002/humu.10221.
11. Jiang L et al. Development and validation of a 4-color multiplexing spinal muscular atrophy (SMA) genotyping assay on a novel integrated digital PCR instrument. Sci Rep. 2020 Nov 16;10(1):19892. doi: 10.1038/s41598-020-76893-7.
12. Stabley DL et al.. Establishing a reference dataset for the authentication of spinal muscular atrophy cell lines using STR profiling and digital PCR. Neuromuscul Disord. 2017 May;27(5):439-446. doi: 10.1016/j.nmd.2017.02.002.
13. Stabley DL et al. SMN1 and SMN2 copy numbers in cell lines derived from patients with spinal muscular atrophy as measured by array digital PCR. Mol Genet Genomic Med. 2015 Jul;3(4):248-57. doi: 10.1002/mgg3.141.
14. Vidal-Folch N et al. Multiplex Droplet Digital PCR Method Applicable to Newborn Screening, Carrier Status, and Assessment of Spinal Muscular Atrophy. Clin Chem. 2018 Dec;64(12):1753-1761. doi: 10.1373/clinchem.2018.293712.
15. US 10612081 B2 (Hitchison JB et al). 11 февраля 2011, весь документ.
16. US 9494520 B2 (Link DR). 19 апреля 2013, весь документ.
17. US 9366632 B2 (Link DR et al). 19 апреля 2013, весь документ.
18. US 10808279 B2 (Link DR et al). 2 июля 2019, весь документ.
19. Ye J et al. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. // BMC Bioinformatics. 2012. Vol. 13. P. 134.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3" fileName="ddPCR
SMN1 SMN2 copies.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-10-02">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-09-27</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-09-27</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Научно-исследовательский институт
медицинской генетики (НИИ медицинской генетики) Федерального
государственного бюджетного научного учреждения "Томский
национальный исследовательский медицинский центр Российской академии
наук" (Томский НИМЦ)</ApplicantName>
<ApplicantNameLatin>Research Institute of Medical Genetics of Tomsk
National Research Medical Center of the Russian Academy of
Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ определения делеции 7
экзона в гене SMN1 с определением количества копий гена SMN2 в
образцах ДНК с помощью цифровой полимеразной цепной
реакции</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaatgtcttgtgaaacaaaatgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaatgtgagcaccttccttct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Locked nucleic acid</INSDQualifier_value>
<NonEnglishQualifier_value>замкнутая нуклеиновая
кислота</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>9..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Locked nucleic acid</INSDQualifier_value>
<NonEnglishQualifier_value>замкнутая нуклеиновая
кислота</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Locked nucleic acid</INSDQualifier_value>
<NonEnglishQualifier_value>замкнутая нуклеиновая
кислота</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attttgtctgaaaccctgtaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Locked nucleic acid</INSDQualifier_value>
<NonEnglishQualifier_value>замкнутая нуклеиновая
кислота</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>11..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Locked nucleic acid</INSDQualifier_value>
<NonEnglishQualifier_value>замкнутая нуклеиновая
кислота</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Locked nucleic acid</INSDQualifier_value>
<NonEnglishQualifier_value>замкнутая нуклеиновая
кислота</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgattttgtctaaaaccctgtaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Молекулярно-генетическая система детекции делеции экзона 7 гена SMN1, пригодная для проведения неонатального скрининга | 2021 |
|
RU2796350C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Способ оценки эффективности терапевтических конструкций, направленных на повышение уровня полноразмерных транскриптов SMN | 2022 |
|
RU2811909C1 |
| Тест-система и способ выявления A, B, D мутаций гена NPM1 для количественного определения минимальной остаточной болезни | 2023 |
|
RU2830545C1 |
| АДЕНОАССОЦИИРОВАННЫЙ ВИРУСНЫЙ ВЕКТОР, СОСТОЯЩИЙ ИЗ БЕЛКОВ КАПСИДА РНР.В, НУКЛЕИНОВОЙ КИСЛОТЫ, КОДИРУЮЩЕЙ БЕЛОК SMN, И ЕГО ПРИМЕНЕНИЕ | 2022 |
|
RU2833225C2 |
| Генетическая конструкция, адаптированная для доставки гена SMN1 человека с помощью аденоассоциированного вируса серотипа 2 для обеспечения нейроспецифичной экспрессии | 2022 |
|
RU2801848C1 |
| Способ детекции генотипа Mycobacterium tuberculosis Beijing 14717-15-кластер методом полимеразной цепной реакции в формате реального времени | 2023 |
|
RU2839699C2 |
| Олигонуклеотиды для диагностики эпилепсии методом количественной ПЦР | 2023 |
|
RU2815113C1 |
| Генетическая конструкция, содержащая последовательности химерных направляющих РНК для делеции гена SMN1 человека в культурах клеток человека | 2022 |
|
RU2816897C2 |
| Способ прогностической оценки гепатотоксичности у ВИЧ-инфицированных лиц при антиретровирусной терапии на основе определения делеционного полиморфизма генов биотрансформации ксенобиотиков GSTM1, GSTT1, CYP2D6 человека и набор олигодезоксирибонуклеотидных праймеров и флуоресцентно меченых зондов | 2022 |
|
RU2807530C1 |
Изобретение относится к области молекулярной и клеточной биологии и предназначено для оценки количества копий гена SMN1 и SMN2 с определением делеции 7 экзона гена SMN1 в исследуемых образцах ДНК человека с помощью цифровой капельной полимеразной реакции, основанной на методике аллель специфичной абсолютной квантификации локуса генов SMN1 и SMN2. Технический результат - повышение точности количественной оценки копий гена SMN1 и SMN2 с определением делеции 7 экзона гена SMN1 в интересующих биологических образцах человека. Указанный результат достигается путем использования количественного подхода с применением технологии цифровой ПЦР и мультиплексного набора праймеров и зондов, специфических 7 экзона генов SMN1 и SMN2. 4 ил., 1 табл.
Способ одновременной оценки количества копий 7 экзона генов SMN1 и SMN2 за одну реакцию методом цифровой полимеразной цепной реакции (ПЦР), отличающийся тем, что при проведении цифровой ПЦР используют набор специфичных праймеров и зондов, представленных последовательностями SEQ ID NO: 1-4.
| CN 111118151 A, 08.05.2020 | |||
| CN 112522389 A, 19.03.2021 | |||
| Stabley DL et al., SMN1 and SMN2 copy numbers in cell lines derived from patients with spinal muscular atrophy as measured by array digital PCR | |||
| Mol Genet Genomic Med | |||
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
| PMID: 26247043; PMCID: PMC4521962 | |||
| Молекулярно-генетическая система детекции делеции экзона 7 гена SMN1, пригодная для проведения неонатального скрининга | 2021 |
|
RU2796350C1 |
Авторы
Даты
2025-04-22—Публикация
2024-10-25—Подача