Перекрестная ссылка на родственные заявки
Настоящая заявка заявляет приоритет предварительной заявки на патент США № 62648888, поданной 27 марта 2018 г., и предварительной заявки на патент США № 6268880, поданной 27 марта 2018 г., которые в полном объеме включены здесь посредством ссылки.
Область техники, к которой относится изобретение
Настоящее изобретение, в общем, относится к области направляющих и навигационных контрольных белков (GNC) с мультиспецифическими связывающими активностями с поверхностными молекулами на иммунных клетках и опухолевых клетках, в частности, относится к получению и применению белков GNC.
Уровень техники
У опухолевых клеток развиваются различные механизмы, чтобы уклониться от надзора иммунной системы. Одним из основных механизмов избегания надзора иммунной системы является снижение распознавания опухолевых клеток иммунной системой. Дефектная презентация опухолеспецифических антигенов или ее отсутствие приводит к иммунной толерантности и прогрессированию рака. Однако даже при наличии эффективного иммунологического распознавания в опухолях развиваются другие механизмы, позволяющие избежать элиминации под действием иммунной системы. Иммунокомпетентные опухоли создают супрессивную микросреду для подавления иммунного ответа. В формирование супрессивной опухолевой микросреды вовлекаются несколько «игроков», включая опухолевые клетки, регуляторные Т-клетки, миелоидные супрессорные клетки, стромальные клетки и клетки других типов. Подавление иммунного ответа может осуществляться в формате, зависимом от контактирования с клеткой, а также независимо от контактирования, посредством секреции иммуносупрессивных цитокинов или элиминации важных факторов выживаемости из локальной среды. Подавление, зависимое от контактирования с клеткой, основывается на молекулах, экспрессируемых на клеточной поверхности, например лиганда 1 белка запрограммированной гибели (PD-L1), T-лимфоцит-ассоциированного белка 4 (CTLA-4) и других [Dunn, Old et al., 2004; Adachi and Tamada, 2015].
Поскольку механизмы, с помощью которых опухоли избегают распознавания иммунной системой, продолжают лучше пониматься, недавно появились новые способы лечения, нацеленные на эти механизмы. 25 марта 2011 г. Управление по санитарному надзору за качеством пищевых продуктов и медикаментов США (FDA) одобрило для применения инъекционный ипилимумаб (Ервой, Bristol-Myers Squibb) для лечения неоперабельной или метастатической меланомы. Ервой связывается с цитотоксическим Т-лимфоцит-ассоциированным белком 4 (CTLA-4), экспрессируемым на активированных T-клетках, и блокирует взаимодействие CTLA-4 с CD80/86 на антигенпрезентирующих клетках, блокируя тем самым негативный или ингибиторный сигнал, доставляемый в T-клетку через CTLA-4, приводя к реактивации антигенспецифических Т-клеток, что, в свою очередь, приводит к ликвидации опухоли у многих пациентов. Несколько лет спустя в 2014 г. FDA одобрило препарат Кейтруда (пембролизумаб, Merck) и препарат Опдиво (ниволумаб, Bristol-Myers Squibb) для лечения меланомы на поздних стадиях. Эти моноклональные антитела связываются с PD-1, который экспрессируется на активированных и/или истощенных Т-клетках, и блокируют взаимодействие PD-1 с PD-L1, который экспрессируется на опухолях, тем самым элиминируя ингибиторный сигнал через PD-1 в Т-клетку, что приводит к реактивации антигенспецифических Т-клеток, приводя, опять же, у многих пациентов к эрадикации опухоли. С тех пор были проведены дальнейшие клинические испытания, с целью сравнения эффективности одного моноклонального антитела Ервой с комбинацией моноклональных антител Ервой и Опдиво в лечении меланомы на поздних стадиях, которые показали повышение общей выживаемости и выживаемости без прогрессирования заболевания у пациентов, получавших комбинацию антител (Hodi, Chesney et al., 2016, Hellman, Callahan et al., 2018). Однако как показали результаты многих клинических испытаний, существенный положительный эффект лечения больных раком моноклональными антителами, которые специфичны для одной или более молекул иммунных контрольных точек, проявляется только у пациентов с высокой мутационной нагрузкой, в результате которой образуется новый эпитоп(ы) Т-клеток, который распознается антигенспецифическими Т-клетками, и в результате имеет место клинический ответ (Snyder, Makarov et al., 2014). Те пациенты, которые имеют низкую опухолевую мутационную нагрузку, в основном не демонстрируют объективный клинический ответ (Snyder, Makarov et al., Hellman, Callahan et al., 2018).
В последние годы другие группы исследователей разработали альтернативный подход, для которого не требуется наличия презентации неоэпитопа антигенпрезентирующими клетками для активации Т-клеток. Одним примером является разработка биспецифического антитела, в котором связывающий домен антитела, который специфичен для опухоль-ассоциированного антигена, например CD19, связан со связывающим доменом антитела, специфичным для CD3 на Т-клетках, создавая таким образом биспецифический активатор Т-клеток или молекулу BiTe. В 2014 г. FDA одобрило для применения биспецифическое антитело под названием блинатумомаб для лечения острого лимфобластного лейкоза из предшественников B-клеток. Блинатумомаб связывает одноцепочечный вариабельный фрагмент (scFv), специфичный для CD19, экспрессируемым на лейкозных клетках, с scFv, специфичный для CD3, экспрессируемым на Т-клетках (Bejnjamin and Stein 2016). Однако, несмотря на начальную частоту ответа у > 50% пациентов с рецидивирующим или рефрактерным ALL, многие пациенты резистентны к терапии блинатумомабом, или у них развиваются рецидивы после успешного лечения блинатумумабом. Появляются доказательства того, что резистентность к блинатумомабу или развитие рецидивов после лечения блинатумомабом обусловлены экспрессией молекул, ингибирующих иммунные контрольные точки, экспрессируемых на опухолевых клетках, таких как PD-L1, который регулирует ингибиторный сигнал через PD-1, экспрессируемый на активированных Т-клетках (Feucht, Kayser et al., 2016). В случае пациента, участвующего в исследовании, с резистентностью к терапии блинатумомабом, был выполнен второй раунд терапии блинатумомабом, но с добавлением моноклонального антитела, пембролизумаба (Кейтруда, Merck), который специфичен для PD-1, и блокирует взаимодействие PD-1, экспрессируемого Т-клетками, с PD-L1, который экспрессируется опухолевыми клетками, что приводило к высокому ответу и снижению количества опухолевых клеток в костном мозге до уровня от 45% до менее чем 5% у этого одного пациента (Feucht, Kayser et al., 2016). Эти результаты показывают, что объединение биспецифической молекулы BiTe с одним или более моноклональными антителами может существенно повысить клиническую активность по сравнению с каждым одним агентом. Несмотря на многообещающий результат, затраты, ведущая к комбинированной терапии, должны быть высоким за счет многочисленных клинических испытаний и трудностей с набором репрезентативных популяций.
Адоптивная клеточная терапия Т-клетками с химерным антигенным рецептором (CAR-T) является еще одной перспективной иммунотерапией для лечения рака. Клинический успех терапии CAR-T выявил длительные полные ремиссии и длительную выживаемость пациентов с CD19-положительными невосприимчивыми к лечению В-клеточными злокачественными новообразованиями (Gill and June, 2015). Однако стоимость и сложность, связанные с созданием и применением персонализированной и генетически модифицированной иммунотерапии CAR-T, ограничивают их создание и применение специализированными центрами для лечения относительно небольшого числа пациентов. Синдром высвобождения цитокинов (CRS), также известный как цитокиновый шторм, считается основным неблагоприятным эффектом после инфузии сконструированных CAR-T клеток (Bonifant, Jackson et al., 2016). Во многих случаях возникновение и степень тяжести CRS, по-видимому, являются специфическими для пациента. Имеющиеся в настоящее время варианты смягчения CRS в основном сосредоточены на быстром ответе и лечении, поскольку возможности контроля CRS до инфузии Т-клеток ограничены.
В то время как эффективность CAR-T терапии, специфичной для CD19-позитивных В-клеточных новообразованиях, в настоящее время четко установлена, эффективность CAR-T терапии против солидных опухолей до настоящего времени не была однозначно продемонстрирована. В настоящее время проводится много клинических испытаний для изучения разнообразных ассоциированных с солидными опухолями антигенов (TAA), для CAR-T терапии. Неэффективный перенос Т-клеток в опухоли, иммуносупрессивная опухолевая микросреда, субоптимальная специфичность распознавания антигенов и отсутствие контроля побочных эффектов, связанных с лечением, в настоящее время рассматриваются в качестве основных препятствий в CAR-T терапии солидных опухолей (Li, Li et al. 2018). Возможность контролирования терапевтическим эффектом, а также любым неблагоприятным эффектом до и после инфузии CAR-T клеток, ограничена.
Сущность изобретения
Заявка обеспечивает, среди прочего, способы получения терапевтических композиций, содержащих направляющие и навигационные белки (GNC), способы лечения злокачественных заболеваний с использованием направляющих и навигационных контрольных белков (GNC) и терапевтические композиции, содержащие белки GNC или терапевтические клетки, включающие цитотоксические клетки, покрытые (или связанные) белками GNC.
В одном аспекте заявка обеспечивает терапевтические композиции. В одном варианте осуществления терапевтическая композиция содержит цитотоксическую клетку, белок GNC и терапевтическую клетку. Терапевтическая клетка содержит белок GNC, связанный с цитотоксической клеткой посредством связывающего взаимодействия с рецептором цитотоксической клетки, и терапевтическая клеточная композиция по существу не содержит экзогенной вирусной и невирусной ДНК и РНК.
В одном варианте осуществления терапевтическая композиция может дополнительно содержать второй белок GNC, вторую терапевтическую клетку или их комбинацию, где вторая терапевтическая клетка включает цитотоксические клетки со вторым связанным с ними белком GNC или с первым и вторым GNC белки, связанные с этим.
Белок GNC включает цитотоксический связывающий фрагмент и нацеливающий на опухоль фрагмент. Цитотоксический связывающий фрагмент обладает специфичностью связывания с рецептором цитотоксической клетки и конфигурирован для активации цитотоксической клетки посредством связывания с рецептором цитотоксической клетки. Нацеливающий на опухоль фрагмент обладает специфичностью связывания с рецептором опухолевых клеток.
В одном варианте осуществления белок GNC включает связывающий домен для рецепторов Т-клеток. Примеры T-клеточного рецептора включают без ограничения CD3, CD28, PDL1, PD1, OX40, 4-1BB, GITR, TIGIT, TIM-3, LAG-3, CTLA4, CD40L, VISTA, ICOS, BTLA, Light, CD30, NKp30, CD28H, CD27, CD226, CD96, CD112R, A2AR, CD160, CD244, CECAM1, CD200R, TNFRSF25 (DR3) или их комбинации. В одном варианте осуществления белок GNC способен активировать Т-клетку посредством связывания связывающего Т-клетки фрагмента с Т-клеточным рецептором на Т-клетке. В одном варианте осуществления белок GNC способен активировать Т-клетку посредством связывания множества T-клеточных связывающих фрагментов на Т-клетке.
В одном варианте осуществления белок GNC включает связывающий домен для рецептора NK-клетки. Примеры рецепторов NK-клеток включают, без ограничения, активационные рецепторы NK-клеток, такие как CD16, NKG2D, KIR2DS1, KIR2DS2, KIR2DS4, KIR3DS1, NKG2C, NKG2E, NKG2H; агонистические рецепторы, такие как NKp30a, NKp30b, NKp46, NKp80, DNAM-1, CD96, CD160, 4-1BB, GITR, CD27, OX-40, CRTAM; и антагонистические рецепторы, такие как KIR2DL1, KIR2DL2, KIR2DL3, KIR3DL1, KIR3DL2, KIR3DL3, NKG2A, NKp30c, TIGIT, SIGLEC7, SIGLEC9, LILR, LAIR-1, CD-1, KLRG1, PD-1.
В одном варианте осуществления белок GNC включает связывающий домен для рецептора макрофагов. Примеры рецепторов макрофагов включают, без ограничения, агонистический рецептор макрофагов, такой как TLR2, TLR4, CD16, CD64, CD40, CD80, CD86, TREM-1, TREM-2, ILT-1, ILT-6a, ILT-7, ILT-8, EMR2, дектин-1, CD69; антагонистические рецепторы, такие как CD32b, SIRPα, LAIR-1, VISTA, TIM-3, CD200R, CD300a, CD300f, SIGLEC1, SIGLEC3, SIGLEC5, SIGLEC7, SIGLEC9, ILT-2, ILT-3, ILT-4, ILT-5, LILRB3, LILRB4, DCIR; и другие поверхностные рецепторы, такие как CSF-1R, LOX-1, CCR2, FRβ, CD163, CR3, DC-SIGN, CD206, SR-A, CD36, MARCO.
В одном варианте осуществления белок GNC включает связывающий домен для рецептора дендритных клеток. Примеры рецепторов дендритных клеток включают, без ограничения, агонистические рецепторы на дендритных клетках, такие как TLR, CD16, CD64, CD40, CD80, CD86, HVEM, CD70; антагонистические рецепторы, такие как VISTA, TIM-3, LAG-3, BTLA; и другие поверхностные рецепторы, такие как CSF-1R, LOX-1, CCR7, DC-SIGN, GM-CSF-R, IL-4R, IL-10R, CD36, CD206, DCIR, RIG-1, CLEC9A, CXCR4.
В одном варианте осуществления белок GNC может включать связывающий с T-клетками фрагмент и нацеливающий на опухоль фрагмент. В одном варианте осуществления связывающийся с Т-клетками фрагмент обладает специфичностью связывания с T-клеточным рецептором, включающим CD3, CD28, PDL1, PDL2, PD1, OX40, 4-1BB, GITR, TIGIT, TIM-3, LAG-3, CTLA4, CD40L, VISTA, ICOS, BTLA, Light, CD30, CD27 или их комбинации. В одном варианте осуществления нацеливающий на опухоль фрагмент обладает специфичностью связывания с рецептором опухолевых клеток. В одном варианте осуществления рецептор опухолевых клеток может включать BCMA, CD19, CD20, CD33, CD123, CD22, CD30, ROR1, CEA, HER2, EGFR, EGFRvIII, LMP1, LMP2A, мезотелин, PSMA, EpCAM, глипикан-3, gpA33, GD2, TROP2, другие еще не открытые опухоль-ассоциированные антигены или их комбинации.
В одном варианте осуществления белок GNC может обладать мультиспецифической антигенсвязывающей активностью с поверхностными молекулами Т-клетки и опухолевой клетки. В одном варианте осуществления направляющий и навигационный контрольный белок (GNC) содержит связывающий домен для активирующего рецептора Т-клеток, связывающий домен для опухоль-ассоциированного антигена, связывающий домен для рецептора иммунных контрольных точек и связывающий домен для костимулирующего рецептора Т-клеток.
В одном варианте осуществления связывающий домен для опухоль-ассоциированного антигена не является смежным со связывающим доменом для костимулирующего рецептора Т-клеток. В одном варианте осуществления связывающий домен для активирующего рецептора Т-клеток, является смежным со связывающим доменом для опухоль-ассоциированного антигена (TAA). Активирующий рецептор Т-клеток может включать, без ограничения, CD3. Костимулирующий рецептор Т-клеток может включать, без ограничения, 4-1BB, CD28, OX40, GITR, CD40L, ICOS, Light, CD27, CD30 или их комбинацию. Рецептор иммунных контрольных точек может включать, без ограничения, PD-L1, PD-1, TIGIT, TIM-3, LAG-3, CTLA4, BTLA, VISTA, PDL2 или их комбинацию.
Опухоль-ассоциированный антиген (TAA) может включать, без ограничения, ROR1, CD19, EGFRVIII, BCMA, CD20, CD33, CD123, CD22, CD30, CEA, HER2, EGFR, LMP1, LMP2A, мезотелин, PSMA, EpCAM, глипикан-3, gpA33, GD2, TROP2 или их комбинацию. В одном варианте осуществления опухоль-ассоциированный антиген может представлять собой ROR1. В одном варианте осуществления опухоль-ассоциированный антиген может представлять CD19. В одном варианте осуществления опухоль-ассоциированный антиген может представлять собой EGFRVIII.
В одном варианте осуществления направляющий и навигационный контрольный белок (GNC) может представлять антитело или мономер антитела или его фрагмент. В одном варианте осуществления белок GNC может представлять триспецифическое антитело. В одном варианте осуществления белок GNC может представлять собой тетраспецифическое антитело. В одном варианте осуществления белок GNC включает домен Fc или его фрагмент. Можно использовать любой домен Fc антитела. Примеры доменов Fc могут включать домены Fc из IgG, IgA, IgD, IgM, IgE или их фрагмент или комбинацию. Домен Fc может быть природным или сконструированным. В одном варианте осуществления домен Fc может содержать антигенсвязывающий сайт.
В одном варианте осуществления белок GNC включает биспецифическое антитело, триспецифическое антитело, тетраспецифическое антитело или их комбинацию, обеспечивающую до восьми связывающих мотивов белка GNC. Примеры антител, мономеров антител, их антигенсвязывающих фрагментов раскрыты здесь. В одном варианте осуществления белки GNC могут включать фрагмент иммуноглобулина G (IgG) с двумя тяжелыми цепями и двумя легкими цепями, и, по меньшей мере, два фрагмента scFv, ковалентно связанных с C- или N-концами тяжелой или легкой цепей. IgG фрагмент может обеспечивать стабильность фрагмента scFv, и триспецифический белок GNC может иметь два фрагмента для связывания с поверхностными молекулами на Т-клетках.
В одном варианте осуществления направляющий и навигационный контрольный белок (GNC) может представлять собой антитело. В одном варианте осуществления опухоль-ассоциированный антиген включает ROR1, CD19 или EGRFVIII. В одном варианте осуществления активирующий рецептор Т-клеток, включает CD3, и связывающий домен для CD3 может быть связан со связывающим доменом для опухоль-ассоциированного антигена (TAA), через линкер, с образованием пары CD3-TAA. В одном варианте осуществления домен Fc IgG может связывать пару CD3-TAA и связывающий домен для рецептора иммунных контрольных точек. В одном варианте осуществления рецептор иммунных контрольных точек может представлять PD-L1.
Линкер может представлять собой ковалентную связь или пептидный линкер. В одном варианте осуществления пептидный линкер может содержать примерно от 2 до примерно 100 аминокислотных остатков.
В одном варианте осуществления направляющий и навигационный контрольный белок (GNC) имеет N-конец и C-конец, и включает последовательно расположенные от N-конца к C-концу связывающий домен для CD3, связывающий домен для EGFRVIII, домен Fc IgG, связывающий домен для PD-L1 и связывающий домен для 4-1BB. В одном варианте осуществления направляющий и навигационный контрольный белок (GNC) имеет N-конец и C-конец, и включает последовательно расположенные от N-конца к C-концу связывающий домен для 4-1BB, связывающий домен для PD-L1, домен Fc IgG, связывающий домен для ROR1 и связывающий домен для CD3. В одном варианте осуществления направляющий и навигационный контрольный белок (GNC) имеет N-конец и C-конец, и включает последовательно расположенные от N-конца к C-концу, связывающий домен для CD3, связывающий домен для CD19, домен IgG Fc, связывающий домен для PD-L1 и связывающий домен для 4-1BB.
В одном варианте осуществления белок GNC содержит аминокислотную последовательность, имеющую процентную гомологию с SEQ ID NO: 50, 52, 80, 82, 84, 86, 88, 90, 92, 94, 96, 98, 100, 102, 104, 106, 108 и 110. Процентная гомология составляет не менее 70%, 80%, 90%, 95%, 98% или 99%.
В еще одном аспекте заявка обеспечивает последовательности нуклеиновых кислот, кодирующие белок GNC или его фрагменты, раскрытые здесь. В одном варианте осуществления нуклеиновая кислота имеет процентную гомологию с SEQ ID NO: 49, 51, 79, 81, 83, 85, 87, 89, 91, 93, 95, 97, 99, 101, 103, 105, 107 и 109. Процентная гомология составляет не менее 70%, 80%, 90%, 95%, 98% или 99%.
В еще одном аспекте заявка обеспечивает способы получения терапевтической композиции. В одном варианте осуществления способ может включать стадии обеспечения клеточного материала, содержащего цитотоксическую клетку, инкубации клеточного материала с первым белком GNC с получением активированной клеточной композиции и составления активированной клеточной композиции с получением терапевтической композиции. Активированная клеточная композиция содержит первую терапевтическую клетку. Первая терапевтическая клетка содержит первый белок GNC, связанный с цитотоксической клеткой посредством связывающего взаимодействия с рецептором первой цитотоксической клетки. Терапевтическая композиция по существу не содержит экзогенной вирусной и невирусной ДНК или РНК.
В одном варианте осуществления клеточный материал может содержать или может быть получен из РВМС.
Первый белок GNC может включать первый цитотоксический связывающий фрагмент и первый нацеливающий на опухоль фрагмент. Первый цитотоксический связывающий фрагмент обладает специфичностью к рецептору первой цитотоксической клетки и конфигурирован для активации первой цитотоксической клетки посредством связывания с рецептором первой цитотоксической клетки. Первый нацеливающий на опухоль фрагмент обладает специфичностью к рецептору первой опухолевой клетки.
В одном варианте осуществления в способе могут повторяться стадии инкубации посредством инкубирования второго белка GNC с активированной клеточной композицией. Второй белок GNC содержит второй цитотоксический связывающий фрагмент и второй нацеливающий на опухоль фрагмент, где второй цитотоксический связывающий фрагмент обладает специфичностью к рецептору второй цитотоксической клетки, и второй нацеливающий на опухоль фрагмент обладает специфичностью к рецептору второй опухолевой клетки. Активированная клеточная композиция содержит вторую терапевтическую клетку, и вторая терапевтическая клетка содержит второй белок GNC, связанный с цитотоксической клеткой или первой терапевтической клеткой посредством связывающего взаимодействия с рецептором второй цитотоксической клетки.
В одном варианте осуществления первый и второй нацеливающий на опухоль фрагмент независимо имеют специфичность для CD19, PDL1 или их комбинации. В одном варианте осуществления первый и второй цитотоксический связывающий фрагмент независимо имеет специфичность для CD3, PDL1, 41BB или их комбинации.
Способ может дополнительно включать повторные стадии инкубации посредством инкубирования дополнительных белков GNC с активированной композицией. Дополнительные белки GNC могут представлять третий белок GNC, четвертый белок GNC и т. д. для обеспечения дополнительных терапевтических клеток, каждая из которых имеет дополнительный белок, связанный с цитотоксической клеткой.
Первый, второй и дополнительный белок GNC могут быть одинаковыми или могут быть различными. Терапевтические клетки могут иметь один белок GNC, несколько одинаковых белков GNC или несколько различных белков GNC, связанных с ними. В одном варианте осуществления терапевтическая клетка может иметь первый белок GNC, связанный с ней. В одном варианте осуществления терапевтическая клетка может иметь как первый, так и второй белки GNC, связанные с ней. В одном варианте осуществления терапевтическая клетка может иметь первый, второй и дополнительный белок GNC, связанные с ней.
В одном варианте осуществления терапевтическая клетка содержит цитотоксическую клетку, имеющую, по меньшей мере, один связанный белок GNC. В одном варианте осуществления терапевтическая клетка содержит цитотоксическую клетку, имеющую, по меньшей мере, 10, 20, 50, 100, 200, 300, 400 связанных белков GNC.
Терапевтическая композиция может включать первую терапевтическую клетку, первый белок GNC, цитотоксическую клетку или их комбинацию. В одном варианте осуществления терапевтическая композиция может включать вторую терапевтическую клетку, второй белок GNC, содержит первую терапевтическую клетку, первый белок GNC, цитотоксическую клетку или их комбинацию. В одном варианте осуществления терапевтическая композиция может включать дополнительные белки GNC и дополнительные терапевтические клетки.
В одном варианте осуществления стадия инкубации может служить для экспансии терапевтических клеток. В одном варианте осуществления экспансия терапевтической клетки может включать инкубацию терапевтических клеток с дополнительным количеством белка GNC для обеспечения экспансированной клеточной популяции. В одном варианте осуществления экспансированная клеточная популяция включает, по меньшей мере, 102, по меньшей мере, 103, по меньшей мере, 104, по меньшей мере, 105, по меньшей мере, 106, по меньшей мере, 107, по меньшей мере, 108, по меньшей мере, 109, по меньшей мере, 1010 клеток на мл. В одном варианте осуществления экспансированная клеточная популяция включает связанную с GNC клетку, белок GNC, цитотоксическую клетку или их комбинацию. В одном варианте осуществления для истощения PD-1+ Т-клеток в культуру для экспансии может быть добавлен белок GNC, который перенаправляет индукцию гибели на PD-1+ Т-клетки, что приводит к снижению числа истощенных PD-1+ Т-клеток. В одном варианте осуществления, чтобы преимущественно поддерживать PD-1+ T-клетки, белок GNC может быть добавлен к культуре экспансии, которая снимает сигнальный путь контрольных точек через PD-1 на T-клетках, что приводит к функциональному улучшению PD-1+ T-клеток. В одном варианте осуществления, чтобы изолировать опосредованную 4-1BB костимуляцию посредством CAR-T 3-го поколения, белок GNC может быть добавлен в экспансионную культуру, которая перенаправляет уничтожение 4-1BB+ T-клеток или приводит к получению терапевтической композиции с контролируемым уровнем стимуляция 4-1BB в терапевтических клетках, таких как CAR-T клетки.
В одном варианте осуществления нацеливающий на опухоль фрагмент обладает специфичностью по отношению к B-клетке, и терапевтическая композиция по существу не содержит B-клеток. Следовательно, способы, раскрытые здесь, объединяют функции активации и очистки для терапевтических клеток, что позволяет с помощью раскрытых способов получать терапевтическую композицию, не содержащую В-клеток, без необходимости введения каких-либо посторонних материалов (таких как шарики) или каких-либо посторонних генетических материалов (таких как вирусные и невирусные ДНК- или РНК-векторы).
В одном варианте осуществления соотношение белка GNC и цитотоксической клетки составляет, по меньшей мере, 30 к 1 при инкубации клеточного материала с белком GNC.
В одном варианте осуществления терапевтическая композиция может включать, по меньшей мере, 107 клеток на мл.
В дополнительном аспекте заявка обеспечивает способы применения направляющих и навигационных контрольных белков (GNC) для лечения рака. В одном варианте осуществления способ лечения субъекта, страдающего раком, включает обеспечение цитотоксической клетки, объединение белка GNC с цитотоксической клеткой с получением терапевтической клетки, необязательно экспансию терапевтической клетки с обеспечением экспансированной клеточной популяции и введение терапевтической клетки или экспансированной клеточной популяции субъекту.
В одном варианте осуществления способ включает стадию обеспечения клеточного материала, содержащего цитотоксическую клетку, инкубации клеточного материала с первым белком GNC с получением активированной клеточной композиции, где активированная клеточная композиция содержит первую терапевтическую клетку, составления активированной клеточной композиции с обеспечением терапевтической композиции, где терапевтическая композиция по существу не содержит экзогенной вирусной и невирусной ДНК или РНК, и введение терапевтической композиции субъекту.
В одном варианте осуществления способ может дополнительно включать стадии инкубации второго белка GNC с активированной клеточной композицией с получением активированной клеточной композиции, дополнительно содержащей вторую терапевтическую клетку. В одном варианте осуществления способ может дополнительно включать стадию инкубации дополнительных белков GNC с активированной клеточной композицией с получением активированной клеточной композиции, дополнительно содержащей дополнительные терапевтические клетки.
В одном варианте осуществления способ может дополнительно включать выделение цитотоксической клетки из мононуклеарных клеток периферической крови (РВМС) перед обеспечением цитотоксической клетки. В одном варианте осуществления способ может дополнительно включать выделение мононуклеарных клеток периферической крови (РВМС) из крови. В одном варианте осуществления кровь взята у субъекта. В одном варианте кровь взята не у субъекта. В одном варианте осуществления цитотоксические клетки могут происходить от пациента, который подвергается лечению, или от другого субъекта, такого как универсальный донор.
В одном варианте осуществления цитотоксическая клетка может быть аутологичной Т-клеткой, аллореактивной Т-клеткой или универсальной донорской Т-клеткой. В одном варианте осуществления, когда используются аутологичные донорские Т-клетки, для предотвращения инфузии загрязняющих опухолевых клеток белок GNC может быть добавлен в экспансионную культуру, которая перенаправляет уничтожение на опухолевые антигены, например, опухолевый антиген может включать CD19 для В-клеточных новообразований, Epcam для рака молочной железы, MCP1 для меланомы.
В одном варианте осуществления способ включает стадии обеспечения крови от субъекта, выделения мононуклеарных клеток периферической крови (PBMC) из крови, выделения цитотоксической клетки из PBMC, объединения белка GNC с цитотоксической клеткой с получением терапевтической клетки, необязательно экспансию терапевтической клетки с обеспечением экспансированной клеточной популяции и введение терапевтической клетки или экспансированной клеточной популяции субъекту.
В одном варианте осуществления способ дополнительно включает введение дополнительного белка GNC субъекту после введения терапевтической композиции субъекту. В одном варианте осуществления цитотоксическая клетка может включать CD3+ T-клетку, NK-клетку или их комбинацию.
В одном варианте осуществления выделение цитотоксической клетки включает выделение, по меньшей мере, одной субпопуляции цитотоксических клеток для обеспечения терапевтических Т-клеток. В одном варианте осуществления субпопуляция цитотоксических клеток включает CD4+ клетки, CD8+ клетки, CD56+ клетки, CD69+ клетки, CD107a+ клетки, CD45RA+ клетки, CD45RO+ клетки, CD2+ клетки, CD178+ клетки, гранзим+ клетки или их комбинацию.
В одном варианте осуществления объединение белка GNC с цитотоксической клеткой включает инкубацию белка GNC с цитотоксической клеткой в течение периода времени примерно от 2 ч до примерно 14 суток, примерно от 1 суток до примерно 7 суток, примерно от 8 ч до примерно 24 ч, примерно от 4 суток до примерно 7 суток или примерно от 10 суток до примерно 14 суток. В одном варианте осуществления период инкубирования может составлять более 14 суток. В одном варианте осуществления инкубационный период может составлять менее 2 ч.
В одном варианте осуществления соотношение между белком GNC и цитотоксической клеткой составляет, по меньшей мере, 600 к 1, 500 к 1, 400 к 1, 300 к 1, 200 к 1, 100 к 1 или 1 к 1. В одном варианте осуществления соотношение между белком GNC и цитотоксической клеткой составляет примерно 1 к 1, 10 к 1, 100 к 1 или примерно 1000 к 1.
В одном варианте осуществления способ может дополнительно включать оценку терапевтической эффективности после стадии введения. В одном варианте осуществления оценка терапевтической эффективности включает анализ одного или более опухолевых биомаркеров, мониторинг продолжительности жизни терапевтических клеток или их комбинации. В одном варианте осуществления оценка терапевтической эффективности включает анализ одного или более опухолевых биомаркеров, мониторинг продолжительности жизни терапевтических клеток или их комбинации. В одном варианте осуществления биомаркер включает опухолевый антиген, высвобождение цитокинов, например гамма-интерферона, IL-2, IL-8 и/или хемокинов, и/или маркеры CD на поверхности клеток различных типов, например CD69, PD-1, TIGIT и/или мутированную нуклеиновую кислоту, высвобождаемую в кровоток опухолями после гибели, циркулирующие опухолевые клетки и связанную с ними нуклеиновую кислоту, или нуклеиновую кислоту, ассоциированну с экзосомами, медиаторы воспаления хозяина или аналиты, полученные из опухоли, или их комбинации. В одном варианте осуществления биомаркер включает опухолевый антиген, опухоль-ассоциированные апоптотические тельца, низкомолекулярные метаболиты, высвобождение цитокинов, экспрессию поверхностного маркера лимфоцитов, фосфорилированные/дефосфорилированные сигнальные молекулы, факторы транскрипции или их комбинацию
Способ, описанный здесь, не включает стадии трансфекции цитотоксической клетки ДНК-вектором или вирусным вектором. В одном варианте осуществления терапевтическая клетка или экспансированная клеточная популяция по существу не содержат ДНК-вектора или вирусного вектора.
Способ можно использовать для лечения человека, страдающего раком. В одном варианте осуществления злокачественная опухоль включает клетки, экспрессирующие ROR1, CEA, HER2, EGFR, EGFRvIII, LMP1, LMP2A, мезотелин, PSMA, EpCAM, глипикан-3, gpA33, GD2, TROP2, BCMA, CD20, CD33, CD123, CD22, CD30 CD19, опухоль-ассоциированные антигены, которые еще предстоит идентифицировать или их комбинации. В одном варианте осуществления способ можно использовать для лечения млекопитающих.
Можно лечить различные типы рака с использованием способов, раскрытых здесь. Примерные злокачественные заболевания включают без ограничения рак молочной железы, колоректальный рак, рак анального канала, рак поджелудочной железы, рак желчного пузыря, рак желчных протоков, рак головы и шеи, рак носоглотки, рак кожи, меланому, рак яичника, рак предстательной железы, рак уретры, рак легкого, немелкоклеточный рак легкого, мелкоклеточный рак легкого, опухоль головного мозга, глиому, нейробластому, рак пищевода, рак желудка, рак печени, рак почки, рак мочевого пузыря, рак шейки матки, рак эндометрия, рак щитовидной железы, рак глаза, саркому, рак кости, лейкоз, миелому или лимфому.
В одном варианте осуществления способ может дополнительно включать введение эффективного количества терапевтического агента после введения терапевтической клетки или экспансированной клеточной популяции субъекту. В одном варианте осуществления терапевтический агент включает моноклональное антитело, химиотерапевтический агент, фермент, белок, костимулятор или их комбинацию. В одном варианте осуществления костимулятор конфигурирован для увеличения количества цитотоксических Т-клеток у субъекта.
Заявка также обеспечивает раствор, содержащий эффективную концентрацию белка GNC. В одном варианте осуществления раствор представляет собой плазму крови субъекта, который подвергается лечению. В одном варианте осуществления раствор включает клетки, связанные с белком GNC. В одном варианте осуществления раствор включает кластер GNC, включающий белок GNC, T-клетку, связанную с Т-клеточным связывающим фрагментом белка GNC, и опухолевую клетку связанную с нацеливающим на опухоль фрагментом белка GNC.
Цели и преимущества раскрытия могут стать понятными из следующего подробного описания предпочтительных вариантов его осуществления в сочетании с прилагаемыми фигурами.
Краткое описание фигур
Вышеизложенные и другие признаки настоящего раскрытия станут более понятными из следующего описания и прилагаемой формулы изобретения, в сочетании с прилагаемыми фигурами. Очевидно, понятно, что эти фигуры иллюстрируют только несколько вариантов осуществления, расположенных в соответствии с раскрытием, и, следовательно, они не должны рассматриваться как ограничивающие его объем, раскрытие может быть описано с дополнительной специфичностью и подробностями посредством использования прилагаемых фигур, на которых:
На фиг. 1 показан белок GNC, содержащий четыре антигенспецифических связывающих домена в структуре антитела с нацеливающей специфичностью к CD19-позитивным клеткам.
На фиг. 2 показано, что тетраспецифическое антитело GNC опосредует мультиспецифическое связывание между Т-клеткой и опухолевой клеткой.
На фиг. 3 представлена блок-схема, сравнивающая способ получения для GNC-T-клеточной терапии (слева) и CAR-T-клеточной терапии (справа).
На фиг. 4 представлена схема, показывающая источники клеточного материала для приготовления GNC-активированной терапевтической клеточной композиции.
На фиг. 5 представлена схема, показывающая источники селектированных Т-клеток для приготовления GNC-активированной терапевтической композиции.
На фиг. 6 представлена схема, показывающая приготовление GNC-активированной терапевтической Т-клеточной композиции.
На фиг. 7 представлена схема, показывающая стадии инкубации и составления для приготовления первый GNC-активированных T-клеток для GNC-T-клеточной терапии.
На фиг. 8 показано, что белки GNC (группа SI-35E) индуцируют секрецию IL-2 из PBMC.
На фиг. 9 показано, что белки GNC (группа SI-35E) индуцируют секрецию гранзима B из PBMC.
На фиг. 10 показано, что белки GNC (группа SI-35E) индуцируют экспрессию маркера активации CD69 на CD4+ Т-клетках.
На фиг. 11 показано, что белки GNC (группа SI-35E) индуцируют экспрессию маркера активации CD69 на CD8+ Т-клетках.
На фиг. 12 показано, что белки GNC (группа SI-35E) индуцируют экспрессию маркера активации CD69 на CD56+ NK-клетках.
На фиг. 13 показано, что белки GNC (группа SI-35E) индуцируют экспрессию маркера цитотоксической дегрануляции CD107a на CD4+ Т-клетках.
На фиг. 14 показано, что белки GNC (группа SI-35E) индуцируют экспрессию маркера цитотоксической дегрануляции CD107a на CD8+ Т-клетках.
На фиг. 15 показано, что белки GNC (группа SI-35E) индуцируют экспрессию маркера цитотоксической дегрануляции CD107a на CD56+ NK-клетках.
На фиг. 16 показано, что белки GNC (группа SI-35E) активируют CD3+ T-клетки к пролиферации.
На фиг. 17 показано, что белки GNC (группа SI-35E) активируют CD3+ T-клетки к секреции гамма-интерферона.
На фиг. 18 показано, что белки GNC (группа SI-35E) активируют наивные CD8+/CD45RA+ T-клетки к пролиферации.
На фиг. 19 показано, что белки GNC (группа SI-35E) активируют наивные CD8+/CD45RA+ T-клетки к секреции гамма-интерферона.
На фиг. 20 приведены изображения роста клеток, активированных GNC, в 6-луночных планшетах G-Rex во времени.
На фиг.21 показан примерный способ приготовления терапевтической композиции, как здесь раскрыто (A), и жизнеспособность клеток PBMC, GET и GNC-T-клеток после оттаивания (B).
На фиг. 22 приведены результаты анализа проточной цитометрией полученной из РВМС первой терапевтической клеточной композиции, активированной GNC (SI-38E17) (продукт A) (22A), второй терапевтической клеточной композиции, покрытой GNC (SI-38E17) (продукт B) (22B) и входного клеточного материала PBMC (22C).
На фиг. 23 показана GNC-T-клеточная терапевтическая композиция из клеток GET и формулированных GNC-T-клеток из биореактора G-Rex 100M после оттаивания.
На фиг.24 приведены результаты RTCC клеток CHO-ROR1 с использованием клеток PBMC, покрытых GNC (группа SI-35E).
На фиг. 25 показана кинетика полученных из PBMC, SI-38E17 GNC-активированных терапевтических клеток в индукции гибели клеток линии лейкоза из предшественников В-клеток Kasumi во времени.
На фиг.26 показана эффективность индукции гибели клеток Nalm-6, MEC-1, Daudi и Jurkat с использованием полученных из PMBC, SI-38E17 GNC-активированных терапевтических клеток.
На фиг. 27 показана гибель лейкозных клеток Nalm-6, MEC-1, Daudi и Jurkat с использованием полученных из PBMC, SI-38E17 GNC-активированных терапевтических клеток на спайк-модели.
Подробное описание изобретения
В последующем подробном описании делается ссылка на прилагаемые фигуры, которые составляют его часть. На фигурах аналогичные символы обычно идентифицируют аналогичные компоненты, если контекст не требует иного. Иллюстративные варианты осуществления, описанные в подробном описании, на фигурах и в формуле изобретения, не предназначены для ограничения. Могут быть использованы другие варианты осуществления и могут быть внесены другие изменения без отклонения от сущности или объема предмета изобретения, представленного в данном документе. Можно легко понять, что аспекты настоящего раскрытия, как, в общем, здесь описано и иллюстрировано на фигурах, могут быть расположены, заменены, объединены, разделены и выполнены в широком разнообразии различных конфигураций, которые все здесь явно предусмотрены.
В одном варианте осуществления направляющие и навигационные контрольные белки (GNC) отличаются своим составом многочисленных антигенспецифических связывающих доменов (AgBD) и способностью направлять Т-клетки (или другие эффекторные клетки) к опухолевым клеткам (или другим клеткам-мишеням, таким как в качестве клеток-супрессоров) посредством связывания с многочисленными поверхностными молекулами на Т-клетке и опухолевой клетке. В одном варианте осуществления белки GNC состоят из фрагмента 1 для связывания, по меньшей мере, с одной поверхностной молекулой на Т-клетке и фрагмента 2 для связывания, по меньшей мере, с одним поверхностным антигеном на опухолевой клетке, как показано в таблице 1. На фиг. 1 показана структура примерного тетраспецифического антитела GNC, содержащего AgBD для связывания как с T-клеткой, экспрессирующей CD3, PD-L1 и/или 4-1BB, так и с B-клеткой-мишенью, экспрессирующей CD19, как показано на фиг. 2.
В Т-клеточной терапии цитотоксические Т-клетки регулируются белками рецепторных комплексов Т-клеток, а также сигнальными белками костимуляции через агонистические рецепторы или антагонистические рецепторы на их поверхности. Для регуляции данного сигнального пути, а также взаимодействия между Т-клеткой и опухолевой клеткой, многочисленные AgBD могут составлять фрагмент 1 и фрагмент 2, соответственно. Примеры молекул, на которые могут быть нацелены агонистические или антагонистически связывающие домены во фрагменте 1 и 2, приведены в таблице 1. В одном варианте осуществления белки GNC могут содержать, по меньшей мере, один линкер, связывающий фрагмент 1 и фрагмент 2. В одном примере белка GNC любую линкерную молекулу можно использовать для связывания двух или более AgBD вместе in vitro или in vivo с использованием комплементарных линкеров на основе взаимодействия ДНК/РНК или белок-белок, включая, не ограничиваясь этим, биотин-авидин, лейциновую застежку-молнию, и любой двухгибридный позитивный белок. В некоторых вариантах осуществления линкеры могут представлять структуру скелета антитела или фрагментов антитела, так что белок GNC и антитело GNC могут иметь одинаковое значение, например, структура примерного тетраспецифического антитела GNC на фиг. 1.
Белки или антитела GNC способны направлять Т-клетку к опухолевой клетке in vivo или ex vivo посредством функции связывания многочисленных AgBD (фиг.2). Т-клетки можно получить от одного и того же пациента или разных субъектов, и опухолевая клетка может существовать in vivo, in vitro или ex vivo. Примеры, представленные в настоящей заявке, позволяют использовать белки GNC в качестве праймирующего агента в Т-клеточной терапии, т. е. GNC-T-клеточной терапии, для активации и контролирования цитотоксических T-клеток ex vivo до адоптивного переноса.
Настоящая заявка относится к способам получения GNC-активированной терапевтической клеточной композиции. Многочисленные AgBD можно разделить на фрагмент 1 и фрагмент 2 на основе их взаимодействия с Т-клеткой и опухолевой клеткой, соответственно (таблица 1). Белок GNC с двумя AgBD может одновременно связываться с поверхностной молекулой, такой как CD3 на Т-клетке, и опухолевым антигеном, таким как ROR1 на опухолевой клетке, для перенаправления Т-клетки к опухолевой клетке.
Добавление третьего AgBD, например, такого, который специфически связывается с 41BB, может способствовать усилению анти-CD3-индуцированной активации T-клеток, поскольку 41BB является фактором костимуляции, и связывание стимулирует его агонистическую активность в отношении активированных T-клеток. Добавление четвертого AgBD к белку GNC, например, такого, который специфически связывается с PD-L1 на опухолевой клетке, может блокировать ингибиторный путь PD-L1 на опухолевых клетках или который опосредуется через его связывание с PD-1 на Т-клетках.
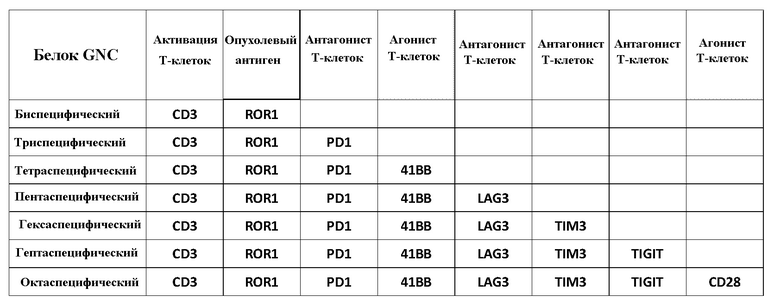
В некоторых вариантах осуществления с использованием этих основных принципов GNC-белки конструируются таким, чтобы содержать многочисленные AgBD, в частности, для связывания неодинакового количества антагонистов и агонистов Т-клеток, не только для перенаправления активированных Т-клеток к опухолевым клеткам, но также для контролирования их активности in vivo (таблица 2). Следовательно, в некоторых вариантах осуществления белки GNC могут быть биспецифическими, триспецифическими, тетраспецифическими, пентаспецифическими, гексаспецифическими, гептаспецифическими или октаспецифическими белками.
В одном варианте осуществления заявка относится к GNC-T-клеточной терапии, где белки GNC используются для экспансии T-клеток ex vivo до адоптивного переноса (фиг. 3). Праймирование ex vivo автономных Т-клеток обеспечивает направляющий и навигационный контроль цитотоксических Т-клеток. Например, мононуклеарные клетки периферической крови (РВМС) или специфические типы клеточных популяций в РВМС, например CD8+, CD45RO+ Т-клетки памяти, можно выделить и праймировать ex vivo белками GNC. Эти экспансированные цитотоксические Т-клетки можно формулировать и ввести обратно пациенту посредством адоптивного переноса. При лечении рака in vivo пациенту можно вводить дополнительные белки GNC для контролирования эффективности и продолжительности наличия цитотоксичности. Таким образом, GNC-T-клеточная терапия отличается от иммунотерапии на основе белков GNC, где белки GNC вводят непосредственно пациентам. Однако GNC-T-клеточная терапия не исключает прямого введения белков GNC для контролирования эффективности инфузированных цитотоксических T-клеток in vivo контролируемым образом. Дополнительный белок GNC может обеспечить цитолитическую активность и стимулировать пролиферацию Т-клеток в зависимости от конфигурации AgBD.
В одном аспекте заявка относится к получению терапевтических GNC-T-клеток. Для сравнения получения терапевтических CAR-T-клеток и их отличий общие процессы показаны на фиг.3 для целей сравнения. При CAR-T-терапии клеточный материал, например лейкоциты пациента, собирают аферезом, и субпопуляцию CD3+ T-клеток селектируют и активируют для облегчения переноса гена в клеточный материал, который затем экспансируют в количестве введением чужеродного каркасного материала для поддержания популяций Т-клеток, например, с использованием гранул, покрытых анти-CD3/анти-CD28 антителом. Преимущественно GNC-T-клеточный материал не требует введения каркасных примесей для экспансии Т-клеток из лейкоцитов пациента.
Клеточный материал для CAR-T терапии должен подвергаться переносу гена, который включает приготовление и трансфекцию ДНК-вектора CAR-T, что приводит к генетической модификации генома Т-клеток. Кроме того, такие генетически модифицированные Т-клетки могут подвергаться еще одному циклу экспансии Т-клеток перед их переносом обратно в организм пациента. Случайная интеграция ДНК-вектора CAR-T несет риск трансформации Т-клеток, что может привести к развитию первичного лейкоза или введению вектора CAR-T в лейкозные клетки, увеличивая риск рецидива по механизму внутренней секвестрации антигена-мишени CAR (Zhang, Lu et al., 2017).
В отличие от этого, GNC-T-клеточная терапия имеет преимущества, заключающиеся в том, что она не включает трансфекцию любой векторной ДНК, и, следовательно, отсутствует риск генетической модификации до адоптивного переноса, что обеспечивает одно из существенных преимуществ и технических усовершенствований по сравнению с существующей CAR-T терапией. Кроме того, преимуществом GNC-T-клеточной терапии является то, что она не включает экзогенного загрязнения родового материала и риска развития рака, эффективность GNC-T-клеточной терапии может быть улучшена, когда PBMC или другие субпопуляции Т-клеток праймируются и активируются ex vivo, как показано на фиг. 5 и 6. Аналогичные подходы были исследованы при использовании CAR-T терапии, где выбранные конкретные соотношения некоторых субпопуляций Т-клеток могут быть перенесены в организм пациента обратно (Turtle, Hanafi et al. 2016, Turtle, Hanafi et al. 2016).
В некоторых вариантах осуществления может быть полезным удалить лейкозные или другие опухолевые клетки из клеточного материала до экспансии клеток (фиг. 7). РВМС пациента с циркулирующими лейкозными клетками, в частности, из злокачественной В-клеточной опухоли, могут существенно изменить клеточную композицию и, таким образом, повлиять на пригодность конечных терапевтических клеточных продуктов. Например, при высоком уровне циркулирующих лейкозных бластных клеток (более 10% от WBC) может потребоваться истощение лейкозных клеток перед GNC-опосредованной экспансией клеток. Процент лейкозных клеток в РВМС, полученных от пациента, можно снизить с использованием методов фракционирования клеток. Эти способы могут включать стадии, включающие разделение клеток в градиенте плотности, или разделение иммунофлуоресцентных клеток, или сортинг флуоресцентно активированных клеток, иммуномагнитную сепарацию клеток или проточные микрофлюидные камеры. Этим методам могут предшествовать или следовать за ними центрифугирование, промывание клеток, инкубация или температурная модуляция. В таких методах можно использовать неклеточные субстраты (магнитные шарики, пластик, полимеры), модификацию неклеточных субстратов (белок, антитела, заряд), обработку антителом, обработку многочисленными антителами, мультиспецифичные антигенсвязывающие белки и антигены клеточной поверхности на основе клеточной связи. В этих методах можно использовать ферментативное расщепление или хелатирование ионов, или механическое перемешивание или вращение клеточного сосуда. В способе снижения количества лейкозных бластов можно использовать конъюгаты антитело-лекарственное средство или агенты, сенсибилизирующие лейкозные клетки. Метод может состоять из комбинации этих подходов.
В одном варианте осуществления для обеспечения продукции терапевтических Т-клеток, праймированных (или покрытых или связанных) белками GNC, получают тетраспецифическое антитело и используют в качестве белка GNC. В одном варианте осуществления тетраспецифическое антитело/белок GNC содержит 4 разных связывающих домена, связанных фрагментами антитела, в качестве его остова. Один связывающий домен специфичен для CD3 на Т-клетках, второй связывающий домен специфичен для опухоль-ассоциированного антигена, включая, не ограничиваясь этим, ROR1, CEA, HER2, EGFR, EGFRvIII, LMP1, LMP2A, мезотелин, PSMA, EpCAM, глипикан-3, gpA33, GD2, TROP2, BCMA, CD19, CD20, CD33, CD123, CD22, CD30, и третий и четвертый связывающие домены являются специфичными для двух различных модуляторов иммунных контрольных точек, таких как PD-L1, PD-L2, PD-1, OX40, 4-1BB, GITR, TIGIT, TIM-3, LAG-3, CTLA4, CD40L, VISTA, ICOS, BTLA, Light и другие.
Не желая связываться с какой-либо теорией, преимущества белок GNC-опосредованной терапии GNC-T-клетками по сравнению с традиционными CAR-T терапиями включают, не ограничиваясь этим, во-первых, включение домена Fc IgG может придавать свойство более длительного периода полураспада в сыворотке по сравнению с биспецифической молекулой BiTe; во-вторых, включение двух связывающих доменов, специфичных для модуляторов иммунных контрольных точек, может ингибировать супрессивные пути и одновременно активировать костимуляторные пути; в-третьих, перекрестное связывание CD3 на Т-клетках с опухоль-ассоциированными антигенами, перенаправляет и направляет Т-клетки на индукцию гибели опухолевых клеток без необходимости удаления Т-клеток из организма пациента и их генетической модификации, чтобы они были специфичны для опухолевых клеток, прежде чем будут повторно введены обратно пациенту, также известное как терапия химерными антигенными рецепторами (CAR-T); и в-четвертых, терапия антителами, опосредованными белком GNC, или Т-клеточная терапия не включают генетическую модификацию Т-клеток, последнее может нести риск трансформации модифицированных Т-клеток в клональную экспансию, т. е. перерождения в лейкозные Т-клетки.
Настоящее раскрытие может быть более легко понятным посредством ссылки на следующее подробное описание конкретных вариантов осуществления и примеров, включенных в настоящий документ. Несмотря на то, что настоящее раскрытие было описано со ссылкой на специфические подробности некоторых его вариантов осуществления, не предполагается, что такие подробности следует рассматривать в качестве ограничений объема раскрытия.
Примеры
Несмотря на то, что следующие примеры приведены только для иллюстрации, а не для ограничения, специалисты в данной области техники легко распознают множество некритических параметров, которые могут быть изменены или модифицированы для получения по существу одинаковых или сходных результатов.
Пример 1: GNC белки и тетраспецифические антитела GNC
В настоящей заявке примерами белков GNC являются группы тетраспецифических антител GNC, из которых 4 AgBD ковалентно связаны с использованием антитела IgG в качестве его остова (фиг. 1). С N-конца этого белка первый scFv связан с доменом Fab константных доменов CH1, 2 и 3 IgG антитела, который затем связан с другим scFv на С-конце. Поскольку каждый из доменов scFv имеет независимую специфичность связывания, то связывание этих AgBD не должно выполняться с использованием константных доменов антитела IgG. Имеющий структуру в виде тетраспецифического антитела GNC, белок GNC может напрямую связываться с опухоль-ассоциированным антигеном (TAA) и активировать эндогенные Т-клетки хозяина, для индукции гибели опухолевых клеток независимо от презентации опухолевого антигена МНС антигенспецифическим рецепторам Т-клеток (фиг. 2). Как показано на фиг.1, CD19 представляет TAA, нацеленный на CD19-позитивные В-клетки и опухолевые клетки. Кроме того, PD-L1 является примером компонента, модулирующего иммунные контрольные точки, для тетраспецифических антител GNC, которые могут преодолевать иммуносупрессивную микросреду опухоли и полностью активировать истощенные Т-клетки в микросреде опухоли.
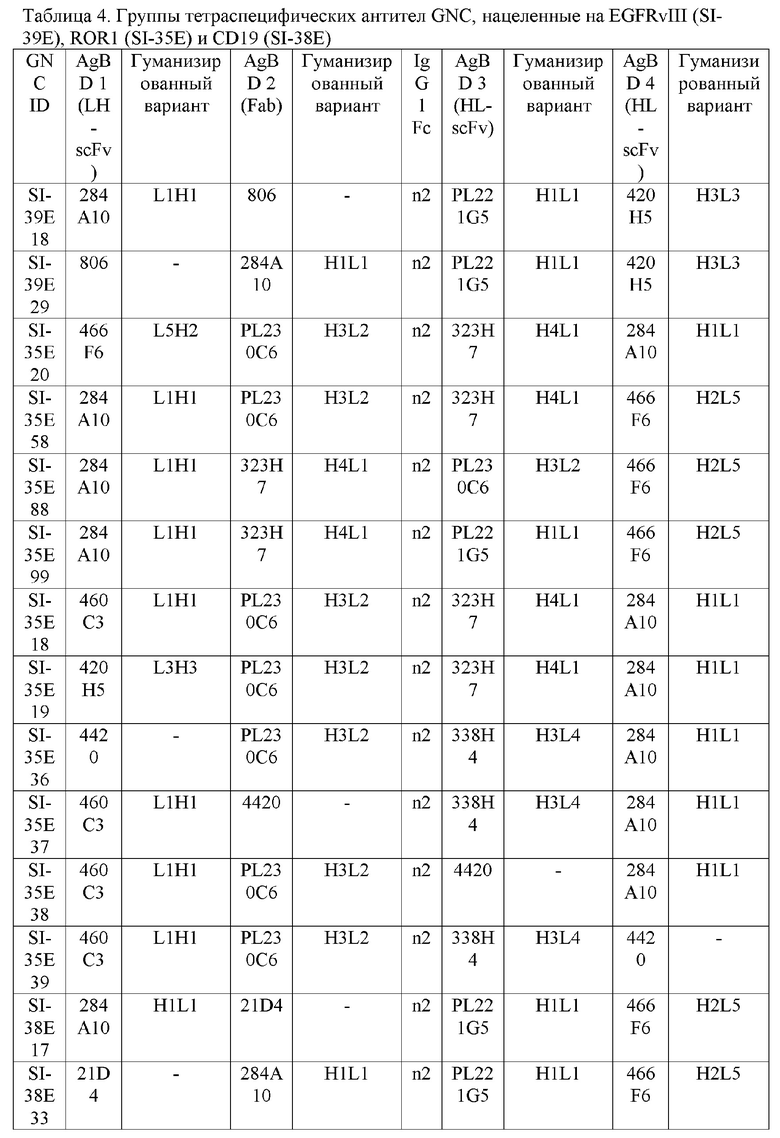
Из тетраспецифических антител GNC группа SI-35E включает мишени античеловеческого CD3-связывающего домена (SEQ ID NO: 1-4), античеловеческого PD-L1 (SEQ ID NO: 5-12), античеловеческого 4-1BB (SEQ ID NO: 13-24) и мишени ROR1 человека (SEQ ID NO: 25-32), т. е. TAA. В данном контексте группы SI-38E и SI-39E нацелены на CD19 (SEQ ID NO: 47-50) и EGFR (SEQ ID NO: 51-54) соответственно.
Для конструирования тетраспецифических антител GNC AgBD были преобразованы в scFv и VLVH для размещения в N-концевом домене 1 (D1) или scFv и VHVL для размещения в C-концевых доменах 3 (D3) и 4 (D4) белка GNC. Все молекулы scFv, описанные здесь, содержат линкер gly-gly-gly-gly-ser (G4S)×4 из 20 аминокислот, который функционально связывает VH и VL, независимо от ориентации V-области (LH или HL). Оставшееся положение в тетраспецифическом антителе GNC, домен 2 (D2), состоит из тяжелой цепи IgG1, VH-CH1-шарнирная область-CH2-CH3, и соответствующей ему легкой цепи, VL-CL, которая может представлять каппа или лямбда цепь. D1 и D2 генетически связаны посредством (G4S) × 2 линкера из 10 аминокислот, так же как и D2, D3 и D4, что приводит к образованию непрерывного мономерного пептида тяжелой цепью ~150 кДа. При котрансфекции с соответствующей легкой цепью конечный симметричный тетраспецифический пептид GNC можно очистить с помощью Fc IgG1 (белок A/белок G) и анализировать для оценки функциональной активности. «Кассеты» генов тяжелой и легкой цепей были сконструированы ранее так, чтобы V-области можно было клонировать с использованием сайтов рестрикции (HindIII/NheI для тяжелой цепи и HindIII/BsiWI для легкой цепи) или «клонирования без рестрикции», такого как Gibson Assembly (SGI-DNA, La Jolla, CA), Infusion (Takara Bio USA) или NEBuilder (NEB, Ipswich, MA), последний из которых использовался здесь.
Тетраспецифические белки получают с помощью способа, который включает конструирование интактной молекулы, синтез и клонирование нуклеотидных последовательностей для каждого домена, экспрессию в клетках млекопитающих и очистку конечного продукта. В данном случае нуклеотидные последовательности собирали с использованием пакета программного обеспечения Geneious 10.2.3 (Biomatters, Auckland, NZ) и разбивали на составляющие их домены-компоненты для синтеза генов (Genewiz, South Plainsfield, NJ). В данном примере SI-35E18 (SEQ ID NO: 65 и 67) расщепляли на составляющие его домены, где анти-4-1BB scFv, VLVH, занимает D1, клон античеловеческий PD-L1 PL230C6 занимает D2 (положение Fab), клон античеловеческий ROR1, Ig домен-специфический 323H7, VHVL scFv занимает положение D3, и античеловеческий CD3 scFv, VHVL, занимает С-концевой D4. Используя веб-инструменты NEBuilder, 5' и 3' нуклеотиды были добавлены к каждому из доменов в зависимости от их положения в более крупном белке, так что каждый домен перекрывает свои фланкирующие домены на 20-30 нуклеотидов, которые направляют сайт-специфическую рекомбинацию, тем самым генетически сливая каждый домен на одной стадии сборки гена. За счет большого количества гомологичных областей в тетраспецифической нуклеотидной последовательности N-концевые домены 1 и 2 собираются отдельно от С-концевых D3 и D4. Затем N- и С-концевые фрагменты собирали вместе во второй реакции NEBuilder. Небольшую аликвоту трансформировали в E.coli DH10b (Invitrogen, Carlsbad, CA) и высевали в планшеты со средой TB+ карбенициллин из расчета 100 мкг/мл (Teknova, Hollister, CA) и инкубировали при 37°C в течение ночи. Полученные колонии селектировали и инокулировали 2 мл ночных культур в TB+ карбенициллин среду. ДНК получали (Thermo-Fisher, Carlsbad, CA) из ночных культур и затем секвенировали (Genewiz, South Plainsfield, NJ), используя праймеры для секвенирования (Sigma, St. Louis, MO), фланкирующие каждый домен. В некоторых вариантах осуществления последовательности ДНК собирали и анализировали в Geneious.
В другом тетраспецифическом белке GNC, SI-38E17, нацеленном на CD19 человека (SEQ ID NO: 47-50), многочисленные AgBD несут античеловеческий 4-1BB- (scFv 466F6, SEQ ID NO: 17-20), а также античеловеческий PD-L1- (scFv PL221G5 SEQ ID NO: 9-13) и античеловеческий CD3-связывающий домен (SEQ ID NO: 1-4). Способы и процедуры получения этого тетраспецифического антитела были аналогичными, как описано выше.
Белки GNC состоят из фрагмента 1 для связывания, по меньшей мере, с одной поверхностной молекулой на Т-клетке и фрагмента 2 для связывания, по меньшей мере, с одним поверхностным антигеном на опухолевой клетке (таблица 1А). Тетраспецифические антитела GNC можно использовать для непосредственной активации эндогенных Т-клеток организма, для индукции гибели опухолевых клеток независимо от презентации опухолевого антигена МНС антигенспецифическим рецепторам Т-клеток. Это отличается от способов лечения, основанных исключительно на блокировании иммунных контрольных точек, которые ограничиваются распознаванием антигена. В данном контексте, компонент, модулирующий иммунные контрольные точки, может быть сконструирован в виде участка тетраспецифических антител GNC, что может обеспечить преимущества, аналогичные тем, которые применяются в стандартной терапии на основе блокирования иммунных контрольных точек.
В дополнение к Т-клеткам другие цитотоксические клетки могут быть нацелены белками GNC для индукции гибели клеток опухолей или для профилактических целей. В таблице 1В приведены примерные композиции функциональных фрагментов (фрагмент 1 и фрагмент 2) и антигенсвязывающий домен в белках GNC со связывающими доменами для NK-клеток. В таблице 1С приведены примерные композиции функциональных фрагментов (фрагмент 1 и фрагмент 2) и антигенсвязывающий домен в белках GNC со связывающими доменами для макрофагов. В таблице 1D приведены примерные композиции функциональных фрагментов (фрагмент 1 и фрагмент 2) и антигенсвязывающий домен в белках GNC со связывающими доменами для дендритных клеток.
Белки GNC конструируют таким образом, что они содержат многочисленные AgBD, в частности, для связывания неодинакового количества антагонистов и агонистов Т-клеток. Таким образом, белки GNC могут перенаправлять активированные Т-клетки к опухолевым клеткам с определенными уровнями контроля их активности in vivo (таблица 2). Следовательно, белки GNC могут быть биспецифическими, триспецифическими, тетраспецифическими, пентаспецифическими, гексаспецифическими, гептаспецифическими или даже октаспецифическими белками. В настоящем изобретении три группы тетраспецифических антител GNC, а именно SI-39E, SI-35E и SI-38E, были созданы для обеспечения GNC-T-клеточной терапии, домены этих антител и их специфичность приведены в таблице 3 Структуры тетраспецифических антител GNC, нацеленные на EGFRvIII (SI-39E), ROR1 (SI-35E) и CD19 (SI-38E), приведены в таблице 4.
Пример 2: GNC-активированная, полученная из PBMS клеточная композиция
Группу SI-35, приведенную в таблице 4, тестировали на ее способность активировать и индуцировать пролиферацию различных типов клеток, таких как CD4+ и/или CD8+ T-клетки и/или CD56+ натуральные клетки-киллеры (NK) в PBMC. Тетраспецифические антитела GNC готовили в конечной концентрации 2× и титровали в последовательных разведениях 1:10 в 6 лунках 96-луночного планшета в 200 мкл RPMI+10% FBS. РВМС человека выделяли в обычном градиенте плотности фиколла из «лейкопака», который представляет собой обогащенный продукт лейкафереза, собранный из нормальной периферической крови человека. В конечном 96-луночном планшете PBMC и серийные разведения белков GNC объединяли добавлением 100 мкл PBMC (100000) и 100 мкл каждого разведения антитела в каждой лунке планшета для анализа. Планшет для анализа инкубировали при 37°С в течение примерно 72 ч и затем содержимое каждой лунки собирали и анализировали с помощью FACS на количество CD4+ T-клеток, CD8+ T-клеток и CD56+ NK-клеток. Клетки собирали из каждой лунки и переносили в другой 96-луночный планшет с V-образным дном, затем центрифугировали при 400 g в течение 3 мин. Супернатант переносили в 96-луночный планшет для анализа IL-2 и гранзима B. Клетки ресуспендировали в 200 мкл 2% FBS/PBS антител FACS и инкубировали на льду в течение 30 мин. Планшет центрифугировали при 400 × g в течение 3 мин и супернатант аспирировали. Эту стадию промывания повторяли еще раз, и затем клетки ресуспендировали в 100 мкл 2% FBS/PBS и анализировали на BD LSR FORTESSA.
Как показано на фиг. 8, все SI-35E тетраспецифические антитела GNC, за исключением тех, которые имели связывающий домен scFv, замененный на FITC в положениях 2 (SI-35E37) и 4 (SI-35E39), индуцировали продукцию IL-2 из РВМС. В этих двух белка отсутствовали домены связывания с PD-L1 или CD3 соответственно. Секреция гранзима B в культуральный супернатант происходила аналогично тому, что имело место для продуцирования IL-2, как показано на фиг.9, и SI-35E37, и SI-35E3 также были существенно менее эффективными в индукции экспрессии на клеточной поверхности маркера активации CD69 на CD4+ клетках (фиг. 10), CD8+ клетках (фиг. 11) и CD56+ клетках (фиг. 12) в культуре РВМС. Поверхностная экспрессия маркера цитотоксической дегрануляции CD107a (LAMP-1) индуцировалась всеми тестированными белками GNC, за исключением тех, у которых отсутствует связывание в положениях 2 и 4 на CD4+ (фиг. 13), CD8+ (фиг. 14), но менее последовательно на CD56+ (фиг. 15) в культуре. D более низких концентрациях 3 из белков GNC (SI-35E42, SI-35E43 и SI-35E46) индуцировали экспрессию CD69 на CD4+ T-клетках, CD8+ T-клетках и CD56+ NK-клетках, что хорошо коррелировало с уровнем IL-2 и секрецией гранзима B (фиг. 8 и 9), индуцированной этими GNC.
Пролиферацию и продукцию гамма-интерферона измеряли на культурах CD3+ клеток или наивных CD8+ Т-клеток (70000 клеток/лунку), стимулированных в течение 5 суток панелью антител группы SI-35. Человеческие CD3+ или CD8+ CD45RA+ наивные Т-клетки обогащали из мононуклеарных клеток периферической крови нормального донора с использованием наборов для выделения CD3+ или наивных CD8+ Т-клеток человека EasySep™ (StemCell Technologies) в соответствии с протоколами изготовителя. Конечная популяция клеток была определена как содержащая > 98% CD3+ или CD8+ CD45RA+ T-клеток методом проточной цитометрии. Пролиферацию в культуре измеряли после окрашивания красителем аламарским синим (ThermoFisher Cat. № DAL1100) в течение 1 ч при 37°С, и затем анализировали на ридере для планшетов Spectramax plus 384 well (Molecular Devices). Пролиферация GNC-экспансированных CD3+ T-клеток выражалась в кратном увеличении числа клеток по сравнению с фоновым уровнем CD3+ T-клеток в клеточной культуре без GNC (фиг. 16). Пролиферация индуцировалась всеми тестированными конструкциями, за исключением той, в которой отсутствует CD3-связывающий домен. Культуральные супернатанты также собирали из этих культур и анализировали на наличие гамма-интерферона с помощью ELISA. Секреция гамма-интерферона (фиг. 17) была высокой, если только CD3- или ROR1-связывающие домены не были заменены на FITC в конструкциях GNC. Пролиферация наивных CD8+ CD45RA+ T-клеток (фиг.18) была более чувствительной к присутствию или отсутствию домена связывания с 4-1BB по сравнению с общим количеством CD3+ T-клеток, что показано добавлением растворимого моноклонального антитела против 4-1BB к культуре, в которой связывание 4-1BB с GNC отсутствовало. Аналогичную картину обнаруживали для секреции гамма-интерферона из наивных CD8+ Т-клеток (фиг. 19).
Пример 3: масштабирование и составление первой GNC-активированной терапевтической клеточной композиции
Получение GNC-активированных и покрытых T-клеток в клинически значимой дозе 10E9 достигалось через 7 суток культивирования. РВМС человека выделяли из конуса лейкоцитов LRS в обычном градиенте плотности фиколла из «лейкопака», который представляет собой обогащенный продукт лейкафереза, собранный из нормальной периферической крови человека. После сбора клетки замораживали при -80°С, и затем оттаивали перед помещением в культуру. С использованием культуральных систем в планшете G-Rex и биореакторах рост культур PBMC, стимулированных SI-38E17 GNC, контролировали в течение 14 суток. Культуральная среда состояла из RPMI 1640, 10% эмбриональной телячьей сыворотки, 1% незаменимых аминокислот, 1% GlutaMax, 0,6% добавки глутамин-аланин, 15 нг/мл человеческого IL-2 и 1 нМ белка GNC. В 6-луночных культурах G-Rex плотность посева составляла 25-100 млн. РВМС/лунку в течение шести суток, что значительно превышало рекомендованные количества, но, в общем, переносилось клетками в системе с одной 50% заменой среды на сутки 7. Кластеризация клеток указывала на их активацию в культуре (фиг. 20). По меньшей мере, 250 млн. клеток от одного донора, полученных лейкаферезом, высевали в два биореактора G-Rex 100M и культивировали в 1 л культуральной среды в течение семи суток. Больший объем среды позволял продолжать культивирование без необходимости в обмене культуральной среды. Выход клеток в каждом из 100М биореакторов составлял 1,2-1,4 миллиарда клеток с жизнеспособностью на уровне более 88%.
Пример 4: вторая GNC-активированная терапевтическая клеточная композиция
Клетки из биореактора собирали в качестве первой GNC-активированной терапевтической клеточной композиции, которую необязательно концентрировали с использованием LOVO Automated Cell Processing System (Fresenius Kabi). Один образец (продукт B) был подвергнут воздействию 1 нМ SI-38E17, который идентичен первому GNC в этом случае для приготовления второй терапевтической клеточной композиции, активированной GNC, что потенциально может быть использовано для лечения пациентов, имеющих CD19-позитивные злокачественные новообразования (фиг. 21А).
После второй стадии концентрирования (объем 100 мл) во время обработки в системе LOVO вторые GNC-активированные терапевтические клетки дважды промывали перед элюцией до конечного объема 54 мл в стерильном пакете для обработки. Другой образец (продукт А) подвергали воздействию только первого белка GNC во время фазы культивирования и не подвергали повторному воздействию во время обработки в системе LOVO (фиг. 21А). Клетки извлекали из пакетов, смешивали 1:1 с реагентом CryoStor CS10 и замораживали до -80°C. Обработанные клетки оттаивали и сравнивали с оттаявшими нестимулированными РВМС от того же донора перед культивированием.
Жизнеспособность клеток в культуре GNC-экспансированных Т-клеток (GET) составляла > 75%, и на нее не оказывало влияния воздействие дополнительного реагента GNC (GNC-T, продукт B) во время обработки (фиг. 21B). Средний диаметр клеток увеличивался во время культивирования, что свидетельствует об активации клеток. Проточную цитометрию выполняли на входном клеточном материале РВМС и двух составах после оттаивания с использованием многоцветной панели антител для окрашивания: живые/мертвые (e780), CD45, TCRα/β, CD56, CD4, CD8, CD14, TCRα/β и CD20. Гейтирование для количественного определения различных субклеточных популяций показано на GNC-активированных T-клетках (продукт A) и дополнительных GNC-покрытых T-клетках (продукт B) (фиг. 22A и 22B). Процентное содержание каждой субпопуляции клеток было одинаковым для продукта A и продукта B, но существенно отличалось от входных PBMC (фиг. 22C). На фиг. 23 представлено суммарное общее количество и процент каждой субпопуляции клеток. По сравнению с входным клеточным материалом РВМС, хотя общее количество лейкоцитов увеличилось с 250 до 1000 млн. или в четыре раза, общее количество каждой субпопуляции Т-клеток достоверно повысилось в 55 раз для α/β Т-клеток, в 45 раз для CD4+ Т-клеток и в 78 раз для CD8+ Т-клеток. В этом контексте увеличение количества γ/δ T-клеток было умеренным в 5 раз, и TCR2α/β, γ/δ+, CD8+ T-клетки казались наиболее многочисленными. Наконец, характерной особенностью клеточных композиций продукта A и продукта B является тот факт, что не было обнаружено B-клеток.
В данном примере показан ряд преимуществ GNC-T-клеток по сравнению с препаратами CAR-T-клеток. Во-первых, клеточный состав исходного материала представлял собой свежие PBMC от донора, и его не нужно было предварительно селектировать в отношении конкретных клеточных субпопуляций или требовать добавления питающих клеток или синтетических шариков. Белок GNC был на 100% ненуклеотидным биологическим материалом и не требовал переноса РНК или ДНК в клетки или трансфекции вирусным вектором. GNC-индуцированная экспансия обеспечивала терапевтическую дозу за 9 суток по сравнению со средним временем 40 суток для экспансии CAR-T-клеток. В полученных клетках отсутствовали B-клетки, и они были высоко обогащены активированными CD4+ и CD8+ T-клетками, которые обладали высоким потенциалом индукции гибели их специфических мишеней. Терапевтическая композиция GNC была жизнеспособной и биологически активной при оттаивании от -80°C. В совокупности эти преимущества, как ожидается, значительно сократят время ожидания, затраты и проблемы, связанные с инфраструктурой и обучением, характерными для CAR-T-клеточной терапии. Повышение чистоты, безопасности и количества конечного продукта будут значительными преимуществами для пациента.
Пример 5: PBMC, предварительно активированные белками GNC, перенаправляются для эффективной индукции гибели опухолевых клеток
Шесть белков GNC группы SI-35, приведенных в таблице 4, тестировали на способность активировать PBMC для перенаправленной цитотоксичности T-клеток (RTCC) против клеток линии CHO, трансфектированной ROR1 человека (фиг. 24). Белки GNC готовили в конечной концентрации 2× и титровали 1:3 в 10 лунках 96-луночного планшета в 200 мкл RPMI+10% FBS. В конечном 96-луночном планшете PBMC и серийные разведения антител объединяли добавлением 100 мкл PBMC (200000) и 100 мкл каждого разведения антитела в каждой лунке планшета для анализа. Планшет для анализа инкубировали при 37°С в течение примерно 72 ч перед добавлением CFSE-меченных клеток CHO-ROR1. Клетки-мишени CHO-ROR1, 5×10e6, метили CFSE (Invitrogen, # C34554) при 0,5 мкМ в 10 мл культуральной среды в течение 20 мин при 37°С. Клетки CHO-ROR1 промывали 3 раза по 50 мл культуральной среды, после чего ресуспендировали в 10 мл, снова подсчитывали и затем в каждую лунку с GNC-активированными PBMC добавляли 5000 CFSE-меченных клеток CHO-ROR1. Клетки инкубировали в течение еще 72 ч, и затем содержимое каждой лунки собирали и анализировали на количество оставшихся CFSE-меченных клеток-мишеней. Как показано на фиг. 24, все тестированные белки GNC направляли активность RTCC, где SI-35E42, SI-35E43 и SI-35E46 были наиболее эффективными в снижении количества клеток CHO-ROR1 в лунке.
Для дальнейшей демонстрации губительных эффектов GNC-меченных PBMC против опухолевых клеток человека, проводили эксперимент с повышением дозы GNC и соотношения эффектором:мишень с использованием системы для визуализации живых клеток IncuCyte S3 (Sartorius) (Sartorius/Essen Biosciences) для мониторинга клеток во времени. PBMC от здорового донора метили белком GNC SI-38E17 в 10-кратных серийных дозах в диапазоне от 0,01 до 100 нМ в течение 30 мин при 37°C и затем промывали перед культивированием. GNC SI-38E17 нацелен на антиген CD19, экспрессируемый на поверхностях B-клеток, и поэтому в качестве клетки-мишени были выбраны клетки лейкозной линии из B-клеток-предшественников Kasumi-2. Используемые клетки Kasumi-2 трансфектировали для экспрессии зеленого флуоресцентного белка (GFP), и поэтому присутствие опухолевых клеток отслеживали измерением средней зеленой флуоресценции на 4 изображениях/лунку, собранных 9 раз в течение шестидневного периода. Соотношения эффектор:мишень (E:T) увеличивали добавлением GNC-меченных PBMC в 2-кратных серийных разведениях от 5000 (1:1) до 160000 (32:1) клеток для дублирования лунок. Как показано на фиг. 25, количество клеток Kasumi-2 увеличилось в лунках с соотношением E:T немеченых РВМС от 1:1 до 8:1. Действие всего лишь 0,1 нМ GNC приводило к снижению роста Kasumi-2 в культуре 1:1 с увеличением супрессии при каждом 2-кратном увеличении отношения E:T. Покрытие РВМС 1 нМ или более высокими концентрациями GNC приводило к почти полной элиминации клеток Kasumi-2 после 42 ч культивирования при всех соотношениях E:T.
В следующем эксперименте в качестве клеток-мишеней использовали три другие трансформированные В-клеточные линии: NALM-6, MEC-1 и Daudi и линию острого Т-клеточного лейкоза Jurkat. Эти клетки-мишени предварительно трансфектировали лентивирусом для конститутивной экспрессии молекулы NucRed 647. В этом анализе PBMC подвергали воздействию 10-кратных доз белка GNC SI-38E17 в течение 30 мин при 37°C и затем промывали, как описано выше. РВМС высевали в количестве 1,2×106 клеток/лунку и добавляли 50000 опухолевых клеток-мишеней. Клетки помещали в систему IncuCyte S3 для сбора красных флуоресцентных изображений (4 изображения на лунку), собранных на 10 временных точек в течение 5,5-дневного периода (фиг. 26). Кривые роста были получены для всех четырех линий опухолевых клеток в отсутствии РВМС (ноль). Мечение PBMC с 1 нМ или более высокой концентрацией белка GNC SI-38E17 приводило к остановке роста всех трех В-клеточных линий, но не лейкозных Т-клеток Jurkat. В-клеточные линии варьировали по своей чувствительности к клеткам РВМС, предварительно подвергнутым воздействию 0,1 нМ белка GNC.
В качестве еще одного метода количественного определения результатов культивирования GNC-T-клеток с опухолевыми клетками установили предел количественного определения (LOQ) для детектирования методом проточной цитометрии. Готовили серию 10-кратных разведений клеток Daudi-Red в диапазоне от 200000 до 20 клеток, и затем смешивали 1:1 с 1 млн. РВМС для получения образцов с 10%, 1,0%, 0,1%, 0,01% и 0,001% опухолевых клеток, которые затем анализировали проточной цитометрией (фиг. 27). Затем клетки собирали из 15-дневной культуры в 6-луночном планшете G-Rex из экспансированных 1 нМ GNC Т-клеток, в которые были добавлены 10%, 1% или 0,1% NALM-6, MEC-1, Daudi или Jurkat (все NucRed-трансдуцированные) опухолевые клетки на временную точку 0 и анализировали с использованием тех же настроек проточной цитометрии, которые описаны выше. Количество опухолевых клеток снижалось до менее чем 0,001% во всех условиях, за исключением культуры, в которой опухолевые клетки линии MEC-1 были внесены на уровне 10%, при этом было обнаружено 44 клетки. В этих условиях количество клеток MEC-1 в культуре снижалось до <0,01%.
Несмотря на то, что настоящее раскрытие было описано со ссылкой на конкретные варианты осуществления или примеры, можно понять, что варианты осуществления являются иллюстративными и что объем раскрытия не ограничен таким образом. Альтернативные варианты осуществления настоящего раскрытия могут стать очевидными для специалистов в данной области техники, к которой относится настоящее раскрытие. Такие альтернативные варианты осуществления включаются в объем настоящего раскрытия. Следовательно, объем настоящего раскрытия определяется прилагаемой формулой изобретения и подтверждается вышеприведенным описанием. Все ссылки, цитированные или упомянутые в этом раскрытии, в полном объеме включены здесь посредством ссылки.
Таблицы
Таблица 1A. Состав примерных белков GNC с доменами связывания Т-клеток
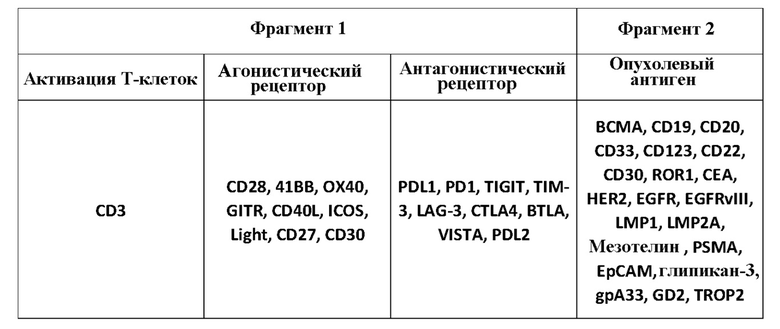
Таблица 2. Примеры возможных комбинаций активации Т-клеток, агониста Т-клеток, антагониста Т-клеток и доменов, связывающих опухолевый антиген, в одном белке GNC
480C8
420H5
466F6
Домен IgD
Домен Kringle
Домен Frizzled
330F11
338H4
324C6
Список последовательностей
GNC-T Список последовательностей тетраспецифических антител GNC
CDR подчеркнуты в аминокислотных последовательностях
>SEQ ID 01 анти-CD3 284A10 VHv1 нуклеотидная последовательность
GAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCA
>SEQ ID 02 анти-CD3 284A10 VHv1 аминокислотная последовательность
EVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSS
>SEQ ID 03 анти-CD3 284A10 VLv1 нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAGTTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 04 анти-CD3 284A10 VLv1 аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIK
>SEQ ID 05 анти-PD-L1 PL230C6 VHv3 нуклеотидная последовательность
CAGTCGGTGGAGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGAATCGACCTTAATACCTACGACATGATCTGGGTCCGCCAGGCTCCAGGCAAGGGGCTAGAGTGGGTTGGAATCATTACTTATAGTGGTAGTAGATACTACGCGAACTGGGCGAAAGGCCGATTCACCATCTCCAAAGACAATACCAAGAACACGGTGTATCTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCCAGAGATTATATGAGTGGTTCCCACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTAGT
>SEQ ID 06 анти-PD-L1 PL230C6 VHv3 аминокислотная последовательность
QSVEESGGGLVQPGGSLRLSCTASGIDLNTYDMIWVRQAPGKGLEWVGIITYSGSRYYANWAKGRFTISKDNTKNTVYLQMNSLRAEDTAVYYCARDYMSGSHLWGQGTLVTVSS
>SEQ ID 07 анти-PD-L1 PL230C6 VLv2 нуклеотидная последовательность
GCCTATGATATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCAAGTGTCAGGCCAGTGAGGACATTTATAGCTTCTTGGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCCATTCTGCATCCTCTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTACTATTGTCAACAGGGTTATGGTAAAAATAATGTTGATAATGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 08 анти-PD-L1 PL230C6 VLv2 аминокислотная последовательность
AYDMTQSPSSVSASVGDRVTIKCQASEDIYSFLAWYQQKPGKAPKLLIHSASSLASGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQGYGKNNVDNAFGGGTKVEIK
>SEQ ID 09 анти-PD-L1 PL221G5 VHv1 нуклеотидная последовательность
GAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCGGGTACGACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTGCTGCTGGTAGTGCTGGTATCACTTACGACGCGAACTGGGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGATCGGCGTTTTCGTTCGACTACGCCATGGACCTCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGC
>SEQ ID 10 анти-PD-L1 PL221G5 VHv1 аминокислотная последовательность
EVQLLESGGGLVQPGGSLRLSCAASGFSFSSGYDMCWVRQAPGKGLEWIACIAAGSAGITYDANWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARSAFSFDYAMDLWGQGTLVTVSS
>SEQ ID 11 анти-PD-L1 PL221G5 VLv1 нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGGCCAGTCAGAGCATTAGTTCCCACTTAAACTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATAAGGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAACAGGGTTATAGTTGGGGTAATGTTGATAATGTTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 12 анти-PD-L1 PL221G5 VLv1 аминокислотная последовательность
DIQMTQSPSTLSASVGDRVTITCQASQSISSHLNWYQQKPGKAPKLLIYKASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQQGYSWGNVDNVFGGGTKVEIK
>SEQ ID 13 анти-4-1BB 420H5 VHv3 нуклеотидная последовательность
CAGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCAACTACTGGATATGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTTATGTTGGTAGTAGTGGTGACACTTACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGAGATAGTAGTAGTTATTATATGTTTAACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGC
>SEQ ID 14 анти-4-1BB 420H5 VHv3 аминокислотная последовательность
QSLVESGGGLVQPGGSLRLSCAASGFSFSSNYWICWVRQAPGKGLEWIACIYVGSSGDTYYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDSSSYYMFNLWGQGTLVTVSS
>SEQ ID 15 анти-4-1BB 420H5 VLv3 нуклеотидная последовательность
GCCCTTGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAGGCCAGTGAGGACATTGATACCTATTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTTTTATGCATCCGATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCGGTTACTATACTAGTAGTGCTGATACGAGGGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 16 анти-4-1BB 420H5 VLv3 аминокислотная последовательность
ALVMTQSPSTLSASVGDRVTINCQASEDIDTYLAWYQQKPGKAPKLLIFYASDLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGGYYTSSADTRGAFGGGTKVEIK
>SEQ ID 17 анти-4-1BB 466F6 VHv2 нуклеотидная последовательность
CGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCGAGCTCCGCGAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGC
>SEQ ID 18 анти-4-1BB 466F6 VHv2 аминокислотная последовательность
RSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSS
>SEQ ID 19 анти-4-1BB 466F6 VLv5 нуклеотидная последовательность
GACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 20 анти-4-1BB 466F6 VLv5 аминокислотная последовательность
DVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIK
>SEQ ID 21 анти-4-1BB 460C3 VHv1 нуклеотидная последовательность
GAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGAATCGACTTCAGTAGGAGATACTACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATATATACTGGTAGCCGCGATACTCCTCACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGAGAAGGTAGCCTGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGC
>SEQ ID 22 анти-4-1BB 460C3 VHv1 аминокислотная последовательность
EVQLLESGGGLVQPGGSLRLSCAASGIDFSRRYYMCWVRQAPGKGLEWIACIYTGSRDTPHYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCAREGSLWGQGTLVTVSS
>SEQ ID 23 анти-4-1BB 460C3 VLv1 нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAGTAACTGGTTCTCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATTCTGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCGCAGGCGGTTACAATACTGTTATTGATACTTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 24 анти-4-1BB 460C3 VLv1 аминокислотная последовательность
DIQMTQSPSTLSASVGDRVTITCQSSQSVYSNWFSWYQQKPGKAPKLLIYSASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCAGGYNTVIDTFAFGGGTKVEIK
>SEQ ID 25 анти-ROR1 323H7 VHv4 нуклеотидная последовательность
GAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTCGCTACCACATGACTTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGACATATTTATGTTAATAATGATGACACAGACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCACCTATTTCTGTGCGAGATTGGATGTTGGTGGTGGTGGTGCTTATATTGGGGACATCTGGGGCCAGGGAACTCTGGTTACCGTCTCTTCA
>SEQ ID 26 анти-ROR1 323H7 VHv4 аминокислотная последовательность
EVQLLESGGGLVQPGGSLRLSCAASGFTISRYHMTWVRQAPGKGLEWIGHIYVNNDDTDYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTATYFCARLDVGGGGAYIGDIWGQGTLVTVSS
>SEQ ID 27 анти-ROR1 323H7 VLv1 нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAACAACAACGACTTAGCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCTCCTGATCTATTATGCTTCCACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTGCAGGCGGTTATGATACGGATGGTCTTGATACGTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 28 анти-ROR1 323H7 VLv1 аминокислотная последовательность
DIQMTQSPSSLSASVGDRVTITCQSSQSVYNNNDLAWYQQKPGKVPKLLIYYASTLASGVPSRFSGSGSGTDFTLTISSLQPEDVATYYCAGGYDTDGLDTFAFGGGTKVEIK
>SEQ ID 29 анти-ROR1 338H4 VHv3 нуклеотидная последовательность
GAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACTGCCTCTGGATTCTCCCTCAGTAGCTATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAGGGGGCTGGAGTGGATCGGAATCATTTATGCTAGTGGTAGCACATACTACGCGAGCTCGGCGAAAGGCAGATTCACCATCTCCAAAGACAATACCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAATTTATGACGGCATGGACCTCTGGGGCCAGGGAACTCTGGTTACCGTCTCTTCA
>SEQ ID 30 анти-ROR1 338H4 VHv3 аминокислотная последовательность
EVQLVESGGGLVQPGGSLRLSCTASGFSLSSYAMSWVRQAPGRGLEWIGIIYASGSTYYASSAKGRFTISKDNTKNTVDLQMNSLRAEDTAVYYCARIYDGMDLWGQGTLVTVSS
>SEQ ID 31 анти-ROR1 338H4 VLv4 нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAGGCCAGTCAGAACATTTACAGCTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCGCCTGATCTATCTGGCATCTACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTACACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTCAAAGCAATTATAACGGTAATTATGGTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 32 анти-ROR1 338H4 VLv4 аминокислотная последовательность
DIQMTQSPSSLSASVGDRVTINCQASQNIYSYLSWYQQKPGKVPKRLIYLASTLASGVPSRFSGSGSGTDYTLTISSLQPEDVATYYCQSNYNGNYGFGGGTKVEIK
>SEQ ID 33 анти-FITC 4420 VH нуклеотидная последовательность
GAGGTGAAGCTGGATGAGACTGGAGGAGGCTTGGTGCAACCTGGGAGGCCCATGAAACTCTCCTGTGTTGCCTCTGGATTCACTTTTAGTGACTACTGGATGAACTGGGTCCGCCAGTCTCCAGAGAAAGGACTGGAGTGGGTAGCACAAATTAGAAACAAACCTTATAATTATGAAACATATTATTCAGATTCTGTGAAAGGCAGATTCACCATCTCAAGAGATGATTCCAAAAGTAGTGTCTACCTGCAAATGAACAACTTAAGAGTTGAAGACATGGGTATCTATTACTGTACGGGTTCTTACTATGGTATGGACTACTGGGGTCAAGGAACCTCAGTCACCGTCTCCTCA
>SEQ ID 34 анти-FITC 4420 VH аминокислотная последовательность
EVKLDETGGGLVQPGRPMKLSCVASGFTFSDYWMNWVRQSPEKGLEWVAQIRNKPYNYETYYSDSVKGRFTISRDDSKSSVYLQMNNLRVEDMGIYYCTGSYYGMDYWGQGTSVTVSS
>SEQ ID 35 анти-FITC 4420 VL нуклеотидная последовательность
GATGTCGTGATGACCCAAACTCCACTCTCCCTGCCTGTCAGTCTTGGAGATCAAGCCTCCATCTCTTGCAGATCTAGTCAGAGCCTTGTACACAGTAATGGAAACACCTATTTACGTTGGTACCTGCAGAAGCCAGGCCAGTCTCCAAAGGTCCTGATCTACAAAGTTTCCAACCGATTTTCTGGGGTCCCAGACAGGTTCAGTGGCAGTGGATCAGGGACAGATTTCACACTCAAGATCAGCAGAGTGGAGGCTGAGGATCTGGGAGTTTATTTCTGCTCTCAAAGTACACATGTTCCGTGGACGTTCGGTGGAGGCACCAAGCTGGAAATCAAA
>SEQ ID 36 анти-FITC 4420 VL аминокислотная последовательность
DVVMTQTPLSLPVSLGDQASISCRSSQSLVHSNGNTYLRWYLQKPGQSPKVLIYKVSNRFSGVPDRFSGSGSGTDFTLKISRVEAEDLGVYFCSQSTHVPWTFGGGTKLEIK
>SEQ ID 37 человеческий IgG1 с нулевой мутацией (G1m-fa с нулевой мутацией ADCC/CDC) нуклеотидная последовательность
GCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGT
>SEQ ID 38 человеческий IgG1 с нулевой мутацией (G1m-fa с нулевой мутацией ADCC/CDC) аминокислотная последовательность
ASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPG
>SEQ ID 39 каппа цепь человеческого Ig нуклеотидная последовательность
CGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 40 каппа цепь человеческого Ig аминокислотная последовательность
RTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 41 SI-35E18 (460C3-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) тяжелая цепь нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAGTAACTGGTTCTCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATTCTGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCGCAGGCGGTTACAATACTGTTATTGATACTTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGAATCGACTTCAGTAGGAGATACTACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATATATACTGGTAGCCGCGATACTCCTCACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGAGAAGGTAGCCTGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCAGTCGGTGGAGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGAATCGACCTTAATACCTACGACATGATCTGGGTCCGCCAGGCTCCAGGCAAGGGGCTAGAGTGGGTTGGAATCATTACTTATAGTGGTAGTAGATACTACGCGAACTGGGCGAAAGGCCGATTCACCATCTCCAAAGACAATACCAAGAACACGGTGTATCTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCCAGAGATTATATGAGTGGTTCCCACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTAGTGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTATACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTCGCTACCACATGACTTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGACATATTTATGTTAATAATGATGACACAGACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCACCTATTTCTGTGCGAGATTGGATGTTGGTGGTGGTGGTGCTTATATTGGGGACATCTGGGGCCAGGGAACTCTGGTTACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAACAACAACGACTTAGCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCTCCTGATCTATTATGCTTCCACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTGCAGGCGGTTATGATACGGATGGTCTTGATACGTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAGTTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 42 SI-35E18 (460C3-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) тяжелая цепь аминокислотная последовательность DIQMTQSPSTLSASVGDRVTITCQSSQSVYSNWFSWYQQKPGKAPKLLIYSASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCAGGYNTVIDTFAFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGIDFSRRYYMCWVRQAPGKGLEWIACIYTGSRDTPHYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCAREGSLWGQGTLVTVSSGGGGSGGGGSQSVEESGGGLVQPGGSLRLSCTASGIDLNTYDMIWVRQAPGKGLEWVGIITYSGSRYYANWAKGRFTISKDNTKNTVYLQMNSLRAEDTAVYYCARDYMSGSHLWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFTISRYHMTWVRQAPGKGLEWIGHIYVNNDDTDYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTATYFCARLDVGGGGAYIGDIWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSSLSASVGDRVTITCQSSQSVYNNNDLAWYQQKPGKVPKLLIYYASTLASGVPSRFSGSGSGTDFTLTISSLQPEDVATYYCAGGYDTDGLDTFAFGGGTKVEIKGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIK
>SEQ ID 43 SI-35E18 (460C3-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) легкая цепь нуклеотидная последовательность
GCCTATGATATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCAAGTGTCAGGCCAGTGAGGACATTTATAGCTTCTTGGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCCATTCTGCATCCTCTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTACTATTGTCAACAGGGTTATGGTAAAAATAATGTTGATAATGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 44 SI-35E18 (460C3-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) легкая цепь аминокислотная последовательность
AYDMTQSPSSVSASVGDRVTIKCQASEDIYSFLAWYQQKPGKAPKLLIHSASSLASGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQGYGKNNVDNAFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 45 анти-CD3 284A10 VHv1b нуклеотидная последовательность
GAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACGGTGGTTCTTCTGCTATTACTAGTAACAACATTTGGGGCCAGGGAACCCTGGTCACCGTGTCGACA
>SEQ ID 46 анти-CD3 284A10 VHv1b аминокислотная последовательность
EVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVST
>SEQ ID 47 анти-huCD19 21D4 VH нуклеотидная последовательность
GAGGTGCAGCTGGTGCAGTCTGGAGCAGAGGTGAAGAAACCAGGAGAGTCTCTGAAGATCTCCTGTAAGGGTTCTGGATACAGCTTTAGCAGTTCATGGATCGGCTGGGTGCGCCAGGCACCTGGGAAAGGCCTGGAATGGATGGGGATCATCTATCCTGATGACTCTGATACCAGATACAGTCCATCCTTCCAAGGCCAGGTCACCATCTCAGCCGACAAGTCCATCAGGACTGCCTACCTGCAGTGGAGTAGCCTGAAGGCCTCGGACACCGCTATGTATTACTGTGCGAGACATGTTACTATGATTTGGGGAGTTATTATTGACTTCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCA
>SEQ ID 48 анти-huCD19 21D4 VH аминокислотная последовательность
EVQLVQSGAEVKKPGESLKISCKGSGYSFSSSWIGWVRQAPGKGLEWMGIIYPDDSDTRYSPSFQGQVTISADKSIRTAYLQWSSLKASDTAMYYCARHVTMIWGVIIDFWGQGTLVTVSS
>SEQ ID 49 анти-huCD19 21D4 VL нуклеотидная последовательность
GCCATCCAGTTGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCGGGCAAGTCAGGGCATTAGCAGTGCTTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCTCCTAAGCTCCTGATCTATGATGCCTCCAGTTTGGAAAGTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTATTACTGTCAACAGTTTAATAGTTACCCATTCACTTTCGGCCCTGGGACCAAAGTGGATATCAAA
>SEQ ID 50 анти-huCD19 21D4 VL аминокислотная последовательность
AIQLTQSPSSLSASVGDRVTITCRASQGISSALAWYQQKPGKAPKLLIYDASSLESGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQFNSYPFTFGPGTKVDIK
>SEQ ID 51 анти-huEGFRvIII 806 VH нуклеотидная последовательность GATGTGCAGCTTCAGGAGTCGGGACCTAGCCTGGTGAAACCTTCTCAGTCTCTGTCCCTCACCTGCACTGTCACTGGCTACTCAATCACCAGTGATTTTGCCTGGAACTGGATTCGGCAGTTTCCAGGAAACAAGCTGGAGTGGATGGGCTACATAAGTTATAGTGGTAACACTAGGTACAACCCATCTCTCAAAAGTCGAATCTCTATCACTCGCGACACATCCAAGAACCAATTCTTCCTGCAGTTGAACTCTGTGACTATTGAGGACACAGCCACATATTACTGTGTAACGGCGGGACGCGGGTTTCCTTATTGGGGCCAAGGGACTCTGGTCACTGTCTCTGCA
>SEQ ID 52 анти-huEGFRvIII 806 VH аминокислотная последовательность
DVQLQESGPSLVKPSQSLSLTCTVTGYSITSDFAWNWIRQFPGNKLEWMGYISYSGNTRYNPSLKSRISITRDTSKNQFFLQLNSVTIEDTATYYCVTAGRGFPYWGQGTLVTVSA
>SEQ ID 53 анти-huEGFRvIII 806 VL нуклеотидная последовательность
GACATCCTGATGACCCAATCTCCATCCTCCATGTCTGTATCTCTGGGAGACACAGTCAGCATCACTTGCCATTCAAGTCAGGACATTAACAGTAATATAGGGTGGTTGCAGCAGAGACCAGGGAAATCATTTAAGGGCCTGATCTATCATGGAACCAACTTGGACGATGAAGTTCCATCAAGGTTCAGTGGCAGTGGATCTGGAGCCGATTATTCTCTCACCATCAGCAGCCTGGAATCTGAAGATTTTGCAGACTATTACTGTGTACAGTATGCTCAGTTTCCGTGGACGTTCGGTGGAGGCACCAAGCTGGAAATCAAA
>SEQ ID 54 анти-huEGFRvIII 806 VL аминокислотная последовательность
DILMTQSPSSMSVSLGDTVSITCHSSQDINSNIGWLQQRPGKSFKGLIYHGTNLDDEVPSRFSGSGSGADYSLTISSLESEDFADYYCVQYAQFPWTFGGGTKLEIK
>SEQ ID 55 линкер GGGGSGGGGSG нуклеотидная последовательность
GGCGGTGGAGGGTCCGGCGGTGGTGGCTCCGGA
>SEQ ID 56 линкер GGGGSGGGGSG аминокислотная последовательность GGGGSGGGGSG
>SEQ ID 57 линкер 01 GGGGSGGGGS нуклеотидная последовательность
GGCGGTGGAGGGTCCGGCGGTGGTGGATCA
>SEQ ID 58 линкер 01 GGGGSGGGGS аминокислотная последовательность
GGGGSGGGGS
>SEQ ID 59 линкер 02 GGGGSGGGGS нуклеотидная последовательность
GGCGGTGGAGGGTCCGGCGGTGGTGGATCC
>SEQ ID 60 линкер 02 GGGGSGGGGS аминокислотная последовательность
GGGGSGGGGS
>SEQ ID 61 линкер GGGGSGGGGSGGGGSGGGGS нуклеотидная последовательность
GGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCA
>SEQ ID 62 линкер GGGGSGGGGSGGGGSGGGGS аминокислотная последовательность
>SEQ ID 63 SI-39E18 (284A10-L1H1-scFv x 806-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) тяжелая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAGTTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGATGTGCAGCTTCAGGAGTCGGGACCTAGCCTGGTGAAACCTTCTCAGTCTCTGTCCCTCACCTGCACTGTCACTGGCTACTCAATCACCAGTGATTTTGCCTGGAACTGGATTCGGCAGTTTCCAGGAAACAAGCTGGAGTGGATGGGCTACATAAGTTATAGTGGTAACACTAGGTACAACCCATCTCTCAAAAGTCGAATCTCTATCACTCGCGACACATCCAAGAACCAATTCTTCCTGCAGTTGAACTCTGTGACTATTGAGGACACAGCCACATATTACTGTGTAACGGCGGGACGCGGGTTTCCTTATTGGGGCCAAGGGACTCTGGTCACTGTCTCTGCAGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCGGGTACGACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTGCTGCTGGTAGTGCTGGTATCACTTACGACGCGAACTGGGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGATCGGCGTTTTCGTTCGACTACGCCATGGACCTCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGGCCAGTCAGAGCATTAGTTCCCACTTAAACTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATAAGGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAACAGGGTTATAGTTGGGGTAATGTTGATAATGTTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCAGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCAACTACTGGATATGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGTATTTATGTTGGTAGTAGTGGTGACACTTACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGAGATAGTAGTAGTTATTATATGTTTAACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGCCCTTGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAGGCCAGTGAGGACATTGATACCTATTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTTTTACGCATCCGATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCGGTTACTATACTAGTAGTGCTGATACGAGGGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 64 SI-39E18 (284A10-L1H1-scFv x 806-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) тяжелая цепь аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSSGGGGSGGGGSDVQLQESGPSLVKPSQSLSLTCTVTGYSITSDFAWNWIRQFPGNKLEWMGYISYSGNTRYNPSLKSRISITRDTSKNQFFLQLNSVTIEDTATYYCVTAGRGFPYWGQGTLVTVSAASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFSFSSGYDMCWVRQAPGKGLEWIACIAAGSAGITYDANWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARSAFSFDYAMDLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSTLSASVGDRVTITCQASQSISSHLNWYQQKPGKAPKLLIYKASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQQGYSWGNVDNVFGGGTKVEIKGGGGSGGGGSQSLVESGGGLVQPGGSLRLSCAASGFSFSSNYWICWVRQAPGKGLEWIACIYVGSSGDTYYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDSSSYYMFNLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSALVMTQSPSTLSASVGDRVTINCQASEDIDTYLAWYQQKPGKAPKLLIFYASDLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGGYYTSSADTRGAFGGGTKVEIK
>SEQ ID 65 SI-39E18 (284A10-L1H1-scFv x 806-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) легкая цепь нуклеотидная последовательность
GACATCCTGATGACCCAATCTCCATCCTCCATGTCTGTATCTCTGGGAGACACAGTCAGCATCACTTGCCATTCAAGTCAGGACATTAACAGTAATATAGGGTGGTTGCAGCAGAGACCAGGGAAATCATTTAAGGGCCTGATCTATCATGGAACCAACTTGGACGATGAAGTTCCATCAAGGTTCAGTGGCAGTGGATCTGGAGCCGATTATTCTCTCACCATCAGCAGCCTGGAATCTGAAGATTTTGCAGACTATTACTGTGTACAGTATGCTCAGTTTCCGTGGACGTTCGGTGGAGGCACCAAGCTGGAAATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 66 SI-39E18 (284A10-L1H1-scFv x 806-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) легкая цепь аминокислотная последовательность
DILMTQSPSSMSVSLGDTVSITCHSSQDINSNIGWLQQRPGKSFKGLIYHGTNLDDEVPSRFSGSGSGADYSLTISSLESEDFADYYCVQYAQFPWTFGGGTKLEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 67 SI-39E29 (806-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) тяжелая цепь нуклеотидная последовательность
GACATCCTGATGACCCAATCTCCATCCTCCATGTCTGTATCTCTGGGAGACACAGTCAGCATCACTTGCCATTCAAGTCAGGACATTAACAGTAATATAGGGTGGTTGCAGCAGAGACCAGGGAAATCATTTAAGGGCCTGATCTATCATGGAACCAACTTGGACGATGAAGTTCCATCAAGGTTCAGTGGCAGTGGATCTGGAGCCGATTATTCTCTCACCATCAGCAGCCTGGAATCTGAAGATTTTGCAGACTATTACTGTGTACAGTATGCTCAGTTTCCGTGGACGTTCGGTGGAGGCACCAAGCTGGAAATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGATGTGCAGCTTCAGGAGTCGGGACCTAGCCTGGTGAAACCTTCTCAGTCTCTGTCCCTCACCTGCACTGTCACTGGCTACTCAATCACCAGTGATTTTGCCTGGAACTGGATTCGGCAGTTTCCAGGAAACAAGCTGGAGTGGATGGGCTACATAAGTTATAGTGGTAACACTAGGTACAACCCATCTCTCAAAAGTCGAATCTCTATCACTCGCGACACATCCAAGAACCAATTCTTCCTGCAGTTGAACTCTGTGACTATTGAGGACACAGCCACATATTACTGTGTAACGGCGGGACGCGGGTTTCCTTATTGGGGCCAAGGGACTCTGGTCACTGTCTCTGCAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCAGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCGGGTACGACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTGCTGCTGGTAGTGCTGGTATCACTTACGACGCGAACTGGGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGATCGGCGTTTTCGTTCGACTACGCCATGGACCTCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGGCCAGTCAGAGCATTAGTTCCCACTTAAACTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATAAGGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAACAGGGTTATAGTTGGGGTAATGTTGATAATGTTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCAGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCAACTACTGGATATGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGTATTTATGTTGGTAGTAGTGGTGACACTTACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGAGATAGTAGTAGTTATTATATGTTTAACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGCCCTTGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAGGCCAGTGAGGACATTGATACCTATTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTTTTACGCATCCGATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCGGTTACTATACTAGTAGTGCTGATACGAGGGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 68 SI-39E29 (806-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) тяжелая цепь аминокислотная последовательность
DILMTQSPSSMSVSLGDTVSITCHSSQDINSNIGWLQQRPGKSFKGLIYHGTNLDDEVPSRFSGSGSGADYSLTISSLESEDFADYYCVQYAQFPWTFGGGTKLEIKGGGGSGGGGSGGGGSGGGGSDVQLQESGPSLVKPSQSLSLTCTVTGYSITSDFAWNWIRQFPGNKLEWMGYISYSGNTRYNPSLKSRISITRDTSKNQFFLQLNSVTIEDTATYYCVTAGRGFPYWGQGTLVTVSAGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFSFSSGYDMCWVRQAPGKGLEWIACIAAGSAGITYDANWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARSAFSFDYAMDLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSTLSASVGDRVTITCQASQSISSHLNWYQQKPGKAPKLLIYKASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQQGYSWGNVDNVFGGGTKVEIKGGGGSGGGGSQSLVESGGGLVQPGGSLRLSCAASGFSFSSNYWICWVRQAPGKGLEWIACIYVGSSGDTYYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDSSSYYMFNLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSALVMTQSPSTLSASVGDRVTINCQASEDIDTYLAWYQQKPGKAPKLLIFYASDLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGGYYTSSADTRGAFGGGTKVEIK
>SEQ ID 69 SI-39E29 (806-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) легкая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 70 SI-39E29 (806-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 420H5-H3L3-scFv) легкая цепь n аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 71 SI-35E20 (466F6-L5H2-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) тяжелая цепь нуклеотидная последовательность
GACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCACGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCGAGCTCCGCGAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCAGTCGGTGGAGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGAATCGACCTTAATACCTACGACATGATCTGGGTCCGCCAGGCTCCAGGCAAGGGGCTAGAGTGGGTTGGAATCATTACTTATAGTGGTAGTAGATACTACGCGAACTGGGCGAAAGGCCGATTCACCATCTCCAAAGACAATACCAAGAACACGGTGTATCTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCCAGAGATTATATGAGTGGTTCCCACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTAGTGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTATACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTCGCTACCACATGACTTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGACATATTTATGTTAATAATGATGACACAGACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCACCTATTTCTGTGCGAGATTGGATGTTGGTGGTGGTGGTGCTTATATTGGGGACATCTGGGGCCAGGGAACTCTGGTTACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAACAACAACGACTTAGCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCTCCTGATCTATTATGCTTCCACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTGCAGGCGGTTATGATACGGATGGTCTTGATACGTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAGTTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 72 SI-35E20 (466F6-L5H2-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) тяжелая цепь аминокислотная последовательность
DVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSRSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSSGGGGSGGGGSQSVEESGGGLVQPGGSLRLSCTASGIDLNTYDMIWVRQAPGKGLEWVGIITYSGSRYYANWAKGRFTISKDNTKNTVYLQMNSLRAEDTAVYYCARDYMSGSHLWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFTISRYHMTWVRQAPGKGLEWIGHIYVNNDDTDYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTATYFCARLDVGGGGAYIGDIWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSSLSASVGDRVTITCQSSQSVYNNNDLAWYQQKPGKVPKLLIYYASTLASGVPSRFSGSGSGTDFTLTISSLQPEDVATYYCAGGYDTDGLDTFAFGGGTKVEIKGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIK
>SEQ ID 73 SI-35E20 (466F6-L5H2-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) легкая цепь нуклеотидная последовательность
GCCTATGATATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCAAGTGTCAGGCCAGTGAGGACATTTATAGCTTCTTGGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCCATTCTGCATCCTCTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTACTATTGTCAACAGGGTTATGGTAAAAATAATGTTGATAATGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 74 SI-35E20 (466F6-L5H2-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 284A10-H1L1-scFv) легкая цепь аминокислотная последовательность
AYDMTQSPSSVSASVGDRVTIKCQASEDIYSFLAWYQQKPGKAPKLLIHSASSLASGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQGYGKNNVDNAFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 75 SI-35E58 (284A10-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 466F6-H2L5-scFv) тяжелая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACGGTGGTTCTTCTGCTATTACTAGTAACAACATTTGGGGCCAGGGAACCCTGGTCACCGTGTCGACAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCAGTCGGTGGAGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACCGCCTCTGGAATCGACCTTAATACCTACGACATGATCTGGGTCCGCCAGGCTCCAGGCAAGGGGCTAGAGTGGGTTGGAATCATTACTTATAGTGGTAGTAGATACTACGCGAACTGGGCGAAAGGCCGATTCACCATCTCCAAAGACAATACCAAGAACACGGTGTATCTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCGAGAGATTATATGAGTGGTTCCCACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCAGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTATACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGGTCCGGAGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTCGCTACCACATGACTTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGACATATTTATGTTAATAATGATGACACAGACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCACCTATTTCTGTGCGAGATTGGATGTTGGTGGTGGTGGTGCTTATATTGGGGACATCTGGGGCCAGGGAACTCTGGTTACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAACAACAACGACTTAGCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCTCCTGATCTATTATGCTTCCACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTGCAGGCGGTTATGATACGGATGGTCTTGATACGTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGGTCCGGACGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACTGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCAAGCTCCGCTAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 76 SI-35E58 (284A10-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 466F6-H2L5-scFv) тяжелая цепь аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSTGGGGSGGGGSQSVEESGGGLVQPGGSLRLSCTASGIDLNTYDMIWVRQAPGKGLEWVGIITYSGSRYYANWAKGRFTISKDNTKNTVYLQMNSLRAEDTAVYYCARDYMSGSHLWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSGEVQLLESGGGLVQPGGSLRLSCAASGFTISRYHMTWVRQAPGKGLEWIGHIYVNNDDTDYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTATYFCARLDVGGGGAYIGDIWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSSLSASVGDRVTITCQSSQSVYNNNDLAWYQQKPGKVPKLLIYYASTLASGVPSRFSGSGSGTDFTLTISSLQPEDVATYYCAGGYDTDGLDTFAFGGGTKVEIKGGGGSGGGGSGRSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIK
>SEQ ID 77 SI-35E58 (284A10-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 466F6-H2L5-scFv) легкая цепь нуклеотидная последовательность
GCCTATGATATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCAAGTGTCAGGCCAGTGAGGACATTTATAGCTTCTTGGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCCATTCTGCATCCTCTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTACTATTGTCAACAGGGTTATGGTAAAAATAATGTTGATAATGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 78 SI-35E58 (284A10-L1H1-scFv x PL230C6-Fab x 323H7-H4L1-scFv x 466F6-H2L5-scFv) легкая цепь аминокислотная последовательность
AYDMTQSPSSVSASVGDRVTIKCQASEDIYSFLAWYQQKPGKAPKLLIHSASSLASGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQGYGKNNVDNAFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 79 SI-35E88 (284A10-L1H1-scFv x 323H7-Fab x PL230C6-H3L2-scFv x 466F6-H2L5-scFv) тяжелая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACGGTGGTTCTTCTGCTATTACTAGTAACAACATTTGGGGCCAGGGAACCCTGGTCACCGTGTCGACAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTCGCTACCACATGACTTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGACATATTTATGTTAATAATGATGACACAGACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCACCTATTTCTGTGCGAGATTGGATGTTGGTGGTGGTGGTGCTTATATTGGGGACATCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTATACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCAGTCGGTGGAGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACCGCCTCTGGAATCGACCTTAATACCTACGACATGATCTGGGTCCGCCAGGCTCCAGGCAAGGGGCTAGAGTGGGTTGGAATCATTACTTATAGTGGTAGTAGATACTACGCGAACTGGGCGAAAGGCCGATTCACCATCTCCAAAGACAATACCAAGAACACGGTGTATCTGCAAATGAACAGCCTGAGAGCTGAGGACACGGCTGTGTATTACTGTGCGAGAGATTATATGAGTGGTTCCCACTTGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCCGGTGGAGGCGGTTCAGGCGGAGGTGGAAGTGGTGGTGGCGGCTCTGGAGGCGGCGGATCTGCCTATGATATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCAAGTGTCAGGCCAGTGAGGACATTTATAGCTTCTTGGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCCATTCTGCATCCTCTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTACTATTGTCAACAGGGTTATGGTAAAAATAATGTTGATAATGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGGTCCGGACGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACTGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCAAGCTCCGCTAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 80 SI-35E88 (284A10-L1H1-scFv x 323H7-Fab x PL230C6-H3L2-scFv x 466F6-H2L5-scFv) тяжелая цепь аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSTGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFTISRYHMTWVRQAPGKGLEWIGHIYVNNDDTDYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTATYFCARLDVGGGGAYIGDIWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSQSVEESGGGLVQPGGSLRLSCTASGIDLNTYDMIWVRQAPGKGLEWVGIITYSGSRYYANWAKGRFTISKDNTKNTVYLQMNSLRAEDTAVYYCARDYMSGSHLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSAYDMTQSPSSVSASVGDRVTIKCQASEDIYSFLAWYQQKPGKAPKLLIHSASSLASGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQGYGKNNVDNAFGGGTKVEIKGGGGSGGGGSGRSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIK
>SEQ ID 81 SI-35E88 (284A10-L1H1-scFv x 323H7-Fab x PL230C6-H3L2-scFv x 466F6-H2L5-scFv) легкая цепь нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAACAACAACGACTTAGCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCTCCTGATCTATTATGCATCCACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTGCAGGCGGTTATGATACGGATGGTCTTGATACGTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 82 SI-35E88 (284A10-L1H1-scFv x 323H7-Fab x PL230C6-H3L2-scFv x 466F6-H2L5-scFv) легкая цепь аминокислотная последовательность
DIQMTQSPSSLSASVGDRVTITCQSSQSVYNNNDLAWYQQKPGKVPKLLIYYASTLASGVPSRFSGSGSGTDFTLTISSLQPEDVATYYCAGGYDTDGLDTFAFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 83 SI-35E99 (284A10-L1H1-scFv x 323H7-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) тяжелая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACGGTGGTTCTTCTGCTATTACTAGTAACAACATTTGGGGCCAGGGAACCCTGGTCACCGTGTCGACAGGCGGTGGAGGGTCCGGCGGTGGTGGATCAGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTCGCTACCACATGACTTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGACATATTTATGTTAATAATGATGACACAGACTACGCGAGCTCCGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCACCTATTTCTGTGCGAGATTGGATGTTGGTGGTGGTGGTGCTTATATTGGGGACATCTGGGGCCAGGGAACTCTGGTTACCGTCTCTTCAGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCGGGTACGACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTGCTGCTGGTAGTGCTGGTATCACTTACGACGCGAACTGGGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGATCGGCGTTTTCGTTCGACTACGCCATGGACCTCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGTGGAGGCGGATCTGGCGGAGGTGGTTCCGGCGGTGGCGGCTCCGGTGGAGGCGGCTCTGACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGGCCAGTCAGAGCATTAGTTCCCACTTAAACTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATAAGGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAACAGGGTTATAGTTGGGGTAATGTTGATAATGTTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGCTCCGGACGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACTGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCAAGCTCCGCTAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCTTCAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 84 SI-35E99 (284A10-L1H1-scFv x 323H7-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) тяжелая цепь аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSTGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFTISRYHMTWVRQAPGKGLEWIGHIYVNNDDTDYASSAKGRFTISRDNSKNTLYLQMNSLRAEDTATYFCARLDVGGGGAYIGDIWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFSFSSGYDMCWVRQAPGKGLEWIACIAAGSAGITYDANWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARSAFSFDYAMDLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSTLSASVGDRVTITCQASQSISSHLNWYQQKPGKAPKLLIYKASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQQGYSWGNVDNVFGGGTKVEIKGGGGSGGGGSGRSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIK
>SEQ ID 85 SI-35E99 (284A10-L1H1-scFv x 323H7-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) легкая цепь нуклеотидная последовательность
GACATCCAGATGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGTCCAGTCAGAGTGTTTATAACAACAACGACTTAGCCTGGTATCAGCAGAAACCAGGGAAAGTTCCTAAGCTCCTGATCTATTATGCATCCACTCTGGCATCTGGGGTCCCATCTCGGTTCAGTGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATGTTGCAACTTATTACTGTGCAGGCGGTTATGATACGGATGGTCTTGATACGTTTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 86 SI-35E99 (284A10-L1H1-scFv x 323H7-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) легкая цепь аминокислотная последовательность
DIQMTQSPSSLSASVGDRVTITCQSSQSVYNNNDLAWYQQKPGKVPKLLIYYASTLASGVPSRFSGSGSGTDFTLTISSLQPEDVATYYCAGGYDTDGLDTFAFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 87 SI-38E17 (284A10-L1H1-scFv x 21D4-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) тяжелая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAGTTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGGTGCAGTCTGGAGCAGAGGTGAAGAAACCAGGAGAGTCTCTGAAGATCTCCTGTAAGGGTTCTGGATACAGCTTTAGCAGTTCATGGATCGGCTGGGTGCGCCAGGCACCTGGGAAAGGCCTGGAATGGATGGGGATCATCTATCCTGATGACTCTGATACCAGATACAGTCCATCCTTCCAAGGCCAGGTCACCATCTCAGCCGACAAGTCCATCAGGACTGCCTACCTGCAGTGGAGTAGCCTGAAGGCCTCGGACACCGCTATGTATTACTGTGCGAGACATGTTACTATGATTTGGGGAGTTATTATTGACTTCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCAGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTATACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCGGGTACGACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTGCTGCTGGTAGTGCTGGTATCACTTACGACGCGAACTGGGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGATCGGCGTTTTCGTTCGACTACGCCATGGACCTCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGTGGAGGCGGATCTGGCGGAGGTGGTTCCGGCGGTGGCGGCTCCGGTGGAGGCGGCTCTGACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGGCCAGTCAGAGCATTAGTTCCCACTTAAACTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATAAGGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAACAGGGTTATAGTTGGGGTAATGTTGATAATGTTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCGAGCTCCGCGAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 88 SI-38E17 (284A10-L1H1-scFv x 21D4-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) тяжелая цепь аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKGGGGSGGGGSGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSSGGGGSGGGGSEVQLVQSGAEVKKPGESLKISCKGSGYSFSSSWIGWVRQAPGKGLEWMGIIYPDDSDTRYSPSFQGQVTISADKSIRTAYLQWSSLKASDTAMYYCARHVTMIWGVIIDFWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFSFSSGYDMCWVRQAPGKGLEWIACIAAGSAGITYDANWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARSAFSFDYAMDLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSTLSASVGDRVTITCQASQSISSHLNWYQQKPGKAPKLLIYKASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQQGYSWGNVDNVFGGGTKVEIKGGGGSGGGGSRSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIK
>SEQ ID 89 SI-38E17 (284A10-L1H1-scFv x 21D4-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) легкая цепь нуклеотидная последовательность
GCCATCCAGTTGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCGGGCAAGTCAGGGCATTAGCAGTGCTTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCTCCTAAGCTCCTGATCTATGATGCCTCCAGTTTGGAAAGTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTATTACTGTCAACAGTTTAATAGTTACCCATTCACTTTCGGCCCTGGGACCAAAGTGGATATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
>SEQ ID 90 SI-38E17 (284A10-L1H1-scFv x 21D4-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) легкая цепь аминокислотная последовательность
AIQLTQSPSSLSASVGDRVTITCRASQGISSALAWYQQKPGKAPKLLIYDASSLESGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQFNSYPFTFGPGTKVDIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
>SEQ ID 91 SI-38E33 (21D4-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) тяжелая цепь нуклеотидная последовательность
GCCATCCAGTTGACCCAGTCTCCATCCTCCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCGGGCAAGTCAGGGCATTAGCAGTGCTTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCTCCTAAGCTCCTGATCTATGATGCCTCCAGTTTGGAAAGTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCAGCCTGCAGCCTGAAGATTTTGCAACTTATTACTGTCAACAGTTTAATAGTTACCCATTCACTTTCGGCCCTGGGACCAAAGTGGATATCAAAGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGAGGTGCAGCTGGTGCAGTCTGGAGCAGAGGTGAAGAAACCAGGAGAGTCTCTGAAGATCTCCTGTAAGGGTTCTGGATACAGCTTTAGCAGTTCATGGATCGGCTGGGTGCGCCAGGCACCTGGGAAAGGCCTGGAATGGATGGGGATCATCTATCCTGATGACTCTGATACCAGATACAGTCCATCCTTCCAAGGCCAGGTCACCATCTCAGCCGACAAGTCCATCAGGACTGCCTACCTGCAGTGGAGTAGCCTGAAGGCCTCGGACACCGCTATGTATTACTGTGCGAGACATGTTACTATGATTTGGGGAGTTATTATTGACTTCTGGGGCCAGGGAACCCTGGTCACCGTCTCCTCAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCACCATCAGTACCAATGCAATGAGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGGAGTCATTACTGGTCGTGATATCACATACTACGCGAGCTGGGCGAAAGGCAGATTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGCGCGACGGTGGATCATCTGCTATTACTAGTAACAACATTTGGGGCCAAGGAACTCTGGTCACCGTTTCTTCAGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGCCCAAATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAAGCCGCGGGGGCACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCGCGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTATACCCTGCCCCCATCCCGGGATGAGCTGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTATAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTGGCGGTGGAGGGTCCGGCGGTGGTGGATCCGAGGTGCAGCTGTTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGGGGGTCCCTGAGACTCTCCTGTGCAGCCTCTGGATTCTCCTTCAGTAGCGGGTACGACATGTGCTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTGGATCGCATGCATTGCTGCTGGTAGTGCTGGTATCACTTACGACGCGAACTGGGCGAAAGGCCGGTTCACCATCTCCAGAGACAATTCCAAGAACACGCTGTATCTGCAAATGAACAGCCTGAGAGCCGAGGACACGGCCGTATATTACTGTGCGAGATCGGCGTTTTCGTTCGACTACGCCATGGACCTCTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGTGGAGGCGGATCTGGCGGAGGTGGTTCCGGCGGTGGCGGCTCCGGTGGAGGCGGCTCTGACATCCAGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCACTTGCCAGGCCAGTCAGAGCATTAGTTCCCACTTAAACTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATAAGGCATCCACTCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTTACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAACAGGGTTATAGTTGGGGTAATGTTGATAATGTTTTCGGCGGAGGGACCAAGGTGGAGATCAAAGGCGGTGGAGGGTCCGGCGGTGGTGGATCCCGGTCGCTGGTGGAGTCTGGGGGAGGCTTGGTCCAGCCTGGGGGGTCCCTGAGACTCTCCTGTACAGCCTCTGGATTCACCATCAGTAGCTACCACATGCAGTGGGTCCGCCAGGCTCCAGGGAAGGGGCTGGAGTACATCGGAACCATTAGTAGTGGTGGTAATGTATACTACGCGAGCTCCGCGAGAGGCAGATTCACCATCTCCAGACCCTCGTCCAAGAACACGGTGGATCTTCAAATGAACAGCCTGAGAGCCGAGGACACGGCTGTGTATTACTGTGCGAGAGACTCTGGTTATAGTGATCCTATGTGGGGCCAGGGAACCCTGGTCACCGTCTCGAGCGGCGGTGGCGGTAGTGGGGGAGGCGGTTCTGGCGGCGGAGGGTCCGGCGGTGGAGGATCAGACGTTGTGATGACCCAGTCTCCATCTTCCGTGTCTGCATCTGTAGGAGACAGAGTCACCATCACCTGTCAGGCCAGTCAGAACATTAGGACTTACTTATCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGCTGCAGCCAATCTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGATTTCACTCTCACCATCAGCGACCTGGAGCCTGGCGATGCTGCAACTTACTATTGTCAGTCTACCTATCTTGGTACTGATTATGTTGGCGGTGCTTTCGGCGGAGGGACCAAGGTGGAGATCAAA
>SEQ ID 92 SI-38E33 (21D4-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) тяжелая цепь аминокислотная последовательность
AIQLTQSPSSLSASVGDRVTITCRASQGISSALAWYQQKPGKAPKLLIYDASSLESGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQFNSYPFTFGPGTKVDIKGGGGSGGGGSGGGGSGGGGSEVQLVQSGAEVKKPGESLKISCKGSGYSFSSSWIGWVRQAPGKGLEWMGIIYPDDSDTRYSPSFQGQVTISADKSIRTAYLQWSSLKASDTAMYYCARHVTMIWGVIIDFWGQGTLVTVSSGGGGSGGGGSEVQLVESGGGLVQPGGSLRLSCAASGFTISTNAMSWVRQAPGKGLEWIGVITGRDITYYASWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARDGGSSAITSNNIWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKRVEPKSCDKTHTCPPCPAPEAAGAPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCAVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGGGGGSGGGGSEVQLLESGGGLVQPGGSLRLSCAASGFSFSSGYDMCWVRQAPGKGLEWIACIAAGSAGITYDANWAKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARSAFSFDYAMDLWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSTLSASVGDRVTITCQASQSISSHLNWYQQKPGKAPKLLIYKASTLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQQGYSWGNVDNVFGGGTKVEIKGGGGSGGGGSRSLVESGGGLVQPGGSLRLSCTASGFTISSYHMQWVRQAPGKGLEYIGTISSGGNVYYASSARGRFTISRPSSKNTVDLQMNSLRAEDTAVYYCARDSGYSDPMWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDVVMTQSPSSVSASVGDRVTITCQASQNIRTYLSWYQQKPGKAPKLLIYAAANLASGVPSRFSGSGSGTDFTLTISDLEPGDAATYYCQSTYLGTDYVGGAFGGGTKVEIK
SEQ ID 93 SI-38E33 (21D4-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) легкая цепь нуклеотидная последовательность
GACGTCGTGATGACCCAGTCTCCTTCCACCCTGTCTGCATCTGTAGGAGACAGAGTCACCATCAATTGCCAAGCCAGTGAGAGCATTAGCAGTTGGTTAGCCTGGTATCAGCAGAAACCAGGGAAAGCCCCTAAGCTCCTGATCTATGAAGCATCCAAACTGGCATCTGGGGTCCCATCAAGGTTCAGCGGCAGTGGATCTGGGACAGAATTCACTCTCACCATCAGCAGCCTGCAGCCTGATGATTTTGCAACTTATTACTGCCAAGGCTATTTTTATTTTATTAGTCGTACTTATGTAAATTCTTTCGGCGGAGGGACCAAGGTGGAGATCAAACGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGT
SEQ ID 94 SI-38E33 (21D4-LH-scFv x 284A10-Fab x PL221G5-H1L1-scFv x 466F6-H2L5-scFv) легкая цепь аминокислотная последовательность
DVVMTQSPSTLSASVGDRVTINCQASESISSWLAWYQQKPGKAPKLLIYEASKLASGVPSRFSGSGSGTEFTLTISSLQPDDFATYYCQGYFYFISRTYVNSFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
--->
Список последовательностей
<110> SICHUAN BAILI PHARMACEUTICAL CO. LTD.
<120> СПОСОБЫ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ НАПРАВЛЯЮЩИХ И НАВИГАЦИОННЫХ
КОНТРОЛЬНЫХ БЕЛКОВ
<130> SIBA104PCT
<150> US62648888
<151> 2018-03-27
<150> US62648880
<151> 2018-03-27
<160> 94
<170> PatentIn version 3.5
<210> 1
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 1
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccatcagt accaatgcaa tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg gatcggagtc attactggtc gtgatatcac atactacgcg 180
agctgggcga aaggcagatt caccatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgcg cgacggtgga 300
tcatctgcta ttactagtaa caacatttgg ggccaaggaa ctctggtcac cgtttcttca 360
<210> 2
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 2
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Thr Asn
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn Ile Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 3
<211> 336
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 3
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagag ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaa 336
<210> 4
<211> 112
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 4
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 5
<211> 345
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 5
cagtcggtgg aggagtctgg gggaggcttg gtccagcctg gggggtccct gagactctcc 60
tgtacagcct ctggaatcga ccttaatacc tacgacatga tctgggtccg ccaggctcca 120
ggcaaggggc tagagtgggt tggaatcatt acttatagtg gtagtagata ctacgcgaac 180
tgggcgaaag gccgattcac catctccaaa gacaatacca agaacacggt gtatctgcaa 240
atgaacagcc tgagagctga ggacacggct gtgtattact gtgccagaga ttatatgagt 300
ggttcccact tgtggggcca gggaaccctg gtcaccgtct ctagt 345
<210> 6
<211> 115
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 6
Gln Ser Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
1 5 10 15
Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Asp Leu Asn Thr Tyr Asp
20 25 30
Met Ile Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
35 40 45
Ile Ile Thr Tyr Ser Gly Ser Arg Tyr Tyr Ala Asn Trp Ala Lys Gly
50 55 60
Arg Phe Thr Ile Ser Lys Asp Asn Thr Lys Asn Thr Val Tyr Leu Gln
65 70 75 80
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
85 90 95
Asp Tyr Met Ser Gly Ser His Leu Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 7
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 7
gcctatgata tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcaagtgtc aggccagtga ggacatttat agcttcttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatccattct gcatcctctc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag ggttatggta aaaataatgt tgataatgct 300
ttcggcggag ggaccaaggt ggagatcaaa 330
<210> 8
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 8
Ala Tyr Asp Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Lys Cys Gln Ala Ser Glu Asp Ile Tyr Ser Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
His Ser Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Gly Lys Asn Asn
85 90 95
Val Asp Asn Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 9
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 9
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt ctccttcagt agcgggtacg acatgtgctg ggtccgccag 120
gctccaggga aggggctgga gtggatcgca tgcattgctg ctggtagtgc tggtatcact 180
tacgacgcga actgggcgaa aggccggttc accatctcca gagacaattc caagaacacg 240
ctgtatctgc aaatgaacag cctgagagcc gaggacacgg ccgtatatta ctgtgcgaga 300
tcggcgtttt cgttcgacta cgccatggac ctctggggcc agggaaccct ggtcaccgtc 360
tcgagc 366
<210> 10
<211> 122
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 10
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser Gly
20 25 30
Tyr Asp Met Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Ala Cys Ile Ala Ala Gly Ser Ala Gly Ile Thr Tyr Asp Ala Asn
50 55 60
Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Ser Ala Phe Ser Phe Asp Tyr Ala Met Asp Leu Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 11
<211> 330
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 11
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggccagtca gagcattagt tcccacttaa actggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcatccactc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa tttactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag ggttatagtt ggggtaatgt tgataatgtt 300
ttcggcggag ggaccaaggt ggagatcaaa 330
<210> 12
<211> 110
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 12
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser His
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Ser Trp Gly Asn
85 90 95
Val Asp Asn Val Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 13
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 13
cagtcgctgg tggagtctgg gggaggcttg gtacagcctg gggggtccct gagactctcc 60
tgtgcagcct ctggattctc cttcagtagc aactactgga tatgctgggt ccgccaggct 120
ccagggaagg ggctggagtg gatcgcatgc atttatgttg gtagtagtgg tgacacttac 180
tacgcgagct ccgcgaaagg ccggttcacc atctccagag acaattccaa gaacacgctg 240
tatctgcaaa tgaacagcct gagagccgag gacacggccg tatattactg tgcgagagat 300
agtagtagtt attatatgtt taacttgtgg ggccagggaa ccctggtcac cgtctcgagc 360
<210> 14
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 14
Gln Ser Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
1 5 10 15
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser Asn Tyr
20 25 30
Trp Ile Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Ala Cys Ile Tyr Val Gly Ser Ser Gly Asp Thr Tyr Tyr Ala Ser Ser
50 55 60
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Ser Tyr Tyr Met Phe Asn Leu Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 15
<211> 336
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 15
gcccttgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aggccagtga ggacattgat acctatttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatcttttat gcatccgatc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc ggttactata ctagtagtgc tgatacgagg 300
ggtgctttcg gcggagggac caaggtggag atcaaa 336
<210> 16
<211> 112
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 16
Ala Leu Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Asp Ile Asp Thr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Phe Tyr Ala Ser Asp Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Gly Tyr Tyr Thr Ser Ser
85 90 95
Ala Asp Thr Arg Gly Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 17
<211> 345
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 17
cggtcgctgg tggagtctgg gggaggcttg gtccagcctg gggggtccct gagactctcc 60
tgtacagcct ctggattcac catcagtagc taccacatgc agtgggtccg ccaggctcca 120
gggaaggggc tggagtacat cggaaccatt agtagtggtg gtaatgtata ctacgcgagc 180
tccgcgagag gcagattcac catctccaga ccctcgtcca agaacacggt ggatcttcaa 240
atgaacagcc tgagagccga ggacacggct gtgtattact gtgcgagaga ctctggttat 300
agtgatccta tgtggggcca gggaaccctg gtcaccgtct cgagc 345
<210> 18
<211> 115
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 18
Arg Ser Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
1 5 10 15
Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Ile Ser Ser Tyr His
20 25 30
Met Gln Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Tyr Ile Gly
35 40 45
Thr Ile Ser Ser Gly Gly Asn Val Tyr Tyr Ala Ser Ser Ala Arg Gly
50 55 60
Arg Phe Thr Ile Ser Arg Pro Ser Ser Lys Asn Thr Val Asp Leu Gln
65 70 75 80
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
85 90 95
Asp Ser Gly Tyr Ser Asp Pro Met Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 19
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 19
gacgttgtga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacctgtc aggccagtca gaacattagg acttacttat cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcagccaatc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcga cctggagcct 240
ggcgatgctg caacttacta ttgtcagtct acctatcttg gtactgatta tgttggcggt 300
gctttcggcg gagggaccaa ggtggagatc aaa 333
<210> 20
<211> 111
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 20
Asp Val Val Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asn Ile Arg Thr Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ala Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asp Leu Glu Pro
65 70 75 80
Gly Asp Ala Ala Thr Tyr Tyr Cys Gln Ser Thr Tyr Leu Gly Thr Asp
85 90 95
Tyr Val Gly Gly Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 21
<211> 345
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 21
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggaat cgacttcagt aggagatact acatgtgctg ggtccgccag 120
gctccaggga aggggctgga gtggatcgca tgcatatata ctggtagccg cgatactcct 180
cactacgcga gctccgcgaa aggccggttc accatctcca gagacaattc caagaacacg 240
ctgtatctgc aaatgaacag cctgagagcc gaggacacgg ccgtatatta ctgtgcgaga 300
gaaggtagcc tgtggggcca gggaaccctg gtcaccgtct cgagc 345
<210> 22
<211> 115
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 22
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Asp Phe Ser Arg Arg
20 25 30
Tyr Tyr Met Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Ala Cys Ile Tyr Thr Gly Ser Arg Asp Thr Pro His Tyr Ala Ser
50 55 60
Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Ala Arg Glu Gly Ser Leu Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 23
<211> 333
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 23
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc agtccagtca gagtgtttat agtaactggt tctcctggta tcagcagaaa 120
ccagggaaag cccctaagct cctgatctat tctgcatcca ctctggcatc tggggtccca 180
tcaaggttca gcggcagtgg atctgggaca gaattcactc tcaccatcag cagcctgcag 240
cctgatgatt ttgcaactta ttactgcgca ggcggttaca atactgttat tgatactttt 300
gctttcggcg gagggaccaa ggtggagatc aaa 333
<210> 24
<211> 111
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 24
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser Val Tyr Ser Asn
20 25 30
Trp Phe Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Ala Gly Gly Tyr Asn Thr Val
85 90 95
Ile Asp Thr Phe Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 25
<211> 366
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 25
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccatcagt cgctaccaca tgacttgggt ccgccaggct 120
ccagggaagg ggctggagtg gatcggacat atttatgtta ataatgatga cacagactac 180
gcgagctccg cgaaaggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccacct atttctgtgc gagattggat 300
gttggtggtg gtggtgctta tattggggac atctggggcc agggaactct ggttaccgtc 360
tcttca 366
<210> 26
<211> 122
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 26
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Arg Tyr
20 25 30
His Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly His Ile Tyr Val Asn Asn Asp Asp Thr Asp Tyr Ala Ser Ser Ala
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Phe Cys
85 90 95
Ala Arg Leu Asp Val Gly Gly Gly Gly Ala Tyr Ile Gly Asp Ile Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 27
<211> 339
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 27
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc agtccagtca gagtgtttat aacaacaacg acttagcctg gtatcagcag 120
aaaccaggga aagttcctaa gctcctgatc tattatgctt ccactctggc atctggggtc 180
ccatctcggt tcagtggcag tggatctggg acagatttca ctctcaccat cagcagcctg 240
cagcctgaag atgttgcaac ttattactgt gcaggcggtt atgatacgga tggtcttgat 300
acgtttgctt tcggcggagg gaccaaggtg gagatcaaa 339
<210> 28
<211> 113
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 28
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser Val Tyr Asn Asn
20 25 30
Asn Asp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu
35 40 45
Leu Ile Tyr Tyr Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe
50 55 60
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
65 70 75 80
Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Ala Gly Gly Tyr Asp Thr
85 90 95
Asp Gly Leu Asp Thr Phe Ala Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys
<210> 29
<211> 345
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 29
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtactg cctctggatt ctccctcagt agctatgcaa tgagctgggt ccgccaggct 120
ccagggaggg ggctggagtg gatcggaatc atttatgcta gtggtagcac atactacgcg 180
agctcggcga aaggcagatt caccatctcc aaagacaata ccaagaacac ggtggatctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag aatttatgac 300
ggcatggacc tctggggcca gggaactctg gttaccgtct cttca 345
<210> 30
<211> 115
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 30
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Arg Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Tyr Ala Ser Gly Ser Thr Tyr Tyr Ala Ser Ser Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Lys Asp Asn Thr Lys Asn Thr Val Asp Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Ile Tyr Asp Gly Met Asp Leu Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 31
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 31
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aggccagtca gaacatttac agctacttat cctggtatca gcagaaacca 120
gggaaagttc ctaagcgcct gatctatctg gcatctactc tggcatctgg ggtcccatct 180
cggttcagtg gcagtggatc tgggacagat tacactctca ccatcagcag cctgcagcct 240
gaagatgttg caacttatta ctgtcaaagc aattataacg gtaattatgg tttcggcgga 300
gggaccaagg tggagatcaa a 321
<210> 32
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 32
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Gln Asn Ile Tyr Ser Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Arg Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Ser Asn Tyr Asn Gly Asn Tyr
85 90 95
Gly Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 33
<211> 354
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 33
gaggtgaagc tggatgagac tggaggaggc ttggtgcaac ctgggaggcc catgaaactc 60
tcctgtgttg cctctggatt cacttttagt gactactgga tgaactgggt ccgccagtct 120
ccagagaaag gactggagtg ggtagcacaa attagaaaca aaccttataa ttatgaaaca 180
tattattcag attctgtgaa aggcagattc accatctcaa gagatgattc caaaagtagt 240
gtctacctgc aaatgaacaa cttaagagtt gaagacatgg gtatctatta ctgtacgggt 300
tcttactatg gtatggacta ctggggtcaa ggaacctcag tcaccgtctc ctca 354
<210> 34
<211> 118
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 34
Glu Val Lys Leu Asp Glu Thr Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Pro Met Lys Leu Ser Cys Val Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Trp Met Asn Trp Val Arg Gln Ser Pro Glu Lys Gly Leu Glu Trp Val
35 40 45
Ala Gln Ile Arg Asn Lys Pro Tyr Asn Tyr Glu Thr Tyr Tyr Ser Asp
50 55 60
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Ser Ser
65 70 75 80
Val Tyr Leu Gln Met Asn Asn Leu Arg Val Glu Asp Met Gly Ile Tyr
85 90 95
Tyr Cys Thr Gly Ser Tyr Tyr Gly Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser
115
<210> 35
<211> 336
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 35
gatgtcgtga tgacccaaac tccactctcc ctgcctgtca gtcttggaga tcaagcctcc 60
atctcttgca gatctagtca gagccttgta cacagtaatg gaaacaccta tttacgttgg 120
tacctgcaga agccaggcca gtctccaaag gtcctgatct acaaagtttc caaccgattt 180
tctggggtcc cagacaggtt cagtggcagt ggatcaggga cagatttcac actcaagatc 240
agcagagtgg aggctgagga tctgggagtt tatttctgct ctcaaagtac acatgttccg 300
tggacgttcg gtggaggcac caagctggaa atcaaa 336
<210> 36
<211> 112
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 36
Asp Val Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Arg Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Val Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser
85 90 95
Thr His Val Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 37
<211> 987
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 37
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 60
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 120
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 240
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 300
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc cgcgggggca 360
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 420
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 480
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 540
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 600
gagtacaagt gcgcggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 660
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 720
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 780
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 840
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 900
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 960
cagaagagcc tctccctgtc tccgggt 987
<210> 38
<211> 329
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 38
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly
325
<210> 39
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 39
cgtacggtgg ctgcaccatc tgtcttcatc ttcccgccat ctgatgagca gttgaaatct 60
ggaactgcct ctgttgtgtg cctgctgaat aacttctatc ccagagaggc caaagtacag 120
tggaaggtgg ataacgccct ccaatcgggt aactcccagg agagtgtcac agagcaggac 180
agcaaggaca gcacctacag cctcagcagc accctgacgc tgagcaaagc agactacgag 240
aaacacaaag tctacgcctg cgaagtcacc catcagggcc tgagctcgcc cgtcacaaag 300
agcttcaaca ggggagagtg t 321
<210> 40
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 40
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 41
<211> 3681
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 41
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc agtccagtca gagtgtttat agtaactggt tctcctggta tcagcagaaa 120
ccagggaaag cccctaagct cctgatctat tctgcatcca ctctggcatc tggggtccca 180
tcaaggttca gcggcagtgg atctgggaca gaattcactc tcaccatcag cagcctgcag 240
cctgatgatt ttgcaactta ttactgcgca ggcggttaca atactgttat tgatactttt 300
gctttcggcg gagggaccaa ggtggagatc aaaggcggtg gcggtagtgg gggaggcggt 360
tctggcggcg gagggtccgg cggtggagga tcagaggtgc agctgttgga gtctggggga 420
ggcttggtac agcctggggg gtccctgaga ctctcctgtg cagcctctgg aatcgacttc 480
agtaggagat actacatgtg ctgggtccgc caggctccag ggaaggggct ggagtggatc 540
gcatgcatat atactggtag ccgcgatact cctcactacg cgagctccgc gaaaggccgg 600
ttcaccatct ccagagacaa ttccaagaac acgctgtatc tgcaaatgaa cagcctgaga 660
gccgaggaca cggccgtata ttactgtgcg agagaaggta gcctgtgggg ccagggaacc 720
ctggtcaccg tctcgagcgg cggtggaggg tccggcggtg gtggatccca gtcggtggag 780
gagtctgggg gaggcttggt ccagcctggg gggtccctga gactctcctg tacagcctct 840
ggaatcgacc ttaataccta cgacatgatc tgggtccgcc aggctccagg caaggggcta 900
gagtgggttg gaatcattac ttatagtggt agtagatact acgcgaactg ggcgaaaggc 960
cgattcacca tctccaaaga caataccaag aacacggtgt atctgcaaat gaacagcctg 1020
agagctgagg acacggctgt gtattactgt gccagagatt atatgagtgg ttcccacttg 1080
tggggccagg gaaccctggt caccgtctct agtgctagca ccaagggccc atcggtcttc 1140
cccctggcac cctcctccaa gagcacctct gggggcacag cggccctggg ctgcctggtc 1200
aaggactact tccccgaacc ggtgacggtg tcgtggaact caggcgccct gaccagcggc 1260
gtgcacacct tcccggctgt cctacagtcc tcaggactct actccctcag cagcgtggtg 1320
accgtgccct ccagcagctt gggcacccag acctacatct gcaacgtgaa tcacaagccc 1380
agcaacacca aggtggacaa gagagttgag cccaaatctt gtgacaaaac tcacacatgc 1440
ccaccgtgcc cagcacctga agccgcgggg gcaccgtcag tcttcctctt ccccccaaaa 1500
cccaaggaca ccctcatgat ctcccggacc cctgaggtca catgcgtggt ggtggacgtg 1560
agccacgaag accctgaggt caagttcaac tggtacgtgg acggcgtgga ggtgcataat 1620
gccaagacaa agccgcggga ggagcagtac aacagcacgt accgtgtggt cagcgtcctc 1680
accgtcctgc accaggactg gctgaatggc aaggagtaca agtgcgcggt ctccaacaaa 1740
gccctcccag cccccatcga gaaaaccatc tccaaagcca aagggcagcc ccgagaacca 1800
caggtgtata ccctgccccc atcccgggat gagctgacca agaaccaggt cagcctgacc 1860
tgcctggtca aaggcttcta tcccagcgac atcgccgtgg agtgggagag caatgggcag 1920
ccggagaaca actacaagac cacgcctccc gtgctggact ccgacggctc cttcttcctc 1980
tatagcaagc tcaccgtgga caagagcagg tggcagcagg ggaacgtctt ctcatgctcc 2040
gtgatgcatg aggctctgca caaccactac acgcagaaga gcctctccct gtctccgggt 2100
ggcggtggag ggtccggcgg tggtggatcc gaggtgcagc tgttggagtc tgggggaggc 2160
ttggtacagc ctggggggtc cctgagactc tcctgtgcag cctctggatt caccatcagt 2220
cgctaccaca tgacttgggt ccgccaggct ccagggaagg ggctggagtg gatcggacat 2280
atttatgtta ataatgatga cacagactac gcgagctccg cgaaaggccg gttcaccatc 2340
tccagagaca attccaagaa cacgctgtat ctgcaaatga acagcctgag agccgaggac 2400
acggccacct atttctgtgc gagattggat gttggtggtg gtggtgctta tattggggac 2460
atctggggcc agggaactct ggttaccgtc tcttcaggcg gtggcggtag tgggggaggc 2520
ggttctggcg gcggagggtc cggcggtgga ggatcagaca tccagatgac ccagtctcca 2580
tcctccctgt ctgcatctgt aggagacaga gtcaccatca cttgccagtc cagtcagagt 2640
gtttataaca acaacgactt agcctggtat cagcagaaac cagggaaagt tcctaagctc 2700
ctgatctatt atgcttccac tctggcatct ggggtcccat ctcggttcag tggcagtgga 2760
tctgggacag atttcactct caccatcagc agcctgcagc ctgaagatgt tgcaacttat 2820
tactgtgcag gcggttatga tacggatggt cttgatacgt ttgctttcgg cggagggacc 2880
aaggtggaga tcaaaggcgg tggagggtcc ggcggtggtg gatccgaggt gcagctggtg 2940
gagtctgggg gaggcttggt ccagcctggg gggtccctga gactctcctg tgcagcctct 3000
ggattcacca tcagtaccaa tgcaatgagc tgggtccgcc aggctccagg gaaggggctg 3060
gagtggatcg gagtcattac tggtcgtgat atcacatact acgcgagctg ggcgaaaggc 3120
agattcacca tctccagaga caattccaag aacacgctgt atcttcaaat gaacagcctg 3180
agagccgagg acacggctgt gtattactgt gcgcgcgacg gtggatcatc tgctattact 3240
agtaacaaca tttggggcca aggaactctg gtcaccgttt cttcaggcgg tggcggtagt 3300
gggggaggcg gttctggcgg cggagggtcc ggcggtggag gatcagacgt cgtgatgacc 3360
cagtctcctt ccaccctgtc tgcatctgta ggagacagag tcaccatcaa ttgccaagcc 3420
agtgagagca ttagcagttg gttagcctgg tatcagcaga aaccagggaa agcccctaag 3480
ctcctgatct atgaagcatc caaactggca tctggggtcc catcaaggtt cagcggcagt 3540
ggatctggga cagagttcac tctcaccatc agcagcctgc agcctgatga ttttgcaact 3600
tattactgcc aaggctattt ttattttatt agtcgtactt atgtaaattc tttcggcgga 3660
gggaccaagg tggagatcaa a 3681
<210> 42
<211> 1227
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 42
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser Val Tyr Ser Asn
20 25 30
Trp Phe Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Ser Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Ala Gly Gly Tyr Asn Thr Val
85 90 95
Ile Asp Thr Phe Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly
100 105 110
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
130 135 140
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Asp Phe
145 150 155 160
Ser Arg Arg Tyr Tyr Met Cys Trp Val Arg Gln Ala Pro Gly Lys Gly
165 170 175
Leu Glu Trp Ile Ala Cys Ile Tyr Thr Gly Ser Arg Asp Thr Pro His
180 185 190
Tyr Ala Ser Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
195 200 205
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
210 215 220
Ala Val Tyr Tyr Cys Ala Arg Glu Gly Ser Leu Trp Gly Gln Gly Thr
225 230 235 240
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
245 250 255
Gln Ser Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
260 265 270
Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Asp Leu Asn Thr Tyr Asp
275 280 285
Met Ile Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
290 295 300
Ile Ile Thr Tyr Ser Gly Ser Arg Tyr Tyr Ala Asn Trp Ala Lys Gly
305 310 315 320
Arg Phe Thr Ile Ser Lys Asp Asn Thr Lys Asn Thr Val Tyr Leu Gln
325 330 335
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
340 345 350
Asp Tyr Met Ser Gly Ser His Leu Trp Gly Gln Gly Thr Leu Val Thr
355 360 365
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
370 375 380
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
385 390 395 400
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
405 410 415
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
420 425 430
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
435 440 445
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
450 455 460
Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
465 470 475 480
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu
485 490 495
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
500 505 510
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
515 520 525
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
530 535 540
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
545 550 555 560
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala
565 570 575
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
580 585 590
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
595 600 605
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
610 615 620
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
625 630 635 640
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
645 650 655
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
660 665 670
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
675 680 685
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly
690 695 700
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly
705 710 715 720
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
725 730 735
Phe Thr Ile Ser Arg Tyr His Met Thr Trp Val Arg Gln Ala Pro Gly
740 745 750
Lys Gly Leu Glu Trp Ile Gly His Ile Tyr Val Asn Asn Asp Asp Thr
755 760 765
Asp Tyr Ala Ser Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
770 775 780
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
785 790 795 800
Thr Ala Thr Tyr Phe Cys Ala Arg Leu Asp Val Gly Gly Gly Gly Ala
805 810 815
Tyr Ile Gly Asp Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
820 825 830
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
835 840 845
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
850 855 860
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser
865 870 875 880
Val Tyr Asn Asn Asn Asp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
885 890 895
Val Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Thr Leu Ala Ser Gly Val
900 905 910
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
915 920 925
Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Ala Gly
930 935 940
Gly Tyr Asp Thr Asp Gly Leu Asp Thr Phe Ala Phe Gly Gly Gly Thr
945 950 955 960
Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
965 970 975
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
980 985 990
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Thr Asn Ala
995 1000 1005
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
1010 1015 1020
Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala Ser Trp Ala
1025 1030 1035
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
1040 1045 1050
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
1055 1060 1065
Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
1070 1075 1080
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
1085 1090 1095
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
1100 1105 1110
Gly Ser Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala
1115 1120 1125
Ser Val Gly Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser
1130 1135 1140
Ile Ser Ser Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala
1145 1150 1155
Pro Lys Leu Leu Ile Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val
1160 1165 1170
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu
1175 1180 1185
Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys
1190 1195 1200
Gln Gly Tyr Phe Tyr Phe Ile Ser Arg Thr Tyr Val Asn Ser Phe
1205 1210 1215
Gly Gly Gly Thr Lys Val Glu Ile Lys
1220 1225
<210> 43
<211> 651
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 43
gcctatgata tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcaagtgtc aggccagtga ggacatttat agcttcttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatccattct gcatcctctc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag ggttatggta aaaataatgt tgataatgct 300
ttcggcggag ggaccaaggt ggagatcaaa cgtacggtgg ctgcaccatc tgtcttcatc 360
ttcccgccat ctgatgagca gttgaaatct ggaactgcct ctgttgtgtg cctgctgaat 420
aacttctatc ccagagaggc caaagtacag tggaaggtgg ataacgccct ccaatcgggt 480
aactcccagg agagtgtcac agagcaggac agcaaggaca gcacctacag cctcagcagc 540
accctgacgc tgagcaaagc agactacgag aaacacaaag tctacgcctg cgaagtcacc 600
catcagggcc tgagctcgcc cgtcacaaag agcttcaaca ggggagagtg t 651
<210> 44
<211> 217
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> синтезированная
<400> 44
Ala Tyr Asp Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Lys Cys Gln Ala Ser Glu Asp Ile Tyr Ser Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
His Ser Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Gly Lys Asn Asn
85 90 95
Val Asp Asn Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr
100 105 110
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
115 120 125
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
130 135 140
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
145 150 155 160
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
165 170 175
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
180 185 190
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
195 200 205
Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 45
<211> 360
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 45
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt caccatcagt accaatgcaa tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg gatcggagtc attactggtc gtgatatcac atactacgcg 180
agctgggcga aaggcagatt caccatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag agacggtggt 300
tcttctgcta ttactagtaa caacatttgg ggccagggaa ccctggtcac cgtgtcgaca 360
<210> 46
<211> 120
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 46
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Thr Asn
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn Ile Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Thr
115 120
<210> 47
<211> 363
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 47
gaggtgcagc tggtgcagtc tggagcagag gtgaagaaac caggagagtc tctgaagatc 60
tcctgtaagg gttctggata cagctttagc agttcatgga tcggctgggt gcgccaggca 120
cctgggaaag gcctggaatg gatggggatc atctatcctg atgactctga taccagatac 180
agtccatcct tccaaggcca ggtcaccatc tcagccgaca agtccatcag gactgcctac 240
ctgcagtgga gtagcctgaa ggcctcggac accgctatgt attactgtgc gagacatgtt 300
actatgattt ggggagttat tattgacttc tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 48
<211> 121
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 48
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Ser Ser Ser
20 25 30
Trp Ile Gly Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Asp Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Arg Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg His Val Thr Met Ile Trp Gly Val Ile Ile Asp Phe Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 49
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 49
gccatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattagc agtgctttag cctggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag tttaatagtt acccattcac tttcggccct 300
gggaccaaag tggatatcaa a 321
<210> 50
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 50
Ala Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Ala
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 51
<211> 348
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 51
gatgtgcagc ttcaggagtc gggacctagc ctggtgaaac cttctcagtc tctgtccctc 60
acctgcactg tcactggcta ctcaatcacc agtgattttg cctggaactg gattcggcag 120
tttccaggaa acaagctgga gtggatgggc tacataagtt atagtggtaa cactaggtac 180
aacccatctc tcaaaagtcg aatctctatc actcgcgaca catccaagaa ccaattcttc 240
ctgcagttga actctgtgac tattgaggac acagccacat attactgtgt aacggcggga 300
cgcgggtttc cttattgggg ccaagggact ctggtcactg tctctgca 348
<210> 52
<211> 116
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 52
Asp Val Gln Leu Gln Glu Ser Gly Pro Ser Leu Val Lys Pro Ser Gln
1 5 10 15
Ser Leu Ser Leu Thr Cys Thr Val Thr Gly Tyr Ser Ile Thr Ser Asp
20 25 30
Phe Ala Trp Asn Trp Ile Arg Gln Phe Pro Gly Asn Lys Leu Glu Trp
35 40 45
Met Gly Tyr Ile Ser Tyr Ser Gly Asn Thr Arg Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Ile Ser Ile Thr Arg Asp Thr Ser Lys Asn Gln Phe Phe
65 70 75 80
Leu Gln Leu Asn Ser Val Thr Ile Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Val Thr Ala Gly Arg Gly Phe Pro Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ala
115
<210> 53
<211> 321
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 53
gacatcctga tgacccaatc tccatcctcc atgtctgtat ctctgggaga cacagtcagc 60
atcacttgcc attcaagtca ggacattaac agtaatatag ggtggttgca gcagagacca 120
gggaaatcat ttaagggcct gatctatcat ggaaccaact tggacgatga agttccatca 180
aggttcagtg gcagtggatc tggagccgat tattctctca ccatcagcag cctggaatct 240
gaagattttg cagactatta ctgtgtacag tatgctcagt ttccgtggac gttcggtgga 300
ggcaccaagc tggaaatcaa a 321
<210> 54
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 54
Asp Ile Leu Met Thr Gln Ser Pro Ser Ser Met Ser Val Ser Leu Gly
1 5 10 15
Asp Thr Val Ser Ile Thr Cys His Ser Ser Gln Asp Ile Asn Ser Asn
20 25 30
Ile Gly Trp Leu Gln Gln Arg Pro Gly Lys Ser Phe Lys Gly Leu Ile
35 40 45
Tyr His Gly Thr Asn Leu Asp Asp Glu Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Val Gln Tyr Ala Gln Phe Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 55
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 55
ggcggtggag ggtccggcgg tggtggctcc gga 33
<210> 56
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 56
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10
<210> 57
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 57
ggcggtggag ggtccggcgg tggtggatca 30
<210> 58
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 58
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 59
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 59
ggcggtggag ggtccggcgg tggtggatcc 30
<210> 60
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 60
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 61
<211> 60
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 61
ggcggtggcg gtagtggggg aggcggttct ggcggcggag ggtccggcgg tggaggatca 60
<210> 62
<211> 20
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 62
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 63
<211> 3693
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 63
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagag ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaaggcg gtggcggtag tgggggaggc 360
ggttctggcg gcggagggtc cggcggtgga ggatcagagg tgcagctggt ggagtctggg 420
ggaggcttgg tccagcctgg ggggtccctg agactctcct gtgcagcctc tggattcacc 480
atcagtacca atgcaatgag ctgggtccgc caggctccag ggaaggggct ggagtggatc 540
ggagtcatta ctggtcgtga tatcacatac tacgcgagct gggcgaaagg cagattcacc 600
atctccagag acaattccaa gaacacgctg tatcttcaaa tgaacagcct gagagccgag 660
gacacggctg tgtattactg tgcgcgcgac ggtggatcat ctgctattac tagtaacaac 720
atttggggcc aaggaactct ggtcaccgtt tcttcaggcg gtggagggtc cggcggtggt 780
ggatccgatg tgcagcttca ggagtcggga cctagcctgg tgaaaccttc tcagtctctg 840
tccctcacct gcactgtcac tggctactca atcaccagtg attttgcctg gaactggatt 900
cggcagtttc caggaaacaa gctggagtgg atgggctaca taagttatag tggtaacact 960
aggtacaacc catctctcaa aagtcgaatc tctatcactc gcgacacatc caagaaccaa 1020
ttcttcctgc agttgaactc tgtgactatt gaggacacag ccacatatta ctgtgtaacg 1080
gcgggacgcg ggtttcctta ttggggccaa gggactctgg tcactgtctc tgcagctagc 1140
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 1200
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 1260
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 1320
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 1380
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agagagttga gcccaaatct 1440
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagccgcggg ggcaccgtca 1500
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 1560
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 1620
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 1680
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 1740
aagtgcgcgg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 1800
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga tgagctgacc 1860
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1920
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1980
tccgacggct ccttcttcct ctatagcaag ctcaccgtgg acaagagcag gtggcagcag 2040
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 2100
agcctctccc tgtctccggg tggcggtgga gggtccggcg gtggtggatc cgaggtgcag 2160
ctgttggagt ctgggggagg cttggtacag cctggggggt ccctgagact ctcctgtgca 2220
gcctctggat tctccttcag tagcgggtac gacatgtgct gggtccgcca ggctccaggg 2280
aaggggctgg agtggatcgc atgcattgct gctggtagtg ctggtatcac ttacgacgcg 2340
aactgggcga aaggccggtt caccatctcc agagacaatt ccaagaacac gctgtatctg 2400
caaatgaaca gcctgagagc cgaggacacg gccgtatatt actgtgcgag atcggcgttt 2460
tcgttcgact acgccatgga cctctggggc cagggaaccc tggtcaccgt ctcgagcggc 2520
ggtggcggta gtgggggagg cggttctggc ggcggagggt ccggcggtgg aggatcagac 2580
atccagatga cccagtctcc ttccaccctg tctgcatctg taggagacag agtcaccatc 2640
acttgccagg ccagtcagag cattagttcc cacttaaact ggtatcagca gaaaccaggg 2700
aaagccccta agctcctgat ctataaggca tccactctgg catctggggt cccatcaagg 2760
ttcagcggca gtggatctgg gacagaattt actctcacca tcagcagcct gcagcctgat 2820
gattttgcaa cttattactg ccaacagggt tatagttggg gtaatgttga taatgttttc 2880
ggcggaggga ccaaggtgga gatcaaaggc ggtggagggt ccggcggtgg tggatcccag 2940
tcgctggtgg agtctggggg aggcttggta cagcctgggg ggtccctgag actctcctgt 3000
gcagcctctg gattctcctt cagtagcaac tactggatat gctgggtccg ccaggctcca 3060
gggaaggggc tggagtggat cgcatgtatt tatgttggta gtagtggtga cacttactac 3120
gcgagctccg cgaaaggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 3180
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gagagatagt 3240
agtagttatt atatgtttaa cttgtggggc cagggaaccc tggtcaccgt ctcttcaggc 3300
ggtggcggta gtgggggagg cggttctggc ggcggagggt ccggcggtgg aggatcagcc 3360
cttgtgatga cccagtctcc ttccaccctg tctgcatctg taggagacag agtcaccatc 3420
aattgccagg ccagtgagga cattgatacc tatttagcct ggtatcagca gaaaccaggg 3480
aaagccccta agctcctgat cttttacgca tccgatctgg catctggggt cccatcaagg 3540
ttcagcggca gtggatctgg gacagaattt actctcacca tcagcagcct gcagcctgat 3600
gattttgcaa cttattactg ccaaggcggt tactatacta gtagtgctga tacgaggggt 3660
gctttcggcg gagggaccaa ggtggagatc aaa 3693
<210> 64
<211> 1231
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 64
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
130 135 140
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
145 150 155 160
Ile Ser Thr Asn Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
165 170 175
Leu Glu Trp Ile Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala
180 185 190
Ser Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
195 200 205
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
210 215 220
Tyr Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
225 230 235 240
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
245 250 255
Ser Gly Gly Gly Gly Ser Asp Val Gln Leu Gln Glu Ser Gly Pro Ser
260 265 270
Leu Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Thr Val Thr Gly
275 280 285
Tyr Ser Ile Thr Ser Asp Phe Ala Trp Asn Trp Ile Arg Gln Phe Pro
290 295 300
Gly Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Ser Gly Asn Thr
305 310 315 320
Arg Tyr Asn Pro Ser Leu Lys Ser Arg Ile Ser Ile Thr Arg Asp Thr
325 330 335
Ser Lys Asn Gln Phe Phe Leu Gln Leu Asn Ser Val Thr Ile Glu Asp
340 345 350
Thr Ala Thr Tyr Tyr Cys Val Thr Ala Gly Arg Gly Phe Pro Tyr Trp
355 360 365
Gly Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro
370 375 380
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
385 390 395 400
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
405 410 415
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
420 425 430
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
435 440 445
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
450 455 460
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
465 470 475 480
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
485 490 495
Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
500 505 510
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
515 520 525
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
530 535 540
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
545 550 555 560
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
565 570 575
Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro
580 585 590
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
595 600 605
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
610 615 620
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
625 630 635 640
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
645 650 655
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
660 665 670
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
675 680 685
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
690 695 700
Ser Pro Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln
705 710 715 720
Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
725 730 735
Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser Gly Tyr Asp Met
740 745 750
Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala Cys
755 760 765
Ile Ala Ala Gly Ser Ala Gly Ile Thr Tyr Asp Ala Asn Trp Ala Lys
770 775 780
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
785 790 795 800
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
805 810 815
Arg Ser Ala Phe Ser Phe Asp Tyr Ala Met Asp Leu Trp Gly Gln Gly
820 825 830
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
835 840 845
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr
850 855 860
Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
865 870 875 880
Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser His Leu Asn Trp Tyr Gln
885 890 895
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Ala Ser Thr
900 905 910
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
915 920 925
Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr
930 935 940
Tyr Tyr Cys Gln Gln Gly Tyr Ser Trp Gly Asn Val Asp Asn Val Phe
945 950 955 960
Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
965 970 975
Gly Gly Ser Gln Ser Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
980 985 990
Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser
995 1000 1005
Ser Asn Tyr Trp Ile Cys Trp Val Arg Gln Ala Pro Gly Lys Gly
1010 1015 1020
Leu Glu Trp Ile Ala Cys Ile Tyr Val Gly Ser Ser Gly Asp Thr
1025 1030 1035
Tyr Tyr Ala Ser Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp
1040 1045 1050
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala
1055 1060 1065
Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Ser Ser Ser Tyr
1070 1075 1080
Tyr Met Phe Asn Leu Trp Gly Gln Gly Thr Leu Val Thr Val Ser
1085 1090 1095
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1100 1105 1110
Ser Gly Gly Gly Gly Ser Ala Leu Val Met Thr Gln Ser Pro Ser
1115 1120 1125
Thr Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Asn Cys Gln
1130 1135 1140
Ala Ser Glu Asp Ile Asp Thr Tyr Leu Ala Trp Tyr Gln Gln Lys
1145 1150 1155
Pro Gly Lys Ala Pro Lys Leu Leu Ile Phe Tyr Ala Ser Asp Leu
1160 1165 1170
Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
1175 1180 1185
Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala
1190 1195 1200
Thr Tyr Tyr Cys Gln Gly Gly Tyr Tyr Thr Ser Ser Ala Asp Thr
1205 1210 1215
Arg Gly Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1220 1225 1230
<210> 65
<211> 642
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 65
gacatcctga tgacccaatc tccatcctcc atgtctgtat ctctgggaga cacagtcagc 60
atcacttgcc attcaagtca ggacattaac agtaatatag ggtggttgca gcagagacca 120
gggaaatcat ttaagggcct gatctatcat ggaaccaact tggacgatga agttccatca 180
aggttcagtg gcagtggatc tggagccgat tattctctca ccatcagcag cctggaatct 240
gaagattttg cagactatta ctgtgtacag tatgctcagt ttccgtggac gttcggtgga 300
ggcaccaagc tggaaatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 66
<211> 214
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 66
Asp Ile Leu Met Thr Gln Ser Pro Ser Ser Met Ser Val Ser Leu Gly
1 5 10 15
Asp Thr Val Ser Ile Thr Cys His Ser Ser Gln Asp Ile Asn Ser Asn
20 25 30
Ile Gly Trp Leu Gln Gln Arg Pro Gly Lys Ser Phe Lys Gly Leu Ile
35 40 45
Tyr His Gly Thr Asn Leu Asp Asp Glu Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Val Gln Tyr Ala Gln Phe Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 67
<211> 3678
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 67
gacatcctga tgacccaatc tccatcctcc atgtctgtat ctctgggaga cacagtcagc 60
atcacttgcc attcaagtca ggacattaac agtaatatag ggtggttgca gcagagacca 120
gggaaatcat ttaagggcct gatctatcat ggaaccaact tggacgatga agttccatca 180
aggttcagtg gcagtggatc tggagccgat tattctctca ccatcagcag cctggaatct 240
gaagattttg cagactatta ctgtgtacag tatgctcagt ttccgtggac gttcggtgga 300
ggcaccaagc tggaaatcaa aggcggtggc ggtagtgggg gaggcggttc tggcggcgga 360
gggtccggcg gtggaggatc agatgtgcag cttcaggagt cgggacctag cctggtgaaa 420
ccttctcagt ctctgtccct cacctgcact gtcactggct actcaatcac cagtgatttt 480
gcctggaact ggattcggca gtttccagga aacaagctgg agtggatggg ctacataagt 540
tatagtggta acactaggta caacccatct ctcaaaagtc gaatctctat cactcgcgac 600
acatccaaga accaattctt cctgcagttg aactctgtga ctattgagga cacagccaca 660
tattactgtg taacggcggg acgcgggttt ccttattggg gccaagggac tctggtcact 720
gtctctgcag gcggtggagg gtccggcggt ggtggatccg aggtgcagct ggtggagtct 780
gggggaggct tggtccagcc tggggggtcc ctgagactct cctgtgcagc ctctggattc 840
accatcagta ccaatgcaat gagctgggtc cgccaggctc cagggaaggg gctggagtgg 900
atcggagtca ttactggtcg tgatatcaca tactacgcga gctgggcgaa aggcagattc 960
accatctcca gagacaattc caagaacacg ctgtatcttc aaatgaacag cctgagagcc 1020
gaggacacgg ctgtgtatta ctgtgcgcgc gacggtggat catctgctat tactagtaac 1080
aacatttggg gccaaggaac tctggtcacc gtttcttcag ctagcaccaa gggcccatcg 1140
gtcttccccc tggcaccctc ctccaagagc acctctgggg gcacagcggc cctgggctgc 1200
ctggtcaagg actacttccc cgaaccggtg acggtgtcgt ggaactcagg cgccctgacc 1260
agcggcgtgc acaccttccc ggctgtccta cagtcctcag gactctactc cctcagcagc 1320
gtggtgaccg tgccctccag cagcttgggc acccagacct acatctgcaa cgtgaatcac 1380
aagcccagca acaccaaggt ggacaagaga gttgagccca aatcttgtga caaaactcac 1440
acatgcccac cgtgcccagc acctgaagcc gcgggggcac cgtcagtctt cctcttcccc 1500
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacatg cgtggtggtg 1560
gacgtgagcc acgaagaccc tgaggtcaag ttcaactggt acgtggacgg cgtggaggtg 1620
cataatgcca agacaaagcc gcgggaggag cagtacaaca gcacgtaccg tgtggtcagc 1680
gtcctcaccg tcctgcacca ggactggctg aatggcaagg agtacaagtg cgcggtctcc 1740
aacaaagccc tcccagcccc catcgagaaa accatctcca aagccaaagg gcagccccga 1800
gaaccacagg tgtacaccct gcccccatcc cgggatgagc tgaccaagaa ccaggtcagc 1860
ctgacctgcc tggtcaaagg cttctatccc agcgacatcg ccgtggagtg ggagagcaat 1920
gggcagccgg agaacaacta caagaccacg cctcccgtgc tggactccga cggctccttc 1980
ttcctctata gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca 2040
tgctccgtga tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct 2100
ccgggtggcg gtggagggtc cggcggtggt ggatccgagg tgcagctgtt ggagtctggg 2160
ggaggcttgg tacagcctgg ggggtccctg agactctcct gtgcagcctc tggattctcc 2220
ttcagtagcg ggtacgacat gtgctgggtc cgccaggctc cagggaaggg gctggagtgg 2280
atcgcatgca ttgctgctgg tagtgctggt atcacttacg acgcgaactg ggcgaaaggc 2340
cggttcacca tctccagaga caattccaag aacacgctgt atctgcaaat gaacagcctg 2400
agagccgagg acacggccgt atattactgt gcgagatcgg cgttttcgtt cgactacgcc 2460
atggacctct ggggccaggg aaccctggtc accgtctcga gcggcggtgg cggtagtggg 2520
ggaggcggtt ctggcggcgg agggtccggc ggtggaggat cagacatcca gatgacccag 2580
tctccttcca ccctgtctgc atctgtagga gacagagtca ccatcacttg ccaggccagt 2640
cagagcatta gttcccactt aaactggtat cagcagaaac cagggaaagc ccctaagctc 2700
ctgatctata aggcatccac tctggcatct ggggtcccat caaggttcag cggcagtgga 2760
tctgggacag aatttactct caccatcagc agcctgcagc ctgatgattt tgcaacttat 2820
tactgccaac agggttatag ttggggtaat gttgataatg ttttcggcgg agggaccaag 2880
gtggagatca aaggcggtgg agggtccggc ggtggtggat cccagtcgct ggtggagtct 2940
gggggaggct tggtacagcc tggggggtcc ctgagactct cctgtgcagc ctctggattc 3000
tccttcagta gcaactactg gatatgctgg gtccgccagg ctccagggaa ggggctggag 3060
tggatcgcat gtatttatgt tggtagtagt ggtgacactt actacgcgag ctccgcgaaa 3120
ggccggttca ccatctccag agacaattcc aagaacacgc tgtatctgca aatgaacagc 3180
ctgagagccg aggacacggc cgtatattac tgtgcgagag atagtagtag ttattatatg 3240
tttaacttgt ggggccaggg aaccctggtc accgtctctt caggcggtgg cggtagtggg 3300
ggaggcggtt ctggcggcgg agggtccggc ggtggaggat cagcccttgt gatgacccag 3360
tctccttcca ccctgtctgc atctgtagga gacagagtca ccatcaattg ccaggccagt 3420
gaggacattg atacctattt agcctggtat cagcagaaac cagggaaagc ccctaagctc 3480
ctgatctttt acgcatccga tctggcatct ggggtcccat caaggttcag cggcagtgga 3540
tctgggacag aatttactct caccatcagc agcctgcagc ctgatgattt tgcaacttat 3600
tactgccaag gcggttacta tactagtagt gctgatacga ggggtgcttt cggcggaggg 3660
accaaggtgg agatcaaa 3678
<210> 68
<211> 1226
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 68
Asp Ile Leu Met Thr Gln Ser Pro Ser Ser Met Ser Val Ser Leu Gly
1 5 10 15
Asp Thr Val Ser Ile Thr Cys His Ser Ser Gln Asp Ile Asn Ser Asn
20 25 30
Ile Gly Trp Leu Gln Gln Arg Pro Gly Lys Ser Phe Lys Gly Leu Ile
35 40 45
Tyr His Gly Thr Asn Leu Asp Asp Glu Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Val Gln Tyr Ala Gln Phe Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp
115 120 125
Val Gln Leu Gln Glu Ser Gly Pro Ser Leu Val Lys Pro Ser Gln Ser
130 135 140
Leu Ser Leu Thr Cys Thr Val Thr Gly Tyr Ser Ile Thr Ser Asp Phe
145 150 155 160
Ala Trp Asn Trp Ile Arg Gln Phe Pro Gly Asn Lys Leu Glu Trp Met
165 170 175
Gly Tyr Ile Ser Tyr Ser Gly Asn Thr Arg Tyr Asn Pro Ser Leu Lys
180 185 190
Ser Arg Ile Ser Ile Thr Arg Asp Thr Ser Lys Asn Gln Phe Phe Leu
195 200 205
Gln Leu Asn Ser Val Thr Ile Glu Asp Thr Ala Thr Tyr Tyr Cys Val
210 215 220
Thr Ala Gly Arg Gly Phe Pro Tyr Trp Gly Gln Gly Thr Leu Val Thr
225 230 235 240
Val Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln
245 250 255
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
260 265 270
Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Thr Asn Ala Met Ser
275 280 285
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile
290 295 300
Thr Gly Arg Asp Ile Thr Tyr Tyr Ala Ser Trp Ala Lys Gly Arg Phe
305 310 315 320
Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn
325 330 335
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Gly
340 345 350
Gly Ser Ser Ala Ile Thr Ser Asn Asn Ile Trp Gly Gln Gly Thr Leu
355 360 365
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
370 375 380
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
385 390 395 400
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
405 410 415
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
420 425 430
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
435 440 445
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
450 455 460
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
465 470 475 480
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val
485 490 495
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
500 505 510
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
515 520 525
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
530 535 540
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
545 550 555 560
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
565 570 575
Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
580 585 590
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
595 600 605
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
610 615 620
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
625 630 635 640
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
645 650 655
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
660 665 670
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
675 680 685
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly
690 695 700
Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly
705 710 715 720
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala
725 730 735
Ser Gly Phe Ser Phe Ser Ser Gly Tyr Asp Met Cys Trp Val Arg Gln
740 745 750
Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala Cys Ile Ala Ala Gly Ser
755 760 765
Ala Gly Ile Thr Tyr Asp Ala Asn Trp Ala Lys Gly Arg Phe Thr Ile
770 775 780
Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu
785 790 795 800
Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Ser Ala Phe Ser
805 810 815
Phe Asp Tyr Ala Met Asp Leu Trp Gly Gln Gly Thr Leu Val Thr Val
820 825 830
Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
835 840 845
Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Thr
850 855 860
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser
865 870 875 880
Gln Ser Ile Ser Ser His Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys
885 890 895
Ala Pro Lys Leu Leu Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val
900 905 910
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr
915 920 925
Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
930 935 940
Gly Tyr Ser Trp Gly Asn Val Asp Asn Val Phe Gly Gly Gly Thr Lys
945 950 955 960
Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser
965 970 975
Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
980 985 990
Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser Asn Tyr Trp Ile
995 1000 1005
Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala
1010 1015 1020
Cys Ile Tyr Val Gly Ser Ser Gly Asp Thr Tyr Tyr Ala Ser Ser
1025 1030 1035
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
1040 1045 1050
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
1055 1060 1065
Tyr Tyr Cys Ala Arg Asp Ser Ser Ser Tyr Tyr Met Phe Asn Leu
1070 1075 1080
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
1085 1090 1095
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1100 1105 1110
Ser Ala Leu Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser
1115 1120 1125
Val Gly Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Asp Ile
1130 1135 1140
Asp Thr Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
1145 1150 1155
Lys Leu Leu Ile Phe Tyr Ala Ser Asp Leu Ala Ser Gly Val Pro
1160 1165 1170
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr
1175 1180 1185
Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln
1190 1195 1200
Gly Gly Tyr Tyr Thr Ser Ser Ala Asp Thr Arg Gly Ala Phe Gly
1205 1210 1215
Gly Gly Thr Lys Val Glu Ile Lys
1220 1225
<210> 69
<211> 657
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 69
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaacgta cggtggctgc accatctgtc 360
ttcatcttcc cgccatctga tgagcagttg aaatctggaa ctgcctctgt tgtgtgcctg 420
ctgaataact tctatcccag agaggccaaa gtacagtgga aggtggataa cgccctccaa 480
tcgggtaact cccaggagag tgtcacagag caggacagca aggacagcac ctacagcctc 540
agcagcaccc tgacgctgag caaagcagac tacgagaaac acaaagtcta cgcctgcgaa 600
gtcacccatc agggcctgag ctcgcccgtc acaaagagct tcaacagggg agagtgt 657
<210> 70
<211> 219
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 70
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 71
<211> 3681
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 71
gacgttgtga tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcacctgtc aggccagtca gaacattagg acttacttat cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcagccaatc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcga cctggagcct 240
ggcgatgctg caacttacta ttgtcagtct acctatcttg gtactgatta tgttggcggt 300
gctttcggcg gagggaccaa ggtggagatc aaaggcggtg gcggtagtgg gggaggcggt 360
tctggcggcg gagggtccgg cggtggagga tcacggtcgc tggtggagtc tgggggaggc 420
ttggtccagc ctggggggtc cctgagactc tcctgtacag cctctggatt caccatcagt 480
agctaccaca tgcagtgggt ccgccaggct ccagggaagg ggctggagta catcggaacc 540
attagtagtg gtggtaatgt atactacgcg agctccgcga gaggcagatt caccatctcc 600
agaccctcgt ccaagaacac ggtggatctt caaatgaaca gcctgagagc cgaggacacg 660
gctgtgtatt actgtgcgag agactctggt tatagtgatc ctatgtgggg ccagggaacc 720
ctggtcaccg tctcgagcgg cggtggaggg tccggcggtg gtggatccca gtcggtggag 780
gagtctgggg gaggcttggt ccagcctggg gggtccctga gactctcctg tacagcctct 840
ggaatcgacc ttaataccta cgacatgatc tgggtccgcc aggctccagg caaggggcta 900
gagtgggttg gaatcattac ttatagtggt agtagatact acgcgaactg ggcgaaaggc 960
cgattcacca tctccaaaga caataccaag aacacggtgt atctgcaaat gaacagcctg 1020
agagctgagg acacggctgt gtattactgt gccagagatt atatgagtgg ttcccacttg 1080
tggggccagg gaaccctggt caccgtctct agtgctagca ccaagggccc atcggtcttc 1140
cccctggcac cctcctccaa gagcacctct gggggcacag cggccctggg ctgcctggtc 1200
aaggactact tccccgaacc ggtgacggtg tcgtggaact caggcgccct gaccagcggc 1260
gtgcacacct tcccggctgt cctacagtcc tcaggactct actccctcag cagcgtggtg 1320
accgtgccct ccagcagctt gggcacccag acctacatct gcaacgtgaa tcacaagccc 1380
agcaacacca aggtggacaa gagagttgag cccaaatctt gtgacaaaac tcacacatgc 1440
ccaccgtgcc cagcacctga agccgcgggg gcaccgtcag tcttcctctt ccccccaaaa 1500
cccaaggaca ccctcatgat ctcccggacc cctgaggtca catgcgtggt ggtggacgtg 1560
agccacgaag accctgaggt caagttcaac tggtacgtgg acggcgtgga ggtgcataat 1620
gccaagacaa agccgcggga ggagcagtac aacagcacgt accgtgtggt cagcgtcctc 1680
accgtcctgc accaggactg gctgaatggc aaggagtaca agtgcgcggt ctccaacaaa 1740
gccctcccag cccccatcga gaaaaccatc tccaaagcca aagggcagcc ccgagaacca 1800
caggtgtata ccctgccccc atcccgggat gagctgacca agaaccaggt cagcctgacc 1860
tgcctggtca aaggcttcta tcccagcgac atcgccgtgg agtgggagag caatgggcag 1920
ccggagaaca actacaagac cacgcctccc gtgctggact ccgacggctc cttcttcctc 1980
tatagcaagc tcaccgtgga caagagcagg tggcagcagg ggaacgtctt ctcatgctcc 2040
gtgatgcatg aggctctgca caaccactac acgcagaaga gcctctccct gtctccgggt 2100
ggcggtggag ggtccggcgg tggtggatcc gaggtgcagc tgttggagtc tgggggaggc 2160
ttggtacagc ctggggggtc cctgagactc tcctgtgcag cctctggatt caccatcagt 2220
cgctaccaca tgacttgggt ccgccaggct ccagggaagg ggctggagtg gatcggacat 2280
atttatgtta ataatgatga cacagactac gcgagctccg cgaaaggccg gttcaccatc 2340
tccagagaca attccaagaa cacgctgtat ctgcaaatga acagcctgag agccgaggac 2400
acggccacct atttctgtgc gagattggat gttggtggtg gtggtgctta tattggggac 2460
atctggggcc agggaactct ggttaccgtc tcttcaggcg gtggcggtag tgggggaggc 2520
ggttctggcg gcggagggtc cggcggtgga ggatcagaca tccagatgac ccagtctcca 2580
tcctccctgt ctgcatctgt aggagacaga gtcaccatca cttgccagtc cagtcagagt 2640
gtttataaca acaacgactt agcctggtat cagcagaaac cagggaaagt tcctaagctc 2700
ctgatctatt atgcttccac tctggcatct ggggtcccat ctcggttcag tggcagtgga 2760
tctgggacag atttcactct caccatcagc agcctgcagc ctgaagatgt tgcaacttat 2820
tactgtgcag gcggttatga tacggatggt cttgatacgt ttgctttcgg cggagggacc 2880
aaggtggaga tcaaaggcgg tggagggtcc ggcggtggtg gatccgaggt gcagctggtg 2940
gagtctgggg gaggcttggt ccagcctggg gggtccctga gactctcctg tgcagcctct 3000
ggattcacca tcagtaccaa tgcaatgagc tgggtccgcc aggctccagg gaaggggctg 3060
gagtggatcg gagtcattac tggtcgtgat atcacatact acgcgagctg ggcgaaaggc 3120
agattcacca tctccagaga caattccaag aacacgctgt atcttcaaat gaacagcctg 3180
agagccgagg acacggctgt gtattactgt gcgcgcgacg gtggatcatc tgctattact 3240
agtaacaaca tttggggcca aggaactctg gtcaccgttt cttcaggcgg tggcggtagt 3300
gggggaggcg gttctggcgg cggagggtcc ggcggtggag gatcagacgt cgtgatgacc 3360
cagtctcctt ccaccctgtc tgcatctgta ggagacagag tcaccatcaa ttgccaagcc 3420
agtgagagca ttagcagttg gttagcctgg tatcagcaga aaccagggaa agcccctaag 3480
ctcctgatct atgaagcatc caaactggca tctggggtcc catcaaggtt cagcggcagt 3540
ggatctggga cagagttcac tctcaccatc agcagcctgc agcctgatga ttttgcaact 3600
tattactgcc aaggctattt ttattttatt agtcgtactt atgtaaattc tttcggcgga 3660
gggaccaagg tggagatcaa a 3681
<210> 72
<211> 1227
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 72
Asp Val Val Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asn Ile Arg Thr Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ala Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asp Leu Glu Pro
65 70 75 80
Gly Asp Ala Ala Thr Tyr Tyr Cys Gln Ser Thr Tyr Leu Gly Thr Asp
85 90 95
Tyr Val Gly Gly Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly
100 105 110
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Arg Ser Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Ile Ser
145 150 155 160
Ser Tyr His Met Gln Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Tyr Ile Gly Thr Ile Ser Ser Gly Gly Asn Val Tyr Tyr Ala Ser Ser
180 185 190
Ala Arg Gly Arg Phe Thr Ile Ser Arg Pro Ser Ser Lys Asn Thr Val
195 200 205
Asp Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
210 215 220
Cys Ala Arg Asp Ser Gly Tyr Ser Asp Pro Met Trp Gly Gln Gly Thr
225 230 235 240
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
245 250 255
Gln Ser Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
260 265 270
Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Asp Leu Asn Thr Tyr Asp
275 280 285
Met Ile Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
290 295 300
Ile Ile Thr Tyr Ser Gly Ser Arg Tyr Tyr Ala Asn Trp Ala Lys Gly
305 310 315 320
Arg Phe Thr Ile Ser Lys Asp Asn Thr Lys Asn Thr Val Tyr Leu Gln
325 330 335
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
340 345 350
Asp Tyr Met Ser Gly Ser His Leu Trp Gly Gln Gly Thr Leu Val Thr
355 360 365
Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
370 375 380
Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val
385 390 395 400
Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala
405 410 415
Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly
420 425 430
Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly
435 440 445
Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys
450 455 460
Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys
465 470 475 480
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu
485 490 495
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
500 505 510
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
515 520 525
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
530 535 540
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
545 550 555 560
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala
565 570 575
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
580 585 590
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
595 600 605
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
610 615 620
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
625 630 635 640
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
645 650 655
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
660 665 670
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
675 680 685
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly
690 695 700
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly
705 710 715 720
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
725 730 735
Phe Thr Ile Ser Arg Tyr His Met Thr Trp Val Arg Gln Ala Pro Gly
740 745 750
Lys Gly Leu Glu Trp Ile Gly His Ile Tyr Val Asn Asn Asp Asp Thr
755 760 765
Asp Tyr Ala Ser Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
770 775 780
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
785 790 795 800
Thr Ala Thr Tyr Phe Cys Ala Arg Leu Asp Val Gly Gly Gly Gly Ala
805 810 815
Tyr Ile Gly Asp Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
820 825 830
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
835 840 845
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
850 855 860
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser
865 870 875 880
Val Tyr Asn Asn Asn Asp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
885 890 895
Val Pro Lys Leu Leu Ile Tyr Tyr Ala Ser Thr Leu Ala Ser Gly Val
900 905 910
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
915 920 925
Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Ala Gly
930 935 940
Gly Tyr Asp Thr Asp Gly Leu Asp Thr Phe Ala Phe Gly Gly Gly Thr
945 950 955 960
Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
965 970 975
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
980 985 990
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Thr Asn Ala
995 1000 1005
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
1010 1015 1020
Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala Ser Trp Ala
1025 1030 1035
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
1040 1045 1050
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
1055 1060 1065
Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
1070 1075 1080
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
1085 1090 1095
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
1100 1105 1110
Gly Ser Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala
1115 1120 1125
Ser Val Gly Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser
1130 1135 1140
Ile Ser Ser Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala
1145 1150 1155
Pro Lys Leu Leu Ile Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val
1160 1165 1170
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu
1175 1180 1185
Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys
1190 1195 1200
Gln Gly Tyr Phe Tyr Phe Ile Ser Arg Thr Tyr Val Asn Ser Phe
1205 1210 1215
Gly Gly Gly Thr Lys Val Glu Ile Lys
1220 1225
<210> 73
<211> 651
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 73
gcctatgata tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcaagtgtc aggccagtga ggacatttat agcttcttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatccattct gcatcctctc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag ggttatggta aaaataatgt tgataatgct 300
ttcggcggag ggaccaaggt ggagatcaaa cgtacggtgg ctgcaccatc tgtcttcatc 360
ttcccgccat ctgatgagca gttgaaatct ggaactgcct ctgttgtgtg cctgctgaat 420
aacttctatc ccagagaggc caaagtacag tggaaggtgg ataacgccct ccaatcgggt 480
aactcccagg agagtgtcac agagcaggac agcaaggaca gcacctacag cctcagcagc 540
accctgacgc tgagcaaagc agactacgag aaacacaaag tctacgcctg cgaagtcacc 600
catcagggcc tgagctcgcc cgtcacaaag agcttcaaca ggggagagtg t 651
<210> 74
<211> 217
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 74
Ala Tyr Asp Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Lys Cys Gln Ala Ser Glu Asp Ile Tyr Ser Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
His Ser Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Gly Lys Asn Asn
85 90 95
Val Asp Asn Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr
100 105 110
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
115 120 125
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
130 135 140
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
145 150 155 160
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
165 170 175
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
180 185 190
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
195 200 205
Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 75
<211> 3687
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 75
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa tttactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaaggcg gtggcggtag tgggggaggc 360
ggttctggcg gcggagggtc cggcggtgga ggatcagagg tgcagctggt ggagtctggg 420
ggaggcttgg tccagcctgg ggggtccctg agactctcct gtgcagcctc tggattcacc 480
atcagtacca atgcaatgag ctgggtccgc caggctccag ggaaggggct ggagtggatc 540
ggagtcatta ctggtcgtga tatcacatac tacgcgagct gggcgaaagg cagattcacc 600
atctccagag acaattccaa gaacacgctg tatcttcaaa tgaacagcct gagagccgag 660
gacacggctg tgtattactg tgcgagagac ggtggttctt ctgctattac tagtaacaac 720
atttggggcc agggaaccct ggtcaccgtg tcgacaggcg gtggagggtc cggcggtggt 780
ggatcccagt cggtggagga gtctggggga ggcttggtcc agcctggggg gtccctgaga 840
ctctcctgta ccgcctctgg aatcgacctt aatacctacg acatgatctg ggtccgccag 900
gctccaggca aggggctaga gtgggttgga atcattactt atagtggtag tagatactac 960
gcgaactggg cgaaaggccg attcaccatc tccaaagaca ataccaagaa cacggtgtat 1020
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gagagattat 1080
atgagtggtt cccacttgtg gggccaggga accctggtca ccgtctcttc agctagcacc 1140
aagggcccat cggtcttccc cctggcaccc tcctccaaga gcacctctgg gggcacagcg 1200
gccctgggct gcctggtcaa ggactacttc cccgaaccgg tgacggtgtc gtggaactca 1260
ggcgccctga ccagcggcgt gcacaccttc ccggctgtcc tacagtcctc aggactctac 1320
tccctcagca gcgtggtgac cgtgccctcc agcagcttgg gcacccagac ctacatctgc 1380
aacgtgaatc acaagcccag caacaccaag gtggacaaga gagttgagcc caaatcttgt 1440
gacaaaactc acacatgccc accgtgccca gcacctgaag ccgcgggggc accgtcagtc 1500
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 1560
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 1620
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 1680
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 1740
tgcgcggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 1800
gggcagcccc gagaaccaca ggtgtatacc ctgcccccat cccgggatga gctgaccaag 1860
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 1920
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 1980
gacggctcct tcttcctcta tagcaagctc accgtggaca agagcaggtg gcagcagggg 2040
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 2100
ctctccctgt ctccgggtgg cggtggaggg tccggcggtg gtgggtccgg agaggtgcag 2160
ctgttggagt ctgggggagg cttggtacag cctggggggt ccctgagact ctcctgtgca 2220
gcctctggat tcaccatcag tcgctaccac atgacttggg tccgccaggc tccagggaag 2280
gggctggagt ggatcggaca tatttatgtt aataatgatg acacagacta cgcgagctcc 2340
gcgaaaggcc ggttcaccat ctccagagac aattccaaga acacgctgta tctgcaaatg 2400
aacagcctga gagccgagga cacggccacc tatttctgtg cgagattgga tgttggtggt 2460
ggtggtgctt atattgggga catctggggc cagggaactc tggttaccgt ctcttcaggc 2520
ggtggcggta gtgggggagg cggttctggc ggcggagggt ccggcggtgg aggatcagac 2580
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 2640
acttgccagt ccagtcagag tgtttataac aacaacgact tagcctggta tcagcagaaa 2700
ccagggaaag ttcctaagct cctgatctat tatgcttcca ctctggcatc tggggtccca 2760
tctcggttca gtggcagtgg atctgggaca gatttcactc tcaccatcag cagcctgcag 2820
cctgaagatg ttgcaactta ttactgtgca ggcggttatg atacggatgg tcttgatacg 2880
tttgctttcg gcggagggac caaggtggag atcaaaggcg gtggagggtc cggcggtggt 2940
gggtccggac ggtcgctggt ggagtctggg ggaggcttgg tccagcctgg ggggtccctg 3000
agactctcct gtactgcctc tggattcacc atcagtagct accacatgca gtgggtccgc 3060
caggctccag ggaaggggct ggagtacatc ggaaccatta gtagtggtgg taatgtatac 3120
tacgcaagct ccgctagagg cagattcacc atctccagac cctcgtccaa gaacacggtg 3180
gatcttcaaa tgaacagcct gagagccgag gacacggctg tgtattactg tgcgagagac 3240
tctggttata gtgatcctat gtggggccag ggaaccctgg tcaccgtctc ttcaggcggt 3300
ggcggtagtg ggggaggcgg ttctggcggc ggagggtccg gcggtggagg atcagacgtt 3360
gtgatgaccc agtctccatc ttccgtgtct gcatctgtag gagacagagt caccatcacc 3420
tgtcaggcca gtcagaacat taggacttac ttatcctggt atcagcagaa accagggaaa 3480
gcccctaagc tcctgatcta tgctgcagcc aatctggcat ctggggtccc atcaaggttc 3540
agcggcagtg gatctgggac agatttcact ctcaccatca gcgacctgga gcctggcgat 3600
gctgcaactt actattgtca gtctacctat cttggtactg attatgttgg cggtgctttc 3660
ggcggaggga ccaaggtgga gatcaaa 3687
<210> 76
<211> 1229
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 76
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
130 135 140
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
145 150 155 160
Ile Ser Thr Asn Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
165 170 175
Leu Glu Trp Ile Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala
180 185 190
Ser Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
195 200 205
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
210 215 220
Tyr Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
225 230 235 240
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Thr Gly Gly Gly Gly
245 250 255
Ser Gly Gly Gly Gly Ser Gln Ser Val Glu Glu Ser Gly Gly Gly Leu
260 265 270
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile
275 280 285
Asp Leu Asn Thr Tyr Asp Met Ile Trp Val Arg Gln Ala Pro Gly Lys
290 295 300
Gly Leu Glu Trp Val Gly Ile Ile Thr Tyr Ser Gly Ser Arg Tyr Tyr
305 310 315 320
Ala Asn Trp Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Asn Thr Lys
325 330 335
Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Ala Arg Asp Tyr Met Ser Gly Ser His Leu Trp Gly
355 360 365
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
370 375 380
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
385 390 395 400
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
405 410 415
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
420 425 430
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
435 440 445
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
450 455 460
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
465 470 475 480
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
485 490 495
Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
500 505 510
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
515 520 525
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
530 535 540
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
545 550 555 560
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
565 570 575
Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
580 585 590
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
595 600 605
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
610 615 620
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
625 630 635 640
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
645 650 655
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
660 665 670
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
675 680 685
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
690 695 700
Pro Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Glu Val Gln
705 710 715 720
Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
725 730 735
Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser Arg Tyr His Met Thr
740 745 750
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Gly His Ile
755 760 765
Tyr Val Asn Asn Asp Asp Thr Asp Tyr Ala Ser Ser Ala Lys Gly Arg
770 775 780
Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met
785 790 795 800
Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Phe Cys Ala Arg Leu
805 810 815
Asp Val Gly Gly Gly Gly Ala Tyr Ile Gly Asp Ile Trp Gly Gln Gly
820 825 830
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
835 840 845
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr
850 855 860
Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
865 870 875 880
Thr Cys Gln Ser Ser Gln Ser Val Tyr Asn Asn Asn Asp Leu Ala Trp
885 890 895
Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Tyr Ala
900 905 910
Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
915 920 925
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val
930 935 940
Ala Thr Tyr Tyr Cys Ala Gly Gly Tyr Asp Thr Asp Gly Leu Asp Thr
945 950 955 960
Phe Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly
965 970 975
Ser Gly Gly Gly Gly Ser Gly Arg Ser Leu Val Glu Ser Gly Gly Gly
980 985 990
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly
995 1000 1005
Phe Thr Ile Ser Ser Tyr His Met Gln Trp Val Arg Gln Ala Pro
1010 1015 1020
Gly Lys Gly Leu Glu Tyr Ile Gly Thr Ile Ser Ser Gly Gly Asn
1025 1030 1035
Val Tyr Tyr Ala Ser Ser Ala Arg Gly Arg Phe Thr Ile Ser Arg
1040 1045 1050
Pro Ser Ser Lys Asn Thr Val Asp Leu Gln Met Asn Ser Leu Arg
1055 1060 1065
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Ser Gly Tyr
1070 1075 1080
Ser Asp Pro Met Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1085 1090 1095
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1100 1105 1110
Gly Gly Gly Gly Ser Asp Val Val Met Thr Gln Ser Pro Ser Ser
1115 1120 1125
Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Gln Ala
1130 1135 1140
Ser Gln Asn Ile Arg Thr Tyr Leu Ser Trp Tyr Gln Gln Lys Pro
1145 1150 1155
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ala Asn Leu Ala
1160 1165 1170
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
1175 1180 1185
Phe Thr Leu Thr Ile Ser Asp Leu Glu Pro Gly Asp Ala Ala Thr
1190 1195 1200
Tyr Tyr Cys Gln Ser Thr Tyr Leu Gly Thr Asp Tyr Val Gly Gly
1205 1210 1215
Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1220 1225
<210> 77
<211> 651
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 77
gcctatgata tgacccagtc tccatcttcc gtgtctgcat ctgtaggaga cagagtcacc 60
atcaagtgtc aggccagtga ggacatttat agcttcttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatccattct gcatcctctc tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttacta ttgtcaacag ggttatggta aaaataatgt tgataatgct 300
ttcggcggag ggaccaaggt ggagatcaaa cgtacggtgg ctgcaccatc tgtcttcatc 360
ttcccgccat ctgatgagca gttgaaatct ggaactgcct ctgttgtgtg cctgctgaat 420
aacttctatc ccagagaggc caaagtacag tggaaggtgg ataacgccct ccaatcgggt 480
aactcccagg agagtgtcac agagcaggac agcaaggaca gcacctacag cctcagcagc 540
accctgacgc tgagcaaagc agactacgag aaacacaaag tctacgcctg cgaagtcacc 600
catcagggcc tgagctcgcc cgtcacaaag agcttcaaca ggggagagtg t 651
<210> 78
<211> 217
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 78
Ala Tyr Asp Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Lys Cys Gln Ala Ser Glu Asp Ile Tyr Ser Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
His Ser Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Gly Lys Asn Asn
85 90 95
Val Asp Asn Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr
100 105 110
Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu
115 120 125
Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro
130 135 140
Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly
145 150 155 160
Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr
165 170 175
Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His
180 185 190
Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val
195 200 205
Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 79
<211> 3675
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 79
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa tttactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaaggcg gtggcggtag tgggggaggc 360
ggttctggcg gcggagggtc cggcggtgga ggatcagagg tgcagctggt ggagtctggg 420
ggaggcttgg tccagcctgg ggggtccctg agactctcct gtgcagcctc tggattcacc 480
atcagtacca atgcaatgag ctgggtccgc caggctccag ggaaggggct ggagtggatc 540
ggagtcatta ctggtcgtga tatcacatac tacgcgagct gggcgaaagg cagattcacc 600
atctccagag acaattccaa gaacacgctg tatcttcaaa tgaacagcct gagagccgag 660
gacacggctg tgtattactg tgcgagagac ggtggttctt ctgctattac tagtaacaac 720
atttggggcc agggaaccct ggtcaccgtg tcgacaggcg gtggagggtc cggcggtggt 780
ggatccgagg tgcagctgtt ggagtctggg ggaggcttgg tacagcctgg ggggtccctg 840
agactctcct gtgcagcctc tggattcacc atcagtcgct accacatgac ttgggtccgc 900
caggctccag ggaaggggct ggagtggatc ggacatattt atgttaataa tgatgacaca 960
gactacgcga gctccgcgaa aggccggttc accatctcca gagacaattc caagaacacg 1020
ctgtatctgc aaatgaacag cctgagagcc gaggacacgg ccacctattt ctgtgcgaga 1080
ttggatgttg gtggtggtgg tgcttatatt ggggacatct ggggccaggg aaccctggtc 1140
accgtctcga gcgctagcac caagggccca tcggtcttcc ccctggcacc ctcctccaag 1200
agcacctctg ggggcacagc ggccctgggc tgcctggtca aggactactt ccccgaaccg 1260
gtgacggtgt cgtggaactc aggcgccctg accagcggcg tgcacacctt cccggctgtc 1320
ctacagtcct caggactcta ctccctcagc agcgtggtga ccgtgccctc cagcagcttg 1380
ggcacccaga cctacatctg caacgtgaat cacaagccca gcaacaccaa ggtggacaag 1440
agagttgagc ccaaatcttg tgacaaaact cacacatgcc caccgtgccc agcacctgaa 1500
gccgcggggg caccgtcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 1560
tcccggaccc ctgaggtcac atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc 1620
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccgcgggag 1680
gagcagtaca acagcacgta ccgtgtggtc agcgtcctca ccgtcctgca ccaggactgg 1740
ctgaatggca aggagtacaa gtgcgcggtc tccaacaaag ccctcccagc ccccatcgag 1800
aaaaccatct ccaaagccaa agggcagccc cgagaaccac aggtgtatac cctgccccca 1860
tcccgggatg agctgaccaa gaaccaggtc agcctgacct gcctggtcaa aggcttctat 1920
cccagcgaca tcgccgtgga gtgggagagc aatgggcagc cggagaacaa ctacaagacc 1980
acgcctcccg tgctggactc cgacggctcc ttcttcctct atagcaagct caccgtggac 2040
aagagcaggt ggcagcaggg gaacgtcttc tcatgctccg tgatgcatga ggctctgcac 2100
aaccactaca cgcagaagag cctctccctg tctccgggtg gcggtggagg gtccggcggt 2160
ggtggatccc agtcggtgga ggagtctggg ggaggcttgg tccagcctgg ggggtccctg 2220
agactctcct gtaccgcctc tggaatcgac cttaatacct acgacatgat ctgggtccgc 2280
caggctccag gcaaggggct agagtgggtt ggaatcatta cttatagtgg tagtagatac 2340
tacgcgaact gggcgaaagg ccgattcacc atctccaaag acaataccaa gaacacggtg 2400
tatctgcaaa tgaacagcct gagagctgag gacacggctg tgtattactg tgcgagagat 2460
tatatgagtg gttcccactt gtggggccag ggaaccctgg tcaccgtctc ttccggtgga 2520
ggcggttcag gcggaggtgg aagtggtggt ggcggctctg gaggcggcgg atctgcctat 2580
gatatgaccc agtctccatc ttccgtgtct gcatctgtag gagacagagt caccatcaag 2640
tgtcaggcca gtgaggacat ttatagcttc ttggcctggt atcagcagaa accagggaaa 2700
gcccctaagc tcctgatcca ttctgcatcc tctctggcat ctggggtccc atcaaggttc 2760
agcggcagtg gatctgggac agatttcact ctcaccatca gcagcctgca gcctgaagat 2820
tttgcaactt actattgtca acagggttat ggtaaaaata atgttgataa tgctttcggc 2880
ggagggacca aggtggagat caaaggcggt ggagggtccg gcggtggtgg gtccggacgg 2940
tcgctggtgg agtctggggg aggcttggtc cagcctgggg ggtccctgag actctcctgt 3000
actgcctctg gattcaccat cagtagctac cacatgcagt gggtccgcca ggctccaggg 3060
aaggggctgg agtacatcgg aaccattagt agtggtggta atgtatacta cgcaagctcc 3120
gctagaggca gattcaccat ctccagaccc tcgtccaaga acacggtgga tcttcaaatg 3180
aacagcctga gagccgagga cacggctgtg tattactgtg cgagagactc tggttatagt 3240
gatcctatgt ggggccaggg aaccctggtc accgtctctt caggcggtgg cggtagtggg 3300
ggaggcggtt ctggcggcgg agggtccggc ggtggaggat cagacgttgt gatgacccag 3360
tctccatctt ccgtgtctgc atctgtagga gacagagtca ccatcacctg tcaggccagt 3420
cagaacatta ggacttactt atcctggtat cagcagaaac cagggaaagc ccctaagctc 3480
ctgatctatg ctgcagccaa tctggcatct ggggtcccat caaggttcag cggcagtgga 3540
tctgggacag atttcactct caccatcagc gacctggagc ctggcgatgc tgcaacttac 3600
tattgtcagt ctacctatct tggtactgat tatgttggcg gtgctttcgg cggagggacc 3660
aaggtggaga tcaaa 3675
<210> 80
<211> 1225
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 80
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
130 135 140
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
145 150 155 160
Ile Ser Thr Asn Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
165 170 175
Leu Glu Trp Ile Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala
180 185 190
Ser Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
195 200 205
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
210 215 220
Tyr Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
225 230 235 240
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Thr Gly Gly Gly Gly
245 250 255
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly
260 265 270
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
275 280 285
Phe Thr Ile Ser Arg Tyr His Met Thr Trp Val Arg Gln Ala Pro Gly
290 295 300
Lys Gly Leu Glu Trp Ile Gly His Ile Tyr Val Asn Asn Asp Asp Thr
305 310 315 320
Asp Tyr Ala Ser Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
325 330 335
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
340 345 350
Thr Ala Thr Tyr Phe Cys Ala Arg Leu Asp Val Gly Gly Gly Gly Ala
355 360 365
Tyr Ile Gly Asp Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
370 375 380
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
385 390 395 400
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
405 410 415
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
420 425 430
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
435 440 445
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
450 455 460
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
465 470 475 480
Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
485 490 495
Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro
500 505 510
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
515 520 525
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
530 535 540
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
545 550 555 560
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
565 570 575
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn
580 585 590
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
595 600 605
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
610 615 620
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
625 630 635 640
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
645 650 655
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
660 665 670
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
675 680 685
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
690 695 700
Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser Gly Gly
705 710 715 720
Gly Gly Ser Gln Ser Val Glu Glu Ser Gly Gly Gly Leu Val Gln Pro
725 730 735
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Ile Asp Leu Asn
740 745 750
Thr Tyr Asp Met Ile Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
755 760 765
Trp Val Gly Ile Ile Thr Tyr Ser Gly Ser Arg Tyr Tyr Ala Asn Trp
770 775 780
Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Asn Thr Lys Asn Thr Val
785 790 795 800
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
805 810 815
Cys Ala Arg Asp Tyr Met Ser Gly Ser His Leu Trp Gly Gln Gly Thr
820 825 830
Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
835 840 845
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Tyr Asp Met Thr Gln
850 855 860
Ser Pro Ser Ser Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Lys
865 870 875 880
Cys Gln Ala Ser Glu Asp Ile Tyr Ser Phe Leu Ala Trp Tyr Gln Gln
885 890 895
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile His Ser Ala Ser Ser Leu
900 905 910
Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
915 920 925
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
930 935 940
Tyr Cys Gln Gln Gly Tyr Gly Lys Asn Asn Val Asp Asn Ala Phe Gly
945 950 955 960
Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
965 970 975
Gly Ser Gly Arg Ser Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
980 985 990
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Ile Ser
995 1000 1005
Ser Tyr His Met Gln Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
1010 1015 1020
Glu Tyr Ile Gly Thr Ile Ser Ser Gly Gly Asn Val Tyr Tyr Ala
1025 1030 1035
Ser Ser Ala Arg Gly Arg Phe Thr Ile Ser Arg Pro Ser Ser Lys
1040 1045 1050
Asn Thr Val Asp Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
1055 1060 1065
Ala Val Tyr Tyr Cys Ala Arg Asp Ser Gly Tyr Ser Asp Pro Met
1070 1075 1080
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
1085 1090 1095
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1100 1105 1110
Ser Asp Val Val Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser
1115 1120 1125
Val Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asn Ile
1130 1135 1140
Arg Thr Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
1145 1150 1155
Lys Leu Leu Ile Tyr Ala Ala Ala Asn Leu Ala Ser Gly Val Pro
1160 1165 1170
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
1175 1180 1185
Ile Ser Asp Leu Glu Pro Gly Asp Ala Ala Thr Tyr Tyr Cys Gln
1190 1195 1200
Ser Thr Tyr Leu Gly Thr Asp Tyr Val Gly Gly Ala Phe Gly Gly
1205 1210 1215
Gly Thr Lys Val Glu Ile Lys
1220 1225
<210> 81
<211> 660
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 81
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc agtccagtca gagtgtttat aacaacaacg acttagcctg gtatcagcag 120
aaaccaggga aagttcctaa gctcctgatc tattatgcat ccactctggc atctggggtc 180
ccatctcggt tcagtggcag tggatctggg acagatttca ctctcaccat cagcagcctg 240
cagcctgaag atgttgcaac ttattactgt gcaggcggtt atgatacgga tggtcttgat 300
acgtttgctt tcggcggagg gaccaaggtg gagatcaaac gtacggtggc tgcaccatct 360
gtcttcatct tcccgccatc tgatgagcag ttgaaatctg gaactgcctc tgttgtgtgc 420
ctgctgaata acttctatcc cagagaggcc aaagtacagt ggaaggtgga taacgccctc 480
caatcgggta actcccagga gagtgtcaca gagcaggaca gcaaggacag cacctacagc 540
ctcagcagca ccctgacgct gagcaaagca gactacgaga aacacaaagt ctacgcctgc 600
gaagtcaccc atcagggcct gagctcgccc gtcacaaaga gcttcaacag gggagagtgt 660
<210> 82
<211> 220
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 82
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser Val Tyr Asn Asn
20 25 30
Asn Asp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu
35 40 45
Leu Ile Tyr Tyr Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe
50 55 60
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
65 70 75 80
Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Ala Gly Gly Tyr Asp Thr
85 90 95
Asp Gly Leu Asp Thr Phe Ala Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 83
<211> 3696
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 83
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa tttactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaaggcg gtggcggtag tgggggaggc 360
ggttctggcg gcggagggtc cggcggtgga ggatcagagg tgcagctggt ggagtctggg 420
ggaggcttgg tccagcctgg ggggtccctg agactctcct gtgcagcctc tggattcacc 480
atcagtacca atgcaatgag ctgggtccgc caggctccag ggaaggggct ggagtggatc 540
ggagtcatta ctggtcgtga tatcacatac tacgcgagct gggcgaaagg cagattcacc 600
atctccagag acaattccaa gaacacgctg tatcttcaaa tgaacagcct gagagccgag 660
gacacggctg tgtattactg tgcgagagac ggtggttctt ctgctattac tagtaacaac 720
atttggggcc agggaaccct ggtcaccgtg tcgacaggcg gtggagggtc cggcggtggt 780
ggatcagagg tgcagctgtt ggagtctggg ggaggcttgg tacagcctgg ggggtccctg 840
agactctcct gtgcagcctc tggattcacc atcagtcgct accacatgac ttgggtccgc 900
caggctccag ggaaggggct ggagtggatc ggacatattt atgttaataa tgatgacaca 960
gactacgcga gctccgcgaa aggccggttc accatctcca gagacaattc caagaacacg 1020
ctgtatctgc aaatgaacag cctgagagcc gaggacacgg ccacctattt ctgtgcgaga 1080
ttggatgttg gtggtggtgg tgcttatatt ggggacatct ggggccaggg aactctggtt 1140
accgtctctt cagctagcac caagggccca tcggtcttcc ccctggcacc ctcctccaag 1200
agcacctctg ggggcacagc ggccctgggc tgcctggtca aggactactt ccccgaaccg 1260
gtgacggtgt cgtggaactc aggcgccctg accagcggcg tgcacacctt cccggctgtc 1320
ctacagtcct caggactcta ctccctcagc agcgtggtga ccgtgccctc cagcagcttg 1380
ggcacccaga cctacatctg caacgtgaat cacaagccca gcaacaccaa ggtggacaag 1440
agagttgagc ccaaatcttg tgacaaaact cacacatgcc caccgtgccc agcacctgaa 1500
gccgcggggg caccgtcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 1560
tcccggaccc ctgaggtcac atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc 1620
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccgcgggag 1680
gagcagtaca acagcacgta ccgtgtggtc agcgtcctca ccgtcctgca ccaggactgg 1740
ctgaatggca aggagtacaa gtgcgcggtc tccaacaaag ccctcccagc ccccatcgag 1800
aaaaccatct ccaaagccaa agggcagccc cgagaaccac aggtgtacac cctgccccca 1860
tcccgggatg agctgaccaa gaaccaggtc agcctgacct gcctggtcaa aggcttctat 1920
cccagcgaca tcgccgtgga gtgggagagc aatgggcagc cggagaacaa ctacaagacc 1980
acgcctcccg tgctggactc cgacggctcc ttcttcctct atagcaagct caccgtggac 2040
aagagcaggt ggcagcaggg gaacgtcttc tcatgctccg tgatgcatga ggctctgcac 2100
aaccactaca cgcagaagag cctctccctg tctccgggtg gcggtggagg gtccggcggt 2160
ggtggatccg aggtgcagct gttggagtct gggggaggct tggtacagcc tggggggtcc 2220
ctgagactct cctgtgcagc ctctggattc tccttcagta gcgggtacga catgtgctgg 2280
gtccgccagg ctccagggaa ggggctggag tggatcgcat gcattgctgc tggtagtgct 2340
ggtatcactt acgacgcgaa ctgggcgaaa ggccggttca ccatctccag agacaattcc 2400
aagaacacgc tgtatctgca aatgaacagc ctgagagccg aggacacggc cgtatattac 2460
tgtgcgagat cggcgttttc gttcgactac gccatggacc tctggggcca gggaaccctg 2520
gtcaccgtct cgagcggtgg aggcggatct ggcggaggtg gttccggcgg tggcggctcc 2580
ggtggaggcg gctctgacat ccagatgacc cagtctcctt ccaccctgtc tgcatctgta 2640
ggagacagag tcaccatcac ttgccaggcc agtcagagca ttagttccca cttaaactgg 2700
tatcagcaga aaccagggaa agcccctaag ctcctgatct ataaggcatc cactctggca 2760
tctggggtcc catcaaggtt cagcggcagt ggatctggga cagaatttac tctcaccatc 2820
agcagcctgc agcctgatga ttttgcaact tattactgcc aacagggtta tagttggggt 2880
aatgttgata atgttttcgg cggagggacc aaggtggaga tcaaaggcgg tggagggtcc 2940
ggcggtggtg gctccggacg gtcgctggtg gagtctgggg gaggcttggt ccagcctggg 3000
gggtccctga gactctcctg tactgcctct ggattcacca tcagtagcta ccacatgcag 3060
tgggtccgcc aggctccagg gaaggggctg gagtacatcg gaaccattag tagtggtggt 3120
aatgtatact acgcaagctc cgctagaggc agattcacca tctccagacc ctcgtccaag 3180
aacacggtgg atcttcaaat gaacagcctg agagccgagg acacggctgt gtattactgt 3240
gcgagagact ctggttatag tgatcctatg tggggccagg gaaccctggt caccgtctct 3300
tcaggcggtg gcggtagtgg gggaggcggt tctggcggcg gagggtccgg cggtggagga 3360
tcagacgttg tgatgaccca gtctccatct tccgtgtctg catctgtagg agacagagtc 3420
accatcacct gtcaggccag tcagaacatt aggacttact tatcctggta tcagcagaaa 3480
ccagggaaag cccctaagct cctgatctat gctgcagcca atctggcatc tggggtccca 3540
tcaaggttca gcggcagtgg atctgggaca gatttcactc tcaccatcag cgacctggag 3600
cctggcgatg ctgcaactta ctattgtcag tctacctatc ttggtactga ttatgttggc 3660
ggtgctttcg gcggagggac caaggtggag atcaaa 3696
<210> 84
<211> 1232
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 84
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
130 135 140
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
145 150 155 160
Ile Ser Thr Asn Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
165 170 175
Leu Glu Trp Ile Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala
180 185 190
Ser Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
195 200 205
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
210 215 220
Tyr Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
225 230 235 240
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Thr Gly Gly Gly Gly
245 250 255
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly
260 265 270
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
275 280 285
Phe Thr Ile Ser Arg Tyr His Met Thr Trp Val Arg Gln Ala Pro Gly
290 295 300
Lys Gly Leu Glu Trp Ile Gly His Ile Tyr Val Asn Asn Asp Asp Thr
305 310 315 320
Asp Tyr Ala Ser Ser Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
325 330 335
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
340 345 350
Thr Ala Thr Tyr Phe Cys Ala Arg Leu Asp Val Gly Gly Gly Gly Ala
355 360 365
Tyr Ile Gly Asp Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
370 375 380
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
385 390 395 400
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
405 410 415
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
420 425 430
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
435 440 445
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
450 455 460
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
465 470 475 480
Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
485 490 495
Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro
500 505 510
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
515 520 525
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
530 535 540
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
545 550 555 560
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
565 570 575
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn
580 585 590
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
595 600 605
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
610 615 620
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
625 630 635 640
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
645 650 655
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
660 665 670
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
675 680 685
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
690 695 700
Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser Gly Gly
705 710 715 720
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
725 730 735
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe
740 745 750
Ser Ser Gly Tyr Asp Met Cys Trp Val Arg Gln Ala Pro Gly Lys Gly
755 760 765
Leu Glu Trp Ile Ala Cys Ile Ala Ala Gly Ser Ala Gly Ile Thr Tyr
770 775 780
Asp Ala Asn Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
785 790 795 800
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
805 810 815
Ala Val Tyr Tyr Cys Ala Arg Ser Ala Phe Ser Phe Asp Tyr Ala Met
820 825 830
Asp Leu Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
835 840 845
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
850 855 860
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
865 870 875 880
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
885 890 895
His Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
900 905 910
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
915 920 925
Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
930 935 940
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Ser Trp Gly
945 950 955 960
Asn Val Asp Asn Val Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly
965 970 975
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Arg Ser Leu Val Glu Ser
980 985 990
Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Thr
995 1000 1005
Ala Ser Gly Phe Thr Ile Ser Ser Tyr His Met Gln Trp Val Arg
1010 1015 1020
Gln Ala Pro Gly Lys Gly Leu Glu Tyr Ile Gly Thr Ile Ser Ser
1025 1030 1035
Gly Gly Asn Val Tyr Tyr Ala Ser Ser Ala Arg Gly Arg Phe Thr
1040 1045 1050
Ile Ser Arg Pro Ser Ser Lys Asn Thr Val Asp Leu Gln Met Asn
1055 1060 1065
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp
1070 1075 1080
Ser Gly Tyr Ser Asp Pro Met Trp Gly Gln Gly Thr Leu Val Thr
1085 1090 1095
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
1100 1105 1110
Gly Gly Ser Gly Gly Gly Gly Ser Asp Val Val Met Thr Gln Ser
1115 1120 1125
Pro Ser Ser Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
1130 1135 1140
Cys Gln Ala Ser Gln Asn Ile Arg Thr Tyr Leu Ser Trp Tyr Gln
1145 1150 1155
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ala
1160 1165 1170
Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
1175 1180 1185
Gly Thr Asp Phe Thr Leu Thr Ile Ser Asp Leu Glu Pro Gly Asp
1190 1195 1200
Ala Ala Thr Tyr Tyr Cys Gln Ser Thr Tyr Leu Gly Thr Asp Tyr
1205 1210 1215
Val Gly Gly Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1220 1225 1230
<210> 85
<211> 660
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 85
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc agtccagtca gagtgtttat aacaacaacg acttagcctg gtatcagcag 120
aaaccaggga aagttcctaa gctcctgatc tattatgcat ccactctggc atctggggtc 180
ccatctcggt tcagtggcag tggatctggg acagatttca ctctcaccat cagcagcctg 240
cagcctgaag atgttgcaac ttattactgt gcaggcggtt atgatacgga tggtcttgat 300
acgtttgctt tcggcggagg gaccaaggtg gagatcaaac gtacggtggc tgcaccatct 360
gtcttcatct tcccgccatc tgatgagcag ttgaaatctg gaactgcctc tgttgtgtgc 420
ctgctgaata acttctatcc cagagaggcc aaagtacagt ggaaggtgga taacgccctc 480
caatcgggta actcccagga gagtgtcaca gagcaggaca gcaaggacag cacctacagc 540
ctcagcagca ccctgacgct gagcaaagca gactacgaga aacacaaagt ctacgcctgc 600
gaagtcaccc atcagggcct gagctcgccc gtcacaaaga gcttcaacag gggagagtgt 660
<210> 86
<211> 220
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 86
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ser Ser Gln Ser Val Tyr Asn Asn
20 25 30
Asn Asp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu
35 40 45
Leu Ile Tyr Tyr Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe
50 55 60
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
65 70 75 80
Gln Pro Glu Asp Val Ala Thr Tyr Tyr Cys Ala Gly Gly Tyr Asp Thr
85 90 95
Asp Gly Leu Asp Thr Phe Ala Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 87
<211> 3690
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 87
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagag ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaaggcg gtggcggtag tgggggaggc 360
ggttctggcg gcggagggtc cggcggtgga ggatcagagg tgcagctggt ggagtctggg 420
ggaggcttgg tccagcctgg ggggtccctg agactctcct gtgcagcctc tggattcacc 480
atcagtacca atgcaatgag ctgggtccgc caggctccag ggaaggggct ggagtggatc 540
ggagtcatta ctggtcgtga tatcacatac tacgcgagct gggcgaaagg cagattcacc 600
atctccagag acaattccaa gaacacgctg tatcttcaaa tgaacagcct gagagccgag 660
gacacggctg tgtattactg tgcgcgcgac ggtggatcat ctgctattac tagtaacaac 720
atttggggcc aaggaactct ggtcaccgtt tcttcaggcg gtggagggtc cggcggtggt 780
ggatccgagg tgcagctggt gcagtctgga gcagaggtga agaaaccagg agagtctctg 840
aagatctcct gtaagggttc tggatacagc tttagcagtt catggatcgg ctgggtgcgc 900
caggcacctg ggaaaggcct ggaatggatg gggatcatct atcctgatga ctctgatacc 960
agatacagtc catccttcca aggccaggtc accatctcag ccgacaagtc catcaggact 1020
gcctacctgc agtggagtag cctgaaggcc tcggacaccg ctatgtatta ctgtgcgaga 1080
catgttacta tgatttgggg agttattatt gacttctggg gccagggaac cctggtcacc 1140
gtctcctcag ctagcaccaa gggcccatcg gtcttccccc tggcaccctc ctccaagagc 1200
acctctgggg gcacagcggc cctgggctgc ctggtcaagg actacttccc cgaaccggtg 1260
acggtgtcgt ggaactcagg cgccctgacc agcggcgtgc acaccttccc ggctgtccta 1320
cagtcctcag gactctactc cctcagcagc gtggtgaccg tgccctccag cagcttgggc 1380
acccagacct acatctgcaa cgtgaatcac aagcccagca acaccaaggt ggacaagaga 1440
gttgagccca aatcttgtga caaaactcac acatgcccac cgtgcccagc acctgaagcc 1500
gcgggggcac cgtcagtctt cctcttcccc ccaaaaccca aggacaccct catgatctcc 1560
cggacccctg aggtcacatg cgtggtggtg gacgtgagcc acgaagaccc tgaggtcaag 1620
ttcaactggt acgtggacgg cgtggaggtg cataatgcca agacaaagcc gcgggaggag 1680
cagtacaaca gcacgtaccg tgtggtcagc gtcctcaccg tcctgcacca ggactggctg 1740
aatggcaagg agtacaagtg cgcggtctcc aacaaagccc tcccagcccc catcgagaaa 1800
accatctcca aagccaaagg gcagccccga gaaccacagg tgtataccct gcccccatcc 1860
cgggatgagc tgaccaagaa ccaggtcagc ctgacctgcc tggtcaaagg cttctatccc 1920
agcgacatcg ccgtggagtg ggagagcaat gggcagccgg agaacaacta caagaccacg 1980
cctcccgtgc tggactccga cggctccttc ttcctctata gcaagctcac cgtggacaag 2040
agcaggtggc agcaggggaa cgtcttctca tgctccgtga tgcatgaggc tctgcacaac 2100
cactacacgc agaagagcct ctccctgtct ccgggtggcg gtggagggtc cggcggtggt 2160
ggatccgagg tgcagctgtt ggagtctggg ggaggcttgg tacagcctgg ggggtccctg 2220
agactctcct gtgcagcctc tggattctcc ttcagtagcg ggtacgacat gtgctgggtc 2280
cgccaggctc cagggaaggg gctggagtgg atcgcatgca ttgctgctgg tagtgctggt 2340
atcacttacg acgcgaactg ggcgaaaggc cggttcacca tctccagaga caattccaag 2400
aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggccgt atattactgt 2460
gcgagatcgg cgttttcgtt cgactacgcc atggacctct ggggccaggg aaccctggtc 2520
accgtctcga gcggtggagg cggatctggc ggaggtggtt ccggcggtgg cggctccggt 2580
ggaggcggct ctgacatcca gatgacccag tctccttcca ccctgtctgc atctgtagga 2640
gacagagtca ccatcacttg ccaggccagt cagagcatta gttcccactt aaactggtat 2700
cagcagaaac cagggaaagc ccctaagctc ctgatctata aggcatccac tctggcatct 2760
ggggtcccat caaggttcag cggcagtgga tctgggacag aatttactct caccatcagc 2820
agcctgcagc ctgatgattt tgcaacttat tactgccaac agggttatag ttggggtaat 2880
gttgataatg ttttcggcgg agggaccaag gtggagatca aaggcggtgg agggtccggc 2940
ggtggtggat cccggtcgct ggtggagtct gggggaggct tggtccagcc tggggggtcc 3000
ctgagactct cctgtacagc ctctggattc accatcagta gctaccacat gcagtgggtc 3060
cgccaggctc cagggaaggg gctggagtac atcggaacca ttagtagtgg tggtaatgta 3120
tactacgcga gctccgcgag aggcagattc accatctcca gaccctcgtc caagaacacg 3180
gtggatcttc aaatgaacag cctgagagcc gaggacacgg ctgtgtatta ctgtgcgaga 3240
gactctggtt atagtgatcc tatgtggggc cagggaaccc tggtcaccgt ctcgagcggc 3300
ggtggcggta gtgggggagg cggttctggc ggcggagggt ccggcggtgg aggatcagac 3360
gttgtgatga cccagtctcc atcttccgtg tctgcatctg taggagacag agtcaccatc 3420
acctgtcagg ccagtcagaa cattaggact tacttatcct ggtatcagca gaaaccaggg 3480
aaagccccta agctcctgat ctatgctgca gccaatctgg catctggggt cccatcaagg 3540
ttcagcggca gtggatctgg gacagatttc actctcacca tcagcgacct ggagcctggc 3600
gatgctgcaa cttactattg tcagtctacc tatcttggta ctgattatgt tggcggtgct 3660
ttcggcggag ggaccaaggt ggagatcaaa 3690
<210> 88
<211> 1230
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 88
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
130 135 140
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
145 150 155 160
Ile Ser Thr Asn Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
165 170 175
Leu Glu Trp Ile Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala
180 185 190
Ser Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
195 200 205
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
210 215 220
Tyr Tyr Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn
225 230 235 240
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
245 250 255
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Gln Ser Gly Ala Glu
260 265 270
Val Lys Lys Pro Gly Glu Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly
275 280 285
Tyr Ser Phe Ser Ser Ser Trp Ile Gly Trp Val Arg Gln Ala Pro Gly
290 295 300
Lys Gly Leu Glu Trp Met Gly Ile Ile Tyr Pro Asp Asp Ser Asp Thr
305 310 315 320
Arg Tyr Ser Pro Ser Phe Gln Gly Gln Val Thr Ile Ser Ala Asp Lys
325 330 335
Ser Ile Arg Thr Ala Tyr Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp
340 345 350
Thr Ala Met Tyr Tyr Cys Ala Arg His Val Thr Met Ile Trp Gly Val
355 360 365
Ile Ile Asp Phe Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
370 375 380
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
385 390 395 400
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
405 410 415
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
420 425 430
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
435 440 445
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
450 455 460
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg
465 470 475 480
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
485 490 495
Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys
500 505 510
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
515 520 525
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
530 535 540
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
545 550 555 560
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
565 570 575
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys
580 585 590
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
595 600 605
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
610 615 620
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
625 630 635 640
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
645 650 655
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
660 665 670
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
675 680 685
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
690 695 700
Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Gly Ser Gly Gly Gly
705 710 715 720
Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro
725 730 735
Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser
740 745 750
Ser Gly Tyr Asp Met Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
755 760 765
Glu Trp Ile Ala Cys Ile Ala Ala Gly Ser Ala Gly Ile Thr Tyr Asp
770 775 780
Ala Asn Trp Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
785 790 795 800
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
805 810 815
Val Tyr Tyr Cys Ala Arg Ser Ala Phe Ser Phe Asp Tyr Ala Met Asp
820 825 830
Leu Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
835 840 845
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
850 855 860
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
865 870 875 880
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser His
885 890 895
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
900 905 910
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
915 920 925
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
930 935 940
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gly Tyr Ser Trp Gly Asn
945 950 955 960
Val Asp Asn Val Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly
965 970 975
Gly Gly Ser Gly Gly Gly Gly Ser Arg Ser Leu Val Glu Ser Gly Gly
980 985 990
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser
995 1000 1005
Gly Phe Thr Ile Ser Ser Tyr His Met Gln Trp Val Arg Gln Ala
1010 1015 1020
Pro Gly Lys Gly Leu Glu Tyr Ile Gly Thr Ile Ser Ser Gly Gly
1025 1030 1035
Asn Val Tyr Tyr Ala Ser Ser Ala Arg Gly Arg Phe Thr Ile Ser
1040 1045 1050
Arg Pro Ser Ser Lys Asn Thr Val Asp Leu Gln Met Asn Ser Leu
1055 1060 1065
Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Asp Ser Gly
1070 1075 1080
Tyr Ser Asp Pro Met Trp Gly Gln Gly Thr Leu Val Thr Val Ser
1085 1090 1095
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1100 1105 1110
Ser Gly Gly Gly Gly Ser Asp Val Val Met Thr Gln Ser Pro Ser
1115 1120 1125
Ser Val Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Gln
1130 1135 1140
Ala Ser Gln Asn Ile Arg Thr Tyr Leu Ser Trp Tyr Gln Gln Lys
1145 1150 1155
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ala Asn Leu
1160 1165 1170
Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
1175 1180 1185
Asp Phe Thr Leu Thr Ile Ser Asp Leu Glu Pro Gly Asp Ala Ala
1190 1195 1200
Thr Tyr Tyr Cys Gln Ser Thr Tyr Leu Gly Thr Asp Tyr Val Gly
1205 1210 1215
Gly Ala Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1220 1225 1230
<210> 89
<211> 642
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 89
gccatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattagc agtgctttag cctggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag tttaatagtt acccattcac tttcggccct 300
gggaccaaag tggatatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 90
<211> 214
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 90
Ala Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Ala
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 91
<211> 3675
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 91
gccatccagt tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattagc agtgctttag cctggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacag tttaatagtt acccattcac tttcggccct 300
gggaccaaag tggatatcaa aggcggtggc ggtagtgggg gaggcggttc tggcggcgga 360
gggtccggcg gtggaggatc agaggtgcag ctggtgcagt ctggagcaga ggtgaagaaa 420
ccaggagagt ctctgaagat ctcctgtaag ggttctggat acagctttag cagttcatgg 480
atcggctggg tgcgccaggc acctgggaaa ggcctggaat ggatggggat catctatcct 540
gatgactctg ataccagata cagtccatcc ttccaaggcc aggtcaccat ctcagccgac 600
aagtccatca ggactgccta cctgcagtgg agtagcctga aggcctcgga caccgctatg 660
tattactgtg cgagacatgt tactatgatt tggggagtta ttattgactt ctggggccag 720
ggaaccctgg tcaccgtctc ctcaggcggt ggagggtccg gcggtggtgg atccgaggtg 780
cagctggtgg agtctggggg aggcttggtc cagcctgggg ggtccctgag actctcctgt 840
gcagcctctg gattcaccat cagtaccaat gcaatgagct gggtccgcca ggctccaggg 900
aaggggctgg agtggatcgg agtcattact ggtcgtgata tcacatacta cgcgagctgg 960
gcgaaaggca gattcaccat ctccagagac aattccaaga acacgctgta tcttcaaatg 1020
aacagcctga gagccgagga cacggctgtg tattactgtg cgcgcgacgg tggatcatct 1080
gctattacta gtaacaacat ttggggccaa ggaactctgg tcaccgtttc ttcagctagc 1140
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 1200
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 1260
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 1320
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 1380
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agagagttga gcccaaatct 1440
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagccgcggg ggcaccgtca 1500
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 1560
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 1620
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 1680
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 1740
aagtgcgcgg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 1800
aaagggcagc cccgagaacc acaggtgtat accctgcccc catcccggga tgagctgacc 1860
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1920
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1980
tccgacggct ccttcttcct ctatagcaag ctcaccgtgg acaagagcag gtggcagcag 2040
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 2100
agcctctccc tgtctccggg tggcggtgga gggtccggcg gtggtggatc cgaggtgcag 2160
ctgttggagt ctgggggagg cttggtacag cctggggggt ccctgagact ctcctgtgca 2220
gcctctggat tctccttcag tagcgggtac gacatgtgct gggtccgcca ggctccaggg 2280
aaggggctgg agtggatcgc atgcattgct gctggtagtg ctggtatcac ttacgacgcg 2340
aactgggcga aaggccggtt caccatctcc agagacaatt ccaagaacac gctgtatctg 2400
caaatgaaca gcctgagagc cgaggacacg gccgtatatt actgtgcgag atcggcgttt 2460
tcgttcgact acgccatgga cctctggggc cagggaaccc tggtcaccgt ctcgagcggt 2520
ggaggcggat ctggcggagg tggttccggc ggtggcggct ccggtggagg cggctctgac 2580
atccagatga cccagtctcc ttccaccctg tctgcatctg taggagacag agtcaccatc 2640
acttgccagg ccagtcagag cattagttcc cacttaaact ggtatcagca gaaaccaggg 2700
aaagccccta agctcctgat ctataaggca tccactctgg catctggggt cccatcaagg 2760
ttcagcggca gtggatctgg gacagaattt actctcacca tcagcagcct gcagcctgat 2820
gattttgcaa cttattactg ccaacagggt tatagttggg gtaatgttga taatgttttc 2880
ggcggaggga ccaaggtgga gatcaaaggc ggtggagggt ccggcggtgg tggatcccgg 2940
tcgctggtgg agtctggggg aggcttggtc cagcctgggg ggtccctgag actctcctgt 3000
acagcctctg gattcaccat cagtagctac cacatgcagt gggtccgcca ggctccaggg 3060
aaggggctgg agtacatcgg aaccattagt agtggtggta atgtatacta cgcgagctcc 3120
gcgagaggca gattcaccat ctccagaccc tcgtccaaga acacggtgga tcttcaaatg 3180
aacagcctga gagccgagga cacggctgtg tattactgtg cgagagactc tggttatagt 3240
gatcctatgt ggggccaggg aaccctggtc accgtctcga gcggcggtgg cggtagtggg 3300
ggaggcggtt ctggcggcgg agggtccggc ggtggaggat cagacgttgt gatgacccag 3360
tctccatctt ccgtgtctgc atctgtagga gacagagtca ccatcacctg tcaggccagt 3420
cagaacatta ggacttactt atcctggtat cagcagaaac cagggaaagc ccctaagctc 3480
ctgatctatg ctgcagccaa tctggcatct ggggtcccat caaggttcag cggcagtgga 3540
tctgggacag atttcactct caccatcagc gacctggagc ctggcgatgc tgcaacttac 3600
tattgtcagt ctacctatct tggtactgat tatgttggcg gtgctttcgg cggagggacc 3660
aaggtggaga tcaaa 3675
<210> 92
<211> 1225
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 92
Ala Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Ser Ser Ala
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Phe Asn Ser Tyr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
115 120 125
Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu Ser
130 135 140
Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Ser Ser Ser Trp
145 150 155 160
Ile Gly Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met Gly
165 170 175
Ile Ile Tyr Pro Asp Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe Gln
180 185 190
Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Arg Thr Ala Tyr Leu
195 200 205
Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys Ala
210 215 220
Arg His Val Thr Met Ile Trp Gly Val Ile Ile Asp Phe Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ile Ser
275 280 285
Thr Asn Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Ile Gly Val Ile Thr Gly Arg Asp Ile Thr Tyr Tyr Ala Ser Trp
305 310 315 320
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
325 330 335
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
340 345 350
Cys Ala Arg Asp Gly Gly Ser Ser Ala Ile Thr Ser Asn Asn Ile Trp
355 360 365
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
370 375 380
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
385 390 395 400
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
405 410 415
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
420 425 430
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
435 440 445
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
450 455 460
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
465 470 475 480
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
485 490 495
Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
500 505 510
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
515 520 525
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
530 535 540
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
545 550 555 560
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
565 570 575
Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro
580 585 590
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
595 600 605
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
610 615 620
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
625 630 635 640
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
645 650 655
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
660 665 670
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
675 680 685
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
690 695 700
Ser Pro Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln
705 710 715 720
Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg
725 730 735
Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser Gly Tyr Asp Met
740 745 750
Cys Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Ala Cys
755 760 765
Ile Ala Ala Gly Ser Ala Gly Ile Thr Tyr Asp Ala Asn Trp Ala Lys
770 775 780
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
785 790 795 800
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
805 810 815
Arg Ser Ala Phe Ser Phe Asp Tyr Ala Met Asp Leu Trp Gly Gln Gly
820 825 830
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
835 840 845
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr
850 855 860
Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile
865 870 875 880
Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser His Leu Asn Trp Tyr Gln
885 890 895
Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Ala Ser Thr
900 905 910
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
915 920 925
Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr
930 935 940
Tyr Tyr Cys Gln Gln Gly Tyr Ser Trp Gly Asn Val Asp Asn Val Phe
945 950 955 960
Gly Gly Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
965 970 975
Gly Gly Ser Arg Ser Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
980 985 990
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Thr Ile Ser
995 1000 1005
Ser Tyr His Met Gln Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
1010 1015 1020
Glu Tyr Ile Gly Thr Ile Ser Ser Gly Gly Asn Val Tyr Tyr Ala
1025 1030 1035
Ser Ser Ala Arg Gly Arg Phe Thr Ile Ser Arg Pro Ser Ser Lys
1040 1045 1050
Asn Thr Val Asp Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
1055 1060 1065
Ala Val Tyr Tyr Cys Ala Arg Asp Ser Gly Tyr Ser Asp Pro Met
1070 1075 1080
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
1085 1090 1095
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1100 1105 1110
Ser Asp Val Val Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser
1115 1120 1125
Val Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Asn Ile
1130 1135 1140
Arg Thr Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
1145 1150 1155
Lys Leu Leu Ile Tyr Ala Ala Ala Asn Leu Ala Ser Gly Val Pro
1160 1165 1170
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
1175 1180 1185
Ile Ser Asp Leu Glu Pro Gly Asp Ala Ala Thr Tyr Tyr Cys Gln
1190 1195 1200
Ser Thr Tyr Leu Gly Thr Asp Tyr Val Gly Gly Ala Phe Gly Gly
1205 1210 1215
Gly Thr Lys Val Glu Ile Lys
1220 1225
<210> 93
<211> 657
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 93
gacgtcgtga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcaattgcc aagccagtga gagcattagc agttggttag cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgaa gcatccaaac tggcatctgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaaggc tatttttatt ttattagtcg tacttatgta 300
aattctttcg gcggagggac caaggtggag atcaaacgta cggtggctgc accatctgtc 360
ttcatcttcc cgccatctga tgagcagttg aaatctggaa ctgcctctgt tgtgtgcctg 420
ctgaataact tctatcccag agaggccaaa gtacagtgga aggtggataa cgccctccaa 480
tcgggtaact cccaggagag tgtcacagag caggacagca aggacagcac ctacagcctc 540
agcagcaccc tgacgctgag caaagcagac tacgagaaac acaaagtcta cgcctgcgaa 600
gtcacccatc agggcctgag ctcgcccgtc acaaagagct tcaacagggg agagtgt 657
<210> 94
<211> 219
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтезированная
<400> 94
Asp Val Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Gln Ala Ser Glu Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gly Tyr Phe Tyr Phe Ile Ser
85 90 95
Arg Thr Tyr Val Asn Ser Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| БЕЛКИ УПРАВЛЕНИЯ, НАВИГАЦИИ И КОНТРОЛЯ И СПОСОБ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2019 |
|
RU2811457C2 |
| АНТИТЕЛА ПРОТИВ CD19 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ И ПОЛУЧЕНИЯ | 2021 |
|
RU2834996C1 |
| МУЛЬТИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2018 |
|
RU2811477C2 |
| МУЛЬТИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2018 |
|
RU2837770C2 |
| МУЛЬТИВАЛЕНТНЫЕ FV-АНТИТЕЛА | 2016 |
|
RU2785766C2 |
| АГЕНТЫ ДЛЯ ЛЕЧЕНИЯ ЭКСПРЕССИРУЮЩИХ КЛАУДИН РАКОВЫХ ЗАБОЛЕВАНИЙ | 2013 |
|
RU2798990C2 |
| МОНОСПЕЦИФИЧЕСКИЕ И БИСПЕЦИФИЧЕСКИЕ БЕЛКИ С РЕГУЛИРОВАНИЕМ ИММУННОЙ КОНТРОЛЬНОЙ ТОЧКИ ДЛЯ ЛЕЧЕНИЯ РАКА | 2018 |
|
RU2793167C2 |
| ТЕРАПЕВТИЧЕСКИЕ СОЕДИНЕНИЯ И СПОСОБЫ | 2016 |
|
RU2770001C2 |
| НОВОЕ АНТИ-С-МЕТ АНТИТЕЛО И ЕГО ПРИМЕНЕНИЕ | 2018 |
|
RU2751720C2 |
| Биспецифическое антитело анти-HER2/анти-4-1BB и его применение | 2020 |
|
RU2831838C2 |
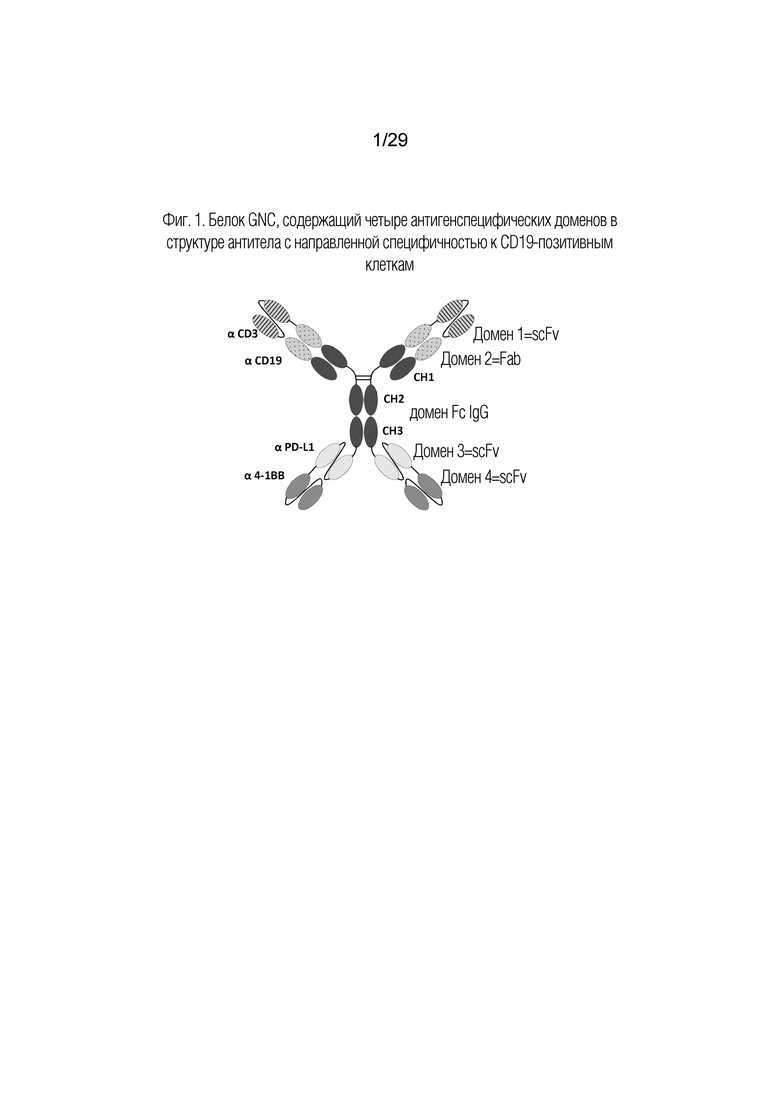
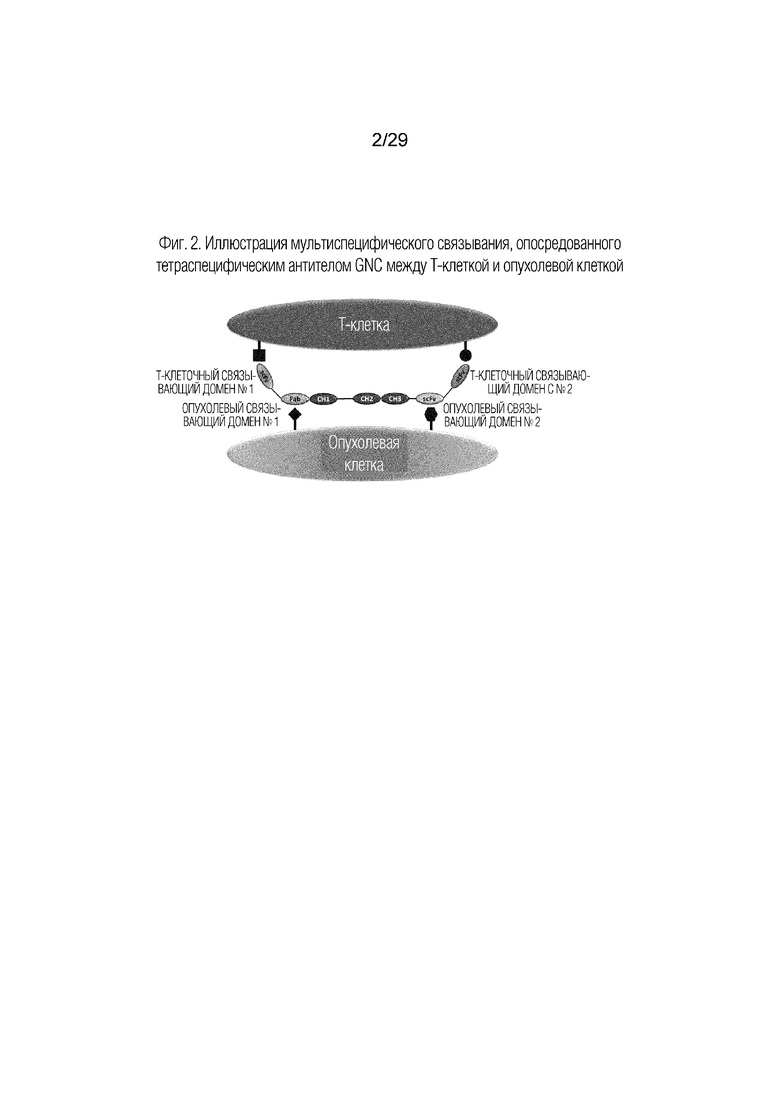
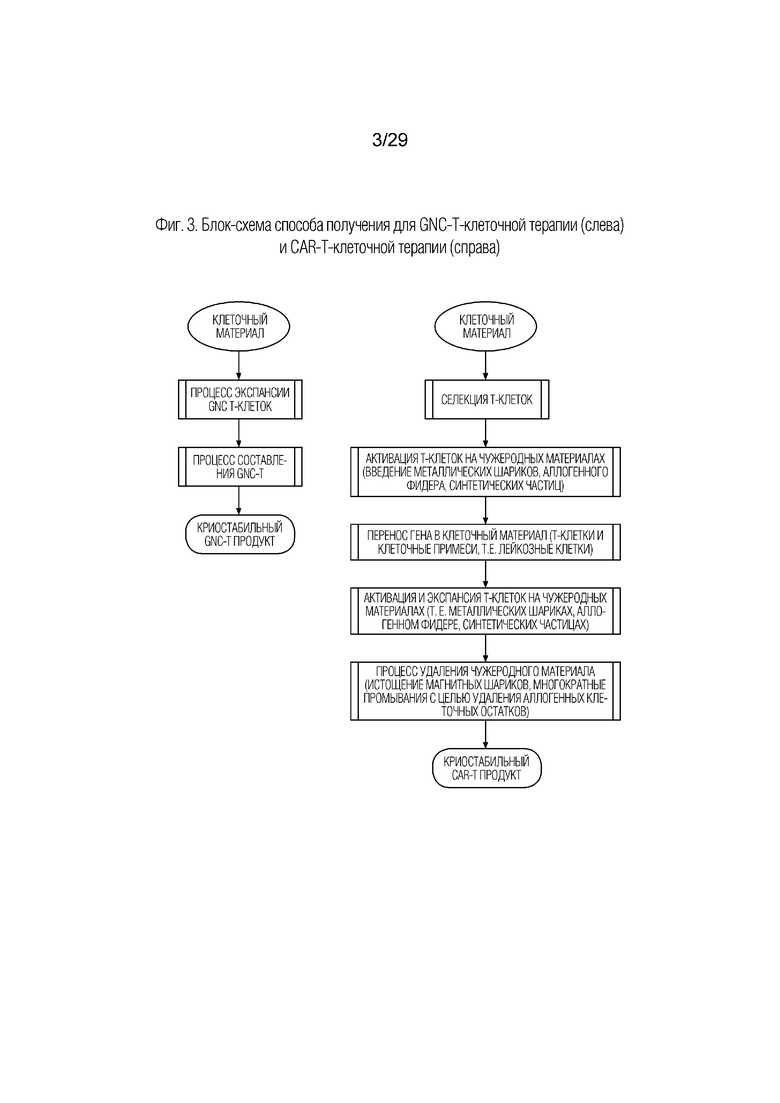
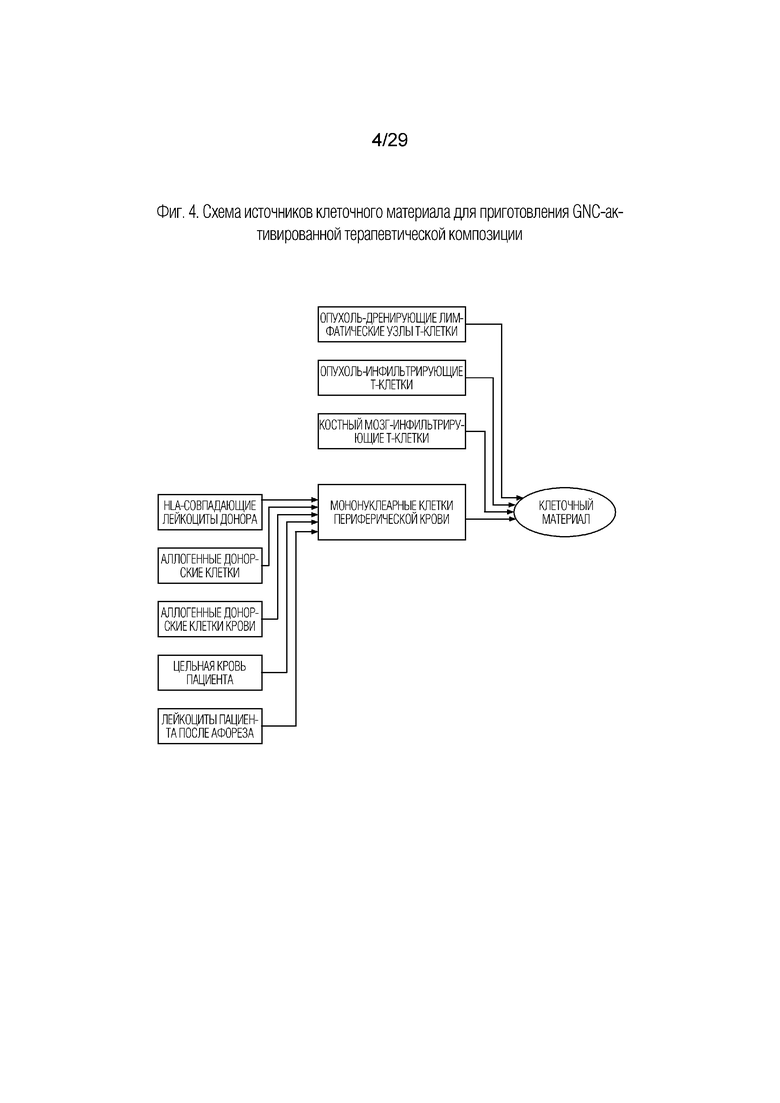
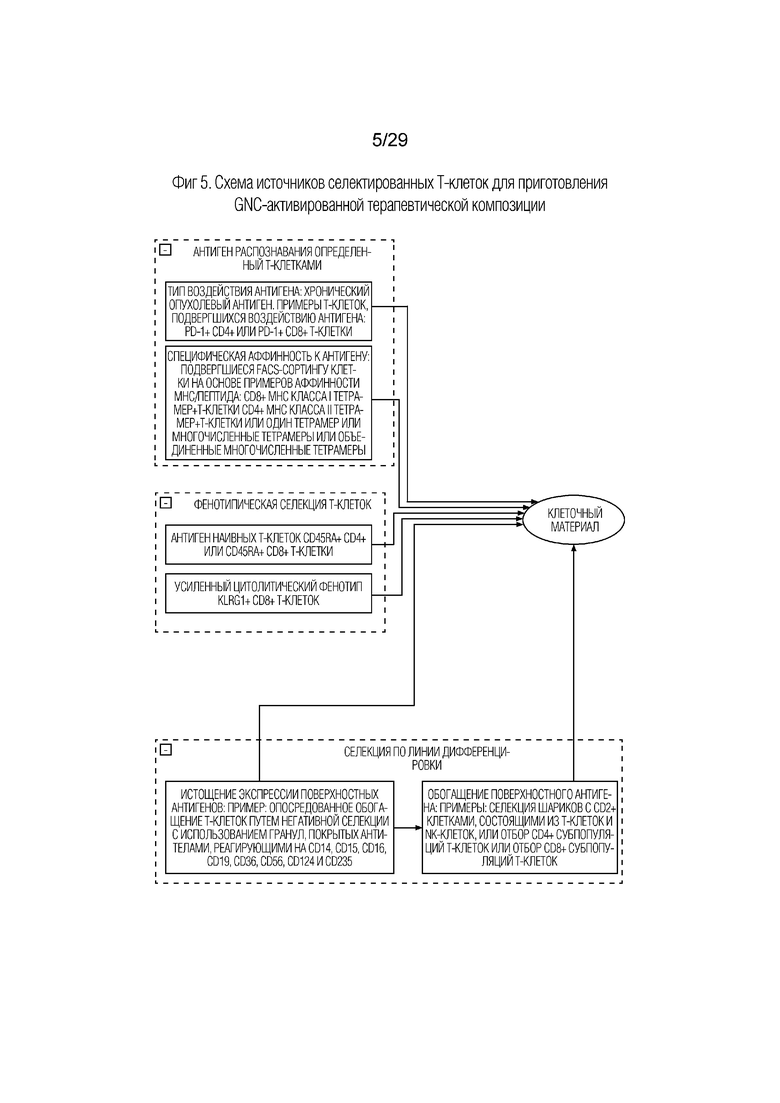

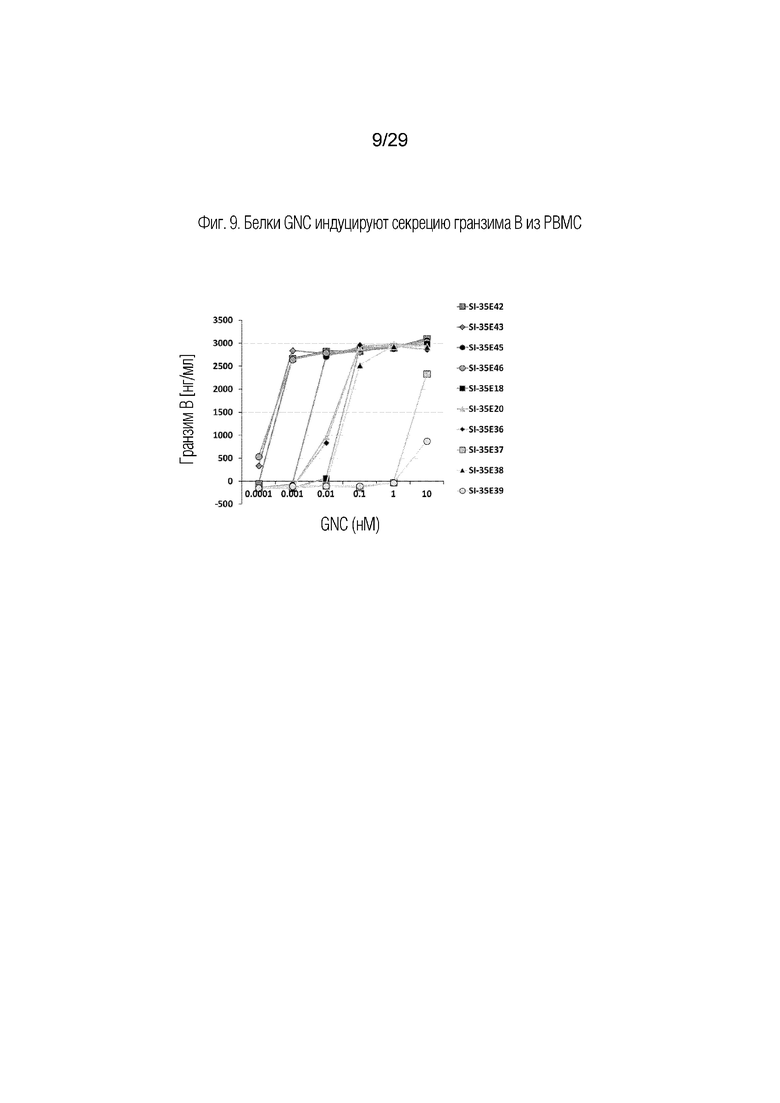
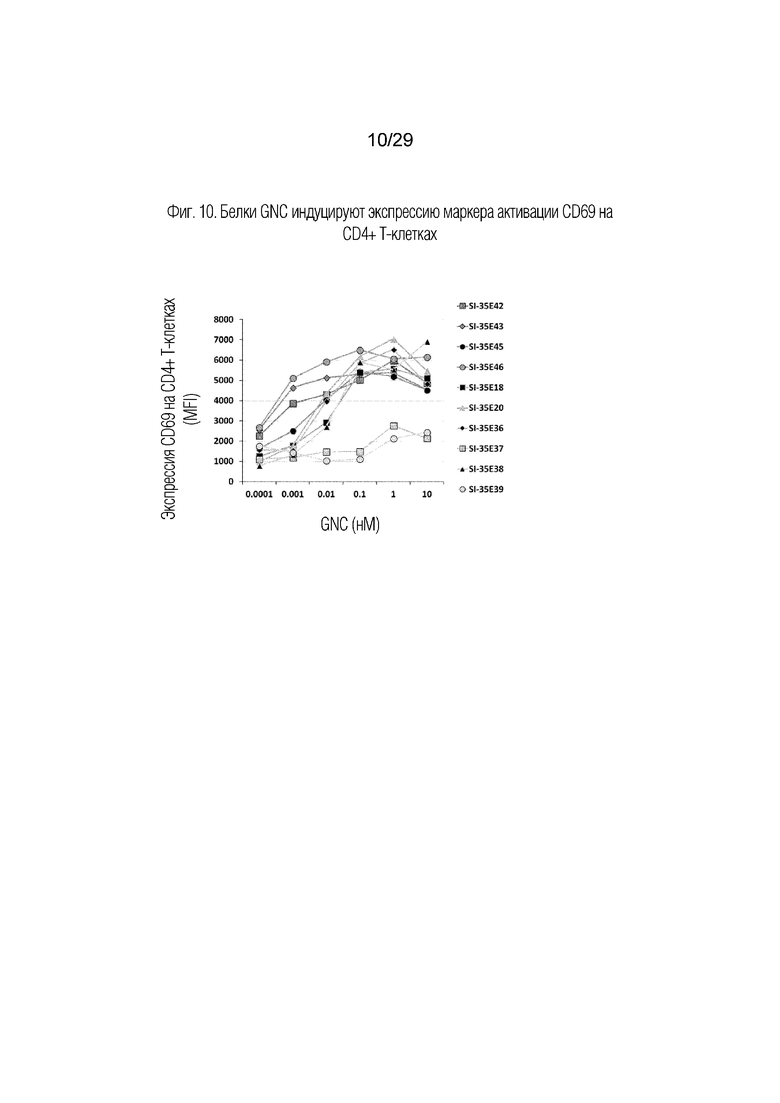


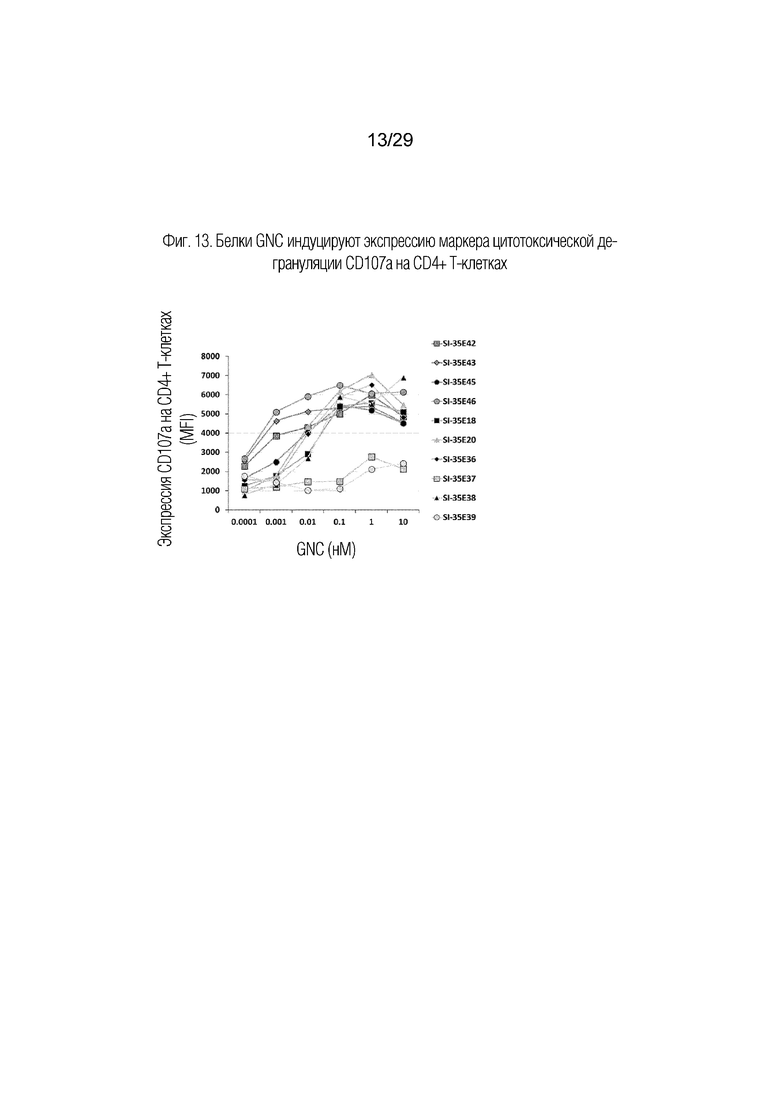
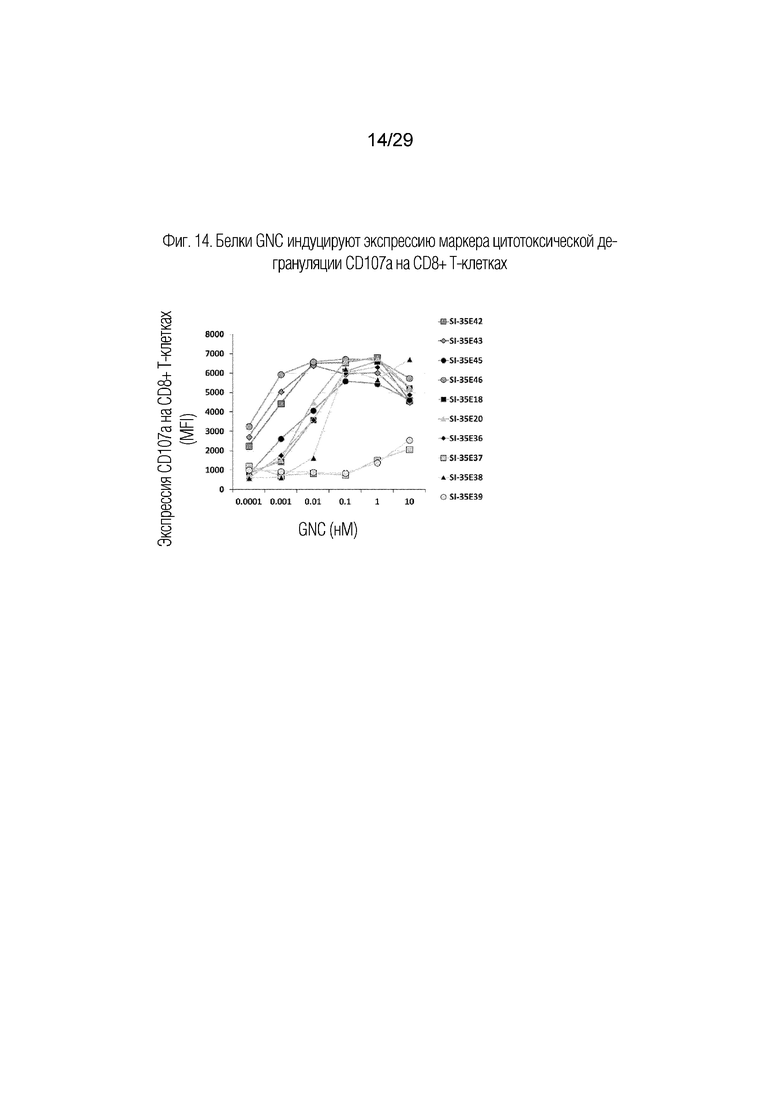
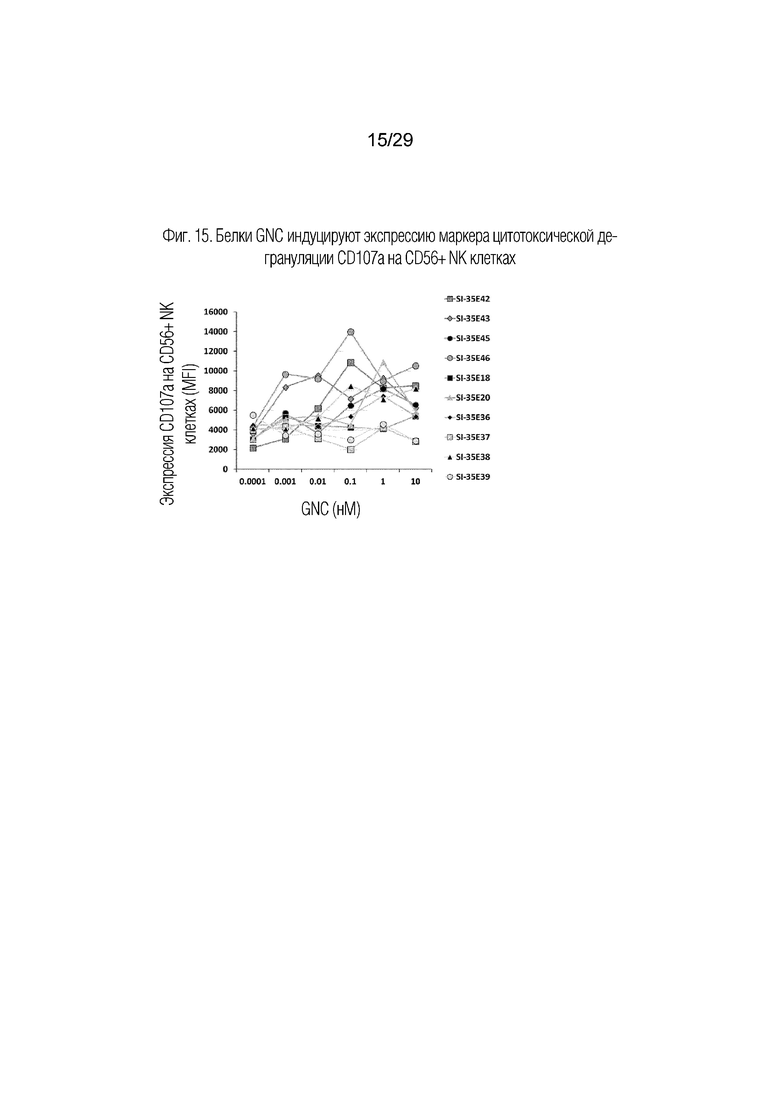




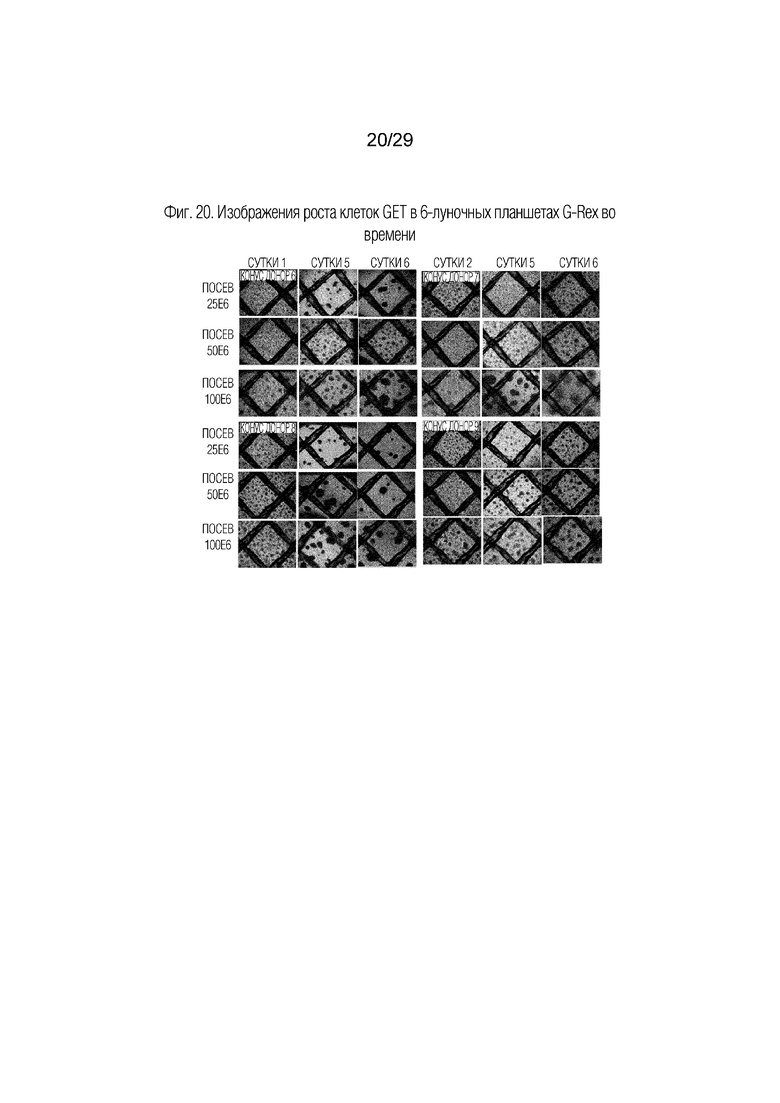


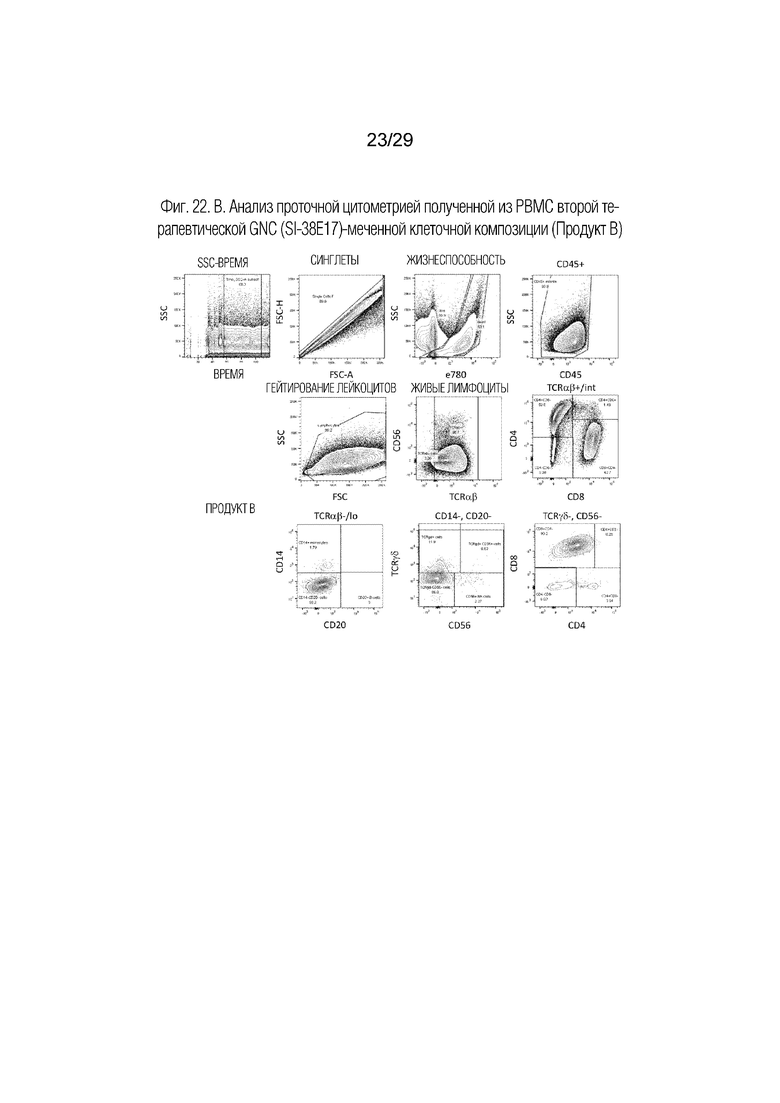


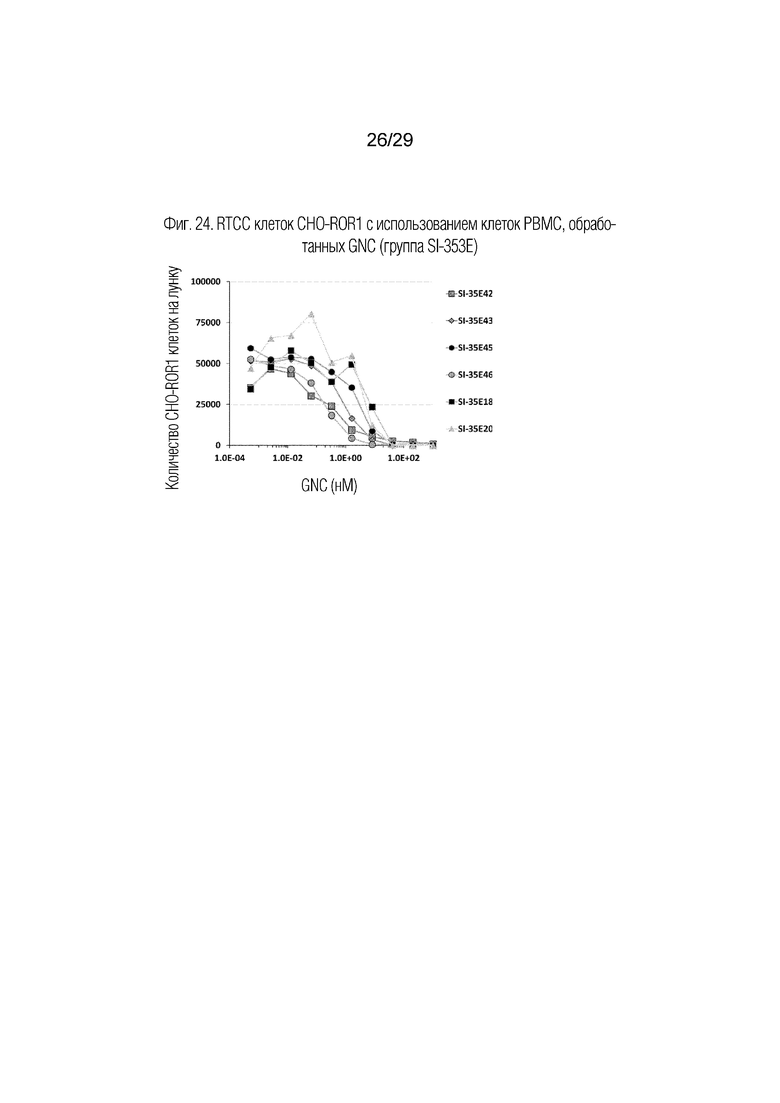
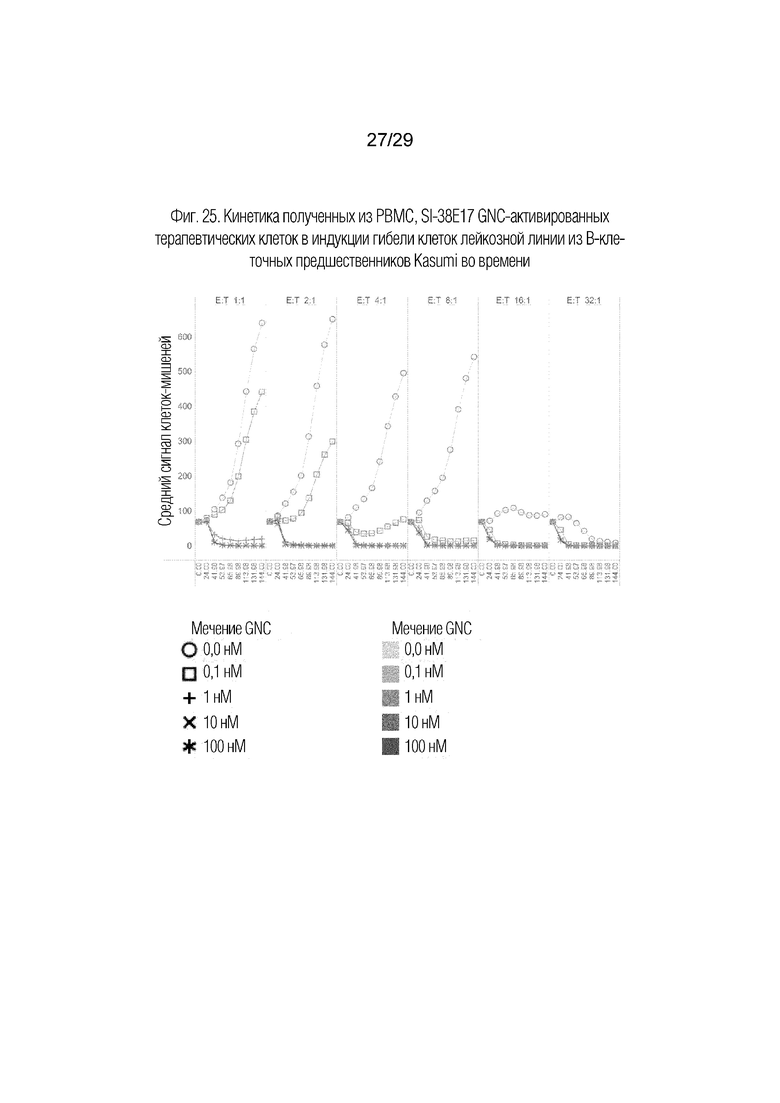
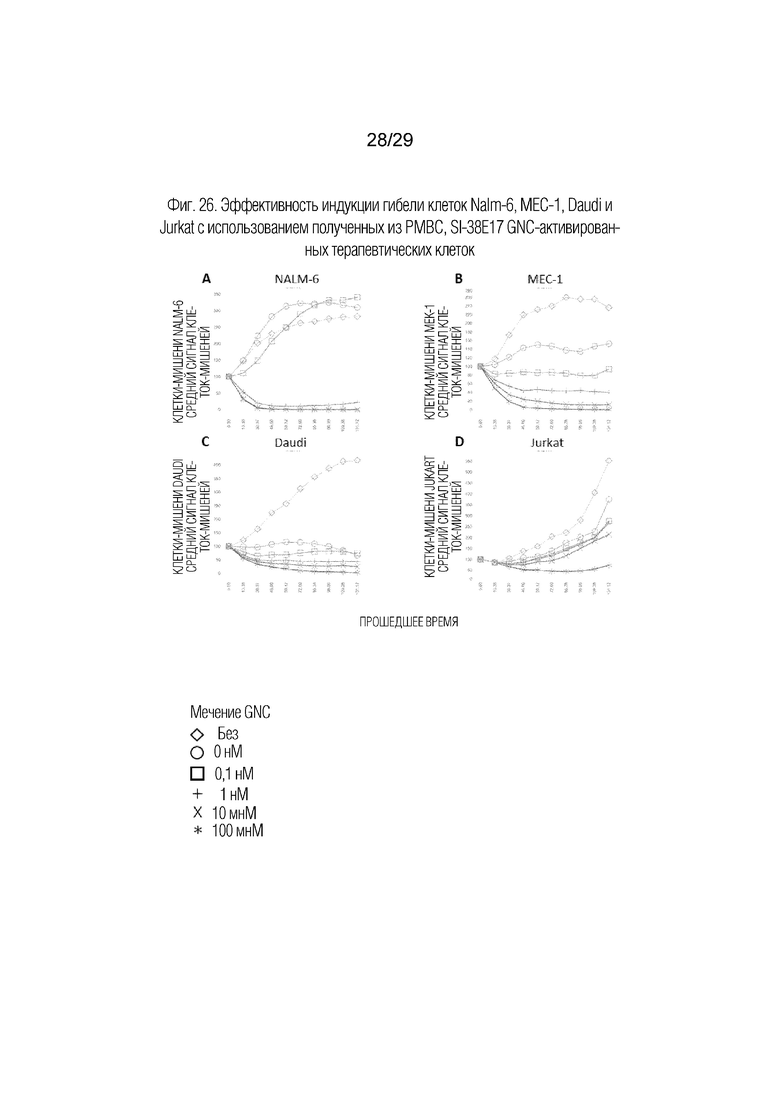

Изобретение относится к биотехнологии и генной инженерии и представляет собой способ получения терапевтической композиции для лечения опухоли, включающей клетки, экспрессирующие ROR1, CD19 или их комбинацию, включающий: обеспечение клеточного материала, содержащего цитотоксическую клетку, инкубацию клеточного материала с первым белком GNC с получением активированной клеточной композиции, где активированная клеточная композиция содержит первую терапевтическую клетку, где первый белок GNC содержит первый цитотоксический связывающий фрагмент и первый нацеливающий на опухоль фрагмент, где первый цитотоксический связывающий фрагмент обладает специфичностью к рецептору первой цитотоксической клетки и конфигурирован для активации первой цитотоксической клетки посредством связывания с рецептором первой цитотоксической клетки, и где первый нацеливающий на опухоль фрагмент обладает специфичностью к рецептору первой опухолевой клетки, и где первая терапевтическая клетка содержит первый белок GNC, связанный с цитотоксической клеткой посредством связывающего взаимодействия с рецептором первой цитотоксической клетки, и составление активированной клеточной композиции с получением терапевтической композиции, где терапевтическая композиция по существу не содержит экзогенной вирусной и невирусной ДНК или РНК. Изобретение позволяет лечить субъекта, имеющего опухоль, с более высокой степенью эффективности. 3 н. и 35 з.п.ф-лы, 27 ил., 4 табл., 5 пр.
1. Способ получения терапевтической композиции для лечения опухоли, включающей клетки, экспрессирующие ROR1, CD19 или их комбинацию, включающий:
обеспечение клеточного материала, содержащего цитотоксическую клетку,
инкубацию клеточного материала с первым белком GNC с получением активированной клеточной композиции, где активированная клеточная композиция содержит первую терапевтическую клетку,
где первый белок GNC содержит первый цитотоксический связывающий фрагмент и первый нацеливающий на опухоль фрагмент, где первый цитотоксический связывающий фрагмент обладает специфичностью к рецептору первой цитотоксической клетки и конфигурирован для активации первой цитотоксической клетки посредством связывания с рецептором первой цитотоксической клетки, и где первый нацеливающий на опухоль фрагмент обладает специфичностью к рецептору первой опухолевой клетки, и
где первая терапевтическая клетка содержит первый белок GNC, связанный с цитотоксической клеткой посредством связывающего взаимодействия с рецептором первой цитотоксической клетки, и
составление активированной клеточной композиции с получением терапевтической композиции, где терапевтическая композиция по существу не содержит экзогенной вирусной и невирусной ДНК или РНК.
2. Способ по п.1, где стадию инкубации повторяют путем инкубирования второго белка GNC с активированной клеточной композицией,
где второй белок GNC содержит второй цитотоксический связывающий фрагмент и второй нацеливающий на опухоль фрагмент, где второй цитотоксический связывающий фрагмент имеет специфичность к рецептору второй цитотоксической клетки, и где второй нацеливающий на опухоль фрагмент имеет специфичность к рецептору второй опухолевой клетки,
где активированная клеточная композиция дополнительно содержит вторую терапевтическую клетку и
где вторая терапевтическая клетка содержит второй белок GNC, связанный с цитотоксической клеткой или первой терапевтической клеткой посредством связывающего взаимодействия с рецептором второй цитотоксической клетки.
3. Способ по п.2, где второй белок GNC является таким же, как первый белок GNC.
4. Способ по п.2, где второй белок GNC отличается от первого белка GNC.
5. Способ по п.1 или 2, где первый или второй нацеленный на опухоль фрагмент обладает специфичностью по отношению к В-клетке, и где терапевтическая композиция по существу не содержит В-клетки.
6. Способ по п.1, где рецептор цитотоксической клетки содержит рецептор Т-клеток, рецептор NK-клеток, рецептор макрофагов, рецептор дендритных клеток или их комбинацию.
7. Способ по п.1, где молярное соотношение клеток к первому белку GNC и цитотоксической клетке составляет, по меньшей мере, 30:1 при инкубации клеточного материала с первым белком GNC.
8. Способ по п.1, где терапевтическая композиция содержит, по меньшей мере, 106 клеток на мл.
9. Способ по п.1, где терапевтическая композиция содержит первую терапевтическую клетку, первый белок GNC, цитотоксическую клетку или их комбинацию.
10. Способ по п.2, где терапевтическая композиция содержит вторую терапевтическую клетку, второй белок GNC, содержит первую терапевтическую клетку, первый белок GNC, цитотоксическую клетку или их комбинацию.
11. Способ по п.1, где клеточный материал содержит РВМС.
12. Способ по п.1, где первый и второй нацеливающий на опухоль фрагмент независимо имеет специфичность к CD19, PDL1 или их комбинации.
13. Способ по п.1, где первый и второй цитотоксический связывающий фрагмент независимо имеет специфичность к CD3, PDL1, 41BB или их комбинации.
14. Способ лечения субъекта, имеющего опухоль, включающую клетки, экспрессирующие ROR1, CD19 или их комбинацию, включающий:
обеспечение клеточного материала, содержащего цитотоксическую клетку,
инкубацию клеточного материала с первым белком GNC с получением активированной клеточной композиции, где активированная клеточная композиция содержит первую терапевтическую клетку,
где первый белок GNC содержит первый цитотоксический связывающий фрагмент и первый нацеливающий на опухоль фрагмент, где первый цитотоксический связывающий фрагмент обладает специфичностью к рецептору первой цитотоксической клетки и конфигурирован для активации первой цитотоксической клетки посредством связывания с рецептором первой цитотоксической клетки, и где первый нацеливающий на опухоль фрагмент обладает специфичностью к рецептору первой опухолевой клетки, и
где первая терапевтическая клетка содержит первый белок GNC, связанный с цитотоксической клеткой посредством связывающего взаимодействия с рецептором первой цитотоксической клетки, и
составление активированной клеточной композиции с получением терапевтической композиции, где терапевтическая композиция по существу не содержит экзогенной вирусной и невирусной ДНК или РНК, и
введение терапевтической композиции субъекту.
15. Способ по п.14, где стадию инкубации повторяют путем инкубирования второго белка GNC с активированной клеточной композицией,
где второй белок GNC содержит второй цитотоксический связывающий фрагмент и второй нацеливающий на опухоль фрагмент, где второй цитотоксический связывающий фрагмент имеет специфичность к рецептору второй цитотоксической клетки, и где второй нацеливающий на опухоль фрагмент имеет специфичность к рецептору второй опухолевой клетки,
где активированная клеточная композиция дополнительно содержит вторую терапевтическую клетку, и
где вторая терапевтическая клетка содержит второй белок GNC, связанный с цитотоксической клеткой или первой терапевтической клеткой посредством связывающего взаимодействия с рецептором второй цитотоксической клетки.
16. Способ по п.14, где второй белок GNC является таким же, как первый белок GNC.
17. Способ по п.14, где второй белок GNC отличается от первого белка GNC.
18. Способ по п.14 или 15, где первый или второй нацеленный на опухоль фрагмент обладает специфичностью по отношению к В-клетке, и где терапевтическая композиция по существу не содержит В-клетки.
19. Способ по п.14, дополнительно включающий выделение цитотоксической клетки из мононуклеарных клеток периферической крови (РВМС) перед обеспечением клеточного материала.
20. Способ по п.19, дополнительно включающий выделение мононуклеарных клеток периферической крови (РВМС) из крови.
21. Способ по п.20, где кровь взята у субъекта.
22. Способ по п.20, где кровь взята не у субъекта.
23. Способ по п.14, дополнительно включающий введение дополнительного белка GNC субъекту после введения терапевтической композиции субъекту.
24. Способ по п.14, где цитотоксическая клетка включает Т-клетку, NK-клетку или их комбинацию.
25. Способ по п.19, где выделение цитотоксической клетки включает выделение, по меньшей мере, одной субпопуляции цитотоксической клетки для получения терапевтических Т-клеток.
26. Способ по п.25, где субпопуляция цитотоксической клетки включает CD3+ клетки, CD4+ клетки, CD8+ клетки, CD56+ клетки, CD28+ клетки, CD69+ клетки, CD107a+ клетки, CD45RA+ клетки, CD45RO+ клетки, γδ TCR+ клетки, αβ TCR+ клетки, CD25+ клетки, CD127 lo/- клетки, CCR7+ клетки, PD-1+ клетки или их комбинации.
27. Способ по п.14, дополнительно включающий оценку терапевтической эффективности после стадии введения.
28. Способ по п.26, где оценка терапевтической эффективности включает анализ одного или более опухолевых биомаркеров, мониторинг продолжительности жизни терапевтических клеток или их комбинации.
29. Способ по п.28, где биомаркер включает опухолевый антиген, высвобождение цитокинов, например гамма-интерферона, IL-2, IL-8 и/или хемокинов, и/или маркеры CD на поверхности клеток различных типов, например, CD69, PD-1, TIGIT и/или мутантную нуклеиновую кислоту, высвобождаемую в кровоток опухолями после гибели, циркулирующие опухолевые клетки и связанную с ними нуклеиновую кислоту, или связанную с экзосомами нуклеиновую кислоту, медиаторы воспаления хозяина или аналиты, полученные из опухоли, или их комбинацию.
30. Способ по п.14, где субъектом является человек.
31. Способ по п.14, где рак включает рак молочной железы, колоректальный рак, рак анального канала, рак поджелудочной железы, рак желчного пузыря, рак желчных протоков, рак головы и шеи, рак носоглотки, рак кожи, меланому, рак яичников, рак предстательной железы, рак уретры, рак легких, немелкоклеточный рак легкого, мелкоклеточный рак легкого, опухоль головного мозга, глиому, нейробластому, рак пищевода, рак желудка, рак печени, рак почки, рак мочевого пузыря, рак шейки матки, рак эндометрия, рак щитовидной железы, рак глаза, саркому, рак кости, лейкоз, миелому или лимфому.
32. Способ по п.14, где злокачественная опухоль является CD19-позитивной.
33. Способ по п.14, дополнительно включающий введение эффективного количества терапевтического агента после введения терапевтической композиции субъекту.
34. Способ по п.33, где терапевтический агент включает моноклональное антитело, мультиспецифическое антитело, химиотерапевтический агент, фермент, белок, костимулятор, сенсибилизатор для апоптоза, антиангиогенный агент или их комбинацию.
35. Способ по п.34, где костимулятор конфигурирован для увеличения количества цитотоксических Т-клеток у субъекта.
36. Терапевтическая композиция для лечения опухоли, включающей клетки экспрессирующие ROR1, CD19 или их комбинацию, содержащая цитотоксическую клетку, белок GNC и терапевтическую клетку,
где белок GNC содержит цитотоксический связывающий фрагмент и нацеленный на опухоль фрагмент, где цитотоксический связывающий фрагмент обладает специфичностью к рецептору цитотоксических клеток, где нацеленный на опухоль фрагмент обладает специфичностью к рецептору опухолевых клеток, и где цитотоксический связывающий фрагмент конфигурирован для активации цитотоксической клетки посредством связывания с рецептором цитотоксической клетки,
где терапевтическая клетка содержит белок GNC, связанный с цитотоксической клеткой посредством связывающего взаимодействия с рецептором цитотоксической клетки, и
где терапевтическая клеточная композиция по существу не содержит экзогенной вирусной и невирусной ДНК и РНК.
37. Терапевтическая композиция по п.36, где нацеливающий на опухоль фрагмент обладает специфичностью к В-клетке, и терапевтическая композиция по существу не содержит В-клетки.
38. Терапевтическая композиция по п.36, дополнительно содержащая второй белок GNC, вторую терапевтическую клетку или их комбинацию, где вторая терапевтическая клетка содержит второй белок GNC, связанный с цитотоксической клеткой или первой терапевтической клеткой.
| US 20170210819 A1, 27.07.2017 | |||
| WO 2017064221 A1, 20.04.2017 | |||
| ТРИ- ИЛИ ТЕТРАСПЕЦИФИЧЕСКИЕ АНТИТЕЛА | 2010 |
|
RU2570633C2 |
Авторы
Даты
2024-08-15—Публикация
2019-03-26—Подача