Область техники
Изобретение относится к методу быстрой и высокоэффективной детекции микроорганизмов, вовлеченных в коррозию металлов, и может быть использовано для мониторинга бактериального заражения нефтепромысловых объектов и биокоррозии.
Уровень техники
Аварии на трубопроводах, перекачивающих горючие энергоносители, приводят к значительным финансовым потерям и серьезному, а иногда даже необратимому ущербу окружающей среды. Основная причина аварий на магистральных нефтепроводах – коррозия, которая помимо основных причин может вызываться сообществом микроорганизмов. Быстрое и эффективное обнаружение микроорганизмов, являющихся причиной коррозии, позволяет своевременно принять необходимые меры по подавлению их роста. Однако из-за чрезвычайно большого разнообразия организмов обнаружение, характеристика и количественная оценка микробных сообществ в экологических или промышленных образцах являются огромными задачами для биологов-экологов. Традиционные культуральные методы обогащения для изучения микробных сообществ оказались сложными и, в конечном итоге, дают крайне ограниченное представление о разнообразии и динамике микробных сообществ, поскольку большинство встречающихся в природе видов невозможно культивировать. Даже для бактерий, которые можно культивировать, скорость роста основных бактерий, вызывающих коррозию (например, сульфатвосстанавливающх бактерий (СВБ)), обычно требует до трех - пяти недель культивирования. Разработка и применение методов на основе нуклеиновых кислот в значительной степени устранили зависимость от методов, зависящих от культивирования, и значительно продвинули обнаружение и характеристику микроорганизмов в естественной среде обитания.
В последние годы для анализа нуклеиновых кислот в образце (например, с целью анализа экспрессии генов, связывания транскрипционных факторов, генотипирования и др.) все шире используют технологию биологических микрочипов (биочипов), которые, в отличие от технологий ПЦР в реальном времени или масс-спектрометрии, позволяют анализировать множество фрагментов ДНК в одной пробе одновременно. Применение технологии биочипов в анализе микроорганизмов в образцах позволяет существенно повысить скорость и точность анализа, являясь высокопроизводительным и экономически эффективным инструментом оценки для мониторинга сложных микробных сообществ, особенно в контексте микроорганизмов, вовлеченных в коррозию. Однако, несмотря на очевидные преимущества методики, в настоящее время известно относительно немного тест-систем для детектирования микроорганизмов, вовлеченных в микробно-индуцированную коррозию, основанных на технологии биочипов.
Наиболее близким аналогом предлагаемого изобретения является биочип низкой плотности для быстрого обнаружения бактериальных организмов, вовлеченных в микробиологическую коррозию нефтепроводов, описанный в патенте US11035009 В1 (опубл. 15.06.2021). Однако, поскольку большая часть ДНК проб, входящих в биочип, конструировалась на основе нуклеотидной последовательности только одного представителя каждого вида бактерий , это приводит к сужению спектра детектируемых бактерий и является причиной их низкой эффективности при мониторинге биокоррозии. Также существенным ограничением известного метода являются трудности изготовления матрикса и его высокая себестоимость.
Таким образом, актуальным является разработка новых методов анализа микробных сообществ, вовлеченных в микробно-индуцированную коррозию, на основе биочипов, которые позволят проводить быстрый качественный и количественный анализ, имеющий высокую чувствительность.
Раскрытие изобретения
Задачей изобретения является создание олигонуклеотидных проб (зондов) и биочипов на их основе для анализа основных групп микроорганизмов, вовлеченных в микробно-индуцированную коррозию (МИK).
Решение поставленной задачи осуществляется путем разработки набора олигонуклеотидных зондов для обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию, включающего по меньшей мере одну из совокупностей зондов i – vi, при этом каждая из совокупностей состоит из зондов, имеющих последовательности:
i. SEQ ID NO: 4, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 21, SEQ ID NO: 23, SEQ ID NO: 29; а также включающая SEQ ID NO: 60 или SEQ ID NO: 61; включающая SEQ ID NO: 5 или SEQ ID NO: 6; и включающая SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 или SEQ ID NO: 28;
ii. SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 16; и также включающая SEQ ID NO: 13, SEQ ID NO: 14 или SEQ ID NO: 15;
iii. SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 37;
iv. SEQ ID NO: 46, SEQ ID NO: 47, SEQ ID NO: 50 и SEQ ID NO: 40; и также включающая SEQ ID NO: 38 или SEQ ID NO: 39; и включающая SEQ ID NO: 18 или SEQ ID NO: 19;
v. SEQ ID NO: 43, SEQ ID NO: 44 и SEQ ID NO: 45;
vi. SEQ ID NO: 56 и SEQ ID NO: 58, а также включающая SEQ ID NO: 52, SEQ ID NO: 53 или SEQ ID NO: 54.
Опционально набор также может включать олигонуклеотидные зонды, имеющие последовательности SEQ ID NO: 62 и/или SEQ ID NO: 63.
В некоторых вариантах изобретения набор олигонуклеотидных зондов включает совокупность всех зондов i – vi, а также зонды, имеющие последовательности SEQ ID NO: 62 и SEQ ID NO: 63.
В некоторых вариантах изобретения совокупности зондов i – vi состоят из следующих последовательностей:
i. SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 21, SEQ ID NO: 23, SEQ ID NO: 27, SEQ ID NO: 29 и SEQ ID NO: 61;
ii. SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12 и SEQ ID NO: 16; а также SEQ ID NO: 13, SEQ ID NO: 14 или SEQ ID NO: 15;
iii. SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 37;
iv. SEQ ID NO: 19, SEQ ID NO: 39, SEQ ID NO: 40, SEQ ID NO: 46, SEQ ID NO: 47 и SEQ ID NO: 50;
v. SEQ ID NO: 43, SEQ ID NO: 44 и SEQ ID NO: 45;
vi. SEQ ID NO: 54; SEQ ID NO: 56 и SEQ ID NO: 58.
В некоторых вариантах изобретения микроорганизмы, вызывающие биокоррозию, представляют собой сульфатвосстанавливающие бактерии, а совокупность зондов для обнаружения, идентификации и/или количественного определения микроорганизмов представлена зондами, имеющими последовательности SEQ ID NO: 4, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 21, SEQ ID NO: 23, SEQ ID NO: 29и SEQ ID NO: 61, а также включающая SEQ ID NO: 5 или SEQ ID NO: 6 SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 или SEQ ID NO: 28.
В некоторых частных вариантах изобретения сульфатвосстанавливающие бактерии представляют собой бактерии рода Desulfovibrio, Desulfotomaculum, Desulfobacterium, Desulfobacter, Desulfococcus, Desulfomicrobium и Desulfuromonadales.
В некоторых вариантах изобретения микроорганизмы, вызывающие биокоррозию, представляют собой металлвосстанавливающие бактерии, а совокупность зондов для обнаружения, идентификации и/или количественного определения микроорганизмов представлена зондами, имеющими последовательности SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 16; и также включающая SEQ ID NO: 13, SEQ ID NO: 14 или SEQ ID NO: 15.
В некоторых частных вариантах изобретения металлвосстанавливающие бактерии представляют собой бактерии рода Shewanella и Geobacter.
В некоторых вариантах изобретения микроорганизмы, вызывающие биокоррозию, представляют собой Arhaea, а совокупность зондов для обнаружения, идентификации и/или количественного определения микроорганизмов представлена зондами, имеющими последовательности SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 37.
В некоторых частных вариантах изобретения Arhaea представляют собой метаногенные археи и арехеи рода Geobacter.
В некоторых вариантах изобретения микроорганизмы, вызывающие биокоррозию, представляют собой бактерии типа Firmicutes, а совокупность зондов для обнаружения, идентификации и/или количественного определения микроорганизмов представлена зондами, имеющими последовательности SEQ ID NO: 46, SEQ ID NO: 47, SEQ ID NO: 50, и SEQ ID NO: 40; и также включающая SEQ ID NO: 38 или SEQ ID NO: 39; и включающая SEQ ID NO: 18 или SEQ ID NO: 19.
В некоторых частных вариантах изобретения бактерии типа Firmicutes представляют собой ацетогенные бактерии, углеводород- деградирующие и бактерии классов Bacillus, Clostridium или Desulfobulbus.
В некоторых вариантах изобретения микроорганизмы, вызывающие биокоррозию, представляют собой нитритредуцирующие бактерии, а совокупность зондов для обнаружения, идентификации и/или количественного определения микроорганизмов представлена зондами, имеющими последовательности SEQ ID NO: 43, SEQ ID NO: 44 и SEQ ID NO: 45.
В некоторых частных вариантах изобретения нитритредуцирующие бактерии представляют собой бактерии рода Pseudomonas.
В некоторых вариантах изобретения микроорганизмы, вызывающие биокоррозию, представляют собой бактерии, усиливающие рост сульфатвосстанавливающих бактерий, а совокупность зондов для обнаружения, идентификации и/или количественного определения микроорганизмов представлена зондами, имеющими последовательности SEQ ID NO: 56 и SEQ ID NO: 58, а также включающая SEQ ID NO: 52, SEQ ID NO: 53 или SEQ ID NO: 54.
В некоторых частных вариантах изобретения бактерии, усиливающие рост сульфатвосстанавливающих бактерий, представляют собой бактерии семейства Enterobacteriaceae и бактерии семейства Hafniaceae.
В некоторых вариантах изобретения зонды иммобилизованы в идентифицируемых местах на биочипе.
Решение поставленной задачи также осуществляется путем разработки биочипа для обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию, включающего любой из описанных выше наборов олигонуклеотидных зондов, иммобилизированных на твердой подложке.
В некоторых вариантах изобретения твердая подложка биочипа включает 3D гидрогель.
Решение поставленной задачи также осуществляется путем разработки способа обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию, включающего следующие этапы:
амплификация ДНК из образца, полученного для анализа;
фрагментация амплифицированной ДНК;
флюоресцентное мечение ДНК;
заливка флуоресцентно-меченой ДНК на биочип и гибридизация с элементами биочипа;
регистрация и интерпретация результатов гибридизации на биочипе, проведенной на стадии (d).
В некоторых вариантах изобретения образец для анализа представляет собой смыв или соскоб с внутренней поверхности трубы из нефте- или газораспределительной системы.
В результате осуществления изобретения достигаются следующие технические результаты:
– разработан набор олигонуклеотидных проб (зондов), специфичных для различных групп микроорганизмов, вовлеченных в микробно-индуцированную коррозию (МИK), в частности, проб, направленных на выявление сульфатвосстанавливающиx прокариот, метаногенныx и термофильных археи, металлвосстанавливающиx, денитрифицирующиx, ферментативныx, ацетoгенныx бактерий, углеводород-деградирующиx бактерий, Enterobacteriaceae;
- разработанные олигонуклеотидные пробы сконструированы на основе анализа нуклеотидных последовательностей различных представителей микроорганизмов, входящих в каждую из перечисленных групп, что позволяет существенно повысить эффективность мониторинга биокоррозии;
- разработанные олигонуклеотидные пробы (зонды), специфичные для различных групп микроорганизмов, могут быть использованы как комплексно, для выявления всех перечисленных групп микроорганизмов, так и раздельно, для выявления только одной или одной и более из перечисленных групп, а также в комбинации с другими наборами олигонуклеотидных проб (или индивидуальными пробами);
– разработаны биочипы, содержащие олигонуклеотидные пробы для детекции функциональных и структурных генов различных групп микроорганизмов, вовлеченных в микробно-индуцированную коррозию (МИK); мультиплексность разработанных биочипов позволяет использовать их для одновременного определения на одном биочипе практически всех известных групп коррозионно-опасных микроорганизмов;
- разработанные олигонуклеотидные пробы (зонды) и биочипы на их основе позволяют значительно расширить спектр и количество детектируемых микроорганизмов, вовлеченных в коррозию металлов;
- разработан способ идентификации и количественного определения микроорганизмов, относящихся к различным группам, вовлеченных в МИК, с использованием созданного набора(-ов) олигонуклеотидных зондов, а также с использованием созданного биочипа(-ов), который может быть использован для мониторинга бактериального заражения нефтепромысловых объектов и биокоррозии как нефтепромысловых, так и любых других объектов, содержащих металлоконструкции; определение микроорганизмов в образце не требует длительной пробоподготовки, а также детекции микроорганизмов микробиологическими методами.
- разработанные наборы, биочипы и способы позволяют проводить быстрый качественный и количественный анализ микробных сообществ, вовлеченных в микробно-индуцированную коррозию, и обеспечивают высокую чувствительность анализа.
Термины и определения
Если иное не оговаривается, все технические и научные термины, используемые в данной заявке, имеют то же самое значение, которое понятно для специалистов в данной области. Ссылки на методики, используемые при описании данного изобретения, относятся к хорошо известным методам, включая изменения этих методов и замену их эквивалентными методами, известными специалистам.
В документах данного изобретения термины «включает», «включающий» и т.п., а также «содержит», «содержащий» и т.п. интерпретируются как означающие «включает, помимо всего прочего» (или «содержит, помимо всего прочего»). Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из».
Термин «и/или» означает один, несколько или все перечисленные элементы.
Под «образцом» в настоящем изобретении подразумевается вода (смывы с труб) из нефте/газо распределительной системы, промысловые воды нефтепромыслов, пластовые воды, либо соскобы из внутренней поверхности труб.
Под «количественным определением» подразумевается количество бактерий в 1 мл водного образца либо в 1 г соскоба. Такое определение возможно путем использования количественного стандарта, нанесённого на биочип, и программного обеспечения для анализа эффективности флюоресцентного свечения.
Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Краткое описание рисунков
Фиг.1 – Схема биочип-мониторинга микроорганизмов, вовлеченных в коррозию металлов.
Фиг.2 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 1. Проба FeoB2 - позиции A1-A2, проба FeoB3 - позиции A3-A4, проба FeoB4a - позиции A5-A6, проба FeoB4b – позиции A7-A8, проба MtrB1 – позиции A9-A10, проба gltA-4T - позиции B1-B2, проба gltA-4C - позиции B3-B4, проба gltA-4G - позиции B5-B6. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.3 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 2. Проба FTHFS-Ac - позиции B7-A8, проба FTHFS-c1 - позиция B9-B10, проба FTHFS-c2 - позиции C1-C2, проба Geog1090 – позиции C3-C4, проба Arg830– позиции C5-C6, проба Arg1597- позиции C7-C8, проба FTHFS-c3 – позиции I9-I10, проба hydfob1 - позиции K7-K8, проба hydfob2 - позиции K9-K10. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.4 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 3. Проба assA1 - позиции C9-C10, проба assA2 - позиции D1-D2, проба nirD233 - позиции D3-D4, проба nirK – позиции D5-D6, проба nirS – позиции D7-D8, проба nirD265 - позиции D9-D10, проба nirK1- позиции I7-I8, проба hydssmic3 - позиции J5-J6. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижних и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.5 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 4. Проба McrA992 - позиция E1-E2, проба McrA994 - позиции E3-E4, проба McrA985 - позиции E5-E6, проба aprAvib1 - позиции E7-E8, проба aprAvib2 - позиции E9-E10, проба aprAvib4 - позиции F1-F2, проба aprAvib3 - позиции L1-L2. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.6 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 5. Проба hybOE156 - позиции F3-F4, проба hybOE784 - позиции F5-F6, проба uidA2m - позиции F7-F8, проба hybOH754 - позиции F9-F10, проба hybOH36y - позиции G1-G2, проба hybOH36 - позиции G3-G4, проба hybOE783 - позиции L3-L4. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.7 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 6. Проба hypBfoc1 - позиции G5-G6, проба hypBfoc2 - позиции G7- G8, проба nifHbac1 - позиции G9-G10, проба nifHbac2 – позиции H1-H2, проба nifHbac3 - позиции H3-H4, проба nifHbac4 - позиции H5-H6. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.8 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 7. Проба nirK2 - позиции D5-D6, проба hynBbam - позиции H7- H8, проба hydsstom27 - позиции H9-H10, проба dsrAtom709 - позиции I1-I2, проба dsrAtom710 - позиции I3-I4, проба dsrAtom883 - позиции I5-I6, проба nir K1 - позиции I7-I8. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.9 (A). Изображение биочипа, иллюстрирующего результаты гибридизациис кассетой 8. Проба FTHFS-c1 - позиции B9-B10, проба FTHFS-c2 - позиции C1-C2, проба nirD233 – позиции D3-D4, проба FTHFS-c3 - позиции I9-I10, проба hydAmic1 – позиции J1-J2, hydssmic2 - позиции J3-J4, hydssmic3 – позиции J5-J6, hydssmic4 – позиции J7-J8, hydsmic5 - позиции J9-J10. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.10 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 9. Проба Geog1090 - позиции C3-C4, проба bac1618 - позиции K1-K2, проба bac1620 - позиции K3-K4, проба bac325 – позиции K5-K6, проба hydfob1– позиции K7-K8, проба hydfob2 - позиции K9-K10. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.11 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 10. Проба aprAvib2 - позиции E9-E10, проба aprAvib4 - позиции F1-F2, проба hybOE784 - позиции F5-F6, проба aprAvib3 – позиции L1-L2, проба hybOE783 – позиции L3-L4, проба hyalGeob - позиции L5-L6, проба nifD1333 – позиции L7-L8, проба nifD915 – позиции L9-L10. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Фиг.12 (A). Изображение биочипа, иллюстрирующего результаты гибридизации с кассетой 11. Проба glt 4T - позиции B1-B2, проба glt 4C - позиции B3-B4, проба glt 4G – позиции B5-B6, проба aprAvib4 - позиции F1-F2, проба 16SCons - позиции M1- M2, проба 16SArch - позиции M3-M4. Маркеры, 3 точки Texas red-меченный олигонуклеотид, для ориентации и идентификации положения проб находятся в правом и в левом нижнем и в левом верхнем углах. (B). Диаграмма интенсивности флюоресцентныx сигналов указанных проб/зондов. Изображения на биочипе были проанализированы с использованием программы ImaGel 2.0.
Подробное описание изобретения
Сущность созданного технического решения заключается в создании диагностического инструмента для быстрой эффективной детекции биологической коррозии, что подразумевает разработку ДНК проб - мишеней (зондов) для различных генов основных групп микроорганизмов, вовлеченных в МИK, для включения их в биочип.
Микробно-индуцированная коррозия (МИK) вызывается разнообразными микроорганизмами, относящимися к различным таксонам. Долгое время считалось, что сульфатвосстанавливающие бактерии (СВБ) рода Desulfovibrio являются главными разрушителями стали, что объяснялось лёгкостью их детекции простыми микробиологическими методами. Это заблуждение было развеяно открытием громадного количества микроорганизмов, которые также вовлечены в коррозию металлов, и их вклад, учитывая их разнообразие и присутствие практически в любом месте окружающей среды, скорее всего выше, чем СВБ. Недооценка их роли была вызвана трудностью их обнаружения, а также невозможностью культивирования большинства из них в лабораторных условиях.
Все микроорганизмы, вовлеченные в биокоррозию, объединены в несколько групп по наличию у них метаболических путей, существенных для коррозивных реакций (Beech, 2002; Little and Lee, 2007). Это такие группы как сульфатвосстанавливающие бактерии (СВБ) и археи, ацетогенные бактерии, метаногенные археи, ферментативные бактерии, Archaeoglobus, Geoglobus, металл-восстанавливающие микроорганизмы, нитрат/нитрит восстанавливающие, углеводород деградирующие бактерии.
С целью быстрой и эффективной идентификации и количественного определения микроорганизмов, вовлеченных в МИК, в рамках изобретения был разработан набор олигонуклеотидных проб (зондов), специфичных для различных групп микроорганизмов, вовлеченных в микробно-индуцированную коррозию (МИK).
1. Разработка олигонуклеотидных проб
Разработка ДНК проб для детекции сульфатвосстанавливающих (СВБ) бактерий и архей
Сульфатвосстанавливающие (сульфатредуцирующие) прокариоты (бактерии и археи) – одна из самых значительных и агрессивных групп микроорганизмов, вовлеченных в коррозию металлов. Характерной особенностью сульфатвосстанавливающих микроорганизмов является наличие у них диссимиляционного пути восстановления сульфатов и элементный серы. Эти анаэробные организмы способны полностью окислять различные источники углерода до CO2 и восстанавливать сульфаты до сероводорода. СВБ осуществляют диссимиляционную сульфатредукцию благодаря наличию трёх ферментов. Процесс начинается с активации сульфата ферментом АТФ-сульфурилазой, которая использует АТФ и сульфат для создания аденозин-5’-фосфосульфата (APS), затем APS-редуктаза превращает аденозин-5’–фосфосульфат в аденозинмонофосфат и сульфит, который, в итоге, превращается в сульфид (сероводород) под действием диссимиляционной сульфитредуктазы DSR. Гены, кодирующие аденозин-5’–фосфосульфат-редуктазу (aprAB) и диссимиляционную сульфит-редуктазу (dsrAB), являются консервативными и присутствуют у всех СВБ, поэтому авторами изобретения было принято решение использовать их в качестве биомаркеров для обнаружения СВБ, как минимум, рода Desulfovibrio. Полные нуклеотидные последовательности aprAB генов бактерий Desulfovibrio vulgaris RCH1(NC_017310.1), D. vulgaris Hildenborough (AE017285.1), D. vulgaris (Z69372.1), D. gigas (KF113859.1), D. piger (ABXU01000081.1) и (DS996360.1), D. alaskensis (AJ271652.1), извлечённые из базы данных, были сохранены в FASTA формате. Области гомологии были найдены путем множественного выравнивания последовательностей в программе SeaView (Sievers and Higgins, 2014). Выявленные консервативные участки последовательностей каждого гена использовали для конструирования ДНК проб. Специфичность этих проб была проверена в программах BLASTn microbe. После тонкой настройки биочипа (более подробная информация приведена ниже) проба aprAvib4 была признана наиболее специфичной для детекции бактерий рода Desulfovibrio и была отобрана для включения в биочип. Структура этой пробы представлена в таблицах 1 и 15.
Для детекции других представителей СВБ, таких как Desulfotomaculum spp, принадлежащих к филуму Firmicutes, был выбран ген диссимиляционной сульфит-редуктазы (dsrAB). Для этого нуклеотидные последовательности dsrAB генов бактерий Desulfotomaculum ruminis DSM 2154 (CP002780.1), D. ruminis (U58118.2), D. putei, D. geothermicum (AF273029.1), D. halophilum DSM 11559 (AY626024.1) были загружены из базы данных NCBI (National Center for Biotechnology Information), консервативные участки были найдены путем множественного выравнивания последовательностей в программе SeaView и далее были использованы для разработки ДНК проб. После тонкой настройки биочипа проба dsrAtom710 была признана наиболее специфичной для детекции Desulfotomaculum spp и была отобрана для включения в биочип. Cконструированная проба dsrAtom710 представлена в таблицах 1 и 15. Также для детекции Desulfotomaculum выбран ген Fe-Fe гидрогеназы группы A. Для этого нуклеотидные последовательности гена hydA Desulfotomaculum aeronauticum DSM1034 (NZ_FRAR01000005.1) и D. njgrificans DSM574 (NZ_KI912183.1) были загружены из базы данных NCBI, консервативные участки были найдены путем множественного выравнивания последовательностей в программе SeaView и далее были использованы для разработки ДНК проб. После тонкой настройки биочипа проба hydsstom27 была отобрана для включения в биочип. Cконструированная проба hydsstom27 представлена в таблицах 1 и 15.
Разработка ДНК проб для детекции металл-восстанавливающих бактерий (МВБ)
Металл-восстанавливающие бактерии усиливают скорость микробной коррозии, они восстанавливают Fe3+ до растворимого Fe2+. Ионы трехвалентного железа нерастворимы, и его соли защищают поверхность металла от дальнейшей коррозии. Восстановление Fe3+ до Fe2+ приводит к растворению защитного окисного слоя и ускорению коррозии. Многие металл-восстанавливающие организмы филогенетически тесно связаны с СВБ, так как ацетат, используемый Fe (III)-редуцирующими организмами, зачастую является продуктом метаболизма СВБ. Род бактерий Shewanella в настоящее время представлен 50 видами, более 20 из которых способны восстанавливать металлы (Konstantinidis, et al. 2009, Lovley, 1991). Эти факультативно анаэробные бактерии обладают комплексной электрон-транспортной системой, которая позволяет им использовать большое разнообразие соединений в качестве конечных акцепторов электронов для анаэробного дыхания. Они окисляют различные источники углерода, используя диссимиляционные восстановительные реакции, растворяют Fe(III)-содержащие минералы. В диссимиляционном восстановлении железа перенос электронов от железа происходит как часть ферментативного или дыхательного пути. Бактерии этого рода играют важную роль в деструктивном закислении грубой нефти (Semple & Westlake, 1987), в восстановлении оксидов марганца и железа (Myers & Nealson, 1988), а также характеризуются способностью образовывать большие количества полиненасыщенных жирных кислот (Russell & Nichols, 1999).
mtrABC оперон электрон-транспортной системы, играющий существенную роль в восстановлении металлов, консервативен у всех Shewanella, за исключением S. denitrificans OS217 и S. violacea DSS12 (Shi L et al., 2012), и может быть использован для обнаружения этих бактерий.
Кроме того, feoA–feoB оперон, вовлеченный в транспорт железа, также является общим для всех штаммов Shewanella и примыкает к mtr–omc кластеру. В металл- восстановительном пути метаболизма электроны проходят от внутренней мембраны через периплазму и наружную мембрану к внеклеточным минералам посредством белков, кодируемых mtr–omc кластером.
На основании этого, mtrB ген, кодирующий внешний мембранный белок, а также ген feoB, кодирующий мембранный транспортный белок (транспортёр пермеазной субъединицы FeoB), были выбраны для конструирования проб для детекции Shewanella с применением технологии биочипов. Для проведения множественного выравнивания последовательностей были использованы последовательности mtrB и feoB генов 24 видов бактерий рода Shewanella, а именно, на наличие консервативных участков анализировались полные последовательности перечисленных штаммов различных видов Shewanella : S. baltica OS155, S. baltica OS117, S. baltica OS223, S. baltica OS678, S. baltica OS195, S. baltica OS185, S. baltica NCTC, S. putrefaciens 200, S. putrefaciens SN32, S. sp. MR-7, S. sp. MR-4, S. sp. ANA-3, S. oneidensis MR-1, S. denitrificans OS217, S. violacea DSS12, S. sediminis HAW-EB3, S. halifaxensis HAW-EB4, S. pealeana ATCC 700345, S. piezotolerans WP3, S. loihica PV-4, S. woodyi ATCC700345, S. xiamenensis DCB2-1, S. bicestrii JAB-1, S.fidelis ATCCBAA-318. В результате, после тонкой настройки биочипа, для включения в биочип были отобраны пробы FeoB2, FeoB3, FeoB4a, FeoB4b, созданные на основе гена feoB, кодирующего Fe(2+)-транспортёр пермеазной субъединицы FeoB, и проба MtrB1, созданная на основе гена MtrB, кодирующего белок наружной мембраны бактерий Shewanella, ассоциированный с декагемом (decaheme) и входящий в семейство белков MtrB/PioB, связанных с мультигемовыми цитохромами с-типа, участвующими в переносе электронов, признанные наиболее специфичными для детекции бактерий рода Shewanella. Структуры указанных проб представлены в таблицах 1 и 15.
Члены семейства бактерий Geobacteraceae также относятся к микроорганизмам, восстанавливающим металлы. Geobacter подавляет другие Fe(III)-восстанавливающие микробы в подземных средах, содержащих Fe(III). Эти бактерии способны создавать электрический заряд и продуцировать электричество, разрушать более сложные соединения на составляющие, которые могут быть использованы метаногенами с образованием метана. Ключевым фактором в их конкурентоспособности является их способность полностью окислять ацетат и другие органические соединения до CO2. Процесс окисления ацетата они совмещают с восстановлением внешних акцепторов электронов, таких как нерастворимые окислы Fe(III). Главным ферментом метаболизма ацетата у Geobacteraceae является цитрат-синтаза, катализирующая конденсацию Коэнзим A и оксалоацетата с образованием лимонной кислоты. Ген gltA, кодирующий цитрат-синтазу, является уникальным в метаболизме всех представителей рода Geobacteraceae (Bond et al., 2005), в связи с чем было принято решение использовать пробы к консервативным участкам этого гена для детекции этих микроорганизмов.
Для поиска консервативных участков гена gltA были использованы полные геномы штаммов различных видов бактерий Geobacter: G. sulfurreducens AM-1, G. sulfurreducens YM18, G. sulfurreducens KN400, G. sulfurreducens PCA, G. metallireducens GS-15, G. uranium, G. bemidjiensis, G. pickeringii G13. В результате проведённого множественного выравнивания были обнаружены консервативные последовательности, на основании которых были созданы пробы к гену gltA, после тонкой настройки биочипа проба gltA была признана наиболее специфичной для обнаружения представителей Geobacteraceae с применением технологии биочипов. Данная проба представлена в таблицах 1 и 15.
Кроме того, для повышения эффективности обнаружения Geobacter была создана проба hyaLGeob на основе полной нуклеотидной последовательности hyaL гена, кодирующего большую субъединицу периплазматической мембрансвязанной [NiFe] гидрогеназы, следующих геобактерий: G. metallireducens GS-15, G. sulfurreducens KN400, G. sulfurreducens PCA, G. bemidjiensis Bem. После тонкой настройки биочипа проба hyaLGeob также была признана эффективной, и была включена в биочип. Данная проба представлена в таблицах 1 и 15.
ДНК пробы на основе генов гидрогеназ для детекции отдельных представителей СВБ
Вовлеченные в МИК микроорганизмы обладают способностью использовать в качестве метаболита водород, благодаря наличию у них ферментов гидрогеназ, которые катализируют процесс разложения молекулярного H2 или образуют H2, восстанавливая протоны. Гидрогеназы подразделяются на три различных типа в зависимости от содержания металла в активном центре: [FeFe] гидрогеназа, [NiFe] гидрогеназа и Fe гидрогеназа. [FeFe] гидрогеназы обычно восстанавливают протоны, тогда как [NiFe] гидрогеназы окисляют водород.
[NiFe] гидрогеназы, наиболее распространённые таксономически, состоят, по крайней мере, из двух субъединиц большой (L), с каталитическим центром, и малой (S), содержащей три консервативных Fe-S кластера, которые служат в качестве переносчика электронов между наружным носителем электронов и NiFe сайтом. Различают периплазматические, цитоплазматические и цитоплазматические мембраносвязанные гидрогеназы. [NiFe] гидрогеназы присутствуют у разнообразных бактерий, а также обнаруживаются у археев и цианобактерий.
[FeFe] гидрогеназы обнаружены у анаэробных бактерий, они превалируют у ферментативных бактерий (Firmicutes) и отсутствуют у археев. [Fe]-гидрогеназа содержит только железосодержащий кофактор. [Fe]-гидрогеназы обнаруживаются только у некоторых метаногенных архей.
Гидрогеназы, используя катодной водород, ускоряют биокоррозию. Коррозия подземных металлических конструкций в основном вызывается бактериями рода Desulfovibrio, который приводит к наиболее вредной “питтинговой” коррозии. Все изученные Desulfovibrio spp имеют гены периплазматических [NiFe] гидрогеназ. Гены hynAB, их кодирующие, содержат консервативные домены. [NiFe] гидрогеназы устойчивы к ингибиторам, таким как CO и NO2-, являющимися главными продуктами метаболизма микробного сообщества в анаэробных условиях. [NiFe] гидрогеназы Desulfobacterium spp. имеют высокую степень гомологии с Desulfovibrio spp, легко различающимися филогенетически.
Наличие гидрогеназ у различных анаэробных СВБ позволяет использовать гены, кодирующие эти ферменты, для разработки ДНК проб для идентификации этих микроорганизмов.
В связи с этим полные нуклеотидные последовательности hynB гена, кодирующего малую субъединицу HynB периплазматической [NiFe] гидрогеназы, бактерий Desulfobacterium autotrophicum HRM2 (CP001087.1), D. vacuolatum DSM 3385 (NZ_FWXY01000006.1), извлеченные из базы данных, были сохранены в FASTA формате, затем, путем множественного выравнивания последовательностей в программе SeaView, в них были найдены консервативные участки и использованы далее для разработки ДНК проб. Специфичность этих проб была проверена в программах BLASTn microbe. Проба hynBbam после тонкой настройки биочипа была признана высокоспецифичной для детекции бактерий рода Desulfobacterium и была отобрана для включения в биочип. Структура этой пробы представлена в таблицах 1 и 15.
ДНК пробы для идентификации бактерий Desulfobacter были разработаны на основании сравнения полных нуклеотидных последовательностей nifH гена Fe-гидрогеназы из штаммов различных видов бактерий Desulfobacter: D. latus AcRS2 (NZ JACADJ010000004.1), D. hydrogenophilus AcRS1 (NZ CP036313.1), D. vibrioformis DSM8776 (NZ JQKJ01000011.1), D. postgatei 2ac9 (NZ CM001488.1) и гена, кодирующего малую субъединицу гидрогеназы бактерий Desulfobacter из D. postgatei 2ac9 (NZ CM001488.1), Desulfobacter curvatus DSM 3379 (NZ_KB893090.1), D. hydrogenophilus AcRS1 (NZ CP036313.1). Две пробы nifHbac2 и nifHbac4 после тонкой настройки биочипа были признаны наиболее специфичными для детекции бактерий рода Desulfobacter и были отобраны для включения в биочип. Структуры этих проб представлены в таблицах 1 и 15.
Для детекции бактерий Desulfomicrobium были использованы полные нуклеотидные последовательности hydA гена, кодирующего малую субъединицу [NiFe] гидрогеназы из следующих штаммов различных видов бактерий Desulfomicrobium: D. norvegicum DSM 1741 (FOTO01000008.1), D. apsheronum DSM 5918 (FORX01000005.1), D. baculatum DSM 4028 (CP001629.1). ДНК проба hydssmic4 после тонкой настройки биочипа, была признана наиболее специфичной для детекции бактерий рода Desulfomicrobium с применением технологии биочипов. Структура этой пробы представлена в таблицах 1 и 15.
Для детекции бактерий Desulfobulbus ДНК пробы были разработаны на основании полных нуклеотидных последовательностей hydA гена, кодирующего альфа субъединицу [NiFe] гидрогеназы из Desulfobulbus elongatus DSM 2908 (NZ_JHZB01000004.1), Desulfobulbus mediterraneus DSM 13871 (NZ_KE387007.1) и Desulfobulbus propionicus DCM 2032 (NC 014972.1). Mножественный alignment этих генов использовали для поиска консервативных участков и создания ДНК проб hydfob1 и hydfob2. После тонкой настройки биочипа обе пробы показали одинаковую интенсивность свечения поэтому для детекции бактерий рода Desulfobulbus. можно использовать любую из них. hydfob2 была выбрара для включения в биочип. Cтруктура этой пробы представлена в таблицах 1 и 15.
Группа бактерий рода Desulfococcus – анаэробные термофильные СВБ, относящиеся к Desulfobacterales, которые могут полностью окислять ацетат, используя сульфат в качестве акцептора электронов в анаэробных условиях. Эти бактерии играют важную роль в цикле соединений серы в морской среде. Desulfococcus oleovorans играет важную роль в накоплении сульфидов (Rueter et al., 1994) и был выделен из нефтехранилища. Эта группа преимущественно использует органические субстраты в качестве доноров электронов и тем самым не ускоряет катодную деполяризацию, на основании чего предполагается их меньший вклад в биокоррозию. Группу Desulfococcus можно распознать по обнаружению hypB гена, кодирующего гидрогеназный вспомогательный акцессорный белок HypB. В связи с этим для разработки проб, специфичных для Desulfococcus, были использованы полные нуклеотидные последовательности hypB гена. Полные нуклеотидные последовательности штаммов Desulfococcus oleovorans Hxd3 (CP000859.1), Desulfococcus multivorans DSM 2059 (CP019913.2) были загружены из базы данных NCBI. Применение множественного выравнивания этих последовательностей позволило выявить гомологию, на основании которой были сконструированы ДНК пробы. Проба hypBfoc1 после тонкой настройки биочипа, была признана наиболее специфичной для детекции бактерий рода Desulfococcus с применением технологии биочипов. Структура пробы hypBfoc1 представлена в таблицах 1 и 15.
Дизайн ДНК проб для детекции Desulfuromonadales: Бактерии Desulfuromonas, Desulfuromusa, Pelobacter, Malonomonas, Geobacter относятся к семейству Desulfuromonadales. Изолированные из корродированных стальных трубопроводов и промысловых вод, эти бактерии вовлечены в МИК. Способность к фиксации азота позволяет им эффективно конкурировать с другими бактериями в бедной азотом среде и заселять места с высоким солевым содержанием. nifD ген, кодирующий альфа субьединицу динитрогеназы (NIFD), катализирует фиксацию азота. Этот процесс сцеплен с восстановлением Fe(III) в Geobacteraceae и некоторых видах Desulfuromonadales. nifD ген является полезным маркером для обнаружения различных видов Desulfuromonadales из промысловых вод, связанных с нефтедобычей (Zapata-Peñasco et al., 2013). Полные нуклеотидные последовательности nifD гена, кодирующего альфа субъединицу нитрогеназного Mo-Fe белка, из Desulfuromonas acetexigens 2873 1 (VJVV01000001.1), Desulfuromusa kysingii DSM 7343, (NZ_FNQN01000010.1), Pelobacter carbinolicus DSM 2380 (NC_007498.2) были загружены из базы данных NCBI. Применение множественного выравнивания этих последовательностей позволило выявить гомологию, на основании которой были сконструированы ДНК пробы nifD915 и nifD1333. После тонкой настройки биочипа, пробы nifD915 и nifD1333 были признаны наиболее специфичными для детекции различных видов Desulfuromonadales с применением технологии биочипов. Структуры этих проб представлены в таблицах 1 и 15.
Разработка ДНК проб для детекции метанoгенных археи
Метанoгены – это гетерогенная группа микроорганизмов, которые объединены в одну группу на основании двух общих для всех её представителей свойств: строгий анаэробиоз и способность производить метан. Метанoгены осуществляют карбонатное дыхание, окисляя молекулярный водород, при этом конечным акцептором электронов является CO2. Производимый ими метан используется анаэробными микроорганизмами в качестве топлива. Метаногенные археи, сосуществуя вместе с СВБ, используют водород, накапливающийся на металле в результате катодной деполяризации, и являются активными участниками биокоррозии. Они способны превращать органические субстраты в метан, используя, по крайней мере, четыре различных вида метаболических путей. Виды Methanosarcina, например, представляют наиболее метаболически разнообразную группу археи, которая может использовать водород + CO2, водород с метанолом, СО, метанол, метиламин, метилсульфиды и ацетат в качестве субстратов для метаногенеза (Rother et al., 2005). Общим этапом в этом процессе является восстановление метил-коэнзим М до метана, который катализирует метил-CoM редуктаза.
Функциональный ген (mcrA), кодирующий альфа субъединицу метил-Коэнзим М редуктазы, также называемую коэнзим- B-сульфоэтилтиотрансферразой, катализирует последний этап в метаногенезе и присутствует у всех метаногенных микроорганизмов. На основании вышеизложенного mcrA ген был выбран для разработки ДНК проб, позволяющих детектировать метаногенные археи. Полные нуклеотидные последовательности mcrA гена бактерий Methanosarcina barkeri (NZ_CP009526.1), Methanosarcina barkeri Wiesmoor (NC_007355.1), Methanosarcina mazei zm-15 (CP042908.1), Methanosarcina acetivorans C2A (NC_003552.1), Methanothermobacter thermautotrophicus Delta H (NC_000916.1), Methanothermobacter marburgensis Marburg (NC_014408.1) были загружены из базы данных NCBI. Множественное выравнивание этих последовательностей и поиск гомологичных участков mcrA гена указанных бактерий позволили разработать ДНК пробы mcrA994 и mcrA985. После тонкой настройки биочипа данные пробы были признаны наиболее специфичными для обнаружения метаногенов с применением технологии биочипов и представлены в таблицах 1 и 15.
Разработка ДНК проб для детекции термофильных архей семейства Archaeoglobae Archaeoglobus относятся к термофильным сульфатредуцирующим археям, филум Euryarchaeota. Они обнаружены на нефтяных полях, как вносящие свой вклад в закисление нефти. В анаэробных условиях термофильные Archaeoglobus fulgidus получают энергию, восстанавливая окисленные соединения серы (сульфаты, сульфиты, тиосульфаты) до сероводорода. Оптимальная температура для их роста 85 °C. Известно, что этот археон вызывает коррозию железа и стали в нефтяной и газовой процессинговой системе, продуцируя сульфид железа. А Archaeoglobus profundus растут литотрофно и гетеротрофно, используя ацетат и CO2.
Geoglobus также относится к гипертермофильным археям семейства Archaeoglobaea. В настоящее время известны нуклеотидные последовательности только двух видов – Geoglobus acetivorans и Geoglobus ahangari. Уникальная особенность этих археобактерий заключается в их строгой зависимости от восстановления окислов Fe(III) до магнетита. Для этого они используют прямой контакт с металлом (Manzella, Reguera, Kashefi 2013), кроме того, Geoglobus acetivorans способны использовать ацетат, который является главным продуктом анаэробный деградации органических веществ. Геномный анализ этого микроорганизма показал также наличие метаболических путей для анаэробной деградации ароматических соединений и n-алканов (Mardanov et al., 2015). Почти все анаэробные археи содержат одну или несколько [NiFe]-гидрогеназ, что свидетельствует о важной роли H2 в их метаболических процессах, протекающих в экстремальных условиях (Vignais & Billoud, 2007). В связи с этим hydA ген был выбран для создания ДНК проб для обнаружения археобактерий Geoglobus. Полные нуклеотидные последовательности hydA гена, кодирующего малую субъединицу [Ni/Fe] гидрогеназы Geoglobus acetivorans SBH6 (NZ_CP009552.1) и Geoglobus ahangari 234 (NZ_CP011267.1) были использованы для поиска консервативных участков и конструирования ДНК проб. Проба Geog1090 после тонкой настройки биочипа оказалась наиболее эффективной для обнаружения археобактерий Geoglobus и была включена в биочип. Структура пробы указана в таблицах 1 и 15.
В связи с тем, что археoбактерии Archaeoglobus являются сульфатредуцирующими, aprA ген, кодирующий альфа субъединицу аденозин-5’-фосфосульфат-редуктазы (aprAB), был выбран для дизайна проб для их обнаружения. Полные нуклеотидные последовательности aprAB гена Archaeoglobus fulgidus DSM 4304 (NC_000917.1), A. profundus DSM 5631(NC_013741.1), A. veneficus SNP6 (NC_015320.1), A. sulfaticallidus PM70-1(CP005290.1) были использовали для множественного выравнивания и поиска гомологичных областей, на основании которых были созданы пробы для обнаружения Archaeoglobus. После тонкой настройки биочипа наиболее эффективной для обнаружения археобактерий Archaeoglobus с применением технологии биочипов была признана проба Arg1597. Структура пробы указана в таблицах 1 и 15.
Разработка ДНК проб для детекции Firmicutes бактерий
Firmicutes включают представителей как факультативно аэробных Bacillus, так и анаэробных бактерий Clostridium, которые в процессе своей жизнедеятельности образуют метаболиты, используемые другими членами микробного сообщества, участвующими в МИК. Анализ микробного консорциума на коррозионных сайтах показал, что Proteobacteria, Firmicutes и Bacteroidetes являются доминирующими и составляют до 89% всех микроорганизмов (Xiaohong Li et al., 2017). Среди них до 46% составляют Desulfovibrio spp., Desulfobacter spp. и Desulfotomaculum spp., вызывающие коррозию стали.
Для создания ДНК проб с целью обнаружения некоторых видов Firmicutes были использованы полные нуклеотидные последовательности гена, кодирующего белок биогенеза цитохрома C бактерий Bacillus pumilus 145 (CP027116.1), Bacillus pumilus ZB201701(CP029464.1) и Bacillus pumilus 150a (CP027034.1), а также последовательности гена, кодирующего цитохром C, бактерий Alkalihalobacillus halodurans LB-1 (NZ_CP040441.1) и Bacillus halodurans C-125 (NC_002570.2). После тонкой настройки биочипа наиболее эффективными для обнаружения были признаны пробы bac1620 и bac325. Структуры проб указаны в таблицах 1 и 15.
Разработка ДНК проб для детекции ацетoгенных бактерий
Ацетoгенные бактерии являются строгими анаэробами, способными расти автотрoфнo – используя водород и CO2, и гетеротрофнo – при использовании органических соединений, таких как сахара, спирты, ароматические соединения и C1 (одноуглеродные) соединения, конечным и единственным продуктом их метаболизма является уксусная кислота. Синтез уксусной кислоты происходит при участии фермента ацетил-КоA синтетазы, являющегося ключевым ферментом ацетогенеза (восстановительного ацетил-КоA пути). Создавая кислую среду, ацетoгены способствуют коррозии стали, так, Acetobacterium woodii в присутствии Desulfovibrio vulgaris вызывает более высокий уровень коррозии, поставляя источник углерода (ацетат) для СВБ.
Сообщалось, что последовательности генов формилтетрагидрофолат синтетазы (FTHFS), кодирующих АТФ-зависимую активацию, являются консервативными (Lovell et al., 1990). Этот ген был выбран для создания проб для обнаружения ацетогенных бактерий. Однако найти гомологичные участки в нуклеотидных последовательностях для разнообразных ацетогенов оказалось невозможно, несмотря на большую аминокислотную гомологию. Для поиска гомологичных участков были использованы полные нуклеотидные последовательности fthfs гена Acetobacterium woodii (NC016894.1) и A. woodii (CP002987.1), на основе которых была разработана ДНК проба FTHFS-Ac, показавшая свою эффективность после тонкой настройки биочипа для детекции ацетогенных бактерий.
Для поиска ферментативных бактерий группы Clostridium были разработаны ДНК пробы на основании полных последовательностей fthfs гена штаммов Clostridium perfringens ATCC13124 (NC008261.1), C. perfringens 13 (BA000016.3), C. cylindrosporum (L12465.10), C. paraputrificum AGR2156(NZ AUJC01000009.1) и C. magnum DSM2767 (NZ FQXL01000004.1). После тонкой настройки биочипа была отобрана проба FTHFS-c1, признанная наиболее эффективный для обнаружения этой группы бактерий. Структуры проб FTHFS-Ac и FTHFS-c1 указаны в таблицах 1 и 15.
Разработка ДНК проб для детекции бактерий, деградирующих углеводороды Углеводороды широко распространены в природе и являются потенциальными субстратами для анаэробной деградации микроорганизмами. Их деградация в глубоко залегающих нефтяных резервуарах приводит к ухудшению качества и количества сырой нефти (Rabus et al., 2016). Предполагается, что деградация нефти осуществляется сообществом анаэробных и аэробных бактерий (Da Cruz et al., 2011). Микробное сообщество расщепляет углеводороды, такие как длинноцепочечные алканы, до метана и углекислого газа (Jones et al., 2008). Анаэробные организмы могут окислять метан в морских осадках, содержащих сульфат, уменьшая выброс метана в атмосферу. Предполагается, что метаболизм окисления алканов, спиртов, органических кислот и насыщенных жирных кислот у этих бактерий сцеплен с восстановлением сульфата и их способностью расти химо-лито-автотрофно (Callaghan et al., 2012). Штамм Desulfatibacillum alkenivorans AK-01 способен расти на n-алканах (C13 -C18) и алкенах в сульфат-восстанавливающих условиях (So and Young, 1999), а Geobacter metallireducens GS15 – на бензоате. Активация алканов осуществляется ферментом алкилсукцинат синтазой (glycyl radical enzyme). assA ген, кодирующий алкилсукцинат синтазу, является полезным биомаркером анаэробного метаболизма алканов (Callaghan et al., 2010).
В связи с этим полные нуклеотидные последовательности гена, кодирующего белок глицил-радикал штаммов Desulfatibacillum alkenivorans AK-01 (DQ826035.1) и Geobacter metallireducens GS15 использовали для поиска консервативных участков и разработки на их основе ДНК проб. Проба assA1 после тонкой настройки биочипа была признана наиболее эффективной для обнаружения микробов, осуществляющих анаэробную деградацию алканов. Структура пробы указана в таблицах 1 и 15.
Разработка ДНК проб для детекции нитритредуцирующих бактерий (НРБ)
Нитритредуцирующие (денитрифицирующие) бактерии являются важным звеном в круговороте азота в природе. В большей степени способность к денитрификации распространена у бактерий родов Bacillus и Pseudomonas. В настоящее время показано, что изученные штаммы Pseudomonas из морской среды усиливают и приводят к появлению коррозии на металлах. Конечными акцепторами электронов при нитратном дыхании являются нитраты (NO3-) или нитриты (NO2-). В результате их восстановления образуются газообразные продукты NО, N2O или N2. Ключевые реакции денитрификации осуществляются ферментами нитрит- и N2O-редуктазами. Эти реакции катализируют два структурно различных, но функционально эквивалентных фермента – Cu-редуктаза и цитохром cd1-содержащая редуктаза, которые кодируются генами nirK и nirS соответственно.
В связи с этим полные нуклеотидные последовательности nirK гена, кодирующегo медь-нитрит редуктазу из штаммов Pseudomonas nitroreducens HBP1 (CP049140.1) и P. denitrificans BG1(CP043626.1), а также nirS гена, кодирующегo цитохром cd1-нитрит редуктазу из штаммов Pseudomonas stutzeri A1501 (CP000304), P. stutzeri DSM 4166 (CP002622) и P. stutzeri NCTC10475 (LR134482.1) использовали для поиска консервативных участков и разработки на их основе ДНК проб. После тонкой настройки биочипа пробы nirK2 и nirS были признаны наиболее эффективными для обнаружения нитритредуцирующих бактерий и были включены в биочип.
Для повышения эффективности обнаружения нитритредуцирующих бактерий дополнительно была разработана проба на основании полной нуклеотидной последовательности nirD гена, кодирующего малую субъединицу нитрит редуктазы четырёх видов Pseudomonas stutzeri: штамм SGAir0442 (CP025149), штамм 1W1-1A(CP027664), штамм DW1 (CP027543) и штамм 273 (CP015641). Множественное выравнивание этих последовательностей и поиск гомологичных участков nirD гена указанных бактерий позволил разработать ДНК пробу nirD265Ps. После тонкой настройки биочипа проба nirD265Ps была признана наиболее эффективной для обнаружения нитритредуцирующих бактерий и была включена в биочип. Структуры проб nirK2, nirS и nirD265Ps представлены в таблицах 1 и 15.
Разработка ДНК проб для детекции бактерий семейства Enterobacteriaceae
Коррозия металлов в морских биоплёнках может быть инициирована бактериями, принадлежащими к семейству Enterobacteriaceae (Videla et al. 1996). Члены этого семейства, принадлежащие к γ-протеобактериям, вовлечены в коррозию металла в морской среде (Bermont-Bouis et al., 2006). Авторами изобретения также было показано, что ряд факультативных анаэробов способствует росту СВБ, приводящему к повышению на 3–4 порядка продукцию сероводорода.
Ген hybO Escherichia coli, кодирующий малую субъединицу гидрогеназы 2, использовали для создания проб для детекции энтеробактерий. Полные нуклеотидные последовательности этого гена из штаммов E. coli K12 MC4100 (HG738867.1), E. coli JE86-ST05 (AP022815.1), E. coli O25b:H4 (CP015085.1), E. coli RH-045-MS (NS_CP050203.1) и E. coli O157:H7 Sakai (NC_002695.2) были загружены из базы данных NCBI. Для поиска гомологичных структур, на основании которых в дальнейшем были созданы пробы использовалось множественное выравнивание этих последовательностей. После тонкой настройки биочипа проба hybOE784 была признана наиболее эффективной для обнаружения энтеробактерий и была включена в биочип.
Для повышения эффективности обнаружения энтеробактерий дополнительно была создана модифицированная проба (путем введения в известную пробу замены одного нуклеотида, что позволило повысить эффективность обнаружения энтеробактерий) uidA2m к гену uidA, кодирующему β-D- глюкуронидазу, на основе опубликованной пробы к этому гену (Tantawiwat S et al., 2005).
Кроме того, также была разработана проба на основе hybO гена, кодирующего малую субъединицу гидрогеназы 2, для обнаружения бактерий рода Hafnia. Полные нуклеотидные последовательности этого гена из штаммов Hafnia alvei CBA 7135 (CP021971.1), H. alvei PCM 1220 (CP036514.1) и H. paralvei FDAARGOS_158 (CP014031.2), извлеченные из базы данных NCBI, использовали для поиска консервативных участков. Множественное выравнивание этих последовательностей показало большую гомологию, на основании которой были разработаны ДНК пробы. Проба hybOH36y после тонкой настройки биочипа была признана наиболее эффективной для обнаружения бактерий, принадлежащих к семейству Enterobacteriaceae, способствующих росту СВБ, и была включена в биочип. Структуры проб hybOE784, uidA2m, hybOH36y указаны в таблицах 1 и 15.
Структурные пробы к генам 16S рРНК для детекции суммарных бактерий (16SCons) и суммарных археи (16SArch) были использованы из литературных источников (Muyzer et al., 1993; Loy et al., 2002).
Таблица 1. Список сконструированных проб
гидрогеназы
(глицил-радикал белок)
гидрогеназы 2
гидрогеназы 2
Обозначения: Y=T/C, B=G/T/C, W=A/T, M=A/C, S=G/C; * - пробы, известные ранее.
Номер (№) последовательности в таблице 1 соответствует ее номеру в Перечне последовательностей (SEQ ID NO) согласно Стандарту ST.26, являющемуся частью настоящего описания изобретения. В случае наличия разночтений в структуре последовательностей между таблицей 1 и соответствующей последовательностью в Перечне последовательностей согласно стандарту ST.26, приоритетными являются данные, представленные в таблице 1.
2. Последовательность операций способа технического решения
2.1. Получение образца и выделение геномной ДНК
В качестве образца может быть использована, например, вода из нефте/ газораспределительной системы, соскобы из внутренней поверхности труб. Для выделения геномной ДНК из клеток микроорганизмов используют известные методики (например, описанные в патенте RU2177035). Количество и качество выделенной ДНК оценивают спектрофотометрически, более конкретно, количество выделенной ДНК оценивают по оптической плотности раствора при длине волны 260 нм (А260), а качество - по соотношению оптических плотностей А260/А280, которое для чистых растворов нуклеиновых кислот должно быть равным 1.8–1.9.
2.2. Амплификация геномной ДНК
Амплификацию геномной ДНК осуществляют методом изотермической амплификации со множественным замещением цепи (MDA – Multiple displacement amplification). Для этого 10 нг высокомолекулярной ДНК смешивали с буфером и случайными гексамерными праймерами, представляющими собой смесь олигонуклеотидов всех возможных структур для этого размера, денатурировали при 95°C в течение 3 минут и немедленно помещали на лед. После охлаждения в течение 5 минут добавляли 10 единиц ДНК-полимеразы phi29 (NEB), объем доводили до 50 мкл, смесь инкубировали при 30°C в течение 4 часов. Реакцию останавливали прогреванием при 65°C в течение 10 минут. Амплифицированная таким образом ДНК пригодна для фрагментации ДНКазой I без дополнительной очистки. Выход ДНК составлял порядка 20 мкг.
2.3. Фрагментация амплифицированной ДНК
Фрагментацию ДНК проводили ДНКазой I. K 10 мкл амплифицированной ДНК добавляли 1 мкл разведённой в соотношении 1:20 ДНКазы I, 4 мкл 1x ДНК-азного буфера и инкубировали при 37°C в течение 20 минут. Время инкубации выбирается экспериментально, так чтобы пул получаемых фрагментов был в размере 50-200 пар оснований, при более длительной инкубации ДНК полностью разрушается. Реакцию останавливали прогреванием при 65°C.
2.4. Флюоресцентное мечение ДНК
Реакция проводилась в объёме 50 мкл. К 1 мкг фрагментированной ДНК добавляли dH2O и случайные октамеры (октамерные праймеры). ДНК денатурировали прогреванием (95°C 5 мин), быстро охлаждали во льду для отжига праймеров, после чего добавляли 5 мкл 10х буфера, по 200 мкмолей дАТФ, дТТФ, дГТФ, 180 мкмолей дЦТФ и 20 мкмолей сульфо-Cy5TM-дУТФ (флюоресцентная метка) и 50 единиц Exo-Klenow ДНК-полимеразы. Флуоресцентная метка встраивается ДНК-полимеразой в растущую цепь ДНК в ходе ПЦР. Реакцию проводили в течение 2 часов при 37°C, останавливали добавлением стоп-буфера (0.5 M ЭДТА pH 8.0). Полученную флюоресцентно меченую ДНК очищали на колонках, элюировали с колонок в объеме 50 мкл и хранили в защищенном от света месте в тёмных пробирках замороженной при -20°C. Выход ДНК после мечения оценивали по формуле: ДНК (мкг) = (A260-A320) x 50 мкг/мл x объем (мл). Включение Cy5TM метки в ДНК оценивали по формуле: Cy5TM (пмол) = (A650-A750)/0.25 x объем (мкл).
2.5. Заливка меченой ДНК на биочип и гибридизация с элементами биочипа
Гибридизацию олигонуклеотидных зондов, иммобилизованных в ячейках биочипа, с флюоресцентно мечеными ДНК проводили в объеме 30 мкл. Флюоресцентно меченый образец (2 мкл) смешивали с 7.5 мкл гибридизационного буфера, содержащего 4M GuSCN (гуанидин тиоцианат), 300 ммоль HEPES, pH 7.5, 30 ммоль ЭДТА, объем доводили до 30 мкл деионизованной водой, после чего образец заливали в гибридизационную камеру, содержащую биочип, и инкубировали при 25°C в течение 18 часов. Для удаления неспецифического связывания биочип отмывали при перемешивании по 30 сек однократно буфером 4 x SSC (standard saline citrate, цитратно-солевой буфер), 7.2% натрий лауроилсаркозина (саркозил), затем буфером 0.2 x SSC.
2.6. Сканирование биочипа лазером
Визуализацию флюоресцентного изображения биочипа при возбуждении лазерами с длинами волны 532 нм и 655 нм осуществляли на приборе ImaGeWare в ООО "БИОЧИП-ИМБ", используя программное обеспечение «ImaGeWare». Данные флюоресценции хранили в Excel формате.
2.7. Анализ полученных данных
Наличие искомого микроорганизма определяется по сигналу соответствующего зонда. Количественное определение микроорганизмов может осуществляться по интенсивности сигнала (свечения) соответствующего зонда. Реализация анализа данных посредством программно-аппаратного комплекса позволяет проводить автоматическую регистрацию и интерпретацию результатов посредством регистрации картины гибридизации и вычисления сигнала в каждой ячейке и выдавать отчет о присутствии в исследуемом образце специфичных молекулярных мишеней, информирующих о наличии искомых микроорганизмов.
3. Разработка синтеза проб (зондов) и кассет для тонкой настройки лабораторного образца биочипа
3.1. Синтез олигонуклеотидов и ДНК кассет для лабораторного образца.
При разработке синтеза проб учитывалась возможность введения модификации как с 5', так и с 3'-конца. Введение модификации с 3'-конца усиливало эффективность ковалентного связывания с матриксом биочипа, поэтому для иммобилизации на биочипе все пробы были разработаны с введением модификации в виде C6-спейсера со свободной аминогруппой на 3'-конец. Спейсерный участок уменьшает стерические препятствия при иммобилизации зонда на подложке, тем самым способствуя гибридизации. Были синтезированы 62 олигонуклеотидные пробы для иммобилизации на биочипе (таблица 1).
Для тонкой настройки биочипа были разработаны 11 комплементарных одноцепочечных ДНК кассет длиной от 112 до 134 нуклеотидов. Для их конструирования использовались нуклеотидные последовательности, комплементарные пробам. Кассеты были составлены из комплементарных блоков так, чтобы их длина не превышала 134 нуклеотида. Порядок блоков может варьировать в зависимости от целей анализа. При синтезе кассет на 5'-конец вводилась флюоресцентная метка. В качестве флюорофора использовали Cy5, который обладает большим квантовом выходом по сравнению с Cy3. Композиция кассет представлена ниже.
Кассета 1 длиной 131 нуклеотид предназначена для детекции бактерий Shewanella и Geobacter, содержит шесть проб (FeoB2, FeoB3, FeoB4a, FeoB4b, MtrB1, gltA-4B) к генам feoB, mtrB, gltA:
ATGGGCATAGAGCAGGATAACTGTCGATGTTAGTGGCATCGACGCAGGCCAGTTATCTTGCTCTAGGCAGTGAGCGTGAGCGTATAGACAAGGTATCACTGGCGACTACGGAGTGVAGGATGAAGTACATC (SEQ ID NO: 64).
Кассета 2 длиной 134 нуклеотида предназначена для детекции бактерий Archaeoglobus, Geoglobus и ацетогенных бактерий, содержит шесть проб (aprArg1597, Arg830, Geog1090, FTHFS-Ac, FTHFS-c1, FTHFS-c2) к генам aprA, hydA, fthfs:
TCGTCCATGATCTTCTGSAGAGGAACCATGCACCGACTGTGTAGTGGTGTATCAGCGTRCCGCCATTATTTCTGATGCAACAGTGGCCATTATTTCTGAWGCAACWGTGGACGGTCATTCATGTCAACACAACG (SEQ ID NO: 65).
Кассета 3 длиной 132 нуклеотида предназначена для детекции углеводород деградирующих и нитрит редуцирующих бактерий (НРБ), содержит шесть проб (nirK2, nirS, nirD256Ps, nirD233, assA2H, assA1H) к генам nirK2, nirS, nirD, assA:
AGGCCGTGGCGGATGCACTCAAATCGTCATTGCCCCATTTGGGACATCGCGATCTTCCGCACCTCAAGTGCTGCTTGTACAGCGGTGGGTCTTGACGAACAGGTTGCCTCGATCACCATGCGGAACTGCACG (SEQ ID NO: 66).
Кассета 4 длиной 132 нуклеотида предназначена для детекции Mетанoгенных архебактерий и Desulfovibrio видов, содержит шесть проб (mcrA992, mcrA994, mcrA985, aprA vib1, aprA vib2, aprA vib42) к генам mcrA, aprA:
GTATTGATGGCGGAVAGGCCCTGGGAGTTCACGAAGCACTTCCACTTGCACCGTTGATCATGATCTGCCAGAGCCAGATCTGGTCGTAGAGACATGTAGGAWCCGAGCCAGACATGTAKGAKCCGAGCCAGA (SEQ ID NO:67).
Кассета 5 длиной 130 нуклеотидов предназначена для детекции Enterobacteriacea, содержит шесть проб (hybOE156, hybOE784, uidA2m, hybO H754, hybO H36y, hybO H36) к генам hybO и uidA:
CTTCATGAAGTCACGACGGTTCCTTCATGAARTCACGACGGTTCAGGTGATACAAACACCAGCCTTGGCCGTTTTCGTCGGTAATCACCATTGCAGCGTTGAGCAGTTGCACTCCTGCGCGCCAATCCAG (SEQ ID NO: 68).
Кассета 6 длиной 130 нуклеотидов предназначена для детекции Desulfococcus и Desulfobacter, содержит шесть проб (hypBfoc1, hypBfoc2, nifHbac1, nifHbac2, nifHbac3, nifHbac4) к генам hypB и nifH:
TCTCCAAAGAAGAGCATGGGACCCTTGCAGATGTTGTTGGCTACCCTTGCAGATGTTGTTGGCATRCCCTTGCAGATGTTGTTGGCATGGGCCGCCAGGTGGCAGTCACATGGGCCGCCAGGTGGCAGTC (SEQ ID NO: 69).
Кассета 7 длиной 127 нуклеотидов предназначена для детекции Desulfotomaculum, Desulfobacterium и нитрат редуцирующих микроорганизмов, содержит шесть проб (hydsstom27, dsrAtom709, dsrAtom710, dsrAtom883, hynBbam, nirK1) к генам hydss, dsrA, hynB, nirK:
TCCTCGATCACCATGCGGAACTGCACGTAGGCGCGSACCGCCATCTCTTGATGCAGTGCATGCAACGACGTTGATGCAGTGCATGCACGTTGATGCAGTGCATGCAAAGCCCCGCCTTGATACTGAT (SEQ ID NO: 70).
Кассета 8 длиной 129 нуклеотидов предназначена для детекции Desulfomicrobium/ Clostridium групп Firmicutes, содержит шесть проб (hydAmic1, hydssmic2, hydssmic3, hydssmic4, hydssmic5, FTHFS-c3) к генам hydA, hydss, fthfs:
CCATTATTTCTGATGCAACAGTCAACTGGTGTGTCGAGAACGCATCCGGACTGGATGGTCGGATCCGGACTGGATGGTCGGCACAGTCTTAGCAGACGTGAGTTCGTCGTCTGG TTGAACTTGACCTTG (SEQ ID NO: 71).
Кассета 9 длиной 111 нуклеотидов предназначена для детекции бактерий Firmicutes классов Bacillus/Clostridium, содержит шесть проб (Bac1618, Bac1620, Bac325, hydfob1, hydfob2) к гену, кодирующему цитохром C, и гену hydD:
TAGTGGTGGATCAGGGTGCCTAGTGGTGGATCAGGGTGCCGATCGCATCTGCCTCTGCATCATCTGCTGATACTTGTTGTATCAGGCTGCTGATACTTGTTGTATCAGGCG (SEQ ID NO: 72).
Кассета 10 длиной 132 нуклеотидa предназначена для детекции Desulfuromonadales, E. coli, СВБ, Geobacter, содержит пять проб (nifD1333, nifD915, hybOE783, aprAvib3, hyaLGeob) к генам nifD, hybO, aprA, hyaL:
GGATGGCGTGGATCTTGATGAGAGTTCACGAAGCACTTCCACTTGGTATTGATGGCGGACAGGCCCTGGTTGCAGCGTTGAGCAGTTGCCATCTTKGTSGGWCCGAAGAAGTGTGTGCATCTGKCGGAAGGG (SEQ ID NO: 73).
Кассета 11 длиной 105 нуклеотидов предназначена для детекции Total Bacteria, Total Archaea, Geobacter, содержит пять проб (16SCons, 16SArch, gltA-4C, gltA-4T, gltA-4G) к генам 16S rRNA бактерий и 16S rRNA археи, gltA:
ATGAATTGGCGGGGGAGCACCTGCTGCCTCCCGTAGGCGGAGTGCAGGATGAAGTACATCCGGAGTGGAGGATGAAGTACATCCGGAGTGAAGGATGAAGTACAT (SEQ ID NO: 74).
3.2. Нанесение олигонуклеотидов на матрицу лабораторного образца биочипа и заливка синтезированных ДНК кассет для гибридизации
Одним из ключевых компонентов биочипа является его матрикс. Элементы биочипа могут быть сформированы посредством нанесения водных растворов олигонуклеотидов непосредственно на подложку, с получением варианта так называемого «планарного» биочипа (например, согласно Matson et al., 2009), либо представлять собой массив трехмерных гидрогелевых ячеек полусферической формы, расположенных на подложке из стекла или пластика и содержащих ковалентно иммобилизованные олигонуклеотиды. Такой биочип может быть изготовлен, например, методом сополимеризационной иммобилизации (патенты RU2216547, ЕР1437368; Rubina et al., 2004). Для получения матрицы биочипа растворы олигонуклеотидных зондов смешивают с гелеобразующими добавками. Капли смеси наносят на поверхность полимерной подложки будущего чипа с помощью робота и облучают ультрафиолетовым светом, под действием которого идет полимеризация. В ходе реакции молекулярные зонды присоединяются к растущим полимерным цепям геля и в итоге равномерно распределяются по всему объему ячейки. При разработке биочипов согласно изобретению в качестве матрикса использовались трехмерные гидрогелевые ячейки. Макропористая структура гидрогеля образуется в результате сополимеризации мономера – производного метакриловой кислоты, с иммобилизованными молекулами ДНК-зонда, что приводит к равномерному распределению молекул во всем объеме гелевой подушечки. Иммобилизация ДНК в 3D гидрогеле в 10 - 100 раз повышает чувствительность и эффективность детекции анализируемого генома (Gryadunov et al., 2018). Кроме того, гель – насыщенное водой желеобразное вещество, исключает возможность взаимодействия зондов друг с другом и с твердой поверхностью подложки, а также обеспечивает отличную изоляцию отдельных ячеек на биочипе.
Биочип представляет собой миниатюрную платформу ячеек, содержащих ДНК-пробы-зонды, которые специфичны к различным участкам анализируемого генома. Наличие на биочипе большого количества ДНК-проб обеспечивает одновременное тестирование множества геномных мишеней, “тем самым реализуя принцип многопараметрического анализа биологического образца (Gryadunov et al., 2018). Это позволяет использовать биочипы в качестве высокоэффективного инструмента для одновременного выявления большого разнообразия микроорганизмов, вовлеченных в биокоррозию.
Лиофилизированные олигонуклеотидные пробы-зонды растворяли в 3 мкл деионизованной воды и смешивали в соотношении 1:10 с компонентами гидрогеля. В результате исходные растворы всех ДНК проб были получены в различных концентрациях, что позволило легко выбрать оптимальное количество материала на каждый спот на биочипе для получения одинаковой интенсивности сигнала (Таблицы 3–14). ДНК пробы в дубликате наносились на подложку в автоматическом режиме (Genetix Qarray2) с использованием пина диаметром 150 микрометров. Объем наносимого материала составлял 0.88 нанолитров. Пробы были иммобилизованы в миниатюрные трёхмерные гелевые подушечки на пластиковой подложке методом фотоиндуцируемой совместной полимеризации молекулярных зондов и компонентов акриламидного геля (Nasedkina et al., 2009). В результате был получен биочип, содержащий 62 олигонуклеотидные пробы, сконструированные к специфическим генам микроорганизмов, вовлеченных в биокоррозию. Расположение проб на гриде представлено в Таблице 2, все пробы нанесены в дубликате.

4.1. Tонкая настройка биочипа с использованием ДНК кассет
Для тонкой настройки биочипа были использованы 11 сконструированных кассет, содержащих Cy5 флюоресцентный краситель на 5’ конце. В таблице 3 представлена композиция проб во всех разработанных кассетах. Каждую из 11 кассет гибридизовали с индивидуальным образцом биочипа. Количество гибридизационного материала рассчитывалось для каждой кассеты индивидуально, исходя из эквимолярного соотношения ДНК кассет и суммарного количества ДНК проб на биочипе, которые должны гибридизоваться с данной кассетой при 50% эффективности связывания ДНК с матриксом (Nasedkina et al., 2009).
Таблица 3. Композиция проб в кассетax 1–11
Desulfobulbus
gltA-4B* – “B” в названии обозначает вырожденную последовательность ДНК, комплементарную для трёх ДНК проб - gltA-4T, gltA-4C и gltA-4G
4.1.1. Гибридизация и тонкая настройка биочипа с кассетой 1.
Количество всех ДНК проб, доступное для гибридизации с кассетой 1, было рассчитано и представлено в таблице 4. Кассета 1 была синтезирована в количестве 0.39 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.85 пмоль/мкл, кассета была растворена в 211 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 4).
Tаблица 4. Расчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 1. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Kассета 1 содержит 4 пробы к гену FeoB и 1 пробу к гену MtrB для детекции различных штаммов бактерий рода Shewanella, а также 1 пробу к гену gltA для детекции бактерий рода Geobacter. Каждая из проб для Shewanella разработана для детекции различных групп (видов) этих бактерий и позволяет диагностировать наличие всех возможных Shewanella в нефтепромысловых образцах.
Результаты гибридизации и эффективности флюоресцентного сигнала представлены на Фиг.2. Как видно на Фиг.2, все пробы, за исключением FeoB4a (позиция A5-A6), MtrB1 (позиция A9-A10) и gltA-4T (позиция B1-B2), показали примерно одинаковую интенсивность флюоресцентного свечения. Наименьший уровень гибридизации показала проба FeoB4a (позиция A5-A6), чуть больше была у пробы MtrB (позиция A9-A10). Из таблицы 4 видно, что эти пробы имеют намного меньшее количество ДНК в каждом пятне. В связи с этим, для выравнивания свечения с другими ДНК пробами, нужно повысить эффективность свечения этих проб добавлением большего количества ДНК на маткрикс биочипа, например до 0.25-0.3 пикомолей по сравнению с указанным в таблице 4.
Пробы gltA-4T, gltA-4C и gltA-4G были разработаны для детекции одной и той же группы бактерий (таблица 4). Небольшие различия были сделаны для поиска наиболее подходящей пробы, дающей при гибридизации примерно одинаковое по интенсивности свечение, сравнимое со свечением других проб. Анализ гибридизаций 4 и 11 и иx сравнение с гибридизацией 1 показали, что эти три пробы имеют очень похожие уровни интенсивности флюоресцентного свечения (Фиг.2 и 12 позиции B1-B2, B3-B4, B5-B6). Проба gltA-4T имеет наименьшую интенсивность свечения. В связи с этим в дальнейшем пробу gltA-4T можно исключить из нанесения на маткрикс биочипа. Проба gltA-4C в свою очередь имеет самую высокую эффективность гибридизации, что позволяет в дальнейшем использовать только её одну.
4.1.2. Гибридизация и тонкая настройка биочипа с кассетой 2.
Количество всех ДНК проб, доступное для гибридизации с кассетой 2, было рассчитано и представлено в таблице 5. Кассета 2 была синтезирована в количестве 0.67 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 0.97 пмоль/мкл, кассета была растворена в 649 мкл dH2O. 1 мкл базового раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 5).
Tаблица 5. Pасчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 2. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
Кассета 2 содержит три ДНК пробы к гену формилтетрагидрофолат синтетазы FTHFS для детекции ацетогенных бактерий: проба FTHFS-Ac – для детекции Acetobacterium, пробы FTHFS-c1, FTHFS-c2 – для детекции бактерий Clostridium, проба Geog1090 к гену hydA [NiFe] гидрогеназы – для детекции рода Geoglobus, а также пробы Arg830, Arg1597, к гену aprA аденозин-5’-фосфосульфат редуктазы для детекции рода Archaeoglobus.
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.3. Как видно на Фиг.3, все шесть проб - зондов FTHFS-Ac, FTHFS-c1, FTHFS-c2, Geog1090, Arg830, Arg1597, иммобилизованные на биочипе, гибридизуются с кассетой 2 и показали довольно высокую и примерно одинаковую интенсивность флюоресцентного свечения. Все эти пробы были разработаны для детекции различных архей, вовлечённых в биокоррозию и растущих при экстремально высоких температурах, с последующим выбором после тонкой настройки наиболее подходящей пробы.
Пробы FTHFS-c1 (позиции B9-B10) и FTHFS-c2 (позиции C1-C2) демонстрируют флюоресцентные сигналы слегка слабее, чем проба FTHFS-Ac (позиции B7-В8). Кроме того, кассета гибридизуется ещё с тремя пробами на биочипе. Одна из них – проба FTHFS-c3 (позиции I9-I10), которая представляет собой укороченный на два нуклеотида вариант проб FTHFS-c1 и FTHFS-c2, все пробы находятся на биочипе в примерно равном количестве. Несмотря на то, что кассета 8 имеет полностью комплементарную последовательность к пробе FTHFS-c3 (позиции I9-I10), эффективность гибридизации выше с пробами FTHFS-c1 (позиции B9-B10) и FTHFS-c2 (позиции C1-C2) (Фиг.13). Также следует отметить, что пробы FTHFS-c1 (позиции B9-B10) и FTHFS-c2 (позиции C1-C2) имеют практически идентичную интенсивность свечения (Фиг.3 и 9). В тоже время, проба FTHFS-c1 имеет вырожденную последовательность ДНК, что позволяет ей узнавать большее количество различных штаммов бактерий. Таким образом, в дальнейшем, при конструировании окончательного варианта диагностического биочипа, пробы FTHFS-c2 и FTHFS-c3 можно не включать.
Кассета 2 содержит комплементарную последовательность к пробе Geog1090, но на изображении биочипа, иллюстрирующего результаты гибридизации с кассетой (Фиг.3 A, B), также хорошо видно, что она довольно эффективно гибридизуется с пробами hydfob 1 (позиции K7-K8) и hydfob2 (позиции K9-K10). Расчёт интенсивности флюоресцентныx сигналов указанных проб показал, что она только в 2.5–3 раза меньше, чем интенсивность сигнала с пробой Geog1090 (Фиг.3 B). Пробы hydfob1, hydfob2 сконструированы к гену, кодирующему альфа субъединицу [NiFe] гидрогиназы Desulfobulbus, и oни отличаются только на один нуклеотид, проба hydfob2 более короткая. Сравнение последовательностей проб hydfob1, hydfob2 с последовательностью кассеты 2 показало 85.7% идентичности с пробой Geog1090, входящей в состав кассеты 2, что и объясняет такой уровень гибридизации ДНК (Pairwise Sequence Alignment, LALIGN – ebi.ac.uk). Для большего уменьшения свечения или устранения видимой гибридизации вообще можно увеличить температуру гибридизации на 5-10°C.
Пробы Arg830 и Arg1597, сконструированные к различным областям гена, кодирующего альфа-субьединицу aprA редуктазы, дают примерно одинаковый сильный флюоресцентный сигнал. Pасчёт интенсивности флюоресцентныx сигналов указанных проб показал, что разница составляет не более 8% (Фиг.3 B). Таким образом, при формировании биочипа, в зависимости, например, от себестоимости биочипа и его характеристик, возможно использование как одной из этих проб, так и обеих одновременно.
4.1.3. Гибридизация и тонкая настройка биочипа с кассетой 3.
Kоличество всех ДНК проб, доступное для гибридизации с кассетой 3, было рассчитано и представлено в таблице 6. Kассета 3 была синтезирована в количестве 0.8 наномолей, Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.11 пмоль/мкл, кассета была растворена в 720 мкл dH2O. 1 мкл базового раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 6).
Tаблица 6. Pасчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 3. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Нитрит редуцирующие
Кассета 3 содержит две ДНК пробы assA1 и assA2 к гену алкил-сукцинат синтазы для детекции углеводород деградирующих бактерий, и 4 пробы nirD233, nirK2, nirS, nirD265Ps для детекции нитрит редуцирующих бактерий. Пробы сконструированы к генам различных нитрит редуктаз, продукты которых являются ключевыми ферментами в диссимиляционном пути денитрификации: nirD ген, к малoй субъединице нитрит редуктазы, nirK - к гену Cu нитрит редуктазы и nirS - к гену цитохром cd1 содержащей редуктазы. Гены nirK и nirS никогда не встречаются вместе в одной бактерии.
Все эти пробы, за исключением assA2 (позиция D1-D2) и hydssmic3 – (позиции J5-J6), показали примерно одинаковую интенсивность флюоресцентного свечения (Фиг.8). Пробы assA1 и assA2 разработаны для детекции и идентификации одной и той же группы микроорганизмов для выбора наиболее подходящей пробы после тонкой настройки биочипа. Эффективность гибридизации кассеты 3 с пробой assA1 (позиции C9-C10) намного выше, чем с пробой assA2 (позиции D1-D2). Pасчёт интенсивности флюоресцентныx сигналов указанных проб показал, что у пробы assA1 он более чем в три раза выше (Фиг. 4 B). Таким образом, несмотря на то, что обе пробы специфичны, для уменьшения себестоимости биочипа целесообразнее включать в него пробу assA1.
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.4. Все комплементарные ДНК пробы, находящиеся на биочипе, гибридизуются с кассетой 3 и показали примерно одинаковую эффективность флюоресцентного свечения. Пробы nirD233, nirK1 и nirK2 также разработаны для детекции и идентификации одной и той же группы нитрит-редуцирующих микроорганизмов. Проба nirK2 представляет собой укороченную на 3 нуклеотида последовательность nirK1. Поэтому неудивительно, что кассета 3 с очень высокой эффективностью гибридизуется с пробой nirK1 (позиции D5-D6), находящейся на биочипе. Pасчёт интенсивности флюоресцентныx сигналов показал, что у пробы nirK2 он самый высокий, поэтому в будущем только пробу nirK2 целесообразно включать в новый биочип.
Кассета 3 также гибридизуется на биочипе с невысокой эффективностью с пробой hydssmic3 (позиции J5-J6), хотя на кассете и отсутствует комплементарная ДНК к этой пробе. Расчёт интенсивности флюоресцентныx сигналов показал, что эффективность свечения примерно в 10 раз меньше, чем производят другие пробы. Флюоресцентный сигнал этой пробы обусловлен тем, что она имеет 22% идентичности с ДНК кассеты, что было обнаружено сравнением последовательностей их ДНК (Pairwise Sequence Alignment, LALIGN – ebi.ac.uk). Для устранения неспецифической гибридизации нужно увеличить температуру гибридизации на 5-10°C.
4.1.4. Гибридизация и тонкая настройка биочипа с кассетой 4.
Количество всех ДНК проб, доступное для гибридизации с кассетой 4, было рассчитано и представлено в таблице 7. Кассета 4 была синтезирована в количестве 0.62 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.41 пмоль/мкл, кассета была растворена в 439 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 7).
Tаблица 7. Расчёт количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 4. Объем наносимой пробы, составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Кассета 4 содержит три ДНК пробы McrA992, McrA994, McrA985, сконструированные к гену альфа субъединицы метил коэнзим М редуктазы для детекции метаногенных Archaea, и три ДНК пробы aprAvib1, aprAvib2 и aprAvib4 к гену аденозин-5’-фосфосульфат редуктазы aprA для детекции бактерий рода Desulfovibrio – представители этого рода являются наиболее часто встречающимися бактериями, вовлечёнными в биокоррозию. Пробы для детекции одного и того же гена и принадлежащего к одной и той же группе микроорганизмов разрабатывались авторами изобретения в нескольких версиях для того, чтобы при тонкой настройке биочипа в случае неудачных вариантов можно было бы легко выбрать другой вариант. Такой подход значительно сокращает время и стоимость изготовления биочипа.
Результаты гибридизации и эффективности флюоресцентного сигнала представлены на Фиг.5. Все комплементарные ДНК пробы, находящиеся на биочипе, гибридизуются с кассетой 4, и показали примерно одинаковую интенсивность флюоресцентного свечения, за исключением пробы aprAvib1 (Фиг.5, позиции E7-E8), которая имеет на представленном биочипе самую низкую эффективность свечения – 11341 пикселей. Пробы aprAvib4 и aprAvib3 имеют идентичную нуклеотидную последовательность за исключением одного вырожденного нуклеотида в пробе aprAvib4 (5'CCA GGG CCT BTC CGC CAT CAA TAC, где B = T/C/G), поэтому кассета 4 также узнает на биочипе пробу aprAvib3 (Фиг.5, позиции L1-L2), хотя ее флюоресцентный сигнал и слабее. На основании изложенного, пробы aprAvib1, aprAvib2 и aprAvib3, показавшие самые низкие интенсивности флюоресценции, было принято решение не включать в финальный вариант биочипа, а включить только aprAvib4.
Пробы McrA994 (TGG CTC GGW TCC TAC ATG TC, где W = A/T) и McrA992 (5' TC TGG CTC GGM TCM TAC ATG TC, где M = A/C) имеют идентичную нуклеотидную последовательность за исключением нескольких вырожденных нуклеотидов в каждой из них, и проба McrA994 короче на два нуклеотида. Более сильный сигнал McrA994 по сравнению с McrA992 можно объяснить в полтора раза большим количеством ДНК этой пробы в пятне (таблица 7). Проба Mcr985 сконструирована к другой области гена альфа субъединицы метил- коэнзим-М редуктазы и узнаёт более широкий спектр бактерий, чем проба McrA994. Поэтому из разрабатываемого биочипа целесообразно исключить пробу McrA992.
4.1.5. Гибридизация и тонкая настройка биочипа с кассетой 5.
Количество всех ДНК проб, доступное для гибридизации с кассетой 5, было рассчитано и представлено в таблице 8. Кассета 5 была синтезирована в количестве 0.76 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания, для получения раствора с концентрацией 1.17 пмоль/мкл, кассета была растворена в 650 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 8).
Tаблица 8. Pасчёт количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 5. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.6.
Кассета 5 содержит последовательности ДНК, комплементарные последовательностям пяти ДНК проб для детекции Enterobacteriaceae, Hafnia и E. coli (таблица 8). Кассета гибридизуется со всеми этими ДНК пробами на биочипе примерно с одинаковой эффективностью.
Проба uidA2m к гену β-Д-глюкуронидазы была сконструирована для обнаружения E. coli, и являясь высоко специфичной для её детекции, также может детектировать, но с меньшой эффективностью, бактерии рода Shigella (E-value 0.033). Представители этих двух видов бактерий индуцируют рост сульфат восстанавливающих бактерий и на несколько порядков усиливают продукцию сероводорода, значительно повышая уровень биокоррозии. Высокая эффективность свечения, 36182 пикселей, указывает на необходимость её включения в финальную версию биочипа.
Пробы hybOE156 и hybOE784 к гену малой субъединицы гидрогеназы 2, сконструированные для детекции некоторых видов энтеробактерий, демонстрируют примерно одинаковую интенсивность гибридизации с кассетой 5. Эта кассета также демонстрирует одинаковую интенсивность гибридизации с пробой hybOE783, находящейся на биочипе. Обе пробы имеют практически идентичную последовательность, hybOE784 короче на один нуклеотид на 5’- конце. Поэтому для включения в диагностический биочип для детекции энтеробактерий можно использовать любую из этих проб.
Пробы hybOH754, hybOH36y, hybOH36 к гену малой субъединицы гидрогеназы 2, были сконструированы для детекции Hafnia. Все эти пробы проявляют примерно одинаковую эффективность флюоресцентного свечения, имеют одинаковую длину, а нуклеотидная последовательность пробы hybOH36y отличается от пробы hybOH36 только тем, что имеет один вырожденный нуклеотид 5' GAA CCG TCG TGA YTT CAT GAA G, где Y = T/C. Проба hybOH36y имеет сравнимый уровень свечения с другими пробами и была выбрана для окончательной версии биочипа.
4.1.6. Гибридизация и тонкая настройка биочипа с кассетой 6.
Количество всех ДНК проб, доступное для гибридизации с кассетой 6, было рассчитано и представлено в таблице 9. Кассета 6 была синтезирована в количестве 0.55 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.42 пмоль/мкл, кассета была растворена в 387 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 9).
Таблица 9. Расчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 6. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.7.
В кассете 6 находятся две ДНК-пробы hypBfoc1 и hypBfoc2 к гену вспомогательного белка гидрогеназы HypB для детекции рода Desulfococcus и 4 ДНК-пробы nifHbac1, nifHbac2, nifHbac3, nifHbac4 к гену Fe гидрогеназы для детекции бактерий рода Desulfobacter. Кассета гибридизуется со всеми комплементарными ДНК пробами на биочипе примерно с одинаковой эффективностью. Расчёты показали, что для окончательной версии биочипа для получения близкого уровня свечения всех проб более всего подходят пробы hypBfoc1, nifHbac2 и nifHbac4.
4.1.7. Гибридизация и тонкая настройка биочипа с кассетой 7.
Количество всех ДНК проб, доступное для гибридизации с кассетой 7, было рассчитано и представлено в таблице 10. Кассета 7 была синтезирована в количестве 0.77 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.36 пмоль/мкл, кассета была растворена в 566 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 10).
Таблица 10. Расчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 7. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.8.
Все комплементарные ДНК пробы, находящиеся на биочипе, гибридизуются с кассетой 7 и показали примерно одинаковую интенсивность флюоресцентного свечения. Кассета 7 содержит одну ДНК пробу hynBbam к гену малой субединицы периплазматической [NiFe] гидрогеназы для детекции бактерий рода Desulfobacterium. Эффективность её гибридизации сравнима с эффективностью гибридизации других проб и указывает, что проба hynBbam подходит для включения в новый биочип.
Кассета 7 также содержит 4 ДНК пробы для детекции бактерий рода Desulfotomaculum, среди них 1 проба hydsstom27 к гену Fe-гидрогеназы, три пробы dsrAtom709, dsrAtom710, dsrAtom883 к гену диссимиляционной сульфит редуктазы, а также пробу nirK1 к гену Cu-нитрит редуктазы, характерную для нитрит редуцирующих микроорганизмов. Кассета 7 гибридизуется со всеми указанными пробами, а также с высокой эффективностью с пробой nirK2, хотя на кассете и отсутствует комплементарная ДНК к этой пробе (позиции D5-D6). В разделе “гибридизация с кассетой 3” выше описан анализ проб, разработанных для детекции нитрит-редуцирующих бактерий. Расчёт интенсивности флюоресцентныx сигналов, полученных при гибридизации кассеты 7, показал, что проба nirK2 является самой эффективной, и подтвердил правильность выбора этой пробы для включения в новый биочип.
Пробы dsrAtom709, dsrAtom710 имеют идентичную последовательность, отличие заключается лишь в том, что проба dsrAtom710 короче на один нуклеотид на 5’-конце. Проба dsrAtom883 сконструирована к другой области гена диссимиляционной сульфит редуктазы. Эффективность их гибридизации практически идентична, поэтому две из них можно исключить из биочипа.
4.1.8. Гибридизация и тонкая настройка биочипа с кассетой 8.
Количество всех ДНК проб, доступное для гибридизации с кассетой 8, было рассчитано и представлено в таблице 11. Кассета 8 была синтезирована в количестве 0.63 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.25 пмоль/мкл, кассета была растворена в 504 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 11).
Таблица 11. Расчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 1. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.9.
Kассета 8 содержит одну пробу FTHFS-c3 к гену формилтетрагидрофолат синтетазы FTHFS для детекции ацетогенных микроорганизмов и пять проб для детекции бактерии рода Desulfomicrobium. Проба hydAmic1 сконструирована к гену альфа субъединицы [NiFe] гидрогеназы, а пробы hydssmic2, hydssmic3, hydssmic4, hydssmic5 – к различным областям малой субъединицы гидрогеназы. Пробы hydssmic3, hydssmic4 отличаются только длиной, последняя короче на три нуклеотида на 5’-конце.
Все шесть проб гибридизуются с биочипом и показали примерно одинаковую эффективность флюоресцентного свечения (Фиг.9). Также кассета 8 гибридизуется с двумя другими пробами на биочипе к гену формилтетрагидрофолат синтетазы, FTHFS-c1 (позиции B9-B10) и FTHFS-c2 (позиции C1-C2). Пробы FTHFS-c3, FTHFS-c2, FTHFS-c1 настроены на одну область гена и имеют небольшие различия. Пробы FTHFS-c1 и FTHFS-c2 длиной 24 нуклеотида отличаются наличием вырожденности в структуре ДНК первой пробы, что позволяет ей узнавать большее количество различных штаммов бактерий, а проба FTHFS-c3, находящаяся в кассете 8, короче их на два нуклеотида. Проба FTHFS-c3 (позиции I9-I10), несмотря на то, что она комплементарна структуре ДНК на кассете, показала наименьшую интенсивность свечения, 22890 пикселей против 36766 (проба FTHFS-c1) и 38870 (проба FTHFS-c2). В связи с этим она не будет включаться в окончательную конструкцию биочипа.
Все пробы, hydAmic1, hydssmic2, hydssmic3, hydssmic4, hydssmic5, к генам альфа и малой субъединицы [NiFe] гидрогеназы показали довольно высокую и примерно одинаковую эффективность гибридизации, поэтому для окончательной версии биочипа может быть выбрана только одна проба, имеющая более близкую эффективность свечения с остальными пробами на биочипе.
Гибридизация и тонкая настройка биочипа с кассетой 9.
Количество всех ДНК проб, доступное для гибридизации с кассетой 9, было рассчитано и представлено в таблице 12. Кассета 9 была синтезирована в количестве 1.29 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.04 пмоль/мкл, кассета была растворена в 620 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 12).
Tаблица 12. Pасчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 9. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Bacillus,
Clostridium
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.10.
Пробы bac1618, bac1620, bac325, сконструированные к гену, кодирующему белок биогенеза цитохрома C, для детекции бактерий, относящихся к классам Bacillus/Clostridium ферментативных бактерий Firmicutes. Пробы bac1618, bac1620 отличаются только длиной, bac1620 короче на 2 нуклеотида на 5’-конце. Пробa bac325 создана к другой области того же гена, но узнает другую группу бактерий. Все перечисленные пробы, включая Geog1090, имеют примерно одинаковую интенсивность гибридизации и могут быть использованы в биочипе.
Пробы hydfob1 и hydfob2, сконструированные к гену hydA, кодирующему альфа субьединицу [NiFe] гидрогеназы, имеют идентичную последовательность, за исключением дополнительного C нуклеотида на 5’-конце у пробы hydfob1. Учитывая, что пробы hydfob1 и hydfob2 показали примерно одинаковую интенсивность свечения и имеют одинаковый круг узнаваемых микроорганизмов, то для биочипа можно использовать любую из них.
Кроме того, кассета 9 гибридизуется с пробой Geog1090, находящейся на биочипе и сконструированной к hydA гену, кодирующему альфа субьединицу нитрат гидрогеназы Geoglobus, относящегося к семейству Archaeoglobaceae. Сравнение нуклеотидныx последовательностей кассеты 9 и пробы Geog1090 обнаружило 85% идентичности c пробой hydfob2, входящей в состав кассеты 9. При необходимости дискриминации этих бактерий можно повысить температуру гибридизации.
Пробы bac1618 и bac1620 показали примерно одинаковую интенсивность свечения и так как они сконструированы для детекции одних и тех же бактерий, то любую из них можно использовать для биочипа.
Гибридизация и тонкая настройка биочипа с кассетой 10.
Количество всех ДНК проб, доступное для гибридизации с кассетой 10, было рассчитано и представлено в таблице 13. Кассета 10 была синтезирована в количестве 0.44 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.1 пмоль/мкл, кассета была растворена в 400 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 13).
Таблица 13. Расчёт суммарного количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 10. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
(0.88 нл)
Enterobacteriaceae
Geobacter
Результаты гибридизации и эффективности флюоресцентного сигнала представленны на Фиг.11.
Все ДНК пробы, входящие в состав кассеты 10, гибридизуются с комплементарными последовательностями проб, находящихся на биочипе. Kассета 10 содержит две пробы nifD1333 и nifD915 к разным областям альфа субъединицы [MoFe] белка нитрогеназного комплекса, который катализирует ключевые энзиматические реакции фиксации азота, сконструирована для обнаружения рода Desulfuromonadales, который эффективно конкурирует с другими микроорганизмами в бедной азотом среде и заселяет места с высоким солевым содержанием, что характерно для промысловых вод нефтепромыслов. Обе пробы имеют примерно одинаковую эффективность гибридизации, поэтому для иммобилизации на биочип можно использовать любую из них.
Проба hyaLGeob сконструированая к гену, кодирующему большую субъединицу периплазматической мембрансвязанной гидрогеназы Geobacter, показала сравнимую с другими пробами, иммобилизованными на биочипе, эффективность свечения (Фиг.11, позиции L5-L6), поэтому она подходит без каких- либо изменений для иммобилизации на биочип.
Проба hybOE783 сконструирована к гену, кодирующему малую субъединицу гидрогеназы 2, для детекции E. coli, и она отличается от пробы hybOE784, находящейся на биочипе, только на одно основание по длине. Кассета 10 содержащая комплементарную ДНК к пробе hybOE783, а также кассета 5 содержащая комплементарную ДНК к пробе hybOE784, показали практически идентичную эффективность гибридизации с обеими пробами (Фиг 11 и 6, позиции F5-F6 и L3-L4). Таким образом, разница по длине не повлияла на гибридизацию, и любую из них можно использовать для иммобилизации на биочипе.
Kассета 10 эффективно гибридизуется с пробой aprAvib3, сконструированой к гену аденозин-5’-фосфосульфат редуктазы aprA для детекции сульфат восстанавливающих бактерий рода Desulfovibrio. Кроме того, она гибридизуется с пробами aprAvib2 и aprAvib4. Гибридизация с пробой aprAvib4 на биочипе не удивительна, так как последовательности aprAvib4 и aprAvib3 практически идентичны, поэтому любую из этих проб можно использовать для биочипа. Удивительным оказалась эффективная гибридизация с пробой aprAvib2 (Фиг.11, позиции E9-E10), так как структура этой пробы значительно отличается от пробы aprAvib3. Было обнаружено сравнением нуклеотидных последовательностей этих проб и кассеты 10 (Pairwise Sequence Alignment, LALIGN – ebi.ac.uk), что при синтезе в кассету 10 по ошибке была введена структура ДНК пробы aprAvib2.
4.1.11. Гибридизация и тонкая настройка биочипа с кассетой 11.
Количество всех ДНК проб, доступное для гибридизации с кассетой 11, было рассчитано и представлено в таблице 14. Кассета 11 была синтезирована в количестве 1.95 наномолей. Учитывая суммарное количество ДНК проб на матриксе биочипа при 50% эффективности связывания для получения раствора с концентрацией 1.19 пмоль/мкл, кассета была растворена в 819 мкл dH2O. 1 мкл полученного раствора обеспечивал эквимолярное соотношение с расчётным суммарным количеством ДНК проб на биочипе (таблица 14).
Таблица 14. Расчёт количества ДНК проб, находящихся на биочипе, для гибридизации c кассетой 11. Объем наносимой пробы составлял 0.88 нл и был рассчитан исходя из объема полусферы капли наносимого раствора.
нмоль / мкл
(0.88 нл)
Результаты гибридизации и эффективности флюоресцентного сигнала представлены на Фиг.12.
В кассете 11 находятся три пробы к гену gltA для детекции Geobacter, а также ДНК пробы к гену 16S рPНК для детекции суммарных бактерий и суммарных Aрхей. Kассета 11 проявляет эффективную гибридизацию со всеми пятью комплементарными пробами на биочипе.
Три gltA пробы на биочипе, отличающиеся одним нуклеотидом (T, C, G) в последовательности 5' GAT GTA CTT CAT CCT T/C/G CAC TCC G дали примерно одинаковый сильный флюоресцентной сигнал при гибридизации с кассетой 11 (Фиг.12, позиции B1-B2, B3-B4, B5-B6), которая содержит вырожденную gltA последовательность 5' GAT GTA CTT CAT CCT BCA CTC CG, B = T/C/G. В связи с этим для иммобилизации на биочип было принято решение использовать пробу с вырожденной структурой в этой позиции (таблица 14).
Пробы для оценки суммарных бактерий 16SCons и суммарных археи 16SArch эффективно гибридизуются с комплиментарными последовательностями на биочипе, показывая сильный флюоресцентный сигнал, и обе пробы подходят для иммобилизации на биочипе.
Кассета 11 также показала слабую гибридизацию с находящейся на биочипе пробой aprAvib4, (позиции F1-F2). Это обусловлено некоторым сходством нуклеотидной последовательности aprAvib4 с пробой 16SArch в составе кассеты, что было обнаружено сравнением их последовательностей (Pairwise Sequence Alignment, LALIGN – ebi.ac.uk).
4.2. Финальная конструкция биочипа для мониторинга микроорганизмов, вовлеченных в биокоррозию
Все пробы, иммобилизованные на лабораторном биочипе для мониторинга микроорганизмов, индуцирующих биокоррозию, дали устойчивую гибридизацию и эффективное свечение, превышающее уровень фона в 100–450 раз, что позволяет этот фон просто не учитывать.
Пробы, имеющие сравнимый уровень гибридизации, были отобраны для производства рабочего прототипа биочипа для мониторинга микроорганизмов, вовлечённых в биокоррозию, и представлены в таблице 15. При отборе проб также учитывались температура плавления, ГЦ состав (содержание остатков гуанина и цитозина в нуклеотидной последовательности) и специфичность к различным группам микроорганизмов, играющих важную роль в биокоррозии.
Таблица 15. Список ДНК проб, отобранных для создания лабораторного образца биочипа.
Bacillus/Clostridium
Обозначения: Y=T/C, B=G/T/C, W=A/T, M=A/C, S=G/C
Из 34 ДНК проб, приведенных в таблице 15, 31 пробы нет в приведенном аналоге (пробы, присутствующие в аналоге, отмечены звездочкой). Основное отличие новых проб заключается в том, что они разработаны к другим генам, к другим бактериям или к другим группам микроорганизмов, что позволило в разработанном биочипе значительно расширить спектр и количество детектируемых микроорганизмов, вовлеченных в коррозию металлов. Использование новой Российской технологии производства матрикса для биочипа в совокупности с новыми структурами ДНК проб, позволило существенно расширить список детектируемых микроорганизмов и повысить уровень детекции микроорганизмов более чем в 100 раз.
Таким образом, в результате проведенных исследований были созданы высокочувствительные биочипы для быстрой и одновременной детекции основных групп микроорганизмов, вовлеченных в микробиологическую коррозию металлов. Для биочипов разработаны уникальные олигонуклеотидные пробы - мишени с расширенным спектром узнавания для следующих групп микроорганизмов: сульфат восстанавливающие бактерии и археи, метаногенные археи, термофильные археи металл восстанавливающие, денитрифицирующие, ферментативные бактерии, ацетoгенные бактерии, углеводород деградирующие бактерии и бактерии, принадлежащие к семейству Enterobacteriaceae. Это позволяет расширить спектр детектируемых бактерий и повысить эффективность мониторинга биокоррозии.
В одном из вариантов изобретения разработан диагностический биочип, включающий 34 олигонуклеотидные пробы размером 17–25 нуклеотидов, созданный к функциональным генам, кодирующим ферменты, которые играют важную роль в разрушении металлов. Следующие гены использованы для дизайна проб мишеней для биочипа:
aprAB (аденозин-5’ – фосфосульфат редуктаза или APS – редуктаза), dsrAB (диссимиляционная сульфитредуктаза), hydA ([NiFe] гидрогеназа), hypB (гидрогеназа [Ni] содержащая), hynB ([NiFe] гидрогеназа, альфа субъединица), hyaL (периплазматическая мембрансвязанная [NiFe] гидрогеназа, большая субъединица), nifD (нитрогеназный Mo-Fe белок, альфа субъединица), nirS (цитохром cd1 нитрит редуктаза), nirD (нитрит редуктаза малая субъединица), nirK (Cu нитрит редуктаза), hyb0 (гидрогеназа 2 малая субъединица), assA (алкилсукцинат синтаза альфа субъединица), mcrA (коэнзим B сульфoэтилтиoтрансфераза альфа субъединица), fthts (формилтетрагидро фолатсинтетаза), gltA (цитрат синтаза), mtrB/PioB (MtrB/PioB family decaheme-associated outer membrane protein), feoB (Fe(2+) transporter permease subunit FeoB), uidA (бета-D- глюкуронидаза).
Сконструированные пробы (таблица 1) были использованы для изготовления лабораторного прототипа биочипа и опробованы с помощью ДНК кассет (см. разделы 3 и 4 выше), как описано в работе Zinkevich et al., 2014, что позволило сравнить и быстро оценить их эффективность и выбрать наиболее пригодные для включения в биочип.
Благодаря высокой степени точности и быстроте определения, разработанный набор олигонуклеотидных зондов и биочипы могут найти широкое применение в детекции биозараженности нефтегазовых объектов, для оценки коррозионной активности грунтов при проектировании, прокладке и эксплуатации нефте- и газопроводов, микробиологического контроля загрязнения нефти и нефтепродуктов, мониторинга коррозионных разрушений резервуаров, танкеров и закрытых коммуникаций, мониторинга пластовых вод и др. Кроме того, разработанные зонды и биочипы могут быть использованы при проектировании техногенных объектов в коммунальном и промышленном строительстве закрытых промышленных систем (таких как нефтяные танкеры, заводские тепловые системы, железобетонные сооружения электростанций) и подземных сооружений (газо- и нефтепроводы, туннели метрополитена). Разработанные зонды и биочипы также применимы при прогнозировании последствий возникающей техногенной нагрузки; разработке и определении эффективности новых ингибиторов биокоррозии, определении биостойкости красок и материалов для защиты от коррозии.
Использование разработанного набора олигонуклеотидных зондов и биочипов позволяет быстро и эффективно определять места возникновения потенциальных коррозионно опасных ситуаций и коррозионную агрессивность грунтов, выяснять микробиологический фактор коррозионных повреждений, что, в свою очередь позволяет подбирать методы противокоррозионной защиты, в частности, выбор и эффективную концентрацию ингибиторов биокоррозии и биоцидов.
Как результат, использование разработанного набора олигонуклеотидных зондов и биочипов позволит снизить риск возникновения аварийных ситуаций при эксплуатации нефтегазовых объектов и избежать тяжелых последствий их преждевременного разрушения.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные эксперименты приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
--->
<ST26SequenceListing dtdVersion="V1_3" fileName="Технология для
детекции биозараженности нефтегазовых объектов.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-06-22">
<ApplicantFileReference>48</ApplicantFileReference>
<ApplicantName languageCode="ru">ООО "Фоско Био"</ApplicantName>
<ApplicantNameLatin>Fosco Bio LLC</ApplicantNameLatin>
<InventorName languageCode="ru">Зинкевич Виталий
Евгеньевич</InventorName>
<InventorNameLatin>Zinkevich Vitaly Evgenievich</InventorNameLatin>
<InventionTitle languageCode="ru">Технология для детекции
биозараженности нефтегазовых объектов</InventionTitle>
<SequenceTotalQuantity>74</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcagatcatgatcaacggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caagtggaagtgcttcgtgaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagggcctgtccgccatcaatac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagggcctbtccgccatcaatac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcatgcactgcatcaacgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcatgcactgcatcaacgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcagtatcaaggcggggctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagttatcctgctctatgcccat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgatgccactaacatcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcaagataactggcctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atacgctcacgctcactgccta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtcgccagtgataccttgtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgtacttcatccttcactccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgtacttcatcctccactccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgtacttcatcctgcactccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcatcaagatccacgccatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagatggcggtscgcgccta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggcaccctgatccaccacta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcaccctgatccaccacta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccaacaacatctgcaagggyat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccaacaacatctgcaagggtat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccaacaacatctgcaagggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccatgctcttctttggaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q49">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaggtcaagttcaaccagacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaactcacgtctgctaagact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgccgaccatccagtccggat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q55">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgaccatccagtccggat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q57">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgttctcgacacaccagttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q59">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactgccacctggcggcccatgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q61">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actgcacactggcggcccatgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q63">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactgccacctggcggcccat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q65">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctggctcggmtcmtacatgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q67">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggctcggwtcctacatgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q69">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctacgaccagatctggctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q71">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagtcggtgcatggttcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q73">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctscagaagatcatggacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q75">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggyacgctgatacaccactaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q77">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcctgatacaacaagtatcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q79">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcctgatacaacaagtatcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q81">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgatgcagaggcagatgcgat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q83">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggtgcggaagatcgcgatgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q85">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgcagttccgcatggtgatcgagga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgcagttccgcatggtgatcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcaacctgttcgtcaagaccca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgctgtacaagcagcactt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgttgtgttgacatgaatgaccgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacwgttgcwtcagaaataatggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cactgttgcatcagaaataatggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actgttgcatcagaaataatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttgagtgcatccgccacggcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaaatggggcaatgacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggattggcgcgcaggagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcaactgctcaacgctgcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaactgctcaacgctgcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggtaattaccgacgaaaacggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggtgattaccgacgaaaacggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaggctggtgtttgtatcacct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaccgtcgtgayttcatgaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaccgtcgtgacttcatgaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacttcttcggmccsacmaaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccttccgmcagatgcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctacgggaggcagcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgctcccccgccaattcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>131</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..131</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggcatagagcaggataactgtcgatgttagtggcatcgacgcaggccag
ttatcttgctctaggcagtgagcgtgagcgtatagacaaggtatcactggcgactacggagtgvaggatg
aagtacatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>134</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..134</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q132">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtccatgatcttctgsagaggaaccatgcaccgactgtgtagtggtgtat
cagcgtrccgccattatttctgatgcaacagtggccattatttctgawgcaacwgtggacggtcattcat
gtcaacacaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>132</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..132</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggccgtggcggatgcactcaaatcgtcattgccccatttgggacatcgcga
tcttccgcacctcaagtgctgcttgtacagcggtgggtcttgacgaacaggttgcctcgatcaccatgcg
gaactgcacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>132</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..132</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q136">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtattgatggcggavaggccctgggagttcacgaagcacttccacttgcacc
gttgatcatgatctgccagagccagatctggtcgtagagacatgtaggawccgagccagacatgtakgak
ccgagccaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>130</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..130</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttcatgaagtcacgacggttccttcatgaartcacgacggttcaggtgata
caaacaccagccttggccgttttcgtcggtaatcaccattgcagcgttgagcagttgcactcctgcgcgc
caatccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="69">
<INSDSeq>
<INSDSeq_length>130</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..130</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q140">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctccaaagaagagcatgggacccttgcagatgttgttggctacccttgcag
atgttgttggcatrcccttgcagatgttgttggcatgggccgccaggtggcagtcacatgggccgccagg
tggcagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="70">
<INSDSeq>
<INSDSeq_length>127</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..127</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctcgatcaccatgcggaactgcacgtaggcgcgsaccgccatctcttgat
gcagtgcatgcaacgacgttgatgcagtgcatgcacgttgatgcagtgcatgcaaagccccgccttgata
ctgat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="71">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q151">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccattatttctgatgcaacagtcaactggtgtgtcgagaacgcatccggact
ggatggtcggatccggactggatggtcggcacagtcttagcagacgtgagttcgtcgtctggttgaactt
gaccttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="72">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtggtggatcagggtgcctagtggtggatcagggtgccgatcgcatctgc
ctctgcatcatctgctgatacttgttgtatcaggctgctgatacttgttgtatcaggcg</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="73">
<INSDSeq>
<INSDSeq_length>132</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..132</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatggcgtggatcttgatgagagttcacgaagcacttccacttggtattga
tggcggacaggccctggttgcagcgttgagcagttgccatcttkgtsggwccgaagaagtgtgtgcatct
gkcggaaggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="74">
<INSDSeq>
<INSDSeq_length>105</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..105</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaattggcgggggagcacctgctgcctcccgtaggcggagtgcaggatga
agtacatccggagtggaggatgaagtacatccggagtgaaggatgaagtacat</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---




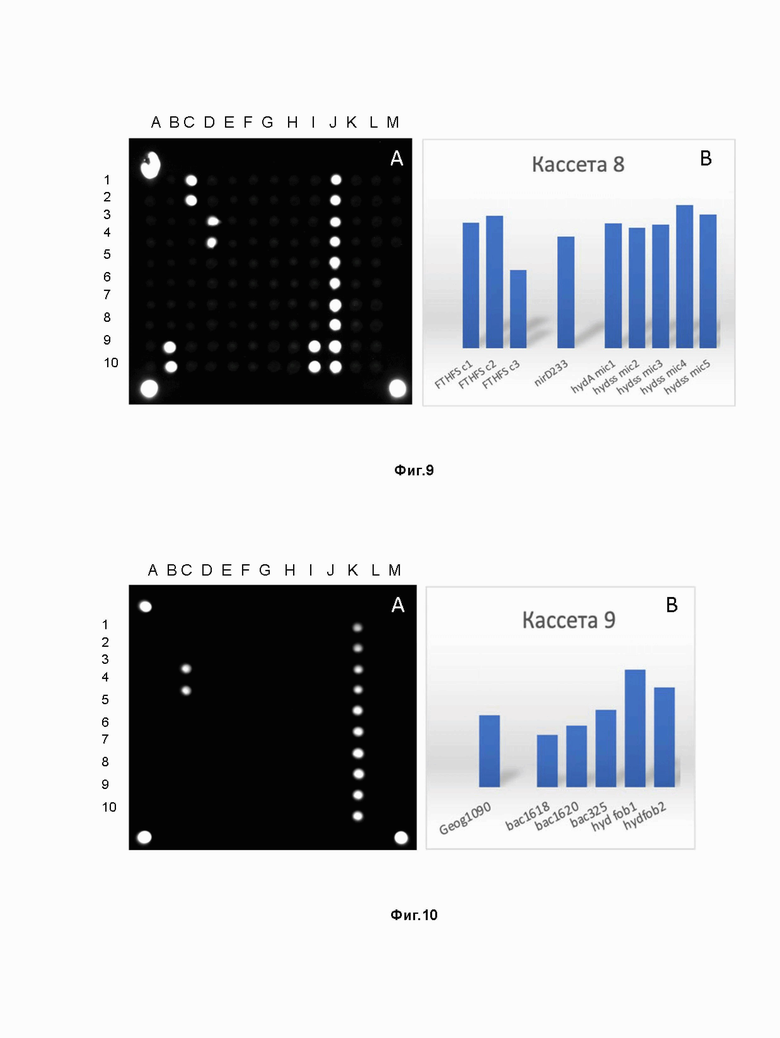

Изобретение относится к биотехнологии, а именно к методу быстрой и высокоэффективной детекции микроорганизмов, вовлеченных в коррозию металлов, и может быть использовано для мониторинга биокоррозии как нефтепромысловых, так и любых других объектов, содержащих металлоконструкции. Предложен набор олигонуклеотидных зондов и биочип для идентификации основных групп микроорганизмов, вовлеченных в коррозию металлов, таких как сульфатвосстанавливающие бактерии и археи, метаногенные археи, термофильные археи металлвосстанавливающие, денитрифицирующие, ферментативные бактерии, ацетoгенные бактерии, углеводород деградирующие бактерии и бактерии, принадлежащие к семейству Enterobacteriaceae. Предложен способ обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию. Изобретение позволяет значительно расширить спектр и количество детектируемых микроорганизмов, вовлеченных в коррозию металлов и повысить эффективность мониторинга биокоррозии. 3 н. и 17 з.п. ф-лы, 12 ил., 15 табл.
1. Набор олигонуклеотидных зондов для обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию, включающий по меньшей мере одну из совокупностей зондов i–vi, при этом каждая из совокупностей состоит из зондов, имеющих последовательности:
i. SEQ ID NO: 4, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 21, SEQ ID NO: 23, SEQ ID NO: 29; а также включающая SEQ ID NO: 60 или SEQ ID NO: 61; включающая SEQ ID NO: 5 или SEQ ID NO: 6; и включающая SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 или SEQ ID NO: 28;
ii. SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 16; и также включающая SEQ ID NO: 13, SEQ ID NO: 14 или SEQ ID NO: 15;
iii. SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 37;
iv. SEQ ID NO: 46, SEQ ID NO: 47, SEQ ID NO: 50 и SEQ ID NO: 40; и также включающая SEQ ID NO: 38 или SEQ ID NO: 39; и включающая SEQ ID NO: 18 или SEQ ID NO: 19;
v. SEQ ID NO: 43, SEQ ID NO: 44 и SEQ ID NO: 45;
vi. SEQ ID NO: 56 и SEQ ID NO: 58, а также включающая SEQ ID NO: 52, SEQ ID NO: 53 или SEQ ID NO: 54;
и опционально включающий зонды, имеющие последовательности SEQ ID NO: 62 и/или SEQ ID NO: 63.
2. Набор по п.1, включающий совокупность всех зондов i–vi, а также зонды, имеющие последовательности SEQ ID NO: 62 и SEQ ID NO: 63.
3. Набор по п.2, в котором совокупности зондов i–vi состоят из следующих последовательностей:
i. SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 21, SEQ ID NO: 23, SEQ ID NO: 27, SEQ ID NO: 29 и SEQ ID NO: 61;
ii. SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12 и SEQ ID NO: 16; а также SEQ ID NO: 13, SEQ ID NO: 14 или SEQ ID NO: 15;
iii. SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 37;
iv. SEQ ID NO: 19, SEQ ID NO: 39, SEQ ID NO: 40, SEQ ID NO: 46, SEQ ID NO: 47 и SEQ ID NO: 50;
v. SEQ ID NO: 43, SEQ ID NO: 44 и SEQ ID NO: 45;
vi. SEQ ID NO: 54; SEQ ID NO: 56 и SEQ ID NO: 58.
4. Набор по п.1, в котором микроорганизмы, вызывающие биокоррозию, представляют собой сульфатвосстанавливающие бактерии, а совокупность зондов представлена зондами, имеющими последовательности SEQ ID NO: 4, SEQ ID NO: 7, SEQ ID NO: 17, SEQ ID NO: 21, SEQ ID NO: 23, SEQ ID NO: 29и SEQ ID NO: 61, а также включающая SEQ ID NO: 5 или SEQ ID NO: 6 SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 или SEQ ID NO: 28.
5. Набор по п.4, в котором сульфатвосстанавливающие бактерии представляют собой бактерии рода Desulfovibrio, Desulfotomaculum, Desulfobacterium, Desulfobacter, Desulfococcus, Desulfomicrobium и Desulfuromonadales.
6. Набор по п.1, в котором микроорганизмы, вызывающие биокоррозию, представляют собой металлвосстанавливающие бактерии, а совокупность зондов представлена зондами, имеющими последовательности SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 16; и также включающая SEQ ID NO: 13, SEQ ID NO: 14 или SEQ ID NO: 15.
7. Набор по п.6, в котором металлвосстанавливающие бактерии представляют собой бактерии рода Shewanella и Geobacter.
8. Набор по п.1, в котором микроорганизмы, вызывающие биокоррозию, представляют собой Arhaea, а совокупность зондов представлена зондами, имеющими последовательности SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 37.
9. Набор по п.8, в котором Arhaea представляют собой метаногенные археи и арехеи рода Geobacter.
10. Набор по п.1, в котором микроорганизмы, вызывающие биокоррозию, представляют собой бактерии типа Firmicutes, а совокупность зондов представлена зондами, имеющими последовательности SEQ ID NO: 46, SEQ ID NO: 47, SEQ ID NO: 50, и SEQ ID NO: 40; и также включающая SEQ ID NO: 38 или SEQ ID NO: 39; и включающая SEQ ID NO: 18 или SEQ ID NO: 19.
11. Набор по п.10, в котором бактерии типа Firmicutes представляют собой ацетогенные бактерии, углеводород- деградирующие и бактерии классов Bacillus, Clostridium или Desulfobulbus.
12. Набор по п.1, в котором микроорганизмы, вызывающие биокоррозию, представляют собой нитритредуцирующие бактерии, а совокупность зондов представлена зондами, имеющими последовательности SEQ ID NO: 43, SEQ ID NO: 44 и SEQ ID NO: 45.
13. Набор по п.12, в котором нитритредуцирующие бактерии представляют собой бактерии рода Pseudomonas.
14. Набор по п.1, в котором микроорганизмы, вызывающие биокоррозию, представляют собой бактерии, усиливающие рост сульфатвосстанавливающих бактерий, а совокупность зондов представлена зондами, имеющими последовательности SEQ ID NO: 56 и SEQ ID NO: 58, а также включающая SEQ ID NO: 52, SEQ ID NO: 53 или SEQ ID NO: 54.
15. Набор по п.14, в котором бактерии, усиливающие рост сульфатвосстанавливающих бактерий, представляют собой бактерии семейства Enterobacteriaceae и бактерии семейства Hafniaceae.
16. Набор по любому из пп.1-15, в котором зонды иммобилизованы в идентифицируемых местах на биочипе.
17. Биочип для обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию, включающий набор олигонуклеотидных зондов по любому из пп.1-16, иммобилизированных на твердой подложке.
18. Биочип по п.17, в котором твердая подложка включает 3D-гидрогель.
19. Способ обнаружения, идентификации и/или количественного определения микроорганизмов, вызывающих биокоррозию, включающий следующие этапы:
a) амплификация ДНК из образца, полученного для анализа;
b) фрагментация амплифицированной ДНК;
c) флюоресцентное мечение ДНК;
d) заливка флуоресцентно-меченой ДНК на биочип по любому из пп.17, 18 и гибридизация с элементами биочипа;
e) регистрация и интерпретация результатов гибридизации на биочипе, проведенной на стадии (d).
20. Способ по п.19, в котором образец представляет собой смыв или соскоб с внутренней поверхности трубы из нефте- или газораспределительной системы.
| WO 2018191222 A1, 18.10.2018 | |||
| WO 202078273 A1, 06.02.2020 | |||
| О.Ю | |||
| Сурсимова и др | |||
| ИСПОЛЬЗОВАНИЕ БИОЛОГИЧЕСКИХ ЧИПОВ В ДЕТЕКЦИИ МИКРОБИОЛОГИЧЕСКОЙ КОРРОЗИИ, Вестник Тверского государственного университета | |||
| Серия "География и геоэкология" | |||
| Электромагнитный прерыватель | 1924 |
|
SU2023A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Машина для изготовления проволочных гвоздей | 1922 |
|
SU39A1 |
| А.В | |||
| Рязанов и др | |||
| Биокоррозия металлов, теоретические представления, методы | |||
Авторы
Даты
2024-01-30—Публикация
2023-07-27—Подача