ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к новым выделенным антителам и их антигенсвязывающим фрагментам, которые связываются с ранее не охарактеризованным эпитопом липазы, стимулируемой солями жёлчных кислот (Bile Salt-Stimulated Lipase, BSSL), расположенным в N-концевой части белка BSSL. Настоящий документ также относится к медицинскому применению антител и их антигенсвязывающих фрагментов, в частности, при лечении воспалительных состояний, а также к родственным фармацевтическим композициям. В настоящем документе также описано применение антител или их антигенсвязывающих фрагментов в качестве молекулярных инструментов при обнаружении BSSL и/или для диагностики заболеваний, связанных с BSSL.
УРОВЕНЬ ТЕХНИКИ
Воспалительные состояния, включая аутоиммунные и аутовоспалительные заболевания, по-прежнему представляют значительную угрозу для здоровья человека. Несмотря на успехи в лечении воспалительных состояний, все еще ведутся поиски оптимизированных методов лечения.
Воспалительные состояния включают широкий спектр нарушений и заболеваний, которые характеризуются воспалением, включая аутоиммунные заболевания и аутовоспалительные заболевания. Воспаление может, например, возникать как реакция на инфекции, травмы, аллергены и/или токсины или как реакция на сам организм, например, аутоиммунные процессы. Аутоиммунное заболевание возникает, когда иммунная система организма атакует и по ошибке разрушает здоровые ткани тела. Сообщается, что известно более 80 аутоиммунных заболеваний.
Некоторые воспалительные состояния носят хронический характер. Хроническое воспаление возникает, когда воспалительная реакция затягивается, оставляя организм в постоянном состоянии готовности. Примерами воспалительных заболеваний и состояний, которые включают хроническое воспаление, являются ревматоидный артрит (RA), ювенильный идиопатический артрит (JIA), псориатический артрит (PsA) и воспалительное заболевание кишечника (IBD), такое как язвенный колит (UC) и болезнь Крона (CD).
RA представляет собой хроническое воспалительное системное аутоиммунное заболевание. Современные методы лечения RA включают нестероидные противовоспалительные препараты (NSAID) для лечения боли, модифицирующие заболевание противоревматические препараты (DMARD) и биологические агенты, нацеленные на специфические провоспалительные цитокины или рецепторы клеточной поверхности различных типов клеток.
JIA, также известный как ювенильный ревматоидный артрит (JRA), является наиболее распространенной формой артрита у детей и подростков. JIA развивается в возрасте до 16 лет, и при этом причина JIA в основном неизвестна. Основное внимание при лечении JIA уделяется тому, чтобы помочь ребенку вернуться к нормальному уровню физической и социальной активности. Большинство детей получают NSAID и внутрисуставные инъекции кортикостероидов. Метотрексат, DMARD, является мощным лекарственным средством, которое помогает подавить воспаление суставов у большинства пациентов с JIA с полиартритом, хотя, как сообщается, этот препарат менее полезен при системном артрите, и многие дети получают лекарственные средства, ингибирующие TNFα, такие как этанерцепт.
IBD представляет собой термин, применяемый для описания нарушений, которые включают хроническое воспаление пищеварительного тракта. IBD включает UC и CD.
Цель лечения IBD – уменьшить выраженность воспаления, которое обуславливает признаки и симптомы. В лучшем случае это может привести не только к облегчению симптомов, но и к долговременной ремиссии и снижению риска осложнений. Лечение IBD обычно включает медикаментозную терапию, такую как противовоспалительные препараты (NSAID), супрессоры иммунной системы и/или биологические агенты, а также хирургическое вмешательство.
Липаза, стимулируемая солями жёлчных кислот (BSSL), также известная как липаза, зависимая от солей желчных кислот (BSDL), карбоксилэфирная липаза (CEL) или липаза, активируемая солями желчных кислот (BAL), представляет собой липолитический фермент, кодируемый геном CEL и экспрессируемый в экзокринной поджелудочной железе, а также секретируемый в просвет кишечника у всех исследованных до настоящего времени видов и способствующий перевариванию липидов.
У некоторых видов, включая людей, приматов, собак, кошек и мышей, BSSL также экспрессируется в лактирующих молочных железах и секретируется с молоком. Более того, было обнаружено, что BSSL находится на низких, но значимых уровнях в сыворотке крови здоровых людей и участвует в метаболизме липопротеинов и модуляции атеросклероза. Также было обнаружено, что BSSL играет роль в воспалительных процессах.
BSSL можно выделить из пригодной ткани, такой как грудное молоко. В альтернативном варианте рекомбинантный BSSL можно получить стандартными способами путем выделения ДНК, кодирующей BSSL.
ДНК, кодирующую BSSL, можно с успехом выделить из коммерчески доступных РНК, библиотек cДНК, геномной ДНК или библиотек геномной ДНК с помощью обычных методов молекулярной биологии, таких как скрининг библиотек и/или полимеразная цепная реакция (PCR).
Способы очистки BSSL из различных тканей и трансфицированных клеточных линий известны в данной области техники [1].
В документе [2] описаны антигенсвязывающие соединения, которые связывают BSSL или фето-ацинарный панкреатический белок (FAPP). Описано, что соединения распознают С-концевой пептид (эпитоп J28) BSSL. FAPP представляет собой онкофетальную форму BSSL, характеризующуюся углеводзависимым эпитопом J28. Утверждается, что антигенсвязывающие соединения вызывают апоптоз и/или замедляют пролиферацию опухолевых клеток, экспрессирующих полипептид BSSL или FAPP. В документе [2] описаны соединения, которые способны напрямую воздействовать на опухолевые клетки, в частности, на опухолевые клетки поджелудочной железы, экспрессирующие BSSL или FAPP, и вызывать их гибель посредством апоптоза и/или останавливать их пролиферацию.
В документах [3, 4] описывается открытие, что BSSL играет роль в воспалительных процессах и что ингибирование или элиминация BSSL защищает от развития хронического артрита на моделях животных. Документы [3, 4] описывают, что белок BSSL присутствует в воспалительных клетках и воспаленной ткани и что мыши с дефицитом BSSL защищены от развития воспалительного заболевания, примером которого является индуцированный коллагеном артрит (CIA).
Хотя методы лечения воспалительных состояний, таких как аутовоспалительное заболевание и аутоиммунное заболевание, значительно усовершенствовались за последние годы за счет внедрения в практику новых лекарственных средств и классов лекарственных препаратов, таких как антитела, большинство схем и лекарственных средств все еще объединяет то, что они направлены на подавление иммунной системы, как, например, в случае со всеми ингибиторами TNFα и кортикостероидами. Это, в свою очередь, увеличивает риск вторичных инфекций и осложнений.
Следовательно, все еще существует значительная клиническая потребность в новых селективных, предпочтительно биологических, лекарственных средствах для лечения, профилактики и предотвращения воспалительных заболеваний, которые направлены на различные и/или новые мишени, участвующие в воспалительных сигналах и процессах, и которые в первую очередь не действуют посредством подавления иммунной системы, и поэтому ожидается, что они будут иметь меньшее количество и/или менее серьезные нежелательные эффекты.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Общей целью настоящего изобретения является получение антител или их антигенсвязывающих фрагментов, которые специфически связываются с липазой, стимулируемой солями жёлчных кислот (BSSL), такой как BSSL человека (hBSSL).
Данная и другие цели достигаются с помощью вариантов осуществления, описанных в данном документе.
Настоящее изобретение определено в независимых пунктах формулы изобретения. Дополнительные варианты осуществления изобретения определены в зависимых пунктах формулы изобретения.
Аспект настоящего изобретения относится к выделенному антителу или его антигенсвязывающему фрагменту, содержащему три определяющих комплементарность области (CDR) вариабельной области тяжелой цепи (HCVR), обозначенных HCDR, и три CDR вариабельной области легкой цепи (LCVR), обозначенных LCDR. В этом аспекте первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, имеющей по меньшей мере 87% идентичности с SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8 или аминокислотной последовательности, имеющей по меньшей мере 75% идентичности с SEQ ID NO: 8, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9, или аминокислотной последовательности, имеющей по меньшей мере 83% идентичности с SEQ ID NO: 9. Кроме того, первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10 или аминокислотной последовательности, имеющей по меньшей мере 80% идентичности с SEQ ID NO: 10, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS или аминокислотной последовательности, имеющей по меньшей мере 66% идентичности с аминокислотной последовательностью ATS, предпочтительно AAS, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности, имеющей по меньшей мере 87% идентичности с SEQ ID NO: 11.
Другой аспект настоящего изобретения относится к выделенному антителу или его антигенсвязывающему фрагменту, содержащему вариабельную область тяжелой цепи (HCVR), состоящую из аминокислотной последовательности, выбранной из ZH1-[GYTFTSYN]-ZH2-[X53GVIX57PGDGX64TSYX68QKFX72]-ZH3-[ARDYYGSSPLGY]-ZH4 и вариабельной области легкой цепи (LCVR), состоящей из аминокислотной последовательности, выбранной из ZL1-[X24ASX27SISYX39N]-ZL2-[AX57SX66LX68]-ZL3-[HQRSSX115PT]-ZL4. В этом аспекте каждая область из ZH1, ZH2, ZH3 и ZH4 независимо представляет ноль, один или несколько независимо выбранных аминокислотных остатков, X53 выбран из I и M, X57 выбран из N и Y, X64 выбран из A и S, X68 выбран из A и N, и X72 выбран из K и Q. Кроме того, каждая область из ZL1, ZL2, ZL3 и ZL4 независимо представляет ноль, один или несколько независимо выбранных аминокислотных остатков, X24 выбран из S и R, X27 выбран из S и P, X39 выбран из M и L, X57 выбран из A и T, X66 выбран из K и S, X68 выбран из A и P, и X115 выбран из S, T и Y.
Дополнительный аспект настоящего изобретения относится к выделенному антителу или его антигенсвязывающему фрагменту, которое специфически связывается с эпитопом BSSL, предпочтительно hBSSL. Эпитоп содержит первую поверхность, содержащую аминокислотную последовательность согласно SEQ ID NO: 1, или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 83% идентичности с SEQ ID NO: 1, и вторую поверхность, содержащую аминокислотную последовательность согласно SEQ ID NO: 2, или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 85% или по меньшей мере 92% идентичности с SEQ ID NO: 2.
Еще один аспект настоящего изобретения относится к фармацевтической композиции, содержащей выделенное антитело и/или его антигенсвязывающий фрагмент, как указано выше, и фармацевтически приемлемый носитель или вспомогательное вещество.
Дополнительные аспекты настоящего изобретения относятся к выделенному антителу или его антигенсвязывающему фрагменту согласно вышеизложенному или к фармацевтической композиции согласно вышеизложенному для применения в качестве лекарственного средства и для применения в лечении и/или профилактике воспалительного заболевания.
Связанный аспект настоящего изобретения определяет применение выделенного антитела или его антигенсвязывающего фрагмента согласно вышеизложенному или к фармацевтической композиции согласно вышеизложенному для изготовления лекарственного средства для лечения и/или профилактики воспалительного заболевания.
Другой связанный аспект настоящего изобретения определяет способ лечения и/или облегчения, и/или предотвращения, и/или профилактики воспалительного заболевания. Указанный способ включает введение терапевтически эффективного количества выделенного антитела или его антигенсвязывающего фрагмента согласно вышеизложенному или фармацевтической композиции согласно вышеизложенному, субъекту, нуждающемуся в этом.
Дополнительные аспекты настоящего изобретения относятся к полинуклеотиду, кодирующему антитело или его антигенсвязывающий фрагмент, согласно вышеизложенному, экспрессионному вектору, содержащему такой полинуклеотид, и клетке, содержащей антитело или его антигенсвязывающий фрагмент, согласно вышеизложенному, a также полинуклеотиду согласно вышеизложенному и/или экспрессионному вектору согласно вышеизложенному.
Другой аспект настоящего изобретения относится к способу обнаружения присутствия или отсутствия BSSL и/или количественного определения количества BSSL в образце. Указанный способ включает приведение образца в контакт с выделенным антителом или его антигенсвязывающим фрагментом, как описано выше, и определение присутствия или отсутствия BSSL в образце и/или определение количества BSSL в образце на основе количества выделенного антитела или его антигенсвязывающего фрагмента, связанного с BSSL.
Другой аспект настоящего изобретения относится к способу диагностики нарушения, связанного с BSSL. Указанный способ включает приведение в контакт образца от субъекта с выделенным антителом или его антигенсвязывающим фрагментом в соответствии с вышеизложенным и обнаружение присутствия или отсутствия BSSL и/или определение количества BSSL в образце на основе количества выделенного антитела или его антигенсвязывающего фрагмента, связанного с BSSL. Указанный способ также включает заключение, основанное на результатах обнаружения и/или количественной оценки, страдает ли субъект нарушением, связанным с BSSL, или нет.
Еще один аспект настоящего изобретения относится к эпитопу BSSL, содержащему первую поверхность, содержащую аминокислотную последовательность согласно SEQ ID NO: 1, или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 83% идентичности с SEQ ID NO: 1, и вторую поверхность, содержащую аминокислотную последовательность согласно SEQ ID NO: 2, или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 85% или по меньшей мере 92% идентичности с SEQ ID NO: 2.
Антитела и их антигенсвязывающие фрагменты по настоящему изобретению связываются с ранее не охарактеризованным эпитопом в hBSSL, отличным от активного сайта фермента. Следовательно, антитела и их антигенсвязывающие фрагменты могут связываться с hBSSL, не конкурируя с ферментативной активностью hBSSL. Антитела и их антигенсвязывающие фрагменты по настоящему изобретению являются полезными для лечения и/или предотвращения воспалительных состояний, облегчая при этом вышеупомянутые и другие недостатки существующих методов лечения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Варианты осуществления вместе с другими назначениями и их преимуществами можно лучше понять, ознакомившись с последующим описанием вместе с прилагаемыми графическими материалами, на которых:
На Фигуре 1 продемонстрировано взаимодействие между hBSSL и иммобилизованным AS20 mIgG1. Сенсорограмма подогнана к модели привязки 1:1. Из-за хорошей подгонки сенсограмму и подогнанную линию невозможно отличить.
На Фигуре 2 продемонстрирован стационарный анализ взаимодействия между mBSSL и иммобилизованным AS20 mIgG1. Значение KD взято из половины максимального отклика, где max был автоматически скорректирован на большой объемный эффект со смещением -173 RU (сверху). Объемный эффект на фигуре не удален.
На Фигуре 3а продемонстрирован график, иллюстрирующий связывание AS20 scFv ELISA с небиотинилированной и биотинилированной BSSL человека. Отображаемые значения абсорбции (ось Y) являются средними для дубликатов. На Фигуре 3b продемонстрирован график, иллюстрирующий связывание AS20 scFv ELISA с BSSL мыши и человека, а также с нерелевантным белком. Отображаемые значения абсорбции (ось Y) являются средними для дубликатов.
На Фигуре 4 продемонстрирована гистограмма результатов мультиплексного анализа на основе гранул (LUMINEX®), в котором анализируется способность AS20 scFv (левые столбцы) связываться с BSSL человека и с 30 различными нерелевантными белками. Положительный контроль scFv, нерелевантный scFv_1 (правые столбцы), предположительно связывающийся с b-нерелевантным белком_1, также был включен в анализ.
На Фигуре 5 продемонстрированы результаты конкурентного анализа на основе HTRF, в котором анализируется связывание AS20 scFv с BSSL мыши и человека.
Фигура 6 представляет собой сравнение последовательностей между трансплантатом AS20, CDR AS20 и каркасом библиотеки гуманизации AS20. * указывает на остановку в рамке считывания, введенную в LCDR3, чтобы гарантировать, что на фаге отображаются только клоны, мутагенизированные в этой области. X указывает на мутагенизированные положения в библиотеке гуманизации AS20. Границы для CDR определены по Kabat, а нумерация остатков определена по номенклатуре IMGT [5]. eHCDR2, eLCDR1 и eLCDR2 обозначают удлиненные области HCDR2, LCDR1 и LCDR2, включая положение аминокислоты за пределами соответствующей области CDR в соответствии с IMGT.
На Фигуре 7 представлены графики, демонстрирующие тенденции данных эксклюзионной хроматографии при A) + 40 °C и B) +4 °C, как описано в Примере 12.
На Фигуре 8 представлены графики, демонстрирующие средние значения Tm1 (круг) и Tm2 (квадрат), нанесенные для каждого кандидата. Столбцы указывают на стандартное отклонение; а) + 4 °С b) +40°C.
На Фигуре 9 представлены результаты анализа DLS, демонстрирующие зависимость интенсивности от размера для кандидатов. Образцы анализировали после хранения в течение 30 дней при -80 °C (заштрихованная стрелка), + 4 °C (пунктирная стрелка) и + 40 °C (полная стрелка) соответственно.
На Фигуре 10 приведено графическое представление структуры BSSL с указанными эпитопными областями предполагаемых антител-кандидатов (аминокислотные остатки 7-12 для S-SL048-11, S-SL048-46, S-SL048-106, S-SL048-116, S-SL048-118; aa 42-55 для S-SL048-11, S-SL048-46, S-SL048-106, S-SL048-116, S-SL048-118; aa 84-101 для S-SL048- 46; aa 174-180 для S-SL048-116; aa 283-295 для S-SL048-11).
На Фигуре 11 продемонстрирован svFv S-SL048-106 с выделенными потенциальными посттрансляционными предрасположенностями.
Фигура 12 представляет собой эскизное изображение t-hBSSL, демонстрирующее вид «прихватки», и эпитоп, обведенный пунктирной линией и окрашенный в светло-серый цвет вокруг задней части «прихватки». Нити показаны стрелками, а спирали – завитками.
На Фигуре 13, на левой панели, продемонстрирован t-hBSSL в поверхностном представлении, в темно-сером цвете, с последовательностями, которые взаимодействуют с AS20-Fab, в светло-сером цвете. Вариабельные области тяжелой и легкой цепи продемонстрированы в виде ленты (легкая цепь окрашена в серый цвет, а тяжелая цепь – в черный). На правой панели продемонстрирован тот же вид, но t-hBSSL представлен в виде «палочек» с триадой активных сайтов, выделенной черным цветом. На левой панели активный сайт скрыт под поверхностью.
Фигура 14 представляет собой таблицу, демонстрирующую различия в аминокислотной последовательности между 38 кандидатами scFv.
Фигуры 15A и 15B представляют собой краткое изложение дизайна комбинаторной библиотеки scFv для вариабельной области тяжелой цепи, как описано в Примере 5.
Фигуры 16A и 16B представляют собой краткое изложение дизайна комбинаторной библиотеки scFv для вариабельной области легкой цепи, как описано в Примере 5.
Фигура 17 представляет собой графики, демонстрирующие анализы активности BSSL согласно Примеру 20. A) и B) демонстрируют результаты анализа гидролиза триглицеридов, а C) и D) демонстрируют результаты анализа гидролиза сложного эфира холестерина. Следует обратить внимание, что химерный AS20 обозначен на Фигуре как AS20.
Фигура 18 иллюстрирует тяжесть артрита. Развитие CAIA у мышей после инъекции CIA-MAB-50 и введения (A) изотипического контроля анти-NP hIgG1 LALA-PG (90 мг/кг), AS20 hIgG1 LALA-PG (90, 30 и 10 мг/кг) каждый 4й день со дня -1 по день 15. (B) Изотипический контроль и AS20 hIgG1 LALA-PG 90 мг/кг. (С) Изотипический контроль и AS20 IgG1 LALA-PG 30 мг/кг. (D) Изотипический контроль и AS20 hIgG1 LALA-PG 10 мг/кг. Два животных, одно в группе AS20 hIgG1 LALA-PG 10 мг/кг и одно в группе изотипического контроля были исключены из исследования досрочно (День 12) из этических соображений (высокий балл). Данные этих животных были включены в результаты до дня исключения их из исследования. Данные представлены как среднее ±SEM. * р <0,05; ** р <0,01.
На Фигуре 19 продемонстрированы графики параметров заболевания. Параметры заболевания CAIA, включая животных, получавших i.p. с изотипическим контролем анти-NP hIgG1 LALA-PG (90 мг/кг), AS20 hIgG1 LALA-PG (90, 30 и 10 мг/кг) каждый 4й день со дня -1 по день 15. (A) Средний балл CAIA (сумма баллов во время эксперимента, деленная на количество дней подсчета баллов). (B) Максимальный балл CAIA. (C) Общее бремя болезни (AUC). (D) Процент ингибирования. Два животных, одно в группе AS20 hIgG1 LALA-PG 10 мг/кг и одно в группе изотипического контроля были исключены из исследования досрочно (День 12) из этических соображений (высокий балл). Эти животные были включены только в максимальный балл. Данные представлены как среднее ±SEM. * р <0,05; ** р <0,01.
На Фигуре 20 продемонстрированы графики подмножества клеток в общем количестве. Данные представлены как среднее ± SD.
На Фигуре 21 продемонстрированы графики подмножества клеток в процентах. Данные представлены как среднее ± SD.
На Фигуре 22 продемонстрирована структура комплекса Fab-BSSL, нарисованная в виде эскиза. Димерный комплекс S-SL048-116 Fab с легкой цепью и тяжелой цепью и BSSL. Этот же комплекс повернут вправо на 180°.
На Фигуре 23 продемонстрированы взаимодействия между BSSL и S-SL048-116 Fab. Вариабельные Ig домены Fab с легкой цепью и тяжелой цепью, взаимодействующие с BSSL. Две жизненно важные аминокислоты для эпитопа Arg 176 и Gln 52 нарисованы в виде шарика и палки.
Фигура 24 иллюстрирует тяжесть артрита. Развитие CAIA у мышей после инъекции CIA-MAB-50 и введения (A) носителя и S-SL048-116 (SOL-116) (90, 30 и 10 мг/кг) каждый 4йдень со дня -1 по день 15. (B) Носитель и S-SL048-116, 90 мг/кг. (C) Носитель и S-SL048-116, 30 мг/кг. (D) Носитель и S-SL048-116, 10 мг/кг. Три животных были исключены из исследования досрочно из этических соображений, двое – в группе носителя (день 7 и день 15) и одно – в группе 90 мг/кг (день 12). Данные этих животных были включены в результаты до дня их исключения из исследования, а данные животного, исключенного на 7 день, полностью исключены из анализа. Данные представлены как среднее ± SEM.
На Фигуре 25 продемонстрированы параметры заболевания CAIA, включая животных, получавших i.p. с носителем и S-SL048-116 (SOL-116) в различных дозах (90, 30 и 10 мг/кг). (A) Средний балл CAIA (сумма баллов во время эксперимента, деленная на количество дней подсчета баллов). (B) Максимальный балл CAIA. (C) Общее бремя болезни (AUC). (D) Процент ингибирования. Три животных были исключены из исследования досрочно из этических соображений, двое – в группе носителя (день 7 и день 15) и одно – в группе 90 мг/кг (день 12). Эти животные были включены только в максимальный балл. Данные представлены как среднее ± SEM.
На Фигуре 26 продемонстрировано общее количество лейкоцитов в селезенке (А) и мезентериальных лимфатических узлах (В). Данные представлены как среднее ±SEM. ** р <0,01.
На Фигуре 27 продемонстрировано соотношение NK-клеток из CD45 + клеток в (A) селезенке, (B) крови и (C) мезентериальных лимфатических узлах. Данные представлены как среднее ± SEM. * ***p<0,001.
ПОДРОБНОЕ ОПИСАНИЕ
Определения
Термин «выделенный» при применении в связи с антителами, например, в выражении «выделенное антитело» и т.п., означает, что антитело было выделено из своего исходного окружения. В контексте данного документа термин «выделенное антитело» предназначено для обозначения антитела, которое по существу не содержит других антител, обладающих другой антигенной специфичностью, например, выделенное антитело, которое специфически связывает BSSL, в частности BSSL человека (hBSSL), по существу не содержит антител, которые специфически связывают антигены, отличные от BSSL. Однако выделенное антитело, которое специфически связывает hBSSL, может обладать перекрестной реактивностью с другими антигенами, такими как молекулы BSSL других видов, такие как BSSL мыши или крысы (mBSSL). Более того, выделенное антитело может по существу не содержать другой клеточный материал и/или химических веществ. Например, выделенное антитело или его антигенсвязывающий фрагмент может быть очищен до чистоты более 95% или 99%, что определяется, например, с помощью электрофореза, например, электрофореза в полиакриламидном геле с додецилсульфатом натрия (SDS-PAGE), изоэлектрического фокусирования (IEF), капиллярного электрофореза или хроматографии, например, ионообменной или обращенно-фазовой высокоэффективной жидкостной хроматографии (HPLC). Квалифицированному специалисту будет понятно, что в данном документе упоминаются выделенные антитела или их антигенсвязывающие фрагменты, даже если слово «выделенный» явно не упоминается каждый раз, когда применяется термин «антитела или их антигенсвязывающие фрагменты» и т.п.
В контексте данного документа термин «выделенное гуманизированное антитело» и т.п. относится к выделенному антителу, которое предварительно гуманизировали.
В контексте данного документа термин «антигенсвязывающий фрагмент» предназначен для обозначения фрагмента или части антитела, которые по существу сохраняют антигенсвязывающие свойства. Антигенсвязывающий фрагмент представляет собой часть или область молекулы антитела или его производного, которая сохраняет все или значительную часть, связывающую антиген, соответствующего полноразмерного антитела. Антигенсвязывающий фрагмент может содержать одну или большее количество последовательностей области антитела, определяющей комплементарность (CDR), или часть этих последовательностей CDR, часть или всю вариабельную область тяжелой цепи (HCVR), часть или всю вариабельную область легкой цепи (LCVR) или их комбинацию. В одном из вариантов осуществления антигенсвязывающий фрагмент антитела может состоять из последовательной аминокислотной последовательности антитела, из которого он получен, или может состоять из различных частей аминокислотной последовательности антитела, соединенных вместе посредством линкера (линкеров) или без него. Примерами антигенсвязывающих фрагментов являются одноцепочечные вариабельные фрагменты (scFv), фрагменты Fab, фрагменты F(ab')2, фрагменты F(ab’)3, фрагменты Fab', фрагменты Fd, фрагменты Fv, фрагменты dAb, выделенные области, определяющие комплементарность (CDR), а также нанотела.
«Вариабельный одноцепочечный фрагмент» или «одноцепочечный вариабельный фрагмент» («scFv») представляет собой слитый белок вариабельных областей тяжелой и легкой цепей иммуноглобулинов, связанный посредством короткого линкерного пептида, как правило, от около 10 до 25 аминокислот. scFv одного или разных типов (имеющих аффинность к одним и тем же или различным эпитопам) можно комбинировать разными способами, как известно специалисту в данной области техники. Неограничивающими примерами таких комбинаций являются тандемные ди-scFv, диатела, тандемные три-scFv или три(а)тела.
Термин «эпитоп» относится к той части антигена, которая распознается иммунной системой, например, антителами. Эпитоп также называют антигенной детерминантой.
Термин «паратоп» относится к части антитела, которая связывается с эпитопом.
В контексте данного документа термины «который связывается с», «имеющий аффинность к», «аффинность» и т.п. относятся к свойству антитела или его антигенсвязывающего фрагмента связываться с молекулой-мишенью. Стандартные анализы для оценки связывающей способности антитела или антигенсвязывающего фрагмента по отношению к молекуле-мишени включают, например, иммуноферментные анализы (EIA), такие как иммуноферментный анализ (ELISA), вестерн-блоттинг, радиоиммуноанализ (RIA), анализ поверхностного плазмонного резонанса (SPR), мультиплексный анализ LUMINEX® и анализ методом проточной цитометрии. Как проиллюстрировано в экспериментальном разделе, кинетику связывания, например, аффинность связывания, антител также можно оценить с помощью стандартных анализов, известных в данной области техники, таких как системный анализ BIACORE®.
Под терминами «специфически связывается с», «специфически связывающийся с» и т.п. подразумевается, что рассматриваемая молекула, такая как антитело или его антигенсвязывающий фрагмент, специфически связывается с антигеном-мишенью без какого-либо значительного связывания с другими молекулами. Специфичность антитела или его антигенсвязывающего фрагмента можно определить на основе аффинности и/или авидности. Аффинность, представленная равновесной константой для диссоциации антигена с антителом или его антигенсвязывающим фрагментом (KD), является мерой силы связывания между антигенной детерминантой, т.е. эпитопом, и антигенсвязывающим сайтом антитела или его антигенсвязывающим фрагментом. Чем ниже значение KD, тем сильнее сила связывания между антигенной детерминантой и антителом или его антигенсвязывающим фрагментом. В альтернативном варианте, аффинность также может быть выражена как константа аффинности (KA), которая составляет 1/KD. Как будет понятно квалифицированному специалисту, аффинность может быть определена известным способом, в зависимости от конкретного антигена, представляющего интерес.
Обычно антитела или их антигенсвязывающие фрагменты связываются со своим антигеном с равновесной константой диссоциации (KD) от 10-5 до 10-12 моль/литр (М) или меньше, и предпочтительно от 10-7 до 10-12 M, или меньше, и более предпочтительно от 10-8 до 10-12 M, т.е. с константой аффинности (KA) от 105 до 1012 M-1 или большее, и предпочтительно от 107 до 1012 M-1 или большее, и более предпочтительно от 108 до 1012 M-1. Обычно считается, что любое значение KD больше 10-4 M (или любое значение KA ниже 104 M-1) указывает на неспецифическое связывание. Предпочтительно антитело или его антигенсвязывающий фрагмент согласно вариантам осуществления будет связываться с BSSL с аффинностью менее 500 нМ, предпочтительно менее 200 нМ, более предпочтительно менее 10 нМ, например, менее 5 нМ.
Термины «обнаружение», «детекция» и т.п. включают любые средства обнаружения, включая прямое и косвенное обнаружение.
В данном документе все аминокислоты в вариабельных областях, включая описанные в данном документе CDR, следовательно, пронумерованы в соответствии с уникальной нумерацией IMGT, как определено в публикации Marie-Paule Lefranc [5].
Термин «нумерация по Kabat» и т.п. относится к схеме нумерации аминокислотных остатков в антителах на основе вариабельных областей.
В контексте данного документа термины «моноклональное антитело», «моноклональные антитела» и т.п. относятся к антителу/антителам, имеющим моновалентную аффинность, что означает, что каждая молекула антитела в образце моноклонального антитела связывается с одним и тем же эпитопом на антигене. Моноклональные антитела производятся идентичными иммунными клетками, которые являются клонами уникальной родительской клетки, например, линии клеток гибридомы.
В контексте данного документа термин «поликлональные антитела» относится к набору антител, которые реагируют против конкретного антигена, но в этом наборе могут быть разные молекулы антител, например, идентифицирующие разные эпитопы на антигене. Поликлональные антитела обычно продуцируют путем инокуляции подходящего млекопитающего и впоследствии очищают из сыворотки этого млекопитающего.
Термин «производные антитела человека» относится к любой модифицированной форме антитела человека, например, конъюгату антитела и другого агента или антитела.
В контексте данного документа термин «полноразмерное антитело» относится к антителу любого класса, такого как иммуноглобулин D (IgD), IgE, IgG, IgA, IgM или IgY, или к любому его подклассу. Структуры субъединиц и трехмерные конфигурации различных классов антител являются хорошо известными.
В контексте данного документа термин «химерное антитело» относится к рекомбинантному или генно-инженерному антителу, такому как, например, моноклональное антитело мыши, которое содержит полипептиды или домены другого вида, например, человека, введенные для снижения иммуногенности указанного антитела.
В контексте данного документа термин «по меньшей мере один» следует интерпретировать как один или большее количество.
Как понятно специалисту в данной области техники, ссылка на «около» значение или параметр в данном документе включает (и описывает) варианты осуществления, которые направлены на это значение или параметр как таковые. Например, описание, в котором употреблено «около X», включает описание «X».
Термины «полинуклеотид» и «нуклеиновая кислота» применяются в контексте данного документа взаимозаменяемо, относятся к полимерам нуклеотидов любой длины и включают дезоксирибонуклеиновую кислоту (ДНК) и рибонуклеиновую кислоту (РНК). Нуклеотиды могут представлять собой дезоксирибонуклеотиды, рибонуклеотиды, модифицированные нуклеотиды или основания и/или их аналоги, или любой субстрат, который может быть включен в полимер посредством ДНК- или РНК-полимеразы, или с помощью синтетической реакции.
В контексте данного документа термин «клетка-хозяин» включает отдельную клетку или клеточную культуру, которая может быть или была реципиентом любого вектора из этого документа. Клетки-хозяева включают потомство одной клетки-хозяина, и это потомство не обязательно может быть полностью идентичным, по морфологии или по общему количеству комплемента ДНК, исходной родительской клетке из-за естественной, случайной или преднамеренной мутации и/или изменения. Клетка-хозяин включает клетки, трансфицированные или инфицированные вектором, содержащим нуклеиновую кислоту по настоящему документу. Клетки-хозяева могут быть прокариотическими или эукариотическими клетками.
Термин «выделенный» при применении в связи с полинуклеотидами, полипептидами и т.п., означает, что молекула или полипептид были удалены из своего исходного окружения.
В контексте данного документа термины «% идентичность» или «% идентичности» могут быть определены с помощью способов, хорошо известных в данной области техники. Например, % идентичности рассчитывается следующим образом. Запрашиваемая последовательность выравнивается с целевой последовательностью с помощью алгоритма CLUSTAL W [6]. Сравнение выполняется в окне, соответствующем самой короткой из выровненных последовательностей. Самая короткая из выровненных последовательностей в некоторых случаях может быть целевой последовательностью. В других случаях запрашиваемая последовательность может составлять самую короткую из выровненных последовательностей. В каждом положении сравнивают аминокислотные остатки или нуклеотиды, и процент положений в запрашиваемой последовательности, которые имеют идентичные соответствия в целевой последовательности, указывают как % идентичности.
В контексте данного документа «терапевтически эффективное количество» агента относится к количеству, эффективному в дозах и в течение периодов времени, необходимых для достижения желаемого терапевтического или профилактического результата.
Следует понимать, что аспект и варианты осуществления, описанные в данном документе, включают «состоящий» и/или «состоящий по существу из» аспектов и вариантов осуществления. В контексте данного документа, слова в единственном числе означают также и множественное число, если не указано иное.
«hIgG1 LALA-PG», как описано в [7].
«hIgG4 S228P, hIgG4 S241P», как описано в [8].
«G-SP140-8», клон, связывающийся с SP140, отрицательный контроль.
«Клетки expiHEK293», клетки человека, полученные из линии клеток 293, и основной компонент экспрессионной системы Expi293™.
Целью настоящего изобретения является предоставление антител и/или их антигенсвязывающих фрагментов, которые могут применяться для лечения и/или предотвращения воспалительных состояний, в то же время облегчая ранее упомянутые и другие недостатки существующих методов лечения. Кроме того, целью настоящего изобретения является обеспечение агентов для применения в диагностике воспалительных состояний и для изучения белка липазы, стимулируемой солями жёлчных кислот (BSSL).
Более подробно, настоящее изобретение относится к новым выделенным антителам и их антигенсвязывающим фрагментам, которые связываются с ранее не охарактеризованным эпитопом BSSL, расположенным в N-концевой части белка BSSL. Настоящий документ также относится к медицинскому применению антител и их антигенсвязывающих фрагментов, в частности, при лечении воспалительных состояний, а также к родственным фармацевтическим композициям. В настоящем документе также описано применение антител или их антигенсвязывающих фрагментов в качестве молекулярных инструментов при обнаружении BSSL и/или для диагностики заболеваний, связанных с BSSL.
В варианте осуществления, в каждом случае, когда упоминается BSSL, он также включает BSSL человека (hBSSL), если только из контекста не ясно, что hBSSL не предназначен для включения.
В настоящем документе описана новая группа антител против BSSL, включая их антигенсвязывающие фрагменты, которые связываются с ранее неидентифицированным эпитопом на hBSSL. Антитела могут быть гуманизированы или их последовательности CDR могут быть привиты к каркасу, не являющемуся человеческим. Антитела или их антигенсвязывающие фрагменты также могут связываться с BSSL мыши или крысы (mBSSL), хотя аффинность к hBSSL и mBSSL может отличаться из-за разницы в аминокислотах в одном из эпитопов, с которыми связываются антитела и/или их антигенсвязывающие фрагменты.
Как хорошо известно, антитела представляют собой молекулы иммуноглобулина, способные к специфическому связыванию с мишенью (антигеном), такой как углевод, полинуклеотид, липид, полипептид или другой, по меньшей мере через один сайт распознавания антигена, расположенный в вариабельной области молекулы иммуноглобулина. Антитело представляет собой гликопротеин, содержащий по меньшей мере две тяжелые (H) цепи (HC) и две легкие (L) цепи (LC), соединенные дисульфидными связями. Вариабельные области тяжелой цепи (HCVR) и вариабельные области легкой цепи (LCVR) содержат связывающий домен, который взаимодействует с антигеном. Константные области антител могут опосредовать связывание иммуноглобулина с тканями или факторами хозяина, включая различные клетки иммунной системы, например эффекторные клетки, и первый компонент классической системы комплемента, то есть компонент комплемента 1q (C1q).
Описанные в данном документе антитела или их антигенсвязывающие фрагменты можно применять для ингибирования или снижения по меньшей мере некоторых биологических активностей белка BSSL, связанного с ним. Связывание может, например, значительно или полностью ингибировать некоторые биологические активности белка BSSL. Эти эффекты антител или их антигенсвязывающих фрагментов по настоящему изобретению были в высшей степени неожиданными, учитывая, что указанные антитела или их антигенсвязывающие фрагменты не связываются с активным сайтом BSSL. Таким образом, предпочтительно, чтобы антитела или их антигенсвязывающие фрагменты по настоящему изобретению существенно не ингибировали или не снижали ферментативную активность BSSL, например, существенно не ингибировали или не снижали способность BSSL гидролизовать сложные эфиры холестерина (EC 3.1. 1.13).
Антитела или их антигенсвязывающие фрагменты можно применять для снижения провоспалительных эффектов BSSL у субъектов, нуждающихся в этом. Таким образом, антитела или их антигенсвязывающие фрагменты можно применять для лечения и/или профилактики различных воспалительных заболеваний, как дополнительно описано в данном документе. Эти медицинские применения антител или их антигенсвязывающих фрагментов по настоящему изобретению могут быть достигнуты без блокирования ферментативной активности BSSL. Следовательно, антитела или их антигенсвязывающие фрагменты по настоящему изобретению не вносят вклад в отрицательные эффекты, вызванные ингибированием ферментативной активности BSSL, которые могут иметь другие антитела анти-BSSL, связывающиеся с активным сайтом BSSL или связанные с ним.
Кроме того, описанные в настоящем документе антитела или их антигенсвязывающие фрагменты можно применять в диагностических целях для диагностики патологических состояний, связанных с BSSL, таких как воспалительные состояния, связанные с BSSL.
Как продемонстрировано в экспериментальном разделе, применяли несколько подходов, чтобы попытаться разработать гуманизированное антитело BSSL или его антигенсвязывающий фрагмент, но, несмотря на исходное количество около 1000000 кандидатов scFv, было обнаружено, что неожиданно низкое количество демонстрирует достаточную аффинность связывания с белком hBSSL. Из 1 000 000 кандидатов было определено 68 первоначальных кандидатов, которые затем были сокращены до 38 кандидатов и, в конечном итоге, до 5 кандидатов, выбранных для дальнейшей оценки и характеристики. Соответственно, антитела анти-BSSL или их антигенсвязывающие фрагменты не могут быть легко гуманизированы с помощью стандартных протоколов и все же имеют достаточную аффинность связывания с hBSSL. Таким образом, гуманизация антител анти-BSSL или антигенсвязывающих фрагментов была большой проблемой, которую удалось преодолеть, как описано в данном документе, для получения гуманизированных антител или их антигенсвязывающих фрагментов, которые все еще обладают достаточной аффинностью связывания с hBSSL. Эти антитела или их антигенсвязывающие фрагменты по настоящему изобретению имеют общие структурные и функциональные особенности, как описано в другом месте в данном документе.
Генерирование антител и антигенсвязывающих фрагментов
Антитела и их антигенсвязывающие фрагменты по настоящему изобретению получали многоэтапным способом с целью поиска антител и их антигенсвязывающих фрагментов, которые имели достаточно хорошую аффиность связывания с hBSSL. Также целью было предоставить гуманизированные антитела и их антигенсвязывающие фрагменты. Было также обнаружено, что некоторые из идентифицированных антител и их антигенсвязывающие фрагменты связываются с достаточной аффинностью связывания с mBSSL. Хотя настоящее изобретение не ограничивается гуманизированными антителами, связывающимися с BSSL, в данном документе описаны гуманизированные антитела, связывающие BSSL, и их антигенсвязывающие фрагменты.
Антитела и их антигенсвязывающие фрагменты были получены на основе последовательности нечеловеческого моноклонального антитела mBSSL. ДНК, кодирующую иммуноглобулины тяжелой и легкой цепей, получали из нечеловеческой гибридомы, экспрессирующей это антитело, и конструировали так, чтобы содержать немышиные, например, человеческие, иммуноглобулиновые последовательности с применением стандартных методов молекулярной биологии.
Однако неожиданно было обнаружено, что CDR-трансплантат, сконструированный, как описано в Примере 6, не имел такой высокой аффинности связывания, как можно было бы ожидать, на основе антитела mBSSL, которое применяли для обеспечения последовательностей CDR. Таким образом, чтобы получить антитела и их антигенсвязывающие фрагменты с достаточно высокой аффинностью связывания с hBSSL, необходимо было провести дальнейшие модификации каркасной области (FW) антитела путем введения мутаций как в CDR, так и в соседние области FW, как описано в примере 5. Это было достигнуто путем конструирования библиотеки гуманизации, применяемой для отбора и выделения фрагментов scFv, связывающих мышиный и человеческий BSSL, с применением фагового дисплея. Такие методы фагового дисплея для выделения антител человека известны в данной области техники, см., например, US 5 223 409; US 5 403 484; US 5 571 698; US 5 427 908; US 5 580 717; US 5 969 108; US 6 172 197; US 5 885 793; US 6 521 404; US 6 544 731; US 6 555 313; US 6 582 915 и US 6 593 081.
Полученное выделенное антитело и его антигенсвязывающий фрагмент имели минимизированное содержание CDR животного происхождения, при этом допускались только основные остатки нечеловеческой зародышевой линии. Остальные CDR были преобразованы в последовательность v-гена человека, за исключением нескольких новых вариаций путем введения нейтральных по отношению к виду существенных остатков de-novo, таких как аминокислотные остатки IMGT 62, 64, 68, 27, 66, 68, 115 и 116 (см. Фигуры 15 и 16). Эти последовательности CDR могут быть привиты к человеческому или нечеловеческому каркасу для получения гуманизированного или негуманизированного антитела и/или его антигенсвязывающего фрагмента, в зависимости от предполагаемого применения антитела.
В гуманизированных антителах константные области и часть вариабельных областей, за исключением последовательностей CDR, имеют каркас, который происходит из последовательностей иммуноглобулинов зародышевой линии человека. Однако в таких гуманизированных антителах последовательности CDR происходят из зародышевой линии другого вида млекопитающих, такого как мышь, которые были привиты к каркасным последовательностям человека. Такие гуманизированные антитела в контексте настоящего изобретения могут также называться CDR-трансплантатами. Преимущество применения гуманизированных антител состоит в том, что они снижают риск иммуногенных реакций, которые могут возникать, когда применяется каркас из другого вида, если такое антитело вводят человеку. Это открывает возможности для их применения в медицине на людях. Антитела или их антигенсвязывающие фрагменты, описанные в данном документе, являются полезны при применении в диагностических целях и для обнаружения белка BSSL, такого как hBSSL, в различных типах образцов, как более подробно описано в другом месте в данном документе.
В данном документе, таким образом, также описан способ получения выделенного антитела или его антигенсвязывающего фрагмента согласно настоящему изобретению. Указанный способ включает культивирование клетки-хозяина, экспрессирующей антитело или его антигенсвязывающий фрагмент, в условиях, допускающих экспрессию антитела или его антигенсвязывающего фрагмента, из экспрессионного вектора, содержащегося в клетке-хозяине и содержащего полинуклеотид, кодирующий антитело или его антигенсвязывающий фрагмент. Указанный способ также включает выделение антитела или его антигенсвязывающего фрагмента из клетки-хозяина или из культуральной среды, в которой культивируется клетка-хозяин.
Связывание эпитопа антителами или их антигенсвязывающими фрагментами
Было обнаружено, что выделенные антитела или их антигенсвязывающие фрагменты связываются с ранее нераспознанным эпитопом белка BSSL человека, который расположен в N-концевой части белка BSSL. Эпитоп может образовывать конформационный эпитоп BSSL.
Неожиданно было обнаружено, что антитела и их антигенсвязывающие фрагменты, полученные в соответствии с настоящим изобретением, связываются с ранее нераспознанным эпитопом белка hBSSL. Еще более неожиданно было обнаружено, что эпитоп находится не рядом с активным сайтом BSSL для метаболизма липидов, а скорее в N-концевой части BSSL. Это обуславливает несколько полезных эффектов. Один из них заключается в том, что антитела или их антигенсвязывающие фрагменты с меньшей вероятностью вызывают отрицательные побочные эффекты, поскольку они не оказывают значительного влияния на ферментативную липазную активность BSSL. Другое преимущество состоит в том, что антитела или их антигенсвязывающие фрагменты являются пригодными для изучения белка BSSL и его липазной активности, поскольку на нее не оказывают существенного влияния антитела или их антигенсвязывающие фрагменты по настоящему изобретению.
Структура BSSL была описана как имеющая большую сердцевинную область, состоящую из скрученного 11-цепочечного бета-листа, окруженного альфа-спиралями и соединительными петлями ([9], Фигура 12). На N-конце имеется 3-цепочечный бета-лист меньшего размера. Структуру можно сравнить с перчаткой для левой руки, в которой ладонь содержит триаду активного центра рядом с «большим пальцем». При таком сходстве небольшой N-концевой бета-лист расположен на тыльной стороне руки рядом с «мизинцем», см. Фиг. 12. Часть структуры BSSL, которая взаимодействует с молекулой Fab, расположена на небольшом N-концевом бета-листе и в C-концевой части альфа-C, третьей альфа-спирали в структуре, см. Фиг. 13. Другими словами, область связывания антитела находится не рядом с активным сайтом BBSL, а на противоположной стороне от BSSL.
Идентифицированная область эпитопа включает остатки 7-12 (цепь 1 и 2) и 42-55 (область петли, ведущая в цепь 3 листа). Указанный эпитоп довольно плоский, с выступом лишь нескольких характерных остатков, а именно Tyr7, Phe12 и Gln52 (основные взаимодействия перечислены в Таблице 25). Область петли 47-54 хорошо определена и образует однородную поверхность. Пролин 47 важен для стэкинг-взаимодействия с Tyr31 антитела, но в целом поверхность здесь плоская. В предпочтительном варианте осуществления область эпитопа также содержит остатки 174-180 (С-конец альфа-С).
Аспект настоящего изобретения относится к выделенному антителу или его антигенсвязывающему фрагменту, которое специфически связывается с эпитопом BSSL, предпочтительно hBSSL. Указанный эпитоп содержит первую поверхность, содержащую или определяемую аминокислотной последовательностью согласно SEQ ID NO: 1, или аминокислотной последовательностью, имеющей по меньшей мере 80%, например, по меньшей мере 83% идентичности с SEQ ID NO: 1. Указанный эпитоп также содержит вторую поверхность,содержащую или определяемую аминокислотной последовательностью согласно SEQ ID NO: 2, или аминокислотной последовательностью, имеющей по меньшей мере 80%, например, по меньшей мере 85% или по меньшей мере 92% идентичности с SEQ ID NO: 2.
Первый пептид, содержащий аминокислотную последовательность SEQ ID NO: 1, определяет первую поверхность эпитопа в BSSL, а второй пептид, содержащий аминокислотную последовательность SEQ ID NO: 2, определяет вторую поверхность эпитопа в BSSL. Следовательно, первая поверхность содержит или, скорее, определяется аминокислотной последовательностью SEQ ID NO: 1, а вторая поверхность содержит или, скорее, определяется аминокислотной последовательностью SEQ ID NO: 2.
В одном из вариантов осуществления первый пептид содержит аминокислотную последовательность согласно SEQ ID NO: 3 или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 83% и более предпочтительно по меньшей мере 91% идентичности с SEQ ID NO: 3. SEQ ID NO: 3 представляет собой более длинную аминокислотную последовательность, содержащую в качестве части аминокислотную последовательность согласно SEQ ID NO: 1.
Выделенное антитело или его антигенсвязывающий фрагмент может дополнительно специфически связываться с другим пептидом и поверхностью, то есть третьим пептидом и третьей поверхностью BSSL, такой как hBSSL. В одном из вариантов осуществления этот третий пептид содержит аминокислотную последовательность согласно SEQ ID NO: 5 или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 85% идентичности с SEQ ID NO: 5. В другом варианте осуществления третий пептид содержит аминокислоту согласно SEQ ID NO: 4 или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 83%, более предпочтительно по меньшей мере 88%, например, по меньшей мере 94% идентичности с SEQ ID NO: 4. В другом варианте осуществления третий пептид содержит аминокислотную последовательность согласно SEQ ID NO: 6 или аминокислотную последовательность, имеющую по меньшей мере 80%, предпочтительно по меньшей мере 84% и более предпочтительно по меньшей мере 92% идентичности с SEQ ID NO: 6.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент может специфически связываться с первым пептидом, содержащим, например, состоящим из SEQ ID NO: 1, со вторым пептидом, содержащим, например, состоящим из SEQ ID NO: 2 и с третьим пептидом, содержащим, например, состоящим из SEQ ID NO: 4, или с аминокислотной последовательностью, имеющей определенные в данном документе соответствующие идентичности. В альтернативном варианте, вместо связывания с более короткой аминокислотной последовательностью согласно SEQ ID NO: 1 такое антитело или его антигенсвязывающий фрагмент может связываться с более длинной аминокислотной последовательностью согласно SEQ ID NO: 3 или с аминокислотной последовательностью, имеющей указанную в данном документе идентичность.
В другом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент специфически связывается с первым пептидом, содержащим, например, состоящим из SEQ ID NO: 1, со вторым пептидом, содержащим например, состоящим из SEQ ID NO: 2 и с третьим пептидом, содержащим, например, состоящим из SEQ ID NO: 5, или с аминокислотной последовательностью, имеющей определенные в данном документе соответствующие идентичности. В альтернативном варианте, вместо связывания с более короткой аминокислотной последовательностью согласно SEQ ID NO: 1 такое антитело или его антигенсвязывающий фрагмент может специфически связываться с более длинной аминокислотной последовательностью согласно SEQ ID NO: 3 или с аминокислотной последовательностью, имеющей указанную в данном документе идентичность.
В дополнительном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент специфически связывается с первым пептидом, содержащим, например, состоящим из SEQ ID NO: 1, со вторым пептидом, содержащим например, состоящим из SEQ ID NO: 2 и с третьим пептидом, содержащим, например, состоящим из SEQ ID NO: 6, или с аминокислотной последовательностью, имеющей определенные в данном документе соответствующие идентичности. В альтернативном варианте, вместо связывания с более короткой аминокислотной последовательностью согласно SEQ ID NO: 1 такое антитело или его антигенсвязывающий фрагмент может связываться с более длинной аминокислотной последовательностью согласно SEQ ID NO: 3 или с аминокислотной последовательностью, имеющей указанную в данном документе идентичность.
Другой аспект настоящего изобретения направлен на эпитоп BSSL, такой как эпитоп hBSSL, содержащий первый пептид, содержащий, например, состоящий из аминокислотной последовательности согласно SEQ ID NO: 1 или аминокислотной последовательности, имеющей по меньшей мере 80 %, предпочтительно по меньшей мере 83% идентичности с SEQ ID NO: 1. Эпитоп BSSL также содержит второй пептид, содержащий, например, состоящий из аминокислотной последовательности согласно SEQ ID NO: 2 или аминокислотной последовательности, имеющей по меньшей мере 80%, предпочтительно по меньшей мере 85% или по меньшей мере 92% идентичности с SEQ ID NO: 2.
В одном из вариантов осуществления первый пептид содержит, например, состоит из аминокислотной последовательности согласно SEQ ID NO: 3 или аминокислотной последовательности, имеющей определенную в данном документе идентичность.
Эпитоп может дополнительно содержать третий пептид, содержащий, например, состоящий из аминокислотной последовательности согласно SEQ ID NO: 4 или аминокислотной последовательности, имеющей определенную идентичность, аминокислотной последовательности согласно SEQ ID NO: 5. или аминокислотной последовательности, имеющей определенную в данном документе идентичность, или аминокислотной последовательности согласно SEQ ID NO: 6, или аминокислотной последовательности, имеющей определенную в данном документе идентичность.
Такие эпитопы могут быть полезны для разработки антител или их антигенсвязывающих фрагментов, связывающихся с белком BSSL, таким как hBSSL, например, для применения при лечении и/или предотвращении состояний, связанных с BSSL, и/или для применения в качестве молекулярных инструментов для изучения белка BSSL. Таким образом, настоящее изобретение также направлено на применение таких эпитопов для разработки антитела или его антигенсвязывающего фрагмента. Поскольку эти эпитопы не расположены в каталитическом центре липазы белков BSSL, они являются привлекательной мишенью для выработки антител анти-BSSL или антигенсвязывающих фрагментов.
Выделенное антитело или его антигенсвязывающий фрагмент согласно настоящему изобретению может специфически связываться с эпитопом (эпитопами), как определено в данном документе.
Антигенсвязывающие части антител и их антигенсвязывающие фрагменты
Полноразмерное антитело содержит две тяжелые цепи и две легкие цепи, соединенные дисульфидными связями. Каждая тяжелая цепь содержит вариабельную область тяжелой цепи (HVCR) и первую, вторую и третью константные области (CH1, CH2 и CH3). В этом документе термины VH, VH и HCVR применяются взаимозаменяемо. Каждая легкая цепь содержит вариабельную область легкой цепи (LVCR) и константную область легкой цепи (CL). В этом документе термины VL, VL и LCVR применяются взаимозаменяемо.
Области HCVR и LCVR могут быть дополнительно подразделены на области гипервариабельности, называемые областями, определяющими комплементарность (CDR), с вкраплениями более консервативных областей, называемых каркасными областями (FR или FW). Каждая HCVR и LCVR состоит из трех CDR и четырех FR/FW, расположенных от N-конца к С-концу в следующем порядке: FW1, CDR1, FW2, CDR2, FW3, CDR3, FW4.
Удлиненная CDR (eCDR) в контексте настоящего описания относится к аминокислотной последовательности, которая содержит по меньшей мере один дополнительный аминокислотный остаток помимо аминокислот CDR, как определено в соответствии с номенклатурой IMGT.
В данной области техники хорошо известно, что паратоп, также известный как антигенсвязывающий сайт, является частью антитела или его антигенсвязывающего фрагмента, который распознает антиген и связывается с ним. Это небольшая область Fv-области антитела, которая содержит части тяжелой и легкой цепей антитела. Каждое плечо Y-образной формы мономера антитела имеет паратоп, который представляет собой набор из 6 CDR. Паратоп состоит из трех CDR легкой цепи (LCDR) и трех CDR тяжелой цепи (HCDR), которые отходят от складки антипараллельных бета-листов.
В следующих разделах выделенное антитело или его антигенсвязывающий фрагмент по настоящему изобретению определяется структурными особенностями его CDR, другими словами, аминокислотной последовательностью его HCDR и/или LCDR, или аминокислотой структурой областей, содержащих HCDR и/или LCDR. Квалифицированному специалисту будет понятно, что незначительные изменения, такие как замены одного, двух, трех, четырех или даже большего количества аминокислотных остатков в аминокислотной последовательности, могут происходить без влияния на функциональные свойства, такие как его связывающая способность или аффинность связывания с BSSL, таким как hBSSL, выделенного антитела или его антигенсвязывающего фрагмента. Следует понимать, что первая HCDR, вторая HCDR, третья HCDR, первая LCDR, вторая LCDR и третья LCDR могут быть независимо выбраны из перечисленных аминокислотных последовательностей.
Таким образом, один из аспектов изобретения относится к выделенному антителу или его антигенсвязывающему фрагменту, содержащему три CDR HCVR (HCDR) и три CDR LCHV (LCDR). В этом аспекте первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, имеющей по меньшей мере 87%, например, по меньшей мере 87,5% идентичности с SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8 или аминокислотной последовательности, имеющей по меньшей мере 75% идентичности с SEQ ID NO: 8, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9, или аминокислотной последовательности, имеющей по меньшей мере 83%, например, по меньшей мере 91,6% идентичности с SEQ ID NO: 9. Кроме того, в этом аспекте первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10 или аминокислотной последовательности, имеющей по меньшей мере 80% идентичности с SEQ ID NO: 10, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS или аминокислотной последовательности, имеющей по меньшей мере 66% идентичности с аминокислотной последовательностью ATS, например, AAS, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности, имеющей по меньшей мере 87% идентичности с SEQ ID NO: 11.
Экспериментальные данные, представленные в Примере 22, демонстрируют, что вторая LCDR может быть не так важна для взаимодействия с BSSL. Следовательно, в одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит первую HCDR, содержащую, предпочтительно состоящую из, аминокислотной последовательности согласно SEQ ID NO: 7, или аминокислотной последовательности, имеющей по меньшей мере 87%, например, по меньшей мере 87,5% идентичности с SEQ ID NO: 7, вторую HCDR, содержащую, предпочтительно состоящую из, аминокислотной последовательности согласно SEQ ID NO: 8 или аминокислотной последовательности, имеющей по меньшей мере 75% идентичности с SEQ ID NO: 8, третью HCDR, содержащую, предпочтительно состоящую из, аминокислотной последовательности согласно SEQ ID NO: 9, или аминокислотной последовательности, имеющей по меньшей мере 83%, например, по меньшей мере 91,6%, идентичности с SEQ ID NO: 9, первую LCDR, содержащую, предпочтительно состоящую из, аминокислотной последовательности согласно SEQ ID NO: 10, или аминокислотную последовательность, имеющую по меньшей мере 80% идентичности с SEQ ID NO: 10, и третью LCDR, содержащую, предпочтительно состоящую из, аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности, имеющей по меньшей мере 87% идентичности с SEQ ID NO: 11.
В одном из вариантов осуществления первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 8, SEQ ID NO: 18 и SEQ ID NO: 19, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 10 и SEQ ID NO: 20, вторая LCDR содержит, предпочтительно состоит из, аминокислотной последовательности, выбранной из группы, состоящей из ATS и AAS, а третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 11, SEQ ID NO: 21 и SEQ ID NO: 22.
В одном варианте осуществления первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит удлиненную вторую HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 12, удлиненную первую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 14 и удлиненную вторую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 15.
В другом конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит удлиненную вторую HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 12, удлиненную первую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 16 и удлиненную вторую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 17.
В одном варианте осуществления первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 18, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 21.
В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит удлиненную вторую HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 23, расширенную первую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 16 и удлиненную вторую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 15.
В одном варианте осуществления первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 20, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности AAS и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит удлиненную вторую HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 24, расширенную первую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 27 и удлиненную вторую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 29.
В одном варианте осуществления первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 19, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 20, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 22.
В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит удлиненную вторую HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 25, удлиненную первую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 26 и удлиненную вторую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 28.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит первую HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 7, удлиненную вторую HCDR, содержащую, предпочтительно состоящую из одной аминокислотой последовательности, выбранной из группы, состоящей из SEQ ID NO: 12, 23, 24 и 25, и третью HCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит удлиненную первую LCDR, содержащую, предпочтительно состоящую из одной аминокислотной последовательности, выбранной из SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 26 и SEQ ID NO: 27, удлиненную вторую LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 28 и SEQ ID NO: 29, и третью LCDR, содержащую, предпочтительно состоящую из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 11, SEQ ID NO: 21 и SEQ ID NO: 22.
В одном варианте осуществления, когда первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, она также включает аминокислотную последовательность, которая по меньшей мере на 87,5% идентична SEQ ID NO: 7.
В одном варианте осуществления, когда вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8, она также включает аминокислотную последовательность, которая по меньшей мере на 75%, например, по меньшей мере на 87% или по меньшей мере на 87,5% идентична SEQ ID NO: 8.
В одном варианте осуществления, когда третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9, она также включает аминокислотную последовательность, которая по меньшей мере на 83%, например, по меньшей мере на 91,6% идентична SEQ ID NO: 9.
В одном варианте осуществления, когда первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10, она также включает аминокислотную последовательность, которая по меньшей мере на 80% идентична SEQ ID NO: 10.
В одном варианте осуществления, когда вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно ATS или AAS, она также включает аминокислотную последовательность, которая по меньшей мере на 66% идентична любой из аминокислотных последовательностей ATS и AAS.
В одном варианте осуществления, когда третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11, она также включает аминокислотную последовательность, которая по меньшей мере на 87,5% идентична SEQ ID NO: 11.
В одном варианте осуществления, когда третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 21, она также включает аминокислотную последовательность, которая по меньшей мере на 75%, например, по меньшей мере на 87,5% идентична SEQ ID NO: 21.
В одном варианте осуществления, когда третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 22, она также включает аминокислотную последовательность, которая по меньшей мере на 75%, например, по меньшей мере на 87,5% идентична SEQ ID NO: 22.
В одном варианте осуществления, когда удлиненная вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 12, она также включает аминокислотную последовательность, которая по меньшей мере на 77,8%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3%, например, по меньшей мере на 88%, например, по меньшей мере на 88,9%, например, по меньшей мере на 94%, например, по меньшей мере на 94,4% идентична SEQ ID NO: 12.
В одном варианте осуществления, когда удлиненная третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 18, она также включает аминокислотную последовательность, которая по меньшей мере на 75%, например, по меньшей мере на 87,5% идентична SEQ ID NO: 18.
В одном варианте осуществления, когда удлиненная третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 19, она также включает аминокислотную последовательность, которая по меньшей мере на 75%, например, по меньшей мере на 87,5% идентична SEQ ID NO: 19.
В одном варианте осуществления, когда удлиненная вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 23, она также включает аминокислотную последовательность, которая по меньшей мере на 77,8%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3%, например, по меньшей мере на 88%, например, по меньшей мере на 88,9%, например, по меньшей мере на 94%, например, по меньшей мере на 94,4% идентична SEQ ID NO: 23.
В одном варианте осуществления, когда удлиненная вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 24, она также включает аминокислотную последовательность, которая по меньшей мере на 77,8%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3%, например, по меньшей мере на 88%, например, по меньшей мере на 88,9%, например, по меньшей мере на 94%, например, по меньшей мере на 94,4% идентична SEQ ID NO: 24.
В одном варианте осуществления, когда удлиненная вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 25, она также включает аминокислотную последовательность, которая по меньшей мере на 77,8%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3%, например, по меньшей мере на 88%, например, по меньшей мере на 88,9%, например, по меньшей мере на 94%, например, по меньшей мере на 94,4% идентична SEQ ID NO: 25.
В одном варианте осуществления, когда удлиненная первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 14, она также включает аминокислотную последовательность, которая по меньшей мере на 80%, например, по меньшей мере на 90% идентична SEQ ID NO: 14.
В одном варианте осуществления, когда удлиненная первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 16, она также включает аминокислотную последовательность, которая по меньшей мере на 80%, например, по меньшей мере на 90% идентична SEQ ID NO: 16.
В одном варианте осуществления, когда удлиненная первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 20, она также включает аминокислотную последовательность, которая по меньшей мере на 80% идентична SEQ ID NO: 20.
В одном варианте осуществления, когда удлиненная первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 26, она также включает аминокислотную последовательность, которая по меньшей мере на 80%, например, по меньшей мере на 90% идентична SEQ ID NO: 26.
В одном варианте осуществления, когда удлиненная первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 27, она также включает аминокислотную последовательность, которая по меньшей мере на 80%, например, по меньшей мере на 90% идентична SEQ ID NO: 27.
В одном варианте осуществления, когда удлиненная вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 15, она также включает аминокислотную последовательность, которая по меньшей мере на 66,7%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3% идентична SEQ ID NO: 15.
В одном варианте осуществления, когда удлиненная вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 17, она также включает аминокислотную последовательность, которая по меньшей мере на 66,7%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3% идентична SEQ ID NO: 17.
В одном варианте осуществления, когда удлиненная вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 28, она также включает аминокислотную последовательность, которая по меньшей мере на 66,7%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3% идентична SEQ ID NO: 28.
В одном варианте осуществления, когда удлиненная вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 29, она также включает аминокислотную последовательность, которая по меньшей мере на 66,7%, например, по меньшей мере на 83%, например, по меньшей мере на 83,3% идентична SEQ ID NO: 29.
Выделенные антитела или их антигенсвязывающие фрагменты, как описано в данном документе, также или в альтернативном варианте могут быть структурно описаны аминокислотной последовательностью их HCVR и/или LCVR. Квалифицированному специалисту будет понятно, что HCVR и LCVR можно независимо выбрать из перечисленных аминокислотных последовательностей. Как объяснено выше, квалифицированному специалисту будет понятно, что незначительные изменения, такие как замены, включая делецию или добавление аминокислот, одного, двух, трех, четырех или даже большего количества аминокислотных остатков в аминокислотной последовательности могут происходить без воздействия на функциональные свойства выделенного антитела или его антигенсвязывающего фрагмента, такие как его способность связываться с hBSSL. Изменение может быть в аминокислотной последовательности CDR, в аминокислотной последовательности за пределами CDR-областей, которые в данном документе называются каркасными областями, или как в аминокислотной последовательности CDR, так и в аминокислотной последовательности за пределами CDR-областей HCVR или LCVR.
Следовательно, в одном из вариантов осуществления антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34 и SEQ ID NO: 36, и аминокислотной последовательности, являющейся по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34 и SEQ ID NO: 36.
В конкретном варианте осуществления аминокислотную последовательность HVCR выбирают из группы, состоящей из SEQ ID NO: 30, SEQ ID NO: 34 и SEQ ID NO: 36, и аминокислотной последовательности, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 30, SEQ ID NO: 34 и SEQ ID NO: 36. В другом варианте осуществления аминокислотную последовательность HVCR выбирают из группы, состоящей из SEQ ID NO: 34 и SEQ ID NO: 36, и аминокислотной последовательности, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 34 и SEQ ID NO: 36. Например, HCVR может содержать аминокислотную последовательность согласно SEQ ID NO: 36 или аминокислотную последовательность, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 36.
В одном из вариантов осуществления антитело или его антигенсвязывающий фрагмент содержит LCVR, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35, SEQ ID NO: 37. и SEQ ID NO: 38, и аминокислотную последовательность, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38.
В конкретном варианте осуществления аминокислотную последовательность LVCR выбирают из группы, состоящей из SEQ ID NO: 31, SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38, и аминокислотной последовательности, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 31, SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38. В другом конкретном варианте осуществления аминокислотную последовательность LVCR выбирают из группы, состоящей из SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38, и аминокислотной последовательности, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38; например, выбирают из группы, состоящей из SEQ ID NO: 37 и SEQ ID NO: 38, и аминокислотной последовательности, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 37 и SEQ ID NO: 38. Например, HCVR может содержать аминокислотную последовательность согласно SEQ ID NO: 37 и аминокислотную последовательность, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной любой из SEQ ID NO: 37.
В одном из вариантов осуществления антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34 и SEQ ID NO: 36, и аминокислотную последовательность, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99 % идентичной любой из SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34 и SEQ ID NO: 36. Антитело или его антигенсвязывающий фрагмент также содержит LCVR, содержащую аминокислотную последовательность, независимо выбранную из группы, состоящей из SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35, SEQ ID NO: 37. и SEQ ID NO: 38, и аминокислотную последовательность, которая является по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99 % идентичной любой из SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38.
В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 36, и LCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 37.
В другом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 36, и LCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 38.
В дополнительном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 30, и LCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 31.
В еще одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 32, и LCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 33.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит HCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 34, и LCVR, содержащую, предпочтительно состоящую из аминокислотной последовательности согласно SEQ ID NO: 35.
Один аспект настоящего изобретения относится к выделенному антителу или его антигенсвязывающему фрагменту, содержащему HCVR, состоящую из аминокислотной последовательности, выбранной из i) ZH1-[GYTFTSYN]-ZH2-[X53GVIX57PGDGX64TSYX68QKFX72]-ZH3-[ARDYYGSSPLGY]-ZH4, или аминокислотной последовательности, являющейся по меньшей мере на 92% идентичной последовательности, определенной в i), например, на 93% или больше, например, на 94% или больше, например, на 95% или больше, например, на 96% или больше, например, на 97% или больше, например, на 98% или больше, например, на 99% или больше идентичностей последовательности, определенная в i), и LCVR, состоящая из аминокислотной последовательности, выбранной из ii) ZL1-[X24ASX27SISYX39N]-ZL2-[AX57SX66LX68]-ZL3-[HQRSSX115PT]-ZL4, или аминокислотной последовательности, являющейся по меньшей мере на 87 % идентичной последовательности, определенной в ii), например, на 88% или больше, например, на 89% или больше, например, на 90% или больше, например, на 91% или больше, например, на 92% или больше, например, на 93% или больше, например, на 94% или больше, например, на 95% или больше, например, на 96% или больше, например, на 97% или больше, например, на 98% или больше, например, на 99% или больше идентичной последовательности, определенной в ii).
В этом аспекте каждая область из ZH1, ZH2, ZH3 и ZH4 независимо представляет ноль, один или несколько независимо выбранных аминокислотных остатков, и каждая область из ZL1, ZL2, ZL3 и ZL4 независимо представляет ноль, один или несколько независимо выбранных аминокислотных остатков. X53 выбирают из I и M, X57 выбирают из N и Y, X64 выбирают из A и S, X68 выбирают из A и N, X72 выбирают из K и Q, X24 выбирают из S и R, X27 выбирают из S и P, X39 выбирают из M и L, X57 выбирают из A и T, X66 выбирают из K и S, X68 выбирают из A и P, и X115 выбирают из S, T и Y. Нумерация аминокислотных остатков соответствует стандарту нумерации IMTG, т.е. для положения "Xn" в последовательности i) или ii), как определено выше, n является целым числом и обозначает положение аминокислотного остатка X согласно нумерации IMGT.
GYTFTSYN представлен в SEQ ID NO: 7, X53GVIX57PGDGX64TSYX68QKFX72 представлен в SEQ ID NO: 169, ARDYYGSSPLGY представлен в SEQ ID NO: 9, X24ASX27SISYX39N представлен в SEQ ID NO: 170, AX57SX66LX68 представлен в SEQ ID NO: 171 и HQRSSX115PT представлен в SEQ ID NO: 172.
В данном документе представлены антитела или их антигенсвязывающие фрагменты, где Xn в последовательности i) независимо выбирают из группы возможных остатков, перечисленных ниже в перечне A. Квалифицированному специалисту будет понятно, что Xn можно выбрать из любой из перечисленных групп возможных остатков и что этот выбор не зависит от выбора аминокислот в Xm, где n≠m. Таким образом, любой из перечисленных возможных остатков в положении Xn можно независимо объединить с любым из перечисленных возможных остатков в любом другом вариабельном положении согласно перечню A.
Перечень A: Список возможных аминокислотных остатков в последовательности i)
X53 может представлять собой I;
X53 может представлять собой M;
X57 может представлять собой N;
X57 может представлять собой Y;
X59 может представлять собой G;
X59 может представлять собой S;
X64 может представлять собой A;
X64 может представлять собой S;
X68 может быть выбран из А и Т;
X68 может быть выбран из А и N;
X68 может быть выбран из T и N;
X68 может представлять собой A;
X68 может представлять собой N;
X68 может представлять собой T;
X72 может представлять собой K; и
X72 может представлять собой Q.
Аналогичным образом, в данном документе представлены антитела или их антигенсвязывающие фрагменты, где Xk в последовательности ii) в данном случае независимо выбирают из группы возможных остатков согласно перечню В, приведенному ниже. Квалифицированному специалисту будет понятно, что Xk можно выбрать из любой из перечисленных групп возможных остатков и что этот выбор не зависит от выбора аминокислот в Xl, где k≠l. Таким образом, любой из перечисленных возможных остатков в положении Xk из перечня А можно независимо объединить с любым из перечисленных возможных остатков в любом другом вариабельном положении согласно перечню В.
Перечень В: Список возможных аминокислотных остатков в последовательности ii)
X24 может представлять собой S;
X24 может представлять собой R;
X27 может представлять собой S;
X27 может представлять собой P;
X39 может представлять собой M;
X39 может представлять собой L;
X40 может представлять собой H;
X40 может представлять собой N;
X57 может представлять собой A;
X57 может представлять собой T;
X66 может быть выбран из K и S;
X66 может быть выбран из R и S;
X66 может быть выбран из R и K;
X66 может представлять собой K;
X66 может представлять собой R;
X66 может представлять собой S;
X68 может быть выбран из А и P;
X68 может быть выбран из А и Q;
X68 может быть выбран из P и Q;
X68 может представлять собой A;
X68 может представлять собой P;
X68 может представлять собой Q.
X105 может представлять собой H;
X105 может представлять собой Q.
X115 может быть выбран из S и Т;
X115 может быть выбран из S и Y;
X115 может быть выбран из T и Y;
X115 может представлять собой S;
X115 может представлять собой Y; и
X115 может представлять собой T.
С целью пояснения следует указать, что выбор аминокислотных остатков для аминокислотных положений в последовательности i) из перечня A не зависит от выбора аминокислотных остатков для аминокислотных положений в последовательности ii) из перечня B. Во избежание сомнений, перечень A и перечень B приводит несколько конкретных и индивидуальных примеров в соответствии с настоящим изобретением, и приведенные примеры можно свободно комбинировать.
Как определено выше, каждая область из ZH1, ZH2, ZH3 и ZH4 может представлять ноль, один или несколько независимо выбранных аминокислотных остатков. Идентичность и количество аминокислотных остатков в каждой из указанных областей ZH1, ZH2, ZH3 и ZH4 могут быть выбраны независимо.
Аналогично, каждый из ZL1, ZL2, ZL3 и ZL4 может представлять собой ноль, один или несколько независимо выбранных аминокислотных остатков. Идентичность и количество аминокислотных остатков в каждом из указанных ZL1, ZL2, ZL3 и ZL4 могут быть выбраны независимо. Кроме того, ZL1 может быть связана через аминокислотный линкер или другой линкер с ZH1 или ZH4. Также, ZL4 может быть связана через аминокислотный линкер или другой линкер с ZH1 или ZH4. Таким образом, последовательность, определенная в i), и последовательность, определенная в ii), могут быть частью одной аминокислотной последовательности, другими словами, могут быть частью одного полипептида.
В одном варианте осуществления ZH1 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 39 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 39.
В одном варианте осуществления ZH2 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 40 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 40.
В одном варианте осуществления ZH3 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 41 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 41.
В одном варианте осуществления ZH4 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 42 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 42.
В одном варианте осуществления ZL1 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 43 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 43.
В одном варианте осуществления ZL2 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 44 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 44.
В одном варианте осуществления ZL3 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 45 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 45.
В одном варианте осуществления ZL4 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 46 или аминокислотной последовательности, которая является по меньшей мере на 90%, например, по меньшей мере на 91%, например, по меньшей мере на 92%, например, по меньшей мере на 93%, например, по меньшей мере на 94%, например, по меньшей мере на 95%, например, по меньшей мере на 96%, например, по меньшей мере на 97%, например, по меньшей мере на 98%, например, по меньшей мере на 99% идентичной последовательности SEQ ID NO: 46.
Квалифицированному специалисту будет понятно, что % идентичности каждой области из ZH1, ZH2, ZH3, ZH4, ZL1, ZL2, ZL3 и ZL4 с аминокислотной последовательностью согласно соответствующей SEQ ID NO: приведенной выше не зависит от % идентичности любой другой области из ZH1, ZH2, ZH3, ZH4, ZL1, ZL2, ZL3 и ZL4 с ее соответствующей SEQ ID NO:. Таким образом, например, ZH1 может проявлять 95% идентичности с SEQ ID NO: 39, а ZH2 может проявлять 99% идентичности с SEQ ID NO: 40.
В ходе процесса гуманизации, в результате которого получают представленные в данном документе новые антитела анти-BSSL или их антигенсвязывающие фрагменты, несколько мутаций были введены как в CDR, так и в соседние области FW. Для целей настоящего изобретения каждое антитело HCVR и LCVR состоит из трех CDR, из которых одна, две или три CDR могут быть удлиненными CDRS (eCDR), и четырех FR/FW, расположенных от N-конца до C-конца. в следующем порядке, как определено в Таблице 1.
Таблица 1 – Нумерация IMGT
Три CDR в HCRV фланкированы каркасными областями, которые могут быть областями ZH1, ZH2, ZH3 и ZH4, описанными выше. Три CDR в LCRV фланкированы каркасными областями, которые могут быть областями ZL1, ZL2, ZL3 и ZL4, описанными выше.
HVCR антител или их антигенсвязывающих фрагментов, сконструированных в рамках настоящего изобретения, перечислены среди аминокислотных последовательностей в группе, состоящей из SEQ ID NO: 30, 32, 34, 36 и 47-84. Соответственно, LVCR антител или их антигенсвязывающих фрагментов, сконструированных в рамках настоящего изобретения, перечислены среди аминокислотных последовательностей в группе, состоящей из SEQ ID NO: 31, 33, 35, 37, 38 и 86-123. Конкретные примеры этих антител или их антигенсвязывающих фрагментов содержат, предпочтительно состоят из следующих комбинаций HVCR и LVCR: SEQ ID NO: 30 и 31; SEQ ID NO: 32 и 33; SEQ ID NO: 34 и 35; SEQ ID NO: 36 и 37; SEQ ID NO: 36 и 38; SEQ ID NO: 47 и 81; SEQ ID NO: 48 и 82; SEQ ID NO: 49 и 83; SEQ ID NO: 50 и 84; SEQ ID NO: 51 и 85; SEQ ID NO: 52 и 86; SEQ ID NO: 53 и 87; SEQ ID NO: 54 и 88; SEQ ID NO: 55 и 89 ; SEQ ID NO: 56 и 90; SEQ ID NO: 57 и 91; SEQ ID NO: 58 и 92; SEQ ID NO: 59 и 93; SEQ ID NO: 60 и 94; SEQ ID NO: 61 и 95; SEQ ID NO: 62 и 96; SEQ ID NO: 63 и 97; SEQ ID NO: 64 и 98; SEQ ID NO: 65 и 99; SEQ ID NO: 66 и 100; SEQ ID NO: 67 и 101; SEQ ID NO: 68 и 102; SEQ ID NO: 69 и 103; SEQ ID NO: 70 и 104; SEQ ID NO: 71 и 105; SEQ ID NO: 72 и 106; SEQ ID NO: 73 и 107; SEQ ID NO: 74 и 108; SEQ ID NO: 85 и 109; SEQ ID NO: 86 и 110; SEQ ID NO: 77 и 111; SEQ ID NO: 78 и 112; SEQ ID NO: 79 и 113 или SEQ ID NO: 80 и SEQ ID NO: 114.
Квалифицированному специалисту будет понятно, что различные модификации и/или добавления могут быть осуществлены для антитела или его антигенсвязывающего фрагмента, как описано в данном документе, для адаптации указанного антитела или его антигенсвязывающего фрагмента к конкретному применению без отклонения от объем настоящего изобретения. Например, антитело или его антигенсвязывающий фрагмент может иметь аминокислотную последовательность, которая была удлинена и/или содержит дополнительные аминокислоты на С-конце и/или N-конце, например, на С-конце и/или на N-конце его тяжелой или легкой цепи. Таким образом, антитело или его антигенсвязывающий фрагмент может содержать любое подходящее количество дополнительных аминокислотных остатков, например, по меньшей мере один дополнительный аминокислотный остаток. Каждый дополнительный аминокислотный остаток может быть добавлен индивидуально или вместе, с целью, например, оптимизации и/или упрощения продуцирования, очистки, стабилизации in vivo или in vitro,связывания или обнаружения полипептида. Такие дополнительные аминокислотные остатки могут включать один или большее количество аминокислотных остатков, добавленных с целью химического связывания. Примером может служить добавление остатка цистеина. Дополнительные аминокислотные остатки могут также обеспечивать «метку» для очистки или обнаружения антитела или его антигенсвязывающего фрагмента, такого как метка His6, метка (HisGlu)3, метка "myc" (c-myc) или метка FLAG.
Выделенное антитело или его антигенсвязывающий фрагмент по настоящему изобретению можно выбрать, помимо прочего, из полноразмерных антител, комбинаций последовательностей CDR, одноцепочечных вариабельных фрагментов, фрагментов Fab, фрагментов F(ab')2, фрагментов F(ab’)3, фрагментов Fab' , фрагментов Fd, фрагментов Fv, фрагментов dAb, выделенных областей, определяющих комплементарность (CDR), и нанотел.
В одном из вариантов осуществления антитело или его антигенсвязывающий фрагмент выбирают из группы, состоящей из человеческого антитела, гуманизированного антитела и химерного антитела или их антигенсвязывающего фрагмента.
Может быть желательно снизить или устранить эффекторную функцию антител или их антигенсвязывающих фрагментов, например, для предотвращения гибели клеток-мишеней или нежелательной секреции цитокинов. Это может быть особенно подходящим, когда антитела или их антигенсвязывающие фрагменты предназначены для взаимодействия с рецепторами клеточной поверхности и предотвращения взаимодействий рецептор-лиганд, то есть антагонистов. Другие примеры, когда может потребоваться снижение эффекторной функции, включают предотвращение взаимодействия конъюгатов «антитело-лекарственное средство» с рецепторами Fc (FcγRs), приводящего к нецелевой цитотоксичности.
Следовательно, в одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит по меньшей мере одну Fc сайлесинг-мутацию, ингибирующую взаимодействие с FcγR. Например, антитело или его антигенсвязывающий фрагмент на основе класса изотипа IgG1 может содержать по меньшей мере одну, предпочтительно по меньшей мере две и большее количество, предпочтительно все три Fc сайлесинг-мутации L234A, L235A и P329G.
Кроме того, антитела или их антигенсвязывающие фрагменты изотипа IgG4 считаются потенциальными кандидатами для иммунотерапии, когда желательны пониженные эффекторные функции. Антитела IgG4, как известно, представляют собой динамические молекулы, способные подвергаться процессу, известному как обмен Fab-плеча (FAE), и, не ограничиваясь теорией, считается, что это приводит к продуцированию функционально моновалентных биспецифических антител (bsAb) с неизвестной специфичностью и, следовательно, потенциально, снижению терапевтической эффективности. Это может привести к нежелательной непрогнозируемости фармакодинамики для иммунотерапии человека.
Следовательно, в одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит по меньшей мере одну стабилизирующую мутацию, которая предотвращает или снижает замену плеча Fab in vivo. Например, в данной области техники было высказано предположение, что одной аминокислотной мутации (S228P) в области ядра-шарнира IgG4 достаточно для предотвращения FAE in vivo [8]. В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент относится к подклассу изотипа IgG4, и по меньшей мере одна стабилизирующая мутация представляет собой S228P.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент имеет класс изотипа, выбранный из группы, состоящей из IgG, IgA, IgM, IgD и IgE. В конкретном варианте осуществления классом изотипа является IgG. Например, выделенное антитело или его антигенсвязывающий фрагмент можно выбрать из группы, состоящей из подкласса изотипов IgG1 и IgG4.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент представляет собой моноклональное антитело или его антигенсвязывающий фрагмент. Моноклональное антитело или его антигенсвязывающий фрагмент предпочтительно представляет собой гуманизированное моноклональное антитело или его антигенсвязывающий фрагмент.
В настоящее время предпочтительные антитела или их антигенсвязывающие фрагменты обозначаются S-SL048-11 (в данном документе также обозначается клон 11, тяжелая цепь SEQ ID NO: 119 и легкая цепь SEQ ID NO: 120), S-SL048-46 (в данном документе также обозначается клон 46, тяжелая цепь SEQ ID NO: 121 и легкая цепь SEQ ID NO: 122), S-SL048-106 (в данном документе также обозначается клон 106, тяжелая цепь SEQ ID NO: 123 и легкая цепь SEQ ID NO: 124), S -SL048-116 (в данном документе также обозначается клон 116, тяжелая цепь SEQ ID NO: 125 и легкая цепь SEQ ID NO: 126) и S-SL048-118 (в данном документе также обозначается клон 118, тяжелая цепь SEQ ID NO: 127 и легкая цепь SEQ ID NO: 128).
Стабильность этих пяти кандидатов оценивали с помощью эксклюзионной хроматографии (SEC), SDS-PAGE, нано-дифференциальной сканирующей флуориметрии (нано-DSF) и дифференциального светорассеяния. В результате объединения этих данных в общее ранжирование, был сделан вывод, что наиболее стабильными кандидатами в формате hIgG4 S228P были кандидаты 106, 118 и 116.
Аффинность связывания
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент согласно данному документу может иметь аффинность к hBSSL не более чем KD 1×10-7 M, предпочтительно не более чем KD 1×10-8 M. Например, выделенное антитело или его антигенсвязывающий фрагмент может иметь аффинность к hBSSL не более чем KD 5 нM, например, не более чем KD 3 нM.
Как продемонстрировано в экспериментальном разделе, выделенное антитело или его антигенсвязывающий фрагмент может иметь аффинность к hBSSL не более чем KD 1.7 нM, например, не более чем KD 1,7, 1,6, 1,5, 1,4, 1,3, 1,2, 1,1, 1,0, 0,9, 0,8, 0,7 или не более чем 0,6 нМ. В одном примере выделенное антитело или его антигенсвязывающий фрагмент, который связывается с hBSSL в соответствии с настоящим описанием, имеет аффинность к hBSSL в диапазоне KD 0,6-1,7 нМ, например, в диапазоне KD 0,6-1,0, 0,7-0,9, 0,8-1,6, 0,9-1,5, 1,0-1,7, 1,1-1,6, 1,2-1,7, 1,3-1,5, 1,0-1,4, 0,7-1,5, 0,7-1,6 или 1,0-1,7 нМ.
Фармацевтические композиции
Термин «фармацевтическая композиция» относится к препарату, который находится в такой форме, чтобы обеспечить эффективность биологической активности содержащегося в нем активного ингредиента, и который не содержит дополнительных компонентов, которые являются неприемлемо токсичными для субъекта, которому будет вводиться данный состав. Фармацевтические композиции по настоящему изобретению содержат антитело и/или его антигенсвязывающий фрагмент, такой как scFv, как определено в данном документе, и фармацевтически приемлемый носитель или вспомогательное вещество.
Антитело или его антигенсвязывающий фрагмент, такой как scFv, как определено в данном документе, можно приготовить с помощью известных в данной области техники способов в форме, например, таблеток, капсул, водных или масляных растворов, суспензий, эмульсий, кремов, мази, гелей, назальных спреев, суппозиториев, тонко измельченных порошков или аэрозолей для ингаляций и для парентерального применения (включая внутривенную, подкожную или внутримышечное введение), стерильных водных или масляных растворов или суспензий или стерильных эмульсий.
Таким образом, в настоящем документе предлагается композиция, содержащая выделенное антитело или его антигенсвязывающий фрагмент, как описано в данном документе, и по меньшей мере одно фармацевтически приемлемое вспомогательное вещество или носитель. Например, вспомогательное вещество может представлять собой разбавитель. В одном примере фармацевтическая композиция может дополнительно содержать по меньшей мере один дополнительный активный агент, например, по меньшей мере два дополнительных активных агента, например, по меньшей мере три дополнительных активных агента. Неограничивающими примерами дополнительных активных агентов, которые могут оказаться полезными в такой комбинации, являются агенты, модифицирующие иммунный ответ.
«Фармацевтически приемлемый носитель» относится к ингредиенту в фармацевтическом составе, отличному от активного ингредиента, который является нетоксичным для субъекта. Фармацевтически приемлемый носитель включает, без ограничения, буфер, вспомогательное вещество, стабилизатор или консервант.
В контексте данного документа термин «фармацевтически приемлемый носитель» включает любые и все растворители, дисперсионные среды, покрытия, антибактериальные и противогрибковые агенты, изотонические и замедляющие абсорбцию агенты и тому подобное, которые являются физиологически совместимыми. Предпочтительно носитель подходит для перорального, а также внутривенного, внутримышечного, подкожного, спинномозгового или эпидермального введения (например, путем инъекции или инфузии).
Фармацевтическая композиция согласно настоящему изобртению также может включать фармацевтически приемлемый антиоксидант. Примеры фармацевтически приемлемых антиоксидантов включают в себя: (1) водорастворимые антиоксиданты, такие как аскорбиновая кислота, гидрохлорид цистеина, бисульфат натрия, метабисульфит натрия, сульфит натрия и тому подобное; (2) маслорастворимые антиоксиданты, такие как аскорбилпальмитат, бутилированный гидроксианизол (ВНА), бутилированный гидрокситолуол (ВНТ), лецитин, пропилгаллат, альфа-токоферол и тому подобное; и (3) металлохелатирующие агенты, такие как лимонная кислота, этилендиаминтетрауксусная кислота (ЭДТК), сорбит, винная кислота, фосфорная кислота и тому подобное.
Примеры подходящих водных и неводных носителей, которые могут применяться в фармацевтических композициях по настоящему изобретению, включают воду, этанол, полиолы (такие как глицерин, пропиленгликоль, полиэтиленгликоль и тому подобное) и их подходящие смеси, растительные масла, такие как оливковое масло и инъекционные органические сложные эфиры, такие как этилолеат. Надлежащая текучесть может поддерживаться, например, путем применения материалов для покрытия, таких как лецитин, путем поддержания требуемого размера частиц в случае дисперсий и путем применения поверхностно-активных веществ.
Данные композиции также могут содержать адъюванты, такие как консерванты, смачивающие агенты, эмульгирующие агенты и диспергирующие агенты. Предотвращение присутствия микроорганизмов может быть обеспечено как с помощью процедур стерилизации, так и путем включения различных антибактериальных и противогрибковых агентов, например, парабена, хлорбутанола, фенолсорбиновой кислоты и тому подобного. Также может быть желательно включать в композиции изотонические агенты, такие как сахара, хлорид натрия и тому подобное. Кроме того, продленную абсорбцию инъекционной фармацевтической формы можно обеспечивать путем включения агентов, которые замедляют абсорбцию, таких как моностеарат алюминия и желатин.
Фармацевтически приемлемые носители включают стерильные водные растворы или дисперсии и стерильные порошки для предусмотренного для немедленного приема приготовления стерильных растворов или дисперсий для инъекций. Применение таких сред и агентов в отношении фармацевтически активных веществ известно в данной области техники.
В настоящем изобретении также предлагается набор частей, включающих антитело или его антигенсвязывающий фрагмент, или фармацевтическую композицию согласно настоящему изобретению, средства для введения антитела или его антигенсвязывающего фрагмента, или фармацевтическую композицию, и, необязательно, вкладыш в упаковку с инструкцией по применению. Средством для введения антитела или его антигенсвязывающего фрагмента или фармацевтической композиции может быть, например, шприц. Термин «вкладыш в упаковку» применяется для обозначения инструкций, обычно включаемых в коммерческие упаковки терапевтических продуктов, которые содержат информацию о показаниях, применении, дозах, введении, комбинированной терапии, противопоказаниях и/или предупреждениях относительно применения таких терапевтических продуктов.
Медицинское применение выделенных антител или их антигенсвязывающих фрагментов.
Антитело или его антигенсвязывающий фрагмент согласно настоящему изобретению можно применять для эффективного снижения провоспалительного эффекта BSSL у субъекта, такого как человек.
Преимущество антител и их антигенсвязывающих фрагментов по настоящему изобретению состоит в том, что они не связываются с активным сайтом белка BSSL, который отвечает за липазную активность BSSL. Это продемонстрировано и подробно описано в экспериментальной части. Таким образом, снижается риск негативных побочных эффектов, поскольку активность липазы существенно не изменяется.
Таким образом, настоящее изобретение направлено на выделенное антитело или его антигенсвязывающий фрагмент, такой как scFv, или фармацевтическую композицию, как определено в данном документе, для применения в качестве лекарственного средства.
Настоящее изобретение также направлено на выделенное антитело или его антигенсвязывающий фрагмент, или на фармацевтическую композицию, как определено в данном документе, для применения при лечении и/или предотвращении воспалительного заболевания. Настоящее изобретение также направлено на применение выделенного антитела или его антигенсвязывающего фрагмента, такого как scFv, или фармацевтической композиции, как определено в данном документе, для изготовления лекарственного средства для лечения и/или предотвращения воспалительного заболевания. Настоящее изобретение также направлено на способ лечения и/или облегчения, и/или предотвращения, и/или профилактики воспалительного заболевания. Указанный способ включает введение терапевтически эффективного количества выделенного антитела или его антигенсвязывающего фрагмента, такого как scFv, или фармацевтической композиции нуждающемуся в этом субъекту.
Различные модели in vivo можно применять для прогнозирования эффекта антител и антигенсвязывающих фрагментов при лечении и/или предотвращении воспалительного заболевания. Обычно в этих моделях используют мышей и вводят различные вещества, чтобы вызвать иммунный ответ. Таким образом, действие антител или их антигенсвязывающих фрагментов на такой иммунный ответ может быть изучено после введения антител/антигенсвязывающих фрагментов.
Одной из моделей, которые можно применять, является модель так называемого «коллаген-индуцированного артрита» (CIA). В этой модели коллаген типа II (CII) в полном адъюванте Фрейнда (CFA) обычно вводят с бустерной дозой CII в неполном адъюванте Фрейнда (IFA) в день 21. Эта модель индуцирует аутоиммунный артрит. Артрит обычно возникает через 21-28 дней после первой инъекции CII. Эта модель является зависимой от В- и Т-клеток (адаптивный иммунитет).
Другой моделью является «артрит, индуцированный антителами к коллагену» (CAIA). В этой модели вводят смесь антител CII, обычно с бустерной дозой липополисахаридов (LPS) в день 5. Эта модель не является зависимой от B- и T-клеток (врожденный иммунитет). Протокол тестирования in vivo эффективности антител и их антигенсвязывающих фрагментов по настоящему изобретению при лечении и/или предотвращении воспалительного заболевания с помощью модели CAIA описан в экспериментальном разделе этого документа.
Еще одной моделью является модель «артрита, индуцированного глюкозо-6-фосфат-изомеразой». В этой модели вводят пептид, соответствующий последовательности глюкозо-6-фосфат-изомеразы, чтобы индуцировать иммунный ответ. Эта модель является зависимой от Т-лимфоцитов.
Другой моделью является модель «пристан-индуцированного артрита» (ПИА), в которой вводят пристан. Эта модель является зависимой от Т-лимфоцитов.
Также может применяться модель «колита, индуцированного декстрансульфатом натрия», в которой декстрансульфат натрия (DSS) вводят с питьевой водой.
Все вышеперечисленные способы хорошо известны специалисту в данной области техники. Указанные способы можно применять для тестирования in vivo эффективности антител или их антигенсвязывающих фрагментов по настоящему изобретению.
Воспалительное заболевание, которое следует лечить и/или предотвращать в соответствии с настоящим изобретением, может, например, быть хроническим воспалительным заболеванием. Воспалительное заболевание может быть местным или системным воспалительным заболеванием.
Воспалительное заболевание может быть, например, аутоиммунным заболеванием или аутовоспалительным заболеванием. Другой тип воспалительного заболевания представляет собой воспалительное заболевание, опосредованное клетками-естественными киллерами (NK). Такие воспалительные заболевания, опосредованные NK-клетками, включают ревматоидный артрит (RA), системный ювенильный идиопатический артрит (sJIA), синдром активации макрофагов (MAS), системную красную волчанку (SLE), системный склероз, множественный склероз (MS), синдром Шегрена и воспалительное заболевание кишечника (IBD).
В одном из вариантов осуществления воспалительное заболевание выбирают из группы, состоящей из RA, JIA псориатического артрита, IBD, например, болезни Крона или язвенного колита (UC), стеатоза печени, также называемого стеатозом печени, и гипервоспаления.
В конкретном варианте осуществления воспалительное заболевание представляет собой воспалительное состояние, вызванное патогеном, таким как бактерия или вирус. Примеры таких вирусов включают коронавирусы, такие как коронавирус 1, вызывающий тяжелый острый респираторный синдром (SARS-CoV-1) или SARS-CoV-2. Этот вирус вызывает коронавирусную болезнь 2019 (COVID-19). Пациенты с тяжелой формой COVID-19 часто страдают системным гипервоспалением, включая повышенные уровни различных воспалительных цитокинов, таких как интерлейкин 2 (IL-2), IL-7, IL-6, гранулоцитарно-макрофагальный колониестимулирующий фактор (GM-CSF), интерферон-γ индуцибельный белок 10 (IP-10), моноцитарный хемоаттрактантный белок 1 (MCP-1), макрофагальный воспалительный белок 1-α (MIP-1α) и TNF-α.
RA в первую очередь поражает суставы, но также может быть системным воспалительным заболеванием, которое может характеризоваться внесуставными проявлениями в нескольких органах. Следовательно, RA можно рассматривать как системное воспалительное заболевание.
Кроме того, описанное в данном документе выделенное антитело или его антигенсвязывающий фрагмент, такой как scFv-фрагмент, или фармацевтическая композиция может быть альтернативой существующим биологическим методам лечения пациентов, не отвечающих или временно отвечающих на существующие ингибиторы фактора некроза опухоли альфа (TNFα), что снижает потребность в применении кортикостероидов и/или иммунодепрессантов и/или фармацевтических препаратов. Таким образом, применение описанного в данном документе выделенного антитела или его антигенсвязывающего фрагмента, такого как scFv-фрагмент, предотвращает и/или снижает нежелательные эффекты и/или побочные эффекты альтернативных схем лечения у пациентов, что является ключевым вопросом при оказании медицинской помощи в целом и особенно важно для молодых пациентов и детей, а также для пациентов с ослабленным иммунитетом и/или пациентов пожилого возраста.
Лечение и/или предотвращение с применением выделенного антитела и/или его антигенсвязывающего фрагмента или фармацевтической композиции, как описано в данном документе, обычно представляет собой пассивную иммунотерапию, что означает, что антитело или его антигенсвязывающий фрагмент, или фармацевтическую композицию, содержащую такие антитела и/или их антигенсвязывающие фрагменты, вводят нуждающемуся в этом субъекту. Однако также можно применять другие типы иммунотерапевтических методов, такие как генная терапия, при которой вместо введения антитела или его антигенсвязывающего фрагмента субъекту вводят непосредственно генную конструкцию, способную экспрессировать такое антитело или его антигенсвязывающий фрагмент.
Субъектом согласно настоящему изобретению может быть человек или животное, не относящееся к человеку. Термин «животное, не относящееся к человеку» включает всех позвоночных, например, млекопитающих и немлекопитающих, таких как приматы, не относящиеся к человеку, овцы, собаки, кошки, лошади, коровы, куры, земноводные, рептилии и т.д. Млекопитающие включают, помимо прочего, домашних животных (например, коров, овец, кошек, собак и лошадей), приматов (например, людей и приматов, не относящихся к человеку, таких как обезьяны), кроликов и грызунов (например, мышей и крыс). Термин «субъект» может применяться в настоящем документе взаимозаменяемо с термином «пациент». Субъект может представлять собой человека.
В контексте данного документа термин «лечение» (и его грамматические вариации, такие как «лечить» или «воздействие») относится к клиническому вмешательству в попытке изменить естественное течение болезни у индивидуума, получающего лечение, и может проводиться либо для профилактики, либо в ходе клинического патологического процесса. Желаемые эффекты лечения включают, помимо прочего, предотвращение возникновения или рецидива заболевания, облегчение симптомов (улучшение качества жизни), уменьшение любых прямых или косвенных патологических последствий заболевания, предотвращение метастазирования, снижение скорости прогрессирования заболевания, улучшение или временное облегчение состояния болезни, и ремиссию или улучшенный прогноз. Антитело или его антигенсвязывающий фрагмент согласно настоящему изобретению можно применять для задержки развития заболевания или для замедления прогрессирования заболевания.
Под «снижением» или «ингибированием» подразумевается способность вызывать общее снижение на 20%, 30%, 40%, 50%, 60%, 70%, 75%, 80%, 85%, 90%, 95% или больше. Термин «снижение» или «ингибирование» может относиться к симптомам заболевания, которое лечится. Термин «снижение» или «ингибирование» также включает задержку начала заболевания, в частности воспалительного заболевания.
Способы введения
Выделенное антитело или его антигенсвязывающий фрагмент, такой как scFv, или фармацевтическая композиция по настоящему изобретению может вводиться стандартным способом при патологическом состоянии, которое желательно лечить и/или предотвращать, например, путем перорального, местного, парентерального, внутривенного, подкожного, буккального, назального или ректального введения или путем ингаляции. Например, антитело или его антигенсвязывающий фрагмент, такой как scFv, или фармацевтическая композиция для применения, как описано в данном документе, могут быть составлены для парентерального введения, такого как внутривенное или подкожное введение, в частности подкожное введение.
Обычно выделенное антитело или его антигенсвязывающий фрагмент, такой как scFv, или фармацевтическую композицию, как определено в данном документе, вводят системно. Способ введения может быть, например, парентеральным, например, внутривенным или подкожным, в частности подкожным.
Хотя схема введения может быть адаптирована к конкретному заболеванию и субъекту, подлежащему лечению, обычно выделенное антитело или его антигенсвязывающий фрагмент, такой как scFv, или фармацевтическую композицию, как определено в данном документе, вводят 1-3 раза в неделю, например, 1-2 раза в неделю, например, 1 раз в неделю, хотя также возможны и другие схемы введения.
Антитела, их антигенсвязывающие фрагменты и/или фармацевтические композиции по настоящему изобретению также можно вводить в комбинированной терапии, то есть в комбинации с другими агентами. Например, комбинированная терапия может включать антитело или его антигенсвязывающий фрагмент, такой как scFv, согласно настоящему изобретению, в комбинации с по меньшей мере одним другим противовоспалительным или иммунодепрессантным агентом. Следует понимать, что комбинированная терапия включает как последовательное, так и одновременное введение. В контексте данного документе термин «одновременное» применяется для обозначения введения двух или большего количества терапевтических агентов, когда по меньшей мере часть введения накладывается во времени. Соответственно, одновременное введение включает схему введения, когда введение одного или большего количества агентов продолжается после прекращения введения одного или большего количества других агентов.
Схемы введения можно корректировать, чтобы обеспечить оптимальный необходимый ответ, например, терапевтический ответ. Например, может быть введена одноразовая доза, несколько разделенных доз могут вводиться с течением времени, или доза может быть пропорционально уменьшена или увеличена, как показано в зависимости от терапевтической ситуации. Особенно выгодно составлять парентеральные композиции в виде стандартной лекарственной формы для простоты введения и однородности дозирования
Терапевтически эффективное количество антитела или его антигенсвязывающего фрагмента, такого как scFv, может варьироваться в зависимости от таких факторов, как патологическое состояние, возраст, пол и вес индивидуума, а также способность антитела или его антигенсвязывающего фрагмента, такого как scFv, вызывать желаемый ответ у субъекта. Терапевтически эффективное количество также является таким, в котором терапевтически благоприятные эффекты преобладают над любыми токсичными или вредными эффектами вводимого вещества. Когда антитело или его антигенсвязывающий фрагмент, такой как scFv, применяется для предотвращения патологического состояния (профилактические цели), обычно, но не обязательно, профилактически эффективное количество меньше терапевтически эффективного количества, поскольку профилактическая доза применяется у субъектов до заболевания или на более ранней его стадии.
Фармацевтически эффективное количество, т.е. доза, антитела или его антигенсвязывающего фрагмента, такого как scFv, согласно настоящему изобретению обычно находится в диапазоне от около 0,0001 до 100 мг/кг, и чаще от 0,01 до 5 мг/кг веса тела хозяина. Однако точную дозу необходимо корректировать в зависимости, например, от патологического состояния, которое необходимо лечить или предотвращать, возраста и/или пола субъекта и от того, предназначена ли эта доза для лечения или предотвращения патологического состояния.
Экспрессионная система
Настоящее изобретение также относится к полинуклеотиду, такому как выделенный полинуклеотид, при этом указанный полинуклеотид кодирует антитело или его антигенсвязывающий фрагмент согласно настоящему изобретению.
Примеры полинуклеотидов согласно вариантам осуществления указаны в SEQ ID NO: 174-202, где продемонстрированы последовательности ДНК, кодирующие HC и LC пяти различных фрагментов антител: S-SL048-11 HC (SEQ ID NO: 174; 185; 196) и LC (SEQ ID NO: 175; 186; 197), S-SL048-46 HC (SEQ ID NO: 176; 187) и LC (SEQ ID NO: 177; 188), S-SL048-106 HC (SEQ ID NO: 178; 189; 198) и LC (SEQ ID NO: 179; 190; 199), S-SL048-116 HC (SEQ ID NO: 180; 191; 200) и LC (SEQ ID NO: 181; 192; 201) и S-SL048-118 HC (SEQ ID NO: 180; 191; 200) и LC (SEQ ID NO: 182; 193; 202) и AS20 HC SEQ ID NO: 183) и LC (SEQ ID NO: 184), а также CDR трансплантата HC (SEQ ID NO: 194) и LC (SEQ ID NO: 195).
Следовательно, в одном из вариантов осуществления указанный полинуклеотид выбирают из группы, состоящей из SEQ ID NO: 174-202 и любой их комбинации и/или варианта. Применяемый в настоящем изобретении вариант любой из SEQ ID NO: 174-202 включает полинуклеотид, кодирующий такое же антитело или его антигенсвязывающий фрагмент, что и полинуклеотид, определенный в любой из SEQ ID NO: 174-202, но может иметь по меньшей мере одну синонимичную замену, т.е. замену по меньшей мере одного основания на другое, так что полученная аминокислотная последовательность не модифицируется. Следовательно, такая синонимичная замена заменяет по меньшей мере одно основание в кодоне в указанном полинуклеотиде на другой кодон, которые оба кодируют аминокислотный остаток. Например, полинуклеотид согласно любой из SEQ ID NO: 174-202 или их комбинация может быть кодон- оптимизированной для экспрессии в конкретной клетке-хозяине.
Полинуклеотид, кодирующий антитело или его антигенсвязывающий фрагмент, как описано в данном документе, может быть введен в экспрессионный вектор. Экспрессионный вектор делает возможным воспроизведение введенного в него полинуклеотида. Указанный вектор может быть самореплицирующейся структурой нуклеиновой кислоты, а также вектором, включенным в геном клетки-хозяина, в которую он был введен. Таким образом, настоящее изобретение также направлено на такой экспрессионный вектор, содержащий полинуклеотид, кодирующий антитело или его антигенсвязывающий фрагмент.
Экспрессионный вектор предпочтительно содержит полинуклеотид, кодирующий антитело или его антигенсвязывающий фрагмент, функционально связанный по меньшей мере с одним регуляторным элементом. В некоторых вариантах осуществления регуляторный элемент представляет собой промотор или содержит его. Промотор представляет собой последовательность ДНК, с которой связываются белки, инициирующие транскрипцию молекулы РНК от ДНК (гена), расположенной в нисходящем направлении. Другой пример регуляторного элемента представляет собой энхансер. Энхансер представляет собой короткую область ДНК, которая может быть связана активаторами для увеличения вероятности того, что произойдет транскрипция определенного гена.
Примеры экспрессионных векторов включают молекулу ДНК, молекулу РНК, плазмиду, эписомальную плазмиду и вирусный вектор. Неограничивающие, но иллюстративные примеры вирусных векторов включают лентивирусный вектор, аденовирусный вектор, аденоассоциированный вирусный вектор, ретровирусный вектор, вирус леса Семлики, вирус полиомиелита и гибридный вектор.
Экспрессионный вектор может быть введен в клетку-хозяина для экспрессии и/или воспроизведения вектора, содержащего полинуклеотид. В частности, экспрессионный вектор предназначен для применения при лечении и/или предотвращении воспалительного заболевания, будучи экспрессированным в субъекте, чтобы таким образом продуцировать антитела или их антигенсвязывающие фрагменты у указанного субъекта.
Таким образом, в настоящем изобретении также предлагается клетка-хозяин, содержащая экспрессионный вектор. Применяемая клетка-хозяин может быть клеткой-хозяином любого типа, включая как эукариотические, так и прокариотические клетки-хозяева. Клетки-хозяева включают «трансформанты» и «трансформированные клетки», которые включают первичную трансформированную клетку и потомство, полученное от нее, независимо от количества пассажей.
Настоящее изобретение также относится к клетке, содержащей антитело или его антигенсвязывающий фрагмент согласно изобретению, полинуклеотид согласно изобретению и/или экспрессионный вектор согласно изобретению.
Клетка может быть выделенной клеткой, включая клетку клеточной линии. Клетка может быть выбрана из бактериальной клетки, эукариотической клетки, такой как дрожжевая клетка, клетка млекопитающего, человеческая клетка или нечеловеческая клетка.
Антитела или их антигенсвязывающие фрагменты можно получить путем введения их последовательности в экспрессионный вектор и предоставления возможности экспрессионному вектору экспрессировать антитело или его антигенсвязывающий фрагмент в клетке-хозяине, после чего продуцируемые антитела или антигенсвязывающие фрагменты выделяют/очищают перед применением, например, в лечебных или диагностических целях, как описано в другом месте в данном документе. Кроме того, сам вектор можно ввести субъекту для прямой экспрессии антитела или его антигенсвязывающего фрагмента у субъекта, подлежащего лечению. Кроме того, экспрессионный вектор предпочтительно содержит промотор, контролирующий экспрессию полинуклеотида, кодирующего антитело, или его антигенсвязывающий фрагмент.
Следовательно, настоящее изобретение также относится к способу продуцирования антитела или его антигенсвязывающего фрагмента. Указанный способ включает культивирование клетки согласно изобретению, содержащей экспрессионный вектор согласно изобретению, в условиях, когда антитело или его антигенсвязывающий фрагмент экспрессируется клеткой. В одном варианте осуществления указанный способ необязательно включает выделение антитела или его антигенсвязывающего фрагмента из клетки или из культуральной среды, в которой культивируется клетка.
Диагностическое применение выделенных антител или их антигенсвязывающих фрагментов.
Антитела или их антигенсвязывающие фрагменты по настоящему изобретению также можно применять для обнаружения BSSL, такого как hBSSL, в образце с помощью стандартных методов, таких как, помимо прочего, ELISA, вестерн-блоттинг, RIA, поверхностный плазмонный резонанс (SPR) и анализ проточной цитометрии.
Преимущество применения антител и их антигенсвязывающих фрагментов по настоящему изобретению состоит в том, что они не связываются с активным сайтом BSSL и, таким образом, не ингибируют липазную активность белка. Таким образом, можно применять антитела или их антигенсвязывающие фрагменты для изучения белка BSSL без значительного влияния на активность липазы, как продемонстрировано в экспериментальном разделе. Таким образом, антитела или их антигенсвязывающие фрагменты являются полезными в качестве молекулярных инструментов при изучении белка BSSL и/или его ферментативной активности in vitro/ex vivo и/или in vivo.
Таким образом, настоящее изобретение описывает способ обнаружения присутствия или отсутствия BSSL и/или количественного определения BSSL, например, hBSSL, в образце. Указанный способ включает приведение в контакт образца с выделенным антителом или его антигенсвязывающим фрагментом согласно настоящему изобретению. Указанный способ также включает обнаружение присутствия или отсутствия BSSL и/или количественное определение BSSL в образце на основе количества выделенного антитела или его антигенсвязывающего фрагмента, связанного с BSSL. Обнаружение или количественную оценку можно, например, выполнять с помощью ELISA, вестерн-блоттинга, RIA, поверхностного плазмонного резонанса (SPR), метода близкого лигирования (PLA) или анализа проточной цитометрии. Одно или множество, то есть по меньшей мере два антитела или их антигенсвязывающих фрагмента по настоящему изобретению можно применять в таком обнаружении.
Вышеописанный способ может быть в форме способа ex vivo или in vitro. В таком случае указанный способ включает приведение в контакт ex vivo или in vitro образца с выделенным антителом или его антигенсвязывающим фрагментом в соответствии с настоящим изобретением.
В одном варианте осуществления указанный способ также включает предоставление образца, потенциально содержащего BSSL.
Настоящее изобретение также описывает способ диагностики нарушения, связанного с BSSL. Указанный способ включает a) приведение образца в контакт с выделенным антителом или его антигенсвязывающим фрагментом согласно настоящему изобретению и b) определение присутствия или отсутствия BSSL и/или количественное определение BSSL в образце на основе количества выделенного антитела или его антигенсвязывающего фрагмента, связанного с BSSL. Обнаружение или количественную оценку можно, например, выполнять с помощью ELISA, вестерн-блоттинга, RIA, SPR, PLA или анализа проточной цитометрии. Указанный способ также включает c) заключение на основании результатов этапа b) о том, диагностировано ли у субъекта нарушение, связанное с BSSL.
В одном из вариантов осуществления указанный способ также включает получение образца от субъекта, у которого подозревается заболевание, связанное с BSSL.
В конкретном варианте осуществления указанный способ включает сравнение количественно определенного уровня BSSL в образце с пороговым значением. В таком конкретном варианте осуществления этап c) включает заключение о том, диагностировано ли у субъекта нарушение, связанное с BSSL, или нет, на основе сравнения между количественно определенным уровнем BSSL в образце и пороговым значением. Например, если количество BSSL в BSSL превышает пороговое значение, можно сделать вывод о том, диагностировано ли у субъекта нарушение, связанное с BSSL, или нет.
Показатель порогового значения зависит от конкретного нарушения, связанного с BSSL, и может быть определен путем количественной оценки уровня BSSL в образцах, взятых у субъектов, у которых уже диагностировано конкретное нарушение, связанное с BSSL, и/или путем определения количества BSSL в образцах, взятых у здоровых субъектов, не страдающих конкретным нарушением, связанным с BSSL. Затем пороговое значение может быть определено на основе этих количественных показателей BSSL от субъектов, страдающих конкретным нарушением, связанным с BSSL, и предпочтительно на основе количественных показателей BSSL от здоровых субъектов.
Нарушение, связанное с BSSL, обычно представляет собой воспалительное состояние, как описано в другом месте в настоящем документе. Воспалительное состояние может быть, например, хроническим или системным воспалительным состоянием, таким как воспалительное заболевание, ауто-воспалительное заболевание и/или аутоиммунное заболевание. Воспалительное состояние может быть, например, ревматоидным артритом, ювенильным артритом, псориатическим артритом, атерогенезом, болезнью Крона или язвенным колитом.
Образец, потенциально содержащий BSSL, может быть любым типом образца, например, образцом, полученным от субъекта. Следовательно, в одном варианте осуществления указанный образец представляет собой биологический образец. Примером такого биологического образца является образец жидкости организма, например, образец крови, образец плазмы крови или образец сыворотки. Другим примером биологического образца является образец ткани тела, такой как биоптат. Образец может быть естественным образцом или образцом in vitro, потенциально содержащим BSSL. Способы обнаружения BSSL и/или диагностики патологических состояний, связанных с BSSL, включают как способы in vitro, так и способы in vivo, такие как гибридизация in situ .
Как упоминалось в другом месте в данном документе, антитела или их антигенсвязывающие фрагменты могут быть гуманизированы или их последовательности CDR (или их части) могут быть привиты к остову, не являющемуся человеческим. Последнее может быть полезным при применении антител или их антигенсвязывающих фрагментов в качестве молекулярного инструмента для изучения белка BSSL у других видов, кроме людей, чтобы, например, уменьшить негативные иммуногенные реакции на антитела и/или их антигенсвязывающие фрагменты.
Иллюстративные варианты осуществления
Вариант осуществления относится к выделенному антителу или его антигенсвязывающему фрагменту, которое специфически связывается с липазой, стимулируемой солями жёлчных кислот (BSSL), такой как BSSL человека (hBSSL). Антитело или его антигенсвязывающий фрагмент связывается по меньшей мере с одним из следующих эпитопов: первым эпитопом или вторым эпитопом, идентифицированным на BSSL. Первый эпитоп содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 1 или аминокислотной последовательности, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96%, 97%, 98%. или 99% идентичности с SEQ ID NO: 1, а второй эпитоп содержит аминокислотную последовательность согласно SEQ ID NO: 2 или аминокислотную последовательность, имеющую по меньшей мере 80%, например, 85%, 90%, 95%, 96 %, 97%, 98% или 99% идентичности с SEQ ID NO: 2.
В одном варианте осуществления первый эпитоп содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 3 или аминокислотной последовательности, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99% идентичности с SEQ ID NO: 3.
В одном варианте осуществления антитело или его антигенсвязывающий фрагмент специфически связывается как с первым эпитопом, так и со вторым эпитопом.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент дополнительно специфически связывается с аминокислотной последовательностью согласно SEQ ID NO: 4 или аминокислотной последовательностью, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99% идентичность с ней.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент дополнительно специфически связывается с аминокислотной последовательностью согласно SEQ ID NO: 5 или аминокислотной последовательностью, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99% идентичность с ней.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент дополнительно специфически связывается с аминокислотной последовательностью согласно SEQ ID NO: 6 или аминокислотной последовательностью, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99% идентичность с ней.
Вариант осуществления относится к выделенному антителу или его антигенсвязывающему фрагменту. Выделенное антитело или его антигенсвязывающий фрагмент содержит три области, определяющие комплементарность (CDR), вариабельной области тяжелой цепи (HCVR) (HCDR). Первая HCDR содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 7, вторая HCDR содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 8 или аминокислотной последовательности, которая по меньшей мере на 75% идентична SEQ ID NO: 8, и третья HCDR содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 9 или аминокислотной последовательности которая по меньшей мере на 90% идентична SEQ ID NO: 9. Выделенное антитело или его антигенсвязывающий фрагмент содержит три CDR вариабельной области легкой цепи (LCVR) (LCDR). Первая LCDR содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 10 или аминокислотной последовательности, которая по меньшей мере на 80% идентична SEQ ID NO: 10, вторая LCDR содержит или состоит из аминокислотной последовательности ATS или аминокислотной последовательности, которая по меньшей мере на 66% идентична аминокислотной последовательности ATS, такой как AAS, и третья LCDR содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности которая по меньшей мере на 87% идентична SEQ ID NO: 11.
В одном из вариантов осуществления первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 8, 18 и 19, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 10 и 20, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из ATS и AAS, а третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 11, 21 и 22.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 18, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 21.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 10, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 19, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 20, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности ATS, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 22.
В одном из вариантов осуществления антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 8, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 20, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности AAS, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 23 или аминокислотной последовательности, которая по меньшей мере на 77% идентична SEQ ID NO: 23, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9 или аминокислотной последовательности, которая по меньшей мере на 83% идентична SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 16 или аминокислотной последовательности, которая по меньшей мере на 80% идентична SEQ ID NO: 16, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 15 или аминокислотной последовательности, которая по меньшей мере на 66% идентична SEQ ID NO: 15, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 21 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 21.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три определяющие комплементарность области тяжелой цепи (HCDR) вариабельной области тяжелой цепи (HCVR). Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 24 или аминокислотной последовательности, которая по меньшей мере на 77% идентична SEQ ID NO: 24, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9 или аминокислотной последовательности, которая по меньшей мере на 83% идентична SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три CDR вариабельной области легкой цепи (LCVR). Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 27 или аминокислотной последовательности, которая по меньшей мере на 70% идентична SEQ ID NO: 27, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 29 или аминокислотной последовательности, которая по меньшей мере на 50% идентична SEQ ID NO: 29, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 25 или аминокислотной последовательности, которая по меньшей мере на 83% идентична SEQ ID NO: 25, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9 или аминокислотной последовательности, которая по меньшей мере на 83% идентична SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 26 или аминокислотной последовательности, которая по меньшей мере на 90% идентична SEQ ID NO: 26, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 28 или аминокислотной последовательности, которая по меньшей мере на 66% идентична SEQ ID NO: 28, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 22 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 22.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 12 или аминокислотной последовательности, которая по меньшей мере на 77% идентична SEQ ID NO: 12, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9 или аминокислотной последовательности, которая по меньшей мере на 83% идентична SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 14 или аминокислотной последовательности, которая по меньшей мере на 70% идентична SEQ ID NO: 14, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 15 или аминокислотной последовательности, которая по меньшей мере на 66% идентична SEQ ID NO: 15, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 12 или аминокислотной последовательности, которая по меньшей мере на 77% идентична SEQ ID NO: 12, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9 или аминокислотной последовательности, которая по меньшей мере на 83% идентична SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 16 или аминокислотной последовательности, которая по меньшей мере на 80% идентична SEQ ID NO: 16, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 17 или аминокислотной последовательности, которая по меньшей мере на 50% идентична SEQ ID NO: 17, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11 или аминокислотной последовательности, которая по меньшей мере на 87% идентична SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из одной аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 12, 23, 24 и 25, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из одной аминокислотной последовательности, выбранной из SEQ ID NO: 14, 16, 26 и 27, и вторая LCDR содержит, предпочтительно состоит из одной аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 15, 17, 28 и 29, и третья LCDR содержит, предпочтительно состоит из одной аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 11, 21 и 22.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 23, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 16, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 15, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 21.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 24, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 27, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 29, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 25, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 26, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 28, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 22.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 12, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 14, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности, содержащей SEQ ID NO: 15, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три HCDR HCVR. Первая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 7, вторая HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 12, и третья HCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 9. В этом варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит три LCDR LCVR. Первая LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 16, вторая LCDR содержит, предпочтительно состоит из аминокислотной последовательности, содержащей SEQ ID NO: 17, и третья LCDR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
В одном из вариантов осуществления HCVR содержит, предпочтительно состоит из одной аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 30, 32, 34 и 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 98%; например, выбранной из группы, состоящей из SEQ ID NO: 30, 34 и 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%; например, выбранной из группы, состоящей из SEQ ID NO: 34 и 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%.
В одном варианте осуществления HCVR содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 36 или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%.
В одном из вариантов осуществления LCVR содержит, предпочтительно состоит из одной аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 31, 33, 35, 37 и 38, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%; например, выбранной из группы, состоящей из SEQ ID NO: 31, 35, 37 и 38, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%; например, выбранной из группы, состоящей из SEQ ID NO: 35, 37 и 38, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%; например, выбранной из группы, состоящей из SEQ ID NO: 37 и 38, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%.
В одном варианте осуществления LCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 37 или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%.
В одном из вариантов осуществления HCVR содержит, предпочтительно состоит из аминокислотной последовательности, независимо выбранной из группы, состоящей из SEQ ID NO: 30, 32, 34 и 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%, и LCVR содержит аминокислотную последовательность, независимо выбранную из группы, состоящей из SEQ ID NO: 31, 33, 35, 37 и 38, или аминокислотную последовательность, которая идентична ей по меньшей мере на 96%. В конкретном варианте осуществления HCVR содержит, предпочтительно состоит из аминокислотной последовательности, независимо выбранной из группы, состоящей из SEQ ID NO: 30, 34 и 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%, и LCVR содержит, предпочтительно состоит из аминокислотной последовательности, независимо выбранной из группы, состоящей из SEQ ID NO: 31, 35, 37 и 38, или аминокислотную последовательность, которая идентична ей по меньшей мере на 96%. В другом варианте осуществления HCVR содержит, предпочтительно состоит из аминокислотной последовательности, независимо выбранной из группы, состоящей из SEQ ID NO: 34 и 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%, и LCVR содержит, предпочтительно состоит из аминокислотной последовательности, независимо выбранной из группы, состоящей из SEQ ID NO: 35, 37 и 38, или аминокислотную последовательность, которая идентична ей по меньшей мере на 96%. В дополнительном варианте осуществления HCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 36, или аминокислотной последовательности, которая идентична ей по меньшей мере на 96%, и LCVR содержит, предпочтительно состоит из аминокислотной последовательности, независимо выбранной из группы, состоящей из SEQ ID NO: 37 и 38, или аминокислотную последовательность, которая идентична ей по меньшей мере на 96%.
В одном из вариантов осуществления выделенное антитело или антигенсвязывающий фрагмент содержит, предпочтительно состоит из HCVR, содержащей аминокислотную последовательность согласно SEQ ID NO: 36, или аминокислотную последовательность, которая идентична ей по меньшей мере на 96%, и LCVR, содержащей аминокислотную последовательность согласно SEQ ID NO: 37 или 38, или аминокислотную последовательность, которая идентична ей по меньшей мере на 96%.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит HCVR и LCVR. HCVR и LCVR представляют собой пару аминокислотных последовательностей, которая по меньшей мере на 96% идентична паре аминокислотных последовательностей, выбранной из группы, состоящей из пары аминокислотных последовательностей SEQ ID NO: 30 и 31; пары аминокислотных последовательностей SEQ ID NO: 32 и 33; пары аминокислотных последовательностей SEQ ID NO: 34 и 35; пары аминокислотных последовательностей SEQ ID NO: 36 и 37; и пары аминокислотных последовательностей SEQ ID NO: 36 и 38; например, пары аминокислотных последовательностей, выбранной из группы, состоящей из пары аминокислотных последовательностей SEQ ID NO: 36 и 37; и пары аминокислотных последовательностей SEQ ID NO: 36 и 38.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи. Вариабельная область тяжелой цепи содержит аминокислотную последовательность ZH1-[CDR-H1]-ZH2-[eCDR-H2]- ZH3-[CDR-H3]-ZH4, где каждый из ZH1, ZH2, ZH3 и ZH4 представляет собой ноль, один или несколько независимо выбранных аминокислотных остатков. В одном из вариантов осуществления вариабельная область тяжелой цепи состоит из аминокислотной последовательности, выбранной из i) ZH1-[GYTFTSYN]-ZH2-[X53GVIX57PGDGX64TSYX68QKFX72]-ZH3- [ARDYYGSSPLGY]-ZH4, где независимо друг от друга X53 выбран из I и M; X57 выбран из N и Y; X64 выбран из A и S; X68 выбран из A и N; и X72 выбран из K и Q, и ii) аминокислотной последовательности, которая имеет по меньшей мере 92% идентичности с последовательностью,
определенной в i). Вариабельная область легкой цепи содержит аминокислотную последовательность ZL1-[eCDR-L1]-ZL2-[eCDR-L2]-ZL3-[CDR-L3]-ZL4, где каждый из ZL1, ZL2, ZL3 и ZL4 представляет собой ноль, один или несколько независимо выбранных аминокислотных остатков. В одном из вариантов осуществления вариабельная область легкой цепи состоит из аминокислотной последовательности, выбранной из iii) ZL1-[X24ASX27SISYX39N] -ZL2- [AX57SX66LX68] -ZL2- [HQRSSX115PT]-ZL4, где независимо друг от друга X24 выбран из S и R; X27 выбран из S и P; X39 выбран из M и L; X57 выбран из A и T; X66 выбран из K и S; X68 выбран из A и P; и X115 выбран из S, T и Y, и iv) аминокислотной последовательности, которая имеет по меньшей мере 87% идентичности с последовательностью, определенной в iii).
В одном варианте осуществления ZH1 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 39 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 39.
В одном варианте осуществления ZH2 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 40 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 40.
В одном варианте осуществления ZH3 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 41 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 41.
В одном варианте осуществления ZH4 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 42 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 42.
В одном варианте осуществления ZL1 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 43 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 43.
В одном варианте осуществления ZL2 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 44 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 44.
В одном варианте осуществления ZL3 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 45 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 45.
В одном варианте осуществления ZL4 содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 46 или аминокислотной последовательности, которая идентична по меньшей мере на 90% с SEQ ID NO: 46.
В одном варианте осуществления антитело представляет собой полноразмерное антитело.
В одном варианте осуществления антитело выбирают из группы, состоящей из человеческих антител, гуманизированных антител и химерных антител.
В одном варианте осуществления антигенсвязывающий фрагмент представляет собой антигенсвязывающий фрагмент, такой как вариабельный одноцепочечный фрагмент, фрагмент Fab, фрагмент F(ab')2, фрагмент F(ab’)3, фрагмент Fab' , фрагмент Fd, фрагмент Fv, фрагмент dAb, выделенная определяющая комплементарность область (CDR) и нанотело. В конкретном варианте осуществления антигенсвязывающий фрагмент представляет собой фрагмент scFv.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент представляет собой моноклональное антитело или его антигенсвязывающий фрагмент. В конкретном варианте осуществления моноклональное антитело или его антигенсвязывающий фрагмент представляет собой гуманизированное моноклональное антитело или его антигенсвязывающее фрагмент.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент выбирают из группы, состоящей из класса изотипов IgG, IgA, IgM, IgD и IgE; например, IgG. В конкретном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент выбирают из группы, состоящей из подкласса изотипов IgG1 и IgG4.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит одну или большее количество Fc сайлесинг-мутаций. В конкретном варианте осуществления IgG1 содержит Fc сайлесинг-мутации L234A, L235A и P329G.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент содержит одну или большее количество стабилизирующих мутаций, которые предотвращают или снижают замену плеча Fab in vivo. В конкретном варианте осуществления IgG4 содержит стабилизирующую мутацию S228P.
В одном из вариантов осуществления выделенное антитело или его антигенсвязывающий фрагмент представляет собой вариабельный одноцепочечный фрагмент (scFv), который специфически связывается с hBSSL и который содержит домен HCVR, содержащий первый HCDR, второй HCDR и третий HCDR, содержащий или состоящий из аминокислотных последовательностей, которые по меньшей мере на 80% идентичны SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 9, соответственно, и домен LCVR, содержащий первый LCDR, второй LCDR и третий LCDR, содержащий или состоящий из аминокислотных последовательностей, которые по меньшей мере на 80% идентичны SEQ ID NO: 10, аминокислотной последовательности ATS и SEQ ID NO: 11 соответственно.
В одном варианте осуществления первая HCDR, вторая HCDR и третья HCDR состоят из аминокислотных последовательностей согласно SEQ ID NO: 7, SEQ ID NO: 8 и SEQ ID NO: 9 соответственно, а первая LCDR, вторая LCDR и третья LCDR состоят из аминокислотных последовательностей согласно SEQ ID NO: 10, аминокислотной последовательности ATS и SEQ ID NO: 11 соответственно.
В одном варианте осуществления антитело представляет собой гуманизированное антитело.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент имеет аффинность к hBSSL не более чем KD 1,7 нМ.
В одном варианте осуществления выделенное антитело или его антигенсвязывающий фрагмент способны замещать связывание hBSSL с моноцитами, предпочтительно CD14+ моноцитами.
Один вариант осуществления относится к фармацевтической композиции, содержащей выделенное антитело и/или его антигенсвязывающий фрагмент согласно настоящему изобретению, и фармацевтически приемлемый носитель или вспомогательное вещество.
Один вариант осуществления относится к выделенному антителу и/или его антигенсвязывающему фрагменту или к фармацевтической композиции согласно настоящему изобретению для применения в качестве лекарственного средства.
Один вариант осуществления относится к выделенному антителу и/или его антигенсвязывающему фрагменту или к фармацевтической композиции согласно настоящему изобретению для применения при лечении и/или предотвращении воспалительного заболевания.
Один вариант осуществления относится к применению выделенного антитела и/или его антигенсвязывающего фрагмента или к фармацевтической композиции согласно настоящему изобретению для изготовления фармацевтической композиции, предназначенной для лечения и/или предотвращения воспалительного заболевания.
Один вариант осуществления относится к способу лечения и/или облегчения, и/или предотвращения, и/или профилактики воспалительного заболевания. В этом способе терапевтически эффективное количество выделенного антитела и/или его антигенсвязывающего фрагмента или фармацевтической композиции согласно настоящего изобретению вводят нуждающемуся в этом субъекту.
В одном варианте осуществления воспалительное заболевание представляет собой хроническое воспалительное заболевание.
В одном варианте осуществления воспалительное заболевание представляет собой системное воспалительное заболевание.
В одном варианте осуществления воспалительное заболевание представляет собой аутоиммунное заболевание. В конкретном варианте осуществления аутоиммунное заболевание представляет собой ревматоидный артрит или ювенильный ревматоидный артрит. В другом конкретном варианте осуществления аутоиммунное заболевание представляет собой воспалительное заболевание кишечника (IBD), такое как болезнь Крона или язвенный колит.
В одном варианте осуществления воспалительное заболевание представляет собой аутовоспалительное заболевание. В конкретном варианте осуществления аутовоспалительное заболевание представляет собой псориатический артрит.
В одном варианте осуществления воспалительное заболевание представляет собой стеатоз печени.
В одном варианте осуществления выделенное антитело и/или его антигенсвязывающий фрагмент или фармацевтическую композицию вводят системно.
В одном варианте осуществления выделенное антитело и/или его антигенсвязывающий фрагмент или фармацевтическую композицию вводят парентерально, например, подкожно. Следовательно, в конкретном варианте осуществления выделенное антитело и/или его антигенсвязывающий фрагмент или фармацевтическая композиция составлены для парентерального введения, такого как подкожное введение.
В одном варианте осуществления выделенное антитело и/или его антигенсвязывающий фрагмент или фармацевтическую композицию вводят 1-3 раза в неделю, например, 1-2 раза в неделю, например, 1 раз в неделю.
В одном варианте осуществления лечение и/или предотвращение осуществляется с помощью пассивной иммунотерапии.
Варианты осуществления относятся к полинуклеотиду, кодирующему выделенное антитело или его антигенсвязывающий фрагмент, как определено согласно изобретению, экспрессионному вектору, содержащему полинуклеотид согласно изобретению, и клетке-хозяину, содержащей экспрессионный вектор согласно изобретению.
Вариант осуществления относится к способу получения выделенного антитела или его антигенсвязывающего фрагмента согласно изобретению. Указанный способ включает культивирование клетки-хозяина согласно изобретению в условиях, допускающих экспрессию антитела или его антигенсвязывающего фрагмента, и выделение антитела или его антигенсвязывающего фрагмента.
Вариант осуществления относится к способу обнаружения присутствия или отсутствия BSSL и/или количественного определения уровня BSSL в образце. Указанный способ включает следующие этапы: а) получение образца, потенциально содержащего BSSL, b) приведение в контакт образца с выделенным антителом или его антигенсвязывающим фрагментом согласно изобретению, и c) обнаружение присутствия или отсутствия BSSL и/или количественное определение уровня BSSL в указанном образце.
Вариант осуществления относится к способу диагностики нарушения, связанного с BSSL. Указанный способ включает следующие этапы: а) получение образца от субъекта, у которого подозревают заболевание, связанное с BSSL, b) приведение в контакт образца с выделенным антителом или его антигенсвязывающим фрагментом согласно изобретению, с) обнаружение присутствия или отсутствия BSSL и/или количественное определение уровня BSSL в образце, и d) заключение на основании результатов этапа c), диагностировано ли у субъекта нарушение, связанное с BSSL, или нет.
В одном варианте осуществления нарушение, связанное с BSSL, представляет собой воспалительное заболевание, такое как хроническое воспалительное заболевание; системное воспалительное заболевание; аутоиммунное заболевание, такое как ревматоидный артрит, ювенильный ревматоидный артрит, воспалительное заболевание кишечника, такое как болезнь Крона, и язвенный колит; аутовоспалительное заболевание, такое как псориатический артрит; или стеатоз печени.
Вариант осуществления относится к способу определения ферментативной активности BSSL. Указанный способ включает следующие этапы: а) получение образца, потенциально содержащего BSSL, b) приведение в контакт образца с выделенным антителом или его антигенсвязывающим фрагментом согласно изобретению, и c) определение ферментативной активности BSSL в образце.
Вариант осуществления относится к эпитопу BSSL, содержащему или состоящему из первого эпитопа и второго эпитопа. Первый эпитоп содержит или состоит из аминокислотной последовательности согласно SEQ ID NO: 1 или аминокислотной последовательности, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96%, 97%, 98%. или 99% идентичности с SEQ ID NO: 1. Второй эпитоп состоит из второй поверхности, содержащей или состоящей из аминокислотной последовательности согласно SEQ ID NO: 2, или аминокислотной последовательности, имеющей по меньшей мере 80%, например, 85%, 90%, 95%, 96 %, 97%, 98% или 99% идентичности с SEQ ID NO: 2.
В одном варианте осуществления первый эпитоп содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 3 или аминокислотной последовательности, идентичной ей по меньшей мере на 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99%.
В одном варианте осуществления эпитоп дополнительно содержит аминокислотную последовательность согласно SEQ ID NO: 4 или аминокислотную последовательность, идентичную ей по меньшей мере на 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99%.
В одном варианте осуществления эпитоп дополнительно содержит аминокислотную последовательность согласно SEQ ID NO: 5 или аминокислотную последовательность, идентичную ей по меньшей мере на 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99%.
В одном варианте осуществления эпитоп дополнительно содержит аминокислотную последовательность согласно SEQ ID NO: 6 или аминокислотную последовательность, идентичную ей по меньшей мере на 80%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99%.
Хотя настоящее изобретение было описано со ссылкой на различные типовые аспекты и варианты осуществления, специалистам в данной области техники будет понятно, что могут быть выполнены различные изменения и элементы могут быть заменены их эквивалентами без отступления от объема настоящего изобретения. Кроме того, может быть выполнено множество модификаций для адаптации конкретной ситуации или молекулы к идеям настоящего изобретения без отступления от его основного объема. Следовательно, предполагается, что настоящее изобретение не ограничивается каким-либо конкретным предполагаемым вариантом осуществления, но что это изобретение будет включать все варианты осуществления, попадающие в объем прилагаемой формулы изобретения.
ПРИМЕРЫ
Таблица 2 - Материал BSSL, примененный в примерах
Пример 1. Связывание IgG AS20 с BSSL человека и мыши (SPR)
В этом примере связывание антитела AS20 – IgG1 (AS20 mIgG) мыши с BSSL человека и мыши исследовали с помощью поверхностного плазмонного резонанса (SPR). Уровни AS20 mIgG (вариабельная область тяжелой цепи (HCVR) SEQ ID NO: 80 и вариабельная область легкой цепи (LCVR) SEQ ID NO: 114) были повышены у мышей по сравнению с полноразмерным белком BSSL (SEQ ID NO: 138), очищенным из грудного молока. Реактивность к BSSL мыши была неизвестна.
Материал и способы
hBSSL и mBSSL (Таблица 2) применяли вместе с IgG1 AS20 (AS20 mIgG1) мыши (собственного производства, HCVR SEQ ID NO: 80 и LCVR SEQ ID NO: 114) в экспериментах, описанных в этом Примере.
Измерения SPR проводили с помощью прибора BIACORE® T200 (GE Healthcare). Чтобы свести к минимуму эффекты авидности, антитело иммобилизовали на поверхности сенсора и в качестве аналита вводили BSSL. Иммобилизацию AS20 mIgG1 проводили путем аминного связывания с сенсорным чипом BIACORE® CM5 (поверхность карбоксилированного декстрана). Чипы активировали путем введения 1:1 смеси 0,2 M N-этил-N’-[(диметиламино)пеопил]карбодиимида (EDC) и 0,05 M N-гидроксисукциминимда (NHS) при времени контакта 7 минут. Антитело вводили в течение 0,2-2,8 минут, разбавленным до 14-50 мкг/мл в 10 мМ ацетат-HCl, pH 5,0 (набор один) или pH 6,0 (набор два), для достижения конечных уровней иммобилизации от 560 до 830 RU. Оставшиеся активированные карбоксильные группы на поверхности сенсора деактивировали введением 1 М этаноламина в течение 7 минут.
В качестве рабочего буфера в первом наборе экспериментов применяли буфер PBS pH 7,4 (10 мМ фосфат, 2,5 мМ KCl, 137 мМ NaCl) с добавлением 0,05% (об./об.) Tween 20. Во втором наборе экспериментов рабочий буфер представлял собой 25 мМ TrisHCl, pH 7,5, 150 мМ NaCl. 146 мМ H3PO4 применяли в качестве стандартного раствора для регенерации. Кинетические исследования и анализ нелинейной регрессии выполняли в соответствии с методом кинетики одного цикла (SCK) для прибора BIACORE® T200 и программного обеспечения для оценки. Взаимодействие между mBSSL и AS20 mIgG1 анализировали с помощью модели аффинности в стационарном состоянии.
В первом наборе экспериментов провели три эксперимента SCK с наивысшими концентрациями hBSSL в серии концентраций, составляющих 300, 100 и 50 нМ соответственно. Серии концентраций были сделаны с разведениями 1:3, 1: 3,16 (полулогарифмический) и 1:2.
Во втором наборе экспериментов hBSSL разводили до 20 нМ начальной концентрации в рабочем буфере с последующим серийным разведением 1:1 в том же буфере, в результате чего получали 5-точечные концентрации в диапазоне от 20 нМ до 1,25 нМ. mBSSL разбавляли до исходной концентрации 2000 нМ в рабочем буфере с последующим серийным разведением 1:1 в том же буфере, в результате чего получали 5-точечные концентрации в диапазоне от 2000 нМ до 125 нМ.
Результаты
Было обнаружено, что AS20 mIgG1 связывается с BSSL как человека, так и мыши. Аффинность к BSSL человека была сильной с низкой наномолярной аффинностью. Взаимодействие хорошо охарактеризовано моделью связывания 1:1 (Фиг. 1). Константы скорости ассоциации и диссоциации, а также равновесная константа диссоциации из нелинейного регрессионного анализа экспериментов SCK представлены в Таблице 3. В первом наборе экспериментов измерения были выполнены в трех повторениях, и, таким образом, представлены средние значения и стандартные отклонения.
Также было отмечено, что BSSL мыши взаимодействует с иммобилизованным AS20 mIgG1, однако почти в 100 раз более слабой аффинностью. Для определения аффинности проводили анализ стационарного состояния (Фиг. 2 и Таблица 3).
Таблица 3 - Кинетические параметры взаимодействия между AS20 mIgG1 и hBSSL, а также AS20 mIgG1 и mBSSL
*Кинетика одного цикла;
**Анализ стационарного состояния;
Н.О. Не определено;
1 Первый набор экспериментов;
2 Второй набор экспериментов.
Пример 2 - Характеризация продукции и связывания AS20 scFv с BSSL человека и мыши (ELISA, SPR и LUMINEX®)
В этом примере версию одноцепочечного вариабельного фрагмента (scFv) на основе AS20 mIgG1, обозначенная AS20 scFv (содержащая HCVR SEQ ID NO: 80 и LCVR SEQ ID NO: 114), генерировали с сохраненным связыванием по отношению к BSSL человека, что оценивалось с помощью иммуноферментного анализа (ELISA), SPR и LUMINEX®.
Материал и способы
Мелкомасштабное продуцирование и очистка
Путем слияния HCVR (SEQ ID NO: 80) с LCVR (SEQ ID NO: 114) через глицин-сериновый линкер был сформирован ген, кодирующий соответствующую конструкцию scFv. Ген scFv субклонировали в скрининговый вектор pHAT-6 (SciLifeLab, Стокгольм, Швеция), обеспечивая сигнал для секреции scFv вместе с меткой тройного FLAG и меткой гексагистидина (His) на С-конце. Впоследствии конструкцию трансформировали в TOP10 Escherichia coli. Бактериальный супернатант лизированных клеток очищали с помощью магнитных гранул, конъюгированных с антителом α-FLAG (Sigma Aldrich, #M8823). Очищенный scFv анализировали гель-электрофорезом в восстанавливающих условиях для определения его чистоты и целостности, а концентрацию белка определяли с помощью набора для анализа BCA (бицинхониновая кислота) (Pierce).
ELISA
Небиотинилированную BSSL человека (hBSSL) и биотинилированную BSSL человека (b-hBSSL) наносили либо непосредственно, либо через стрептавидин на 384-луночный планшет для ELISA в двух различных концентрациях, 1 μг/мл и 0,5 μг/мл в PBS при 4°C в течение ночи. Очищенный scFv AS20 серийно разбавляли в 3 раза блокирующим буфером (фосфатно-солевой буферный раствор (PBS) с добавлением 0,5% бычьего сывороточного альбумина (BSA) и 0,05% Tween 20) с концентрациями в диапазоне от 1 μг/мл до 4 нг/мл. Обнаружение связывания осуществляли с помощью антитела α-FLAG M2, конъюгированного с пероксидазой хрена (HRP) (Sigma-Aldrich), с последующей инкубацией с хромогенным субстратом Ultra 3,3’, 5,5’-тетраметилбензидин (TMB) ELISA. Развитие сигнала останавливали добавлением 1 М серной кислоты и измеряли оптическую плотность при 450 нм.
Во втором анализе ELISA, 1 μг/мл BSSL человека и мыши, а также белок отрицательного контроля наносили непосредственно на 384-луночный планшет ELISA и инкубировали при 4°C в течение ночи. Очищенный scFv AS20 и scFv отрицательного контроля добавляли в двух различных концентрациях: 1 μг/мл и 0,2 μг/мл. Обнаружение сигнала связывания выполняли, как описано выше. Все образцы в обоих наборах ELISA анализировали в двух повторениях. hBSSL, b-hBSSL и mBSSL (Таблица 2) применяли вместе с scFv AS20 (собственного производства, HCVR SEQ ID NO: 80 и LCVR SEQ ID NO: 114) в экспериментах, описанных в этом примере.
Измерения SPR
Оценку аффинности клонов scFv проводили с помощью SPR с помощью BIACORE® T200 (GE Healthcare). Антитело α-FLAG M2 иммобилизовали на чипе CM5 S посредством связывания первичным амином с использованием химического состава NHS-EDC, что позволило захватить AS20 scFv через его метку 3xFLAG. Серию 3-кратных разведений, состоящую из пяти различных концентраций, от 200 нМ до 2 нМ, hBSSL и b-hBSSL, последовательно вводили в проточные кюветы, обеспечивая связывание с захваченным scFv AS20. Регенерация поверхности осуществлялась в кислых условиях с применением 10 мМ глицин-HCl при pH 2,2. Полученные данные кинетики одного цикла подгоняли к модели связывания Ленгмюра 1:1, а кинетические параметры, ka (1/Mс), kd (1/с) и KD (M) получали с помощью программного обеспечения BIAevaluation.
Анализы LUMINEX®
Биотинилированную hBSSL инкубировали с гранулами LUMINEX®, связанными с нейтравидином, и смешивали с 30 различными гранулами ID, каждая из которых была конъюгирована с нерелевантным белком. Смешанный пул гранул инкубировали с AS20 scFv, присутствующим в бактериальном супернатанте, разведенном 1:10 в буфере для анализа (PBS с добавлением 3% BSA, 0,05% Tween 20 и 10 μг/мл нейтравидина). Также был включен один положительный контроль scFv, т.е. scFv, как ожидается, будет связываться с гранулами, покрытыми одним из нерелевантных белков. Связывание клонов scFv с конкретной конъюгированной с белком гранулой стало возможным посредством применения R-PE-конъюгированного антитела анти-FLAG M2 с последующим анализом на приборе FlexMAP 3D.
Результаты
Мелкомасштабное продуцирование и очистка
Анализ посредством гель-электрофореза бактериально экспрессированного и очищенного scFv AS20 продемонстрировал высокую чистоту с одной полосой основного белка, хорошо соответствующей ожидаемой молекулярной массе scFv (данные не приведены).
ELISA
AS20 scFv проявлял зависящее от концентрации связывание как с небиотинилированной, так и с биотинилированной BSSL человека (Фиг. 3a). Интенсивность сигнала при конкретной концентрации scFv была намного выше в отношении биотинилированной BSSL, чем в отношении небиотинилированной BSSL, что может быть связано с различиями в условиях покрытия.
AS20 scFv также демонстрировал связывание с BSSL мыши, но интенсивность сигнала была намного слабее, чем с BSSL человека (Фиг. 3b), что может указывать на более слабую аффинность AS20 scFv к BSSL мыши. Связывания scFv AS20 с отрицательным контролем не обнаружено.
Измерения SPR
Подход с кинетикой одного цикла применяли для определения аффинности AS20 scFv к небиотинилированной и биотинилированной BSSL человека. AS20 scFv проявлял очень сходную аффинность к небиотинилированной BSSL человека (KD = 0,6 нМ) и к биотинилированной BSSL человека (KD = 0,8 нМ) (Таблица 4).
Таблица 4 - Кинетические параметры взаимодействия между AS20 scFv и hBSSL, нативной и биотинилированной
Анализы Luminex
Для того чтобы определить, подвержен ли scFv AS20 неспецифическому взаимодействию с нерелевантными белками, проводили анализ Luminex, в котором scFv AS20 анализировали на 30 различных нерелевантных белках, а также на его родственной мишени. AS20 scFv продемонстрировал связывание только с BSSL человека без связывания или с очень низким связыванием со всеми другими белками, включенными в анализ (Фиг. 4).
Заключение
Было обнаружено, что scFv AS20 связывается как с небиотинилированной BSSL человека, так и с биотинилированной BSSL человека со сходной аффинностью (сходными значениями KD) в субнаномолярном диапазоне. Полученное значение KD для небиотинилированной BSSL хорошо согласуется с тем, что было сообщено в Примере 1 для полноразмерного антитела IgG. AS20 scFv также демонстрировал низкое нецелевое связывание с 30 нерелевантными белками при анализе с применением подхода на основе Luminex. Как показывают результаты ELISA, scFv AS20 демонстрирует связывание с BSSL мыши, что было продемонстрировано для полноразмерного IgG в Примере 1.
Пример 3 - Характеризация связывания AS20 scFv с BSSL мыши посредством HTRF
Гомогенная флуоресценция с временным разрешением (HTRF) представляет собой спектрофотометрический метод, основанный на явлении резонансного переноса энергии флуоресценции (FRET) между двумя разными молекулами, которые известны как донор и акцептор соответственно. В этом примере описывается разработка и применение конкурентного анализа на основе HTRF в качестве альтернативного способа для характеризации взаимодействия между scFv AS20 и BSSL мыши.
Материал и способы
Для изучения взаимодействия между AS20 scFv и нативной BSSL мыши был разработан конкурентный анализ. Обнаружение связывания стало возможным с помощью донорной молекулы α-FLAG-антитела, конъюгированного с тербием (Cisbio # 611FG2TL), которое взаимодействует с расположенным на С-конце FLAG-меткой scFv, и акцепторной молекулы XL665, конъюгированной со стрептавидином (Cisbio # 610SAXL), которая взаимодействует с биотиновыми фрагментами на BSSL человека. Эксперименты проводили с применением диапазона концентраций небиотинилированных белков; 0-500 нМ для mBSSL и 0-80 нМ для hBSSL. hBSSL, b-hBSSL и mBSSL (Таблица 2) применяли вместе с scFv AS20 (собственного производства, HCVR SEQ ID NO: 80 и LCVR SEQ ID NO: 114) в экспериментах, описанных в этом Примере.
2,5 нМ scFv AS20 предварительно инкубировали в течение 2 часов с ортологом BSSL мыши или человека, после чего добавляли 5 нМ b-hBSSL и FRET донорную и акцепторную молекулы. Наконец, смесь инкубировали при комнатной температуре в течение 16 часов и сигнал связывания (665 нм) и сигнал фон/шум (615 нм) измеряли с помощью EnVision (PerkinElmer). Выходные данные эксперимента Delta R рассчитывали для каждой точки с применением четырех повторений и двух холостых опытов.
Результаты
Данные продемонстрировали, что BSSL мыши в зависимости от концентрации конкурировал с человеческим BSSL-биотином за связывание с scFv AS20 (Фиг. 5). То же самое наблюдение было характерным для нативной BSSL человека. Снижение Delta R на 50% достигали с 150-200 нМ BSSL мыши и примерно 2 нМ BSSL человека, что позволяет предположить, что scFv AS20 имеет примерно в 100 раз меньшую аффинность к BSSL мыши по сравнению с человеческим ортологом.
Заключение
Полученные данные продемонстрировали, что AS20 scFv имел примерно в 100 раз меньшую аффинность к BSSL мыши по сравнению с человеческим ортологом. Это очень соответствовало аффинности, полученной для полноразмерных форматов антител того же клона с применением SPR; AS20 mIgG1 (Пример 1) и химерного AS20 (Пример 4).
Пример 4 - Продуцирование химерного AS20
В этом примере получают химерный AS20. Более конкретно, был сконструирован химер подкласса IgG4 человека.
Материалы и способы
Для изготовления материала была привлечена компания GenScript (Пискатауэй, штат Нью-Джерси, США). Применяли последовательности вариабельных доменов тяжелой (VH) и легкой (VL) цепей мышиного антитела AS20, соответствующие SEQ ID NO: 80 и SEQ ID NO: 114 соответственно. Гены, кодирующие тяжелую и легкую цепи, синтезировали и клонировали в вектор, кодирующий подкласс IgG4 человека. Конструкции трансфицировали и временно экспрессировали в клетках FreeStyle 293-F. Экспрессированное антитело затем очищали с помощью аффинной хроматографии с применением протеина A с последующей препаративной эксклюзионной хроматографией (SEC); чистоту определяли с помощью высокоэффективной жидкостной хроматографии (HPLC), а концентрацию определяли спектрофотометрически при 280 нм.
Результаты
Чистоту определяли с помощью HPLC и при этом она составляла >98%. Последовательность химерного AS20 определяли как SEQ ID NO: 139 для тяжелой цепи и SEQ ID NO: 140 для легкой цепи.
Химерный AS20 сохранял такую же аффинность связывания (данные не приведены) с BSSL мыши и человека, что и антитело IgG1 мыши AS20 в Примере 1.
Пример 5 - Дизайн и конструкция антитела AS20 CDR-трансплантата и библиотеки гуманизации AS20
AS20 представляет собой мышиное антитело, AS20 mIgG, см. Пример 1. Было продемонстрировано, что нечеловеческие антитела вызывают иммунные ответы у человека, которые могут приводить к нейтрализации введенного антитела и, в свою очередь, ограничивают эффект антитела при лечении заболевания. С целью преодоления этой потенциальной проблемы осуществляли гуманизацию антитела. В этом Примере описаны две стратегии гуманизации AS20, а именно прививание определяющих комплементарность областей (CDR) и подход на основе библиотеки. Полученное в результате антитело трансплантата CDR в настоящем документе называется AS20 CDR-трансплантатом или CDR трансплантатом, а созданная библиотека называется в настоящем документе библиотекой гуманизации AS20. Библиотеку впоследствии применяли для отбора и выделения AS20-связывающих scFv фрагментов с помощью фагового дисплея (см. Пример 6).
Материалы и способы
Фигуры 15A и 15B представляют собой краткое изложение дизайна комбинаторной библиотеки scFv для вариабельной области тяжелой цепи, а Фигуры 16A и 16B представляют собой краткое изложение дизайна комбинаторной библиотеки scFv для вариабельной области легкой цепи.
Формат scFv был выбран в качестве каркаса как для CDR трансплантата, так и для библиотеки гуманизации. Данные, представленные в Примере 2, продемонстрировали, что AS20 scFv полностью сохранял связывающую способность своего полноразмерного родительского аналога IgG, позволяя предположить, что ген scFv будет хорошим каркасным форматом как для CDR трансплантата, так и для построения комбинаторных библиотек.
Ген вариабельной области тяжелой цепи зародышевой линии иммуноглобулина человека (IHGV) с наивысшей гомологией последовательности с вариабельной областью тяжелой цепи AS20, согласно IMGT/DomainGapAlign, (http://www.imgt.org/3Dstructure-DB/cgi/DomainGapAlign.cgi), IGHV1-46 с гомологией 73,5% (остатки 1-104) был выбран в качестве каркаса IHGV. При выборе последовательности легкой цепи учитывалась гомология, но также учитывались пары тяжелых и легких цепей с предпочтительными биофизическими свойствами [10]. Все вместе это обусловило выбор гена зародышевой линии человека IGKV1-39. Также на основе поиска домена IMGT/DomainGapAlign, IGHJ4 и IKVJ2 были выбраны в качестве соединяющих фрагментов для получения полных вариабельных доменов тяжелой и легкой цепи соответственно.
AS20 CDR трансплантат получали путем прививания шести петель CDR мыши в гены зародышевой линии человека. Для тяжелой цепи в каркас IGHV1-46 прививали следующие области: определяющую комплементарность область 1 тяжелой цепи (HCDR1) (SEQ ID NO: 7); удлиненную HCDR2 (eHCDR2) (SEQ ID NO: 141); и HCDR3 (SEQ ID NO: 9). В результате получали CDR трансплантат на HCVR согласно SEQ ID NO: 144. Для легких цепей следующие области прививали в каркас IGKV1-39: определяющую комплементарность удлиненную область 1 легкой цепи, (eLCDR1) (SEQ ID NO: 142), eLCDR2 (SEQ ID NO: 143) и LCDR3 (SEQ ID NO: 21), в результате получали LCVR согласно SEQ ID NO: 145. Путем слияния HCVR с LCVR через глицин-сериновый линкер ((Gly4Ser)3), был сформирован ген, кодирующий соответствующую конструкцию scFv. Две дополнительные аминокислоты (Arg и Thr, обе части домена CL) добавляли в конец LCVR с целью включения сайта рестрикции BsiWI. Для синтеза и субклонирования гена scFv привлекали компанию GenScript (Пискатауэй, штат Нью-Джерси, США). После синтеза ген scFv клонировали во внутреннюю фагмиду с применением рестрикционных ферментов SfiI и BsiWII.
Тот же каркас HCVR и LCVR, который применяли в CDR трансплантате, также применяли для конструирования каркаса библиотеки гуманизации AS20 (Фиг. 6). Стратегия мутагенеза для библиотеки гуманизации AS20 обобщена в Таблице 5. HCDR3 считается наиболее важной областью для связывания антигена. Было высказано предположение, что эта петля, скорее всего, также важна для взаимодействия AS20-BSSL и поэтому оставалась постоянной. Вместо этого для вариации были выбраны 22 положения, различающиеся между AS20 и каркасом гуманизации AS20 в других пяти областях CDR (Фиг. 6). В этом случае в первую очередь была предпринята попытка осуществления двойного разнообразия, то есть допущение в процесс остатков, обнаруженных в AS20 и генах зародышевой линии человека, которые создают каркас гуманизации в определенном положении. Однако не все пары аминокислот могут быть образованы посредством олиго NNS без введения дополнительных аминокислот, поэтому в шести положениях было добавлено дополнительное химическое разнообразие (три в VH: 62, 64, 68 и три в VL: 27, 66, 68). Для LCDR3 была предпринята альтернативная стратегия. В этом случае было учтено разнообразие реаранжированных функциональных антител, кодируемых геном IGVK1-39/IKV1D-39, как обнаружено в базе данных IMGT (http://www.imgt.org/ligmdb/). Более конкретно, последовательности антител, имеющие длину LCDR3 из 8 аминокислот, длину LCDR3 в AS20, применяли в качестве руководства для того, какое разнообразие вводить. Полученная консенсусная последовательность представляла собой QQSYSTPT (аминокислотные остатки 105-117, SEQ ID NO: 173). На основе AS20 мыши и консенсусных последовательностей человека в положения 105, 107 и 108 было введено двойное разнообразие. В положение 115, опять же из-за ограничения олиго NNS, были введены четыре аминокислоты. В результате процесса присоединения гена VJ наибольшее разнообразие в LCDR3 обнаруживалось в положении 116. В попытке имитировать эту изменчивость, но также ограничиваясь применением кодонов NNS, в этом случает в процесс были вовлечены шесть аминокислот (P, H, L, Y, S и F). Эта стратегия позволяет захватить >50% разнообразия, обнаруженного среди антител в этом положении. В целом описанная выше процедура создает комбинаторное теоретическое разнообразие из примерно 1.2×109 различных вариантов.
Таблица 5 - Положения, предназначенные для мутагенеза, в библиотеке гуманизации AS20. Положения в HCVR и LCVR указаны в верхней и нижней части таблицы, соответственно, аминокислоты, выделенные жирным шрифтом, представляют собой аминокислоты, обнаруженные в AS20, тогда как подчеркнутые аминокислоты представляют собой соответствующее разнообразие, обнаруженное в гене зародышевой линии человека. Нумерация определяется номенклатурой IMGT, а для определения кодона применяется код нуклеотида IUPAC. Для введения разнообразия применяли по одному праймеру для пяти целевых областей (HCDR1, HCDR2, LCDR1, LCDR2 и LCDR3)
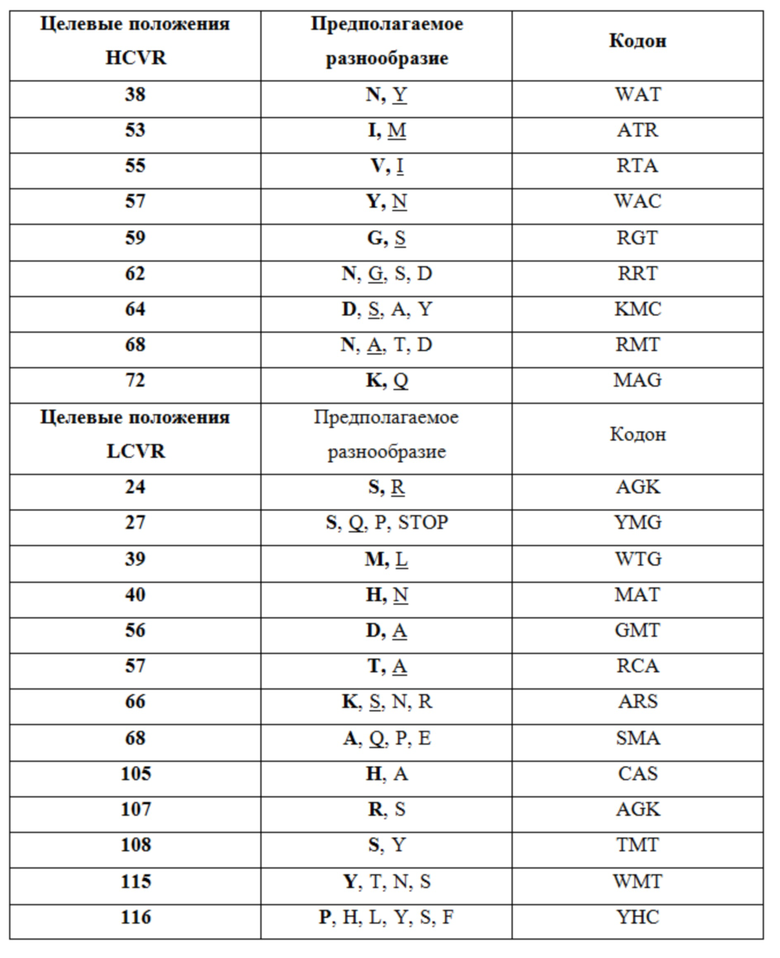
Разнообразие вносили в ген каркаса библиотеки с помощью оптимизированной методологии мутагенеза Кункеля, в основном, как описано в [11], с применением гена каркаса библиотеки гуманизации AS20 (Фиг. 6) вместе с пятью мутагенными олигонуклеотидами (Таблица 6). Для того, чтобы оценить, было ли включено предполагаемое разнообразие, клетки E. Coli TOP10 химически трансформировали небольшой аликвотой ДНК, полученной с помощью методологии мутагенеза Кункеля, и 96 клонов отбирали и отправляли на секвенирование (GATC, Германия). Оставшуюся ДНК затем подвергали электропорации в клетки SS320 (Lucigen, Миддлтон, штат Висконсин, США), получая очень разнообразную библиотеку, содержащую примерно 1,7×1010 клонов, что измерено по количеству бактериальных колоний, полученных после трансформации. Трансформированные клетки SS320 собирали и хранили с 15% глицерином при -80 °C. Исходный раствор бактериального глицерина применяли для инокуляции в общей сложности 600 мл 2×YT с антибиотиками, селективными как для фагмиды, так и для F'-эписомы. Бактерии выращивали до экспоненциальной фазы, а затем инфицировали фагами-хелперами M13KO7 (New England Biolabs, Ипсвич, штат Массачусетс, США) с количеством инфицирований, кратным пяти. Культуру размножали в течение ночи и фаги, отображающие scFv, собирали путем стандартного осаждения полиэтиленгликолем PEG/NaCl. Конечный исходный материал библиотеки растворяли в PBS с добавлением 0,5% BSA, 0,05% Tween-20.
Таблица 6 - Олигонуклеотидные праймеры, применяемые для создания библиотеки гуманизации AS20. Последовательности отформатированы с применением нуклеотидного кода IUPAC.
Результаты
Гены, кодирующие AS20 CDR трансплантат и каркас библиотеки гуманизации AS20, синтезировали и клонировали в фагмидный вектор pHAT4. Библиотеку гуманизации AS20 конструировали с помощью оптимизированной процедуры по Кункелю,в результате которой получали 1,7×1010 трансформантов. Секвенирование 96 случайно выбранных клонов подтвердило внесение заданного разнообразия (данные не приведены).
Заключение
AS20 CDR трансплантат и библиотека гуманизации AS20 были успешно сконструированы. В обоих случаях IGHV1-46 и IGKV1-39 применяли в качестве человеческих генов структурного каркаса. Связывание AS20 CDR трансплантата с BSSL оценивали как в scFv (Пример 6), так и в формате IgG (Примеры 9 и 11). Библиотеку гуманизации AS20 применяли для выделения гуманизированных фрагментов scFv, связывающих BSSL, с помощью фагового дисплея и различных анализов скрининга связывания (Пример 6). Некоторые из выбранных клонов продемонстрировали связывание с родственной мишенью (BSSL человека) с показателями аффинности и специфичности, эквивалентными родительскому IgG, а при связывании с ортологом мыши продемонстрировали даже лучшую аффинность (Пример 9). При анализе последовательностей выбранных клонов (Пример 11) становится ясно, что в определенных положениях наблюдается обогащение специфических остатков. Представляет интерес тот факт, что в нескольких положениях (например, VH: 62, 64 и VL: 115) существует предпочтительный отбор аминокислот за пределами двойного разнообразия, что указывает на то, что введение дополнительного разнообразия было успешной стратегией.
Пример 6 - Отбор фагового дисплея на BSSL человека и мыши и последующий скрининг и секвенирование
В этом примере отбор фагового дисплея выполняли для обеспечения выделения фрагментов scFv, специфичных для BSSL человека и мыши.
Материал и способы
Антигены
Во время отбора фагового дисплея в качестве антигенов-мишеней применяли BSSL мыши и небиотинилированную и биотинилированную BSSL человека. Более конкретно, были изготовлены два варианта с разной степенью биотинилирования и разными химическими характеристиками связывания. Это были амин BSSL-b и глико BSSL-b. hBSSL, b-hBSSL, hBSSL-b амин и mBSSL (Таблица 2) применяли в качестве мишени для отбора фагового дисплея в этом примере.
Отбор фагового дисплея
Для всех антигенов фаговый дисплей выполняли с четырьмя циклами обогащения с применением двух сконструированных на собственном производстве синтетических фаговых библиотек человека scFv: библиотеки SciLifeLib2 и библиотеки гуманизации AS20 (см. Пример 5). SciLifeLib2 представляет собой наивную синтетическую библиотеку scFv человека, аналогичную по дизайну и конструкции ранее описанным [12]. Давление отбора увеличивалось путем постепенного уменьшения количества антигена и увеличения количества и интенсивности промывок между различными циклами. Для двух биотинилированных образцов BSSL человека отбор проводили путем иммобилизации на парамагнитных гранулах, покрытых стрептавидином (Dynabeads M-280, ThermoFisher Scientific, # 11206D), и большинство этапов процесса отбора были автоматизированы и выполнены с помощью робота Kingfisher Flex. Отбор нативных антигенов проводили путем нанесения на 96-луночный планшет (NUNC Maxisorp # 442404). В некоторых из дорожек, чтобы предпочтительно выбрать межвидовой реактивный scFv, антиген чередовали между BSSL человека и мыши в разных циклах. Элюцию фагов проводили трипсином или путем конкурентной элюции с применением BSSL мыши для отбора на BSSL человека и наоборот. В результате применения комбинации этих различных параметров получили схему, охватывающую в общей сложности пять различных дорожек отбора для ScilifeLib2 и девять для библиотеки гуманизации AS20. Выделенные фаги размножали в Top10F’ E. coli, либо на чашках с агаром при 37 °C в течение ночи (циклы 1 и 2), либо в растворе при 30 °C в течение ночи (циклы 3 и 4). Исходный материал фага получали путем инфицирования избытком фага-хелпера M13K07 (New England Biolabs, # N0315S) и экспрессии scFv, индуцированной добавлением IPTG. Культуры, выдержанные в течение ночи, осаждали PEG/NaCl, ресуспендировали в буфере для отбора и применяли для следующего цикла отбора. В Таблице 7 приведены дорожки отбора фагового дисплея.
Таблица 7 - Дорожки фагового дисплея
1 – отбор, выполненный на биотинилированной BSSL (амино-связанная, 10X), связанной с магнитными гранулами SAV (M280);
2 – отбор, выполненный на биотинилированной BSSL (на углеводах, BH8520), связанной с магнитными гранулами SAV (M280);
3 – отбор, выполненный на нативном антигене, нанесенном на поверхность иммунотрубок;
4 – элюция фага путем конкуренции с другими видами BSSL (мыши или человека) (во всех других случаях для элюции будет применяться трипсин).
Повторное клонирование
Чтобы обеспечить продукцию растворимого scFv, выделяли фагмидную ДНК из третьего и четвертого циклов каждой дорожки отбора. В пулах, гены, кодирующие фрагменты scFv, субклонировали в скрининговый вектор, обеспечивая сигнал для секреции scFv вместе с меткой тройного FLAG и меткой гексагистидина (His) на С-конце. Впоследствии конструкцию трансформировали в TOP10 E.coli.
Первичный анализ ELISA и секвенирование
Для каждой дорожки отбора, от 89 до 222 колоний в целом из цикла 3 и 4 отбирали и культивировали в 96-луночных планшетах и выращивали в течение ночи. Экспрессированные scFv (супернатанты) подвергали скринингу с помощью ELISA на связывание с нативной формой соответствующей мишени отбора. Эксперименты в этом процессе скрининга включали scFv AS20, который был ранее сконструирован, продуцирован и охарактеризован в отношении связывания с BSSL как человека, так и мыши (см. Пример 2). Также был включен AS20 CDR трансплантат в качестве scFv (см. Пример 5). Клоны, расцененные как положительными в ELISA, подвергали секвенированию ДНК (GATC Biotech, Кельн, Германия).
Вторичный анализ ELISA и HTRF
Все клоны с уникальной последовательностью, идентифицированные для каждой дорожки отбора, дополнительно анализировали во втором скрининге с помощью ELISA. В этом эксперименте количество антигенов было увеличено, чтобы включить нативную BSSL человека и мыши, а также биотинилированную BSSL человека. Стрептавидин применяли как нерелевантный антиген. Связывание всех scFv также оценивали с помощью HTRF (гомогенная флуоресценция с временным разрешением).
Результаты
В общей сложности 14 дорожек отбора фага были выполнены параллельно на четырех формах BSSL с применением библиотеки SciLifeLib 2 и библиотеки гуманизации AS20. С каждой из 14 дорожек были отобраны и проанализированы от 89 до 222 клонов. В результате скрининга связывания ELISA и HTRF и секвенирования обнаружили в общей сложности 68 уникальных клонов scFv, способных связываться с ортологом BSSL, для которого они были отобраны. Неожиданно не было обнаружено связывания AS20 CDR трансплантата scFv.
Заключение
Первичный скрининг в общей сложности 2365 клонов с помощью ELISA привел к тому, что всего 467 scFv-фрагментов с потенциальной аффинностью связывания для BSSL человека и мыши были отправлены на секвенирование. В результате вторичного скрининга ELISA с последующим HTRF и повторным секвенированием получили в общей сложности 68 клонов scFv с уникальной последовательностью. По данным их связывания можно предположить, что их относительное связывание с BSSL человека и мыши можно разделить на три группы с характеристиками распознавания одного или обоих этих ортологов.
64 изолятов scFv получили из библиотеки гуманизации AS20, в то время как только четыре из них получили из SciLifeLib2. Это указывает на то, что BSSL является сложной мишенью для отбора на фаговом дисплее с применением библиотеки наивных антител.
Пример 7 - ELISA и ранжирование аффинности 68 анти-BSSL scFv по SPR
В этом Примере 68 уникальных scFv, сгенерированных в Примере 6, анализировали с помощью ELISA и, кроме того, ранжировали на основе аффинности с помощью SPR. Вместе с более ранними данными связывания (ELISA и HTRF), эти результаты учитывали при принятии решения относительно выбора кандидатов для дальнейшей разработки.
Материал и способы
hBSSL и mBSSL (Таблица 2) применяли в качестве реагентов BSSL в этом Примере.
ELISA
BSSL человека и мыши покрывали при 4°C в течение ночи, 1 μг/мл в PBS, на 384-луночном планшете для ELISA. Также были включены два отрицательных контрольных белка, стрептавидин и BSA. FLAG-меченные клоны scFv, присутствующие в бактериальном супернатанте, разбавляли 1:2 и 1:20 в буфере для анализа (PBS + 0,5% BSA + 0,05% Tween 20) и оставляли для связывания с покрытыми белками. Все образцы анализировали в двух повторениях. Обнаружение связывания осуществляли с помощью HRP -конъюгированного антитела α-FLAG M2 (Sigma-Aldrich # A8592) с последующей инкубацией с субстратом TMB ELISA (ThermoFisher Scientific # 34029). Развитие колориметрического сигнала останавливали добавлением 1 М серной кислоты, и планшет анализировали при 450 нм.
SPR
Кинетический скрининг проводили на биосенсорном приборе BIACORE® T200 (GE Healthcare). Антитело α-FLAG M2 (Sigma-Aldrich #F1804), функционирующее как захватывающий лиганд, иммобилизовали на всех 4 поверхностях сенсорного чипа CM5-S в соответствии с рекомендациями производителя.
FLAG-меченные клоны scFv, присутствующие в бактериальном супернатанте, инъецировали и захватывали на поверхности чипа с последующей инъекцией BSSL либо человека или мыши при 50 нМ и 200 нМ соответственно. Поверхность регенерировали 10 мМ глицин-HCl pH 2,2. Все эксперименты проводили при 25°C в рабочем буфере (PBS + 0,1% BSA + 0,05% Tween 20 pH 7,5 для BSSL человека и 25 мМ Tris-HCl + 150 мМ NaCl pH 7,5 для BSSL мыши).
Результаты
ELISA
Связывание 68 scFv-клонов с непосредственно покрытой BSSL человека и мыши можно было подтвердить для большинства клонов. Как продемонстрировано в Примере 6, BSSL-связывающие клоны можно разделить на три группы с характеристиками распознавания либо BSSL человека, либо BSSL мыши, либо BSSL человека и мыши. Большинство клонов демонстрируют предпочтительное связывание с BSSL человека (данные не приведены).
SPR
Анализ данных проводили путем визуального осмотра сенсограмм (не продемонстрированы). При их изучении стало очевидно, что большинство клонов имели значительно более высокую аффинность, более низкое значение KD (M) к BSSL человека, чем к BSSL мыши. Изучение сенсограмм также продемонстрировало, что многие из гуманизированных клонов проявляли аффинности в том же диапазоне, что и для scFv AS20. Примерами таких клонов являются S-SL048-11 (содержащий HCVR SEQ ID NO: 30 и LCVR SEQ ID NO: 31), S-SL048-14 (содержащий HCVR SEQ ID NO: 50 и LCVR SEQ ID NO: 84), S-SL048-106 (содержащий HCVR SEQ ID NO: 34 и LCVR SEQ ID NO: 35), S-SL048-108 (содержащий HCVR SEQ ID NO: 72, LCVR SEQ ID NO: 106), S-SL048-109 (содержащий HCVR SEQ ID NO: 73 и LCVR SEQ ID NO: 107), S-SL048-116 (содержащий HCVR SEQ ID NO: 36 и LCVR SEQ ID NO: 37) и S-SL048-125 (содержащий HCVR SEQ ID NO: 77, LCVR SEQ ID NO: 111). Аффинность к BSSL мыши для этих конкретных клонов также была сходной с той, что наблюдается для AS20.
Несколько клонов, полученных из селекционных дорожек фагового дисплея, сортированных по отношению к BSSL мыши, продемонстрировали предпочтительное связывание с BSSL мыши, чем с BSSL человека, примером которого является клон S-SL048-66 (содержащий SEQ ID NO: 61 HCVR и SEQ ID NO: 95 LCVR)
Заключение
В этом Примере связывание ранее идентифицированных 68 scFv-клонов с BSSL было подтверждено с помощью ELISA. Кинетический скрининг был также выполнен на всех 68 клонах с применением одной концентрации BSSL человека и мыши. Как было обнаружено для AS20, подавляющее большинство клонов продемонстрировало более высокую расчетную аффинность связывания, определяемую как равновесие константы диссоциации,KD (M), по отношению к BSSL человека, чем к BSSL мыши. Расчетная аффинность к BSSL человека находилось в диапазоне от низких наномолярных до наномолярных значений, при этом эталонный AS20 scFv имел значение KD, равное 2 нМ. Небольшой набор клонов scFv проявлял более высокую аффинность к BSSL мыши, чем к BSSL человека, с самым высокой аффинностью, соответствующей значению KD, равному 43 нМ.
Из всех собранных данных, 38 клонов (содержащих HCVR SEQ ID NO: 30, 32, 34, 36 и 47-79 и LCVR SEQ ID NO: 31, 33, 35, 37, 38 и 81-113) и эталонный scFv AS20 ( HCVR SEQ ID NO: 80 и LCVR SEQ ID NO: 114) были выбраны для преобразования в IgG4 S228P человека. Все клоны-кандидаты происходили из библиотеки гуманизации AS20 и ни один из SciLifeLib.
Пример 8 - Преобразование в формат hIgG4 S228P и мелкомасштабная временная экспрессия 38 гуманизированных BSSL-специфических антител
В этом эксперименте, 38 наиболее многообещающих гуманизированных клонов scFv (содержащих HCVR SEQ ID NO: 30, 32, 34, 36 и 47-79 и LCVR SEQ ID NO: 31, 33, 35, 37, 38 и 81-113) из фагового отбора и последующего скрининга связывания в Примере 7 были преобразованы в формат антитела IgG4 S228P человека. Кроме того, AS20 (родительский клон) (содержащий HCVR SEQ NO: 80 и LCVR SEQ ID NO: 114) и AS20 CDR трансплантат (содержащий HCVR SEQ NO: 144 и LCVR SEQ ID NO: 145) аналогичным образом были преобразованы в IgG4 S228P.
Вкратце, IgG4 человека считается самым Fc-сайлесинговым естественным подклассом IgG у человека, т.е. он не опосредует основные эффекторные функции через Fc-часть антитела. Подобно IgG1, IgG4 имеет период полужизни в сыворотке 21 день. Однако IgG4 естественным образом имеет тенденцию диссоциировать in vivo на половину молекулы IgG4 и затем может объединяться с другими циркулирующими молекулами IgG4. Этого обмена половинами молекул можно избежать путем введения стабилизирующей мутации в шарнирную область, а именно S228P (нумерация Eu; она идентична нумерации Kabat S241P) [13].
Гены, кодирующие VH и VL 38 клонов scFv, AS20 и AS20 CDR трансплантата, успешно переносили в вектор, кодирующий подкласс IgG4 S228P человека. Клетки ExpiHEK293 временно трансфицировали, антитела экспрессировали в мелком масштабе (4 мл) и очищали с протеином А. Чистоту и целостность/содержание мономеров анализировали с помощью SDS-PAGE и аналитической эксклюзионной хроматографии (SEC).
Материал и способы
Секвенирование
Аминокислотные последовательности HCVR и LCVR scFv, выбранные для преобразования IgG, представлены в перечне последовательностей и для ясности в Таблице 8 вместе с антителом против гаптена (4-гидрокси-3-нитрофенилацетил, NP) (анти-NP). .
Таблица 8 - Антитела, примененные в этом Примере
HCVR
LCVR
hIgG4 S228 P тяжелая цепь (HC)
hIgG4 S228 P легкая цепь (LC)
На Фиг. 14 продемонстрированы различия в последовательностях 38 гуманизированных клонов, преобразованных в hIgG4 S228P. В библиотеке гуманизации AS20 всего 20 положений были нацелены на диверсификацию в CDR1 и CDR2 тяжелой цепи и CDR1, CDR2 и CDR3 легкой цепи. Для сравнения на фигуре продемонстрированы AS20 и конструкция CDR трансплантата.
Клонирование In-Fusion
Плазмидную ДНК 38 BSSL-специфичных scFv и AS20 очищали из бактериальной культуры с помощью стандартной процедуры Mini-Prep. Ген для AS20 CDR трансплантата синтезировали в компании Genscript. Области VH и VL амплифицировали с помощью PCR и вставляли в сконструированный на собственном производстве вектор pHAT-hIgG4-S241P с применением набора для клонирования In-Fusion HD Plus (Clontech # 638909). Одним из репрезентативных примеров полученной полноразмерной последовательности IgG является последовательность тяжелой цепи S-SL048-11 hIgG4 S228P (VH-CH1-шарнир-CH2-CH3), соответствующая SEQ ID NO: 119, и последовательность легкой цепи S-SL048-11 hIgG4 S228P (VL-CL), соответствующая SEQ ID NO: 120.
Трансфекция в HEK293, экспрессия и очистка
Трансфекцию плазмидной ДНК в клетки expiHEK293 проводили с применением набора для трансфекции ExpiFectamineTM 293 (ThermoFisher Scientific # A14525) в 4 мл культур на 24-луночном планшете. После 5 дней культивирования при 37°C, 6% CO2, 80% относительной влажности и 400 об/мин, супернатант среды смешивали с конъюгированными с протеином А магнитными гранулами и очищали на приборе KingFisher Flex. Сразу после элюирования в 0,1 М глицине, pH 2,7, выполняли нейтрализацию добавлением 1 М трис-HCl, pH 8,8, а замену буфера на PBS выполняли с применением 96-луночного вращающегося планшета для обессоливания. SDS-PAGE выполняли для определения чистоты и целостности очищенного IgG и концентраций, определенных с помощью спектрофотометра Implen NP80 UV-Vis (Fisher Scientific).
Результаты
Клонирование In-Fusion
38 уникальных scFvs, AS20 и AS20 CDR трансплантат были успешно преобразованы в полноразмерные антитела IgG4 человека, что подтверждено секвенированием.
Трансфекция в HEK293, экспрессия и очистка
Антитела экспрессировали в клетках expiHEK293 и очищали из супернатанта очисткой с протеином А. Чистоту и целостность очищенного IgG подтверждали с помощью SDS-PAGE (данные не приведены).
Заключение
Все BSSL-связывающие антитела были успешно повторно клонированы в формат hIgG4, экспрессированы в клетках HEK293 и очищены с помощью магнитных гранул, конъюгированных с протеином А, на приборе Kingfisher Flex. Все продемонстрировали приемлемый уровень чистоты при оценке с помощью SDS-PAGE.
Пример 9 – Связывание 38 клонов hIgG4 S228P с BSSL человека и мыши
В этом примере описан анализ связывания с мишенью 38 клонов hIgG4 S228P (Пример 8, Таблица 8) с помощью поверхностного плазмонного резонанса (SPR), который проводили для верификации того, что связывание с BSSL человека и мыши сохраняется после преобразования scFv в формат IgG.
Материал и способы
Кинетические параметры клонов hIgG4 S228P определяли посредством SPR с помощью системы BIACORE® T200 (GE Healthcare). Кинетику одного цикла учитывали для измерения аффинности очищенных молекул hIgG4 к BSSL человека и мыши. Антитело анти-Fab (GE Healthcare, # 28958325) иммобилизовали на сенсорном чипе CM5 S посредством связывания первичным амином с использованием химического состава NHS-EDC. hIgG4 захватывался антителом анти-Fab, и впоследствии на поверхность наносили пять различных концентраций hBSSL (разведения 1:5, начиная с 50 нМ) или mBSSL (разведения 1:5, начиная с 500 нМ). Поверхность сенсорного чипа регенерировали 10 мМ глицин-HCl pH 2,1. Применяли реагенты BSSL, перечисленные в Таблице 2. Для связывания с BSSL человека, кинетические данные одного цикла подгоняли к модели связывания 1:1, и получали кинетические параметры с помощью программного обеспечения BIAevaluation. Для BSSL мыши, анализ стационарного состояния выполняли путем построения графика зависимости уровня ответа в равновесии по отношению к каждой концентрации, и значения KD получали с помощью программного обеспечения BIAevalution.
Результаты
Для определения аффинности преобразованных антител к небиотинилированной BSSL человека и BSSL мыши применяли подход с кинетикой одного цикла. Полученные равновесные константы диссоциации (KD) перечислены в Таблице 9. Для hBSSL, данные, как правило, успешно подгоняли под модели связывания 1:1. Для BSSL мыши, данные не всегда очень хорошо соответствовали модели связывания 1:1. Вместо этого данные анализировали с помощью анализа стационарного состояния. Определенные значения KD для связывания с BSSL человека, а также с BSSL мыши находились в том же диапазоне, что и значения KD, определенные для тех же клонов в формате scFv (см. Пример 7). Для клона AS20 CDR трансплантата, наблюдалась низкая степень связывания с BSSL человека (данные не приведены). Однако подгонка модели не была сочтена точной, и поэтому значение KD не было получено для этого конкретного клона.
Таблица 9 - Измеренные равновесные константы диссоциации (KD). В некоторых случаях качество данных было сочтено слишком низким, чтобы быть надежным, и обозначено в данном документе как «н.о.» (не определено)
Заключение
В целом результаты демонстрируют, что на аффинность связывания антител с BSSL человека и мыши не влияло повторное клонирование в формат hIgG4. Однако AS20 CDR трансплантат вёл себя по-другому. Как описано в Примере 6, AS20 CDR трансплантат не демонстрировал связывания с BSSL при экспрессии в формате scFv. Однако в формате IgG наблюдали сигнал связывания с BSSL человека.
Пример 10 - Функциональный тест 28 клонов hIgG4 S228P с помощью анализа замещения на основе проточной цитометрии.
В этом примере, 28 антител hIgG4 S228P с наивысшей аффинностью связывания с BSSL человека и/или мыши в Примере 9 (содержащие HCVR SEQ ID NO: 30, 32, 34, 36, 47, 50-56, 59-65, 68, 69, 71-73, 75, 77 и 78 и LCVR SEQ ID NO:31, 33, 35, 37, 38, 81, 84-90, 93-99, 102, 103,105-107, 109, 111 и 112) были протестированы на их способность блокировать связывание BSSL человека с CD14+ моноцитами с помощью анализа замещения на основе проточной цитометрии. Пять антител были включены в качестве эталонных; AS20 mIgG1 (HC SEQ ID NO: 135 и LC SEQ ID NO: 136), AS20 hIgG4 (HC SEQ ID NO: 129 и LC SEQ ID NO: 130), AS20 CDR трансплантат (HC SEQ ID NO: 131 и LC SEQ ID NO: 132), античеловеческий альфа-синуклеин mIgG1 и анти-NP hIgG4 (HC SEQ ID NO: 133 и LC SEQ ID NO: 134).
Материал и способы
Приготовление лейкотромбоцитарной плёнки
Человеческую кровь была взята у одного здорового донора в пробирки Vacutainer с добавлением цитратного антикоагулянта (BD Vacutainer). Лейкотромбоцитарную плёнку, состоящую из лейкоцитов и тромбоцитов, выделяли после центрифугирования при 1300×g в течение 10 мин при комнатной температуре в центрифужном роторе со стаканами.
Анализ замещения на основе проточной цитометрии
28 BSSL-специфичных антител к hIgG4 и пять эталонных антител в различных концентрациях (от 0,5 μг до 3,0 μг на реакцию; см. Таблицу 10) добавляли к b-hBSSL (1 μг на реакцию, см. Таблицу 2) в пробирки из полистирола с круглым дном, 1× PBS (pH 7,4) добавляли до конечного объема 20 μл и смеси «антитело/b-hBSSL» инкубировали в течение 30 мин при +4°C для облегчения связывания антител с BSSL. Затем к каждой смеси антитело/b-hBSSL добавляли лейкотромбоцитарную пленку (50 μл) и инкубацию продолжали еще 30 мин при +4°C. После этого добавляли 2 мл лизирующего раствора FACS (BD Biosciences) и клетки инкубировали в течение 10 минут при комнатной температуре для лизиса эритроцитов и фиксации лейкоцитов. Затем клетки центрифугировали 5 минут при 200× g, полученные осадки промывали добавлением 2 мл буфера FACS (1× PBS с добавлением 1% FCS и 0,1% NaN3) и снова центрифугировали. Наконец, супернатанты сливали и клетки ресуспендировали в последней капле жидкости, примерно 50 μл.
5 μл BV421-меченного антитела против CD14 человека (BD Biosciences) и 5 μл BB515-меченного стрептавидина (BD Biosciences) добавляли в каждую пробирку и инкубировали в течение 30 мин при +4°C в защищенном от света месте. После этого клетки дважды промывали буфером FACS, ресуспендировали в 500 μл буфера FACS и прогоняли на проточном цитометре BD LSRII (BD Biosciences). Наконец, данные анализировали с помощью программного обеспечения FlowJo (BD Biosciences).
Результаты
Клетки CD14+ сначала выделяли, чтобы очертить популяцию моноцитов. Затем связывание b-hBSSL с выделенными моноцитами CD14+ обнаруживали с помощью BB515-меченного стрептавидина, и количественно определяли как среднюю интенсивность флуоресценции (MFI) в канале BB515. Способность BSSL-специфического hIgG4 и эталонных антител замещать связывание b-hBSSL с моноцитами CD14+ количественно оценивали как снижение BB515 MFI в моноцитах после инкубации с повышенными концентрациями BSSL-специфического hIgG4 или эталонных антител.
Таблица 10. Способность 28 BSSL-специфических hIgG4 S228P и эталонных антител блокировать связывание BSSL с моноцитами CD14+ человека. Следует обратить внимание, что не все антитела были протестированы во всех концентрациях
Заключение
Молекулярная масса hBSSL (76 кДа) составляет примерно половину молекулы IgG (150 кДа). Следовательно, в этом примере 1 μг BSSL и 2 μг IgG примерно соответствовали молярному соотношению 1:1. Применяя самую высокую концентрацию антител (3 μг на реакцию), авторы продемоснстрировали, что 9 из 28 BSSL-специфических антител hIgG4 S228P ингибировали (вытесняли) по меньшей мере 60% BSSL (1 μг на реакцию) от связывания с моноцитами. Наиболее эффективным антителом для замещения связывания было AS20 mIgG1, тогда как AS20 CDR трансплантат hIgG4, анти-NP hIgG4 и анти-α-синуклеин mIgG1 вообще не влияли на связывание.
Пример 11 - Получение 5 ранее указанных антител hIgG4 S228P и контролей
На основании результатов, полученных в анализах связывания, выполненных в Примере 9, и функциональных исследований in vitro в Примере 10, а также содержания последовательности, пять гуманизированных клонов hIgG4 S228P были выбраны для крупномасштабного производства (10 мг), а именно S-SL048-11, S-SL048- 46, S-SL048-106, S-SL048-116 и S-SL048-118.
Также были включены два контроля: AS20 и клон AS20 CDR трансплантата. Кроме того, был включен изотипический контроль. Для этой цели у компании Absolute Antibody было заказано антитело против гаптена (4-гидрокси-3-нитрофенилацетил, NP) (клон B1-8). Выбранный формат подкласса hIgG4 S228P такой же, как ранее применяли для оценки 38 BSSL-специфичных клонов в Примере 8.
Материал и способы
Продуцирование антитела
Для изготовления материала была привлечена компания Absolute Antibody (Оксфорд, Великобритания). Информацию о последовательностях VH и VL семи клонов отправляли в компанию, где синтезировали гены и клонировали их в векторы, кодирующие подкласс IgG4-S228P человека. Антитела временно экспрессировали в клетках HEK293 млекопитающих и затем очищали аффинной хроматографией с применением протеина A. Чистоту и целостность оценивали с помощью SDS-PAGE, а уровни эндотоксина определяли с помощью хромогенного анализа эндотоксина LAL.
Аминокислотные последовательности восьми антител соответствуют SEQ ID NO, как продемонстрировано в Таблице 11.
Таблица 11 - Антитела, применяемые в этом Примере
Поверхностный плазмонный резонанс (SPR)
Кинетические параметры клонов hIgG4 S228P определяли посредством SPR с помощью системы BIACORE® T200 (GE Healthcare). Антитело анти-Fab (GE # 28958325) иммобилизовали на сенсорном чипе CM5 S посредством связывания первичным амином с использованием химического состава NHS-EDC. Антитела hIgG4 захватывались антителом анти-Fab, а затем на поверхность вводили пять различных концентраций hBSSL (разведения 1:5, 0,08-50 нМ) или mBSSL (разведения 1:2, 50-800 нМ). Поверхность сенсорного чипа регенерировали 10 мМ глицин-HCl pH 2,1. Для связывания с BSSL человека, кинетические данные одного цикла подгоняли к модели связывания 1:1, и получали кинетические параметры с помощью программного обеспечения BIAevaluation. Для BSSL мыши, анализ стационарного состояния выполняли путем построения графика зависимости уровня ответа в равновесии по отношению к каждой концентрации, и значения KD получали с помощью программного обеспечения BIAevalution.
Результаты
Продуцирование антитела
Десять миллиграммов в концентрации 4,6-5 мг/мл восьми антител в 25 мМ гистидине, 150 мМ NaCl, 0,02% P80 (pH 6,0) получали от компании Absolute Antibody. Чистоту определяли с помощью SDS-PAGE до > 98%, а уровни эндотоксина определяли с помощью хромогенного анализа эндотоксина LAL до < 0,05 ЕЭ/мг. Содержание мономеров в каждом из клонов определяли с помощью аналитической эксклюзионной хроматографии до > 98%.
SPR
SPR применяли для определения аффинности антител к BSSL человека и мыши. В таблице 12 приведены различные значения KD ранее указанных клонов для связывания hBSSL и mBSSL. Для сравнения также включены значения KD, определенные для тех же клонов, произведенных на своих мощностях (эти эксперименты описаны в Примере 9).
Таблица 12 - Обобщенные результаты SPR-анализов антител в формате hIgG4 S228P. *Эти значения были получены в результате экспериментов, представленных в Примере 9. Числа, отмеченные знаком #, считаются ненадежными, и их следует рассматривать с осторожностью. В некоторых случаях качество данных было сочтено слишком низким, чтобы быть надежным, и обозначено в данном документе как «н.о.» (не определено)
(нМ)
(нМ)
(нМ)
Заключение
Пять ранее указанных кандидатов и три контроля, изготовленные компанией Absolute Antibody, анализировали с помощью ELISA (данные не приведены) и SPR на предмет связывания с BSSL. Полученные результаты продемонстрировали, что связывание с BSSL было, в общих чертах, очень сходным с тем, которое наблюдалось для соответствующих клонов, изготовленных на своих мощностях в том же формате IgG (см. Пример 9). Полученные результаты продемонстрировали, что контрольное изотипическое антитело анти-NP hIgG4 S228P не связывается с BSSL и, следовательно, может быть пригодно для применения в качестве отрицательного контроля в будущих анализах. Функциональность этих партий антител также оценивали в анализе замещения (Пример 10). Очень похожие результаты были получены для различных клонов, как описано в Примере 10 (данные не приведены).
В этом примере авторы впервые смогли измерить аффинность CDR-трансплантата. В эксперименте AS20 CDR трансплантат hIgG4 S228P (содержащий HC SEQ ID NO: 131 и LC SEQ ID NO: 132) демонстрировал четкое связывание с hBSSL при более высоких концентрациях, хотя связывание было значительно ниже по сравнению с AS20 hsIgG S228P (содержащим HC SEQ ID NO: 129 и LC SEQ ID NO: 130). Аффинность для hBSSL, измеренная в этом анализе, была ниже примерно в 100 раз, при этом для mBSSL аффинность было слишком низкой, чтобы ее можно было определить. Таким образом, аффинность к мишени была значительно ниже, когда CDR прививали на новый каркас. Наблюдаемое более сильное связывание BSSL посредством AS20 CDR трансплантата hIgG4 S228P по сравнению с scFv AS20 CDR трансплантатом (содержащим HCVR SEQ ID NO: 144 и LCVR SEQ ID NO: 145, см. Пример 6) может быть обусловлено разными форматами антител.
Пример 12 - Исследования стабильности пяти кандидатных антител
В этом примере описано исследование стабильности S-SL048-11, S-SL048-46, S-SL048-106, S-SL048-116 и S-SL048-118 в формате hIgG4 S228P, проводимое с целью характеризации биофизической стабильности антител. Для сравнения были включены AS20 как hIgG4 S228P, так и hIgG1 LALA-PG (см. Пример 17). Также были включены: AS20 CDR трансплантат hIgG4 S228P и анти-NP hIgG1 LALA-PG изотипический контроль. Анализ выполняли с помощью SDS-PAGE, аналитической SEC, нано-DSF и DLS.
Материалы и способы
Девять различных антител, включенных в исследование стабильности, и их соответствующие SEQ ID NO перечислены в Таблице 13. Антитела аликвотировали во флаконы и инкубировали при -80 °C, + 4 °C и + 40 °C при 5 мг/мл в 25 мМ гистидине, 150 мМ NaCl, 0,02% P80, pH 6,0. Образцы отбирали и анализировали в соответствии с графиком, приведенным в Таблице 14, где день 0 является начальным днем.
Таблица 13 - Антитела, примененные в этом Примере
Таблица 14 - График анализа образцов
Аналитическая эксклюзионная хроматография (SEC) выполнялась с применением колонки BioSEC (300A, 7,8×300 мм; πPN 5190-2511, Agillent), подключенной к системе Agilent 1100, и с применением рабочего буфера 0,15 M фосфата натрия (NaxHyPO4), pH 6,8 при скорости потока 1 мл/мин. Белки определяли путем измерения оптической плотности при A280 и A220. В колонку загружали 20 мкг каждого образца. SDS-PAGE выполняли с использованием гелей NuPAGE 4-12% Bis-Tris (InVitrogen NP0321BOX), рабочего буфера NuPAGE MES SDS (InVItrogen NP000202) в соответствии с инструкциями производителя. Образцы обрабатывали либо в восстанавливающих, либо в невосстанавливающих условиях. Полосы визуализировали с помощью красителя SimplyBlue (Invitrogen LC6065) и количественно оценивали с помощью денситометрического сканера (Oddyssey, Li-cor). В каждую лунку загружали 5 мкг каждого образца.
Nano-DSF (дифференциальную сканирующую флуориметрию) выполняли с помощью прибора Prometheus (NanoTemper), применяя температурный градиент от 20 до 95 °C с линейным изменением 1 градус в минуту. Регистрировали эмиссию флуоресценции при 300 нм и 350 нм, а также сигнал обратного рассеяния. Для каждой пробы загружали 10 мкл образца с концентрацией 5 мг/мл. Данные анализировали с помощью программного обеспечения PR.Stability analysis v1.02 от компании Nanotemper.
Динамическое рассеяние света выполняли с помощью Zetasizer Pro (Malvern). Рассеянный свет регистрировали и анализировали с помощью программного обеспечения ZS Explorer v 1.0.0.436 и встроенного алгоритма. Образцы анализировали при концентрации 5 мг/ мл.
Результаты
Эксклюзионная хроматография (SEC)
Хроматограммы получали для каждого образца в соответствии с графиком, представленным в Таблице 14. Было отмечено, что дополнительные пики, появляющиеся с течением времени, имели более низкий молекулярный вес, что свидетельствует о разложении материала. График общей интегрированной площади с течением времени продемонстрировал, что никакой материал не был потерян в предфильтре или на колонке.
Данные эксклюзионной хроматографии представлены на Фиг. 7. Если посмотреть на процент общей площади, состоящей из основного пика, можно наблюдать только небольшие изменения при + 4 °C. При + 40 °C эффекты были более выражены.
Наибольшее снижение наблюдалось для S-SL048-46 hIgG4 S228P и анти-NP hIgG1 LALA-PG. S-SL048-118 hIgG4 S228P, AS20 в hIgG4 S228P и AS20 hIgG1 LALA-PG продемонстрировали промежуточное снижение, в то время как CDR трансплантат практически не продемонстрировал снижения.
SDS PAGE
Образцы анализировали в восстанавливающих и невосстанавливающих условиях, как указано в Таблице 14. Образцы -80 °C не демонстрировали дополнительных полос, за исключением S-SL048-46 hIgG4 S228P и анти-NP hIgG1 LALA-PG, которые демонстрировали одну слабую полосу, каждая из которых составляла менее 1% в восстановленных условиях. Это было почти идентично тому, что наблюдалось для образцов дня 0. Фактически, образец дня 0 продемонстрировал дополнительные полосы, которые не наблюдались при температуре -80 °C, проанализированные на день 30, что отражает изменчивость анализа. Невосстановленные образцы были почти идентичны образцам дня 0. При + 4 °C на день 9 или день 30 в восстанавливающих или невосстанавливающих условиях не наблюдалось никакой дополнительной значительной полосы. При + 40 °C все образцы продемонстрировали дополнительные полосы, появляющиеся на день 9 и день 30. В восстанавливающих условиях все кандидаты продемонстрировали полосы с более низкой молекулярной массой, т.е. менее 25 кДа, тогда как AS20 hIgG4 S228P, AS20 hIgG1 LALA-PG и анти-NP hIgG1 LALA-PG продемонстрировали полосы с более высокой молекулярной массой (т.е. более 50 кДа на день 9 и на день 30). В обоих случаях эти полосы со временем стали более выраженными. В невосстанавливающих условиях можно было увидеть четкие различия с появлением дополнительных полос во всех образцах. Полученные результаты указывают на то, что кандидаты S-SL048-46 и S-SL048-106 могут быть более подвержены разложению.
Нано-DSF
Образцы анализировали в соответствии с графиком, приведенным в Таблице 14, и результаты представлены на Фиг. 8. Основным результатом наблюдений на основе данных нано-DSF были изменения, происходящие при применении формата AS20 hIgG4 S228P, AS20 hIgG1 LALA-PG и анти-NP hIgG1 LALA-PG, когда основные сдвиги температуры плавления Tm можно было наблюдать в условиях хранения при + 40 °C.
Для кандидатов основным результатом наблюдений была уменьшенная амплитуда второй Tm (данные не приведены), которая может отражать основной вклад Fab-фрагмента антитела. Кандидаты S-SL048-46 и S-SL048-106 продемонстрировали несколько большее изменение, а S-SL048-11, S-SL048-116 и S-SL048-118 продемонстрировали сходные, но несколько меньшие эффекты. Эти отличия считаются незначительными для всех кандидатов. CDR-трансплантат оказался наиболее стабильным из протестированных образцов.
Динамическое рассеяние света (DLS)
Образцы анализировали методом DLS на день 30. Результаты при трех различных температурах для каждого образца приведены на Фиг. 9. При -80 и +4 °C все образцы демонстрируют один пик с кажущимся диаметром около 10-11 нм, что соответствует ожидаемым результатам для мономерных антител. Ни в одном из образцов не наблюдали более крупных частиц, т.е. можно было обнаружить только молекулы белка.
При + 40 °C более крупные частицы отчетливо видны в разной степени. С учетом среднего значения Z и числа полидисперсности в качестве индикаторов однородности по размеру (данные не приведены), определено, что S-SL048-106 демонстрировал наименьшее количество частиц с более высокой Mw. Напротив, AS20 hIgG1 LALA-PG и анти-NP hIgG1 LALA-PG продемонстрировали наибольшее количество частиц с более высокой Mw, что указывает на то, что эти две молекулы были более склонны к агрегации.
Заключение
Таким образом, при объединении данных в общий рейтинг, наиболее стабильными кандидатами в формате hIgG4 S228P определены: S-SL048-106, S-SL048-118 и S-SL048-116 в указанном порядке.
Пример 13 - Картирование эпитопа AS20 с помощью HDX-MS
В этом примере масс-спектрометрию с водородно-дейтериевым обменом (HDX-MS) проводили на химерном антителе AS20 IgG4 вместе с BSSL человека для картирования его эпитопа.
Способы и материалы
40 мкл раствора 2 мг/мл BSSL человека (SEQ ID NO: 138) в PBS смешивали с 92,5 мкл 1,7 мг/мл химерного антитела AS20 hIgG4 (содержащего HC SEQ ID NO: 139 и LC SEQ ID NO: 140) для мольного соотношения 1:1. Комплекс BSSL/антитело концентрировали до 40 мкл с помощью установки центробежных фильтров 10K (Amicon Ultra, Merck). Параллельно аналогичным образом готовили образец, содержащий только BSSL, без добавления антител. Образцы анализировали в автоматизированной системе HDX-MS (CTC PAL/Biomotif HDX), в которой образцы автоматически помечали, гасили, расщепляли, очищали и разделяли при 2 °C. Более конкретно, образцы помечали путем смешивания 3 мкл BSSL (или комплекса BSSL/антитело) с 22 мкл дейтерированного PBS и инкубировали при 4 °C в течение четырех моментов времени мечения: 5 минут, 30 минут, 90 минут и 180 минут. Реакцию мечения останавливали/гасили снижением pH до ~2,3 и температуры до ~4 °C путем добавления 20 мкл раствора, содержащего 6 М мочевины, 100 мМ TCEP и 0,5% TFA. Образцы расщепляли на колонке с иммобилизованным пепсином (колонка 2,1 (2,1 × 30 мм) при 250 мкл/мин с последующей стадией обессоливания на линии с использованием преколонки C-18 с внутренним диаметром 2 мм и длиной × 10 мм (колонка ACE HPLC, Aberdeen , Великобритания) с применением 0,05% TFA при 350 мкл/мин в течение 3 минут. Затем пептиды разделяли 18-мин. 8-55% линейным градиентом ACN в 0,1% муравьиной кислоте с помощью аналитической колонки HALO C18/1,8 мкм с внутренним диаметром 2 мм и длиной × 50 мм, работающей при 95 мкл/мин. Для анализа применяли масс-спектрометр Orbitrap Q Exactive (Thermo Fisher Scientific), работающий с 70000 разрешением и m/z 400. Для идентификации пептидов применяли программное обеспечение Mascot, а для обработки всех данных HDX-MS применяли программное обеспечение HDExaminer (Sierra Analytics, США). Статистический анализ проводили с применением доверительного интервала 95%.
Результаты
За кинетикой дейтерирования 66 пептидов следили с помощью HDX-MS, покрывая 57,9% белка. Рассчитывали мечение дейтерием (5, 30, 90 и 180 мин) и дифференциальную кинетику поглощения дейтерирования между BSSL отдельно и в присутствии AS20. Несколько пептидов, близких к N-концу, продемонстрировали статистически более низкое поглощение дейтерия в присутствии антитела AS20 с четким картированием эпитопа в этой области. При рассмотрении перекрывающихся пептидов пространственное разрешение можно уменьшить, что приводит к появлению двух пептидных областей, продемонстрированных в Таблице 15.
Таблица 15 - Перечень областей эпитопа AS20, предполагаемый в эксперименте HDX-MS
Заключение
Таким образом, эпитоп AS20 был успешно картирован в N-концевой области BSSL. Был идентифицирован кластер из трех прерывистых пептидных областей со значительно более низким поглощением дейтерия в присутствии AS20. Эти области вместе картируются в трехмерной структуре BSSL, указывая на то, что эти пептиды вместе составляют конформационный эпитоп AS20. В Примере 21 авторы описывают кристаллическую структуру Fab-фрагмента AS20 в комплексе с hBSSL. Анализ трехмерной структуры подтверждает результаты данных HDX-MS и может конкретно определить аминокислоты, участвующие во взаимодействии.
Пример 14 - Картирование эпитопа ранее указанных антител-кандидатов с помощью HDX-MS
В этом примере была проведена масс-спектрометрия с водородно-дейтериевым обменом (HDX-MS) для картирования эпитопов BSSL пяти ранее указанных антител-кандидатов S-SL048-11 hIgG4 S228P (mAb11, HC SEQ ID NO: 119, LC SEQ ID NO: 120), S-SL048-46 hIgG4 S228P (mAb46, HC SEQ ID NO: 121, LC SEQ ID NO: 122), S-SL048-106 hIgG4 S228P (mAb106, HC SEQ ID NO: 123, LC SEQ ID NO: 124), S-SL048-116 hIgG4 S228P (mAb116, HC SEQ ID NO: 125, LC SEQ ID NO: 126) и S-SL048-118 hIgG4 S228P (mAb11,8 HC SEQ ID NO: 127, LC SEQ ID NO: 128).
Способы и материалы.
Раствор BSSL 2 мг/мл в PBS смешивали с различными антителами (в 5 мг/мл в 25 мМ гистидина, 150 мМ NaCl, 0,02% P80, pH 6,0) для молярного отношения 1:1. Образцы концентрировали и буфер заменяли на PBS с помощью установки центробежных фильтров 10K (Amicon Ultra, Merck). Параллельно образец, содержащий только BSSL, без добавления антител, подвергали той же процедуре.
Образцы анализировали в автоматизированной системе HDX-MS (CTC PAL/Biomotif HDX), в которой указанные образцы автоматически помечали, гасили, расщепляли, очищали и разделяли при 2 °C. Более конкретно, образцы помечали путем смешивания 3 мкл BSSL (или комплекса BSSL/антитело) с 22 мкл дейтерированного PBS и инкубировали при 4 °C в течение четырех моментов времени мечения: 5 минут, 30 минут, 90 минут и 180 минут. Реакцию мечения останавливали/гасили снижением pH до ~2,3 и температуры до ~4 °C путем добавления 20 мкл раствора, содержащего 2 М мочевины, 100 мМ TCEP и 0,5% TFA. Образцы расщепляли на колонке с иммобилизованным пепсином (колонка 2,1 (2,1 × 30 мм) при 250 мкл/мин с последующей стадией обессоливания на линии с использованием преколонки C-18 с внутренним диаметром 2 мм и длиной ×10 мм (колонка ACE HPLC, Aberdeen, Великобритания) с применением 0,05% TFA при 350 мкл/мин в течение 3 минут. Затем пептиды разделяли 15-мин. 8-60% линейным градиентом ACN в 0,1% муравьиной кислоте с помощью аналитической колонки HALO C18/1,8 мкм с внутренним диаметром 2 мм и длиной × 50 мм, работающей при 95 мкл/мин. Для анализа применяли масс-спектрометр Orbitrap Q Exactive (Thermo Fisher Scientific), работающий с 70000 разрешением и m/z 400. Для идентификации пептидов применяли программное обеспечение Mascot, а для обработки всех данных HDX-MS применяли программное обеспечение HDExaminer (Sierra Analytics, США). Статистический анализ проводили с применением доверительного интервала 95%.
Результаты
За кинетикой дейтерирования 54 пептидов следили с помощью HDX-MS, покрывая 64% белка. Рассчитывали мечение дейтерием (5, 30, 90 и 180 мин) и дифференциальную кинетику поглощения дейтерирования между только BSSL и в присутствии любого из пяти антител. Было замечено, что несколько пептидов около N-конца BSSL продемонстрировали статистически более низкое поглощение дейтерия в присутствии антитела для каждого из ранее указанных кандидатов. При рассмотрении перекрывающихся пептидов пространственное разрешение можно уменьшить, что приводит к появлению двух пептидных областей, продемонстрированных в Таблице 16.
Таблица 16 - Перечень наиболее частых областей эпитопа, предложенных в эксперименте HDX-MS
В дополнение к областям эпитопа, общим для всех ранее указанных антител, один дополнительный пептид был идентифицирован как имеющий статистически более низкое поглощение дейтерия, в трех из антител, а именно пептид 10 (аа 84-101; NIWVPQGRKQVSRDLPVM (SEQ ID NO: 4)) для S- SL048-46, пептид 24 для S-SL048-116 (аминокислотные остатки 174-180; VKRNIAA (SEQ ID NO: 5)) и пептид 39 (аминокислотные остатки 283-295; HYVGFVPVIDGDF (SEQ ID NO: 6)) для S-SL048-11 (Таблица 17). Однако сигналы их были относительно недельными.
Можно отметить, что две области эпитопа, общие для всех пяти антител (aa 1-12 и aa 42-55), хотя и рассеяны по первичной последовательности, группируются вместе в трехмерной структуре (Фиг. 10). Пептид 24 (174–180 аминокислотных остатков) расположен относительно близко к этой области ядра, тогда как пептиды 10 (aa 84-101) и 39 (aa 283-295) расположены более рассеяно.
Таблица 17 - Обобщенная информация по пептидам, имеющим дифференциальную кинетику поглощения дейтерирования между одной только BSSL и в присутствии любого из пяти антител
(SEQ ID NO:)
(148)
(4)
(5)
Заключение
Эпитопы пяти ранее указанных антител-кандидатов были успешно картированы в N-концевой области BSSL. Были идентифицированы две прерывистые пептидные области со значительно более низким поглощением дейтерия в присутствии всех пяти антител.
Перекрывающиеся пептиды могут способствовать снижению пространственного разрешения. Например, в эксперименте AS20 (Пример 13) было обнаружено, что пептид, соответствующий aa 1-6, не изменил дейтериевый обмен, тогда как более длинный перекрывающийся пептид действительно изменил указанный обмен (AKLGAVYTEGGF, aa 1-12, SEQ ID NO: 3). Другими словами, остатки эпитопа могут быть восстановлены до YTEGGF (aa 7-12) (SEQ ID NO: 1). В текущем примере, напротив, такое восстановление невозможно, поскольку пептид, соответствующий аминокислотным aa 1-6, не обнаружен.
В дополнение к двум общим областям эпитопа, каждый дополнительный пептид был идентифицирован как потенциальная область эпитопа в трех из ранее указанных кандидатов (S-SL048-46, S-SL048-116 и S-SL048-11). Несмотря на статистическую значимость, величина изменения сигнала этих пептидов была относительно низкой и может представлять меньшую важность для взаимодействия. Тем не менее, учитывая тот факт, что пептид VKRNIAA (aa 174-180, SEQ ID NO: 5) кластеризуется в центральную область в трехмерном пространстве, можно предполагать, что эта область также является частью эпитопа. Следует отметить, что этот пептид также перекрывается с одним пептидом, обнаруженным в исходном картировании AS20. Изменение сигнала еще двух периферических пептидов 10 (aa 84-101, SEQ ID NO: 4) и пептида 39 (aa 283-295, SEQ ID NO: 6) можно объяснить стабилизацией при связывании антител («аллостерические» эффекты).
В своей совокупности эти данные убедительно указывают на наличие общего ядра, состоящего из двух пептидных областей где-то в пределах последовательностей 1-12 (SEQ ID NO: 3) и 42-55 (SEQ ID NO: 2) для всех ранее указанных кандидатов. Эти области также были идентифицированы в предыдущих экспериментах по картированию эпитопов AS20, как описано в Примере 13, и, как видно, они являются важными для взаимодействия в кристаллической структуре комплекса AS20, описанного в Примере 21.
Пример 15 - Оценка иммуногенности
В этом примере описана оценка иммуногенности кандидатов SL048-11 (HC SEQ ID NO: 119 и LC SEQ ID NO: 120), S-SL048-46 (HC SEQ ID NO: 121 и LC SEQ ID NO: 122), S -SL048-106 (HC SEQ ID NO: 123 и HC SEQ ID NO: 124), S-SL048-116 (HC SEQ ID NO: 125 и LC SEQ ID NO: 126) и S-SL048-118 (HC SEQ ID NO: 127 и HC SEQ ID NO: 128) и AS20 hIgG4 S228P (HC SEQ ID NO: 129 и LC SEQ ID NO: 130), выполненная с помощью алгоритма Abzena.
Способы и материалы.
С помощью алгоритма прогнозирования iTope™ MHC класса II Abzena in silico, последовательности, предоставленные Lipum, анализировали на предмет потенциала иммуногенности. Программное обеспечение iTope™ прогнозирует полезные взаимодействия между аминокислотными боковыми цепями пептида и специфическими связывающими карманами (положения карманов; p1, p4, p6, p7 и p9) внутри открытых связывающих борозд 34 аллелей MHC класса II человека. Эти аллели представляют собой наиболее распространенные аллели HLA-DR, встречающиеся повсеместно, без оценки тех, которые наиболее часто встречаются в какой-либо этнической популяции. Двадцать аллелей содержат «открытую» конфигурацию p1, а 14 содержат «закрытую» конфигурацию, в которой глицин в положении 83 заменен на валин. Расположение ключевых связывающих остатков достигается in silico генерацией 9-мерных пептидов, которые перекрываются восемью аминокислотами, охватывающими последовательность тестируемого белка.
Результаты следует оценивать в свете того факта, что все способы прогнозирования связывания MHC класса II по своей природе дают завышенный прогноз количества эпитопов Т-клеток, поскольку они не учитывают другие важные процессы во время презентации антигена, такие как процессинг белка/пептида, распознавание рецептора Т-клеток или толерантность Т-клеток к пептиду.
Положение якорных остатков p1 (включающих первый остаток 9-мерного лиганда ядра MHC класса II) выделено в S-SL048-11, S-SL048-46, S-SL048-106, S-SL048-116, S-SL048-118 и AS20 в формате hIgG4 S228P.
Если ≥50% пептидов, связывающих MHC класса II (т.е. ≥17 из 34 аллелей), имели высокую аффинность связывания (показатель >0,6), такие пептиды определялись как пептиды, связывающие MHC класса II с «неизбирательной высокой аффинностью».
Пептиды, связывающие ≥50% аллелей MHC класса II с показателем >0,55 (но без связывания подавляющего большинства >0,6), определялись как пептиды с «неизбирательной умеренной аффинностью».
Эти критерии изменялись в случае, если большая ароматическая аминокислота (то есть F, W, Y) находилась в якорном положении p1, при этом открытый карман p1 у 20 из 34 аллелей обеспечивал возможность связываться с большим ароматическим остатком. Когда это происходит, неизбирательный пептид определяется как связывающийся с 10 или большим количеством из поднабора из 20 аллелей.
Также были проанализированы якорное положение p1 связывающих пептидов зародышевой линии с умеренной и высокой аффинностью, и при этим не ожидалось, что они будут проблематичными для здоровых индивидуумов из-за толерантности к Т-клеткам.
Положительные попадания iTope™ подвергали поиску методом BLAST в базе данных TCED™, содержащей известные положительные пептиды и при этом были указаны области, представляющие близкие гомологичные пептиды из базы данных Т-клеточных эпитопов (т.е. пептиды, которые, как известно, индуцируют активацию Т-клеток в анализе картирования Т-клеточных эпитопов ex vivo EpiScreen™). Для мечения последовательностей антител применяли нумерацию по Kabat.
Результаты
TCED™-анализ пептидов, ранее протестированных в Т-клеточных анализах EpiScreen™ ex vivo, выявил сильную гомологию с рядом пептидов, связывающих MHC iTope™ . В таблице 18, «+» указывает на несовпадающие остатки, где замены представляют собой аминокислоты со сходными физико-химическими свойствами, а «-» указывает на другие несовпадающие остатки. Положения ключевых положений карманов MHC класса II для последовательностей iTope™ и TCED™ приведены в нижнем ряду Таблицы 18.
Таблица 18 - Результаты анализа TCED™
В таблице 19 продемонстрировано общее количество смешанных пептидов iTope™, связывающих MHC класса II со средней и высокой аффинностью, и попаданий TCED™ для каждой кандидатной последовательности-(AS20 продемонстрирован только для информации).
Таблица 19 - Обобщенные результаты анализа иммуногенности
Все пять гуманизированных кандидатных клонов содержали меньше пептидов не зародышевой линии, неизбирательно связывающихся с МНС класса II, по сравнению с AS20 hIgG4 S228P. Последовательности всех вариантов содержали по меньшей мере два попадания TCED ™ с частичной (минимум 6 из 9 положений) гомологией с пептидами, которые, как известно, индуцируют Т-клеточную активацию в анализах EpiScreen™ ex vivo. Из пяти вариантов, S-SL048-106 hIgG4 S228P представлял самый низкий риск иммуногенности, на основе количества пептидов не зародышевой линии, неизбирательно связывающихся с МНС класса II с высокой и умеренной аффинностью. Кандидаты были ранжированы следующим образом: меньше неизбирательных попаданий, 106 < 46 < 11 = 118 < 116, больше неизбирательных попаданий.
Следует отметить, что анализ иммуногенности in silico по своей природе дает завышенный прогноз.
Пример 16 - Оценка выполнимости изготовления
В этом примере описана оценка выполнимости изготовления кандидатов in silico S-SL048-11 (HC SEQ ID NO: 119 и LC SEQ ID NO: 120), S-SL048-46 (HC SEQ ID NO: 121 и LC SEQ ID NO: 122), S-SL048-106 (HC SEQ ID NO: 123 и LC SEQ ID NO: 124), S-SL048-116 (HC SEQ ID NO: 125 и LC SEQ ID NO: 126) и S-SL048-118 (HC SEQ ID NO: 127 и LC SEQ ID NO: 128) в формате hIgG4 S228P, выполненная с помощью алгоритма Abzena.
Способы и материалы.
Аминокислотные последовательности S-SL048-11, S-SL048-46, S-SL048-106, S-SL048-116 и S-SL048-118 в формате hIgG4 S228P анализировали с помощью алгоритма прогнозирования предрасположенности Abzena in silico. Выявленные предрасположенности впоследствии анализировали в структурном контексте. Вкратце, для последовательности каждого домена V анализировали следующее:
• Наличие сайтов дезамидирования;
• Наличие сайтов изомеризации;
• Потенциальные сайты окисления;
• Наличие сайтов N-связанного гликозилирования
• Наличие свободных цистеинов.
В данном документе применяются определения и нумерация IMGT CDR, если не указано иное.
Наличие сайтов дезамидирования
Деамидирование остатков аспарагина может привести к структурным изменениям, изменениям фармакокинетики, эффективности и потенциальной иммуногенности. Остатки аспарагина как потенциальные сайты дезамидирования были проанализированы в контексте как амино-, так и карбокси- соседних аминокислот с помощью способа, основанного в [14].
Наличие сайтов изомеризации
Наличие сайтов изомеризации аспартата прогнозировали путем анализа последовательности известных мотивов изомеризации (DG, DS, DT или DD), фокусирующихся в областях CDR.
Потенциальные сайты окисления
Создавали структурные модели вариабельных доменов как тяжелой, так и легкой цепи, и идентифицировали и оценивали остатки метионина и триптофана с целью определение того, могут ли подвергаться их поверхность воздействию и, следовательно, являются ли они кандидатами на окисление. Окисление отдельных остатков может влиять на биологическую активность антител и может иметь биологические последствия, например, снижение эффективности или изменение фармакокинетики.
Наличие сайтов N-связанного гликозилирования
Последовательности VH и Vκ анализировали на основе консенсусного N-связанного мотива гликозилирования: - N-X-S/T -, где X может быть любой аминокислотой, кроме пролина.
Наличие свободных цистеинов
Последовательности анализировали для выявления неспаренных остатков цистеина. Каждый из доменов VH и Vκ обычно содержит два канонических цистеина, которые образуют внутрицепочечную дисульфидную связь в свернутой молекуле. Ожидается, что дополнительные цистеины будут негативно влиять на сворачивание и потенциально могут вызывать такие проблемы, как агрегация.
Результаты
Был проведен анализ, и не было идентифицировано никаких потенциальных сайтов N-связанного гликозилирования ни в последовательности VH, ни в последовательности Vκ для пяти кандидатных антител; непарные цистеины не были идентифицированы ни в последовательности VH, ни в последовательности Vκ для пяти кандидатных антител; не было идентифицировано потенциальных сайтов окисления ни в последовательности VH, ни в последовательности Vκ для пяти кандидатных антител, а также не было идентифицировано сайтов с высоким (t1/2<10 дней) или средним (t1/2<25 дней) потенциалом дезамидирования ни в последовательности VH, ни в последовательности Vκ для любого из пяти кандидатных антител.
В пределах VH для всех пяти клонов в областях CDR или близко к ним были идентифицированы два потенциальных сайта изомеризации.
- Asp 54 (DG) расположен внутри VH CDR2, поэтому изомеризация может влиять на связывание антигена;
- Asp72 расположен рядом с CDR в области, иногда называемой «CDR4», и поэтому изомеризация может влиять на связывание антигена.
Таблица 20 - Обобщенная информация об предрасположенности последовательности
- Asp 54 (VH CDR2) и Asp 72 (FW2, примыкающий к области CDR)
- Asp 54 (VH CDR2) и Asp 72 (FW2, примыкающий к области CDR)
Один потенциальный сайт дезамидирования был идентифицирован в Asn 60 в VH CDR2.
- Asp 54 (VH CDR2) и Asp 72 (FW2, примыкающий к области CDR)
Один потенциальный сайт дезамидирования был идентифицирован в Asn 60 в VH CDR2.
- Asp 54 (VH CDR2) и Asp 72 (FW2, примыкающий к области CDR)
- Asp 54 (VH CDR2) и Asp 72 (FW2, примыкающий к области CDR)
На Фигуре 11 продемонстрирован S-SL048-106 Fv с выделенными потенциальными посттрансляционными предрасположенностями.
Таким образом, ни в одном из пяти основных кандидатов не было выявлено свободных цистеинов, сайтов окисления или сайтов N-связанного гликозилирования. Сайты с высоким потенциалом дезамидирования не были идентифицированы в S-SL048-11, S-SL048-116 или S-SL048-118. S-SL048-46 и S-SL048-106 содержат потенциальный сайт дезамидирования в VH CDR2. Два потенциальных сайта изомеризации были идентифицированы в тяжелой цепи каждого из пяти основных вариантов.
Пример 17 - Характеризация продуцирования и связывания AS20 hIgG1 LALA-PG и анти-NP hIgG1 LALA-PG
В этом примере представлены характеристики продуцирования и связывания AS20 и антител анти-NP (клон B1-8) подкласса человеческого IgG1-LALA-PG, в дальнейшем называемого AS20 hIgG1 LALA-PG (содержащего HC SEQ ID NO: 115 и LC SEQ ID NO: 116) и анти-NP hIgG1 LALA-PG (содержащего HC SEQ ID NO: 117 и LC SEQ ID NO: 118). Антитело анти-NP применяли в качестве изотипического контроля.
Материал и способы
Экспрессия и очистка
IgG4 является наиболее Fc инертным естественным подклассом человека. Однако несколько публикаций продемонстрировали, что IgG4 может взаимодействовать с FcR, а также с комплементом как у мышей [15], так и у людей. Таким образом, IgG1 человека с мутациями в трех положениях, а именно L234A, L235A и P329G (hIgG1 LALA-PG для краткости), был выбран в этом исследовании, поскольку, как сообщается, он является наиболее Fc «молчащим» из доступных вариантов [7, 16], т.е. не имеющий иммунных эффекторных функций.
Для изготовления материала была привлечена компания Absolute Antibody (Оксфорд, Великобритания). Информацию о последовательностях VH и VL AS20 отправляли в компанию, где синтезировали гены и клонировали их в векторы, кодирующие подкласс IgG1-LALA-PG человека. Антитела временно экспрессировали в клетках HEK293 млекопитающих и затем очищали аффинной хроматографией с применением протеина A. Чистоту и целостность оценивали с помощью SDS-PAGE, а уровни эндотоксина определяли с помощью хромогенного анализа эндотоксина LAL.
Поверхностный плазмонный резонанс (SPR)
Кинетику одного цикла использовали для измерения аффинности антител. AS20 hIgG1 LALA-PG и анти-NP hIgG1 LALA-PG иммобилизовали на сенсорном чипе CM5 S посредством связывания первичным амином с использованием химического состава NHS-EDC. На поверхность вводили пять различных концентраций антигена (разведения 1:3, начиная с 50 нМ для hBSSL и разведения 1:2, начиная с 800 нМ для mBSSL). Поверхность сенсорного чипа регенерировали 10 мМ глицин-HCl pH 2,1. Полученные кинетические данные одного цикла подгоняли к модели связывания Ленгмюра 1:1, и получали кинетические параметры с помощью программного обеспечения BIAevaluation. Для BSSL мыши, анализ стационарного состояния также выполняли путем построения графика зависимости уровня ответа в равновесии по отношению к каждой концентрации, и значения KD получали с помощью программного обеспечения BIAevalution.
Результаты
Успешно продуцировали АS20 hIgG1 LALA-PG и анти-NP hIgG1 LALA-PG (данные не продемонстрированы).
Кинетику SPR одного цикла использовали для определения аффинности IgG. Для связывания AS20 hIgG1 LALA-PG с hBSSL рассчитывали равновесную константу диссоциации (KD), которая составила 0,91 нМ. Для mBSSL было определено, что KD составляет 361 нМ. Как и ожидалось, связывания анти-NP hIgG1 LALA-PG с BSSL не наблюдалось.
Заключение
В этом Примере описана проверка качества, выполняемая для верификации того, что AS20 hIgG1 LALA-PG является функциональным, т.е. что связывание с BSSL сравнимо со связыванием AS20 в формате hIgG4 S228P.
SPR-анализ LALA-PG AS20 hIgG1 продемонстрировал, что связывание с BSSL было очень сходным с тем, которое наблюдается для AS20 в других форматах IgG. KD для hBSSL определяли с помощью SPR до 0,91 нМ, что сопоставимо со значениями KD 0,6-3 нМ, которые были определены для других форматов IgG AS20 (как описано в Примерах 1, 2 и 4). Аффинность связывания AS20 hIgG1 LALA-PG с mBSSL оценивали в анализе аффинности в стационарном состоянии до значения KD 361 нМ, которое находится в том же порядке величины, что и KD, ранее оцененное с помощью того же анализа (155 нМ, Пример 1), а также внешний вид сенсограмм был аналогичным (не продемонстрированы).
Таким образом, полученные данные продемонстрировали, что на связывание AS20 hIgG1 LALA-PG не повлияло изменение подкласса. Полученные результаты продемонстрировали, что контрольное изотипическое антитело анти-NP hIgG1 LALA-PG не связывается с BSSL и, следовательно, может быть пригодно для применения в качестве отрицательного контроля в будущих анализах.
Пример 18 - Подтверждение эффективности AS20 hIgG1 LALA-PG в мышиной модели ревматоидного артрита
В этом Примере, эффект AS20 hIgG1 LALA-PG (тяжелая цепь SEQ ID NO: 115 и легкая цепь SEQ ID NO: 116) исследовали в мышиной модели ревматоидного артрита (RA) in vivo, то есть артрита, индуцированного антителами к коллагену (CAIA). Антитело анти-NP hIgG1 LALA-PG (тяжелая цепь SEQ ID NO: 117 и легкая цепь SEQ ID NO: 118) было включено в исследование в качестве изотипического контроля.
Описание модели - CAIA у мышей
Артрит, индуцированный антителами к коллагену (CAIA) у мышей, представляет собой модель артрита, независимую как от В-, так и от Т-клеток. Заболевание индуцировали введением антител к коллагену типа II (CII) внутривенно (в/в) с последующим внутрибрюшинным (в/бр) введением LPS через 3-5 дней для ускорения развития заболевания. Введенные антитела связываются с хрящом, тем самым активируя иммунную систему и привлекая макрофаги и гранулоциты к суставам. Для достижения значительной степени тяжести и частоты развития заболевания требуется бустерная инъекция LPS. Течение болезни очень предсказуемо и начинается после бустерной дозы LPS. Заболевание достигает максимальной степени тяжести примерно ко дню 15, после чего его тяжесть уменьшается до полного выздоровления. Исследование было одобрено местным комитетом по этике животных Мальме/Лунд, Швеция (M118-15).
Материал и способы
Индукция заболевания
Мышам DBA/1 (самцы, 8-9 недель) в день 0 вводили внутривенно смесь моноклональных антител анти-CII в дозе 2 мг/мышь (CIA-MAB-50, MD Bioproducts). В 5 день мышам внутрибрюшинно вводили LPS (50 мкг/мышь) для усиления заболевания.
Экспериментальные группы и ведение исследуемых продуктов
AS20 hIgG1 LALA-PG и антитела изотипического контроля доставляли в концентрации 5 мг/мл и дополнительно разбавляли носителем (25 мМ гистидин, 150 мМ NaCl, 0,02% P80, pH 6,0). Исследуемые продукты (антитела) вводили внутрибрюшинно каждые 4 дня , начиная за день до индукции заболевания (день -1), а затем на 3, 7, 11 и 15 день. AS20 hIgG1 LALA-PG вводили в трех различных дозах, то есть 10, 30 и 90 мг/кг, а изотипический контроль (анти-NP hIgG1 LALA-PG) в дозе 90 мг/кг, исходя из среднего веса животных в день -2. Относительно высокие дозы были выбраны для компенсации низкой аффинности AS20 hIgG1 LALA-PG к BSSL мыши по сравнению с BSSL человека, как описано в Примере 17. Экспериментальные группы представлены в Таблице 21. Первое введение в день -1 представляло собой болюсную дозу, т. е. все исследуемые продукты вводили в виде двойных доз, разделенных на две инъекции, первую инъекцию выполняли утром, а вторую инъекцию - во второй половине дня. Следующие введения (день 3, 7, 11 и 15) осуществляли в виде однократных доз. Животных взвешивали перед каждым введением, и объем дозы 20 мл/кг определяли исходя из индивидуального веса животных.
Таблица 21 - Экспериментальные группы
hIgG1 LALA-PG
hIgG1 LALA-PG
hIgG1 LALA-PG
*Два сателлитных животных на группу.
Оценка заболевания
Заболевание оценивали ежедневно, начиная со дня 3, заслепленным методом с использованием макроскопической балльной оценки для четырех конечностей в диапазоне от 0 до 15 (1 балл за каждый опухший или гиперемированный палец лапы, 1 балл за опухший или гиперемированный средний палец или сустав стопы, 5 баллов за опухшую лодыжку) с максимальным количеством баллов 60 для каждой мыши. Из-за этических ограничений животные с баллом выше 45 были исключены из эксперимента.
Забор крови
Кровь собирали у всех мышей на день прекращения исследования 19 путем пункции сердца под глубокой анестезией (96 часов после последней инъекции на день 15) в микропробирки, содержащие LiHeparin. Образцы немедленно помещали на лед и центрифугировали (2000 ×g в течение 5 минут, при 4 °C) в течение 20 минут после отбора. Образцы плазмы разделяли на аликвоты и замораживали на сухом льду. Аликвоты хранили при -80°C до анализа воздействия AS20 IgG1 LALA-PG и антител против лекарственных средств (ADA) (все группы, n = 70), а также анализа панели биомаркеров безопасности (группы 1-3, n = 42).
Заборы крови для изучения PK у сателлитных животных
С целью получения информации о PK-профиле и ADA, в каждую группу дозирования животных, получавших антитела, были включены два сателлитных животных. Образцы крови брали из подъязычной вены за 1 час до введения в день 7 и через 24 часа после введения (т.е. в день 8) (по два животных на группу дозирования). Образцы крови также отбирали за 1 час до введения дозы в день 15 и через 24 часа после введения (т.е. в день 16) (по два животных на группу дозирования).
Забор ткани
При прекращении исследования у всех животных вырезали и взвешивали селезенки. 14 селезенок из группы, получавшей изотипический контроль (группа 2), и от 14 животных в группе 90 мг/кг AS20 hIgG1 LALA-PG (группа 3) гомогенизировали и анализировали с помощью FACS, как описано в Примере 19.
Экспозиция препарата в плазме
Экспозицию AS20 IgG1 LALA-PG анализировали в образцах плазмы, собранных у сателлитных животных (см. выше) и у всех животных при прекращении исследования (день 19) с помощью жидкостной хроматографии-тандемной масс-спектрометрии (LC-MS/MS).
Антитела против лекарственных средств (ADA)
Оценку иммуногенности проводили путем анализа ADA в образцах плазмы, собранных у сателлитных животных (см. выше) и у всех животных при прекращении исследования (день 19).
ADA измеряли с помощью иммуноферментного анализа (ELISA). Вкратце, планшеты maxisorp покрывали AS20 hIgG1 LALA-20 или антителами изотипического контроля и блокировали 5% BSA, 0,05% Tween-20 в 1× PBS. Добавляли образцы плазмы или мышиную плазму с введенными антителами положительного контроля, и инкубировали планшеты в течение 2 часов при комнатной температуре. Планшеты промывали, добавляли вторичные антитела к пероксидазе AffiniPure козьи анти-мышиные IgG + IgM (H + L) (Jackson ImmunoResearch) и после инкубации в течение 1 ч при комнатной температуре планшеты тщательно промывали и, наконец, добавляли субстрат TMD (Sigma-Aldrich), применяемый для обнаружения.
Измерение биомаркеров безопасности
Образцы плазмы, собранные в группе 1 (контроль с носителем), группе 2 (изотипический контроль, 90 мг/кг) и группе 3 (AS20 hIgG1 LALA-PG, 90 мг/кг), анализировали с помощью способов клинической биохимии на 14 биомаркеров безопасности (альбумин, аланинаминотрансфераза, щелочная фосфатаза, амилаза, билирубин, азот мочевины крови, кальций, креатинин, глобулин, глюкоза, фосфат, калий, натрий и общий белок). Анализы выполняли в отделении химической и фармацевтической безопасности, RISE, Седертелье, Швеция, с применением системы Abaxis Vetscan с кассетой № 500-0038.
Результаты
Степень тяжести артрита
У животных c индуцированным CAIA и получавших носитель, развилось заболевание от умеренной до тяжелой степени со 100% частотой. Сходные результаты наблюдались у животных, получавших изотипический контроль. Эффективность AS20 hIgG1 LALA-PG, вводимого внутрибрюшинно каждый четвертый день от дня -1 до прекращения лечения оценивали в трех дозах (10, 30 и 90 мг/кг). AS20 hIgG1 LALA-PG в дозе 90 мг/кг и 30 мг/кг продемонстрировал значительный положительный эффект на течение заболевания по сравнению с изотипическим контролем в дни 7-14, 16, 18 и 7-12 соответственно. Небольшое снижение тяжести заболевания наблюдалось при введении AS20 hIgG1 LALA-PG в дозе 90 мг/кг при сравнении с носителем, хотя и не было статистически значимым (Фиг. 18 и 19).
Два животных, одно в группе AS20 hIgG1 LALA-PG 10 мг/кг и одно в группе изотипического контроля были исключены из исследования досрочно (День 12) из этических соображений (высокий балл). Эти животные были включены в анализ максимального балла, но исключены из анализа среднего значения, AUC и % ингибирования (Фиг. 19, таблица 22).
Статистика параметров заболевания представлена в Таблице 22.
Таблица 22 - Статистика параметра тяжести CAIA
1Кумулятивная частота. Считалось, что мышь заболела, если в течение двух дней подряд набирала балл 1 или выше.
2Средний балл CAIA для всех оцениваемых моментов времени.
3Площадь под кривой.
4Процентное изменение общего бремени болезни у каждого животного по сравнению с группой, получавшей изотипический контроль. Рассчитано путем определения разницы между средним значением AUC группы, получавшей изотипический контроль, и AUC для каждого отдельного животного, деленным на AUC группы, получавшей изотипический контроль, и умноженным на 100 *(-1). * р <0,05; ** p <0,01 по сравнению с изотипическим контролем.
Оценка состояния здоровья
Общую оценку состояния здоровья проводили ежедневно вместе с оценкой заболевания со дня 3 до прекращения исследования. Мышей взвешивали два раза в неделю в ходе общей оценки состояния здоровья. У животных не наблюдалось нежелательных эффектов в ходе лечения (данные не приведены).
Экспозиция препарата в плазме
Экспозиция в плазме AS20 hIgG1 LALA-PG в конце исследования в день 19 в целом находилась в ожидаемом диапазоне концентраций, на основании предыдущей фармакокинетической оценки однократной дозы. Средняя концентрация в группе, получавшей дозу 10 мг/кг, составляла 199 мкг/мл (~1,3 мкМ, 58 - 309 мкг/мл), в группе, получавшей дозу 30 мг/кг – до 614 мкг/мл (~4,1 мкМ, 229 - 970 мкг/мл), а в группе, получавшей дозу 90 мг/мл, соответствующая средняя концентрация составляла 1408 мкг/мл (~9,4 мкМ, 520 - 2557 мкг/мл). Таким образом, наблюдалась несколько нелинейная зависимость дозы от воздействия. Признаков изменения экспозиции в плазме в ходе лечения из-за иммуногенности обнаружено не было. Средняя концентрация hIgG1 LALA-PG в образцах плазмы мышей, получавших изотипический контроль, составила 1815 (~12 мкМ, 1224-2511 мкг/мл).
Антитела против лекарственных средств (ADA)
В исследуемых образцах не было обнаружено ADA, вызывающих обеспокоенность. Хотя большинство образцов продемонстрировали положительный результат, титры в группах AS20 hIgG1 LALA-PG не были выше, чем в контрольных группах. Принимая во внимание раннее начало ответа (~ день 7) и неизменное экспозицию лекарственного средства в динамике, возможно, что обнаруженный сигнал против лекарственного средства в образцах был вызван в основном неспецифическими/низкоаффинными IgM.
Биомаркеры безопасности
Уровень глюкозы в плазме увеличивался после введения как AS20 hIgG1 LALA-PG, так и антитела изотипического контроля. При применении высокой дозы AS20 hIgG1 LALA-PG не повышался ни один из биомаркеров поражения печени. Наблюдалась тенденция к повышению креатинина в плазме в группе, получавшей AS20 hIgG1 LALA-PG, по сравнению с группой, получавшей изотипический контроль, хотя и не была статистически значимой (p<0,06). Другие маркеры поражения почек, то есть мочевина крови, электролиты или общий белок, не отличались между мышами, получавшими AS20 hIgG1 LALA-PG и изотипический контроль.
Заключение
У животных c индуцированным CAIA и получавших изотипический контроль, развивалось заболевание от умеренной до тяжелой степени со 100% частотой. Такие же результаты наблюдались у мышей, которым вводили носитель.
Введение AS20 hIgG1 LALA-PG в дозе 90 мг/кг и 30 мг/кг продемонстрировало значительный положительный эффект на течение заболевания по сравнению с изотипическим контролем в дни 7-14, 16-18 и 7-12 соответственно. Небольшое снижение тяжести заболевания наблюдалось при введении AS20 hIgG1 LALA-PG в дозе 90 мг/кг при сравнении с носителем, хотя и не было статистически значимым.
Экспозиция в плазме AS20 IgG1 LALA-PG в конце исследования в целом определялась в ожидаемом диапазоне концентраций. Признаков изменения экспозиции в плазме в ходе лечения из-за иммуногенности обнаружено не было. В исследуемых образцах не было обнаружено ADA, вызывающих обеспокоенность.
При применении высокой дозы AS20 IgG1 LALA-PG не повышался ни один из биомаркеров поражения печени. Наблюдалась тенденция к повышению креатинина в плазме в группе, получавшей AS20 hIgG1 LALA-PG, хотя и не была статистически значимой. Ни один из других маркеров поражения почек, то есть мочевина в крови, электролиты или общий белок, не отличались между AS20 hIgG1 LAL.
Пример 19 - FACS-анализ селезенки из эксперимента CAIA
В этом Примере эффект AS20 hIgG1 LALA-PG (тяжелая цепь SEQ ID NO: 115 и легкая цепь SEQ ID NO: 116) на клеточные субпопуляции в селезенке мышей с артритом, индуцированным антителами к коллагену (CAIA), исследовали с помощью сортировки клеток с активацией флуоресценции (FACS). Антитело анти-NP hIgG1 LALA-PG (тяжелая цепь SEQ ID NO: 117 и легкая цепь SEQ ID NO: 118) было включено в исследование в качестве изотипического контроля.
Материал и способы
Это исследование является частью экспериментального исследования in vivo, описанного выше (Пример 18). Вкратце, CAIA индуцировали у мышей DBA/1 (самцы, 8-9 недель) путем внутривенного (в/в) введения антител к коллагену типа II с последующим внутрибрюшинным (в/бр) введением LPS для ускорения развития заболевания. Мышам в/бр вводили AS20 hIgG1 LALA-PG или антител изотипического контроля (анти-NP hIgG1 LALA-PG) каждые 4 дня, начиная за день до индукции заболевания (день -1). При прекращении исследования (день 19) селезенки животных, получавших наивысшую дозу AS20 hIgG1 LALA-PG (90 мг/кг) или изотипический контроль (90 мг/кг), вырезали, взвешивали и гомогенизировали, как описано ниже. Для идентификации Т-клеток, В-клеток, NKT-клеток, нейтрофилов, эозинофилов, NK-клеток, дендритных клеток, моноцитов и макрофагов была разработана панель FACS.
Суспензии отдельных клеток
Селезенки пропускали через 70-μм сетчатый фильтр в культуральной среде RPMI-1640 (Thermo Fisher, HyClone) для получения суспензий отдельных клеток. Клетки осаждали и ресуспендировали в 1 мл воды MilliQ для лизирования эритроцитов (10 сек), добавляли 1 мл 2×BS, а затем 10 мл 1× PBS. Клетки промывали 10 мл HBSS (Thermo Fisher, HyClone) и, наконец, ресуспендировали в соответствующем объеме HBSS, чтобы получить 10×106 клеток/мл.
Протокол FACS
Клетки в количестве 1×106 высевали в каждую лунку 96-луночного планшета круглым дном лунок и центрифугировали в течение 1 мин при 850×g, 4°C, промывали буфером FACS (1× PBS, 3% FBS, 2 мМ EDTA) и снова осаждали. Очищенные крысиные антимышиные CD16/CD32 (блок Fc), разведенные 1:50 в буфере FACS, добавляли в каждую лунку и инкубировали клетки в течение 15 минут, после чего снова промывали буфером FACS. Смесь антител готовили путем добавления следующих антител к соответствующему объему буфера FACS:
FITC хомяковый антимышиный CD3ε (1:100)
PE крысиный антимышиный CD86 (1:100)
PE-CF594 крысиный антимышиный Ly-6C (1:50)
PE-Cy7 крысиный антимышиный Ly-6G (1:200)
Биотиновый хомяковый антимышиный CD49b (1:200)
APC-Cy7 хомяковый антимышиный CD11c (1:50)
Alexa Fluor 647 крысиный антимышиный I-A/I-E (MHCII) (1:200)
Alexa Fluor 700 мышиный антимышиный CD45.2 (1:100)
BV421 крысиный антимышиный Siglec-F (1:200)
BV605 крысиный анти-CD11b (1:200)
BV650 крысиный антимышиный CD19 (1:100)
BV786 хомяковый антимышиный CD80 (1:200)
eF506 Восстановимый краситель для определения жизнеспособности (1:400).
К клеткам в каждую лунку добавляли 25 μл смеси антител и инкубировали в течение 20 мин на льду в защищенном от света месте. Клетки промывали буфером FACS, добавляли 25 μл смеси вторичных антител (PerCP-Cy5.5 стрептавидин, разведенный 1: 200 в буфере FACS) и инкубировали клетки в течение 20 мин на льду в защищенном от света месте. Затем клетки дважды промывали буфером FACS, ресуспендировали в 250 μл буфера FACS, переносили в пробирки для FACS и, наконец, анализировали на платформе проточного цитометра CytoFLEX (Beckman Coulter).
Стратегия гейтирования
Подмножества клеток были идентифицированы следующим образом:
T-клетки: CD45.2+, CD11b-, CD3+
B-клетки: CD45.2+, CD11b-, CD19+
NKT-клетки: CD45.2+, CD11b-, CD3+, CD49b+
Нейтрофилы: CD45.2+, CD11b+, Ly6G+
Эозинофилы: CD45.2+, CD11b+, Ly6G-, Siglec F+
NK-клетки: CD45.2+, CD11b+, Ly6G-, Siglec F-, CD49b+
Дендритные клетки: CD45.2+, CD11b+, Ly6G-, Siglec F-, CD11c+, MHCII+
Моноциты: CD45.2+, CD11b+, Ly6G-, Siglec F-, CD11C-, Ly6C+
Макрофаги: CD45.2+, CD11b+, Ly6G-, Siglec F-, CD11C-, Ly6C-, MHCII+
Кроме того, анализировали экспрессию CD80 и CD86 на дендритных клетках.
Результаты
Не было значимой разницы в массе тела между группой, получавшей AS20 hIgG1 LALA-PG (группа 3, Пример 18), и группой, получавшей изотипический контроль (группа 2, Пример 18), ни за два дня до индукции заболевания (AS20, 23,9 ± 1,4 г; изотипический контроль, 23,8 ± 1,2 г), ни на день 19 прекращения исследования (AS20, 22,5 ± 1,7 г; изотипический контроль, 22,1 ± 1,0 г). Однако вес селезенки был значительно выше в группе, получавшей AS20 hIgG1 LALA-PG, по сравнению с группой, получавшей изотипический контроль (110 ± 23 мг против 88 ± 21 мг; p = 0,02), а общее количество лейкоцитов CD45+ также было выше в группе AS20 по сравнению с группой, получавшей изотипический контроль (44,5×106 против 39,0×106; p = 0,008).
Общее количество В-клеток в селезенке было выше у мышей, получавших AS20 hIgG1 LALA-PG, по сравнению с мышами, получавшими изотипический контроль (27,1 ± 3,5 × 106 против 21,0 ± 5,8×106; p = 0,004). Напротив, общее количество NK-клеток было ниже у мышей, получавших AS20 hIgG1 LALA-PG, по сравнению с мышами, получавшими изотипический контроль (0,44 ± 0,09×106 против 0,54 ± 0,09×106; p = 0,01). Общее количество NKT-клеток было также ниже у мышей, получавших AS20 hIgG1 LALA-PG, по сравнению с мышами, получавшими изотипический контроль, хотя и без статистической значимости (0,14 ± 0,03×106 против 0,16 ± 0,03 × 106; p = 0,08). Не было обнаружено значимой разницы в общем количестве нейтрофилов, эозинофилов, моноцитов, макрофагов, дендритных клеток или Т-клеток между мышами, получавшими AS20 IgG1 LALA-PG, и мышами, получавшими изотипический контроль (Фиг. 20).
Доля В-клеток из CD45+ клеток в селезенке была выше у мышей, получавших AS20 IgG1 LALA-PG, по сравнению с мышами, получавшими изотипический контроль (60,9 ± 5,4% против 53,0 ± 10,0%, p = 0,02). Напротив, доля Т-клеток, NKT-клеток и NK-клеток из CD45+ клеток была ниже у мышей, получавших AS20 hIgG1 LALA-PG, по сравнению с мышами, получавшими изотипический контроль (Т-клетки: 15,2 ± 3,3% против 18,9 ± 4,1%, p = 0,02; NKT-клетки: 0,31 ± 0,05% против 0,40 ± 0,07%, p = 0,0003; NK-клетки: 0,99 ± 0,22% против 1,4 ± 0,23%, p = 0,0001). Не было обнаружено значимой разницы в доле нейтрофилов, эозинофилов, моноцитов, макрофагов или дендритных клеток из лейкоцитов CD45+ между мышами, получавшими AS20 IgG1 LALA-PG, и мышами, получавшими изотипический контроль (Фиг. 21). Не было значимой разницы между мышами, получавшими AS20 IgG1 LALA-PG, и мышами, получавшими изотипический контроль, в клетках, экспрессирующих CD80 или CD86, ни по общему количеству, ни по процентному соотношению CD45+ клеток (данные не приведены).
Заключение
Применение AS20 hIgG1 LALA-PG (доза 90 мг/кг) значимо снижало общее количество NK-клеток и долю T-клеток, NKT-клеток и NK-клеток из CD45+ в селезенке мышей с CAIA.
Пример 20 - Анализ влияния ранее указанных антител-кандидатов на активность фермента BSSL
В этом примере изучали влияние пяти ранее указанных антител-кандидатов, а именно S-SL048-11 (тяжелая цепь SEQ ID NO: 119 и легкая цепь SEQ ID NO: 120), S-SL048-46 (тяжелая цепь SEQ ID NO: 121 и легкая цепь SEQ ID NO: 122), S-SL048-106 (тяжелая цепь SEQ ID NO: 123 и легкая цепь SEQ ID NO: 124), S-SL048-116 (тяжелая цепь SEQ ID NO: 125 и легкая цепь SEQ ID NO: 126) и S-SL048-118 (тяжелая цепь SEQ ID NO: 127 и легкая цепь SEQ ID NO: 128) в формате hIgG4 S228P, на ферментативную активность BSSL. Сравнения проводили с AS20 CDR трансплантатом (тяжелая цепь SEQ ID NO: 131 и легкая цепь SEQ ID NO: 132), химерным AS20 (тяжелая цепь SEQ ID NO: 129 и легкая цепь SEQ ID NO: 130) и антителом анти-NP изотипического контроля (тяжелая цепь SEQ ID NO: 133 и легкая цепь SEQ ID NO: 134) в формате hIgG4 S228P.
Способы и материалы
Преинкубация BSSL с ранее указанными антителами-кандидатами
В этом примере применяли нативную BSSL человека (SEQ ID NO: 138; Таблица 2). Основной раствор BSSL разбавляли 10× в MilliQ (MQ)-H2O до концентрации 0,42 мг/мл.
Антитела разбавляли до исходной концентрации 0,3 μг/μл в MQ-H2O,с последующим серийным разведением 1:1 в MQ-H2O, в результате чего получали 6-точечные концентрации в диапазоне от 0,3 μг/μл до 0,009 μг/μл. Из каждого разведения антител 20 μл добавляли к 2,4 μл BSSL (1μг), и смеси антитело/BSSL инкубировали при + +4°C в течение 1 часа. После инкубации к каждой реакции BSSL/антитело добавляли 20 μл MQ-H2O
Анализ гидролиза триглицеридов
Эмульсию триглицеридов (TG) получали путем смешивания 25 мг немеченого триолеина (Sigma, № по каталогу 92860) с 50 мкл 3H-меченного триолеина (триолеин [9,10-3H (N)], 91 Ки/ммоль) NET431001MC Perkin Elmer (Уолтем, штат Массачусетс) в стеклянном сосуде с круглым дном диаметром 30 мм, пригодном для обработки ультразвуком; выпаривания растворителя в атмосфере азота (N2) при комнатной температуре; добавления в сосуд 1,0 мл 10% гуммиарабика (Sigma№ по каталогу G-9752), 1,25 мл 1,0 M трис-HCl pH 9,0 и 2,0 мл MQ-H2O; охлаждения сосуда в ледяной воде и обработки ультразвуком в течение 10 мин в режиме 50% -ного импульса с помощью Soniprep 150 (MSE, Великобритания) с зондом с плоским наконечником диаметром 9 мм при средних настройках, помещенным на несколько мм ниже поверхности жидкости до получения эмульсии; и добавления 2,5 мл 18,7% BSA (Sigma, № по каталогу A7906), 2,5 мл 1,0 M NaCl и 3,25 мл MQ-H2O к указанной эмульсии. Эмульсию использовали в тот же день, когда она была приготовлена. Затем 10 мкл предварительно инкубированного раствора BSSL/антитело (см. выше) смешивали с 150 мкл эмульсии TG и 10 мМ холата натрия (Sigma, № по каталогу C-1254) и MQ-H2O в общем объеме 200 мкл в стеклянных пробирках 13×100 мм. Образцы готовили в двух повторностях. Пробирки инкубировали при 37°C в течение 15 минут, а затем реакции останавливали добавлением 3,25 мл метанола/хлороформа/гептана (объем/объем/объем, 760/680/540) и 1,0 мл 0,1 М карбоната натрия, pH 10,5, с последующим центрифугирование при 3500× g в течение 10 мин. Из верхней водорастворимой фазы, содержащей гидролизованные свободные жирные кислоты, извлекали 1400 мкл и смешивали с 2,0 мл сцинтилляционного смеси Optiphase Hisafe 3 (Perkin Elmer, Уолтем, штат Массачусетс) в 6 мл полиэтиленовых флаконах (Perkin Elmer) и количество гидролизованных 3H-меченных свободных жирных кислот измеряли с помощью жидкостной сцинтилляционной спектрометрии на приборе WinSpectral 1414 (Wallac, Турку, Финляндия).
Анализ гидролиза сложного эфира холестерина
Эмульсию сложного эфира холестерина (CE) готовили путем добавления 40 мкл 14C-меченного холестерилолеата (олеат-1-14C, NEC6380050UC, Perkin Elmer) в круглодонный стеклянный сосуд диаметром 30 мм, пригодный для обработки ультразвуком и выпаривания растворителя в атмосфере азота при комнатной температуре; добавления в сосуд 2,0 мл 0,2 M трис-HCl pH 7,5 и 0,85 мл MQ-H2O; охлаждения сосуда в ледяной воде и обработки ультразвуком в течение 10 мин в режиме 50%-ного импульса с помощью Soniprep 150 (MSE) с зондом с плоским наконечником диаметром 9 мм при средних настройках, помещенным на несколько мм ниже поверхности жидкости до получения эмульсии; и добавления к эмульсии 1,65 мл 200 мМ холата натрия и 1,0 мл MQ-H2O. Эмульсию использовали в тот же день, когда она была приготовлена. Затем 10 мкл предварительно инкубированного раствора BSSL/антитело (см. выше) смешивали с 100 мкл эмульсии CE и MQ-H2O в общем объеме 200 мкл в стеклянных пробирках 13 × 100 мм. Образцы готовили в двух повторностях. Пробирки инкубировали при 37°C в течение 30 минут, а затем реакцию останавливали добавлением 3,25 мл метанола/хлороформа/гептана (объем/объем/объем, 760/680/540) и 1,0 мл 0,1 М карбоната натрия, pH 10,5, с последующим центрифугированием при 3500 ×g в течение 10 минут. Из верхней водорастворимой фазы, содержащей гидролизованные свободные жирные кислоты, извлекали 400 мкл и смешивали с 2,0 мл сцинтилляционной смеси Optiphase Hisafe 3 (Perkin Elmer) в 6 мл полиэтиленовых флаконах (Perkin Elmer), и количество гидролизованных 14C-меченных свободных жирных кислот измеряли с помощью жидкостной сцинтилляционной спектрометрии на приборе WinSpectral 1414 (Wallac).
Результаты
Ферментативную активность оценивали путем измерения высвобождения свободных жирных кислот (радиоактивно меченных) после 15 мин инкубации (анализ гидролиза триглицеридов) или 30 мин инкубации (анализ гидролиза сложного эфира холестерина), соответственно, см. Фигуру 17. По техническим причинам образцы должны были быть проанализированы в двух последовательных наборах, но два антитела (AS20 и антитело изотипического контроля анти-NP) были включены в оба набора. Данные выражены в виде относительных значений, причем значение, полученное без добавления антител в реакцию, установлено на 100% (Таблицы 23 и 24).
Таблица 23 - Влияние пяти BSSL-специфичных hIgG4 S228P и контрольных антител подтипа hIgG4 S228P на ферментативную активность BSSL (гидролиз триглицеридов)
(относительные значения; значения, полученные без антител в реакции, принимают за 100%)
Таблица 24 - Влияние пяти BSSL-специфичных hIgG4 S228P и контрольных антител подтипа hIgG4 S228P на ферментативную активность BSSL (гидролиз сложных эфиров холестерина)
Заключение
Ни одно из протестированных антител, то есть пять ранее указанных лекарственных препаратов-кандидатов, AS20, AS20 CDR трансплантат и анти-NP в формате hIgG4 S228P, не проявили какого-либо значительного влияния на ферментативную активность BSSL.
Пример 21 - Картирование эпитопа AS20 с помощью рентгеновской кристаллографии
Рентгеновскую кристаллографию применяли для определения трехмерной структуры AS20 в комплексе с hBSSL. Для этого эксперимента антитело расщепляли с образованием Fab-фрагментов, и создавали новую конструкцию hBSSL (t-hBSSL), усеченную на С-конце, соответствующую аминокислотам 1-530 hBSSL, за которыми следует AHHHHHH (SEQ ID NO: 146).
Материал и способы
Клонирование, экспрессия и очистка t-hBSSL
BSSL человека ранее кристаллизовали в усеченной форме без гибкой С-концевой части. Таким образом, для получения кристаллов AS20 Fab и hBSSL была создана новая усеченная конструкция BSSL, включающая C-концевую метку his для очистки. Конструкцию gp67-BSSL-6×H состояла из аминокислот 1-530 + AHHHHHH (нумерация BSSL на основе последовательности после удаления сигнального пептида), заказывали в GeneArt и клонировали в вектор pFastBac tGFP Dual, который был подготовлен для клонирования, независимого от лигирования (LIC). LIC выполняли с помощью набора для клонирования InFusion и трансформировали в компетентные клетки Stellar.
Рекомбинантную бакмидную ДНК генерировали в клетках DH10Bac E. coli. Трансфекцию бакмиды в клетки Sf9 проводили в течение 120 ч при 27 °C. Вирус P1 собирали из питательной среды и впоследствии применяли для генерирования исходного материала вируса P2. Через 96 ч исходный материал вируса Р2 собирали с помощью центрифугирования. 400 мл клеток Sf9 (1,5 × 106 клеток/мл) инфицировали 2 мл исходного материала вируса P2 и выращивали в течение 72 часов после инфицирования. Среду (исходный материал P3) собирали с помощью центрифугирования и фильтровали. Для крупномасштабной экспрессии 35 мл исходного материала вируса P3 добавляли к 2130 мл клеток Sf9 (1,6 × 106 клеток/ мл) в колбу Thomson Optimum Growth Flask (5 л). Экспрессию проводили при 27 °C в течение 72 часов. При сборе плотность клеток составляла 2,34×106 клеток/мл с 75%-ой экспрессией GFP и 89%-ой жизнеспособностью. Культуру центрифугировали для удаления клеток. t-hBSSL очищали из среды с помощью периодической IMAC с применением смолы Ni Sepharose Fast Flow и элюировали с 50 мМ Трис pH 7,5, 500 мМ NaCl, 500 мМ имидазолом. Дополнительную очистку проводили с помощью эксклюзионной хроматографии на колонке Superdex 200 16/60 в 50 мМ Hepes pH 7,0, 500 мМ NaCl. Белок элюировался в виде единственного мономерного пика, который объединяли и концентрировали.
Комплексное образование Fab-фрагмента AS20 и t-hBSSL
Производство антитела AS20 осуществляли в компании Absolute Antibody (Оксфорд, Великобритания). Fab-фрагменты получали, следуя протоколу расщепления папаина от поставщика иммобилизованного папаина (Thermo Scientific, продукт № 20341). AS20, всего 15 мг, концентрировали до 17 мг/мл и буфер заменяли на 20 мМ фосфат натрия, 10 мМ EDTA, pH 7, 20 мМ цистеин. Антитело инкубировали с иммобилизованным папаином (0,6 мл суспензии) при 37 °C в течение 3 часов с последующей инкубацией при 4 °C в течение ночи. Поскольку расщепление не было полным, образец инкубировали при 37 °C в течение дополнительных 3 часов, а затем при комнатной температуре в течение выходных. Антитело элюировали 10 мМ Трис, pH 7,5, и добавляли очищенную t-hBSSL. Смесь инкубировали в течение около 30 мин при комнатной температуре, после чего ее загружали на колонку Superdex 200 в 50 мМ Hepes pH 7,0, 500 мМ NaCl. Первый основной пик при объеме элюирования около 60 мл содержал комплекс t-hBSSL и Fab-фрагмента AS20. Релевантные фракции концентрировали (10K MWCO) и концентрацию соли снижали до 250 мМ путем разбавления 50 мМ буфером Hepes pH 7,0.
Кристаллизация AS20 Fab в комплексе с t-hBSSL и структурирование раствора
После очистки комплекса Fab t-hBSSL образец выдерживали на льду при 4°C в течение примерно 36 часов при транспортировке между лабораториями. Затем комплекс концентрировали с применением 500 мкл Vivaspin с 30 000 MWCO (Vivaspin 500 от Sartorius, VS0122) от 4 мг/мл до конечной концентрации 17,3 мг/мл. Концентрацию измеряли при A280 на Nanodrop с применением коэффициента экстинкции 174 375 и Mw 106,242 кДа.
Эксперименты по кристаллизации проводили с помощью робота для обработки жидкостей Mosquito в 3-луночных планшетах Swiss CI XTAL SD-3 с 35 мкл резервуарного раствора и каплями 150 + 50, 100 + 100, 50 + 150 нл белка + резервуар. Экран Morpheus от Molecular Dimensions давал кристаллы при 20 градусах в условиях B9 и B10. Кристаллы стержневидной морфологии появлялись в первые 12 ч и увеличивались в течение следующих 24 ч. После быстрого переноса в резервуарный раствор, содержащий дополнительно 10% глицерина, их подвергали криоохлаждению, погружая в жидкий азот. Данные собирали в канале синхротронного излучения Biomax в MaxIV. Файл с автоматически обработанными данными использовали для решения структуры путем молекулярной замены в Phaser с 1f6w.pdb (hBSSL) и 4n0y.pdb (Fab-фрагмент) в качестве моделей поиска. Молекулы воды удаляли, как и самую С-концевую часть BSSL, а для 4n0y.pdb сохраняли только цепи Fab, превращенные в модель полиаланина. Ручное построение модели выполняли в Coot, и оптимизировали модель с помощью Refmac 5 (все для комплекта CCP4).
Результаты
Кристаллизация и определение структуры AS20 Fab t-hBSSL
Fab-фрагмент AS20 может быть кристаллизован вместе с t-hBSSL в условиях Morpheus B9, предусматривающих наличие 30 мМ NaBr, 30 мМ NaFl, 30 мМ NaI, 0,1 М трис (основание), 0,1 М бицин (буфер установлен на pH 8,5 с этими двумя буферами), 20% PEG550 MME и 10% PEG 20K. Кристаллы принадлежали к пространственной группе C2 с параметрами элементарной ячейки 323, 67, 123, 90, 101,7, 90 и дифрагировались до 2,5 Å. Структура была сформирована путем молекулярного замещения, и в асимметричной единице присутствовали два полных комплекса. Кристаллическая упаковка вокруг двух копий различается, что приводит к небольшим вариациям структур t-hBSSL. Особенно последние остатки, включая метку 6-His, видны в молекуле A, но не в молекуле B из-за того, что B имеет меньше контактов с кристаллами в этой области и больше места для гибкости C-конца. Область между остатками 272 и 283 также сильно разупорядочена в B и не может быть смоделирована. Помимо этих несоответствий, две модели t-hBSSL очень хорошо накладываются друг на друга.
Анализ поверхности взаимодействия AS20 Fab и t-hBSSL
Структура BSSL была описана как имеющая большую сердцевинную область, состоящую из скрученного 11-цепочечного бета-листа, окруженного альфа-спиралями и соединительными петлями [9]. На N-конце имеется 3-цепочечный бета-лист меньшего размера. Структуру можно сравнить с перчаткой для левой руки, в которой ладонь содержит триаду активного центра рядом с «большим пальцем». При таком сходстве небольшой N-концевой бета-лист расположен на тыльной стороне руки рядом с «мизинцем», см. Фиг. 12. Часть структуры BSSL, которая взаимодействует с молекулой Fab, расположена на небольшом N-концевом бета-листе и в C-концевой части альфа-C [17], третьей альфа-спирали в структуре. Другими словами, область связывания антитела находится не рядом с активным сайтом, а на противоположной стороне антигена. На Фиг. 13 продемонстрировано, как вариабельные цепи AS20 связываются с t-hBSSL с последовательностью эпитопа, выделенной светло-серым цветом.
Области эпитопа перечислены в Таблице 25 и содержат остатки 7-12 (цепи 1 и 2, SEQ ID NO: 1), 42-55 (область петли, ведущая в цепь 3 листа, часть SEQ ID NO: 2), и 174-180 (С-конец альфа-С, SEQ ID NO: 5). Эпитоп является довольно плоским, с выступающими только несколькими характерными остатками, а именно Tyr7, Phe12 и Gln52 (основные взаимодействия, перечисленные в Таблице 245). Область петли 47-54 хорошо определена и образует однородную поверхность. Пролин 47 важен для стэкинг-взаимодействия с Tyr31 Fab, но в целом поверхность здесь плоская. Многие остатки в последовательности эпитопа важны для сворачивания BSSL, но не взаимодействуют специфическим образом с AS20.
Таблица 25 - Перечень остатков BSSL, участвующих во взаимодействии между BSSL и Fab AS20, ранжированных с наиболее важными взаимодействиями в верхнем ряду. Также перечислены остатки BSSL, которые важны для сворачивания BSSL в области эпитопа.
P47, Q52, T54
R176
P47, H48, P49, G50, W51, Q52, G53, T54, K56
W173, V174, K175, R176, N177, A180
Q46, P47, H48, P49, G50, W51, Q52, G53, T54, L55, K56
W173, V174, K175, R176, N177, I178, A179, A180
Q46, P47, H48, P49, G50, W51, Q52, G53, T54, L55, K56
H168, M169, I171, A172, W173, V174, K175, R176, N177, I178, A179, A180
Заключение
В этом Примере описывается кристаллизация и структурирование раствора AS20 Fab и t-hBSSL. Структура демонстрирует, что область эпитопа расположена в той же области, что и ранее идентифицированная с помощью HDX-MS, описанная в Примере 13. Это трехмерный эпитоп, состоящий из небольшого бета-листа, хорошо упорядоченной области петли, ведущей к третьей цепи листа, и С-концевой части соседней спирали. Последовательности эпитопа следующие: 7-12 (YTEGGF, SEQ ID NO: 1), 42-55 (LENPQPHPGWQGTL, SEQ ID NO: 2) и 174-180 (VKRNIAA, SEQ ID NO: 5); распространяются в последовательности, но сближаются в структуре. Наиболее определяющими остатками являются Tyr7, Phe12 и Gln52, которые выступают над уровнем поверхности.
Последовательности пяти ранен указанных антител анализировали в контексте структуры AS20 Fab t-hBSSL. Различия в последовательностях, которые в основном являются консервативными, и результаты картирования посредством HDX-MS (Пример 14) демонстрируют, что все пять антител будут связывать один и тот же эпитоп на BSSL.
Пример 22 - Комплексообразование, кристаллизация, определение структуры и анализ эпитопа комплекса S-SL048-116 Fab-BSSL
Материалы и способы
Расщепление S-SL048-116 с помощью FabRICATOR
Расщепляли антитело S-SL048-116 (тяжелая цепь SEQ ID NO: 125 и легкая цепь SEQ ID NO: 126) в PBS и очищали F(ab’)2 с применением набора FragIT (Genovis) в соответствии с инструкциями производителя.
Восстановление F(ab’)2 - фрагмента и очистка Fab’-фрагмента
Крупномасштабное восстановление очищенного F(ab’)2 -фрагмента проводили с применением цистеамина в конечной концентрации 50 мМ при комнатной температуре в течение 2 часов в PBS, pH 7,2, содержащем 5 мМ EDTA. Четыре пятых полученного образца очищали на приборе HiLoad 26/60 Superdex 200 (GE Healthcare) с PBS pH 7,2, 2 мМ EDTA в качестве подвижной фазы. Фракции из пика, представляющего интерес, объединяли, концентрировали и хранили при -80 °C для последующих применений. Общий выход составил 7,2 мг.
Алкилирование Fab’-фрагмента и последующая очистка
Оставшуюся пятую часть восстановленного образца обрабатывали равным объемом 375 мМ йодацетамида при комнатной температуре в течение 30 минут, чтобы заблокировать свободные цистеины путем алкилирования. Полученный алкилированный образец очищали точно так же, как неалкилированный образец. Общий выход составил 2 мг.
Экспрессия и очистка BSSL
Исходный материал вируса P2, применяемый для экспрессии, получали от компании SciLifeLab (Стокгольм, Швеция). Клеточную линию насекомых ExpiSf9 применяли в среде ExpiSf CD. Экспрессию с применением набора для экспрессии белка ExpiSf (ThermoFisher Scientific) проводили в соответствии с рекомендациями производителя с использованием вирусной нагрузки, рекомендованной SciLife Lab.
К супернатанту культуры от экспрессии 2 л добавляли таблетки ингибитора протеазы, не содержащие EDTA (Merck Millipore), NiSO4 и имидазол (конечная концентрация 15 мМ). Любые частицы удаляли центрифугированием при 13000 ×g. Супернатант загружали в колонку HisTrap Excel на 5 мл (GE Healthcare) на ночь при 4 °C со скоростью потока 2 мл/мин. Колонку промывали 6 объемами колонки (CV) IMAC-буфера A (50 мМ трис-HCl, 500 мМ NaCl; pH 7,5), содержащим 15 мМ имидазол. Колонку снова промывали 10 CV IMAC-буфера A, содержащего 25 мМ имидазол. Связанный белок элюировали линейным градиентом 20 CV до 100% IMAC-буфера B (50 мМ трис-HCl, 500 мМ NaCl, 0,5 М имидазол, pH 7,5). Элюированный белок концентрировали, центрифугировали и прогоняли на колонке для гель-фильтрации HiLoad 26/600 Superdex 200 (GE Healthcare), предварительно уравновешенной буфером SEC (50 мМ HEPES, 500 мМ NaCl, 2 мМ EDTA; pH 7). Фракции из пика, представляющего интерес (1E6-1F8), объединяли и концентрировали. Общий выход составлял 6,2 мг, а чистота образца составляла около 85%.
Образование и очистка комплекса BSSL:Fab’
Перед генерирвоанием комплекса, BSSL оценивали с помощью SEC на полупрепаративной колонке HiLoad 26/600 Superdex 200 prep grade, чтобы установить отсутствие олигомеризации, вызванной концентрацией/хранением. Комплекс смешивали при молярном соотношении 1:1,3 BSSL:Fab’ и инкубировали на льду в течение 1 часа. Инкубированный комплекс запускали на той же колонке SEC, применяя тот же буфер, что и для BSSL. Представляющие интерес фракции (B3-C4) объединяли и заменяли буфер на 50 мМ HEPES, 250 мМ NaCl, 2 мМ EDTA; pH 7. Образец с замененным буфером концентрировали до 17 мг/мл, быстро замораживали в жидком азоте и хранили при -80 °C для кристаллизации. Чистота образца составляла > 90% согласно анализу SDS-PAGE.
Образование и очистка комплекса BSSL:Alk-Fab’
Комплекс смешивали при молярном соотношении 1,1:1 BSSL:Alk-Fab’ и инкубировали на льду в течение одного часа. Инкубированный комплекс запускали на той же колонке SEC, применяя тот же буфер, что и для комплекса BSSL:Fab’. Представляющие интерес фракции объединяли и заменяли буфер на 50 мМ HEPES, 250 мМ NaCl, 2 мМ EDTA; pH 7. Образец с замененным буфером концентрировали до 14,5 мг/мл, быстро замораживали в жидком азоте и хранили при -80 °C для кристаллизации.
Кристаллизация и замораживание комплекса Fab-BSSL
Лучше всего дифрагирующие кристаллы выращивали при 20 °C из 14,5 мг/мл комплекса Fab-BSSL в буфере (50 мМ HEPES, 250 мМ NaCl, 2 мМ EDTA, pH 7,0) и смешивали с резервуаром (16% (мас./об.) PEG 4000, 0,1 М цитрат натрия pH 5 и 6% (об./об.) этанол) и затравочный раствор, как описано ниже:
150 нл комплекса + 37 нл затравки (измельченные кристаллы в 0,1 М цитрате натрия pH 5,0, 20% (мас./об.) PEG 4000 и 0,2 М сульфата аммония) + 113 нл резервуара
Сидящую каплю пипеткой наносили на планшет MRC с резервуаром 40 мкл. Кристаллы появлялись в течение нескольких дней и замораживались в криорастворе, содержащем 20% (об./об.) глицерина, 16% (мас./об.) PEG 4000, 0,1 М цитрата натрия, pH 5,0 и 3% (об./об.) этанол.
Сбор данных
Набор данных собирали при 100 К на станции BioMAX, MAX IV, Лунд, Швеция (λ = 0,97625 Å), оснащенной гибридным пиксельным детектором Eiger 16M. Набор данных собирали с временем экспозиции 0,011 с и колебанием 0,1° на изображение, всего 360 °. Данные обрабатывали с помощью программного конвейера autoPROC до 2,5 Å в пространственной группе P21.
Результаты
Определение структуры
Структуру Fab-BSSL определяли с применением молекулярной замены с помощью программного обеспечения Phaser со структурой 2,3 Å каталитического домена липазы BSSL, активированной солями желчных кислот человека (из PDB: 1F6W) и гомологичной структурой Fab 2,86 Å (из PDB: 3NFP) в качестве матриц. Один комплекс обнаруживали в асимметричном блоке. Структура была доработана в Refmac5, затем в Buster, а построение модели было выполнено в Coot. Окончательная модель включала три белковые цепи с BSSL (цепь A), а также тяжелую цепь (H) и легкую цепь (L) Fab S-SL048-116 (Фиг. 22). Окончательная модель включала аминокислоты 1 - 531 в цепи A, за исключением гибкой петли, не видимой в электронной плотности (аминокислоты 117 - 123). Модель включала аминокислоты 1 - 227 в цепи H и аминокислоты 1-211 в цепи L. Первая аминокислота в цепи H, Gln 1, была смоделирована как PCA, пироглутаминовая кислота, чтобы наилучшим образом соответствовать электронной плотности. В цепи H, два свободных цистеина (Cys 133 и Cys 225) были смоделированы как неалкилированные цистеины, хотя обработка S-SL048-116 Fab была необходима для кристаллизации. Никакой дополнительной плотности для метиламидной группы, предположительно присоединенной к атому серы цистеина, не было. Вероятно, остатки цистеина были, по меньшей мере, частично алкилированы, но присоединенные атомы были настолько гибкими, что они не были четко видны на карте электронной плотности. Кроме того, смоделировано 87 молекул воды.
Анализ эпитопа
Анализ эпитопа проводили с использованием координат комплекса Fab-BSSL. Анализ выполняли с помощью программного обеспечения CONTACT в составе пакета программ CCP4. S-SL048-116-Fab связывается с BSSL как через тяжелую, так и через легкую цепи. В тяжелой цепи задействованы все три петли CDR (CDR1, CDR2 и CDR3), тогда как в легкой цепи только CDR1 и CDR3 контактируют с BSSL (Фиг. 23, Таблица 26). Также Ile 2 близко к N-концу легкой цепи обеспечивает гидрофобное ван-дер-ваальсовое взаимодействие с BSSL. Сеть водородных связей стабилизирует взаимодействие между тяжелой цепью и BSSL (всего 7 водородных связей) и BSSL и легкой цепью (всего 5 водородных связей), рассчитанное с помощью анализа PISA (QT). Анализ PISA (QT) демонстрирует, что 426 Å2 доступной для растворителя области скрыты на границе контакта между BSSL и тяжелой цепью, а 468 Å2 скрыты на границе контакта BSSL и легкой цепью.
Таблица 26 - Обобщенные данные всех взаимодействующих остатков между S-SL048-116-Fab и BSSL между 0 и 4 Å
Заключение
Результаты, полученные в данном Примере для S-SCL048-116, согласуются с результатами, полученными в исследовании с анализом HDX-MS в Примере 14.
Пример 23 - Подтверждение эффективности S-SL048-116 в мышиной модели ревматоидного артрита
В этом Примере, эффект S-SL048-116 (тяжелая цепь SEQ ID NO: 125 и легкая цепь SEQ ID NO: 126) исследовали в мышиной модели ревматоидного артрита (RA) in vivo, то есть артрита, индуцированного антителами к коллагену (CAIA). Модель CAIA описана в Примере 18. Исследование было одобрено местным комитетом по этике животных Мальме/Лунд, Швеция (02896-20).
Материал и способы
Индукция заболевания
Мышам DBA/1 (самцы, 8 недель) в день 0 вводили внутривенно смесь моноклональных антител анти-CII в дозе 2 мг/мышь (CIA-MAB-50, MD Bioproducts). В 5 день мышам внутрибрюшинно вводили LPS (50 мкг/мышь) для усиления заболевания.
Экспериментальные группы и ведение исследуемых продуктов
S-SL048-116 доставляли в концентрации 5 мг/мл и дополнительно разбавляли носителем (25 мМ гистидин, 150 мМ NaCl, 0,02% P80, pH 6,0). Исследуемые продукты (антитела и носитель) вводили внутрибрюшинно каждые 4 дня, начиная за день до индукции заболевания (день -1), а затем на 3, 7, 11 и 15 день. S-SL048-116 вводили в трех разных дозах, т.е. 10, 30 и 90 мг/кг, исходя из среднего веса животных в день -2. Относительно высокие дозы были выбраны для компенсации низкой аффинности S-SL048-116 к BSSL мыши (KD = 40 нМ) по сравнению с BSSL человека (KD = 0,5 нМ), как описано в Примере 11. Экспериментальные группы представлены в Таблице 27. Первое введение в день -1 представляло собой болюсную дозу, т. е. исследуемые продукты вводили в виде двойных доз, разделенных на две инъекции, первую инъекцию выполняли утром, а вторую инъекцию - во второй половине дня. Следующие введения (день 3, 7, 11 и 15) осуществляли в виде однократных доз. Животных взвешивали перед каждым введением, и объем дозы 20 мл/кг определяли исходя из индивидуального веса животных.
Таблица 27 - Экспериментальные группы
Оценка заболевания
Заболевание оценивали ежедневно, начиная со дня 3, заслепленным методом с использованием макроскопической балльной оценки для четырех конечностей в диапазоне от 0 до 15 (1 балл за каждый опухший или гиперемированный палец лапы, 1 балл за опухший или гиперемированный средний палец или сустав стопы, 5 баллов за опухшую лодыжку) с максимальным количеством баллов 60 для каждой мыши. Из-за этических ограничений животные с баллом выше 45 были исключены из эксперимента.
Результаты
Степень тяжести артрита
У животных c индуцированным CAIA и получавших носитель, развилось заболевание от умеренной до тяжелой степени со 100% частотой. Эффективность S-SL048-116 (SOL-116), вводимого внутрибрюшинно каждый четвертый день от дня -1 до прекращения лечения оценивали в трех дозах (10, 30 и 90 мг/кг). S-SL048-116, вводимый в дозе 90 мг/кг, продемонстрировал положительный эффект относительно снижения тяжести заболевания по сравнению с носителем (Фиг. 24 и 25).
Два животных были исключены из исследования досрочно из этических соображений (высокий балл), одно – в группе носителя (день 15) и одно – в группе 90 мг/кг (день 12). Эти животные были включены в анализ максимального балла, но исключены из анализа среднего значения, AUC и % ингибирования (Фиг. 25, Таблица 28). Одна мышь в группе носителя не восстановилась после бустерной дозы LPS, т.е. была гипотермической, имела кифотическую позу и очень низкий вес. Эту мышь удалили из эксперементальной группы до начала заболевания (день 7) и поэтому ее данные исключили из всех результатов.
Статистика параметров заболевания представлена в Таблице 28.
Таблица 28 - Статистика параметра тяжести CAIA
1 Кумулятивная частота. Считалось, что мышь заболела, если в течение двух дней подряд набирала балл 1 или выше.
2 Средний балл CAIA для всех оцениваемых моментов времени.
3 Площадь под кривой.
4 Процентное изменение общего бремени болезни у каждого животного по сравнению с группой, получавшей контрольный носитель. Рассчитано путем определения разницы между средним значением AUC группы, получавшей контрольный носитель, и AUC для каждого отдельного животного, деленным на AUC группы, получавшей контрольный носитель, и умноженным на 100 *(-1).
Оценка состояния здоровья
Общую оценку состояния здоровья проводили ежедневно вместе с оценкой заболевания со дня 3 до прекращения исследования. Мышей взвешивали два раза в неделю в ходе общей оценки состояния здоровья. У животных не наблюдалось нежелательных эффектов в ходе лечения.
Заключение
У животных c индуцированным CAIA и получавших носитель, развивалось заболевание от умеренной до тяжелой степени со 100% частотой. Оценивали эффективность S-SL048-116 в трех различных дозах, то есть в дозах 10, 30 и 90 мг/кг внутрибрюшинно, с начальным днем лечения -1. S-SL048-116, вводимый в дозе 90 мг/кг, продемонстрировал положительный эффект на снижение тяжести заболевания по сравнению с носителем. У животных не наблюдалось нежелательных эффектов в ходе лечения с применением S-SL048-116 или только носителя.
Пример 24 - Анализ клеточности в крови, селезенке и мезентериальных лимфатических узлах мышей с нокаутом BSSL (KO) и мышей дикого типа (WT)
В этом примере количество различных субпопуляций лейкоцитов в крови, селезенке и брыжеечных лимфатических узлах (MLN), собранных от мышей с нокаутом BSSL (KO) и однопометных мышей дикого типа исследовали с помощью сортировки клеток с активацией флуоресценции (FACS).
Материал и способы
Лейкоциты выделяли для анализа из крови, селезенки и MLN от 10 мышей BSSL KO и 10 однопометных мышей дикого типа (15-19 недель). Общее количество лейкоцитов определяли для селезенки и MLN с помощью ручного подсчета в камере Burcher. Клетки, выделенные из крови, селезенки и MLN, инкубировали с FC-блоком (CD16/C032 клон 2.4G2) с последующим применением двух различных смесей антител, Окрашивания A и Окрашивания B.
Окрашивание A
Антитела/клоны/ флуорохромы, использованные для Окрашивания A, были следующими: CD19 (ID3/BB515}, γδTCR (GL3/PE), CD8a (53-6.7/PerCP-Cy5.5), CD45.2 (104/PE-Cy7), NK1.×(PK136/APC), CD4 (GK1.5/APC-H7), TCRβ (H57-597/BV421) и восстановимый краситель для определения жизнеспособности (FVD) (Horizon 510).
Популяции клеток, идентифицированные окрашиванием A, были следующие:
αβ T-клетки: CD45.2+, TCRβ+
CD4 αβ T-клетки: CD45.2+, TCRβ+, CD4+
CD8 αβ T-клетки: CD45.2+, TCRβ+, CD8+
γδ T-клетки: CD45.2+, TCRγδ+
NKT-клетки: CD45.2+, TCRβ+, NK1.1+
B-клетки: CD45.2+, CD19+
NK-клетки: CD45.2+, TCRβ-, TCRγδ-, NK1.1+
Окрашивание В
Антитела/клоны/флуорохромы, использованные для Окрашивания В, были следующими: MHC II (MS/114.15.2/Alexa488), FcεRlα (MAR-1/PE), Siglec F (E50-2440/PE-CF594), Ly6G (1A8/PerCP-Cy5.5), CD45.2 (104/PE-Cy7), Ly6C (AL-21/APC), CD11b (M1/70/APC-Cy7), CD117 (2B8/BV421) и восстановимый краситель для определения жизнеспособности (FVD) (Horizon 510).
Популяции клеток, идентифицированные окрашиванием B, были следующие:
Миелоидные клетки: CD45.2+, CD11b+
Нейтрофилы: CD45.2+, CD11b+, Ly6G+
Эозинофилы: CD45.2+, CD11b+, Ly6G-, Siglec F+, SSCвыс
Моноциты: CD45.2+, CD11b+, Ly6G-, Siglec F-, SSCнизк, Ly6C+, MHCII-
Макрофаги: CD45.2+, CD11b+, Ly6G-, Siglec F-, SSCнизк, Ly6C-, MHCII+
Тучные клетки: CD45.2+, CD11b+, FcεRlα+, CD117+
Базофилы: CD45.2+, CD11b+, FcεRlα+, CD117-
После инкубации (20 мин на льду) клетки промывали, ресуспендировали в буфере FACS, и с помощью FACS идентифицировали различные клеточные субпопуляции.
Результаты
Общее количество лейкоцитов в селезенке и MLN
Было обнаружено, что количество лейкоцитов в селезенке мышей KO значительно снизилось примерно до 50% от количества лейкоцитов у мышей WT. Значимой разницы в количестве лейкоцитов в MLN между мышами KO и WT не наблюдалось (Фиг. 26).
αβТ-клетки
Было обнаружено, что общее количество αβТ-клеток в селезенке было снижено у мышей KO по сравнению с мышами WT. Снижение было аналогично тому, которое наблюдалось для общего количества лейкоцитов CD45+. Значимой разницы в количестве αβ Т-клеток в MLN не наблюдалось. Было обнаружено, что процент αβТ-клеток от общего количества CD45+ клеток был выше в селезенке и крови мышей KO по сравнению с мышами WT, но без наблюдаемой разницы в MLN. В селезенке соотношение CD4+/CD8+ было выше у мышей KO по сравнению с мышами WT.
γδТ-клетки
Было обнаружено, что общее количество γδТ-клеток в селезенке было снижено у мышей KO по сравнению с мышами WT. Снижение было аналогично тому, которое наблюдалось для общего количества лейкоцитов CD45+. В MLN это снижение было даже более выражено, чем в селезенке. Доля γδТ-клеток из CD45+ клеток в MLN также была ниже у мышей KO по сравнению с мышами WT. В селезенке и крови такой разницы не наблюдалось.
NKT-клетки
Было обнаружено, что общее количество NKT-клеток в селезенке было снижено у мышей KO по сравнению с мышами WT. Снижение было аналогично тому, которое наблюдалось для общего количества лейкоцитов CD45+. Значимой разницы в количестве NKT-клеток в MLN не наблюдалось. Доля NKT-клеток из CD45+ клеток в крови также была ниже у мышей KO по сравнению с мышами WT. В селезенке и MLN такой разницы не наблюдалось.
В-клетки
Было обнаружено, что общее количество B-клеток в селезенке было снижено у мышей KO по сравнению с мышами WT. Снижение было аналогично тому, которое наблюдалось для общего количества лейкоцитов CD45+. Значимой разницы в количестве B-клеток в MLN не наблюдалось. Доля B-клеток из CD45+ клеток в селезенке и крови также была ниже у мышей KO по сравнению с мышами WT. В MLN такой разницы не наблюдалось.
NK-клетки
Было обнаружено, что общее количество NK-клеток в селезенке было снижено у мышей KO по сравнению с мышами WT. Было обнаружено, что снижение было более выраженным для NK-клеток по сравнению с тем, которое наблюдалось для общего количества лейкоцитов CD45+ и других лимфоидных субпопуляций. Значимой разницы в количестве NK-клеток в MLN не наблюдалось. Доля NK-клеток из CD45+ клеток в селезенке и крови также была ниже у мышей KO по сравнению с мышами WT. В MLN такой разницы не наблюдалось (Фиг. 27).
Миелоидные клетки
Общее количество миелоидных клеток было снижено у мышей KO по сравнению с мышами WT как в селезенке, так и в MLN. Было обнаружено, что доля миелоидных клеток из CD45+ клеток в селезенке и MLN также была ниже у мышей KO по сравнению с мышами WT. В крови такой разницы не наблюдалось.
Нейтрофилы
Значимой разницы в количестве нейтрофилов в селезенке и MLN между мышами KO и WT не наблюдалось. Доля нейтрофилов в крови была выше у мышей KO, но не было отмечено значимых различий в селезенке и MLN.
Эозинофилы
Значимой разницы в количестве эозинофилов в селезенке и MLN между мышами KO и WT не наблюдалось. Было обнаружено, что доля эозинофилов в крови была выше у мышей KO, но не было отмечено значимых различий в селезенке и MLN.
Моноциты
Было обнаружено, что общее количество моноцитов было ниже у мышей KO по сравнению с мышами WT как в селезенке, так и в MLN. Никакой значимой разницы в доле моноцитов между мышами KO и WT не наблюдалось ни в одном из исследованных органов.
Макрофаги
Было обнаружено, что общее количество макрофагов было ниже у мышей KO по сравнению с мышами WT как в селезенке, так и в MLN. Никакой значимой разницы в доле макрофагов между мышами KO и WT не наблюдалось ни в одном из исследованных органов.
Тучные клетки
Значимой разницы в количестве тучных клеток в селезенке и MLN между мышами KO и WT не наблюдалось. Доля тучных клеток в селезенке была выше у мышей KO по сравнению с мышами WT. В крови и MLN такой разницы не наблюдалось.
Базофилы
Было обнаружено, что общее количество базофилов в селезенке было снижено у мышей KO по сравнению с мышами WT. Никаких различий в количестве базофилов в MLN не наблюдалось. Было обнаружено, что доля базофилов в селезенке была больше у мышей KO по сравнению с мышами WT. В селезенке или MLN такой разницы не наблюдалось.
Заключение
В дополнение к общему эффекту на лейкоциты CD45+, полученные данные свидетельствуют о дополнительном воздействии конкретно на NK-клетки, т. е. как общее количество, так и доля NK-клеток из CD45+ клеток были ниже в крови и селезенке наивных мышей с дефицитом BSSL по сравнению с однопометными мышами дикого типа. Эти данные подтверждают вывод о том, что применение AS20 hIgG1 LALA-PG (доза 90 мг/кг) значимо снижало общее количество NK-клеток и долю T-клеток, NKT-клеток и NK-клеток из CD45+ в селезенке мышей с CAIA (Пример 19).
Таблица 29 - Перечень последовательностей и SEQ ID NO:, применяемых в настоящем изобретении
ССЫЛКИ
1 European Journal of Biochemistry (1981) 116(2): 221-225
2 Заявка на патент США № 2010/0124555
3 Заявка на патент США № 8 597 650
4 Заявка на патент США № 9 168 299
5 Developmental and Comparative Immunology (2003) 27(1): 55-77
6 Nucleic Acids Research (1994) 22: 4673-4680
7 Protein Engineering, Design and Selection (2016) 29(10): 457-466
8 The Journal of Biological Chemistry (2015) 290(9): 5462–5469
9 Journal of Molecular Biology (2001) 312: 511-523
10 MAbs (2013) 5(3): 445-470
11 Journal of Molecular Biology (2007) 373: 924–940
12 Protein Engineering, Design and Selection (2016) 29: 427-437
13 Molecular Immunoogy.(1993) 30(1): 105–1058
14 PNAS (2001) 98(3): 944-949
15 The Journal of Immunology (2012) 189(7): 3430-3438
16 The Journal of Biological Chemistry (2017) 292(9): 3900-3908
17 Protein Science (2000) 9: 1783–1790 .
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Lipum AB
<120> НОВЫЕ АНТИТЕЛА К BSSL
<130> HSJ106660P.WOP
<150> SE 1950888-6
<151> 2019-07-12
<160> 202
<170> PatentIn версия 3.5
<210> 1
<211> 6
<212> БЕЛОК
<213> Homo sapiens
<400> 1
Tyr Thr Glu Gly Gly Phe
1 5
<210> 2
<211> 14
<212> БЕЛОК
<213> Homo sapiens
<400> 2
Leu Glu Asn Pro Gln Pro His Pro Gly Trp Gln Gly Thr Leu
1 5 10
<210> 3
<211> 12
<212> БЕЛОК
<213> Homo sapiens
<400> 3
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe
1 5 10
<210> 4
<211> 18
<212> БЕЛОК
<213> Homo sapiens
<400> 4
Asn Ile Trp Val Pro Gln Gly Arg Lys Gln Val Ser Arg Asp Leu Pro
1 5 10 15
Val Met
<210> 5
<211> 7
<212> БЕЛОК
<213> Homo sapiens
<400> 5
Val Lys Arg Asn Ile Ala Ala
1 5
<210> 6
<211> 13
<212> БЕЛОК
<213> Homo sapiens
<400> 6
His Tyr Val Gly Phe Val Pro Val Ile Asp Gly Asp Phe
1 5 10
<210> 7
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HCDR1
<400> 7
Gly Tyr Thr Phe Thr Ser Tyr Asn
1 5
<210> 8
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HCDR2
<400> 8
Ile Asn Pro Gly Asp Gly Ala Thr
1 5
<210> 9
<211> 12
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HCDR3
<400> 9
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr
1 5 10
<210> 10
<211> 5
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCDR1
<400> 10
Pro Ser Ile Ser Tyr
1 5
<210> 11
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCDR3
<400> 11
His Gln Arg Ser Ser Ser Pro Thr
1 5
<210> 12
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный HCDR2
<400> 12
Met Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys
1 5 10 15
Phe Lys
<210> 13
<211> 9
<212> БЕЛОК
<213> Homo sapiens
<400> 13
Ala Trp Val Lys Arg Asn Ile Ala Ala
1 5
<210> 14
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR1
<400> 14
Arg Ala Ser Pro Ser Ile Ser Tyr Met Asn
1 5 10
<210> 15
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR2
<400> 15
Ala Thr Ser Ser Leu Ala
1 5
<210> 16
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR1
<400> 16
Ser Ala Ser Pro Ser Ile Ser Tyr Met Asn
1 5 10
<210> 17
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR2
<400> 17
Ala Thr Ser Ser Leu Pro
1 5
<210> 18
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HCDR2
<400> 18
Ile Tyr Pro Gly Asp Gly Ala Thr
1 5
<210> 19
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HCDR2
<400> 19
Ile Tyr Pro Gly Asp Gly Ser Thr
1 5
<210> 20
<211> 5
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCDR1
<400> 20
Ser Ser Ile Ser Tyr
1 5
<210> 21
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCDR3
<400> 21
His Gln Arg Ser Ser Tyr Pro Thr
1 5
<210> 22
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCDR3
<400> 22
His Gln Arg Ser Ser Thr Pro Thr
1 5
<210> 23
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный HCDR2
<400> 23
Met Gly Val Ile Tyr Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys
1 5 10 15
Phe Lys
<210> 24
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный HCDR2
<400> 24
Met Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys
1 5 10 15
Phe Gln
<210> 25
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный HCDR2
<400> 25
Ile Gly Val Ile Tyr Pro Gly Asp Gly Ser Thr Ser Tyr Asn Gln Lys
1 5 10 15
Phe Gln
<210> 26
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR1
<400> 26
Ser Ala Ser Ser Ser Ile Ser Tyr Met Asn
1 5 10
<210> 27
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR1
<400> 27
Arg Ala Ser Ser Ser Ile Ser Tyr Leu Asn
1 5 10
<210> 28
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR2
<400> 28
Ala Thr Ser Lys Leu Pro
1 5
<210> 29
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR2
<400> 29
Ala Ala Ser Ser Leu Ala
1 5
<210> 30
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HVCR
<400> 30
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Tyr Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 31
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LVCR
<400> 31
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 32
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HVCR
<400> 32
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 33
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCVR
<400> 33
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 34
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HVCR
<400> 34
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asp Gly Ser Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 35
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCVR
<400> 35
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Lys Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 36
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HVCR
<400> 36
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 37
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCVR
<400> 37
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 38
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCVR
<400> 38
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 39
<211> 25
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZH1
<400> 39
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser
20 25
<210> 40
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZH2
<400> 40
Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp
1 5 10
<210> 41
<211> 31
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZH3
<400> 41
Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr Met
1 5 10 15
Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
20 25 30
<210> 42
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZH4
<400> 42
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 43
<211> 23
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZL1
<400> 43
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 44
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZL2
<400> 44
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 45
<211> 33
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZL3
<400> 45
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
1 5 10 15
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
20 25 30
Cys
<210> 46
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> ZL4
<400> 46
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 47
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 47
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Tyr Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 48
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 48
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 49
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 49
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 50
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 50
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 51
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 51
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Tyr Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 52
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 52
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 53
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 53
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 54
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 54
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 55
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 55
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 56
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 56
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 57
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 57
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 58
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 58
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Gly Gly Tyr Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 59
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 59
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 60
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 60
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 61
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 61
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 62
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 62
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 63
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 63
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Tyr Pro Gly Gly Gly Tyr Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 64
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 64
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 65
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 65
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 66
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 66
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 67
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 67
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Ser Gly Ser Thr Ser Tyr Thr Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 68
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 68
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 69
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 69
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asp Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 70
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 70
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 71
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 71
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 72
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 72
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 73
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 73
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 74
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 74
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 75
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 75
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 76
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 76
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 77
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 77
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 78
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 78
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 79
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 79
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Ser Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 80
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VH
<400> 80
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Lys Gln Thr Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 81
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 81
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 82
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 82
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Ser Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 83
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 83
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 84
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 84
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 85
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 85
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Lys Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 86
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 86
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 87
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 87
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 88
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 88
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 89
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 89
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 90
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 90
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Ser Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 91
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 91
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 92
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 92
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 93
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 93
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 94
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 94
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Asp Ala Ser Ser Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 95
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 95
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Asp Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 96
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 96
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 97
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 97
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 98
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 98
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 99
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 99
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 100
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 100
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 101
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 101
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Asn Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 102
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 102
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 103
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 103
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Leu
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 104
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 104
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Lys Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 105
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 105
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 106
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 106
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 107
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 107
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 108
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 108
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Gln Ser Ile Ser Tyr Leu
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 109
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 109
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Arg Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 110
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 110
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Lys Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 111
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 111
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Leu
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Arg Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 112
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 112
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 113
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 113
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Lys Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 114
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> VL
<400> 114
Gln Ile Val Leu Thr Gln Ser Pro Ala Val Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Thr Ser Pro Lys Arg Trp Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 115
<211> 449
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> IgG1 LALA-PG HC
<400> 115
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Lys Gln Thr Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 116
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG1 LALA-PG LC
<400> 116
Gln Ile Val Leu Thr Gln Ser Pro Ala Val Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Thr Ser Pro Lys Arg Trp Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 117
<211> 450
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG1 LALA-PG HC
<400> 117
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Trp Met His Trp Val Lys Gln Arg Pro Gly Arg Gly Leu Glu Trp Ile
35 40 45
Gly Arg Ile Asp Pro Asn Ser Gly Gly Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Ser Lys Ala Thr Leu Thr Val Asp Lys Pro Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Asp Tyr Tyr Gly Ser Ser Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 118
<211> 213
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG1 LALA-PG LC
<400> 118
Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu
1 5 10 15
Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly
35 40 45
Leu Ile Gly Gly Thr Asn Asn Arg Ala Pro Gly Val Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Ile Gly Asn Lys Ala Ala Leu Thr Ile Thr Gly Ala
65 70 75 80
Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn
85 90 95
His Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gln Pro Lys
100 105 110
Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln
115 120 125
Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly
130 135 140
Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly
145 150 155 160
Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala
165 170 175
Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser
180 185 190
Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val
195 200 205
Ala Pro Thr Glu Cys
210
<210> 119
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 119
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Tyr Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 120
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 120
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 121
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 121
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 122
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 122
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Ser Ser Ile Ser Tyr Leu
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Ala Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 123
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 123
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asp Gly Ser Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 124
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 124
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Lys Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Thr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 125
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 125
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 126
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 126
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 127
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 127
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Asp Gly Ala Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 128
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 128
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Pro Ser Ile Ser Tyr Met
20 25 30
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Ala Thr Ser Ser Leu Pro Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Ser Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 129
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 129
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Lys Gln Thr Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 130
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 130
Gln Ile Val Leu Thr Gln Ser Pro Ala Val Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Thr Ser Pro Lys Arg Trp Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 131
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 131
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 132
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 132
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 133
<211> 447
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P HC
<400> 133
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Trp Met His Trp Val Lys Gln Arg Pro Gly Arg Gly Leu Glu Trp Ile
35 40 45
Gly Arg Ile Asp Pro Asn Ser Gly Gly Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Ser Lys Ala Thr Leu Thr Val Asp Lys Pro Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Asp Tyr Tyr Gly Ser Ser Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 134
<211> 216
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> hIgG4 S228P LC
<400> 134
Gln Ala Val Val Thr Gln Glu Ser Ala Leu Thr Thr Ser Pro Gly Glu
1 5 10 15
Thr Val Thr Leu Thr Cys Arg Ser Ser Thr Gly Ala Val Thr Thr Ser
20 25 30
Asn Tyr Ala Asn Trp Val Gln Glu Lys Pro Asp His Leu Phe Thr Gly
35 40 45
Leu Ile Gly Gly Thr Asn Asn Arg Ala Pro Gly Val Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Ile Gly Asn Lys Ala Ala Leu Thr Ile Thr Gly Ala
65 70 75 80
Gln Thr Glu Asp Glu Ala Ile Tyr Phe Cys Ala Leu Trp Tyr Ser Asn
85 90 95
His Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Arg Thr Val
100 105 110
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
115 120 125
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
130 135 140
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
145 150 155 160
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
165 170 175
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
180 185 190
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
195 200 205
Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 135
<211> 138
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> IgG HC
<400> 135
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys
20 25 30
Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe
35 40 45
Thr Ser Tyr Asn Met His Trp Val Lys Gln Thr Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn
65 70 75 80
Gln Lys Phe Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser
85 90 95
Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp
115 120 125
Gly Gln Gly Thr Thr Leu Thr Val Ser Ser
130 135
<210> 136
<211> 127
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> IgG LC
<400> 136
Met Asp Phe Gln Val Gln Ile Phe Ser Phe Leu Leu Ile Ser Ala Ser
1 5 10 15
Val Ile Leu Ser Arg Gly Gln Ile Val Leu Thr Gln Ser Pro Ala Val
20 25 30
Met Ser Ala Ser Pro Gly Glu Lys Val Thr Met Thr Cys Ser Ala Ser
35 40 45
Ser Ser Ile Ser Tyr Met His Trp Tyr Gln Gln Lys Pro Gly Thr Ser
50 55 60
Pro Lys Arg Trp Ile Tyr Asp Thr Ser Lys Leu Ala Ser Gly Val Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile
85 90 95
Ser Ser Met Glu Ala Glu Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg
100 105 110
Ser Ser Tyr Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
115 120 125
<210> 137
<211> 579
<212> БЕЛОК
<213> Mus musculus
<400> 137
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe Val Glu Gly Val
1 5 10 15
Asn Lys Lys Leu Ser Leu Leu Gly Gly Asp Ser Val Asp Ile Phe Lys
20 25 30
Gly Ile Pro Phe Ala Thr Ala Lys Thr Leu Glu Asn Pro Gln Arg His
35 40 45
Pro Gly Trp Gln Gly Thr Leu Lys Ala Thr Asn Phe Lys Lys Arg Cys
50 55 60
Leu Gln Ala Thr Ile Thr Gln Asp Asn Thr Tyr Gly Gln Glu Asp Cys
65 70 75 80
Leu Tyr Leu Asn Ile Trp Val Pro Gln Gly Arg Lys Gln Val Ser His
85 90 95
Asn Leu Pro Val Met Val Trp Ile Tyr Gly Gly Ala Phe Leu Met Gly
100 105 110
Ser Gly Gln Gly Ala Asn Phe Leu Lys Asn Tyr Leu Tyr Asp Gly Glu
115 120 125
Glu Ile Ala Thr Arg Gly Asn Val Ile Val Val Thr Phe Asn Tyr Arg
130 135 140
Val Gly Pro Leu Gly Phe Leu Ser Thr Gly Asp Ala Asn Leu Pro Gly
145 150 155 160
Asn Phe Gly Leu Arg Asp Gln His Met Ala Ile Ala Trp Val Lys Arg
165 170 175
Asn Ile Ala Ala Phe Gly Gly Asp Pro Asp Asn Ile Thr Ile Phe Gly
180 185 190
Glu Ser Ala Gly Ala Ala Ser Val Ser Leu Gln Thr Leu Ser Pro Tyr
195 200 205
Asn Lys Gly Leu Ile Arg Arg Ala Ile Ser Gln Ser Gly Met Ala Leu
210 215 220
Ser Pro Trp Ala Ile Gln Lys Asn Pro Leu Phe Trp Ala Lys Thr Ile
225 230 235 240
Ala Lys Lys Val Gly Cys Pro Thr Glu Asp Thr Gly Lys Met Ala Ala
245 250 255
Cys Leu Lys Ile Thr Asp Pro Arg Ala Leu Thr Leu Ala Tyr Lys Leu
260 265 270
Pro Val Lys Lys Gln Glu Tyr Pro Val Val His Tyr Leu Ala Phe Ile
275 280 285
Pro Val Ile Asp Gly Asp Phe Ile Pro Asp Asp Pro Ile Asn Leu Tyr
290 295 300
Asn Asn Thr Ala Asp Ile Asp Tyr Ile Ala Gly Ile Asn Asn Met Asp
305 310 315 320
Gly His Leu Phe Ala Thr Ile Asp Val Pro Ala Val Asp Lys Thr Lys
325 330 335
Gln Thr Val Thr Glu Glu Asp Phe Tyr Arg Leu Val Ser Gly His Thr
340 345 350
Val Ala Lys Gly Leu Lys Gly Ala Gln Ala Thr Phe Asp Ile Tyr Thr
355 360 365
Glu Ser Trp Ala Gln Asp Pro Ser Gln Glu Asn Met Lys Lys Thr Val
370 375 380
Val Ala Phe Glu Thr Asp Val Leu Phe Leu Ile Pro Thr Glu Ile Ala
385 390 395 400
Leu Ala Gln His Lys Ala His Ala Lys Ser Ala Lys Thr Tyr Ser Tyr
405 410 415
Leu Phe Ser His Pro Ser Arg Met Pro Ile Tyr Pro Lys Trp Met Gly
420 425 430
Ala Asp His Ala Asp Asp Leu Gln Tyr Val Phe Gly Lys Pro Phe Ala
435 440 445
Thr Pro Leu Gly Tyr Arg Pro Gln Asp Arg Ala Val Ser Lys Ala Met
450 455 460
Ile Ala Tyr Trp Thr Asn Phe Ala Arg Ser Gly Asp Pro Asn Met Gly
465 470 475 480
Asn Ser Pro Val Pro Thr His Trp Tyr Pro Tyr Thr Leu Glu Asn Gly
485 490 495
Asn Tyr Leu Asp Ile Thr Lys Thr Ile Thr Ser Ala Ser Met Lys Glu
500 505 510
His Leu Arg Glu Lys Phe Leu Lys Phe Trp Ala Val Thr Phe Glu Val
515 520 525
Leu Pro Thr Val Thr Gly Asp Gln Asp Thr Leu Thr Pro Pro Glu Asp
530 535 540
Asp Ser Glu Val Ala Pro Asp Pro Pro Ser Asp Asp Ser Gln Val Val
545 550 555 560
Pro Val Pro Pro Thr Asp Asp Ser Val Glu Ala Gln Met Pro Ala Thr
565 570 575
Ile Gly Phe
<210> 138
<211> 722
<212> БЕЛОК
<213> Homo sapiens
<400> 138
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe Val Glu Gly Val
1 5 10 15
Asn Lys Lys Leu Gly Leu Leu Gly Asp Ser Val Asp Ile Phe Lys Gly
20 25 30
Ile Pro Phe Ala Ala Pro Thr Lys Ala Leu Glu Asn Pro Gln Pro His
35 40 45
Pro Gly Trp Gln Gly Thr Leu Lys Ala Lys Asn Phe Lys Lys Arg Cys
50 55 60
Leu Gln Ala Thr Ile Thr Gln Asp Ser Thr Tyr Gly Asp Glu Asp Cys
65 70 75 80
Leu Tyr Leu Asn Ile Trp Val Pro Gln Gly Arg Lys Gln Val Ser Arg
85 90 95
Asp Leu Pro Val Met Ile Trp Ile Tyr Gly Gly Ala Phe Leu Met Gly
100 105 110
Ser Gly His Gly Ala Asn Phe Leu Asn Asn Tyr Leu Tyr Asp Gly Glu
115 120 125
Glu Ile Ala Thr Arg Gly Asn Val Ile Val Val Thr Phe Asn Tyr Arg
130 135 140
Val Gly Pro Leu Gly Phe Leu Ser Thr Gly Asp Ala Asn Leu Pro Gly
145 150 155 160
Asn Tyr Gly Leu Arg Asp Gln His Met Ala Ile Ala Trp Val Lys Arg
165 170 175
Asn Ile Ala Ala Phe Gly Gly Asp Pro Asn Asn Ile Thr Leu Phe Gly
180 185 190
Glu Ser Ala Gly Gly Ala Ser Val Ser Leu Gln Thr Leu Ser Pro Tyr
195 200 205
Asn Lys Gly Leu Ile Arg Arg Ala Ile Ser Gln Ser Gly Val Ala Leu
210 215 220
Ser Pro Trp Val Ile Gln Lys Asn Pro Leu Phe Trp Ala Lys Lys Val
225 230 235 240
Ala Glu Lys Val Gly Cys Pro Val Gly Asp Ala Ala Arg Met Ala Gln
245 250 255
Cys Leu Lys Val Thr Asp Pro Arg Ala Leu Thr Leu Ala Tyr Lys Val
260 265 270
Pro Leu Ala Gly Leu Glu Tyr Pro Met Leu His Tyr Val Gly Phe Val
275 280 285
Pro Val Ile Asp Gly Asp Phe Ile Pro Ala Asp Pro Ile Asn Leu Tyr
290 295 300
Ala Asn Ala Ala Asp Ile Asp Tyr Ile Ala Gly Thr Asn Asn Met Asp
305 310 315 320
Gly His Ile Phe Ala Ser Ile Asp Met Pro Ala Ile Asn Lys Gly Asn
325 330 335
Lys Lys Val Thr Glu Glu Asp Phe Tyr Lys Leu Val Ser Glu Phe Thr
340 345 350
Ile Thr Lys Gly Leu Arg Gly Ala Lys Thr Thr Phe Asp Val Tyr Thr
355 360 365
Glu Ser Trp Ala Gln Asp Pro Ser Gln Glu Asn Lys Lys Lys Thr Val
370 375 380
Val Asp Phe Glu Thr Asp Val Leu Phe Leu Val Pro Thr Glu Ile Ala
385 390 395 400
Leu Ala Gln His Arg Ala Asn Ala Lys Ser Ala Lys Thr Tyr Ala Tyr
405 410 415
Leu Phe Ser His Pro Ser Arg Met Pro Val Tyr Pro Lys Trp Val Gly
420 425 430
Ala Asp His Ala Asp Asp Ile Gln Tyr Val Phe Gly Lys Pro Phe Ala
435 440 445
Thr Pro Thr Gly Tyr Arg Pro Gln Asp Arg Thr Val Ser Lys Ala Met
450 455 460
Ile Ala Tyr Trp Thr Asn Phe Ala Lys Thr Gly Asp Pro Asn Met Gly
465 470 475 480
Asp Ser Ala Val Pro Thr His Trp Glu Pro Tyr Thr Thr Glu Asn Ser
485 490 495
Gly Tyr Leu Glu Ile Thr Lys Lys Met Gly Ser Ser Ser Met Lys Arg
500 505 510
Ser Leu Arg Thr Asn Phe Leu Arg Tyr Trp Thr Leu Thr Tyr Leu Ala
515 520 525
Leu Pro Thr Val Thr Asp Gln Glu Ala Thr Pro Val Pro Pro Thr Gly
530 535 540
Asp Ser Glu Ala Thr Pro Val Pro Pro Thr Gly Asp Ser Glu Thr Ala
545 550 555 560
Pro Val Pro Pro Thr Gly Asp Ser Gly Ala Pro Pro Val Pro Pro Thr
565 570 575
Gly Asp Ser Gly Ala Pro Pro Val Pro Pro Thr Gly Asp Ser Gly Ala
580 585 590
Pro Pro Val Pro Pro Thr Gly Asp Ser Gly Ala Pro Pro Val Pro Pro
595 600 605
Thr Gly Asp Ser Gly Ala Pro Pro Val Pro Pro Thr Gly Asp Ser Gly
610 615 620
Ala Pro Pro Val Pro Pro Thr Gly Asp Ser Gly Ala Pro Pro Val Pro
625 630 635 640
Pro Thr Gly Asp Ala Gly Pro Pro Pro Val Pro Pro Thr Gly Asp Ser
645 650 655
Gly Ala Pro Pro Val Pro Pro Thr Gly Asp Ser Gly Ala Pro Pro Val
660 665 670
Thr Pro Thr Gly Asp Ser Glu Thr Ala Pro Val Pro Pro Thr Gly Asp
675 680 685
Ser Gly Ala Pro Pro Val Pro Pro Thr Gly Asp Ser Glu Ala Ala Pro
690 695 700
Val Pro Pro Thr Asp Asp Ser Lys Glu Ala Gln Met Pro Ala Val Ile
705 710 715 720
Arg Phe
<210> 139
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> IgG4 HC
<400> 139
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Lys Gln Thr Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro
210 215 220
Cys Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val
260 265 270
Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 140
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> IgG4 LC
<400> 140
Gln Ile Val Leu Thr Gln Ser Pro Ala Val Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Thr Ser Pro Lys Arg Trp Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 141
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный HCDR2
<400> 141
Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys
1 5 10 15
Phe Lys
<210> 142
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR1
<400> 142
Ser Ala Ser Ser Ser Ile Ser Tyr Met His
1 5 10
<210> 143
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Расширенный LCDR2
<400> 143
Asp Thr Ser Lys Leu Ala
1 5
<210> 144
<211> 119
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HCVR
<400> 144
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Tyr Tyr Gly Ser Ser Pro Leu Gly Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 145
<211> 105
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LCVR
<400> 145
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Ile Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
65 70 75 80
Asp Phe Ala Thr Tyr Tyr Cys His Gln Arg Ser Ser Tyr Pro Thr Phe
85 90 95
Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 146
<211> 537
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> t-hBSSL
<400> 146
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe Val Glu Gly Val
1 5 10 15
Asn Lys Lys Leu Gly Leu Leu Gly Asp Ser Val Asp Ile Phe Lys Gly
20 25 30
Ile Pro Phe Ala Ala Pro Thr Lys Ala Leu Glu Asn Pro Gln Pro His
35 40 45
Pro Gly Trp Gln Gly Thr Leu Lys Ala Lys Asn Phe Lys Lys Arg Cys
50 55 60
Leu Gln Ala Thr Ile Thr Gln Asp Ser Thr Tyr Gly Asp Glu Asp Cys
65 70 75 80
Leu Tyr Leu Asn Ile Trp Val Pro Gln Gly Arg Lys Gln Val Ser Arg
85 90 95
Asp Leu Pro Val Met Ile Trp Ile Tyr Gly Gly Ala Phe Leu Met Gly
100 105 110
Ser Gly His Gly Ala Asn Phe Leu Asn Asn Tyr Leu Tyr Asp Gly Glu
115 120 125
Glu Ile Ala Thr Arg Gly Asn Val Ile Val Val Thr Phe Asn Tyr Arg
130 135 140
Val Gly Pro Leu Gly Phe Leu Ser Thr Gly Asp Ala Asn Leu Pro Gly
145 150 155 160
Asn Tyr Gly Leu Arg Asp Gln His Met Ala Ile Ala Trp Val Lys Arg
165 170 175
Asn Ile Ala Ala Phe Gly Gly Asp Pro Asn Asn Ile Thr Leu Phe Gly
180 185 190
Glu Ser Ala Gly Gly Ala Ser Val Ser Leu Gln Thr Leu Ser Pro Tyr
195 200 205
Asn Lys Gly Leu Ile Arg Arg Ala Ile Ser Gln Ser Gly Val Ala Leu
210 215 220
Ser Pro Trp Val Ile Gln Lys Asn Pro Leu Phe Trp Ala Lys Lys Val
225 230 235 240
Ala Glu Lys Val Gly Cys Pro Val Gly Asp Ala Ala Arg Met Ala Gln
245 250 255
Cys Leu Lys Val Thr Asp Pro Arg Ala Leu Thr Leu Ala Tyr Lys Val
260 265 270
Pro Leu Ala Gly Leu Glu Tyr Pro Met Leu His Tyr Val Gly Phe Val
275 280 285
Pro Val Ile Asp Gly Asp Phe Ile Pro Ala Asp Pro Ile Asn Leu Tyr
290 295 300
Ala Asn Ala Ala Asp Ile Asp Tyr Ile Ala Gly Thr Asn Asn Met Asp
305 310 315 320
Gly His Ile Phe Ala Ser Ile Asp Met Pro Ala Ile Asn Lys Gly Asn
325 330 335
Lys Lys Val Thr Glu Glu Asp Phe Tyr Lys Leu Val Ser Glu Phe Thr
340 345 350
Ile Thr Lys Gly Leu Arg Gly Ala Lys Thr Thr Phe Asp Val Tyr Thr
355 360 365
Glu Ser Trp Ala Gln Asp Pro Ser Gln Glu Asn Lys Lys Lys Thr Val
370 375 380
Val Asp Phe Glu Thr Asp Val Leu Phe Leu Val Pro Thr Glu Ile Ala
385 390 395 400
Leu Ala Gln His Arg Ala Asn Ala Lys Ser Ala Lys Thr Tyr Ala Tyr
405 410 415
Leu Phe Ser His Pro Ser Arg Met Pro Val Tyr Pro Lys Trp Val Gly
420 425 430
Ala Asp His Ala Asp Asp Ile Gln Tyr Val Phe Gly Lys Pro Phe Ala
435 440 445
Thr Pro Thr Gly Tyr Arg Pro Gln Asp Arg Thr Val Ser Lys Ala Met
450 455 460
Ile Ala Tyr Trp Thr Asn Phe Ala Lys Thr Gly Asp Pro Asn Met Gly
465 470 475 480
Asp Ser Ala Val Pro Thr His Trp Glu Pro Tyr Thr Thr Glu Asn Ser
485 490 495
Gly Tyr Leu Glu Ile Thr Lys Lys Met Gly Ser Ser Ser Met Lys Arg
500 505 510
Ser Leu Arg Thr Asn Phe Leu Arg Tyr Trp Thr Leu Thr Tyr Leu Ala
515 520 525
Leu Pro Ala His His His His His His
530 535
<210> 147
<211> 5
<212> БЕЛОК
<213> Homo sapiens
<400> 147
Tyr Thr Glu Gly Gly
1 5
<210> 148
<211> 7
<212> БЕЛОК
<213> Homo sapiens
<400> 148
Ala Lys Leu Gly Ala Val Tyr
1 5
<210> 149
<211> 11
<212> БЕЛОК
<213> Homo sapiens
<400> 149
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly
1 5 10
<210> 150
<211> 25
<212> БЕЛОК
<213> Homo sapiens
<400> 150
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe Val Glu Gly Val
1 5 10 15
Asn Lys Lys Leu Gly Leu Leu Gly Asp
20 25
<210> 151
<211> 26
<212> БЕЛОК
<213> Homo sapiens
<400> 151
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe Val Glu Gly Val
1 5 10 15
Asn Lys Lys Leu Gly Leu Leu Gly Asp Ser
20 25
<210> 152
<211> 28
<212> БЕЛОК
<213> Homo sapiens
<400> 152
Ala Lys Leu Gly Ala Val Tyr Thr Glu Gly Gly Phe Val Glu Gly Val
1 5 10 15
Asn Lys Lys Leu Gly Leu Leu Gly Asp Ser Val Asp
20 25
<210> 153
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 153
Val Lys Pro Gly Ala Ser Val Lys Met
1 5
<210> 154
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 154
Tyr Asn Met His Trp Val Arg Gln Ala
1 5
<210> 155
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 155
Tyr Ala Gln Lys Phe Lys Gly Arg Val
1 5
<210> 156
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 156
Trp Gly Gln Gly Thr Thr Leu Thr Val
1 5
<210> 157
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 157
Val Thr Met Thr Cys Ser Ala Ser Ser
1 5
<210> 158
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 158
Val Thr Ile Thr Cys Arg Ala Ser Ser
1 5
<210> 159
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 159
Val Thr Ile Thr Cys Ser Ala Ser Ser
1 5
<210> 160
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 160
Leu Leu Ile Tyr Ala Thr Ser Lys Leu
1 5
<210> 161
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 161
Ile Tyr Ala Thr Ser Ser Leu Ala Ser
1 5
<210> 162
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 162
Ile Tyr Ala Ala Ser Ser Leu Ala Ser
1 5
<210> 163
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> iTope остатки
<400> 163
Phe Gly Gly Gly Thr Lys Leu Glu Ile
1 5
<210> 164
<211> 37
<212> DNA
<213> Искусственная последовательность
<220>
<223> Oligonucleotide primer
<400> 164
gatacacctt caccagctac watatgcact gggtgcg 37
<210> 165
<211> 83
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 165
Gly Ala Cys Ala Ala Gly Gly Gly Cys Thr Thr Gly Ala Gly Thr Gly
1 5 10 15
Gly Ala Thr Arg Gly Gly Ala Arg Thr Ala Ala Thr Cys Trp Ala Cys
20 25 30
Cys Cys Thr Arg Gly Thr Arg Arg Thr Gly Gly Thr Lys Met Cys Ala
35 40 45
Cys Ala Ala Gly Cys Thr Ala Cys Arg Met Thr Cys Ala Gly Ala Ala
50 55 60
Gly Thr Thr Cys Met Ala Gly Gly Gly Cys Cys Gly Cys Gly Thr Cys
65 70 75 80
Ala Cys Cys
<210> 166
<211> 62
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 166
cgtcaccatc acctgcagkg caagtymgag cattagctat wtgmattggt atcagcagaa 60
ac 62
<210> 167
<211> 52
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 167
cctaagctcc tgatctatgm trcatccars ttgsmaagtg gggtcccatc ac 52
<210> 168
<211> 57
<212> DNA
<213> Искусственная последовательность
<220>
<223> Олигонуклеотидный праймер
<400> 168
gattttgcaa cttattactg tcascagagk tmtagtwmty hcacttttgg ccagggg 57
<210> 169
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> HVCR часть 2
<220>
<221> Прочий_признак
<222> (1)..(1)
<223> X=I или M
<220>
<221> Прочий_признак
<222> (5)..(5)
<223> X=N или Y
<220>
<221> Прочий_признак
<222> (10)..(10)
<223> X=A или S
<220>
<221> Прочий_признак
<222> (14)..(14)
<223> X=A или N
<220>
<221> Прочий_признак
<222> (18)..(18)
<223> X=K или Q
<400> 169
Xaa Gly Val Ile Xaa Pro Gly Asp Gly Xaa Thr Ser Tyr Xaa Gln Lys
1 5 10 15
Phe Xaa
<210> 170
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LVCR часть 1
<220>
<221> Прочий_признак
<222> (1)..(1)
<223> X=S или R
<220>
<221> Прочий_признак
<222> (4)..(4)
<223> X=S или P
<220>
<221> Прочий_признак
<222> (9)..(9)
<223> X=M или L
<400> 170
Xaa Ala Ser Xaa Ser Ile Ser Tyr Xaa Asn
1 5 10
<210> 171
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LVCR часть 2
<220>
<221> Прочий_признак
<222> (2)..(2)
<223> X=A или T
<220>
<221> Прочий_признак
<222> (4)..(4)
<223> X=K или S
<220>
<221> Прочий_признак
<222> (6)..(6)
<223> X=A или P
<400> 171
Ala Xaa Ser Xaa Leu Xaa
1 5
<210> 172
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> LVCR часть 3
<220>
<221> Прочий_признак
<222> (6)..(6)
<223> X=S, T или Y
<400> 172
His Gln Arg Ser Ser Xaa Pro Thr
1 5
<210> 173
<211> 8
<212> БЕЛОК
<213> Homo sapiens
<400> 173
Gln Gln Ser Tyr Ser Thr Pro Thr
1 5
<210> 174
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 174
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctacaata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagta atctaccctg gtgatggtgc cacaagctac 180
gctcagaagt tcaagggccg cgtcaccatg acccgcgaca cgtccacgag cacagtctac 240
atggagctga gcagcctgcg ctctgaggac acggctgtgt attactgtgc gagagattac 300
tacggtagta gcccccttgg ctactggggc caaggaaccc tggtcaccgt ctcctca 357
<210> 175
<211> 315
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 175
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacctgca gtgcaagtcc gagcattagc tatatgaatt ggtatcagca gaaaccaggg 120
aaagccccta agctcctgat ctatgctaca tccagcttgg caagtggggt cccatcacgt 180
ttcagtggca gtggaagcgg gacagatttc actctcacca tcagcagtct gcaacctgaa 240
gattttgcaa cttattactg tcaccagagg tctagttatc ccacttttgg ccaggggacc 300
aagctggaga tcaaa 315
<210> 176
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 176
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctacaata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagta atcaaccctg gtgatggtgc cacaagctac 180
aatcagaagt tccagggccg cgtcaccatg acccgcgaca cgtccacgag cacagtctac 240
atggagctga gcagcctgcg ctctgaggac acggctgtgt attactgtgc gagagattac 300
tacggtagta gcccccttgg ctactggggc caaggaaccc tggtcaccgt ctcctca 357
<210> 177
<211> 315
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 177
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacctgca gggcaagttc gagcattagc tatttgaatt ggtatcagca gaaaccaggg 120
aaagccccta agctcctgat ctatgctgca tccagcttgg caagtggggt cccatcacgt 180
ttcagtggca gtggaagcgg gacagatttc actctcacca tcagcagtct gcaacctgaa 240
gattttgcaa cttattactg tcaccagagg tctagttctc ccacttttgg ccaggggacc 300
aagctggaga tcaaa 315
<210> 178
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 178
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctacaata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gataggagta atctaccctg gtgatggttc cacaagctac 180
aatcagaagt tccagggccg cgtcaccatg acccgcgaca cgtccacgag cacagtctac 240
atggagctga gcagcctgcg ctctgaggac acggctgtgt attactgtgc gagagattac 300
tacggtagta gcccccttgg ctactggggc caaggaaccc tggtcaccgt ctcctca 357
<210> 179
<211> 315
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 179
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacctgca gtgcaagttc gagcattagc tatatgaatt ggtatcagca gaaaccaggg 120
aaagccccta agctcctgat ctatgctaca tccaagttgc caagtggggt cccatcacgt 180
ttcagtggca gtggaagcgg gacagatttc actctcacca tcagcagtct gcaacctgaa 240
gattttgcaa cttattactg tcaccagagg tctagtactc ccacttttgg ccaggggacc 300
aagctggaga tcaaa 315
<210> 180
<211> 357
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 180
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctacaata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggagta atcaaccctg gtgatggtgc cacaagctac 180
gctcagaagt tcaagggccg cgtcaccatg acccgcgaca cgtccacgag cacagtctac 240
atggagctga gcagcctgcg ctctgaggac acggctgtgt attactgtgc gagagattac 300
tacggtagta gcccccttgg ctactggggc caaggaaccc tggtcaccgt ctcctca 357
<210> 181
<211> 315
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 181
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacctgca gggcaagtcc gagcattagc tatatgaatt ggtatcagca gaaaccaggg 120
aaagccccta agctcctgat ctatgctaca tccagcttgg caagtggggt cccatcacgt 180
ttcagtggca gtggaagcgg gacagatttc actctcacca tcagcagtct gcaacctgaa 240
gattttgcaa cttattactg tcaccagagg tctagttctc ccacttttgg ccaggggacc 300
aagctggaga tcaaa 315
<210> 182
<211> 315
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 182
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacctgca gtgcaagtcc gagcattagc tatatgaatt ggtatcagca gaaaccaggg 120
aaagccccta agctcctgat ctatgctaca tccagcttgc caagtggggt cccatcacgt 180
ttcagtggca gtggaagcgg gacagatttc actctcacca tcagcagtct gcaacctgaa 240
gattttgcaa cttattactg tcaccagagg tctagttctc ccacttttgg ccaggggacc 300
aagctggaga tcaaa 315
<210> 183
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 183
caagttcagc tgcagcagcc cggtgccgag ctggtgaaac ccggtgcctc tgtgaagatg 60
agctgcaagg ccagcggcta cacctttacc agctacaaca tgcactgggt gaagcagaca 120
cccggacaag gtttagagtg gatcggcgtg atctaccccg gcaacggcga cacctcttac 180
aaccagaagt tcaagggcaa ggccacactg accgccgaca agagcagcag caccgcctac 240
atgcagctga gctctttaac cagcgaggac tccgccgtgt actactgcgc tcgtgattac 300
tacggcagca gccctttagg ctattgggga caaggtacca ctttaaccgt ctcgagcgcc 360
tcaaccaaag gaccctccgt gtttcccctc gccccctgtt cccgctccac atccgagtca 420
accgcggcgc tgggctgcct cgtgaaggac tacttccctg agcccgtcac tgtgtcgtgg 480
aactccgggg ccctgacctc cggcgtgcac accttccctg ccgtgcttca atcctccgga 540
ctgtactccc tgtcctcggt ggtcaccgtg ccgtcgagct cgttgggaac caagacttac 600
acttgcaacg tggaccacaa gccaagcaac accaaagtgg acaagagagt cgaatctaag 660
tacggaccgc cctgcccgcc ttgccccgcc cctgagtttc tcggcggtcc tagcgtgttc 720
ctgttcccac ccaagcccaa ggacactctg atgatctccc ggacccctga agtgacctgt 780
gtggtcgtgg acgtgtcgca ggaagatccg gaggtccagt tcaattggta cgtggatggg 840
gtggaggtcc acaacgccaa gacgaagccg agagaagaac agttcaactc aacttaccgg 900
gtggtgtccg tgctgaccgt gctgcatcag gattggctca acggaaagga gtacaagtgc 960
aaagtgtcca acaagggcct gcctagctca atcgaaaaga ccatttccaa ggccaagggc 1020
cagccgaggg aaccacaggt ctatactctg ccaccgagcc aagaagagat gaccaagaac 1080
caagtgtccc tgacttgcct ggtcaagggg ttctacccgt cggacatcgc agtggagtgg 1140
gagagcaacg gacagcctga aaacaattac aagaccaccc cgcccgtgct ggatagcgac 1200
ggttccttct tcctttactc gcgcctcacc gtcgacaaga gccggtggca ggagggcaac 1260
gtgttctcct gctccgtgat gcacgaagct ctgcataacc actacactca gaagtccttg 1320
tcgctgagcc tcggaaag 1338
<210> 184
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 184
cagatcgtgc tgacccagag ccccgctgtg atgagcgcct ctcccggtga gaaggtgacc 60
atgacttgta gcgccagcag cagcatcagc tacatgcact ggtaccagca gaagcccggc 120
accagcccta agaggtggat ctacgacacc agcaagctgg ccagcggcgt gcccgctagg 180
ttcagcggaa gcggcagcgg caccagctac tctttaacca tcagcagcat ggaggccgag 240
gatgccgcca cctactactg ccaccagaga agcagctacc ccaccttcgg cggcggcacc 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 185
<211> 1340
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 185
ctcaagttca gctcgtgcag agcggtgctg aagtgaagaa gcccggtgcc tctgtgaagg 60
tgagctgcaa ggccagcggc tacaccttca ccagctacaa catgcactgg gtgagacaag 120
ctcccggtca aggtttagag tggatgggcg tgatctaccc cggtgatggt gctaccagct 180
acgcccagaa gttcaagggt cgtgtgacca tgaccagaga caccagcacc agcaccgtgt 240
acatggagct gagctcttta aggagcgagg acaccgccgt gtactactgc gctcgtgact 300
actacggcag cagcccttta ggctattggg gacaaggtac tttagtgacc gtctcgagcg 360
cctcaaccaa aggaccctcc gtgtttcccc tcgccccctg ttcccgctcc acatccgagt 420
caaccgcggc gctgggctgc ctcgtgaagg actacttccc tgagcccgtc actgtgtcgt 480
ggaactccgg ggccctgacc tccggcgtgc acaccttccc tgccgtgctt caatcctccg 540
gactgtactc cctgtcctcg gtggtcaccg tgccgtcgag ctcgttggga accaagactt 600
acacttgcaa cgtggaccac aagccaagca acaccaaagt ggacaagaga gtcgaatcta 660
agtacggacc gccctgcccg ccttgccccg cccctgagtt tctcggcggt cctagcgtgt 720
tcctgttccc acccaagccc aaggacactc tgatgatctc ccggacccct gaagtgacct 780
gtgtggtcgt ggacgtgtcg caggaagatc cggaggtcca gttcaattgg tacgtggatg 840
gggtggaggt ccacaacgcc aagacgaagc cgagagaaga acagttcaac tcaacttacc 900
gggtggtgtc cgtgctgacc gtgctgcatc aggattggct caacggaaag gagtacaagt 960
gcaaagtgtc caacaagggc ctgcctagct caatcgaaaa gaccatttcc aaggccaagg 1020
gccagccgag ggaaccacag gtctatactc tgccaccgag ccaagaagag atgaccaaga 1080
accaagtgtc cctgacttgc ctggtcaagg ggttctaccc gtcggacatc gcagtggagt 1140
gggagagcaa cggacagcct gaaaacaatt acaagaccac cccgcccgtg ctggatagcg 1200
acggttcctt cttcctttac tcgcgcctca ccgtcgacaa gagccggtgg caggagggca 1260
acgtgttctc ctgctccgtg atgcacgaag ctctgcataa ccactacact cagaagtcct 1320
tgtcgctgag cctcggaaag 1340
<210> 186
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 186
gacatccaga tgacccagag ccctagctct ttaagcgcct ctgtgggcga tcgtgtgacc 60
atcacttgta gcgccagccc cagcatcagc tacatgaact ggtaccagca gaagcccggc 120
aaggccccca agctgctgat ctacgccaca agctctttag ccagcggcgt gcctagcaga 180
tttagcggca gcggtagcgg cacagacttc actttaacca tcagctcttt acagccagaa 240
gacttcgcca cctactactg ccaccagagg agcagctacc ccaccttcgg ccaaggtacc 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 187
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 187
caagttcagc tcgtgcagag cggtgctgaa gtgaagaagc ccggtgcctc tgtgaaggtg 60
agctgcaagg ccagcggcta caccttcacc agctacaaca tgcactgggt gagacaagct 120
cccggtcaag gtttagagtg gatgggcgtg atcaaccccg gtgatggtgc taccagctac 180
aaccagaagt tccagggtcg tgtgaccatg accagagaca ccagcaccag caccgtgtac 240
atggagctga gctctttaag gagcgaggac accgccgtgt actactgcgc tcgtgactac 300
tacggcagca gccctttagg ctattgggga caaggtactt tagtgaccgt ctcgagcgcc 360
tcaaccaaag gaccctccgt gtttcccctc gccccctgtt cccgctccac atccgagtca 420
accgcggcgc tgggctgcct cgtgaaggac tacttccctg agcccgtcac tgtgtcgtgg 480
aactccgggg ccctgacctc cggcgtgcac accttccctg ccgtgcttca atcctccgga 540
ctgtactccc tgtcctcggt ggtcaccgtg ccgtcgagct cgttgggaac caagacttac 600
acttgcaacg tggaccacaa gccaagcaac accaaagtgg acaagagagt cgaatctaag 660
tacggaccgc cctgcccgcc ttgccccgcc cctgagtttc tcggcggtcc tagcgtgttc 720
ctgttcccac ccaagcccaa ggacactctg atgatctccc ggacccctga agtgacctgt 780
gtggtcgtgg acgtgtcgca ggaagatccg gaggtccagt tcaattggta cgtggatggg 840
gtggaggtcc acaacgccaa gacgaagccg agagaagaac agttcaactc aacttaccgg 900
gtggtgtccg tgctgaccgt gctgcatcag gattggctca acggaaagga gtacaagtgc 960
aaagtgtcca acaagggcct gcctagctca atcgaaaaga ccatttccaa ggccaagggc 1020
cagccgaggg aaccacaggt ctatactctg ccaccgagcc aagaagagat gaccaagaac 1080
caagtgtccc tgacttgcct ggtcaagggg ttctacccgt cggacatcgc agtggagtgg 1140
gagagcaacg gacagcctga aaacaattac aagaccaccc cgcccgtgct ggatagcgac 1200
ggttccttct tcctttactc gcgcctcacc gtcgacaaga gccggtggca ggagggcaac 1260
gtgttctcct gctccgtgat gcacgaagct ctgcataacc actacactca gaagtccttg 1320
tcgctgagcc tcggaaag 1338
<210> 188
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 188
gacatccaga tgacccagag ccctagctct ttaagcgcct ctgtgggcga tcgtgtgacc 60
atcacttgtc gtgccagcag cagcatcagc tatttaaact ggtaccagca gaagcccggc 120
aaggccccca agctgctgat ctacgccgcc tcttctttag cctctggcgt gccttctcgt 180
ttcagcggaa gcggcagcgg caccgacttc actttaacca tcagctcttt acagccagaa 240
gacttcgcca cctactactg ccaccagagg agcagcagcc ccaccttcgg acaaggtacc 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 189
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 189
caagttcagc tggtgcagag cggagccgag gtgaaaaagc ccggtgcctc tgtgaaggtg 60
agctgcaagg ccagcggcta cacctttacc agctacaaca tgcactgggt gaggcaagct 120
cccggtcaag gtctggagtg gatcggcgtg atctaccccg gcgacggcag cacctcttac 180
aaccagaagt tccaaggtcg tgtgaccatg actcgtgaca ccagcaccag caccgtgtac 240
atggagctga gctctttaag gagcgaggat accgccgtgt actactgcgc tcgtgactac 300
tacggcagca gccctctggg ctattggggc caaggtactt tagtgaccgt ctcgagcgcc 360
tcaaccaaag gaccctccgt gtttcccctc gccccctgtt cccgctccac atccgagtca 420
accgcggcgc tgggctgcct cgtgaaggac tacttccctg agcccgtcac tgtgtcgtgg 480
aactccgggg ccctgacctc cggcgtgcac accttccctg ccgtgcttca atcctccgga 540
ctgtactccc tgtcctcggt ggtcaccgtg ccgtcgagct cgttgggaac caagacttac 600
acttgcaacg tggaccacaa gccaagcaac accaaagtgg acaagagagt cgaatctaag 660
tacggaccgc cctgcccgcc ttgccccgcc cctgagtttc tcggcggtcc tagcgtgttc 720
ctgttcccac ccaagcccaa ggacactctg atgatctccc ggacccctga agtgacctgt 780
gtggtcgtgg acgtgtcgca ggaagatccg gaggtccagt tcaattggta cgtggatggg 840
gtggaggtcc acaacgccaa gacgaagccg agagaagaac agttcaactc aacttaccgg 900
gtggtgtccg tgctgaccgt gctgcatcag gattggctca acggaaagga gtacaagtgc 960
aaagtgtcca acaagggcct gcctagctca atcgaaaaga ccatttccaa ggccaagggc 1020
cagccgaggg aaccacaggt ctatactctg ccaccgagcc aagaagagat gaccaagaac 1080
caagtgtccc tgacttgcct ggtcaagggg ttctacccgt cggacatcgc agtggagtgg 1140
gagagcaacg gacagcctga aaacaattac aagaccaccc cgcccgtgct ggatagcgac 1200
ggttccttct tcctttactc gcgcctcacc gtcgacaaga gccggtggca ggagggcaac 1260
gtgttctcct gctccgtgat gcacgaagct ctgcataacc actacactca gaagtccttg 1320
tcgctgagcc tcggaaag 1338
<210> 190
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 190
gacatccaga tgacccagag ccctagctct ttaagcgcct ctgtgggcga tcgtgtgacc 60
atcacttgta gcgccagcag cagcatcagc tacatgaact ggtaccagca gaagcccggc 120
aaggccccca agctgctgat ctacgccacc agcaagctgc ctagcggcgt gccctccaga 180
ttttctggca gcggctctgg caccgacttt actttaacca tcagctcttt acagccagaa 240
gacttcgcca cctactactg ccaccagagg agcagcaccc ctaccttcgg ccaaggtacc 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 191
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 191
caagttcagc tcgtgcagag cggtgctgaa gtgaagaagc ccggtgcctc tgtgaaggtg 60
agctgcaagg ccagcggcta caccttcacc agctacaaca tgcactgggt gagacaagct 120
cccggtcaag gtttagagtg gatgggcgtg atcaaccccg gtgatggtgc taccagctac 180
gcccagaagt tcaagggtcg tgtgaccatg accagagaca ccagcaccag caccgtgtac 240
atggagctga gctctttaag gagcgaggac accgccgtgt actactgcgc tcgtgactac 300
tacggcagca gccctttagg ctattgggga caaggtactt tagtgaccgt ctcgagcgcc 360
tcaaccaaag gaccctccgt gtttcccctc gccccctgtt cccgctccac atccgagtca 420
accgcggcgc tgggctgcct cgtgaaggac tacttccctg agcccgtcac tgtgtcgtgg 480
aactccgggg ccctgacctc cggcgtgcac accttccctg ccgtgcttca atcctccgga 540
ctgtactccc tgtcctcggt ggtcaccgtg ccgtcgagct cgttgggaac caagacttac 600
acttgcaacg tggaccacaa gccaagcaac accaaagtgg acaagagagt cgaatctaag 660
tacggaccgc cctgcccgcc ttgccccgcc cctgagtttc tcggcggtcc tagcgtgttc 720
ctgttcccac ccaagcccaa ggacactctg atgatctccc ggacccctga agtgacctgt 780
gtggtcgtgg acgtgtcgca ggaagatccg gaggtccagt tcaattggta cgtggatggg 840
gtggaggtcc acaacgccaa gacgaagccg agagaagaac agttcaactc aacttaccgg 900
gtggtgtccg tgctgaccgt gctgcatcag gattggctca acggaaagga gtacaagtgc 960
aaagtgtcca acaagggcct gcctagctca atcgaaaaga ccatttccaa ggccaagggc 1020
cagccgaggg aaccacaggt ctatactctg ccaccgagcc aagaagagat gaccaagaac 1080
caagtgtccc tgacttgcct ggtcaagggg ttctacccgt cggacatcgc agtggagtgg 1140
gagagcaacg gacagcctga aaacaattac aagaccaccc cgcccgtgct ggatagcgac 1200
ggttccttct tcctttactc gcgcctcacc gtcgacaaga gccggtggca ggagggcaac 1260
gtgttctcct gctccgtgat gcacgaagct ctgcataacc actacactca gaagtccttg 1320
tcgctgagcc tcggaaag 1338
<210> 192
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 192
gacatccaga tgacccagag ccctagctct ttaagcgcca gcgtgggaga tcgtgtgacc 60
atcacttgtc gtgccagccc cagcatcagc tacatgaact ggtaccagca gaagcccggc 120
aaggccccca agctgctgat ctacgccacc agctctttag cctctggcgt gcctagcaga 180
ttcagcggca gcggaagcgg caccgacttc actttaacca tcagctcttt acagccagaa 240
gacttcgcca cctactactg ccaccagagg agcagcagcc ctaccttcgg ccaaggtacc 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 193
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 193
gacatccaga tgacccagag ccctagctct ttaagcgcct ctgtgggcga tcgtgtgacc 60
atcacttgta gcgccagccc cagcatcagc tacatgaact ggtaccagca gaagcccggc 120
aaggccccca agctgctgat ctacgccaca agctctttac ccagcggcgt gcctagcaga 180
ttcagcggca gcggaagcgg caccgacttc actttaacca tcagctcttt acagccagaa 240
gacttcgcca cctactactg ccaccagaga agcagcagcc ccaccttcgg ccaaggtaca 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 194
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 194
caagttcagc tggtgcagag cggtgctgag gtgaaaaaac ccggtgcttc cgtgaaggtg 60
agctgcaagg ccagcggcta cacctttacc agctacaaca tgcactgggt gagacaagcc 120
cccggtcaag gtttagagtg gatcggcgtg atctaccccg gcaacggcga cacctcttac 180
aaccagaagt tcaagggtcg tgtgaccatg actcgtgaca cctccaccag caccgtgtac 240
atggagctga gctctttaag gagcgaggac acagccgtgt actactgcgc tcgtgactac 300
tacggcagca gccctctggg ctattggggc caaggtactt tagtgaccgt ctcgagcgcc 360
tcaaccaaag gaccctccgt gtttcccctc gccccctgtt cccgctccac atccgagtca 420
accgcggcgc tgggctgcct cgtgaaggac tacttccctg agcccgtcac tgtgtcgtgg 480
aactccgggg ccctgacctc cggcgtgcac accttccctg ccgtgcttca atcctccgga 540
ctgtactccc tgtcctcggt ggtcaccgtg ccgtcgagct cgttgggaac caagacttac 600
acttgcaacg tggaccacaa gccaagcaac accaaagtgg acaagagagt cgaatctaag 660
tacggaccgc cctgcccgcc ttgccccgcc cctgagtttc tcggcggtcc tagcgtgttc 720
ctgttcccac ccaagcccaa ggacactctg atgatctccc ggacccctga agtgacctgt 780
gtggtcgtgg acgtgtcgca ggaagatccg gaggtccagt tcaattggta cgtggatggg 840
gtggaggtcc acaacgccaa gacgaagccg agagaagaac agttcaactc aacttaccgg 900
gtggtgtccg tgctgaccgt gctgcatcag gattggctca acggaaagga gtacaagtgc 960
aaagtgtcca acaagggcct gcctagctca atcgaaaaga ccatttccaa ggccaagggc 1020
cagccgaggg aaccacaggt ctatactctg ccaccgagcc aagaagagat gaccaagaac 1080
caagtgtccc tgacttgcct ggtcaagggg ttctacccgt cggacatcgc agtggagtgg 1140
gagagcaacg gacagcctga aaacaattac aagaccaccc cgcccgtgct ggatagcgac 1200
ggttccttct tcctttactc gcgcctcacc gtcgacaaga gccggtggca ggagggcaac 1260
gtgttctcct gctccgtgat gcacgaagct ctgcataacc actacactca gaagtccttg 1320
tcgctgagcc tcggaaag 1338
<210> 195
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 195
gacatccaga tgacccagag ccctagctct ttaagcgcct ctgtgggcga tcgtgtgacc 60
atcacttgta gcgccagcag cagcatcagc tacatgcact ggtaccagca gaagcccggc 120
aaggccccca agctgctgat ctacgacacc agcaagctgg ccagcggcgt gcctagcaga 180
ttcagcggca gcggaagcgg caccgacttc actttaacca tcagctcttt acagccagaa 240
gacttcgcca cctactactg ccaccagagg agcagctacc ccaccttcgg ccaaggtacc 300
aagctcgaga tcaagagaac tgtggccgcg ccgtcagtgt ttatcttccc tccatcggat 360
gaacagctta agtccggcac ggcgtctgtg gtctgcctgc tcaataactt ttaccctagg 420
gaagctaaag tccaatggaa agtggataac gccctgcagt caggaaacag ccaggaatcg 480
gttaccgaac aggacagcaa ggacagcact tactccttgt cgtcgactct tactctgagc 540
aaggccgatt acgagaagca caaggtctac gcctgcgagg tcacccatca gggactctcg 600
tccccggtga ccaaatcctt caatagaggc gaatgc 636
<210> 196
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 196
caagtgcagc tcgtgcagtc cggagccgaa gtcaagaagc ccggagcgtc agtgaaagtg 60
tcctgcaagg cctcgggcta cactttcaca agctacaaca tgcactgggt cagacaggca 120
cctgggcagg gtctggagtg gatgggagtg atctacccgg gcgacggcgc cacttcctac 180
gcccaaaagt tcaagggccg cgtgaccatg actagggaca cctcgacctc aaccgtgtac 240
atggaactga gctccctgcg gtccgaggat accgccgtgt actattgtgc tcgggactac 300
tacgggtcca gcccactggg atactgggga cagggtaccc ttgtcacggt gtcgtcagct 360
tccaccaagg gcccatccgt cttccccctg gcgccctgct ccaggagcac ctccgagagc 420
acagccgccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac gaagacctac 600
acctgcaatg tagatcacaa gcccagcaac accaaggtgg acaagagagt tgagtccaaa 660
tatggtcccc catgcccacc atgcccagca cctgagttcc tggggggacc atcagtcttc 720
ctgttccccc caaaacccaa ggacactctc atgatctccc ggacccctga ggtcacgtgc 780
gtggtggtgg acgtgagcca ggaagacccc gaggtccagt tcaactggta cgtggatggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agttcaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga acggcaagga gtacaagtgc 960
aaggtctcca acaaaggcct cccgtcctcc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag agccacaggt gtacaccctg cccccatccc aggaggagat gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caggctaacc gtggacaaga gcaggtggca ggaggggaat 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacaca gaagagcctc 1320
tccctgtctc tgggtaaa 1338
<210> 197
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 197
gacatccaaa tgactcagtc cccgtcatcc ctgtcggcat ccgtgggaga cagagtcacc 60
attacgtgca gcgcgagccc gagcatttcc tatatgaact ggtaccagca gaagcccggg 120
aaagccccta agctgttgat ctacgccact tcctcactgg cttccggcgt gccatcccgg 180
ttctcgggtt ccggctcggg aaccgatttt acccttacta tctcgtccct gcaacccgag 240
gacttcgcca cctactactg tcaccagcgc tctagctacc ctacattcgg acagggcacc 300
aagctcgaaa tcaaacgaac tgtggctgca ccatctgtct tcatcttccc gccatctgat 360
gagcagttga aatctggaac tgcctctgtt gtgtgcctgc tgaataactt ctatcccaga 420
gaggccaaag tacagtggaa ggtggataac gccctccaat cgggtaactc ccaggagagt 480
gtcacagagc aggacagcaa ggacagcacc tacagcctca gcagcaccct gacgctgagc 540
aaagcagact acgagaaaca caaagtctac gcctgcgaag tcacccatca gggcctgagc 600
tcgcccgtca caaagagctt caacagggga gagtgt 636
<210> 198
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 198
caagtgcagc tcgtgcagtc cggagccgaa gtcaagaagc ccggagcgtc agtgaaagtg 60
tcctgcaagg cctcgggcta cactttcaca agctacaaca tgcactgggt cagacaggca 120
cctgggcagg gtctggagtg gattggagtg atctacccgg gcgacggctc cacttcctac 180
aaccaaaagt tccagggccg cgtgaccatg actagggaca cctcgacctc aaccgtgtac 240
atggaactga gctccctgcg gtccgaggat accgccgtgt actattgtgc tcgggactac 300
tacgggtcca gcccactggg atactgggga cagggtaccc ttgtcacggt gtcgtcagct 360
tccaccaagg gcccatccgt cttccccctg gcgccctgct ccaggagcac ctccgagagc 420
acagccgccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac gaagacctac 600
acctgcaatg tagatcacaa gcccagcaac accaaggtgg acaagagagt tgagtccaaa 660
tatggtcccc catgcccacc atgcccagca cctgagttcc tggggggacc atcagtcttc 720
ctgttccccc caaaacccaa ggacactctc atgatctccc ggacccctga ggtcacgtgc 780
gtggtggtgg acgtgagcca ggaagacccc gaggtccagt tcaactggta cgtggatggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agttcaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga acggcaagga gtacaagtgc 960
aaggtctcca acaaaggcct cccgtcctcc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag agccacaggt gtacaccctg cccccatccc aggaggagat gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caggctaacc gtggacaaga gcaggtggca ggaggggaat 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacaca gaagagcctc 1320
tccctgtctc tgggtaaa 1338
<210> 199
<211> 635
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 199
acatccaaat gactcagtcc ccgtcatccc tgtcggcatc cgtgggagac agagtcacca 60
ttacgtgcag cgcgagctca agcatttcct atatgaactg gtaccagcag aagcccggga 120
aagcccctaa gctgttgatc tacgccactt ccaagctgcc gtccggcgtg ccatcccggt 180
tctcgggttc cggctcggga accgatttta cccttactat ctcgtccctg caacccgagg 240
acttcgccac ctactactgt caccagcgct ctagcacccc tacattcgga cagggcacca 300
agctcgaaat caaacgaact gtggctgcac catctgtctt catcttcccg ccatctgatg 360
agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc tatcccagag 420
aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc caggagagtg 480
tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg acgctgagca 540
aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag ggcctgagct 600
cgcccgtcac aaagagcttc aacaggggag agtgt 635
<210> 200
<211> 1338
<212> ДНК
<213> Искусственная последовательность
<220>
<223> HC
<400> 200
caagtgcagc tcgtgcagtc cggagccgaa gtcaagaagc ccggagcgtc agtgaaagtg 60
tcctgcaagg cctcgggcta cactttcaca agctacaaca tgcactgggt cagacaggca 120
cctgggcagg gtctggagtg gatgggagtg atcaacccgg gcgacggcgc cacttcctac 180
gcccaaaagt tcaagggccg cgtgaccatg actagggaca cctcgacctc aaccgtgtac 240
atggaactga gctccctgcg gtccgaggat accgccgtgt actattgtgc tcgggactac 300
tacgggtcca gcccactggg atactgggga cagggtaccc ttgtcacggt gtcgtcagct 360
tccaccaagg gcccatccgt cttccccctg gcgccctgct ccaggagcac ctccgagagc 420
acagccgccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac gaagacctac 600
acctgcaatg tagatcacaa gcccagcaac accaaggtgg acaagagagt tgagtccaaa 660
tatggtcccc catgcccacc atgcccagca cctgagttcc tggggggacc atcagtcttc 720
ctgttccccc caaaacccaa ggacactctc atgatctccc ggacccctga ggtcacgtgc 780
gtggtggtgg acgtgagcca ggaagacccc gaggtccagt tcaactggta cgtggatggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agttcaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga acggcaagga gtacaagtgc 960
aaggtctcca acaaaggcct cccgtcctcc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag agccacaggt gtacaccctg cccccatccc aggaggagat gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caggctaacc gtggacaaga gcaggtggca ggaggggaat 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacaca gaagagcctc 1320
tccctgtctc tgggtaaa 1338
<210> 201
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 201
gacatccaaa tgactcagtc cccgtcatcc ctgtcggcat ccgtgggaga cagagtcacc 60
attacgtgcc gcgcgagccc gagcatttcc tatatgaact ggtaccagca gaagcccggg 120
aaagccccta agctgttgat ctacgccact tcctcactgg cttccggcgt gccatcccgg 180
ttctcgggtt ccggctcggg aaccgatttt acccttacta tctcgtccct gcaacccgag 240
gacttcgcca cctactactg tcaccagcgc tctagcagcc ctacattcgg acagggcacc 300
aagctcgaaa tcaaacgaac tgtggctgca ccatctgtct tcatcttccc gccatctgat 360
gagcagttga aatctggaac tgcctctgtt gtgtgcctgc tgaataactt ctatcccaga 420
gaggccaaag tacagtggaa ggtggataac gccctccaat cgggtaactc ccaggagagt 480
gtcacagagc aggacagcaa ggacagcacc tacagcctca gcagcaccct gacgctgagc 540
aaagcagact acgagaaaca caaagtctac gcctgcgaag tcacccatca gggcctgagc 600
tcgcccgtca caaagagctt caacagggga gagtgt 636
<210> 202
<211> 636
<212> ДНК
<213> Искусственная последовательность
<220>
<223> LC
<400> 202
gacatccaaa tgactcagtc cccgtcatcc ctgtcggcat ccgtgggaga cagagtcacc 60
attacgtgca gcgcgagccc gagcatttcc tatatgaact ggtaccagca gaagcccggg 120
aaagccccta agctgttgat ctacgccact tcctcactgc cgtccggcgt gccatcccgg 180
ttctcgggtt ccggctcggg aaccgatttt acccttacta tctcgtccct gcaacccgag 240
gacttcgcca cctactactg tcaccagcgc tctagcagcc ctacattcgg acagggcacc 300
aagctcgaaa tcaaacgaac tgtggctgca ccatctgtct tcatcttccc gccatctgat 360
gagcagttga aatctggaac tgcctctgtt gtgtgcctgc tgaataactt ctatcccaga 420
gaggccaaag tacagtggaa ggtggataac gccctccaat cgggtaactc ccaggagagt 480
gtcacagagc aggacagcaa ggacagcacc tacagcctca gcagcaccct gacgctgagc 540
aaagcagact acgagaaaca caaagtctac gcctgcgaag tcacccatca gggcctgagc 600
tcgcccgtca caaagagctt caacagggga gagtgt 636
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ТЕРАПЕВТИЧЕСКИЕ АНТИТЕЛА ПРОТИВ ЛИГАНДА CD40 | 2018 |
|
RU2770209C2 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2837821C1 |
| АНТИТЕЛО К CLDN-18.2 И ЕГО ПРИМЕНЕНИЕ | 2021 |
|
RU2829997C1 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2808030C2 |
| МУЛЬТИСПЕЦИФИЧЕСКАЯ АНТИГЕНСВЯЗЫВАЮЩАЯ МОЛЕКУЛА, ОБЛАДАЮЩАЯ ЗАМЕЩАЮЩЕЙ ФУНКЦИОНАЛЬНОЙ АКТИВНОСТЬЮ КОФАКТОРА КОАГУЛИРУЮЩЕГО ФАКТОРА КРОВИ VIII, И ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ, СОДЕРЖАЩАЯ УКАЗАННУЮ МОЛЕКУЛУ В КАЧЕСТВЕ АКТИВНОГО ИНГРЕДИЕНТА | 2018 |
|
RU2812909C2 |
| ПРОКОАГУЛЯНТНЫЕ АНТИТЕЛА | 2018 |
|
RU2810094C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ, СОДЕРЖАЩИЕ АНТИ-FAP КЛОН 212 | 2019 |
|
RU2799429C2 |
| АМАТОКСИНОВЫЕ КОНЪЮГАТЫ АНТИТЕЛА С ЛЕКАРСТВЕННЫМ СРЕДСТВОМ И ИХ ПРИМЕНЕНИЕ | 2020 |
|
RU2826004C2 |
| АНТИ-CD137 АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2829536C2 |
| АНТИТЕЛО ПРОТИВ СТОЛБНЯЧНОГО ТОКСИНА И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2815280C1 |
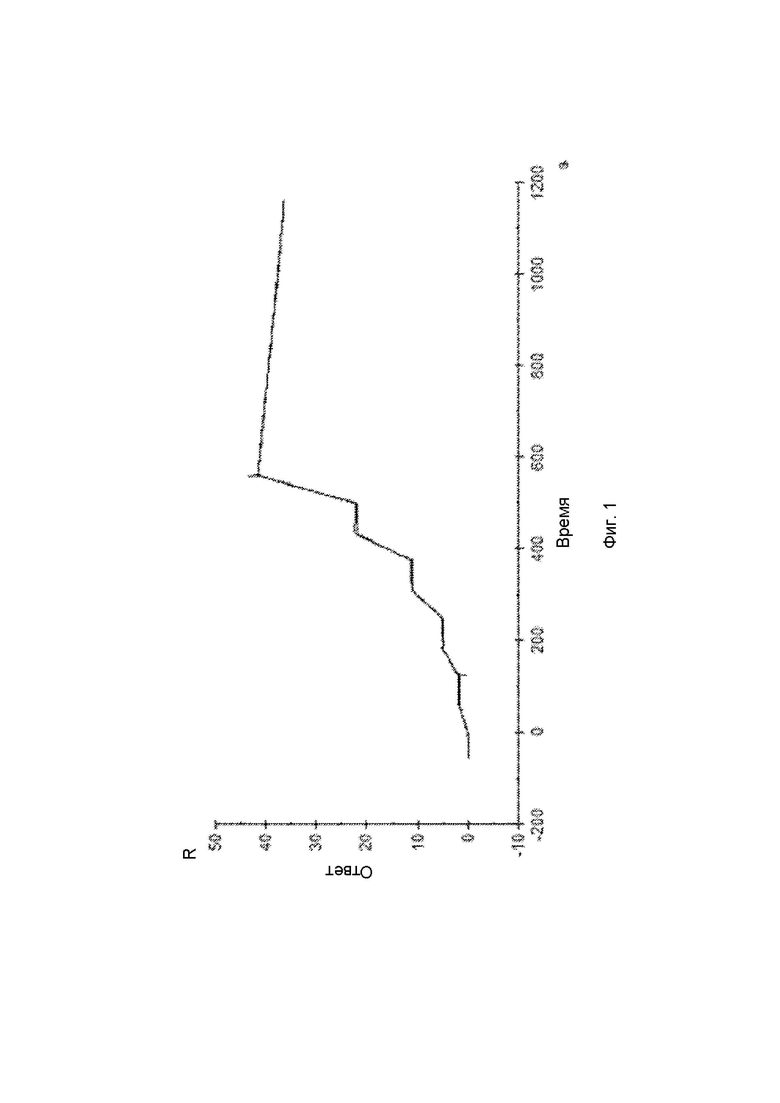
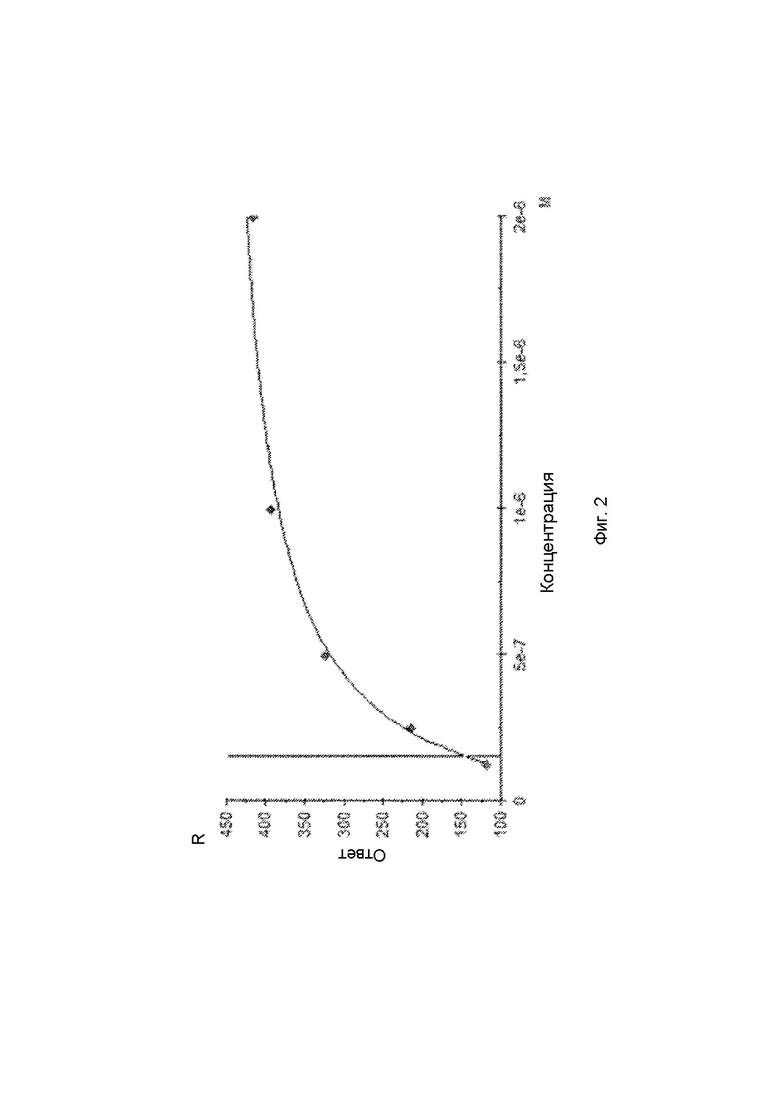

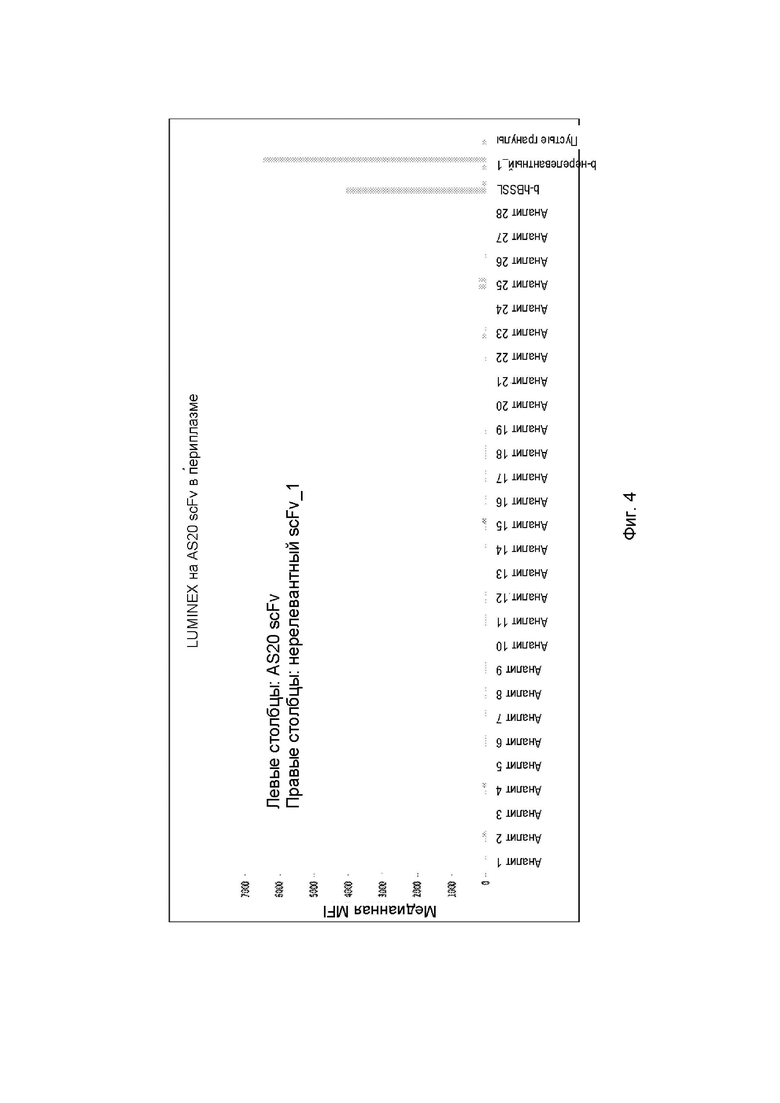
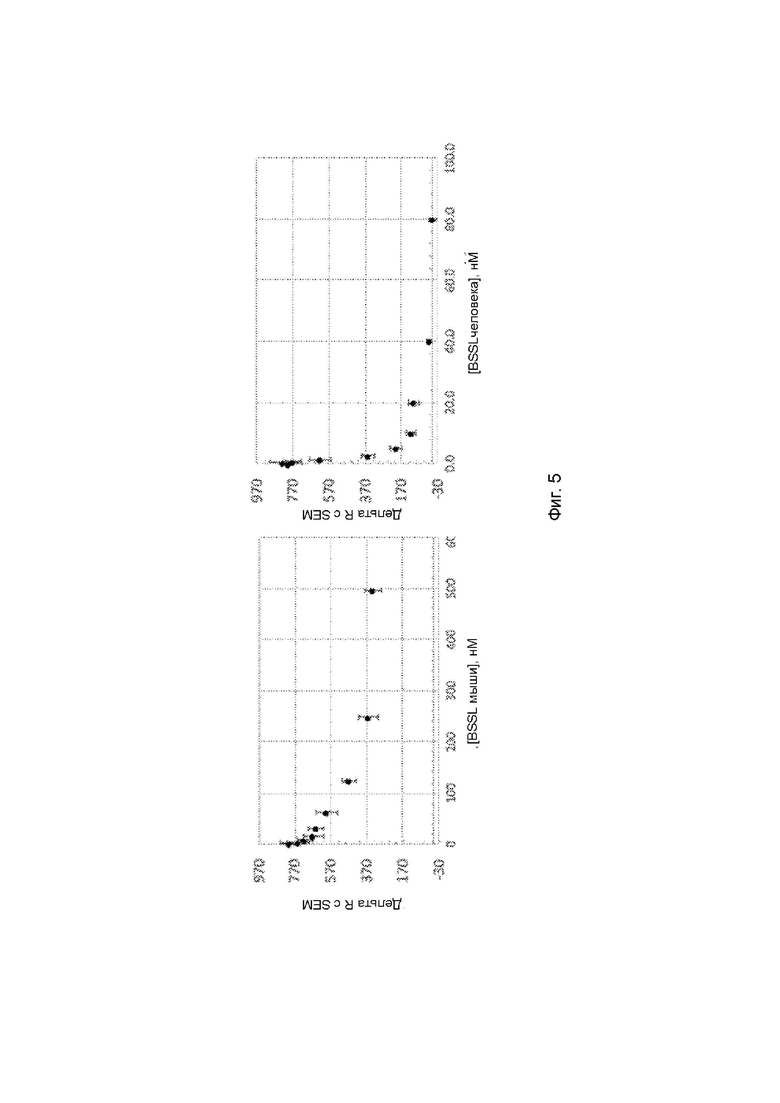

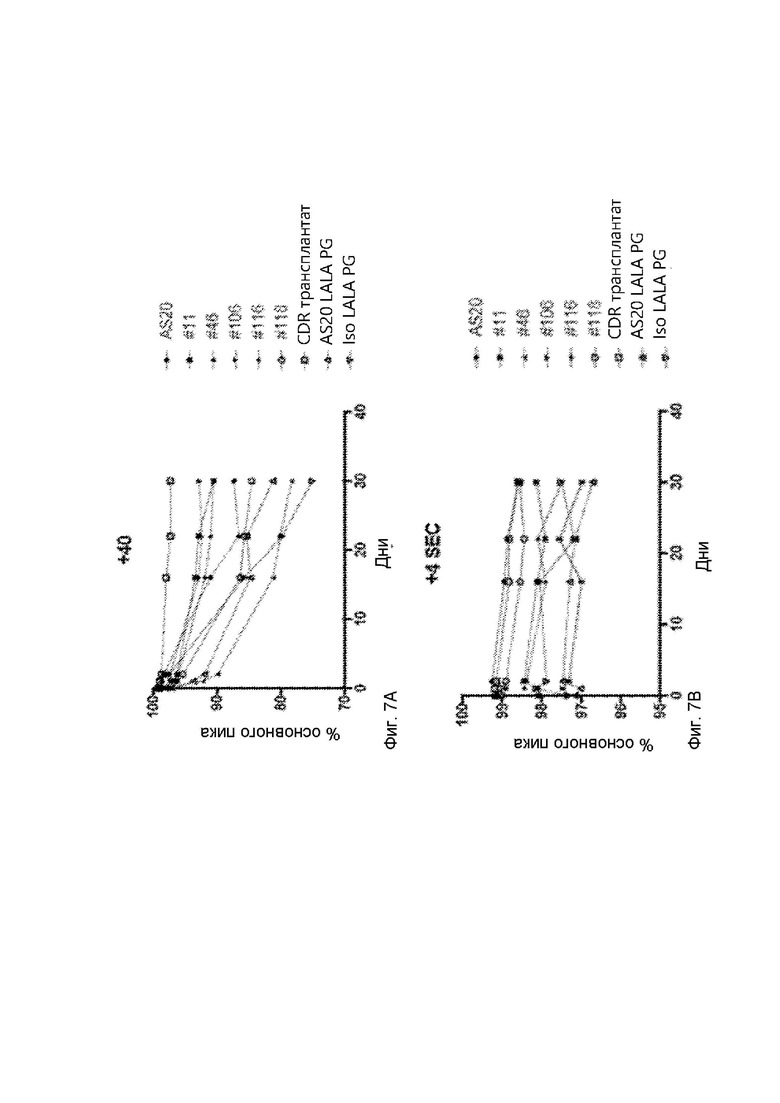
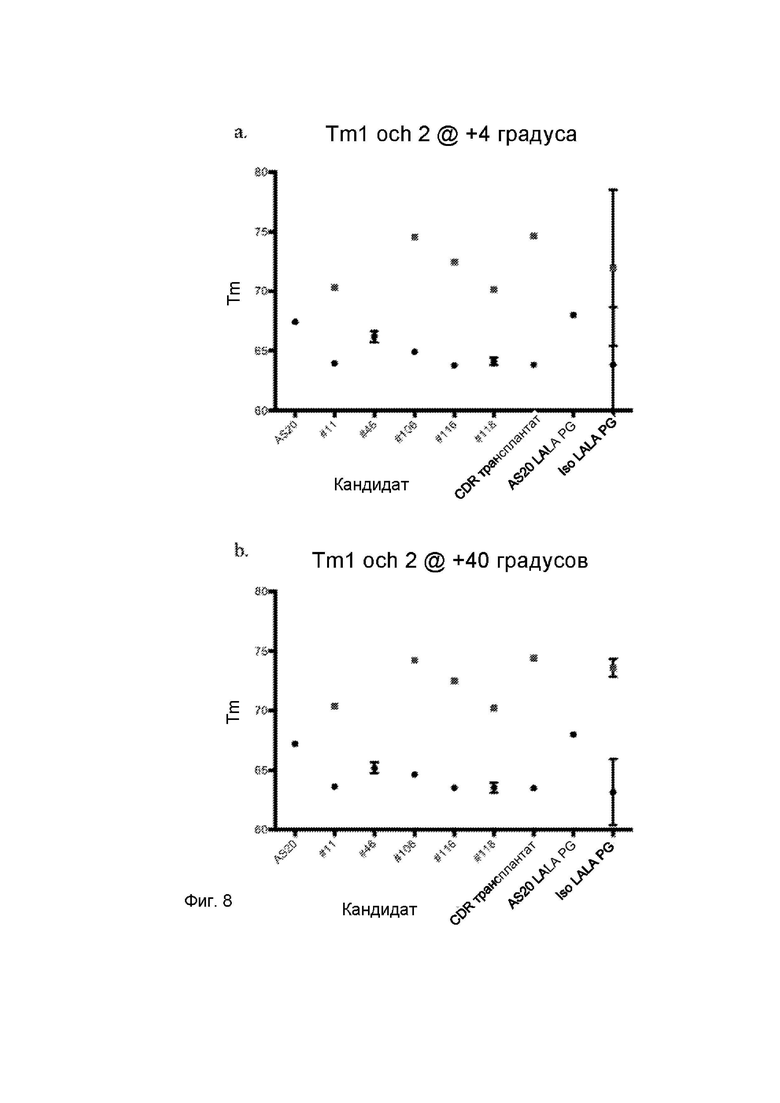
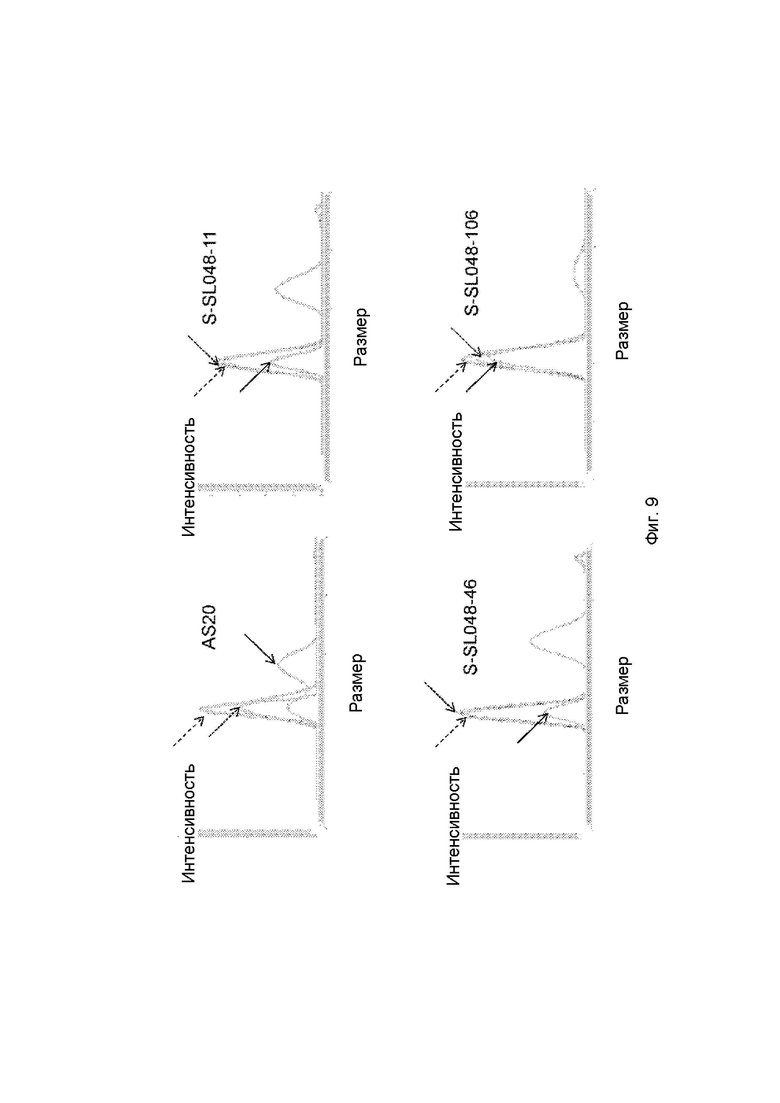
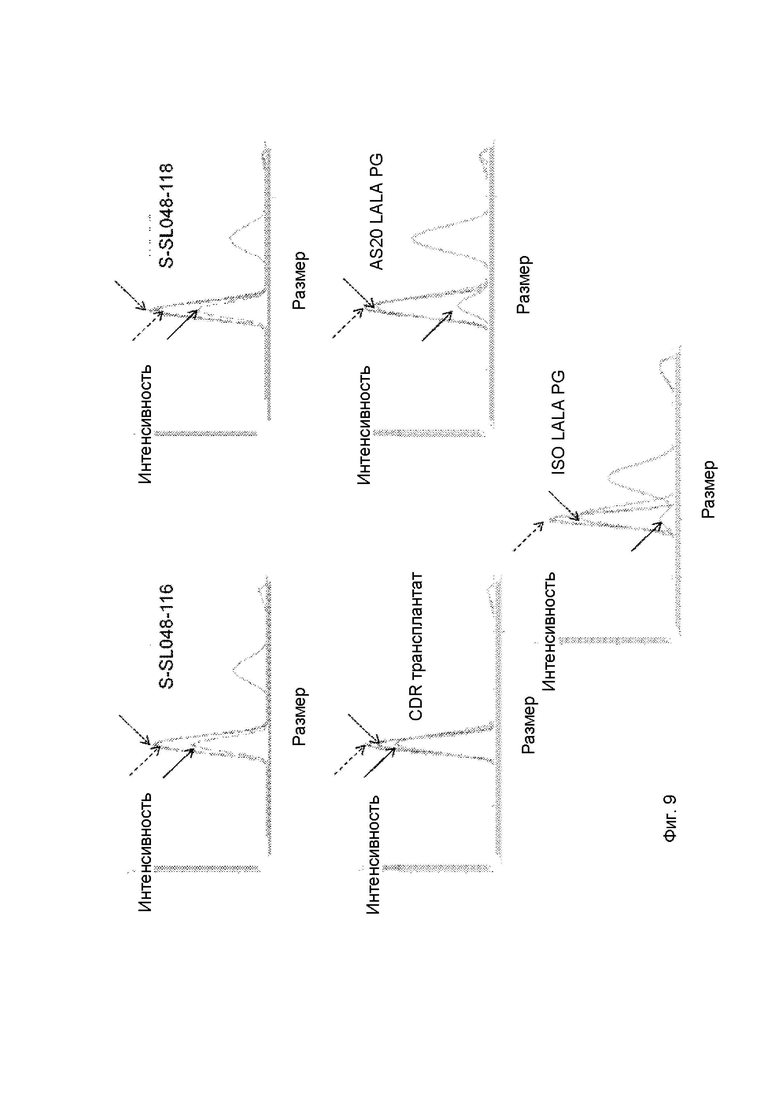
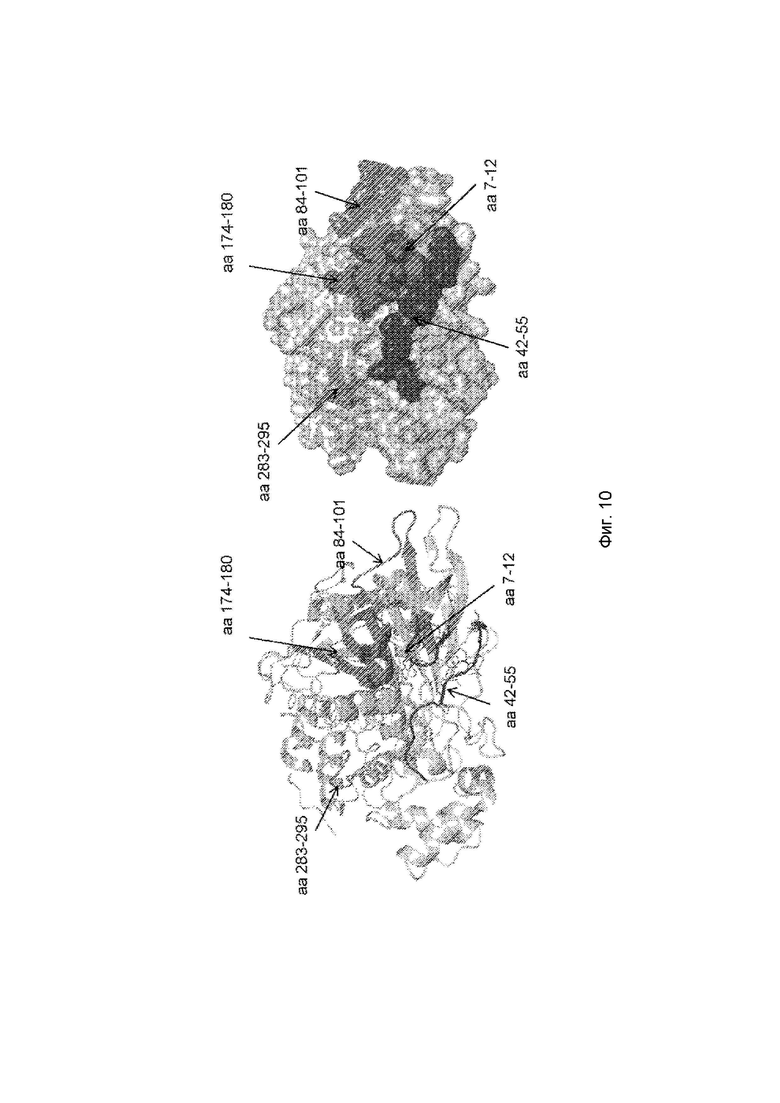

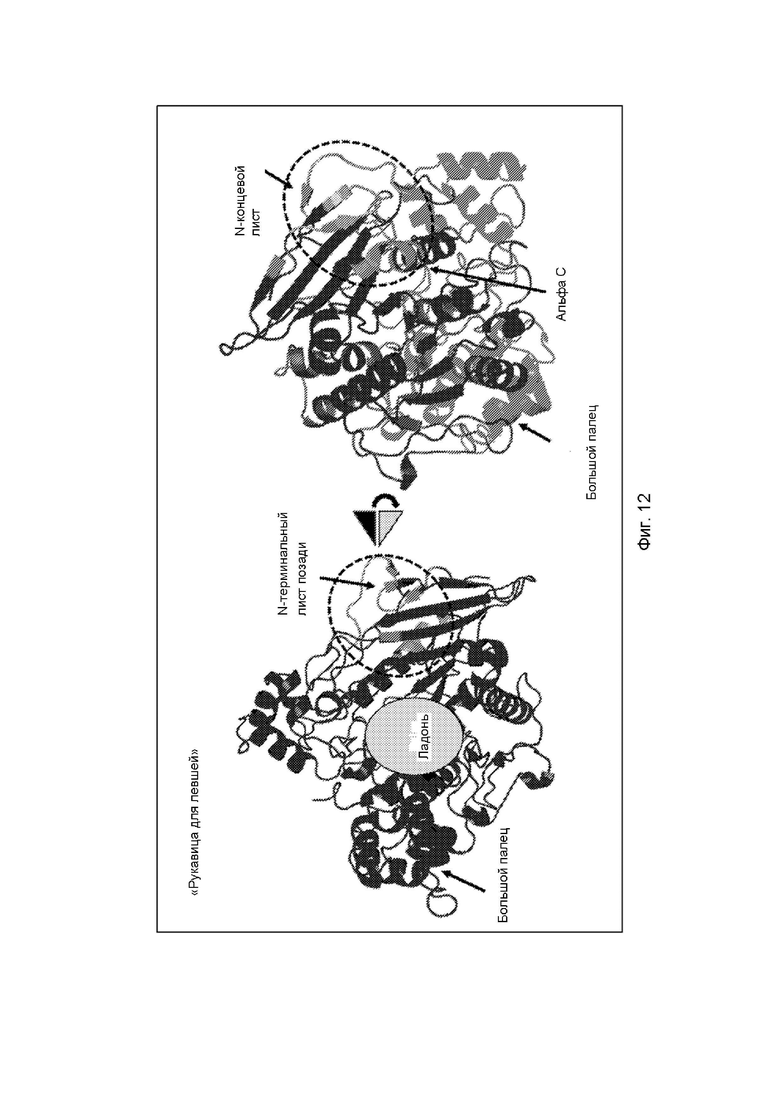
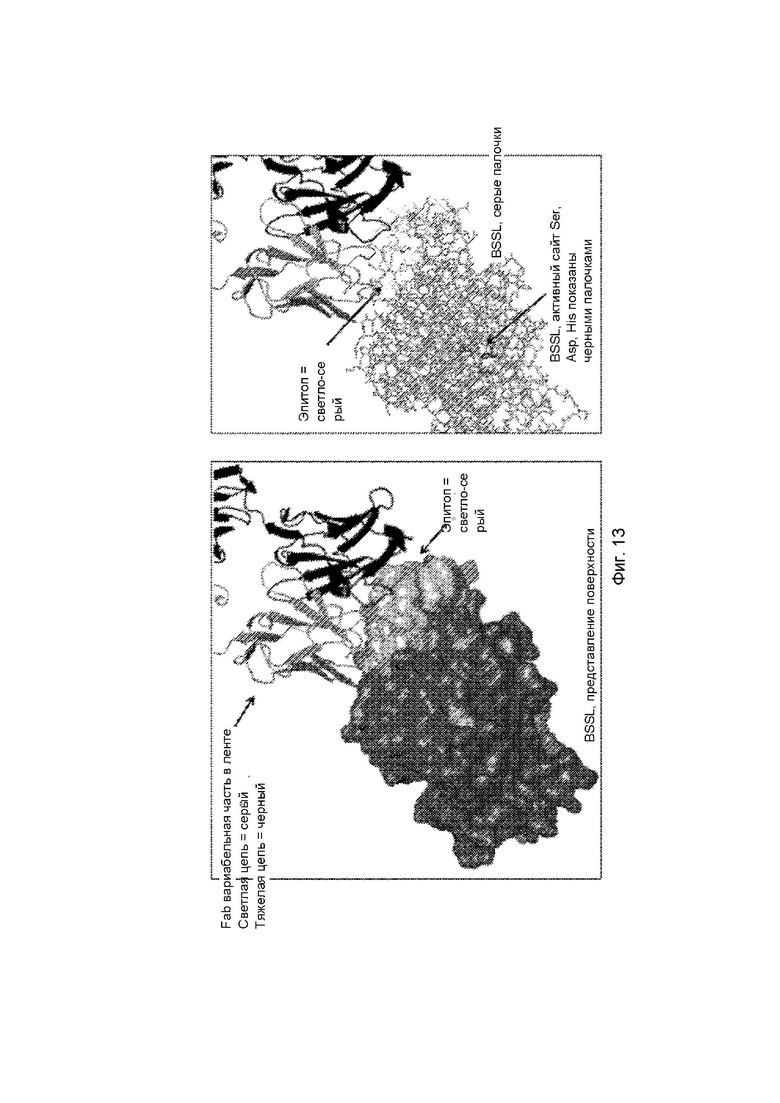
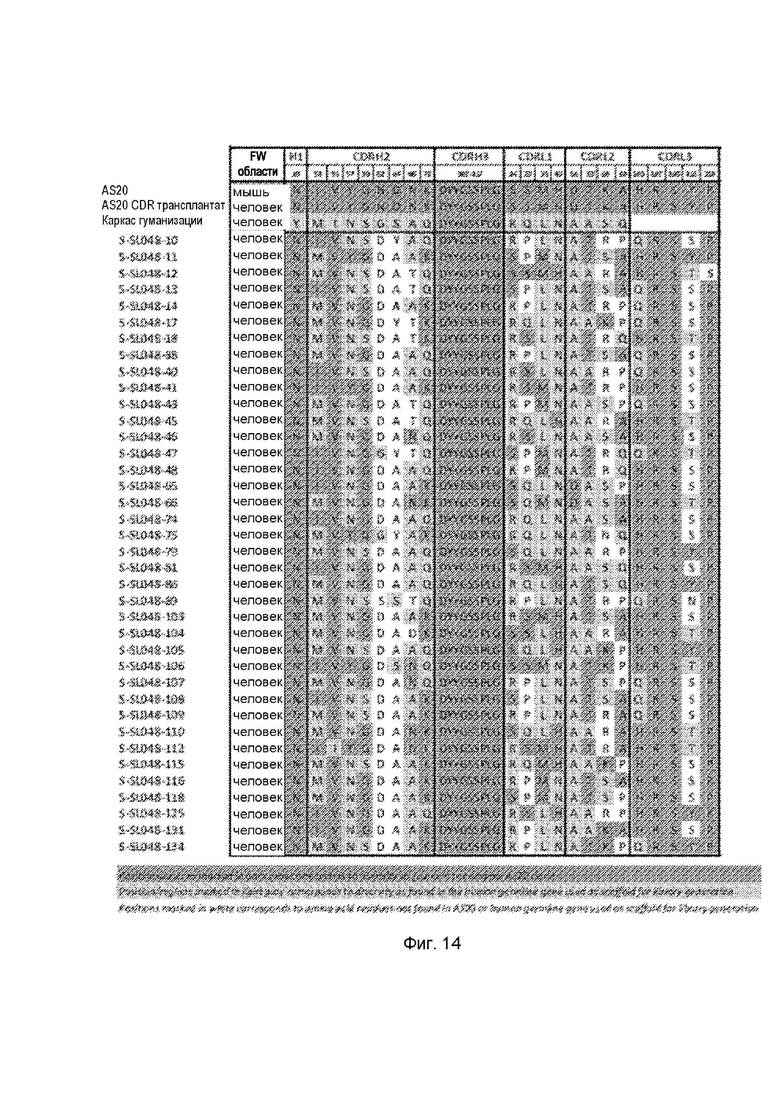
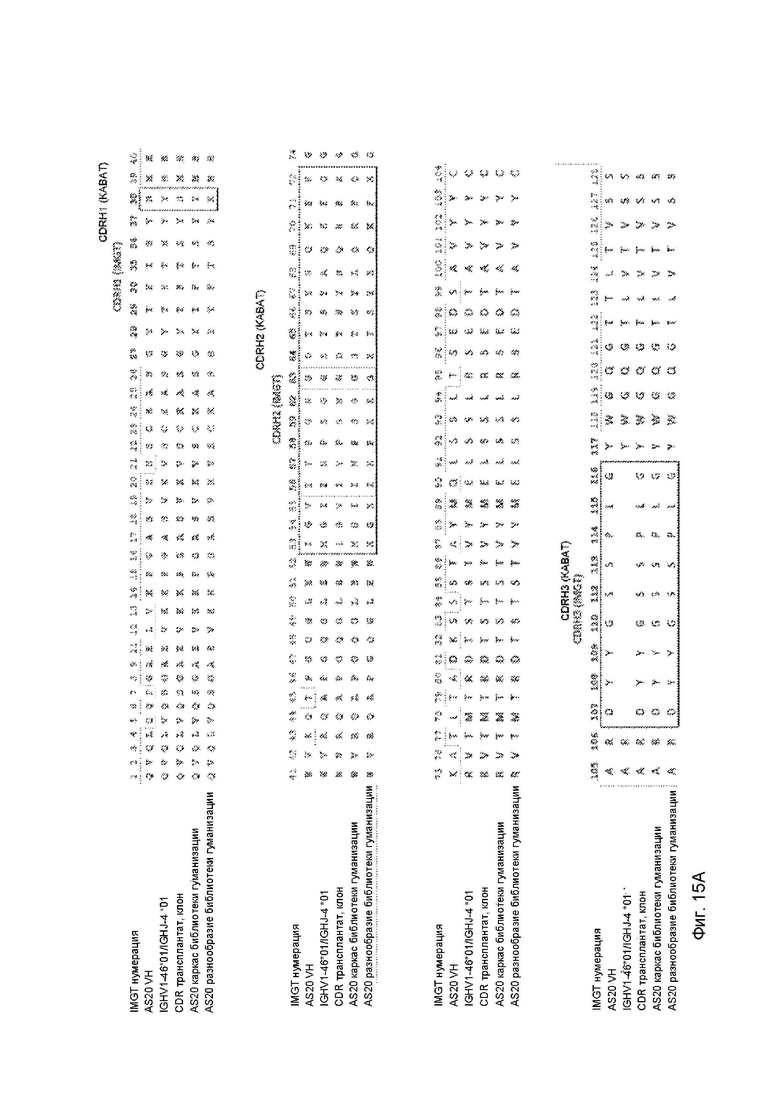

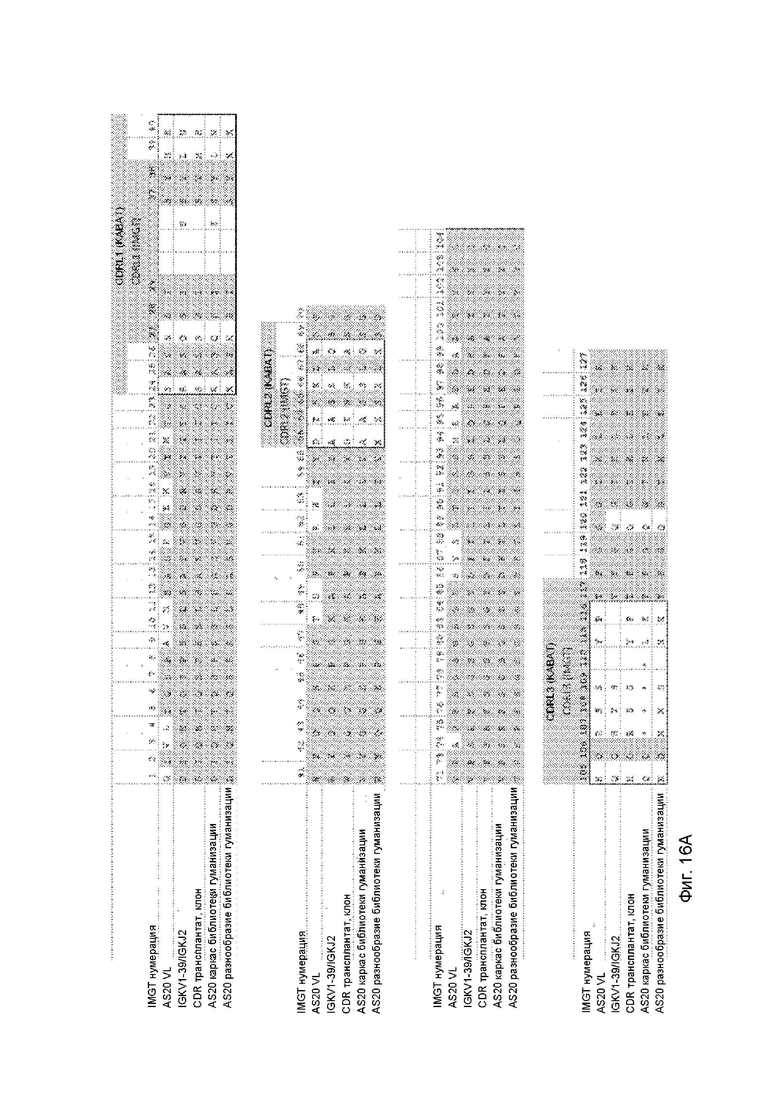
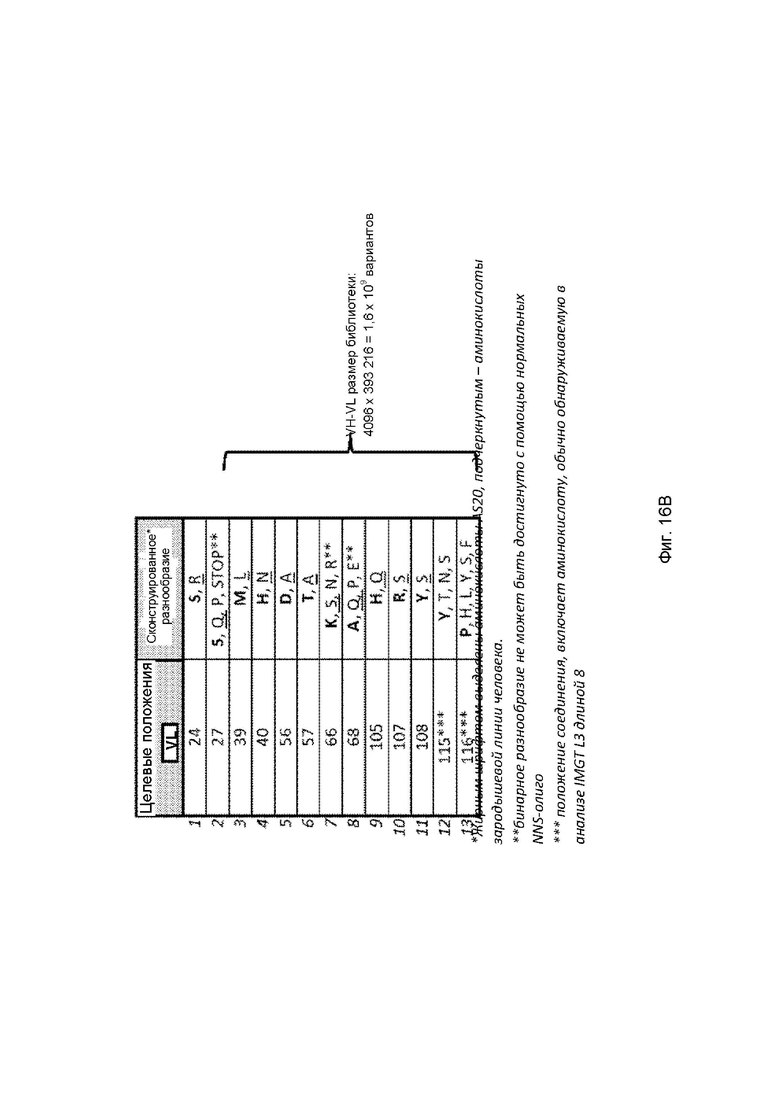
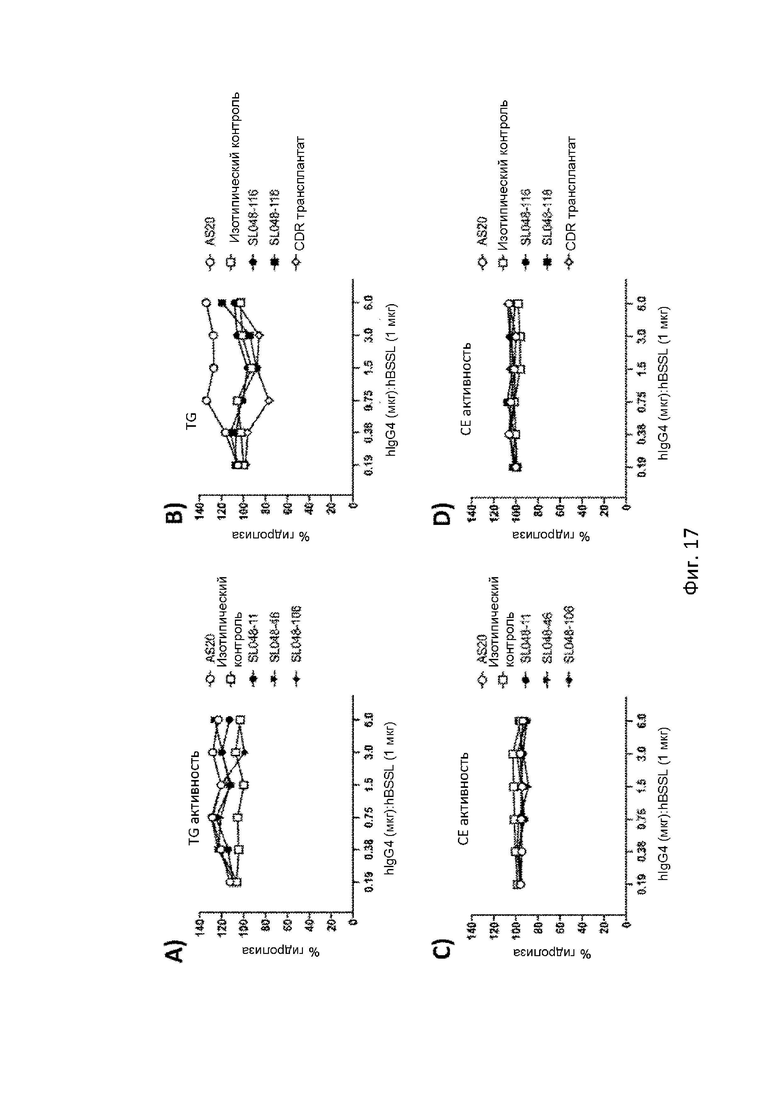
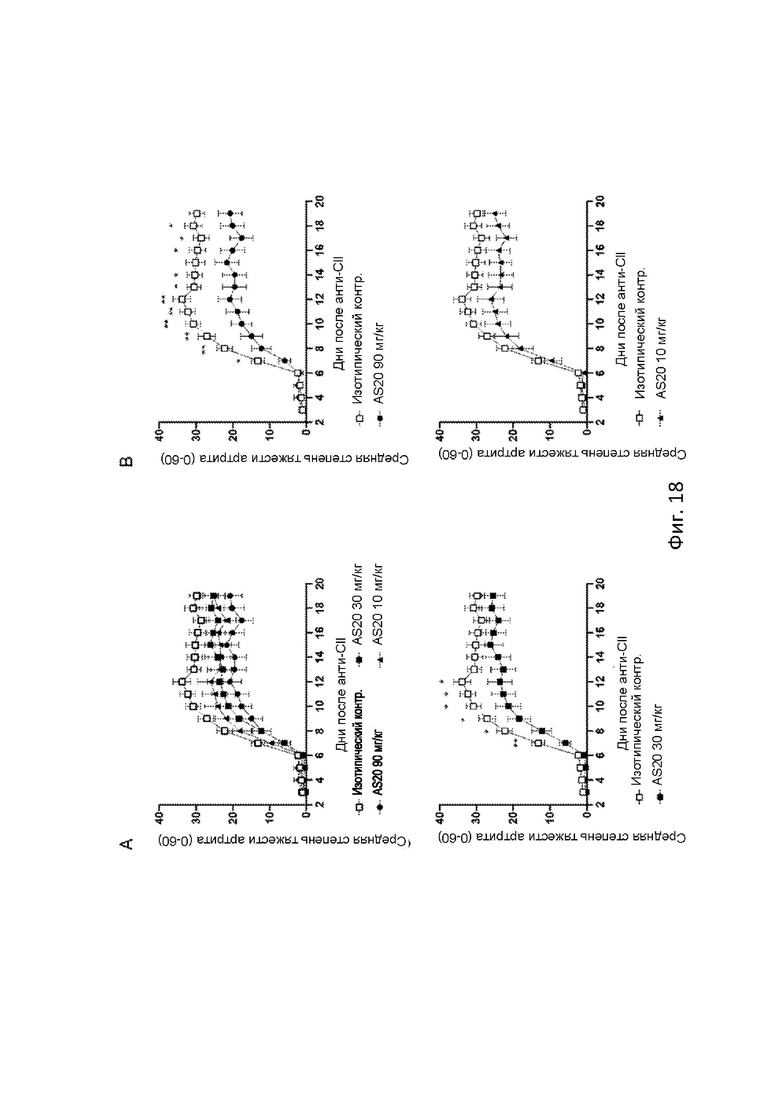

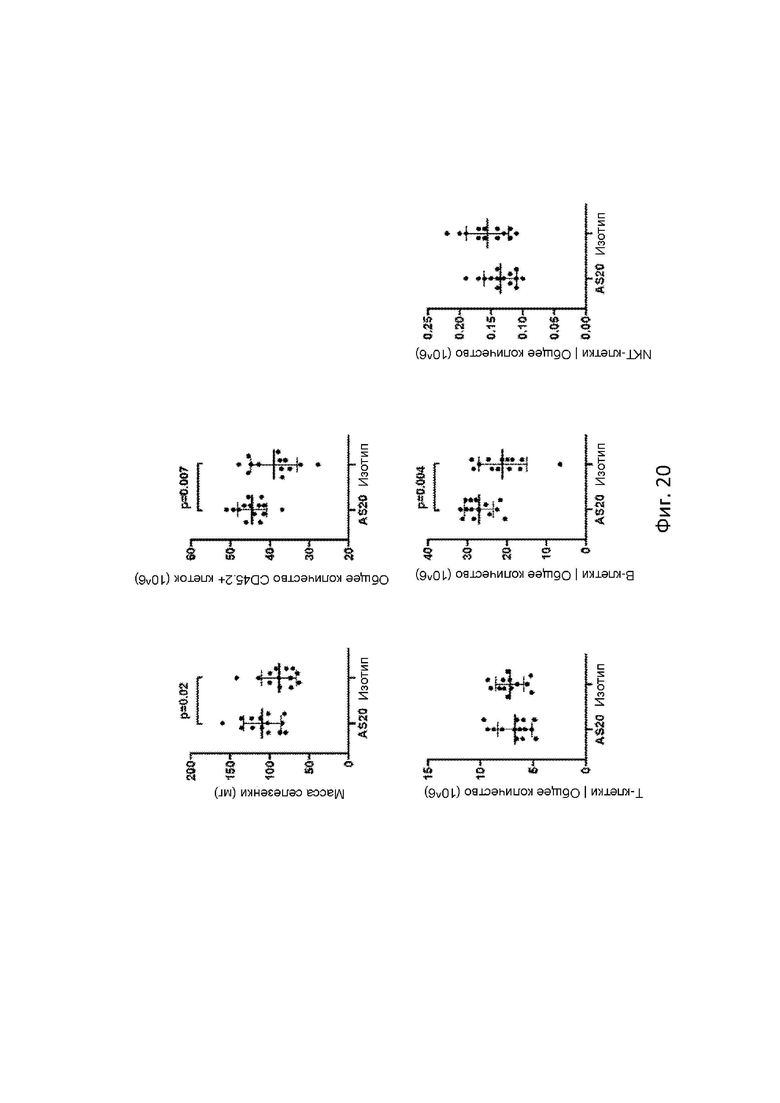
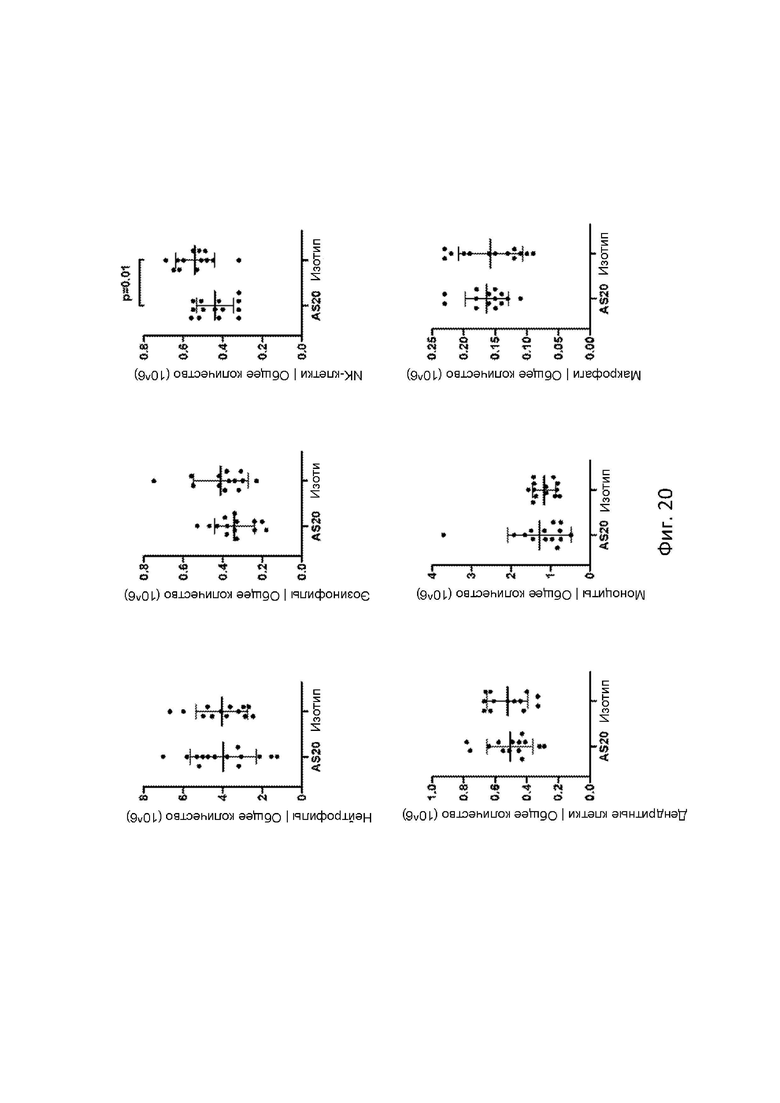
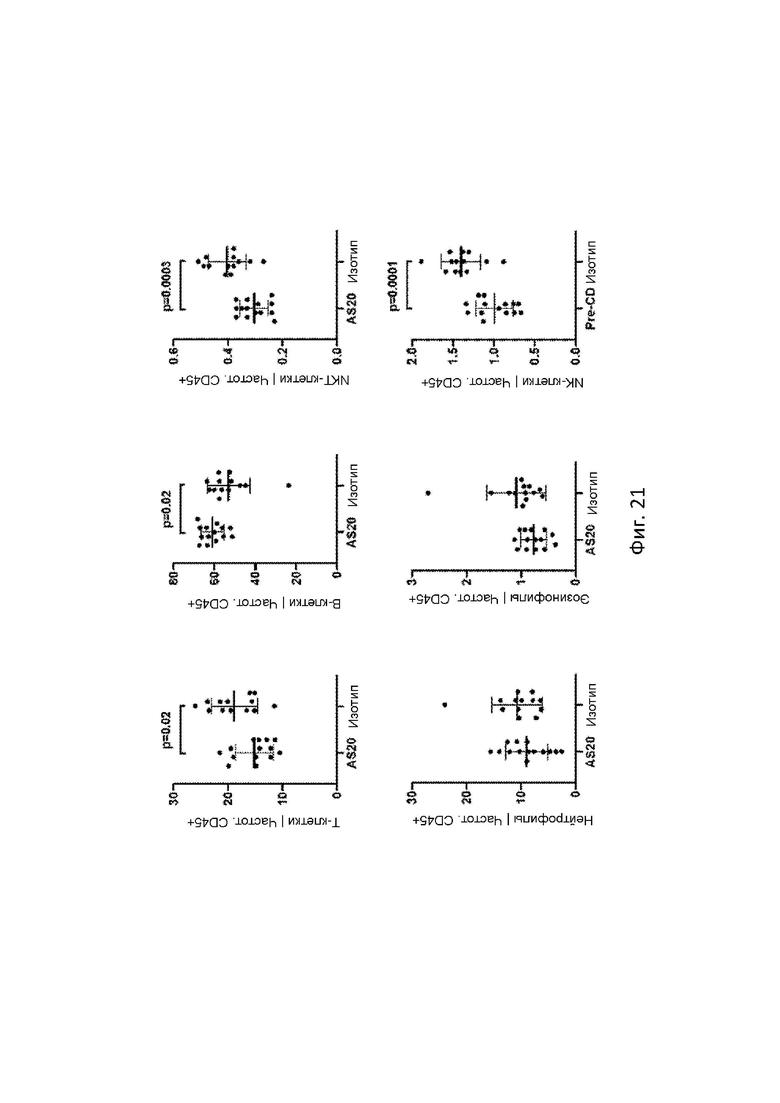
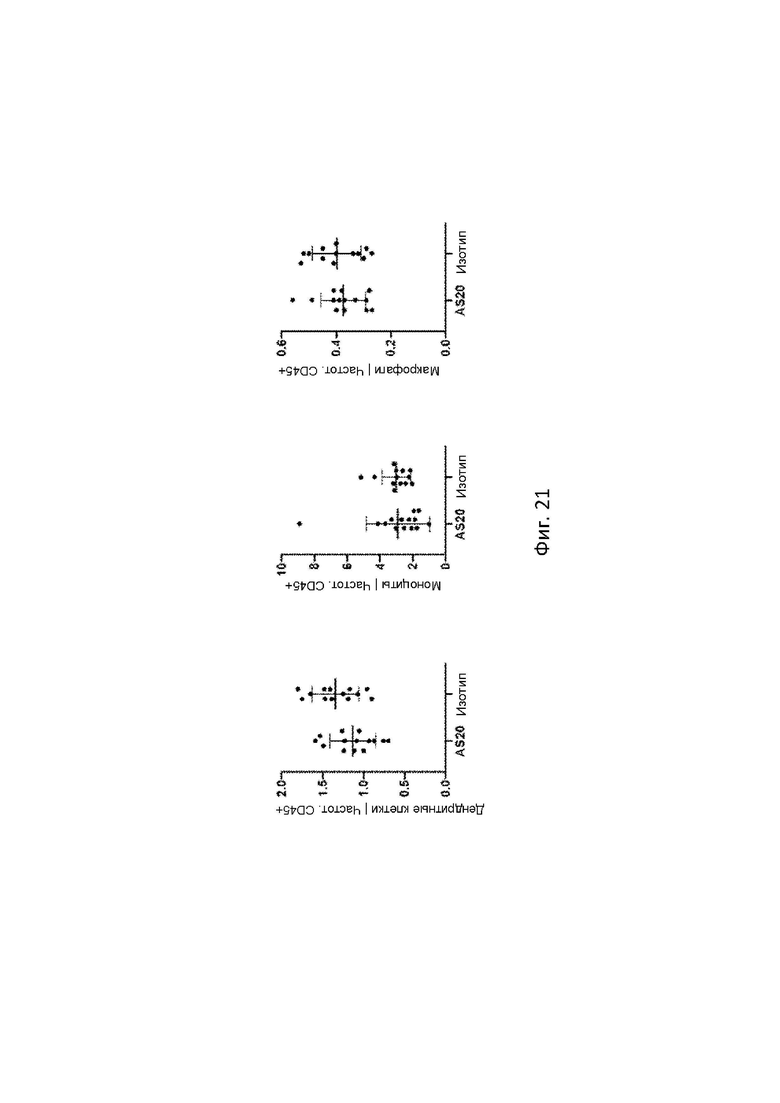
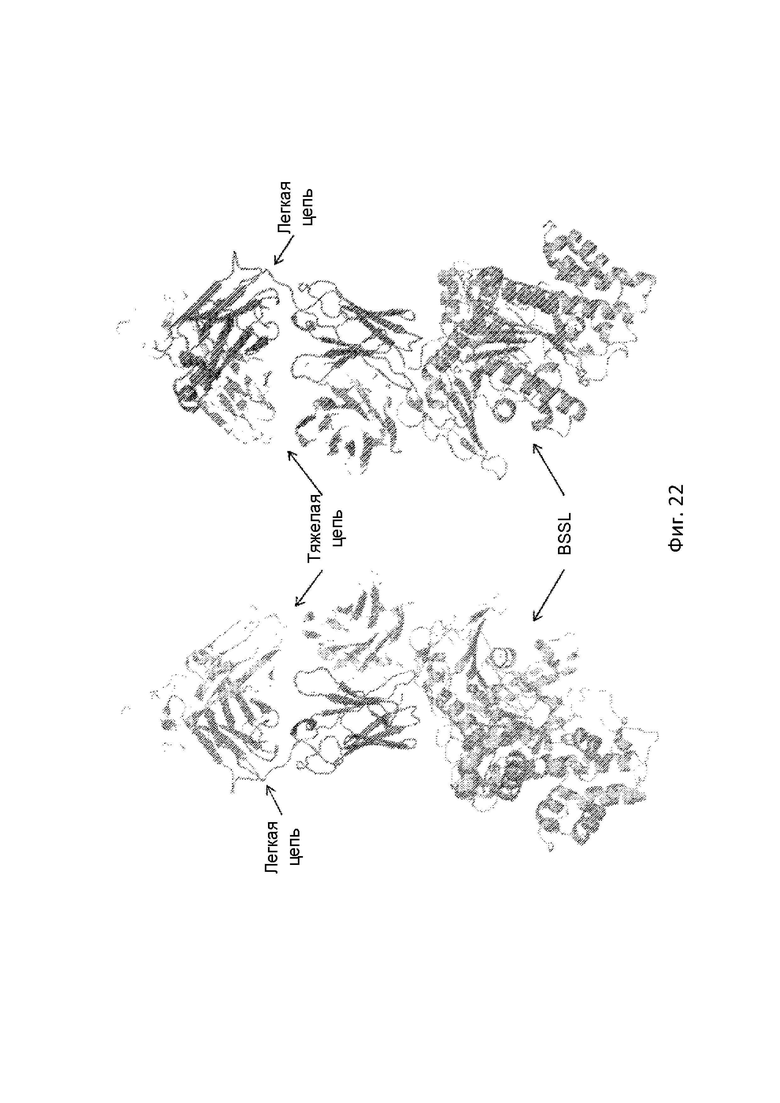
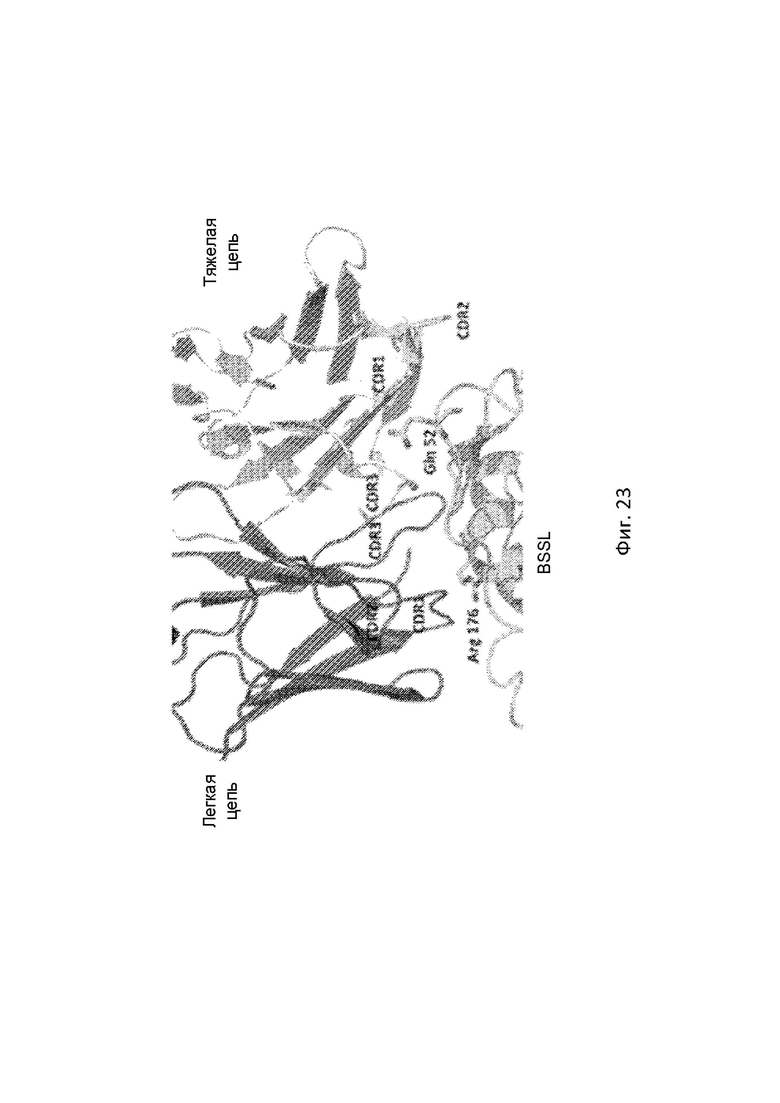
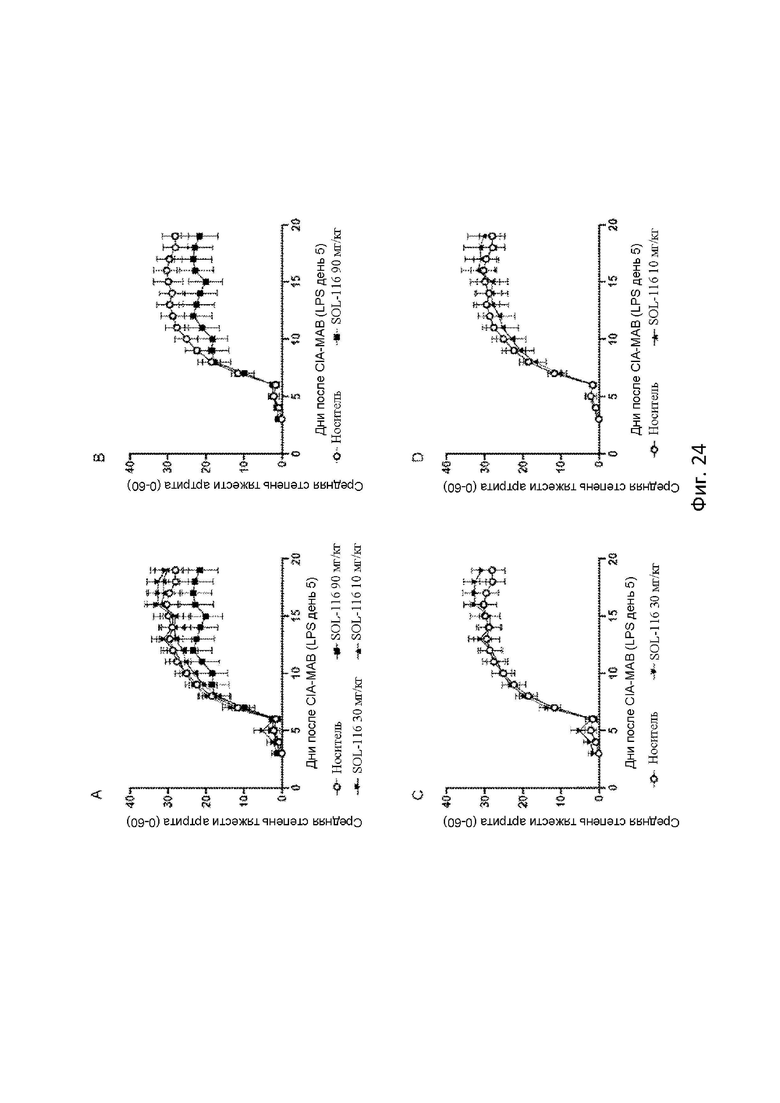
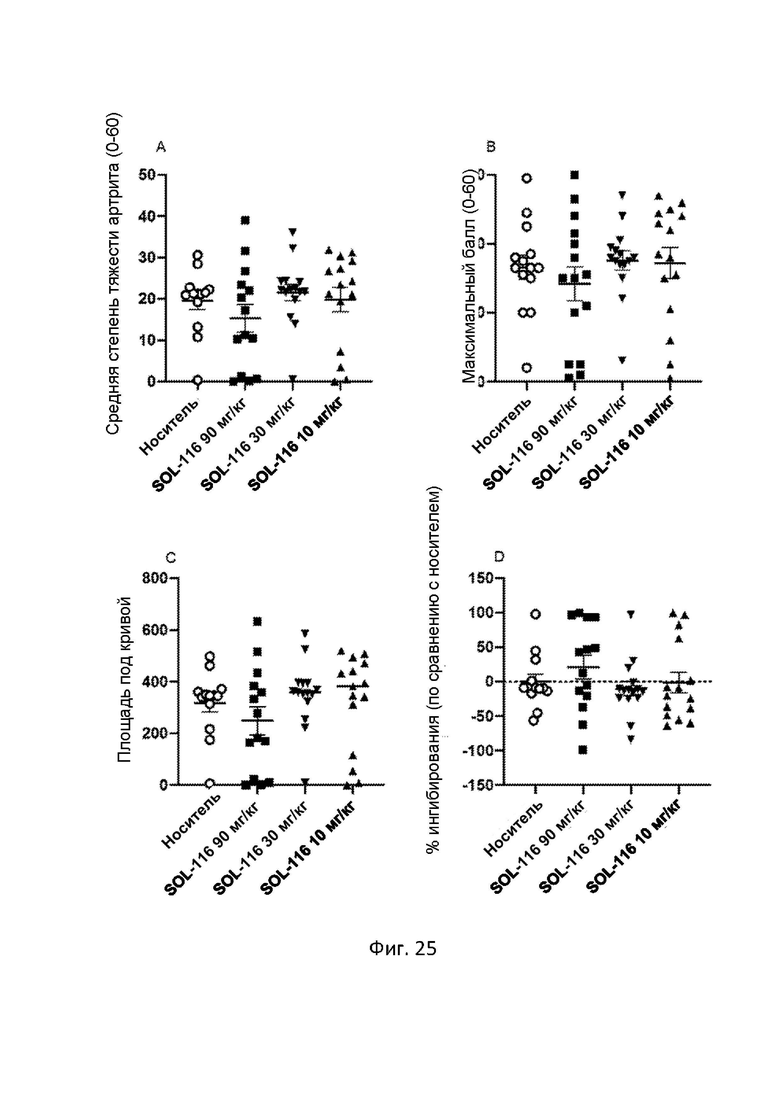
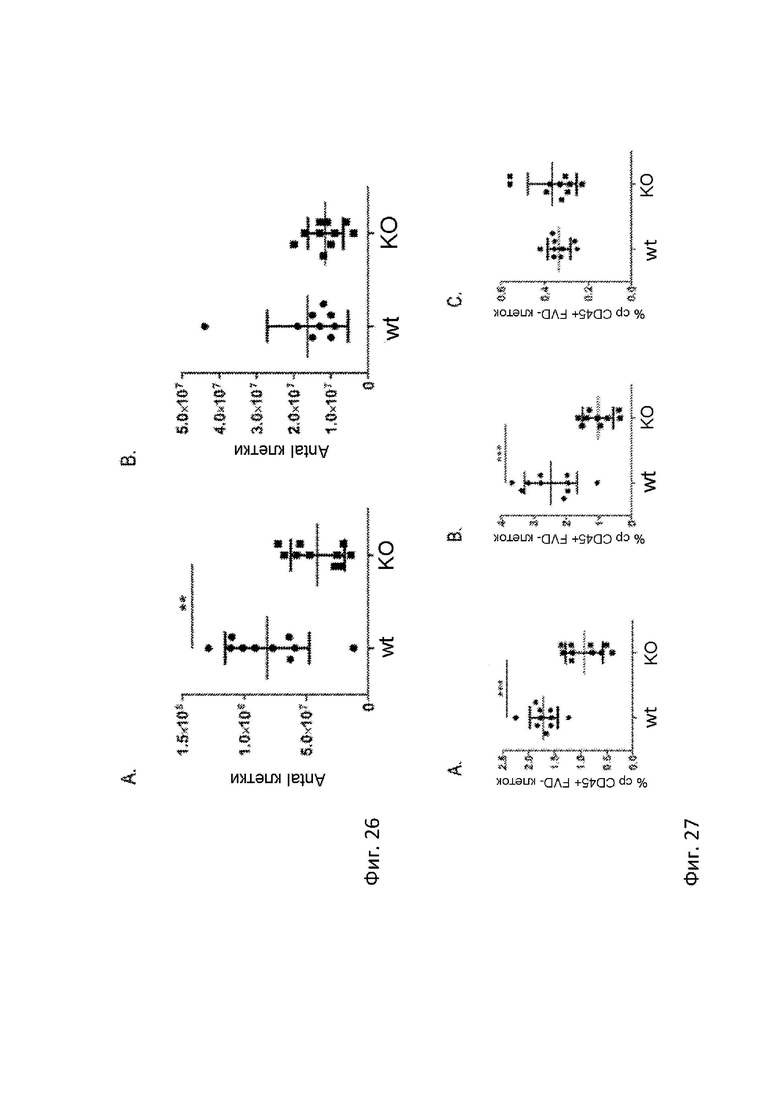
Изобретение относится к области биотехнологии. Предложены выделенные антитела и их антигенсвязывающие фрагменты, которые связываются с липазой человека, стимулируемой солями желчных кислот (hBSSL). Антитела и их антигенсвязывающие фрагменты связываются с эпитопом, расположенным в N-концевой части hBSSL и идентифицированным как содержащий аминокислотные остатки 7-12 и аминокислотные остатки 42-55. Также предложены полинуклеотиды, кодирующие указанные антитела или их фрагменты, экспрессионные векторы и клетки-хозяева, содержащие указанные полинуклеотиды, способ получения антител. Кроме того, изобретение также относится к медицинскому применению антител и/или их антигенсвязывающих фрагментов, в частности, при лечении или диагностике воспалительных состояний, а также к родственным фармацевтическим композициям. Изобретение обеспечивает специфическое связывание BSSL без значительного влияния на ферментативную активность и с низким риском иммуногенности. 10 н. и 31 з.п. ф-лы, 33 ил., 29 табл., 24 пр.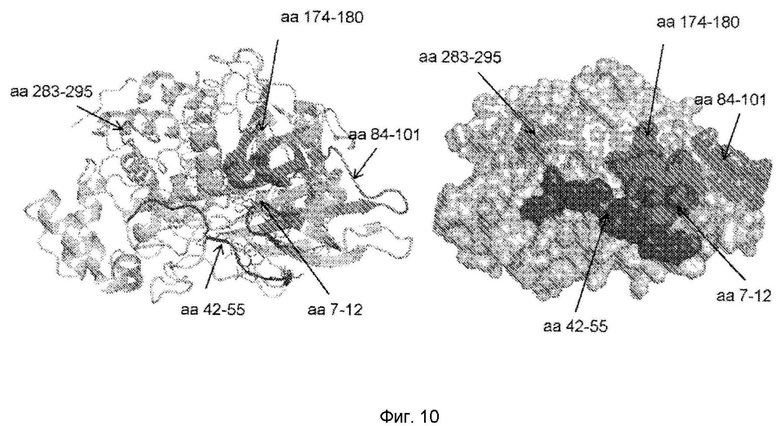
1. Выделенное антитело или его антигенсвязывающий фрагмент, специфически связывающийся с липазой, стимулированной солями желчных кислот (BSSL), и содержащий:
три определяющих комплементарность области (CDR) вариабельной области тяжелой цепи (HCVR) (HCDR); и
три CDR вариабельной области легкой цепи (LCVR) (LCDR), при этом
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7,
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 8,
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9,
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 10,
вторая LCDR состоит из аминокислотной последовательности ATS, и третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 11; или
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7,
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 18,
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9,
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 10,
вторая LCDR состоит из аминокислотной последовательности ATS, и третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 21; или
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7,
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 8,
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9,
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 20,
вторая LCDR состоит из аминокислотной последовательности AAS, и третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 11; или
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7,
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 19,
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9,
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 20,
вторая LCDR состоит из аминокислотной последовательности ATS, и третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 22.
2. Выделенное антитело или его антигенсвязывающий фрагмент по п. 1, в котором
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7;
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 8;
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9;
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 10;
вторая LCDR состоит из аминокислотной последовательности ATS; и
третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
3. Выделенное антитело или его антигенсвязывающий фрагмент по п. 2, причем выделенное антитело или его антигенсвязывающий фрагмент содержит:
удлиненную вторую HCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 12;
удлиненную первую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 14 и
удлиненную вторую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 15.
4. Выделенное антитело или его антигенсвязывающий фрагмент по п. 2 или 3, в котором
HCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 36 и/или
LCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 37.
5. Выделенное антитело или его антигенсвязывающий фрагмент по п. 2, причем выделенное антитело или его антигенсвязывающий фрагмент содержит:
удлиненную вторую HCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 12;
удлиненную первую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 16 и
удлиненную вторую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 17.
6. Выделенное антитело или его антигенсвязывающий фрагмент по п. 2 или 5, в котором
HCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 36 и/или
LCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 38.
7. Выделенное антитело или его антигенсвязывающий фрагмент по п. 1, в котором
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7;
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 18;
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9;
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 10;
вторая LCDR состоит из аминокислотной последовательности ATS; и
третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 21.
8. Выделенное антитело или его антигенсвязывающий фрагмент по п. 7, причем выделенное антитело или его антигенсвязывающий фрагмент содержит:
удлиненную вторую HCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 23;
удлиненную первую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 16 и
удлиненную вторую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 15.
9. Выделенное антитело или его антигенсвязывающий фрагмент по п. 7 или 8, в котором
HCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 30 и/или
LCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 31.
10. Выделенное антитело или его антигенсвязывающий фрагмент по п. 1, в котором
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7;
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 8;
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9;
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 20;
вторая LCDR состоит из аминокислотной последовательности AAS; и
третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 11.
11. Выделенное антитело или его антигенсвязывающий фрагмент по п. 10, причем выделенное антитело или его антигенсвязывающий фрагмент содержит:
удлиненную вторую HCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 24;
удлиненную первую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 27 и
удлиненную вторую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 29.
12. Выделенное антитело или его антигенсвязывающий фрагмент по п. 10 или 11, в котором
HCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 32 и/или
LCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 33.
13. Выделенное антитело или его антигенсвязывающий фрагмент по п. 1, в котором
первая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 7;
вторая HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 19;
третья HCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 9;
первая LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 20;
вторая LCDR состоит из аминокислотной последовательности ATS; и
третья LCDR состоит из аминокислотной последовательности согласно SEQ ID NO: 22.
14. Выделенное антитело или его антигенсвязывающий фрагмент по п. 13, причем выделенное антитело или его антигенсвязывающий фрагмент содержит:
удлиненную вторую HCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 25;
удлиненную первую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 26 и
удлиненную вторую LCDR, которая состоит из аминокислотной последовательности согласно SEQ ID NO: 28.
15. Выделенное антитело или его антигенсвязывающий фрагмент по п. 13 или 14, в котором
HCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 34 и/или
LCVR содержит, предпочтительно состоит из аминокислотной последовательности согласно SEQ ID NO: 35.
16. Выделенное антитело или его антигенсвязывающий фрагмент по п. 1, в котором HCVR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34 и SEQ ID NO: 36.
17. Выделенное антитело или его антигенсвязывающий фрагмент по п. 1, в котором LCVR содержит, предпочтительно состоит из аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35, SEQ ID NO: 37 и SEQ ID NO: 38.
18. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем антитело или его антигенсвязывающий фрагмент выбрано из группы, состоящей из человеческого антитела, гуманизированного антитела и химерного антитела или их антигенсвязывающего фрагмента.
19. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем антигенсвязывающий фрагмент выбран из группы, состоящей из вариабельного одноцепочечного фрагмента (scFv), фрагмента Fab, фрагмента F(ab')2, фрагмента F(ab’)3, фрагмента Fab', фрагмента Fd, фрагмента Fv, фрагмента dAb, выделенной определяющей комплементарность области (CDR) и нанотела, предпочтительно scFv.
20. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем выделенное антитело или его антигенсвязывающий фрагмент представляет собой моноклональное антитело или его антигенсвязывающий фрагмент.
21. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем выделенное антитело или его антигенсвязывающий фрагмент имеет класс изотипа, выбранный из группы, состоящей из IgG, IgA, IgM, IgD и IgE, предпочтительно IgG и более предпочтительно IgG1 или IgG4.
22. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем выделенное антитело или его антигенсвязывающий фрагмент содержит по меньшей мере одну Fc сайлесинг-мутацию, ингибирующую взаимодействие с Fc-рецепторами.
23. Выделенное антитело или его антигенсвязывающий фрагмент по п. 22, причем выделенное антитело или его антигенсвязывающий фрагмент относится к классу изотипа IgG, и по меньшей мере одна Fc сайлесинг-мутация выбрана из группы, состоящей из L234A, L235A и P329G.
24. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем выделенное антитело или его антигенсвязывающий фрагмент содержит по меньшей мере одну стабилизирующую мутацию, которая предотвращает или снижает обмен Fab-плеча in vivo.
25. Выделенное антитело или его антигенсвязывающий фрагмент по п. 24, причем выделенное антитело или его антигенсвязывающий фрагмент относится к подклассу изотипа IgG4, и по меньшей мере одна стабилизирующая мутация представляет собой S228P.
26. Выделенное антитело или его антигенсвязывающий фрагмент по любому из предшествующих пунктов, причем выделенное антитело или его антигенсвязывающий фрагмент специфически связывается с BSSL человека (hBSSL).
27. Фармацевтическая композиция для снижения провоспалительных эффектов BSSL, содержащая выделенное антитело и/или его антигенсвязывающий фрагмент по любому из предшествующих пунктов и фармацевтически приемлемый носитель или вспомогательное вещество.
28. Применение выделенного антитела или его антигенсвязывающего фрагмента по любому из пп. 1-26 или фармацевтической композиции по п. 27 при лечении и/или предотвращении воспалительного заболевания, выбранного из группы, состоящей из ревматоидного артрита (RA), ювенильного идиопатического артрита (JIA), псориатического артрита, воспалительное заболевание кишечника (IBD), такого как болезнь Крона или язвенный колит (UC), и стеатоза печени.
29. Применение выделенного антитела или его антигенсвязывающего фрагмента по любому из пп. 1-26 или фармацевтической композиции по п. 27 при лечении и/или предотвращении воспалительного заболевания, выбранного из группы, состоящей из хронического воспалительного заболевания, системного воспалительного заболевания, аутоиммунного заболевания, аутовоспалительного заболевания и воспалительного заболевания, опосредованного клетками-естественными киллерами (NK).
30. Применение по п. 29, где воспалительное заболевание представляет собой хроническое воспалительное заболевание.
31. Применение по п. 29, где воспалительное заболевание представляет собой системное воспалительное заболевание.
32. Применение по п. 29, где воспалительное заболевание представляет собой аутоиммунное заболевание.
33. Применение по п. 29, где воспалительное заболевание представляет собой аутовоспалительное заболевание.
34. Применение по п. 29, где воспалительное заболевание представляет собой воспалительное заболевание, опосредованное клетками-естественными киллерами (NK).
35. Применение по п. 28, где воспалительное заболевание выбрано из группы, состоящей из ревматоидного артрита (RA), ювенильного идиопатического артрита (JIA) и псориатического артрита.
36. Полинуклеотид, кодирующий антитело или его антигенсвязывающий фрагмент по любому из пп. 1-26.
37. Экспрессионный вектор, содержащий полинуклеотид по п. 36.
38. Клетка-хозяин для получения антитела или его антигенсвязывающего фрагмента по любому из пп. 1-26, причем клетка-хозяин включает экспрессионный вектор по п. 37.
39. Способ получения антитела или его антигенсвязывающего фрагмента, включающий:
культивирование клетки по п. 38, содержащей экспрессионный вектор по п. 37, в условиях, когда антитело или его антигенсвязывающий фрагмент экспрессируется клеткой; и
необязательно, выделение антитела или его антигенсвязывающего фрагмента из клетки или из культуральной среды, в которой культивируется клетка.
40. Способ обнаружения наличия или отсутствия липазы, стимулируемой солями желчных кислот (BSSL) и/или количественного определения уровня BSSL в образце, включающий:
приведение образца в контакт с выделенным антителом или его антигенсвязывающим фрагментом по любому из пп. 1-26; и
обнаружение наличия или отсутствия BSSL в образце и/или количественное определение уровня BSSL в образце на основе количества выделенного антитела или его антигенсвязывающего фрагмента, связанного с BSSL.
41. Способ диагностики нарушения, связанного с липазой, стимулируемой солями желчных кислот (BSSL), включающий:
a) приведение образца, полученного от субъекта, в контакт с выделенным антителом или его антигенсвязывающим фрагментом по любому из пп. 1-26;
b) обнаружение наличия или отсутствия BSSL и/или количественное определение уровня BSSL в образце на основе количества выделенного антитела или его антигенсвязывающего фрагмента, связанного с BSSL; и
c) заключение на основании результатов этапа b) о том, имеет ли субъект нарушение, связанное с BSSL, или нет.
| WO 2010117325 A1, 14.10.2010 | |||
| LINDQUIST S | |||
| et al., Bile Salt-Stimulated Lipase Plays an Unexpected Role in Arthritis Development in Rodents, PLOS ONE, 2012, vol.7(10): e47006 | |||
| WANG X | |||
| et al., The crystal structure of bovine bile salt activated lipase: insights into the bile salt activation mechanism, Structure, 1997, vol | |||
| Кипятильник для воды | 1921 |
|
SU5A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
Авторы
Даты
2024-09-19—Публикация
2020-07-10—Подача