ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к антигенсвязывающим белкам и их фрагментам, которые специфически связывают CD96, и в частности, CD96 человека. Настоящее изобретение также относится к способам лечения заболеваний или нарушений указанными антигенсвязывающими фрагментами, фармацевтическими композициями, содержащими указанные антигенсвязывающие фрагменты, а также к способам их получения. Другие варианты осуществления настоящего изобретения станут очевидными из следующего далее описания.
ПРЕДПОСЫЛКИ СОЗДАНИЯ ИЗОБРЕТЕНИЯ
CD96/TACTILE («T-клеточной активации повышенной поздней экспрессии») представляет собой клеточный поверхностный рецептор в суперсемействе иммуноглобулинов, который экспрессируется в основном на T-клетках, клетках - естественных киллерах (NK) и T-клетках - естественных киллерах (NKT). CD96 принадлежит к семейству рецепторов, включающему CD226 и TIGIT («T-клеточный иммунорецептор с доменами Ig и ITIM»), которые, как известно, взаимодействуют с нектином и нектин-подобными лигандами. CD155/NECL5 («нектин-подобный белок-5») представляет собой основной лиганд для всех трех рецепторов (CD96, TIGIT и CD226). TIGIT связывает CD155 с более высокой аффинностью (3,15 нМ), чем CD226 (119 нМ), и связывание CD96 является промежуточным (37,6 нМ) (Martinet L. & Smyth M.J. Nat Rev Immunol. 2015 Apr; 15(4): 243-54). Помимо CD155, как TIGIT, так и CD226, также связывают другой лиганд CD112 с гораздо меньшей аффинностью. Недавно был обнаружен новый рецептор CD112R, который также связывает CD112 (Zhu Y. et al. J Exp Med. 2016 Feb 8; 213(2): 167-76).
Среди рецепторов в этой оси CD226 (DNAM-1) является одним из основных активирующих рецепторов NK-клеток. Сообщалось, что CD226 потенцирует цитотоксичность NK-клеток в отношении раковых клеток, и играет решающую роль в иммунологическом надзоре за опухолями (Lakshmikanth T. et al. J Clin Invest. 2009; 119(5): 1251-63; Chan C.J., et al. J Immunol. 2010; 184(2): 902-11; Gilfillan S. et al. J Exp Med. 2008; 205(13): 2965-73; Iguchi-Manaka A. et al. J Exp Med. 2008; 205(13): 2959-64). Напротив, и CD96 (Chan C.J. et al. Nat Immunol. 2014; 15(5): 431-8), и TIGIT (Lozano E. et al. J Immunol. 2012; 188(8): 3869-75), как известно, подавляют иммунные ответы за счет ингибирования функции NK-клеток и/или T-клеток. Экспрессия TIGIT связана с истощением T-клеток (Lozano E. et al. 2012; Kurtulus S. et al. J Clin Invest. 2015; 125(11): 4053-62) и истощением NK-клеток (Zhang Q. et al. Nat Immunol. 2018; 19(7): 723-32), и несколько анти-TIGIT антител находятся в стадии клинической разработки.
В целом, существует значительно больше литературы и понимания механизма действия в случае CD226 и TIGIT, чем CD96. CD226 не имеет классического мотива ITAM как другие иммунные активирующие рецепторы. При связывании лиганда и димеризации рецептора он передает положительный сигнал посредством серии событий фосфорилирования белков, включая белки PKC и Vav1. Цитоплазматический фрагмент TIGIT содержит мотив ITT и классический ингибирующий мотив ITIM. При связывании CD155 происходит фосфорилирование тирозина в мотиве ITT, и сигнал иммунного ингибирования передается далее, с вовлечением SHIP1. Напротив, сигналы для CD96 пока не выявлены. Известно, что существует потенциально ингибирующий мотив ITIM в цитоплазматическом фрагменте CD96, а также потенциально активирующий мотив YXXM, который имеется во многих иммунных активирующих рецепторах (например, ICOS и CD28) (Georgiev H. et al. Front Immunol. 2018; 9: 1072).
Although CD96 был открыт более 25 лет назад (Wang P.L. et al. J Immunol. 1992; 148(8): 2600-8), мало что было известно о функции CD96, кроме того, что он является членом семейства иммуноглобулинов, который имеет общий лиганд CD155 с CD226 и TIGIT (Fuchs A., et al. J Immunol. 2004; 172(7): 3994-8). Последующие публикации, связывающие CD96 с раком, в основном касались CD96 в качестве маркера лейкозных стволовых клеток (ЛСК). Первая работа, указывающая на CD96 как на потенциальную мишень при иммунотерапии рака, была опубликована сотрудниками лаборатории профессора Марка Смита в 2014 г.; было показано, что CD96 конкурирует с CD226 за связывание CD155 в NK-клетках, и CD96 отрицательно регулирует продуцирование провоспалительных цитокинов, включая IFNγ (гамма), у мышей после активации ЛПС (Chan C.J. et al. 2014). Мыши с нокаутом CD96, а также мыши, которым вводили анти-CD96 антитело, были менее подвержены образованию MCA-индуцируемой саркомы (Id.). В том же исследовании блокада CD226 или CD155 приводила к худшему исходу, и предполагается, что это связано с потерей активирующего пути CD155:CD226 (Id.).
Затем были опубликованы результаты дальнейших исследований in vivo, свидетельствующие в пользу ингибирования CD96 для лечения рака (смотри, например, Blake S.J. et al. Cancer Discov. 2016; 6(4): 446-59; Brooks J. et al. 2018; 78(2): 475-88; Harjunpää H. et al. Oncoimmunology. 2018; 7(7): e1445949). Соответственно, существует потребность в усовершенствованных антигенсвязывающих белках, и их фрагментах, направленных на CD96, для применения в лечении заболеваний. Такие композиции и соответствующие способы предложены в настоящем документе.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
В первом аспекте настоящее изобретение относится к CD96-связывающим белкам. Во втором аспекте настоящее изобретение также относится к конструкциям нуклеиновой кислоты, кодирующим CD96-связывающие белки. В третьем аспекте настоящее изобретение относится к экспрессионным векторам, содержащим нуклеиновую кислоту по второму аспекту. В четвертом аспекте настоящее изобретение относится к рекомбинантной клетке-хозяину, содержащей нуклеиновую кислоту или экспрессионный вектор, описанные в предшествующих аспектах. Кроме того, в пятом аспекте настоящее изобретение относится к способам получения CD96-связывающего белка, включающим культивирование клетки-хозяина, описанной в предшествующем аспекте, в условиях, подходящих для экспрессии последовательности(ей) указанной нуклеиновой кислоты или вектора(ов), за счет чего продуцируется полипептид, представляющий собой CD96-связывающий белок. Шестой аспект изобретения относится к CD96-связывающему белку, полученному способом получения, описанным в предшествующем аспекте. В седьмом аспекте настоящее изобретение также относится к фармацевтическим композициям, содержащим CD96-связывающий белок, описанный в любом из предшествующих аспектов, и фармацевтически приемлемый эксципиент. Другой аспект изобретения относится к способу лечения заболевания у субъекта, который нуждается в этом, включающему введение указанному субъекту терапевтически эффективного количества CD96-связывающего белка или фармацевтической композиции, описанных в любом из предшествующих аспектов. Следующий аспект изобретения относится к способу лечения, описанному в предшествующем аспекте, дополнительно включающему определение того, экспрессируется ли у субъекта CD96. Другой аспект изобретения относится к CD96-связывающему белку или фармацевтической композиции, описанным в любом из предшествующих аспектов, для применения в терапии или для применения в лечении заболевания.
Следующее далее подробное описание предпочтительных вариантов осуществления изобретения будет более понятным при прочтении в сочетании с прилагаемыми чертежами. Для целей иллюстрации изобретения на чертежах представлены варианты осуществления, которые в настоящее время являются предпочтительными. Следует понимать, однако, что изобретение не ограничено конкретными механизмами и инструментарием вариантов осуществления, показанными на чертежах.
ОПИСАНИЕ ЧЕРТЕЖЕЙ/ФИГУР
На ФИГ. 1 показано связывание CD96-связывающих белков с человеческими CD3+T-клетками (A), а также способность CD96-связывающих белков предотвращать связывание CD155 с клетками CHO, экспрессирующими CD96 человека (B).
На ФИГ. 2 показаны данные титрования при равновесии в растворе (SET) для связывания CD96-связывающого белка с CD96 человека (a), яванского макака (b) или мыши (c).
На ФИГ. 3 показано связывание CD96-связывающего белка с клетками HEK, временно трансфицированными изоформами CD96 человека или яванского макака.
На ФИГ. 4 показано связывание CD96-связывающего белка (в сравнении с контролем по изотипу) с первичными человеческими T-клетками (суммарные CD3+ T-клетки в сравнении с подмножествами CD4+ или CD8+ клеток).
На ФИГ. 5 показано связывание CD96-связывающего белка с активированными первичными T-клетками яванского макака.
На ФИГ. 6 показана интернализация CD96 в CD8+T-клетках после связывания с человеческими МКПК с использованием метода визуализирующей цитометрии; репрезентативные изображения CD8+ T-клеток (серый цвет=окрашивание CD8, белый цвет=окрашивание комплекса CD96-связывающий белок-PE).
На ФИГ. 7 показано, что CD96-связывающий белок после предварительного образования комплекса с мембранным CD96 в первичных человеческих T-клетках предотвращает связывание CD155:Fc с указанными T-клетками.
На ФИГ. 8 показано вытеснение CD155:Fc, предварительно связанного с человеческими T-клетками, CD96-связывающим белком.
На ФИГ. 9 представлены данные анализа человеческого Fcγ-репортера, показывающие, что связывание CD96-связывающих белков с CD96 на первичных человеческих T-клетках не индуцирует перекрестное связывание и/или сигнализацию через Fcγ-рецепторы.
На ФИГ. 10 представлены данные анализа уничтожения за счет ADCC человеческих клеток-мишеней в CD4+ и CD8+T-клетках; не наблюдалось никаких признаков повышенной клеточной гибели ни CD4+, ни CD8+, T-клеток в присутствии CD96-связывающих белков.
На ФИГ. 11 представлены данные анализа уничтожения за счет CDC человеческих клеток-мишеней в CD4+T-клетках; не наблюдалось никаких признаков комплемент-зависимой клеточной токсичности, индуцированной CD96-связывающим белком.
На ФИГ. 12 представлены данные в виде EC50 для высвобождения IFNγ за счет CD96-связывающих белков (экспрессируемых клетками CHO или HEK) в анализе МКПК-СКЛ.
На ФИГ. 13 показано влияние CD96-связывающего белка (экспрессируемого клетками HEK) на продуцирование IFNγ в анализе МКПК-СКЛ, с добавлением, или без добавления, CD4+(A) или CD8+(B) T-клеток.
На ФИГ. 14 показан эффект CD96-связывающего белка на секрецию IFNγ и гранзима B в анализе МКПК-СКЛ.
На ФИГ. 15 показана частота встречаемости IFNγ+клеток в разных клеточных популяциях в день 3 в анализе МКПК-СКЛ, в присутствии CD96-связывающего белка и контролей.
На ФИГ. 16 показана частота встречаемости CD96+клеток в популяциях CD4+, CD8+ и NK-клеток после обработки CD96-связывающим белком и контролями в анализе МКПК-СКЛ.
На ФИГ. 17 показан уровень экспрессии CD96 в популяциях CD4+, CD8+ и NK-клеток после обработки CD96-связывающим белком и контролями в анализе МКПК-СКЛ.
На ФИГ. 18 представлены полученные методом FACS характеристики, демонстрирующие эффект CD96-связывающего белка на соотношение однократно положительных CD226+ и дважды положительных CD226+CD96+NK-клеток в анализе МКПК-СКЛ.
На ФИГ. 19 представлены полученные методом FACS характеристики, демонстрирующие эффект CD96-связывающего белка на количество дважды положительных IFNγ+GrzB+клеток среди общей популяции NK-клеток в анализе МКПК-СКЛ.
На ФИГ. 20 показан ингибирующий эффект связанного с планшетом CD155-Fc на продуцирование IFNγ в анализе с человеческими МКПК.
На ФИГ. 21 показана активность CD96-связывающего белка в функциональных анализах с ОИЛ при раке почки, отдельно или в сочетании с анти-PD1 или анти-TIGIT антителами.
На ФИГ. 22 представлены данные исследования методом биолюминесцентной визуализации CD96-связывающего белка в модели зависимой от NK-клеток колонизации легких клетками меланомы B16F10, с демонстрацией репрезентативных биолюминесцентных изображений, полученных приблизительно через 15 минут после инъекции клеток меланомы B16F10 RFluc.
На ФИГ. 23 показан биолюминесцентный сигнал в легких в день 14 после инъекции клеток B16F10 мышам; без истощения, с истощением CD4, CD8 или NK-клеток.
На ФИГ. 24 показаны in vivo биолюминесцентные сигналы в легких в день 14 и день 20, демонстрирующие эффект CD96-связывающих белков (в сравнении с контролем) на метастазирование в легких в различных группах.
На ФИГ. 25 представлены изображения обработанных CD96-связывающим белком истощенных по CD4+/CD8+ клеткам мышиных легких в сравнении с контролем в день 20 (окончание исследования).
На ФИГ. 26 показано продуцирование IFNγ в анализе с покрытыми CD155 МКПК в присутствии CD96-связывающих белков или контроля по изотипу в присутствии анти-TIGIT мАт, при оценке методом MSD.
На ФИГ. 27 показано продуцирование TNFα в анализе с покрытыми CD155 МКПК в присутствии CD96-связывающих белков или контроля по изотипу в присутствии анти-TIGIT мАт, при оценке методом MSD.
На ФИГ. 28 показано продуцирование IFNγ в анализе с покрытыми CD155 МКПК в присутствии CD96-связывающих белков или контроля по изотипу в присутствии или в отсутствие анти-TIGIT мАт в течение 3 дней, при оценке методом MSD.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к CD96-связывающим белкам, нуклеиновым кислотам, кодирующим указанные белки, и связанным с ними объектам изобретения.
При использовании в настоящем описании и в формуле изобретения форма единственного числа терминов включает соответствующую форму множественного числа, если из контекста явно не следует иное. Таким образом, например, ссылка на «пептидную цепь» является ссылкой на одну или более пептидных цепей и охватывает их эквиваленты, известные специалистам в данной области.
При использовании в настоящем описании и в формуле изобретения термин «содержащие» охватывает термины «включающие» или «состоящие из», например, композиция, «содержащая» X, может состоять исключительно из X, или может включать некоторые дополнительные компоненты, например, X+Y.
Термин «состоящие в основном из» ограничивает область применения признака указанными материалами или стадиями, а также теми, которые не влияют материально на основную характеристику(и) заявленного признака.
Термин «состоящие из» исключает присутствие каких-либо дополнительных компонентов.
Если нет иных указаний, все технические и научные термины, используемые в настоящем описании, имеют то значение, которое им обычно придают специалисты в области, к которой относится настоящее изобретение. Хотя любые композиции и способы, аналогичные или эквивалентные тем, которые описаны в настоящем описании, могут быть использованы при осуществлении на практике или при тестировании способов по изобретению, иллюстративные композиции и способы описаны в настоящем описании. Любые из аспектов и вариантов осуществления изобретения, описанных в настоящем описании, также могут быть объединены. Например, объект изобретения по любому зависимому или независимому пункту формулы изобретения, раскрытый в настоящем описании, может быть многократно объединен (например, один или более перечисленных признаков каждого зависимого пункта могут быть объединены в один пункт на основании независимого пункта, от которого они зависят).
Диапазоны, представленные в настоящем описании, включают все значения в пределах конкретного описанного диапазона и значения около конечной точки для конкретного диапазона. На фигурах и в таблицах настоящего документа также описаны диапазоны и дискретные значения, которые могут представлять собой элемент любого из способов, раскрытых в настоящем описании.
Концентрации, описанные в настоящем описании, определены при температуре и давлении окружающей среды. Это может быть, например, комнатная температура и давление, или температура и давление в пределах определенной части технологического потока. Предпочтительно, концентрации определены в стандартных условиях 25°C и давления 1 бар.
Термин «приблизительно» означает величину в пределах двух стандартных отклонений от среднего значения для любого конкретного измеренного значения.
«Аффинность» представляет собой силу связывания одной молекулы с другой. Аффинность связывания антигенсвязывающего белка с его мишенью можно определять равновесными методами (например, в твердофазным иммуноферментном анализе (ELISA) или радиоиммунном анализе (RIA)), или путем измерения кинетики (например, на приборе BIACORE™).
Используемый в настоящем описании термин «антиген» означает структуру макромолекулы, которая избирательно узнается антигенсвязывающим белком. Антигены включают, но не ограничиваются ими, белок (с полисахаридами или без них) или белковую композицию, содержащую один или более T-клеточных эпитопов. В настоящем описании предусмотрено, что связывающие мишень домены антигенсвязывающего белка могут узнавать сахарную боковую цепь гликопротеина, а не конкретную аминокислотную последовательность или макромолекулу. Таким образом, сахарный фрагмент, или сульфатированный сахарный фрагмент, служит в качестве антигена.
Используемый в настоящем описании термин «антигенсвязывающий белок» относится к выделенным белкам, антителам, фрагментам антител (например, Fab-фрагментам и так далее) и другим полученным из антител белковым конструкциям, таким как те, которые содержат домены (например, домен антитела и так далее), способным связывать CD96. Такие альтернативные форматы антител включают триатело, тетратело, мини-антитело и мини-тело. Также включены альтернативные каркасы, в которых одна или более областей CDR любых молекул по изобретению могут быть сгруппированы в подходящем, не являющемся иммуноглобулином белковом каркасе или скелете, например, аффитело, каркас SpA, домен рецептора LDL класса A, авимер (смотри, например, публикации патентных заявок США №№2005/0053973, 2005/0089932, 2005/0164301) или домен EGF. АСБ также включает антигенсвязывающие фрагменты таких антител или других молекул. Кроме того, АСБ может включать области VH по изобретению, переведенные в формат полноразмерного антитела, (Fab’)2-фрагмента, Fab-фрагмента, биспецифической или бипаратопной молекулы, или их эквивалента (например, scFV, би- три- или тетратела, Tandab и так далее), спаренные с соответствующей легкой цепью. АСБ могут включать антитело, которое представляет собой IgG1, IgG2, IgG3 или IgG4; или IgM; IgA, IgE или IgD, или их модифицированный вариант.Константный домен тяжелой цепи антитела может быть выбран соответствующим образом. Константный домен легкой цепи может представлять собой константный домен каппа или лямбда. АСБ может также представлять собой химерное антитело такого типа, который описан в WO86/01533, содержащее антигенсвязывающую область и область не иммуноглобулина. Антигенсвязывающие белки по изобретению могут быть предоставлены в виде лиофилизированного порошка, содержащего антитело и эксципиенты, который может быть восстановлен фармацевтически приемлемым носителем (например, стерильной водой). Эту восстановленную фармацевтическую композицию затем можно вводить либо подкожно, либо внутривенно (например, с дополнительным разведением). Антигенсвязывающие белки по изобретению также могут быть предоставлены в виде жидкого препарата, содержащего антитело, эксципиенты и фармацевтически приемлемый носитель. Эту жидкую фармацевтическую композицию затем можно вводить либо подкожно, либо внутривенно (например, с дополнительным разведением). В настоящем описании термины «АСБ», «антигенсвязывающий белок» и «связывающий белок» используются взаимозаменяемо. В настоящем описании термины «пептид», «полипептид» и «белок» используются взаимозаменяемо и относятся к соединению, состоящему из аминокислотных остатков, ковалентно связанных пептидными связями. Белок или пептид должен содержать по меньшей мере две аминокислоты, и максимальное количество аминокислот, которые могут составлять последовательность белка или пептида, не имеет ограничений. Полипептиды включают любой пептид или белок, содержащий две или более аминокислот, соединенных между собой пептидными связями. Используемый в настоящем описании термин относится как к коротким цепям, которые в данной области обычно называют пептидами, олигопептидами и олигомерами, например, так и к более длинным цепям, которые в данной области обычно называют белками, которых существует множество видов. Термин «полипептиды» охватывает, например, биологически активные фрагменты, в значительной степени гомологичные полипептиды, олигопептиды, гомодимеры, гетеродимеры, варианты полипептидов, модифицированные полипептиды, производные, аналоги, слитые белки, в числе прочих. Полипептиды включают природные пептиды, рекомбинантные пептиды, синтетические пептиды или их сочетание.
Используемый в настоящем описании термин «вариант антитела» означает антитело, которое отличается от исходного антитела по меньшей мере одной аминокислотной модификацией (например, наличием иной аминокислотной боковой цепи), посттрансляционной модификацией или другой модификацией в по меньшей мере одной тяжелой цепи, легкой цепи, или в их сочетании, которая приводит к структурному изменению (например, иной аминокислотной боковой цепи, иной посттрансляционной модификации или другой модификации) относительно исходного антитела. Структурные изменения можно определять непосредственно различными способами, хорошо известными в данной области, такими как ЖХ-МС, прямое секвенирование, или опосредованно такими способами, как изоэлектрическое фокусирование и тому подобное. Такие способы хорошо известны специалистам в данной области.
Используемый в настоящем описании термин «эпитоп» относится к части антигена, которая вступает в контакт с конкретным связывающим доменом. Эпитоп может быть линейным или конформационным/прерывистым. Конформационный или прерывистый эпитоп содержит аминокислотные остатки, которые разделены другими последовательностями, то есть, не занимают соседние положения в первичной последовательности антигена. Хотя остатки могут быть из разных областей пептидной цепи, они находятся в непосредственной близости друг от друга в трехмерной структуре антигена. В случае мультимерных антигенов конформационный или прерывистый эпитоп может включать остатки из разных пептидных цепей. Конкретные остатки, входящие в состав эпитопа, могут быть определены при помощи компьютерных программ моделирования или с помощью трехмерных структур, полученных способами, известными в данной области, такими как рентгеновская кристаллография. Используемый в настоящем описании термин «эпитоп» охватывает посттрансляционную модификацию полипептида, которая может быть узнана антигенсвязывающим белком или доменом, такую как сахарный фрагмент гликозилированного белка.
Используемый в настоящем описании термин «выделенные» означает измененные или извлеченные из своего естественного окружения. Например, нуклеиновая кислота или пептид, естественным образом присутствующие в живом животном, не являются «выделенными», но та же самая нуклеиновая кислота или пептид, частично или полностью отделенные от присутствующих вместе с ними материалов в их естественном окружении, являются «выделенными». Выделенная нуклеиновая кислота или белок могут существовать в практически очищенной форме, или могут существовать в неестественном окружении, таком как, например, клетка-хозяин.
CD96 (кластер дифференцировки 96), также известный как TACTILE, представляет собой рецептор, экспрессируемый на T-клетках и NK-клетках, который имеет сходство последовательности с CD226 (DNAM01) и представляет собой трансмембранный гликопротеин 1 типа, принадлежащий к суперсемейству иммуноглобулинов.
CD96 человека имеет 3 изоформы: две мембранные (V1; V2) формы и растворимую форму. Самая длинная изоформа V1 представляет собой белок из 585 аминокислот с ММ 65634 Да. Более короткая изоформа V2 (569 ак) имеет ММ 63888 Да. V2 отличается от V1 наличием короткой делеции Ig-складки второго домена. Первый домен CD96, как известно, содержит эпитоп(ы), необходимый для связывания CD155, в то время как второй домен модулирует степень/силу связывания. CD96V2 связывается более сильно с CD155, чем CD96V1, и также является преимущественно экспрессируемой формой, за исключением клеток острого миелоидного лейкоза AML. О растворимой форме CD96 (sCD96) известно мало. Сообщалось, что sCD96 была обнаружена в крови здорового донора крови (1-3 нг/мл), и на более высоких уровнях была обнаружена у пациентов с инфекцией вируса гепатита B (HBV), HBV/циррозом печени.
Исходя из результатов анализа мРНК нормальных человеческих тканей, наивысшая экспрессия CD96 (с зондом, обнаруживающим как V1, так и V2, форму CD96) наблюдается в гемопоэтических клетках. Экспрессия CD96 также наблюдается в тканях, содержащих большое количество лимфоцитов, таких как селезенка, легкие, щитовидная железа и тонкий кишечник. В гемопоэтических клетках CD96 в наибольших количествах присутствует в T-клетках и NK-клетках, с меньшим уровнем экспрессии в некоторых B-клетках. Как и CD96, TIGIT и CD226 в наибольших количествах присутствуют в гемопоэтических клетках, и экспрессируются как в T-клетках, так и в NK-клетках. CD226 также экспрессируется в обычных DC клетках. Лиганд CD155 демонстрирует широкий паттерн экспрессии в нормальных человеческих тканях, и также экспрессируется в DC клетках и макрофагах (антигенпредставляющие клетки). CD155 не экспрессируется в T-клетках или NK-клетках.
Аффинность связывания (KD) при взаимодействии антигенсвязывающий белок - целевой антиген может составлять 1 мМ или менее, 100 нМ или менее, 10 нМ или менее, 2 нМ или менее, или 1 нМ или менее. Альтернативно, значение KD может составлять от 5 до 10 нМ; или от 1 до 2 нМ. Значение KD может составлять от 1 пМ до 500 пМ; или от 500 пМ до 1 нМ. Например, некоторые такие полезные варианты имеют аффинность связывания (KD), составляющую по меньшей мере приблизительно 40 нМ или по меньшей мере приблизительно 35 нМ, или по меньшей мере приблизительно 30 нМ, например, от приблизительно 10 пМ до приблизительно 30 нМ.
Аффинность связывания антигенсвязывающего белка определяют на основании константы ассоциации (Ka) и константы диссоциации (Kd) (KD=Kd/Ka). Аффинность связывания можно измерять на приборе BIACORE™, например, захватывая тестируемое антитело на покрытую белком A поверхность сенсора и пропуская целевой антиген над этой поверхностью. Альтернативно, аффинность связывания можно измерять при помощи FORTEBIO, например, захватывая тестируемое антитело на покрытую белком A иглу и пропуская целевой антиген над этой поверхностью. Альтернативно, аффинность связывания (KD) можно измерять с использованием анализа MSD-SET (MSD титрование при равновесии в растворе), например, с тестируемым антителом, тестируемом на стандартном связывающем MSD планшете и обнаружением при помощи MSD SECTOR IMAGER. MSD-SET позволяет определять аффинность антител при равновесии в фазе раствора. В этом известном методе обнаруживают свободный антиген в состоянии равновесия при титруемых серийных концентрациях антитела.
Значение Kd может составлять 1×10-3 мс-1 или менее, 1×10-4 мс-1 или менее, или 1×10-5 мс-1 или менее. Значение Kd может составлять от 1×10-5 мс-1 до 1×10-4 мс-1; или от 1×10-4 мс-1 до 1×10-3 мс-1. Низкоскоростная Kd может приводить к медленной диссоциации комплекса антигенсвязывающий белок - целевой антиген и лучшей нейтрализации целевого антигена.
Используемый в настоящем описании термин «специфическая активность связывания антигена» означает активность связывания антигена, измеренную, например, методом поверхностного плазмонного резонанса (ППР). Специфическую активность связывания CD96 можно определять методом ППР с использованием прибора BIACORE™, например, работающего в режиме связывания. Она равна активности связывания, деленной на общее содержание белка в образце.
Используемые в настоящем описании термины «VH» и «VL» означают вариабельную область тяжелой цепи и вариабельную область легкой цепи, соответственно, антигенсвязывающего белка.
«Области CDR» определяют как аминокислотные последовательности определяющей комплементарность области антигенсвязывающего белка. Они представляют собой гипервариабельные области тяжелой и легкой цепей иммуноглобулина. В вариабельной области иммуноглобулина имеются три области CDR (или CDR) тяжелой цепи и три области CDR легкой цепи. Таким образом, используемый в настоящем описании термин «области CDR» относится ко всем трем областям CDR тяжелой цепи, всем трем областям CDR легкой цепи, всем областям CDR тяжелой и легкой цепи, или по меньшей мере одной области CDR, где по меньшей мере одна область CDR представляет собой CDRH3. Каркасные области следуют за каждой из этих областей CDR. Приемлемые области каркаса 1, каркаса 2 и каркаса 3 вариабельной области тяжелой цепи и вариабельной области легкой цепи известны специалистам в данной области. Приемлемые константные области тяжелой цепи (включая шарнирные области) и константные области легкой цепи также известны специалистам в данной области. Приемлемые изотипы антитела также известны специалистам в данной области.
В тексте настоящего описания аминокислотные остатки в последовательностях вариабельных доменов и последовательностях полноразмерного антитела пронумерованы в соответствии с системой нумерации Kabat. Аналогично, термины «CDR», «CDRL1», «CDRL2», «CDRL3», «CDRH1», «CDRH2», «CDRH3», используемые в спецификации, соответствуют системе нумерации Kabat.
Специалистам в данной области понятно, что существуют альтернативные системы нумерации для аминокислотных остатков в последовательностях вариабельных доменов и последовательностях полноразмерного антитела. Существуют также альтернативные системы нумерации для последовательностей CDR, например, система нумерации Chothia. Структура и сворачивание белка антитела могут приводить к тому, что другие остатки считают частью последовательностей CDR, и это понятно квалифицированным специалистам.
Другие системы нумерации последовательностей CDR, доступные для специалистов, включают систему «AbM» (University of Bath) и «контактную» (University College London) систему. Минимальную перекрывающуюся область при использовании по меньшей мере двух из систем Kabat, Chothia, AbM и контактной можно определять для выявления «минимальной связывающей единицы». Минимальная связывающая единица может представлять собой субфрагмент CDR.
В табл. 1, ниже, приведено определение для каждой CDR или связывающей единицы с использованием каждой из систем нумерации. Система нумерации Kabat использована в табл. 1 для нумерации аминокислотной последовательности вариабельных доменов. Следует отметить, что некоторые из определений CDR могут варьироваться в зависимости от используемой отдельной публикации.
Один вариант осуществления изобретения относится к CD96-связывающему белку, содержащему: (a) (i) любую одну, или сочетание, из областей CDR, выбранных из CDRH1, CDRH2, CDRH3 с SEQ ID NO: 2, 6, 10, 14, 18, 22, 26, 30, 34, 38, 42, 46, 50, 54, 58, 62, 66, 70, 74, 78, 82, 86, 90 и 94, и/или CDRL1, CDRL2, CRDL3 с SEQ ID NO: 1, 5, 9, 13, 17, 21, 25, 29, 33, 37, 41, 45, 49, 53, 57, 61, 65, 69, 73, 77, 81, 85, 89 и 93; или (ii) вариант CDR, где (i) вариант имеет 1, 2 или 3 аминокислотные модификации; или (b) область VH, содержащую последовательность, по меньшей мере на 80% идентичную (или по меньшей мере на 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5% идентичную) последовательности SEQ ID NO: 2, 6, 10, 14, 18, 22, 26, 30, 34, 38, 42, 46, 50, 54, 58, 62, 66, 70, 74, 78, 82, 86, 90 или 94; и/или область VL, содержащую последовательность, по меньшей мере на 80% идентичную последовательности SEQ ID NO: 1, 5, 9, 13, 17, 21, 25, 29, 33, 37, 41, 45, 49, 53, 57, 61, 65, 69, 73, 77, 81, 85, 89 или 93.
В другом варианте осуществления CD96-связывающий белок содержит: (a) (i) любую одну, или сочетание, из областей CDR, выбранных из CDRH1, CDRH2, CDRH3 с SEQ ID NO: 2, 6, 10, 14, 18, 22, 26, 30, 34, 38, 42, 46, 50, 54, 58, 62, 66, 70, 74, 78, 82, 86, 90 и 94, и/или CDRL1, CDRL2, CRDL3 с SEQ ID NO: 1, 5, 9, 13, 17, 21, 25, 29, 33, 37, 41, 45, 49, 53, 57, 61, 65, 69, 73, 77, 81, 85, 89 и 93; или (ii) вариант CDR, где (i) вариант имеет 1, 2 или 3 аминокислотные модификации; или (b) область VH, содержащую последовательность SEQ ID NO: 2, 6, 10, 14, 18, 22, 26, 30, 34, 38, 42, 46, 50, 54, 58, 62, 66, 70, 74, 78, 82, 86, 90 или 94; и/или область VL, содержащую последовательность, по меньшей мере на 80% идентичную (или по меньшей мере на 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5% идентичную) последовательности SEQ ID NO: 1, 5, 9, 13, 17, 21, 25, 29, 33, 37, 41, 45, 49, 53, 57, 61, 65, 69, 73, 77, 81, 85, 89 или 93.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в предшествующем аспекте, содержащему CDRH1, выбранную из SEQ ID NO: 105-125; CDRH2, выбранную из SEQ ID NO: 126-146; и/или CDRH3, выбранную из SEQ ID NO: 147-150; CDRL1, выбранную из SEQ ID NO: 97-98; CDRL2, выбранную из SEQ ID NO: 99-100; и/или CDRL3, выбранную из SEQ ID NO: 101-104.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, где связывающий белок содержит CDRH3, которая на 100% идентична SEQ ID NO: 147, 148, 149 или 150.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, содержащему CDRH1, которая на 100% идентична SEQ ID NO: 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116, 117, 118, 119, 120, 121, 122, 123, 124 или 125; CDRH2, которая на 100% идентична SEQ ID NO: 126, 127, 128, 129, 130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142, 143, 144, 145 или 146; и/или CDRH3, которая на 100% идентична SEQ ID NO: 147, 148, 149 или 150; CDRL1, которая на 100% идентична SEQ ID NO: 97 или 98; CDRL2, которая на 100% идентична SEQ ID NO: 99 или 100; и/или CDRL3, которая на 100% идентична SEQ ID NO: 101, 102, 103 или 104.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, в котором присутствуют все 6 областей CDR.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, содержащему CDRH1 с SEQ ID NO: 115; CDRH2 с SEQ ID NO: 145 и CDRH3 с SEQ ID NO: 147; и/или CDRL1 с SEQ ID NO: 97; CDRL2 с SEQ ID NO: 99 и CDRL3 с SEQ ID NO: 101.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, где связывающий белок содержит: область VH, которая на 75% идентична SEQ ID NO: 86; и/или область VL, которая на 75% идентична SEQ ID NO: 85 (или по меньшей мере на 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5% идентична этим последовательностям). Другой аспект изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, где связывающий белок содержит: область VH, которая на 100% идентична SEQ ID NO: 86; и/или область VL, которая на 100% идентична SEQ ID NO: 85.
В одном варианте осуществления изобретение относится к CD96-связывающему белку, который содержит любую из областей CDR, описанных в настоящем описании (отдельно или в описанных сочетаниях), и который также содержит Fc-область, которая способна связываться с рецептором Fc-гамма, и/или способна стимулировать высвобождение IFN-гамма. Такая Fc-область может представлять собой IgG1 Fc дикого типа.
В одном варианте осуществления изобретение относится к CD96-связывающему белку, который содержит любую из областей VH, описанных в настоящем описании (отдельно или в описанных сочетаниях), и который также содержит Fc-область, которая способна связываться с рецептором Fc-гамма, и/или способна стимулировать высвобождение IFN-гамма. Такая Fc-область может представлять собой IgG1 Fc дикого типа.
В одном варианте осуществления изобретение относится к CD96-связывающему белку, который содержит любую из областей VL, описанных в настоящем описании (отдельно или в описанных сочетаниях), и который также содержит Fc-область, которая способна связываться с рецептором Fc-гамма, и/или способна стимулировать высвобождение IFN-гамма. Такая Fc-область может представлять собой IgG1 Fc дикого типа.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, который представляет собой антитело, где связывающий белок содержит: (полную) тяжелую цепь, которая на 75% идентична (или по меньшей мере на 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5% идентична) SEQ ID NO: 165; и/или (полную) легкую цепь, которая на 75% идентична (или по меньшей мере на 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5% идентична) SEQ ID NO: 166.
Другой аспект изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, где связывающий белок содержит: область VH, которая на 100% идентична SEQ ID NO: 86; и/или область VL, которая на 100% идентична SEQ ID NO: 85.
Другой аспект изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, который представляет собой антитело, где связывающий белок содержит: (полную) тяжелую цепь, которая на 100% идентична аминокислотной последовательности SEQ ID NO: 165; и/или (полную) легкую цепь, которая на 100% идентична аминокислотной последовательности SEQ ID NO: 166 (области CDR подчеркнуты в SEQ ID NO: 165 и 166); или представляет собой антитело, которое связывает CD96, где тяжелая цепь закодирована последовательностью нуклеиновой кислоты SEQ ID NO: 167 и/или легкая цепь закодирована последовательностью нуклеиновой кислоты SEQ ID NO: 168.
Другой аспект изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, который представляет собой антитело, где связывающий белок содержит: (полную) тяжелую цепь, которая на 100% идентична аминокислотной последовательности SEQ ID NO: 170; и/или (полную) легкую цепь, которая на 100% идентична аминокислотной последовательности SEQ ID NO: 169 (области CDR подчеркнуты), или представляет собой антитело, которое связывает CD96, где тяжелая цепь закодирована последовательностью нуклеиновой кислоты SEQ ID NO: 172 и/или легкая цепь закодирована последовательностью нуклеиновой кислоты SEQ ID NO: 171.
Другой аспект изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, который представляет собой антитело, где связывающий белок содержит: (полную) тяжелую цепь, которая на 100% идентична аминокислотной последовательности SEQ ID NO: 174; и/или (полную) легкую цепь, которая на 100% идентична аминокислотной последовательности SEQ ID NO: 173 (области CDR подчеркнуты), или представляет собой антитело, которое связывает CD96, где тяжелая цепь закодирована последовательностью нуклеиновой кислоты SEQ ID NO: 176 и/или легкая цепь закодирована последовательностью нуклеиновой кислоты SEQ ID NO: 175.
Изобретение относится к связывающим белкам, которые на 100% идентичны любой из аминокислотных последовательностей, описанных в настоящем описании, а также к белкам, которые представляют собой варианты аминокислотных последовательностей, описанных в настоящем описании, например, последовательности, которые по меньшей мере на 75% идентичны, или по меньшей мере на 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5%, идентичны последовательностям, описанным в настоящем описании.
Изобретение также относится к нуклеиновым кислотам, кодирующим связывающие белки по изобретению, включая нуклеиновые кислоты, которые на 100% идентичны любой из последовательностей нуклеиновых кислот, описанных в настоящем описании, а также нуклеиновые кислоты, которые представляют собой варианты последовательностей, описанных в настоящем описании, например, последовательности, которые по меньшей мере на 75% идентичны, или по меньшей мере на 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5%, идентичны последовательностям, описанным в настоящем описании.
Области CDR и вариабельные области с аминокислотными последовательностями, предложенными в настоящей заявке, имеют аффинность связывания с CD96, которая является такой же или лучшей, чем аффинность CD155, и/или представляют собой вариабельные области, которые предотвращают связывание CD155, или вытесняют CD155 из связывания, с CD96 (примеры анализов связывания, которые можно использовать для определения этого, приведены в примерах 1 и 2 настоящего документа). Полезными вариантами последовательностей, описанных в настоящем описании, являются варианты, в которых вариабельные области имеют аффинность связывания с CD96, которая является такой же или лучшей, чем аффинность CD155, или которые могут предотвращать связывание CD155, или вытеснять CD155 из связывания, с CD96 (примеры анализов связывания, которые можно использовать для определения этого, приведены в примерах 1 и 2 настоящего документа). Например, некоторые такие полезные варианты имеют аффинность связывания (KD), которая составляет по меньшей мере приблизительно 40 нМ или по меньшей мере приблизительно 35 нМ, или по меньшей мере приблизительно 30 нМ, например, от приблизительно 10 пМ до приблизительно 30 нМ, когда, например, аффинность связывания определяют в анализах MSD-SET, подробно описанных в настоящем описании.
Полезные CD96-связывающие белки (такие как антитела) по настоящему изобретению могут содержать такие вариабельные области, а также Fc-область, описанную в настоящем описании, где указанная Fc-область может связывать рецептор Fc-гамма и/или может стимулировать высвобождение IFN-гамма. Такая Fc-область по изобретению представляет собой IgG1 Fc дикого типа или ее функциональный вариант (например, инактивированный вариант Fc-области, которая может связывать рецептор Fc-гамма и/или может стимулировать высвобождение IFN-гамма). Таким образом, любые из областей CDR и/или вариабельных областей по изобретению можно объединять с Fc-областью по изобретению, например, IgG1 Fc дикого типа или ее функциональным вариантом.
Другой вариант осуществления изобретения относится к CD96-связывающему белку, описанному в любом из предшествующих аспектов, где связывающий белок содержит синтетический полипептид, гуманизированную последовательность или химерную последовательность.
Используемый в настоящем описании термин «консервативные модификации последовательности» означает аминокислотные модификации, которые существенно не влияют на, или не изменяют, характеристики связывания антитела или фрагмента антитела, содержащего аминокислотную последовательность. Такие консервативные модификации включают аминокислотные замены, добавления и делеции. Модификации могут быть внесены в антитело или фрагмент антитела по изобретению стандартными методами, известными в данной области, такими как сайт-направленный мутагенез и ПЦР-опосредованный мутагенез. Консервативные аминокислотные замены представляют собой замены, при которых аминокислотный остаток заменен аминокислотным остатком, имеющим аналогичную боковую цепь. Семейства аминокислотных остатков, имеющих аналогичные боковые цепи, определены в данной области. Эти семейства включают аминокислоты с основными боковыми цепями (например, лизин, аргинин, гистидин), кислотными боковыми цепями (например, аспарагиновая кислота, глутаминовая кислота), незаряженными полярными боковыми цепями (например, глицин, аспарагин, глутамин, серин, треонин, тирозин, цистеин, триптофан), неполярными боковыми цепями (например, аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин), бета-разветвленными боковыми цепями (например, треонин, валин, изолейцин) и ароматическими боковыми цепями (например, тирозин, фенилаланин, триптофан, гистидин).
Термин «домен» относится к структуре свернутого белка, которая сохраняет свою третичную структуру независимо от остальной части белка. Как правило, домены ответственны за отдельные функциональные свойства белков, и во многих случаях, могут быть добавлены, удалены или перенесены в другие белки без потери функции остальной части белка и/или домена.
Термин «нуклеиновая кислота», или «полинуклеотид», относится к дезоксирибонуклеиновым кислотам (ДНК) или рибонуклеиновым кислотам (РНК), а также их полимерам в одно- или двухцепочечной форме. Если конкретные ограничения отсутствуют, термин охватывает нуклеиновые кислоты, содержащие известные аналоги природных нуклеотидов, которые имеют свойства связывания, аналогичные свойствам эталонной нуклеиновой кислоты, и метаболизируются аналогично природным нуклеотидам. Если нет иных указаний, конкретная последовательность нуклеиновой кислоты также косвенно охватывает ее консервативно модифицированные варианты (например, замены вырожденных кодонов), аллели, ортологи, ОНП и комплементарные последовательности, а также непосредственно указанную последовательность. В частности, замены вырожденных кодонов могут быть получены путем создания последовательностей, в которых третье положение одного или более выбранных (или всех) кодонов замещено смешанным основанием и/или остатками дезоксиинозина (Batzer et al. Nucleic Acid Res. 19:5081 (1991); Ohtsuka et al. J. Biol. Chem. 260:2605-2608 (1985); и Rossolini et al. Mol. Cell. Probes 8:91-98 (1994)).
Один вариант осуществления изобретения относится к последовательности нуклеиновой кислоты, кодирующей CD96-связывающий белок, описанный в любом из предшествующих вариантов осуществления.
Следующий вариант осуществления изобретения относится к последовательности нуклеиновой кислоты, описанной в предшествующем варианте осуществления, которая включает SEQ ID NO 4, 8, 12, 16, 20, 24, 28, 32, 36, 40, 44, 48, 52, 56, 60, 64, 68, 72, 76, 80, 84, 88, 92 или 96, кодирующие тяжелую цепь; и/или SEQ ID NO: 3, 7, 11, 15, 19, 23, 27, 31, 35, 39, 43, 47, 51, 55, 59, 63, 67, 71, 75, 79, 83, 87, 91 или 95, кодирующие легкую цепь. Другой вариант осуществления изобретения относится к экспрессионному вектору, содержащему нуклеиновую кислоту, описанную в любом из предшествующих вариантов осуществления.
Композиции, описанные в настоящем описании, могут быть получены различными общепринятыми методами. Например, композиции могут быть экспрессированы в рекомбинантных экспрессионных системах и очищены из них. В одном варианте осуществления композиция получена способом культивирования клетки-хозяина в условиях, подходящих для экспрессии CD96-связывающего белка, описанного в любом из предшествующих вариантов осуществления, при этом композиция экспрессирована и, необязательно, очищена и, необязательно, сформулирована в фармацевтическую композицию.
Целый ряд различных экспрессионных систем и методов очистки можно использовать для получения композиций. Как правило, клетки-хозяева трансформируют рекомбинантным экспрессионным вектором, кодирующим антитело. Можно использовать широкий спектр клеток-хозяев, включая линии эукариотических клеток, полученных от млекопитающих (например, CHO, Perc6, HEK293, HeLa, NS0). Подходящие клетки-хозяева включают такие клетки млекопитающих, как CHO (например, CHOK1 и CHO-DG44).
Клетка-хозяин может представлять собой выделенную клетку-хозяина. Клетка-хозяин, как правило, не является частью многоклеточного организма (например, растения или животного). Клетка-хозяин может представлять собой не принадлежащую человеку клетку-хозяина.
Соответствующие клонирующие и экспрессионные векторы для использования с эукариотическими или полученными от млекопитающих клетками-хозяевами и способы клонирования известны в данной области.
Клетки можно культивировать в условиях, стимулирующих экспрессию антитела. Например, для культивирования клеток используют производственный биореактор. Объем производственного биореактора может составлять: (i) приблизительно 20000 литров, приблизительно 10000 литров; приблизительно 5000 литров; приблизительно 2000 литров; приблизительно 1000 литров; или приблизительно 500 литров; или (ii) от 500 до 20000 литров; от 500 до 10000 литров; от 500 до 5000 литров; от 1000 до 10000 литров или от 2000 до 10000 литров. Например, клетки можно культивировать в производственном биореакторе при значении pH, составляющем от приблизительно 6,75 до 7,00. Альтернативно, клетки можно культивировать в производственном биореакторе в течение срока от приблизительно 12 до приблизительно 18 дней. Альтернативно, клетки можно культивировать в производственном биореакторе при значении pH, составляющем от приблизительно 6,75 до 7,00, в течение срока от приблизительно 12 до приблизительно 18 дней. Этот этап культивирования может помогать контролировать уровень дезамидированных вариантов антител, например, снижать уровень дезамидированных вариантов антител.
Композицию можно извлекать и очищать общепринятыми методами очистки белков. Например, композицию можно собирать непосредственно из культуральной среды. Сбор из клеточной культуральной среды можно осуществлять путем осветления, например, путем центрифугирования и/или глубинного фильтрования. После извлечения композиции производят ее очистку для обеспечения адекватной степени чистоты.
Следующий вариант осуществления изобретения относится к рекомбинантной клетке-хозяину, содержащей последовательности нуклеиновых кислот или экспрессионный вектор, описанный в любом из предшествующих вариантов осуществления. Другой вариант осуществления изобретения относится к способу получения CD96-связывающего белка, включающему культивирование клетки-хозяина, описанной в предшествующем варианте осуществления, в условиях, подходящих для экспрессии указанной последовательности нуклеиновой кислоты(кислот) или вектора(векторов), с получением полипептида, представляющего собой CD96-связывающий белок. Следующий вариант осуществления изобретения относится к CD96-связывающему белку, полученному способом получения, описанным в предшествующем варианте осуществления. Другой вариант осуществления изобретения относится к линии клеток, генетически модифицированных для экспрессии CD96-связывающего белка, описанного в любом из предшествующих вариантов осуществления.
«Процент идентичности» между искомой последовательностью нуклеиновой кислоты и предметной последовательностью нуклеиновой кислоты представляет собой показатель «идентичности», выраженный в процентах, который рассчитывается алгоритмом BLASTN, когда предметная последовательность нуклеиновой кислоты имеет 100% покрытие по запросу искомой последовательностью нуклеиновой кислоты после выполнения попарного выравнивая BLASTN. Такие попарные выравнивания BLASTN между искомой последовательностью нуклеиновой кислоты и предметной последовательностью нуклеиновой кислоты проводят с использованием параметров по умолчанию алгоритма BLASTN, доступного на вебсайте Национального центра биотехнологической информации, с отключенным фильтром для областей низкой сложности. Важно отметить, что искомая последовательность нуклеиновой кислоты может быть описана последовательностью нуклеиновой кислоты, идентифицированной в одном или более пунктов формулы изобретения в настоящем описании.
«Процент идентичности» между искомой аминокислотной последовательностью и предметной аминокислотной последовательностью представляет собой показатель «идентичности», выраженный в процентах, который рассчитывается алгоритмом BLASTP, когда предметная аминокислотная последовательность имеет 100% покрытие по запросу искомой аминокислотной последовательностью после выполнения попарного выравнивая BLASTP. Такие попарные выравнивания BLASTP между искомой аминокислотной последовательностью и предметной аминокислотной последовательностью проводят с использованием параметров по умолчанию алгоритма BLASTP, доступного на вебсайте Национального центра биотехнологической информации, с отключенным фильтром для областей низкой сложности. Важно отметить, что искомая аминокислотная последовательность может быть описана аминокислотной последовательностью, идентифицированной в одном или более пунктов формулы изобретения в настоящем описании.
Искомая последовательность может быть на 100% идентичной предметной последовательности, или может включать определенное целочисленное количество аминокислотных или нуклеотидных изменений в сравнении с предметной последовательностью, так что % идентичности составляет менее 100%. Например, искомая последовательность по меньшей мере на 50, 60, 70, 75, 80, 85, 90, 95, 96, 97, 98 или 99% идентична предметной последовательности. Такие изменения включают по меньшей мере одну аминокислотную делецию, замену (включая консервативную и не консервативную замену) или вставку, и при этом указанные изменения могут иметь место в амино-концевых или карбокси-концевых положениях искомой последовательности, или в любом участке между этими концевыми положениями, вкрапленные либо отдельно среди аминокислот или нуклеотидов в искомой последовательности, либо одной или более непрерывными группами в искомой последовательности.
Используемый в настоящем описании термин «идентичность последовательностей» означает степень сходства между двумя или более аминокислотными последовательностями, или двумя или более нуклеотидными последовательностями, определяемую путем сравнения последовательностей. Сравнение последовательностей и определение идентичности последовательностей можно выполнять с использованием математического алгоритма; специалистам в данной области известны компьютерные программы, доступные для выравнивания двух последовательностей и определения процента идентичности между ними. Специалистам известно, что разные алгоритмы могут приводить к слегка отличающимся результатам.
Используемый в настоящем описании термин «специфически связывает», и его грамматические вариации, применительно к антителу означает, что антитело, или фрагмент антитела, узнает и связывает определенный антиген, но практически не узнает или не связывает другие молекулы в образце. Например, антитело, которое специфически связывает антиген из одного биологического вида, также может связывать этот антиген из одного или более других биологических видов. Однако такая межвидовая реакционная способность сама по себе не изменяет классификацию антитела как специфического. В другом примере антитело, которое специфически связывает антиген, может также связывать другие аллельные формы антигена. Однако такая перекрестная реакционная способность сама по себе не изменяет классификацию антитела как специфического. В некоторых случаях термины «специфическое связывание» или «специфически связывающее» могут быть использованы применительно к взаимодействию антитела, белка или пептида со второй химической молекулой для указания на то, что взаимодействие зависит от присутствия конкретной структуры (например, антигенной детерминанты или эпитопа) на химической молекуле; например, антитело узнает и связывает конкретную белковую структуру, а не сами белки в целом. Если антитело является специфическим для эпитопа «A», присутствие молекулы, содержащей эпитоп A (или свободного, немеченого «A»), в реакционной смеси, содержащей меченый «A» и антитело, будет уменьшать количество меченого «A», связанного с антителом.
Используемый в настоящем описании термин «фармацевтическая композиция» означает композицию, подходящую для введения пациенту.
Фармацевтические композиции, описанные в настоящем описании, могут содержать очищенные препараты CD96-связывающих белков, описанных в настоящем описании.
Например, фармацевтический препарат может содержать очищенный препарат CD96-связывающего белка, описанного в настоящем описании, в сочетании с фармацевтически приемлемым носителем.
Как правило, такие фармацевтические композиции содержат фармацевтически приемлемый носитель, известный, и считающийся таковым, в общепринятой фармацевтической практике. Примеры таких носителей включают стерилизованные носители, такие как солевой раствор, раствор Рингера или раствор декстрозы, необязательно, буферизованный соответствующими буферами до значения pH в диапазоне от 5 до 8.
Фармацевтические композиции можно вводить путем инъекции или инфузии (например, внутривенной, внутрибрюшинной, внутрикожной, подкожной, внутримышечной или интрапортальной). Такие композиции предпочтительно не содержат видимых частиц. Фармацевтические композиции могут содержать от 1 мг до 10 г антигенсвязывающего белка, например, от 5 мг до 1 г антигенсвязывающего белка. Альтернативно, композиция может содержать от 5 мг до 500 мг антигенсвязывающего белка, например, от 5 мг до 50 мг.
Способы получения таких фармацевтических композиций хорошо известны специалистам в данной области. Фармацевтические композиции могут содержать от 1 мг до 10 г антигенсвязывающего белка в стандартной лекарственной форме, необязательно, вместе с инструкциями по применению. Фармацевтические композиции могут быть лиофилизированными (сублимированными) для восстановления перед введением способами, хорошо известными или очевидными для специалистов в данной области. Если антитела имеют изотип IgG1, в фармацевтическую композицию можно добавлять хелатор меди, такой как цитрат (например, цитрат натрия) или ЭДТА, или гистидин, для уменьшения степени опосредуемой медью деградации антител этого изотипа. Фармацевтические композиции могут также содержать солюбилизатор, такой как аргинин, сурфактант/средство против агрегации, например, полисорбат 80, и инертный газ, такой как азот, для замены кислорода в свободном пространстве над продуктом во флаконе.
Один вариант осуществления изобретения относится к фармацевтической композиции, содержащей CD96-связывающий белок, описанный в любом из предшествующих вариантов осуществления, и фармацевтически приемлемый эксципиент. Следующий вариант осуществления изобретения относится к фармацевтической композиции, содержащей терапевтически эффективное количество CD96-связывающего белка, описанного в любом из предшествующих вариантов осуществления.
Используемый в настоящем описании термин «противоопухолевый эффект» означает биологический эффект, который может проявляться в виде снижения скорости роста опухоли, уменьшения объема опухоли, уменьшения количества опухолевых клеток, уменьшения количества метастазов, увеличения продолжительности жизни или ослабления различных физиологических симптомов, связанных с онкологическим состоянием. «Противоопухолевый эффект» также может проявляться в виде способности пептидов, полинуклеотидов, клеток и антител по изобретению к предотвращению изначального возникновения опухоли.
В настоящем описании термины «рак», «новообразование» и «опухоль» используются взаимозаменяемо и, в единственном или во множественном числе, относятся к клеткам, которые подверглись злокачественной трансформации, которая сделала их патологическими для организма-хозяина. Иллюстративные примеры клеток, на которые могут быть направлены композиции и способы, предусмотренные в конкретных вариантах осуществления, включают, но без ограничения, следующие виды рака: синовиальную саркому, немелкоклеточную карциному легких (NSCLC), миксоидную круглоклеточную липосаркому (MRCLS) и множественную миелому (MM). Первичные раковые клетки можно с легкостью отличать от не злокачественных клеток с помощью хорошо зарекомендовавших себя методов, в частности, гистологического анализа. Используемое в настоящем описании определение раковой клетки охватывает не только первичную раковую клетку, но и любую клетку, происходящую от раковой клетки-предка. Сюда относятся метастатические раковые клетки, а также in vitro культуры и линии клеток, полученные из раковых клеток. Когда речь идет о типе рака, который обычно проявляется в виде солидной опухоли, «клинически выявляемая» опухоль представляет собой опухоль, которую обнаруживают по опухолевой массе; например, с помощью таких методов, как компьютерная томография (КТ), магнитно-резонансная томография (МРТ), рентгеноскопия, ультразвуковое обследование или пальпация при физическом осмотре, и/или которую обнаруживают вследствие экспрессии одного или более специфических для рака антигенов в образце, полученном от пациента. Опухоли могут представлять собой гемопоэтический рак (или гематологический рак или рак крови), например, виды рака, происходящего из клеток крови или иммунных клеток, такие опухоли могут быть названы «жидкими опухолями». Конкретные примеры клинических состояний на основе гематологических опухолей включают лейкозы, такие как хронический миелоцитарный лейкоз, острый миелоцитарный лейкоз, хронический лимфоцитарный лейкоз и острый лимфоцитарный лейкоз; злокачественные новообразования из плазматических клеток, такие как множественная миелома, MGUS и макроглобулинемия Вальденстрема; лимфомы, такие как неходжскинская лимфома, лимфома Ходжкина, и тому подобное.
Рак может быть любым раком, при котором имеет место аномальное количество бластных клеток или нежелательная пролиферация клеток, или который диагностируют как гематологический рак, включая как лимфоидные, так и миелоидные, злокачественные новообразования. Миелоидные злокачественные новообразования включают, но не ограничиваются ими, острый миелоидный (или миелоцитарный, или миелогенный, или миелобластный) лейкоз (недифференцированный или дифференцированный), острый промиелоидный (или промиелоцитарный, или промиелогенный, или промиелобластный) лейкоз, острый миеломоноцитарный (или миеломонобластный) лейкоз, острый моноцитарный (или монобластный) лейкоз, эритролейкоз и мегакариоцитарный (или мегакариобластный) лейкоз. Эти лейкозы могут быть совместно названы острым миелоидным (или миелоцитарным, или миелогенным) лейкозом (AML). Миелоидные злокачественные новообразования также включают миелопролиферативные заболевания (МПЗ), которые включают, но не ограничиваются ими, хронический миелогенный (или миелоидный) лейкоз (CML), хронический миеломоноцитарный лейкоз (CMML), эссенциальную тромбоцитемию (или тромбоцитоз) и истинную полицитемию (PCV). Миелоидные злокачественные новообразования также включают миелодисплазию (или миелодиспластический синдром, или MDS), которая может быть названа рефрактерной анемией (RA), рефрактерной анемией с избытком бластов (RAEB) и рефрактерной анемией с избытком бластов при трансформации (RAEBT); а также миелофиброз (MFS) с агногенной миелоидной метаплазией или без нее.
Гемопоэтические виды рака также включают лимфоидные злокачественные новообразования, которые могут затрагивать лимфатические узлы, селезенку, костный мозг, периферическую кровь и/или экстранодальные участки. Лимфоидный рак включает B-клеточные злокачественные новообразования, которые включают, но без ограничения, B-клеточные неходжскинские лимфомы (B-NHL). B-NHL могут быть медленно прогрессирующими (или низкой степени злокачественности), средней степени злокачественности (или агрессивными) или высокой степени злокачественности (очень агрессивными). Медленно прогрессирующие B-клеточные лимфомы включают фолликулярную лимфому (FL); мелколимфоцитарную лимфому (SLL); лимфому из клеток маргинальной зоны (MZL), включая нодальную MZL, экстранодальную MZL, селезеночную MZL и селезеночную MZL с ворсинчатыми лимфоцитами; лимфоплазматическую лимфому (LPL) и лимфому лимфоидной ткани, ассоциированной со слизистой оболочкой (MALT или экстранодальную лимфому из клеток маргинальной зоны). B-NHL средней степени злокачественности включают лимфому из клеток мантийной зоны (MCL) с лейкозным поражением или без него, диффузную крупноклеточную лимфому (DLBCL), фолликулярную крупноклеточную (категории 3 или категории 3B) лимфому и первичную медиастинальную лимфому (PML). B-NHL высокой степени злокачественности включают лимфому Беркитта (BL), подобную лимфоме Беркитта лимфому, мелкоклеточную лимфому с нерассеченными ядрами (SNCCL) и лимфобластную лимфому. Другие B-NHL включают иммунобластную лимфому (или иммуноцитому), первичную выпотную лимфому, ВИЧ-ассоциированные (или связанные со СПИДом) лимфомы и посттрансплантационное лимфопролиферативное заболевание (PTLD) или лимфому. B-клеточные злокачественные новообразования также включают, но без ограничения, хронический лимфоцитарный лейкоз (CLL), пролимфоцитарный лейкоз (PLL), макроглобулинемию Вальденстрема (WM), волосатоклеточный лейкоз (HCL), лейкоз из больших гранулярных лимфоцитов (LGL), острый лимфоидный (или лимфоцитарный, или лимфобластный) лейкоз и болезнь Кастлемана. NHL также могут включать T-клеточные неходжскинские лимфомы (T-NHL), которые включают, но без ограничения, T-клеточную неходжскинскую лимфому неуточненного генеза (NOS), периферическую T-клеточную лимфому (PTCL), анапластическую крупноклеточную лимфому (ALCL), ангиоиммунобластное лимфоидное заболевание (AILD), назальную лимфому из клеток-естественных киллеров (NK)/T-клеток, гамма/дельта лимфому, кожную T-клеточную лимфому, фунгоидный микоз и синдром Сезари.
Гемопоэтические виды рака также включают лимфому (или болезнь) Ходжкина, включая классическую лимфому Ходжкина, нодулярную склерозирующую лимфому Ходжкина, лимфому Ходжкина из смешанных клеток, лимфому Ходжкина с преобладанием лимфоцитов (LP), нодулярную LP лимфому Ходжкина и лимфому Ходжкина с истощением лимфоцитов. Гемопоэтические виды рака также включают болезни, или формы рака, плазматических клеток, такие как множественная миелома (MM), включая вялотекущую MM, моноклональная гаммопатия неопределенного (или неизвестного, или неясного) генеза (MGUS), плазмацитома (костей, экстрамедуллярная), лимфоплазматическая лимфома (LPL), макроглобулинемия Вальденстрема, плазмоклеточный лейкоз и первичный амилоидоз (AL). Гемопоэтические виды рака также могут включать другие виды рака из других гемопоэтических клеток, включая полиморфноядерные лейкоциты (или нейтрофилы), базофилы, эозинофилы, дендритные клетки, тромбоциты, эритроциты и клетки-естественные киллеры. Ткани, содержащие гемопоэтические клетки, в настоящем описании называемые «тканями из гемопоэтических клеток», включают костный мозг; периферическую кровь; тимус и периферические лимфоидные ткани, такие как селезенка, лимфатические узлы, лимфоидные ткани, связанные со слизистой оболочкой (например, лимфоидные ткани, связанные со слизистой оболочкой кишечника), миндалины, Пейеровы бляшки и аппендикс, а также лимфоидные ткани, связанные с другими слизистыми оболочками, например, с выстилкой бронхов.
В одном из вариантов осуществления виды рака, которые можно лечить связывающими белками по изобретению, могут включать солидные опухоли (например, рецидивирующие, метастатические или прогрессирующие солидные опухоли). Примеры таких солидных опухолей включают рак яичников, легких (например, NSCLC), желудка, мочевого пузыря, колоректальный рак, рак печени (например, HCC), почки (например, RCC), а также плоскоклеточную карциному головы и шеи (HNSCC).
Используемый в настоящем описании термин «терапевтическое применение» означает лечение и/или профилактику. Терапевтический эффект достигается за счет подавления, ремиссии или устранения болезненного состояния.
Используемый в настоящем описании термин «терапевтически эффективное количество», или «эффективное количество», означает количество, которое обеспечивает терапевтическую или профилактическую пользу, или вызывает биологический или медицинский ответ в ткани, системе или организме субъекта, которого добивается исследователь, ветеринар, врач или другой клиницист. Терапевтически эффективные количества и режимы лечения, как правило, определяют эмпирически, и они могут зависеть от таких факторов, как возраст, масса тела и состояние здоровья пациента, а также заболевание или нарушение, подвергаемое лечению. Такие факторы находятся в компетенции лечащего врача.
Используемый в настоящем описании термин «лечение», и его грамматические вариации, означает терапевтическое вмешательство. Применительно к конкретному состоянию лечение означает: (1) облегчение или предотвращение состояния, или одного или более из биологических проявлений состояния, (2) вмешательство (a) в функцию одного или более узловых пунктов в биологическом каскаде, которые являются причиной, или ответственны за развитие, состояния, или (b) в одно или более из биологических проявлений состояния, (3) ослабление одного или более из симптомов, эффектов или побочных эффектов, связанных с состоянием, или их лечение, (4) замедление прогрессирования состояния, или одного или более из биологических проявлений состояния, и/или (5) исцеление указанного состояния, или одного или более из биологических проявлений состояния, путем элиминации или уменьшения до неподдающихся обнаружению уровней одного или более из биологических проявлений состояния в течение периода времени, который считается состоянием ремиссии данного проявления, без дополнительного лечения в течение периода ремиссии. Специалисту в данной области известна продолжительность времени, считающаяся ремиссией для конкретного заболевания или состояния. Профилактическое лечение также охвачено данным понятием. Квалифицированный специалист понимает, что «предотвращение» не является абсолютным термином. В области медицины «предотвращение» понимают как профилактическое введение лекарственного средства для существенного снижения вероятности или степени тяжести заболевания, или его биологического проявления, или для отсрочки начала такого состояния, или его биологического проявления. Профилактическое лечение уместно, например, когда считается, что субъект имеет высокий риск развития рака, например, когда субъект имеет рак в семейном анамнезе или когда субъект был подвергнут воздействию канцерогенов.
В настоящем описании термины «индивидуум», «субъект» и «пациент» используют взаимозаменяемо. В одном варианте осуществления субъект является млекопитающим, таким как примат, например, мартышка или обезьяна, или человек. В следующем варианте осуществления субъект является человеком.
Доза антигенсвязывающего белка, вводимая субъекту, как правило, составляет от 1 мкг/кг до 150 мг/кг, от 0,1 мг/кг до 100 мг/кг, от 0,5 мг/кг до 50 мг/кг, от 1 до 25 мг/кг, от приблизительно 0,3 мг/кг до приблизительно 3 мг/кг или от 1 до 10 мг/кг массы тела субъекта. Например, доза может составлять 10 мг/кг, 30 мг/кг или 60 мг/кг. Доза также может составлять от 10 мг/кг до 110 мг/мг, от 15 мг/кг до 25 мг/кг или от 15 мг/кг до 100 мг/кг. Антигенсвязывающий белок можно вводить, например, парентерально, подкожно, внутривенно или внутримышечно. Дозы также можно вводить в расчете на субъекта, например, от приблизительно 20 мг на субъекта до приблизительно 750 мг на субъекта, от приблизительно 75 мг на субъекта до приблизительно 750 мг на субъекта, от приблизительно 20 мг на субъекта до приблизительно 200 мг на субъекта. Доза может находиться в пределах любого дискретного поддиапазона в таких диапазонах. Например, дозу также можно вводить подкожно в расчете на субъекта, например, приблизительно 100 мг на субъекта (например, один раз в четыре недели) или 300 мг на субъекта (или другие подкожно вводимые дозы, обеспечивающие достижение приблизительно такой же, или сопоставимой, биодоступности, как при внутривенном введении - например, три дозы по 100 мг на субъекта для достижения суммарной подкожно вводимой дозы 300 мг на субъекта).
Диапазоны любого вида, приведенные в настоящем описании, включают все значения в конкретном описанном диапазоне, а также значения около конечных точек конкретного диапазона.
При желании эффективную суточную дозу антитела или антигенсвязывающего белка по изобретению (например, в виде фармацевтической композиции) можно вводить в виде двух, трех, четырех, пяти, шести или более доз, вводимых раздельно через соответствующие промежутки времени в течение суток, необязательно, в стандартных лекарственных формах.
Введение дозы можно выполнять путем медленной непрерывной инфузии в течение периода времени от 2 до 24 часов, например, от 2 до 12 часов или от 2 до 6 часов. Такое введение может приводить к снижению побочных эффектов.
Введение дозы можно повторять один или более раз по мере необходимости, например, три раза в сутки, раз в сутки, раз в 2 дня, раз в неделю, раз в 14 дней, раз в месяц, раз в 3 месяца, раз в 4 месяца, раз в 6 месяцев или раз в 12 месяцев. Антигенсвязывающие белки можно вводить в виде поддерживающей терапии, например, один раз в неделю в течение 6 месяцев или более. Антигенсвязывающие белки можно вводить в виде прерывистой терапии, например, в течение 3-6 месяцев, затем без введения доз в течение 3-6 месяцев, с последующим введением антигенсвязывающих белков вновь в течение 3-6 месяцев, и так далее, циклически.
Например, дозу можно вводить подкожно, раз в 14 или 28 дней, в форме нескольких доз в каждый день введения. В одном варианте осуществления доза композиции составляет 100 мг один раз в 4 недели (28 дней).
Антигенсвязывающий белок можно вводить субъекту таким образом, чтобы терапия была направлена на конкретный участок.
CD96-связывающий белок в способах по изобретению может быть использован в сочетании, или при совместном введении, с одним или более другими терапевтически активными средствами, такими как антитела, низкомолекулярные ингибиторы, или в сочетании с клеточной терапией. Используемый в настоящем описании термин «совместное введение» означает либо одновременное введение, либо любой вариант раздельного последовательного введения, CD96-связывающего белка, описанного в настоящем описании, и дополнительного активного средства или средств, которые, как известно, полезны в лечении рака, включая химиотерапию и радиационную терапию. Используемый в настоящем описании термин «дополнительное активное средство или средства» охватывает любое соединение или лекарственное средство, которое, как известно или продемонстрировано, обладает полезными свойствами при введении пациенту, который нуждается в лечении рака. Предпочтительно, если введение не является одновременным, соединения вводят в непосредственной временной близости друг от друга. Более того, не важно, вводят ли соединения в одной и той же лекарственной форме, например, одно соединение можно вводить инъекцией, а другое соединение можно вводить перорально.
Как правило, любое анти-неопластическое средство, обладающее активностью против восприимчивой опухоли, подвергаемой лечению, может быть совместно введено при лечении рака по настоящему изобретению. Примеры таких средств можно найти в публикации Cancer Principles and Practice of Oncology, V.T. Devita, T.S. Lawrence, and S.A. Rosenberg (редакторы), 10-е издание (5 декабря 2014 г.), издательства Lippincott Williams & Wilkins. Специалист в данной области может определять, какие сочетания средств могут быть полезны, исходя из конкретных характеристик лекарственных средств и подвергаемого лечению рака. Типичные анти-неопластические средства, полезные по настоящему изобретению, включают, но без ограничения, нарушающие сборку микротрубочек или антимитотические средства; комплексные соединения платины; алкилирующие средства; антибиотики; ингибиторы топоизомеразы I; ингибиторы топоизомеразы II; антиметаболиты; гормоны и аналоги гормонов; ингибиторы путей сигнальной трансдукции; ингибиторы нерецепторных тирозинкиназ, связанных с ангиогенезом; иммунотерапевтические средства; проапоптотические средства; ингибиторы сигнализации клеточного цикла; ингибиторы протеасом; ингибиторы белков теплового шока; ингибиторы метаболизма рака и средства генной терапии рака.
Примерами дополнительного активного ингредиента или ингредиентов для применения в сочетании или совместном введении с раскрытыми в настоящем описании CD96-связывающими белками являются анти-неопластические средства. Примеры анти-неопластических средств включают, но без ограничения, химиотерапевтические средства; иммуномодулирующие средства; иммуномодуляторы и иммуностимулирующие адъюванты.
Раскрытые в настоящем описании CD96-связывающие белки могут также быть использованы в сочетании с анти-TIGIT антителами. Такое сочетание может дополнительно повышать активацию CD155/CD226. Сочетание с анти-TIGIT антителом можно использовать для лечения солидных опухолей, таких как опухоли почек, например, почечноклеточный рак (RCC). Примером такого анти-TIGIT антитела является тираголумаб.
Нарушающие сборку микротрубочек или анти-митотические средства представляют собой специфические для фазы клеточного цикла средства, активные в отношении микротрубочек в клетках опухолей в процессе M или фазы митоза клеточного цикла. Примеры нарушающих сборку микротрубочек средств включают, но без ограничения, дитерпеноиды и алкалоиды барвинка.
Комплексные соединения платины представляют собой не зависящие от фазы клеточного цикла противораковые средства, которые взаимодействуют с ДНК. Комплексы платины проникают в опухолевые клетки, подвергаются гидратации и образуют внутри- и межцепочечные перекрестные сшивки с ДНК, вызывая неблагоприятные биологические эффекты в опухоли. Примеры комплексных соединений платины включают, но без ограничения, цисплатин и карбоплатин.
Алкилирующие средства представляют собой не зависящие от фазы клеточного цикла противораковые средства и сильные электрофилы. Как правило, алкилирующие средства образуют путем алкилирования ковалентные связи с ДНК через нуклеофильные фрагменты молекулы ДНК, такие как фосфат, амино, сульфгидрильные, гидроксильные, карбоксильные и имидазольные группы. Такое алкилирование нарушает функцию нуклеиновой кислоты, приводя к клеточной гибели. Примеры алкилирующих средств включают, но без ограничения, азотистые иприты, такие как циклофосфамид, мелфалан и хлорамбуцил; алкилсульфонаты, такие как бусульфан; нитрозомочевины, такие как кармустин; и триазены, такие как дакарбазин.
Анти-неопластические антибиотики представляют собой не зависящие от фазы клеточного цикла средства, которые связываются с, или встраиваются в, ДНК. Такое действие нарушает обычную функцию нуклеиновых кислот, приводя к клеточной гибели. Примеры анти-неопластических антибиотиков включают, но без ограничения, актиномицины, такие как дактиномицин; антрациклины, такие как даунорубицин и доксорубицин; и блеомицины.
Ингибиторы топоизомеразы I включают, но без ограничения, камптотецины. Цитотоксическая активность камптотецинов, как считают, связана с их активностью ингибирования топоизомеразы I.
Ингибиторы топоизомеразы II включают, но без ограничения, эпиподофиллотоксины. Эпиподофиллотоксины представляют собой специфические для фазы клеточного цикла анти-неопластические средства, полученные из растения мандрагоры. Эпиподофиллотоксины, как правило, влияют на клетки в фазах S и G2 клеточного цикла, образуя тройной комплекс с топоизомеразой II и ДНК, и вызывая разрывы цепей ДНК. Разрывы цепей накапливаются, и наступает клеточная гибель. Примеры эпиподофиллотоксинов включают, но без ограничения, этопозид и тенипозид.
Анти-неопластические антиметаболиты представляют собой специфические для фазы клеточного цикла анти-неопластические средства, которые действуют в фазе S (синтез ДНК) клеточного цикла, ингибируя синтез ДНК или ингибируя синтез пуриновых или пиримидиновых оснований, и за счет этого ограничивая синтез ДНК. Как следствие, фаза S не продолжается, и наступает клеточная гибель. Примеры анти-неопластических антиметаболитов включают, но без ограничения, фторурацил, метотрексат, цитарабин, меркаптопурин, тиогуанин и гемцитабин.
Гормоны и аналоги гормонов представляет собой полезные соединения для лечения рака, при котором существует взаимосвязь между гормоном(ами) и ростом и/или отсутствием роста раковых клеток. Примеры гормонов и аналогов гормонов, полезных для лечения рака, включают, но без ограничения, адренокортикостероиды, такие как преднизон и преднизолон; аминоглутетимид и другие ингибиторы ароматазы, такие как анастрозол, летразол, воразол и экземестан; прогестрины, такие как магестрол ацетат; эстрогены, андрогены, а также анти-андрогены, такие как флутамид, нилутамид, бикалутамид, ципротерон ацетат и 5αα-редуктазы, такие как финастерид и дутастерид; анти-эстрогены, такие как тамоксифен, торемифен, ралоксифен, дролоксифен иодоксифен, и избирательные модуляторы рецепторов эстрогенов (SERMS); а также гонадотропин-рилизинг гормон (GnRH) и его аналоги, которые стимулируют высвобождение лютеинизирующего гормона (LH) и/или фолликулостимулирующего гормона (FSH), агонисты и антагонисты LHRH, такие как гозерелин ацетат и леупролид.
Ингибиторы путей сигнальной трансдукции представляют собой ингибиторы, которые блокируют или ингибируют химический процесс, который вызывает внутриклеточное изменение. В настоящем описании данное изменение представляет собой клеточную пролиферацию или дифференциацию. Ингибиторы сигнальной трансдукции, полезные по настоящему изобретению, включают, но без ограничения, ингибиторы рецепторных тирозинкиназ, нерецепторных тирозинкиназ, блокаторы доменов SH2/SH3, серин/треонин киназ, фосфатидилинозитол-3-киназ, сигнализации мио-инозитола и онкогенов Ras.
Некоторые протеинтирозинкиназы катализируют фосфорилирование определенных остатков тирозина в различных белках, участвующих в регуляции клеточного роста. Такие протеинтирозинкиназы могут быть в широком смысле классифицированы как рецепторные или нерецепторные киназы.
Рецепторные тирозинкиназы представляют собой трансмембранные белки, имеющие внеклеточный лиганд-связывающий домен, трансмембранный домен и домен тирозинкиназы. Рецепторные тирозинкиназы участвуют в регуляции клеточного роста, и их обычно называют рецепторами фактора роста. Несоответствующая или неконтролируемая активация многих таких киназ, то есть, аберрантная активность киназного рецептора фактора роста, например, вследствие избыточной экспрессии или мутации, как показано, приводит к неконтролируемому клеточному росту. Соответственно, аберрантная активность таких киназ была соотнесена с ростом злокачественной ткани. Следовательно, использование ингибиторов таких киназ может быть способом лечения рака. Рецепторы фактора роста включают, например, рецептор эпидермального фактора роста (EGFr), рецептор тромбоцитарного фактора роста (PDGFr), erbB2, erbB4, рецептор фактора роста эндотелия сосудов (VEGFR), тирозинкиназу с иммуноглобулиноподобным и гомологичным эпидермальному фактору роста доменами (TIE-2), рецептор инсулинопобного фактора роста I (IGFI), макрофагальный колониестимулирующий фактор (Cfms), BTK, ckit, cmet, рецепторы фактора роста фибробластов (FGF), рецепторы Trk (TrkA, TrkB, и TrkC), рецепторы эфрина (eph) и протоонкоген RET. Некоторые ингибиторы рецепторов факторов роста находятся в стадии разработки и включают антагонисты лигандов, антитела, ингибиторы тирозинкиназ и антисмысловые олигонуклеотиды. Рецепторы факторов роста и средства, которые ингибируют функцию рецепторов факторов роста, описаны, например, в публикациях Kath J.C., Exp.Opin. Ther. Patents, 10(6):803-818 (2000); Shawver L.K. et al. Drug Discov. Today, 2(2): 50-63 (1997); и Lofts, F.J. and Gullick W.J. «Growth factor receptors as targets» in New Molecular Targets for Cancer Chemotherapy, Kerr D.J. and Workman P. (редакторы), (27 июня 1994 г.), CRC Press. Неограничивающие примеры ингибиторов рецепторов факторов роста включают пазопаниб и сорафениб.
Тирозинкиназы, которые не являются рецепторными киназами факторов роста, называют нерецепторными тирозинкиназами. Нерецепторные тирозинкиназы, полезные по настоящему изобретению, которые являются мишенями или потенциальными мишенями противораковых лекарственных средств, включают cSrc, Lck, Fyn, Yes, Jak, cAbl, FAK (киназу фокальной адгезии), тирозинкиназу Брутона и Bcr-Abl. Такие нерецепторные киназы и средства, ингибирующие функцию нерецепторных тирозинкиназ, описаны в Sinha S. and Corey S.J., J. Hematother. Stem Cell Res., 8(5): 465-480 (2004) и Bolen, J.B., Brugge, J.S., Annu. Rev. Immunol., 15: 371-404 (1997).
Блокаторы доменов SH2/SH3 представляют собой средства, которые нарушают связывание домена SH2 или SH3 в различных ферментах или адаптерных белках, включая p85 субъединицу PI3-K, киназы семейства Src, адаптерные молекулы (Shc, Crk, Nck, Grb2) и Ras-GAP. Домены SH2/SH3 в качестве мишеней для противораковых лекарственных средств описаны в Smithgall T.E., J. Pharmacol. Toxicol. Methods, 34(3): 125-32 (1995).
Ингибиторы серин/треонин киназ включают, но без ограничения, блокаторы каскада MAP-киназы, которые включают блокаторы Raf-киназ (rafk), регулируемых митогенами или внеклеточными сигналами киназ (MEK) и регулируемых внеклеточными сигналами киназ (ERK); блокаторы членов семейства протеинкиназ C, включая блокаторы PKC (альфа, бета, гамма, эпсилон, мю, лямбда, йота, зета); IkB-киназ (IKKa, IKKb); киназ семейства PKB; членов семейства AKT-киназ; киназных рецепторов TGF-бета; а также ингибиторы мишени рапамицина у млекопитающих (mTOR), включая, но без ограничения, рапамицин (FK506) и рапалоги, RAD001 или эверолимус (афинитор®), CCI-779 или темсиролимус, AP23573, AZD8055, WYE-354, WYE-600, WYE-687 и Pp121. Примеры ингибиторов серин/треонин киназ включают, но без ограничения, траметиниб, дабрафениб, а также ингибиторы Akt афуресертиб и N-{(1S)-2-амино-1-[(3,4-дифторфенил)метил]этил}-5-хлор-4-(4-хлор-1-метил-1H-пиразол-5-ил)-2-фуранкарбоксамид.
Ингибиторы членов семейства фосфатидилинозитол-3-киназ, включая блокаторы PI3-киназы, ATM, DNA-PK и Ku, также могут быть использованы по настоящему изобретению. Такие киназы описаны в публикациях Abraham R.T., Curr. Opin. Immunol., 8(3): 412-418 (1996); Canman C.E., and Lim D.S., Oncogene, 17(25): 3301-3308 (1998); Jackson S.P., Int. J. Biochem. Cell Biol., 29(7): 935-938 (1997); и Zhong H. et al. Cancer Res., 60(6): 1541-1545 (2000).
По настоящему изобретению также могут быть использованы ингибиторы сигнализации мио-инозитола, такие как блокаторы фосфолипазы C и аналоги мио-инозитола. Такие ингибиторы сигналов описаны в Powis G., and Kozikowski A., «Inhibitors of Myo-Inositol Signaling», в: New Molecular Targets for Cancer Chemotherapy, Kerr D.J. and Workman P. (редакторы), (27 июня 1994 г.), CRC Press.
Другой группой ингибиторов путей сигнальной трансдукции являются ингибиторы онкогена Ras. Такие ингибиторы включают ингибиторы фарнезилтрансферазы, геранилгеранилтрансферазы и CAAX-протеаз, а также антисмысловые олигонуклеотиды, рибозимы и другие иммунотерапевтические средства. Показано, что такие ингибиторы блокируют активацию ras в клетках, содержащих мутант ras дикого типа, тем самым действуя в качестве антипролиферативных средств. Ингибирование онкогена Ras описано в Scharovsky O.G. et al. J. Biomed. Sci., 7(4): 292-298 (2000); Ashby M.N., Curr. Opin. Lipidol., 9(2): 99-102 (1998); и Bennett C.F. and Cowsert L.M., Biochem. Biophys. Acta., 1489(1): 19-30 (1999).
Антагонисты связывания лиганда рецепторных киназ также могут служить ингибиторами сигнальной трансдукции. К этой группе ингибиторов путей сигнальной трансдукции относятся гуманизированные антитела или другие антагонисты связывания внеклеточного лиганда с доменом рецепторных тирозинкиназ. Примеры антител или других антагонистов связывания лиганда рецепторной киназой включают, но без ограничения, цетуксимаб (эрбитукс®), трастузумаб (герцептин®); трастузумаб эмтанзин (кадсила®); пертузумаб (перьета®); ингибиторы ErbB, включая лапатиниб, эрлотиниб и гефитиниб; а также специфическое антитело к 2C3 VEGFR2 (смотри Brekken R.A. et al. Cancer Res., 60(18): 5117-5124 (2000)).
Ингибиторы нерецепторных киназ, связанных с ангиогенезом, также могут быть использованы по настоящему изобретению. Ингибиторы связанных с ангиогенезом VEGFR и TIE2 описаны выше применительно к ингибиторам сигнальной трансдукции (оба рецептора являются рецепторными тирозинкиназами). Ангиогенез, как правило, связан с сигнализацией erbB2/EGFR, поскольку показано, что ингибиторы erbB2 и EGFR ингибируют ангиогенез, в основном экспрессию VEGF. Соответственно, ингибиторы нерецепторных тирозинкиназ могут быть использованы в сочетании с ингибиторами EGFR/erbB2 по настоящему изобретению. Например, анти-VEGF антитела, которые не узнают VEGFR (рецепторную тирозинкиназу), но связываются с лигандом; низкомолекулярные ингибиторы интегрина (альфа-бета 3), ингибирующие ангиогенез; эндостатин и ангиостатин (не-RTK) также могут быть полезны в сочетании с раскрытыми соединениями. (Смотри Bruns C.J. et al. Cancer Res., 60(11): 2926-2935 (2000); Schreiber A.B. et al. Science, 232(4755): 1250-1253 (1986); Yen L. et al. Oncogene, 19(31): 3460-3469 (2000)).
Средства, используемые в режимах иммунотерапии, также могут быть полезны в сочетании по настоящему изобретению. Существует ряд иммунологических стратегий для вызывания иммунного ответа против erbB2 или EGFR. Эти стратегии, как правило, относятся к области противоопухолевых вакцин. Эффективность иммунологических подходов может быть значительно повышена за счет комбинированного ингибирования сигнальных путей erbB2/EGFR с использованием низкомолекулярного ингибитора. Описание подхода с использованием иммунологических/противоопухолевых вакцин против erbB2/EGFR приведено в Reilly R.T. et al. Cancer Res., 60(13): 3569-3576 (2000); и Chen Y. et al. Cancer Res., 58(9): 1965-1971 (1998).
Средства, используемые в проапоптотических режимах лечения (например, антисмысловые олигонуклеотиды для Bcl-2) также могут быть использованы в сочетании по настоящему изобретению. Члены семейства белков Bcl-2 блокируют апоптоз. Вследствие этого повышенная экспрессия Bcl-2 была связана с резистентностью к химиотерапии. Исследования показали, что эпидермальный фактор роста (EGF) стимулирует антиапоптотические члены семейства Bcl-2 то есть, Mcl-1). Таким образом, стратегии, разработанные для снижения экспрессии Bcl-2 в опухолях, продемонстрировали клиническую пользу. Такие проапоптотические стратегии с использованием антисмысловых олигонуклеотидов для Bcl-2 описаны в Waters J.S. et al. J. Clin. Oncol., 18(9): 1812-1823 (2000); и Kitada S. et al. Antisense Res. Dev., 4(2): 71-79 (1994).
Ингибиторы сигнализации клеточного цикла ингибируют молекулы, вовлеченные в контроль клеточного цикла. Члены семейства белков киназ, называемых циклин-зависимыми киназами (CDK), и их взаимодействие с членами семейства белков, называемых циклинами, контролируют продвижение по клеточному циклу эукариотических клеток. Координация активации и инактивации разных комплексов циклин/CDK необходима для нормального продвижения по клеточному циклу. Несколько ингибиторов сигнализации клеточного цикла находятся в стадии разработки. Например, примеры циклин-зависимых киназ, включая CDK2, CDK4 и CDK6, а также их ингибиторов, описаны, например, в Rosania G.R., and Chang Y.T., Exp.Opin. Ther. Patents, 10(2): 215-230 (2000). Кроме того, p21WAF1/CIP1 был описан как сильный и универсальный ингибитор циклин-зависимых киназ (Cdk) (Ball K.L., Prog. Cell Cycle Res., 3: 125-134 (1997)). Соединения, которые, как известно, индуцируют экспрессию p21WAF1/CIP1, связаны с подавлением клеточной пролиферации и обладают активностью подавления роста опухолей (Richon V.M. et al. Proc. Natl. Acad. Sci. USA, 97(18): 10014-10019 (2000)), и их используют в качестве ингибиторов сигнализации клеточного цикла. Ингибиторы гистондеацетилазы (HDAC) участвуют в транскрипционной активации p21WAF1/CIP1 (Vigushin D.M., and Coombes R.C., Anticancer Drugs, 13(1): 1-13 (2002)) и являются подходящими ингибиторами сигнализации клеточного цикла для использования в сочетании по настоящему изобретению. Примеры таких ингибиторов HDAC включают, но без ограничения, вориностат, ромидепсин, панобиностат, вальпроевую кислоту и моцетиностат.
Ингибиторы протеасом представляют собой лекарственные средства, которые блокируют действие протеасом, клеточных комплексов, разрушающих белки, такие как белок p53. Несколько ингибиторов протеасом имеются в продаже или изучаются для лечения рака. Подходящие ингибиторы протеасом для использования в сочетании по настоящему изобретению, включают, но без ограничения, бортезомиб, дисульфирам, эпигаллокатехин галлат, салиноспорамид A и карфилзомиб.
Белки теплового шока с массой 70 килодальтон (Hsp70) и белки теплового шока с массой 90 килодальтон (Hsp90) относятся к семейству повсеместно экспрессируемых белков теплового шока. Hsp70 и Hsp90 избыточно экспрессируются при некоторых видах рака. Несколько ингибиторов Hsp70 и Hsp90 находятся в стадии изучения для лечения рака. Примеры ингибиторов Hsp70 и Hsp90 для использования в сочетании по настоящему изобретению включают, но без ограничения, танеспимицин и радицикол.
Метаболизм многих опухолевых клеток заметно отличается от метаболизма нормальных тканей. Например, скорость гликолиза, метаболического процесса, который преобразует глюкозу в пируват, увеличивается, а образующийся пируват восстанавливается до лактата, вместо того чтобы быть далее окисленным в митохондриях через цикл трикарбоновых кислот (ТСА). Этот эффект часто наблюдается даже в аэробных условиях и известен как эффект Варбурга.
Лактатдегидрогеназа А (LDH-A), изоформа лактатдегидрогеназы, экспрессируемая в мышечных клетках, играет ключевую роль в метаболизме опухолевых клеток, осуществляя восстановление пирувата до лактата, который затем может быть выведен из клетки. Было показано, что экспрессия этого фермента повышена в опухолях многих типов. Изменение метаболизма глюкозы, описанное в эффекте Варбурга, является критическим для роста и пролиферации раковых клеток, и было показано, что нокдаун LDH-A с помощью РНКи приводит к снижению пролиферации клеток и роста опухоли в моделях ксенотрансплантатов (Tennant D.A. et al. Nat. Rev. Cancer, 10(4): 267-277 (2010); Fantin V.R. et al. Cancer Cell, 9(6): 425-434 (2006)).
Высокие уровни синтазы жирных кислот (FAS) были обнаружены в предшественниках раковых опухолей. Фармакологическое ингибирование FAS влияет на экспрессию ключевых онкогенов, участвующих как в развитии, так и в поддержании рака. Alli P.M. et al. Oncogene, 24(1): 39-46 (2005).
Ингибиторы метаболизма рака, включая ингибиторы LDH-A и ингибиторы биосинтеза жирных кислот (или ингибиторы FAS), подходят для использования в сочетании по настоящему изобретению.
Генная терапия рака включает избирательный перенос рекомбинантной ДНК/РНК с помощью вирусных или невирусных доставляющих гены векторов для модификации раковых клеток в терапевтических целях. Примеры генной терапии рака включают, но не ограничиваются ими, терапию суицидальными и онколитическими генами, а также адоптивную Т-клеточную терапию.
Используемый в настоящем описании термин «иммуномодуляторы» относится к любому веществу, включая моноклональные антитела, которое оказывает влияние на иммунную систему. CD96-связывающие белки по настоящему изобретению можно считать иммуномодуляторами. Иммуномодуляторы могут быть использованы в качестве анти-неопластических средств для лечения рака. Например, иммуномодуляторы включают, но без ограничения, антитела или другие антагонисты CTLA-4, такие как ипилимумаб (ервой®), и PD-1, такие как достарлимаб, ниволумаб (опдиво®), пембролизумаб (кейтруда®) и цемиплимаб (либтайо®). Другие иммуномодуляторы включают, но без ограничения, антитела или другие антагонисты PD-L1, OX-40, LAG3, TIM-3, 41BB и GITR.
Используемый в настоящем описании термин «антагонист PD-1» означает любое химическое соединение, или биологическую молекулу, которое блокирует связывание PD-L1, экспрессируемого на раковой клетке, с PD-1, экспрессируемым на иммунной клетке (T-клетке, B-клетке или NKT-клетке), и предпочтительно также блокирует связывание PD-L2, экспрессируемого на раковой клетке, с PD-1, экспрессируемым на иммунной клетке. Альтернативные названия или синонимы для PD-1 и его лигандов включают: PDCD1, PD1, CD279 и SLEB2 для PD-1; PDCD1L1, PDL1, B7H1, B7-4, CD274 и B7-H для PD-L1; а также PDCD1L2, PDL2, B7-DC, Btdc и CD273 для PD-L2. Аминокислотные последовательности PD-1 человека можно найти в NCBI локусе №: NP_005009. Аминокислотные последовательности PD-L1 и PD-L2 человека можно найти в NCBI локусе №: NP_054862 и NP_079515, соответственно.
Антагонисты PD-1, полезные в любом из аспектов настоящего изобретения, включают моноклональное антитело (мАт), или его антигенсвязывающий фрагмент, которое специфически связывает PD-1 или PD-L1, и предпочтительно, специфически связывает PD-1 человека или PD-L1 человека. мАт может представлять собой человеческое антитело, гуманизированное антитело или химерное антитело, и может содержать человеческую константную область. В некоторых вариантах осуществления человеческая константная область выбрана из группы, состоящей из константных областей IgG1, IgG2, IgG3 и IgG4, и в предпочтительных вариантах осуществления, человеческая константная область представляет собой константную область IgG1 или IgG4. В некоторых вариантах осуществления антигенсвязывающий фрагмент выбран из группы, состоящей из Fab, Fab'-SH, F(ab')2, scFv и Fv-фрагментов.
Примеры мАт, которые связывают PD-1 человека и полезны в различных аспектах и вариантах осуществления настоящего изобретения, описаны в патенте США №8552154; патенте США № 8354509; патенте США № 8168757; патенте США № 8008449; патенте США № 7521051; патенте США № 7488802; WO 2004072286; WO 2004056875 и WO 2004004771.
Другие антагонисты PD-1, полезные в любом из аспектов и вариантов осуществления настоящего изобретения, включают иммуноадгезин, который специфически связывает PD-1, и предпочтительно, специфически связывает PD-1 человека, например, слитый белок, содержащий внеклеточный или PD-1-связывающий фрагмент PD-L1 или PD-L2, слитый с константной областью, такой как Fc-область молекулы иммуноглобулина. Примеры молекул иммуноадгезина, которые специфически связывают PD-1, описаны в WO2010027827 и WO2011066342. Специфические слитые белки, полезные в качестве антагониста PD-1 в способе лечения, лечебных препаратах и вариантах применения по настоящему изобретению, включают AMP-224 (также известный как B7-DCIg), который представляет собой слитый белок PD-L2-FC и связывает PD-1 человека.
Ниволумаб представляет собой гуманизированное моноклональное анти-PD-1 антитело, коммерчески доступное под названием опдиво®. Ниволумаб показан для лечения некоторых неоперабельных или метастатических меланом. Ниволумаб связывает и блокирует активацию PD-1, трансмембранного белка суперсемейства Ig, его лигандами PD-L1 и PD-L2, что приводит к активации T-клеток и опосредованным клетками иммунным ответам против опухолевых клеток или патогенов. Активированный PD-1 отрицательно регулирует активацию и эффекторную функцию T-клеток за счет подавления активации пути P13k/Akt. Другие названия ниволумаба включают: BMS-936558, MDX-1106 и ONO-4538. Аминокислотная последовательность ниволумаба, а также способы его применения и получения описаны в патенте США №US 8008449.
Пембролизумаб представляет собой гуманизированное моноклональное анти-PD-1 антитело, коммерчески доступное под названием кейтруда®. Пембролизумаб показан для лечения некоторых неоперабельных или метастатических меланом. Аминокислотная последовательность пембролизумаба, а также способы его применения описаны в патенте США №8168757.
В одном варианте осуществления антагонист PD-1 содержит любую одну, или сочетание, из следующих областей CDR:
CDRH1: SYDMS (SEQ ID NO: 151)
CDRH2: TISGGGSYTYYQDSVKG (SEQ ID NO: 152)
CDRH3: PYYAMDY (SEQ ID NO: 153)
CDRL1: KASQDVGTAVA (SEQ ID NO: 154)
CDRL2: WASTLHT (SEQ ID NO: 155)
CDRL3: QHYSSYPWT (SEQ ID NO: 156).
В одном варианте осуществления антагонист PD-1 содержит CDR1 вариабельной области тяжелой цепи («CDRH1»), содержащую аминокислотную последовательность с одной или двумя аминокислотными вариациями («вариант CDR») относительно аминокислотной последовательности, приведенной в SEQ ID NO: 151.
В одном варианте осуществления антагонист PD-1 содержит CDR2 вариабельной области тяжелой цепи («CDRH2»), содержащую аминокислотную последовательность с пятью или менее, например, четырьмя или менее, тремя или менее, двумя или менее, или одной аминокислотной вариацией («вариант CDR») относительно аминокислотной последовательности, приведенной в SEQ ID NO: 152. В следующем варианте осуществления CDRH2 содержит аминокислотную последовательность с одной или двумя аминокислотными вариациями относительно аминокислотной последовательности, приведенной в SEQ ID NO: 152.
В одном варианте осуществления антагонист PD-1 содержит CDR3 вариабельной области тяжелой цепи («CDRH3»), содержащую аминокислотную последовательность с одной или двумя аминокислотными вариациями («вариант CDR») относительно аминокислотной последовательности, приведенной в SEQ ID NO: 153.
В одном варианте осуществления антагонист PD-1 содержит CDR1 вариабельной области легкой цепи («CDRL1»), содержащую аминокислотную последовательность с тремя или менее, например, с одной или двумя аминокислотными вариациями («вариант CDR») относительно аминокислотной последовательности, приведенной в SEQ ID NO: 154.
В одном варианте осуществления антагонист PD-1 содержит CDR2 вариабельной области легкой цепи («CDRL2»), содержащую аминокислотную последовательность с одной или двумя аминокислотными вариациями («вариант CDR») относительно аминокислотной последовательности, приведенной в SEQ ID NO: 155.
В одном варианте осуществления антагонист PD-1 содержит CDR3 вариабельной области легкой цепи («CDRL3»), содержащую аминокислотную последовательность с тремя или менее, например, с одной или двумя аминокислотными вариациями («вариант CDR») относительно аминокислотной последовательности, приведенной в SEQ ID NO: 156. В конкретном варианте осуществления CDRL3 содержит аминокислотную последовательность с одной аминокислотной вариацией относительно аминокислотной последовательности, приведенной в SEQ ID NO: 156. В следующем варианте осуществления вариант CDRL3 содержит аминокислотную последовательность, приведенную в SEQ ID NO: 157.
В одном варианте осуществления антагонист PD-1 содержит CDRH1, содержащую аминокислотную последовательность, имеющую одну аминокислотную вариацию относительно аминокислотной последовательности, приведенной в SEQ ID NO: 151; CDRH2, содержащую аминокислотную последовательность, имеющую до пяти аминокислотных вариаций относительно аминокислотной последовательности, приведенной в SEQ ID NO: 152; CDRH3, содержащую аминокислотную последовательность, имеющую одну аминокислотную вариацию относительно аминокислотной последовательности, приведенной в SEQ ID NO: 153; CDRL1, содержащую аминокислотную последовательность, имеющую до трех аминокислотных вариаций относительно аминокислотной последовательности, приведенной в SEQ ID NO: 154; CDRL2, содержащую аминокислотную последовательность, имеющую одну аминокислотную вариацию относительно аминокислотной последовательности, приведенной в SEQ ID NO: 155; и/или CDRL3, содержащую аминокислотную последовательность, имеющую до трех аминокислотных вариаций относительно аминокислотной последовательности, приведенной в SEQ ID NO: 156.
В одном варианте осуществления настоящего изобретения антагонист PD-1 содержит CDRH1 (SEQ ID NO: 151), CDRH2 (SEQ ID NO: 151) и CDRH3 (SEQ ID NO: 153) в вариабельной области тяжелой цепи, имеющей аминокислотную последовательность, приведенную в SEQ ID NO: 158. В некоторых вариантах осуществления анти-PD-1 антитела по настоящему изобретению содержат вариабельную область тяжелой цепи, имеющую по меньшей мере 90% идентичности последовательности с SEQ ID NO: 158. Предпочтительно, антагонисты PD-1 по настоящему изобретению могут содержать вариабельную область тяжелой цепи, имеющую приблизительно 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с SEQ ID NO: 158.
Вариабельная область тяжелой цепи (VH) антагониста PD-1:
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSS (SEQ ID NO: 158)
В одном варианте осуществления антагонист PD-1 содержит вариабельную область тяжелой цепи («VH»), содержащую аминокислотную последовательность с по меньшей мере приблизительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности относительно аминокислотной последовательности, приведенной в SEQ ID NO: 158. В одном варианте осуществления VH содержит аминокислотную последовательность с по меньшей мере одной аминокислотной вариацией относительно аминокислотной последовательности, приведенной в SEQ ID NO: 158, например, 1-5, например, 1-3, в частности, 2 аминокислотными вариациями относительно аминокислотной последовательности, приведенной в SEQ ID NO: 158.
В одном варианте осуществления настоящего изобретения антагонист PD-1 содержит CDRL1 (SEQ ID NO: 154), CDRL2 (SEQ ID NO: 155) и CDRL3 (SEQ ID NO: 156) в вариабельной области легкой цепи, имеющей аминокислотную последовательность, приведенную в SEQ ID NO: 159. В одном варианте осуществления антагонист PD-1 по настоящему изобретению содержит вариабельную область тяжелой цепи с SEQ ID NO: 158 и вариабельную область легкой цепи с SEQ ID NO: 159.
В некоторых вариантах осуществления антагонисты PD-1 по настоящему изобретению содержат вариабельную область легкой цепи, имеющую по меньшей мере 90% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 159. Предпочтительно, антагонисты PD-1 по настоящему изобретению могут содержать вариабельную область легкой цепи, имеющую приблизительно 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с SEQ ID NO: 159.
Вариабельная область легкой цепи (VL) антагониста PD-1:
DIQLTQSPSFLSAYVGDRVTITCKASQDVGTAVAWYQQKPGKAPKLLIYWASTLHTGVPSRFSGSGSGTEFTLTISSLQPEDFATYYCQHYSSYPWTFGQGTKLEIK (SEQ ID NO: 159)
В одном варианте осуществления антагонист PD-1 содержит вариабельную область легкой цепи («VL»), содержащую аминокислотную последовательность с по меньшей мере приблизительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности относительно аминокислотной последовательности, приведенной в SEQ ID NO: 159. В одном варианте осуществления VL содержит аминокислотную последовательность с по меньшей мере одной аминокислотной вариацией относительно аминокислотной последовательности, приведенной в SEQ ID NO: 159, например, 1-5, например, 1-3, в частности, 2 аминокислотными вариациями относительно аминокислотной последовательности, приведенной в SEQ ID NO: 159.
В одном варианте осуществления антагонист PD-1 содержит VH, содержащую аминокислотную последовательность с по меньшей мере приблизительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 158; и VL, содержащую аминокислотную последовательность с по меньшей мере приблизительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 159. В одном варианте осуществления антагонист PD-1 содержит VH, которая по меньшей мере на приблизительно 90% идентична аминокислотной последовательности SEQ ID NO: 158, и/или VL, которая по меньшей мере на приблизительно 90% идентична аминокислотной последовательности SEQ ID NO: 159.
В одном варианте осуществления антагонист PD-1 содержит VH с аминокислотной последовательностью, приведенной в SEQ ID NO: 158, и VL с аминокислотной последовательностью, приведенной в SEQ ID NO: 159.
В одном варианте осуществления антагонист PD-1 представляет собой моноклональное антитело, содержащее аминокислотную последовательность тяжелой цепи (HC), имеющую по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 160.
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK (SEQ ID NO: 160)
В одном варианте осуществления HC содержит аминокислотную последовательность с по меньшей мере одной аминокислотной вариацией относительно аминокислотной последовательности, приведенной в SEQ ID NO: 160, например, 1-10, например, 1-7, в частности, 6 аминокислотными вариациями относительно аминокислотной последовательности, приведенной в SEQ ID NO: 160. В следующем варианте осуществления HC содержит одну, две, три, четыре, пять, шесть или семь аминокислотных вариаций относительно аминокислотной последовательности, приведенной в SEQ ID NO: 160.
В одном варианте осуществления цепь HC имеет вариацию в положении 380 и/или 385 в SEQ ID NO: 160. Остатки аспарагина в этих положениях могут быть модифицированы, например, путем дезамидирования (превращение остатка аспарагина (N) в остаток аспартата (D)). Таким образом, в одном варианте осуществления HC содержит аминокислотную последовательность SEQ ID NO: 162 (N380D), SEQ ID NO: 163 (N385D) или SEQ ID NO: 164 (N380D и N385D).
В одном варианте осуществления антагонист PD-1 представляет собой моноклональное антитело, содержащее аминокислотную последовательность легкой цепи (LC), имеющую по меньшей мере 90%, 91%, 92,%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 161.
DIQLTQSPSFLSAYVGDRVTITCKASQDVGTAVAWYQQKPGKAPKLLIYWASTLHTGVPSRFSGSGSGTEFTLTISSLQPEDFATYYCQHYSSYPWTFGQGTKLEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC (SEQ ID NO: 161)
В одном варианте осуществления LC содержит аминокислотную последовательность с по меньшей мере одной аминокислотной вариацией относительно аминокислотной последовательности, приведенной в SEQ ID NO: 161, например, 1-10, например, 1-5, в частности, 3 аминокислотными вариациями относительно аминокислотной последовательности, приведенной в SEQ ID NO: 161. В следующем варианте осуществления LC имеет одну, две или три аминокислотные вариации относительно аминокислотной последовательности, приведенной в SEQ ID NO: 161.
В одном варианте осуществления антагонист PD-1 содержит HC, содержащую аминокислотную последовательность с по меньшей мере приблизительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 160; и LC, содержащую аминокислотную последовательность с по меньшей мере приблизительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности последовательности с аминокислотной последовательностью, приведенной в SEQ ID NO: 161. Таким образом, антитело представляет собой антитело с тяжелой цепью, по меньшей мере на приблизительно 90% идентичной аминокислотной последовательности тяжелой цепи SEQ ID NO: 160, и/или с легкой цепью, по меньшей мере на приблизительно 90% идентичной аминокислотной последовательности легкой цепи SEQ ID NO: 161.
В одном варианте осуществления антагонист PD-1 содержит аминокислотную последовательность тяжелой цепи, по меньшей мере на приблизительно 90% идентичную аминокислотной последовательности SEQ ID NO: 160, и/или аминокислотную последовательность легкой цепи, по меньшей мере на приблизительно 90% идентичную аминокислотной последовательности SEQ ID NO: 161.
В одном варианте осуществления антагонист PD-1 содержит последовательность тяжелой цепи SEQ ID NO: 160 и последовательность легкой цепи SEQ ID NO: 161. В одном варианте осуществления антитело представляет собой достарлимаб, содержащий последовательность тяжелой цепи SEQ ID NO: 160 и последовательность легкой цепи SEQ ID NO: 162.
Анти-PD-L1 антитела и способы их получения известны в данной области. Такие антитела к PD-L1 могут быть поликлональными или моноклональными и/или рекомбинантными, и/или гуманизированными. Анти-PD-L1 антитела находятся в стадии разработки в качестве иммуномодулирующих средств для лечения рака.
Иллюстративные анти-PD-L1 антитела раскрыты в патенте США № 9212224; патенте США № 8779108; патенте США № 8552154; патенте США № 8383796; патенте США № 8217149; публикации патента США № 20110280877; WO 2013079174 и WO 2013019906. Дополнительные иллюстративные антитела к PD-L1 (также называемому CD274 или B7-H1) и способы их применения раскрыты в патенте США № 8168179; патенте США № 7943743; патенте США № 7595048; WO 2014055897; WO 2013019906 и WO 2010077634. Специфические моноклональные антитела против PD-L1 человека, полезные в качестве антагонистов PD-1 в способе лечения, лекарственных препаратах и вариантах применения по настоящему изобретению, включают MPDL3280A, BMS-936559, MEDI4736, MSB0010718C.
Атезолизумаб представляет собой полностью гуманизированное моноклональное анти-PD-L1 антитело, коммерчески доступное под названием тецентрик®. Атезолизумаб показан для лечения некоторых местнораспространенных или метастатических уротелиальных карцином. Атезолизумаб блокирует взаимодействие PD-L1 с PD-1 и CD80. Другие иллюстративные анти-PD-L1 антитела включают авелумаб (бавенсио®), дурвалумаб (имфинзи®).
CD134, также известный как OX40, является членом суперсемейства рецепторов TNFR, который не экспрессируется конститутивно на покоящихся необученных T-клетках, в отличие от CD28. OX40 представляет собой вторичную костимулирующую молекулу, экспрессируемую через 24-72 часа после активации; его лиганд, OX40L, также не экспрессируется на покоящихся антигенпредставляющих клетках, но экспрессируется после их активации. Экспрессия OX40 зависит от полной активации T-клетки; без CD28 экспрессия OX40 задерживается или имеет в четыре раза более низкие уровни. Анти-OX-40 антитела, анти-OX-40 слитые белки и способы их применения раскрыты в патентах США №№: US 7504101; US 7758852; US 7858765; US 7550140; US 7960515; WO2012027328; WO2013028231.
Дополнительными примерами другого активного ингредиента или ингредиентов (анти-неопластических средств) для применения в сочетании, или совместном введении, с раскрытыми в настоящем описании CD96-связывающими белками являются антитела или другие антагонисты CD20, ретиноиды или другие ингибиторы киназ. Примеры таких антител или антагонистов включают, но без ограничения, ритуксимаб (ритуксан® и мабтера®), офатумумаб (арзерра®) и бексаротен (таргретин®).
Дополнительными примерами другого активного ингредиента или ингредиентов (анти-неопластических средств) для применения в сочетании, или совместном введении, с раскрытыми в настоящем описании CD96-связывающими белками являются антагонисты Toll-подобного рецептора 4 (TLR4), включая, но без ограничения, аминоалкил-глюкозаминид фосфаты (AGP).
Известно, что AGP полезны в качестве вакцинных адъювантов и иммуностимулирующих средств для стимуляции продуцирования цитокинов, активации макрофагов, стимуляции врожденного иммунного ответа и повышения выработки антител у иммунизированных животных. AGP представляют собой синтетические лиганды TLR4. AGP и их иммуномодулирующие эффекты через TLR4 раскрыты в патентных публикациях, таких как WO 2006016997, WO 2001090129, и/или патенте США № 6113918, и описаны в литературе. Дополнительные производные AGP раскрыты в патенте США №7129219, патенте США № 6911434 и патенте США №6525028. Некоторые AGP действуют как агонисты TLR4, в то время как другие являются признанными антагонистами TLR4.
Дополнительными неограничивающими примерами другого активного ингредиента или ингредиентов (анти-неопластических средств) для применения в сочетании, или совместном введении, с раскрытыми в настоящем описании CD96-связывающими белками являются антитела к ICOS.
Области CDR для мышиных антител к ICOS человека, имеющих активность агониста, описаны в PCT/EP2012/055735 (WO 2012131004). Антитела к ICOS также раскрыты в WO 2008137915, WO 2010056804, EP 1374902, EP1374901 и EP1125585.
Дополнительными примерами другого активного ингредиента или ингредиентов (анти-неопластических средств) для применения в сочетании, или совместном введении, с раскрытыми в настоящем описании CD96-связывающими белками являются ингибиторы поли(АДФ-рибоза)-полимеразы (PARP). Неограничивающие примеры таких ингибиторов включают нирапариб, олапариб, рукапариб и талазопариб.
Дополнительными неограничивающими примерами другого активного ингредиента или ингредиентов (анти-неопластических средств) для применения в сочетании, или совместном введении, с раскрытыми в настоящем описании CD96-связывающими белками являются модулирующие STING соединения, ингибиторы CD39, а также антагонисты A2a и A2a рецепторов аденозина.
Избранные анти-неопластические средства, которые могут быть использованы в сочетании с CD96-связывающими белками, или их фармацевтически приемлемые соли, включают, но не ограничиваются ими: абареликс, абемациклиб, абиратерон, афатиниб, афлиберцепт, альдоксорубицин, алектиниб, алемтузумаб, триоксид мышьяка, аспарагиназу, акситиниб, AZD-9291, белиностат, бендамустин, бевацизумаб, блинатумомаб, босутиниб, брентуксимаб ведотин, кабазитаксел, кабозантиниб, капецитабин, церитиниб, клофарабин, кобиметиниб, кризотиниб, даратумумаб, дазатиниб, дегареликс, деносумаб, динутуксимаб, доцетаксел, элотузумаб, энтиностат, энзалутамид, эпирубицин, эрибулин, филграстим, флуматиниб, фулвестрант, фруктиниб, гемтузумаб озогамицин, ибритумомаб, ибрутиниб, иделалисиб, иматиниб, иринотекан, иксабепилон, иксазомиб, леналидомид, ленватиниб, лейковорин, мехлорэтамин, нецитумумаб, неларабин, нетупитант, нилотиниб, обинутузумаб, олапариб, омацетаксин, осимертиниб, оксалиплатин, паклитаксел, палбоциклиб, палоносетрон, панитумумаб, пегфилграстим, пегинтерферон альфа-2b, пеметрексед, плериксафор, помалидомид, понатиниб, пралатрексат, квизартиниб, радий-223, рамуцирумаб, регорафениб, ролапитант, рукапариб, сипулейцел-Т, сонидегиб, сунитиниб, талимоген лахерпарепвек, типирацил, топотекан, трабедецин, трифлуридин, трипторелин, уридин, вандетаниб, велапариб, вемурафениб, венетоклакс, винкристин, висмодегиб и золедроновую кислоту.
Лечение может быть терапевтическим, профилактическим или превентивным. Субъект будет субъектом, который нуждается в этом. Субъекты, которые нуждаются в лечении, могут включать индивидуумов, уже страдающих от конкретного заболевания, в дополнение к тем, у кого заболевание может развиться в будущем.
Таким образом, способы, антигенсвязывающие белки и композиции по изобретению, описанные в настоящем описании, могут быть использованы для профилактического лечения или превентивного лечения, если указано. В этом случае способы, антигенсвязывающие белки и композиции по изобретению могут быть использованы для предотвращения или отсрочки начала одного или более аспектов или симптомов заболевания. Субъект может не иметь симптомов. Субъект может иметь генетическую предрасположенность к заболеванию. Такому индивидууму вводят профилактически эффективное количество антигенсвязывающего белка. Профилактически эффективное количество представляет собой количество, которое предотвращает или отсрочивает начало одного или более аспектов или симптомов заболевания, описанного в настоящем описании.
Способы, антигенсвязывающие белки и композиции по изобретению не обязательно должны полностью излечивать или устранять каждый симптом или проявление заболевания, чтобы быть полезным терапевтическим средством. Как признано в данной области, лекарственные средства, используемые в качестве терапевтических средств в способах лечения, могут приводить к снижению степени тяжести конкретного болезненного состояния, но не обязательно устранять каждое проявление заболевания, чтобы их считали полезными терапевтическими средствами. Аналогично, профилактически вводимое терапевтическое средство не обязательно должно быть полностью эффективным для предотвращения начала заболевания, чтобы являться полезным профилактическим средством. Простое уменьшение воздействия заболевания (например, за счет уменьшения количества или степени тяжести его симптомов, или за счет повышения эффективности другого метода лечения, или за счет оказания другого полезного эффекта) или снижение вероятности развития заболевания (например, за счет отсрочки начала развития заболевания) или ухудшения состояния субъекта, является достаточным.
Другой аспект изобретения относится к способу лечения заболевания у субъекта, который нуждается в этом, включающему введение указанному субъекту терапевтически эффективного количества CD96-связывающего белка или фармацевтической композиции, описанных в любом из предшествующих аспектов. Следующий аспект изобретения относится к способу лечения, описанному в предшествующем аспекте, дополнительно включающему определение того, экспрессируется ли у субъекта CD96.
Другой аспект изобретения относится к CD96-связывающему белку или фармацевтической композиции, описанным в любом из предшествующих аспектов, для применения в терапии или для применения в лечении заболевания.
Другой аспект изобретения относится к применению CD96-связывающего белка или фармацевтической композиции, описанных в одном из предшествующих аспектов, в производстве лекарственного препарата для применения в лечении заболевания.
Другой аспект изобретения относится к фармацевтической композиции, содержащей терапевтически эффективное количество CD96-связывающего белка, описанного в любом из предшествующих аспектов.
Один вариант осуществления изобретения относится к способу лечения заболевания у субъекта (такого как субъект-человек), который нуждается в этом, включающему введение указанному субъекту терапевтически эффективного количества CD96-связывающего белка, описанного в любом из предшествующих вариантов осуществления, или фармацевтической композиции, описанной в любом из предшествующих вариантов осуществления.
Один вариант осуществления настоящего изобретения относится к способу лечения заболевания у субъекта, который нуждается в этом, включающему введение указанному субъекту терапевтически эффективного количества CD96-связывающего белка или фармацевтической композиции, описанных в любом из предшествующих вариантов осуществления. Следующий вариант осуществления настоящего изобретения относится к способу лечения заболевания у субъекта, который нуждается в этом, дополнительно включающему определение того, экспрессируется ли у субъекта CD96. Дополнительный вариант осуществления настоящего изобретения относится к CD96-связывающему белку или фармацевтической композиции, описанным в любом из предшествующих вариантов осуществления, для применения в терапии. Дополнительный вариант осуществления настоящего изобретения относится к CD96-связывающему белку или фармацевтической композиции, описанным в любом из предшествующих вариантов осуществления, для применения в лечении заболевания. Дополнительный вариант осуществления относится к применению CD96-связывающего белка или фармацевтической композиции, описанных в любом из предшествующих вариантов осуществления, в производстве лекарственного препарата для применения в лечении заболевания. Дополнительный вариант осуществления настоящего изобретения относится к способу лечения заболевания, или терапии, включающему введение указанному субъекту терапевтически эффективного количества CD96-связывающего белка или фармацевтической композиции, описанных в любом из предшествующих вариантов осуществления, где заболевание, подвергаемое лечению, представляет собой рак. В следующем аспекте рак представляет собой солидную опухоль (например, рецидивирующую, метастатическую или прогрессирующую солидную опухоль). Рак может представлять собой: рак печени (например, HCC), рак яичника, рак легких, такой как немелкоклеточный рак легких (NSCLC), рак почки (например, RCC), рак толстого кишечника, рак желудка, рак мочевого пузыря, плоскоклеточную карциному головы и шеи (HNSCC), или он может представлять собой лейкоз, и/или любое B-клеточное злокачественное новообразование.
ПРИМЕРЫ
Пример 1
Получение CD96-связывающих белков
Получение связывающего белка
Полностью человеческие антитела, специфические для CD96 человека, были выделены из не подвергнутых воздействию эукариотических библиотек с использованием биотинилированного рекомбинантного CD96 человека и CD96 яванского макака, в сочетании с сортировкой клеток и методами селекции на основе FACS. Полученные из не подвергнутой воздействию эукариотической библиотеки варианты тяжелой цепи были перетасованы с вариантами из библиотеки легких цепей, и был проведен дополнительный отбор для определения оптимальных пар с легкими цепями.
Проводили созревание аффинности для повышения аффинности/эффективности антител. Для этого интегрировали CDRH3 антитела 42Y073-86F08-1 (GAGYYGDKDPMDV в SEQ ID NO: 2) в предварительно полученные библиотеки с разнообразными CDRH1 или CDRH2. Выполняли дополнительный отбор для данной диверсифицированной библиотеки, лучшие молекулы идентифицировали и секвенировали. Аминокислотные и нуклеотидные последовательности вариабельных областей легкой и тяжелой цепей для лучших молекул приведены в SEQ ID NO: 1-96. Аминокислотные последовательности областей CDR приведены в SEQ ID NO: 97-150.
Выбор Fc-области
В процессе выбора лучших молекул связывающие белки оценивали в качестве молекул человеческого IgG1 ДТ или в качестве молекул человеческого IgG1 с инактивированной Fc-областью. Для получения молекул с инактивированной Fc-областью проводили мутацию аминокислотных остатков в положениях 234, 235 и 237, в частности, L234 и L235 или L235 и G237, на аланин, получая, таким образом, чIgG1 «LALA» (L234A/L235A) и чIgG1 «LAGA» (L235A/G237A) молекулы человеческого IgG1 с инактивированной Fc-областью. Эти связывающие белки проявляли свойства связывания CD96 и нейтрализации связывания CD155. Три CD96-связывающих белка, 42Y073-86F08-66 (86F08-66), 42Y073-86F08-16 (86F08-16) и 42Y073-86F04-23 (86F04-23), были отобраны для дальнейшей оценки. Связывающий белок 42Y073-86F08-66 также известен как GSK 6097608 (моноклональное антитело). Эти три CD96-связывающих белка оценивали в виде разных вариантов IgG (человеческих IgG1, IgG2, IgG4PE или IgG1 с инактивированной Fc-областью) на связывание первичных T-клеток, а также активность нейтрализации CD155. Изменения в Fc-области не влияли на связывание с CD96-экспрессирующими клетками или нейтрализацию связывания CD155 с CD96-экспрессирующими клетками, как показано на фиг. 1.
Пример 2
Определение характеристик CD96-связывающих белков
Связывание с клетками CHO, экспрессирующими CD96 человека или яванского макака
Клетки CHO, экспрессирующие CD96v2 человека или CD96v2 яванского макака, готовили в PBS. Экспрессирующие CD96v2 яванского макака клетки окрашивали 1 мкМ фиолетовым красителем на пролиферацию (VPD450). Клетки разводили в предварительно подогретой среде (RPMI, содержащей 10% эмбриональной телячьей сыворотки) и инкубировали в течение 10 минут при 37°C, с последующим центрифугированием при 400 g, 5 минут, и ресуспендировали в проточном буфере (PBS+0,5% БСА+2 мМ ЭДТА) до плотности 1,5×106/мл. Окрашенные CHO-cyCD96 и неокрашенные CHO-huCD96 клетки объединяли и добавляли в лунки 384-луночного аналитического планшета вместе с разведенными тестируемыми анти-CD96-связывающими белками на 1 ч при 4°C. Клетки дополнительно инкубировали с конъюгатом анти-IgG антитела с APC, и количество антитела, связанного на поверхности клеток CHO-cyCD96 и CHO-huCD96, определяли методом проточной цитометрии. Значения средней интенсивности флуоресценции (СИФ) были подогнаны с помощью 4-х параметрической логистической модели для расчета EC50 связывания. Данные приведены в табл. 2.
Нейтрализация связывания CD155 человека с клетками CHO, экспрессирующими CD96v2 человека
CD96-связывающие белки готовили в PBS в 96-луночных планшетах с U-образным дном. Добавляли клетки CHO, экспрессирующие CD96v2 человека, и инкубировали в течение 30 минут при комнатной температуре. После промывания клеток три раза путем центрифугирования добавляли конъюгат человеческого CD155-Fc-AF647 еще на 30 минут при комнатной температуре. После трех промываний клетки фиксировали и анализировали на уровень окрашивания CD155-Fc-AF647 методом проточной цитометрии. Значения средней интенсивности флуоресценции (СИФ) были подогнаны с помощью 4-параметрической логистической модели для расчета значений IC50. Данные приведены в табл. 2.
Связывание CD96-связывающих белков с первичными человеческими T-клетками
Человеческие МКПК выделяли из лейкоредукционных фильтров с использованием центрифугирования в градиенте плотности гистопак-1077. Затем CD3+T-клетки выделяли с использованием набора для выделения пан-T-клеток (Miltenyi), следуя инструкциям производителя. Тестируемые CD96-связывающие белки в серийных разведениях инкубировали с человеческими T-клетками в течение 1 ч при 4°C. После трех промываний в буфере добавляли конъюгат антител против человеческих IgG-APC на 1 ч при 4°C. После трех промываний клетки фиксировали и анализировали на окрашивание APC методом проточной цитометрии. Значения средней интенсивности флуоресценции (СИФ) были подогнаны с помощью 4-х параметрической логистической модели для расчета EC50 связывания. Данные приведены в табл. 2
Выбор 42Y073-86F08-66 для дальнейшего определения характеристик
С целью выбора лучших молекул оценивали биофизические свойства анти-CD96 связывающих белков. А именно иммуногенность, дезамидирование, гликозилирование, окисление, изомеризация аспартата были предсказаны с помощью in silico анализа, и свойства агрегации, фрагментации и химической деградации были оценены в следующих условиях: в 50 мМ фосфате натрия, pH 7,5, и 50 мМ ацетате натрия, pH 5,0, при концентрации либо 10 мг/мл, либо 1 мг/мл, а также в стрессовых или не стрессовых (тепловой стресс при 40°C в течение 2 недель) условиях. Анти-CD96 связывающие белки были экспрессированы во временной системе HEK и очищены с использованием аффинной хроматографии на белке A и эксклюзионной хроматографии. Анти-CD96 связывающие белки были оценены следующим образом:
Связывание антигена с использованием BIAcore:
Изменение связывания антигена в пределах ±10% между не подвергнутыми стрессу и подвергнутыми стрессу образцами считали приемлемым диапазоном (низкий риск). Молекулы за пределами данного диапазона могли указывать на потенциальный риск вследствие потери активности связывания.
Данные приведены в табл. 6 и 7. В табл. 6 идентифицировано положение предсказанного риска в последовательности (нумерация Kabat). В табл. 7 использованы следующие сокращения для наблюдаемых рисков и условий, в которых они наблюдались: Агг=агрегация; увел. масс%=увеличение гетерогенности; потеря связывания=наблюдается потеря связывания, превышающая предел анализа; увел. связывания=увеличение связывания (указывает на возможную склонность к агрегированию); ВМС=высокомолекулярные соединения; НМС=низкомолекулярные соединения; Rh=гидродинамический радиус (где больший радиус указывает на возможность агрегации); PBS=фосфатно-солевой буферный раствор; Н/С=не подвергнутые стрессу; С=подвергнутые стрессу; A=50 мМ ацетат натрия pH 5,0; и P=50 мМ фосфат натрия pH 7,5.
42Y073-86F08-66, 42Y073-86F08-16 и 42Y073-86F04-23 продемонстрировали минимальные недостатки разработки и минимальные in silico риски. Однако 42Y073-86F08-66 было выбрано для дальнейшей работы и дополнительного определения характеристик, поскольку были отмечены следующие признаки:
- приемлемый уровень процентного общего содержания высокомолекулярных соединений (ВМС;<5%), низкомолекулярных соединений (НМС;<2,5%) и мономеров (95%) при определении методом аналитической эксклюзионной хроматографии (аЭХ);
- ожидаемые значения для распределения по размерам (>98% в пике 2 при ЭХ) и гидродинамического радиуса (4,5-7 нм) при определении методом динамического рассеяния света (ДРС);
- минимальные неожиданные изменения химической гетерогенности между не подвергнутыми и подвергнутыми стрессу молекулами при определении методом капиллярного изоэлектрического фокусирования (кИЭФ); и
- для связывания антигена<10% изменения было определено в анализе связывания антигена.
(DP-мотив H_cdr3:100C)
(DP-мотив H_cdr3:100C)
(DP-мотив H_cdr3:100C)
(DP-мотив H_cdr3:100C)
(DP-мотив H_cdr3:100C)
(H_cdr2:52)
(DP-мотив H_cdr3:101)
(DP-мотив H_cdr3:101)
(DP-мотив H_cdr3:101)
(DP-мотив H_cdr3:101)
(H_cdr3:100E, H_fr2:48)
(DP-мотив H_cdr3:100C)
(DP-мотив H_cdr3:100C)
(H_cdr2:54)
(Агг в U, S, A и P, и увел. масс% в U, S и P)
(Агг и увел. масс% в U, S и P)
(11% потеря связывания в S и A)
(ВМС в S и A)
(37% потеря связывания в S и A)
(9,57% увел. связывания в S и P)
(ВМС в S и A)
(41% потеря связывания в S и A)
(34% увел. связывания в S и P)
(уменьшение выхода в PBS)
(ВМС и НМС в U, S и A)
(Агг и увел. масс% в S, A)
(36% потеря связывания в S и A)
(9,99% потеря связывания в S и P)
(13% потеря связывания в S и P)
(12% потеря связывания в S и A)
(потеря связывания в A)
(74,3% извление в U и A)
(НМС в S)
(Rh в U, S и P)
Эпитоп-специфическая сортировка CD96-связывающих белков
Проводили конкурентные анализы для эпитоп-специфической сортировки с целью определения эпитопов CD96, с которыми связываются анти-CD96 связывающие белки, представленные в настоящем описании. Вкратце, HuCD96-His инкубировали с первым CD96-связывающим белком при комнатной температуре в течение 1 часа, с последующим измерением связывания второго связывающего белка, захваченного на сенсоре с белком A при помощи BLI. Если наблюдали связывание второго CD96-связывающего белка, эти два связывающих белка считали неконкурирующими и относили к разным подгруппам эпитопов. Если можно было наблюдать отсутствие связывания второго CD96-связывающего белка, эти два связывающих белка считали конкурирующими и относили к одной и той же подгруппе эпитопов. Контроль с само-конкуренцией включали для каждого CD96-связывающего белка, используя один и тот же связывающий белок в качестве первого и второго связывающего белка.
Наблюдали конкуренцию между всеми CD96-связывающими белками, за исключением 42Y073-2B04-46, это указывало на то, что все белки, за исключением 42Y073-2B04-46, связывают аналогичные эпитопы CD96. Эти данные свидетельствуют о том, что, за исключением 42Y073-2B04-46, все CD96-связывающие белки, описанные в настоящем описании, связывают один и тот же эпитоп CD96 или расположенные близко в пространстве эпитопы, так что связывание других CD96-связывающих белков ингибируется (то есть, они принадлежат к одной и той же группе эпитопов). Однако тот факт, что 42Y073-2B04-46 не конкурирует с другими CD96-связывающими белками, описанными в настоящем описании, указывает на то, что белок этого клона связывает иной эпитоп CD96 (и, следовательно, принадлежит к иной подгруппе эпитопов). Таким образом, в совокупности с данными, приведенными в табл. 2, понятно, что связывание, и нейтрализация связывания CD155 человека, hCD96v2-экспрессирующих клеток CHO CD96-связывающими белками, описанными в настоящем описании, не ограничено связыванием какого-либо конкретного эпитопа CD96.
Дополнительные исследования аффинности связывания CD96-связывающих белков с изоформами CD96
Аффинность связывания 42Y073-86F08-66, полностью человеческого IgG1 антитела с Fc-областью дикого типа, экспрессированного и очищенного из клеток CHO, с рекомбинантным CD96 определяли в анализах титрования при равновесии в растворе (MSD-SET). Белок 42Y073-86F08-66 связывал рекомбинантный CD96v2 человека со средним значением KD, равным 20 пМ, и рекомбинантный CD96v2 яванского макака со средним значением KD, равным 278 пМ (фиг. 2). Кроме того, было показано, что 42Y073-86F08-66 связывал рекомбинантный мышиный CD96v2 со средним значением KD, равным 479 пМ.
В анализах поверхностного плазмонного резонанса (ППР) 42Y073-86F08-66 не связывал рекомбинантный CD155 человека, CD226 человека, TIGIT человека или нектин-1 человека, которые являются гомологами в пути.
ПРОЦЕДУРА АНАЛИЗА MSD-SET
Резюме: Анализ MSD-SET (MSD титрование при равновесии в растворе) использовали для определения аффинности этих антител в отношении человеческих и мышиных белков CD96 при 25°C, поскольку скорости диссоциации были слишком низкими для измерения методом BIACORE при данной температуре. Методом MSD-SET определяют аффинность антител при равновесии в фазе раствора. Способ позволяет определять свободный антиген в условиях равновесия при использовании серии титруемых концентраций антитела.
Процедура
Биотинилированный белок CD96 человека использовали в постоянной концентрации 1,5 нМ, и биотинилированный белок CD96 яванского макака в концентрации 3 нМ. Образцы антител титровали 1 к 5 для построения кривой по 22 точкам, от 6 нМ в случае CD96 человека и CD96 яванского макака. Титруемое антитело и белок CD96 инкубировали в течение 24 ч при комнатной температуре. Через 24 ч 5 нМ антитела наносили на стандартные связывающие MSD планшеты (Meso Scale Discovery, L15XA) на 30 мин при комнатной температуре. Затем планшеты блокировали блокирующим буфером STARTING BLOCK (Thermo Scientific, №37542) в течение 30 мин со встряхиванием при 700 об/мин, с последующими тремя промываниями промывающим буфером. Инкубируемые растворы добавляли в MSD планшеты на 150 с со встряхиванием при 700 об/мин, с последующим одним промыванием. Антиген, захваченный на планшете, обнаруживали при помощи SULFOTAG-меченого стрептавидина (Meso Scale Discovery, R32AD-1) путем его инкубации в планшете в течение 3 мин. Планшеты промывали три раза промывающим буфером, а затем снимали показания на приборе MSD SECTOR IMAGER с использованием 1x проявляющего буфера T с сурфактантом (Meso Scale Discovery, R92TC-1). Строили график зависимости процентного содержания свободного антигена от титруемого антитела с использованием программы GRAPHPAD PRISM, и подгоняли кривую под квадратичное уравнение.
Связывание CD96-связывающих белков с клетками HEK, избыточно экспрессирующими белки CD96
Связывание 42Y073-86F08-66 с известными изоформами CD96 человека или яванского макака на клеточных мембранах определяли методом проточной цитометрии с использованием клеток HEK, трансфицированных тремя разными изоформами (фиг. 3). Несмотря на различия в максимальной реакции связывания с экспрессируемыми изоформами, и между экспериментами (скорее всего, из-за различий в эффективности экспрессии), были продемонстрированы сопоставимые активности связывания, на что указывали значения EC50 для 42Y073-86F08-66 с этими изоформами CD96 (табл. 8). В совокупности эти данные свидетельствуют о том, что белок 42Y073-86F08-66 способен узнавать все мембранные формы CD96, представленные у человека и яванского макака.
(3,13-8,89)
(4,72-8,78)
(6,83-12,3)
(5,54-11,21)
(5,27-10,23)
Аффинность связывания CD96-связывающих белков с природным CD96 на T-клетках человека и яванского макака
Для подтверждения того, что 42Y073-86F08-66 связывает природный CD96, экспрессируемый на первичных клетках человека и обезьян, определяли связывание 42Y073-86F08-66 с CD3+T-клетками и подмножествами (CD4+ или CD8+) клеток от обоих биологических видов методом проточной цитометрии. 42Y073-86F08-66 связывал с высокой аффинностью CD4+ и CD8+ T-клетки человека (значения EC50 составляли 47 пМ и 45 пМ, соответственно) (фиг. 4 и табл. 9). 42Y073-86F08-66 имел более высокую эффективность (пМ) связывания с первичными человеческими T-клетками в сравнении с клетками HEK, которые избыточно экспрессируют CD96 (нМ), вследствие относительно низкой экспрессии CD96 на первичных клетках в сравнении с экспрессией CD96 после временной трансфекции изоформами CD96 под контролем сильного конститутивного промотора.
(39,6-62,6)
(37,7-58,5)
(31,6-63,3)
Для подтверждения того, что 42Y073-86F08-66 связывает CD96 яванского макака, определяли связывание с первичными T-клетками обезьян. Исходя из результатов связывания 42Y073-86F08-66 и коммерчески доступного анти-CD96 антитела 6F9, было установлено, что T-клетки яванского макака экспрессируют CD96 на гораздо более низких уровнях, чем T-клетки человека. Аналогичное наблюдение было сделано для T-клеток макак резусов. Для надежной количественной оценки связывания целевого CD96 очищенные CD3+T-клетки яванского макака активировали с использованием анти-CD2/3/28 гранул в течение 17 дней для повышения экспрессии CD96. Связывание 42Y073-86F08-66 определяли для 3 разных образцов активированных T-клеток от яванских макак, и связывание 42Y073-86F08-66 было подтверждено во всех 3 образцах. В то время как среди образцов наблюдались различия максимального сигнала, значения EC50 были очень сходными (в среднем, 76,6 пМ), и в пределах 2-кратных значений от того, что наблюдалось для T-клеток человека (49,9 пМ) (фиг. 5).
Интернализация CD96-связывающих белков при связывании с T-клетками и NK-клетками человека
Судьбу 42Y073-86F08-66 после связывания определяли в культурах человеческих МКПК от 3 доноров с использованием визуализирующей цитометрии. Сильное точечное окрашивание за счет 42Y073-86F08-66 наблюдали на клеточной мембране во всех 3 клеточных популяциях (CD4+ T-клетки, CD8+ T-клетки и NK-клетки) от всех 3 доноров в исходном состоянии, и этот паттерн окрашивания сохранялся в течение 45 часов (фиг. 6). Во всех трех анализируемых клеточных популяциях (CD4+, CD8+ и NK-клетки) интернализация 42Y073-86F08-66-PE была медленной, и не завершалась за 45 часов. Эти данные позволяют предположить, что интернализация 42Y073-86F08-66 является медленной в сравнении с другими антителами, направленными на T-клеточные поверхностные рецепторы.
CD96-связывающие белки ингибируют связывание лиганда CD96 CD155 с CD96-экспрессирующими клетками
На первичных человеческих T-клетках было продемонстрировано, что предварительное образование комплекса 42Y073-86F08-66 с CD96 на мембране ингибировало связывание последнего с его лигандом (рекомбинантным человеческим CD155:Fc) (IC50 0,16 нМ) (фиг. 7).
Нарушение установленных взаимодействий CD96:CD155 за счет CD96-связывающих белков
Когда CD155:Fc был предварительно связан с первичными человеческими T-клетками, белок 42Y073-86F08-66 мог конкурентно вытеснять CD155 зависимым от дозы образом (IC50 1,93 нМ) (фиг. 8), это указывало на то, что 42Y073-86F08-66 может вытеснять связанный с CD96 CD155, естественный лиганд для CD96.
Связывание Fc-рецептора CD96-связывающими белками
Поскольку 42Y073-86F08-66 является полностью человеческим IgG1 антителом с Fc-областью ДТ, ожидается, что он будет связывать соответствующие Fc-рецепторы.
Используя панель рекомбинантных линий клеток (Promega), экспрессирующих активирующие человеческие Fcγ-рецепторные репортеры (FcγRI, FcγRIIa(R), FcγRIIa(H), FcγRIIIa(V) и FcγRIIIa(F)), авторы изобретения показали, что связывание 42Y073-86F08-66 с CD96 на первичных человеческих T-клетках не приводит к активации человеческих Fcγ-рецепторов (фиг. 9). Напротив, контрольное анти-CD52 антитело (кампат) эффективно вызывало сигналы активации FcγR в случае всех протестированных экспрессирующих Fcγ-рецепторы клеток.
Было важно подтвердить результаты анализов с репортерами в анализах уничтожения клеток с использованием первичных человеческих клеток. В культурах свежих человеческих МКПК, содержащих NK-клетки, отсутствовали признаки повышенной клеточной гибели как CD4+, так и CD8+, T-клеток в присутствии 42Y073-86F08-66 (фиг. 10). В тех же экспериментах как CD4+, так и CD8+, T-клетки были эффективно истощены в присутствии анти-CD52 антитело (кампат).
Первый этап классического пути активации комплемента опосредуется связыванием компонента комплемента C1q с клетками, которые опсонизированы антителами. Аффинность связывания человеческого компонента комплемента C1q с 42Y073-86F08-66 определяли с использованием ППР, и она была выше (KD 94,4 нМ), чем таковая для контрольного по изотипу IgG1 ДТ (KD 643,5 нМ). Потенциальную способность 42Y073-86F08-66 индуцировать комплементзависимую цитотоксичность (CDC) изучали с использованием первичных человеческих T-клеток в качестве мишеней.
В анализах CDC с использованием человеческой сыворотки в качестве источника комплемента отсутствовали признаки истощения как CD4+, так и CD8+, T-клеток за счет 42Y073-86F08-66, в то время как контрольное анти-CD52 антитело эффективно опосредовало истощение обоих подмножеств (фиг. 11). В совокупности эти данные свидетельствуют о том, что риск ADCC или CDC-опосредованного истощения CD96-экспрессирующих T-клеток со связавшимися 42Y073-86F08-66 является незначительным.
Пример 3
In vitro исследования эффективности и механизма действия CD96-связывающих белков
In vitro эффективность 42Y073-86F08-66 в анализе со смешанной культурой первичных человеческих лимфоцитов (МКПК-СКЛ)
Эффект 42Y073-86F08-66, экспрессированного и очищенного из клеток CHO, а также из клеток HEK, тестировали в анализе с первичными МКПК без добавления CD155, анти-CD3 или анти-TIGIT. Для этого анализа МКПК от 8 разных людей-доноров смешивали вместе и добавляли в лунки с разными антителами в растворе. После инкубации в течение 3 дней определяли содержание IFNγ в супернатанте с использованием гомогенной флуоресценции с временным разрешением (HTRF) в качестве метода детекции. В этом анализе анти-CD3 не требуется, поскольку МКПК были активированы в результате несовпадения MHC у разных доноров. Эффективность была аналогичной в случае обоих CD96-связывающих белков (экспрессируемых в клетках CHO или HEK) (фиг. 12). Оба оказались более эффективными, чем тецентрик (EC50 49 пМ). В отдельном повторении того же эксперимента значения EC50 составляли 22 пМ, 10 пМ и 140 пМ для 42Y073-86F08-66 (HEK), 42Y073-86F08-66 (CHO) и тецентрика, соответственно (фиг. 12). Жизнеспособность клеток также измеряли после завершения 3 дней, и лишь в случае тецентрика была обнаружена несколько сниженная жизнеспособность клеток. Ни одно из анти-CD96 мАт или контролей по изотипу не приводили к снижению жизнеспособности клеток, в соответствии с наблюдаемым отсутствием истощения клеток в анализах ADCC.
CD4+ T-клетки являются основным источником 42Y073-86F08-66-индуцируемого IFNγ в анализе со смешанной культурой человеческих лимфоцитов (МКПК-СКЛ)
Для выяснения того, какие клеточные популяции ответственны за наблюдаемое увеличение уровня секретируемого IFNγ в супернатанте при воздействии 42Y073-86F08-66 (HEK) и 42Y073-86F08-66 (CHO) в анализах с МКПК, проводили исследования по истощению клеток. МКПК от 4 разных доноров истощали отдельно по CD4+ T-клеткам или CD8+ T-клеткам перед смешиванием для проведения активации лимфоцитов и воздействием антитела. Микрогранулы с антителами против CD4 или CD8 человека (Miltenyi) использовали для проведения истощения, и чистоту истощения CD4+ или CD8+ T-клеток проверяли методом проточной цитометрии. CD4+-клетки были истощены из МКПК на 98,5-99,5%, и CD8+-клетки были истощены из МКПК на 97,2-100%. В сравнении с не истощенными клетками данные, полученные в анализе со смешанной культурой лимфоцитов МКПК-СКЛ с истощением T-клеток, указывали на то, что CD4+ T-клетки, но не CD8+ T-клетки, были основным подмножеством, ответственным за индукцию IFNγ за счет 42Y073-86F08-66 (HEK) в данном анализе (фиг. 13).
Исследование методом проточной цитометрии для понимания механизма действия CD96-связывающих белков
Для более полного понимания механизма действия анти-CD96 антител проводили исследования методом проточной цитометрии с целью изучения изменений в экспрессии клеточных поверхностных рецепторов, а также внутриклеточных цитокинов, в разных клеточных популяциях в человеческих МКПК. Использовали ту же систему смешанной культуры лимфоцитов, МКПК-СКЛ. МКПК от 8 доноров смешивали вместе для активации клеток за счет несовпадения MHC. После добавления анти-CD96 или контрольных по изотипу антител МКПК инкубировали в течение 0 дней (исходное состояние) или 3 дней, а затем клетки фиксировали и количественно определяли различные маркеры методом проточной цитометрии. Для подтверждения активности 42Y073-86F08-66 в данном эксперименте также измеряли секретируемые IFNγ и гранзим B в супернатанте методами MSD и ELISA после дня 3. Как было замечено ранее, белок 42Y073-86F08-66 приводил к усилению высвобождения IFNγ и гранзима B (фиг. 14). Увеличение секреции IFNγ или гранзима B отсутствовало в случае анти-TIGIT мАт клона 1F4 (Roche, чIgG1-ДТ).
Для понимания того, какие клеточные популяции вносят вклад в увеличение секреции IFNγ за счет 42Y073-86F08-66, анализировали внутриклеточное окрашивание IFNγ в разных клеточных популяциях методом проточной цитометрии. Даже в группе обработки контрольным по изотипу IgG1 наблюдалось значительное увеличение экспрессии (данные не представлены), а также частоты встречаемости подмножеств IFNγ+CD4+, CD8+ и NK-клеток в день 3 в сравнении с днем 0, это указывало на активацию лимфоцитов за счет смешивания МКПК от разных доноров (фиг. 15). Обработка 42Y073-86F08-66 дополнительно приводила к значительному увеличению частоты встречаемости IFNγ+клеток во всех трех подмножествах иммунных клеток, а именно, CD4+, CD8+ и NK-клетках (фиг. 15). Анти-TIGIT антитело (клон 1F4) приводило к увеличению частоты встречаемости IFNγ+клеток в популяции NK-клеток.
Кроме того, экспрессия CD96 была значительно повышена во всех трех популяциях клеток после активации клеток в день 3 в сравнении с днем 0, профиль, обычно наблюдаемый для белков контрольных точек. Обработка анти-CD96 антителом приводила к значительному снижению СИФ для CD96 и частоты встречаемости CD96+клеток во всех 3 подмножествах, CD4+, CD8+ и NK-клетках, в сравнении с контрольной группой с совпадением по изотипу. Обработка антителом 86F08-66-LAGA с инактивированной Fc-областью также приводила к снижению профиля CD96, хотя снижение экспрессии CD96 в группе обработки 42Y073-86F08-66 было гораздо более значительным (фиг. 16-17). Анти-TIGIT Ат клона 1F4 не оказывало никакого эффекта. Это появляющееся снижение экспрессии CD96 при обработке 42Y073-86F08-66 могло быть следствием конкуренции между анти-CD96 обнаруживающим антителом и терапевтическим Ат, на что указывает занятость рецепторов терапевтическим антителом, или могло быть следствием интернализации рецептора CD96 при связывании 42Y073-86F08-66. Обнаруживающим антителом, используемым в анализе, было коммерческое Ат клона 6F9 (BD Biosciences).
CD226 является одним из основных активирующих рецепторов в NK-клетках, однако его роль в T-клетках не так хорошо изучена. Для более детального понимания механизма авторы изобретения сосредоточились на определении характеристик CD226+популяции NK-клеток в анализе МКПК-СКЛ. Повышение экспрессии CD96 после активации клеток в день 3 приводило к увеличению частоты встречаемости CD226+CD96+NK-клеток. Обработка 42Y073-86F08-66 приводила к более высокому относительному содержанию CD226+ однократно положительных, чем CD226+CD96+ дважды положительных NK-клеток в день 3 (26,2%/25,2%=1), в сравнении с обоими контролями по изотипу (5,91%/46,4%=0,127), а также 86F08-66 IgG1 LAGA антителом с инактивированной Fc-областью (13,8%/47,2%=0,29), потенциально за счет занятости мишеней и/или интернализации CD96 (фиг. 18). Поскольку CD226 является активирующим рецептором в оси, и CD96 является предполагаемым рецептором - контрольной точкой в той же оси, CD226+однократно положительные клетки могут представлять собой более активированные NK-клетки, чем CD226+CD96+ дважды положительные клетки.
Обработка 42Y073-86F08-66 приводила к более высокой частоте встречаемости IFNγ+клеток и GrzB+клеток среди CD226+NK-клеток в день 3 после обработки 42Y073-86F08-66. В случае ведущего IgG1-LAGA антитела с инактивированной Fc-областью (86F08-66-LAGA) эффект отсутствовал.
В целом, обработка 42Y073-86F08-66 приводила к большему количеству IFNγ+GrzB+дважды положительных клеток в общем количестве NK-клеток в день 3 в сравнении с контрольными по изотипу Ат или ведущим IgG1-LAGA антителом с инактивированной Fc-областью (86F08-66-LAGA) (фиг. 19).
CD155-МКПК анализ для скрининга CD96-связывающих белков
Для определения ингибирующего эффекта связанного с планшетом конъюгата CD155-Fc на продуцирование IFNγ в человеческих МКПК круглодонный 96-лучночный не-TC планшет (№ 351177) покрывали rhCD155-Fc (каталожный №9174-CD-050, R&D Systems) в разных дозах в течение ночи при 4°C и блокировали средой AIM-V (каталожный №12055-091, Therrmo Fisher), содержащей 5% БСА (каталожный № 9576, Sigma), в течение 30 мин при комнатной температуре. МКПК (каталожный №70025, Stemcell Technologies), предварительно обрабатывали CD96-связывающими белками, анти-TIGIT мАт (каталожный №MAB7898, R&D Systems) или контролем по изотипу при комнатной температуре в течение 10 мин, добавляли в лунки при плотности 2×105 клеток/лунку в среде AIM-V, содержащей 0,01 мкг/мл анти-CD3 мАт, и культивировали в течение 3 дней. Супернатанты собирали и хранили при -20°C для количественного определения IFNγ и гранзима B методами MSD или ELISA. Ингибирующий эффект связанного с планшетом конъюгата CD155-Fc на продуцирование IFNγ в человеческих МКПК показан на фиг. 20. CD96-связывающие белки, судя по всему, уменьшают опосредованное CD155-Fc ингибирование продуцирования IFNγ (в сравнении с контрольным по изотипу антителом IgG1-ДТ) (фиг. 26). Проводили дополнительные анализы, определяя способность CD96-связывающих белков уменьшать опосредованную CD155-Fc супрессию TNFαα (в присутствии анти-TIGIT мАт) (фиг. 27). Дополнительные анализы, проведенные с, и без, анти-TIGIT мАт, свидетельствуют о том, что способность CD96-связывающих белков уменьшать опосредованное CD155-Fc ингибирование продуцирования IFNγ имеет место как в присутствии, так и в отсутствие, анти-TIGIT мАт (фиг. 28).
Активность CD96-связывающих белков в анализах с человеческими опухоль-инфильтрирующими лимфоцитами (ОИЛ)
Микроокружение опухоли (МОО) является иммуносупрессивным, и опухоль-инфильтрирующие лимфоциты (ОИЛ), обнаруженные в опухолях, часто имеют нарушенную иммунную функцию или являются «истощенными». Анализ с первичными человеческими ОИЛ был разработан для оценки потенциального терапевтического эффекта 42Y073-86F08-66 in vitro. Свежие первичные резецированные опухоли механически и с помощью ферментов диссоциировали на суспензии отдельных клеток, содержащие как опухолевые клетки, так и ОИЛ. ОИЛ в клеточной суспензии слабо активировали неоптимальной дозой растворимого анти-CD3 (клон HIT3a) и высевали в тройном повторе в 96-луночные круглодонные планшеты со сверхслабым прикреплением для формирования сфероидов. Затем в лунки добавляли антитела, и IFNγ в супернатанте количественно определяли через 6 дней. 42Y073-86F08-66 оценивали в данном анализе при 3 концентрациях (10, 2 и 0,5 мкг/мл) отдельно или в сочетании либо с анти-PD-1 (кейтруда/пембролизумаб) (10 мкг/мл) или анти-TIGIT (100 мкг/мл). Анти-PD-1 отдельно также тестировали в качестве положительного контроля для индукции IFNγ, превышающей индукцию только за счет анти-CD3. Соответствующие контроли по изотипу также были включены.
В данном анализе тестировали всего 6 опухолей (4 опухоли эндометрия и 2 опухоли почки). В случае 4 опухолей эндометрия анти-PD-1 совместно с обработкой анти-CD3 индуцировали IFNγ на уровнях, эквивалентных уровням при использовании одного анти-CD3, это указывало на то, что стимуляция анти-CD3 может быть слишком высокой и может потребоваться дальнейшая техническая разработка. В одной из двух почечных опухолей (образец 5001063) обработка анти-PD-1 приводила к повышению (p<0,05 при использовании одностороннего Anova) продуцирования IFNγ до уровня, превышающего уровень при использовании одного анти-CD3. Кроме того, обработка сочетанием анти-PD-1+42Y073-86F08-66 (10 мкг/мл) приводила к значительному (p<0,001 при использовании одностороннего Anova) повышению уровней IFNγ до 7764 пг/мл, выше чем отдельно 42Y073-86F08-66 в концентрации 10 мкг/мл (599 пг/мл), отдельно анти-PD-1 (2692 пг/мл) или простой аддитивный эффект обоих (фиг. 21). В случае второй почечной опухоли (образец 1002273), обработка анти-PD-1 не приводила к увеличению продуцирования IFNγ до уровней, превышающих стимуляцию одним только анти-CD3. Эти опухоли не проходили предварительный скрининг на экспрессию оси PD-1 или экспрессию оси CD96. Сложность этих анализов с ОИЛ широко признана, и наблюдались вариации в ответ на обработку анти-PD-1 среди опухолей разных пациентов. Это неудивительно, учитывая частоту общего ответа для анти-PD-1 (кейтруда) у не проходивших скрининг пациентов, составляющую ниже 25%.
Пример 4
In vivo исследования эффективности и механизма действия
Исследование методом визуализации биолюминесценции CD96-связывающего белка в модели зависимой от NK-клеток колонизации легких клетками меланомы B16F10
NK-клетки относятся к семейству врожденных лимфоцитов и играют важную роль в контроле роста опухоли на ранних стадиях и распространения метастазов за счет цитотоксической активности и высвобождения воспалительных цитокинов. Часто используемой in vivo моделью для изучения зависимой от NK-клеток противораковой активности является модель колонизации легких клетками меланомы B16F10. Эта модель также была описана в публикациях, относящихся к связанному с CD96 раку (Blake S.J. et al. 2016). Авторы изобретения решили использовать данную модель для исследования активности 42Y073-86F08-66 (продуцируемого в клетках HEK) in vivo. Для определения эффективности анти-CD96 мАт в борьбе с метастазами в легких в режиме реального времени, и в более количественном выражении, вместо использования обычных клеток меланомы B16F10 были использованы клетки B16F10 с закодированной красной люциферазой светляков (RFluc) для получения возможности in vivo визуализации люциферазного сигнала из легких, являющегося показателем опухолевого бремени.
После введения в хвостовую вену ~500000 клеток B16F10 RFluc метастатической меланомы проводили in vivo биолюминесцентную визуализацию в день 0 (через ~15 минут после инъекции клеток), день 7, 10, 14, 17 и 20. (Начальная колонизация клеток происходит в легких уже через 15 минут после инъекции, согласно результатам предыдущих исследований по созданию модели). Частота дозирования составляла два раза в неделю (фиг. 22). После визуализации in vivo в день 20 у мышей извлекали легкие для ex vivo биолюминесцентной визуализации.
Для дальнейшей оценки роли определенных типов иммунных клеток (CD4+, CD8+ и NK-клеток) в данной системе CD4+ клетки, CD8+ клетки, NK-клетки, или как CD4+, так и CD8+, клетки истощали с использованием хорошо известного метода с обработкой антителом. Последующий анализ методом проточной цитометрии подтвердил истощение NK-клеток, а также T-клеток в группе истощения CD4+/CD8+ клеток.
Биолюминесцентная визуализация в день 14 выявила значительно повышенный сигнал в легких у животных в группе с истощенными NK-клетками (фиг. 23). Более слабый сигнал в легких (фотон/сек) соответствует меньшему количеству метастазов. Действительно, 19 из 20 мышей не выжили до конца исследования, предположительно из-за тяжелого опухолевого бремени в легких. Эти данные четко подтверждают крайне важную роль NK-клеток как клеток, в первую очередь ответственных за подавление легочных метастазов в данной модели.
In vivo биолюминесцентная визуализация, проведенная в день 14, когда мыши с истощенными NK-клетками все еще были живы, показала, что в группах животных с истощенными NK-клетками введение 42Y073-86F08-66 (продуцируемого в клетках HEK) приводило к значительному уменьшению количества легочных метастазов (*P<0,05) в сравнении с введением контроля по изотипу (фиг. 24, A). В группе животных с неистощенными клетками тенденция также наблюдалась, но не была статистически значимой. В день 20 в группах животных с неистощенными клетками сигнал в легких был снижен в группе получения 42Y073-86F08-66, однако результаты не были статистически значимыми (н/з). В случае групп животных с истощенными T-клетками 42Y073-86F08-66 приводил к значительному снижению (**P<0,01) сигнала в легких в сравнении с контролем по изотипу (фиг. 24, B). Более слабый сигнал в легких (фотон/сек) соответствует меньшему количеству метастазов. Цифровое изображение легких после умерщвления мышей в день 20 подтверждало эффект 42Y073-86F08-66 (фиг. 25).
Пример 5
Краткое описание данных по эффективности и механизму действия
Доказательства активности 42Y073-86F08-66 в отношении T-клеток
Человек: CD96 экспрессируется на легко поддающемся обнаружению уровне как в CD4+, так и в CD8+, T-клетках в МКПК, а также в микроокружении опухоли. В ОИЛ, помимо NK-клеток, CD96 был обнаружен на CD8+, CD4+Teff клетках, а также Treg. В соответствии с паттерном экспрессии, анализ методом проточной цитометрии показал, что обработка 42Y073-86F08-66 приводила к увеличению процентного содержания IFNγ+CD4+ и CD8+ T-клеток в анализе со смешанной культурой лимфоцитов МКПК-СКЛ. Исследования по истощению показали резкое снижение высвобождения IFNγ при использовании 42Y073-86F08-66 в анализе со смешанной культурой лимфоцитов МКПК-СКЛ, когда CD4+ T-клетки были истощены. Однако истощение CD8+ клеток не оказывало существенного эффекта в данном конкретном анализе.
Мышь: В условиях in vivo при проведении анализа колонизации легких клетками B16F10 в день 14 после инъекции опухолевых клеток, когда мыши с истощенными NK-клетками все еще были живы, после истощения NK-клеток введение 42Y073-86F08-66 все еще приводило к статистически значимому снижению количества легочных метастазов в сравнении с контролем по изотипу, предположительно за счет активации T-клеток.
Доказательства активности NK-клеток
Человек: CD96 экспрессируется на легко поддающемся обнаружению уровне на NK-клетках в МКПК, а также в микроокружении опухоли. Действительно, среди субпопуляций ОИЛ наивысший уровень экспрессии CD96 был обнаружен на NK-клетках. CD226 является одним из основных активирующих рецепторов для NK-клеток, и убедительные данные по влиянию CD96 на активность NK-клеток также приведены в литературе. В условиях in vitro исследования с внутриклеточным окрашиванием цитокинов и использованием проточной цитометрии показали увеличение содержания IFNγ+GrzB+NK-клеток под воздействием 42Y073-86F08-66 в анализах со смешанной культурой лимфоцитов МКПК-СКЛ.
Мышь: В условиях in vivo при использовании признанной, в высокой степени зависимой от NK-клеток модели, модели колонизации легких клетками B16F10, 42Y073-86F08-66 был способен значительно подавлять развитие метастазов в легких, когда и CD4+, и CD8+T-клетки были истощены, убедительно свидетельствуя в пользу активности NK-клеток.
Пример 6
Анти-CD96 мАт - доклинические токсикологические исследования
GSK6097608B (далее в настоящем описании называемый GSK6097608, что является синонимом 42Y073-86F08-66) представляет собой моноклональное антитело (мАт), направленное на кластер дифференцировки (CD)96, которое находится в стадии разработки для лечения рака. Исследования для изучения диапазона доз при внутривенном (в/в) введении, и 4-недельные исследования токсичности повторных доз, анти-CD96 мАт были проведены на яванских макаках и мышах BALB/c. Кроме того, ФК и ФД исследования с введением одной дозы были проведены на обезьянах. Проводили in vitro АВЦ в образцах человеческой крови и АВЦ для сочетания с достарлимабом (анти-PD-1 мАт). Оценку профиля связывания проводили с использованием микрочипов с человеческими белками, с последующими подтверждающими анализами связывания. Предварительное иммуногистохимическое (IHC) исследование проводили с использованием избранных тканей человека и яванского макака.
Мыши BALB/c и яванские макаки были выбраны в качестве видов для доклинических исследований для оценки профиля безопасности анти-CD96 мАт.Обезьяна была признана подходящим для доклинических исследований видом, на котором можно оценивать потенциальную токсичность анти-CD96 мАт, поскольку анти-CD96 мАт перекрестно реагирует с аналогичной аффинностью с рецепторами CD96 у человека и обезьян. Дополнительные доклинические исследования безопасности проводили на мышах BALB/c, исходя из начальных результатов ФК и эффективности, а также in vitro связывания анти-CD96 мАт с мышиными спленоцитами и продуцирования цитокинов мышиными спленоцитами, предварительно стимулированными CD3/CD28 и инкубированными с анти-CD96 мАт.
В токсикологических исследованиях на обезьянах анти-CD96 мАт хорошо переносилось после 4 еженедельных доз вплоть до 100 мг/кг/неделю. Занятость рецепторов (ЗР) CD96 или связывание мишени на CD8+T-клетках, CD4+T-клетках и NK-клетках сохранялось на протяжении всего исследования. Наблюдали низкую частоту встречаемости антител против лекарственного средства (ADA) с низкими титрами, что не влияло на связывание с рецепторами или системное воздействие. Не было никаких изменений в количестве циркулирующих клеток, экспрессирующих CD96, и никаких гистопатологических отклонений в тканях, включая первичные и вторичные иммунные ткани, что указывало на низкий риск зависимого от кристаллизуемого фрагмента (Fc) истощения CD96-экспрессирующих эффекторных клеток. Анти-CD96 мАт хорошо переносились в течение 4 недель при введении мышам доз 100 мг/кг/неделю; однако более низкая доза 10 мг/кг/неделю вызывала опосредованную иммунной системой анафилаксию вследствие ADA или циркулирующих иммунных комплексов после введения третьей еженедельной дозы.
Проводили in vitro АВЦ, оценивая эффект отдельно анти-CD96 мАт, отдельно достарлимаба, и сочетания анти-CD96 мАт с достарлимабом с использованием МКПК, полученных от здоровых доноров - 5 мужчин и 5 женщин. Относительно контролей со средой наблюдалось минимальное увеличение содержания интерлейкина (IL)-10, иммунорегуляторного противовоспалительного цитокина, у большинства доноров при воздействии каждого антитела, и слабое или умеренное связанное с воздействием увеличение содержания IL-6 и/или фактора некроза опухоли альфа (TNF-αα) у 1 донора в каждом из вариантов с использованием только анти-CD96 мАт и только достарлимаба. Эти уровни были ниже, чем в случае анти-CD3 и анти-CD28 положительных контролей, которые индуцировали пан-цитокиновый ответ. В целом, не наблюдалось увеличения продуцирования цитокинов при комбинированном лечении; однако 1 донор, для которого было отмечено повышенное содержание IL-6 и TNF-αα при использовании только анти-CD96 мАт, также оказался более чувствительным к комбинированному лечению. Результаты указывают на общий низкий риск возникновения синдрома высвобождения цитокинов (СВЦ), однако отдельные участники могут отличаться в отношении индукции цитокинов.
На основании переносимости и отсутствия соответствующих побочных явлений уровень, не вызывающий наблюдаемых нежелательных явлений (NOAEL) был определен как 100 мг/кг/неделю (наивысшая протестированная доза) как у обезьян (усредненная для обоих полов в неделю 4 средняя площадь под кривой [AUC]0-168 ч: 580 мг⋅ч/мл [диапазон: 483-647 мг⋅ч/мл] и Cmax: 5,54 мг/мл [диапазон: 5,10-6,54 мг/мл]), так и у мышей (усредненная для обоих полов в неделю 4 средняя площадь под кривой [AUC]0-168 ч: 319 мг⋅ч/мл и Cmax: 2,98 мг/мл).
Пример 7
Впервые проводимое на людях исследование анти-CD96 мАт в качестве монотерапии и в сочетании с достарлимабом (также известным как TSR-042)
GSK6097608B (далее в настоящем описании называемый GSK6097608, что является синонимом 42Y073-86F08-66) представляет собой моноклональное антитело (мАт), направленное на кластер дифференцировки (CD)96 (анти-CD96 мАт), которое находится в стадии разработки для лечения рака. Связывание CD96 соответствующим рецептором, CD155, действует в качестве «выключателя», или иммунной контрольной точки, с подавлением иммунных ответов. Было разработано антагонистическое антитело к CD96 для блокирования ингибирующей оси CD96:CD155 и повышения противоопухолевой активности T-клеток и клеток - естественных киллеров (NK). В данном впервые проводимом на людях (FTIH) исследовании будет оценена безопасность, переносимость, фармакокинетика (ФК), фармакодинамика (ФД) и предварительная клиническая эффективность GSK6097608, вводимого в качестве монотерапии и в сочетании с достарлимабом. Достарлимаб (также известный как TSR-042) является экспериментальным гуманизированным мАт изотипа иммуноглобулина (Ig) G4 (IgG4); оно имеет высокую аффинность связывания с белком 1 программируемой гибели клеток (PD-1), что приводит к ингибированию связывания с лигандом программируемой гибели клеток (PD-L)1 и PD-L2. На основании наблюдаемой противоопухолевой активности других антител того же класса, ожидается, что достарлимаб будет проявлять клиническую активность при широком спектре раковых заболеваний.
На основе доказательств, подтверждающих молекулярное взаимодействие между путями и полезность обращения к различным иммунным популяциям, GSK6097608 может не только действовать совместно, с ингибированием PD-(L)1 (белка 1 программируемой гибели клеток и/или лиганда 1 программируемой гибели клеток), но также приносить пользу пациентам, которые являются рефрактерными к, или выработали устойчивость к, современным терапевтическим средствам на основе T-клеток.
Научное обоснование для сочетания с анти-PD-1
Несмотря на терапевтическую пользу от блокирования путей иммунных контрольных точек PD-(L)1 и CTLA-4 в опухолях различных видов, большинство пациентов не отвечают на монотерапию препаратами ингибиторов контрольных точек, и активно изучаются стратегии повышения их активности с помощью комбинированных подходов. Обоснование для сочетания анти-CD96 мАт (GSK6097608) со средством, предназначенным для блокирования пути PD-(L)1 (достарлимаб, анти-PD-1 мАт), основано на доказательствах, подтверждающих молекулярное взаимодействие между путями, полезности обращения к различным иммунным популяциям, а также терапевтической комплементарности между стратегиями вмешательства.
На механистическом уровне было показано, что сигнализация PD-1 приводит к дефосфорилированию внутриклеточного домена CD226, ослабляя потенциал костимуляции после CD96-опосредованного перенаправления CD155. Примечательно, что экспрессия CD96 повышается в опухолевой ткани меланомы после лечения ниволумабом (анти-PD-1), что указывает на ингибирующую ось CD96 как на возможный механизм адаптивной резистентности к блокаде PD-1. При блокаде CD96 также может наблюдаться обратное повышение регуляции компонентов пути, поскольку опосредованная GSK6097608 индукция интерферона гамма (IFNγ) обладает потенциалом для повышения экспрессии PD-L1. В совокупности, эти наблюдения позволяют предположить, что совместная блокада CD96 и PD-(L)1 может быть необходима для обеспечения эффективных противоопухолевых иммунных ответов.
Клинический опыт применения достарлимаба
Достарлимаб в настоящее время разрабатывают в качестве монотерапии для пациентов с рецидивирующими или прогрессирующими солидными опухолями, включая рак эндометрия (опухоли с микросателлитной стабильностью и высокой частотой микросателлитной нестабильности [MSI-H]), немелкоклеточный рак легких (NSCLC) и не эндометриальные MSI-H солидные опухоли, а также рак с мутантной полимеразой ε. Кроме того, достарлимаб разрабатывают в качестве комбинированной терапии с другими терапевтическими средствами для пациентов с прогрессирующими солидными опухолями (включая меланому, NSCLC и колоректальный рак), либо прогрессирующим или метастатическим раком (включая эндотелиальный рак яичника, трижды негативный рак молочной железы и уротелиальную карциному).
По состоянию на 21 января 2019 года проводилось 4 исследования фазы 1, 2 исследования фазы 2 и 1 исследование фазы 3 с достарлимабом.
Безопасность и переносимость достарлимаба были оценены у более чем 627 участников с прогрессирующим раком, которые получили по меньшей мере 1 дозу достарлимаба. Из 335 участников, получавших монотерапию достарлимабом в FTIH исследовании 4010-01-001 (GARNET), 93,7% сообщали о по меньшей мере 1 нежелательном явлении, возникшем в ходе лечения (НЯВЛ), с наиболее часто упоминаемыми явлениями усталости, тошноты и диареи. Возникающие в ходе исследуемого вмешательства НЯВЛ ≥3 степени были зарегистрированы у 36 участников (10,7%). Большинство из связанных с исследуемым вмешательством НЯВЛ ≥3 степени произошло только у 2 участников каждое, за исключением усталости (6 участников), повышения уровня аланинаминотрансферазы (ALT) (4 участника), анемии (4 участника), повышения уровня аспартатаминотрансферазы (AST) (3 участника) и повышения уровня липазы (3 участника). Серьезные нежелательные явления (СНЯ) имели место у 106 участников (31,6%); у 21 из этих участников данные СНЯ рассматривались как связанные с исследуемым вмешательством. Все связанные с исследуемым вмешательством СНЯ возникали у 1 участника каждое, за исключением пневмонии (4 участника), диспноэ (2 участника), пирексии (2 участника) и макулопапулезной сыпи (2 участника). Двадцать три участника (6,9%), получавшие монотерапию достарлимабом, испытали по меньшей мере 1 связанное с иммунитетом нежелательное явление (иНЯ) со степенью тяжести ≥3; для 18 из 23 участников с иНЯ ≥3 степени нежелательные явления (НЯ) были оценены исследователями как связанные с исследуемым вмешательством. Большинство из иНЯ ≥3 степени были зарегистрированы у ≤2 участников каждое. Зарегистрированные у>2 участников иНЯ ≥3 степени представляли собой повышение уровня AST (4 участника), повышение уровня ALT (4 участника), повышение уровня липазы (4 участника) и сыпь (3 участника).
Из 292 участников, получавших достарлимаб в сочетании с другими терапевтическими средствами, 94,5% сообщали о по меньшей мере 1 НЯВЛ, с наиболее часто упоминаемыми явлениями усталости, тошноты и диспноэ. СНЯ имели место у 108 участников (37,0%); у 20 из этих 108 участников СНЯ были связаны с исследуемым вмешательством.
Исходя из данных по безопасности из человеческого опыта и имеющейся доклинической информации о фармакологии и токсикологии, достарлимаб продемонстрировал приемлемый клинический и доклинический профиль безопасности, который, по-видимому, сопоставим с профилем безопасности одобренных ингибиторов PD-1, мАт пембролизумаба и ниволумаба.
Исходя из наблюдаемой противоопухолевой активности других антител того же класса, ожидается, что достарлимаб будет проявлять клиническую активность при широком спектре раковых заболеваний. Предварительные данные по эффективности, полученные у 15 участников с раком эндометрия MSI-H и 24 участников с NSCLC, у которых была проведена по меньшей мере 1 оценка опухоли, были представлены на ежегодном собрании Американской ассоциации исследований рака в апреле 2018 года. Ответы были оценены исследователями с использованием связанных с иммунитетом критериев оценки объективного ответа при солидных опухолях (иRECIST). Среди 15 участников с раком эндометрия MSI-H частота общего ответа составляла 47% и состояла полностью из частичных ответов (ЧО); 20% имели стабильное заболевание (СЗ) и 33% имели прогрессирующее заболевание. Среди 24 участников с NSCLC ЧОО составляла 29% и состояла полностью из ЧО; 42% имели СЗ и 17% имели прогрессирующее заболевание. Таким образом, предварительные данные по эффективности от участников с NSCLC, получавших лечение достарлимабом, по-видимому, сопоставимы с заявленной эффективностью других ингибиторов PD-(L)1, таких как пембролизумаб, у участников с прогрессирующим и рецидивирующим раком (ЧОО: 19%) [Herbst, 2016].
Цели и конечные критерии оценки
Данное FTIH открытое исследование с эскалацией дозы позволит оценить безопасность, переносимость, ФК, ФД и предварительную клиническую эффективность GSK6097608 у участников с местнораспространенными, рецидивирующими или метастатическими солидными опухолями в качестве монотерапии (группа A) или в сочетании с достарлимабом (группа B); исследование будет использовано для определения рекомендуемой дозы фазы 2 (РД2Ф).
- Частота, продолжительность и тяжесть НЯ и СНЯ
- Изменения дозы (например, снижение или отсрочка дозы)
- Выведение из исследования вследствие НЯ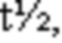 насколько позволяют данные
насколько позволяют данные
- Достарлимаб (группа B): концентрации в плазме, ФК параметры, такие как Cmax, Cmin, AUC, 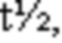 , насколько позволяют данные
, насколько позволяют данные
- ЧКЗ, ВДО, ДО и ВДП на основании RECIST 1.1 и иRECIST, насколько позволяют данные
- ФК (концентрации в плазме, ФК параметры, такие как Cmax, Cmin, AUC, 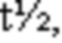 насколько позволяют данные)
насколько позволяют данные)
- ФД биомаркеры (например, фенотипы иммунных клеток, показатели иммунной функции)
- ЧОО на основании критериев RECIST 1.1
- Безопасность (например, лабораторные показатели, НЯ и СНЯ)
- Ответа, включая использование GSK6097608 отдельно или в сочетании с любыми сопутствующими лекарственными препаратами
- Восприимчивость к заболеванию, тяжесть, прогрессирование и связанные с этим состояния
- Корреляция между использованием антибиотиков/пробиотиков до лечения и противоопухолевой активностью
Сокращения: ADA=антитела против лекарственного средства; НЯ=нежелательное явление; AUC=площадь под кривой; Cmax=максимальная концентрация; Cmin=минимальная концентрация; ЧКЗ=частота контроля заболевания; ДЛТ=дозолимитирующая токсичность; ДНК=дезоксирибонуклеиновая кислота; ДО=длительность ответа; ИГХ=иммуногистохимия; иRECIST=модифицированные критерии оценки объективного ответа при солидных опухолях, версия 1.1 для иммунных терапевтических средств; в/в=внутривенно(е); ЧОО=частота общего ответа; ФД=фармакодинамика; ФК=фармакокинетика; РНК=рибонуклеиновая кислота; RECIST 1.1=Критерии оценки объективного ответа при солидных опухолях, версия 1.1; РД2Ф=рекомендованная доза фазы 2; СНЯ=серьезное нежелательное явление; 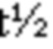 =период полувыведения; ВДП=время до прогрессирования; ВДО=время до ответа.
=период полувыведения; ВДП=время до прогрессирования; ВДО=время до ответа.
Общий дизайн исследования
Данное исследование представляет собой FTIH открытое, не рандомизированное, многоцентровое исследование, предназначенное для изучения безопасности, переносимости, ФК, ФД и предварительной клинической эффективности GSK6097608 в возрастающих дозах, вводимого в/в в качестве монотерапии (группа A) или в сочетании с достарлимабом (группа B) участникам с местнораспространенными, рецидивирующими или метастатическими солидными опухолями.
Препарат GSK6097608, вводимый внутривенно каждые 3 недели (Q3W), будет оценен в качестве монотерапии в возрастающих дозах (группа A) под руководством Комитета по эскалации дозы (DEC). Для дальнейшей оценки ФК и ФД дополнительные участники будут включены в группу ФК/ФД с введением РД2Ф (до 15 участников) при следующих типах опухолей: немелкоклеточный рак легких (NSCLC), плоскоклеточная карцинома головы и шеи (HNSCC), или опухолях других типов, определенных на основании появляющихся неклинических и/или клинических данных. Дополнительные участники также могут быть включены в группы ФК/ФД на ранее разрешенных уровнях дозы (до 15 участников на группу) для изучения доз. Эти дополнительные участники внесут вклад в оценку безопасности и предварительной противораковый активности, а также общего профиля данных ФК/ФД.
После определения дозы GSK6097608, которая является переносимой и обеспечивает адекватное воздействие лекарственного средства на основании данных ФК, может быть начат набор участников в группу комбинированного лечения (группа B). В группе B будут оценены возрастающие дозы GSK6097608 наряду с фиксированным режимом дозирования достарлимаба. Для дальнейшей оценки ФК и ФД дополнительные участники будут включены в группу ФК/ФД с введением РД2Ф (до 15 участников) при следующих типах опухолей: NSCLC, HNSCC, или опухолях других типов, определенных на основании появляющихся неклинических и/или клинических данных. Помимо группы, получающей РД2Ф, участники также могут быть включены в группы ФК/ФД на ранее разрешенных уровнях дозы (до 15 участников на группу) для изучения доз.
Эти дополнительные участники внесут вклад в оценку безопасности и предварительной противораковый активности, а также общего профиля данных ФК/ФД.
Оценку статуса заболевания при скрининге и при посещениях во время исследуемого вмешательства будет проводить исследователь в соответствии с критериями оценки объективного ответа при солидных опухолях, версии 1.1 (RECIST 1.1) и модифицированного варианта RECIST для иммунных терапевтических средств (иRECIST). Решение о прекращении лечения в связи с прогрессированием заболевания будет основано на иRECIST; однако некоторые вторичные и исследовательские анализы противораковой активности будут основаны на RECIST 1.1. Результаты сканирования будут собраны централизованно и храниться для возможности централизованной проверки.
Анализ
После формирования каждой группы дозирования будет использован метод непрерывной переоценки Ноеншвандера (N-CRM) для определения эскалации доз при монотерапии и комбинированной терапии. Решения об эскалации дозы будут основаны в первую очередь на дозолимитирующей токсичности (ДЛТ); однако будет также рассмотрена совокупность оценок клинической безопасности, а также данных ФК и ФД. Формальные статистические гипотезы проверяться не будут, и анализы будут носить описательный характер.
Данное исследование представляет собой открытое исследование лечения с 2 группами.
Количество участников
Приблизительно 100 участников будут зачислены для получения исследуемого лечения. Общее количество участников, которые будут набраны, является примерным, и будет зависеть от количества, необходимого для адекватной характеристики профиля ДЛТ и определения РД2Ф.
Группы лечения и продолжительность исследования
В группе A (монотерапии) GSK6097608 будет введен в/в на протяжении приблизительно 30 минут участникам под медицинским наблюдением исследователя или уполномоченного лица, имеющего соответствующую квалификацию.
В группе B (комбинированной терапии) GSK6097608 сначала будет введен в/в. Затем достарлимаб будет введен внутривенно в течение 30 минут после окончания инфузии GSK6097608. Оба вмешательства в рамках исследования будут проведены под медицинским наблюдением исследователя или уполномоченного лица.
Исследование включает 3 периода: скрининг (оценки за 28 дней до введения первой дозы), лечение (до прогрессирования заболевания, неприемлемой токсичности, смерти или отзыва согласия), а также прекращение лечения и последующее наблюдение (90 дней). Общая продолжительность участия в исследовании начинается с момента подписания формы информированного согласия (ICF) и продолжается в течение установленного протоколом заключительного периода последующей оценки. Максимальная продолжительность участия в исследовании будет зависеть от продолжительности получения участником исследуемого лечения, ориентировочная продолжительность составляет до 2 лет.
Визуализация опухоли и оценки заболевания
Метод и сроки оценки очага поражения, оценки заболевания, прогрессирования заболевания и ответа будут соответствовать критериям оценки объективного ответа при солидных опухолях, варианта 1.1 (RECIST 1.1). RECIST 1.1 будет использован при оценке бремени заболевания (определение целевых и нецелевых очагов поражения) во время скрининга и в качестве основного критерия оценки опухолевого ответа. иRECIST будет использован исследователями для оценки реакции и прогрессирования опухоли и для принятия решений о лечении.
СПИСКИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Области CDR, определенные по Kabat, подчеркнуты.
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-1
SEQ ID NO: 1
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-1
SEQ ID NO: 2
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGIIPIFGTASYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-1
SEQ ID NO: 3
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-1
SEQ ID NO: 4
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCAGCTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTTTCGGCACCGCCAGCTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-3
SEQ ID NO: 5
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-3
SEQ ID NO: 6
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSYNAISWVRQAPGQGLEWMGGIIPIMGTARYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-3
SEQ ID NO: 7
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-3
SEQ ID NO: 8
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCTACAACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTATGGGCACCGCCAGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-4
SEQ ID NO: 9
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-4
SEQ ID NO: 10
QVQLVQSGAEVKKPGSSVKVSCKASGGTFESEAISWVRQAPGQGLEWMGGIIPIFGRARYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-4
SEQ ID NO: 11
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-4
SEQ ID NO: 12
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCGAGAGCGAGGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTTTCGGCAGGGCCAGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-5
SEQ ID NO: 13
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-5
SEQ ID NO: 14
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSHAISWVRQAPGQGLEWMGGIIPIFGRGKYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-5
SEQ ID NO: 15
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-5
SEQ ID NO: 16
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCAGCCACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTTTCGGCAGGGGCAAGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-6
SEQ ID NO: 17
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-6
SEQ ID NO: 18
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSGHAISWVRQAPGQGLEWMGGIIPIFGRARYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-6
SEQ ID NO: 19
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-6
SEQ ID NO: 20
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCGGCCACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTTTCGGCAGGGCCAGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-18
SEQ ID NO: 21
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-18
SEQ ID NO: 22
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSRAISWVRQAPGQGLEWMGGIIPIMGTARYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-18
SEQ ID NO: 23
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-18
SEQ ID NO: 24
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCAGCAGGGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTATGGGCACCGCCAGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-33
SEQ ID NO: 25
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-33
SEQ ID NO: 26
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSYAISWVRQAPGQGLEWMGGILPIFGRANYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-33
SEQ ID NO: 27
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-33
SEQ ID NO: 28
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCAGCTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCCTGCCCATTTTCGGCAGGGCCAACTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-88
SEQ ID NO: 29
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-88
SEQ ID NO: 30
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSSSAISWVRQAPGQGLEWMGGIIPIFGRANYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-88
SEQ ID NO: 31
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-88
SEQ ID NO: 32
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCAGCAGCGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTTTCGGCAGGGCCAACTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-3
SEQ ID NO: 33
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-3
SEQ ID NO: 34
QVQLVQSGAEVKKPGSSVKVSCKASGGTFVNYAISWVRQAPGQGLEWMGGIIPALGTANYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-3
SEQ ID NO: 35
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-3
SEQ ID NO: 36
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCGTGAACTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCGCCCTGGGCACCGCCAACTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-4
SEQ ID NO: 37
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-4
SEQ ID NO: 38
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSEYAIHWVRQAPGQGLEWMGNIIPIFGTAGYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-4
SEQ ID NO: 39
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-4
SEQ ID NO: 40
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCGAGTACGCCATCCACTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCAACATCATCCCCATTTTCGGCACCGCCGGCTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-8
SEQ ID NO: 41
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-8
SEQ ID NO: 42
QVQLVQSGAEVKKPGSSVKVSCKASGGTFWLYAISWVRQAPGQGLEWMGGIIPQLGTANYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-8
SEQ ID NO: 43
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-8
SEQ ID NO: 44
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCTGGCTGTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCCAGCTGGGCACCGCCAACTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-17
SEQ ID NO: 45
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-17
SEQ ID NO: 46
QVQLVQSGAEVKKPGSSVKVSCKASGGTFREYAISWVRQAPGQGLEWMGGIIPVFGTANYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-17
SEQ ID NO: 47
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-17
SEQ ID NO: 48
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGGGAGTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCGTGTTCGGCACCGCCAACTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-22
SEQ ID NO: 49
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-22
SEQ ID NO: 50
QVQLVQSGAEVKKPGASVKVSCKASGYTFDSYAMHWVRQAPGQGLEWMGGIIPIFGTAWYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-22
SEQ ID NO: 51
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-22
SEQ ID NO: 52
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCGCCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCTACACCTTCGACAGCTACGCCATGCACTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTTTCGGCACCGCCTGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-47
SEQ ID NO: 53
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-47
SEQ ID NO: 54
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSQYAIHWVRQAPGQGLEWMGVIIPIFGKANYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-47
SEQ ID NO: 55
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-47
SEQ ID NO: 56
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCCAGTACGCCATCCACTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGTGATCATCCCCATTTTCGGCAAGGCCAACTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-2B04-46
SEQ ID NO: 57
EIVLTQSPATLSLSPGERATLSCRASQSVSSYLAWYQQKPGQAPRLLIYDASKRATGIPARFSGSGSGTDFTLTISSLEPEDFAVYYCQQLDNWPITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-2B04-46
SEQ ID NO: 58
QVQLQQWGAGLLKPSETLSLTCAVYGGSFRRYYWSWIRQPPGKGLEWIGEIDGWGSTNYNPSLKSRVTISVDTSKNQFSLKLSSVTAADTAVYYCARGGSVDFWSGSDYYYYMDVWGKGATVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-2B04-46
SEQ ID NO: 59
GAGATCGTGCTGACCCAGAGCCCCGCAACCCTGTCCCTGAGCCCCGGCGAAAGGGCCACTCTGAGCTGCAGGGCCAGCCAGAGCGTGAGCAGCTACCTCGCCTGGTACCAGCAGAAGCCCGGCCAGGCCCCTAGGCTGCTGATCTACGACGCCAGCAAGAGGGCCACCGGCATTCCCGCCAGGTTCAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTGGAGCCCGAGGACTTCGCCGTCTACTACTGCCAGCAGCTGGACAACTGGCCCATCACCTTCGGGGGCGGCACCAAGGTGGAGATCAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-2B04-46
SEQ ID NO: 60
CAGGTGCAGCTGCAGCAGTGGGGCGCCGGACTGCTGAAGCCCAGCGAGACCCTGAGCCTGACCTGCGCCGTGTACGGCGGGTCCTTCAGGAGGTACTACTGGAGCTGGATCAGGCAGCCCCCCGGCAAAGGCCTGGAGTGGATCGGCGAGATCGACGGCTGGGGCAGCACCAACTACAACCCCAGCCTCAAGAGCAGGGTGACCATCAGCGTGGACACCAGCAAGAACCAGTTCAGCCTGAAGCTGAGCAGCGTGACCGCCGCCGACACCGCCGTGTACTATTGCGCCAGGGGCGGCAGCGTGGACTTCTGGAGCGGCAGCGACTACTACTACTACATGGACGTGTGGGGCAAGGGCGCCACCGTCACCGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-1A01-85
SEQ ID NO: 61
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSLYPPRTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-1A01-85
SEQ ID NO: 62
QVQLVQSGAEVKKPGASVKVSCKASGYTFTSYPMHWVRQAPGQGLEWMGIINPSGGFTSYAQKFQGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCARETAYYTTKGNWFDPWGQGTLVTVSA
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-1A01-85
SEQ ID NO: 63
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGAGCCTGTACCCCCCCAGGACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-1A01-85
SEQ ID NO: 64
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAAGTGAAAAAGCCCGGCGCCAGCGTGAAGGTCAGCTGCAAGGCCTCCGGGTACACCTTCACCAGCTACCCCATGCACTGGGTGAGGCAGGCCCCCGGCCAGGGCCTCGAGTGGATGGGCATCATCAACCCCAGCGGAGGCTTCACCAGCTACGCCCAGAAGTTCCAGGGCAGGGTGACCATGACAAGGGACACCAGCACCAGCACCGTGTACATGGAGCTGAGCAGCCTGAGGAGCGAGGACACCGCCGTGTATTACTGCGCAAGGGAGACCGCCTACTACACCACCAAGGGCAACTGGTTCGACCCCTGGGGCCAGGGCACCCTGGTGACCGTGAGCGCC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-1A01-97
SEQ ID NO: 65
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSLYPPRTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-1A01-97
SEQ ID NO: 66
QVQLVQSGAEVKKPGASVKVSCKASGYTFTRAAMHWVRQAPGQGLEWMGIINPAGGYTSYAQKFQGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCARETAYYTTKGNWFDPWGQGTLVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-1A01-97
SEQ ID NO: 67
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGAGCCTGTACCCCCCCAGGACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-1A01-97
SEQ ID NO: 68
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAAGTGAAAAAGCCCGGCGCCAGCGTGAAGGTCAGCTGCAAGGCCTCCGGGTACACCTTCACCAGGGCCGCCATGCACTGGGTGAGGCAGGCCCCCGGCCAGGGCCTCGAGTGGATGGGCATCATCAACCCCGCCGGAGGCTACACCAGCTACGCCCAGAAGTTCCAGGGCAGGGTGACCATGACAAGGGACACCAGCACCAGCACCGTGTACATGGAGCTGAGCAGCCTGAGGAGCGAGGACACCGCCGTGTATTACTGCGCAAGGGAGACCGCCTACTACACCACCAAGGGCAACTGGTTCGACCCCTGGGGCCAGGGCACCCTGGTGACCGTGAGCTCT
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-1A01-100
SEQ ID NO: 69
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSLYPPRTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-1A01-100
SEQ ID NO: 70
QVQLVQSGAEVKKPGASVKVSCKASGYTFTTYRMHWVRQAPGQGLEWMGIINPSGGLTQYAQKFQGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCARETAYYTTKGNWFDPWGQGTLVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-1A01-100
SEQ ID NO: 71
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGAGCCTGTACCCCCCCAGGACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-1A01-100
SEQ ID NO: 72
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAAGTGAAAAAGCCCGGCGCCAGCGTGAAGGTCAGCTGCAAGGCCTCCGGGTACACCTTCACCACCTACAGGATGCACTGGGTGAGGCAGGCCCCCGGCCAGGGCCTCGAGTGGATGGGCATCATCAACCCCAGCGGAGGCCTGACCCAGTACGCCCAGAAGTTCCAGGGCAGGGTGACCATGACAAGGGACACCAGCACCAGCACCGTGTACATGGAGCTGAGCAGCCTGAGGAGCGAGGACACCGCCGTGTATTACTGCGCAAGGGAGACCGCCTACTACACCACCAAGGGCAACTGGTTCGACCCCTGGGGCCAGGGCACCCTGGTGACCGTGAGCTCT
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-1A01-103
SEQ ID NO: 73
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSLYPPRTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-1A01-103
SEQ ID NO: 74
QVQLVQSGAEVKKPGASVKVSCKASGYTFTAYQMHWVRQAPGQGLEWMGIINPAGGWTSYAQKFQGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCARETAYYTTKGNWFDPWGQGTLVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-1A01-103
SEQ ID NO: 75
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGAGCCTGTACCCCCCCAGGACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-1A01-103
SEQ ID NO: 76
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAAGTGAAAAAGCCCGGCGCCAGCGTGAAGGTCAGCTGCAAGGCCTCCGGGTACACCTTCACCGCCTACCAGATGCACTGGGTGAGGCAGGCCCCCGGCCAGGGCCTCGAGTGGATGGGCATCATCAACCCCGCCGGAGGCTGGACCAGCTACGCCCAGAAGTTCCAGGGCAGGGTGACCATGACAAGGGACACCAGCACCAGCACCGTGTACATGGAGCTGAGCAGCCTGAGGAGCGAGGACACCGCCGTGTATTACTGCGCAAGGGAGACCGCCTACTACACCACCAAGGGCAACTGGTTCGACCCCTGGGGCCAGGGCACCCTGGTGACCGTGAGCTCT
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-1A01-126
SEQ ID NO: 77
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSLYPPRTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-1A01-126
SEQ ID NO: 78
QVQLVQSGAEVKKPGASVKVSCKASGYTFTRYNMHWVRQAPGQGLEWMGWINPAGGSTSYAQKFQGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCARETAYYTTKGNWFDPWGQGTLVTVSA
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-1A01-126
SEQ ID NO: 79
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGAGCCTGTACCCCCCCAGGACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-1A01-126
SEQ ID NO: 80
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAAGTGAAAAAGCCCGGCGCCAGCGTGAAGGTCAGCTGCAAGGCCTCCGGGTACACCTTCACCAGGTACAACATGCACTGGGTGAGGCAGGCCCCCGGCCAGGGCCTCGAGTGGATGGGCTGGATCAACCCCGCCGGAGGCAGCACCAGCTACGCCCAGAAGTTCCAGGGCAGGGTGACCATGACAAGGGACACCAGCACCAGCACCGTGTACATGGAGCTGAGCAGCCTGAGGAGCGAGGACACCGCCGTGTATTACTGCGCAAGGGAGACCGCCTACTACACCACCAAGGGCAACTGGTTCGACCCCTGGGGCCAGGGCACCCTGGTGACCGTGAGCGCC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-1A01-191
SEQ ID NO: 81
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSLYPPRTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-1A01-191
SEQ ID NO: 82
QVQLVQSGAEVKKPGASVKVSCKASGYTFTKYRMHWVRQAPGQGLEWMGIINPQGGSTSYAQKFQGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCARETAYYTTKGNWFDPWGQGTLVTVSA
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-1A01-191
SEQ ID NO: 83
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGAGCCTGTACCCCCCCAGGACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-1A01-191
SEQ ID NO: 84
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAAGTGAAAAAGCCCGGCGCCAGCGTGAAGGTCAGCTGCAAGGCCTCCGGGTACACCTTCACCAAGTACAGGATGCACTGGGTGAGGCAGGCCCCCGGCCAGGGCCTCGAGTGGATGGGCATCATCAACCCCCAGGGAGGCAGCACCAGCTACGCCCAGAAGTTCCAGGGCAGGGTGACCATGACAAGGGACACCAGCACCAGCACCGTGTACATGGAGCTGAGCAGCCTGAGGAGCGAGGACACCGCCGTGTATTACTGCGCAAGGGAGACCGCCTACTACACCACCAAGGGCAACTGGTTCGACCCCTGGGGCCAGGGCACCCTGGTGACCGTGAGCGCC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-66
SEQ ID NO: 85
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-66
SEQ ID NO: 86
QVQLVQSGAEVKKPGSSVKVSCKASGGTFVEYAISWVRQAPGQGLEWMGGIIPAFGTAQYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-66
SEQ ID NO: 87
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-66
SEQ ID NO: 88
CAGGTGCAGCTGGTGCAGAGCGGCGCAGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCGTGGAGTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCGCCTTCGGCACCGCCCAGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGAGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCAC CGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F08-16
SEQ ID NO: 89
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F08-16
SEQ ID NO: 90
QVQLVQSGAEVKKPGSSVKVSCKASGGTFNEYAISWVRQAPGQGLEWMGGIVPVFGTAKYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F08-16
SEQ ID NO: 91
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F08-16
SEQ ID NO: 92
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAACGAGTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCGTGCCCGTGTTCGGCACCGCCAAGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
Аминокислотная последовательность вариабельной области легкой цепи 42Y073-86F04-23
SEQ ID NO: 93
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIK
Аминокислотная последовательность вариабельной области тяжелой цепи 42Y073-86F04-23
SEQ ID NO: 94
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSGYPISWVRQAPGQGLEWMGGIIPIMGTARYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSS
Нуклеотидная последовательность вариабельной области легкой цепи 42Y073-86F04-23
SEQ ID NO: 95
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAG
Нуклеотидная последовательность вариабельной области тяжелой цепи 42Y073-86F04-23
SEQ ID NO: 96
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCGGCTACCCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTATGGGCACCGCCAGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGC
CDRL1, аминокислотная последовательность 1
SEQ ID NO: 97
RASQSISSYLN
CDRL1, аминокислотная последовательность 2
SEQ ID NO: 98
RASQSVSSYLA
CDRL2, аминокислотная последовательность 1
SEQ ID NO: 99
AASSLQS
CDRL2, аминокислотная последовательность 2
SEQ ID NO: 100
DASKRAT
CDRL3, аминокислотная последовательность 1
SEQ ID NO: 101
QQVLHTIT
CDRL3, аминокислотная последовательность 2
SEQ ID NO: 102
QQPYFSPPT
CDRL3, аминокислотная последовательность 3
SEQ ID NO: 103
QQLDNWPIT
CDRL3, аминокислотная последовательность 4
SEQ ID NO: 104
QQSLYPPRT
CDRH1, аминокислотная последовательность 1
SEQ ID NO: 105
SYAIS
CDRH1, аминокислотная последовательность 2
SEQ ID NO: 106
YNAIS
CDRH1, аминокислотная последовательность 3
SEQ ID NO: 107
SEAIS
CDRH1, аминокислотная последовательность 4
SEQ ID NO: 108
SHAIS
CDRH1, аминокислотная последовательность 5
SEQ ID NO: 109
GHAIS
CDRH1, аминокислотная последовательность 6
SEQ ID NO: 110
SRAIS
CDRH1, аминокислотная последовательность 7
SEQ ID NO: 111
SSAIS
CDRH1, аминокислотная последовательность 8
SEQ ID NO: 112
NYAIS
CDRH1, аминокислотная последовательность 9
SEQ ID NO: 113
EYAIH
CDRH1, аминокислотная последовательность 10
SEQ ID NO: 114
LYAIS
CDRH1, аминокислотная последовательность 11
SEQ ID NO: 115
EYAIS
CDRH1, аминокислотная последовательность 12
SEQ ID NO: 116
SYAMH
CDRH1, аминокислотная последовательность 13
SEQ ID NO: 117
QYAIH
CDRH1, аминокислотная последовательность 14
SEQ ID NO: 118
RYYWS
CDRH1, аминокислотная последовательность 15
SEQ ID NO: 119
SYPMH
CDRH1, аминокислотная последовательность 16
SEQ ID NO: 120
RAAMH
CDRH1, аминокислотная последовательность 17
SEQ ID NO: 121
TYRMH
CDRH1, аминокислотная последовательность 18
SEQ ID NO: 122
AYQMH
CDRH1, аминокислотная последовательность 19
SEQ ID NO: 123
RYNMH
CDRH1, аминокислотная последовательность 20
SEQ ID NO: 124
KYRMH
CDRH1, аминокислотная последовательность 21
SEQ ID NO: 125
GYPIS
CDRH2, аминокислотная последовательность 1
SEQ ID NO: 126
GIIPIFGTASYAQKFQG
CDRH2, аминокислотная последовательность 2
SEQ ID NO: 127
GIIPIMGTARYAQKFQG
CDRH2, аминокислотная последовательность 3
SEQ ID NO: 128
GIIPIFGRARYAQKFQG
CDRH2, аминокислотная последовательность 4
SEQ ID NO: 129
GIIPIFGRGKYAQKFQG
CDRH2, аминокислотная последовательность 5
SEQ ID NO: 130
GILPIFGRANYAQKFQG
CDRH2, аминокислотная последовательность 6
SEQ ID NO: 131
GIIPIFGRANYAQKFQG
CDRH2, аминокислотная последовательность 7
SEQ ID NO: 132
GIIPALGTANYAQKFQG
CDRH2, аминокислотная последовательность 8
SEQ ID NO: 133
NIIPIFGTAGYAQKFQG
CDRH2, аминокислотная последовательность 9
SEQ ID NO: 134
GIIPQLGTANYAQKFQG
CDRH2, аминокислотная последовательность 10
SEQ ID NO: 135
GIIPVFGTANYAQKFQG
CDRH2, аминокислотная последовательность 11
SEQ ID NO: 136
GIIPIFGTAWYAQKFQG
CDRH2, аминокислотная последовательность 12
SEQ ID NO: 137
VIIPIFGKANYAQKFQG
CDRH2, аминокислотная последовательность 13
SEQ ID NO: 138
EIDGWGSTNYNPSLKS
CDRH2, аминокислотная последовательность 14
SEQ ID NO: 139
IINPSGGFTSYAQKFQG
CDRH2, аминокислотная последовательность 15
SEQ ID NO: 140
IINPAGGYTSYAQKFQG
CDRH2, аминокислотная последовательность 16
SEQ ID NO: 141
IINPSGGLTQYAQKFQG
CDRH2, аминокислотная последовательность 17
SEQ ID NO: 142
IINPAGGWTSYAQKFQG
CDRH2, аминокислотная последовательность 18
SEQ ID NO: 143
WINPAGGSTSYAQKFQG
CDRH2, аминокислотная последовательность 19
SEQ ID NO: 144
IINPQGGSTSYAQKFQG
CDRH2, аминокислотная последовательность 20
SEQ ID NO: 145
GIIPAFGTAQYAQKFQG
CDRH2, аминокислотная последовательность 21
SEQ ID NO: 146
GIVPVFGTAKYAQKFQG
CDRH3, аминокислотная последовательность 1
SEQ ID NO: 147
GAGYYGDKDPMDV
CDRH3, аминокислотная последовательность 2
SEQ ID NO: 148
LLGESGMDV
CDRH3, аминокислотная последовательность 3
SEQ ID NO: 149
GGSVDFWSGSDYYYYMDV
CDRH3, аминокислотная последовательность 4
SEQ ID NO: 150
ETAYYTTKGNWFDP
CDRH1 антагониста PD-1
SEQ ID NO: 151
SYDMS
CDRH2 антагониста PD-1
SEQ ID NO: 152
TISGGGSYTYYQDSVKG
CDRH3 антагониста PD-1
SEQ ID NO: 153
PYYAMDY
CDRL1 антагониста PD-1
SEQ ID NO: 154
KASQDVGTAVA
CDRL2 антагониста PD-1
SEQ ID NO: 155
WASTLHT
CDRL3 антагониста PD-1
SEQ ID NO: 156
QHYSSYPWT
Альтернативная CDRL3 антагониста PD-1
SEQ ID NO: 157
QHYNSYPWT
Вариабельная область тяжелой цепи антагониста PD-1
SEQ ID NO: 158
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSS
Вариабельная область легкой цепи антагониста PD-1
SEQ ID NO: 159
DIQLTQSPSFLSAYVGDRVTITCKASQDVGTAVAWYQQKPGKAPKLLIYWASTLHTGVPSRFSGSGSGTEFTLTISSLQPEDFATYYCQHYSSYPWTFGQGTKLEIK
Тяжелая цепь моноклонального антитела - антагониста PD-1
SEQ ID NO: 160
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
Легкая цепь моноклонального антитела - антагониста PD-1
SEQ ID NO: 161
DIQLTQSPSFLSAYVGDRVTITCKASQDVGTAVAWYQQKPGKAPKLLIYWASTLHTGVPSRFSGSGSGTEFTLTISSLQPEDFATYYCQHYSSYPWTFGQGTKLEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность тяжелой цепи антагониста PD-1 с модификацией N380D
SEQ NO: 162
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESDGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
Последовательность легкой цепи антагониста PD-1 с модификацией N385D
SEQ NO: 163
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPEDNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
Последовательность тяжелой цепи антагониста PD-1 с модификациями N380D и N385D
SEQ NO: 164
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYDMSWVRQAPGKGLEWVSTISGGGSYTYYQDSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCASPYYAMDYWGQGTTVTVSSASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESDGQPEDNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
Аминокислотная последовательность полноразмерной тяжелой цепи 42Y073-86F08-66
SEQ ID NO: 165
QVQLVQSGAEVKKPGSSVKVSCKASGGTFVEYAISWVRQAPGQGLEWMGGIIPAFGTAQY
AQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTV
SSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQ
SSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELL
GGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQ
YNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSR
DELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKS
RWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Аминокислотная последовательность полноразмерной легкой цепи 42Y073-86F08-66
SEQ ID NO: 166
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPS
RFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIKRTVAAPSVFIFPPS
DEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTL
SKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Последовательность ДНК полноразмерной тяжелой цепи 42Y073-86F08-66
SEQ ID NO: 167
CAGGTGCAGCTGGTGCAGAGCGGCGCAGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCGTGGAGTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCGCCTTCGGCACCGCCCAGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGAGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGCGCCAGCACCAAGGGCCCCAGCGTGTTCCCCCTGGCCCCCAGCAGCAAGAGCACCAGCGGCGGCACAGCCGCCCTGGGCTGCCTGGTGAAGGACTACTTCCCCGAGCCCGTGACCGTGTCCTGGAACAGCGGAGCCCTGACCAGCGGCGTGCACACCTTCCCCGCCGTGCTGCAGAGCAGCGGCCTGTACAGCCTGAGCAGCGTGGTGACCGTGCCCAGCAGCAGCCTGGGCACCCAGACCTACATCTGTAACGTGAACCACAAGCCCAGCAACACCAAGGTGGACAAGAAGGTGGAGCCCAAGAGCTGTGACAAGACCCACACCTGCCCCCCCTGCCCTGCCCCCGAGCTGCTGGGAGGCCCCAGCGTGTTCCTGTTCCCCCCCAAGCCTAAGGACACCCTGATGATCAGCAGAACCCCCGAGGTGACCTGTGTGGTGGTGGATGTGAGCCACGAGGACCCTGAGGTGAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCACAATGCCAAGACCAAGCCCAGGGAGGAGCAGTACAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAACGGCAAGGAGTACAAGTGTAAGGTGTCCAACAAGGCCCTGCCTGCCCCTATCGAGAAAACCATCAGCAAGGCCAAGGGCCAGCCCAGAGAGCCCCAGGTGTACACCCTGCCCCCTAGCAGAGATGAGCTGACCAAGAACCAGGTGTCCCTGACCTGCCTGGTGAAGGGCTTCTACCCCAGCGACATCGCCGTGGAGTGGGAGAGCAACGGCCAGCCCGAGAACAACTACAAGACCACCCCCCCTGTGCTGGACAGCGATGGCAGCTTCTTCCTGTACAGCAAGCTGACCGTGGACAAGAGCAGATGGCAGCAGGGCAACGTGTTCAGCTGCTCCGTGATGCACGAGGCCCTGCACAATCACTACACCCAGAAGAGCCTGAGCCTGTCCCCTGGCAAG
Последовательность ДНК полноразмерной легкой цепи 42Y073-86F08-66
SEQ ID NO: 168
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAGCGTACGGTGGCCGCCCCCAGCGTGTTCATCTTCCCCCCCAGCGATGAGCAGCTGAAGAGCGGCACCGCCAGCGTGGTGTGTCTGCTGAACAACTTCTACCCCCGGGAGGCCAAGGTGCAGTGGAAGGTGGACAATGCCCTGCAGAGCGGCAACAGCCAGGAGAGCGTGACCGAGCAGGACAGCAAGGACTCCACCTACAGCCTGAGCAGCACCCTGACCCTGAGCAAGGCCGACTACGAGAAGCACAAGGTGTACGCCTGTGAGGTGACCCACCAGGGCCTGTCCAGCCCCGTGACCAAGAGCTTCAACCGGGGCGAGTGC
Аминокислотная последовательность полноразмерной легкой цепи 42Y073-86F08-16
SEQ ID NO: 169
DIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQVLHTITFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Аминокислотная последовательность полноразмерной тяжелой цепи 42Y073-86F08-16
SEQ ID NO: 170
QVQLVQSGAEVKKPGSSVKVSCKASGGTFNEYAISWVRQAPGQGLEWMGGIVPVFGTAKYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARGAGYYGDKDPMDVWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность ДНК полноразмерной легкой цепи 42Y073-86F08-16
SEQ ID NO: 171
GACATCCAGCTGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGGTGCTGCACACCATCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAGCGTACGGTGGCCGCCCCCAGCGTGTTCATCTTCCCCCCCAGCGATGAGCAGCTGAAGAGCGGCACCGCCAGCGTGGTGTGTCTGCTGAACAACTTCTACCCCCGGGAGGCCAAGGTGCAGTGGAAGGTGGACAATGCCCTGCAGAGCGGCAACAGCCAGGAGAGCGTGACCGAGCAGGACAGCAAGGACTCCACCTACAGCCTGAGCAGCACCCTGACCCTGAGCAAGGCCGACTACGAGAAGCACAAGGTGTACGCCTGTGAGGTGACCCACCAGGGCCTGTCCAGCCCCGTGACCAAGAGCTTCAACCGGGGCGAGTGC
Последовательность ДНК полноразмерной тяжелой цепи 42Y073-86F08-16
SEQ ID NO: 172
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAACGAGTACGCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCGTGCCCGTGTTCGGCACCGCCAAGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGGGCGCCGGCTACTACGGCGACAAGGACCCCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGCGCCAGCACCAAGGGCCCCAGCGTGTTCCCCCTGGCCCCCAGCAGCAAGAGCACCAGCGGCGGCACAGCCGCCCTGGGCTGCCTGGTGAAGGACTACTTCCCCGAGCCCGTGACCGTGTCCTGGAACAGCGGAGCCCTGACCAGCGGCGTGCACACCTTCCCCGCCGTGCTGCAGAGCAGCGGCCTGTACAGCCTGAGCAGCGTGGTGACCGTGCCCAGCAGCAGCCTGGGCACCCAGACCTACATCTGTAACGTGAACCACAAGCCCAGCAACACCAAGGTGGACAAGAAGGTGGAGCCCAAGAGCTGTGACAAGACCCACACCTGCCCCCCCTGCCCTGCCCCCGAGCTGCTGGGAGGCCCCAGCGTGTTCCTGTTCCCCCCCAAGCCTAAGGACACCCTGATGATCAGCAGAACCCCCGAGGTGACCTGTGTGGTGGTGGATGTGAGCCACGAGGACCCTGAGGTGAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCACAATGCCAAGACCAAGCCCAGGGAGGAGCAGTACAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAACGGCAAGGAGTACAAGTGTAAGGTGTCCAACAAGGCCCTGCCTGCCCCTATCGAGAAAACCATCAGCAAGGCCAAGGGCCAGCCCAGAGAGCCCCAGGTGTACACCCTGCCCCCTAGCAGAGATGAGCTGACCAAGAACCAGGTGTCCCTGACCTGCCTGGTGAAGGGCTTCTACCCCAGCGACATCGCCGTGGAGTGGGAGAGCAACGGCCAGCCCGAGAACAACTACAAGACCACCCCCCCTGTGCTGGACAGCGATGGCAGCTTCTTCCTGTACAGCAAGCTGACCGTGGACAAGAGCAGATGGCAGCAGGGCAACGTGTTCAGCTGCTCCGTGATGCACGAGGCCCTGCACAATCACTACACCCAGAAGAGCCTGAGCCTGTCCCCTGGCAAG
Аминокислотная последовательность полноразмерной легкой цепи 42Y073-86F04-23
SEQ ID NO: 173
DIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQPYFSPPTFGGGTKVEIKRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC
Аминокислотная последовательность полноразмерной тяжелой цепи 42Y073-86F04-23
SEQ ID NO: 174
QVQLVQSGAEVKKPGSSVKVSCKASGGTFSGYPISWVRQAPGQGLEWMGGIIPIMGTARYAQKFQGRVTITADESTSTAYMELSSLRSEDTAVYYCARLLGESGMDVWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Последовательность ДНК полноразмерной легкой цепи 42Y073-86F04-23
SEQ ID NO: 175
GACATCCAGATGACCCAGAGCCCTAGCAGCCTGAGCGCCAGCGTGGGAGACAGGGTGACCATCACCTGCAGGGCCAGCCAGTCCATCAGCAGCTACCTGAACTGGTACCAGCAGAAGCCCGGCAAGGCCCCCAAGCTGCTGATCTACGCCGCAAGCTCACTGCAGAGCGGCGTGCCCTCTAGGTTTAGCGGCAGCGGCAGCGGCACCGACTTCACCCTGACCATCAGCAGCCTCCAGCCCGAGGACTTCGCCACCTACTACTGCCAGCAGCCCTACTTCAGCCCCCCCACTTTCGGCGGCGGCACCAAGGTGGAGATTAAGCGTACGGTGGCCGCCCCCAGCGTGTTCATCTTCCCCCCCAGCGATGAGCAGCTGAAGAGCGGCACCGCCAGCGTGGTGTGTCTGCTGAACAACTTCTACCCCCGGGAGGCCAAGGTGCAGTGGAAGGTGGACAATGCCCTGCAGAGCGGCAACAGCCAGGAGAGCGTGACCGAGCAGGACAGCAAGGACTCCACCTACAGCCTGAGCAGCACCCTGACCCTGAGCAAGGCCGACTACGAGAAGCACAAGGTGTACGCCTGTGAGGTGACCCACCAGGGCCTGTCCAGCCCCGTGACCAAGAGCTTCAACCGGGGCGAGTGC
Последовательность ДНК полноразмерной тяжелой цепи 42Y073-86F04-23
SEQ ID NO: 176
CAGGTGCAGCTGGTGCAGAGCGGCGCCGAGGTGAAAAAGCCCGGCAGCAGCGTGAAGGTGAGCTGCAAGGCCTCCGGCGGGACCTTCAGCGGCTACCCCATCAGCTGGGTGAGGCAGGCTCCCGGACAGGGCCTGGAGTGGATGGGCGGCATCATCCCCATTATGGGCACCGCCAGGTACGCCCAGAAGTTCCAGGGAAGGGTCACCATCACCGCCGACGAGAGCACCAGCACCGCCTACATGGAACTCAGCAGCCTGAGGAGCGAGGACACCGCCGTGTACTATTGCGCCAGGCTGCTGGGCGAGAGCGGCATGGACGTGTGGGGCCAGGGCACCACCGTGACTGTGAGCAGCGCCAGCACCAAGGGCCCCAGCGTGTTCCCCCTGGCCCCCAGCAGCAAGAGCACCAGCGGCGGCACAGCCGCCCTGGGCTGCCTGGTGAAGGACTACTTCCCCGAGCCCGTGACCGTGTCCTGGAACAGCGGAGCCCTGACCAGCGGCGTGCACACCTTCCCCGCCGTGCTGCAGAGCAGCGGCCTGTACAGCCTGAGCAGCGTGGTGACCGTGCCCAGCAGCAGCCTGGGCACCCAGACCTACATCTGTAACGTGAACCACAAGCCCAGCAACACCAAGGTGGACAAGAAGGTGGAGCCCAAGAGCTGTGACAAGACCCACACCTGCCCCCCCTGCCCTGCCCCCGAGCTGCTGGGAGGCCCCAGCGTGTTCCTGTTCCCCCCCAAGCCTAAGGACACCCTGATGATCAGCAGAACCCCCGAGGTGACCTGTGTGGTGGTGGATGTGAGCCACGAGGACCCTGAGGTGAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCACAATGCCAAGACCAAGCCCAGGGAGGAGCAGTACAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAACGGCAAGGAGTACAAGTGTAAGGTGTCCAACAAGGCCCTGCCTGCCCCTATCGAGAAAACCATCAGCAAGGCCAAGGGCCAGCCCAGAGAGCCCCAGGTGTACACCCTGCCCCCTAGCAGAGATGAGCTGACCAAGAACCAGGTGTCCCTGACCTGCCTGGTGAAGGGCTTCTACCCCAGCGACATCGCCGTGGAGTGGGAGAGCAACGGCCAGCCCGAGAACAACTACAAGACCACCCCCCCTGTGCTGGACAGCGATGGCAGCTTCTTCCTGTACAGCAAGCTGACCGTGGACAAGAGCAGATGGCAGCAGGGCAACGTGTTCAGCTGCTCCGTGATGCACGAGGCCCTGCACAATCACTACACCCAGAAGAGCCTGAGCCTGTCCCCTGGCAAG
Аминокислотная последовательность CD155-fc, используемого в примерах (последовательность, выделенная жирным шрифтом, представляет собой последовательность зрелого CD155 ECD (внеклеточного домена) без последовательности лидера, остальное представляет собой Fc-область IgG1 человека)
SEQ ID NO: 177
WPPPGTGDVVVQAPTQVPGFLGDSVTLPCYLQVPNMEVTHVSQLTWARHGESGSMAVFHQTQGPSYSESKRLEFVAARLGAELRNASLRMFGLRVEDEGNYTCLFVTFPQGSRSVDIWLRVLAKPQNTAEVQKVQLTGEPVPMARCVSTGGRPPAQITWHSDLGGMPNTSQVPGFLSGTVTVTSLWILVPSSQVDGKNVTCKVEHESFEKPQLLTVNLTVYYPPEVSISGYDNNWYLGQNEATLTCDARSNPEPTGYNWSTTMGPLPPFAVAQGAQLLIRPVDKPINTTLICNVTNALGARQAELTVQVKEGPPSEHSGISRNSGENLYFQGDPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Аминокислотная последовательность CD155 (природная последовательность лидера подчеркнута, трансмембранный и цитоплазматический фрагменты выделены жирным шрифтом)
SEQ ID NO: 178
MARAMAAAWPLLLVALLVLSWPPPGTGDVVVQAPTQVPGFLGDSVTLPCYLQVPNMEVTHVSQLTWARHGESGSMAVFHQTQGPSYSESKRLEFVAARLGAELRNASLRMFGLRVEDEGNYTCLFVTFPQGSRSVDIWLRVLAKPQNTAEVQKVQLTGEPVPMARCVSTGGRPPAQITWHSDLGGMPNTSQVPGFLSGTVTVTSLWILVPSSQVDGKNVTCKVEHESFEKPQLLTVNLTVYYPPEVSISGYDNNWYLGQNEATLTCDARSNPEPTGYNWSTTMGPLPPFAVAQGAQLLIRPVDKPINTTLICNVTNALGARQAELTVQVKEGPPSEHSGISRNAIIFLVLGILVFLILLGIGIYFYWSKCSREVLWHCHLCPSSTEHASASANGHVSYSAVSRENSSSQDPQTEGTR
Аминокислотная последовательность Fc-области IgG1 человека
SEQ ID NO: 179
ASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK
Нуклеотидная последовательность Fc-области IgG1 человека
SEQ ID NO: 180
GCCAGCACCAAGGGCCCCAGCGTGTTCCCCCTGGCCCCCAGCAGCAAGAGCACCAGCGGCGGCACAGCCGCCCTGGGCTGCCTGGTGAAGGACTACTTCCCCGAGCCCGTGACCGTGTCCTGGAACAGCGGAGCCCTGACCAGCGGCGTGCACACCTTCCCCGCCGTGCTGCAGAGCAGCGGCCTGTACAGCCTGAGCAGCGTGGTGACCGTGCCCAGCAGCAGCCTGGGCACCCAGACCTACATCTGTAACGTGAACCACAAGCCCAGCAACACCAAGGTGGACAAGAAGGTGGAGCCCAAGAGCTGTGACAAGACCCACACCTGCCCCCCCTGCCCTGCCCCCGAGCTGCTGGGAGGCCCCAGCGTGTTCCTGTTCCCCCCCAAGCCTAAGGACACCCTGATGATCAGCAGAACCCCCGAGGTGACCTGTGTGGTGGTGGATGTGAGCCACGAGGACCCTGAGGTGAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCACAATGCCAAGACCAAGCCCAGGGAGGAGCAGTACAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAACGGCAAGGAGTACAAGTGTAAGGTGTCCAACAAGGCCCTGCCTGCCCCTATCGAGAAAACCATCAGCAAGGCCAAGGGCCAGCCCAGAGAGCCCCAGGTGTACACCCTGCCCCCTAGCAGAGATGAGCTGACCAAGAACCAGGTGTCCCTGACCTGCCTGGTGAAGGGCTTCTACCCCAGCGACATCGCCGTGGAGTGGGAGAGCAACGGCCAGCCCGAGAACAACTACAAGACCACCCCCCCTGTGCTGGACAGCGATGGCAGCTTCTTCCTGTACAGCAAGCTGACCGTGGACAAGAGCAGATGGCAGCAGGGCAACGTGTTCAGCTGCTCCGTGATGCACGAGGCCCTGCACAATCACTACACCCAGAAGAGCCTGAGCCTGTCCCCTGGCAAG
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110>GlaxoSmithKline Intellectual Property Development Limited
<120>АНТИГЕНСВЯЗЫВАЮЩИЕ БЕЛКИ
<130>PU66651
<150>US 63/057508
<151>2020-07-28
<150>US 62/906876
<151>2019-09-27
<160>180
<170>PatentIn version 3.5
<210>1
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-1
<400>1
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>2
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-1
<400>2
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>3
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-1
<400>3
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>4
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-1
<400>4
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc agctacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttttcggcac cgccagctac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>5
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-3
<400>5
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>6
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-3
<400>6
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Tyr Asn
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Met Gly Thr Ala Arg Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>7
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-3
<400>7
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>8
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-3
<400>8
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc tacaacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttatgggcac cgccaggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>9
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-4
<400>9
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>10
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-4
<400>10
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Glu Ser Glu
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Arg Ala Arg Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>11
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-4
<400>11
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>12
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-4
<400>12
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcgag agcgaggcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttttcggcag ggccaggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>13
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-5
<400>13
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>14
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-5
<400>14
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Arg Gly Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>15
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-5
<400>15
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>16
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-5
<400>16
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc agccacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttttcggcag gggcaagtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>17
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-6
<400>17
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>18
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-6
<400>18
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Gly His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Arg Ala Arg Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>19
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-6
<400>19
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>20
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-6
<400>20
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc ggccacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttttcggcag ggccaggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>21
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-18
<400>21
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>22
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-18
<400>22
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Arg
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Met Gly Thr Ala Arg Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>23
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-18
<400>23
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>24
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-18
<400>24
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc agcagggcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttatgggcac cgccaggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>25
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-33
<400>25
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>26
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-33
<400>26
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Leu Pro Ile Phe Gly Arg Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>27
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-33
<400>27
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>28
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-33
<400>28
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc agctacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcctgccca ttttcggcag ggccaactac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>29
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-88
<400>29
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>30
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-88
<400>30
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Arg Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>31
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-88
<400>31
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>32
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-88
<400>32
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc agcagcgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttttcggcag ggccaactac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>33
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-3
<400>33
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>34
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-3
<400>34
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Val Asn Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ala Leu Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>35
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-3
<400>35
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>36
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-3
<400>36
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcgtg aactacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatccccg ccctgggcac cgccaactac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>37
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-4
<400>37
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>38
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-4
<400>38
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Glu Tyr
20 25 30
Ala Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Ile Pro Ile Phe Gly Thr Ala Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>39
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-4
<400>39
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>40
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-4
<400>40
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc gagtacgcca tccactgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcaac atcatcccca ttttcggcac cgccggctac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>41
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-8
<400>41
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>42
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-8
<400>42
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Trp Leu Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Gln Leu Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>43
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-8
<400>43
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>44
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-8
<400>44
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttctgg ctgtacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatccccc agctgggcac cgccaactac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>45
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-17
<400>45
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>46
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-17
<400>46
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Arg Glu Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Val Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>47
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-17
<400>47
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>48
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-17
<400>48
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagg gagtacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatccccg tgttcggcac cgccaactac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>49
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-22
<400>49
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>50
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-22
<400>50
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Asp Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Trp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>51
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-22
<400>51
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>52
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-22
<400>52
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcgccag cgtgaaggtg 60
agctgcaagg cctccggcta caccttcgac agctacgcca tgcactgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttttcggcac cgcctggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>53
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-47
<400>53
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>54
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-47
<400>54
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Gln Tyr
20 25 30
Ala Ile His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Ile Pro Ile Phe Gly Lys Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>55
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-47
<400>55
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>56
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-47
<400>56
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc cagtacgcca tccactgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcgtg atcatcccca ttttcggcaa ggccaactac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>57
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-2B04-46
<400>57
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Lys Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Leu Asp Asn Trp Pro Ile
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>58
<211>126
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-2B04-46
<400>58
Gln Val Gln Leu Gln Gln Trp Gly Ala Gly Leu Leu Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Tyr Gly Gly Ser Phe Arg Arg Tyr
20 25 30
Tyr Trp Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asp Gly Trp Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Gly Gly Ser Val Asp Phe Trp Ser Gly Ser Asp Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Ala Thr Val Thr Val Ser Ser
115 120 125
<210>59
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-2B04-46
<400>59
gagatcgtgc tgacccagag ccccgcaacc ctgtccctga gccccggcga aagggccact 60
ctgagctgca gggccagcca gagcgtgagc agctacctcg cctggtacca gcagaagccc 120
ggccaggccc ctaggctgct gatctacgac gccagcaaga gggccaccgg cattcccgcc 180
aggttcagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctggagccc 240
gaggacttcg ccgtctacta ctgccagcag ctggacaact ggcccatcac cttcgggggc 300
ggcaccaagg tggagatcaa g 321
<210>60
<211>377
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-2B04-46
<400>60
caggtgcagc tgcagcagtg gggcgccgga ctgctgaagc ccagcgagac cctgagcctg 60
acctgcgccg tgtacggcgg gtccttcagg aggtactact ggagctggat caggcagccc 120
cccggcaaag gcctggagtg gatcggcgag atcgacggct ggggcagcac caactacaac 180
cccagcctca agagcagggt gaccatcagc gtggacacca gcaagaacca gttcagcctg 240
aagctgagca gcgtgaccgc cgccgacacc gccgtgtact attgcgccag gggcggcagc 300
gtggacttct ggagcggcag cgactactac tactacatgg acgtgtgggg caagggcgcc 360
accgtcaccg tgagcag 377
<210>61
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-1A01-85
<400>61
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Leu Tyr Pro Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>62
<211>123
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-1A01-85
<400>62
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Pro Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Phe Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
115 120
<210>63
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-1A01-85
<400>63
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag agcctgtacc cccccaggac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>64
<211>369
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-1A01-85
<400>64
caggtgcagc tggtgcagag cggcgccgaa gtgaaaaagc ccggcgccag cgtgaaggtc 60
agctgcaagg cctccgggta caccttcacc agctacccca tgcactgggt gaggcaggcc 120
cccggccagg gcctcgagtg gatgggcatc atcaacccca gcggaggctt caccagctac 180
gcccagaagt tccagggcag ggtgaccatg acaagggaca ccagcaccag caccgtgtac 240
atggagctga gcagcctgag gagcgaggac accgccgtgt attactgcgc aagggagacc 300
gcctactaca ccaccaaggg caactggttc gacccctggg gccagggcac cctggtgacc 360
gtgagcgcc 369
<210>65
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-1A01-97
<400>65
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Leu Tyr Pro Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>66
<211>123
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-1A01-97
<400>66
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Arg Ala
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ala Gly Gly Tyr Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210>67
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-1A01-97
<400>67
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag agcctgtacc cccccaggac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>68
<211>369
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-1A01-97
<400>68
caggtgcagc tggtgcagag cggcgccgaa gtgaaaaagc ccggcgccag cgtgaaggtc 60
agctgcaagg cctccgggta caccttcacc agggccgcca tgcactgggt gaggcaggcc 120
cccggccagg gcctcgagtg gatgggcatc atcaaccccg ccggaggcta caccagctac 180
gcccagaagt tccagggcag ggtgaccatg acaagggaca ccagcaccag caccgtgtac 240
atggagctga gcagcctgag gagcgaggac accgccgtgt attactgcgc aagggagacc 300
gcctactaca ccaccaaggg caactggttc gacccctggg gccagggcac cctggtgacc 360
gtgagctct 369
<210>69
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-1A01-100
<400>69
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Leu Tyr Pro Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>70
<211>123
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-1A01-100
<400>70
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Thr Tyr
20 25 30
Arg Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Leu Thr Gln Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210>71
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-1A01-100
<400>71
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag agcctgtacc cccccaggac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>72
<211>369
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-1A01-100
<400>72
caggtgcagc tggtgcagag cggcgccgaa gtgaaaaagc ccggcgccag cgtgaaggtc 60
agctgcaagg cctccgggta caccttcacc acctacagga tgcactgggt gaggcaggcc 120
cccggccagg gcctcgagtg gatgggcatc atcaacccca gcggaggcct gacccagtac 180
gcccagaagt tccagggcag ggtgaccatg acaagggaca ccagcaccag caccgtgtac 240
atggagctga gcagcctgag gagcgaggac accgccgtgt attactgcgc aagggagacc 300
gcctactaca ccaccaaggg caactggttc gacccctggg gccagggcac cctggtgacc 360
gtgagctct 369
<210>73
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-1A01-103
<400>73
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Leu Tyr Pro Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>74
<211>123
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-1A01-103
<400>74
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ala Tyr
20 25 30
Gln Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ala Gly Gly Trp Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210>75
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-1A01-103
<400>75
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag agcctgtacc cccccaggac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>76
<211>369
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-1A01-103
<400>76
caggtgcagc tggtgcagag cggcgccgaa gtgaaaaagc ccggcgccag cgtgaaggtc 60
agctgcaagg cctccgggta caccttcacc gcctaccaga tgcactgggt gaggcaggcc 120
cccggccagg gcctcgagtg gatgggcatc atcaaccccg ccggaggctg gaccagctac 180
gcccagaagt tccagggcag ggtgaccatg acaagggaca ccagcaccag caccgtgtac 240
atggagctga gcagcctgag gagcgaggac accgccgtgt attactgcgc aagggagacc 300
gcctactaca ccaccaaggg caactggttc gacccctggg gccagggcac cctggtgacc 360
gtgagctct 369
<210>77
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-1A01-126
<400>77
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Leu Tyr Pro Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>78
<211>123
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-1A01-126
<400>78
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Arg Tyr
20 25 30
Asn Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Trp Ile Asn Pro Ala Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
115 120
<210>79
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-1A01-126
<400>79
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag agcctgtacc cccccaggac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>80
<211>369
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-1A01-126
<400>80
caggtgcagc tggtgcagag cggcgccgaa gtgaaaaagc ccggcgccag cgtgaaggtc 60
agctgcaagg cctccgggta caccttcacc aggtacaaca tgcactgggt gaggcaggcc 120
cccggccagg gcctcgagtg gatgggctgg atcaaccccg ccggaggcag caccagctac 180
gcccagaagt tccagggcag ggtgaccatg acaagggaca ccagcaccag caccgtgtac 240
atggagctga gcagcctgag gagcgaggac accgccgtgt attactgcgc aagggagacc 300
gcctactaca ccaccaaggg caactggttc gacccctggg gccagggcac cctggtgacc 360
gtgagcgcc 369
<210>81
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-1A01-191
<400>81
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Leu Tyr Pro Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>82
<211>123
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-1A01-191
<400>82
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Lys Tyr
20 25 30
Arg Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Gln Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ala
115 120
<210>83
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-1A01-191
<400>83
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag agcctgtacc cccccaggac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>84
<211>369
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-1A01-191
<400>84
caggtgcagc tggtgcagag cggcgccgaa gtgaaaaagc ccggcgccag cgtgaaggtc 60
agctgcaagg cctccgggta caccttcacc aagtacagga tgcactgggt gaggcaggcc 120
cccggccagg gcctcgagtg gatgggcatc atcaaccccc agggaggcag caccagctac 180
gcccagaagt tccagggcag ggtgaccatg acaagggaca ccagcaccag caccgtgtac 240
atggagctga gcagcctgag gagcgaggac accgccgtgt attactgcgc aagggagacc 300
gcctactaca ccaccaaggg caactggttc gacccctggg gccagggcac cctggtgacc 360
gtgagcgcc 369
<210>85
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-66
<400>85
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>86
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-66
<400>86
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Val Glu Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ala Phe Gly Thr Ala Gln Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>87
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-66
<400>87
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>88
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-66
<400>88
caggtgcagc tggtgcagag cggcgcagag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcgtg gagtacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatccccg ccttcggcac cgcccagtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggagcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>89
<211>106
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F08-16
<400>89
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>90
<211>122
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F08-16
<400>90
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Asn Glu Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Val Pro Val Phe Gly Thr Ala Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210>91
<211>318
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F08-16
<400>91
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaag 318
<210>92
<211>366
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F08-16
<400>92
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcaac gagtacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcgtgcccg tgttcggcac cgccaagtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagc 366
<210>93
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области легкой цепи
42Y073-86F04-23
<400>93
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210>94
<211>118
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность вариабельной области тяжелой цепи
42Y073-86F04-23
<400>94
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Gly Tyr
20 25 30
Pro Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Met Gly Thr Ala Arg Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210>95
<211>321
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области легкой цепи
42Y073-86F04-23
<400>95
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa g 321
<210>96
<211>354
<212>ДНК
<213>Искусственная
<220>
<223>Нуклеотидная последовательность вариабельной области тяжелой цепи
42Y073-86F04-23
<400>96
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc ggctacccca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttatgggcac cgccaggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagc 354
<210>97
<211>11
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL1, аминокислотная последовательность 1
<400>97
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210>98
<211>11
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL1, аминокислотная последовательность 2
<400>98
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210>99
<211>7
<212>БЕЛОК
<213>Искусственная
<220>
<223>
CDRL2, аминокислотная последовательность 1
<400>99
Ala Ala Ser Ser Leu Gln Ser
1 5
<210>100
<211>7
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL2, аминокислотная последовательность 2
<400>100
Asp Ala Ser Lys Arg Ala Thr
1 5
<210>101
<211>8
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL3, аминокислотная последовательность 1
<400>101
Gln Gln Val Leu His Thr Ile Thr
1 5
<210>102
<211>9
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL3, аминокислотная последовательность 2
<400>102
Gln Gln Pro Tyr Phe Ser Pro Pro Thr
1 5
<210>103
<211>9
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL3, аминокислотная последовательность 3
<400>103
Gln Gln Leu Asp Asn Trp Pro Ile Thr
1 5
<210>104
<211>9
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL3, аминокислотная последовательность 4
<400>104
Gln Gln Ser Leu Tyr Pro Pro Arg Thr
1 5
<210>105
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 1
<400>105
Ser Tyr Ala Ile Ser
1 5
<210>106
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 2
<400>106
Tyr Asn Ala Ile Ser
1 5
<210>107
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 3
<400>107
Ser Glu Ala Ile Ser
1 5
<210>108
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 4
<400>108
Ser His Ala Ile Ser
1 5
<210>109
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 5
<400>109
Gly His Ala Ile Ser
1 5
<210>110
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 6
<400>110
Ser Arg Ala Ile Ser
1 5
<210>111
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 7
<400>111
Ser Ser Ala Ile Ser
1 5
<210>112
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 8
<400>112
Asn Tyr Ala Ile Ser
1 5
<210>113
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 9
<400>113
Glu Tyr Ala Ile His
1 5
<210>114
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 10
<400>114
Leu Tyr Ala Ile Ser
1 5
<210>115
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 11
<400>115
Glu Tyr Ala Ile Ser
1 5
<210>116
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 12
<400>116
Ser Tyr Ala Met His
1 5
<210>117
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 13
<400>117
Gln Tyr Ala Ile His
1 5
<210>118
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 14
<400>118
Arg Tyr Tyr Trp Ser
1 5
<210>119
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 15
<400>119
Ser Tyr Pro Met His
1 5
<210>120
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 16
<400>120
Arg Ala Ala Met His
1 5
<210>121
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 17
<400>121
Thr Tyr Arg Met His
1 5
<210>122
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 18
<400>122
Ala Tyr Gln Met His
1 5
<210>123
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 19
<400>123
Arg Tyr Asn Met His
1 5
<210>124
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 20
<400>124
Lys Tyr Arg Met His
1 5
<210>125
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1, аминокислотная последовательность 21
<400>125
Gly Tyr Pro Ile Ser
1 5
<210>126
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 1
<400>126
Gly Ile Ile Pro Ile Phe Gly Thr Ala Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>127
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 2
<400>127
Gly Ile Ile Pro Ile Met Gly Thr Ala Arg Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>128
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 3
<400>128
Gly Ile Ile Pro Ile Phe Gly Arg Ala Arg Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>129
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 4
<400>129
Gly Ile Ile Pro Ile Phe Gly Arg Gly Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>130
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 5
<400>130
Gly Ile Leu Pro Ile Phe Gly Arg Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>131
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 6
<400>131
Gly Ile Ile Pro Ile Phe Gly Arg Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>132
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 7
<400>132
Gly Ile Ile Pro Ala Leu Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>133
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 8
<400>133
Asn Ile Ile Pro Ile Phe Gly Thr Ala Gly Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>134
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 9
<400>134
Gly Ile Ile Pro Gln Leu Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>135
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 10
<400>135
Gly Ile Ile Pro Val Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>136
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 11
<400>136
Gly Ile Ile Pro Ile Phe Gly Thr Ala Trp Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>137
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 12
<400>137
Val Ile Ile Pro Ile Phe Gly Lys Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>138
<211>16
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 13
<400>138
Glu Ile Asp Gly Trp Gly Ser Thr Asn Tyr Asn Pro Ser Leu Lys Ser
1 5 10 15
<210>139
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 14
<400>139
Ile Ile Asn Pro Ser Gly Gly Phe Thr Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>140
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 15
<400>140
Ile Ile Asn Pro Ala Gly Gly Tyr Thr Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>141
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 16
<400>141
Ile Ile Asn Pro Ser Gly Gly Leu Thr Gln Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>142
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 17
<400>142
Ile Ile Asn Pro Ala Gly Gly Trp Thr Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>143
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 18
<400>143
Trp Ile Asn Pro Ala Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>144
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 19
<400>144
Ile Ile Asn Pro Gln Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>145
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 20
<400>145
Gly Ile Ile Pro Ala Phe Gly Thr Ala Gln Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>146
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2, аминокислотная последовательность 21
<400>146
Gly Ile Val Pro Val Phe Gly Thr Ala Lys Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210>147
<211>13
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH3, аминокислотная последовательность 1
<400>147
Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val
1 5 10
<210>148
<211>9
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH3, аминокислотная последовательность 2
<400>148
Leu Leu Gly Glu Ser Gly Met Asp Val
1 5
<210>149
<211>18
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH3, аминокислотная последовательность 3
<400>149
Gly Gly Ser Val Asp Phe Trp Ser Gly Ser Asp Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210>150
<211>14
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH3, аминокислотная последовательность 4
<400>150
Glu Thr Ala Tyr Tyr Thr Thr Lys Gly Asn Trp Phe Asp Pro
1 5 10
<210>151
<211>5
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH1 антагониста PD-1
<400>151
Ser Tyr Asp Met Ser
1 5
<210>152
<211>17
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH2 антагониста PD-1
<400>152
Thr Ile Ser Gly Gly Gly Ser Tyr Thr Tyr Tyr Gln Asp Ser Val Lys
1 5 10 15
Gly
<210>153
<211>7
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRH3 антагониста PD-1
<400>153
Pro Tyr Tyr Ala Met Asp Tyr
1 5
<210>154
<211>11
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL1 антагониста PD-1
<400>154
Lys Ala Ser Gln Asp Val Gly Thr Ala Val Ala
1 5 10
<210>155
<211>7
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL2 антагониста PD-1
<400>155
Trp Ala Ser Thr Leu His Thr
1 5
<210>156
<211>9
<212>БЕЛОК
<213>Искусственная
<220>
<223>CDRL3 антагониста PD-1
<400>156
Gln His Tyr Ser Ser Tyr Pro Trp Thr
1 5
<210>157
<211>9
<212>БЕЛОК
<213>Искусственная
<220>
<223>Альтернативная CDRL3 антагониста PD-1
<400>157
Gln His Tyr Asn Ser Tyr Pro Trp Thr
1 5
<210>158
<211>116
<212>БЕЛОК
<213>Искусственная
<220>
<223>Вариабельная область тяжелой цепи антагониста PD-1
<400>158
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Ser Tyr Thr Tyr Tyr Gln Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210>159
<211>107
<212>БЕЛОК
<213>Искусственная
<220>
<223>Вариабельная область легкой цепи антагониста PD-1
<400>159
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Tyr Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Trp Ala Ser Thr Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Tyr Ser Ser Tyr Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210>160
<211>443
<212>БЕЛОК
<213>Искусственная
<220>
<223>Тяжелая цепь моноклонального антитела - антагониста PD-1
<400>160
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Ser Tyr Thr Tyr Tyr Gln Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
340 345 350
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210>161
<211>214
<212>БЕЛОК
<213>Искусственная
<220>
<223>Легкая цепь моноклонального антитела - антагониста PD-1
<400>161
Asp Ile Gln Leu Thr Gln Ser Pro Ser Phe Leu Ser Ala Tyr Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Trp Ala Ser Thr Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Tyr Ser Ser Tyr Pro Trp
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210>162
<211>443
<212>БЕЛОК
<213>Искусственная
<220>
<223>Последовательность тяжелой цепи антагониста PD-1 с модификацией N380D
<400>162
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Ser Tyr Thr Tyr Tyr Gln Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
340 345 350
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asp Gly Gln Pro Glu
370 375 380
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210>163
<211>443
<212>БЕЛОК
<213>Искусственная
<220>
<223>Последовательность легкой цепи антагониста PD-1 с модификацией N385D
<400>163
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Ser Tyr Thr Tyr Tyr Gln Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
340 345 350
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
370 375 380
Asp Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210>164
<211>443
<212>БЕЛОК
<213>Искусственная
<220>
<223>Последовательность тяжелой цепи антагониста PD-1 с модификациями N380D и N385D
<400>164
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Asp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Thr Ile Ser Gly Gly Gly Ser Tyr Thr Tyr Tyr Gln Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro
210 215 220
Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
225 230 235 240
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
245 250 255
Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn
260 265 270
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
275 280 285
Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
290 295 300
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
305 310 315 320
Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys
325 330 335
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
340 345 350
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
355 360 365
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asp Gly Gln Pro Glu
370 375 380
Asp Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
385 390 395 400
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
405 410 415
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
420 425 430
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210>165
<211>452
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность полноразмерной тяжелой цепи
42Y073-86F08-66
<400>165
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Val Glu Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ala Phe Gly Thr Ala Gln Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210>166
<211>213
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность полноразмерной легкой цепи
42Y073-86F08-66
<400>166
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210>167
<211>1356
<212>ДНК
<213>Искусственная
<220>
<223>Последовательность ДНК полноразмерной тяжелой цепи 42Y073-86F08-66
<400>167
caggtgcagc tggtgcagag cggcgcagag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcgtg gagtacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatccccg ccttcggcac cgcccagtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggagcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagcgcca gcaccaaggg ccccagcgtg ttccccctgg cccccagcag caagagcacc 420
agcggcggca cagccgccct gggctgcctg gtgaaggact acttccccga gcccgtgacc 480
gtgtcctgga acagcggagc cctgaccagc ggcgtgcaca ccttccccgc cgtgctgcag 540
agcagcggcc tgtacagcct gagcagcgtg gtgaccgtgc ccagcagcag cctgggcacc 600
cagacctaca tctgtaacgt gaaccacaag cccagcaaca ccaaggtgga caagaaggtg 660
gagcccaaga gctgtgacaa gacccacacc tgccccccct gccctgcccc cgagctgctg 720
ggaggcccca gcgtgttcct gttccccccc aagcctaagg acaccctgat gatcagcaga 780
acccccgagg tgacctgtgt ggtggtggat gtgagccacg aggaccctga ggtgaagttc 840
aactggtacg tggacggcgt ggaggtgcac aatgccaaga ccaagcccag ggaggagcag 900
tacaacagca cctaccgggt ggtgtccgtg ctgaccgtgc tgcaccagga ttggctgaac 960
ggcaaggagt acaagtgtaa ggtgtccaac aaggccctgc ctgcccctat cgagaaaacc 1020
atcagcaagg ccaagggcca gcccagagag ccccaggtgt acaccctgcc ccctagcaga 1080
gatgagctga ccaagaacca ggtgtccctg acctgcctgg tgaagggctt ctaccccagc 1140
gacatcgccg tggagtggga gagcaacggc cagcccgaga acaactacaa gaccaccccc 1200
cctgtgctgg acagcgatgg cagcttcttc ctgtacagca agctgaccgt ggacaagagc 1260
agatggcagc agggcaacgt gttcagctgc tccgtgatgc acgaggccct gcacaatcac 1320
tacacccaga agagcctgag cctgtcccct ggcaag 1356
<210>168
<211>639
<212>ДНК
<213>Искусственная
<220>
<223>Последовательность ДНК полноразмерной легкой цепи 42Y073-86F08-66
<400>168
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaagcg tacggtggcc gcccccagcg tgttcatctt cccccccagc 360
gatgagcagc tgaagagcgg caccgccagc gtggtgtgtc tgctgaacaa cttctacccc 420
cgggaggcca aggtgcagtg gaaggtggac aatgccctgc agagcggcaa cagccaggag 480
agcgtgaccg agcaggacag caaggactcc acctacagcc tgagcagcac cctgaccctg 540
agcaaggccg actacgagaa gcacaaggtg tacgcctgtg aggtgaccca ccagggcctg 600
tccagccccg tgaccaagag cttcaaccgg ggcgagtgc 639
<210>169
<211>213
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность полноразмерной легкой цепи
42Y073-86F08-16
<400>169
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Leu His Thr Ile Thr
85 90 95
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210>170
<211>452
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность полноразмерной тяжелой цепи
42Y073-86F08-16
<400>170
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Asn Glu Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Val Pro Val Phe Gly Thr Ala Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Ala Gly Tyr Tyr Gly Asp Lys Asp Pro Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210>171
<211>639
<212>ДНК
<213>Искусственная
<220>
<223>Последовательность ДНК полноразмерной легкой цепи 42Y073-86F08-16
<400>171
gacatccagc tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag gtgctgcaca ccatcacttt cggcggcggc 300
accaaggtgg agattaagcg tacggtggcc gcccccagcg tgttcatctt cccccccagc 360
gatgagcagc tgaagagcgg caccgccagc gtggtgtgtc tgctgaacaa cttctacccc 420
cgggaggcca aggtgcagtg gaaggtggac aatgccctgc agagcggcaa cagccaggag 480
agcgtgaccg agcaggacag caaggactcc acctacagcc tgagcagcac cctgaccctg 540
agcaaggccg actacgagaa gcacaaggtg tacgcctgtg aggtgaccca ccagggcctg 600
tccagccccg tgaccaagag cttcaaccgg ggcgagtgc 639
<210>172
<211>1356
<212>ДНК
<213>Искусственная
<220>
<223>Последовательность ДНК полноразмерной тяжелой цепи 42Y073-86F08-16
<400>172
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcaac gagtacgcca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcgtgcccg tgttcggcac cgccaagtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggggcgcc 300
ggctactacg gcgacaagga ccccatggac gtgtggggcc agggcaccac cgtgactgtg 360
agcagcgcca gcaccaaggg ccccagcgtg ttccccctgg cccccagcag caagagcacc 420
agcggcggca cagccgccct gggctgcctg gtgaaggact acttccccga gcccgtgacc 480
gtgtcctgga acagcggagc cctgaccagc ggcgtgcaca ccttccccgc cgtgctgcag 540
agcagcggcc tgtacagcct gagcagcgtg gtgaccgtgc ccagcagcag cctgggcacc 600
cagacctaca tctgtaacgt gaaccacaag cccagcaaca ccaaggtgga caagaaggtg 660
gagcccaaga gctgtgacaa gacccacacc tgccccccct gccctgcccc cgagctgctg 720
ggaggcccca gcgtgttcct gttccccccc aagcctaagg acaccctgat gatcagcaga 780
acccccgagg tgacctgtgt ggtggtggat gtgagccacg aggaccctga ggtgaagttc 840
aactggtacg tggacggcgt ggaggtgcac aatgccaaga ccaagcccag ggaggagcag 900
tacaacagca cctaccgggt ggtgtccgtg ctgaccgtgc tgcaccagga ttggctgaac 960
ggcaaggagt acaagtgtaa ggtgtccaac aaggccctgc ctgcccctat cgagaaaacc 1020
atcagcaagg ccaagggcca gcccagagag ccccaggtgt acaccctgcc ccctagcaga 1080
gatgagctga ccaagaacca ggtgtccctg acctgcctgg tgaagggctt ctaccccagc 1140
gacatcgccg tggagtggga gagcaacggc cagcccgaga acaactacaa gaccaccccc 1200
cctgtgctgg acagcgatgg cagcttcttc ctgtacagca agctgaccgt ggacaagagc 1260
agatggcagc agggcaacgt gttcagctgc tccgtgatgc acgaggccct gcacaatcac 1320
tacacccaga agagcctgag cctgtcccct ggcaag 1356
<210>173
<211>214
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность полноразмерной легкой цепи
42Y073-86F04-23
<400>173
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Pro Tyr Phe Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210>174
<211>448
<212>БЕЛОК
<213>Искусственная
<220>
<223>Аминокислотная последовательность полноразмерной тяжелой цепи
42Y073-86F04-23
<400>174
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Gly Tyr
20 25 30
Pro Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Met Gly Thr Ala Arg Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Leu Gly Glu Ser Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210>175
<211>642
<212>ДНК
<213>Искусственная
<220>
<223>Последовательность ДНК полноразмерной легкой цепи 42Y073-86F04-23
<400>175
gacatccaga tgacccagag ccctagcagc ctgagcgcca gcgtgggaga cagggtgacc 60
atcacctgca gggccagcca gtccatcagc agctacctga actggtacca gcagaagccc 120
ggcaaggccc ccaagctgct gatctacgcc gcaagctcac tgcagagcgg cgtgccctct 180
aggtttagcg gcagcggcag cggcaccgac ttcaccctga ccatcagcag cctccagccc 240
gaggacttcg ccacctacta ctgccagcag ccctacttca gcccccccac tttcggcggc 300
ggcaccaagg tggagattaa gcgtacggtg gccgccccca gcgtgttcat cttccccccc 360
agcgatgagc agctgaagag cggcaccgcc agcgtggtgt gtctgctgaa caacttctac 420
ccccgggagg ccaaggtgca gtggaaggtg gacaatgccc tgcagagcgg caacagccag 480
gagagcgtga ccgagcagga cagcaaggac tccacctaca gcctgagcag caccctgacc 540
ctgagcaagg ccgactacga gaagcacaag gtgtacgcct gtgaggtgac ccaccagggc 600
ctgtccagcc ccgtgaccaa gagcttcaac cggggcgagt gc 642
<210>176
<211>1344
<212>ДНК
<213>Искусственная
<220>
<223>Последовательность ДНК полноразмерной тяжелой цепи 42Y073-86F04-23
<400>176
caggtgcagc tggtgcagag cggcgccgag gtgaaaaagc ccggcagcag cgtgaaggtg 60
agctgcaagg cctccggcgg gaccttcagc ggctacccca tcagctgggt gaggcaggct 120
cccggacagg gcctggagtg gatgggcggc atcatcccca ttatgggcac cgccaggtac 180
gcccagaagt tccagggaag ggtcaccatc accgccgacg agagcaccag caccgcctac 240
atggaactca gcagcctgag gagcgaggac accgccgtgt actattgcgc caggctgctg 300
ggcgagagcg gcatggacgt gtggggccag ggcaccaccg tgactgtgag cagcgccagc 360
accaagggcc ccagcgtgtt ccccctggcc cccagcagca agagcaccag cggcggcaca 420
gccgccctgg gctgcctggt gaaggactac ttccccgagc ccgtgaccgt gtcctggaac 480
agcggagccc tgaccagcgg cgtgcacacc ttccccgccg tgctgcagag cagcggcctg 540
tacagcctga gcagcgtggt gaccgtgccc agcagcagcc tgggcaccca gacctacatc 600
tgtaacgtga accacaagcc cagcaacacc aaggtggaca agaaggtgga gcccaagagc 660
tgtgacaaga cccacacctg ccccccctgc cctgcccccg agctgctggg aggccccagc 720
gtgttcctgt tcccccccaa gcctaaggac accctgatga tcagcagaac ccccgaggtg 780
acctgtgtgg tggtggatgt gagccacgag gaccctgagg tgaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcacaa tgccaagacc aagcccaggg aggagcagta caacagcacc 900
taccgggtgg tgtccgtgct gaccgtgctg caccaggatt ggctgaacgg caaggagtac 960
aagtgtaagg tgtccaacaa ggccctgcct gcccctatcg agaaaaccat cagcaaggcc 1020
aagggccagc ccagagagcc ccaggtgtac accctgcccc ctagcagaga tgagctgacc 1080
aagaaccagg tgtccctgac ctgcctggtg aagggcttct accccagcga catcgccgtg 1140
gagtgggaga gcaacggcca gcccgagaac aactacaaga ccaccccccc tgtgctggac 1200
agcgatggca gcttcttcct gtacagcaag ctgaccgtgg acaagagcag atggcagcag 1260
ggcaacgtgt tcagctgctc cgtgatgcac gaggccctgc acaatcacta cacccagaag 1320
agcctgagcc tgtcccctgg caag 1344
<210>177
<211>564
<212>БЕЛОК
<213>Искусственная Sequence
<220>
<223>Аминокислотная последовательность CD155-fc, используемого в примерах
<400>177
Trp Pro Pro Pro Gly Thr Gly Asp Val Val Val Gln Ala Pro Thr Gln
1 5 10 15
Val Pro Gly Phe Leu Gly Asp Ser Val Thr Leu Pro Cys Tyr Leu Gln
20 25 30
Val Pro Asn Met Glu Val Thr His Val Ser Gln Leu Thr Trp Ala Arg
35 40 45
His Gly Glu Ser Gly Ser Met Ala Val Phe His Gln Thr Gln Gly Pro
50 55 60
Ser Tyr Ser Glu Ser Lys Arg Leu Glu Phe Val Ala Ala Arg Leu Gly
65 70 75 80
Ala Glu Leu Arg Asn Ala Ser Leu Arg Met Phe Gly Leu Arg Val Glu
85 90 95
Asp Glu Gly Asn Tyr Thr Cys Leu Phe Val Thr Phe Pro Gln Gly Ser
100 105 110
Arg Ser Val Asp Ile Trp Leu Arg Val Leu Ala Lys Pro Gln Asn Thr
115 120 125
Ala Glu Val Gln Lys Val Gln Leu Thr Gly Glu Pro Val Pro Met Ala
130 135 140
Arg Cys Val Ser Thr Gly Gly Arg Pro Pro Ala Gln Ile Thr Trp His
145 150 155 160
Ser Asp Leu Gly Gly Met Pro Asn Thr Ser Gln Val Pro Gly Phe Leu
165 170 175
Ser Gly Thr Val Thr Val Thr Ser Leu Trp Ile Leu Val Pro Ser Ser
180 185 190
Gln Val Asp Gly Lys Asn Val Thr Cys Lys Val Glu His Glu Ser Phe
195 200 205
Glu Lys Pro Gln Leu Leu Thr Val Asn Leu Thr Val Tyr Tyr Pro Pro
210 215 220
Glu Val Ser Ile Ser Gly Tyr Asp Asn Asn Trp Tyr Leu Gly Gln Asn
225 230 235 240
Glu Ala Thr Leu Thr Cys Asp Ala Arg Ser Asn Pro Glu Pro Thr Gly
245 250 255
Tyr Asn Trp Ser Thr Thr Met Gly Pro Leu Pro Pro Phe Ala Val Ala
260 265 270
Gln Gly Ala Gln Leu Leu Ile Arg Pro Val Asp Lys Pro Ile Asn Thr
275 280 285
Thr Leu Ile Cys Asn Val Thr Asn Ala Leu Gly Ala Arg Gln Ala Glu
290 295 300
Leu Thr Val Gln Val Lys Glu Gly Pro Pro Ser Glu His Ser Gly Ile
305 310 315 320
Ser Arg Asn Ser Gly Glu Asn Leu Tyr Phe Gln Gly Asp Pro Lys Ser
325 330 335
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
340 345 350
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
355 360 365
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
370 375 380
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
385 390 395 400
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
405 410 415
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
420 425 430
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
435 440 445
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
450 455 460
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
465 470 475 480
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
485 490 495
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
500 505 510
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
515 520 525
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
530 535 540
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
545 550 555 560
Ser Pro Gly Lys
<210>178
<211>417
<212>БЕЛОК
<213>Homo sapiens
<400>178
Met Ala Arg Ala Met Ala Ala Ala Trp Pro Leu Leu Leu Val Ala Leu
1 5 10 15
Leu Val Leu Ser Trp Pro Pro Pro Gly Thr Gly Asp Val Val Val Gln
20 25 30
Ala Pro Thr Gln Val Pro Gly Phe Leu Gly Asp Ser Val Thr Leu Pro
35 40 45
Cys Tyr Leu Gln Val Pro Asn Met Glu Val Thr His Val Ser Gln Leu
50 55 60
Thr Trp Ala Arg His Gly Glu Ser Gly Ser Met Ala Val Phe His Gln
65 70 75 80
Thr Gln Gly Pro Ser Tyr Ser Glu Ser Lys Arg Leu Glu Phe Val Ala
85 90 95
Ala Arg Leu Gly Ala Glu Leu Arg Asn Ala Ser Leu Arg Met Phe Gly
100 105 110
Leu Arg Val Glu Asp Glu Gly Asn Tyr Thr Cys Leu Phe Val Thr Phe
115 120 125
Pro Gln Gly Ser Arg Ser Val Asp Ile Trp Leu Arg Val Leu Ala Lys
130 135 140
Pro Gln Asn Thr Ala Glu Val Gln Lys Val Gln Leu Thr Gly Glu Pro
145 150 155 160
Val Pro Met Ala Arg Cys Val Ser Thr Gly Gly Arg Pro Pro Ala Gln
165 170 175
Ile Thr Trp His Ser Asp Leu Gly Gly Met Pro Asn Thr Ser Gln Val
180 185 190
Pro Gly Phe Leu Ser Gly Thr Val Thr Val Thr Ser Leu Trp Ile Leu
195 200 205
Val Pro Ser Ser Gln Val Asp Gly Lys Asn Val Thr Cys Lys Val Glu
210 215 220
His Glu Ser Phe Glu Lys Pro Gln Leu Leu Thr Val Asn Leu Thr Val
225 230 235 240
Tyr Tyr Pro Pro Glu Val Ser Ile Ser Gly Tyr Asp Asn Asn Trp Tyr
245 250 255
Leu Gly Gln Asn Glu Ala Thr Leu Thr Cys Asp Ala Arg Ser Asn Pro
260 265 270
Glu Pro Thr Gly Tyr Asn Trp Ser Thr Thr Met Gly Pro Leu Pro Pro
275 280 285
Phe Ala Val Ala Gln Gly Ala Gln Leu Leu Ile Arg Pro Val Asp Lys
290 295 300
Pro Ile Asn Thr Thr Leu Ile Cys Asn Val Thr Asn Ala Leu Gly Ala
305 310 315 320
Arg Gln Ala Glu Leu Thr Val Gln Val Lys Glu Gly Pro Pro Ser Glu
325 330 335
His Ser Gly Ile Ser Arg Asn Ala Ile Ile Phe Leu Val Leu Gly Ile
340 345 350
Leu Val Phe Leu Ile Leu Leu Gly Ile Gly Ile Tyr Phe Tyr Trp Ser
355 360 365
Lys Cys Ser Arg Glu Val Leu Trp His Cys His Leu Cys Pro Ser Ser
370 375 380
Thr Glu His Ala Ser Ala Ser Ala Asn Gly His Val Ser Tyr Ser Ala
385 390 395 400
Val Ser Arg Glu Asn Ser Ser Ser Gln Asp Pro Gln Thr Glu Gly Thr
405 410 415
Arg
<210>179
<211>330
<212>БЕЛОК
<213>Homo sapiens
<400>179
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210>180
<211>990
<212>ДНК
<213>Homo sapiens
<400>180
gccagcacca agggccccag cgtgttcccc ctggccccca gcagcaagag caccagcggc 60
ggcacagccg ccctgggctg cctggtgaag gactacttcc ccgagcccgt gaccgtgtcc 120
tggaacagcg gagccctgac cagcggcgtg cacaccttcc ccgccgtgct gcagagcagc 180
ggcctgtaca gcctgagcag cgtggtgacc gtgcccagca gcagcctggg cacccagacc 240
tacatctgta acgtgaacca caagcccagc aacaccaagg tggacaagaa ggtggagccc 300
aagagctgtg acaagaccca cacctgcccc ccctgccctg cccccgagct gctgggaggc 360
cccagcgtgt tcctgttccc ccccaagcct aaggacaccc tgatgatcag cagaaccccc 420
gaggtgacct gtgtggtggt ggatgtgagc cacgaggacc ctgaggtgaa gttcaactgg 480
tacgtggacg gcgtggaggt gcacaatgcc aagaccaagc ccagggagga gcagtacaac 540
agcacctacc gggtggtgtc cgtgctgacc gtgctgcacc aggattggct gaacggcaag 600
gagtacaagt gtaaggtgtc caacaaggcc ctgcctgccc ctatcgagaa aaccatcagc 660
aaggccaagg gccagcccag agagccccag gtgtacaccc tgccccctag cagagatgag 720
ctgaccaaga accaggtgtc cctgacctgc ctggtgaagg gcttctaccc cagcgacatc 780
gccgtggagt gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 840
ctggacagcg atggcagctt cttcctgtac agcaagctga ccgtggacaa gagcagatgg 900
cagcagggca acgtgttcag ctgctccgtg atgcacgagg ccctgcacaa tcactacacc 960
cagaagagcc tgagcctgtc ccctggcaag 990
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИТЕЛО ПРОТИВ LILRB1 И ЕГО ПРИМЕНЕНИЯ | 2021 |
|
RU2813373C1 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2837821C1 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА ПОДОБНЫЙ FC-РЕЦЕПТОРУ БЕЛОК 5, И ИХ ПРИМЕНЕНИЕ | 2015 |
|
RU2779747C2 |
| БИСПЕЦИФИЧНЫЙ СЛИТЫЙ БЕЛОК И ЕГО ПРИМЕНЕНИЕ | 2021 |
|
RU2801528C2 |
| ИНДУЦИРУЮЩИЙ ЦИТОТОКСИЧНОСТЬ ТЕРАПЕВТИЧЕСКИЙ АГЕНТ | 2015 |
|
RU2743464C2 |
| АНТИТЕЛА, НАПРАВЛЕННЫЕ ПРОТИВ FC-РЕЦЕПТОР-ПОДОБНОГО БЕЛКА 5, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2774158C2 |
| АНТИТЕЛО К CLDN-18.2 И ЕГО ПРИМЕНЕНИЕ | 2021 |
|
RU2829997C1 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2808030C2 |
| АНТИТЕЛА К РЕЦЕПТОРУ IL4 ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2837141C2 |
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО ПРОТИВ ВИРУСА БЕШЕНСТВА И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2764740C1 |
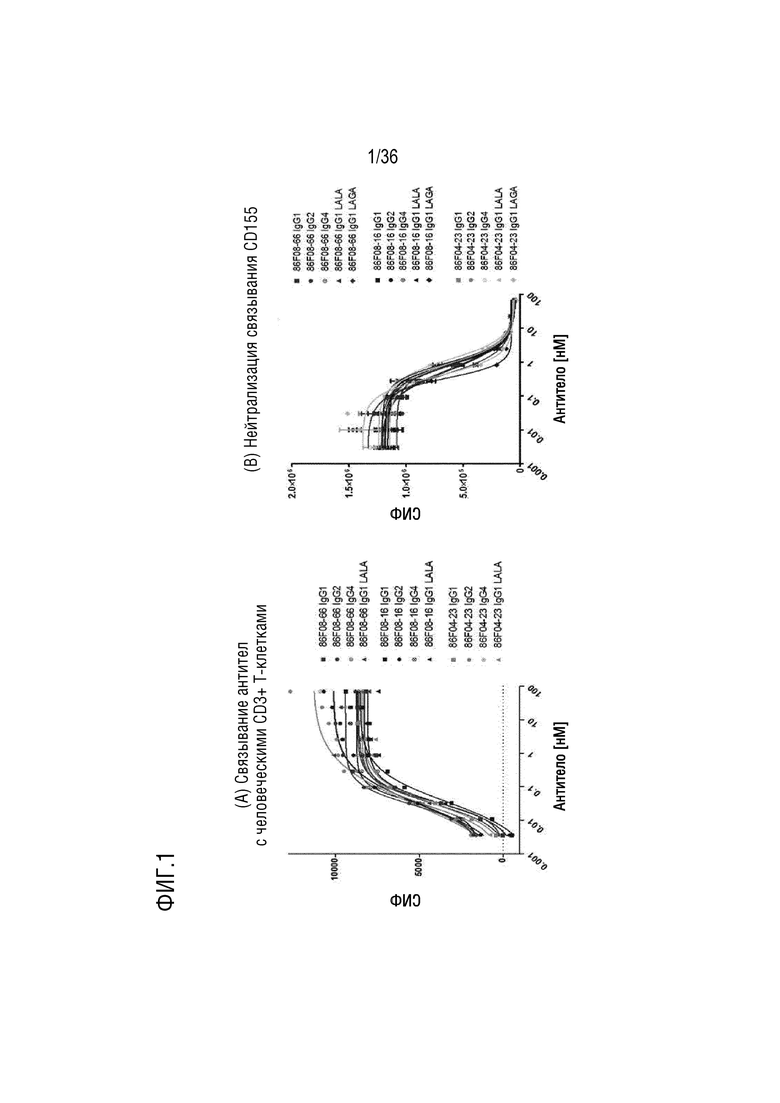
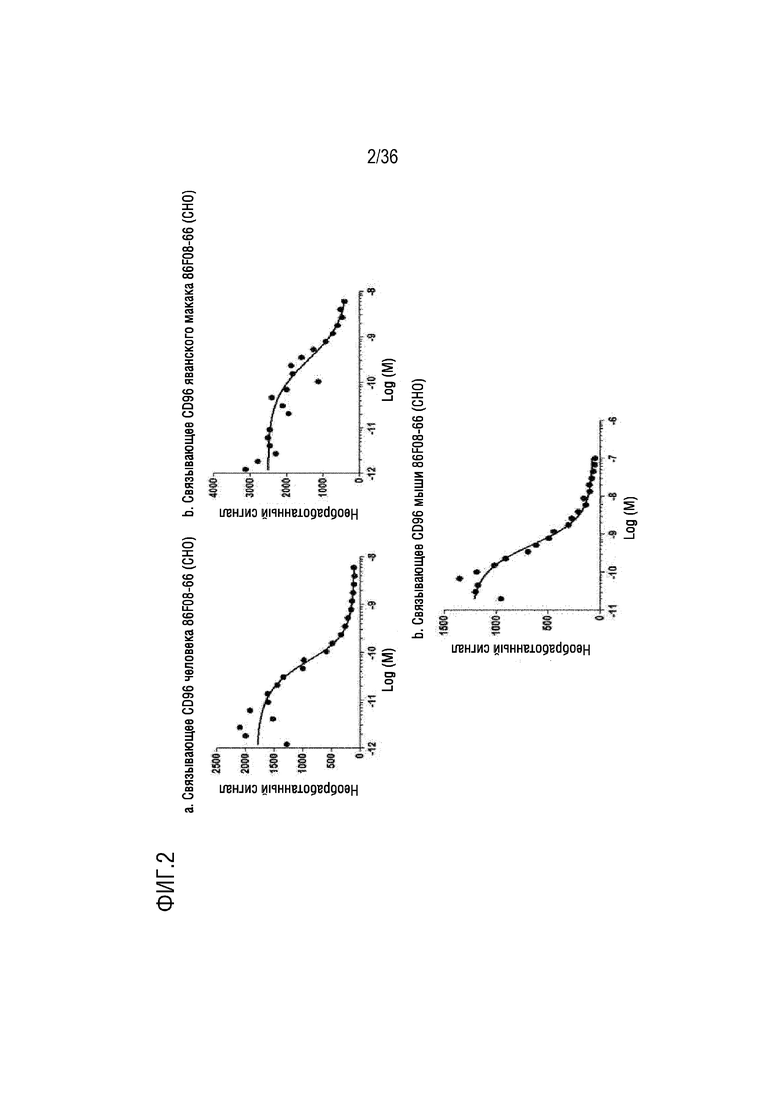
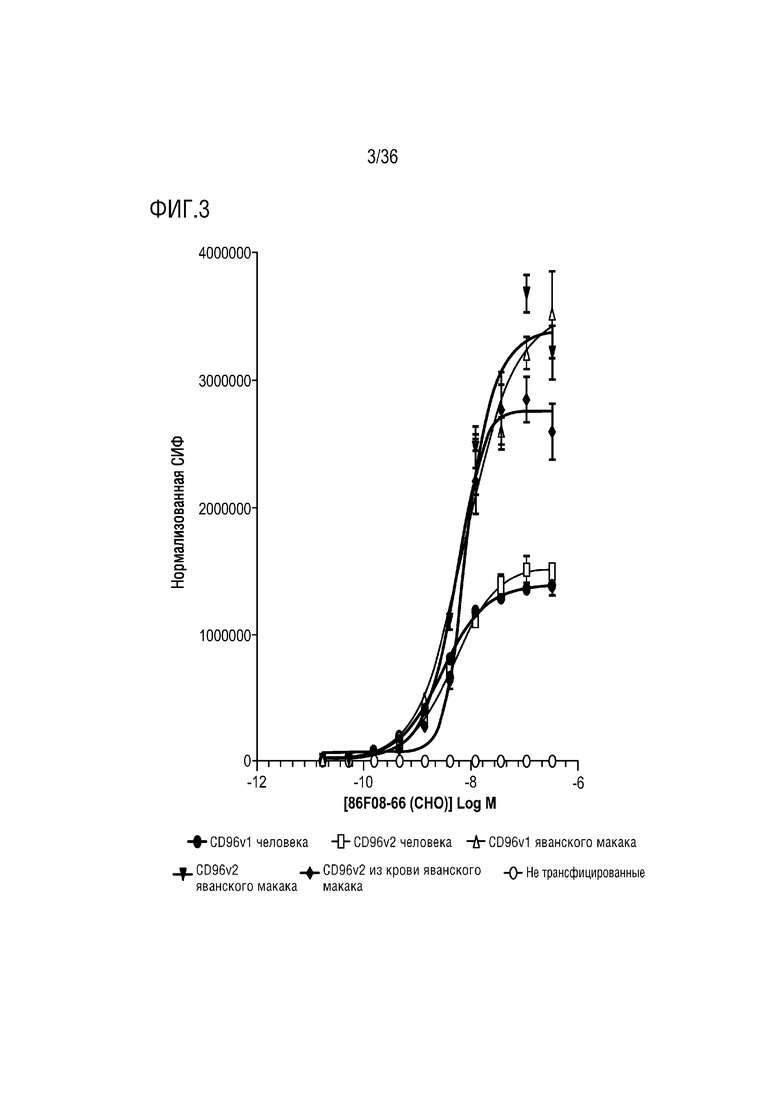
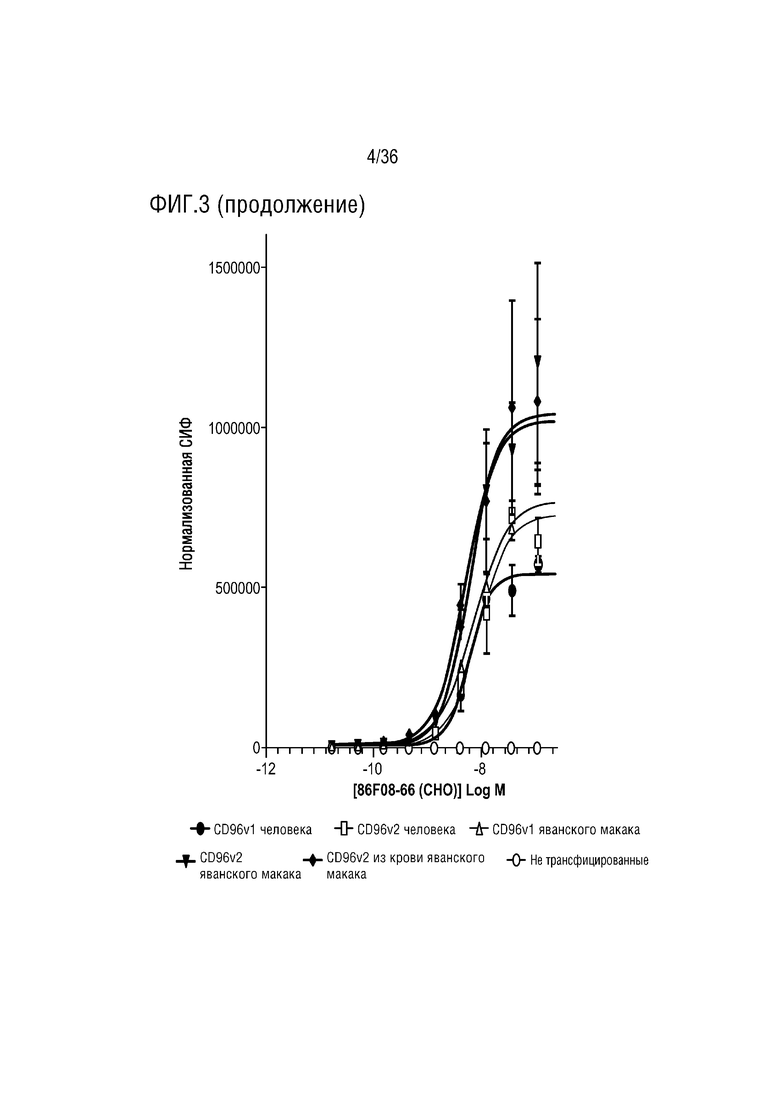
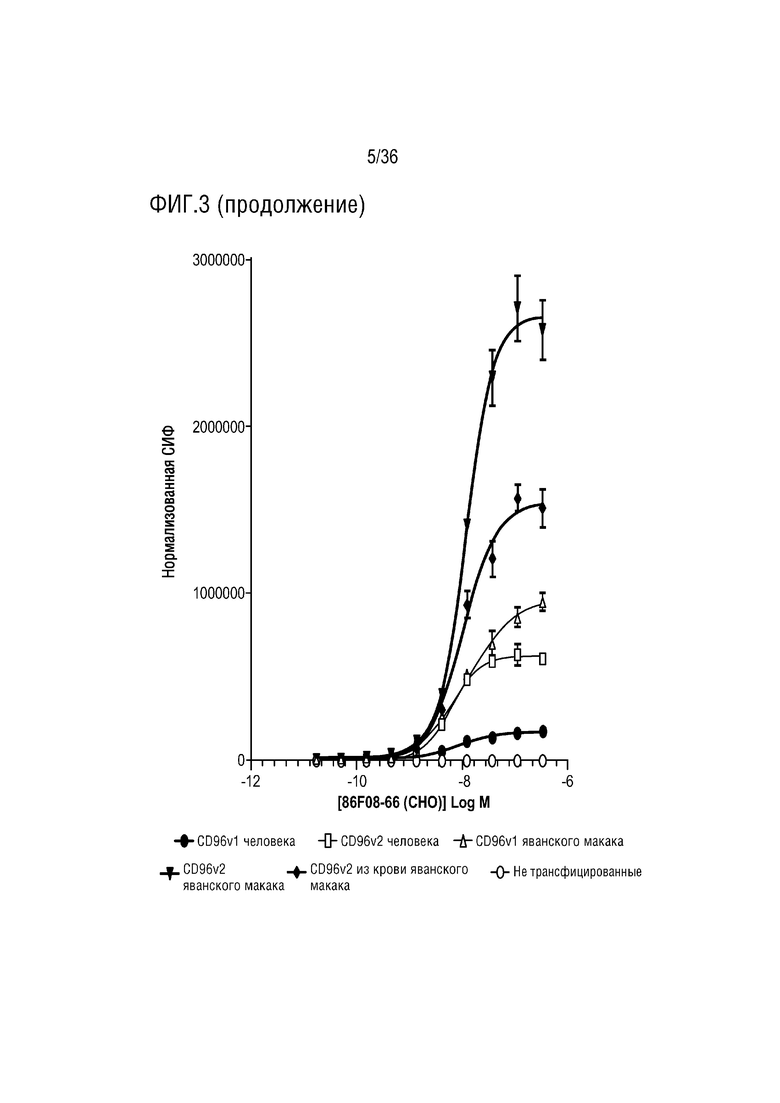
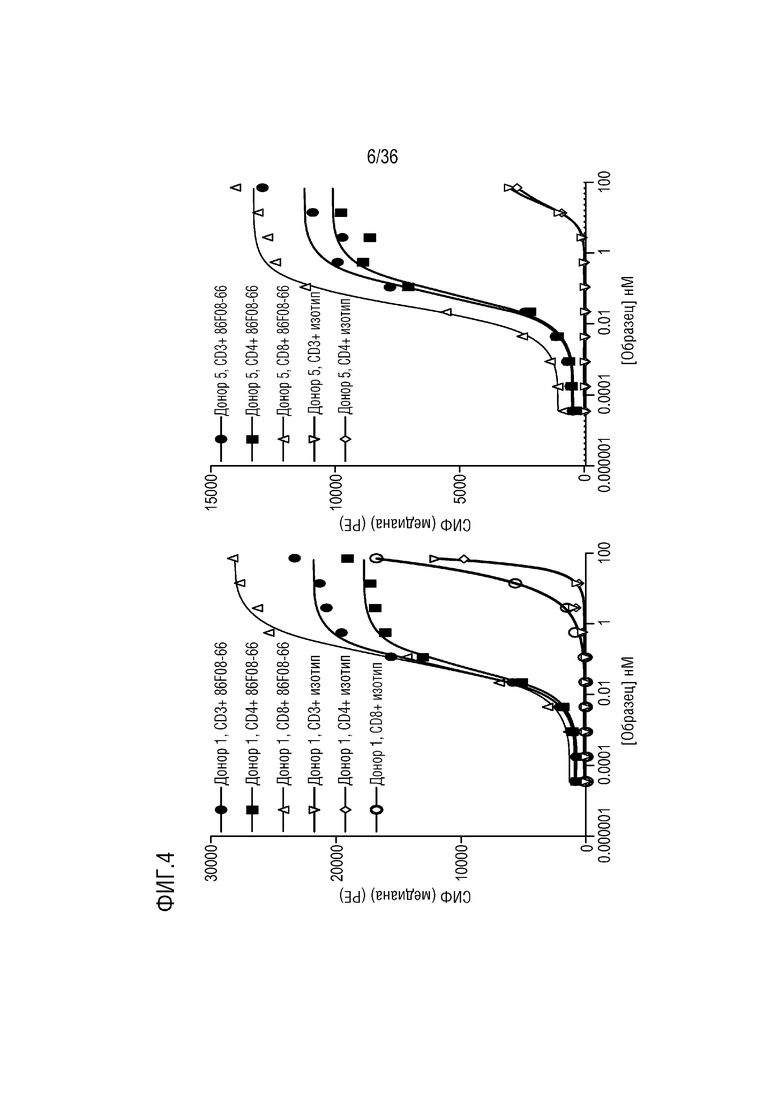
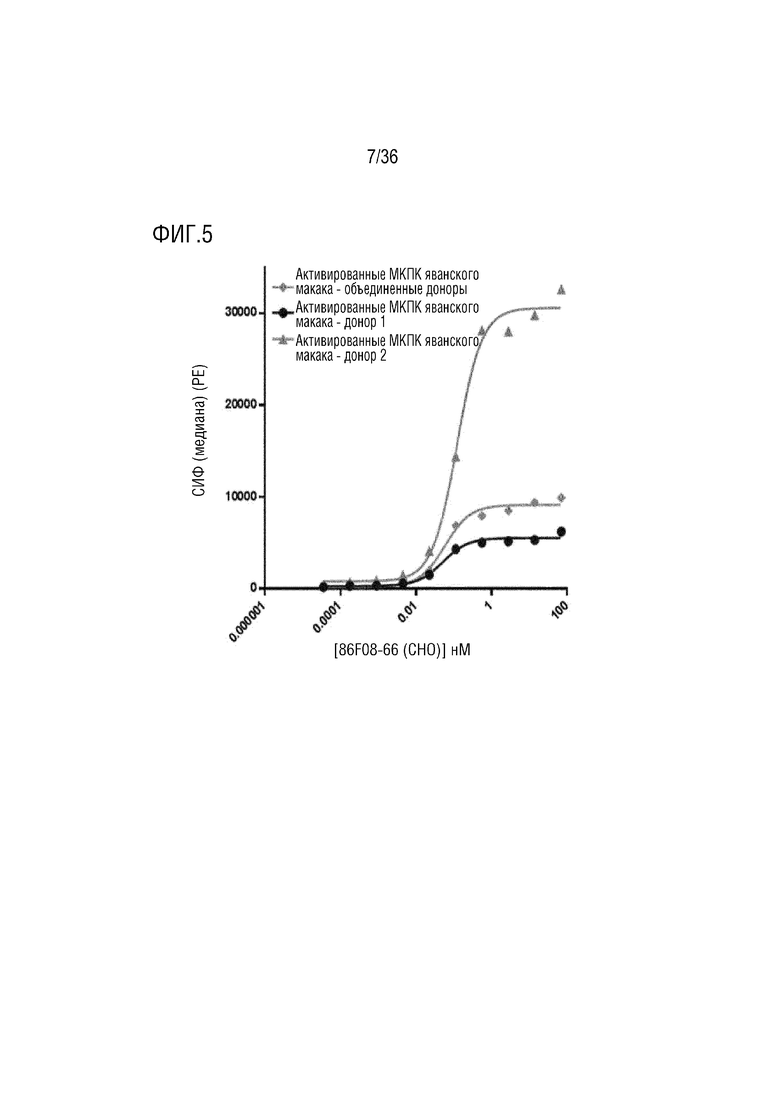

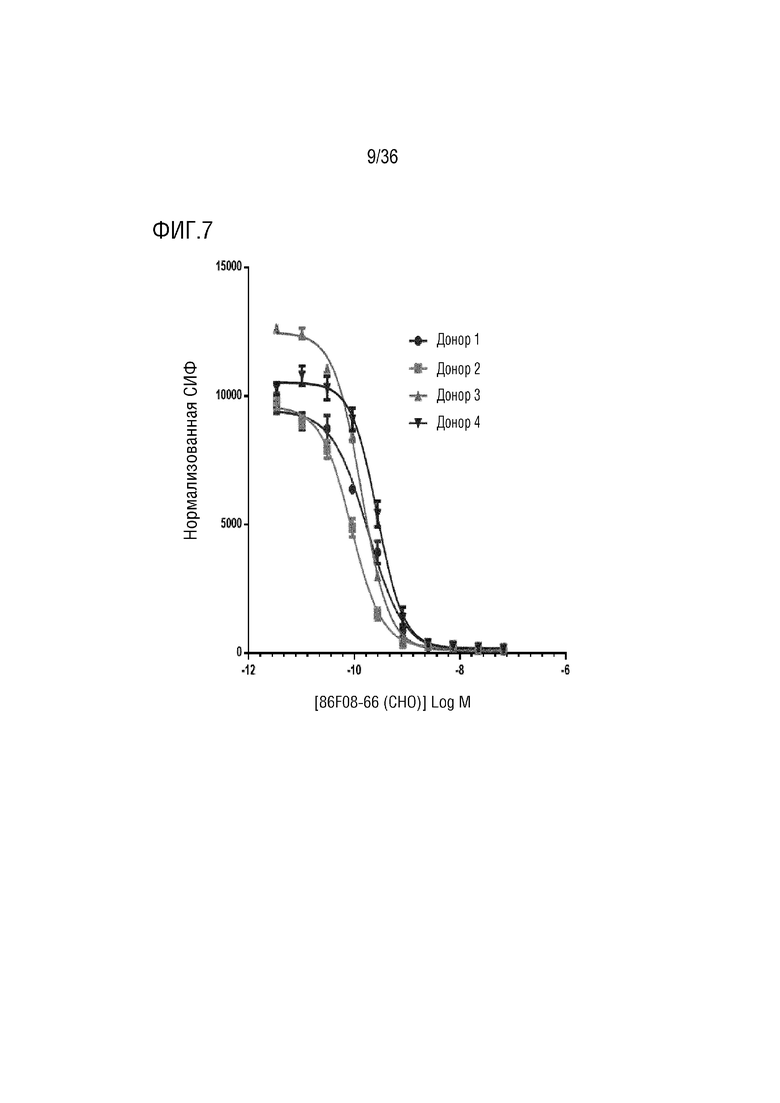
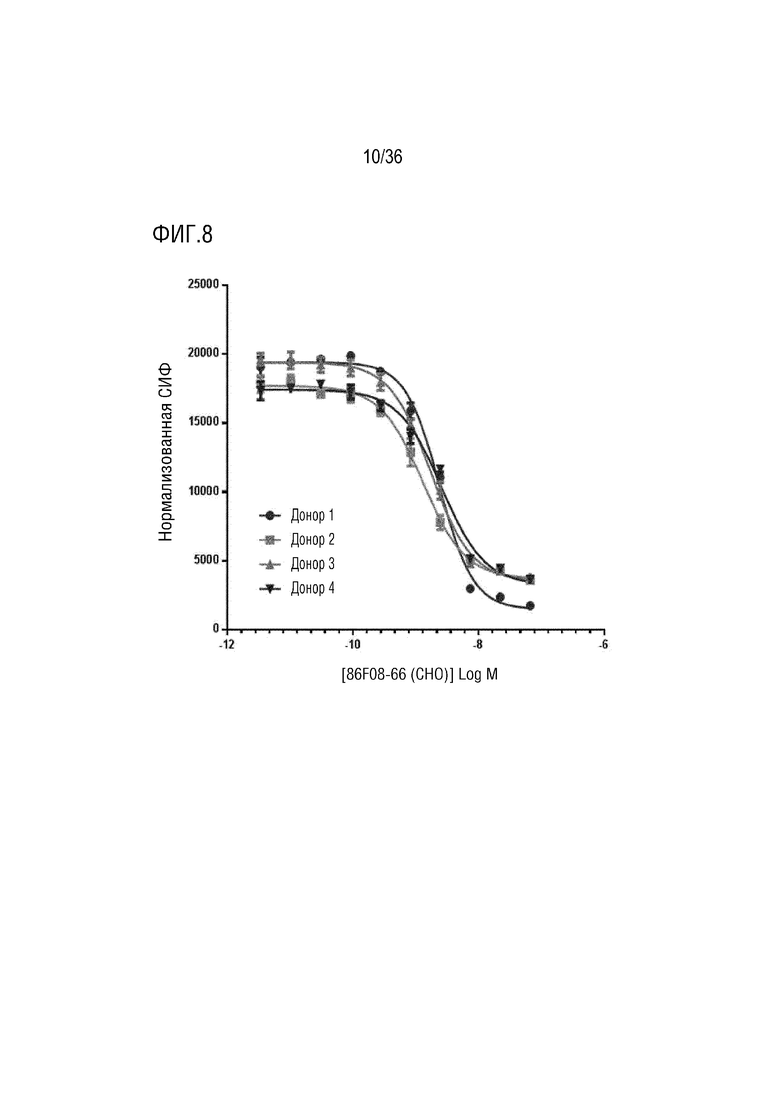
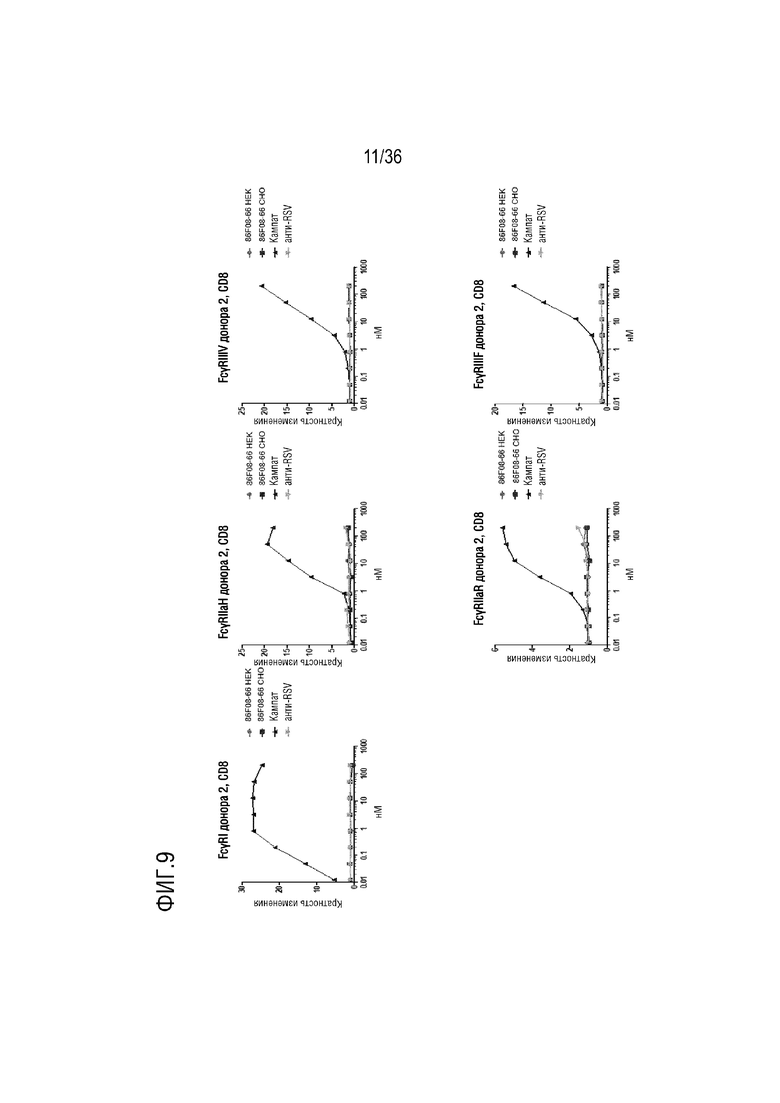
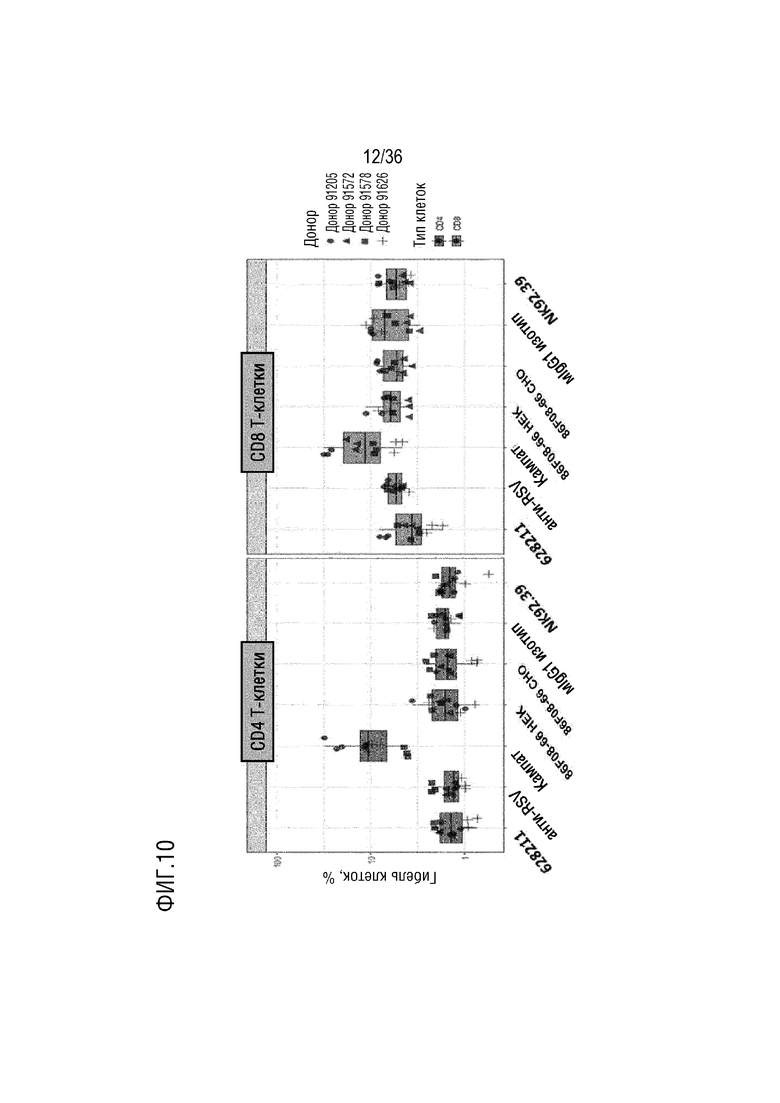
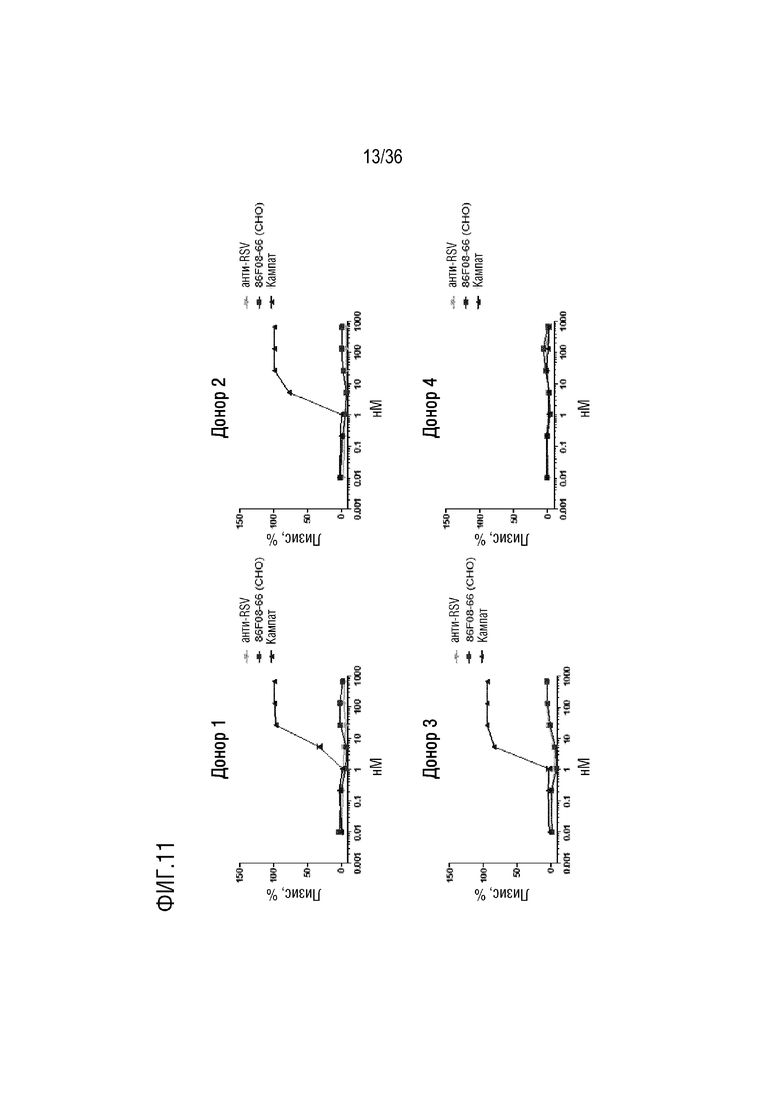
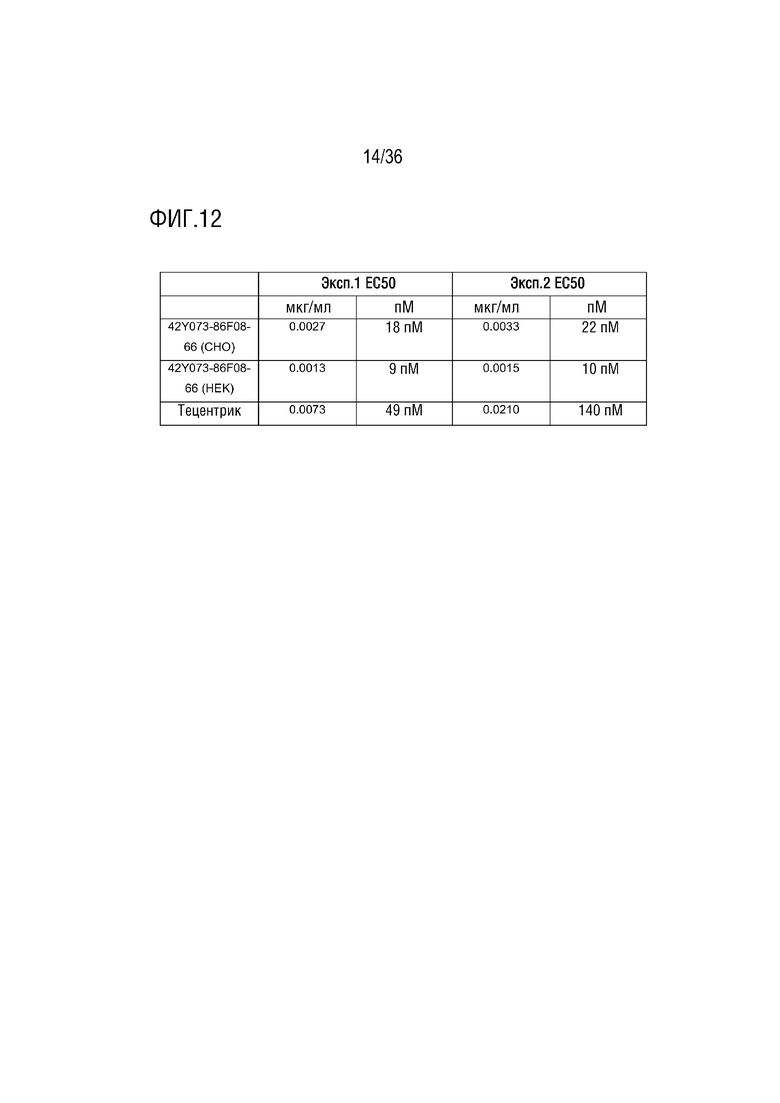
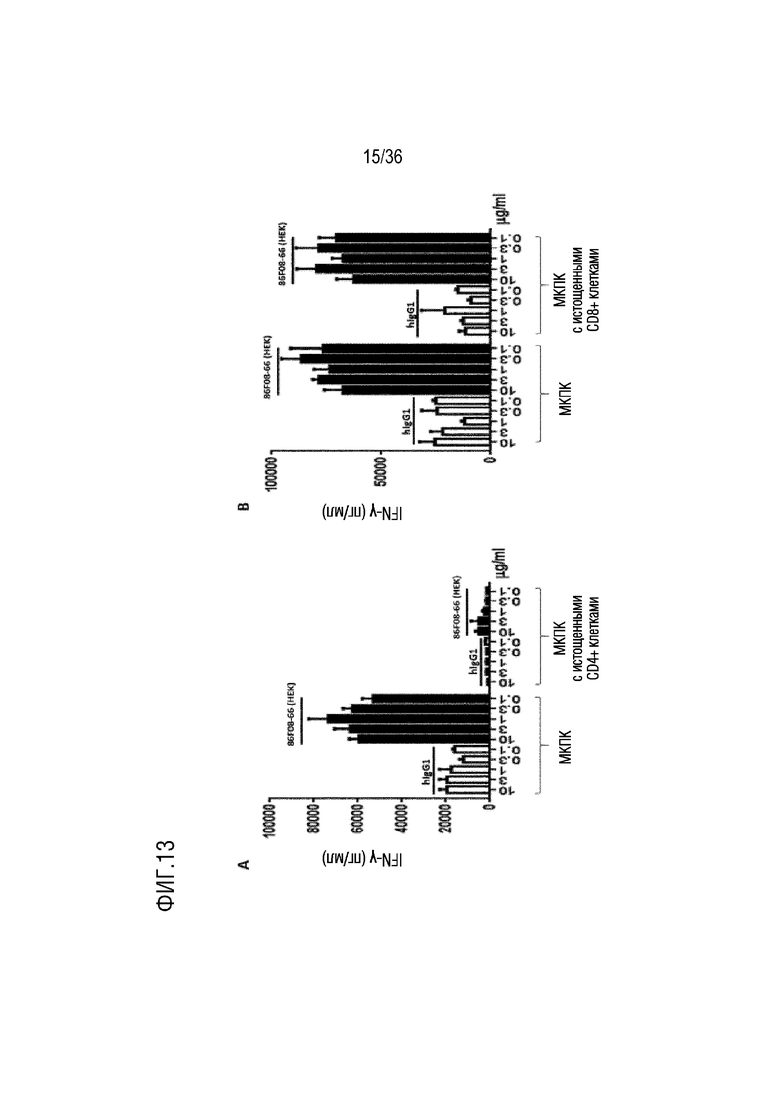
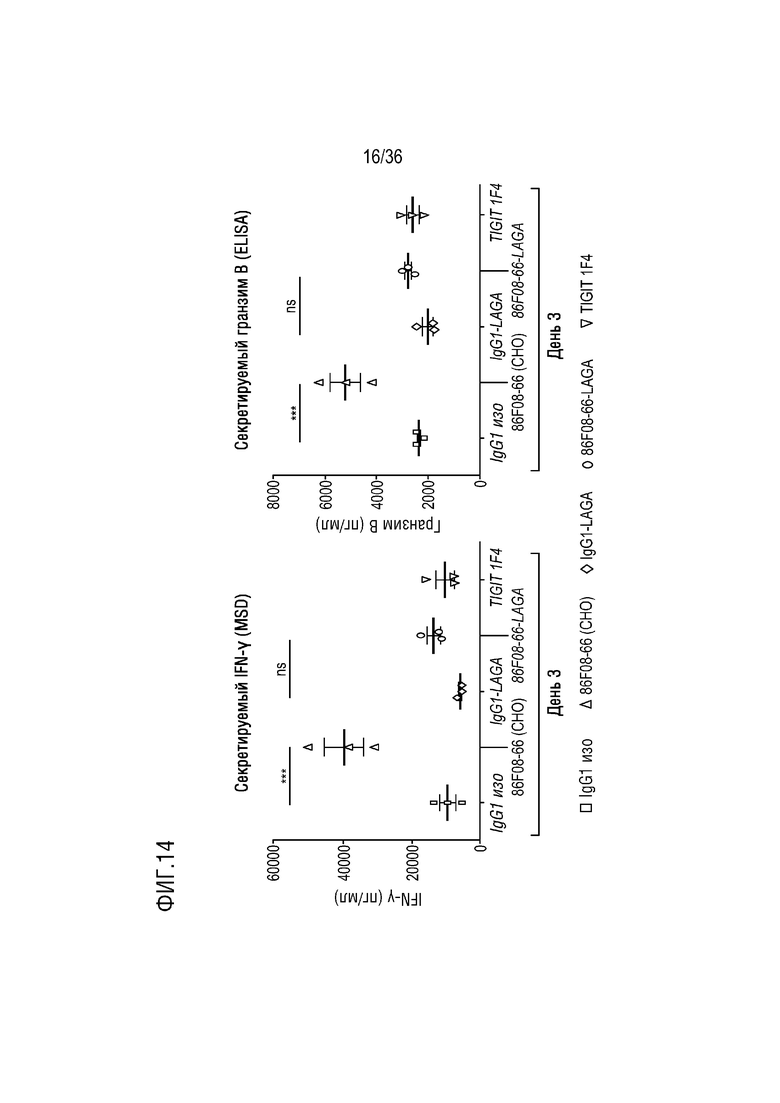
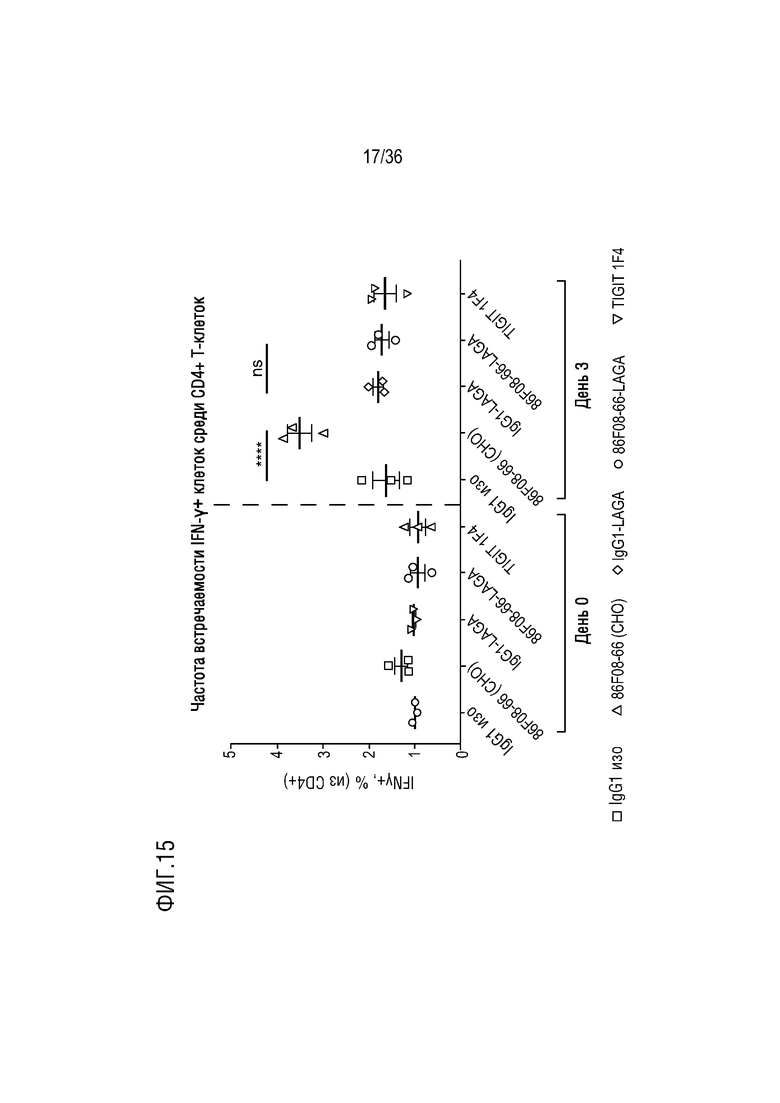

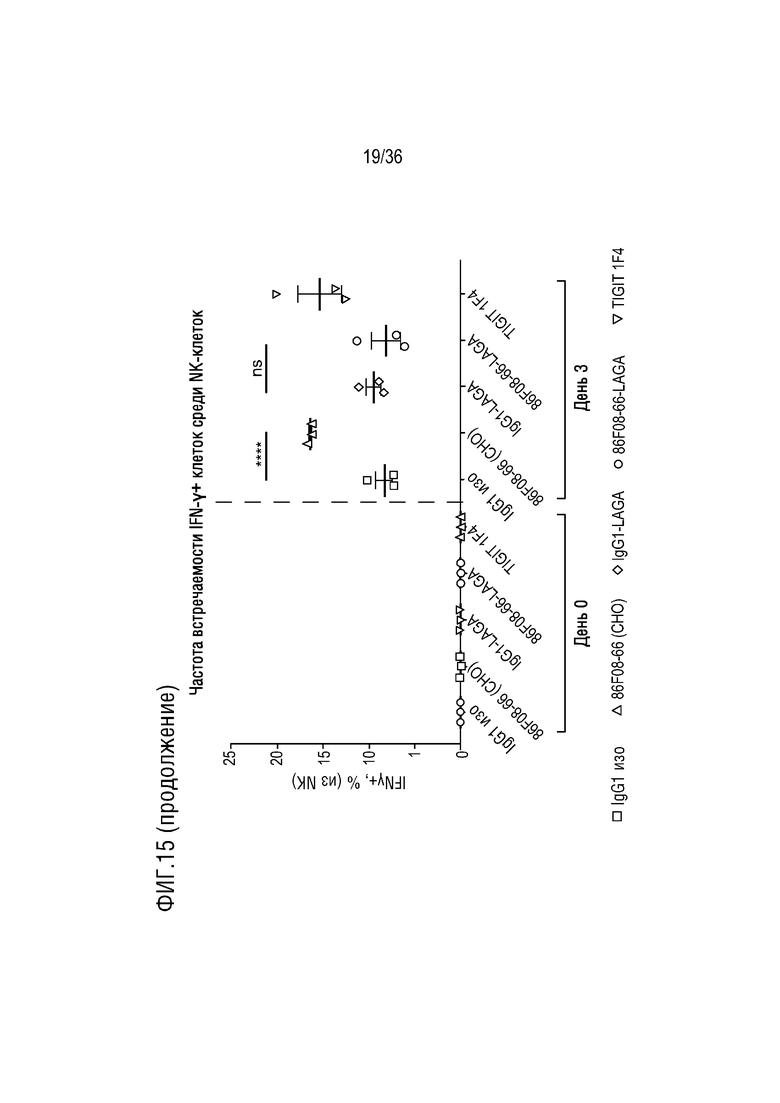


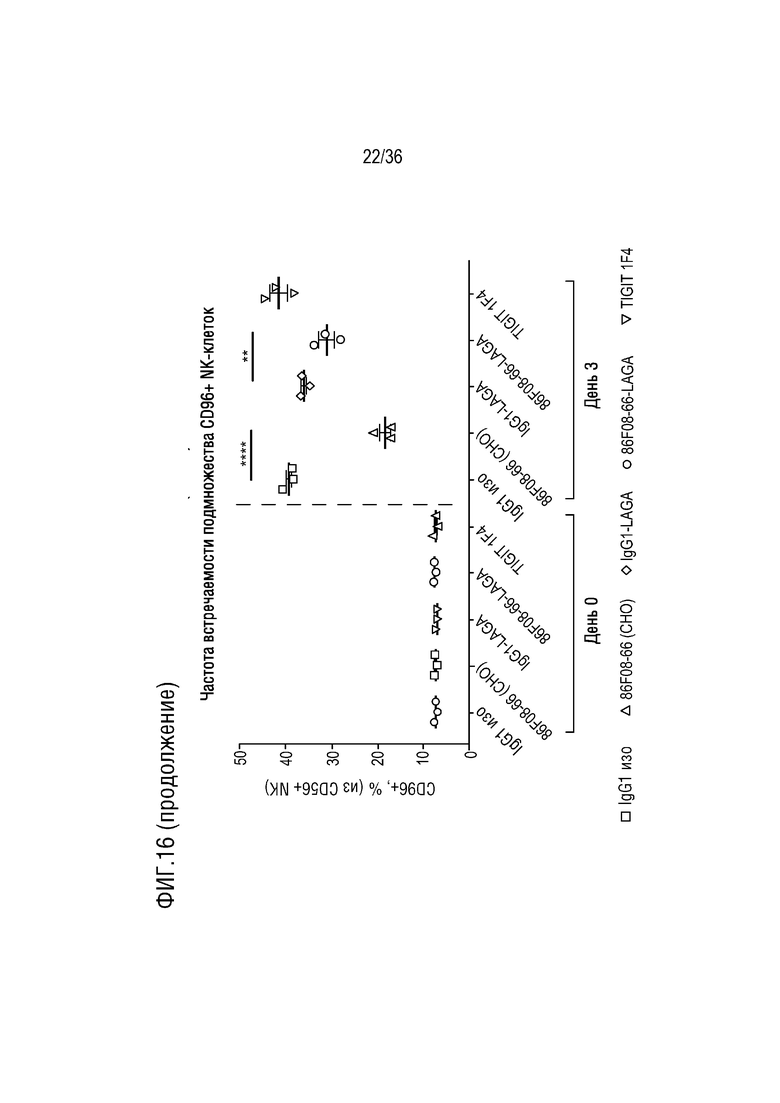
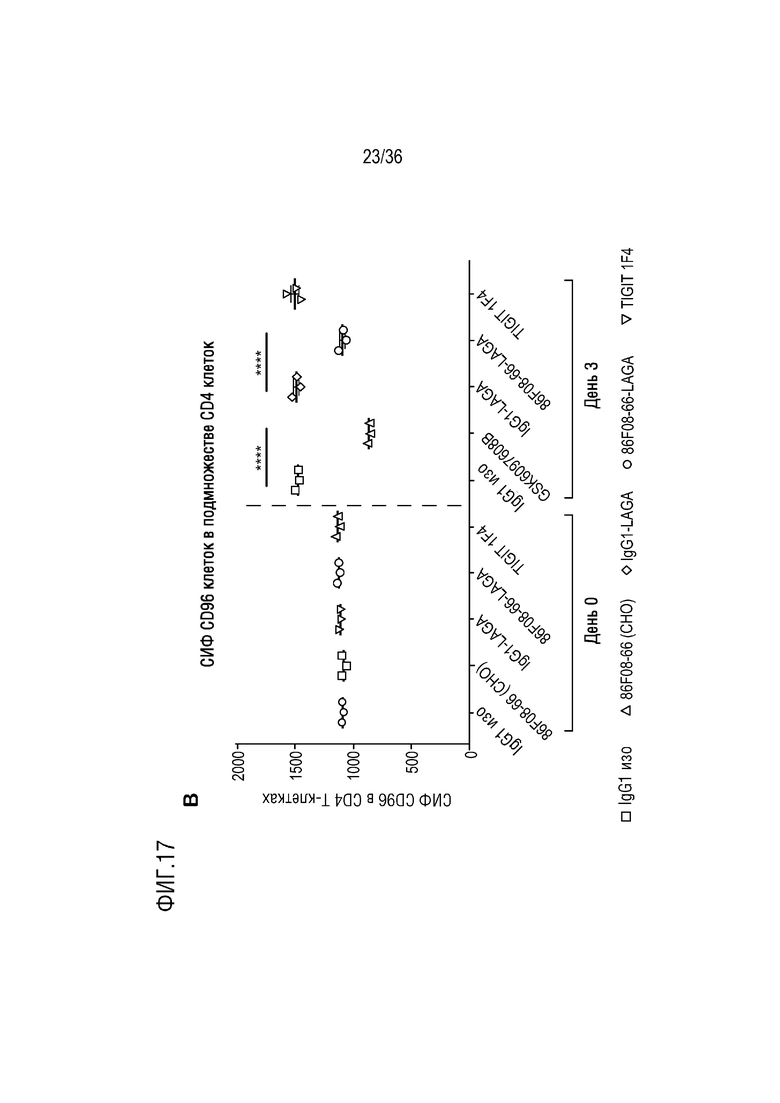
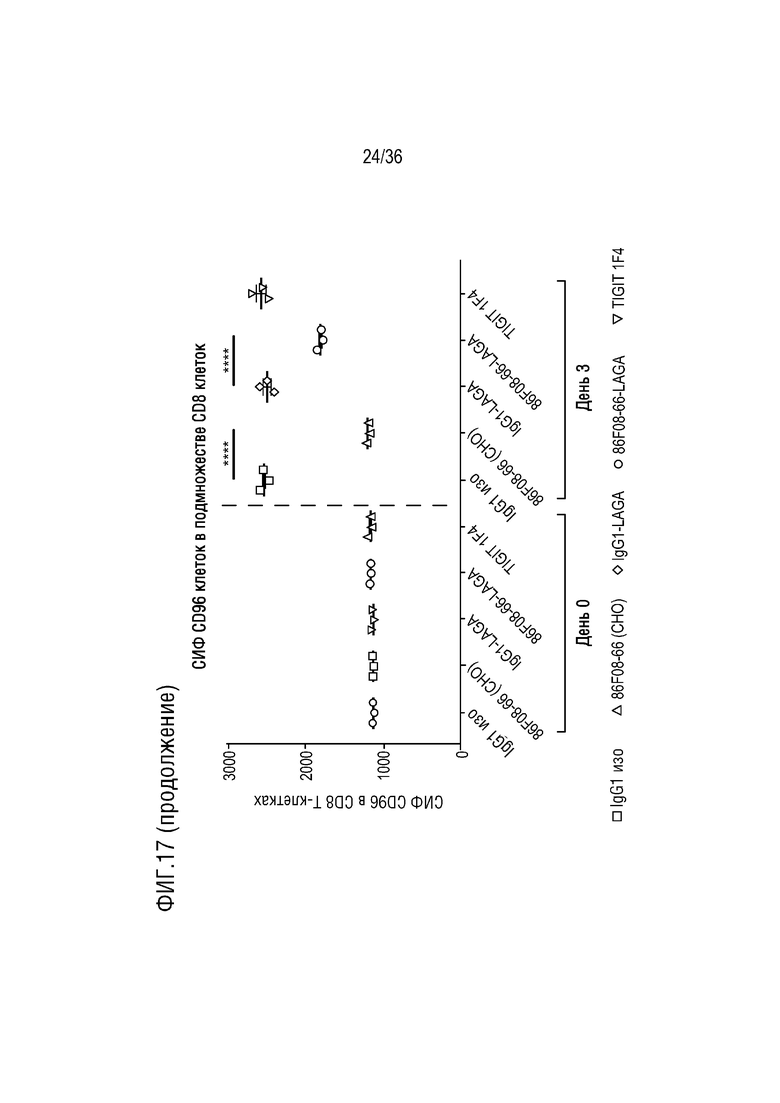
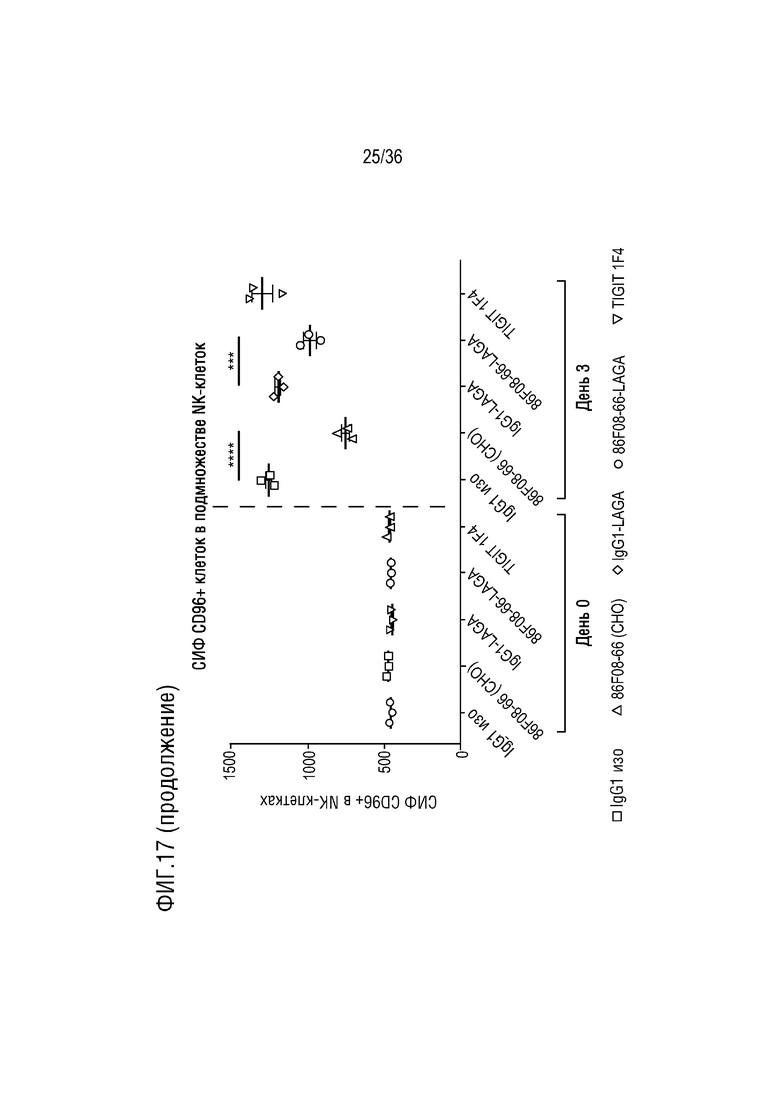
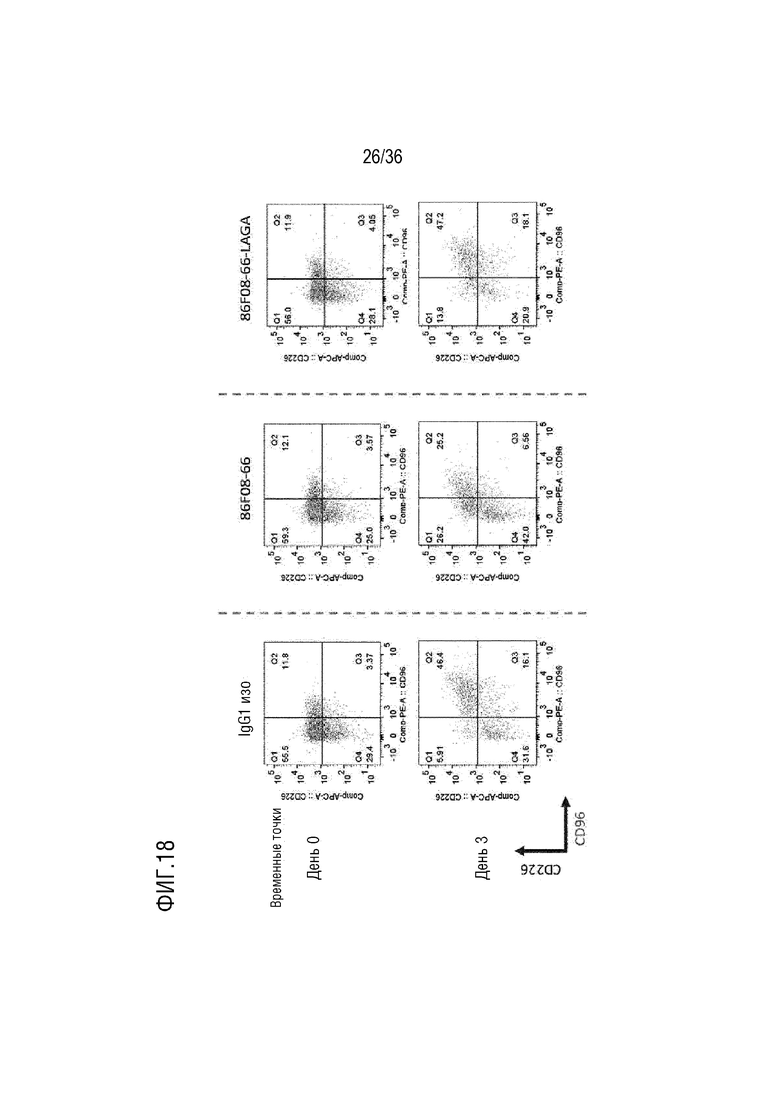
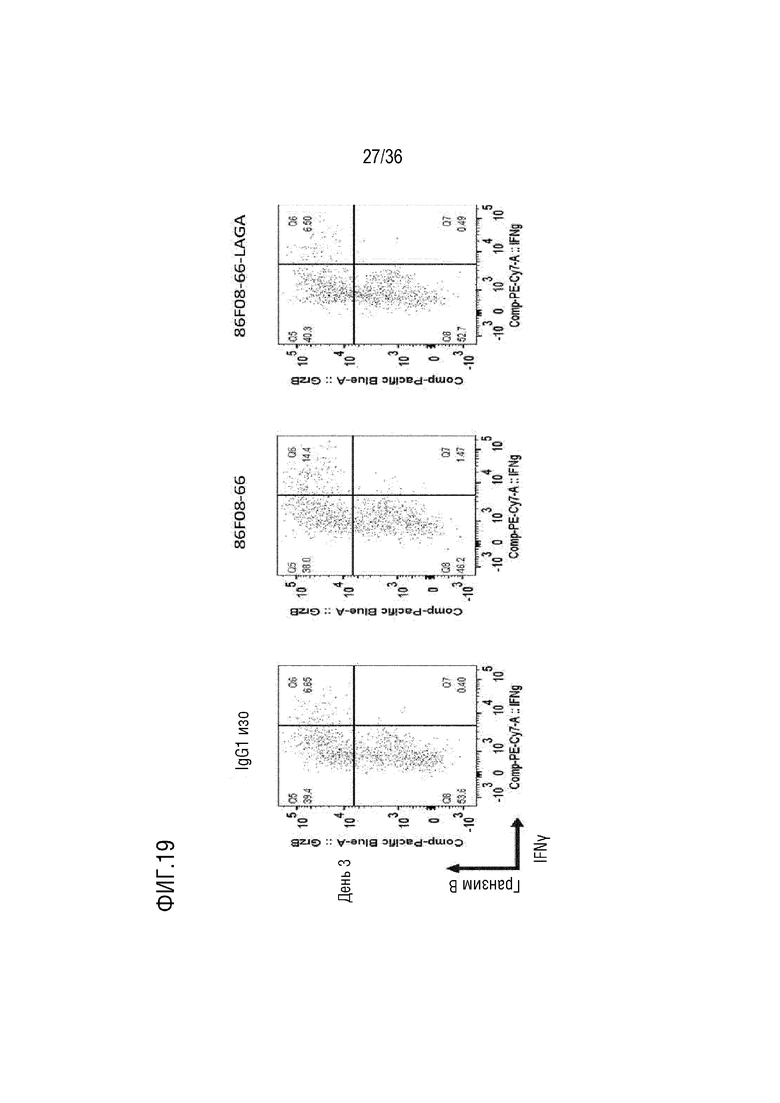
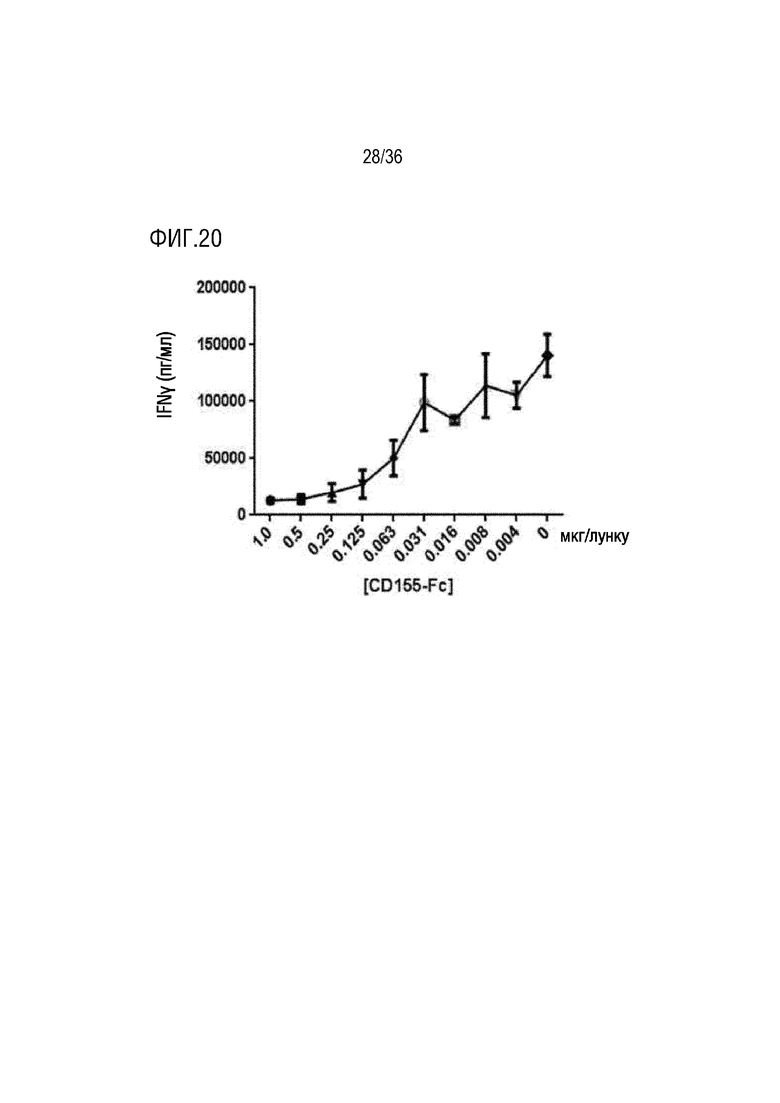
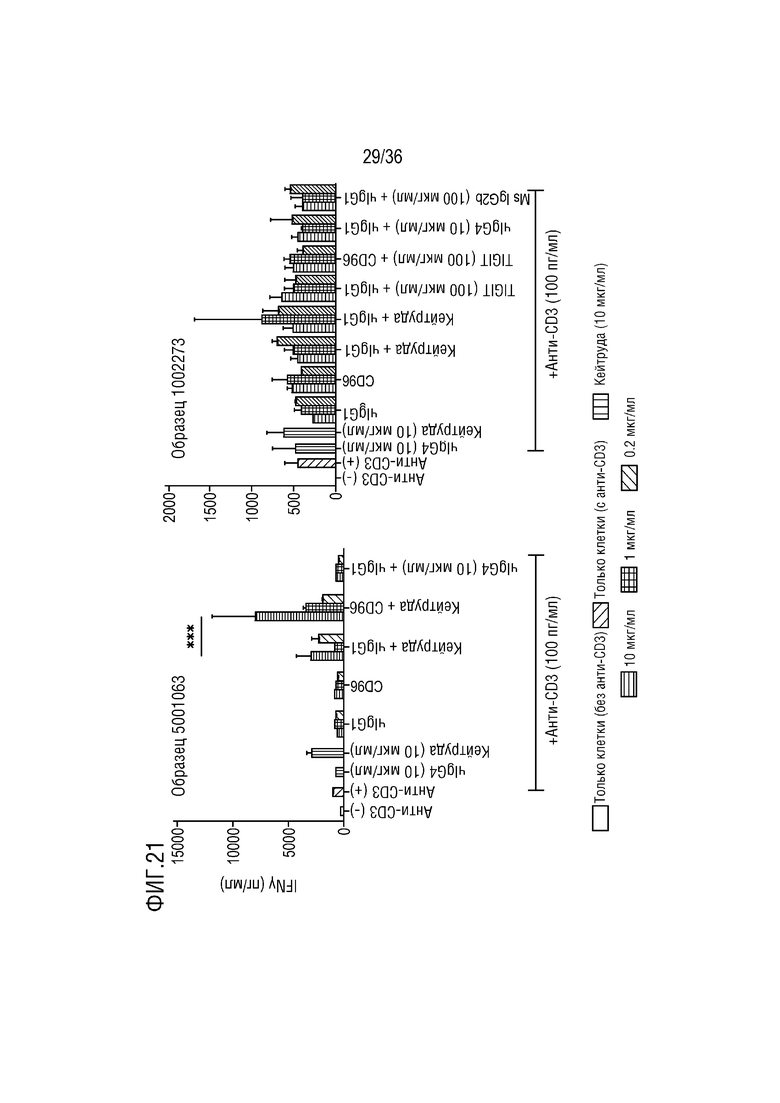

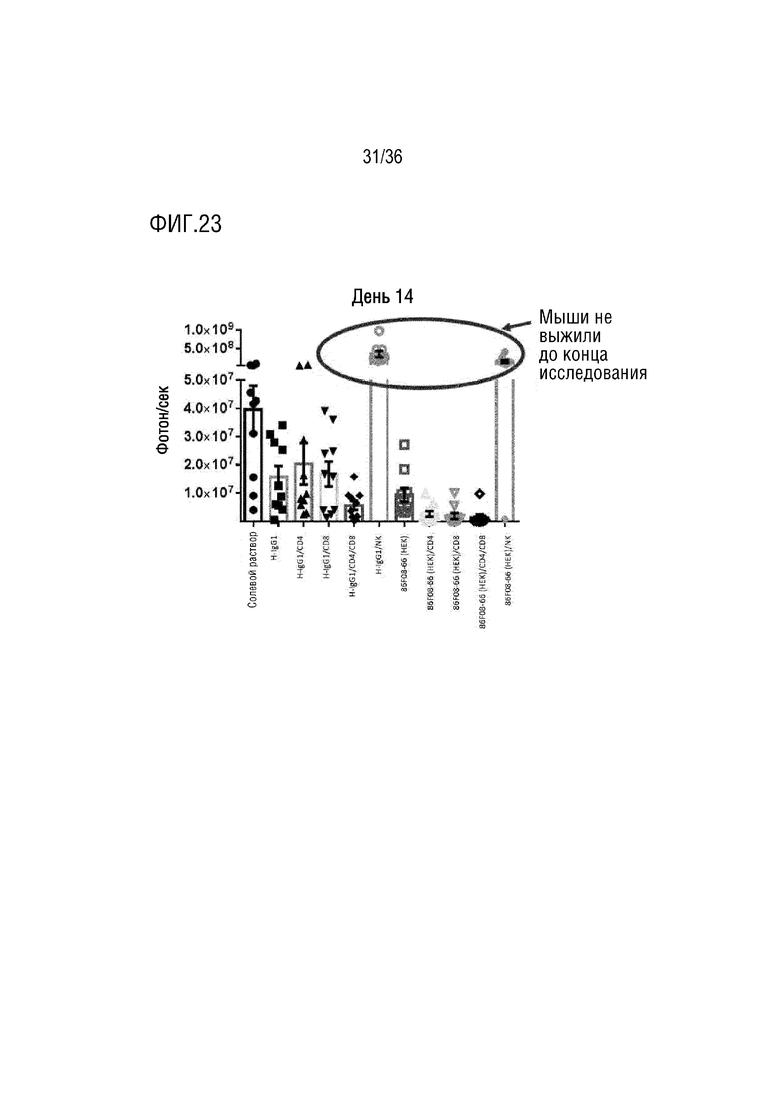
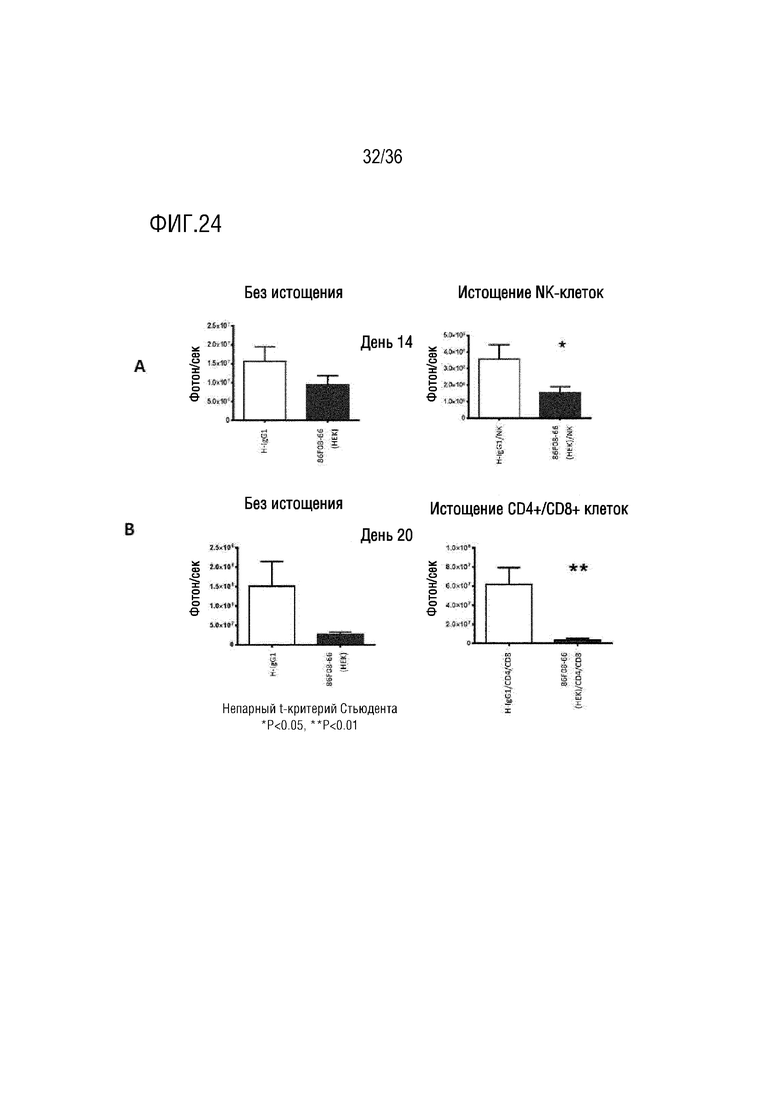

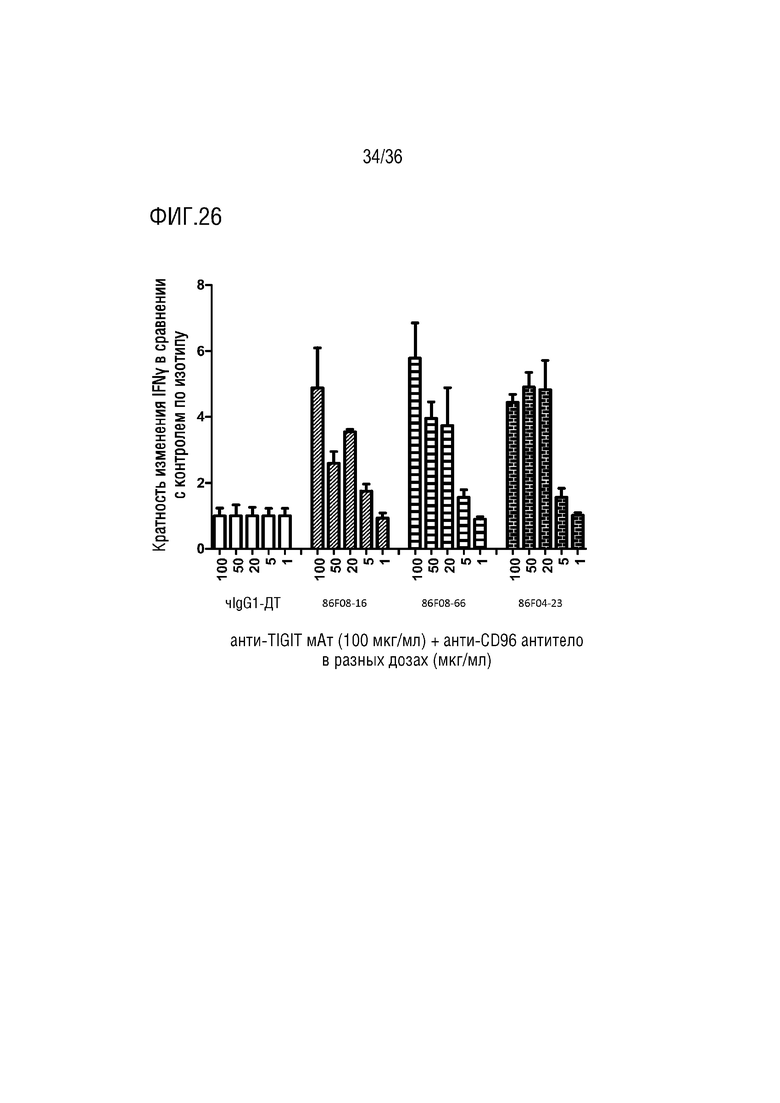
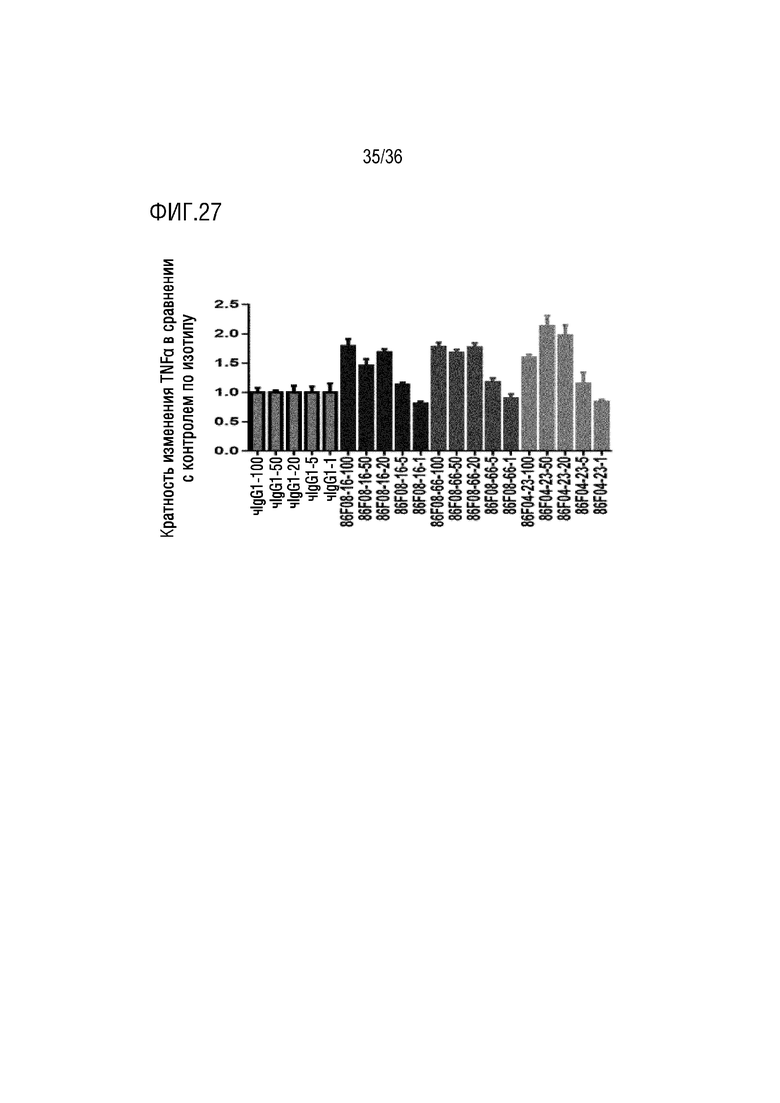
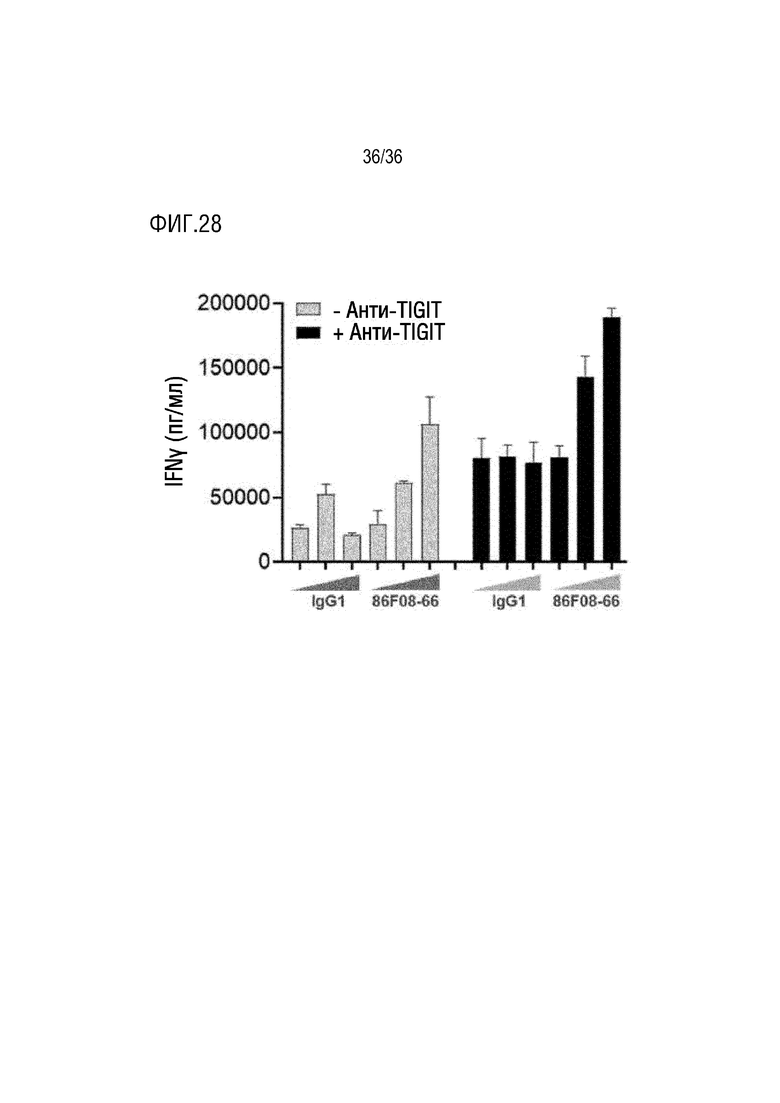
Изобретение относится к области биотехнологии, конкретно к рекомбинантным антителам, связывающим CD96 (кластер дифференцировки 96), и может быть использовано в медицине для лечения рака. Предложенный CD96-связывающий белок, который представляет собой человеческое IgG1 антитело к CD96 с улучшенными характеристиками, способен стимулировать противоопухолевую эффективность, опосредованную NK-клетками. 6 н. и 3 з.п. ф-лы, 28 ил., 10 табл., 7 пр.
1. CD96 (кластер дифференцировки 96)-связывающий белок, содержащий следующие 6 определяющих комплементарность областей (CDR):
CDRH1 с SEQ ID NO: 115;
CDRH2 с SEQ ID NO: 145; и
CDRH3 с SEQ ID NO: 147; и
CDRL1 с SEQ ID NO: 97;
CDRL2 с SEQ ID NO: 99; и
CDRL3 с SEQ ID NO: 101,
причем выделенный CD96-связывающий белок представляет собой человеческое IgG1 антитело, которое специфически связывает CD96 и содержит вариабельную область тяжелой цепи (VH) SEQ ID NO: 86 и вариабельную область легкой цепи (VL) SEQ ID NO: 85.
2. Молекула нуклеиновой кислоты, которая кодирует белок, связывающий CD96, как определено в п.1.
3. Молекула нуклеиновой кислоты по п.2, где последовательность молекулы нуклеиновой кислоты содержит SEQ ID NO: 88, кодирующую тяжелую цепь; и SEQ ID NO: 87, кодирующую легкую цепь.
4. Экспрессионный вектор, содержащий молекулу нуклеиновой кислоты, как определено в п.2 или 3.
5. Рекомбинантная клетка-хозяин, содержащая экспрессионный вектор, как определено в п.4, где клетка-хозяин представляет собой клетку млекопитающего и направлена на экспрессию человеческого IgG1 антитела, которое специфически связывает CD96, по п.1.
6. Фармацевтическая композиция для лечения рака, содержащая эффективное количество CD96-связывающего белка, как определено в п.1, и фармацевтически приемлемый эксципиент.
7. Применение CD96-связывающего белка, как определено в п.1, или фармацевтической композиции, как определено в п.6, для лечения рака.
8. Применение по п.7, где рак представляет собой рак печени, рак яичников, немелкоклеточный рак легкого, рак почек, колоректальный рак, рак мочевого пузыря или плоскоклеточную карциному головы и шеи (HNSCC).
9. Применение по п.8, где рак представляет собой немелкоклеточный рак легкого.
| WO 2019030377 A1, 14.02.2019 | |||
| WO 2019091449 A1, 16.05.2019 | |||
| BLAKE S | |||
| J | |||
| et al | |||
| Приспособление в пере для письма с целью увеличения на нем запаса чернил и уменьшения скорости их высыхания | 1917 |
|
SU96A1 |
| Машина для добывания торфа и т.п. | 1922 |
|
SU22A1 |
| Выбрасывающий ячеистый аппарат для рядовых сеялок | 1922 |
|
SU21A1 |
| Водяной двигатель | 1926 |
|
SU5183A1 |
| WO 2018129559 A1, 12.07.2018 | |||
| RU 2016104880 A, 21.08.2017 | |||
| VAN DEN BEUCKEN T | |||
| et al | |||
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
Авторы
Даты
2024-09-23—Публикация
2020-09-25—Подача