Группа изобретений относится к биотехнологии и молекулярной биологии, а именно к способу выделения дезоксирибонуклеиновой кислоты (ДНК) и средству для осуществления способа – лизирующей смеси. Изобретения могут быть использованы в лабораторной и исследовательской практике для выделения высокомолекулярных ДНК из образцов почвы, клеток грамположительных и грамотрицательных бактерий, грибов, растений и клеток животных, пригодных для их последующего использования при постановке полимеразной цепной реакции (ПЦР).
Выбор метода выделения ДНК – это один из важнейших этапов в проведении исследования, поскольку от качества и количества выделенной ДНК зависит успех дальнейших исследований. Существующие методы выделения метагеномной (тотальной) ДНК часто предлагают выбор между высокой эффективностью выделения, затрачиваемым временем на выделение и доступностью компонентов, но не все сразу. Известные способы выделения ДНК из биоматериалов, в основном, отличаются использованием различных поверхностно-активных веществ (ПАВ) с целью достижения наилучшего разрушения клеток и диссоциации биополимеров. Выход ДНК при использовании этих методов, как правило, невелик, что приводит к увеличению массы используемых образцов, повышению трудозатрат и расходу реактивов. В связи с этим исследователями постоянно предпринимаются попытки увеличить выход ДНК, предлагаются различные методы и наборы реактивов.
Например, известны коммерческие наборы реактивов для выделения метагеномной почвенной ДНК. Выделение ДНК в данном случае проводится в соответствии с протоколом производителей. Набор для выделения геномной ДНК из почв FastDNA Spin Kit For Soil (MP Biomedicals) [FastDNA Spin Kit For Soil, MP Biomedicals. Электронный ресурс: https://www.dia-m.ru/catalog/lab/gomogenizatory/gomogenizatory-fastprep/116560200-nabor-dlya-vydeleniya-genomnoj-dnk-iz-pochvennyh-obrazcovfastdna-spin-kit-for-soil/ дата обращения: 25.12.2021] используется с гомогенизатором FastPrep или с альтернативным прибором. До 500 мг почвенной пробы помещают в 2х мл пробирки, содержащие лизирующую матрицу Е – смесь стеклянных (4 мм в диаметре), кварцевых (0.1 мм в диаметре) и керамических (1.4 мм в диаметре) шариков, способных разрушать все почвенные организмы. Гомогенизация происходит в присутствии МТ буфера и фосфата натрия. По завершению лизиса образцы центрифугируют. Полученную ДНК очищают с использованием технологии Geneclean, включающую применение силикагелевых фильтров SPIN и устранение гуминовых кислот и полифенолов. Однако этот набор имеет высокую стоимость, которая составляет примерно 400 долларов на 50 выделений, выход ДНК низкий и составляет порядка 1-5 мкг из 500 мг почвы.
В патенте [RU2753768 С1, 23.08.2021] предложен набор для выделения внеклеточной ДНК, состоящий из связывающего буфера на основе 5,5 М гуанидинтиоцианата, 5 мМ Трилона Б, 10 мМ Трис-HCl pH 8,0, отмывочного буфера на основе 80% этанола и элюирующего буфера на основе 10 мМ Трис-HCl и 1 мМ Трилона Б с pH 8,5, ДНК-аффинного полипептида (термический полилизин с молекулярной массой 10-30 килодальтон) и суспензии сорбента – частиц магнетита, покрытых слоем силикагеля, общим диаметром 1,5 микрона. Заявленный метод выделения ДНК основан на аффинном связывании ДНК с термическим полипептидом с полилизином и осаждении комплекса ДНК-полилизин центрифугированием. Изобретение позволяет повысить выход внеклеточной ДНК из биологических жидкостей, чистоту выделяемой ДНК и сократить время ее выделения. В данном случае выделение ДНК происходит из физиологических жидкостей, которые не обеспечивают достаточную очистку образов почвы, грибов или растений в виду отсутствия солей и детергенов. Полипептиды в присутствии высоких концентраций солей и детергенов теряют свои ДНК-связывающие свойства.
Еще одним способом является метод выделения ДНК из образцов почвы [Выделение ДНК из образцов почвы: Методические указания. Российская академия сельскохозяйственных наук. Всероссийский научно исследовательский институт сельскохозяйственной микробиологии. – СПб., 2011. – С. 15-17], в котором в качестве основного детергента для выделения ДНК используется додецилсульфат натрия – ДСН. К навеске 0.2 г замороженной почвы, помещенной в пробирку, добавляют количество стеклянных бусин, по объему примерно равное почве, и вносят раствор гуанидина – гуанидина изотиоцианат 240 mM, натрий-фосфатный буфер 200 mM, рН 7.0, и 350 мкл 1% раствора SDS - Tris-HCl 500 mM, ДСН 1% (вес/об.), рН 7.9, а также 400 мкл смеси фенол-хлороформ. Пробирку помещают во встряхиватель и гомогенизируют образец в течение 1-15 мин, в зависимости от мощности прибора (FastPrep 24-1 мин при максимальной мощности, Vortex Genie® 2-15 мин при максимальной скорости). Затем центрифугируют при ускорении 10-15 × 103g при максимальной скорости в течение 5 мин. Водную фазу отбирают, добавляют 400 мкл хлороформа, центрифугируют также, как и на предыдущей стадии, и отбирают водную фазу. К неочищенному экстракту ДНК добавляют равный объем изопропилового спирта, центрифугируют на максимальной скорости в течение 5 мин, промывают 70% (об./об.) этанолом, слегка подсушивают на воздухе и растворяют осадок при 65°C в течение 5-10 мин в 100 мкл воды. Этот метод является более дешевым и эффективным, однако предполагает использование гомогенизатора, а также раствора гуанидина, который может искажать результаты измерения спектрофотометра.
Описан [RU2696052 С1, 30.07.2019] способ выделения ДНК из почвы, включающий гомогенизацию образца почвы путем вибрации (при помощи вибромельницы в диапазоне частот 25-35 Гц) в присутствии мелющих тел (смесь стеклянных бусин диаметром 0.5 мм и 1.0 мм и керамических бусин диаметром 2.0 мм в весовом соотношении 3:1:1 соответственно) в растворе, содержащем гуанидин НCl, натрий-фосфатный буфер, TrisHCl, додецилсульфат натрия (SDS), смесь фенол-хлороформ, с последующим центрифугированием и осаждением ДНК изопропанолом, обенностью которого является дополнительное введение в раствор для гомогенизации почвы детергент лаурилсаркозинат натрия при следующем соотношении исходных компонентов, вес.%: лаурилсаркозинат натрия – 2-6, додецилсульфат натрия – 1-4. Недостатком указанного способа также можно назвать использование раствора гуанидина, который может искажать результаты измерения спектрофотометра.
Что касается выделения нуклеиновых кислот из растений, то известен способ выделения НК с использованием сорбента [Rapid and Simple Method for Purification of Nucleic Acids / R. BOOM [and others] // JOURNAL OF CLINICAL MICROBIOLOGY. – 1990. – Vol. 28, No. 3. – p. 495-503]. Принцип метода заключается в обработке биологического материла хаотропными агентами – высококонцентрированными растворами в присутствии суспензии силики (двуокись кремния). Хаотропный агент растворяет клетки и вирусные частицы, приводя к высвобождению нуклеиновых кислот в раствор, которые под действием того же хаотропного агента избирательно сорбируются на поверхности силики. Лизирующий раствор включает в себя 140М GuSCN, 0.1М Tris-HCl, 20% 0.2М EDTA, 16.2М Triton Х-100. Ингибиторы и другие компоненты клинического материала остаются в растворе. С помощью центрифугирования силика с ДНК осаждаются, а супернатант с ингибиторами ПЦР удаляется. Серия последующих отмывок обеспечивает получение высоко очищенного препарата ДНК, который состоит из 140М GuSCN и 0.1М Tris-HCl с последующей сушкой. Элюция НК осуществляется в ТЕ-буфере и в дальнейшем используют в ПЦР. К недостаткам данного метода можно отнести высокую стоимость сорбента и ограниченный количественный выход НК в связи с ограничением активной площади сорбирующего вещества. А также наличие большого количества внеядерного генома и поврежденных НК.
Из уровня техники известен [RU2672378 С1, 14.11.2018] метод выделения ДНК из растений, пригодный для постановки ПЦР. Осуществляют гомогенизацию исследуемого образца в нелизирующем экстрагирующем буфере, содержащем ТЕ-буфер и поливинилпирролидон (ПВП). Далее проводят первичный лизис с использованием TRITON Х-10. Ресуспендируют осадок в ТЕ-буфере с ПВП с последующим центрифугированием. Удаляют супернатант. Этап повторяют 2-3 раза. Затем к осадку добавляют лизирующий раствор, содержащий GuSCN, DTT, EDTA, TRIS-HCI, перемешивают и добавляют протеиназу К и/или РНКазу А. Далее проводят экстракцию хлороформом и концентрирование ДНК изопропанолом. После осаждения ДНК осадок промывают двукратно, сначала 70% этанолом, а затем ацетоном. Высушивают и элюируют ДНК в ТЕ-буфере. Чистота выделенной ДНК, свободной от ингибиторов ПЦР, относительно белка А260/А280 составляет 2,0±0,09. Изобретение позволяет получить раствор ДНК высокого качества и высокой концентрации преимущественно ядерного генома, пригодный для лабораторных исследований, в том числе проведения ПЦР. Данный метод не адаптирован для выделения ДНК из грибов, бактерий или клеток животных.
Предложенное в [RU2185440 С2, 20.07.2002] изобретение относится к методу выделения ДНК, и может быть использовано в лабораторной и исследовательской практике для выделения высокомолекулярных ДНК или их фрагментов (макрорестриктов) из клеток дрожжей и грамположительных микроорганизмов, пригодных для их последующего анализа с помощью техники пульс-электрофореза, а также при постановке полимеразной цепной реакции (ПЦР) и для клонирования. Дрожжи или грамположительные бактерии обрабатывают буферным раствором с нейтральным значением рН, содержащим соль – хаотроп. Соль выбирают из группы производных гуанидиния в концентрации 4-8 М. Клетки инкубируют в экстракционном буфере при температуре на 5-10°С ниже температуры плавления ДНК в данном буфере. Затем выделяют геномную ДНК, прочно ассоциированную с клеточными оболочками микроорганизмов. Проводят гидролиз компонентов клеточных стенок микроорганизмов литическими ферментами. Изобретение позволяет упростить процедуру, снизить трудоемкость и удешевить способ выделения интактных геномных ДНК из клеток дрожжей и других микроорганизмов с прочной клеточной стенкой за счет того, что депротеинизация и очистка ДНК от белков, липидов и полисахаридов, а также низкомолекулярных компонентов проводится без предварительного разрушения клеточных оболочек.
В способе выделения ДНК микроорганизмов, раскрытом в [RU2230120 С2, 10.06.2004], ДНК выделяют путем разрушения микробных клеток лизирующим раствором, содержащим 80% фенола, 18,8% Н2О и 1,2% NaOH (рН 9,3-9,5). Способ позволяет получить препараты микробиальной ДНК высокой степени чистоты и нативности. Метод не пригоден для лизиса клеток растений.
В способе выделения ДНК из грамположительных и грамотрицательных бактерий и дрожжей [Cheng H. R., Jiang N. Extremely rapid extraction of DNA from bacteria and yeasts // Biotechnology letters. – 2006. – Т. 28. – №. 1. – С. 55-59] в качестве единственного лизирующего агента выступает фенол. К образцу, помещенному в пробирку, добавляют 200 мкл TE буфера (10 мМ Tris-HCl, 1 мМ EDTA, pH 8,0), стеклянные крошки (при выделении ДНК из дрожжей) и 100 мкл фенола. Затем пробирки встряхивают на встряхивателе в течение 60 с для бактерий и 120-200 с для дрожжей и центрифугируют при 13000g в течение 5 мин при 4°C для разделения фаз. Водную фазу отбирают, добавляют 0,5 объема хлороформа и центрифугируют 5 мин при 13000g и 4°C. Очистка хлороформом может повторяться до тех пор, пока не исчезнет белый осадок в интерфазе. После этого к раствору добавляют 5 мкл РНКазы (10 мг/мл) и инкубируют 10 мин при 4°C. Повторяют очистку хлороформом. После чего водный раствор ДНК готов для использования в дальнейших экспериментах. Вышеуказанный метод обладает рядом недостатков:
1) Отказ от детергентов приводит к снижению чистоты образца и снижению эффективности лизиса,
2) Встряхивание клеток на встряхивателе является недостаточно эффективным методом механического воздействия для лизиса,
3) Многократные этапы промывания образцов в хлороформе неизбежно приводят к потерям части водной фазы,
4) Отсутствие этапа осаждения ДНК делает невозможным ее концентрирование в необходимом объеме.
Предложен способ выделения ДНК из костного материала [RU2724506 С1, 23.06.2020], который позволяет повысить качество получаемого препарата нуклеиновых кислот и сократить время его получения. Согласно заявленному способу измельчение костного материала проводят в среде жидкого азота, приготовление лизата осуществляют путем инкубации в термостате с последующим центрифугированием, причем инкубацию осуществляют в два этапа, где на первом этапе навеску измельченного костного материала с добавлением буфера 50 мМ Tris-HCl, 50 мМ EDTA, рН 8,0, 1% SDS и 20 мкл протеиназы К, инкубируют с перемешиванием в течение 8-12 часов при температуре 56°С, на втором этапе добавляют буфер 4М Gu-HCl и инкубируют в течение 10 минут при температуре 70°С, затем проводят стадию отделения супернатанта центрифугированием, выделение нуклеиновых кислот из лизата осуществляют путем пропускания лизата через силикатную мембрану, отмывку осуществляют путем добавления в центр силикатной мембраны буфера 10 мМ Tris-HCl, рН 7,5, 80% этанол и последующего ее центрифугирования, при этом отмывку осуществляют дважды, а элюцию осуществляют путем добавления в центр силикатной мембраны буфера 10 мМ Tris-HCl, 0,5 мМ EDTA, рН 8,0 и инкубирования при комнатной температуре в течение 5 минут, с последующим центрифугированием силикатной мембраны. Метод не предназначен для выделения из растительной, грибной или бактериальной биомассы.
Изобретение, описанное в [RU2659197 С2, 28.06.2018], может быть использовано в лабораторной диагностике туберкулеза легких, его сущность – способ выделения ДНК клинических изолятов Mycobacterium tuberculosis из ткани легкого – заключается в добавлении к образцу ткани легкого, находящемуся в пробирке, содержащей стеклянные шарики, деконтаминирующего лизирующего буфера состава 5 М GuSCN, 100 мМ TRIS HCl рН 8.0, 10 мМ EDTA рН 8.0, 100 мМ NaCl, 0,5% SDS, с последующими центрифугированием и осаждением спиртом. Изобретение обеспечивает высокую эффективность выделения ДНК, характеризующуюся концентрацией образца в среднем 116,031 нг/мкл, высокое качество выделения ДНК, характеризующееся отношением А260/А280, равным 1,83, и быстроту процесса выделения ДНК. Метод не предназначен для выделения из растительной или грибной биомассы. Кроме того, данный метод требует предварительного перемалывания образца в жидком азоте.
Описан [RU2485178 С2, 20.06.2013] способ выделения ДНК из биологических объектов на предметах-носителях – марле, бумаге, синтетических тканях. Измельчают биологический объект – костную ткань, роговые ткани. Помещают в пробирку с лизирующим буферным раствором состава 0,1 М Трис-HCl, 0,1 М ЭДТА, 0,1 М NaCl, 0,5% N-лаурилсаркозил Na и протеиназы K, рН 6-7. Получают клеточный лизат и к нему добавляют сорбент магнитных наночастиц оксида железа Fе3О4, модифицированных хитозаном. Смесь перемешивают и инкубируют в течение 25-35 мин. Пробирку помещают на магнитный штатив и разделяют смесь на фракции – сорбент, связанный с ДНК, и надосадочная жидкость. Надосадок удаляют. К осадку приливают элюирующий буферный раствор состава: 10 мМ трис-HCl, рН 7,4; 100 мМ NaCl; 1 мМ ЭДТА и инкубируют. Пробирки помещают на магнитный штатив. Разделяют смесь на фракции сорбент - осадок и надосадок - ДНК, растворенная в элюирующем буферном растворе. Осадок удаляют. В надосадке остается конечный продукт ДНК. Изобретение позволяет получить ДНК из различных биологических объектов и увеличить выход выделяемой ДНК не менее чем в 1,5 раза. Метод требует наличия специального оборудования (магнитного штатива) и применение специальных магнитных наночастиц оксида железа Fе3О4, модифицированных хитозаном, которые не являются общедоступным реактивом.
Из [RU2129610 С1, 27.04.1999] известен способ выделения ДНК из микроорганизмов и животных клеток (крови, тканей, органов) с целью последующей ПЦР-амплификации специфического участка изолированной ДНК. Для выделения ДНК исследуемый образец в количестве 5-10 мкл помещают в 182 мкл лизирующе-осаждающего раствора, включающего в себя 38 мкл лизирующего раствора: 3,45 М NaOH 0,36 M NaCl и 144 мкл 96% этанола, перемешивают и оставляют при комнатной температуре в течение 10-15 мин. Затем центрифугируют в течение 5 мин при 12000-14000 об/мин. Супернатант отбрасывают, а осадок промывают 500 мкл 80% этанола однократно. Затем вновь центрифугируют 2 мин при 12000-14000 об/мин, супернатант отбрасывают, а осадок сушат при комнатной температуре в течение 5-10 мин. К сухому осадку добавляют 15-30 мкл ТЕ-буфера и перемешивают. В результате чего достигаются эффективность выделения ДНК за счет лизиса клеток, обладающих клеточной стенкой, устойчивой к действию большинства лизирующих агентов, удешевление способа за счет использования доступных и недорогих реактивов, снижение трудоемкости способа путем сокращения этапов способа, экологическая безопасность за счет исключения использования токсических химреактивов.
Предшествующий уровень техники показывает, что описанные способы выделения ДНК не обладают универсальностью, не предназначены для выделения ДНК из образцов почв, растений, бактерий, грибов, или клеток животных. Кроме того, имеющиеся методы, как правило, достаточно трудоемки, имеют много этапов.
В связи с вышесказанным техническая проблема, на решение которой направлено изобретение, состоит в расширении арсенала способов выделения ДНК универсальным способом, подходящим для выделения ДНК из различных образцов – почвы, клеток грамположительных и грамотрицательных бактерий, грибов, растений и животных клеток, позволяющим при этом сохранить высокие значения концентрации и чистоты конечного продукта и уменьшить количество этапов при выделении ДНК, что способствует сокращению времени и снижению трудозатратности способа. Также изобретение решает проблему расширения ассортимента средств для выделения ДНК.
Технический результат состоит в реализации указанного назначения, результативном выделении ДНК из различных образцов за счет повышения эффективности процессов разрушения клеток и десорбции биополимеров, в том числе лизиса клеток, обладающих клеточной стенкой, устойчивой к действию большинства лизирующих агентов.
Указанная техническая проблема решается, и технический результат достигается тем, что в качестве детергента используется лизирующая смесь –буфер ЦТАБ (2% ЦТАБ, 1,4 M NaCl, 20 мM ЭДТА, 100 мM Tris-HCl pH=8,0), способствующий более эффективному лизису клеток, в смеси с фенолом в объемном соотношении буфер ЦТАБ : фенол 1-1,5:1.
Заявляемая лизирующая смесь способствует эффективности процессов разрушения клеток и десорбции биополимеров, в том числе лизиса клеток, обладающих клеточной стенкой, устойчивой к действию большинства лизирующих агентов, и подходит для выделения ДНК из различных образцов – почвы, клеток грамположительных и грамотрицательных бактерий, грибов, растений и животных клеток, при сохранении высокого значения выделяемой ДНК и ее чистоты. Указанное соотношение компонентов в лизирующей смеси является оптимальным, поскольку обеспечивает качественный лизис клеток и очистку препарата. Меньшее количество ЦТАБ приводит к менее качественному разделению ДНК и белков, в то время как меньшее количество фенола приводят к загрязнению проб белками из-за недостаточной депротеинизации, большее количество фенола приводит к загрязнению проб ДНК фенолом.
Также указанная техническая проблема решается, и технический результат достигается заявляемым способом выделения ДНК из образца почвы, грибов, бактерий, растений и животных клеток, включающим гомогенизацию образца путем последовательных этапов промораживания и нагрева образца в лизирующей смеси, содержащей ЦТАБ (2% ЦТАБ, 1,4 M NaCl, 20 мM ЭДТА, 100 мM Tris-HCl pH=8,0) с фенолом в объемном соотношении 1-1,5:1 соответственно, при объемном соотношении образец : лизирующая смесь 1:6,5-10,0, экстракцию хлороформом, добавляемым непосредственно в лизирующую смесь, с последующим центрифугированием, осаждением ДНК изопропанолом и промыванием осадка ДНК этанолом.
Заявленное соотношение образца и лизирующей смеси оптимально, поскольку меньшее количество лизирующей смеси приводит к снижению чистоты ДНК, а большее – к загрязнению проб ДНК фенолом.
Экстракцию хлороформом преимущественно проводят при добавлении хлороформа, в два раза по объему превышающего количество фенола в лизирующей смеси для эффективного разделения фаз и удаления фенола.
В указанной пропорции выделение с использованием стандартных пробирок типа «Eppendorf» объемом 2,1 мл может быть исполнено в следующих объемах – образец – 100 мкл, ЦТАБ – 500 мкл, фенол – 350 мкл, хлороформ – 700 мкл.
В заявляемом способе для более эффективного лизиса образцы замораживают при температуре (-80)-(-40)°C в ЦТАБ-фенольной смеси с последующим прогреванием при 80-95°C. В отличие от большинства существующих методов, хлороформ добавляют непосредственно в ЦТАБ-фенольную смесь, после чего следует этап разделения фаз, что значительно сокращает время и количество используемых пробирок. Отсутствие многократного промывания образца в хлороформе позволяет избежать потерь водной фазы, а значит, количественных потерь ДНК. Осаждение ДНК в изопропаноле и промывка этиловым спиртом позволяют получить более концентрированные и чистые от примесей образцы. Кроме того, время встряхивания образцов на встряхивателе сокращается в 4 раза в случае с бактериальными клетками.
При реализации изобретения используют следующие реактивы и оборудование:
ЦТАБ (ДиаЭМ, РФ)
Фенол (ДиаЭМ, РФ)
Хлорофом (Реахим, РФ)
Изопропанол (Реахим, РФ)
Этанол (Реахим, РФ)
Деионизованная вода – получена с помощью установки АквалабХХ
Степень чистоты всех реактивов, использованных в экспериментах – «ч.д.а.».
Пробирки типа «Eppendorf» объемом 1,5 мл (SSI, Китай).
Встряхиватель (D-lab, КНР)
Морозильник MHT(-80°С), (МТН, Россия)
Термостат твердотельный Гном (ДНК-технологии, РФ)
центрифуга MiniSpin Plus Eppendorf (Eppendorf, ФРГ)
Спектрофотометр NanoPhotometer® P 360 Implen (Thermo, США).
Все образцы были отобраны для выделения в количестве 0.1 мг. Образцы почвы были отобраны с корней пшеницы сорта «Универсиада» и помещены в лизирующую смесь. Образцы мицелия гриба Fusarium oxysporum ZUM2704 были получены лабораторно при выращивании на чашке Петри со средой Сабуро (Difco, США), собраны микробиологической петлей и помещены в лизирующую смесь. Биомасса бактериального штамма Bacillus mojavensis PS17 (коллекция Лаборатории молекулярно-генетических и микробиологических методов ФИЦ КазНЦ РАН) получена при выращивании в жидкой среде LB (Difco, США), осаждена центрифугированием при скорости не менее 5 000 об/мин. Супернатант был вылит, а к осадку приливали лизирующий раствор. Для выделения растительной ДНК использовали недельные растения томатов сорта «Дубрава» (Гавриш, РФ). Для выделения ДНК животных использовали культуру клеток линии свиньи (SPEV), почка эмбриона, компании (Биолот, РФ).
Заявляемый способ осуществляют следующим образом:
В пробирку помещают ЦТАБ 2%-буфера (2% ЦТАБ, 1,4 M NaCI, 20 мM ЭДТА, 100 мM Tris-HCl pH=8,0) и фенол в объемном соотношении ЦТАБ : фенол 1-1,5:1, туда же помещают образец – биомассу или образец почвы – в объемном соотношении образец : лизирующая смесь 1:6,5-10,0, тщательно встряхивают на встряхивателе и замораживают при (-80)-(-40)°C. Далее инкубируют 5-10 мин при 80-95°C, снова встряхивают на встряхивателе. Затем в пробирку вносят хлороформ по объему в два раза больше, чем фенола. Смесь встряхивают на встряхивателе, затем центрифугируют на центрифуге до полного разделения фаз. Отбирают водную фазу в новую пробирку и добавляют к ней изопропанол. Аккуратно перемешивают и центрифугируют до получения осадка ДНК. Осадок промывают этанолом для удаления солей, воды и изопропанола, после чего осадок нуклеиновых кислот растворяют в деионизованной воде. Концентрацию ДНК определяют спектрофотометрическим способом при помощи спектрофотометра NanoPhotometer® P 360 (Implen).
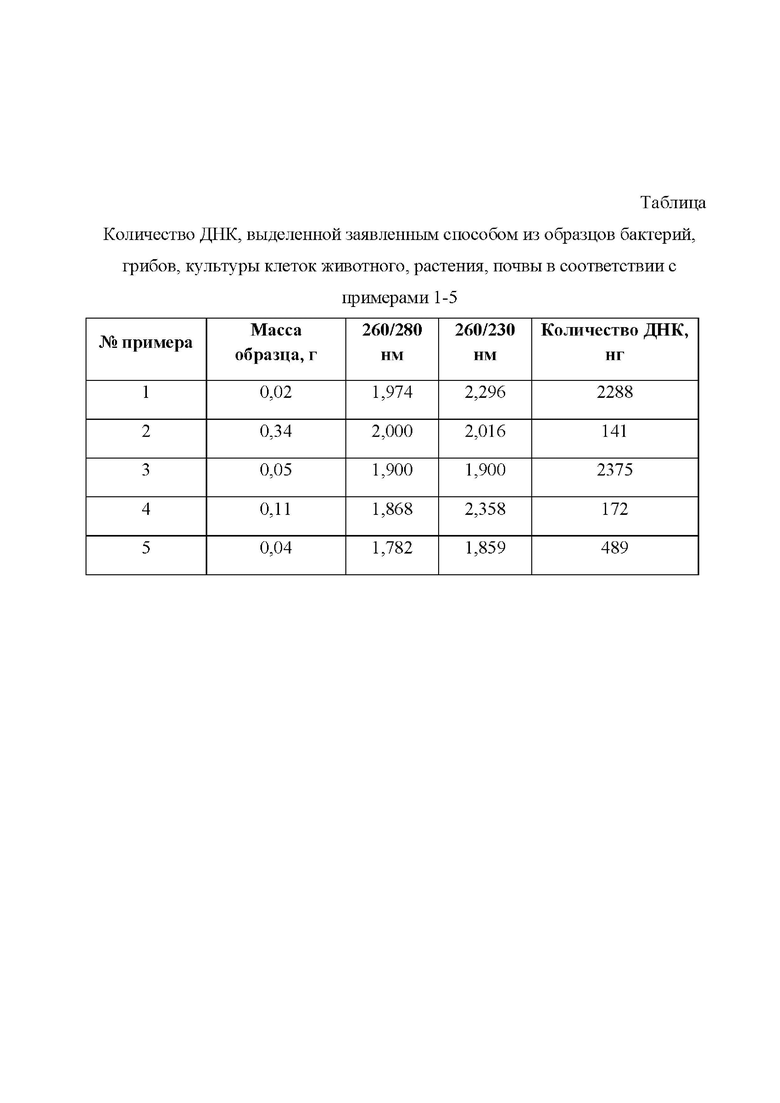
В таблице приведено количество (в нг) ДНК, выделенной из образцов бактерий, грибов, культуры клеток животного, растения, почвы заявленным способом в соответствии с примерами, иллюстрирующими изобретение. Данные таблицы показывают, что заявляемый универсальный способ обеспечивает выход ДНК от 141 нг/мкл до 2375 нг/мкл (2288 для бактериальной культуры, 141 для культуры гриба, 2375 для культуры клеток животного, 172 для растения, 489 для почвы) и чистоте, равной 1,782-2,000 при отношении 260/280 нм и 1,859-2,358 при отношении 260/230 нм из 1 г образца. Отношение показателей поглощения 260/280 служит надежным индикатором загрязнения белками: значение ≥ 1,8 указывает на чистый образец ДНК. Отношение показателей поглощения 260/230 меньше 1,8 указывает на загрязнение, вероятно, вызванное органическими соединениями или хаотропными агентами, которые поглощают свет на длине волны 230 нм.
Для сравнения чистоты образцов производят выделение ДНК с помощью стандартного метода для бактерий [Wright, M.H., Adelskov, J. and Greene, A.C., 2017. Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. Journal of microbiology & biology education, 18(2), pp.18-2]. Средняя чистота образцов ДНК, полученных указанным методом, составила 1,680 (соотношение измерений 260/280 нм).
Как видно из данных, приведенных в таблице, заявляемый способ обеспечивает высокие значения эффективности выделения ДНК, сохраняя относительную быстроту и дешевизну.
Для сравнения эффективности амплификации проводят ПЦР-РВ для ДНК, выделенных заявляемым способом и способом сравнения. Для этого реакцию проводят на образцах с пошаговым разведением в 10 раз (от 10 нг/мкл ДНК на реакцию до 0,001 нг/мкл). Стандартные кривые ПЦР-РВ бактериальной ДНК, полученной способом сравнения – стандартным – и заявляемым способом, приведены на фигурах 1 и 2 соответственно. Эффективность реакции ПЦР-РВ (Е) с ДНК, выделенной стандартным и заявленным способами, составила 104,7 % и 110,2 % соответственно, с наклоном кривой – 3,213 – для реакции ПЦР-РВ с ДНК, выделенной стандартным способом и – 3,099 – заявляемым способом, что соответствует приемлемым уровням эффективности ПЦР реакции – 90-110% [Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M.W. and Kubista, M., 2015. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular detection and quantification, 3, pp.9-16; Tellinghuisen, J. and Spiess, A.N., 2015. Bias and imprecision in analysis of real-time quantitative polymerase chain reaction data. Analytical chemistry, 87(17), pp.8925-8931].
Изобретение иллюстрируется примерами конкретного выполнения.
Пример 1. Выделение ДНК из образца бактериальной культуры Bacillus mojavensis PS17 заявленным способом
В пробирку объемом 2 мл с 500 мкл ЦТАБ 2%-буфера (2% ЦТАВ, 1,4 M NaCI, 20 мM ЭДТА, 100 мM Tris-HCl pH=8,0) и 350 мкл фенола помещают образец бактериальной культуры 100 мкл, тщательно встряхивают на встряхивателе 15 с и замораживают при -80°C. Далее инкубируют 10 мин при 80°C, снова встряхивают на встряхивателе 15 с. Затем в пробирку вносят 700 мкл хлороформа. Смесь встряхивают на встряхивателе 5 с, затем центрифугируют на центрифуге MiniSpin Plus Eppendorf в течение 5 минут при ускорении 14000 g. Отбирают водную фазу и добавляют к ней 0,8 объема изопропанола. Аккуратно перемешивают и центрифугируют в течение 5 минут при ускорении 14000 g. Осадок 2 раза промывают 70% этанолом (об./об.) и растворяют в 50 мкл деионизованной воды. Концентрацию ДНК определяют спектрофотометрическим способом при помощи спектрофотометра NanoPhotometer® P 360 (Implen).
Пример 2. Выделение ДНК из образца Fusarium oxysporum ZUM2704 заявленным способом
Пример 2 осуществляют в условиях примера 1, за исключением того, что выделение ДНК осуществляют из образца гриба Fusarium oxysporum ZUM2704.
Пример 3. Выделение ДНК из образца культуры клеток животного – свиньи линии (SPEV), почка эмбриона, (Биолот, РФ) заявленным способом
Пример 3 осуществляют в условиях примера 1, за исключением того, что выделение ДНК осуществляют из образца культуры клеток животного – свиньи линии (SPEV), почка эмбриона, (Биолот, РФ).
Пример 4. Выделение ДНК из образца проростка томатов (недельные растения томатов сорта «Дубрава» (Гавриш, РФ)) заявленным способом
Пример 4 осуществляют в условиях примера 1, за исключением того, что выделение ДНК осуществляют из образца проростка томатов и используют лизирующую смесь при объемном соотношении ЦТАБ : фенол, равном 1:1, и объемном отношении образец : лизирующая смесь 1:6,7.
Пример 5. Выделение ДНК из образца почвы, отобранного с корней пшеницы сорта «Универсиада», заявленным способом
Пример 5 осуществляют в условиях примера 1, за исключением того, что выделение ДНК осуществляют из образца почвы.
Таким образом предложены лизирующая смесь и универсальный способ, подходящий для выделения ДНК из различных образцов – почвы, клеток грамположительных и грамотрицательных бактерий, грибов, растений и животных клеток, позволяющий при этом сохранить высокие значения концентрации и чистоты выделяемой ДНК и уменьшить количество этапов при выделении, что способствует сокращению времени и снижению трудозатратности способа. Предложенные технические решения расширяют арсенал средств и способов выделения ДНК.
Изобретение обладает универсальностью в сочетании с дешевизной, простотой и скоростью, какими не обладает ни один из известных способов для выделения ДНК из образцов почвы, клеток грамположительных и грамотрицательных бактерий, грибов, растений и животных. Заявляемый способ выделения ДНК пригоден для постановки ПЦР.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ВЫДЕЛЕНИЯ ДНК COCCIDIOIDES IMMITIS ДЛЯ ПРОВЕДЕНИЯ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2005 |
|
RU2295569C1 |
| СПОСОБ ВЫДЕЛЕНИЯ ТОТАЛЬНОЙ ДНК БАКТЕРИЙ ИЗ ОБРАЗЦОВ ПОЧВЫ, СПОСОБ ОЦЕНКИ БАКТЕРИАЛЬНОГО СОСТАВА ПОЧВ ПОСРЕДСТВОМ МЕТАГЕНОМНОГО СЕКВЕНИРОВАНИЯ И НАБОРЫ ДЛЯ ОСУЩЕСТВЛЕНИЯ СПОСОБОВ | 2024 |
|
RU2829656C1 |
| Способ выделения ДНК из почвы | 2018 |
|
RU2696052C1 |
| Способ выделения ДНК из растений, пригодный для постановки ПЦР | 2017 |
|
RU2672378C1 |
| СПОСОБ ВЫДЕЛЕНИЯ И ОЧИСТКИ ДЕЗОКСИРИБОНУКЛЕИНОВЫХ КИСЛОТ | 2008 |
|
RU2400537C2 |
| СПОСОБ ВЫДЕЛЕНИЯ НУКЛЕИНОВЫХ КИСЛОТ | 2014 |
|
RU2584346C2 |
| Бесклеточная система синтеза белка на основе клеток Staphylococcus aureus, способ синтеза белка на основе клеток Staphylococcus aureus с использованием бесклеточной системы синтеза белка на основе клеток Staphylococcus aureus и способ выявления ингибиторов синтеза белка с ее использованием | 2022 |
|
RU2802080C1 |
| СПОСОБ ВЫСОКОПРОИЗВОДИТЕЛЬНОГО ВЫДЕЛЕНИЯ ДНК ИЗ ПРОРОСТКОВ СОИ | 2024 |
|
RU2833968C1 |
| СПОСОБ ВЫДЕЛЕНИЯ ПЛАЗМИД BURKHOLDERIA PSEUDOMALLEI | 2002 |
|
RU2218401C2 |
| СПОСОБ ВЫДЕЛЕНИЯ ДНК ИЗ МИКРООРГАНИЗМОВ И КЛЕТОК ЖИВОТНЫХ, ПРИГОДНОЙ ДЛЯ ПОСТАНОВКИ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 1997 |
|
RU2129610C1 |

Изобретение относится к области биотехнологии. Описана группа изобретений, включающая лизирующую смесь для выделения ДНК из образцов бактерий, грибов, растений, животных и почвы, и способ выделения ДНК из образцов бактерий, грибов, растений, животных и почвы. Лизирующая смесь для выделения ДНК, представляет из себя смесь буфера ЦТАБ, содержащего 2% ЦТАБ, 1,4 M NaCl, 20 мM ЭДТА, 100 мM Tris-HCl, pH=8,0, с фенолом в объемном соотношении 1-1,5:1, соответственно. Изобретение расширяет арсенал средств для выделения ДНК из образцов бактерий, грибов, растений, животных и почвы. 2 н. и 4 з.п. ф-лы, 2 ил., 1 табл., 5 пр.
1. Лизирующая смесь для выделения ДНК из образцов бактерий, грибов, растений, животных и почвы, представляющая собой смесь буфера ЦТАБ, содержащего 2% ЦТАБ, 1,4 M NaCl, 20 мM ЭДТА, 100 мM Tris-HCl, pH=8,0, с фенолом в объемном соотношении 1-1,5:1 соответственно.
2. Способ выделения ДНК из образцов бактерий, грибов, растений, животных и почвы, включающий гомогенизацию образца путем последовательных этапов промораживания и прогрева образца в лизирующей смеси, включающей буфер ЦТАБ, содержащий 2% ЦТАБ, 1,4 M NaCl, 20 мM ЭДТА, 100 мM Tris-HCl pH=8,0, с фенолом в объемном соотношении 1-1,5:1 соответственно, при объемном соотношении образец : лизирующая смесь 1:6,5-10, экстракцию хлороформом, добавляемым непосредственно в лизирующую смесь, с последующим центрифугированием, осаждением ДНК изопропанолом и промыванием осадка ДНК этанолом.
3. Способ по п. 2, отличающийся тем, что этап промораживания образца в лизирующей смеси осуществляют при температуре (-80)-(-40)°C.
4. Способ по п. 2, отличающийся тем, что этап прогревания образца в лизирующей смеси осуществляют при температуре при 80-95°C.
5. Способ по п. 2, отличающийся тем, что для экстракции преимущественно используют количество хлороформа, в два раза по объему превышающего количество фенола в лизирующей смеси.
6. Способ по п. 2, отличающийся тем, что промывание осадка выделенной ДНК осуществляют 70% этанолом, предпочтительно дважды.
| Minas K | |||
| et al | |||
| Optimization of a high-throughput CTAB-based protocol for the extraction of qPCR-grade DNA from rumen fluid, plant and bacterial pure cultures //FEMS microbiology letters, 2011, vol | |||
| Водяной двигатель | 1921 |
|
SU325A1 |
| Деревянное стыковое скрепление | 1920 |
|
SU162A1 |
| Dairawan M., Shetty P | |||
| J | |||
| The evolution of DNA extraction methods //Am | |||
| J | |||
| Biomed | |||
| Sci | |||
| Res., 2020, vol | |||
| Топка с несколькими решетками для твердого топлива | 1918 |
|
SU8A1 |
| Машина для изготовления проволочных гвоздей | 1922 |
|
SU39A1 |
| (p | |||
| Устройство для усиления микрофонного тока с применением самоиндукции | 1920 |
|
SU42A1 |
Авторы
Даты
2023-11-13—Публикация
2022-12-26—Подача