Ссылка на родственные заявки
[1] Согласно настоящей заявке испрашивается приоритет в соответствии с предварительной заявкой США № 62/827018, поданной 30 марта 2019 года, которая полностью включена в данный документ посредством ссылки.
Предшествующий уровень техники настоящего изобретения
[2] Адоптивная иммунотерапия или адоптивная клеточная терапия лимфоцитами (ACT) представляет собой перенос генетически модифицированных T-лимфоцитов субъекту для лечения заболевания. Адоптивная иммунотерапия еще не реализовала свой потенциал для лечения широкого спектра заболеваний, включая рак, инфекционное заболевание, аутоиммунное заболевание, воспалительное заболевание и иммунодефицит. Однако для большинства, если не для всех стратегий адоптивной иммунотерапии требуются этапы активации и размножения Т-клеток для получения клинически эффективной терапевтической дозы T-клеток. Существующие стратегии получения клеток пациентов и активации, размножения и извлечения ex vivo эффективного количества клеток для ACT являются длительным, громоздким и сложным по своей сути процессом и представляют собой серьезную проблему. Соответственно, остается потребность в разработке композиций и способов размножения и индукции антигенспецифических T-клеток с благоприятным фенотипом и функцией и в течение более короткого периода времени.
Сущность настоящего изобретения
[3] В данном документе представлен способ лечения рака у нуждающегося в этом субъекта, предусматривающий: выбор по меньшей мере одной эпитопной последовательности из библиотеки эпитопных последовательностей, причем каждая эпитопная последовательность в библиотеке соответствует белку, кодируемому аллелем HLA субъекта; и введение в контакт T-клетки субъекта или аллогенной T-клетки с одним или несколькими пептидами, содержащими по меньшей мере одну выбранную эпитопную последовательность, причем каждую по меньшей мере одну выбранную эпитопную последовательность предварительно проверяют на соответствие по меньшей мере трем следующим критериям: связывается с белком, кодируемым аллелем HLA субъекта, является иммуногенной согласно анализу иммуногенности, презентируются антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности.
[4] В некоторых вариантах осуществления по меньшей мере одна выбранная эпитопная последовательность содержит мутацию, а способ предусматривает идентификацию раковых клеток субъекта на кодирование эпитопа с мутацией; по меньшей мере одна выбранная эпитопная последовательность находится в белке, сверхэкспрессируемом раковыми клетками субъекта, а способ предусматривает идентификацию раковых клеток субъекта на сверхэкспрессию белка, содержащего эпитоп; или по меньшей мере одна эпитопная последовательность содержит белок, экспрессируемый клетками в микросреде опухоли.
[5] В некоторых вариантах осуществления одна или более из по меньшей одной выбранной эпитопной последовательности содержит эпитоп, который не экспрессируется раковыми клетками субъекта.
[6] В некоторых вариантах осуществления эпитоп, который не экспрессируется раковыми клетками субъекта, экспрессируют клетки в микросреде опухоли субъекта.
[7] В некоторых вариантах осуществления эпитоп, который связывается с белком, кодируемым аллелем HLA субъекта, связывается с молекулой MHC, кодируемой аллелем HLA, с аффинностью 500 нМ или менее согласно анализу связывания.
[8] В некоторых вариантах осуществления связывание эпитопа, который связывается с белком, кодируемым аллелем HLA субъекта, с молекулой MHC, кодируемой аллелем HLA, с аффинностью 500 нМ или менее прогнозируют с использованием программы прогнозирования эпитопа MHC, выполняемой на компьютере.
[9] В некоторых вариантах осуществления программой прогнозирования эпитопа MHC, выполняемой на компьютере, является NetMHCpan. В некоторых вариантах осуществления программой прогнозирования эпитопа MHC, выполняемой на компьютере, является NetMHCpan версия 4.0.
[10] В некоторых вариантах осуществления эпитоп, который презентируют антигенпрезентирующие клетки (APC) согласно масс-спектрометрическому анализу, обнаруживают посредством масс-спектрометрии после элюирования из APC с точностью массы обнаруженного пептида, составляющей менее 15 Да, 10 Да или 5 Да или менее 10000 или 5000 частей на миллион (ч/млн).
[11] В некоторых вариантах осуществления эпитоп, который является иммуногенным согласно анализу иммуногенности, является иммуногенным согласно мультимерному анализу или функциональному анализу.
[12] В некоторых вариантах осуществления мультимерный анализ представляет собой анализ проточной цитометрии.
[13] В некоторых вариантах осуществления мультимерный анализ включает обнаружение T-клеток, связанных с мультимером пептид-MHC, содержащим по меньшей мере одну выбранную эпитопную последовательность и соответствующий аллель HLA, причем T-клетки были стимулированы APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность.
[14] В некоторых вариантах осуществления эпитоп является иммуногенным согласно мультимерному анализу (i) при обнаружении по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,1% или 0,01% или 0,005% проанализированных CD8+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ T-клеток, обнаруженных в контрольном образце.
[15] В некоторых вариантах осуществления эпитоп является иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в одной из шести стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность.
[16] В некоторых вариантах осуществления контрольный образец содержит T-клетки, стимулированные APC, которые (i) не содержат пептида, содержащего по меньшей мере одну выбранную эпитопную последовательность, (ii) содержат пептид, полученный из другого белка, чем по меньшей мере одна выбранная эпитопная последовательность, или (iii) содержат пептид со случайной последовательностью.
[17] В некоторых вариантах осуществления T-клетки были стимулированы APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, в течение по меньшей мере 3, 4, 5, 6, 7, 8,9, 10, 11, 12, 13, 14, 15, 16, 7, 18, 19, 20 или более дней.
[18] В некоторых вариантах осуществления антигенспецифические T-клетки размножили по меньшей мере в 5, 10, 20, 50, 100, 500 или 1000 раз или более в присутствии APC, содержащих пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность.
[19] В некоторых вариантах осуществления функциональный анализ представляет собой иммуноанализ.
[20] В некоторых вариантах осуществления функциональный анализ включает обнаружение T-клеток с помощью внутриклеточного окрашивания IFNγ или TNFα или экспрессии CD107a и/или CD107b на поверхности клеток, причем T-клетки были стимулированы APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность.
[21] В некоторых вариантах осуществления эпитоп является иммуногенным согласно функциональному анализу (i) при обнаружении по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,1% или 0,01% или 0,005% проанализированных CD8+ или CD4+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ или CD4+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ или CD4+ T-клеток, обнаруженных в контрольном образце.
[22] В некоторых вариантах осуществления T-клетками со стимулированной цитотоксичностью согласно анализу цитотоксичности являются T-клетки, стимулированные APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, которые убивают клетки, презентирующие эпитоп.
[23] В некоторых вариантах осуществления количество презентирующих эпитоп клеток, уничтожаемых Т-клетками, по меньшей мере в 1,1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 50, 100, 500 или 1000 раз выше количества не презентирующих эпитопа клеток, уничтожаемых Т-клетками.
[24] В некоторых вариантах осуществления количество презентирующих эпитоп клеток, уничтожаемых Т-клетками, по меньшей мере в 1,1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 50, 100, 500 или 1000 раз выше количества презентирующих эпитоп клеток, уничтожаемых Т-клетками, стимулированными APC, которые (i) не содержат пептида, содержащего по меньшей мере одну выбранную эпитопную последовательность, (ii) содержат пептид, полученный из другого белка, чем по меньшей мере одна выбранная эпитопная последовательность, или (iii) содержат пептид со случайной последовательностью.
[25] В некоторых вариантах осуществления количество презентирующих мутантный эпитоп клеток, уничтожаемых Т-клетками, по меньшей мере в 1,1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 50, 100, 500 или 1000 раз выше количества презентирующих соответствующий эпитоп дикого типа клеток, уничтожаемых Т-клетками.
[26] В некоторых вариантах осуществления T-клетками со стимулированной цитотоксичностью согласно анализу цитотоксичности являются T-клетки со стимулированной специфической цитотоксичностью согласно анализу цитотоксичности.
[27] В некоторых вариантах осуществления способ предусматривает выбор субъекта с использованием анализа циркулирующей опухолевой ДНК.
[28] В некоторых вариантах осуществления способ предусматривает выбор субъекта с использованием панели генов.
[29] В некоторых вариантах осуществления T-клетку получают из биологического образца субъекта.
[30] В некоторых вариантах осуществления T-клетку получают из образца для афереза или для лейкафереза субъекта.
[31] В некоторых вариантах осуществления T-клетка является аллогенной T-клеткой.
[32] В некоторых вариантах осуществления каждую по меньшей мере одну выбранную эпитопную последовательность предварительно проверяют на соответствие каждому из следующих критериев: связывается с белком, кодируемым аллелем HLA субъекта, является иммуногенной согласно анализу иммуногенности, презентируются антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности.
[33] В некоторых вариантах осуществления по меньшей мере один из одного или нескольких пептидов представляет собой синтезированный пептид или пептид, экспрессированный из последовательности нуклеиновой кислоты.
[34] В некоторых вариантах осуществления способ предусматривает идентификацию белка, кодируемого аллелем HLA субъекта, или идентификацию аллеля HLA в геноме субъекта.
[35] В некоторых вариантах осуществления по меньшей мере одну выбранную эпитопную последовательность выбирают из одной или нескольких эпитопных последовательностей таблицы 1A-1F, таблицы 2A-2C, таблицы 3, таблицы 4A-4M, таблицы 5, таблицы 6, таблицы 7, таблицы 8, таблицы 11, таблицы 12, таблицы 13 и таблицы 14.
[36] В некоторых вариантах осуществления способ предусматривает размножение T-клетки, контактировавшей с одним или несколькими пептидами, in vitro или ex vivo для получения популяции T-клеток, специфичных по меньшей мере к одной выбранной эпитопной последовательности в комплексе с белком MHC.
[37] В некоторых вариантах осуществления способ дополнительно предусматривает введение популяции T-клеток субъекту.
[38] В некоторых вариантах осуществления белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, экспрессирует раковая клетка субъекта.
[39] В некоторых вариантах осуществления белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, экспрессируют клетки в микросреде опухоли субъекта.
[40] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит мутацию.
[41] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит опухолеспецифичную мутацию.
[42] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка, сверхэкспрессируемого раковой клеткой субъекта.
[43] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит драйверную мутацию.
[44] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит мутацию устойчивости к лекарственным средствам.
[45] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из тканеспецифического белка.
[46] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из раково-тестикулярного белка.
[47] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности является вирусным эпитопом.
[48] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности является минорным эпитопом гистосовместимости.
[49] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка RAS.
[50] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка GATA3.
[51] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка EGFR.
[52] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка BTK.
[53] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка p53.
[54] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из полипептида слияния TMPRSS2::ERG.
[55] В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка Myc.
[56] В некоторых вариантах осуществления по меньшей мере одну из по меньшей мере одной выбранной эпитопной последовательности получают из белка, кодируемого геном, выбранным из группы, состоящей из ANKRD30A, COL10A1, CTCFL, PPIAL4G, POTEE, DLL3, MMP13, SSX1, DCAF4L2, MAGEA4, MAGEA11, MAGEC2, MAGEA12, PRAME, CLDN6, EPYC, KLK3, KLK2, KLK4, TGM4, POTEG, RLN1, POTEH, SLC45A2, TSPAN10, PAGE5, CSAG1, PRDM7, TG, TSHR, RSPH6A, SCXB, HIST1H4K, ALPPL2, PRM2, PRM1, TNP1, LELP1, HMGB4, AKAP4, CETN1, UBQLN3, ACTL7A, ACTL9, ACTRT2, PGK2, C2orf53, KIF2B, ADAD1, SPATA8, CCDC70, TPD52L3, ACTL7B, DMRTB1, SYCN, CELA2A, CELA2B, PNLIPRP1, CTRC, AMY2A, SERPINI2, RBPJL, AQP12A, IAPP, KIRREL2, G6PC2, AQP12B, CYP11B1, CYP11B2, STAR, CYP11A1 и MC2R.
[57] В некоторых вариантах осуществления по меньшей мере одну из по меньшей мере одной выбранной эпитопной последовательности получают из тканеспецифического белка, уровень экспрессии которого в ткани-мишени субъекта по меньшей мере в 2 раза превышает уровень экспрессии тканеспецифического белка в каждой ткани из множества не являющихся мишенью тканей, которые отличаются от ткани-мишени.
[58] В некоторых вариантах осуществления введение в контакт T-клетки субъекта или аллогенной T-клетки с одним или несколькими пептидами, содержащими по меньшей мере одну выбранную эпитопную последовательность, предусматривает введение в контакт T-клетки с APC, презентирующими эпитоп.
[59] В некоторых вариантах осуществления APC, презентирующие эпитоп, содержат один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность или полинуклеиновую кислоту, которая кодирует один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность.
[60] В некоторых вариантах осуществления способ предусматривает истощение CD14+ клеток и CD25+ клеток из популяции иммунных клеток, содержащей антигенпрезентирующие клетки (APC) и T-клетки, образуя за счет этого популяцию иммунных клеток с истощенными CD14/CD25, содержащую первую популяцию APC и T-клеток.
[61] В некоторых вариантах осуществления популяцию иммунных клеток получают из биологического образца субъекта.
[62] В некоторых вариантах осуществления способ дополнительно предусматривает (b) инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей первую популяцию APC и T-клеток, в течение первого периода времени в присутствии лиганда рецептора FMS-подобной тирозинкиназы 3 (FLT3L) и (A) полипептида, содержащего по меньшей мере одну выбранную эпитопную последовательность, или (B) полинуклеотида, кодирующего полипептид; образуя за счет этого популяцию клеток, содержащую стимулированные T-клетки.
[63] В некоторых вариантах осуществления способ дополнительно предусматривает (c) размножение популяции клеток, содержащей стимулированные T-клетки, образуя за счет этого размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки, причем специфические в отношении опухолевых антигенов T-клетки включают в себя T-клетки, которые являются специфичными к комплексу, содержащему (i) по меньшей мере одну выбранную эпитопную последовательность и (ii) белок MHC, экспрессируемый раковыми клетками или APC субъекта.
[64] В некоторых вариантах осуществления T-клетки размножают менее 28 дней.
[65] В некоторых вариантах осуществления доля CD8+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD8+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, по меньшей мере в два раза превышает долю CD8+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD8+ T-клеток в биологическом образце.
[66] В некоторых вариантах осуществления доля CD4+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD4+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, по меньшей мере в два раза превышает долю CD4+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD4+ T-клеток в биологическом образце.
[67] В некоторых вариантах осуществления по меньшей мере 0,1% CD8+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD8+ T-клетки, полученные из наивных CD8+ T-клеток.
[68] В некоторых вариантах осуществления по меньшей мере 0,1% CD4+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD4+ T-клетки, полученные из наивных CD4+ T-клеток.
[69] В некоторых вариантах осуществления размножение предусматривает введение в контакт популяции клеток, содержащей стимулированные T-клетки, со второй популяцией зрелых APC, причем вторая популяция зрелых APC инкубирована с FLT3L и презентирует по меньшей мере одну выбранную эпитопную последовательность; и размножение популяции клеток, содержащей стимулированные T-клетки, в течение второго периода времени, образуя за счет этого размноженную популяцию T-клеток.
[70] В некоторых вариантах осуществления вторую популяцию зрелых APC инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт популяции клеток, содержащей стимулированные T-клетки, со второй популяцией зрелых APC.
[71] В некоторых вариантах осуществления размножение дополнительно предусматривает (C) введение в контакт размноженной популяции T-клеток с третьей популяцией зрелых APC, причем третья популяция зрелых APC (i) инкубирована с FLT3L и (ii) презентирует по меньшей мере одну выбранную эпитопную последовательность; и (D) размножение размноженной популяции T-клеток в течение третьего периода времени, образуя за счет этого размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки.
[72] В некоторых вариантах осуществления третью популяцию зрелых APC инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт размноженной популяции T-клеток с третьей популяцией зрелых APC.
[73] В некоторых вариантах осуществления биологический образец представляет собой образец периферической крови, образец для лейкафереза или образец для афереза.
[74] В некоторых вариантах осуществления способ дополнительно предусматривает сбор размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, криоконсервацию размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, или получение фармацевтической композиции, содержащей размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки.
[75] В некоторых вариантах осуществления инкубация представляет собой инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей первую популяцию APC и T-клеток, в течение первого периода времени в присутствии FLT3L и РНК, кодирующей полипептид.
[76] В некоторых вариантах осуществления способ дополнительно предусматривает введение субъекту-человеку с раком фармацевтической композиции, содержащей размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки.
[77] В некоторых вариантах осуществления субъектом-человеком с раком является субъект-человек, у которого был получен биологический образец.
[78] В некоторых вариантах осуществления полипептид имеет длину от 8 до 50 аминокислот.
[79] В некоторых вариантах осуществления полипептид содержит по меньшей мере две выбранные эпитопные последовательности, каждую из которых экспрессируют раковые клетки субъекта-человека с раком.
[80] В некоторых вариантах осуществления истощение CD14+ клеток и CD25+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток, предусматривает введение в контакт популяции иммунных клеток, содержащей первую популяцию APC и T-клеток, со связывающим CD14 средством и связывающим CD25 средством.
[81] В некоторых вариантах осуществления истощение дополнительно представляет собой истощение CD19+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток.
[82] В некоторых вариантах осуществления истощение дополнительно представляет собой истощение CD11b+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток.
[83] В некоторых вариантах осуществления способ предусматривает получение нуклеиновых кислот раковых клеток из первого биологического образца, содержащего раковые клетки, полученные у субъекта, и получение нуклеиновых кислот нераковых клеток из второго биологического образца, содержащего нераковые клетки, полученные у того же субъекта.
[84] В некоторых вариантах осуществления белок, кодируемый аллелем HLA субъекта, представляет собой белок, кодируемый аллелем HLA, выбранным из группы, состоящей из HLA-A01:01, HLA-A02:01, HLA-A03:01, HLA-A11:01, HLA-A24:01, HLA-A30:01, HLA-A31:01, HLA-A32:01, HLA-A33:01, HLA-A68:01, HLA-B07:02, HLA-B08:01, HLA-B15:01, HLA-B44:03, HLA-C07:01 и HLA-C07:02.
[85] В некоторых вариантах осуществления способ предусматривает идентификацию одного или двух, или более различных белков, которые содержат по меньшей мере одну выбранную эпитопную последовательность и которые экспрессируются раковыми клетками субъекта.
[86] В некоторых вариантах осуществления способ предусматривает идентификацию одного или двух, или более различных белков, которые содержат по меньшей мере одну выбранную эпитопную последовательность и которые экспрессируются раковыми клетками субъекта, путем измерения уровней РНК, кодирующей один или два, или более различных белков в раковых клетках.
[87] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности имеет длину от 8 до 12 аминокислот.
[88] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности имеет длину 13-25 аминокислот.
[89] В некоторых вариантах осуществления способ предусматривает выделение геномной ДНК или РНК из раковых клеток и нераковых клеток субъекта.
[90] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит точечную мутацию или последовательность, кодируемую точечной мутацией.
[91] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую мутацией neoORF.
[92] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую мутацией гена слияния.
[93] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую инсерционно-делеционной мутацией.
[94] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую мутацией сайта сплайсинга.
[95] В некоторых вариантах осуществления по меньшей мере две из по меньшей мере одной выбранной эпитопной последовательности получены из одного и того же белка.
[96] В некоторых вариантах осуществления по меньшей мере две из по меньшей мере одной выбранной эпитопной последовательности содержат совпадающую последовательность.
[97] В некоторых вариантах осуществления по меньшей мере две из по меньшей мере одной выбранной эпитопной последовательности получены из разных белков.
[98] В некоторых вариантах осуществления один или несколько пептидов содержат по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9 или 10 или более пептидов.
[99] В некоторых вариантах осуществления раковые клетки субъекта представляют собой раковые клетки солидного рака.
[100] В некоторых вариантах осуществления раковые клетки субъекта представляют собой раковые клетки лейкоза или лимфомы.
[101] В некоторых вариантах осуществления мутация представляет собой мутацию, которая возникает у множества онкологических пациентов.
[102] В некоторых вариантах осуществления MHC представляет собой MHC I класса.
[103] В некоторых вариантах осуществления MHC представляет собой MHC II класса.
[104] В некоторых вариантах осуществления T-клетка представляет собой CD8 T-клетку.
[105] В некоторых вариантах осуществления T-клетка представляет собой CD4 T-клетку.
[106] В некоторых вариантах осуществления T-клетка представляет собой цитотоксичную T-клетку.
[107] В некоторых вариантах осуществления T-клетка представляет собой T-клетку памяти.
[108] В некоторых вариантах осуществления T-клетка представляет собой наивную T-клетку.
[109] В некоторых вариантах осуществления способ дополнительно предусматривает выбор одной или нескольких субпопуляций клеток из размноженной популяции T-клеток перед введением субъекту.
[110] В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает индукцию выработки IL2 в культуре T-клеток при контакте с пептидом.
[111] В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает индукцию выработки цитокина в культуре T-клеток при контакте с пептидом, причем цитокин представляет собой интерферон гамма (IFN-γ), фактор некроза опухоли (TNF) альфа (α) и/или бета (β) или их комбинации.
[112] В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает индукцию уничтожения культурой Т-клеток клетки, экспрессирующей пептид.
[113] В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает обнаружение экспрессии лиганда Fas, гранзима, перфоринов, IFN, TNF или их комбинации в культуре T-клеток.
[114] В некоторых вариантах осуществления очищают один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность.
[115] В некоторых вариантах осуществления лиофилизируют один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность.
[116] В некоторых вариантах осуществления один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность, находится в растворе.
[117] В некоторых вариантах осуществления один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность, находится в таких условиях хранения, чтобы целостность пептида составляла ≥99%.
[118] В некоторых вариантах осуществления способ предусматривает стимуляцию цитотоксичности T-клеток против клеток, нагруженных по меньшей мере одной выбранной эпитопной последовательностью, согласно анализу цитотоксичности.
[119] В некоторых вариантах осуществления способ предусматривает стимуляцию цитотоксичности T-клеток против раковых клеток, экспрессирующих белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, согласно анализу цитотоксичности.
[120] В некоторых вариантах осуществления способ предусматривает стимуляцию цитотоксичности T-клеток против ассоциированной с раком клетки, экспрессирующей белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, согласно анализу цитотоксичности.
[121] В некоторых вариантах осуществления по меньшей мере один выбранный эпитоп экспрессирует раковая клетка, а дополнительный выбранный эпитоп экспрессирует ассоциированная с раком клетка.
[122] В некоторых вариантах осуществления дополнительный выбранный эпитоп экспрессирует ассоциированная с раком клетка фибробласта.
[123] В некоторых вариантах осуществления дополнительный выбранный эпитоп выбирают из таблицы 8.
[124] Также в данном документе представлена фармацевтическая композиция, содержащая T-клетку, полученную с помощью способа, представленного в данном документе.
[125] Также в данном документе представлена библиотека полипептидов, содержащая эпитопные последовательности или полинуклеотиды, кодирующие полипептиды, причем каждая эпитопная последовательность в библиотеке соответствует белку, кодируемому аллелем HLA; и при этом каждую эпитопную последовательность в библиотеке предварительно проверяют на соответствие по меньшей мере трем следующим критериям: связывается с белком, кодируемым аллелем HLA субъекта с раком, подлежащего лечению, является иммуногенной согласно иммуногенному анализу, презентируется антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и/или стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности.
[126] Также в данном документе представлен способ лечения рака у субъекта, включающий введение субъекту (i) полипептида, содержащего эпитоп G12R RAS, или (ii) полинуклеотида, кодирующего полипептид; причем: (a) эпитоп G12R RAS представляет собой vvgaRgvgk (SEQ ID NO: 1), а субъект экспрессирует белок, кодируемый аллелем HLA-A03:01; (b) эпитоп G12R RAS представляет собой eyklvvvgaR (SEQ ID NO: 2), а субъект экспрессирует белок, кодируемый аллелем HLA-A33:03; (c) эпитоп G12R RAS представляет собой vvvgaRgvgk (SEQ ID NO: 3), а субъект экспрессирует белок, кодируемый аллелем HLA-A11:01; или (d) эпитоп G12R RAS представляет собой aRgvgksal (SEQ ID NO: 4), а субъект экспрессирует белок, кодируемый аллелем HLA, выбранным из группы, состоящей из HLA-C07:02, HLA-B39:01 и HLA-C07:01.
Краткое описание чертежей
[127] На фиг. 1A представлено схематичное изображение иллюстративного способа, представленного в данном документе, для примирования, активации и размножения антигенспецифических T-клеток.
[128] На фиг. 1B представлено схематичное изображение иллюстративного способа, представленного в данном документе, для примирования, активации и размножения антигенспецифических T-клеток.
[129] На фиг. 2 представлено схематичное изображение иллюстративного способа автономного получения характеристики общих эпитопов.
[130] На фиг. 3A представлены данные, иллюстрирующие, что прогнозирование эпитопа in silico идентифицировало множество неоантигенов, полученных в результате мутаций RAS G12D, которые имеются согласно масс-спектрометрии. На фигуре раскрыто SEQ ID NOS 1420, 1421, 1147, 1245 и 1247, соответственно, в порядке возникновения.
[131] На фиг. 3B представлены данные, иллюстрирующие, что прогнозирование эпитопа in silico идентифицировало множество неоантигенов, полученных в результате мутаций RAS G12V, которые имеются согласно масс-спектрометрии. На фигуре раскрыто SEQ ID NOS 1422, 1423, 162, 163 и 1148, соответственно, в порядке возникновения.
[132] На фиг. 3C представлены данные, иллюстрирующие, что прогнозирование эпитопа in silico идентифицировало множество неоантигенов, полученных в результате мутаций RAS G12C, которые имеются согласно масс-спектрометрии. На фигуре раскрыто SEQ ID NO 1424
[133] На фиг. 3D представлены данные, иллюстрирующие, что прогнозирование эпитопа in silico идентифицировало множество неоантигенов, полученных в результате мутаций RAS G12R, которые имеются согласно масс-спектрометрии. На фигуре раскрыто SEQ ID NOS 1425, 1426, 1253 и 2, соответственно, в порядке возникновения.
[134] На фиг. 4A представлены данные, иллюстрирующие, что презентация общих эпитопов неоантигенов может быть прямо подтверждена посредством масс-спектрометрии и что неоантигены RAS можно нацеливать в определенных популяциях пациентов.
[135] На фиг. 4B представлен детальный график спектра MS/MS для эндогенно процессированного мутантного эпитопа пептида RAS VVVGAVGVGK (SEQ ID NO: 5) (сверху) и его соответствующего тяжелого пептида (снизу). клетки 293T подвергли лентивирусной трансдукции как с помощью полипептида, содержащего мутантный пептид RASG12V, так и с помощью гена HLA-A*03:01.
[136] На фиг. 4C представлен детальный график спектра MS/MS для эндогенно процессированного мутантного эпитопа пептида RAS VVVGAVGVGK (SEQ ID NO: 5) (сверху) и его соответствующего тяжелого пептида (снизу). клетки SW620, которые в естественных условиях экспрессируют мутант RASG12V, трансдуцировали лентивирусным вектором, кодирующим ген HLA-A*03:01.
[137] На фиг. 4D представлен детальный график спектра MS/MS для эндогенно процессированного мутантного эпитопа пептида RAS VVVGAVGVGK (SEQ ID NO: 5) (сверху) и его соответствующего тяжелого пептида (снизу). для этого эксперимента использовали клетки NCI-H441, экспрессирующие в естественных условиях как мутацию RASG12V, так и ген HLA-A*03:01.
[138] На фиг. 4E представлен детальный график спектра MS/MS для эндогенно процессированного эпитопа SMLTGPPARV (SEQ ID NO: 6) пептида GATA3 neoORF. Спектр эндогенного пептида показан на верхней панели, а соответствующий световой синтетический спектр показан на нижних панелях.
[139] На фиг. 5 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигенов RAS G12 на HLA-A11:01 и HLA-A03:01.
[140] На фиг. 6 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует многомерные CD8 T-клеточные ответы de novo против неоантигена RAS G12V на HLA-A11:01. Как показано на круговых диаграммах, показана частота отдельных клонов Т-клеток, индуцированных против неоантигена RAS G12V на HLA-A11:01 у 3 независимых здоровых доноров.
[141] На фиг. 7 представлены данные, иллюстрирующие, что активированные RASG12V T-клетки, полученные ex vivo, могут убивать клетки-мишени. В клетки-мишени A375, экспрессирующие GFP, загружали 2 мкМ антигена RASG12V, антиген RAS дикого типа или без пептида в качестве контрольных GFP+ клеток. Специфические к RASG12V CD8 T-клетки (эффекторные клетки) инкубировали с контрольными клетками или клетками-мишенями в соотношении 0,05:1. В присутствии эффекторных клеток клетки-мишени лизировали и истощали легче, чем контрольные клетки, которые либо презентируют антиген RASWT, либо не содержат антиген. График гибели конкретных клеток, нормализованный по росту клеток-мишеней без пептида, показан на левой схеме. Типичные изображения показаны на правой.
[142] На фиг. 8 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения специфических к RAS G12V T-клеток с помощью неоантигенов RAS G12V на HLA-11:01, но не соответствующих антигенов дикого типа, индуцирует возникновение цитотоксичности T-клеток с использованием указанных соотношений эффектор:клетка-мишень и возрастающей концентрации пептида.
[143] На фиг. 9 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток с помощью одного цикла (1x стимуляция) или двух циклов (2 стимуляции) обработанных FLT3L PBMC, презентирующих эпитоп с мутацией RASG12V, индуцирует возникновение цитотоксичности T-клеток при измерении с помощью положительных по AnnexinV клеток с течением времени после совместного культивирования этих T-клеток с клетками SW620 (в естественных условиях экспрессируют мутант RASG12V), трансдуцированных лентивирусным вектором, кодирующим ген HLA-A*11:01.
[144] На фиг. 10 представлен график положительных по AnnexinV клеток с течением времени после совместного культивирования клеток NCI-H441, экспрессирующих в естественных условиях и мутацию RASG12V и ген HLA-A*03:01, с T-клетками, которые примировали и активировали и размножали с пептидом, содержащим эпитоп с мутацией RASG12V, в указанном соотношении эффекторы:клетки-мишени.
[145] На фиг. 11A представлен график концентрации IL-2 (пг/мл) для клеток-мишеней, нагруженных мутантным пептидом или дикого типа RAS-G12V (A375-A11:01), после инкубации в присутствии клеток Jurkat, трансдуцированных TCR, который связывается с эпитопом RAS-G12V, связанным с MHC, кодируемым аллелем HLA-A11:01.
[146] На фиг. 11B представлены графики положительных по AnnexinV клеток с течением времени после совместного культивирования трансдуцированых TCR PBMC с 5000 клеток SNGM с природными G12V и HLA-A11:01 в диапазоне соотношений эффектор: клетки-мишени.
[147] На фиг. 11C представлен график концентрации IL-2 (пг/мл) для клеток-мишеней, нагруженных мутантным пептидом или дикого типа RAS-G12V (A375-A03:01), после инкубации в присутствии клеток Jurkat, трансдуцированных TCR, который связывается с RAS-G12V, связанных с MHC, кодируемым аллелем HLA-A03:01.
[148] На фиг. 11D представлен график положительных по AnnexinV клеток с течением времени (сверху) после совместного культивирования трансдуцированых TCR PBMC с клетками с природными G12V и HLA-A03:01 с использованием соотношения эффекторы:клетки-мишени 0,75:1 и график концентрации IFNγ (пг/мл) через 24 часа совместного культивирования трансдуцированых TCR PBMC с клетками с природными G12V и HLA-A03:01 с использованием соотношения эффекторы:клетки-мишени 0,75:1.
[149] На фиг. 12A представлен график концентрации IL-2 (пг/мл) для обработанных FLT3L PBMC, контактировавших с возрастающими количествами указанных мутантных пептидов RAS-G12V после совместного культивирования с клетками Jurkat, трансдуцированными TCR, который связывается с подчеркнутым эпитопом RAS-G12V, связанным с MHC, кодируемым аллелем HLA-A11:01. На фигуре раскрыто SEQ ID NOS 164, 1427 и 1428, соответственно, в порядке возникновения.
[150] На фиг. 12B представлены данные, иллюстрирующие иммуногенность указанных мутантных пептидов RAS-G12V на фиг. 12A как in vitro с использованием PBMC здоровых доноров (сверху), так и in vivo с использованием трансгенных мышей HLA-A11:01, иммунизированных пептидами (снизу).
[151] На фиг. 13 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигена RAS G12V на HLA-02:01.
[152] На фиг. 14 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигенов RAS G12 на HLA-A68:01.
[153] На фиг. 15 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигенов RAS G12 на HLA-B07:02
[154] На фиг. 16 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигенов RAS G12 на HLA-B08:01.
[155] На фиг. 17 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигена RAS G12D на HLA-C08:02.
[156] На фиг. 18 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD4 T-клеточные ответы de novo против неоантигенов RAS.
[157] На фиг. 19A представлены данные, иллюстрирующие данные проточной цитометрии, демонстрирующие, что процедуры обогащения можно использовать перед дальнейшим размножением антигенспецифических Т-клеток. Клетки, активирующие 4-1BB, обогащали с использованием магнитного разделения клеток (MACS; Miltenyi). T-клетки, окрашенные мультимерами, обогащали с помощью MACS на 14 день стимуляции. Этот подход обеспечил обогащение множества популяций антигенспецифических Т-клеток.
[158] На фиг. 19B представлена иллюстративная гистограмма, количественно отображающая результаты на фиг. 19A.
[159] На фиг. 20 представлена сводная информация по экспериментам, иллюстрирующая, что спрогнозированные эпитопы GATA3 neoORF обладают сильной аффинностью (<500 нМ), длительной стабильностью (>0,5 ч) и/или могут быть обнаружены с помощью масс-спектрометрического анализа эпитопов, элюированных из молекул HLA из клеток, экспрессирующих GATA3 neoORF. На фигуре раскрыто SEQ ID NOS 1081, 6, 1088, 1097, 1089, 1085, 1089, 1078, 1093, 1095, 1082, 1079, 1091, 1075, 1078, 1097, 1092, 1079, 1094 и 1096, соответственно, в порядке возникновения.
[160] На фиг. 21 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигенов GATA3 neoORF на HLA-A02:01, HLA-A03:01, HLA-A11:01, HLA-B07:02 и HLA-B08:01. На фигуре раскрыто SEQ ID NOS 1081, 1089, 1089, 1095 и 1091, соответственно, в порядке возникновения.
[161] На фиг. 22 представлены данные, иллюстрирующие, что активированные эпитопом GATA3 neoORF T-клетки, полученные ex vivo, могут убивать клетки-мишени. в клетки-мишени 293T, экспрессирующие GFP, загружали 2 мкМ антигена GATA3 neoORF или оставляли без загрузки в качестве контрольных GFP+ клеток. Специфические к GATA3-neoORF CD8 T-клетки (эффекторные клетки) инкубировали с контрольными клетками или клетками-мишенями в соотношении 1:10. В присутствии эффекторных клеток клетки-мишени лизировали и истощали легче, чем контрольные клетки, которые действительно презентируют неоантиген GATA3. На верхней схеме показан график GFP+ клеток в течение 100 часов. Показаны изображения контрольных (нижнее левое изображение) и GFP+ клеток-мишеней (нижнее правое изображение) в присутствии активированных неоантигеном GATA3 CD8 клеток.
[162] На фиг. 23 представлен график сравнения доли положительных по каспазе-3 живых клеток-мишеней в клетках HEK 293T, трансдуцированных неоантигеном GATA3, по сравнению с нетрансдуцированными клетками HEK 293T. PBMC двух разных индуцированных GATA3 здоровых доноров совместно культивировали с трансдуцированными неоантигеном GATA3 клетками HEK 293T или с нетрансдуцированными клетками HEK 293T в качестве группы отрицательного контроля.
[163] На фиг. 24 представлены данные проточной цитометрии, иллюстрирующие индукцию антигенспецифических CD4+ T-клеток специфическим пептидом GATA3 neoORF после 20 дней культивирования, включая две стимуляции. Антигенспецифические T-клетки обнаруживают по увеличению IFNγ и/или TNFα после инкубации с пептидами GATA3 neoORF (справа) относительно отсутствия пептидов (слева).
[164] На фиг. 25A представлено схематичное изображение этапов, сопровождаемых обнаружением и проверкой пептидов, находящихся в линиях клеток рака предстательной железы или ткани предстательной железы доноров-людей, и получением проверенных пептидов для тщательно отобранной проверенной библиотеки пептидов.
[165] На фиг. 25B представлены данные, иллюстрирующие получение специфических к эпитопу CD8 T-клеток in vitro. Пептиды прогнозировали с использованием программного обеспечения для прогнозирования эпитопов T-клеток в белках, специфичных к раку предстательной железы. На фигуре раскрыто SEQ ID NOS 1403, 1405 и 7, соответственно, в порядке возникновения.
[166] На фиг. 25C представлены данные, иллюстрирующие, то Т-клетки, активированные эпитопом KLK4, полученные ex vivo, являются иммуногенными и убивают клетки-мишени. в клетки-мишени 293T, экспрессирующие GFP, загружали 2 мкМ антигена KLK4 (LLAНГRMPTV (SEQ ID NO: 7)) или оставляли без загрузки в качестве контрольных GFP+ клеток. Специфические к KLK4 CD8 T-клетки (эффекторные клетки) инкубировали с контрольными клетками или клетками-мишенями в соотношении 1:10. В присутствии эффекторных клеток рост клеток-мишеней регулировали более легко, чем контрольных клеток, которые не экспрессируют KLK4. Также представлен график GFP+ клеток в течение 100 часов (снизу). Показаны изображения контрольных (нижнее левое изображение) и GFP+ клеток-мишеней (нижнее правое изображение) в присутствии активированных KLK4 CD8 клеток.
[167] На фиг. 26 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигена BTK C481S на HLA-02:01.
[168] На фиг. 27 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8 T-клеточные ответы de novo против неоантигенов EGFR T790M на HLA-02:01.
[169] На фиг. 28A представлено схематичное изображение иллюстративного способа, представленного в данном документе, для применения T-клеточной терапии.
[170] На фиг. 28B представлено схематичное изображение иллюстративного способа, представленного в данном документе, для применения T-клеточной терапии.
[171] На фиг. 29 представлено схематичное изображение иллюстративного способа прогнозирования эпитопов T-клеток in silico. PPV определяли для заданного количества n попаданий и 5000 непопаданий, какая доля из n пептидов с самым высоким рейтингом была попаданием.
[172] На фиг. 30 представлено схематичное изображение аллельного охвата лигандома MHC с использованием прогнозирования эпитопов in silico.
[173] На фиг. 31 представлено схематичное сравнение моделей прогнозирования эпитопов T-клеток in silico.
[174] На фиг. 32 представлено схематичное изображение, иллюстрирующее идентификацию и проверку иммуногенных пептидов с использованием прогнозирования эпитопов T-клеток in silico и представленного в данном документе иллюстративного способа примирования, активации и размножения антигенспецифических T-клеток.
[175] На фиг. 33 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток может индуцировать и размножать популяции CD8+ T-клеток с множеством неоантигенов. Представленные данные являются типичными данными из образца пациента с меланомой.
[176] На фиг. 34 представлены данные, иллюстрирующие, что с представленным в данном документе иллюстративным способом примирования, активации и размножения антигенспецифических T-клеток получено три CD4+ популяции у одного и того же пациента. Представленные данные являются типичными данными из образца пациента с меланомой.
[177] На фиг. 35 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток неоднократно демонстрирует индукцию Т-клеток в образцах пациентов с меланомой.
[178] На фиг. 36 представлены реперезентативные данные для образца пациента с меланомой, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует T-клетки, высоко специфические для мутантных эпитопов.
[179] На фиг. 37 представлены реперезентативные данные для образца пациента с меланомой, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует T-клетки, которые являются высоко функциональными.
[180] На фиг. 38 представлены данные, иллюстрирующие, что представленный в данном документе иллюстративный способ примирования, активации и размножения антигенспецифических T-клеток индуцирует CD8+ T-клетки, способные убивать опухолевые клетки.
Подробное описание
[181] Хотя многие эпитопы обладают потенциалом связываться с молекулой MHC, лишь немногие из них способны связываться с молекулой MHC при экспериментальном тестировании. Хотя многие эпитопы также обладают потенциалом презентации молекулой MHC, которую можно, например, обнаружить посредством масс-спектрометрии, только определенное количество этих эпитопов может быть презентировано и обнаружено посредством масс-спектрометрии. Хотя многие эпитопы также обладают потенциалом иммуногенности, при экспериментальном тестировании многие из этих эпитопов не являются иммуногенными, несмотря на то, что было продемонстрировано, что их презентируют антигенпрезентирующие клетки. Многие эпитопы также обладают потенциалом активации возникновения цитотоксичности T-клеток; однако многие эпитопы, которые, как было продемонстрировано, презентируются антигенпрезентирующими клетками и/или являются иммуногенными, все еще не способны активировать возникновение цитотоксичности T-клеток.
[182] В данном документе представлены антигены, содержащие эпитопы Т-клеток, которые были идентифицированы и подтверждены как связывающиеся с одной или несколькими молекулами MHC, презентируемые одной или несколькими молекулами MHC, являющиеся иммуногенными и способные активировать возникновение цитотоксичности T-клеток. Проверенные антигены и полинуклеотиды, кодирующие эти антигены, можно использовать для получения антигенспецифических T-клеток для терапевтического применения. В некоторых вариантах осуществления проверенные антигены и полинуклеотиды, кодирующие эти антигены, можно заранее получать и хранить для использования в способе получения T-клеток для терапевтического применения. Например, проверенные антигены и полинуклеотиды, кодирующие эти антигены, можно заранее получать или быстро получать для быстрого получения терапевтических композиций T-клеток для пациентов. Используя проверенные антигены с эпитопами Т-клеток, можно получать иммуногены, такие как пептиды, обладающие активностью связывания HLA, или РНК, кодирующую такие пептиды. Множество иммуногенов можно идентифицировать, проверять и заранее получать в библиотеке. В некоторых вариантах осуществления пептиды можно получать в масштабе, подходящем для хранения, архивирования и использования для фармакологического вмешательства у подходящего пациента в подходящее время.
[183] Некоторые, если не все виды рака имеют антигены, которые являются потенциальными мишенями для иммунотерапии. Каждый пептидный антиген может быть презентирован для активации Т-клеток на антигенпрезентирующих клетках в ассоциации со специфической молекулой MHC, кодируемой HLA. С другой стороны, в данном документе представлен потенциально универсальный подход, при котором определенные эпитопы предварительно идентифицируют и предварительно проверяют на определенные HLA, и эти эпитопы можно заранее получать для процесса получения клеточного терапевтического средства. Например, количество эпитопов KRAS с мутациями G12, G13 и Q61 можно идентифицировать с использованием надежной модели прогнозирования презентации эпитопа Т-клеток (см., например, PCT/US2018/017849, поданный 12 февраля 2018 года, и PCT/US2019/068084, поданный 20 декабря 2019 года, каждая из которых полностью включена посредством ссылки), с проверкой иммуногенности этих эпитопов, процессинга и презентации этих эпитопов с использованием масс-спектрометрии и способности создавать цитотоксичные T-клетки с TCR против этих эпитопов и MHC, кодируемых разными HLA. Каждый эпитоп проверяют на его специфическую аминокислотную последовательность и релевантный HLA. После проверки этих эпитопов можно создать библиотеку, содержащую заранее полученные иммуногены, такие как пептиды, содержащие эпитопы или РНК, кодирующую пептиды, содержащие эти эпитопы.
[184] Антигены могут представлять собой немутантные антигены или мутантные антигены. Например, антигены могут представлять собой опухолеассоциированные антигены, мутантные антигены, тканеспецифические антигены или неоантигены. В некоторых вариантах осуществления антигены представляют собой опухолеассоциированные антигены. В некоторых вариантах осуществления антигены представляют собой мутантные антигены. В некоторых вариантах осуществления антигены представляют собой тканеспецифические антигены. В некоторых вариантах осуществления антигены представляют собой неоантигены. Неоантигены находят при раке или в опухоли у субъекта и не проявляются в зародышевой линии и не экспрессируются в здоровой ткани субъекта. Следовательно, чтобы генная мутация при раке соответствовала критериям получения неоантигена, генная мутация при раке не должна быть молчащей мутацией, которая транслируется в измененный белковый продукт. Измененный белок продукт содержит аминокислотную последовательность с мутацией, которой может быть мутантный эпитоп для T-клетки. Мутантный эпитоп может связываться с молекулой MHC. Мутантный эпитоп также может презентировать молекула MHC, которую можно, например, обнаружить посредством масс-спектрометрии. Кроме того, мутантный эпитоп может быть иммуногенным. Кроме того, мутантный эпитоп может активировать возникновение цитотоксичности T-клеток.
[185] В данном документе представлен способ лечения рака у нуждающегося в этом субъекта, предусматривающий выбор по меньшей мере одной эпитопной последовательности из библиотеки эпитопных последовательностей, причем каждая эпитопная последовательность в библиотеке соответствует белку, кодируемому аллелем HLA субъекта; и введение в контакт T-клетки субъекта или аллогенной T-клетки с одним или несколькими пептидами, содержащими по меньшей мере одну выбранную эпитопную последовательность, причем каждую по меньшей мере одну выбранную эпитопную последовательность предварительно проверяют на соответствие по меньшей мере двум или трем, или четырем следующим критериям: связывается с белком, кодируемым аллелем HLA субъекта, является иммуногенной согласно анализу иммуногенности, презентируются антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности. В некоторых вариантах осуществления способ дополнительно предусматривает введение популяции T-клеток субъекту.
[186] В некоторых вариантах осуществления по меньшей мере одна выбранная эпитопная последовательность содержит мутацию, а способ предусматривает идентификацию раковых клеток субъекта на кодирование эпитопа с мутацией; по меньшей мере одна выбранная эпитопная последовательность находится в белке, сверхэкспрессируемом раковыми клетками субъекта, а способ предусматривает идентификацию раковых клеток субъекта на сверхэкспрессию белка, содержащего эпитоп; или по меньшей мере одна эпитопная последовательность содержит белок, экспрессируемый клетками в микросреде опухоли. В некоторых вариантах осуществления одна или более из по меньшей одной выбранной эпитопной последовательности содержит эпитоп, который не экспрессируется раковыми клетками субъекта. В некоторых вариантах осуществления эпитоп, который не экспрессируется раковыми клетками субъекта, экспрессируют клетки в микросреде опухоли субъекта. В некоторых вариантах осуществления способ предусматривает выбор субъекта с использованием анализа циркулирующей опухолевой ДНК. В некоторых вариантах осуществления способ предусматривает выбор субъекта с использованием панели генов.
[187] В некоторых вариантах осуществления T-клетку получают из биологического образца субъекта. В некоторых вариантах осуществления T-клетку получают из образца для афереза или для лейкафереза субъекта. В некоторых вариантах осуществления T-клетка является аллогенной T-клеткой.
[188] В некоторых вариантах осуществления каждую по меньшей мере одну выбранную эпитопную последовательность предварительно проверяют на соответствие одному или нескольким, или каждому из следующих критериев: связывается с белком, кодируемым аллелем HLA субъекта, является иммуногенной согласно анализу иммуногенности, презентируются антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности.
[189] В некоторых вариантах осуществления эпитоп, который связывается с белком, кодируемым аллелем HLA субъекта, связывается с молекулой MHC, кодируемой аллелем HLA, с аффинностью 500 нМ или менее согласно анализу связывания. Например, эпитоп, который связывается с белком, кодируемым аллелем HLA субъекта, может связываться с молекулой MHC, кодируемой аллелем HLA, с аффинностью 400 нМ, 300 нМ, 200 нМ, 150 нМ, 100 нМ, 75 нМ, 50 нМ или 25 нМ или менее согласно анализу связывания. В некоторых вариантах осуществления связывание эпитопа, который связывается с белком, кодируемым аллелем HLA субъекта, с молекулой MHC, кодируемой аллелем HLA, с аффинностью 500 нМ или менее прогнозируют с использованием программы прогнозирования эпитопа MHC, выполняемой на компьютере. Например, можно спрогнозировать связывание эпитопа, который связывается с белком, кодируемым аллелем HLA субъекта, с молекулой MHC, кодируемой аллелем HLA, с аффинностью 400 нМ, 300 нМ, 200 нМ, 150 нМ, 100 нМ, 75 нМ, 50 нМ или 25 нМ или менее с использованием программы прогнозирования эпитопа MHC, выполняемой на компьютере. В некоторых вариантах осуществления программой прогнозирования эпитопа MHC, выполняемой на компьютере, является NetMHCpan. В некоторых вариантах осуществления программой прогнозирования эпитопа MHC, выполняемой на компьютере, является NetMHCpan версия 4.0.
[190] В некоторых вариантах осуществления эпитоп, который презентируют антигенпрезентирующие клетки (APC) согласно масс-спектрометрическому анализу, обнаруживают посредством масс-спектрометрии после элюирования из APC с точностью массы обнаруженного пептида, составляющей менее 15 Да. Например, эпитоп, который презентируют антигенпрезентирующие клетки (APC) согласно масс-спектрометрическому анализу, можно обнаружить посредством масс-спектрометрии после элюирования из APC с точностью массы обнаруженного пептида, составляющей менее 14 Да, 13 Да, 12 Да, 11 Да, 10 Да, 9 Да, 8 Да, 7 Да, 6 Да, 5 Да, 4 Да, 3 Да, 2 Да или 1 Да. В некоторых вариантах осуществления эпитоп, который презентируют антигенпрезентирующие клетки (APC) согласно масс-спектрометрическому анализу, обнаруживают посредством масс-спектрометрии после элюирования из APC с точностью массы обнаруженного пептида, составляющей менее 10000 частей на миллион (ч/млн). Например, эпитоп, который презентируют антигенпрезентирующие клетки (APC) согласно масс-спектрометрическому анализу, можно обнаружить посредством масс-спектрометрии после элюирования из APC с точностью массы обнаруженного пептида, составляющей менее 7500 ч/млн; 5000 ч/млн; 2500 ч/млн; 1000 ч/млн; 900 ч/млн; 800 ч/млн; 700 ч/млн; 600 ч/млн; 500 ч/млн; 400 ч/млн; 300 ч/млн; 200 ч/млн или 100 ч/млн.
[191] В некоторых вариантах осуществления эпитоп, который является иммуногенным согласно анализу иммуногенности, является иммуногенным согласно мультимерному анализу. В некоторых вариантах осуществления мультимерный анализ представляет собой анализ проточной цитометрии. В некоторых вариантах осуществления мультимерный анализ включает обнаружение T-клеток, связанных с мультимером пептид-MHC, содержащим по меньшей мере одну выбранную эпитопную последовательность и соответствующий аллель HLA, причем T-клетки были стимулированы APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. В некоторых вариантах осуществления эпитоп является иммуногенным согласно мультимерному анализу (i) при обнаружении по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,005% проанализированных CD8+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ T-клеток, обнаруженных в контрольном образце. Например, эпитоп может быть иммуногенным согласно мультимерному анализу, когда (i) обнаруживают по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) обнаруженные T-клетки составляют по меньшей мере 0,005% проанализированных CD8+ клеток, и (iii) процентное значение обнаруженных T-клеток из CD8+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ T-клеток, обнаруженных в контрольном образце. Например, эпитоп может быть иммуногенным согласно мультимерному анализу (i) при обнаружении по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,01%, 0,05%, 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% проанализированных CD8+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ T-клеток, обнаруженных в контрольном образце. Например, эпитоп может быть иммуногенным согласно мультимерному анализу, когда (i) обнаруживают по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) обнаруженные T-клетки составляют по меньшей мере 0,01%, 0,05%, 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% проанализированных CD8+ клеток, и (iii) процентное значение обнаруженных T-клеток из CD8+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ T-клеток, обнаруженных в контрольном образце.
[192] В некоторых вариантах осуществления эпитоп является иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в одной из шести стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в одной из шести стимуляций из одного и того же начального образца по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2 из 6, 7, 8, 9, 10, 11 или 12 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5 или 6 из 6 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5, 6 или 7 из 7 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5, 6, 7 или 8 из 8 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5, 6, 7, 8 или 9 из 9 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5, 6, 7, 8, 9 или 10 из 10 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5, 6, 7, 8, 9, 10 или 11 из 11 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 2, 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 из 12 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 3 из 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в 4 из 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24 стимуляций из одного и того же начального образца по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере в одной из шести стимуляций из одного и того же начального образца по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. Например, эпитоп может быть иммуногенным согласно мультимерному анализу при обнаружении по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, по меньшей мере в 2 из 6, 7, 8, 9, 10, 11 или 12 стимуляций из одного и того же начального образца или по меньшей мере в 3 из 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18 стимуляций из одного и того же начального образца или по меньшей мере в 4 из 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24 стимуляций из одного и того же начального образца. В некоторых вариантах осуществления контрольный образец содержит T-клетки, стимулированные APC, которые (i) не содержат пептида, содержащего по меньшей мере одну выбранную эпитопную последовательность, (ii) содержат пептид, полученный из другого белка, чем по меньшей мере одна выбранная эпитопная последовательность, или (iii) содержат пептид со случайной последовательностью. В некоторых вариантах осуществления T-клетки были стимулированы APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, в течение по меньшей мере 3, 4, 5, 6, 7, 8,9, 10, 11, 12, 13, 14, 15, 16, 7, 18, 19, 20 или более дней. В некоторых вариантах осуществления антигенспецифические T-клетки размножили по меньшей мере в 5, 10, 20, 50, 100, 500 или 1000 раз или более в присутствии APC, содержащих пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность.
[193] В некоторых вариантах осуществления эпитоп, который является иммуногенным согласно анализу иммуногенности, является иммуногенным согласно функциональному анализу. В некоторых вариантах осуществления функциональный анализ представляет собой иммуноанализ. В некоторых вариантах осуществления функциональный анализ включает обнаружение T-клеток с помощью внутриклеточного окрашивания IFNγ или TNFα или экспрессии CD107a и/или CD107b на поверхности клеток, причем T-клетки были стимулированы APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность. В некоторых вариантах осуществления эпитоп является иммуногенным согласно функциональному анализу (i) при обнаружении по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,005% проанализированных CD8+ или CD4+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ или CD4+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ или CD4+ T-клеток, обнаруженных в контрольном образце. Например эпитоп может быть иммуногенным согласно функциональному анализу, когда (i) обнаруживают по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) обнаруженные T-клетки составляют по меньшей мере 0,005% проанализированных CD8+ или CD4+ клеток, и (iii) процентное значение обнаруженных T-клеток из CD8+ или CD4+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ или CD4+ T-клеток, обнаруженных в контрольном образце. Например эпитоп может быть иммуногенным согласно функциональному анализу (i) при обнаружении по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,01%, 0,05%, 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% проанализированных CD8+ или CD4+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ или CD4+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ или CD4+ T-клеток, обнаруженных в контрольном образце. Например эпитоп может быть иммуногенным согласно функциональному анализу, когда (i) обнаруживают по меньшей мере 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900 или более T-клеток, стимулированных APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, (ii) когда обнаруженные T-клетки составляют по меньшей мере 0,01%, 0,05%, 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% проанализированных CD8+ или CD4+ клеток, и (iii) когда процентное значение обнаруженных T-клеток из CD8+ или CD4+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ или CD4+ T-клеток, обнаруженных в контрольном образце.
[194] В некоторых вариантах осуществления T-клетками со стимулированной цитотоксичностью согласно анализу цитотоксичности являются T-клетки, стимулированные APC, содержащими пептид, содержащий по меньшей мере одну выбранную эпитопную последовательность, которые убивают клетки, презентирующие эпитоп. В некоторых вариантах осуществления количество презентирующих эпитоп клеток, уничтожаемых Т-клетками, по меньшей мере в 1,1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 50, 100, 500 или 1000 раз выше количества не презентирующих эпитопа клеток, уничтожаемых Т-клетками. В некоторых вариантах осуществления количество презентирующих эпитоп клеток, уничтожаемых Т-клетками, по меньшей мере в 1,1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 50, 100, 500 или 1000 раз выше количества презентирующих эпитоп клеток, уничтожаемых Т-клетками, стимулированными APC, которые (i) не содержат пептида, содержащего по меньшей мере одну выбранную эпитопную последовательность, (ii) содержат пептид, полученный из другого белка, чем по меньшей мере одна выбранная эпитопная последовательность, или (iii) содержат пептид со случайной последовательностью В некоторых вариантах осуществления количество презентирующих мутантный эпитоп клеток, уничтожаемых Т-клетками, по меньшей мере в 1,1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 50, 100, 500 или 1000 раз выше количества презентирующих соответствующий эпитоп дикого типа клеток, уничтожаемых Т-клетками. В некоторых вариантах осуществления T-клетками со стимулированной цитотоксичностью согласно анализу цитотоксичности являются T-клетки со стимулированной специфической цитотоксичностью согласно анализу цитотоксичности.
[195] В некоторых вариантах осуществления по меньшей мере один из одного или нескольких пептидов представляет собой синтезированный пептид или пептид, экспрессированный из последовательности нуклеиновой кислоты.
[196] В некоторых вариантах осуществления способ предусматривает идентификацию белка, кодируемого аллелем HLA субъекта, или идентификацию аллеля HLA в геноме субъекта.
[197] В некоторых вариантах осуществления по меньшей мере одну выбранную эпитопную последовательность выбирают из одной или нескольких эпитопных последовательностей таблицы 1-8 и 11-14.
[198] В некоторых вариантах осуществления способ предусматривает размножение T-клетки, контактировавшей с одним или несколькими пептидами, in vitro или ex vivo для получения популяции T-клеток, специфичных по меньшей мере к одной выбранной эпитопной последовательности в комплексе с белком MHC.
[199] В некоторых вариантах осуществления белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, экспрессирует раковая клетка субъекта. В некоторых вариантах осуществления белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, экспрессируют клетки в микросреде опухоли субъекта.
[200] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит мутацию. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит опухолеспецифичную мутацию. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка, сверхэкспрессируемого раковой клеткой субъекта. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит драйверную мутацию. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит мутацию устойчивости к лекарственным средствам. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из тканеспецифического белка. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из раково-тестикулярного белка. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности является вирусным эпитопом. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности является минорным эпитопом гистосовместимости. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка RAS. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка GATA3. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка EGFR. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка BTK. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка p53. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из полипептида слияния TMPRSS2::ERG. В некоторых вариантах осуществления одну или несколько из по меньшей мере одной выбранной эпитопной последовательности получают из белка Myc. В некоторых вариантах осуществления по меньшей мере одну из по меньшей мере одной выбранной эпитопной последовательности получают из белка, кодируемого геном, выбранным из группы, состоящей из ANKRD30A, COL10A1, CTCFL, PPIAL4G, POTEE, DLL3, MMP13, SSX1, DCAF4L2, MAGEA4, MAGEA11, MAGEC2, MAGEA12, PRAME, CLDN6, EPYC, KLK3, KLK2, KLK4, TGM4, POTEG, RLN1, POTEH, SLC45A2, TSPAN10, PAGE5, CSAG1, PRDM7, TG, TSHR, RSPH6A, SCXB, HIST1H4K, ALPPL2, PRM2, PRM1, TNP1, LELP1, HMGB4, AKAP4, CETN1, UBQLN3, ACTL7A, ACTL9, ACTRT2, PGK2, C2orf53, KIF2B, ADAD1, SPATA8, CCDC70, TPD52L3, ACTL7B, DMRTB1, SYCN, CELA2A, CELA2B, PNLIPRP1, CTRC, AMY2A, SERPINI2, RBPJL, AQP12A, IAPP, KIRREL2, G6PC2, AQP12B, CYP11B1, CYP11B2, STAR, CYP11A1 и MC2R.
[201] В некоторых вариантах осуществления по меньшей мере одну из по меньшей мере одной выбранной эпитопной последовательности получают из тканеспецифического белка, уровень экспрессии которого в ткани-мишени субъекта по меньшей мере в 2 раза превышает уровень экспрессии тканеспецифического белка в каждой ткани из множества не являющихся мишенью тканей, которые отличаются от ткани-мишени.
[202] В некоторых вариантах осуществления введение в контакт T-клетки субъекта или аллогенной T-клетки с одним или несколькими пептидами, содержащими по меньшей мере одну выбранную эпитопную последовательность, предусматривает введение в контакт T-клетки с APC, презентирующими эпитоп.
[203] В некоторых вариантах осуществления APC, презентирующие эпитоп, содержат один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность, или полинуклеиновую кислоту, которая кодирует один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность. В некоторых вариантах осуществления полипептид содержит по меньшей мере две выбранные эпитопные последовательности, каждую из которых экспрессируют раковые клетки субъекта-человека с раком.
[204] В некоторых вариантах осуществления способ предусматривает истощение CD14+ клеток и CD25+ клеток из популяции иммунных клеток, содержащей антигенпрезентирующие клетки (APC) и T-клетки, образуя за счет этого популяцию иммунных клеток с истощенными CD14/CD25, содержащую первую популяцию APC и T-клеток. В некоторых вариантах осуществления популяцию иммунных клеток получают из биологического образца субъекта. В некоторых вариантах осуществления способ дополнительно включает инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей первую популяцию APC и T-клеток, в течение первого периода времени в присутствии лиганда рецептора FMS-подобной тирозинкиназы 3 (FLT3L) и полипептида, содержащего по меньшей мере одну выбранную эпитопную последовательность, или полинуклеотида, кодирующего полипептид; образуя за счет этого популяцию клеток, содержащую стимулированные T-клетки. В некоторых вариантах осуществления способ дополнительно предусматривает размножение популяции клеток, содержащей стимулированные T-клетки, образуя за счет этого размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки, причем специфические в отношении опухолевых антигенов T-клетки включают в себя T-клетки, которые являются специфичными к комплексу, содержащему по меньшей мере одну выбранную эпитопную последовательность и белок MHC, экспрессируемый раковыми клетками или APC субъекта. В некоторых вариантах осуществления размножение проводят менее 28 дней. В некоторых вариантах осуществления инкубация представляет собой инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей первую популяцию APC и T-клеток, в течение первого периода времени в присутствии FLT3L и РНК, кодирующей полипептид. В некоторых вариантах осуществления истощение CD14+ клеток и CD25+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток, предусматривает введение в контакт популяции иммунных клеток, содержащей первую популяцию APC и T-клеток, со связывающим CD14 средством и связывающим CD25 средством. В некоторых вариантах осуществления истощение дополнительно представляет собой истощение CD19+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток. В некоторых вариантах осуществления истощение дополнительно представляет собой истощение CD11b+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток.
[205] В некоторых вариантах осуществления способ дополнительно предусматривает введение субъекту-человеку с раком фармацевтической композиции, содержащей размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки. В некоторых вариантах осуществления субъектом-человеком с раком является субъект-человек, у которого был получен биологический образец.
[206] В некоторых вариантах осуществления доля CD8+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD8+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, по меньшей мере в два раза превышает долю CD8+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD8+ T-клеток в биологическом образце. В некоторых вариантах осуществления доля CD4+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD4+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, по меньшей мере в два раза превышает долю CD4+ специфических в отношении опухолевых антигенов T-клеток от общего количества CD4+ T-клеток в биологическом образце. В некоторых вариантах осуществления по меньшей мере 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% CD8+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD8+ T-клетки, полученные из наивных CD8+ T-клеток. В некоторых вариантах осуществления по меньшей мере 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% CD8+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD8+ T-клетки, полученные из CD8+ T-клеток памяти. В некоторых вариантах осуществления по меньшей мере 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% CD4+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD4+ T-клетки, полученные из наивных CD4+ T-клеток. В некоторых вариантах осуществления по меньшей мере 0,1%, 0,2%, 0,3%, 0,4%, 0,5%, 0,6%, 0,7%, 0,8%, 0,9%, 1%, 1,5%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10% CD4+ T-клеток в размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD4+ T-клетки, полученные из CD4+ T-клеток памяти.
[207] В некоторых вариантах осуществления размножение предусматривает введение в контакт популяции клеток, содержащей стимулированные T-клетки, со второй популяцией зрелых APC, причем вторая популяция зрелых APC инкубирована с FLT3L и презентирует по меньшей мере одну выбранную эпитопную последовательность; и размножение популяции клеток, содержащей стимулированные T-клетки, в течение второго периода времени, образуя за счет этого размноженную популяцию T-клеток. В некоторых вариантах осуществления вторую популяцию зрелых APC инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт популяции клеток, содержащей стимулированные T-клетки, со второй популяцией зрелых APC. В некоторых вариантах осуществления размножение дополнительно предусматривает введение в контакт размноженной популяции T-клеток с третьей популяцией зрелых APC, причем третья популяция зрелых APC инкубирована с FLT3L и презентирует по меньшей мере одну выбранную эпитопную последовательность; и размножение размноженной популяции T-клеток в течение третьего периода времени, образуя за счет этого размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки. В некоторых вариантах осуществления третью популяцию зрелых APC инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт размноженной популяции T-клеток с третьей популяцией зрелых APC. В некоторых вариантах осуществления биологический образец представляет собой образец периферической крови, образец для лейкафереза или образец для афереза.
[208] В некоторых вариантах осуществления способ дополнительно предусматривает сбор размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, криоконсервацию размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, или получение фармацевтической композиции, содержащей размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки.
[209] В некоторых вариантах осуществления способ предусматривает получение нуклеиновых кислот раковых клеток из первого биологического образца, содержащего раковые клетки, полученные у субъекта, и получение нуклеиновых кислот нераковых клеток из второго биологического образца, содержащего нераковые клетки, полученные у того же субъекта.
[210] В некоторых вариантах осуществления белок, кодируемый аллелем HLA субъекта, представляет собой белок, кодируемый аллелем HLA, выбранным из группы, состоящей из HLA-A01:01, HLA-A02:01, HLA-A03:01, HLA-A11:01, HLA-A24:01, HLA-A30:01, HLA-A31:01, HLA-A32:01, HLA-A33:01, HLA-A68:01, HLA-B07:02, HLA-B08:01, HLA-B15:01, HLA-B44:03, HLA-C07:01 и HLA-C07:02.
[211] В некоторых вариантах осуществления способ предусматривает идентификацию одного или двух, или более различных белков, которые содержат по меньшей мере одну выбранную эпитопную последовательность и которые экспрессируются раковыми клетками субъекта. В некоторых вариантах осуществления способ предусматривает идентификацию одного или двух, или более различных белков, которые содержат по меньшей мере одну выбранную эпитопную последовательность и которые экспрессируются раковыми клетками субъекта, путем измерения уровней РНК, кодирующей один или два, или более различных белков в раковых клетках. В некоторых вариантах осуществления способ предусматривает выделение геномной ДНК или РНК из раковых клеток и нераковых клеток субъекта.
[212] В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит точечную мутацию или последовательность, кодируемую точечной мутацией. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую мутацией neoORF. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую мутацией гена слияния. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую инсерционно-делеционной мутацией. В некоторых вариантах осуществления одна или несколько из по меньшей мере одной выбранной эпитопной последовательности содержит последовательность, кодируемую мутацией сайта сплайсинга. В некоторых вариантах осуществления по меньшей мере две из по меньшей мере одной выбранной эпитопной последовательности получены из одного и того же белка. В некоторых вариантах осуществления по меньшей мере две из по меньшей мере одной выбранной эпитопной последовательности содержат совпадающую последовательность. В некоторых вариантах осуществления по меньшей мере две из по меньшей мере одной выбранной эпитопной последовательности получены из разных белков. В некоторых вариантах осуществления один или несколько пептидов содержат по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9 или 10 или более пептидов.
[213] В некоторых вариантах осуществления раковые клетки субъекта представляют собой раковые клетки солидного рака. В некоторых вариантах осуществления раковые клетки субъекта представляют собой раковые клетки лейкоза или лимфомы.
[214] В некоторых вариантах осуществления мутация представляет собой мутацию, которая возникает у множества онкологических пациентов.
[215] В некоторых вариантах осуществления MHC представляет собой MHC I класса. В некоторых вариантах осуществления MHC представляет собой MHC II класса.
[216] В некоторых вариантах осуществления T-клетка представляет собой CD8 T-клетку. В некоторых вариантах осуществления T-клетка представляет собой CD4 T-клетку. В некоторых вариантах осуществления T-клетка представляет собой цитотоксичную T-клетку. В некоторых вариантах осуществления T-клетка представляет собой T-клетку памяти. В некоторых вариантах осуществления T-клетка представляет собой наивную T-клетку.
[217] В некоторых вариантах осуществления способ дополнительно предусматривает выбор одной или нескольких субпопуляций клеток из размноженной популяции T-клеток перед введением субъекту.
[218] В некоторых вариантах осуществления индукция иммунного ответ в культуре T-клеток включает индукцию выработки IL2 в культуре T-клеток при контакте с пептидом. В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает индукцию выработки цитокина в культуре T-клеток при контакте с пептидом, причем цитокин представляет собой интерферон гамма (IFN-γ), фактор некроза опухоли (TNF) альфа (α) и/или бета (β) или их комбинации. В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает индукцию уничтожения культурой Т-клеток клетки, экспрессирующей пептид. В некоторых вариантах осуществления индукция иммунного ответа в культуре T-клеток включает обнаружение экспрессии лиганда Fas, гранзима, перфоринов, IFN, TNF или их комбинации в культуре T-клеток.
[219] В некоторых вариантах осуществления очищают один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность. В некоторых вариантах осуществления лиофилизируют один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность. В некоторых вариантах осуществления один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность, находится в растворе. В некоторых вариантах осуществления один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность, находится в таких условиях хранения, чтобы целостность пептида составляла ≥99%.
[220] В некоторых вариантах осуществления способ предусматривает стимуляцию цитотоксичности T-клеток против клеток, нагруженных по меньшей мере одной выбранной эпитопной последовательностью, согласно анализу цитотоксичности. В некоторых вариантах осуществления способ предусматривает стимуляцию цитотоксичности T-клеток против раковых клеток, экспрессирующих белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, согласно анализу цитотоксичности. В некоторых вариантах осуществления способ предусматривает стимуляцию цитотоксичности T-клеток против ассоциированной с раком клетки, экспрессирующей белок, содержащий по меньшей мере одну выбранную эпитопную последовательность, согласно анализу цитотоксичности.
[221] В некоторых вариантах осуществления по меньшей мере один выбранный эпитоп экспрессирует раковая клетка, а дополнительный выбранный эпитоп экспрессирует ассоциированная с раком клетка. В некоторых вариантах осуществления дополнительный выбранный эпитоп экспрессирует ассоциированная с раком клетка фибробласта. В некоторых вариантах осуществления дополнительный выбранный эпитоп выбирают из таблицы 8.
[222] В некоторых вариантах осуществления представленный в данном документе способ представляет собой способ лечения рака у нуждающегося в этом субъекта, предусматривающий: выбор по меньшей мере одной эпитопной последовательности из библиотеки эпитопных последовательностей, причем каждая эпитопная последовательность в библиотеке соответствует белку, кодируемому аллелем HLA; и введение в контакт T-клетки субъекта или аллогенной T-клетки с одним или несколькими пептидами, содержащими по меньшей мере одну выбранную эпитопную последовательность, причем каждая из по меньшей мере одной выбранной эпитопной последовательности: связывается с белком, кодируемым аллелем HLA субъекта; является иммуногенной согласно иммуногенному анализу; презентируется антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности.
[223] В некоторых вариантах осуществления способ предусматривает выбор субъекта с использованием анализа циркулирующей опухолевой ДНК. В некоторых вариантах осуществления способ предусматривает выбор субъекта с использованием панели генов.
[224] В некоторых вариантах осуществления T-клетку получают из биологического образца субъекта. В некоторых вариантах осуществления T-клетку получают из образца для афереза или для лейкафереза субъекта.
[225] В некоторых вариантах осуществления по меньшей мере один из одного или нескольких пептидов представляет собой синтезированный пептид или пептид, экспрессированный из последовательности нуклеиновой кислоты.
[226] В некоторых вариантах осуществления способ предусматривает идентификацию белка, кодируемого аллелем HLA субъекта, или идентификацию аллеля HLA в геноме субъекта. В некоторых вариантах осуществления способ предусматривает идентификацию белка, кодируемого аллелем HLA субъекта, который экспрессирует субъект. В некоторых вариантах осуществления способ предусматривает введение в контакт T-клетки субъекта с одним или несколькими пептидами, выбранными из одной или нескольких таблиц пептидов, представленных в данном документе. В некоторых вариантах осуществления способ предусматривает введение в контакт T-клетки субъекта с одним или несколькими пептидами, содержащими эпитоп, выбранный из таблиц эпитопов, представленных в данном документе. В некоторых вариантах осуществления способ дополнительно предусматривает размножение in vitro или ex vivo T-клетки, контактировавшей с одним или несколькими пептидами, для получения популяции T-клеток. В некоторых вариантах осуществления способ дополнительно предусматривает введение популяции T-клеток субъекту в такой дозе и временном интервале, чтобы уменьшить или устранить рак.
[227] В некоторых вариантах осуществления по меньшей мере один из одного или нескольких пептидов экспрессирует раковая клетка субъекта. В некоторых вариантах осуществления по меньшей мере один из эпитопов одного или нескольких пептидов содержит мутацию.
[228] В некоторых вариантах осуществления по меньшей мере один из эпитопов одного или нескольких пептидов содержит опухолеспецифичную мутацию. В некоторых вариантах осуществления по меньшей мере один из эпитопов одного или нескольких пептидов получают из белка, сверхэкспрессируемого раковой клеткой субъекта. В некоторых вариантах осуществления по меньшей мере один из эпитопов одного или нескольких пептидов получают из белка, кодируемого геном, выбранным из группы, состоящей из ANKRD30A, COL10A1, CTCFL, PPIAL4G, POTEE, DLL3, MMP13, SSX1, DCAF4L2, MAGEA4, MAGEA11, MAGEC2, MAGEA12, PRAME, CLDN6, EPYC, KLK3, KLK2, KLK4, TGM4, POTEG, RLN1, POTEH, SLC45A2, TSPAN10, PAGE5, CSAG1, PRDM7, TG, TSHR, RSPH6A, SCXB, HIST1H4K, ALPPL2, PRM2, PRM1, TNP1, LELP1, HMGB4, AKAP4, CETN1, UBQLN3, ACTL7A, ACTL9, ACTRT2, PGK2, C2orf53, KIF2B, ADAD1, SPATA8, CCDC70, TPD52L3, ACTL7B, DMRTB1, SYCN, CELA2A, CELA2B, PNLIPRP1, CTRC, AMY2A, SERPINI2, RBPJL, AQP12A, IAPP, KIRREL2, G6PC2, AQP12B, CYP11B1, CYP11B2, STAR, CYP11A1 и MC2R.
[229] В некоторых вариантах осуществления по меньшей мере один из одного или нескольких пептидов получают из белка, кодируемого геном тканеспецифического антигенного эпитопа, уровень экспрессии которого в ткани-мишени субъекта по меньшей мере в 2 раза превышает уровень экспрессии гена тканеспецифического антигена в каждой ткани из множества не являющихся мишенью тканей, которые отличаются от ткани-мишени.
[230] В некоторых вариантах осуществления способ предусматривает: инкубацию одного или нескольких препаратов антигенпрезентирующих клеток (APC) с популяцией иммунных клеток из биологического образца с истощенными клетками, экспрессирующими CD14 и CD25, в течение одного или нескольких отдельных периодов времени; инкубацию одного или нескольких препаратов APC с популяцией иммунных клеток из биологического образца в течение одного или нескольких отдельных периодов времени, причем одна или несколько APC включают в себя одну или несколько APC, стимулированных лигандом рецептора FMS-подобной тирозинкиназы 3 (FLT3L); или инкубацию FLT3L и по меньшей мере одного пептида с популяцией иммунных клеток из биологического образца, при этом FLT3L инкубируют с популяцией иммунных клеток в течение первого периода времени, и при этом по меньшей мере один пептид инкубируют с популяцией иммунных клеток в течение первого периода времени стимуляции пептида, получая за счет этого первый образец стимулированных T-клеток, в котором популяция иммунных клеток содержит по меньшей мере одну T-клетку и по меньшей мере одну APC; причем размножают по меньшей мере одну антигенспецифическую T-клетку памяти, или индуцируют по меньшей мере одну антигенспецифическую наивную T-клетку.
[231] В некоторых вариантах осуществления способ предусматривает инкубацию популяция иммунных клеток из биологического образца с одним или несколькими препаратами APC в течение одного или нескольких отдельных периодов времени менее 28 дней с момента инкубации популяции иммунных клеток с первым препаратом APC из одного или нескольких препаратов APC. В некоторых вариантах осуществления способ предусматривает инкубацию популяции иммунных клеток из биологического образца с 3 или менее препаратами APC в течение 3 или менее отдельных периодов времени. В некоторых вариантах осуществления способ предусматривает инкубацию популяции иммунных клеток из биологического образца с 2 или менее препаратами APC в течение 2 или менее отдельных периодов времени. В некоторых вариантах осуществления способ предусматривает инкубацию популяции иммунных клеток из биологического образца с одним или несколькими препаратами APC в течение одного или нескольких отдельных периодов времени менее 28 дней с момента инкубации популяции иммунных клеток с первым препаратом APC из одного или нескольких препаратов APC. В некоторых вариантах осуществления общий период получения T-клеток, стимулированных антигеном, путем инкубации популяции иммунных клеток из биологического образца с одним или несколькими препаратами APC в течение одного или нескольких отдельных периодов времени составляет менее 28 дней.
[232] В некоторых вариантах осуществления по меньшей мере два из одного или нескольких препаратов APC содержат стимулированные FLT3L APC. В некоторых вариантах осуществления по меньшей мере три из одного или нескольких препаратов APC содержат стимулированные FLT3L APC. В некоторых вариантах осуществления инкубация предусматривает инкубацию первого препарата APC из препаратов APC с T-клетками в течение более 7 дней. В некоторых вариантах осуществления APC из препаратов APC содержит APC с загруженным одним или несколькими антигенными пептидами, содержащими одну или несколько из по меньшей мере одной последовательности антигенного пептида. В некоторых вариантах осуществления APC из препаратов APC являются аутологичными APC или аллогенными APC. В некоторых вариантах осуществления APC из препаратов APC представляет собой дендритную клетку (DC). В некоторых вариантах осуществления DC представляет собой CD141+ DC. В некоторых вариантах осуществления способ предусматривает истощение клеток, экспрессирующих CD14 и CD25, из биологического образца, получая за счет этого популяцию иммунных клеток из биологического образца с истощенными клетками, экспрессирующими CD14 и CD25. В некоторых вариантах осуществления способ дополнительно предусматривает истощение клеток, экспрессирующих CD19. В некоторых вариантах осуществления способ дополнительно предусматривает истощение клеток, экспрессирующих CD11b. В некоторых вариантах осуществления истощение клеток, экспрессирующих CD14 и CD25, предусматривает связывание связывающего CD14 или CD25 средства с APC одного или нескольких препаратов APC. В некоторых вариантах осуществления способ дополнительно предусматривает введение одной или нескольких из по меньшей мере одной антигенспецифической T-клетки субъекту.
[233] В некоторых вариантах осуществления инкубация предусматривает инкубацию первого препарата APC из одного или нескольких препаратов APC с T-клетками в течение более 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 дней. В некоторых вариантах осуществления способ предусматривает инкубацию по меньшей мере одного из одного или нескольких препаратов APC с первой средой, содержащей по меньшей мере один цитокин или фактор роста в течение первого периода времени. В некоторых вариантах осуществления способ предусматривает инкубацию по меньшей мере одного из одного или нескольких препаратов APC со второй средой, содержащей один или несколько цитокинов или факторов роста в течение третьего периода времени, получая за счет этого созревшие APC. В некоторых вариантах осуществления способ дополнительно предусматривает удаление одного или нескольких цитокинов или факторов роста из второй среды после третьего периода времени. В некоторых вариантах осуществления APC из препаратов APC стимулируют одним или несколькими цитокинами или факторами роста. В некоторых вариантах осуществления один или несколько цитокинов или факторов роста включают в себя GM-CSF, IL-4, FLT3L, TNF-α, IL-1β, PGE1, IL-6, IL-7, IFN-α, R848, LPS, ss-РНК40, поли I:C или их комбинации.
[234] В некоторых вариантах осуществления антиген представляет собой неоантиген, опухолеассоциированный антиген, вирусный антиген, минoрный антиген гистосовместимости или их комбинации.
[235] В некоторых вариантах осуществления способ проводят ex vivo.
[236] В некоторых вариантах осуществления способ предусматривает инкубацию популяции иммунных клеток из биологического образца с истощенными клетками, экспрессирующими CD14 и CD25, с FLT3L в течение первого периода времени. В некоторых вариантах осуществления способ предусматривает инкубацию по меньшей мере одного пептида с популяцией иммунных клеток из биологического образца с истощенными клетками, экспрессирующими CD14 и CD25, в течение второго периода времени, получая за счет этого первый образец с загруженным пептидом созревших APC. В некоторых вариантах осуществления способ предусматривает истощение клеток, экспрессирующих CD14, клеток, экспрессирующих CD19, и клеток, экспрессирующих CD25, из популяции иммунных клеток. В некоторых вариантах осуществления способ предусматривает истощение клеток, экспрессирующих CD14, клеток, экспрессирующих CD11b, и клеток, экспрессирующих CD25, из популяции иммунных клеток. В некоторых вариантах осуществления способ предусматривает истощение клеток, экспрессирующих CD14, клеток, экспрессирующих CD11b, клеток, экспрессирующих CD19, и клеток, экспрессирующих CD25. В некоторых вариантах осуществления способ предусматривает истощение по меньшей мере CD14, CD11b, CD19 и CD25. В некоторых вариантах осуществления способ предусматривает истощение клеток, экспрессирующих по меньшей мере один из CD14, CD11b, CD19 и CD25, и по меньшей мере пятого типа клеток, экспрессирующих пятый маркер клеточной поверхности. В некоторых вариантах осуществления способ предусматривает выборочное истощение клеток, экспрессирующих CD14 и CD25, из популяции иммунных клеток и клеток, экспрессирующих любой один или несколько из CD19, CD11b, из популяции иммунных клеток, в первый инкубационный период, во второй инкубационный период и/или в третий инкубационный период.
[237] В некоторых вариантах осуществления способа, описанного в данном документе, введение в контакт T-клетки субъекта или аллогенной T-клетки с одним или несколькими пептидами, содержащими по меньшей мере одну выбранную эпитопную последовательность, предусматривает введение в контакт T-клетки с APC, презентирующими эпитоп.
[238] В некоторых вариантах осуществления способа, описанного в данном документе, APC, презентирующие эпитоп, содержат один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность, или полинуклеиновую кислоту, которая кодирует один или несколько пептидов, содержащих по меньшей мере одну выбранную эпитопную последовательность.
[239] В некоторых вариантах осуществления способ предусматривает истощение CD14+ клеток и CD25+ клеток из популяции иммунных клеток, содержащей антигенпрезентирующие клетки (APC) и T-клетки, образуя за счет этого популяцию иммунных клеток с истощенными CD14/CD25, содержащую первую популяцию APC и T-клеток. В некоторых вариантах осуществления популяцию иммунных клеток получают из биологического образца субъекта. В некоторых вариантах осуществления способа, описанного в данном документе, способ дополнительно включает инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей первую популяцию APC и T-клеток, в течение первого периода времени в присутствии лиганда рецептора FMS-подобной тирозинкиназы 3 (FLT3L) и полипептида, содержащего по меньшей мере один выбранный эпитопные последовательности или полинуклеотида, кодирующего полипептид; образуя за счет этого популяцию клеток, содержащую стимулированные T-клетки. В некоторых вариантах осуществления способ дополнительно предусматривает размножение популяции клеток, содержащей стимулированные T-клетки, образуя за счет этого размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки, причем специфические в отношении опухолевых антигенов T-клетки включают в себя T-клетки, которые являются специфичными к комплексу, содержащему по меньшей мере одну из выбранных эпитопных последовательностей и белок MHC, экспрессируемый раковыми клетками или APC субъекта.
[240] В некоторых вариантах осуществления способа, описанного в данном документе, размножение предусматривает введение в контакт популяции клеток, содержащей стимулированные T-клетки, со второй популяцией зрелых APC, причем вторая популяция зрелых APC инкубирована с FLT3L и презентирует по меньшей мере одну выбранную эпитопную последовательность, и размножение популяции клеток, содержащей стимулированные T-клетки, в течение второго периода времени, образуя за счет этого размноженную популяцию T-клеток. В некоторых вариантах осуществления вторую популяцию зрелых APC инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт популяции клеток, содержащей стимулированные T-клетки, со второй популяцией зрелых APC. В некоторых вариантах осуществления размножение дополнительно предусматривает введение в контакт размноженной популяции T-клеток с третьей популяцией зрелых APC, причем третья популяция зрелых APC инкубирована с FLT3L и презентирует по меньшей мере одну выбранную эпитопную последовательность; и размножение размноженной популяции T-клеток в течение третьего периода времени, образуя за счет этого размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки. В некоторых вариантах осуществления третью популяцию зрелых APC инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт размноженной популяции T-клеток с третьей популяцией зрелых APC. В некоторых вариантах осуществления способа, описанного в данном документе, способ дополнительно предусматривает сбор размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, криоконсервацию размноженной популяции клеток, содержащей специфические в отношении опухолевых антигенов T-клетки, или получение фармацевтической композиции, содержащей размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки. В некоторых вариантах осуществления инкубация представляет собой инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей первую популяцию APC и T-клеток, в течение первого периода времени в присутствии FLT3L и РНК, кодирующей полипептид.
[241] В некоторых вариантах осуществления способ дополнительно предусматривает введение субъекту-человеку с раком фармацевтической композиции, содержащей размноженную популяцию клеток, содержащую специфические в отношении опухолевых антигенов T-клетки. В некоторых вариантах осуществления субъектом-человеком с раком является субъект-человек, у которого был получен биологический образец. В некоторых вариантах осуществления полипептид имеет длину от 8 до 50 аминокислот. В некоторых вариантах осуществления полипептид содержит по меньшей мере две выбранные эпитопные последовательности, каждую из которых экспрессируют раковые клетки субъекта-человека с раком.
[242] В некоторых вариантах осуществления истощение CD14+ клеток и CD25+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток, предусматривает введение в контакт популяции иммунных клеток, содержащей первую популяцию APC и T-клеток, со связывающим CD14 средством и связывающим CD25 средством. В некоторых вариантах осуществления истощение дополнительно представляет собой истощение CD19+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток. В некоторых вариантах осуществления способ дополнительно предусматривает введение в контакт популяции иммунных клеток со связывающим CD19 средством. В некоторых вариантах осуществления истощение дополнительно представляет собой истощение CD11b+ клеток из популяции иммунных клеток, содержащей первую популяцию APC и T-клеток. В некоторых вариантах осуществления способ дополнительно предусматривает введение в контакт популяции иммунных клеток со связывающим CD11b средством.
[243] В некоторых вариантах осуществления способ предусматривает инкубацию образца с загруженным пептидом первых созревших APC по меньшей мере с одной T-клеткой в течение третьего периода времени, получая за счет этого образец стимулированных T-клеток. В некоторых вариантах осуществления способ предусматривает инкубацию T-клеток из первого образца стимулированных T-клеток со стимулированными FLT3L APC из образца созревших APC в течение четвертого периода времени, FLT3L и вторым образцом с загруженным пептидом APC из образца созревших APC в течение четвертого периода времени или FLT3L и стимулированными FLT3L APC из образца созревших APC в течение четвертого периода времени, получая за счет этого образец стимулированных T-клеток. В некоторых вариантах осуществления способ предусматривает инкубацию T-клеток из второго образца стимулированных T-клеток со стимулированными FLT3L APC из образца созревших APC в течение пятого периода времени, FLT3L и третьим образцом с загруженным пептидом APC из образца созревших APC в течение пятого периода времени или FLT3L и третьим образцом с загруженным пептидом APC из образца созревших APC в течение пятого периода времени, получая за счет этого образец стимулированных T-клеток.
[244] В некоторых вариантах осуществления один или несколько отдельных периодов времени, 3 или менее отдельных периодов времени, первый период времени, второй период времени, третий период времени, четвертый период времени или пятый период времени составляет по меньшей мере 1 час, по меньшей мере 2 часа, по меньшей мере 3 часа, по меньшей мере 4 часа, по меньшей мере 5 часов, по меньшей мере 6 часов, по меньшей мере 7 часов, по меньшей мере 8 часов, по меньшей мере 9 часов, по меньшей мере 10 часов, по меньшей мере 11 часов, по меньшей мере 12 часов, по меньшей мере 13 часов, по меньшей мере 14 часов, по меньшей мере 15 часов, по меньшей мере 16 часов, по меньшей мере 17 часов, по меньшей мере 18 часов, по меньшей мере 19 часов, по меньшей мере 20 часов, по меньшей мере 21 час, по меньшей мере 22 часа, по меньшей мере 23 часа, по меньшей мере 24 часа, по меньшей мере 25 часов, по меньшей мере 26 часов, по меньшей мере 27 часов, по меньшей мере 28 часов, по меньшей мере 29 часов, по меньшей мере 30 часов, по меньшей мере 31 час, по меньшей мере 32 часа, по меньшей мере 33 часа, по меньшей мере 34 часа, по меньшей мере 35 часов, по меньшей мере 36 часов, по меньшей мере 37 часов, по меньшей мере 38 часов, по меньшей мере 39 часов или по меньшей мере 40 часов.
[245] В некоторых вариантах осуществления один или несколько отдельных периодов времени, 3 или менее отдельных периодов времени, первый период времени, второй период времени, третий период времени, четвертый период времени или пятый период времени составляет от 1 до 4 часов, от 1 до 3 часов, от 1 до 2 часов, от 4 до 40 часов, от 7 до 40 часов, от 4 до 35 часов, от 4 до 32 часов, от 7 до 35 часов или от 7 до 32 часов.
[246] В некоторых вариантах осуществления популяция иммунных клеток содержит APC или по меньшей мере один из одного или нескольких препаратов APC. В некоторых вариантах осуществления популяция иммунных клеток не содержит APC, и/или популяция иммунных клеток не содержит один из одного или нескольких препаратов APC.
[247] В некоторых вариантах осуществления способ предусматривает инкубацию FLT3L и по меньшей мере одного пептида с популяцией иммунных клеток из биологического образца, при этом FLT3L инкубируют с популяцией иммунных клеток в течение первого периода времени, и при этом по меньшей мере один пептид инкубируют с популяцией иммунных клеток в течение первого периода времени стимуляции пептида, получая за счет этого первый образец стимулированных T-клеток, в котором популяция иммунных клеток содержит по меньшей мере одну T-клетку и по меньшей мере одну APC. В некоторых вариантах осуществления способ предусматривает инкубацию FLT3L и по меньшей мере одного пептида по меньшей мере с одной APC, при этом FLT3L инкубируют по меньшей мере с одной APC в течение второго периода времени, и при этом по меньшей мере один пептид инкубируют по меньшей мере с одной APC в течение второго периода времени стимуляции пептида, получая за счет этого первый образец с загруженным пептидом созревших APC; и инкубацию первого образца с загруженным пептидом созревших APC с первым образцом стимулированных T-клеток, получая за счет этого второй образец стимулированных T-клеток. В некоторых вариантах осуществления способ предусматривает инкубацию FLT3L и по меньшей мере одного пептида по меньшей мере с одной APC, при этом FLT3L инкубируют по меньшей мере с одной APC в течение третьего периода времени, и при этом по меньшей мере один пептид инкубируют по меньшей мере с одной APC в течение третьего периода времени стимуляции пептида, получая за счет этого второй образец с загруженным пептидом созревших APC; и инкубацию второго образца с загруженным пептидом созревших APC со вторым образцом стимулированных T-клеток, получая за счет этого третий образец стимулированных T-клеток.
[248] В некоторых вариантах осуществления способ дополнительно предусматривает выделение первой стимулированной T-клетки из образца стимулированных T-клеток. В некоторых вариантах осуществления выделение, описанное в предыдущем предложении, включает обогащение стимулированных Т-клеток из популяции иммунных клеток, которые контактировали по меньшей мере с одной APC, инкубированной по меньшей мере с одним пептидом. В некоторых вариантах осуществления обогащение включает определение экспрессии одного или нескольких клеточных маркеров по меньшей мере одной стимулированной T-клетки и выделение стимулированной T-клетки, экспрессирующей один или несколько клеточных маркеров. В некоторых вариантах осуществления маркером клеточной поверхности может быть, но без ограничения, один или несколько из TNF-α, IFN-γ, LAMP-1, 4-1BB, IL-2, IL-17A, гранзима B, PD-1, CD25, CD69, TIM3, LAG3, CTLA-4, CD62L, CD45RA, CD45RO, FoxP3 или любая их комбинация. В некоторых вариантах осуществления один или несколько клеточных маркеров содержат цитокин.
[249] В некоторых вариантах осуществления способ предусматривает введение по меньшей мере одной T-клетки из первого или второго, или третьего образца стимулированных T-клеток нуждающемуся в этом субъекту.
[250] В некоторых вариантах осуществления способ предусматривает: получение биологического образца субъекта, содержащего по меньшей мере одну антигенпрезентирующую клетку (APC); обогащение клеток, экспрессирующих CD14, из биологического образца, получая за счет этого обогащенный CD14+ клетками образец; инкубацию обогащенного CD14+ клетками образца по меньшей мере с одним цитокином или фактором роста в течение первого периода времени; инкубацию по меньшей мере одного пептида с обогащенным CD14+ клетками образцом в течение второго периода времени, получая за счет этого образец с загруженным пептидом APC; инкубацию образца с загруженным пептидом APC с одним или несколькими цитокинами или факторами роста в течение третьего периода времени, получая за счет этого образец созревших APC; инкубацию APC из образца созревших APC с образцом с истощенными CD14 и CD25, содержащим T-клетки в течение четвертого периода времени; инкубацию T-клеток с APC из образца созревших APC в течение пятого периода времени; инкубацию T-клеток с APC из образца созревших APC в течение шестого периода времени; и введение по меньшей мере одной T-клетки из этих T-клеток нуждающемуся в этом субъекту.
[251] В некоторых вариантах осуществления способ предусматривает: получение биологического образца субъекта, содержащего по меньшей мере одну APC и по меньшей мере одну T-клетку; истощение клеток, экспрессирующих CD14 и CD25, из биологического образца, получая за счет этого образец с истощенными CD14 и CD25 клетками; инкубацию образца с истощенными CD14 и CD25 клетками с FLT3L в течение первого периода времени; инкубацию по меньшей мере одного пептида с образцом с истощенными CD14 и CD25 клетками в течение второго периода времени, получая за счет этого образец с загруженным пептидом APC; инкубацию образца с загруженным пептидом APC по меньшей мере с одной T-клеткой в течение третьего периода времени, получая за счет этого первый образец стимулированных T-клеток; инкубацию T-клетки из первого образца стимулированных T-клеток с APC из образца созревших APC в течение четвертого периода времени, получая за счет этого второй образец стимулированных T-клеток; необязательно, инкубацию T-клетки из второго образца стимулированных T-клеток с APC из образца созревших APC в течение пятого периода времени, получая за счет этого третий образец стимулированных T-клеток; введение по меньшей мере одной T-клетки из первого, второго или третьего образца стимулированных T-клеток нуждающемуся в этом субъекту.
[252] В некоторых вариантах осуществления способ предусматривает: получение биологического образца субъекта, содержащего по меньшей мере одну APC и по меньшей мере одну T-клетку; истощение клеток, экспрессирующих CD14 и CD25, из биологического образца, получая за счет этого образец с истощенными CD14 и CD25 клетками; инкубацию образца с истощенными CD14 и CD25 клетками с FLT3L в течение первого периода времени; инкубацию по меньшей мере одного пептида с образцом с истощенными CD14 и CD25 клетками в течение второго периода времени, получая за счет этого образец с загруженным пептидом APC; инкубацию образца с загруженным пептидом APC по меньшей мере с одной T-клеткой в течение третьего периода времени, получая за счет этого первый образец стимулированных T-клеток; необязательно, инкубацию T-клетки из первого образца стимулированных T-клеток со стимулированными FLT3L APC из образца созревших APC в течение четвертого периода времени, получая за счет этого второй образец стимулированных T-клеток; необязательно, инкубацию T-клетки из второго образца стимулированных T-клеток со стимулированными FLT3L APC из образца созревших APC в течение пятого периода времени, получая за счет этого третий образец стимулированных T-клеток; введение по меньшей мере одной T-клетки из первого, второго или третьего образца стимулированных T-клеток нуждающемуся в этом субъекту.
[253] В некоторых вариантах осуществления способ предусматривает: получение биологического образца субъекта, содержащего по меньшей мере одну APC и по меньшей мере одну T-клетку; истощение клеток, экспрессирующих CD14 и CD25, из биологического образца, получая за счет этого образец с истощенными CD14 и CD25 клетками; инкубацию образца с истощенными CD14 и CD25 клетками с FLT3L в течение первого периода времени; инкубацию по меньшей мере одного пептида с образцом с истощенными CD14 и CD25 клетками в течение второго периода времени, получая за счет этого первый образец с загруженным пептидом APC; инкубацию первого образца с загруженным пептидом APC по меньшей мере с одной T-клеткой в течение третьего периода времени, получая за счет этого первый образец стимулированных T-клеток; необязательно, инкубацию T-клетки из первого образца стимулированных T-клеток с FLT3L и вторым образцом с загруженным пептидом APC из образца созревших APC в течение четвертого периода времени, получая за счет этого второй образец стимулированных T-клеток; необязательно, инкубацию T-клетки из второго образца стимулированных T-клеток с FLT3L и третьим образцом с загруженным пептидом APC из образца созревших APC в течение пятого периода времени, получая за счет этого третий образец стимулированных T-клеток; введение по меньшей мере одной T-клетки из первого, второго или третьего образца стимулированных T-клеток нуждающемуся в этом субъекту.
[254] В некоторых вариантах осуществления способ предусматривает: получение биологического образца субъекта, содержащего по меньшей мере одну APC и по меньшей мере одну T-клетку; истощение клеток, экспрессирующих CD14 и CD25, из биологического образца, получая за счет этого образец с истощенными CD14 и CD25 клетками; инкубацию образца с истощенными CD14 и CD25 клетками с FLT3L в течение первого периода времени; инкубацию по меньшей мере одного пептида с образцом с истощенными CD14 и CD25 клетками в течение второго периода времени, получая за счет этого первый образец с загруженным пептидом APC; инкубацию первого образца с загруженным пептидом APC по меньшей мере с одной T-клеткой в течение третьего периода времени, получая за счет этого первый образец стимулированных T-клеток; необязательно, инкубацию T-клетки из первого образца стимулированных T-клеток с FLT3L и стимулированными FLT3L APC из образца созревших APC в течение четвертого периода времени, получая за счет этого второй образец стимулированных T-клеток; необязательно, инкубацию T-клетки из второго образца стимулированных T-клеток с FLT3L и стимулированными FLT3L APC из образца созревших APC в течение пятого периода времени, получая за счет этого третий образец стимулированных T-клеток; введение по меньшей мере одной T-клетки из первого, второго или третьего образца стимулированных T-клеток нуждающемуся в этом субъекту.
[255] В некоторых вариантах осуществления способ предусматривает: инкубацию FLT3L и по меньшей мере одного пептида с популяцией иммунных клеток из биологического образца, при этом FLT3L инкубируют с популяцией иммунных клеток в течение первого периода времени, и при этом по меньшей мере один пептид инкубируют с популяцией иммунных клеток в течение первого периода времени стимуляции пептида, получая за счет этого первый образец стимулированных T-клеток, в котором популяция иммунных клеток содержит по меньшей мере одну T-клетку и по меньшей мере одну APC; необязательно, инкубацию FLT3L и по меньшей мере одного пептида по меньшей мере с одной APC, при этом FLT3L инкубируют по меньшей мере с одной APC в течение второго периода времени, и при этом по меньшей мере один пептид инкубируют по меньшей мере с одной APC в течение второго периода времени стимуляции пептида, получая за счет этого первый образец с загруженным пептидом созревших APC; и инкубацию первого образца с загруженным пептидом созревших APC с первым образцом стимулированных T-клеток, получая за счет этого второй образец стимулированных T-клеток; необязательно, инкубацию FLT3L и по меньшей мере одного пептида по меньшей мере с одной APC, при этом FLT3L инкубируют по меньшей мере с одной APC в течение третьего периода времени, и при этом по меньшей мере один пептид инкубируют по меньшей мере с одной APC в течение третьего периода времени стимуляции пептида, получая за счет этого второй образец с загруженным пептидом созревших APC; и инкубацию второго образца с загруженным пептидом созревших APC со вторым образцом стимулированных T-клеток, получая за счет этого третий образец стимулированных T-клеток; и введение по меньшей мере одной T-клетки из первого образца стимулированных T-клеток, второго образца стимулированных T-клеток или третьего образца стимулированных T-клеток нуждающемуся в этом субъекту.
[256] В некоторых вариантах осуществления способ предусматривает получение нуклеиновых кислот раковых клеток из первого биологического образца, содержащего раковые клетки, полученные у субъекта, и получение нуклеиновых кислот нераковых клеток из второго биологического образца, содержащего нераковые клетки, полученные у того же субъекта.
[257] В некоторых вариантах осуществления способ предусматривает секвенирование нуклеиновых кислот раковых клеток путем полногеномного секвенирования или полноэкзомного секвенирования, получая за счет этого первое множество последовательностей нуклеиновых кислот, содержащее последовательности нуклеиновых кислот раковых клеток; и секвенирование нуклеиновых кислот нераковых клеток путем полногеномного секвенирования или полноэкзомного секвенирования, получая за счет этого второе множество последовательностей нуклеиновых кислот, содержащее последовательности нуклеиновых кислот нераковых клеток. В некоторых вариантах осуществления способ предусматривает идентификацию множества специфических к раку последовательностей нуклеиновых кислот из первого множества последовательностей нуклеиновых кислот, которые являются уникальными для раковых клеток субъекта и которые не включают последовательностей нуклеиновых кислот из второго множества последовательностей нуклеиновых кислот из нераковых клеток субъекта.
[258] В некоторых вариантах осуществления способ дополнительно предусматривает выбор одной или нескольких субпопуляций клеток из размноженной популяции T-клеток перед введением субъекту. В некоторых вариантах осуществления выбор одной или нескольких субпопуляций проводят путем сортировки клеток на основании экспрессии одного или нескольких маркеров клеточной поверхности, представленных в данном документе. В некоторых вариантах осуществления активированные T-клетки можно сортировать на основании маркеров клеточной поверхности, в том числе, но без ограничения, любого одного или нескольких из следующих: CD27, CD274, CD276, CD8A, CMKLR1, CXCL9, CXCR6, HLA-DQA1, HLA-DRB1, HLA-E, IDO1, LAG3, NKG7, PDCD1LG2, PSMB10, STAT1, CD45RO, CCR7, FLT3LG, IL-6 и других.
[259] В некоторых вариантах осуществления способ дополнительно предусматривает истощение одной или нескольких клеток у субъекта перед введением популяции T-клеток.
[260] В некоторых вариантах осуществления в данном документе представлена одна или несколько субпопуляций клеток, экспрессирующих маркер клеточной поверхности.
[261] В некоторых вариантах осуществления аминокислотную последовательность пептида, представленного в данном документе, проверяют путем секвенирования пептида. В некоторых вариантах осуществления аминокислотную последовательность пептида, представленного в данном документе, проверяют посредством масс-спектрометрии.
[262] Также в данном документе представлена фармацевтическая композиция, содержащая T-клетку, полученную путем размножения T-клетки в присутствии антигенпрезентирующей клетки, презентирующей одну или несколько последовательностей эпитопа по любой из таблиц 1-8 и 11-14.
[263] Также в данном документе представлена библиотека полипептидов, содержащая эпитопные последовательности или полинуклеотиды, кодирующие полипептиды, причем каждая эпитопная последовательность в библиотеке соответствует белку, кодируемому аллелем HLA; и при этом каждую эпитопную последовательность в библиотеке предварительно проверяют на соответствие по меньшей мере двум или трем, или четырем следующим критериям: связывается с белком, кодируемым аллелем HLA субъекта с раком, подлежащего лечению, является иммуногенной согласно иммуногенному анализу, презентируется антигенпрезентирующими клетками (APC) согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности. В некоторых вариантах осуществления библиотека содержит одну или две, или более пептидных последовательностей, содержащих эпитопную последовательность по любой из таблиц 1-8 и 11-14.
[264] пептиды и полинуклеотиды, представленные в данном документе, могут быть предназначены для получения антигенспецифических T-клеток и включают рекомбинантные пептиды и полинуклеотиды и синтетические пептиды, содержащие эпитопы, такие как опухолеспецифические неоэпитопы, которые были идентифицированы и подтверждены как связывающиеся с одной или несколькими молекулами MHC, презентируемые одной или несколькими молекулами MHC, являющиеся иммуногенными и/или способные активировать возникновение цитотоксичности T-клеток. Пептиды можно получать для использования в способе примирования T-клеток ex vivo. Пептиды можно получать для использования в способе активации T-клеток ex vivo. Пептиды можно получать для использования в способе размножения антигенспецифических T-клеток. Пептиды можно получать для использования в способе индукции CD8 T-клеточных ответов de novo ex vivo. Пептиды можно получать для использования в способе индукции CD4 T-клеточных ответов de novo ex vivo. Пептиды можно получать для использования в способе стимуляции ответов CD8 T-клеток памяти ex vivo. Пептиды можно получать для использования в способе стимуляции ответов CD4 T-клеток памяти ex vivo. T-клетки можно получать у субъекта-человека. T-клетки могут быть аллогенными T-клетками. T-клетки могут быть T-клеточными линиями.
[265] Эпитопы могут содержать по меньшей мере 8 смежных аминокислот аминокислотной последовательности, кодируемой геномом раковой клетки. Эпитопы могут состоять из 8-12 смежных аминокислот аминокислотной последовательности, кодируемой геномом раковой клетки. Эпитопы могут состоять из 13-25 смежных аминокислот аминокислотной последовательности, кодируемой геномом раковой клетки. Эпитопы могут состоять из 8-50 смежных аминокислот аминокислотной последовательности, кодируемой геномом раковой клетки. В некоторых вариантах осуществления эпитоп имеет длину от приблизительно 8 до приблизительно 30 аминокислот. В некоторых вариантах осуществления эпитоп имеет длину от приблизительно 8 до приблизительно 25 аминокислот. В некоторых вариантах осуществления эпитоп имеет длину от приблизительно 15 до приблизительно 24 аминокислот. В некоторых вариантах осуществления эпитоп имеет длину от приблизительно 9 до приблизительно 15 аминокислот. В некоторых вариантах осуществления эпитоп имеет длину 8 аминокислот. В некоторых вариантах осуществления эпитоп имеет длину 9 аминокислот. В некоторых вариантах осуществления эпитоп имеет длину 10 аминокислот.
[266] В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину самое большее 500, самое большее 250, самое большее 150, самое большее 125 или самое большее 100 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину по меньшей мере 8, по меньшей мере 50, по меньшей мере 100, по меньшей мере 200 или по меньшей мере 300 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 500 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 100 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 50 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 15 до приблизительно 35 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 15 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 11 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину 9 или 10 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 30 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 8 до приблизительно 25 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 15 до приблизительно 24 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет длину от приблизительно 9 до приблизительно 15 аминокислот.
[267] В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет общую длину по меньшей мере 8, по меньшей мере 9, по меньшей мере 10, по меньшей мере 11, по меньшей мере 12, по меньшей мере 13, по меньшей мере 14, по меньшей мере 15, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18, по меньшей мере 19, по меньшей мере 20, по меньшей мере 21, по меньшей мере 22, по меньшей мере 23, по меньшей мере 24, по меньшей мере 25, по меньшей мере 26, по меньшей мере 27, по меньшей мере 28, по меньшей мере 29, по меньшей мере 30, по меньшей мере 40, по меньшей мере 50, по меньшей мере 60, по меньшей мере 70, по меньшей мере 80, по меньшей мере 90, по меньшей мере 100, по меньшей мере 150, по меньшей мере 200, по меньшей мере 250, по меньшей мере 300, по меньшей мере 350, по меньшей мере 400, по меньшей мере 450 или по меньшей мере 500 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, имеет общую длину самое большее 8, самое большее 9, самое большее 10, самое большее 11, самое большее 12, самое большее 13, самое большее 14, самое большее 15, самое большее 16, самое большее 17, самое большее 18, самое большее 19, самое большее 20, самое большее 21, самое большее 22, самое большее 23, самое большее 24, самое большее 25, самое большее 26, самое большее 27, самое большее 28, самое большее 29, самое большее 30, самое большее 40, самое большее 50, самое большее 60, самое большее 70, самое большее 80, самое большее 90, самое большее 100, самое большее 150, самое большее 200, самое большее 250, самое большее 300, самое большее 350, самое большее 400, самое большее 450 или самое большее 500 аминокислот. В некоторых вариантах осуществления пептид, содержащий эпитоп, содержит первый неоэпитопный пептид, связанный по меньшей мере со вторым неоэпитопом.
[268] В некоторых вариантах осуществления пептид содержит проверенный эпитоп из одного или нескольких из: ABL1, AC011997, ACVR2A, AFP, AKT1, ALK, ALPPL2, ANAPC1, APC, ARID1A, AR, AR-v7, ASCL2, β2M, BRAF, BTK, C15ORF40, CDH1, CLDN6, CNOT1, CT45A5, CTAG1B, DCT, DKK4, EEF1B2, EEF1DP3, EGFR, EIF2B3, env, EPHB2, ERBB3, ESR1, ESRP1, FAM111B, FGFR3, FRG1B, GAGE1, GAGE10, GATA3, GBP3, HER2, IDH1, JAK1, KIT, KRAS, LMAN1, MABEB16, MAGEA1, MAGEA10, MAGEA4, MAGEA8, MAGEB17, MAGEB4, MAGEC1, MEK, MLANA, MLL2, MMP13, MSH3, MSH6, MYC, NDUFC2, NRAS, PAGE2, PAGE5, PDGFRa, PIK3CA, PMEL, белка pol, POLE, PTEN, RAC1, RBM27, RNF43, RPL22, RUNX1, SEC31A, SEC63, SF3B1, SLC35F5, SLC45A2, SMAP1, SMAP1, SPOP, TFAM, TGFBR2, THAP5, TP53, TTK, TYR, UBR5, VHL, XPOT, полипептида слияния EEF1DP3:FRY, полипептида слияния EGFR:SEPT14, полипептида с делецией EGFRVIII, полипептида слияния EML4:ALK, полипептида слияния NDRG1:ERG, полипептида слияния AC011997.1:LRRC69, полипептида слияния RUNX1(ex5)-RUNX1T1, полипептида слияния TMPRSS2:ERG, полипептида слияния NAB:STAT6, полипептида слияния NDRG1:ERG, полипептида слияния PML:RARA, полипептида слияния PPP1R1B:STARD3, полипептида слияния MAD1L1:MAFK, полипептида слияния FGFR3:TAC, полипептида слияния FGFR3:TACC3, полипептида слияния BCR:ABL, полипептида слияния C11orf95:RELA, полипептида слияния CBFB:MYH11, полипептида слияния CBFB:MYH11, полипептида слияния CD74:ROS1, полипептида слияния CD74:ROS1, ERVE-4: протеазы, ERVE-4: обратной транскриптазы, ERVE-4: обратной транскриптазы, ERVE-4: неизвестно, ERVH-2 матричного белка, ERVH-2: gag, ERVH-2: ретровирусной матрицы, ERVH48-1: белка оболочки, ERVH48-1: синцитина, ERVI-1 белка оболочки, ERVK-5 gag, ERVK-5 env, ERVK-5 pol, EBV A73, EBV BALF3, EBV BALF4, EBV BALF5, EBV BARF0, EBV LF2, EBV RPMS1, HPV-16, HPV-16 E7 и HPV-16 E6. В некоторых вариантах осуществления неоэпитоп содержит мутацию, вызванную событием мутации в β2M, BTK, EGFR, GATA3, KRAS, MLL2, полипептиде слияния TMPRSS2:ERG или TP53, или Myc.
[269] В некоторых вариантах осуществления эпитоп связывает молекулу главного комплекса гистосовместимости (MHC) I класса. В некоторых вариантах осуществления эпитоп связывает молекулу MHC I класса с аффинностью связывания приблизительно 500 нМ или менее. В некоторых вариантах осуществления эпитоп связывает молекулу MHC I класса с аффинностью связывания приблизительно 250 нМ или менее. В некоторых вариантах осуществления эпитоп связывает молекулу MHC I класса с аффинностью связывания приблизительно 150 нМ или менее. В некоторых вариантах осуществления эпитоп связывает молекулу MHC I класса с аффинностью связывания приблизительно 50 нМ или менее.
[270] В некоторых вариантах осуществления эпитоп связывает молекулу MHC I класса, а пептид, содержащий эпитоп I класса, связывается с молекулой MHC II класса.
[271] В некоторых вариантах осуществления эпитоп связывает молекулу MHC II класса. В некоторых вариантах осуществления эпитоп связывается с антигеном лейкоцита человека (HLA) -A, -B, -C, -DP, -DQ или -DR. В некоторых вариантах осуществления эпитоп связывает молекулу MHC II класса с аффинностью связывания 1000 нМ или менее. В некоторых вариантах осуществления эпитоп связывает MHC II класса с аффинностью связывания 500 нМ или менее. В некоторых вариантах осуществления эпитоп связывает молекулу MHC II класса с аффинностью связывания приблизительно 250 нМ или менее. В некоторых вариантах осуществления эпитоп связывает молекулу MHC II класса с аффинностью связывания приблизительно 150 нМ или менее. В некоторых вариантах осуществления эпитоп связывает молекулу MHC II класса с аффинностью связывания приблизительно 50 нМ или менее.
[272] В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит одну или несколько аминокислот, фланкирующих C-конец эпитопа. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит одну или несколько аминокислот, фланкирующих N-конец эпитопа. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит одну или несколько аминокислот, фланкирующих C-конец эпитопа, и одну или несколько аминокислот, фланкирующих N-конец эпитопа. В некоторых вариантах осуществления фланкирующие аминокислоты являются не нативными фланкирующими аминокислотами. В некоторых вариантах осуществления первый эпитоп использованный в способе, описанном в данном документе, связывает молекулу MHC I класса, а второй эпитоп связывает молекулу MHC II класса. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая увеличивает время полужизни пептида in vivo. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая увеличивает нацеливание пептида на клетку. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая увеличивает клеточное поглощение пептида. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая повышает процессинг пептида. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая повышает аффинность эпитопа MHC. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая повышает стабильность эпитопа MHC. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая повышает презентацию эпитопа молекулой MHC I класса и/или молекулой MHC II класса.
[273] В некоторых вариантах осуществления способы секвенирования используют для идентификации опухолеспецифических мутаций. Можно использовать любой подходящий способ секвенирования согласно изобретению, например, технологии секвенирования следующего поколения (NGS). В будущем методы секвенирования третьего поколения могут заменить технологию NGS, чтобы ускорить этап секвенирования метода. В целях пояснения: термины «Секвенирование следующего поколения» или «NGS» в контексте настоящего изобретения означают все новые технологии высокопроизводительного секвенирования, которые, в отличие от «традиционной» методологии секвенирования, известной как химия Сэнгера, считывают матрицы нуклеиновых кислот случайным образом, параллельно по всему геному, разбивая весь геном на небольшие части. Такие технологии NGS (также известные как технологии массового параллельного секвенирования) способны доставлять информацию о последовательностях нуклеиновых кислот всего генома, экзома, транскриптома (всех транскрибируемых последовательностей генома) или метилома (всех метилированных последовательностей генома) за очень короткое периоды времени, например в течение 1-2 недель, например, в течение 1-7 дней или в течение менее 24 часов, и в принципе допускают подходы к секвенированию отдельных клеток. В контексте изобретения можно использовать несколько платформ NGS, которые коммерчески доступны или упомянуты в литературе, например, те, что подробно описаны в WO 2012/159643.
[274] В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, связан по меньшей мере со вторым пептидом, например, с помощью полиглицинового или полисеринового линкера. В некоторых вариантах осуществления модификация представляет собой конъюгацию с белком-носителем, конъюгацию с лигандом, конъюгацию с антителом, пегилирование, полисиалирование, гесилирование, рекомбинантные миметики ПЭГ, слияние Fc, слияние альбумина, прикрепление наночастиц, инкапсуляцию наночастиц, слияние холестерина, слияние железа, ацилирование, амидирование, гликозилирование, окисление боковой цепи, фосфорилирование, биотинилирование, добавление поверхностно-активного материала, добавление миметиков аминокислот или добавление неприродных аминокислот. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, дополнительно содержит модификацию, которая увеличивает клеточное нацеливание на конкретные органы, ткани или типы клеток. В некоторых вариантах осуществления пептид, содержащий проверенный эпитоп, содержит антигенпрезентирующий фрагмент или маркер, нацеливающий на клетку. В некоторых вариантах осуществления антигенпрезентирующие клетки представляют собой дендритные клетки. В некоторых вариантах осуществления дендритные клетки нацеливают с использованием DEC205, XCR1, CD197, CD80, CD86, CD123, CD209, CD273, CD283, CD289, CD184, CD85h, CD85j, CD85k, CD85d, CD85g, CD85a, CD141, CD11c, CD83, рецептора TSLP, маркера Clec9a или CD1a. В некоторых вариантах осуществления дендритные клетки нацеливают с использованием маркера CD141, DEC205, Clec9a или XCR1. В некоторых вариантах осуществления дендритные клетки являются аутологичными клетками. В некоторых вариантах осуществления одна или несколько дендритных клеток связаны с Т-клеткой.
[275] В некоторых вариантах осуществления способ, описанный в данном документе, предусматривает крупномасштабное получение и хранение совпадающих с HLA пептидов, соответствующих общим антигенам для лечения рака или опухоли.
[276] В некоторых вариантах осуществления способ, описанный в данном документе, предусматривает способы лечения, включающие введение субъекту с раком антигенспецифических T-клеток, которые являются специфичными к проверенному эпитопу, выбранному из репертуара пептидов, совпадающих с HLA, находящихся в любой из таблиц 1-8 и 11-14. В некоторых вариантах осуществления эпитопспецифические T-клетки вводят пациенту путем инфузии. В некоторых вариантах осуществления T-клетки вводят пациенту путем прямой внутривенной инъекции. В некоторых вариантах осуществления T-клетка является аутологичной T-клеткой. В некоторых вариантах осуществления T-клетка является аллогенной T-клеткой.
[277] Способы согласно раскрытию можно использовать для лечения любого типа рака, известного в данной области. В некоторых вариантах осуществления способ лечения рака включает лечение рака молочной железы, рака предстательной железы, рака яичников, рака шейки матки, рака кожи, рака поджелудочной железы, колоректального рака, рака почки, рака печени, рака мозга, рака легкого, метастатической меланомы, тимомы, лимфомы, саркомы, мезотелиомы, почечно-клеточной карциномы, рака желудка, рака яичников, NHL, лейкоза, рака матки, рака толстой кишки, рака мочевого пузыря или рака эндометрия. В некоторых вариантах осуществления рак выбирают из группы, состоящей из карциномы, лимфомы, бластомы, саркомы, лейкоза, плоскоклеточного рака, рака легкого (в том числе мелкоклеточного рака легкого, немелкоклеточного рака легкого, аденокарциномы легкого и плоскоклеточного рака легкого), рака брюшины, гепатоцеллюлярного рака, рака желудка (в том числе рака желудочно-кишечного тракта), рака поджелудочной железы, глиобластомы, рака шейки матки, рака яичников, рака печени, рак мочевого пузыря, гепатомы, рака молочной железы, рака толстой кишки, меланомы, рака эндометрия или матки, карциномы слюнной железы, рака почки, рака печени, рака предстательной железы, рака вульвы, рака щитовидной железы, карциномы печени, рака головы и шеи, колоректального рака, рака прямой кишки, саркомы мягких тканей, саркомы Капоши, B-клеточной лимфомы (в том числе фолликулярной неходжкинской лимфомы (NHL) низкой степени, мелкоклеточной лимфоцитарной (SL) NHL, фолликулярной NHL промежуточной степени, диффузной NHL промежуточной степени, иммунобластной NHL высокой степени, лимфобластной NHL высокой степени, мелкоклеточной NHL высокой степени с нерассеченными ядрами, NHL с массивным поражением, мантийноклеточной лимфомы, лимфомы, связанной со СПИДом, и макроглобулинемии Вальденстрема), хронического лимфоцитарного лейкоза (CLL), острого лимфоцитарного лейкоза (ALL), миеломы, волосатоклеточного лейкоза, хронического лейкоза миелобластов и посттрансплантационного лимфопролиферативного расстройства (PTLD), аномальной пролиферации сосудов, связанной с факоматозами, отека, синдрома Мейгса. В неограничивающие примеры видов рака, подлежащего лечению с помощью способов согласно настоящему раскрытию, входит меланома (например, метастатическая злокачественная меланома), рак почки (например, светлоклеточная карцинома), рак предстательной железы (например, гормонорезистентная аденокарцинома предстательной железы), аденокарцинома поджелудочной железы, рак молочной железы, рак толстой кишки, рак легкого (например, немелкоклеточный рак легкого), рак пищевода, плоскоклеточная карцинома головы и шеи, рак печени, рак яичников, рак шейки матки, рак щитовидной железы, глиобластома, глиома, лейкоз, лимфома и другие злокачественные новообразования. В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов лечения согласно настоящему раскрытию, выбирают из группы, состоящей из карциномы, плоскоклеточного рака, аденокарциномы, саркомы, рака эндометрия, рака молочной железы, рака яичников, рака шейки матки, рака маточной трубы, первичного рака брюшин, рака толстой кишки, колоректального рака, плоскоклеточной карциномы аногенитальной области, меланомы, почечно-клеточной карциномы, рака легкого, немелкоклеточного рака легкого, плоскоклеточной карциномы легкого, рака желудка, рака мочевого пузыря, рака желчного пузыря, рака печени, рака щитовидной железы, рака гортани, рака слюнной железы, рака пищевода, рака головы и шеи, глиобластомы, глиомы, плоскоклеточной карциномы головы и шеи, рака предстательной железы, рака поджелудочной железы, мезотелиомы, саркомы, гематологического рака, лейкоза, лимфомы, невромы и их комбинаций. В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов согласно настоящему раскрытию, включает, например, карциному, плоскоклеточный рак (например, цервикального канала, века, конъюнктивы оболочки, влагалища, легкого, полости рта, кожи, мочевого пузыря, языка, гортани и пищевода) и аденокарциному (например, предстательной железы, тонкого кишечника, эндометрия, цервикального канала, толстого кишечника, легкого, поджелудочной железы, пищевода, прямой кишки, матки, желудка, молочной железы и яичника). В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов согласно настоящему раскрытию, дополнительно включает саркому (например, миогенную саркому), лейкоз, неврому, меланому и лимфому. В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов согласно настоящему раскрытию, представляет собой рак молочной железы. В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов лечения согласно настоящему раскрытию, представляет собой тройной негативный рак молочной железы (TNBC). В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов лечения согласно настоящему раскрытию, представляет собой рак предстательной железы. В некоторых вариантах осуществления рак, подлежащий лечению с помощью способов лечения согласно настоящему раскрытию, представляет собой колоректальный рак. В некоторых вариантах осуществления пациент или популяция пациентов, подлежащих лечению фармацевтической композицией согласно настоящему раскрытию, имеют солидную опухоль. В некоторых вариантах осуществления солидная опухоль представляет собой меланому, почечно-клеточную карциному, рак легкого, рак мочевого пузыря, рак молочной железы, рака шейки матки, рак толстой кишки, рак желчного пузыря, рак гортани, рак печени, рак щитовидной железы, рак желудка, рак слюнной железы, рак предстательной железы, рак поджелудочной железы или карциному из клеток Меркеля. В некоторых вариантах осуществления пациент или популяция пациентов, подлежащих лечению фармацевтической композицией согласно настоящему раскрытию, имеют гематологический рак. В некоторых вариантах осуществления у пациента имеется гематологический рак, такой как диффузная крупноклеточная В-клеточная лимфома («DLBCL»), ходжкинская лимфома («HL»), неходжкинская лимфома («NHL»), фолликулярная лимфома («FL»), острый миелоидный лейкоз («AML») или множественная миелома («MM»). В некоторых вариантах осуществления пациент или популяция пациентов, подлежащих лечению, имеют рак, выбранный из группы, состоящей из рака яичников, рака легкого и меланомы.
[278] Фармацевтические композиции, представленные в данном документе, можно использовать отдельно или в комбинации с обычными схемами лечения, такими как хирургия, облучение, химиотерапия и/или трансплантация костного мозга (аутологичная, сингенная, аллогенная или неродственная). В некоторых вариантах осуществления по меньшей мере одно или несколько химиотерапевтических средств можно вводить в дополнение к фармацевтической композиции, содержащей иммуногенное терапевтическое средство. В некоторых вариантах осуществления одно или несколько химиотерапевтических средств могут принадлежать к разным классам химиотерапевтических средств. При практическом применении или использовании способов лечения, представленных в данном документе, субъекту, имеющему заболевание или состояние, можно вводить терапевтически эффективные количества фармацевтических композиций. терапевтически эффективное количество может широко варьироваться в зависимости от тяжести заболевания, возраста и относительного здоровья субъекта, эффективности используемых соединений и других факторов.
[279] В некоторых вариантах осуществления способы лечения включают один или несколько циклов лейкафереза перед трансплантацией T-клеток. лейкаферез может включать сбор мононуклеарных клеток периферической крови (PBMC). Лейкаферез может включать мобилизацию PBMC перед сбором. Альтернативно, можно собирать немобилизованные PBMC. за один цикл у субъекта можно собирать большой объем PBMC. Альтернативно, субъект может пройти два или более циклов лейкафереза. объем афереза может зависеть от количества клеток, необходимых для трансплантации. Например, за один цикл у субъекта можно собирать 12-15 литров немобилизованных PBMC. количество PBMC, собираемых у субъекта, может составлять от 1×108 до 5×1010 клеток. количество PBMC, собираемых у субъекта, может составлять 1×108, 5×108, 1×109, 5×109, 1×1010 или 5×1010 клеток. Минимальное количество PBMC, собираемых у субъекта, может составлять 1×106/кг веса субъекта. Минимальное количество PBMC, собираемых у субъекта, может составлять 1×106/кг, 5×106/кг, 1×107/кг, 5×107/кг, 1×108/кг, 5×108/кг веса субъекта.
[280] Разовая инфузия может содержать дозу от 1×106 клеток на квадратный метр поверхности тела субъекта (клеток/м2) и 5×109 клеток/м2. разовая инфузия может содержать от приблизительно 2,5×106 до приблизительно 5×109 клеток/м2. разовая инфузия может содержать от по меньшей мере приблизительно 2,5×106 клеток/м2. разовая инфузия может содержать самое большее 5×109 клеток/м2. разовая инфузия может содержать от 1×106 до 2,5×106, от 1×106 до 5×106, от 1×106 до 7,5×106, от 1×106 до 1×107, от 1×106 до 5×107, от 1×106 до 7,5×107, от 1×106 до 1×108, от 1×106 до 2,5×108, от 1×106 до 5×108, от 1×106 до 1×109, от 1×106 до 5×109, от 2,5×106 до 5×106, от 2,5×106 до 7,5×106, от 2,5×106 до 1×107, от 2,5×106 до 5×107, от 2,5×106 до 7,5×107, от 2,5×106 до 1×108, от 2,5×106 до 2,5×108, от 2,5×106 до 5×108, от 2,5×106 до 1×109, от 2,5×106 до 5×109, от 5×106 до 7,5×106, от 5×106 до 1×107, от 5×106 до 5×107, от 5×106 до 7,5×107, от 5×106 до 1×108, от 5×106 до 2,5×108, от 5×106 до 5×108, от 5×106 до 1×109, от 5×106 до 5×109, от 7,5×106 до 1×107, от 7,5×106 до 5×107, от 7,5×106 до 7,5×107, от 7,5×106 до 1×108, от 7,5×106 до 2,5×108, от 7,5×106 до 5×108, от 7,5×106 до 1×109, от 7,5×106 до 5×109, от 1×107 до 5×107, от 1×107 до 7,5×107, от 1×107 до 1×108, от 1×107 до 2,5×108, от 1×107 до 5×108, от 1×107 до 1×109, от 1×107 до 5×109, от 5×107 до 7,5×107, от 5×107 до 1×108, от 5×107 до 2,5×108, от 5×107 до 5×108, от 5×107 до 1×109, от 5×107 до 5×109, от 7,5×107 до 1×108, от 7,5×107 до 2,5×108, от 7,5×107 до 5×108, от 7,5×107 до 1×109, от 7,5×107 до 5×109, от 1×108 до 2,5×108, от 1×108 до 5×108, от 1×108 до 1×109, от 1×108 до 5×109, от 2,5×108 до 5×108, от 2,5×108 до 1×109, от 2,5×108 до 5×109, от 5×108 до 1×109, от 5×108 до 5×109 или от 1×109 до 5×109 клеток/м2. разовая инфузия может содержать 1×106 клеток/м2, 2,5×106 клеток/м2, 5×106 клеток/м2, 7,5×106 клеток/м2, 1×107 клеток/м2, 5×107 клеток/м2, 7,5×107 клеток/м2, 1×108 клеток/м2, 2,5×108 клеток/м2, 5×108 клеток/м2, 1×109 клеток/м2 или 5×109 клеток/м2.
[281] Способы могут включать применение химиотерапии субъекту, в том числе противолимфоцитарной химиотерапии с использованием высоких доз миелоаблативных средств. В некоторых вариантах осуществления способы включают введение субъекту прекондиционирующего средства, такого как противолимфоцитарное или химиотерапевтическое средство, такое как циклофосфамид, флударабин или их комбинации, перед первой или последующей дозой. Например, субъекту можно вводить прекондиционирующее средство по меньшей мере за 2 дня перед, например, по меньшей мере за 3, 4, 5, 6, 7, 8, 9 или 10 дней перед первой или последующей дозой. В некоторых вариантах осуществления субъекту вводят прекондиционирующее средство не более чем за 10 дней перед, например, не более чем за 9, 8, 7, 6, 5, 4, 3 или за 2 дня перед первой или последующей дозой.
[282] В некоторых вариантах осуществления, где противолимфоцитарное средство содержит циклофосфамид, субъекту вводят циклофосфамид от 0,3 грамм на квадратный метр поверхности тела субъекта (г/м2) до 5 г/м2. В некоторых случаях количество циклофосфамида, вводимого субъекту, составляет приблизительно по меньшей мере 0,3 г/м2. В некоторых случаях количество циклофосфамида, вводимого субъекту, составляет приблизительно самое большее 5 г/м2. В некоторых случаях количество циклофосфамида, вводимого субъекту, составляет приблизительно от 0,3 г/м2 до 0,4 г/м2, от 0,3 г/м2 до 0,5 г/м2, от 0,3 г/м2 до 0,6 г/м2, от 0,3 г/м2 до 0,7 г/м2, от 0,3 г/м2 до 0,8 г/м2, от 0,3 г/м2 до 0,9 г/м2, от 0,3 г/м2 до 1 г/м2, от 0,3 г/м2 до 2 г/м2, от 0,3 г/м2 до 3 г/м2, от 0,3 г/м2 до 4 г/м2, от 0,3 г/м2 до 5 г/м2, от 0,4 г/м2 до 0,5 г/м2, от 0,4 г/м2 до 0,6 г/м2, от 0,4 г/м2 до 0,7 г/м2, от 0,4 г/м2 до 0,8 г/м2, от 0,4 г/м2 до 0,9 г/м2, от 0,4 г/м2 до 1 г/м2, от 0,4 г/м2 до 2 г/м2, от 0,4 г/м2 до 3 г/м2, от 0,4 г/м2 до 4 г/м2, от 0,4 г/м2 до 5 г/м2, от 0,5 г/м2 до 0,6 г/м2, от 0,5 г/м2 до 0,7 г/м2, от 0,5 г/м2 до 0,8 г/м2, от 0,5 г/м2 до 0,9 г/м2, от 0,5 г/м2 до 1 г/м2, от 0,5 г/м2 до 2 г/м2, от 0,5 г/м2 до 3 г/м2, от 0,5 г/м2 до 4 г/м2, от 0,5 г/м2 до 5 г/м2, от 0,6 г/м2 до 0,7 г/м2, от 0,6 г/м2 до 0,8 г/м2, от 0,6 г/м2 до 0,9 г/м2, от 0,6 г/м2 до 1 г/м2, от 0,6 г/м2 до 2 г/м2, от 0,6 г/м2 до 3 г/м2, от 0,6 г/м2 до 4 г/м2, от 0,6 г/м2 до 5 г/м2, от 0,7 г/м2 до 0,8 г/м2, от 0,7 г/м2 до 0,9 г/м2, от 0,7 г/м2 до 1 г/м2, от 0,7 г/м2 до 2 г/м2, от 0,7 г/м2 до 3 г/м2, от 0,7 г/м2 до 4 г/м2, от 0,7 г/м2 до 5 г/м2, от 0,8 г/м2 до 0,9 г/м2, от 0,8 г/м2 до 1 г/м2, от 0,8 г/м2 до 2 г/м2, от 0,8 г/м2 до 3 г/м2, от 0,8 г/м2 до 4 г/м2, от 0,8 г/м2 до 5 г/м2, от 0,9 г/м2 до 1 г/м2, от 0,9 г/м2 до 2 г/м2, от 0,9 г/м2 до 3 г/м2, от 0,9 г/м2 до 4 г/м2, от 0,9 г/м2 до 5 г/м2, от 1 г/м2 до 2 г/м2, от 1 г/м2 до 3 г/м2, от 1 г/м2 до 4 г/м2, от 1 г/м2 до 5 г/м2, от 2 г/м2 до 3 г/м2, от 2 г/м2 до 4 г/м2, от 2 г/м2 до 5 г/м2, от 3 г/м2 до 4 г/м2, от 3 г/м2 до 5 г/м2 или от 4 г/м2 до 5 г/м2. В некоторых случаях количество циклофосфамида, вводимого субъекту, составляет приблизительно 0,3 г/м2, 0,4 г/м2, 0,5 г/м2, 0,6 г/м2, 0,7 г/м2, 0,8 г/м2, 0,9 г/м2, 1 г/м2, 2 г/м2, 3 г/м2, 4 г/м2 или 5 г/м2. В некоторых вариантах осуществления субъекту предварительно вводят циклофосфамид в дозе между или от приблизительно 20 мг/кг до 100 мг/кг, например, между или от приблизительно 40 мг/кг до 80 мг/кг. В некоторых аспектах субъекту предварительно вводят приблизительно 60 мг/кг циклофосфамида.
[283] В некоторых вариантах осуществления, где противолимфоцитарное средство содержит флударабин, субъекту вводят флударабин в дозе между или от приблизительно 1 миллиграмм на квадратный метр поверхности тела субъекта (мг/м2) до 100 мг/м2. В некоторых случаях количество флударабина, вводимого субъекту, составляет приблизительно по меньшей мере 1 мг/м2. В некоторых случаях количество флударабина, вводимого субъекту, составляет приблизительно самое большее 100 мг/м2. В некоторых случаях количество флударабина, вводимого субъекту, составляет приблизительно от 1 мг/м2 до 5 мг/м2, от 1 мг/м2 до 10 мг/м2, от 1 мг/м2 до 15 мг/м2, от 1 мг/м2 до 20 мг/м2, от 1 мг/м2 до 30 мг/м2, от 1 мг/м2 до 40 мг/м2, от 1 мг/м2 до 50 мг/м2, от 1 мг/м2 до 70 мг/м2, от 1 мг/м2 до 90 мг/м2, от 1 мг/м2 до 100 мг/м2, от 5 мг/м2 до 10 мг/м2, от 5 мг/м2 до 15 мг/м2, от 5 мг/м2 до 20 мг/м2, от 5 мг/м2 до 30 мг/м2, от 5 мг/м2 до 40 мг/м2, от 5 мг/м2 до 50 мг/м2, от 5 мг/м2 до 70 мг/м2, от 5 мг/м2 до 90 мг/м2, от 5 мг/м2 до 100 мг/м2, от 10 мг/м2 до 15 мг/м2, от 10 мг/м2 до 20 мг/м2, от 10 мг/м2 до 30 мг/м2, от 10 мг/м2 до 40 мг/м2, от 10 мг/м2 до 50 мг/м2, от 10 мг/м2 до 70 мг/м2, от 10 мг/м2 до 90 мг/м2, от 10 мг/м2 до 100 мг/м2, от 15 мг/м2 до 20 мг/м2, от 15 мг/м2 до 30 мг/м2, от 15 мг/м2 до 40 мг/м2, от 15 мг/м2 до 50 мг/м2, от 15 мг/м2 до 70 мг/м2, от 15 мг/м2 до 90 мг/м2, от 15 мг/м2 до 100 мг/м2, от 20 мг/м2 до 30 мг/м2, от 20 мг/м2 до 40 мг/м2, от 20 мг/м2 до 50 мг/м2, от 20 мг/м2 до 70 мг/м2, от 20 мг/м2 до 90 мг/м2, от 20 мг/м2 до 100 мг/м2, от 30 мг/м2 до 40 мг/м2, от 30 мг/м2 до 50 мг/м2, от 30 мг/м2 до 70 мг/м2, от 30 мг/м2 до 90 мг/м2, от 30 мг/м2 до 100 мг/м2, от 40 мг/м2 до 50 мг/м2, от 40 мг/м2 до 70 мг/м2, от 40 мг/м2 до 90 мг/м2, от 40 мг/м2 до 100 мг/м2, от 50 мг/м2 до 70 мг/м2, от 50 мг/м2 до 90 мг/м2, от 50 мг/м2 до 100 мг/м2, от 70 мг/м2 до 90 мг/м2, от 70 мг/м2 до 100 мг/м2 или от 90 мг/м2 до 100 мг/м2. В некоторых случаях количество флударабина, вводимого субъекту, составляет приблизительно 1 мг/м2, 5 мг/м2, 10 мг/м2, 15 мг/м2, 20 мг/м2, 30 мг/м2, 40 мг/м2, 50 мг/м2, 70 мг/м2, 90 мг/м2 или 100 мг/м2. В некоторых вариантах осуществления флударабин можно вводить в виде разовой дозы или можно вводить в виде множества доз, например, ежедневно, через день или каждые три дня. Например, в некоторых случаях, средство, например, флударабин, вводят между или приблизительно от 1 до 5 раз, например, между или приблизительно от 3 до 5 раз. В некоторых вариантах осуществления такое множество доз вводят в один день, например, от 1 до 5 раз или от 3 до 5 раз ежедневно.
[284] В некоторых вариантах осуществления противолимфоцитарное средство содержит комбинацию средств, например, комбинацию циклофосфамида и флударабина. Таким образом, комбинация средств может включать циклофосфамид в любой дозе или схеме введения, например, как описано выше, и флударабин в любой дозе или схеме введения, например, как описано выше. Например, в некоторых аспектах субъекту вводят 400 мг/м2 циклофосфамида и одну или несколько доз по 20 мг/м2 флударабина перед первой или последующей дозой T-клеток. В некоторых примерах субъекту вводят 500 мг/м2 циклофосфамида и одну или несколько доз по 25 мг/м2 флударабина перед первой или последующей дозой T-клеток. В некоторых примерах субъекту вводят 600 мг/м2 циклофосфамида и одну или несколько доз по 30 мг/м2 флударабина перед первой или последующей дозой T-клеток. В некоторых примерах субъекту вводят 700 мг/м2 циклофосфамида и одну или несколько доз по 35 мг/м2 флударабина перед первой или последующей дозой T-клеток. В некоторых примерах субъекту вводят 700 мг/м2 циклофосфамида и одну или несколько доз по 40 мг/м2 флударабина перед первой или последующей дозой T-клеток. В некоторых примерах субъекту вводят 800 мг/м2 циклофосфамида и одну или несколько доз по 45 мг/м2 флударабина перед первой или последующей дозой T-клеток.
[285] Флударабин и циклофосфамид можно вводить поочередно. В некоторых случаях флударабин и циклофосфамид можно вводить одновременно. В некоторых случаях за первоначальной дозой флударабина следует доза циклофосфамида. В некоторых случаях за первоначальной дозой циклофосфамида может следовать первоначальная доза флударабина. В некоторых примерах схема лечения может включать лечение субъекта первоначальной дозой флударабина 10 дней перед трансплантацией с последующим лечением первоначальной дозой циклофосфамида, вводимой 9 дней перед трансплантацией клеток, одновременно со второй дозой флударабина. В некоторых примерах схема лечения может включать лечение субъекта первоначальной дозой флударабина 8 дней перед трансплантацией, с последующим лечением первоначальной дозой циклофосфамида введенный 7 дней перед трансплантацией одновременно со второй дозой флударабина.
[286] В некоторых вариантах осуществления пептид содержит эпитопную последовательность согласно любой из таблиц 1-8 и 11-14. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 1. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 2. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 3. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4A. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4B. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4C. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4D. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4E. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4F. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4G. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4H. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4I. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4J. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4K. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4L. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 4M. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 5. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 6. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 7. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 8. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 11. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 12. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 13. В некоторых вариантах осуществления пептид содержит эпитопную последовательность в соответствии с таблицей 14.
LVVVGACGV (SEQ ID NO: 155) (A02.01)
VVGACGVGK (SEQ ID NO: 156) (A03.01, A11.01)
VVVGACGVGK (SEQ ID NO: 157) (A03.01)
VVVGADGVGK (SEQ ID NO: 159) (A11.01)
KLVVVGADGV (SEQ ID NO: 160) (A02.01)
LVVVGADGV (SEQ ID NO: 161) (A02.01)
LVVVGAVGV (SEQ ID NO: 163) (A02.01)
VVGAVGVGK (SEQ ID NO: 164) (A03.01, A11.01)
VVVGAVGVGK (SEQ ID NO: 5) (A03.01, A11.01)
LLDILDTAGL (SEQ ID NO: 167) (A02.01)
MANGSLLNY (SEQ ID NO: 171) (A01.01, A03.01, A11.01)
MANGSLLNYL (SEQ ID NO: 172) (A02.01, B07.02, B08.01)
SLLNYLREM (SEQ ID NO: 173) (A02.01, B07.02, B08.01)
YMANGSLLN (SEQ ID NO: 174) (A02.01)
YMANGSLLNY (SEQ ID NO: 175) (A01.01, A03.01, A11.01)
IMQLMPFGC (SEQ ID NO: 178) (A02.01)
IMQLMPFGCL (SEQ ID NO: 179) (A02.01, A24.02, B08.01)
LIMQLMPFG (SEQ ID NO: 180) (A02.01)
LIMQLMPFGC (SEQ ID NO: 181) (A02.01)
LTSTVQLIM (SEQ ID NO: 182) (A01.01)
MQLMPFGCL (SEQ ID NO: 183) (A02.01, B07.02, B08.01)
MQLMPFGCLL (SEQ ID NO: 184) (A02.01, A24.02, B08.01)
QLIMQLMPF (SEQ ID NO: 185) (A02.01, A24.02, B08.01)
QLIMQLMPFG (SEQ ID NO: 186) (A02.01)
STVQLIMQL (SEQ ID NO: 187) (A02.01)
VQLIMQLMPF (SEQ ID NO: 188) (A02.01, A24.02, B08.01)
GQYGKVYEGV (SEQ ID NO: 190) (A02.01)
KLGGGQYGK (SEQ ID NO: 191) (A03.01)
KLGGGQYGKV (SEQ ID NO: 192) (A02.01)
KVYEGVWKK (SEQ ID NO: 193) (A02.01, A03.01)
KVYEGVWKKY (SEQ ID NO: 194) (A03.01)
QYGKVYEGV (SEQ ID NO: 195) (A24.02)
QYGKVYEGVW (SEQ ID NO: 196) (A24.02)
GQYGVVYEGV (SEQ ID NO: 198) (A02.01)
KLGGGQYGV (SEQ ID NO: 199) (A02.01)
KLGGGQYGVV (SEQ ID NO: 200) (A02.01)
QYGVVYEGV (SEQ ID NO: 201) (A24.02)
QYGVVYEGVW (SEQ ID NO: 202) (A24.02)
VVYEGVWKK (SEQ ID NO: 203) (A02.01, A03.01)
VVYEGVWKKY (SEQ ID NO: 204) (A03.01)
ISSATEYLEK (SEQ ID NO: 206) (A03.01)
SSATEYLEK (SEQ ID NO: 207) (A03.01)
TQISSATEYL (SEQ ID NO: 208) (A02.01)
YMATQISSAT (SEQ ID NO: 209) (A02.01)
IIEFMTYGNL (SEQ ID NO: 211) (A02.01)
IIIEFMTYG (SEQ ID NO: 212) (A02.01)
IIIEFMTYGN (SEQ ID NO: 213) (A02.01)
YIIIEFMTYG (SEQ ID NO: 214) (A02.01)
KLGGGQHGEV (SEQ ID NO: 216) (A02.01)
RVAKIADFGM (SEQ ID NO: 218) (A02.01, B07.02)
ILMELMAGG (SEQ ID NO: 220) (A02.01)
ILMELMAGGD (SEQ ID NO: 221) (A02.01)
LMELMAGGDL (SEQ ID NO: 222) (A02.01)
LPRFILMEL (SEQ ID NO: 223) (B07.02, B08.01)
LPRFILMELM (SEQ ID NO: 224) (B07.02)
LQSLPRFILM (SEQ ID NO: 225) (A02.01, B08.01)
SLPRFILMEL (SEQ ID NO: 226) (A02.01, A24.02, B07.02, B08.01)
LATEKSRWSG (SEQ ID NO: 228) (A02.01, B08.01)
LYGLLLEML (SEQ ID NO: 231) (A24.02)
NVVPLYGLL (SEQ ID NO: 232) (A02.01)
PLYGLLLEM (SEQ ID NO: 233) (A02.01)
PLYGLLLEML (SEQ ID NO: 234) (A02.01, A24.02)
VPLYGLLLEM (SEQ ID NO: 235) (B07.02)
VVPLYGLLL (SEQ ID NO: 236) (A02.01, A24.02)
GVYTFLPST (SEQ ID NO: 238) (A02.01)
GVYTFLPSTL (SEQ ID NO: 239) (A02.01, A24.02)
TFLPSTLKSL (SEQ ID NO: 240) (A24.02)
VYTFLPSTL (SEQ ID NO: 241) (A24.02)
YTFLPSTLK (SEQ ID NO: 242) (A03.01)
NVVPLCDLLL (SEQ ID NO: 244) (A02.01)
PLCDLLLEM (SEQ ID NO: 245) (A02.01)
PLCDLLLEML (SEQ ID NO: 246) (A02.01)
VPLCDLLLEM (SEQ ID NO: 247) (B07.02)
VVPLCDLLL (SEQ ID NO: 248) (A02.01, A24.02)
NVVPLNDLLL (SEQ ID NO: 250) (A02.01)
PLNDLLLEM (SEQ ID NO: 251) (A02.01)
PLNDLLLEML (SEQ ID NO: 252) (A02.01)
VPLNDLLLEM (SEQ ID NO: 253) (B07.02)
NVVPLSDLLL (SEQ ID NO: 255) (A02.01)
PLSDLLLEM (SEQ ID NO: 256) (A02.01)
PLSDLLLEML (SEQ ID NO: 257) (A02.01)
VPLSDLLLEM (SEQ ID NO: 258) (B07.02)
VVPLSDLLL (SEQ ID NO: 259) (A02.01, A24.02)
YTLDVLERC (SEQ ID NO: 261) (A02.01)
(Устойчивость)
TIGGPTLVII (SEQ ID NO: 269) (A02.01)
VIIEYCCYG (SEQ ID NO: 270) (A02.01)
IANLLGACTI (SEQ ID NO: 272) (A02.01)
MNIANLLGA (SEQ ID NO: 273) (A02.01)
YLGNHMNIA (SEQ ID NO: 274) (A02.01, B08.01)
YLGNHMNIAN (SEQ ID NO: 275) (A02.01)
VLHESNSPYI (SEQ ID NO: 277) (A02.01)
LYIVGFYGAF (SEQ ID NO: 279) (A24.02)
NSLYIVGFY (SEQ ID NO: 280) (A01.01)
QVLHECNSL (SEQ ID NO: 281) (A02.01, B08.01)
SLYIVGFYG (SEQ ID NO: 282) (A02.01)
SLYIVGFYGA (SEQ ID NO: 283) (A02.01)
VLHECNSLY (SEQ ID NO: 284) (A03.01)
VLHECNSLYI (SEQ ID NO: 285) (A02.01, A03.01)
QQQSDLQPPA (SEQ ID NO: 287) (A02.01)
QQSDLQPPA (SEQ ID NO: 288) (A02.01)
YQQQQQSDL (SEQ ID NO: 289) (A02.01, B08.01)
LLSTPPLSPS (SEQ ID NO: 291) (A02.01)
IWKKFELLPI (SEQ ID NO: 293) (A24.02)
LLPIPPLSPS (SEQ ID NO: 294) (A02.01, B07.02)
LPIPPLSPS (SEQ ID NO: 295) (B07.02)
IIIEYCFYG (SEQ ID NO: 297) (A02.01)
IYIIIEYCF (SEQ ID NO: 298) (A24.02)
IYIIIEYCFY (SEQ ID NO: 299) (A24.02)
YIIIEYCFYG (SEQ ID NO: 300) (A02.01)
DPLSEITK (SEQ ID NO: 303) (A03.01)
YMCNSSCMGS (SEQ ID NO: 308) (A02.01)
ALKYIFVAVR (SEQ ID NO: 313) (A03.01)
AVRAICVMK (SEQ ID NO: 314) (A03.01)
AVRAICVMKS (SEQ ID NO: 315) (A03.01)
CVEKKTALK (SEQ ID NO: 316) (A03.01)
CVEKKTALKY (SEQ ID NO: 317) (A01.01)
CVMKSFLIF (SEQ ID NO: 318) (A24.02, B08.01)
CVMKSFLIFR (SEQ ID NO: 319) (A03.01)
FLIFRRWKS (SEQ ID NO: 320) (A02.01, B08.01)
FRRWKSHSPL (SEQ ID NO: 321) (B08.01)
FVAVRAICV (SEQ ID NO: 322) (A02.01, B08.01)
FVAVRAICVM (SEQ ID NO: 323) (B08.01)
IQLHLSHPI (SEQ ID NO: 324) (A02.01)
KSFLIFRRWK (SEQ ID NO: 325) (A03.01)
KTALKYIFV (SEQ ID NO: 326) (A02.01)
KYIFVAVRAI (SEQ ID NO: 327) (A24.02)
RWKSHSPLQI (SEQ ID NO: 328) (A24.02)
TALKYIFVAV (SEQ ID NO: 329) (A02.01, B08.01)
VAVRAICVMK (SEQ ID NO: 330) (A03.01)
VMKSFLIFR (SEQ ID NO: 331) (A03.01)
VMKSFLIFRR (SEQ ID NO: 332) (A03.01)
YIFVAVRAI (SEQ ID NO: 333) (A02.01)
ALFFFFFETK (SEQ ID NO: 335) (A03.01)
AQAGVQWRSL (SEQ ID NO: 336) (A02.01)
CLANFCIFNR (SEQ ID NO: 337) (A03.01)
CLSFLSSWDY (SEQ ID NO: 338) (A01.01, A03.01)
FFETKSCSV (SEQ ID NO: 339) (B08.01)
FFFETKSCSV (SEQ ID NO: 340) (A02.01)
FKLFSCLSFL (SEQ ID NO: 341) (A02.01)
FLSSWDYRRM (SEQ ID NO: 342) (A02.01)
GFKLFSCLSF (SEQ ID NO: 343) (A24.02)
KLFSCLSFL (SEQ ID NO: 344) (A02.01, A03.01)
KLFSCLSFLS (SEQ ID NO: 345) (A02.01, A03.01)
LALFFFFFET (SEQ ID NO: 346) (A02.01)
LFFFFFETK (SEQ ID NO: 347) (A03.01)
LSFLSSWDY (SEQ ID NO: 348) (A01.01)
LSFLSSWDYR (SEQ ID NO: 349) (A03.01)
RMPPCLANF (SEQ ID NO: 350) (A24.02)
RRMPPCLANF (SEQ ID NO: 351) (A24.02)
SLQPPPPGFK (SEQ ID NO: 352) (A03.01)
VQWRSLGSL (SEQ ID NO: 353) (A02.01)
MSVCFFFFSV (SEQ ID NO: 355) (A02.01)
SVCFFFFSV (SEQ ID NO: 356) (A02.01, B08.01)
SVCFFFFSVI (SEQ ID NO: 357) (A02.01)
MSVCFFFFCY (SEQ ID NO: 359) (A01.01)
SVCFFFFCYI (SEQ ID NO: 360) (A02.01)
VLWMPTSTV (SEQ ID NO: 362) (A02.01)
QLKWTPHILK (SEQ ID NO: 368) (A03.01)
IVSEKNQQLK (SEQ ID NO: 369) (A03.01)
QLKWTPHILK (SEQ ID NO: 368) (A03.01)
QQLKWTPHI (SEQ ID NO: 367) (A24.02)
NQQLKWTPHIL (SEQ ID NO: 370) (B08.01)
NQQLKWTPHI (SEQ ID NO: 371) (B08.01)
QLKWTPHIL (SEQ ID NO: 372) (B08.01)
PPRPPRAAC (SEQ ID NO: 374) (B07.02)
CLIVSRTLL (SEQ ID NO: 378) (B08.01)
CLIVSRTLLL (SEQ ID NO: 379) (A02.01, B08.01)
FLLALWECS (SEQ ID NO: 380) (A02.01)
FLLALWECSL (SEQ ID NO: 381) (A02.01, B08.01)
IVSRTLLLV (SEQ ID NO: 382) (A02.01)
LIVSRTLLL (SEQ ID NO: 383) (A02.01, B08.01)
LIVSRTLLLV (SEQ ID NO: 384) (A02.01)
LLALWECSL (SEQ ID NO: 385) (A02.01, B08.01)
LPQARLCLI (SEQ ID NO: 386) (B08.01, B07.02)
LPQARLCLIV (SEQ ID NO: 387) (B08.01)
NVRDKKRATF (SEQ ID NO: 388) (B08.01)
SLPQARLCLI (SEQ ID NO: 389) (A02.01, B08.01)
FFFCWILSCK (SEQ ID NO: 391) (A03.01)
ITAFFFCWI (SEQ ID NO: 392) (A02.01)
LYITAFFFCW (SEQ ID NO: 393) (A24.02)
YITAFFFCWI (SEQ ID NO: 394) (A02.01)
LLYITAFFL (SEQ ID NO: 396) (A02.01, B08.01)
LLYITAFFLL (SEQ ID NO: 397) (A02.01, A24.02)
LYITAFFLL (SEQ ID NO: 398) (A24.02)
LYITAFFLLD (SEQ ID NO: 399) (A24.02)
YITAFFLLDI (SEQ ID NO: 400) (A02.01)
MPKDVNIQV (SEQ ID NO: 402) (B07.02)
TGSNEVTTRY (SEQ ID NO: 403) (A01.01)
KLMLLRLNL (SEQ ID NO: 405) (A02.01, A03.01, B07.02, B08.01)
KLMLLRLNLR (SEQ ID NO: 406) (A03.01)
LLRLNLRKM (SEQ ID NO: 407) (B08.01)
LMLLRLNL (SEQ ID NO: 408) (B08.01)
LMLLRLNLRK (SEQ ID NO: 409) (A03.01)
LNLRKMCGPF (SEQ ID NO: 410) (B08.01)
MLLRLNLRK (SEQ ID NO: 411) (A03.01)
MLLRLNLRKM (SEQ ID NO: 412) (A02.01, A03.01, B08.01)
NLRKMCGPF (SEQ ID NO: 413) (B08.01)
NYCQKKLMLL (SEQ ID NO: 414) (A24.02)
YCQKKLMLL (SEQ ID NO: 415) (B08.01)
ITTVKATETK (SEQ ID NO: 417) (A03.01)
KSKKKETFK (SEQ ID NO: 418) (A03.01)
KSKKKETFKK (SEQ ID NO: 419) (A03.01)
KTYTCAITTV (SEQ ID NO: 420) (A02.01, A24.02)
TFKKKTYTC (SEQ ID NO: 421) (B08.01)
TYTCAITTV (SEQ ID NO: 422) (A24.02)
TYTCAITTVK (SEQ ID NO: 423) (A03.01)
YTCAITTVK (SEQ ID NO: 424) (A03.01)
SLKKVPAL (SEQ ID NO: 428) (B08.01)
RKISNWSLKK (SEQ ID NO: 429) (A03.01)
VPALKKLRSPL (SEQ ID NO: 430) (B07.02)
MTKKKRVNTA (SEQ ID NO: 432) (B08.01)
RVNTAWKTK (SEQ ID NO: 433) (A03.01)
RVNTAWKTKK (SEQ ID NO: 434) (A03.01)
TKKKRVNTA (SEQ ID NO: 435) (B08.01)
WKTKKTSFSL (SEQ ID NO: 436) (B08.01)
AMTTSSSQK (SEQ ID NO: 438) (A03.01, A11.01)
AMTTSSSQKN (SEQ ID NO: 439) (A03.01)
CIMKEKKSL (SEQ ID NO: 440) (B08.01)
CIMKEKKSLV (SEQ ID NO: 441) (B08.01)
IMKEKKSL (SEQ ID NO: 442) (B08.01)
IMKEKKSLV (SEQ ID NO: 443) (B08.01)
KSLVRLSSCV (SEQ ID NO: 444) (A02.01)
LVRLSSCVPV (SEQ ID NO: 445) (A02.01)
RLSSCVPVA (SEQ ID NO: 446) (A02.01, A03.01)
RLSSCVPVAL (SEQ ID NO: 447) (A02.01)
SAMTTSSSQK (SEQ ID NO: 448) (A03.01, A11.01)
SLVRLSSCV (SEQ ID NO: 449) (A02.01)
VPVALMSAM (SEQ ID NO: 450) (B07.02)
VRLSSCVPVA (SEQ ID NO: 451) (A02.01)
FVMSDTTYKI (SEQ ID NO: 454) (A02.01)
KTFEKKGEK (SEQ ID NO: 455) (A03.01)
LFVMSDTTYK (SEQ ID NO: 456) (A03.01)
MSDTTYKIY (SEQ ID NO: 457) (A01.01)
VMSDTTYKI (SEQ ID NO: 458) (A02.01)
VMSDTTYKIY (SEQ ID NO: 459) (A01.01)
F1354fs
Q1378fs
S1398fs
FRRPHSCLA (SEQ ID NO: 462) (B08.01)
LIVLRVVRL (SEQ ID NO: 463) (B08.01)
LLSVHLIVL (SEQ ID NO: 464) (A02.01, B08.01)
R1435fs
T1438fs
P1442fs
P1443fs
V1452fs
P1453fs
K1462fs
E1464fs
HLHHLLKQLK (SEQ ID NO: 466) (A03.01)
HLLLKRERV (SEQ ID NO: 467) (B08.01)
KIKHLLLKR (SEQ ID NO: 468) (A03.01)
KPSEKYLKI (SEQ ID NO: 469) (B07.02)
KYLKIKHLL (SEQ ID NO: 470) (A24.02)
KYLKIKHLLL (SEQ ID NO: 471) (A24.02)
LLKQLKPSEK (SEQ ID NO: 472) (A03.01)
LLKRERVDL (SEQ ID NO: 473) (B08.01)
LLLKRERVDL (SEQ ID NO: 474) (B08.01)
QLKPSEKYLK (SEQ ID NO: 475) (A03.01)
YLKIKHLLL (SEQ ID NO: 476) (A02.01, B08.01)
YLKIKHLLLK (SEQ ID NO: 477) (A03.01)
H1490fs
L1488fs
KVLQMDFLV (SEQ ID NO: 479) (A02.01, A24.02)
LPRKVLQMDF (SEQ ID NO: 480) (B07.02, B08.01)
LQMDFLVHPA (SEQ ID NO: 481) (A02.01)
QMDFLVHPA (SEQ ID NO: 482) (A02.01)
YILPRKVLQM (SEQ ID NO: 483) (A02.01, B08.01)
S1316fs
Y1324fs
T1348fs
G1351fs
G1378fs
P1467fs
HPLAPMPSKT (SEQ ID NO: 485) (B07.02)
ILPLPQLLL (SEQ ID NO: 486) (A02.01)
LPTQTRGCIL (SEQ ID NO: 487) (B07.02)
RISCLPTQTR (SEQ ID NO: 488) (A03.01)
SLAETVSLH (SEQ ID NO: 489) (A03.01)
TPRSSSSSS (SEQ ID NO: 490) (B07.02)
TPRSSSSSSS (SEQ ID NO: 491) (B07.02)
P725fs
R727fs
I736fs
ALPPVLLSLA (SEQ ID NO: 493) (A02.01)
ALPRPLLAL (SEQ ID NO: 494) (A02.01)
ASRTASCIL (SEQ ID NO: 495) (B07.02)
EALPRPLLAL (SEQ ID NO: 496) (B08.01)
HLGHPVASR (SEQ ID NO: 497) (A03.01)
HPVASRTAS (SEQ ID NO: 498) (B07.02)
HPVASRTASC (SEQ ID NO: 499) (B07.02)
IIQLSLLSLL (SEQ ID NO: 500) (A02.01)
IQLSLLSLL (SEQ ID NO: 501) (A02.01)
IQLSLLSLLI (SEQ ID NO: 502) (A02.01, A24.02)
LLALPPVLL (SEQ ID NO: 503) (A02.01)
LLIPPLTCL (SEQ ID NO: 504) (A02.01)
LLIPPLTCLA (SEQ ID NO: 505) (A02.01)
LLSLLIPPL (SEQ ID NO: 506) (A02.01)
LLSLLIPPLT (SEQ ID NO: 507) (A02.01)
LPRPLLALPP (SEQ ID NO: 508) (B07.02)
QLSLLSLLI (SEQ ID NO: 509) (A02.01)
RLLQCQATR (SEQ ID NO: 510) (A03.01)
RPLLALPPV (SEQ ID NO: 511) (B07.02)
RPLLALPPVL (SEQ ID NO: 512) (B07.02)
SLAQDHSRL (SEQ ID NO: 513) (A02.01)
SLAQDHSRLL (SEQ ID NO: 514) (A02.01)
SLLIPPLTCL (SEQ ID NO: 515) (A02.01)
SLLSLLIPP (SEQ ID NO: 516) (A02.01)
SLLSLLIPPL (SEQ ID NO: 517) (A02.01, B08.01)
Q473fs
H477fs
S499fs
P504fs
Q548fs
P549fs
AAIPASTSAV (SEQ ID NO: 519) (B07.02)
AIPASTSAV (SEQ ID NO: 520) (A02.01)
ALPAGCVSSA (SEQ ID NO: 521) (A02.01)
APLLTATGSV (SEQ ID NO: 522) (B07.02)
APVLSASIL (SEQ ID NO: 523) (B07.02)
ATLLPATTV (SEQ ID NO: 524) (A02.01)
ATVSTTTAPV (SEQ ID NO: 525) (A02.01)
AVPANCLFPA (SEQ ID NO: 526) (A02.01)
CLFPAALPST (SEQ ID NO: 527) (A02.01)
CPTFVSAAA (SEQ ID NO: 528) (B07.02)
FPAALPSTA (SEQ ID NO: 529) (B07.02)
FPAALPSTAG (SEQ ID NO: 530) (B07.02)
GAECHGRPL (SEQ ID NO: 531) (B07.02)
GAISRFIWV (SEQ ID NO: 532) (A02.01)
ILPAAIPAST (SEQ ID NO: 533) (A02.01)
IWVSGILSPL (SEQ ID NO: 534) (A24.02)
LLTATGSVSL (SEQ ID NO: 535) (A02.01)
LLYTADSSL (SEQ ID NO: 536) (A02.01)
LPAAATSAA (SEQ ID NO: 537) (B07.02)
LPAAATSAAS (SEQ ID NO: 538) (B07.02)
LPAAIPAST (SEQ ID NO: 539) (B07.02)
LPAGCVSSA (SEQ ID NO: 540) (B07.02)
LPAGCVSSAP (SEQ ID NO: 541) (B07.02)
LYTADSSLW (SEQ ID NO: 542) (A24.02)
QPAKSSPSA (SEQ ID NO: 543) (B07.02)
QPAKSSPSAA (SEQ ID NO: 544) (B07.02)
RFIWVSGIL (SEQ ID NO: 545) (A24.02)
RPQRVWSTG (SEQ ID NO: 546) (B07.02)
RVWSTGPDSI (SEQ ID NO: 547) (A02.01)
SAVPGSIPL (SEQ ID NO: 548) (B07.02)
SILPAAIPA (SEQ ID NO: 549) (A02.01)
SLPAAATSA (SEQ ID NO: 550) (A02.01)
SLPAAATSAA (SEQ ID NO: 551) (A02.01)
SLWTTRPQR (SEQ ID NO: 552) (A03.01)
SLWTTRPQRV (SEQ ID NO: 553) (A02.01)
SPSAAAATL (SEQ ID NO: 554) (B07.02)
SPSAAAATLL (SEQ ID NO: 555) (B07.02)
TLDALPAGCV (SEQ ID NO: 556) (A02.01)
TVSTTTAPV (SEQ ID NO: 557) (A02.01)
VLSASILPA (SEQ ID NO: 558) (A02.01)
VLSASILPAA (SEQ ID NO: 559) (A02.01)
VPANCLFPA (SEQ ID NO: 560) (B07.02)
VPANCLFPAA (SEQ ID NO: 561) (B07.02)
VPDPSCPTF (SEQ ID NO: 562) (B07.02)
VPGSIPLPA (SEQ ID NO: 563) (B07.02)
VPGSIPLPAV (SEQ ID NO: 564) (B07.02)
WVSGILSPL (SEQ ID NO: 565) (A02.01)
YTADSSLWTT (SEQ ID NO: 566) (A02.01)
A441fs
Y447fs
P483fs
P484fs
P504fs
S519fs
H544fs
P549fs
P554fs
Q563fs
ASLHRRSYL (SEQ ID NO: 568) (B08.01)
ASLHRRSYLK (SEQ ID NO: 569) (A03.01)
FLLMDNKAPA (SEQ ID NO: 570) (A02.01)
HPRRSPSRL (SEQ ID NO: 571) (B07.02, B08.01)
HPSLHISSP (SEQ ID NO: 572) (B07.02)
HRRSYLKIHL (SEQ ID NO: 573) (B08.01)
HSRFLLMDNK (SEQ ID NO: 574) (A03.01)
KLPIPSSASL (SEQ ID NO: 575) (A02.01)
KVLTLSSSSH (SEQ ID NO: 576) (A03.01)
LIHSRFLLM (SEQ ID NO: 577) (B08.01)
LLMDNKAPA (SEQ ID NO: 578) (A02.01)
LMDNKAPAGM (SEQ ID NO: 579) (A02.01)
LPIPSSASL (SEQ ID NO: 580) (B07.02)
MPNLRISSS (SEQ ID NO: 581) (B07.02, B08.01)
MPNLRISSSH (SEQ ID NO: 582) (B07.02)
NLRISSSHSL (SEQ ID NO: 583) (B07.02, B08.01)
PPTHSHRLSL (SEQ ID NO: 584) (B07.02)
RAGRRVPWAA (SEQ ID NO: 585) (B08.01)
RARLHITTSK (SEQ ID NO: 586) (A03.01)
RISSSHSLNH (SEQ ID NO: 587) (A03.01)
RLHTPPSSR (SEQ ID NO: 588) (A03.01)
RLHTPPSSRR (SEQ ID NO: 589) (A03.01)
RLRILSPSL (SEQ ID NO: 590) (A02.01, B07.02, B08.01)
RPLMPNLRI (SEQ ID NO: 591) (B07.02)
RPRPLMPNL (SEQ ID NO: 592) (B07.02)
SASLHRRSYL (SEQ ID NO: 593) (B07.02, B08.01)
SLHISSPRL (SEQ ID NO: 594) (A02.01)
SLHRRSYLK (SEQ ID NO: 595) (A03.01)
SLHRRSYLKI (SEQ ID NO: 596) (B08.01)
SLIHSRFLL (SEQ ID NO: 597) (A02.01)
SLIHSRFLLM (SEQ ID NO: 598) (A02.01, B08.01)
SLLTSSSNL (SEQ ID NO: 599) (A02.01)
SLNHHSSSPL (SEQ ID NO: 600) (A02.01, B07.02, B08.01)
SLSSPSKLPI (SEQ ID NO: 601) (A02.01)
SPLSLHTPS (SEQ ID NO: 602) (B07.02)
SPLSLHTPSS (SEQ ID NO: 603) (B07.02)
SPPTHSHRL (SEQ ID NO: 604) (B07.02)
SPRLHTPPS (SEQ ID NO: 605) (B07.02)
SPRLHTPPSS (SEQ ID NO: 606) (B07.02)
SPSLSSPSKL (SEQ ID NO: 607) (B07.02)
SYLKIHLGL (SEQ ID NO: 608) (A24.02)
TPSNHRPRPL (SEQ ID NO: 609) (B07.02, B08.01)
TPSSHPSLHI (SEQ ID NO: 610) (B07.02)
P2139fs
L1970fs
V1994fs
HCVPFWTGRIL (SEQ ID NO: 612) (B07.02)
ILLPSAASV (SEQ ID NO: 613) (A02.01)
ILLPSAASVC (SEQ ID NO: 614) (A02.01)
LLPSAASVCPI (SEQ ID NO: 615) (A02.01)
LPSAASVCPI (SEQ ID NO: 616) (B07.02)
MRPHCVPF (SEQ ID NO: 617) (B08.01)
RILLPSAASV (SEQ ID NO: 618) (A02.01)
RMRPHCVPF (SEQ ID NO: 619) (A24.02, B07.02, B08.01)
RMRPHCVPFW (SEQ ID NO: 620) (A24.02)
RTNPTVRMR (SEQ ID NO: 621) (A03.01)
SVCPIPFEA (SEQ ID NO: 622) (A02.01)
TVRMRPHCV (SEQ ID NO: 623) (B08.01)
TVRMRPHCVPF (SEQ ID NO: 624) (B08.01)
VPFWTGRIL (SEQ ID NO: 625) (B07.02)
VPFWTGRILL (SEQ ID NO: 626) (B07.02)
VRMRPHCVPF (SEQ ID NO: 627) (B08.01)
S764fs
T783fs
Q799fs
A817fs
AMVPRGVSMA (SEQ ID NO: 629) (A02.01)
AWAPTSRTPW (SEQ ID NO: 630) (A24.02)
CPMPTTPVQA (SEQ ID NO: 631) (B07.02)
CPSTVPPSPA (SEQ ID NO: 632) (B07.02)
GAMVPRGVSM (SEQ ID NO: 633) (B07.02, B08.01)
MPCPMPTTPV (SEQ ID NO: 634) (B07.02)
MPTTPVQAW (SEQ ID NO: 635) (B07.02)
MPTTPVQAWL (SEQ ID NO: 636) (B07.02)
SLPEDRYTQA (SEQ ID NO: 637) (A02.01)
SPAQPYLRV (SEQ ID NO: 638) (B07.02)
SPAQPYLRVS (SEQ ID NO: 639) (B07.02)
TIMPCPMPT (SEQ ID NO: 640) (A02.01)
TPVQAWLEA (SEQ ID NO: 641) (B07.02)
TSRTPWGAM (SEQ ID NO: 642) (B07.02)
VPPSPAQPYL (SEQ ID NO: 643) (B07.02)
VPRGVSMAH (SEQ ID NO: 644) (B07.02)
E67fs
L74fs
F82fs
T91fs
E94fs
CTTLNSPPLK (SEQ ID NO: 646) (A03.01)
GLSISCTTL (SEQ ID NO: 647) (A02.01)
SPPLKKMSM (SEQ ID NO: 648) (B07.02, B08.01)
TLNSPPLKK (SEQ ID NO: 649) (A03.01)
TTLNSPPLK (SEQ ID NO: 650) (A03.01)
TTLNSPPLKK (SEQ ID NO: 651) (A03.01)
S14fs
LQRFRFTHVI (SEQ ID NO: 653) (B08.01)
RLSSVLQRF (SEQ ID NO: 654) (A24.02)
RLSSVLQRFR (SEQ ID NO: 655) (A03.01)
VLQRFRFTHV (SEQ ID NO: 656) (A02.01, B08.01)
P708fs
L711fs
KFLPFWGFL (SEQ ID NO: 658) (A24.02)
LASVGRHSL (SEQ ID NO: 659) (B07.02)
LPFWGFLEEF (SEQ ID NO: 660) (B07.02)
PFWGFLEEF (SEQ ID NO: 661) (A24.02)
SVGRHSLSK (SEQ ID NO: 662) (A03.01)
P126fs
H128fs
N144fs
V157fs
P159fs
N166fs
N181fs
F189fs
P201fs
F205fs
APRPIRPPFL (SEQ ID NO: 664) (B07.02)
AQKMKKAHFL (SEQ ID NO: 665) (B08.01)
FLPSAAQKM (SEQ ID NO: 666) (A02.01)
GLFLPSAAQK (SEQ ID NO: 667) (A03.01)
HPLLVSLLL (SEQ ID NO: 668) (B07.02)
KAHFLKTWFR (SEQ ID NO: 669) (A03.01)
KARFSTASL (SEQ ID NO: 670) (B07.02)
KMKKAHFLK (SEQ ID NO: 671) (A03.01)
KTWFRSNPTK (SEQ ID NO: 672) (A03.01)
LAKELTHPL (SEQ ID NO: 673) (B07.02, B08.01)
LAKELTHPLL (SEQ ID NO: 674) (B08.01)
NPTKTKKARF (SEQ ID NO: 675) (B07.02)
QKMKKAHFL (SEQ ID NO: 676) (B08.01)
RFSTASLAK (SEQ ID NO: 677) (A03.01)
RPIRPPFLES (SEQ ID NO: 678) (B07.02)
RSNPTKTKK (SEQ ID NO: 679) (A03.01)
SLAKELTHPL (SEQ ID NO: 680) (A02.01, B08.01)
TKKARFSTA (SEQ ID NO: 681) (B08.01)
P127fs
V132fs
P160fs
ISQLLSWPQK (SEQ ID NO: 683) (A03.01)
RIAHISQLL (SEQ ID NO: 684) (A02.01)
RLGYSSHQL (SEQ ID NO: 685) (A02.01)
SQLLSWPQK (SEQ ID NO: 686) (A03.01)
SRIAHISQL (SEQ ID NO: 687) (B08.01)
WPQKTEERL (SEQ ID NO: 688) (B07.02)
YSSHQLPRK (SEQ ID NO: 689) (A03.01)
R749fs
E757fs
G759fs
EAEKRTRTL (SEQ ID NO: 691) (B08.01)
GTTFITMMK (SEQ ID NO: 692) (A03.01)
GTTFITMMKK (SEQ ID NO: 693) (A03.01)
ITMMKKEAEK (SEQ ID NO: 694) (A03.01)
RMTPGTTFI (SEQ ID NO: 695) (A02.01)
SPYCPQRMT (SEQ ID NO: 696) (B07.02)
TMMKKEAEK (SEQ ID NO: 697) (A03.01)
TPGTTFITM (SEQ ID NO: 698) (B07.02)
TPGTTFITMM (SEQ ID NO: 699) (B07.02)
TTFITMMKK (SEQ ID NO: 700) (A03.01)
E24fs
S36fs
CPGATWREAA (SEQ ID NO: 702) (B07.02)
RSRCPGATWR (SEQ ID NO: 703) (A03.01)
TPRATRSRC (SEQ ID NO: 704) (B07.02)
P387fs
S398fs
H400fs
M401fs
S408fs
P409fs
S408fs
P409fs
T419fs
H424fs
P425fs
S427fs
F431fs
S430fs
H434fs
H435fs
S438fs
M443fs
G444fs
*445fs
RPLQTHVLPE (SEQ ID NO: 706) (B07.02)
VLWTTPPLQH (SEQ ID NO: 707) (A03.01)
H434fs
P433fs
T441fs
CPLDHTTPPA (SEQ ID NO: 709) (B07.02)
FLQEQYHEA (SEQ ID NO: 710) (A02.01, B08.01)
RLAFLQEQYH (SEQ ID NO: 711) (A03.01)
SPCSPFRLAF (SEQ ID NO: 712) (B07.02)
SPPWVRALL (SEQ ID NO: 713) (B07.02)
YPACPLDHTT (SEQ ID NO: 714) (B07.02)
E524fs
P647fs
S654fs
L656fs
R755fs
L761fs
Q773fs
CLHHRRHLV (SEQ ID NO: 716) (B08.01)
CLHHRRHLVC (SEQ ID NO: 717) (B08.01)
CLHRKSHPHL (SEQ ID NO: 718) (B08.01)
CLRSHACPP (SEQ ID NO: 719) (B08.01)
CLRSHTCPP (SEQ ID NO: 720) (B08.01)
CLWCHACLH (SEQ ID NO: 721) (A03.01)
CPHHLKNHL (SEQ ID NO: 722) (B07.02)
CPHHLKNLL (SEQ ID NO: 723) (B07.02)
CPHHLRTRL (SEQ ID NO: 724) (B07.02, B08.01)
CPLHLRSLPF (SEQ ID NO: 725) (B07.02, B08.01)
CPLPRHLKHL (SEQ ID NO: 726) (B07.02, B08.01)
CPLSLRSHPC (SEQ ID NO: 727) (B07.02)
CPRHLRNCPL (SEQ ID NO: 728) (B07.02, B08.01)
FPHHLRHHA (SEQ ID NO: 729) (B07.02, B08.01)
FPHHLRHHAC (SEQ ID NO: 730) (B07.02, B08.01)
GLRSRTCPP (SEQ ID NO: 731) (B08.01)
HACLHRLRNL (SEQ ID NO: 732) (B08.01)
HLACLHHLR (SEQ ID NO: 733) (A03.01)
HLCPPHLRY (SEQ ID NO: 734) (A03.01)
HLCPPHLRYR (SEQ ID NO: 735) (A03.01)
HLKHLACLH (SEQ ID NO: 736) (A03.01)
HLKHRPCPH (SEQ ID NO: 737) (B08.01)
HLKNHLCPP (SEQ ID NO: 738) (B08.01)
HLKSHPCLH (SEQ ID NO: 739) (A03.01)
HLKSLLCPL (SEQ ID NO: 740) (A02.01, B08.01)
HLLHLRRLY (SEQ ID NO: 741) (A03.01)
HLRNCPLPR (SEQ ID NO: 742) (A03.01)
HLRNCPLPRH (SEQ ID NO: 743) (A03.01)
HLRRLYPHHL (SEQ ID NO: 744) (B08.01)
HLRSHPCPL (SEQ ID NO: 745) (B07.02, B08.01)
HLRSHPCPLH (SEQ ID NO: 746) (A03.01)
HLRSLPFPH (SEQ ID NO: 747) (A03.01)
HLRTRLCPH (SEQ ID NO: 748) (A03.01, B08.01)
HLVCSHHLK (SEQ ID NO: 749) (A03.01)
HPCLHHRRHL (SEQ ID NO: 750) (B07.02, B08.01)
HPGLRSRTC (SEQ ID NO: 751) (B07.02)
HPHLLHLRRL (SEQ ID NO: 752) (B07.02, B08.01)
HRKSHPHLL (SEQ ID NO: 753) (B08.01)
HRRTRSCPC (SEQ ID NO: 754) (B08.01)
KSHPHLLHLR (SEQ ID NO: 755) (A03.01)
KSLLCPLHLR (SEQ ID NO: 756) (A03.01)
LLCPLHLRSL (SEQ ID NO: 757) (A02.01, B08.01)
LLHLRRLYPH (SEQ ID NO: 758) (B08.01)
LPRHLKHLA (SEQ ID NO: 759) (B07.02)
LPRHLKHLAC (SEQ ID NO: 760) (B07.02, B08.01)
LRRLRSHTC (SEQ ID NO: 761) (B08.01)
LRRLYPHHL (SEQ ID NO: 762) (B08.01)
LVCSHHLKSL (SEQ ID NO: 763) (B08.01)
NLRNHTCPPS (SEQ ID NO: 764) (B08.01)
PLHLRSLPF (SEQ ID NO: 765) (B08.01)
RLCPHHLKNH (SEQ ID NO: 766) (A03.01)
RLYPHHLKH (SEQ ID NO: 767) (A03.01)
RLYPHHLKHR (SEQ ID NO: 768) (A03.01)
RPCPHHLKNL (SEQ ID NO: 769) (B07.02)
RSHPCPLHLK (SEQ ID NO: 770) (A03.01)
RSLPFPHHLR (SEQ ID NO: 771) (A03.01)
RTRLCPHHL (SEQ ID NO: 772) (B07.02)
RTRLCPHHLK (SEQ ID NO: 773) (A03.01)
SLLCPLHLR (SEQ ID NO: 774) (A03.01)
SLRSHACPP (SEQ ID NO: 775) (B08.01)
SPLRSQANA (SEQ ID NO: 776) (B07.02)
YLRRLRSHT (SEQ ID NO: 777) (B08.01)
YPHHLKHRPC (SEQ ID NO: 778) (B07.02, B08.01)
I135fs
A148fs
L152fs
D162fs
I168fs
IFITSGQIF (SEQ ID NO: 780) (A24.02)
SQSEALCVL (SEQ ID NO: 781) (A02.01)
SQSEALCVLL (SEQ ID NO: 782) (A02.01)
K266fs
E40fs
V45fs
R47fs
N48fs
DLKAYTGTIL (SEQ ID NO: 784) (B08.01)
T321fs
K327fs
A328fs
A333fs
KMILTKQIK (SEQ ID NO: 786) (A03.01)
KPTDTFLQI (SEQ ID NO: 787) (B07.02)
KPTDTFLQIL (SEQ ID NO: 788) (B07.02)
MILTKQIKTK (SEQ ID NO: 789) (A03.01)
E73fs
A86fs
N94fs
LIAELHNIL (SEQ ID NO: 791) (A02.01)
LIAELHNILL (SEQ ID NO: 792) (A02.01)
MTPPNLIAEL (SEQ ID NO: 793) (A02.01)
NLIAELHNI (SEQ ID NO: 794) (A02.01)
NLIAELHNIL (SEQ ID NO: 795) (A02.01)
RYTIFVLKDI (SEQ ID NO: 796) (A24.02)
TITRYTIFVL (SEQ ID NO: 797) (A02.01)
TPPNLIAEL (SEQ ID NO: 798) (B07.02)
G209fs
C211fs
I224fs
G230fs
P231fs
R233fs
D236fs
LPAKGEDIFL (SEQ ID NO: 800) (B07.02)
LQFRTHTTGR (SEQ ID NO: 801) (A03.01)
NLQSSVCGL (SEQ ID NO: 802) (A02.01)
SSVCGLPAK (SEQ ID NO: 803) (A03.01)
VQWRNLQSSV (SEQ ID NO: 804) (A02.01)
E256fs
K260fs
Q261fs
L265fs
M270fs
H272fs
T286fs
E288fs
HTRTRGNLRK (SEQ ID NO: 806) (A03.01)
ILHTRTRGNL (SEQ ID NO: 807) (B08.01)
KGQNVSLLGK (SEQ ID NO: 808) (A03.01)
LLGKYILHT (SEQ ID NO: 809) (A02.01)
LRKSRKWKSM (SEQ ID NO: 810) (B08.01)
SLLGKYILH (SEQ ID NO: 811) (A03.01)
SLLGKYILHT (SEQ ID NO: 812) (A02.01)
P72fs
A76fs
A79fs
P89fs
W91fs
S96fs
V97fs
V97fs
G108fs
G117fs
S121fs
V122fs
C124fs
K139fs
V143fs
FPENLPGQL (SEQ ID NO: 814) (B07.02)
GLLAFWDSQV (SEQ ID NO: 815) (A02.01)
IFCPFPENL (SEQ ID NO: 816) (A24.02)
LLAFWDSQV (SEQ ID NO: 817) (A02.01)
LLAPVIFCP (SEQ ID NO: 818) (A02.01)
LLAPVIFCPF (SEQ ID NO: 819) (A02.01, A24.02)
LPCPQQDVL (SEQ ID NO: 820) (B07.02)
RFPSGLLAF (SEQ ID NO: 821) (A24.02)
RFPSGLLAFW (SEQ ID NO: 822) (A24.02)
SPLLAPVIF (SEQ ID NO: 823) (B07.02)
SPRGPCTSS (SEQ ID NO: 824) (B07.02)
SPRGPCTSSS (SEQ ID NO: 825) (B07.02)
SQVCDLHVL (SEQ ID NO: 826) (A02.01)
VIFCPFPENL (SEQ ID NO: 827) (A02.01)
H178fs
D186fs
H193fs
L194fs
E198fs
V203fs
E204fs
L206fs
D207fs
N210fs
T211fs
F212fs
V225fs
S241fs
AMVWPLLSIL (SEQ ID NO: 829) (A02.01)
AQIAMVWPL (SEQ ID NO: 830) (A02.01, A24.02)
AQIAMVWPLL (SEQ ID NO: 831) (A02.01)
CPMSRLRLA (SEQ ID NO: 832) (B07.02, B08.01)
CPMSRLRLAL (SEQ ID NO: 833) (B07.02, B08.01)
IAMVWPLLSI (SEQ ID NO: 834) (A02.01, A24.02, B08.01)
ILSEWKEICV (SEQ ID NO: 835) (A02.01)
IVWWCPMSR (SEQ ID NO: 836) (A03.01)
IVWWCPMSRL (SEQ ID NO: 837) (A02.01)
IWMTETLFDI (SEQ ID NO: 838) (A24.02)
LLSILSEWK (SEQ ID NO: 839) (A03.01)
MSAAQIAMV (SEQ ID NO: 840) (A02.01)
MSRLRLALT (SEQ ID NO: 841) (B08.01)
MSRLRLALTV (SEQ ID NO: 842) (B08.01)
MVWPLLSIL (SEQ ID NO: 843) (A02.01)
RLALTVPPST (SEQ ID NO: 844) (A02.01)
TLFDIVWWC (SEQ ID NO: 845) (A02.01)
TLFDIVWWCP (SEQ ID NO: 846) (A02.01)
TMSAAQIAMV (SEQ ID NO: 847) (A02.01)
VWSIWMTETL (SEQ ID NO: 848) (A24.02)
WMTETLFDI (SEQ ID NO: 849) (A02.01, A24.02)
WMTETLFDIV (SEQ ID NO: 850) (A01.01, A02.01)
P250fs
S260fs
N263fs
G266fs
N268fs
V272fs
V274fs
P278fs
D281fs
R282fs
T284fs
E285fs
L289fs
K292fs
P301fs
S303fs
T312fs
S314fs
K319fs
K320fs
P322fs
Y327fs
F328fs
L330fs
R333fs
R335fs
R337fs
E339fs
ALRCVFVPVL (SEQ ID NO: 852) (A02.01, B08.01)
ALSEHCPTT (SEQ ID NO: 853) (A02.01)
AQRKRISARK (SEQ ID NO: 854) (A03.01)
GAQRKRISA (SEQ ID NO: 855) (B08.01)
HWMENISPF (SEQ ID NO: 856) (A24.02)
LPSQRRNHW (SEQ ID NO: 857) (B07.02)
LPSQRRNHWM (SEQ ID NO: 858) (B07.02, B08.01)
NISPFRSVGV (SEQ ID NO: 859) (A02.01)
RISARKGSL (SEQ ID NO: 860) (B07.02, B08.01)
SPFRSVGVSA (SEQ ID NO: 861) (B07.02)
SPSSHWKTPV (SEQ ID NO: 862) (B07.02, B08.01)
TALRCVFVPV (SEQ ID NO: 863) (A02.01)
VIYWDGTAL (SEQ ID NO: 864) (A02.01)
VIYWDGTALR (SEQ ID NO: 865) (A03.01)
VLGETGAQRK (SEQ ID NO: 866) (A03.01)
P151fs
P152fs
V157fs
Q165fs
S166fs
H168fs
V173fs
HPRPRHGHLQ (SEQ ID NO: 868) (B07.02)
RPRHGHLQA (SEQ ID NO: 869) (B07.02)
RPRHGHLQAV (SEQ ID NO: 870) (B07.02, B08.01)
D48fs
D49fs
Q52fs
F54fs
E56fs
P58fs
P60fs
E62fs
M66fs
P72fs
V73fs
P75fs
A78fs
P82fs
P85fs
S96fs
P98fs
T102fs
Y103fs
G108fs
F109fs
R110fs
G117fs
PPGPCHLLSL (SEQ ID NO: 872) (B07.02)
RTILNNGSLK (SEQ ID NO: 873) (A03.01)
SLKTQVQMK (SEQ ID NO: 874) (A03.01)
SLKTQVQMKL (SEQ ID NO: 875) (B08.01)
TILNNGSLK (SEQ ID NO: 876) (A03.01)
P27fs
P34fs
P36fs
A39fs
Q38fs
TTFCPPCRPK (SEQ ID NO: 878) (A03.01)
L130fs
N131fs
C135fs
K139fs
A138fs
T140fs
V143fs
Q144fs
V147fs
T150fs
P151fs
P152fs
G154fs
R156fs
R158fs
A161fs
FANWPRPAL (SEQ ID NO: 880) (B07.02, B08.01)
GLIPHPRPA (SEQ ID NO: 881) (A02.01)
HPRPAPASA (SEQ ID NO: 882) (B07.02, B08.01)
HPRPAPASAP (SEQ ID NO: 883) (B07.02)
IPHPRPAPA (SEQ ID NO: 884) (B07.02, B08.01)
IPHPRPAPAS (SEQ ID NO: 885) (B07.02)
RPALCSCGL (SEQ ID NO: 886) (B07.02)
RPALCSCGLI (SEQ ID NO: 887) (B07.02)
TPLPSTRCF (SEQ ID NO: 888) (B07.02)
WPRPALCSC (SEQ ID NO: 889) (B07.02)
WPRPALCSCG (SEQ ID NO: 890) (B07.02)
D179fs
L184fs
T202fs
R205fs
D213fs
G212fs
GLVPSLVSK (SEQ ID NO: 892) (A03.01)
KISVETYTV (SEQ ID NO: 893) (A02.01)
LLMVLMSLDL (SEQ ID NO: 894) (A02.01, B08.01)
LMSLDLDTGL (SEQ ID NO: 895) (A02.01)
LMVLMSLDL (SEQ ID NO: 896) (A02.01)
LVSKCLILRV (SEQ ID NO: 897) (A02.01)
QLLMVLMSL (SEQ ID NO: 898) (A02.01, B08.01)
RPGAADTGA (SEQ ID NO: 899) (B07.02)
RPGAADTGAH (SEQ ID NO: 900) (B07.02)
SLDLDTGLV (SEQ ID NO: 901) (A02.01)
SLVSKCLIL (SEQ ID NO: 902) (A02.01, B08.01)
SQLLMVLMSL (SEQ ID NO: 903) (A02.01)
TVSSQLLMV (SEQ ID NO: 904) (A02.01)
TYTVSSQLL (SEQ ID NO: 905) (A24.02)
TYTVSSQLLM (SEQ ID NO: 906) (A24.02)
VLMSLDLDT (SEQ ID NO: 907) (A02.01)
VPSLVSKCL (SEQ ID NO: 908) (B07.02)
VSKCLILRVK (SEQ ID NO: 909) (A03.01)
YTVSSQLLM (SEQ ID NO: 910) (A01.01)
YTVSSQLLMV (SEQ ID NO: 911) (A02.01)
K159fs
R161fs
Q164fs
I147fs
F148fs
L158fs
S72fs
I75fs
S80fs
P86fs
P97fs
I109fs
H115fs
L116fs
G123fs
T124fs
N131fs
L135fs
V137fs
G144fs
D143fs
I147fs
CPYGSTSTAS (SEQ ID NO: 914) (B07.02)
LARAAASTAT (SEQ ID NO: 915) (B07.02)
MLTDSLFLP (SEQ ID NO: 916) (A02.01)
PPRSSSAIAV (SEQ ID NO: 917) (B07.02)
RAAASTATEV (SEQ ID NO: 918) (B07.02)
SPPRSSSAI (SEQ ID NO: 919) (B07.02)
SPPRSSSAIA (SEQ ID NO: 920) (B07.02)
SPTQRCRLA (SEQ ID NO: 921) (B07.02)
TQRCRLARA (SEQ ID NO: 922) (B08.01)
TQRCRLARAA (SEQ ID NO: 923) (B08.01)
P172fs
N174fs
L178fs
D179fs
L188fs
WTSSGRSTK (SEQ ID NO: 925) (A03.01)
V66fs
Q73fs
V84fs
F91fs
T100fs
P103fs
S111fs
L116fs
H115fs
D126fs
AQLRRRAAA (SEQ ID NO: 927) (B08.01)
AQLRRRAAAL (SEQ ID NO: 928) (B08.01)
ARRAARMAQL (SEQ ID NO: 929) (B08.01)
HPQLPRSPL (SEQ ID NO: 930) (B07.02, B08.01)
HPQLPRSPLA (SEQ ID NO: 931) (B07.02)
LARALPGHL (SEQ ID NO: 932) (B07.02)
LARALPGHLL (SEQ ID NO: 933) (B07.02)
MAQLRRRAA (SEQ ID NO: 934) (B07.02, B08.01)
MAQLRRRAAA (SEQ ID NO: 935) (B07.02, B08.01)
QLRRRAAAL (SEQ ID NO: 936) (B07.02, B08.01)
RAAALPNAAA (SEQ ID NO: 937) (B07.02)
RMAQLRRRAA (SEQ ID NO: 938) (B07.02, B08.01)
SQSARRAARM (SEQ ID NO: 939) (B08.01)
RVGNCKHLK (SEQ ID NO: 941) (A03.01)
*вне рамки
KICNESASRK (SEQ ID NO: 943) (A03.01)
IQVLNVSLK (SEQ ID NO: 945) (A03.01)
KSSSNVISY (SEQ ID NO: 946) (A01.01, A03.01)
KYGWSLLRV (SEQ ID NO: 947) (A24.02)
RSWKYGWSL (SEQ ID NO: 948) (A02.01)
SLKSSSNVI (SEQ ID NO: 949) (B08.01)
SWKYGWSLL (SEQ ID NO: 950) (A24.02)
TVANGRSWK (SEQ ID NO: 951) (A03.01)
VPQVNGIQV (SEQ ID NO: 952) (B07.02)
VPQVNGIQVL (SEQ ID NO: 953) (B07.02)
VTVANGRSWK (SEQ ID NO: 954) (A03.01)
WSLLRVPQV (SEQ ID NO: 955) (B08.01)
KLRVPGSSV (SEQ ID NO: 957) (B07.02)
KLRVPGSSVL (SEQ ID NO: 958) (B07.02)
RVPGSSVLV (SEQ ID NO: 959) (A02.01)
SVLVTVPGL (SEQ ID NO: 960) (A02.01)
VPGSSVLVTV (SEQ ID NO: 961) (B07.02)
ALSALLLRPR (SEQ ID NO: 963) (A03.01)
ATGFKQSSK (SEQ ID NO: 966) (A03.01)
KGPELFPLI (SEQ ID NO: 968) (A02.01, A24.02)
KGPELFPLIF (SEQ ID NO: 969) (A24.02)
QLQDKFEHL (SEQ ID NO: 973) (A02.01, B08.01)
QLQDKFEHLK (SEQ ID NO: 974) (A03.01)
YLVIQLQDKF (SEQ ID NO: 975) (A02.01, A24.02)
STREKNSQV (SEQ ID NO: 977) (B08.01)
VYRRKHQEL (SEQ ID NO: 978) (A24.02, B08.01)
VLTVTSTDVK (SEQ ID NO: 980) (A03.01)
IMSLWGLVSK (SEQ ID NO: 982) (A03.01)
KLKQEATSK (SEQ ID NO: 983) (A03.01)
QIMSLWGLV (SEQ ID NO: 984) (A02.01)
SQIMSLWGL (SEQ ID NO: 985) (A02.01, A24.02, B08.01)
SQIMSLWGLV (SEQ ID NO: 986) (A02.01)
TSKSQIMSL (SEQ ID NO: 987) (B08.01)
LQEFDVQEAL (SEQ ID NO: 989) (A02.01)
RNRTEKHSTM (SEQ ID NO: 991) (B08.01)
ALNSEALSVV (SEQ ID NO: 993) (A02.01)
MALNSEALSV (SEQ ID NO: 994) (A02.01, B08.01)
WLHKRGKYI (SEQ ID NO: 1006) (A02.01, B07.02, B08.01)
WLHKRGKYIK (SEQ ID NO: 1007) (A03.01)
GVSAPKPLSK (SEQ ID NO: 1009) (A03.01)
VSAPKPLSK (SEQ ID NO: 1010) (A03.01)
KLSDSRTALK (SEQ ID NO: 1013) (A03.01)
LSDSRTALK (SEQ ID NO: 1014) (A01.01, A03.01)
RTALKSGYGK (SEQ ID NO: 1015) (A03.01)
TALKSGYGK (SEQ ID NO: 1016) (A03.01)
ALSASNSCFI (SEQ ID NO: 1018) (A02.01)
FQNGKMALSA (SEQ ID NO: 1019) (A02.01, B08.01)
(Устойчивость)
LVVVGACGV (SEQ ID NO: 155) (A02.01)
VVGACGVGK (SEQ ID NO: 156) (A03.01, A11.01)
VVVGACGVGK (SEQ ID NO: 157) (A03.01)
VVVGADGVGK (SEQ ID NO: 159) (A11.01)
KLVVVGADGV (SEQ ID NO: 160) (A02.01)
LVVVGADGV (SEQ ID NO: 161) (A02.01)
LVVVGAVGV (SEQ ID NO: 163) (A02.01)
VVGAVGVGK (SEQ ID NO: 164) (A03.01, A11.01)
VVVGAVGVGK (SEQ ID NO: 5) (A03.01, A11.01)
LLDILDTAGL (SEQ ID NO: 167) (A02.01)
TTNAFSGEY (SEQ ID NO: 1025) (A01.01)
YTTNAFSGEY (SEQ ID NO: 1026) (A01.01)
GSMNRRPIL (SEQ ID NO: 1031) (B08.01)
MGSMNRRPI (SEQ ID NO: 1032) (B08.01)
MGSMNRRPIL (SEQ ID NO: 1033) (B08.01)
SMNRRPILTI (SEQ ID NO: 1034) (A02.01, A24.02, B08.01)
GMNQRPILTI (SEQ ID NO: 1036) (A02.01, B08.01)
NQRPILTII (SEQ ID NO: 1037) (A02.01, B08.01)
GMNWRPILTI (SEQ ID NO: 1039) (A02.01, B08.01)
MNWRPILTI (SEQ ID NO: 1040) (A02.01, A24.02, B08.01)
MNWRPILTII (SEQ ID NO: 1041) (A02.01, A24.02)
VVVPCEPPEV (SEQ ID NO: 1045) (A02.01)
LLPEDTPPL (SEQ ID NO: 1047) (A02.01)
CLADVLLSV (SEQ ID NO: 1049) (A02.01)
FLQERNLPPK (SEQ ID NO: 1050) (A03.01)
HLIVLRVVRL (SEQ ID NO: 1051) (A02.01, B08.01)
HPKVHLNTM (SEQ ID NO: 1052) (B07.02, B08.01)
HPKVHLNTMF (SEQ ID NO: 1053) (B07.02, B08.01)
KVHLNTMFR (SEQ ID NO: 1054) (A03.01)
KVHLNTMFRR (SEQ ID NO: 1055) (A03.01)
LPAPFRVNHA (SEQ ID NO: 1056) (B07.02)
MFRRPHSCL (SEQ ID NO: 1057) (B07.02, B08.01)
MFRRPHSCLA (SEQ ID NO: 1058) (B08.01)
NTMFRRPHSC (SEQ ID NO: 1059) (B08.01)
RPHSCLADV (SEQ ID NO: 1060) (B07.02)
RPHSCLADVL (SEQ ID NO: 1061) (B07.02)
RVVRLPAPFR (SEQ ID NO: 1062) (A03.01)
SVHLIVLRV (SEQ ID NO: 1063) (A02.01)
TMFRRPHSC (SEQ ID NO: 1064) (B08.01)
TMFRRPHSCL (SEQ ID NO: 1065) (A02.01, B08.01)
VLLSVHLIV (SEQ ID NO: 1066) (A02.01)
VLLSVHLIVL (SEQ ID NO: 1067) (A02.01)
VLRVVRLPA (SEQ ID NO: 1068) (B08.01)
VVRLPAPFR (SEQ ID NO: 1069) (A03.01)
[287] Синтезировали подгруппу пептидов из таблицы 1 (n=562), и их аффинность к данной молекуле HLA I класса измеряли, как описано. значения показаны в таблице 3. Эти данные показывают сильную корреляцию между прогнозом и измерением (пунктирная линия представляет наилучшее соответствие, R2=0,45), демонстрируя значение прогнозов. Однако выбросы демонстрируют важность этих измерений. Толстые вертикальные и горизонтальные линии показаны при 500 нМ для прогнозируемой аффинности и наблюдаемой аффинности, соответственно. 500 нМ обычно принимают в данной области в качестве максимальной аффинности к эпитопу, который является «слабым связующим» с HLA I класса. Следовательно, точки в нижнем правом квадрате (прогноз более 500 нМ измерение менее 500 нМ) представляют собой эпитопы, которые считались очень слабыми связующими, но наблюдали связывание в приемлемых пределах. Эпитопы в этом квадрате (n=75) представляют 30,5% эпитопов, не считающихся связующими согласно прогнозам (комбинация нижнего правого и верхнего правого квадратов, n=246).
(IC50; (нМ))
(IC50; (нМ))
[288] В таблице 4B-4M представлены пептидные последовательности, содержащие мутации RAS, соответствующие аллелю HLA, с которым он связан, и соответствующая оценка прогнозируемой аффинности связывания с наименьшим числом (например, 1), имеющая наивысшую аффинность, и наоборот.
[289] Также в данном документе представлен способ лечения рака у субъекта, включающий введение субъекту (i) полипептида, содержащего эпитоп G12R RAS, или (ii) полинуклеотида, кодирующего полипептид; причем: (a) эпитоп G12R RAS представляет собой vvgaRgvgk (SEQ ID NO: 1), а субъект экспрессирует белок, кодируемый аллелем HLA-A03:01; (b) эпитоп G12R RAS представляет собой eyklvvvgaR(SEQ ID NO: 2), а субъект экспрессирует белок, кодируемый аллелем HLA-A33:03; (c) эпитоп G12R RAS представляет собой vvvgaRgvgk(SEQ ID NO: 3), а субъект экспрессирует белок, кодируемый аллелем HLA-A11:01; или (d) эпитоп G12R RAS представляет собой aRgvgksal(SEQ ID NO: 4), а субъект экспрессирует белок, кодируемый аллелем HLA, выбранным из группы, состоящей из HLA-C07:02, HLA-B39:01 и HLA-C07:01.
[290] В таблице 5 представлены пептиды GATA и их партнеры по связыванию HLA.
N334fs
CQWGPCLQCL (SEQ ID NO: 1300) (A02.01)
QWGPCLQCL (SEQ ID NO: 1301) (A24.02)
QWGPCLQCLW (SEQ ID NO: 1302) (A24.02)
S408fs
S408fs
S430fs
H434fs
H435fs
ALQPLQPHA (SEQ ID NO: 1304) (A02.01)
DLHFCRSSIM (SEQ ID NO: 1305) (B08.01)
EPHLALQPL (SEQ ID NO: 1306) (B07.02, B08.01)
ESKIMFATL (SEQ ID NO: 1307) (B08.01)
FATLQRSSL (SEQ ID NO: 1308) (B07.02, B08.01)
FLKAESKIM (SEQ ID NO: 1309) (B08.01)
FLKAESKIMF (SEQ ID NO: 1310) (B08.01)
GPPARVPAV (SEQ ID NO: 1311) (B07.02)
IMKPKRDGYM (SEQ ID NO: 1312) (B08.01)
KIMFATLQR (SEQ ID NO: 1313) (A03.01)
KPKRDGYMF (SEQ ID NO: 1314) (B07.02)
KPKRDGYMFL (SEQ ID NO: 1315) (B07.02)
LHFCRSSIM (SEQ ID NO: 1316) (B08.01)
LQHGHRHGL (SEQ ID NO: 1317) (B08.01)
MFATLQRSSL (SEQ ID NO: 1318) (B07.02, B08.01)
MFLKAESKI (SEQ ID NO: 1319) (A24.02)
MLTGPPARV (SEQ ID NO: 1320) (A02.01)
QPVLWTTPPL (SEQ ID NO: 1321) (B07.02)
SMLTGPPARV (SEQ ID NO: 1322) (A02.01)
TLQRSSLWCL (SEQ ID NO: 1323) (A02.01)
VLPEPHLAL (SEQ ID NO: 1324) (A02.01)
VPAVPFDLHF (SEQ ID NO: 1325) (B07.02)
YMFLKAESK (SEQ ID NO: 1326) (A03.01)
YMFLKAESKI (SEQ ID NO: 1327) (A02.01, A03.01, A24.02, B08.01)
[291] В таблице 6 представлена аффинность HLA и стабильность выбранных пептидов BTK:
[292] В таблице 7 представлена аффинность HLA и стабильность выбранных пептидов EGFR:
(время полужизни (ч))
Опухолевые антигены, ассоциированные с микросредой опухоли
[293] Во многих случаях в микросреде опухоли клетки экспрессируют преобладающие антигены, которые не только служат отличными биомаркерами заболевания, но также могут быть важными кандидатами на вакцины для иммунотерапии. Такие антигены, ассоциированные с опухолью (ТА) не обязательно присутствуют на поверхности опухолевых клеток, но на клетках, расположенных рядом с опухолью, которые могут быть стромальными клетками, клетками соединительной ткани, фибробластами и так далее. Это клетки, которые часто вносят вклад в структурную целостность опухоли, питают опухоль и поддерживает рост опухоли. В большинстве случаев TAA являются сверхэкспрессируемыми антигенами в микросреде опухоли, однако некоторые антигены в микросреде опухоли также могут быть уникальными в клетках, ассоциированных с опухолью. Например, обратная транскриптаза теломеразы (TERT) представляет собой TAA, которая не присутствует в большинстве нормальных тканей, но активируется в большинстве опухолей человека. С другой стороны, тканевые калликреин-родственные пептидазы или калликреины (KLK) сверхэкспрессируются при различных раковых заболеваниях и составляют большое семейство секретируемых трипсин- или химотрипсиноподобных сериновых протеаз. Калликреины активируются при раке предстательной железы, яичников и молочной железы. Некоторые TAA специфичны для определенных видов рака, некоторые экспрессируются в большом количестве видов рака. Карциноэмбриональный антиген (CEA) сверхэкспрессируется в карциномах молочной железы, толстой кишки, легких и поджелудочной железы, тогда как MUC-1 представляет собой рак груди, легких, предстательной железы, толстой кишки. Некоторые TAA являются дифференцированными или тканеспецифичными, например, MART-1/melan-A и gp100 экспрессируются в нормальных меланоцитах и меланоме, а простатоспецифический мембранный антиген (PSMA) и простатоспецифический антиген (PSA) экспрессируются эпителиальными клетками предстательной железы, а также карциномой предстательной железы.
[294] В некоторых вариантах осуществления для адоптивной терапии конструируют T-клетки, которые направлены на сверхэкспрессируемые тканеспецифические или опухолеассоциированные антигены, такие как специфические для предстательной железы калликреиновые белки KLK2, KLK3, KLK4 в случае терапии рака предстательной железы или трансглутамазный белок 4, TGM4 для аденокарциномы.
[295] В некоторых вариантах осуществления антигенные пептиды, нацеленные на адоптивную терапию в способах, раскрытых в настоящем документе, эффективны для модуляции микросреды опухоли. Т-клетки примируют антигенами, экспрессируемыми клетками в TME, так что терапия направлена на ослабление и/или разрушение опухоли, способствующей TME, часто в дополнение к прямому нацеливанию на опухолевые клетки для опосредованного Т-клетками лизиса.
[296] Микросреда опухоли содержит фибробласты, стромальные клетки, эндотелиальные клетки и клетки соединительной ткани, которые составляют большую часть клеток, которые индуцируют или влияют на рост опухоли. Так же, как Т-клетки можно стимулировать и направлять атаку на опухолевые клетки в иммуносупрессивной среде опухоли, можно использовать определенные пептиды и антигены, чтобы направлять Т-клетки против клеток рядом с опухолью, которые помогают в распространении опухоли, можно получать CD8+ и CD4+ Т-клетки ex vivo, которые направлены против антигенов на поверхности неопухолевых клеток в микросреде опухоли, которые способствуют поддержанию и распространению опухоли. Связанные с раком/опухолью фибробласты (CAF) являются отличительной чертой рака поджелудочной железы, такого как аденокарцинома поджелудочной железы (PDAC). CAF экспрессируют антиген Col10a1. CAF представляют собой клетки, которые могут способствовать сохранению опухоли. Col10A1 часто дает отрицательный прогноз для опухоли. В некоторых вариантах осуществления Col10A1 можно рассматривать как биомаркер питания и прогрессирования опухоли. Это гетеродимерный белок длиной 680 аминокислот, связанный с плохим прогнозом при раке молочной железы и колоректальном раке.
[297] Активация Col10a1-специфических CD8+ Т-клеток и CD4+ T-клеток может способствовать атаке и разрушению Col10A1-специфических фибробластов и способствовать разрушению тканевого матрикса солидных опухолей.
[298] Т-клетки можно получать ex vivo с использованием описанного в данном документе способа, так чтобы активировать Т-клетки против фибробластов, связанных с раком (CAF). Для этого были получены пептиды Col10a1, содержащие эпитопы, которые могут специфически активировать Т-клетки, и определен партнер по связыванию HLA с использованием высоконадежных данных, полученных с помощью собственного программного обеспечения для презентации эпитопа с машинным обучением, описанного ранее, как описано в таблице 8.
[299] Неоантигенные пептиды, представленные в настоящем документе, предварительно проверяют на иммуногенность связывания HLA (таблицы 1-8 и 11-14). В некоторых вариантах осуществления неоантигенные пептиды, полученные и сохраненные ранее, использованы для контакта с антигенпрезентирующей клеткой (APC), чтобы затем обеспечить презентацию Т-клетке in vitro для получения неоантиген-специфической активированной Т-клетки. В некоторых вариантах осуществления от 2 до 80 или более неоантигенных пептидов использованы для одновременной стимуляции Т-клеток пациента.
[300] В некоторых вариантах осуществления APC является аутологичной APC. В некоторых вариантах осуществления APC не является аутологичной APC. В некоторых вариантах осуществления APC представляет собой синтетическую клетку, предназначенную для функционирования в качестве APC. В некоторых вариантах осуществления Т-клетка представляет собой аутологичную клетку. В некоторых вариантах осуществления антигенпрезентирующая клетка представляет собой клетку, экспрессирующую антиген. Например, антигенпрезентирующая клетка может быть фагоцитарной клеткой, такой как дендритная клетка или миелоидная клетка, которая обрабатывает антиген после клеточного поглощения и презентирует антиген в ассоциации с MHC для активации Т-клеток. В рамках настоящего изобретения для определенных целей APC представляет собой клетку, которая обычно представляет антиген на своей поверхности. В несвязывающем или неограничивающем примере, относящемся к определенным анализам цитотоксичности, как описано в данном документе, опухолевая клетка представляет собой антигенпрезентирующую клетку, при этом Т-клетка может распознавать антигенпрезентирующую клетку (опухолевую клетку). Аналогично, клетка или линия клеток, экспрессирующих антиген, для определенных целей в рамках настоящего изобретения может быть антигенпрезентирующей клеткой.
[301] В некоторых вариантах осуществления один или несколько полинуклеотидов, кодирующих один или несколько неоантигенных пептидов, можно использовать для экспрессии в клетке и презентации Т-клетке для активации in vitro. Один или несколько полинуклеотидов, кодирующих один или несколько неоантигенных пептидов, кодируют в векторе. В некоторых вариантах осуществления композиция содержит от приблизительно 2 до приблизительно 80 неоантигенных полинуклеотидов. В вариантах осуществления изобретения по меньшей мере один из дополнительных неоантигенных пептидов является специфическим для опухоли отдельного субъекта. В вариантах осуществления настоящего изобретения неоантигенный пептид, специфичный для данного субъекта, выбирают путем выявления различий в последовательностях между геномом, экзомом и/или транскриптомом образца опухоли субъекта и геномом, экзомом и/или транскриптомом неопухолевого образца. В вариантах осуществления изобретения образцы представляют собой свежие или зафиксированные в формалине залитые парафином опухолевые ткани, свежевыделенные клетки или циркулирующие опухолевые клетки. В вариантах осуществления различия последовательностей определяют путем секвенирования следующего поколения.
[302] В некоторых вариантах осуществления способ и композиции, представленные в настоящем документе, можно использовать для идентификации или выделения Т-клеточного рецептора (TCR), способного связывать по меньшей мере один неоантигенный пептид, описанный в данном документе, или комплекс MHC-пептид, содержащий по меньшей мере один неоантигенный пептид, описанный в данном документе. В вариантах осуществления MHC комплекса MHC-пептид представляет собой MHC I класса или II класса. В вариантах осуществления TCR представляет собой биспецифический TCR, дополнительно содержащий домен, содержащий антитело или фрагмент антитела, способный связывать антиген. В вариантах осуществления антиген представляет собой антиген, специфический для Т-клеток. В вариантах осуществления антиген представляет собой CD3. В вариантах осуществления антитело или фрагмент антитела представляет собой scFv против CD3.
[303] В некоторых вариантах осуществления способ и композиции, представленные в настоящем документе, можно использовать для получения рецептора химерного антигена, содержащего: (i) молекулу активации Т-клеток; (ii) трансмембранную область; и (iii) фрагмент распознавания антигена, способный связывать по меньшей мере один неоантигенный пептид, описанный в данном документе, или комплекс MHC-пептид, содержащий по меньшей мере один неоантигенный пептид, описанный в данном документе. В вариантах осуществления молекулой активации Т-клеток является CD3-дзета. В вариантах осуществления рецептор химерного антигена дополнительно содержит по меньшей мере один костимулирующий сигнальный домен. В вариантах осуществления сигнальный домен представляет собой CD28, 4-1BB, ICOS, OX40, ITAM или Fc-эпсилон RI-гамма. В вариантах осуществления фрагмент распознавания антигена способен связывать выделенный неоантигенный пептид в контексте MHC I класса или II класса. В вариантах осуществления трансмембранная область CD3-дзета, CD28, CTLA-4, ICOS, BTLA, KIR, LAG3, CD137, OX40, CD27, CD40L, Tim-3, A2aR или PD-1. В вариантах осуществления неоантигенный пептид расположен во внеклеточном домене полипептида, ассоциированного с опухолью. В вариантах осуществления MHC комплекса MHC-пептида представляет собой MHC I класса или II Класса.
[304] В данном документе представлена T-клетка, содержащая рецептор T-клетки или химерный антигенный рецептор, описанный в данном документе, причем необязательно, T-клетка представляет собой хелперную или цитотоксичную T-клетку. В вариантах осуществления T-клетка представляет собой T-клетку субъекта.
[305] В данном документе представлена T-клетка, содержащая рецептор T-клетки (TCR), способный связывать по меньшей мере один неоантигенный пептид, описанный в данном документе, или комплекс MHC-пептид, содержащий по меньшей мере один неоантигенный пептид, описанный в данном документе, причем T-клетка представляет собой T-клетку, выделенную из популяции T-клеток субъекта, которые инкубировали с антигенпрезентирующими клетками и одним или несколькими из по меньшей мере одного неоантигенного пептида, описанного в данном документе, в течение достаточного времени для активации T-клеток. В вариантах осуществления T-клетка представляет собой CD8+ T-клетку, хелперную T-клетку или цитотоксичную T-клетку. В вариантах осуществления популяция T-клеток субъекта представляет собой популяцию CD8+ T-клеток субъекта. В вариантах осуществления один или несколько из по меньшей мере одного неоантигенного пептида, описанного в данном документе, представляет собой специфичный для субъекта неоантигенный пептид. В вариантах осуществления специфичный для субъекта неоантигенный пептид имеет другой неоэпитоп опухоли, который является эпитопом, специфичным для опухоли субъекта. В вариантах осуществления специфический для субъекта неоантигенный пептид представляет собой продукт экспрессии опухолеспецифической немолчащей мутации, которая отсутствует в неопухолевом образце субъекта. В вариантах осуществления специфичный для субъекта неоантигенный пептид связывается с белком HLA субъекта. В вариантах осуществления специфичный для субъекта неоантигенный пептид связывается с белком HLA субъекта с IC50 менее 500 нМ. В вариантах осуществления активированные CD8+ Т-клетки отделяют от антигенпрезентирующих клеток. В вариантах осуществления антигенпрезентирующие клетки представляют собой дендритные клетки или В-клетки с увеличенным CD40L. В вариантах осуществления антигенпрезентирующие клетки представляют собой нетрансформированные клетки. В вариантах осуществления антигенпрезентирующие клетки представляют собой неинфицированные клетки. В вариантах осуществления антигенпрезентирующие клетки являются аутологичными. В вариантах осуществления антигенпрезентирующие клетки обрабатывали для удаления эндогенных пептидов, ассоциированных с MHC, с их поверхности. В вариантах осуществления обработка для удаления эндогенных пептидов, ассоциированных с MHC, включает культивирование клеток приблизительно при 26°C. В вариантах осуществления обработка для удаления эндогенных пептидов, ассоциированных с MHC, включает обработку клеток слабым кислотным раствором. В вариантах осуществления в антигенпрезентирующие клетки вводят по меньшей мере один неоантигенный пептид, описанный в данном документе. В вариантах осуществления введение включает инкубацию антигенпрезентирующих клеток в присутствии по меньшей мере приблизительно 2 мкг/мл каждого из по меньшей мере одного неоантигенного пептида, описанного в данном документе. В вариантах осуществления соотношение выделенных Т-клеток и антигенпрезентирующих клеток составляет приблизительно от 30:1 до 300:1. В вариантах осуществления инкубация выделенной популяции Т-клеток происходит в присутствии IL-2 и IL-7. В вариантах осуществления MHC комплекса MHC-пептид представляет собой MHC I класса или II класса.
[306] В данном документе представлен способ активации опухолеспецифических T-клеток, включающий: выделение популяции T-клеток субъекта; и инкубацию выделенной популяции T-клеток с антигенпрезентирующими клетками и по меньшей мере одним неоантигенным пептидом, описанным в данном документе, в течение достаточного времени активации T-клеток. В вариантах осуществления T-клетка представляет собой CD8+ T-клетку, хелперную T-клетку или цитотоксичную T-клетку. В вариантах осуществления популяция T-клеток субъекта представляет собой популяцию CD8+ T-клеток субъекта. В вариантах осуществления один или несколько из по меньшей мере одного неоантигенного пептида, описанного в данном документе, представляет собой специфичный для субъекта неоантигенный пептид. В вариантах осуществления специфичный для субъекта неоантигенный пептид имеет другой неоэпитоп опухоли, который является эпитопом, специфичным для опухоли субъекта. В вариантах осуществления специфичный для субъекта неоантигенный пептид представляет собой продукт экспрессии опухолеспецифической немолчащей мутации, которая отсутствует в неопухолевом образце субъекта. В вариантах осуществления специфичный для субъекта неоантигенный пептид связывается с белком HLA субъекта. В вариантах осуществления специфичный для субъекта неоантигенный пептид связывается с белком HLA субъекта с IC50 менее 500 нМ. В вариантах осуществления способ дополнительно предусматривает отделение активированных T-клеток от антигенпрезентирующих клеток. В вариантах осуществления способ дополнительно предусматривает тестирование активированных T-клеток для подтверждения реактивности против по меньшей мере одного неоантигенного пептида, описанного в данном документе. В вариантах осуществления антигенпрезентирующие клетки являются дендритными клетками или B клетками с увеличенным CD40L. В вариантах осуществления антигенпрезентирующие клетки являются нетрансформированными клетками. В вариантах осуществления антигенпрезентирующие клетки являются незараженными клетками. В вариантах осуществления антигенпрезентирующие клетки являются аутологичными. В вариантах осуществления антигенпрезентирующие клетки обрабатывали для удаления эндогенных пептидов, ассоциированных с MHC, с их поверхности. В вариантах осуществления обработка для удаления эндогенных пептидов, ассоциированных с MHC, включает культивирование клеток приблизительно при 26°C. В вариантах осуществления обработка для удаления эндогенных пептидов, ассоциированных с MHC, включает обработку клеток слабым кислотным раствором. В вариантах осуществления в антигенпрезентирующие клетки вводят по меньшей мере один неоантигенный пептид, описанный в данном документе. В вариантах осуществления введение включает инкубацию антигенпрезентирующих клеток в присутствии по меньшей мере приблизительно 2 мкг/мл каждого из по меньшей мере одного неоантигенного пептида, описанного в данном документе. В вариантах осуществления соотношение выделенных Т-клеток и антигенпрезентирующих клеток составляет приблизительно от 30:1 до 300:1. В вариантах осуществления инкубация выделенной популяции Т-клеток происходит в присутствии IL-2 и IL-7. В вариантах осуществления MHC комплекса MHC-пептид представляет собой MHC I класса или II класса.
[307] В данном документе представлена композиция, содержащая активированные опухолеспецифические T-клетки, полученные с помощью способа, описанного в данном документе.
[308] В данном документе представлен способ лечения рака у субъекта, включающий введение субъекту терапевтически эффективного количества активированных опухолеспецифических T-клеток, описанных в данном документе или полученных с помощью способа, описанного в данном документе. В вариантах осуществления введение предусматривает введение приблизительно от 106 до 1012, приблизительно от 108 до 1011 или приблизительно от 109 до 1010 активированных опухолеспецифических T-клеток.
[309] В данном документе представлена нуклеиновая кислота, содержащая промотор, функционально связанный с полинуклеотидом, кодирующим рецептор T-клетки, описанный в данном документе. В вариантах осуществления TCR способен связывать по меньшей мере один неоантигенный пептид в контексте главного комплекса гистосовместимости (MHC) I класса или II класса.
[310] В данном документе представлена нуклеиновая кислота, содержащая промотор, функционально связанный с полинуклеотидом, кодирующим химерный антигенный рецептор, описанный в данном документе. В вариантах осуществления фрагмент распознавания антигена способен связывать по меньшей мере один неоантигенный пептид в контексте главного комплекса гистосовместимости (MHC) I класса или II класса. В вариантах осуществления неоантигенный пептид расположен во внеклеточном домене опухолеассоциированного полипептида. В вариантах осуществления нуклеиновая кислота содержит трансмембранную область CD3-дзета, CD28, CTLA-4, ICOS, BTLA, KIR, LAG3, CD137, OX40, CD27, CD40L, Tim-3, A2aR или PD-1.
[311] В некоторых вариантах осуществления аутологичные иммунные клетки из периферической крови пациента представляют собой мононуклеарные клетки периферической крови (PBMC). В некоторых вариантах осуществления аутологичные иммунные клетки из периферической крови пациента собирают посредством процедуры афереза. В некоторых вариантах осуществления PBMC собирают в результате более чем одной процедуры афереза или более чем одного отбора периферической крови.
[312] В некоторых вариантах осуществления и CD25+ клетки и CD14+ клетки истощают перед добавлением пептидов. В некоторых вариантах осуществления либо CD25+ клетки, либо CD14+ клетки истощают перед добавлением пептидов. В некоторых вариантах осуществления перед добавлением пептидов истощают CD25+ клетки, а не CD14+ клетки.
[313] В некоторых вариантах осуществления за процедурой истощения следует добавление лиганда рецептора FMS-подобной тирозинкиназы 3 (FLT3L) для стимуляции антигенпрезентирующих клеток (APC), состоящих из моноцитов, макрофагов или дендритных клеток (DC), перед добавлением пептидов. В некоторых вариантах осуществления за процедурой истощения следует выбор DC в качестве подходящих PAC для презентации пептида Т-клеткам, и зрелые макрофаги и другие антигенпрезентирующие клетки удаляют из аутологичных иммунных клеток пациента. В некоторых вариантах осуществления за процедурой истощения следует выбор незрелых DC в качестве подходящих PAC для презентации пептида Т-клеткам.
[314] В некоторых вариантах осуществления результат выбора количества ‘n’ неоантигенных пептидов вводят в контакт с APC для стимуляции APC для презентации антигена Т-клеткам.
[315] В некоторых вариантах осуществления выбор первого уровня количества ‘n’ неоантигенных пептидов основан на способности каждого из пептидов связываться по меньшей мере с гаплотипом HLA, который, как это заранее определено, имеется у пациента-реципиента. Для того, чтобы определить гаплотип HLA, который, как это заранее определено, имеется у пациента-реципиента, как известно специалисту в данной области, пациента подвергают анализу гаплотипирования HLA по образцу крови до начала процедуры лечения. В некоторых вариантах осуществления за выбором первого уровня количества ‘n’ неоантигенных пептидов следует выбор второго уровня на основании определения, соответствует ли мутация, имеющаяся в неоантигенном пептиде (пептидах), неоантигенам, которые, как известно, обнаружены по меньшей мере у 5% пациентов, которые имеют рак (или приводящим к ним мутациям). В некоторых вариантах осуществления второй уровень выбора включает дальнейшее определение, очевидна ли мутация у пациента.
[316] В некоторых вариантах осуществления за выбором первого и второго уровня количества ‘n’ неоантигенных пептидов для введения в контакт с APC следует третий уровень выбора на основании аффинности связывания пептида с HLA, с которым пептид способен связываться, и которая составляет по меньшей мере менее 500 нМ, с определением, что чем выше аффинность связывания, тем лучше выбор пептида, который надо отобрать. В некоторых вариантах осуществления в итоге выбранное количество ‘n’ пептидов может колебаться в диапазоне 1-200 пептидов, которые находятся в смеси, для предъявления APC пептидам в культуральной среде и введения в контакт с APC.
[317] В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 10-190 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 20-180 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 30-170 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 40-160 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 50-150 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 60-140 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 70-130 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 80-120 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 50-100 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 50-90 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов может колебаться в диапазоне 50-80 неоантигенных пептидов. В некоторых вариантах осуществления количество ‘n’ пептидов составляет по меньшей мере 60 неоантигенных пептидов. В некоторых вариантах осуществления в количество ‘n’ пептидов входит смесь (a) коротких неоантигенных пептидов, длиной 8-15 аминокислот, содержащих мутантную аминокислоту, описанную ранее, согласно формуле AxByCz; для целей этой заявки эти пептиды взаимозаменяемо называются короткомерными или короткими пептидами; и (b) длинных пептидов длиной 15, 30, 50, 60, 80, 100-300 аминокислот и любой промежуточной длиной, которые дендритные клетки подвергают эндогенному процессингу для более хорошей презентации антигена; для целей этой заявки эти пептиды взаимозаменяемо называются длинномерными или длинными пептидами. В некоторых вариантах осуществления по меньшей мере 60 неоантигенных пептидов содержат по меньшей мере 30 короткомерных и по меньшей мере 30 длинномерных или их вариантов. Их иллюстративные варианты включают, но без ограничения, следующее: в некоторых вариантах осуществления по меньшей мере 60 неоантигенных пептидов содержат по меньшей мере 32 короткомерных и по меньшей мере 32 длинномерных или их вариантов. В некоторых вариантах осуществления по меньшей мере 60 неоантигенных пептидов содержат по меньшей мере 34 короткомерных и по меньшей мере 30 длинномерных или их вариантов. В некоторых вариантах осуществления по меньшей мере 60 неоантигенных пептидов содержат по меньшей мере 28 короткомерных и по меньшей мере 34 длинномерных или их вариантов.
[318] В некоторых вариантах осуществления «n» пептидов инкубируют в среде, содержащей APC в культуре, где APC (DC) были выделены из PBMC и предварительно стимулированы FLT3L. В некоторых вариантах осуществления «n» пептидов инкубируют с APC в присутствии FLT3L. В некоторых вариантах осуществления после стадии инкубации APC с FLT3L в клетки добавляют свежую среду, содержащую FL3TL, для инкубации с пептидами. В некоторых вариантах осуществления созревание APC до зрелых нагруженных пептидом DC может включать несколько этапов культивирования DC до созревания, изучения состояния созревания путем анализа одного или нескольких высвобождаемых веществ (например, цитокинов, хемокинов) в культуральной среде или получения время от времени аликвот DC в виде культуры. В некоторых вариантах осуществления для созревания DC в культуре требуется по меньшей мере 5 дней от начала культивирования. В некоторых вариантах осуществления для созревания DC в культуре требуется не менее 7 дней от начала культивирования. В некоторых вариантах осуществления для созревания DC в культуре требуется по меньшей мере 11 дней от начала культивирования или любое количество дней между ними.
[319] В некоторых вариантах осуществления DC вводят в контакт с T-клетками после подтверждения наличия или отсутствия факторов созревания и анализа пептидного тетрамера для проверки репертуара презентированных антигенов.
[320] В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 2 дней для первой индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 3 дней для первой индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 4 дней для первой индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение по меньшей мере приблизительно 2 дней для второй индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение по меньшей мере приблизительно 3 дней для второй индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение по меньшей мере приблизительно 4 дней для второй индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение 5 дней для второй индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 6 дней для второй индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 7, 8, 9 или 10 дней для второй индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно менее 1 дня для третьей индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение по меньшей мере приблизительно 2 или 3 дней для третьей индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение по меньшей мере приблизительно 4 дней для третьей индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение 5 дней для третьей индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 6 дней для третьей индукции. В некоторых вариантах осуществления DC вводят в контакт с T-клетками в среде Т-клеток в течение приблизительно 7, 8, 9 или 10 дней для второй индукции.
[321] В некоторых вариантах осуществления T-клетки вводят в дополнительный контакт с одним или несколькими короткомерными пептидами во время инкубации с DC (и в дополнение к DC) либо на первой фазе индукции, либо на второй фазе индукции, либо на третьей фазе индукции. В некоторых вариантах осуществления T-клетки вводят в дополнительный контакт с одним или несколькими короткомерными пептидами во время инкубации с DC на первой фазе индукции и второй фазе индукции. В некоторых вариантах осуществления T-клетки вводят в дополнительный контакт с одним или несколькими короткомерными пептидами во время инкубации с DC во второй фазе индукции и третьей фазе индукции. В некоторых вариантах осуществления T-клетки вводят в дополнительный контакт с одним или несколькими короткомерными пептидами во всех трех фазах индукции.
[322] В некоторых вариантах осуществления APC и T-клетки находятся в одних и тех же аутологичных иммунных клетках из периферической крови пациента взятой у пациента на первом этапе. T-клетки выделяют и сохраняют на время активации DC в конце фазы созревания DC. В некоторых вариантах осуществления T-клетки совместно культивируют в присутствии подходящей среды для активации на время активации DC в конце фазы созревания DC. В некоторых вариантах осуществления T-клетки представляют собой ранее криоконсервированные клетки пациента, которые берут и культивируют в течение от по меньшей мере 4 часов до приблизительно 48 часов для индукции во время активации DC в конце фазы созревания DC.
[323] В некоторых вариантах осуществления APC и T-клетки находятся в одних и тех же аутологичных иммунных клетках из периферической крови пациента, взятых у пациента в разные периоды времени, например, в разных процедурах афереза. В некоторых вариантах осуществления время от афереза пациента до времени сбора занимает от приблизительно 20 дней до приблизительно менее 26 дней. В некоторых вариантах осуществления время от афереза пациента до времени сбора занимает от приблизительно 21 дня до приблизительно менее 25 дней. В некоторых вариантах осуществления время от афереза пациента до времени сбора занимает от приблизительно 21 дня до приблизительно менее 24 дней. В некоторых вариантах осуществления время от афереза пациента до времени сбора занимает от приблизительно 21 дня до приблизительно менее 23 дней. В некоторых вариантах осуществления время от афереза пациента до времени сбора занимает приблизительно 21 день. В некоторых вариантах осуществления время от афереза пациента до времени сбора занимает приблизительно менее 21 дня.
[324] В некоторых вариантах осуществления критерии высвобождения для активированных T-клеток (лекарственное вещество) включают любое одно или несколько из следующего: стерильность, эндотоксин, фенотип клеток, количество TNC, жизнеспособность, концентрация клеток, эффективность. В некоторых вариантах осуществления критерии высвобождения для активированных T-клеток (лекарственное вещество) включают каждое из следующего: стерильность, эндотоксин, фенотип клеток, количество TNC, жизнеспособность, концентрация клеток, эффективность.
[325] В некоторых вариантах осуществления общее количество клеток составляет 2×1010. В некоторых вариантах осуществления общее количество клеток составляет 2×109. В некоторых вариантах осуществления общее количество клеток составляет 5×108. В некоторых вариантах осуществления общее количество клеток составляет 2×108. В некоторых вариантах осуществления итоговая концентрация ресуспендированных Т-клеток составляет 2×105 клеток/мл или более. В некоторых вариантах осуществления итоговая концентрация ресуспендированных Т-клеток составляет 1×106 клеток/мл или более. В некоторых вариантах осуществления итоговая концентрация ресуспендированных Т-клеток составляет 2×106 клеток/мл или более.
[326] Следующие критерии высвобожденных клеток описаны как иллюстративные неограничивающие условия, в частности, по той причине, что критерии для клеточной популяции и субпопуляций в лекарственном веществе (DS) могут варьироваться в зависимости от рака, состояния рака, состояния пациента, доступности подходящего гаплотипа HLA и потенциала роста APC и T-клеток в присутствии пептида. В некоторых вариантах осуществления активированные T-клетки (лекарственное вещество) содержат по меньшей мере 2% или по меньшей мере 3% или по меньшей мере 4% или по меньшей мере 5% CD8+ T-клеток, реагирующих на конкретный неоантиген по тетрамерному анализу. В некоторых вариантах осуществления активированные T-клетки (лекарственное вещество) содержат по меньшей мере 2% или по меньшей мере 3% или по меньшей мере 4% или по меньшей мере 5% CD4+ T-клеток, реагирующих на конкретный неоантиген по тетрамерному анализу. В некоторых вариантах осуществления активированные T-клетки (лекарственное вещество) содержат по меньшей мере 5% или по меньшей мере 6% или по меньшей мере 7% или по меньшей мере 8% или по меньшей мере 9% или по меньшей мере 10% клеток, положительных в отношении фенотипа T-клеток памяти.
[327] В некоторых вариантах осуществления активированные T-клетки (лекарственное вещество) выбирают на основании одного или нескольких маркеров. В некоторых вариантах осуществления активированные T-клетки (лекарственное вещество) не выбирают на основании одного или нескольких маркеров. В некоторых вариантах осуществления аликвоту активированных T-клеток (лекарственное вещество) тестируют на наличие или отсутствие одного или нескольких следующих маркеров и доли клеток, демонстрирующих каждый из тестируемых маркеров, один или несколько маркеров выбирают из группы, состоящей из: CD19, CD20, CD21, CD22, CD24, CD27, CD38, CD40, CD72, CD3, CD79a, CD79b, IGKC, IGHD, MZB1, TNFRSF17, MS4A1, CD138, TNFRSR13B, GUSPB11, BAFFR, AID, IGHM, IGHE, IGHA1, IGHA2, IGHA3, IGHA4, BCL6, FCRLA CCR7, CD27, CD45RO, FLT3LG, GRAP2, IL16, IL7R, LTB, S1PR1, SELL, TCF7, CD62L, PLAC8, SORL1, MGAT4A, FAM65B, PXN, A2M, ATM, C20orf112, GPR183, EPB41, ADD3, GRAP2, KLRG1, GIMAP5, TC2N, TXNIP, GIMAP2, TNFAIP8, LMNA, NR4A3, CDKN1A, KDM6B, ELL2, TIPARP, SC5D, PLK3, CD55, NR4A1, REL, PBX4, RGCC, FOSL2, SIK1, CSRNP1, GPR132, GLUL, KIAA1683, RALGAPA1, PRNP, PRMT10, FAM177A1, CHMP1B, ZC3H12A, TSC22D2, P2RY8, NEU1, ZNF683, MYADM, ATP2B1, CREM, OAT, NFE2L2, DNAJB9, SKIL, DENND4A, SERTAD1, YPEL5, BCL6, EGR1, PDE4B, ANXA1, SOD2, RNF125, GADD45B, SELK, RORA, MXD1, IFRD1, PIK3R1, TUBB4B, HECA, MPZL3, USP36, INSIG1, NR4A2, SLC2A3, PER1, S100A10, AIM1, CDC42EP3, NDEL1, IDI1, EIF4A3, BIRC3, TSPYL2, DCTN6, HSPH1, CDK17, DDX21, PPP1R15B, ZNF331, BTG2, AMD1, SLC7A5 POLR3E, JMJD6, CHD1, TAF13, VPS37B, GTF2B, PAF1, BCAS2, RGPD6, TUBA4A, TUBA1A, RASA3, GPCPD1, RASGEF1B, DNAJA1, FAM46C, PTP4A1, KPNA2, ZFAND5, SLC38A2, PLIN2, HEXIM1, TMEM123, JUND, MTRNR2L1, GABARAPL1, STAT4, ALG13, FOSB, GPR65, SDCBP, HBP1, MAP3K8, RANBP2, FAM129A, FOS, DDIT3, CCNH, RGPD5, TUBA1C, ATP1B3, GLIPR1, PRDM2, EMD, HSPD1, MORF4L2, IL21R, NFKBIA, LYAR, DNAJB6, TMBIM1, PFKFB3, MED29, B4GALT1, NXF1, BIRC2, ARHGAP26, SYAP1, DNTTIP2, ETF1, BTG1, PBXIP1, MKNK2, DEDD2, AKIRIN1, HLA-DMA, HLA-DNB, HLA-DOA, HLA-DPA1, HLA-DPB1, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5, CCL18, CCL19, CCL21, CXCL13, LAMP3, LTB, IL7R, MS4A1, CCL2, CCL3, CCL4, CCL5, CCL8, CXCL10, CXCL11, CXCL9, CD3, LTA, IL17, IL23, IL21, IL7, CCL5, CD27, CD274, CD276, CD8A, CMKLR1, CXCL9, CXCR6, HLA-DQA1, HLA-DRB1, HLA-E, IDO1, LAG3, NKG7, PDCD1LG2, PSMB10, STAT1, TIGIT, CD56, CCL2, CCL3, CCL4, CCL5, CXCL8, IFN, IL-2, IL-12, IL-15, IL-18, NCR1, XCL1, XCL2, IL21R, KIR2DL3, KIR3DL1, KIR3DL2, NCAM1, HLA-DMA, HLA-DNB, HLA-DOA, HLA-DPA1, HLA-DPB1, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4 и HLA-DRB5.
[328] В некоторых вариантах осуществления по меньшей мере 0,01% наивных Т-клеток, которые были получены путем получения аутологичных иммунных клеток из периферической крови пациента, стимулировали в ответ на неоантиген и амплифицировали в конце процедуры и собирали. В некоторых вариантах осуществления более 0,01% наивных Т-клеток, которые были получены путем получения яаутологичных иммунных клеток из периферической крови пациента, стимулировали в ответ на неоантиген и амплифицировали в конце процедуры и собирали. В некоторых вариантах осуществления более 0,1% наивных Т-клеток, которые были получены путем получения аутологичных иммунных клеток из периферической крови пациента, стимулировали в ответ на неоантиген и амплифицировали в конце процедуры и собирали. В некоторых вариантах осуществления более 1% наивных Т-клеток, которые были получены путем получения аутологичных иммунных клеток из периферической крови пациента, стимулировали в ответ на неоантиген и амплифицировали в конце процедуры и собирали.
[329] В некоторых вариантах осуществления общее количество клеток собирают из 1, 2 или 3 циклов процесса созревания DC и активации T-клеток.
[330] В некоторых вариантах осуществления собранные клетки криоконсервируют в паровой фазе жидкого азота в мешках.
[331] Как известно специалисту в данной области, все варианты применения, описанные в предыдущих абзацах этого раздела, от получения аутологичных иммунных клеток из периферической крови пациента до сбора клеток, выполняют в асептической замкнутой системе за исключением этапов, когда аликвоты среды или клеток отбирают для исследования с помощью проточной цитометрии, масс-спектроскопии, подсчета клеток, сортировки клеток или любых функциональных анализов, которые являются конечными для клеток или материалов, взятых в виде аликвот. В некоторых вариантах осуществления закрытая система для асептического культивирования до сбора урожая запатентована для способа заявителя.
[332] В некоторых вариантах осуществления изобретения Т-клетки являются результатом способа культивирования и размножения активированных Т-клеток, включающего этапы, описанные выше, начиная с получения аутологичных иммунных клеток из периферической крови пациента и заканчивая сбором, причем способ можно масштабировать с помощью асептической процедуры. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 1 литра клеток DC. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 1-2 литров Т-клеток. В некоторых вариантах осуществления одновременно проводят культивирование по меньшей мере 5 литров клеток DC. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 5-10 литров Т-клеток. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 10 литров клеток DC. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 10-40 литров Т-клеток. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 10 литров культуры клеток DC. В некоторых вариантах осуществления за один раз проводят культивирование по меньшей мере 10-50 литров Т-клеток. В некоторых вариантах осуществления проводят одновременное периодическое культивирование, и тестируют в системе, которая является замкнутой системой, и которой можно манипулировать и вмешиваться извне без использования неасептических средств. В некоторых вариантах осуществления описанная в данном документе замкнутая система полностью автоматизирована.
[333] Когда введение осуществляют путем инъекции, активное средство может быть составлено в виде водных растворов, в частности, в физиологически совместимых буферах, таких как раствор Хенкса, раствор Рингера или физиологический солевой буфер. Раствор может содержать средства для приготовления готовой формы, такие как суспендирующие, стабилизирующие и/или диспергирующие средства. Альтернативно, активное соединение может быть в форме порошка для смешивания перед применением с подходящим носителем, например, стерильной апирогенной водой. В другом варианте осуществления лекарственный продукт содержит вещество, которое дополнительно активирует или ингибирует компонент иммунного ответа хозяина, например, вещество для снижения или устранения иммунного ответа хозяина на пептид.
[334] Раскрытие, представленное в данном документе, демонстрирует, что общие неоантигены могут быть использованы для быстрого терапевтического введения пациенту, тем самым значительно сокращая временную задержку от исследования до пациента. Композиция и способы, описанные в данном документе, обеспечивают инновационные достижения в области лечения рака.
ПРИМЕРЫ
Пример 1. Прецизионный клинический процесс NEOSTIM
[335] В настоящем документе представлена адоптивная Т-клеточная терапия, при которой субъекту вводят Т-клетки, примированные и вызывающие ответ против тщательно отобранных предварительно проверенных, отложенных антигенных пептидов, специфичных для рака субъекта. В этом примере представлен способ обхода длительного секвенирования, идентификации и получения специфичных для субъекта неоантигенных пептидов и последующего создания Т-клеток, имеющих специфические для субъекта TCR, для иммунотерапии рака по меньшей мере на время, когда субъект проходит процесс такой оценки и подготовки к индивидуальной терапии. Преимущество этого процесса состоит в том, что он быстрый, адресный и надежный. Как показано на фиг. 1А, пациенту, у которого выявлен рак или опухоль, можно вводить Т-клетки, которые активируют ex vivo с помощью отложенных тщательно отобранных пептидов, имеющих отобранную предварительно проверенную коллекцию эпитопов, созданных из библиотеки общих антигенов, известных для идентифицированного рака. Процесс от выбора пациента до терапии Т-клетками может занять менее 6 недель. На фиг. 1B показан способ создания Т-клеток, специфичных для рака, ex vivo путем примирования Т-клеток антигенпрезентирующими клетками (APC), экспрессирующими предполагаемые Т-клеточные эпитопы, и размножения активированных Т-клеток для получения специфичных к эпитопу CD8+ и CD4+, включая популяцию этих клеток, проявляющих фенотип памяти (см., например, WO2019094642, полностью включенную посредством ссылки). Библиотеку предварительно проверенных эпитопов создают заранее. Такие эпитопы собирают на основе предшествующих знаний в данной области, общих драйверных мутаций, общих мутаций устойчивости к лекарственным средствам, тканеспецифических антигенов и опухолеассоциированных антигенов. С помощью эффективной компьютерной программы для прогнозирования эпитопов, связывания HLA и характеристик презентации предварительно проверенные пептиды создают для хранения и запасания, как показано на диаграмме на фиг. 2. На фиг. 3А-3D показаны иллюстративные прогнозы для общих мутаций RAS G12. Проверки выполняют с использованием систематического процесса, описанного в примерах 2-5. Т-клетки, реагирующие на антигены клеток опухоли-мишени, создают ex vivo, и иммуногенность подтверждают с использованием анализа антигенспецифических Т-клеток in vitro (пример 2). Масс-спектрометрию используют для подтверждения того, что клетки, экспрессирующие интересующий антиген, могут процессировать и презентировать пептиды на соответствующих молекулах HLA (Пример 3). Кроме того, способность этих Т-клеток убивать клетки, презентирующие пептид, подтверждают с помощью анализа цитотоксичности (Пример 4). Приведенные в данном документе иллюстративные данные демонстрируют этот процесс проверки для неоантигенов RAS и GATA3 и могут быть легко применены к другим антигенам.
Пример 2. Получение Т-клеток, реагирующих на антигены клеток опухоли-мишени ex vivo
[336] Материалы:
Среда AIM V (Invitrogen)
FLT3L человека, доклинический CellGenix #1415-050 маточный раствор 50 нг/мкл
TNF-α, доклинический CellGenix #1406-050 маточный раствор 10 нг/мкл
IL-1β, доклинический CellGenix #1411-050 маточный раствор 10 нг/мкл
PGE1 или алпростадил - Cayman из Чехии маточный раствор 0,5 мкг/мкл
среда R10- RPMI 1640 глутамакс+10% человеческая сыворотка+ 1% пенстреп
среда 20/80- 18% AIM V+72% RPMI 1640 глутамакс+10% человеческая сыворотка+1% пенстреп
IL7 маточный раствор 5 нг/мкл
IL15 маточный раствор 5 нг/мкл
Процедура:
Этап 1: Посев 5 миллионов PBMC (или интересующих клеток) в каждой лунке 24 луночного планшета с FLT3L в 2 мл среды AIM V
Этап 2: Загрузка и созревание пептида - в AIMV
1. Смешивание интересующего пула пептидов (за исключением условия без пептидов) с PBMC (или интересующими клетками) в соответствующих лунках.
2. Инкубация в течение 1 ч.
3. Примешивание созревающего коктейля (в том числе TNF-α, IL-1β, PGE1 и IL-7) в каждую лунку после инкубации.
Этап 3: Добавление человеческой сыворотки в каждую лунку с итоговой концентрацией 10% по объему и перемешивание.
Этап 4: Замена среды свежей средой RPMI+ 10% HS, дополненной IL7+IL15.
Этап 5: Замена среды свежей средой 20/80, дополненной IL7+IL15, во время периода инкубации каждые 1-6 дней.
Этап 6: посев 5 миллионов PBMC (или интересующих клеток) в каждой лунке нового 6-луночного планшета с FLT3L в 2 мл среды AIM V
Этап 7: Загрузка и созревание пептида для повторной стимуляции- (новые планшеты)
1. Смешивание интересующего пула пептидов (за исключением условия без пептидов) с PBMC (или интересующими клетками) в соответствующих лунках
2. Инкубация в течение 1 ч.
3. Примешивание созревающего коктейля в каждую лунку после инкубации
Этап 8: Повторная стимуляция:
1. Подсчет культур FLT3L первой стимуляции и добавление 5 миллионов культивируемых клеток на новые планшеты для повторной стимуляции.
2. Доведение объема культуры до 5 мл (AIM V) и добавление 500 мкл человеческой сыворотки (10% по объему)
Этап 9: Удаление 3 мл среды и добавление 6 мл среды RPMI+ 10% HS, дополненной IL7+IL15.
Этап 10: Замена 75% среды свежей средой 20/80, дополненной IL7+IL15.
Этап 11: При необходимости повторение повторной стимуляции.
Анализ антигенспецифической индукции
[337] Тетрамеры MHC приобретают или производят на месте согласно способам, известным рядовому специалисту, и используют для измерения размножения пептидспецифических T-клеток в анализах иммуногенности. для оценки тетрамер добавляют к 1×105 клеткам в PBS, содержащей 1% FCS и 0,1% азид натрия (буфер FACS) согласно инструкции производителя. Клетки инкубируют в темноте в течение 20 минут при комнатной температуре. Затем антитела, специфические для T-клеточных маркеров, таких как CD8, доводят до итоговой концентрации, предложенной производителем, и клетки инкубируют в темноте при 4°C в течение 20 минут. Клетки промывают холодным буфером FACS и ресуспендируют в буфере, содержащем 1% формальдегид. Клетки помещают на приборе LSR Fortessa (Becton Dickinson) и анализируют путем использования программного обеспечения FlowJo (Becton Dickinson). для анализа тетрамер-положительных клеток ворота лимфоцитов берут из графиков прямого и бокового рассеяния. Данные выводят в виде процентного значения клеток, которые представляют собой CD8+/тетрамер+.
[338] На фиг. 5 показаны иллюстративные данные для неоантигенов RAS на HLA-A03:01 и HLA-A11:01. На фиг. 6 показаны иллюстративные данные для нескольких здоровых доноров для неоантигенов RAS G12V на HLA-A11:01. На фиг. 13 показаны иллюстративные данные для неоантигенов RAS G12V на HLA-A02:01. На фиг. 14 показаны иллюстративные данные для неоантигенов RAS на HLA-A68:01. На фиг. 15 показаны иллюстративные данные для неоантигенов RAS на HLA-B07:02. На фиг. 16 показаны иллюстративные данные для неоантигенов RAS на HLA-B08:01. На фиг. 17 показаны иллюстративные данные для неоантигенов RAS G12D на HLA-C08:02. На фиг. 21 показаны иллюстративные данные для неоантиген GATA3ы на HLA-A02:01, HLA-A03:01, HLA-A11:01, HLA-B07:02 и HLA-B08:01. На фиг. 26 показаны иллюстративные данные для неоантиген BTK C481S на HLA-A02:01. На фиг. 27 показаны иллюстративные данные для неоантигенов EGFR T790M на HLA-A02:01.
[339] Ответы CD4+ T-клеток на неоантигены можно индуцировать с использованием протокола индукции ex vivo. В этом примере ответы CD4+ T-клеток идентифицировали путем отслеживания выработки IFNγ и/или TNFα антигенспецифическим образом. На фиг.18 представлены типичные примеры такого проточного цитометрического анализа для CD4+ T-клеток, реагирующих на неоантиген RAS G12D. На фиг.24 представлены типичные примеры такого проточного цитометрического анализа для CD4+ T-клеток, реагирующих на неоантиген GATA3.
Пример 3. Оценка презентации антигенов
[340] Для подгруппы спрогнозированных антигенов определяли аффинность неоэпитопов к указанным аллелям HLA и стабильность неоэпитопов с аллелями HLA. Иллюстративные данные для подгруппы неоантигенов RAS и неоантигенов GATA3 показаны на фиг. 4А и 20, соответственно.
[341] Иллюстративное подробное описание протокола, используемого для измерения аффинности связывания пептидов с MHC I класса, было опубликовано (Sette et al., Mol. Immunol. 31 (11): 813-22, 1994). Вкратце, комплексы MHCI получали и связывали с радиоактивно меченными эталонными пептидами. Пептиды инкубировали при различных концентрациях с этими комплексами в течение 2 дней, и количество оставшегося радиоактивно меченного пептида, связанного с MHCI, измеряли с помощью эксклюзионной гель-фильтрации. Более низкая концентрация тестируемого пептида, необходимая для замещения эталонного радиоактивно меченного пептида, демонстрирует более сильную аффинность тестового пептида к MHCI. Пептиды с аффинностью к MHCI <50 нМ обычно считаются сильными связывающими веществами, в то время как пептиды с аффинностью <150 нМ считаются промежуточными связывающими веществами, а пептиды <500 нМ считаются слабыми связывающими веществами (Fritsch et al, 2014).
[342] Иллюстративное подробное описание протокола, используемого для измерения стабильности связывания пептидов с MHC I класса, было опубликовано (Harndahl et al. J. Immunol Methods. 374: 5-12, 2011). Вкратце, синтетические гены, кодирующие биотинилированные тяжелые и легкие цепи MHC-I, экспрессируют в E. coli и очищают от телец включения с использованием стандартных методов. Легкую цепь (β2m) метят радиоактивным йодом (125I) и объединяют с очищенной тяжелой цепью MHC-I и представляющим интерес пептидом при 18°C, чтобы инициировать образование комплекса pMHC-I. Эти реакции проводят в микропланшетах, покрытых стрептавидином, для связывания биотинилированных тяжелых цепей MHC-I с поверхностью и для измерения радиоактивно меченой легкой цепи для мониторинга образования комплекса. Диссоциацию инициируют путем добавления более высоких концентраций немеченой легкой цепи и инкубации при 37°C. Стабильность определяют как время в часах, необходимое для диссоциации половины комплексов, при измерении путем сцинтилляционного подсчета.
[343] Чтобы оценить, можно ли антигены процессировать и презентировать в контексте более крупных полипептидов, пептиды, элюированные из молекул HLA, выделенных из клеток, экспрессирующих интересующие гены, анализировали с помощью тандемной масс-спектрометрии (MS/MS).
[344] Для анализа презентации неоантигенов RAS использовали клеточные линии, которые имеют естественные мутации RAS или были трансдуцированы лентивирусом для экспрессии мутантного гена RAS. Молекулы HLA либо выделяли на основе естественной экспрессии клеточных линий, либо клеточные линии трансдуцировали лентивирусом или временно трансфицировали для экспрессии представляющего интерес HLA. Клетки 293T трансдуцировали лентивирусным вектором, кодирующим различные области мутантного пептида RAS. культивировали более 50 миллионов клеток, экспрессирующих пептиды, кодируемые мутантным пептидом RAS, и пептиды элюировали из комплексов HLA-пептид с использованием кислотной промывки. Затем элюированные пептиды анализировали с помощью направленной MS/MS с параллельным мониторингом реакции (PRM). Для клеток 293T, трансдуцированных лентивирусом как c мутацией RASG12V, так и c геном HLA-A*03:01, с помощью масс-спектрометрии был обнаружен пептид с аминокислотной последовательностью vvvgaVgvgk (SEQ ID NO: 5). Спектральное сравнение с его соответствующим стабильным меченным тяжелым изотопом синтетическим пептидом (фиг. 4B) показало, что точность определения массы обнаруженного пептида составляет менее 5 частей на миллион (ч/млн). Спектры эндогенных пептидов показаны на верхних панелях, а соответствующие спектры, меченные тяжелыми изотопами, показаны на нижних панелях. Для клеток SW620, естественно экспрессирующих мутацию RASG12V и трансдуцированных лентивирусом геном HLA-A*03:01, с помощью масс-спектрометрии был обнаружен пептид с аминокислотной последовательностью vvvgaVgvgk(SEQ ID NO: 5). Спектральное сравнение с его соответствующим стабильным меченным тяжелым изотопом синтетическим пептидом показало, что точность определения массы обнаруженного пептида составляет менее 5 ч/млн (фиг. 4C). Спектры эндогенных пептидов показаны на верхних панелях, а соответствующие спектры, меченные тяжелыми изотопами, показаны на нижних панелях. Для клеток NCI-H441, естественно экспрессирующих как мутацию RASG12V, так и ген HLA-A*03:01, с помощью масс-спектрометрии был обнаружен пептид с аминокислотной последовательностью vvvgaVgvgk(SEQ ID NO: 5). Спектральное сравнение с его соответствующим стабильным меченным тяжелым изотопом синтетическим пептидом показало, что точность определения массы обнаруженного пептида составляет менее 5 ч/млн (фиг. 4D). Спектры эндогенных пептидов показаны на верхних панелях, а соответствующие спектры, меченные тяжелыми изотопами, показаны на нижних панелях. Аналогичная процедура была проведена для анализа пептидов, полученных в результате множественных мутаций RASG12 в HLA-A*03:01, HLA-A*11:01, HLA-A*30:01, HLA-A*68:01 и HLA-B*07:02 и в таблице 13 перечислены те пептиды, которые были обнаружены с помощью масс-спектрометрии.
[345] Для анализа презентации неоантигенов GATA3 клетки 293T трансдуцировали лентивирусным вектором, кодирующим различные области пептидов, кодируемых GATA3 neoORF. культивировали от 50 до 700 миллионов трансдуцированных клеток, экспрессирующих пептиды, кодируемые последовательностью GATA3 neoORF, и пептиды элюировали из комплексов HLA-пептид с использованием кислотной промывки. Затем элюированные пептиды анализировали с помощью направленной МС/МС с использованием PRM. Для подтверждения каждого обнаружения проводили спектральное сравнение пептидов, полученных из GATA3 neoORF, и соответствующих синтетических пептидов. Для клеток 293T, экспрессирующих белок HLA-A*02:01, с помощью масс-спектрометрии были обнаружены пептиды VLPEPHLAL(SEQ ID NO: 1084), SMLTGPPARV (SEQ ID NO: 6) и MLTGPPARV(SEQ ID NO: 1081) (таблица 14 и фиг.20). Спектральное сравнение с соответствующими синтетическими пептидами показало, что точность определения массы обнаруженного пептида (SMLTGPPARV(SEQ ID NO: 6)) составляет менее 5 ч/млн (фиг. 4E). Для клеток 293T, экспрессирующих белок HLA-A*03:01 или HLA-A*11:01, с помощью масс-спектрометрии был обнаружен пептид KIMFATLQR(SEQ ID NO: 1089) (фиг. 20). Для клеток 293T, экспрессирующих белок HLA-A*30:02, с помощью масс-спектрометрии были обнаружены пептиды IMKPKRDGY(SEQ ID NO: 139) и SIMKPKRDGY(SEQ ID NO: 1391) (таблица 14). Для клеток 293T, экспрессирующих белок HLA-B*07:02, с помощью масс-спектрометрии были обнаружены пептиды KPKRDGYMF(SEQ ID NO: 1093) и KPKRDGYMFL(SEQ ID NO: 1095) (таблица 14 и фиг.20). Для клеток 293T, экспрессирующих белок HLA-B*08 01, с помощью масс-спектрометрии был обнаружен пептид ESKIMFATL(SEQ ID NO: 1091) (таблица 14 и фиг.20). Для клеток 293T, экспрессирующих белок HLA-B*40:02, с помощью масс-спектрометрии были обнаружены пептиды AESKIMFATL(SEQ ID NO: 1392) и AESKIMFAT(SEQ ID NO: 1393) (таблица 14). Для клеток 293T, экспрессирующих белок HLA-C*03:03, с помощью масс-спектрометрии был обнаружен пептид FATLQRSSL(SEQ ID NO: 1078) (таблица 14).
Связывание и стабильность HLA I класса
[346] Подмножество пептидов, используемых для измерения аффинности, также использовали для измерения стабильности с использованием описанного анализа (n=275). Эти данные показаны в Таблице 3. Менее 50 нМ считалось в этой области сильным связующим, 50-150 нМ считалось промежуточным связующим, 150-500 нМ считалось слабым связующим, а более 500 нМ считалось очень слабым связующим. Связь между наблюдаемой стабильностью и наблюдаемой аффинностью была очевидна по снижению медианной стабильности в этих сгруппированных интервалах стабильности. Однако существует значительное перекрытие между позициями, и, что важно, во всех позициях есть эпитопы с наблюдаемой стабильностью в многочасовом диапазоне, включая очень слабые связующие.
[347] Анализы иммуногенности используют для проверки способности каждого тестируемого пептида размножать Т-клетки. Зрелые профессиональные APC готовят для этих анализов следующим образом. Моноциты обогащают из PBMC здоровых доноров-людей с использованием набора на основе шариков (Miltenyi). Обогащенные клетки высевают в GM-CSF и IL-4 для индукции незрелых DC. Через 5 дней незрелые DC инкубируют при 37°C с каждым пептидом в течение 1 часа перед добавлением коктейля созревания цитокинов (GM-CSF, IL-1β, IL-4, IL-6, TNFα, PGE1β). Клетки инкубируют при 37°C до созревания DC. В некоторых вариантах осуществления нужно, чтобы пептиды при введении пациенту вызывали иммунный ответ.
[348] В таблице 4А показаны пептидные последовательности, содержащие мутации RAS, соответствующие аллелю HLA, с которым он связывается, а также измеренные стабильность и аффинность.
Пример 4. Оценка цитотоксической способности антигенспецифических Т-клеток in vitro
[349] Цитотоксическая активность может быть измерена путем обнаружения расщепленной каспазы 3 в клетках-мишенях с помощью проточной цитометрии. Раковые клетки-мишени конструируют так, чтобы экспрессировать мутантный пептид и соответствующий аллель MHC-I. Ложно-трансдуцированные клетки-мишени (то есть не экспрессирующие мутантный пептид) используют в качестве отрицательного контроля. Клетки метят CFSE, чтобы отличить их от стимулированных PBMC, используемых в качестве эффекторных клеток. Перед сбором клетки-мишени и эффекторные клетки совместно культивируют в течение 6 часов. Для обнаружения расщепленной формы каспазы 3 в CFSE-положительных раковых клетках-мишенях проводят внутриклеточное окрашивание. Процентное значение специфического лизиса рассчитывают следующим образом: экспериментальное расщепление каспазы 3/спонтанное расщепление каспазы 3 (измеренное в отсутствие экспрессии мутантного пептида) x 100. На фиг. 23 показаны иллюстративные данные, показывающие, что Т-клетки, индуцированные против неоантигенов GATA3, могут убивать клетки-мишени, экспрессирующие GATA3 neoORF.
[350] В некоторых примерах цитотоксическую активность оценивают путем совместного культивирования индуцированных Т-клеток с популяцией антигенспецифических Т-клеток с клетками-мишенями, экспрессирующими соответствующий HLA, и путем определения относительного роста клеток-мишеней вместе с специфическим измерением апоптотического маркера аннексина V в раковых клетках-мишенях. Раковые клетки-мишени конструируют для экспрессии мутантного пептида, или пептид загружают экзогенно. Ложно-трансдуцированные клетки-мишени (то есть не экспрессирующие мутантный пептид), клетки-мишени, нагруженные пептидами дикого типа, или клетки-мишени без нагруженного пептида, используют в качестве отрицательного контроля. Клетки также трансдуцируют для стабильной экспрессии GFP, что позволяет отслеживать рост клеток-мишеней. Сигнал GFP или сигнал аннексина V измеряют с течением времени с помощью устройства IncuCyte S3. Сигнал аннексина V, исходящий от эффекторных клеток, отфильтровывают методом разделения по размеру. Рост и гибель клеток-мишеней выражают как площадь GFP и Аннексина-V (мм2) с течением времени, соответственно.
[351] На фиг. 7 показаны иллюстративные данные, демонстрирующие, что Т-клетки, стимулированные для распознавания неоантигена RASG12V на HLA-A11:01, специфически распознают и убивают клетки-мишени, нагруженные мутантным пептидом, но не пептидом дикого типа. На фиг. 8 показаны иллюстративные данные, демонстрирующие, что Т-клетки, стимулированные для распознавания неоантигена RASG12V на HLA-A11:01, убивают клетки-мишени, нагруженные наномолярными количествами пептида при соотношении E:T <0,2:1. На фиг. 9 показаны иллюстративные данные, демонстрирующие, что Т-клетки, стимулированные для распознавания неоантигена RASG12V на HLA-A11:01, убивают клетки SW620, которые в естесственных условиях имеют мутацию RASG12V и трансдуцированы HLA-A11:01. На фиг. 10 показаны иллюстративные данные, демонстрирующие, что Т-клетки, стимулированные для распознавания неоантигена RASG12V на HLA-A03:01, убивают клетки NCI-H441, которые в естесственных условиях имеют мутацию RASG12V и HLA-A03:01. На фиг. 22 и 23 показаны иллюстративные данные, демонстрирующие, что Т-клетки, стимулированные для распознавания неоантигена GATA3 на HLA-A02:01, убивают клетки 293T, которые в естесственных условиях имеют HLA-A02:01 и трансдуцированы GATA3 neoORF.
Пример 5. Прецизионный процесс NEO-STIM, применяемый к тканеспецифическим антигенам
[352] Антигены, которые специфически экспрессируются в неосновной ткани, могут стать мишенью, если в такой ткани возникает опухоль. Например, антигены, специфически экспрессируемые в тканях предстательной железы, могут стать мишенью в контексте метастатического рака предстательной железы, при котором была резецирована первичная опухоль, потому что единственными клетками, экспрессирующими эти антигены, являются метастатические раковые клетки. Таких неосновных тканей множество. Например, клетки предстательной железы оценивали с использованием двух методик для обнаружения потенциальных антигенов, специфичных для предстательной железы. В одном подходе ткани предстательной железы или клеточные линии рака предстательной железы оценивали с использованием HLA-MS, как показано в примере 3. Этот подход может привести к идентификации антигенов, проверенных на процессинг и презентацию. Иллюстративные данные этого подхода показаны на фиг. 25А. В другом подходе гены, о которых известно, что они специфически экспрессируются в клетках предстательной железы, могут быть оценены с помощью одной или нескольких программ для прогнозирования связывания и презентации MHC. Был создан алгоритм прогнозирования комплекса пептид-MHC, который использовали в этих исследованиях. Как и в примерах 2, 3 и 4, масс-спектрометрия, клеточные и иммунологические анализы дополнительно помогают подтвердить спрогнозированную пару пептид-HLA. Иллюстративные результаты этого анализа 4 генов, которые, как известно, специфически экспрессируются в простате (KLK2, KLK3, KLK4, TGM4), показаны в таблице ниже. Эти эпитопы были дополнительно подвергнуты исследованиям иммуногенности, как в примере 2. Было показано, что эпитопы с префиксом «*» индуцируют положительный ответ CD8+ Т-лимфоцитов у одного или обоих доноров (отмечены цифрами 1 или 2 в столбце 6 соответственно). И также показано на фиг. 25B.
[353] В дополнительном анализе T-клетки, специфические для пептидов, указанных в таблице, тестировали на способность убивать клетки-мишени, как описано в примере 4. Иллюстративные данные представлены на фиг. 25C, где KLK4-специфический эпитоп предстательной железы совместно культивировали с клетками 293T-GFP либо загруженными 2 мкМ пептида, либо не загруженными. Клетки-мишени, нагруженные пептидом, погибали в гораздо большей степени (правое изображение) по сравнению с контролем без пептида (левое изображение).
Пример 6. Обогащение Т-клеток, активированных антигеном-мишенью
[354] Т-клетки, чувствительные к опухолевому антигену, могут быть дополнительно обогащены. В этом примере исследуются несколько способов обогащения антиген-чувствительных Т-клеток и представлены результаты. После начальной стимуляции антиген-специфических Т-клеток (пример 2, этапы 1-5) можно использовать процедуру обогащения до дальнейшего размножения этих клеток. Например, стимулированные культуры и введение тех же пептидов, которые использовали для начальной стимуляции на 13 день, и клетки, активирующие 4-1BB, обогащают с помощью магнитно-вспомогательного разделения клеток (MACS; Miltenyi). Затем эти клетки могут быть дополнительно размножены, например, с использованием микрошариков против CD3 и против CD28 и низких доз IL-2. Как показано на фиг. 19A (средний ряд) и фиг. 19B (средний столбец), этот подход может обогатить несколько популяций антигенспецифических Т-клеток. В качестве другого примера, Т-клетки, окрашенные мультимерами, могут быть обогащены MACS на 14 день стимуляции и дополнительно размножены, например, с использованием микрошариков против CD3 и CD28 и низких доз IL-2. Как показано на фиг. 19A (нижний ряд) и на фиг. 19B (правый столбец), этот подход может обогатить несколько популяций антигенспецифических Т-клеток.
Пример 7. Анализ иммуногенности выбранных пептидов
[355] После созревания DC к зрелым дендритным клеткам с цитокинами пролиферации добавляют PBMC (либо объемные, либо обогащенные Т-клетками). Культуры контролируют на наличие пептид-специфичных Т-клеток с использованием комбинации функциональных анализов и/или окрашивания тетрамерами. Параллельные анализы иммуногенности с модифицированными и исходными пептидами позволили сравнить относительную эффективность, с которой пептиды размножали пептид-специфические Т-клетки. В некоторых вариантах реализации пептиды вызывают иммунный ответ в культуре Т-клеток, включая обнаружение экспрессии лиганда FAS, гранзима, перфоринов, IFN, TNF или их комбинации в культуре Т-клеток.
[356] Иммуногенность можно измерить с помощью тетрамерного анализа. Тетрамеры MHC приобретают или производят на месте и используют для измерения экспансии пептид-специфичных Т-клеток в анализах иммуногенности. Для оценки тетрамер добавляют к 1×105 клеток в PBS, содержащем 1% FCS и 0,1% азида натрия (буфер FACS) в соответствии с инструкциями производителя. Клетки инкубируют в темноте 20 минут при комнатной температуре. Затем добавляют антитела, специфичные для маркеров Т-клеток, таких как CD8, до конечной концентрации, предложенной производителем, и клетки инкубируют в темноте при 4 градусах Цельсия в течение 20 минут. Клетки промывают холодным буфером FACS и ресуспендируют в буфере, содержащем 1% формальдегида. Клетки помещают на прибор FACS Calibur (Becton Dickinson) и анализируют с использованием программного обеспечения Cellquest (Becton Dickinson). Для анализа тетрамер-положительных клеток ворота лимфоцитов берут из графиков прямого и бокового рассеяния. Данные представлены как процент клеток, которые представляли собой CD8+/тетрамер+.
[357] Иммуногенность можно измерить путем окрашивания внутриклеточных цитокинов. В отсутствие хорошо зарекомендовавшего себя окрашивания тетрамеров для идентификации популяций антигенспецифических Т-клеток, антиген-специфичность можно оценить с помощью оценки выработки цитокинов с использованием хорошо зарекомендовавших себя анализов проточной цитометрии. Вкратце, Т-клетки стимулируют интересующим пептидом и сравнивают с контролем. После стимуляции выработку цитокинов CD4+ Т-клетками (например, IFNγ и TNFα) оценивают по внутриклеточному окрашиванию. Эти цитокины, особенно IFNγ, используют для идентификации стимулированных клеток.
[358] В некоторых вариантах осуществления иммуногенность измеряют путем измерения белка или пептида, экспрессируемого Т-клеткой, с использованием анализа ELISpot. Пептид-специфические Т-клетки функционально подсчитывают с помощью анализа ELISpot (BD Biosciences), который измеряет высвобождение IFNγ из Т-клеток на основе отдельных клеток. Клетки-мишени (C1R, трансфицированные T2 или HLA-A0201) обрабатывали 10 мкМ пептида в течение одного часа при 37°C и трижды промывали. 1×105 мишеней, обработанных пептидом, совместно культивируют в лунках планшета ELISPOT с различными концентрациями Т-клеток (от 5×102 до 2×103), взятых из культуры иммуногенности. Планшеты разрабатывают в соответствии с протоколом производителя и анализируют на считывателе ELISPOT (Cellular Technology Ltd.) с помощью сопровождающего программного обеспечения. Пятна, соответствующие количеству Т-клеток, продуцирующих IFN-гамма, представлены как абсолютное количество пятен на количество высеянных Т-клеток. Т-клетки, размноженные на модифицированных пептидах, тестируют не только на их способность распознавать мишени, обработанные модифицированным пептидом, но также на их способность распознавать мишени, обработанные исходным пептидом.
[359] CD107a и b экспрессируют на клеточной поверхности CD8+ Т-клеток после активации родственным пептидом. Литические гранулы Т-клеток имеют липидный бислой, который содержит связанные с лизосомами мембранные гликопротеины («LAMP»), которые содержат молекулы CD107a и b. Когда цитотоксические Т-клетки активируются посредством Т-клеточного рецептора, мембраны этих литических гранул мобилизуются и сливаются с плазматической мембраной Т-клетки. Содержимое гранул высвобождается, и это приводит к гибели клетки-мишени. Когда мембрана гранул сливается с плазматической мембраной, C107a и b открываются на поверхности клетки и, следовательно, являются маркерами дегрануляции. Поскольку дегрануляцию, измеренную с помощью окрашивания CD107a и b, описывают на основе отдельных клеток, используют анализ для функционального подсчета пептид-специфичных Т-клеток. Для проведения анализа пептид добавляют к трансфицированным HLA-A0201 клеткам C1R до конечной концентрации 20 мкМ, клетки инкубируют в течение 1 часа при 37°C и трижды промывают. Аликвоты 1×105 клеток C1R, обработанных пептидом, помещали в пробирки и добавляли антитела, специфичные для CD107a и b, до конечной концентрации, предложенной производителем (Becton Dickinson). Антитела добавляют до добавления Т-клеток, чтобы «захватить» молекулы CD107, поскольку они временно появляются на поверхности в ходе анализа. Затем добавляли 1×105 Т-клеток из культуры иммуногенности, и образцы инкубировали в течение 4 часов при 37°C. Т-клетки дополнительно окрашивали на дополнительные молекулы клеточной поверхности, такие как CD8, и помещали на прибор FACS Calibur (Becton Dickinson). Данные анализируют с помощью прилагаемого программного обеспечения Cellquest, и результаты представляют, как процентное содержание CD8+ CD107 a и b+ клеток.
[360] Цитотоксическую активность измеряют с использованием анализа высвобождения хрома. Клетки-мишени T2 метят в течение 1 часа при 37°C с помощью Na51Cr, и промытые 5×103 клеток-мишеней T2 затем добавляют к разному количеству Т-клеток из культуры иммуногенности. Высвобождение хрома измеряют в супернатанте, собранном после 4 часов инкубации при 37°C. Процент специфического лизиса рассчитывают как:
Экспериментальное высвобождение - спонтанное высвобождение/Общее высвобождение - спонтанное высвобождение × 100
[361] Анализы иммуногенности проводили для оценки того, может ли каждый пептид вызывать Т-клеточный ответ путем размножения антиген-специфических клеток. Хотя существующие методы несовершенны, и поэтому отрицательные результаты не означают, что пептид не способен вызывать ответ, положительный результат демонстрирует, что пептид может вызывать ответ Т-клеток. Несколько пептидов из таблицы 3 были протестированы на их способность вызывать ответы CD8+ Т-клеток с считыванием мультимеров, как описано. Каждый положительный результат был измерен с помощью второго мультимерного препарата, чтобы избежать каких-либо погрешностей препаратов. В иллюстративном анализе Т-клетки HLA-A02:01+ совместно культивировали с дендритными клетками, полученными из моноцитов, нагруженными неоэпитопом слияния TMPRSS2::ERG (ALNSEALSV; HLA-A02:01) в течение 10 дней. CD8+ Т-клетки анализировали на антиген-специфичность для неоэпитопа слияния TMPRSS2::ERG с использованием мультимеров (исходные: BV421 и PE; проверка: APC и BUV396).
[362] В то время как антиген-специфические ответы CD8+ Т-клеток легко оценивать с использованием хорошо зарекомендовавшей себя мультимерной технологии HLA класса I, ответы CD4+ Т-клеток требуют отдельного анализа для оценки, поскольку технология мультимеров HLA класса II еще недостаточно отработана. Чтобы оценить ответы CD4+ Т-клеток, Т-клетки повторно стимулировали представляющим интерес пептидом и сравнивали с контролем. В случае полностью новой последовательности (например, возникающей в результате сдвига рамки считывания или слияния) в контроле не было пептида. В случае точечной мутации контролем был пептид WT. После стимуляции выработку цитокинов (например, IFNγ и TNFα) CD4+ Т-клетками оценивали по внутриклеточному окрашиванию. Эти цитокины, особенно IFNγ, используют для идентификации стимулированных клеток. Антиген-специфические ответы CD4+ Т-клеток показали повышенную выработку цитокинов по сравнению с контролем.
Пример 8. Размножение и получение клеток
[363] Для получения APC использовали следующий метод (а) получение аутологичных иммунных клеток из периферической крови пациента; обогащение моноцитов и дендритных клеток в культуре; загрузка пептидов и зрелых DC.
Индукция Т-клеток (Протокол 1)
[364] Первая индукция: (а) получение аутологичных Т-клеток из пакета для афереза; (b) истощение CD25+ клеток и CD14+ клеток, альтернативно, истощение только CD25+ клеток; (c) промывание нагруженных пептидом и зрелых DC клеток, ресуспендирование в среде для культивирования Т-клеток; (d) инкубация Т-клеток со зрелыми DC.
[365] Вторая индукция: (а) промывание Т-клеток и ресуспендирование в среде Т-клеток и, необязательно, оценка небольшой аликвоты из культуры клеток для определения роста клеток, сравнительного роста и индукции подтипов Т-клеток, и антигенной специфичности, и мониторинг потери клеточной популяции; (b) инкубация Т-клеток со зрелыми DC.
[366] Третья индукция: (а) промывание Т-клеток и ресуспендирование в среде Т-клеток и, необязательно, оценка небольшой аликвоты из культуры клеток для определения роста клеток, сравнительного роста и индукции подтипов Т-клеток, и специфичности антигена, и мониторинг потери. клеточной популяции; (b) инкубация Т-клеток со зрелыми DC.
[367] Для сбора активированных пептидом Т-клеток и криоконсервации Т-клеток был использован следующий метод (а) промывание и ресуспендирование конечного состава, содержащего активированные Т-клетки с оптимальным количеством клеток и соотношением типов клеток, которые составляют желаемые характеристики лекарственного вещества (DS). Тестирование критериев высвобождения включает, среди прочего, стерильность, эндотоксин, фенотип клеток, количество TNC, жизнеспособность, концентрацию клеток, эффективность; (b) заполнение лекарственного вещества в подходящие закрытые инфузионные пакеты; (c) сохранение до использования.
Пример 9. Методы получения функциональной характеристики CD4+ и CD8+ неоантиген-специфических Т-клеток
[368] Неоантигены, которые возникают в раковых клетках в результате соматических мутаций, которые изменяют последовательности генов, кодирующих белок, становятся привлекательной мишенью для иммунотерапии. Они уникально экспрессируются на опухолевых клетках, а не на здоровых тканях, и могут распознаваться иммунной системой как чужеродные антигены, повышая иммуногенность. Способы получения Т-клеток были разработаны для увеличения ответов Т-клеток памяти и CD4+ и CD8+ Т-клеток de novo на специфичные для пациента неоантигены с помощью нескольких циклов стимуляции Т-клеток ex-vivo, генерируя выработку реагирующих на неоантиген Т-клеток для использования в адоптивной клеточной терапии. Подробная характеристика выработки стимулированных Т-клеток может быть использована для тестирования многих потенциальных переменных, которые используются в этих способах.
[369] Чтобы исследовать функциональность и/или специфичность Т-клеток, был разработан анализ для одновременного обнаружения антиген-специфических ответов Т-клеток и характеристики их величины и функции. В этом анализе использованы следующие этапы. Первые совместные культуры Т-клеток и АРС использовали для выявления реактивности антиген-специфических Т-клеток. Необязательно применяют мультиплексирование образцов с использованием штрих-кодирования флуоресцентных клеток. Чтобы идентифицировать антиген-специфические CD8+ Т-клетки и изучить функциональность Т-клеток, одновременно с использованием анализа FACS зондировали окрашивание мультимеров пептид-MHC и многопараметрическое окрашивание маркером клеток внутриклеточной и/или клеточной поверхности. Результаты этого оптимизированного анализа продемонстрировали его применение для изучения ответов Т-клеток, индуцированных у здорового донора. У здорового донора были идентифицированы неоантиген-специфические Т-клеточные ответы, индуцированные в отношении пептидов. Также сравнивали величину, специфичность и функциональность индуцированных Т-клеточных ответов. Вкратце, разные образцы Т-клеток были закодированы разными флуоресцентными красителями в разных концентрациях (см., например, пример 19). Каждый образец получил разную концентрацию флуоресцентного красителя или комбинацию нескольких красителей в разных концентрациях. Образцы ресуспендировали в фосфатно-солевом буфере (PBS), а затем добавляли флуорофоры, растворенные в DMSO (обычно при разведении 1:50) до максимальной конечной концентрации 5 мкМ. После мечения в течение 5 мин при 37°C избыток флуоресцентного красителя гасили добавлением белковой среды (например, среды RPMI, содержащей 10% объединенной сыворотки человека типа AB). Культуры Т-клеток с уникальным штрих-кодом заражали аутологичными APC, обработанными антигенными пептидами, как описано выше.
[370] Дифференциально меченые образцы объединяли в одну пробирку или лунку для FACS и снова осаждали, если полученный объем превышал 100 мкл. Объединенный образец со штрих-кодом (обычно 100 мкл) окрашивали антителами к поверхностным маркерам, включая мультимеры пептид-MHC, конъюгированный с флуорохромом. После фиксации и пермеабилизации образец дополнительно внутриклеточно окрашивали антителами, нацеленными на TNF-α и IFN-γ.
[371] Профиль клеточного маркера и окрашивание тетрамера MHC объединенного образца Т-клеток со штрих-кодом затем анализировали одновременно с помощью проточной цитометрии на проточном цитометре. В отличие от других методов, которые анализируют профили клеточных маркеров и окрашивание тетрамерами MHC образца Т-клеток по отдельности, одновременный анализ профиля клеточных маркеров и окрашивание тетрамера MHC образца Т-клеток, описанный в этом примере, дает информацию о процентном содержании Т-клеток, которые являются как антигенспецифическими, так и имеют повышенное окрашивание клеточных маркеров. Другие методы, которые анализируют профили клеточных маркеров и окрашивание образца Т-клеток тетрамером MHC, отдельно определяют процент Т-клеток образца, которые являются антигенспецифическими, и отдельно определяют процент Т-клеток, которые имеют повышенное окрашивание маркеров клеток, допуская только корреляцию. Этих частот.
[372] Одновременный анализ профиля клеточных маркеров и окрашивания тетрамером MHC образца Т-клеток, описанный в этом примере, не основан на корреляции частоты антигенспецифических Т-клеток и частоты Т-клеток, которые имеют повышенное окрашивание клеточных маркеров; вместо этого он обеспечивает частоту Т-клеток, которые являются как антигенспецифическими, так и имеют повышенное окрашивание клеточных маркеров. Одновременный анализ профиля клеточных маркеров и окрашивания тетрамера MHC образца Т-клеток, описанный в этом примере, позволяет определять на уровне отдельных клеток те клетки, которые являются как антигенспецифическими, так и имеют повышенное окрашивание клеточных маркеров.
[373] Чтобы оценить успех данного способа индукции, использовали анализ вторичного ответа с последующим мультиплексным панельным анализом многопараметрической проточной цитометрии. Образец, взятый из индукционной культуры, был помечен уникальным двухцветным флуоресцентным клеточным штрих-кодом. Меченые клетки инкубировали на нагруженных антигеном DC или ненагруженных DC в течение ночи для стимуляции функционального ответа в антиген-специфических клетках. На следующий день уникально меченые клетки объединяли перед окрашиванием антителом и мультимером в соответствии с таблицей 9 ниже.
[374] Неоантигены, специфичные для пациента, прогнозировали с использованием механизма биоинформатики. Синтетические длинные пептиды, покрывающие прогнозируемые неоантигены, использовали в качестве иммуногенов в протоколе стимуляции для оценки иммуногенной способности. Протокол стимуляции включает загрузку этих неоантиген-кодирующих пептидов в АРС, полученные от пациента, которые затем совместно культивируют с Т-клетками, полученными от пациента, для примирования неоантиген-специфических Т-клеток.
[375] Множественные циклы стимуляции включены в протокол стимуляции для примирования, активации и увеличения ответов Т-клеток памяти и de novo. Специфичность, фенотип и функциональность этих неоантиген-специфичных Т-клеток анализировали, характеризуя эти ответы с помощью следующих анализов. Комбинаторный анализ кодирования с использованием мультимеров pMHC использовали для обнаружения множественных неоантиген-специфичных ответов CD8+ Т-клеток. Анализ вторичного ответа с использованием мультиплексной многопараметрической проточной цитометрии использовался для идентификации и подтверждения ответов CD4+ Т-клеток. Функциональность CD8+ и CD4+ Т-клеточных ответов оценивали путем измерения выработки прововоспалительных цитокинов, включая IFN-γ и TNFα, и активации CD107a в качестве маркера дегрануляции. Анализ цитотоксичности с использованием линий опухолей, экспрессирующих неоантиген, был использован для понимания способности ответов CD8+ Т-клеток распознавать и уничтожать клетки-мишени в ответ на процессируемый и презентируемый в естественных условиях антиген. Цитотоксичность измеряли по повышающей регуляции CD107a на клеточной поверхности Т-клеток и повышающей регуляции активной каспазы 3 в опухолевых клетках, экспрессирующих неоантиген. Протокол стимуляции был успешным в увеличении ранее существовавших CD8+ Т-клеточных ответов, а также в индукции de novo CD8+ Т-клеточных ответов (Таблица 10).
ARAP1Y>H
PKDREJG>R
Ankyrin Repeat And PH Domain
Polycystin Family Receptor For Egg Jelly
CREB Binding Protein
Two Pore Segment Channel 1
ACTN4K>N CSNK1A1S>L DHX40neoORF GLI3P>L,
QARSR>W FAM178BP>L
RPS26P>
Actinin Alpha 4
Casein Kinase 1 Alpha 1
DEAH-Box Helicase 40
GLI Family Zinc Finger 3
Glutaminyl-TRNA Synthetase
Family With Sequence Similarity 178 Member В
Ribosomal Protein S26
[376] При использовании PBMC пациента с меланомой в клиническом исследовании, проведенном группой заявителя, наблюдали увеличение ранее существовавшего CD8+ Т-клеточного ответа с 4,5% CD8+ Т-клеток до 72,1% CD8+ Т-клеток (SRSF1E>K). Более того, протокол стимуляции был эффективен в индукции двух предполагаемых de novo CD8+ Т-клеточных ответов на специфические для пациента неоантигены (иллюстративные de novo CD8+ Т-клеточные ответы: ARAP1Y>H: 6,5% CD8+ Т-клеток и PKDREJG>R: 13,4% CD8+ Т-клетки; до процесса стимуляции клетки не обнаруживались). Протокол стимуляции успешно индуцировал семь de novo CD8+ Т-клеточных ответов как на ранее описанные, так и на новые модельные неоантигены с использованием PBMC от другого пациента с меланомой, NV6, до различной степени (ACTN4K>N CSNK1A1S>L DHX40neoORF 7, GLI3P>L, QARSR>W , FAM178BP>L и RPS26P>L, диапазон: от 0,2% CD8+ Т-клеток до 52% CD8+ Т-клеток). Кроме того, был увеличен ответ CD8+ Т-клеток памяти на специфический для пациента неоантиген (AASDHneoORF, до 13% CD8+ Т-клеток после стимуляции).
[377] Индуцированные CD8+ Т-клетки пациента были охарактеризованы более подробно. При повторном заражении DC, нагруженными мутантным пептидом, неоантиген-специфические CD8+ Т-клетки проявляли одну, две и/или все три функции (16,9% и 65,5% функциональных CD8+ pMHC+Т-клеток для SRSF1E>K и ARAP1Y>H, соответственно. Подвергнутые повторному воздействию различных концентраций неоантигенных пептидов, индуцированные CD8+ Т-клетки в значительной степени отвечали на мутантный неоантигенный пептид, но не на пептид дикого типа. У указанного пациента CD4+ Т-клеточные ответы были идентифицированы с использованием анализа вторичного ответа с мутантными неоантигенами, нагруженными DC. Идентифицировали три CD4+ Т-клеточных ответа (MKRN1S>L, CREBBPS>L и TPCN1K>E) на основании реактивности к DC, нагруженным мутантным неоантигенным пептидом. Эти CD4+ Т-клеточные ответы также показали полифункциональный профиль при повторном заражении мутантным неоантигенным пептидом. 31.3%, 34,5% и 41,9% CD4+ Т-клеток проявляли одну, две и/или три функции; ответы MKRN1S>L, CREBBPS>L и TPCN1K>E соответственно.
[378] Также оценивали цитотоксическую способность индуцированных ответов CD8+ указанного пациента. Оба ответа SRSF1E>K и ARAP1Y>H показали значительную повышающую регуляцию CD107a на CD8+ Т-клетках и активной каспазы 3 на опухолевых клетках, трансдуцированных мутантной конструкцией после совместного культивирования.
[379] Используя протокол стимуляции, на материале пациента было подтверждено, что прогнозируемые специфичные для пациента неоантигены, а также модельные неоантигены являются иммуногенными путем индукции множественных неоантиген-специфических CD8+ и CD4+ Т-клеточных ответов. Способность индуцировать полифункциональные и специфичные для мутантов CD8+ и CD4+ Т-клеточные ответы доказывает возможность прогнозировать высококачественные неоантигены и вызывать сильные Т-клеточные ответы. Присутствие множества обогащенных популяций неоантиген-специфических Т-клеток (памяти и de novo) в конце процесса стимуляции демонстрирует способность вызывать новые Т-клеточные ответы и создавать эффективные иммунотерапевтические методы лечения рака для лечения больных раком.
[380] Иллюстративные материалы для культуры Т-клеток представлены ниже:
Материалы: среда AIM V (Invitrogen), FLT3L человека; доклинический CellGenix # 1415-050 маточный раствор 50 нг/мкл TNFα; доклинический CellGenix # 1406-050 маточный раствор 10 нг/мкл; IL-1β, доклинический CellGenix № 1411-050 маточный раствор 10 нг/мкл; PGE1 или Алпростадил - Cayman из Чешской республики маточный раствор 0,5 мкг/мкл; Среда R10 - RPMI 1640 глутамакс+10% сыворотка человека+1% PenStrep; среда 20/80-18% AIM V+72% RPMI 1640 глутамакс+10% сыворотка человека+1% PenStrep; IL7 маточный раствор 5 нг/мкл; IL15 Stock 5 нг/мкл; среда DC (Cellgenix); Микрошарики CD14, человек, Miltenyi # 130-050-201, цитокины и/или факторы роста, Т-клеточная среда (AIM V+RPMI 1640 глутамакс+сыворотка+PenStrep), маточные растворы пептидов - 1 мМ на пептид (HIV A02-5-10 пептиды, HIV B07-5-10 пептидов, DOM - 4-8 пептидов, PIN - 6-12 пептидов).
--->
Список последовательностей
<110> NEON THERAPEUTICS, INC.
<120> КОМПОЗИЦИИ И СПОСОБЫ ПОЛУЧЕНИЯ КОМПОЗИЦИЙ T-КЛЕТОК И ИХ ПРИМЕНЕНИЕ
<130> 50401-744.601
<140> PCT/US2020/025796
<141> 2020-03-30
<150> 62/827,018
<151> 2019-03-30
<160> 1428
<170> PatentIn version 3.5
<210> 1
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1
Val Val Gly Ala Arg Gly Val Gly Lys
1 5
<210> 2
<211> 10
<212> PRT
<213> Homo sapiens
<400> 2
Glu Tyr Lys Leu Val Val Val Gly Ala Arg
1 5 10
<210> 3
<211> 10
<212> PRT
<213> Homo sapiens
<400> 3
Val Val Val Gly Ala Arg Gly Val Gly Lys
1 5 10
<210> 4
<211> 9
<212> PRT
<213> Homo sapiens
<400> 4
Ala Arg Gly Val Gly Lys Ser Ala Leu
1 5
<210> 5
<211> 10
<212> PRT
<213> Homo sapiens
<400> 5
Val Val Val Gly Ala Val Gly Val Gly Lys
1 5 10
<210> 6
<211> 10
<212> PRT
<213> Homo sapiens
<400> 6
Ser Met Leu Thr Gly Pro Pro Ala Arg Val
1 5 10
<210> 7
<211> 10
<212> PRT
<213> Homo sapiens
<400> 7
Leu Leu Ala Asn Gly Arg Met Pro Thr Val
1 5 10
<210> 8
<211> 62
<212> PRT
<213> Homo sapiens
<400> 8
Met Thr Glu Tyr Lys Leu Val Val Val Gly Ala Cys Gly Val Gly Lys
1 5 10 15
Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn His Phe Val Asp Glu Tyr
20 25 30
Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys Gln Val Val Ile Asp Gly
35 40 45
Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr Ala Gly Gln Glu
50 55 60
<210> 9
<211> 62
<212> PRT
<213> Homo sapiens
<400> 9
Met Thr Glu Tyr Lys Leu Val Val Val Gly Ala Asp Gly Val Gly Lys
1 5 10 15
Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn His Phe Val Asp Glu Tyr
20 25 30
Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys Gln Val Val Ile Asp Gly
35 40 45
Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr Ala Gly Gln Glu
50 55 60
<210> 10
<211> 62
<212> PRT
<213> Homo sapiens
<400> 10
Met Thr Glu Tyr Lys Leu Val Val Val Gly Ala Val Gly Val Gly Lys
1 5 10 15
Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn His Phe Val Asp Glu Tyr
20 25 30
Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys Gln Val Val Ile Asp Gly
35 40 45
Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr Ala Gly Gln Glu
50 55 60
<210> 11
<211> 101
<212> PRT
<213> Homo sapiens
<400> 11
Ala Gly Gly Val Gly Lys Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn
1 5 10 15
His Phe Val Asp Glu Tyr Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys
20 25 30
Gln Val Val Ile Asp Gly Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr
35 40 45
Ala Gly His Glu Glu Tyr Ser Ala Met Arg Asp Gln Tyr Met Arg Thr
50 55 60
Gly Glu Gly Phe Leu Cys Val Phe Ala Ile Asn Asn Thr Lys Ser Phe
65 70 75 80
Glu Asp Ile His His Tyr Arg Glu Gln Ile Lys Arg Val Lys Asp Ser
85 90 95
Glu Asp Val Pro Met
100
<210> 12
<211> 101
<212> PRT
<213> Homo sapiens
<400> 12
Ala Gly Gly Val Gly Lys Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn
1 5 10 15
His Phe Val Asp Glu Tyr Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys
20 25 30
Gln Val Val Ile Asp Gly Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr
35 40 45
Ala Gly Leu Glu Glu Tyr Ser Ala Met Arg Asp Gln Tyr Met Arg Thr
50 55 60
Gly Glu Gly Phe Leu Cys Val Phe Ala Ile Asn Asn Thr Lys Ser Phe
65 70 75 80
Glu Asp Ile His His Tyr Arg Glu Gln Ile Lys Arg Val Lys Asp Ser
85 90 95
Glu Asp Val Pro Met
100
<210> 13
<211> 101
<212> PRT
<213> Homo sapiens
<400> 13
Ala Gly Gly Val Gly Lys Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn
1 5 10 15
His Phe Val Asp Glu Tyr Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys
20 25 30
Gln Val Val Ile Asp Gly Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr
35 40 45
Ala Gly Lys Glu Glu Tyr Ser Ala Met Arg Asp Gln Tyr Met Arg Thr
50 55 60
Gly Glu Gly Phe Leu Cys Val Phe Ala Ile Asn Asn Ser Lys Ser Phe
65 70 75 80
Ala Asp Ile Asn Leu Tyr Arg Glu Gln Ile Lys Arg Val Lys Asp Ser
85 90 95
Asp Asp Val Pro Met
100
<210> 14
<211> 101
<212> PRT
<213> Homo sapiens
<400> 14
Ala Gly Gly Val Gly Lys Ser Ala Leu Thr Ile Gln Leu Ile Gln Asn
1 5 10 15
His Phe Val Asp Glu Tyr Asp Pro Thr Ile Glu Asp Ser Tyr Arg Lys
20 25 30
Gln Val Val Ile Asp Gly Glu Thr Cys Leu Leu Asp Ile Leu Asp Thr
35 40 45
Ala Gly Arg Glu Glu Tyr Ser Ala Met Arg Asp Gln Tyr Met Arg Thr
50 55 60
Gly Glu Gly Phe Leu Cys Val Phe Ala Ile Asn Asn Ser Lys Ser Phe
65 70 75 80
Ala Asp Ile Asn Leu Tyr Arg Glu Gln Ile Lys Arg Val Lys Asp Ser
85 90 95
Asp Asp Val Pro Met
100
<210> 15
<211> 101
<212> PRT
<213> Homo sapiens
<400> 15
Met Ile Lys Glu Gly Ser Met Ser Glu Asp Glu Phe Ile Glu Glu Ala
1 5 10 15
Lys Val Met Met Asn Leu Ser His Glu Lys Leu Val Gln Leu Tyr Gly
20 25 30
Val Cys Thr Lys Gln Arg Pro Ile Phe Ile Ile Thr Glu Tyr Met Ala
35 40 45
Asn Gly Ser Leu Leu Asn Tyr Leu Arg Glu Met Arg His Arg Phe Gln
50 55 60
Thr Gln Gln Leu Leu Glu Met Cys Lys Asp Val Cys Glu Ala Met Glu
65 70 75 80
Tyr Leu Glu Ser Lys Gln Phe Leu His Arg Asp Leu Ala Ala Arg Asn
85 90 95
Cys Leu Val Asn Asp
100
<210> 16
<211> 101
<212> PRT
<213> Homo sapiens
<400> 16
Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser
1 5 10 15
Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn
20 25 30
Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys
35 40 45
Ile Ile Arg Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val
50 55 60
Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg
65 70 75 80
Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp
85 90 95
Lys Cys Asn Leu Leu
100
<210> 17
<211> 101
<212> PRT
<213> Homo sapiens
<400> 17
Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser Pro Lys Ala
1 5 10 15
Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser Val Asp Asn
20 25 30
Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser Thr Val Gln
35 40 45
Leu Ile Met Gln Leu Met Pro Phe Gly Cys Leu Leu Asp Tyr Val Arg
50 55 60
Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn Trp Cys Val
65 70 75 80
Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg Leu Val His
85 90 95
Arg Asp Leu Ala Ala
100
<210> 18
<211> 101
<212> PRT
<213> Homo sapiens
<400> 18
Val Ala Asp Gly Leu Ile Thr Thr Leu His Tyr Pro Ala Pro Lys Arg
1 5 10 15
Asn Lys Pro Thr Val Tyr Gly Val Ser Pro Asn Tyr Asp Lys Trp Glu
20 25 30
Met Glu Arg Thr Asp Ile Thr Met Lys His Lys Leu Gly Gly Gly Gln
35 40 45
Tyr Gly Lys Val Tyr Glu Gly Val Trp Lys Lys Tyr Ser Leu Thr Val
50 55 60
Ala Val Lys Thr Leu Lys Glu Asp Thr Met Glu Val Glu Glu Phe Leu
65 70 75 80
Lys Glu Ala Ala Val Met Lys Glu Ile Lys His Pro Asn Leu Val Gln
85 90 95
Leu Leu Gly Val Cys
100
<210> 19
<211> 101
<212> PRT
<213> Homo sapiens
<400> 19
Val Ala Asp Gly Leu Ile Thr Thr Leu His Tyr Pro Ala Pro Lys Arg
1 5 10 15
Asn Lys Pro Thr Val Tyr Gly Val Ser Pro Asn Tyr Asp Lys Trp Glu
20 25 30
Met Glu Arg Thr Asp Ile Thr Met Lys His Lys Leu Gly Gly Gly Gln
35 40 45
Tyr Gly Val Val Tyr Glu Gly Val Trp Lys Lys Tyr Ser Leu Thr Val
50 55 60
Ala Val Lys Thr Leu Lys Glu Asp Thr Met Glu Val Glu Glu Phe Leu
65 70 75 80
Lys Glu Ala Ala Val Met Lys Glu Ile Lys His Pro Asn Leu Val Gln
85 90 95
Leu Leu Gly Val Cys
100
<210> 20
<211> 101
<212> PRT
<213> Homo sapiens
<400> 20
Leu Leu Gly Val Cys Thr Arg Glu Pro Pro Phe Tyr Ile Ile Thr Glu
1 5 10 15
Phe Met Thr Tyr Gly Asn Leu Leu Asp Tyr Leu Arg Glu Cys Asn Arg
20 25 30
Gln Glu Val Asn Ala Val Val Leu Leu Tyr Met Ala Thr Gln Ile Ser
35 40 45
Ser Ala Thr Glu Tyr Leu Glu Lys Lys Asn Phe Ile His Arg Asp Leu
50 55 60
Ala Ala Arg Asn Cys Leu Val Gly Glu Asn His Leu Val Lys Val Ala
65 70 75 80
Asp Phe Gly Leu Ser Arg Leu Met Thr Gly Asp Thr Tyr Thr Ala His
85 90 95
Ala Gly Ala Lys Phe
100
<210> 21
<211> 101
<212> PRT
<213> Homo sapiens
<400> 21
Ser Leu Thr Val Ala Val Lys Thr Leu Lys Glu Asp Thr Met Glu Val
1 5 10 15
Glu Glu Phe Leu Lys Glu Ala Ala Val Met Lys Glu Ile Lys His Pro
20 25 30
Asn Leu Val Gln Leu Leu Gly Val Cys Thr Arg Glu Pro Pro Phe Tyr
35 40 45
Ile Ile Ile Glu Phe Met Thr Tyr Gly Asn Leu Leu Asp Tyr Leu Arg
50 55 60
Glu Cys Asn Arg Gln Glu Val Asn Ala Val Val Leu Leu Tyr Met Ala
65 70 75 80
Thr Gln Ile Ser Ser Ala Met Glu Tyr Leu Glu Lys Lys Asn Phe Ile
85 90 95
His Arg Asp Leu Ala
100
<210> 22
<211> 101
<212> PRT
<213> Homo sapiens
<400> 22
Ser Thr Val Ala Asp Gly Leu Ile Thr Thr Leu His Tyr Pro Ala Pro
1 5 10 15
Lys Arg Asn Lys Pro Thr Val Tyr Gly Val Ser Pro Asn Tyr Asp Lys
20 25 30
Trp Glu Met Glu Arg Thr Asp Ile Thr Met Lys His Lys Leu Gly Gly
35 40 45
Gly Gln His Gly Glu Val Tyr Glu Gly Val Trp Lys Lys Tyr Ser Leu
50 55 60
Thr Val Ala Val Lys Thr Leu Lys Glu Asp Thr Met Glu Val Glu Glu
65 70 75 80
Phe Leu Lys Glu Ala Ala Val Met Lys Glu Ile Lys His Pro Asn Leu
85 90 95
Val Gln Leu Leu Gly
100
<210> 23
<211> 101
<212> PRT
<213> Homo sapiens
<400> 23
Ser Ser Leu Ala Met Leu Asp Leu Leu His Val Ala Arg Asp Ile Ala
1 5 10 15
Cys Gly Cys Gln Tyr Leu Glu Glu Asn His Phe Ile His Arg Asp Ile
20 25 30
Ala Ala Arg Asn Cys Leu Leu Thr Cys Pro Gly Pro Gly Arg Val Ala
35 40 45
Lys Ile Ala Asp Phe Gly Met Ala Arg Asp Ile Tyr Arg Ala Ser Tyr
50 55 60
Tyr Arg Lys Gly Gly Cys Ala Met Leu Pro Val Lys Trp Met Pro Pro
65 70 75 80
Glu Ala Phe Met Glu Gly Ile Phe Thr Ser Lys Thr Asp Thr Trp Ser
85 90 95
Phe Gly Val Leu Leu
100
<210> 24
<211> 101
<212> PRT
<213> Homo sapiens
<400> 24
Gln Val Ala Val Lys Thr Leu Pro Glu Val Cys Ser Glu Gln Asp Glu
1 5 10 15
Leu Asp Phe Leu Met Glu Ala Leu Ile Ile Ser Lys Phe Asn His Gln
20 25 30
Asn Ile Val Arg Cys Ile Gly Val Ser Leu Gln Ser Leu Pro Arg Phe
35 40 45
Ile Leu Met Glu Leu Met Ala Gly Gly Asp Leu Lys Ser Phe Leu Arg
50 55 60
Glu Thr Arg Pro Arg Pro Ser Gln Pro Ser Ser Leu Ala Met Leu Asp
65 70 75 80
Leu Leu His Val Ala Arg Asp Ile Ala Cys Gly Cys Gln Tyr Leu Glu
85 90 95
Glu Asn His Phe Ile
100
<210> 25
<211> 101
<212> PRT
<213> Homo sapiens
<400> 25
Met Ile Lys Leu Ile Asp Ile Ala Arg Gln Thr Ala Gln Gly Met Asp
1 5 10 15
Tyr Leu His Ala Lys Ser Ile Ile His Arg Asp Leu Lys Ser Asn Asn
20 25 30
Ile Phe Leu His Glu Asp Leu Thr Val Lys Ile Gly Asp Phe Gly Leu
35 40 45
Ala Thr Glu Lys Ser Arg Trp Ser Gly Ser His Gln Phe Glu Gln Leu
50 55 60
Ser Gly Ser Ile Leu Trp Met Ala Pro Glu Val Ile Arg Met Gln Asp
65 70 75 80
Lys Asn Pro Tyr Ser Phe Gln Ser Asp Val Tyr Ala Phe Gly Ile Val
85 90 95
Leu Tyr Glu Leu Met
100
<210> 26
<211> 93
<212> PRT
<213> Homo sapiens
<400> 26
Met Gly Phe Gly Asp Leu Lys Ser Pro Ala Gly Leu Gln Val Leu Asn
1 5 10 15
Asp Tyr Leu Ala Asp Lys Ser Tyr Ile Glu Gly Tyr Val Pro Ser Gln
20 25 30
Ala Asp Val Ala Val Phe Glu Ala Val Ser Gly Pro Pro Pro Ala Asp
35 40 45
Leu Cys His Ala Leu Arg Trp Tyr Asn His Ile Lys Ser Tyr Glu Lys
50 55 60
Glu Lys Ala Ser Leu Pro Gly Val Lys Lys Ala Leu Gly Lys Tyr Gly
65 70 75 80
Pro Ala Asp Val Glu Asp Thr Thr Gly Ser Gly Ala Thr
85 90
<210> 27
<211> 101
<212> PRT
<213> Homo sapiens
<400> 27
Glu Arg Cys Glu Val Val Met Gly Asn Leu Glu Ile Val Leu Thr Gly
1 5 10 15
His Asn Ala Asp Leu Ser Phe Leu Gln Trp Ile Arg Glu Val Thr Gly
20 25 30
Tyr Val Leu Val Ala Met Asn Glu Phe Ser Thr Leu Pro Leu Pro Asn
35 40 45
Leu Arg Met Val Arg Gly Thr Gln Val Tyr Asp Gly Lys Phe Ala Ile
50 55 60
Phe Val Met Leu Asn Tyr Asn Thr Asn Ser Ser His Ala Leu Arg Gln
65 70 75 80
Leu Arg Leu Thr Gln Leu Thr Glu Ile Leu Ser Gly Gly Val Tyr Ile
85 90 95
Glu Lys Asn Asp Lys
100
<210> 28
<211> 101
<212> PRT
<213> Homo sapiens
<400> 28
His Leu Met Ala Lys Ala Gly Leu Thr Leu Gln Gln Gln His Gln Arg
1 5 10 15
Leu Ala Gln Leu Leu Leu Ile Leu Ser His Ile Arg His Met Ser Asn
20 25 30
Lys Gly Met Glu His Leu Tyr Ser Met Lys Cys Lys Asn Val Val Pro
35 40 45
Leu Tyr Gly Leu Leu Leu Glu Met Leu Asp Ala His Arg Leu His Ala
50 55 60
Pro Thr Ser Arg Gly Gly Ala Ser Val Glu Glu Thr Asp Gln Ser His
65 70 75 80
Leu Ala Thr Ala Gly Ser Thr Ser Ser His Ser Leu Gln Lys Tyr Tyr
85 90 95
Ile Thr Gly Glu Ala
100
<210> 29
<211> 101
<212> PRT
<213> Homo sapiens
<400> 29
Asn Gln Gly Lys Cys Val Glu Gly Met Val Glu Ile Phe Asp Met Leu
1 5 10 15
Leu Ala Thr Ser Ser Arg Phe Arg Met Met Asn Leu Gln Gly Glu Glu
20 25 30
Phe Val Cys Leu Lys Ser Ile Ile Leu Leu Asn Ser Gly Val Tyr Thr
35 40 45
Phe Leu Pro Ser Thr Leu Lys Ser Leu Glu Glu Lys Asp His Ile His
50 55 60
Arg Val Leu Asp Lys Ile Thr Asp Thr Leu Ile His Leu Met Ala Lys
65 70 75 80
Ala Gly Leu Thr Leu Gln Gln Gln His Gln Arg Leu Ala Gln Leu Leu
85 90 95
Leu Ile Leu Ser His
100
<210> 30
<211> 101
<212> PRT
<213> Homo sapiens
<400> 30
Ile His Leu Met Ala Lys Ala Gly Leu Thr Leu Gln Gln Gln His Gln
1 5 10 15
Arg Leu Ala Gln Leu Leu Leu Ile Leu Ser His Ile Arg His Met Ser
20 25 30
Asn Lys Gly Met Glu His Leu Tyr Ser Met Lys Cys Lys Asn Val Val
35 40 45
Pro Leu Cys Asp Leu Leu Leu Glu Met Leu Asp Ala His Arg Leu His
50 55 60
Ala Pro Thr Ser Arg Gly Gly Ala Ser Val Glu Glu Thr Asp Gln Ser
65 70 75 80
His Leu Ala Thr Ala Gly Ser Thr Ser Ser His Ser Leu Gln Lys Tyr
85 90 95
Tyr Ile Thr Gly Glu
100
<210> 31
<211> 101
<212> PRT
<213> Homo sapiens
<400> 31
Ile His Leu Met Ala Lys Ala Gly Leu Thr Leu Gln Gln Gln His Gln
1 5 10 15
Arg Leu Ala Gln Leu Leu Leu Ile Leu Ser His Ile Arg His Met Ser
20 25 30
Asn Lys Gly Met Glu His Leu Tyr Ser Met Lys Cys Lys Asn Val Val
35 40 45
Pro Leu Asn Asp Leu Leu Leu Glu Met Leu Asp Ala His Arg Leu His
50 55 60
Ala Pro Thr Ser Arg Gly Gly Ala Ser Val Glu Glu Thr Asp Gln Ser
65 70 75 80
His Leu Ala Thr Ala Gly Ser Thr Ser Ser His Ser Leu Gln Lys Tyr
85 90 95
Tyr Ile Thr Gly Glu
100
<210> 32
<211> 101
<212> PRT
<213> Homo sapiens
<400> 32
Ile His Leu Met Ala Lys Ala Gly Leu Thr Leu Gln Gln Gln His Gln
1 5 10 15
Arg Leu Ala Gln Leu Leu Leu Ile Leu Ser His Ile Arg His Met Ser
20 25 30
Asn Lys Gly Met Glu His Leu Tyr Ser Met Lys Cys Lys Asn Val Val
35 40 45
Pro Leu Ser Asp Leu Leu Leu Glu Met Leu Asp Ala His Arg Leu His
50 55 60
Ala Pro Thr Ser Arg Gly Gly Ala Ser Val Glu Glu Thr Asp Gln Ser
65 70 75 80
His Leu Ala Thr Ala Gly Ser Thr Ser Ser His Ser Leu Gln Lys Tyr
85 90 95
Tyr Ile Thr Gly Glu
100
<210> 33
<211> 101
<212> PRT
<213> Homo sapiens
<400> 33
His Arg Ile Gly Gly Ile Lys Leu Arg His Gln Gln Trp Ser Leu Val
1 5 10 15
Met Glu Ser Val Val Pro Ser Asp Arg Gly Asn Tyr Thr Cys Val Val
20 25 30
Glu Asn Lys Phe Gly Ser Ile Arg Gln Thr Tyr Thr Leu Asp Val Leu
35 40 45
Glu Arg Cys Pro His Arg Pro Ile Leu Gln Ala Gly Leu Pro Ala Asn
50 55 60
Gln Thr Ala Val Leu Gly Ser Asp Val Glu Phe His Cys Lys Val Tyr
65 70 75 80
Ser Asp Ala Gln Pro His Ile Gln Trp Leu Lys His Val Glu Val Asn
85 90 95
Gly Ser Lys Val Gly
100
<210> 34
<211> 101
<212> PRT
<213> Homo sapiens
<400> 34
Ala Val Lys Leu Ser Asp Ser Arg Ile Ala Leu Lys Ser Gly Tyr Gly
1 5 10 15
Lys Tyr Leu Gly Ile Asn Ser Asp Glu Leu Val Gly His Ser Asp Ala
20 25 30
Ile Gly Pro Arg Glu Gln Trp Glu Pro Val Phe Gln Asn Gly Lys Met
35 40 45
Ala Leu Ser Ala Ser Asn Ser Cys Phe Ile Arg Cys Asn Glu Ala Gly
50 55 60
Asp Ile Glu Ala Lys Ser Lys Thr Ala Gly Glu Glu Glu Met Ile Lys
65 70 75 80
Ile Arg Ser Cys Ala Glu Lys Glu Thr Lys Lys Lys Asp Asp Ile Pro
85 90 95
Glu Glu Asp Lys Gly
100
<210> 35
<211> 101
<212> PRT
<213> Homo sapiens
<400> 35
Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys Gly Ile Trp Ile Pro Asp
1 5 10 15
Gly Glu Asn Val Lys Ile Pro Val Ala Ile Lys Val Leu Arg Glu Asn
20 25 30
Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met
35 40 45
Ala Gly Leu Gly Ser Pro Tyr Val Ser Arg Leu Leu Gly Ile Cys Leu
50 55 60
Thr Ser Thr Val Gln Leu Val Thr Gln Leu Met Pro Tyr Gly Cys Leu
65 70 75 80
Leu Asp His Val Arg Glu Asn Arg Gly Arg Leu Gly Ser Gln Asp Leu
85 90 95
Leu Asn Trp Cys Met
100
<210> 36
<211> 101
<212> PRT
<213> Homo sapiens
<400> 36
Arg Val Glu Glu Phe Lys Leu Lys Gln Met Trp Lys Ser Pro Asn Gly
1 5 10 15
Thr Ile Arg Asn Ile Leu Gly Gly Thr Val Phe Arg Glu Ala Ile Ile
20 25 30
Cys Lys Asn Ile Pro Arg Leu Val Ser Gly Trp Val Lys Pro Ile Ile
35 40 45
Ile Gly His His Ala Tyr Gly Asp Gln Tyr Arg Ala Thr Asp Phe Val
50 55 60
Val Pro Gly Pro Gly Lys Val Glu Ile Thr Tyr Thr Pro Ser Asp Gly
65 70 75 80
Thr Gln Lys Val Thr Tyr Leu Val His Asn Phe Glu Glu Gly Gly Gly
85 90 95
Val Ala Met Gly Met
100
<210> 37
<211> 101
<212> PRT
<213> Homo sapiens
<400> 37
Arg Val Glu Glu Phe Lys Leu Lys Gln Met Trp Lys Ser Pro Asn Gly
1 5 10 15
Thr Ile Arg Asn Ile Leu Gly Gly Thr Val Phe Arg Glu Ala Ile Ile
20 25 30
Cys Lys Asn Ile Pro Arg Leu Val Ser Gly Trp Val Lys Pro Ile Ile
35 40 45
Ile Gly Cys His Ala Tyr Gly Asp Gln Tyr Arg Ala Thr Asp Phe Val
50 55 60
Val Pro Gly Pro Gly Lys Val Glu Ile Thr Tyr Thr Pro Ser Asp Gly
65 70 75 80
Thr Gln Lys Val Thr Tyr Leu Val His Asn Phe Glu Glu Gly Gly Gly
85 90 95
Val Ala Met Gly Met
100
<210> 38
<211> 101
<212> PRT
<213> Homo sapiens
<400> 38
Arg Val Glu Glu Phe Lys Leu Lys Gln Met Trp Lys Ser Pro Asn Gly
1 5 10 15
Thr Ile Arg Asn Ile Leu Gly Gly Thr Val Phe Arg Glu Ala Ile Ile
20 25 30
Cys Lys Asn Ile Pro Arg Leu Val Ser Gly Trp Val Lys Pro Ile Ile
35 40 45
Ile Gly Gly His Ala Tyr Gly Asp Gln Tyr Arg Ala Thr Asp Phe Val
50 55 60
Val Pro Gly Pro Gly Lys Val Glu Ile Thr Tyr Thr Pro Ser Asp Gly
65 70 75 80
Thr Gln Lys Val Thr Tyr Leu Val His Asn Phe Glu Glu Gly Gly Gly
85 90 95
Val Ala Met Gly Met
100
<210> 39
<211> 101
<212> PRT
<213> Homo sapiens
<400> 39
Arg Val Glu Glu Phe Lys Leu Lys Gln Met Trp Lys Ser Pro Asn Gly
1 5 10 15
Thr Ile Arg Asn Ile Leu Gly Gly Thr Val Phe Arg Glu Ala Ile Ile
20 25 30
Cys Lys Asn Ile Pro Arg Leu Val Ser Gly Trp Val Lys Pro Ile Ile
35 40 45
Ile Gly Ser His Ala Tyr Gly Asp Gln Tyr Arg Ala Thr Asp Phe Val
50 55 60
Val Pro Gly Pro Gly Lys Val Glu Ile Thr Tyr Thr Pro Ser Asp Gly
65 70 75 80
Thr Gln Lys Val Thr Tyr Leu Val His Asn Phe Glu Glu Gly Gly Gly
85 90 95
Val Ala Met Gly Met
100
<210> 40
<211> 101
<212> PRT
<213> Homo sapiens
<400> 40
Val Ala Val Lys Met Leu Lys Pro Ser Ala His Leu Thr Glu Arg Glu
1 5 10 15
Ala Leu Met Ser Glu Leu Lys Val Leu Ser Tyr Leu Gly Asn His Met
20 25 30
Asn Ile Val Asn Leu Leu Gly Ala Cys Thr Ile Gly Gly Pro Thr Leu
35 40 45
Val Ile Ile Glu Tyr Cys Cys Tyr Gly Asp Leu Leu Asn Phe Leu Arg
50 55 60
Arg Lys Arg Asp Ser Phe Ile Cys Ser Lys Gln Glu Asp His Ala Glu
65 70 75 80
Ala Ala Leu Tyr Lys Asn Leu Leu His Ser Lys Glu Ser Ser Cys Ser
85 90 95
Asp Ser Thr Asn Glu
100
<210> 41
<211> 101
<212> PRT
<213> Homo sapiens
<400> 41
Val Glu Ala Thr Ala Tyr Gly Leu Ile Lys Ser Asp Ala Ala Met Thr
1 5 10 15
Val Ala Val Lys Met Leu Lys Pro Ser Ala His Leu Thr Glu Arg Glu
20 25 30
Ala Leu Met Ser Glu Leu Lys Val Leu Ser Tyr Leu Gly Asn His Met
35 40 45
Asn Ile Ala Asn Leu Leu Gly Ala Cys Thr Ile Gly Gly Pro Thr Leu
50 55 60
Val Ile Thr Glu Tyr Cys Cys Tyr Gly Asp Leu Leu Asn Phe Leu Arg
65 70 75 80
Arg Lys Arg Asp Ser Phe Ile Cys Ser Lys Gln Glu Asp His Ala Glu
85 90 95
Ala Ala Leu Tyr Lys
100
<210> 42
<211> 101
<212> PRT
<213> Homo sapiens
<400> 42
Ile Ser Glu Leu Gly Ala Gly Asn Gly Gly Val Val Phe Lys Val Ser
1 5 10 15
His Lys Pro Ser Gly Leu Val Met Ala Arg Lys Leu Ile His Leu Glu
20 25 30
Ile Lys Pro Ala Ile Arg Asn Gln Ile Ile Arg Glu Leu Gln Val Leu
35 40 45
His Glu Ser Asn Ser Pro Tyr Ile Val Gly Phe Tyr Gly Ala Phe Tyr
50 55 60
Ser Asp Gly Glu Ile Ser Ile Cys Met Glu His Met Asp Gly Gly Ser
65 70 75 80
Leu Asp Gln Val Leu Lys Lys Ala Gly Arg Ile Pro Glu Gln Ile Leu
85 90 95
Gly Lys Val Ser Ile
100
<210> 43
<211> 101
<212> PRT
<213> Homo sapiens
<400> 43
Leu Gly Ala Gly Asn Gly Gly Val Val Phe Lys Val Ser His Lys Pro
1 5 10 15
Ser Gly Leu Val Met Ala Arg Lys Leu Ile His Leu Glu Ile Lys Pro
20 25 30
Ala Ile Arg Asn Gln Ile Ile Arg Glu Leu Gln Val Leu His Glu Cys
35 40 45
Asn Ser Leu Tyr Ile Val Gly Phe Tyr Gly Ala Phe Tyr Ser Asp Gly
50 55 60
Glu Ile Ser Ile Cys Met Glu His Met Asp Gly Gly Ser Leu Asp Gln
65 70 75 80
Val Leu Lys Lys Ala Gly Arg Ile Pro Glu Gln Ile Leu Gly Lys Val
85 90 95
Ser Ile Ala Val Ile
100
<210> 44
<211> 89
<212> PRT
<213> Homo sapiens
<400> 44
Met Pro Leu Asn Val Ser Phe Thr Asn Arg Asn Tyr Asp Leu Asp Tyr
1 5 10 15
Asp Ser Val Gln Pro Tyr Phe Tyr Cys Asp Glu Glu Glu Asn Phe Tyr
20 25 30
Gln Gln Gln Gln Gln Ser Asp Leu Gln Pro Pro Ala Pro Ser Glu Asp
35 40 45
Ile Trp Lys Lys Phe Glu Leu Leu Pro Thr Pro Pro Leu Ser Pro Ser
50 55 60
Arg Arg Ser Gly Leu Cys Ser Pro Ser Tyr Val Ala Val Thr Pro Phe
65 70 75 80
Ser Leu Arg Gly Asp Asn Asp Gly Gly
85
<210> 45
<211> 101
<212> PRT
<213> Homo sapiens
<400> 45
Phe Thr Asn Arg Asn Tyr Asp Leu Asp Tyr Asp Ser Val Gln Pro Tyr
1 5 10 15
Phe Tyr Cys Asp Glu Glu Glu Asn Phe Tyr Gln Gln Gln Gln Gln Ser
20 25 30
Glu Leu Gln Pro Pro Ala Pro Ser Glu Asp Ile Trp Lys Lys Phe Glu
35 40 45
Leu Leu Ser Thr Pro Pro Leu Ser Pro Ser Arg Arg Ser Gly Leu Cys
50 55 60
Ser Pro Ser Tyr Val Ala Val Thr Pro Phe Ser Leu Arg Gly Asp Asn
65 70 75 80
Asp Gly Gly Gly Gly Ser Phe Ser Thr Ala Asp Gln Leu Glu Met Val
85 90 95
Thr Glu Leu Leu Gly
100
<210> 46
<211> 101
<212> PRT
<213> Homo sapiens
<400> 46
Thr Asn Arg Asn Tyr Asp Leu Asp Tyr Asp Ser Val Gln Pro Tyr Phe
1 5 10 15
Tyr Cys Asp Glu Glu Glu Asn Phe Tyr Gln Gln Gln Gln Gln Ser Glu
20 25 30
Leu Gln Pro Pro Ala Pro Ser Glu Asp Ile Trp Lys Lys Phe Glu Leu
35 40 45
Leu Pro Ile Pro Pro Leu Ser Pro Ser Arg Arg Ser Gly Leu Cys Ser
50 55 60
Pro Ser Tyr Val Ala Val Thr Pro Phe Ser Leu Arg Gly Asp Asn Asp
65 70 75 80
Gly Gly Gly Gly Ser Phe Ser Thr Ala Asp Gln Leu Glu Met Val Thr
85 90 95
Glu Leu Leu Gly Gly
100
<210> 47
<211> 101
<212> PRT
<213> Homo sapiens
<400> 47
Val Ala Val Lys Met Leu Lys Pro Thr Ala Arg Ser Ser Glu Lys Gln
1 5 10 15
Ala Leu Met Ser Glu Leu Lys Ile Met Thr His Leu Gly Pro His Leu
20 25 30
Asn Ile Val Asn Leu Leu Gly Ala Cys Thr Lys Ser Gly Pro Ile Tyr
35 40 45
Ile Ile Ile Glu Tyr Cys Phe Tyr Gly Asp Leu Val Asn Tyr Leu His
50 55 60
Lys Asn Arg Asp Ser Phe Leu Ser His His Pro Glu Lys Pro Lys Lys
65 70 75 80
Glu Leu Asp Ile Phe Gly Leu Asn Pro Ala Asp Glu Ser Thr Arg Ser
85 90 95
Tyr Val Ile Leu Ser
100
<210> 48
<211> 101
<212> PRT
<213> Homo sapiens
<400> 48
Ile Glu Glu His Ala Asn Trp Ser Val Ser Arg Glu Ala Gly Phe Ser
1 5 10 15
Tyr Ser His Ala Gly Leu Ser Asn Arg Leu Ala Arg Asp Asn Glu Leu
20 25 30
Arg Glu Asn Asp Lys Glu Gln Leu Lys Ala Ile Ser Thr Arg Asp Pro
35 40 45
Leu Ser Lys Ile Thr Glu Gln Glu Lys Asp Phe Leu Trp Ser His Arg
50 55 60
His Tyr Cys Val Thr Ile Pro Glu Ile Leu Pro Lys Leu Leu Leu Ser
65 70 75 80
Val Lys Trp Asn Ser Arg Asp Glu Val Ala Gln Met Tyr Cys Leu Val
85 90 95
Lys Asp Trp Pro Pro
100
<210> 49
<211> 101
<212> PRT
<213> Homo sapiens
<400> 49
His Ala Asn Trp Ser Val Ser Arg Glu Ala Gly Phe Ser Tyr Ser His
1 5 10 15
Ala Gly Leu Ser Asn Arg Leu Ala Arg Asp Asn Glu Leu Arg Glu Asn
20 25 30
Asp Lys Glu Gln Leu Lys Ala Ile Ser Thr Arg Asp Pro Leu Ser Glu
35 40 45
Ile Thr Lys Gln Glu Lys Asp Phe Leu Trp Ser His Arg His Tyr Cys
50 55 60
Val Thr Ile Pro Glu Ile Leu Pro Lys Leu Leu Leu Ser Val Lys Trp
65 70 75 80
Asn Ser Arg Asp Glu Val Ala Gln Met Tyr Cys Leu Val Lys Asp Trp
85 90 95
Pro Pro Ile Lys Pro
100
<210> 50
<211> 72
<212> PRT
<213> Homo sapiens
<400> 50
Leu Phe Ile Asn Leu Phe Ser Met Met Leu Gly Ser Gly Met Pro Glu
1 5 10 15
Leu Gln Ser Phe Asp Asp Ile Ala Tyr Ile Arg Lys Thr Leu Ala Leu
20 25 30
Asp Lys Thr Glu Gln Glu Ala Leu Glu Tyr Phe Met Lys Gln Met Asn
35 40 45
Asp Ala Arg His Gly Gly Trp Thr Thr Lys Met Asp Trp Ile Phe His
50 55 60
Thr Ile Lys Gln His Ala Leu Asn
65 70
<210> 51
<211> 101
<212> PRT
<213> Homo sapiens
<400> 51
Gln Arg Gly Gly Val Ile Thr Asp Glu Glu Glu Thr Ser Lys Lys Ile
1 5 10 15
Ala Asp Gln Leu Asp Asn Ile Val Asp Met Arg Glu Tyr Asp Val Pro
20 25 30
Tyr His Ile Arg Leu Ser Ile Asp Ile Glu Thr Thr Lys Leu Pro Leu
35 40 45
Lys Phe Arg Asp Ala Glu Thr Asp Gln Ile Met Met Ile Ser Tyr Met
50 55 60
Ile Asp Gly Gln Gly Tyr Leu Ile Thr Asn Arg Glu Ile Val Ser Glu
65 70 75 80
Asp Ile Glu Asp Phe Glu Phe Thr Pro Lys Pro Glu Tyr Glu Gly Pro
85 90 95
Phe Cys Val Phe Asn
100
<210> 52
<211> 101
<212> PRT
<213> Homo sapiens
<400> 52
Lys Phe Asn Cys Arg Val Ala Gln Tyr Pro Phe Glu Asp His Asn Pro
1 5 10 15
Pro Gln Leu Glu Leu Ile Lys Pro Phe Cys Glu Asp Leu Asp Gln Trp
20 25 30
Leu Ser Glu Asp Asp Asn His Val Ala Ala Ile His Cys Lys Ala Gly
35 40 45
Lys Gly Gln Thr Gly Val Met Ile Cys Ala Tyr Leu Leu His Arg Gly
50 55 60
Lys Phe Leu Lys Ala Gln Glu Ala Leu Asp Phe Tyr Gly Glu Val Arg
65 70 75 80
Thr Arg Asp Lys Lys Gly Val Thr Ile Pro Ser Gln Arg Arg Tyr Val
85 90 95
Tyr Tyr Tyr Ser Tyr
100
<210> 53
<211> 79
<212> PRT
<213> Homo sapiens
<400> 53
Met Gln Ala Ile Lys Cys Val Val Val Gly Asp Gly Ala Val Gly Lys
1 5 10 15
Thr Cys Leu Leu Ile Ser Tyr Thr Thr Asn Ala Phe Ser Gly Glu Tyr
20 25 30
Ile Pro Thr Val Phe Asp Asn Tyr Ser Ala Asn Val Met Val Asp Gly
35 40 45
Lys Pro Val Asn Leu Gly Leu Trp Asp Thr Ala Gly Gln Glu Asp Tyr
50 55 60
Asp Arg Leu Arg Pro Leu Ser Tyr Pro Gln Thr Val Gly Glu Thr
65 70 75
<210> 54
<211> 101
<212> PRT
<213> Homo sapiens
<400> 54
Ile Arg Val Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg Asn
1 5 10 15
Thr Phe Arg His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val Gly
20 25 30
Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser Cys
35 40 45
Met Gly Ser Met Asn Arg Arg Pro Ile Leu Thr Ile Ile Thr Leu Glu
50 55 60
Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val Arg Val
65 70 75 80
Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu Arg
85 90 95
Lys Lys Gly Glu Pro
100
<210> 55
<211> 101
<212> PRT
<213> Homo sapiens
<400> 55
Thr Tyr Ser Pro Ala Leu Asn Lys Met Phe Cys Gln Leu Ala Lys Thr
1 5 10 15
Cys Pro Val Gln Leu Trp Val Asp Ser Thr Pro Pro Pro Gly Thr Arg
20 25 30
Val Arg Ala Met Ala Ile Tyr Lys Gln Ser Gln His Met Thr Glu Val
35 40 45
Val Arg His Cys Pro His His Glu Arg Cys Ser Asp Ser Asp Gly Leu
50 55 60
Ala Pro Pro Gln His Leu Ile Arg Val Glu Gly Asn Leu Arg Val Glu
65 70 75 80
Tyr Leu Asp Asp Arg Asn Thr Phe Arg His Ser Val Val Val Pro Tyr
85 90 95
Glu Pro Pro Glu Val
100
<210> 56
<211> 101
<212> PRT
<213> Homo sapiens
<400> 56
Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg Asn Thr Phe Arg
1 5 10 15
His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val Gly Ser Asp Cys
20 25 30
Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser Cys Met Gly Gly
35 40 45
Met Asn Gln Arg Pro Ile Leu Thr Ile Ile Thr Leu Glu Asp Ser Ser
50 55 60
Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val Arg Val Cys Ala Cys
65 70 75 80
Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu Arg Lys Lys Gly
85 90 95
Glu Pro His His Glu
100
<210> 57
<211> 101
<212> PRT
<213> Homo sapiens
<400> 57
Glu Gly Asn Leu Arg Val Glu Tyr Leu Asp Asp Arg Asn Thr Phe Arg
1 5 10 15
His Ser Val Val Val Pro Tyr Glu Pro Pro Glu Val Gly Ser Asp Cys
20 25 30
Thr Thr Ile His Tyr Asn Tyr Met Cys Asn Ser Ser Cys Met Gly Gly
35 40 45
Met Asn Trp Arg Pro Ile Leu Thr Ile Ile Thr Leu Glu Asp Ser Ser
50 55 60
Gly Asn Leu Leu Gly Arg Asn Ser Phe Glu Val Arg Val Cys Ala Cys
65 70 75 80
Pro Gly Arg Asp Arg Arg Thr Glu Glu Glu Asn Leu Arg Lys Lys Gly
85 90 95
Glu Pro His His Glu
100
<210> 58
<211> 101
<212> PRT
<213> Homo sapiens
<400> 58
Pro Glu Val Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys
1 5 10 15
Asn Ser Ser Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile
20 25 30
Ile Thr Leu Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe
35 40 45
Glu Val Cys Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu
50 55 60
Glu Asn Leu Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly
65 70 75 80
Ser Thr Lys Arg Ala Leu Pro Asn Asn Thr Ser Ser Ser Pro Gln Pro
85 90 95
Lys Lys Lys Pro Leu
100
<210> 59
<211> 53
<212> PRT
<213> Homo sapiens
<400> 59
Gly Val Glu Pro Cys Tyr Gly Asp Lys Asp Lys Arg Arg His Cys Phe
1 5 10 15
Ala Thr Trp Lys Asn Ile Ser Gly Ser Ile Glu Ile Val Lys Gln Gly
20 25 30
Cys Trp Leu Asp Asp Ile Asn Cys Tyr Asp Arg Thr Asp Cys Val Glu
35 40 45
Lys Lys Arg Gln Pro
50
<210> 60
<211> 103
<212> PRT
<213> Homo sapiens
<400> 60
Gly Val Glu Pro Cys Tyr Gly Asp Lys Asp Lys Arg Arg His Cys Phe
1 5 10 15
Ala Thr Trp Lys Asn Ile Ser Gly Ser Ile Glu Ile Val Lys Gln Gly
20 25 30
Cys Trp Leu Asp Asp Ile Asn Cys Tyr Asp Arg Thr Asp Cys Val Glu
35 40 45
Lys Lys Thr Ala Leu Lys Tyr Ile Phe Val Ala Val Arg Ala Ile Cys
50 55 60
Val Met Lys Ser Phe Leu Ile Phe Arg Arg Trp Lys Ser His Ser Pro
65 70 75 80
Leu Gln Ile Gln Leu His Leu Ser His Pro Ile Thr Thr Ser Cys Ser
85 90 95
Ile Pro Trp Cys His Leu Cys
100
<210> 61
<211> 118
<212> PRT
<213> Homo sapiens
<400> 61
Thr Ala Glu Ala Val Asn Val Ala Ile Ala Ala Pro Pro Ser Glu Gly
1 5 10 15
Glu Ala Asn Ala Glu Leu Cys Arg Tyr Leu Ser Lys Val Leu Glu Leu
20 25 30
Arg Lys Ser Asp Val Val Leu Asp Lys Val Gly Leu Ala Leu Phe Phe
35 40 45
Phe Phe Phe Glu Thr Lys Ser Cys Ser Val Ala Gln Ala Gly Val Gln
50 55 60
Trp Arg Ser Leu Gly Ser Leu Gln Pro Pro Pro Pro Gly Phe Lys Leu
65 70 75 80
Phe Ser Cys Leu Ser Phe Leu Ser Ser Trp Asp Tyr Arg Arg Met Pro
85 90 95
Pro Cys Leu Ala Asn Phe Cys Ile Phe Asn Arg Asp Gly Val Ser Pro
100 105 110
Cys Trp Ser Gly Trp Ser
115
<210> 62
<211> 71
<212> PRT
<213> Homo sapiens
<400> 62
Leu Ser Val Ile Ile Phe Phe Phe Val Tyr Ile Trp His Trp Ala Leu
1 5 10 15
Pro Leu Ile Leu Asn Asn His His Ile Cys Leu Met Ser Ser Ile Ile
20 25 30
Leu Asp Cys Asn Ser Val Arg Gln Ser Ile Met Ser Val Cys Phe Phe
35 40 45
Phe Phe Ser Val Ile Phe Ser Thr Arg Cys Leu Thr Asp Ser Arg Tyr
50 55 60
Pro Asn Ile Cys Trp Phe Lys
65 70
<210> 63
<211> 60
<212> PRT
<213> Homo sapiens
<400> 63
Leu Ser Val Ile Ile Phe Phe Phe Val Tyr Ile Trp His Trp Ala Leu
1 5 10 15
Pro Leu Ile Leu Asn Asn His His Ile Cys Leu Met Ser Ser Ile Ile
20 25 30
Leu Asp Cys Asn Ser Val Arg Gln Ser Ile Met Ser Val Cys Phe Phe
35 40 45
Phe Phe Cys Tyr Ile Leu Asn Thr Met Phe Asp Arg
50 55 60
<210> 64
<211> 106
<212> PRT
<213> Homo sapiens
<400> 64
Val Leu Val Leu Ser Cys Asp Leu Ile Thr Asp Val Ala Leu His Glu
1 5 10 15
Val Val Asp Leu Phe Arg Ala Tyr Asp Ala Ser Leu Ala Met Leu Met
20 25 30
Arg Lys Gly Gln Asp Ser Ile Glu Pro Val Pro Gly Gln Lys Gly Lys
35 40 45
Lys Lys Gln Trp Ser Ser Val Thr Ser Leu Glu Trp Thr Ala Gln Glu
50 55 60
Arg Gly Cys Ser Ser Trp Leu Met Lys Gln Thr Trp Met Lys Ser Trp
65 70 75 80
Ser Leu Arg Asp Pro Ser Tyr Arg Ser Ile Leu Glu Tyr Val Ser Thr
85 90 95
Arg Val Leu Trp Met Pro Thr Ser Thr Val
100 105
<210> 65
<211> 112
<212> PRT
<213> Homo sapiens
<400> 65
Ser Ile Gln Val Met Arg Ala Gln Met Asn Gln Ile Gln Ser Val Glu
1 5 10 15
Gly Gln Pro Leu Ala Arg Arg Pro Arg Ala Thr Gly Arg Thr Lys Arg
20 25 30
Cys Gln Pro Arg Asp Val Thr Lys Lys Thr Cys Asn Ser Asn Asp Gly
35 40 45
Lys Lys Arg Glu Trp Glu Lys Arg Lys Gln Ile Leu Gly Gly Gly Gly
50 55 60
Lys Tyr Lys Glu Tyr Phe Leu Lys Arg Ile Leu Ile Arg Lys Ala Met
65 70 75 80
Thr Val Leu Ala Gly Asp Lys Lys Gly Leu Gly Arg Phe Met Arg Cys
85 90 95
Val Gln Ser Glu Thr Lys Ala Val Ser Leu Gln Leu Pro Leu Gly Arg
100 105 110
<210> 66
<211> 57
<212> PRT
<213> Homo sapiens
<400> 66
Leu Asp Phe Leu Gly Glu Phe Ala Thr Asp Ile Arg Thr His Gly Val
1 5 10 15
His Met Val Leu Asn His Gln Gly Arg Pro Ser Gly Asp Ala Phe Ile
20 25 30
Gln Met Lys Ser Ala Asp Arg Ala Phe Met Ala Ala Gln Lys Cys His
35 40 45
Lys Lys Lys His Glu Gly Gln Ile Cys
50 55
<210> 67
<211> 51
<212> PRT
<213> Homo sapiens
<400> 67
Leu Asp Phe Leu Gly Glu Phe Ala Thr Asp Ile Arg Thr His Gly Val
1 5 10 15
His Met Val Leu Asn His Gln Gly Arg Pro Ser Gly Asp Ala Phe Ile
20 25 30
Gln Met Lys Ser Ala Asp Arg Ala Phe Met Ala Ala Gln Lys Cys His
35 40 45
Lys Lys Thr
50
<210> 68
<211> 75
<212> PRT
<213> Homo sapiens
<400> 68
Gly Ala Leu Cys Lys Asp Gly Arg Phe Arg Ser Asp Ile Gly Glu Phe
1 5 10 15
Glu Trp Lys Leu Lys Glu Gly His Lys Lys Ile Tyr Gly Lys Gln Ser
20 25 30
Met Val Asp Glu Val Ser Gly Lys Val Leu Glu Met Asp Ile Ser Lys
35 40 45
Lys Lys His Tyr Asn Arg Lys Ile Ser Ile Lys Lys Leu Asn Arg Met
50 55 60
Lys Val Pro Leu Met Lys Leu Ile Thr Arg Val
65 70 75
<210> 69
<211> 58
<212> PRT
<213> Homo sapiens
<400> 69
Arg Glu Arg Ala Gln Leu Leu Glu Glu Gln Glu Lys Thr Leu Thr Ser
1 5 10 15
Lys Leu Gln Glu Gln Ala Arg Val Leu Lys Glu Arg Cys Gln Gly Glu
20 25 30
Ser Thr Gln Leu Gln Asn Glu Ile Gln Lys Leu Gln Lys Thr Leu Lys
35 40 45
Lys Lys Pro Arg Asp Ile Cys Arg Ile Ser
50 55
<210> 70
<211> 53
<212> PRT
<213> Homo sapiens
<400> 70
Val Asn Thr Leu Lys Glu Gly Lys Arg Leu Pro Cys Pro Pro Asn Cys
1 5 10 15
Pro Asp Glu Val Tyr Gln Leu Met Arg Lys Cys Trp Glu Phe Gln Pro
20 25 30
Ser Asn Arg Thr Ser Phe Gln Asn Leu Ile Glu Gly Phe Glu Ala Leu
35 40 45
Leu Lys Thr Ser Asn
50
<210> 71
<211> 65
<212> PRT
<213> Homo sapiens
<400> 71
Cys Arg Pro Val Thr Pro Ser Cys Lys Glu Leu Ala Asp Leu Met Thr
1 5 10 15
Arg Cys Met Asn Tyr Asp Pro Asn Gln Arg Pro Phe Phe Arg Ala Ile
20 25 30
Met Arg Asp Ile Asn Lys Leu Glu Glu Gln Asn Pro Asp Ile Val Ser
35 40 45
Glu Lys Asn Gln Gln Leu Lys Trp Thr Pro His Ile Leu Lys Ser Ala
50 55 60
Ser
65
<210> 72
<211> 69
<212> PRT
<213> Homo sapiens
<400> 72
Asp Asp His Asp Val Leu Ser Phe Leu Thr Phe Gln Leu Thr Glu Pro
1 5 10 15
Gly Lys Glu Pro Pro Thr Pro Asp Lys Glu Ile Ser Glu Lys Glu Lys
20 25 30
Glu Lys Tyr Gln Glu Glu Phe Glu His Phe Gln Gln Glu Leu Asp Lys
35 40 45
Lys Lys Arg Gly Ile Pro Glu Gly Pro Pro Arg Pro Pro Arg Ala Ala
50 55 60
Cys Gly Gly Asn Ile
65
<210> 73
<211> 71
<212> PRT
<213> Homo sapiens
<400> 73
Asp Asp His Asp Val Leu Ser Phe Leu Thr Phe Gln Leu Thr Glu Pro
1 5 10 15
Gly Lys Glu Pro Pro Thr Pro Asp Lys Glu Ile Ser Glu Lys Glu Lys
20 25 30
Glu Lys Tyr Gln Glu Glu Phe Glu His Phe Gln Gln Glu Leu Asp Lys
35 40 45
Lys Lys Arg Asn Ser Arg Arg Ala Thr Pro Thr Ser Lys Gly Ser Leu
50 55 60
Arg Arg Lys Tyr Leu Arg Val
65 70
<210> 74
<211> 68
<212> PRT
<213> Homo sapiens
<400> 74
Thr Lys Ser Thr Leu Ile Gly Glu Asp Val Asn Pro Leu Ile Lys Leu
1 5 10 15
Asp Asp Ala Val Asn Val Asp Glu Ile Met Thr Asp Thr Ser Thr Ser
20 25 30
Tyr Leu Leu Cys Ile Ser Glu Asn Lys Glu Asn Val Arg Asp Lys Lys
35 40 45
Lys Gly Gln His Phe Tyr Trp His Cys Gly Ser Ala Ala Cys His Arg
50 55 60
Arg Gly Cys Val
65
<210> 75
<211> 81
<212> PRT
<213> Homo sapiens
<400> 75
Leu Tyr Thr Lys Ser Thr Leu Ile Gly Glu Asp Val Asn Pro Leu Ile
1 5 10 15
Lys Leu Asp Asp Ala Val Asn Val Asp Glu Ile Met Thr Asp Thr Ser
20 25 30
Thr Ser Tyr Leu Leu Cys Ile Ser Glu Asn Lys Glu Asn Val Arg Asp
35 40 45
Lys Lys Arg Ala Thr Phe Leu Leu Ala Leu Trp Glu Cys Ser Leu Pro
50 55 60
Gln Ala Arg Leu Cys Leu Ile Val Ser Arg Thr Leu Leu Leu Val Gln
65 70 75 80
Ser
<210> 76
<211> 58
<212> PRT
<213> Homo sapiens
<400> 76
Leu Pro Pro Pro Lys Leu Thr Asp Pro Arg Leu Leu Tyr Ile Gly Phe
1 5 10 15
Leu Gly Tyr Cys Ser Gly Leu Ile Asp Asn Leu Ile Arg Arg Arg Pro
20 25 30
Ile Ala Thr Ala Gly Leu His Arg Gln Leu Leu Tyr Ile Thr Ala Phe
35 40 45
Phe Phe Cys Trp Ile Leu Ser Cys Lys Thr
50 55
<210> 77
<211> 56
<212> PRT
<213> Homo sapiens
<400> 77
Ser Leu Pro Pro Pro Lys Leu Thr Asp Pro Arg Leu Leu Tyr Ile Gly
1 5 10 15
Phe Leu Gly Tyr Cys Ser Gly Leu Ile Asp Asn Leu Ile Arg Arg Arg
20 25 30
Pro Ile Ala Thr Ala Gly Leu His Arg Gln Leu Leu Tyr Ile Thr Ala
35 40 45
Phe Phe Leu Leu Asp Ile Ile Leu
50 55
<210> 78
<211> 112
<212> PRT
<213> Homo sapiens
<400> 78
Asn Gln Ser Gly Gly Ala Gly Glu Asp Cys Gln Ile Phe Ser Thr Pro
1 5 10 15
Gly His Pro Lys Met Ile Tyr Ser Ser Ser Asn Leu Lys Thr Pro Ser
20 25 30
Lys Leu Cys Ser Gly Ser Lys Ser His Asp Val Gln Glu Val Leu Lys
35 40 45
Lys Lys Thr Gly Ser Asn Glu Val Thr Thr Arg Tyr Glu Glu Lys Lys
50 55 60
Thr Gly Ser Val Arg Lys Ala Asn Arg Met Pro Lys Asp Val Asn Ile
65 70 75 80
Gln Val Arg Lys Lys Gln Lys His Glu Thr Arg Arg Lys Ser Lys Tyr
85 90 95
Asn Glu Asp Phe Glu Arg Ala Trp Arg Glu Asp Leu Thr Ile Lys Arg
100 105 110
<210> 79
<211> 23
<212> PRT
<213> Homo sapiens
<400> 79
Met Ala Pro Val Lys Lys Leu Val Val Lys Gly Gly Lys Lys Lys Glu
1 5 10 15
Ala Ser Ser Glu Val His Ser
20
<210> 80
<211> 18
<212> PRT
<213> Homo sapiens
<400> 80
Met Ala Pro Val Lys Lys Leu Val Val Lys Gly Gly Lys Lys Arg Ser
1 5 10 15
Lys Phe
<210> 81
<211> 51
<212> PRT
<213> Homo sapiens
<400> 81
Met Pro Ser His Gln Gly Ala Glu Gln Gln Gln Gln Gln His His Val
1 5 10 15
Phe Ile Ser Gln Val Val Thr Glu Lys Glu Phe Leu Ser Arg Ser Asp
20 25 30
Gln Leu Gln Gln Ala Val Gln Ser Gln Gly Phe Ile Asn Tyr Cys Gln
35 40 45
Lys Lys Asn
50
<210> 82
<211> 65
<212> PRT
<213> Homo sapiens
<400> 82
Met Pro Ser His Gln Gly Ala Glu Gln Gln Gln Gln Gln His His Val
1 5 10 15
Phe Ile Ser Gln Val Val Thr Glu Lys Glu Phe Leu Ser Arg Ser Asp
20 25 30
Gln Leu Gln Gln Ala Val Gln Ser Gln Gly Phe Ile Asn Tyr Cys Gln
35 40 45
Lys Lys Leu Met Leu Leu Arg Leu Asn Leu Arg Lys Met Cys Gly Pro
50 55 60
Phe
65
<210> 83
<211> 79
<212> PRT
<213> Homo sapiens
<400> 83
Ala Glu Val Phe Glu Lys Glu Gln Ser Ile Cys Ala Ala Glu Glu Gln
1 5 10 15
Pro Ala Glu Asp Gly Gln Gly Glu Thr Asn Lys Asn Arg Thr Lys Gly
20 25 30
Gly Trp Gln Gln Lys Ser Lys Gly Pro Lys Lys Thr Ala Lys Ser Lys
35 40 45
Lys Lys Glu Thr Phe Lys Lys Lys Thr Tyr Thr Cys Ala Ile Thr Thr
50 55 60
Val Lys Ala Thr Glu Thr Lys Ala Gly Lys Trp Ser Arg Trp Glu
65 70 75
<210> 84
<211> 53
<212> PRT
<213> Homo sapiens
<400> 84
Met Ala Glu Val Phe Glu Lys Glu Gln Ser Ile Cys Ala Ala Glu Glu
1 5 10 15
Gln Pro Ala Glu Asp Gly Gln Gly Glu Thr Asn Lys Asn Arg Thr Lys
20 25 30
Gly Gly Trp Gln Gln Lys Ser Lys Gly Pro Lys Lys Thr Ala Lys Ser
35 40 45
Lys Lys Arg Asn Leu
50
<210> 85
<211> 71
<212> PRT
<213> Homo sapiens
<400> 85
Asn Ile Met Glu Ile Arg Gln Leu Pro Ser Ser His Ala Leu Glu Ala
1 5 10 15
Lys Leu Ser Arg Met Ser Tyr Pro Val Lys Glu Gln Glu Ser Ile Leu
20 25 30
Lys Thr Val Gly Lys Leu Thr Ala Thr Gln Val Ala Lys Ile Ser Phe
35 40 45
Phe Phe Ala Leu Cys Gly Phe Trp Gln Ile Cys His Ile Lys Lys His
50 55 60
Phe Gln Thr His Lys Leu Leu
65 70
<210> 86
<211> 68
<212> PRT
<213> Homo sapiens
<400> 86
Tyr Glu Lys Lys Lys Tyr Tyr Asp Lys Asn Ala Ile Ala Ile Thr Asn
1 5 10 15
Ile Ser Ser Ser Asp Ala Pro Leu Gln Pro Leu Val Ser Ser Pro Ser
20 25 30
Leu Gln Ala Ala Val Asp Lys Asn Lys Leu Glu Lys Glu Lys Glu Lys
35 40 45
Lys Lys Gly Arg Glu Lys Glu Arg Lys Gly Ala Arg Lys Ala Gly Lys
50 55 60
Thr Thr Tyr Ser
65
<210> 87
<211> 97
<212> PRT
<213> Homo sapiens
<400> 87
Lys Tyr Glu Lys Lys Lys Tyr Tyr Asp Lys Asn Ala Ile Ala Ile Thr
1 5 10 15
Asn Ile Ser Ser Ser Asp Ala Pro Leu Gln Pro Leu Val Ser Ser Pro
20 25 30
Ser Leu Gln Ala Ala Val Asp Lys Asn Lys Leu Glu Lys Glu Lys Glu
35 40 45
Lys Lys Arg Lys Arg Lys Arg Glu Lys Arg Ser Gln Lys Ser Arg Gln
50 55 60
Asn His Leu Gln Leu Lys Ser Cys Arg Arg Lys Ile Ser Asn Trp Ser
65 70 75 80
Leu Lys Lys Val Pro Ala Leu Lys Lys Leu Arg Ser Pro Leu Trp Ile
85 90 95
Phe
<210> 88
<211> 65
<212> PRT
<213> Homo sapiens
<400> 88
Ile Tyr Gln Asp Ala Tyr Arg Ala Glu Trp Gln Val Tyr Lys Glu Glu
1 5 10 15
Ile Ser Arg Phe Lys Glu Gln Leu Thr Pro Ser Gln Ile Met Ser Leu
20 25 30
Glu Lys Glu Ile Met Asp Lys His Leu Lys Arg Lys Ala Met Thr Lys
35 40 45
Lys Lys Arg Val Asn Thr Ala Trp Lys Thr Lys Lys Thr Ser Phe Ser
50 55 60
Leu
65
<210> 89
<211> 51
<212> PRT
<213> Homo sapiens
<400> 89
Ile Tyr Gln Asp Ala Tyr Arg Ala Glu Trp Gln Val Tyr Lys Glu Glu
1 5 10 15
Ile Ser Arg Phe Lys Glu Gln Leu Thr Pro Ser Gln Ile Met Ser Leu
20 25 30
Glu Lys Glu Ile Met Asp Lys His Leu Lys Arg Lys Ala Met Thr Lys
35 40 45
Lys Lys Ser
50
<210> 90
<211> 52
<212> PRT
<213> Homo sapiens
<400> 90
Lys Pro Gln Glu Val Cys Val Ala Val Trp Arg Lys Asn Asp Glu Asn
1 5 10 15
Ile Thr Leu Glu Thr Val Cys His Asp Pro Lys Leu Pro Tyr His Asp
20 25 30
Phe Ile Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile Met Lys Glu Lys
35 40 45
Lys Lys Ala Trp
50
<210> 91
<211> 84
<212> PRT
<213> Homo sapiens
<400> 91
Glu Lys Pro Gln Glu Val Cys Val Ala Val Trp Arg Lys Asn Asp Glu
1 5 10 15
Asn Ile Thr Leu Glu Thr Val Cys His Asp Pro Lys Leu Pro Tyr His
20 25 30
Asp Phe Ile Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile Met Lys Glu
35 40 45
Lys Lys Ser Leu Val Arg Leu Ser Ser Cys Val Pro Val Ala Leu Met
50 55 60
Ser Ala Met Thr Thr Ser Ser Ser Gln Lys Asn Ile Thr Pro Ala Ile
65 70 75 80
Leu Thr Cys Cys
<210> 92
<211> 74
<212> PRT
<213> Homo sapiens
<400> 92
Val Pro Ser Lys Tyr Gln Phe Leu Cys Ser Asp His Phe Thr Pro Asp
1 5 10 15
Ser Leu Asp Ile Arg Trp Gly Ile Arg Tyr Leu Lys Gln Thr Ala Val
20 25 30
Pro Thr Ile Phe Ser Leu Pro Glu Asp Asn Gln Gly Lys Asp Pro Ser
35 40 45
Lys Lys Asn Pro Arg Arg Lys Thr Trp Lys Met Arg Lys Lys Tyr Ala
50 55 60
Gln Lys Pro Ser Gln Lys Asn His Leu Tyr
65 70
<210> 93
<211> 88
<212> PRT
<213> Homo sapiens
<400> 93
Gly Thr Thr Glu Glu Met Lys Tyr Val Leu Gly Gln Leu Val Gly Leu
1 5 10 15
Asn Ser Pro Asn Ser Ile Leu Lys Ala Ala Lys Thr Leu Tyr Glu His
20 25 30
Tyr Ser Gly Gly Glu Ser His Asn Ser Ser Ser Ser Lys Thr Phe Glu
35 40 45
Lys Lys Gly Glu Lys Asn Asp Leu Gln Leu Phe Val Met Ser Asp Thr
50 55 60
Thr Tyr Lys Ile Tyr Trp Thr Val Ile Leu Leu Asn Pro Cys Gly Asn
65 70 75 80
Leu His Leu Lys Thr Thr Ser Leu
85
<210> 94
<211> 55
<212> PRT
<213> Homo sapiens
<400> 94
Gln Gln Leu Ile Arg Glu Thr Leu Ile Ser Trp Leu Gln Ala Gln Met
1 5 10 15
Leu Asn Pro Gln Pro Glu Lys Thr Phe Ile Arg Asn Lys Ala Ala Gln
20 25 30
Val Phe Ala Leu Leu Phe Val Thr Glu Tyr Leu Thr Lys Trp Pro Lys
35 40 45
Phe Phe Leu Thr Phe Ser Gln
50 55
<210> 95
<211> 93
<212> PRT
<213> Homo sapiens
<400> 95
Ala Lys Phe Gln Gln Cys His Ser Thr Leu Glu Pro Asn Pro Ala Asp
1 5 10 15
Cys Arg Val Leu Val Tyr Leu Gln Asn Gln Pro Gly Thr Lys Leu Leu
20 25 30
Asn Phe Leu Gln Glu Arg Asn Leu Pro Pro Lys Val Val Leu Arg His
35 40 45
Pro Lys Val His Leu Asn Thr Met Phe Arg Arg Pro His Ser Cys Leu
50 55 60
Ala Asp Val Leu Leu Ser Val His Leu Ile Val Leu Arg Val Val Arg
65 70 75 80
Leu Pro Ala Pro Phe Arg Val Asn His Ala Val Glu Trp
85 90
<210> 96
<211> 53
<212> PRT
<213> Homo sapiens
<400> 96
Ala Pro Val Ile Phe Gln Ile Ala Leu Asp Lys Pro Cys His Gln Ala
1 5 10 15
Glu Val Lys His Leu His His Leu Leu Lys Gln Leu Lys Pro Ser Glu
20 25 30
Lys Tyr Leu Lys Ile Lys His Leu Leu Leu Lys Arg Glu Arg Val Asp
35 40 45
Leu Ser Lys Leu Gln
50
<210> 97
<211> 33
<212> PRT
<213> Homo sapiens
<400> 97
Met Leu Gln Phe Arg Gly Ser Arg Phe Phe Gln Met Leu Ile Leu Tyr
1 5 10 15
Tyr Ile Leu Pro Arg Lys Val Leu Gln Met Asp Phe Leu Val His Pro
20 25 30
Ala
<210> 98
<211> 179
<212> PRT
<213> Homo sapiens
<400> 98
Ala Leu Gly Pro His Ser Arg Ile Ser Cys Leu Pro Thr Gln Thr Arg
1 5 10 15
Gly Cys Ile Leu Leu Ala Ala Thr Pro Arg Ser Ser Ser Ser Ser Ser
20 25 30
Ser Asn Asp Met Ile Pro Met Ala Ile Ser Ser Pro Pro Lys Ala Pro
35 40 45
Leu Leu Ala Ala Pro Ser Pro Ala Ser Arg Leu Gln Cys Ile Asn Ser
50 55 60
Asn Ser Arg Ile Thr Ser Gly Gln Trp Met Ala His Met Ala Leu Leu
65 70 75 80
Pro Ser Gly Thr Lys Gly Arg Cys Thr Ala Cys His Thr Ala Leu Gly
85 90 95
Arg Gly Ser Leu Ser Ser Ser Ser Cys Pro Gln Pro Ser Pro Ser Leu
100 105 110
Pro Ala Ser Asn Lys Leu Pro Ser Leu Pro Leu Ser Lys Met Tyr Thr
115 120 125
Thr Ser Met Ala Met Pro Ile Leu Pro Leu Pro Gln Leu Leu Leu Ser
130 135 140
Ala Asp Gln Gln Ala Ala Pro Arg Thr Asn Phe His Ser Ser Leu Ala
145 150 155 160
Glu Thr Val Ser Leu His Pro Leu Ala Pro Met Pro Ser Lys Thr Cys
165 170 175
His His Lys
<210> 99
<211> 79
<212> PRT
<213> Homo sapiens
<400> 99
Ala His Gln Gly Phe Pro Ala Ala Lys Glu Ser Arg Val Ile Gln Leu
1 5 10 15
Ser Leu Leu Ser Leu Leu Ile Pro Pro Leu Thr Cys Leu Ala Ser Glu
20 25 30
Ala Leu Pro Arg Pro Leu Leu Ala Leu Pro Pro Val Leu Leu Ser Leu
35 40 45
Ala Gln Asp His Ser Arg Leu Leu Gln Cys Gln Ala Thr Arg Cys His
50 55 60
Leu Gly His Pro Val Ala Ser Arg Thr Ala Ser Cys Ile Leu Pro
65 70 75
<210> 100
<211> 222
<212> PRT
<213> Homo sapiens
<400> 100
Pro Ile Leu Ala Ala Thr Gly Thr Ser Val Arg Thr Ala Ala Arg Thr
1 5 10 15
Trp Val Pro Arg Ala Ala Ile Arg Val Pro Asp Pro Ala Ala Val Pro
20 25 30
Asp Asp His Ala Gly Pro Gly Ala Glu Cys His Gly Arg Pro Leu Leu
35 40 45
Tyr Thr Ala Asp Ser Ser Leu Trp Thr Thr Arg Pro Gln Arg Val Trp
50 55 60
Ser Thr Gly Pro Asp Ser Ile Leu Gln Pro Ala Lys Ser Ser Pro Ser
65 70 75 80
Ala Ala Ala Ala Thr Leu Leu Pro Ala Thr Thr Val Pro Asp Pro Ser
85 90 95
Cys Pro Thr Phe Val Ser Ala Ala Ala Thr Val Ser Thr Thr Thr Ala
100 105 110
Pro Val Leu Ser Ala Ser Ile Leu Pro Ala Ala Ile Pro Ala Ser Thr
115 120 125
Ser Ala Val Pro Gly Ser Ile Pro Leu Pro Ala Val Asp Asp Thr Ala
130 135 140
Ala Pro Pro Glu Pro Ala Pro Leu Leu Thr Ala Thr Gly Ser Val Ser
145 150 155 160
Leu Pro Ala Ala Ala Thr Ser Ala Ala Ser Thr Leu Asp Ala Leu Pro
165 170 175
Ala Gly Cys Val Ser Ser Ala Pro Val Ser Ala Val Pro Ala Asn Cys
180 185 190
Leu Phe Pro Ala Ala Leu Pro Ser Thr Ala Gly Ala Ile Ser Arg Phe
195 200 205
Ile Trp Val Ser Gly Ile Leu Ser Pro Leu Asn Asp Leu Gln
210 215 220
<210> 101
<211> 185
<212> PRT
<213> Homo sapiens
<400> 101
Pro Cys Arg Ala Gly Arg Arg Val Pro Trp Ala Ala Ser Leu Ile His
1 5 10 15
Ser Arg Phe Leu Leu Met Asp Asn Lys Ala Pro Ala Gly Met Val Asn
20 25 30
Arg Ala Arg Leu His Ile Thr Thr Ser Lys Val Leu Thr Leu Ser Ser
35 40 45
Ser Ser His Pro Thr Pro Ser Asn His Arg Pro Arg Pro Leu Met Pro
50 55 60
Asn Leu Arg Ile Ser Ser Ser His Ser Leu Asn His His Ser Ser Ser
65 70 75 80
Pro Leu Ser Leu His Thr Pro Ser Ser His Pro Ser Leu His Ile Ser
85 90 95
Ser Pro Arg Leu His Thr Pro Pro Ser Ser Arg Arg His Ser Ser Thr
100 105 110
Pro Arg Ala Ser Pro Pro Thr His Ser His Arg Leu Ser Leu Leu Thr
115 120 125
Ser Ser Ser Asn Leu Ser Ser Gln His Pro Arg Arg Ser Pro Ser Arg
130 135 140
Leu Arg Ile Leu Ser Pro Ser Leu Ser Ser Pro Ser Lys Leu Pro Ile
145 150 155 160
Pro Ser Ser Ala Ser Leu His Arg Arg Ser Tyr Leu Lys Ile His Leu
165 170 175
Gly Leu Arg His Pro Gln Pro Pro Gln
180 185
<210> 102
<211> 58
<212> PRT
<213> Homo sapiens
<400> 102
Arg Thr Asn Pro Thr Val Arg Met Arg Pro His Cys Val Pro Phe Trp
1 5 10 15
Thr Gly Arg Ile Leu Leu Pro Ser Ala Ala Ser Val Cys Pro Ile Pro
20 25 30
Phe Glu Ala Cys His Leu Cys Gln Ala Met Thr Leu Arg Cys Pro Asn
35 40 45
Thr Gln Gly Cys Cys Ser Ser Trp Ala Ser
50 55
<210> 103
<211> 90
<212> PRT
<213> Homo sapiens
<400> 103
Thr Asn Gln Ala Leu Pro Lys Ile Glu Val Ile Cys Arg Gly Thr Pro
1 5 10 15
Arg Cys Pro Ser Thr Val Pro Pro Ser Pro Ala Gln Pro Tyr Leu Arg
20 25 30
Val Ser Leu Pro Glu Asp Arg Tyr Thr Gln Ala Trp Ala Pro Thr Ser
35 40 45
Arg Thr Pro Trp Gly Ala Met Val Pro Arg Gly Val Ser Met Ala His
50 55 60
Lys Val Ala Thr Pro Gly Ser Gln Thr Ile Met Pro Cys Pro Met Pro
65 70 75 80
Thr Thr Pro Val Gln Ala Trp Leu Glu Ala
85 90
<210> 104
<211> 41
<212> PRT
<213> Homo sapiens
<400> 104
Arg Met Glu Arg Glu Leu Lys Lys Trp Ser Ile Gln Thr Cys Leu Ser
1 5 10 15
Ala Arg Thr Gly Leu Ser Ile Ser Cys Thr Thr Leu Asn Ser Pro Pro
20 25 30
Leu Lys Lys Met Ser Met Pro Ala Val
35 40
<210> 105
<211> 35
<212> PRT
<213> Homo sapiens
<400> 105
Leu Cys Ser Arg Tyr Ser Leu Phe Leu Ala Trp Arg Leu Ser Ser Val
1 5 10 15
Leu Gln Arg Phe Arg Phe Thr His Val Ile Gln Gln Arg Met Glu Ser
20 25 30
Gln Ile Ser
35
<210> 106
<211> 39
<212> PRT
<213> Homo sapiens
<400> 106
Arg Ser Ala Cys Val Thr Val Lys Gly Pro Leu Ala Ser Val Gly Arg
1 5 10 15
His Ser Leu Ser Lys Gln Asp Cys Lys Phe Leu Pro Phe Trp Gly Phe
20 25 30
Leu Glu Glu Phe Leu Leu Cys
35
<210> 107
<211> 97
<212> PRT
<213> Homo sapiens
<400> 107
Ile Gln Trp Gly Thr Thr Thr Ala Pro Arg Pro Ile Arg Pro Pro Phe
1 5 10 15
Leu Glu Ser Lys Gln Asn Cys Ser His Phe Pro Thr Pro Leu Leu Ala
20 25 30
Ser Glu Asp Arg Arg Glu Thr Gly Leu Phe Leu Pro Ser Ala Ala Gln
35 40 45
Lys Met Lys Lys Ala His Phe Leu Lys Thr Trp Phe Arg Ser Asn Pro
50 55 60
Thr Lys Thr Lys Lys Ala Arg Phe Ser Thr Ala Ser Leu Ala Lys Glu
65 70 75 80
Leu Thr His Pro Leu Leu Val Ser Leu Leu Leu Lys Glu Lys Gln Asp
85 90 95
Gly
<210> 108
<211> 73
<212> PRT
<213> Homo sapiens
<400> 108
Pro Thr Asp Pro Phe Leu Gly Leu Arg Leu Gly Leu His Leu Gln Lys
1 5 10 15
Val Phe His Gln Ser His Ala Glu Tyr Ser Gly Ala Pro Pro Pro Pro
20 25 30
Pro Ala Pro Ser Gly Leu Arg Phe Trp Asn Pro Ser Arg Ile Ala His
35 40 45
Ile Ser Gln Leu Leu Ser Trp Pro Gln Lys Thr Glu Glu Arg Leu Gly
50 55 60
Tyr Ser Ser His Gln Leu Pro Arg Lys
65 70
<210> 109
<211> 45
<212> PRT
<213> Homo sapiens
<400> 109
Phe Cys Cys Ser Cys Cys Phe Phe Gly Gly Glu Arg Trp Ser Lys Ser
1 5 10 15
Pro Tyr Cys Pro Gln Arg Met Thr Pro Gly Thr Thr Phe Ile Thr Met
20 25 30
Met Lys Lys Glu Ala Glu Lys Arg Thr Arg Thr Leu Thr
35 40 45
<210> 110
<211> 95
<212> PRT
<213> Homo sapiens
<400> 110
Trp Arg Arg Asn Cys Lys Ala Pro Val Ser Leu Arg Lys Ser Val Gln
1 5 10 15
Thr Pro Ala Arg Ser Ser Pro Ala Arg Pro Asp Arg Thr Arg Arg Leu
20 25 30
Pro Ser Leu Gly Val Pro Gly Gln Pro Trp Ala Leu Gly Ala Ala Ala
35 40 45
Ser Arg Arg Cys Cys Cys Cys Cys Arg Ser Pro Leu Gly Ser Ala Arg
50 55 60
Ser Arg Ser Pro Ala Thr Leu Ala Leu Thr Pro Arg Ala Thr Arg Ser
65 70 75 80
Arg Cys Pro Gly Ala Thr Trp Arg Glu Ala Ala Ser Trp Ala Glu
85 90 95
<210> 111
<211> 113
<212> PRT
<213> Homo sapiens
<400> 111
Pro Gly Arg Pro Leu Gln Thr His Val Leu Pro Glu Pro His Leu Ala
1 5 10 15
Leu Gln Pro Leu Gln Pro His Ala Asp His Ala His Ala Asp Ala Pro
20 25 30
Ala Ile Gln Pro Val Leu Trp Thr Thr Pro Pro Leu Gln His Gly His
35 40 45
Arg His Gly Leu Glu Pro Cys Ser Met Leu Thr Gly Pro Pro Ala Arg
50 55 60
Val Pro Ala Val Pro Phe Asp Leu His Phe Cys Arg Ser Ser Ile Met
65 70 75 80
Lys Pro Lys Arg Asp Gly Tyr Met Phe Leu Lys Ala Glu Ser Lys Ile
85 90 95
Met Phe Ala Thr Leu Gln Arg Ser Ser Leu Trp Cys Leu Cys Ser Asn
100 105 110
His
<210> 112
<211> 57
<212> PRT
<213> Homo sapiens
<400> 112
Pro Arg Pro Arg Arg Cys Thr Arg His Pro Ala Cys Pro Leu Asp His
1 5 10 15
Thr Thr Pro Pro Ala Trp Ser Pro Pro Trp Val Arg Ala Leu Leu Asp
20 25 30
Ala His Arg Ala Pro Ser Glu Ser Pro Cys Ser Pro Phe Arg Leu Ala
35 40 45
Phe Leu Gln Glu Gln Tyr His Glu Ala
50 55
<210> 113
<211> 494
<212> PRT
<213> Homo sapiens
<400> 113
Thr Arg Arg Cys His Cys Cys Pro His Leu Arg Ser His Pro Cys Pro
1 5 10 15
His His Leu Arg Asn His Pro Arg Pro His His Leu Arg His His Ala
20 25 30
Cys His His His Leu Arg Asn Cys Pro His Pro His Phe Leu Arg His
35 40 45
Cys Thr Cys Pro Gly Arg Trp Arg Asn Arg Pro Ser Leu Arg Arg Leu
50 55 60
Arg Ser Leu Leu Cys Leu Pro His Leu Asn His His Leu Phe Leu His
65 70 75 80
Trp Arg Ser Arg Pro Cys Leu His Arg Lys Ser His Pro His Leu Leu
85 90 95
His Leu Arg Arg Leu Tyr Pro His His Leu Lys His Arg Pro Cys Pro
100 105 110
His His Leu Lys Asn Leu Leu Cys Pro Arg His Leu Arg Asn Cys Pro
115 120 125
Leu Pro Arg His Leu Lys His Leu Ala Cys Leu His His Leu Arg Ser
130 135 140
His Pro Cys Pro Leu His Leu Lys Ser His Pro Cys Leu His His Arg
145 150 155 160
Arg His Leu Val Cys Ser His His Leu Lys Ser Leu Leu Cys Pro Leu
165 170 175
His Leu Arg Ser Leu Pro Phe Pro His His Leu Arg His His Ala Cys
180 185 190
Pro His His Leu Arg Thr Arg Leu Cys Pro His His Leu Lys Asn His
195 200 205
Leu Cys Pro Pro His Leu Arg Tyr Arg Ala Tyr Pro Pro Cys Leu Trp
210 215 220
Cys His Ala Cys Leu His Arg Leu Arg Asn Leu Pro Cys Pro His Arg
225 230 235 240
Leu Arg Ser Leu Pro Arg Pro Leu His Leu Arg Leu His Ala Ser Pro
245 250 255
His His Leu Arg Thr Pro Pro His Pro His His Leu Arg Thr His Leu
260 265 270
Leu Pro His His Arg Arg Thr Arg Ser Cys Pro Cys Arg Trp Arg Ser
275 280 285
His Pro Cys Cys His Tyr Leu Arg Ser Arg Asn Ser Ala Pro Gly Pro
290 295 300
Arg Gly Arg Thr Cys His Pro Gly Leu Arg Ser Arg Thr Cys Pro Pro
305 310 315 320
Gly Leu Arg Ser His Thr Tyr Leu Arg Arg Leu Arg Ser His Thr Cys
325 330 335
Pro Pro Ser Leu Arg Ser His Ala Tyr Ala Leu Cys Leu Arg Ser His
340 345 350
Thr Cys Pro Pro Arg Leu Arg Asp His Ile Cys Pro Leu Ser Leu Arg
355 360 365
Asn Cys Thr Cys Pro Pro Arg Leu Arg Ser Arg Thr Cys Leu Leu Cys
370 375 380
Leu Arg Ser His Ala Cys Pro Pro Asn Leu Arg Asn His Thr Cys Pro
385 390 395 400
Pro Ser Leu Arg Ser His Ala Cys Pro Pro Gly Leu Arg Asn Arg Ile
405 410 415
Cys Pro Leu Ser Leu Arg Ser His Pro Cys Pro Leu Gly Leu Lys Ser
420 425 430
Pro Leu Arg Ser Gln Ala Asn Ala Leu His Leu Arg Ser Cys Pro Cys
435 440 445
Ser Leu Pro Leu Gly Asn His Pro Tyr Leu Pro Cys Leu Glu Ser Gln
450 455 460
Pro Cys Leu Ser Leu Gly Asn His Leu Cys Pro Leu Cys Pro Arg Ser
465 470 475 480
Cys Arg Cys Pro His Leu Gly Ser His Pro Cys Arg Leu Ser
485 490
<210> 114
<211> 53
<212> PRT
<213> Homo sapiens
<400> 114
Ser Trp Lys Gly Thr Asn Trp Cys Asn Asp Met Cys Ile Phe Ile Thr
1 5 10 15
Ser Gly Gln Ile Phe Lys Gly Thr Arg Gly Pro Arg Phe Leu Trp Gly
20 25 30
Ser Lys Asp Gln Arg Gln Lys Gly Ser Asn Tyr Ser Gln Ser Glu Ala
35 40 45
Leu Cys Val Leu Leu
50
<210> 115
<211> 9
<212> PRT
<213> Homo sapiens
<400> 115
Lys Arg Thr Lys Cys Phe Thr Phe Gly
1 5
<210> 116
<211> 27
<212> PRT
<213> Homo sapiens
<400> 116
Pro Ile Phe Ile Gln Thr Leu Leu Leu Trp Asp Phe Leu Gln Lys Asp
1 5 10 15
Leu Lys Ala Tyr Thr Gly Thr Ile Leu Met Met
20 25
<210> 117
<211> 22
<212> PRT
<213> Homo sapiens
<400> 117
Gln Lys Met Ile Leu Thr Lys Gln Ile Lys Thr Lys Pro Thr Asp Thr
1 5 10 15
Phe Leu Gln Ile Leu Arg
20
<210> 118
<211> 43
<212> PRT
<213> Homo sapiens
<400> 118
Gly Phe Trp Ile Gln Ser Ile Lys Thr Ile Thr Arg Tyr Thr Ile Phe
1 5 10 15
Val Leu Lys Asp Ile Met Thr Pro Pro Asn Leu Ile Ala Glu Leu His
20 25 30
Asn Ile Leu Leu Lys Thr Ile Thr His His Ser
35 40
<210> 119
<211> 40
<212> PRT
<213> Homo sapiens
<400> 119
Asn Tyr Ser Asn Val Gln Trp Arg Asn Leu Gln Ser Ser Val Cys Gly
1 5 10 15
Leu Pro Ala Lys Gly Glu Asp Ile Phe Leu Gln Phe Arg Thr His Thr
20 25 30
Thr Gly Arg Gln Val His Val Leu
35 40
<210> 120
<211> 44
<212> PRT
<213> Homo sapiens
<400> 120
Tyr Gln Ser Arg Val Leu Pro Gln Thr Glu Gln Asp Ala Lys Lys Gly
1 5 10 15
Gln Asn Val Ser Leu Leu Gly Lys Tyr Ile Leu His Thr Arg Thr Arg
20 25 30
Gly Asn Leu Arg Lys Ser Arg Lys Trp Lys Ser Met
35 40
<210> 121
<211> 85
<212> PRT
<213> Homo sapiens
<400> 121
Ser Ser Gln Asn Ala Arg Gly Cys Ser Pro Arg Gly Pro Cys Thr Ser
1 5 10 15
Ser Ser Tyr Thr Gly Gly Pro Cys Thr Ser Pro Leu Leu Ala Pro Val
20 25 30
Ile Phe Cys Pro Phe Pro Glu Asn Leu Pro Gly Gln Leu Arg Phe Pro
35 40 45
Ser Gly Leu Leu Ala Phe Trp Asp Ser Gln Val Cys Asp Leu His Val
50 55 60
Leu Pro Cys Pro Gln Gln Asp Val Leu Pro Thr Gly Gln Asp Leu Pro
65 70 75 80
Cys Ala Ala Val Gly
85
<210> 122
<211> 72
<212> PRT
<213> Homo sapiens
<400> 122
Gly Ala Ala Pro Thr Met Ser Ala Ala Gln Ile Ala Met Val Trp Pro
1 5 10 15
Leu Leu Ser Ile Leu Ser Glu Trp Lys Glu Ile Cys Val Trp Ser Ile
20 25 30
Trp Met Thr Glu Thr Leu Phe Asp Ile Val Trp Trp Cys Pro Met Ser
35 40 45
Arg Leu Arg Leu Ala Leu Thr Val Pro Pro Ser Thr Thr Thr Thr Cys
50 55 60
Val Thr Val Pro Ala Trp Ala Ala
65 70
<210> 123
<211> 97
<212> PRT
<213> Homo sapiens
<400> 123
Thr Gly Gly Pro Ser Ser Pro Ser Ser His Trp Lys Thr Pro Val Val
1 5 10 15
Ile Tyr Trp Asp Gly Thr Ala Leu Arg Cys Val Phe Val Pro Val Leu
20 25 30
Gly Glu Thr Gly Ala Gln Arg Lys Arg Ile Ser Ala Arg Lys Gly Ser
35 40 45
Leu Thr Thr Ser Cys Pro Gln Gly Ala Leu Ser Glu His Cys Pro Thr
50 55 60
Thr Pro Ala Pro Leu Pro Ser Gln Arg Arg Asn His Trp Met Glu Asn
65 70 75 80
Ile Ser Pro Phe Arg Ser Val Gly Val Ser Ala Ser Arg Cys Ser Glu
85 90 95
Ser
<210> 124
<211> 31
<212> PRT
<213> Homo sapiens
<400> 124
Phe His Thr Pro Ala Arg His Pro Arg Pro Arg His Gly His Leu Gln
1 5 10 15
Ala Val Thr Ala His Asp Gly Gly Cys Glu Ala Leu Pro Pro Pro
20 25 30
<210> 125
<211> 78
<212> PRT
<213> Homo sapiens
<400> 125
Cys Cys Pro Arg Thr Ile Leu Asn Asn Gly Ser Leu Lys Thr Gln Val
1 5 10 15
Gln Met Lys Leu Pro Glu Cys Gln Arg Leu Leu Pro Pro Trp Pro Leu
20 25 30
His Gln Gln Leu Leu His Arg Arg Pro Leu His Gln Pro Pro Pro Gly
35 40 45
Pro Cys His Leu Leu Ser Leu Pro Arg Lys Pro Thr Arg Ala Ala Thr
50 55 60
Val Ser Val Trp Ala Ser Cys Ile Leu Gly Gln Pro Ser Leu
65 70 75
<210> 126
<211> 28
<212> PRT
<213> Homo sapiens
<400> 126
Val Arg Lys His Phe Gln Thr Tyr Gly Asn Tyr Phe Leu Lys Thr Thr
1 5 10 15
Phe Cys Pro Pro Cys Arg Pro Lys Gln Trp Met Ile
20 25
<210> 127
<211> 46
<212> PRT
<213> Homo sapiens
<400> 127
Leu Ala Arg Thr Pro Leu Pro Ser Thr Arg Cys Phe Ala Asn Trp Pro
1 5 10 15
Arg Pro Ala Leu Cys Ser Cys Gly Leu Ile Pro His Pro Arg Pro Ala
20 25 30
Pro Ala Ser Ala Pro Trp Pro Ser Thr Ser Ser His Ser Thr
35 40 45
<210> 128
<211> 81
<212> PRT
<213> Homo sapiens
<400> 128
Glu Leu Gln Glu Thr Gly His Arg Gln Val Ala Leu Arg Arg Ser Gly
1 5 10 15
Arg Pro Pro Lys Cys Ala Glu Arg Pro Gly Ala Ala Asp Thr Gly Ala
20 25 30
His Cys Thr Ser Thr Asp Gly Arg Leu Lys Ile Ser Val Glu Thr Tyr
35 40 45
Thr Val Ser Ser Gln Leu Leu Met Val Leu Met Ser Leu Asp Leu Asp
50 55 60
Thr Gly Leu Val Pro Ser Leu Val Ser Lys Cys Leu Ile Leu Arg Val
65 70 75 80
Lys
<210> 129
<211> 10
<212> PRT
<213> Homo sapiens
<400> 129
Lys Ser Asp Ala Ser Arg Leu Ser Gly Ala
1 5 10
<210> 130
<211> 29
<212> PRT
<213> Homo sapiens
<400> 130
Arg Thr Ala Tyr Phe Cys Gln Tyr His Thr Ala Ser Val Tyr Ser Glu
1 5 10 15
Arg Ala Met Pro Pro Gly Cys Pro Glu Pro Ser Gln Ala
20 25
<210> 131
<211> 91
<212> PRT
<213> Homo sapiens
<400> 131
Thr Arg Ala Ser Pro Pro Arg Ser Ser Ser Ala Ile Ala Val Arg Ala
1 5 10 15
Ser Cys Cys Pro Tyr Gly Ser Thr Ser Thr Ala Ser Arg Ser Pro Thr
20 25 30
Gln Arg Cys Arg Leu Ala Arg Ala Ala Ala Ser Thr Ala Thr Glu Val
35 40 45
Thr Phe Gly Ser Ser Glu Met Gln Gly His Thr Met Gly Phe Trp Leu
50 55 60
Thr Lys Leu Asn Tyr Leu Cys His Leu Ser Met Leu Thr Asp Ser Leu
65 70 75 80
Phe Leu Pro Ile Ser His Cys Gln Cys Ile Leu
85 90
<210> 132
<211> 31
<212> PRT
<213> Homo sapiens
<400> 132
Ser Ser Leu Arg Ile Thr Gly Asp Trp Thr Ser Ser Gly Arg Ser Thr
1 5 10 15
Lys Ile Trp Lys Thr Thr Gln Met Cys Arg Lys Thr Trp Ser Gly
20 25 30
<210> 133
<211> 104
<212> PRT
<213> Homo sapiens
<400> 133
Arg Arg Arg Arg Gly Gly Val Gly Arg Arg Gly Val Arg Pro Gly Arg
1 5 10 15
Val Arg Pro Gly Gly Thr Gly Arg Arg Gly Gly Asp Gly Gly Arg Ala
20 25 30
Ala Ala Ala Arg Ala Ala Leu Gly Glu Leu Ala Arg Ala Leu Pro Gly
35 40 45
His Leu Leu Gln Ser Gln Ser Ala Arg Arg Ala Ala Arg Met Ala Gln
50 55 60
Leu Arg Arg Arg Ala Ala Ala Leu Pro Asn Ala Ala Ala Trp His Gly
65 70 75 80
Pro Pro His Pro Gln Leu Pro Arg Ser Pro Leu Ala Leu Gln Arg Cys
85 90 95
Arg Asp Thr Arg Trp Ala Ser Gly
100
<210> 134
<211> 66
<212> PRT
<213> Homo sapiens
<400> 134
Ser Cys Lys Val Phe Phe Lys Arg Ala Ala Glu Gly Lys Gln Lys Tyr
1 5 10 15
Leu Cys Ala Ser Arg Asn Asp Cys Thr Ile Asp Lys Phe Arg Arg Lys
20 25 30
Asn Cys Pro Ser Cys Arg Leu Arg Lys Cys Tyr Glu Ala Gly Met Thr
35 40 45
Leu Gly Glu Lys Phe Arg Val Gly Asn Cys Lys His Leu Lys Met Thr
50 55 60
Arg Pro
65
<210> 135
<211> 49
<212> PRT
<213> Homo sapiens
<400> 135
Met Ala Gly Ala Pro Pro Pro Ala Ser Leu Pro Pro Cys Ser Leu Ile
1 5 10 15
Ser Asp Cys Cys Ala Ser Asn Gln Arg Asp Ser Val Gly Val Gly Pro
20 25 30
Ser Glu Pro Gly Asn Asn Ile Lys Ile Cys Asn Glu Ser Ala Ser Arg
35 40 45
Lys
<210> 136
<211> 61
<212> PRT
<213> Homo sapiens
<400> 136
His Gly Trp Arg Pro Phe Leu Pro Val Arg Ala Arg Ser Arg Trp Asn
1 5 10 15
Arg Arg Leu Asp Val Thr Val Ala Asn Gly Arg Ser Trp Lys Tyr Gly
20 25 30
Trp Ser Leu Leu Arg Val Pro Gln Val Asn Gly Ile Gln Val Leu Asn
35 40 45
Val Ser Leu Lys Ser Ser Ser Asn Val Ile Ser Tyr Glu
50 55 60
<210> 137
<211> 65
<212> PRT
<213> Homo sapiens
<400> 137
Arg Leu Lys Glu Val Phe Gln Thr Lys Ile Gln Glu Phe Arg Lys Ala
1 5 10 15
Cys Tyr Thr Leu Thr Gly Tyr Gln Ile Asp Ile Thr Thr Glu Asn Gln
20 25 30
Tyr Arg Leu Thr Ser Leu Tyr Ala Glu His Pro Gly Asp Cys Leu Ile
35 40 45
Phe Lys Leu Arg Val Pro Gly Ser Ser Val Leu Val Thr Val Pro Gly
50 55 60
Leu
65
<210> 138
<211> 107
<212> PRT
<213> Homo sapiens
<400> 138
Ala Glu Val Leu Lys Val Ile Arg Gln Ser Ala Gly Gln Lys Thr Thr
1 5 10 15
Cys Gly Gln Gly Leu Glu Gly Pro Trp Glu Arg Pro Pro Pro Leu Asp
20 25 30
Glu Ser Glu Arg Asp Gly Gly Ser Glu Asp Gln Val Glu Asp Pro Ala
35 40 45
Leu Ser Ala Leu Leu Leu Arg Pro Arg Pro Pro Arg Pro Glu Val Gly
50 55 60
Ala His Gln Asp Glu Gln Ala Ala Gln Gly Ala Asp Pro Arg Leu Gly
65 70 75 80
Ala Gln Pro Ala Cys Arg Gly Leu Pro Gly Leu Leu Thr Val Pro Gln
85 90 95
Pro Glu Pro Leu Leu Ala Pro Pro Ser Ala Ala
100 105
<210> 139
<211> 100
<212> PRT
<213> Homo sapiens
<400> 139
Glu Arg Ala Glu Trp Arg Glu Asn Ile Arg Glu Gln Gln Lys Lys Cys
1 5 10 15
Phe Arg Ser Phe Ser Leu Thr Ser Val Glu Leu Gln Met Leu Thr Asn
20 25 30
Ser Cys Val Lys Leu Gln Thr Val His Ser Ile Pro Leu Thr Ile Asn
35 40 45
Lys Glu Glu Ala Leu Gln Arg Pro Val Ala Ser Asp Phe Glu Pro Gln
50 55 60
Gly Leu Ser Glu Ala Ala Arg Trp Asn Ser Lys Glu Asn Leu Leu Ala
65 70 75 80
Gly Pro Ser Glu Asn Asp Pro Asn Leu Phe Val Ala Leu Tyr Asp Phe
85 90 95
Val Ala Ser Gly
100
<210> 140
<211> 101
<212> PRT
<213> Homo sapiens
<400> 140
Glu Leu Gln Met Leu Thr Asn Ser Cys Val Lys Leu Gln Thr Val His
1 5 10 15
Ser Ile Pro Leu Thr Ile Asn Lys Glu Asp Asp Glu Ser Pro Gly Leu
20 25 30
Tyr Gly Phe Leu Asn Val Ile Val His Ser Ala Thr Gly Phe Lys Gln
35 40 45
Ser Ser Lys Ala Leu Gln Arg Pro Val Ala Ser Asp Phe Glu Pro Gln
50 55 60
Gly Leu Ser Glu Ala Ala Arg Trp Asn Ser Lys Glu Asn Leu Leu Ala
65 70 75 80
Gly Pro Ser Glu Asn Asp Pro Asn Leu Phe Val Ala Leu Tyr Asp Phe
85 90 95
Val Ala Ser Gly Asp
100
<210> 141
<211> 101
<212> PRT
<213> Homo sapiens
<400> 141
Ile Ser Asn Ser Trp Asp Ala His Leu Gly Leu Gly Ala Cys Gly Glu
1 5 10 15
Ala Glu Gly Leu Gly Val Gln Gly Ala Glu Glu Glu Glu Glu Glu Glu
20 25 30
Glu Glu Glu Glu Glu Glu Gly Ala Gly Val Pro Ala Cys Pro Pro Lys
35 40 45
Gly Pro Glu Leu Phe Pro Leu Ile Phe Pro Ala Glu Pro Ala Gln Ala
50 55 60
Ser Gly Pro Tyr Val Glu Ile Ile Glu Gln Pro Lys Gln Arg Gly Met
65 70 75 80
Arg Phe Arg Tyr Lys Cys Glu Gly Arg Ser Ala Gly Ser Ile Pro Gly
85 90 95
Glu Arg Ser Thr Asp
100
<210> 142
<211> 100
<212> PRT
<213> Homo sapiens
<400> 142
Leu Gln Arg Leu Asp Gly Met Gly Cys Leu Glu Phe Asp Glu Glu Arg
1 5 10 15
Ala Gln Gln Glu Asp Ala Leu Ala Gln Gln Ala Phe Glu Glu Ala Arg
20 25 30
Arg Arg Thr Arg Glu Phe Glu Asp Arg Asp Arg Ser His Arg Glu Glu
35 40 45
Met Glu Val His Glu Leu Glu Lys Ser Lys Arg Ala Leu Glu Thr Gln
50 55 60
Met Glu Glu Met Lys Thr Gln Leu Glu Glu Leu Glu Asp Glu Leu Gln
65 70 75 80
Ala Thr Glu Asp Ala Lys Leu Arg Leu Glu Val Asn Met Gln Ala Leu
85 90 95
Lys Gly Gln Phe
100
<210> 143
<211> 100
<212> PRT
<213> Homo sapiens
<400> 143
Lys Gly Ser Phe Pro Glu Asn Leu Arg His Leu Lys Asn Thr Met Glu
1 5 10 15
Thr Ile Asp Trp Lys Val Phe Glu Ser Trp Met His His Trp Leu Leu
20 25 30
Phe Glu Met Ser Arg His Ser Leu Glu Gln Lys Pro Thr Asp Ala Pro
35 40 45
Pro Lys Ala Gly Val Pro Asn Lys Pro Gly Ile Pro Lys Leu Leu Glu
50 55 60
Gly Ser Lys Asn Ser Ile Gln Trp Glu Lys Ala Glu Asp Asn Gly Cys
65 70 75 80
Arg Ile Thr Tyr Tyr Ile Leu Glu Ile Arg Lys Ser Thr Ser Asn Asn
85 90 95
Leu Gln Asn Gln
100
<210> 144
<211> 80
<212> PRT
<213> Homo sapiens
<400> 144
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Gly Asn Tyr
20 25 30
Val Val Thr Asp His Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser
35 40 45
Tyr Glu Met Glu Glu Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly
50 55 60
Pro Cys Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp
65 70 75 80
<210> 145
<211> 100
<212> PRT
<213> Homo sapiens
<400> 145
Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr Met Ile Met Val
1 5 10 15
Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys Phe Arg Glu Leu
20 25 30
Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln Arg Tyr Leu Val
35 40 45
Ile Gln Leu Gln Asp Lys Phe Glu His Leu Lys Met Ile Gln Gln Glu
50 55 60
Glu Ile Arg Lys Leu Glu Glu Glu Lys Lys Gln Leu Glu Gly Glu Ile
65 70 75 80
Ile Asp Phe Tyr Lys Met Lys Ala Ala Ser Glu Ala Leu Gln Thr Gln
85 90 95
Leu Ser Thr Asp
100
<210> 146
<211> 101
<212> PRT
<213> Homo sapiens
<400> 146
Ser Trp Glu Asn Ser Asp Asp Ser Arg Asn Lys Leu Ser Lys Ile Pro
1 5 10 15
Ser Thr Pro Lys Leu Ile Pro Lys Val Thr Lys Thr Ala Asp Lys His
20 25 30
Lys Asp Val Ile Ile Asn Gln Ala Lys Met Ser Thr Arg Glu Lys Asn
35 40 45
Ser Gln Val Tyr Arg Arg Lys His Gln Glu Leu Gln Ala Met Gln Met
50 55 60
Glu Leu Gln Ser Pro Glu Tyr Lys Leu Ser Lys Leu Arg Thr Ser Thr
65 70 75 80
Ile Met Thr Asp Tyr Asn Pro Asn Tyr Cys Phe Ala Gly Lys Thr Ser
85 90 95
Ser Ile Ser Asp Leu
100
<210> 147
<211> 100
<212> PRT
<213> Homo sapiens
<400> 147
Glu Gly His Arg Met Asp Lys Pro Ala Asn Cys Thr His Asp Leu Tyr
1 5 10 15
Met Ile Met Arg Glu Cys Trp His Ala Ala Pro Ser Gln Arg Pro Thr
20 25 30
Phe Lys Gln Leu Val Glu Asp Leu Asp Arg Val Leu Thr Val Thr Ser
35 40 45
Thr Asp Val Lys Ala Thr Gln Glu Glu Asn Arg Glu Leu Arg Ser Arg
50 55 60
Cys Glu Glu Leu His Gly Lys Asn Leu Glu Leu Gly Lys Ile Met Asp
65 70 75 80
Arg Phe Glu Glu Val Val Tyr Gln Ala Met Glu Glu Val Gln Lys Gln
85 90 95
Lys Glu Leu Ser
100
<210> 148
<211> 107
<212> PRT
<213> Homo sapiens
<400> 148
Arg Asp Asn Thr Leu Leu Leu Arg Arg Val Glu Leu Phe Ser Leu Ser
1 5 10 15
Arg Gln Val Ala Arg Glu Ser Thr Tyr Leu Ser Ser Leu Lys Gly Ser
20 25 30
Arg Leu His Pro Glu Glu Leu Gly Gly Pro Pro Leu Lys Lys Leu Lys
35 40 45
Gln Glu Ala Thr Ser Lys Ser Gln Ile Met Ser Leu Trp Gly Leu Val
50 55 60
Ser Lys Met Pro Pro Glu Lys Val Gln Arg Leu Tyr Val Asp Phe Pro
65 70 75 80
Gln His Leu Arg His Leu Leu Gly Asp Trp Leu Glu Ser Gln Pro Trp
85 90 95
Glu Phe Leu Val Gly Ser Asp Ala Phe Cys Cys
100 105
<210> 149
<211> 83
<212> PRT
<213> Homo sapiens
<400> 149
Met Ser Arg Glu Met Gln Asp Val Asp Leu Ala Glu Val Lys Pro Leu
1 5 10 15
Val Glu Lys Gly Glu Thr Ile Thr Gly Leu Leu Gln Glu Phe Asp Val
20 25 30
Gln Glu Ala Leu Ser Val Val Ser Glu Asp Gln Ser Leu Phe Glu Cys
35 40 45
Ala Tyr Gly Thr Pro His Leu Ala Lys Thr Glu Met Thr Ala Ser Ser
50 55 60
Ser Ser Asp Tyr Gly Gln Thr Ser Lys Met Ser Pro Arg Val Pro Gln
65 70 75 80
Gln Asp Trp
<210> 150
<211> 101
<212> PRT
<213> Homo sapiens
<400> 150
Val Leu Asp Met His Gly Phe Leu Arg Gln Ala Leu Cys Arg Leu Arg
1 5 10 15
Gln Glu Glu Pro Gln Ser Leu Gln Ala Ala Val Arg Thr Asp Gly Phe
20 25 30
Asp Glu Phe Lys Val Arg Leu Gln Asp Leu Ser Ser Cys Ile Thr Gln
35 40 45
Gly Lys Ala Ile Glu Thr Gln Ser Ser Ser Ser Glu Glu Ile Val Pro
50 55 60
Ser Pro Pro Ser Pro Pro Pro Leu Pro Arg Ile Tyr Lys Pro Cys Phe
65 70 75 80
Val Cys Gln Asp Lys Ser Ser Gly Tyr His Tyr Gly Val Ser Ala Cys
85 90 95
Glu Gly Cys Lys Gly
100
<210> 151
<211> 101
<212> PRT
<213> Homo sapiens
<400> 151
Arg Ser Ser Pro Glu Gln Pro Arg Pro Ser Thr Ser Lys Ala Val Ser
1 5 10 15
Pro Pro His Leu Asp Gly Pro Pro Ser Pro Arg Ser Pro Val Ile Gly
20 25 30
Ser Glu Val Phe Leu Pro Asn Ser Asn His Val Ala Ser Gly Ala Gly
35 40 45
Glu Ala Ala Ile Glu Thr Gln Ser Ser Ser Ser Glu Glu Ile Val Pro
50 55 60
Ser Pro Pro Ser Pro Pro Pro Leu Pro Arg Ile Tyr Lys Pro Cys Phe
65 70 75 80
Val Cys Gln Asp Lys Ser Ser Gly Tyr His Tyr Gly Val Ser Ala Cys
85 90 95
Glu Gly Cys Lys Gly
100
<210> 152
<211> 101
<212> PRT
<213> Homo sapiens
<400> 152
Val Ala Arg Phe Asn Asp Leu Arg Phe Val Gly Arg Ser Gly Arg Gly
1 5 10 15
Lys Ser Phe Thr Leu Thr Ile Thr Val Phe Thr Asn Pro Pro Gln Val
20 25 30
Ala Thr Tyr His Arg Ala Ile Lys Ile Thr Val Asp Gly Pro Arg Glu
35 40 45
Pro Arg Asn Arg Thr Glu Lys His Ser Thr Met Pro Asp Ser Pro Val
50 55 60
Asp Val Lys Thr Gln Ser Arg Leu Thr Pro Pro Thr Met Pro Pro Pro
65 70 75 80
Pro Thr Thr Gln Gly Ala Pro Arg Thr Ser Ser Phe Thr Pro Thr Thr
85 90 95
Leu Thr Asn Gly Thr
100
<210> 153
<211> 55
<212> PRT
<213> Homo sapiens
<400> 153
Met Ala Leu Asn Ser Glu Ala Leu Ser Val Val Ser Glu Asp Gln Ser
1 5 10 15
Leu Phe Glu Cys Ala Tyr Gly Thr Pro His Leu Ala Lys Thr Glu Met
20 25 30
Thr Ala Ser Ser Ser Ser Asp Tyr Gly Gln Thr Ser Lys Met Ser Pro
35 40 45
Arg Val Pro Gln Gln Asp Trp
50 55
<210> 154
<211> 10
<212> PRT
<213> Homo sapiens
<400> 154
Lys Leu Val Val Val Gly Ala Cys Gly Val
1 5 10
<210> 155
<211> 9
<212> PRT
<213> Homo sapiens
<400> 155
Leu Val Val Val Gly Ala Cys Gly Val
1 5
<210> 156
<211> 9
<212> PRT
<213> Homo sapiens
<400> 156
Val Val Gly Ala Cys Gly Val Gly Lys
1 5
<210> 157
<211> 10
<212> PRT
<213> Homo sapiens
<400> 157
Val Val Val Gly Ala Cys Gly Val Gly Lys
1 5 10
<210> 158
<211> 9
<212> PRT
<213> Homo sapiens
<400> 158
Val Val Gly Ala Asp Gly Val Gly Lys
1 5
<210> 159
<211> 10
<212> PRT
<213> Homo sapiens
<400> 159
Val Val Val Gly Ala Asp Gly Val Gly Lys
1 5 10
<210> 160
<211> 10
<212> PRT
<213> Homo sapiens
<400> 160
Lys Leu Val Val Val Gly Ala Asp Gly Val
1 5 10
<210> 161
<211> 9
<212> PRT
<213> Homo sapiens
<400> 161
Leu Val Val Val Gly Ala Asp Gly Val
1 5
<210> 162
<211> 10
<212> PRT
<213> Homo sapiens
<400> 162
Lys Leu Val Val Val Gly Ala Val Gly Val
1 5 10
<210> 163
<211> 9
<212> PRT
<213> Homo sapiens
<400> 163
Leu Val Val Val Gly Ala Val Gly Val
1 5
<210> 164
<211> 9
<212> PRT
<213> Homo sapiens
<400> 164
Val Val Gly Ala Val Gly Val Gly Lys
1 5
<210> 165
<211> 10
<212> PRT
<213> Homo sapiens
<400> 165
Ile Leu Asp Thr Ala Gly His Glu Glu Tyr
1 5 10
<210> 166
<211> 10
<212> PRT
<213> Homo sapiens
<400> 166
Ile Leu Asp Thr Ala Gly Leu Glu Glu Tyr
1 5 10
<210> 167
<211> 10
<212> PRT
<213> Homo sapiens
<400> 167
Leu Leu Asp Ile Leu Asp Thr Ala Gly Leu
1 5 10
<210> 168
<211> 10
<212> PRT
<213> Homo sapiens
<400> 168
Ile Leu Asp Thr Ala Gly Lys Glu Glu Tyr
1 5 10
<210> 169
<211> 10
<212> PRT
<213> Homo sapiens
<400> 169
Ile Leu Asp Thr Ala Gly Arg Glu Glu Tyr
1 5 10
<210> 170
<211> 9
<212> PRT
<213> Homo sapiens
<400> 170
Glu Tyr Met Ala Asn Gly Ser Leu Leu
1 5
<210> 171
<211> 9
<212> PRT
<213> Homo sapiens
<400> 171
Met Ala Asn Gly Ser Leu Leu Asn Tyr
1 5
<210> 172
<211> 10
<212> PRT
<213> Homo sapiens
<400> 172
Met Ala Asn Gly Ser Leu Leu Asn Tyr Leu
1 5 10
<210> 173
<211> 9
<212> PRT
<213> Homo sapiens
<400> 173
Ser Leu Leu Asn Tyr Leu Arg Glu Met
1 5
<210> 174
<211> 9
<212> PRT
<213> Homo sapiens
<400> 174
Tyr Met Ala Asn Gly Ser Leu Leu Asn
1 5
<210> 175
<211> 10
<212> PRT
<213> Homo sapiens
<400> 175
Tyr Met Ala Asn Gly Ser Leu Leu Asn Tyr
1 5 10
<210> 176
<211> 11
<212> PRT
<213> Homo sapiens
<400> 176
Ile Ile Arg Asn Arg Gly Glu Asn Ser Cys Lys
1 5 10
<210> 177
<211> 10
<212> PRT
<213> Homo sapiens
<400> 177
Cys Leu Thr Ser Thr Val Gln Leu Ile Met
1 5 10
<210> 178
<211> 9
<212> PRT
<213> Homo sapiens
<400> 178
Ile Met Gln Leu Met Pro Phe Gly Cys
1 5
<210> 179
<211> 10
<212> PRT
<213> Homo sapiens
<400> 179
Ile Met Gln Leu Met Pro Phe Gly Cys Leu
1 5 10
<210> 180
<211> 9
<212> PRT
<213> Homo sapiens
<400> 180
Leu Ile Met Gln Leu Met Pro Phe Gly
1 5
<210> 181
<211> 10
<212> PRT
<213> Homo sapiens
<400> 181
Leu Ile Met Gln Leu Met Pro Phe Gly Cys
1 5 10
<210> 182
<211> 9
<212> PRT
<213> Homo sapiens
<400> 182
Leu Thr Ser Thr Val Gln Leu Ile Met
1 5
<210> 183
<211> 9
<212> PRT
<213> Homo sapiens
<400> 183
Met Gln Leu Met Pro Phe Gly Cys Leu
1 5
<210> 184
<211> 10
<212> PRT
<213> Homo sapiens
<400> 184
Met Gln Leu Met Pro Phe Gly Cys Leu Leu
1 5 10
<210> 185
<211> 9
<212> PRT
<213> Homo sapiens
<400> 185
Gln Leu Ile Met Gln Leu Met Pro Phe
1 5
<210> 186
<211> 10
<212> PRT
<213> Homo sapiens
<400> 186
Gln Leu Ile Met Gln Leu Met Pro Phe Gly
1 5 10
<210> 187
<211> 9
<212> PRT
<213> Homo sapiens
<400> 187
Ser Thr Val Gln Leu Ile Met Gln Leu
1 5
<210> 188
<211> 10
<212> PRT
<213> Homo sapiens
<400> 188
Val Gln Leu Ile Met Gln Leu Met Pro Phe
1 5 10
<210> 189
<211> 9
<212> PRT
<213> Homo sapiens
<400> 189
Gly Gln Tyr Gly Lys Val Tyr Glu Gly
1 5
<210> 190
<211> 10
<212> PRT
<213> Homo sapiens
<400> 190
Gly Gln Tyr Gly Lys Val Tyr Glu Gly Val
1 5 10
<210> 191
<211> 9
<212> PRT
<213> Homo sapiens
<400> 191
Lys Leu Gly Gly Gly Gln Tyr Gly Lys
1 5
<210> 192
<211> 10
<212> PRT
<213> Homo sapiens
<400> 192
Lys Leu Gly Gly Gly Gln Tyr Gly Lys Val
1 5 10
<210> 193
<211> 9
<212> PRT
<213> Homo sapiens
<400> 193
Lys Val Tyr Glu Gly Val Trp Lys Lys
1 5
<210> 194
<211> 10
<212> PRT
<213> Homo sapiens
<400> 194
Lys Val Tyr Glu Gly Val Trp Lys Lys Tyr
1 5 10
<210> 195
<211> 9
<212> PRT
<213> Homo sapiens
<400> 195
Gln Tyr Gly Lys Val Tyr Glu Gly Val
1 5
<210> 196
<211> 10
<212> PRT
<213> Homo sapiens
<400> 196
Gln Tyr Gly Lys Val Tyr Glu Gly Val Trp
1 5 10
<210> 197
<211> 9
<212> PRT
<213> Homo sapiens
<400> 197
Gly Gln Tyr Gly Val Val Tyr Glu Gly
1 5
<210> 198
<211> 10
<212> PRT
<213> Homo sapiens
<400> 198
Gly Gln Tyr Gly Val Val Tyr Glu Gly Val
1 5 10
<210> 199
<211> 9
<212> PRT
<213> Homo sapiens
<400> 199
Lys Leu Gly Gly Gly Gln Tyr Gly Val
1 5
<210> 200
<211> 10
<212> PRT
<213> Homo sapiens
<400> 200
Lys Leu Gly Gly Gly Gln Tyr Gly Val Val
1 5 10
<210> 201
<211> 9
<212> PRT
<213> Homo sapiens
<400> 201
Gln Tyr Gly Val Val Tyr Glu Gly Val
1 5
<210> 202
<211> 10
<212> PRT
<213> Homo sapiens
<400> 202
Gln Tyr Gly Val Val Tyr Glu Gly Val Trp
1 5 10
<210> 203
<211> 9
<212> PRT
<213> Homo sapiens
<400> 203
Val Val Tyr Glu Gly Val Trp Lys Lys
1 5
<210> 204
<211> 10
<212> PRT
<213> Homo sapiens
<400> 204
Val Val Tyr Glu Gly Val Trp Lys Lys Tyr
1 5 10
<210> 205
<211> 10
<212> PRT
<213> Homo sapiens
<400> 205
Ala Thr Gln Ile Ser Ser Ala Thr Glu Tyr
1 5 10
<210> 206
<211> 10
<212> PRT
<213> Homo sapiens
<400> 206
Ile Ser Ser Ala Thr Glu Tyr Leu Glu Lys
1 5 10
<210> 207
<211> 9
<212> PRT
<213> Homo sapiens
<400> 207
Ser Ser Ala Thr Glu Tyr Leu Glu Lys
1 5
<210> 208
<211> 10
<212> PRT
<213> Homo sapiens
<400> 208
Thr Gln Ile Ser Ser Ala Thr Glu Tyr Leu
1 5 10
<210> 209
<211> 10
<212> PRT
<213> Homo sapiens
<400> 209
Tyr Met Ala Thr Gln Ile Ser Ser Ala Thr
1 5 10
<210> 210
<211> 10
<212> PRT
<213> Homo sapiens
<400> 210
Phe Tyr Ile Ile Ile Glu Phe Met Thr Tyr
1 5 10
<210> 211
<211> 10
<212> PRT
<213> Homo sapiens
<400> 211
Ile Ile Glu Phe Met Thr Tyr Gly Asn Leu
1 5 10
<210> 212
<211> 9
<212> PRT
<213> Homo sapiens
<400> 212
Ile Ile Ile Glu Phe Met Thr Tyr Gly
1 5
<210> 213
<211> 10
<212> PRT
<213> Homo sapiens
<400> 213
Ile Ile Ile Glu Phe Met Thr Tyr Gly Asn
1 5 10
<210> 214
<211> 10
<212> PRT
<213> Homo sapiens
<400> 214
Tyr Ile Ile Ile Glu Phe Met Thr Tyr Gly
1 5 10
<210> 215
<211> 10
<212> PRT
<213> Homo sapiens
<400> 215
Gly Gln His Gly Glu Val Tyr Glu Gly Val
1 5 10
<210> 216
<211> 10
<212> PRT
<213> Homo sapiens
<400> 216
Lys Leu Gly Gly Gly Gln His Gly Glu Val
1 5 10
<210> 217
<211> 9
<212> PRT
<213> Homo sapiens
<400> 217
Lys Ile Ala Asp Phe Gly Met Ala Arg
1 5
<210> 218
<211> 10
<212> PRT
<213> Homo sapiens
<400> 218
Arg Val Ala Lys Ile Ala Asp Phe Gly Met
1 5 10
<210> 219
<211> 10
<212> PRT
<213> Homo sapiens
<400> 219
Phe Ile Leu Met Glu Leu Met Ala Gly Gly
1 5 10
<210> 220
<211> 9
<212> PRT
<213> Homo sapiens
<400> 220
Ile Leu Met Glu Leu Met Ala Gly Gly
1 5
<210> 221
<211> 10
<212> PRT
<213> Homo sapiens
<400> 221
Ile Leu Met Glu Leu Met Ala Gly Gly Asp
1 5 10
<210> 222
<211> 10
<212> PRT
<213> Homo sapiens
<400> 222
Leu Met Glu Leu Met Ala Gly Gly Asp Leu
1 5 10
<210> 223
<211> 9
<212> PRT
<213> Homo sapiens
<400> 223
Leu Pro Arg Phe Ile Leu Met Glu Leu
1 5
<210> 224
<211> 10
<212> PRT
<213> Homo sapiens
<400> 224
Leu Pro Arg Phe Ile Leu Met Glu Leu Met
1 5 10
<210> 225
<211> 10
<212> PRT
<213> Homo sapiens
<400> 225
Leu Gln Ser Leu Pro Arg Phe Ile Leu Met
1 5 10
<210> 226
<211> 10
<212> PRT
<213> Homo sapiens
<400> 226
Ser Leu Pro Arg Phe Ile Leu Met Glu Leu
1 5 10
<210> 227
<211> 9
<212> PRT
<213> Homo sapiens
<400> 227
Leu Ala Thr Glu Lys Ser Arg Trp Ser
1 5
<210> 228
<211> 10
<212> PRT
<213> Homo sapiens
<400> 228
Leu Ala Thr Glu Lys Ser Arg Trp Ser Gly
1 5 10
<210> 229
<211> 11
<212> PRT
<213> Homo sapiens
<400> 229
Gly Pro Pro Pro Ala Asp Leu Cys His Ala Leu
1 5 10
<210> 230
<211> 9
<212> PRT
<213> Homo sapiens
<400> 230
Gly Leu Leu Leu Glu Met Leu Asp Ala
1 5
<210> 231
<211> 9
<212> PRT
<213> Homo sapiens
<400> 231
Leu Tyr Gly Leu Leu Leu Glu Met Leu
1 5
<210> 232
<211> 9
<212> PRT
<213> Homo sapiens
<400> 232
Asn Val Val Pro Leu Tyr Gly Leu Leu
1 5
<210> 233
<211> 9
<212> PRT
<213> Homo sapiens
<400> 233
Pro Leu Tyr Gly Leu Leu Leu Glu Met
1 5
<210> 234
<211> 10
<212> PRT
<213> Homo sapiens
<400> 234
Pro Leu Tyr Gly Leu Leu Leu Glu Met Leu
1 5 10
<210> 235
<211> 10
<212> PRT
<213> Homo sapiens
<400> 235
Val Pro Leu Tyr Gly Leu Leu Leu Glu Met
1 5 10
<210> 236
<211> 9
<212> PRT
<213> Homo sapiens
<400> 236
Val Val Pro Leu Tyr Gly Leu Leu Leu
1 5
<210> 237
<211> 9
<212> PRT
<213> Homo sapiens
<400> 237
Phe Leu Pro Ser Thr Leu Lys Ser Leu
1 5
<210> 238
<211> 9
<212> PRT
<213> Homo sapiens
<400> 238
Gly Val Tyr Thr Phe Leu Pro Ser Thr
1 5
<210> 239
<211> 10
<212> PRT
<213> Homo sapiens
<400> 239
Gly Val Tyr Thr Phe Leu Pro Ser Thr Leu
1 5 10
<210> 240
<211> 10
<212> PRT
<213> Homo sapiens
<400> 240
Thr Phe Leu Pro Ser Thr Leu Lys Ser Leu
1 5 10
<210> 241
<211> 9
<212> PRT
<213> Homo sapiens
<400> 241
Val Tyr Thr Phe Leu Pro Ser Thr Leu
1 5
<210> 242
<211> 9
<212> PRT
<213> Homo sapiens
<400> 242
Tyr Thr Phe Leu Pro Ser Thr Leu Lys
1 5
<210> 243
<211> 9
<212> PRT
<213> Homo sapiens
<400> 243
Asn Val Val Pro Leu Cys Asp Leu Leu
1 5
<210> 244
<211> 10
<212> PRT
<213> Homo sapiens
<400> 244
Asn Val Val Pro Leu Cys Asp Leu Leu Leu
1 5 10
<210> 245
<211> 9
<212> PRT
<213> Homo sapiens
<400> 245
Pro Leu Cys Asp Leu Leu Leu Glu Met
1 5
<210> 246
<211> 10
<212> PRT
<213> Homo sapiens
<400> 246
Pro Leu Cys Asp Leu Leu Leu Glu Met Leu
1 5 10
<210> 247
<211> 10
<212> PRT
<213> Homo sapiens
<400> 247
Val Pro Leu Cys Asp Leu Leu Leu Glu Met
1 5 10
<210> 248
<211> 9
<212> PRT
<213> Homo sapiens
<400> 248
Val Val Pro Leu Cys Asp Leu Leu Leu
1 5
<210> 249
<211> 9
<212> PRT
<213> Homo sapiens
<400> 249
Asn Val Val Pro Leu Asn Asp Leu Leu
1 5
<210> 250
<211> 10
<212> PRT
<213> Homo sapiens
<400> 250
Asn Val Val Pro Leu Asn Asp Leu Leu Leu
1 5 10
<210> 251
<211> 9
<212> PRT
<213> Homo sapiens
<400> 251
Pro Leu Asn Asp Leu Leu Leu Glu Met
1 5
<210> 252
<211> 10
<212> PRT
<213> Homo sapiens
<400> 252
Pro Leu Asn Asp Leu Leu Leu Glu Met Leu
1 5 10
<210> 253
<211> 10
<212> PRT
<213> Homo sapiens
<400> 253
Val Pro Leu Asn Asp Leu Leu Leu Glu Met
1 5 10
<210> 254
<211> 9
<212> PRT
<213> Homo sapiens
<400> 254
Asn Val Val Pro Leu Ser Asp Leu Leu
1 5
<210> 255
<211> 10
<212> PRT
<213> Homo sapiens
<400> 255
Asn Val Val Pro Leu Ser Asp Leu Leu Leu
1 5 10
<210> 256
<211> 9
<212> PRT
<213> Homo sapiens
<400> 256
Pro Leu Ser Asp Leu Leu Leu Glu Met
1 5
<210> 257
<211> 10
<212> PRT
<213> Homo sapiens
<400> 257
Pro Leu Ser Asp Leu Leu Leu Glu Met Leu
1 5 10
<210> 258
<211> 10
<212> PRT
<213> Homo sapiens
<400> 258
Val Pro Leu Ser Asp Leu Leu Leu Glu Met
1 5 10
<210> 259
<211> 9
<212> PRT
<213> Homo sapiens
<400> 259
Val Val Pro Leu Ser Asp Leu Leu Leu
1 5
<210> 260
<211> 10
<212> PRT
<213> Homo sapiens
<400> 260
Val Leu Glu Arg Cys Pro His Arg Pro Ile
1 5 10
<210> 261
<211> 9
<212> PRT
<213> Homo sapiens
<400> 261
Tyr Thr Leu Asp Val Leu Glu Arg Cys
1 5
<210> 262
<211> 9
<212> PRT
<213> Homo sapiens
<400> 262
Phe Gln Asn Gly Lys Met Ala Leu Ser
1 5
<210> 263
<211> 10
<212> PRT
<213> Homo sapiens
<400> 263
Val Met Ala Gly Leu Gly Ser Pro Tyr Val
1 5 10
<210> 264
<211> 9
<212> PRT
<213> Homo sapiens
<400> 264
Lys Pro Ile Ile Ile Gly His His Ala
1 5
<210> 265
<211> 9
<212> PRT
<213> Homo sapiens
<400> 265
Lys Pro Ile Ile Ile Gly Cys His Ala
1 5
<210> 266
<211> 9
<212> PRT
<213> Homo sapiens
<400> 266
Lys Pro Ile Ile Ile Gly Gly His Ala
1 5
<210> 267
<211> 9
<212> PRT
<213> Homo sapiens
<400> 267
Lys Pro Ile Ile Ile Gly Ser His Ala
1 5
<210> 268
<211> 10
<212> PRT
<213> Homo sapiens
<400> 268
Ile Ile Glu Tyr Cys Cys Tyr Gly Asp Leu
1 5 10
<210> 269
<211> 10
<212> PRT
<213> Homo sapiens
<400> 269
Thr Ile Gly Gly Pro Thr Leu Val Ile Ile
1 5 10
<210> 270
<211> 9
<212> PRT
<213> Homo sapiens
<400> 270
Val Ile Ile Glu Tyr Cys Cys Tyr Gly
1 5
<210> 271
<211> 10
<212> PRT
<213> Homo sapiens
<400> 271
His Met Asn Ile Ala Asn Leu Leu Gly Ala
1 5 10
<210> 272
<211> 10
<212> PRT
<213> Homo sapiens
<400> 272
Ile Ala Asn Leu Leu Gly Ala Cys Thr Ile
1 5 10
<210> 273
<211> 9
<212> PRT
<213> Homo sapiens
<400> 273
Met Asn Ile Ala Asn Leu Leu Gly Ala
1 5
<210> 274
<211> 9
<212> PRT
<213> Homo sapiens
<400> 274
Tyr Leu Gly Asn His Met Asn Ile Ala
1 5
<210> 275
<211> 10
<212> PRT
<213> Homo sapiens
<400> 275
Tyr Leu Gly Asn His Met Asn Ile Ala Asn
1 5 10
<210> 276
<211> 9
<212> PRT
<213> Homo sapiens
<400> 276
Val Leu His Glu Ser Asn Ser Pro Tyr
1 5
<210> 277
<211> 10
<212> PRT
<213> Homo sapiens
<400> 277
Val Leu His Glu Ser Asn Ser Pro Tyr Ile
1 5 10
<210> 278
<211> 10
<212> PRT
<213> Homo sapiens
<400> 278
Leu Gln Val Leu His Glu Cys Asn Ser Leu
1 5 10
<210> 279
<211> 10
<212> PRT
<213> Homo sapiens
<400> 279
Leu Tyr Ile Val Gly Phe Tyr Gly Ala Phe
1 5 10
<210> 280
<211> 9
<212> PRT
<213> Homo sapiens
<400> 280
Asn Ser Leu Tyr Ile Val Gly Phe Tyr
1 5
<210> 281
<211> 9
<212> PRT
<213> Homo sapiens
<400> 281
Gln Val Leu His Glu Cys Asn Ser Leu
1 5
<210> 282
<211> 9
<212> PRT
<213> Homo sapiens
<400> 282
Ser Leu Tyr Ile Val Gly Phe Tyr Gly
1 5
<210> 283
<211> 10
<212> PRT
<213> Homo sapiens
<400> 283
Ser Leu Tyr Ile Val Gly Phe Tyr Gly Ala
1 5 10
<210> 284
<211> 9
<212> PRT
<213> Homo sapiens
<400> 284
Val Leu His Glu Cys Asn Ser Leu Tyr
1 5
<210> 285
<211> 10
<212> PRT
<213> Homo sapiens
<400> 285
Val Leu His Glu Cys Asn Ser Leu Tyr Ile
1 5 10
<210> 286
<211> 10
<212> PRT
<213> Homo sapiens
<400> 286
Phe Tyr Gln Gln Gln Gln Gln Ser Asp Leu
1 5 10
<210> 287
<211> 10
<212> PRT
<213> Homo sapiens
<400> 287
Gln Gln Gln Ser Asp Leu Gln Pro Pro Ala
1 5 10
<210> 288
<211> 9
<212> PRT
<213> Homo sapiens
<400> 288
Gln Gln Ser Asp Leu Gln Pro Pro Ala
1 5
<210> 289
<211> 9
<212> PRT
<213> Homo sapiens
<400> 289
Tyr Gln Gln Gln Gln Gln Ser Asp Leu
1 5
<210> 290
<211> 9
<212> PRT
<213> Homo sapiens
<400> 290
Phe Glu Leu Leu Ser Thr Pro Pro Leu
1 5
<210> 291
<211> 10
<212> PRT
<213> Homo sapiens
<400> 291
Leu Leu Ser Thr Pro Pro Leu Ser Pro Ser
1 5 10
<210> 292
<211> 9
<212> PRT
<213> Homo sapiens
<400> 292
Phe Glu Leu Leu Pro Ile Pro Pro Leu
1 5
<210> 293
<211> 10
<212> PRT
<213> Homo sapiens
<400> 293
Ile Trp Lys Lys Phe Glu Leu Leu Pro Ile
1 5 10
<210> 294
<211> 10
<212> PRT
<213> Homo sapiens
<400> 294
Leu Leu Pro Ile Pro Pro Leu Ser Pro Ser
1 5 10
<210> 295
<211> 9
<212> PRT
<213> Homo sapiens
<400> 295
Leu Pro Ile Pro Pro Leu Ser Pro Ser
1 5
<210> 296
<211> 10
<212> PRT
<213> Homo sapiens
<400> 296
Ile Ile Glu Tyr Cys Phe Tyr Gly Asp Leu
1 5 10
<210> 297
<211> 9
<212> PRT
<213> Homo sapiens
<400> 297
Ile Ile Ile Glu Tyr Cys Phe Tyr Gly
1 5
<210> 298
<211> 9
<212> PRT
<213> Homo sapiens
<400> 298
Ile Tyr Ile Ile Ile Glu Tyr Cys Phe
1 5
<210> 299
<211> 10
<212> PRT
<213> Homo sapiens
<400> 299
Ile Tyr Ile Ile Ile Glu Tyr Cys Phe Tyr
1 5 10
<210> 300
<211> 10
<212> PRT
<213> Homo sapiens
<400> 300
Tyr Ile Ile Ile Glu Tyr Cys Phe Tyr Gly
1 5 10
<210> 301
<211> 10
<212> PRT
<213> Homo sapiens
<400> 301
Lys Ile Thr Glu Gln Glu Lys Asp Phe Leu
1 5 10
<210> 302
<211> 11
<212> PRT
<213> Homo sapiens
<400> 302
Ser Thr Arg Asp Pro Leu Ser Glu Ile Thr Lys
1 5 10
<210> 303
<211> 8
<212> PRT
<213> Homo sapiens
<400> 303
Asp Pro Leu Ser Glu Ile Thr Lys
1 5
<210> 304
<211> 10
<212> PRT
<213> Homo sapiens
<400> 304
Leu Pro Leu Lys Phe Arg Asp Ala Glu Thr
1 5 10
<210> 305
<211> 10
<212> PRT
<213> Homo sapiens
<400> 305
Gln Thr Gly Val Met Ile Cys Ala Tyr Leu
1 5 10
<210> 306
<211> 10
<212> PRT
<213> Homo sapiens
<400> 306
Ala Phe Ser Gly Glu Tyr Ile Pro Thr Val
1 5 10
<210> 307
<211> 9
<212> PRT
<213> Homo sapiens
<400> 307
Ser Met Asn Arg Arg Pro Ile Leu Thr
1 5
<210> 308
<211> 10
<212> PRT
<213> Homo sapiens
<400> 308
Tyr Met Cys Asn Ser Ser Cys Met Gly Ser
1 5 10
<210> 309
<211> 9
<212> PRT
<213> Homo sapiens
<400> 309
Gly Met Asn Gln Arg Pro Ile Leu Thr
1 5
<210> 310
<211> 9
<212> PRT
<213> Homo sapiens
<400> 310
Gly Met Asn Trp Arg Pro Ile Leu Thr
1 5
<210> 311
<211> 10
<212> PRT
<213> Homo sapiens
<400> 311
Leu Leu Gly Arg Asn Ser Phe Glu Val Cys
1 5 10
<210> 312
<211> 9
<212> PRT
<213> Homo sapiens
<400> 312
Ala Leu Lys Tyr Ile Phe Val Ala Val
1 5
<210> 313
<211> 10
<212> PRT
<213> Homo sapiens
<400> 313
Ala Leu Lys Tyr Ile Phe Val Ala Val Arg
1 5 10
<210> 314
<211> 9
<212> PRT
<213> Homo sapiens
<400> 314
Ala Val Arg Ala Ile Cys Val Met Lys
1 5
<210> 315
<211> 10
<212> PRT
<213> Homo sapiens
<400> 315
Ala Val Arg Ala Ile Cys Val Met Lys Ser
1 5 10
<210> 316
<211> 9
<212> PRT
<213> Homo sapiens
<400> 316
Cys Val Glu Lys Lys Thr Ala Leu Lys
1 5
<210> 317
<211> 10
<212> PRT
<213> Homo sapiens
<400> 317
Cys Val Glu Lys Lys Thr Ala Leu Lys Tyr
1 5 10
<210> 318
<211> 9
<212> PRT
<213> Homo sapiens
<400> 318
Cys Val Met Lys Ser Phe Leu Ile Phe
1 5
<210> 319
<211> 10
<212> PRT
<213> Homo sapiens
<400> 319
Cys Val Met Lys Ser Phe Leu Ile Phe Arg
1 5 10
<210> 320
<211> 9
<212> PRT
<213> Homo sapiens
<400> 320
Phe Leu Ile Phe Arg Arg Trp Lys Ser
1 5
<210> 321
<211> 10
<212> PRT
<213> Homo sapiens
<400> 321
Phe Arg Arg Trp Lys Ser His Ser Pro Leu
1 5 10
<210> 322
<211> 9
<212> PRT
<213> Homo sapiens
<400> 322
Phe Val Ala Val Arg Ala Ile Cys Val
1 5
<210> 323
<211> 10
<212> PRT
<213> Homo sapiens
<400> 323
Phe Val Ala Val Arg Ala Ile Cys Val Met
1 5 10
<210> 324
<211> 9
<212> PRT
<213> Homo sapiens
<400> 324
Ile Gln Leu His Leu Ser His Pro Ile
1 5
<210> 325
<211> 10
<212> PRT
<213> Homo sapiens
<400> 325
Lys Ser Phe Leu Ile Phe Arg Arg Trp Lys
1 5 10
<210> 326
<211> 9
<212> PRT
<213> Homo sapiens
<400> 326
Lys Thr Ala Leu Lys Tyr Ile Phe Val
1 5
<210> 327
<211> 10
<212> PRT
<213> Homo sapiens
<400> 327
Lys Tyr Ile Phe Val Ala Val Arg Ala Ile
1 5 10
<210> 328
<211> 10
<212> PRT
<213> Homo sapiens
<400> 328
Arg Trp Lys Ser His Ser Pro Leu Gln Ile
1 5 10
<210> 329
<211> 10
<212> PRT
<213> Homo sapiens
<400> 329
Thr Ala Leu Lys Tyr Ile Phe Val Ala Val
1 5 10
<210> 330
<211> 10
<212> PRT
<213> Homo sapiens
<400> 330
Val Ala Val Arg Ala Ile Cys Val Met Lys
1 5 10
<210> 331
<211> 9
<212> PRT
<213> Homo sapiens
<400> 331
Val Met Lys Ser Phe Leu Ile Phe Arg
1 5
<210> 332
<211> 10
<212> PRT
<213> Homo sapiens
<400> 332
Val Met Lys Ser Phe Leu Ile Phe Arg Arg
1 5 10
<210> 333
<211> 9
<212> PRT
<213> Homo sapiens
<400> 333
Tyr Ile Phe Val Ala Val Arg Ala Ile
1 5
<210> 334
<211> 9
<212> PRT
<213> Homo sapiens
<400> 334
Ala Leu Phe Phe Phe Phe Phe Glu Thr
1 5
<210> 335
<211> 10
<212> PRT
<213> Homo sapiens
<400> 335
Ala Leu Phe Phe Phe Phe Phe Glu Thr Lys
1 5 10
<210> 336
<211> 10
<212> PRT
<213> Homo sapiens
<400> 336
Ala Gln Ala Gly Val Gln Trp Arg Ser Leu
1 5 10
<210> 337
<211> 10
<212> PRT
<213> Homo sapiens
<400> 337
Cys Leu Ala Asn Phe Cys Ile Phe Asn Arg
1 5 10
<210> 338
<211> 10
<212> PRT
<213> Homo sapiens
<400> 338
Cys Leu Ser Phe Leu Ser Ser Trp Asp Tyr
1 5 10
<210> 339
<211> 9
<212> PRT
<213> Homo sapiens
<400> 339
Phe Phe Glu Thr Lys Ser Cys Ser Val
1 5
<210> 340
<211> 10
<212> PRT
<213> Homo sapiens
<400> 340
Phe Phe Phe Glu Thr Lys Ser Cys Ser Val
1 5 10
<210> 341
<211> 10
<212> PRT
<213> Homo sapiens
<400> 341
Phe Lys Leu Phe Ser Cys Leu Ser Phe Leu
1 5 10
<210> 342
<211> 10
<212> PRT
<213> Homo sapiens
<400> 342
Phe Leu Ser Ser Trp Asp Tyr Arg Arg Met
1 5 10
<210> 343
<211> 10
<212> PRT
<213> Homo sapiens
<400> 343
Gly Phe Lys Leu Phe Ser Cys Leu Ser Phe
1 5 10
<210> 344
<211> 9
<212> PRT
<213> Homo sapiens
<400> 344
Lys Leu Phe Ser Cys Leu Ser Phe Leu
1 5
<210> 345
<211> 10
<212> PRT
<213> Homo sapiens
<400> 345
Lys Leu Phe Ser Cys Leu Ser Phe Leu Ser
1 5 10
<210> 346
<211> 10
<212> PRT
<213> Homo sapiens
<400> 346
Leu Ala Leu Phe Phe Phe Phe Phe Glu Thr
1 5 10
<210> 347
<211> 9
<212> PRT
<213> Homo sapiens
<400> 347
Leu Phe Phe Phe Phe Phe Glu Thr Lys
1 5
<210> 348
<211> 9
<212> PRT
<213> Homo sapiens
<400> 348
Leu Ser Phe Leu Ser Ser Trp Asp Tyr
1 5
<210> 349
<211> 10
<212> PRT
<213> Homo sapiens
<400> 349
Leu Ser Phe Leu Ser Ser Trp Asp Tyr Arg
1 5 10
<210> 350
<211> 9
<212> PRT
<213> Homo sapiens
<400> 350
Arg Met Pro Pro Cys Leu Ala Asn Phe
1 5
<210> 351
<211> 10
<212> PRT
<213> Homo sapiens
<400> 351
Arg Arg Met Pro Pro Cys Leu Ala Asn Phe
1 5 10
<210> 352
<211> 10
<212> PRT
<213> Homo sapiens
<400> 352
Ser Leu Gln Pro Pro Pro Pro Gly Phe Lys
1 5 10
<210> 353
<211> 9
<212> PRT
<213> Homo sapiens
<400> 353
Val Gln Trp Arg Ser Leu Gly Ser Leu
1 5
<210> 354
<211> 9
<212> PRT
<213> Homo sapiens
<400> 354
Phe Phe Phe Ser Val Ile Phe Ser Thr
1 5
<210> 355
<211> 10
<212> PRT
<213> Homo sapiens
<400> 355
Met Ser Val Cys Phe Phe Phe Phe Ser Val
1 5 10
<210> 356
<211> 9
<212> PRT
<213> Homo sapiens
<400> 356
Ser Val Cys Phe Phe Phe Phe Ser Val
1 5
<210> 357
<211> 10
<212> PRT
<213> Homo sapiens
<400> 357
Ser Val Cys Phe Phe Phe Phe Ser Val Ile
1 5 10
<210> 358
<211> 10
<212> PRT
<213> Homo sapiens
<400> 358
Phe Phe Cys Tyr Ile Leu Asn Thr Met Phe
1 5 10
<210> 359
<211> 10
<212> PRT
<213> Homo sapiens
<400> 359
Met Ser Val Cys Phe Phe Phe Phe Cys Tyr
1 5 10
<210> 360
<211> 10
<212> PRT
<213> Homo sapiens
<400> 360
Ser Val Cys Phe Phe Phe Phe Cys Tyr Ile
1 5 10
<210> 361
<211> 9
<212> PRT
<213> Homo sapiens
<400> 361
Lys Gln Trp Ser Ser Val Thr Ser Leu
1 5
<210> 362
<211> 9
<212> PRT
<213> Homo sapiens
<400> 362
Val Leu Trp Met Pro Thr Ser Thr Val
1 5
<210> 363
<211> 9
<212> PRT
<213> Homo sapiens
<400> 363
Ile Leu Ile Arg Lys Ala Met Thr Val
1 5
<210> 364
<211> 8
<212> PRT
<213> Homo sapiens
<400> 364
Arg Met Lys Val Pro Leu Met Lys
1 5
<210> 365
<211> 9
<212> PRT
<213> Homo sapiens
<400> 365
Thr Leu Lys Lys Lys Pro Arg Asp Ile
1 5
<210> 366
<211> 10
<212> PRT
<213> Homo sapiens
<400> 366
Leu Ile Glu Gly Phe Glu Ala Leu Leu Lys
1 5 10
<210> 367
<211> 9
<212> PRT
<213> Homo sapiens
<400> 367
Gln Gln Leu Lys Trp Thr Pro His Ile
1 5
<210> 368
<211> 10
<212> PRT
<213> Homo sapiens
<400> 368
Gln Leu Lys Trp Thr Pro His Ile Leu Lys
1 5 10
<210> 369
<211> 10
<212> PRT
<213> Homo sapiens
<400> 369
Ile Val Ser Glu Lys Asn Gln Gln Leu Lys
1 5 10
<210> 370
<211> 11
<212> PRT
<213> Homo sapiens
<400> 370
Asn Gln Gln Leu Lys Trp Thr Pro His Ile Leu
1 5 10
<210> 371
<211> 10
<212> PRT
<213> Homo sapiens
<400> 371
Asn Gln Gln Leu Lys Trp Thr Pro His Ile
1 5 10
<210> 372
<211> 9
<212> PRT
<213> Homo sapiens
<400> 372
Gln Leu Lys Trp Thr Pro His Ile Leu
1 5
<210> 373
<211> 10
<212> PRT
<213> Homo sapiens
<400> 373
Gly Pro Pro Arg Pro Pro Arg Ala Ala Cys
1 5 10
<210> 374
<211> 9
<212> PRT
<213> Homo sapiens
<400> 374
Pro Pro Arg Pro Pro Arg Ala Ala Cys
1 5
<210> 375
<211> 9
<212> PRT
<213> Homo sapiens
<400> 375
Ser Leu Arg Arg Lys Tyr Leu Arg Val
1 5
<210> 376
<211> 10
<212> PRT
<213> Homo sapiens
<400> 376
Ser Ala Ala Cys His Arg Arg Gly Cys Val
1 5 10
<210> 377
<211> 10
<212> PRT
<213> Homo sapiens
<400> 377
Ala Leu Trp Glu Cys Ser Leu Pro Gln Ala
1 5 10
<210> 378
<211> 9
<212> PRT
<213> Homo sapiens
<400> 378
Cys Leu Ile Val Ser Arg Thr Leu Leu
1 5
<210> 379
<211> 10
<212> PRT
<213> Homo sapiens
<400> 379
Cys Leu Ile Val Ser Arg Thr Leu Leu Leu
1 5 10
<210> 380
<211> 9
<212> PRT
<213> Homo sapiens
<400> 380
Phe Leu Leu Ala Leu Trp Glu Cys Ser
1 5
<210> 381
<211> 10
<212> PRT
<213> Homo sapiens
<400> 381
Phe Leu Leu Ala Leu Trp Glu Cys Ser Leu
1 5 10
<210> 382
<211> 9
<212> PRT
<213> Homo sapiens
<400> 382
Ile Val Ser Arg Thr Leu Leu Leu Val
1 5
<210> 383
<211> 9
<212> PRT
<213> Homo sapiens
<400> 383
Leu Ile Val Ser Arg Thr Leu Leu Leu
1 5
<210> 384
<211> 10
<212> PRT
<213> Homo sapiens
<400> 384
Leu Ile Val Ser Arg Thr Leu Leu Leu Val
1 5 10
<210> 385
<211> 9
<212> PRT
<213> Homo sapiens
<400> 385
Leu Leu Ala Leu Trp Glu Cys Ser Leu
1 5
<210> 386
<211> 9
<212> PRT
<213> Homo sapiens
<400> 386
Leu Pro Gln Ala Arg Leu Cys Leu Ile
1 5
<210> 387
<211> 10
<212> PRT
<213> Homo sapiens
<400> 387
Leu Pro Gln Ala Arg Leu Cys Leu Ile Val
1 5 10
<210> 388
<211> 10
<212> PRT
<213> Homo sapiens
<400> 388
Asn Val Arg Asp Lys Lys Arg Ala Thr Phe
1 5 10
<210> 389
<211> 10
<212> PRT
<213> Homo sapiens
<400> 389
Ser Leu Pro Gln Ala Arg Leu Cys Leu Ile
1 5 10
<210> 390
<211> 9
<212> PRT
<213> Homo sapiens
<400> 390
Phe Phe Cys Trp Ile Leu Ser Cys Lys
1 5
<210> 391
<211> 10
<212> PRT
<213> Homo sapiens
<400> 391
Phe Phe Phe Cys Trp Ile Leu Ser Cys Lys
1 5 10
<210> 392
<211> 9
<212> PRT
<213> Homo sapiens
<400> 392
Ile Thr Ala Phe Phe Phe Cys Trp Ile
1 5
<210> 393
<211> 10
<212> PRT
<213> Homo sapiens
<400> 393
Leu Tyr Ile Thr Ala Phe Phe Phe Cys Trp
1 5 10
<210> 394
<211> 10
<212> PRT
<213> Homo sapiens
<400> 394
Tyr Ile Thr Ala Phe Phe Phe Cys Trp Ile
1 5 10
<210> 395
<211> 9
<212> PRT
<213> Homo sapiens
<400> 395
Ile Thr Ala Phe Phe Leu Leu Asp Ile
1 5
<210> 396
<211> 9
<212> PRT
<213> Homo sapiens
<400> 396
Leu Leu Tyr Ile Thr Ala Phe Phe Leu
1 5
<210> 397
<211> 10
<212> PRT
<213> Homo sapiens
<400> 397
Leu Leu Tyr Ile Thr Ala Phe Phe Leu Leu
1 5 10
<210> 398
<211> 9
<212> PRT
<213> Homo sapiens
<400> 398
Leu Tyr Ile Thr Ala Phe Phe Leu Leu
1 5
<210> 399
<211> 10
<212> PRT
<213> Homo sapiens
<400> 399
Leu Tyr Ile Thr Ala Phe Phe Leu Leu Asp
1 5 10
<210> 400
<211> 10
<212> PRT
<213> Homo sapiens
<400> 400
Tyr Ile Thr Ala Phe Phe Leu Leu Asp Ile
1 5 10
<210> 401
<211> 9
<212> PRT
<213> Homo sapiens
<400> 401
Gly Ser Asn Glu Val Thr Thr Arg Tyr
1 5
<210> 402
<211> 9
<212> PRT
<213> Homo sapiens
<400> 402
Met Pro Lys Asp Val Asn Ile Gln Val
1 5
<210> 403
<211> 10
<212> PRT
<213> Homo sapiens
<400> 403
Thr Gly Ser Asn Glu Val Thr Thr Arg Tyr
1 5 10
<210> 404
<211> 10
<212> PRT
<213> Homo sapiens
<400> 404
Lys Lys Leu Met Leu Leu Arg Leu Asn Leu
1 5 10
<210> 405
<211> 9
<212> PRT
<213> Homo sapiens
<400> 405
Lys Leu Met Leu Leu Arg Leu Asn Leu
1 5
<210> 406
<211> 10
<212> PRT
<213> Homo sapiens
<400> 406
Lys Leu Met Leu Leu Arg Leu Asn Leu Arg
1 5 10
<210> 407
<211> 9
<212> PRT
<213> Homo sapiens
<400> 407
Leu Leu Arg Leu Asn Leu Arg Lys Met
1 5
<210> 408
<211> 8
<212> PRT
<213> Homo sapiens
<400> 408
Leu Met Leu Leu Arg Leu Asn Leu
1 5
<210> 409
<211> 10
<212> PRT
<213> Homo sapiens
<400> 409
Leu Met Leu Leu Arg Leu Asn Leu Arg Lys
1 5 10
<210> 410
<211> 10
<212> PRT
<213> Homo sapiens
<400> 410
Leu Asn Leu Arg Lys Met Cys Gly Pro Phe
1 5 10
<210> 411
<211> 9
<212> PRT
<213> Homo sapiens
<400> 411
Met Leu Leu Arg Leu Asn Leu Arg Lys
1 5
<210> 412
<211> 10
<212> PRT
<213> Homo sapiens
<400> 412
Met Leu Leu Arg Leu Asn Leu Arg Lys Met
1 5 10
<210> 413
<211> 9
<212> PRT
<213> Homo sapiens
<400> 413
Asn Leu Arg Lys Met Cys Gly Pro Phe
1 5
<210> 414
<211> 10
<212> PRT
<213> Homo sapiens
<400> 414
Asn Tyr Cys Gln Lys Lys Leu Met Leu Leu
1 5 10
<210> 415
<211> 9
<212> PRT
<213> Homo sapiens
<400> 415
Tyr Cys Gln Lys Lys Leu Met Leu Leu
1 5
<210> 416
<211> 10
<212> PRT
<213> Homo sapiens
<400> 416
Phe Lys Lys Lys Thr Tyr Thr Cys Ala Ile
1 5 10
<210> 417
<211> 10
<212> PRT
<213> Homo sapiens
<400> 417
Ile Thr Thr Val Lys Ala Thr Glu Thr Lys
1 5 10
<210> 418
<211> 9
<212> PRT
<213> Homo sapiens
<400> 418
Lys Ser Lys Lys Lys Glu Thr Phe Lys
1 5
<210> 419
<211> 10
<212> PRT
<213> Homo sapiens
<400> 419
Lys Ser Lys Lys Lys Glu Thr Phe Lys Lys
1 5 10
<210> 420
<211> 10
<212> PRT
<213> Homo sapiens
<400> 420
Lys Thr Tyr Thr Cys Ala Ile Thr Thr Val
1 5 10
<210> 421
<211> 9
<212> PRT
<213> Homo sapiens
<400> 421
Thr Phe Lys Lys Lys Thr Tyr Thr Cys
1 5
<210> 422
<211> 9
<212> PRT
<213> Homo sapiens
<400> 422
Thr Tyr Thr Cys Ala Ile Thr Thr Val
1 5
<210> 423
<211> 10
<212> PRT
<213> Homo sapiens
<400> 423
Thr Tyr Thr Cys Ala Ile Thr Thr Val Lys
1 5 10
<210> 424
<211> 9
<212> PRT
<213> Homo sapiens
<400> 424
Tyr Thr Cys Ala Ile Thr Thr Val Lys
1 5
<210> 425
<211> 9
<212> PRT
<213> Homo sapiens
<400> 425
Thr Ala Lys Ser Lys Lys Arg Asn Leu
1 5
<210> 426
<211> 9
<212> PRT
<213> Homo sapiens
<400> 426
Phe Ala Leu Cys Gly Phe Trp Gln Ile
1 5
<210> 427
<211> 8
<212> PRT
<213> Homo sapiens
<400> 427
Leu Lys Lys Leu Arg Ser Pro Leu
1 5
<210> 428
<211> 8
<212> PRT
<213> Homo sapiens
<400> 428
Ser Leu Lys Lys Val Pro Ala Leu
1 5
<210> 429
<211> 10
<212> PRT
<213> Homo sapiens
<400> 429
Arg Lys Ile Ser Asn Trp Ser Leu Lys Lys
1 5 10
<210> 430
<211> 11
<212> PRT
<213> Homo sapiens
<400> 430
Val Pro Ala Leu Lys Lys Leu Arg Ser Pro Leu
1 5 10
<210> 431
<211> 10
<212> PRT
<213> Homo sapiens
<400> 431
Lys Arg Val Asn Thr Ala Trp Lys Thr Lys
1 5 10
<210> 432
<211> 10
<212> PRT
<213> Homo sapiens
<400> 432
Met Thr Lys Lys Lys Arg Val Asn Thr Ala
1 5 10
<210> 433
<211> 9
<212> PRT
<213> Homo sapiens
<400> 433
Arg Val Asn Thr Ala Trp Lys Thr Lys
1 5
<210> 434
<211> 10
<212> PRT
<213> Homo sapiens
<400> 434
Arg Val Asn Thr Ala Trp Lys Thr Lys Lys
1 5 10
<210> 435
<211> 9
<212> PRT
<213> Homo sapiens
<400> 435
Thr Lys Lys Lys Arg Val Asn Thr Ala
1 5
<210> 436
<211> 10
<212> PRT
<213> Homo sapiens
<400> 436
Trp Lys Thr Lys Lys Thr Ser Phe Ser Leu
1 5 10
<210> 437
<211> 9
<212> PRT
<213> Homo sapiens
<400> 437
Ala Leu Met Ser Ala Met Thr Thr Ser
1 5
<210> 438
<211> 9
<212> PRT
<213> Homo sapiens
<400> 438
Ala Met Thr Thr Ser Ser Ser Gln Lys
1 5
<210> 439
<211> 10
<212> PRT
<213> Homo sapiens
<400> 439
Ala Met Thr Thr Ser Ser Ser Gln Lys Asn
1 5 10
<210> 440
<211> 9
<212> PRT
<213> Homo sapiens
<400> 440
Cys Ile Met Lys Glu Lys Lys Ser Leu
1 5
<210> 441
<211> 10
<212> PRT
<213> Homo sapiens
<400> 441
Cys Ile Met Lys Glu Lys Lys Ser Leu Val
1 5 10
<210> 442
<211> 8
<212> PRT
<213> Homo sapiens
<400> 442
Ile Met Lys Glu Lys Lys Ser Leu
1 5
<210> 443
<211> 9
<212> PRT
<213> Homo sapiens
<400> 443
Ile Met Lys Glu Lys Lys Ser Leu Val
1 5
<210> 444
<211> 10
<212> PRT
<213> Homo sapiens
<400> 444
Lys Ser Leu Val Arg Leu Ser Ser Cys Val
1 5 10
<210> 445
<211> 10
<212> PRT
<213> Homo sapiens
<400> 445
Leu Val Arg Leu Ser Ser Cys Val Pro Val
1 5 10
<210> 446
<211> 9
<212> PRT
<213> Homo sapiens
<400> 446
Arg Leu Ser Ser Cys Val Pro Val Ala
1 5
<210> 447
<211> 10
<212> PRT
<213> Homo sapiens
<400> 447
Arg Leu Ser Ser Cys Val Pro Val Ala Leu
1 5 10
<210> 448
<211> 10
<212> PRT
<213> Homo sapiens
<400> 448
Ser Ala Met Thr Thr Ser Ser Ser Gln Lys
1 5 10
<210> 449
<211> 9
<212> PRT
<213> Homo sapiens
<400> 449
Ser Leu Val Arg Leu Ser Ser Cys Val
1 5
<210> 450
<211> 9
<212> PRT
<213> Homo sapiens
<400> 450
Val Pro Val Ala Leu Met Ser Ala Met
1 5
<210> 451
<211> 10
<212> PRT
<213> Homo sapiens
<400> 451
Val Arg Leu Ser Ser Cys Val Pro Val Ala
1 5 10
<210> 452
<211> 9
<212> PRT
<213> Homo sapiens
<400> 452
Lys Met Arg Lys Lys Tyr Ala Gln Lys
1 5
<210> 453
<211> 9
<212> PRT
<213> Homo sapiens
<400> 453
Phe Val Met Ser Asp Thr Thr Tyr Lys
1 5
<210> 454
<211> 10
<212> PRT
<213> Homo sapiens
<400> 454
Phe Val Met Ser Asp Thr Thr Tyr Lys Ile
1 5 10
<210> 455
<211> 9
<212> PRT
<213> Homo sapiens
<400> 455
Lys Thr Phe Glu Lys Lys Gly Glu Lys
1 5
<210> 456
<211> 10
<212> PRT
<213> Homo sapiens
<400> 456
Leu Phe Val Met Ser Asp Thr Thr Tyr Lys
1 5 10
<210> 457
<211> 9
<212> PRT
<213> Homo sapiens
<400> 457
Met Ser Asp Thr Thr Tyr Lys Ile Tyr
1 5
<210> 458
<211> 9
<212> PRT
<213> Homo sapiens
<400> 458
Val Met Ser Asp Thr Thr Tyr Lys Ile
1 5
<210> 459
<211> 10
<212> PRT
<213> Homo sapiens
<400> 459
Val Met Ser Asp Thr Thr Tyr Lys Ile Tyr
1 5 10
<210> 460
<211> 10
<212> PRT
<213> Homo sapiens
<400> 460
Tyr Leu Thr Lys Trp Pro Lys Phe Phe Leu
1 5 10
<210> 461
<211> 9
<212> PRT
<213> Homo sapiens
<400> 461
Phe Leu Gln Glu Arg Asn Leu Pro Pro
1 5
<210> 462
<211> 9
<212> PRT
<213> Homo sapiens
<400> 462
Phe Arg Arg Pro His Ser Cys Leu Ala
1 5
<210> 463
<211> 9
<212> PRT
<213> Homo sapiens
<400> 463
Leu Ile Val Leu Arg Val Val Arg Leu
1 5
<210> 464
<211> 9
<212> PRT
<213> Homo sapiens
<400> 464
Leu Leu Ser Val His Leu Ile Val Leu
1 5
<210> 465
<211> 9
<212> PRT
<213> Homo sapiens
<400> 465
Glu Val Lys His Leu His His Leu Leu
1 5
<210> 466
<211> 10
<212> PRT
<213> Homo sapiens
<400> 466
His Leu His His Leu Leu Lys Gln Leu Lys
1 5 10
<210> 467
<211> 9
<212> PRT
<213> Homo sapiens
<400> 467
His Leu Leu Leu Lys Arg Glu Arg Val
1 5
<210> 468
<211> 9
<212> PRT
<213> Homo sapiens
<400> 468
Lys Ile Lys His Leu Leu Leu Lys Arg
1 5
<210> 469
<211> 9
<212> PRT
<213> Homo sapiens
<400> 469
Lys Pro Ser Glu Lys Tyr Leu Lys Ile
1 5
<210> 470
<211> 9
<212> PRT
<213> Homo sapiens
<400> 470
Lys Tyr Leu Lys Ile Lys His Leu Leu
1 5
<210> 471
<211> 10
<212> PRT
<213> Homo sapiens
<400> 471
Lys Tyr Leu Lys Ile Lys His Leu Leu Leu
1 5 10
<210> 472
<211> 10
<212> PRT
<213> Homo sapiens
<400> 472
Leu Leu Lys Gln Leu Lys Pro Ser Glu Lys
1 5 10
<210> 473
<211> 9
<212> PRT
<213> Homo sapiens
<400> 473
Leu Leu Lys Arg Glu Arg Val Asp Leu
1 5
<210> 474
<211> 10
<212> PRT
<213> Homo sapiens
<400> 474
Leu Leu Leu Lys Arg Glu Arg Val Asp Leu
1 5 10
<210> 475
<211> 10
<212> PRT
<213> Homo sapiens
<400> 475
Gln Leu Lys Pro Ser Glu Lys Tyr Leu Lys
1 5 10
<210> 476
<211> 9
<212> PRT
<213> Homo sapiens
<400> 476
Tyr Leu Lys Ile Lys His Leu Leu Leu
1 5
<210> 477
<211> 10
<212> PRT
<213> Homo sapiens
<400> 477
Tyr Leu Lys Ile Lys His Leu Leu Leu Lys
1 5 10
<210> 478
<211> 9
<212> PRT
<213> Homo sapiens
<400> 478
Ile Leu Pro Arg Lys Val Leu Gln Met
1 5
<210> 479
<211> 9
<212> PRT
<213> Homo sapiens
<400> 479
Lys Val Leu Gln Met Asp Phe Leu Val
1 5
<210> 480
<211> 10
<212> PRT
<213> Homo sapiens
<400> 480
Leu Pro Arg Lys Val Leu Gln Met Asp Phe
1 5 10
<210> 481
<211> 10
<212> PRT
<213> Homo sapiens
<400> 481
Leu Gln Met Asp Phe Leu Val His Pro Ala
1 5 10
<210> 482
<211> 9
<212> PRT
<213> Homo sapiens
<400> 482
Gln Met Asp Phe Leu Val His Pro Ala
1 5
<210> 483
<211> 10
<212> PRT
<213> Homo sapiens
<400> 483
Tyr Ile Leu Pro Arg Lys Val Leu Gln Met
1 5 10
<210> 484
<211> 10
<212> PRT
<213> Homo sapiens
<400> 484
Ala Pro Ser Pro Ala Ser Arg Leu Gln Cys
1 5 10
<210> 485
<211> 10
<212> PRT
<213> Homo sapiens
<400> 485
His Pro Leu Ala Pro Met Pro Ser Lys Thr
1 5 10
<210> 486
<211> 9
<212> PRT
<213> Homo sapiens
<400> 486
Ile Leu Pro Leu Pro Gln Leu Leu Leu
1 5
<210> 487
<211> 10
<212> PRT
<213> Homo sapiens
<400> 487
Leu Pro Thr Gln Thr Arg Gly Cys Ile Leu
1 5 10
<210> 488
<211> 10
<212> PRT
<213> Homo sapiens
<400> 488
Arg Ile Ser Cys Leu Pro Thr Gln Thr Arg
1 5 10
<210> 489
<211> 9
<212> PRT
<213> Homo sapiens
<400> 489
Ser Leu Ala Glu Thr Val Ser Leu His
1 5
<210> 490
<211> 9
<212> PRT
<213> Homo sapiens
<400> 490
Thr Pro Arg Ser Ser Ser Ser Ser Ser
1 5
<210> 491
<211> 10
<212> PRT
<213> Homo sapiens
<400> 491
Thr Pro Arg Ser Ser Ser Ser Ser Ser Ser
1 5 10
<210> 492
<211> 9
<212> PRT
<213> Homo sapiens
<400> 492
Ala Leu Pro Pro Val Leu Leu Ser Leu
1 5
<210> 493
<211> 10
<212> PRT
<213> Homo sapiens
<400> 493
Ala Leu Pro Pro Val Leu Leu Ser Leu Ala
1 5 10
<210> 494
<211> 9
<212> PRT
<213> Homo sapiens
<400> 494
Ala Leu Pro Arg Pro Leu Leu Ala Leu
1 5
<210> 495
<211> 9
<212> PRT
<213> Homo sapiens
<400> 495
Ala Ser Arg Thr Ala Ser Cys Ile Leu
1 5
<210> 496
<211> 10
<212> PRT
<213> Homo sapiens
<400> 496
Glu Ala Leu Pro Arg Pro Leu Leu Ala Leu
1 5 10
<210> 497
<211> 9
<212> PRT
<213> Homo sapiens
<400> 497
His Leu Gly His Pro Val Ala Ser Arg
1 5
<210> 498
<211> 9
<212> PRT
<213> Homo sapiens
<400> 498
His Pro Val Ala Ser Arg Thr Ala Ser
1 5
<210> 499
<211> 10
<212> PRT
<213> Homo sapiens
<400> 499
His Pro Val Ala Ser Arg Thr Ala Ser Cys
1 5 10
<210> 500
<211> 10
<212> PRT
<213> Homo sapiens
<400> 500
Ile Ile Gln Leu Ser Leu Leu Ser Leu Leu
1 5 10
<210> 501
<211> 9
<212> PRT
<213> Homo sapiens
<400> 501
Ile Gln Leu Ser Leu Leu Ser Leu Leu
1 5
<210> 502
<211> 10
<212> PRT
<213> Homo sapiens
<400> 502
Ile Gln Leu Ser Leu Leu Ser Leu Leu Ile
1 5 10
<210> 503
<211> 9
<212> PRT
<213> Homo sapiens
<400> 503
Leu Leu Ala Leu Pro Pro Val Leu Leu
1 5
<210> 504
<211> 9
<212> PRT
<213> Homo sapiens
<400> 504
Leu Leu Ile Pro Pro Leu Thr Cys Leu
1 5
<210> 505
<211> 10
<212> PRT
<213> Homo sapiens
<400> 505
Leu Leu Ile Pro Pro Leu Thr Cys Leu Ala
1 5 10
<210> 506
<211> 9
<212> PRT
<213> Homo sapiens
<400> 506
Leu Leu Ser Leu Leu Ile Pro Pro Leu
1 5
<210> 507
<211> 10
<212> PRT
<213> Homo sapiens
<400> 507
Leu Leu Ser Leu Leu Ile Pro Pro Leu Thr
1 5 10
<210> 508
<211> 10
<212> PRT
<213> Homo sapiens
<400> 508
Leu Pro Arg Pro Leu Leu Ala Leu Pro Pro
1 5 10
<210> 509
<211> 9
<212> PRT
<213> Homo sapiens
<400> 509
Gln Leu Ser Leu Leu Ser Leu Leu Ile
1 5
<210> 510
<211> 9
<212> PRT
<213> Homo sapiens
<400> 510
Arg Leu Leu Gln Cys Gln Ala Thr Arg
1 5
<210> 511
<211> 9
<212> PRT
<213> Homo sapiens
<400> 511
Arg Pro Leu Leu Ala Leu Pro Pro Val
1 5
<210> 512
<211> 10
<212> PRT
<213> Homo sapiens
<400> 512
Arg Pro Leu Leu Ala Leu Pro Pro Val Leu
1 5 10
<210> 513
<211> 9
<212> PRT
<213> Homo sapiens
<400> 513
Ser Leu Ala Gln Asp His Ser Arg Leu
1 5
<210> 514
<211> 10
<212> PRT
<213> Homo sapiens
<400> 514
Ser Leu Ala Gln Asp His Ser Arg Leu Leu
1 5 10
<210> 515
<211> 10
<212> PRT
<213> Homo sapiens
<400> 515
Ser Leu Leu Ile Pro Pro Leu Thr Cys Leu
1 5 10
<210> 516
<211> 9
<212> PRT
<213> Homo sapiens
<400> 516
Ser Leu Leu Ser Leu Leu Ile Pro Pro
1 5
<210> 517
<211> 10
<212> PRT
<213> Homo sapiens
<400> 517
Ser Leu Leu Ser Leu Leu Ile Pro Pro Leu
1 5 10
<210> 518
<211> 10
<212> PRT
<213> Homo sapiens
<400> 518
Ala Ala Ala Thr Ser Ala Ala Ser Thr Leu
1 5 10
<210> 519
<211> 10
<212> PRT
<213> Homo sapiens
<400> 519
Ala Ala Ile Pro Ala Ser Thr Ser Ala Val
1 5 10
<210> 520
<211> 9
<212> PRT
<213> Homo sapiens
<400> 520
Ala Ile Pro Ala Ser Thr Ser Ala Val
1 5
<210> 521
<211> 10
<212> PRT
<213> Homo sapiens
<400> 521
Ala Leu Pro Ala Gly Cys Val Ser Ser Ala
1 5 10
<210> 522
<211> 10
<212> PRT
<213> Homo sapiens
<400> 522
Ala Pro Leu Leu Thr Ala Thr Gly Ser Val
1 5 10
<210> 523
<211> 9
<212> PRT
<213> Homo sapiens
<400> 523
Ala Pro Val Leu Ser Ala Ser Ile Leu
1 5
<210> 524
<211> 9
<212> PRT
<213> Homo sapiens
<400> 524
Ala Thr Leu Leu Pro Ala Thr Thr Val
1 5
<210> 525
<211> 10
<212> PRT
<213> Homo sapiens
<400> 525
Ala Thr Val Ser Thr Thr Thr Ala Pro Val
1 5 10
<210> 526
<211> 10
<212> PRT
<213> Homo sapiens
<400> 526
Ala Val Pro Ala Asn Cys Leu Phe Pro Ala
1 5 10
<210> 527
<211> 10
<212> PRT
<213> Homo sapiens
<400> 527
Cys Leu Phe Pro Ala Ala Leu Pro Ser Thr
1 5 10
<210> 528
<211> 9
<212> PRT
<213> Homo sapiens
<400> 528
Cys Pro Thr Phe Val Ser Ala Ala Ala
1 5
<210> 529
<211> 9
<212> PRT
<213> Homo sapiens
<400> 529
Phe Pro Ala Ala Leu Pro Ser Thr Ala
1 5
<210> 530
<211> 10
<212> PRT
<213> Homo sapiens
<400> 530
Phe Pro Ala Ala Leu Pro Ser Thr Ala Gly
1 5 10
<210> 531
<211> 9
<212> PRT
<213> Homo sapiens
<400> 531
Gly Ala Glu Cys His Gly Arg Pro Leu
1 5
<210> 532
<211> 9
<212> PRT
<213> Homo sapiens
<400> 532
Gly Ala Ile Ser Arg Phe Ile Trp Val
1 5
<210> 533
<211> 10
<212> PRT
<213> Homo sapiens
<400> 533
Ile Leu Pro Ala Ala Ile Pro Ala Ser Thr
1 5 10
<210> 534
<211> 10
<212> PRT
<213> Homo sapiens
<400> 534
Ile Trp Val Ser Gly Ile Leu Ser Pro Leu
1 5 10
<210> 535
<211> 10
<212> PRT
<213> Homo sapiens
<400> 535
Leu Leu Thr Ala Thr Gly Ser Val Ser Leu
1 5 10
<210> 536
<211> 9
<212> PRT
<213> Homo sapiens
<400> 536
Leu Leu Tyr Thr Ala Asp Ser Ser Leu
1 5
<210> 537
<211> 9
<212> PRT
<213> Homo sapiens
<400> 537
Leu Pro Ala Ala Ala Thr Ser Ala Ala
1 5
<210> 538
<211> 10
<212> PRT
<213> Homo sapiens
<400> 538
Leu Pro Ala Ala Ala Thr Ser Ala Ala Ser
1 5 10
<210> 539
<211> 9
<212> PRT
<213> Homo sapiens
<400> 539
Leu Pro Ala Ala Ile Pro Ala Ser Thr
1 5
<210> 540
<211> 9
<212> PRT
<213> Homo sapiens
<400> 540
Leu Pro Ala Gly Cys Val Ser Ser Ala
1 5
<210> 541
<211> 10
<212> PRT
<213> Homo sapiens
<400> 541
Leu Pro Ala Gly Cys Val Ser Ser Ala Pro
1 5 10
<210> 542
<211> 9
<212> PRT
<213> Homo sapiens
<400> 542
Leu Tyr Thr Ala Asp Ser Ser Leu Trp
1 5
<210> 543
<211> 9
<212> PRT
<213> Homo sapiens
<400> 543
Gln Pro Ala Lys Ser Ser Pro Ser Ala
1 5
<210> 544
<211> 10
<212> PRT
<213> Homo sapiens
<400> 544
Gln Pro Ala Lys Ser Ser Pro Ser Ala Ala
1 5 10
<210> 545
<211> 9
<212> PRT
<213> Homo sapiens
<400> 545
Arg Phe Ile Trp Val Ser Gly Ile Leu
1 5
<210> 546
<211> 9
<212> PRT
<213> Homo sapiens
<400> 546
Arg Pro Gln Arg Val Trp Ser Thr Gly
1 5
<210> 547
<211> 10
<212> PRT
<213> Homo sapiens
<400> 547
Arg Val Trp Ser Thr Gly Pro Asp Ser Ile
1 5 10
<210> 548
<211> 9
<212> PRT
<213> Homo sapiens
<400> 548
Ser Ala Val Pro Gly Ser Ile Pro Leu
1 5
<210> 549
<211> 9
<212> PRT
<213> Homo sapiens
<400> 549
Ser Ile Leu Pro Ala Ala Ile Pro Ala
1 5
<210> 550
<211> 9
<212> PRT
<213> Homo sapiens
<400> 550
Ser Leu Pro Ala Ala Ala Thr Ser Ala
1 5
<210> 551
<211> 10
<212> PRT
<213> Homo sapiens
<400> 551
Ser Leu Pro Ala Ala Ala Thr Ser Ala Ala
1 5 10
<210> 552
<211> 9
<212> PRT
<213> Homo sapiens
<400> 552
Ser Leu Trp Thr Thr Arg Pro Gln Arg
1 5
<210> 553
<211> 10
<212> PRT
<213> Homo sapiens
<400> 553
Ser Leu Trp Thr Thr Arg Pro Gln Arg Val
1 5 10
<210> 554
<211> 9
<212> PRT
<213> Homo sapiens
<400> 554
Ser Pro Ser Ala Ala Ala Ala Thr Leu
1 5
<210> 555
<211> 10
<212> PRT
<213> Homo sapiens
<400> 555
Ser Pro Ser Ala Ala Ala Ala Thr Leu Leu
1 5 10
<210> 556
<211> 10
<212> PRT
<213> Homo sapiens
<400> 556
Thr Leu Asp Ala Leu Pro Ala Gly Cys Val
1 5 10
<210> 557
<211> 9
<212> PRT
<213> Homo sapiens
<400> 557
Thr Val Ser Thr Thr Thr Ala Pro Val
1 5
<210> 558
<211> 9
<212> PRT
<213> Homo sapiens
<400> 558
Val Leu Ser Ala Ser Ile Leu Pro Ala
1 5
<210> 559
<211> 10
<212> PRT
<213> Homo sapiens
<400> 559
Val Leu Ser Ala Ser Ile Leu Pro Ala Ala
1 5 10
<210> 560
<211> 9
<212> PRT
<213> Homo sapiens
<400> 560
Val Pro Ala Asn Cys Leu Phe Pro Ala
1 5
<210> 561
<211> 10
<212> PRT
<213> Homo sapiens
<400> 561
Val Pro Ala Asn Cys Leu Phe Pro Ala Ala
1 5 10
<210> 562
<211> 9
<212> PRT
<213> Homo sapiens
<400> 562
Val Pro Asp Pro Ser Cys Pro Thr Phe
1 5
<210> 563
<211> 9
<212> PRT
<213> Homo sapiens
<400> 563
Val Pro Gly Ser Ile Pro Leu Pro Ala
1 5
<210> 564
<211> 10
<212> PRT
<213> Homo sapiens
<400> 564
Val Pro Gly Ser Ile Pro Leu Pro Ala Val
1 5 10
<210> 565
<211> 9
<212> PRT
<213> Homo sapiens
<400> 565
Trp Val Ser Gly Ile Leu Ser Pro Leu
1 5
<210> 566
<211> 10
<212> PRT
<213> Homo sapiens
<400> 566
Tyr Thr Ala Asp Ser Ser Leu Trp Thr Thr
1 5 10
<210> 567
<211> 9
<212> PRT
<213> Homo sapiens
<400> 567
Ala Pro Ala Gly Met Val Asn Arg Ala
1 5
<210> 568
<211> 9
<212> PRT
<213> Homo sapiens
<400> 568
Ala Ser Leu His Arg Arg Ser Tyr Leu
1 5
<210> 569
<211> 10
<212> PRT
<213> Homo sapiens
<400> 569
Ala Ser Leu His Arg Arg Ser Tyr Leu Lys
1 5 10
<210> 570
<211> 10
<212> PRT
<213> Homo sapiens
<400> 570
Phe Leu Leu Met Asp Asn Lys Ala Pro Ala
1 5 10
<210> 571
<211> 9
<212> PRT
<213> Homo sapiens
<400> 571
His Pro Arg Arg Ser Pro Ser Arg Leu
1 5
<210> 572
<211> 9
<212> PRT
<213> Homo sapiens
<400> 572
His Pro Ser Leu His Ile Ser Ser Pro
1 5
<210> 573
<211> 10
<212> PRT
<213> Homo sapiens
<400> 573
His Arg Arg Ser Tyr Leu Lys Ile His Leu
1 5 10
<210> 574
<211> 10
<212> PRT
<213> Homo sapiens
<400> 574
His Ser Arg Phe Leu Leu Met Asp Asn Lys
1 5 10
<210> 575
<211> 10
<212> PRT
<213> Homo sapiens
<400> 575
Lys Leu Pro Ile Pro Ser Ser Ala Ser Leu
1 5 10
<210> 576
<211> 10
<212> PRT
<213> Homo sapiens
<400> 576
Lys Val Leu Thr Leu Ser Ser Ser Ser His
1 5 10
<210> 577
<211> 9
<212> PRT
<213> Homo sapiens
<400> 577
Leu Ile His Ser Arg Phe Leu Leu Met
1 5
<210> 578
<211> 9
<212> PRT
<213> Homo sapiens
<400> 578
Leu Leu Met Asp Asn Lys Ala Pro Ala
1 5
<210> 579
<211> 10
<212> PRT
<213> Homo sapiens
<400> 579
Leu Met Asp Asn Lys Ala Pro Ala Gly Met
1 5 10
<210> 580
<211> 9
<212> PRT
<213> Homo sapiens
<400> 580
Leu Pro Ile Pro Ser Ser Ala Ser Leu
1 5
<210> 581
<211> 9
<212> PRT
<213> Homo sapiens
<400> 581
Met Pro Asn Leu Arg Ile Ser Ser Ser
1 5
<210> 582
<211> 10
<212> PRT
<213> Homo sapiens
<400> 582
Met Pro Asn Leu Arg Ile Ser Ser Ser His
1 5 10
<210> 583
<211> 10
<212> PRT
<213> Homo sapiens
<400> 583
Asn Leu Arg Ile Ser Ser Ser His Ser Leu
1 5 10
<210> 584
<211> 10
<212> PRT
<213> Homo sapiens
<400> 584
Pro Pro Thr His Ser His Arg Leu Ser Leu
1 5 10
<210> 585
<211> 10
<212> PRT
<213> Homo sapiens
<400> 585
Arg Ala Gly Arg Arg Val Pro Trp Ala Ala
1 5 10
<210> 586
<211> 10
<212> PRT
<213> Homo sapiens
<400> 586
Arg Ala Arg Leu His Ile Thr Thr Ser Lys
1 5 10
<210> 587
<211> 10
<212> PRT
<213> Homo sapiens
<400> 587
Arg Ile Ser Ser Ser His Ser Leu Asn His
1 5 10
<210> 588
<211> 9
<212> PRT
<213> Homo sapiens
<400> 588
Arg Leu His Thr Pro Pro Ser Ser Arg
1 5
<210> 589
<211> 10
<212> PRT
<213> Homo sapiens
<400> 589
Arg Leu His Thr Pro Pro Ser Ser Arg Arg
1 5 10
<210> 590
<211> 9
<212> PRT
<213> Homo sapiens
<400> 590
Arg Leu Arg Ile Leu Ser Pro Ser Leu
1 5
<210> 591
<211> 9
<212> PRT
<213> Homo sapiens
<400> 591
Arg Pro Leu Met Pro Asn Leu Arg Ile
1 5
<210> 592
<211> 9
<212> PRT
<213> Homo sapiens
<400> 592
Arg Pro Arg Pro Leu Met Pro Asn Leu
1 5
<210> 593
<211> 10
<212> PRT
<213> Homo sapiens
<400> 593
Ser Ala Ser Leu His Arg Arg Ser Tyr Leu
1 5 10
<210> 594
<211> 9
<212> PRT
<213> Homo sapiens
<400> 594
Ser Leu His Ile Ser Ser Pro Arg Leu
1 5
<210> 595
<211> 9
<212> PRT
<213> Homo sapiens
<400> 595
Ser Leu His Arg Arg Ser Tyr Leu Lys
1 5
<210> 596
<211> 10
<212> PRT
<213> Homo sapiens
<400> 596
Ser Leu His Arg Arg Ser Tyr Leu Lys Ile
1 5 10
<210> 597
<211> 9
<212> PRT
<213> Homo sapiens
<400> 597
Ser Leu Ile His Ser Arg Phe Leu Leu
1 5
<210> 598
<211> 10
<212> PRT
<213> Homo sapiens
<400> 598
Ser Leu Ile His Ser Arg Phe Leu Leu Met
1 5 10
<210> 599
<211> 9
<212> PRT
<213> Homo sapiens
<400> 599
Ser Leu Leu Thr Ser Ser Ser Asn Leu
1 5
<210> 600
<211> 10
<212> PRT
<213> Homo sapiens
<400> 600
Ser Leu Asn His His Ser Ser Ser Pro Leu
1 5 10
<210> 601
<211> 10
<212> PRT
<213> Homo sapiens
<400> 601
Ser Leu Ser Ser Pro Ser Lys Leu Pro Ile
1 5 10
<210> 602
<211> 9
<212> PRT
<213> Homo sapiens
<400> 602
Ser Pro Leu Ser Leu His Thr Pro Ser
1 5
<210> 603
<211> 10
<212> PRT
<213> Homo sapiens
<400> 603
Ser Pro Leu Ser Leu His Thr Pro Ser Ser
1 5 10
<210> 604
<211> 9
<212> PRT
<213> Homo sapiens
<400> 604
Ser Pro Pro Thr His Ser His Arg Leu
1 5
<210> 605
<211> 9
<212> PRT
<213> Homo sapiens
<400> 605
Ser Pro Arg Leu His Thr Pro Pro Ser
1 5
<210> 606
<211> 10
<212> PRT
<213> Homo sapiens
<400> 606
Ser Pro Arg Leu His Thr Pro Pro Ser Ser
1 5 10
<210> 607
<211> 10
<212> PRT
<213> Homo sapiens
<400> 607
Ser Pro Ser Leu Ser Ser Pro Ser Lys Leu
1 5 10
<210> 608
<211> 9
<212> PRT
<213> Homo sapiens
<400> 608
Ser Tyr Leu Lys Ile His Leu Gly Leu
1 5
<210> 609
<211> 10
<212> PRT
<213> Homo sapiens
<400> 609
Thr Pro Ser Asn His Arg Pro Arg Pro Leu
1 5 10
<210> 610
<211> 10
<212> PRT
<213> Homo sapiens
<400> 610
Thr Pro Ser Ser His Pro Ser Leu His Ile
1 5 10
<210> 611
<211> 10
<212> PRT
<213> Homo sapiens
<400> 611
Cys Val Pro Phe Trp Thr Gly Arg Ile Leu
1 5 10
<210> 612
<211> 11
<212> PRT
<213> Homo sapiens
<400> 612
His Cys Val Pro Phe Trp Thr Gly Arg Ile Leu
1 5 10
<210> 613
<211> 9
<212> PRT
<213> Homo sapiens
<400> 613
Ile Leu Leu Pro Ser Ala Ala Ser Val
1 5
<210> 614
<211> 10
<212> PRT
<213> Homo sapiens
<400> 614
Ile Leu Leu Pro Ser Ala Ala Ser Val Cys
1 5 10
<210> 615
<211> 11
<212> PRT
<213> Homo sapiens
<400> 615
Leu Leu Pro Ser Ala Ala Ser Val Cys Pro Ile
1 5 10
<210> 616
<211> 10
<212> PRT
<213> Homo sapiens
<400> 616
Leu Pro Ser Ala Ala Ser Val Cys Pro Ile
1 5 10
<210> 617
<211> 8
<212> PRT
<213> Homo sapiens
<400> 617
Met Arg Pro His Cys Val Pro Phe
1 5
<210> 618
<211> 10
<212> PRT
<213> Homo sapiens
<400> 618
Arg Ile Leu Leu Pro Ser Ala Ala Ser Val
1 5 10
<210> 619
<211> 9
<212> PRT
<213> Homo sapiens
<400> 619
Arg Met Arg Pro His Cys Val Pro Phe
1 5
<210> 620
<211> 10
<212> PRT
<213> Homo sapiens
<400> 620
Arg Met Arg Pro His Cys Val Pro Phe Trp
1 5 10
<210> 621
<211> 9
<212> PRT
<213> Homo sapiens
<400> 621
Arg Thr Asn Pro Thr Val Arg Met Arg
1 5
<210> 622
<211> 9
<212> PRT
<213> Homo sapiens
<400> 622
Ser Val Cys Pro Ile Pro Phe Glu Ala
1 5
<210> 623
<211> 9
<212> PRT
<213> Homo sapiens
<400> 623
Thr Val Arg Met Arg Pro His Cys Val
1 5
<210> 624
<211> 11
<212> PRT
<213> Homo sapiens
<400> 624
Thr Val Arg Met Arg Pro His Cys Val Pro Phe
1 5 10
<210> 625
<211> 9
<212> PRT
<213> Homo sapiens
<400> 625
Val Pro Phe Trp Thr Gly Arg Ile Leu
1 5
<210> 626
<211> 10
<212> PRT
<213> Homo sapiens
<400> 626
Val Pro Phe Trp Thr Gly Arg Ile Leu Leu
1 5 10
<210> 627
<211> 10
<212> PRT
<213> Homo sapiens
<400> 627
Val Arg Met Arg Pro His Cys Val Pro Phe
1 5 10
<210> 628
<211> 9
<212> PRT
<213> Homo sapiens
<400> 628
Ala Met Val Pro Arg Gly Val Ser Met
1 5
<210> 629
<211> 10
<212> PRT
<213> Homo sapiens
<400> 629
Ala Met Val Pro Arg Gly Val Ser Met Ala
1 5 10
<210> 630
<211> 10
<212> PRT
<213> Homo sapiens
<400> 630
Ala Trp Ala Pro Thr Ser Arg Thr Pro Trp
1 5 10
<210> 631
<211> 10
<212> PRT
<213> Homo sapiens
<400> 631
Cys Pro Met Pro Thr Thr Pro Val Gln Ala
1 5 10
<210> 632
<211> 10
<212> PRT
<213> Homo sapiens
<400> 632
Cys Pro Ser Thr Val Pro Pro Ser Pro Ala
1 5 10
<210> 633
<211> 10
<212> PRT
<213> Homo sapiens
<400> 633
Gly Ala Met Val Pro Arg Gly Val Ser Met
1 5 10
<210> 634
<211> 10
<212> PRT
<213> Homo sapiens
<400> 634
Met Pro Cys Pro Met Pro Thr Thr Pro Val
1 5 10
<210> 635
<211> 9
<212> PRT
<213> Homo sapiens
<400> 635
Met Pro Thr Thr Pro Val Gln Ala Trp
1 5
<210> 636
<211> 10
<212> PRT
<213> Homo sapiens
<400> 636
Met Pro Thr Thr Pro Val Gln Ala Trp Leu
1 5 10
<210> 637
<211> 10
<212> PRT
<213> Homo sapiens
<400> 637
Ser Leu Pro Glu Asp Arg Tyr Thr Gln Ala
1 5 10
<210> 638
<211> 9
<212> PRT
<213> Homo sapiens
<400> 638
Ser Pro Ala Gln Pro Tyr Leu Arg Val
1 5
<210> 639
<211> 10
<212> PRT
<213> Homo sapiens
<400> 639
Ser Pro Ala Gln Pro Tyr Leu Arg Val Ser
1 5 10
<210> 640
<211> 9
<212> PRT
<213> Homo sapiens
<400> 640
Thr Ile Met Pro Cys Pro Met Pro Thr
1 5
<210> 641
<211> 9
<212> PRT
<213> Homo sapiens
<400> 641
Thr Pro Val Gln Ala Trp Leu Glu Ala
1 5
<210> 642
<211> 9
<212> PRT
<213> Homo sapiens
<400> 642
Thr Ser Arg Thr Pro Trp Gly Ala Met
1 5
<210> 643
<211> 10
<212> PRT
<213> Homo sapiens
<400> 643
Val Pro Pro Ser Pro Ala Gln Pro Tyr Leu
1 5 10
<210> 644
<211> 9
<212> PRT
<213> Homo sapiens
<400> 644
Val Pro Arg Gly Val Ser Met Ala His
1 5
<210> 645
<211> 10
<212> PRT
<213> Homo sapiens
<400> 645
Cys Leu Ser Ala Arg Thr Gly Leu Ser Ile
1 5 10
<210> 646
<211> 10
<212> PRT
<213> Homo sapiens
<400> 646
Cys Thr Thr Leu Asn Ser Pro Pro Leu Lys
1 5 10
<210> 647
<211> 9
<212> PRT
<213> Homo sapiens
<400> 647
Gly Leu Ser Ile Ser Cys Thr Thr Leu
1 5
<210> 648
<211> 9
<212> PRT
<213> Homo sapiens
<400> 648
Ser Pro Pro Leu Lys Lys Met Ser Met
1 5
<210> 649
<211> 9
<212> PRT
<213> Homo sapiens
<400> 649
Thr Leu Asn Ser Pro Pro Leu Lys Lys
1 5
<210> 650
<211> 9
<212> PRT
<213> Homo sapiens
<400> 650
Thr Thr Leu Asn Ser Pro Pro Leu Lys
1 5
<210> 651
<211> 10
<212> PRT
<213> Homo sapiens
<400> 651
Thr Thr Leu Asn Ser Pro Pro Leu Lys Lys
1 5 10
<210> 652
<211> 9
<212> PRT
<213> Homo sapiens
<400> 652
Leu Gln Arg Phe Arg Phe Thr His Val
1 5
<210> 653
<211> 10
<212> PRT
<213> Homo sapiens
<400> 653
Leu Gln Arg Phe Arg Phe Thr His Val Ile
1 5 10
<210> 654
<211> 9
<212> PRT
<213> Homo sapiens
<400> 654
Arg Leu Ser Ser Val Leu Gln Arg Phe
1 5
<210> 655
<211> 10
<212> PRT
<213> Homo sapiens
<400> 655
Arg Leu Ser Ser Val Leu Gln Arg Phe Arg
1 5 10
<210> 656
<211> 10
<212> PRT
<213> Homo sapiens
<400> 656
Val Leu Gln Arg Phe Arg Phe Thr His Val
1 5 10
<210> 657
<211> 10
<212> PRT
<213> Homo sapiens
<400> 657
Ala Ser Val Gly Arg His Ser Leu Ser Lys
1 5 10
<210> 658
<211> 9
<212> PRT
<213> Homo sapiens
<400> 658
Lys Phe Leu Pro Phe Trp Gly Phe Leu
1 5
<210> 659
<211> 9
<212> PRT
<213> Homo sapiens
<400> 659
Leu Ala Ser Val Gly Arg His Ser Leu
1 5
<210> 660
<211> 10
<212> PRT
<213> Homo sapiens
<400> 660
Leu Pro Phe Trp Gly Phe Leu Glu Glu Phe
1 5 10
<210> 661
<211> 9
<212> PRT
<213> Homo sapiens
<400> 661
Pro Phe Trp Gly Phe Leu Glu Glu Phe
1 5
<210> 662
<211> 9
<212> PRT
<213> Homo sapiens
<400> 662
Ser Val Gly Arg His Ser Leu Ser Lys
1 5
<210> 663
<211> 9
<212> PRT
<213> Homo sapiens
<400> 663
Ala Pro Arg Pro Ile Arg Pro Pro Phe
1 5
<210> 664
<211> 10
<212> PRT
<213> Homo sapiens
<400> 664
Ala Pro Arg Pro Ile Arg Pro Pro Phe Leu
1 5 10
<210> 665
<211> 10
<212> PRT
<213> Homo sapiens
<400> 665
Ala Gln Lys Met Lys Lys Ala His Phe Leu
1 5 10
<210> 666
<211> 9
<212> PRT
<213> Homo sapiens
<400> 666
Phe Leu Pro Ser Ala Ala Gln Lys Met
1 5
<210> 667
<211> 10
<212> PRT
<213> Homo sapiens
<400> 667
Gly Leu Phe Leu Pro Ser Ala Ala Gln Lys
1 5 10
<210> 668
<211> 9
<212> PRT
<213> Homo sapiens
<400> 668
His Pro Leu Leu Val Ser Leu Leu Leu
1 5
<210> 669
<211> 10
<212> PRT
<213> Homo sapiens
<400> 669
Lys Ala His Phe Leu Lys Thr Trp Phe Arg
1 5 10
<210> 670
<211> 9
<212> PRT
<213> Homo sapiens
<400> 670
Lys Ala Arg Phe Ser Thr Ala Ser Leu
1 5
<210> 671
<211> 9
<212> PRT
<213> Homo sapiens
<400> 671
Lys Met Lys Lys Ala His Phe Leu Lys
1 5
<210> 672
<211> 10
<212> PRT
<213> Homo sapiens
<400> 672
Lys Thr Trp Phe Arg Ser Asn Pro Thr Lys
1 5 10
<210> 673
<211> 9
<212> PRT
<213> Homo sapiens
<400> 673
Leu Ala Lys Glu Leu Thr His Pro Leu
1 5
<210> 674
<211> 10
<212> PRT
<213> Homo sapiens
<400> 674
Leu Ala Lys Glu Leu Thr His Pro Leu Leu
1 5 10
<210> 675
<211> 10
<212> PRT
<213> Homo sapiens
<400> 675
Asn Pro Thr Lys Thr Lys Lys Ala Arg Phe
1 5 10
<210> 676
<211> 9
<212> PRT
<213> Homo sapiens
<400> 676
Gln Lys Met Lys Lys Ala His Phe Leu
1 5
<210> 677
<211> 9
<212> PRT
<213> Homo sapiens
<400> 677
Arg Phe Ser Thr Ala Ser Leu Ala Lys
1 5
<210> 678
<211> 10
<212> PRT
<213> Homo sapiens
<400> 678
Arg Pro Ile Arg Pro Pro Phe Leu Glu Ser
1 5 10
<210> 679
<211> 9
<212> PRT
<213> Homo sapiens
<400> 679
Arg Ser Asn Pro Thr Lys Thr Lys Lys
1 5
<210> 680
<211> 10
<212> PRT
<213> Homo sapiens
<400> 680
Ser Leu Ala Lys Glu Leu Thr His Pro Leu
1 5 10
<210> 681
<211> 9
<212> PRT
<213> Homo sapiens
<400> 681
Thr Lys Lys Ala Arg Phe Ser Thr Ala
1 5
<210> 682
<211> 9
<212> PRT
<213> Homo sapiens
<400> 682
Gly Leu Arg Phe Trp Asn Pro Ser Arg
1 5
<210> 683
<211> 10
<212> PRT
<213> Homo sapiens
<400> 683
Ile Ser Gln Leu Leu Ser Trp Pro Gln Lys
1 5 10
<210> 684
<211> 9
<212> PRT
<213> Homo sapiens
<400> 684
Arg Ile Ala His Ile Ser Gln Leu Leu
1 5
<210> 685
<211> 9
<212> PRT
<213> Homo sapiens
<400> 685
Arg Leu Gly Tyr Ser Ser His Gln Leu
1 5
<210> 686
<211> 9
<212> PRT
<213> Homo sapiens
<400> 686
Ser Gln Leu Leu Ser Trp Pro Gln Lys
1 5
<210> 687
<211> 9
<212> PRT
<213> Homo sapiens
<400> 687
Ser Arg Ile Ala His Ile Ser Gln Leu
1 5
<210> 688
<211> 9
<212> PRT
<213> Homo sapiens
<400> 688
Trp Pro Gln Lys Thr Glu Glu Arg Leu
1 5
<210> 689
<211> 9
<212> PRT
<213> Homo sapiens
<400> 689
Tyr Ser Ser His Gln Leu Pro Arg Lys
1 5
<210> 690
<211> 10
<212> PRT
<213> Homo sapiens
<400> 690
Cys Pro Gln Arg Met Thr Pro Gly Thr Thr
1 5 10
<210> 691
<211> 9
<212> PRT
<213> Homo sapiens
<400> 691
Glu Ala Glu Lys Arg Thr Arg Thr Leu
1 5
<210> 692
<211> 9
<212> PRT
<213> Homo sapiens
<400> 692
Gly Thr Thr Phe Ile Thr Met Met Lys
1 5
<210> 693
<211> 10
<212> PRT
<213> Homo sapiens
<400> 693
Gly Thr Thr Phe Ile Thr Met Met Lys Lys
1 5 10
<210> 694
<211> 10
<212> PRT
<213> Homo sapiens
<400> 694
Ile Thr Met Met Lys Lys Glu Ala Glu Lys
1 5 10
<210> 695
<211> 9
<212> PRT
<213> Homo sapiens
<400> 695
Arg Met Thr Pro Gly Thr Thr Phe Ile
1 5
<210> 696
<211> 9
<212> PRT
<213> Homo sapiens
<400> 696
Ser Pro Tyr Cys Pro Gln Arg Met Thr
1 5
<210> 697
<211> 9
<212> PRT
<213> Homo sapiens
<400> 697
Thr Met Met Lys Lys Glu Ala Glu Lys
1 5
<210> 698
<211> 9
<212> PRT
<213> Homo sapiens
<400> 698
Thr Pro Gly Thr Thr Phe Ile Thr Met
1 5
<210> 699
<211> 10
<212> PRT
<213> Homo sapiens
<400> 699
Thr Pro Gly Thr Thr Phe Ile Thr Met Met
1 5 10
<210> 700
<211> 9
<212> PRT
<213> Homo sapiens
<400> 700
Thr Thr Phe Ile Thr Met Met Lys Lys
1 5
<210> 701
<211> 9
<212> PRT
<213> Homo sapiens
<400> 701
Cys Pro Gly Ala Thr Trp Arg Glu Ala
1 5
<210> 702
<211> 10
<212> PRT
<213> Homo sapiens
<400> 702
Cys Pro Gly Ala Thr Trp Arg Glu Ala Ala
1 5 10
<210> 703
<211> 10
<212> PRT
<213> Homo sapiens
<400> 703
Arg Ser Arg Cys Pro Gly Ala Thr Trp Arg
1 5 10
<210> 704
<211> 9
<212> PRT
<213> Homo sapiens
<400> 704
Thr Pro Arg Ala Thr Arg Ser Arg Cys
1 5
<210> 705
<211> 10
<212> PRT
<213> Homo sapiens
<400> 705
His Val Leu Pro Glu Pro His Leu Ala Leu
1 5 10
<210> 706
<211> 10
<212> PRT
<213> Homo sapiens
<400> 706
Arg Pro Leu Gln Thr His Val Leu Pro Glu
1 5 10
<210> 707
<211> 10
<212> PRT
<213> Homo sapiens
<400> 707
Val Leu Trp Thr Thr Pro Pro Leu Gln His
1 5 10
<210> 708
<211> 10
<212> PRT
<213> Homo sapiens
<400> 708
Ala Pro Ser Glu Ser Pro Cys Ser Pro Phe
1 5 10
<210> 709
<211> 10
<212> PRT
<213> Homo sapiens
<400> 709
Cys Pro Leu Asp His Thr Thr Pro Pro Ala
1 5 10
<210> 710
<211> 9
<212> PRT
<213> Homo sapiens
<400> 710
Phe Leu Gln Glu Gln Tyr His Glu Ala
1 5
<210> 711
<211> 10
<212> PRT
<213> Homo sapiens
<400> 711
Arg Leu Ala Phe Leu Gln Glu Gln Tyr His
1 5 10
<210> 712
<211> 10
<212> PRT
<213> Homo sapiens
<400> 712
Ser Pro Cys Ser Pro Phe Arg Leu Ala Phe
1 5 10
<210> 713
<211> 9
<212> PRT
<213> Homo sapiens
<400> 713
Ser Pro Pro Trp Val Arg Ala Leu Leu
1 5
<210> 714
<211> 10
<212> PRT
<213> Homo sapiens
<400> 714
Tyr Pro Ala Cys Pro Leu Asp His Thr Thr
1 5 10
<210> 715
<211> 9
<212> PRT
<213> Homo sapiens
<400> 715
Ala Leu His Leu Arg Ser Cys Pro Cys
1 5
<210> 716
<211> 9
<212> PRT
<213> Homo sapiens
<400> 716
Cys Leu His His Arg Arg His Leu Val
1 5
<210> 717
<211> 10
<212> PRT
<213> Homo sapiens
<400> 717
Cys Leu His His Arg Arg His Leu Val Cys
1 5 10
<210> 718
<211> 10
<212> PRT
<213> Homo sapiens
<400> 718
Cys Leu His Arg Lys Ser His Pro His Leu
1 5 10
<210> 719
<211> 9
<212> PRT
<213> Homo sapiens
<400> 719
Cys Leu Arg Ser His Ala Cys Pro Pro
1 5
<210> 720
<211> 9
<212> PRT
<213> Homo sapiens
<400> 720
Cys Leu Arg Ser His Thr Cys Pro Pro
1 5
<210> 721
<211> 9
<212> PRT
<213> Homo sapiens
<400> 721
Cys Leu Trp Cys His Ala Cys Leu His
1 5
<210> 722
<211> 9
<212> PRT
<213> Homo sapiens
<400> 722
Cys Pro His His Leu Lys Asn His Leu
1 5
<210> 723
<211> 9
<212> PRT
<213> Homo sapiens
<400> 723
Cys Pro His His Leu Lys Asn Leu Leu
1 5
<210> 724
<211> 9
<212> PRT
<213> Homo sapiens
<400> 724
Cys Pro His His Leu Arg Thr Arg Leu
1 5
<210> 725
<211> 10
<212> PRT
<213> Homo sapiens
<400> 725
Cys Pro Leu His Leu Arg Ser Leu Pro Phe
1 5 10
<210> 726
<211> 10
<212> PRT
<213> Homo sapiens
<400> 726
Cys Pro Leu Pro Arg His Leu Lys His Leu
1 5 10
<210> 727
<211> 10
<212> PRT
<213> Homo sapiens
<400> 727
Cys Pro Leu Ser Leu Arg Ser His Pro Cys
1 5 10
<210> 728
<211> 10
<212> PRT
<213> Homo sapiens
<400> 728
Cys Pro Arg His Leu Arg Asn Cys Pro Leu
1 5 10
<210> 729
<211> 9
<212> PRT
<213> Homo sapiens
<400> 729
Phe Pro His His Leu Arg His His Ala
1 5
<210> 730
<211> 10
<212> PRT
<213> Homo sapiens
<400> 730
Phe Pro His His Leu Arg His His Ala Cys
1 5 10
<210> 731
<211> 9
<212> PRT
<213> Homo sapiens
<400> 731
Gly Leu Arg Ser Arg Thr Cys Pro Pro
1 5
<210> 732
<211> 10
<212> PRT
<213> Homo sapiens
<400> 732
His Ala Cys Leu His Arg Leu Arg Asn Leu
1 5 10
<210> 733
<211> 9
<212> PRT
<213> Homo sapiens
<400> 733
His Leu Ala Cys Leu His His Leu Arg
1 5
<210> 734
<211> 9
<212> PRT
<213> Homo sapiens
<400> 734
His Leu Cys Pro Pro His Leu Arg Tyr
1 5
<210> 735
<211> 10
<212> PRT
<213> Homo sapiens
<400> 735
His Leu Cys Pro Pro His Leu Arg Tyr Arg
1 5 10
<210> 736
<211> 9
<212> PRT
<213> Homo sapiens
<400> 736
His Leu Lys His Leu Ala Cys Leu His
1 5
<210> 737
<211> 9
<212> PRT
<213> Homo sapiens
<400> 737
His Leu Lys His Arg Pro Cys Pro His
1 5
<210> 738
<211> 9
<212> PRT
<213> Homo sapiens
<400> 738
His Leu Lys Asn His Leu Cys Pro Pro
1 5
<210> 739
<211> 9
<212> PRT
<213> Homo sapiens
<400> 739
His Leu Lys Ser His Pro Cys Leu His
1 5
<210> 740
<211> 9
<212> PRT
<213> Homo sapiens
<400> 740
His Leu Lys Ser Leu Leu Cys Pro Leu
1 5
<210> 741
<211> 9
<212> PRT
<213> Homo sapiens
<400> 741
His Leu Leu His Leu Arg Arg Leu Tyr
1 5
<210> 742
<211> 9
<212> PRT
<213> Homo sapiens
<400> 742
His Leu Arg Asn Cys Pro Leu Pro Arg
1 5
<210> 743
<211> 10
<212> PRT
<213> Homo sapiens
<400> 743
His Leu Arg Asn Cys Pro Leu Pro Arg His
1 5 10
<210> 744
<211> 10
<212> PRT
<213> Homo sapiens
<400> 744
His Leu Arg Arg Leu Tyr Pro His His Leu
1 5 10
<210> 745
<211> 9
<212> PRT
<213> Homo sapiens
<400> 745
His Leu Arg Ser His Pro Cys Pro Leu
1 5
<210> 746
<211> 10
<212> PRT
<213> Homo sapiens
<400> 746
His Leu Arg Ser His Pro Cys Pro Leu His
1 5 10
<210> 747
<211> 9
<212> PRT
<213> Homo sapiens
<400> 747
His Leu Arg Ser Leu Pro Phe Pro His
1 5
<210> 748
<211> 9
<212> PRT
<213> Homo sapiens
<400> 748
His Leu Arg Thr Arg Leu Cys Pro His
1 5
<210> 749
<211> 9
<212> PRT
<213> Homo sapiens
<400> 749
His Leu Val Cys Ser His His Leu Lys
1 5
<210> 750
<211> 10
<212> PRT
<213> Homo sapiens
<400> 750
His Pro Cys Leu His His Arg Arg His Leu
1 5 10
<210> 751
<211> 9
<212> PRT
<213> Homo sapiens
<400> 751
His Pro Gly Leu Arg Ser Arg Thr Cys
1 5
<210> 752
<211> 10
<212> PRT
<213> Homo sapiens
<400> 752
His Pro His Leu Leu His Leu Arg Arg Leu
1 5 10
<210> 753
<211> 9
<212> PRT
<213> Homo sapiens
<400> 753
His Arg Lys Ser His Pro His Leu Leu
1 5
<210> 754
<211> 9
<212> PRT
<213> Homo sapiens
<400> 754
His Arg Arg Thr Arg Ser Cys Pro Cys
1 5
<210> 755
<211> 10
<212> PRT
<213> Homo sapiens
<400> 755
Lys Ser His Pro His Leu Leu His Leu Arg
1 5 10
<210> 756
<211> 10
<212> PRT
<213> Homo sapiens
<400> 756
Lys Ser Leu Leu Cys Pro Leu His Leu Arg
1 5 10
<210> 757
<211> 10
<212> PRT
<213> Homo sapiens
<400> 757
Leu Leu Cys Pro Leu His Leu Arg Ser Leu
1 5 10
<210> 758
<211> 10
<212> PRT
<213> Homo sapiens
<400> 758
Leu Leu His Leu Arg Arg Leu Tyr Pro His
1 5 10
<210> 759
<211> 9
<212> PRT
<213> Homo sapiens
<400> 759
Leu Pro Arg His Leu Lys His Leu Ala
1 5
<210> 760
<211> 10
<212> PRT
<213> Homo sapiens
<400> 760
Leu Pro Arg His Leu Lys His Leu Ala Cys
1 5 10
<210> 761
<211> 9
<212> PRT
<213> Homo sapiens
<400> 761
Leu Arg Arg Leu Arg Ser His Thr Cys
1 5
<210> 762
<211> 9
<212> PRT
<213> Homo sapiens
<400> 762
Leu Arg Arg Leu Tyr Pro His His Leu
1 5
<210> 763
<211> 10
<212> PRT
<213> Homo sapiens
<400> 763
Leu Val Cys Ser His His Leu Lys Ser Leu
1 5 10
<210> 764
<211> 10
<212> PRT
<213> Homo sapiens
<400> 764
Asn Leu Arg Asn His Thr Cys Pro Pro Ser
1 5 10
<210> 765
<211> 9
<212> PRT
<213> Homo sapiens
<400> 765
Pro Leu His Leu Arg Ser Leu Pro Phe
1 5
<210> 766
<211> 10
<212> PRT
<213> Homo sapiens
<400> 766
Arg Leu Cys Pro His His Leu Lys Asn His
1 5 10
<210> 767
<211> 9
<212> PRT
<213> Homo sapiens
<400> 767
Arg Leu Tyr Pro His His Leu Lys His
1 5
<210> 768
<211> 10
<212> PRT
<213> Homo sapiens
<400> 768
Arg Leu Tyr Pro His His Leu Lys His Arg
1 5 10
<210> 769
<211> 10
<212> PRT
<213> Homo sapiens
<400> 769
Arg Pro Cys Pro His His Leu Lys Asn Leu
1 5 10
<210> 770
<211> 10
<212> PRT
<213> Homo sapiens
<400> 770
Arg Ser His Pro Cys Pro Leu His Leu Lys
1 5 10
<210> 771
<211> 10
<212> PRT
<213> Homo sapiens
<400> 771
Arg Ser Leu Pro Phe Pro His His Leu Arg
1 5 10
<210> 772
<211> 9
<212> PRT
<213> Homo sapiens
<400> 772
Arg Thr Arg Leu Cys Pro His His Leu
1 5
<210> 773
<211> 10
<212> PRT
<213> Homo sapiens
<400> 773
Arg Thr Arg Leu Cys Pro His His Leu Lys
1 5 10
<210> 774
<211> 9
<212> PRT
<213> Homo sapiens
<400> 774
Ser Leu Leu Cys Pro Leu His Leu Arg
1 5
<210> 775
<211> 9
<212> PRT
<213> Homo sapiens
<400> 775
Ser Leu Arg Ser His Ala Cys Pro Pro
1 5
<210> 776
<211> 9
<212> PRT
<213> Homo sapiens
<400> 776
Ser Pro Leu Arg Ser Gln Ala Asn Ala
1 5
<210> 777
<211> 9
<212> PRT
<213> Homo sapiens
<400> 777
Tyr Leu Arg Arg Leu Arg Ser His Thr
1 5
<210> 778
<211> 10
<212> PRT
<213> Homo sapiens
<400> 778
Tyr Pro His His Leu Lys His Arg Pro Cys
1 5 10
<210> 779
<211> 9
<212> PRT
<213> Homo sapiens
<400> 779
Phe Ile Thr Ser Gly Gln Ile Phe Lys
1 5
<210> 780
<211> 9
<212> PRT
<213> Homo sapiens
<400> 780
Ile Phe Ile Thr Ser Gly Gln Ile Phe
1 5
<210> 781
<211> 9
<212> PRT
<213> Homo sapiens
<400> 781
Ser Gln Ser Glu Ala Leu Cys Val Leu
1 5
<210> 782
<211> 10
<212> PRT
<213> Homo sapiens
<400> 782
Ser Gln Ser Glu Ala Leu Cys Val Leu Leu
1 5 10
<210> 783
<211> 9
<212> PRT
<213> Homo sapiens
<400> 783
Ala Tyr Thr Gly Thr Ile Leu Met Met
1 5
<210> 784
<211> 10
<212> PRT
<213> Homo sapiens
<400> 784
Asp Leu Lys Ala Tyr Thr Gly Thr Ile Leu
1 5 10
<210> 785
<211> 9
<212> PRT
<213> Homo sapiens
<400> 785
Ile Leu Thr Lys Gln Ile Lys Thr Lys
1 5
<210> 786
<211> 9
<212> PRT
<213> Homo sapiens
<400> 786
Lys Met Ile Leu Thr Lys Gln Ile Lys
1 5
<210> 787
<211> 9
<212> PRT
<213> Homo sapiens
<400> 787
Lys Pro Thr Asp Thr Phe Leu Gln Ile
1 5
<210> 788
<211> 10
<212> PRT
<213> Homo sapiens
<400> 788
Lys Pro Thr Asp Thr Phe Leu Gln Ile Leu
1 5 10
<210> 789
<211> 10
<212> PRT
<213> Homo sapiens
<400> 789
Met Ile Leu Thr Lys Gln Ile Lys Thr Lys
1 5 10
<210> 790
<211> 10
<212> PRT
<213> Homo sapiens
<400> 790
Ile Thr Arg Tyr Thr Ile Phe Val Leu Lys
1 5 10
<210> 791
<211> 9
<212> PRT
<213> Homo sapiens
<400> 791
Leu Ile Ala Glu Leu His Asn Ile Leu
1 5
<210> 792
<211> 10
<212> PRT
<213> Homo sapiens
<400> 792
Leu Ile Ala Glu Leu His Asn Ile Leu Leu
1 5 10
<210> 793
<211> 10
<212> PRT
<213> Homo sapiens
<400> 793
Met Thr Pro Pro Asn Leu Ile Ala Glu Leu
1 5 10
<210> 794
<211> 9
<212> PRT
<213> Homo sapiens
<400> 794
Asn Leu Ile Ala Glu Leu His Asn Ile
1 5
<210> 795
<211> 10
<212> PRT
<213> Homo sapiens
<400> 795
Asn Leu Ile Ala Glu Leu His Asn Ile Leu
1 5 10
<210> 796
<211> 10
<212> PRT
<213> Homo sapiens
<400> 796
Arg Tyr Thr Ile Phe Val Leu Lys Asp Ile
1 5 10
<210> 797
<211> 10
<212> PRT
<213> Homo sapiens
<400> 797
Thr Ile Thr Arg Tyr Thr Ile Phe Val Leu
1 5 10
<210> 798
<211> 9
<212> PRT
<213> Homo sapiens
<400> 798
Thr Pro Pro Asn Leu Ile Ala Glu Leu
1 5
<210> 799
<211> 9
<212> PRT
<213> Homo sapiens
<400> 799
Phe Leu Gln Phe Arg Thr His Thr Thr
1 5
<210> 800
<211> 10
<212> PRT
<213> Homo sapiens
<400> 800
Leu Pro Ala Lys Gly Glu Asp Ile Phe Leu
1 5 10
<210> 801
<211> 10
<212> PRT
<213> Homo sapiens
<400> 801
Leu Gln Phe Arg Thr His Thr Thr Gly Arg
1 5 10
<210> 802
<211> 9
<212> PRT
<213> Homo sapiens
<400> 802
Asn Leu Gln Ser Ser Val Cys Gly Leu
1 5
<210> 803
<211> 9
<212> PRT
<213> Homo sapiens
<400> 803
Ser Ser Val Cys Gly Leu Pro Ala Lys
1 5
<210> 804
<211> 10
<212> PRT
<213> Homo sapiens
<400> 804
Val Gln Trp Arg Asn Leu Gln Ser Ser Val
1 5 10
<210> 805
<211> 9
<212> PRT
<213> Homo sapiens
<400> 805
Gly Gln Asn Val Ser Leu Leu Gly Lys
1 5
<210> 806
<211> 10
<212> PRT
<213> Homo sapiens
<400> 806
His Thr Arg Thr Arg Gly Asn Leu Arg Lys
1 5 10
<210> 807
<211> 10
<212> PRT
<213> Homo sapiens
<400> 807
Ile Leu His Thr Arg Thr Arg Gly Asn Leu
1 5 10
<210> 808
<211> 10
<212> PRT
<213> Homo sapiens
<400> 808
Lys Gly Gln Asn Val Ser Leu Leu Gly Lys
1 5 10
<210> 809
<211> 9
<212> PRT
<213> Homo sapiens
<400> 809
Leu Leu Gly Lys Tyr Ile Leu His Thr
1 5
<210> 810
<211> 10
<212> PRT
<213> Homo sapiens
<400> 810
Leu Arg Lys Ser Arg Lys Trp Lys Ser Met
1 5 10
<210> 811
<211> 9
<212> PRT
<213> Homo sapiens
<400> 811
Ser Leu Leu Gly Lys Tyr Ile Leu His
1 5
<210> 812
<211> 10
<212> PRT
<213> Homo sapiens
<400> 812
Ser Leu Leu Gly Lys Tyr Ile Leu His Thr
1 5 10
<210> 813
<211> 9
<212> PRT
<213> Homo sapiens
<400> 813
Cys Thr Ser Pro Leu Leu Ala Pro Val
1 5
<210> 814
<211> 9
<212> PRT
<213> Homo sapiens
<400> 814
Phe Pro Glu Asn Leu Pro Gly Gln Leu
1 5
<210> 815
<211> 10
<212> PRT
<213> Homo sapiens
<400> 815
Gly Leu Leu Ala Phe Trp Asp Ser Gln Val
1 5 10
<210> 816
<211> 9
<212> PRT
<213> Homo sapiens
<400> 816
Ile Phe Cys Pro Phe Pro Glu Asn Leu
1 5
<210> 817
<211> 9
<212> PRT
<213> Homo sapiens
<400> 817
Leu Leu Ala Phe Trp Asp Ser Gln Val
1 5
<210> 818
<211> 9
<212> PRT
<213> Homo sapiens
<400> 818
Leu Leu Ala Pro Val Ile Phe Cys Pro
1 5
<210> 819
<211> 10
<212> PRT
<213> Homo sapiens
<400> 819
Leu Leu Ala Pro Val Ile Phe Cys Pro Phe
1 5 10
<210> 820
<211> 9
<212> PRT
<213> Homo sapiens
<400> 820
Leu Pro Cys Pro Gln Gln Asp Val Leu
1 5
<210> 821
<211> 9
<212> PRT
<213> Homo sapiens
<400> 821
Arg Phe Pro Ser Gly Leu Leu Ala Phe
1 5
<210> 822
<211> 10
<212> PRT
<213> Homo sapiens
<400> 822
Arg Phe Pro Ser Gly Leu Leu Ala Phe Trp
1 5 10
<210> 823
<211> 9
<212> PRT
<213> Homo sapiens
<400> 823
Ser Pro Leu Leu Ala Pro Val Ile Phe
1 5
<210> 824
<211> 9
<212> PRT
<213> Homo sapiens
<400> 824
Ser Pro Arg Gly Pro Cys Thr Ser Ser
1 5
<210> 825
<211> 10
<212> PRT
<213> Homo sapiens
<400> 825
Ser Pro Arg Gly Pro Cys Thr Ser Ser Ser
1 5 10
<210> 826
<211> 9
<212> PRT
<213> Homo sapiens
<400> 826
Ser Gln Val Cys Asp Leu His Val Leu
1 5
<210> 827
<211> 10
<212> PRT
<213> Homo sapiens
<400> 827
Val Ile Phe Cys Pro Phe Pro Glu Asn Leu
1 5 10
<210> 828
<211> 9
<212> PRT
<213> Homo sapiens
<400> 828
Ala Met Val Trp Pro Leu Leu Ser Ile
1 5
<210> 829
<211> 10
<212> PRT
<213> Homo sapiens
<400> 829
Ala Met Val Trp Pro Leu Leu Ser Ile Leu
1 5 10
<210> 830
<211> 9
<212> PRT
<213> Homo sapiens
<400> 830
Ala Gln Ile Ala Met Val Trp Pro Leu
1 5
<210> 831
<211> 10
<212> PRT
<213> Homo sapiens
<400> 831
Ala Gln Ile Ala Met Val Trp Pro Leu Leu
1 5 10
<210> 832
<211> 9
<212> PRT
<213> Homo sapiens
<400> 832
Cys Pro Met Ser Arg Leu Arg Leu Ala
1 5
<210> 833
<211> 10
<212> PRT
<213> Homo sapiens
<400> 833
Cys Pro Met Ser Arg Leu Arg Leu Ala Leu
1 5 10
<210> 834
<211> 10
<212> PRT
<213> Homo sapiens
<400> 834
Ile Ala Met Val Trp Pro Leu Leu Ser Ile
1 5 10
<210> 835
<211> 10
<212> PRT
<213> Homo sapiens
<400> 835
Ile Leu Ser Glu Trp Lys Glu Ile Cys Val
1 5 10
<210> 836
<211> 9
<212> PRT
<213> Homo sapiens
<400> 836
Ile Val Trp Trp Cys Pro Met Ser Arg
1 5
<210> 837
<211> 10
<212> PRT
<213> Homo sapiens
<400> 837
Ile Val Trp Trp Cys Pro Met Ser Arg Leu
1 5 10
<210> 838
<211> 10
<212> PRT
<213> Homo sapiens
<400> 838
Ile Trp Met Thr Glu Thr Leu Phe Asp Ile
1 5 10
<210> 839
<211> 9
<212> PRT
<213> Homo sapiens
<400> 839
Leu Leu Ser Ile Leu Ser Glu Trp Lys
1 5
<210> 840
<211> 9
<212> PRT
<213> Homo sapiens
<400> 840
Met Ser Ala Ala Gln Ile Ala Met Val
1 5
<210> 841
<211> 9
<212> PRT
<213> Homo sapiens
<400> 841
Met Ser Arg Leu Arg Leu Ala Leu Thr
1 5
<210> 842
<211> 10
<212> PRT
<213> Homo sapiens
<400> 842
Met Ser Arg Leu Arg Leu Ala Leu Thr Val
1 5 10
<210> 843
<211> 9
<212> PRT
<213> Homo sapiens
<400> 843
Met Val Trp Pro Leu Leu Ser Ile Leu
1 5
<210> 844
<211> 10
<212> PRT
<213> Homo sapiens
<400> 844
Arg Leu Ala Leu Thr Val Pro Pro Ser Thr
1 5 10
<210> 845
<211> 9
<212> PRT
<213> Homo sapiens
<400> 845
Thr Leu Phe Asp Ile Val Trp Trp Cys
1 5
<210> 846
<211> 10
<212> PRT
<213> Homo sapiens
<400> 846
Thr Leu Phe Asp Ile Val Trp Trp Cys Pro
1 5 10
<210> 847
<211> 10
<212> PRT
<213> Homo sapiens
<400> 847
Thr Met Ser Ala Ala Gln Ile Ala Met Val
1 5 10
<210> 848
<211> 10
<212> PRT
<213> Homo sapiens
<400> 848
Val Trp Ser Ile Trp Met Thr Glu Thr Leu
1 5 10
<210> 849
<211> 9
<212> PRT
<213> Homo sapiens
<400> 849
Trp Met Thr Glu Thr Leu Phe Asp Ile
1 5
<210> 850
<211> 10
<212> PRT
<213> Homo sapiens
<400> 850
Trp Met Thr Glu Thr Leu Phe Asp Ile Val
1 5 10
<210> 851
<211> 9
<212> PRT
<213> Homo sapiens
<400> 851
Ala Leu Arg Cys Val Phe Val Pro Val
1 5
<210> 852
<211> 10
<212> PRT
<213> Homo sapiens
<400> 852
Ala Leu Arg Cys Val Phe Val Pro Val Leu
1 5 10
<210> 853
<211> 9
<212> PRT
<213> Homo sapiens
<400> 853
Ala Leu Ser Glu His Cys Pro Thr Thr
1 5
<210> 854
<211> 10
<212> PRT
<213> Homo sapiens
<400> 854
Ala Gln Arg Lys Arg Ile Ser Ala Arg Lys
1 5 10
<210> 855
<211> 9
<212> PRT
<213> Homo sapiens
<400> 855
Gly Ala Gln Arg Lys Arg Ile Ser Ala
1 5
<210> 856
<211> 9
<212> PRT
<213> Homo sapiens
<400> 856
His Trp Met Glu Asn Ile Ser Pro Phe
1 5
<210> 857
<211> 9
<212> PRT
<213> Homo sapiens
<400> 857
Leu Pro Ser Gln Arg Arg Asn His Trp
1 5
<210> 858
<211> 10
<212> PRT
<213> Homo sapiens
<400> 858
Leu Pro Ser Gln Arg Arg Asn His Trp Met
1 5 10
<210> 859
<211> 10
<212> PRT
<213> Homo sapiens
<400> 859
Asn Ile Ser Pro Phe Arg Ser Val Gly Val
1 5 10
<210> 860
<211> 9
<212> PRT
<213> Homo sapiens
<400> 860
Arg Ile Ser Ala Arg Lys Gly Ser Leu
1 5
<210> 861
<211> 10
<212> PRT
<213> Homo sapiens
<400> 861
Ser Pro Phe Arg Ser Val Gly Val Ser Ala
1 5 10
<210> 862
<211> 10
<212> PRT
<213> Homo sapiens
<400> 862
Ser Pro Ser Ser His Trp Lys Thr Pro Val
1 5 10
<210> 863
<211> 10
<212> PRT
<213> Homo sapiens
<400> 863
Thr Ala Leu Arg Cys Val Phe Val Pro Val
1 5 10
<210> 864
<211> 9
<212> PRT
<213> Homo sapiens
<400> 864
Val Ile Tyr Trp Asp Gly Thr Ala Leu
1 5
<210> 865
<211> 10
<212> PRT
<213> Homo sapiens
<400> 865
Val Ile Tyr Trp Asp Gly Thr Ala Leu Arg
1 5 10
<210> 866
<211> 10
<212> PRT
<213> Homo sapiens
<400> 866
Val Leu Gly Glu Thr Gly Ala Gln Arg Lys
1 5 10
<210> 867
<211> 9
<212> PRT
<213> Homo sapiens
<400> 867
His Pro Arg Pro Arg His Gly His Leu
1 5
<210> 868
<211> 10
<212> PRT
<213> Homo sapiens
<400> 868
His Pro Arg Pro Arg His Gly His Leu Gln
1 5 10
<210> 869
<211> 9
<212> PRT
<213> Homo sapiens
<400> 869
Arg Pro Arg His Gly His Leu Gln Ala
1 5
<210> 870
<211> 10
<212> PRT
<213> Homo sapiens
<400> 870
Arg Pro Arg His Gly His Leu Gln Ala Val
1 5 10
<210> 871
<211> 10
<212> PRT
<213> Homo sapiens
<400> 871
Gly Ser Leu Lys Thr Gln Val Gln Met Lys
1 5 10
<210> 872
<211> 10
<212> PRT
<213> Homo sapiens
<400> 872
Pro Pro Gly Pro Cys His Leu Leu Ser Leu
1 5 10
<210> 873
<211> 10
<212> PRT
<213> Homo sapiens
<400> 873
Arg Thr Ile Leu Asn Asn Gly Ser Leu Lys
1 5 10
<210> 874
<211> 9
<212> PRT
<213> Homo sapiens
<400> 874
Ser Leu Lys Thr Gln Val Gln Met Lys
1 5
<210> 875
<211> 10
<212> PRT
<213> Homo sapiens
<400> 875
Ser Leu Lys Thr Gln Val Gln Met Lys Leu
1 5 10
<210> 876
<211> 9
<212> PRT
<213> Homo sapiens
<400> 876
Thr Ile Leu Asn Asn Gly Ser Leu Lys
1 5
<210> 877
<211> 10
<212> PRT
<213> Homo sapiens
<400> 877
Cys Pro Pro Cys Arg Pro Lys Gln Trp Met
1 5 10
<210> 878
<211> 10
<212> PRT
<213> Homo sapiens
<400> 878
Thr Thr Phe Cys Pro Pro Cys Arg Pro Lys
1 5 10
<210> 879
<211> 10
<212> PRT
<213> Homo sapiens
<400> 879
Cys Phe Ala Asn Trp Pro Arg Pro Ala Leu
1 5 10
<210> 880
<211> 9
<212> PRT
<213> Homo sapiens
<400> 880
Phe Ala Asn Trp Pro Arg Pro Ala Leu
1 5
<210> 881
<211> 9
<212> PRT
<213> Homo sapiens
<400> 881
Gly Leu Ile Pro His Pro Arg Pro Ala
1 5
<210> 882
<211> 9
<212> PRT
<213> Homo sapiens
<400> 882
His Pro Arg Pro Ala Pro Ala Ser Ala
1 5
<210> 883
<211> 10
<212> PRT
<213> Homo sapiens
<400> 883
His Pro Arg Pro Ala Pro Ala Ser Ala Pro
1 5 10
<210> 884
<211> 9
<212> PRT
<213> Homo sapiens
<400> 884
Ile Pro His Pro Arg Pro Ala Pro Ala
1 5
<210> 885
<211> 10
<212> PRT
<213> Homo sapiens
<400> 885
Ile Pro His Pro Arg Pro Ala Pro Ala Ser
1 5 10
<210> 886
<211> 9
<212> PRT
<213> Homo sapiens
<400> 886
Arg Pro Ala Leu Cys Ser Cys Gly Leu
1 5
<210> 887
<211> 10
<212> PRT
<213> Homo sapiens
<400> 887
Arg Pro Ala Leu Cys Ser Cys Gly Leu Ile
1 5 10
<210> 888
<211> 9
<212> PRT
<213> Homo sapiens
<400> 888
Thr Pro Leu Pro Ser Thr Arg Cys Phe
1 5
<210> 889
<211> 9
<212> PRT
<213> Homo sapiens
<400> 889
Trp Pro Arg Pro Ala Leu Cys Ser Cys
1 5
<210> 890
<211> 10
<212> PRT
<213> Homo sapiens
<400> 890
Trp Pro Arg Pro Ala Leu Cys Ser Cys Gly
1 5 10
<210> 891
<211> 10
<212> PRT
<213> Homo sapiens
<400> 891
Ala Leu Arg Arg Ser Gly Arg Pro Pro Lys
1 5 10
<210> 892
<211> 9
<212> PRT
<213> Homo sapiens
<400> 892
Gly Leu Val Pro Ser Leu Val Ser Lys
1 5
<210> 893
<211> 9
<212> PRT
<213> Homo sapiens
<400> 893
Lys Ile Ser Val Glu Thr Tyr Thr Val
1 5
<210> 894
<211> 10
<212> PRT
<213> Homo sapiens
<400> 894
Leu Leu Met Val Leu Met Ser Leu Asp Leu
1 5 10
<210> 895
<211> 10
<212> PRT
<213> Homo sapiens
<400> 895
Leu Met Ser Leu Asp Leu Asp Thr Gly Leu
1 5 10
<210> 896
<211> 9
<212> PRT
<213> Homo sapiens
<400> 896
Leu Met Val Leu Met Ser Leu Asp Leu
1 5
<210> 897
<211> 10
<212> PRT
<213> Homo sapiens
<400> 897
Leu Val Ser Lys Cys Leu Ile Leu Arg Val
1 5 10
<210> 898
<211> 9
<212> PRT
<213> Homo sapiens
<400> 898
Gln Leu Leu Met Val Leu Met Ser Leu
1 5
<210> 899
<211> 9
<212> PRT
<213> Homo sapiens
<400> 899
Arg Pro Gly Ala Ala Asp Thr Gly Ala
1 5
<210> 900
<211> 10
<212> PRT
<213> Homo sapiens
<400> 900
Arg Pro Gly Ala Ala Asp Thr Gly Ala His
1 5 10
<210> 901
<211> 9
<212> PRT
<213> Homo sapiens
<400> 901
Ser Leu Asp Leu Asp Thr Gly Leu Val
1 5
<210> 902
<211> 9
<212> PRT
<213> Homo sapiens
<400> 902
Ser Leu Val Ser Lys Cys Leu Ile Leu
1 5
<210> 903
<211> 10
<212> PRT
<213> Homo sapiens
<400> 903
Ser Gln Leu Leu Met Val Leu Met Ser Leu
1 5 10
<210> 904
<211> 9
<212> PRT
<213> Homo sapiens
<400> 904
Thr Val Ser Ser Gln Leu Leu Met Val
1 5
<210> 905
<211> 9
<212> PRT
<213> Homo sapiens
<400> 905
Thr Tyr Thr Val Ser Ser Gln Leu Leu
1 5
<210> 906
<211> 10
<212> PRT
<213> Homo sapiens
<400> 906
Thr Tyr Thr Val Ser Ser Gln Leu Leu Met
1 5 10
<210> 907
<211> 9
<212> PRT
<213> Homo sapiens
<400> 907
Val Leu Met Ser Leu Asp Leu Asp Thr
1 5
<210> 908
<211> 9
<212> PRT
<213> Homo sapiens
<400> 908
Val Pro Ser Leu Val Ser Lys Cys Leu
1 5
<210> 909
<211> 10
<212> PRT
<213> Homo sapiens
<400> 909
Val Ser Lys Cys Leu Ile Leu Arg Val Lys
1 5 10
<210> 910
<211> 9
<212> PRT
<213> Homo sapiens
<400> 910
Tyr Thr Val Ser Ser Gln Leu Leu Met
1 5
<210> 911
<211> 10
<212> PRT
<213> Homo sapiens
<400> 911
Tyr Thr Val Ser Ser Gln Leu Leu Met Val
1 5 10
<210> 912
<211> 9
<212> PRT
<213> Homo sapiens
<400> 912
Phe Cys Gln Tyr His Thr Ala Ser Val
1 5
<210> 913
<211> 9
<212> PRT
<213> Homo sapiens
<400> 913
Cys Pro Tyr Gly Ser Thr Ser Thr Ala
1 5
<210> 914
<211> 10
<212> PRT
<213> Homo sapiens
<400> 914
Cys Pro Tyr Gly Ser Thr Ser Thr Ala Ser
1 5 10
<210> 915
<211> 10
<212> PRT
<213> Homo sapiens
<400> 915
Leu Ala Arg Ala Ala Ala Ser Thr Ala Thr
1 5 10
<210> 916
<211> 9
<212> PRT
<213> Homo sapiens
<400> 916
Met Leu Thr Asp Ser Leu Phe Leu Pro
1 5
<210> 917
<211> 10
<212> PRT
<213> Homo sapiens
<400> 917
Pro Pro Arg Ser Ser Ser Ala Ile Ala Val
1 5 10
<210> 918
<211> 10
<212> PRT
<213> Homo sapiens
<400> 918
Arg Ala Ala Ala Ser Thr Ala Thr Glu Val
1 5 10
<210> 919
<211> 9
<212> PRT
<213> Homo sapiens
<400> 919
Ser Pro Pro Arg Ser Ser Ser Ala Ile
1 5
<210> 920
<211> 10
<212> PRT
<213> Homo sapiens
<400> 920
Ser Pro Pro Arg Ser Ser Ser Ala Ile Ala
1 5 10
<210> 921
<211> 9
<212> PRT
<213> Homo sapiens
<400> 921
Ser Pro Thr Gln Arg Cys Arg Leu Ala
1 5
<210> 922
<211> 9
<212> PRT
<213> Homo sapiens
<400> 922
Thr Gln Arg Cys Arg Leu Ala Arg Ala
1 5
<210> 923
<211> 10
<212> PRT
<213> Homo sapiens
<400> 923
Thr Gln Arg Cys Arg Leu Ala Arg Ala Ala
1 5 10
<210> 924
<211> 10
<212> PRT
<213> Homo sapiens
<400> 924
Lys Ile Trp Lys Thr Thr Gln Met Cys Arg
1 5 10
<210> 925
<211> 9
<212> PRT
<213> Homo sapiens
<400> 925
Trp Thr Ser Ser Gly Arg Ser Thr Lys
1 5
<210> 926
<211> 9
<212> PRT
<213> Homo sapiens
<400> 926
Ala Leu Gly Glu Leu Ala Arg Ala Leu
1 5
<210> 927
<211> 9
<212> PRT
<213> Homo sapiens
<400> 927
Ala Gln Leu Arg Arg Arg Ala Ala Ala
1 5
<210> 928
<211> 10
<212> PRT
<213> Homo sapiens
<400> 928
Ala Gln Leu Arg Arg Arg Ala Ala Ala Leu
1 5 10
<210> 929
<211> 10
<212> PRT
<213> Homo sapiens
<400> 929
Ala Arg Arg Ala Ala Arg Met Ala Gln Leu
1 5 10
<210> 930
<211> 9
<212> PRT
<213> Homo sapiens
<400> 930
His Pro Gln Leu Pro Arg Ser Pro Leu
1 5
<210> 931
<211> 10
<212> PRT
<213> Homo sapiens
<400> 931
His Pro Gln Leu Pro Arg Ser Pro Leu Ala
1 5 10
<210> 932
<211> 9
<212> PRT
<213> Homo sapiens
<400> 932
Leu Ala Arg Ala Leu Pro Gly His Leu
1 5
<210> 933
<211> 10
<212> PRT
<213> Homo sapiens
<400> 933
Leu Ala Arg Ala Leu Pro Gly His Leu Leu
1 5 10
<210> 934
<211> 9
<212> PRT
<213> Homo sapiens
<400> 934
Met Ala Gln Leu Arg Arg Arg Ala Ala
1 5
<210> 935
<211> 10
<212> PRT
<213> Homo sapiens
<400> 935
Met Ala Gln Leu Arg Arg Arg Ala Ala Ala
1 5 10
<210> 936
<211> 9
<212> PRT
<213> Homo sapiens
<400> 936
Gln Leu Arg Arg Arg Ala Ala Ala Leu
1 5
<210> 937
<211> 10
<212> PRT
<213> Homo sapiens
<400> 937
Arg Ala Ala Ala Leu Pro Asn Ala Ala Ala
1 5 10
<210> 938
<211> 10
<212> PRT
<213> Homo sapiens
<400> 938
Arg Met Ala Gln Leu Arg Arg Arg Ala Ala
1 5 10
<210> 939
<211> 10
<212> PRT
<213> Homo sapiens
<400> 939
Ser Gln Ser Ala Arg Arg Ala Ala Arg Met
1 5 10
<210> 940
<211> 10
<212> PRT
<213> Homo sapiens
<400> 940
Gly Met Thr Leu Gly Glu Lys Phe Arg Val
1 5 10
<210> 941
<211> 9
<212> PRT
<213> Homo sapiens
<400> 941
Arg Val Gly Asn Cys Lys His Leu Lys
1 5
<210> 942
<211> 9
<212> PRT
<213> Homo sapiens
<400> 942
Gly Pro Ser Glu Pro Gly Asn Asn Ile
1 5
<210> 943
<211> 10
<212> PRT
<213> Homo sapiens
<400> 943
Lys Ile Cys Asn Glu Ser Ala Ser Arg Lys
1 5 10
<210> 944
<211> 10
<212> PRT
<213> Homo sapiens
<400> 944
Gly Ile Gln Val Leu Asn Val Ser Leu Lys
1 5 10
<210> 945
<211> 9
<212> PRT
<213> Homo sapiens
<400> 945
Ile Gln Val Leu Asn Val Ser Leu Lys
1 5
<210> 946
<211> 9
<212> PRT
<213> Homo sapiens
<400> 946
Lys Ser Ser Ser Asn Val Ile Ser Tyr
1 5
<210> 947
<211> 9
<212> PRT
<213> Homo sapiens
<400> 947
Lys Tyr Gly Trp Ser Leu Leu Arg Val
1 5
<210> 948
<211> 9
<212> PRT
<213> Homo sapiens
<400> 948
Arg Ser Trp Lys Tyr Gly Trp Ser Leu
1 5
<210> 949
<211> 9
<212> PRT
<213> Homo sapiens
<400> 949
Ser Leu Lys Ser Ser Ser Asn Val Ile
1 5
<210> 950
<211> 9
<212> PRT
<213> Homo sapiens
<400> 950
Ser Trp Lys Tyr Gly Trp Ser Leu Leu
1 5
<210> 951
<211> 9
<212> PRT
<213> Homo sapiens
<400> 951
Thr Val Ala Asn Gly Arg Ser Trp Lys
1 5
<210> 952
<211> 9
<212> PRT
<213> Homo sapiens
<400> 952
Val Pro Gln Val Asn Gly Ile Gln Val
1 5
<210> 953
<211> 10
<212> PRT
<213> Homo sapiens
<400> 953
Val Pro Gln Val Asn Gly Ile Gln Val Leu
1 5 10
<210> 954
<211> 10
<212> PRT
<213> Homo sapiens
<400> 954
Val Thr Val Ala Asn Gly Arg Ser Trp Lys
1 5 10
<210> 955
<211> 9
<212> PRT
<213> Homo sapiens
<400> 955
Trp Ser Leu Leu Arg Val Pro Gln Val
1 5
<210> 956
<211> 10
<212> PRT
<213> Homo sapiens
<400> 956
His Pro Gly Asp Cys Leu Ile Phe Lys Leu
1 5 10
<210> 957
<211> 9
<212> PRT
<213> Homo sapiens
<400> 957
Lys Leu Arg Val Pro Gly Ser Ser Val
1 5
<210> 958
<211> 10
<212> PRT
<213> Homo sapiens
<400> 958
Lys Leu Arg Val Pro Gly Ser Ser Val Leu
1 5 10
<210> 959
<211> 9
<212> PRT
<213> Homo sapiens
<400> 959
Arg Val Pro Gly Ser Ser Val Leu Val
1 5
<210> 960
<211> 9
<212> PRT
<213> Homo sapiens
<400> 960
Ser Val Leu Val Thr Val Pro Gly Leu
1 5
<210> 961
<211> 10
<212> PRT
<213> Homo sapiens
<400> 961
Val Pro Gly Ser Ser Val Leu Val Thr Val
1 5 10
<210> 962
<211> 10
<212> PRT
<213> Homo sapiens
<400> 962
Ala Leu Leu Leu Arg Pro Arg Pro Pro Arg
1 5 10
<210> 963
<211> 10
<212> PRT
<213> Homo sapiens
<400> 963
Ala Leu Ser Ala Leu Leu Leu Arg Pro Arg
1 5 10
<210> 964
<211> 9
<212> PRT
<213> Homo sapiens
<400> 964
Leu Thr Ile Asn Lys Glu Glu Ala Leu
1 5
<210> 965
<211> 9
<212> PRT
<213> Homo sapiens
<400> 965
Ile Val His Ser Ala Thr Gly Phe Lys
1 5
<210> 966
<211> 9
<212> PRT
<213> Homo sapiens
<400> 966
Ala Thr Gly Phe Lys Gln Ser Ser Lys
1 5
<210> 967
<211> 9
<212> PRT
<213> Homo sapiens
<400> 967
Glu Leu Phe Pro Leu Ile Phe Pro Ala
1 5
<210> 968
<211> 9
<212> PRT
<213> Homo sapiens
<400> 968
Lys Gly Pro Glu Leu Phe Pro Leu Ile
1 5
<210> 969
<211> 10
<212> PRT
<213> Homo sapiens
<400> 969
Lys Gly Pro Glu Leu Phe Pro Leu Ile Phe
1 5 10
<210> 970
<211> 11
<212> PRT
<213> Homo sapiens
<400> 970
Lys Pro Thr Asp Ala Pro Pro Lys Ala Gly Val
1 5 10
<210> 971
<211> 10
<212> PRT
<213> Homo sapiens
<400> 971
Ala Leu Glu Glu Lys Lys Gly Asn Tyr Val
1 5 10
<210> 972
<211> 10
<212> PRT
<213> Homo sapiens
<400> 972
Ile Gln Leu Gln Asp Lys Phe Glu His Leu
1 5 10
<210> 973
<211> 9
<212> PRT
<213> Homo sapiens
<400> 973
Gln Leu Gln Asp Lys Phe Glu His Leu
1 5
<210> 974
<211> 10
<212> PRT
<213> Homo sapiens
<400> 974
Gln Leu Gln Asp Lys Phe Glu His Leu Lys
1 5 10
<210> 975
<211> 10
<212> PRT
<213> Homo sapiens
<400> 975
Tyr Leu Val Ile Gln Leu Gln Asp Lys Phe
1 5 10
<210> 976
<211> 10
<212> PRT
<213> Homo sapiens
<400> 976
Gln Val Tyr Arg Arg Lys His Gln Glu Leu
1 5 10
<210> 977
<211> 9
<212> PRT
<213> Homo sapiens
<400> 977
Ser Thr Arg Glu Lys Asn Ser Gln Val
1 5
<210> 978
<211> 9
<212> PRT
<213> Homo sapiens
<400> 978
Val Tyr Arg Arg Lys His Gln Glu Leu
1 5
<210> 979
<211> 9
<212> PRT
<213> Homo sapiens
<400> 979
Val Leu Thr Val Thr Ser Thr Asp Val
1 5
<210> 980
<211> 10
<212> PRT
<213> Homo sapiens
<400> 980
Val Leu Thr Val Thr Ser Thr Asp Val Lys
1 5 10
<210> 981
<211> 9
<212> PRT
<213> Homo sapiens
<400> 981
Ile Met Ser Leu Trp Gly Leu Val Ser
1 5
<210> 982
<211> 10
<212> PRT
<213> Homo sapiens
<400> 982
Ile Met Ser Leu Trp Gly Leu Val Ser Lys
1 5 10
<210> 983
<211> 9
<212> PRT
<213> Homo sapiens
<400> 983
Lys Leu Lys Gln Glu Ala Thr Ser Lys
1 5
<210> 984
<211> 9
<212> PRT
<213> Homo sapiens
<400> 984
Gln Ile Met Ser Leu Trp Gly Leu Val
1 5
<210> 985
<211> 9
<212> PRT
<213> Homo sapiens
<400> 985
Ser Gln Ile Met Ser Leu Trp Gly Leu
1 5
<210> 986
<211> 10
<212> PRT
<213> Homo sapiens
<400> 986
Ser Gln Ile Met Ser Leu Trp Gly Leu Val
1 5 10
<210> 987
<211> 9
<212> PRT
<213> Homo sapiens
<400> 987
Thr Ser Lys Ser Gln Ile Met Ser Leu
1 5
<210> 988
<211> 10
<212> PRT
<213> Homo sapiens
<400> 988
Leu Leu Gln Glu Phe Asp Val Gln Glu Ala
1 5 10
<210> 989
<211> 10
<212> PRT
<213> Homo sapiens
<400> 989
Leu Gln Glu Phe Asp Val Gln Glu Ala Leu
1 5 10
<210> 990
<211> 9
<212> PRT
<213> Homo sapiens
<400> 990
Gly Pro Arg Glu Pro Arg Asn Arg Thr
1 5
<210> 991
<211> 10
<212> PRT
<213> Homo sapiens
<400> 991
Arg Asn Arg Thr Glu Lys His Ser Thr Met
1 5 10
<210> 992
<211> 9
<212> PRT
<213> Homo sapiens
<400> 992
Ala Leu Asn Ser Glu Ala Leu Ser Val
1 5
<210> 993
<211> 10
<212> PRT
<213> Homo sapiens
<400> 993
Ala Leu Asn Ser Glu Ala Leu Ser Val Val
1 5 10
<210> 994
<211> 10
<212> PRT
<213> Homo sapiens
<400> 994
Met Ala Leu Asn Ser Glu Ala Leu Ser Val
1 5 10
<210> 995
<211> 67
<212> PRT
<213> Homo sapiens
<400> 995
Met Ser Asp Val Ala Ile Val Lys Glu Gly Trp Leu His Lys Arg Gly
1 5 10 15
Lys Tyr Ile Lys Thr Trp Arg Pro Arg Tyr Phe Leu Leu Lys Asn Asp
20 25 30
Gly Thr Phe Ile Gly Tyr Lys Glu Arg Pro Gln Asp Val Asp Gln Arg
35 40 45
Glu Ala Pro Leu Asn Asn Phe Ser Val Ala Gln Cys Gln Leu Met Lys
50 55 60
Thr Glu Arg
65
<210> 996
<211> 101
<212> PRT
<213> Homo sapiens
<400> 996
Thr Met Leu Val Leu Glu Gly Ser Gly Asn Leu Val Leu Tyr Thr Gly
1 5 10 15
Val Val Arg Val Gly Lys Val Phe Ile Pro Gly Leu Pro Ala Pro Ser
20 25 30
Leu Thr Met Ser Asn Thr Met Pro Arg Pro Ser Thr Pro Leu Asp Gly
35 40 45
Val Ser Ala Pro Lys Pro Leu Ser Lys Leu Leu Gly Ser Leu Asp Glu
50 55 60
Val Val Leu Leu Ser Pro Val Pro Glu Leu Arg Asp Ser Ser Lys Leu
65 70 75 80
His Asp Ser Leu Tyr Asn Glu Asp Cys Thr Phe Gln Gln Leu Gly Thr
85 90 95
Tyr Ile His Ser Ile
100
<210> 997
<211> 90
<212> PRT
<213> Homo sapiens
<400> 997
Met Arg Glu Pro Ile Tyr Met His Ser Thr Met Val Phe Leu Pro Trp
1 5 10 15
Glu Leu His Thr Lys Lys Gly Pro Ser Pro Pro Glu Gln Phe Met Ala
20 25 30
Val Lys Leu Ser Asp Ser Arg Thr Ala Leu Lys Ser Gly Tyr Gly Lys
35 40 45
Tyr Leu Gly Ile Asn Ser Asp Glu Leu Val Gly His Ser Asp Ala Ile
50 55 60
Gly Pro Arg Glu Gln Trp Glu Pro Val Phe Gln Asn Gly Lys Met Ala
65 70 75 80
Leu Leu Ala Ser Asn Ser Cys Phe Ile Arg
85 90
<210> 998
<211> 101
<212> PRT
<213> Homo sapiens
<400> 998
Ala Met Pro Asn Gln Ala Gln Met Arg Ile Leu Lys Glu Thr Glu Leu
1 5 10 15
Arg Lys Val Lys Val Leu Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys
20 25 30
Gly Ile Trp Ile Pro Asp Gly Glu Asn Val Lys Ile Pro Val Ala Ile
35 40 45
Lys Val Ser Arg Glu Asn Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu
50 55 60
Asp Glu Ala Tyr Val Met Ala Gly Val Gly Ser Pro Tyr Val Ser Arg
65 70 75 80
Leu Leu Gly Ile Cys Leu Thr Ser Thr Val Gln Leu Val Thr Gln Leu
85 90 95
Met Pro Tyr Gly Cys
100
<210> 999
<211> 101
<212> PRT
<213> Homo sapiens
<400> 999
Ala Val Cys Lys Ser Lys Lys Ser Trp Gln Ala Arg His Thr Gly Ile
1 5 10 15
Lys Ile Val Gln Gln Ile Ala Ile Leu Met Gly Cys Ala Ile Leu Pro
20 25 30
His Leu Arg Ser Leu Val Glu Ile Ile Glu His Gly Leu Val Asp Glu
35 40 45
Gln Gln Glu Val Arg Thr Ile Ser Ala Leu Ala Ile Ala Ala Leu Ala
50 55 60
Glu Ala Ala Thr Pro Tyr Gly Ile Glu Ser Phe Asp Ser Val Leu Lys
65 70 75 80
Pro Leu Trp Lys Gly Ile Arg Gln His Arg Gly Lys Gly Leu Ala Ala
85 90 95
Phe Leu Lys Ala Ile
100
<210> 1000
<211> 101
<212> PRT
<213> Homo sapiens
<400> 1000
Tyr Leu Ser Leu Tyr Leu Leu Leu Val Ser Cys Pro Lys Ser Glu Val
1 5 10 15
Arg Ala Lys Phe Lys Phe Ser Ile Leu Asn Ala Lys Gly Glu Glu Thr
20 25 30
Lys Ala Met Glu Ser Gln Arg Ala Tyr Arg Phe Val Gln Gly Lys Asp
35 40 45
Trp Gly Leu Lys Lys Phe Ile Arg Arg Asp Phe Leu Leu Asp Glu Ala
50 55 60
Asn Gly Leu Leu Pro Asp Asp Lys Leu Thr Leu Phe Cys Glu Val Ser
65 70 75 80
Val Val Gln Asp Ser Val Asn Ile Ser Gly Gln Asn Thr Met Asn Met
85 90 95
Val Lys Val Pro Glu
100
<210> 1001
<211> 101
<212> PRT
<213> Homo sapiens
<400> 1001
Tyr Leu Ser Leu Tyr Leu Leu Leu Val Ser Cys Pro Lys Ser Glu Val
1 5 10 15
Arg Ala Lys Phe Lys Phe Ser Ile Leu Asn Ala Lys Gly Glu Glu Thr
20 25 30
Lys Ala Met Glu Ser Gln Arg Ala Tyr Arg Phe Val Gln Gly Lys Asp
35 40 45
Trp Gly Val Lys Lys Phe Ile Arg Arg Asp Phe Leu Leu Asp Glu Ala
50 55 60
Asn Gly Leu Leu Pro Asp Asp Lys Leu Thr Leu Phe Cys Glu Val Ser
65 70 75 80
Val Val Gln Asp Ser Val Asn Ile Ser Gly Gln Asn Thr Met Asn Met
85 90 95
Val Lys Val Pro Glu
100
<210> 1002
<211> 101
<212> PRT
<213> Homo sapiens
<400> 1002
Pro Glu Val Gly Ser Asp Cys Thr Thr Ile His Tyr Asn Tyr Met Cys
1 5 10 15
Asn Ser Ser Cys Met Gly Gly Met Asn Arg Arg Pro Ile Leu Thr Ile
20 25 30
Ile Thr Leu Glu Asp Ser Ser Gly Asn Leu Leu Gly Arg Asn Ser Phe
35 40 45
Glu Val His Val Cys Ala Cys Pro Gly Arg Asp Arg Arg Thr Glu Glu
50 55 60
Glu Asn Leu Arg Lys Lys Gly Glu Pro His His Glu Leu Pro Pro Gly
65 70 75 80
Ser Thr Lys Arg Ala Leu Pro Asn Asn Thr Ser Ser Ser Pro Gln Pro
85 90 95
Lys Lys Lys Pro Leu
100
<210> 1003
<211> 101
<212> PRT
<213> Homo sapiens
<400> 1003
Thr Glu Val Val Arg Arg Cys Pro His His Glu Arg Cys Ser Asp Ser
1 5 10 15
Asp Gly Leu Ala Pro Pro Gln His Leu Ile Arg Val Glu Gly Asn Leu
20 25 30
Arg Val Glu Tyr Leu Asp Asp Arg Asn Thr Phe Arg His Ser Val Val
35 40 45
Val Pro Cys Glu Pro Pro Glu Val Gly Ser Asp Cys Thr Thr Ile His
50 55 60
Tyr Asn Tyr Met Cys Asn Ser Ser Cys Met Gly Gly Met Asn Arg Arg
65 70 75 80
Pro Ile Leu Thr Ile Ile Thr Leu Glu Asp Ser Ser Gly Asn Leu Leu
85 90 95
Gly Arg Asn Ser Phe
100
<210> 1004
<211> 54
<212> PRT
<213> Homo sapiens
<400> 1004
Tyr Asn Phe Asp Lys Asn Tyr Lys Asp Trp Gln Ser Ala Val Glu Cys
1 5 10 15
Ile Ala Val Leu Asp Val Leu Leu Cys Leu Ala Asn Tyr Ser Arg Gly
20 25 30
Gly Asp Gly Pro Met Cys Arg Pro Val Ile Leu Leu Pro Glu Asp Thr
35 40 45
Pro Pro Leu Leu Arg Ala
50
<210> 1005
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1005
Lys Tyr Ile Lys Thr Trp Arg Pro Arg Tyr
1 5 10
<210> 1006
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1006
Trp Leu His Lys Arg Gly Lys Tyr Ile
1 5
<210> 1007
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1007
Trp Leu His Lys Arg Gly Lys Tyr Ile Lys
1 5 10
<210> 1008
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1008
Ala Pro Lys Pro Leu Ser Lys Leu Leu
1 5
<210> 1009
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1009
Gly Val Ser Ala Pro Lys Pro Leu Ser Lys
1 5 10
<210> 1010
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1010
Val Ser Ala Pro Lys Pro Leu Ser Lys
1 5
<210> 1011
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1011
Cys Pro His Arg Pro Ile Leu Gln Ala
1 5
<210> 1012
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1012
Lys Leu Ser Asp Ser Arg Thr Ala Leu
1 5
<210> 1013
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1013
Lys Leu Ser Asp Ser Arg Thr Ala Leu Lys
1 5 10
<210> 1014
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1014
Leu Ser Asp Ser Arg Thr Ala Leu Lys
1 5
<210> 1015
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1015
Arg Thr Ala Leu Lys Ser Gly Tyr Gly Lys
1 5 10
<210> 1016
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1016
Thr Ala Leu Lys Ser Gly Tyr Gly Lys
1 5
<210> 1017
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1017
Ala Leu Ser Ala Ser Asn Ser Cys Phe
1 5
<210> 1018
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1018
Ala Leu Ser Ala Ser Asn Ser Cys Phe Ile
1 5 10
<210> 1019
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1019
Phe Gln Asn Gly Lys Met Ala Leu Ser Ala
1 5 10
<210> 1020
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1020
Lys Val Ser Arg Glu Asn Thr Ser Pro Lys
1 5 10
<210> 1021
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1021
Lys Pro Ile Ile Ile Gly Gly His Ala Tyr
1 5 10
<210> 1022
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1022
Ala Ile Ser Thr Arg Asp Pro Leu Ser Lys
1 5 10
<210> 1023
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1023
Gln Thr Gly Val Met Ile Cys Ala Tyr
1 5
<210> 1024
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1024
Phe Ser Gly Glu Tyr Ile Pro Thr Val
1 5
<210> 1025
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1025
Thr Thr Asn Ala Phe Ser Gly Glu Tyr
1 5
<210> 1026
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1026
Tyr Thr Thr Asn Ala Phe Ser Gly Glu Tyr
1 5 10
<210> 1027
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1027
Gly Leu Val Asp Glu Gln Gln Glu Val
1 5
<210> 1028
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1028
Phe Val Gln Gly Lys Asp Trp Gly Leu
1 5
<210> 1029
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1029
Phe Val Gln Gly Lys Asp Trp Gly Val
1 5
<210> 1030
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1030
Cys Met Gly Ser Met Asn Arg Arg Pro Ile
1 5 10
<210> 1031
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1031
Gly Ser Met Asn Arg Arg Pro Ile Leu
1 5
<210> 1032
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1032
Met Gly Ser Met Asn Arg Arg Pro Ile
1 5
<210> 1033
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1033
Met Gly Ser Met Asn Arg Arg Pro Ile Leu
1 5 10
<210> 1034
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1034
Ser Met Asn Arg Arg Pro Ile Leu Thr Ile
1 5 10
<210> 1035
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1035
Cys Met Gly Gly Met Asn Gln Arg Pro Ile
1 5 10
<210> 1036
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1036
Gly Met Asn Gln Arg Pro Ile Leu Thr Ile
1 5 10
<210> 1037
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1037
Asn Gln Arg Pro Ile Leu Thr Ile Ile
1 5
<210> 1038
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1038
Cys Met Gly Gly Met Asn Trp Arg Pro Ile
1 5 10
<210> 1039
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1039
Gly Met Asn Trp Arg Pro Ile Leu Thr Ile
1 5 10
<210> 1040
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1040
Met Asn Trp Arg Pro Ile Leu Thr Ile
1 5
<210> 1041
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1041
Met Asn Trp Arg Pro Ile Leu Thr Ile Ile
1 5 10
<210> 1042
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1042
Asn Ser Phe Glu Val Cys Val Cys Ala
1 5
<210> 1043
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1043
Asn Ser Phe Glu Val His Val Cys Ala
1 5
<210> 1044
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1044
Val Val Pro Cys Glu Pro Pro Glu Val
1 5
<210> 1045
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1045
Val Val Val Pro Cys Glu Pro Pro Glu Val
1 5 10
<210> 1046
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1046
Ile Leu Leu Pro Glu Asp Thr Pro Pro Leu
1 5 10
<210> 1047
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1047
Leu Leu Pro Glu Asp Thr Pro Pro Leu
1 5
<210> 1048
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1048
Ala Pro Phe Arg Val Asn His Ala Val
1 5
<210> 1049
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1049
Cys Leu Ala Asp Val Leu Leu Ser Val
1 5
<210> 1050
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1050
Phe Leu Gln Glu Arg Asn Leu Pro Pro Lys
1 5 10
<210> 1051
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1051
His Leu Ile Val Leu Arg Val Val Arg Leu
1 5 10
<210> 1052
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1052
His Pro Lys Val His Leu Asn Thr Met
1 5
<210> 1053
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1053
His Pro Lys Val His Leu Asn Thr Met Phe
1 5 10
<210> 1054
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1054
Lys Val His Leu Asn Thr Met Phe Arg
1 5
<210> 1055
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1055
Lys Val His Leu Asn Thr Met Phe Arg Arg
1 5 10
<210> 1056
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1056
Leu Pro Ala Pro Phe Arg Val Asn His Ala
1 5 10
<210> 1057
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1057
Met Phe Arg Arg Pro His Ser Cys Leu
1 5
<210> 1058
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1058
Met Phe Arg Arg Pro His Ser Cys Leu Ala
1 5 10
<210> 1059
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1059
Asn Thr Met Phe Arg Arg Pro His Ser Cys
1 5 10
<210> 1060
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1060
Arg Pro His Ser Cys Leu Ala Asp Val
1 5
<210> 1061
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1061
Arg Pro His Ser Cys Leu Ala Asp Val Leu
1 5 10
<210> 1062
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1062
Arg Val Val Arg Leu Pro Ala Pro Phe Arg
1 5 10
<210> 1063
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1063
Ser Val His Leu Ile Val Leu Arg Val
1 5
<210> 1064
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1064
Thr Met Phe Arg Arg Pro His Ser Cys
1 5
<210> 1065
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1065
Thr Met Phe Arg Arg Pro His Ser Cys Leu
1 5 10
<210> 1066
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1066
Val Leu Leu Ser Val His Leu Ile Val
1 5
<210> 1067
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1067
Val Leu Leu Ser Val His Leu Ile Val Leu
1 5 10
<210> 1068
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1068
Val Leu Arg Val Val Arg Leu Pro Ala
1 5
<210> 1069
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1069
Val Val Arg Leu Pro Ala Pro Phe Arg
1 5
<210> 1070
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1070
Leu Thr Ser Ser Ser Arg Glu Trp Lys
1 5
<210> 1071
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1071
Leu Leu Thr Ser Ser Ser Arg Glu Trp Lys
1 5 10
<210> 1072
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1072
Tyr Pro Ala Tyr Ser Lys Asp Ser Gly Leu
1 5 10
<210> 1073
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1073
Glu Trp Lys Val Lys Phe Pro Glu Leu
1 5
<210> 1074
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1074
Lys Phe Pro Glu Leu Leu Cys Val Trp
1 5
<210> 1075
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1075
Phe Leu Lys Ala Glu Ser Lys Ile Met
1 5
<210> 1076
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1076
Leu Gln His Gly His Arg His Gly Leu
1 5
<210> 1077
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1077
Glu Pro His Leu Ala Leu Gln Pro Leu
1 5
<210> 1078
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1078
Phe Ala Thr Leu Gln Arg Ser Ser Leu
1 5
<210> 1079
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1079
Met Phe Ala Thr Leu Gln Arg Ser Ser Leu
1 5 10
<210> 1080
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1080
Met Phe Leu Lys Ala Glu Ser Lys Ile
1 5
<210> 1081
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1081
Met Leu Thr Gly Pro Pro Ala Arg Val
1 5
<210> 1082
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1082
Gly Pro Pro Ala Arg Val Pro Ala Val
1 5
<210> 1083
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1083
Ala Leu Gln Pro Leu Gln Pro His Ala
1 5
<210> 1084
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1084
Val Leu Pro Glu Pro His Leu Ala Leu
1 5
<210> 1085
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1085
Tyr Met Phe Leu Lys Ala Glu Ser Lys
1 5
<210> 1086
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1086
Val Pro Ala Val Pro Phe Asp Leu His Phe
1 5 10
<210> 1087
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1087
Ala Ile Gln Pro Val Leu Trp Thr Thr
1 5
<210> 1088
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1088
Thr Leu Gln Arg Ser Ser Leu Trp Cys Leu
1 5 10
<210> 1089
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1089
Lys Ile Met Phe Ala Thr Leu Gln Arg
1 5
<210> 1090
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1090
Gln Pro Val Leu Trp Thr Thr Pro Pro Leu
1 5 10
<210> 1091
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1091
Glu Ser Lys Ile Met Phe Ala Thr Leu
1 5
<210> 1092
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1092
Ile Met Lys Pro Lys Arg Asp Gly Tyr Met
1 5 10
<210> 1093
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1093
Lys Pro Lys Arg Asp Gly Tyr Met Phe
1 5
<210> 1094
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1094
Phe Leu Lys Ala Glu Ser Lys Ile Met Phe
1 5 10
<210> 1095
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1095
Lys Pro Lys Arg Asp Gly Tyr Met Phe Leu
1 5 10
<210> 1096
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1096
Leu His Phe Cys Arg Ser Ser Ile Met
1 5
<210> 1097
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1097
Tyr Met Phe Leu Lys Ala Glu Ser Lys Ile
1 5 10
<210> 1098
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1098
Val Met Ala Tyr Val Met Ala Gly Val
1 5
<210> 1099
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1099
Tyr Val Met Ala Tyr Val Met Ala Gly Val
1 5 10
<210> 1100
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1100
Tyr Val Met Ala Tyr Val Met Ala Gly
1 5
<210> 1101
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1101
Ser Leu Met Pro Ala His Trp Ser Ile
1 5
<210> 1102
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1102
Leu Ser Leu Met Pro Ala His Trp Ser Ile
1 5 10
<210> 1103
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1103
Phe Gln Met Gln Pro Leu Ser Leu Met
1 5
<210> 1104
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1104
Leu Leu Gln Val Thr Gln Thr Ser Phe Ala
1 5 10
<210> 1105
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1105
Arg Leu Trp His Leu Leu Leu Gln Val
1 5
<210> 1106
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1106
Leu Gln Val Thr Gln Thr Ser Phe Ala Leu
1 5 10
<210> 1107
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1107
Arg Leu Trp His Leu Leu Leu Gln Val Thr
1 5 10
<210> 1108
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1108
Ala Leu Ala Pro Thr Leu Thr His Met
1 5
<210> 1109
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1109
Ala Leu Ala Pro Thr Leu Thr His Met Leu
1 5 10
<210> 1110
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1110
Ser Leu Gly Asn His Leu Cys Pro Leu
1 5
<210> 1111
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1111
Thr Gln Leu Ala Arg Phe Phe Pro Ile
1 5
<210> 1112
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1112
Lys Ser Arg Gln Asn His Leu Gln Leu Lys
1 5 10
<210> 1113
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1113
Lys Ser Arg Gln Asn His Leu Gln Leu
1 5
<210> 1114
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1114
Lys Leu Arg Ser Pro Leu Trp Ile Phe
1 5
<210> 1115
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1115
Lys Ile Ser Asn Trp Ser Leu Lys Lys
1 5
<210> 1116
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1116
Ser Leu Lys Lys Val Pro Ala Leu Lys
1 5
<210> 1117
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1117
Trp Ser Leu Lys Lys Val Pro Ala Leu Lys
1 5 10
<210> 1118
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1118
Lys Ile Ser Asn Trp Ser Leu Lys Lys Val
1 5 10
<210> 1119
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1119
Ser Gln Lys Ser Arg Gln Asn His Leu
1 5
<210> 1120
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1120
Ala Leu Lys Lys Leu Arg Ser Pro Leu
1 5
<210> 1121
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1121
Trp Ser Leu Lys Lys Val Pro Ala Leu
1 5
<210> 1122
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1122
His Leu Gln Leu Lys Ser Cys Arg Arg Lys
1 5 10
<210> 1123
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1123
Ser Leu Lys Lys Val Pro Ala Leu Lys Lys
1 5 10
<210> 1124
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1124
Leu Pro Arg Lys Pro Thr Arg Ala Ala Thr
1 5 10
<210> 1125
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1125
Leu Pro Arg Lys Pro Thr Arg Ala Ala
1 5
<210> 1126
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1126
Lys Pro Thr Arg Ala Ala Thr Val Ser Val
1 5 10
<210> 1127
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1127
Arg Val Gln Asn Gln Gly His Leu Leu
1 5
<210> 1128
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1128
Met Leu Thr Asp Ser Leu Phe Leu Pro Ile
1 5 10
<210> 1129
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1129
Ser Met Leu Thr Asp Ser Leu Phe Leu
1 5
<210> 1130
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1130
Ala Val Glu Trp
1
<210> 1131
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1131
Val His Pro Ala
1
<210> 1132
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1132
Met Pro Ala Val
1
<210> 1133
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1133
Ser Ile Arg His
1
<210> 1134
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1134
Ser Gln Ile Ser
1
<210> 1135
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1135
Cys Ser Asn His
1
<210> 1136
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1136
Tyr His Glu Ala
1
<210> 1137
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1137
Tyr Val Met Ala
1
<210> 1138
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1138
Leu Ser Pro His
1
<210> 1139
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1139
Cys Arg Leu Ser
1
<210> 1140
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1140
Arg His Thr Pro
1
<210> 1141
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1141
Ala Ala Val Gly
1
<210> 1142
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1142
Ala Trp Ala Ala
1
<210> 1143
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1143
Cys Ser Glu Ser
1
<210> 1144
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1144
Gln Pro Ser Leu
1
<210> 1145
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1145
Ser His Ser Thr
1
<210> 1146
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1146
Gln Cys Ile Leu
1
<210> 1147
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1147
Asp Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1148
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1148
Val Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1149
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1149
Cys Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1150
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1150
Asp Thr Ala Gly His Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1151
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1151
Gly His Glu Glu Tyr Ser Ala Met
1 5
<210> 1152
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1152
Asp Thr Ala Gly His Glu Glu Tyr
1 5
<210> 1153
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1153
Ile Leu Asp Thr Ala Gly His Glu Glu
1 5
<210> 1154
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1154
Ala Gly His Glu Glu Tyr Ser Ala Met
1 5
<210> 1155
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1155
His Glu Glu Tyr Ser Ala Met Arg Asp
1 5
<210> 1156
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1156
Thr Ala Gly His Glu Glu Tyr Ser Ala
1 5
<210> 1157
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1157
Asp Thr Ala Gly His Glu Glu Tyr Ser
1 5
<210> 1158
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1158
Asp Thr Ala Gly His Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1159
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1159
Leu Asp Thr Ala Gly His Glu Glu Tyr
1 5
<210> 1160
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1160
Leu Leu Asp Ile Leu Asp Thr Ala Gly His
1 5 10
<210> 1161
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1161
Thr Ala Gly His Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1162
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1162
Asp Ile Leu Asp Thr Ala Gly His Glu
1 5
<210> 1163
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1163
Asp Ile Leu Asp Thr Ala Gly His Glu Glu Tyr
1 5 10
<210> 1164
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1164
Asp Ile Leu Asp Thr Ala Gly His
1 5
<210> 1165
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1165
Ile Leu Asp Thr Ala Gly His Glu
1 5
<210> 1166
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1166
Asp Thr Ala Gly His Glu Glu Tyr Ser Ala
1 5 10
<210> 1167
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1167
Asp Thr Ala Gly Arg Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1168
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1168
Asp Ile Leu Asp Thr Ala Gly Arg
1 5
<210> 1169
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1169
Asp Thr Ala Gly Arg Glu Glu Tyr
1 5
<210> 1170
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1170
Cys Leu Leu Asp Ile Leu Asp Thr Ala Gly Arg
1 5 10
<210> 1171
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1171
Arg Glu Glu Tyr Ser Ala Met Arg Asp
1 5
<210> 1172
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1172
Gly Arg Glu Glu Tyr Ser Ala Met Arg
1 5
<210> 1173
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1173
Ile Leu Asp Thr Ala Gly Arg Glu Glu
1 5
<210> 1174
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1174
Ala Gly Arg Glu Glu Tyr Ser Ala Met
1 5
<210> 1175
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1175
Asp Ile Leu Asp Thr Ala Gly Arg Glu
1 5
<210> 1176
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1176
Gly Arg Glu Glu Tyr Ser Ala Met
1 5
<210> 1177
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1177
Thr Ala Gly Arg Glu Glu Tyr Ser Ala
1 5
<210> 1178
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1178
Asp Thr Ala Gly Arg Glu Glu Tyr Ser
1 5
<210> 1179
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1179
Ile Leu Asp Thr Ala Gly Arg Glu
1 5
<210> 1180
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1180
Asp Thr Ala Gly Arg Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1181
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1181
Leu Leu Asp Ile Leu Asp Thr Ala Gly Arg
1 5 10
<210> 1182
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1182
Thr Ala Gly Arg Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1183
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1183
Asp Ile Leu Asp Thr Ala Gly Arg Glu Glu Tyr
1 5 10
<210> 1184
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1184
Ala Gly Arg Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1185
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1185
Leu Asp Ile Leu Asp Thr Ala Gly Arg
1 5
<210> 1186
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1186
Leu Asp Thr Ala Gly Arg Glu Glu Tyr
1 5
<210> 1187
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1187
Arg Glu Glu Tyr Ser Ala Met Arg Asp Gln Tyr
1 5 10
<210> 1188
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1188
Asp Thr Ala Gly Lys Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1189
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1189
Cys Leu Leu Asp Ile Leu Asp Thr Ala Gly Lys
1 5 10
<210> 1190
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1190
Asp Thr Ala Gly Lys Glu Glu Tyr
1 5
<210> 1191
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1191
Ala Gly Lys Glu Glu Tyr Ser Ala Met
1 5
<210> 1192
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1192
Asp Ile Leu Asp Thr Ala Gly Lys Glu
1 5
<210> 1193
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1193
Lys Glu Glu Tyr Ser Ala Met Arg Asp
1 5
<210> 1194
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1194
Gly Lys Glu Glu Tyr Ser Ala Met
1 5
<210> 1195
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1195
Ile Leu Asp Thr Ala Gly Lys Glu Glu
1 5
<210> 1196
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1196
Asp Thr Ala Gly Lys Glu Glu Tyr Ser
1 5
<210> 1197
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1197
Leu Asp Thr Ala Gly Lys Glu Glu Tyr
1 5
<210> 1198
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1198
Thr Ala Gly Lys Glu Glu Tyr Ser Ala
1 5
<210> 1199
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1199
Asp Ile Leu Asp Thr Ala Gly Lys
1 5
<210> 1200
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1200
Asp Thr Ala Gly Lys Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1201
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1201
Leu Leu Asp Ile Leu Asp Thr Ala Gly Lys
1 5 10
<210> 1202
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1202
Ile Leu Asp Thr Ala Gly Lys Glu
1 5
<210> 1203
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1203
Ala Gly Lys Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1204
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1204
Thr Ala Gly Lys Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1205
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1205
Gly Leu Glu Glu Tyr Ser Ala Met Arg Asp Gln Tyr
1 5 10
<210> 1206
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1206
Asp Thr Ala Gly Leu Glu Glu Tyr
1 5
<210> 1207
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1207
Asp Thr Ala Gly Leu Glu Glu Tyr Ser Ala Met
1 5 10
<210> 1208
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1208
Ile Leu Asp Thr Ala Gly Leu Glu Glu
1 5
<210> 1209
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1209
Cys Leu Leu Asp Ile Leu Asp Thr Ala Gly Leu
1 5 10
<210> 1210
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1210
Asp Ile Leu Asp Thr Ala Gly Leu
1 5
<210> 1211
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1211
Asp Ile Leu Asp Thr Ala Gly Leu Glu Glu Tyr
1 5 10
<210> 1212
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1212
Asp Thr Ala Gly Leu Glu Glu Tyr Ser
1 5
<210> 1213
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1213
Gly Leu Glu Glu Tyr Ser Ala Met Arg
1 5
<210> 1214
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1214
Ile Leu Asp Thr Ala Gly Leu Glu
1 5
<210> 1215
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1215
Leu Asp Thr Ala Gly Leu Glu Glu Tyr
1 5
<210> 1216
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1216
Asp Thr Ala Gly Leu Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1217
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1217
Ala Gly Leu Glu Glu Tyr Ser Ala Met
1 5
<210> 1218
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1218
Asp Thr Ala Gly Leu Glu Glu Tyr Ser Ala
1 5 10
<210> 1219
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1219
Leu Asp Ile Leu Asp Thr Ala Gly Leu
1 5
<210> 1220
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1220
Ala Gly Leu Glu Glu Tyr Ser Ala Met Arg
1 5 10
<210> 1221
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1221
Leu Glu Glu Tyr Ser Ala Met Arg Asp
1 5
<210> 1222
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1222
Leu Leu Asp Ile Leu Asp Thr Ala Gly Leu Glu
1 5 10
<210> 1223
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1223
Asp Ile Leu Asp Thr Ala Gly Leu Glu
1 5
<210> 1224
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1224
Thr Ala Gly Leu Glu Glu Tyr Ser Ala
1 5
<210> 1225
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1225
Gly Leu Glu Glu Tyr Ser Ala Met
1 5
<210> 1226
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1226
Ala Ala Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1227
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1227
Val Val Val Gly Ala Ala Gly Val Gly Lys
1 5 10
<210> 1228
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1228
Val Val Gly Ala Ala Gly Val Gly Lys
1 5
<210> 1229
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1229
Thr Glu Tyr Lys Leu Val Val Val Gly Ala Ala
1 5 10
<210> 1230
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1230
Gly Ala Ala Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1231
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1231
Leu Val Val Val Gly Ala Ala Gly Val
1 5
<210> 1232
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1232
Gly Ala Ala Gly Val Gly Lys Ser Ala
1 5
<210> 1233
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1233
Lys Leu Val Val Val Gly Ala Ala Gly Val
1 5 10
<210> 1234
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1234
Ala Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1235
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1235
Val Val Val Gly Ala Ala Gly Val
1 5
<210> 1236
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1236
Tyr Lys Leu Val Val Val Gly Ala Ala
1 5
<210> 1237
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1237
Ala Ala Gly Val Gly Lys Ser Ala
1 5
<210> 1238
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1238
Val Gly Ala Ala Gly Val Gly Lys Ser Ala
1 5 10
<210> 1239
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1239
Ala Gly Val Gly Lys Ser Ala Leu Thr Ile
1 5 10
<210> 1240
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1240
Ala Cys Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1241
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1241
Gly Ala Cys Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1242
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1242
Tyr Lys Leu Val Val Val Gly Ala Cys
1 5
<210> 1243
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1243
Gly Ala Cys Gly Val Gly Lys Ser Ala
1 5
<210> 1244
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1244
Gly Ala Asp Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1245
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1245
Ala Asp Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1246
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1246
Gly Ala Asp Gly Val Gly Lys Ser Ala
1 5
<210> 1247
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1247
Gly Ala Asp Gly Val Gly Lys Ser
1 5
<210> 1248
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1248
Val Gly Ala Asp Gly Val Gly Lys Ser Ala
1 5 10
<210> 1249
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1249
Val Gly Ala Asp Gly Val Gly Lys
1 5
<210> 1250
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1250
Tyr Lys Leu Val Val Val Gly Ala Asp
1 5
<210> 1251
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1251
Asp Gly Val Gly Lys Ser Ala Leu Thr Ile
1 5 10
<210> 1252
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1252
Lys Leu Val Val Val Gly Ala Asp Gly Val Gly Lys
1 5 10
<210> 1253
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1253
Gly Ala Arg Gly Val Gly Lys Ser Ala
1 5
<210> 1254
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1254
Arg Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1255
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1255
Gly Ala Arg Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1256
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1256
Arg Gly Val Gly Lys Ser Ala Leu Thr Ile
1 5 10
<210> 1257
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1257
Leu Val Val Val Gly Ala Arg Gly Val
1 5
<210> 1258
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1258
Lys Leu Val Val Val Gly Ala Arg Gly Val
1 5 10
<210> 1259
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1259
Tyr Lys Leu Val Val Val Gly Ala Arg
1 5
<210> 1260
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1260
Lys Leu Val Val Val Gly Ala Arg
1 5
<210> 1261
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1261
Val Val Val Gly Ala Arg Gly Val
1 5
<210> 1262
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1262
Lys Leu Val Val Val Gly Ala Arg Gly Val Gly Lys
1 5 10
<210> 1263
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1263
Val Val Val Gly Ala Ser Gly Val Gly Lys
1 5 10
<210> 1264
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1264
Val Val Gly Ala Ser Gly Val Gly Lys
1 5
<210> 1265
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1265
Ala Ser Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1266
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1266
Gly Ala Ser Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1267
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1267
Ser Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1268
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1268
Gly Ala Ser Gly Val Gly Lys Ser Ala
1 5
<210> 1269
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1269
Lys Leu Val Val Val Gly Ala Ser Gly Val
1 5 10
<210> 1270
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1270
Leu Val Val Val Gly Ala Ser Gly Val
1 5
<210> 1271
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1271
Ala Ser Gly Val Gly Lys Ser Ala
1 5
<210> 1272
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1272
Ser Gly Val Gly Lys Ser Ala Leu Thr Ile
1 5 10
<210> 1273
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1273
Val Gly Ala Ser Gly Val Gly Lys Ser Ala
1 5 10
<210> 1274
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1274
Lys Leu Val Val Val Gly Ala Ser Gly
1 5
<210> 1275
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1275
Ala Val Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1276
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1276
Gly Ala Val Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1277
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1277
Val Val Val Gly Ala Val Gly Val
1 5
<210> 1278
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1278
Gly Ala Val Gly Val Gly Lys Ser Ala
1 5
<210> 1279
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1279
Val Gly Val Gly Lys Ser Ala Leu Thr Ile
1 5 10
<210> 1280
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1280
Ala Val Gly Val Gly Lys Ser Ala
1 5
<210> 1281
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1281
Tyr Lys Leu Val Val Val Gly Ala Val
1 5
<210> 1282
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1282
Leu Val Val Val Gly Ala Val Gly Val Gly Lys
1 5 10
<210> 1283
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1283
Lys Leu Val Val Val Gly Ala Val Gly Val Gly Lys
1 5 10
<210> 1284
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1284
Val Val Val Gly Ala Gly Cys Val Gly Lys
1 5 10
<210> 1285
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1285
Val Val Gly Ala Gly Cys Val Gly Lys
1 5
<210> 1286
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1286
Ala Gly Cys Val Gly Lys Ser Ala Leu
1 5
<210> 1287
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1287
Cys Val Gly Lys Ser Ala Leu Thr Ile
1 5
<210> 1288
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1288
Gly Cys Val Gly Lys Ser Ala Leu Thr Ile
1 5 10
<210> 1289
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1289
Lys Leu Val Val Val Gly Ala Gly Cys
1 5
<210> 1290
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1290
Gly Cys Val Gly Lys Ser Ala Leu
1 5
<210> 1291
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1291
Ala Gly Asp Val Gly Lys Ser Ala Leu
1 5
<210> 1292
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1292
Val Val Gly Ala Gly Asp Val Gly Lys
1 5
<210> 1293
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1293
Val Val Val Gly Ala Gly Asp Val Gly Lys
1 5 10
<210> 1294
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1294
Gly Ala Gly Asp Val Gly Lys Ser Ala
1 5
<210> 1295
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1295
Gly Ala Gly Asp Val Gly Lys Ser Ala Leu
1 5 10
<210> 1296
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1296
Asp Val Gly Lys Ser Ala Leu Thr Ile
1 5
<210> 1297
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1297
Gly Asp Val Gly Lys Ser Ala Leu
1 5
<210> 1298
<211> 48
<212> PRT
<213> Homo sapiens
<400> 1298
Ala Gln Ala Lys Ala Val Cys Ser Gln Glu Ser Arg Asp Val Leu Cys
1 5 10 15
Glu Leu Ser Asp His His Asn His Thr Leu Glu Glu Glu Cys Gln Trp
20 25 30
Gly Pro Cys Leu Gln Cys Leu Trp Ala Leu Leu Gln Ala Ser Gln Tyr
35 40 45
<210> 1299
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1299
Cys Leu Gln Cys Leu Trp Ala Leu Leu
1 5
<210> 1300
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1300
Cys Gln Trp Gly Pro Cys Leu Gln Cys Leu
1 5 10
<210> 1301
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1301
Gln Trp Gly Pro Cys Leu Gln Cys Leu
1 5
<210> 1302
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1302
Gln Trp Gly Pro Cys Leu Gln Cys Leu Trp
1 5 10
<210> 1303
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1303
Ala Ile Gln Pro Val Leu Trp Thr Thr
1 5
<210> 1304
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1304
Ala Leu Gln Pro Leu Gln Pro His Ala
1 5
<210> 1305
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1305
Asp Leu His Phe Cys Arg Ser Ser Ile Met
1 5 10
<210> 1306
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1306
Glu Pro His Leu Ala Leu Gln Pro Leu
1 5
<210> 1307
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1307
Glu Ser Lys Ile Met Phe Ala Thr Leu
1 5
<210> 1308
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1308
Phe Ala Thr Leu Gln Arg Ser Ser Leu
1 5
<210> 1309
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1309
Phe Leu Lys Ala Glu Ser Lys Ile Met
1 5
<210> 1310
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1310
Phe Leu Lys Ala Glu Ser Lys Ile Met Phe
1 5 10
<210> 1311
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1311
Gly Pro Pro Ala Arg Val Pro Ala Val
1 5
<210> 1312
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1312
Ile Met Lys Pro Lys Arg Asp Gly Tyr Met
1 5 10
<210> 1313
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1313
Lys Ile Met Phe Ala Thr Leu Gln Arg
1 5
<210> 1314
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1314
Lys Pro Lys Arg Asp Gly Tyr Met Phe
1 5
<210> 1315
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1315
Lys Pro Lys Arg Asp Gly Tyr Met Phe Leu
1 5 10
<210> 1316
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1316
Leu His Phe Cys Arg Ser Ser Ile Met
1 5
<210> 1317
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1317
Leu Gln His Gly His Arg His Gly Leu
1 5
<210> 1318
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1318
Met Phe Ala Thr Leu Gln Arg Ser Ser Leu
1 5 10
<210> 1319
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1319
Met Phe Leu Lys Ala Glu Ser Lys Ile
1 5
<210> 1320
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1320
Met Leu Thr Gly Pro Pro Ala Arg Val
1 5
<210> 1321
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1321
Gln Pro Val Leu Trp Thr Thr Pro Pro Leu
1 5 10
<210> 1322
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1322
Ser Met Leu Thr Gly Pro Pro Ala Arg Val
1 5 10
<210> 1323
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1323
Thr Leu Gln Arg Ser Ser Leu Trp Cys Leu
1 5 10
<210> 1324
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1324
Val Leu Pro Glu Pro His Leu Ala Leu
1 5
<210> 1325
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1325
Val Pro Ala Val Pro Phe Asp Leu His Phe
1 5 10
<210> 1326
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1326
Tyr Met Phe Leu Lys Ala Glu Ser Lys
1 5
<210> 1327
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1327
Tyr Met Phe Leu Lys Ala Glu Ser Lys Ile
1 5 10
<210> 1328
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1328
Phe Thr Cys Gln Ile Pro Gly Ile Tyr Tyr
1 5 10
<210> 1329
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1329
Gly Ser Asp Gly Lys Pro Gly Tyr
1 5
<210> 1330
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1330
Asn Ala Glu Ser Asn Gly Leu Tyr
1 5
<210> 1331
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1331
Leu Thr Glu Asn Asp Gln Val Trp Leu
1 5
<210> 1332
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1332
Gly Thr His Val Trp Val Gly Leu Tyr
1 5
<210> 1333
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1333
Thr Tyr Asp Glu Tyr Thr Lys Gly Tyr
1 5
<210> 1334
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1334
Tyr Thr Tyr Asp Glu Tyr Thr Lys Gly Tyr
1 5 10
<210> 1335
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1335
Phe Thr Cys Gln Ile Pro Gly Ile Tyr
1 5
<210> 1336
<211> 12
<212> PRT
<213> Homo sapiens
<400> 1336
Asn Ala Glu Ser Asn Gly Leu Tyr Ser Ser Glu Tyr
1 5 10
<210> 1337
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1337
Tyr Leu Asp Gln Ala Ser Gly Ser Ala
1 5
<210> 1338
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1338
Phe Leu Leu Leu Val Ser Leu Asn Leu
1 5
<210> 1339
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1339
Phe Leu Leu Leu Val Ser Leu Asn Leu Val
1 5 10
<210> 1340
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1340
Gly Leu Tyr Lys Asn Gly Thr Pro Val
1 5
<210> 1341
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1341
Gly Leu Asp Gly Pro Lys Gly Asn Pro Gly Leu
1 5 10
<210> 1342
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1342
Leu Leu Leu Val Ser Leu Asn Leu Val
1 5
<210> 1343
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1343
Ser Leu Ser Gly Thr Pro Leu Val Ser Ala
1 5 10
<210> 1344
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1344
Gly Leu Tyr Ser Ser Glu Tyr Val
1 5
<210> 1345
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1345
Ser Leu Ser Gly Thr Pro Leu Val
1 5
<210> 1346
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1346
Met Leu Pro Gln Ile Pro Phe Leu Leu
1 5
<210> 1347
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1347
Gly Leu Pro Gly Pro Pro Gly Pro Ser Ala
1 5 10
<210> 1348
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1348
Ser Ala Phe Thr Val Ile Leu Ser Lys
1 5
<210> 1349
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1349
Ala Val Met Pro Glu Gly Phe Ile Lys
1 5
<210> 1350
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1350
Gly Leu Tyr Lys Asn Gly Thr Pro Val Met Tyr
1 5 10
<210> 1351
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1351
Ala Ile Gly Thr Pro Ile Pro Phe Asp Lys
1 5 10
<210> 1352
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1352
Gly Leu Pro Gly Gly Pro Gly Ala Lys
1 5
<210> 1353
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1353
Ile Leu Tyr Asn Arg Gln Gln His Tyr
1 5
<210> 1354
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1354
Ala Gly Pro Pro Gly Pro Pro Gly Phe Gly Lys
1 5 10
<210> 1355
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1355
Gly Ile Pro Gly Phe Pro Gly Ser Lys
1 5
<210> 1356
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1356
Gly Thr His Val Trp Val Gly Leu Tyr Lys
1 5 10
<210> 1357
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1357
Gly Val Pro Gly Gln Pro Gly Ile Lys
1 5
<210> 1358
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1358
Val Ser Ala Phe Thr Val Ile Leu Ser Lys
1 5 10
<210> 1359
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1359
Ala Val Met Pro Glu Gly Phe Ile Lys Ala
1 5 10
<210> 1360
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1360
Ser Ser Phe Ser Gly Phe Leu Val Ala
1 5
<210> 1361
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1361
Pro Val Ser Ala Phe Thr Val Ile Leu Ser Lys
1 5 10
<210> 1362
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1362
Gly Val Pro Gly Met Asn Gly Gln Lys
1 5
<210> 1363
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1363
Ala Tyr Pro Ala Ile Gly Thr Pro Ile Pro Phe
1 5 10
<210> 1364
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1364
Ile Gly Pro Pro Gly Ile Pro Gly Phe
1 5
<210> 1365
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1365
His Tyr Asp Pro Arg Thr Gly Ile Phe
1 5
<210> 1366
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1366
Glu Tyr Val His Ser Ser Phe Ser Gly Phe
1 5 10
<210> 1367
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1367
Ala Gly Pro Pro Gly Pro Pro Gly Phe
1 5
<210> 1368
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1368
Tyr Tyr Phe Ser Tyr His Val His Val
1 5
<210> 1369
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1369
Ala Tyr Pro Ala Ile Gly Thr Pro Ile
1 5
<210> 1370
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1370
Pro Leu Pro Asn Thr Lys Thr Gln Phe
1 5
<210> 1371
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1371
Cys Gln Ile Pro Gly Ile Tyr Tyr Phe
1 5
<210> 1372
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1372
Arg Pro Ser Leu Ser Gly Thr Pro Leu
1 5
<210> 1373
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1373
Leu Pro Gln Ile Pro Phe Leu Leu Leu
1 5
<210> 1374
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1374
Ile Pro Phe Leu Leu Leu Val Ser Leu
1 5
<210> 1375
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1375
Leu Pro Gly Pro Pro Gly Pro Ser Ala Val
1 5 10
<210> 1376
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1376
Gly Pro Ile Gly Pro Pro Gly Ile Pro Gly Phe
1 5 10
<210> 1377
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1377
Ile Pro Gly Pro Ala Gly Ile Ser Val
1 5
<210> 1378
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1378
Tyr Pro Ala Ile Gly Thr Pro Ile Pro Phe
1 5 10
<210> 1379
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1379
Ser Pro Gly Pro Pro Gly Pro Ala Gly Ile
1 5 10
<210> 1380
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1380
Leu Pro Gly Pro Pro Gly Pro Ser Ala
1 5
<210> 1381
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1381
Ser Pro Gly Pro Pro Gly Pro Ala Gly
1 5
<210> 1382
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1382
Thr Ile Lys Ser Lys Gly Ile Ala Val
1 5
<210> 1383
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1383
His Val His Val Lys Gly Thr His Val
1 5
<210> 1384
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1384
Leu Pro Asn Thr Lys Thr Gln Phe
1 5
<210> 1385
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1385
Leu Pro Gln Ile Pro Phe Leu Leu
1 5
<210> 1386
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1386
Pro Phe Leu Leu Leu Val Ser Leu
1 5
<210> 1387
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1387
Ser Leu Asn Leu Val His Gly Val
1 5
<210> 1388
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1388
Thr Gly Met Pro Val Ser Ala Phe
1 5
<210> 1389
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1389
Thr Pro Ile Pro Phe Asp Lys Ile Leu
1 5
<210> 1390
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1390
Ile Met Lys Pro Lys Arg Asp Gly Tyr
1 5
<210> 1391
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1391
Ser Ile Met Lys Pro Lys Arg Asp Gly Tyr
1 5 10
<210> 1392
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1392
Ala Glu Ser Lys Ile Met Phe Ala Thr Leu
1 5 10
<210> 1393
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1393
Ala Glu Ser Lys Ile Met Phe Ala Thr
1 5
<210> 1394
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1394
Ser Leu Gln Cys Val Ser Leu His Leu
1 5
<210> 1395
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1395
Leu Val Leu Ser Ile Ala Leu Ser Val
1 5
<210> 1396
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1396
Val Ile Leu Gly Val His Leu Ser Val
1 5
<210> 1397
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1397
Val Leu Ala Pro Gln Glu Ser Ser Val
1 5
<210> 1398
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1398
Ser Leu Gln Cys Val Ser Leu His Leu Leu
1 5 10
<210> 1399
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1399
Met Leu Leu Arg Leu Ser Glu Pro Ala
1 5
<210> 1400
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1400
Leu Thr Met Pro Ala Leu Pro Met Val
1 5
<210> 1401
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1401
Phe Leu Thr Leu Ser Val Thr Trp Ile Ala
1 5 10
<210> 1402
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1402
Lys Leu Gln Cys Val Asp Leu His Val
1 5
<210> 1403
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1403
Phe Leu Thr Pro Lys Lys Leu Gln Cys Val
1 5 10
<210> 1404
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1404
Phe Leu Arg Pro Gly Asp Asp Ser Thr Leu
1 5 10
<210> 1405
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1405
Phe Leu Gly Tyr Leu Ile Leu Gly Val
1 5
<210> 1406
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1406
Leu Leu Ala Asn Asp Leu Met Leu Ile
1 5
<210> 1407
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1407
Phe Gln Asn Ser Tyr Thr Ile Gly Leu
1 5
<210> 1408
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1408
Met Leu Ile Lys Leu Asp Glu Ser Val
1 5
<210> 1409
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1409
Val Leu Gln Cys Val Asn Val Ser Val
1 5
<210> 1410
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1410
Ile Leu Asn Asp Thr Gly Cys His Tyr Val
1 5 10
<210> 1411
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1411
Phe Gln Tyr Pro Glu Phe Ser Ile Glu Leu
1 5 10
<210> 1412
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1412
Ile Leu Gly Lys Tyr Gln Leu Asn Val
1 5
<210> 1413
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1413
Leu Leu Gly Asn Ser Val Asn Phe Thr Val
1 5 10
<210> 1414
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1414
Val Leu Asp Cys Cys Ile Ser Leu Leu
1 5
<210> 1415
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1415
Ile Leu Gly Ser Phe Glu Leu Gln Leu
1 5
<210> 1416
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1416
Arg Leu Ile Trp Leu Val Lys Met Val
1 5
<210> 1417
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1417
Val Leu Leu Gly Asn Ser Val Asn Phe Thr Val
1 5 10
<210> 1418
<211> 9
<212> PRT
<213> Homo sapiens
<400> 1418
Thr Leu Ala Ile Pro Leu Thr Asp Val
1 5
<210> 1419
<211> 4
<212> PRT
<213> Homo sapiens
<400> 1419
Asp Glu Ala His
1
<210> 1420
<211> 10
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> May or may not be present
<400> 1420
Val Val Val Gly Ala Asp Gly Val Gly Lys
1 5 10
<210> 1421
<211> 10
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (10)..(10)
<223> May or may not be present
<400> 1421
Gly Ala Asp Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1422
<211> 10
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> May or may not be present
<400> 1422
Val Val Val Gly Ala Val Gly Val Gly Lys
1 5 10
<210> 1423
<211> 10
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> May or may not be present
<400> 1423
Gly Ala Val Gly Val Gly Lys Ser Ala Leu
1 5 10
<210> 1424
<211> 10
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> May or may not be present
<400> 1424
Val Val Val Gly Ala Cys Gly Val Gly Lys
1 5 10
<210> 1425
<211> 10
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> May or may not be present
<400> 1425
Val Val Val Gly Ala Arg Gly Val Gly Lys
1 5 10
<210> 1426
<211> 9
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> May or may not be present
<400> 1426
Ala Arg Gly Val Gly Lys Ser Ala Leu
1 5
<210> 1427
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1427
Thr Glu Tyr Lys Leu Val Val Val Gly Ala Val Gly Val Gly Lys
1 5 10 15
<210> 1428
<211> 20
<212> PRT
<213> Homo sapiens
<400> 1428
Thr Glu Tyr Lys Leu Val Val Val Gly Ala Val Gly Val Gly Lys Ser
1 5 10 15
Ala Leu Thr Ile
20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НЕОАНТИГЕНЫ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2813924C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ИММУНООНКОЛОГИИ | 2016 |
|
RU2771624C2 |
| ПЕПТИД, ПОЛУЧЕННЫЙ ИЗ GPC3, ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ ДЛЯ ЛЕЧЕНИЯ ИЛИ ПРЕДОТВРАЩЕНИЯ РАКА С ЕГО ИСПОЛЬЗОВАНИЕМ, ИНДУКТОР ИММУНИТЕТА И СПОСОБ ПОЛУЧЕНИЯ АНТИГЕН-ПРЕЗЕНТИРУЮЩИХ КЛЕТОК | 2016 |
|
RU2714117C2 |
| АНТИ-PRE-S1 HBV АНТИТЕЛА | 2016 |
|
RU2739955C2 |
| ИММУНОДОМИНАНТНЫЕ БЕЛКИ И ФРАГМЕНТЫ ПРИ РАССЕЯННОМ СКЛЕРОЗЕ | 2019 |
|
RU2828590C2 |
| НОВЫЕ МОЛЕКУЛЫ АГОНИСТИЧЕСКИХ АНТИ-TNFR2 АНТИТЕЛ | 2019 |
|
RU2840049C2 |
| АНТИТЕЛА, СВЯЗЫВАЮЩИЕ CTLA-4, И ИХ ПРИМЕНЕНИЕ | 2018 |
|
RU2756100C1 |
| СПЕЦИФИЧЕСКИЕ АНТИТЕЛА К PD-L1 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2749109C2 |
| ИСКУССТВЕННЫЕ АНТИГЕНПРЕЗЕНТИРУЮЩИЕ КЛЕТКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2763798C1 |
| НЕОАНТИГЕНЫ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2805196C2 |
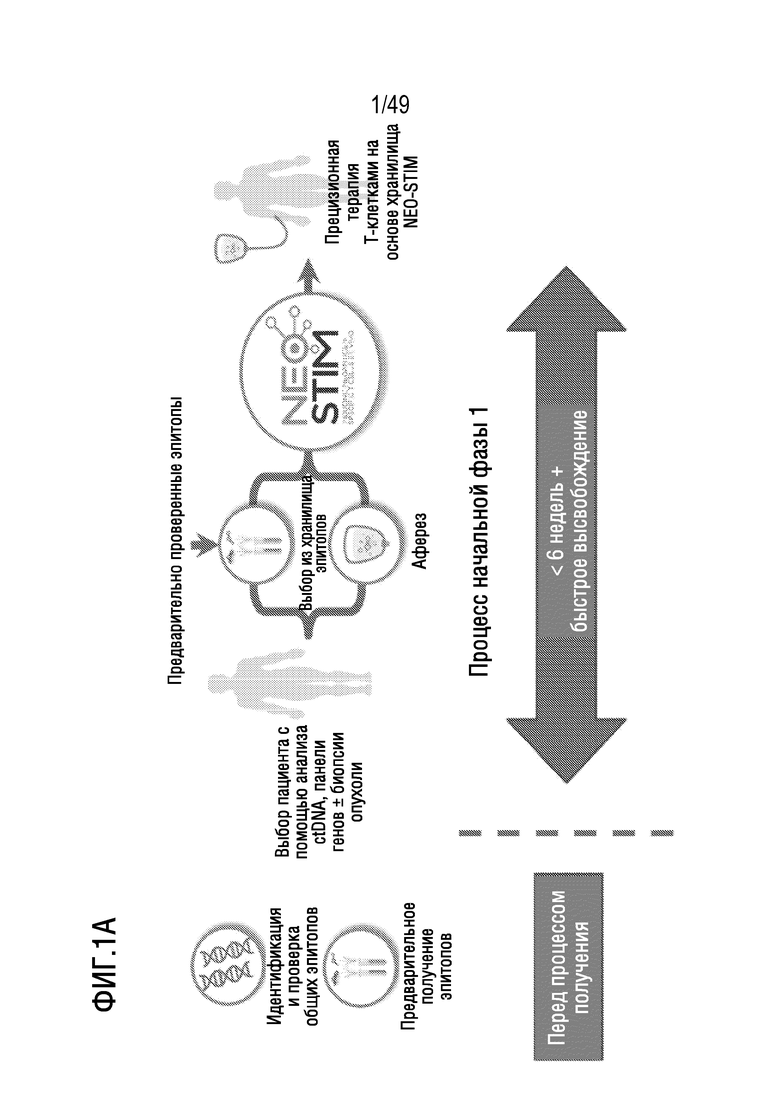
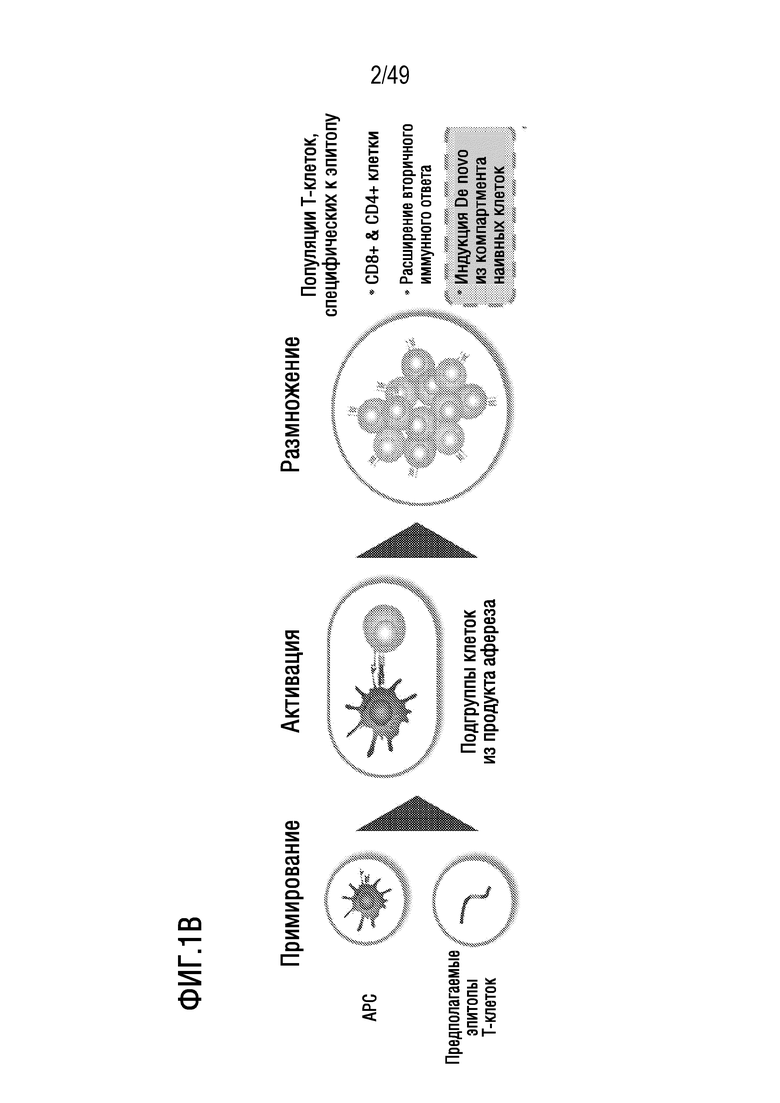
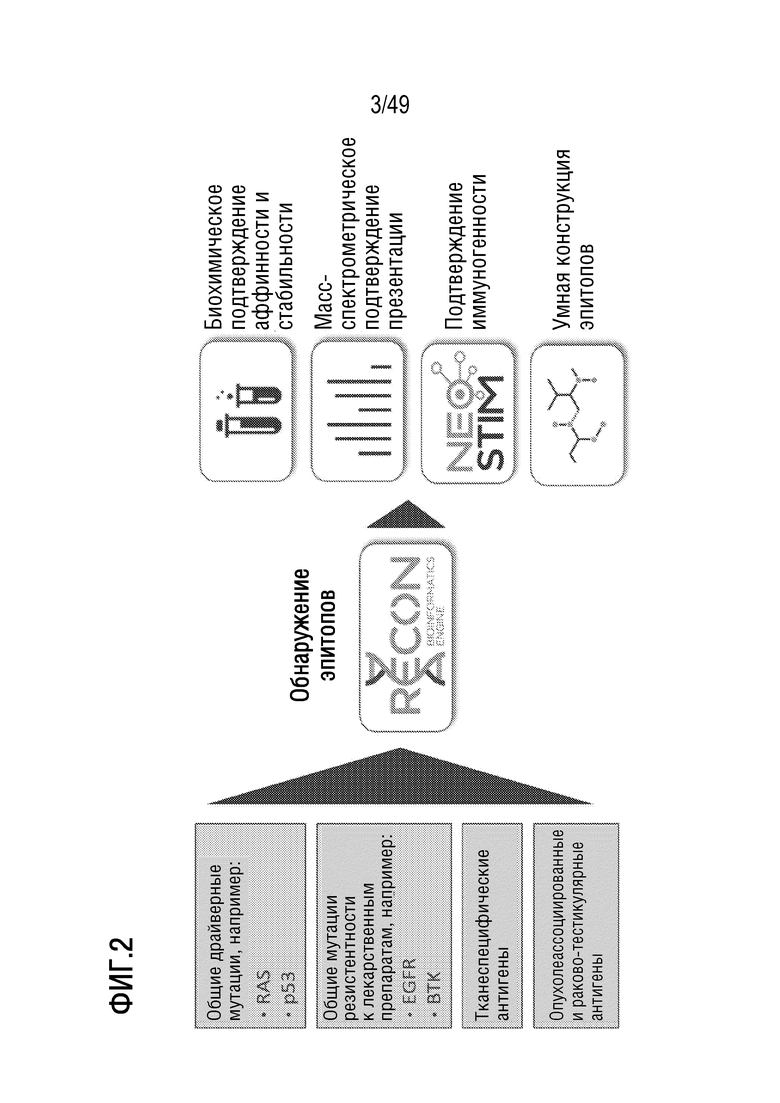
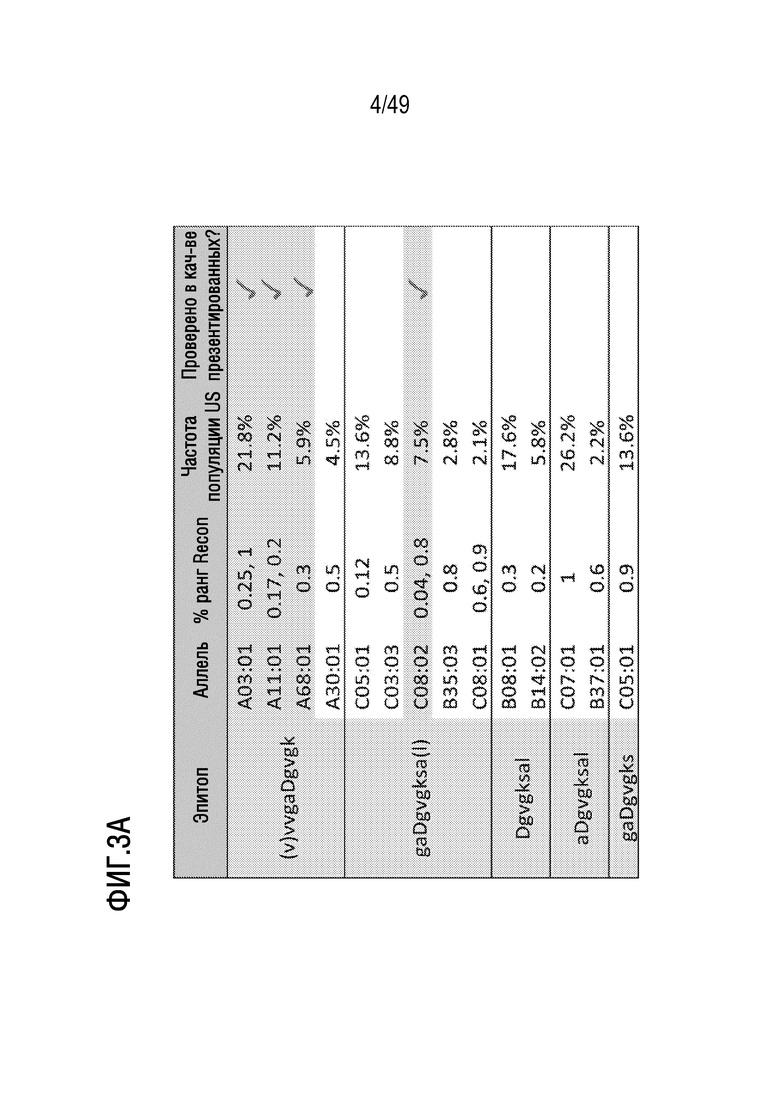
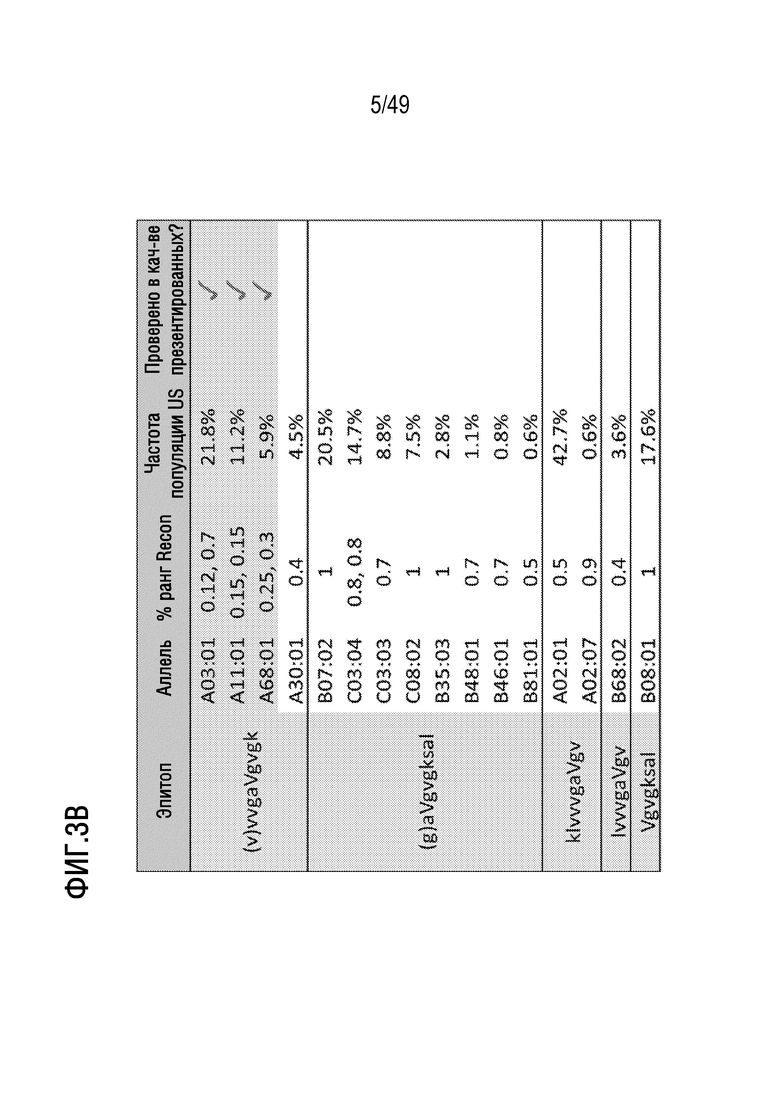
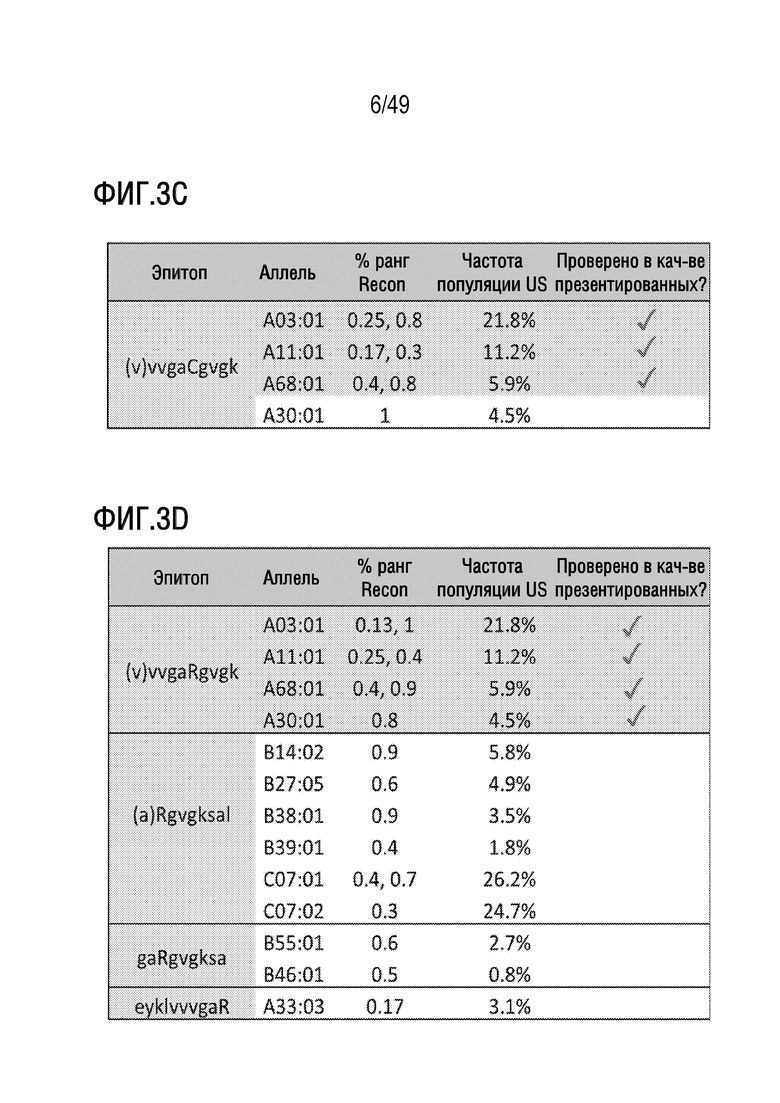
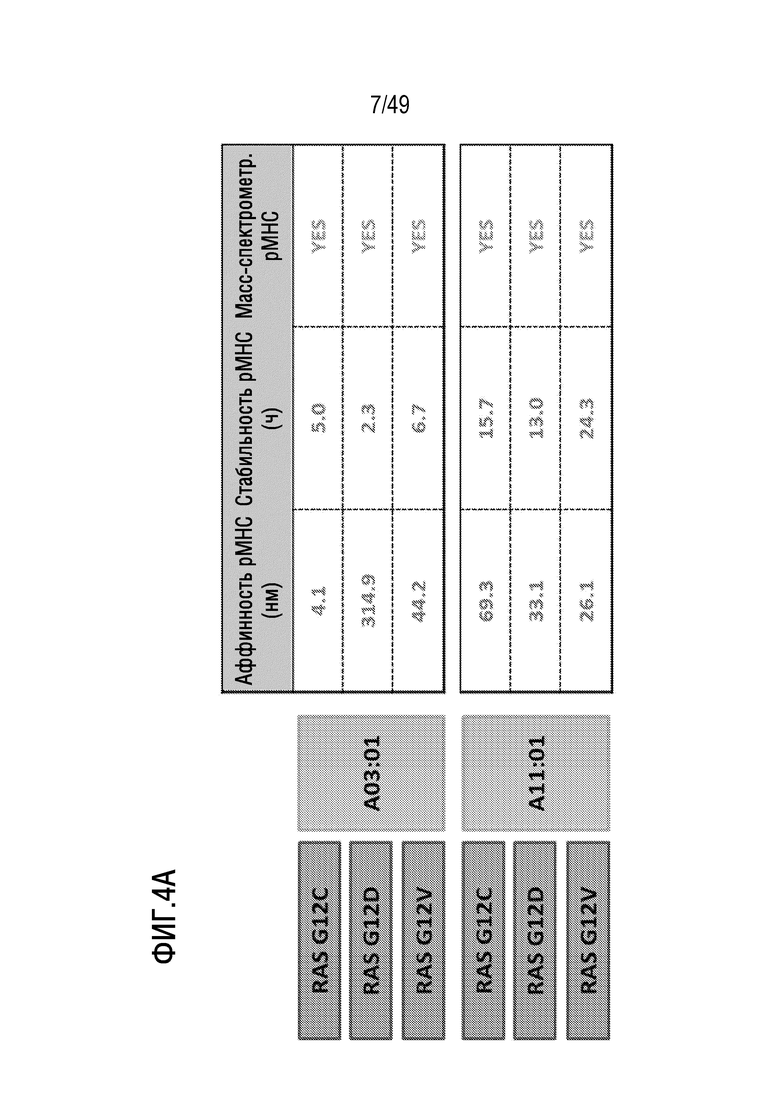
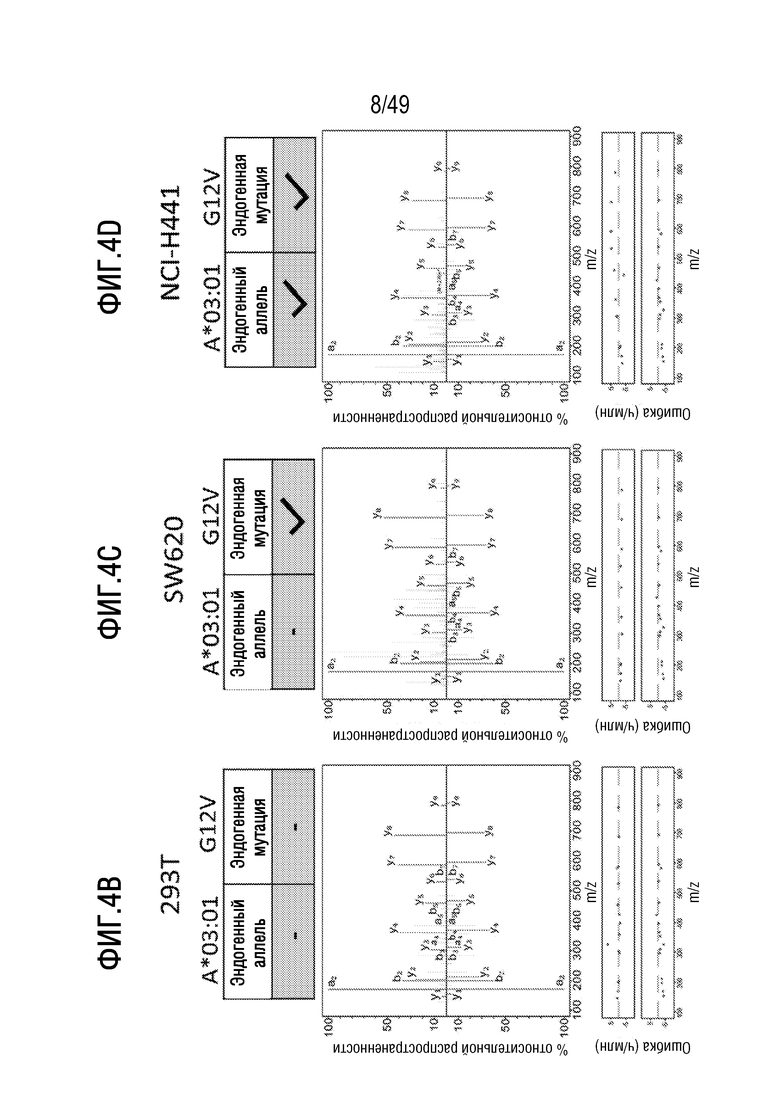
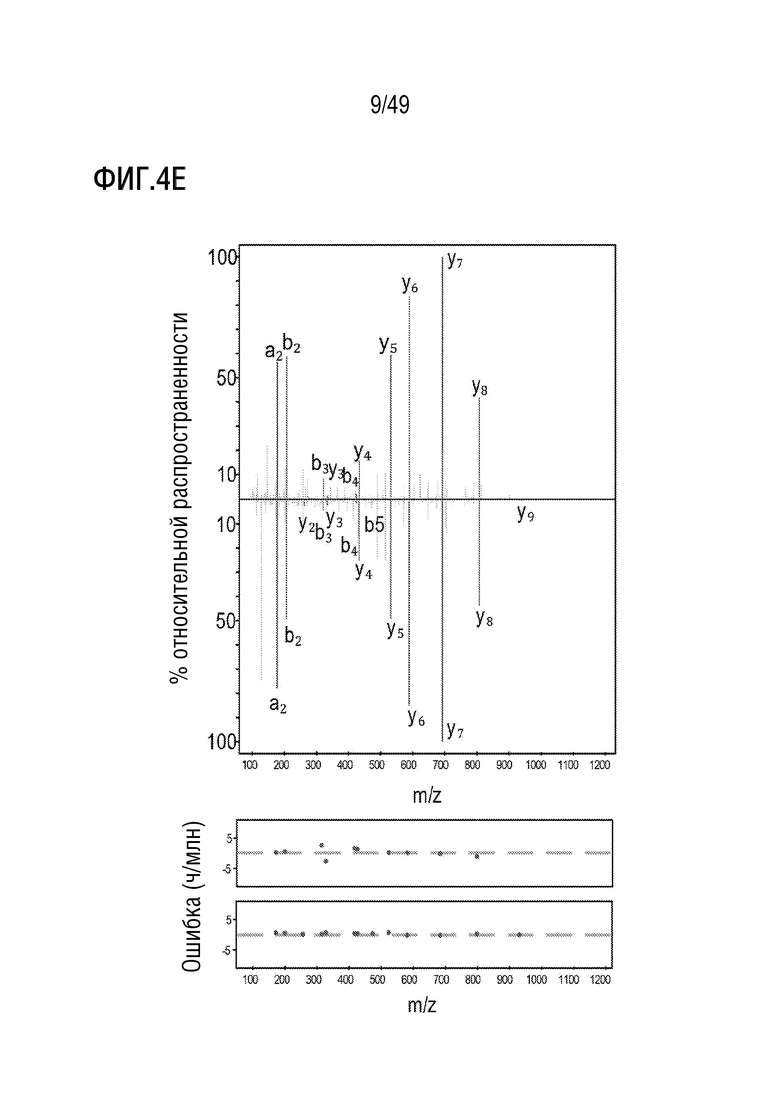
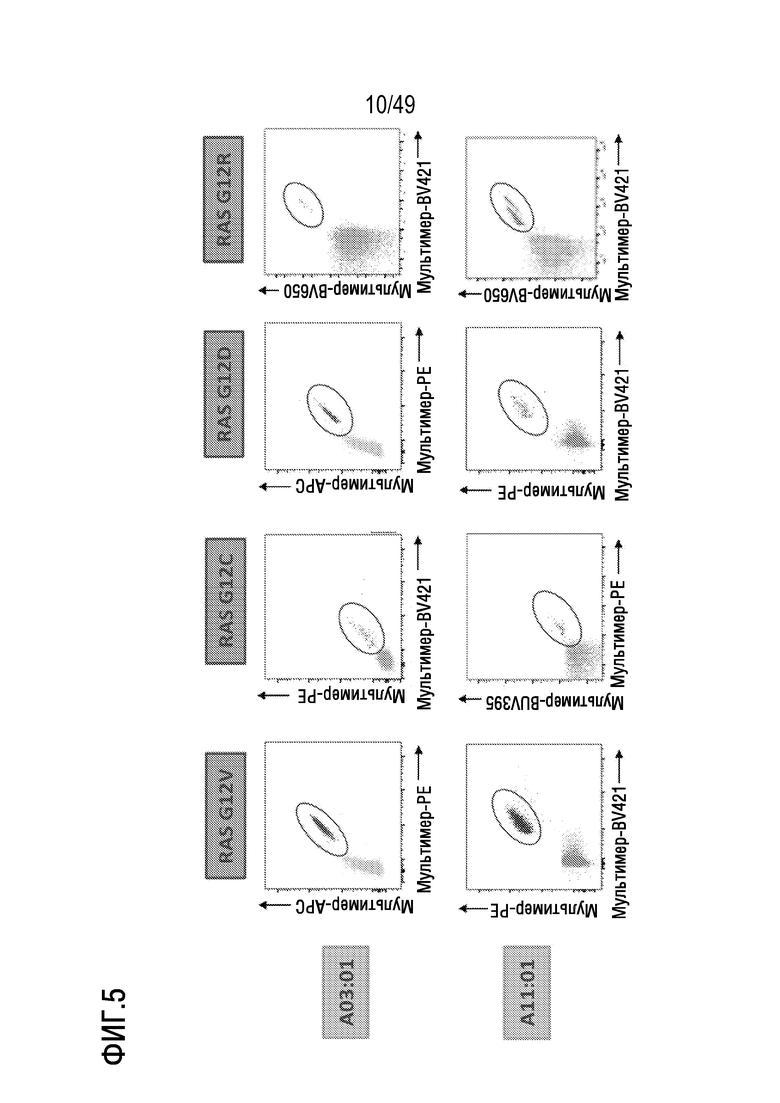
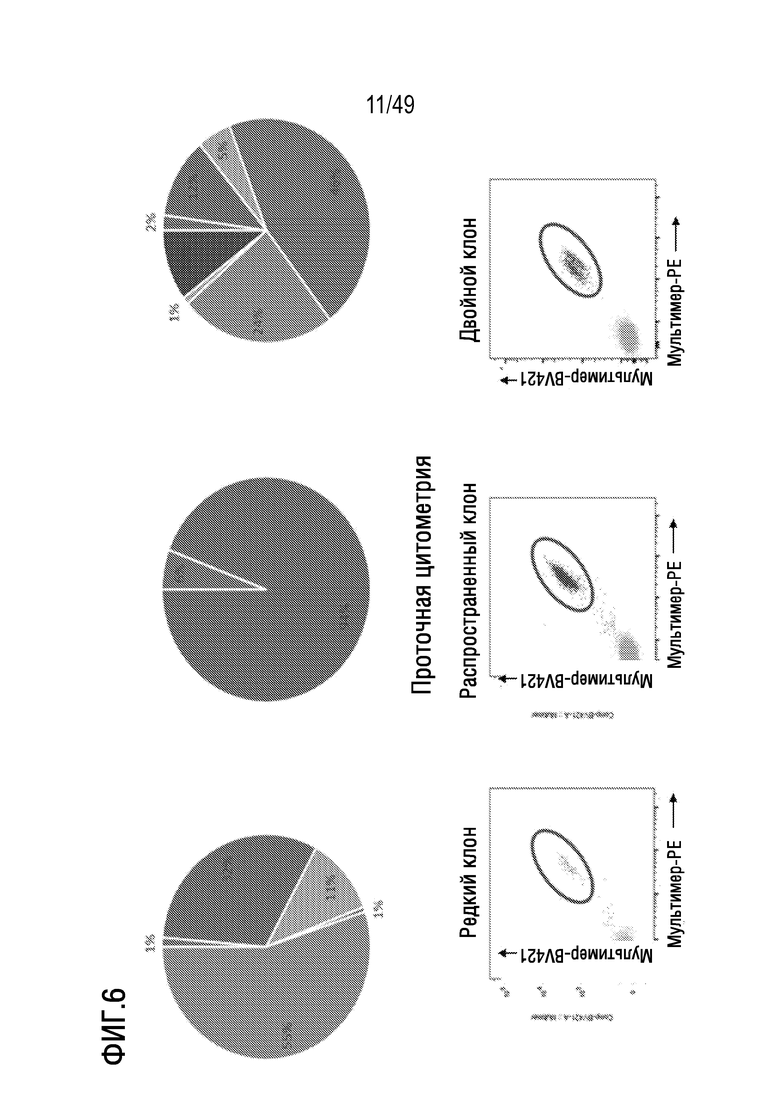
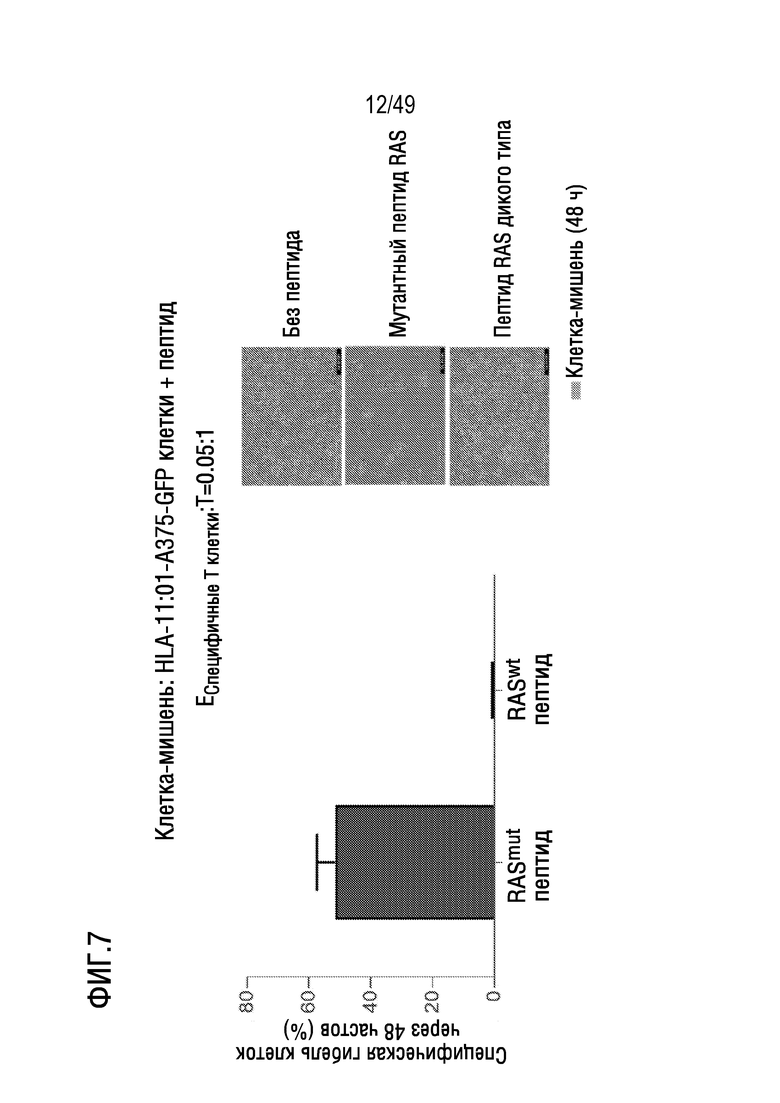
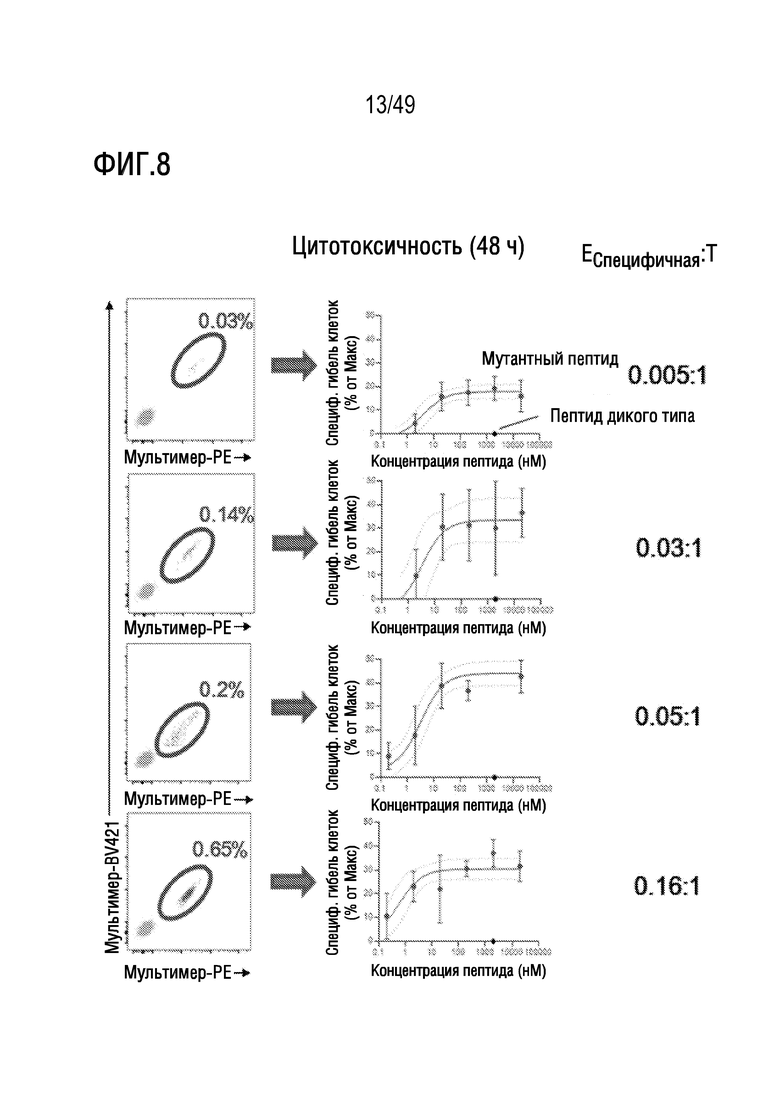
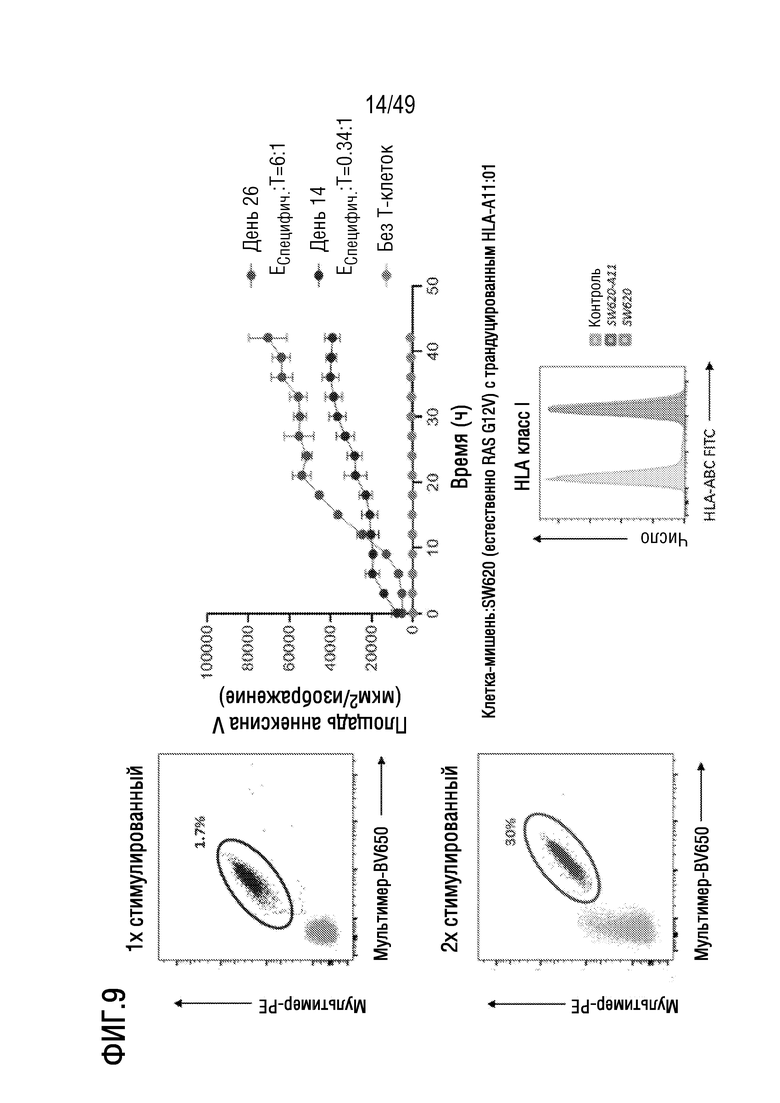
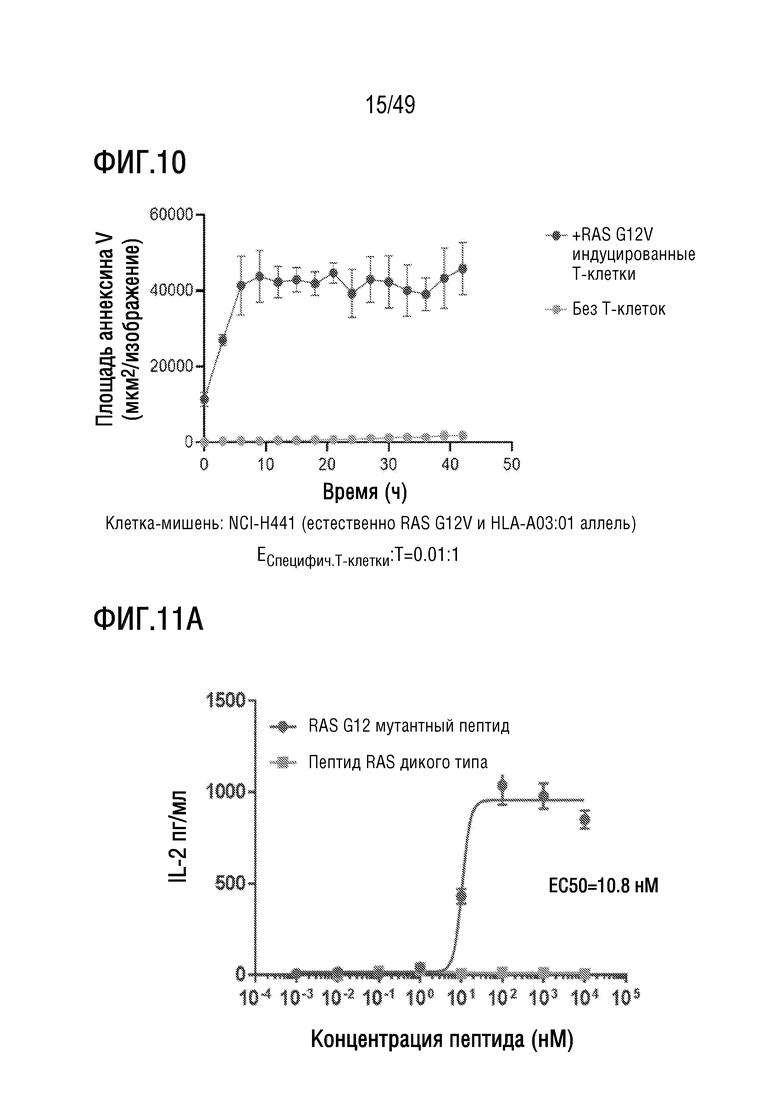
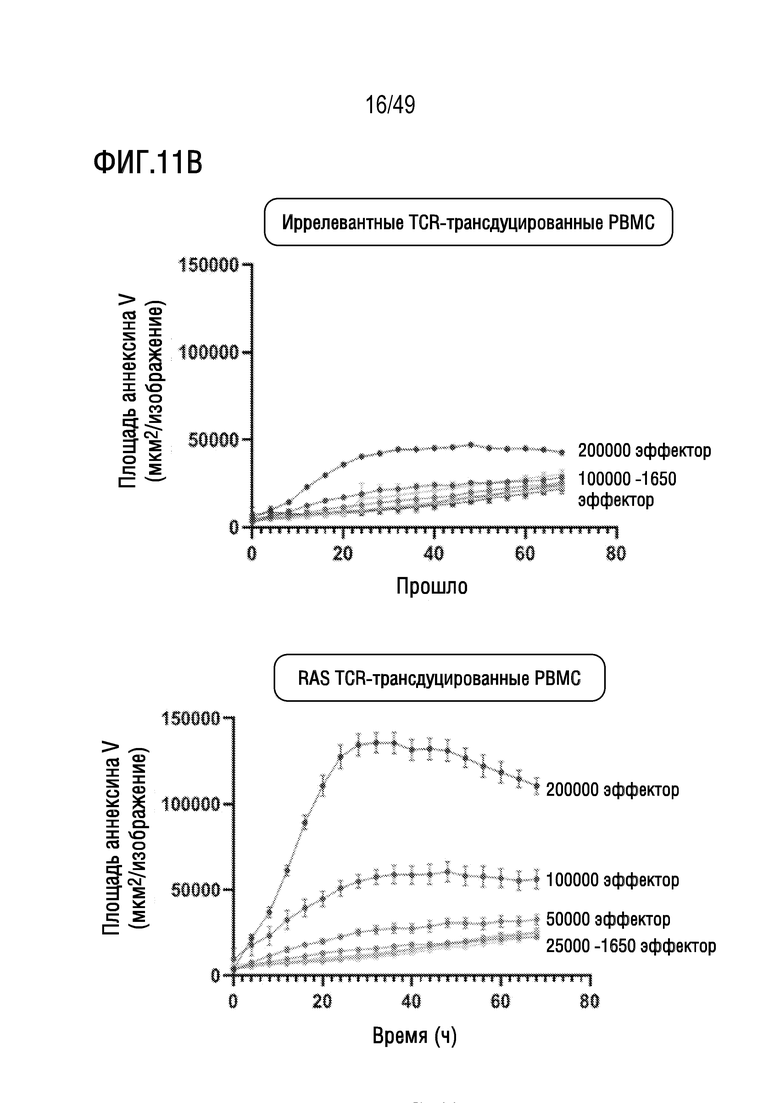
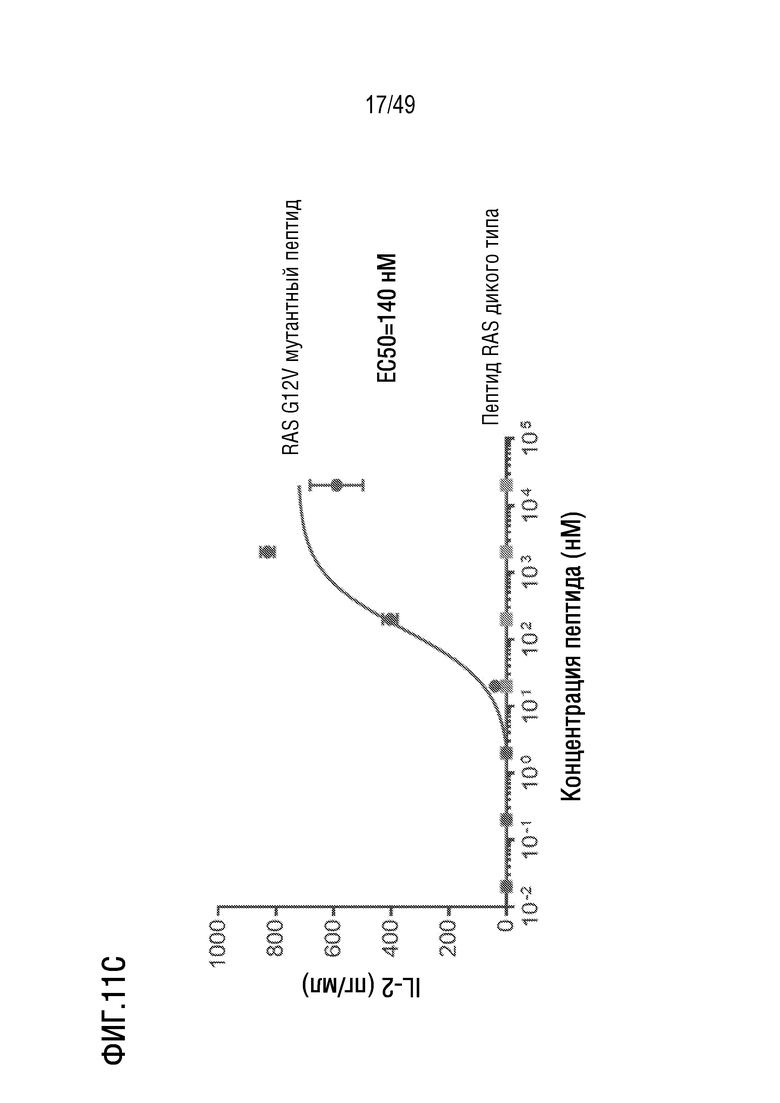
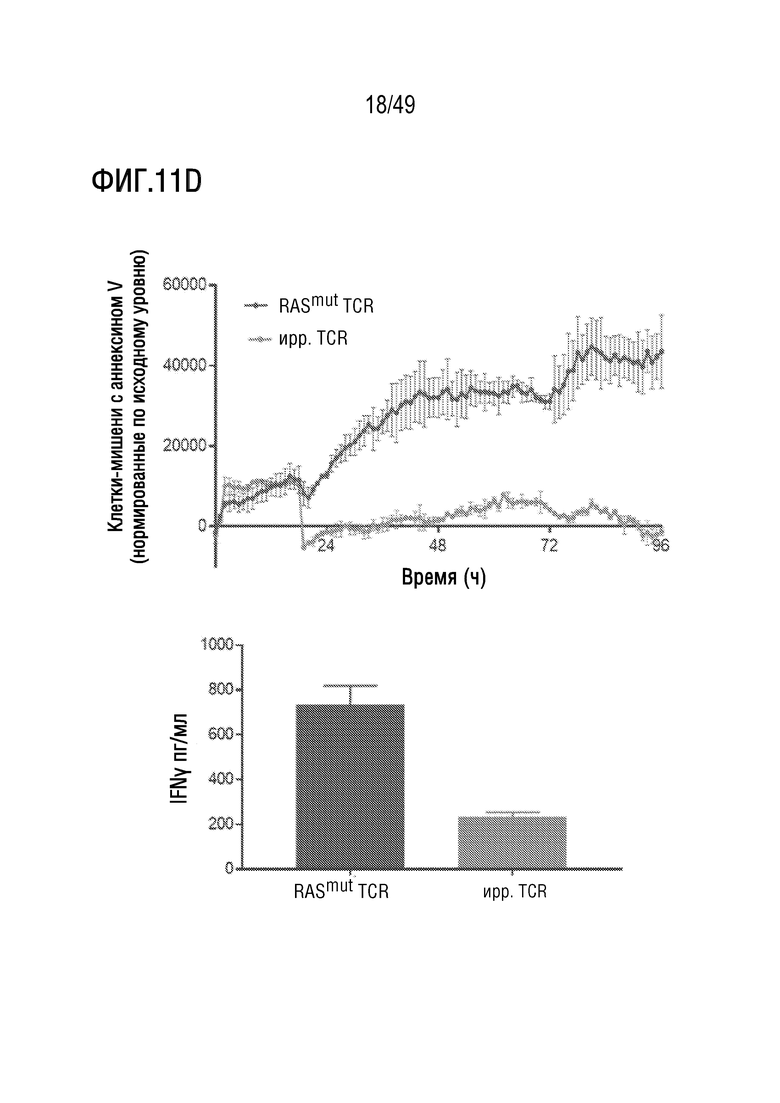
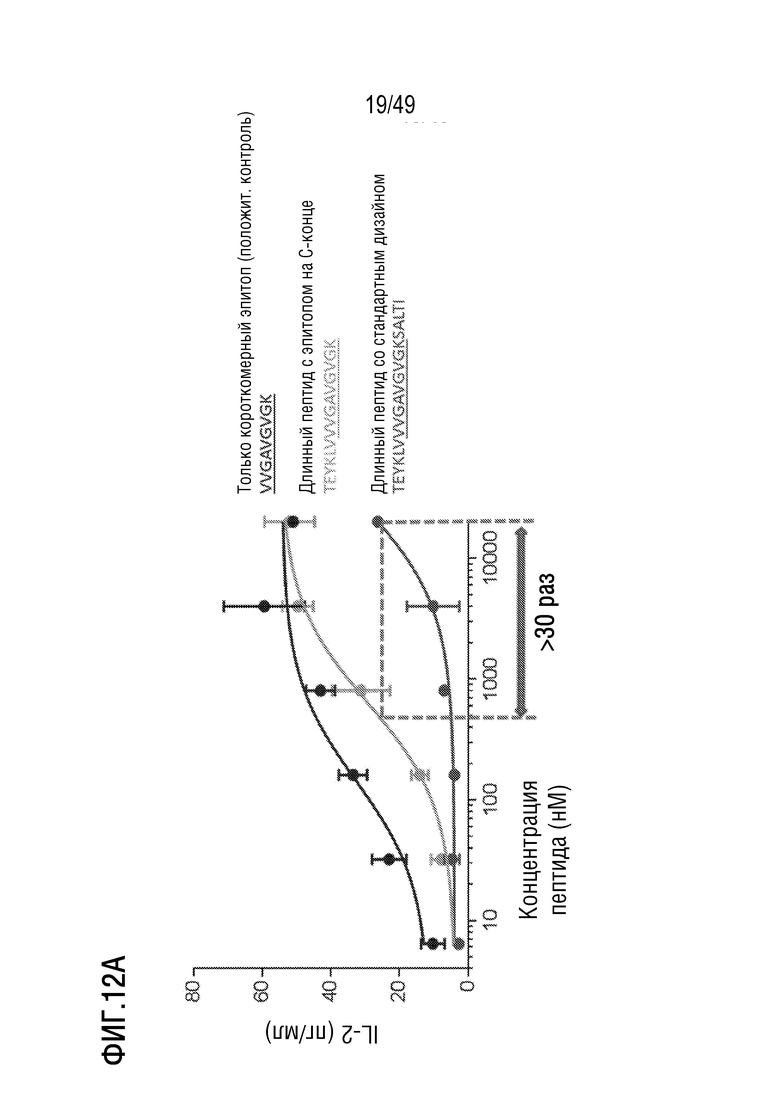
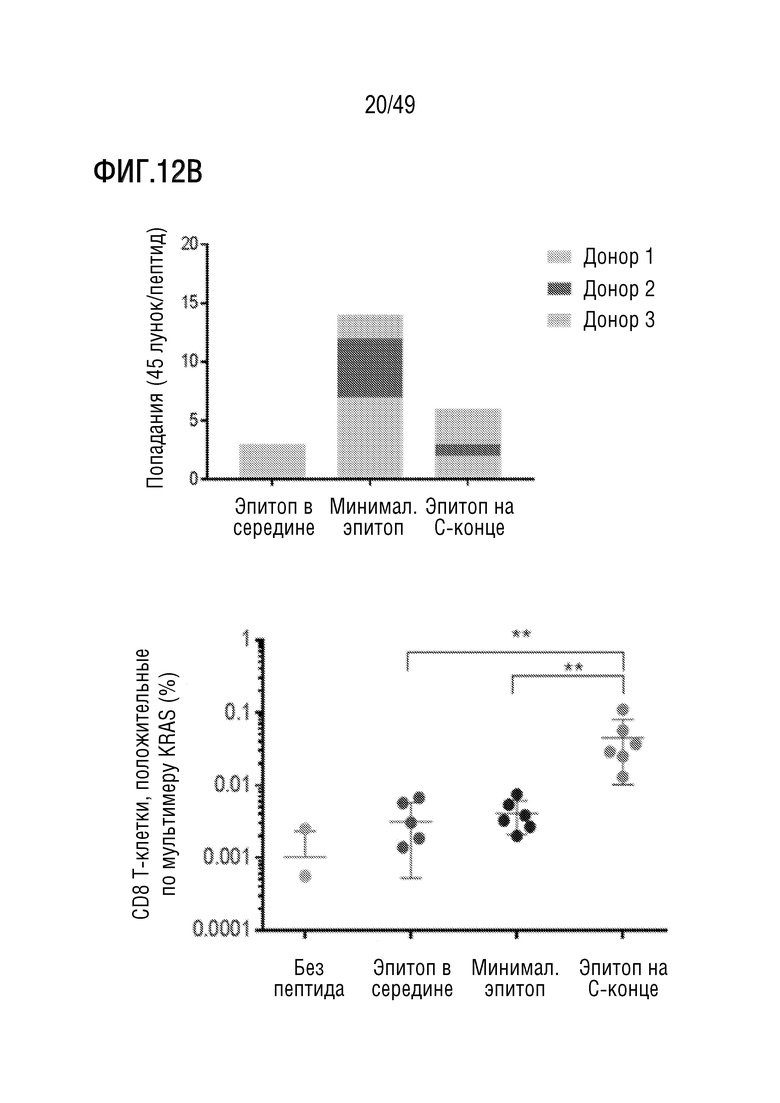
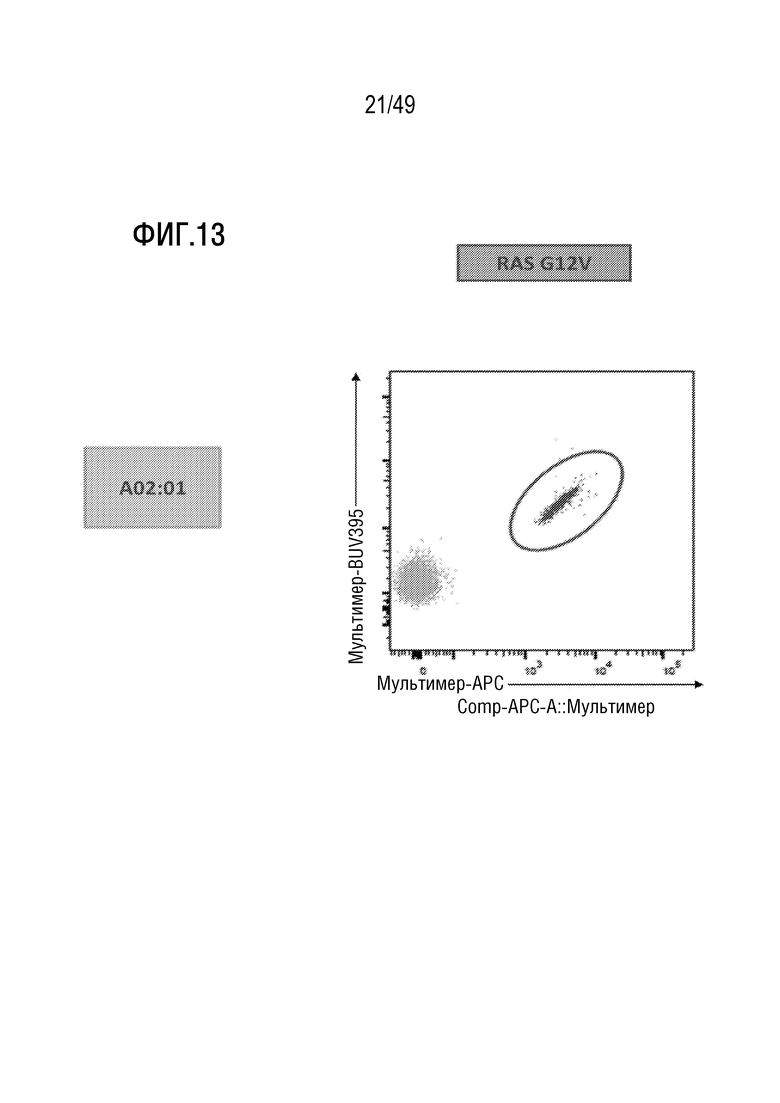


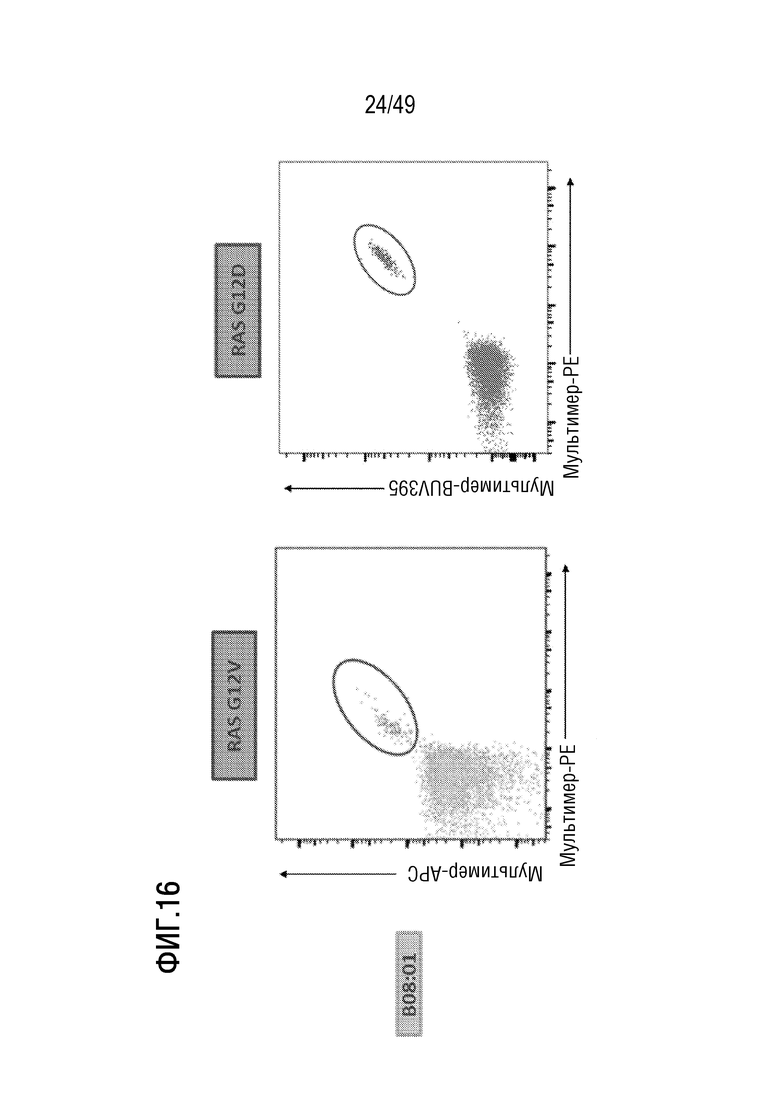
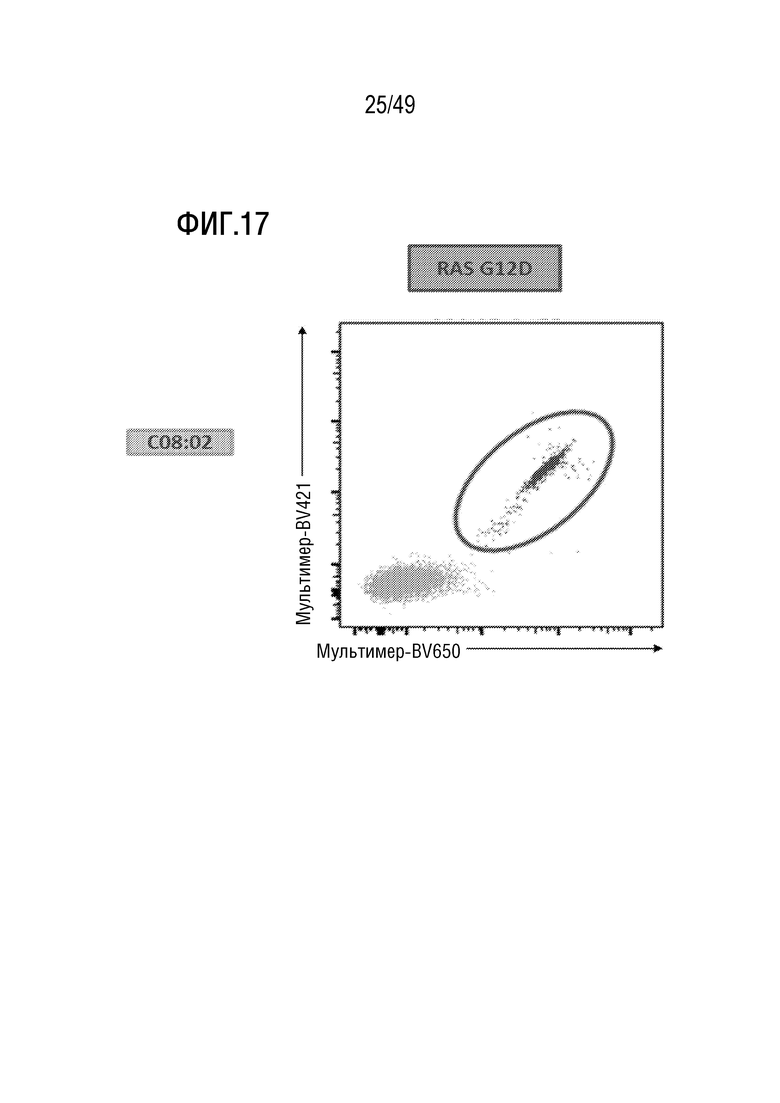
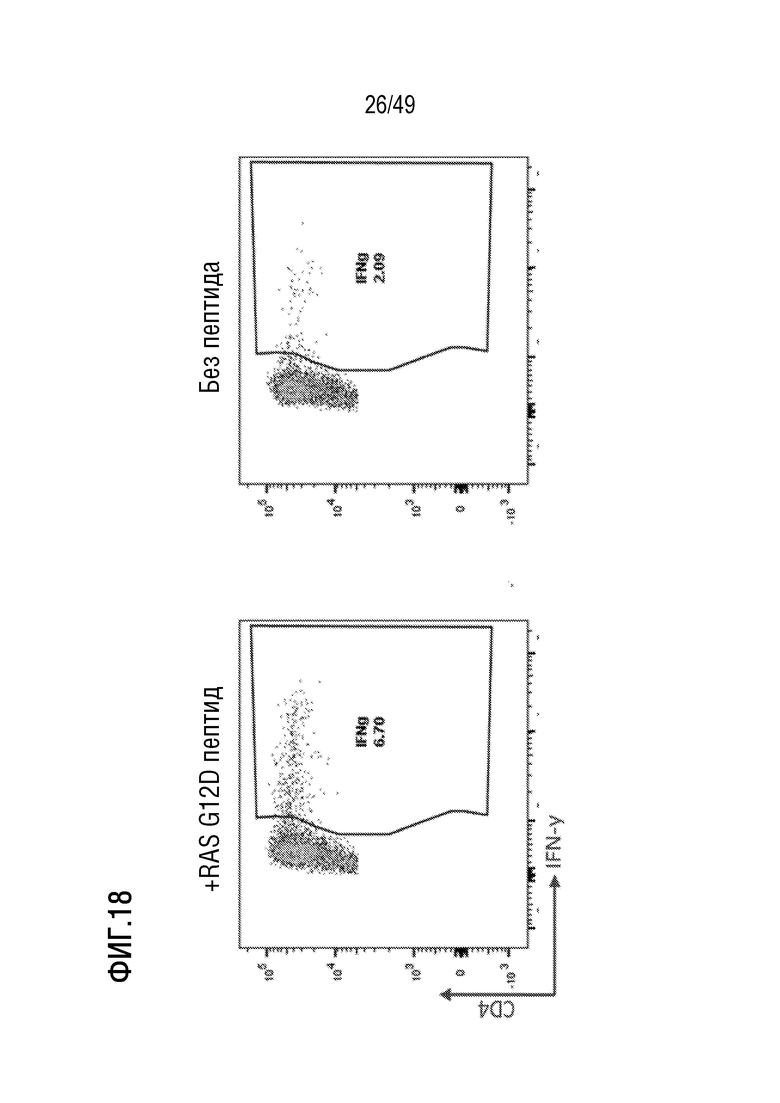
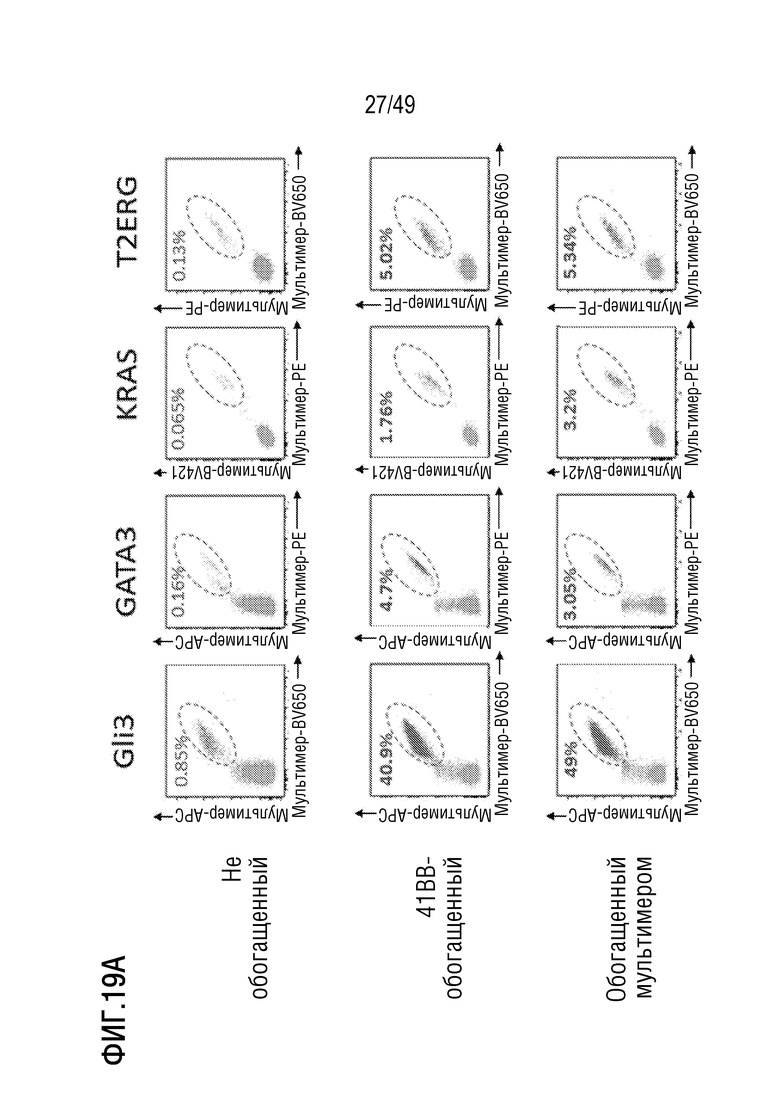
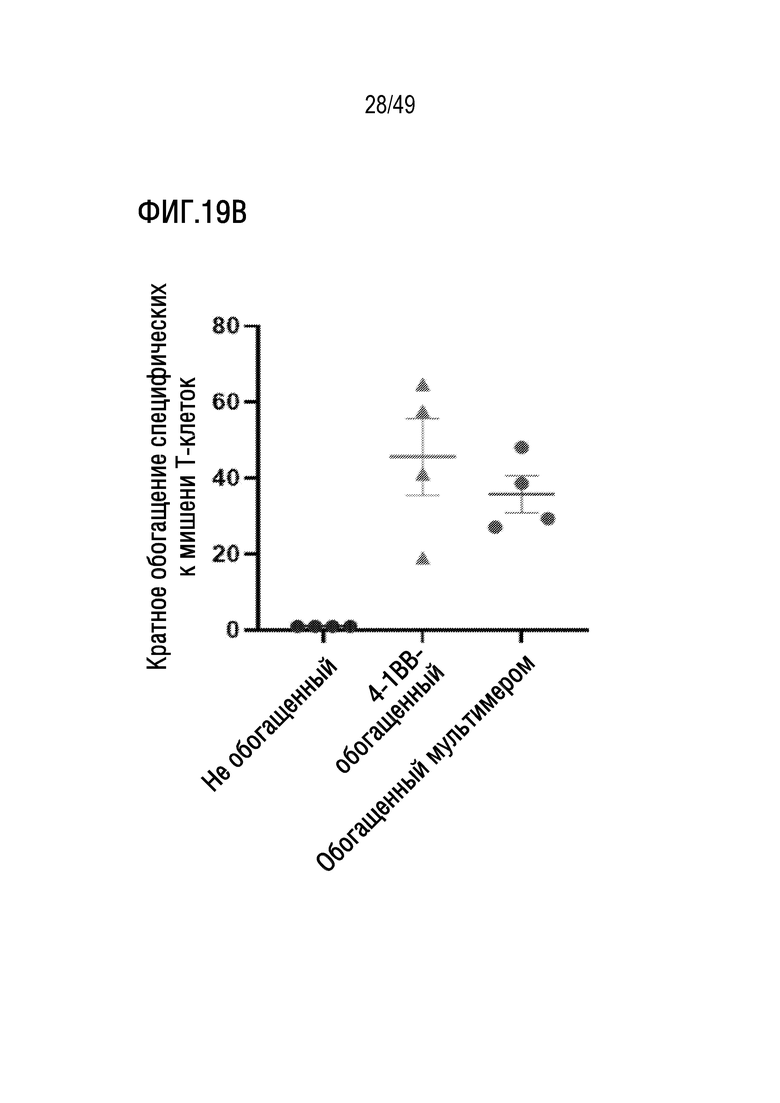
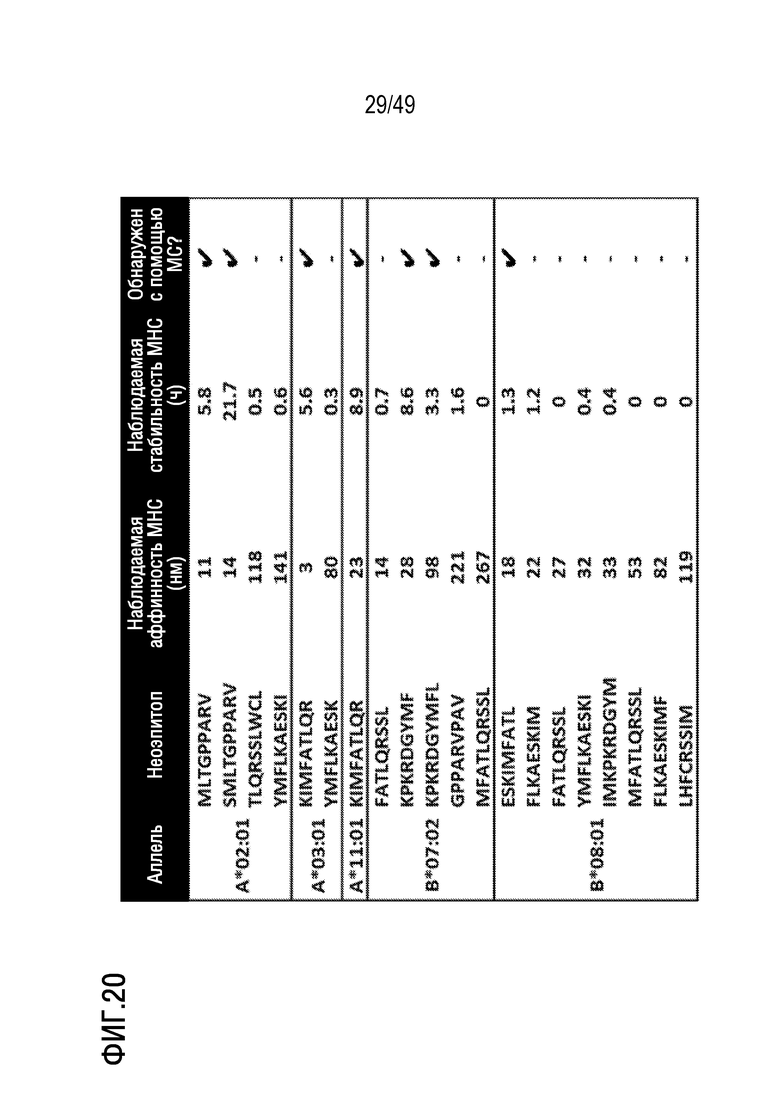
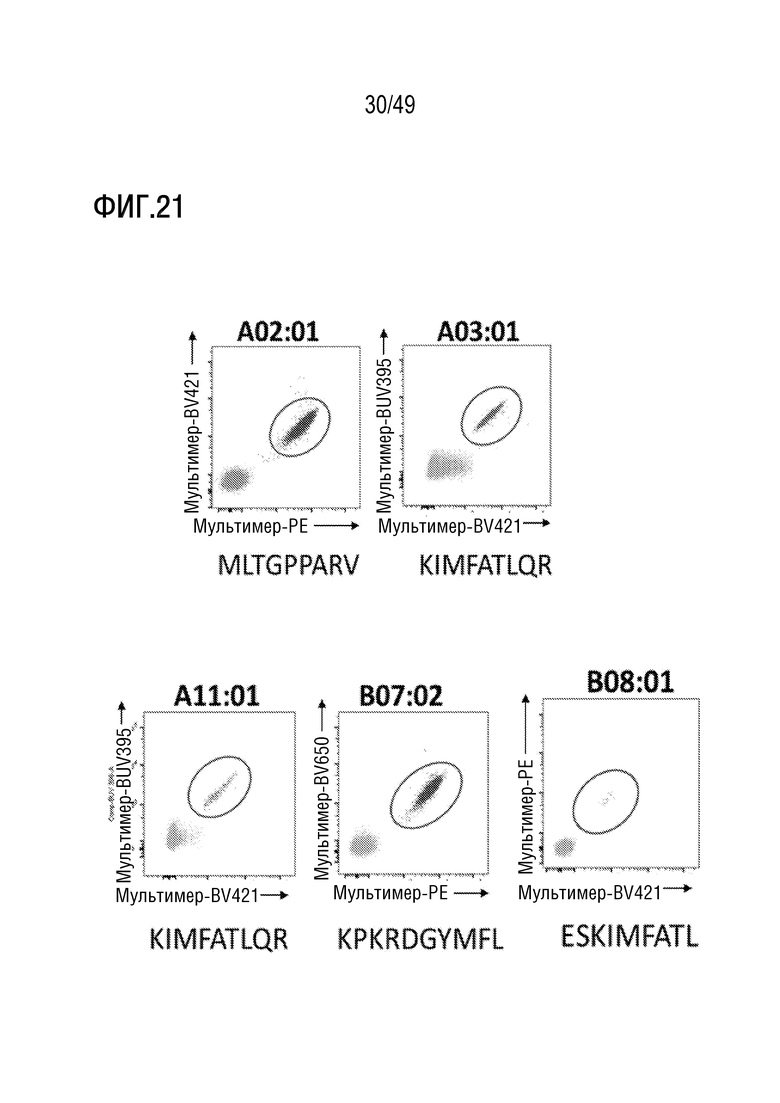
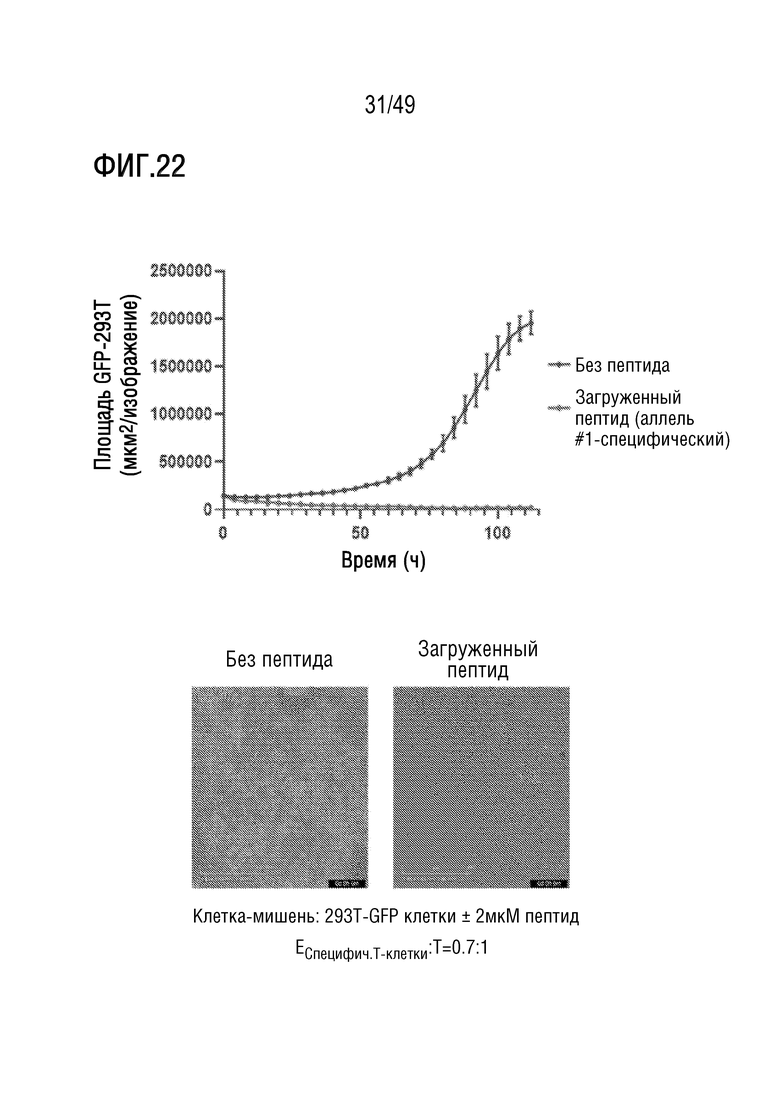
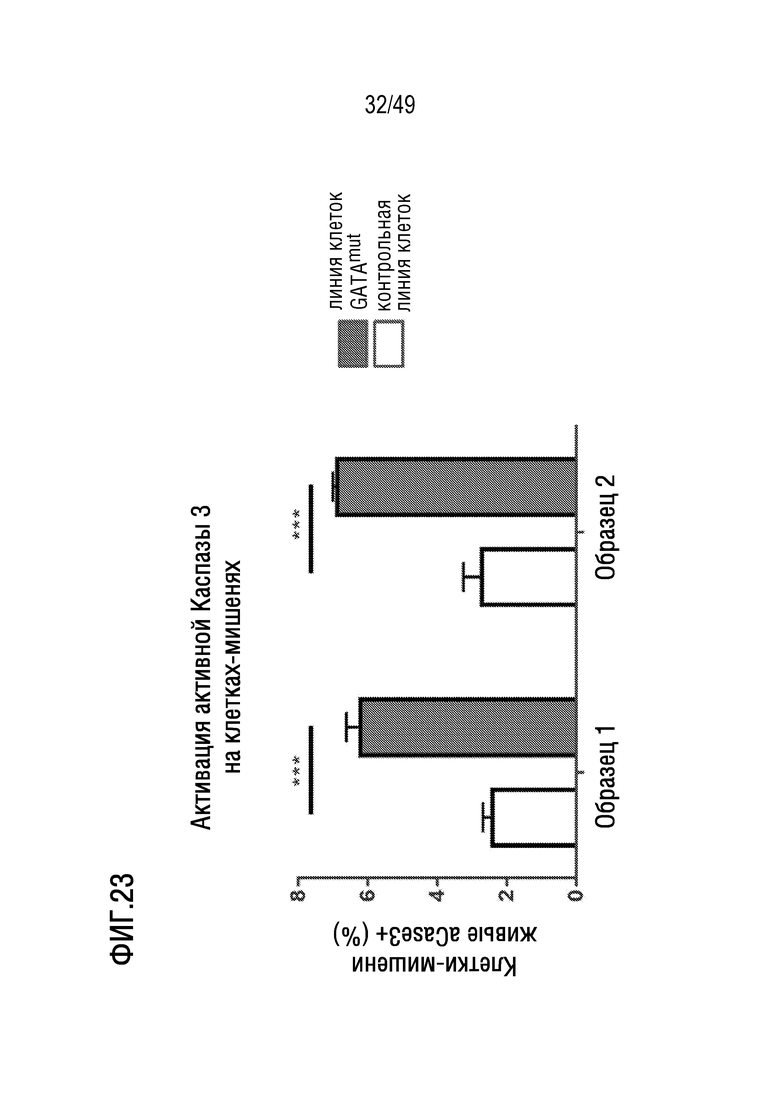
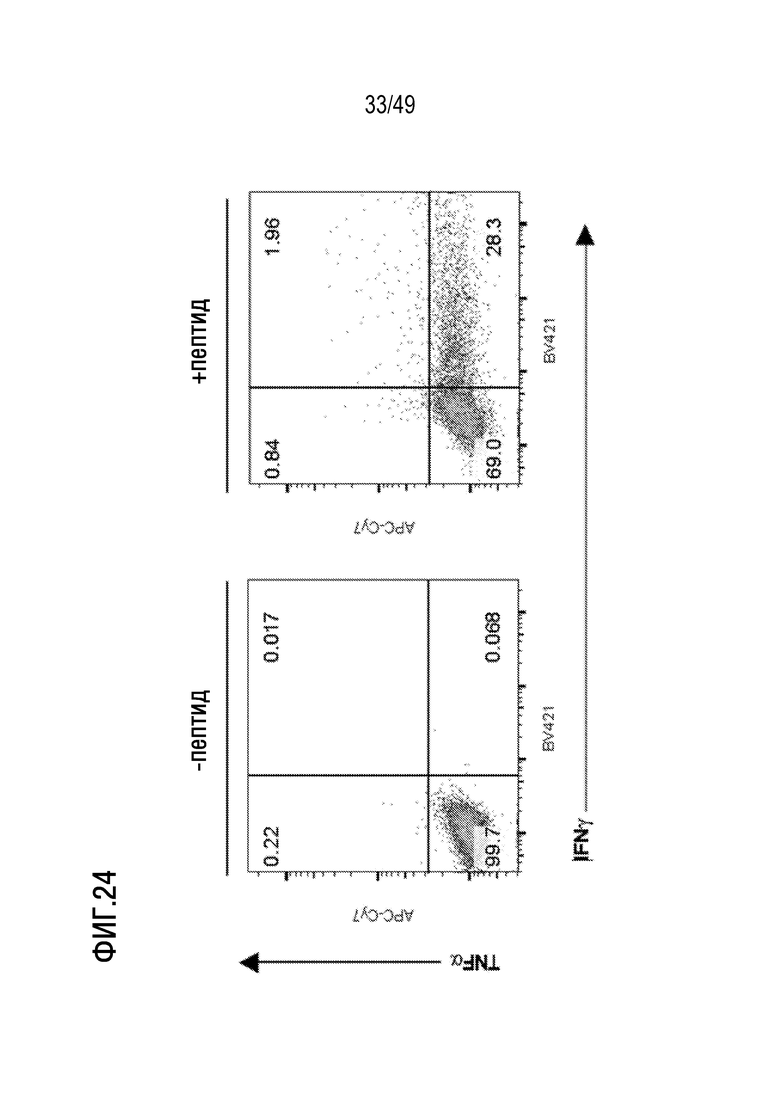
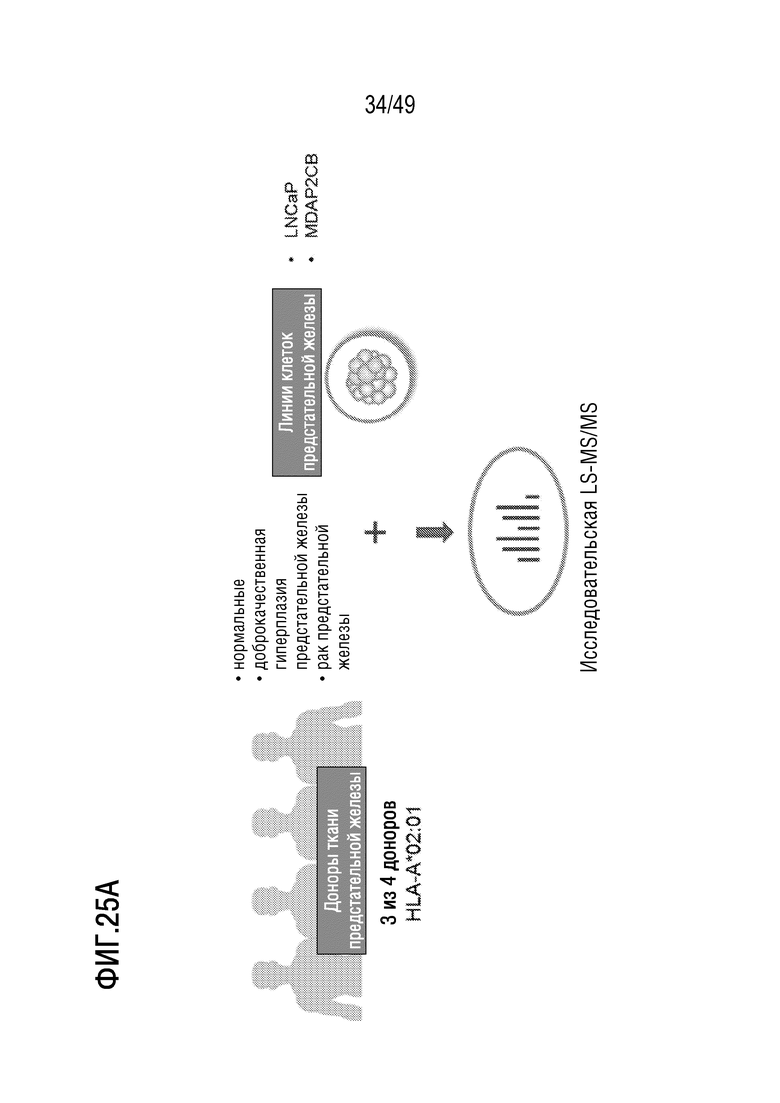

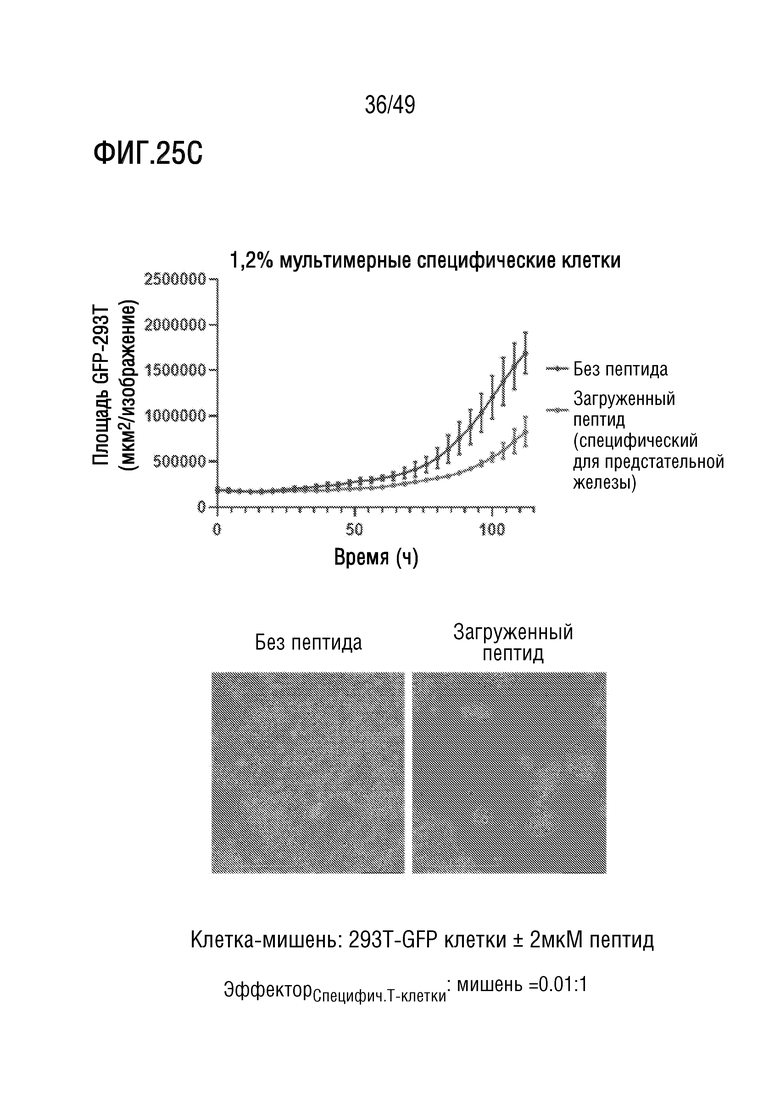
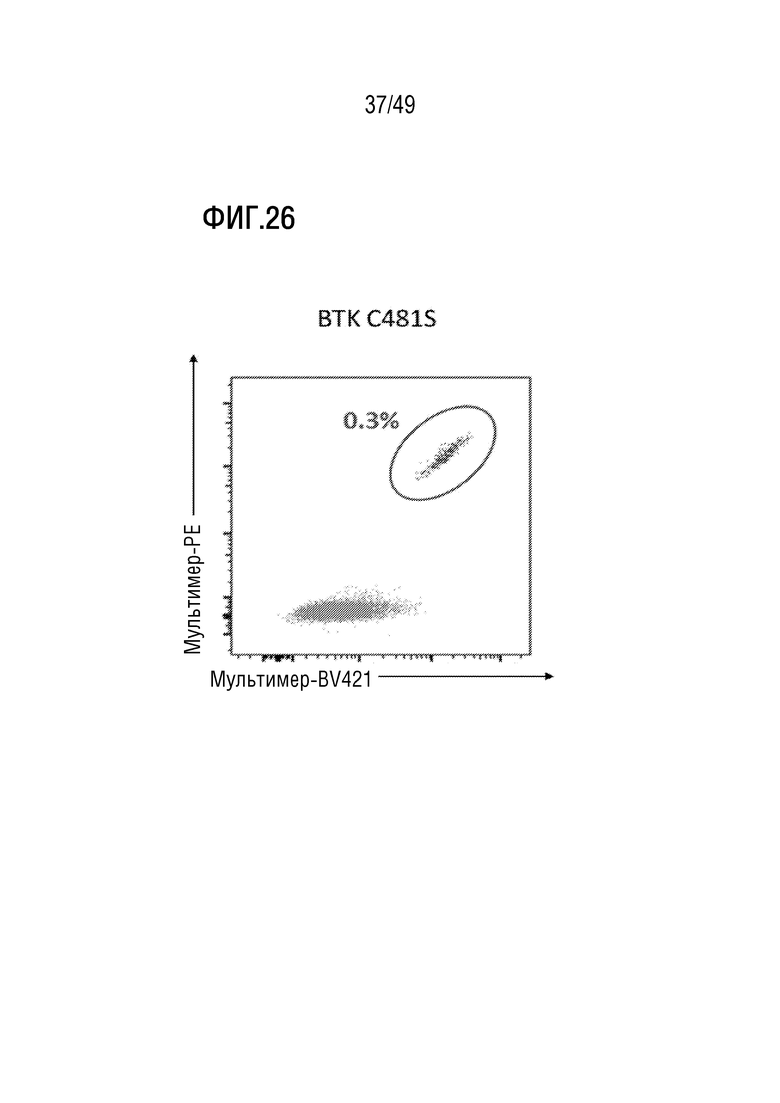
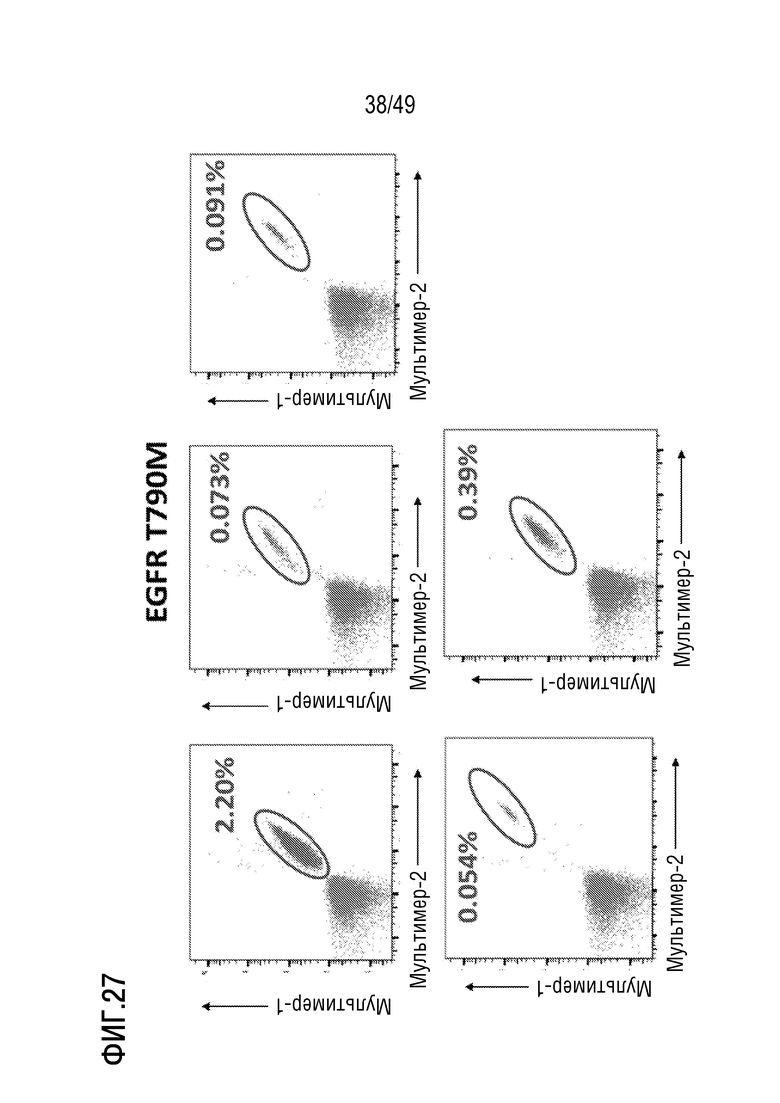
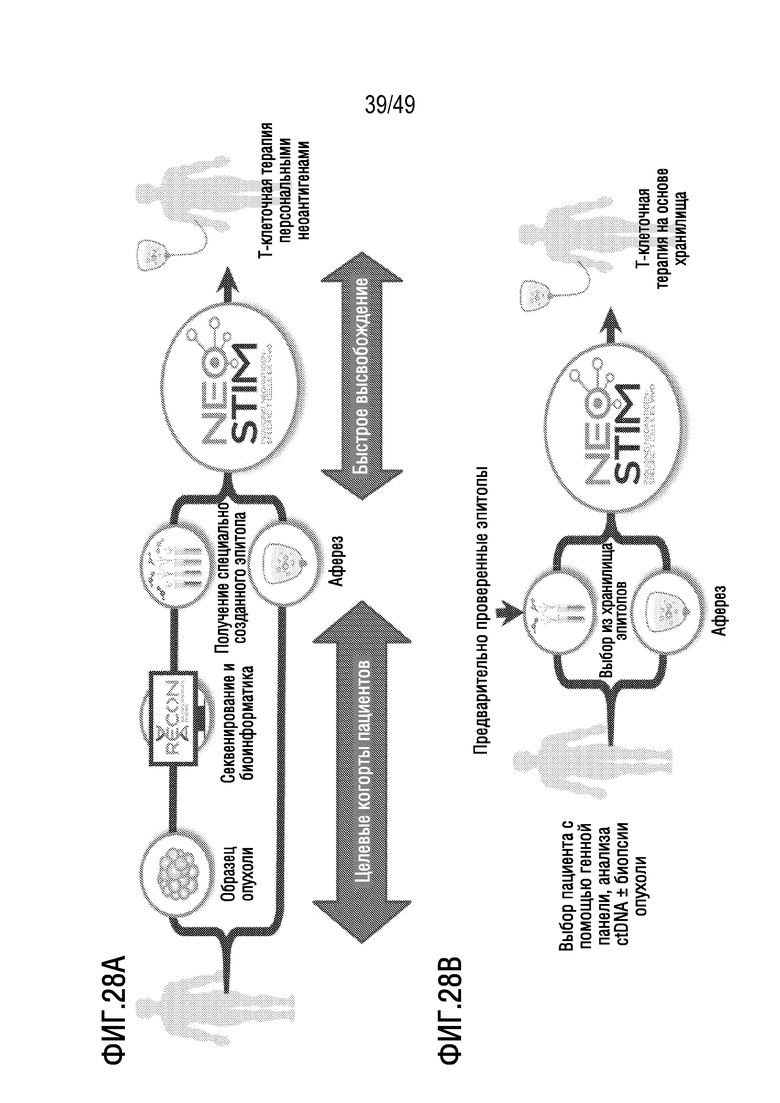
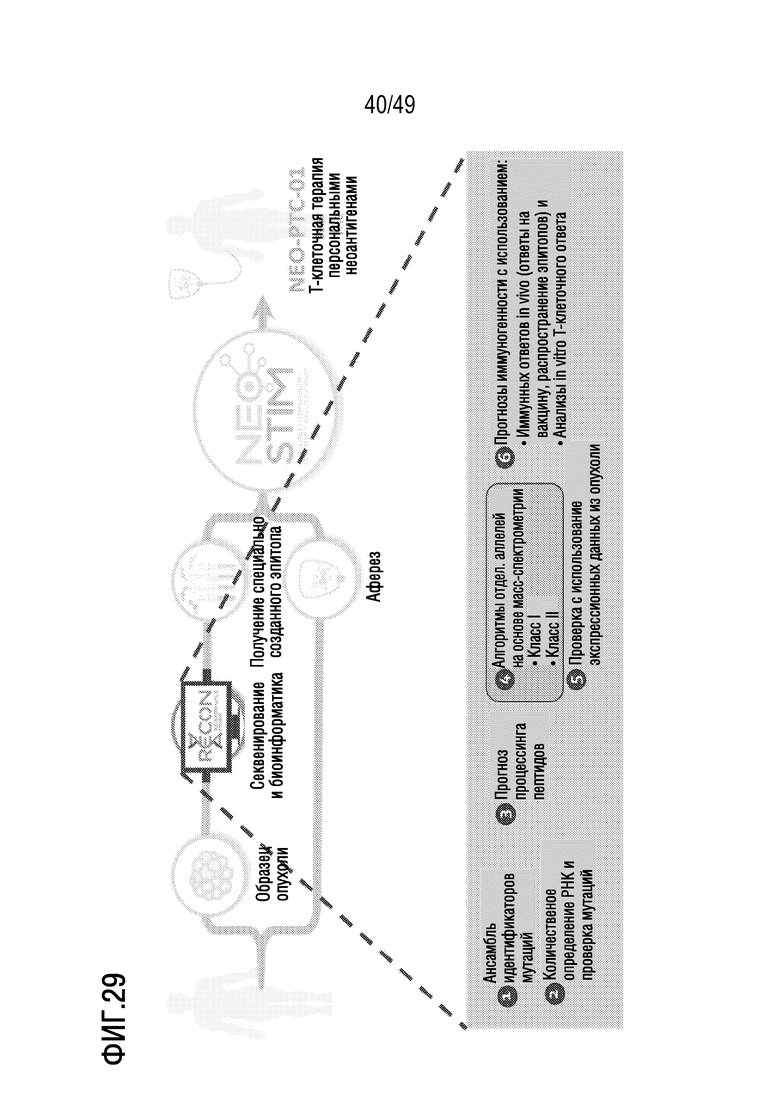
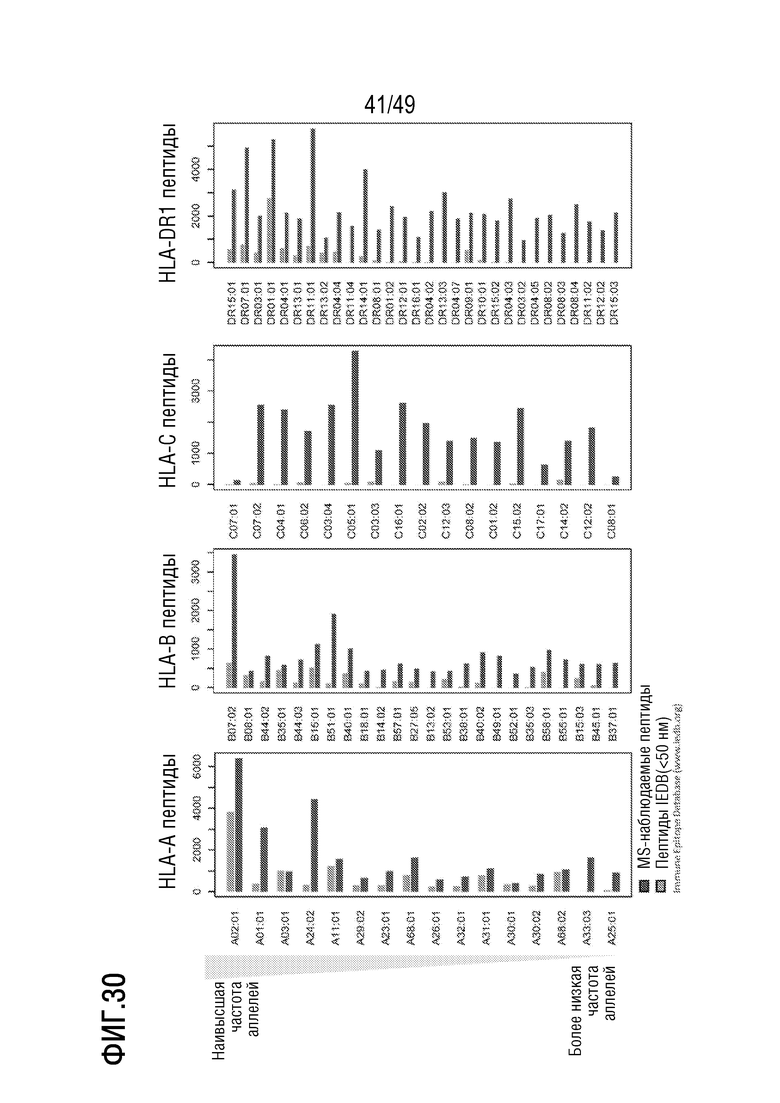
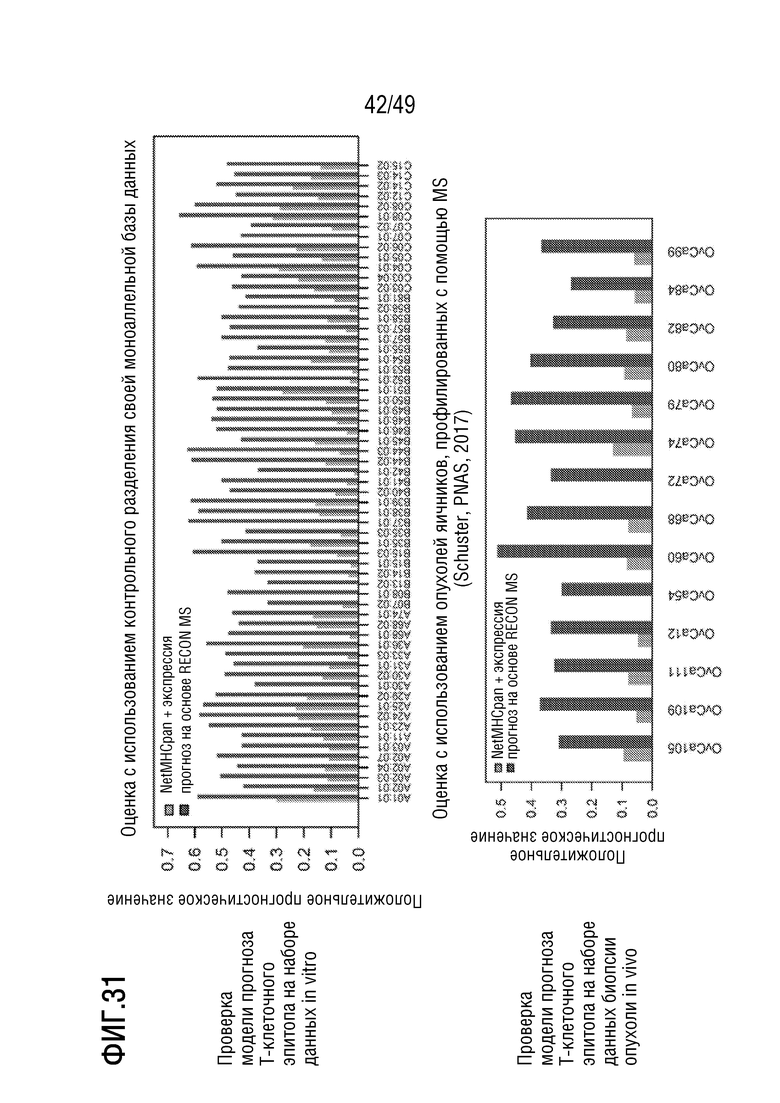
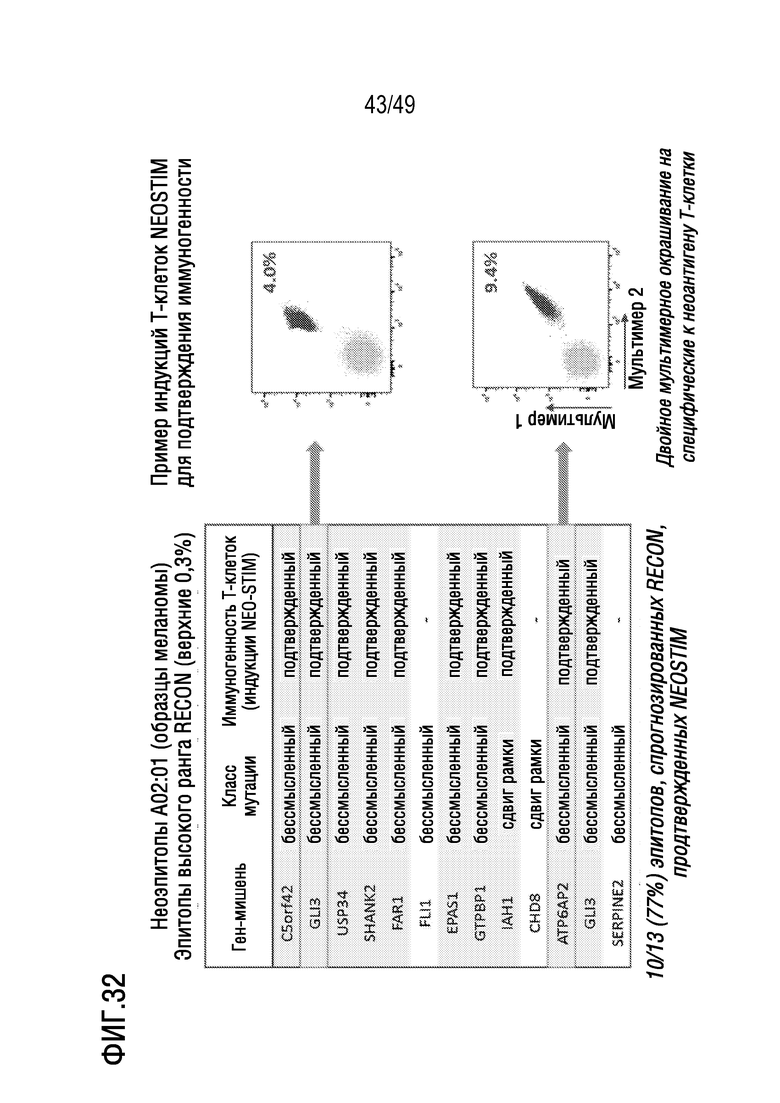
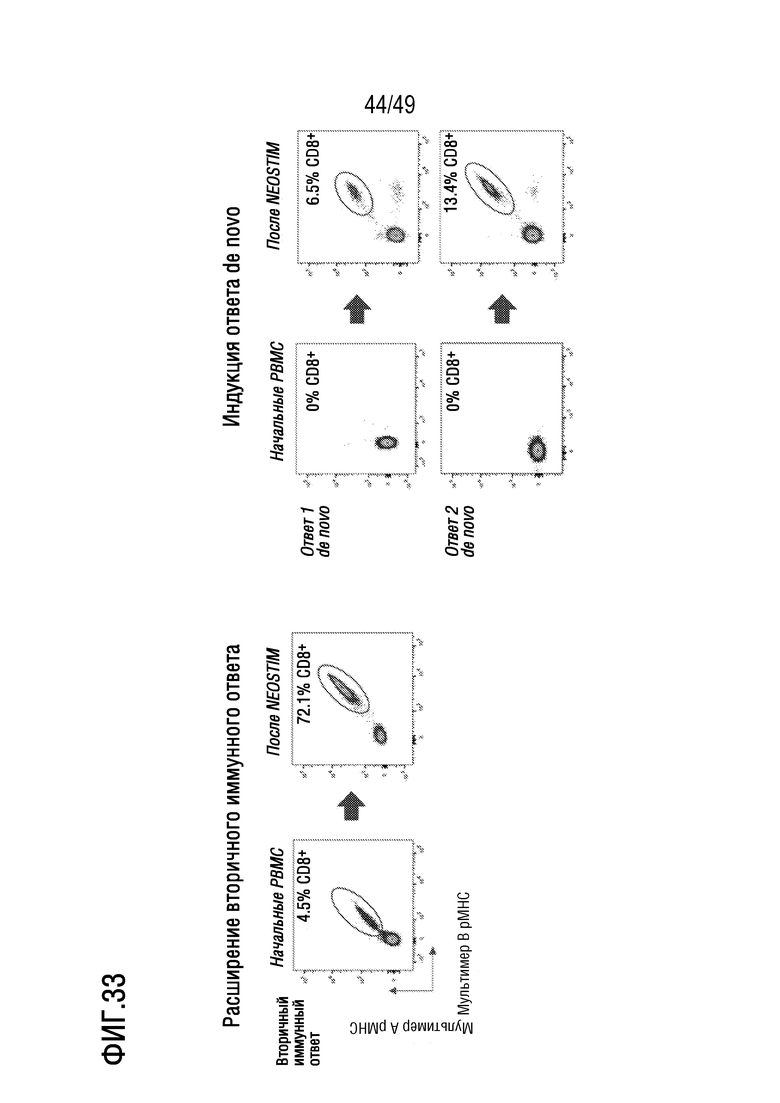
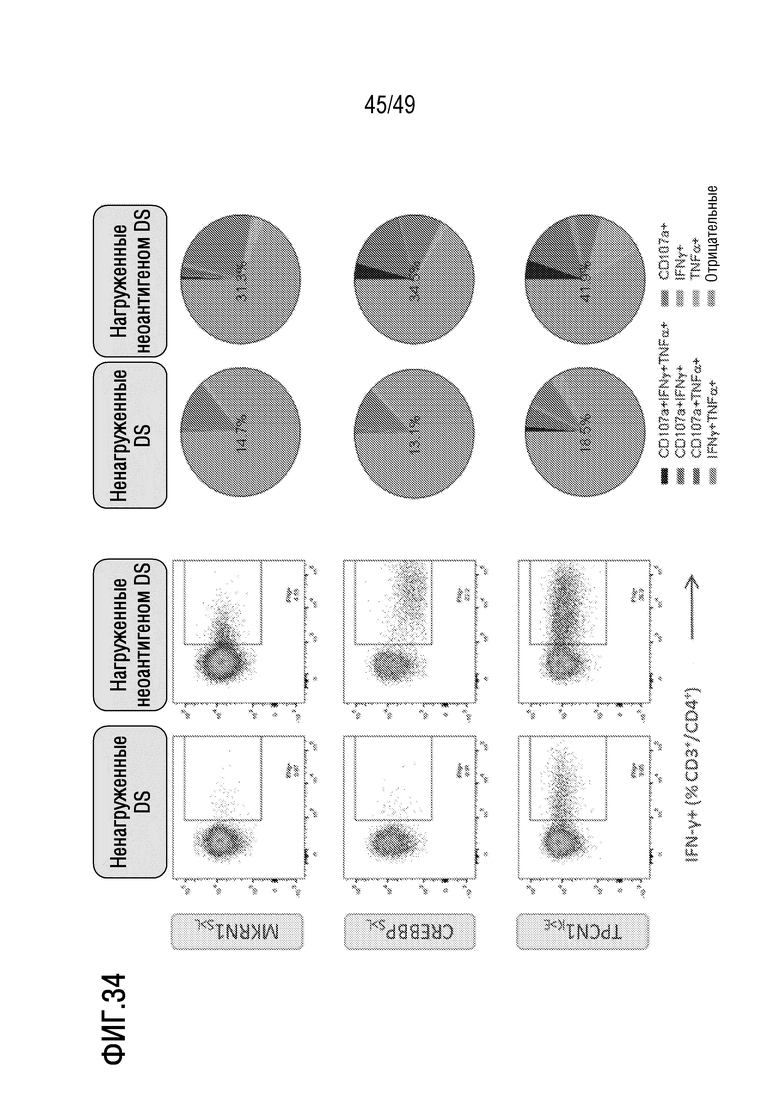
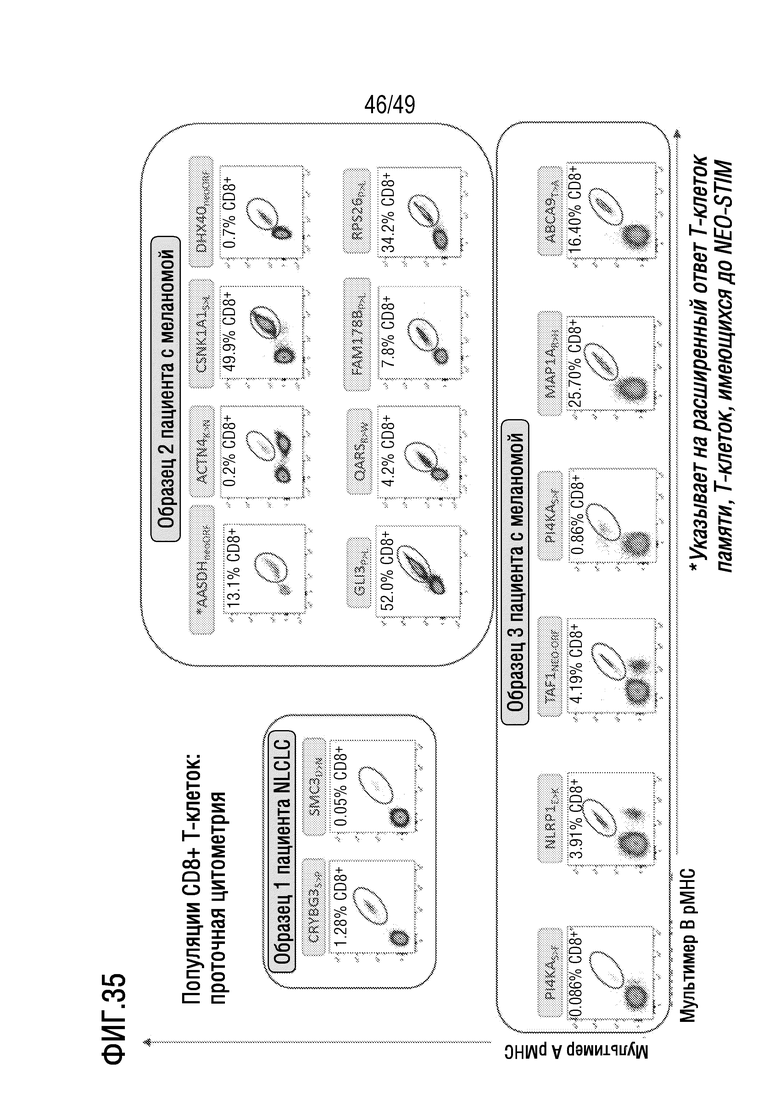
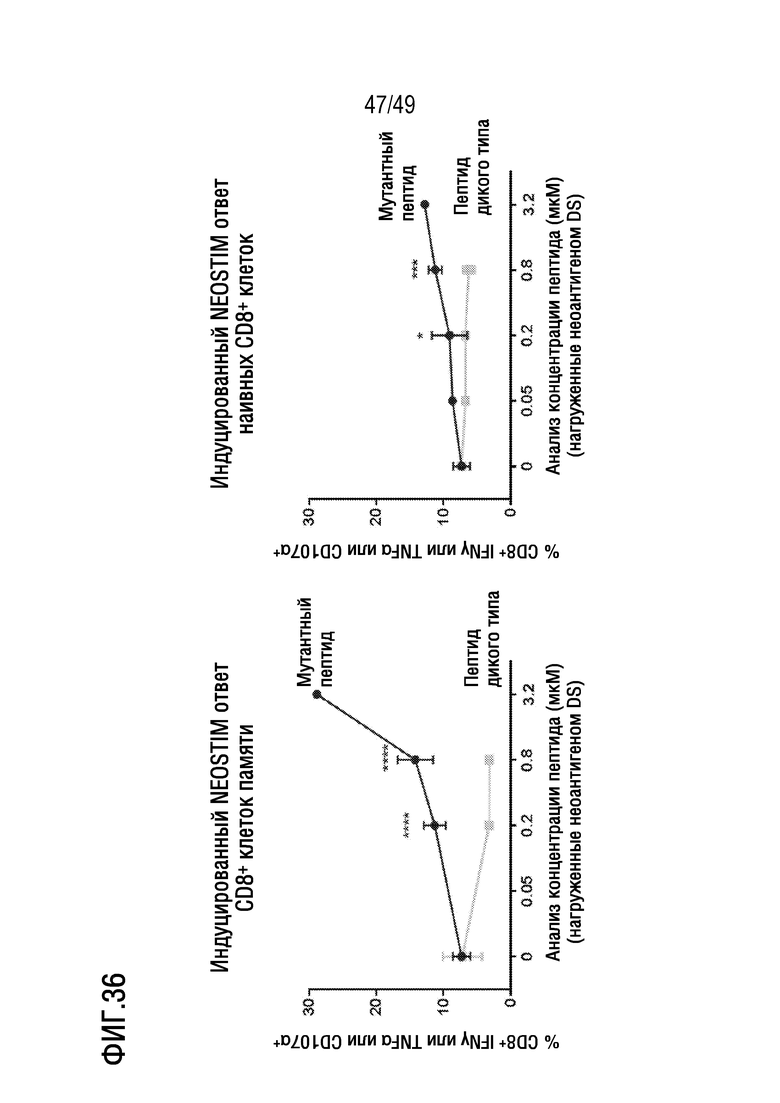
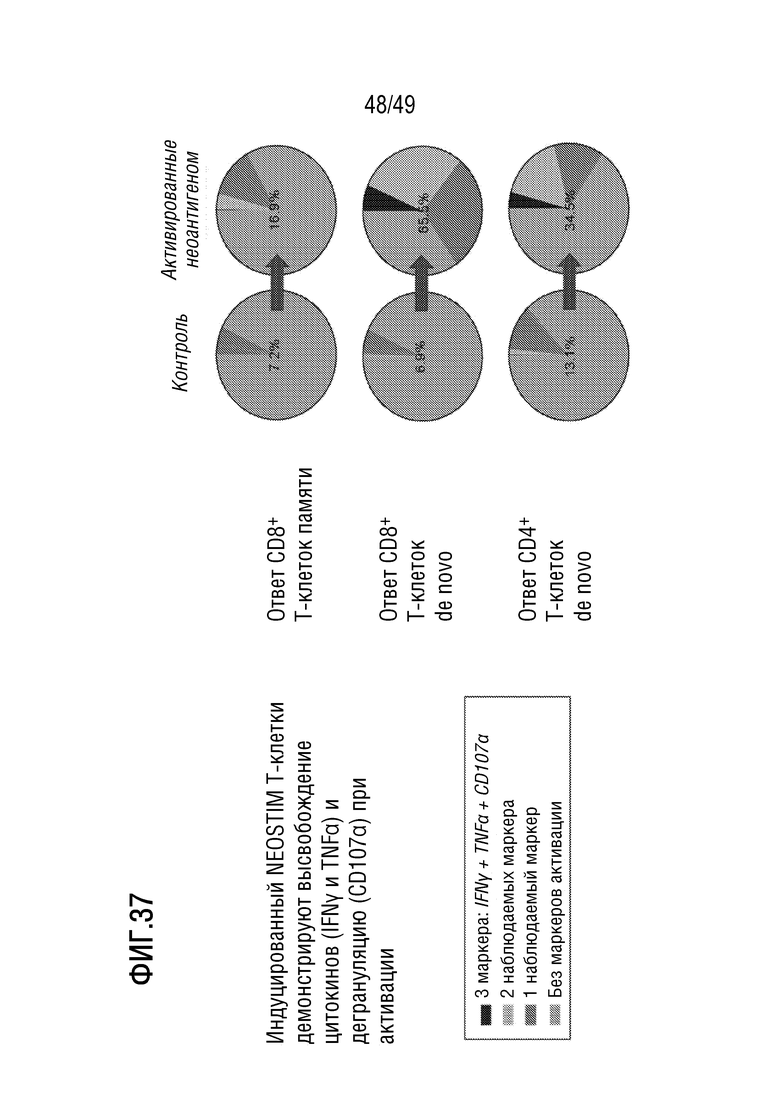
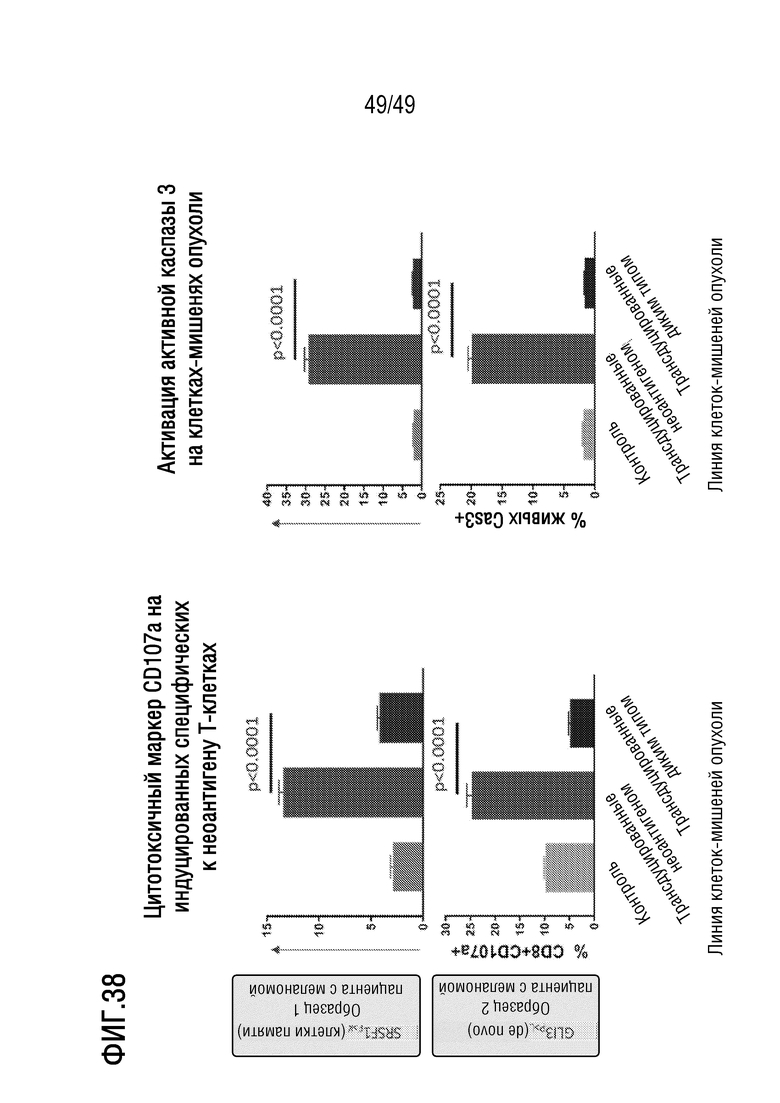
Изобретение относится к области биотехнологии. Предложен cпособ получения популяции клеток ex vivo, содержащей антигенспецифические T-клетки, для применения в лечении рака у человека. Способ включает индукцию T-клеток пептидами, содержащими эпитопную последовательность из библиотеки эпитопных последовательностей. Каждая эпитопная последовательность в библиотеке соответствует белку, кодируемому аллелем HLA, и связывается с белком, кодируемым аллелем HLA субъекта, является иммуногенной согласно иммуногенному анализу, презентируется антигенпрезентирующими клетками согласно масс-спектрометрическому анализу и стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности. 40 з.п. ф-лы, 54 ил., 25 табл., 9 пр.
1. Способ получения популяции клеток ex vivo, содержащей антигенспецифические T-клетки, для применения в лечении рака у человека, предусматривающий:
(a) отбор по меньшей мере одной последовательности эпитопа из заранее проверенной библиотеки из хранилища отобранных последовательностей эпитопов, известных для указанного рака,
где каждая последовательность эпитопа в заранее проверенной библиотеке соответствует белку, кодируемому аллелем HLA; и
(b) приведение клеточной популяции, содержащей T-клетки, в контакт с антиген-презентирующими клетками (APC), содержащими один или несколько пептидов, содержащих по меньшей мере одну выбранную последовательность эпитопа,
где по меньшей мере каждая выбранная последовательность эпитопа удовлетворяет каждому из следующих критериев:
(i) связывается с белком, кодируемым аллелем HLA человека,
(ii) является иммуногенной согласно анализу иммуногенности,
(iii) презентируется APC согласно масс-спектрометрическому анализу и
(iv) стимулирует цитотоксичность T-клеток согласно анализу цитотоксичности; и
где
(a) по меньшей мере одна выбранная последовательность эпитопа содержит мутацию, и способ предусматривает идентификацию раковых клеток человека, кодирующих эпитоп с этой мутацией;
(b) по меньшей мере одна выбранная последовательность эпитопа находится в белке, сверхэкспрессируемом раковыми клетками человека, и способ предусматривает идентификацию раковых клеток человека, которые сверхэкспрессируют белок, содержащий этот эпитоп; или
(c) по меньшей мере одна последовательность эпитопа содержит белок, экспрессируемый клеткой в микросреде опухоли,
где по меньшей мере 0,01% CD8+ T-клеток из популяции клеток обнаруживают иммуногенность по меньшей мере в отношении одной выбранной последовательности эпитопа согласно мультимерному анализу; и процент обнаруженных CD8+ Т-клеток выше процента CD8+ Т-клеток, обнаруженных в контрольном образце,
где контрольный образец содержит Т-клетки, которые приводили в контакт с APC, которые
(i) не содержат пептид, содержащий по меньшей мере одну выбранную последовательность эпитопа,
(ii) содержат пептид, полученный из белка, отличающегося по меньшей мере от одной выбранной последовательности эпитопа, или
(iii) содержат пептид со случайной последовательностью.
2. Способ по п.1, дополнительно предусматривающий введение антигенспецифических T-клеток нуждающемуся в этом человеку.
3. Способ по п.1 или 2, где T-клетка взята у этого человека или является аллогенной T-клеткой.
4. Способ по п.1 или 2, где по меньшей мере одна выбранная последовательность эпитопа содержит мутацию, экспрессируемую клетками этого рака и не экспрессируемую нераковыми клетками.
5. Способ по п.1 или 2, где по меньшей мере одна выбранная последовательность эпитопа находится в белке, сверхэкспрессируемом раковыми клетками; или в белке, экспрессируемом клеткой в микросреде опухоли.
6. Способ по п.1 или 2, где эпитоп, который связывается с белком, кодируемым аллелем HLA человека, связывается с MHC I класса или MHC II класса, кодируемыми аллелем HLA, с аффинностью 500 нМ или менее согласно анализу связывания или согласно программе прогнозирования эпитопа MHC, выполняемой на компьютере.
7. Способ по п.1 или 2, где белок, кодируемый аллелем HLA, представляет собой белок, кодируемый аллелем HLA, выбранным из группы, состоящей из HLA-A01:01, HLA-A02:01, HLA-A03:01, HLA-A11: 01, HLA-A24:01, HLA-A30:01, HLA-A31:01, HLA-A32:01, HLA-A33:01, HLA-A68:01, HLA-B07:02, HLA-B08:01, HLA-B15:01, HLA-B44:03, HLA-C07:01 и HLA-C07:02.
8. Способ по п.1 или 2, где эпитоп, который презентирован антиген-презентирующими клетками (APC) согласно масс-спектрометрическому анализу, обнаруживают посредством масс-спектрометрии после элюирования из APC с точностью определения массы обнаруженного пептида, составляющей менее 15 Да, 10 Да или 5 Да или менее 10000 или 5000 частей на миллион (ppm).
9. Способ по п.1 или 2, где эпитоп, который является иммуногенным согласно анализу иммуногенности, является иммуногенным согласно мультимерному анализу или функциональному анализу.
10. Способ по п.9, где мультимерный анализ представляет собой анализ проточной цитометрии,
где мультимерный анализ предусматривает обнаружение T-клеток, связанных с мультимером пептид-MHC, содержащим по меньшей мере одну выбранную последовательность эпитопа и соответствующий аллель HLA,
где T-клетки были стимулированы APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа.
11. Способ по п.9 или 10, где эпитоп является иммуногенным согласно мультимерному анализу
(i) если обнаружено по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа,
(ii) если обнаруженные T-клетки составляют по меньшей мере 0,1% или 0,01% или 0,005% от проанализированных CD8+ клеток, и
(iii) если процент обнаруженных T-клеток из CD8+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ T-клеток, обнаруженных в контрольном образце.
12. Способ по п.9, где эпитоп является иммуногенным согласно мультимерному анализу, если по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа, обнаружено по меньшей мере в одной из шести стимуляций из стартового образца, содержащего популяцию клеток, содержащих Т-клетки.
13. Способ по п.9, где образец содержит T-клетки, который стимулировали APC, которые
(i) не содержат пептида, содержащего по меньшей мере одну выбранную последовательность эпитопа,
(ii) содержат пептид, полученный из белка, отличающегося по меньшей мере от одной из выбранных последовательностей эпитопа, или
(iii) содержат пептид со случайной последовательностью.
14. Способ по любому из пп.10-12, где T-клетки стимулировали APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа, в течение по меньшей мере 3 дней.
15. Способ по любому из пп.10-12, где число антигенспецифических T-клеток увеличивали по меньшей мере в 5 раз в присутствии APC, содержащих пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа.
16. Способ по п.9, где функциональный анализ представляет собой иммуноанализ.
17. Способ по п.9 или 16, где функциональный анализ предусматривает обнаружение T-клеток с помощью внутриклеточного окрашивания IFNγ или TNFα или экспрессии CD107a и/или CD107b на поверхности клеток,
где T-клетки стимулировали APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа.
18. Способ по п.9, где эпитоп является иммуногенным согласно функциональному анализу
(i) если обнаружено по меньшей мере 10 T-клеток, стимулированных APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа,
(ii) если обнаруженные T-клетки составляют по меньшей мере 0,1% или 0,01% или 0,005% от проанализированных CD8+ или CD4+ клеток, и
(iii) если процент обнаруженных T-клеток из CD8+ или CD4+ T-клеток выше процентного значения обнаруженных T-клеток из CD8+ или CD4+ T-клеток, обнаруженных в контрольном образце.
19. Способ по п.1, где T-клетки, стимулированные на цитотоксичность согласно анализу цитотоксичности, являются T-клетками, которые стимулировали APC, содержащими пептид, который содержит по меньшей мере одну выбранную последовательность эпитопа, который уничтожает клетки, презентирующие эпитоп.
20. Способ по п.19, где число клеток, презентирующих эпитоп, который уничтожается Т-клетками, по меньшей мере в 2 раза выше числа клеток, которые не презентируют эпитоп, который уничтожается Т-клетками;
где число клеток, презентирующих эпитоп, который уничтожается Т-клетками, по меньшей мере в 2 раза выше числа клеток, презентирующих эпитоп, который уничтожается Т-клетками, стимулированными APC, которые
(i) не содержат пептида, содержащего по меньшей мере одну выбранную последовательность эпитопа,
(ii) содержат пептид, полученный из белка, отличающегося по меньшей мере от одной из выбранных последовательностей эпитопа, или
(iii) содержат пептид со случайной последовательностью.
21. Способ по п.20, где число клеток, презентирующих мутантный эпитоп, который уничтожается Т-клетками, по меньшей мере в 2 раза выше числа клеток, презентирующих соответствующий эпитоп дикого типа, который уничтожается Т-клетками.
22. Способ по п.1, где T-клетку получают из образца, содержащего мононуклеарные клетки периферической крови (PBMC), образца для афереза или образца для лейкофереза.
23. Способ по любому из пп.1-22, где по меньшей мере одна выбранная последовательность эпитопа выбрана из одной или нескольких последовательностей эпитопов таблицы 1A-1F, таблицы 2A-2C, таблицы 3, таблицы 4A-4M, таблицы 5, таблицы 6, таблицы 7, таблицы 8, таблицы 12, таблицы 13 и таблицы 14.
24. Способ по п.1, где способ предусматривает увеличение числа T-клеток, которые приводили в контакт с одним или несколькими пептидами, in vitro или ex vivo для получения популяции T-клеток, специфичных по меньшей мере в отношении одной выбранной последовательности эпитопа в комплексе с белком MHC.
25. Способ по п.1, где одна или несколько из по меньшей мере одной выбранной последовательности эпитопа происходит из белка, сверхэкспрессируемого раковой клеткой человека, происходит из тканеспецифического белка, происходит из раково-тестикулярного белка, содержит драйверную мутацию, содержит мутацию устойчивости к лекарственным средствам, содержит опухолеспецифичную мутацию, является вирусным эпитопом, или является минорным эпитопом гистосовместимости.
26. Способ по п.1, где по меньшей мере одна из по меньшей мере одной выбранной последовательности эпитопа происходит из белка RAS и содержит мутацию.
27. Способ по п.26, где по меньшей мере одна из по меньшей мере одной выбранной последовательности эпитоп происходит из белка RAS и содержит мутацию, выбранную из G12R, G12C, G12D и G12V.
28. Способ по п.26, где по меньшей мере одна из по меньшей мере одной выбранной последовательности эпитопа выбрана из группы, состоящей из VVGAVGVGK (SEQ ID NO: 164), TEYKLVVVGAVGVGK (SEQ ID NO: 1427) или TEYKLVVVGAVGVGKSALTI (SEQ ID NO: 1428),
где по меньшей мере один выбранный эпитоп связывается с HLA-A11:01; и GAVGVGKSA (SEQ ID NO: 1278), где по меньшей мере один выбранный эпитоп связывается с HLA-C03:04; и AVGVGKSAL (SEQ ID NO: 1275), где по меньшей мере один выбранный эпитоп связывается с HLA-C01:02.
29. Способ по любому из пп.1-28, где по меньшей мере одну по меньшей мере из одной выбранной последовательности эпитопа получают из тканеспецифического белка, уровень экспрессии которого в ткани-мишени субъекта по меньшей мере в 2 раза выше уровня экспрессии тканеспецифического белка в каждой ткани из множества немишеневых тканей, которые отличаются от ткани-мишени.
30. Способ по п.1, где APC, презентирующие эпитоп, содержат один или несколько пептидов, содержащих по меньшей мере одну выбранную последовательность эпитопа, или нуклеиновую кислоту, которая кодирует один или несколько пептидов, содержащих по меньшей мере одну выбранную последовательность эпитопа.
31. Способ по п.1, где способ предусматривает истощение CD14+ клеток и CD25+ клеток из популяции клеток, содержащей T-клетки, образуя за счет этого популяцию иммунных клеток с истощенными CD14/CD25, содержащую APC и T-клетки.
32. Способ по п.31, где способ дополнительно предусматривает (b) инкубацию популяции иммунных клеток с истощенными CD14/CD25, содержащей APC и T-клетки, в присутствии
(i) лиганда рецептора FMS-подобной тирозинкиназы 3 (FLT3L) и
(ii) (A) полипептида, содержащего по меньшей мере одну выбранную последовательность эпитопа, или
(B) полинуклеотида, кодирующего полипептид; образуя за счет этого популяцию клеток, содержащую стимулированные T-клетки.
33. Способ по п.32, где способ дополнительно предусматривает увеличение числа клеток в популяции, содержащей стимулированные T-клетки, образуя за счет этого увеличенную в числе клеток популяцию, содержащую специфические в отношении опухолевых антигенов T-клетки,
где специфические в отношении опухолевых антигенов T-клетки включают T-клетки, которые специфичны к комплексу, содержащему
(i) по меньшей мере одну выбранную последовательность эпитопа и
(ii) белок MHC, экспрессируемый раковыми клетками или APC человека.
34. Способ по п.33, где увеличение числа клеток предусматривает:
(A) приведение популяции клеток, содержащей стимулированные T-клетки, в контакт со второй популяцией зрелых APC, где вторую популяцию зрелых APC
(i) инкубировали с FLT3L и
(ii) презентировали по меньшей мере в отношении одной выбранной последовательности эпитопа; и
(B) увеличение числа клеток в популяции, содержащей стимулированные T-клетки, в течение второго периода времени, образуя за счет этого увеличенную в числе популяцию T-клеток.
35. Способ по любому из пп.32-34, где стадии инкубирования и увеличения числа клеток проводят менее 28 дней.
36. Способ по любому из пп.22, 30-35, где доля CD8+ специфических в отношении опухолевых антигенов T-клеток к общему числу CD8+ T-клеток в увеличенной в числе клеток популяции, содержащей специфические в отношении опухолевых антигенов T-клетки, по меньшей мере в два раза превышает долю CD8+ специфических в отношении опухолевых антигенов T-клеток к общему числу CD8+ T-клеток в биологическом образце.
37. Способ по любому из пп.22, 30-35, где доля CD4+ специфических в отношении опухолевых антигенов T-клеток к общему числу CD4+ T-клеток в увеличенной в числе клеток популяции, содержащей специфические в отношении опухолевых антигенов T-клетки, по меньшей мере в два раза превышает долю CD4+ специфических в отношении опухолевых антигенов T-клеток к общему числу CD4+ T-клеток в биологическом образце.
38. Способ по любому из пп.22, 30-35, где по меньшей мере 0,1% CD8+ T-клеток в увеличенной в числе клеток популяции, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD8+ T-клетки, полученные из наивных CD8+ T-клеток.
39. Способ по любому из пп.22, 30-35, где по меньшей мере 0,1% CD4+ T-клеток в увеличенной в числе клеток популяции, содержащей специфические в отношении опухолевых антигенов T-клетки, составляют специфические в отношении опухолевых антигенов CD4+ T-клетки, полученные из наивных CD4+ T-клеток.
40. Способ по любому из пп.33-35, где популяцию иммунных клеток с истощенными CD14/CD25, содержащую APC и Т-клетки, инкубируют с FLT3L в течение по меньшей мере 1 дня перед введением в контакт с полипептидом, содержащим по меньшей мере одну выбранную последовательность эпитопа, или полинуклеотидом, кодирующим указанный полипептид.
41. Способ по п.32 или 34, дополнительно предусматривающий сбор увеличенной в числе клеток популяции, содержащей специфические в отношении опухолевых антигенов T-клетки, криоконсервацию увеличенной в числе клеток популяции, содержащей специфические в отношении опухолевых антигенов T-клетки, или получение фармацевтической композиции, содержащей увеличенную в числе клеток популяцию, содержащую специфические в отношении опухолевых антигенов T-клетки.
| US 2018043003 A1, 15.02.2018 | |||
| WO 2018005556 A1, 04.01.2018 | |||
| WO 2017173321 A1, 05.10.2017 | |||
| US 2011274723 A1, 10.11.2011 | |||
| САРАНЦЕВА К.А | |||
| и др | |||
| Иммунология: формирование иммунного ответа как ведущего фактора противоопухолевой защиты; ЗЛОКАЧЕСТВЕННЫЕ ОПУХОЛИ, 2016, Номер 2 (18), с.4-12. |
Авторы
Даты
2025-02-21—Публикация
2020-03-30—Подача