Область техники
Настоящее изобретение относится к новому варианту цитратсинтазы, к микроорганизму, включающему этот вариант, и к способу получения L-аминокислот с использованием этого микроорганизма.
Предшествующий уровень техники
С целью получения L-аминокислот и других полезных веществ были выполнены различные исследования по созданию микроорганизмов с высокоэффективным продуцированием и технологий для процессов ферментации. Например, широко использовались мишень-специфичные подходы, такие как способ увеличения экспрессии гена, кодирующего фермент, вовлеченный в биосинтез L-валина, или способ удаления гена, ненужного для биосинтеза (US 8465962 В2 и KR 10-2153534 В1).
В то же время цитратсинтаза (CS) представляет собой фермент, который продуцирует цитрат посредством катализа конденсации ацетил-КоА и оксалоацетата, которые образуются в процессе гликолиза микроорганизма, и она также является важным ферментом для направления потока углерода в метаболический путь ТСА (трикарбоновые кислоты).
Ранее в литературе (Ooyen et al., Biotechnol. Bioeng., 109(8):2070-2081, 2012) сообщалось о фенотипических изменениях в штаммах, продуцирующих L-лизин, вследствие делеции гена gltA, кодирующего цитратсинтазу. Однако недостатками этих штаммов с делецией гена gltA является не только то, что ингибируется их рост, но также значительно снижается их уровень потребления сахара, что приводит к низкому продуцированию лизина в единицу времени. Соответственно, все еще необходимы дальнейшие исследования, учитывающие как эффективное увеличение продуктивности в отношении L-аминокислот, так и рост штаммов.
Описание изобрения
Техническая задача
В результате интенсивных усилий по получению L-аминокислот с высоким выходом авторы настоящего изобретения завершили эту заявку, подтвердив, что новый вариант цитратсинтазы повышает способность продуцировать L-амиинокислоты.
Техническое решение
Целью настоящего изобретения является предложение варианта цитратсинтазы, в котором лизин, который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином.
Другая цель настоящего изобретения заключается в предложении полинуклеотида, кодирующего вариант по настоящему изобретению.
Еще одна цель настоящего изобретения заключается в предложении микроорганизма рода Corynebacterium, включающего вариант по настоящему изобретению или полинуклеотид, кодирующий этот вариант.
Еще одна цель настоящего изобретения заключается в предложении способа получения L-аминокислот с применением микроорганизма по настоящему изобретению.
Еще одна цель настоящего изобретения заключается в предложении композиции для получения L-аминокислот, включающей микроорганизм по настоящему изобретению; среду, на которой выращивают микроорганизм по настоящему изобретению; или их комбинацию.
Полезные эффекты
При использовании варианта цитратсинтазы по настоящему изобретению L-аминокислоты могут быть получены с высоким выходом.
Подробное описание предпочтительных воплощений
Далее настоящее изобретение будет описано подробно. В то же время, каждое описание и воплощение, раскрытые в данном документе, могут быть применены к другим описаниям и воплощениям в отношении общих признаков. То есть все комбинации различных элементов, раскрытых в данном документе, входят в объем настоящего изобретения. Кроме того, объем настоящего изобретения не ограничен конкретным описанием, приведенным ниже. Более того, в настоящем описании изобретения были сделаны ссылки на и процитированы ряд статей и патентных документов. Содержание цитируемых статей и патентных документов во всей их полноте включено в данное описание изобретения посредством ссылки, и уровень области техники, к которой относится настоящее изобретение, и содержание настоящего изобретения будут описаны более определенно.
В одном аспекте настоящего изобретения предлагается вариант цитратсинтазы, в котором лизин, который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином.
Вариант по настоящему изобретению может представлять собой вариант, в котором аминокислота, соответствующая положению 415 на основе аминокислотной последовательности SEQ ID NO: 1 в аминокислотной последовательности, представленной SEQ ID NO: 1, представляет собой гистидин, и который имеет гомологию или идентичность по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7% или 99,9% или более с аминокислотной последовательностью, представленной SEQ ID NO: 1. Например, вариант по настоящему изобретению может представлять собой вариант, в котором аминокислота, соответствующая положению 415 на основе аминокислотной последовательности SEQ ID NO: 1 в аминокислотной последовательности, представленной SEQ ID NO: 1, представляет собой гистидин, и может иметь или включать аминокислотную последовательность, имеющую гомологию или идентичность по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7% или 99,9% или более с аминокислотной последовательностью, представленной SEQ ID NO: 1, или может состоять или по существу состоять из данной аминокислотной последовательности. Кроме того, очевидно, что любой вариант, имеющий аминокислотную последовательность, в которой часть последовательности удалена, модифицирована, замещена, консервативно замещена или добавлена, также может входить в объем настоящего изобретения, пока аминокислотная последовательность имеет такую гомологию или идентичность и проявляет эффективность, соответствующую таковой у варианта по настоящему изобретению.
Например, это может быть случай, где N-конец, С-конец и/или внутренняя часть аминокислотной последовательности добавлен(а) или удален(а), с последовательностью, которая не изменяет функцию варианта по настоящему изобретению, естественной мутацией, молчащей мутацией или консервативной заменой.
При использовании в данном документе термин "консервативная замена" относится к замене аминокислоты другой аминокислотой, имеющей аналогичные структурные и/или химические свойства. Такая аминокислотная замена, в общем случае, может иметь место на основе подобия полярности, заряда, растворимости, гидрофобности, гидрофильности и/или амфипатической природы остатка. Обычно консервативные замены могут оказывать незначительное влияние или вообще не оказывать никакого влияния на активность белка или полипептида.
При использовании в данном документе термин "вариант" относится к полипептиду, имеющему одну или более аминокислот, отличных от аминокислотной последовательности этого варианта до мутации посредством консервативных замен и/или модификаций таким образом, что функции и свойства полипептида сохраняются. Такие варианты, в общем случае, могут быть идентифицированы путем модификации одной или более указанных выше аминокислотных последовательностей полипептида и оценки свойств модифицированного полипептида. То есть способность вариантов может быть усилена, не изменена или снижена по сравнению с полипептидом до мутации. Кроме того, некоторые варианты могут включать те, в которых одна или несколько областей, таких как N-концевая лидерная последовательность или трансмембранный домен, удалены. Кроме того, другие варианты могут включать те, в которых удалена область с N- и/или С-конца зрелого белка. Термин "вариант" можно использовать взаимозаменяемо с такими терминами, как модификация, модифицированный полипептид, модифицированный белок, мутант, мутеин, дивергент и т.д., если эти термины используются для обозначения мутации. Для цели настоящего изобретения вариант может представлять собой вариант, в котором лизин (Lys, K), который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином (His, Н).
Кроме того, варианты могут также включать удаление или добавление аминокислот, которые оказывают минимальное влияние на свойства и вторичную структуру полипептида. Например, варианты могут быть конъюгированы с сигнальной (или лидерной) последовательностью на N-конце, вовлеченной в транслокацию белков, ко-трансляционно или посттрансляционно. Кроме того, варианты также могут быть конъюгированы с другой последовательностью или линкером для идентификации, очистки или синтеза полипептида.
При использовании в данном документе термин "гомология" или "идентичность" относится к степени соответствия двух взятых аминокислотных последовательностей или нуклеотидных последовательностей и может быть выражен в процентах. Термины "гомология" и "идентичность" часто могут быть использованы взаимозаменяемо друг с другом.
Гомология последовательностей или идентичность консервативных полинуклеотидов или полипептидов может быть определена с помощью стандартных алгоритмов выравнивания и может использоваться со штрафом за пробелы по умолчанию, установленным используемой программой. По существу, обычно ожидается, что гомологичные или идентичные последовательности будут гибридизоваться со всеми последовательностями или с частью их в умеренных или очень жестких условиях. Очевидно, что в гибризацию полинуклеотидов также включена гибридизация с полинуклеотидами, содержащими обычный кодон или вырожденные кодоны в гибридизуемых полинуклеотидах.
Имеют ли какие-либо две полинуклеотидные или полипептидные последовательности гомологию, сходство или идентичность, можно, например, определить посредством известного компьютерного алгоритма, такого как программа "FASTA" (Pearson et al., (1988) [Proc. Natl. Acad. Sci. USA 85]: 2444) с использованием параметров по умолчанию. Альтернативно, это может быть определено посредством алгоритма Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443-453), который выполняют с использованием программы Needleman из пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000, Trends Genet. 16: 276-277) (предпочтительно версия 5.0.0 или последующие версии) (пакет программ GCG (Devereux, J., et al., Nucleic Acids Research 12: 387 (1984)), BLASTP, BLASTN, FASTA (Atschul, [S.] [F.,] [et al/, J. MOLEC BIOL 215]: 403 (1990); Guide to Huge Computers, Мартин Дж. Бишоп, [ED.,] Academic Press, San Diego, 1994, и [CARILLO et al.] (1988) SIAM J Applied Math 48: 1073). Например, гомология, сходство или идентичность могут быть определены с помощью BLAST или ClustalW Национального центра биотехнологической информации (NCBI).
Гомология, сходство или идентичность полинуклеотидов или полипептидов могут быть определены, например, путем сравнения информации о последовательностях с использованием, например, компьютерной программы GAP, такой как Needleman et al. (1970), J. Mol Biol. 48: 443, как раскрыто в Smith and Waterman, Adv. Appl. Math (1981) 2:482. В кратком изложении, программа GAP определяет гомологию, сходство или идентичность в виде значения, полученного путем деления количества аналогично выровненных символов (т.е. нуклеотидов или аминокислот) на общее количество символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать (1) двоичную матрицу сравнения (содержащую значение 1 для идентичностей и 0 для неидентичностей) и взвешенную матрицу сравнения Gribskov et al. (1986), Nucl. Acids Res. 14:6745, как описано в Schwartz and Dayhoff, eds., Atlas of Protein Sequence and Structure, Национального фонда биомедицинских исследований, стр. 353-358 (1979) (или матрицу замещения EDNAFULL (EMBOSS version of NCBI NUC4.4)); (2) штраф 3,0 за каждый пробел и дополнительный штраф 0,10 за каждый символ в каждом пробеле (или штраф за открытие пробела в размере 10 и штраф за продление пробела в размере 0,5); и (3) отсутствие штрафа за конечные пробелы.
При использовании в данном документе термин "соответствующий" относится к аминокислотному остатку в положении, указанном в пептиде, или аминокислотному остатку, который подобен, идентичен или гомологичен остатку, указанному в пептиде. Идентификация аминокислоты в соответствующем положении может представлять собой определение конкретной аминокислоты в последовательности, которая относится к конкретной последовательности. При использовании в данном документе термин "соответствующая область" обычно относится к аналогичному или соответствующему положению в родственном белке или в эталонном белке.
Например, любую аминокислотную последовательность выравнивают с SEQ ID NO: 1, и на основе этого выравнивания каждый аминокислотный остаток аминокислотной последовательности может быть пронумерован со ссылкой на пронумерованное положение аминокислотного остатка, соответствующего аминокислотному остатку в SEQ ID NO: 1. Например, алгоритм выравнивания последовательностей, такой как описано в данном документе, может идентифицировать положение аминокислоты или положение, где имеют место модификации, такие как замены, вставки или делеции, по сравнению с запрашиваемой последовательностью (также называемой "эталонной последовательностью").
Пример выравнивания может быть определен с помощью алгоритма Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443-453), который выполняют с использованием программы Needleman из пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000, Trends Genet. 16: 276-277) и т.д., но без ограничения этим, и могут быть соответствующим образом использованы программы выравнивания последовательностей, такие как алгоритмы попарного сравнения последовательностей и т.д., известные в данной области техники.
При использовании в данном документе термин "цитратсинтаза" относится к ферменту, который продуцирует цитрат посредством катализа конденсации ацетил-КоА и оксалоацетата, которые образуются в процессе гликолиза микроорганизма. Кроме того, цитратсинтаза катализирует реакцию конденсации двухуглеродного ацетатного остатка от ацетил-КоА и молекулы 4-углеродного оксалоацетата с образованием 6-углеродного цитрата. Термин "цитратсинтаза" можно использовать взаимозаменяемо с "ферментом для синтеза цитрата", "CS", "GltA-белок" или "GltA". В настоящем раскрытии последовательность GltA может быть получена из известной базы данных GenBank NCBI. Кроме того, GltA может представлять собой полипептид, имеющий цитратсинтазную активность, кодируемый геном gltA, но без ограничения этим.
Вариант по настоящему изобретению может иметь активность увеличения способности продуцирования L-аминокислот по сравнению с полипептидом дикого типа.
Вариант по настоящему изобретению может иметь идентичность последовательности 80% или более с аминокислотной последовательностью SEQ ID NO: 1.
Кроме того, вариант по настоящему изобретению может включать полипептид, представленный аминокислотной последовательностью SEQ ID NO: 3. Аминокислотная последовательность SEQ ID NO: 3 может представлять собой аминокислотную последовательность, в которой лизин, соответствующий положению 415 в аминокислотной последовательности от положения 362 до положения 415 от N-конца аминокислотной последовательности SEQ ID NO: 1, заменен гистидином.
Вариант по настоящему изобретению может включать аминокислотную последовательность общей формулы 1, приведенной ниже:
X1N HGGDATX2FMN KVKNKEDGVR LMGFGHRVYK NYDPRAAIVK ETAHEILEHL GGDDLLDLAI KLEEIALADD X3FISRKLYPN VDFYTGLIYR AMGFPTDFFT VLFAIGRLPG WIAHYREQLG AAGNH (SEQ ID NO: 51),
где в общей формуле 1
X1 представляет собой аспарагин или серии;
Х2 представляет собой аланин или глутаминовую кислоту; и
Х3 представляет собой тирозин или цистеин.
Вариант по настоящему изобретения может иметь идентичность последовательности 90% или более с аминокислотной последовательностью SEQ ID NO: 8, 10 или 12. Кроме того, вариант по настоящему изобретению может включать, состоять или по существу состоять из аминокислотной последовательности, имеющей идентичность последовательности 90% или более с аминокислотной последовательностью SEQ ID NO: 8, 10 или 12. Например вариант по настоящему изобретению может иметь идентичность последовательности 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, 99,5%, или 99,7% с аминокислотной последовательностью SEQ ID NO: 8, 10 или 12, может включать аминокислотную последовательность, имеющую указанную идентичность последовательности, или может состоять или по существу состоять из аминокислотной последовательности, имеющей указанную идентичность последовательности.
В другом аспекте настоящего изобретения предлагается полинуклеотид, кодирующий вариант по настоящему изобретению.
При использовании в данном документе термин "полинуклеотид", который представляет собой полимер из нуклеотидов, состоящий из нуклеотидных мономеров, соединенных в длинную цепь посредством ковалентной связи, представляет собой нить ДНК или РНК, имеющую, по меньшей мере, определенную длину. Более конкретно, он может относиться к фрагменту полинуклеотида, кодирующему этот вариант.
В полинуклеотиде по настоящему изобретению нуклеотиды, соответствующие положениям с 1243 по 1245 на основе последовательности нуклеиновой кислоты SEQ ID NO: 2, представляют собой САС, и может быть включен любой полинуклеотид, представленный последовательностью нуклеиновой кислоты, имеющей гомологию или идентичность по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7%, или 99,9% или более и менее 100%, с последовательностью нуклеиновой кислоты, представленной SEQ ID NO: 2. Кроме того, очевидно, что любой полинуклеотид, представленный нуклеинов о кислотной последовательностью, в которой часть последовательности удалена, модифицирована, замещена, консервативно замещена или добавлена, также может входить в объем настоящего изобретения, до тех пор пока последовательность имеет такую гомологию или идентичность и кодирует полипептид или белок, проявляющий эффективность, соответствующую эффективности варианта по настоящему изобретению.
Полинуклеотид по настоящему изобретению может подвергаться различным модификациям в кодирующей области в пределах объема, который не изменяет аминокислотную последовательность варианта по настоящему изобретению, вследствие вырождения кодонов или с учетом кодонов, предпочтительных в организме, в котором должен экспрессироваться вариант по настоящему изобретению. Здесь, в последовательности, имеющей гомологию или идентичность, кодон, кодирующий аминокислоту, соответствующую положению 415 SEQ ID NO: 1, может представлять собой один из кодонов, кодирующих гистидин.
Кроме того, полинуклеотид по настоящему изобретению может включать зонд, который может быть получен из известной последовательности гена, например любой последовательности, которая может гибридизоваться с последовательностью, комплементарной всей или части полинуклеотид ной последовательности по настоящему изобретению в жестких условиях без ограничения. "Жесткие условия" относятся к условиям, в которых обеспечивается специфическая гибридизация между полинуклеотидами. Такие условия конкретно описаны в литературе (J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York, 9.50-9.51, 11.7-11.8). Например жесткие условия могут включать условия, в которых полинуклеотиды, имеющие высокую гомологию или идентичность 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более, или 99% или более, гибридизуются друг с другом, и полинуклеотиды, имеющие гомологию или идентичность ниже указанных выше гомологий или идентичностей, не гибридизуются друг с другом, или условия промывки саузерн-гибридизации, то есть промывка один раз, конкретно два или три раза при концентрации соли и температуре, соответствующих 60°С, 1 × SSC (раствор хлорида натрия и цитрат натрия), 0,1% SDS (додецилсульфат натрия), в частности 60°С, 0,1 × SSC, 0,1% SDS и, более конкретно 68°С, 0,1 × SSC, 0,1% SDS.
Гибридизация требует, чтобы две нуклеиновые кислоты содержали комплементарные последовательности, хотя возможны ошибки спаривания между основаниями, в зависимости от жесткости гибридизации. Термин "комплементарный" используют для описания взаимоотношения между нуклеотидными основаниями, которые могут гибридизоваться друг с другом. Например, в контексте ДНК аденин комплементарен тимину и цитозин комплементарен гуанину. Таким образом, полинуклеотид по настоящему изобретению может включать выделенные фрагменты нуклеотидов, комплементарные всей последовательности, а также последовательности нуклеиновых кислот, по существу аналогичные им.
Конкретно, полинуклеотиды, имеющие гомологию или идентичность с полинуклеотидом по настоящему изобретению, могут быть обнаружены с использованием условий гибридизации, включающих стадию гибридизации при значении Tm 55°С в описанных выше условиях. Кроме того, значение Tm может составлять 60°С, 63°С или 65°С, но не ограничено этим, и может быть соответствующим образом скорректировано специалистами в данной области техники в зависимости от их цели.
Подходящая жесткость для гибридизации полинуклеотидов зависит от длины полинуклеотидов и степени комплементации, и эти переменные хорошо известны в данной области техники (например Sambrook et al.).
В одном примере полинуклеотид по настоящему изобретению может включать полинуклеотид, представленный нуклеиновокислотной последовательностью в положениях с 1084 по 1245 на основе нуклеиновокислотной последовательности SEQ ID NO: 9, 11 или 13; или полинуклеотид, представленный нуклеиновокислотной последовательностью SEQ ID NO: 9, 11, 13 или 15.
В полинуклеотиде по настоящему изобретению вариант является таким, как описано в других аспектах выше.
В еще одном аспекте настоящего изобретения предложен вектор, содержащий полинуклеотид по настоящему изобретению. Вектор может представлять собой экспрессионный вектор для экспрессии полинуклеотида в клетке-хозяине, но не ограничивается этим.
Вектор по настоящему изобретению может включать конструкцию ДНК, содержащую нуклеотидную последовательность полинуклеотида, кодирующего целевой полипептид, функционально связанную с подходящей областью регуляции экспрессии (регуляторной последовательностью экспрессии) таким образом, чтобы обеспечивалась возможность экспрессировать целевой полипептид в подходящей клетке-хозяине. Область регуляции экспрессии может включать промотор, способный инициировать транскрипцию, любую последовательность оператора для регуляции транскрипции, последовательность, кодирующую подходящий сайт связывания мРНК с рибосомой, и последовательность регуляции окончания транскрипции и трансляции. После трансформации в подходящую клетку-хозяина вектор может реплицироваться или функционировать независимо от генома хозяина или может интегрироваться в его геном.
Вектор, используемый в настоящем изобретении, конкретно не ограничен и можно использовать любой вектор, известный в данной области техники. Примеры обычно используемого вектора могут включать природные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, в качестве фагового вектора или космидного вектора можно использовать pWE15, М13, MBL3, MBL4, IXII, ASHII, APII, t10, t11, Charon4A и Charon21A и т.д.; и в качестве плазмидного вектора можно использовать векторы на основе pDZ, pBR, pUC, pBluescriptII, pGEM, pTZ, pCL и pET и т.д. Конкретно, можно использовать векторы pDZ, pDC, pDCM2, pACYC177, pACYC184, pCL, pECCG117 (Biotechnology letters vol 13, No. 10, p.721-726 (1991), корейский патент №10-1992-0007401), pUC19, pBR322, pMW118, pCC1BAC и т.д.
В одном примере полинуклеотид, кодирующий целевой полипептид, может быть встроен в хромосому посредством вектора для внутриклеточной хромосомной вставки. Встраивание полинуклеотида в хромосому можно выполнять посредством любого способа, известного в данной области, например путем гомологичной рекомбинации, но без ограничения этим. Вектор может дополнительно включать селективный маркер для подтверждения встраивания в хромосому. Селективный маркер предназначен для отбора клеток, трансформированных вектором, то есть для подтверждения, была ли встроена целевая молекула нуклеиновой кислоты, и можно использовать маркеры, которые обеспечивают селектируемые фенотипы, такие как лекарственная устойчивость, ауксотрофия, устойчивость к клеточным токсичным агентам или экспрессия поверхностных полипептидов. Только клетки, экспрессирующие селективный маркер, способны выживать или демонстрировать разные фенотипы в среде, обработанной селективным агентом, и таким образом могут быть отобраны трансформированные клетки.
При использовании в данном документе термин "трансформация" относится к введению вектора, содержащего полинуклеотид, кодирующий целевой полипептид, в клетку-хозяина или микроорганизм таким образом, что полипептид, кодируемый полинуклеотидом, может экспрессироваться в клетке-хозяине. До тех пор, пока трансформированный полинуклеотид может экспрессироваться в клетке-хозяине, не имеет значения, интегрирован ли трансформированный полинуклеотид в хромосому клетки-хозяина и локализован в ней или расположен вне хромосомы, и могут быть включены оба случая. Кроме того, полинуклеотид может включать ДНК и/или РНК, кодирующие целевой полипептид. Полинуклеотид может быть введен в любой форме, при условии, что он может быть введен в клетку-хозяина и экспрессироваться в ней. Например, полинуклеотид может быть введен в клетку-хозяина в форме экспрессионной кассеты, которая представляет собой генную конструкцию, включающую все элементы, необходимые для ее автономной экспрессии. Обычно экспрессионная кассета может включать промотор, функционально связанный с полинуклеотидом, терминатор транскрипции, сайт связывания рибосомы или терминатор трансляции. Экспрессионная кассета может находиться в форме самореплицирующегося вектора экспрессии. Кроме того, полинуклеотид может быть введен в клетку-хозяина как есть и функционально связан с последовательностями, необходимыми для экспрессии в клетке-хозяине, но без ограничения этим.
Кроме того, при использовании в данном документе термин "функционально связанный" означает, что полинуклеотидная последовательность функционально связана с промоторной последовательностью, которая инициирует и опосредует транскрипцию полинуклеотида, кодирующего целевой вариант по настоящему изобретению.
В векторе по настоящему изобретению вариант и полинуклеотид являются такими, как описано в других аспектах выше.
В еще одном аспекте настоящего изобретения предлагается микроорганизм рода Corynebacterium, включающий вариант по настоящему изобретению или полинуклеотид по настоящему изобретению.
Микроорганизм по настоящему изобретению может включать вариант по настоящему изобретению, полинуклеотид, кодирующий вариант, или вектор, содержащий полинуклеотид по настоящему изобретению.
При использовании в данном документе термин "микроорганизм (или штамм)" включает все микроорганизмы дикого типа, или естественно или искусственно генетически модифицированные микроорганизмы, и он может представлять собой микроорганизм, в котором конкретный механизм ослаблен или усилен вследствие введения чужого гена, или усиления или инактивации активности эндогенного гена и т.д., и может представлять собой микроорганизм, включающий генетическую модификацию для получения целевого полипептида, белка или продукта.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, включающий любой один или более вариантов по настоящему изобретению, полинуклеотид по настоящему изобретения и вектор, содержащий полинуклеотид по настоящему изобретению; микроорганизм, модифицированный для экспрессии варианта по настоящему изобретению или полинуклеотида по настоящему изобретению; микроорганизм (например рекомбинантный штамм), экспрессирующий вариант по настоящему изобретению или полинуклеотид по настоящему изобретению; или микроорганизм (например рекомбинантный штамм), обладающий вариантной активностью по настоящему изобретению, но без ограничения этим.
Микроорганизм по настоящему изобретению может иметь способность продуцировать L-аминокислоты. Конкретно, в микроорганизме по настоящему изобретению способность продуцировать L-аминокислоты может представлять собой способность продуцировать L-валин или О-ацетил-L-гомосерин.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, который естественным образом обладает способностью продуцировать GltA или L-аминокислоты, микроорганизм, в который был введен вариант по настоящему изобретению или полинуклеотид, кодирующий то же самое (или вектор, содержащий полинуклеотид), в родительский штамм, который естественным образом не обладает способностью продуцировать GltA или L-аминокислоты и/или микроорганизм, которому была придана способность продуцировать GltA или L-аминокислоты, но без ограничения этим.
В одном примере микроорганизм по настоящему изобретению представляет собой клетку или микроорганизм, трансформированные с помощью полинуклеотида по настоящему изобретению или вектора, содержащего полинуклеотид по настоящему изобретению, для экспрессии варианта по настоящему изобретению, и для целей настоящего изобретения микроорганизм по настоящему изобретению может включать все микроорганизмы, способные продуцировать L-аминокислоты, включая вариант по настоящему изобретению. Например, штамм по настоящему изобретению может представлять собой рекомбинантный штамм, способность которого продуцировать L-аминокислоты повышена путем введения полинуклеотида, кодирующего вариант по настоящему изобретению, в природный микроорганизм дикого типа или микроорганизм, продуцирующий L-аминокислоты. Рекомбинантный штамм с повышенной способностью продуцировать L-аминокислоты может представлять собой микроорганизм, имеющий повышенную способность продуцировать L-аминокислоты по сравнению с природным микроорганизмом дикого типа или немодифицированным микроорганизмом цитратсинтазы (т.е. микроорганизмом, экспрессирующим белок дикого типа (SEQ ID NO: 1), или микроорганизмом, не экспрессирующий вариант по настоящему изобретению), но без ограничения этим. Например, немодифицированный микроорганизм цитратсинтазы, который представляет собой целевой штамм для сравнения увеличения способности продуцировать L-аминокислоты, может представлять собой штамм АТСС14067, штамм АТСС13032, штамм АТСС13869, штамм Corynebacterium glutamicum CJ7V, Corynebacterium glutamicum CJ8V или CA08-0072, но без ограничения этим.
В одном примере рекомбинантный штамм, имеющий повышенную продуктивную способность, может иметь способность продуцировать L-аминокислоты, повышенную примерно на 1% или более, 5% или более, 7% или более, примерно 10% или более, примерно 20% или более, или примерно 30% или более (верхний предел конкретно не ограничен, например примерно 200% или менее, примерно 150% или менее, примерно 100% или менее, примерно 50% или менее, примерно 45% или менее, примерно 40% или менее, или примерно 30% или менее) по сравнению со способностью продуцирования L-аминокислот у родительского штамма до модификации или немодифицированного микроорганизма, но без ограничения этим, пока он имеет повышенное значение + по сравнению с продуктивной способностью родительского штамма до модификации или немодифицированного микроорганизма. В другом примере рекомбинантный штамм, имеющий повышенную продуктивную способность, может иметь способность продуцировать L-аминокислоты примерно в 1,01 раза или более, примерно в 1,05 раза или более, примерно в 1,07 раза или более, примерно в 1,1 раза или более, примерно в 1,2 раза или более, или примерно в 1,3 раза или более (верхний предел конкретно не ограничен, например примерно в 10 раз или менее, примерно в 5 раз или менее, примерно в 3 раза или менее, или примерно в 2 раза или менее) по сравнению с родительским штаммом до модификации или немодифицированным микроорганизмом.
При использовании в данном документе термин "примерно" относится к диапазону, который включает в себя все из ±0,5, ±0,4, ±0,3, ±0,2, ±0.1, и т.д., и включает в себя все значения, которые эквивалентны или подобны тем, которые следуют за этими значениями, но диапазон не ограничивается этим.
При использовании в данном документе термин "немодифицированный микроорганизм" не исключает штамм, содержащий мутацию, которая может возникать в микроорганизме естественным образом, и может относиться к штамму дикого типа или к самому штамму природного типа, или к штамму до изменения признака вследствие генетической модификации, вызванной природными или искусственными факторами. Например немодифицированный микроорганизм может относиться к штамму, в который не введен вариант белка, описанный в данном документе, или к штамму до его введения. "Немодифицированный микроорганизм" можно использовать взаимозаменяемо со "штаммом до модификации", "микроорганизмом до модификации", "немутантным штаммом", "немодифицированным штаммом", "немутантным микроорганизмом" или "эталонным микроорганизмом".
В другом примере настоящего изобретения микроорганизм по настоящему изобретению может представлять собой Corynebacterium glutamicum, Corynebacterium crudilactis, Corynebacterium deserti, Corynebacterium efficiens, Corynebacterium callunae, Corynebacterium stationis, Corynebacterium singulare, Corynebacterium halotolerans, Corynebacterium striatum, Corynebacterium ammoniagenes, Corynebacterium pollutisoli, Corynebacterium imitans, Corynebacterium testudinoris или Corynebacterium flavescens.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, в котором дополнительно ослаблен белок NCgl2335. Кроме того, микроорганизм по настоящему изобретению может представлять собой микроорганизм у которого дополнительно усилена активность белка, выбранного из группы, состоящей из малой субъединицы изофермента 1 ацетолактатсинтазы (IlvN) или экспортера L-метионина/аминокислот с разветвленной цепью (YjeH).
Конкретно, микроорганизм по настоящему изобретению, продуцирующий L-валин, может представлять собой микроорганизм, в котором активность IlvN дополнительно усилена и/или дополнительно ослаблен белок NCgl2335. Кроме того, микроорганизм, продуцирующий О-ацетил-Lгомосерин по настоящему изобретению, может представлять собой микроорганизм, в котором дополнительно усилена активность экспортера L-метионина/аминокислот с разветвленной цепью (YjeH).
При использовании в данном документе термин "ослабление" полипептида представляет собой расширенное понятие, включающее как сниженную активность, так и ее отсутствие по сравнению с его эндогенной активностью. Ослабление можно использовать взаимозаменяемо с такими терминами, как инактивация, дефицит, понижающая регуляция, уменьшение, снижение, подавление и т.д.
Ослабление может также включать случай, когда саму полипептидную активность снижают или удаляют по сравнению с активностью полипептида, которой первоначально обладал микроорганизм, вследствие мутации полинуклеотида, кодирующего полипептид; случай, когда общий уровень внутриклеточной полипептидной активности и/или концентрации (уровень экспрессии) снижают по сравнению с природным штаммом вследствие ингибирования экспрессии гена полинуклеотида, кодирующего полипептид, или ингибирования трансляции в полипептид и т.д.; случай, когда полинуклеотид не экспрессируется вообще; и/или случай, когда полипептидная активность не наблюдается даже когда полинуклеотид экспрессируется. При использовании в данном документе термин "эндогенная активность" относится к активности конкретного полипептида, которой первоначально обладал родительский штамм до трансформации, микроорганизм дикого типа или немодифицированный микроорганизм, когда признак изменяется посредством генетической модификации, вызванной естественными или искусственными факторами, и может использоваться взаимозаменяемо с "активностью до модификации". Выражение, что активность полипептида "инактивирована, недостаточна, понижена, нерегулируема, снижена или аттенуирована" по сравнению с его эндогенной активностью, означает, что активность полипептида понижена по сравнению с активностью конкретного полипептида, которой первоначально обладал родительский штамм до трансформации или немодифицированный микроорганизм.
Ослабление активности полипептида может быть выполнено любым способом, известным в данной области техники, но способ не ограничивается ими, и может быть достигнуто посредством применения различных способов, хорошо известных в данной области техники (например, Nakashima N et al., Bacterial cellular engineering by genome editing and gene silencing. Int J Mol Sci. 2014; 15(2):2773-2793, Sambrook et al. Molecular Cloning 2012, и др.).
Конкретно, ослабление активности полипептида по настоящему изобретению может представлять собой:
1) делетирование части или всего гена, кодирующего полипептид;
2) модификацию области регуляции экспрессии (регуляторной последовательности экспрессии) таким образом, что экспрессия гена, кодирующего полипептид, снижается;
3) модификацию аминокислотной последовательности, составляющей полипептид, таким образом, что активность полипептида ликвидируется или ослабляется (например делеция/замена/добавление одной или более аминокислот в аминокислотной последовательности);
4) модификацию последовательности гена, кодирующего полипептид, таким образом, что активность полипептида ликвидируется или ослабляется (например делеция/замена/добавление одного или более нуклеотидов в нуклеотидной последовательности гена полипептида для кодирования полипептида, который был модифицирован для ликвидации или ослабления активности полипептида);
5) модификацию нуклеотидной последовательности, кодирующей инициирующий кодон, или 5'-UTR, транскрипта гена, кодирующего полипептид;
6) введение антисмыслового олигонуклеотида (например антисмысловой РНК), который комплементарно связывается с транскриптом гена, кодирующего полипептид;
7) добавление последовательности, комплементарной последовательности Шайна-Дальгарно (SD), на переднем конце последовательности SD гена, кодирующего полипептид, для образования вторичной структуры, тем самым ингибируя связывание рибосом;
8) инженерию обратной транскрипции (RTE), которая добавляет промотор, подлежащий обратной транскрипции, на З'-конце открытой рамки считывания (ORF) последовательности гена, кодирующей полипептид; или
9) комбинацию из двух или более, выбранных из приведенных выше (1)-(8), но без конкретного ограничения ими.
Например:
1) Делеция части или всего гена, кодирующего полипептид, может представлять собой делецию всего полинуклеотида, кодирующего эндогенный целевой полипептид в хромосоме, или замену полинуклеотида на полинуклеотид, имеющий частично делетированный нуклеотид, или на маркерный ген.
2) Модификация области регуляции экспрессии (регуляторной последовательности экспрессии) может представлять собой индуцирование модификации регуляторной области экспрессии (регуляторной последовательности экспрессии) посредством делеции, вставки, неконсервативной замены или консервативной замены или их комбинации; или замену последовательности на последовательность, имеющую более слабую активность. Область регуляции экспрессии может включать промотор, последовательность оператора, последовательность, кодирующую сайт связывания с рибосомой, и последовательность регулирования окончания транскрипции и трансляции, но не ограничивается этим.
3) и 4) Модификация аминокислотной последовательности или полинуклеотидной последовательности может представлять собой индуцирование модификации последовательности посредством делеции, вставки, неконсервативной или консервативной замены аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей полипептид, или их комбинацию для ослабления активности полипептида, или замену последовательности на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную так, чтобы обладать более слабой активностью, или на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную так, чтобы не обладать активностью, но без ограничения этим. Например, экспрессию гена можно ингибировать или ослаблять путем введения мутации в полинуклеотидную последовательность с образованием терминирующего кодона, но без ограничения этим.
5) Модификация нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR транскрипта гена, кодирующего полипептид, может представлять собой, например, замену нуклеотидной последовательности на нуклеотидную последовательность, кодирующую другой инициирующий кодон, имеющий более низкую скорость экспрессии полипептида, чем эндогенный инициирующий кодон, но без ограничения этим.
6) Введение антисмыслового олигонуклеотида (например антисмысловой РНК), который комплементарно связывается с транскриптом гена, кодирующего полипептид, можно найти в литературе [Weintraub, Н. et al., Antisense -RNA as a molecular tool for genetic analysis, Reviews - Trends in Genetics, Vol.1(1) 1986].
7) Добавление последовательности, комплементарной последовательности Шайна-Дальгарно (SD) на переднем конце последовательности гена SD, кодирующего полипептид, для образования вторичной структуры, тем самым ингибируя прикрепление рибосомы, может ингибировать трансляцию мРНК или снижать ее скорость.
Кроме того, 8) инженерия обратной транскрипции (RTE), которая добавляет промотор, который подлежит обратной транскрипции, на 3'-конце открытой рамки считывания (ORF) последовательности гена, кодирующей полипептид, может образовывать антисмысловой нуклеотид, комплементарный транскрипту гена, кодирующему полипептид, для ослабления активности.
Используемый здесь термин "усиление" активности полипептида означает, что активность полипептида повышается по сравнению с его эндогенной активностью. Усиление можно использовать взаимозаменяемо с такими терминами, как активация, ап-регуляция, сверхэкспрессия, увеличение и т.д. В частности, активация, усиление, ап-регуляция, сверхэкспрессия и увеличение могут включать оба случая, в которых проявляется изначально отсутствующая активность, или активность усиливается по сравнению с эндогенной активностью или активностью до модификации. "Эндогенная активность" относится к активности конкретного полипептида, которой первоначально обладал родительский штамм до трансформации или немодифицированный микроорганизм, где признак изменяют посредством генетической модификации, вызванной естественными или искусственными факторами, и может использоваться взаимозаменяемо с термином "активность до модификации". "Усиление", "ап-регуляция", "сверхэкспрессия" или "увеличение" активности полипептида по сравнению с его эндогенной активностью означает, что активность и/или концентрация (уровень экспрессии) полипептида усиливаются по сравнению с активностью конкретного полипептида, которой первоначально обладал родительский штамм до трансформации, или немодифицированный микроорганизм.
Усиление может быть достигнуто посредством введения чужеродного полипептида или усиления активности и/или концентрации (уровня экспрессии) эндогенного полипептида. Усиление активности полипептида можно подтвердить по повышению уровня активности полипептида, уровня экспрессии или количества продукта, выделяемого от полипептида.
Усиление активности полипептида может проводиться посредством различных способов, хорошо известных в данной области техники, и способ не ограничен до тех пор, пока он может усиливать активность целевого полипептида по сравнению с активностью микроорганизма до модификации. Конкретно, можно использовать генную инженерию и/или белковую инженерию, хорошо известную специалистам в данной области техники, которая представляет собой обычный способ молекулярной биологии, но способ не ограничивается этим (например, Sitnicka et al. Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol.2. 1-16, Sambrook et al. Molecular Cloning 2012 и др.).
Конкретно, усиление активности полипептида по настоящему изобретению может представлять собой:
1) увеличение числа внутриклеточных копий полинуклеотида, кодирующего полипептид;
2) замену области регуляции экспрессии гена, кодирующего полипептид, на хромосоме, последовательностью, имеющей более сильную активность;
3) модификацию нуклеотидной последовательности, кодирующей инициирующий кодон, или 5'-UTR, транскрипта гена, кодирующего полипептид;
4) модификацию аминокислотной последовательности полипептида таким образом, что активность полипептида усиливается;
5) модификацию полинуклеотидной последовательности, кодирующей полипептид, таким образом, что активность полипептида усиливается (например модификацию полинуклеотидной последовательности гена полипептида для кодирования полипептида, который был модифицирован для усиления активности полипептида);
6) введение чужеродного полипептида, проявляющего полипептидную активность, или чужеродного полинуклеотида, кодирующего его;
7) кодон-оптимизацию полинуклеотида, кодирующего полипептид;
8) анализ третичной структуры полипептида и, тем самым, выбор и модификацию экспонированного участка или его химическая модификация; или
9) комбинацию из двух или более выбранных из приведенных (1)-(8), но конкретно этим не ограничивается.
Более конкретно,
1) Увеличение числа внутриклеточных копий полинуклеотида, кодирующего полипептид, может представлять собой введение в клетку-хозяина вектора, который функционально связан с полинуклеотидом, кодирующим полипептид, и способен реплицироваться и функционировать независимо от клетки-хозяина. Альтернативно, оно может представлять собой введение одной копии или двух копий полинуклеотидов, кодирующих полипептид, в хромосому клетки-хозяина. Введение в хромосому можно выполнять путем введения вектора, который способен встраивать полинуклеотид в хромосому клетки-хозяина, в клетку-хозяина, но не ограничивается этим. Вектор является таким, как описано выше.
2) Замена регулирующей экспрессию области (или последовательности, регулирующей экспрессию) гена, кодирующего полипептид на хромосоме, на последовательность, имеющую сильную активность, может представлять собой, например, индуцирование модификации последовательности посредством делеции, вставки, неконсервативной или консервативной замены или их комбинации для дальнейшего усиления активности регулирующей экспрессию области, или замену последовательности на последовательность, имеющую более сильную активность. Регулирующая экспрессию область может включать, но без конкретного ограничения этим, промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, и последовательность, регулирующую окончание транскрипции и трансляции и т.д. В одном примере это может представлять собой замену исходного промотора на сильный промотор, но без ограничения этим.
Примеры известных сильных промоторов могут включать промоторы CJ1-CJ7 (US 7662943 В2), промотор lac, промотор trp, промотор trc, промотор tac, промотор PR лямбда-фага, промотор PL, промотор tet, промотор gapA, промотор SPL7, промотор SPL13 (sm3) (US 10584338 В2), промотор 02 (US 10273491 В2), промотор tkt, промотор уссА и т.д., но сильные промоторы не ограничиваются ими.
3) Модификация нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR транскрипт гена, кодирующего полипептид, может, например, представлять собой замену нуклеотидной последовательности на нуклеотидную последовательность, кодирующую другой инициирующий кодон, имеющий более высокую степень экспрессии полипептида по сравнению с эндогенным инициирующим кодоном, но без ограничения этим.
4) и 5) Модификация аминокислотной последовательности или полинуклеотидной последовательности может представлять собой индуцирование модификации последовательности посредством делеции, вставки, неконсервативной или консервативной замены аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей полипептид, или их комбинацию для усиления активности полипептида, или замену последовательности на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную для придания более сильной активности, или на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную для усиления активности, но без ограничения этим. Замену конкретно можно выполнять путем встраивания полинуклеотида в хромосому посредством гомологичной рекомбинации, но без ограничения этим. Вектор, используемый здесь, может дополнительно включать селективный маркер для подтверждения встраивания в хромосому. Селективный маркер является таким, как описано выше.
6) Введение чужеродного полинуклеотида, проявляющего активность полипептида, может представлять собой введение в клетку-хозяина чужеродного полинуклеотида, кодирующего полипептид, который проявляет такую же/аналогичную активность, что и полипептид. Чужеродный полинуклеотид можно использовать без ограничения, независимо от его происхождения или последовательности, пока он проявляет такую же активность, что и полипептид. Введение может быть выполнено обычными специалистами в данной области техники посредством соответствующего выбора известного в данной области техники метода трансформации, и экспрессия введенного полинуклеотида в клетке-хозяине дает возможность продуцировать полипептид, тем самым повышая его активность.
7) Оптимизация кодонов полинуклеотида, кодирующего полипептид, может представлять собой оптимизацию кодонов эндогенного полинуклеотида для увеличения транскрипции или трансляции внутри клетки-хозяина или оптимизацию его кодонов так, чтобы можно было достичь оптимизированной транскрипции и трансляции чужеродного полинуклеотида внутри клетки-хозяина.
Кроме того, 8) анализ третичной структуры полипептида и, посредством этого, выбор и модификация экспонированного участка или его химическая модификация могут представлять собой, например, сравнение информации о последовательности подлежащего анализу полипептида с базой данных, в которой хранится информация о последовательности известных белков, для определения кандидатов матричных белков в соответствии со степенью сходства последовательностей и, таким образом, подтверждения структуры на основе информации, таким образом отбирая и трансформируя или модифицируя экспонированный участок, подлежащий модификации или химической модификации.
Такое усиление активности полипептида может означать, что активность или концентрация (уровень экспрессии) соответствующего полипептида повышается относительно активности или концентрации (уровня экспрессии) полипептида, экспрессируемого в штамме дикого типа или в микроорганизме до модификации, или что количество продукта, полученного из полипептида, увеличивается, но без ограничения этим.
Модификации части или всего полинуклеотида в микроорганизме по настоящему изобретению (например модификация для кодирования варианта белка, описанного выше) можно достичь посредством (а) гомологичной рекомбинации с использованием вектора для хромосомной вставки в микроорганизм или редактирования генома с использованием сконструированной нуклеазы (например CRISPR-Cas9), и/или (б) она может быть индуцирована светом, таким как ультрафиолетовые лучи и радиация и т.д., и/или химической обработкой, но без ограничения этим. Способ модификации части или всего гена может включать способ с использованием технологии рекомбинантной ДНК. Например, часть или весь ген могут быть делетированы посредством инъекции нуклеотидной последовательности или вектора, содержащего нуклеотидную последовательность, гомологичную целевому гену, в микроорганизм для индуцирования гомологичной рекомбинации. Введенная нуклеотидная последовательность или вектор могут включать доминантный селективный маркер, но без ограничения этим.
Более конкретно, микроорганизм, продуцирующий L-валин по настоящему изобретению, может представлять собой микроорганизм, включающий полипептид, представленный аминокислотной последовательностью SEQ ID NO: 27 и/или полинуклеотид, представленный нуклеотидной последовательностью SEQ ID NO: 28. Кроме того, микроорганизм, продуцирующий О-ацетил-L-гомосерин по настоящему изобретению, может представлять собой микроорганизм, включающий полипептид, представленный аминокислотной последовательностью SEQ ID NO: 47 и/или полинуклеотид, представленный нуклеотидной последовательностью SEQ ID NO: 48; и может представлять собой микроорганизм, включающий мутацию, выбранную из группы, состоящей из инактивации полипептида, представленного аминокислотной последовательностью SEQ ID NO: 37, и/или делецию полинуклеотида, представленного нуклеотидной последовательностью SEQ ID NO: 38.
В микроорганизмах по настоящему изобретению вариант, полинуклеотид и т.д. являются такими, как описано в других аспектах выше.
В еще одном аспекте настоящего изобретения предлагается способ получения L-аминокислот, включающий: культивирование в среде микроорганизма рода Corynebacterium, который включает вариант по настоящему изобретению или полинуклеотид по настоящему изобретению.
Способ получения L-аминокислот по настоящему изобретению может включать культивирование в среде штамма Corynebacterium glutamicum, который включает вариант по настоящему изобретению, полинуклеотид по настоящему изобретению или вектор по настоящему изобретению.
Кроме того, в способе получения L-аминокислот по настоящему изобретению L-аминокислота может представлять собой L-валин, О-ацетил-L-гомосерин или L-метионин.
При использовании здесь термин "культивирование" означает, что микроорганизм рода Corynebacterium по настоящему изобретению выращивают в надлежащим образом контролируемых условиях окружающей среды. Процесс культивирования по настоящему изобретению можно выполнять в подходящей культуральной среде и в условиях культивирования, известных в данной области техники. Такой процесс культивирования можно легко адаптировать для использования специалистами в данной области техники в соответствии с выбранным штаммом. Конкретно, культивирование может представлять собой периодическое культивирование, непрерывное культивирование и/или периодическое культивирование с подпиткой, но без ограничения этим.
При использовании здесь термин "среда" относится к смеси веществ, которая содержит питательные вещества, необходимые для культивирования микроорганизма рода Corynebacterium по настоящему изобретению, в качестве основного ингредиента, и она доставляет питательные вещества и факторы роста вместе с водой, которая является необходимой для выживания и роста. Конкретно, среда и другие условия культивирования, используемые для культивирования микроорганизма рода Corynebacterium по настоящему изобретению, могут представлять собой любую среду, используемую для обычного культивирования микроорганизмов без каких-либо конкретных ограничений. Однако микроорганизм рода Corynebacterium по настоящему изобретению можно культивировать в аэробных условиях в обычной среде, содержащей соответствующий источник углерода, источник азота, источник фосфора, неорганическое соединение, аминокислоту и/или витамин, при регулировании температуры, рН и т.д.
Конкретно, питательную среду для микроорганизма рода Corynebacterium можно найти в литературе ["Manual of Methods for General Bacteriology" by the American Society for Bacteriology (Washington D.C., USA, 1981)].
В настоящем изобретении источник углерода может включать углеводы, такие как глюкоза, сахароза, лактоза, фруктоза, сахароза, мальтоза и т.д.; сахарные спирты, такие как маннит, сорбит и т.д.; органические кислоты, такие как пировиноградная кислота, молочная кислота, лимонная кислота и т.д.; аминокислоты, такие как глутаминовая кислота, метионин, лизин и т.д. Кроме того, источник углерода может включать натуральные органические питательные вещества, такие как гидролизат крахмала, меласса, черная меласса, рисовые отруби, маниока, меласса из сахарного тростника, кукурузный сироп и т.д. Конкретно, можно использовать углеводы, такие как глюкоза и стерилизованная предварительно обработанная меласса (т.е. меласса, превращенная в редуцирующий сахар), и, кроме того, можно использовать различные другие источники углерода в соответствующем количестве без ограничения. Эти источники углерода можно использовать по отдельности или в комбинации двух или более видов, но без ограничения этим.
Источник азота может включать в себя неорганические источники азота, такие как аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония, нитрат аммония и т.д.; аминокислоты, такие как глутаминовая кислота, метионин, глутамин и т.д.; и органические источники азота, такие как пептон, NZ-амин, мясной экстракт, дрожжевой экстракт, солодовый экстракт, кукурузный сироп, гидролизат казеина, рыба или продукт ее разложения, обезжиренный соевый жмых или продукт его разложения и т.д. Эти источники азота можно использовать по отдельности или в комбинации двух или более видов, но без ограничения ими.
Источник фосфора может включать монокалийфосфат, дикалийфосфат или соответствующие натрийсодержащие соли и т.д. Примеры неорганических соединений могут включать хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и т.д. Кроме того, могут быть включены аминокислоты, витамины и/или подходящие предшественники. Эти составляющие ингредиенты или предшественники можно добавлять в среду периодическим или непрерывным способом, но этими источники фосфора не ограничены перечисленным.
Кроме того, рН среды можно регулировать подходящим образом путем добавления соединения, такого как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота, серная кислота и т.д., во время культивирования штамма Corynebacterium glutamicum по настоящему изобретению. Кроме того, образование пузырьков во время культивирования можно предотвращать с помощью пеногасителя, такого как полигликолевый эфир жирных кислот. Кроме того, с целью поддержания аэробных условий среды можно вводить в среду газообразный кислород или газ, содержащий кислород; или для поддержания анаэробных или микроаэробных условий можно вводить газообразный азот, газообразный водород или диоксид углерода, без введения газа, но газ не ограничивается этим.
Температура во время культивирования по настоящему изобретению может находиться в диапазоне от 20°С до 45°С, конкретно от 25°С до 40°С, и культивирование можно выполнять в течение примерно от 10 до 160 часов, но культивирование не ограничивается этим.
L-аминокислоты, полученные путем культивирования по настоящему изобретению, могут высвобождаться в среду или оставаться в клетках.
Способ получения L-аминокислот по настоящему изобретению может дополнительно включать стадию получения микроорганизма рода Corynebacterium по настоящему изобретению, стадию приготовления среды для культивирования микроорганизма или их комбинацию (независимо от порядка, в любом порядке), например, перед стадией культивирования.
Способ получения L-валина по настоящему изобретению может дополнительно включать стадию выделения L-валина из культуральной среды (среды, на которой выращивали культуру) или микроорганизма рода Corynebacterium по настоящему изобретению. Стадия выделения может быть дополнительно включена после стадии культивирования.
На стадии выделения нужные L-аминокислоты можно собирать с использованием способа культивирования микроорганизма по настоящему изобретению, например, с использованием подходящего способа, известного в данной области техники, в соответствии со способом периодического культивирования, непрерывного культивирования или способа периодического культивирования с подпиткой. Например, можно использовать такие методы, как центрифугирование, фильтрация, обработка осадителем для кристаллизации белка (метод высаливания), экстракция, ультразвуковое разрушение, ультрафильтрация, диализ, различные виды хроматографии, такие как хроматография на молекулярных ситах (гель-фильтрация), адсорбционная хроматография, ионообменная хроматография, аффинная хроматография и т.д., ВЭЖХ, или их комбинацию, и нужный L-валин можно выделять из среды или микроорганизмов с использованием подходящих методов, известных в данной области техники.
Кроме того, способ получения L-аминокислот по настоящему изобретению может дополнительно включать стадию очистки, которую можно выполнять с использованием соответствующего способа, известного в данной области техники. В одном примере, когда способ получения L-аминокислот по настоящему изобретению включает как стадию выделения, так и стадию очистки, стадию выделения и стадию очистки можно выполнять непрерывно или с перерывами независимо от порядка или одновременно, или их можно объединять в одну стадию, но способ этим не ограничивается.
Кроме того, способ получения L-метионина по настоящему изобретению может дополнительно включать стадию превращения О-ацетил-L-гомосерина в L-метионин. В способ получения L-метионина по настоящему изобретению можно дополнительно включать стадию конверсии после стадии культивирования или стадии выделения. Стадию превращения можно выполнять, используя подходящий способ, известный в данной области техники (US 8426171 В2). В одном воплощении способ получения L-метионина по настоящему изобретению может включать стадию получения L-метионина посредством приведения О-ацетил-L-гомосерин и метилмеркаптана в контакт с О-ацетилгомосерин-сульфгидрилазой, цистатионин-гамма-синтазой или О-сукцинилгомосерин-сульфгидрилазой.
В способе по настоящему изобретению вариант, полинуклеотид, вектор, микроорганизм и т.д. представляют собой такие, как описано в других аспектах выше.
Кроме того, в другом аспекте настоящего изобретения предлагается композиция для получения L-аминокислот, включающая: микроорганизм рода Corynebacterium, который включает вариант по настоящему изобретению, полинуклеотид, кодирующий вариант по настоящему изобретению, или вектор, содержащий полинуклеотид по настоящему изобретению; среду, на которой выращивают микроорганизм; или их комбинацию.
Композиция по настоящему изобретению может дополнительно включать любой подходящий эксципиент, обычно используемый в композициях для получения аминокислот, и такие эксципиент включают, например, консерванты, смачивающие агенты, диспергирующие агенты, суспендирующие агенты, буферы, стабилизаторы или изотонические агенты и т.д., но без ограничения этим.
В композиции для получения L-аминокислот по настоящему изобретению L-аминокислота может представлять собой L-валин, О-ацетил-L-гомосерин или L-метионин.
В композиции по настоящему изобретению вариант, полинуклеотид, вектор, штамм, среда и т.д. представляют собой такие, как описано в других аспектах выше.
Вариант осуществления изобретения
Далее настоящее изобретение будет подробно описано с помощью Примеров.
Однако эти Примеры представляют собой всего лишь предпочтительные Примеры, представленные в иллюстративных целях, и, таким образом, объем настоящего изобретения не предназначен для ограничения этими примерами или посредством этих Примеров. Вместе с тем, технические признаки, которы не описаны здесь, могут быть достаточно понятны и легко реализованы специалистами в области техники настоящего изобретения или в аналогичной технической области.
Пример 1: Конструирование вектора для варианта цитратсинтазы (GltA)
Авторы настоящего изобретения обнаружили 415-ый аминокислотный остаток gltA в качестве сайта связывания ацетил-КоА и предположили, что при замене этой аминокислоты другой аминокислотой цитратсинтазная активность будет ослабевать по мере увеличения значения Km ацетил-КоА.
Соответственно, был сконструирован вектор, в котором лизин, 415-ая аминокислота в GltA, заменен другой аминокислотой. Конкретно, сконструировали вектор, содержащий мутации, в котором лизин, 415-ая аминокислота в GltA, заменен гистидином (К415Н), триптофаном (K415W) и глицином (K415G).
ПЦР выполняли с использованием пар праймеров SEQ ID NO: 15 и 17 и SEQ ID NO: 16 и 18, пар праймеров SEQ ID NO: 15 и 20 и SEQ ID NO: 18 и 19 и пар праймеров SEQ ID NO: 15 и 22 и SEQ ID NO: 18 и 21 на основе гДНК Corynebacterium glutamicum АТСС 14067 дикого типа в качестве матрицы. ПЦР с перекрывающимися праймерами выполняли на основе смеси двух фрагментов, полученных выше, в качестве матрицы, используя пару праймеров с SEQ ID NO: 15 и 18 с получением трех фрагментов. ПЦР выполняли в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд и затем полимеризации при 72°С в течение 5 минут.Вектор pDCM2 (SEQ ID №: 14, публикация корейской заявки №10-2020-0136813) обрабатывали smal и каждый из трех продуктов ПЦР, полученных выше, подвергали клонированию способом слияния. Клонирование способом слияния выполняли, используя набор для клонирования In-Fusion® HD (Clontech). Полученная в результате клонирования выше плазмиды назвали pDCM2-GltA(K415H), pDCM2-GltA(K415W) и pDCM2-GltA(K415G) соответственно. Последовательности праймеров, использованных в данном Примере, показаны в Таблице 1 ниже.


Пример 2: Введение варианта GltA в штаммы, продуцирующие L-валин, и их оценка
2-1. Конструирование штаммов на основании продуцирования L-валина и их оценка
Один вид мутации [ilvN(A42V); Biotechnology and Bioprocess Engineering, June 2014, Volume 19, Issue 3, pp 456-467] (SEQ ID NO: 27) вводили в малую субъединицу изофермента 1 ацетолактатсинтазы (IlvN) Corynebacterium glutamicum АТСС 14067 и АТСС 13869 дикого типа для конструирования штаммов, обладающих усиленной способностью продуцировать L-валин (KR 10-1947945 В1).
Конкретно, ПЦР выполняли с использованием пар праймеров SEQ ID NO: 29 и 31 и SEQ ID NO: 30 и 32, основанных на гДНК АТСС14067 Corynebacterium glutamicum дикого типа в качестве матрицы. Перекрывающуюся ПЦР выполняли на основе смеси двух фрагментов, полученных выше, в качестве матрицы, используя пару праймеров с SEQ IS NO: 29 и 32 с получением трех фрагментов. Здесь ПЦР выполняли в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут.Вектор pDCM2 обрабатывали с помощью smal, и каждый из трех продуктов ПЦР, полученных выше, подвергали клонированию способом слияния. Плазмиду, полученную в результате описанного выше клонирования, назвали pDCM2-ilvN(A42V). После этого pDCM2-ilvN(A42V) трансформировали в штаммы АТСС 14067 и АТСС13869 Corynebacterium glutamicum дикого типа для индуцирования гомологичной рекомбинации на хромосоме (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Штаммы с вектором, введенным в хромосому посредством рекомбинации гомологичных последовательностей, отбирали в среде, содержащей 25 мг/л канамицина. Фрагменты генов амплифицировали на основе выбранных трансформантов Corynebacterium glutamicum методом ПЦР с использованием пары праймеров SEQ ID NO: 33 и 34, и введение мутации подтверждали посредством анализа секвенирования генов. Рекомбинантные штаммы назвали Corynebacterium glutamicum CJ7V и CJ8V соответственно. Последовательности праймеров, используемых в данном Примере, показаны в Таблице 2 ниже.

После этого выполняли эксперимент по определению титра ферментации на основе Corynebacterium glutamicum АТСС 14067 и АТСС13869 дикого типа и штаммов CJ7V и CJ8V, сконструированных выше. Каждый штамм подвергали субкультивированию в питательной среде и затем высевали в колбу 250 мл с угловой перегородкой, содержащую 25 мл среды для продуцирования, и культивировали при 30°С в течение 72 часов при 200 об/мин при встряхивании. После этого концентрацию L-валина анализировали, используя ВЭЖХ, и проанализированная концентрация L-валина показана в Таблице 3 ниже
Питательная среда (рН 7,2)
Глюкоза 10 г, мясной экстракт 5 г, полипептон 10 г, хлорид натрия 2,5 г, дрожжевой экстракт 5 г, агар 20 г, мочевина 2 г (из расчета на 1 л дистиллированной воды)
Среда для продуцирования (рН 7,0)
Глюкоза 100 г, (NH4)2SO4 40 г, соевый белок 2,5 г, кукурузный крахмал 5 г, мочевина 3 г, K2HPO4 1 г, MgSO4⋅7H2O 0,5 г, биотин 100 мкг, тиамин-HCl 1 мг, кальций-пантотеновая кислота 2 мг, никотинамид 3 мг, СаСО3 30 г (из расчета на 1 л дистиллированной воды).
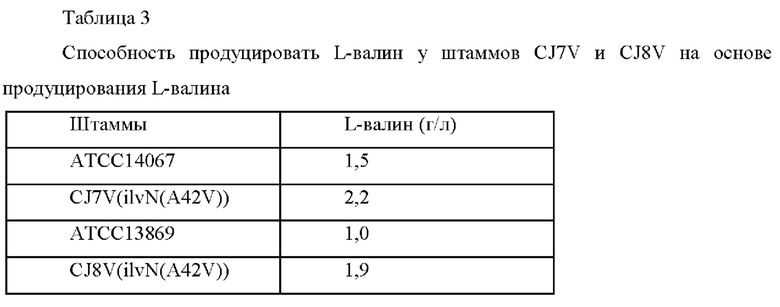
Как показано в результатах, было подтверждено, что способность продуцировать L-валин была повышена у штаммов CJ7V и CJ8V с введенной мутацией гена ilvN(A42V) по сравнению со штаммами Corynebacterium glutamicum АТСС 14067 и АТСС 13869 дикого типа.
2-2. Введение вариантов с ослабленным GltA (K415H, K415W, K415G) в штаммы, продуцирующие L-валин, и их оценка
Способность продуцировать L-валин оценивали путем введения вариантов GltA в штаммы, продуцирующие L-валин. Векторы pDCM2-gltA(K415H), pDCM2-GltA (K415W) и pDCM2-GltA (K415G), сконструированные в Примере 1, трансформировали в каждый из продуцирующих L-валин штаммов CJ7V и CJ8V и СА08-0072 (КССМ11201Р, US 8465962 В2) посредством гомологичной рекомбинации на хромосоме. Штаммы с векторами, введенными в хромосому путем рекомбинации гомологичных последовательностей, отбирали в среде, содержащей 25 мг/л канамицина.
После этого фрагменты генов амплифицировали на основе трансформантов Corynebacterium glutamicum, в которых завершена вторичная рекомбинация, посредством ПЦР с использованием пары праймеров SEQ ID NO: 23 и 24 (Таблица 4) и затем штаммы с введенной мутацией подтверждали путем анализа секвенирования генов. Рекомбинантные штаммы названы на основе Corynebacterium glutamicum, как показано ниже, и оценку титра проводили таким же образом, как в Примере 2-1.
Результаты приведены в Таблице 5.
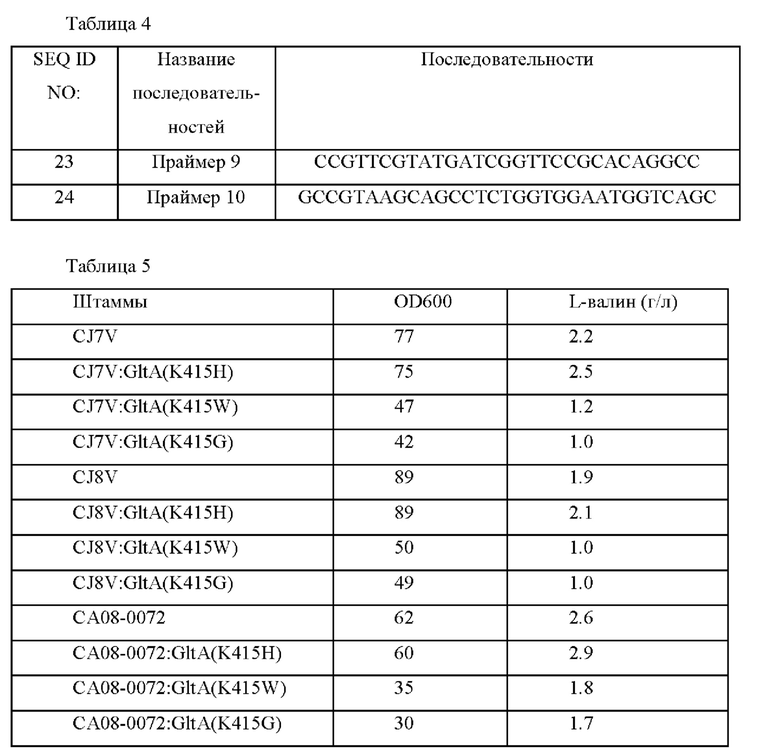
Как показано в результатах, вариант K415H обладал повышенной способностью продуцировать L-валин без снижения роста.
СА08-0072: gltA(K415H) назвали СА08-1688 и депонировали в Корейском центре культур микроорганизмов (КССМ) в соответствии с Будапештским договором от 28 сентября 2020 года с регистрационным номером КССМ12795Р.
Пример 3: Конструирование штамма с усиленным продуцированием О-ацетил-L-гомосерина и оценка способности продуцировать О-ацетил-L-гомосерин
3-1. Конструирование штамма, с введенным вариантом чужеродного мембранного белка YjeH
Для определения эффективности варианта YjeH, который представляет собой чужеродный мембранный белок и экспортер О-ацетилгомосерина, введенного в Corynebacterium glutamicum АТСС 13032, конструировали вектор для хромосомного введения, содержащий ген (SEQ ID NO: 48), кодирующий вариант YjeH (SEQ ID NO: 47).
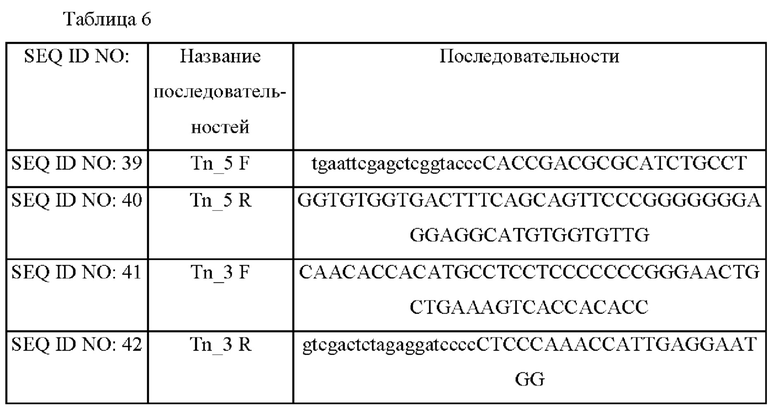
Конкретно, для конструирования вектора с делетированной транспозазой, разработали пару праймеров для амплификации участка выше 5'-конца (SEQ ID NO: 39 и 40) и пару праймеров для амплификации участка ниже 3'-конца (SEQ ID NO: 41 и 42) вокруг гена, кодирующего транспозазу (SEQ ID NO: 38, номер гена NCgl2335). Сайт фермента рестрикции Xbal встраивали на каждом конце праймеров SEQ ID NO: 39 и 42 и праймеры SEQ ID NO: 40 и 41 конструировали таким образом, чтобы они пересекали друг друга таким образом, чтобы последовательность фермента рестрикции Smal располагалась в созданном сайте. Последовательности праймеров приведены в Таблице 6.
ПЦР выполняли с использованием пар праймеров SEQ ID NO: 39 и 40 и SEQ ID NO: 41 и 42 на основе хромосомы АТСС13032 дикого типа в качестве матрицы. ПЦР выполняли в условиях денатурации при 95°С в течение 5 минут с последующими 30 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 30 секунд и затем полимеризации при 72°С в течение 7 минут. В результате получали фрагмент ДНК (851 п.н.) в области выше 5'-конца и фрагмент ДНК (847 п.н.) в области ниже 3'-конца вокруг области с делетированным геном NCgl2335.
ПЦР выполняли с использованием пары праймеров SEQ ID NO: 39 и 42 на основе двух амплифицированных фрагментов ДНК в качестве матрицы. ПЦР выполняли в условиях денатурации при 95°С в течение 5 минут с последующими 30 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 90 секунд и затем полимеризации при 72°С в течение 7 минут. В результате амплифицировали фрагмент ДНК (1648 п.н.), включающий сайт, способный удалять ген, кодирующий транспозазу (SEQ ID NO: 38, ген No. NCgl2335).
Полученные таким образом продукты ПЦР подвергали клонированию способом слияния в векторе pDCM2, обработанном ферментом рестрикции Smal, используя набор для клонирования In-Fusion ® HD (Clontech). Клонированный вектор трансформировали в Е. coli DH5a и трансформированную Е. coli высевали на твердую среду LB, содержащую 25 мг/л канамицина. Колонии, трансформированные плазмидой, в которую встроен ген-мишень, отбирали посредством ПЦР и затем получали плазмиду путем экстракции плазмиды. В итоге был сконструирован рекомбинантный вектор pDCM2-ΔNCgl2335, в котором клонируют кассету с делетированным NCgl2335.
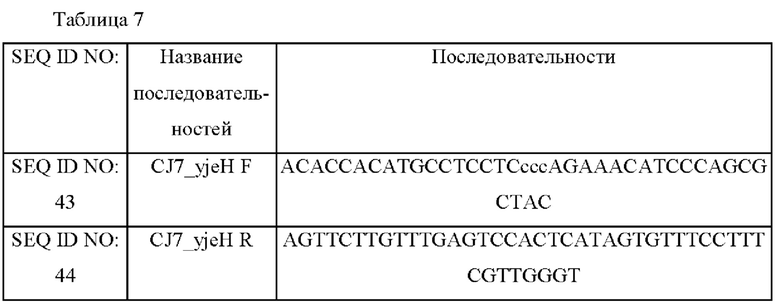
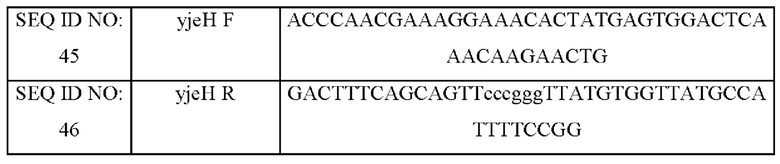
Для определения эффективности экспортера О-ацетилгомосерина конструировали вектор для хромосомного введения, содержащий ген (SEQ ID NO: 48), кодирующий вариант YjeH, полученный изЕ. coli. С этой целью конструировали вектор, экспрессирующий ген yjeH, с использованием промотора CJ7 (US 7662943 В2). Были разработаны пара праймеров (SEQ ID NO: 43 и 44) для амплификации области промотора CJ7 и пара праймеров (SEQ ID NO: 45 и 46) для амплификации области yjeH Е. coli. Последовательности праймеров приведены в Таблице 7 ниже.
ПЦР выполняли с использованием пары праймеров SEQ ID NO: 43 и 44 на основе pECCG117-PCJ7-gfp (US 7662943 В2, p117-Pcj7-gfp) в качестве матрицы и используя пару праймеров SEQ ID NO: 45 и 46 на основе хромосомы Е. coli дикого типа в качестве матрицы. ПЦР выполняли в условиях денатурации при 95°С в течение 5 минут с последующими 30 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 90 секунд и затем полимеризации при 72°С в течение 7 минут. В результате получали фрагмент ДНК (360 п. н.) области промотора CJ7 и фрагмент ДНК (1297 п.н.) области теиа yjeH Е. coli.
ПЦР выполняли с использованием пары праймеров SEQ ID NO: 43 и 46 на основе двух амплифицированных фрагментов ДНК в качестве матрицы. ПЦР выполняли в условиях денатурации при 95°С в течение 5 минут с последующими 30 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 90 секунд и затем полимеризации при 72°С в течение 7 минут. В результате амплифицировали фрагмент ДНК (1614 п.н.), включающий участки, в которые введены промотор CJ7 и ген yjeH.
Фрагмент ДНК с удаленным геном, полученный посредством ПЦР, клонировали в вектор pDCM2-ΔNCgl2335, обработанный ферментом рестрикции Smal, используя набор для клонирования In-Fusion® HD (Clontech), и конструировали рекомбинантный вектор pDCM2-ΔNCgl2335::PCJ7-yjeH(eco, WT).
Кроме того, конструировали рекомбинантный вектор для введения мутантного TeuayjeH (eco, F351L).
Конкретно, фенилаланин, который представляет собой 351-ую аминокислоту аминокислотной последовательности YjeH, заменяли лейцином (F351L) на основе плазмиды pDCM2-ΔNCgl2335::PCJ7-yjeH(eco, WT) в качестве матрицы с использованием пары праймеров SEQ ID NO: 49 и 50. Плазмида, содержащая сконструированный таким образом ген, кодирующий мутацию YjeH(F351L), была названа pDCM2-ΔNCgl2335::PCJ7-yjeH(eco,F351L). Последовательности праймеров приведены в Таблице 8 ниже.

Полученные таким образом pDCM2-ΔNCgl2335 и pDCM2-ΔNCgl2335::PCJ7-yjeH(eco,F351L) трансформировали в штамм АТСС13032 электроимпульсным методом. Посредством вторичного перекрывания на хромосоме получали АТСС13032 -ΔNCgl2335 с делетированным NCgl2335 и АТСС13032 -ΔNCgl2335::PCJ7-yjeH(eco,F351L). Делецию гена NCgl2335 и встраивание гена, кодирующего вариант YjeH, окончательно подтверждали посредством ПЦР с использованием пары праймеров SEQ ID NO: 39 и 42 и последующего сравнения с АТСС13032.
3-2. Оценка способности продуцировать О-ацетилгомосерин
Для сравнения способности продуцировать О-ацетилгомосерин (О-АН) у штаммов АТСС13032 -ΔNCgl2335 и АТСС13032 -ΔNCgl2335::PCJ7-yjeH(eco,F351L), сконструированных в Примере 3-1, и штамма АТСС 13032 дикого типа, штаммы для анализа О-ацетилгомосерин в растворе среды культивировали следующим образом.
Одну платиновую петлю штаммов инокулировали в колбу объемом 250 мл с угловой перегородкой, содержащую 25 мл среды для продуцирования О-ацетилгомосерина, приведенной ниже, и затем культивировали при встряхивании при 200 об/мин при 33°С в течение 20 часов. Концентрацию О-ацетилгомосерина анализировали посредством ВЭЖХ, и проанализированные концентрации приведены в Таблице 9.
Среда для продуцирования О-ацетилгомосерина (рН 7.2)
Глюкоза 30 г, KH2PO4 2 г, мочевина 3 г, (NH4)2SO4 40 г, пептон 2,5 г, кукурузный крахмал (CSL, Sigma) 5 г (10 мл), MgSO4⋅7H2O 0,5 г, СаСО3 20 г (из расчета на 1 л дистиллированной воды).
Таблица 9

В результате, как показано в Таблице 9, при культивировании контрольного штамма АТСС 13032 накопление О-ацетил-L-гомосерина составляло 0,3 г/л, и было подтверждено, что даже когда ген транспозазы NCgl2335 был удален, это не оказало никакого влияния на продуцирование О-ацетил-L-гомосерина. В частности, было подтверждено, что при экспрессии мутантного гена yjeH накопление О-ацетил-L-гомосерина составляло 1,0 г/л.
3-3. Введение вариантов GltA (K415H) в штаммы, продуцирующие О-ацетил-L-гомосерин, и их оценка
Способность продуцировать О-ацетил-L-гомосерин оценивали посредством введения варианта GltA в штаммы, продуцирующие О-ацетил-L-гомосерин, из Примера 3-2. Вектор pDCM2-GltA(K415H), сконструированный в Примере 1, трансформировали в каждый из штаммов дикого типа АТСС13032 и АТСС13032 -ΔNCgl2335, а также в штамм, продуцирующий О-ацетил-L-гомосерин АТСС13032 -ΔNCgl2335::PCJ7-yjeH(eco,F351L). Штаммы с вектором, введенным в хромосому путем рекомбинации гомологичных последовательностей, отбирали в среде, содержащей 25 мг/л канамицина.
После этого фрагменты гена амплифицировали на основе трансформантов Corynebacterium glutamicum, в которых вторичная рекомбинация была завершена, посредством ПЦР с использованием пары праймеров SEQ ID NO: 23 и 24, и штаммы с введенной мутацией GltA(K415H) подтверждали путем анализа секвенирования. Рекомбинантные штаммы назвали на основе Corynebacterium glutamicum, как показано ниже, и оценку титра проводили таким же образом, как в Примере 3-2. Результаты приведены в Таблице 10.


Как можно видеть из этих результатов, все штаммы с введенным вариантом GltA K415H демонстрировали увеличение способности продуцировать О-ацетил-Ь-гомосерин по сравнению с родительским штаммом, в который этот вариант не был введен.
АТСС13032 -ΔNCgl2335::PCJ7-yjeH(eco,F351L) GltA(K415H) был назван СМ04-1006 и депонирован в Корейском центре культур микроорганизмов (КССМ) в соответствии с Будапештским договором от 21 октября 2020 года, с регистрационным номером КССМ12809Р.
Исходя из вышеизложенного, специалист в области техники, к которой относится настоящее изобретение, может понять, что настоящее изобретение может быть воплощено в других конкретных формах без изменения технических концепций или существенных характеристик настоящего изобретения. Таким образом, примеры осуществления изобретения, раскрытые здесь, предназначены только для иллюстративных целей и не должны истолковываться как ограничивающие объем настоящего изобретения. Напротив, предполагается, что настоящее изобретение охватывает не только указанные примеры осуществления изобретения, но также различные альтернативы, модификации, эквиваленты и другие воплощения, которые могут быть включены в соответствии с сущностью и объемом настоящего изобретения, как определено в прилагаемой формуле изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> CJ CheilJedang Corporation
<120> Novel citrate synthase variant and method for producing L-amino
acids using same
<130> OPA21402
<150> KR 10-2021-0031641
<151> 2021-03-10
<160> 51
<170> KoPatentIn 3.0
<210> 1
<211> 437
<212> PRT
<213> Unknown
<220>
<223> ATCC 14067 GltA AA
<400> 1
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Asn Asn His Gly Gly Asp Ala Thr Ala Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 2
<211> 1314
<212> DNA
<213> Unknown
<220>
<223> ATCC 14067 GltA NT
<400> 2
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggatca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aacgcgcgtg agaacttcct gcgcatgatg ttcggttacc caactgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagaacaac cacggtggcg acgcaaccgc gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aattacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 3
<211> 54
<212> PRT
<213> Unknown
<220>
<223> ATCC 14067 GltA K415H 362~415 AA
<400> 3
Phe Ile Ser Arg Lys Leu Tyr Pro Asn Val Asp Phe Tyr Thr Gly Leu
1 5 10 15
Ile Tyr Arg Ala Met Gly Phe Pro Thr Asp Phe Phe Thr Val Leu Phe
20 25 30
Ala Ile Gly Arg Leu Pro Gly Trp Ile Ala His Tyr Arg Glu Gln Leu
35 40 45
Gly Ala Ala Gly Asn His
50
<210> 4
<211> 437
<212> PRT
<213> Unknown
<220>
<223> ATCC 13032 GltA AA
<400> 4
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 5
<211> 1314
<212> DNA
<213> Unknown
<220>
<223> ATCC13032 GltA NT
<400> 5
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 6
<211> 437
<212> PRT
<213> Unknown
<220>
<223> ATCC13869 GltA AA
<400> 6
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Asn Asn His Gly Gly Asp Ala Thr Ala Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Cys Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Lys Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 7
<211> 1314
<212> DNA
<213> Unknown
<220>
<223> ATCC13869 GltA NT
<400> 7
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggatca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aacgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagaacaac cacggtggcg acgcaaccgc gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tgcttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaagga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 8
<211> 437
<212> PRT
<213> Artificial Sequence
<220>
<223> ATCC 14067 GltA K415H AA
<400> 8
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Asn Asn His Gly Gly Asp Ala Thr Ala Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn His Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 9
<211> 1314
<212> DNA
<213> Artificial Sequence
<220>
<223> ATCC 14067 GltA K415H NT
<400> 9
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggatca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aacgcgcgtg agaacttcct gcgcatgatg ttcggttacc caactgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagaacaac cacggtggcg acgcaaccgc gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aattacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca accacatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 10
<211> 437
<212> PRT
<213> Artificial Sequence
<220>
<223> ATCC13032 GltA K415H AA
<400> 10
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn His Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 11
<211> 1314
<212> DNA
<213> Artificial Sequence
<220>
<223> ATCC13032 GltA K415H NT
<400> 11
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca accacatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 12
<211> 437
<212> PRT
<213> Artificial Sequence
<220>
<223> ATCC13869 GltA K415H AA
<400> 12
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Asn Asn His Gly Gly Asp Ala Thr Ala Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Cys Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn His Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Lys Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 13
<211> 1314
<212> DNA
<213> Artificial Sequence
<220>
<223> ATCC13869 GltA K415H NT
<400> 13
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggatca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aacgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagaacaac cacggtggcg acgcaaccgc gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tgcttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca accacatcaa ccgcccacgc 1260
caggtctaca ccggcaagga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 14
<211> 5803
<212> DNA
<213> Artificial Sequence
<220>
<223> pDCM2
<400> 14
gttcgcttgc tgtccataaa accgcccagt ctagctatcg ccatgtaagc ccactgcaag 60
ctacctgctt tctctttgcg cttgcgtttt cccttgtcca gatagcccag tagctgacat 120
tcatccgggg tcagcaccgt ttctgcggac tggctttcta cgtgttccgc ttcctttagc 180
agcccttgcg ccctgagtgc ttgcggcagc gtgaagctag cttttatcgc cattcgccat 240
tcaggctgcg caactgttgg gaagggcgat cggtgcgggc ctcttcgcta ttacgccagc 300
tggcgaaagg gggatgtgct gcaaggcgat taagttgggt aacgccaggg ttttcccagt 360
cacgacgttg taaaacgacg gccagtgaat tcgagctcgg tacccgggga tcctctagag 420
tcgacctgca ggcatgcaag cttggcgtaa tcatggtcat agctgtttcc tgtgtgaaat 480
tgttatccgc tcacaattcc acacaacata cgagccggaa gcataaagtg taaagcctgg 540
ggtgcctaat gagtgagcta actcacatta attgcgttgc gctcactgcc cgctttccag 600
tcgggaaacc tgtcgtgcca gctgcattaa tgaatcggcc aacgcgcggg gagaggcggt 660
ttgcgtattg ggcgctcttc cgcttcctcg ctcactgact cgctgcgctc ggtcgttcgg 720
ctgcggcgag cggtatcagc tcactcaaag gcggtaatac ggttatccac agaatcaggg 780
gataacgcag gaaagaacat gtgagcaaaa ggccagcaaa aggccaggaa ccgtaaaaag 840
gccgcgttgc tggcgttttt ccataggctc cgcccccctg acgagcatca caaaaatcga 900
cgctcaagtc agaggtggcg aaacccgaca ggactataaa gataccaggc gtttccccct 960
ggaagctccc tcgtgcgctc tcctgttccg accctgccgc ttaccggata cctgtccgcc 1020
tttctccctt cgggaagcgt ggcgctttct caatgctcac gctgtaggta tctcagttcg 1080
gtgtaggtcg ttcgctccaa gctgggctgt gtgcacgaac cccccgttca gcccgaccgc 1140
tgcgccttat ccggtaacta tcgtcttgag tccaacccgg taagacacga cttatcgcca 1200
ctggcagcag ccactggtaa caggattagc agagcgaggt atgtaggcgg tgctacagag 1260
ttcttgaagt ggtggcctaa ctacggctac actagaagga cagtatttgg tatctgcgct 1320
ctgctgaagc cagttacctt cggaaaaaga gttggtagct cttgatccgg caaacaaacc 1380
accgctggta gcggtggttt ttttgtttgc aagcagcaga ttacgcgcag aaaaaaagga 1440
tctcaagaag atcctttgat cttttctacg gggtctgacg ctcagtggaa cgaaaactca 1500
cgttaaggga ttttggtcat gagattatca aaaaggatct tcacctagat ccttttgggg 1560
tgggcgaaga actccagcat gagatccccg cgctggagga tcatccagcc ctgatagaaa 1620
cagaagccac tggagcacct caaaaacacc atcatacact aaatcagtaa gttggcagca 1680
tcacccgacg cactttgcgc cgaataaata cctgtgacgg aagatcactt cgcagaataa 1740
ataaatcctg gtgtccctgt tgataccggg aagccctggg ccaacttttg gcgaaaatga 1800
gacgttgatc ggcacgtaag aggttccaac tttcaccata atgaaataag atcactaccg 1860
ggcgtatttt ttgagttatc gagattttca ggagctgata gaaacagaag ccactggagc 1920
acctcaaaaa caccatcata cactaaatca gtaagttggc agcatcaccc gacgcacttt 1980
gcgccgaata aatacctgtg acggaagatc acttcgcaga ataaataaat cctggtgtcc 2040
ctgttgatac cgggaagccc tgggccaact tttggcgaaa atgagacgtt gatcggcacg 2100
taagaggttc caactttcac cataatgaaa taagatcact accgggcgta ttttttgagt 2160
tatcgagatt ttcaggagct ctttggcatc gtctctcgcc tgtcccctca gttcagtaat 2220
ttcctgcatt tgcctgtttc cagtcggtag atattccaca aaacagcagg gaagcagcgc 2280
ttttccgctg cataaccctg cttcggggtc attatagcga ttttttcggt atatccatcc 2340
tttttcgcac gatatacagg attttgccaa agggttcgtg tagactttcc ttggtgtatc 2400
caacggcgtc agccgggcag gataggtgaa gtaggcccac ccgcgagcgg gtgttccttc 2460
ttcactgtcc cttattcgca cctggcggtg ctcaacggga atcctgctct gcgaggctgg 2520
ccggctaccg ccggcgtaac agatgagggc aagcggatgg ctgatgaaac caagccaacc 2580
aggaagggca gcccacctat caaggtgtac tgccttccag acgaacgaag agcgattgag 2640
gaaaaggcgg cggcggccgg catgagcctg tcggcctacc tgctggccgt cggccagggc 2700
tacaaaatca cgggcgtcgt ggactatgag cacgtccgcg agggcgtccc ggaaaacgat 2760
tccgaagccc aacctttcat agaaggcggc ggtggaatcg aaatctcgtg atggcaggtt 2820
gggcgtcgct tggtcggtca tttcgaaaaa ggttaggaat acggttagcc atttgcctgc 2880
ttttatatag ttcantatgg gattcacctt tatgttgata agaaataaaa gaaaatgcca 2940
ataggatatc ggcattttct tttgcgtttt tatttgttaa ctgttaattg tccttgttca 3000
aggatgctgt ctttgacaac agatgttttc ttgcctttga tgttcagcag gaagctcggc 3060
gcaaacgttg attgtttgtc tgcgtagaat cctctgtttg tcatatagct tgtaatcacg 3120
acattgtttc ctttcgcttg aggtacagcg aagtgtgagt aagtaaaggt tacatcgtta 3180
ggcggatcaa gatccatttt taacacaagg ccagttttgt tcagcggctt gtatgggcca 3240
gttaaagaat tagaaacata accaagcatg taaatatcgt tagacgtaat gccgtcaatc 3300
gtcatttttg atccgcggga gtcagtgaac aggtaccatt tgccgttcat tttaaagacg 3360
ttcgcgcgtt caatttcatc tgttactgtg ttagatgcaa tcagcggttt catcactttt 3420
ttcagtgtgt aatcatcgtt tagctcaatc ataccgagag cgccgtttgc taactcagcc 3480
gtgcgttttt tatcgctttg cagaagtttt tgactttctt gacggaagaa tgatgtgctt 3540
ttgccatagt atgctttgtt aaataaagat tcttcgcctt ggtagccatc ttcagttcca 3600
gtgtttgctt caaatactaa gtatttgtgg cctttatctt ctacgtagtg aggatctctc 3660
agcgtatggt tgtcgcctga gctgtagttg ccttcatcga tgaactgctg tacattttga 3720
tacgtttttc cgtcaccgtc aaagattgat ttataatcct ctacaccgtt gatgttcaaa 3780
gagctgtctg atgctgatac gttaacttgt gcagttgtca gtgtttgttt gccgtaatgt 3840
ttaccggaga aatcagtgta gaataaacgg atttttccgt cagatgtaaa tgtggctgaa 3900
cctgaccatt cttgtgtttg gtcttttagg atagaatcat ttgcatcgaa tttgtcgctg 3960
tctttaaaga cgcggccagc gtttttccag ctgtcaatag aagtttcgcc gactttttga 4020
tagaacatgt aaatcgatgt gtcatccgca tttttaggat ctccggctaa tgcaaagacg 4080
atgtggtagc cgtgatagtt tgcgacagtg ccgtcagcgt tttgtaatgg ccagctgtcc 4140
caaacgtcca ggccttttgc agaagagata tttttaattg tggacgaatc aaattcagaa 4200
acttgatatt tttcattttt ttgctgttca gggatttgca gcatatcatg gcgtgtaata 4260
tgggaaatgc cgtatgtttc cttatatggc ttttggttcg tttctttcgc aaacgcttga 4320
gttgcgcctc ctgccagcag tgcggtagta aaggttaata ctgttgcttg ttttgcaaac 4380
tttttgatgt tcatcgttca tgtctccttt tttatgtact gtgttagcgg tctgcttctt 4440
ccagccctcc tgtttgaaga tggcaagtta gttacgcaca ataaaaaaag acctaaaata 4500
tgtaaggggt gacgccaaag tatacacttt gccctttaca cattttaggt cttgcctgct 4560
ttatcagtaa caaacccgcg cgatttactt ttcgacctca ttctattaga ctctcgtttg 4620
gattgcaact ggtctatttt cctcttttgt ttgatagaaa atcataaaag gatttgcaga 4680
ctacgggcct aaagaactaa aaaatctatc tgtttctttt cattctctgt attttttata 4740
gtttctgttg catgggcata aagttgcctt tttaatcaca attcagaaaa tatcataata 4800
tctcatttca ctaaataata gtgaacggca ggtatatgtg atgggttaaa aaggatcacc 4860
ccagagtccc gctcagaaga actcgtcaag aaggcgatag aaggcgatgc gctgcgaatc 4920
gggagcggcg ataccgtaaa gcacgaggaa gcggtcagcc cattcgccgc caagctcttc 4980
agcaatatca cgggtagcca acgctatgtc ctgatagcgg tccgccacac ccagccggcc 5040
acagtcgatg aatccagaaa agcggccatt ttccaccatg atattcggca agcaggcatc 5100
gccatgggtc acgacgagat cctcgccgtc gggcatccgc gccttgagcc tggcgaacag 5160
ttcggctggc gcgagcccct gatgctcttc gtccagatca tcctgatcga caagaccggc 5220
ttccatccga gtacgtgctc gctcgatgcg atgtttcgct tggtggtcga atgggcaggt 5280
agccggatca agcgtatgca gccgccgcat tgcatcagcc atgatggata ctttctcggc 5340
aggagcaagg tgagatgaca ggagatcctg ccccggcact tcgcccaata gcagccagtc 5400
ccttcccgct tcagtgacaa cgtcgagaca gctgcgcaag gaacgcccgt cgtggccagc 5460
cacgatagcc gcgctgcctc gtcttggagt tcattcaggg caccggacag gtcggtcttg 5520
acaaaaagaa ccgggcgccc ctgcgctgac agccggaaca cggcggcatc agagcagccg 5580
attgtctgtt gtgcccagtc atagccgaat agcctctcca cccaagcggc cggagaacct 5640
gcgtgcaatc catcttgttc aatcatgcga aacgatcctc atcctgtctc ttgatcagat 5700
cttgatcccc tgcgccatca gatccttggc ggcaagaaag ccatccagtt tactttgcag 5760
ggcttcccaa ccttaccaga gggcgcccca gctggcaatt ccg 5803
<210> 15
<211> 44
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 1
<400> 15
tcgagctcgg tacccccgtt cgtatgatcg gttccgcaca ggcc 44
<210> 16
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 2
<400> 16
gtgcagcagg caaccacatc aaccgcccac g 31
<210> 17
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 3
<400> 17
cgtgggcggt tgatgtggtt gcctgctgca c 31
<210> 18
<211> 46
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 4
<400> 18
ctctagagga tccccgccgt aagcagcctc tggtggaatg gtcagc 46
<210> 19
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 5
<400> 19
gtgcagcagg caactggatc aaccgcccac g 31
<210> 20
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 6
<400> 20
cgtgggcggt tgatccagtt gcctgctgca c 31
<210> 21
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 7
<400> 21
gtgcagcagg caacggcatc aaccgcccac g 31
<210> 22
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 8
<400> 22
cgtgggcggt tgatgccgtt gcctgctgca c 31
<210> 23
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 9
<400> 23
ccgttcgtat gatcggttcc gcacaggcc 29
<210> 24
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 10
<400> 24
gccgtaagca gcctctggtg gaatggtcag c 31
<210> 25
<211> 172
<212> PRT
<213> Unknown
<220>
<223> IlvN AA
<400> 25
Met Ala Asn Ser Asp Val Thr Arg His Ile Leu Ser Val Leu Val Gln
1 5 10 15
Asp Val Asp Gly Ile Ile Ser Arg Val Ser Gly Met Phe Thr Arg Arg
20 25 30
Ala Phe Asn Leu Val Ser Leu Val Ser Ala Lys Thr Glu Thr Leu Gly
35 40 45
Ile Asn Arg Ile Thr Val Val Val Asp Ala Asp Glu Leu Asn Ile Glu
50 55 60
Gln Ile Thr Lys Gln Leu Asn Lys Leu Ile Pro Val Leu Lys Val Val
65 70 75 80
Arg Leu Asp Glu Glu Thr Thr Ile Ala Arg Ala Ile Met Leu Val Lys
85 90 95
Val Ser Ala Asp Ser Thr Asn Arg Pro Gln Ile Val Asp Ala Ala Asn
100 105 110
Ile Phe Arg Ala Arg Val Val Asp Val Ala Pro Asp Ser Val Val Ile
115 120 125
Glu Ser Thr Gly Thr Pro Gly Lys Leu Arg Ala Leu Leu Asp Val Met
130 135 140
Glu Pro Phe Gly Ile Arg Glu Leu Ile Gln Ser Gly Gln Ile Ala Leu
145 150 155 160
Asn Arg Gly Pro Lys Thr Met Ala Pro Ala Lys Ile
165 170
<210> 26
<211> 520
<212> DNA
<213> Unknown
<220>
<223> IlvN NT
<400> 26
atggctaatt ctgacgtcac ccgccacatc ctgtccgtac tcgttcagga cgtagacgga 60
atcatttccc gcgtatcagg tatgttcacc cgacgcgcat tcaacctcgt gtccctcgtg 120
tctgcaaaga ccgaaacact cggcatcaac cgcatcacgg ttgttgtcga cgccgacgag 180
ctcaacattg agcagatcac caagcagctc aacaagctga tccccgtgct caaagtcgtg 240
cgacttgatg aagagaccac catcgcccgc gcaatcatgc tggttaaggt ctctgcggat 300
agcaccaacc gtccgcagat cgtcgacgcc gcgaacatct tccgcgcccg agtcgtcgac 360
gtggctccag actctgtggt tattgaatcc acaggcaccc caggcaagct ccgcgcactg 420
cttgatgtga tggaaccatt cggaatccgc gaactgatcc aatccggaca gattgcactc 480
aaccgcggtc cgaagaccat ggctccggcc aagatctaaa 520
<210> 27
<211> 172
<212> PRT
<213> Artificial Sequence
<220>
<223> IlvN A42V AA
<400> 27
Met Ala Asn Ser Asp Val Thr Arg His Ile Leu Ser Val Leu Val Gln
1 5 10 15
Asp Val Asp Gly Ile Ile Ser Arg Val Ser Gly Met Phe Thr Arg Arg
20 25 30
Ala Phe Asn Leu Val Ser Leu Val Ser Val Lys Thr Glu Thr Leu Gly
35 40 45
Ile Asn Arg Ile Thr Val Val Val Asp Ala Asp Glu Leu Asn Ile Glu
50 55 60
Gln Ile Thr Lys Gln Leu Asn Lys Leu Ile Pro Val Leu Lys Val Val
65 70 75 80
Arg Leu Asp Glu Glu Thr Thr Ile Ala Arg Ala Ile Met Leu Val Lys
85 90 95
Val Ser Ala Asp Ser Thr Asn Arg Pro Gln Ile Val Asp Ala Ala Asn
100 105 110
Ile Phe Arg Ala Arg Val Val Asp Val Ala Pro Asp Ser Val Val Ile
115 120 125
Glu Ser Thr Gly Thr Pro Gly Lys Leu Arg Ala Leu Leu Asp Val Met
130 135 140
Glu Pro Phe Gly Ile Arg Glu Leu Ile Gln Ser Gly Gln Ile Ala Leu
145 150 155 160
Asn Arg Gly Pro Lys Thr Met Ala Pro Ala Lys Ile
165 170
<210> 28
<211> 519
<212> DNA
<213> Artificial Sequence
<220>
<223> IlvN A42V NT
<400> 28
atggctaatt ctgacgtcac ccgccacatc ctgtccgtac tcgttcagga cgtagacgga 60
atcatttccc gcgtatcagg tatgttcacc cgacgcgcat tcaacctcgt gtccctcgtg 120
tctgtaaaga ccgaaacact cggcatcaac cgcatcacgg ttgttgtcga cgccgacgag 180
ctcaacattg agcagatcac caagcagctc aacaagctga tccccgtgct caaagtcgtg 240
cgacttgatg aagagaccac catcgcccgc gcaatcatgc tggttaaggt ctctgcggat 300
agcaccaacc gtccgcagat cgtcgacgcc gcgaacatct tccgcgcccg agtcgtcgac 360
gtggctccag actctgtggt tattgaatcc acaggcaccc caggcaagct ccgcgcactg 420
cttgatgtga tggaaccatt cggaatccgc gaactgatcc aatccggaca gattgcactc 480
aaccgcggtc cgaagaccat ggctccggcc aagatctaa 519
<210> 29
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 11
<400> 29
tcgagctcgg tacccccgcg tcaccaaagc gga 33
<210> 30
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 12
<400> 30
gtccctcgtg tctgtaaaga ccgaaacact 30
<210> 31
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 13
<400> 31
agtgtttcgg tctttacaga cacgagggac 30
<210> 32
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 14
<400> 32
ctctagagga tccccttaga tcttggccgg agcca 35
<210> 33
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 15
<400> 33
ccgcgtcacc aaagcgga 18
<210> 34
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер 16
<400> 34
ttagatcttg gccggagcca 20
<210> 35
<211> 418
<212> PRT
<213> Unknown
<220>
<223> YjeH AA
<400> 35
Met Ser Gly Leu Lys Gln Glu Leu Gly Leu Ala Gln Gly Ile Gly Leu
1 5 10 15
Leu Ser Thr Ser Leu Leu Gly Thr Gly Val Phe Ala Val Pro Ala Leu
20 25 30
Ala Ala Leu Val Ala Gly Asn Asn Ser Leu Trp Ala Trp Pro Val Leu
35 40 45
Ile Ile Leu Val Phe Pro Ile Ala Ile Val Phe Ala Ile Leu Gly Arg
50 55 60
His Tyr Pro Ser Ala Gly Gly Val Ala His Phe Val Gly Met Ala Phe
65 70 75 80
Gly Ser Arg Leu Glu Arg Val Thr Gly Trp Leu Phe Leu Ser Val Ile
85 90 95
Pro Val Gly Leu Pro Ala Ala Leu Gln Ile Ala Ala Gly Phe Gly Gln
100 105 110
Ala Met Phe Gly Trp His Ser Trp Gln Leu Leu Leu Ala Glu Leu Gly
115 120 125
Thr Leu Ala Leu Val Trp Tyr Ile Gly Thr Arg Gly Ala Ser Ser Ser
130 135 140
Ala Asn Leu Gln Thr Val Ile Ala Gly Leu Ile Val Ala Leu Ile Val
145 150 155 160
Ala Ile Trp Trp Ala Gly Asp Ile Lys Pro Ala Asn Ile Pro Phe Pro
165 170 175
Ala Pro Gly Asn Ile Glu Leu Thr Gly Leu Phe Ala Ala Leu Ser Val
180 185 190
Met Phe Trp Cys Phe Val Gly Leu Glu Ala Phe Ala His Leu Ala Ser
195 200 205
Glu Phe Lys Asn Pro Glu Arg Asp Phe Pro Arg Ala Leu Met Ile Gly
210 215 220
Leu Leu Leu Ala Gly Leu Val Tyr Trp Gly Cys Thr Val Val Val Leu
225 230 235 240
His Phe Asp Ala Tyr Gly Glu Lys Met Ala Ala Ala Ala Ser Leu Pro
245 250 255
Lys Ile Val Val Gln Leu Phe Gly Val Gly Ala Leu Trp Ile Ala Cys
260 265 270
Val Ile Gly Tyr Leu Ala Cys Phe Ala Ser Leu Asn Ile Tyr Ile Gln
275 280 285
Ser Phe Ala Arg Leu Val Trp Ser Gln Ala Gln His Asn Pro Asp His
290 295 300
Tyr Leu Ala Arg Leu Ser Ser Arg His Ile Pro Asn Asn Ala Leu Asn
305 310 315 320
Ala Val Leu Gly Cys Cys Val Val Ser Thr Leu Val Ile His Ala Leu
325 330 335
Glu Ile Asn Leu Asp Ala Leu Ile Ile Tyr Ala Asn Gly Ile Phe Ile
340 345 350
Met Ile Tyr Leu Leu Cys Met Leu Ala Gly Cys Lys Leu Leu Gln Gly
355 360 365
Arg Tyr Arg Leu Leu Ala Val Val Gly Gly Leu Leu Cys Val Leu Leu
370 375 380
Leu Ala Met Val Gly Trp Lys Ser Leu Tyr Ala Leu Ile Met Leu Ala
385 390 395 400
Gly Leu Trp Leu Leu Leu Pro Lys Arg Lys Thr Pro Glu Asn Gly Ile
405 410 415
Thr Thr
<210> 36
<211> 1257
<212> DNA
<213> Unknown
<220>
<223> YjeH NT
<400> 36
atgagtggac tcaaacaaga actggggctg gcccagggca ttggcctgct atcgacgtca 60
ttattaggca ctggcgtgtt tgccgttcct gcgttagctg cgctggtagc gggcaataac 120
agcctgtggg cgtggcccgt tttgattatc ttagtgttcc cgattgcgat tgtgtttgcg 180
attctgggtc gccactatcc cagcgcaggc ggcgtcgcgc acttcgtcgg tatggcgttt 240
ggttcgcggc ttgagcgagt caccggctgg ctgtttttat cggtcattcc cgtgggtttg 300
cctgccgcac tacaaattgc cgccgggttc ggccaggcga tgtttggctg gcatagctgg 360
caactgttgt tggcagaact cggtacgctg gcgctggtgt ggtatatcgg tactcgcggt 420
gccagttcca gtgctaatct acaaaccgtt attgccggac ttatcgtcgc gctgattgtc 480
gctatctggt gggcgggcga tatcaaacct gcgaatatcc cctttccggc acctggtaat 540
atcgaactta ccgggttatt tgctgcgtta tcagtgatgt tctggtgttt tgtcggtctg 600
gaggcatttg cccatctcgc ctcggaattt aaaaatccag agcgtgattt tcctcgtgct 660
ttgatgattg gtctgctgct ggcaggatta gtctactggg gctgtacggt agtcgtctta 720
cacttcgacg cctatggtga aaaaatggcg gcggcagcat cgcttccaaa aattgtagtg 780
cagttgttcg gtgtaggagc gttatggatt gcctgcgtga ttggctatct ggcctgcttt 840
gccagtctca acatttatat acagagcttc gcccgcctgg tctggtcgca ggcgcaacat 900
aatcctgacc actacctggc acgcctctct tctcgccata tcccgaataa tgccctcaat 960
gcggtgctcg gctgctgtgt ggtgagcact ttggtgattc atgctttaga gatcaatctg 1020
gacgctctta ttatttatgc caatggcatc tttattatga tttatctgtt atgcatgctg 1080
gcaggctgta aattattgca aggacgttat cgactactgg cggtggttgg cgggctgtta 1140
tgcgttctgt tactggcaat ggtcggctgg aaaagtctct atgcgctgat catgctggcg 1200
gggttatggc tgttgctgcc aaaacgaaaa acgccggaaa atggcataac cacataa 1257
<210> 37
<211> 88
<212> PRT
<213> Unknown
<220>
<223> NCgl2335 AA
<400> 37
Met Ala Tyr Thr Phe Asp His Val Val Ala Trp Arg Trp Cys Thr Lys
1 5 10 15
Glu Asp Ala Tyr Asn Tyr Thr His Leu Phe Asp Gln Leu Gln Pro Pro
20 25 30
Leu Ile Val Thr Thr Asp Gly Gln Lys Arg Arg Thr Gln Ser His His
35 40 45
His Asp Leu Ala Asp Asn Glu Asn Pro Thr Leu Pro Arg Pro Arg Gln
50 55 60
Thr Gln Arg Pro Lys Thr Arg His Pro Lys Thr Arg Ala Glu Leu Ala
65 70 75 80
Glu Lys His Ser Gly Val Ser Pro
85
<210> 38
<211> 267
<212> DNA
<213> Unknown
<220>
<223> NCgl2335 NT
<400> 38
gtggcctaca ccttcgacca cgtcgtcgcc tggcgctggt gcaccaaaga agacgcctac 60
aactacaccc acctcttcga tcaactccaa ccacccttaa tcgtgaccac cgacggacaa 120
aaaaggcgca ctcaaagcca tcaccacgac ctggccgaca acgaaaatcc aacgctgcct 180
cgtccacgtc aaacgcaacg tccaaaaaca cgtcacccta agacccgtgc tgagctcgcc 240
gaaaagcact ccggggtctc tccttga 267
<210> 39
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Tn_5 F
<400> 39
tgaattcgag ctcggtaccc caccgacgcg catctgcct 39
<210> 40
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Tn_5 R
<400> 40
ggtgtggtga ctttcagcag ttcccggggg ggaggaggca tgtggtgttg 50
<210> 41
<211> 50
<212> DNA
<213> Artificial Sequence
<220>
<223> Tn_3 F
<400> 41
caacaccaca tgcctcctcc cccccgggaa ctgctgaaag tcaccacacc 50
<210> 42
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> Tn_3 R
<400> 42
gtcgactcta gaggatcccc ctcccaaacc attgaggaat gg 42
<210> 43
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> CJ7_yjeH F
<400> 43
acaccacatg cctcctcccc agaaacatcc cagcgctac 39
<210> 44
<211> 43
<212> DNA
<213> Artificial Sequence
<220>
<223> CJ7_yjeH R
<400> 44
agttcttgtt tgagtccact catagtgttt cctttcgttg ggt 43
<210> 45
<211> 44
<212> DNA
<213> Artificial Sequence
<220>
<223> yjeH F
<400> 45
acccaacgaa aggaaacact atgagtggac tcaaacaaga actg 44
<210> 46
<211> 44
<212> DNA
<213> Artificial Sequence
<220>
<223> yjeH R
<400> 46
gactttcagc agttcccggg ttatgtggtt atgccatttt ccgg 44
<210> 47
<211> 418
<212> PRT
<213> Artificial Sequence
<220>
<223> yjeH F351L AA
<400> 47
Met Ser Gly Leu Lys Gln Glu Leu Gly Leu Ala Gln Gly Ile Gly Leu
1 5 10 15
Leu Ser Thr Ser Leu Leu Gly Thr Gly Val Phe Ala Val Pro Ala Leu
20 25 30
Ala Ala Leu Val Ala Gly Asn Asn Ser Leu Trp Ala Trp Pro Val Leu
35 40 45
Ile Ile Leu Val Phe Pro Ile Ala Ile Val Phe Ala Ile Leu Gly Arg
50 55 60
His Tyr Pro Ser Ala Gly Gly Val Ala His Phe Val Gly Met Ala Phe
65 70 75 80
Gly Ser Arg Leu Glu Arg Val Thr Gly Trp Leu Phe Leu Ser Val Ile
85 90 95
Pro Val Gly Leu Pro Ala Ala Leu Gln Ile Ala Ala Gly Phe Gly Gln
100 105 110
Ala Met Phe Gly Trp His Ser Trp Gln Leu Leu Leu Ala Glu Leu Gly
115 120 125
Thr Leu Ala Leu Val Trp Tyr Ile Gly Thr Arg Gly Ala Ser Ser Ser
130 135 140
Ala Asn Leu Gln Thr Val Ile Ala Gly Leu Ile Val Ala Leu Ile Val
145 150 155 160
Ala Ile Trp Trp Ala Gly Asp Ile Lys Pro Ala Asn Ile Pro Phe Pro
165 170 175
Ala Pro Gly Asn Ile Glu Leu Thr Gly Leu Phe Ala Ala Leu Ser Val
180 185 190
Met Phe Trp Cys Phe Val Gly Leu Glu Ala Phe Ala His Leu Ala Ser
195 200 205
Glu Phe Lys Asn Pro Glu Arg Asp Phe Pro Arg Ala Leu Met Ile Gly
210 215 220
Leu Leu Leu Ala Gly Leu Val Tyr Trp Gly Cys Thr Val Val Val Leu
225 230 235 240
His Phe Asp Ala Tyr Gly Glu Lys Met Ala Ala Ala Ala Ser Leu Pro
245 250 255
Lys Ile Val Val Gln Leu Phe Gly Val Gly Ala Leu Trp Ile Ala Cys
260 265 270
Val Ile Gly Tyr Leu Ala Cys Phe Ala Ser Leu Asn Ile Tyr Ile Gln
275 280 285
Ser Phe Ala Arg Leu Val Trp Ser Gln Ala Gln His Asn Pro Asp His
290 295 300
Tyr Leu Ala Arg Leu Ser Ser Arg His Ile Pro Asn Asn Ala Leu Asn
305 310 315 320
Ala Val Leu Gly Cys Cys Val Val Ser Thr Leu Val Ile His Ala Leu
325 330 335
Glu Ile Asn Leu Asp Ala Leu Ile Ile Tyr Ala Asn Gly Ile Leu Ile
340 345 350
Met Ile Tyr Leu Leu Cys Met Leu Ala Gly Cys Lys Leu Leu Gln Gly
355 360 365
Arg Tyr Arg Leu Leu Ala Val Val Gly Gly Leu Leu Cys Val Leu Leu
370 375 380
Leu Ala Met Val Gly Trp Lys Ser Leu Tyr Ala Leu Ile Met Leu Ala
385 390 395 400
Gly Leu Trp Leu Leu Leu Pro Lys Arg Lys Thr Pro Glu Asn Gly Ile
405 410 415
Thr Thr
<210> 48
<211> 1257
<212> DNA
<213> Artificial Sequence
<220>
<223> yjeH F351L NT
<400> 48
atgagtggac tcaaacaaga actggggctg gcccagggca ttggcctgct atcgacgtca 60
ttattaggca ctggcgtgtt tgccgttcct gcgttagctg cgctggtagc gggcaataac 120
agcctgtggg cgtggcccgt tttgattatc ttagtgttcc cgattgcgat tgtgtttgcg 180
attctgggtc gccactatcc cagcgcaggc ggcgtcgcgc acttcgtcgg tatggcgttt 240
ggttcgcggc ttgagcgagt caccggctgg ctgaatttat cggtcattcc cgtgggtttg 300
cctgccgcac tacaaattgc cgccgggttc ggccaggcga tgtttggctg gcatagctgg 360
caactgttgt tggcagaact cggtacgctg gcgctggtgt ggtatatcgg tactcgcggt 420
gccagttcca gtgctaatct acaaaccgtt attgccggac ttatcgtcgc gctgattgtc 480
gctatctggt gggcgggcga tatcaaacct gcgaatatcc cctttccggc acctggtaat 540
atcgaactta ccgggttatt tgctgcgtta tcagtgatgt tctggtgttt tgtcggtctg 600
gaggcatttg cccatctcgc ctcggaattt aaaaatccag agcgtgattt tcctcgtgct 660
ttgatgattg gtctgctgct ggcaggatta gtctactggg gctgtacggt agtcgtctta 720
cacttcgacg cctatggtga aaaaatggcg gcggcagcat cgcttccaaa aattgtagtg 780
cagttgttcg gtgtaggagc gttatggatt gcctgcgtga ttggctatct ggcctgcttt 840
gccagtctca acatttatat acagagcttc gcccgcctgg tctggtcgca ggcgcaacat 900
aatcctgacc actacctggc acgcctctct tctcgccata tcccgaataa tgccctcaat 960
gcggtgctcg gctgctgtgt ggtgagcact ttggtgattc atgctttaga gatcaatctg 1020
gacgctctta ttatttatgc caatggcatc cttattatga tttatctgtt atgcatgctg 1080
gcaggctgta aattattgca aggacgttat cgactactgg cggtggttgg cgggctgtta 1140
tgcgttctgt tactggcaat ggtcggctgg aaaagtctct atgcgctgat catgctggcg 1200
gggttatggc tgttgctgcc aaaacgaaaa acgccggaaa atggcataac cacataa 1257
<210> 49
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> F351L F
<400> 49
caatggcatc cttattatga ttt 23
<210> 50
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> F351L R
<400> 50
aaatcataat aaggatgcca ttg 23
<210> 51
<211> 127
<212> PRT
<213> Artificial Sequence
<220>
<223> Обшая формула 1(GltA 289~415)
<220>
<221> MISC_FEATURE
<222> (1)
<223> Xaa представляет собой N или S.
<220>
<221> MISC_FEATURE
<222> (9)
<223> Xaa представляет собой A или E.
<220>
<221> MISC_FEATURE
<222> (73)
<223> Xaa представляет собой Y или C.
<400> 51
Xaa Asn His Gly Gly Asp Ala Thr Xaa Phe Met Asn Lys Val Lys Asn
1 5 10 15
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
20 25 30
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
35 40 45
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
50 55 60
Glu Glu Ile Ala Leu Ala Asp Asp Xaa Phe Ile Ser Arg Lys Leu Tyr
65 70 75 80
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
85 90 95
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
100 105 110
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn His
115 120 125
<---
Изобретение относится к биотехнологии и представляет собой полипептид, вовлеченный в получение L-аминокислоты, в котором лизин, который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином, где полипептид имеет идентичность последовательности 80% или более с аминокислотной последовательностью SEQ ID NO: 1. Изобретение относится также к способу получения L-аминокислот с использованием микроорганизма, трансформированного полинуклеотидом, кодирующим указанный пептид. Изобретение позволяет получать L-аминокислоты с высокой степенью эффективности. 5 н. и 9 з.п. ф-лы, 10 табл.
1. Полипептид, вовлеченный в получение L-аминокислоты, в котором лизин, который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином, где полипептид имеет идентичность последовательности 80% или более с аминокислотной последовательностью SEQ ID NO: 1.
2. Полипептид по п. 1, который имеет идентичность последовательности 90% или более с аминокислотной последовательностью SEQ ID NO: 1.
3. Полипептид по п. 1, содержащий аминокислотную последовательность SEQIDNO: 3.
4. Полипептид по п. 1, содержащий аминокислотную последовательность общей формулы 1, приведенной ниже:
общая формула 1
X1N_HGGDATX2FMN_KVKNKEDGVR_LMGFGHRVYK_NYDPRAAIVK_ETAHEILEHLGGDDLLDLAI_KLEEIALADD_X3FISRKLYPN_VDFYTGLIYR_AMGFPTDFFT_VLFAIGRLPGWIAHYREQLGAAGNH (SEQ ID NO: 51),
где в приведенной выше общей формуле 1:
X1 представляет собой аспарагин или серин;
X2 представляет собой аланин или глутаминовую кислоту; и
X3 представляет собой тирозин или цистеин.
5. Полипептид по п. 1, который имеет идентичность последовательности 90% или более с аминокислотной последовательностью SEQ ID NO: 8, 10 или 12.
6. Полинуклеотид, кодирующий полипептид по любому из пп. 1-5.
7. Микроорганизм рода Corynebacterium для получения L-аминокислоты, содержащий полипептид, вовлеченный в получение L-аминокислоты, в котором лизин, который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином, где полипептид имеет идентичность последовательности 80% или более с аминокислотной последовательностью SEQ ID NO: 1, или полинуклеотид, кодирующий данный полипептид.
8. Микроорганизм по п. 7, который обладает способностью продуцировать L-валин или O-ацетил-L-гомосерин.
9. Микроорганизм по п. 7, который представляет собой Corynebacterium glutamicum.
10. Способ получения L-аминокислот, включающий: культивирование в среде микроорганизма рода Corynebacterium, который содержит полипептид, вовлеченный в получение L-аминокислоты, в котором лизин, который представляет собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином, где полипептид имеет идентичность последовательности 80% или более с аминокислотной последовательностью SEQ ID NO: 1, или полинуклеотид, кодирующий этот полипептид.
11. Способ по п. 10, дополнительно включающий выделение L-аминокислот из культуральной среды или культивируемого микроорганизма.
12. Способ по п. 10, где L-аминокислота представляет собой L-валин, O-ацетил-L-гомосерин или L-метионин.
13. Композиция для получения L-аминокислот, содержащая:
(1) микроорганизм рода Corynebacterium, который содержит полипептид, вовлеченный в получение L-аминокислоты, в котором лизин, представляющий собой аминокислоту, соответствующую положению 415 в аминокислотной последовательности SEQ ID NO: 1, заменен гистидином, где полипептид имеет идентичность последовательности 80% или более с аминокислотной последовательностью SEQ ID NO: 1, или полинуклеотид, кодирующий этот полипептид; и
(2) среду, на которой выращивают микроорганизм; или эксципиент.
14. Композиция по п. 13, где L-аминокислота представляет собой L-валин, O-ацетил-L-гомосерин или L-метионин.
| US 20080014618 A1, 17.01.2008 | |||
| EP 3783096 A1, 24.02.2021 | |||
| KR 101915433 B1, 05.11.2018 | |||
| Модифицированный полипептид с пониженной активностью цитратсинтазы и способ получения L-аминокислоты с использованием этого полипептида | 2019 |
|
RU2732815C1 |
Авторы
Даты
2025-03-20—Публикация
2022-03-10—Подача