Настоящее изобретение относится к лечению неврологических нарушений.
Неврологические нарушения включают нейронные повреждения, нейродегенеративные нарушения, сенсорные нарушения и вегетативные нарушения.
Нейронные повреждения, такие как травмы спинного мозга (SCI), вызывают дегенерацию поврежденных аксонов, препятствующую нормальной сенсорной, моторной и вегетативной функции Восстановление может происходить за счет эндогенных механизмов, таких как регенерация поврежденных аксонов и коллатеральное прорастание неповрежденных аксонов, что приводит к реиннервации денервированных мишеней. Однако регенеративная способность поврежденных нейронов (особенно спинного мозга) ограничена у взрослых млекопитающих, и пациенты могут страдать различными нарушениями, которые сильно влияют на качество жизни.
Обычные методы лечения нейронных повреждений включают интерлейкин-6 (IL-6) и трансплантацию стволовых клеток, однако немногие из них находятся на стадии разработки для использования в клинике. Поэтому сохраняется потребность в терапевтических средствах для лечения нейронных повреждений, способных стимулировать рост или восстановление нейронов.
Бактерии рода Clostridia продуцируют сильнодействующие и специфические белковые токсины, которые могут отравлять нейроны и другие клетки, в которые они доставляются Примеры таких клостридиальных токсинов включают нейротоксины, продуцируемые C. tetani (TeNT) и C. botulinum (BoNT) серотипы AG и X (см. WO 2018/009903 A2), а также продуцируемые C. baratii и C. butyricum.
Среди клостридиальных нейротоксинов имеются некоторые из наиболее сильнодействующих известных токсинов. Например, ботулинические нейротоксины имеют значения средней летальной дозы (LD50) для мышей в диапазоне от 0,5 до 5 нг/кг, в зависимости от серотипа. И столбнячные, и ботулинические токсины действуют путем ингибирования функции пораженных нейронов, в частности высвобождения нейротрансмиттеров. В то время как ботулинический токсин действует на нервно-мышечное соединение и ингибирует холинергическую трансмиссию в периферической нервной системе, столбнячный токсин действует в центральной нервной системе.
В природе клостридиальные нейротоксины синтезируются в виде одноцепочечного полипептида, который пост-трансляционно модифицируется в результате протеолитического расщепления с образованием двух полипептидных цепей, соединенных дисульфидной связью. Расщепление происходит в определенном сайте расщепления, часто называемом сайтом активации, который расположен между цистеиновыми остатками, которые обеспечивают межцепочечную дисульфидную связь. Именно эта двухцепочечная форма является активной формой токсина. Две цепи называются тяжелой цепью (Н-цепью), которая имеет молекулярную массу примерно 100 кДа, и легкой цепью (L-цепь), которая имеет молекулярную массу примерно 50 кДа. Н-цепь содержит N-концевой транслокационный компонент (HN домен) и С-концевой таргетный компонент (HC домен). Сайт расщепления расположен между компонентами L-цепи и домена транслокации. После связывания HC домена с его нейроном-мишенью и интернализации связанного токсина в клетку через эндосому, HN домен перемещает L-цепь через эндосомальную мембрану в цитозоль, и L-цепь обеспечивает функцию протеазы (также известной как не цитотоксическая протеаза).
Не цитотоксические протеазы действуют путем протеолитического расщепления внутриклеточных транспортных белков, известных как белки SNARE (например, SNAP-25, VAMP или Syntaxin). Аббревиатура SNARE происходит от термина «растворимый NSF рецептор стыковочных белков», где NSF означает «чувствительный к N-этилмалеимиду». Белки SNARE являются неотъемлемой частью слияния внутриклеточных везикул и, таким образом, секреции молекул через везикулярный транспорт из клетки. Функция протеазы представляет собой цинк-зависимую эндопептидазную активность и проявляет высокую субстратную специфичность для белков SNARE. Соответственно, после доставки в желаемую клетку-мишень, не цитотоксическая протеаза способна ингибировать клеточную секрецию из клетки-мишени. Протеазы L-цепи клостридиальных нейротоксинов представляют собой не цитотоксические протеазы, которые расщепляют белки SNARE.
Ввиду повсеместной природы белков SNARE, клостридиальные нейротоксины, такие как ботулинический токсин, успешно применяют в широком диапазоне методов лечения.
В WO 2016/170501 A1 описано применение каталитически активного полноразмерного BoNT/A (содержащего L-цепь и полную H-цепь, включая домены HN и HC) для лечения паралича, вызванного повреждением спинного мозга. В WO 2016 /170501 A1 показано, что каждый из функциональных доменов BoNT/A необходим для наблюдаемых терапевтических эффектов, включая возможности связывания и транслокации H-цепи и не цитотоксическую протеазную активность L-цепи. Как описано выше, полноразмерные клостридиальные нейротоксины являются чрезвычайно сильнодействующими, что требует принятия специальных мер безопасности при обращении с токсином. Кроме того, считается, что распространение токсина из ткани-мишени является причиной нежелательных побочных эффектов, которые, в крайних случаях, могут быть опасными для жизни. Это может вызывать особую озабоченность при использовании лекарственных средств на основе клостридиальных нейротоксинов (таких как BoNT лекарственные средства) в высоких дозах, концентрациях и объемах введения. Нежелательные эффекты, связанные с этой проблемой, о которых сообщалось в отношении коммерческих терапевтических средств BoNT/A, относятся астения, общая мышечная слабость, диплопия, птоз, дисфагия, дисфония, дизартрия, недержание мочи и затрудненное дыхание. Затруднения при глотании и дыхании могут быть опасными для жизни, и были зарегистрированы случаи смерти, связанные с распространением эффектов токсина. Таким образом, существует потребность в более безопасном терапевтическом средстве для стимуляции роста или восстановления нейронов.
Учитывая их размер, использование полноразмерных клостридиальных нейротоксинов (~150 кДа) или их полных Н-цепей (~100 кДа) связано с повышенным риском возникновения иммунного ответа у субъекта, получающего лечение указанным полипептидом. Более того, присутствие всей Н-цепи (и, в частности, НС домена) приводит к связыванию полипептида с рецепторами-мишенями клостридиального нейротоксина, что может быть связано с нежелательными эффектами вне мишени у субъекта, которому вводят указанный полипептид.
Настоящее изобретение преодолевает одну или более вышеупомянутых проблем.
Авторы настоящего изобретения неожиданно обнаружили, что полипептид, содержащий L-цепь клостридиального нейротоксина и/или фрагмент Н-цепи клостридиального нейротоксина (например, домен транслокации (HN) или рецептор-связывающий домен (HC) способствует росту или восстановления нейронов, и, таким образом, находит применение в лечении неврологических нарушений. Преимущественно, это позволяет использовать не токсичные (или по существу не токсичные) фрагменты клостридиальных нейротоксинов, которые, учитывая меньший размер (по сравнению с полноразмерной Н-цепью или полноразмернымй клостридиальным нейротоксином), с меньшей вероятностью вызывают иммунный ответ у субъекта, которому вводят указанные фрагменты. Более того, не токсичные (или по существу не токсичные) фрагменты менее дорогие и/или менее сложные в производстве, чем полноразмерные клостридиальные нейротоксины. Дополнительно, не токсичные (или по существу не токсичные) фрагменты представляют собой более четко определенные терапевтические средства, чем полноразмерные клостридиальные токсины, и, учитывая более короткую длину полипептидов, снижается вероятность, например, шаффлинга цистеина между доменами.
Таким образом, в одном аспекте, изобретение относится к полипептиду для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит:
легкую цепь клостридиального нейротоксина (L-цепь) или ее фрагмент; и/или
фрагмент тяжелой цепи клостридиального нейротоксина (Н-цепь).
В родственном аспекте изобретение относится к способу стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, включающему введение субъекту полипептида, где полипептид содержит:
L-цепь клостридиального нейротоксина или ее фрагмент; и/или
фрагмент Н-цепи клостридиального нейротоксина.
В другом аспекте изобртение относится к применению полипептида в получении лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит:
L-цепь клостридиального нейротоксина или ее фрагмент и/или
фрагмент Н-цепи клостридиального нейротоксина.
В одном аспекте, изобретение относится к полипептиду для применения в лечении неврологического нарушения у субъекта, где полипептид содержит:
легкую цепь клостридиального нейротоксина (L-цепь) или ее фрагмент; и/или
фрагмент тяжелой цепи клостридиального нейротоксина (Н-цепь).
В родственном аспекте изобртение относится к способу лечения неврологического нарушения у субъекта, включающему введение субъекту полипептида, где полипептид содержит:
L-цепь клостридиального нейротоксина или ее фрагмент; и/или
фрагмент Н-цепи клостридиального нейротоксина.
В другом аспекте изобртение относится к применению полипептида в получении лекарственного средства для лечения неврологического нарушения у субъекта, где полипептид содержит:
L-цепь клостридиального нейротоксина или ее фрагмент и/или
фрагмент Н-цепи клостридиального нейротоксина.
В одном варианте осуществления, полипептид по изобретению содержит L-цепь клостридиального нейротоксина. Предпочтительно, чтобы L-цепь была каталитически неактивной.
Таким образом, в одном аспекте изобретение относится к полипептиду для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит каталитически неактивную L-цепь клостридиального нейротоксина.
В родственном аспекте, изобретение относится к способу стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где способ включает введение субъекту полипептида, где полипептид содержит каталитически неактивную L-цепь клостридиального нейротоксина.
В другом родственном аспекте, изобретение относится к применению полипептида, содержащего каталитически неактивную L-цепь клостридиального нейротоксина, в получении лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта.
В одном аспекте, изобретение относится к полипептиду для лечения неврологического нарушения у субъекта, где полипептид содержит каталитически неактивную L-цепь клостридиального нейротоксина.
В родственном аспекте, изобретение относится к способу лечения неврологического нарушения у субъекта, включающему введение субъекту полипептида, где полипептид содержит каталитически неактивную L-цепь клостридиального нейротоксина.
В другом родственном аспекте, изобретение относится к применению полипептида, содержащего каталитически неактивную L-цепь клостридиального нейротоксина, в получении лекарственного средства для лечения неврологического нарушения у субъекта.
Авторы настоящего изобретения были первыми, кто показал, что каталитическая активность L-цепи клостридиального нейротоксина не является необходимой для стимуляции роста или восстановления нейронов. Таким образом, настоящее изобретение позволяет обеспечить более безопасное (менее токсичное) терапевтическое средство.
Активная L-цепь клостридиального нейротоксина обладает не цитотоксической протеазной активностью. В частности, активная L-цепь клостридиального нейротоксина обладает эндопептидазной активностью и способна расщеплять белок аппарата экзоцитарного слияния в клетке-мишени. Белком аппарата экзоцитарного слияния предпочтительно является белок SNARE, такой как SNAP-25, синаптобревин/ VAMP или синтаксин.
Термин «каталитически неактивный», используемый в настоящем изобретении в отношении L-цепи клостридиального нейротоксина, означает, что указанная L-цепь по существу не проявляет не цитотоксической протеазную активность, предпочтительно термин «каталитически неактивный», используемый в настоящем изобретении в отношении L-цепи клостридиального нейротоксина означает, что указанная L-цепь не проявляет не цитотоксическую протеазную активность. В одном варианте осуществления, каталитически неактивная L-цепь клостридиального нейротоксина представляет собой цепь, которая не расщепляет белок аппарата экзоцитарного слияния в клетке-мишени. Термин «по существу отсутствие не цитотоксической протеазной активности» означает, что L-цепь клостридиального нейротоксина имеет менее 5% не цитотоксической протеазной активности каталитически активной L-цепи клостридиального нейротоксина, например, менее 2%, 1% или, предпочтительно, менее 0,1% не цитотоксической протеазной активности каталитически активной L-цепи клостридиального нейротоксина. Не цитотоксическую протеазную активность можно определить in vitro путем инкубации L-цепи тестируемого клостридиального нейротоксина с белком SNARE и сравнения количества белка SNARE, расщепленного L-цепью тестируемого клостридиального нейротоксина, по сравнению с количеством белка SNARE, расщепленного каталитически активной L-цепью клостридиального нейротоксина в тех же условиях. Обычные методики, такие как SDS-PAGE и вестерн-блоттинг могут применяться для количественного определения количества расщепленного белка SNARE. Подходящие анализы in vitro описаны в WO 2019/145577 A1, которая включена в настоящее описание посредством ссылки.
Анализы на основе клеток и in vivo также можно использовать для определения того, обладает ли клостридиальный нейротоксин, содержащий L-цепь и функциональный домен клеточного связывания и транслокации, не цитотоксической протеазной активностью. Анализы, такие как Шкала отведения пальцев (DAS), анализ дорсальных корешковых ганглиев (DRG), анализ нейронов спинного мозга (SCN) и анализ гемидиафрагмы диафрагмального нерва мыши (PNHD) являются обычными в данной области техники. Подходящим анализом для определения не цитотоксической протеазной активности может быть анализ, описанный в Donald et al. (2018), Pharmacol Res Perspect, e00446, 1-14, которая включена сюда посредством ссылки.
Каталитически неактивная L-цепь может иметь одну или более мутаций, которые инактивируют указанную каталитическую активность. Например, каталитически неактивная L-цепь BoNT/A может содержать мутацию остатка активного сайта, такого как His223, Glu224, His227, Glu262, и/или Tyr366. Нумерация положений соответствует положениям аминокислот SEQ ID NO: 62 и может быть определена выравниванием полипептида с SEQ ID NO: 62. Так как наличие метионинового остатка в положении 1 SEQ ID NO: 62 является необязательным, специалист в данной области техники примет во внимание наличие/отсутствие метионинового остатка при определении нумерации аминокислотных остатков. Например, если SEQ ID NO: 62 включает метионин, нумерация положений будет такой, как определено выше (например, His223 будет His223 из SEQ ID NO: 62). Аналогично, если метионин отсутствует в SEQ ID NO: 62, нумерация аминокислотных остатков должна быть изменена на -1 (например, His223 будет His222 из SEQ ID NO: 62). Те же соображения применяют, когда метионин в положение 1 других полипептидных последовательностей, описанных в настоящем изобретении, присутствует/отсутствует, и специалист в данной области техники без труда определит правильную нумерацию аминокислотных остатков, используя методики, общепринятые в данной области техники.
В особенно предпочтительном варианте осуществления, полипептид по настоящему изобретению может содержать модифицированный BoNT/A или его фрагмент (предпочтительно НC домен BoNT/A или его фрагмент). Модифицированный BoNT/A или его фрагмент может содержать модификацию в один или более аминокислотных остатков, выбранных из: ASN 886, ASN 905, GLN 915, ASN 918, GLU 920, ASN 930, ASN 954, SER 955, GLN 991, GLU 992, GLN 995, ASN 1006, ASN 1025, ASN 1026, ASN 1032, ASN 1043, ASN 1046, ASN 1052, ASP 1058, HIS 1064, ASN 1080, GLU 1081, GLU 1083, ASP 1086, ASN 1188, ASP 1213, GLY 1215, ASN 1216, GLN 1229, ASN 1242, ASN 1243, SER 1274 и THR 1277. Такой модифицированный BoNT/A или его фрагмент может демонстрировать снижение или отсутствие побочных эффектов по сравнению с использованием известного BoNT/A. Повышенные свойства удержания в тканях модифицированного BoNT/A по настоящему изобретению может также обеспечивать повышенную активность и/или продолжительность действия, и может позволять использовать меньшие дозы по сравнению с известными терапевтическими средствами на основе клостридиального токсина (или повышенные дозы без каких-либо дополнительных побочных эффектов), что дает дополнительные преимущества.
Модификация может быть модификацией по сравнению с не модифицированным BoNT/A, показанным как SEQ ID NO: 62, где нумерация аминокислотных остатков определяется путем выравнивания с SEQ ID NO: 62. Поскольку наличие метионинового остатка в положении 1 SEQ ID NO: 62 (а также SEQ ID NO, соответствующих модифицированным полипептидам BoNT/A или их фрагментам, описанным в настоящем изобретении) является необязательным, специалист в данной области техники примет во внимание наличие/отсутствие метионинового остатка при определении нумерации аминокислотных остатков. Например, если SEQ ID NO: 62 включает метионин, нумерация положений будет такой, как определено выше (например, ASN 886 будет ASN 886 SEQ ID NO: 62). Альтернативно, если метионин отсутствует в SEQ ID NO: 2, нумерация аминокислотных остатков должна быть изменена на -1 (например, ASN 886 будет ASN 885 SEQ ID NO: 62). Аналогичные соображения применимы, когда метионин в положении 1 других полипептидных последовательностей, описанных в настоящем изобретении, присутствует/отсутствует, и специалист в данной области техники сможет легко определить правильную нумерацию аминокислотных остатков, используя методы, известные в данной области техники.
Аминокислотные остатки, указанные выше для модификации, представляют собой аминокислотные остатки, экспонированные на поверхности.
Модифицированный BoNT/A или его фрагмент может содержать модификацию одного или более аминокислотных остатков, выбранных из: ASN 886, ASN 930, ASN 954, SER 955, GLN 991, ASN 1025, ASN 1026, ASN 1052, ASN 1188, ASP 1213, GLY 1215, ASN 1216, GLN 1229, ASN 1242, ASN 1243, SER 1274 и THR 1277.
Термин «один или более аминокислотных остатков» при использовании в контексте модифицированного BoNT/A или его фрагмента предпочтительно означает по меньшей мере 2, 3, 4, 5, 6 или 7 указанных аминокислотных остатков. Таким образом, модифицированный BoNT/A может содержать по меньшей мере 2, 3, 4, 5, 6 или 7 (предпочтительно, 7) модификаций на указанных аминокислотных остатках. Модифицированный BoNT/A или его фрагмент может содержать 1-30, 3-20 или 5-10 аминокислотных модификаций. Более предпочтительно, термин «один или более аминокислотных остатков» при использовании в контексте модифицированного BoNT/A или его фрагмента означает все указанные аминокислотные остатки.
Предпочтительно, кроме одной или нескольких аминокислотных модификаций в указанном(ых) аминокислотном(ых) остатке(ах), модифицированный BoNT/A или его фрагмент не содержит каких-либо дополнительных аминокислотных модификаций по сравнению с SEQ ID NO: 62.
Модификация может быть выбрана из:
i. замена кислого аминокислотного остатка, экспонированного на поверхности, основным аминокислотным остатком;
ii. замена кислого аминокислотного остатка, экспонированного на поверхности, незаряженным аминокислотным остатком;
iii. замена незаряженного аминокислотного остатка, экспонированного на поверхности, основным аминокислотным остатком;
iv. вставка основного аминокислотного остатка и
v. делеция кислого аминокислотного остатка, экспонированного на поверхности.
Модификация, как указано выше, приводит к модифицированному BoNT/A или его фрагменту, который имеет повышенный положительный поверхностный заряд и повышенную изоэлектрическую точку по сравнению с соответствующим немодифицированным BoNT/A или его фрагментом.
Изоэлектрическая точка (pI) является специфическим свойством данного белка. Как хорошо известно в данной области техники, белки состоят из определенной последовательности аминокислот (также называемой в белке аминокислотными остатками). Каждая из аминокислот стандартного набора из двадцати имеет разные боковые цепи (или группу R), что означает, что каждый аминокислотный остаток в белке проявляет различные химические свойства, такие как заряд и гидрофобность. На эти свойства может влиять окружающая химическая среда, такая как температура и pH. Общие химические характеристики белка будут зависеть от суммы этих различных факторов.
Некоторые аминокислотные остатки (подробно описанные ниже) обладают ионизируемыми боковыми цепями, которые могут проявлять электрический заряд в зависимости от окружающего pH. Заряжена ли или нет такая боковая цепь при данном pH, зависит от pKa соответствующей ионизируемой группы, где pKa представляет собой отрицательный логарифм константы диссоциации кислоты (Ka) для указанного протона из конъюгированного основания.
Например, кислотные остатки, такие как аспарагиновая кислота и глутаминовая кислота, имеют карбоксильные группы боковой цепи со значениями pKa приблизительно 4,1 (точные значения pKa могут зависеть от температуры, ионной силы и микроокружения ионизируемой группы). Таким образом, эти боковые цепи имеют отрицательный заряд при рН 7,4 (часто называемом «физиологическим рН»). При низких значениях рН эти боковые цепи становятся протонированными и теряют свой заряд.
И наоборот, основные остатки, такие как лизин и аргинин, имеют азотсодержащие группы боковых цепей со значениями pKa приблизительно 10-12. Следовательно, эти боковые цепи демонстрируют положительный заряд при pH 7,4. Эти боковые цепи станут депротонированными и теряют заряд при высоких значениях рН.
Таким образом, общий (суммарный) заряд белковой молекулы зависит от количества кислотных и основных остатков, присутствующих в белке (и степени их экспонирования на поверхности), а также от pH окружающей среды. Изменение pH окружающей среды изменяет общий заряд белка. Соответственно, для каждого белка существует заданный рН, при котором количество положительных и отрицательных зарядов равно, и белок не проявляет общий суммарный заряд. Эта точка известна как изоэлектрическая точка (pI). Изоэлектрическая точка является стандартным понятием в биохимии белков, с которым специалист в данной области техники должен быть знаком.
Таким образом, изоэлектрическая точка (pI) определяется как значение pH, при котором суммарный заряд белка равен нулю. Увеличение pI означает, что для того, чтобы белок имел нулевой суммарный заряд, требуется более высокое значение pH. Таким образом, увеличение pI представляет собой увеличение суммарного положительного заряда белка при заданном pH. Наоборот, уменьшение pI означает, что для того, чтобы белок имел нулевой суммарный заряд, требуется более низкое значение pH. Таким образом, уменьшение pI представляет собой уменьшение суммарного положительного заряда белка при данном рН.
Способы определения pI белка известны в данной области техники и должны быть знакомы специалисту в данной области техники. Например, pI белка можно рассчитать из средних значений pKa каждой аминокислоты, присутствующей в белке («расчетный pI»). Такие расчеты могут быть выполнены с использованием компьютерных программ, известных в данной области техники, таких как Compute pI/MW Tool от ExPASy (https://web.expasy.org/compute_pi/), который является предпочтительным способом расчета pI в соответствии с настоящим изобретением. Сравнения значений pI между различными молекулами следует проводить с использованием одной и той же методики/программы расчета.
При необходимости, расчетный pI белка может быть подтвержден экспериментально с использованием метода изоэлектрического фокусирования («наблюдаемый pI»). В этом методе используют электрофорез для разделения белков в соответствии с их pI. Изоэлектрическое фокусирование обычно выполняют с использованием геля с иммобилизованным pH градиентом. Когда применяют электрическое поле, белок мигрирует через pH градиент, пока не достигнет pH, при котором он имеет нулевой суммарный заряд, эта точка является pI белка. Результаты, полученные с помощью изоэлектрического фокусирования, обычно имеют относительно низкое разрешение в природе, и таким образом, авторы настоящего изобретения считают, что результаты, полученные с помощью расчетного pI (как описано выше), являются более подходящими для использования.
В настоящем описании «pI» означает «расчетный pI», если не указано иное.
pI белка можно увеличить или уменьшить, изменив количество основных и/или кислотных групп, находящихся на его поверхности. Этого можно добиться путем модификации одной или нескольких аминокислот белка. Например, увеличение pI может быть получено за счет уменьшения количества кислотных остатков или за счет увеличения количества основных остатков.
Модифицированный BoNT/A или его фрагмент по изобретению может иметь значение pI, которое, по меньшей мере, на 0,2, 0,4, 0,5 или 1 единицу pI выше, чем у не модифицированного BoNT/A (например, SEQ ID NO: 62) или его фрагмента. Предпочтительно, модифицированный BoNT/A или его фрагмент может иметь pI, по меньшей мере, 6,6, например, по меньшей мере, 6,8.
Свойства 20 стандартных аминокислот указаны в таблице ниже:
Следующие аминокислоты считаются заряженными аминокислотами: аспарагиновая кислота (отрицательная), глутаминовая кислота (отрицательная), аргинин (положительная) и лизин (положительная).
При pH 7,4 боковые цепи аспарагиновой кислоты (pKa 3,1) и глутаминовой кислоты (pKa 4,1) имеют отрицательный заряд, в то время как боковые цепи аргинина (pKa 12,5) и лизина (pKa 10,8) имеют положительный заряд. Аспарагиновая кислота и глутаминовая кислота относятся к кислым аминокислотным остаткам. Аргинин и лизин относятся к основным аминокислотным остаткам.
Следующие аминокислоты считаются незаряженными, полярными (то есть могут участвовать в связывании водорода) аминокислотами: аспарагин, глутамин, гистидин, серин, треонин, тирозин, цистеин, метионин и триптофан.
Следующие аминокислоты считаются незаряженными, гидрофобными аминокислотами: аланин, валин, лейцин, изолейцин, фенилаланин, пролин и глицин.
При аминокислотной вставке, дополнительный аминокислотный остаток (который обычно не присутствует) включается в последовательность полипептида BoNT/A или его фрагмент, таким образом увеличивая общее количество аминокислотных остатков в указанной последовательности. При аминокислотной делеции, аминокислотный остаток удаляется из аминокислотной последовательности клостридиального токсина, тем самым уменьшая общее количество аминокислотных остатков в указанной последовательности.
Предпочтительно, модификация представляет собой замену, при которой предпочтительно сохраняется такое же количество аминокислотных остатков в модифицированном BoNT/A или его фрагменте. При аминокислотной замене, аминокислотный остаток, который образует часть полипептидной последовательности BoNT/A или фрагмента, который заменяется другим аминокислотным остатком. Заменяющий аминокислотный остаток может быть одной из 20 стандартных аминокислот, как описано выше. Альтернативно, замещающая аминокислота при аминокислотной замене может быть не стандартной аминокислотой (аминокислотой, которая не является частью стандартного набора из 20, описанного выше). Например, замещающая аминокислота может представлять собой основную не стандартную аминокислоту, например L-орнитин, L-2-амино-3-гуанидинопропионовую кислоту или D-изомеры лизина, аргинина и орнитина). Способы введения не стандартных аминокислот в белки известны в данной области техники и включают синтез рекомбинантного белка с использованием E.coli ауксотрофных хозяев экспрессии.
В одном варианте осуществления, замена выбрана из: замены кислого аминокислотного остатка на основной аминокислотный остаток, замены кислого аминокислотного остатка на незаряженный аминокислотный остаток и замены незаряженного аминокислотного остатка на основной аминокислотный остаток. В одном варианте осуществления, где замена представляет собой замену кислого аминокислотного остатка незаряженным аминокислотным остатком, кислый аминокислотный остаток заменяется соответствующим ему незаряженным амидным аминокислотным остатком (т.е. аспарагиновая кислота заменяется на аспарагин, и глутаминовая кислота заменяется на глутамин).
Предпочтительно, основной аминокислотный остаток представляет собой лизиновый остаток или аргининовый остаток. Другими словами, замена представляет собой замену на лизин или аргинин. Наиболее предпочтительно, модификация представляет собой замену на лизин.
Предпочтительно, модифицированный BoNT/A или его фрагмент для использования в изобретении содержит от 4 до 40 аминокислотных модификаций, расположенных в домене HCN клостридиального токсина. Указанный модифицированный BoNT/A или его фрагмент предпочтительно также имеет pI, по меньшей мере, 6,6. Указанный модифицированный BoNT/A предпочтительно содержит модификации по меньшей мере 4 аминокислот, выбранных из: ASN 886, ASN 930, ASN 954, SER 955, GLN 991, ASN 1025, ASN 1026 и ASN 1052, где указанная модификация включает замену аминокислот с лизиновым остатком или аргининовым остатком. Например, указанный модифицированный BoNT/A или его фрагмент могут содержать модификации по меньшей мере 5 аминокислот, выбранных из: ASN 886, ASN 930, ASN 954, SER 955, GLN 991, ASN 1025, ASN 1026, ASN 1052 и GLN 1229, где указанная модификация включает замену аминокислот лизиновым остатком или аргининовым остатком.
Способы модификации белков путем замены, вставки или делеции аминокислотных остатков известны в данной области техники. Например, аминокислотные модификации могут быть введены путем модификации последовательности ДНК, кодирующей полипептид (например, кодирующей не модифицированный BoNT/A или его фрагмент). Этого можно достичь с помощью стандартных методов молекулярного клонирования, например, путем сайт-направленного мутагенеза, при котором короткие цепи ДНК (олигонуклеотиды), кодирующие желаемую аминокислоту(ы), используют для замены исходной кодирующей последовательности с использованием фермента полимеразы, или путем вставки/делеции частей гена с помощью различных ферментов (например, лигаз и рестрикционных эндонуклеаз). Альтернативно, можно химически синтезировать модифицированную последовательность гена.
В одном аспекте, изобретение относится к полипептиду для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
В родственном аспекте, представлен способ стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где способ включает введение субъекту полипептида, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
В еще одном родственном аспекте изобретение относится к применению полипептида в получении лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или при этом полипептид содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
В одном аспекте, изобретение относится к полипептиду для применения в лечении неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере на 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
В родственном аспекте изобретение относится к способу лечения неврологического нарушения у субъекта, включающему введение субъекту полипептида, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
В еще одном родственном аспекте изобретение относится к применению полипептида в получении лекарственного средства для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
В одном варианте осуществления, полипептид для применения по изобретению содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к SEQ ID NO: 42. Предпочтительно, полипептид для применения по изобретению содержит полипептидную последовательность, показанную как SEQ ID NO: 42.
В одном варианте осуществления, полипептид для применения согласно изобретению содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к SEQ ID NO: 41. Предпочтительно, полипептид для применения по изобретению содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, показанной как SEQ ID NO: 41.
В одном варианте осуществления, полипептид для применения по изобретению (например, содержащий SEQ ID NO: 42 или кодируемый SEQ ID NO: 41) может представлять собой часть полипептида, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 61 или 65. Таким образом, в одном варианте осуществления, полипептид для применения по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к SEQ ID NO: 61 или 65. Предпочтительно, полипептид для применения по изобретению может содержать (более предпочтительно, состоять из) SEQ ID NO: 61 или 65. В одном варианте осуществления, полипептид содержит каталитически неактивную L-цепь (например, по SEQ ID NO: 65).
В одном варианте осуществления, полипептид для применения по изобретению (например, содержащий SEQ ID NO: 42 или кодируемый SEQ ID NO: 41) может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 60. Таким образом, в одном варианте осуществления, полипептид для применения по изобретению может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к SEQ ID NO: 60. Предпочтительно, полипептид для применения по изобретению может кодироваться нуклеотидной последовательностью, содержащей (более предпочтительно, состоящей из) SEQ ID NO: 60. В одном варианте осуществления, полипептид содержит каталитически неактивную L-цепь.
SEQ ID NO: 42 представляет собой пример модифицированного фрагмента BoNT/A, и SEQ ID NO: 61 и 65 представляют собой примеры модифицированных полипептидов BoNT/A, которые являются каталитически активными и неактивными, соответственно. Такие модифицированные полипептиды и фрагменты BoNT/A особенно предпочтительны для применения по настоящему изобретению. Полипептиды, показанные как SEQ ID NO: 42, 61 и 62, имеют ряд аминокислотных модификаций (например, замен) по сравнению с BoNT/A дикого типа, которые увеличивают изоэлектрическую точку полипептида. Не желая быть связанными теорией, полагают, что повышенный суммарный положительный заряд способствует электростатическим взаимодействиям между полипептидом и анионными внеклеточными компонентами, тем самым способствуя связыванию между полипептидом и клеточной поверхностью, тем самым увеличивая удержание в месте введения и/или продолжительность действия. таким образом, предусмотрено, что свойства SEQ ID NO: 42, 61 и 65 в отношении роста и/или восстановления нейронов будут улучшены по сравнению с эквивалентными полипептидами, в которых отсутствуют указанные модификации.
Для каталитически активных модифицированных полипептидов BoNT/A, описанных выше (например, SEQ ID NO: 61), одним из способов определения этих преимущественных свойств (которые представляют собой увеличение терапевтического индекса) является коэффициент безопасности модифицированного BoNT/A. В связи с этим, нежелательные эффекты клостридиального нейротоксина (вызванные диффузией токсина из места введения) можно оценить экспериментально путем измерения доли потери массы тела на соответствующей животной модели (например, на мышах, у которых потеря массы тела определена в течение семи дней после введения). И наоборот, желаемые эффекты вне мишени клостридиального нейротоксина можно оценить экспериментально с помощью анализа Шкалы отведения пальцев (DAS), измерения мышечного паралича. Анализ DAS можно проводить путем инъекции 20 мкл клостридиального нейротоксина, составленного в желатин-фосфатном буфере, в икроножно/камбаловидный комплекс мыши с последующей оценкой балла Шкалы отведения пальцев с использованием способа Aoki (Aoki KR, Toxicon 39: 1815-1820; 2001). В анализе DAS, мышей ненадолго подвешивают за хвост, чтобы вызвать характерную реакцию испуга, при которой мышь вытягивает задние конечности и отводит задние пальцы. После инъекции клостридиального нейротоксина, различные степени отведения пальцев оценивают по пятибалльной шкале (от 0=норма до 4=максимальное уменьшение отведения пальцев и разгибания ног).
Затем коэффициент безопасности клостридиального нейротоксина может быть выражен как отношение между количеством токсина, необходимого для снижения массы тела на 10% (измерено при пиковом эффекте в течение первых семи дней после дозирования у мыши), и количеством токсина, требуемым для оценки DAS 2. Таким образом, желательны высокие оценки отношения безопасности, которые указывают на токсин, способный эффективно парализовать мышцу-мишень с небольшими нежелательными побочными эффектами. Каталитически активный модифицированный BoNT/A по настоящему изобретению может иметь коэффициент безопасности, который выше, чем коэффициент безопасности эквивалентного не модифицированного (нативного) ботулинического токсина (например, SEQ ID NO: 62).
Таким образом, в одном варианте осуществления, каталитически активный модифицированный BoNT/A по настоящему изобретению имеет коэффициент безопасности по меньшей мере 8 (например, по меньшей мере 8, 9, 10, 15, 20, 25, 30, 35, 40, 45 или 50), где коэффициент безопасности рассчитывается как: доза токсина, необходимая для изменения массы тела на -10% (пг/мышь), деленная на DAS ED50 (пг/мышь) [ED50=доза, необходимая для получения оценки DAS, равной 2].
В одном варианте осуществления, каталитически активный модифицированный BoNT/A по настоящему изобретению имеет коэффициент безопасности по меньшей мере 10. В одном варианте осуществления, модифицированный BoNT/A или его фрагмент по настоящему изобретению имеет коэффициент безопасности, по меньшей мере 15.
Полипептиды, имеющие по меньшей мере 70% идентичность последовательности к SEQ ID NO: 61, описаны в WO 2015/004461 A1, которая полностью включена в настоящее описание посредством ссылки.
В одном варианте осуществления, полипептид, содержащий полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, 61 или 65, и/или содержащий полипептидную последовательность, которая кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41 или 60, содержит замену в одном или более (предпочтительно, двух или нескольких, трех или нескольких, четырех или нескольких, пяти или нескольких или шести или нескольких, более предпочтительно, во всех) положениях 930, 955, 991, 1026, 1052, 1229 и 886. Нумерация положений соответствует положениям SEQ ID NO: 62 и может быть определена путем сопоставления полипептидной последовательности с SEQ ID NO: 62 (BoNT/A не модифицированный/дикого типа). Так как присутствие метионинового остатка в положении 1 SEQ ID NO: 62 является необязательным, специалист в данной области техники будет учитывать наличие/отсутствие метионинового остатка при определении нумерации аминокислотных остатков. Например, если SEQ ID NO: 62 содержит метионин, нумерация положений будет такая, как определена выше (например, положение 886 будет ASN 886 из SEQ ID NO: 62). Альтернативно, если метионин отсутствует в SEQ ID NO: 62, нумерация аминокислотных остатков должна быть модифицирована на -1 (например, положение 886 будет ASN 885 из SEQ ID NO: 62). Аналогичные соображения применимы, когда метионин, находящийся в положении 1 других полипептидных последовательностей, описанных в настоящем изобретении, присутствует/отсутствует, и специалист в данной области техники легко определит правильную нумерацию аминокислотных остатков, используя методики, общепринятые в данной области.
Предпочтительно, полипептид, содержащий полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, 61 или 65, и/или содержащий полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41 или 60, содержит лизин или аргинин (более предпочтительно, лизин) в одном или более положениях 930, 955, 991, 1026, 1052, 1229 и 886. В одном варианте осуществления, полипептид содержит лизин или аргинин (более предпочтительно, лизин) в по меньшей мере двух, трех, четырех, пяти, шести или всех положениях 930, 955, 991, 1026, 1052, 1229 и 886. Наиболее предпочтительно, полипептид содержит лизин или аргинин (более предпочтительно, лизин) во всех положениях 930, 955, 991, 1026, 1052, 1229 и 886.
Полипептиды по настоящему изобретению стимулируют рост и/или восстановление нейронов. Таким образом, указанные полипептиды находят применение в лечении неврологических нарушений. Термин «неврологическое нарушение», используемый в настоящем изобретении, представляет собой нарушение, которое можно лечить, стимулируя рост и/или восстановление нейронов у субъекта.
Таким образом, в одном аспекте, изобретение относится к способу стимуляции роста и/или восстановления нейронов, где способ включает введение субъекту полипептида, где полипептид, содержит легкую цепь клостридиального нейротоксина (L-цепь) или ее фрагмент; и/или фрагмент тяжелой цепи клостридиального нейротоксина (Н-цепь). В другом аспекте, изобретение относится к способу стимуляции роста и/или восстановления нейронов, где способ включает введение полипептида субъекту, полипептид содержит L-цепь каталитически неактивного клостридиального нейротоксина. В другом аспекте изобретение относится к способу стимуляции роста нейронов или восстановления нейронов, где способ включает введение субъекту полипептида, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42 и/или где полипептид содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41. В другом аспекте изобретение относится к способу стимуляции роста нейронов или восстановления нейронов, включающий введение полипептида субъекту, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63.
Термин «способствует росту нейронов и/или восстановлению нейронов» может означать, что полипептид по изобретению инициирует рост нейронов и/или восстановление нейронов, например, там, где рост нейронов и/или восстановление нейронов не происходило. В других вариантах осуществления, термин «способствует росту нейронов и/или восстановлению нейронов» может означать, что полипептид по изобретению увеличивает скорость роста нейронов и/или восстановления нейронов. Указанное увеличение может быть увеличением по сравнению со скоростью роста нейронов и/или восстановления нейронов в отсутствие полипептида по изобретению. В одном варианте осуществления, рост нейронов и/или восстановление нейронов позволяет восстанавливать поврежденные нейронные цепи, тем самым восстанавливая активность и/или нейронную связь в сети или популяции нейронов. Таким образом, термин «восстановление нейронов», используемый в настоящем изобретении, может включать восстановление конкретного нейрона, а также восстановление нейронной цепи.
Термин «рост нейронов и/или восстановление нейронов» может также охватывать пластичность нейронов. Таким образом, в одном варианте осуществления, полипептид по настоящему изобретению способствует пластичности нейронов. Термин «пластичность нейронов» в данном контексте охватывает спрутинг аксонов, спрутинг дендритов, нейрогенез (например, образование новых нейронов), созревание, дифференциацию и/или синаптическую пластичность (например, включая изменения синаптической силы, активности, анатомии и/или проводимости). В одном варианте осуществления, полипептид по изобретению способствует установлению функциональных синапсов (например, на месте повреждения или рядом с ним).
Рост и/или восстановление нейронов могут быть усилены по меньшей мере на 10%, 20%, 30%, 40%, 50%, 60% или 70% (предпочтительно, по меньшей мере на 80%) в присутствии полипептида по изобретению по сравнению с ростом и/или восстановлением нейронов в отсутствие полипептида по изобретению или в присутствии альтернативного полипептида. В некоторых вариантах осуществления, рост и/или восстановление нейронов может быть увеличено на по меньшей мере 100%, 150% или 200% в присутствии полипептида по изобретению по сравнению с ростом и/или восстановлением нейронов в отсутствие полипептида по изобретению или в присутствии альтернативного полипептида.
В одном варианте осуществления, полипептид по изобретению способствует росту нейронов. Термин «рост нейронов», используемый в данном документе, охватывает рост любой части нейрона, включая рост аксонов и/или дендритов. Полипептид по изобретению может увеличивать длину нейритов, количество нейритов (например, количество нейритов на клетку) и/или может увеличивать длину и/или количество выступов тела клетки или клеточной мембраны нейрона. Предпочтительно, полипептид по изобретению способствует росту аксонов нейрона, например, нейрона у субъекта. Другими словами, предпочтительно, полипептид по изобретению увеличивает рост аксонов, например, разрастание аксонов. Указанный рост аксонов может стимулировать связи и/или химическую коммуникацию между нейронами.
Неврологическое нарушение, которое лечат полипептидом по изобретению, может представлять собой повреждение нейронов, нейродегенеративное нарушение, сенсорное нарушение или вегетативное нарушение.
Неврологическое нарушение может представлять собой повреждение нейронов. В одном варианте осуществления, повреждение нейронов может представлять собой травму нерва, невропатию (например, периферическую невропатию), повреждение спинного мозга, иссечение нерва, повреждение головного мозга (например, черепно-мозговую травму), не травматическое повреждение (например, инсульт или инфаркт спинного мозга) или повреждение плечевого сплетения, например, паралич Эрба или паралич Клюмпке.
В одном варианте осуществления, травма нерва может быть результатом рубцевания и/или перелома кости. В таких случаях травмы нерва повреждаются нервные окончания. Полипептид по изобретению, предпочтительно, позволяет восстанавливать указанные нервные окончания или дистальные нервные окончания, позволяя лечить травму нерва.
Повреждением нейронов может быть паралич, такой как паралич, вызванный повреждением спинного мозга (например, вызванным сдавлением, сужением и/или растяжением). В одном варианте осуществления, повреждением спинного мозга является параплегия или тетраплегия.
Неврологическое нарушение может представлять собой сенсорное нарушение. В одном варианте осуществления, сенсорное нарушение представляет собой сенсорную невропатию, сенсомоторную полинейропатию, диабетическую невропатию, боль, синдром Брауна-Секара, болезнь Шарко-Мари-Тута или синдром Девика. Предпочтительно, описанное сенсорное нарушение не является болью. Другими словами, предпочтительно, описанное здесь неврологическое нарушение не является болью.
Неврологическое нарушение может представлять собой вегетативное нарушение. В одном варианте осуществления, вегетативное нарушение представляет собой вегетативную невропатию, множественную системную атрофию, острую идиопатическую полинейропатию, вегетативную дистонию, семейную вегетативную дистонию, диабетическую вегетативную недостаточность, истинную вегетативную недостаточность, нарушения терморегуляции, гипергидроз, нейромедиаторный обморок (вазовагальный, мочеиспускание, кашель, глотание и другие ситуативные формы), эректильная дисфункция, ортостатическая гипотензия, синдром постуральной тахикардии (PoTS) или синдром Гийена-Барре.
Неврологическое нарушение может представлять собой нейродегенеративное нарушение. В одном варианте осуществления, нейродегенеративное нарушение представляет собой болезнь Альцгеймера, болезнь Паркинсона, нарушения, связанные с болезнью Паркинсона, заболевание мотонейронов, периферическую невропатию, моторную невропатию, прионную болезнь, болезнь Хантингтона, спиноцеребеллярную атаксию, спинальную мышечную атрофию, амиотрофию одной конечности, атаксию Фридрейха, болезнь Халлервордена-Шпатца или лобно-височную лобарную дегенерацию. Предпочтительно, нейродегенеративное нарушение представляет собой болезнь Паркинсона или заболевание мотонейронов. Преимущественно, полипептиды по изобретению находят применение в лечении нейродегенеративных нарушений, обусловленных их способностью стимулировать рост нейронов (например, включая пластичность нейронов) и/или восстановление нейронов, и, кроме того, благодаря их способности восстанавливать поврежденные нейронные цепи, тем самым восстанавливая активность и/или нейронную связь в сети или популяции нейронов.
Полипептиды по настоящему изобретению могут считаться нейротрофическими полипептидами ввиду их способности стимулировать рост нейронов и/или восстановление нейронов. Описанный в настоящем изобретении нейрон может быть одним или несколькими, выбранными из: мотонейрона (включая автономный нейрон), сенсорного нейрона, спинального интернейрона и церебрального интернейрона. Таким образом, в одном варианте осуществления, полипептид по изобретению способствует росту и/или восстановлению мотонейрона, сенсорного нейрона и/или интернейрона. Предпочтительно, полипептид по изобретению способствует росту и/или восстановлению моторного нейрона.
Используемый в настоящем изобретении термин «субъект» может быть млекопитающим, таким как человек или другое млекопитающее. Предпочтительно, «субъект» означает человека.
Используемый в настоящем изобретении термин «нарушение» также включает «заболевание». В одном варианте осуществления, нарушение представляет собой заболевание.
Используемый в настоящем изобретении термин «лечить» или «лечение» охватывает профилактическое лечение (например, для профилактики возникновения нарушения), а также корректирующее лечение (лечение субъекта, уже страдающего нарушением). Предпочтительно, «лечить» или «лечение» в настоящем изобретении означает корректирующее лечение.
Используемый в настоящем изобретении термин «лечить» или «лечение» относится к нарушению и/или его симптому.
Следовательно, полипептид по изобретению можно вводить субъекту в терапевтически эффективном количестве или в профилактически эффективном количестве. Предпочтительно, полипептид по изобретению вводят субъекту в терапевтически эффективном количестве.
«Терапевтически эффективное количество» представляет собой любое количество полипептида, которое при введении отдельно или в комбинации субъекту для лечения указанного нарушения (или его симптома) является достаточным для осуществления такого лечения нарушения или его симптома.
«Профилактически эффективное количество» представляет собой любое количество полипептида, которое, при введении субъекту отдельно или в комбинации, ингибирует или отсрочивает начало или рецидив нарушения (или его симптома). В некоторых вариантах осуществления, профилактически эффективное количество полностью предотвращает начало или рецидив нарушения. «Ингибирование» начала означает либо уменьшение вероятности возникновения нарушения (или его симптома), либо полную профилактику возникновения.
Полипептиды по изобретению могут быть составлены любым подходящим способом для введения субъекту, например, как часть фармацевтической композиции. Таким образом, в одном аспекте, изобретение относится к фармацевтической композиции, содержащей полипептид по изобретению и фармацевтически приемлемый носитель, эксципиент, адъювант, пропеллент и/или соль. В некоторых вариантах осуществления, полипептид по изобретению может быть в одноцепочечной форме, в то время как в других вариантах осуществления, полипептид может быть в двухцепочечной форме, например, где две цепи соединены дисульфидным мостиком. Предпочтительно, полипептид находится в двухцепочечной форме.
Полипептиды по настоящему изобретению могут быть составлены для перорального, парентерального введения, непрерывной инфузии, ингаляции или местного нанесения. Композиции, подходящие для инъекций, могут быть в форме растворов, суспензий или эмульсий или сухих порошков, которые растворяют или суспендируют в подходящем носителе перед использованием.
В случае полипептида, который должен быть доставлен локально, полипептид может быть составлен в виде крема (например, для местного применения), или для подкожной инъекции.
Средства местной доставки могут включать аэрозоль или другой спрей (например, небулайзер). В этом отношении, аэрозольный состав полипептида представляет доставку в легкие и/или другие носовые и/или бронхиальные или дыхательные пути.
Полипептиды по изобретению можно вводить субъекту путем интратекальной или эпидуральной инъекции в позвоночник на уровне сегмента позвоночника, участвующего в иннервации пораженного органа.
Путь введения может представлять собой лапароскопическую и/или локальную инъекцию. В одном варианте осуществления, полипептид по изобретению вводят в место повреждения или рядом с ним, предпочтительно в место повреждения. Например, если повреждение представляет собой спинной мозг, полипептид может быть введен интратекально или интраспинально (предпочтительно, интратекально). В одном варианте осуществления, путь введения полипептида по изобретению может быть периневральным, интраневральным, интраспинальным и/или интратекальным.
Диапазоны доз для введения полипептидов по настоящему изобретению являются такими, чтобы вызвать желаемый терапевтический и/или профилактический эффект. Следует понимать, что требуемый диапазон дозировки зависит от точной природы клостридиального нейротоксина или композиции, пути введения, природы состава, возраста субъекта, природы, степени или тяжести состояния субъекта, противопоказаний, если таковые имеются, и решения лечащего врача. Вариации в этих уровнях дозировки могут быть скорректированы с использованием стандартных эмпирических процедур для оптимизации.
В одном варианте осуществления, доза полипептида представляет собой базовую дозу. Базовая доза может составлять от 50 пг до 250 мкг, предпочтительно, от 100 пг до 100 мкг. В одном варианте осуществления, базовая доза может составлять по меньшей мере 50 пг, 100 пг, 500 пг, 1 нг, 50 нг, 100 нг, 500 нг, 1 мкг или 50 мкг. Указанная доза может представлять собой разовую базовую дозу.
Жидкие лекарственные формы обычно готовят с использованием полипептида и апирогенного стерильного носителя. Клостридиальный нейротоксин, в зависимости от используемого носителя и концентрации, может быть либо растворен, либо суспендирован в носителе. При приготовлении растворов, полипептид может быть растворен в носителе, при необходимости раствор делают изотоническим путем добавления хлорида натрия и стерилизуют фильтрованием через стерильный фильтр с использованием асептических методов перед наполнением в подходящие стерильные флаконы или ампулы и запечатыванием. Альтернативно, если стабильность раствора является не пригодной, раствор в герметично закрытых контейнерах может быть стерилизован автоклавированием. Преимущественно, в носителе могут быть растворены такие добавки, как буферные, солюбилизирующие, стабилизирующие, консервирующие или бактерицидные, суспендирующие или эмульгирующие агенты и/или местные анестезирующие агенты.
Сухие порошки, которые растворяют или суспендируют в подходящем носителе перед использованием, можно получить путем заполнения предварительно стерилизованных ингредиентов в стерильном контейнере с использованием асептической методики в стерильной зоне. Альтернативно, ингредиенты могут быть растворены в подходящих контейнерах с применением асептической методики в стерильной зоне. Продукт затем лиофилизируют, и контейнеры асептически закрывают.
Парентеральные суспензии, подходящие для способа введения, описанного в настоящем изобретении, готовят по существу таким же образом, за исключением того, что стерильные компоненты суспендируют в стерильном носителе, а не растворяют, и стерилизация не может быть проведена путем фильтрации. Компоненты могут быть выделены в стерильном состоянии или, альтернативно, они могут быть стерилизованы после выделения, например, гамма-облучением.
Преимущественно, в композицию(и) включают суспендирующий агент, например поливинилпирролидон, для облегчения равномерного распределения компонентов.
При введении в соответствии с настоящим изобретением могут использоваться различные технологии доставки, включая инкапсуляцию микрочастиц или воздействие аэрозоля под высоким давлением.
Полипептид по изобретению может представлять собой клостридиальный нейротоксин или его фрагмент, предпочтительно его фрагмент.
В одном варианте осуществления, полипептид по изобретению может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60. В одном варианте осуществления, полипептид по изобретению может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60. Предпочтительно, полипептид по изобретению может кодироваться нуклеотидной последовательностью, содержащей любую из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60.
В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности с любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65. В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65. Предпочтительно, полипептид по изобретению может содержать полипептидную последовательность любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
В одном варианте осуществления, настоящее изобретение охватывает применение полноразмерных клостридиальных нейротоксинов, содержащих L-цепь клостридиального нейротоксина и Н-цепь клостридиального нейротоксина, при условии, что указанная L-цепь клостридиального нейротоксина является каталитически неактивной.
Термин «клостридиальный нейротоксин» охватывает токсины, продуцируемые C. botulinum (ботулинический нейротоксин серотипов A, B, C1, D, E, F, G и X), C. tetani (столбнячный нейротоксин), C. butyricum (ботулинический нейротоксин серотипа E) и C. baratii (ботулинический нейротоксин серотипа F), а также модифицированные клостридиальные нейротоксины или производные любого из вышеперечисленных.
Ботулинический нейротоксин (BoNT) продуцируется C. botulinum в виде большого белкового комплекса, состоящего из самого BoNT в комплексе с рядом вспомогательных белков. В настоящее время существует восемь различных классов ботулинического нейротоксина, а именно: ботулинический нейротоксин серотипов A, B, C1, D, E, F, G и X, все из которых имеют сходную структуру и способы действия. Различные серотипы ботулотоксина можно различить на основе инактивации специфическими нейтрализующими антисыворотками, при этом такая классификация по серотипу коррелирует с долей идентичности последовательности на уровне аминокислот. BoNT белки данного серотипа далее делятся на различные подтипы на основе доли идентичности аминокислотной последовательности.
BoNT всасываются в желудочно-кишечном тракте и, после попадания в общий кровоток, связываются с пресинаптической мембраной холинергических нервных окончаний и препятствуют высвобождению их нейротрансмиттера ацетилхолина. BoNT/B, BoNT/D, BoNT/F и BoNT/G расщепляют синаптобревин/везикуло-ассоциированный мембранный белок (VAMP), BoNT/C1, BoNT/A и BoNT/E расщепляют синаптосомально-ассоциированный белок 25 кДа (SNAP-25), и BoNT/C1 расщепляет синтаксин. Было обнаружено, что BoNT/X расщепляет SNAP-25, VAMP1, VAMP2, VAMP3, VAMP4, VAMP5, Ykt6 и синтаксин 1.
Столбнячный токсин продуцируется в одном серотипе C. tetani, C. butyricum продуцирует BoNT/E, и C. baratii продуцирует BoNT/F.
Термин «клостридиальный нейротоксин» также охватывает модифицированные клостридиальные нейротоксины и их производные, включая, но не ограничиваясь ими, описанные ниже. Модифицированный клостридиальный нейротоксин или производное может содержать одну или более аминокислот, которые были модифицированы по сравнению с нативной (не модифицированной) формой клостридиального нейротоксина, или может содержать одну или более вставленных аминокислот, которые не присутствуют в нативной (не модифицированной) форме клостридиального нейротоксина. Например, модифицированный клостридиальный нейротоксин может иметь модифицированные аминокислотные последовательности в одном или более доменах относительно нативной (не модифицированной) последовательности клостридиального нейротоксина. Такие модификации могут модифицировать функциональные аспекты токсина, например, биологическую активность или жизнестойкость. Таким образом, в одном варианте осуществления, клостридиальный нейротоксин по изобретению представляет собой модифицированный клостридиальный нейротоксин, или модифицированное производное клостридиального нейротоксина или производное клостридиального нейротоксина.
Модифицированный клостридиальный нейротоксин может иметь одну или более модификаций в аминокислотной последовательности тяжелой цепи (например, модифицированный HC домен), где указанная модифицированная тяжелая цепь связывается с нервными клетками-мишенями с более высокой или более низкой аффинностью, чем нативный (не модифицированный) клостридиальный нейротоксин. Такие модификации в HC домене могут включать модифицирующие остатки в сайте связывания ганглиозидов HC домена или в сайте связывания белка (SV2 или синаптотагмина), которые изменяют связывание с ганглиозидным рецептором и/или белковым рецептором нервной клетки-мишени. Примеры таких модифицированных клостридиальных нейротоксинов описаны в WO 2006/027207 и WO 2006/114308, которые полностью включены в настоящее описание посредством ссылки.
Модифицированный клостридиальный нейротоксин может иметь одну или более модификаций в аминокислотной последовательности легкой цепи, например, модификации в субстратсвязывающем или каталитическом домене, которые могут изменять или модифицировать специфичность к белку SNARE модифицированной L-цепи. Примеры таких клостридиальных нейротоксинов описаны в WO 2010/120766 и US 2011/0318385, которые полностью включены в настоящее описание посредством ссылки.
Модифицированный клостридиальный нейротоксин может содержать одну или более модификаций, которые повышают или снижают биологическую активность и/или биологическую жизнестойкость модифицированного клостридиального нейротоксина. Например, модифицированный клостридиальный нейротоксин может содержать мотив на основе лейцина или тирозина, где указанный мотив увеличивает или биологическую активность и/или биологическую жизнестойкость модифицированного клостридиального нейротоксина. Подходящие мотивы на основе лейцина включают xDxxxLL, xExxxLL, xExxxIL и xExxxLM (где x представляет собой любую аминокислоту). Подходящие мотивы на основе тирозина включают Yxx-Hy (где Hy представляет собой гидрофобную аминокислоту). Примеры модифицированных клостридиальных нейротоксинов, содержащих мотивы на основе лейцина и тирозина, описаны в WO 2002/08268, которая полностью включена в настоящее описание посредством ссылки.
Как описано выше, модифицированный клостридиальный нейротоксин (или фрагмент клостридиального нейротоксина) может включать одну или более модификаций, повышающих изоэлектрическую точку клостридиального нейротоксина по сравнению с эквивалентным не модифицированным клостридиальным нейротоксином, не имеющим указанной одной или нескольких модификаций. Подходящие модифицированные клостридиальные нейротоксины описаны выше и в WO 2015/004461 A1 и WO 2016/110662 A1, которые включены в настоящее описание посредством ссылки. Типовые последовательности включают SEQ ID NO: 61 и 42, описанные в настоящем изобретении.
Термин «клостридиальный нейротоксин» охватывает гибридные и химерные клостридиальные нейротоксины. Гибридный клостридиальный нейротоксин содержит по меньшей мере часть легкой цепи одного клостридиального нейротоксина или его подтипа и, по меньшей мере часть тяжелой цепи другого клостридиального нейротоксина или подтипа клостридиального нейтротоксина. В другом варианте осуществления, гибридный клостридиальный нейротоксин может содержать полную легкую цепь легкой цепи из одного подтипа клостидиального нейротоксина, и тяжелую цепь из другого подтипа клостридиального нейротоксина. В другом варианте осуществления, химерный клостридиальный нейротоксин может содержать часть (например, связывающий домен) тяжелой цепи одного подтипа клостридиального нейротоксина, и другая часть тяжелой цепи относится из другого подтипа клостридиального нейротоксина. Аналогично или альтернативно, терапевтический элемент может содержат части легкой цепи различных клостридиальных нейротоксинов. Такие гибридные или химерные клостридиальные нейротоксины могут быть использованы, например, в качестве средства доставки терапевтических преимуществ таких клостридиальных нейротоксинов субъектам, которые иммунологически резистентны к данному подтипу клостридиального нейротоксина, субъектам, которые могут иметь концентрацию рецепторов ниже средней по отношению к данному домену, связывающему тяжелую цепь клостридиального нейротоксина, или субъектам, у которых может быть резистентный к протеазе вариант субстрата мембранного или везикулярного токсина (например, SNAP-25, VAMP и синтаксин). Гибридные и химерные клостридиальные нейротоксины описаны в US 8,071,110, публикация которого полностью включена в настоящее описание посредством ссылки. Таким образом, в одном варианте осуществления, клостридиальный нейротоксин (или его фрагмент) по изобретению представляет собой гибридный клостридиальный нейротоксин или химерный клостридиальный нейротоксин.
В особенно предпочтительном варианте осуществления, полипептид по изобретению может представлять собой химерный клостридиальный нейротоксин, содержащий (предпочтительно, состоящий из) легкую цепь и домен транслокации BoNT/A, а также рецептор-связывающий домен BoNT/B (HC домен) или его часть. Подходящий химерный и/или гибридный клостридиальный нейротоксин может быть описан в WO 2017/191315 A1, которая включена в настоящее описание посредством ссылки. Такие предпочтительные последовательности включают SEQ ID NO: 44, 63 и 64.
Домен LHN BoNT/A может быть ковалентно связан с HC доменом BoNT/B. Указанный химерный BoNT/A также упоминается в настоящем изобретении как «BoNT/AB» или «химера BoNT/AB».
С-концевой аминокислотный остаток LHN домена может соответствовать первому аминокислотному остатку спирали 310, разделяющей LHN и HC домены BoNT/A, и N-концевой аминокислотный остаток HC домена может соответствовать второму аминокислотному остатку спирали 310, разделяющей LHN и HC домены в BoNT/B.
Ссылка в настоящем изобретении на «первый аминокислотный остаток спирали 310, разделяющий LHN и HC домены BoNT/A», означает N-концевой остаток спирали 310, разделяющий LHN и HC домены.
Ссылка в настоящем изобретении на «второй аминокислотный остаток спирали 310, разделяющий домены LHN и HC BoNT/В», означает аминокислотный остаток после N-концевого остатка спирали 310, разделяющий LHN и HC домены.
«Спираль 310» представляет собой тип вторичной структуры, обнаруженный в белках и полипептидах, наряду с α-спиралями, β-листами и обратными витками. Аминокислоты в спирали 310 расположены в правозакрученной спиральной структуре, где каждый полный оборот завершают три остатка и десять атомов, разделяющих внутримолекулярную водородную связь между ними. Каждой аминокислоте соответствует 120° поворот спирали (т.е. спираль имеет три остатка на виток) и трансляция 2,0 Å (= 0,2 нм) вдоль оси спирали, и она имеет 10 атомов в кольце, образованном за счет образования водородной связи. Наиболее важно, группа N-H аминокислоты образует водородную связь с группой C=O аминокислоты на три остатка раньше; эта повторяющаяся i+3 → i водородная связь определяет спираль 310. Спираль 310 является стандартной концепцией в структурной биологии, с которой знаком специалист в данной области техники.
Эта спираль 310 соответствует четырем остаткам, которые образуют настоящую спираль, и двум кэпирующим (или переходным) остаткам, по одному на каждом конце этих четырех остатков. Термин «спираль 310, разделяющая LHN и HC домены», используемый в настоящем изобретении, состоит из этих 6 остатков.
Путем проведения структурных анализов и выравнивания последовательностей, была идентифицирована спираль 310, разделяющая LHN и HC домены. Эта спираль 310 окружена α-спиралью на своем N-конце (т.е. в С-концевой части LHN домена), и β-цепью на его С-конце (т. е. в N-концевой части HC домена). Первый (N-концевой) остаток (кэп или переходный остаток) спирали 310 также соответствует С-концевому остатку этой α-спирали.
Спираль 310, разделяющая LHN и HC домены, может быть, например, определена из общедоступных кристаллических структур ботулинических нейротоксинов, например, 3BTA (http://www.rcsb.org/pdb/explore/explore.do?structureId=3BTA) и 1EPW (http://www.rcsb.org/pdb/explore/explore.do?structureId=1EPW) для ботулинических нейротоксинов A1 и B1, соответственно.
In silico моделирование и инструменты выравнивания, которые являются общедоступными, также можно использовать для определения местоположения спирали 310, разделяющей LHN и HC домены в других нейротоксинах, например, серверы моделирования гомологии LOOPP (Learning, Observing and Outputting Protein Patterns, http://loopp.org), PHYRE (Protein Homology/analogY Recognition Engine, http://www.sbg.bio.ic.ac.uk/phyre2/) и Rosetta (https://www.rosettacommons.org), сервер наложения белков SuperPose (http://wishart.biology.ualberta.ca/superpose/), программа выравнивания Clustal Omega (http://www.clustal.org/omega/) и ряд других инструментов/услуг, перечисленных на Internet Resources for Molecular and Cell Biologists (http://molbiol-tools.ca). В частности, то, что область вокруг соединения «HN/HCN» структурно высококонсервативна, делает ее идеальной областью для наложения различных серотипов.
Например, для определения последовательности этой спирали 310 в других нейротоксинах можно использовать следующую методологию:
1. Инструмент моделирования структурной гомологии LOOP (http://loopp.org) используют для получения спрогнозированной структуры других серотипов BoNT на основе кристаллической структуры BoNT/A1 (3BTA.pdb);
2. Структурные (pdb) файлы, полученные таким образом, редактируют для включения только N-конца HCN домена и около 80 остатков перед ним (которые являются частью HN домена), тем самым сохраняя «HN/HCN» область, которая является структурно высококонсервативной;
3. Сервер наложения белков SuperPose (http://wishart.biology.ualberta.ca/superpose/) используют для наложения каждого серотипа на структуру 3BTA.pdb;
4. Наложенные друг на друга файлы pdb проверяют, чтобы расположить спираль 310 в начале HC домена BoNT/A1, и затем идентифицируют соответствующие остатки в другом серотипе;
5. Последовательности других серотипов BoNT выравнивают с Clustal Omega, чтобы проверить правильность соответствующих остатков.
Примеры доменов LHN, HC и 310 спирали, определенных этим способом, представлены ниже:
(он же SEQ ID NO: 52)
С помощью структурного анализа и выравнивания последовательностей, было обнаружено, что β-цепь, следующая за спиралью 310, разделяющей LHN и HC домены, является консервативной структурой во всех ботулинических и столбнячных нейротоксинах и начинается с 8 остатка, если начинать с первого остатка спирали 310, разделяющий домены LHN и HC (например, на остатке 879 для BoNT/A1).
Химера BoNT/AB может содержать LHN домен из BoNT/A, ковалентно связанный с HC доменом из BoNT/B,
- где С-концевой аминокислотный остаток LHN домена соответствует восьмому аминокислотному остатку на N-конце β-цепи, расположенной в начале (N-конце) HC домена BoNT/A, и
- где N-концевой аминокислотный остаток HC домена соответствует седьмому аминокислотному остатку на N-конце β-цепи, расположенной в начале (N-конце) HC домена BoNT/B.
Химера BoNT/AB может содержать LHN домен из BoNT/A, ковалентно связанный с HC доменом из BoNT/B,
- где С-концевой аминокислотный остаток LHN домена соответствует С-концевому аминокислотному остатку α-спирали, расположенному на конце (С-конце) LHN домена BoNT/A, и
- где N-концевой аминокислотный остаток НС домена соответствует аминокислотному остатку, непосредственно расположенному на С-конце по отношению к С-концевому аминокислотному остатку α-спирали, расположенному на конце (С-конце) LHN домена BoNT/B.
Смысл процесса конструирования химеры BoNT/AB заключается в том, чтобы попытаться гарантировать, что вторичная структура не будет нарушена, и тем самым свести к минимуму любые изменения третичной структуры и функции каждого домена. Не желая быть связанными теорией, полагают, что отсутствие разрушения четырех центральных аминокислотных остатков спирали 310 в химере BoNT/AB обеспечивает оптимальную конформацию химерного нейротоксина, тем самым позволяя химерному нейротоксину выполнять свои функции в полную силу.
LHN домен из BoNT/A может соответствовать аминокислотным остаткам 1-872 SEQ ID NO: 62 или полипептидной последовательности, имеющей по меньшей мере 70% идентичность с ней. LHN домен из BoNT/A может соответствовать аминокислотным остаткам 1-872 SEQ ID NO: 62, или полипептидной последовательности, имеющей по меньшей мере 80%, 90% или 95% идентичность с ней. Предпочтительно, LHN домен из BoNT/A соответствует аминокислотным остаткам 1-872 SEQ ID NO: 62.
НС домен из BoNT/B может соответствовать аминокислотным остаткам 860-1291 SEQ ID NO: 52 или полипептидной последовательности, имеющей по меньшей мере 70% идентичность с ней. НС домен из BoNT/B может соответствовать аминокислотным остаткам 860-1291 SEQ ID NO: 52 или полипептидной последовательности, имеющей по меньшей мере 80%, 90% или 95% идентичность с ней. Предпочтительно, НС домен из BoNT/B соответствует аминокислотным остаткам 860-1291 SEQ ID NO: 52.
Химера BoNT/AB предпочтительно содержит LHN домен из BoNT/A и НС домен из BoNT/B. Более предпочтительно, LHN домен соответствует аминокислотным остаткам 1-872 BoNT/A (SEQ ID NO: 62) и НС домен соответствует аминокислотным остаткам 860-1291 BoNT/B (SEQ ID NO: 52).
Предпочтительно, НС домен из BoNT/B дополнительно содержит по меньшей мере одну аминокислотную замену, добавление или делецию в HCC субдомене, что приводит к увеличению аффинности связывания нейротоксина BoNT/B с Syt II человека по сравнению с природной последовательностью BoNT/B. Подходящие аминокислотные замены, добавления или делеции в HCC субдомене BoNT/BH описаны в WO 2013/180799 и WO 2016/154534 (обе включены в настоящее описание посредством ссылки).
Подходящая аминокислотная замена, добавление или делеция в HCC субдомене BoNT/BH включает мутации замены, выбранные из группы, состоящей из: V1118M; Y1183M; E1191M; E1191I; E1191Q; E1191T; S1199Y; S1199F; S1199L; S1201V; E1191C, E1191V, E1191L, E1191Y, S1199W, S1199E, S1199H, W1178Y, W1178Q, W1178A, W1178S, Y1183C, Y1183P и их комбинаций.
Подходящая аминокислотная замена, добавление или делеция в HCC субдомене BoNT/BH дополнительно включает комбинации двух мутаций замены, выбранных из группы, состоящей из: E1191M и S1199L, E1191M и S1199Y, E1191M и S1199F, E1191Q и S1199L, E1191Q и S1199Y, E1191Q и S1199F, E1191M и S1199W, E1191M и W1178Q, E1191C и S1199W, E1191C и S1199Y, E1191C и W1178Q, E1191Q и S1199W, E1191V и S1199W, E1191V и S1199Y или E1191V и W1178Q.
Подходящая аминокислотная замена, добавление или делеция в HCC субдомене BoNT/BH также включает комбинацию трех мутаций замены, которые представляют собой E1191M, S1199W и W1178Q.
Предпочтительно, подходящая аминокислотная замена, добавление или делеция в HCC субдомене BoNT/BH включает комбинацию двух мутаций замены, которые представляют собой E1191M и S1199Y.
Модификация может быть модификацией по сравнению с не модифицированным BoNT/B, показанным как SEQ ID NO: 52, где нумерация аминокислотных остатков определяется путем выравнивания с SEQ ID NO: 52. Поскольку наличие метионинового остатка в положении 1 SEQ ID NO: 52 является необязательным, специалист в данной области техники примет во внимание наличие/отсутствие метионинового остатка при определении нумерации аминокислотных остатков. Например, если SEQ ID NO: 52 включает метионин, нумерация позиций будет такой, как определено выше (например, E1191 будет представлять собой E1191 в SEQ ID NO: 52). Альтернативно, если метионин отсутствует в SEQ ID NO: 52, нумерация аминокислотных остатков должна быть модифицирована на -1 (например, E1191 будет представлять собой E1190 в SEQ ID NO: 52). Аналогичные соображения применимы, когда метионин в положении 1 других полипептидных последовательностей, описанных в данном документе, присутствует/отсутствует, и специалист в данной области техники легко определит правильную нумерацию аминокислотных остатков, используя методы, общепринятые в данной области техники.
Таким образом, в одном аспекте изобретения представлен полипептид для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
В родственном аспекте изобретение относится к способу стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, включающему введение субъекту полипептида, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
В еще одном родственном аспекте изобретение относится к применению полипептида в получении лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
В одном аспекте, изобретение относится к полипептиду для применения в лечении неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
В родственном аспекте изобретение относится к способу лечения неврологического нарушения у субъекта, включающему введение субъекту полипептида, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
В еще одном родственном аспекте изобретение относится к применению полипептида в получении лекарственного средства для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
В одном варианте осуществления, полипептид для применения по изобретению содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к SEQ ID NO: 63 или 64. Предпочтительно, полипептид для применения по изобретению содержит (более предпочтительно, состоит из) полипептидную последовательность, показанную как SEQ ID NO: 63 или 64.
Предпочтительно, полипептид, содержащий полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности с SEQ ID NO:63, содержит каталитически неактивную L-цепь, такую как SEQ ID NO:64.
Химерный и/или гибридный клостридиальный нейротоксин для применения в настоящем изобретении может содержать часть полипептида BoNT/A и часть полипептида BoNT/B, пример которого включает полипептид, описанный в настоящем изобретении как SEQ ID NO: 44.
Подходящие химерные клостридиальные нейротоксины могут включать BoNT/FA. Действительно, в особенно предпочтительном варианте осуществления, полипептид по изобретению может содержать BoNT/FA или его фрагмент. Каталитически неактивные формы BoNT/FA описаны в настоящем изобретении как SEQ ID NO: 26 и 34. Подходящие фрагменты BoNT/FA также описаны в настоящем изобретении как SEQ ID NO: 28, 30 и 32.
Термин «клостридиальный нейротоксин» может также охватывать недавно открытые члены семейства белков ботулинического нейротоксина, экспрессируемые не клостридиальными микроорганизмами, такие как токсин, кодируемый Enterococcus, который имеет наиболее близкую идентичность по последовательности с BoNT/X, токсин, кодируемый Weissella oryzae, называемый BoNT/Wo (NCBI Ref Seq: WP_027699549.1), который расщепляет VAMP2 на W89-W90, токсин, кодируемый Enterococcus faecium (GenBank: OTO22244.1), который расщепляет VAMP2 и SNAP25, и токсин, кодируемый Chryseobacterium pipero (NCBI Ref.Seq: WP_034687872.1).
В полипептиде по настоящему изобретению может отсутствовать функциональный HC домен клостридиального нейротоксина, а также отсутствует какой-либо функционально эквивалентная таргетная группа экзогенного лиганда (TM).
Таким образом, в особенно предпочтительном варианте осуществления, клостридиальный нейротоксин по изобретению не является ре-таргетирующим клостридиальным нейротоксином. В ре-таргетирующем клостридиальном нейротоксине, клостридиальный нейротоксин модифицирован для включения экзогенного лиганда, известного как таргетная группа (TM). ТМ выбирают для обеспечения специфичности связывания с желаемой клеткой-мишенью, и как часть процесса ре-таргетирования, может быть удалена нативная связывающая часть клостридиального нейротоксина (например, НС домен или НСС домен). Технология ре-таргетирования описана, например, в: EP-B-0689459; WO 1994/021300; EP-B-0939818; US 6,461,617; US 7,192,596; WO 1998/007864; EP-B-0826051; US 5,989,545; US 6,395,513; US 6,962,703; WO 1996/033273; EP-B-0996468; US 7,052,702; WO 1999/017806; EP-B-1107794; US 6,632,440; WO 2000/010598; WO 2001/21213; WO 2006/059093; WO 2000/62814; WO 2000/04926; WO 1993/15766; WO 2000/61192; и WO 1999/58571; все они полностью включены в настоящее описание посредством ссылки.
Как обсуждалось выше, (полноразмерные) клостридиальные нейротоксины образуются из двух полипептидных цепей: тяжелой цепи (Н-цепи), имеющей молекулярную массу приблизительно 100 кДа, и легкой цепи (L-цепи), имеющей молекулярную массу приблизительно 50 кДа. Н-цепь содержит С-концевой таргетный компонент (рецептор-связывающий домен или НС домен) и N-концевой транслокационный компонент (HN домен).
Клостридиальный нейротоксин может быть выбран из BoNT/A, BoNT/B, BoNT/C, BoNT/D, BoNT/E, BoNT/F, BoNT/G, BoNT/X и TeNT (столбнячного нейротоксина). Предпочтительно, клостридиальный нейротоксин представляет собой ботулинический нейротоксин, такой как ботулинический нейротоксин, выбранный из BoNT/A, BoNT/B, BoNT/C, BoNT/D, BoNT/E, BoNT/F, BoNT/G и BoNT/X.
В одном варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/A. Эталонная последовательность BoNT/A показана как SEQ ID NO: 51. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/B. Эталонная последовательность BoNT/B показана как SEQ ID NO: 52. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/C. Эталонная последовательность BoNT/С показана как SEQ ID NO: 53. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/D. Эталонная последовательность BoNT/D показана как SEQ ID NO: 54. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/E. Эталонная последовательность BoNT/E показана как SEQ ID NO: 55. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/F. Эталонная последовательность BoNT/F показана как SEQ ID NO: 56. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/G. Эталонная последовательность BoNT/G показана как SEQ ID NO: 57. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой TeNT. Эталонная последовательность TeNT показана как SEQ ID NO: 58. В другом варианте осуществления, клостридиальный нейротоксин может представлять собой BoNT/X. Эталонная последовательность BoNT/X показана как SEQ ID NO: 59.
В другом варианте осуществления полипептид по изобретению содержит фрагмент BoNT/A или фрагмент BoNT/F. В другом варианте осуществления, полипептид по изобретению содержит каталитически неактивную L-цепь BoNT/A или BoNT/F.
В вариантах осуществления, где полипептид, описанный в настоящем изобретении, имеет метку для очистки (например, His-метку) и/или линкер, указанная метка и/или линкер являются необязательными.
Подходящие полноразмерные клостридиальные нейротоксины описаны в настоящем изобретении.
В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичности последовательности к любой из SEQ ID NO: 22, 10, 12, 14, 16, 18, 26, 34, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной. В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной. Предпочтительно, полипептид по изобретению может содержать полипептидную последовательность, содержащую любую одну из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65 с при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной.
В одном варианте осуществления, полипептид по изобретению может быть полипептидом, кодируемым нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25, 33 или 60, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной. В одном варианте осуществления, полипептид по изобретению, кодированный нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25, 33 или 60, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной. Предпочтительно, полипептидом по изобретению является такой, который кодируется нуклеотидной последовательностью, содержащей любую из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25, 33 или 60, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной.
В одном варианте осуществления полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65 при условии что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной. В одном варианте осуществления, полипептид по изобретению содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной. Предпочтительно, полипептид по изобретению содержит любую из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65, при условии, что L-цепь клостридиального нейротоксина указанного полипептида является каталитически неактивной.
В одном варианте осуществления полипептид по изобретению представляет собой полноразмерный клостридиальный нейротоксин, выбранный из BoNT/B, BoNT/C, BoNT/D, BoNT/E, BoNT/F, BoNT/G, BoNT/X и TeNT.
В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 52-59, 61 или 63. В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 52-59, 61 или 63. В одном варианте осуществления, полипептид по изобретению может содержать полипептидную последовательность, имеющую по меньшей мере 99% или 99,9% идентичность последовательности к любой из SEQ ID NO: 52-59, 61 или 63. Предпочтительно, полипептид по изобретению может содержать (более предпочтительно, состоять из) полипептидную последовательность, содержащую любую из SEQ ID NO: 52-59, 61 или 63.
В особенно предпочтительном варианте осуществления, полипептид по изобретению не является полноразмерным каталитически активным клостридиальным нейротоксином, например, не является каталитически активным полноразмерным BoNT/A.
Полипептид по настоящему изобретению может содержать (или состоять из) фрагмент клостридиального нейротоксина, например, фрагмент любого описанного в настоящем изобретении полноразмерного клостридиального нейротоксина.
В одном варианте осуществления, полипептид по изобретению может содержать фрагмент последовательности полипептида, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 22, 10, 12, 14, 16, 18, 26, 34, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65. В одном варианте осуществления, полипептид по изобретению может содержать фрагмент полипептидной последовательности, имеющей по меньшей мере 80%, 90% 95% или 98% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65. Предпочтительно, полипептид по изобретению может содержать фрагмент полипептидной последовательности, содержащей любую из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
В одном варианте осуществления, полипептид по изобретению содержит (или состоит из) L-цепь клостиридиального нейротоксина или ее фрагмент. Фрагмент L-цепи клостридиального нейротоксина может иметь ≤400, ≤350, ≤300, ≤250, ≤200, ≤150, ≤100 или ≤50 аминокислотных остатков L-цепи клостридиального нейротоксина. В одном варианте осуществления, фрагмент L-цепи клостридиального нейротоксина содержит по меньшей мере 20, 30, 40, 50, 60, 70, 80, 90, 100, 120, 150 или 200 аминокислотных остатков L-цепи клостридиального нейротоксина. Например, фрагмент L-цепи клостридиального нейротоксина может иметь 20-400, 50-300 или 100-200 аминокислотных остатков L-цепи клостридиального нейротоксина.
Примеры эталонных последовательностей L-цепи включают:
Ботулинический нейротоксин типа А: аминокислотные остатки 1-448
Ботулинический нейротоксин типа В: аминокислотные остатки 1-440
Ботулинический нейротоксин типа С1: аминокислотные остатки 1-441
Ботулинический нейротоксин типа D: аминокислотные остатки 1-445
Ботулинический нейротоксин типа Е: аминокислотные остатки 1-422
Ботулинический нейротоксин типа F: аминокислотные остатки 1-439
Ботулинический нейротоксин типа G: аминокислотные остатки 1-441
Нейротоксин столбняка: аминокислотные остатки 1-457
Сообщается, что для недавно идентифицированного BoNT/X L-цепь соответствует аминокислотам 1-439, при этом граница L-цепи потенциально варьируется примерно на 25 аминокислот (например, 1-414 или 1-464).
Идентифицированные выше эталонные последовательности следует рассматривать как ориентир, поскольку в зависимости от под-серотипов могут возникать незначительные вариации. Например, в US 2007/0166332 (полностью включенном в настоящее описание посредством ссылки) цитируются слегка отличающиеся клостридиальные последовательности:
Ботулинический нейротоксин типа А: аминокислотные остатки M1-K448
Ботулинический нейротоксин типа В: аминокислотные остатки M1-K441
Ботулинический нейротоксин типа С1: аминокислотные остатки M1-K449
Ботулинический нейротоксин типа D: аминокислотные остатки M1-R445
Ботулинический нейротоксин типа Е: аминокислотные остатки M1-R422
Ботулинический нейротоксин типа F: аминокислотные остатки M1-K439
Ботулинический нейротоксин типа G: аминокислотные остатки M1-K446
Столбнячный нейротоксин: аминокислотные остатки M1-A457
Подходящие L-цепи клостридиального нейротоксина описаны в настоящем изобретении.
L-цепь клостридиального нейротоксина может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 6, 24, 32 или 40, или ее фрагмент. В одном варианте осуществления, L-цепь клостридиального нейротоксина содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 6, 24, 32 или 40, или ее фрагмент. Предпочтительно, L-цепь клостридиального нейротоксина содержит (более предпочтительно, состоит из из) полипептидной последовательности, содержащей любую из SEQ ID NO: 6, 24, 32 или 40, или ее фрагмента.
L-цепь клостридиального нейротоксина может быть цепью, кодируемой нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 5, 23, 31 или 39, или ее фрагмент. L-цепь клостридиального нейротоксина представляет собой цепь, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 5, 23, 31 или 39, или ее фрагментом. Предпочтительно, L-цепью клостридиального нейротоксина является такая, которая кодируется нуклеотидной последовательностью, содержащей любую из SEQ ID NO: 5, 23, 31 или 39, или ее фрагментом.
В одном варианте осуществления, полипептид по изобретению содержит (или состоит из) фрагмент Н-цепи клостридиального нейротоксина. Фрагмент Н-цепи клостридиального нейротоксина может иметь ≤800, ≤700, ≤600, ≤500, ≤400, ≤350, ≤300, ≤250, ≤200, ≤150, ≤100 или ≤50 аминокислотных остатков Н-цепи клостридиального нейротоксина. В одном варианте осуществления, фрагмент Н-цепи клостридиального нейротоксина имеет по меньшей мере 20, 30, 40, 50, 60, 70, 80, 90, 100, 120, 150 или 200 аминокислотных остатков Н-цепи клостридиального нейротоксина Например, фрагмент Н-цепи клостридиального нейротоксина может иметь 20-800, 30-600, 40-400, 50-300 или 100-200 аминокислотных остатков Н-цепи клостридиального нейротоксина.
H-цепь клостридиального нейротоксина включает два структурных/функциональных домена: домен транслокации (HN) и рецептор-связывающий домен (HC).
В одном варианте осуществления, полипептид по изобретению содержит (или состоит из) домен клостридиального нейротоксина или его фрагмента. Фрагмент домена транслокации клостридиального нейротоксина может иметь ≤400, ≤350, ≤300, ≤250, ≤200, ≤150, ≤100 или ≤50 аминокислотных остатков домена транслокации клостридиального нейротоксина. В одном варианте осуществления, фрагмент домена транслокации клостридиального нейротоксина содержит по меньшей мере 20, 30, 40, 50, 60, 70, 80, 90, 100, 120, 150 или 200 аминокислотных остатков домена транслокации клостридиального нейротоксина. Например, фрагмент домен транслокации клостридиального нейротоксина может иметь 20-400, 50-300 или 100-200 аминокислотных остатков домена транслокации клостридиального нейротоксина.
Домен транслокации представляет собой Н-цепь клостридиального нейротоксина, приблизительно эквивалентный амино-концевой половине Н-цепи, или домен, соответствующий этому фрагменту в интактной Н-цепи. В одном варианте осуществления, НС функцию H-цепи можно удалить путем делеции аминокислотной последовательности HC (либо на уровне синтеза ДНК, либо на уровне пост-синтеза обработкой нуклеазой или протеазой). Альтернативно, функция HC может быть инактивирована химической или биологической обработкой. Таким образом, в некоторых вариантах осуществления, Н-цепь может быть неспособна связываться с сайтом связывания на клетке-мишени, с которой связывается нативный клостридиальный нейротоксин (т.е. голотоксин).
Примеры подходящих (эталонных) доменов транслокации включают:
Ботулинический нейротоксин типа А - аминокислотные остатки (449-871)
Ботулинический нейротоксин типа В - аминокислотные остатки (441-858)
Ботулинический нейротоксин типа С - аминокислотные остатки (442-866)
Ботулинический нейротоксин типа D - аминокислотные остатки (446-862)
Ботулинический нейротоксин типа Е - аминокислотные остатки (423-845)
Ботулинический нейротоксин типа F - аминокислотные остатки (440-864)
Ботулинический нейротоксин типа G - аминокислотные остатки (442-863)
Столбнячный нейротоксин - аминокислотные остатки (458-879)
Указанную выше эталонную последовательность следует рассматривать как ориентир, поскольку в зависимости от суб-серотипа могут возникать незначительные вариации. Например, в патенте США 2007/0166332 (включенном в настоящее описание посредством ссылки) цитируются слегка отличающиеся клостридиальные последовательности:
Ботулинический нейротоксин типа А - аминокислотные остатки (A449-K871)
Ботулинический нейротоксин типа В - аминокислотные остатки (A442-S858)
Ботулинический нейротоксин типа С - аминокислотные остатки (T450-N866)
Ботулинический нейротоксин типа D - аминокислотные остатки (D446-N862)
Ботулинический нейротоксин типа Е - аминокислотные остатки (K423-K845)
Ботулинический нейротоксин типа F - аминокислотные остатки (A440-K864)
Ботулинический нейротоксин типа G - аминокислотные остатки (S447-S863)
Столбнячный нейротоксин - аминокислотные остатки (S458-V879)
В контексте настоящего изобретения, различные HN области клостридиального нейротоксина, содержащие домен транслокации, могут быть полезны в аспектах настоящего изобретения. В одном варианте осуществления, эти активные фрагменты могут способствовать выделению не цитотоксической протеазы (например, клостридиальной L-цепи) из внутриклеточных везикул в цитоплазму клетки-мишени и, таким образом, участвуют в выполнении общего клеточного механизма, посредством которого клостридиальный нейротоксин протеолитически расщепляет субстрат. HN области тяжелых цепей клостридиальных нейротоксинов имеют длину приблизительно 410-430 аминокислот, и содержат домен транслокации. Исследования показали, что полная длина HN области тяжелой цепи клостридиального нейротоксина не является необходимой для транслоцирующей активности домена транслокации. Таким образом, аспекты этого варианта осуществления могут включать HN области клостридиального нейротоксина, содержащие домен транслокации, имеющий длину, например, по меньшей мере 350 аминокислот, по меньшей мере 375 аминокислот, по меньшей мере 400 аминокислот и по меньшей мере 425 аминокислот. Другие аспекты этого варианта осуществления могут включать HN области клостридиального нейротоксина, содержащие домен транслокации, имеющий длину, например, не более 350 аминокислот, не более 375 аминокислот, не более 400 аминокислот и не более 425 аминокислот.
Дополнительную информацию о генетической основе продуцирования токсинов в Clostridium botulinum и C. tetani см. Henderson et al. (1997) in The Clostridia: Molecular Biology and Pathogenesis, Academic press.
Термин HN охватывает HN части встречающегося в природе нейротоксина и модифицированные HN части, имеющие аминокислотные последовательности, не встречающиеся в природе, и/или синтетические аминокислотные остатки. В одном варианте осуществления, указанные модифицированные HN части все еще демонстрируют вышеупомянутую транслокационную функцию.
В предпочтительно варианте осуществления, полипептид по изобретению содержит (или состоит из) рецептор-связывающего домена (НС) клостридиального нейротоксина или его фрагмента. Фрагмент рецептор-связывающего домена, клостридиального нейротоксина (HC) может иметь ≤350, ≤300, ≤250, ≤200, ≤150, ≤100 или ≤50 аминокислотных остатков рецептор-связывающего домена клостридиального нейротоксина (HC). В одном варианте осуществления, фрагмент рецептор-связывающего домена клостридиального нейротоксина (НС) содержит по меньшей мере 20, 30, 40, 50, 60, 70, 80, 90, 100, 120, 150 или 200 аминокислотных остатков рецептор-связывающего домена клостридиального нейротоксина (НС). Например, фрагмент рецептор-связывающего домена клостридиального нейротоксина (НС) может иметь 20-350, 50-300 или 100-200 аминокислотных остатков рецептор-связывающего домена клостридиального нейротоксина (НС).
Примеры эталонных последовательностей рецептор-связывающего домена клостридиального нейротоксина (HC) включают:
BoNT/A - N872-L1296
BoNT/B - E859-E1291
BoNT/C1 - N867-E1291
BoNT/D - S863-E1276
BoNT/E - R846-K1252
BoNT/F - K865-E1274
BoNT/G - N864-E1297
TeNT - I880-D1315
Сообщалось, что для недавно идентифицированного BoNT/X, HC домен соответствует аминокислотам 893-1306, при этом граница домена потенциально варьируется примерно на 25 аминокислот (например, 868-1306 или 918-1306).
H-цепь клостридиального нейротоксина может дополнительно содержать домен, облегчающий транслокацию. Указанный домен облегчает доставку L-цепи в цитозоль клетки-мишени и описан, например, в WO 08/008803 и WO 08/008805, каждый из которых включен в настоящее описание посредством ссылки.
Например, домен, облегчающий транслокацию, может содержать HCN домен клостридиального нейротоксина или его фрагмент или вариант. Более подробно, HCN домен клостридиального нейротоксина, облегчающий транслокацию, может иметь длину по меньшей мере 200 аминокислот, по меньшей мере 225 аминокислот, по меньшей мере 250 аминокислот, по меньшей мере 275 аминокислот. В этом отношении, HCN домен клостридиального нейротоксина, облегчающий транслокацию, предпочтительно имеет длину не более 200 аминокислот, не более 225 аминокислот, не более 250 аминокислот, или не более 275 аминокислот. Конкретные (эталонные) примеры включают:
Ботулинический нейротоксин типа А - аминокислотные остатки (872-1110)
Ботулинический нейротоксин типа В - аминокислотные остатки (859-1097)
Ботулинический нейротоксин типа С - аминокислотные остатки (867-1111)
Ботулинический нейротоксин типа D - аминокислотные остатки (863-1098)
Ботулинический нейротоксин типа Е - аминокислотные остатки (846-1085)
Ботулинический нейротоксин типа F - аминокислотные остатки (865-1105)
Ботулинический нейротоксин типа G - аминокислотные остатки (864-1105)
Столбнячный нейротоксин - аминокислотные остатки (880-1127)
Приведенные выше положения последовательности могут немного варьироваться в зависимости от серотипа/подтипа, и дополнительные примеры подходящих (эталонных) HCN доменов клостридиального нейротоксина включают:
Ботулинический нейротоксин типа А - аминокислотные остатки (874-1110)
Ботулинический нейротоксин типа В - аминокислотные остатки (861-1097)
Ботулинический нейротоксин типа С - аминокислотные остатки (869-1111)
Ботулинический нейротоксин типа D - аминокислотные остатки (865-1098)
Ботулинический нейротоксин типа Е - аминокислотные остатки (848-1085)
Ботулинический нейротоксин типа F - аминокислотные остатки (867-1105)
Ботулинический нейротоксин типа G - аминокислотные остатки (866-1105)
Столбнячный нейротоксин - аминокислотные остатки (882-1127)
Подходящие HC домены клостридиального нейротоксина описаны в настоящем изобретении.
HC домен клостридиального нейротоксина может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 8, 22, 30, 38, 42, 44, 46, 48 или 50, или ее фрагмент. В одном варианте осуществления, HC домен клостридиального нейротоксина содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 8, 22, 30, 38, 42, 44, 46, 48 или 50, или ее фрагмент. Предпочтительно, HC домен клостридиального нейротоксина содержит (более предпочтительно, состоит из) полипептидную последовательность, содержащую любую из SEQ ID NO: 8, 22, 30, 38, 42, 44, 46, 48 или 50, или ее фрагмент.
HC домен клостридиального нейротоксина может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 7, 21, 29, 37, 41, 43, 45, 47 или 49, или ее фрагментом. В одном варианте осуществления, HC домен клостридиального нейротоксина кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 7, 21, 29, 37, 41, 43, 45, 47 или 49, или ее фрагментом. Предпочтительно, HC домен клостридиального нейротоксина кодируется нуклеотидной последовательностью, содержащей любую из SEQ ID NO: 7, 21, 29, 37, 41, 43, 45, 47 или 49 или ее фрагментом.
В одном варианте осуществления, НС домен клостридиального нейротоксина для применения по изобретению представляет собой НС домен BoNT/A. Указанный вариант НС домена BoNT/A может содержать модификацию одного или более аминокислотных остатков, выбранных из Y1117, F1252, H1253 и L1278. Например, вариант НС домена BoNT/A может содержать один или более (предпочтительно, два или несколько) следующих модификаций Y1117V, F1252Y, H1253K и L1278F или L1278H.
В одном варианте осуществления, вариант НС домен BoNT/A содержит следующие модификации: Y1117V и H1253K; или Y1117V, F1252Y, H1253K и L1278F; или Y1117V, F1252Y, H1253K и L1278H.
Предпочтительно, вариант НС домена BoNT/A содержит следующие модификации: Y1117V и H1253K; или Y1117V, F1252Y, H1253K и L1278H.
Модификация может быть модификацией по сравнению с не модифицированным BoNT/A, показанным как SEQ ID NO: 62, где нумерация аминокислотных остатков определяется путем выравнивания с SEQ ID NO: 62. Поскольку присутствие метионинового остатка в положении 1 SEQ ID NO: 62 является необязательным, специалист в данной области техники примет во внимание наличие/отсутствие метионинового остатка при определении нумерации аминокислотных остатков. Например, если SEQ ID NO: 62 включает метионин, нумерация положений будет такой, как определено выше (например, Y1117 будет соответствовать Y1117 из SEQ ID NO: 62). Альтернативно, если метионин отсутствует в SEQ ID NO: 62, нумерация аминокислотных остатков должна быть модифицирована на -1 (например, Y1117 будет соответствовать Y1116 из SEQ ID NO: 52). Аналогичные соображения применимы, когда метионин в положении 1 других полипептидных последовательностей, описанных в настоящем изобретении, присутствует/отсутствует, и специалист в данной области техники легко определит правильную нумерацию аминокислотных остатков, используя методы, общепринятые в данной области техники.
Вариант НС домена BoNT/A может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 46, 48 или 50, или ее фрагмент, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 46, 48 или 50, или ее фрагмент, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A содержит полипептидную последовательность, имеющую по меньшей мере 99% или 99,9% идентичность последовательности к любой из SEQ ID NO: 46, 48 или 50, или его фрагмент, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. Предпочтительно, вариант НС домена BoNT/A содержит (более предпочтительно, состоит из) полипептидную последовательность, содержащую любую из SEQ ID NO: 46, 48 или 50, или ее фрагмент.
Вариант НС домена BoNT/A может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 46 или 50, или ее фрагмент, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 46 или 50, или ее фрагмент, при условии что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A содержит полипептидную последовательность, имеющую по меньшей мере 99% или 99,9% идентичность последовательности к любой из SEQ ID NO: 46 или 50, или ее фрагмент, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. Предпочтительно, вариант НС домена BoNT/A содержит (более предпочтительно, состоит из) полипептидную последовательность, содержащую любую из SEQ ID NO: 46 или 50, или ее фрагмент.
Вариант НС домена BoNT/A может быть кодирован нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 45, 47 или 49, или ее фрагментом, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 45, 47 или 49, или ее фрагментом, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% или 99,9% идентичность последовательности к любой из SEQ ID NO: 45, 47 или 49, или ее фрагментом, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. Предпочтительно, вариант НС домена BoNT/A кодирован любой из SEQ ID NO: 45, 47 или 49, или ее фрагментом.
Вариант НС домена BoNT/A может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательность к любой из SEQ ID NO: 45 или 49, или ее фрагментом, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 45 или 49, или его фрагментом, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. В одном варианте осуществления, вариант НС домена BoNT/A кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% или 99,9% идентичность последовательности к любой из SEQ ID NO: 45 или 49, или ее фрагментом, при условии, что вариант НС домена BoNT/A содержит модификацию, как описано выше. Вариант НС домена BoNT/A кодируется любой из SEQ ID NO: 45 или 49 или ее фрагментом.
Любой из вышеописанных облегчающих доменов может быть объединен с любым из ранее описанных пептидов домена транслокации, подходящих для использования в настоящем изобретении. Таким образом, например, не клостридиальный облегчающий домен может быть объединен с пептидом не клостридиального домена транслокации. Альтернативно, HCN облегчающий транслокацию домен клостридиального нейротоксина может быть объединен с пептидом домена неклостридиальной транслокации. Альтернативно, облегчающий домен клостридиального нейротоксина H CN может быть объединен с пептидом клостридиального домена транслокации, примеры которого включают:
Ботулинический нейротоксин типа А - аминокислотные остатки (449-1110)
Ботулинический нейротоксин типа В - аминокислотные остатки (442-1097)
Ботулинический нейротоксин типа С - аминокислотные остатки (450-1111)
Ботулинический нейротоксин типа D - аминокислотные остатки (446-1098)
Ботулинический нейротоксин типа Е - аминокислотные остатки (423-1085)
Ботулинический нейротоксин типа F - аминокислотные остатки (440-1105)
Ботулинический нейротоксин типа G - аминокислотные остатки (447-1105)
Нейротоксин столбняка - аминокислотные остатки (458-1127)
В некоторых вариантах осуществления, в клостридиальных нейротоксинах по настоящему изобретению может отсутствовать функциональный HC домен клостридиального нейротоксина. В одном варианте осуществления, в клостридиальных нейротоксинах предпочтительно отсутствуют последние 50 С-концевых аминокислот голотоксина клостридиального нейротоксина. В другом варианте осуществления, в клостридиальных нейротоксинах предпочтительно отсутствуют последние 100, предпочтительно, последние 150, более предпочтительно, последние 200, особенно предпочтительно, последние 250 и наиболее предпочтительно, последние 300 С -концевых аминокислотных остатков голотоксина клостридиального нейротоксина. Альтернативно, связывающая активность НС может быть сведена на нет/уменьшена путем мутагенеза - например, ссылаясь на BoNT/A для удобства, модификация одной или двух мутаций аминокислотного остатка (W1266 на L и Y1267 на F) в кармане связывания ганглиозида приводит к тому, что HC область теряет свою рецептор-связывающую функцию. Аналогичные мутации могут быть сделаны в компонентах клостридиального пептида, не относящегося к серотипу А, например, в конструкции на основе ботулинического В с мутациями (W 1262 на L и Y1263 на F) или ботулинического E (W1224 на L и Y1225 на F). Другие мутации в активном сайте приводят к такому же устранению активности связывания HC рецептора, например, Y1267S в ботулиническом токсине типа А и соответствующем высококонсервативном остатке в других клостридиальных нейротоксинах. Подробности этой и других мутаций описаны в Rummel et al. (2004) (Molecular Microbiol. 51:631-634), которая включена в настоящее описание посредством ссылки.
НС пептид нативного клостридиального нейротоксина содержит приблизительно 400-440 аминокислотных остатков и состоит из двух функционально различных доменов с молекулярной массой приблизительно 25 кДа каждый, а именно, N-концевой области (обычно называемой HCN пептидом или доменом) и С-концевой области (обычно называемой НСС пептидом или доменом). Этот факт подтверждается следующими публикациями, каждая из которых полностью включена в настоящее описание посредством ссылки: Umland TC (1997) Nat. Struct. Biol. 4: 788-792; Herreros J (2000) Biochem. J. 347: 199-204; Halpern J (1993) J. Biol. Chem. 268: 15, pp. 11188-11192; Rummel A (2007) PNAS 104: 359-364; Lacey DB (1998) Nat. Struct. Biol. 5: 898-902; Knapp (1998) Am. Cryst. Assoc. Abstract Papers 25: 90; Swaminathan and Eswaramoorthy (2000) Nat. Struct. Biol. 7: 1751-1759; и Rummel A (2004) Mol. Microbiol. 51(3), 631-643. Кроме того, хорошо задокументировано, что С-концевая область (НСС), которая составляет 160-200 С-концевых аминокислотных остатков, отвечает за связывание клостридиального нейротоксина с его естественными клеточными рецепторами, а именно с нервными окончаниями в нервно-мышечном соединении - этот факт также подтверждается вышеуказанными публикациями. Таким образом, ссылка в данном описании на клостридиальную тяжелую цепь, не содержащую функциональный HC пептид (или домен) тяжелой цепи, такой, что тяжелая цепь не способна связываться с рецепторами клеточной поверхности, с которыми связывается нативный клостридиальный нейротоксин, означает, что в клостридиальной тяжелой цепи просто отсутствует функциональный HCC пептид. Другими словами, область HCC пептида может быть либо частично, либо полностью удалена, либо иным образом модифицирована (например, с помощью обычной химической или протеолитической обработки) для снижения его нативной способности связываться с нервными окончаниями в нервно-мышечном соединении.
Таким образом, в одном варианте осуществления, HN пептид клостридиального нейротоксина по настоящему изобретению лишен части С-концевой пептидной части (HCC) клостридиального нейротоксина и, таким образом, он лишен функции связывания НС нативного клостридиального нейротоксина. В качестве примера, в одном варианте осуществления, в удлиненном на С-конце клостридиальном HN пептиде отсутствуют 40 С-концевых аминокислотных остатков, или 60 С-концевых аминокислотных остатков, или 80 С-концевых аминокислотных остатков, или 100 С-концевых аминокислотных остатков, или 120 С-концевых аминокислотных остатков, или 140 С-концевых аминокислотных остатков, или 150 С-концевых аминокислотных остатков, или 160 С-концевых аминокислотных остатков тяжелой цепи клостридиального нейротоксина. В другом варианте осуществления, в клостридиальном HN пептиде по настоящему изобретению отсутствует вся С-концевая пептидная часть (HCC) клостридиального нейротоксина и, таким образом, он лишен функции связывания HC нативного клостридиального нейротоксина. В качестве примера, в одном варианте осуществления, клостридиальный HN пептид не содержит 165 С-концевых аминокислотных остатков, или 170 С-концевых аминокислотных остатков, или 175 С-концевых аминокислотных остатков, или 180 С-концевых аминокислотных остатков, или 185 С-концевых аминокислотных остатков, или 190 С-концевых аминокислотных остатков, или 195 С-концевых аминокислотных остатков тяжелой цепи клостридиального нейротоксина. В качестве другого примера, клостридиальный HN пептид по изобретению не содержит клостридиальную НСС эталонную последовательность, выбранную из группы, состоящей из:
Ботулинического нейротоксина типа А - аминокислотные остатки (Y1111-L1296)
Ботулинического нейротоксина типа В - аминокислотные остатки (Y1098-E1291)
Ботулинического нейротоксина типа С - аминокислотные остатки (Y1112-E1291)
Ботулинического нейротоксина типа D - аминокислотные остатки (Y1099-E1276)
Ботулинического нейротоксина типа Е - аминокислотные остатки (Y1086-K1252)
Ботулинического нейротоксина типа F - аминокислотные остатки (Y1106-E1274)
Ботулинического нейротоксина типа G - аминокислотные остатки (Y1106-E1297)
Столбнячного нейротоксина - аминокислотные остатки (Y1128-D1315).
Вышеуказанные эталонные последовательности следует рассматривать как руководство, поскольку, в зависимости от суб-серотипа могут возникать небольшие вариации.
В предпочтительном варианте осуществления, полипептид по изобретению содержит (или состоит из) L-цепи клостридиального нейротоксина или ее фрагмента и фрагмента H-цепи клостридиального нейротоксина. Например, полипептид может содержать (или состоять из) L-цепи клостридиального нейротоксина или ее фрагмента и домена транслокации клостридиального нейротоксина (HN). Предпочтительно, полипептид дополнительно не содержит рецептор-связывающий домен клостридиального нейротоксина (HC), или по меньшей мере С-концевую часть рецептор-связывающего домена клостридиального нейротоксина (HCC). Таким образом, в одном варианте осуществления, полипептид по настоящему изобретению лишен С-концевой части рецептор-связывающего домена клостридиального нейротоксина (HCC). Преимущественно, такие полипептиды лишены рецептор-связывающей способности эндогенного клостридиального нейротоксина и, таким образом, проявляют меньше эффектов вне мишени у субъекта, которому вводят указанный полипептид.
В одном варианте осуществления, полипептид по изобретению по существу состоит из L-цепи клостридиального нейротоксина или ее фрагмента и/или фрагмента Н-цепи клостридиального нейротоксина. Термин «по существу состоит из», используемый в данном контексте, означает, что полипептид дополнительно не содержит один или более аминокислотных остатков, которые придают полипептиду дополнительную функциональность, например, при введении субъекту. Другими словами, полипептид, который «по существу состоит из» L-цепи клостридиального нейротоксина или ее фрагмента и/или фрагмента Н-цепи клостридиального нейротоксина, может дополнительно содержать один или более аминокислотных остатков (по сравнению с остатками L-цепи клостридиального нейротоксина или ее фрагмента и/или фрагмента Н-цепи клостридиального нейротоксина), но указанный один или более дополнительных аминокислотных остатков не придают полипептиду дополнительную функциональность, например, при введении субъекту. Дополнительная функциональность может включать ферментативную активность, связывающую активность и/или любую физиологическую активность, как бы то ни было.
В одном варианте осуществления, полипептид может содержать последовательности не клостридиального нейротоксина в дополнение к любым последовательностям клостридиального нейротоксина. Последовательности не клостридиального нейротоксина предпочтительно не нарушают способность полипептида по изобретению стимулировать рост или восстановление нейронов. Предпочтительно, последовательность не клостридиального нейротоксина не обладает каталитической активностью, например ферментативной активностью. Предпочтительно, не клостридиальная последовательность не связывается с клеточным рецептором. Другими словами, наиболее предпочтительно, чтобы не клостридиальная последовательность не являлась лигандом для клеточного рецептора Клеточный рецептор может быть белковым клеточным рецептором, таким как интегральный мембранный белок. Примеры клеточных рецепторов можно найти в IUPHAR Guide to Pharmacology Database, version 2019.4, доступной по адресу https://www.guidetopharmacology.org/download.jsp#db_reports. Последовательности не клостридиального нейротоксина могут включать метки, помогающие очистке, такие как His-метки. Предпочтительно, чтобы любые последовательности клостридиального нейротоксина, содержащиеся в указанном полипептиде, состояли из L-цепи клостридиального нейротоксина или ее фрагмента и/или фрагмента Н-цепи клостридиального нейротоксина. В одном варианте осуществления, последовательность клостридиального нейротоксина, содержащаяся в указанном полипептиде, может состоять из L-цепи клостридиального нейротоксина. В одном варианте осуществления, последовательность клостридиального нейротоксина, содержащаяся в указанном полипептиде, может состоять из домена транслокации клостридиального нейротоксина. В одном варианте осуществления, последовательность клостридиального нейротоксина, содержащаяся в указанном полипептиде, может состоять из рецептор-связывающего домена клостридиального нейротоксина. В одном варианте осуществления, последовательность клостридиального нейротоксина, содержащаяся в указанном полипептиде, может состоять из L-цепи клостридиального нейротоксина и домена транслокации клостридиального нейротоксина.
Подходящие полипептиды, содержащие (или состоящие из) L-цепи клостридиального нейротоксина и домена транслокации, описаны в настоящем изобретении.
Клостридиальный нейротоксин, содержащий (или состоящий из) L-цепи клостридиального нейротоксина и домена транслокации, может содержать полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 4, 20, 28 или 36, или ее фрагмент. В одном клостридиальный нейротоксин, содержащий (или состоящий из) L-цепи клостридиального нейротоксина и домена транслокации, содержит полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 4, 20, 28 или 36, или ее фрагмент. Предпочтительно, клостридиальный нейротоксин, содержащий (или состоящий из) L-цепи клостридиального нейротоксина и домена транслокации, содержит (более предпочтительно, состоит из) полипептидную последовательность, содержащую любую из SEQ ID NO: 4, 20, 28 или 36, или ее фрагмент.
Клостридиальный нейротоксин, содержащий (или состоящий из) L-цепи клостридиального нейротоксина и домена транслокации, может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 3, 19, 27 или 35, или ее фрагментом. В одном варианте осуществления, клостридиальный нейротоксин, содержащий (или состоящий из) L-цепи клостридиального нейротоксина и домена транслокации, может кодироваться нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичность последовательности к любой из SEQ ID NO: 3, 19, 27 или 35, или ее фрагментом. Предпочтительно, клостридиальный нейротоксин, содержащий (или состоящий из) L-цепи клостридиального нейротоксина и домена транслокации, представляет собой такой, который кодируется нуклеотидной последовательностью, любой из SEQ ID NO: 3, 19, 27 или 35, или ее фрагментом.
Полипептиды по настоящему изобретению могут быть свободны от комплексообразующих белков, которые присутствуют в природном комплексе клостридиального нейротоксина.
Полипептиды по настоящему изобретению могут быть получены с использованием технологий рекомбинантных нуклеиновых кислот. Таким образом, в одном варианте осуществления, полипептид (как описано выше) представляет собой рекомбинантный полипептид.
В одном варианте осуществления, представлена нуклеиновая кислота (например, ДНК), содержащая последовательность нуклеиновой кислоты, кодирующую полипептид. В одном варианте осуществления, последовательность нуклеиновой кислоты получают как часть ДНК вектора, содержащего промотор и терминатор.
В предпочтительном варианте осуществления, вектор имеет промотор, выбранный из:
В другом предпочтительном варианте осуществления, вектор имеет промотор, выбранный из:
Молекулы нуклеиновых кислот могут быть получены с использованием любого подходящего способа, известного в данной области техники. Таким образом, молекулы нуклеиновых кислот могут быть получены с использованием методов химического синтеза. Альтернативно, молекулы нуклеиновых кислот по изобретению могут быть получены с использованием методов молекулярной биологии.
Конструкция ДНК по настоящему изобретению предпочтительно создается in silico, и затем синтезируется с помощью обычных методов синтеза ДНК.
Вышеупомянутая информация о последовательности нуклеиновой кислоты необязательно модифицируется для смещения кодонов в соответствии с конечной системой экспрессии клетки-хозяина (например, E. coli), которую следует использовать.
Термины «нуклеотидная последовательность» и «нуклеиновая кислота» используются в настоящем изобретении как синонимы. Предпочтительно, нуклеотидная последовательность представляет собой последовательность ДНК.
Полипептид по настоящему изобретению (и особенно любая его часть клостридиального нейротоксина) может быть представлен в одноцепочечной или двухцепочечной форме.
Изобретение относится к способу получения одноцепочечного полипептида, имеющего легкую цепь и тяжелую цепь, способу, включающему экспрессию нуклеиновой кислоты, описанной в настоящем изобретении, в хозяине экспрессии, лизис клетки-хозяина с получением гомогената клетки-хозяина, содержащего одноцепочечный полипептид, и выделение одноцепочечного полипептида. В одном аспекте, настоящее изобретение относится к способу активации полипептида, описанного в настоящем изобретении, где способ включает контакт полипептида с протеазой, которая гидролизует пептидную связь в петле активации полипептида, тем самым превращая (одноцепочечный) полипептид в соответствующий двухцепочечный полипептид (например, где легкая цепь и тяжелая цепь соединены дисульфидной связью).
Таким образом, настоящее изобретение относится к двухцепочечному полипептиду, который можно получить способом по изобретению.
Варианты осуществления, относящиеся к различным терапевтическим применениям изобретения, предназначены для равного применения к способам лечения, полипептидам по изобретению, и наоборот.
ГОМОЛОГИЯ ПОСЛЕДОВАТЕЛЬНОСТИ
Для определения доли идентичности можно использовать любой из множества способов выравнивания последовательностей, включая, помимо прочего, глобальные методы, локальные методы и гибридные методы, такие как, например, методы сегментного подхода. Протоколы для определения доли идентичности представляют собой рутинные процедуры, доступные специалисту в данной области техники. Глобальные способы выравнивают последовательности от начала до конца молекулы и определяют наилучшее выравнивание путем суммирования оценок отдельных пар остатков и наложения штрафов за гэпы. Неограничивающие способы включают, например, CLUSTAL W, см., например, Julie D. Thompson et al., CLUSTAL W: Improving the Sensitivity of Progressive Multiple Sequence Alignment Through Sequence Weighting, Position- Specific Gap Penalties and Weight Matrix Choice, 22(22) Nucleic Acids Research 4673-4680 (1994); и итерационное уточнение, см., например, Osamu Gotoh, Significant Improvement in Accuracy of Multiple Protein. Sequence Alignments by Iterative Refinement as Assessed by Reference to Structural Alignments, 264(4) J. MoI. Biol. 823-838 (1996). Локальные способы выравнивают последовательности путем идентификации одного или более консервативных мотивов, общих для всех входных последовательностей. Неограничивающие способы включают, например, Match-box, см. например, Eric Depiereux and Ernest Feytmans, Match-Box: A Fundamentally New Algorithm for the Simultaneous Alignment of Several Protein Sequences, 8(5) CABIOS 501 -509 (1992); выборку Гиббса, см., например, C. E. Lawrence et al., Detecting Subtle Sequence Signals: A Gibbs Sampling Strategy for Multiple Alignment, 262(5131) Science 208-214 (1993); Align-M, см., например, Ivo Van WaIIe et al., Align-M - A New Algorithm for Multiple Alignment of Highly Divergent Sequences, 20(9) Bioinformatics:1428-1435 (2004).
Таким образом, долю идентичности последовательности определяют обычными способами. См., например, Altschul et al., Bull. Math. Bio. 48: 603-16, 1986 и Henikoff and Henikoff, Proc. Natl. Acad. Sci. USA 89:10915-19, 1992. Коротко, две аминокислотные последовательности выравнивают для оптимизации оценок выравнивания с использованием штрафа за открытие гэпа, равного 10, штрафа за удлинение гэпа, равного 1, и матрицы оценки «blosum 62» Henikoff and Henikoff (там же), как показано ниже (аминокислоты обозначены стандартными однобуквенными кодами).
«Доля идентичности последовательности» между двумя или несколькими последовательностями нуклеиновых кислот или аминокислот является функцией количества идентичных положений, общих для последовательностей. Таким образом, % идентичности можно рассчитать как количество идентичных нуклеотидов/аминокислот, деленное на общее число нуклеотидов/аминокислот, умноженное на 100. При расчете % идентичности последовательности также можно учитывать количество гэпов и длину каждого гэпа, которые необходимо ввести для оптимизации выравнивания двух или нескольких последовательностей. Сравнение последовательностей и определение доли идентичности между двумя или несколькими последовательностями можно проводить с использованием конкретных математических алгоритмов, таких как BLAST, которые знакомы специалисту в данной области техники.
ОЦЕНКИ ВЫРАВНИВАНИЯ ДЛЯ ОПРЕДЕЛЕНИЯ ИДЕНТИЧНОСТИ ПОСЛЕДОВАТЕЛЬНОСТИ
A R N D C Q E G H I L K M F P S T W Y V
A 4
R -1 5
N -2 0 6
D -2 -2 1 6
C 0 -3 -3 -3 9
Q -1 1 0 0 -3 5
E -1 0 0 2 -4 2 5
G 0 -2 0 -1 -3 -2 -2 6
H -2 0 1 -1 -3 0 0 -2 8
I -1 -3 -3 -3 -1 -3 -3 -4 -3 4
L -1 -2 -3 -4 -1 -2 -3 -4 -3 2 4
K -1 2 0 -1 -3 1 1 -2 -1 -3 -2 5
M -1 -1 -2 -3 -1 0 -2 -3 -2 1 2 -1 5
F -2 -3 -3 -3 -2 -3 -3 -3 -1 0 0 -3 0 6
P -1 -2 -2 -1 -3 -1 -1 -2 -2 -3 -3 -1 -2 -4 7
S 1 -1 1 0 -1 0 0 0 -1 -2 -2 0 -1 -2 -1 4
T 0 -1 0 -1 -1 -1 -1 -2 -2 -1 -1 -1 -1 -2 -1 1 5
W -3 -3 -4 -4 -2 -2 -3 -2 -2 -3 -2 -3 -1 1 -4 -3 -2 11
Y -2 -2 -2 -3 -2 -1 -2 -3 2 -1 -1 -2 -1 3 -3 -2 -2 2 7
V 0 -3 -3 -3 -1 -2 -2 -3 -3 3 1 -2 1 -1 -2 -2 0 -3 -1 4
Затем долю идентичности рассчитывают как:
Общее количество одинаковых совпадений
__________________________________________ х 100
[длина более длинной последовательности плюс
количество гэпов, введенных в более длинную
последовательность, чтобы выровнять две последовательности]
По существу гомологичные полипептиды характеризуются наличием одной или нескольких аминокислотных замен, делеций или добавлений. Эти изменения, предпочтительно, носят незначительный характер, т.е. консервативные аминокислотные замены (см. ниже) и другие замены, которые существенно не влияют на свертывание или активность полипептида; небольшие делеции, обычно от одной до примерно 30 аминокислот; и небольшие удлинения на амино- или карбоксильных концах, такие как амино-концевой метиониновый остаток, небольшой линкерный пептид, содержащий вплоть до примерно 20-25 остатков, или метка аффинности.
КОНСЕРВАТИВНЫЕ ЗАМЕНЫ АМИНОКИСЛОТ
Основные: аргинин
лизин
гистидин
Кислые: глутаминовая кислота
аспарагиновая кислота
Полярные: глютамин
аспарагин
Гидрофобные: лейцин
изолейцин
валин
Ароматические: фенилаланин
триптофан
тирозин
Малые: глицин
аланин
серин
треонин
метионин
В дополнение к 20 стандартным аминокислотам, не стандартные аминокислоты (такие как 4-гидроксипролин, 6-N-метиллизин, 2-аминоизомасляная кислота, изовалин и α-метилсерин), могут быть замещены аминокислотными остатками или полипептидами по настоящему изобретению. Ограниченное количество не консервативных аминокислот, аминокислот, которые не кодированы генетическим кодом, и не природных аминокислот могут быть заменены полипептидными аминокислотными остатками. Полипептиды по настоящему изобретению могут также содержать не существующие в природе аминокислотные остатки.
Не существующие в природе аминокислоты включают, без ограничений, транс-3-метилпролин, 2,4-метанопролин, цис-4-гидроксипролин, транс-4-гидроксипролин, N-метилглицин, алло-треонин, метил-треонин, гидрокси-этилцистеин, гидроксиэтилгомо-цистеин, нитро-глутамин, гомоглутамин, пипеколиновую кислоту, трет-лейцин, норвалин, 2-азафенилаланин, 3-азафенил-аланин, 4-азафенил-аланин и 4-фторфенилаланин. Множество способов известны в данной области техники для включения не существующих в природе аминокислотных остатков в белки. Например, можно использовать систему in vitro, в которой нонсенс-мутации подавляются с помощью химически аминоацилированных супрессорных тРНК. Способы синтеза аминокислот и аминоацилирования тРНК известны в данной области техники. Транскрипцию и трансляцию плазмид, содержащих нонсенс-мутации, проводят в бесклеточной системе, содержащей экстракт E. coli S30 и коммерчески доступные ферменты и другие реагенты. Белки очищают хроматографией. См., например, Robertson et al., J. Am. Chem. Soc. 113:2722, 1991; Ellman et al., Methods Enzymol. 202:301, 1991; Chung et al., Science 259:806-9, 1993; and Chung et al., Proc. Natl. Acad. Sci. USA 90:10145-9, 1993. Во втором способе, трансляцию проводят в ооцитах Xenopus путем микроинъекции мутантной мРНК и химически аминоацилированных супрессорных тРНК (Turcatti et al., J. Biol. Chem. 271:19991-8, 1996). В рамках третьего способа, клетки E.coli культивируют в отсутствие природной аминокислоты, которую необходимо заменить (например, фенилаланин), и в присутствии желаемых не существующих в природе аминокислот (например, 2-азафенилаланина, 3-азафенилаланина, 4-азафенилаланина или 4-фторфенилаланина). Не существующую в природе аминокислоту вводят в полипептид вместо ее природного аналога. См. Koide et al., Biochem. 33:7470-6, 1994. Встречающиеся в природе аминокислотные остатки могут быть превращены в не встречающиеся в природе виды химической модификацией in vitro. Химическая модификация может быть скомбинирована с сайт-направленным мутагенезом для дальнейшего расширения диапазона замен (Wynn and Richards, Protein Sci. 2:395-403, 1993).
Ограниченное число не консервативных аминокислот, аминокислот, которые не кодируются генетическим кодом, не встречающихся в природе аминокислот и не природных аминокислот может быть заменено аминокислотными остатки полипептидов по настоящему изобретению.
Незаменимые аминокислоты в полипептидах по настоящему изобретению могут быть идентифицированы в соответствии с методами, известными в данной области техники, такими как сайт-направленный мутагенез или аланин-сканирующий мутагенез (Cunningham and Wells, Science 244: 1081-5, 1989). Сайты биологического взаимодействия также могут быть определены с помощью физического анализа структуры, определяемого такими методами, как ядерный магнитный резонанс, кристаллография, электронная дифракция или фотоаффинное мечение, в сочетании с мутацией аминокислот предполагаемого сайта контакта. См., например, de Vos et al., Science 255:306-12, 1992; Smith et al., J. Mol. Biol. 224:899-904, 1992; Wlodaver et al., FEBS Lett. 309:59-64, 1992. Идентичность аминокислот также можно вывести из анализа гомологии с родственными компонентами (например, транслокационными или протеазными компонентами) полипептидов по настоящему изобретению.
Множественные аминокислотные замены могут быть сделаны и протестированы с использованием известных способов мутагенеза и скрининга, таких как описаны в Reidhaar-Olson and Sauer (Science 241:53-7, 1988) or Bowie and Sauer (Proc. Natl. Acad. Sci. USA 86:2152-6, 1989). Коротко, эти авторы описывают способы одновременной рандомизации двух или нескольких положений в полипептиде для селекции функционального полипептида, и последующего секвенирования мутагенизированных полипептидов для определения спектра допустимых замен в каждом положении. Другие способы, которые могут быть использованы, включают фаговый дисплей (например, Lowman et al., Biochem. 30:10832-7, 1991; Ladner et al., патент США № 5,223,409; Huse, публикация WIPO WO 92/06204) и область-направленный мутагенез (Derbyshire et al., Gene 46:145, 1986; Ner et al., DNA 7:127, 1988).
Если не указано иное, все технические и научные термины, используемые в данном документе, имеют то же значение, которое обычно понимается специалистом в области, к которой относится данное описание. Singleton, et al., DICTIONARY OF MICROBIOLOGY AND MOLECULAR BIOLOGY, 20 ED., John Wiley and Sons, New York (1994), and Hale & Marham, THE HARPER COLLINS DICTIONARY OF BIOLOGY, Harper Perennial, NY (1991) предоставляют специалистам в данной области техники общий словарь многих терминов, используемых в данном описании.
Это описание не ограничено типовыми способами и материалами, описанными в настоящем изобретении, и любые способы и материалы, аналогичные или эквивалентные описанным в настоящем изобретении, могут использоваться на практике или при тестировании вариантов осуществления этого описания. Числовые диапазоны являются включительными для чисел, определяющих диапазон. Если не указано иное, любые последовательности нуклеиновых кислот записываются слева направо с ориентацией от 5' к 3'; аминокислотные последовательности записываются слева направо с ориентацией от амино к карбокси, соответственно.
Представленные в настоящем изобретении заголовки не являются ограничениями различных аспектов или вариантов осуществления настоящего описания.
Аминокислоты упоминаются в настоящем изобретении с использованием названия аминокислоты, трехбуквенной аббревиатуры или однобуквенной аббревиатуры. Термин «белок», используемый в настоящем изобретении, включает белки, полипептиды и пептиды. Используемый в настоящем изобретении термин «аминокислотная последовательность» является синонимом термина «полипептид» и/или термина «белок». В некоторых случаях, термин «аминокислотная последовательность» является синонимом термина «пептид». В некоторых случаях, термин «аминокислотная последовательность» является синонимом термина «фермент». Термины «белок» и «полипептид» используются в настоящем изобретении взаимозаменяемо. В настоящем описании и формуле изобретения, могут применяться общепринятые однобуквенные и трехбуквенные коды аминокислотных остатков. Используется трехбуквенный код аминокислот, определенный в соответствии с Объединенной комиссией IUPACIUB Joint Commission on Biochemical Nomenclature (JCBN). Также понятно, что полипептид может кодироваться более чем одной нуклеотидной последовательностью из-за вырожденности генетического кода.
В описании могут встречаться другие определения терминов. Прежде чем будут описаны более подробно типовые варианты осуществления, следует понимать, что это описание не ограничено конкретными описанными вариантами осуществления и, как таковое, может варьироваться. Также следует понимать, что терминология, используемая в настоящем изобретении, предназначена только для целей описания конкретных вариантов осуществления и не предназначена для ограничения, поскольку объем настоящего описания будет определяться только прилагаемой формулой изобретения.
Там, где указан диапазон значений, подразумевается, что каждое промежуточное значение, с точностью до десятой доли единицы нижнего предела, если контекст явно не указывает иное, между верхним и нижним пределами этого диапазона также конкретно описывается. Каждый меньший диапазон между любым заявленным значением или промежуточным значением в указанном диапазоне и любым другим заявленным или промежуточным значением в указанном диапазоне охватывается настоящим описанием. Верхний и нижний пределы этих меньших диапазонов могут независимо включаться или исключаться из диапазона, и каждый диапазон, в котором один или оба предела не включены в меньшие диапазоны, также охватывается данным описанием, с учетом любого специально исключенного предела в указанном диапазоне. Если указанный диапазон включает один или оба предела, диапазоны, исключающие один или оба из этих включенных пределов, также включены в данное описание.
Следует отметить, что используемые в настоящем изобретении и в прилагаемой формуле изобретения формы единственного числа включают ссылки во множественном числе, если из контекста явно не следует иное. Так, например, ссылка на «клостридиальный нейротоксин» включает множество таких агентов-кандидатов, и ссылка на «клостридиальный нейротоксин» включает ссылку на один или более клостридиальных нейротоксинов и их эквивалентов, известных специалистам в данной области техники, и так далее.
Публикации, обсуждаемые в настоящем изобретении, представлены исключительно для их описания до даты подачи настоящей заявки. Ничто в данном документе не должно быть истолковано как допущение того, что такие публикации представляют собой предшествующий уровень техники для прилагаемой формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
Далее будут описаны варианты осуществления изобретения только в качестве примера со ссылкой на следующие фигуры и примеры.
На фигуре 1 показан нейротрофический эффект различных рекомбинантно экспрессируемых каталитически неактивных серотипов BoNT по сравнению с положительным контролем нейротрофического фактора головного мозга (BDNF) в мотонейрон-подобной клеточной линии NSC34. * р<0,05 к необработанному контролю, однофакторный ANOVA с последующим критерием множественного сравнения Даннета. Данные представляют собой среднее значение ± стандартная ошибка трех независимых экспериментов, каждый из которых выполнен в шести повторяющихся лунках.
На фигуре 2 показан нейротрофический эффект фрагментов ботулинического нейротоксина серотипа А в мотонейрон-подобной клеточной линии NSC34 и эффект рекомбинантно экспрессированного каталитически неактивного BoNT/A. В качестве положительного контроля используют BDNF. * р<0,05 к необработанному контролю, однофакторный ANOVA с последующим критерием множественного сравнения Даннета. Данные представляют собой среднее значение ± стандартная ошибка трех независимых экспериментов, каждый из которых выполнен в шести повторяющихся лунках.
На фигуре 3 показан нейротрофический эффект отрицательных контролей по сравнению с рекомбинантно экспрессированным каталитически неактивным BoNT/A (BoNT/A (0)) в мотонейрон-подобной клеточной линии NSC34. В качестве положительного контроля используют BDNF. * р<0,05 к необработанному контролю, однофакторный ANOVA с последующим критерием множественного сравнения Даннета. Данные представляют собой среднее значение ± стандартная ошибка трех независимых экспериментов, каждый из которых выполнен в шести повторяющихся лунках.
На фигуре 4 показаны результаты теста с горизонтальной лестницей для мышей, которым вводили контрольный носитель (PBS) или rBoNT/A(0) в количестве 100 пг, 100 нг или 50 мкг.
На фигуре 5 показаны: (A) иммуногистохимия с использованием антител, связывающихся с нейрофиламентом 200 (NF200) через 4 недели после введения носителя (PBS) (левая панель) или 100 нг rBoNT/A(0) (правая панель); и (B) иммуногистохимия с использованием антител, связывающихся с MAP1b через 4 недели после введения носителя (PBS) (левая панель) или 100 нг rBoNT/A(0) (правая панель). Места поражения обозначены * (и на фигуре 5B обозначены белыми стрелками).
На фигуре 6 показано влияние (A) каталитически неактивного BoNT/A(0), (B) легкой цепи BoNT/A плюс фрагмент домена транслокации (LHN/A), (C) легкой цепи BoNT/A (LC/A, т.е. L/A), и (D) рецептор-связывающего домена BoNT/A (HC/A) на количество нейритов на клетку. BoNT или фрагмент BoNT сравнивают с BSA (отрицательный контроль), BDNF (положительный контроль) и тестируют при концентрациях 0,1 нМ, 1 нМ и 10 нМ. *p<0,05 по сравнению с контролем BSA, однофакторный ANOVA с последующим апостериорным тестом Даннетта. Данные представляют собой среднее ± с.о. среднего.
На фигуре 7 показано влияние (A) каталитически неактивного BoNT/FA(0), (B) легкой цепи BoNT/FA плюс фрагмент домена транслокации (LHN/FA), (C) легкой цепи BoNT/FA (LC/FA, т.е. L/FA), и (D) рецептор-связывающего домена BoNT/FA (HC/FA) на количество нейритов на клетку. BoNT или фрагмент BoNT сравнивают с BSA (отрицательный контроль), BDNF (положительный контроль) и тестируют при концентрациях 0,1 нМ, 1 нМ и 10 нМ. *p<0,05 по сравнению с контролем BSA, однофакторный ANOVA с последующим апостериорным тестом Даннетта. Данные представляют собой среднее ± с.о. среднего.
На фигуре 8 показано влияние (A) легкой цепи BoNT/F плюс фрагмент домена транслокации (LHN/F), (B) легкой цепи BoNT/F (LC/F, т.е. L/F) и (C) рецептор-связывающего домена BoNT/F (HC/F) по количеству нейритов на клетку. Фрагмент BoNT или BoNT сравнивают с BSA (отрицательный контроль), BDNF (положительный контроль) и тестируют при концентрациях 0,1 нМ, 1 нМ и 10 нМ. *p<0,05 по сравнению с контролем BSA, однофакторный ANOVA с последующим апостериорным тестом Даннетта. Данные представляют собой среднее ± с.о. среднего.
На фигуре 9 показано влияние катионного rHC/A (т.е. mrHC/A) на количество нейритов на клетку. Катионный фрагмент BoNT сравнивают с BSA (отрицательный контроль), BDNF (положительный контроль) и тестируют при концентрациях 0,1 нМ, 1 нМ и 10 нМ. *p<0,05 по сравнению с контролем BSA, однофакторный ANOVA с последующим апостериорным тестом Даннетта. Данные представляют собой среднее ± с.о. среднего.
На фигуре 10 показано влияние (A) toxHC/A YH (т.е. rHC/A варианта Y1117V H1253K) и (B) toxHC/A YFHL (L - H) (т.е. rHC/A варианта Y1117V F1252Y H1253K L1278H) на количество нейритов на клетку. Варианты фрагментов BoNT сравнивают с BSA (отрицательный контроль), BDNF (положительный контроль) и тестируют в концентрациях 0,1 нМ, 1 нМ и 10 нМ. *p<0,05 по сравнению с контролем BSA, однофакторный ANOVA с последующим апостериорным тестом Даннетта. Данные представляют собой среднее ± с.о. среднего.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Если начальный аминокислотный остаток Met или соответствующий начальный кодон указаны в любой из следующих SEQ ID NO, указанный остаток/кодон является необязательным.
SEQ ID NO: 1 - Нуклеотидная последовательность рекомбинантного каталитически неактивного BoNT/A (rBoNT/A(0))
SEQ ID NO: 2 - полипептидная последовательность rBoNT/A(0)
SEQ ID NO: 3 - Нуклеотидная последовательность rLHN/A (только легкая цепь плюс домен транслокации).
SEQ ID NO: 4 - Полипептидная последовательность rLHН/А
SEQ ID NO: 5 - Нуклеотидная последовательность rL/A (только легкая цепь)
SEQ ID NO: 6 - Полипептидная последовательность rL/A.
SEQ ID NO: 7 - Нуклеотидная последовательность rHC/A
SEQ ID NO: 8 - Полипептидная последовательность rHC/A
SEQ ID NO:9 - Нуклеотидная последовательность rBoNT/B(0)
SEQ ID NO: 10 - Полипептидная последовательность rBoNT/B(0)
SEQ ID NO: 11 - Нуклеотидная последовательность rBoNT/C(0)
SEQ ID NO: 12 - Полипептидная последовательность rBoNT/C(0)
SEQ ID NO: 13 - Нуклеотидная последовательность rBoNT/E(0)
SEQ ID NO: 14 - Полипептидная последовательность rBoNT/E(0)
SEQ ID NO: 15 - Нуклеотидная последовательность rBoNT/F(0)
SEQ ID NO: 16 - Полипептидная последовательность rBoNT/F(0)
SEQ ID NO: 17 - Нуклеотидная последовательность rBoNT/A(0) (His-меченная)
SEQ ID NO: 18 - Полипептидная последовательность rBoNT/A(0) (His-меченная)
SEQ ID NO: 19 - Нуклеотидная последовательность rLHN/A (His-меченная)
SEQ ID NO: 20 - Полипептидная последовательность rLHN/A (His-меченная)
SEQ ID NO: 21 - Нуклеотидная последовательность rHC/A (His-меченная)
SEQ ID NO: 22 - полипептидная последовательность rHC/A (His-меченная)
SEQ ID NO: 23 - Нуклеотидная последовательность rLC/A (His-меченная)
SEQ ID NO: 24 - Полипептидная последовательность rLC/A (His-меченная)
SEQ ID NO: 25 - Нуклеотидная последовательность rBoNT/FA(0) (His-меченная)
SEQ ID NO: 26 - Полипептидная последовательность rBoNT/FA(0) (His-меченная)
SEQ ID NO: 27 - Нуклеотидная последовательность rLHN/FA (His-меченная)
SEQ ID NO: 28 - Полипептидная последовательность rLHN/FA (His-меченная)
SEQ ID NO: 29 - Нуклеотидная последовательность rHC/FA (His-меченая)
SEQ ID NO: 30 - Полипептидная последовательность rHC/FA (His-меченная)
SEQ ID NO: 31 - Нуклеотидная последовательность rLC/FA (His-меченная)
SEQ ID NO: 32 - Полипептидная последовательность rLC/FA (His-меченная)
SEQ ID NO: 33 - Нуклеотидная последовательность rBoNT/F(0) (His-меченная)
SEQ ID NO: 34 - Полипептидная последовательность rBoNT/F(0) (His-меченная)
SEQ ID NO: 35 - Нуклеотидная последовательность rLHN/F (His-меченная)
SEQ ID NO: 36 - Полипептидная последовательность rLHN/F (His-меченная)
SEQ ID NO: 37 - Нуклеотидная последовательность rHC/F (His-меченная)
SEQ ID NO: 38 - Полипептидная последовательность rHC/F (His-меченная)
SEQ ID NO: 39 - Нуклеотидная последовательность rLC/F (His-меченная)
SEQ ID NO: 40 - Полипептидная последовательность rLC/F (His-меченная)
SEQ ID NO: 41 - Нуклеотидная последовательность катионного rHC/A (His-меченная)
SEQ ID NO: 42 - Полипептидная последовательность катионного rHC/A (His-меченная)
SEQ ID NO: 43 - Нуклеотидная последовательность rHC/AB (His-меченая)
SEQ ID NO: 44 - Полипептидная последовательность rHC/AB (His-меченная)
SEQ ID NO: 45 - Нуклеотидная последовательность варианта rHC/A Y1117V H1253K (His-меченная)
SEQ ID NO: 46 - Полипептидная последовательность варианта rHC/A Y1117V H1253K (His-меченная)
SEQ ID NO: 47 - Нуклеотидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278F (His-меченная)
SEQ ID NO: 48 - Полипептидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278F (His-меченная)
SEQ ID NO: 49 - Нуклеотидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278H (His-меченная)
SEQ ID NO: 50 - Полипептидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278H (His-меченная)
SEQ ID NO: 51 - Полипептидная последовательность BoNT/A - UniProt P10845
SEQ ID NO: 52 - Полипептидная последовательность BoNT/B - UniProt P10844
SEQ ID NO: 53 - Полипептидная последовательность BoNT/C - UniProt P18640
SEQ ID NO: 54 - Полипептидная последовательность BoNT/D - UniProt P19321
SEQ ID NO: 55 - Полипептидная последовательность BoNT/E - UniProt Q00496
SEQ ID NO: 56 - Полипептидная последовательность BoNT/F - UniProt A7GBG3
SEQ ID NO: 57 - Полипептидная последовательность BoNT/G - UniProt Q60393
SEQ ID NO: 58 - Полипептидная последовательность TeNT - UniProt P04958
SEQ ID NO: 59 - Полипептидная последовательность BoNT/X
SEQ ID NO: 60 - Нуклеотидная последовательность mrBoNT/A
SEQ ID NO: 61 - полипептидная последовательность mrBoNT/A.
SEQ ID NO: 62 - Полипептидная последовательность не модифицированного BoNT/A1
SEQ ID NO: 63 - Полипептидная последовательность mrBoNT/AB
SEQ ID NO: 64 - Полипептидная последовательность mrBoNT/AB(0)
SEQ ID NO: 65 - Полипептидная последовательность mrBoNT/A(0)
SEQ ID NO: 1 - Нуклеотидная последовательность rBoNT/A(0)
ATGCCATTCGTCAACAAGCAATTCAACTACAAAGACCCAGTCAACGGCGTCGACATCGCATACATCAAGATTCCGAACGCCGGTCAAATGCAGCCGGTTAAGGCTTTTAAGATCCACAACAAGATTTGGGTTATCCCGGAGCGTGACACCTTCACGAACCCGGAAGAAGGCGATCTGAACCCGCCACCGGAAGCGAAGCAAGTCCCTGTCAGCTACTACGATTCGACGTACCTGAGCACGGATAACGAAAAAGATAACTACCTGAAAGGTGTGACCAAGCTGTTCGAACGTATCTACAGCACGGATCTGGGTCGCATGCTGCTGACTAGCATTGTTCGCGGTATCCCGTTCTGGGGTGGTAGCACGATTGACACCGAACTGAAGGTTATCGACACTAACTGCATTAACGTTATTCAACCGGATGGTAGCTATCGTAGCGAAGAGCTGAATCTGGTCATCATTGGCCCGAGCGCAGACATTATCCAATTCGAGTGCAAGAGCTTTGGTCACGAGGTTCTGAATCTGACCCGCAATGGCTATGGTAGCACCCAGTACATTCGTTTTTCGCCGGATTTTACCTTCGGCTTTGAAGAGAGCCTGGAGGTTGATACCAATCCGTTGCTGGGTGCGGGCAAATTCGCTACCGATCCGGCTGTCACGCTGGCCCATcAACTGATCtACGCAGGCCACCGCCTGTACGGCATTGCCATCAACCCAAACCGTGTGTTCAAGGTTAATACGAATGCATACTACGAGATGAGCGGCCTGGAAGTCAGCTTCGAAGAACTGCGCACCTTCGGTGGCCATGACGCTAAATTCATTGACAGCTTGCAAGAGAATGAGTTCCGTCTGTACTACTATAACAAATTCAAAGACATTGCAAGCACGTTGAACAAGGCCAAAAGCATCGTTGGTACTACCGCGTCGTTGCAGTATATGAAGAATGTGTTTAAAGAGAAGTACCTGCTGTCCGAGGATACCTCCGGCAAGTTTAGCGTTGATAAGCTGAAGTTTGACAAACTGTACAAGATGCTGACCGAGATTTACACCGAGGACAACTTTGTGAAATTCTTCAAAGTGTTGAATCGTAAAACCTATCTGAATTTTGACAAAGCGGTTTTCAAGATTAACATCGTGCCGAAGGTGAACTACACCATCTATGACGGTTTTAACCTGCGTAACACCAACCTGGCGGCGAACTTTAACGGTCAGAATACGGAAATCAACAACATGAATTTCACGAAGTTGAAGAACTTCACGGGTCTGTTCGAGTTCTATAAGCTGCTGTGCGTGCGCGGTATCATCACCAGCAAAACCAAAAGCCTGGACAAAGGCTACAACAAGGCGCTGAATGACCTGTGCATTAAGGTAAACAATTGGGATCTGTTCTTTTCGCCATCCGAAGATAATTTTACCAACGACCTGAACAAGGGTGAAGAAATCACCAGCGATACGAATATTGAAGCAGCGGAAGAGAATATCAGCCTGGATCTGATCCAGCAGTACTATCTGACCTTTAACTTCGACAATGAACCGGAGAACATTAGCATTGAGAATCTGAGCAGCGACATTATCGGTCAGCTGGAACTGATGCCGAATATCGAACGTTTCCCGAACGGCAAAAAGTACGAGCTGGACAAGTACACTATGTTCCATTACCTGCGTGCACAGGAGTTTGAACACGGTAAAAGCCGTATCGCGCTGACCAACAGCGTTAACGAGGCCCTGCTGAACCCGAGCCGTGTCTATACCTTCTTCAGCAGCGACTATGTTAAGAAAGTGAACAAAGCCACTGAGGCCGCGATGTTCCTGGGCTGGGTGGAACAGCTGGTATATGACTTCACGGACGAGACGAGCGAAGTGAGCACTACCGACAAAATTGCTGATATTACCATCATTATCCCGTATATTGGTCCGGCACTGAACATTGGCAACATGCTGTACAAAGACGATTTTGTGGGTGCCCTGATCTTCTCCGGTGCCGTGATTCTGCTGGAGTTCATTCCGGAGATTGCGATCCCGGTGTTGGGTACCTTCGCGCTGGTGTCCTACATCGCGAATAAGGTTCTGACGGTTCAGACCATCGATAACGCGCTGTCGAAACGTAATGAAAAATGGGACGAGGTTTACAAATACATTGTTACGAATTGGCTGGCGAAAGTCAATACCCAGATCGACCTGATCCGTAAGAAAATGAAAGAGGCGCTGGAGAATCAGGCGGAGGCCACCAAAGCAATTATCAACTACCAATACAACCAGTACACGGAAGAAGAGAAGAATAACATTAACTTCAATATCGATGATTTGAGCAGCAAGCTGAATGAATCTATCAACAAAGCGATGATCAATATCAACAAGTTTTTGAATCAGTGTAGCGTTTCGTACCTGATGAATAGCATGATTCCGTATGGCGTCAAACGTCTGGAGGACTTCGACGCCAGCCTGAAAGATGCGTTGCTGAAATACATTTACGACAATCGTGGTACGCTGATTGGCCAAGTTGACCGCTTGAAAGACAAAGTTAACAATACCCTGAGCACCGACATCCCATTTCAACTGAGCAAGTATGTTGATAATCAACGTCTGTTGAGCACTTTCACCGAGTATATCAAAAACATCATCAATACTAGCATTCTGAACCTGCGTTACGAGAGCAATCATCTGATTGATCTGAGCCGTTATGCAAGCAAGATCAACATCGGTAGCAAGGTCAATTTTGACCCGATCGATAAGAACCAGATCCAGCTGTTTAATCTGGAATCGAGCAAAATTGAGGTTATCCTGAAAAACGCCATTGTCTACAACTCCATGTACGAGAATTTCTCCACCAGCTTCTGGATTCGCATCCCGAAATACTTCAACAGCATTAGCCTGAACAACGAGTATACTATCATCAACTGTATGGAGAACAACAGCGGTTGGAAGGTGTCTCTGAACTATGGTGAGATCATTTGGACCTTGCAGGACACCCAAGAGATCAAGCAGCGCGTCGTGTTCAAGTACTCTCAAATGATCAACATTTCCGATTACATTAATCGTTGGATCTTCGTGACCATTACGAATAACCGTCTGAATAACAGCAAGATTTACATCAATGGTCGCTTGATCGATCAGAAACCGATTAGCAACCTGGGTAATATCCACGCAAGCAACAACATTATGTTCAAATTGGACGGTTGCCGCGATACCCATCGTTATATCTGGATCAAGTATTTCAACCTGTTTGATAAAGAACTGAATGAGAAGGAGATCAAAGATTTGTATGACAACCAATCTAACAGCGGCATTTTGAAGGACTTCTGGGGCGATTATCTGCAATACGATAAGCCGTACTATATGCTGAACCTGTATGATCCGAACAAATATGTGGATGTCAATAATGTGGGTATTCGTGGTTACATGTATTTGAAGGGTCCGCGTGGCAGCGTTATGACGACCAACATTTACCTGAACTCTAGCCTGTACCGTGGTACGAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAACGATCGTGTCTACATCAACGTGGTCGTGAAGAATAAAGAGTACCGTCTGGCGACCAACGCTTCGCAGGCGGGTGTTGAGAAAATTCTGAGCGCGTTGGAGATCCCTGATGTCGGTAATCTGAGCCAAGTCGTGGTTATGAAGAGCAAGAACGACCAGGGTATCACTAACAAGTGCAAGATGAACCTGCAAGACAACAATGGTAACGACATCGGCTTTATTGGTTTCCACCAGTTCAACAATATTGCTAAACTGGTAGCGAGCAATTGGTACAATCGTCAGATTGAGCGCAGCAGCCGTACTTTGGGCTGTAGCTGGGAGTTTATCCCGGTCGATGATGGTTGGGGCGAACGTCCGCTG
SEQ ID NO: 2 - полипептидная последовательность rBoNT/A(0)
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHQLIYAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
SEQ ID NO: 3 - Нуклеотидная последовательность rLHN/A
atggagttcgttaacaaacagttcaactataaagacccagttaacggtgttgacattgcttacatcaaaatcccgaacgctggccagatgcagccggtaaaggcattcaaaatccacaacaaaatctgggttatcccggaacgtgatacctttactaacccggaagaaggtgacctgaacccgccaccggaagcgaaacaggtgccggtatcttactatgactccacctacctgtctaccgataacgaaaaggacaactacctgaaaggtgttactaaactgttcgagcgtatttactccaccgacctgggccgtatgctgctgactagcatcgttcgcggtatcccgttctggggcggttctaccatcgataccgaactgaaagtaatcgacactaactgcatcaacgttattcagccggacggttcctatcgttccgaagaactgaacctggtgatcatcggcccgtctgctgatatcatccagttcgagtgtaagagctttggtcacgaagttctgaacctcacccgtaacggctacggttccactcagtacatccgtttctctccggacttcaccttcggttttgaagaatccctggaagtagacacgaacccactgctgggcgctggtaaattcgcaactgatcctgcggttaccctggctcacgaactgattcatgcaggccaccgcctgtacggtatcgccatcaatccgaaccgtgtcttcaaagttaacaccaacgcgtattacgagatgtccggtctggaagttagcttcgaagaactgcgtacttttggcggtcacgacgctaaattcatcgactctctgcaagaaaacgagttccgtctgtactactataacaagttcaaagatatcgcatccaccctgaacaaagcgaaatccatcgtgggtaccactgcttctctccagtacatgaagaacgtttttaaagaaaaatacctgctcagcgaagacacctccggcaaattctctgtagacaagttgaaattcgataaactttacaaaatgctgactgaaatttacaccgaagacaacttcgttaagttctttaaagttctgaaccgcaaaacctatctgaacttcgacaaggcagtattcaaaatcaacatcgtgccgaaagttaactacactatctacgatggtttcaacctgcgtaacaccaacctggctgctaattttaacggccagaacacggaaatcaacaacatgaacttcacaaaactgaaaaacttcactggtctgttcgagttttacaagctgctgtgcGTCGACGGCATCATTACCTCCAAAACTAAATCTGACGATGACGATAAAAACAAAGCGCTGAACCTGCAGtgtatcaaggttaacaactgggatttattcttcagcccgagtgaagacaacttcaccaacgacctgaacaaaggtgaagaaatcacctcagatactaacatcgaagcagccgaagaaaacatctcgctggacctgatccagcagtactacctgacctttaatttcgacaacgagccggaaaacatttctatcgaaaacctgagctctgatatcatcggccagctggaactgatgccgaacatcgaacgtttcccaaacggtaaaaagtacgagctggacaaatataccatgttccactacctgcgcgcgcaggaatttgaacacggcaaatcccgtatcgcactgactaactccgttaacgaagctctgctcaacccgtcccgtgtatacaccttcttctctagcgactacgtgaaaaaggtcaacaaagcgactgaagctgcaatgttcttgggttgggttgaacagcttgtttatgattttaccgacgagacgtccgaagtatctactaccgacaaaattgcggatatcactatcatcatcccgtacatcggtccggctctgaacattggcaacatgctgtacaaagacgacttcgttggcgcactgatcttctccggtgcggtgatcctgctggagttcatcccggaaatcgccatcccggtactgggcacctttgctctggtttcttacattgcaaacaaggttctgactgtacaaaccatcgacaacgcgctgagcaaacgtaacgaaaaatgggatgaagtttacaaatatatcgtgaccaactggctggctaaggttaatactcagatcgacctcatccgcaaaaaaatgaaagaagcactggaaaaccaggcggaagctaccaaggcaatcattaactaccagtacaaccagtacaccgaggaagaaaaaaacaacatcaacttcaacatcgacgatctgtcctctaaactgaacgaatccatcaacaaagctatgatcaacatcaacaagttcctgaaccagtgctctgtaagctatctgatgaactccatgatcccgtacggtgttaaacgtctggaggacttcgatgcgtctctgaaagacgccctgctgaaatacatttacgacaaccgtggcactctgatcggtcaggttgatcgtctgaaggacaaagtgaacaataccttatcgaccgacatcccttttcagctcagtaaatatgtcgataaccaacgccttttgtccactctagaagcaCACCATCATCACcaccatcaccatcaccat
SEQ ID NO: 4 - Полипептидная последовательность rLHN/A
MEFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVDGIITSKTKSDDDDKNKALNLQCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTLEAHHHHHHHHHH
SEQ ID NO: 5 - Нуклеотидная последовательность rL/A
ATGCCATTCGTCAACAAGCAATTCAACTACAAAGACCCAGTCAACGGCGTCGACATCGCATACATCAAGATTCCGAACGCCGGTCAAATGCAGCCGGTTAAGGCTTTTAAGATCCACAACAAGATTTGGGTTATCCCGGAGCGTGACACCTTCACGAACCCGGAAGAAGGCGATCTGAACCCGCCACCGGAAGCGAAGCAAGTCCCTGTCAGCTACTACGATTCGACGTACCTGAGCACGGATAACGAAAAAGATAACTACCTGAAAGGTGTGACCAAGCTGTTCGAACGTATCTACAGCACGGATCTGGGTCGCATGCTGCTGACTAGCATTGTTCGCGGTATCCCGTTCTGGGGTGGTAGCACGATTGACACCGAACTGAAGGTTATCGACACTAACTGCATTAACGTTATTCAACCGGATGGTAGCTATCGTAGCGAAGAGCTGAATCTGGTCATCATTGGCCCGAGCGCAGACATTATCCAATTCGAGTGCAAGAGCTTTGGTCACGAGGTTCTGAATCTGACCCGCAATGGCTATGGTAGCACCCAGTACATTCGTTTTTCGCCGGATTTTACCTTCGGCTTTGAAGAGAGCCTGGAGGTTGATACCAATCCGTTGCTGGGTGCGGGCAAATTCGCTACCGATCCGGCTGTCACGCTGGCCCATGAACTGATCCACGCAGGCCACCGCCTGTACGGCATTGCCATCAACCCAAACCGTGTGTTCAAGGTTAATACGAATGCATACTACGAGATGAGCGGCCTGGAAGTCAGCTTCGAAGAACTGCGCACCTTCGGTGGCCATGACGCTAAATTCATTGACAGCTTGCAAGAGAATGAGTTCCGTCTGTACTACTATAACAAATTCAAAGACATTGCAAGCACGTTGAACAAGGCCAAAAGCATCGTTGGTACTACCGCGTCGTTGCAGTATATGAAGAATGTGTTTAAAGAGAAGTACCTGCTGTCCGAGGATACCTCCGGCAAGTTTAGCGTTGATAAGCTGAAGTTTGACAAACTGTACAAGATGCTGACCGAGATTTACACCGAGGACAACTTTGTGAAATTCTTCAAAGTGTTGAATCGTAAAACCTATCTGAATTTTGACAAAGCGGTTTTCAAGATTAACATCGTGCCGAAGGTGAACTACACCATCTATGACGGTTTTAACCTGCGTAACACCAACCTGGCGGCGAACTTTAACGGTCAGAATACGGAAATCAACAACATGAATTTCACGAAGTTGAAGAACTTCACGGGTCTGTTCGAGTTCTATAAGCTGCTGggtctagaagcaCACCATCATCACcaccatcaccatcaccat
SEQ ID NO: 6 - полипептидная последовательность rL/A
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLGLEAHHHHHHHHHH
SEQ ID NO: 7 - Нуклеотидная последовательность rHC/A
ATGCATCATCACCATCACCACAAAAACATCATCAATACTAGCATTCTGAACCTGCGTTACGAGAGCAATCATCTGATTGATCTGAGCCGTTATGCAAGCAAGATCAACATCGGTAGCAAGGTCAATTTTGACCCGATCGATAAGAACCAGATCCAGCTGTTTAATCTGGAATCGAGCAAAATTGAGGTTATCCTGAAAAACGCCATTGTCTACAACTCCATGTACGAGAATTTCTCCACCAGCTTCTGGATTCGCATCCCGAAATACTTCAACAGCATTAGCCTGAACAACGAGTATACTATCATCAACTGTATGGAGAACAACAGCGGTTGGAAGGTGTCTCTGAACTATGGTGAGATCATTTGGACCTTGCAGGACACCCAAGAGATCAAGCAGCGCGTCGTGTTCAAGTACTCTCAAATGATCAACATTTCCGATTACATTAATCGTTGGATCTTCGTGACCATTACGAATAACCGTCTGAATAACAGCAAGATTTACATCAATGGTCGCTTGATCGATCAGAAACCGATTAGCAACCTGGGTAATATCCACGCAAGCAACAACATTATGTTCAAATTGGACGGTTGCCGCGATACCCATCGTTATATCTGGATCAAGTATTTCAACCTGTTTGATAAAGAACTGAATGAGAAGGAGATCAAAGATTTGTATGACAACCAATCTAACAGCGGCATTTTGAAGGACTTCTGGGGCGATTATCTGCAATACGATAAGCCGTACTATATGCTGAACCTGTATGATCCGAACAAATATGTGGATGTCAATAATGTGGGTATTCGTGGTTACATGTATTTGAAGGGTCCGCGTGGCAGCGTTATGACGACCAACATTTACCTGAACTCTAGCCTGTACCGTGGTACGAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAACGATCGTGTCTACATCAACGTGGTCGTGAAGAATAAAGAGTACCGTCTGGCGACCAACGCTTCGCAGGCGGGTGTTGAGAAAATTCTGAGCGCGTTGGAGATCCCTGATGTCGGTAATCTGAGCCAAGTCGTGGTTATGAAGAGCAAGAACGACCAGGGTATCACTAACAAGTGCAAGATGAACCTGCAAGACAACAATGGTAACGACATCGGCTTTATTGGTTTCCACCAGTTCAACAATATTGCTAAACTGGTAGCGAGCAATTGGTACAATCGTCAGATTGAGCGCAGCAGCCGTACTTTGGGCTGTAGCTGGGAGTTTATCCCGGTCGATGATGGTTGGGGCGAACGTCCGCTG
SEQ ID NO: 8 - Полипептидная последовательность rHC/A
MHHHHHHKNIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
SEQ ID NO:9 - Нуклеотидная последовательность rBoNT/B(0)
ATGCCGGTGACGATTAACAACTTCAACTACAACGACCCGATTGACAACAACAACATTATCATGATGGAACCGCCGTTTGCACGCGGCACGGGCCGTTATTACAAAGCGTTTAAAATCACCGATCGTATTTGGATTATCCCGGAACGCTACACGTTTGGTTATAAACCGGAAGACTTCAACAAAAGCTCTGGCATCTTCAACCGTGATGTTTGCGAATACTACGATCCGGACTACCTGAACACCAACGATAAGAAAAACATTTTTCTGCAAACGATGATCAAACTGTTCAATCGCATTAAAAGCAAACCGCTGGGTGAAAAACTGCTGGAAATGATTATCAATGGCATTCCGTATCTGGGTGATCGTCGCGTGCCGCTGGAAGAATTTAACACCAATATCGCGAGTGTTACGGTCAACAAACTGATTTCCAATCCGGGTGAAGTCGAACGTAAAAAAGGCATCTTCGCCAACCTGATCATCTTCGGCCCGGGTCCGGTGCTGAACGAAAATGAAACCATTGATATCGGTATTCAGAACCATTTTGCCTCACGCGAAGGCTTCGGCGGTATTATGCAAATGAAATTTTGCCCGGAATATGTGTCGGTTTTCAACAATGTTCAGGAAAACAAAGGTGCAAGCATCTTTAATCGTCGCGGCTATTTCTCTGATCCGGCTCTGATCCTGATGCACcAACTGATTtATGTGCTGCACGGCCTGTATGGTATCAAAGTGGATGACCTGCCGATCGTTCCGAACGAGAAAAAATTTTTCATGCAGAGCACCGACGCAATTCAAGCTGAAGAACTGTATACGTTTGGCGGTCAGGACCCGTCTATTATCACCCCGAGCACCGACAAAAGCATCTACGATAAAGTGCTGCAAAACTTTCGTGGCATTGTTGACCGCCTGAATAAAGTCCTGGTGTGTATCTCTGATCCGAACATCAACATCAACATCTACAAAAACAAATTCAAAGACAAATACAAATTCGTTGAAGATTCTGAAGGCAAATATAGTATTGACGTCGAATCCTTTGATAAACTGTACAAAAGTCTGATGTTCGGTTTCACCGAAACGAACATCGCGGAAAACTACAAAATCAAAACCCGCGCCTCCTATTTCAGCGACTCTCTGCCGCCGGTTAAAATCAAAAATCTGCTGGATAACGAAATTTATACGATCGAAGAAGGTTTCAACATCAGCGATAAAGACATGGAAAAAGAATACCGTGGCCAGAATAAAGCAATCAACAAACAGGCGTATGAAGAAATTAGTAAAGAACATCTGGCGGTCTACAAAATTCAGATGTGCAAATCCGTGAAAGCCCCGGGTATTTGTATCGATGTTGACAATGAAGACCTGTTTTTCATCGCCGATAAAAACAGTTTTTCCGATGACCTGTCAAAAAATGAACGCATCGAATACAACACCCAATCGAACTACATCGAAAACGATTTCCCGATCAACGAACTGATTCTGGATACGGACCTGATTAGTAAAATCGAACTGCCGTCAGAAAACACCGAATCGCTGACGGACTTTAATGTTGATGTCCCGGTGTATGAAAAACAGCCGGCAATTAAGAAAATTTTTACCGATGAAAACACGATCTTCCAGTACCTGTACAGCCAAACCTTTCCGCTGGACATTCGCGATATCTCTCTGACGAGTTCCTTTGATGACGCACTGCTGTTCAGCAACAAAGTGTACTCCTTTTTCTCAATGGATTACATCAAAACCGCTAACAAAGTGGTTGAAGCGGGCCTGTTTGCCGGTTGGGTGAAACAGATCGTTAACGATTTCGTCATCGAAGCCAACAAAAGTAACACGATGGATAAAATTGCTGATATCTCCCTGATTGTCCCGTATATTGGCCTGGCACTGAATGTGGGTAACGAAACGGCGAAAGGCAATTTTGAAAACGCCTTCGAAATTGCAGGCGCTTCAATCCTGCTGGAATTTATTCCGGAACTGCTGATCCCGGTCGTGGGTGCGTTCCTGCTGGAATCTTACATCGACAACAAAAACAAAATCATCAAAACCATTGATAACGCGCTGACGAAACGTAACGAAAAATGGTCAGATATGTACGGCCTGATTGTTGCCCAGTGGCTGAGCACCGTCAACACGCAATTTTACACCATCAAAGAAGGTATGTACAAAGCGCTGAATTATCAGGCGCAAGCCCTGGAAGAAATCATCAAATACCGCTACAACATCTACAGCGAAAAAGAAAAATCTAACATCAACATCGACTTTAATGATATCAACAGCAAACTGAACGAAGGTATCAACCAGGCAATCGATAACATCAACAACTTCATCAACGGCTGCTCAGTGTCGTATCTGATGAAGAAAATGATCCCGCTGGCTGTTGAAAAACTGCTGGATTTTGACAACACCCTGAAGAAAAACCTGCTGAACTACATCGATGAAAACAAACTGTACCTGATCGGCTCAGCCGAATACGAAAAATCGAAAGTGAACAAATACCTGAAAACCATCATGCCGTTTGACCTGAGTATTTACACCAACGATACGATCCTGATCGAAATGTTCAACAAATACAACTCCGAAATTCTGAACAATATTATCCTGAACCTGCGTTACAAAGACAACAATCTGATCGATCTGAGCGGCTATGGTGCAAAAGTTGAAGTCTACGACGGTGTCGAACTGAACGATAAAAACCAGTTCAAACTGACCTCATCGGCTAACTCAAAAATTCGTGTGACGCAGAACCAAAACATCATCTTCAACTCGGTCTTTCTGGACTTCAGCGTGTCTTTCTGGATTCGCATCCCGAAATATAAAAATGATGGCATCCAGAACTACATCCATAACGAATACACCATCATCAACTGTATGAAAAACAACAGTGGTTGGAAAATTTCCATCCGTGGCAACCGCATTATCTGGACCCTGATTGATATCAATGGTAAAACGAAAAGCGTGTTTTTCGAATACAACATCCGTGAAGATATCTCTGAATACATCAATCGCTGGTTTTTCGTGACCATTACGAACAATCTGAACAATGCGAAAATCTATATCAACGGCAAACTGGAAAGTAATACCGACATCAAAGATATTCGTGAAGTTATCGCCAACGGTGAAATCATCTTCAAACTGGATGGCGACATCGATCGCACCCAGTTCATTTGGATGAAATACTTCTCCATCTTCAACACGGAACTGAGTCAGTCCAATATCGAAGAACGCTACAAAATCCAATCATACTCGGAATACCTGAAAGATTTCTGGGGTAACCCGCTGATGTACAACAAAGAATACTACATGTTCAACGCGGGCAACAAAAACTCATACATCAAACTGAAAAAAGATTCGCCGGTGGGTGAAATCCTGACCCGTAGCAAATACAACCAGAACTCTAAATACATCAACTATCGCGATCTGTACATTGGCGAAAAATTTATTATCCGTCGCAAAAGCAACTCTCAGAGTATTAATGATGACATCGTGCGTAAAGAAGACTACATCTATCTGGATTTCTTTAATCTGAACCAAGAATGGCGCGTTTATACCTACAAATACTTCAAAAAAGAAGAAGAGAAACTGTTCCTGGCCCCGATTAGCGACAGCGATGAATTTTACAACACCATCCAGATCAAAGAATACGATGAACAGCCGACGTATAGTTGCCAACTGCTGTTCAAAAAAGACGAAGAATCCACCGATGAAATTGGCCTGATTGGTATCCACCGTTTCTATGAAAGCGGTATCGTTTTCGAAGAATACAAAGATTACTTCTGTATCTCTAAATGGTATCTGAAAGAAGTCAAACGCAAACCGTACAACCTGAAACTGGGCTGCAACTGGCAATTTATCCCGAAAGACGAAGGCTGGACCGAA
SEQ ID NO: 10 - Полипептидная последовательность rBoNT/B(0)
MPVTINNFNYNDPIDNNNIIMMEPPFARGTGRYYKAFKITDRIWIIPERYTFGYKPEDFNKSSGIFNRDVCEYYDPDYLNTNDKKNIFLQTMIKLFNRIKSKPLGEKLLEMIINGIPYLGDRRVPLEEFNTNIASVTVNKLISNPGEVERKKGIFANLIIFGPGPVLNENETIDIGIQNHFASREGFGGIMQMKFCPEYVSVFNNVQENKGASIFNRRGYFSDPALILMHQLIYVLHGLYGIKVDDLPIVPNEKKFFMQSTDAIQAEELYTFGGQDPSIITPSTDKSIYDKVLQNFRGIVDRLNKVLVCISDPNININIYKNKFKDKYKFVEDSEGKYSIDVESFDKLYKSLMFGFTETNIAENYKIKTRASYFSDSLPPVKIKNLLDNEIYTIEEGFNISDKDMEKEYRGQNKAINKQAYEEISKEHLAVYKIQMCKSVKAPGICIDVDNEDLFFIADKNSFSDDLSKNERIEYNTQSNYIENDFPINELILDTDLISKIELPSENTESLTDFNVDVPVYEKQPAIKKIFTDENTIFQYLYSQTFPLDIRDISLTSSFDDALLFSNKVYSFFSMDYIKTANKVVEAGLFAGWVKQIVNDFVIEANKSNTMDKIADISLIVPYIGLALNVGNETAKGNFENAFEIAGASILLEFIPELLIPVVGAFLLESYIDNKNKIIKTIDNALTKRNEKWSDMYGLIVAQWLSTVNTQFYTIKEGMYKALNYQAQALEEIIKYRYNIYSEKEKSNINIDFNDINSKLNEGINQAIDNINNFINGCSVSYLMKKMIPLAVEKLLDFDNTLKKNLLNYIDENKLYLIGSAEYEKSKVNKYLKTIMPFDLSIYTNDTILIEMFNKYNSEILNNIILNLRYKDNNLIDLSGYGAKVEVYDGVELNDKNQFKLTSSANSKIRVTQNQNIIFNSVFLDFSVSFWIRIPKYKNDGIQNYIHNEYTIINCMKNNSGWKISIRGNRIIWTLIDINGKTKSVFFEYNIREDISEYINRWFFVTITNNLNNAKIYINGKLESNTDIKDIREVIANGEIIFKLDGDIDRTQFIWMKYFSIFNTELSQSNIEERYKIQSYSEYLKDFWGNPLMYNKEYYMFNAGNKNSYIKLKKDSPVGEILTRSKYNQNSKYINYRDLYIGEKFIIRRKSNSQSINDDIVRKEDYIYLDFFNLNQEWRVYTYKYFKKEEEKLFLAPISDSDEFYNTIQIKEYDEQPTYSCQLLFKKDEESTDEIGLIGIHRFYESGIVFEEYKDYFCISKWYLKEVKRKPYNLKLGCNWQFIPKDEGWTE
SEQ ID NO: 11 - Нуклеотидная последовательность rBoNT/C(0)
ATGCCGATCACGATTAATAATTTCAACTATAGCGATCCGGTGGACAATAAGAATATTCTGTATCTGGATACTCATCTGAATACGCTGGCTAACGAACCGGAGAAAGCGTTCCGCATCACAGGCAACATCTGGGTTATTCCCGATCGCTTTTCACGCAACAGCAACCCTAATCTGAACAAACCTCCTCGTGTCACCAGTCCTAAATCCGGTTATTACGACCCAAACTATCTGAGTACGGATAGCGATAAAGATCCCTTTCTGAAAGAGATCATTAAGCTGTTCAAACGCATTAACTCTCGCGAAATTGGGGAAGAGCTGATCTATCGGCTTTCGACAGATATCCCGTTCCCAGGTAACAATAATACCCCGATTAATACTTTCGACTTTGATGTTGATTTCAATTCTGTGGATGTGAAAACGCGTCAAGGCAATAATTGGGTGAAAACTGGTAGCATTAACCCGAGTGTAATTATCACAGGTCCCCGTGAGAACATCATCGACCCGGAAACCTCTACCTTCAAGCTGACGAACAACACGTTTGCTGCACAGGAAGGGTTTGGTGCCCTGTCAATCATTTCCATCTCACCGCGTTTCATGTTAACCTACTCCAATGCCACAAATGATGTTGGCGAAGGACGTTTTAGCAAATCAGAATTTTGCATGGACCCAATTCTCATTCTGATGggCacGCTGAACaATGCGATGCACAACTTGTATGGCATTGCTATTCCAAACGATCAAACCATTAGCTCCGTTACCAGTAATATCTTCTATAGCCAGTATAATGTCAAATTGGAGTATGCCGAAATTTACGCCTTTGGAGGCCCGACCATTGACCTGATTCCGAAATCTGCACGCAAATACTTCGAAGAAAAGGCGTTAGATTACTATCGCAGCATCGCGAAACGCCTGAACTCGATTACCACGGCCAATCCGTCGTCGTTCAACAAATACATTGGTGAATATAAACAGAAACTGATTCGCAAATATCGGTTTGTCGTAGAAAGCTCTGGTGAAGTGACTGTAAACCGCAACAAATTTGTCGAACTCTACAACGAGTTGACCCAAATCTTTACCGAGTTTAACTACGCAAAGATCTATAACGTACAGAACCGCAAGATTTATCTTAGCAATGTATACACACCGGTTACTGCGAACATCTTAGACGACAATGTGTATGATATTCAGAATGGCTTTAACATCCCGAAATCAAATCTGAACGTTCTGTTTATGGGCCAGAACCTGAGTCGTAATCCAGCACTGCGTAAAGTGAACCCGGAAAATATGCTCTACTTGTTTACCAAATTTTGCCACAAAGCGATTGATGGCCGCTCTCTCTATAACAAAACGCTGGATTGTCGTGAGTTACTTGTGAAGAACACTGATTTACCGTTCATTGGGGATATCTCCGACGTGAAAACCGATATCTTCCTGCGCAAAGACATTAATGAAGAAACGGAAGTCATCTATTACCCCGACAATGTGAGCGTTGATCAGGTCATTTTATCGAAGAACACCTCCGAACATGGTCAGTTGGATTTGCTGTACCCTAGCATTGACTCGGAGAGTGAAATCCTTCCGGGCGAAAATCAAGTGTTTTACGACAACCGTACCCAAAATGTTGATTATTTGAATTCTTATTACTACCTGGAATCTCAGAAATTGAGCGACAATGTGGAAGATTTCACGTTCACACGCTCCATTGAGGAAGCGCTGGATAATAGCGCGAAAGTGTATACGTATTTCCCTACCTTGGCGAATAAAGTAAATGCTGGTGTCCAGGGAGGCTTATTTCTGATGTGGGCGAATGATGTGGTAGAAGATTTTACGACCAATATTTTGCGTAAGGACACCTTAGATAAAATTAGCGATGTTAGCGCCATCATCCCCTATATTGGCCCAGCACTGAATATCTCGAACTCTGTGCGTCGCGGAAACTTCACCGAAGCATTTGCGGTGACCGGGGTTACTATTCTGTTGGAAGCCTTTCCGGAGTTTACTATTCCGGCGCTGGGTGCGTTTGTGATTTATTCGAAAGTACAAGAACGCAATGAAATTATCAAAACCATCGATAATTGCCTGGAACAACGCATTAAACGCTGGAAGGATTCTTATGAATGGATGATGGGCACCTGGTTATCCCGTATTATCACACAGTTTAACAACATCTCGTATCAGATGTACGATTCACTGAACTACCAAGCAGGGGCGATCAAAGCCAAGATCGACTTAGAATACAAGAAATATTCAGGTAGCGATAAAGAGAATATTAAAAGCCAGGTTGAAAACCTGAAGAACTCTCTGGATGTCAAAATTTCAGAGGCTATGAACAACATTAACAAATTTATCCGCGAATGTAGCGTCACGTATCTGTTTAAAAACATGCTCCCGAAAGTGATTGATGAGCTCAACGAGTTTGATCGCAACACAAAGGCCAAACTGATTAACCTGATTGATAGTCACAATATTATTTTAGTCGGTGAAGTTGACAAGCTGAAGGCTAAGGTCAATAACAGCTTTCAGAACACTATTCCGTTTAATATTTTCTCCTATACGAACAATAGTCTGCTGAAAGACATTATCAACGAATACTTCAACAATATTAATGACAGCAAAATTCTGAGCCTGCAGAATCGTAAGAATACGCTGGTAGATACCAGTGGATATAATGCGGAAGTCTCAGAAGAGGGTGATGTACAGCTGAACCCGATCTTTCCGTTCGACTTTAAACTGGGGTCTAGTGGTGAAGATCGCGGTAAAGTGATCGTTACCCAAAACGAGAACATTGTGTATAACAGCATGTACGAGAGTTTCTCAATTTCTTTCTGGATTCGCATCAATAAATGGGTTTCTAATTTGCCTGGCTATACCATCATTGATAGCGTCAAAAACAACTCGGGCTGGTCGATTGGCATTATTAGCAACTTTCTGGTGTTTACCCTGAAACAGAATGAGGATTCGGAACAGAGCATTAACTTCTCCTACGACATCAGCAACAATGCACCAGGGTATAACAAATGGTTCTTCGTAACGGTGACGAACAATATGATGGGCAATATGAAAATCTACATTAACGGGAAACTTATCGACACCATTAAAGTGAAAGAGCTTACTGGGATCAATTTTAGTAAAACCATTACCTTTGAGATCAACAAAATTCCGGACACGGGTCTGATTACCTCCGATTCGGATAATATCAATATGTGGATTCGCGACTTTTATATCTTCGCCAAAGAACTTGATGGCAAAGATATCAACATTTTGTTTAATTCCCTGCAGTATACCAATGTCGTTAAGGACTATTGGGGCAATGATCTCCGCTACAATAAAGAATACTACATGGTTAACATCGACTATCTCAATCGCTACATGTATGCTAACTCGCGTCAAATTGTGTTTAACACACGTCGTAACAACAACGATTTTAACGAAGGTTATAAAATCATTATCAAACGGATCCGCGGCAATACGAACGATACTCGTGTTCGTGGCGGTGACATTCTGTATTTCGACATGACGATTAATAATAAAGCGTACAATCTGTTCATGAAGAACGAAACCATGTACGCCGATAACCATTCCACTGAAGATATCTACGCAATCGGACTTCGCGAACAGACCAAAGACATTAACGACAACATCATCTTTCAGATTCAACCGATGAATAATACCTACTACTATGCCTCCCAGATCTTCAAAAGTAATTTCAACGGCGAAAACATTTCAGGCATTTGCTCAATCGGCACTTATCGGTTCCGGTTAGGTGGTGATTGGTATCGTCACAACTACCTTGTTCCCACAGTGAAACAAGGCAACTATGCATCGCTCTTAGAAAGCACATCTACGCATTGGGGTTTTGTGCCAGTCAGTGAA
SEQ ID NO: 12 - полипептидная последовательность rBoNT/C(0)
MPITINNFNYSDPVDNKNILYLDTHLNTLANEPEKAFRITGNIWVIPDRFSRNSNPNLNKPPRVTSPKSGYYDPNYLSTDSDKDPFLKEIIKLFKRINSREIGEELIYRLSTDIPFPGNNNTPINTFDFDVDFNSVDVKTRQGNNWVKTGSINPSVIITGPRENIIDPETSTFKLTNNTFAAQEGFGALSIISISPRFMLTYSNATNDVGEGRFSKSEFCMDPILILMGTLNNAMHNLYGIAIPNDQTISSVTSNIFYSQYNVKLEYAEIYAFGGPTIDLIPKSARKYFEEKALDYYRSIAKRLNSITTANPSSFNKYIGEYKQKLIRKYRFVVESSGEVTVNRNKFVELYNELTQIFTEFNYAKIYNVQNRKIYLSNVYTPVTANILDDNVYDIQNGFNIPKSNLNVLFMGQNLSRNPALRKVNPENMLYLFTKFCHKAIDGRSLYNKTLDCRELLVKNTDLPFIGDISDVKTDIFLRKDINEETEVIYYPDNVSVDQVILSKNTSEHGQLDLLYPSIDSESEILPGENQVFYDNRTQNVDYLNSYYYLESQKLSDNVEDFTFTRSIEEALDNSAKVYTYFPTLANKVNAGVQGGLFLMWANDVVEDFTTNILRKDTLDKISDVSAIIPYIGPALNISNSVRRGNFTEAFAVTGVTILLEAFPEFTIPALGAFVIYSKVQERNEIIKTIDNCLEQRIKRWKDSYEWMMGTWLSRIITQFNNISYQMYDSLNYQAGAIKAKIDLEYKKYSGSDKENIKSQVENLKNSLDVKISEAMNNINKFIRECSVTYLFKNMLPKVIDELNEFDRNTKAKLINLIDSHNIILVGEVDKLKAKVNNSFQNTIPFNIFSYTNNSLLKDIINEYFNNINDSKILSLQNRKNTLVDTSGYNAEVSEEGDVQLNPIFPFDFKLGSSGEDRGKVIVTQNENIVYNSMYESFSISFWIRINKWVSNLPGYTIIDSVKNNSGWSIGIISNFLVFTLKQNEDSEQSINFSYDISNNAPGYNKWFFVTVTNNMMGNMKIYINGKLIDTIKVKELTGINFSKTITFEINKIPDTGLITSDSDNINMWIRDFYIFAKELDGKDINILFNSLQYTNVVKDYWGNDLRYNKEYYMVNIDYLNRYMYANSRQIVFNTRRNNNDFNEGYKIIIKRIRGNTNDTRVRGGDILYFDMTINNKAYNLFMKNETMYADNHSTEDIYAIGLREQTKDINDNIIFQIQPMNNTYYYASQIFKSNFNGENISGICSIGTYRFRLGGDWYRHNYLVPTVKQGNYASLLESTSTHWGFVPVSE
SEQ ID NO: 13 - Нуклеотидная последовательность rBoNT/E(0)
atgccgaaaatcaactctttcaactacaacgacccggttaacgaccgtaccatcctgtatatcaaaccgggtggttgccaggagttctacaaatctttcaacatcatgaaaaacatctggatcatcccggaacgtaacgttatcggtaccaccccgcaggacttccacccgccgacctctctgaaaaacggtgactcttcttactacgacccgaactacctccagtctgacgaagaaaaagaccgtttcctgaaaatcgttaccaaaatcttcaaccgtatcaacaacaacctgtctggtggtatcctgctggaagaactgtctaaagctaacccgtacctgggtaacgacaacaccccggacaaccagttccacatcggtgacgcttctgctgttgaaatcaaattctctaacggttctcaggacatcctgctgccgaacgttatcatcatgggtgctgaaccggacctgttcgaaaccaactcttctaacatctctctgcgtaacaactacatgccgtctaaccacggtttcggttctatcgctatcgttaccttctctccggaatactctttccgtttcaacgacaacagcatgaacgagttcatccaggacccggctctgaccctgatgcaccaactgatctactctctgcacggtctgtacggtgctaaaggtatcaccaccaaatacaccatcacccagaaacagaacccgctgatcaccaacatccgtggtaccaacatcgaagagttcctgaccttcggtggtaccgacctgaacatcatcacctctgctcagtctaacgacatctacaccaacctgctggctgactacaaaaaaatcgcttctaaactgtctaaagttcaggtttctaacccgctgctgaacccgtacaaagacgttttcgaagctaaatacggtctggacaaagacgcttctggtatctactctgttaacatcaacaaattcaacgacatcttcaaaaaactgtactctttcaccgagttcgacctggcgaccaaattccaggttaaatgccgtcagacctacatcggtcagtacaaatacttcaaactgtctaacctgctgaacgactctatctacaacatctctgaaggttacaacatcaacaacctgaaagttaacttccgtggtcagaacgctaacctgaacccgcgtatcatcaccccgatcaccggtcgtggtctggttaaaaaaatcatccgtttctgcAAGAATATTGTAAGCGTTAAAGGAATAAGAAAAAGTATCtgcatcgaaatcaacaacggtgaactgttcttcgttgcttctgaaaactcttacaacgacgacaacatcaacaccccgaaagaaatcgacgacaccgttacctctaacaacaactacgaaaacgacctggaccaggttatcctgaacttcaactctgaatctgctccgggtctgtctgacgaaaaactgaacctgaccatccagaacgacgcttacatcccgaaatacgactctaacggtacctctgacatcgaacagcacgacgttaacgaactgaacgttttcttctacctggacgctcagaaagttccggaaggtgaaaacaacgttaacctgacctcttctatcgacaccgctctgctggaacagccgaaaatctacaccttcttctcttctgagttcatcaacaacgttaacaaaccggttcaggctgctctgttcgtttcttggattcagcaggttctggttgacttcaccaccgaagctaaccagaaatctaccgttgacaaaatcgctgacatctctatcgttgttccgtacatcggtctggctctgaacatcggtaacgaagctcagaaaggtaacttcaaagacgctctggaactgctgggtgctggtatcctgctggagttcgaaccggaactgctgatcccgaccatcctggttttcaccatcaaatctttcctgggttcttctgacaacaaaaacaaagttatcaaagctatcaacaacgctctgaaagaacgtgacgaaaaatggaaagaagtttactctttcatcgtttctaactggatgaccaaaatcaacacccagttcaacaaacgtaaagaacagatgtaccaggctctccagaaccaggttaacgctatcaaaaccatcatcgaatctaaatacaactcttacaccctggaagaaaaaaacgaactgaccaacaaatacgacatcaaacagatcgaaaacgaactgaaccagaaagtttctatcgctatgaacaacatcgaccgtttcctgaccgaatcttctatctcttacctgatgaaactcatcaacgaagttaaaatcaacaaactgcgtgaatacgacgaaaacgttaaaacctacctgctgaactacatcatccagcacggttctatcctgggtgaatctcagcaggaactgaactctatggttaccgacaccctgaacaactctatcccgttcaaactgtcttcttacaccgacgacaaaatcctGATCTCTTACTTCAACAAATTCTTTAAAcgcATTAAGAGTTCATCGGTTctgaatATGCGGTACAAAAATGATAAAtatGTCGATACTTCTGGATATgatAGCAATATCAACATTAACGGCGACGTGTATAAATATccgACAAATAAAAACCAGTTTGGGATATATAACGACAAGctgTCGGAGGTCAATattTCTCAAAACGACtatATCattTACGATAATaaaTATAAAAACTTTAGCATTAGTtttTGGGTTcgtATACCTAATtatGACAATaaaattGTAAATGTGAATAACGAGTATACCATTATAAACTGTATGcgcGACAATAACAGTGGTTGGAAGGTATCGctgAACCATAATGAGATTATCTGGACCctgcagGATAATgcaGGTATAAACCAGAAACTGGCTTTTAACTATGGAAACGCAAATGGGATCTCAGATTACATTaataaaTGGatttttGTTaccATTACGAACGATcgcTTAGGCGACTCAAAACTTTATATTAATggcAATctgATAGATCAGAAATCAATCTTAAATTTGGGCAATATTCATGTCTCTgatAACATCTTGTTCAAGATCGTTAATTGCAGTTACACTcgtTATATTGGCATTCGTTACTTTAATATCTTCgataaaGAActgGACGAGACGGAAATCcagACTCTGTATTCAAACGAGCCCAATACTAATATATTGAAAGATTTTTGGGGTAACTATCTTTTATATGATAAAGAATACTATCTCCTGaatGTATTGAAGCCAAACAATTTCATAGATAGACGCAAGGATAGCACATTAAGTATCAACAATATCAGATCTACTATActgttaGCAAATCGCCTcTACTCCggtATTAAAGTGAAGATTcagCGGGTTAATAACTCCAGTACCAATGATAATCTGGTCCGTAAGAACGATCAGGTATACATCaatTTCGTCGCGAGCAAAACTcatCTCTTCCCGCTTTACGCCgatACAGCTACGACAAACAAGGAAAAAACCATAAAAATTTCCAGCTCCGGAAACAGATTCAATCAAGTAGTTGTAATGAACTCTGTGGGTaatAATTGTACGATGAACTTTaagAATAACAATGGGAACAATattGGACTTTTGGGCTTcAAAGCCGACACAGTGGTGGCGTCCACCTGGTATTACACGcacATGcggGACCATACGAATTCGAACGGTTGCTTCTGGAACTTTATCTCGGAAgaaCACGGGTGGCAAGAAAAA
SEQ ID NO: 14 - Полипептидная последовательность rBoNT/E(0)
MPKINSFNYNDPVNDRTILYIKPGGCQEFYKSFNIMKNIWIIPERNVIGTTPQDFHPPTSLKNGDSSYYDPNYLQSDEEKDRFLKIVTKIFNRINNNLSGGILLEELSKANPYLGNDNTPDNQFHIGDASAVEIKFSNGSQDILLPNVIIMGAEPDLFETNSSNISLRNNYMPSNHGFGSIAIVTFSPEYSFRFNDNSMNEFIQDPALTLMHQLIYSLHGLYGAKGITTKYTITQKQNPLITNIRGTNIEEFLTFGGTDLNIITSAQSNDIYTNLLADYKKIASKLSKVQVSNPLLNPYKDVFEAKYGLDKDASGIYSVNINKFNDIFKKLYSFTEFDLATKFQVKCRQTYIGQYKYFKLSNLLNDSIYNISEGYNINNLKVNFRGQNANLNPRIITPITGRGLVKKIIRFCKNIVSVKGIRKSICIEINNGELFFVASENSYNDDNINTPKEIDDTVTSNNNYENDLDQVILNFNSESAPGLSDEKLNLTIQNDAYIPKYDSNGTSDIEQHDVNELNVFFYLDAQKVPEGENNVNLTSSIDTALLEQPKIYTFFSSEFINNVNKPVQAALFVSWIQQVLVDFTTEANQKSTVDKIADISIVVPYIGLALNIGNEAQKGNFKDALELLGAGILLEFEPELLIPTILVFTIKSFLGSSDNKNKVIKAINNALKERDEKWKEVYSFIVSNWMTKINTQFNKRKEQMYQALQNQVNAIKTIIESKYNSYTLEEKNELTNKYDIKQIENELNQKVSIAMNNIDRFLTESSISYLMKLINEVKINKLREYDENVKTYLLNYIIQHGSILGESQQELNSMVTDTLNNSIPFKLSSYTDDKILISYFNKFFKRIKSSSVLNMRYKNDKYVDTSGYDSNININGDVYKYPTNKNQFGIYNDKLSEVNISQNDYIIYDNKYKNFSISFWVRIPNYDNKIVNVNNEYTIINCMRDNNSGWKVSLNHNEIIWTLQDNAGINQKLAFNYGNANGISDYINKWIFVTITNDRLGDSKLYINGNLIDQKSILNLGNIHVSDNILFKIVNCSYTRYIGIRYFNIFDKELDETEIQTLYSNEPNTNILKDFWGNYLLYDKEYYLLNVLKPNNFIDRRKDSTLSINNIRSTILLANRLYSGIKVKIQRVNNSSTNDNLVRKNDQVYINFVASKTHLFPLYADTATTNKEKTIKISSSGNRFNQVVVMNSVGNNCTMNFKNNNGNNIGLLGFKADTVVASTWYYTHMRDHTNSNGCFWNFISEEHGWQEK
SEQ ID NO: 15 - Нуклеотидная последовательность rBoNT/F(0)
ATGCCGGTGGTCATCAACAGCTTCAACTACAACGACCCAGTAAACGACGACACGATCCTGTATATGCAAATCCCGTATGAAGAGAAGAGCAAGAAGTACTATAAGGCCTTTGAAATCATGCGCAATGTGTGGATTATTCCGGAGCGTAATACGATTGGTACTGACCCAAGCGACTTCGATCCACCTGCGTCTTTGGAAAACGGCTCGTCCGCATATTACGACCCGAATTACCTGACCACCGATGCGGAGAAAGATCGTTATTTGAAAACCACCATCAAGCTGTTCAAACGCATTAACAGCAATCCGGCAGGTGAGGTCCTGCTGCAAGAGATTAGCTACGCAAAGCCTTATCTGGGTAATGAGCATACGCCTATTAACGAGTTTCACCCGGTTACCCGCACTACCAGCGTTAACATCAAGTCCTCGACCAACGTGAAGTCTAGCATTATCCTGAACCTGCTGGTTCTGGGTGCCGGTCCGGACATCTTCGAAAACTCTAGCTACCCGGTGCGTAAACTGATGGATAGCGGCGGTGTTTATGACCCGAGCAATGACGGTTTTGGCAGCATCAATATCGTGACGTTTAGCCCGGAGTACGAGTACACCTTCAATGATATCAGCGGTGGTTACAATTCTTCTACCGAGAGCTTCATCGCCGACCCGGCGATCAGCCTGGCACACCAACTGATCTATGCATTGCATGGCTTGTACGGTGCCCGTGGTGTGACGTATAAAGAGACTATCAAGGTTAAGCAGGCACCTCTGATGATTGCGGAAAAGCCGATTCGCCTGGAAGAGTTCCTGACCTTCGGCGGTCAAGATTTGAACATCATTACCTCGGCCATGAAAGAGAAAATCTATAACAATTTGCTGGCCAACTATGAAAAGATTGCAACGCGCTTGTCTCGTGTTAACTCCGCTCCGCCGGAATACGACATTAATGAGTACAAAGACTACTTTCAATGGAAATATGGCCTGGACAAAAATGCGGATGGTTCTTATACCGTGAATGAAAACAAATTCAATGAAATCTACAAGAAACTGTACAGCTTCACCGAAATCGATCTGGCGAACAAGTTCAAAGTCAAATGTCGTAATACCTACTTCATCAAATATGGCTTCCTGAAAGTCCCGAACCTGCTGGACGATGACATCTATACCGTCAGCGAAGGCTTCAACATCGGCAATCTGGCCGTGAATAATCGTGGTCAGAACATCAAACTGAATCCGAAAATCATTGACTCCATCCCAGACAAGGGCCTGGTTGAGAAAATCGTGAAGTTCTGCAAAAGCGTTATTCCGCGTAAAGGTACGAAAGCACCGCCTCGCCTGTGCATTCGCGTTAACAACCGTGAGTTGTTCTTTGTGGCATCTGAAAGCAGCTACAACGAGAACGACATCAACACCCCTAAAGAAATTGATGATACCACGAACCTGAATAACAATTATCGCAACAATCTGGACGAGGTGATCCTGGATTACAATTCGGAAACCATTCCGCAAATTAGCAATCAGACGCTGAACACCCTGGTTCAGGACGATAGCTACGTTCCGCGTTACGACTCCAATGGTACTAGCGAGATTGAAGAACACAACGTAGTGGACTTGAACGTTTTCTTTTATCTGCACGCCCAGAAGGTTCCGGAGGGCGAAACCAATATTAGCCTGACCAGCTCGATCGACACCGCGCTGTCTGAGGAGAGCCAAGTCTACACCTTTTTCAGCAGCGAGTTTATCAACACTATTAACAAGCCAGTTCATGCTGCATTGTTTATCTCTTGGATTAACCAGGTGATTCGCGACTTTACGACGGAGGCGACCCAGAAGTCTACCTTCGACAAAATTGCAGACATCTCCCTGGTCGTCCCATACGTCGGCCTGGCGTTGAATATTGGCAATGAAGTTCAAAAAGAGAACTTCAAAGAAGCGTTCGAGCTGCTGGGTGCAGGCATCCTGCTGGAGTTCGTGCCGGAACTGTTGATCCCGACCATCCTGGTGTTCACCATTAAGAGCTTCATTGGATCCTCCGAGAATAAGAACAAGATCATCAAGGCGATCAATAACAGCCTGATGGAGCGTGAAACGAAGTGGAAAGAAATCTATAGCTGGATTGTTAGCAATTGGCTGACTCGTATTAACACGCAATTCAACAAGCGTAAAGAGCAAATGTACCAAGCCCTGCAAAACCAAGTTGACGCCATCAAAACGGTAATTGAATACAAGTACAACAATTACACGAGCGATGAGCGCAACCGCCTGGAAAGCGAATACAACATCAACAACATTCGCGAAGAATTGAACAAGAAAGTGAGCCTGGCGATGGAGAACATTGAGCGTTTTATCACCGAAAGCAGCATCTTTTACCTGATGAAATTGATTAATGAGGCGAAAGTCTCGAAACTGCGTGAGTACGACGAAGGTGTGAAAGAGTATCTGCTGGATTACATTAGCGAGCACCGTAGCATCTTGGGTAACTCGGTTCAGGAGCTGAACGATCTGGTGACCTCTACCCTGAACAATAGCATCCCGTTCGAACTGAGCAGCTATACCAATGACAAGATTCTGATTCTGTATTTCAATAAACTGTATAAGAAGATCAAGGATAACAGCATTCTGGATATGCGTTACGAAAACAATAAGTTTATCGACATTTCTGGTTACGGCAGCAACATTTCCATCAATGGCGATGTCTACATCTACAGCACCAATCGCAACCAGTTCGGCATCTACTCTAGCAAACCGAGCGAAGTTAACATCGCACAGAACAATGATATTATTTATAACGGTCGTTATCAAAACTTCTCTATCAGCTTTTGGGTCCGTATCCCGAAGTACTTCAATAAAGTCAATCTGAATAATGAATACACGATCATCGACTGCATTCGCAATAACAACAGCGGTTGGAAAATCAGCCTGAATTACAACAAAATTATTTGGACCCTGCAAGATACGGCGGGTAACAATCAGAAACTGGTGTTTAACTACACGCAAATGATCAGCATTTCTGACTATATCAACAAGTGGATCTTTGTTACCATCACCAATAATCGTCTGGGCAATAGCCGTATTTACATCAACGGTAACCTGATTGATGAGAAAAGCATCAGCAACCTGGGCGATATTCACGTCAGCGACAACATTCTGTTCAAAATTGTTGGTTGTAACGATACCCGTTACGTCGGCATCCGTTATTTCAAGGTTTTCGATACGGAGCTGGGTAAAACGGAAATCGAAACGTTGTACTCCGATGAACCAGATCCGAGCATTCTGAAGGACTTTTGGGGTAACTACTTGCTGTACAATAAACGTTACTATCTGCTGAATCTGTTGCGCACCGACAAGAGCATTACCCAAAACAGCAATTTCCTGAACATTAATCAGCAACGCGGCGTATACCAAAAACCGAACATCTTCAGCAATACGCGCCTGTATACTGGTGTTGAAGTGATCATTCGTAAGAACGGTAGCACCGACATTAGCAACACGGACAATTTCGTCCGTAAGAATGACCTGGCGTACATTAACGTCGTGGACCGTGATGTCGAGTATCGTCTGTACGCAGACATCAGCATTGCGAAACCGGAAAAGATTATCAAGCTGATCCGTACCAGCAACAGCAACAACAGCCTGGGTCAGATCATTGTGATGGACAGCATTGGTAATAACTGCACGATGAACTTCCAGAACAACAATGGTGGTAATATCGGTCTGCTGGGTTTTCACAGCAATAATCTGGTTGCTTCCAGCTGGTACTACAATAACATTCGTAAAAACACGTCTAGCAATGGTTGTTTTTGGAGCTTTATCAGCAAAGAGCACGGCTGGCAAGAAAAT
SEQ ID NO: 16 - Полипептидная последовательность rBoNT/F(0)
MPVVINSFNYNDPVNDDTILYMQIPYEEKSKKYYKAFEIMRNVWIIPERNTIGTDPSDFDPPASLENGSSAYYDPNYLTTDAEKDRYLKTTIKLFKRINSNPAGEVLLQEISYAKPYLGNEHTPINEFHPVTRTTSVNIKSSTNVKSSIILNLLVLGAGPDIFENSSYPVRKLMDSGGVYDPSNDGFGSINIVTFSPEYEYTFNDISGGYNSSTESFIADPAISLAHQLIYALHGLYGARGVTYKETIKVKQAPLMIAEKPIRLEEFLTFGGQDLNIITSAMKEKIYNNLLANYEKIATRLSRVNSAPPEYDINEYKDYFQWKYGLDKNADGSYTVNENKFNEIYKKLYSFTEIDLANKFKVKCRNTYFIKYGFLKVPNLLDDDIYTVSEGFNIGNLAVNNRGQNIKLNPKIIDSIPDKGLVEKIVKFCKSVIPRKGTKAPPRLCIRVNNRELFFVASESSYNENDINTPKEIDDTTNLNNNYRNNLDEVILDYNSETIPQISNQTLNTLVQDDSYVPRYDSNGTSEIEEHNVVDLNVFFYLHAQKVPEGETNISLTSSIDTALSEESQVYTFFSSEFINTINKPVHAALFISWINQVIRDFTTEATQKSTFDKIADISLVVPYVGLALNIGNEVQKENFKEAFELLGAGILLEFVPELLIPTILVFTIKSFIGSSENKNKIIKAINNSLMERETKWKEIYSWIVSNWLTRINTQFNKRKEQMYQALQNQVDAIKTVIEYKYNNYTSDERNRLESEYNINNIREELNKKVSLAMENIERFITESSIFYLMKLINEAKVSKLREYDEGVKEYLLDYISEHRSILGNSVQELNDLVTSTLNNSIPFELSSYTNDKILILYFNKLYKKIKDNSILDMRYENNKFIDISGYGSNISINGDVYIYSTNRNQFGIYSSKPSEVNIAQNNDIIYNGRYQNFSISFWVRIPKYFNKVNLNNEYTIIDCIRNNNSGWKISLNYNKIIWTLQDTAGNNQKLVFNYTQMISISDYINKWIFVTITNNRLGNSRIYINGNLIDEKSISNLGDIHVSDNILFKIVGCNDTRYVGIRYFKVFDTELGKTEIETLYSDEPDPSILKDFWGNYLLYNKRYYLLNLLRTDKSITQNSNFLNINQQRGVYQKPNIFSNTRLYTGVEVIIRKNGSTDISNTDNFVRKNDLAYINVVDRDVEYRLYADISIAKPEKIIKLIRTSNSNNSLGQIIVMDSIGNNCTMNFQNNNGGNIGLLGFHSNNLVASSWYYNNIRKNTSSNGCFWSFISKEHGWQEN
SEQ ID NO: 17 - Нуклеотидная последовательность rBoNT/A(0) (His-меченная)
ATGCCGTTTGTGAACAAGCAGTTCAACTATAAAGATCCGGTTAATGGTGTGGATATCGCCTATATCAAAATTCCGAATGCAGGTCAGATGCAGCCGGTTAAAGCCTTTAAAATCCATAACAAAATTTGGGTGATTCCGGAACGTGATACCTTTACCAATCCGGAAGAAGGTGATCTGAATCCGCCTCCGGAAGCAAAACAGGTTCCGGTTAGCTATTATGATAGCACCTATCTGAGCACCGATAACGAGAAAGATAACTATCTGAAAGGTGTGACCAAACTGTTTGAACGCATTTATAGTACCGATCTGGGTCGTATGCTGCTGACCAGCATTGTTCGTGGTATTCCGTTTTGGGGTGGTAGCACCATTGATACCGAACTGAAAGTTATTGACACCAACTGCATTAATGTGATTCAGCCGGATGGTAGCTATCGTAGCGAAGAACTGAATCTGGTTATTATTGGTCCGAGCGCAGATATCATTCAGTTTGAATGTAAAAGCTTTGGCCACGAAGTTCTGAATCTGACCCGTAATGGTTATGGTAGTACCCAGTATATTCGTTTCAGTCCGGATTTTACCTTTGGCTTTGAAGAAAGCCTGGAAGTTGATACAAATCCGCTGTTAGGTGCAGGTAAATTTGCAACCGATCCGGCAGTTACCCTGGCACACCAGCTGATTTATGCCGGTCATCGTCTGTATGGTATTGCCATTAATCCGAATCGTGTGTTCAAAGTGAATACCAACGCCTATTATGAAATGAGCGGTCTGGAAGTGAGTTTTGAAGAACTGCGTACCTTTGGTGGTCATGATGCCAAATTTATCGATAGCCTGCAAGAAAATGAATTTCGCCTGTACTACTATAACAAATTCAAGGATATTGCGAGCACCCTGAATAAAGCCAAAAGCATTGTTGGCACCACCGCAAGCCTGCAGTATATGAAAAATGTGTTTAAAGAAAAATATCTGCTGAGCGAAGATACCAGCGGTAAATTTAGCGTTGACAAACTGAAATTCGATAAACTGTACAAGATGCTGACCGAGATTTATACCGAAGATAACTTCGTGAAGTTTTTCAAAGTGCTGAACCGCAAAACCTACCTGAACTTTGATAAAGCCGTGTTCAAAATCAACATCGTGCCGAAAGTGAACTATACCATCTATGATGGTTTTAACCTGCGCAATACCAATCTGGCAGCAAACTTTAATGGTCAGAACACCGAAATCAACAACATGAACTTTACCAAACTGAAGAACTTCACCGGTCTGTTCGAATTTTACAAACTGCTGTGTGTTCGTGGCATTATTACCAGCAAAACCAAAAGTCTGGATAAAGGCTACAATAAAGCCCTGAATGATCTGTGCATTAAGGTGAATAATTGGGACCTGTTTTTTAGCCCGAGCGAGGATAATTTCACCAACGATCTGAACAAAGGCGAAGAAATTACCAGCGATACCAATATTGAAGCAGCCGAAGAAAACATTAGCCTGGATCTGATTCAGCAGTATTATCTGACCTTCAACTTCGATAATGAGCCGGAAAATATCAGCATTGAAAACCTGAGCAGCGATATTATTGGCCAGCTGGAACTGATGCCGAATATTGAACGTTTTCCGAACGGCAAAAAATACGAGCTGGATAAATACACCATGTTCCATTATCTGCGTGCCCAAGAATTTGAACATGGTAAAAGCCGTATTGCACTGACCAATAGCGTTAATGAAGCACTGCTGAACCCGAGCCGTGTTTATACCTTTTTTAGCAGCGATTACGTGAAAAAGGTTAACAAAGCAACCGAAGCAGCCATGTTTTTAGGTTGGGTTGAACAGCTGGTTTATGATTTCACCGATGAAACCAGCGAAGTTAGCACCACCGATAAAATTGCAGATATTACCATCATCATCCCGTATATCGGTCCGGCACTGAATATTGGCAATATGCTGTATAAAGACGATTTTGTGGGTGCCCTGATTTTTAGCGGTGCAGTTATTCTGCTGGAATTTATTCCGGAAATTGCCATTCCGGTTCTGGGCACCTTTGCACTGGTGAGCTATATTGCAAATAAAGTTCTGACCGTGCAGACCATCGATAATGCACTGAGCAAACGTAACGAAAAATGGGATGAAGTGTACAAGTATATCGTGACCAATTGGCTGGCAAAAGTTAACACCCAGATTGACCTGATTCGCAAGAAGATGAAAGAAGCACTGGAAAATCAGGCAGAAGCAACCAAAGCCATTATCAACTATCAGTATAACCAGTACACCGAAGAAGAGAAAAATAACATCAACTTCAACATCGACGATCTGTCCAGCAAACTGAACGAAAGCATCAACAAAGCCATGATTAACATTAACAAATTTCTGAACCAGTGCAGCGTGAGCTATCTGATGAATAGCATGATTCCGTATGGTGTGAAACGTCTGGAAGATTTTGATGCAAGCCTGAAAGATGCCCTGCTGAAATATATCTATGATAATCGTGGCACCCTGATTGGTCAGGTTGATCGTCTGAAAGATAAAGTGAACAACACCCTGAGTACCGATATTCCTTTTCAGCTGAGCAAATATGTGGATAATCAGCGTCTGCTGTCAACCTTTACCGAATACATTAAGAACATCATCAACACCAGCATTCTGAACCTGCGTTATGAAAGCAATCATCTGATTGATCTGAGCCGTTATGCCAGCAAAATCAATATAGGCAGCAAGGTTAACTTCGACCCGATTGACAAAAATCAGATACAGCTGTTTAATCTGGAAAGCAGCAAAATTGAGGTGATCCTGAAAAACGCCATTGTGTATAATAGCATGTACGAGAATTTCTCGACCAGCTTTTGGATTCGTATCCCGAAATACTTTAATAGCATCAGCCTGAACAACGAGTACACCATTATTAACTGCATGGAAAACAATAGCGGCTGGAAAGTTAGCCTGAATTATGGCGAAATTATCTGGACCCTGCAGGATACCCAAGAAATCAAACAGCGTGTGGTTTTCAAATACAGCCAGATGATTAATATCAGCGACTATATCAACCGCTGGATTTTTGTGACCATTACCAATAATCGCCTGAATAACAGCAAGATCTATATTAACGGTCGTCTGATTGACCAGAAACCGATTAGTAATCTGGGTAATATTCATGCGAGCAACAACATCATGTTTAAACTGGATGGTTGTCGTGATACCCATCGTTATATTTGGATCAAGTACTTCAACCTGTTCGATAAAGAGTTGAACGAAAAAGAAATTAAAGACCTGTATGATAACCAGAGCAACAGCGGTATTCTGAAGGATTTTTGGGGAGATTATCTGCAGTATGACAAACCGTATTATATGCTGAATCTGTACGACCCGAATAAATACGTGGATGTGAATAATGTTGGCATCCGTGGTTATATGTACCTGAAAGGTCCGCGTGGTAGCGTTATGACCACAAACATTTATCTGAATAGCAGCCTGTATCGCGGAACCAAATTCATCATTAAAAAGTATGCCAGCGGCAACAAGGATAATATTGTGCGTAATAATGATCGCGTGTACATTAACGTTGTGGTGAAGAATAAAGAATATCGCCTGGCAACCAATGCAAGCCAGGCAGGCGTTGAAAAAATTCTGAGTGCCCTGGAAATTCCGGATGTTGGTAATCTGAGCCAGGTTGTTGTGATGAAAAGCAAAAATGATCAGGGCATCACCAACAAGTGCAAAATGAATCTGCAGGACAATAACGGCAACGATATTGGTTTTATTGGCTTCCACCAGTTCAACAATATTGCGAAACTGGTTGCAAGCAATTGGTATAATCGTCAGATTGAACGTAGCAGTCGTACCCTGGGTTGTAGCTGGGAATTTATCCCTGTGGATGATGGTTGGGGTGAACGTCCGCTGGAAAACCTGTATTTTCAAGGTGCAAGTCATCATCACCATCACCACCATCATTAA
SEQ ID NO: 18 - полипептидная последовательность rBoNT/A(0) (His-меченная)
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHQLIYAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPLENLYFQGASHHHHHHHH
SEQ ID NO: 19 - Нуклеотидная последовательность rLHN/A (His-меченная)
ATGCCGTTTGTGAACAAGCAGTTCAACTATAAAGATCCGGTTAATGGTGTGGATATCGCCTATATCAAAATTCCGAATGCAGGTCAGATGCAGCCGGTTAAAGCCTTTAAAATCCATAACAAAATTTGGGTGATTCCGGAACGTGATACCTTTACCAATCCGGAAGAAGGTGATCTGAATCCGCCTCCGGAAGCAAAACAGGTTCCGGTTAGCTATTATGATAGCACCTATCTGAGCACCGATAACGAGAAAGATAACTATCTGAAAGGTGTGACCAAACTGTTTGAACGCATTTATAGTACCGATCTGGGTCGTATGCTGCTGACCAGCATTGTTCGTGGTATTCCGTTTTGGGGTGGTAGCACCATTGATACCGAACTGAAAGTTATTGACACCAACTGCATTAATGTGATTCAGCCGGATGGTAGCTATCGTAGCGAAGAACTGAATCTGGTTATTATTGGTCCGAGCGCAGATATCATTCAGTTTGAATGTAAATCCTTTGGCCACGAAGTTCTGAATCTGACCCGTAATGGTTATGGTAGTACCCAGTATATTCGTTTCAGTCCGGATTTTACCTTTGGCTTTGAAGAAAGCCTGGAAGTTGATACAAATCCGCTGTTAGGTGCAGGTAAATTTGCAACCGATCCGGCAGTTACCCTGGCACATGAACTGATTCATGCCGGTCATCGTCTGTATGGTATTGCAATTAATCCGAACCGTGTGTTCAAAGTGAATACCAACGCATATTATGAAATGAGCGGTCTGGAAGTGTCATTTGAAGAACTGCGTACCTTTGGTGGTCATGATGCCAAATTTATCGATAGCCTGCAAGAAAATGAATTTCGCCTGTACTACTATAACAAATTCAAGGATATTGCGAGCACCCTGAATAAAGCCAAAAGCATTGTTGGCACCACCGCAAGCCTGCAGTATATGAAAAATGTGTTTAAAGAAAAATATCTGCTGAGCGAAGATACCAGCGGTAAATTTAGCGTTGACAAACTGAAATTCGATAAACTGTACAAGATGCTGACCGAGATTTATACCGAAGATAACTTCGTGAAGTTTTTCAAAGTGCTGAACCGCAAAACCTACCTGAACTTTGATAAAGCCGTGTTCAAAATCAACATCGTGCCGAAAGTGAACTATACCATCTATGATGGTTTTAACCTGCGCAATACCAATCTGGCAGCAAACTTTAATGGTCAGAACACCGAAATCAACAACATGAACTTTACCAAACTGAAGAACTTCACCGGTCTGTTCGAATTTTACAAACTGCTGTGTGTTCGTGGCATTATTACCAGCAAAACCAAAAGTCTGGATAAAGGCTACAATAAAGCCCTGAATGATCTGTGCATTAAGGTGAATAATTGGGACCTGTTTTTTAGCCCGAGCGAGGATAATTTCACCAACGATCTGAACAAAGGCGAAGAAATTACCAGCGATACCAATATTGAAGCAGCCGAAGAAAACATTAGCCTGGATCTGATTCAGCAGTATTATCTGACCTTCAACTTCGATAATGAGCCGGAAAATATCAGCATTGAAAACCTGAGCAGCGATATTATTGGCCAGCTGGAACTGATGCCGAATATTGAACGTTTTCCGAACGGCAAAAAATACGAGCTGGATAAATACACCATGTTCCATTATCTGCGTGCCCAAGAATTTGAACATGGTAAAAGCCGTATTGCACTGACCAATAGCGTTAATGAAGCACTGCTGAACCCGAGCCGTGTTTATACCTTTTTTAGCAGCGATTACGTGAAAAAGGTTAACAAAGCAACCGAAGCAGCCATGTTTTTAGGTTGGGTTGAACAGCTGGTTTATGATTTCACCGATGAAACCAGCGAAGTTAGCACCACCGATAAAATTGCAGATATTACCATCATCATCCCGTATATCGGTCCGGCACTGAATATTGGCAATATGCTGTATAAAGACGATTTTGTGGGTGCCCTGATTTTTAGCGGTGCAGTTATTCTGCTGGAATTTATTCCGGAAATTGCCATTCCGGTTCTGGGCACCTTTGCACTGGTGAGCTATATTGCAAATAAAGTTCTGACCGTGCAGACCATCGATAATGCACTGAGCAAACGTAACGAAAAATGGGATGAAGTGTACAAGTATATCGTGACCAATTGGCTGGCAAAAGTTAACACCCAGATTGACCTGATTCGCAAGAAGATGAAAGAAGCACTGGAAAATCAGGCAGAAGCAACCAAAGCCATTATCAACTATCAGTATAACCAGTACACCGAAGAAGAGAAAAATAACATCAACTTCAACATCGACGATCTGTCCAGCAAACTGAACGAAAGCATCAACAAAGCCATGATTAACATTAACAAATTTCTGAACCAGTGCAGCGTGAGCTATCTGATGAATAGCATGATTCCGTATGGTGTGAAACGTCTGGAAGATTTTGATGCAAGCCTGAAAGATGCCCTGCTGAAATATATCTATGATAATCGTGGCACCCTGATTGGTCAGGTTGATCGTCTGAAAGATAAAGTGAACAACACCCTGAGTACCGATATTCCTTTTCAGCTGAGCAAATATGTGGATAATCAGCGTCTGCTGTCAACCGAAAATCTGTATTTCCAGGGTGCAAGTCATCATCACCATCACCACCATCATTAA
SEQ ID NO: 20 - Полипептидная последовательность rLHN/A (His-меченная)
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTENLYFQGASHHHHHHHH
SEQ ID NO: 21 - Нуклеотидная последовательность rHC/A (His-меченная)
atgcatcatcaccatcaccacGAAAATCTATACTTCCAAGGAaaaaacatcatcaatactagcattctgaacctgcgttacgagagcaatcatctgattgatctgagccgttatgcaagcaagatcaacatcggtagcaaggtcaattttgacccgatcgataagaaccagatccagctgtttaatctggaatcgagcaaaattgaggttatcctgaaaaacgccattgtctacaactccatgtacgagaatttctccaccagcttctggattcgcatcccgaaatacttcaacagcattagcctgaacaacgagtatactatcatcaactgtatggagaacaacagcggttggaaggtgtctctgaactatggtgagatcatttggaccttgcaggacacccaagagatcaagcagcgcgtcgtgttcaagtactctcaaatgatcaacatttccgattacattaatcgttggatcttcgtgaccattacgaataaccgtctgaataacagcaagatttacatcaatggtcgcttgatcgatcagaaaccgattagcaacctgggtaatatccacgcaagcaacaacattatgttcaaattggacggttgccgcgatacccatcgttatatctggatcaagtatttcaacctgtttgataaagaactgaatgagaaggagatcaaagatttgtatgacaaccaatctaacagcggcattttgaaggacttctggggcgattatctgcaatacgataagccgtactatatgctgaacctgtatgatccgaacaaatatgtggatgtcaataatgtgggtattcgtggttacatgtatttgaagggtccgcgtggcagcgttatgacgaccaacatttacctgaactctagcctgtaccgtggtacgaaattcatcattaagaaatatgccagcggcaacaaagataacattgtgcgtaataacgatcgtgtctacatcaacgtggtcgtgaagaataaagagtaccgtctggcgaccaacgcttcgcaggcgggtgttgagaaaattctgagcgcgttggagatccctgatgtcggtaatctgagccaagtcgtggttatgaagagcaagaacgaccagggtatcactaacaagtgcaagatgaacctgcaagacaacaatggtaacgacatcggctttattggtttccaccagttcaacaatattgctaaactggtagcgagcaattggtacaatcgtcagattgagcgcagcagccgtactttgggctgtagctgggagtttatcccggtcgatgatggttggggcgaacgtccgctgtaa
SEQ ID NO: 22 - полипептидная последовательность rHC/A (His-меченная)
MHHHHHHENLYFQGKNIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
SEQ ID NO: 23 - Нуклеотидная последовательность rLC/A (His-меченная)
ATGCCGTTTGTGAACAAGCAGTTCAACTATAAAGATCCGGTTAATGGTGTGGATATCGCCTATATCAAAATTCCGAATGCAGGTCAGATGCAGCCGGTTAAAGCCTTTAAAATCCATAACAAAATTTGGGTGATTCCGGAACGTGATACCTTTACCAATCCGGAAGAAGGTGATCTGAATCCGCCTCCGGAAGCAAAACAGGTTCCGGTTAGCTATTATGATAGCACCTATCTGAGCACCGATAACGAGAAAGATAACTATCTGAAAGGTGTGACCAAACTGTTTGAACGCATTTATAGTACCGATCTGGGTCGTATGCTGCTGACCAGCATTGTTCGTGGTATTCCGTTTTGGGGTGGTAGCACCATTGATACCGAACTGAAAGTTATTGACACCAACTGCATTAATGTGATTCAGCCGGATGGTAGCTATCGTAGCGAAGAACTGAATCTGGTTATTATTGGTCCGAGCGCAGATATCATTCAGTTTGAATGTAAATCCTTTGGCCACGAAGTTCTGAATCTGACCCGTAATGGTTATGGTAGTACCCAGTATATTCGTTTCAGTCCGGATTTTACCTTTGGCTTTGAAGAAAGCCTGGAAGTTGATACAAATCCGCTGTTAGGTGCAGGTAAATTTGCAACCGATCCGGCAGTTACCCTGGCACATGAACTGATTCATGCCGGTCATCGTCTGTATGGTATTGCAATTAATCCGAACCGTGTGTTCAAAGTGAATACCAACGCATATTATGAAATGAGCGGTCTGGAAGTGTCATTTGAAGAACTGCGTACCTTTGGTGGTCATGATGCCAAATTTATCGATAGCCTGCAAGAAAATGAATTTCGCCTGTACTACTATAACAAATTCAAGGATATTGCGAGCACCCTGAATAAAGCCAAAAGCATTGTTGGCACCACCGCAAGCCTGCAGTATATGAAAAATGTGTTTAAAGAAAAATATCTGCTGAGCGAAGATACCAGCGGTAAATTTAGCGTTGACAAACTGAAATTCGATAAACTGTACAAGATGCTGACCGAGATTTATACCGAAGATAACTTCGTGAAGTTTTTCAAAGTGCTGAACCGCAAAACCTACCTGAACTTTGATAAAGCCGTGTTCAAAATCAACATCGTGCCGAAAGTGAACTATACCATCTATGATGGTTTTAACCTGCGCAATACCAATCTGGCAGCAAACTTTAATGGTCAGAACACCGAAATCAACAACATGAACTTTACCAAACTGAAGAACTTCACCGGTCTGTTTGAAGAGAATCTGTATTTCCAGGGTGCAAGTCATCATCACCATCACCACCATCATTAA
SEQ ID NO: 24 - Полипептидная последовательность rLC/A (His-меченная)
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEENLYFQGASHHHHHHHH
SEQ ID NO: 25 - нуклеотидная последовательность rBoNT/FA(0) (His-меченная)
ATGCCGGTTGTGATTAACAGCTTCAATTATGATGATCCGGTGAACGATAACACCATCATTTATATCCGTCCGCCTTATTATGAAACCAGCAACACCTATTTCAAAGCCTTCCAGATTATGGATAACGTGTGGATTATTCCGGAACGTTATCGTCTGGGTATTGATCCGAGCCTGTTTAATCCGCCTGTTAGCCTGAAAGCAGGTAGTGATGGTTATTTTGATCCGAATTATCTGAGCACCAACACCGAGAAAAACAAATACCTGCAGATTATGATCAAGCTGTTCAAACGCATTAATAGCAAACCGGCAGGTCAGATTCTGCTGGAAGAAATCAAAAATGCAATTCCGTATCTGGGCAACAGCTATACCCAAGAAGAACAGTTTACCACCAATAATCGTACCGTGAGCTTTAATGTTAAACTGGCCAATGGTAATATCGTTCAGCAGATGGCAAATCTGATTATTTGGGGTCCGGGTCCTGATCTGACCACAAATAAAACCGGTGGTATCATCTATAGCCCGTATCAGAGCATGGAAGCAACCCCGTATAAAGATGGTTTTGGTAGCATTATGACCGTGGAATTTAGTCCGGAATATGCAACCGCCTTTAACGATATTTCAATTGCAAGCCATAGTCCGTCGCTGTTTATCAAAGATCCGGCACTGATTCTGATGCACCAGCTGATTTATGTTCTGCATGGTCTGTATGGCACCTATATCACCGAATACAAAATTACCCCGAATGTGGTTCAGAGCTATATGAAAGTTACCAAACCGATTACCAGCGCAGAATTTCTGACCTTTGGTGGTCGTGATCGCAATATTGTTCCGCAGAGCATTCAGAGCCAGCTGTATAACAAAGTTCTGAGCGATTATAAACGTATTGCCAGCCGTCTGAATAAAGTTAATACCGCAACCGCACTGATCAACATCGATGAATTCAAAAACCTGTACGAGTGGAAATACCAGTTTGCCAAAGATAGCAATGGTGTGTATAGCGTGGATCTGAACAAATTTGAGCAGCTGTACAAAAAAATCTATAGCTTCACCGAATTCAACCTGGCCTATGAGTTTAAAATCAAAACCCGTCTGGGTTATCTGGCCGAAAATTTTGGTCCGTTTTATCTGCCGAATCTGCTGGATGATAGCATTTATACCGAAGTGGATGGTTTTAACATTGGTGCACTGAGCATTAACTATCAGGGTCAGAATATTGGCAGCGATATCAACAGCATCAAAAAACTGCAAGGTCAGGGTGTTGTTAGCCGTGTTGTTCGTCTGTGTAGCAATAGCAATACCAAAAACAGCCTGTGCATTACCGTTAATAATCGCGACCTGTTTTTTATCGCAAGCCAAGAAAGCTATGGCGAGAATACCATTAACACCTATAAAGAGATTGACGATACCACCACACTGGATCCGAGCTTTGAAGATATTCTGGATAAAGTGATCCTGAACTTCAACGAACAGGTTATTCCGCAGATGCCGAATCGTAATGTTAGCACCGATATTCAGAAAGACAACTACATCCCGAAATACGATTATAACCGCACCGACATTATCGATAGCTATGAAGTTGGTCGCAACTACAACACCTTTTTCTATCTGAATGCCCAGAAATTTAGCCCGAACGAAAGCAATATTACCCTGACCAGCAGCTTTGATACAGGTCTGTTAGAAGGTAGCAAAGTGTATACCTTTTTCAGCAGCGATTTCATTAACAACATCAACAAACCGGTTCAGGCCCTGCTGTTTATTGAATGGGTTAAACAGGTGATTCGCGATTTTACCACCGAAGCAACCAAAACCTCAACCGTTGATAAACTGAAAGATATTAGCCTGGTGGTGCCGTATATTGGTCTGGCACTGAATATTGGTGATGAGATCTACAAACAGCATTTTGCAGAAGCAGTTGAACTGGTTGGTGCAGGTCTGCTGCTGGAATTTTCACCGGAATTTCTTATTCCGACGCTGCTGATTTTTACCATCAAAGGTTATCTGACCGGTAGCATTCGCGATAAAGACAAAATCATTAAAACCCTGGATAACGCCCTGAATGTTCGTGATCAGAAATGGAAAGAACTGTATCGTTGGGTTGTTAGCAAATGGCTGACCACCATTAATACGCAGTTCAACAAACGCAAAGAACAAATGTACAAAGCCCTGAAAAATCAGGCCACCGCCATTAAAAAGATCATCGAGAACAAATATAACAACTATACCACCGATGAAAAAAGCAAGATCGATAGCAGCTATAACATCAACGAAATTGAACGCACCCTGAACGAAAAAATCAATCTGGCCATGAAAAACATCGAGCAGTTTATTACCGAAAGCAGCATTGCCTATCTGATCAATATCATCAACAACGAAACGATCCAGAAACTGAAAAGCTATGATGACCTGGTTCGTCGTTATCTGCTGGGTTATATTCGTAATCATAGCAGCATTCTGGGCAATAGCGTTGAAGAACTGAATTCCAAAGTGAACAACCATCTGGATAATGGCATTCCGTTTGAACTGAGCAGTTATACCAATGATAGCCTGCTGATCCGCTACTTCAATAAAAACTATGGCGAACTGAAGTACAACTGCATTCTGAACATCAAATATGAGATGGATCGTGACAAACTGGTTGATAGCAGCGGTTATCGTAGCCGTATCAATATTGGTACAGGCGTCAAATTTAGCGAGATCGATAAAAATCAAGTGCAGCTGAGCAATCTGGAATCCAGCAAAATTGAAGTCATTCTGAATAACGGCGTCATCTATAACAGCATGTATGAAAACTTTTCGACCAGCTTTTGGATTCGCATTCCGAAATACTTTCGCAACATCAATAACGAGTACAAGATCATCAGCTGTATGCAGAATAATAGCGGTTGGGAAGTGAGCCTGAATTTTAGCAATATGAACTCGAAAATCATCTGGACCCTGCAGGATACCGAAGGTATCAAAAAAACCGTTGTGTTTCAGTACACCCAGAACATTAACATTAGCGACTATATCAACCGCTGGATCTTTGTGACCATTACAAATAATCGTCTGAGCAACAGCAAAATCTACATTAATGGTCGCCTGATCAACGAAGAAAGCATTAGCGATCTGGGTAATATCCATGCCAGCAACAACATTATGTTTAAACTGGATGGTTGCCGTGATCCGCATCGTTATATCTGGATTAAATACTTTAACCTGTTTGACAAAGAGCTGAACAAGAAAGAAATTAAAGATCTGTACGACAACCAGAGCAATAGCGGTATTCTGAAAGATTTCTGGGGTGATTATCTGCAGTATGACAAACCGTATTATATGCTGAATCTGTATGACCCGAATAAGTATCTGGATGTGAATAATGTTGGCATCCGTGGCTATATGTATCTGAAAGGTCCGCGTGGTCGTATTGTGACCACCAACATTTATCTGAATAGCACCCTGTATATGGGCACCAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAATGATCGCGTGTATATTAACGTGGTGGTGAAGAATAAAGAATATCGCCTGGCAACCAATGCAAGCCAGGCAGGCGTTGAAAAAATTCTGAGCGCAGTTGAAATCCCGGATGTTGGTAATCTGAGCCAGGTTGTTGTGATGAAAAGCGAAAATGATCAGGGCATTCGCAACAAGTGTAAAATGAATCTGCAAGACAATAACGGCAACGATATTGGCTTTATCGGCTTTCACCAGTTTAATAACATTGCAAAACTGGTGGCCAGCAACTGGTATAACCGTCAGATTGGTAAAGCAAGCCGTACCTTTGGTTGTAGCTGGGAATTTATCCCGGTTGATGATGGTTGGGGTGAAAGCAGCCTGGAAAATCTGTATTTCCAGGGTGCCAGTCATCATCACCACCATCACCATCACTGA
SEQ ID NO: 26 - Полипептидная последовательность rBoNT/FA(0) (His-меченная)
MPVVINSFNYDDPVNDNTIIYIRPPYYETSNTYFKAFQIMDNVWIIPERYRLGIDPSLFNPPVSLKAGSDGYFDPNYLSTNTEKNKYLQIMIKLFKRINSKPAGQILLEEIKNAIPYLGNSYTQEEQFTTNNRTVSFNVKLANGNIVQQMANLIIWGPGPDLTTNKTGGIIYSPYQSMEATPYKDGFGSIMTVEFSPEYATAFNDISIASHSPSLFIKDPALILMHQLIYVLHGLYGTYITEYKITPNVVQSYMKVTKPITSAEFLTFGGRDRNIVPQSIQSQLYNKVLSDYKRIASRLNKVNTATALINIDEFKNLYEWKYQFAKDSNGVYSVDLNKFEQLYKKIYSFTEFNLAYEFKIKTRLGYLAENFGPFYLPNLLDDSIYTEVDGFNIGALSINYQGQNIGSDINSIKKLQGQGVVSRVVRLCSNSNTKNSLCITVNNRDLFFIASQESYGENTINTYKEIDDTTTLDPSFEDILDKVILNFNEQVIPQMPNRNVSTDIQKDNYIPKYDYNRTDIIDSYEVGRNYNTFFYLNAQKFSPNESNITLTSSFDTGLLEGSKVYTFFSSDFINNINKPVQALLFIEWVKQVIRDFTTEATKTSTVDKLKDISLVVPYIGLALNIGDEIYKQHFAEAVELVGAGLLLEFSPEFLIPTLLIFTIKGYLTGSIRDKDKIIKTLDNALNVRDQKWKELYRWVVSKWLTTINTQFNKRKEQMYKALKNQATAIKKIIENKYNNYTTDEKSKIDSSYNINEIERTLNEKINLAMKNIEQFITESSIAYLINIINNETIQKLKSYDDLVRRYLLGYIRNHSSILGNSVEELNSKVNNHLDNGIPFELSSYTNDSLLIRYFNKNYGELKYNCILNIKYEMDRDKLVDSSGYRSRINIGTGVKFSEIDKNQVQLSNLESSKIEVILNNGVIYNSMYENFSTSFWIRIPKYFRNINNEYKIISCMQNNSGWEVSLNFSNMNSKIIWTLQDTEGIKKTVVFQYTQNINISDYINRWIFVTITNNRLSNSKIYINGRLINEESISDLGNIHASNNIMFKLDGCRDPHRYIWIKYFNLFDKELNKKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYLDVNNVGIRGYMYLKGPRGRIVTTNIYLNSTLYMGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSAVEIPDVGNLSQVVVMKSENDQGIRNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIGKASRTFGCSWEFIPVDDGWGESSLENLYFQGASHHHHHHHH
SEQ ID NO: 27 - Нуклеотидная последовательность rLHN/FA (His-меченная)
ATGCCGGTTGTGATTAACAGCTTCAATTATGATGATCCGGTGAACGATAACACCATCATTTATATCCGTCCGCCTTATTATGAAACCAGCAACACCTATTTCAAAGCCTTCCAGATTATGGATAACGTGTGGATTATTCCGGAACGTTATCGTCTGGGTATTGATCCGAGCCTGTTTAATCCGCCTGTTAGCCTGAAAGCAGGTAGTGATGGTTATTTTGATCCGAATTATCTGAGCACCAACACCGAGAAAAACAAATACCTGCAGATTATGATCAAGCTGTTCAAACGCATTAATAGCAAACCGGCAGGTCAGATTCTGCTGGAAGAAATCAAAAATGCAATTCCGTATCTGGGCAACAGCTATACCCAAGAAGAACAGTTTACCACCAATAATCGTACCGTGAGCTTTAATGTTAAACTGGCCAATGGTAATATCGTTCAGCAGATGGCAAATCTGATTATTTGGGGTCCGGGTCCTGATCTGACCACAAATAAAACCGGTGGTATCATCTATAGCCCGTATCAGAGCATGGAAGCAACCCCGTATAAAGATGGTTTTGGTAGCATTATGACCGTGGAATTTAGTCCGGAATATGCAACCGCCTTTAACGATATTTCAATTGCAAGCCATAGTCCGTCGCTGTTTATCAAAGATCCGGCACTGATTCTGATGCATGAACTGATTCATGTTCTGCATGGTCTGTATGGCACCTATATTACCGAATACAAAATTACCCCGAATGTGGTGCAGAGCTATATGAAAGTTACCAAACCGATTACCAGCGCAGAATTTCTGACCTTTGGTGGTCGTGATCGCAATATTGTTCCGCAGAGCATTCAGAGCCAGCTGTATAACAAAGTTCTGAGCGATTATAAACGTATTGCCAGCCGTCTGAATAAAGTTAATACCGCAACCGCACTGATCAACATCGATGAATTCAAAAACCTGTACGAGTGGAAATACCAGTTTGCCAAAGATAGCAATGGTGTGTATAGCGTGGATCTGAACAAATTTGAGCAGCTGTACAAAAAAATCTATAGCTTCACCGAATTCAACCTGGCCTATGAGTTTAAAATCAAAACCCGTCTGGGTTATCTGGCCGAAAATTTTGGTCCGTTTTATCTGCCGAATCTGCTGGATGATAGCATTTATACCGAAGTGGATGGTTTTAACATTGGTGCACTGAGCATTAACTATCAGGGTCAGAATATTGGCAGCGATATCAACAGCATCAAAAAACTGCAAGGTCAGGGTGTTGTTAGCCGTGTTGTTCGTCTGTGTAGCAATAGCAATACCAAAAACAGCCTGTGCATTACCGTTAATAATCGCGACCTGTTTTTTATCGCAAGCCAAGAAAGCTATGGCGAGAATACCATTAACACCTATAAAGAGATTGACGATACCACCACACTGGATCCGAGCTTTGAAGATATTCTGGATAAAGTGATCCTGAACTTCAACGAACAGGTTATTCCGCAGATGCCGAATCGTAATGTTAGCACCGATATTCAGAAAGACAACTACATCCCGAAATACGATTATAACCGCACCGACATTATCGATAGCTATGAAGTTGGTCGCAACTACAACACCTTTTTCTATCTGAATGCCCAGAAATTTAGCCCGAACGAAAGCAATATTACCCTGACCAGCAGCTTTGATACAGGTCTGTTAGAAGGTAGCAAAGTGTATACCTTTTTCAGCAGCGATTTCATTAACAACATCAACAAACCGGTTCAGGCCCTGCTGTTTATTGAATGGGTTAAACAGGTGATTCGCGATTTTACCACCGAAGCAACCAAAACCTCAACCGTTGATAAACTGAAAGATATTAGCCTGGTGGTGCCGTATATTGGTCTGGCACTGAATATTGGTGATGAGATCTACAAACAGCATTTTGCAGAAGCAGTTGAACTGGTTGGTGCAGGTCTGCTGCTGGAATTTTCACCGGAATTTCTTATTCCGACGCTGCTGATTTTTACCATCAAAGGTTATCTGACCGGTAGCATTCGCGATAAAGACAAAATCATTAAAACCCTGGATAACGCCCTGAATGTTCGTGATCAGAAATGGAAAGAACTGTATCGTTGGGTTGTTAGCAAATGGCTGACCACCATTAATACGCAGTTCAACAAACGCAAAGAACAAATGTACAAAGCCCTGAAAAATCAGGCCACCGCCATTAAAAAGATCATCGAGAACAAATATAACAACTATACCACCGATGAAAAAAGCAAGATCGATAGCAGCTATAACATCAACGAAATTGAACGCACCCTGAACGAAAAAATCAATCTGGCCATGAAAAACATCGAGCAGTTTATTACAGAAAGCAGCATTGCCTACCTGATCAATATCATCAACAACGAAACCATTCAGAAACTGAAAAGCTATGATGACCTGGTTCGTCGTTATCTGCTGGGTTATATTCGTAATCATAGCAGCATTCTGGGCAATAGCGTTGAAGAACTGAATTCCAAAGTGAACAACCATCTGGATAATGGCATTCCGTTTGAACTGAGCAGTTATACCAATGATAGCCTGCTGATCCGCTACTTCAATAAAAACTATGGCGAAGAGAACCTGTATTTCCAGGGTGCCAGTCATCATCACCACCATCACCATCACTGA
SEQ ID NO: 28 - Полипептидная последовательность rLHN/FA (His-меченная)
MPVVINSFNYDDPVNDNTIIYIRPPYYETSNTYFKAFQIMDNVWIIPERYRLGIDPSLFNPPVSLKAGSDGYFDPNYLSTNTEKNKYLQIMIKLFKRINSKPAGQILLEEIKNAIPYLGNSYTQEEQFTTNNRTVSFNVKLANGNIVQQMANLIIWGPGPDLTTNKTGGIIYSPYQSMEATPYKDGFGSIMTVEFSPEYATAFNDISIASHSPSLFIKDPALILMHELIHVLHGLYGTYITEYKITPNVVQSYMKVTKPITSAEFLTFGGRDRNIVPQSIQSQLYNKVLSDYKRIASRLNKVNTATALINIDEFKNLYEWKYQFAKDSNGVYSVDLNKFEQLYKKIYSFTEFNLAYEFKIKTRLGYLAENFGPFYLPNLLDDSIYTEVDGFNIGALSINYQGQNIGSDINSIKKLQGQGVVSRVVRLCSNSNTKNSLCITVNNRDLFFIASQESYGENTINTYKEIDDTTTLDPSFEDILDKVILNFNEQVIPQMPNRNVSTDIQKDNYIPKYDYNRTDIIDSYEVGRNYNTFFYLNAQKFSPNESNITLTSSFDTGLLEGSKVYTFFSSDFINNINKPVQALLFIEWVKQVIRDFTTEATKTSTVDKLKDISLVVPYIGLALNIGDEIYKQHFAEAVELVGAGLLLEFSPEFLIPTLLIFTIKGYLTGSIRDKDKIIKTLDNALNVRDQKWKELYRWVVSKWLTTINTQFNKRKEQMYKALKNQATAIKKIIENKYNNYTTDEKSKIDSSYNINEIERTLNEKINLAMKNIEQFITESSIAYLINIINNETIQKLKSYDDLVRRYLLGYIRNHSSILGNSVEELNSKVNNHLDNGIPFELSSYTNDSLLIRYFNKNYGEENLYFQGASHHHHHHHH
SEQ ID NO: 29 - Нуклеотидная последовательность rHC/FA (His-меченая)
ATGCTGAAGTATAACTGCATCCTGAACATCAAATATGAGATGGATCGTGATAAACTGGTTGATAGCAGCGGTTATCGTAGCCGTATCAATATTGGCACCGGTGTGAAATTTAGCGAGATCGATAAAAATCAGGTGCAGCTGAGCAATCTGGAAAGCAGCAAAATTGAAGTGATTCTGAATAACGGCGTGATCTACAATAGCATGTATGAAAACTTTTCGACCAGCTTCTGGATTCGCATTCCGAAATACTTTCGCAACATCAACAACGAGTACAAGATTATCAGCTGTATGCAGAATAATAGCGGTTGGGAAGTTAGCCTGAATTTCAGCAATATGAACAGCAAAATCATTTGGACCCTGCAGGATACCGAAGGTATCAAAAAAACCGTTGTGTTTCAGTACACCCAGAACATTAACATCAGCGATTACATTAACCGCTGGATCTTTGTGACCATTACCAATAATCGTCTGAGCAACAGCAAGATCTATATTAACGGTCGCCTGATTAACGAAGAGAGCATTAGCGATCTGGGTAATATTCATGCCAGCAACAACATCATGTTTAAACTGGATGGTTGTCGTGATCCGCATCGTTATATTTGGATCAAATACTTCAACCTGTTTGATAAAGAACTGAACAAAAAAGAAATCAAAGACCTGTATGATAACCAGAGCAATAGCGGCATTCTGAAAGATTTTTGGGGTGATTATCTGCAGTATGACAAACCGTATTACATGCTGAATCTGTACGATCCGAACAAATATCTGGATGTGAATAATGTGGGTATCCGTGGCTATATGTATCTGAAAGGTCCGCGTGGTCGTATTGTTACCACCAACATTTATCTGAATAGCACCCTGTATATGGGCACCAAATTCATCATTAAAAAGTATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAATGATCGCGTGTATATCAATGTGGTGGTGAAGAATAAAGAATATCGTCTGGCCACCAATGCAAGCCAGGCAGGCGTTGAAAAAATTCTGAGCGCAGTTGAAATTCCGGATGTTGGTAATCTGAGCCAGGTTGTTGTTATGAAAAGCGAAAATGATCAGGGCATTCGCAACAAATGCAAAATGAATCTGCAGGACAATAACGGCAACGATATTGGTTTTATTGGCTTCCACCAGTTCAACAACATTGCAAAACTGGTGGCGAGCAATTGGTATAATCGTCAGATTGGTAAAGCAAGCCGTACCTTTGGTTGTAGCTGGGAATTTATTCCGGTTGATGATGGTTGGGGTGAAAGCAGCCTGGAAAATCTGTATTTTCAGGGTGCAAGTCATCATCACCACCATCACCATCATTAA
SEQ ID NO: 30 - Полипептидная последовательность rHC/FA (His-меченная)
MLKYNCILNIKYEMDRDKLVDSSGYRSRINIGTGVKFSEIDKNQVQLSNLESSKIEVILNNGVIYNSMYENFSTSFWIRIPKYFRNINNEYKIISCMQNNSGWEVSLNFSNMNSKIIWTLQDTEGIKKTVVFQYTQNINISDYINRWIFVTITNNRLSNSKIYINGRLINEESISDLGNIHASNNIMFKLDGCRDPHRYIWIKYFNLFDKELNKKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYLDVNNVGIRGYMYLKGPRGRIVTTNIYLNSTLYMGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSAVEIPDVGNLSQVVVMKSENDQGIRNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIGKASRTFGCSWEFIPVDDGWGESSLENLYFQGASHHHHHHHH
SEQ ID NO: 31 - Нуклеотидная последовательность rLC/FA (His-меченная)
ATGCCGGTTGTGATTAACAGCTTCAATTATGATGATCCGGTGAACGATAACACCATCATTTATATCCGTCCGCCTTATTATGAAACCAGCAACACCTATTTCAAAGCCTTCCAGATTATGGATAACGTGTGGATTATTCCGGAACGTTATCGTCTGGGTATTGATCCGAGCCTGTTTAATCCGCCTGTTAGCCTGAAAGCAGGTAGTGATGGTTATTTTGATCCGAATTATCTGAGCACCAACACCGAGAAAAACAAATACCTGCAGATTATGATCAAGCTGTTCAAACGCATTAATAGCAAACCGGCAGGTCAGATTCTGCTGGAAGAAATCAAAAATGCAATTCCGTATCTGGGCAACAGCTATACCCAAGAAGAACAGTTTACCACCAATAATCGTACCGTGAGCTTTAATGTTAAACTGGCCAATGGTAATATCGTTCAGCAGATGGCAAATCTGATTATTTGGGGTCCGGGTCCTGATCTGACCACAAATAAAACCGGTGGTATCATCTATAGCCCGTATCAGAGCATGGAAGCAACCCCGTATAAAGATGGTTTTGGTAGCATTATGACCGTGGAATTTAGTCCGGAATATGCAACCGCCTTTAACGATATTTCAATTGCAAGCCATAGTCCGTCGCTGTTTATCAAAGATCCGGCACTGATTCTGATGCATGAACTGATTCATGTTCTGCATGGTCTGTATGGCACCTATATTACCGAATACAAAATTACCCCGAATGTGGTGCAGAGCTATATGAAAGTTACCAAACCGATTACCAGCGCAGAATTTCTGACCTTTGGTGGTCGTGATCGCAATATTGTTCCGCAGAGCATTCAGAGCCAGCTGTATAACAAAGTTCTGAGCGATTATAAACGTATTGCCAGCCGTCTGAATAAAGTTAATACCGCAACCGCACTGATCAACATCGATGAATTCAAAAACCTGTACGAGTGGAAATACCAGTTTGCCAAAGATAGCAATGGTGTGTATAGCGTGGATCTGAACAAATTTGAGCAGCTGTACAAAAAAATCTATAGCTTCACCGAATTCAACCTGGCCTATGAGTTTAAAATCAAAACCCGTCTGGGTTATCTGGCCGAAAATTTTGGTCCGTTTTATCTGCCGAATCTGCTGGATGATAGCATTTATACCGAAGTGGATGGTTTTAACATTGGTGCACTGAGCATTAACTATCAGGGTCAGAATATTGGCAGCGATATCAACAGCATCAAAAAACTGCAAGGTCAGGGTGTTGTTAGCCGTGTTGTTCGTCTGTGTAGCAATAGCGAAAATCTGTATTTTCAGGGTGCCAGTCATCATCACCACCATCACCATCACTGA
SEQ ID NO: 32 - полипептидная последовательность rLC/FA (His-меченная)
MPVVINSFNYDDPVNDNTIIYIRPPYYETSNTYFKAFQIMDNVWIIPERYRLGIDPSLFNPPVSLKAGSDGYFDPNYLSTNTEKNKYLQIMIKLFKRINSKPAGQILLEEIKNAIPYLGNSYTQEEQFTTNNRTVSFNVKLANGNIVQQMANLIIWGPGPDLTTNKTGGIIYSPYQSMEATPYKDGFGSIMTVEFSPEYATAFNDISIASHSPSLFIKDPALILMHELIHVLHGLYGTYITEYKITPNVVQSYMKVTKPITSAEFLTFGGRDRNIVPQSIQSQLYNKVLSDYKRIASRLNKVNTATALINIDEFKNLYEWKYQFAKDSNGVYSVDLNKFEQLYKKIYSFTEFNLAYEFKIKTRLGYLAENFGPFYLPNLLDDSIYTEVDGFNIGALSINYQGQNIGSDINSIKKLQGQGVVSRVVRLCSNSENLYFQGASHHHHHHHH
SEQ ID NO: 33 - нуклеотидная последовательность rBoNT/F(0) (His-меченная)
ATGCCGGTTGTGATTAACAGCTTCAATTATAACGATCCGGTGAACGATGATACCATCCTGTATATGCAGATTCCGTATGAAGAGAAAAGCAAAAAGTACTACAAAGCCTTTGAGATCATGCGCAACGTTTGGATTATTCCGGAACGTAATACCATTGGCACCGATCCGAGCGATTTTGATCCGCCTGCAAGCCTGGAAAATGGTAGCAGCGCATATTATGATCCGAATTATCTGACCACCGATGCCGAAAAAGATCGTTATCTGAAAACCACCATCAAACTGTTCAAACGCATTAATAGCAATCCGGCAGGCGAAGTTCTGCTGCAAGAAATTAGCTATGCAAAACCGTATCTGGGCAATGAACATACCCCGATTAATGAATTTCATCCGGTTACACGTACCACGAGCGTTAACATTAAAAGCAGCACCAATGTGAAGTCCAGCATTATTCTGAATCTGCTGGTTTTAGGTGCAGGTCCGGATATTTTTGAAAATTCAAGCTATCCGGTGCGCAAACTGATGGATAGCGGTGGTGTGTATGATCCGTCAAATGATGGTTTTGGCAGCATTAACATTGTGACCTTTAGTCCGGAATATGAATACACCTTCAACGATATTAGCGGTGGCTATAATAGCAGCACCGAAAGTTTTATTGCAGATCCGGCAATTAGCCTGGCACACCAGCTGATTTATGCACTGCATGGTCTGTATGGTGCACGTGGTGTTACCTATAAAGAAACCATTAAAGTTAAACAGGCACCGCTGATGATTGCGGAAAAACCGATTCGTCTGGAAGAATTTCTGACCTTTGGTGGTCAGGATCTGAACATTATTACCAGCGCAATGAAAGAGAAAATCTATAATAACCTGCTGGCCAACTATGAGAAAATTGCAACCCGTCTGAGCCGTGTTAATAGCGCACCTCCTGAATATGATATCAACGAGTATAAAGACTATTTTCAGTGGAAATACGGCCTGGATAAAAATGCAGATGGTAGCTATACCGTGAACGAGAACAAATTTAACGAGATCTACAAAAAACTGTATAGCTTCACCGAAATCGATCTGGCCAACAAATTCAAAGTGAAATGCCGCAACACCTACTTCATCAAATATGGCTTTCTGAAAGTTCCGAACCTGCTTGATGATGATATCTATACCGTTAGCGAAGGCTTTAACATTGGTAATCTGGCCGTTAATAATCGCGGTCAGAACATTAAACTGAACCCGAAAATTATCGATAGCATCCCGGATAAAGGCCTGGTTGAAAAAATTGTGAAATTCTGCAAAAGCGTGATTCCGCGTAAAGGCACCAAAGCACCGCCTCGTCTGTGTATTCGTGTGAATAATCGTGAACTGTTTTTTGTTGCAAGCGAGAGCAGCTATAACGAGAATGATATTAACACCCCGAAAGAGATTGACGATACCACCAATCTGAATAACAACTATCGCAACAATCTGGATGAAGTGATCCTGGATTATAACAGCGAAACCATTCCGCAGATTAGCAATCAGACCCTGAATACCCTGGTTCAGGATGATAGCTATGTTCCGCGTTATGATAGCAATGGCACCAGCGAAATTGAAGAACATAATGTGGTTGATCTGAACGTGTTCTTTTATCTGCATGCACAGAAAGTGCCGGAAGGTGAAACCAATATTAGCCTGACCAGCAGCATTGATACCGCACTGAGCGAAGAAAGCCAGGTTTATACCTTTTTTAGCAGCGAATTCATCAACACCATTAACAAACCGGTTCATGCAGCACTGTTTATTAGCTGGATTAATCAGGTGATTCGCGATTTTACCACCGAAGCAACCCAGAAAAGCACCTTTGATAAAATTGCCGATATTAGTCTGGTGGTGCCGTATGTTGGTCTGGCACTGAATATTGGTAATGAAGTGCAGAAAGAGAACTTTAAAGAAGCCTTCGAACTGTTAGGTGCCGGTATTCTGCTGGAATTTGTGCCGGAACTGCTGATTCCGACCATTCTGGTTTTTACCATTAAGAGCTTTATTGGCAGCAGCGAGAACAAGAACAAAATCATTAAAGCCATCAACAACAGCCTGATGGAACGCGAAACCAAATGGAAAGAAATTTACAGCTGGATTGTGAGCAATTGGCTGACCCGTATCAATACCCAGTTTAACAAACGCAAAGAACAAATGTATCAGGCCCTGCAGAATCAGGTTGATGCAATTAAAACCGTGATCGAATACAAATACAACAACTATACCAGCGACGAACGTAATCGCCTGGAAAGCGAATACAACATTAATAACATTCGCGAAGAACTGAACAAAAAAGTGAGCCTGGCAATGGAAAACATCGAACGTTTTATTACCGAAAGCAGCATCTTCTACCTGATGAAACTGATTAACGAAGCCAAAGTTAGCAAACTGCGCGAATATGATGAAGGCGTTAAAGAATATCTGCTGGACTATATTAGCGAACATCGTAGCATTCTGGGTAATAGCGTTCAAGAGCTGAATGATCTGGTTACCAGCACACTGAATAATAGCATTCCGTTTGAACTGAGCAGCTACACCAACGATAAAATCCTGATCCTGTACTTCAACAAACTGTACAAGAAGATCAAGGACAACAGCATACTGGATATGCGCTATGAAAACAACAAGTTCATTGATATCAGCGGCTATGGTAGCAACATTAGCATTAATGGTGATGTGTATATCTACAGCACCAACCGCAATCAGTTTGGTATTTATAGCAGCAAACCGAGCGAAGTTAATATTGCGCAGAATAACGATATCATCTACAACGGTCGCTATCAGAACTTTAGCATTAGCTTTTGGGTTCGCATTCCGAAATACTTTAACAAGGTGAACCTGAACAACGAGTACACCATTATTGATTGCATTCGCAATAATAACAGCGGCTGGAAAATCAGCCTGAACTATAACAAAATTATCTGGACCCTGCAGGATACCGCAGGTAATAATCAGAAACTGGTGTTTAACTACACCCAGATGATTAGCATCAGCGACTATATCAACAAATGGATCTTTGTGACCATTACCAACAATCGTCTGGGTAACAGCCGCATTTATATCAATGGCAATCTGATCGACGAAAAAAGCATTTCAAATCTGGGCGATATTCACGTGAGCGATAACATTCTGTTCAAAATTGTTGGCTGCAACGATACCCGTTATGTTGGTATTCGTTACTTCAAAGTGTTTGATACGGAACTGGGCAAAACGGAAATTGAAACCCTGTATAGTGATGAACCGGATCCGAGCATTCTGAAAGATTTTTGGGGTAATTATCTGCTGTACAACAAACGCTACTATCTGCTGAACCTGCTGCGTACCGATAAAAGCATTACACAGAATAGCAACTTTCTGAACATCAATCAGCAGCGTGGTGTTTATCAGAAACCGAACATTTTTAGCAACACCCGTCTGTATACCGGTGTGGAAGTTATTATTCGTAAAAACGGTAGCACCGATATCAGCAACACCGATAACTTTGTGCGTAAAAATGACCTGGCCTATATTAACGTTGTTGATCGTGATGTTGAGTATCGTCTGTATGCGGATATTAGCATTGCCAAACCGGAAAAGATTATCAAACTGATCCGTACCAGCAACAGCAATAATTCACTGGGTCAGATTATCGTGATGGACAGCATTGGTAACAATTGCACCATGAATTTCCAGAACAATAACGGTGGTAATATTGGCCTGCTGGGCTTTCATAGCAATAATCTGGTTGCAAGCAGCTGGTATTACAACAACATCCGTAAAAATACCAGCAGTAATGGTTGCTTTTGGAGCTTTATCAGTAAAGAACATGGCTGGCAAGAAAACGAGAACCTGTATTTTCAGGGTGCAAGTCATCATCACCATCACCACCATCATTAA
SEQ ID NO: 34 - Полипептидная последовательность rBoNT/F(0) (His-меченная)
MPVVINSFNYNDPVNDDTILYMQIPYEEKSKKYYKAFEIMRNVWIIPERNTIGTDPSDFDPPASLENGSSAYYDPNYLTTDAEKDRYLKTTIKLFKRINSNPAGEVLLQEISYAKPYLGNEHTPINEFHPVTRTTSVNIKSSTNVKSSIILNLLVLGAGPDIFENSSYPVRKLMDSGGVYDPSNDGFGSINIVTFSPEYEYTFNDISGGYNSSTESFIADPAISLAHQLIYALHGLYGARGVTYKETIKVKQAPLMIAEKPIRLEEFLTFGGQDLNIITSAMKEKIYNNLLANYEKIATRLSRVNSAPPEYDINEYKDYFQWKYGLDKNADGSYTVNENKFNEIYKKLYSFTEIDLANKFKVKCRNTYFIKYGFLKVPNLLDDDIYTVSEGFNIGNLAVNNRGQNIKLNPKIIDSIPDKGLVEKIVKFCKSVIPRKGTKAPPRLCIRVNNRELFFVASESSYNENDINTPKEIDDTTNLNNNYRNNLDEVILDYNSETIPQISNQTLNTLVQDDSYVPRYDSNGTSEIEEHNVVDLNVFFYLHAQKVPEGETNISLTSSIDTALSEESQVYTFFSSEFINTINKPVHAALFISWINQVIRDFTTEATQKSTFDKIADISLVVPYVGLALNIGNEVQKENFKEAFELLGAGILLEFVPELLIPTILVFTIKSFIGSSENKNKIIKAINNSLMERETKWKEIYSWIVSNWLTRINTQFNKRKEQMYQALQNQVDAIKTVIEYKYNNYTSDERNRLESEYNINNIREELNKKVSLAMENIERFITESSIFYLMKLINEAKVSKLREYDEGVKEYLLDYISEHRSILGNSVQELNDLVTSTLNNSIPFELSSYTNDKILILYFNKLYKKIKDNSILDMRYENNKFIDISGYGSNISINGDVYIYSTNRNQFGIYSSKPSEVNIAQNNDIIYNGRYQNFSISFWVRIPKYFNKVNLNNEYTIIDCIRNNNSGWKISLNYNKIIWTLQDTAGNNQKLVFNYTQMISISDYINKWIFVTITNNRLGNSRIYINGNLIDEKSISNLGDIHVSDNILFKIVGCNDTRYVGIRYFKVFDTELGKTEIETLYSDEPDPSILKDFWGNYLLYNKRYYLLNLLRTDKSITQNSNFLNINQQRGVYQKPNIFSNTRLYTGVEVIIRKNGSTDISNTDNFVRKNDLAYINVVDRDVEYRLYADISIAKPEKIIKLIRTSNSNNSLGQIIVMDSIGNNCTMNFQNNNGGNIGLLGFHSNNLVASSWYYNNIRKNTSSNGCFWSFISKEHGWQENENLYFQGASHHHHHHHH
SEQ ID NO: 35 - Нуклеотидная последовательность rLHN/F (His-меченная)
ATGCCGGTTGTGATTAACAGCTTCAATTATAACGATCCGGTGAACGATGATACCATCCTGTATATGCAGATTCCGTATGAAGAGAAAAGCAAAAAGTACTACAAAGCCTTTGAGATCATGCGCAACGTTTGGATTATTCCGGAACGTAATACCATTGGCACCGATCCGAGCGATTTTGATCCGCCTGCAAGCCTGGAAAATGGTAGCAGCGCATATTATGATCCGAATTATCTGACCACCGATGCCGAAAAAGATCGTTATCTGAAAACCACCATCAAACTGTTCAAACGCATTAATAGCAATCCGGCAGGCGAAGTTCTGCTGCAAGAAATTAGCTATGCAAAACCGTATCTGGGCAATGAACATACCCCGATTAATGAATTTCATCCGGTTACACGTACCACGAGCGTTAACATTAAAAGCAGCACCAATGTGAAGTCCAGCATTATTCTGAATCTGCTGGTTTTAGGTGCAGGTCCGGATATTTTTGAAAATTCAAGCTATCCGGTGCGCAAACTGATGGATAGCGGTGGTGTGTATGATCCGTCAAATGATGGTTTTGGCAGCATTAACATTGTGACCTTTAGTCCGGAATATGAATACACCTTCAACGATATTAGCGGTGGCTATAATAGCAGCACCGAAAGTTTTATTGCAGATCCGGCAATTAGCCTGGCACATGAACTGATTCATGCACTGCATGGTCTGTATGGTGCACGTGGTGTTACCTATAAAGAAACCATTAAAGTTAAACAGGCACCGCTGATGATTGCGGAAAAACCGATTCGTCTGGAAGAATTTCTGACCTTTGGTGGTCAGGATCTGAACATTATTACCAGCGCAATGAAAGAGAAAATCTATAATAACCTGCTGGCCAACTATGAGAAAATTGCAACCCGTCTGAGCCGTGTTAATAGCGCACCTCCTGAATATGATATCAACGAGTATAAAGACTATTTTCAGTGGAAATACGGCCTGGATAAAAATGCAGATGGTAGCTATACCGTGAACGAGAACAAATTTAACGAGATCTACAAAAAACTGTATAGCTTCACCGAAATCGATCTGGCCAACAAATTCAAAGTGAAATGCCGCAACACCTACTTCATCAAATATGGCTTTCTGAAAGTTCCGAACCTGCTTGATGATGATATCTATACCGTTAGCGAAGGCTTTAACATTGGTAATCTGGCCGTTAATAATCGCGGTCAGAACATTAAACTGAACCCGAAAATTATCGATAGCATCCCGGATAAAGGCCTGGTTGAAAAAATTGTGAAATTCTGCAAAAGCGTGATTCCGCGTAAAGGCACCAAAGCACCGCCTCGTCTGTGTATTCGTGTGAATAATCGTGAACTGTTTTTTGTTGCAAGCGAGAGCAGCTATAACGAGAATGATATTAACACCCCGAAAGAGATTGACGATACCACCAATCTGAATAACAACTATCGCAACAATCTGGATGAAGTGATCCTGGATTATAACAGCGAAACCATTCCGCAGATTAGCAATCAGACCCTGAATACCCTGGTTCAGGATGATAGCTATGTTCCGCGTTATGATAGCAATGGCACCAGCGAAATTGAAGAACATAATGTGGTTGATCTGAACGTGTTCTTTTATCTGCATGCACAGAAAGTGCCGGAAGGTGAAACCAATATTAGCCTGACCAGCAGCATTGATACCGCACTGAGCGAAGAAAGCCAGGTTTATACCTTTTTTAGCAGCGAATTCATCAACACCATTAACAAACCGGTTCATGCAGCACTGTTTATTAGCTGGATTAATCAGGTGATTCGCGATTTTACCACCGAAGCAACCCAGAAAAGCACCTTTGATAAAATTGCCGATATTAGTCTGGTGGTGCCGTATGTTGGTCTGGCACTGAATATTGGTAATGAAGTGCAGAAAGAGAACTTTAAAGAAGCCTTCGAACTGTTAGGTGCCGGTATTCTGCTGGAATTTGTGCCGGAACTGCTGATTCCGACCATTCTGGTTTTTACCATTAAGAGCTTTATTGGCAGCAGCGAGAACAAGAACAAAATCATTAAAGCCATCAACAACAGCCTGATGGAACGCGAAACCAAATGGAAAGAAATTTACAGCTGGATTGTGAGCAATTGGCTGACCCGTATCAATACCCAGTTTAACAAACGCAAAGAACAAATGTATCAGGCCCTGCAGAATCAGGTTGATGCAATTAAAACCGTGATCGAATACAAATACAACAACTATACCAGCGACGAACGTAATCGCCTGGAAAGCGAATACAACATTAATAACATTCGCGAAGAACTGAACAAAAAAGTGAGCCTGGCAATGGAAAACATCGAACGTTTTATTACCGAAAGCAGCATCTTCTACCTGATGAAACTGATTAACGAAGCCAAAGTTAGCAAACTGCGCGAATATGATGAAGGCGTTAAAGAATATCTGCTGGACTATATTAGCGAACATCGTAGCATTCTGGGTAATAGCGTTCAAGAGCTGAATGATCTGGTTACCAGCACACTGAATAATAGCATTCCGTTTGAACTGAGCAGCTACACCAACGATAAAATCCTGATCCTGTACTTCAACAAACTGTACAAGAAAGAAAACCTGTATTTTCAGGGTGCAAGCCATCATCACCACCATCACCATCATTAA
SEQ ID NO: 36 - Полипептидная последовательность rLHN/F (His-меченная)
MPVVINSFNYNDPVNDDTILYMQIPYEEKSKKYYKAFEIMRNVWIIPERNTIGTDPSDFDPPASLENGSSAYYDPNYLTTDAEKDRYLKTTIKLFKRINSNPAGEVLLQEISYAKPYLGNEHTPINEFHPVTRTTSVNIKSSTNVKSSIILNLLVLGAGPDIFENSSYPVRKLMDSGGVYDPSNDGFGSINIVTFSPEYEYTFNDISGGYNSSTESFIADPAISLAHELIHALHGLYGARGVTYKETIKVKQAPLMIAEKPIRLEEFLTFGGQDLNIITSAMKEKIYNNLLANYEKIATRLSRVNSAPPEYDINEYKDYFQWKYGLDKNADGSYTVNENKFNEIYKKLYSFTEIDLANKFKVKCRNTYFIKYGFLKVPNLLDDDIYTVSEGFNIGNLAVNNRGQNIKLNPKIIDSIPDKGLVEKIVKFCKSVIPRKGTKAPPRLCIRVNNRELFFVASESSYNENDINTPKEIDDTTNLNNNYRNNLDEVILDYNSETIPQISNQTLNTLVQDDSYVPRYDSNGTSEIEEHNVVDLNVFFYLHAQKVPEGETNISLTSSIDTALSEESQVYTFFSSEFINTINKPVHAALFISWINQVIRDFTTEATQKSTFDKIADISLVVPYVGLALNIGNEVQKENFKEAFELLGAGILLEFVPELLIPTILVFTIKSFIGSSENKNKIIKAINNSLMERETKWKEIYSWIVSNWLTRINTQFNKRKEQMYQALQNQVDAIKTVIEYKYNNYTSDERNRLESEYNINNIREELNKKVSLAMENIERFITESSIFYLMKLINEAKVSKLREYDEGVKEYLLDYISEHRSILGNSVQELNDLVTSTLNNSIPFELSSYTNDKILILYFNKLYKKENLYFQGASHHHHHHHH
SEQ ID NO: 37 - Нуклеотидная последовательность rHC/F (His-меченная)
ATGATCAAGGATAACAGCATTCTGGATATGCGCTATGAGAACAACAAATTCATTGATATTAGCGGCTATGGCAGCAACATTAGCATTAATGGTGATGTGTATATCTACAGCACCAACCGTAATCAGTTTGGCATTTATAGCAGCAAACCGAGCGAAGTTAATATTGCCCAGAACAACGATATCATCTATAACGGTCGCTATCAGAACTTCAGCATTAGCTTTTGGGTTCGCATTCCGAAATACTTCAATAAGGTGAACCTGAACAACGAGTATACCATCATTGATTGCATTCGCAATAATAACAGCGGCTGGAAAATTAGCCTGAACTACAACAAAATTATCTGGACCCTGCAGGATACCGCAGGTAATAATCAGAAACTGGTGTTTAACTACACCCAGATGATTAGCATCAGCGACTATATCAACAAATGGATCTTTGTGACCATTACCAATAATCGCCTGGGTAATAGCCGCATTTATATCAATGGTAACCTGATCGATGAGAAAAGCATTAGCAATCTGGGTGATATTCATGTGAGCGATAACATCCTGTTTAAAATCGTGGGTTGTAACGATACCCGTTATGTTGGTATTCGCTACTTCAAAGTGTTTGATACCGAACTGGGTAAAACCGAAATTGAAACCCTGTATAGTGATGAACCGGATCCGAGCATTCTGAAAGATTTTTGGGGTAATTATCTGCTGTACAACAAACGCTACTATCTGCTGAATCTGCTGCGTACCGATAAATCAATTACCCAGAATAGCAACTTCCTGAACATTAATCAGCAGCGTGGTGTTTATCAGAAACCGAACATTTTTAGCAACACCCGTCTGTATACCGGTGTGGAAGTTATTATTCGTAAAAATGGCAGCACCGATATCAGCAACACCGATAACTTTGTTCGCAAAAATGATCTGGCGTATATCAACGTTGTTGATCGTGATGTTGAATATCGTCTGTATGCCGATATTAGCATTGCCAAACCGGAAAAAATCATCAAACTGATCCGTACCAGCAACAGCAATAATTCACTGGGTCAGATTATTGTGATGGATAGCATTGGTAATAACTGCACCATGAACTTTCAGAACAATAACGGTGGTAATATTGGTCTGCTGGGCTTTCATAGTAATAATCTGGTTGCAAGCAGCTGGTATTATAACAACATCCGTAAAAATACCAGCAGCAATGGTTGCTTTTGGAGCTTTATTAGCAAAGAACATGGCTGGCAAGAAAACGAGAATCTGTATTTTCAGGGTGCAAGTCATCATCACCACCATCACCATCATTAA
SEQ ID NO: 38 - Полипептидная последовательность rHC/F (His-меченная)
MIKDNSILDMRYENNKFIDISGYGSNISINGDVYIYSTNRNQFGIYSSKPSEVNIAQNNDIIYNGRYQNFSISFWVRIPKYFNKVNLNNEYTIIDCIRNNNSGWKISLNYNKIIWTLQDTAGNNQKLVFNYTQMISISDYINKWIFVTITNNRLGNSRIYINGNLIDEKSISNLGDIHVSDNILFKIVGCNDTRYVGIRYFKVFDTELGKTEIETLYSDEPDPSILKDFWGNYLLYNKRYYLLNLLRTDKSITQNSNFLNINQQRGVYQKPNIFSNTRLYTGVEVIIRKNGSTDISNTDNFVRKNDLAYINVVDRDVEYRLYADISIAKPEKIIKLIRTSNSNNSLGQIIVMDSIGNNCTMNFQNNNGGNIGLLGFHSNNLVASSWYYNNIRKNTSSNGCFWSFISKEHGWQENENLYFQGASHHHHHHHH
SEQ ID NO: 39 - Нуклеотидная последовательность rLC/F (His-меченная)
ATGCCGGTTGTGATTAACAGCTTCAATTATAACGATCCGGTGAACGATGATACCATCCTGTATATGCAGATTCCGTATGAAGAGAAAAGCAAAAAGTACTACAAAGCCTTTGAGATCATGCGCAACGTTTGGATTATTCCGGAACGTAATACCATTGGCACCGATCCGAGCGATTTTGATCCGCCTGCAAGCCTGGAAAATGGTAGCAGCGCATATTATGATCCGAATTATCTGACCACCGATGCCGAAAAAGATCGTTATCTGAAAACCACCATCAAACTGTTCAAACGCATTAATAGCAATCCGGCAGGCGAAGTTCTGCTGCAAGAAATTAGCTATGCAAAACCGTATCTGGGCAATGAACATACCCCGATTAATGAATTTCATCCGGTTACACGTACCACGAGCGTTAACATTAAAAGCAGCACCAATGTGAAGTCCAGCATTATTCTGAATCTGCTGGTTTTAGGTGCAGGTCCGGATATTTTTGAAAATTCAAGCTATCCGGTGCGCAAACTGATGGATAGCGGTGGTGTGTATGATCCGTCAAATGATGGTTTTGGCAGCATTAACATTGTGACCTTTAGTCCGGAATATGAATACACCTTCAACGATATTAGCGGTGGCTATAATAGCAGCACCGAAAGTTTTATTGCAGATCCGGCAATTAGCCTGGCACATGAACTGATTCATGCACTGCATGGTCTGTATGGTGCACGTGGTGTTACCTATAAAGAAACCATTAAAGTTAAACAGGCACCGCTGATGATTGCGGAAAAACCGATTCGTCTGGAAGAATTTCTGACCTTTGGTGGTCAGGATCTGAACATTATTACCAGCGCAATGAAAGAGAAAATCTATAATAACCTGCTGGCCAACTATGAGAAAATTGCAACCCGTCTGAGCCGTGTTAATAGCGCACCTCCTGAATATGATATCAACGAGTATAAAGACTATTTTCAGTGGAAATACGGCCTGGATAAAAATGCAGATGGTAGCTATACCGTGAACGAGAACAAATTTAACGAGATCTACAAAAAACTGTATAGCTTCACCGAAATCGATCTGGCCAACAAATTCAAAGTGAAATGCCGCAACACCTACTTCATCAAATATGGCTTTCTGAAAGTTCCGAACCTGCTTGATGATGATATCTATACCGTTAGCGAAGGCTTTAACATTGGTAATCTGGCCGTTAATAATCGCGGTCAGAACATTAAACTGAACCCGAAAATTATCGATAGCATCCCGGATAAAGGCCTGGTTGAAAAAATTGTGAAATTCTGCAAAAGCGAGAACCTGTATTTTCAGGGTGCAAGTCATCATCACCATCACCACCATCATTAA
SEQ ID NO: 40 - полипептидная последовательность rLC/F (His-меченная)
MPVVINSFNYNDPVNDDTILYMQIPYEEKSKKYYKAFEIMRNVWIIPERNTIGTDPSDFDPPASLENGSSAYYDPNYLTTDAEKDRYLKTTIKLFKRINSNPAGEVLLQEISYAKPYLGNEHTPINEFHPVTRTTSVNIKSSTNVKSSIILNLLVLGAGPDIFENSSYPVRKLMDSGGVYDPSNDGFGSINIVTFSPEYEYTFNDISGGYNSSTESFIADPAISLAHELIHALHGLYGARGVTYKETIKVKQAPLMIAEKPIRLEEFLTFGGQDLNIITSAMKEKIYNNLLANYEKIATRLSRVNSAPPEYDINEYKDYFQWKYGLDKNADGSYTVNENKFNEIYKKLYSFTEIDLANKFKVKCRNTYFIKYGFLKVPNLLDDDIYTVSEGFNIGNLAVNNRGQNIKLNPKIIDSIPDKGLVEKIVKFCKSENLYFQGASHHHHHHHH
SEQ ID NO: 41 - Нуклеотидная последовательность катионного rHC/A (His-меченная)
ATGATCATCAACACCAGCATTCTGAACCTGCGTTATGAAAGCAAACATCTGATTGATCTGAGCCGTTATGCCAGCAAAATCAATATAGGCAGCAAGGTTAACTTCGACCCGATTGACAAAAATCAGATACAGCTGTTTAATCTGGAAAGCAGCAAAATTGAGGTGATCCTGAAAAAAGCGATCGTGTATAATAGCATGTACGAGAATTTTTCGACCAGCTTTTGGATTCGCATCCCGAAATACTTTAACAAGATTAGCCTGAACAACGAGTATACCATCATTAACTGCATGGAAAACAATAGCGGTTGGAAAGTCAGCCTGAATTATGGCGAAATTATCTGGACCCTGCAGGATACCAAAGAAATCAAACAGCGTGTGGTGTTCAAATACAGCCAGATGATTAATATCAGCGACTATATCAACCGCTGGATTTTTGTGACCATTACCAATAATCGGCTGAACAAGAGCAAGATCTATATTAACGGTCGTCTGATTGACCAGAAACCGATTAGTAATCTGGGTAATATTCATGCGAGCAACAAAATCATGTTTAAACTGGATGGTTGCCGTGATACCCATCGTTATATTTGGATCAAATACTTCAACCTGTTCGATAAAGAGTTGAACGAAAAAGAAATTAAAGACCTGTACGATAACCAGAGCAATAGCGGCATACTGAAAGATTTTTGGGGAGATTATCTGCAGTATGACAAACCGTATTATATGCTGAATCTGTACGACCCGAATAAATACGTGGATGTTAATAATGTGGGCATCCGTGGTTATATGTACCTGAAAGGTCCGCGTGGTAGCGTTATGACCACAAACATTTATCTGAATAGCAGCCTGTATCGCGGAACCAAATTCATCATTAAAAAGTATGCCAGCGGCAACAAGGATAATATTGTGCGTAATAATGATCGCGTGTACATTAACGTTGTGGTGAAGAATAAAGAATATCGCCTGGCAACCAATGCAAGCCAGGCAGGCGTTGAAAAAATTCTGAGTGCCCTGGAAATTCCGGATGTTGGTAATCTGAGCCAGGTTGTTGTGATGAAAAGCAAAAACGATAAAGGCATCACCAACAAATGCAAGATGAATCTGCAGGACAATAACGGCAATGATATTGGCTTCATTGGCTTTCACCAGTTTAACAACATTGCAAAACTGGTTGCGAGCAATTGGTATAATCGTCAGATTGAACGTAGCAGTCGTACCCTGGGTTGTAGCTGGGAATTTATCCCTGTGGATGATGGTTGGGGTGAACGTCCGCTGAAGCTTGCGGCCGCACTCGAGCACCACCACCACCACCACTGA
SEQ ID NO: 42 - Полипептидная последовательность катионного rHC/A (His-меченная)
MIINTSILNLRYESKHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKKAIVYNSMYENFSTSFWIRIPKYFNKISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTKEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNKSKIYINGRLIDQKPISNLGNIHASNKIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDKGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPLKLAAALEHHHHHH
SEQ ID NO: 43 - Нуклеотидная последовательность rHC/AB (His-меченая)
ATGATTCTGAACAATATTATCCTGAACCTGCGTTACAAAGACAACAATCTGATCGATCTGAGCGGCTATGGTGCAAAAGTTGAAGTCTACGACGGTGTCGAACTGAACGATAAAAACCAGTTCAAACTGACCTCATCGGCTAACTCAAAAATTCGTGTGACGCAGAACCAAAACATCATCTTCAACTCGGTCTTTCTGGACTTCAGCGTGTCTTTCTGGATTCGCATCCCGAAATATAAAAATGATGGCATCCAGAACTACATCCATAACGAATACACCATCATCAACTGTATGAAAAACAACAGTGGTTGGAAAATTTCCATCCGTGGCAACCGCATTATCTGGACCCTGATTGATATCAATGGTAAAACGAAAAGCGTGTTTTTCGAATACAACATCCGTGAAGATATCTCTGAATACATCAATCGCTGGTTTTTCGTGACCATTACGAACAATCTGAACAATGCGAAAATCTATATCAACGGCAAACTGGAAAGTAATACCGACATCAAAGATATTCGTGAAGTTATCGCCAACGGTGAAATCATCTTCAAACTGGATGGCGACATCGATCGCACCCAGTTCATTTGGATGAAATACTTCTCCATCTTCAACACGGAACTGAGTCAGTCCAATATCGAAGAACGCTACAAAATCCAATCATACTCGGAATACCTGAAAGATTTCTGGGGTAACCCGCTGATGTACAACAAAGAATACTACATGTTCAACGCGGGCAACAAAAACTCATACATCAAACTGAAAAAAGATTCGCCGGTGGGTGAAATCCTGACCCGTAGCAAATACAACCAGAACTCTAAATACATCAACTATCGCGATCTGTACATTGGCGAAAAATTTATTATCCGTCGCAAAAGCAACTCTCAGAGTATTAATGATGACATCGTGCGTAAAGAAGACTACATCTATCTGGATTTCTTTAATCTGAACCAAGAATGGCGCGTTTATACCTACAAATACTTCAAAAAAGAAGAAATGAAACTGTTCCTGGCCCCGATTTACGACAGCGATGAATTTTACAACACCATCCAGATCAAAGAATACGATGAACAGCCGACGTATAGTTGCCAACTGCTGTTCAAAAAAGACGAAGAATCCACCGATGAAATTGGCCTGATTGGTATCCACCGTTTCTATGAAAGCGGTATCGTTTTCGAAGAATACAAAGATTACTTCTGTATCTCTAAATGGTATCTGAAAGAAGTCAAACGCAAACCGTACAACCTGAAACTGGGCTGCAACTGGCAATTTATCCCGAAAGACGAAGGCTGGACCGAAAAGCTTGCGGCCGCACTCGAGCACCACCACCACCACCACTGA
SEQ ID NO: 44 - Полипептидная последовательность rHC/AB (His-меченная)
MILNNIILNLRYKDNNLIDLSGYGAKVEVYDGVELNDKNQFKLTSSANSKIRVTQNQNIIFNSVFLDFSVSFWIRIPKYKNDGIQNYIHNEYTIINCMKNNSGWKISIRGNRIIWTLIDINGKTKSVFFEYNIREDISEYINRWFFVTITNNLNNAKIYINGKLESNTDIKDIREVIANGEIIFKLDGDIDRTQFIWMKYFSIFNTELSQSNIEERYKIQSYSEYLKDFWGNPLMYNKEYYMFNAGNKNSYIKLKKDSPVGEILTRSKYNQNSKYINYRDLYIGEKFIIRRKSNSQSINDDIVRKEDYIYLDFFNLNQEWRVYTYKYFKKEEMKLFLAPIYDSDEFYNTIQIKEYDEQPTYSCQLLFKKDEESTDEIGLIGIHRFYESGIVFEEYKDYFCISKWYLKEVKRKPYNLKLGCNWQFIPKDEGWTEKLAAALEHHHHHH
SEQ ID NO: 45 - нуклеотидная последовательность варианта rHC/A Y1117V H1253K (His-меченная)
ATGATCATCAATACTAGCATTCTGAACCTGCGTTACGAGAGCAATCATCTGATTGATCTGAGCCGTTATGCAAGCAAGATCAACATCGGTAGCAAGGTCAATTTTGACCCGATCGATAAGAACCAGATCCAGCTGTTTAATCTGGAATCGAGCAAAATTGAGGTTATCCTGAAAAACGCCATTGTCTACAACTCCATGTACGAGAATTTCTCCACCAGCTTCTGGATTCGCATCCCGAAATACTTCAACAGCATTAGCCTGAACAACGAGTATACTATCATCAACTGTATGGAGAACAACAGCGGTTGGAAGGTGTCTCTGAACTATGGTGAGATCATTTGGACCTTGCAGGACACCCAAGAGATCAAGCAGCGCGTCGTGTTCAAGTACTCTCAAATGATCAACATTTCCGATTACATTAATCGTTGGATCTTCGTGACCATTACGAATAACCGTCTGAATAACAGCAAGATTTACATCAATGGTCGCTTGATCGATCAGAAACCGATTAGCAACCTGGGTAATATCCACGCAAGCAACAACATTATGTTCAAATTGGACGGTTGCCGCGATACCCATCGTTATATCTGGATCAAGTATTTCAACCTGTTTGATAAAGAACTGAATGAGAAGGAGATCAAAGATTTGTATGACAACCAATCTAACAGCGGCATTTTGAAGGACTTCTGGGGCGATTATCTGCAATACGATAAGCCGTACTATATGCTGAACCTGgtTGATCCGAACAAATATGTGGATGTCAATAATGTGGGTATTCGTGGTTACATGTATTTGAAGGGTCCGCGTGGCAGCGTTATGACGACCAACATTTACCTGAACTCTAGCCTGTACCGTGGTACGAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAACGATCGTGTCTACATCAACGTGGTCGTGAAGAATAAAGAGTACCGTCTGGCGACCAACGCTTCGCAGGCGGGTGTTGAGAAAATTCTGAGCGCGTTGGAGATCCCTGATGTCGGTAATCTGAGCCAAGTCGTGGTTATGAAGAGCAAGAACGACCAGGGTATCACTAACAAGTGCAAGATGAACCTGCAAGACAACAATGGTAACGACATCGGCTTTATTGGTTTCaAaCAGTTCAACAATATTGCTAAACTGGTAGCGAGCAATTGGTACAATCGTCAGATTGAGCGCAGCAGCCGTACTTTGGGCTGTAGCTGGGAGTTTATCCCGGTCGATGATGGTTGGGGCGAACGTCCGCTGCaCcatcaccatCaCcatcaccatCaCcatT
SEQ ID NO: 46 - полипептидная последовательность варианта rHC/A Y1117V H1253K (His-меченная)
MIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLVDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFKQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPLHHHHHHHHHH
SEQ ID NO: 47 - Нуклеотидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278F (His-меченная)
ATGATCATCAATACTAGCATTCTGAACCTGCGTTACGAGAGCAATCATCTGATTGATCTGAGCCGTTATGCAAGCAAGATCAACATCGGTAGCAAGGTCAATTTTGACCCGATCGATAAGAACCAGATCCAGCTGTTTAATCTGGAATCGAGCAAAATTGAGGTTATCCTGAAAAACGCCATTGTCTACAACTCCATGTACGAGAATTTCTCCACCAGCTTCTGGATTCGCATCCCGAAATACTTCAACAGCATTAGCCTGAACAACGAGTATACTATCATCAACTGTATGGAGAACAACAGCGGTTGGAAGGTGTCTCTGAACTATGGTGAGATCATTTGGACCTTGCAGGACACCCAAGAGATCAAGCAGCGCGTCGTGTTCAAGTACTCTCAAATGATCAACATTTCCGATTACATTAATCGTTGGATCTTCGTGACCATTACGAATAACCGTCTGAATAACAGCAAGATTTACATCAATGGTCGCTTGATCGATCAGAAACCGATTAGCAACCTGGGTAATATCCACGCAAGCAACAACATTATGTTCAAATTGGACGGTTGCCGCGATACCCATCGTTATATCTGGATCAAGTATTTCAACCTGTTTGATAAAGAACTGAATGAGAAGGAGATCAAAGATTTGTATGACAACCAATCTAACAGCGGCATTTTGAAGGACTTCTGGGGCGATTATCTGCAATACGATAAGCCGTACTATATGCTGAACCTGgtTGATCCGAACAAATATGTGGATGTCAATAATGTGGGTATTCGTGGTTACATGTATTTGAAGGGTCCGCGTGGCAGCGTTATGACGACCAACATTTACCTGAACTCTAGCCTGTACCGTGGTACGAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAACGATCGTGTCTACATCAACGTGGTCGTGAAGAATAAAGAGTACCGTCTGGCGACCAACGCTTCGCAGGCGGGTGTTGAGAAAATTCTGAGCGCGTTGGAGATCCCTGATGTCGGTAATCTGAGCCAAGTCGTGGTTATGAAGAGCAAGAACGACCAGGGTATCACTAACAAGTGCAAGATGAACCTGCAAGACAACAATGGTAACGACATCGGCTTTATTGGTTaCaAaCAGTTCAACAATATTGCTAAACTGGTAGCGAGCAATTGGTACAATCGTCAGATTGAGCGCAGCAGCCGTACTTTtGGCTGTAGCTGGGAGTTTATCCCGGTCGATGATGGTTGGGGCGAACGTCCGCTGCaCcatcaccatCaCcatcaccatCaCcatTAA
SEQ ID NO: 48 - Полипептидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278F (His-меченная)
MIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLVDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGYKQFNNIAKLVASNWYNRQIERSSRTFGCSWEFIPVDDGWGERPLHHHHHHHHHH
SEQ ID NO: 49 - нуклеотидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278H (His-меченная)
ATGATCATCAATACTAGCATTCTGAACCTGCGTTACGAGAGCAATCATCTGATTGATCTGAGCCGTTATGCAAGCAAGATCAACATCGGTAGCAAGGTCAATTTTGACCCGATCGATAAGAACCAGATCCAGCTGTTTAATCTGGAATCGAGCAAAATTGAGGTTATCCTGAAAAACGCCATTGTCTACAACTCCATGTACGAGAATTTCTCCACCAGCTTCTGGATTCGCATCCCGAAATACTTCAACAGCATTAGCCTGAACAACGAGTATACTATCATCAACTGTATGGAGAACAACAGCGGTTGGAAGGTGTCTCTGAACTATGGTGAGATCATTTGGACCTTGCAGGACACCCAAGAGATCAAGCAGCGCGTCGTGTTCAAGTACTCTCAAATGATCAACATTTCCGATTACATTAATCGTTGGATCTTCGTGACCATTACGAATAACCGTCTGAATAACAGCAAGATTTACATCAATGGTCGCTTGATCGATCAGAAACCGATTAGCAACCTGGGTAATATCCACGCAAGCAACAACATTATGTTCAAATTGGACGGTTGCCGCGATACCCATCGTTATATCTGGATCAAGTATTTCAACCTGTTTGATAAAGAACTGAATGAGAAGGAGATCAAAGATTTGTATGACAACCAATCTAACAGCGGCATTTTGAAGGACTTCTGGGGCGATTATCTGCAATACGATAAGCCGTACTATATGCTGAACCTGgtTGATCCGAACAAATATGTGGATGTCAATAATGTGGGTATTCGTGGTTACATGTATTTGAAGGGTCCGCGTGGCAGCGTTATGACGACCAACATTTACCTGAACTCTAGCCTGTACCGTGGTACGAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAACGATCGTGTCTACATCAACGTGGTCGTGAAGAATAAAGAGTACCGTCTGGCGACCAACGCTTCGCAGGCGGGTGTTGAGAAAATTCTGAGCGCGTTGGAGATCCCTGATGTCGGTAATCTGAGCCAAGTCGTGGTTATGAAGAGCAAGAACGACCAGGGTATCACTAACAAGTGCAAGATGAACCTGCAAGACAACAATGGTAACGACATCGGCTTTATTGGTTaCaAaCAGTTCAACAATATTGCTAAACTGGTAGCGAGCAATTGGTACAATCGTCAGATTGAGCGCAGCAGCCGTACTcatGGCTGTAGCTGGGAGTTTATCCCGGTCGATGATGGTTGGGGCGAACGTCCGCTGCaCcatcaccatCaCcat
SEQ ID NO: 50 - Полипептидная последовательность варианта rHC/A Y1117V F1252Y H1253K L1278H (His-меченная)
MIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLVDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGYKQFNNIAKLVASNWYNRQIERSSRTHGCSWEFIPVDDGWGERPLHHHHHH
SEQ ID NO: 51 - Полипептидная последовательность BoNT/A - UniProt P10845
MPFVNKQFNYKDPVNGVDIAYIKIPNVGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
SEQ ID NO: 52 - Полипептидная последовательность BoNT/B - UniProt P10844
MPVTINNFNYNDPIDNNNIIMMEPPFARGTGRYYKAFKITDRIWIIPERYTFGYKPEDFNKSSGIFNRDVCEYYDPDYLNTNDKKNIFLQTMIKLFNRIKSKPLGEKLLEMIINGIPYLGDRRVPLEEFNTNIASVTVNKLISNPGEVERKKGIFANLIIFGPGPVLNENETIDIGIQNHFASREGFGGIMQMKFCPEYVSVFNNVQENKGASIFNRRGYFSDPALILMHELIHVLHGLYGIKVDDLPIVPNEKKFFMQSTDAIQAEELYTFGGQDPSIITPSTDKSIYDKVLQNFRGIVDRLNKVLVCISDPNININIYKNKFKDKYKFVEDSEGKYSIDVESFDKLYKSLMFGFTETNIAENYKIKTRASYFSDSLPPVKIKNLLDNEIYTIEEGFNISDKDMEKEYRGQNKAINKQAYEEISKEHLAVYKIQMCKSVKAPGICIDVDNEDLFFIADKNSFSDDLSKNERIEYNTQSNYIENDFPINELILDTDLISKIELPSENTESLTDFNVDVPVYEKQPAIKKIFTDENTIFQYLYSQTFPLDIRDISLTSSFDDALLFSNKVYSFFSMDYIKTANKVVEAGLFAGWVKQIVNDFVIEANKSNTMDKIADISLIVPYIGLALNVGNETAKGNFENAFEIAGASILLEFIPELLIPVVGAFLLESYIDNKNKIIKTIDNALTKRNEKWSDMYGLIVAQWLSTVNTQFYTIKEGMYKALNYQAQALEEIIKYRYNIYSEKEKSNINIDFNDINSKLNEGINQAIDNINNFINGCSVSYLMKKMIPLAVEKLLDFDNTLKKNLLNYIDENKLYLIGSAEYEKSKVNKYLKTIMPFDLSIYTNDTILIEMFNKYNSEILNNIILNLRYKDNNLIDLSGYGAKVEVYDGVELNDKNQFKLTSSANSKIRVTQNQNIIFNSVFLDFSVSFWIRIPKYKNDGIQNYIHNEYTIINCMKNNSGWKISIRGNRIIWTLIDINGKTKSVFFEYNIREDISEYINRWFFVTITNNLNNAKIYINGKLESNTDIKDIREVIANGEIIFKLDGDIDRTQFIWMKYFSIFNTELSQSNIEERYKIQSYSEYLKDFWGNPLMYNKEYYMFNAGNKNSYIKLKKDSPVGEILTRSKYNQNSKYINYRDLYIGEKFIIRRKSNSQSINDDIVRKEDYIYLDFFNLNQEWRVYTYKYFKKEEEKLFLAPISDSDEFYNTIQIKEYDEQPTYSCQLLFKKDEESTDEIGLIGIHRFYESGIVFEEYKDYFCISKWYLKEVKRKPYNLKLGCNWQFIPKDEGWTE
SEQ ID NO: 53 - Полипептидная последовательность BoNT/C - UniProt P18640
MPITINNFNYSDPVDNKNILYLDTHLNTLANEPEKAFRITGNIWVIPDRFSRNSNPNLNKPPRVTSPKSGYYDPNYLSTDSDKDPFLKEIIKLFKRINSREIGEELIYRLSTDIPFPGNNNTPINTFDFDVDFNSVDVKTRQGNNWVKTGSINPSVIITGPRENIIDPETSTFKLTNNTFAAQEGFGALSIISISPRFMLTYSNATNDVGEGRFSKSEFCMDPILILMHELNHAMHNLYGIAIPNDQTISSVTSNIFYSQYNVKLEYAEIYAFGGPTIDLIPKSARKYFEEKALDYYRSIAKRLNSITTANPSSFNKYIGEYKQKLIRKYRFVVESSGEVTVNRNKFVELYNELTQIFTEFNYAKIYNVQNRKIYLSNVYTPVTANILDDNVYDIQNGFNIPKSNLNVLFMGQNLSRNPALRKVNPENMLYLFTKFCHKAIDGRSLYNKTLDCRELLVKNTDLPFIGDISDVKTDIFLRKDINEETEVIYYPDNVSVDQVILSKNTSEHGQLDLLYPSIDSESEILPGENQVFYDNRTQNVDYLNSYYYLESQKLSDNVEDFTFTRSIEEALDNSAKVYTYFPTLANKVNAGVQGGLFLMWANDVVEDFTTNILRKDTLDKISDVSAIIPYIGPALNISNSVRRGNFTEAFAVTGVTILLEAFPEFTIPALGAFVIYSKVQERNEIIKTIDNCLEQRIKRWKDSYEWMMGTWLSRIITQFNNISYQMYDSLNYQAGAIKAKIDLEYKKYSGSDKENIKSQVENLKNSLDVKISEAMNNINKFIRECSVTYLFKNMLPKVIDELNEFDRNTKAKLINLIDSHNIILVGEVDKLKAKVNNSFQNTIPFNIFSYTNNSLLKDIINEYFNNINDSKILSLQNRKNTLVDTSGYNAEVSEEGDVQLNPIFPFDFKLGSSGEDRGKVIVTQNENIVYNSMYESFSISFWIRINKWVSNLPGYTIIDSVKNNSGWSIGIISNFLVFTLKQNEDSEQSINFSYDISNNAPGYNKWFFVTVTNNMMGNMKIYINGKLIDTIKVKELTGINFSKTITFEINKIPDTGLITSDSDNINMWIRDFYIFAKELDGKDINILFNSLQYTNVVKDYWGNDLRYNKEYYMVNIDYLNRYMYANSRQIVFNTRRNNNDFNEGYKIIIKRIRGNTNDTRVRGGDILYFDMTINNKAYNLFMKNETMYADNHSTEDIYAIGLREQTKDINDNIIFQIQPMNNTYYYASQIFKSNFNGENISGICSIGTYRFRLGGDWYRHNYLVPTVKQGNYASLLESTSTHWGFVPVSE
SEQ ID NO: 54 - Полипептидная последовательность BoNT/D - UniProt P19321
MTWPVKDFNYSDPVNDNDILYLRIPQNKLITTPVKAFMITQNIWVIPERFSSDTNPSLSKPPRPTSKYQSYYDPSYLSTDEQKDTFLKGIIKLFKRINERDIGKKLINYLVVGSPFMGDSSTPEDTFDFTRHTTNIAVEKFENGSWKVTNIITPSVLIFGPLPNILDYTASLTLQGQQSNPSFEGFGTLSILKVAPEFLLTFSDVTSNQSSAVLGKSIFCMDPVIALMHELTHSLHQLYGINIPSDKRIRPQVSEGFFSQDGPNVQFEELYTFGGLDVEIIPQIERSQLREKALGHYKDIAKRLNNINKTIPSSWISNIDKYKKIFSEKYNFDKDNTGNFVVNIDKFNSLYSDLTNVMSEVVYSSQYNVKNRTHYFSRHYLPVFANILDDNIYTIRDGFNLTNKGFNIENSGQNIERNPALQKLSSESVVDLFTKVCLRLTKNSRDDSTCIKVKNNRLPYVADKDSISQEIFENKIITDETNVQNYSDKFSLDESILDGQVPINPEIVDPLLPNVNMEPLNLPGEEIVFYDDITKYVDYLNSYYYLESQKLSNNVENITLTTSVEEALGYSNKIYTFLPSLAEKVNKGVQAGLFLNWANEVVEDFTTNIMKKDTLDKISDVSVIIPYIGPALNIGNSALRGNFNQAFATAGVAFLLEGFPEFTIPALGVFTFYSSIQEREKIIKTIENCLEQRVKRWKDSYQWMVSNWLSRITTQFNHINYQMYDSLSYQADAIKAKIDLEYKKYSGSDKENIKSQVENLKNSLDVKISEAMNNINKFIRECSVTYLFKNMLPKVIDELNKFDLRTKTELINLIDSHNIILVGEVDRLKAKVNESFENTMPFNIFSYTNNSLLKDIINEYFNSINDSKILSLQNKKNALVDTSGYNAEVRVGDNVQLNTIYTNDFKLSSSGDKIIVNLNNNILYSAIYENSSVSFWIKISKDLTNSHNEYTIINSIEQNSGWKLCIRNGNIEWILQDVNRKYKSLIFDYSESLSHTGYTNKWFFVTITNNIMGYMKLYINGELKQSQKIEDLDEVKLDKTIVFGIDENIDENQMLWIRDFNIFSKELSNEDINIVYEGQILRNVIKDYWGNPLKFDTEYYIINDNYIDRYIAPESNVLVLVQYPDRSKLYTGNPITIKSVSDKNPYSRILNGDNIILHMLYNSRKYMIIRDTDTIYATQGGECSQNCVYALKLQSNLGNYGIGIFSIKNIVSKNKYCSQIFSSFRENTMLLADIYKPWRFSFKNAYTPVAVTNYETKLLSTSSFWKFISRDPGWVE
SEQ ID NO: 55 - Полипептидная последовательность BoNT/E - UniProt Q00496
MPKINSFNYNDPVNDRTILYIKPGGCQEFYKSFNIMKNIWIIPERNVIGTTPQDFHPPTSLKNGDSSYYDPNYLQSDEEKDRFLKIVTKIFNRINNNLSGGILLEELSKANPYLGNDNTPDNQFHIGDASAVEIKFSNGSQDILLPNVIIMGAEPDLFETNSSNISLRNNYMPSNHRFGSIAIVTFSPEYSFRFNDNCMNEFIQDPALTLMHELIHSLHGLYGAKGITTKYTITQKQNPLITNIRGTNIEEFLTFGGTDLNIITSAQSNDIYTNLLADYKKIASKLSKVQVSNPLLNPYKDVFEAKYGLDKDASGIYSVNINKFNDIFKKLYSFTEFDLRTKFQVKCRQTYIGQYKYFKLSNLLNDSIYNISEGYNINNLKVNFRGQNANLNPRIITPITGRGLVKKIIRFCKNIVSVKGIRKSICIEINNGELFFVASENSYNDDNINTPKEIDDTVTSNNNYENDLDQVILNFNSESAPGLSDEKLNLTIQNDAYIPKYDSNGTSDIEQHDVNELNVFFYLDAQKVPEGENNVNLTSSIDTALLEQPKIYTFFSSEFINNVNKPVQAALFVSWIQQVLVDFTTEANQKSTVDKIADISIVVPYIGLALNIGNEAQKGNFKDALELLGAGILLEFEPELLIPTILVFTIKSFLGSSDNKNKVIKAINNALKERDEKWKEVYSFIVSNWMTKINTQFNKRKEQMYQALQNQVNAIKTIIESKYNSYTLEEKNELTNKYDIKQIENELNQKVSIAMNNIDRFLTESSISYLMKIINEVKINKLREYDENVKTYLLNYIIQHGSILGESQQELNSMVTDTLNNSIPFKLSSYTDDKILISYFNKFFKRIKSSSVLNMRYKNDKYVDTSGYDSNININGDVYKYPTNKNQFGIYNDKLSEVNISQNDYIIYDNKYKNFSISFWVRIPNYDNKIVNVNNEYTIINCMRDNNSGWKVSLNHNEIIWTFEDNRGINQKLAFNYGNANGISDYINKWIFVTITNDRLGDSKLYINGNLIDQKSILNLGNIHVSDNILFKIVNCSYTRYIGIRYFNIFDKELDETEIQTLYSNEPNTNILKDFWGNYLLYDKEYYLLNVLKPNNFIDRRKDSTLSINNIRSTILLANRLYSGIKVKIQRVNNSSTNDNLVRKNDQVYINFVASKTHLFPLYADTATTNKEKTIKISSSGNRFNQVVVMNSVGNCTMNFKNNNGNNIGLLGFKADTVVASTWYYTHMRDHTNSNGCFWNFISEEHGWQEK
SEQ ID NO: 56 - Полипептидная последовательность BoNT/F - UniProt A7GBG3
MPVVINSFNYNDPVNDDTILYMQIPYEEKSKKYYKAFEIMRNVWIIPERNTIGTDPSDFDPPASLENGSSAYYDPNYLTTDAEKDRYLKTTIKLFKRINSNPAGEVLLQEISYAKPYLGNEHTPINEFHPVTRTTSVNIKSSTNVKSSIILNLLVLGAGPDIFENSSYPVRKLMDSGGVYDPSNDGFGSINIVTFSPEYEYTFNDISGGYNSSTESFIADPAISLAHELIHALHGLYGARGVTYKETIKVKQAPLMIAEKPIRLEEFLTFGGQDLNIITSAMKEKIYNNLLANYEKIATRLSRVNSAPPEYDINEYKDYFQWKYGLDKNADGSYTVNENKFNEIYKKLYSFTEIDLANKFKVKCRNTYFIKYGFLKVPNLLDDDIYTVSEGFNIGNLAVNNRGQNIKLNPKIIDSIPDKGLVEKIVKFCKSVIPRKGTKAPPRLCIRVNNRELFFVASESSYNENDINTPKEIDDTTNLNNNYRNNLDEVILDYNSETIPQISNQTLNTLVQDDSYVPRYDSNGTSEIEEHNVVDLNVFFYLHAQKVPEGETNISLTSSIDTALSEESQVYTFFSSEFINTINKPVHAALFISWINQVIRDFTTEATQKSTFDKIADISLVVPYVGLALNIGNEVQKENFKEAFELLGAGILLEFVPELLIPTILVFTIKSFIGSSENKNKIIKAINNSLMERETKWKEIYSWIVSNWLTRINTQFNKRKEQMYQALQNQVDAIKTVIEYKYNNYTSDERNRLESEYNINNIREELNKKVSLAMENIERFITESSIFYLMKLINEAKVSKLREYDEGVKEYLLDYISEHRSILGNSVQELNDLVTSTLNNSIPFELSSYTNDKILILYFNKLYKKIKDNSILDMRYENNKFIDISGYGSNISINGDVYIYSTNRNQFGIYSSKPSEVNIAQNNDIIYNGRYQNFSISFWVRIPKYFNKVNLNNEYTIIDCIRNNNSGWKISLNYNKIIWTLQDTAGNNQKLVFNYTQMISISDYINKWIFVTITNNRLGNSRIYINGNLIDEKSISNLGDIHVSDNILFKIVGCNDTRYVGIRYFKVFDTELGKTEIETLYSDEPDPSILKDFWGNYLLYNKRYYLLNLLRTDKSITQNSNFLNINQQRGVYQKPNIFSNTRLYTGVEVIIRKNGSTDISNTDNFVRKNDLAYINVVDRDVEYRLYADISIAKPEKIIKLIRTSNSNNSLGQIIVMDSIGNNCTMNFQNNNGGNIGLLGFHSNNLVASSWYYNNIRKNTSSNGCFWSFISKEHGWQEN
SEQ ID NO: 57 - Полипептидная последовательность BoNT/G - UniProt Q60393
MPVNIKXFNYNDPINNDDIIMMEPFNDPGPGTYYKAFRIIDRIWIVPERFTYGFQPDQFNASTGVFSKDVYEYYDPTYLKTDAEKDKFLKTMIKLFNRINSKPSGQRLLDMIVDAIPYLGNASTPPDKFAANVANVSINKKIIQPGAEDQIKGLMTNLIIFGPGPVLSDNFTDSMIMNGHSPISEGFGARMMIRFCPSCLNVFNNVQENKDTSIFSRRAYFADPALTLMHELIHVLHGLYGIKISNLPITPNTKEFFMQHSDPVQAEELYTFGGHDPSVISPSTDMNIYNKALQNFQDIANRLNIVSSAQGSGIDISLYKQIYKNKYDFVEDPNGKYSVDKDKFDKLYKALMFGFTETNLAGEYGIKTRYSYFSEYLPPIKTEKLLDNTIYTQNEGFNIASKNLKTEFNGQNKAVNKEAYEEISLEHLVIYRIAMCKPVMYKNTGKSEQCIIVNNEDLFFIANKDSFSKDLAKAETIAYNTQNNTIENNFSIDQLILDNDLSSGIDLPNENTEPFTNFDDIDIPVYIKQSALKKIFVDGDSLFEYLHAQTFPSNIENLQLTNSLNDALRNNNKVYTFFSTNLVEKANTVVGASLFVNWVKGVIDDFTSESTQKSTIDKVSDVSIIIPYIGPALNVGNETAKENFKNAFEIGGAAILMEFIPELIVPIVGFFTLESYVGNKGHIIMTISNALKKRDQKWTDMYGLIVSQWLSTVNTQFYTIKERMYNALNNQSQAIEKIIEDQYNRYSEEDKMNINIDFNDIDFKLNQSINLAINNIDDFINQCSISYLMNRMIPLAVKKLKDFDDNLKRDLLEYIDTNELYLLDEVNILKSKVNRHLKDSIPFDLSLYTKDTILIQVFNNYISNISSNAILSLSYRGGRLIDSSGYGATMNVGSDVIFNDIGNGQFKLNNSENSNITAHQSKFVVYDSMFDNFSINFWVRTPKYNNNDIQTYLQNEYTIISCIKNDSGWKVSIKGNRIIWTLIDVNAKSKSIFFEYSIKDNISDYINKWFSITITNDRLGNANIYINGSLKKSEKILNLDRINSSNDIDFKLINCTDTTKFVWIKDFNIFGRELNATEVSSLYWIQSSTNTLKDFWGNPLRYDTQYYLFNQGMQNIYIKYFSKASMGETAPRTNFNNAAINYQNLYLGLRFIIKKASNSRNINNDNIVREGDYIYLNIDNISDESYRVYVLVNSKEIQTQLFLAPINDDPTFYDVLQIKKYYEKTTYNCQILCEKDTKTFGLFGIGKFVKDYGYVWDTYDNYFCISQWYLRRISENINKLRLGCNWQFIPVDEGWTE
SEQ ID NO: 58 - Полипептидная последовательность TeNT - UniProt P04958
MPITINNFRYSDPVNNDTIIMMEPPYCKGLDIYYKAFKITDRIWIVPERYEFGTKPEDFNPPSSLIEGASEYYDPNYLRTDSDKDRFLQTMVKLFNRIKNNVAGEALLDKIINAIPYLGNSYSLLDKFDTNSNSVSFNLLEQDPSGATTKSAMLTNLIIFGPGPVLNKNEVRGIVLRVDNKNYFPCRDGFGSIMQMAFCPEYVPTFDNVIENITSLTIGKSKYFQDPALLLMHELIHVLHGLYGMQVSSHEIIPSKQEIYMQHTYPISAEELFTFGGQDANLISIDIKNDLYEKTLNDYKAIANKLSQVTSCNDPNIDIDSYKQIYQQKYQFDKDSNGQYIVNEDKFQILYNSIMYGFTEIELGKKFNIKTRLSYFSMNHDPVKIPNLLDDTIYNDTEGFNIESKDLKSEYKGQNMRVNTNAFRNVDGSGLVSKLIGLCKKIIPPTNIRENLYNRTASLTDLGGELCIKIKNEDLTFIAEKNSFSEEPFQDEIVSYNTKNKPLNFNYSLDKIIVDYNLQSKITLPNDRTTPVTKGIPYAPEYKSNAASTIEIHNIDDNTIYQYLYAQKSPTTLQRITMTNSVDDALINSTKIYSYFPSVISKVNQGAQGILFLQWVRDIIDDFTNESSQKTTIDKISDVSTIVPYIGPALNIVKQGYEGNFIGALETTGVVLLLEYIPEITLPVIAALSIAESSTQKEKIIKTIDNFLEKRYEKWIEVYKLVKAKWLGTVNTQFQKRSYQMYRSLEYQVDAIKKIIDYEYKIYSGPDKEQIADEINNLKNKLEEKANKAMININIFMRESSRSFLVNQMINEAKKQLLEFDTQSKNILMQYIKANSKFIGITELKKLESKINKVFSTPIPFSYSKNLDCWVDNEEDIDVILKKSTILNLDINNDIISDISGFNSSVITYPDAQLVPGINGKAIHLVNNESSEVIVHKAMDIEYNDMFNNFTVSFWLRVPKVSASHLEQYGTNEYSIISSMKKHSLSIGSGWSVSLKGNNLIWTLKDSAGEVRQITFRDLPDKFNAYLANKWVFITITNDRLSSANLYINGVLMGSAEITGLGAIREDNNITLKLDRCNNNNQYVSIDKFRIFCKALNPKEIEKLYTSYLSITFLRDFWGNPLRYDTEYYLIPVASSSKDVQLKNITDYMYLTNAPSYTNGKLNIYYRRLYNGLKFIIKRYTPNNEIDSFVKSGDFIKLYVSYNNNEHIVGYPKDGNAFNNLDRILRVGYNAPGIPLYKKMEAVKLRDLKTYSVQLKLYDDKNASLGLVGTHNGQIGNDPNRDILIASNWYFNHLKDKILGCDWYFVPTDEGWTND
SEQ ID NO: 59 - Полипептидная последовательность BoNT/X
MKLEINKFNYNDPIDGINVITMRPPRHSDKINKGKGPFKAFQVIKNIWIVPERYNFTNNTNDLNIPSEPIMEADAIYNPNYLNTPSEKDEFLQGVIKVLERIKSKPEGEKLLELISSSIPLPLVSNGALTLSDNETIAYQENNNIVSNLQANLVIYGPGPDIANNATYGLYSTPISNGEGTLSEVSFSPFYLKPFDESYGNYRSLVNIVNKFVKREFAPDPASTLMHELVHVTHNLYGISNRNFYYNFDTGKIETSRQQNSLIFEELLTFGGIDSKAISSLIIKKIIETAKNNYTTLISERLNTVTVENDLLKYIKNKIPVQGRLGNFKLDTAEFEKKLNTILFVLNESNLAQRFSILVRKHYLKERPIDPIYVNILDDNSYSTLEGFNISSQGSNDFQGQLLESSYFEKIESNALRAFIKICPRNGLLYNAIYRNSKNYLNNIDLEDKKTTSKTNVSYPCSLLNGCIEVENKDLFLISNKDSLNDINLSEEKIKPETTVFFKDKLPPQDITLSNYDFTEANSIPSISQQNILERNEELYEPIRNSLFEIKTIYVDKLTTFHFLEAQNIDESIDSSKIRVELTDSVDEALSNPNKVYSPFKNMSNTINSIETGITSTYIFYQWLRSIVKDFSDETGKIDVIDKSSDTLAIVPYIGPLLNIGNDIRHGDFVGAIELAGITALLEYVPEFTIPILVGLEVIGGELAREQVEAIVNNALDKRDQKWAEVYNITKAQWWGTIHLQINTRLAHTYKALSRQANAIKMNMEFQLANYKGNIDDKAKIKNAISETEILLNKSVEQAMKNTEKFMIKLSNSYLTKEMIPKVQDNLKNFDLETKKTLDKFIKEKEDILGTNLSSSLRRKVSIRLNKNIAFDINDIPFSEFDDLINQYKNEIEDYEVLNLGAEDGKIKDLSGTTSDINIGSDIELADGRENKAIKIKGSENSTIKIAMNKYLRFSATDNFSISFWIKHPKPTNLLNNGIEYTLVENFNQRGWKISIQDSKLIWYLRDHNNSIKIVTPDYIAFNGWNLITITNNRSKGSIVYVNGSKIEEKDISSIWNTEVDDPIIFRLKNNRDTQAFTLLDQFSIYRKELNQNEVVKLYNYYFNSNYIRDIWGNPLQYNKKYYLQTQDKPGKGLIREYWSSFGYDYVILSDSKTITFPNNIRYGALYNGSKVLIKNSKKLDGLVRNKDFIQLEIDGYNMGISADRFNEDTNYIGTTYGTTHDLTTDFEIIQRQEKYRNYCQLKTPYNIFHKSGLMSTETSKPTFHDYRDWVYSSAWYFQNYENLNLRKHTKTNWYFIPKDEGWDED
SEQ ID NO: 60 - Нуклеотидная последовательность mrBoNT/A
ATGCCATTCGTCAACAAGCAATTCAACTACAAAGACCCAGTCAACGGCGTCGACATCGCATACATCAAGATTCCGAACGCCGGTCAAATGCAGCCGGTTAAGGCTTTTAAGATCCACAACAAGATTTGGGTTATCCCGGAGCGTGACACCTTCACGAACCCGGAAGAAGGCGATCTGAACCCGCCACCGGAAGCGAAGCAAGTCCCTGTCAGCTACTACGATTCGACGTACCTGAGCACGGATAACGAAAAAGATAACTACCTGAAAGGTGTGACCAAGCTGTTCGAACGTATCTACAGCACGGATCTGGGTCGCATGCTGCTGACTAGCATTGTTCGCGGTATCCCGTTCTGGGGTGGTAGCACGATTGACACCGAACTGAAGGTTATCGACACTAACTGCATTAACGTTATTCAACCGGATGGTAGCTATCGTAGCGAAGAGCTGAATCTGGTCATCATTGGCCCGAGCGCAGACATTATCCAATTCGAGTGCAAGAGCTTTGGTCACGAGGTTCTGAATCTGACCCGCAATGGCTATGGTAGCACCCAGTACATTCGTTTTTCGCCGGATTTTACCTTCGGCTTTGAAGAGAGCCTGGAGGTTGATACCAATCCGTTGCTGGGTGCGGGCAAATTCGCTACCGATCCGGCTGTCACGCTGGCCCATGAACTGATCCACGCAGGCCACCGCCTGTACGGCATTGCCATCAACCCAAACCGTGTGTTCAAGGTTAATACGAATGCATACTACGAGATGAGCGGCCTGGAAGTCAGCTTCGAAGAACTGCGCACCTTCGGTGGCCATGACGCTAAATTCATTGACAGCTTGCAAGAGAATGAGTTCCGTCTGTACTACTATAACAAATTCAAAGACATTGCAAGCACGTTGAACAAGGCCAAAAGCATCGTTGGTACTACCGCGTCGTTGCAGTATATGAAGAATGTGTTTAAAGAGAAGTACCTGCTGTCCGAGGATACCTCCGGCAAGTTTAGCGTTGATAAGCTGAAGTTTGACAAACTGTACAAGATGCTGACCGAGATTTACACCGAGGACAACTTTGTGAAATTCTTCAAAGTGTTGAATCGTAAAACCTATCTGAATTTTGACAAAGCGGTTTTCAAGATTAACATCGTGCCGAAGGTGAACTACACCATCTATGACGGTTTTAACCTGCGTAACACCAACCTGGCGGCGAACTTTAACGGTCAGAATACGGAAATCAACAACATGAATTTCACGAAGTTGAAGAACTTCACGGGTCTGTTCGAGTTCTATAAGCTGCTGTGCGTGCGCGGTATCATCACCAGCAAAACCAAAAGCCTGGACAAAGGCTACAACAAGGCGCTGAATGACCTGTGCATTAAGGTAAACAATTGGGATCTGTTCTTTTCGCCATCCGAAGATAATTTTACCAACGACCTGAACAAGGGTGAAGAAATCACCAGCGATACGAATATTGAAGCAGCGGAAGAGAATATCAGCCTGGATCTGATCCAGCAGTACTATCTGACCTTTAACTTCGACAATGAACCGGAGAACATTAGCATTGAGAATCTGAGCAGCGACATTATCGGTCAGCTGGAACTGATGCCGAATATCGAACGTTTCCCGAACGGCAAAAAGTACGAGCTGGACAAGTACACTATGTTCCATTACCTGCGTGCACAGGAGTTTGAACACGGTAAAAGCCGTATCGCGCTGACCAACAGCGTTAACGAGGCCCTGCTGAACCCGAGCCGTGTCTATACCTTCTTCAGCAGCGACTATGTTAAGAAAGTGAACAAAGCCACTGAGGCCGCGATGTTCCTGGGCTGGGTGGAACAGCTGGTATATGACTTCACGGACGAGACGAGCGAAGTGAGCACTACCGACAAAATTGCTGATATTACCATCATTATCCCGTATATTGGTCCGGCACTGAACATTGGCAACATGCTGTACAAAGACGATTTTGTGGGTGCCCTGATCTTCTCCGGTGCCGTGATTCTGCTGGAGTTCATTCCGGAGATTGCGATCCCGGTGTTGGGTACCTTCGCGCTGGTGTCCTACATCGCGAATAAGGTTCTGACGGTTCAGACCATCGATAACGCGCTGTCGAAACGTAATGAAAAATGGGACGAGGTTTACAAATACATTGTTACGAATTGGCTGGCGAAAGTCAATACCCAGATCGACCTGATCCGTAAGAAAATGAAAGAGGCGCTGGAGAATCAGGCGGAGGCCACCAAAGCAATTATCAACTACCAATACAACCAGTACACGGAAGAAGAGAAGAATAACATTAACTTCAATATCGATGATTTGAGCAGCAAGCTGAATGAATCTATCAACAAAGCGATGATCAATATCAACAAGTTTTTGAATCAGTGTAGCGTTTCGTACCTGATGAATAGCATGATTCCGTATGGCGTCAAACGTCTGGAGGACTTCGACGCCAGCCTGAAAGATGCGTTGCTGAAATACATTTACGACAATCGTGGTACGCTGATTGGCCAAGTTGACCGCTTGAAAGACAAAGTTAACAATACCCTGAGCACCGACATCCCATTTCAACTGAGCAAGTATGTTGATAATCAACGTCTGTTGAGCACTTTCACCGAGTATATCAAAAACATCATCAATACTAGCATTCTGAACCTGCGTTACGAGAGCAAGCATCTGATTGATCTGAGCCGTTATGCTAGCAAGATCAACATCGGTAGCAAGGTCAATTTTGACCCGATCGATAAGAACCAGATCCAGCTGTTTAATCTGGAATCGAGCAAAATTGAGGTTATCCTGAAAAAGGCCATTGTCTACAACTCCATGTACGAGAATTTCTCCACCAGCTTCTGGATTCGCATCCCGAAATACTTCAACAAGATTAGCCTGAACAACGAGTATACTATCATCAACTGTATGGAGAACAACAGCGGTTGGAAGGTGTCTCTGAACTATGGTGAGATCATTTGGACCTTGCAGGACACCAAAGAGATCAAGCAGCGCGTCGTGTTCAAGTACTCTCAAATGATCAACATTTCCGATTACATTAATCGTTGGATCTTCGTGACCATTACGAATAACCGTCTGAATAAGAGCAAGATTTACATCAATGGTCGCTTGATCGATCAGAAACCGATTAGCAACCTGGGTAATATCCACGCAAGCAACAAGATTATGTTCAAATTGGACGGTTGCCGCGATACCCATCGTTATATCTGGATCAAGTATTTCAACCTGTTTGATAAAGAACTGAATGAGAAGGAGATCAAAGATTTGTATGACAACCAATCTAACAGCGGCATTTTGAAGGACTTCTGGGGCGATTATCTGCAATACGATAAGCCGTACTATATGCTGAACCTGTATGATCCGAACAAATATGTGGATGTCAATAATGTGGGTATTCGTGGTTACATGTATTTGAAGGGTCCGCGTGGCAGCGTTATGACGACCAACATTTACCTGAACTCTAGCCTGTACCGTGGTACGAAATTCATCATTAAGAAATATGCCAGCGGCAACAAAGATAACATTGTGCGTAATAACGATCGTGTCTACATCAACGTGGTCGTGAAGAATAAAGAGTACCGTCTGGCGACCAACGCTTCGCAGGCGGGTGTTGAGAAAATTCTGAGCGCGTTGGAGATCCCTGATGTCGGTAATCTGAGCCAAGTCGTGGTTATGAAGAGCAAGAACGACAAGGGTATCACTAACAAGTGCAAGATGAACCTGCAAGACAACAATGGTAACGACATCGGCTTTATTGGTTTCCACCAGTTCAACAATATTGCTAAACTGGTAGCGAGCAATTGGTACAATCGTCAGATTGAGCGCAGCAGCcGTACTTTGGGCTGTAGCTGGGAGTTTATCCCGGTCGATGATGGTTGGGGCGAACGTCCGCTG
SEQ ID NO: 61 - полипептидная последовательность mrBoNT/A.
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNIINTSILNLRYESKHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKKAIVYNSMYENFSTSFWIRIPKYFNKISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTKEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNKSKIYINGRLIDQKPISNLGNIHASNKIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDKGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
SEQ ID NO: 62 - Полипептидная последовательность не модифицированного BoNT/A1
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNIINTSILNLRYESNHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKNAIVYNSMYENFSTSFWIRIPKYFNSISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTQEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNNSKIYINGRLIDQKPISNLGNIHASNNIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDQGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
SEQ ID NO: 63 - Полипептидная последовательность mrBoNT/AB
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHELIHAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNILNNIILNLRYKDNNLIDLSGYGAKVEVYDGVELNDKNQFKLTSSANSKIRVTQNQNIIFNSVFLDFSVSFWIRIPKYKNDGIQNYIHNEYTIINCMKNNSGWKISIRGNRIIWTLIDINGKTKSVFFEYNIREDISEYINRWFFVTITNNLNNAKIYINGKLESNTDIKDIREVIANGEIIFKLDGDIDRTQFIWMKYFSIFNTELSQSNIEERYKIQSYSEYLKDFWGNPLMYNKEYYMFNAGNKNSYIKLKKDSPVGEILTRSKYNQNSKYINYRDLYIGEKFIIRRKSNSQSINDDIVRKEDYIYLDFFNLNQEWRVYTYKYFKKEEMKLFLAPIYDSDEFYNTIQIKEYDEQPTYSCQLLFKKDEESTDEIGLIGIHRFYESGIVFEEYKDYFCISKWYLKEVKRKPYNLKLGCNWQFIPKDEGWTE
SEQ ID NO: 64 - Полипептидная последовательность mrBoNT/AB(0)
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHQLIYAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNILNNIILNLRYKDNNLIDLSGYGAKVEVYDGVELNDKNQFKLTSSANSKIRVTQNQNIIFNSVFLDFSVSFWIRIPKYKNDGIQNYIHNEYTIINCMKNNSGWKISIRGNRIIWTLIDINGKTKSVFFEYNIREDISEYINRWFFVTITNNLNNAKIYINGKLESNTDIKDIREVIANGEIIFKLDGDIDRTQFIWMKYFSIFNTELSQSNIEERYKIQSYSEYLKDFWGNPLMYNKEYYMFNAGNKNSYIKLKKDSPVGEILTRSKYNQNSKYINYRDLYIGEKFIIRRKSNSQSINDDIVRKEDYIYLDFFNLNQEWRVYTYKYFKKEEMKLFLAPIYDSDEFYNTIQIKEYDEQPTYSCQLLFKKDEESTDEIGLIGIHRFYESGIVFEEYKDYFCISKWYLKEVKRKPYNLKLGCNWQFIPKDEGWTE
SEQ ID NO: 65 - Полипептидная последовательность mrBoNT/A(0)
MPFVNKQFNYKDPVNGVDIAYIKIPNAGQMQPVKAFKIHNKIWVIPERDTFTNPEEGDLNPPPEAKQVPVSYYDSTYLSTDNEKDNYLKGVTKLFERIYSTDLGRMLLTSIVRGIPFWGGSTIDTELKVIDTNCINVIQPDGSYRSEELNLVIIGPSADIIQFECKSFGHEVLNLTRNGYGSTQYIRFSPDFTFGFEESLEVDTNPLLGAGKFATDPAVTLAHQLIYAGHRLYGIAINPNRVFKVNTNAYYEMSGLEVSFEELRTFGGHDAKFIDSLQENEFRLYYYNKFKDIASTLNKAKSIVGTTASLQYMKNVFKEKYLLSEDTSGKFSVDKLKFDKLYKMLTEIYTEDNFVKFFKVLNRKTYLNFDKAVFKINIVPKVNYTIYDGFNLRNTNLAANFNGQNTEINNMNFTKLKNFTGLFEFYKLLCVRGIITSKTKSLDKGYNKALNDLCIKVNNWDLFFSPSEDNFTNDLNKGEEITSDTNIEAAEENISLDLIQQYYLTFNFDNEPENISIENLSSDIIGQLELMPNIERFPNGKKYELDKYTMFHYLRAQEFEHGKSRIALTNSVNEALLNPSRVYTFFSSDYVKKVNKATEAAMFLGWVEQLVYDFTDETSEVSTTDKIADITIIIPYIGPALNIGNMLYKDDFVGALIFSGAVILLEFIPEIAIPVLGTFALVSYIANKVLTVQTIDNALSKRNEKWDEVYKYIVTNWLAKVNTQIDLIRKKMKEALENQAEATKAIINYQYNQYTEEEKNNINFNIDDLSSKLNESINKAMININKFLNQCSVSYLMNSMIPYGVKRLEDFDASLKDALLKYIYDNRGTLIGQVDRLKDKVNNTLSTDIPFQLSKYVDNQRLLSTFTEYIKNIINTSILNLRYESKHLIDLSRYASKINIGSKVNFDPIDKNQIQLFNLESSKIEVILKKAIVYNSMYENFSTSFWIRIPKYFNKISLNNEYTIINCMENNSGWKVSLNYGEIIWTLQDTKEIKQRVVFKYSQMINISDYINRWIFVTITNNRLNKSKIYINGRLIDQKPISNLGNIHASNKIMFKLDGCRDTHRYIWIKYFNLFDKELNEKEIKDLYDNQSNSGILKDFWGDYLQYDKPYYMLNLYDPNKYVDVNNVGIRGYMYLKGPRGSVMTTNIYLNSSLYRGTKFIIKKYASGNKDNIVRNNDRVYINVVVKNKEYRLATNASQAGVEKILSALEIPDVGNLSQVVVMKSKNDKGITNKCKMNLQDNNGNDIGFIGFHQFNNIAKLVASNWYNRQIERSSRTLGCSWEFIPVDDGWGERPL
ПРИМЕРЫ
ПРИМЕР 1
Множественные каталитически неактивные серотипы BoNT увеличивают общую длину нейритов по сравнению с необработанными контрольными клетками
Материалы и методы
Пять каталитически неактивных (т.е. эндопептидаза-неактивных) серотипов ботулинического нейротоксина (BoNT) рекомбинантно экспрессируют в E. coli, а именно соответствующие серотипам A, B, C, E и F, и обозначают как rBoNT/A(0), rBoNT/B(0), rBoNT/C(0), rBoNT/E(0) и rBoNT/F(0). В результате своей каталитической неактивности эти молекулы не способны расщеплять свои соответствующие (SNARE) белковые субстраты.
Мотонейрон-подобную гибридную клеточную линию (клетки NSC34) (Tebu-Bio, Cedarlane laboratories, France), культивируют в черных многолуночных планшетах, покрытых поли-D-лизином, по 5000 клеток/лунку и культивируют в среде DMEM с добавлением 10% FCS и пенициллина/стрептомицин. После посева клетки дифференцируют в моторнейроны путем воздействия 1 мкМ ретиноевой кислоты и низкой сыворотки в течение 4 дней, затем клетки обрабатывают rBoNT/A(0), rBoNT/B(0), rBoNT/C(0), rBoNT/E(0) и rBoNT/F(0) в 3 различных концентрациях: 0,1, 1 и 10 нМ в течение 4 дней и фиксируют 4% параформальдегидом-4% сахарозой. Нейротрофический фактор головного мозга (BDNF) (коммерчески доступный от ReproTech EC Ltd, London, UK) 1 нг/мл используют в качестве положительного контроля роста нейронов. Клетки фиксируют 4% параформальдегидом-4% сахарозой, затем окрашивают соответствующими антителами. В частности, анти-βIII тубулин mAb (Promega G7121) разводят (1:1000) в 1xPBS+2% BSA+0,3% TritonX-100 и планшеты инкубируют при 37°C в течение 3 часов. Затем вводят вторичное анти-мышиное IgG антитело козы Alexa Fluor 488 Goat anti-Mouse IgG (H+L) (Life Tech, кат. A-11001) (1:2000 в 1x PBS+2% BSA+0,3% TritonX-100) в течение 1 ч при 37°C. Ядра окрашивают DAPI. Анализ изображения: 6 изображений на лунку получают с помощью ArrayScan XTI HCA Reader (Thermo Fisher Scientific) с 10x объективом. Весь анализ проводят с использованием программного обеспечения Image J (открытое программное обеспечение от NIH, Maryland, USA). Проводят три независимых эксперимента. Каждый независимый эксперимент включает 6 повторов.
Результаты
Клетки подвергают воздействию различных каталитически неактивных серотипов BoNT в течение 4 дней (фигура 1). На фигуре 1 показано среднее значение роста нейритов клеток NSC34, обработанных тремя различными концентрациями. На графике представлены средние значения трех независимых экспериментальных циклов. Данные среднего роста нейритов подтверждает, что rBoNT/A(0) увеличивает длину нейритов на клетку NSC34 по сравнению с необработанным контролем, аналогично положительному контролю BDNF. Также обнаружено, что rBoNT/B(0), rBoNT/C(0), rBoNT/E(0) и rBoNT/F(0) увеличивает длину нейритов на клетку NSC34.
Таким образом, эти данные подтверждают, что нейротрофические свойства BoNT/A можно экстраполировать и на другие серотипы BoNT.
ПРИМЕР 2
L-цепь BoNT и LHN увеличивают общую длину нейритов по сравнению с контролем
Материалы и методы
Каталитически неактивный ботулинический токсин rBoNT/A(0) рекомбинантно экспрессируют в E. coli. Фрагменты BoNT/A экспрессируют в E. coli и обозначают как легкую цепь (L/A), легкую цепь и домен транслокации (LHN/A) и фрагмент клеточно-связывающего домена (HC/A) тяжелой цепи. Клетки NSC34 подвергают воздействию фрагментов BoNT/A, а также полноразмерного rBoNT/A(0), как в примере 1.
Результаты
На фигуре 2 показано среднее значение роста нейритов клеток NSC34, обработанных тремя различными концентрациями rBoNT/A(0), rL/A, rLHN/A и rHc/A. На графике представлено среднее значение трех независимых экспериментальных циклов.
Подобно rHC/A, было обнаружено, что как rL/A, так и rLHN/A увеличивают длину нейритов на клетку NSC34 при каждой концентрации по сравнению с необработанным контролем, аналогично положительному контролю BDNF. Особенно неожиданно, что фрагменты rL/A и rLHN/A являются нейротрофическими, поскольку в обоих отсутствует рецептор-связывающий домен клостридиального токсина (присутствует в rHC/A).
ПРИМЕР 3
Другие белки, вводимые в концентрации, аналогичной BoNT/A(0) или его фрагментам, не увеличивают рост нейритов
Материалы и методы
Клетки NSC34 дифференцируют, затем культивируют в течение 4 дней при следующих экспериментальных условиях: (1) контроль из необработанных клеток: клетки подвергают такому же количеству манипуляций, т.е. промывок/подкормок, как и клетки, обработанные соединением, однако необработанные контрольные клетки подвергают воздействию только среды для выращивания клеток, (2) BDNF - положительный аналитический контроль, 1 нг/мл, (3) BoNT/A(0) в 3 дозах (0,1, 1 и 10 нМ), (4) отрицательный контроль (белковый контроль): 1. A7030, Sigma, бычий сывороточный альбумин (BSA), 2. NBP1-37082, Bio-techne, рекомбинантный белок человека аннексин A4, 3. U-100AT, Bio-techne, рекомбинантный растительный белок убиквитин, 4. лизат экспрессионный E. coli, который не содержат ботулинические нейротоксины или их фрагменты. Все белки отрицательного контроля тестируют в конечной концентрации 1,5 мкг/мл. Эта концентрация соответствует 10 нМ BoNT/A(0). Белковые растворы находятся PBS, за исключением аннексина 4-20 мМ Tris-HCl-буфера (рН 8,0), содержащего 20% глицерина, 0,2 М NaCl. Все растворы белка имеют концентрацию 1 мг/мл. Клетки окрашивают разбавителем Anti-Beta III Tubulin 1:1000 в 1xPBS-4%BSA-0,3% TritonX100 и вторичным анти-мышиным антителом Alexa Fluor 488; DAPI используют в качестве красителя ядра. Все исходные изображения сигнала бета-3-тубулина обрабатывают с помощью NeurphologyJ (макрос Image J, NIH, Maryland, USA).
Результаты
Клетки подвергают воздействию различных экспериментальных условий. На фигуре 3 показана средняя длина нейритов в клетках NSC34. На графике представлено среднее значение трех независимых экспериментальных циклов. Данные по среднему росту нейритов подтверждают, что хотя rBoNT/A(0) увеличивает длину нейритов на клетку NSC34 по сравнению с необработанным контролем, то же самое отмечают для положительного контроля BDNF. Напротив, ни одно из других условий «отрицательного контроля» не увеличивает длину нейритов. Это подтверждает нейротрофические эффекты, наблюдаемые при воздействии rL/A и rLHN/A (а также различных серотипов BoNT и rHC/A), и демонстрирует, что эффекты возникают не просто из-за воздействия на клетки NSC34 белков или предполагаемых остаточных компонентов E. coli, присутствующих в препаратах ботулинического токсина.
ПРИМЕР 4
Лечение повреждения нейронов in vivo
Исследование разработано для изучения эффективности каталитически неактивного ботулинического токсина rBoNT/A(0) в усилении функционального восстановления и нейрорегенерации с использованием модели повреждения заднего столба мыши in vivo. Модель полезна для анализа эффективности молекул, которые вызывают локальное прорастание и/или регенерации аксонов длинных путей. Как хорошо известно, сдавливание является частым сценарием при повреждении спинного мозга, и поэтому модель имитирует большинство патологических изменений, происходящих в спинном мозге после травмы (см. Lagord et al, 2002; Molecular and Cellular Neuroscience 20:69; Esmaelli et al., 2014; Neural Regeneration Research 9:1653; Surey et al., 2014; Neuroscience 275C:62; Almutiri et al., 2018; Scientific Reports 8:10707 для деталей модели и ответов на травму).
Материалы и методы
Мышиная модель повреждения спинного мозга
Перед операцией мышам C57/BL подкожно вводят бупренорфин и анестезируют 5% изофлураном в 1,8 мл/л O2, при этом температуру тела и частоту сердечных сокращений контролируют на протяжении всей операции. После частичной ламинэктомии на грудном уровне 8 (T8) восходящий сенсорный, нисходящий моторный и сегментарный проприоцептивный аксоны (SPA) заднего столба спинного мозга (SDC) сдавливают с двух сторон с помощью калиброванных часовых щипцов глубиной 1 мм и шириной 1 мм.
Введение лекарственного средства
Введение rBoNT/A(0) осуществляют путем однократной интратекальной 10 мкл инъекции (в CSF позвоночного канала) одной из 3 доз (100 пг, 100 нг и 50 мкг/мышь) во время операции. Группы лечения для каждой из 3 доз следующие:
1. Носитель (физиологический раствор с фосфатным буфером [PBS]), т. е. поражение SDC плюс немедленная однократная интратекальная 10 мкл инъекция носителя; n=6 мышей.
2. Лечение BoNT, т. е. поражение SDC плюс немедленная однократная интратекальная 10 мкл инъекция одной из 3 доз BoNT (100 пг, 100 нг и 50 мкг/мышь), 3x n=6/группу, 18 мышей.
Интратекальную инъекцию BoNT проводят следующим образом. Мышей помещают в положение лежа, и инъекцию делают между позвонками L5 и S1. Остистые отростки иссекают и отражают рострально для выявления желтой связки под углом 60° к горизонтали, и доступ в интратекальное пространство подтверждают рефлюксом спинномозговой жидкости (CSF) и наличием «подергивания хвоста». Затем 10 мкл вводимого раствора медленно инъецируют в течение 1 мин, и экспрессию CSF усиливают осторожным подъемом хвоста.
Измеренные конечные точки
1. Двигательную функцию измеряют с помощью теста ходьбы по горизонтальной лестнице на исходном уровне (до травмы), и затем еще раз на 2 д, 1 нед, 2 нед, 3 нед и 4 нед после травмы SDC.
2. Качественная гистологическая оценка в момент времени 4 нед прорастания и регенерации моторных и сенсорных нейронов/аксонов, т. е. рост аксонов на короткие (<1 мм) и длинные (~5 мм) расстояния. Срезы тканей, окрашенные нейрофиламентом 200 (NF200), определяют зрелые аксоны. Фосфорилированный MAP1b присутствует в растущих аксонах и конусах роста, где он поддерживает динамический баланс между компонентами цитоскелета и регулирует стабильность и взаимодействие микротрубочек и актина, чтобы способствовать росту аксонов, нейронной проводимости и регенерации в центральной нервной системе. Окрашивание MAP1b показывает области активного разрастания аксонов.
Тест горизонтальной лестницы
Этот тест проверяет двигательную функцию и выполняется на горизонтальной лестнице длиной 0,6 метра с шириной 8 см и произвольно отрегулированными перекладинами с переменными промежутками 1-2 см. До травмы, затем снова на 2 д, 1 нед, 2 нед, 3 нед и 4 нед неделе после травмы SDC мышей оценивают при пересечении лестницы, и соскальзывание левой и правой задней лапы, вместе с общим количеством шагов, регистрирует индивидуум, который не знает о группе лечения. Для расчета среднего коэффициента ошибок, количество промахов делят на общее количество шагов.
Подготовка тканей и криосрезы
Через 4 недели после повреждения SDC мышам вводят интракардиальную перфузию 4% формальдегидом (Raymond A Lamb, Peterborough, UK) и иссеченные сегменты спинного мозга T8, содержащие участки повреждения DC (место поражения+5 мм с каждой стороны) вместе с большеберцовыми мышцами фиксируют в течение 2 часов при КТ, подвергают криозащите в градуированной серии сахарозы, блокируют в среде с оптимальной температурой резки (OCT; Raymond A Lamb) и делают срезы толщиной 15 мкм с использованием криостата Bright.
Иммуногистохимия
Срезы размораживают при комнатной температуре в течение 30 мин, затем дважды промывают в 0,1 М физиологическом растворе с фосфатным буфером, рН 7,4 (PBS; Raymond A Lamb), затем срезы пропитывают 0,1% Triton X-100 в PBS (Sigma) в течение 10 мин и блокируют в PBS, содержащем 0,5% бычьего сывороточного альбумина (BSA) и 0,1% Triton-X100 (все от Sigma) в течение 30 минут при комнатной температуре. Срезы затем инкубируют с подходящим первичным антителом, разведенным буфером для разведения антител (ADB; PBS, содержащим 0,5% BSA и 0,05% Tween-20 (все от Sigma)) и инкубируют в течение ночи при 4°C во влажной камере. Затем срезы промывают в PBS и инкубируют с соответствующим флуоресцентно-меченым вторичным антителом, разведенным в ADB. Затем срезы промывают в PBS, и покровные стекла заделывают с использованием Vectashield, содержащего DAPI (Vector Laboratories, Peterborough, UK). Отрицательные контроли включают в каждый прогон, который включает отсутствие первичного антитела, и их используют для установки фоновых пороговых уровней для получения изображения. Срезы просматривают, и изображения получают с использованием эпифлуоресцентного микроскопа Axioplan 2, оборудованного Axiocam HRc с программным обеспечением Axiovision.
Используемые первичные антитела следующие:
- анти-NF200 кролика, Sigma, Poole, UK (разведение 1:300)
- MAP1b кролика Abcam, Cambridge, UK (разведение 1:400)
Используемые вторичные антитела следующие:
- Alexa 488 анти-кроличье IgG, Invitrogen, Paisley, UK (разведение 1:400)
- Alexa 594 анти-кроличье IgG, Invitrogen, Paisley, UK (разведение 1:400)
Статистика
Статистический анализ функциональных данных проводят с использованием SPSS 20 (IBM, USA). Для определения наиболее подходящего статистического анализа для сравнения методов лечения проводят тесты нормального распределения. Статистическая значимость определена при p<0,05.
Результаты
На фигуре 4 показано, что введение rBoNT/A(0) уменьшает степень двигательного дефицита, вызванного повреждением заднего столба, на 2 день, по сравнению с контрольным носителем для доз 100 пг и 100 нг. Введение rBoNT/A(0) значительно снижает двигательный дефицит, вызванный повреждением заднего столба, через 4 недели, и скорость восстановления, по сравнению с контрольным носителем при всех протестированных дозах. Кроме того, эффекты более выражены при интратекальном введении rBoNT/A(0), чем при интраспинальном введении (данные не показаны).
В иммуногистохимической оценке используют антитела к нейрофиламенту 200 (NF200) и MAP1b. Нейрофиламент 200 (NF200) экспрессируется в зрелых аксонах, и антитело pMAP1b выявляет нейрофиламенты в окончаниях активно разрастающихся аксонов, показывая, что аксоны все еще активно разрастаются вокруг и внутри участка поражения.
На фигуре 5A показано, что многие аксоны, окрашенные NF200, видны вокруг участка поражения у животных, получавших носитель, при этом небольшое количество NF200+ аксонов присутствует в центре участка поражения у не леченных животных. Напротив, многие аксоны, окрашенные NF200, были видны вокруг участка поражения у животных, получавших rBoNT/A(0), при этом многочисленные NF200+ аксоны также были видны в центре участка поражения.
На фигуре 5B показано, что умеренное количество прорастающих аксонов, окрашенных MAP1b, видно вокруг участка поражения у животных, получавших носитель, при этом небольшое количество MAP1b аксонов присутствует в центре участка поражения. Напротив, окрашивание MAP1b выявило ярко выраженное прорастание аксонов вокруг места поражения, а также разветвление из сердцевины участка поражения у животных, получавших rBoNT/A(0).
Быстрота начала улучшения показателей в функциональном тесте показывает, что rBoNT/A(0) вызывает прорастание аксонов с образованием полезных функциональных синапсов под очагом поражения. Количественная иммуногистохимия представляет доказательство индуцированного BoNT ярко выраженного прорастания аксонов локально, через участок поражения SDC.
Эти данные in vivo являются четкими доказательствами, подтверждающими роль rBoNT/A(0) в лечении неврологических нарушений.
ПРИМЕР 5
Влияние полноразмерных каталитически неактивных рекомбинантных BoNT, фрагментов и вариантов BoNT на количество нейритов в клетке
Ряд полноразмерных каталитически неактивных рекомбинантных серотипов BoNT, а также фрагменты и варианты BoNT тестируют на их модулирующее действие на рост нейритов in vitro.
Материалы и методы
Клетки, подвергшиеся воздействию полипептидов, сравнивают с клетками, подвергшимися воздействию положительного контроля (1 нг/мл BDNF). Гибридные мотонейрон-подобные клетки мыши (NSC34) дифференцируют и подвергают воздействию в течение 4 дней in vitro (DIV) различными полипептидами в 3 различных дозах (0,1 нМ, 1 нМ и 10 нМ).
Клетки NSC34 получают путем слияния обогащенных мотонейронами эмбриональных клеток спинного мозга мыши и нейробластомы мыши (Cashman et al. Dev Dyn. 1992 Jul;194(3):209-21, которая включена в настоящее описание посредством ссылки). Указанные клетки имитируют многие свойства мотонейронов, включая холинацетилтрансферазу, синтез, хранение и высвобождение ацетилхолина и триплетных белков нейрофиламента. Кроме того, мотонейроны спинного мозга NSC34 экспрессируют белки рецептора глутамата и создают потенциалы действия. Нейроны NSC34 широко используют для изучения механизмов передачи сигналов нейронов и дегенерации нейронов.
Принимают следующую экспериментальную схему: Скрининг на клеточной линии нейронов (NSC34):
Клетки NSC34 культивируют на покровных стеклах, покрытых поли-D-лизином, в среде DMEM плюс 10% FCS.
После посева клетки дифференцируют в мотонейроны при обработке ретиноевой кислотой и низких уровнях в сыворотке в течение 4 дней. Клетки культивируют в присутствии/отсутствии полипептидов в определенный момент времени (т.е. 4 DIV). Данные испытаний сравнивают с эффектами, наблюдаемыми на данных положительного (BDNF), а также отрицательного (BSA) контроля.
Через 4 дня in vitro (DIV) клетки фиксируют в 4% параформальдегиде, окрашивают специфическими маркерами нейронов (бета-тубулином) и количественно анализируют на наличие отростков нейритов (удлинение нейритов, удлинение аксонов, разветвление). Получение изображений проводят с использованием микроскопа Operetta CLS HCS (PerkinElmer) с 20x объективом. На каждую лунку получают шесть (6) полей зрения. Проводят анализ роста нейритов и оценивают среднее количество нейритов на клетку.
Результаты
Фигуры 6-10 представляют среднее значение количества нейритов, подсчитанных на каждой клетке, оцененное в трех независимых экспериментальных сессиях. Данные нормализуют на необработанных контрольных клетках. Полипептиды статистически значимо увеличивают количество нейритов на клетку по сравнению с BSA.
Для BoNT/A, фрагмент LHN/A (легкая цепь плюс домен транслокации) обладает улучшенной активностью по сравнению с фрагментом клеточно-связывающего домена (HC домена) (см. фигуру 6).
Как для BoNT/FA, так и для BoNT/F, фрагменты LHN и LC (только легкая цепь) показывают улучшенную активность по сравнению с фрагментами HC домена (см. фигуры 7 и 8).
Наконец, было показано, что все вариантные фрагменты HC домена обладают высокой эффективностью (фигуры 9 и 10), и катионный домен HC/A (SEQ ID NO: 42 - фигура 9) демонстрирует исключительную активность, которая при 2 из 3 концентраций является улучшенной по сравнению с BDNF. Ожидается, что высокая активность катионного домена HC/A также будет очевидна в полноразмерных полипептидах, содержащих указанный домен (каталитически неактивный или активный).
Все публикации, упомянутые в вышеприведенном описании, включены в настоящее описание посредством ссылки. Различные модификации и вариации описанных способов и систем по настоящему изобретению будут очевидны специалистам в данной области техники без отклонения от объема и сущности настоящего изобретения. Хотя настоящее изобретение было описано в связи с конкретными предпочтительными вариантами осуществления, следует понимать, что заявленное изобретение не должно быть ненадлежащим образом ограничено такими конкретными вариантами осуществления. Действительно, различные модификации описанных способов осуществления изобретения, которые очевидны для специалистов в области биохимии и биотехнологии или в смежных областях, как предполагается, входят в объем следующей формулы изобретения.
ЧАСТИ ФОРМУЛЫ ИЗОБРЕТЕНИЯ
1. Полипептид для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит:
легкую цепь клостридиального нейротоксина (L-цепь) или ее фрагмент; и/или
фрагмент тяжелой цепи клостридиального нейротоксина (H-цепь).
2. Способ стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, включающий введение субъекту полипептида, где полипептид содержит:
L-цепь клостридиального нейротоксина или ее фрагмент; и/или
фрагмент Н-цепь клостридиального нейротоксина.
3. Применение полипептида в производстве лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит:
L-цепь клостридиального нейротоксина или ее фрагмент; и/или
фрагмент Н-цепи клостридиального нейротоксина.
4. Полипептид для применения по пункту 1, способ по пункту 2 или применение по пункту 3, отличающийся тем, что L-цепь является каталитически неактивной.
5. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид состоит по существу из легкой цепи клостридиального нейротоксина (L-цепь) или ее фрагмента; и/или фрагмента тяжелой цепи клостридиального нейротоксина (H -цепь).
6. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид состоит из легкой цепи клостридиального нейротоксина (L-цепь) или ее фрагмента; и/или фрагмента тяжелой цепи клостридиального нейротоксина (H-цепь).
7. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что фрагмент Н-цепи клостридиального нейротоксина содержит: домен транслокации (HN) или его фрагмент; или рецептор-связывающий домен клостридиального нейротоксина (HC) или его фрагмент.
8. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что фрагмент Н-цепи клостридиального нейротоксина содержит HN домен или его фрагмент.
9. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что фрагмент Н-цепи клостридиального нейротоксина состоит из HN домена или его фрагмента.
10. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что фрагмент Н-цепи клостридиального нейротоксина содержит HC домен или его фрагмент.
11. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что фрагмент Н-цепи клостридиального нейротоксина состоит из HC домена или его фрагмента.
12. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что в полипептиде отсутствует С-концевая часть рецептор-связывающего домена клостридиального нейротоксина (HCC).
13. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что указанный полипептид не содержит HN домен и HC домен клостридиального нейротоксина.
14. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид дополнительно не содержит не клостридиальный каталитический домен.
15. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид содержит: L-цепь клостридиального нейротоксина или ее фрагмент и HN домен или его фрагмент.
16. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид состоит из: L-цепи клостридиального нейротоксина или ее фрагмента и HN домена или его фрагмента.
17. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид состоит из L-цепи клостридиального нейротоксина и HN домена.
18. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 19, 21, 23, 27, 29, 31, 35, 37, 39, 41, 43, 45, 47 или 49; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 4, 6, 8, 20, 22, 24, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48 или 50.
19. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 19, 21, 23, 27, 29, 31, 35, 37, 39, 41, 43, 45, 47 или 49; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 80% идентичность последовательности к любой из SEQ ID NO: 4, 6, 8, 20, 22, 24, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48 или 50.
20. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 90% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 19, 21, 23, 27, 29, 31, 35, 37, 39, 41, 43, 45, 47 или 49; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 90% идентичность последовательности к любой из SEQ ID NO: 4, 6, 8, 20, 22, 24, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48 или 50.
21. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 95% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 19, 21, 23, 27, 29, 31, 35, 37, 39, 41, 43, 45, 47 или 49; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 95% идентичность последовательности к любой из SEQ ID NO: 4, 6, 8, 20, 22, 24, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48 или 50.
22. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 19, 21, 23, 27, 29, 31, 35, 37, 39, 41, 43, 45, 47 или 49; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, по меньшей мере 99% идентичность последовательности к любой из SEQ ID NO: 4, 6, 8, 20, 22, 24, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48 или 50.
23. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99,9% идентичность последовательности к любой из SEQ ID NO: 3, 5, 7, 19, 21, 23, 27, 29, 31, 35, 37, 39, 41, 43, 45, 47 или 49; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99,9% идентичность последовательности к любой из SEQ ID NO: 4, 6, 8, 20, 22, 24, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48 или 50.
24. Полипептид для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит каталитически неактивную L-цепь клостридиального нейротоксина.
25. Способ стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, включающий введение субъекту полипептида, где полипептид содержит каталитически неактивную L-цепь клостридиального нейротоксина.
26. Применение полипептида, содержащего каталитически неактивную L-цепь клостридиального нейротоксина, в производстве лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта.
27. Полипептид для применения в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
28. Способ стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, включающий введение субъекту полипептида, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42 и/или где полипептид содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
29. Применение полипептида в производстве лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 42, и/или где полипептид содержит полипептидную последовательность, кодируемую нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 41.
30. Полипептид для применения, способ или применение по любому из пунктов 27-29, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 80% идентичность последовательности к SEQ ID NO: 42, и/или где указанный полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80% идентичность последовательности к SEQ ID NO: 41.
31. Полипептид для применения, способ или применение по любому из пунктов 27-30, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 90% идентичность последовательности к SEQ ID NO: 42, и/или где указанный полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 90% идентичность последовательности к SEQ ID NO: 41.
32. Полипептид для применения, способ или применение по любому из пунктов 27-31, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 95% идентичность последовательности к SEQ ID NO: 42, и/или где указанный полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 95% идентичность последовательности к SEQ ID NO: 41.
33. Полипептид для применения, способ или применение по любому из пунктов 27-32, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99% идентичность последовательности к SEQ ID NO: 42, и/или где указанный полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% идентичность последовательности к SEQ ID NO: 41.
34. Полипептид для применения, способ или применение по любому из пунктов 27-33, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99,9% идентичность последовательности к SEQ ID NO: 42, и/или где указанный полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99,9% идентичность последовательности к SEQ ID NO: 41.
35. Полипептид для применения, способ или применение по любому из пунктов 27-34, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 61 или 65, и/или где полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к SEQ ID NO: 60.
36. Полипептид для применения, способ или применение по любому из пунктов 27-35, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 80% идентичность последовательности к SEQ ID NO: 61 или 65, и/или где полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80% идентичность последовательности к SEQ ID NO: 60.
37. Полипептид для применения, способ или применение по любому из пунктов 27-36, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 90% идентичность последовательности к SEQ ID NO: 61 или 65, и/или где полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 90% идентичность последовательности к SEQ ID NO: 60.
38. Полипептид для применения, способ или применение по любому из пунктов 27-37, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 95% идентичность последовательности к SEQ ID NO: 61 или 65, и/или где полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 95% идентичность последовательности к SEQ ID NO: 60.
39. Полипептид для применения, способ или применение по любому из пунктов 27-38, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99% идентичность последовательности к SEQ ID NO: 61 или 65, и/или где полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% идентичность последовательности к SEQ ID NO: 60.
40. Полипептид для применения, способ или применение по любому из пунктов 27-39, отличающийся тем, что указанный полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99,9% идентичность последовательности к SEQ ID NO: 61 или 65, и/или где полипептид кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99,9% идентичность последовательности к SEQ ID NO: 60.
41. Полипептид для применения для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
42. Способ стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, включающий введение полипептида субъекту, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
43. Применение полипептида в получении лекарственного средства для стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к SEQ ID NO: 63 или 64.
44. Полипептид для применения, способ или применение по любому из пунктов 41-43, отличающийся тем, что полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 80% идентичность последовательности к SEQ ID NO: 63 или 64.
45. Полипептид для применения, способ или применение по любому из пунктов 41-44, отличающийся тем, что полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 90% идентичность последовательности к SEQ ID NO: 63 или 64.
46. Полипептид для применения, способ или применение по любому из пунктов 41-45, отличающийся тем, что полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 95% идентичность последовательности к SEQ ID NO: 63 или 64.
47. Полипептид для применения, способ или применение по любому из пунктов 41-46, отличающийся тем, что полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99% идентичность последовательности к SEQ ID NO: 63 или 64.
48. Полипептид для применения, способ или применение по любому из пунктов 41-47, отличающийся тем, что полипептид содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99,9% идентичность последовательности к SEQ ID NO: 63 или 64.
49. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что указанный полипептид не содержит Н-цепь нативного клостридиального нейротоксина.
50. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид является нейротрофическим.
51. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где полипептид способствует росту и/или восстановлению нейронов.
52. Полипептид для применения, способа или применения по любому из предыдущих пунктов, отличающийся тем, что неврологическое нарушение представляет собой нарушение, которое можно лечить путем стимуляции роста и/или восстановления нейронов.
53. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное нарушение, сенсорное нарушение или вегетативное нарушение.
54. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где неврологическое нарушение представляет собой повреждение нейронов, выбранное из: травмы нерва (например, в результате рубцевания и/или перелома кости), невропатии (например, периферической невропатии), повреждения спинного мозга (например, включая паралич), иссечения нерва, повреждения головного мозга (например, черепно-мозговой травмы), не травматического повреждения (например, инсульта или инфаркта спинного мозга) и повреждения плечевого сплетения, например паралича Эрба или паралича Клюмпке.
55. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где неврологическое нарушение представляет собой нейродегенеративное нарушение, выбранное из: болезни Альцгеймера, болезни Паркинсона, нарушений, связанных с болезнью Паркинсона, заболевания мотонейронов, периферической невропатии, моторной невропатии, прионовой болезни, болезни Хантингтона, спиноцеребеллярной атаксии, спинальной мышечной атрофии, амиотрофии одной конечности, атаксии Фридрейха, болезни Халлервордена-Шпатца и лобно-височной лобулярной дегенерации.
56. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где полипептид способствует росту или восстановлению мотонейрона.
57. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где полипептид представляет собой модифицированный клостридиальный нейротоксин, такой как химерный клостридиальный нейротоксин или гибридный клостридиальный нейротоксин.
58. Полипептид для применения, способ или применение по любому из пунктов 24-34 или 49-57, отличающийся тем, что полипептид является каталитически неактивным, и:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
59. Полипептид для применения, способ или применение по любому из пунктов 24-34 или 49-58, отличающийся тем, что полипептид является каталитически неактивным, и:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80% идентичность последовательности к любой из SEQ ID NO: 11, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 80% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
60. Полипептид для применения, способ или применение по любому из пунктов 24-34 или 49-59, отличающийся тем, что полипептид является каталитически неактивным, и:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 90% идентичность последовательности к любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 90% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
61. Полипептид для применения, способ или применение по любому из пунктов 24-34 или 49-60, отличающийся тем, что полипептид является каталитически неактивным, и:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 95% идентичность последовательности к любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
b. содержит (предпочтительно состоит из) полипептидную последовательность, имеющую по меньшей мере 95% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
62. Полипептид для применения, способ или применение по любому из пунктов 24-34 или 49-61, отличающийся тем, что полипептид является каталитически неактивным, и:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% идентичность последовательности к любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
b. содержит (предпочтительно состоит из) полипептидную последовательность, имеющую по меньшей мере 99% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
63. Полипептид для применения, способ или применение по любому из пунктов 24-34 или 49-62, отличающийся тем, что полипептид является каталитически неактивным, и:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99,9% идентичность последовательности к любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99,9% идентичность последовательности к любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62, 63, 64 или 65.
64. Полипептид для применения, способ или применение по любому из пунктов 24-26 или 49-63, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25 или 33; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 70% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65.
65. Полипептид для применения, способ или применение по любому из пунктов 24-26 или 49-64, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25 или 33; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 80% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65.
66. Полипептид для применения, способ или применение по любому из пунктов 24-26 или 49-65, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 90% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25 или 33; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 90% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65.
67. Полипептид для применения, способ или применение по любому из пунктов 24-26 или 49-66, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 95% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25 или 33; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 95% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65.
68. Полипептид для применения, способ или применение по любому из пунктов 24-26 или 49-67, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25 или 33; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65.
69. Полипептид для применения, способ или применение по любому из пунктов 24-26 или 49-68, отличающийся тем, что полипептид:
а. кодируется нуклеотидной последовательностью, имеющей по меньшей мере 99,9% идентичность последовательности к любой из SEQ ID NO: 1, 9, 11, 13, 15, 17, 25 или 33; или
b. содержит (предпочтительно, состоит из) полипептидную последовательность, имеющую по меньшей мере 99,9% идентичность последовательности к любой из SEQ ID NO: 2, 10, 12, 14, 16, 18, 26, 34, 64 или 65.
70. Полипептид для применения, способ или применение в соответствии с любым из предыдущих пунктов, где полипептид предпочтительно вводят в или рядом с местом повреждения, предпочтительно, где полипептид вводят интратекально.
71. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где полипептид дополнительно не содержит домен, который связывается с клеточным рецептором.
72. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что в полипептиде отсутствует функциональный HC домен клостридиального нейротоксина, а также отсутствует какой-либо функционально эквивалентная таргетная группа (TM) экзогенного лиганда.
73. Полипептид для применения, способ или применение по любому из предыдущих пунктов, где полипептид не экспрессируется в клетке субъекта.
74. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что клостридиальные последовательности полипептида состоят из легкой цепи клостридиального нейротоксина (L-цепи) или ее фрагмента; и/или фрагмента тяжелой цепи клостридиального нейротоксина (H-цепи).
75. Полипептид для применения, способ или применение по любому из предыдущих пунктов, отличающийся тем, что полипептид дополнительно содержит одну или более последовательностей не клостридиального нейротоксина.
76. Полипептид для применения, способ или применение по пункту 75, отличающийся тем, что одна или более последовательностей не клостридиального нейротоксина не связываются с клеточным рецептором.
77. Полипептид для применения, способ или применение по пункту 75 или 76, отличающийся тем, что одна или более последовательностей не клостридиального нейротоксина не содержат лиганд для клеточного рецептора.
78. Полипептид для применения, способ или применение по любому из пунктов 1-40 или 49-77, отличающийся тем, что полипептид представляет собой модифицированный BoNT/A или его фрагмент, содержащий модификацию одного или более аминокислотных остатков, выбранных из: ASN 886, ASN 905, GLN 915, ASN 918, GLU 920, ASN 930, ASN 954, SER 955, GLN 991, GLU 992, GLN 995, ASN 1006, ASN 1025, ASN 1026, ASN 1032, ASN 1043, ASN 1046, ASN 1052, ASP 1058, HIS 1064, ASN 1080, GLU 1081, GLU 1083, ASP 1086, ASN 1188, ASP 1213, GLY 1215, ASN 1216, GLN 1229, ASN 1242, ASN 1243, SER 1274 и THR 1277, где модификация выбрана из:
i. замены экспонированного на поверхности кислого аминокислотного остатка на основной аминокислотный остаток;
ii. замены экспонированного на поверхности кислого аминокислотного остатка на незаряженный аминокислотный остаток;
iii. замены экспонированного на поверхности незаряженного аминокислотного остатка на основной аминокислотный остаток;
iv. вставки основного аминокислотного остатка; и
v. делеции экспонированного на поверхности кислого аминокислотного остатка.
79. Полипептид для применения, способ или применение по любому из пунктов 1-26 или 41-77, отличающийся тем, что полипептид представляет собой химерный BoNT, содержащий легкую цепь BoNT/A и домен транслокации, и рецептор-связывающий домен BoNT/B (HC домен).
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> IPSEN BIOPHARM LIMITED
<120> НЕ ТОКСИЧЕСКИЕ ПОЛИПЕПТИДЫ КЛОСТРИДИАЛЬНОГО НЕЙРОТОКСИНА ДЛЯ
ПРИМЕНЕНИЯ В ЛЕЧЕНИИ НЕВРОЛОГИЧЕСКИХ НАРУШЕНИЙ
<130> P60073WO
<150> GB1914034.2
<151> 2019-09-30
<160> 76
<170> PatentIn version 3.5
<210> 1
<211> 3888
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/A(0)
<400> 1
atgccattcg tcaacaagca attcaactac aaagacccag tcaacggcgt cgacatcgca 60
tacatcaaga ttccgaacgc cggtcaaatg cagccggtta aggcttttaa gatccacaac 120
aagatttggg ttatcccgga gcgtgacacc ttcacgaacc cggaagaagg cgatctgaac 180
ccgccaccgg aagcgaagca agtccctgtc agctactacg attcgacgta cctgagcacg 240
gataacgaaa aagataacta cctgaaaggt gtgaccaagc tgttcgaacg tatctacagc 300
acggatctgg gtcgcatgct gctgactagc attgttcgcg gtatcccgtt ctggggtggt 360
agcacgattg acaccgaact gaaggttatc gacactaact gcattaacgt tattcaaccg 420
gatggtagct atcgtagcga agagctgaat ctggtcatca ttggcccgag cgcagacatt 480
atccaattcg agtgcaagag ctttggtcac gaggttctga atctgacccg caatggctat 540
ggtagcaccc agtacattcg tttttcgccg gattttacct tcggctttga agagagcctg 600
gaggttgata ccaatccgtt gctgggtgcg ggcaaattcg ctaccgatcc ggctgtcacg 660
ctggcccatc aactgatcta cgcaggccac cgcctgtacg gcattgccat caacccaaac 720
cgtgtgttca aggttaatac gaatgcatac tacgagatga gcggcctgga agtcagcttc 780
gaagaactgc gcaccttcgg tggccatgac gctaaattca ttgacagctt gcaagagaat 840
gagttccgtc tgtactacta taacaaattc aaagacattg caagcacgtt gaacaaggcc 900
aaaagcatcg ttggtactac cgcgtcgttg cagtatatga agaatgtgtt taaagagaag 960
tacctgctgt ccgaggatac ctccggcaag tttagcgttg ataagctgaa gtttgacaaa 1020
ctgtacaaga tgctgaccga gatttacacc gaggacaact ttgtgaaatt cttcaaagtg 1080
ttgaatcgta aaacctatct gaattttgac aaagcggttt tcaagattaa catcgtgccg 1140
aaggtgaact acaccatcta tgacggtttt aacctgcgta acaccaacct ggcggcgaac 1200
tttaacggtc agaatacgga aatcaacaac atgaatttca cgaagttgaa gaacttcacg 1260
ggtctgttcg agttctataa gctgctgtgc gtgcgcggta tcatcaccag caaaaccaaa 1320
agcctggaca aaggctacaa caaggcgctg aatgacctgt gcattaaggt aaacaattgg 1380
gatctgttct tttcgccatc cgaagataat tttaccaacg acctgaacaa gggtgaagaa 1440
atcaccagcg atacgaatat tgaagcagcg gaagagaata tcagcctgga tctgatccag 1500
cagtactatc tgacctttaa cttcgacaat gaaccggaga acattagcat tgagaatctg 1560
agcagcgaca ttatcggtca gctggaactg atgccgaata tcgaacgttt cccgaacggc 1620
aaaaagtacg agctggacaa gtacactatg ttccattacc tgcgtgcaca ggagtttgaa 1680
cacggtaaaa gccgtatcgc gctgaccaac agcgttaacg aggccctgct gaacccgagc 1740
cgtgtctata ccttcttcag cagcgactat gttaagaaag tgaacaaagc cactgaggcc 1800
gcgatgttcc tgggctgggt ggaacagctg gtatatgact tcacggacga gacgagcgaa 1860
gtgagcacta ccgacaaaat tgctgatatt accatcatta tcccgtatat tggtccggca 1920
ctgaacattg gcaacatgct gtacaaagac gattttgtgg gtgccctgat cttctccggt 1980
gccgtgattc tgctggagtt cattccggag attgcgatcc cggtgttggg taccttcgcg 2040
ctggtgtcct acatcgcgaa taaggttctg acggttcaga ccatcgataa cgcgctgtcg 2100
aaacgtaatg aaaaatggga cgaggtttac aaatacattg ttacgaattg gctggcgaaa 2160
gtcaataccc agatcgacct gatccgtaag aaaatgaaag aggcgctgga gaatcaggcg 2220
gaggccacca aagcaattat caactaccaa tacaaccagt acacggaaga agagaagaat 2280
aacattaact tcaatatcga tgatttgagc agcaagctga atgaatctat caacaaagcg 2340
atgatcaata tcaacaagtt tttgaatcag tgtagcgttt cgtacctgat gaatagcatg 2400
attccgtatg gcgtcaaacg tctggaggac ttcgacgcca gcctgaaaga tgcgttgctg 2460
aaatacattt acgacaatcg tggtacgctg attggccaag ttgaccgctt gaaagacaaa 2520
gttaacaata ccctgagcac cgacatccca tttcaactga gcaagtatgt tgataatcaa 2580
cgtctgttga gcactttcac cgagtatatc aaaaacatca tcaatactag cattctgaac 2640
ctgcgttacg agagcaatca tctgattgat ctgagccgtt atgcaagcaa gatcaacatc 2700
ggtagcaagg tcaattttga cccgatcgat aagaaccaga tccagctgtt taatctggaa 2760
tcgagcaaaa ttgaggttat cctgaaaaac gccattgtct acaactccat gtacgagaat 2820
ttctccacca gcttctggat tcgcatcccg aaatacttca acagcattag cctgaacaac 2880
gagtatacta tcatcaactg tatggagaac aacagcggtt ggaaggtgtc tctgaactat 2940
ggtgagatca tttggacctt gcaggacacc caagagatca agcagcgcgt cgtgttcaag 3000
tactctcaaa tgatcaacat ttccgattac attaatcgtt ggatcttcgt gaccattacg 3060
aataaccgtc tgaataacag caagatttac atcaatggtc gcttgatcga tcagaaaccg 3120
attagcaacc tgggtaatat ccacgcaagc aacaacatta tgttcaaatt ggacggttgc 3180
cgcgataccc atcgttatat ctggatcaag tatttcaacc tgtttgataa agaactgaat 3240
gagaaggaga tcaaagattt gtatgacaac caatctaaca gcggcatttt gaaggacttc 3300
tggggcgatt atctgcaata cgataagccg tactatatgc tgaacctgta tgatccgaac 3360
aaatatgtgg atgtcaataa tgtgggtatt cgtggttaca tgtatttgaa gggtccgcgt 3420
ggcagcgtta tgacgaccaa catttacctg aactctagcc tgtaccgtgg tacgaaattc 3480
atcattaaga aatatgccag cggcaacaaa gataacattg tgcgtaataa cgatcgtgtc 3540
tacatcaacg tggtcgtgaa gaataaagag taccgtctgg cgaccaacgc ttcgcaggcg 3600
ggtgttgaga aaattctgag cgcgttggag atccctgatg tcggtaatct gagccaagtc 3660
gtggttatga agagcaagaa cgaccagggt atcactaaca agtgcaagat gaacctgcaa 3720
gacaacaatg gtaacgacat cggctttatt ggtttccacc agttcaacaa tattgctaaa 3780
ctggtagcga gcaattggta caatcgtcag attgagcgca gcagccgtac tttgggctgt 3840
agctgggagt ttatcccggt cgatgatggt tggggcgaac gtccgctg 3888
<210> 2
<211> 1296
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/A(0)
<400> 2
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Gln
210 215 220
Leu Ile Tyr Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Ile Asn Thr Ser Ile Leu Asn
865 870 875 880
Leu Arg Tyr Glu Ser Asn His Leu Ile Asp Leu Ser Arg Tyr Ala Ser
885 890 895
Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys Asn
900 905 910
Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu
915 920 925
Lys Asn Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser
930 935 940
Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Ser Ile Ser Leu Asn Asn
945 950 955 960
Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys Val
965 970 975
Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Gln Glu
980 985 990
Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile Ser
995 1000 1005
Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
1010 1015 1020
Leu Asn Asn Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln
1025 1030 1035
Lys Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Asn Ile
1040 1045 1050
Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp
1055 1060 1065
Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu
1070 1075 1080
Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met
1100 1105 1110
Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
1115 1120 1125
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
1130 1135 1140
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr
1145 1150 1155
Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile
1160 1165 1170
Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn
1175 1180 1185
Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
1190 1195 1200
Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser
1205 1210 1215
Gln Val Val Val Met Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn
1220 1225 1230
Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly
1235 1240 1245
Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
1250 1255 1260
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu
1265 1270 1275
Gly Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu
1280 1285 1290
Arg Pro Leu
1295
<210> 3
<211> 2634
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLHN/A
<400> 3
atggagttcg ttaacaaaca gttcaactat aaagacccag ttaacggtgt tgacattgct 60
tacatcaaaa tcccgaacgc tggccagatg cagccggtaa aggcattcaa aatccacaac 120
aaaatctggg ttatcccgga acgtgatacc tttactaacc cggaagaagg tgacctgaac 180
ccgccaccgg aagcgaaaca ggtgccggta tcttactatg actccaccta cctgtctacc 240
gataacgaaa aggacaacta cctgaaaggt gttactaaac tgttcgagcg tatttactcc 300
accgacctgg gccgtatgct gctgactagc atcgttcgcg gtatcccgtt ctggggcggt 360
tctaccatcg ataccgaact gaaagtaatc gacactaact gcatcaacgt tattcagccg 420
gacggttcct atcgttccga agaactgaac ctggtgatca tcggcccgtc tgctgatatc 480
atccagttcg agtgtaagag ctttggtcac gaagttctga acctcacccg taacggctac 540
ggttccactc agtacatccg tttctctccg gacttcacct tcggttttga agaatccctg 600
gaagtagaca cgaacccact gctgggcgct ggtaaattcg caactgatcc tgcggttacc 660
ctggctcacg aactgattca tgcaggccac cgcctgtacg gtatcgccat caatccgaac 720
cgtgtcttca aagttaacac caacgcgtat tacgagatgt ccggtctgga agttagcttc 780
gaagaactgc gtacttttgg cggtcacgac gctaaattca tcgactctct gcaagaaaac 840
gagttccgtc tgtactacta taacaagttc aaagatatcg catccaccct gaacaaagcg 900
aaatccatcg tgggtaccac tgcttctctc cagtacatga agaacgtttt taaagaaaaa 960
tacctgctca gcgaagacac ctccggcaaa ttctctgtag acaagttgaa attcgataaa 1020
ctttacaaaa tgctgactga aatttacacc gaagacaact tcgttaagtt ctttaaagtt 1080
ctgaaccgca aaacctatct gaacttcgac aaggcagtat tcaaaatcaa catcgtgccg 1140
aaagttaact acactatcta cgatggtttc aacctgcgta acaccaacct ggctgctaat 1200
tttaacggcc agaacacgga aatcaacaac atgaacttca caaaactgaa aaacttcact 1260
ggtctgttcg agttttacaa gctgctgtgc gtcgacggca tcattacctc caaaactaaa 1320
tctgacgatg acgataaaaa caaagcgctg aacctgcagt gtatcaaggt taacaactgg 1380
gatttattct tcagcccgag tgaagacaac ttcaccaacg acctgaacaa aggtgaagaa 1440
atcacctcag atactaacat cgaagcagcc gaagaaaaca tctcgctgga cctgatccag 1500
cagtactacc tgacctttaa tttcgacaac gagccggaaa acatttctat cgaaaacctg 1560
agctctgata tcatcggcca gctggaactg atgccgaaca tcgaacgttt cccaaacggt 1620
aaaaagtacg agctggacaa atataccatg ttccactacc tgcgcgcgca ggaatttgaa 1680
cacggcaaat cccgtatcgc actgactaac tccgttaacg aagctctgct caacccgtcc 1740
cgtgtataca ccttcttctc tagcgactac gtgaaaaagg tcaacaaagc gactgaagct 1800
gcaatgttct tgggttgggt tgaacagctt gtttatgatt ttaccgacga gacgtccgaa 1860
gtatctacta ccgacaaaat tgcggatatc actatcatca tcccgtacat cggtccggct 1920
ctgaacattg gcaacatgct gtacaaagac gacttcgttg gcgcactgat cttctccggt 1980
gcggtgatcc tgctggagtt catcccggaa atcgccatcc cggtactggg cacctttgct 2040
ctggtttctt acattgcaaa caaggttctg actgtacaaa ccatcgacaa cgcgctgagc 2100
aaacgtaacg aaaaatggga tgaagtttac aaatatatcg tgaccaactg gctggctaag 2160
gttaatactc agatcgacct catccgcaaa aaaatgaaag aagcactgga aaaccaggcg 2220
gaagctacca aggcaatcat taactaccag tacaaccagt acaccgagga agaaaaaaac 2280
aacatcaact tcaacatcga cgatctgtcc tctaaactga acgaatccat caacaaagct 2340
atgatcaaca tcaacaagtt cctgaaccag tgctctgtaa gctatctgat gaactccatg 2400
atcccgtacg gtgttaaacg tctggaggac ttcgatgcgt ctctgaaaga cgccctgctg 2460
aaatacattt acgacaaccg tggcactctg atcggtcagg ttgatcgtct gaaggacaaa 2520
gtgaacaata ccttatcgac cgacatccct tttcagctca gtaaatatgt cgataaccaa 2580
cgccttttgt ccactctaga agcacaccat catcaccacc atcaccatca ccat 2634
<210> 4
<211> 878
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLHN/A
<400> 4
Met Glu Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Asp
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Asp Asp Asp Asp Lys Asn Lys
435 440 445
Ala Leu Asn Leu Gln Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Leu Glu Ala His His His His His His His His His His
865 870 875
<210> 5
<211> 1329
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rL/A
<400> 5
atgccattcg tcaacaagca attcaactac aaagacccag tcaacggcgt cgacatcgca 60
tacatcaaga ttccgaacgc cggtcaaatg cagccggtta aggcttttaa gatccacaac 120
aagatttggg ttatcccgga gcgtgacacc ttcacgaacc cggaagaagg cgatctgaac 180
ccgccaccgg aagcgaagca agtccctgtc agctactacg attcgacgta cctgagcacg 240
gataacgaaa aagataacta cctgaaaggt gtgaccaagc tgttcgaacg tatctacagc 300
acggatctgg gtcgcatgct gctgactagc attgttcgcg gtatcccgtt ctggggtggt 360
agcacgattg acaccgaact gaaggttatc gacactaact gcattaacgt tattcaaccg 420
gatggtagct atcgtagcga agagctgaat ctggtcatca ttggcccgag cgcagacatt 480
atccaattcg agtgcaagag ctttggtcac gaggttctga atctgacccg caatggctat 540
ggtagcaccc agtacattcg tttttcgccg gattttacct tcggctttga agagagcctg 600
gaggttgata ccaatccgtt gctgggtgcg ggcaaattcg ctaccgatcc ggctgtcacg 660
ctggcccatg aactgatcca cgcaggccac cgcctgtacg gcattgccat caacccaaac 720
cgtgtgttca aggttaatac gaatgcatac tacgagatga gcggcctgga agtcagcttc 780
gaagaactgc gcaccttcgg tggccatgac gctaaattca ttgacagctt gcaagagaat 840
gagttccgtc tgtactacta taacaaattc aaagacattg caagcacgtt gaacaaggcc 900
aaaagcatcg ttggtactac cgcgtcgttg cagtatatga agaatgtgtt taaagagaag 960
tacctgctgt ccgaggatac ctccggcaag tttagcgttg ataagctgaa gtttgacaaa 1020
ctgtacaaga tgctgaccga gatttacacc gaggacaact ttgtgaaatt cttcaaagtg 1080
ttgaatcgta aaacctatct gaattttgac aaagcggttt tcaagattaa catcgtgccg 1140
aaggtgaact acaccatcta tgacggtttt aacctgcgta acaccaacct ggcggcgaac 1200
tttaacggtc agaatacgga aatcaacaac atgaatttca cgaagttgaa gaacttcacg 1260
ggtctgttcg agttctataa gctgctgggt ctagaagcac accatcatca ccaccatcac 1320
catcaccat 1329
<210> 6
<211> 443
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rL/A
<400> 6
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Gly Leu Glu
420 425 430
Ala His His His His His His His His His His
435 440
<210> 7
<211> 1299
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/A
<400> 7
atgcatcatc accatcacca caaaaacatc atcaatacta gcattctgaa cctgcgttac 60
gagagcaatc atctgattga tctgagccgt tatgcaagca agatcaacat cggtagcaag 120
gtcaattttg acccgatcga taagaaccag atccagctgt ttaatctgga atcgagcaaa 180
attgaggtta tcctgaaaaa cgccattgtc tacaactcca tgtacgagaa tttctccacc 240
agcttctgga ttcgcatccc gaaatacttc aacagcatta gcctgaacaa cgagtatact 300
atcatcaact gtatggagaa caacagcggt tggaaggtgt ctctgaacta tggtgagatc 360
atttggacct tgcaggacac ccaagagatc aagcagcgcg tcgtgttcaa gtactctcaa 420
atgatcaaca tttccgatta cattaatcgt tggatcttcg tgaccattac gaataaccgt 480
ctgaataaca gcaagattta catcaatggt cgcttgatcg atcagaaacc gattagcaac 540
ctgggtaata tccacgcaag caacaacatt atgttcaaat tggacggttg ccgcgatacc 600
catcgttata tctggatcaa gtatttcaac ctgtttgata aagaactgaa tgagaaggag 660
atcaaagatt tgtatgacaa ccaatctaac agcggcattt tgaaggactt ctggggcgat 720
tatctgcaat acgataagcc gtactatatg ctgaacctgt atgatccgaa caaatatgtg 780
gatgtcaata atgtgggtat tcgtggttac atgtatttga agggtccgcg tggcagcgtt 840
atgacgacca acatttacct gaactctagc ctgtaccgtg gtacgaaatt catcattaag 900
aaatatgcca gcggcaacaa agataacatt gtgcgtaata acgatcgtgt ctacatcaac 960
gtggtcgtga agaataaaga gtaccgtctg gcgaccaacg cttcgcaggc gggtgttgag 1020
aaaattctga gcgcgttgga gatccctgat gtcggtaatc tgagccaagt cgtggttatg 1080
aagagcaaga acgaccaggg tatcactaac aagtgcaaga tgaacctgca agacaacaat 1140
ggtaacgaca tcggctttat tggtttccac cagttcaaca atattgctaa actggtagcg 1200
agcaattggt acaatcgtca gattgagcgc agcagccgta ctttgggctg tagctgggag 1260
tttatcccgg tcgatgatgg ttggggcgaa cgtccgctg 1299
<210> 8
<211> 433
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/A
<400> 8
Met His His His His His His Lys Asn Ile Ile Asn Thr Ser Ile Leu
1 5 10 15
Asn Leu Arg Tyr Glu Ser Asn His Leu Ile Asp Leu Ser Arg Tyr Ala
20 25 30
Ser Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys
35 40 45
Asn Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile
50 55 60
Leu Lys Asn Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr
65 70 75 80
Ser Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Ser Ile Ser Leu Asn
85 90 95
Asn Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys
100 105 110
Val Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Gln
115 120 125
Glu Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile
130 135 140
Ser Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
145 150 155 160
Leu Asn Asn Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln Lys
165 170 175
Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Asn Ile Met Phe
180 185 190
Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp Ile Lys Tyr
195 200 205
Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu Ile Lys Asp Leu
210 215 220
Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys Asp Phe Trp Gly Asp
225 230 235 240
Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met Leu Asn Leu Tyr Asp Pro
245 250 255
Asn Lys Tyr Val Asp Val Asn Asn Val Gly Ile Arg Gly Tyr Met Tyr
260 265 270
Leu Lys Gly Pro Arg Gly Ser Val Met Thr Thr Asn Ile Tyr Leu Asn
275 280 285
Ser Ser Leu Tyr Arg Gly Thr Lys Phe Ile Ile Lys Lys Tyr Ala Ser
290 295 300
Gly Asn Lys Asp Asn Ile Val Arg Asn Asn Asp Arg Val Tyr Ile Asn
305 310 315 320
Val Val Val Lys Asn Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln
325 330 335
Ala Gly Val Glu Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly
340 345 350
Asn Leu Ser Gln Val Val Val Met Lys Ser Lys Asn Asp Gln Gly Ile
355 360 365
Thr Asn Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile
370 375 380
Gly Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
385 390 395 400
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu Gly
405 410 415
Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu Arg Pro
420 425 430
Leu
<210> 9
<211> 3873
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/B(0)
<400> 9
atgccggtga cgattaacaa cttcaactac aacgacccga ttgacaacaa caacattatc 60
atgatggaac cgccgtttgc acgcggcacg ggccgttatt acaaagcgtt taaaatcacc 120
gatcgtattt ggattatccc ggaacgctac acgtttggtt ataaaccgga agacttcaac 180
aaaagctctg gcatcttcaa ccgtgatgtt tgcgaatact acgatccgga ctacctgaac 240
accaacgata agaaaaacat ttttctgcaa acgatgatca aactgttcaa tcgcattaaa 300
agcaaaccgc tgggtgaaaa actgctggaa atgattatca atggcattcc gtatctgggt 360
gatcgtcgcg tgccgctgga agaatttaac accaatatcg cgagtgttac ggtcaacaaa 420
ctgatttcca atccgggtga agtcgaacgt aaaaaaggca tcttcgccaa cctgatcatc 480
ttcggcccgg gtccggtgct gaacgaaaat gaaaccattg atatcggtat tcagaaccat 540
tttgcctcac gcgaaggctt cggcggtatt atgcaaatga aattttgccc ggaatatgtg 600
tcggttttca acaatgttca ggaaaacaaa ggtgcaagca tctttaatcg tcgcggctat 660
ttctctgatc cggctctgat cctgatgcac caactgattt atgtgctgca cggcctgtat 720
ggtatcaaag tggatgacct gccgatcgtt ccgaacgaga aaaaattttt catgcagagc 780
accgacgcaa ttcaagctga agaactgtat acgtttggcg gtcaggaccc gtctattatc 840
accccgagca ccgacaaaag catctacgat aaagtgctgc aaaactttcg tggcattgtt 900
gaccgcctga ataaagtcct ggtgtgtatc tctgatccga acatcaacat caacatctac 960
aaaaacaaat tcaaagacaa atacaaattc gttgaagatt ctgaaggcaa atatagtatt 1020
gacgtcgaat cctttgataa actgtacaaa agtctgatgt tcggtttcac cgaaacgaac 1080
atcgcggaaa actacaaaat caaaacccgc gcctcctatt tcagcgactc tctgccgccg 1140
gttaaaatca aaaatctgct ggataacgaa atttatacga tcgaagaagg tttcaacatc 1200
agcgataaag acatggaaaa agaataccgt ggccagaata aagcaatcaa caaacaggcg 1260
tatgaagaaa ttagtaaaga acatctggcg gtctacaaaa ttcagatgtg caaatccgtg 1320
aaagccccgg gtatttgtat cgatgttgac aatgaagacc tgtttttcat cgccgataaa 1380
aacagttttt ccgatgacct gtcaaaaaat gaacgcatcg aatacaacac ccaatcgaac 1440
tacatcgaaa acgatttccc gatcaacgaa ctgattctgg atacggacct gattagtaaa 1500
atcgaactgc cgtcagaaaa caccgaatcg ctgacggact ttaatgttga tgtcccggtg 1560
tatgaaaaac agccggcaat taagaaaatt tttaccgatg aaaacacgat cttccagtac 1620
ctgtacagcc aaacctttcc gctggacatt cgcgatatct ctctgacgag ttcctttgat 1680
gacgcactgc tgttcagcaa caaagtgtac tcctttttct caatggatta catcaaaacc 1740
gctaacaaag tggttgaagc gggcctgttt gccggttggg tgaaacagat cgttaacgat 1800
ttcgtcatcg aagccaacaa aagtaacacg atggataaaa ttgctgatat ctccctgatt 1860
gtcccgtata ttggcctggc actgaatgtg ggtaacgaaa cggcgaaagg caattttgaa 1920
aacgccttcg aaattgcagg cgcttcaatc ctgctggaat ttattccgga actgctgatc 1980
ccggtcgtgg gtgcgttcct gctggaatct tacatcgaca acaaaaacaa aatcatcaaa 2040
accattgata acgcgctgac gaaacgtaac gaaaaatggt cagatatgta cggcctgatt 2100
gttgcccagt ggctgagcac cgtcaacacg caattttaca ccatcaaaga aggtatgtac 2160
aaagcgctga attatcaggc gcaagccctg gaagaaatca tcaaataccg ctacaacatc 2220
tacagcgaaa aagaaaaatc taacatcaac atcgacttta atgatatcaa cagcaaactg 2280
aacgaaggta tcaaccaggc aatcgataac atcaacaact tcatcaacgg ctgctcagtg 2340
tcgtatctga tgaagaaaat gatcccgctg gctgttgaaa aactgctgga ttttgacaac 2400
accctgaaga aaaacctgct gaactacatc gatgaaaaca aactgtacct gatcggctca 2460
gccgaatacg aaaaatcgaa agtgaacaaa tacctgaaaa ccatcatgcc gtttgacctg 2520
agtatttaca ccaacgatac gatcctgatc gaaatgttca acaaatacaa ctccgaaatt 2580
ctgaacaata ttatcctgaa cctgcgttac aaagacaaca atctgatcga tctgagcggc 2640
tatggtgcaa aagttgaagt ctacgacggt gtcgaactga acgataaaaa ccagttcaaa 2700
ctgacctcat cggctaactc aaaaattcgt gtgacgcaga accaaaacat catcttcaac 2760
tcggtctttc tggacttcag cgtgtctttc tggattcgca tcccgaaata taaaaatgat 2820
ggcatccaga actacatcca taacgaatac accatcatca actgtatgaa aaacaacagt 2880
ggttggaaaa tttccatccg tggcaaccgc attatctgga ccctgattga tatcaatggt 2940
aaaacgaaaa gcgtgttttt cgaatacaac atccgtgaag atatctctga atacatcaat 3000
cgctggtttt tcgtgaccat tacgaacaat ctgaacaatg cgaaaatcta tatcaacggc 3060
aaactggaaa gtaataccga catcaaagat attcgtgaag ttatcgccaa cggtgaaatc 3120
atcttcaaac tggatggcga catcgatcgc acccagttca tttggatgaa atacttctcc 3180
atcttcaaca cggaactgag tcagtccaat atcgaagaac gctacaaaat ccaatcatac 3240
tcggaatacc tgaaagattt ctggggtaac ccgctgatgt acaacaaaga atactacatg 3300
ttcaacgcgg gcaacaaaaa ctcatacatc aaactgaaaa aagattcgcc ggtgggtgaa 3360
atcctgaccc gtagcaaata caaccagaac tctaaataca tcaactatcg cgatctgtac 3420
attggcgaaa aatttattat ccgtcgcaaa agcaactctc agagtattaa tgatgacatc 3480
gtgcgtaaag aagactacat ctatctggat ttctttaatc tgaaccaaga atggcgcgtt 3540
tatacctaca aatacttcaa aaaagaagaa gagaaactgt tcctggcccc gattagcgac 3600
agcgatgaat tttacaacac catccagatc aaagaatacg atgaacagcc gacgtatagt 3660
tgccaactgc tgttcaaaaa agacgaagaa tccaccgatg aaattggcct gattggtatc 3720
caccgtttct atgaaagcgg tatcgttttc gaagaataca aagattactt ctgtatctct 3780
aaatggtatc tgaaagaagt caaacgcaaa ccgtacaacc tgaaactggg ctgcaactgg 3840
caatttatcc cgaaagacga aggctggacc gaa 3873
<210> 10
<211> 1291
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/B(0)
<400> 10
Met Pro Val Thr Ile Asn Asn Phe Asn Tyr Asn Asp Pro Ile Asp Asn
1 5 10 15
Asn Asn Ile Ile Met Met Glu Pro Pro Phe Ala Arg Gly Thr Gly Arg
20 25 30
Tyr Tyr Lys Ala Phe Lys Ile Thr Asp Arg Ile Trp Ile Ile Pro Glu
35 40 45
Arg Tyr Thr Phe Gly Tyr Lys Pro Glu Asp Phe Asn Lys Ser Ser Gly
50 55 60
Ile Phe Asn Arg Asp Val Cys Glu Tyr Tyr Asp Pro Asp Tyr Leu Asn
65 70 75 80
Thr Asn Asp Lys Lys Asn Ile Phe Leu Gln Thr Met Ile Lys Leu Phe
85 90 95
Asn Arg Ile Lys Ser Lys Pro Leu Gly Glu Lys Leu Leu Glu Met Ile
100 105 110
Ile Asn Gly Ile Pro Tyr Leu Gly Asp Arg Arg Val Pro Leu Glu Glu
115 120 125
Phe Asn Thr Asn Ile Ala Ser Val Thr Val Asn Lys Leu Ile Ser Asn
130 135 140
Pro Gly Glu Val Glu Arg Lys Lys Gly Ile Phe Ala Asn Leu Ile Ile
145 150 155 160
Phe Gly Pro Gly Pro Val Leu Asn Glu Asn Glu Thr Ile Asp Ile Gly
165 170 175
Ile Gln Asn His Phe Ala Ser Arg Glu Gly Phe Gly Gly Ile Met Gln
180 185 190
Met Lys Phe Cys Pro Glu Tyr Val Ser Val Phe Asn Asn Val Gln Glu
195 200 205
Asn Lys Gly Ala Ser Ile Phe Asn Arg Arg Gly Tyr Phe Ser Asp Pro
210 215 220
Ala Leu Ile Leu Met His Gln Leu Ile Tyr Val Leu His Gly Leu Tyr
225 230 235 240
Gly Ile Lys Val Asp Asp Leu Pro Ile Val Pro Asn Glu Lys Lys Phe
245 250 255
Phe Met Gln Ser Thr Asp Ala Ile Gln Ala Glu Glu Leu Tyr Thr Phe
260 265 270
Gly Gly Gln Asp Pro Ser Ile Ile Thr Pro Ser Thr Asp Lys Ser Ile
275 280 285
Tyr Asp Lys Val Leu Gln Asn Phe Arg Gly Ile Val Asp Arg Leu Asn
290 295 300
Lys Val Leu Val Cys Ile Ser Asp Pro Asn Ile Asn Ile Asn Ile Tyr
305 310 315 320
Lys Asn Lys Phe Lys Asp Lys Tyr Lys Phe Val Glu Asp Ser Glu Gly
325 330 335
Lys Tyr Ser Ile Asp Val Glu Ser Phe Asp Lys Leu Tyr Lys Ser Leu
340 345 350
Met Phe Gly Phe Thr Glu Thr Asn Ile Ala Glu Asn Tyr Lys Ile Lys
355 360 365
Thr Arg Ala Ser Tyr Phe Ser Asp Ser Leu Pro Pro Val Lys Ile Lys
370 375 380
Asn Leu Leu Asp Asn Glu Ile Tyr Thr Ile Glu Glu Gly Phe Asn Ile
385 390 395 400
Ser Asp Lys Asp Met Glu Lys Glu Tyr Arg Gly Gln Asn Lys Ala Ile
405 410 415
Asn Lys Gln Ala Tyr Glu Glu Ile Ser Lys Glu His Leu Ala Val Tyr
420 425 430
Lys Ile Gln Met Cys Lys Ser Val Lys Ala Pro Gly Ile Cys Ile Asp
435 440 445
Val Asp Asn Glu Asp Leu Phe Phe Ile Ala Asp Lys Asn Ser Phe Ser
450 455 460
Asp Asp Leu Ser Lys Asn Glu Arg Ile Glu Tyr Asn Thr Gln Ser Asn
465 470 475 480
Tyr Ile Glu Asn Asp Phe Pro Ile Asn Glu Leu Ile Leu Asp Thr Asp
485 490 495
Leu Ile Ser Lys Ile Glu Leu Pro Ser Glu Asn Thr Glu Ser Leu Thr
500 505 510
Asp Phe Asn Val Asp Val Pro Val Tyr Glu Lys Gln Pro Ala Ile Lys
515 520 525
Lys Ile Phe Thr Asp Glu Asn Thr Ile Phe Gln Tyr Leu Tyr Ser Gln
530 535 540
Thr Phe Pro Leu Asp Ile Arg Asp Ile Ser Leu Thr Ser Ser Phe Asp
545 550 555 560
Asp Ala Leu Leu Phe Ser Asn Lys Val Tyr Ser Phe Phe Ser Met Asp
565 570 575
Tyr Ile Lys Thr Ala Asn Lys Val Val Glu Ala Gly Leu Phe Ala Gly
580 585 590
Trp Val Lys Gln Ile Val Asn Asp Phe Val Ile Glu Ala Asn Lys Ser
595 600 605
Asn Thr Met Asp Lys Ile Ala Asp Ile Ser Leu Ile Val Pro Tyr Ile
610 615 620
Gly Leu Ala Leu Asn Val Gly Asn Glu Thr Ala Lys Gly Asn Phe Glu
625 630 635 640
Asn Ala Phe Glu Ile Ala Gly Ala Ser Ile Leu Leu Glu Phe Ile Pro
645 650 655
Glu Leu Leu Ile Pro Val Val Gly Ala Phe Leu Leu Glu Ser Tyr Ile
660 665 670
Asp Asn Lys Asn Lys Ile Ile Lys Thr Ile Asp Asn Ala Leu Thr Lys
675 680 685
Arg Asn Glu Lys Trp Ser Asp Met Tyr Gly Leu Ile Val Ala Gln Trp
690 695 700
Leu Ser Thr Val Asn Thr Gln Phe Tyr Thr Ile Lys Glu Gly Met Tyr
705 710 715 720
Lys Ala Leu Asn Tyr Gln Ala Gln Ala Leu Glu Glu Ile Ile Lys Tyr
725 730 735
Arg Tyr Asn Ile Tyr Ser Glu Lys Glu Lys Ser Asn Ile Asn Ile Asp
740 745 750
Phe Asn Asp Ile Asn Ser Lys Leu Asn Glu Gly Ile Asn Gln Ala Ile
755 760 765
Asp Asn Ile Asn Asn Phe Ile Asn Gly Cys Ser Val Ser Tyr Leu Met
770 775 780
Lys Lys Met Ile Pro Leu Ala Val Glu Lys Leu Leu Asp Phe Asp Asn
785 790 795 800
Thr Leu Lys Lys Asn Leu Leu Asn Tyr Ile Asp Glu Asn Lys Leu Tyr
805 810 815
Leu Ile Gly Ser Ala Glu Tyr Glu Lys Ser Lys Val Asn Lys Tyr Leu
820 825 830
Lys Thr Ile Met Pro Phe Asp Leu Ser Ile Tyr Thr Asn Asp Thr Ile
835 840 845
Leu Ile Glu Met Phe Asn Lys Tyr Asn Ser Glu Ile Leu Asn Asn Ile
850 855 860
Ile Leu Asn Leu Arg Tyr Lys Asp Asn Asn Leu Ile Asp Leu Ser Gly
865 870 875 880
Tyr Gly Ala Lys Val Glu Val Tyr Asp Gly Val Glu Leu Asn Asp Lys
885 890 895
Asn Gln Phe Lys Leu Thr Ser Ser Ala Asn Ser Lys Ile Arg Val Thr
900 905 910
Gln Asn Gln Asn Ile Ile Phe Asn Ser Val Phe Leu Asp Phe Ser Val
915 920 925
Ser Phe Trp Ile Arg Ile Pro Lys Tyr Lys Asn Asp Gly Ile Gln Asn
930 935 940
Tyr Ile His Asn Glu Tyr Thr Ile Ile Asn Cys Met Lys Asn Asn Ser
945 950 955 960
Gly Trp Lys Ile Ser Ile Arg Gly Asn Arg Ile Ile Trp Thr Leu Ile
965 970 975
Asp Ile Asn Gly Lys Thr Lys Ser Val Phe Phe Glu Tyr Asn Ile Arg
980 985 990
Glu Asp Ile Ser Glu Tyr Ile Asn Arg Trp Phe Phe Val Thr Ile Thr
995 1000 1005
Asn Asn Leu Asn Asn Ala Lys Ile Tyr Ile Asn Gly Lys Leu Glu
1010 1015 1020
Ser Asn Thr Asp Ile Lys Asp Ile Arg Glu Val Ile Ala Asn Gly
1025 1030 1035
Glu Ile Ile Phe Lys Leu Asp Gly Asp Ile Asp Arg Thr Gln Phe
1040 1045 1050
Ile Trp Met Lys Tyr Phe Ser Ile Phe Asn Thr Glu Leu Ser Gln
1055 1060 1065
Ser Asn Ile Glu Glu Arg Tyr Lys Ile Gln Ser Tyr Ser Glu Tyr
1070 1075 1080
Leu Lys Asp Phe Trp Gly Asn Pro Leu Met Tyr Asn Lys Glu Tyr
1085 1090 1095
Tyr Met Phe Asn Ala Gly Asn Lys Asn Ser Tyr Ile Lys Leu Lys
1100 1105 1110
Lys Asp Ser Pro Val Gly Glu Ile Leu Thr Arg Ser Lys Tyr Asn
1115 1120 1125
Gln Asn Ser Lys Tyr Ile Asn Tyr Arg Asp Leu Tyr Ile Gly Glu
1130 1135 1140
Lys Phe Ile Ile Arg Arg Lys Ser Asn Ser Gln Ser Ile Asn Asp
1145 1150 1155
Asp Ile Val Arg Lys Glu Asp Tyr Ile Tyr Leu Asp Phe Phe Asn
1160 1165 1170
Leu Asn Gln Glu Trp Arg Val Tyr Thr Tyr Lys Tyr Phe Lys Lys
1175 1180 1185
Glu Glu Glu Lys Leu Phe Leu Ala Pro Ile Ser Asp Ser Asp Glu
1190 1195 1200
Phe Tyr Asn Thr Ile Gln Ile Lys Glu Tyr Asp Glu Gln Pro Thr
1205 1210 1215
Tyr Ser Cys Gln Leu Leu Phe Lys Lys Asp Glu Glu Ser Thr Asp
1220 1225 1230
Glu Ile Gly Leu Ile Gly Ile His Arg Phe Tyr Glu Ser Gly Ile
1235 1240 1245
Val Phe Glu Glu Tyr Lys Asp Tyr Phe Cys Ile Ser Lys Trp Tyr
1250 1255 1260
Leu Lys Glu Val Lys Arg Lys Pro Tyr Asn Leu Lys Leu Gly Cys
1265 1270 1275
Asn Trp Gln Phe Ile Pro Lys Asp Glu Gly Trp Thr Glu
1280 1285 1290
<210> 11
<211> 3873
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/C(0)
<400> 11
atgccgatca cgattaataa tttcaactat agcgatccgg tggacaataa gaatattctg 60
tatctggata ctcatctgaa tacgctggct aacgaaccgg agaaagcgtt ccgcatcaca 120
ggcaacatct gggttattcc cgatcgcttt tcacgcaaca gcaaccctaa tctgaacaaa 180
cctcctcgtg tcaccagtcc taaatccggt tattacgacc caaactatct gagtacggat 240
agcgataaag atccctttct gaaagagatc attaagctgt tcaaacgcat taactctcgc 300
gaaattgggg aagagctgat ctatcggctt tcgacagata tcccgttccc aggtaacaat 360
aataccccga ttaatacttt cgactttgat gttgatttca attctgtgga tgtgaaaacg 420
cgtcaaggca ataattgggt gaaaactggt agcattaacc cgagtgtaat tatcacaggt 480
ccccgtgaga acatcatcga cccggaaacc tctaccttca agctgacgaa caacacgttt 540
gctgcacagg aagggtttgg tgccctgtca atcatttcca tctcaccgcg tttcatgtta 600
acctactcca atgccacaaa tgatgttggc gaaggacgtt ttagcaaatc agaattttgc 660
atggacccaa ttctcattct gatgggcacg ctgaacaatg cgatgcacaa cttgtatggc 720
attgctattc caaacgatca aaccattagc tccgttacca gtaatatctt ctatagccag 780
tataatgtca aattggagta tgccgaaatt tacgcctttg gaggcccgac cattgacctg 840
attccgaaat ctgcacgcaa atacttcgaa gaaaaggcgt tagattacta tcgcagcatc 900
gcgaaacgcc tgaactcgat taccacggcc aatccgtcgt cgttcaacaa atacattggt 960
gaatataaac agaaactgat tcgcaaatat cggtttgtcg tagaaagctc tggtgaagtg 1020
actgtaaacc gcaacaaatt tgtcgaactc tacaacgagt tgacccaaat ctttaccgag 1080
tttaactacg caaagatcta taacgtacag aaccgcaaga tttatcttag caatgtatac 1140
acaccggtta ctgcgaacat cttagacgac aatgtgtatg atattcagaa tggctttaac 1200
atcccgaaat caaatctgaa cgttctgttt atgggccaga acctgagtcg taatccagca 1260
ctgcgtaaag tgaacccgga aaatatgctc tacttgttta ccaaattttg ccacaaagcg 1320
attgatggcc gctctctcta taacaaaacg ctggattgtc gtgagttact tgtgaagaac 1380
actgatttac cgttcattgg ggatatctcc gacgtgaaaa ccgatatctt cctgcgcaaa 1440
gacattaatg aagaaacgga agtcatctat taccccgaca atgtgagcgt tgatcaggtc 1500
attttatcga agaacacctc cgaacatggt cagttggatt tgctgtaccc tagcattgac 1560
tcggagagtg aaatccttcc gggcgaaaat caagtgtttt acgacaaccg tacccaaaat 1620
gttgattatt tgaattctta ttactacctg gaatctcaga aattgagcga caatgtggaa 1680
gatttcacgt tcacacgctc cattgaggaa gcgctggata atagcgcgaa agtgtatacg 1740
tatttcccta ccttggcgaa taaagtaaat gctggtgtcc agggaggctt atttctgatg 1800
tgggcgaatg atgtggtaga agattttacg accaatattt tgcgtaagga caccttagat 1860
aaaattagcg atgttagcgc catcatcccc tatattggcc cagcactgaa tatctcgaac 1920
tctgtgcgtc gcggaaactt caccgaagca tttgcggtga ccggggttac tattctgttg 1980
gaagcctttc cggagtttac tattccggcg ctgggtgcgt ttgtgattta ttcgaaagta 2040
caagaacgca atgaaattat caaaaccatc gataattgcc tggaacaacg cattaaacgc 2100
tggaaggatt cttatgaatg gatgatgggc acctggttat cccgtattat cacacagttt 2160
aacaacatct cgtatcagat gtacgattca ctgaactacc aagcaggggc gatcaaagcc 2220
aagatcgact tagaatacaa gaaatattca ggtagcgata aagagaatat taaaagccag 2280
gttgaaaacc tgaagaactc tctggatgtc aaaatttcag aggctatgaa caacattaac 2340
aaatttatcc gcgaatgtag cgtcacgtat ctgtttaaaa acatgctccc gaaagtgatt 2400
gatgagctca acgagtttga tcgcaacaca aaggccaaac tgattaacct gattgatagt 2460
cacaatatta ttttagtcgg tgaagttgac aagctgaagg ctaaggtcaa taacagcttt 2520
cagaacacta ttccgtttaa tattttctcc tatacgaaca atagtctgct gaaagacatt 2580
atcaacgaat acttcaacaa tattaatgac agcaaaattc tgagcctgca gaatcgtaag 2640
aatacgctgg tagataccag tggatataat gcggaagtct cagaagaggg tgatgtacag 2700
ctgaacccga tctttccgtt cgactttaaa ctggggtcta gtggtgaaga tcgcggtaaa 2760
gtgatcgtta cccaaaacga gaacattgtg tataacagca tgtacgagag tttctcaatt 2820
tctttctgga ttcgcatcaa taaatgggtt tctaatttgc ctggctatac catcattgat 2880
agcgtcaaaa acaactcggg ctggtcgatt ggcattatta gcaactttct ggtgtttacc 2940
ctgaaacaga atgaggattc ggaacagagc attaacttct cctacgacat cagcaacaat 3000
gcaccagggt ataacaaatg gttcttcgta acggtgacga acaatatgat gggcaatatg 3060
aaaatctaca ttaacgggaa acttatcgac accattaaag tgaaagagct tactgggatc 3120
aattttagta aaaccattac ctttgagatc aacaaaattc cggacacggg tctgattacc 3180
tccgattcgg ataatatcaa tatgtggatt cgcgactttt atatcttcgc caaagaactt 3240
gatggcaaag atatcaacat tttgtttaat tccctgcagt ataccaatgt cgttaaggac 3300
tattggggca atgatctccg ctacaataaa gaatactaca tggttaacat cgactatctc 3360
aatcgctaca tgtatgctaa ctcgcgtcaa attgtgttta acacacgtcg taacaacaac 3420
gattttaacg aaggttataa aatcattatc aaacggatcc gcggcaatac gaacgatact 3480
cgtgttcgtg gcggtgacat tctgtatttc gacatgacga ttaataataa agcgtacaat 3540
ctgttcatga agaacgaaac catgtacgcc gataaccatt ccactgaaga tatctacgca 3600
atcggacttc gcgaacagac caaagacatt aacgacaaca tcatctttca gattcaaccg 3660
atgaataata cctactacta tgcctcccag atcttcaaaa gtaatttcaa cggcgaaaac 3720
atttcaggca tttgctcaat cggcacttat cggttccggt taggtggtga ttggtatcgt 3780
cacaactacc ttgttcccac agtgaaacaa ggcaactatg catcgctctt agaaagcaca 3840
tctacgcatt ggggttttgt gccagtcagt gaa 3873
<210> 12
<211> 1291
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/C(0)
<400> 12
Met Pro Ile Thr Ile Asn Asn Phe Asn Tyr Ser Asp Pro Val Asp Asn
1 5 10 15
Lys Asn Ile Leu Tyr Leu Asp Thr His Leu Asn Thr Leu Ala Asn Glu
20 25 30
Pro Glu Lys Ala Phe Arg Ile Thr Gly Asn Ile Trp Val Ile Pro Asp
35 40 45
Arg Phe Ser Arg Asn Ser Asn Pro Asn Leu Asn Lys Pro Pro Arg Val
50 55 60
Thr Ser Pro Lys Ser Gly Tyr Tyr Asp Pro Asn Tyr Leu Ser Thr Asp
65 70 75 80
Ser Asp Lys Asp Pro Phe Leu Lys Glu Ile Ile Lys Leu Phe Lys Arg
85 90 95
Ile Asn Ser Arg Glu Ile Gly Glu Glu Leu Ile Tyr Arg Leu Ser Thr
100 105 110
Asp Ile Pro Phe Pro Gly Asn Asn Asn Thr Pro Ile Asn Thr Phe Asp
115 120 125
Phe Asp Val Asp Phe Asn Ser Val Asp Val Lys Thr Arg Gln Gly Asn
130 135 140
Asn Trp Val Lys Thr Gly Ser Ile Asn Pro Ser Val Ile Ile Thr Gly
145 150 155 160
Pro Arg Glu Asn Ile Ile Asp Pro Glu Thr Ser Thr Phe Lys Leu Thr
165 170 175
Asn Asn Thr Phe Ala Ala Gln Glu Gly Phe Gly Ala Leu Ser Ile Ile
180 185 190
Ser Ile Ser Pro Arg Phe Met Leu Thr Tyr Ser Asn Ala Thr Asn Asp
195 200 205
Val Gly Glu Gly Arg Phe Ser Lys Ser Glu Phe Cys Met Asp Pro Ile
210 215 220
Leu Ile Leu Met Gly Thr Leu Asn Asn Ala Met His Asn Leu Tyr Gly
225 230 235 240
Ile Ala Ile Pro Asn Asp Gln Thr Ile Ser Ser Val Thr Ser Asn Ile
245 250 255
Phe Tyr Ser Gln Tyr Asn Val Lys Leu Glu Tyr Ala Glu Ile Tyr Ala
260 265 270
Phe Gly Gly Pro Thr Ile Asp Leu Ile Pro Lys Ser Ala Arg Lys Tyr
275 280 285
Phe Glu Glu Lys Ala Leu Asp Tyr Tyr Arg Ser Ile Ala Lys Arg Leu
290 295 300
Asn Ser Ile Thr Thr Ala Asn Pro Ser Ser Phe Asn Lys Tyr Ile Gly
305 310 315 320
Glu Tyr Lys Gln Lys Leu Ile Arg Lys Tyr Arg Phe Val Val Glu Ser
325 330 335
Ser Gly Glu Val Thr Val Asn Arg Asn Lys Phe Val Glu Leu Tyr Asn
340 345 350
Glu Leu Thr Gln Ile Phe Thr Glu Phe Asn Tyr Ala Lys Ile Tyr Asn
355 360 365
Val Gln Asn Arg Lys Ile Tyr Leu Ser Asn Val Tyr Thr Pro Val Thr
370 375 380
Ala Asn Ile Leu Asp Asp Asn Val Tyr Asp Ile Gln Asn Gly Phe Asn
385 390 395 400
Ile Pro Lys Ser Asn Leu Asn Val Leu Phe Met Gly Gln Asn Leu Ser
405 410 415
Arg Asn Pro Ala Leu Arg Lys Val Asn Pro Glu Asn Met Leu Tyr Leu
420 425 430
Phe Thr Lys Phe Cys His Lys Ala Ile Asp Gly Arg Ser Leu Tyr Asn
435 440 445
Lys Thr Leu Asp Cys Arg Glu Leu Leu Val Lys Asn Thr Asp Leu Pro
450 455 460
Phe Ile Gly Asp Ile Ser Asp Val Lys Thr Asp Ile Phe Leu Arg Lys
465 470 475 480
Asp Ile Asn Glu Glu Thr Glu Val Ile Tyr Tyr Pro Asp Asn Val Ser
485 490 495
Val Asp Gln Val Ile Leu Ser Lys Asn Thr Ser Glu His Gly Gln Leu
500 505 510
Asp Leu Leu Tyr Pro Ser Ile Asp Ser Glu Ser Glu Ile Leu Pro Gly
515 520 525
Glu Asn Gln Val Phe Tyr Asp Asn Arg Thr Gln Asn Val Asp Tyr Leu
530 535 540
Asn Ser Tyr Tyr Tyr Leu Glu Ser Gln Lys Leu Ser Asp Asn Val Glu
545 550 555 560
Asp Phe Thr Phe Thr Arg Ser Ile Glu Glu Ala Leu Asp Asn Ser Ala
565 570 575
Lys Val Tyr Thr Tyr Phe Pro Thr Leu Ala Asn Lys Val Asn Ala Gly
580 585 590
Val Gln Gly Gly Leu Phe Leu Met Trp Ala Asn Asp Val Val Glu Asp
595 600 605
Phe Thr Thr Asn Ile Leu Arg Lys Asp Thr Leu Asp Lys Ile Ser Asp
610 615 620
Val Ser Ala Ile Ile Pro Tyr Ile Gly Pro Ala Leu Asn Ile Ser Asn
625 630 635 640
Ser Val Arg Arg Gly Asn Phe Thr Glu Ala Phe Ala Val Thr Gly Val
645 650 655
Thr Ile Leu Leu Glu Ala Phe Pro Glu Phe Thr Ile Pro Ala Leu Gly
660 665 670
Ala Phe Val Ile Tyr Ser Lys Val Gln Glu Arg Asn Glu Ile Ile Lys
675 680 685
Thr Ile Asp Asn Cys Leu Glu Gln Arg Ile Lys Arg Trp Lys Asp Ser
690 695 700
Tyr Glu Trp Met Met Gly Thr Trp Leu Ser Arg Ile Ile Thr Gln Phe
705 710 715 720
Asn Asn Ile Ser Tyr Gln Met Tyr Asp Ser Leu Asn Tyr Gln Ala Gly
725 730 735
Ala Ile Lys Ala Lys Ile Asp Leu Glu Tyr Lys Lys Tyr Ser Gly Ser
740 745 750
Asp Lys Glu Asn Ile Lys Ser Gln Val Glu Asn Leu Lys Asn Ser Leu
755 760 765
Asp Val Lys Ile Ser Glu Ala Met Asn Asn Ile Asn Lys Phe Ile Arg
770 775 780
Glu Cys Ser Val Thr Tyr Leu Phe Lys Asn Met Leu Pro Lys Val Ile
785 790 795 800
Asp Glu Leu Asn Glu Phe Asp Arg Asn Thr Lys Ala Lys Leu Ile Asn
805 810 815
Leu Ile Asp Ser His Asn Ile Ile Leu Val Gly Glu Val Asp Lys Leu
820 825 830
Lys Ala Lys Val Asn Asn Ser Phe Gln Asn Thr Ile Pro Phe Asn Ile
835 840 845
Phe Ser Tyr Thr Asn Asn Ser Leu Leu Lys Asp Ile Ile Asn Glu Tyr
850 855 860
Phe Asn Asn Ile Asn Asp Ser Lys Ile Leu Ser Leu Gln Asn Arg Lys
865 870 875 880
Asn Thr Leu Val Asp Thr Ser Gly Tyr Asn Ala Glu Val Ser Glu Glu
885 890 895
Gly Asp Val Gln Leu Asn Pro Ile Phe Pro Phe Asp Phe Lys Leu Gly
900 905 910
Ser Ser Gly Glu Asp Arg Gly Lys Val Ile Val Thr Gln Asn Glu Asn
915 920 925
Ile Val Tyr Asn Ser Met Tyr Glu Ser Phe Ser Ile Ser Phe Trp Ile
930 935 940
Arg Ile Asn Lys Trp Val Ser Asn Leu Pro Gly Tyr Thr Ile Ile Asp
945 950 955 960
Ser Val Lys Asn Asn Ser Gly Trp Ser Ile Gly Ile Ile Ser Asn Phe
965 970 975
Leu Val Phe Thr Leu Lys Gln Asn Glu Asp Ser Glu Gln Ser Ile Asn
980 985 990
Phe Ser Tyr Asp Ile Ser Asn Asn Ala Pro Gly Tyr Asn Lys Trp Phe
995 1000 1005
Phe Val Thr Val Thr Asn Asn Met Met Gly Asn Met Lys Ile Tyr
1010 1015 1020
Ile Asn Gly Lys Leu Ile Asp Thr Ile Lys Val Lys Glu Leu Thr
1025 1030 1035
Gly Ile Asn Phe Ser Lys Thr Ile Thr Phe Glu Ile Asn Lys Ile
1040 1045 1050
Pro Asp Thr Gly Leu Ile Thr Ser Asp Ser Asp Asn Ile Asn Met
1055 1060 1065
Trp Ile Arg Asp Phe Tyr Ile Phe Ala Lys Glu Leu Asp Gly Lys
1070 1075 1080
Asp Ile Asn Ile Leu Phe Asn Ser Leu Gln Tyr Thr Asn Val Val
1085 1090 1095
Lys Asp Tyr Trp Gly Asn Asp Leu Arg Tyr Asn Lys Glu Tyr Tyr
1100 1105 1110
Met Val Asn Ile Asp Tyr Leu Asn Arg Tyr Met Tyr Ala Asn Ser
1115 1120 1125
Arg Gln Ile Val Phe Asn Thr Arg Arg Asn Asn Asn Asp Phe Asn
1130 1135 1140
Glu Gly Tyr Lys Ile Ile Ile Lys Arg Ile Arg Gly Asn Thr Asn
1145 1150 1155
Asp Thr Arg Val Arg Gly Gly Asp Ile Leu Tyr Phe Asp Met Thr
1160 1165 1170
Ile Asn Asn Lys Ala Tyr Asn Leu Phe Met Lys Asn Glu Thr Met
1175 1180 1185
Tyr Ala Asp Asn His Ser Thr Glu Asp Ile Tyr Ala Ile Gly Leu
1190 1195 1200
Arg Glu Gln Thr Lys Asp Ile Asn Asp Asn Ile Ile Phe Gln Ile
1205 1210 1215
Gln Pro Met Asn Asn Thr Tyr Tyr Tyr Ala Ser Gln Ile Phe Lys
1220 1225 1230
Ser Asn Phe Asn Gly Glu Asn Ile Ser Gly Ile Cys Ser Ile Gly
1235 1240 1245
Thr Tyr Arg Phe Arg Leu Gly Gly Asp Trp Tyr Arg His Asn Tyr
1250 1255 1260
Leu Val Pro Thr Val Lys Gln Gly Asn Tyr Ala Ser Leu Leu Glu
1265 1270 1275
Ser Thr Ser Thr His Trp Gly Phe Val Pro Val Ser Glu
1280 1285 1290
<210> 13
<211> 3756
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/E(0)
<400> 13
atgccgaaaa tcaactcttt caactacaac gacccggtta acgaccgtac catcctgtat 60
atcaaaccgg gtggttgcca ggagttctac aaatctttca acatcatgaa aaacatctgg 120
atcatcccgg aacgtaacgt tatcggtacc accccgcagg acttccaccc gccgacctct 180
ctgaaaaacg gtgactcttc ttactacgac ccgaactacc tccagtctga cgaagaaaaa 240
gaccgtttcc tgaaaatcgt taccaaaatc ttcaaccgta tcaacaacaa cctgtctggt 300
ggtatcctgc tggaagaact gtctaaagct aacccgtacc tgggtaacga caacaccccg 360
gacaaccagt tccacatcgg tgacgcttct gctgttgaaa tcaaattctc taacggttct 420
caggacatcc tgctgccgaa cgttatcatc atgggtgctg aaccggacct gttcgaaacc 480
aactcttcta acatctctct gcgtaacaac tacatgccgt ctaaccacgg tttcggttct 540
atcgctatcg ttaccttctc tccggaatac tctttccgtt tcaacgacaa cagcatgaac 600
gagttcatcc aggacccggc tctgaccctg atgcaccaac tgatctactc tctgcacggt 660
ctgtacggtg ctaaaggtat caccaccaaa tacaccatca cccagaaaca gaacccgctg 720
atcaccaaca tccgtggtac caacatcgaa gagttcctga ccttcggtgg taccgacctg 780
aacatcatca cctctgctca gtctaacgac atctacacca acctgctggc tgactacaaa 840
aaaatcgctt ctaaactgtc taaagttcag gtttctaacc cgctgctgaa cccgtacaaa 900
gacgttttcg aagctaaata cggtctggac aaagacgctt ctggtatcta ctctgttaac 960
atcaacaaat tcaacgacat cttcaaaaaa ctgtactctt tcaccgagtt cgacctggcg 1020
accaaattcc aggttaaatg ccgtcagacc tacatcggtc agtacaaata cttcaaactg 1080
tctaacctgc tgaacgactc tatctacaac atctctgaag gttacaacat caacaacctg 1140
aaagttaact tccgtggtca gaacgctaac ctgaacccgc gtatcatcac cccgatcacc 1200
ggtcgtggtc tggttaaaaa aatcatccgt ttctgcaaga atattgtaag cgttaaagga 1260
ataagaaaaa gtatctgcat cgaaatcaac aacggtgaac tgttcttcgt tgcttctgaa 1320
aactcttaca acgacgacaa catcaacacc ccgaaagaaa tcgacgacac cgttacctct 1380
aacaacaact acgaaaacga cctggaccag gttatcctga acttcaactc tgaatctgct 1440
ccgggtctgt ctgacgaaaa actgaacctg accatccaga acgacgctta catcccgaaa 1500
tacgactcta acggtacctc tgacatcgaa cagcacgacg ttaacgaact gaacgttttc 1560
ttctacctgg acgctcagaa agttccggaa ggtgaaaaca acgttaacct gacctcttct 1620
atcgacaccg ctctgctgga acagccgaaa atctacacct tcttctcttc tgagttcatc 1680
aacaacgtta acaaaccggt tcaggctgct ctgttcgttt cttggattca gcaggttctg 1740
gttgacttca ccaccgaagc taaccagaaa tctaccgttg acaaaatcgc tgacatctct 1800
atcgttgttc cgtacatcgg tctggctctg aacatcggta acgaagctca gaaaggtaac 1860
ttcaaagacg ctctggaact gctgggtgct ggtatcctgc tggagttcga accggaactg 1920
ctgatcccga ccatcctggt tttcaccatc aaatctttcc tgggttcttc tgacaacaaa 1980
aacaaagtta tcaaagctat caacaacgct ctgaaagaac gtgacgaaaa atggaaagaa 2040
gtttactctt tcatcgtttc taactggatg accaaaatca acacccagtt caacaaacgt 2100
aaagaacaga tgtaccaggc tctccagaac caggttaacg ctatcaaaac catcatcgaa 2160
tctaaataca actcttacac cctggaagaa aaaaacgaac tgaccaacaa atacgacatc 2220
aaacagatcg aaaacgaact gaaccagaaa gtttctatcg ctatgaacaa catcgaccgt 2280
ttcctgaccg aatcttctat ctcttacctg atgaaactca tcaacgaagt taaaatcaac 2340
aaactgcgtg aatacgacga aaacgttaaa acctacctgc tgaactacat catccagcac 2400
ggttctatcc tgggtgaatc tcagcaggaa ctgaactcta tggttaccga caccctgaac 2460
aactctatcc cgttcaaact gtcttcttac accgacgaca aaatcctgat ctcttacttc 2520
aacaaattct ttaaacgcat taagagttca tcggttctga atatgcggta caaaaatgat 2580
aaatatgtcg atacttctgg atatgatagc aatatcaaca ttaacggcga cgtgtataaa 2640
tatccgacaa ataaaaacca gtttgggata tataacgaca agctgtcgga ggtcaatatt 2700
tctcaaaacg actatatcat ttacgataat aaatataaaa actttagcat tagtttttgg 2760
gttcgtatac ctaattatga caataaaatt gtaaatgtga ataacgagta taccattata 2820
aactgtatgc gcgacaataa cagtggttgg aaggtatcgc tgaaccataa tgagattatc 2880
tggaccctgc aggataatgc aggtataaac cagaaactgg cttttaacta tggaaacgca 2940
aatgggatct cagattacat taataaatgg atttttgtta ccattacgaa cgatcgctta 3000
ggcgactcaa aactttatat taatggcaat ctgatagatc agaaatcaat cttaaatttg 3060
ggcaatattc atgtctctga taacatcttg ttcaagatcg ttaattgcag ttacactcgt 3120
tatattggca ttcgttactt taatatcttc gataaagaac tggacgagac ggaaatccag 3180
actctgtatt caaacgagcc caatactaat atattgaaag atttttgggg taactatctt 3240
ttatatgata aagaatacta tctcctgaat gtattgaagc caaacaattt catagataga 3300
cgcaaggata gcacattaag tatcaacaat atcagatcta ctatactgtt agcaaatcgc 3360
ctctactccg gtattaaagt gaagattcag cgggttaata actccagtac caatgataat 3420
ctggtccgta agaacgatca ggtatacatc aatttcgtcg cgagcaaaac tcatctcttc 3480
ccgctttacg ccgatacagc tacgacaaac aaggaaaaaa ccataaaaat ttccagctcc 3540
ggaaacagat tcaatcaagt agttgtaatg aactctgtgg gtaataattg tacgatgaac 3600
tttaagaata acaatgggaa caatattgga cttttgggct tcaaagccga cacagtggtg 3660
gcgtccacct ggtattacac gcacatgcgg gaccatacga attcgaacgg ttgcttctgg 3720
aactttatct cggaagaaca cgggtggcaa gaaaaa 3756
<210> 14
<211> 1252
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/E(0)
<400> 14
Met Pro Lys Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp Arg
1 5 10 15
Thr Ile Leu Tyr Ile Lys Pro Gly Gly Cys Gln Glu Phe Tyr Lys Ser
20 25 30
Phe Asn Ile Met Lys Asn Ile Trp Ile Ile Pro Glu Arg Asn Val Ile
35 40 45
Gly Thr Thr Pro Gln Asp Phe His Pro Pro Thr Ser Leu Lys Asn Gly
50 55 60
Asp Ser Ser Tyr Tyr Asp Pro Asn Tyr Leu Gln Ser Asp Glu Glu Lys
65 70 75 80
Asp Arg Phe Leu Lys Ile Val Thr Lys Ile Phe Asn Arg Ile Asn Asn
85 90 95
Asn Leu Ser Gly Gly Ile Leu Leu Glu Glu Leu Ser Lys Ala Asn Pro
100 105 110
Tyr Leu Gly Asn Asp Asn Thr Pro Asp Asn Gln Phe His Ile Gly Asp
115 120 125
Ala Ser Ala Val Glu Ile Lys Phe Ser Asn Gly Ser Gln Asp Ile Leu
130 135 140
Leu Pro Asn Val Ile Ile Met Gly Ala Glu Pro Asp Leu Phe Glu Thr
145 150 155 160
Asn Ser Ser Asn Ile Ser Leu Arg Asn Asn Tyr Met Pro Ser Asn His
165 170 175
Gly Phe Gly Ser Ile Ala Ile Val Thr Phe Ser Pro Glu Tyr Ser Phe
180 185 190
Arg Phe Asn Asp Asn Ser Met Asn Glu Phe Ile Gln Asp Pro Ala Leu
195 200 205
Thr Leu Met His Gln Leu Ile Tyr Ser Leu His Gly Leu Tyr Gly Ala
210 215 220
Lys Gly Ile Thr Thr Lys Tyr Thr Ile Thr Gln Lys Gln Asn Pro Leu
225 230 235 240
Ile Thr Asn Ile Arg Gly Thr Asn Ile Glu Glu Phe Leu Thr Phe Gly
245 250 255
Gly Thr Asp Leu Asn Ile Ile Thr Ser Ala Gln Ser Asn Asp Ile Tyr
260 265 270
Thr Asn Leu Leu Ala Asp Tyr Lys Lys Ile Ala Ser Lys Leu Ser Lys
275 280 285
Val Gln Val Ser Asn Pro Leu Leu Asn Pro Tyr Lys Asp Val Phe Glu
290 295 300
Ala Lys Tyr Gly Leu Asp Lys Asp Ala Ser Gly Ile Tyr Ser Val Asn
305 310 315 320
Ile Asn Lys Phe Asn Asp Ile Phe Lys Lys Leu Tyr Ser Phe Thr Glu
325 330 335
Phe Asp Leu Ala Thr Lys Phe Gln Val Lys Cys Arg Gln Thr Tyr Ile
340 345 350
Gly Gln Tyr Lys Tyr Phe Lys Leu Ser Asn Leu Leu Asn Asp Ser Ile
355 360 365
Tyr Asn Ile Ser Glu Gly Tyr Asn Ile Asn Asn Leu Lys Val Asn Phe
370 375 380
Arg Gly Gln Asn Ala Asn Leu Asn Pro Arg Ile Ile Thr Pro Ile Thr
385 390 395 400
Gly Arg Gly Leu Val Lys Lys Ile Ile Arg Phe Cys Lys Asn Ile Val
405 410 415
Ser Val Lys Gly Ile Arg Lys Ser Ile Cys Ile Glu Ile Asn Asn Gly
420 425 430
Glu Leu Phe Phe Val Ala Ser Glu Asn Ser Tyr Asn Asp Asp Asn Ile
435 440 445
Asn Thr Pro Lys Glu Ile Asp Asp Thr Val Thr Ser Asn Asn Asn Tyr
450 455 460
Glu Asn Asp Leu Asp Gln Val Ile Leu Asn Phe Asn Ser Glu Ser Ala
465 470 475 480
Pro Gly Leu Ser Asp Glu Lys Leu Asn Leu Thr Ile Gln Asn Asp Ala
485 490 495
Tyr Ile Pro Lys Tyr Asp Ser Asn Gly Thr Ser Asp Ile Glu Gln His
500 505 510
Asp Val Asn Glu Leu Asn Val Phe Phe Tyr Leu Asp Ala Gln Lys Val
515 520 525
Pro Glu Gly Glu Asn Asn Val Asn Leu Thr Ser Ser Ile Asp Thr Ala
530 535 540
Leu Leu Glu Gln Pro Lys Ile Tyr Thr Phe Phe Ser Ser Glu Phe Ile
545 550 555 560
Asn Asn Val Asn Lys Pro Val Gln Ala Ala Leu Phe Val Ser Trp Ile
565 570 575
Gln Gln Val Leu Val Asp Phe Thr Thr Glu Ala Asn Gln Lys Ser Thr
580 585 590
Val Asp Lys Ile Ala Asp Ile Ser Ile Val Val Pro Tyr Ile Gly Leu
595 600 605
Ala Leu Asn Ile Gly Asn Glu Ala Gln Lys Gly Asn Phe Lys Asp Ala
610 615 620
Leu Glu Leu Leu Gly Ala Gly Ile Leu Leu Glu Phe Glu Pro Glu Leu
625 630 635 640
Leu Ile Pro Thr Ile Leu Val Phe Thr Ile Lys Ser Phe Leu Gly Ser
645 650 655
Ser Asp Asn Lys Asn Lys Val Ile Lys Ala Ile Asn Asn Ala Leu Lys
660 665 670
Glu Arg Asp Glu Lys Trp Lys Glu Val Tyr Ser Phe Ile Val Ser Asn
675 680 685
Trp Met Thr Lys Ile Asn Thr Gln Phe Asn Lys Arg Lys Glu Gln Met
690 695 700
Tyr Gln Ala Leu Gln Asn Gln Val Asn Ala Ile Lys Thr Ile Ile Glu
705 710 715 720
Ser Lys Tyr Asn Ser Tyr Thr Leu Glu Glu Lys Asn Glu Leu Thr Asn
725 730 735
Lys Tyr Asp Ile Lys Gln Ile Glu Asn Glu Leu Asn Gln Lys Val Ser
740 745 750
Ile Ala Met Asn Asn Ile Asp Arg Phe Leu Thr Glu Ser Ser Ile Ser
755 760 765
Tyr Leu Met Lys Leu Ile Asn Glu Val Lys Ile Asn Lys Leu Arg Glu
770 775 780
Tyr Asp Glu Asn Val Lys Thr Tyr Leu Leu Asn Tyr Ile Ile Gln His
785 790 795 800
Gly Ser Ile Leu Gly Glu Ser Gln Gln Glu Leu Asn Ser Met Val Thr
805 810 815
Asp Thr Leu Asn Asn Ser Ile Pro Phe Lys Leu Ser Ser Tyr Thr Asp
820 825 830
Asp Lys Ile Leu Ile Ser Tyr Phe Asn Lys Phe Phe Lys Arg Ile Lys
835 840 845
Ser Ser Ser Val Leu Asn Met Arg Tyr Lys Asn Asp Lys Tyr Val Asp
850 855 860
Thr Ser Gly Tyr Asp Ser Asn Ile Asn Ile Asn Gly Asp Val Tyr Lys
865 870 875 880
Tyr Pro Thr Asn Lys Asn Gln Phe Gly Ile Tyr Asn Asp Lys Leu Ser
885 890 895
Glu Val Asn Ile Ser Gln Asn Asp Tyr Ile Ile Tyr Asp Asn Lys Tyr
900 905 910
Lys Asn Phe Ser Ile Ser Phe Trp Val Arg Ile Pro Asn Tyr Asp Asn
915 920 925
Lys Ile Val Asn Val Asn Asn Glu Tyr Thr Ile Ile Asn Cys Met Arg
930 935 940
Asp Asn Asn Ser Gly Trp Lys Val Ser Leu Asn His Asn Glu Ile Ile
945 950 955 960
Trp Thr Leu Gln Asp Asn Ala Gly Ile Asn Gln Lys Leu Ala Phe Asn
965 970 975
Tyr Gly Asn Ala Asn Gly Ile Ser Asp Tyr Ile Asn Lys Trp Ile Phe
980 985 990
Val Thr Ile Thr Asn Asp Arg Leu Gly Asp Ser Lys Leu Tyr Ile Asn
995 1000 1005
Gly Asn Leu Ile Asp Gln Lys Ser Ile Leu Asn Leu Gly Asn Ile
1010 1015 1020
His Val Ser Asp Asn Ile Leu Phe Lys Ile Val Asn Cys Ser Tyr
1025 1030 1035
Thr Arg Tyr Ile Gly Ile Arg Tyr Phe Asn Ile Phe Asp Lys Glu
1040 1045 1050
Leu Asp Glu Thr Glu Ile Gln Thr Leu Tyr Ser Asn Glu Pro Asn
1055 1060 1065
Thr Asn Ile Leu Lys Asp Phe Trp Gly Asn Tyr Leu Leu Tyr Asp
1070 1075 1080
Lys Glu Tyr Tyr Leu Leu Asn Val Leu Lys Pro Asn Asn Phe Ile
1085 1090 1095
Asp Arg Arg Lys Asp Ser Thr Leu Ser Ile Asn Asn Ile Arg Ser
1100 1105 1110
Thr Ile Leu Leu Ala Asn Arg Leu Tyr Ser Gly Ile Lys Val Lys
1115 1120 1125
Ile Gln Arg Val Asn Asn Ser Ser Thr Asn Asp Asn Leu Val Arg
1130 1135 1140
Lys Asn Asp Gln Val Tyr Ile Asn Phe Val Ala Ser Lys Thr His
1145 1150 1155
Leu Phe Pro Leu Tyr Ala Asp Thr Ala Thr Thr Asn Lys Glu Lys
1160 1165 1170
Thr Ile Lys Ile Ser Ser Ser Gly Asn Arg Phe Asn Gln Val Val
1175 1180 1185
Val Met Asn Ser Val Gly Asn Asn Cys Thr Met Asn Phe Lys Asn
1190 1195 1200
Asn Asn Gly Asn Asn Ile Gly Leu Leu Gly Phe Lys Ala Asp Thr
1205 1210 1215
Val Val Ala Ser Thr Trp Tyr Tyr Thr His Met Arg Asp His Thr
1220 1225 1230
Asn Ser Asn Gly Cys Phe Trp Asn Phe Ile Ser Glu Glu His Gly
1235 1240 1245
Trp Gln Glu Lys
1250
<210> 15
<211> 3834
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/F(0)
<400> 15
atgccggtgg tcatcaacag cttcaactac aacgacccag taaacgacga cacgatcctg 60
tatatgcaaa tcccgtatga agagaagagc aagaagtact ataaggcctt tgaaatcatg 120
cgcaatgtgt ggattattcc ggagcgtaat acgattggta ctgacccaag cgacttcgat 180
ccacctgcgt ctttggaaaa cggctcgtcc gcatattacg acccgaatta cctgaccacc 240
gatgcggaga aagatcgtta tttgaaaacc accatcaagc tgttcaaacg cattaacagc 300
aatccggcag gtgaggtcct gctgcaagag attagctacg caaagcctta tctgggtaat 360
gagcatacgc ctattaacga gtttcacccg gttacccgca ctaccagcgt taacatcaag 420
tcctcgacca acgtgaagtc tagcattatc ctgaacctgc tggttctggg tgccggtccg 480
gacatcttcg aaaactctag ctacccggtg cgtaaactga tggatagcgg cggtgtttat 540
gacccgagca atgacggttt tggcagcatc aatatcgtga cgtttagccc ggagtacgag 600
tacaccttca atgatatcag cggtggttac aattcttcta ccgagagctt catcgccgac 660
ccggcgatca gcctggcaca ccaactgatc tatgcattgc atggcttgta cggtgcccgt 720
ggtgtgacgt ataaagagac tatcaaggtt aagcaggcac ctctgatgat tgcggaaaag 780
ccgattcgcc tggaagagtt cctgaccttc ggcggtcaag atttgaacat cattacctcg 840
gccatgaaag agaaaatcta taacaatttg ctggccaact atgaaaagat tgcaacgcgc 900
ttgtctcgtg ttaactccgc tccgccggaa tacgacatta atgagtacaa agactacttt 960
caatggaaat atggcctgga caaaaatgcg gatggttctt ataccgtgaa tgaaaacaaa 1020
ttcaatgaaa tctacaagaa actgtacagc ttcaccgaaa tcgatctggc gaacaagttc 1080
aaagtcaaat gtcgtaatac ctacttcatc aaatatggct tcctgaaagt cccgaacctg 1140
ctggacgatg acatctatac cgtcagcgaa ggcttcaaca tcggcaatct ggccgtgaat 1200
aatcgtggtc agaacatcaa actgaatccg aaaatcattg actccatccc agacaagggc 1260
ctggttgaga aaatcgtgaa gttctgcaaa agcgttattc cgcgtaaagg tacgaaagca 1320
ccgcctcgcc tgtgcattcg cgttaacaac cgtgagttgt tctttgtggc atctgaaagc 1380
agctacaacg agaacgacat caacacccct aaagaaattg atgataccac gaacctgaat 1440
aacaattatc gcaacaatct ggacgaggtg atcctggatt acaattcgga aaccattccg 1500
caaattagca atcagacgct gaacaccctg gttcaggacg atagctacgt tccgcgttac 1560
gactccaatg gtactagcga gattgaagaa cacaacgtag tggacttgaa cgttttcttt 1620
tatctgcacg cccagaaggt tccggagggc gaaaccaata ttagcctgac cagctcgatc 1680
gacaccgcgc tgtctgagga gagccaagtc tacacctttt tcagcagcga gtttatcaac 1740
actattaaca agccagttca tgctgcattg tttatctctt ggattaacca ggtgattcgc 1800
gactttacga cggaggcgac ccagaagtct accttcgaca aaattgcaga catctccctg 1860
gtcgtcccat acgtcggcct ggcgttgaat attggcaatg aagttcaaaa agagaacttc 1920
aaagaagcgt tcgagctgct gggtgcaggc atcctgctgg agttcgtgcc ggaactgttg 1980
atcccgacca tcctggtgtt caccattaag agcttcattg gatcctccga gaataagaac 2040
aagatcatca aggcgatcaa taacagcctg atggagcgtg aaacgaagtg gaaagaaatc 2100
tatagctgga ttgttagcaa ttggctgact cgtattaaca cgcaattcaa caagcgtaaa 2160
gagcaaatgt accaagccct gcaaaaccaa gttgacgcca tcaaaacggt aattgaatac 2220
aagtacaaca attacacgag cgatgagcgc aaccgcctgg aaagcgaata caacatcaac 2280
aacattcgcg aagaattgaa caagaaagtg agcctggcga tggagaacat tgagcgtttt 2340
atcaccgaaa gcagcatctt ttacctgatg aaattgatta atgaggcgaa agtctcgaaa 2400
ctgcgtgagt acgacgaagg tgtgaaagag tatctgctgg attacattag cgagcaccgt 2460
agcatcttgg gtaactcggt tcaggagctg aacgatctgg tgacctctac cctgaacaat 2520
agcatcccgt tcgaactgag cagctatacc aatgacaaga ttctgattct gtatttcaat 2580
aaactgtata agaagatcaa ggataacagc attctggata tgcgttacga aaacaataag 2640
tttatcgaca tttctggtta cggcagcaac atttccatca atggcgatgt ctacatctac 2700
agcaccaatc gcaaccagtt cggcatctac tctagcaaac cgagcgaagt taacatcgca 2760
cagaacaatg atattattta taacggtcgt tatcaaaact tctctatcag cttttgggtc 2820
cgtatcccga agtacttcaa taaagtcaat ctgaataatg aatacacgat catcgactgc 2880
attcgcaata acaacagcgg ttggaaaatc agcctgaatt acaacaaaat tatttggacc 2940
ctgcaagata cggcgggtaa caatcagaaa ctggtgttta actacacgca aatgatcagc 3000
atttctgact atatcaacaa gtggatcttt gttaccatca ccaataatcg tctgggcaat 3060
agccgtattt acatcaacgg taacctgatt gatgagaaaa gcatcagcaa cctgggcgat 3120
attcacgtca gcgacaacat tctgttcaaa attgttggtt gtaacgatac ccgttacgtc 3180
ggcatccgtt atttcaaggt tttcgatacg gagctgggta aaacggaaat cgaaacgttg 3240
tactccgatg aaccagatcc gagcattctg aaggactttt ggggtaacta cttgctgtac 3300
aataaacgtt actatctgct gaatctgttg cgcaccgaca agagcattac ccaaaacagc 3360
aatttcctga acattaatca gcaacgcggc gtataccaaa aaccgaacat cttcagcaat 3420
acgcgcctgt atactggtgt tgaagtgatc attcgtaaga acggtagcac cgacattagc 3480
aacacggaca atttcgtccg taagaatgac ctggcgtaca ttaacgtcgt ggaccgtgat 3540
gtcgagtatc gtctgtacgc agacatcagc attgcgaaac cggaaaagat tatcaagctg 3600
atccgtacca gcaacagcaa caacagcctg ggtcagatca ttgtgatgga cagcattggt 3660
aataactgca cgatgaactt ccagaacaac aatggtggta atatcggtct gctgggtttt 3720
cacagcaata atctggttgc ttccagctgg tactacaata acattcgtaa aaacacgtct 3780
agcaatggtt gtttttggag ctttatcagc aaagagcacg gctggcaaga aaat 3834
<210> 16
<211> 1278
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/F(0)
<400> 16
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp
1 5 10 15
Asp Thr Ile Leu Tyr Met Gln Ile Pro Tyr Glu Glu Lys Ser Lys Lys
20 25 30
Tyr Tyr Lys Ala Phe Glu Ile Met Arg Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Asn Thr Ile Gly Thr Asp Pro Ser Asp Phe Asp Pro Pro Ala Ser
50 55 60
Leu Glu Asn Gly Ser Ser Ala Tyr Tyr Asp Pro Asn Tyr Leu Thr Thr
65 70 75 80
Asp Ala Glu Lys Asp Arg Tyr Leu Lys Thr Thr Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Asn Pro Ala Gly Glu Val Leu Leu Gln Glu Ile Ser
100 105 110
Tyr Ala Lys Pro Tyr Leu Gly Asn Glu His Thr Pro Ile Asn Glu Phe
115 120 125
His Pro Val Thr Arg Thr Thr Ser Val Asn Ile Lys Ser Ser Thr Asn
130 135 140
Val Lys Ser Ser Ile Ile Leu Asn Leu Leu Val Leu Gly Ala Gly Pro
145 150 155 160
Asp Ile Phe Glu Asn Ser Ser Tyr Pro Val Arg Lys Leu Met Asp Ser
165 170 175
Gly Gly Val Tyr Asp Pro Ser Asn Asp Gly Phe Gly Ser Ile Asn Ile
180 185 190
Val Thr Phe Ser Pro Glu Tyr Glu Tyr Thr Phe Asn Asp Ile Ser Gly
195 200 205
Gly Tyr Asn Ser Ser Thr Glu Ser Phe Ile Ala Asp Pro Ala Ile Ser
210 215 220
Leu Ala His Gln Leu Ile Tyr Ala Leu His Gly Leu Tyr Gly Ala Arg
225 230 235 240
Gly Val Thr Tyr Lys Glu Thr Ile Lys Val Lys Gln Ala Pro Leu Met
245 250 255
Ile Ala Glu Lys Pro Ile Arg Leu Glu Glu Phe Leu Thr Phe Gly Gly
260 265 270
Gln Asp Leu Asn Ile Ile Thr Ser Ala Met Lys Glu Lys Ile Tyr Asn
275 280 285
Asn Leu Leu Ala Asn Tyr Glu Lys Ile Ala Thr Arg Leu Ser Arg Val
290 295 300
Asn Ser Ala Pro Pro Glu Tyr Asp Ile Asn Glu Tyr Lys Asp Tyr Phe
305 310 315 320
Gln Trp Lys Tyr Gly Leu Asp Lys Asn Ala Asp Gly Ser Tyr Thr Val
325 330 335
Asn Glu Asn Lys Phe Asn Glu Ile Tyr Lys Lys Leu Tyr Ser Phe Thr
340 345 350
Glu Ile Asp Leu Ala Asn Lys Phe Lys Val Lys Cys Arg Asn Thr Tyr
355 360 365
Phe Ile Lys Tyr Gly Phe Leu Lys Val Pro Asn Leu Leu Asp Asp Asp
370 375 380
Ile Tyr Thr Val Ser Glu Gly Phe Asn Ile Gly Asn Leu Ala Val Asn
385 390 395 400
Asn Arg Gly Gln Asn Ile Lys Leu Asn Pro Lys Ile Ile Asp Ser Ile
405 410 415
Pro Asp Lys Gly Leu Val Glu Lys Ile Val Lys Phe Cys Lys Ser Val
420 425 430
Ile Pro Arg Lys Gly Thr Lys Ala Pro Pro Arg Leu Cys Ile Arg Val
435 440 445
Asn Asn Arg Glu Leu Phe Phe Val Ala Ser Glu Ser Ser Tyr Asn Glu
450 455 460
Asn Asp Ile Asn Thr Pro Lys Glu Ile Asp Asp Thr Thr Asn Leu Asn
465 470 475 480
Asn Asn Tyr Arg Asn Asn Leu Asp Glu Val Ile Leu Asp Tyr Asn Ser
485 490 495
Glu Thr Ile Pro Gln Ile Ser Asn Gln Thr Leu Asn Thr Leu Val Gln
500 505 510
Asp Asp Ser Tyr Val Pro Arg Tyr Asp Ser Asn Gly Thr Ser Glu Ile
515 520 525
Glu Glu His Asn Val Val Asp Leu Asn Val Phe Phe Tyr Leu His Ala
530 535 540
Gln Lys Val Pro Glu Gly Glu Thr Asn Ile Ser Leu Thr Ser Ser Ile
545 550 555 560
Asp Thr Ala Leu Ser Glu Glu Ser Gln Val Tyr Thr Phe Phe Ser Ser
565 570 575
Glu Phe Ile Asn Thr Ile Asn Lys Pro Val His Ala Ala Leu Phe Ile
580 585 590
Ser Trp Ile Asn Gln Val Ile Arg Asp Phe Thr Thr Glu Ala Thr Gln
595 600 605
Lys Ser Thr Phe Asp Lys Ile Ala Asp Ile Ser Leu Val Val Pro Tyr
610 615 620
Val Gly Leu Ala Leu Asn Ile Gly Asn Glu Val Gln Lys Glu Asn Phe
625 630 635 640
Lys Glu Ala Phe Glu Leu Leu Gly Ala Gly Ile Leu Leu Glu Phe Val
645 650 655
Pro Glu Leu Leu Ile Pro Thr Ile Leu Val Phe Thr Ile Lys Ser Phe
660 665 670
Ile Gly Ser Ser Glu Asn Lys Asn Lys Ile Ile Lys Ala Ile Asn Asn
675 680 685
Ser Leu Met Glu Arg Glu Thr Lys Trp Lys Glu Ile Tyr Ser Trp Ile
690 695 700
Val Ser Asn Trp Leu Thr Arg Ile Asn Thr Gln Phe Asn Lys Arg Lys
705 710 715 720
Glu Gln Met Tyr Gln Ala Leu Gln Asn Gln Val Asp Ala Ile Lys Thr
725 730 735
Val Ile Glu Tyr Lys Tyr Asn Asn Tyr Thr Ser Asp Glu Arg Asn Arg
740 745 750
Leu Glu Ser Glu Tyr Asn Ile Asn Asn Ile Arg Glu Glu Leu Asn Lys
755 760 765
Lys Val Ser Leu Ala Met Glu Asn Ile Glu Arg Phe Ile Thr Glu Ser
770 775 780
Ser Ile Phe Tyr Leu Met Lys Leu Ile Asn Glu Ala Lys Val Ser Lys
785 790 795 800
Leu Arg Glu Tyr Asp Glu Gly Val Lys Glu Tyr Leu Leu Asp Tyr Ile
805 810 815
Ser Glu His Arg Ser Ile Leu Gly Asn Ser Val Gln Glu Leu Asn Asp
820 825 830
Leu Val Thr Ser Thr Leu Asn Asn Ser Ile Pro Phe Glu Leu Ser Ser
835 840 845
Tyr Thr Asn Asp Lys Ile Leu Ile Leu Tyr Phe Asn Lys Leu Tyr Lys
850 855 860
Lys Ile Lys Asp Asn Ser Ile Leu Asp Met Arg Tyr Glu Asn Asn Lys
865 870 875 880
Phe Ile Asp Ile Ser Gly Tyr Gly Ser Asn Ile Ser Ile Asn Gly Asp
885 890 895
Val Tyr Ile Tyr Ser Thr Asn Arg Asn Gln Phe Gly Ile Tyr Ser Ser
900 905 910
Lys Pro Ser Glu Val Asn Ile Ala Gln Asn Asn Asp Ile Ile Tyr Asn
915 920 925
Gly Arg Tyr Gln Asn Phe Ser Ile Ser Phe Trp Val Arg Ile Pro Lys
930 935 940
Tyr Phe Asn Lys Val Asn Leu Asn Asn Glu Tyr Thr Ile Ile Asp Cys
945 950 955 960
Ile Arg Asn Asn Asn Ser Gly Trp Lys Ile Ser Leu Asn Tyr Asn Lys
965 970 975
Ile Ile Trp Thr Leu Gln Asp Thr Ala Gly Asn Asn Gln Lys Leu Val
980 985 990
Phe Asn Tyr Thr Gln Met Ile Ser Ile Ser Asp Tyr Ile Asn Lys Trp
995 1000 1005
Ile Phe Val Thr Ile Thr Asn Asn Arg Leu Gly Asn Ser Arg Ile
1010 1015 1020
Tyr Ile Asn Gly Asn Leu Ile Asp Glu Lys Ser Ile Ser Asn Leu
1025 1030 1035
Gly Asp Ile His Val Ser Asp Asn Ile Leu Phe Lys Ile Val Gly
1040 1045 1050
Cys Asn Asp Thr Arg Tyr Val Gly Ile Arg Tyr Phe Lys Val Phe
1055 1060 1065
Asp Thr Glu Leu Gly Lys Thr Glu Ile Glu Thr Leu Tyr Ser Asp
1070 1075 1080
Glu Pro Asp Pro Ser Ile Leu Lys Asp Phe Trp Gly Asn Tyr Leu
1085 1090 1095
Leu Tyr Asn Lys Arg Tyr Tyr Leu Leu Asn Leu Leu Arg Thr Asp
1100 1105 1110
Lys Ser Ile Thr Gln Asn Ser Asn Phe Leu Asn Ile Asn Gln Gln
1115 1120 1125
Arg Gly Val Tyr Gln Lys Pro Asn Ile Phe Ser Asn Thr Arg Leu
1130 1135 1140
Tyr Thr Gly Val Glu Val Ile Ile Arg Lys Asn Gly Ser Thr Asp
1145 1150 1155
Ile Ser Asn Thr Asp Asn Phe Val Arg Lys Asn Asp Leu Ala Tyr
1160 1165 1170
Ile Asn Val Val Asp Arg Asp Val Glu Tyr Arg Leu Tyr Ala Asp
1175 1180 1185
Ile Ser Ile Ala Lys Pro Glu Lys Ile Ile Lys Leu Ile Arg Thr
1190 1195 1200
Ser Asn Ser Asn Asn Ser Leu Gly Gln Ile Ile Val Met Asp Ser
1205 1210 1215
Ile Gly Asn Asn Cys Thr Met Asn Phe Gln Asn Asn Asn Gly Gly
1220 1225 1230
Asn Ile Gly Leu Leu Gly Phe His Ser Asn Asn Leu Val Ala Ser
1235 1240 1245
Ser Trp Tyr Tyr Asn Asn Ile Arg Lys Asn Thr Ser Ser Asn Gly
1250 1255 1260
Cys Phe Trp Ser Phe Ile Ser Lys Glu His Gly Trp Gln Glu Asn
1265 1270 1275
<210> 17
<211> 3942
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/A(0) (His-меченая)
<400> 17
atgccgtttg tgaacaagca gttcaactat aaagatccgg ttaatggtgt ggatatcgcc 60
tatatcaaaa ttccgaatgc aggtcagatg cagccggtta aagcctttaa aatccataac 120
aaaatttggg tgattccgga acgtgatacc tttaccaatc cggaagaagg tgatctgaat 180
ccgcctccgg aagcaaaaca ggttccggtt agctattatg atagcaccta tctgagcacc 240
gataacgaga aagataacta tctgaaaggt gtgaccaaac tgtttgaacg catttatagt 300
accgatctgg gtcgtatgct gctgaccagc attgttcgtg gtattccgtt ttggggtggt 360
agcaccattg ataccgaact gaaagttatt gacaccaact gcattaatgt gattcagccg 420
gatggtagct atcgtagcga agaactgaat ctggttatta ttggtccgag cgcagatatc 480
attcagtttg aatgtaaaag ctttggccac gaagttctga atctgacccg taatggttat 540
ggtagtaccc agtatattcg tttcagtccg gattttacct ttggctttga agaaagcctg 600
gaagttgata caaatccgct gttaggtgca ggtaaatttg caaccgatcc ggcagttacc 660
ctggcacacc agctgattta tgccggtcat cgtctgtatg gtattgccat taatccgaat 720
cgtgtgttca aagtgaatac caacgcctat tatgaaatga gcggtctgga agtgagtttt 780
gaagaactgc gtacctttgg tggtcatgat gccaaattta tcgatagcct gcaagaaaat 840
gaatttcgcc tgtactacta taacaaattc aaggatattg cgagcaccct gaataaagcc 900
aaaagcattg ttggcaccac cgcaagcctg cagtatatga aaaatgtgtt taaagaaaaa 960
tatctgctga gcgaagatac cagcggtaaa tttagcgttg acaaactgaa attcgataaa 1020
ctgtacaaga tgctgaccga gatttatacc gaagataact tcgtgaagtt tttcaaagtg 1080
ctgaaccgca aaacctacct gaactttgat aaagccgtgt tcaaaatcaa catcgtgccg 1140
aaagtgaact ataccatcta tgatggtttt aacctgcgca ataccaatct ggcagcaaac 1200
tttaatggtc agaacaccga aatcaacaac atgaacttta ccaaactgaa gaacttcacc 1260
ggtctgttcg aattttacaa actgctgtgt gttcgtggca ttattaccag caaaaccaaa 1320
agtctggata aaggctacaa taaagccctg aatgatctgt gcattaaggt gaataattgg 1380
gacctgtttt ttagcccgag cgaggataat ttcaccaacg atctgaacaa aggcgaagaa 1440
attaccagcg ataccaatat tgaagcagcc gaagaaaaca ttagcctgga tctgattcag 1500
cagtattatc tgaccttcaa cttcgataat gagccggaaa atatcagcat tgaaaacctg 1560
agcagcgata ttattggcca gctggaactg atgccgaata ttgaacgttt tccgaacggc 1620
aaaaaatacg agctggataa atacaccatg ttccattatc tgcgtgccca agaatttgaa 1680
catggtaaaa gccgtattgc actgaccaat agcgttaatg aagcactgct gaacccgagc 1740
cgtgtttata ccttttttag cagcgattac gtgaaaaagg ttaacaaagc aaccgaagca 1800
gccatgtttt taggttgggt tgaacagctg gtttatgatt tcaccgatga aaccagcgaa 1860
gttagcacca ccgataaaat tgcagatatt accatcatca tcccgtatat cggtccggca 1920
ctgaatattg gcaatatgct gtataaagac gattttgtgg gtgccctgat ttttagcggt 1980
gcagttattc tgctggaatt tattccggaa attgccattc cggttctggg cacctttgca 2040
ctggtgagct atattgcaaa taaagttctg accgtgcaga ccatcgataa tgcactgagc 2100
aaacgtaacg aaaaatggga tgaagtgtac aagtatatcg tgaccaattg gctggcaaaa 2160
gttaacaccc agattgacct gattcgcaag aagatgaaag aagcactgga aaatcaggca 2220
gaagcaacca aagccattat caactatcag tataaccagt acaccgaaga agagaaaaat 2280
aacatcaact tcaacatcga cgatctgtcc agcaaactga acgaaagcat caacaaagcc 2340
atgattaaca ttaacaaatt tctgaaccag tgcagcgtga gctatctgat gaatagcatg 2400
attccgtatg gtgtgaaacg tctggaagat tttgatgcaa gcctgaaaga tgccctgctg 2460
aaatatatct atgataatcg tggcaccctg attggtcagg ttgatcgtct gaaagataaa 2520
gtgaacaaca ccctgagtac cgatattcct tttcagctga gcaaatatgt ggataatcag 2580
cgtctgctgt caacctttac cgaatacatt aagaacatca tcaacaccag cattctgaac 2640
ctgcgttatg aaagcaatca tctgattgat ctgagccgtt atgccagcaa aatcaatata 2700
ggcagcaagg ttaacttcga cccgattgac aaaaatcaga tacagctgtt taatctggaa 2760
agcagcaaaa ttgaggtgat cctgaaaaac gccattgtgt ataatagcat gtacgagaat 2820
ttctcgacca gcttttggat tcgtatcccg aaatacttta atagcatcag cctgaacaac 2880
gagtacacca ttattaactg catggaaaac aatagcggct ggaaagttag cctgaattat 2940
ggcgaaatta tctggaccct gcaggatacc caagaaatca aacagcgtgt ggttttcaaa 3000
tacagccaga tgattaatat cagcgactat atcaaccgct ggatttttgt gaccattacc 3060
aataatcgcc tgaataacag caagatctat attaacggtc gtctgattga ccagaaaccg 3120
attagtaatc tgggtaatat tcatgcgagc aacaacatca tgtttaaact ggatggttgt 3180
cgtgataccc atcgttatat ttggatcaag tacttcaacc tgttcgataa agagttgaac 3240
gaaaaagaaa ttaaagacct gtatgataac cagagcaaca gcggtattct gaaggatttt 3300
tggggagatt atctgcagta tgacaaaccg tattatatgc tgaatctgta cgacccgaat 3360
aaatacgtgg atgtgaataa tgttggcatc cgtggttata tgtacctgaa aggtccgcgt 3420
ggtagcgtta tgaccacaaa catttatctg aatagcagcc tgtatcgcgg aaccaaattc 3480
atcattaaaa agtatgccag cggcaacaag gataatattg tgcgtaataa tgatcgcgtg 3540
tacattaacg ttgtggtgaa gaataaagaa tatcgcctgg caaccaatgc aagccaggca 3600
ggcgttgaaa aaattctgag tgccctggaa attccggatg ttggtaatct gagccaggtt 3660
gttgtgatga aaagcaaaaa tgatcagggc atcaccaaca agtgcaaaat gaatctgcag 3720
gacaataacg gcaacgatat tggttttatt ggcttccacc agttcaacaa tattgcgaaa 3780
ctggttgcaa gcaattggta taatcgtcag attgaacgta gcagtcgtac cctgggttgt 3840
agctgggaat ttatccctgt ggatgatggt tggggtgaac gtccgctgga aaacctgtat 3900
tttcaaggtg caagtcatca tcaccatcac caccatcatt aa 3942
<210> 18
<211> 1313
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/A(0) (His-меченая)
<400> 18
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Gln
210 215 220
Leu Ile Tyr Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Ile Asn Thr Ser Ile Leu Asn
865 870 875 880
Leu Arg Tyr Glu Ser Asn His Leu Ile Asp Leu Ser Arg Tyr Ala Ser
885 890 895
Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys Asn
900 905 910
Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu
915 920 925
Lys Asn Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser
930 935 940
Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Ser Ile Ser Leu Asn Asn
945 950 955 960
Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys Val
965 970 975
Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Gln Glu
980 985 990
Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile Ser
995 1000 1005
Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
1010 1015 1020
Leu Asn Asn Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln
1025 1030 1035
Lys Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Asn Ile
1040 1045 1050
Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp
1055 1060 1065
Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu
1070 1075 1080
Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met
1100 1105 1110
Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
1115 1120 1125
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
1130 1135 1140
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr
1145 1150 1155
Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile
1160 1165 1170
Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn
1175 1180 1185
Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
1190 1195 1200
Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser
1205 1210 1215
Gln Val Val Val Met Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn
1220 1225 1230
Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly
1235 1240 1245
Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
1250 1255 1260
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu
1265 1270 1275
Gly Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu
1280 1285 1290
Arg Pro Leu Glu Asn Leu Tyr Phe Gln Gly Ala Ser His His His
1295 1300 1305
His His His His His
1310
<210> 19
<211> 2649
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLHN/A (His-меченая)
<400> 19
atgccgtttg tgaacaagca gttcaactat aaagatccgg ttaatggtgt ggatatcgcc 60
tatatcaaaa ttccgaatgc aggtcagatg cagccggtta aagcctttaa aatccataac 120
aaaatttggg tgattccgga acgtgatacc tttaccaatc cggaagaagg tgatctgaat 180
ccgcctccgg aagcaaaaca ggttccggtt agctattatg atagcaccta tctgagcacc 240
gataacgaga aagataacta tctgaaaggt gtgaccaaac tgtttgaacg catttatagt 300
accgatctgg gtcgtatgct gctgaccagc attgttcgtg gtattccgtt ttggggtggt 360
agcaccattg ataccgaact gaaagttatt gacaccaact gcattaatgt gattcagccg 420
gatggtagct atcgtagcga agaactgaat ctggttatta ttggtccgag cgcagatatc 480
attcagtttg aatgtaaatc ctttggccac gaagttctga atctgacccg taatggttat 540
ggtagtaccc agtatattcg tttcagtccg gattttacct ttggctttga agaaagcctg 600
gaagttgata caaatccgct gttaggtgca ggtaaatttg caaccgatcc ggcagttacc 660
ctggcacatg aactgattca tgccggtcat cgtctgtatg gtattgcaat taatccgaac 720
cgtgtgttca aagtgaatac caacgcatat tatgaaatga gcggtctgga agtgtcattt 780
gaagaactgc gtacctttgg tggtcatgat gccaaattta tcgatagcct gcaagaaaat 840
gaatttcgcc tgtactacta taacaaattc aaggatattg cgagcaccct gaataaagcc 900
aaaagcattg ttggcaccac cgcaagcctg cagtatatga aaaatgtgtt taaagaaaaa 960
tatctgctga gcgaagatac cagcggtaaa tttagcgttg acaaactgaa attcgataaa 1020
ctgtacaaga tgctgaccga gatttatacc gaagataact tcgtgaagtt tttcaaagtg 1080
ctgaaccgca aaacctacct gaactttgat aaagccgtgt tcaaaatcaa catcgtgccg 1140
aaagtgaact ataccatcta tgatggtttt aacctgcgca ataccaatct ggcagcaaac 1200
tttaatggtc agaacaccga aatcaacaac atgaacttta ccaaactgaa gaacttcacc 1260
ggtctgttcg aattttacaa actgctgtgt gttcgtggca ttattaccag caaaaccaaa 1320
agtctggata aaggctacaa taaagccctg aatgatctgt gcattaaggt gaataattgg 1380
gacctgtttt ttagcccgag cgaggataat ttcaccaacg atctgaacaa aggcgaagaa 1440
attaccagcg ataccaatat tgaagcagcc gaagaaaaca ttagcctgga tctgattcag 1500
cagtattatc tgaccttcaa cttcgataat gagccggaaa atatcagcat tgaaaacctg 1560
agcagcgata ttattggcca gctggaactg atgccgaata ttgaacgttt tccgaacggc 1620
aaaaaatacg agctggataa atacaccatg ttccattatc tgcgtgccca agaatttgaa 1680
catggtaaaa gccgtattgc actgaccaat agcgttaatg aagcactgct gaacccgagc 1740
cgtgtttata ccttttttag cagcgattac gtgaaaaagg ttaacaaagc aaccgaagca 1800
gccatgtttt taggttgggt tgaacagctg gtttatgatt tcaccgatga aaccagcgaa 1860
gttagcacca ccgataaaat tgcagatatt accatcatca tcccgtatat cggtccggca 1920
ctgaatattg gcaatatgct gtataaagac gattttgtgg gtgccctgat ttttagcggt 1980
gcagttattc tgctggaatt tattccggaa attgccattc cggttctggg cacctttgca 2040
ctggtgagct atattgcaaa taaagttctg accgtgcaga ccatcgataa tgcactgagc 2100
aaacgtaacg aaaaatggga tgaagtgtac aagtatatcg tgaccaattg gctggcaaaa 2160
gttaacaccc agattgacct gattcgcaag aagatgaaag aagcactgga aaatcaggca 2220
gaagcaacca aagccattat caactatcag tataaccagt acaccgaaga agagaaaaat 2280
aacatcaact tcaacatcga cgatctgtcc agcaaactga acgaaagcat caacaaagcc 2340
atgattaaca ttaacaaatt tctgaaccag tgcagcgtga gctatctgat gaatagcatg 2400
attccgtatg gtgtgaaacg tctggaagat tttgatgcaa gcctgaaaga tgccctgctg 2460
aaatatatct atgataatcg tggcaccctg attggtcagg ttgatcgtct gaaagataaa 2520
gtgaacaaca ccctgagtac cgatattcct tttcagctga gcaaatatgt ggataatcag 2580
cgtctgctgt caaccgaaaa tctgtatttc cagggtgcaa gtcatcatca ccatcaccac 2640
catcattaa 2649
<210> 20
<211> 882
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLHN/A (His-меченая)
<400> 20
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Glu Asn Leu Tyr Phe Gln Gly Ala Ser His His His His His His
865 870 875 880
His His
<210> 21
<211> 1323
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/A (His-меченая)
<400> 21
atgcatcatc accatcacca cgaaaatcta tacttccaag gaaaaaacat catcaatact 60
agcattctga acctgcgtta cgagagcaat catctgattg atctgagccg ttatgcaagc 120
aagatcaaca tcggtagcaa ggtcaatttt gacccgatcg ataagaacca gatccagctg 180
tttaatctgg aatcgagcaa aattgaggtt atcctgaaaa acgccattgt ctacaactcc 240
atgtacgaga atttctccac cagcttctgg attcgcatcc cgaaatactt caacagcatt 300
agcctgaaca acgagtatac tatcatcaac tgtatggaga acaacagcgg ttggaaggtg 360
tctctgaact atggtgagat catttggacc ttgcaggaca cccaagagat caagcagcgc 420
gtcgtgttca agtactctca aatgatcaac atttccgatt acattaatcg ttggatcttc 480
gtgaccatta cgaataaccg tctgaataac agcaagattt acatcaatgg tcgcttgatc 540
gatcagaaac cgattagcaa cctgggtaat atccacgcaa gcaacaacat tatgttcaaa 600
ttggacggtt gccgcgatac ccatcgttat atctggatca agtatttcaa cctgtttgat 660
aaagaactga atgagaagga gatcaaagat ttgtatgaca accaatctaa cagcggcatt 720
ttgaaggact tctggggcga ttatctgcaa tacgataagc cgtactatat gctgaacctg 780
tatgatccga acaaatatgt ggatgtcaat aatgtgggta ttcgtggtta catgtatttg 840
aagggtccgc gtggcagcgt tatgacgacc aacatttacc tgaactctag cctgtaccgt 900
ggtacgaaat tcatcattaa gaaatatgcc agcggcaaca aagataacat tgtgcgtaat 960
aacgatcgtg tctacatcaa cgtggtcgtg aagaataaag agtaccgtct ggcgaccaac 1020
gcttcgcagg cgggtgttga gaaaattctg agcgcgttgg agatccctga tgtcggtaat 1080
ctgagccaag tcgtggttat gaagagcaag aacgaccagg gtatcactaa caagtgcaag 1140
atgaacctgc aagacaacaa tggtaacgac atcggcttta ttggtttcca ccagttcaac 1200
aatattgcta aactggtagc gagcaattgg tacaatcgtc agattgagcg cagcagccgt 1260
actttgggct gtagctggga gtttatcccg gtcgatgatg gttggggcga acgtccgctg 1320
taa 1323
<210> 22
<211> 440
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/A (His-меченая)
<400> 22
Met His His His His His His Glu Asn Leu Tyr Phe Gln Gly Lys Asn
1 5 10 15
Ile Ile Asn Thr Ser Ile Leu Asn Leu Arg Tyr Glu Ser Asn His Leu
20 25 30
Ile Asp Leu Ser Arg Tyr Ala Ser Lys Ile Asn Ile Gly Ser Lys Val
35 40 45
Asn Phe Asp Pro Ile Asp Lys Asn Gln Ile Gln Leu Phe Asn Leu Glu
50 55 60
Ser Ser Lys Ile Glu Val Ile Leu Lys Asn Ala Ile Val Tyr Asn Ser
65 70 75 80
Met Tyr Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile Pro Lys Tyr
85 90 95
Phe Asn Ser Ile Ser Leu Asn Asn Glu Tyr Thr Ile Ile Asn Cys Met
100 105 110
Glu Asn Asn Ser Gly Trp Lys Val Ser Leu Asn Tyr Gly Glu Ile Ile
115 120 125
Trp Thr Leu Gln Asp Thr Gln Glu Ile Lys Gln Arg Val Val Phe Lys
130 135 140
Tyr Ser Gln Met Ile Asn Ile Ser Asp Tyr Ile Asn Arg Trp Ile Phe
145 150 155 160
Val Thr Ile Thr Asn Asn Arg Leu Asn Asn Ser Lys Ile Tyr Ile Asn
165 170 175
Gly Arg Leu Ile Asp Gln Lys Pro Ile Ser Asn Leu Gly Asn Ile His
180 185 190
Ala Ser Asn Asn Ile Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His
195 200 205
Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn
210 215 220
Glu Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile
225 230 235 240
Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr
245 250 255
Met Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
260 265 270
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val Met
275 280 285
Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr Lys Phe
290 295 300
Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile Val Arg Asn
305 310 315 320
Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn Lys Glu Tyr Arg
325 330 335
Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu Lys Ile Leu Ser Ala
340 345 350
Leu Glu Ile Pro Asp Val Gly Asn Leu Ser Gln Val Val Val Met Lys
355 360 365
Ser Lys Asn Asp Gln Gly Ile Thr Asn Lys Cys Lys Met Asn Leu Gln
370 375 380
Asp Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly Phe His Gln Phe Asn
385 390 395 400
Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr Asn Arg Gln Ile Glu
405 410 415
Arg Ser Ser Arg Thr Leu Gly Cys Ser Trp Glu Phe Ile Pro Val Asp
420 425 430
Asp Gly Trp Gly Glu Arg Pro Leu
435 440
<210> 23
<211> 1326
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLC/A (His-меченая)
<400> 23
atgccgtttg tgaacaagca gttcaactat aaagatccgg ttaatggtgt ggatatcgcc 60
tatatcaaaa ttccgaatgc aggtcagatg cagccggtta aagcctttaa aatccataac 120
aaaatttggg tgattccgga acgtgatacc tttaccaatc cggaagaagg tgatctgaat 180
ccgcctccgg aagcaaaaca ggttccggtt agctattatg atagcaccta tctgagcacc 240
gataacgaga aagataacta tctgaaaggt gtgaccaaac tgtttgaacg catttatagt 300
accgatctgg gtcgtatgct gctgaccagc attgttcgtg gtattccgtt ttggggtggt 360
agcaccattg ataccgaact gaaagttatt gacaccaact gcattaatgt gattcagccg 420
gatggtagct atcgtagcga agaactgaat ctggttatta ttggtccgag cgcagatatc 480
attcagtttg aatgtaaatc ctttggccac gaagttctga atctgacccg taatggttat 540
ggtagtaccc agtatattcg tttcagtccg gattttacct ttggctttga agaaagcctg 600
gaagttgata caaatccgct gttaggtgca ggtaaatttg caaccgatcc ggcagttacc 660
ctggcacatg aactgattca tgccggtcat cgtctgtatg gtattgcaat taatccgaac 720
cgtgtgttca aagtgaatac caacgcatat tatgaaatga gcggtctgga agtgtcattt 780
gaagaactgc gtacctttgg tggtcatgat gccaaattta tcgatagcct gcaagaaaat 840
gaatttcgcc tgtactacta taacaaattc aaggatattg cgagcaccct gaataaagcc 900
aaaagcattg ttggcaccac cgcaagcctg cagtatatga aaaatgtgtt taaagaaaaa 960
tatctgctga gcgaagatac cagcggtaaa tttagcgttg acaaactgaa attcgataaa 1020
ctgtacaaga tgctgaccga gatttatacc gaagataact tcgtgaagtt tttcaaagtg 1080
ctgaaccgca aaacctacct gaactttgat aaagccgtgt tcaaaatcaa catcgtgccg 1140
aaagtgaact ataccatcta tgatggtttt aacctgcgca ataccaatct ggcagcaaac 1200
tttaatggtc agaacaccga aatcaacaac atgaacttta ccaaactgaa gaacttcacc 1260
ggtctgtttg aagagaatct gtatttccag ggtgcaagtc atcatcacca tcaccaccat 1320
cattaa 1326
<210> 24
<211> 441
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLC/A (His-меченая)
<400> 24
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Glu Asn Leu Tyr Phe Gln Gly Ala
420 425 430
Ser His His His His His His His His
435 440
<210> 25
<211> 3918
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/FA(0) (His-меченая)
<400> 25
atgccggttg tgattaacag cttcaattat gatgatccgg tgaacgataa caccatcatt 60
tatatccgtc cgccttatta tgaaaccagc aacacctatt tcaaagcctt ccagattatg 120
gataacgtgt ggattattcc ggaacgttat cgtctgggta ttgatccgag cctgtttaat 180
ccgcctgtta gcctgaaagc aggtagtgat ggttattttg atccgaatta tctgagcacc 240
aacaccgaga aaaacaaata cctgcagatt atgatcaagc tgttcaaacg cattaatagc 300
aaaccggcag gtcagattct gctggaagaa atcaaaaatg caattccgta tctgggcaac 360
agctataccc aagaagaaca gtttaccacc aataatcgta ccgtgagctt taatgttaaa 420
ctggccaatg gtaatatcgt tcagcagatg gcaaatctga ttatttgggg tccgggtcct 480
gatctgacca caaataaaac cggtggtatc atctatagcc cgtatcagag catggaagca 540
accccgtata aagatggttt tggtagcatt atgaccgtgg aatttagtcc ggaatatgca 600
accgccttta acgatatttc aattgcaagc catagtccgt cgctgtttat caaagatccg 660
gcactgattc tgatgcacca gctgatttat gttctgcatg gtctgtatgg cacctatatc 720
accgaataca aaattacccc gaatgtggtt cagagctata tgaaagttac caaaccgatt 780
accagcgcag aatttctgac ctttggtggt cgtgatcgca atattgttcc gcagagcatt 840
cagagccagc tgtataacaa agttctgagc gattataaac gtattgccag ccgtctgaat 900
aaagttaata ccgcaaccgc actgatcaac atcgatgaat tcaaaaacct gtacgagtgg 960
aaataccagt ttgccaaaga tagcaatggt gtgtatagcg tggatctgaa caaatttgag 1020
cagctgtaca aaaaaatcta tagcttcacc gaattcaacc tggcctatga gtttaaaatc 1080
aaaacccgtc tgggttatct ggccgaaaat tttggtccgt tttatctgcc gaatctgctg 1140
gatgatagca tttataccga agtggatggt tttaacattg gtgcactgag cattaactat 1200
cagggtcaga atattggcag cgatatcaac agcatcaaaa aactgcaagg tcagggtgtt 1260
gttagccgtg ttgttcgtct gtgtagcaat agcaatacca aaaacagcct gtgcattacc 1320
gttaataatc gcgacctgtt ttttatcgca agccaagaaa gctatggcga gaataccatt 1380
aacacctata aagagattga cgataccacc acactggatc cgagctttga agatattctg 1440
gataaagtga tcctgaactt caacgaacag gttattccgc agatgccgaa tcgtaatgtt 1500
agcaccgata ttcagaaaga caactacatc ccgaaatacg attataaccg caccgacatt 1560
atcgatagct atgaagttgg tcgcaactac aacacctttt tctatctgaa tgcccagaaa 1620
tttagcccga acgaaagcaa tattaccctg accagcagct ttgatacagg tctgttagaa 1680
ggtagcaaag tgtatacctt tttcagcagc gatttcatta acaacatcaa caaaccggtt 1740
caggccctgc tgtttattga atgggttaaa caggtgattc gcgattttac caccgaagca 1800
accaaaacct caaccgttga taaactgaaa gatattagcc tggtggtgcc gtatattggt 1860
ctggcactga atattggtga tgagatctac aaacagcatt ttgcagaagc agttgaactg 1920
gttggtgcag gtctgctgct ggaattttca ccggaatttc ttattccgac gctgctgatt 1980
tttaccatca aaggttatct gaccggtagc attcgcgata aagacaaaat cattaaaacc 2040
ctggataacg ccctgaatgt tcgtgatcag aaatggaaag aactgtatcg ttgggttgtt 2100
agcaaatggc tgaccaccat taatacgcag ttcaacaaac gcaaagaaca aatgtacaaa 2160
gccctgaaaa atcaggccac cgccattaaa aagatcatcg agaacaaata taacaactat 2220
accaccgatg aaaaaagcaa gatcgatagc agctataaca tcaacgaaat tgaacgcacc 2280
ctgaacgaaa aaatcaatct ggccatgaaa aacatcgagc agtttattac cgaaagcagc 2340
attgcctatc tgatcaatat catcaacaac gaaacgatcc agaaactgaa aagctatgat 2400
gacctggttc gtcgttatct gctgggttat attcgtaatc atagcagcat tctgggcaat 2460
agcgttgaag aactgaattc caaagtgaac aaccatctgg ataatggcat tccgtttgaa 2520
ctgagcagtt ataccaatga tagcctgctg atccgctact tcaataaaaa ctatggcgaa 2580
ctgaagtaca actgcattct gaacatcaaa tatgagatgg atcgtgacaa actggttgat 2640
agcagcggtt atcgtagccg tatcaatatt ggtacaggcg tcaaatttag cgagatcgat 2700
aaaaatcaag tgcagctgag caatctggaa tccagcaaaa ttgaagtcat tctgaataac 2760
ggcgtcatct ataacagcat gtatgaaaac ttttcgacca gcttttggat tcgcattccg 2820
aaatactttc gcaacatcaa taacgagtac aagatcatca gctgtatgca gaataatagc 2880
ggttgggaag tgagcctgaa ttttagcaat atgaactcga aaatcatctg gaccctgcag 2940
gataccgaag gtatcaaaaa aaccgttgtg tttcagtaca cccagaacat taacattagc 3000
gactatatca accgctggat ctttgtgacc attacaaata atcgtctgag caacagcaaa 3060
atctacatta atggtcgcct gatcaacgaa gaaagcatta gcgatctggg taatatccat 3120
gccagcaaca acattatgtt taaactggat ggttgccgtg atccgcatcg ttatatctgg 3180
attaaatact ttaacctgtt tgacaaagag ctgaacaaga aagaaattaa agatctgtac 3240
gacaaccaga gcaatagcgg tattctgaaa gatttctggg gtgattatct gcagtatgac 3300
aaaccgtatt atatgctgaa tctgtatgac ccgaataagt atctggatgt gaataatgtt 3360
ggcatccgtg gctatatgta tctgaaaggt ccgcgtggtc gtattgtgac caccaacatt 3420
tatctgaata gcaccctgta tatgggcacc aaattcatca ttaagaaata tgccagcggc 3480
aacaaagata acattgtgcg taataatgat cgcgtgtata ttaacgtggt ggtgaagaat 3540
aaagaatatc gcctggcaac caatgcaagc caggcaggcg ttgaaaaaat tctgagcgca 3600
gttgaaatcc cggatgttgg taatctgagc caggttgttg tgatgaaaag cgaaaatgat 3660
cagggcattc gcaacaagtg taaaatgaat ctgcaagaca ataacggcaa cgatattggc 3720
tttatcggct ttcaccagtt taataacatt gcaaaactgg tggccagcaa ctggtataac 3780
cgtcagattg gtaaagcaag ccgtaccttt ggttgtagct gggaatttat cccggttgat 3840
gatggttggg gtgaaagcag cctggaaaat ctgtatttcc agggtgccag tcatcatcac 3900
caccatcacc atcactga 3918
<210> 26
<211> 1305
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/FA(0) (His-меченая)
<400> 26
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asp Asp Pro Val Asn Asp
1 5 10 15
Asn Thr Ile Ile Tyr Ile Arg Pro Pro Tyr Tyr Glu Thr Ser Asn Thr
20 25 30
Tyr Phe Lys Ala Phe Gln Ile Met Asp Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Tyr Arg Leu Gly Ile Asp Pro Ser Leu Phe Asn Pro Pro Val Ser
50 55 60
Leu Lys Ala Gly Ser Asp Gly Tyr Phe Asp Pro Asn Tyr Leu Ser Thr
65 70 75 80
Asn Thr Glu Lys Asn Lys Tyr Leu Gln Ile Met Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Lys Pro Ala Gly Gln Ile Leu Leu Glu Glu Ile Lys
100 105 110
Asn Ala Ile Pro Tyr Leu Gly Asn Ser Tyr Thr Gln Glu Glu Gln Phe
115 120 125
Thr Thr Asn Asn Arg Thr Val Ser Phe Asn Val Lys Leu Ala Asn Gly
130 135 140
Asn Ile Val Gln Gln Met Ala Asn Leu Ile Ile Trp Gly Pro Gly Pro
145 150 155 160
Asp Leu Thr Thr Asn Lys Thr Gly Gly Ile Ile Tyr Ser Pro Tyr Gln
165 170 175
Ser Met Glu Ala Thr Pro Tyr Lys Asp Gly Phe Gly Ser Ile Met Thr
180 185 190
Val Glu Phe Ser Pro Glu Tyr Ala Thr Ala Phe Asn Asp Ile Ser Ile
195 200 205
Ala Ser His Ser Pro Ser Leu Phe Ile Lys Asp Pro Ala Leu Ile Leu
210 215 220
Met His Gln Leu Ile Tyr Val Leu His Gly Leu Tyr Gly Thr Tyr Ile
225 230 235 240
Thr Glu Tyr Lys Ile Thr Pro Asn Val Val Gln Ser Tyr Met Lys Val
245 250 255
Thr Lys Pro Ile Thr Ser Ala Glu Phe Leu Thr Phe Gly Gly Arg Asp
260 265 270
Arg Asn Ile Val Pro Gln Ser Ile Gln Ser Gln Leu Tyr Asn Lys Val
275 280 285
Leu Ser Asp Tyr Lys Arg Ile Ala Ser Arg Leu Asn Lys Val Asn Thr
290 295 300
Ala Thr Ala Leu Ile Asn Ile Asp Glu Phe Lys Asn Leu Tyr Glu Trp
305 310 315 320
Lys Tyr Gln Phe Ala Lys Asp Ser Asn Gly Val Tyr Ser Val Asp Leu
325 330 335
Asn Lys Phe Glu Gln Leu Tyr Lys Lys Ile Tyr Ser Phe Thr Glu Phe
340 345 350
Asn Leu Ala Tyr Glu Phe Lys Ile Lys Thr Arg Leu Gly Tyr Leu Ala
355 360 365
Glu Asn Phe Gly Pro Phe Tyr Leu Pro Asn Leu Leu Asp Asp Ser Ile
370 375 380
Tyr Thr Glu Val Asp Gly Phe Asn Ile Gly Ala Leu Ser Ile Asn Tyr
385 390 395 400
Gln Gly Gln Asn Ile Gly Ser Asp Ile Asn Ser Ile Lys Lys Leu Gln
405 410 415
Gly Gln Gly Val Val Ser Arg Val Val Arg Leu Cys Ser Asn Ser Asn
420 425 430
Thr Lys Asn Ser Leu Cys Ile Thr Val Asn Asn Arg Asp Leu Phe Phe
435 440 445
Ile Ala Ser Gln Glu Ser Tyr Gly Glu Asn Thr Ile Asn Thr Tyr Lys
450 455 460
Glu Ile Asp Asp Thr Thr Thr Leu Asp Pro Ser Phe Glu Asp Ile Leu
465 470 475 480
Asp Lys Val Ile Leu Asn Phe Asn Glu Gln Val Ile Pro Gln Met Pro
485 490 495
Asn Arg Asn Val Ser Thr Asp Ile Gln Lys Asp Asn Tyr Ile Pro Lys
500 505 510
Tyr Asp Tyr Asn Arg Thr Asp Ile Ile Asp Ser Tyr Glu Val Gly Arg
515 520 525
Asn Tyr Asn Thr Phe Phe Tyr Leu Asn Ala Gln Lys Phe Ser Pro Asn
530 535 540
Glu Ser Asn Ile Thr Leu Thr Ser Ser Phe Asp Thr Gly Leu Leu Glu
545 550 555 560
Gly Ser Lys Val Tyr Thr Phe Phe Ser Ser Asp Phe Ile Asn Asn Ile
565 570 575
Asn Lys Pro Val Gln Ala Leu Leu Phe Ile Glu Trp Val Lys Gln Val
580 585 590
Ile Arg Asp Phe Thr Thr Glu Ala Thr Lys Thr Ser Thr Val Asp Lys
595 600 605
Leu Lys Asp Ile Ser Leu Val Val Pro Tyr Ile Gly Leu Ala Leu Asn
610 615 620
Ile Gly Asp Glu Ile Tyr Lys Gln His Phe Ala Glu Ala Val Glu Leu
625 630 635 640
Val Gly Ala Gly Leu Leu Leu Glu Phe Ser Pro Glu Phe Leu Ile Pro
645 650 655
Thr Leu Leu Ile Phe Thr Ile Lys Gly Tyr Leu Thr Gly Ser Ile Arg
660 665 670
Asp Lys Asp Lys Ile Ile Lys Thr Leu Asp Asn Ala Leu Asn Val Arg
675 680 685
Asp Gln Lys Trp Lys Glu Leu Tyr Arg Trp Val Val Ser Lys Trp Leu
690 695 700
Thr Thr Ile Asn Thr Gln Phe Asn Lys Arg Lys Glu Gln Met Tyr Lys
705 710 715 720
Ala Leu Lys Asn Gln Ala Thr Ala Ile Lys Lys Ile Ile Glu Asn Lys
725 730 735
Tyr Asn Asn Tyr Thr Thr Asp Glu Lys Ser Lys Ile Asp Ser Ser Tyr
740 745 750
Asn Ile Asn Glu Ile Glu Arg Thr Leu Asn Glu Lys Ile Asn Leu Ala
755 760 765
Met Lys Asn Ile Glu Gln Phe Ile Thr Glu Ser Ser Ile Ala Tyr Leu
770 775 780
Ile Asn Ile Ile Asn Asn Glu Thr Ile Gln Lys Leu Lys Ser Tyr Asp
785 790 795 800
Asp Leu Val Arg Arg Tyr Leu Leu Gly Tyr Ile Arg Asn His Ser Ser
805 810 815
Ile Leu Gly Asn Ser Val Glu Glu Leu Asn Ser Lys Val Asn Asn His
820 825 830
Leu Asp Asn Gly Ile Pro Phe Glu Leu Ser Ser Tyr Thr Asn Asp Ser
835 840 845
Leu Leu Ile Arg Tyr Phe Asn Lys Asn Tyr Gly Glu Leu Lys Tyr Asn
850 855 860
Cys Ile Leu Asn Ile Lys Tyr Glu Met Asp Arg Asp Lys Leu Val Asp
865 870 875 880
Ser Ser Gly Tyr Arg Ser Arg Ile Asn Ile Gly Thr Gly Val Lys Phe
885 890 895
Ser Glu Ile Asp Lys Asn Gln Val Gln Leu Ser Asn Leu Glu Ser Ser
900 905 910
Lys Ile Glu Val Ile Leu Asn Asn Gly Val Ile Tyr Asn Ser Met Tyr
915 920 925
Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile Pro Lys Tyr Phe Arg
930 935 940
Asn Ile Asn Asn Glu Tyr Lys Ile Ile Ser Cys Met Gln Asn Asn Ser
945 950 955 960
Gly Trp Glu Val Ser Leu Asn Phe Ser Asn Met Asn Ser Lys Ile Ile
965 970 975
Trp Thr Leu Gln Asp Thr Glu Gly Ile Lys Lys Thr Val Val Phe Gln
980 985 990
Tyr Thr Gln Asn Ile Asn Ile Ser Asp Tyr Ile Asn Arg Trp Ile Phe
995 1000 1005
Val Thr Ile Thr Asn Asn Arg Leu Ser Asn Ser Lys Ile Tyr Ile
1010 1015 1020
Asn Gly Arg Leu Ile Asn Glu Glu Ser Ile Ser Asp Leu Gly Asn
1025 1030 1035
Ile His Ala Ser Asn Asn Ile Met Phe Lys Leu Asp Gly Cys Arg
1040 1045 1050
Asp Pro His Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe Asp
1055 1060 1065
Lys Glu Leu Asn Lys Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln
1070 1075 1080
Ser Asn Ser Gly Ile Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln
1085 1090 1095
Tyr Asp Lys Pro Tyr Tyr Met Leu Asn Leu Tyr Asp Pro Asn Lys
1100 1105 1110
Tyr Leu Asp Val Asn Asn Val Gly Ile Arg Gly Tyr Met Tyr Leu
1115 1120 1125
Lys Gly Pro Arg Gly Arg Ile Val Thr Thr Asn Ile Tyr Leu Asn
1130 1135 1140
Ser Thr Leu Tyr Met Gly Thr Lys Phe Ile Ile Lys Lys Tyr Ala
1145 1150 1155
Ser Gly Asn Lys Asp Asn Ile Val Arg Asn Asn Asp Arg Val Tyr
1160 1165 1170
Ile Asn Val Val Val Lys Asn Lys Glu Tyr Arg Leu Ala Thr Asn
1175 1180 1185
Ala Ser Gln Ala Gly Val Glu Lys Ile Leu Ser Ala Val Glu Ile
1190 1195 1200
Pro Asp Val Gly Asn Leu Ser Gln Val Val Val Met Lys Ser Glu
1205 1210 1215
Asn Asp Gln Gly Ile Arg Asn Lys Cys Lys Met Asn Leu Gln Asp
1220 1225 1230
Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly Phe His Gln Phe Asn
1235 1240 1245
Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr Asn Arg Gln Ile
1250 1255 1260
Gly Lys Ala Ser Arg Thr Phe Gly Cys Ser Trp Glu Phe Ile Pro
1265 1270 1275
Val Asp Asp Gly Trp Gly Glu Ser Ser Leu Glu Asn Leu Tyr Phe
1280 1285 1290
Gln Gly Ala Ser His His His His His His His His
1295 1300 1305
<210> 27
<211> 2634
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLHN/FA (His-меченая)
<400> 27
atgccggttg tgattaacag cttcaattat gatgatccgg tgaacgataa caccatcatt 60
tatatccgtc cgccttatta tgaaaccagc aacacctatt tcaaagcctt ccagattatg 120
gataacgtgt ggattattcc ggaacgttat cgtctgggta ttgatccgag cctgtttaat 180
ccgcctgtta gcctgaaagc aggtagtgat ggttattttg atccgaatta tctgagcacc 240
aacaccgaga aaaacaaata cctgcagatt atgatcaagc tgttcaaacg cattaatagc 300
aaaccggcag gtcagattct gctggaagaa atcaaaaatg caattccgta tctgggcaac 360
agctataccc aagaagaaca gtttaccacc aataatcgta ccgtgagctt taatgttaaa 420
ctggccaatg gtaatatcgt tcagcagatg gcaaatctga ttatttgggg tccgggtcct 480
gatctgacca caaataaaac cggtggtatc atctatagcc cgtatcagag catggaagca 540
accccgtata aagatggttt tggtagcatt atgaccgtgg aatttagtcc ggaatatgca 600
accgccttta acgatatttc aattgcaagc catagtccgt cgctgtttat caaagatccg 660
gcactgattc tgatgcatga actgattcat gttctgcatg gtctgtatgg cacctatatt 720
accgaataca aaattacccc gaatgtggtg cagagctata tgaaagttac caaaccgatt 780
accagcgcag aatttctgac ctttggtggt cgtgatcgca atattgttcc gcagagcatt 840
cagagccagc tgtataacaa agttctgagc gattataaac gtattgccag ccgtctgaat 900
aaagttaata ccgcaaccgc actgatcaac atcgatgaat tcaaaaacct gtacgagtgg 960
aaataccagt ttgccaaaga tagcaatggt gtgtatagcg tggatctgaa caaatttgag 1020
cagctgtaca aaaaaatcta tagcttcacc gaattcaacc tggcctatga gtttaaaatc 1080
aaaacccgtc tgggttatct ggccgaaaat tttggtccgt tttatctgcc gaatctgctg 1140
gatgatagca tttataccga agtggatggt tttaacattg gtgcactgag cattaactat 1200
cagggtcaga atattggcag cgatatcaac agcatcaaaa aactgcaagg tcagggtgtt 1260
gttagccgtg ttgttcgtct gtgtagcaat agcaatacca aaaacagcct gtgcattacc 1320
gttaataatc gcgacctgtt ttttatcgca agccaagaaa gctatggcga gaataccatt 1380
aacacctata aagagattga cgataccacc acactggatc cgagctttga agatattctg 1440
gataaagtga tcctgaactt caacgaacag gttattccgc agatgccgaa tcgtaatgtt 1500
agcaccgata ttcagaaaga caactacatc ccgaaatacg attataaccg caccgacatt 1560
atcgatagct atgaagttgg tcgcaactac aacacctttt tctatctgaa tgcccagaaa 1620
tttagcccga acgaaagcaa tattaccctg accagcagct ttgatacagg tctgttagaa 1680
ggtagcaaag tgtatacctt tttcagcagc gatttcatta acaacatcaa caaaccggtt 1740
caggccctgc tgtttattga atgggttaaa caggtgattc gcgattttac caccgaagca 1800
accaaaacct caaccgttga taaactgaaa gatattagcc tggtggtgcc gtatattggt 1860
ctggcactga atattggtga tgagatctac aaacagcatt ttgcagaagc agttgaactg 1920
gttggtgcag gtctgctgct ggaattttca ccggaatttc ttattccgac gctgctgatt 1980
tttaccatca aaggttatct gaccggtagc attcgcgata aagacaaaat cattaaaacc 2040
ctggataacg ccctgaatgt tcgtgatcag aaatggaaag aactgtatcg ttgggttgtt 2100
agcaaatggc tgaccaccat taatacgcag ttcaacaaac gcaaagaaca aatgtacaaa 2160
gccctgaaaa atcaggccac cgccattaaa aagatcatcg agaacaaata taacaactat 2220
accaccgatg aaaaaagcaa gatcgatagc agctataaca tcaacgaaat tgaacgcacc 2280
ctgaacgaaa aaatcaatct ggccatgaaa aacatcgagc agtttattac agaaagcagc 2340
attgcctacc tgatcaatat catcaacaac gaaaccattc agaaactgaa aagctatgat 2400
gacctggttc gtcgttatct gctgggttat attcgtaatc atagcagcat tctgggcaat 2460
agcgttgaag aactgaattc caaagtgaac aaccatctgg ataatggcat tccgtttgaa 2520
ctgagcagtt ataccaatga tagcctgctg atccgctact tcaataaaaa ctatggcgaa 2580
gagaacctgt atttccaggg tgccagtcat catcaccacc atcaccatca ctga 2634
<210> 28
<211> 877
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLHN/FA (His-меченая)
<400> 28
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asp Asp Pro Val Asn Asp
1 5 10 15
Asn Thr Ile Ile Tyr Ile Arg Pro Pro Tyr Tyr Glu Thr Ser Asn Thr
20 25 30
Tyr Phe Lys Ala Phe Gln Ile Met Asp Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Tyr Arg Leu Gly Ile Asp Pro Ser Leu Phe Asn Pro Pro Val Ser
50 55 60
Leu Lys Ala Gly Ser Asp Gly Tyr Phe Asp Pro Asn Tyr Leu Ser Thr
65 70 75 80
Asn Thr Glu Lys Asn Lys Tyr Leu Gln Ile Met Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Lys Pro Ala Gly Gln Ile Leu Leu Glu Glu Ile Lys
100 105 110
Asn Ala Ile Pro Tyr Leu Gly Asn Ser Tyr Thr Gln Glu Glu Gln Phe
115 120 125
Thr Thr Asn Asn Arg Thr Val Ser Phe Asn Val Lys Leu Ala Asn Gly
130 135 140
Asn Ile Val Gln Gln Met Ala Asn Leu Ile Ile Trp Gly Pro Gly Pro
145 150 155 160
Asp Leu Thr Thr Asn Lys Thr Gly Gly Ile Ile Tyr Ser Pro Tyr Gln
165 170 175
Ser Met Glu Ala Thr Pro Tyr Lys Asp Gly Phe Gly Ser Ile Met Thr
180 185 190
Val Glu Phe Ser Pro Glu Tyr Ala Thr Ala Phe Asn Asp Ile Ser Ile
195 200 205
Ala Ser His Ser Pro Ser Leu Phe Ile Lys Asp Pro Ala Leu Ile Leu
210 215 220
Met His Glu Leu Ile His Val Leu His Gly Leu Tyr Gly Thr Tyr Ile
225 230 235 240
Thr Glu Tyr Lys Ile Thr Pro Asn Val Val Gln Ser Tyr Met Lys Val
245 250 255
Thr Lys Pro Ile Thr Ser Ala Glu Phe Leu Thr Phe Gly Gly Arg Asp
260 265 270
Arg Asn Ile Val Pro Gln Ser Ile Gln Ser Gln Leu Tyr Asn Lys Val
275 280 285
Leu Ser Asp Tyr Lys Arg Ile Ala Ser Arg Leu Asn Lys Val Asn Thr
290 295 300
Ala Thr Ala Leu Ile Asn Ile Asp Glu Phe Lys Asn Leu Tyr Glu Trp
305 310 315 320
Lys Tyr Gln Phe Ala Lys Asp Ser Asn Gly Val Tyr Ser Val Asp Leu
325 330 335
Asn Lys Phe Glu Gln Leu Tyr Lys Lys Ile Tyr Ser Phe Thr Glu Phe
340 345 350
Asn Leu Ala Tyr Glu Phe Lys Ile Lys Thr Arg Leu Gly Tyr Leu Ala
355 360 365
Glu Asn Phe Gly Pro Phe Tyr Leu Pro Asn Leu Leu Asp Asp Ser Ile
370 375 380
Tyr Thr Glu Val Asp Gly Phe Asn Ile Gly Ala Leu Ser Ile Asn Tyr
385 390 395 400
Gln Gly Gln Asn Ile Gly Ser Asp Ile Asn Ser Ile Lys Lys Leu Gln
405 410 415
Gly Gln Gly Val Val Ser Arg Val Val Arg Leu Cys Ser Asn Ser Asn
420 425 430
Thr Lys Asn Ser Leu Cys Ile Thr Val Asn Asn Arg Asp Leu Phe Phe
435 440 445
Ile Ala Ser Gln Glu Ser Tyr Gly Glu Asn Thr Ile Asn Thr Tyr Lys
450 455 460
Glu Ile Asp Asp Thr Thr Thr Leu Asp Pro Ser Phe Glu Asp Ile Leu
465 470 475 480
Asp Lys Val Ile Leu Asn Phe Asn Glu Gln Val Ile Pro Gln Met Pro
485 490 495
Asn Arg Asn Val Ser Thr Asp Ile Gln Lys Asp Asn Tyr Ile Pro Lys
500 505 510
Tyr Asp Tyr Asn Arg Thr Asp Ile Ile Asp Ser Tyr Glu Val Gly Arg
515 520 525
Asn Tyr Asn Thr Phe Phe Tyr Leu Asn Ala Gln Lys Phe Ser Pro Asn
530 535 540
Glu Ser Asn Ile Thr Leu Thr Ser Ser Phe Asp Thr Gly Leu Leu Glu
545 550 555 560
Gly Ser Lys Val Tyr Thr Phe Phe Ser Ser Asp Phe Ile Asn Asn Ile
565 570 575
Asn Lys Pro Val Gln Ala Leu Leu Phe Ile Glu Trp Val Lys Gln Val
580 585 590
Ile Arg Asp Phe Thr Thr Glu Ala Thr Lys Thr Ser Thr Val Asp Lys
595 600 605
Leu Lys Asp Ile Ser Leu Val Val Pro Tyr Ile Gly Leu Ala Leu Asn
610 615 620
Ile Gly Asp Glu Ile Tyr Lys Gln His Phe Ala Glu Ala Val Glu Leu
625 630 635 640
Val Gly Ala Gly Leu Leu Leu Glu Phe Ser Pro Glu Phe Leu Ile Pro
645 650 655
Thr Leu Leu Ile Phe Thr Ile Lys Gly Tyr Leu Thr Gly Ser Ile Arg
660 665 670
Asp Lys Asp Lys Ile Ile Lys Thr Leu Asp Asn Ala Leu Asn Val Arg
675 680 685
Asp Gln Lys Trp Lys Glu Leu Tyr Arg Trp Val Val Ser Lys Trp Leu
690 695 700
Thr Thr Ile Asn Thr Gln Phe Asn Lys Arg Lys Glu Gln Met Tyr Lys
705 710 715 720
Ala Leu Lys Asn Gln Ala Thr Ala Ile Lys Lys Ile Ile Glu Asn Lys
725 730 735
Tyr Asn Asn Tyr Thr Thr Asp Glu Lys Ser Lys Ile Asp Ser Ser Tyr
740 745 750
Asn Ile Asn Glu Ile Glu Arg Thr Leu Asn Glu Lys Ile Asn Leu Ala
755 760 765
Met Lys Asn Ile Glu Gln Phe Ile Thr Glu Ser Ser Ile Ala Tyr Leu
770 775 780
Ile Asn Ile Ile Asn Asn Glu Thr Ile Gln Lys Leu Lys Ser Tyr Asp
785 790 795 800
Asp Leu Val Arg Arg Tyr Leu Leu Gly Tyr Ile Arg Asn His Ser Ser
805 810 815
Ile Leu Gly Asn Ser Val Glu Glu Leu Asn Ser Lys Val Asn Asn His
820 825 830
Leu Asp Asn Gly Ile Pro Phe Glu Leu Ser Ser Tyr Thr Asn Asp Ser
835 840 845
Leu Leu Ile Arg Tyr Phe Asn Lys Asn Tyr Gly Glu Glu Asn Leu Tyr
850 855 860
Phe Gln Gly Ala Ser His His His His His His His His
865 870 875
<210> 29
<211> 1341
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/FA (His-меченая)
<400> 29
atgctgaagt ataactgcat cctgaacatc aaatatgaga tggatcgtga taaactggtt 60
gatagcagcg gttatcgtag ccgtatcaat attggcaccg gtgtgaaatt tagcgagatc 120
gataaaaatc aggtgcagct gagcaatctg gaaagcagca aaattgaagt gattctgaat 180
aacggcgtga tctacaatag catgtatgaa aacttttcga ccagcttctg gattcgcatt 240
ccgaaatact ttcgcaacat caacaacgag tacaagatta tcagctgtat gcagaataat 300
agcggttggg aagttagcct gaatttcagc aatatgaaca gcaaaatcat ttggaccctg 360
caggataccg aaggtatcaa aaaaaccgtt gtgtttcagt acacccagaa cattaacatc 420
agcgattaca ttaaccgctg gatctttgtg accattacca ataatcgtct gagcaacagc 480
aagatctata ttaacggtcg cctgattaac gaagagagca ttagcgatct gggtaatatt 540
catgccagca acaacatcat gtttaaactg gatggttgtc gtgatccgca tcgttatatt 600
tggatcaaat acttcaacct gtttgataaa gaactgaaca aaaaagaaat caaagacctg 660
tatgataacc agagcaatag cggcattctg aaagattttt ggggtgatta tctgcagtat 720
gacaaaccgt attacatgct gaatctgtac gatccgaaca aatatctgga tgtgaataat 780
gtgggtatcc gtggctatat gtatctgaaa ggtccgcgtg gtcgtattgt taccaccaac 840
atttatctga atagcaccct gtatatgggc accaaattca tcattaaaaa gtatgccagc 900
ggcaacaaag ataacattgt gcgtaataat gatcgcgtgt atatcaatgt ggtggtgaag 960
aataaagaat atcgtctggc caccaatgca agccaggcag gcgttgaaaa aattctgagc 1020
gcagttgaaa ttccggatgt tggtaatctg agccaggttg ttgttatgaa aagcgaaaat 1080
gatcagggca ttcgcaacaa atgcaaaatg aatctgcagg acaataacgg caacgatatt 1140
ggttttattg gcttccacca gttcaacaac attgcaaaac tggtggcgag caattggtat 1200
aatcgtcaga ttggtaaagc aagccgtacc tttggttgta gctgggaatt tattccggtt 1260
gatgatggtt ggggtgaaag cagcctggaa aatctgtatt ttcagggtgc aagtcatcat 1320
caccaccatc accatcatta a 1341
<210> 30
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/FA (His-меченая)
<400> 30
Met Leu Lys Tyr Asn Cys Ile Leu Asn Ile Lys Tyr Glu Met Asp Arg
1 5 10 15
Asp Lys Leu Val Asp Ser Ser Gly Tyr Arg Ser Arg Ile Asn Ile Gly
20 25 30
Thr Gly Val Lys Phe Ser Glu Ile Asp Lys Asn Gln Val Gln Leu Ser
35 40 45
Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu Asn Asn Gly Val Ile
50 55 60
Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile
65 70 75 80
Pro Lys Tyr Phe Arg Asn Ile Asn Asn Glu Tyr Lys Ile Ile Ser Cys
85 90 95
Met Gln Asn Asn Ser Gly Trp Glu Val Ser Leu Asn Phe Ser Asn Met
100 105 110
Asn Ser Lys Ile Ile Trp Thr Leu Gln Asp Thr Glu Gly Ile Lys Lys
115 120 125
Thr Val Val Phe Gln Tyr Thr Gln Asn Ile Asn Ile Ser Asp Tyr Ile
130 135 140
Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg Leu Ser Asn Ser
145 150 155 160
Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asn Glu Glu Ser Ile Ser Asp
165 170 175
Leu Gly Asn Ile His Ala Ser Asn Asn Ile Met Phe Lys Leu Asp Gly
180 185 190
Cys Arg Asp Pro His Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe
195 200 205
Asp Lys Glu Leu Asn Lys Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln
210 215 220
Ser Asn Ser Gly Ile Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln Tyr
225 230 235 240
Asp Lys Pro Tyr Tyr Met Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Leu
245 250 255
Asp Val Asn Asn Val Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro
260 265 270
Arg Gly Arg Ile Val Thr Thr Asn Ile Tyr Leu Asn Ser Thr Leu Tyr
275 280 285
Met Gly Thr Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp
290 295 300
Asn Ile Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys
305 310 315 320
Asn Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
325 330 335
Lys Ile Leu Ser Ala Val Glu Ile Pro Asp Val Gly Asn Leu Ser Gln
340 345 350
Val Val Val Met Lys Ser Glu Asn Asp Gln Gly Ile Arg Asn Lys Cys
355 360 365
Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly
370 375 380
Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr
385 390 395 400
Asn Arg Gln Ile Gly Lys Ala Ser Arg Thr Phe Gly Cys Ser Trp Glu
405 410 415
Phe Ile Pro Val Asp Asp Gly Trp Gly Glu Ser Ser Leu Glu Asn Leu
420 425 430
Tyr Phe Gln Gly Ala Ser His His His His His His His His
435 440 445
<210> 31
<211> 1347
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLC/FA (His-меченая)
<400> 31
atgccggttg tgattaacag cttcaattat gatgatccgg tgaacgataa caccatcatt 60
tatatccgtc cgccttatta tgaaaccagc aacacctatt tcaaagcctt ccagattatg 120
gataacgtgt ggattattcc ggaacgttat cgtctgggta ttgatccgag cctgtttaat 180
ccgcctgtta gcctgaaagc aggtagtgat ggttattttg atccgaatta tctgagcacc 240
aacaccgaga aaaacaaata cctgcagatt atgatcaagc tgttcaaacg cattaatagc 300
aaaccggcag gtcagattct gctggaagaa atcaaaaatg caattccgta tctgggcaac 360
agctataccc aagaagaaca gtttaccacc aataatcgta ccgtgagctt taatgttaaa 420
ctggccaatg gtaatatcgt tcagcagatg gcaaatctga ttatttgggg tccgggtcct 480
gatctgacca caaataaaac cggtggtatc atctatagcc cgtatcagag catggaagca 540
accccgtata aagatggttt tggtagcatt atgaccgtgg aatttagtcc ggaatatgca 600
accgccttta acgatatttc aattgcaagc catagtccgt cgctgtttat caaagatccg 660
gcactgattc tgatgcatga actgattcat gttctgcatg gtctgtatgg cacctatatt 720
accgaataca aaattacccc gaatgtggtg cagagctata tgaaagttac caaaccgatt 780
accagcgcag aatttctgac ctttggtggt cgtgatcgca atattgttcc gcagagcatt 840
cagagccagc tgtataacaa agttctgagc gattataaac gtattgccag ccgtctgaat 900
aaagttaata ccgcaaccgc actgatcaac atcgatgaat tcaaaaacct gtacgagtgg 960
aaataccagt ttgccaaaga tagcaatggt gtgtatagcg tggatctgaa caaatttgag 1020
cagctgtaca aaaaaatcta tagcttcacc gaattcaacc tggcctatga gtttaaaatc 1080
aaaacccgtc tgggttatct ggccgaaaat tttggtccgt tttatctgcc gaatctgctg 1140
gatgatagca tttataccga agtggatggt tttaacattg gtgcactgag cattaactat 1200
cagggtcaga atattggcag cgatatcaac agcatcaaaa aactgcaagg tcagggtgtt 1260
gttagccgtg ttgttcgtct gtgtagcaat agcgaaaatc tgtattttca gggtgccagt 1320
catcatcacc accatcacca tcactga 1347
<210> 32
<211> 448
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLC/FA (His-меченая)
<400> 32
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asp Asp Pro Val Asn Asp
1 5 10 15
Asn Thr Ile Ile Tyr Ile Arg Pro Pro Tyr Tyr Glu Thr Ser Asn Thr
20 25 30
Tyr Phe Lys Ala Phe Gln Ile Met Asp Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Tyr Arg Leu Gly Ile Asp Pro Ser Leu Phe Asn Pro Pro Val Ser
50 55 60
Leu Lys Ala Gly Ser Asp Gly Tyr Phe Asp Pro Asn Tyr Leu Ser Thr
65 70 75 80
Asn Thr Glu Lys Asn Lys Tyr Leu Gln Ile Met Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Lys Pro Ala Gly Gln Ile Leu Leu Glu Glu Ile Lys
100 105 110
Asn Ala Ile Pro Tyr Leu Gly Asn Ser Tyr Thr Gln Glu Glu Gln Phe
115 120 125
Thr Thr Asn Asn Arg Thr Val Ser Phe Asn Val Lys Leu Ala Asn Gly
130 135 140
Asn Ile Val Gln Gln Met Ala Asn Leu Ile Ile Trp Gly Pro Gly Pro
145 150 155 160
Asp Leu Thr Thr Asn Lys Thr Gly Gly Ile Ile Tyr Ser Pro Tyr Gln
165 170 175
Ser Met Glu Ala Thr Pro Tyr Lys Asp Gly Phe Gly Ser Ile Met Thr
180 185 190
Val Glu Phe Ser Pro Glu Tyr Ala Thr Ala Phe Asn Asp Ile Ser Ile
195 200 205
Ala Ser His Ser Pro Ser Leu Phe Ile Lys Asp Pro Ala Leu Ile Leu
210 215 220
Met His Glu Leu Ile His Val Leu His Gly Leu Tyr Gly Thr Tyr Ile
225 230 235 240
Thr Glu Tyr Lys Ile Thr Pro Asn Val Val Gln Ser Tyr Met Lys Val
245 250 255
Thr Lys Pro Ile Thr Ser Ala Glu Phe Leu Thr Phe Gly Gly Arg Asp
260 265 270
Arg Asn Ile Val Pro Gln Ser Ile Gln Ser Gln Leu Tyr Asn Lys Val
275 280 285
Leu Ser Asp Tyr Lys Arg Ile Ala Ser Arg Leu Asn Lys Val Asn Thr
290 295 300
Ala Thr Ala Leu Ile Asn Ile Asp Glu Phe Lys Asn Leu Tyr Glu Trp
305 310 315 320
Lys Tyr Gln Phe Ala Lys Asp Ser Asn Gly Val Tyr Ser Val Asp Leu
325 330 335
Asn Lys Phe Glu Gln Leu Tyr Lys Lys Ile Tyr Ser Phe Thr Glu Phe
340 345 350
Asn Leu Ala Tyr Glu Phe Lys Ile Lys Thr Arg Leu Gly Tyr Leu Ala
355 360 365
Glu Asn Phe Gly Pro Phe Tyr Leu Pro Asn Leu Leu Asp Asp Ser Ile
370 375 380
Tyr Thr Glu Val Asp Gly Phe Asn Ile Gly Ala Leu Ser Ile Asn Tyr
385 390 395 400
Gln Gly Gln Asn Ile Gly Ser Asp Ile Asn Ser Ile Lys Lys Leu Gln
405 410 415
Gly Gln Gly Val Val Ser Arg Val Val Arg Leu Cys Ser Asn Ser Glu
420 425 430
Asn Leu Tyr Phe Gln Gly Ala Ser His His His His His His His His
435 440 445
<210> 33
<211> 3888
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rBoNT/F(0) (His-меченая)
<400> 33
atgccggttg tgattaacag cttcaattat aacgatccgg tgaacgatga taccatcctg 60
tatatgcaga ttccgtatga agagaaaagc aaaaagtact acaaagcctt tgagatcatg 120
cgcaacgttt ggattattcc ggaacgtaat accattggca ccgatccgag cgattttgat 180
ccgcctgcaa gcctggaaaa tggtagcagc gcatattatg atccgaatta tctgaccacc 240
gatgccgaaa aagatcgtta tctgaaaacc accatcaaac tgttcaaacg cattaatagc 300
aatccggcag gcgaagttct gctgcaagaa attagctatg caaaaccgta tctgggcaat 360
gaacataccc cgattaatga atttcatccg gttacacgta ccacgagcgt taacattaaa 420
agcagcacca atgtgaagtc cagcattatt ctgaatctgc tggttttagg tgcaggtccg 480
gatatttttg aaaattcaag ctatccggtg cgcaaactga tggatagcgg tggtgtgtat 540
gatccgtcaa atgatggttt tggcagcatt aacattgtga cctttagtcc ggaatatgaa 600
tacaccttca acgatattag cggtggctat aatagcagca ccgaaagttt tattgcagat 660
ccggcaatta gcctggcaca ccagctgatt tatgcactgc atggtctgta tggtgcacgt 720
ggtgttacct ataaagaaac cattaaagtt aaacaggcac cgctgatgat tgcggaaaaa 780
ccgattcgtc tggaagaatt tctgaccttt ggtggtcagg atctgaacat tattaccagc 840
gcaatgaaag agaaaatcta taataacctg ctggccaact atgagaaaat tgcaacccgt 900
ctgagccgtg ttaatagcgc acctcctgaa tatgatatca acgagtataa agactatttt 960
cagtggaaat acggcctgga taaaaatgca gatggtagct ataccgtgaa cgagaacaaa 1020
tttaacgaga tctacaaaaa actgtatagc ttcaccgaaa tcgatctggc caacaaattc 1080
aaagtgaaat gccgcaacac ctacttcatc aaatatggct ttctgaaagt tccgaacctg 1140
cttgatgatg atatctatac cgttagcgaa ggctttaaca ttggtaatct ggccgttaat 1200
aatcgcggtc agaacattaa actgaacccg aaaattatcg atagcatccc ggataaaggc 1260
ctggttgaaa aaattgtgaa attctgcaaa agcgtgattc cgcgtaaagg caccaaagca 1320
ccgcctcgtc tgtgtattcg tgtgaataat cgtgaactgt tttttgttgc aagcgagagc 1380
agctataacg agaatgatat taacaccccg aaagagattg acgataccac caatctgaat 1440
aacaactatc gcaacaatct ggatgaagtg atcctggatt ataacagcga aaccattccg 1500
cagattagca atcagaccct gaataccctg gttcaggatg atagctatgt tccgcgttat 1560
gatagcaatg gcaccagcga aattgaagaa cataatgtgg ttgatctgaa cgtgttcttt 1620
tatctgcatg cacagaaagt gccggaaggt gaaaccaata ttagcctgac cagcagcatt 1680
gataccgcac tgagcgaaga aagccaggtt tatacctttt ttagcagcga attcatcaac 1740
accattaaca aaccggttca tgcagcactg tttattagct ggattaatca ggtgattcgc 1800
gattttacca ccgaagcaac ccagaaaagc acctttgata aaattgccga tattagtctg 1860
gtggtgccgt atgttggtct ggcactgaat attggtaatg aagtgcagaa agagaacttt 1920
aaagaagcct tcgaactgtt aggtgccggt attctgctgg aatttgtgcc ggaactgctg 1980
attccgacca ttctggtttt taccattaag agctttattg gcagcagcga gaacaagaac 2040
aaaatcatta aagccatcaa caacagcctg atggaacgcg aaaccaaatg gaaagaaatt 2100
tacagctgga ttgtgagcaa ttggctgacc cgtatcaata cccagtttaa caaacgcaaa 2160
gaacaaatgt atcaggccct gcagaatcag gttgatgcaa ttaaaaccgt gatcgaatac 2220
aaatacaaca actataccag cgacgaacgt aatcgcctgg aaagcgaata caacattaat 2280
aacattcgcg aagaactgaa caaaaaagtg agcctggcaa tggaaaacat cgaacgtttt 2340
attaccgaaa gcagcatctt ctacctgatg aaactgatta acgaagccaa agttagcaaa 2400
ctgcgcgaat atgatgaagg cgttaaagaa tatctgctgg actatattag cgaacatcgt 2460
agcattctgg gtaatagcgt tcaagagctg aatgatctgg ttaccagcac actgaataat 2520
agcattccgt ttgaactgag cagctacacc aacgataaaa tcctgatcct gtacttcaac 2580
aaactgtaca agaagatcaa ggacaacagc atactggata tgcgctatga aaacaacaag 2640
ttcattgata tcagcggcta tggtagcaac attagcatta atggtgatgt gtatatctac 2700
agcaccaacc gcaatcagtt tggtatttat agcagcaaac cgagcgaagt taatattgcg 2760
cagaataacg atatcatcta caacggtcgc tatcagaact ttagcattag cttttgggtt 2820
cgcattccga aatactttaa caaggtgaac ctgaacaacg agtacaccat tattgattgc 2880
attcgcaata ataacagcgg ctggaaaatc agcctgaact ataacaaaat tatctggacc 2940
ctgcaggata ccgcaggtaa taatcagaaa ctggtgttta actacaccca gatgattagc 3000
atcagcgact atatcaacaa atggatcttt gtgaccatta ccaacaatcg tctgggtaac 3060
agccgcattt atatcaatgg caatctgatc gacgaaaaaa gcatttcaaa tctgggcgat 3120
attcacgtga gcgataacat tctgttcaaa attgttggct gcaacgatac ccgttatgtt 3180
ggtattcgtt acttcaaagt gtttgatacg gaactgggca aaacggaaat tgaaaccctg 3240
tatagtgatg aaccggatcc gagcattctg aaagattttt ggggtaatta tctgctgtac 3300
aacaaacgct actatctgct gaacctgctg cgtaccgata aaagcattac acagaatagc 3360
aactttctga acatcaatca gcagcgtggt gtttatcaga aaccgaacat ttttagcaac 3420
acccgtctgt ataccggtgt ggaagttatt attcgtaaaa acggtagcac cgatatcagc 3480
aacaccgata actttgtgcg taaaaatgac ctggcctata ttaacgttgt tgatcgtgat 3540
gttgagtatc gtctgtatgc ggatattagc attgccaaac cggaaaagat tatcaaactg 3600
atccgtacca gcaacagcaa taattcactg ggtcagatta tcgtgatgga cagcattggt 3660
aacaattgca ccatgaattt ccagaacaat aacggtggta atattggcct gctgggcttt 3720
catagcaata atctggttgc aagcagctgg tattacaaca acatccgtaa aaataccagc 3780
agtaatggtt gcttttggag ctttatcagt aaagaacatg gctggcaaga aaacgagaac 3840
ctgtattttc agggtgcaag tcatcatcac catcaccacc atcattaa 3888
<210> 34
<211> 1295
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rBoNT/F(0) (His-меченая)
<400> 34
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp
1 5 10 15
Asp Thr Ile Leu Tyr Met Gln Ile Pro Tyr Glu Glu Lys Ser Lys Lys
20 25 30
Tyr Tyr Lys Ala Phe Glu Ile Met Arg Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Asn Thr Ile Gly Thr Asp Pro Ser Asp Phe Asp Pro Pro Ala Ser
50 55 60
Leu Glu Asn Gly Ser Ser Ala Tyr Tyr Asp Pro Asn Tyr Leu Thr Thr
65 70 75 80
Asp Ala Glu Lys Asp Arg Tyr Leu Lys Thr Thr Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Asn Pro Ala Gly Glu Val Leu Leu Gln Glu Ile Ser
100 105 110
Tyr Ala Lys Pro Tyr Leu Gly Asn Glu His Thr Pro Ile Asn Glu Phe
115 120 125
His Pro Val Thr Arg Thr Thr Ser Val Asn Ile Lys Ser Ser Thr Asn
130 135 140
Val Lys Ser Ser Ile Ile Leu Asn Leu Leu Val Leu Gly Ala Gly Pro
145 150 155 160
Asp Ile Phe Glu Asn Ser Ser Tyr Pro Val Arg Lys Leu Met Asp Ser
165 170 175
Gly Gly Val Tyr Asp Pro Ser Asn Asp Gly Phe Gly Ser Ile Asn Ile
180 185 190
Val Thr Phe Ser Pro Glu Tyr Glu Tyr Thr Phe Asn Asp Ile Ser Gly
195 200 205
Gly Tyr Asn Ser Ser Thr Glu Ser Phe Ile Ala Asp Pro Ala Ile Ser
210 215 220
Leu Ala His Gln Leu Ile Tyr Ala Leu His Gly Leu Tyr Gly Ala Arg
225 230 235 240
Gly Val Thr Tyr Lys Glu Thr Ile Lys Val Lys Gln Ala Pro Leu Met
245 250 255
Ile Ala Glu Lys Pro Ile Arg Leu Glu Glu Phe Leu Thr Phe Gly Gly
260 265 270
Gln Asp Leu Asn Ile Ile Thr Ser Ala Met Lys Glu Lys Ile Tyr Asn
275 280 285
Asn Leu Leu Ala Asn Tyr Glu Lys Ile Ala Thr Arg Leu Ser Arg Val
290 295 300
Asn Ser Ala Pro Pro Glu Tyr Asp Ile Asn Glu Tyr Lys Asp Tyr Phe
305 310 315 320
Gln Trp Lys Tyr Gly Leu Asp Lys Asn Ala Asp Gly Ser Tyr Thr Val
325 330 335
Asn Glu Asn Lys Phe Asn Glu Ile Tyr Lys Lys Leu Tyr Ser Phe Thr
340 345 350
Glu Ile Asp Leu Ala Asn Lys Phe Lys Val Lys Cys Arg Asn Thr Tyr
355 360 365
Phe Ile Lys Tyr Gly Phe Leu Lys Val Pro Asn Leu Leu Asp Asp Asp
370 375 380
Ile Tyr Thr Val Ser Glu Gly Phe Asn Ile Gly Asn Leu Ala Val Asn
385 390 395 400
Asn Arg Gly Gln Asn Ile Lys Leu Asn Pro Lys Ile Ile Asp Ser Ile
405 410 415
Pro Asp Lys Gly Leu Val Glu Lys Ile Val Lys Phe Cys Lys Ser Val
420 425 430
Ile Pro Arg Lys Gly Thr Lys Ala Pro Pro Arg Leu Cys Ile Arg Val
435 440 445
Asn Asn Arg Glu Leu Phe Phe Val Ala Ser Glu Ser Ser Tyr Asn Glu
450 455 460
Asn Asp Ile Asn Thr Pro Lys Glu Ile Asp Asp Thr Thr Asn Leu Asn
465 470 475 480
Asn Asn Tyr Arg Asn Asn Leu Asp Glu Val Ile Leu Asp Tyr Asn Ser
485 490 495
Glu Thr Ile Pro Gln Ile Ser Asn Gln Thr Leu Asn Thr Leu Val Gln
500 505 510
Asp Asp Ser Tyr Val Pro Arg Tyr Asp Ser Asn Gly Thr Ser Glu Ile
515 520 525
Glu Glu His Asn Val Val Asp Leu Asn Val Phe Phe Tyr Leu His Ala
530 535 540
Gln Lys Val Pro Glu Gly Glu Thr Asn Ile Ser Leu Thr Ser Ser Ile
545 550 555 560
Asp Thr Ala Leu Ser Glu Glu Ser Gln Val Tyr Thr Phe Phe Ser Ser
565 570 575
Glu Phe Ile Asn Thr Ile Asn Lys Pro Val His Ala Ala Leu Phe Ile
580 585 590
Ser Trp Ile Asn Gln Val Ile Arg Asp Phe Thr Thr Glu Ala Thr Gln
595 600 605
Lys Ser Thr Phe Asp Lys Ile Ala Asp Ile Ser Leu Val Val Pro Tyr
610 615 620
Val Gly Leu Ala Leu Asn Ile Gly Asn Glu Val Gln Lys Glu Asn Phe
625 630 635 640
Lys Glu Ala Phe Glu Leu Leu Gly Ala Gly Ile Leu Leu Glu Phe Val
645 650 655
Pro Glu Leu Leu Ile Pro Thr Ile Leu Val Phe Thr Ile Lys Ser Phe
660 665 670
Ile Gly Ser Ser Glu Asn Lys Asn Lys Ile Ile Lys Ala Ile Asn Asn
675 680 685
Ser Leu Met Glu Arg Glu Thr Lys Trp Lys Glu Ile Tyr Ser Trp Ile
690 695 700
Val Ser Asn Trp Leu Thr Arg Ile Asn Thr Gln Phe Asn Lys Arg Lys
705 710 715 720
Glu Gln Met Tyr Gln Ala Leu Gln Asn Gln Val Asp Ala Ile Lys Thr
725 730 735
Val Ile Glu Tyr Lys Tyr Asn Asn Tyr Thr Ser Asp Glu Arg Asn Arg
740 745 750
Leu Glu Ser Glu Tyr Asn Ile Asn Asn Ile Arg Glu Glu Leu Asn Lys
755 760 765
Lys Val Ser Leu Ala Met Glu Asn Ile Glu Arg Phe Ile Thr Glu Ser
770 775 780
Ser Ile Phe Tyr Leu Met Lys Leu Ile Asn Glu Ala Lys Val Ser Lys
785 790 795 800
Leu Arg Glu Tyr Asp Glu Gly Val Lys Glu Tyr Leu Leu Asp Tyr Ile
805 810 815
Ser Glu His Arg Ser Ile Leu Gly Asn Ser Val Gln Glu Leu Asn Asp
820 825 830
Leu Val Thr Ser Thr Leu Asn Asn Ser Ile Pro Phe Glu Leu Ser Ser
835 840 845
Tyr Thr Asn Asp Lys Ile Leu Ile Leu Tyr Phe Asn Lys Leu Tyr Lys
850 855 860
Lys Ile Lys Asp Asn Ser Ile Leu Asp Met Arg Tyr Glu Asn Asn Lys
865 870 875 880
Phe Ile Asp Ile Ser Gly Tyr Gly Ser Asn Ile Ser Ile Asn Gly Asp
885 890 895
Val Tyr Ile Tyr Ser Thr Asn Arg Asn Gln Phe Gly Ile Tyr Ser Ser
900 905 910
Lys Pro Ser Glu Val Asn Ile Ala Gln Asn Asn Asp Ile Ile Tyr Asn
915 920 925
Gly Arg Tyr Gln Asn Phe Ser Ile Ser Phe Trp Val Arg Ile Pro Lys
930 935 940
Tyr Phe Asn Lys Val Asn Leu Asn Asn Glu Tyr Thr Ile Ile Asp Cys
945 950 955 960
Ile Arg Asn Asn Asn Ser Gly Trp Lys Ile Ser Leu Asn Tyr Asn Lys
965 970 975
Ile Ile Trp Thr Leu Gln Asp Thr Ala Gly Asn Asn Gln Lys Leu Val
980 985 990
Phe Asn Tyr Thr Gln Met Ile Ser Ile Ser Asp Tyr Ile Asn Lys Trp
995 1000 1005
Ile Phe Val Thr Ile Thr Asn Asn Arg Leu Gly Asn Ser Arg Ile
1010 1015 1020
Tyr Ile Asn Gly Asn Leu Ile Asp Glu Lys Ser Ile Ser Asn Leu
1025 1030 1035
Gly Asp Ile His Val Ser Asp Asn Ile Leu Phe Lys Ile Val Gly
1040 1045 1050
Cys Asn Asp Thr Arg Tyr Val Gly Ile Arg Tyr Phe Lys Val Phe
1055 1060 1065
Asp Thr Glu Leu Gly Lys Thr Glu Ile Glu Thr Leu Tyr Ser Asp
1070 1075 1080
Glu Pro Asp Pro Ser Ile Leu Lys Asp Phe Trp Gly Asn Tyr Leu
1085 1090 1095
Leu Tyr Asn Lys Arg Tyr Tyr Leu Leu Asn Leu Leu Arg Thr Asp
1100 1105 1110
Lys Ser Ile Thr Gln Asn Ser Asn Phe Leu Asn Ile Asn Gln Gln
1115 1120 1125
Arg Gly Val Tyr Gln Lys Pro Asn Ile Phe Ser Asn Thr Arg Leu
1130 1135 1140
Tyr Thr Gly Val Glu Val Ile Ile Arg Lys Asn Gly Ser Thr Asp
1145 1150 1155
Ile Ser Asn Thr Asp Asn Phe Val Arg Lys Asn Asp Leu Ala Tyr
1160 1165 1170
Ile Asn Val Val Asp Arg Asp Val Glu Tyr Arg Leu Tyr Ala Asp
1175 1180 1185
Ile Ser Ile Ala Lys Pro Glu Lys Ile Ile Lys Leu Ile Arg Thr
1190 1195 1200
Ser Asn Ser Asn Asn Ser Leu Gly Gln Ile Ile Val Met Asp Ser
1205 1210 1215
Ile Gly Asn Asn Cys Thr Met Asn Phe Gln Asn Asn Asn Gly Gly
1220 1225 1230
Asn Ile Gly Leu Leu Gly Phe His Ser Asn Asn Leu Val Ala Ser
1235 1240 1245
Ser Trp Tyr Tyr Asn Asn Ile Arg Lys Asn Thr Ser Ser Asn Gly
1250 1255 1260
Cys Phe Trp Ser Phe Ile Ser Lys Glu His Gly Trp Gln Glu Asn
1265 1270 1275
Glu Asn Leu Tyr Phe Gln Gly Ala Ser His His His His His His
1280 1285 1290
His His
1295
<210> 35
<211> 2649
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLHN/F (His-меченая)
<400> 35
atgccggttg tgattaacag cttcaattat aacgatccgg tgaacgatga taccatcctg 60
tatatgcaga ttccgtatga agagaaaagc aaaaagtact acaaagcctt tgagatcatg 120
cgcaacgttt ggattattcc ggaacgtaat accattggca ccgatccgag cgattttgat 180
ccgcctgcaa gcctggaaaa tggtagcagc gcatattatg atccgaatta tctgaccacc 240
gatgccgaaa aagatcgtta tctgaaaacc accatcaaac tgttcaaacg cattaatagc 300
aatccggcag gcgaagttct gctgcaagaa attagctatg caaaaccgta tctgggcaat 360
gaacataccc cgattaatga atttcatccg gttacacgta ccacgagcgt taacattaaa 420
agcagcacca atgtgaagtc cagcattatt ctgaatctgc tggttttagg tgcaggtccg 480
gatatttttg aaaattcaag ctatccggtg cgcaaactga tggatagcgg tggtgtgtat 540
gatccgtcaa atgatggttt tggcagcatt aacattgtga cctttagtcc ggaatatgaa 600
tacaccttca acgatattag cggtggctat aatagcagca ccgaaagttt tattgcagat 660
ccggcaatta gcctggcaca tgaactgatt catgcactgc atggtctgta tggtgcacgt 720
ggtgttacct ataaagaaac cattaaagtt aaacaggcac cgctgatgat tgcggaaaaa 780
ccgattcgtc tggaagaatt tctgaccttt ggtggtcagg atctgaacat tattaccagc 840
gcaatgaaag agaaaatcta taataacctg ctggccaact atgagaaaat tgcaacccgt 900
ctgagccgtg ttaatagcgc acctcctgaa tatgatatca acgagtataa agactatttt 960
cagtggaaat acggcctgga taaaaatgca gatggtagct ataccgtgaa cgagaacaaa 1020
tttaacgaga tctacaaaaa actgtatagc ttcaccgaaa tcgatctggc caacaaattc 1080
aaagtgaaat gccgcaacac ctacttcatc aaatatggct ttctgaaagt tccgaacctg 1140
cttgatgatg atatctatac cgttagcgaa ggctttaaca ttggtaatct ggccgttaat 1200
aatcgcggtc agaacattaa actgaacccg aaaattatcg atagcatccc ggataaaggc 1260
ctggttgaaa aaattgtgaa attctgcaaa agcgtgattc cgcgtaaagg caccaaagca 1320
ccgcctcgtc tgtgtattcg tgtgaataat cgtgaactgt tttttgttgc aagcgagagc 1380
agctataacg agaatgatat taacaccccg aaagagattg acgataccac caatctgaat 1440
aacaactatc gcaacaatct ggatgaagtg atcctggatt ataacagcga aaccattccg 1500
cagattagca atcagaccct gaataccctg gttcaggatg atagctatgt tccgcgttat 1560
gatagcaatg gcaccagcga aattgaagaa cataatgtgg ttgatctgaa cgtgttcttt 1620
tatctgcatg cacagaaagt gccggaaggt gaaaccaata ttagcctgac cagcagcatt 1680
gataccgcac tgagcgaaga aagccaggtt tatacctttt ttagcagcga attcatcaac 1740
accattaaca aaccggttca tgcagcactg tttattagct ggattaatca ggtgattcgc 1800
gattttacca ccgaagcaac ccagaaaagc acctttgata aaattgccga tattagtctg 1860
gtggtgccgt atgttggtct ggcactgaat attggtaatg aagtgcagaa agagaacttt 1920
aaagaagcct tcgaactgtt aggtgccggt attctgctgg aatttgtgcc ggaactgctg 1980
attccgacca ttctggtttt taccattaag agctttattg gcagcagcga gaacaagaac 2040
aaaatcatta aagccatcaa caacagcctg atggaacgcg aaaccaaatg gaaagaaatt 2100
tacagctgga ttgtgagcaa ttggctgacc cgtatcaata cccagtttaa caaacgcaaa 2160
gaacaaatgt atcaggccct gcagaatcag gttgatgcaa ttaaaaccgt gatcgaatac 2220
aaatacaaca actataccag cgacgaacgt aatcgcctgg aaagcgaata caacattaat 2280
aacattcgcg aagaactgaa caaaaaagtg agcctggcaa tggaaaacat cgaacgtttt 2340
attaccgaaa gcagcatctt ctacctgatg aaactgatta acgaagccaa agttagcaaa 2400
ctgcgcgaat atgatgaagg cgttaaagaa tatctgctgg actatattag cgaacatcgt 2460
agcattctgg gtaatagcgt tcaagagctg aatgatctgg ttaccagcac actgaataat 2520
agcattccgt ttgaactgag cagctacacc aacgataaaa tcctgatcct gtacttcaac 2580
aaactgtaca agaaagaaaa cctgtatttt cagggtgcaa gccatcatca ccaccatcac 2640
catcattaa 2649
<210> 36
<211> 882
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLHN/F (His-меченая)
<400> 36
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp
1 5 10 15
Asp Thr Ile Leu Tyr Met Gln Ile Pro Tyr Glu Glu Lys Ser Lys Lys
20 25 30
Tyr Tyr Lys Ala Phe Glu Ile Met Arg Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Asn Thr Ile Gly Thr Asp Pro Ser Asp Phe Asp Pro Pro Ala Ser
50 55 60
Leu Glu Asn Gly Ser Ser Ala Tyr Tyr Asp Pro Asn Tyr Leu Thr Thr
65 70 75 80
Asp Ala Glu Lys Asp Arg Tyr Leu Lys Thr Thr Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Asn Pro Ala Gly Glu Val Leu Leu Gln Glu Ile Ser
100 105 110
Tyr Ala Lys Pro Tyr Leu Gly Asn Glu His Thr Pro Ile Asn Glu Phe
115 120 125
His Pro Val Thr Arg Thr Thr Ser Val Asn Ile Lys Ser Ser Thr Asn
130 135 140
Val Lys Ser Ser Ile Ile Leu Asn Leu Leu Val Leu Gly Ala Gly Pro
145 150 155 160
Asp Ile Phe Glu Asn Ser Ser Tyr Pro Val Arg Lys Leu Met Asp Ser
165 170 175
Gly Gly Val Tyr Asp Pro Ser Asn Asp Gly Phe Gly Ser Ile Asn Ile
180 185 190
Val Thr Phe Ser Pro Glu Tyr Glu Tyr Thr Phe Asn Asp Ile Ser Gly
195 200 205
Gly Tyr Asn Ser Ser Thr Glu Ser Phe Ile Ala Asp Pro Ala Ile Ser
210 215 220
Leu Ala His Glu Leu Ile His Ala Leu His Gly Leu Tyr Gly Ala Arg
225 230 235 240
Gly Val Thr Tyr Lys Glu Thr Ile Lys Val Lys Gln Ala Pro Leu Met
245 250 255
Ile Ala Glu Lys Pro Ile Arg Leu Glu Glu Phe Leu Thr Phe Gly Gly
260 265 270
Gln Asp Leu Asn Ile Ile Thr Ser Ala Met Lys Glu Lys Ile Tyr Asn
275 280 285
Asn Leu Leu Ala Asn Tyr Glu Lys Ile Ala Thr Arg Leu Ser Arg Val
290 295 300
Asn Ser Ala Pro Pro Glu Tyr Asp Ile Asn Glu Tyr Lys Asp Tyr Phe
305 310 315 320
Gln Trp Lys Tyr Gly Leu Asp Lys Asn Ala Asp Gly Ser Tyr Thr Val
325 330 335
Asn Glu Asn Lys Phe Asn Glu Ile Tyr Lys Lys Leu Tyr Ser Phe Thr
340 345 350
Glu Ile Asp Leu Ala Asn Lys Phe Lys Val Lys Cys Arg Asn Thr Tyr
355 360 365
Phe Ile Lys Tyr Gly Phe Leu Lys Val Pro Asn Leu Leu Asp Asp Asp
370 375 380
Ile Tyr Thr Val Ser Glu Gly Phe Asn Ile Gly Asn Leu Ala Val Asn
385 390 395 400
Asn Arg Gly Gln Asn Ile Lys Leu Asn Pro Lys Ile Ile Asp Ser Ile
405 410 415
Pro Asp Lys Gly Leu Val Glu Lys Ile Val Lys Phe Cys Lys Ser Val
420 425 430
Ile Pro Arg Lys Gly Thr Lys Ala Pro Pro Arg Leu Cys Ile Arg Val
435 440 445
Asn Asn Arg Glu Leu Phe Phe Val Ala Ser Glu Ser Ser Tyr Asn Glu
450 455 460
Asn Asp Ile Asn Thr Pro Lys Glu Ile Asp Asp Thr Thr Asn Leu Asn
465 470 475 480
Asn Asn Tyr Arg Asn Asn Leu Asp Glu Val Ile Leu Asp Tyr Asn Ser
485 490 495
Glu Thr Ile Pro Gln Ile Ser Asn Gln Thr Leu Asn Thr Leu Val Gln
500 505 510
Asp Asp Ser Tyr Val Pro Arg Tyr Asp Ser Asn Gly Thr Ser Glu Ile
515 520 525
Glu Glu His Asn Val Val Asp Leu Asn Val Phe Phe Tyr Leu His Ala
530 535 540
Gln Lys Val Pro Glu Gly Glu Thr Asn Ile Ser Leu Thr Ser Ser Ile
545 550 555 560
Asp Thr Ala Leu Ser Glu Glu Ser Gln Val Tyr Thr Phe Phe Ser Ser
565 570 575
Glu Phe Ile Asn Thr Ile Asn Lys Pro Val His Ala Ala Leu Phe Ile
580 585 590
Ser Trp Ile Asn Gln Val Ile Arg Asp Phe Thr Thr Glu Ala Thr Gln
595 600 605
Lys Ser Thr Phe Asp Lys Ile Ala Asp Ile Ser Leu Val Val Pro Tyr
610 615 620
Val Gly Leu Ala Leu Asn Ile Gly Asn Glu Val Gln Lys Glu Asn Phe
625 630 635 640
Lys Glu Ala Phe Glu Leu Leu Gly Ala Gly Ile Leu Leu Glu Phe Val
645 650 655
Pro Glu Leu Leu Ile Pro Thr Ile Leu Val Phe Thr Ile Lys Ser Phe
660 665 670
Ile Gly Ser Ser Glu Asn Lys Asn Lys Ile Ile Lys Ala Ile Asn Asn
675 680 685
Ser Leu Met Glu Arg Glu Thr Lys Trp Lys Glu Ile Tyr Ser Trp Ile
690 695 700
Val Ser Asn Trp Leu Thr Arg Ile Asn Thr Gln Phe Asn Lys Arg Lys
705 710 715 720
Glu Gln Met Tyr Gln Ala Leu Gln Asn Gln Val Asp Ala Ile Lys Thr
725 730 735
Val Ile Glu Tyr Lys Tyr Asn Asn Tyr Thr Ser Asp Glu Arg Asn Arg
740 745 750
Leu Glu Ser Glu Tyr Asn Ile Asn Asn Ile Arg Glu Glu Leu Asn Lys
755 760 765
Lys Val Ser Leu Ala Met Glu Asn Ile Glu Arg Phe Ile Thr Glu Ser
770 775 780
Ser Ile Phe Tyr Leu Met Lys Leu Ile Asn Glu Ala Lys Val Ser Lys
785 790 795 800
Leu Arg Glu Tyr Asp Glu Gly Val Lys Glu Tyr Leu Leu Asp Tyr Ile
805 810 815
Ser Glu His Arg Ser Ile Leu Gly Asn Ser Val Gln Glu Leu Asn Asp
820 825 830
Leu Val Thr Ser Thr Leu Asn Asn Ser Ile Pro Phe Glu Leu Ser Ser
835 840 845
Tyr Thr Asn Asp Lys Ile Leu Ile Leu Tyr Phe Asn Lys Leu Tyr Lys
850 855 860
Lys Glu Asn Leu Tyr Phe Gln Gly Ala Ser His His His His His His
865 870 875 880
His His
<210> 37
<211> 1296
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/F (His-меченая)
<400> 37
atgatcaagg ataacagcat tctggatatg cgctatgaga acaacaaatt cattgatatt 60
agcggctatg gcagcaacat tagcattaat ggtgatgtgt atatctacag caccaaccgt 120
aatcagtttg gcatttatag cagcaaaccg agcgaagtta atattgccca gaacaacgat 180
atcatctata acggtcgcta tcagaacttc agcattagct tttgggttcg cattccgaaa 240
tacttcaata aggtgaacct gaacaacgag tataccatca ttgattgcat tcgcaataat 300
aacagcggct ggaaaattag cctgaactac aacaaaatta tctggaccct gcaggatacc 360
gcaggtaata atcagaaact ggtgtttaac tacacccaga tgattagcat cagcgactat 420
atcaacaaat ggatctttgt gaccattacc aataatcgcc tgggtaatag ccgcatttat 480
atcaatggta acctgatcga tgagaaaagc attagcaatc tgggtgatat tcatgtgagc 540
gataacatcc tgtttaaaat cgtgggttgt aacgataccc gttatgttgg tattcgctac 600
ttcaaagtgt ttgataccga actgggtaaa accgaaattg aaaccctgta tagtgatgaa 660
ccggatccga gcattctgaa agatttttgg ggtaattatc tgctgtacaa caaacgctac 720
tatctgctga atctgctgcg taccgataaa tcaattaccc agaatagcaa cttcctgaac 780
attaatcagc agcgtggtgt ttatcagaaa ccgaacattt ttagcaacac ccgtctgtat 840
accggtgtgg aagttattat tcgtaaaaat ggcagcaccg atatcagcaa caccgataac 900
tttgttcgca aaaatgatct ggcgtatatc aacgttgttg atcgtgatgt tgaatatcgt 960
ctgtatgccg atattagcat tgccaaaccg gaaaaaatca tcaaactgat ccgtaccagc 1020
aacagcaata attcactggg tcagattatt gtgatggata gcattggtaa taactgcacc 1080
atgaactttc agaacaataa cggtggtaat attggtctgc tgggctttca tagtaataat 1140
ctggttgcaa gcagctggta ttataacaac atccgtaaaa ataccagcag caatggttgc 1200
ttttggagct ttattagcaa agaacatggc tggcaagaaa acgagaatct gtattttcag 1260
ggtgcaagtc atcatcacca ccatcaccat cattaa 1296
<210> 38
<211> 431
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/F (His-меченая)
<400> 38
Met Ile Lys Asp Asn Ser Ile Leu Asp Met Arg Tyr Glu Asn Asn Lys
1 5 10 15
Phe Ile Asp Ile Ser Gly Tyr Gly Ser Asn Ile Ser Ile Asn Gly Asp
20 25 30
Val Tyr Ile Tyr Ser Thr Asn Arg Asn Gln Phe Gly Ile Tyr Ser Ser
35 40 45
Lys Pro Ser Glu Val Asn Ile Ala Gln Asn Asn Asp Ile Ile Tyr Asn
50 55 60
Gly Arg Tyr Gln Asn Phe Ser Ile Ser Phe Trp Val Arg Ile Pro Lys
65 70 75 80
Tyr Phe Asn Lys Val Asn Leu Asn Asn Glu Tyr Thr Ile Ile Asp Cys
85 90 95
Ile Arg Asn Asn Asn Ser Gly Trp Lys Ile Ser Leu Asn Tyr Asn Lys
100 105 110
Ile Ile Trp Thr Leu Gln Asp Thr Ala Gly Asn Asn Gln Lys Leu Val
115 120 125
Phe Asn Tyr Thr Gln Met Ile Ser Ile Ser Asp Tyr Ile Asn Lys Trp
130 135 140
Ile Phe Val Thr Ile Thr Asn Asn Arg Leu Gly Asn Ser Arg Ile Tyr
145 150 155 160
Ile Asn Gly Asn Leu Ile Asp Glu Lys Ser Ile Ser Asn Leu Gly Asp
165 170 175
Ile His Val Ser Asp Asn Ile Leu Phe Lys Ile Val Gly Cys Asn Asp
180 185 190
Thr Arg Tyr Val Gly Ile Arg Tyr Phe Lys Val Phe Asp Thr Glu Leu
195 200 205
Gly Lys Thr Glu Ile Glu Thr Leu Tyr Ser Asp Glu Pro Asp Pro Ser
210 215 220
Ile Leu Lys Asp Phe Trp Gly Asn Tyr Leu Leu Tyr Asn Lys Arg Tyr
225 230 235 240
Tyr Leu Leu Asn Leu Leu Arg Thr Asp Lys Ser Ile Thr Gln Asn Ser
245 250 255
Asn Phe Leu Asn Ile Asn Gln Gln Arg Gly Val Tyr Gln Lys Pro Asn
260 265 270
Ile Phe Ser Asn Thr Arg Leu Tyr Thr Gly Val Glu Val Ile Ile Arg
275 280 285
Lys Asn Gly Ser Thr Asp Ile Ser Asn Thr Asp Asn Phe Val Arg Lys
290 295 300
Asn Asp Leu Ala Tyr Ile Asn Val Val Asp Arg Asp Val Glu Tyr Arg
305 310 315 320
Leu Tyr Ala Asp Ile Ser Ile Ala Lys Pro Glu Lys Ile Ile Lys Leu
325 330 335
Ile Arg Thr Ser Asn Ser Asn Asn Ser Leu Gly Gln Ile Ile Val Met
340 345 350
Asp Ser Ile Gly Asn Asn Cys Thr Met Asn Phe Gln Asn Asn Asn Gly
355 360 365
Gly Asn Ile Gly Leu Leu Gly Phe His Ser Asn Asn Leu Val Ala Ser
370 375 380
Ser Trp Tyr Tyr Asn Asn Ile Arg Lys Asn Thr Ser Ser Asn Gly Cys
385 390 395 400
Phe Trp Ser Phe Ile Ser Lys Glu His Gly Trp Gln Glu Asn Glu Asn
405 410 415
Leu Tyr Phe Gln Gly Ala Ser His His His His His His His His
420 425 430
<210> 39
<211> 1347
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rLC/F (His-меченая)
<400> 39
atgccggttg tgattaacag cttcaattat aacgatccgg tgaacgatga taccatcctg 60
tatatgcaga ttccgtatga agagaaaagc aaaaagtact acaaagcctt tgagatcatg 120
cgcaacgttt ggattattcc ggaacgtaat accattggca ccgatccgag cgattttgat 180
ccgcctgcaa gcctggaaaa tggtagcagc gcatattatg atccgaatta tctgaccacc 240
gatgccgaaa aagatcgtta tctgaaaacc accatcaaac tgttcaaacg cattaatagc 300
aatccggcag gcgaagttct gctgcaagaa attagctatg caaaaccgta tctgggcaat 360
gaacataccc cgattaatga atttcatccg gttacacgta ccacgagcgt taacattaaa 420
agcagcacca atgtgaagtc cagcattatt ctgaatctgc tggttttagg tgcaggtccg 480
gatatttttg aaaattcaag ctatccggtg cgcaaactga tggatagcgg tggtgtgtat 540
gatccgtcaa atgatggttt tggcagcatt aacattgtga cctttagtcc ggaatatgaa 600
tacaccttca acgatattag cggtggctat aatagcagca ccgaaagttt tattgcagat 660
ccggcaatta gcctggcaca tgaactgatt catgcactgc atggtctgta tggtgcacgt 720
ggtgttacct ataaagaaac cattaaagtt aaacaggcac cgctgatgat tgcggaaaaa 780
ccgattcgtc tggaagaatt tctgaccttt ggtggtcagg atctgaacat tattaccagc 840
gcaatgaaag agaaaatcta taataacctg ctggccaact atgagaaaat tgcaacccgt 900
ctgagccgtg ttaatagcgc acctcctgaa tatgatatca acgagtataa agactatttt 960
cagtggaaat acggcctgga taaaaatgca gatggtagct ataccgtgaa cgagaacaaa 1020
tttaacgaga tctacaaaaa actgtatagc ttcaccgaaa tcgatctggc caacaaattc 1080
aaagtgaaat gccgcaacac ctacttcatc aaatatggct ttctgaaagt tccgaacctg 1140
cttgatgatg atatctatac cgttagcgaa ggctttaaca ttggtaatct ggccgttaat 1200
aatcgcggtc agaacattaa actgaacccg aaaattatcg atagcatccc ggataaaggc 1260
ctggttgaaa aaattgtgaa attctgcaaa agcgagaacc tgtattttca gggtgcaagt 1320
catcatcacc atcaccacca tcattaa 1347
<210> 40
<211> 448
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rLC/F (His-меченая)
<400> 40
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp
1 5 10 15
Asp Thr Ile Leu Tyr Met Gln Ile Pro Tyr Glu Glu Lys Ser Lys Lys
20 25 30
Tyr Tyr Lys Ala Phe Glu Ile Met Arg Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Asn Thr Ile Gly Thr Asp Pro Ser Asp Phe Asp Pro Pro Ala Ser
50 55 60
Leu Glu Asn Gly Ser Ser Ala Tyr Tyr Asp Pro Asn Tyr Leu Thr Thr
65 70 75 80
Asp Ala Glu Lys Asp Arg Tyr Leu Lys Thr Thr Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Asn Pro Ala Gly Glu Val Leu Leu Gln Glu Ile Ser
100 105 110
Tyr Ala Lys Pro Tyr Leu Gly Asn Glu His Thr Pro Ile Asn Glu Phe
115 120 125
His Pro Val Thr Arg Thr Thr Ser Val Asn Ile Lys Ser Ser Thr Asn
130 135 140
Val Lys Ser Ser Ile Ile Leu Asn Leu Leu Val Leu Gly Ala Gly Pro
145 150 155 160
Asp Ile Phe Glu Asn Ser Ser Tyr Pro Val Arg Lys Leu Met Asp Ser
165 170 175
Gly Gly Val Tyr Asp Pro Ser Asn Asp Gly Phe Gly Ser Ile Asn Ile
180 185 190
Val Thr Phe Ser Pro Glu Tyr Glu Tyr Thr Phe Asn Asp Ile Ser Gly
195 200 205
Gly Tyr Asn Ser Ser Thr Glu Ser Phe Ile Ala Asp Pro Ala Ile Ser
210 215 220
Leu Ala His Glu Leu Ile His Ala Leu His Gly Leu Tyr Gly Ala Arg
225 230 235 240
Gly Val Thr Tyr Lys Glu Thr Ile Lys Val Lys Gln Ala Pro Leu Met
245 250 255
Ile Ala Glu Lys Pro Ile Arg Leu Glu Glu Phe Leu Thr Phe Gly Gly
260 265 270
Gln Asp Leu Asn Ile Ile Thr Ser Ala Met Lys Glu Lys Ile Tyr Asn
275 280 285
Asn Leu Leu Ala Asn Tyr Glu Lys Ile Ala Thr Arg Leu Ser Arg Val
290 295 300
Asn Ser Ala Pro Pro Glu Tyr Asp Ile Asn Glu Tyr Lys Asp Tyr Phe
305 310 315 320
Gln Trp Lys Tyr Gly Leu Asp Lys Asn Ala Asp Gly Ser Tyr Thr Val
325 330 335
Asn Glu Asn Lys Phe Asn Glu Ile Tyr Lys Lys Leu Tyr Ser Phe Thr
340 345 350
Glu Ile Asp Leu Ala Asn Lys Phe Lys Val Lys Cys Arg Asn Thr Tyr
355 360 365
Phe Ile Lys Tyr Gly Phe Leu Lys Val Pro Asn Leu Leu Asp Asp Asp
370 375 380
Ile Tyr Thr Val Ser Glu Gly Phe Asn Ile Gly Asn Leu Ala Val Asn
385 390 395 400
Asn Arg Gly Gln Asn Ile Lys Leu Asn Pro Lys Ile Ile Asp Ser Ile
405 410 415
Pro Asp Lys Gly Leu Val Glu Lys Ile Val Lys Phe Cys Lys Ser Glu
420 425 430
Asn Leu Tyr Phe Gln Gly Ala Ser His His His His His His His His
435 440 445
<210> 41
<211> 1317
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность Катионного rHC/A (His-меченая)
<400> 41
atgatcatca acaccagcat tctgaacctg cgttatgaaa gcaaacatct gattgatctg 60
agccgttatg ccagcaaaat caatataggc agcaaggtta acttcgaccc gattgacaaa 120
aatcagatac agctgtttaa tctggaaagc agcaaaattg aggtgatcct gaaaaaagcg 180
atcgtgtata atagcatgta cgagaatttt tcgaccagct tttggattcg catcccgaaa 240
tactttaaca agattagcct gaacaacgag tataccatca ttaactgcat ggaaaacaat 300
agcggttgga aagtcagcct gaattatggc gaaattatct ggaccctgca ggataccaaa 360
gaaatcaaac agcgtgtggt gttcaaatac agccagatga ttaatatcag cgactatatc 420
aaccgctgga tttttgtgac cattaccaat aatcggctga acaagagcaa gatctatatt 480
aacggtcgtc tgattgacca gaaaccgatt agtaatctgg gtaatattca tgcgagcaac 540
aaaatcatgt ttaaactgga tggttgccgt gatacccatc gttatatttg gatcaaatac 600
ttcaacctgt tcgataaaga gttgaacgaa aaagaaatta aagacctgta cgataaccag 660
agcaatagcg gcatactgaa agatttttgg ggagattatc tgcagtatga caaaccgtat 720
tatatgctga atctgtacga cccgaataaa tacgtggatg ttaataatgt gggcatccgt 780
ggttatatgt acctgaaagg tccgcgtggt agcgttatga ccacaaacat ttatctgaat 840
agcagcctgt atcgcggaac caaattcatc attaaaaagt atgccagcgg caacaaggat 900
aatattgtgc gtaataatga tcgcgtgtac attaacgttg tggtgaagaa taaagaatat 960
cgcctggcaa ccaatgcaag ccaggcaggc gttgaaaaaa ttctgagtgc cctggaaatt 1020
ccggatgttg gtaatctgag ccaggttgtt gtgatgaaaa gcaaaaacga taaaggcatc 1080
accaacaaat gcaagatgaa tctgcaggac aataacggca atgatattgg cttcattggc 1140
tttcaccagt ttaacaacat tgcaaaactg gttgcgagca attggtataa tcgtcagatt 1200
gaacgtagca gtcgtaccct gggttgtagc tgggaattta tccctgtgga tgatggttgg 1260
ggtgaacgtc cgctgaagct tgcggccgca ctcgagcacc accaccacca ccactga 1317
<210> 42
<211> 438
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность Катионного rHC/A (His-меченая)
<400> 42
Met Ile Ile Asn Thr Ser Ile Leu Asn Leu Arg Tyr Glu Ser Lys His
1 5 10 15
Leu Ile Asp Leu Ser Arg Tyr Ala Ser Lys Ile Asn Ile Gly Ser Lys
20 25 30
Val Asn Phe Asp Pro Ile Asp Lys Asn Gln Ile Gln Leu Phe Asn Leu
35 40 45
Glu Ser Ser Lys Ile Glu Val Ile Leu Lys Lys Ala Ile Val Tyr Asn
50 55 60
Ser Met Tyr Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile Pro Lys
65 70 75 80
Tyr Phe Asn Lys Ile Ser Leu Asn Asn Glu Tyr Thr Ile Ile Asn Cys
85 90 95
Met Glu Asn Asn Ser Gly Trp Lys Val Ser Leu Asn Tyr Gly Glu Ile
100 105 110
Ile Trp Thr Leu Gln Asp Thr Lys Glu Ile Lys Gln Arg Val Val Phe
115 120 125
Lys Tyr Ser Gln Met Ile Asn Ile Ser Asp Tyr Ile Asn Arg Trp Ile
130 135 140
Phe Val Thr Ile Thr Asn Asn Arg Leu Asn Lys Ser Lys Ile Tyr Ile
145 150 155 160
Asn Gly Arg Leu Ile Asp Gln Lys Pro Ile Ser Asn Leu Gly Asn Ile
165 170 175
His Ala Ser Asn Lys Ile Met Phe Lys Leu Asp Gly Cys Arg Asp Thr
180 185 190
His Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu
195 200 205
Asn Glu Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly
210 215 220
Ile Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr
225 230 235 240
Tyr Met Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn
245 250 255
Val Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
260 265 270
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr Lys
275 280 285
Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile Val Arg
290 295 300
Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn Lys Glu Tyr
305 310 315 320
Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu Lys Ile Leu Ser
325 330 335
Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser Gln Val Val Val Met
340 345 350
Lys Ser Lys Asn Asp Lys Gly Ile Thr Asn Lys Cys Lys Met Asn Leu
355 360 365
Gln Asp Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly Phe His Gln Phe
370 375 380
Asn Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr Asn Arg Gln Ile
385 390 395 400
Glu Arg Ser Ser Arg Thr Leu Gly Cys Ser Trp Glu Phe Ile Pro Val
405 410 415
Asp Asp Gly Trp Gly Glu Arg Pro Leu Lys Leu Ala Ala Ala Leu Glu
420 425 430
His His His His His His
435
<210> 43
<211> 1341
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/AB (His-меченая)
<400> 43
atgattctga acaatattat cctgaacctg cgttacaaag acaacaatct gatcgatctg 60
agcggctatg gtgcaaaagt tgaagtctac gacggtgtcg aactgaacga taaaaaccag 120
ttcaaactga cctcatcggc taactcaaaa attcgtgtga cgcagaacca aaacatcatc 180
ttcaactcgg tctttctgga cttcagcgtg tctttctgga ttcgcatccc gaaatataaa 240
aatgatggca tccagaacta catccataac gaatacacca tcatcaactg tatgaaaaac 300
aacagtggtt ggaaaatttc catccgtggc aaccgcatta tctggaccct gattgatatc 360
aatggtaaaa cgaaaagcgt gtttttcgaa tacaacatcc gtgaagatat ctctgaatac 420
atcaatcgct ggtttttcgt gaccattacg aacaatctga acaatgcgaa aatctatatc 480
aacggcaaac tggaaagtaa taccgacatc aaagatattc gtgaagttat cgccaacggt 540
gaaatcatct tcaaactgga tggcgacatc gatcgcaccc agttcatttg gatgaaatac 600
ttctccatct tcaacacgga actgagtcag tccaatatcg aagaacgcta caaaatccaa 660
tcatactcgg aatacctgaa agatttctgg ggtaacccgc tgatgtacaa caaagaatac 720
tacatgttca acgcgggcaa caaaaactca tacatcaaac tgaaaaaaga ttcgccggtg 780
ggtgaaatcc tgacccgtag caaatacaac cagaactcta aatacatcaa ctatcgcgat 840
ctgtacattg gcgaaaaatt tattatccgt cgcaaaagca actctcagag tattaatgat 900
gacatcgtgc gtaaagaaga ctacatctat ctggatttct ttaatctgaa ccaagaatgg 960
cgcgtttata cctacaaata cttcaaaaaa gaagaaatga aactgttcct ggccccgatt 1020
tacgacagcg atgaatttta caacaccatc cagatcaaag aatacgatga acagccgacg 1080
tatagttgcc aactgctgtt caaaaaagac gaagaatcca ccgatgaaat tggcctgatt 1140
ggtatccacc gtttctatga aagcggtatc gttttcgaag aatacaaaga ttacttctgt 1200
atctctaaat ggtatctgaa agaagtcaaa cgcaaaccgt acaacctgaa actgggctgc 1260
aactggcaat ttatcccgaa agacgaaggc tggaccgaaa agcttgcggc cgcactcgag 1320
caccaccacc accaccactg a 1341
<210> 44
<211> 446
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/AB (His-меченая)
<400> 44
Met Ile Leu Asn Asn Ile Ile Leu Asn Leu Arg Tyr Lys Asp Asn Asn
1 5 10 15
Leu Ile Asp Leu Ser Gly Tyr Gly Ala Lys Val Glu Val Tyr Asp Gly
20 25 30
Val Glu Leu Asn Asp Lys Asn Gln Phe Lys Leu Thr Ser Ser Ala Asn
35 40 45
Ser Lys Ile Arg Val Thr Gln Asn Gln Asn Ile Ile Phe Asn Ser Val
50 55 60
Phe Leu Asp Phe Ser Val Ser Phe Trp Ile Arg Ile Pro Lys Tyr Lys
65 70 75 80
Asn Asp Gly Ile Gln Asn Tyr Ile His Asn Glu Tyr Thr Ile Ile Asn
85 90 95
Cys Met Lys Asn Asn Ser Gly Trp Lys Ile Ser Ile Arg Gly Asn Arg
100 105 110
Ile Ile Trp Thr Leu Ile Asp Ile Asn Gly Lys Thr Lys Ser Val Phe
115 120 125
Phe Glu Tyr Asn Ile Arg Glu Asp Ile Ser Glu Tyr Ile Asn Arg Trp
130 135 140
Phe Phe Val Thr Ile Thr Asn Asn Leu Asn Asn Ala Lys Ile Tyr Ile
145 150 155 160
Asn Gly Lys Leu Glu Ser Asn Thr Asp Ile Lys Asp Ile Arg Glu Val
165 170 175
Ile Ala Asn Gly Glu Ile Ile Phe Lys Leu Asp Gly Asp Ile Asp Arg
180 185 190
Thr Gln Phe Ile Trp Met Lys Tyr Phe Ser Ile Phe Asn Thr Glu Leu
195 200 205
Ser Gln Ser Asn Ile Glu Glu Arg Tyr Lys Ile Gln Ser Tyr Ser Glu
210 215 220
Tyr Leu Lys Asp Phe Trp Gly Asn Pro Leu Met Tyr Asn Lys Glu Tyr
225 230 235 240
Tyr Met Phe Asn Ala Gly Asn Lys Asn Ser Tyr Ile Lys Leu Lys Lys
245 250 255
Asp Ser Pro Val Gly Glu Ile Leu Thr Arg Ser Lys Tyr Asn Gln Asn
260 265 270
Ser Lys Tyr Ile Asn Tyr Arg Asp Leu Tyr Ile Gly Glu Lys Phe Ile
275 280 285
Ile Arg Arg Lys Ser Asn Ser Gln Ser Ile Asn Asp Asp Ile Val Arg
290 295 300
Lys Glu Asp Tyr Ile Tyr Leu Asp Phe Phe Asn Leu Asn Gln Glu Trp
305 310 315 320
Arg Val Tyr Thr Tyr Lys Tyr Phe Lys Lys Glu Glu Met Lys Leu Phe
325 330 335
Leu Ala Pro Ile Tyr Asp Ser Asp Glu Phe Tyr Asn Thr Ile Gln Ile
340 345 350
Lys Glu Tyr Asp Glu Gln Pro Thr Tyr Ser Cys Gln Leu Leu Phe Lys
355 360 365
Lys Asp Glu Glu Ser Thr Asp Glu Ile Gly Leu Ile Gly Ile His Arg
370 375 380
Phe Tyr Glu Ser Gly Ile Val Phe Glu Glu Tyr Lys Asp Tyr Phe Cys
385 390 395 400
Ile Ser Lys Trp Tyr Leu Lys Glu Val Lys Arg Lys Pro Tyr Asn Leu
405 410 415
Lys Leu Gly Cys Asn Trp Gln Phe Ile Pro Lys Asp Glu Gly Trp Thr
420 425 430
Glu Lys Leu Ala Ala Ala Leu Glu His His His His His His
435 440 445
<210> 45
<211> 1306
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/A Вариант Y1117V H1253K
(His-меченая)
<400> 45
atgatcatca atactagcat tctgaacctg cgttacgaga gcaatcatct gattgatctg 60
agccgttatg caagcaagat caacatcggt agcaaggtca attttgaccc gatcgataag 120
aaccagatcc agctgtttaa tctggaatcg agcaaaattg aggttatcct gaaaaacgcc 180
attgtctaca actccatgta cgagaatttc tccaccagct tctggattcg catcccgaaa 240
tacttcaaca gcattagcct gaacaacgag tatactatca tcaactgtat ggagaacaac 300
agcggttgga aggtgtctct gaactatggt gagatcattt ggaccttgca ggacacccaa 360
gagatcaagc agcgcgtcgt gttcaagtac tctcaaatga tcaacatttc cgattacatt 420
aatcgttgga tcttcgtgac cattacgaat aaccgtctga ataacagcaa gatttacatc 480
aatggtcgct tgatcgatca gaaaccgatt agcaacctgg gtaatatcca cgcaagcaac 540
aacattatgt tcaaattgga cggttgccgc gatacccatc gttatatctg gatcaagtat 600
ttcaacctgt ttgataaaga actgaatgag aaggagatca aagatttgta tgacaaccaa 660
tctaacagcg gcattttgaa ggacttctgg ggcgattatc tgcaatacga taagccgtac 720
tatatgctga acctggttga tccgaacaaa tatgtggatg tcaataatgt gggtattcgt 780
ggttacatgt atttgaaggg tccgcgtggc agcgttatga cgaccaacat ttacctgaac 840
tctagcctgt accgtggtac gaaattcatc attaagaaat atgccagcgg caacaaagat 900
aacattgtgc gtaataacga tcgtgtctac atcaacgtgg tcgtgaagaa taaagagtac 960
cgtctggcga ccaacgcttc gcaggcgggt gttgagaaaa ttctgagcgc gttggagatc 1020
cctgatgtcg gtaatctgag ccaagtcgtg gttatgaaga gcaagaacga ccagggtatc 1080
actaacaagt gcaagatgaa cctgcaagac aacaatggta acgacatcgg ctttattggt 1140
ttcaaacagt tcaacaatat tgctaaactg gtagcgagca attggtacaa tcgtcagatt 1200
gagcgcagca gccgtacttt gggctgtagc tgggagttta tcccggtcga tgatggttgg 1260
ggcgaacgtc cgctgcacca tcaccatcac catcaccatc accatt 1306
<210> 46
<211> 435
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/A Вариант Y1117V H1253K
(His-меченая)
<400> 46
Met Ile Ile Asn Thr Ser Ile Leu Asn Leu Arg Tyr Glu Ser Asn His
1 5 10 15
Leu Ile Asp Leu Ser Arg Tyr Ala Ser Lys Ile Asn Ile Gly Ser Lys
20 25 30
Val Asn Phe Asp Pro Ile Asp Lys Asn Gln Ile Gln Leu Phe Asn Leu
35 40 45
Glu Ser Ser Lys Ile Glu Val Ile Leu Lys Asn Ala Ile Val Tyr Asn
50 55 60
Ser Met Tyr Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile Pro Lys
65 70 75 80
Tyr Phe Asn Ser Ile Ser Leu Asn Asn Glu Tyr Thr Ile Ile Asn Cys
85 90 95
Met Glu Asn Asn Ser Gly Trp Lys Val Ser Leu Asn Tyr Gly Glu Ile
100 105 110
Ile Trp Thr Leu Gln Asp Thr Gln Glu Ile Lys Gln Arg Val Val Phe
115 120 125
Lys Tyr Ser Gln Met Ile Asn Ile Ser Asp Tyr Ile Asn Arg Trp Ile
130 135 140
Phe Val Thr Ile Thr Asn Asn Arg Leu Asn Asn Ser Lys Ile Tyr Ile
145 150 155 160
Asn Gly Arg Leu Ile Asp Gln Lys Pro Ile Ser Asn Leu Gly Asn Ile
165 170 175
His Ala Ser Asn Asn Ile Met Phe Lys Leu Asp Gly Cys Arg Asp Thr
180 185 190
His Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu
195 200 205
Asn Glu Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly
210 215 220
Ile Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr
225 230 235 240
Tyr Met Leu Asn Leu Val Asp Pro Asn Lys Tyr Val Asp Val Asn Asn
245 250 255
Val Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
260 265 270
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr Lys
275 280 285
Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile Val Arg
290 295 300
Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn Lys Glu Tyr
305 310 315 320
Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu Lys Ile Leu Ser
325 330 335
Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser Gln Val Val Val Met
340 345 350
Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn Lys Cys Lys Met Asn Leu
355 360 365
Gln Asp Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly Phe Lys Gln Phe
370 375 380
Asn Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr Asn Arg Gln Ile
385 390 395 400
Glu Arg Ser Ser Arg Thr Leu Gly Cys Ser Trp Glu Phe Ile Pro Val
405 410 415
Asp Asp Gly Trp Gly Glu Arg Pro Leu His His His His His His His
420 425 430
His His His
435
<210> 47
<211> 1308
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/A Вариант Y1117V F1252Y H1253K
L1278F (His-меченая)
<400> 47
atgatcatca atactagcat tctgaacctg cgttacgaga gcaatcatct gattgatctg 60
agccgttatg caagcaagat caacatcggt agcaaggtca attttgaccc gatcgataag 120
aaccagatcc agctgtttaa tctggaatcg agcaaaattg aggttatcct gaaaaacgcc 180
attgtctaca actccatgta cgagaatttc tccaccagct tctggattcg catcccgaaa 240
tacttcaaca gcattagcct gaacaacgag tatactatca tcaactgtat ggagaacaac 300
agcggttgga aggtgtctct gaactatggt gagatcattt ggaccttgca ggacacccaa 360
gagatcaagc agcgcgtcgt gttcaagtac tctcaaatga tcaacatttc cgattacatt 420
aatcgttgga tcttcgtgac cattacgaat aaccgtctga ataacagcaa gatttacatc 480
aatggtcgct tgatcgatca gaaaccgatt agcaacctgg gtaatatcca cgcaagcaac 540
aacattatgt tcaaattgga cggttgccgc gatacccatc gttatatctg gatcaagtat 600
ttcaacctgt ttgataaaga actgaatgag aaggagatca aagatttgta tgacaaccaa 660
tctaacagcg gcattttgaa ggacttctgg ggcgattatc tgcaatacga taagccgtac 720
tatatgctga acctggttga tccgaacaaa tatgtggatg tcaataatgt gggtattcgt 780
ggttacatgt atttgaaggg tccgcgtggc agcgttatga cgaccaacat ttacctgaac 840
tctagcctgt accgtggtac gaaattcatc attaagaaat atgccagcgg caacaaagat 900
aacattgtgc gtaataacga tcgtgtctac atcaacgtgg tcgtgaagaa taaagagtac 960
cgtctggcga ccaacgcttc gcaggcgggt gttgagaaaa ttctgagcgc gttggagatc 1020
cctgatgtcg gtaatctgag ccaagtcgtg gttatgaaga gcaagaacga ccagggtatc 1080
actaacaagt gcaagatgaa cctgcaagac aacaatggta acgacatcgg ctttattggt 1140
tacaaacagt tcaacaatat tgctaaactg gtagcgagca attggtacaa tcgtcagatt 1200
gagcgcagca gccgtacttt tggctgtagc tgggagttta tcccggtcga tgatggttgg 1260
ggcgaacgtc cgctgcacca tcaccatcac catcaccatc accattaa 1308
<210> 48
<211> 435
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/A Вариант Y1117V F1252Y
H1253K L1278F (His-меченая)
<400> 48
Met Ile Ile Asn Thr Ser Ile Leu Asn Leu Arg Tyr Glu Ser Asn His
1 5 10 15
Leu Ile Asp Leu Ser Arg Tyr Ala Ser Lys Ile Asn Ile Gly Ser Lys
20 25 30
Val Asn Phe Asp Pro Ile Asp Lys Asn Gln Ile Gln Leu Phe Asn Leu
35 40 45
Glu Ser Ser Lys Ile Glu Val Ile Leu Lys Asn Ala Ile Val Tyr Asn
50 55 60
Ser Met Tyr Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile Pro Lys
65 70 75 80
Tyr Phe Asn Ser Ile Ser Leu Asn Asn Glu Tyr Thr Ile Ile Asn Cys
85 90 95
Met Glu Asn Asn Ser Gly Trp Lys Val Ser Leu Asn Tyr Gly Glu Ile
100 105 110
Ile Trp Thr Leu Gln Asp Thr Gln Glu Ile Lys Gln Arg Val Val Phe
115 120 125
Lys Tyr Ser Gln Met Ile Asn Ile Ser Asp Tyr Ile Asn Arg Trp Ile
130 135 140
Phe Val Thr Ile Thr Asn Asn Arg Leu Asn Asn Ser Lys Ile Tyr Ile
145 150 155 160
Asn Gly Arg Leu Ile Asp Gln Lys Pro Ile Ser Asn Leu Gly Asn Ile
165 170 175
His Ala Ser Asn Asn Ile Met Phe Lys Leu Asp Gly Cys Arg Asp Thr
180 185 190
His Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu
195 200 205
Asn Glu Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly
210 215 220
Ile Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr
225 230 235 240
Tyr Met Leu Asn Leu Val Asp Pro Asn Lys Tyr Val Asp Val Asn Asn
245 250 255
Val Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
260 265 270
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr Lys
275 280 285
Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile Val Arg
290 295 300
Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn Lys Glu Tyr
305 310 315 320
Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu Lys Ile Leu Ser
325 330 335
Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser Gln Val Val Val Met
340 345 350
Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn Lys Cys Lys Met Asn Leu
355 360 365
Gln Asp Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly Tyr Lys Gln Phe
370 375 380
Asn Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr Asn Arg Gln Ile
385 390 395 400
Glu Arg Ser Ser Arg Thr Phe Gly Cys Ser Trp Glu Phe Ile Pro Val
405 410 415
Asp Asp Gly Trp Gly Glu Arg Pro Leu His His His His His His His
420 425 430
His His His
435
<210> 49
<211> 1293
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность rHC/A Вариант Y1117V F1252Y H1253K
L1278H (His-меченая)
<400> 49
atgatcatca atactagcat tctgaacctg cgttacgaga gcaatcatct gattgatctg 60
agccgttatg caagcaagat caacatcggt agcaaggtca attttgaccc gatcgataag 120
aaccagatcc agctgtttaa tctggaatcg agcaaaattg aggttatcct gaaaaacgcc 180
attgtctaca actccatgta cgagaatttc tccaccagct tctggattcg catcccgaaa 240
tacttcaaca gcattagcct gaacaacgag tatactatca tcaactgtat ggagaacaac 300
agcggttgga aggtgtctct gaactatggt gagatcattt ggaccttgca ggacacccaa 360
gagatcaagc agcgcgtcgt gttcaagtac tctcaaatga tcaacatttc cgattacatt 420
aatcgttgga tcttcgtgac cattacgaat aaccgtctga ataacagcaa gatttacatc 480
aatggtcgct tgatcgatca gaaaccgatt agcaacctgg gtaatatcca cgcaagcaac 540
aacattatgt tcaaattgga cggttgccgc gatacccatc gttatatctg gatcaagtat 600
ttcaacctgt ttgataaaga actgaatgag aaggagatca aagatttgta tgacaaccaa 660
tctaacagcg gcattttgaa ggacttctgg ggcgattatc tgcaatacga taagccgtac 720
tatatgctga acctggttga tccgaacaaa tatgtggatg tcaataatgt gggtattcgt 780
ggttacatgt atttgaaggg tccgcgtggc agcgttatga cgaccaacat ttacctgaac 840
tctagcctgt accgtggtac gaaattcatc attaagaaat atgccagcgg caacaaagat 900
aacattgtgc gtaataacga tcgtgtctac atcaacgtgg tcgtgaagaa taaagagtac 960
cgtctggcga ccaacgcttc gcaggcgggt gttgagaaaa ttctgagcgc gttggagatc 1020
cctgatgtcg gtaatctgag ccaagtcgtg gttatgaaga gcaagaacga ccagggtatc 1080
actaacaagt gcaagatgaa cctgcaagac aacaatggta acgacatcgg ctttattggt 1140
tacaaacagt tcaacaatat tgctaaactg gtagcgagca attggtacaa tcgtcagatt 1200
gagcgcagca gccgtactca tggctgtagc tgggagttta tcccggtcga tgatggttgg 1260
ggcgaacgtc cgctgcacca tcaccatcac cat 1293
<210> 50
<211> 431
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность rHC/A Вариант Y1117V F1252Y
H1253K L1278H (His-меченая)
<400> 50
Met Ile Ile Asn Thr Ser Ile Leu Asn Leu Arg Tyr Glu Ser Asn His
1 5 10 15
Leu Ile Asp Leu Ser Arg Tyr Ala Ser Lys Ile Asn Ile Gly Ser Lys
20 25 30
Val Asn Phe Asp Pro Ile Asp Lys Asn Gln Ile Gln Leu Phe Asn Leu
35 40 45
Glu Ser Ser Lys Ile Glu Val Ile Leu Lys Asn Ala Ile Val Tyr Asn
50 55 60
Ser Met Tyr Glu Asn Phe Ser Thr Ser Phe Trp Ile Arg Ile Pro Lys
65 70 75 80
Tyr Phe Asn Ser Ile Ser Leu Asn Asn Glu Tyr Thr Ile Ile Asn Cys
85 90 95
Met Glu Asn Asn Ser Gly Trp Lys Val Ser Leu Asn Tyr Gly Glu Ile
100 105 110
Ile Trp Thr Leu Gln Asp Thr Gln Glu Ile Lys Gln Arg Val Val Phe
115 120 125
Lys Tyr Ser Gln Met Ile Asn Ile Ser Asp Tyr Ile Asn Arg Trp Ile
130 135 140
Phe Val Thr Ile Thr Asn Asn Arg Leu Asn Asn Ser Lys Ile Tyr Ile
145 150 155 160
Asn Gly Arg Leu Ile Asp Gln Lys Pro Ile Ser Asn Leu Gly Asn Ile
165 170 175
His Ala Ser Asn Asn Ile Met Phe Lys Leu Asp Gly Cys Arg Asp Thr
180 185 190
His Arg Tyr Ile Trp Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu
195 200 205
Asn Glu Lys Glu Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly
210 215 220
Ile Leu Lys Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr
225 230 235 240
Tyr Met Leu Asn Leu Val Asp Pro Asn Lys Tyr Val Asp Val Asn Asn
245 250 255
Val Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
260 265 270
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr Lys
275 280 285
Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile Val Arg
290 295 300
Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn Lys Glu Tyr
305 310 315 320
Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu Lys Ile Leu Ser
325 330 335
Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser Gln Val Val Val Met
340 345 350
Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn Lys Cys Lys Met Asn Leu
355 360 365
Gln Asp Asn Asn Gly Asn Asp Ile Gly Phe Ile Gly Tyr Lys Gln Phe
370 375 380
Asn Asn Ile Ala Lys Leu Val Ala Ser Asn Trp Tyr Asn Arg Gln Ile
385 390 395 400
Glu Arg Ser Ser Arg Thr His Gly Cys Ser Trp Glu Phe Ile Pro Val
405 410 415
Asp Asp Gly Trp Gly Glu Arg Pro Leu His His His His His His
420 425 430
<210> 51
<211> 1296
<212> БЕЛОК
<213> Clostridium botulinum
<400> 51
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Val Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Ile Asn Thr Ser Ile Leu Asn
865 870 875 880
Leu Arg Tyr Glu Ser Asn His Leu Ile Asp Leu Ser Arg Tyr Ala Ser
885 890 895
Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys Asn
900 905 910
Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu
915 920 925
Lys Asn Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser
930 935 940
Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Ser Ile Ser Leu Asn Asn
945 950 955 960
Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys Val
965 970 975
Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Gln Glu
980 985 990
Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile Ser
995 1000 1005
Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
1010 1015 1020
Leu Asn Asn Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln
1025 1030 1035
Lys Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Asn Ile
1040 1045 1050
Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp
1055 1060 1065
Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu
1070 1075 1080
Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met
1100 1105 1110
Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
1115 1120 1125
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
1130 1135 1140
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr
1145 1150 1155
Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile
1160 1165 1170
Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn
1175 1180 1185
Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
1190 1195 1200
Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser
1205 1210 1215
Gln Val Val Val Met Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn
1220 1225 1230
Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly
1235 1240 1245
Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
1250 1255 1260
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu
1265 1270 1275
Gly Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu
1280 1285 1290
Arg Pro Leu
1295
<210> 52
<211> 1291
<212> БЕЛОК
<213> Clostridium botulinum
<400> 52
Met Pro Val Thr Ile Asn Asn Phe Asn Tyr Asn Asp Pro Ile Asp Asn
1 5 10 15
Asn Asn Ile Ile Met Met Glu Pro Pro Phe Ala Arg Gly Thr Gly Arg
20 25 30
Tyr Tyr Lys Ala Phe Lys Ile Thr Asp Arg Ile Trp Ile Ile Pro Glu
35 40 45
Arg Tyr Thr Phe Gly Tyr Lys Pro Glu Asp Phe Asn Lys Ser Ser Gly
50 55 60
Ile Phe Asn Arg Asp Val Cys Glu Tyr Tyr Asp Pro Asp Tyr Leu Asn
65 70 75 80
Thr Asn Asp Lys Lys Asn Ile Phe Leu Gln Thr Met Ile Lys Leu Phe
85 90 95
Asn Arg Ile Lys Ser Lys Pro Leu Gly Glu Lys Leu Leu Glu Met Ile
100 105 110
Ile Asn Gly Ile Pro Tyr Leu Gly Asp Arg Arg Val Pro Leu Glu Glu
115 120 125
Phe Asn Thr Asn Ile Ala Ser Val Thr Val Asn Lys Leu Ile Ser Asn
130 135 140
Pro Gly Glu Val Glu Arg Lys Lys Gly Ile Phe Ala Asn Leu Ile Ile
145 150 155 160
Phe Gly Pro Gly Pro Val Leu Asn Glu Asn Glu Thr Ile Asp Ile Gly
165 170 175
Ile Gln Asn His Phe Ala Ser Arg Glu Gly Phe Gly Gly Ile Met Gln
180 185 190
Met Lys Phe Cys Pro Glu Tyr Val Ser Val Phe Asn Asn Val Gln Glu
195 200 205
Asn Lys Gly Ala Ser Ile Phe Asn Arg Arg Gly Tyr Phe Ser Asp Pro
210 215 220
Ala Leu Ile Leu Met His Glu Leu Ile His Val Leu His Gly Leu Tyr
225 230 235 240
Gly Ile Lys Val Asp Asp Leu Pro Ile Val Pro Asn Glu Lys Lys Phe
245 250 255
Phe Met Gln Ser Thr Asp Ala Ile Gln Ala Glu Glu Leu Tyr Thr Phe
260 265 270
Gly Gly Gln Asp Pro Ser Ile Ile Thr Pro Ser Thr Asp Lys Ser Ile
275 280 285
Tyr Asp Lys Val Leu Gln Asn Phe Arg Gly Ile Val Asp Arg Leu Asn
290 295 300
Lys Val Leu Val Cys Ile Ser Asp Pro Asn Ile Asn Ile Asn Ile Tyr
305 310 315 320
Lys Asn Lys Phe Lys Asp Lys Tyr Lys Phe Val Glu Asp Ser Glu Gly
325 330 335
Lys Tyr Ser Ile Asp Val Glu Ser Phe Asp Lys Leu Tyr Lys Ser Leu
340 345 350
Met Phe Gly Phe Thr Glu Thr Asn Ile Ala Glu Asn Tyr Lys Ile Lys
355 360 365
Thr Arg Ala Ser Tyr Phe Ser Asp Ser Leu Pro Pro Val Lys Ile Lys
370 375 380
Asn Leu Leu Asp Asn Glu Ile Tyr Thr Ile Glu Glu Gly Phe Asn Ile
385 390 395 400
Ser Asp Lys Asp Met Glu Lys Glu Tyr Arg Gly Gln Asn Lys Ala Ile
405 410 415
Asn Lys Gln Ala Tyr Glu Glu Ile Ser Lys Glu His Leu Ala Val Tyr
420 425 430
Lys Ile Gln Met Cys Lys Ser Val Lys Ala Pro Gly Ile Cys Ile Asp
435 440 445
Val Asp Asn Glu Asp Leu Phe Phe Ile Ala Asp Lys Asn Ser Phe Ser
450 455 460
Asp Asp Leu Ser Lys Asn Glu Arg Ile Glu Tyr Asn Thr Gln Ser Asn
465 470 475 480
Tyr Ile Glu Asn Asp Phe Pro Ile Asn Glu Leu Ile Leu Asp Thr Asp
485 490 495
Leu Ile Ser Lys Ile Glu Leu Pro Ser Glu Asn Thr Glu Ser Leu Thr
500 505 510
Asp Phe Asn Val Asp Val Pro Val Tyr Glu Lys Gln Pro Ala Ile Lys
515 520 525
Lys Ile Phe Thr Asp Glu Asn Thr Ile Phe Gln Tyr Leu Tyr Ser Gln
530 535 540
Thr Phe Pro Leu Asp Ile Arg Asp Ile Ser Leu Thr Ser Ser Phe Asp
545 550 555 560
Asp Ala Leu Leu Phe Ser Asn Lys Val Tyr Ser Phe Phe Ser Met Asp
565 570 575
Tyr Ile Lys Thr Ala Asn Lys Val Val Glu Ala Gly Leu Phe Ala Gly
580 585 590
Trp Val Lys Gln Ile Val Asn Asp Phe Val Ile Glu Ala Asn Lys Ser
595 600 605
Asn Thr Met Asp Lys Ile Ala Asp Ile Ser Leu Ile Val Pro Tyr Ile
610 615 620
Gly Leu Ala Leu Asn Val Gly Asn Glu Thr Ala Lys Gly Asn Phe Glu
625 630 635 640
Asn Ala Phe Glu Ile Ala Gly Ala Ser Ile Leu Leu Glu Phe Ile Pro
645 650 655
Glu Leu Leu Ile Pro Val Val Gly Ala Phe Leu Leu Glu Ser Tyr Ile
660 665 670
Asp Asn Lys Asn Lys Ile Ile Lys Thr Ile Asp Asn Ala Leu Thr Lys
675 680 685
Arg Asn Glu Lys Trp Ser Asp Met Tyr Gly Leu Ile Val Ala Gln Trp
690 695 700
Leu Ser Thr Val Asn Thr Gln Phe Tyr Thr Ile Lys Glu Gly Met Tyr
705 710 715 720
Lys Ala Leu Asn Tyr Gln Ala Gln Ala Leu Glu Glu Ile Ile Lys Tyr
725 730 735
Arg Tyr Asn Ile Tyr Ser Glu Lys Glu Lys Ser Asn Ile Asn Ile Asp
740 745 750
Phe Asn Asp Ile Asn Ser Lys Leu Asn Glu Gly Ile Asn Gln Ala Ile
755 760 765
Asp Asn Ile Asn Asn Phe Ile Asn Gly Cys Ser Val Ser Tyr Leu Met
770 775 780
Lys Lys Met Ile Pro Leu Ala Val Glu Lys Leu Leu Asp Phe Asp Asn
785 790 795 800
Thr Leu Lys Lys Asn Leu Leu Asn Tyr Ile Asp Glu Asn Lys Leu Tyr
805 810 815
Leu Ile Gly Ser Ala Glu Tyr Glu Lys Ser Lys Val Asn Lys Tyr Leu
820 825 830
Lys Thr Ile Met Pro Phe Asp Leu Ser Ile Tyr Thr Asn Asp Thr Ile
835 840 845
Leu Ile Glu Met Phe Asn Lys Tyr Asn Ser Glu Ile Leu Asn Asn Ile
850 855 860
Ile Leu Asn Leu Arg Tyr Lys Asp Asn Asn Leu Ile Asp Leu Ser Gly
865 870 875 880
Tyr Gly Ala Lys Val Glu Val Tyr Asp Gly Val Glu Leu Asn Asp Lys
885 890 895
Asn Gln Phe Lys Leu Thr Ser Ser Ala Asn Ser Lys Ile Arg Val Thr
900 905 910
Gln Asn Gln Asn Ile Ile Phe Asn Ser Val Phe Leu Asp Phe Ser Val
915 920 925
Ser Phe Trp Ile Arg Ile Pro Lys Tyr Lys Asn Asp Gly Ile Gln Asn
930 935 940
Tyr Ile His Asn Glu Tyr Thr Ile Ile Asn Cys Met Lys Asn Asn Ser
945 950 955 960
Gly Trp Lys Ile Ser Ile Arg Gly Asn Arg Ile Ile Trp Thr Leu Ile
965 970 975
Asp Ile Asn Gly Lys Thr Lys Ser Val Phe Phe Glu Tyr Asn Ile Arg
980 985 990
Glu Asp Ile Ser Glu Tyr Ile Asn Arg Trp Phe Phe Val Thr Ile Thr
995 1000 1005
Asn Asn Leu Asn Asn Ala Lys Ile Tyr Ile Asn Gly Lys Leu Glu
1010 1015 1020
Ser Asn Thr Asp Ile Lys Asp Ile Arg Glu Val Ile Ala Asn Gly
1025 1030 1035
Glu Ile Ile Phe Lys Leu Asp Gly Asp Ile Asp Arg Thr Gln Phe
1040 1045 1050
Ile Trp Met Lys Tyr Phe Ser Ile Phe Asn Thr Glu Leu Ser Gln
1055 1060 1065
Ser Asn Ile Glu Glu Arg Tyr Lys Ile Gln Ser Tyr Ser Glu Tyr
1070 1075 1080
Leu Lys Asp Phe Trp Gly Asn Pro Leu Met Tyr Asn Lys Glu Tyr
1085 1090 1095
Tyr Met Phe Asn Ala Gly Asn Lys Asn Ser Tyr Ile Lys Leu Lys
1100 1105 1110
Lys Asp Ser Pro Val Gly Glu Ile Leu Thr Arg Ser Lys Tyr Asn
1115 1120 1125
Gln Asn Ser Lys Tyr Ile Asn Tyr Arg Asp Leu Tyr Ile Gly Glu
1130 1135 1140
Lys Phe Ile Ile Arg Arg Lys Ser Asn Ser Gln Ser Ile Asn Asp
1145 1150 1155
Asp Ile Val Arg Lys Glu Asp Tyr Ile Tyr Leu Asp Phe Phe Asn
1160 1165 1170
Leu Asn Gln Glu Trp Arg Val Tyr Thr Tyr Lys Tyr Phe Lys Lys
1175 1180 1185
Glu Glu Glu Lys Leu Phe Leu Ala Pro Ile Ser Asp Ser Asp Glu
1190 1195 1200
Phe Tyr Asn Thr Ile Gln Ile Lys Glu Tyr Asp Glu Gln Pro Thr
1205 1210 1215
Tyr Ser Cys Gln Leu Leu Phe Lys Lys Asp Glu Glu Ser Thr Asp
1220 1225 1230
Glu Ile Gly Leu Ile Gly Ile His Arg Phe Tyr Glu Ser Gly Ile
1235 1240 1245
Val Phe Glu Glu Tyr Lys Asp Tyr Phe Cys Ile Ser Lys Trp Tyr
1250 1255 1260
Leu Lys Glu Val Lys Arg Lys Pro Tyr Asn Leu Lys Leu Gly Cys
1265 1270 1275
Asn Trp Gln Phe Ile Pro Lys Asp Glu Gly Trp Thr Glu
1280 1285 1290
<210> 53
<211> 1291
<212> БЕЛОК
<213> Clostridium botulinum
<400> 53
Met Pro Ile Thr Ile Asn Asn Phe Asn Tyr Ser Asp Pro Val Asp Asn
1 5 10 15
Lys Asn Ile Leu Tyr Leu Asp Thr His Leu Asn Thr Leu Ala Asn Glu
20 25 30
Pro Glu Lys Ala Phe Arg Ile Thr Gly Asn Ile Trp Val Ile Pro Asp
35 40 45
Arg Phe Ser Arg Asn Ser Asn Pro Asn Leu Asn Lys Pro Pro Arg Val
50 55 60
Thr Ser Pro Lys Ser Gly Tyr Tyr Asp Pro Asn Tyr Leu Ser Thr Asp
65 70 75 80
Ser Asp Lys Asp Pro Phe Leu Lys Glu Ile Ile Lys Leu Phe Lys Arg
85 90 95
Ile Asn Ser Arg Glu Ile Gly Glu Glu Leu Ile Tyr Arg Leu Ser Thr
100 105 110
Asp Ile Pro Phe Pro Gly Asn Asn Asn Thr Pro Ile Asn Thr Phe Asp
115 120 125
Phe Asp Val Asp Phe Asn Ser Val Asp Val Lys Thr Arg Gln Gly Asn
130 135 140
Asn Trp Val Lys Thr Gly Ser Ile Asn Pro Ser Val Ile Ile Thr Gly
145 150 155 160
Pro Arg Glu Asn Ile Ile Asp Pro Glu Thr Ser Thr Phe Lys Leu Thr
165 170 175
Asn Asn Thr Phe Ala Ala Gln Glu Gly Phe Gly Ala Leu Ser Ile Ile
180 185 190
Ser Ile Ser Pro Arg Phe Met Leu Thr Tyr Ser Asn Ala Thr Asn Asp
195 200 205
Val Gly Glu Gly Arg Phe Ser Lys Ser Glu Phe Cys Met Asp Pro Ile
210 215 220
Leu Ile Leu Met His Glu Leu Asn His Ala Met His Asn Leu Tyr Gly
225 230 235 240
Ile Ala Ile Pro Asn Asp Gln Thr Ile Ser Ser Val Thr Ser Asn Ile
245 250 255
Phe Tyr Ser Gln Tyr Asn Val Lys Leu Glu Tyr Ala Glu Ile Tyr Ala
260 265 270
Phe Gly Gly Pro Thr Ile Asp Leu Ile Pro Lys Ser Ala Arg Lys Tyr
275 280 285
Phe Glu Glu Lys Ala Leu Asp Tyr Tyr Arg Ser Ile Ala Lys Arg Leu
290 295 300
Asn Ser Ile Thr Thr Ala Asn Pro Ser Ser Phe Asn Lys Tyr Ile Gly
305 310 315 320
Glu Tyr Lys Gln Lys Leu Ile Arg Lys Tyr Arg Phe Val Val Glu Ser
325 330 335
Ser Gly Glu Val Thr Val Asn Arg Asn Lys Phe Val Glu Leu Tyr Asn
340 345 350
Glu Leu Thr Gln Ile Phe Thr Glu Phe Asn Tyr Ala Lys Ile Tyr Asn
355 360 365
Val Gln Asn Arg Lys Ile Tyr Leu Ser Asn Val Tyr Thr Pro Val Thr
370 375 380
Ala Asn Ile Leu Asp Asp Asn Val Tyr Asp Ile Gln Asn Gly Phe Asn
385 390 395 400
Ile Pro Lys Ser Asn Leu Asn Val Leu Phe Met Gly Gln Asn Leu Ser
405 410 415
Arg Asn Pro Ala Leu Arg Lys Val Asn Pro Glu Asn Met Leu Tyr Leu
420 425 430
Phe Thr Lys Phe Cys His Lys Ala Ile Asp Gly Arg Ser Leu Tyr Asn
435 440 445
Lys Thr Leu Asp Cys Arg Glu Leu Leu Val Lys Asn Thr Asp Leu Pro
450 455 460
Phe Ile Gly Asp Ile Ser Asp Val Lys Thr Asp Ile Phe Leu Arg Lys
465 470 475 480
Asp Ile Asn Glu Glu Thr Glu Val Ile Tyr Tyr Pro Asp Asn Val Ser
485 490 495
Val Asp Gln Val Ile Leu Ser Lys Asn Thr Ser Glu His Gly Gln Leu
500 505 510
Asp Leu Leu Tyr Pro Ser Ile Asp Ser Glu Ser Glu Ile Leu Pro Gly
515 520 525
Glu Asn Gln Val Phe Tyr Asp Asn Arg Thr Gln Asn Val Asp Tyr Leu
530 535 540
Asn Ser Tyr Tyr Tyr Leu Glu Ser Gln Lys Leu Ser Asp Asn Val Glu
545 550 555 560
Asp Phe Thr Phe Thr Arg Ser Ile Glu Glu Ala Leu Asp Asn Ser Ala
565 570 575
Lys Val Tyr Thr Tyr Phe Pro Thr Leu Ala Asn Lys Val Asn Ala Gly
580 585 590
Val Gln Gly Gly Leu Phe Leu Met Trp Ala Asn Asp Val Val Glu Asp
595 600 605
Phe Thr Thr Asn Ile Leu Arg Lys Asp Thr Leu Asp Lys Ile Ser Asp
610 615 620
Val Ser Ala Ile Ile Pro Tyr Ile Gly Pro Ala Leu Asn Ile Ser Asn
625 630 635 640
Ser Val Arg Arg Gly Asn Phe Thr Glu Ala Phe Ala Val Thr Gly Val
645 650 655
Thr Ile Leu Leu Glu Ala Phe Pro Glu Phe Thr Ile Pro Ala Leu Gly
660 665 670
Ala Phe Val Ile Tyr Ser Lys Val Gln Glu Arg Asn Glu Ile Ile Lys
675 680 685
Thr Ile Asp Asn Cys Leu Glu Gln Arg Ile Lys Arg Trp Lys Asp Ser
690 695 700
Tyr Glu Trp Met Met Gly Thr Trp Leu Ser Arg Ile Ile Thr Gln Phe
705 710 715 720
Asn Asn Ile Ser Tyr Gln Met Tyr Asp Ser Leu Asn Tyr Gln Ala Gly
725 730 735
Ala Ile Lys Ala Lys Ile Asp Leu Glu Tyr Lys Lys Tyr Ser Gly Ser
740 745 750
Asp Lys Glu Asn Ile Lys Ser Gln Val Glu Asn Leu Lys Asn Ser Leu
755 760 765
Asp Val Lys Ile Ser Glu Ala Met Asn Asn Ile Asn Lys Phe Ile Arg
770 775 780
Glu Cys Ser Val Thr Tyr Leu Phe Lys Asn Met Leu Pro Lys Val Ile
785 790 795 800
Asp Glu Leu Asn Glu Phe Asp Arg Asn Thr Lys Ala Lys Leu Ile Asn
805 810 815
Leu Ile Asp Ser His Asn Ile Ile Leu Val Gly Glu Val Asp Lys Leu
820 825 830
Lys Ala Lys Val Asn Asn Ser Phe Gln Asn Thr Ile Pro Phe Asn Ile
835 840 845
Phe Ser Tyr Thr Asn Asn Ser Leu Leu Lys Asp Ile Ile Asn Glu Tyr
850 855 860
Phe Asn Asn Ile Asn Asp Ser Lys Ile Leu Ser Leu Gln Asn Arg Lys
865 870 875 880
Asn Thr Leu Val Asp Thr Ser Gly Tyr Asn Ala Glu Val Ser Glu Glu
885 890 895
Gly Asp Val Gln Leu Asn Pro Ile Phe Pro Phe Asp Phe Lys Leu Gly
900 905 910
Ser Ser Gly Glu Asp Arg Gly Lys Val Ile Val Thr Gln Asn Glu Asn
915 920 925
Ile Val Tyr Asn Ser Met Tyr Glu Ser Phe Ser Ile Ser Phe Trp Ile
930 935 940
Arg Ile Asn Lys Trp Val Ser Asn Leu Pro Gly Tyr Thr Ile Ile Asp
945 950 955 960
Ser Val Lys Asn Asn Ser Gly Trp Ser Ile Gly Ile Ile Ser Asn Phe
965 970 975
Leu Val Phe Thr Leu Lys Gln Asn Glu Asp Ser Glu Gln Ser Ile Asn
980 985 990
Phe Ser Tyr Asp Ile Ser Asn Asn Ala Pro Gly Tyr Asn Lys Trp Phe
995 1000 1005
Phe Val Thr Val Thr Asn Asn Met Met Gly Asn Met Lys Ile Tyr
1010 1015 1020
Ile Asn Gly Lys Leu Ile Asp Thr Ile Lys Val Lys Glu Leu Thr
1025 1030 1035
Gly Ile Asn Phe Ser Lys Thr Ile Thr Phe Glu Ile Asn Lys Ile
1040 1045 1050
Pro Asp Thr Gly Leu Ile Thr Ser Asp Ser Asp Asn Ile Asn Met
1055 1060 1065
Trp Ile Arg Asp Phe Tyr Ile Phe Ala Lys Glu Leu Asp Gly Lys
1070 1075 1080
Asp Ile Asn Ile Leu Phe Asn Ser Leu Gln Tyr Thr Asn Val Val
1085 1090 1095
Lys Asp Tyr Trp Gly Asn Asp Leu Arg Tyr Asn Lys Glu Tyr Tyr
1100 1105 1110
Met Val Asn Ile Asp Tyr Leu Asn Arg Tyr Met Tyr Ala Asn Ser
1115 1120 1125
Arg Gln Ile Val Phe Asn Thr Arg Arg Asn Asn Asn Asp Phe Asn
1130 1135 1140
Glu Gly Tyr Lys Ile Ile Ile Lys Arg Ile Arg Gly Asn Thr Asn
1145 1150 1155
Asp Thr Arg Val Arg Gly Gly Asp Ile Leu Tyr Phe Asp Met Thr
1160 1165 1170
Ile Asn Asn Lys Ala Tyr Asn Leu Phe Met Lys Asn Glu Thr Met
1175 1180 1185
Tyr Ala Asp Asn His Ser Thr Glu Asp Ile Tyr Ala Ile Gly Leu
1190 1195 1200
Arg Glu Gln Thr Lys Asp Ile Asn Asp Asn Ile Ile Phe Gln Ile
1205 1210 1215
Gln Pro Met Asn Asn Thr Tyr Tyr Tyr Ala Ser Gln Ile Phe Lys
1220 1225 1230
Ser Asn Phe Asn Gly Glu Asn Ile Ser Gly Ile Cys Ser Ile Gly
1235 1240 1245
Thr Tyr Arg Phe Arg Leu Gly Gly Asp Trp Tyr Arg His Asn Tyr
1250 1255 1260
Leu Val Pro Thr Val Lys Gln Gly Asn Tyr Ala Ser Leu Leu Glu
1265 1270 1275
Ser Thr Ser Thr His Trp Gly Phe Val Pro Val Ser Glu
1280 1285 1290
<210> 54
<211> 1276
<212> БЕЛОК
<213> Clostridium botulinum
<400> 54
Met Thr Trp Pro Val Lys Asp Phe Asn Tyr Ser Asp Pro Val Asn Asp
1 5 10 15
Asn Asp Ile Leu Tyr Leu Arg Ile Pro Gln Asn Lys Leu Ile Thr Thr
20 25 30
Pro Val Lys Ala Phe Met Ile Thr Gln Asn Ile Trp Val Ile Pro Glu
35 40 45
Arg Phe Ser Ser Asp Thr Asn Pro Ser Leu Ser Lys Pro Pro Arg Pro
50 55 60
Thr Ser Lys Tyr Gln Ser Tyr Tyr Asp Pro Ser Tyr Leu Ser Thr Asp
65 70 75 80
Glu Gln Lys Asp Thr Phe Leu Lys Gly Ile Ile Lys Leu Phe Lys Arg
85 90 95
Ile Asn Glu Arg Asp Ile Gly Lys Lys Leu Ile Asn Tyr Leu Val Val
100 105 110
Gly Ser Pro Phe Met Gly Asp Ser Ser Thr Pro Glu Asp Thr Phe Asp
115 120 125
Phe Thr Arg His Thr Thr Asn Ile Ala Val Glu Lys Phe Glu Asn Gly
130 135 140
Ser Trp Lys Val Thr Asn Ile Ile Thr Pro Ser Val Leu Ile Phe Gly
145 150 155 160
Pro Leu Pro Asn Ile Leu Asp Tyr Thr Ala Ser Leu Thr Leu Gln Gly
165 170 175
Gln Gln Ser Asn Pro Ser Phe Glu Gly Phe Gly Thr Leu Ser Ile Leu
180 185 190
Lys Val Ala Pro Glu Phe Leu Leu Thr Phe Ser Asp Val Thr Ser Asn
195 200 205
Gln Ser Ser Ala Val Leu Gly Lys Ser Ile Phe Cys Met Asp Pro Val
210 215 220
Ile Ala Leu Met His Glu Leu Thr His Ser Leu His Gln Leu Tyr Gly
225 230 235 240
Ile Asn Ile Pro Ser Asp Lys Arg Ile Arg Pro Gln Val Ser Glu Gly
245 250 255
Phe Phe Ser Gln Asp Gly Pro Asn Val Gln Phe Glu Glu Leu Tyr Thr
260 265 270
Phe Gly Gly Leu Asp Val Glu Ile Ile Pro Gln Ile Glu Arg Ser Gln
275 280 285
Leu Arg Glu Lys Ala Leu Gly His Tyr Lys Asp Ile Ala Lys Arg Leu
290 295 300
Asn Asn Ile Asn Lys Thr Ile Pro Ser Ser Trp Ile Ser Asn Ile Asp
305 310 315 320
Lys Tyr Lys Lys Ile Phe Ser Glu Lys Tyr Asn Phe Asp Lys Asp Asn
325 330 335
Thr Gly Asn Phe Val Val Asn Ile Asp Lys Phe Asn Ser Leu Tyr Ser
340 345 350
Asp Leu Thr Asn Val Met Ser Glu Val Val Tyr Ser Ser Gln Tyr Asn
355 360 365
Val Lys Asn Arg Thr His Tyr Phe Ser Arg His Tyr Leu Pro Val Phe
370 375 380
Ala Asn Ile Leu Asp Asp Asn Ile Tyr Thr Ile Arg Asp Gly Phe Asn
385 390 395 400
Leu Thr Asn Lys Gly Phe Asn Ile Glu Asn Ser Gly Gln Asn Ile Glu
405 410 415
Arg Asn Pro Ala Leu Gln Lys Leu Ser Ser Glu Ser Val Val Asp Leu
420 425 430
Phe Thr Lys Val Cys Leu Arg Leu Thr Lys Asn Ser Arg Asp Asp Ser
435 440 445
Thr Cys Ile Lys Val Lys Asn Asn Arg Leu Pro Tyr Val Ala Asp Lys
450 455 460
Asp Ser Ile Ser Gln Glu Ile Phe Glu Asn Lys Ile Ile Thr Asp Glu
465 470 475 480
Thr Asn Val Gln Asn Tyr Ser Asp Lys Phe Ser Leu Asp Glu Ser Ile
485 490 495
Leu Asp Gly Gln Val Pro Ile Asn Pro Glu Ile Val Asp Pro Leu Leu
500 505 510
Pro Asn Val Asn Met Glu Pro Leu Asn Leu Pro Gly Glu Glu Ile Val
515 520 525
Phe Tyr Asp Asp Ile Thr Lys Tyr Val Asp Tyr Leu Asn Ser Tyr Tyr
530 535 540
Tyr Leu Glu Ser Gln Lys Leu Ser Asn Asn Val Glu Asn Ile Thr Leu
545 550 555 560
Thr Thr Ser Val Glu Glu Ala Leu Gly Tyr Ser Asn Lys Ile Tyr Thr
565 570 575
Phe Leu Pro Ser Leu Ala Glu Lys Val Asn Lys Gly Val Gln Ala Gly
580 585 590
Leu Phe Leu Asn Trp Ala Asn Glu Val Val Glu Asp Phe Thr Thr Asn
595 600 605
Ile Met Lys Lys Asp Thr Leu Asp Lys Ile Ser Asp Val Ser Val Ile
610 615 620
Ile Pro Tyr Ile Gly Pro Ala Leu Asn Ile Gly Asn Ser Ala Leu Arg
625 630 635 640
Gly Asn Phe Asn Gln Ala Phe Ala Thr Ala Gly Val Ala Phe Leu Leu
645 650 655
Glu Gly Phe Pro Glu Phe Thr Ile Pro Ala Leu Gly Val Phe Thr Phe
660 665 670
Tyr Ser Ser Ile Gln Glu Arg Glu Lys Ile Ile Lys Thr Ile Glu Asn
675 680 685
Cys Leu Glu Gln Arg Val Lys Arg Trp Lys Asp Ser Tyr Gln Trp Met
690 695 700
Val Ser Asn Trp Leu Ser Arg Ile Thr Thr Gln Phe Asn His Ile Asn
705 710 715 720
Tyr Gln Met Tyr Asp Ser Leu Ser Tyr Gln Ala Asp Ala Ile Lys Ala
725 730 735
Lys Ile Asp Leu Glu Tyr Lys Lys Tyr Ser Gly Ser Asp Lys Glu Asn
740 745 750
Ile Lys Ser Gln Val Glu Asn Leu Lys Asn Ser Leu Asp Val Lys Ile
755 760 765
Ser Glu Ala Met Asn Asn Ile Asn Lys Phe Ile Arg Glu Cys Ser Val
770 775 780
Thr Tyr Leu Phe Lys Asn Met Leu Pro Lys Val Ile Asp Glu Leu Asn
785 790 795 800
Lys Phe Asp Leu Arg Thr Lys Thr Glu Leu Ile Asn Leu Ile Asp Ser
805 810 815
His Asn Ile Ile Leu Val Gly Glu Val Asp Arg Leu Lys Ala Lys Val
820 825 830
Asn Glu Ser Phe Glu Asn Thr Met Pro Phe Asn Ile Phe Ser Tyr Thr
835 840 845
Asn Asn Ser Leu Leu Lys Asp Ile Ile Asn Glu Tyr Phe Asn Ser Ile
850 855 860
Asn Asp Ser Lys Ile Leu Ser Leu Gln Asn Lys Lys Asn Ala Leu Val
865 870 875 880
Asp Thr Ser Gly Tyr Asn Ala Glu Val Arg Val Gly Asp Asn Val Gln
885 890 895
Leu Asn Thr Ile Tyr Thr Asn Asp Phe Lys Leu Ser Ser Ser Gly Asp
900 905 910
Lys Ile Ile Val Asn Leu Asn Asn Asn Ile Leu Tyr Ser Ala Ile Tyr
915 920 925
Glu Asn Ser Ser Val Ser Phe Trp Ile Lys Ile Ser Lys Asp Leu Thr
930 935 940
Asn Ser His Asn Glu Tyr Thr Ile Ile Asn Ser Ile Glu Gln Asn Ser
945 950 955 960
Gly Trp Lys Leu Cys Ile Arg Asn Gly Asn Ile Glu Trp Ile Leu Gln
965 970 975
Asp Val Asn Arg Lys Tyr Lys Ser Leu Ile Phe Asp Tyr Ser Glu Ser
980 985 990
Leu Ser His Thr Gly Tyr Thr Asn Lys Trp Phe Phe Val Thr Ile Thr
995 1000 1005
Asn Asn Ile Met Gly Tyr Met Lys Leu Tyr Ile Asn Gly Glu Leu
1010 1015 1020
Lys Gln Ser Gln Lys Ile Glu Asp Leu Asp Glu Val Lys Leu Asp
1025 1030 1035
Lys Thr Ile Val Phe Gly Ile Asp Glu Asn Ile Asp Glu Asn Gln
1040 1045 1050
Met Leu Trp Ile Arg Asp Phe Asn Ile Phe Ser Lys Glu Leu Ser
1055 1060 1065
Asn Glu Asp Ile Asn Ile Val Tyr Glu Gly Gln Ile Leu Arg Asn
1070 1075 1080
Val Ile Lys Asp Tyr Trp Gly Asn Pro Leu Lys Phe Asp Thr Glu
1085 1090 1095
Tyr Tyr Ile Ile Asn Asp Asn Tyr Ile Asp Arg Tyr Ile Ala Pro
1100 1105 1110
Glu Ser Asn Val Leu Val Leu Val Gln Tyr Pro Asp Arg Ser Lys
1115 1120 1125
Leu Tyr Thr Gly Asn Pro Ile Thr Ile Lys Ser Val Ser Asp Lys
1130 1135 1140
Asn Pro Tyr Ser Arg Ile Leu Asn Gly Asp Asn Ile Ile Leu His
1145 1150 1155
Met Leu Tyr Asn Ser Arg Lys Tyr Met Ile Ile Arg Asp Thr Asp
1160 1165 1170
Thr Ile Tyr Ala Thr Gln Gly Gly Glu Cys Ser Gln Asn Cys Val
1175 1180 1185
Tyr Ala Leu Lys Leu Gln Ser Asn Leu Gly Asn Tyr Gly Ile Gly
1190 1195 1200
Ile Phe Ser Ile Lys Asn Ile Val Ser Lys Asn Lys Tyr Cys Ser
1205 1210 1215
Gln Ile Phe Ser Ser Phe Arg Glu Asn Thr Met Leu Leu Ala Asp
1220 1225 1230
Ile Tyr Lys Pro Trp Arg Phe Ser Phe Lys Asn Ala Tyr Thr Pro
1235 1240 1245
Val Ala Val Thr Asn Tyr Glu Thr Lys Leu Leu Ser Thr Ser Ser
1250 1255 1260
Phe Trp Lys Phe Ile Ser Arg Asp Pro Gly Trp Val Glu
1265 1270 1275
<210> 55
<211> 1251
<212> БЕЛОК
<213> Clostridium botulinum
<400> 55
Met Pro Lys Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp Arg
1 5 10 15
Thr Ile Leu Tyr Ile Lys Pro Gly Gly Cys Gln Glu Phe Tyr Lys Ser
20 25 30
Phe Asn Ile Met Lys Asn Ile Trp Ile Ile Pro Glu Arg Asn Val Ile
35 40 45
Gly Thr Thr Pro Gln Asp Phe His Pro Pro Thr Ser Leu Lys Asn Gly
50 55 60
Asp Ser Ser Tyr Tyr Asp Pro Asn Tyr Leu Gln Ser Asp Glu Glu Lys
65 70 75 80
Asp Arg Phe Leu Lys Ile Val Thr Lys Ile Phe Asn Arg Ile Asn Asn
85 90 95
Asn Leu Ser Gly Gly Ile Leu Leu Glu Glu Leu Ser Lys Ala Asn Pro
100 105 110
Tyr Leu Gly Asn Asp Asn Thr Pro Asp Asn Gln Phe His Ile Gly Asp
115 120 125
Ala Ser Ala Val Glu Ile Lys Phe Ser Asn Gly Ser Gln Asp Ile Leu
130 135 140
Leu Pro Asn Val Ile Ile Met Gly Ala Glu Pro Asp Leu Phe Glu Thr
145 150 155 160
Asn Ser Ser Asn Ile Ser Leu Arg Asn Asn Tyr Met Pro Ser Asn His
165 170 175
Arg Phe Gly Ser Ile Ala Ile Val Thr Phe Ser Pro Glu Tyr Ser Phe
180 185 190
Arg Phe Asn Asp Asn Cys Met Asn Glu Phe Ile Gln Asp Pro Ala Leu
195 200 205
Thr Leu Met His Glu Leu Ile His Ser Leu His Gly Leu Tyr Gly Ala
210 215 220
Lys Gly Ile Thr Thr Lys Tyr Thr Ile Thr Gln Lys Gln Asn Pro Leu
225 230 235 240
Ile Thr Asn Ile Arg Gly Thr Asn Ile Glu Glu Phe Leu Thr Phe Gly
245 250 255
Gly Thr Asp Leu Asn Ile Ile Thr Ser Ala Gln Ser Asn Asp Ile Tyr
260 265 270
Thr Asn Leu Leu Ala Asp Tyr Lys Lys Ile Ala Ser Lys Leu Ser Lys
275 280 285
Val Gln Val Ser Asn Pro Leu Leu Asn Pro Tyr Lys Asp Val Phe Glu
290 295 300
Ala Lys Tyr Gly Leu Asp Lys Asp Ala Ser Gly Ile Tyr Ser Val Asn
305 310 315 320
Ile Asn Lys Phe Asn Asp Ile Phe Lys Lys Leu Tyr Ser Phe Thr Glu
325 330 335
Phe Asp Leu Arg Thr Lys Phe Gln Val Lys Cys Arg Gln Thr Tyr Ile
340 345 350
Gly Gln Tyr Lys Tyr Phe Lys Leu Ser Asn Leu Leu Asn Asp Ser Ile
355 360 365
Tyr Asn Ile Ser Glu Gly Tyr Asn Ile Asn Asn Leu Lys Val Asn Phe
370 375 380
Arg Gly Gln Asn Ala Asn Leu Asn Pro Arg Ile Ile Thr Pro Ile Thr
385 390 395 400
Gly Arg Gly Leu Val Lys Lys Ile Ile Arg Phe Cys Lys Asn Ile Val
405 410 415
Ser Val Lys Gly Ile Arg Lys Ser Ile Cys Ile Glu Ile Asn Asn Gly
420 425 430
Glu Leu Phe Phe Val Ala Ser Glu Asn Ser Tyr Asn Asp Asp Asn Ile
435 440 445
Asn Thr Pro Lys Glu Ile Asp Asp Thr Val Thr Ser Asn Asn Asn Tyr
450 455 460
Glu Asn Asp Leu Asp Gln Val Ile Leu Asn Phe Asn Ser Glu Ser Ala
465 470 475 480
Pro Gly Leu Ser Asp Glu Lys Leu Asn Leu Thr Ile Gln Asn Asp Ala
485 490 495
Tyr Ile Pro Lys Tyr Asp Ser Asn Gly Thr Ser Asp Ile Glu Gln His
500 505 510
Asp Val Asn Glu Leu Asn Val Phe Phe Tyr Leu Asp Ala Gln Lys Val
515 520 525
Pro Glu Gly Glu Asn Asn Val Asn Leu Thr Ser Ser Ile Asp Thr Ala
530 535 540
Leu Leu Glu Gln Pro Lys Ile Tyr Thr Phe Phe Ser Ser Glu Phe Ile
545 550 555 560
Asn Asn Val Asn Lys Pro Val Gln Ala Ala Leu Phe Val Ser Trp Ile
565 570 575
Gln Gln Val Leu Val Asp Phe Thr Thr Glu Ala Asn Gln Lys Ser Thr
580 585 590
Val Asp Lys Ile Ala Asp Ile Ser Ile Val Val Pro Tyr Ile Gly Leu
595 600 605
Ala Leu Asn Ile Gly Asn Glu Ala Gln Lys Gly Asn Phe Lys Asp Ala
610 615 620
Leu Glu Leu Leu Gly Ala Gly Ile Leu Leu Glu Phe Glu Pro Glu Leu
625 630 635 640
Leu Ile Pro Thr Ile Leu Val Phe Thr Ile Lys Ser Phe Leu Gly Ser
645 650 655
Ser Asp Asn Lys Asn Lys Val Ile Lys Ala Ile Asn Asn Ala Leu Lys
660 665 670
Glu Arg Asp Glu Lys Trp Lys Glu Val Tyr Ser Phe Ile Val Ser Asn
675 680 685
Trp Met Thr Lys Ile Asn Thr Gln Phe Asn Lys Arg Lys Glu Gln Met
690 695 700
Tyr Gln Ala Leu Gln Asn Gln Val Asn Ala Ile Lys Thr Ile Ile Glu
705 710 715 720
Ser Lys Tyr Asn Ser Tyr Thr Leu Glu Glu Lys Asn Glu Leu Thr Asn
725 730 735
Lys Tyr Asp Ile Lys Gln Ile Glu Asn Glu Leu Asn Gln Lys Val Ser
740 745 750
Ile Ala Met Asn Asn Ile Asp Arg Phe Leu Thr Glu Ser Ser Ile Ser
755 760 765
Tyr Leu Met Lys Ile Ile Asn Glu Val Lys Ile Asn Lys Leu Arg Glu
770 775 780
Tyr Asp Glu Asn Val Lys Thr Tyr Leu Leu Asn Tyr Ile Ile Gln His
785 790 795 800
Gly Ser Ile Leu Gly Glu Ser Gln Gln Glu Leu Asn Ser Met Val Thr
805 810 815
Asp Thr Leu Asn Asn Ser Ile Pro Phe Lys Leu Ser Ser Tyr Thr Asp
820 825 830
Asp Lys Ile Leu Ile Ser Tyr Phe Asn Lys Phe Phe Lys Arg Ile Lys
835 840 845
Ser Ser Ser Val Leu Asn Met Arg Tyr Lys Asn Asp Lys Tyr Val Asp
850 855 860
Thr Ser Gly Tyr Asp Ser Asn Ile Asn Ile Asn Gly Asp Val Tyr Lys
865 870 875 880
Tyr Pro Thr Asn Lys Asn Gln Phe Gly Ile Tyr Asn Asp Lys Leu Ser
885 890 895
Glu Val Asn Ile Ser Gln Asn Asp Tyr Ile Ile Tyr Asp Asn Lys Tyr
900 905 910
Lys Asn Phe Ser Ile Ser Phe Trp Val Arg Ile Pro Asn Tyr Asp Asn
915 920 925
Lys Ile Val Asn Val Asn Asn Glu Tyr Thr Ile Ile Asn Cys Met Arg
930 935 940
Asp Asn Asn Ser Gly Trp Lys Val Ser Leu Asn His Asn Glu Ile Ile
945 950 955 960
Trp Thr Phe Glu Asp Asn Arg Gly Ile Asn Gln Lys Leu Ala Phe Asn
965 970 975
Tyr Gly Asn Ala Asn Gly Ile Ser Asp Tyr Ile Asn Lys Trp Ile Phe
980 985 990
Val Thr Ile Thr Asn Asp Arg Leu Gly Asp Ser Lys Leu Tyr Ile Asn
995 1000 1005
Gly Asn Leu Ile Asp Gln Lys Ser Ile Leu Asn Leu Gly Asn Ile
1010 1015 1020
His Val Ser Asp Asn Ile Leu Phe Lys Ile Val Asn Cys Ser Tyr
1025 1030 1035
Thr Arg Tyr Ile Gly Ile Arg Tyr Phe Asn Ile Phe Asp Lys Glu
1040 1045 1050
Leu Asp Glu Thr Glu Ile Gln Thr Leu Tyr Ser Asn Glu Pro Asn
1055 1060 1065
Thr Asn Ile Leu Lys Asp Phe Trp Gly Asn Tyr Leu Leu Tyr Asp
1070 1075 1080
Lys Glu Tyr Tyr Leu Leu Asn Val Leu Lys Pro Asn Asn Phe Ile
1085 1090 1095
Asp Arg Arg Lys Asp Ser Thr Leu Ser Ile Asn Asn Ile Arg Ser
1100 1105 1110
Thr Ile Leu Leu Ala Asn Arg Leu Tyr Ser Gly Ile Lys Val Lys
1115 1120 1125
Ile Gln Arg Val Asn Asn Ser Ser Thr Asn Asp Asn Leu Val Arg
1130 1135 1140
Lys Asn Asp Gln Val Tyr Ile Asn Phe Val Ala Ser Lys Thr His
1145 1150 1155
Leu Phe Pro Leu Tyr Ala Asp Thr Ala Thr Thr Asn Lys Glu Lys
1160 1165 1170
Thr Ile Lys Ile Ser Ser Ser Gly Asn Arg Phe Asn Gln Val Val
1175 1180 1185
Val Met Asn Ser Val Gly Asn Cys Thr Met Asn Phe Lys Asn Asn
1190 1195 1200
Asn Gly Asn Asn Ile Gly Leu Leu Gly Phe Lys Ala Asp Thr Val
1205 1210 1215
Val Ala Ser Thr Trp Tyr Tyr Thr His Met Arg Asp His Thr Asn
1220 1225 1230
Ser Asn Gly Cys Phe Trp Asn Phe Ile Ser Glu Glu His Gly Trp
1235 1240 1245
Gln Glu Lys
1250
<210> 56
<211> 1278
<212> БЕЛОК
<213> Clostridium botulinum
<400> 56
Met Pro Val Val Ile Asn Ser Phe Asn Tyr Asn Asp Pro Val Asn Asp
1 5 10 15
Asp Thr Ile Leu Tyr Met Gln Ile Pro Tyr Glu Glu Lys Ser Lys Lys
20 25 30
Tyr Tyr Lys Ala Phe Glu Ile Met Arg Asn Val Trp Ile Ile Pro Glu
35 40 45
Arg Asn Thr Ile Gly Thr Asp Pro Ser Asp Phe Asp Pro Pro Ala Ser
50 55 60
Leu Glu Asn Gly Ser Ser Ala Tyr Tyr Asp Pro Asn Tyr Leu Thr Thr
65 70 75 80
Asp Ala Glu Lys Asp Arg Tyr Leu Lys Thr Thr Ile Lys Leu Phe Lys
85 90 95
Arg Ile Asn Ser Asn Pro Ala Gly Glu Val Leu Leu Gln Glu Ile Ser
100 105 110
Tyr Ala Lys Pro Tyr Leu Gly Asn Glu His Thr Pro Ile Asn Glu Phe
115 120 125
His Pro Val Thr Arg Thr Thr Ser Val Asn Ile Lys Ser Ser Thr Asn
130 135 140
Val Lys Ser Ser Ile Ile Leu Asn Leu Leu Val Leu Gly Ala Gly Pro
145 150 155 160
Asp Ile Phe Glu Asn Ser Ser Tyr Pro Val Arg Lys Leu Met Asp Ser
165 170 175
Gly Gly Val Tyr Asp Pro Ser Asn Asp Gly Phe Gly Ser Ile Asn Ile
180 185 190
Val Thr Phe Ser Pro Glu Tyr Glu Tyr Thr Phe Asn Asp Ile Ser Gly
195 200 205
Gly Tyr Asn Ser Ser Thr Glu Ser Phe Ile Ala Asp Pro Ala Ile Ser
210 215 220
Leu Ala His Glu Leu Ile His Ala Leu His Gly Leu Tyr Gly Ala Arg
225 230 235 240
Gly Val Thr Tyr Lys Glu Thr Ile Lys Val Lys Gln Ala Pro Leu Met
245 250 255
Ile Ala Glu Lys Pro Ile Arg Leu Glu Glu Phe Leu Thr Phe Gly Gly
260 265 270
Gln Asp Leu Asn Ile Ile Thr Ser Ala Met Lys Glu Lys Ile Tyr Asn
275 280 285
Asn Leu Leu Ala Asn Tyr Glu Lys Ile Ala Thr Arg Leu Ser Arg Val
290 295 300
Asn Ser Ala Pro Pro Glu Tyr Asp Ile Asn Glu Tyr Lys Asp Tyr Phe
305 310 315 320
Gln Trp Lys Tyr Gly Leu Asp Lys Asn Ala Asp Gly Ser Tyr Thr Val
325 330 335
Asn Glu Asn Lys Phe Asn Glu Ile Tyr Lys Lys Leu Tyr Ser Phe Thr
340 345 350
Glu Ile Asp Leu Ala Asn Lys Phe Lys Val Lys Cys Arg Asn Thr Tyr
355 360 365
Phe Ile Lys Tyr Gly Phe Leu Lys Val Pro Asn Leu Leu Asp Asp Asp
370 375 380
Ile Tyr Thr Val Ser Glu Gly Phe Asn Ile Gly Asn Leu Ala Val Asn
385 390 395 400
Asn Arg Gly Gln Asn Ile Lys Leu Asn Pro Lys Ile Ile Asp Ser Ile
405 410 415
Pro Asp Lys Gly Leu Val Glu Lys Ile Val Lys Phe Cys Lys Ser Val
420 425 430
Ile Pro Arg Lys Gly Thr Lys Ala Pro Pro Arg Leu Cys Ile Arg Val
435 440 445
Asn Asn Arg Glu Leu Phe Phe Val Ala Ser Glu Ser Ser Tyr Asn Glu
450 455 460
Asn Asp Ile Asn Thr Pro Lys Glu Ile Asp Asp Thr Thr Asn Leu Asn
465 470 475 480
Asn Asn Tyr Arg Asn Asn Leu Asp Glu Val Ile Leu Asp Tyr Asn Ser
485 490 495
Glu Thr Ile Pro Gln Ile Ser Asn Gln Thr Leu Asn Thr Leu Val Gln
500 505 510
Asp Asp Ser Tyr Val Pro Arg Tyr Asp Ser Asn Gly Thr Ser Glu Ile
515 520 525
Glu Glu His Asn Val Val Asp Leu Asn Val Phe Phe Tyr Leu His Ala
530 535 540
Gln Lys Val Pro Glu Gly Glu Thr Asn Ile Ser Leu Thr Ser Ser Ile
545 550 555 560
Asp Thr Ala Leu Ser Glu Glu Ser Gln Val Tyr Thr Phe Phe Ser Ser
565 570 575
Glu Phe Ile Asn Thr Ile Asn Lys Pro Val His Ala Ala Leu Phe Ile
580 585 590
Ser Trp Ile Asn Gln Val Ile Arg Asp Phe Thr Thr Glu Ala Thr Gln
595 600 605
Lys Ser Thr Phe Asp Lys Ile Ala Asp Ile Ser Leu Val Val Pro Tyr
610 615 620
Val Gly Leu Ala Leu Asn Ile Gly Asn Glu Val Gln Lys Glu Asn Phe
625 630 635 640
Lys Glu Ala Phe Glu Leu Leu Gly Ala Gly Ile Leu Leu Glu Phe Val
645 650 655
Pro Glu Leu Leu Ile Pro Thr Ile Leu Val Phe Thr Ile Lys Ser Phe
660 665 670
Ile Gly Ser Ser Glu Asn Lys Asn Lys Ile Ile Lys Ala Ile Asn Asn
675 680 685
Ser Leu Met Glu Arg Glu Thr Lys Trp Lys Glu Ile Tyr Ser Trp Ile
690 695 700
Val Ser Asn Trp Leu Thr Arg Ile Asn Thr Gln Phe Asn Lys Arg Lys
705 710 715 720
Glu Gln Met Tyr Gln Ala Leu Gln Asn Gln Val Asp Ala Ile Lys Thr
725 730 735
Val Ile Glu Tyr Lys Tyr Asn Asn Tyr Thr Ser Asp Glu Arg Asn Arg
740 745 750
Leu Glu Ser Glu Tyr Asn Ile Asn Asn Ile Arg Glu Glu Leu Asn Lys
755 760 765
Lys Val Ser Leu Ala Met Glu Asn Ile Glu Arg Phe Ile Thr Glu Ser
770 775 780
Ser Ile Phe Tyr Leu Met Lys Leu Ile Asn Glu Ala Lys Val Ser Lys
785 790 795 800
Leu Arg Glu Tyr Asp Glu Gly Val Lys Glu Tyr Leu Leu Asp Tyr Ile
805 810 815
Ser Glu His Arg Ser Ile Leu Gly Asn Ser Val Gln Glu Leu Asn Asp
820 825 830
Leu Val Thr Ser Thr Leu Asn Asn Ser Ile Pro Phe Glu Leu Ser Ser
835 840 845
Tyr Thr Asn Asp Lys Ile Leu Ile Leu Tyr Phe Asn Lys Leu Tyr Lys
850 855 860
Lys Ile Lys Asp Asn Ser Ile Leu Asp Met Arg Tyr Glu Asn Asn Lys
865 870 875 880
Phe Ile Asp Ile Ser Gly Tyr Gly Ser Asn Ile Ser Ile Asn Gly Asp
885 890 895
Val Tyr Ile Tyr Ser Thr Asn Arg Asn Gln Phe Gly Ile Tyr Ser Ser
900 905 910
Lys Pro Ser Glu Val Asn Ile Ala Gln Asn Asn Asp Ile Ile Tyr Asn
915 920 925
Gly Arg Tyr Gln Asn Phe Ser Ile Ser Phe Trp Val Arg Ile Pro Lys
930 935 940
Tyr Phe Asn Lys Val Asn Leu Asn Asn Glu Tyr Thr Ile Ile Asp Cys
945 950 955 960
Ile Arg Asn Asn Asn Ser Gly Trp Lys Ile Ser Leu Asn Tyr Asn Lys
965 970 975
Ile Ile Trp Thr Leu Gln Asp Thr Ala Gly Asn Asn Gln Lys Leu Val
980 985 990
Phe Asn Tyr Thr Gln Met Ile Ser Ile Ser Asp Tyr Ile Asn Lys Trp
995 1000 1005
Ile Phe Val Thr Ile Thr Asn Asn Arg Leu Gly Asn Ser Arg Ile
1010 1015 1020
Tyr Ile Asn Gly Asn Leu Ile Asp Glu Lys Ser Ile Ser Asn Leu
1025 1030 1035
Gly Asp Ile His Val Ser Asp Asn Ile Leu Phe Lys Ile Val Gly
1040 1045 1050
Cys Asn Asp Thr Arg Tyr Val Gly Ile Arg Tyr Phe Lys Val Phe
1055 1060 1065
Asp Thr Glu Leu Gly Lys Thr Glu Ile Glu Thr Leu Tyr Ser Asp
1070 1075 1080
Glu Pro Asp Pro Ser Ile Leu Lys Asp Phe Trp Gly Asn Tyr Leu
1085 1090 1095
Leu Tyr Asn Lys Arg Tyr Tyr Leu Leu Asn Leu Leu Arg Thr Asp
1100 1105 1110
Lys Ser Ile Thr Gln Asn Ser Asn Phe Leu Asn Ile Asn Gln Gln
1115 1120 1125
Arg Gly Val Tyr Gln Lys Pro Asn Ile Phe Ser Asn Thr Arg Leu
1130 1135 1140
Tyr Thr Gly Val Glu Val Ile Ile Arg Lys Asn Gly Ser Thr Asp
1145 1150 1155
Ile Ser Asn Thr Asp Asn Phe Val Arg Lys Asn Asp Leu Ala Tyr
1160 1165 1170
Ile Asn Val Val Asp Arg Asp Val Glu Tyr Arg Leu Tyr Ala Asp
1175 1180 1185
Ile Ser Ile Ala Lys Pro Glu Lys Ile Ile Lys Leu Ile Arg Thr
1190 1195 1200
Ser Asn Ser Asn Asn Ser Leu Gly Gln Ile Ile Val Met Asp Ser
1205 1210 1215
Ile Gly Asn Asn Cys Thr Met Asn Phe Gln Asn Asn Asn Gly Gly
1220 1225 1230
Asn Ile Gly Leu Leu Gly Phe His Ser Asn Asn Leu Val Ala Ser
1235 1240 1245
Ser Trp Tyr Tyr Asn Asn Ile Arg Lys Asn Thr Ser Ser Asn Gly
1250 1255 1260
Cys Phe Trp Ser Phe Ile Ser Lys Glu His Gly Trp Gln Glu Asn
1265 1270 1275
<210> 57
<211> 1297
<212> БЕЛОК
<213> Clostridium botulinum
<220>
<221> прочий_признак
<222> (7)..(7)
<223> Xaa может быть любой существующей в природе аминокислотой
<400> 57
Met Pro Val Asn Ile Lys Xaa Phe Asn Tyr Asn Asp Pro Ile Asn Asn
1 5 10 15
Asp Asp Ile Ile Met Met Glu Pro Phe Asn Asp Pro Gly Pro Gly Thr
20 25 30
Tyr Tyr Lys Ala Phe Arg Ile Ile Asp Arg Ile Trp Ile Val Pro Glu
35 40 45
Arg Phe Thr Tyr Gly Phe Gln Pro Asp Gln Phe Asn Ala Ser Thr Gly
50 55 60
Val Phe Ser Lys Asp Val Tyr Glu Tyr Tyr Asp Pro Thr Tyr Leu Lys
65 70 75 80
Thr Asp Ala Glu Lys Asp Lys Phe Leu Lys Thr Met Ile Lys Leu Phe
85 90 95
Asn Arg Ile Asn Ser Lys Pro Ser Gly Gln Arg Leu Leu Asp Met Ile
100 105 110
Val Asp Ala Ile Pro Tyr Leu Gly Asn Ala Ser Thr Pro Pro Asp Lys
115 120 125
Phe Ala Ala Asn Val Ala Asn Val Ser Ile Asn Lys Lys Ile Ile Gln
130 135 140
Pro Gly Ala Glu Asp Gln Ile Lys Gly Leu Met Thr Asn Leu Ile Ile
145 150 155 160
Phe Gly Pro Gly Pro Val Leu Ser Asp Asn Phe Thr Asp Ser Met Ile
165 170 175
Met Asn Gly His Ser Pro Ile Ser Glu Gly Phe Gly Ala Arg Met Met
180 185 190
Ile Arg Phe Cys Pro Ser Cys Leu Asn Val Phe Asn Asn Val Gln Glu
195 200 205
Asn Lys Asp Thr Ser Ile Phe Ser Arg Arg Ala Tyr Phe Ala Asp Pro
210 215 220
Ala Leu Thr Leu Met His Glu Leu Ile His Val Leu His Gly Leu Tyr
225 230 235 240
Gly Ile Lys Ile Ser Asn Leu Pro Ile Thr Pro Asn Thr Lys Glu Phe
245 250 255
Phe Met Gln His Ser Asp Pro Val Gln Ala Glu Glu Leu Tyr Thr Phe
260 265 270
Gly Gly His Asp Pro Ser Val Ile Ser Pro Ser Thr Asp Met Asn Ile
275 280 285
Tyr Asn Lys Ala Leu Gln Asn Phe Gln Asp Ile Ala Asn Arg Leu Asn
290 295 300
Ile Val Ser Ser Ala Gln Gly Ser Gly Ile Asp Ile Ser Leu Tyr Lys
305 310 315 320
Gln Ile Tyr Lys Asn Lys Tyr Asp Phe Val Glu Asp Pro Asn Gly Lys
325 330 335
Tyr Ser Val Asp Lys Asp Lys Phe Asp Lys Leu Tyr Lys Ala Leu Met
340 345 350
Phe Gly Phe Thr Glu Thr Asn Leu Ala Gly Glu Tyr Gly Ile Lys Thr
355 360 365
Arg Tyr Ser Tyr Phe Ser Glu Tyr Leu Pro Pro Ile Lys Thr Glu Lys
370 375 380
Leu Leu Asp Asn Thr Ile Tyr Thr Gln Asn Glu Gly Phe Asn Ile Ala
385 390 395 400
Ser Lys Asn Leu Lys Thr Glu Phe Asn Gly Gln Asn Lys Ala Val Asn
405 410 415
Lys Glu Ala Tyr Glu Glu Ile Ser Leu Glu His Leu Val Ile Tyr Arg
420 425 430
Ile Ala Met Cys Lys Pro Val Met Tyr Lys Asn Thr Gly Lys Ser Glu
435 440 445
Gln Cys Ile Ile Val Asn Asn Glu Asp Leu Phe Phe Ile Ala Asn Lys
450 455 460
Asp Ser Phe Ser Lys Asp Leu Ala Lys Ala Glu Thr Ile Ala Tyr Asn
465 470 475 480
Thr Gln Asn Asn Thr Ile Glu Asn Asn Phe Ser Ile Asp Gln Leu Ile
485 490 495
Leu Asp Asn Asp Leu Ser Ser Gly Ile Asp Leu Pro Asn Glu Asn Thr
500 505 510
Glu Pro Phe Thr Asn Phe Asp Asp Ile Asp Ile Pro Val Tyr Ile Lys
515 520 525
Gln Ser Ala Leu Lys Lys Ile Phe Val Asp Gly Asp Ser Leu Phe Glu
530 535 540
Tyr Leu His Ala Gln Thr Phe Pro Ser Asn Ile Glu Asn Leu Gln Leu
545 550 555 560
Thr Asn Ser Leu Asn Asp Ala Leu Arg Asn Asn Asn Lys Val Tyr Thr
565 570 575
Phe Phe Ser Thr Asn Leu Val Glu Lys Ala Asn Thr Val Val Gly Ala
580 585 590
Ser Leu Phe Val Asn Trp Val Lys Gly Val Ile Asp Asp Phe Thr Ser
595 600 605
Glu Ser Thr Gln Lys Ser Thr Ile Asp Lys Val Ser Asp Val Ser Ile
610 615 620
Ile Ile Pro Tyr Ile Gly Pro Ala Leu Asn Val Gly Asn Glu Thr Ala
625 630 635 640
Lys Glu Asn Phe Lys Asn Ala Phe Glu Ile Gly Gly Ala Ala Ile Leu
645 650 655
Met Glu Phe Ile Pro Glu Leu Ile Val Pro Ile Val Gly Phe Phe Thr
660 665 670
Leu Glu Ser Tyr Val Gly Asn Lys Gly His Ile Ile Met Thr Ile Ser
675 680 685
Asn Ala Leu Lys Lys Arg Asp Gln Lys Trp Thr Asp Met Tyr Gly Leu
690 695 700
Ile Val Ser Gln Trp Leu Ser Thr Val Asn Thr Gln Phe Tyr Thr Ile
705 710 715 720
Lys Glu Arg Met Tyr Asn Ala Leu Asn Asn Gln Ser Gln Ala Ile Glu
725 730 735
Lys Ile Ile Glu Asp Gln Tyr Asn Arg Tyr Ser Glu Glu Asp Lys Met
740 745 750
Asn Ile Asn Ile Asp Phe Asn Asp Ile Asp Phe Lys Leu Asn Gln Ser
755 760 765
Ile Asn Leu Ala Ile Asn Asn Ile Asp Asp Phe Ile Asn Gln Cys Ser
770 775 780
Ile Ser Tyr Leu Met Asn Arg Met Ile Pro Leu Ala Val Lys Lys Leu
785 790 795 800
Lys Asp Phe Asp Asp Asn Leu Lys Arg Asp Leu Leu Glu Tyr Ile Asp
805 810 815
Thr Asn Glu Leu Tyr Leu Leu Asp Glu Val Asn Ile Leu Lys Ser Lys
820 825 830
Val Asn Arg His Leu Lys Asp Ser Ile Pro Phe Asp Leu Ser Leu Tyr
835 840 845
Thr Lys Asp Thr Ile Leu Ile Gln Val Phe Asn Asn Tyr Ile Ser Asn
850 855 860
Ile Ser Ser Asn Ala Ile Leu Ser Leu Ser Tyr Arg Gly Gly Arg Leu
865 870 875 880
Ile Asp Ser Ser Gly Tyr Gly Ala Thr Met Asn Val Gly Ser Asp Val
885 890 895
Ile Phe Asn Asp Ile Gly Asn Gly Gln Phe Lys Leu Asn Asn Ser Glu
900 905 910
Asn Ser Asn Ile Thr Ala His Gln Ser Lys Phe Val Val Tyr Asp Ser
915 920 925
Met Phe Asp Asn Phe Ser Ile Asn Phe Trp Val Arg Thr Pro Lys Tyr
930 935 940
Asn Asn Asn Asp Ile Gln Thr Tyr Leu Gln Asn Glu Tyr Thr Ile Ile
945 950 955 960
Ser Cys Ile Lys Asn Asp Ser Gly Trp Lys Val Ser Ile Lys Gly Asn
965 970 975
Arg Ile Ile Trp Thr Leu Ile Asp Val Asn Ala Lys Ser Lys Ser Ile
980 985 990
Phe Phe Glu Tyr Ser Ile Lys Asp Asn Ile Ser Asp Tyr Ile Asn Lys
995 1000 1005
Trp Phe Ser Ile Thr Ile Thr Asn Asp Arg Leu Gly Asn Ala Asn
1010 1015 1020
Ile Tyr Ile Asn Gly Ser Leu Lys Lys Ser Glu Lys Ile Leu Asn
1025 1030 1035
Leu Asp Arg Ile Asn Ser Ser Asn Asp Ile Asp Phe Lys Leu Ile
1040 1045 1050
Asn Cys Thr Asp Thr Thr Lys Phe Val Trp Ile Lys Asp Phe Asn
1055 1060 1065
Ile Phe Gly Arg Glu Leu Asn Ala Thr Glu Val Ser Ser Leu Tyr
1070 1075 1080
Trp Ile Gln Ser Ser Thr Asn Thr Leu Lys Asp Phe Trp Gly Asn
1085 1090 1095
Pro Leu Arg Tyr Asp Thr Gln Tyr Tyr Leu Phe Asn Gln Gly Met
1100 1105 1110
Gln Asn Ile Tyr Ile Lys Tyr Phe Ser Lys Ala Ser Met Gly Glu
1115 1120 1125
Thr Ala Pro Arg Thr Asn Phe Asn Asn Ala Ala Ile Asn Tyr Gln
1130 1135 1140
Asn Leu Tyr Leu Gly Leu Arg Phe Ile Ile Lys Lys Ala Ser Asn
1145 1150 1155
Ser Arg Asn Ile Asn Asn Asp Asn Ile Val Arg Glu Gly Asp Tyr
1160 1165 1170
Ile Tyr Leu Asn Ile Asp Asn Ile Ser Asp Glu Ser Tyr Arg Val
1175 1180 1185
Tyr Val Leu Val Asn Ser Lys Glu Ile Gln Thr Gln Leu Phe Leu
1190 1195 1200
Ala Pro Ile Asn Asp Asp Pro Thr Phe Tyr Asp Val Leu Gln Ile
1205 1210 1215
Lys Lys Tyr Tyr Glu Lys Thr Thr Tyr Asn Cys Gln Ile Leu Cys
1220 1225 1230
Glu Lys Asp Thr Lys Thr Phe Gly Leu Phe Gly Ile Gly Lys Phe
1235 1240 1245
Val Lys Asp Tyr Gly Tyr Val Trp Asp Thr Tyr Asp Asn Tyr Phe
1250 1255 1260
Cys Ile Ser Gln Trp Tyr Leu Arg Arg Ile Ser Glu Asn Ile Asn
1265 1270 1275
Lys Leu Arg Leu Gly Cys Asn Trp Gln Phe Ile Pro Val Asp Glu
1280 1285 1290
Gly Trp Thr Glu
1295
<210> 58
<211> 1315
<212> БЕЛОК
<213> Clostridium tetani
<400> 58
Met Pro Ile Thr Ile Asn Asn Phe Arg Tyr Ser Asp Pro Val Asn Asn
1 5 10 15
Asp Thr Ile Ile Met Met Glu Pro Pro Tyr Cys Lys Gly Leu Asp Ile
20 25 30
Tyr Tyr Lys Ala Phe Lys Ile Thr Asp Arg Ile Trp Ile Val Pro Glu
35 40 45
Arg Tyr Glu Phe Gly Thr Lys Pro Glu Asp Phe Asn Pro Pro Ser Ser
50 55 60
Leu Ile Glu Gly Ala Ser Glu Tyr Tyr Asp Pro Asn Tyr Leu Arg Thr
65 70 75 80
Asp Ser Asp Lys Asp Arg Phe Leu Gln Thr Met Val Lys Leu Phe Asn
85 90 95
Arg Ile Lys Asn Asn Val Ala Gly Glu Ala Leu Leu Asp Lys Ile Ile
100 105 110
Asn Ala Ile Pro Tyr Leu Gly Asn Ser Tyr Ser Leu Leu Asp Lys Phe
115 120 125
Asp Thr Asn Ser Asn Ser Val Ser Phe Asn Leu Leu Glu Gln Asp Pro
130 135 140
Ser Gly Ala Thr Thr Lys Ser Ala Met Leu Thr Asn Leu Ile Ile Phe
145 150 155 160
Gly Pro Gly Pro Val Leu Asn Lys Asn Glu Val Arg Gly Ile Val Leu
165 170 175
Arg Val Asp Asn Lys Asn Tyr Phe Pro Cys Arg Asp Gly Phe Gly Ser
180 185 190
Ile Met Gln Met Ala Phe Cys Pro Glu Tyr Val Pro Thr Phe Asp Asn
195 200 205
Val Ile Glu Asn Ile Thr Ser Leu Thr Ile Gly Lys Ser Lys Tyr Phe
210 215 220
Gln Asp Pro Ala Leu Leu Leu Met His Glu Leu Ile His Val Leu His
225 230 235 240
Gly Leu Tyr Gly Met Gln Val Ser Ser His Glu Ile Ile Pro Ser Lys
245 250 255
Gln Glu Ile Tyr Met Gln His Thr Tyr Pro Ile Ser Ala Glu Glu Leu
260 265 270
Phe Thr Phe Gly Gly Gln Asp Ala Asn Leu Ile Ser Ile Asp Ile Lys
275 280 285
Asn Asp Leu Tyr Glu Lys Thr Leu Asn Asp Tyr Lys Ala Ile Ala Asn
290 295 300
Lys Leu Ser Gln Val Thr Ser Cys Asn Asp Pro Asn Ile Asp Ile Asp
305 310 315 320
Ser Tyr Lys Gln Ile Tyr Gln Gln Lys Tyr Gln Phe Asp Lys Asp Ser
325 330 335
Asn Gly Gln Tyr Ile Val Asn Glu Asp Lys Phe Gln Ile Leu Tyr Asn
340 345 350
Ser Ile Met Tyr Gly Phe Thr Glu Ile Glu Leu Gly Lys Lys Phe Asn
355 360 365
Ile Lys Thr Arg Leu Ser Tyr Phe Ser Met Asn His Asp Pro Val Lys
370 375 380
Ile Pro Asn Leu Leu Asp Asp Thr Ile Tyr Asn Asp Thr Glu Gly Phe
385 390 395 400
Asn Ile Glu Ser Lys Asp Leu Lys Ser Glu Tyr Lys Gly Gln Asn Met
405 410 415
Arg Val Asn Thr Asn Ala Phe Arg Asn Val Asp Gly Ser Gly Leu Val
420 425 430
Ser Lys Leu Ile Gly Leu Cys Lys Lys Ile Ile Pro Pro Thr Asn Ile
435 440 445
Arg Glu Asn Leu Tyr Asn Arg Thr Ala Ser Leu Thr Asp Leu Gly Gly
450 455 460
Glu Leu Cys Ile Lys Ile Lys Asn Glu Asp Leu Thr Phe Ile Ala Glu
465 470 475 480
Lys Asn Ser Phe Ser Glu Glu Pro Phe Gln Asp Glu Ile Val Ser Tyr
485 490 495
Asn Thr Lys Asn Lys Pro Leu Asn Phe Asn Tyr Ser Leu Asp Lys Ile
500 505 510
Ile Val Asp Tyr Asn Leu Gln Ser Lys Ile Thr Leu Pro Asn Asp Arg
515 520 525
Thr Thr Pro Val Thr Lys Gly Ile Pro Tyr Ala Pro Glu Tyr Lys Ser
530 535 540
Asn Ala Ala Ser Thr Ile Glu Ile His Asn Ile Asp Asp Asn Thr Ile
545 550 555 560
Tyr Gln Tyr Leu Tyr Ala Gln Lys Ser Pro Thr Thr Leu Gln Arg Ile
565 570 575
Thr Met Thr Asn Ser Val Asp Asp Ala Leu Ile Asn Ser Thr Lys Ile
580 585 590
Tyr Ser Tyr Phe Pro Ser Val Ile Ser Lys Val Asn Gln Gly Ala Gln
595 600 605
Gly Ile Leu Phe Leu Gln Trp Val Arg Asp Ile Ile Asp Asp Phe Thr
610 615 620
Asn Glu Ser Ser Gln Lys Thr Thr Ile Asp Lys Ile Ser Asp Val Ser
625 630 635 640
Thr Ile Val Pro Tyr Ile Gly Pro Ala Leu Asn Ile Val Lys Gln Gly
645 650 655
Tyr Glu Gly Asn Phe Ile Gly Ala Leu Glu Thr Thr Gly Val Val Leu
660 665 670
Leu Leu Glu Tyr Ile Pro Glu Ile Thr Leu Pro Val Ile Ala Ala Leu
675 680 685
Ser Ile Ala Glu Ser Ser Thr Gln Lys Glu Lys Ile Ile Lys Thr Ile
690 695 700
Asp Asn Phe Leu Glu Lys Arg Tyr Glu Lys Trp Ile Glu Val Tyr Lys
705 710 715 720
Leu Val Lys Ala Lys Trp Leu Gly Thr Val Asn Thr Gln Phe Gln Lys
725 730 735
Arg Ser Tyr Gln Met Tyr Arg Ser Leu Glu Tyr Gln Val Asp Ala Ile
740 745 750
Lys Lys Ile Ile Asp Tyr Glu Tyr Lys Ile Tyr Ser Gly Pro Asp Lys
755 760 765
Glu Gln Ile Ala Asp Glu Ile Asn Asn Leu Lys Asn Lys Leu Glu Glu
770 775 780
Lys Ala Asn Lys Ala Met Ile Asn Ile Asn Ile Phe Met Arg Glu Ser
785 790 795 800
Ser Arg Ser Phe Leu Val Asn Gln Met Ile Asn Glu Ala Lys Lys Gln
805 810 815
Leu Leu Glu Phe Asp Thr Gln Ser Lys Asn Ile Leu Met Gln Tyr Ile
820 825 830
Lys Ala Asn Ser Lys Phe Ile Gly Ile Thr Glu Leu Lys Lys Leu Glu
835 840 845
Ser Lys Ile Asn Lys Val Phe Ser Thr Pro Ile Pro Phe Ser Tyr Ser
850 855 860
Lys Asn Leu Asp Cys Trp Val Asp Asn Glu Glu Asp Ile Asp Val Ile
865 870 875 880
Leu Lys Lys Ser Thr Ile Leu Asn Leu Asp Ile Asn Asn Asp Ile Ile
885 890 895
Ser Asp Ile Ser Gly Phe Asn Ser Ser Val Ile Thr Tyr Pro Asp Ala
900 905 910
Gln Leu Val Pro Gly Ile Asn Gly Lys Ala Ile His Leu Val Asn Asn
915 920 925
Glu Ser Ser Glu Val Ile Val His Lys Ala Met Asp Ile Glu Tyr Asn
930 935 940
Asp Met Phe Asn Asn Phe Thr Val Ser Phe Trp Leu Arg Val Pro Lys
945 950 955 960
Val Ser Ala Ser His Leu Glu Gln Tyr Gly Thr Asn Glu Tyr Ser Ile
965 970 975
Ile Ser Ser Met Lys Lys His Ser Leu Ser Ile Gly Ser Gly Trp Ser
980 985 990
Val Ser Leu Lys Gly Asn Asn Leu Ile Trp Thr Leu Lys Asp Ser Ala
995 1000 1005
Gly Glu Val Arg Gln Ile Thr Phe Arg Asp Leu Pro Asp Lys Phe
1010 1015 1020
Asn Ala Tyr Leu Ala Asn Lys Trp Val Phe Ile Thr Ile Thr Asn
1025 1030 1035
Asp Arg Leu Ser Ser Ala Asn Leu Tyr Ile Asn Gly Val Leu Met
1040 1045 1050
Gly Ser Ala Glu Ile Thr Gly Leu Gly Ala Ile Arg Glu Asp Asn
1055 1060 1065
Asn Ile Thr Leu Lys Leu Asp Arg Cys Asn Asn Asn Asn Gln Tyr
1070 1075 1080
Val Ser Ile Asp Lys Phe Arg Ile Phe Cys Lys Ala Leu Asn Pro
1085 1090 1095
Lys Glu Ile Glu Lys Leu Tyr Thr Ser Tyr Leu Ser Ile Thr Phe
1100 1105 1110
Leu Arg Asp Phe Trp Gly Asn Pro Leu Arg Tyr Asp Thr Glu Tyr
1115 1120 1125
Tyr Leu Ile Pro Val Ala Ser Ser Ser Lys Asp Val Gln Leu Lys
1130 1135 1140
Asn Ile Thr Asp Tyr Met Tyr Leu Thr Asn Ala Pro Ser Tyr Thr
1145 1150 1155
Asn Gly Lys Leu Asn Ile Tyr Tyr Arg Arg Leu Tyr Asn Gly Leu
1160 1165 1170
Lys Phe Ile Ile Lys Arg Tyr Thr Pro Asn Asn Glu Ile Asp Ser
1175 1180 1185
Phe Val Lys Ser Gly Asp Phe Ile Lys Leu Tyr Val Ser Tyr Asn
1190 1195 1200
Asn Asn Glu His Ile Val Gly Tyr Pro Lys Asp Gly Asn Ala Phe
1205 1210 1215
Asn Asn Leu Asp Arg Ile Leu Arg Val Gly Tyr Asn Ala Pro Gly
1220 1225 1230
Ile Pro Leu Tyr Lys Lys Met Glu Ala Val Lys Leu Arg Asp Leu
1235 1240 1245
Lys Thr Tyr Ser Val Gln Leu Lys Leu Tyr Asp Asp Lys Asn Ala
1250 1255 1260
Ser Leu Gly Leu Val Gly Thr His Asn Gly Gln Ile Gly Asn Asp
1265 1270 1275
Pro Asn Arg Asp Ile Leu Ile Ala Ser Asn Trp Tyr Phe Asn His
1280 1285 1290
Leu Lys Asp Lys Ile Leu Gly Cys Asp Trp Tyr Phe Val Pro Thr
1295 1300 1305
Asp Glu Gly Trp Thr Asn Asp
1310 1315
<210> 59
<211> 1306
<212> БЕЛОК
<213> Clostridium botulinum
<400> 59
Met Lys Leu Glu Ile Asn Lys Phe Asn Tyr Asn Asp Pro Ile Asp Gly
1 5 10 15
Ile Asn Val Ile Thr Met Arg Pro Pro Arg His Ser Asp Lys Ile Asn
20 25 30
Lys Gly Lys Gly Pro Phe Lys Ala Phe Gln Val Ile Lys Asn Ile Trp
35 40 45
Ile Val Pro Glu Arg Tyr Asn Phe Thr Asn Asn Thr Asn Asp Leu Asn
50 55 60
Ile Pro Ser Glu Pro Ile Met Glu Ala Asp Ala Ile Tyr Asn Pro Asn
65 70 75 80
Tyr Leu Asn Thr Pro Ser Glu Lys Asp Glu Phe Leu Gln Gly Val Ile
85 90 95
Lys Val Leu Glu Arg Ile Lys Ser Lys Pro Glu Gly Glu Lys Leu Leu
100 105 110
Glu Leu Ile Ser Ser Ser Ile Pro Leu Pro Leu Val Ser Asn Gly Ala
115 120 125
Leu Thr Leu Ser Asp Asn Glu Thr Ile Ala Tyr Gln Glu Asn Asn Asn
130 135 140
Ile Val Ser Asn Leu Gln Ala Asn Leu Val Ile Tyr Gly Pro Gly Pro
145 150 155 160
Asp Ile Ala Asn Asn Ala Thr Tyr Gly Leu Tyr Ser Thr Pro Ile Ser
165 170 175
Asn Gly Glu Gly Thr Leu Ser Glu Val Ser Phe Ser Pro Phe Tyr Leu
180 185 190
Lys Pro Phe Asp Glu Ser Tyr Gly Asn Tyr Arg Ser Leu Val Asn Ile
195 200 205
Val Asn Lys Phe Val Lys Arg Glu Phe Ala Pro Asp Pro Ala Ser Thr
210 215 220
Leu Met His Glu Leu Val His Val Thr His Asn Leu Tyr Gly Ile Ser
225 230 235 240
Asn Arg Asn Phe Tyr Tyr Asn Phe Asp Thr Gly Lys Ile Glu Thr Ser
245 250 255
Arg Gln Gln Asn Ser Leu Ile Phe Glu Glu Leu Leu Thr Phe Gly Gly
260 265 270
Ile Asp Ser Lys Ala Ile Ser Ser Leu Ile Ile Lys Lys Ile Ile Glu
275 280 285
Thr Ala Lys Asn Asn Tyr Thr Thr Leu Ile Ser Glu Arg Leu Asn Thr
290 295 300
Val Thr Val Glu Asn Asp Leu Leu Lys Tyr Ile Lys Asn Lys Ile Pro
305 310 315 320
Val Gln Gly Arg Leu Gly Asn Phe Lys Leu Asp Thr Ala Glu Phe Glu
325 330 335
Lys Lys Leu Asn Thr Ile Leu Phe Val Leu Asn Glu Ser Asn Leu Ala
340 345 350
Gln Arg Phe Ser Ile Leu Val Arg Lys His Tyr Leu Lys Glu Arg Pro
355 360 365
Ile Asp Pro Ile Tyr Val Asn Ile Leu Asp Asp Asn Ser Tyr Ser Thr
370 375 380
Leu Glu Gly Phe Asn Ile Ser Ser Gln Gly Ser Asn Asp Phe Gln Gly
385 390 395 400
Gln Leu Leu Glu Ser Ser Tyr Phe Glu Lys Ile Glu Ser Asn Ala Leu
405 410 415
Arg Ala Phe Ile Lys Ile Cys Pro Arg Asn Gly Leu Leu Tyr Asn Ala
420 425 430
Ile Tyr Arg Asn Ser Lys Asn Tyr Leu Asn Asn Ile Asp Leu Glu Asp
435 440 445
Lys Lys Thr Thr Ser Lys Thr Asn Val Ser Tyr Pro Cys Ser Leu Leu
450 455 460
Asn Gly Cys Ile Glu Val Glu Asn Lys Asp Leu Phe Leu Ile Ser Asn
465 470 475 480
Lys Asp Ser Leu Asn Asp Ile Asn Leu Ser Glu Glu Lys Ile Lys Pro
485 490 495
Glu Thr Thr Val Phe Phe Lys Asp Lys Leu Pro Pro Gln Asp Ile Thr
500 505 510
Leu Ser Asn Tyr Asp Phe Thr Glu Ala Asn Ser Ile Pro Ser Ile Ser
515 520 525
Gln Gln Asn Ile Leu Glu Arg Asn Glu Glu Leu Tyr Glu Pro Ile Arg
530 535 540
Asn Ser Leu Phe Glu Ile Lys Thr Ile Tyr Val Asp Lys Leu Thr Thr
545 550 555 560
Phe His Phe Leu Glu Ala Gln Asn Ile Asp Glu Ser Ile Asp Ser Ser
565 570 575
Lys Ile Arg Val Glu Leu Thr Asp Ser Val Asp Glu Ala Leu Ser Asn
580 585 590
Pro Asn Lys Val Tyr Ser Pro Phe Lys Asn Met Ser Asn Thr Ile Asn
595 600 605
Ser Ile Glu Thr Gly Ile Thr Ser Thr Tyr Ile Phe Tyr Gln Trp Leu
610 615 620
Arg Ser Ile Val Lys Asp Phe Ser Asp Glu Thr Gly Lys Ile Asp Val
625 630 635 640
Ile Asp Lys Ser Ser Asp Thr Leu Ala Ile Val Pro Tyr Ile Gly Pro
645 650 655
Leu Leu Asn Ile Gly Asn Asp Ile Arg His Gly Asp Phe Val Gly Ala
660 665 670
Ile Glu Leu Ala Gly Ile Thr Ala Leu Leu Glu Tyr Val Pro Glu Phe
675 680 685
Thr Ile Pro Ile Leu Val Gly Leu Glu Val Ile Gly Gly Glu Leu Ala
690 695 700
Arg Glu Gln Val Glu Ala Ile Val Asn Asn Ala Leu Asp Lys Arg Asp
705 710 715 720
Gln Lys Trp Ala Glu Val Tyr Asn Ile Thr Lys Ala Gln Trp Trp Gly
725 730 735
Thr Ile His Leu Gln Ile Asn Thr Arg Leu Ala His Thr Tyr Lys Ala
740 745 750
Leu Ser Arg Gln Ala Asn Ala Ile Lys Met Asn Met Glu Phe Gln Leu
755 760 765
Ala Asn Tyr Lys Gly Asn Ile Asp Asp Lys Ala Lys Ile Lys Asn Ala
770 775 780
Ile Ser Glu Thr Glu Ile Leu Leu Asn Lys Ser Val Glu Gln Ala Met
785 790 795 800
Lys Asn Thr Glu Lys Phe Met Ile Lys Leu Ser Asn Ser Tyr Leu Thr
805 810 815
Lys Glu Met Ile Pro Lys Val Gln Asp Asn Leu Lys Asn Phe Asp Leu
820 825 830
Glu Thr Lys Lys Thr Leu Asp Lys Phe Ile Lys Glu Lys Glu Asp Ile
835 840 845
Leu Gly Thr Asn Leu Ser Ser Ser Leu Arg Arg Lys Val Ser Ile Arg
850 855 860
Leu Asn Lys Asn Ile Ala Phe Asp Ile Asn Asp Ile Pro Phe Ser Glu
865 870 875 880
Phe Asp Asp Leu Ile Asn Gln Tyr Lys Asn Glu Ile Glu Asp Tyr Glu
885 890 895
Val Leu Asn Leu Gly Ala Glu Asp Gly Lys Ile Lys Asp Leu Ser Gly
900 905 910
Thr Thr Ser Asp Ile Asn Ile Gly Ser Asp Ile Glu Leu Ala Asp Gly
915 920 925
Arg Glu Asn Lys Ala Ile Lys Ile Lys Gly Ser Glu Asn Ser Thr Ile
930 935 940
Lys Ile Ala Met Asn Lys Tyr Leu Arg Phe Ser Ala Thr Asp Asn Phe
945 950 955 960
Ser Ile Ser Phe Trp Ile Lys His Pro Lys Pro Thr Asn Leu Leu Asn
965 970 975
Asn Gly Ile Glu Tyr Thr Leu Val Glu Asn Phe Asn Gln Arg Gly Trp
980 985 990
Lys Ile Ser Ile Gln Asp Ser Lys Leu Ile Trp Tyr Leu Arg Asp His
995 1000 1005
Asn Asn Ser Ile Lys Ile Val Thr Pro Asp Tyr Ile Ala Phe Asn
1010 1015 1020
Gly Trp Asn Leu Ile Thr Ile Thr Asn Asn Arg Ser Lys Gly Ser
1025 1030 1035
Ile Val Tyr Val Asn Gly Ser Lys Ile Glu Glu Lys Asp Ile Ser
1040 1045 1050
Ser Ile Trp Asn Thr Glu Val Asp Asp Pro Ile Ile Phe Arg Leu
1055 1060 1065
Lys Asn Asn Arg Asp Thr Gln Ala Phe Thr Leu Leu Asp Gln Phe
1070 1075 1080
Ser Ile Tyr Arg Lys Glu Leu Asn Gln Asn Glu Val Val Lys Leu
1085 1090 1095
Tyr Asn Tyr Tyr Phe Asn Ser Asn Tyr Ile Arg Asp Ile Trp Gly
1100 1105 1110
Asn Pro Leu Gln Tyr Asn Lys Lys Tyr Tyr Leu Gln Thr Gln Asp
1115 1120 1125
Lys Pro Gly Lys Gly Leu Ile Arg Glu Tyr Trp Ser Ser Phe Gly
1130 1135 1140
Tyr Asp Tyr Val Ile Leu Ser Asp Ser Lys Thr Ile Thr Phe Pro
1145 1150 1155
Asn Asn Ile Arg Tyr Gly Ala Leu Tyr Asn Gly Ser Lys Val Leu
1160 1165 1170
Ile Lys Asn Ser Lys Lys Leu Asp Gly Leu Val Arg Asn Lys Asp
1175 1180 1185
Phe Ile Gln Leu Glu Ile Asp Gly Tyr Asn Met Gly Ile Ser Ala
1190 1195 1200
Asp Arg Phe Asn Glu Asp Thr Asn Tyr Ile Gly Thr Thr Tyr Gly
1205 1210 1215
Thr Thr His Asp Leu Thr Thr Asp Phe Glu Ile Ile Gln Arg Gln
1220 1225 1230
Glu Lys Tyr Arg Asn Tyr Cys Gln Leu Lys Thr Pro Tyr Asn Ile
1235 1240 1245
Phe His Lys Ser Gly Leu Met Ser Thr Glu Thr Ser Lys Pro Thr
1250 1255 1260
Phe His Asp Tyr Arg Asp Trp Val Tyr Ser Ser Ala Trp Tyr Phe
1265 1270 1275
Gln Asn Tyr Glu Asn Leu Asn Leu Arg Lys His Thr Lys Thr Asn
1280 1285 1290
Trp Tyr Phe Ile Pro Lys Asp Glu Gly Trp Asp Glu Asp
1295 1300 1305
<210> 60
<211> 3888
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность mrBoNT/A
<400> 60
atgccattcg tcaacaagca attcaactac aaagacccag tcaacggcgt cgacatcgca 60
tacatcaaga ttccgaacgc cggtcaaatg cagccggtta aggcttttaa gatccacaac 120
aagatttggg ttatcccgga gcgtgacacc ttcacgaacc cggaagaagg cgatctgaac 180
ccgccaccgg aagcgaagca agtccctgtc agctactacg attcgacgta cctgagcacg 240
gataacgaaa aagataacta cctgaaaggt gtgaccaagc tgttcgaacg tatctacagc 300
acggatctgg gtcgcatgct gctgactagc attgttcgcg gtatcccgtt ctggggtggt 360
agcacgattg acaccgaact gaaggttatc gacactaact gcattaacgt tattcaaccg 420
gatggtagct atcgtagcga agagctgaat ctggtcatca ttggcccgag cgcagacatt 480
atccaattcg agtgcaagag ctttggtcac gaggttctga atctgacccg caatggctat 540
ggtagcaccc agtacattcg tttttcgccg gattttacct tcggctttga agagagcctg 600
gaggttgata ccaatccgtt gctgggtgcg ggcaaattcg ctaccgatcc ggctgtcacg 660
ctggcccatg aactgatcca cgcaggccac cgcctgtacg gcattgccat caacccaaac 720
cgtgtgttca aggttaatac gaatgcatac tacgagatga gcggcctgga agtcagcttc 780
gaagaactgc gcaccttcgg tggccatgac gctaaattca ttgacagctt gcaagagaat 840
gagttccgtc tgtactacta taacaaattc aaagacattg caagcacgtt gaacaaggcc 900
aaaagcatcg ttggtactac cgcgtcgttg cagtatatga agaatgtgtt taaagagaag 960
tacctgctgt ccgaggatac ctccggcaag tttagcgttg ataagctgaa gtttgacaaa 1020
ctgtacaaga tgctgaccga gatttacacc gaggacaact ttgtgaaatt cttcaaagtg 1080
ttgaatcgta aaacctatct gaattttgac aaagcggttt tcaagattaa catcgtgccg 1140
aaggtgaact acaccatcta tgacggtttt aacctgcgta acaccaacct ggcggcgaac 1200
tttaacggtc agaatacgga aatcaacaac atgaatttca cgaagttgaa gaacttcacg 1260
ggtctgttcg agttctataa gctgctgtgc gtgcgcggta tcatcaccag caaaaccaaa 1320
agcctggaca aaggctacaa caaggcgctg aatgacctgt gcattaaggt aaacaattgg 1380
gatctgttct tttcgccatc cgaagataat tttaccaacg acctgaacaa gggtgaagaa 1440
atcaccagcg atacgaatat tgaagcagcg gaagagaata tcagcctgga tctgatccag 1500
cagtactatc tgacctttaa cttcgacaat gaaccggaga acattagcat tgagaatctg 1560
agcagcgaca ttatcggtca gctggaactg atgccgaata tcgaacgttt cccgaacggc 1620
aaaaagtacg agctggacaa gtacactatg ttccattacc tgcgtgcaca ggagtttgaa 1680
cacggtaaaa gccgtatcgc gctgaccaac agcgttaacg aggccctgct gaacccgagc 1740
cgtgtctata ccttcttcag cagcgactat gttaagaaag tgaacaaagc cactgaggcc 1800
gcgatgttcc tgggctgggt ggaacagctg gtatatgact tcacggacga gacgagcgaa 1860
gtgagcacta ccgacaaaat tgctgatatt accatcatta tcccgtatat tggtccggca 1920
ctgaacattg gcaacatgct gtacaaagac gattttgtgg gtgccctgat cttctccggt 1980
gccgtgattc tgctggagtt cattccggag attgcgatcc cggtgttggg taccttcgcg 2040
ctggtgtcct acatcgcgaa taaggttctg acggttcaga ccatcgataa cgcgctgtcg 2100
aaacgtaatg aaaaatggga cgaggtttac aaatacattg ttacgaattg gctggcgaaa 2160
gtcaataccc agatcgacct gatccgtaag aaaatgaaag aggcgctgga gaatcaggcg 2220
gaggccacca aagcaattat caactaccaa tacaaccagt acacggaaga agagaagaat 2280
aacattaact tcaatatcga tgatttgagc agcaagctga atgaatctat caacaaagcg 2340
atgatcaata tcaacaagtt tttgaatcag tgtagcgttt cgtacctgat gaatagcatg 2400
attccgtatg gcgtcaaacg tctggaggac ttcgacgcca gcctgaaaga tgcgttgctg 2460
aaatacattt acgacaatcg tggtacgctg attggccaag ttgaccgctt gaaagacaaa 2520
gttaacaata ccctgagcac cgacatccca tttcaactga gcaagtatgt tgataatcaa 2580
cgtctgttga gcactttcac cgagtatatc aaaaacatca tcaatactag cattctgaac 2640
ctgcgttacg agagcaagca tctgattgat ctgagccgtt atgctagcaa gatcaacatc 2700
ggtagcaagg tcaattttga cccgatcgat aagaaccaga tccagctgtt taatctggaa 2760
tcgagcaaaa ttgaggttat cctgaaaaag gccattgtct acaactccat gtacgagaat 2820
ttctccacca gcttctggat tcgcatcccg aaatacttca acaagattag cctgaacaac 2880
gagtatacta tcatcaactg tatggagaac aacagcggtt ggaaggtgtc tctgaactat 2940
ggtgagatca tttggacctt gcaggacacc aaagagatca agcagcgcgt cgtgttcaag 3000
tactctcaaa tgatcaacat ttccgattac attaatcgtt ggatcttcgt gaccattacg 3060
aataaccgtc tgaataagag caagatttac atcaatggtc gcttgatcga tcagaaaccg 3120
attagcaacc tgggtaatat ccacgcaagc aacaagatta tgttcaaatt ggacggttgc 3180
cgcgataccc atcgttatat ctggatcaag tatttcaacc tgtttgataa agaactgaat 3240
gagaaggaga tcaaagattt gtatgacaac caatctaaca gcggcatttt gaaggacttc 3300
tggggcgatt atctgcaata cgataagccg tactatatgc tgaacctgta tgatccgaac 3360
aaatatgtgg atgtcaataa tgtgggtatt cgtggttaca tgtatttgaa gggtccgcgt 3420
ggcagcgtta tgacgaccaa catttacctg aactctagcc tgtaccgtgg tacgaaattc 3480
atcattaaga aatatgccag cggcaacaaa gataacattg tgcgtaataa cgatcgtgtc 3540
tacatcaacg tggtcgtgaa gaataaagag taccgtctgg cgaccaacgc ttcgcaggcg 3600
ggtgttgaga aaattctgag cgcgttggag atccctgatg tcggtaatct gagccaagtc 3660
gtggttatga agagcaagaa cgacaagggt atcactaaca agtgcaagat gaacctgcaa 3720
gacaacaatg gtaacgacat cggctttatt ggtttccacc agttcaacaa tattgctaaa 3780
ctggtagcga gcaattggta caatcgtcag attgagcgca gcagccgtac tttgggctgt 3840
agctgggagt ttatcccggt cgatgatggt tggggcgaac gtccgctg 3888
<210> 61
<211> 1296
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность mrBoNT/A
<400> 61
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Ile Asn Thr Ser Ile Leu Asn
865 870 875 880
Leu Arg Tyr Glu Ser Lys His Leu Ile Asp Leu Ser Arg Tyr Ala Ser
885 890 895
Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys Asn
900 905 910
Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu
915 920 925
Lys Lys Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser
930 935 940
Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Lys Ile Ser Leu Asn Asn
945 950 955 960
Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys Val
965 970 975
Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Lys Glu
980 985 990
Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile Ser
995 1000 1005
Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
1010 1015 1020
Leu Asn Lys Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln
1025 1030 1035
Lys Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Lys Ile
1040 1045 1050
Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp
1055 1060 1065
Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu
1070 1075 1080
Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met
1100 1105 1110
Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
1115 1120 1125
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
1130 1135 1140
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr
1145 1150 1155
Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile
1160 1165 1170
Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn
1175 1180 1185
Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
1190 1195 1200
Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser
1205 1210 1215
Gln Val Val Val Met Lys Ser Lys Asn Asp Lys Gly Ile Thr Asn
1220 1225 1230
Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly
1235 1240 1245
Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
1250 1255 1260
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu
1265 1270 1275
Gly Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu
1280 1285 1290
Arg Pro Leu
1295
<210> 62
<211> 1296
<212> БЕЛОК
<213> Clostridium botulinum
<400> 62
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Ile Asn Thr Ser Ile Leu Asn
865 870 875 880
Leu Arg Tyr Glu Ser Asn His Leu Ile Asp Leu Ser Arg Tyr Ala Ser
885 890 895
Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys Asn
900 905 910
Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu
915 920 925
Lys Asn Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser
930 935 940
Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Ser Ile Ser Leu Asn Asn
945 950 955 960
Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys Val
965 970 975
Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Gln Glu
980 985 990
Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile Ser
995 1000 1005
Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
1010 1015 1020
Leu Asn Asn Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln
1025 1030 1035
Lys Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Asn Ile
1040 1045 1050
Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp
1055 1060 1065
Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu
1070 1075 1080
Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met
1100 1105 1110
Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
1115 1120 1125
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
1130 1135 1140
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr
1145 1150 1155
Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile
1160 1165 1170
Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn
1175 1180 1185
Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
1190 1195 1200
Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser
1205 1210 1215
Gln Val Val Val Met Lys Ser Lys Asn Asp Gln Gly Ile Thr Asn
1220 1225 1230
Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly
1235 1240 1245
Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
1250 1255 1260
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu
1265 1270 1275
Gly Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu
1280 1285 1290
Arg Pro Leu
1295
<210> 63
<211> 1304
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность mrBoNT/AB
<400> 63
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Glu
210 215 220
Leu Ile His Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Leu Asn Asn Ile Ile Leu Asn
865 870 875 880
Leu Arg Tyr Lys Asp Asn Asn Leu Ile Asp Leu Ser Gly Tyr Gly Ala
885 890 895
Lys Val Glu Val Tyr Asp Gly Val Glu Leu Asn Asp Lys Asn Gln Phe
900 905 910
Lys Leu Thr Ser Ser Ala Asn Ser Lys Ile Arg Val Thr Gln Asn Gln
915 920 925
Asn Ile Ile Phe Asn Ser Val Phe Leu Asp Phe Ser Val Ser Phe Trp
930 935 940
Ile Arg Ile Pro Lys Tyr Lys Asn Asp Gly Ile Gln Asn Tyr Ile His
945 950 955 960
Asn Glu Tyr Thr Ile Ile Asn Cys Met Lys Asn Asn Ser Gly Trp Lys
965 970 975
Ile Ser Ile Arg Gly Asn Arg Ile Ile Trp Thr Leu Ile Asp Ile Asn
980 985 990
Gly Lys Thr Lys Ser Val Phe Phe Glu Tyr Asn Ile Arg Glu Asp Ile
995 1000 1005
Ser Glu Tyr Ile Asn Arg Trp Phe Phe Val Thr Ile Thr Asn Asn
1010 1015 1020
Leu Asn Asn Ala Lys Ile Tyr Ile Asn Gly Lys Leu Glu Ser Asn
1025 1030 1035
Thr Asp Ile Lys Asp Ile Arg Glu Val Ile Ala Asn Gly Glu Ile
1040 1045 1050
Ile Phe Lys Leu Asp Gly Asp Ile Asp Arg Thr Gln Phe Ile Trp
1055 1060 1065
Met Lys Tyr Phe Ser Ile Phe Asn Thr Glu Leu Ser Gln Ser Asn
1070 1075 1080
Ile Glu Glu Arg Tyr Lys Ile Gln Ser Tyr Ser Glu Tyr Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asn Pro Leu Met Tyr Asn Lys Glu Tyr Tyr Met
1100 1105 1110
Phe Asn Ala Gly Asn Lys Asn Ser Tyr Ile Lys Leu Lys Lys Asp
1115 1120 1125
Ser Pro Val Gly Glu Ile Leu Thr Arg Ser Lys Tyr Asn Gln Asn
1130 1135 1140
Ser Lys Tyr Ile Asn Tyr Arg Asp Leu Tyr Ile Gly Glu Lys Phe
1145 1150 1155
Ile Ile Arg Arg Lys Ser Asn Ser Gln Ser Ile Asn Asp Asp Ile
1160 1165 1170
Val Arg Lys Glu Asp Tyr Ile Tyr Leu Asp Phe Phe Asn Leu Asn
1175 1180 1185
Gln Glu Trp Arg Val Tyr Thr Tyr Lys Tyr Phe Lys Lys Glu Glu
1190 1195 1200
Met Lys Leu Phe Leu Ala Pro Ile Tyr Asp Ser Asp Glu Phe Tyr
1205 1210 1215
Asn Thr Ile Gln Ile Lys Glu Tyr Asp Glu Gln Pro Thr Tyr Ser
1220 1225 1230
Cys Gln Leu Leu Phe Lys Lys Asp Glu Glu Ser Thr Asp Glu Ile
1235 1240 1245
Gly Leu Ile Gly Ile His Arg Phe Tyr Glu Ser Gly Ile Val Phe
1250 1255 1260
Glu Glu Tyr Lys Asp Tyr Phe Cys Ile Ser Lys Trp Tyr Leu Lys
1265 1270 1275
Glu Val Lys Arg Lys Pro Tyr Asn Leu Lys Leu Gly Cys Asn Trp
1280 1285 1290
Gln Phe Ile Pro Lys Asp Glu Gly Trp Thr Glu
1295 1300
<210> 64
<211> 1304
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность mrBoNT/AB(0)
<400> 64
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Gln
210 215 220
Leu Ile Tyr Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Leu Asn Asn Ile Ile Leu Asn
865 870 875 880
Leu Arg Tyr Lys Asp Asn Asn Leu Ile Asp Leu Ser Gly Tyr Gly Ala
885 890 895
Lys Val Glu Val Tyr Asp Gly Val Glu Leu Asn Asp Lys Asn Gln Phe
900 905 910
Lys Leu Thr Ser Ser Ala Asn Ser Lys Ile Arg Val Thr Gln Asn Gln
915 920 925
Asn Ile Ile Phe Asn Ser Val Phe Leu Asp Phe Ser Val Ser Phe Trp
930 935 940
Ile Arg Ile Pro Lys Tyr Lys Asn Asp Gly Ile Gln Asn Tyr Ile His
945 950 955 960
Asn Glu Tyr Thr Ile Ile Asn Cys Met Lys Asn Asn Ser Gly Trp Lys
965 970 975
Ile Ser Ile Arg Gly Asn Arg Ile Ile Trp Thr Leu Ile Asp Ile Asn
980 985 990
Gly Lys Thr Lys Ser Val Phe Phe Glu Tyr Asn Ile Arg Glu Asp Ile
995 1000 1005
Ser Glu Tyr Ile Asn Arg Trp Phe Phe Val Thr Ile Thr Asn Asn
1010 1015 1020
Leu Asn Asn Ala Lys Ile Tyr Ile Asn Gly Lys Leu Glu Ser Asn
1025 1030 1035
Thr Asp Ile Lys Asp Ile Arg Glu Val Ile Ala Asn Gly Glu Ile
1040 1045 1050
Ile Phe Lys Leu Asp Gly Asp Ile Asp Arg Thr Gln Phe Ile Trp
1055 1060 1065
Met Lys Tyr Phe Ser Ile Phe Asn Thr Glu Leu Ser Gln Ser Asn
1070 1075 1080
Ile Glu Glu Arg Tyr Lys Ile Gln Ser Tyr Ser Glu Tyr Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asn Pro Leu Met Tyr Asn Lys Glu Tyr Tyr Met
1100 1105 1110
Phe Asn Ala Gly Asn Lys Asn Ser Tyr Ile Lys Leu Lys Lys Asp
1115 1120 1125
Ser Pro Val Gly Glu Ile Leu Thr Arg Ser Lys Tyr Asn Gln Asn
1130 1135 1140
Ser Lys Tyr Ile Asn Tyr Arg Asp Leu Tyr Ile Gly Glu Lys Phe
1145 1150 1155
Ile Ile Arg Arg Lys Ser Asn Ser Gln Ser Ile Asn Asp Asp Ile
1160 1165 1170
Val Arg Lys Glu Asp Tyr Ile Tyr Leu Asp Phe Phe Asn Leu Asn
1175 1180 1185
Gln Glu Trp Arg Val Tyr Thr Tyr Lys Tyr Phe Lys Lys Glu Glu
1190 1195 1200
Met Lys Leu Phe Leu Ala Pro Ile Tyr Asp Ser Asp Glu Phe Tyr
1205 1210 1215
Asn Thr Ile Gln Ile Lys Glu Tyr Asp Glu Gln Pro Thr Tyr Ser
1220 1225 1230
Cys Gln Leu Leu Phe Lys Lys Asp Glu Glu Ser Thr Asp Glu Ile
1235 1240 1245
Gly Leu Ile Gly Ile His Arg Phe Tyr Glu Ser Gly Ile Val Phe
1250 1255 1260
Glu Glu Tyr Lys Asp Tyr Phe Cys Ile Ser Lys Trp Tyr Leu Lys
1265 1270 1275
Glu Val Lys Arg Lys Pro Tyr Asn Leu Lys Leu Gly Cys Asn Trp
1280 1285 1290
Gln Phe Ile Pro Lys Asp Glu Gly Trp Thr Glu
1295 1300
<210> 65
<211> 1296
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Полипептидная последовательность mrBoNT/A(0)
<400> 65
Met Pro Phe Val Asn Lys Gln Phe Asn Tyr Lys Asp Pro Val Asn Gly
1 5 10 15
Val Asp Ile Ala Tyr Ile Lys Ile Pro Asn Ala Gly Gln Met Gln Pro
20 25 30
Val Lys Ala Phe Lys Ile His Asn Lys Ile Trp Val Ile Pro Glu Arg
35 40 45
Asp Thr Phe Thr Asn Pro Glu Glu Gly Asp Leu Asn Pro Pro Pro Glu
50 55 60
Ala Lys Gln Val Pro Val Ser Tyr Tyr Asp Ser Thr Tyr Leu Ser Thr
65 70 75 80
Asp Asn Glu Lys Asp Asn Tyr Leu Lys Gly Val Thr Lys Leu Phe Glu
85 90 95
Arg Ile Tyr Ser Thr Asp Leu Gly Arg Met Leu Leu Thr Ser Ile Val
100 105 110
Arg Gly Ile Pro Phe Trp Gly Gly Ser Thr Ile Asp Thr Glu Leu Lys
115 120 125
Val Ile Asp Thr Asn Cys Ile Asn Val Ile Gln Pro Asp Gly Ser Tyr
130 135 140
Arg Ser Glu Glu Leu Asn Leu Val Ile Ile Gly Pro Ser Ala Asp Ile
145 150 155 160
Ile Gln Phe Glu Cys Lys Ser Phe Gly His Glu Val Leu Asn Leu Thr
165 170 175
Arg Asn Gly Tyr Gly Ser Thr Gln Tyr Ile Arg Phe Ser Pro Asp Phe
180 185 190
Thr Phe Gly Phe Glu Glu Ser Leu Glu Val Asp Thr Asn Pro Leu Leu
195 200 205
Gly Ala Gly Lys Phe Ala Thr Asp Pro Ala Val Thr Leu Ala His Gln
210 215 220
Leu Ile Tyr Ala Gly His Arg Leu Tyr Gly Ile Ala Ile Asn Pro Asn
225 230 235 240
Arg Val Phe Lys Val Asn Thr Asn Ala Tyr Tyr Glu Met Ser Gly Leu
245 250 255
Glu Val Ser Phe Glu Glu Leu Arg Thr Phe Gly Gly His Asp Ala Lys
260 265 270
Phe Ile Asp Ser Leu Gln Glu Asn Glu Phe Arg Leu Tyr Tyr Tyr Asn
275 280 285
Lys Phe Lys Asp Ile Ala Ser Thr Leu Asn Lys Ala Lys Ser Ile Val
290 295 300
Gly Thr Thr Ala Ser Leu Gln Tyr Met Lys Asn Val Phe Lys Glu Lys
305 310 315 320
Tyr Leu Leu Ser Glu Asp Thr Ser Gly Lys Phe Ser Val Asp Lys Leu
325 330 335
Lys Phe Asp Lys Leu Tyr Lys Met Leu Thr Glu Ile Tyr Thr Glu Asp
340 345 350
Asn Phe Val Lys Phe Phe Lys Val Leu Asn Arg Lys Thr Tyr Leu Asn
355 360 365
Phe Asp Lys Ala Val Phe Lys Ile Asn Ile Val Pro Lys Val Asn Tyr
370 375 380
Thr Ile Tyr Asp Gly Phe Asn Leu Arg Asn Thr Asn Leu Ala Ala Asn
385 390 395 400
Phe Asn Gly Gln Asn Thr Glu Ile Asn Asn Met Asn Phe Thr Lys Leu
405 410 415
Lys Asn Phe Thr Gly Leu Phe Glu Phe Tyr Lys Leu Leu Cys Val Arg
420 425 430
Gly Ile Ile Thr Ser Lys Thr Lys Ser Leu Asp Lys Gly Tyr Asn Lys
435 440 445
Ala Leu Asn Asp Leu Cys Ile Lys Val Asn Asn Trp Asp Leu Phe Phe
450 455 460
Ser Pro Ser Glu Asp Asn Phe Thr Asn Asp Leu Asn Lys Gly Glu Glu
465 470 475 480
Ile Thr Ser Asp Thr Asn Ile Glu Ala Ala Glu Glu Asn Ile Ser Leu
485 490 495
Asp Leu Ile Gln Gln Tyr Tyr Leu Thr Phe Asn Phe Asp Asn Glu Pro
500 505 510
Glu Asn Ile Ser Ile Glu Asn Leu Ser Ser Asp Ile Ile Gly Gln Leu
515 520 525
Glu Leu Met Pro Asn Ile Glu Arg Phe Pro Asn Gly Lys Lys Tyr Glu
530 535 540
Leu Asp Lys Tyr Thr Met Phe His Tyr Leu Arg Ala Gln Glu Phe Glu
545 550 555 560
His Gly Lys Ser Arg Ile Ala Leu Thr Asn Ser Val Asn Glu Ala Leu
565 570 575
Leu Asn Pro Ser Arg Val Tyr Thr Phe Phe Ser Ser Asp Tyr Val Lys
580 585 590
Lys Val Asn Lys Ala Thr Glu Ala Ala Met Phe Leu Gly Trp Val Glu
595 600 605
Gln Leu Val Tyr Asp Phe Thr Asp Glu Thr Ser Glu Val Ser Thr Thr
610 615 620
Asp Lys Ile Ala Asp Ile Thr Ile Ile Ile Pro Tyr Ile Gly Pro Ala
625 630 635 640
Leu Asn Ile Gly Asn Met Leu Tyr Lys Asp Asp Phe Val Gly Ala Leu
645 650 655
Ile Phe Ser Gly Ala Val Ile Leu Leu Glu Phe Ile Pro Glu Ile Ala
660 665 670
Ile Pro Val Leu Gly Thr Phe Ala Leu Val Ser Tyr Ile Ala Asn Lys
675 680 685
Val Leu Thr Val Gln Thr Ile Asp Asn Ala Leu Ser Lys Arg Asn Glu
690 695 700
Lys Trp Asp Glu Val Tyr Lys Tyr Ile Val Thr Asn Trp Leu Ala Lys
705 710 715 720
Val Asn Thr Gln Ile Asp Leu Ile Arg Lys Lys Met Lys Glu Ala Leu
725 730 735
Glu Asn Gln Ala Glu Ala Thr Lys Ala Ile Ile Asn Tyr Gln Tyr Asn
740 745 750
Gln Tyr Thr Glu Glu Glu Lys Asn Asn Ile Asn Phe Asn Ile Asp Asp
755 760 765
Leu Ser Ser Lys Leu Asn Glu Ser Ile Asn Lys Ala Met Ile Asn Ile
770 775 780
Asn Lys Phe Leu Asn Gln Cys Ser Val Ser Tyr Leu Met Asn Ser Met
785 790 795 800
Ile Pro Tyr Gly Val Lys Arg Leu Glu Asp Phe Asp Ala Ser Leu Lys
805 810 815
Asp Ala Leu Leu Lys Tyr Ile Tyr Asp Asn Arg Gly Thr Leu Ile Gly
820 825 830
Gln Val Asp Arg Leu Lys Asp Lys Val Asn Asn Thr Leu Ser Thr Asp
835 840 845
Ile Pro Phe Gln Leu Ser Lys Tyr Val Asp Asn Gln Arg Leu Leu Ser
850 855 860
Thr Phe Thr Glu Tyr Ile Lys Asn Ile Ile Asn Thr Ser Ile Leu Asn
865 870 875 880
Leu Arg Tyr Glu Ser Lys His Leu Ile Asp Leu Ser Arg Tyr Ala Ser
885 890 895
Lys Ile Asn Ile Gly Ser Lys Val Asn Phe Asp Pro Ile Asp Lys Asn
900 905 910
Gln Ile Gln Leu Phe Asn Leu Glu Ser Ser Lys Ile Glu Val Ile Leu
915 920 925
Lys Lys Ala Ile Val Tyr Asn Ser Met Tyr Glu Asn Phe Ser Thr Ser
930 935 940
Phe Trp Ile Arg Ile Pro Lys Tyr Phe Asn Lys Ile Ser Leu Asn Asn
945 950 955 960
Glu Tyr Thr Ile Ile Asn Cys Met Glu Asn Asn Ser Gly Trp Lys Val
965 970 975
Ser Leu Asn Tyr Gly Glu Ile Ile Trp Thr Leu Gln Asp Thr Lys Glu
980 985 990
Ile Lys Gln Arg Val Val Phe Lys Tyr Ser Gln Met Ile Asn Ile Ser
995 1000 1005
Asp Tyr Ile Asn Arg Trp Ile Phe Val Thr Ile Thr Asn Asn Arg
1010 1015 1020
Leu Asn Lys Ser Lys Ile Tyr Ile Asn Gly Arg Leu Ile Asp Gln
1025 1030 1035
Lys Pro Ile Ser Asn Leu Gly Asn Ile His Ala Ser Asn Lys Ile
1040 1045 1050
Met Phe Lys Leu Asp Gly Cys Arg Asp Thr His Arg Tyr Ile Trp
1055 1060 1065
Ile Lys Tyr Phe Asn Leu Phe Asp Lys Glu Leu Asn Glu Lys Glu
1070 1075 1080
Ile Lys Asp Leu Tyr Asp Asn Gln Ser Asn Ser Gly Ile Leu Lys
1085 1090 1095
Asp Phe Trp Gly Asp Tyr Leu Gln Tyr Asp Lys Pro Tyr Tyr Met
1100 1105 1110
Leu Asn Leu Tyr Asp Pro Asn Lys Tyr Val Asp Val Asn Asn Val
1115 1120 1125
Gly Ile Arg Gly Tyr Met Tyr Leu Lys Gly Pro Arg Gly Ser Val
1130 1135 1140
Met Thr Thr Asn Ile Tyr Leu Asn Ser Ser Leu Tyr Arg Gly Thr
1145 1150 1155
Lys Phe Ile Ile Lys Lys Tyr Ala Ser Gly Asn Lys Asp Asn Ile
1160 1165 1170
Val Arg Asn Asn Asp Arg Val Tyr Ile Asn Val Val Val Lys Asn
1175 1180 1185
Lys Glu Tyr Arg Leu Ala Thr Asn Ala Ser Gln Ala Gly Val Glu
1190 1195 1200
Lys Ile Leu Ser Ala Leu Glu Ile Pro Asp Val Gly Asn Leu Ser
1205 1210 1215
Gln Val Val Val Met Lys Ser Lys Asn Asp Lys Gly Ile Thr Asn
1220 1225 1230
Lys Cys Lys Met Asn Leu Gln Asp Asn Asn Gly Asn Asp Ile Gly
1235 1240 1245
Phe Ile Gly Phe His Gln Phe Asn Asn Ile Ala Lys Leu Val Ala
1250 1255 1260
Ser Asn Trp Tyr Asn Arg Gln Ile Glu Arg Ser Ser Arg Thr Leu
1265 1270 1275
Gly Cys Ser Trp Glu Phe Ile Pro Val Asp Asp Gly Trp Gly Glu
1280 1285 1290
Arg Pro Leu
1295
<210> 66
<211> 7
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Мотив на основе лейцина
<220>
<221> САЙТ
<222> (1)..(1)
<223> Xaa может быть любой существующей в природе аминокислотой
<220>
<221> САЙТ
<222> (3)..(5)
<223> Xaa может быть любой существующей в природе аминокислотой
<400> 66
Xaa Asp Xaa Xaa Xaa Leu Leu
1 5
<210> 67
<211> 7
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Мотив на основе лейцина
<220>
<221> САЙТ
<222> (1)..(1)
<223> Xaa может быть любой существующей в природе аминокислотой
<220>
<221> САЙТ
<222> (3)..(5)
<223> Xaa может быть любой существующей в природе аминокислотой
<400> 67
Xaa Glu Xaa Xaa Xaa Leu Leu
1 5
<210> 68
<211> 7
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Мотив на основе лейцина
<220>
<221> САЙТ
<222> (1)..(1)
<223> Xaa может быть любой существующей в природе аминокислотой
<220>
<221> САЙТ
<222> (3)..(5)
<223> Xaa может быть любой существующей в природе аминокислотой
<400> 68
Xaa Glu Xaa Xaa Xaa Ile Leu
1 5
<210> 69
<211> 7
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Мотив на основе лейцина
<220>
<221> САЙТ
<222> (1)..(1)
<223> Xaa может быть любой существующей в природе аминокислотой
<220>
<221> САЙТ
<222> (3)..(5)
<223> Xaa может быть любой существующей в природе аминокислотой
<400> 69
Xaa Glu Xaa Xaa Xaa Leu Met
1 5
<210> 70
<211> 4
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Мотив на основе тирозина
<220>
<221> САЙТ
<222> (2)..(3)
<223> Xaa может быть любой существующей в природе аминокислотой
<220>
<221> ВАРИАНТ
<222> (4)..(4)
<223> Xaa является гидрофобной аминокислотой
<400> 70
Tyr Xaa Xaa Xaa
1
<210> 71
<211> 6
<212> БЕЛОК
<213> Clostridium botulinum
<400> 71
Asn Ile Ile Asn Thr Ser
1 5
<210> 72
<211> 6
<212> БЕЛОК
<213> Clostridium botulinum
<400> 72
Asn Ile Val Asn Thr Ser
1 5
<210> 73
<211> 6
<212> БЕЛОК
<213> Clostridium botulinum
<400> 73
Asn Ile Thr Asn Ala Ser
1 5
<210> 74
<211> 6
<212> БЕЛОК
<213> Clostridium botulinum
<400> 74
Asn Ile Thr Asn Thr Ser
1 5
<210> 75
<211> 6
<212> БЕЛОК
<213> Clostridium botulinum
<400> 75
Glu Ile Leu Asn Asn Ile
1 5
<210> 76
<211> 6
<212> БЕЛОК
<213> Clostridium botulinum
<400> 76
Asp Ile Leu Asn Asn Ile
1 5
<---
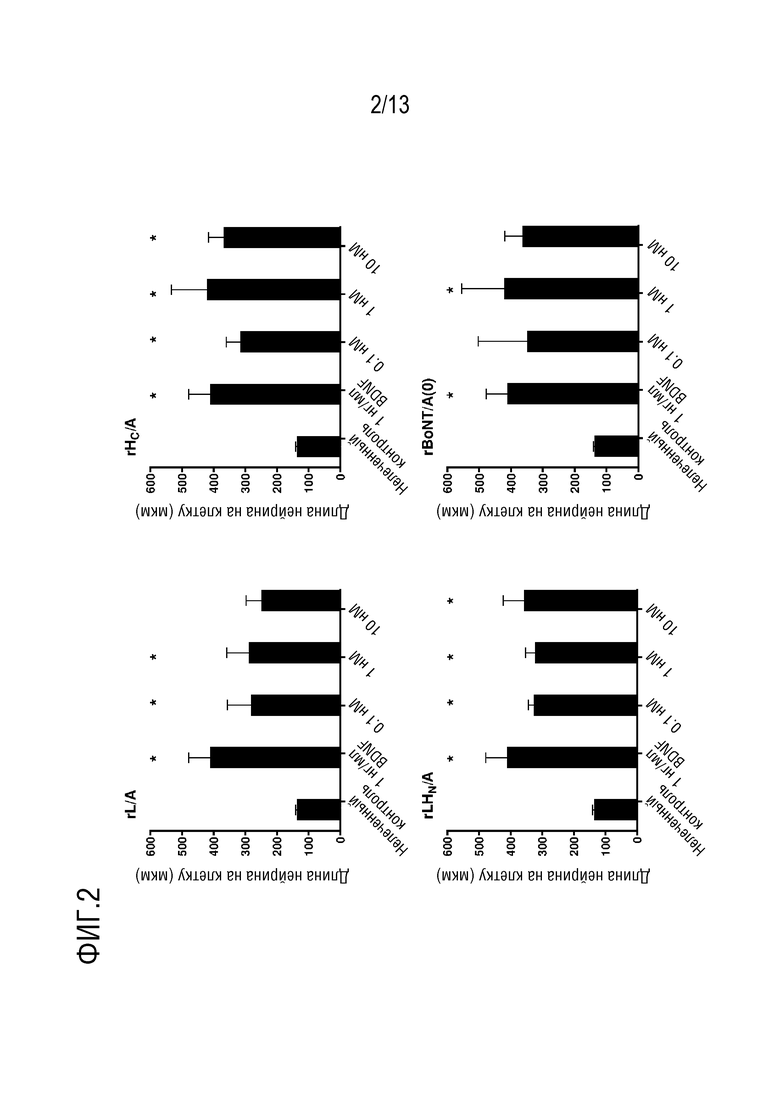
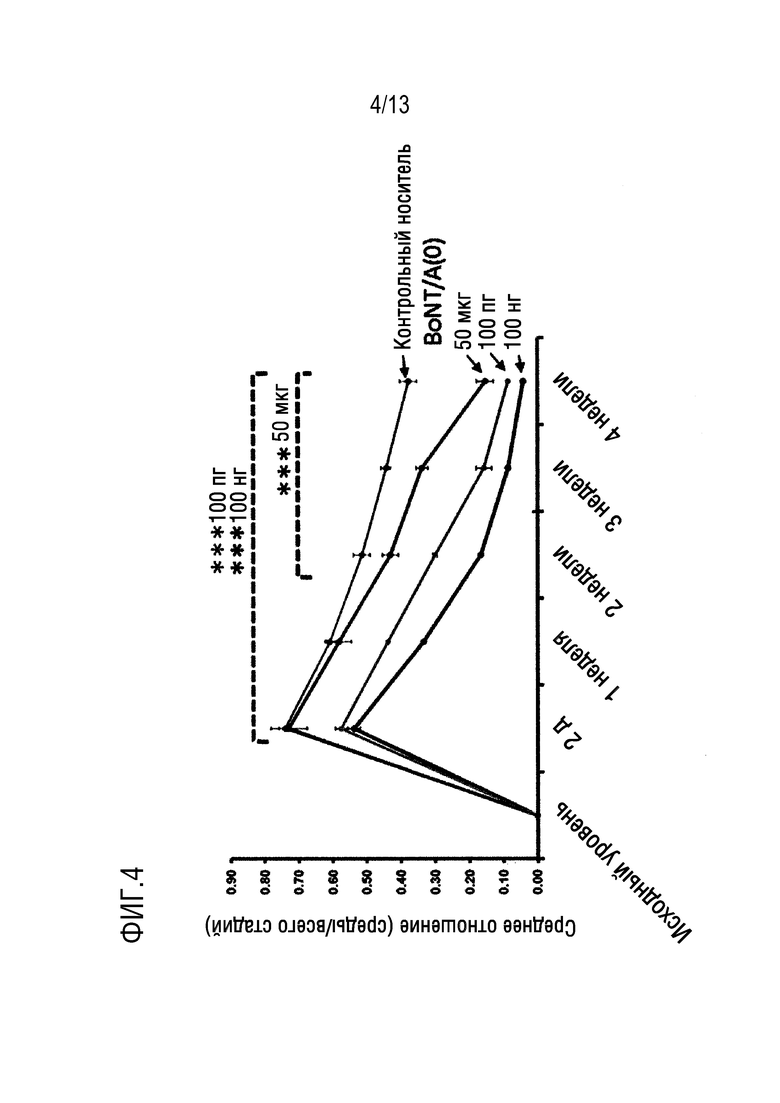
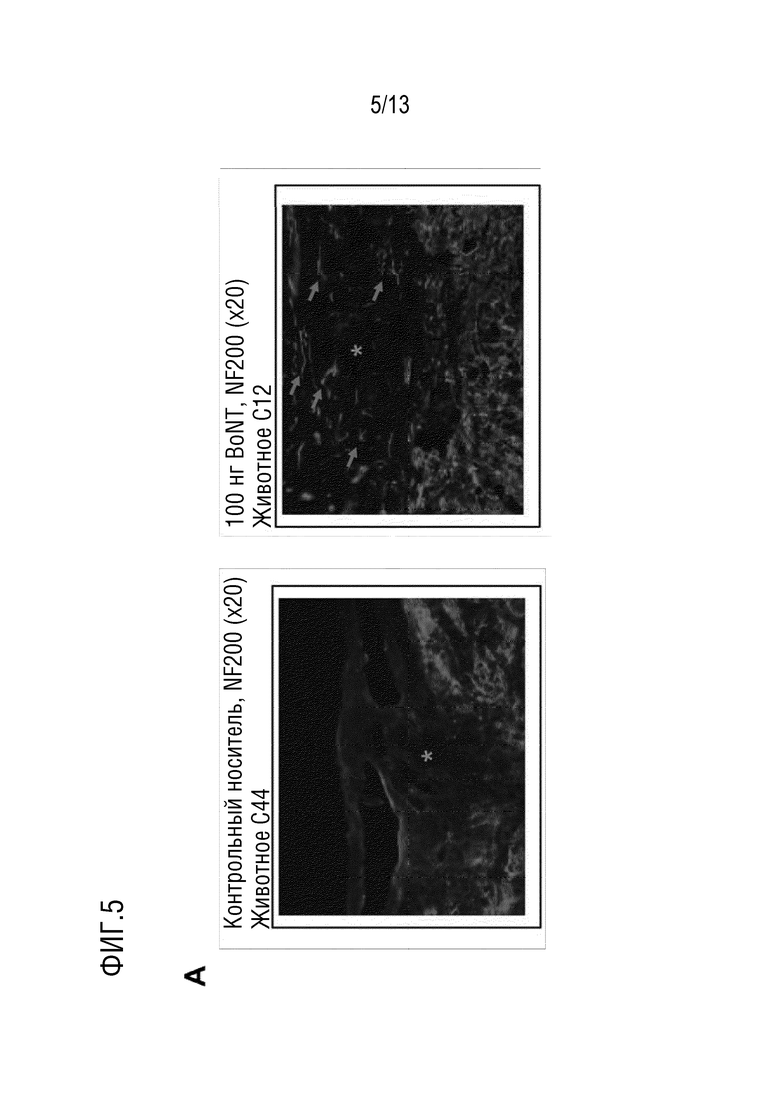


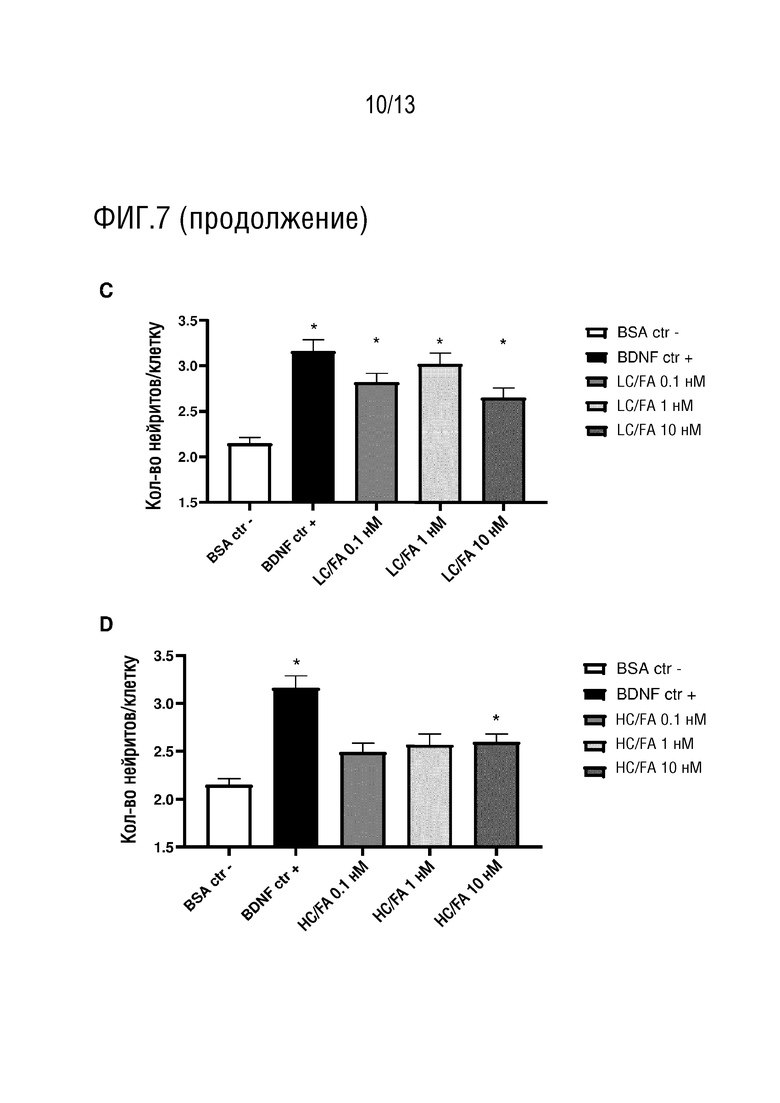
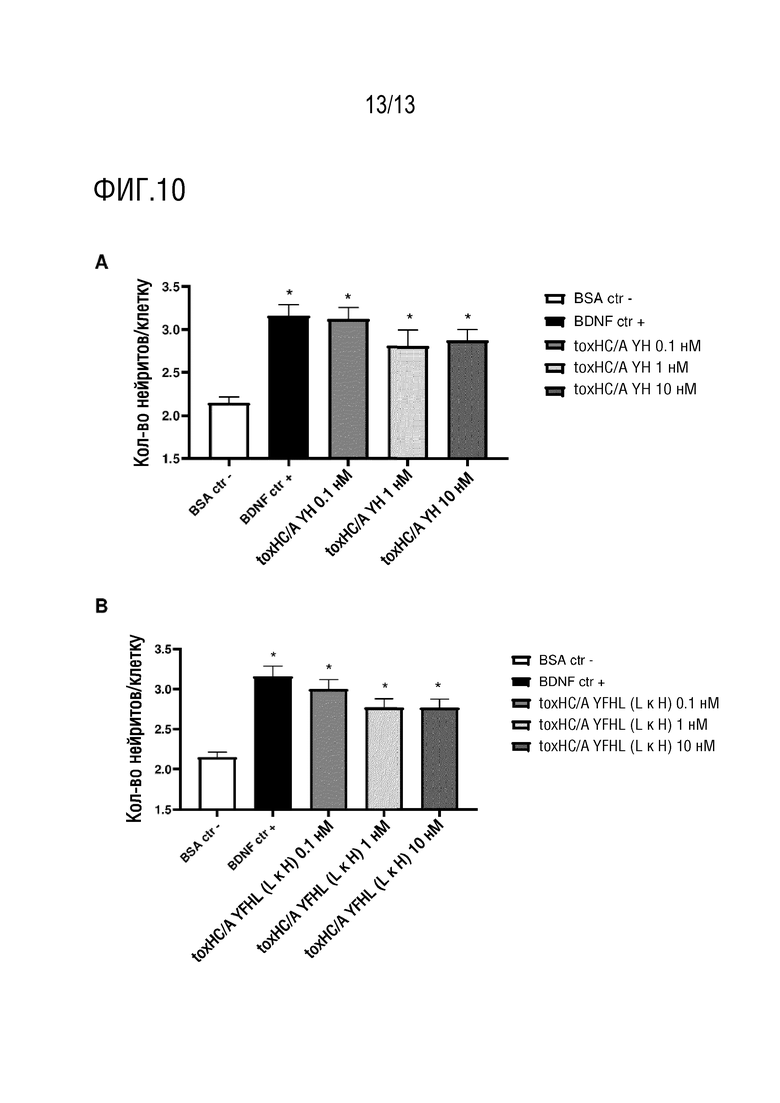
Изобретение относится к применению полипептида в стимуляции роста или восстановления нейронов для лечения неврологического нарушения у субъекта, где полипептид содержит: легкую цепь клостридиального нейротоксина (L-цепь), где L-цепь каталитически неактивна и/или фрагмент тяжелой цепи клостридиального нейротоксина (Н-цепь), и причем полипептид не содержит ни домен HN клостридиального нейротоксина, ни домен HC клостридиального нейротоксина. 8 н. и 10 з.п. ф-лы, 10 ил., 5 пр.
1. Применение полипептида в получении лекарственного средства для лечения неврологического нарушения у субъекта, причем полипептид содержит:
(i) легкую цепь (L-цепь) серотипа А ботулинического нейротоксина (BoNT/A), L-цепь серотипа B ботулинического нейротоксина (BoNT/B), L-цепь серотипа C ботулинического нейротоксина (BoNT/C), L-цепь серотипа D ботулинического нейротоксина (BoNT/D), L-цепь серотипа E ботулинического нейротоксина (BoNT/E), L-цепь серотипа F ботулинического нейротоксина (BoNT/F), L-цепь серотипа G ботулинического нейротоксина (BoNT/G), L-цепь серотипа X ботулинического нейротоксина (BoNT/X), L-цепь серотипа FA ботулинического нейротоксина (BoNT/FA) или L-цепь столбнячного нейротоксина (TeNT), где L-цепь каталитически неактивна; и/или
(ii) фрагмент тяжелой цепи клостридиального нейротоксина (Н-цепь), причем фрагмент H-цепи клостридиального нейротоксина содержит: (а) транслокационный домен (домен HN) BoNT/A, домен HN BoNT/B, домен HN BoNT/C, домен HN BoNT/D, домен HN BoNT/E, домен HN BoNT/F, домен HN BoNT/G, домен HN BoNT/X, домен HN BoNT/FA или домен HN TeNT или (b) рецептор-связывающий домен (домен HC) BoNT/A, домен HC BoNT/B, домен HC BoNT/C, домен HC BoNT/D, домен HC BoNT/E, домен HC BoNT/F, домен HC BoNT/G, домен HC BoNT/X, домен HC BoNT/FA или домен HC TeNT, и причем полипептид не содержит ни домен HN клостридиального нейротоксина, ни домен HC клостридиального нейротоксина;
и причем неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное расстройство, сенсорное расстройство, вегетативное расстройство или расстройство, которое можно лечить путем стимуляции роста нейронов и/или их восстановления у субъекта.
2. Способ лечения неврологического нарушения у субъекта, включающий введение субъекту терапевтически эффективного количества полипептида, причем полипептид содержит:
(i) легкую цепь (L-цепь) серотипа А ботулинического нейротоксина (BoNT/A), L-цепь серотипа B ботулинического нейротоксина (BoNT/B), L-цепь серотипа C ботулинического нейротоксина (BoNT/C), L-цепь серотипа D ботулинического нейротоксина (BoNT/D), L-цепь серотипа E ботулинического нейротоксина (BoNT/E), L-цепь серотипа F ботулинического нейротоксина (BoNT/F), L-цепь серотипа G ботулинического нейротоксина (BoNT/G), L-цепь серотипа X ботулинического нейротоксина (BoNT/X), L-цепь серотипа FA ботулинического нейротоксина (BoNT/FA) или L-цепь столбнячного нейротоксина (TeNT), где L-цепь каталитически неактивна; и/или
(ii) фрагмент тяжелой цепи клостридиального нейротоксина (Н-цепь), причем фрагмент H-цепи клостридиального нейротоксина содержит: (а) транслокационный домен (домен HN) BoNT/A, домен HN BoNT/B, домен HN BoNT/C, домен HN BoNT/D, домен HN BoNT/E, домен HN BoNT/F, домен HN BoNT/G, домен HN BoNT/X, домен HN BoNT/FA или домен HN TeNT или (b) рецептор-связывающий домен (домен HC) BoNT/A, домен HC BoNT/B, домен HC BoNT/C, домен HC BoNT/D, домен HC BoNT/E, домен HC BoNT/F, домен HC BoNT/G, домен HC BoNT/X, домен HC BoNT/FA или домен HC TeNT, и причем полипептид не содержит ни домен HN клостридиального нейротоксина, ни домен HC клостридиального нейротоксина;
и причем неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное расстройство, сенсорное расстройство, вегетативное расстройство или расстройство, которое можно лечить путем стимуляции роста нейронов и/или их восстановления у субъекта.
3. Применение по п. 1 или способ по п. 2, в котором:
(i) полипептид способствует росту нейронов или восстановлению нейронов для лечения неврологического нарушения;
(ii) полипептид состоит из легкой цепи клостридиального нейротоксина (L-цепи) и/или фрагмента тяжелой цепи клостридиального нейротоксина (Н-цепь);
(iii) фрагмент Н-цепи клостридиального нейротоксина содержит домен HN;
(iv) фрагмент Н-цепи клостридиального нейротоксина состоит из домена HN;
(v) фрагмент Н-цепи клостридиального нейротоксина содержит домен HC;
(vi) фрагмент Н-цепи клостридиального нейротоксина состоит из домена HC;
(vii) в полипептиде отсутствует С-концевая часть домена, связывающего рецептор клостридиального нейротоксина (HCC);
(viii) полипептид дополнительно не содержит неклостридиального каталитического домена;
(ix) полипептид содержит: L-цепь клостридиального нейротоксина и домен HN;
(x) полипептид состоит из: L-цепи клостридиального нейротоксина и домена HN.
4. Применение или способ по любому из пп. 1-3, в котором:
(i) полипептид не содержит нативной Н-цепи клостридиального нейротоксина;
(ii) полипептид является нейротрофным;
(iii) полипептид способствует росту нейронов и/или восстановлению нейронов;
(iv) неврологическое нарушение представляет собой нарушение, которое можно лечить путем стимуляции роста и/или восстановления нейронов; и/или
(v) полипептид способствует росту или восстановлению мотонейрона.
5. Применение или способ по любому из пп. 1-4, в котором полипептид:
(i) кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичности последовательности с любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
(ii) содержит (предпочтительно состоит из) полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичности последовательности с любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62 или 63.
6. Применение или способ по любому из пп. 1-5, где полипептид представляет собой модифицированный клостридиальный нейротоксин.
7. Применение или способ по п. 6, где модифицированный клостридиальный нейротоксин представляет собой химерный клостридиальный нейротоксин или гибридный клостридиальный нейротоксин.
8. Применение или способ по любому из пп. 1-7, где домен HC представляет собой модифицированный домен HC.
9. Применение или способ по любому из пп. 1-8, в котором:
(i) полипептид вводят в место повреждения или вблизи него, предпочтительно, в котором полипептид вводят интратекально;
(ii) в полипептиде отсутствует функциональный домен HC клостридиального нейротоксина, а также отсутствует какая-либо функционально эквивалентная экзогенная нацеливающая на лиганд группа (TM); и/или
(iii) клостридиальные последовательности полипептида состоят из легкой цепи клостридиального нейротоксина (L-цепи); и/или фрагмента тяжелой цепи клостридиального нейротоксина (Н-цепь).
10. Применение или способ по любому из пп. 1-9, в котором полипептид представляет собой химерный BoNT, содержащий домен легкой цепи и транслокации BoNT/A, и домен, связывающий рецептор BoNT/B (домен HC).
11. Применение или способ по любому из пп. 1-10, в котором полипептид представляет собой двухцепочечный полипептид, в котором легкая цепь (L-цепь) полипептида связана с тяжелой цепью (Н-цепью) полипептида через дисульфидную связь, и в котором двухцепочечный полипептид может быть получен способом, включающим приведение одноцепочечного полипептида в контакт с протеазой, которая гидролизует пептидную связь в его активационной петле, с превращением тем самым одноцепочечного полипептида в соответствующий двухцепочечный полипептид, предпочтительно, где одноцепочечный полипептид:
(i) кодируется нуклеотидной последовательностью, имеющей по меньшей мере 80%, 90%, 95% или 98% идентичности последовательности с любой из SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43, 45, 47, 49 или 60; или
(ii) содержит (предпочтительно состоит из) полипептидную последовательность, имеющую по меньшей мере 80%, 90%, 95% или 98% идентичности последовательности с любой из SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 61, 62 или 63.
12. Применение полипептида в получении лекарственного средства для стимуляции роста нейронов или восстановления нейронов для лечения неврологического расстройства у субъекта, причем полипептид состоит из:
(i) L-цепи BoNT/A, L-цепи BoNT/B, L-цепи BoNT/C, L-цепи BoNT/D, L-цепи BoNT/E, L-цепи BoNT/F, L-цепи BoNT/G, L-цепи BoNT/X, L-цепи BoNT/FA или L-цепи TeNT; или
(ii) L-цепи BoNT/A, L-цепи BoNT/B, L-цепи BoNT/C, L-цепи BoNT/D, L-цепи BoNT/E, L-цепи BoNT/F, L-цепи BoNT/G, L-цепи BoNT/X, L-цепи BoNT/FA или L-цепи TeNT и домена HN BoNT/A, домена HN BoNT/B, домена HN BoNT/C, домена HN BoNT/D, домена HN BoNT/E, домена HN BoNT/F, домена HN BoNT/G, домена HN BoNT/X, домена HN BoNT/FA или домена HN TeNT;
и причем неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное расстройство, сенсорное расстройство, вегетативное расстройство или расстройство, которое можно лечить путем стимуляции роста нейронов и/или их восстановления у субъекта.
13. Способ стимуляции роста или восстановления нейронов для лечения неврологического расстройства у субъекта, включающий введение субъекту терапевтически эффективного количества полипептида, причем полипептид состоит из:
(i) L-цепи BoNT/A, L-цепи BoNT/B, L-цепи BoNT/C, L-цепи BoNT/D, L-цепи BoNT/E, L-цепи BoNT/F, L-цепи BoNT/G, L-цепи BoNT/X, L-цепи BoNT/FA или L-цепи TeNT; или
(ii) домена HN BoNT/A, BoNT/B, BoNT/C, BoNT/D, BoNT/E, BoNT/F, BoNT/G, BoNT/X, BoNT/FA или L-цепь TeNT и BoNT/A, BoNT/B, BoNT/C, BoNT/D, BoNT/E, BoNT/F, BoNT/G, BoNT/X, BoNT/FA или TeNT;
и причем неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное расстройство, сенсорное расстройство, вегетативное расстройство или расстройство, которое можно лечить путем стимуляции роста нейронов и/или их восстановления у субъекта.
14. Применение или способ по п. 12 или 13, где L-цепь каталитически неактивна.
15. Применение полипептида в получении лекарственного средства для лечения неврологического расстройства у субъекта, причем полипептид:
(i) содержит полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63, 61, 44 или 42;
(ii) содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, по меньшей мере на 80% идентичной SEQ ID NO: 43, 60 или 41; и/или
(iii) представляет собой двухцепочечный полипептид, в котором легкая цепь (L-цепь) полипептида связана с тяжелой цепью (Н-цепь) полипептида посредством дисульфидной связи, и где двухцепочечный полипептид может быть получен способом, включающим приведение в контакт одноцепочечного полипептида, содержащего полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63 или SEQ ID NO: 61, с протеазой, которая гидролизует пептидную связь в его активационной петле, тем самым превращая одноцепочечный полипептид в соответствующий двухцепочечный полипептид;
причем доза полипептида, вводимая субъекту, составляет по меньшей мере 50 нг; и
причем неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное расстройство, сенсорное расстройство или вегетативное расстройство.
16. Способ лечения неврологического расстройства у субъекта, включающий введение субъекту терапевтически эффективного количества полипептида, причем полипептид:
(i) содержит полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63, 61, 44 или 42;
(ii) содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, по меньшей мере на 80% идентичной последовательности с SEQ ID NO: 43, 60 или 41; и/или
(iii) представляет собой двухцепочечный полипептид, в котором легкая цепь (L-цепь) полипептида связана с тяжелой цепью (Н-цепь) полипептида посредством дисульфидной связи, и где двухцепочечный полипептид может быть получен способом, включающим приведение в контакт одноцепочечного полипептида, содержащего полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63 или SEQ ID NO: 61, с протеазой, которая гидролизует пептидную связь в его активационной петле, тем самым превращая одноцепочечный полипептид в соответствующий двухцепочечный полипептид;
причем доза полипептида, вводимая субъекту, составляет по меньшей мере 50 нг; и
причем неврологическое нарушение представляет собой повреждение нейронов, нейродегенеративное расстройство, сенсорное расстройство или вегетативное расстройство.
17. Применение полипептида в получении лекарственного средства для лечения неврологического расстройства у субъекта, причем полипептид:
(i) содержит полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63, 61, 44 или 42;
(ii) содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, по меньшей мере на 80% идентичной SEQ ID NO: 43, 60 или 41; и/или
(iii) представляет собой двухцепочечный полипептид, в котором легкая цепь (L-цепь) полипептида связана с тяжелой цепью (Н-цепь) полипептида посредством дисульфидной связи, и где двухцепочечный полипептид может быть получен способом, включающим приведение в контакт одноцепочечного полипептида, содержащего полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63 или SEQ ID NO: 61, с протеазой, которая гидролизует пептидную связь в его активационной петле, тем самым превращая одноцепочечный полипептид в соответствующий двухцепочечный полипептид;
причем неврологическое расстройство представляет собой сенсорное расстройство, повреждение нейронов или нейродегенеративное расстройство; и
причем сенсорное расстройство не является болью.
18. Способ лечения неврологического расстройства у субъекта, включающий введение субъекту терапевтически эффективного количества полипептида, причем полипептид:
(i) содержит полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63, 61, 44 или 42;
(ii) содержит полипептидную последовательность, которая кодируется нуклеотидной последовательностью, по меньшей мере на 80% идентичной SEQ ID NO: 43, 60 или 41; и/или
(iii) представляет собой двухцепочечный полипептид, в котором легкая цепь (L-цепь) полипептида связана с тяжелой цепью (Н-цепь) полипептида посредством дисульфидной связи, и где двухцепочечный полипептид может быть получен способом, включающим приведение в контакт одноцепочечного полипептида, содержащего полипептидную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 63 или SEQ ID NO: 61, с протеазой, которая гидролизует пептидную связь в его активационной петле, тем самым превращая одноцепочечный полипептид в соответствующий двухцепочечный полипептид;
причем неврологическое расстройство представляет собой сенсорное расстройство, повреждение нейронов или нейродегенеративное расстройство; и
причем сенсорное расстройство не является болью.
| JULIE A | |||
| COFFIELD et al., Neuritogenic Actions of Botulinum Neurotoxin A on Cultured Motor Neurons, JOURNAL OF PHARMACOLOGY AND EXPERIMENTAL THERAPEUTICS, 2009, v | |||
| Катодная трубка Брауна | 1922 |
|
SU330A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Судно | 1918 |
|
SU352A1 |
| YA-FANG WANG et al., The Effect of Botulinum Neurotoxin Serotype a Heavy Chain on the Growth Related Proteins and Neurite Outgrowth after Spinal Cord Injury in | |||
Авторы
Даты
2025-04-30—Публикация
2020-09-30—Подача