Область техники
Настоящее изобретение относится к антителу, биспецифическому антителу и подобному, применимым для лечения опухолей.
Уровень техники
NY-ESO-1 представляет собой молекулу, которая была идентифицирована при раке пищевода способом серологического анализа библиотек экспрессии рекомбинантных кДНК (SEREX) (непатентная литература 1), и LAGE-1 также упоминается как «NY-ESO-2», которая представляет собой молекулу, идентифицированную с помощью типового анализа различий библиотеки кДНК опухоли (непатентная литература 2). Хотя известно, что экспрессия таких молекул локализована в яичках в случае нормальной ткани, ее механизм остается неизвестным. Поскольку сообщалось, что экспрессия NY-ESO-1 и LAGE-1 встречается при самых разных видах рака, таких как меланома, рак легких, рак мочевого пузыря, рак яичников, саркома мягких тканей и миелома (непатентная литература 3), также предполагается ее связь с раком. Кроме того, были сделаны сообщения о корреляции со степенью злокачественности. Например, уровень экспрессии NY-ESO-1 выше в метастатическом очаге меланомы, чем в ее первичном очаге (непатентная литература 4), уровни экспрессии NY-ESO-1 и LAGE-1 выше на поздней стадии уротелиальной карциномы, чем на ранней стадии уротелиальной карциномы (непатентная литература 5), и уровень экспрессии NY-ESO-1 выше при миеломе высокого риска с хромосомными аномалиями, чем при миеломе без хромосомных аномалий (непатентная литература 6). На основе такой информации, NY-ESO-1 и LAGE-1 привлекли внимание как молекулы с высокой специфичностью к раку, и было проведено большое количество исследований и разработок, направленных на открытие лекарственных средств для противораковой вакцинотерапии. Однако до настоящего времени не было утвержденных фармацевтических продуктов в этом отношении.
Известно, что каждый из 9-mer NY-ESO пептидов NY-ESO-1 и LAGE-1 содержит остатки со 157 по 165 (т.е. SLLMWITQC), образуют комплекс с HLA (лейкоцитарный антиген гистосовместимости)-A2 (т.е. пептидный комплекс HLA/NY-ESO) и презентирует комплекс внеклеточно (непатентная литература 7). Как описано выше, экспрессия NY-ESO-1 и LAGE-1 специфична для рака. Это указывает на то, что пептидный комплекс HLA/NY-ESO является терапевтической мишенью, специфичной для рака, которая селективно присутствует на HLA-A2-положительных и NY-ESO-1- или LAGE-1-положительных раковых клетках (непатентная литература 8). Кроме того, сообщалось о молекулах, которые связываются с пептидным комплексом HLA/NY-ESO, таких как TCR (патентный документ 1 и патентный документ 2) и антитела (патентный документ 3 и патентный документ 8).
Применение молекулы, связывающейся с молекулой, таргетирующей рак, представляет собой CD3-биспецифическое антитело, которое функционирует на основе механизма перенаправления Т-клеток, посредством чего оно рекрутирует Т-клетки к раковым клеткам и, таким образом, оказывает противоопухолевое действие за счет цитотоксичности (непатентная литература 9 и непатентная литература 10). Примером доступного в настоящее время CD3-биспецифического антитела является Блинатумомаб, который представляет собой привлекающий Т-клетки CD19-биспецифический активатор (BiTE). Такое лекарственное средство на основе антител одобрено для лечения острого лимфобластного лейкоза (ALL), и в настоящее время проводятся клинические испытания, направленные на другие виды рака крови. Однако формат его биспецифического антитела представляет собой тандемный scFv (taFv) без области Fc, и период полувыведения из крови, при его введении пациенту, значительно короче, чем при использовании антител типа IgG, которые обычно используют в качестве терапевтического антитела (непатентная литература 11).
Что касается биспецифических антител, имеющих гетеродимерную Fc область, демонстрирующих период полужизни в крови эквивалентный таковому антитела типа IgG, исследования и клинические испытания CD3-биспецифических антител с различными форматами антител, такими как выступы во впадины, CrossMAb и DuoBody® (патентные документы 4, 5, 6 и 7), находятся в разработке, и сообщается о CD3-биспецифическом антителе, использующем антитело против пептидного комплекса HLA/NY-ESO (непатентный документ 12).
Перечень цитирования
Патентная литература
Патентная литература 1: WO 2005/113595
Патентная литература 2: WO 2017/109496
Патентная литература 3: WO 2010/106431
Патентная литература 4: WO 1998/050431
Патентная литература 5: WO 2006/106905
Патентная литература 6: WO 2011/028952
Патентная литература 7: WO 2011/131746
Патентная литература 8: WO 2021/003357.
Непатентная литература
Непатентная литература 1: Proc. Natl. Acad. Sci., U.S.A., 94 (5), 1914-8, 1997
Непатентная литература 2: Int. J. Cancer, 76 (6), 903-8, 1998
Непатентная литература 3: Immunol. Cell Biol., 84 (3), 303-17, 2006
Непатентная литература 4: J. Surg. Res., 98 (2), 76-80, 2001
Непатентная литература 5: Cancer Res., 61 (12), 4671-4, 2001
Непатентная литература 6: Blood, 105 (10), 3939-44, 2005
Непатентная литература 7: J. Exp. Med., 187 (2), 265-70, 1998
Непатентная литература 8: J. Immunol., 176 (12), 7308-16, 2006
Непатентная литература 9: Nature, 314 (6012), 628-31, 1985
Непатентная литература 10: Int. Rev. Immunol., 4 (2), 159-73, 1989
Непатентная литература 11: Drug Des. Devel. Ther., 10, 757-765, 2016
Непатентная литература 12: Abstracts of the 21st Annual Meeting of the Japanese Association of Cancer Immunology, O13-4.
Сущность изобретения
Техническая проблема
Настоящее изобретение предлагает новое анти-HLA-A2/NY-ESO антитело, которое можно использовать в качестве противоопухолевого агента, и противоопухолевый агент, содержащий в качестве активного ингредиента молекулу, которая связывается с HLA-A2/NY-ESO, содержащий такое антитело и подобные.
Решение проблемы
Авторы настоящего изобретения провели концентрированные исследования, чтобы решить указанную выше проблему. В результате они обнаружили новое анти-HLA-A2/NY-ESO антитело и молекулу, которая связывается с HLA-A2/NY-ESO, содержащую такое антитело, и подобное. Это привело к завершению настоящего изобретения.
В частности, настоящее изобретение включает следующее.
[1] Антитело, которое специфически связывается с HLA/NY-ESO человека или его связывающим фрагментом, содержащее:
CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:57, или CDRL1 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:57, в которой аминокислота 7 представляет собой W и/или аминокислота 8 представляет собой K,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:59, или CDRL3 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:59, в которой аминокислота 2 представляет собой A или С.
[2] Антитело по [1], содержащее CDRH1-CDRH3 и CDRL1-CDRL3 одной или нескольких групп, выбранных из группы, состоящей из групп (i)-(v), указанных ниже, или его связывающий фрагмент:
(i) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:57,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:59;
(ii) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:57, в которой аминокислота 7 представляет собой W,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:59;
(iii) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:57,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:59, в которой аминокислота 2 представляет собой А;
(iv) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:57,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:59, в которой аминокислота 2 представляет собой S; и
(v) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:57, в которой аминокислота 7 представляет собой W, и аминокислота 8 представляет собой K,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:59.
[3] Антитело или его связывающий фрагмент по [1], содержащее вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности, имеющей 95% или более идентичность последовательности с аминокислотной последовательностью аминокислот 21-140 аминокислотной последовательности SEQ ID NO:27, или аминокислотной последовательности SEQ ID NO:38 или 39, и вариабельной области легкой цепи, состоящей из аминокислотной последовательности, имеющей 95% или более идентичность аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:27 или SEQ ID NO:52, или аминокислотной последовательности SEQ ID NO:40.
[4] Антитело или его связывающий фрагмент по [1], содержащее:
(H1) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:6,
(H2) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:18,
(H3) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:29,
(H4) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:26,
(H5) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:27,
(H6) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:28,
(H7) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:36,
(H8) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:47,
(H9) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:48,
(H10) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:50;
(H11) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:51,
(H12) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:52,
(H13) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:53,
(H14) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 из аминокислотной последовательности SEQ ID NO:30; или
(H15) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 из аминокислотной последовательности SEQ ID NO:156; и
(L1) вариабельную область легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:8,
(L2) вариабельную область легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:20,
(L3) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:29,
(L4) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:26,
(L5) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:27,
(L6) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:28,
(L7) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:36,
(L8) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:47,
(L9) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:48,
(L10) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:50,
(L11) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:51,
(L12) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:52,
(L13) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:53,
(L14) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:30; или
(L15) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 161-271 аминокислотной последовательности SEQ ID NO:156.
[5] Антитело или его связывающий фрагмент по [4], содержащее:
(H1L1) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:6, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:8,
(H2L2) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:18, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:20,
(H3L3) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:29, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:29,
(H4L4) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:26, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:26,
(H5L5) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:27, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:27,
(H6L6) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:28, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:28,
(H7L7) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:36, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:36,
(H8L8) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:47, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:47,
(H9L9) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:48, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:48,
(H10L10) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:50, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:50,
(H11L11) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:51, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:51,
(H12L12) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот от 21 до 140 аминокислотной последовательности SEQ ID NO:52, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:52,
(H13L13) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:53, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:53,
(H14L14) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:30, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:30, или
(H15L14) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:156, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот со 161 по 140 271 аминокислотной последовательности SEQ ID NO:156.
[6] Антитело или его связывающий фрагмент по любому из [1]-[5], которое представляет собой scFv.
[7] Антитело или его связывающий фрагмент по [6], которое представляет собой
(s1) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:70,
(s2) scFv, содержащий вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:18, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:20,
(s3) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:29,
(s4) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 из аминокислотной последовательности SEQ ID NO:26,
(s5) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:27,
(s6) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:28,
(s7) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:36,
(s8) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 из аминокислотной последовательности SEQ ID NO:47,
(s9) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:48,
(s10) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 из аминокислотной последовательности SEQ ID NO:50,
(s11) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 из аминокислотной последовательности SEQ ID NO:51,
(s12) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:52,
(s13) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 из аминокислотной последовательности SEQ ID NO:53,
(s14) scFv, состоящий из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:30, или
(s15) scFv, состоящий из аминокислотной последовательности из аминокислот 21-271 аминокислотной последовательности SEQ ID NO:156.
[8] Полинуклеотид, кодирующий антитело или его связывающий фрагмент по любому из [1]-[7].
[9] Вектор, содержащий полинуклеотид по [8].
[10] Клетка-хозяин, содержащая полинуклеотид по [8] или вектор по [9].
[11] Способ получения антитела, которое специфически связывается с HLA/NY-ESO человека или его связывающего фрагмента, включающий: (i) стадию культивирования клетки-хозяина по [10]; и (ii) стадию очистки антитела или его связывающего фрагмента из культурального продукта, полученного на стадии (i).
[12] Антитело, которое специфически связывается с HLA/NY-ESO человека или его связывающим фрагментом, полученное способом по [11].
[13] Антитело, которое обладает свойствами (i) или (ii) ниже и связывается с HLA-A2/NY-ESO или его связывающим фрагментом:
(i) связывание с сайтом на HLA-A2/NY-ESO, распознаваемым антителом или его связывающим фрагментом по [7]; или
(ii) связывание HLA-A2/NY-ESO человека на конкурентной основе с антителом или его связывающим фрагментом по [7].
[14] Фармацевтическая композиция, содержащая, в качестве активного ингредиента, антитело или его связывающий фрагмент по любому из [1]-[7], [12] и [13], полинуклеотид по [8], вектор по [9], или клетку по [10].
[15] Молекула, которая специфически связывается с HLA/NY-ESO человека, содержащая антитело или его связывающий фрагмент по любому из [1]-[7], [12] и [13].
[16] Молекула по [15], которая представляет собой мультиспецифическое антитело.
[17] Молекула по [15], которая представляет собой биспецифическое антитело.
[18] Молекула по любому из [15]-[17], содержащая антитело, которое специфически связывается с CD3, или его связывающий фрагмент.
[19] Молекула по [18], где антитело, которое специфически связывается с CD3, или его связывающий фрагмент содержит:
(CCH1) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:141;
(CCH2) CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:142, или CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:142, в которой аминокислота 3 представляет собой N или S;
(CCH3) CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:143;
(CCL1) CDRL1 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:144;
(CCL2) CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:145, или CDRL2 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:145, в которой аминокислота 2 представляет собой N; и
(CCL3) CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:146.
[20] Молекула по [19], где антитело, которое специфически связывается с CD3, или его связывающий фрагмент содержит:
(CH1) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:136,
(CH2) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:137,
(CH3) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:147,
(CH4) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:138,
(CH5) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:139,
(CH6) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:140,
(СН7) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 272-389 аминокислотной последовательности SEQ ID NO:155,
(CH8) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:156; или
(CH9) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:157; и
(CL1) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:136,
(CL2) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:137,
(CL3) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:147,
(CL4) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:138,
(CL5) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:139,
(CL6) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:140,
(CL7) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 405-511 аминокислотной последовательности SEQ ID NO:155,
(CL8) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:156, или
(CL9) вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:157.
[21] Молекула по [20], где антитело, которое специфически связывается с CD3, или его связывающий фрагмент содержит:
(CH1CL1) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:136, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:136;
(CH2CL2) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:137, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:137;
(CH3CL3) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:147, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:147;
(CH4CL4) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:138, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:138;
(CH5CL5) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот со 2 по 119 аминокислотной последовательности SEQ ID NO:139, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:139;
(CH6CL6) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:140, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:140;
(CH7CL7) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 272-389 аминокислотной последовательности SEQ ID NO:155, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 272-389 аминокислотной последовательности SEQ ID NO:155;
(CH8CL8) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:156, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:156; или
(CH9CL9) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:157, и вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:157.
[22] Молекула по любому из [18]-[21], где антитело, которое специфически связывается с CD3, или его связывающий фрагмент представляет собой scFv.
[23] Молекула по [22], где scFv содержит:
(CS1) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:136;
(CS2) scFv, состоящий из аминокислотной последовательности из аминокислот 2-241 аминокислотной последовательности SEQ ID NO:137;
(CS3) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:147;
(CS4) scFv, состоящий из аминокислотной последовательности из аминокислот 2-241 аминокислотной последовательности SEQ ID NO:138;
(CS5) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:139;
(CS6) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:140;
(CS7) scFv, состоящий из аминокислотной последовательности из аминокислот 272-511 из аминокислотной последовательности SEQ ID NO:155;
(CS8) scFv, состоящий из аминокислотной последовательности из аминокислот 277-516 из аминокислотной последовательности SEQ ID NO:156; или
(CS9) scFv, состоящий из аминокислотной последовательности из аминокислот 277-516 аминокислотной последовательности SEQ ID NO:157.
[24] Молекула по любому из [18]-[23], содержащая: первый полипептид, содержащий scFv, который специфически связывается с HLA/NY-ESO человека, scFv, который специфически связывается с CD3, и Fc область (i) в этом порядке от N конца к С концу; и второй полипептид, содержащий Fc область (ii), где первый полипептид предпочтительно связан со вторым полипептидом в Fc области (i) и Fc области (ii).
[25] Молекула по [24], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [1], который представляет собой scFv.
[26] Молекула по [24], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [2], который представляет собой scFv.
[27] Молекула по [24], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [3], который представляет собой scFv.
[28] Молекула по [24], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [4], который представляет собой scFv.
[29] Молекула по [24], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [5], который представляет собой scFv.
[30] Молекула по любому из [19]-[24], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [18], который представляет собой scFv.
[31] Молекула по любому из [19]-[24], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [19], который представляет собой scFv.
[32] Молекула по любому из [19]-[24], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [20] или [21], который представляет собой scFv.
[33] Молекула по любому из [19]-[24], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [23], который представляет собой scFv.
[34] Молекула по любому из [24]-[33], содержащая аминокислотную последовательность, выбранную из группы, состоящей из аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:85, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:87, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:88, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:89, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:90, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:91, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:92, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:93, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:94, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:95, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности показанной в SEQ ID NO:96, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:86, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:149, и аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:150.
[35] Молекула по любому из [24]-[33], которая содержит аминокислотную последовательность, выбранную из группы, состоящей из аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:155, аминокислотной последовательности из аминокислот 21-516 аминокислотной последовательности SEQ ID NO:156, и аминокислотной последовательности из аминокислот 21-516 аминокислотной последовательности SEQ ID NO:157.
[36] Молекула по любому из [24]-[34], где первый полипептид содержит аминокислотную последовательность аминокислот 529-745 аминокислотной последовательности SEQ ID NO:85, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 86, 149 или 150.
[37] Молекула по любому из [24]-[33] и [35], где первый полипептид содержит аминокислотную последовательность аминокислот 529-745 аминокислотной последовательности SEQ ID NO:155, аминокислотную последовательность аминокислот 534-750 аминокислотной последовательности SEQ ID NO:156, или аминокислотную последовательность аминокислот 534-750 аминокислотной последовательности SEQ ID NO:157.
[38] Молекула по [34] или [36], где первый полипептид состоит из аминокислотной последовательности, выбранной из группы, состоящей из аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:85, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:87, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:88, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:89, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:90, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:91, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:92, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:93, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:94, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:95, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:96, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:86, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:149, и аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:150.
[39] Молекула по [35] или [37], где первый полипептид состоит из аминокислотной последовательности, выбранной из группы, состоящей из аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:155, аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:156, и аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:157.
[40] Молекула по любому из [24]-[39], где второй полипептид содержит аминокислотную последовательность аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84.
[41] Молекула по любому из [18]-[23], содержащая первый полипептид, второй полипептид и третий полипептид, где
первый полипептид содержит scFv, который специфически связывается с HLA/NY-ESO человека, scFv, который специфически связывается с CD3, и Fc область (i) в указанном порядке от N конца к С концу,
второй полипептид содержит тяжелую цепь иммуноглобулина, содержащую Fc область (ii), и
третий полипептид содержит легкую цепь иммуноглобулина ; и
второй полипептид предпочтительно связан с третьим полипептидом, и
первый полипептид предпочтительно связан со вторым полипептидом в Fc областях.
[42] Молекула по [41], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [1], который представляет собой scFv.
[43] Молекула по [41], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [2], который представляет собой scFv.
[44] Молекула по [41], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [3], который представляет собой scFv.
[45] Молекула по [41], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [4], который представляет собой scFv.
[46] Молекула по [41], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [5], который представляет собой scFv.
[47] Молекула по любому из [42]-[46], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [18], который представляет собой scFv.
[48] Молекула по любому из [42]-[46], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [19], который представляет собой scFv.
[49] Молекула по любому из [42]-[46], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [20] или [21], который представляет собой scFv.
[50] Молекула по любому из [42]-[46], где scFv, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [23], который представляет собой scFv.
[51] Молекула по любому из [41]-[50], где второй полипептид содержит аминокислотную последовательность аминокислот 20-242 аминокислотной последовательности SEQ ID NO:99.
[52] Молекула по любому из [41]-[51], где третий полипептид содержит аминокислотную последовательность, показанную в SEQ ID NO:100.
[53] Молекула по любому из пунктов [41]-[52], где первый полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:85, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:87, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:88, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:89, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:90, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:91, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:92, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:93, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:94, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:95, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:96, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:86, аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:149, и аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:150.
[54] Молекула по любому из [41]-[53], где первый полипептид состоит из аминокислотной последовательности, выбранной из группы, состоящей из аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:85, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:87, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:88, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:89, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:90, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:91, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:92, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:93, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:94, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:95, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:96, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:86, аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:149, и аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:150.
[55] Молекула по любому из [18]-[23], содержащая первый полипептид, содержащий scFv, который специфически связывается с HLA/NY-ESO человека, вариабельную область тяжелой цепи и константную область CH1 антитела, которое специфически связывается с CD3, и Fc область иммуноглобулина (i) в указанном порядке от N конца к C концу, второй полипептид, который содержит шарнирную область и Fc область (ii) иммуноглобулина, и третий полипептид, который содержит легкую цепь антитела, состоящую из вариабельной области и константной области, где первый полипептид предпочтительно связан со вторым полипептидом в Fc области (i) и Fc области (ii), и первый полипептид предпочтительно связан с третьим полипептидом в вариабельной области тяжелой цепи и константной области CH1 антитела.
[56] Молекула по [55], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [1], который представляет собой scFv.
[57] Молекула по [55], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [2], который представляет собой scFv.
[58] Молекула по [55], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [3], который представляет собой scFv.
[59] Молекула по [55], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [4], который представляет собой scFv.
[60] Молекула по [55], где scFv, который специфически связывается с HLA/NY-ESO человека, представляет собой антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент по [5], который представляет собой scFv.
[61] Молекула по любому из [18]-[21], где антитело, которое специфически связывается с CD3 или его связывающим фрагментом, представляет собой Fab.
[62] Молекула по любому из [56]-[61], где Fab, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [18], который представляет собой Fab.
[63] Молекула по любому из [56]-[61], где Fab, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [19], который представляет собой Fab.
[64] Молекула по любому из [56]-[61], где Fab, который специфически связывается с CD3, представляет собой антитело, которое специфически связывается с CD3, или его связывающий фрагмент по [20] или [21], который представляет собой Fab.
[65] Молекула по любому из [55]-[64], где первый полипептид содержит аминокислотную последовательность аминокислот 21-394 аминокислотной последовательности SEQ ID NO:160.
[66] Молекула по любому из [55]-[65], где первый полипептид содержит аминокислотную последовательность по любому из (i)-(iii) ниже:
(i) аминокислотную последовательность аминокислот 20-724 аминокислотной последовательности SEQ ID NO:160;
(ii) аминокислотную последовательность аминокислот 20-719 аминокислотной последовательности SEQ ID NO:197; и
(iii) аминокислотную последовательность аминокислот 20-719 аминокислотной последовательности SEQ ID NO:198.
[67] Молекула по любому из [55]-[66], где второй полипептид содержит аминокислотную последовательность аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84.
[68] Молекула по любому из [56]-[67], где третий полипептид содержит аминокислотную последовательность аминокислот 21-127 аминокислотной последовательности SEQ ID NO:161.
[69] Молекула по любому из [56]-[68], где третий полипептид содержит аминокислотную последовательность аминокислот 21-233 аминокислотной последовательности SEQ ID NO:161.
[70] Молекула по любому из пунктов [15]-[69], где 1 или 2 аминокислоты удалены с карбоксильного конца аминокислотной последовательности, по меньшей мере, одного полипептида, включенного в молекулу.
[71] Полинуклеотид, содержащий нуклеотидную последовательность, кодирующую аминокислотную последовательность, включенную в молекулу по любому из [15]-[70].
[72] Вектор, содержащий полинуклеотид по [71].
[73] Клетка-хозяин, содержащая полинуклеотид по [71] или вектор по [72].
[74] Способ получения молекулы, которая специфически связывается с HLA/NY-ESO человека и CD3 человека, включающий: (i) стадию культивирования клетки-хозяина по [73]; и (ii) стадию очистки антитела или его связывающего фрагмента из культурального продукта, полученного на стадии (i).
[75] Молекула, специфически связывающаяся с HLA/NY-ESO человека и CD3 человека, которую получают способом по [74].
[76] Фармацевтическая композиция, содержащая в качестве активного ингредиента молекулу по любому из [15]-[70] и [75], полинуклеотид по [71], вектор по [72] или клетку-хозяина по [73].
[77] Фармацевтическая композиция по [14] или [76], которая представляет собой противораковый агент.
[78] Фармацевтическая композиция по [77], где рак представляет собой один или несколько видов рака, выбранных из группы, состоящей из рака почки, меланомы, плоскоклеточного рака, базальноклеточного рака, рака конъюнктивы, рака полости рта, рака гортани, рака глотки, рака щитовидной железы, рака легкого (немелкоклеточного рака легкого (аденокарциномы, эпидермоидного рака, крупноклеточного рака) и мелкоклеточного рака легкого), рака молочной железы, рака пищевода, рака желудка, рака двенадцатиперстной кишки, рака тонкой кишки, рака толстой кишки, рака прямой кишки, рака аппендикса, рака анального канала, рака печени, рака желчного пузыря, рака желчных протоков, рака поджелудочной железы, рака надпочечников, рака мочевого пузыря, рака предстательной железы, рака матки, рака влагалища, липосаркомы, ангиосаркомы, хондросаркомы, рабдомиосаркомы, саркомы Юинга, остеосаркомы, недифференцированной плеоморфной саркомы, миксофибросаркомы, злокачественной опухоли оболочек периферических нервов, забрюшинной саркомы, синовиальной саркомы, саркомы матки, стромальной опухоли желудочно-кишечного тракта, лейомиосаркомы, эпителиоидной саркомы, В-клеточной лимфомы, Т/NK-клеточной лимфомы, лимфомы Ходжкина, миелогенного лейкоза, лимфатического лейкоза, миелопролиферативного заболевания, миелодиспластического синдрома, множественной миеломы, карциномы яичек и рака яичников.
[79] Фармацевтическая композиция по любому из [76]-[78], которую применяют в комбинации с другим агентом.
Настоящее описание охватывает содержание, описанное в заявке на патент Японии № 2020-061476, приоритет которой испрашивается по настоящей заявке.
Полезные эффекты изобретения
Согласно настоящему изобретению получают антитело, которое связывается с HLA-A2/NY-ESO, и новое биспецифическое антитело (биспецифическую молекулу), которое связывается с HLA-A2/NY-ESO и с CD3. Также получена новая фармацевтическая композиция, содержащая в качестве активного ингредиента такое антитело (молекулу). Такое антитело или молекула обладают цитотоксичностью и, таким образом, могут быть использованы в качестве агента для лечения или профилактики рака и подобных.
Краткое описание чертежей
[Фиг. 1] На фиг. 1 показана таблица, демонстрирующая стандартизированные gMFI анти-HLA/NY-ESO scFv: NYA-0001, NYA-1143, NYA-1154, NYA-1163, NYA-2023, NYA-2027, NYA-2035, NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060, NYA-2061, NYA-2143, NYC-0003 и NYC-0004 по сравнению с Т2 клетками, дополненными различными точечными мутантными пептидами. *Каждое подчеркнутое значение указывает половину или меньше стандартизированного gMFI каждого scFv по сравнению с T2 клетками, дополненными пептидом NY-ESO.
[Фиг. 2A] На фиг. 2A показана таблица, демонстрирующая информацию, касающуюся выбранных гомологичных пептидов. Аффинность связывания с HLA-A0201, спрогнозированная с использованием NetMHCPan2.8, указана в терминах 50% ингибирующей концентрации (IC50).
[Фиг. 2B] На фиг. 2B показана таблица, демонстрирующая стандартизированные gMFI анти-HLA/NY-ESO scFv: NYA-0001, NYA-1143, NYA-1154, NYA-1163, NYA-2023, NYA-2027, NYA-2035, NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060, NYA-2061, NYA-2143, NYC-0003 и NYC-0004 по сравнению с Т2 клетками, дополненными различными гомологичными пептидами. *Каждое подчеркнутое значение указывает на значение, превышающее стандартизированную gMFI каждого scFv по отношению к таковому для T2 клеток с добавлением ДМСО.
[Фиг. 3] На фиг. 3 показаны форматы антител, продемонстрированные в примерах. (а) показан формат scFv, в котором вариабельная область Н цепи антитела (VH, описанная ниже) лигирована с вариабельной областью L цепи антитела (VL, описана ниже) (оба пустые) с помощью линкера. В примерах, для оценки используют scFv анти-HLA-A2/NY-ESO и анти-CD3 scFv. (b) показан формат Fab, который включает VH (пустой), константную область H цепи антитела (CH1: клетчатый узор), VL (пустой) и константную область L цепи антитела (граничные линии). В примерах, для оценки используют анти-HLA-A2/NY-ESO Fab и подобные. (c) показан формат taFv, в котором конструкции scFv двух типов (пустые и положительные наклоны) лигированы друг с другом с помощью линкера. В примерах, для оценки используют taFv, включая анти-HLA-A2/NY-ESO scFv и анти-CD3 scFv. (d) показан формат Fc типа гетеродимера taFv, в котором Fc (отрицательные наклоны), содержащий образующую гетеродимер мутацию, введенную в С концевой сайт taFv (первый полипептид), гетерологически связан с другим Fc (закрашено: второй полипептид). В примере, для оценки используют taFv гетеродимер Fc, содержащий анти-HLA-A2/NY-ESO scFv и анти-CD3 scFv. (e) показан формат типа Fc гетеродимера taFv-Fab, в котором Fab добавлен к формату типа Fc гетеродимера taFv. В примере, для оценки используют taFv, включая анти-HLA-A2/NY-ESO scFv и анти-CD3 scFv, и taFv-Fab-гетеродимер-Fc, включая HLA-A2/NY-ESO Fab. (f) показан первый полипептид, общий для гетеродимера taFv типа Fc и гетеродимера taFv-Fab типа Fc. Первый полипептид содержит scFv, который специфически связывается с HLA/NY-ESO человека, scFv, который специфически связывается с CD3, и Fc область (i) в указанном порядке от N конца к С концу. (g) показан второй полипептид taFv-гетеродимера Fc типа. Второй полипептид содержит шарнирную область и Fc область (ii). (h) показан второй полипептид taFv-Fab-гетеродимера Fc типа. Второй полипептид содержит тяжелую цепь иммуноглобулина, включающую шарнирную область и Fc область (ii). (i) показан третий полипептид taFv-Fab-гетеродимера Fc типа. Третий полипептид содержит легкую цепь иммуноглобулина.
[Фиг. 4A] Фиг. 4A демонстрирует, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0019, NYF-0022, NYF-0023, NYF-0027, NYF-0035, NYF-0044 и NYF-0047 демонстрируют цитотоксичность в отношении клеток U266B1 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4B] Фиг. 4B демонстрирует, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016 и NYF-0058 демонстрируют цитотоксичность в отношении клеток U266B1 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4C] На фиг. 4C показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0023, NYF-0045, NYF-0048, NYF-0060 и NYF-0061 оказывают цитотоксическое действие на эндогенные клетки U266B1, экспрессирующие NY-ESO человека, эндогенно экспрессирующие NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4D] На фиг. 4D показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0019, NYF-0022, NYF-0023, NYF-0027, NYF-0035, NYF-0044 и NYF-0047 демонстрируют цитотоксичность в отношении клеток NCI-H1703 человека, эндогенно экспрессирующих NY-ESO в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4E] На фиг. 4E показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016 и NYF-0058 демонстрируют цитотоксичность в отношении клеток NCI-H1703 человека, эндогенно экспрессирующих NY-ESO в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4F] На фиг. 4F показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0023, NYF-0045, NYF-0048, NYF-0060 и NYF-0061 демонстрируют цитотоксичность в отношении клеток NCI-H1703 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4G] На фиг. 4G показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0019, NYF-0022, NYF-0023, NYF-0027, NYF-0035, NYF-0044 и NYF-0047 не оказывают цитотоксического действия на клетки AGS человека, эндогенно не экспрессирующие NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4H] На фиг. 4H показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016 и NYF-0058 не демонстрируют цитотоксичности в отношении клеток AGS человека, эндогенно не экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4I] На фиг. 4I показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0023, NYF-0045, NYF-0048, NYF-0060 и NYF-0061 не демонстрируют цитотоксичности в отношении клеток AGS человека, эндогенно не экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4J] На фиг. 4J показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0019, NYF-0022, NYF-0023, NYF-0027, NYF-0035, NYF-0044 и NYF-0047 не оказывают цитотоксического действия на клетки CFPAC-1 человека, эндогенно не экспрессирующие NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4K] На фиг. 4K показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016 и NYF-0058 не демонстрируют цитотоксичности в отношении клеток CFPAC-1 человека, эндогенно не экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 4L] На фиг. 4L показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0023, NYF-0045, NYF-0048, NYF-0060 и NYF-0061 не демонстрируют цитотоксичности в отношении клеток CFPAC-1 человека, эндогенно не экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 5A] На фиг. 5A показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0019, NYF-0044 и NYF-0047 демонстрируют противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5, но только n=4 на 32 день в группе NYF-0044).
[Фиг. 5B] На фиг. 5B показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0022, NYF-0023, NYF-0027, NYF-0035 и NYF-0058 демонстрируют противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5, n=6 только в группе контрольного носителя).
[Фиг. 5C] На фиг. 5C показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYF-0016, NYF-0045, NYF-0048, NYF-0060 и NYF-0061 демонстрируют противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5).
[Фиг. 6A] На фиг. 6A показаны форматы антител, описанные в примерах. (а): формат гибридного типа: Fc (косые линии), содержащий мутацию, образующую гетеродимер, и Fc (сплошной), содержащий мутацию, образующую гетеродимер, добавляют к С концевой области Fab и scFv, соответственно, и две Fc конструкции связывают. В примере, для оценки используют формат гибридного типа, содержащий анти-HLA-A2/NY-ESO Fab и анти-CD3 scFv. (b): Формат двойного типа: Fc (отрицательные наклоны), содержащий мутацию, образующую гетеродимер, и Fc (сплошной), содержащий мутацию, образующую гетеродимер, добавляют к С концевым областям двух различных типов конструкций scFv, и две конструкции Fc гетерологически связывают. В примере, для оценки используют формат двойного типа, включающий анти-HLA-A2/NY-ESO scFv и анти-CD3 scFv. (c): формат scFv-Fab-гетеродимер Fc типа: Fc (отрицательные наклоны), содержащий мутацию, образующую гетеродимер, добавляют в С концевую область, где scFv лигируют с Fab линкером и связывают с другим Fc (сплошной). В примере, для оценки используют формат scFv-Fab-гетеродимер Fc типа, содержащий анти-CD3 scFv (положительные наклоны) и анти-HLA-A2/NY-ESO Fab. Кроме того, для оценки используют scFv-Fab-гетеродимер Fc типа, содержащий анти-HLA-A2/NY-ESO scFv (положительные наклоны) и анти-CD3 Fab.
[Фиг. 6B] На фиг. 6B показаны форматы антител, описанные в примерах. (а): формат taFv-гетеродимера Fc типа: то же, что и на фиг. 3 (d); Fc (отрицательные наклоны), содержащий образующую гетеродимер мутацию, добавляют к С концевой области taFv и гетерологически связывают с другим Fc (сплошной). В примере, для оценки используют taFv-гетеродимер Fc типа, содержащий анти-HLA-A2/NY-ESO scFv и анти-CD3 scFv. (b): формат taFv (инвертированного)-гетеродимера Fc типа: порядок двух конструкций scFv инвертирован в формате taFv-гетеродимер Fc типа. В примере, для оценки используют taFv (инвертированный)-гетеродимер Fc типа, содержащий анти-CD3 scFv и анти-HLA-A2/NY-ESO scFv. (c) показан первый полипептид taFv (инвертированного)-гетеродимера Fc типа. Первый полипептид содержит scFv, который специфически связывается с CD3, scFv, который специфически связывается с HLA/NY-ESO человека, и область Fc (i) в указанном порядке от N конца к С концу. (d) показан второй полипептид taFv (инвертированный)-гетеродимер Fc типа. Второй полипептид содержит шарнирную область и область Fc (ii).
[Фиг. 7A] Фиг. 7A демонстрирует, что различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3; гибридного типа (NYG-3143), двойного типа (NYG-2143) и taFv-гетеродимер типа Fc (NYF-0011) демонстрируют цитотоксичность в отношении клеток U266B1 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 7B] Фиг. 7B демонстрирует, что различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3; scFv-Fab-гетеродимер Fc типа (NYF-0003), taFv-гетеродимер Fc типа (NYF-0010), taFv (инвертированный)-гетеродимер Fc типа (NYF-0004), демонстрируют цитотоксичность в отношении клеток U266B1 человека, эндогенно экспрессирующих NY-ESO в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 8] Аминокислотная последовательность пептида в NY-ESO (SEQ ID NO:1).
[Фиг. 9] Аминокислотная последовательность пептида в MAGEC-1 (SEQ ID NO:2).
[Фиг. 10] Праймер 1 для анализа последовательности scFv (SEQ ID NO:3).
[Фиг. 11] Праймер 2 для анализа последовательности scFv (SEQ ID NO:4).
[Фиг. 12] Нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0001 (SEQ ID NO:5).
[Фиг. 13] Аминокислотная последовательность вариабельной области тяжелой цепи NYA-0001 (SEQ ID NO:6).
[Фиг. 14] Нуклеотидная последовательность вариабельной области легкой цепи NYA-0001 (SEQ ID NO:7).
[Фиг. 15] Аминокислотная последовательность вариабельной области легкой цепи NYA-0001 (SEQ ID NO:8).
[Фиг. 16] Нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0060 (SEQ ID NO:9).
[Фиг. 17] Аминокислотная последовательность вариабельной области тяжелой цепи NYA-0060 (SEQ ID NO:10).
[Фиг. 18] Нуклеотидная последовательность вариабельной области легкой цепи NYA-0060 (SEQ ID NO:11).
[Фиг. 19] Аминокислотная последовательность вариабельной области легкой цепи NYA-0060 (SEQ ID NO:12).
[Фиг. 20] Нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0068 (SEQ ID NO:13).
[Фиг. 21] Аминокислотная последовательность вариабельной области тяжелой цепи NYA-0068 (SEQ ID NO:14).
[Фиг. 22] Нуклеотидная последовательность вариабельной области легкой цепи NYA-0068 (SEQ ID NO:15).
[Фиг. 23] Аминокислотная последовательность вариабельной области легкой цепи NYA-0068 (SEQ ID NO:16).
[Фиг. 24] Нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0082 (SEQ ID NO:17).
[Фиг. 25] Аминокислотная последовательность вариабельной области тяжелой цепи NYA-0082 (SEQ ID NO:18).
[Фиг. 26] Нуклеотидная последовательность вариабельной области легкой цепи NYA-0082 (SEQ ID NO:19).
[Фиг. 27] Аминокислотная последовательность вариабельной области легкой цепи NYA-0082 (SEQ ID NO:20).
[Фиг. 28] Нуклеотидная последовательность аддукта метки NYA-1163 (SEQ ID NO:21).
[Фиг. 29] Нуклеотидная последовательность аддукта метки NYA-2023 (SEQ ID NO:22).
[Фиг. 30] Нуклеотидная последовательность аддукта метки NYA-2027 (SEQ ID NO:23).
[Фиг. 31] Нуклеотидная последовательность аддукта метки NYA-1143 (SEQ ID NO:24).
[Фиг. 32] Нуклеотидная последовательность аддукта метки NYA-2143 (SEQ ID NO:25).
[Фиг. 33] Аминокислотная последовательность аддукта метки NYA-1163 (SEQ ID NO:26); NYA-1163: аминокислоты 21-266.
[Фиг. 34] Аминокислотная последовательность аддукта метки NYA-2023 (SEQ ID NO:27); NYA-2023: аминокислоты 21-266.
[Фиг. 35] Аминокислотная последовательность аддукта метки NYA-2027 (SEQ ID NO:28); NYA-2027: аминокислоты 21-266.
[Фиг. 36] Аминокислотная последовательность аддукта метки NYA-1143 (SEQ ID NO:29); NYA-1143: аминокислоты 21-266.
[Фиг. 37] Аминокислотная последовательность аддукта метки NYA-2143 (SEQ ID NO:30); NYA-2143: аминокислоты 21-266.
[Фиг. 38] Нуклеотидная последовательность аддукта метки NYA-1154 (SEQ ID NO:31).
[Фиг. 39] Аминокислотная последовательность аддукта метки NYA-1154 (SEQ ID NO:32); NYA-1154: аминокислоты 21-266.
[Фиг. 40] Усеченная аминокислотная последовательность HLA-A*0201 (GenBank: ASA47534.1) (SEQ ID NO:33).
[Фиг. 41] Аминокислотная последовательность β2-микроглобина (SEQ ID NO:34).
[Фиг. 42] Нуклеотидная последовательность аддукта метки NYA-2035 (SEQ ID NO:35).
[Фиг. 43] Аминокислотная последовательность аддукта метки NYA-2035 (SEQ ID NO:36); NYA-2035: аминокислоты 21-266.
[Фиг. 44] Аминокислотная последовательность NYA-1143-VH01 (SEQ ID NO:37).
[Фиг. 45] Аминокислотная последовательность NYA-1143-VH02 (SEQ ID NO:38).
[Фиг. 46] Аминокислотная последовательность NYA-1143-VH03 (SEQ ID NO:39).
[Фиг. 47] Аминокислотная последовательность NYA-1143-VL01 (SEQ ID NO:40).
[Фиг. 48] Нуклеотидная последовательность аддукта метки NYA-2044 (SEQ ID NO:41).
[Фиг. 49] Нуклеотидная последовательность аддукта метки NYA-2045 (SEQ ID NO:42).
[Фиг. 50] Нуклеотидная последовательность аддукта метки NYA-2047 (SEQ ID NO:43).
[Фиг. 51] Нуклеотидная последовательность аддукта метки NYA-2048 (SEQ ID NO:44).
[Фиг. 52] Нуклеотидная последовательность аддукта метки NYA-2060 (SEQ ID NO:45).
[Фиг. 53] Нуклеотидная последовательность аддукта метки NYA-2061 (SEQ ID NO:46).
[Фиг. 54] Аминокислотная последовательность аддукта метки NYA-2044 (SEQ ID NO:47); NYA-2044: аминокислоты 21-266.
[Фиг. 55] Аминокислотная последовательность аддукта метки NYA-2045 (SEQ ID NO:48); NYA-2045: аминокислоты 21-266.
[Фиг. 56] Аминокислотная последовательность NYA-0082 (SEQ ID NO:49).
[Фиг. 57] Аминокислотная последовательность аддукта метки NYA-2047 (SEQ ID NO:50); NYA-2047: аминокислоты 21-266.
[Фиг. 58] Аминокислотная последовательность аддукта метки NYA-2048 (SEQ ID NO:51); NYA-2048: аминокислоты 21-266.
[Фиг. 59] Аминокислотная последовательность аддукта метки NYA-2060 (SEQ ID NO:52); NYA-2060: аминокислоты 21-266.
[Фиг. 60] Аминокислотная последовательность аддукта метки NYA-2061 (SEQ ID NO:53); NYA-2061: аминокислоты 21-266.
[Фиг. 61] Аминокислотные последовательности от CDRH1 до CDRH3 тяжелой цепи NYA-0001 и от CDRL1 до CDRL3 легкой цепи NYA-0001 (SEQ ID NO:54-59).
[Фиг. 62] Аминокислотная последовательность CDRL1 NYA-2023 (SEQ ID NO:60).
[Фиг. 63] Аминокислотная последовательность CDRL3 NYA-2027 (SEQ ID NO:61).
[Фиг. 64] Аминокислотные последовательности CDRH3 и CDRL3 NYA-1154 (SEQ ID NO:62 и 63).
[Фиг. 65] Аминокислотная последовательность CDRL1 NYA-0035 (SEQ ID NO:64).
[Фиг. 66] Нуклеотидная последовательность аддукта метки NYC-0003 (SEQ ID NO:65).
[Фиг. 67] Нуклеотидная последовательность аддукта метки NYC-0004 (SEQ ID NO:66).
[Фиг. 68] Аминокислотная последовательность аддукта метки NYC-0003 (SEQ ID NO:67); NYC-0003: аминокислоты с 21 по 263.
[Фиг. 69] Аминокислотная последовательность аддукта метки NYC-0004 (SEQ ID NO:68); NYC-0004: аминокислоты с 21 по 263.
[Фиг. 70] Нуклеотидная последовательность аддукта метки NYA-0001 (SEQ ID NO:69).
[Фиг. 71] Аминокислотная последовательность аддукта метки NYA-0001 (SEQ ID NO:70); NYA-0001: аминокислоты 21-266.
[Фиг. 72] Нуклеотидная последовательность HC1 (SEQ ID NO:71).
[Фиг. 73] Нуклеотидная последовательность NYF-0016-HC2 (SEQ ID NO:72).
[Фиг. 74] Нуклеотидная последовательность NYF-0019-HC2 (SEQ ID NO:73).
[Фиг. 75] Нуклеотидная последовательность NYF-0022-HC2 (SEQ ID NO:74).
[Фиг. 76] Нуклеотидная последовательность NYF-0023-HC2 (SEQ ID NO:75).
[Фиг. 77] Нуклеотидная последовательность NYF-0027-HC2 (SEQ ID NO:76).
[Фиг. 78] Нуклеотидная последовательность NYF-0035-HC2 (SEQ ID NO:77).
[Фиг. 79] Нуклеотидная последовательность NYF-0044-HC2 (SEQ ID NO:78).
[Фиг. 80] Нуклеотидная последовательность NYF-0045-HC2 (SEQ ID NO:79).
[Фиг. 81] Нуклеотидная последовательность NYF-0047-HC2 (SEQ ID NO:80).
[Фиг. 82] Нуклеотидная последовательность NYF-0048-HC2 (SEQ ID NO:81).
[Фиг. 83] Нуклеотидная последовательность NYF-0060-HC2 (SEQ ID NO:82).
[Фиг. 84] Нуклеотидная последовательность NYF-0061-HC2 (SEQ ID NO:83).
[Фиг. 85] Аминокислотная последовательность HC1 (SEQ ID NO:84).
[Фиг. 86] Аминокислотная последовательность NYF-0016-HC2 (SEQ ID NO:85); NYA-1143: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 87] Аминокислотная последовательность NYF-0019-HC2 (SEQ ID NO:86); NYA-2143: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 88] Аминокислотная последовательность NYF-0022-HC2 (SEQ ID NO:87); NYA-1163: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 89] Аминокислотная последовательность NYF-0023-HC2 (SEQ ID NO:88); NYA-2023: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 90] Аминокислотная последовательность NYF-0027-HC2 (SEQ ID NO:89); NYA-2027: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 91] Аминокислотная последовательность NYF-0035-HC2 (SEQ ID NO:90); NYA-2035: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 92] Аминокислотная последовательность NYF-0044-HC2 (SEQ ID NO:91); NYA-2044: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 93] Аминокислотная последовательность NYF-0045-HC2 (SEQ ID NO:92); NYA-2045: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 94] Аминокислотная последовательность NYF-0047-HC2 (SEQ ID NO:93); NYA-2047: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 95] Аминокислотная последовательность NYF-0048-HC2 (SEQ ID NO:94); NYA-2048: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 96] Аминокислотная последовательность NYF-0060-HC2 (SEQ ID NO:95); NYA-2060: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 97] Аминокислотная последовательность NYF-0061-HC2 (SEQ ID NO:96); NYA-2061: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 98] Нуклеотидная последовательность делеции NYA-0001-Fab-HC1-k (SEQ ID NO:97)
[Фиг. 99] Нуклеотидная последовательность NYA-0001-LC (SEQ ID NO:98).
[Фиг. 100] Аминокислотная последовательность делеции NYA-0001-Fab-HC1-k (SEQ ID NO:99); Вариабельная область тяжелой цепи NYA-0001: аминокислоты 20-139.
[Фиг. 101] Аминокислотная последовательность NYA-0001-LC (SEQ ID NO:100); Вариабельная область легкой цепи NYA-0001: аминокислоты 21-131.
[Фиг. 102] Нуклеотидная последовательность делеции NYA-1143-Fab-HC1-k (SEQ ID NO:101).
[Фиг. 103] Нуклеотидная последовательность NYA-1143-LC (SEQ ID NO:102).
[Фиг. 104] Нуклеотидная последовательность C3E-7085-HC2-k с делецией C (SEQ ID NO:103).
[Фиг. 105] Аминокислотная последовательность делеции NYA-1143-Fab-HC1-k (SEQ ID NO:104); вариабельная область тяжелой цепи NYA-1143: аминокислоты 20-139.
[Фиг. 106] Аминокислотная последовательность NYA-1143-LC (SEQ ID NO:105); вариабельная область легкой цепи NYA-1143: аминокислоты 21-131.
[Фиг. 107] Аминокислотная последовательность делеции C3E-7085-HC2-k (SEQ ID NO:106); C3E-7085: аминокислоты 21-260.
[Фиг. 108] Нуклеотидная последовательность делеции NYA-1143-HC1-k (SEQ ID NO:107).
[Фиг. 109] Аминокислотная последовательность делеции NYA-1143-HC1-k (SEQ ID NO:108); NYA-1143: аминокислоты 21-266.
[Фиг. 110] Нуклеотидная последовательность делеции C3E-7085-NYA-1154-Fab-HC2-k (SEQ ID NO:109).
[Фиг. 111] Нуклеотидная последовательность NYA-1154-LC (SEQ ID NO:110).
[Фиг. 112] Нуклеотидная последовательность делеции OAA-HC1-k (SEQ ID NO:111).
[Фиг. 113] Аминокислотная последовательность делеции C3E-7085-NYA-1154-Fab-HC2-k (SEQ ID NO:112); C3E-7085: аминокислоты 21-260; вариабельная область тяжелой цепи NYA-1154: аминокислоты 266-285.
[Фиг. 114] Аминокислотная последовательность NYA-1154-LC (SEQ ID NO:113); вариабельная область легкой цепи NYA-1154: аминокислоты 21-131.
[Фиг. 115] Аминокислотная последовательность делеции OAA-HC1-k (SEQ ID NO:114).
[Фиг. 116] Нуклеотидная последовательность делеции NYF-0010-HC2-k (SEQ ID NO:115).
[Фиг. 117] Нуклеотидная последовательность делеции NYF-0004-HC2-k (SEQ ID NO:116).
[Фиг. 118] Нуклеотидная последовательность делеции NYF-0011-HC2-k (SEQ ID NO:117).
[Фиг. 119] Аминокислотная последовательность делеции NYF-0010-HC2-k (SEQ ID NO:18); NYA-1154: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 120] Аминокислотная последовательность делеции NYF-0004-HC2-k (SEQ ID NO:119); C3E-7085: аминокислоты 21-260; NYA-1154: аминокислоты 272-511.
[Фиг. 121] Аминокислотная последовательность делеции NYF-0011-HC2-k (SEQ ID NO:120); NYA-1143: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 122] Аминокислотная последовательность точечного мутантного пептида 1F NY-ESO (SEQ ID NO:121).
[Фиг. 123] Аминокислотная последовательность точечного мутантного пептида NY-ESO 2M (SEQ ID NO:122).
[Фиг. 124] Аминокислотная последовательность точечного мутантного пептида 3A NY-ESO (SEQ ID NO:123).
[Фиг. 125] Аминокислотная последовательность точечного мутантного пептида 4A NY-ESO (SEQ ID NO:124).
[Фиг. 126] Аминокислотная последовательность точечного мутантного пептида 5A NY-ESO (SEQ ID NO:125).
[Фиг. 127] Аминокислотная последовательность точечного мутантного пептида 6L NY-ESO (SEQ ID NO:126).
[Фиг. 128] Аминокислотная последовательность точечного мутантного пептида 7F NY-ESO (SEQ ID NO:127).
[Фиг. 129] Аминокислотная последовательность точечного мутантного пептида 8A NY-ESO (SEQ ID NO:128).
[Фиг. 130] Аминокислотная последовательность точечного мутантного пептида 9A NY-ESO (SEQ ID NO:129).
[Фиг. 131] Аминокислотная последовательность пептида gp100 (SEQ ID NO:130).
[Фиг. 132] Аминокислотная последовательность гомологичного пептида DOLPP1 (SEQ ID NO:131).
[Фиг. 133] Аминокислотная последовательность гомологичного пептида IL20RB (SEQ ID NO:132).
[Фиг. 134] Аминокислотная последовательность гомологичного пептида PRKD2 (SEQ ID NO:133).
[Фиг. 135] Аминокислотная последовательность гомологичного пептида CD163 (SEQ ID NO:134).
[Фиг. 136] Аминокислотная последовательность гомологичного пептида P2RY8 (SEQ ID NO:135).
[Фиг. 137] Аминокислотная последовательность C3E-7034 (SEQ ID NO:136).
[Фиг. 138] Аминокислотная последовательность C3E-7036 (SEQ ID NO:137).
[Фиг. 139] Аминокислотная последовательность C3E-7085 (SEQ ID NO:138).
[Фиг. 140] Аминокислотная последовательность C3E-7088 (SEQ ID NO:139).
[Фиг. 141] Аминокислотная последовательность C3E-7093 (SEQ ID NO:140).
[Фиг. 142] Аминокислотные последовательности CDRH1-CDRH3 тяжелой цепи C3E-7085 и CDRL1-CDRL3 легкой цепи C3E-7085 (SEQ ID NO:141-146).
[Фиг. 143] Аминокислотная последовательность C3E-7078 (SEQ ID NO:147).
[Фиг. 144] Нуклеотидная последовательность NYF-0014-HC2 (SEQ ID NO:148).
[Фиг. 145] Аминокислотная последовательность NYF-0014-HC2 (SEQ ID NO:149); NYA-0001: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 146] Аминокислотная последовательность NYF-0082-HC2 (SEQ ID NO:150); NYA-0082: аминокислоты 21-266; C3E-7085: аминокислоты 272-511.
[Фиг. 147] Аминокислотная последовательность CD3 ε человека (SEQ ID NO:151).
[Фиг. 148] Нуклеотидная последовательность всего NYZ-0038-HC2 (SEQ ID NO:152).
[Фиг. 149] Нуклеотидная последовательность всего NYZ-0082-HC2 (SEQ ID NO:153).
[Фиг. 150] Нуклеотидная последовательность всего NYZ-0083-HC2 (SEQ ID NO:154).
[Фиг. 151] Аминокислотная последовательность всего NYZ-0038-HC2 (SEQ ID NO:155); NYA-2061: аминокислоты 21-266; C3E-7096: аминокислоты 272-511.
[Фиг. 152] Аминокислотная последовательность всего NYZ-0082-HC2 (SEQ ID NO:156); NYA-3061: аминокислоты 21-271; C3E-7096: аминокислоты 277-516.
[Фиг. 153] Аминокислотная последовательность всего NYZ-0083-HC2 (SEQ ID NO:157); NYA-3061: аминокислоты 21-271; C3E-7097: аминокислоты 277-516.
[Фиг. 154] Нуклеотидная последовательность полноразмерного NYZ-1010-HC2 (SEQ ID NO:158).
[Фиг. 155] Нуклеотидная последовательность полноразмерного C3E-7085-LC (SEQ ID NO:159).
[Фиг. 156] Аминокислотная последовательность полноразмерного NYZ-1010-HC2 (SEQ ID NO:160); NYA-3061: аминокислоты 21-271; вариабельная область тяжелой цепи C3E-7085: аминокислоты 277-394.
[Фиг. 157] Аминокислотная последовательность полноразмерного C3E-7085-LC (SEQ ID NO:161).
[Фиг. 158A] На фиг. 158A показана таблица, демонстрирующая стандартизированную gMFI Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3; NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 относительно нокаутных по CD3e Т2-клеток, дополненных различными точечными мутантными пептидами. *Каждое подчеркнутое значение указывает половину или меньше стандартизированной gMFI каждой Fc-конъюгированной анти-HLA-A2/NY-ESO-анти-CD3 биспецифической молекулы по сравнению с Т2-клетками с нокаутом по CD3e, дополненными пептидом NY-ESO, и *каждое полужирное значение указывает на четверть или меньше стандартизированной gMFI.
[Фиг. 158B] На фиг. 158B представлена таблица, демонстрирующая стандартизированную gMFI Fc конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3; NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 относительно нокаутных по CD3e Т2-клеток, дополненных различными гомологичными пептидами. *Каждое подчеркнутое значение указывает значение каждой биспецифической молекулы анти-HLA-A2/NY-ESO-анти-CD3 с добавлением Fc, превышающее стандартизированную gMFI T2-клеток с нокаутом CD3e, дополненных ДМСО.
[Фиг. 159A] На фиг. 159A показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYZ-0038, NYZ-0082 и NYZ-0083 демонстрируют цитотоксичность в отношении клеток U266B1 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159B] На фиг. 159B показано, что Fc-конъюгированная анти-HLA-A2/NY-ESO-анти-CD3 биспецифическая молекула; т.е. NYZ-1010 проявляет цитотоксичность в отношении клеток U266B1 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159C] На фиг. 159C показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYZ-0082 и NYZ-1010 демонстрируют цитотоксичность в отношении клеток NCI-H1703 человека, эндогенно экспрессирующих NY-ESO в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159D] На фиг. 159D показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYZ-0038 и NYZ-0083 демонстрируют цитотоксичность в отношении клеток NCI-H1703 человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159E] На фиг. 159E показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYZ-0038, NYZ-0082 и NYZ-0083 не демонстрируют цитотоксичности в отношении клеток AGS человека, эндогенно экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159F] На фиг. 159F показано, что Fc-конъюгированная анти-HLA-A2/NY-ESO-анти-CD3 биспецифическая молекула; т.е. NYZ-1010 не оказывает цитотоксического действия на клетки AGS человека, эндогенно не экспрессирующие NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159G] На фиг. 159G показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYZ-0082 и NYZ-1010 не демонстрируют цитотоксичности в отношении клеток CFPAC-1 человека, эндогенно не экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 159H] На фиг. 159H показано, что Fc-конъюгированные анти-HLA-A2/NY-ESO-анти-CD3 биспецифические молекулы; т.е. NYZ-0038 и NYZ-0083 не демонстрируют цитотоксичности в отношении клеток CFPAC-1 человека, эндогенно не экспрессирующих NY-ESO, в присутствии РВМС человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=3).
[Фиг. 160A] На фиг. 160A показано, что Fc-конъюгированная анти-HLA-A2/NY-ESO-анти-CD3 биспецифическая молекула; т.е. NYZ-0038 проявляет противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5).
[Фиг. 160B] На фиг. 160B показано, что Fc-конъюгированная анти-HLA-A2/NY-ESO-анти-CD3 биспецифическая молекула; т.е. NYZ-0082 проявляет противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5).
[Фиг. 160C] На фиг. 160C показано, что Fc-конъюгированная анти-HLA-A2/NY-ESO-анти-CD3 биспецифическая молекула; т.е. NYZ-0083 проявляет противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5).
[Фиг. 160D] На фиг. 160D показано, что Fc-конъюгированная анти-HLA-A2/NY-ESO-анти-CD3 биспецифическая молекула; т.е. NYZ-1010 проявляет противоопухолевую активность на РВМС-трансфицированных моделях человека. Планка погрешности на фигуре указывает на стандартное отклонение (n=5).
[Фиг. 161] Аминокислотная последовательность пептидного линкера (SEQ ID NO:162).
[Фиг. 162] На фиг. 162 показано содержание полимера (%) при обработке кислотой различных Fc-конъюгированных анти-HLA/NY-ESO-анти-CD3 биспецифических молекул.
[Фиг. 163] На фиг. 163 показаны результаты оценки стабильности раствора анти-HLA/NY-ESO scFv-гетеродимера Fc.
[Фиг. 164] Нуклеотидная последовательность полноразмерного NYA-3061 (SEQ ID NO:163).
[Фиг. 165] Аминокислотная последовательность полноразмерного NYA-3061 (SEQ ID NO:164).
[Фиг. 166] Нуклеотидная последовательность полноразмерного NYC-0005 (SEQ ID NO:165).
[Фиг. 167] Аминокислотная последовательность полноразмерного NYC-0005 (SEQ ID NO:166).
[Фиг. 168] Нуклеотидная последовательность полноразмерного NYC-0006 (SEQ ID NO:167).
[Фиг. 169] Аминокислотная последовательность полноразмерного NYC-0006 (SEQ ID NO:168).
[Фиг. 170] Нуклеотидная последовательность полноразмерного NYC-0007 (SEQ ID NO:169).
[Фиг. 171] Аминокислотная последовательность полноразмерного NYC-0007 (SEQ ID NO:170).
[Фиг. 172] Нуклеотидная последовательность полноразмерного NYC-0008 (SEQ ID NO:171).
[Фиг. 173] Аминокислотная последовательность полноразмерного NYC-0008 (SEQ ID NO:172).
[Фиг. 174] Нуклеотидная последовательность полноразмерного NYC-0009 (SEQ ID NO:173).
[Фиг. 175] Аминокислотная последовательность полноразмерного NYC-0009 (SEQ ID NO:174).
[Фиг. 176] Нуклеотидная последовательность полноразмерного NYC-0010 (SEQ ID NO:175).
[Фиг. 177] Аминокислотная последовательность полноразмерного NYC-0010 (SEQ ID NO:176).
[Фиг. 178] Нуклеотидная последовательность полноразмерного HC-h (SEQ ID NO:177).
[Фиг. 179] Аминокислотная последовательность полноразмерного HC-h (SEQ ID NO:178).
[Фиг. 180] Нуклеотидная последовательность полноразмерного NYD-2047-HC-k (SEQ ID NO:179).
[Фиг. 181] Аминокислотная последовательность полноразмерного NYD-2047-HC-k (SEQ ID NO:180).
[Фиг. 182] Нуклеотидная последовательность полноразмерного NYD-2061-HC-k (SEQ ID NO:181).
[Фиг. 183] Аминокислотная последовательность полноразмерного NYD-2061-HC-k (SEQ ID NO:182).
[Фиг. 184] Нуклеотидная последовательность полноразмерного NYD-3061-HC-k (SEQ ID NO:183).
[Фиг. 185] Аминокислотная последовательность полноразмерного NYD-3061-HC-k (SEQ ID NO:184).
[Фиг. 186] Нуклеотидная последовательность полноразмерного NYC-0011-HC-k (SEQ ID NO:185).
[Фиг. 187] Аминокислотная последовательность полноразмерного NYC-0011-HC-k (SEQ ID NO:186).
[Фиг. 188] Нуклеотидная последовательность полноразмерного NYC-0012-HC-k (SEQ ID NO:187).
[Фиг. 189] Аминокислотная последовательность полноразмерного NYC-0012-HC-k (SEQ ID NO:188).
[Фиг. 190] Нуклеотидная последовательность полноразмерного NYC-0013-HC-k (SEQ ID NO:189).
[Фиг. 191] Аминокислотная последовательность полноразмерного NYC-0013-HC-k (SEQ ID NO:190).
[Фиг. 192] Нуклеотидная последовательность полноразмерного NYC-0014-HC-k (SEQ ID NO:191).
[Фиг. 193] Аминокислотная последовательность полноразмерного NYC-0014-HC-k (SEQ ID NO:192).
[Фиг. 194] Нуклеотидная последовательность полноразмерного NYC-0015-HC-k (SEQ ID NO:193).
[Фиг. 195] Аминокислотная последовательность полноразмерного NYC-0015-HC-k (SEQ ID NO:194).
[Фиг. 196] Нуклеотидная последовательность полноразмерного NYC-0016-HC-k (SEQ ID NO:195).
[Фиг. 197] Аминокислотная последовательность полноразмерного NYC-0016-HC-k (SEQ ID NO:196).
[Фиг. 198] Аминокислотная последовательность полноразмерного NYZ-1007-HC2 (SEQ ID NO:197); NYA-2061: аминокислоты 21-266; вариабельная область тяжелой цепи C3E-7085: аминокислоты 272-389.
[Фиг. 199] Аминокислотная последовательность полноразмерного NYZ-1017-HC2 (SEQ ID NO:198); NYA-2047: аминокислоты 21-266; вариабельная область тяжелой цепи C3E-7085: аминокислоты с 277 по 389.
Описание вариантов осуществления
Далее настоящее изобретение описано подробно.
1. Определение
В настоящем изобретении, термин «ген» относится к нуклеотидной цепи, включающей нуклеотидную последовательность, кодирующую аминокислоту белка или его комплементарную цепь. Например, полинуклеотид, олигонуклеотид, ДНК, мРНК, кДНК, кРНК или подобные, которые представляют собой нуклеотидную цепь, включающую нуклеотидную последовательность, кодирующую аминокислоту белка или его комплементарную цепь, входят в объем «гена». Такой ген представляет собой одноцепочечный, двухцепочечный, трех- или более цепочечный нуклеотид, и соединение цепи ДНК и цепи РНК, одноцепочечной цепи нуклеотидов, содержащей как рибонуклеотид (РНК), так и дезоксирибонуклеотид (ДНК), и двухцепочечный или трехцепочечный нуклеотид, содержащий такую нуклеотидную цепь, входят в объем «гена». В настоящем изобретении, термин «последовательность оснований» является синонимом термина «последовательность нуклеотидов».
В настоящем изобретении термины «полинуклеотид», «нуклеотидная цепь», «нуклеиновая кислота» и «молекула нуклеиновой кислоты» являются синонимами. Например, ДНК, РНК, зонд, олигонуклеотид и праймер входят в понятие «полинуклеотид». Такой полинуклеотид состоит из 1, 2, 3 или более цепей, а также соединения цепи ДНК и цепи РНК, одиночной полинуклеотидной цепи, включающей как рибонуклеотид (РНК), так и дезоксирибонуклеотид (ДНК), и двухцепочечного или трехцепочечного нуклеотида, содержащего такую полинуклеотидную цепь, входит в объем «полинуклеотида».
В настоящем изобретении термины «полипептид», «пептид» и «белок» являются синонимами.
В настоящем изобретении, термин «антиген» может относиться к термину «иммуноген» по необходимости.
В настоящем изобретении, термин «клетка» охватывает различные клетки, полученные от животных, клетки субкультуры, клетки первичной культуры, клеточные линии, рекомбинантные клетки и микроорганизмы.
В настоящем изобретении, термин «антитело» является синонимом термина «иммуноглобулин». Однако термин «антитело» в случае анти-HLA/NY-ESO антитела по настоящему изобретению относится к иммуноглобулину, содержащему константную область и вариабельную область. Антитело конкретно не ограничено, и оно может представлять собой встречающийся в природе или частично или полностью синтезированный иммуноглобулин. Анти-HLA/NY-ESO антитело по настоящему изобретению включено в термин «молекула», описанный ниже.
В настоящем изобретении, «NY-ESO пептид» означает пептид, состоящий из 9 аминокислот 157-165 NY-ESO-1 и LAGE-1 (SLLMWITQC: SEQ ID NO:1).
В настоящем изобретении «HLA-A2/NY-ESO» обозначает комплекс пептида NY-ESO и лейкоцитарного антигена-A2 гистосовместимости (HLA-A2), и он также обозначается как «HLA/NY-ESO».
В настоящем изобретении термин «анти-HLA-A2/NY-ESO антитело» относится к антителу, которое связывается с HLA-A2/NY-ESO. Другими словами, этот термин относится к антителу, которое распознает HLA-A2/NY-ESO. Кроме того, термин «анти-HLA-A2/NY-ESO scFv» относится к scFv, который связывается с HLA/NY-ESO. Другими словами, этот термин относится к scFv, который распознает HLA-A2/NY-ESO. Термины «анти-HLA-A2/NY-ESO антитело» и «анти-HLA-A2/NY-ESO scFv» также обозначаются как «анти-HLA/NY-ESO антитело» и «анти-HLA/NY-ESO scFv», соответственно.
Базовая четырехцепочечная структура антитела состоит из двух идентичных легких цепей (L цепей) и двух идентичных тяжелых цепей (Н цепей). Легкая цепь соединяется с тяжелой цепью посредством одинарной ковалентной дисульфидной связи. Две тяжелые цепи связаны друг с другом посредством одной или нескольких дисульфидных связей в соответствии с изотипами тяжелых цепей. Каждая из легкой цепи и тяжелой цепи имеет дисульфидную связь внутри цепи через равные промежутки времени. В легкой цепи и тяжелой цепи имеется константная область, проявляющая очень высокое сходство аминокислотной последовательности, и вариабельная область, проявляющая низкое сходство аминокислотной последовательности. Легкая цепь содержит на своем амино конце вариабельную область (VL), примыкающую к константной области (CL). Тяжелая цепь содержит на своем амино конце вариабельную область (VL), примыкающую к 3 константным областям (СН1/СН2/СН3). VL спаривается с VH, и CL выравнивается с первой константной областью тяжелой цепи (CH1). VL спаривается с VH, образуя один антигенсвязывающий сайт.
Константная область антитела по настоящему изобретению конкретно не ограничена. Антитело по настоящему изобретению, предназначенное для лечения или профилактики заболеваний человека, предпочтительно, содержит константную область антитела человека. Примеры константных областей тяжелой цепи антитела человека включают Cγ1, Cγ2, Cγ3, Cγ4, Cµ, Cδ, Cα1, Cα2 и Cε. Примеры константных областей легкой цепи антитела человека включают Cκ и Cλ.
Fab содержит VH тяжелой цепи, примыкающую к ней CH1, VL легкой цепи и примыкающую к ней CL. Каждая из VH и VL содержит определяющую комплементарность область (CDR).
Fc (также называемый «областью Fc») представляет собой карбокси концевую область константной области тяжелой цепи, он содержит СН2 и СН3 и представляет собой димер. Fc по настоящему изобретению может содержать встречающуюся в природе последовательность или он может содержать последовательность, полученную из встречающейся в природе последовательности путем мутации (называется «мутантным Fc»). В мультиспецифической молекуле и биспецифической молекуле по настоящему изобретению, Fc область предпочтительно представляет собой мутантный Fc, и более предпочтительно, комбинацию Fc областей, способную образовывать гетеродимер. Примером комбинации Fc областей является комбинация Fc (i) в первом полипептиде и Fc (ii) во втором полипептиде, описанная ниже. Комбинация не ограничивается этим при условии, что такая комбинация Fc областей способна к ассоциации (образованию гетеродимера).
Примеры мутантного Fc включают, но не ограничены ими, модифицированную Fc область, включенную в гетерополимер с улучшенной стабильностью (включая гетеродимерную Fc область), описанную в WO 2013/063702, Fc, включающую CD3 область иммуноглобулина, индуцированную из антитела IgG с «выступом» и «впадиной», содержащимися в гетерополимере, описанную в WO 1996/27011, Fc, включающую СН3 домен, включенный в гетеродимер, который становится электростатически выгодным при замене одной или нескольких аминокислот заряженными аминокислотами, описанную в WO 2009/089004, гетеродимерную Fc область, включенную в гетеродимер, включающий пространственную мутацию и/или мутацию pI (изоэлектрической точки), описанную в WO 2014/110601, и гетеродимерную Fc, включающую СН3 домен с модификацией для устранения или уменьшения связывания с белком А, описанную в WO 2010/151792.
Вариабельная область состоит из области с исключительной вариабельностью, называемой гипервариабельной областью (HVR), и относительно невариабельных областей, называемых каркасными областями (FR), разделенных HVR. Встречающиеся в природе вариабельные области тяжелой и легкой цепей содержат 4 FR, соединенных 3 гипервариабельными областями, где гипервариабельная область каждой цепи и гипервариабельная область других цепей находятся очень близко к ним, и такие области способствуют образованию антигенсвязывающего сайта антитела.
Известно, что тяжелая цепь и легкая цепь молекулы антитела содержат 3 определяющие комплементарность области (CDR). Определяющая комплементарность область также называется гипервариабельной областью, она присутствует в вариабельных областях тяжелой цепи и легкой цепи антитела, где вариабельность первичной структуры особенно высока, и, как правило, она разделена в 3 положениях в первичной структуре полипептидной цепи тяжелой цепи и легкой цепи. В настоящем изобретении, определяющие комплементарность области тяжелой цепи антитела обозначены как CDRH1, CDRH2 и CDRH3 от амино конца аминокислотной последовательности тяжелой цепи, и определяющие комплементарность области легкой цепи обозначены как CDRL1, CDRL2 и CDRL3 от амино конца аминокислотной последовательности легкой цепи. Эти области примыкают друг к другу на пространственной структуре и определяют специфичность к антигенам, с которыми они связываются.
В настоящем изобретении, положение и длину CDR определяют в соответствии с определением IMGT (Developmental and Comparative Immunology 27, 2003, 55-77).
FR представляет собой вариабельную область, отличную от CDR. Обычно вариабельная область включает 4 FR; т.е. FR1, FR2, FR3 и FR4.
CDR и FR, включенные в тяжелую и легкую цепи, представлены в порядке FRH1-CDRH1-FRH2-CDRH2-FRH3-CDRH3-FRH4 и FRL1-CDRL1-FRL2-CDRL2-FRL3-CDRL3-FRL4, соответственно, от амино конца к карбоксильному концу.
Положения CDR и FR можно определить в соответствии с различными определениями, хорошо известными в данной области, такими как определения Kabat, Chothia, AbM, Contact, в дополнение к IMGT.
В настоящем изобретении, термин «антигенсвязывающий фрагмент антитела» относится к частичному фрагменту антитела, обладающему активностью связывания с антигеном, который состоит из вариабельной области тяжелой цепи и вариабельной области легкой цепи. Примеры «антигенсвязывающего фрагмента антитела» включают, но не ограничены ими, антигенсвязывающие фрагменты, такие как Fab, F(ab')2, scFv, Fab', Fv и однодоменное антитело (sdAb). Такой антигенсвязывающий фрагмент антитела может быть получен путем обработки полноразмерной молекулы белка антитела ферментом, таким как папаин или пепсин, или он может представлять собой рекомбинантный белок, продуцируемый в адекватной клетке-хозяине с использованием рекомбинантного гена. В настоящем изобретении, термин «связывающий фрагмент антитела» является синонимом термина «антигенсвязывающий фрагмент антитела».
В настоящем изобретении, «сайт», с которым связывается антитело; т.е. «сайт», который распознается антителом, представляет собой частичный пептид или частичную структуру более высокого порядка антигена, с которым связывается антитело или который распознается антителом.
В настоящем изобретении, такой сайт называется эпитопом или сайтом связывания антитела. В настоящем изобретении, «мутантное антитело» относится к полипептиду, имеющему аминокислотную последовательность, полученную из аминокислотной последовательности исходного антитела путем замены, делеции или добавления («добавление» включает «вставку») (далее совместно именуемые как «мутация») аминокислот и связывание с HLA/NY-ESO по настоящему изобретению. Количество мутантных аминокислот в таком мутантном антителе составляет 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 15, 20, 25, 30, 40 или 50. Такое мутантное антитело входит в объем термина «антитело» по настоящему изобретению.
В настоящем изобретении, термин «несколько» в «один или несколько» означает от 2 до 10.
Используемый в настоящем документе термин «молекула» означает молекулу, содержащую антитело или антигенсвязывающий фрагмент антитела, описанные выше. Кроме того, термин «молекула» охватывает мультиспецифическую молекулу, образованную из антитела или множества производных от него антигенсвязывающих фрагментов.
Термин «мультиспецифическая молекула» конкретно не ограничен при условии, что такая молекула способна связываться с множеством различных эпитопов на молекуле и/или с разными эпитопами на двух или более молекулах. Мультиспецифическая молекула включает антитело, содержащее вариабельную область тяжелой цепи (VH) и вариабельную область легкой цепи (VL). Примеры такой мультиспецифической молекулы включают, но не ограничены ими, молекулу полноразмерного антитела, содержащую два или несколько различных типов тяжелых цепей и легких цепей; т.е. мультиспецифическая молекула IgG типа и молекула, содержащая антигенсвязывающий фрагмент, состоящий из двух или нескольких типов VL и VH; т.е. молекула, полученная из комбинации Fab, Fab', Fv, scFv, sdAb или подобных, такая как тандемный scFv, диатело, одноцепочечное диатело или триатитело. Кроме того, молекула, полученная генетическим или химическим лигированием белка без иммуноглобулинового скелета и способная связываться с антигеном с антигенсвязывающим фрагментом, входит в объем мультиспецифической молекулы. В настоящем изобретении, такая мультиспецифическая молекула может называться «мультиспецифическим антителом», за исключением случая, когда молекула не содержит антитело или его антигенсвязывающий фрагмент, по необходимости.
Анти-HLA/NY-ESO антитело по настоящему изобретению, антигенсвязывающий фрагмент такого антитела или молекула по настоящему изобретению обладают, например, биологической активностью и физико-химическими свойствами (которые также могут быть обозначены как физические свойства). Их конкретные примеры включают различную биологическую активность, такую как цитотоксичность, ADCC активность или противоопухолевую активность (описанную ниже), и физические свойства, такие как антиген- или эпитопсвязывающая активность, стабильность при производстве или хранении и термостабильность.
В настоящем изобретении, «гибридизацию в жестких условиях» проводят в растворе, содержащем 5× SSC, при 65°C, в водном растворе, содержащем 2× SSC и 0,1% SDS, при 65°C в течение 20 минут, в водном растворе, содержащем 0,5×SSC и 0,1% SDS при 65°C в течение 20 минут, или в водном растворе, содержащем 0,2×SSC и 0,1% SDS, при 65°C в течение 20 минут в условиях промывки или эквивалентных им условиях. SSC представляет собой водный раствор 150 мМ NaCl и 15 мМ цитрата натрия, и n× SSC представляет собой SSC n-кратной концентрации.
В настоящем изобретении, «повреждение клетки» представляет собой любую форму патологического изменения, произошедшего в клетке. В дополнение к внешнему повреждению, термин «повреждение клетки» относится к любому повреждению, нанесенному клеточной структуре или функциям, такому как расщепление ДНК, образование димеров нуклеотидов, расщепление хромосом, повреждение митотического аппарата или снижение активности различных ферментов. В настоящем изобретении, «цитотоксичность» представляет собой индукцию повреждения клеток.
В настоящем изобретении, термин «антителозависимая клеточная цитотоксичность (ADCC)» относится к активности NK клеток в отношении повреждения клеток-мишеней, таких как опухолевые клетки, опосредованного антителом.
В настоящем изобретении, термин «цитотоксичность, перенаправляющая Т-клетки» относится к индукции цитотоксичности, опосредованной мультиспецифической молекулой, содержащей антитело против опухолевого антигена и анти-HLA/NY-ESO антитело. В частности, антитело против опухолевого антигена связывается с опухолевой клеткой-мишенью, анти-HLA/NY-ESO антитело связывается с Т-клеткой, расстояние между опухолевой клеткой-мишенью и Т-клеткой сокращается, и цитотоксичность индуцируется активацией T-клеток. Такая молекула может быть включена в фармацевтическую композицию.
2. Антиген
2-1. HLA/NY-ESO антиген
В настоящем изобретении, «HLA/NY-ESO» используется в том же смысле, что и «HLA/NY-ESO белок».
HLA/NY-ESO представляет собой тройной комплекс HLA-A2, β2-микроглобулина и NY-ESO пептида. HLA-A2 представляет собой аллель HLA, которая наиболее часто экспрессируется у представителей европеоидной расы. HLA образует тройной комплекс с β2-микроглобулином и пептидным фрагментом аутологичного белка в клеточном эндоплазматическом ретикулуме; комплекс представлен внеклеточно и распознается Т-клеточным рецептором (TCR) Т-клетки. Сообщается, что NY-ESO пептид (SLLMWITQC: SEQ ID NO:1, фиг. 8) представляет собой пептид, состоящий из 9 аминокислот в положениях 157-165 NY-ESO-1 и LAGE-1, который презентируется HLA-А2.
2-2. CD3 антиген
В настоящем изобретении, «CD3» используется в том же смысле, что и «CD3 белок».
CD3 экспрессируется на Т-клетке как часть мультимолекулярного Т-клеточного рецепторного комплекса, и представляет собой комплекс 5 типов полипептидных цепей γ, δ, ε, ζ и η (их молекулярные массы 25000-28000, 21000, 20000, 16000 и 22000, соответственно).
Примеры комплексов CD3 включают цепи γ, δ, ε, ζ и η, которые также называются субъединицами. Когда анти-CD3 антитело связывается с Т-клеткой, повреждение клетки индуцируется активацией Т-клетки. Многие анти-CD3 антитела связываются с CD3 ε.
Нуклеотидная последовательность кДНК, кодирующая CD3 ε человека, зарегистрирована под номером доступа: NM_000733 (NM_000733.3) в NCBI/GenBank, и аминокислотная последовательность CD3 ε человека зарегистрирована под номером доступа: NP_000724 (NM_000724.1) в NCBI/GenPept. Нуклеотидная последовательность кДНК, кодирующая CD3 яванского макака, зарегистрирована под номером доступа: NM_001283615.1 в GenBank. Аминокислотная последовательность CD3 ε человека показана в SEQ ID NO:151 из перечня последовательностей (фиг. 147).
2-3. Получение антигена
Белки-антигены, используемые в настоящем изобретении, описаны выше; т.е., HLA/NY-ESO и CD3 (далее HLA/NY-ESO и CD3 вместе именуются антигенными белками) могут быть получены из тканей животных (включая биологические жидкости), клеток, полученных из тканей животных, или культурального продукта клеток посредством, например, очистки, выделения, рекомбинации генов, трансляции in vitro или химического синтеза.
кДНК антигенного белка может быть получена так называемым способом ПЦР, в котором проводят полимеразную цепную реакцию (далее именуемую «ПЦР») с использованием, в качестве матрицы, библиотеки кДНК органов, экспрессирующих мРНК антигенного белка, и праймеров, специфически амплифицирующих кДНК антигенного белка (Saiki, R. K., et al., Science, 1988, 239, 487-49).
кДНК антигенного белка включает полинуклеотид, гибридизующийся в жестких условиях с полинуклеотидом, состоящим из нуклеотидной последовательности, комплементарной нуклеотидной последовательности, кодирующей антигенный белок, экспрессируемый у человека или крысы, и кодирующий белок, обладающий биологической активностью, эквивалентной биологической активности антигенного белка.
Кроме того, кДНК антигенного белка включает вариант сплайсинга, транскрибируемый из локуса антигенного белка, экспрессированного у человека или крысы, и полинуклеотид, гибридизующийся с ним в жестких условиях и кодирующий белок, обладающий биологической активностью, эквивалентной активности антигенного белка.
Нуклеотидная последовательность, кодирующая белок, содержащий аминокислотную последовательность антигенного белка человека или крысы, или аминокислотную последовательность, полученную из такой аминокислотной последовательности путем замены, делеции или добавления одной или нескольких аминокислот, из которых удалена сигнальная последовательность, и обладающий биологической активностью, эквивалентной активности антигенного белка, включен в нуклеотидную последовательность гена антигенного белка.
Белок, содержащий аминокислотную последовательность, кодируемую вариантом сплайсинга, транскрибированным из генного локуса антигенного белка человека или крысы, или аминокислотную последовательность, полученную из такой аминокислотной последовательности путем замены, делеции или добавления одной или нескольких аминокислот, и обладающий биологической активностью, эквивалентной активности антигенного белка, включен в антигенный белок.
2-4. Специфичность связывания с антигенным белком
Анти-HLA/NY-ESO антитело по настоящему изобретению, его антигенсвязывающий фрагмент и подобные распознают HLA/NY-ESO. В частности, они связываются с HLA/NY-ESO антигеном. Присутствие HLA/NY-ESO у животных, отличных от человека, таких как мыши, крысы и яванские макаки, неизвестно.
Анти-CD3 антитело, включенное в мультиспецифическую молекулу по настоящему изобретению, его связывающие фрагменты и подобные распознают; т.е. связываются с CD3 антигеном. Такое анти-CD3 антитело и подобное, предпочтительно, связывается, например, с CD3 человека и CD3 обезьяны, и более предпочтительно, с CD3 человека и CD3 яванского макака. Напротив, такое предпочтительное анти-CD3 антитело не связывается с CD3 крысы и/или мыши.
Противоопухолевую активность мультиспецифической молекулы по настоящему изобретению можно оценить, например, путем (i) трансплантации раковых клеток человека или раковой ткани человека животным, отличным от человека, которым были трансплантированы лимфоциты периферической крови человека, предпочтительно крысам или мышам, и более предпочтительно, крысам или мышам с дефицитными эндогенными эффекторными функциями (например, крысам или мышам с иммунодефицитом) или (ii) трансплантации раковых клеток мыши, в которые были трансдуцированы гены HLA и NY-ESO, в животных, отличных от человека, с нокаутом гена CD3 человека, и предпочтительно, в крыс или мышей. Выполняя оценку с использованием таких иммунодефицитных животных или нокаутированных животных, можно проводить различные анализы, иммуногистохимические анализы и подобные с использованием тел мышей и/или крыс. Это предпочтительно для лекарственных средств, содержащих мультиспецифическую молекулу по настоящему изобретению, для доклинических разработок и других целей.
В настоящем изобретении «распознавание»; т.е. «связывание» представляет собой связывание, которое не является неспецифической адсорбцией. Распознает ли, т.е. связывает ли антитело или нет, можно оценить на основе, например, константы диссоциации (KD). Предпочтительное значение KD антитела и подобных по настоящему изобретению для HLA/NY-ESO или CD3 составляет 1×10-5 М или меньше, 5×10-6 М или меньше, 2×10-6 М или меньше или 1×10-6 М или меньше, их значение KD для HLA/NY-ESO предпочтительно составляет 5×10-7 М или меньше, 2×10-7 М или меньше, 1×10-7 М или меньше, 5×10-8 М или меньше, 2×10-8 М или меньше, 1×10-8 М или меньше, 5×10-9 М или меньше, или 2×10-9 М или меньше, и более предпочтительно, 1×10-9 М или меньше. Примеры анти-HLA/NY-ESO scFv по настоящему изобретению, обладающего превосходной антигенсвязывающей активностью, включают NYA-1143, NYA-2023, NYA-2143, NYA-2044, NYA-2045, NYA-2060, NYA-2061 и NYA-3061, и их значение KD для HLA/NY-ESO, таких как NYA-1143, NYA-2044, NYA-2045 и NYA-2143, составляет 1×10-9 М или меньше (например, пример 4).
В настоящем изобретении, связывание антиген-антитело можно анализировать или оценивать, например, с помощью системы анализа биомолекулярных взаимодействий, такой как SPR или BLI, ELISA или RIA. Связывание между антигеном и антителом, экспрессированным на клеточной поверхности, можно оценить, например, с помощью проточной цитометрии.
Способ анализа поверхностного плазмонного резонанса (SPR) выполняет кинетический анализ реакции для определения константы скорости ассоциации (значение Ka) и константы скорости диссоциации (значение Kd), и определяет константу диссоциации (значение KD), служащую показателем аффинности. Примеры аппаратов, используемых для SPR анализа, включают Biacore™ (GE Healthcare), ProteOn™ (BioRad), SPR-Navi™ (BioNavis), Spreeta™ (Texas Instruments), SPRi-PlexII™ (Horiba Ltd.) и Autolab SPR™ (Metrohm).
Способ биослойной интерферометрии (BLI) выполняет анализ взаимодействия между биомолекулами с использованием интерференции биослоя. Примером аппарата, используемого для анализа взаимодействия методом BLI, является система Octet (Pall ForteBio).
Способ твердофазного иммуноферментного анализа (ELISA) осуществляет обнаружение и количественное определение антигена или антитела-мишени, содержащихся в растворе образца, путем захвата антигена или антитела-мишени с использованием специфического антитела или антигена, соответственно, с использованием ферментативной реакции. Меченый ферментом антиген или антитело интегрируют в реакционную систему для определения ферментативной активности. Ферментативную активность определяют с использованием субстрата, демонстрирующего спектры поглощения, изменяющиеся в зависимости от реакции, и ферментативную активность определяют количественно на основе анализа абсорбции.
Cell-ELISA выполняет обнаружение и количественную оценку анализируемого вещества-мишени на поверхности клетки путем захвата анализируемого вещества-мишени вместе с клеткой с использованием ферментативной реакции.
Способ радиоиммуноанализа (RIA) заключается в мечении антитела радиоактивным веществом и определении радиоактивности антитела. Таким образом, антитело может быть определено количественно.
В способе проточной цитометрии, клетки диспергируют в жидкости, жидкости дают возможность течь узко, и каждую клетку подвергают оптическому анализу. Антителу, меченному флуоресцентным красителем, позволяют связываться с антигеном клеточной поверхности посредством реакции антиген-антитело, и интенсивность флуоресценции, испускаемой меченым антителом, связанным с клеткой, анализируют для количественной оценки антигенсвязывающей способности антитела.
Примеры анти-HLA/NY-ESO антител по настоящему изобретению, демонстрирующих превосходную антигенсвязывающую специфичность, включают анти-HLA/NY-ESO scFv, такие как NYA-0001, NYA-1143, NYA-1163, NYA-2023, NYA-2027, NYA-2035, NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060, NYA-2061, NYA-2143 и NYA-3061 (например, пример 6).
3. Антитело, специфически связывающееся с HLA/NY-ESO или его связывающим фрагментом.
3-1. Анти-HLA/NY-ESO или его связывающий фрагмент
Настоящее изобретение предлагает антитело, которое распознает и связывается с HLA/NY-ESO или его связывающим фрагментом.
Как описано выше, HLA/NY-ESO представляет собой комплекс, содержащий HLA-A2 и 9-mer пептид NY-ESO (SLLMWITQC: SEQ ID NO:1). NY-ESO пептид представляет собой пептид, полученный из NY-ESO-1 или LAGE-1, который представляет собой внутриклеточный белок и раковый антиген яичка. HLA/NY-ESO экспрессируется на поверхности раковых клеток.
Анти-HLA/NY-ESO антитело по настоящему изобретению и антигенсвязывающий фрагмент антитела (который в дальнейшем может называться «антитело и подобные по настоящему изобретению») могут быть моноклональными или поликлональными антителами. В настоящем изобретении, изотип изотипа моноклонального антитела конкретно не ограничен, и примеры включают IgG, такой как IgG1, IgG2, IgG3 и IgG4, IgM, IgA, такой как IgA1 и IgA2, IgD и Ig. Изотип и подкласс моноклонального антитела можно определить, например, способом Оухтерлони, ELISA или RIA. Примеры моноклональных антител по настоящему изобретению включают антитело, полученное из животного, отличного от человека (антитело животного, отличного от человека), антитело человека, химеризированное антитело (также называемое «химерным антителом») и гуманизированное антитело, где предпочтительным является антитело человека. Антитело по настоящему изобретению включает мутант антитела («мутантное антитело», описанное ниже), и, например, антитело человека включает мутантное антитело человека.
Примеры антител животных, отличных от человека, включают антитела, полученные от позвоночных, таких как млекопитающие и птицы. Примеры антител, происходящих от млекопитающих, включают антитела, полученные от грызунов, такие как антитело мыши и антитело крысы. Примером полученного от птиц антитела является антитело курицы.
Примеры химеризованных антител включают, но не ограничены ими, антитела, содержащие вариабельную область, полученную из антитела животного, отличного от человека, связанного с константной областью, полученной из антитела человека (иммуноглобулина человека).
Примеры гуманизированных антител включают, но не ограничены ими, гуманизированное антитело, полученное путем трансплантации CDR в вариабельной области антитела животного, отличного от человека, в антитело человека (вариабельную область иммуноглобулина человека), гуманизированное антитело, полученное путем трансплантации, в дополнение к CDR, части последовательности каркасной области антитела животного, отличного от человека, в антитело человека, и гуманизированное антитело, полученное заменой 1 или нескольких аминокислот, полученных из антитела животного, отличного от человека, аминокислотами полученными из антитела человека.
Антитело может быть получено различными известными методами. Например, антитело можно получить способом, включающим использование гибридомы, клеточно-опосредованного иммунитета или генетической рекомбинации. Кроме того, можно получить антитело человека, полученное с помощью фагового дисплея, выбранное из библиотеки антител человека. Например, в способе фагового дисплея, вариабельная область антитела человека может быть экспрессирована в виде scFv на поверхности фага, после чего может быть выбран антигенсвязывающий фаг. Ген фага, выбранного при его связывании с антигеном, может быть проанализирован, чтобы можно было определить последовательность ДНК, кодирующую вариабельную область антигена человека, связывающуюся с антигеном. Если выяснена последовательность ДНК антигенсвязывающего scFv, можно получить вектор экспрессии, содержащий такую последовательность, ввести в подходящую клетку-хозяина и экспрессировать в ней. Таким образом, можно получить антитело человека (WO 92/01047, WO 92/20791, WO 93/06213, WO 93/11236, WO 93/19172, WO 95/01438, WO 95/15388, Annu. Rev. Immunol., 1994, 12, 433-455).
Полученное таким образом антитело с высокой активностью можно использовать в качестве ведущего антитела, и ген, кодирующий такое ведущее антитело, можно мутировать, чтобы можно было получить мутант с более высокой активностью («мутантное антитело», описанное ниже).
Предпочтительной комбинацией CDRH1-CDRH3, включенной в тяжелую цепь анти-HLA/NY-ESO антитела по настоящему изобретению, или его антигенсвязывающего фрагмента, является CDRH1, состоящая из аминокислотной последовательности SEQ ID NO:54 (фиг. 61), CDRH2, состоящая из аминокислотной последовательности SEQ ID NO:55 (фиг. 61), и CDRH3, состоящая из аминокислотной последовательности SEQ ID NO:56 (фиг. 61), или аминокислотная последовательность, полученная из аминокислотной последовательности SEQ ID NO:56 (фиг. 61), в которой аминокислота 6 представляет собой N (Asn). Более предпочтительная комбинация CDRH1-CDRH3 включена в вариабельную область тяжелой цепи NYA-0001, состоящую из аминокислотной последовательности SEQ ID NO:6 (фиг. 13), вариабельную область тяжелой цепи NYA-0082, состоящую из аминокислотной последовательности SEQ ID NO:18 (фиг. 25), вариабельную область тяжелой цепи NYA-2023, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:27 (фиг. 34), вариабельную область тяжелой цепи NYA-2027, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:28 (фиг. 35), вариабельную область тяжелой цепи NYA-1143, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:29 (фиг. 36), вариабельную область тяжелой цепи NYA-1163, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:26 (фиг. 33), вариабельную область тяжелой цепи NYA-2023, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:27 (фиг. 34), вариабельную область тяжелой цепи NYA-2027, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:28 (фиг. 35), вариабельную область тяжелой цепи NYA-2035 состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:36 (фиг. 43), вариабельную область тяжелой цепи NYA-2044, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:47 (фиг. 54), вариабельную область тяжелой цепи NYA-2045, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:48 (фиг. 55), вариабельную область тяжелой цепи NYA-2047, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:50 (фиг. 57), вариабельную область тяжелой цепи NYA-2048, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:51 (фиг. 58), вариабельную область тяжелой цепи NYA-2060, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:52 (фиг. 59), вариабельную область тяжелой цепи NYA-2061 состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:53 (фиг. 60), или вариабельную области тяжелой цепи NYA-2143, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:30 (фиг. 37).
Еще один пример комбинации CDRH1-CDRH3 включен в вариабельную область тяжелой цепи NYA-3061, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:156 (фиг. 152).
Предпочтительная комбинация CDRL1-CDRL3, включенная в легкую цепь анти-HLA/NY-ESO антитела по настоящему изобретению, или его антигенсвязывающего фрагмента, представляет собой CDRL1, состоящую из аминокислотной последовательности SEQ ID NO:57. (фиг. 61) или аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:57 (фиг. 61), в которой аминокислота 7 представляет собой W (Trp) или аминокислота 8 представляет собой K (Lys), CDRL2 состоящую из аминокислотной последовательности SEQ ID NO:58 (фиг. 61), и CDRL3, состоящую из аминокислотной последовательности SEQ ID NO:59 (фиг. 61), или аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:59 (фиг. 61), в которой аминокислота 2 представляет собой A (Ala) или S (Ser).
Более предпочтительная комбинация CDRL1-CDRL3 включена в вариабельную область легкой цепи NYA-0001, состоящую из аминокислотной последовательности SEQ ID NO:8 (фиг. 15), вариабельную область легкой цепи NYA-0082, состоящую из аминокислотной последовательности SEQ ID NO:20 (фиг. 27), вариабельную область легкой цепи NYA-1143, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:29 (фиг. 36), вариабельную область легкой цепи NYA-1163, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:26 (фиг. 33), вариабельную область легкой цепи NYA-2023, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:27 (фиг. 34), вариабельную области легкой цепи NYA-2027, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:28 (фиг. 35), вариабельную область легкой цепи NYA-2035, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:36 (фиг. 43), вариабельную область легкой цепи NYA-2044, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:47 (фиг. 54), вариабельную область легкой цепи NYA-2045, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:48 (фиг. 55), вариабельную область легкой цепи NYA-2047, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:50 (фиг. 57), вариабельную область легкой цепи NYA-2048, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:51 (фиг. 58), вариабельную область легкой цепи NYA-2060, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:52 (фиг. 59), вариабельную область легкой цепи NYA-2061, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:53 (фиг. 60), или вариабельную область легкой цепи NYA-2143, состоящую из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:30 (фиг. 37).
Еще один пример комбинации CDRL1-CDRL3 включен в вариабельную область легкой цепи NYA-3061, состоящую из аминокислот 161-271 аминокислотной последовательности SEQ ID NO:156 (фиг. 152).
Предпочтительная комбинация CDRH1-CDRH3 в тяжелой цепи и CDRL1-CDRL3 в легкой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента представляет собой комбинацию CDRH1, состоящей из аминокислотной последовательности SEQ ID NO:54 (фиг. 61), CDRH2, состоящей из аминокислотной последовательности SEQ ID NO:55 (фиг. 61), и CDRH3, состоящей из аминокислотной последовательности SEQ ID NO:56 (фиг. 61) или аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:56 (фиг. 61), в которой аминокислота 6 представляет собой N (Asn), и комбинацию CDRL1, состоящей из аминокислотной последовательности SEQ ID NO:57 (фиг. 61), или аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:57 (фиг. 61), в которой аминокислота 7 представляет собой N (Asn) и/или аминокислота 8 представляет собой K (Lys), CDRL2, состоящей из аминокислотной последовательности SEQ ID NO:58 (фиг. 61), и CDRL3, состоящей из аминокислотной последовательности, как показана в SEQ ID NO:59 (фиг. 61) или аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:59, в которой аминокислота 2 представляет собой A (Ala) или S (Ser). Более предпочтительная комбинация CDRH1-CDRH3 и CDRL1-CDRL3 включена в вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-0001, состоящую из аминокислотных последовательностей, показанных в SEQ ID NO:6 и SEQ ID NO:8, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-1143, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:29 (фиг. 36), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-1163, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:26 (фиг. 33), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2023, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:27 (фиг. 34), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2027, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности показанной в SEQ ID NO:28 (фиг. 35), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2035, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:36 (фиг. 43), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2044, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:47 (фиг. 54), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2045, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:48 (фиг. 55), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2047, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:50 (фиг. 57), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2048, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:51 (фиг. 58), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2060, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:52 (фиг. 59), вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2061, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:53 (фиг. 60), или вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2143, состоящую из аминокислот 21-140 и аминокислот 156-266 аминокислотной последовательности SEQ ID NO:30 (фиг. 37).
Еще один пример комбинации CDRH1-CDRH3 и CDRL1-CDRL3 включен в вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-3061, состоящую из аминокислот 21-140 и аминокислот 161-271 аминокислотной последовательности SEQ ID NO:156 (фиг. 152).
Предпочтительные примеры вариабельной области тяжелой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента включают CDR тяжелой цепи, описанные выше, и вариабельные области, включающие такие CDR тяжелой цепи в соответствующей комбинации. Более предпочтительные примеры включают вариабельную область тяжелой цепи NYA-0001, вариабельную область тяжелой цепи NYA-0082, вариабельную область тяжелой цепи NYA-1143, вариабельную область тяжелой цепи NYA-1163, вариабельную область тяжелой цепи NYA-2023, вариабельная область тяжелой цепи NYA-2027, вариабельную область тяжелой цепи NYA-2035, вариабельную область тяжелой цепи NYA-2044, вариабельную область тяжелой цепи NYA-2045, вариабельную область тяжелой цепи NYA-2047, вариабельную область тяжелой цепи NYA-2048 вариабельную область тяжелой цепи NYA-2060, вариабельную область тяжелой цепи NYA-2061, вариабельную область тяжелой цепи NYA-2143 и вариабельную область тяжелой цепи NYA-3061. Аминокислотная последовательность каждой вариабельной области тяжелой цепи является такой, как описано выше.
Предпочтительные примеры вариабельной области легкой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента включают CDR легкой цепи, описанные выше, и вариабельные области, включающие такие CDR легкой цепи в соответствующей комбинации. Более предпочтительные примеры включают вариабельную область легкой цепи NYA-0001, вариабельную область легкой цепи NYA-0082, вариабельную область легкой цепи NYA-1143, вариабельную область легкой цепи NYA-1163, вариабельную область легкой цепи NYA-2023, вариабельную область легкой цепи NYA-2027, вариабельную область легкой цепи NYA-2035, вариабельную область легкой цепи NYA-2044, вариабельную область легкой цепи NYA-2045, вариабельную область легкой цепи NYA-2047, вариабельную область легкой цепи NYA-2048, вариабельную область легкой цепи NYA-2060, вариабельную область легкой цепи NYA-2061, вариабельную область легкой цепи NYA-2143 и вариабельную область легкой цепи NYA-3061. Аминокислотная последовательность каждой вариабельной области легкой цепи является такой, как описано выше.
Предпочтительные примеры вариабельной области тяжелой цепи и вариабельной области легкой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента включают CDR тяжелой и легкой цепи, описанные выше, или CDR, включающие такие CDR в адекватной комбинации. Более предпочтительные примеры включают вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-0001, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-0082, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-1143, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-1163, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2023, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2027, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2035 вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2044, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2045, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2047, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2048, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2060, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2061, вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-2143, и вариабельную область тяжелой цепи и вариабельную область легкой цепи NYA-3061.
Предпочтительные примеры тяжелой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента включают тяжелые цепи, включая предпочтительные или более предпочтительные вариабельные области тяжелой цепи, описанные выше. Более предпочтительные примеры включают тяжелую цепь NYA-0001, тяжелую цепь NYA-0082, тяжелую цепь NYA-1143, тяжелую цепь NYA-1163, тяжелую цепь NYA-2023, тяжелую цепь NYA-2027, тяжелую цепь NYA-2035, тяжелую цепь NYA-2044, тяжелую цепь NYA-2045, тяжелую цепь NYA-2047, тяжелую цепь NYA-2048, тяжелую цепь NYA-2060, тяжелую цепь NYA-2061, тяжелую цепь NYA-2143 цепь и тяжелую цепь NYA-3061.
Предпочтительные примеры легкой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента включают легкие цепи, включая предпочтительные или более предпочтительные вариабельные области легкой цепи, описанные выше. Более предпочтительные примеры включают легкую цепь NYA-0001, легкую цепь NYA-0082, легкую цепь NYA-1143, легкую цепь NYA-1163, легкую цепь NYA-2023, легкую цепь NYA-2027, легкую цепь NYA-2035, легкую цепь NYA-2044, легкую цепь NYA-2045, легкую цепь NYA-2047, легкую цепь NYA-2048, легкую цепь NYA-2060, легкую цепь NYA-2061, легкую цепь NYA-2143 цепь и легкую цепь NYA-3061.
Предпочтительные примеры тяжелой цепи и легкой цепи анти-HLA/NY-ESO антитела по настоящему изобретению или его антигенсвязывающего фрагмента включают тяжелые цепи и легкие цепи, включая предпочтительные или более предпочтительные вариабельные области тяжелой цепи и вариабельные области легкой цепи, описанные выше. Более предпочтительные примеры включают тяжелую цепь и легкую цепь NYA-0001, тяжелую цепь и легкую цепь NYA-1143, тяжелую цепь и легкую цепь NYA-1163, тяжелую цепь и легкую цепь NYA-2023, тяжелую цепь и легкую цепь NYA-2027, тяжелую цепь и легкую цепь NYA-2035, тяжелую цепь и легкую цепь NYA-2044, тяжелую цепь и легкую цепь NYA-2045, тяжелую цепь и легкую цепь NYA-2047, тяжелую цепь и легкую цепь NYA-2048, тяжелую цепь и легкую цепь NYA-2060, тяжелую цепь и легкую цепь NYA-2061, тяжелую цепь и легкую цепь NYA-2143 и тяжелую цепь и легкую цепь NYA-3061.
Антигенсвязывающий фрагмент антитела представляет собой фрагмент, который сохраняет, по меньшей мере, антигенсвязывающую способность вне функций исходного антитела или его модифицированного продукта. Примеры функций антитела включают, как правило, антигенсвязывающую активность, активность регуляции антигенной активности, антителозависимую клеточную цитотоксичность и комплемент-зависимую цитотоксичность. Примеры функций антитела и подобных по настоящему изобретению и мультиспецифических молекул, содержащих антитело и подобные по настоящему изобретению, включают перенаправление Т-клеток, активацию Т-клеток и противораковую цитотоксичность, вызванную активацией Т-клеток.
Антигенсвязывающий фрагмент антитела конкретно не ограничен при условии, что такой фрагмент получен из антитела, которое сохраняет, по меньшей мере, антигенсвязывающую способность вне функций исходного антитела. Их примеры включают, но не ограничены ими, Fab, Fab', F(ab')2, Fv, одноцепочечный Fv (scFv), содержащий Fv тяжелой цепи, лигированный с Fv легкой цепи посредством соответствующего линкера, и однодоменное антитело (sdAb). Молекула, содержащая область, отличный от антигенсвязывающего фрагмента антитела по настоящему изобретению, как в случае scFv, содержащего линкерную часть, входит в объем антигенсвязывающего фрагмента антитела по настоящему изобретению.
Молекула белка антитела, лишенная, по меньшей мере, одной или нескольких аминокислот с амино конца и/или карбоксильного конца и сохраняющая некоторые функции антитела, входит в объем антигенсвязывающего фрагмента антитела. Модифицированный антигенсвязывающий фрагмент антитела входит в объем антитела по настоящему изобретению, его антигенсвязывающего фрагмента или модифицированного антитела или фрагмента (описано ниже).
Вариантом осуществления антитела по настоящему изобретению или его антигенсвязывающего фрагмента является scFv. scFv получают лигированием вариабельной области тяжелой цепи антитела с вариабельной областью его легкой цепи с помощью полипептидного линкера (Pluckthun A., The Pharmacology of Monoclonal Antibodies 113, Rosenburg and Moore (ed.), Springer Verlag, New York, 269-315, 1994, Nature Biotechnology, 2005, 23, 1126-1136). Кроме того, в качестве биспецифической молекулы можно использовать тандемный scFv, полученный путем соединения двух конструкций scFv с полипептидным линкером. Кроме того, в качестве мультиспецифической молекулы можно использовать триатело или подобное, содержащее 3 или более конструкций scFv.
Предпочтительные примеры HLA/NY-ESO-специфического scFv (также называемого «анти-HLA/NY-ESO scFv») включают scFv, содержащий CDRH1-CDRH3 и CDRL1-CDRL3, описанные выше, их более предпочтительные примеры включают scFv, содержащий вариабельную область тяжелой цепи и вариабельную область легкой цепи, и их дополнительные предпочтительные примеры включают NYA-0001 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:70 (фиг. 71)), NYA-0082 (содержащую аминокислотную последовательность, показанную в SEQ ID NO:18 (фиг. 25), и аминокислотную последовательность, показанную в SEQ ID NO:20 (фиг. 27)), NYA-1143 (аминокислоты 21-266 аминокислотной последовательности показанной в SEQ ID NO:29 (фиг. 36)), NYA-1163 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:26 (фиг. 33)), NYA-2023 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:27 (фиг. 34)), NYA-2027 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:28 (фиг. 35), NYA-2035 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:36 (фиг. 43)), NYA-2044 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:47 (фиг. 54)), NYA-2045 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:48 (фиг. 55)), NYA-2047 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:50 (фиг. 57)), NYA-2048 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:51 (фиг. 58)), NYA-2060 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:52 (фиг. 59)), NYA-2061 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:53 (фиг. 60)), и NYA-2143 (аминокислоты 21-266 аминокислотной последовательности SEQ ID NO:30 (фиг. 37)). Другим примером является NYA-3061 (аминокислоты 21-271 аминокислотной последовательности SEQ ID NO:156 (фиг. 152)).
Предпочтительным вариантом осуществления анти-HLA/NY-ESO scFv является scFv, содержащий метку FLAG-His, слитую с его карбоксильным концом (может называться просто «аддукт метки»). Примеры предпочтительных аддуктов метки включают аддукт метки NYA-0001 (аминокислоты 20-292 SEQ ID NO:70 (фиг. 71)), аддукт метки NYA-1143 (аминокислоты 20-292 SEQ ID NO:29 (фиг. 36)), аддукт метки NYA-1163 (аминокислоты 20-292 SEQ ID NO:26 (фиг. 33)), аддукт метки NYA-2023 (аминокислоты 20-292 SEQ ID NO:27 (фиг. 34) ), аддукт метки NYA-2027 (аминокислоты 20-292 SEQ ID NO:28 (фиг. 35)), аддукт метки NYA-2035 (аминокислоты 20-292 SEQ ID NO:36 (фиг. 43)), аддукт метки NYA-2044 (аминокислоты 20-292 SEQ ID NO:47 (фиг. 54)), аддукт метки NYA-2045 (аминокислоты 20-292 SEQ ID NO:48 (фиг. 55)), аддукт метки NYA-2047 (аминокислоты 20-292 SEQ ID NO:50 (фиг. 57)), аддукт метки NYA-2048 (аминокислоты 20-292 SEQ ID NO:51 (фиг. 58)), аддукт метки NYA-2060 (аминокислоты 20-292 SEQ ID NO:52 (фиг. 59)), аддукт метки NYA-2061 (аминокислоты 20-292 SEQ ID NO:53 (фиг. 60)) и аддукт метки NYA-2143 (аминокислоты 20-292 SEQ ID NO:30 (фиг. 37)).
Из них, NYA-2023 и его аддукт метки, NYA-2047 и его аддукт метки, NYA-2048 и его аддукт метки, NYA-2060 и его аддукт метки, и NYA-2061 и его аддукт метки демонстрируют превосходную биологическую активность, физические свойства и подобные в биспецифических молекулах с добавлением Fc, и поэтому являются более предпочтительными.
Когда анти-HLA/NY-ESO scFv и его аддукт метки должны быть экспрессированы в клетке-хозяине, сигнальный пептид может быть добавлен к его амино концу. Примеры аминокислотных последовательностей аддуктов метки анти-HLA/NY-ESO scFv с добавлением сигнального пептида включают аминокислотные последовательности, показанные в SEQ ID NO:70, 29, 26-28, 36, 47, 48, 50-53, и 30 (фиг. 71, 36, 33-35, 43, 54, 55, 57-60 и 37).
scFv можно получить методом фагового дисплея, при котором вариабельная область антитела экспрессируется в виде одноцепочечного антитела (scFv) на поверхности фага, и затем выбирается антигенсвязывающий фаг (Nature Biotechnology, 2005, 23, (9), pp. 1105-1116). Ген фага, выбранного при его связывании с антигеном, может быть проанализирован, чтобы можно было определить последовательность ДНК, кодирующую вариабельную область человеческого антигена, связывающуюся с антигеном. Если выяснена последовательность ДНК антигенсвязывающего scFv, можно получить вектор экспрессии, содержащий такую последовательность, ввести в подходящую клетку-хозяина и экспрессировать в ней. Таким образом, можно получить антитело человека (WO 92/01047, WO 92/20791, WO 93/06213, WO 93/11236, WO 93/19172, WO 95/01438, WO 95/15388, Annu. Rev. Immunol., 1994, 12, pp. 433-455, Nature Biotechnology, 2005, 23 (9), pp. 1105-1116).
Антитело по настоящему изобретению может содержать одну вариабельную область тяжелой цепи и может не содержать последовательность легкой цепи. Такое антитело называется однодоменным антителом (sdAb) или нанотелом и сохраняет антигенсвязывающую способность (Muyldemans S. et al., Protein Eng., 1994, 7 (9), 1129-35, Hamers-Casterman C. et al., Nature, 1993, 363 (6428), 446-448). Такие антитела входят в объем антигенсвязывающего фрагмента антитела по настоящему изобретению.
Настоящее изобретение также включает одноцепочечный иммуноглобулин, содержащий полноразмерную последовательность тяжелой цепи, лигированную с последовательностью легкой цепи антитела с помощью соответствующего линкера (Lee, H-S, et al., Molecular Immunology, 1999, 36, 61-71; Shirrmann, T. et al., mAbs, 2010, 2 (1), 1-4). Такой одноцепочечный иммуноглобулин может быть димеризован, так что он может сохранять структуру и активность, подобные таковым у антитела, которое по своей природе является тетрамером. Анти-HLA/NY-ESO антитело по настоящему изобретению может представлять собой одноцепочечный иммуноглобулин.
В scFv по настоящему изобретению вариабельная область тяжелой цепи может образовывать дисульфидную связь с вариабельной областью легкой цепи.
Анти-HLA/NY-ESO антитело по настоящему изобретению может состоять из компонентов, полученных из множества различных антител, при условии, что оно связывается с HLA/NY-ESO. Его примеры включают таковые, полученные замещением тяжелых цепей и/или легких цепей среди множества различных антител, замещением полноразмерных последовательностей тяжелых цепей и/или легких цепей, замещением в результате селективной замены любой вариабельной области или константной области, и таковые, полученные в результате селективной замены части или всей CDR. В химеризованном антителе, вариабельная область тяжелой цепи и вариабельная область легкой цепи могут быть получены из другого анти-HLA/NY-ESO антитела по настоящему изобретению. В вариабельных областях тяжелой цепи и легкой цепи гуманизированного антитела, CDR1 тяжелой цепи - CDR3 тяжелой цепи и CDR1 легкой цепи - CDR3 легкой цепи могут быть получены из двух или нескольких типов анти-HLA/NY-ESO антител в соответствии с настоящим изобретением. В вариабельных областях тяжелой цепи и легкой цепи антитела человека, комбинация CDR1 тяжелой цепи - CDR3 тяжелой цепи и CDR1 легкой цепи - CDR3 легкой цепи может быть получена из двух или нескольких типов анти-HLA/NY-ESO антител по настоящему изобретению.
Анти-HLA/NY-ESO антитело по настоящему изобретению включает антитело, которое содержит аминокислотную последовательность, кодируемую нуклеотидной последовательностью, включенной в полинуклеотид, гибридизующийся в жестких условиях с комплементарной цепью полинуклеотида, содержащего нуклеотидную последовательность, кодирующую аминокислотную последовательность, включенную в анти-HLA/NY-ESO антитело по настоящему изобретению и связывающуюся с HLA/NY-ESO.
Это может быть антитело, содержащее аминокислотную последовательность, имеющую, по меньшей мере, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичность с аминокислотной последовательностью, включенной в вариабельную область тяжелой цепи анти-HLA/NY-ESO антитела по настоящему изобретению (предпочтительно аминокислотной последовательностью аминокислот 21-140 аминокислотной последовательности SEQ ID NO:27, аминокислотной последовательностью, показанной в SEQ ID NO:38, аминокислотной последовательностью, показанной в SEQ ID NO:39, или аминокислотной последовательностью аминокислот 21-140 из аминокислотной последовательности SEQ ID NO:160, SEQ ID NO:197 или SEQ ID NO:198) и/или аминокислотной последовательностью, включенной в вариабельную область ее легкой цепи (предпочтительно, аминокислотной последовательностью аминокислот 156-266 аминокислотной последовательности SEQ ID NO:27, аминокислотной последовательностью аминокислот 156-266 из аминокислотной последовательности SEQ ID NO:52, аминокислотной последовательностью, показанной в SEQ ID NO:40, аминокислотной последовательностью аминокислот 161-271 аминокислотной последовательности SEQ ID NO:160, или аминокислотной последовательностью аминокислот 156-266 аминокислотной последовательности SEQ ID NO:197 или SEQ ID NO:198) или его антигенсвязывающий фрагмент.
Когда положение и длину вариабельной области легкой цепи определяют в соответствии с определением, которое отличается от IMGT (например, Kabat, Chothia, AbM, Contact), одна или несколько аминокислот, таких как аргинин или глицин, могут дополнительно включаться в карбоксильный конец аминокислотной последовательности вариабельной области легкой цепи, определенной в соответствии с определением IMGT. Такое антитело или его связывающий фрагмент, содержащий такую вариабельную область легкой цепи, входит в объем антитела по настоящему изобретению или его связывающего фрагмента.
Антитело и подобные по настоящему изобретению можно получить путем введения мутации в связывающий фрагмент анти-HLA/NY-ESO антитела по настоящему изобретению и оптимизации способности связываться с HLA/NY-ESO, и, в частности, с HLA/NY-ESO человека и/или яванского макака. Конкретные примеры способов введения мутации включают случайный мутагенез с использованием ПЦР с ошибками, сайт-направленное введение аминокислот с использованием библиотек NHK, сайт-направленный мутагенез с использованием структурной информации и любую их комбинацию.
3-2. Мутант анти-HLA/NY-ESO антитела (мутантное антитело)
Мутантное антитело анти-HLA/NY-ESO антитела по настоящему изобретению может быть, предпочтительно, предложено с, например, пониженной восприимчивостью к деградации или окислению белка, сохраненной или улучшенной биологической активностью или функциями, подавлением снижения или изменения такой активности или функций, улучшенной или регулируемой антигенсвязывающей способностью, физико-химическими свойствами или функциональными свойствами. Известно, что белок изменяет свои функции или активность при чередовании конкретной боковой цепи аминокислоты на его поверхности, и примеры включают дезамидирование боковой цепи аспарагина и изомеризацию боковой цепи аспарагиновой кислоты. Антитело, полученное в результате замены конкретной аминокислоты другой аминокислотой с целью предотвращения изменения боковой цепи аминокислоты, входит в объем мутантного антитела по настоящему изобретению.
Примером мутантного антитела по настоящему изобретению является антитело, содержащее аминокислотную последовательность, полученную из аминокислотной последовательности исходного антитела путем консервативной аминокислотной замены. Консервативная аминокислотная замена происходит внутри аминокислотной группы, связанной с боковой цепью аминокислоты.
Предпочтительными аминокислотными группами являются следующие: кислотная группа: аспарагиновая кислота и глутаминовая кислота; основная группа: лизин, аргинин и гистидин; не полярная группа: аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин и триптофан; и незаряженное полярное семейство: глицин, аспарагин, глутамин, цистеин, серин, треонин и тирозин. Другими предпочтительными аминокислотными группами являются следующие: алифатическая гидроксильная группа: серин и треонин; амидосодержащая группа: аспарагин и глутамин; алифатическая группа: аланин, валин, лейцин и изолейцин; и ароматическая группа: фенилаланин, триптофан и тирозин. В таком мутантном антителе аминокислотную замену предпочтительно проводят, воздерживаясь от снижения антигенсвязывающей активности исходного антитела.
Анти-HLA/NY-ESO антитело по настоящему изобретению, его антигенсвязывающий фрагмент, его мутант (мутантное антитело или его антигенсвязывающий фрагмент) или молекула по настоящему изобретению включает: мутантное антитело, содержащее аминокислотную последовательность, полученную из аминокислотной последовательности антитела по настоящему изобретению, такого как NYA-2023, путем консервативной аминокислотной замены и/или другой мутации и связывания с HLA/NY-ESO, его антигенсвязывающий фрагмент, и молекулы, содержащие их; химеризированное антитело, гуманизированное антитело или антитело человека, содержащее CDR аминокислотной последовательности, полученной из аминокислотной последовательности любой из CDRH1-CDRH3 и CDRL1-CDRL3, полученной из антитела по настоящему изобретению, включая NYA-2023, путем консервативной аминокислотной замены и/или другой мутации и связывания с HLA/NY-ESO, его антигенсвязывающим фрагментом или молекулой, содержащей его.
3-3. Связывающий фрагмент анти-HLA/NY-ESO антитела
В одном из аспектов настоящего изобретения, предложен антигенсвязывающий фрагмент анти-HLA/NY-ESO антитела по настоящему изобретению (далее он упоминается просто как «связывающий фрагмент»). Связывающие фрагменты анти-HLA/NY-ESO антитела по настоящему изобретению включают связывающие фрагменты химеризованного антитела, гуманизированного антитела и антитела человека. Связывающий фрагмент антитела представляет собой фрагмент, который сохраняет, по меньшей мере, антигенсвязывающую способность функций исходного антитела или его модифицированного продукта. В общем, примеры таких функций антител включают антигенсвязывающую активность, активность регуляции антигенной активности (например, агонистическую активность), активность интернализации антигена в клетку и активность ингибирования или стимуляции взаимодействий между антигеном и веществом, взаимодействующим с ним.
Связывающий фрагмент антитела конкретно не ограничен при условии, что он представляет собой фрагмент фрагмента, сохраняющего, по меньшей мере, антигенсвязывающую способность активности исходного антитела. Примеры такого связывающего фрагмента антитела включают, но не ограничены ими, Fab, Fab', F(ab')2, одноцепочечные Fab (scFab), в которых карбоксильный конец легкой цепи Fab лигирован с амино концом тяжелой цепи Fab через соответствующий линкер, Fv, одноцепочечный Fv (scFv), содержащий Fv тяжелой цепи, лигированный с Fv легкой цепи через соответствующий линкер, и однодоменное антитело (sdAb) с одной вариабельной областью тяжелой цепи, но без последовательности легкой цепи, которое также называют нанотелом (Muyldemans S. et al., Protein Eng., 1994, 7 (9), 1129-35, Hamers-Casterman C. et al., Nature, 1993, 363 (6428), 446-448). Молекула, содержащая области, отличные от связывающего фрагмента антитела по настоящему изобретению, такие как scFab или scFv, содержащие линкерную область, входит в объем связывающего фрагмента антитела по настоящему изобретению.
3-4. Модифицированное анти-HLA/NY-ESO антитело, его модифицированный связывающий фрагмент или их комплекс
Настоящее изобретение предлагает модифицированное антитело или его модифицированный связывающий фрагмент. Модифицированное антитело по настоящему изобретению или его модифицированный связывающий фрагмент подвергают химической или биологической модификации. Примеры химической модификации включают связь химической части с остовом аминокислоты и химическую модификацию N-связанных или O-связанных углеводных цепей. Примеры биологической модификации включают посттрансляционную модификацию (например, добавление сахарной цепи к N- или О-связи, обработку аминоконцевой или карбоксиконцевой области, дезамидирование, изомеризацию аспарагиновой кислоты и окисление метионина) и добавление метионина к амино концу с помощью экспрессии в прокариотической клетке-хозяине. Кроме того, метки, которые позволяют обнаруживать или выделять антитело или антиген по настоящему изобретению, такие как ферментная метка, флуоресцентная метка и аффинная метка, входят в объем модифицированного антитела или антигена, как описано выше. Модифицированное антитело по настоящему изобретению или его связывающий фрагмент, как описано выше, можно использовать для улучшения стабильности и сохраняемости в крови исходного антитела по настоящему изобретению или его связывающего фрагмента, снижения антигенности, обнаружения или выделения антитела или антигена, и другие цели.
Примеры химических частей, включенных в химически модифицированное антитело или фрагмент, включают водорастворимые полимеры, такие как полиэтиленгликоль (PEG), полимер этиленгликоля/пропиленгликоля, карбоксиметилцеллюлозу, декстран и поливиниловый спирт.
Примеры биологической модификации включают обработку ферментами, обработку клеток, добавление других пептидов, таких как метки, посредством генетической рекомбинации и использование клеток-хозяев, экспрессирующих ферменты, модифицированные эндогенными или экзогенными сахарными цепями.
Такая модификация может быть предложена в любом желаемом положении в антителе или его связывающем фрагменте, и в одном или нескольких положениях могут быть предложены одинаковые или два или несколько различных типов модификации.
Однако делеция такой последовательности тяжелой цепи или модификация последовательности тяжелой цепи или легкой цепи не оказывает существенного влияния на антигенсвязывающую способность и эффекторные функции антитела (например, на активацию комплемента или антителозависимую цитотоксичность).
Соответственно, настоящее изобретение охватывает антитело, подвергнутое такой делеции или модификации. Примеры включают мутант с делецией, лишенный 1 или 2 аминокислот на карбоксильном конце тяжелой цепи (Journal of Chromatography A; 705; 129-134, 1995), мутант с делецией, лишенный 2 аминокислот (глицина и лизина) на карбоксильном конце тяжелой цепи и подвергнутый амидированию пролина на карбоксильном конце (Analytical Biochemistry, 360: 75-83, 2007), и антитело, полученное в результате пироглутамилирования амино концевого глутамина или глутаминовой кислоты тяжелой или легкой цепи (WO 2013/147153) (в совокупности они называются «делеционными мутантами»). Пока сохраняются антигенсвязывающая способность и эффекторные функции, антитело по настоящему изобретению, лишенное конца тяжелой цепи и карбоксильного конца легкой цепи, не ограничивается делеционными мутантами, описанными выше. Когда антитело по настоящему изобретению содержит 2 или несколько цепей (например, тяжелых цепей), любая из таких 2 или нескольких цепей (например, тяжелых цепей) или обе могут представлять собой полноразмерную тяжелую цепь или тяжелую цепь, выбранную из группы, состоящей из делеционных мутантов, описанных выше. Хотя на количественное соотношение или числовое соотношение молекул делеционного мутанта будут влиять тип и условия культивирования культивируемых клеток млекопитающих животных, продуцирующих антитело по настоящему изобретению, основными компонентами антитела по настоящему изобретению могут быть обе из 2 тяжелых цепей, лишенных аминокислотного остатка на карбоксильном конце.
Кроме того, антитело по настоящему изобретению или его антигенсвязывающий фрагмент (включая те, которые входят в молекулу, мультиспецифическую молекулу и биспецифическую молекулу по настоящему изобретению), содержащие от одной до нескольких аминокислот, полученных из вектора экспрессии и/или или сигнальную последовательность, добавленную к амино концу и/или карбокси концу (и частично или полностью модифицированную, как описано выше) или подобные, входят в объем модифицированного антитела по настоящему изобретению или его модифицированного антигенсвязывающего фрагмента, пока сохраняется представляющая интерес антигенсвязывающая активность. Молекула, содержащая такое модифицированное антитело или его модифицированный антигенсвязывающий фрагмент, входит в объем молекулы по настоящему изобретению.
В настоящем изобретении «антитело или его связывающий фрагмент» включает «модифицированное антитело или его модифицированный антигенсвязывающий фрагмент». Кроме того, «антитело или его антигенсвязывающий фрагмент», включенные в молекулу, мультиспецифическую молекулу и биспецифическую молекулу по настоящему изобретению, включают «модифицированное антитело или его модифицированный антигенсвязывающий фрагмент».
Антителозависимая клеточная цитотоксичность может быть усилена регулированием (гликозилированием, афукозилированием и подобными) модификации сахарной цепи, связанной с антителом по настоящему изобретению. Известные методы регуляции модификации сахарной цепи антитела описаны, например, в WO 99/54342, WO 00/61739 и WO 02/31140, хотя методы ими не ограничиваются.
Настоящее изобретение охватывает комплекс антитела и других молекул, лигированных друг с другом линкером (т.е. иммуноконъюгатом). Примером комплекса антитело-лекарственное средство, включающего антитело, связанное с радиоактивным веществом, или соединение, обладающее фармакологической активностью, является конъюгат антитело-лекарственное средство (ADC) (Methods Mol. Biol., 2013, 1045: 1-27; Nature Biotechnology, 2005, 23, pp. 1137-1146).
Кроме того, настоящее изобретение охватывает комплекс, содержащий такое антитело, связанное с другим функциональным полипептидом. Примером такого комплекса антитело-пептид является комплекс антитела с альбумин-связанным полипептидом (Protein Eng. Des. Sel., 2012, (2): 81-8).
Модифицированное антитело, антитело с регулируемой модификацией сахарной цепи и комплекс, описанный выше, входят в объем антитела по настоящему изобретению, и связывающие фрагменты модифицированного антитела, антитела с регулируемой модификацией сахарной цепи и комплекса находятся в рамках связывающего фрагмента антитела по настоящему изобретению.
4. Способ получения антител
Антитело по настоящему изобретению может быть получено в клетке в виде рекомбинантного антитела, например, путем встраивания ДНК, кодирующей вариабельную область тяжелой цепи, или ДНК, кодирующей вариабельную область легкой цепи, в вектор экспрессии, трансформации клетки-хозяина для экспрессии с вектором и культивирования клетки-хозяина.
Что касается ДНК, кодирующей антитела, ДНК, кодирующую тяжелую цепь, можно получить путем лигирования ДНК, кодирующей вариабельную область тяжелой цепи, с ДНК, кодирующей константную область тяжелой цепи, и ДНК, кодирующую легкую цепь, можно получить путем лигирования ДНК, кодирующей вариабельную область легкой цепи, с ДНК, кодирующей константную область легкой цепи.
Анти-HLA/NY-ESO антитело по настоящему изобретению может быть получено путем встраивания ДНК, кодирующей тяжелую цепь, и ДНК, кодирующей легкую цепь, упомянутых выше, в вектор экспрессии, трансформации клетки-хозяина вектором и культивирования клетки-хозяина. В таком случае, ДНК, кодирующая тяжелую цепь, и ДНК, кодирующая легкую цепь, указанные выше, могут быть введены в один и тот же вектор экспрессии, и клетка-хозяин может быть трансформирована этим вектором. Альтернативно, ДНК, кодирующая тяжелую цепь, и ДНК, кодирующая легкую цепь, могут быть введены в отдельные векторы, и клетка-хозяин может быть трансформирована двумя векторами. В этом случае ДНК, кодирующие вариабельную область тяжелой цепи и вариабельную область легкой цепи, можно ввести в вектор, в который заранее были введены ДНК, кодирующая константную область тяжелой цепи, и ДНК, кодирующая константную область легкой цепи. Кроме того, вектор может содержать ДНК, кодирующую сигнальный пептид, который способствует секреции антитела из клетки-хозяина. В этом случае, ДНК, кодирующую сигнальный пептид, и ДНК, кодирующую антитело, заранее лигируют в рамке. Антитело может быть получено в виде зрелого белка путем удаления сигнального пептида после образования антитела.
В этом случае, ДНК, кодирующая вариабельную область тяжелой цепи, ДНК, кодирующая вариабельную область легкой цепи, ДНК, содержащая ДНК, кодирующую вариабельную область тяжелой цепи, лигированная с ДНК, кодирующей константную область тяжелой цепи, или ДНК, содержащая ДНК, кодирующую вариабельную область легкой цепи, лигированная с ДНК, кодирующей константную область легкой цепи, может быть функционально лигирована с элементом, таким как промотор, энхансер или сигнал полиаденилирования. Когда ДНК «функционально лигирована» в настоящем документе, ДНК лигируется с элементом так, что элемент может выполнять свои функции.
Вектор экспрессии конкретно не ограничен, при условии, что он может быть реплицирован в клетке животного, клетке бактерии, клетке дрожжей или другом хозяине, и его примеры включают известные плазмиды и фаги. Примеры вектора, используемого для создания вектора экспрессии, включают pcDNA™ (Thermo Fisher Scientific), вектор Flexi® (Promega), pUC19, pUEX2 (Amersham), pGEX-4T, pKK233-2 (Pharmacia) и pMAMneo (Clontech). В качестве клеток-хозяев можно использовать прокариотические клетки, такие как Escherichia coli и Bacillus subtilis, и эукариотические клетки, такие как дрожжи и клетки животных, где использование эукариотических клеток является предпочтительным. Примеры клеток животных включают линию клеток эмбриональной почки человека HEK293 и клеток яичника китайского хомячка (CHO). Достаточно ввести вектор экспрессии в клетку-хозяина известным способом для трансформации клетки-хозяина. Примеры способов включают способ электропорации, способ преципитации фосфатом кальция и способ трансфекции DEAE-декстраном. Полученное антитело можно очистить обычными способами выделения или очистки белка. Например, подходящим образом могут быть выбраны и объединены аффинная хроматография или другие методы хроматографии, фильтрация, ультрафильтрация, высаливание, диализ и подобные.
5. Молекула, которая связывается с HLA/NY-ESO
Молекула по настоящему изобретению содержит анти-HLA/NY-ESO антитело по настоящему изобретению или его антигенсвязывающий фрагмент. Молекула по настоящему изобретению предпочтительно представляет собой мультиспецифическую молекулу, содержащую 2 или несколько сайтов связывания антигена. В частности, молекула по настоящему изобретению может связываться с 2 или несколькими различными эпитопами на молекуле или с разными эпитопами на 2 или нескольких различных молекулах, и такая молекула содержит множество различных антигенсвязывающих фрагментов. Примеры такой мультиспецифической молекулы включают, но не ограничены ими, мультиспецифическую молекулу IgG-типа и мультиспецифическую молекулу, содержащую два или несколько типов вариабельных областей, например, фрагменты антител, такие как тандемное scFv (taFv), одноцепочечное диатело, диатело и триатитело, а также фрагмент антитела, полученный в результате ковалентного или не ковалентного связывания. Мультиспецифическая молекула может содержать Fc.
Мультиспецифическая молекула по настоящему изобретению может содержать, в дополнение к анти-HLA/NY-ESO антителу по настоящему изобретению или его антигенсвязывающему фрагменту, один или два или несколько типов антител или их антигенсвязывающих фрагментов. Примеры таких антигенсвязывающих фрагментов антитела включают Fab, F(ab)', Fv, scFv и sdAb.
Предпочтительная мультиспецифическая молекула по настоящему изобретению дополнительно содержит анти-CD3 антитело или его антигенсвязывающий фрагмент, и она также специфически связывается с CD3.
Анти-CD3 антитело или его антигенсвязывающий фрагмент, включенные в состав мультиспецифической молекулы по настоящему изобретению, конкретно не ограничены, при условии, что это CD3-связывающее антитело человека или его связывающий фрагмент, и оно предпочтительно связывается с CD3 примата, отличного от человека, такого как яванский макак. Более предпочтительным примером анти-CD3 антитела или его антигенсвязывающего фрагмента является антитело или его антигенсвязывающий фрагмент, содержащие вариабельную область тяжелой цепи CDRH1, состоящую из аминокислотной последовательности SEQ ID NO:141, вариабельную область тяжелой цепи CDRH2, состоящую из аминокислотной последовательности SEQ ID NO:142, вариабельную область тяжелой цепи CDRH3, состоящую из аминокислотной последовательности SEQ ID NO:143, вариабельную область легкой цепи CDRL1, состоящую из аминокислотной последовательности SEQ ID NO:144, вариабельную область легкой цепи CDRL2, состоящую из аминокислотной последовательности SEQ ID NO:145, и вариабельную область легкой цепи CDRL3, состоящую из аминокислотной последовательности SEQ ID NO:146 (фиг. 142).
Примером более предпочтительного антитела или его антигенсвязывающего фрагмента, содержащего CDRH1-CDRH3 и CDRL1-CDRL3, является антитело или его антигенсвязывающий фрагмент, содержащий вариабельную область тяжелой цепи C3E-7034, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:136 (фиг. 137), вариабельную область тяжелой цепи C3E-7036, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:137 (фиг. 138), вариабельную область тяжелой цепи C3E-7085, состоящую из аминокислотной последовательности из аминокислот 2-119 SEQ ID NO:138 (фиг. 139), вариабельную область тяжелой цепи C3E-7088, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:139 (фиг. 140), вариабельную область тяжелой цепи C3E-7093, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:140 (фиг. 141), вариабельную область тяжелой цепи C3E-7096, состоящую из аминокислотной последовательности из аминокислот 272-389 аминокислотной последовательности SEQ ID NO:155 (фиг. 151), вариабельную область тяжелой цепи C3E-7096, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:156 (фиг. 152), и вариабельную область тяжелой цепи C3E-7097. область, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:157 (фиг. 153).
Примером более предпочтительного антитела или его антигенсвязывающего фрагмента, содержащего CDRH1-CDRH3 и CDRL1-CDRL3, является антитело или его антигенсвязывающий фрагмент, содержащий вариабельную область легкой цепи C3E-7034, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:136 (фиг. 137), вариабельную область легкой цепи C3E-7036, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:137 (фиг. 138), вариабельную область легкой цепи C3E-7085, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:138 (фиг. 139), вариабельную область легкой цепи C3E-7088, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:139 (фиг. 140), вариабельную область легкой цепи C3E-7093, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:140 (фиг. 141), вариабельную область легкой цепи C3E-7096, состоящую из аминокислотной последовательности из аминокислот 405-511 аминокислотной последовательности SEQ ID NO:155 (фиг. 151), вариабельную область легкой цепи C3E-7096 состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:156 (фиг. 152), и вариабельную области легкой цепи C3E-7097, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:157 (фиг. 153).
Примером более предпочтительного антитела или его антигенсвязывающего фрагмента, содержащего CDRH1-CDRH3 и CDRL1-CDRL3, является антитело или его антигенсвязывающий фрагмент, содержащие комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7034, состоящую из аминокислот 2-119 и аминокислот 135-243 SEQ ID NO:136 (фиг. 137), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7036, состоящую из аминокислот 2-119 и аминокислот 135-241 SEQ ID NO:137 (фиг. 138), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7078, состоящую из аминокислот 2-119 и аминокислот 135-243 SEQ ID NO:147 (фиг. 143), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7085, состоящую из аминокислот 2-119 и аминокислот 135-241 SEQ ID NO:138 (фиг. 139), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7088, состоящую из аминокислот 2-119 и аминокислот 135-243 SEQ ID NO:139 (фиг. 140), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7093, состоящую из аминокислот 2-119 и аминокислот 135-243 SEQ ID NO:140 (фиг. 141), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7096, состоящую из аминокислот 272-389 и аминокислот 405-511 SEQ ID NO:155 (фиг. 151), комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7096, состоящую из аминокислот 277-394 и аминокислот 410-516 SEQ ID NO:156 (фиг. 152), и комбинацию вариабельной области тяжелой цепи и вариабельной области легкой цепи C3E-7097, состоящую из аминокислот 277-394 и аминокислот 410-516 SEQ ID NO:157 (фиг. 153).
Кроме того, примеры более предпочтительного антитела или его антигенсвязывающего фрагмента, содержащего CDRH1-CDRH3 и CDRL1-CDRL3, включают scFv C3E-7034, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:136 (фиг. 137), scFv C3E-7036, состоящий из аминокислотной последовательности из аминокислот 2-241 аминокислотной последовательности SEQ ID NO:137 (фиг. 138), scFv C3E-7078, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:147 (фиг. 143), scFv C3E-7085, состоящей из аминокислотной последовательности из аминокислот 2-241 аминокислотной последовательности SEQ ID NO:138 (фиг. 139), scFv C3E-7088, состоящей из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:139 (фиг. 140), scFv C3E-7093, состоящей из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:140 (фиг. 141), и антитело или его связывающий фрагмент, содержащий любую из таких конструкций scFv. Предпочтительный вариант осуществления CD3-специфического scFv (который может называться «анти-CD3 scFv») включает scFv, содержащий метку FLAG-His, добавленную к его карбоксильному концу (может быть просто назван «аддуктом метки»). Примеры предпочтительных аддуктов метки включают C3E-7034 (SEQ ID NO:136; фиг. 137), C3E-7036 (SEQ ID NO:137; фиг. 138), C3E-7085 (SEQ ID NO:138; фиг. 139), C3E-7088 (SEQ ID NO:139; фиг. 140) и C3E-7093 (SEQ ID NO:140; фиг. 141), где C3E-7085 является более предпочтительным.
Предпочтительным примером мультиспецифической молекулы по настоящему изобретению является биспецифическая молекула. «Биспецифическая» молекула способна связываться с двумя разными эпитопами на одной молекуле или с разными эпитопами на двух молекулах и включает антитело или антигенсвязывающий фрагмент, обладающие такой биспецифичностью. Биспецифическая молекула по настоящему изобретению связывается с HLA/NY-ESO и дополнительно связывается с CD3.
Примеры биспецифической молекулы по настоящему изобретению включают те, которые имеют структуры (форматы), описанные ниже.
В биспецифической молекуле двойного scFv типа, каждый из двух типов конструкций scFv, связывающихся с разными эпитопами, может связываться с одним Fc димера с линкером или может напрямую связываться с ним без линкера. Альтернативно, каждому из двух типов конструкций scFv, связывающихся с разными эпитопами, разрешено связываться с CH и CL с помощью линкеров и, кроме того, им разрешено связываться с одним Fc димера с помощью линкеров. В такой биспецифической молекуле, Fc, содержащий образующую гетеродимер мутацию ниже одного scFv, гетерологически ассоциирован с другим Fc, содержащим образующую гетеродимер мутацию ниже другого scFv. Биспецифическая молекула двойного scFv типа упоминается как биспецифическая молекула двойного типа или просто как молекула двойного типа (фиг. 6A(b)).
В настоящем изобретении, например, можно использовать биспецифическую молекулу двойного типа, состоящую из анти-HLA-A2/NY-ESO scFv и анти-CD3 scFv.
Альтернативно, биспецифическая молекула по настоящему изобретению может содержать Fab и scFv, связывающиеся с разными эпитопами, Fab первого антитела может быть связан с одним Fc димера, и scFv второго антитела может быть связан с другим Fc с помощью линкеров. В такой биспецифической молекуле, Fc, содержащий образующую гетеродимер мутацию ниже Fab, гетерологически связан с Fc, содержащим образующую гетеродимер мутацию ниже scFv. Такую биспецифическую молекулу называют «биспецифической молекулой гибридного типа» или «гибридным типом» (фиг. 6А (а)). В настоящем изобретении, например, можно использовать гибридный тип, состоящий из анти-HLA-A2/NY-ESO Fab и анти-CD3 scFv.
Кроме того, биспецифическая молекула может позволять Fab первого антитела и scFv второго антитела присоединяться к одному Fc димера с линкерами. В таком случае, Fab может быть разрешено присоединяться к Fc, и scFv может быть разрешено присоединяться к такому Fab. Альтернативно, scFv может быть позволено присоединяться к Fc, и Fab может быть позволено присоединяться к такому scFv. Предпочтительно, Fab можно присоединить к Fc, и scFv можно присоединить к такому Fab. scFv может присоединяться к вариабельной области Fab с помощью линкера. В такой биспецифической молекуле, Fc, содержащий мутацию, образующую гетеродимер, связан с сайтом ниже того сайта, где scFv лигируется с Fab. Такая биспецифическая молекула называется биспецифической молекулой scFv-Fab-гетеродимер Fc типа или scFv-Fab-гетеродимер Fc типа (фиг. 6A(c)).
В настоящем изобретении, например, можно использовать scFv-Fab-гетеродимер типа Fc, состоящий из анти-CD3 scFv и анти-HLA-A2/NY-ESO Fab.
Кроме того, taFv (фиг. 3(с)), содержащий конструкции scFv двух типов (первого антитела и второго антитела), лигированные друг с другом линкером, может быть присоединен к одному Fc димера с линкером или непосредственно, без линкера. Такая биспецифическая молекула называется биспецифической молекулой taFv-гетеродимера Fc типа или taFv-гетеродимера Fc типа (фиг. 3(d)). В такой биспецифической молекуле Fc, содержащий мутацию, образующую гетеродимер, гетерологически связан с другим Fc, содержащим мутацию, образующую гетеродимер, в сайте ниже taFv. Порядок лигирования первого антитела и второго антитела к taFv не ограничен. Ранее упомянутая биспецифическая молекула упоминается как taFv-гетеродимер Fc типа, но когда порядок лигирования первого антитела и второго антитела с taFv инвертируется, биспецифическая молекула упоминается как taFv (инвертированный)-гетеродимер Fc типа (taFv (инвертированный)-Fc тип).
На фиг. 6А (а) показана структура биспецифической молекулы гибридного типа, на фиг. 6А (b) показана структура биспецифической молекулы двойного типа, и на фиг. 6А (с) показана структура биспецифической молекулы scFv-Fab-гетеродимера Fc типа. На фиг. 3 (а) представлена структура scFv, на фиг. 3 (b) представлена структура Fab, на фиг. 3 (с) представлена структура taFv, на фиг. 3 (d) представлена структура биспецифической молекулы taFv-гетеродимера Fc типа, и на фиг. 3(e) показана структура биспецифической молекулы taFv-Fab-гетеродимера Fc типа. На фиг. 6 В (а) представлена структура биспецифической молекулы taFv-гетеродимера Fc типа (такой же, как на фиг. 3 (d)), на фиг. 6 В (b) представлена структура биспецифической молекулы taFv (инвертированного)-гетеродимера Fc типа, на фиг. 6B (c) показана структура первого полипептида, включенного в состав биспецифической молекулы taFv (инвертированного)-гетеродимера Fc типа, и на фиг. 6 (d) показана структура второго полипептида, включенного в состав биспецифической молекулы taFv (инвертированного)-гетеродимера Fc типа.
В биспецифической молекуле по настоящему изобретению ассоциировано множество полипептидов.
В настоящем изобретении, можно использовать taFv, например, состоящий из scFv анти-HLA-A2/NY-ESO и scFv анти-CD3. Биспецифическая молекула taFv-гетеродимера Fc типа предпочтительно содержит (а) первый полипептид, содержащий scFv, который специфически связывается с HLA/NY-ESO, scFv, который специфически связывается с CD3, и Fc область (i) иммуноглобулина в указанном порядке от N конца к С концу, и второй полипептид, содержащий шарнирную область и Fc область (ii) иммуноглобулина. Более предпочтительно, он включает (b) первый полипептид, связанный со вторым полипептидом в Fc области (i) и Fc области (ii). Fc области первого полипептида и второго полипептида могут содержать мутацию, образующую гетеродимер. На фиг. 3 (d) показан пример биспецифической молекулы taFv-гетеродимера Fc типа. Как показано на фиг. 3(d), Fc область (i) первого полипептида связывается с Fc областью (ii) второго полипептида (сплошной), и, таким образом, первый полипептид связывается со вторым полипептидом. На фиг. 3(f) показан первый полипептид, и на фиг. 3(g) показан второй полипептид. На фиг. 3(d), например, scFv, показанный пустым, представляет собой анти-HLA-A2/NY-ESO scFv, и scFv, указанный с положительным наклоном, представляет собой анти-CD3 scFv.
Первый полипептид, включенный в более предпочтительную биспецифическую молекулу taFv-гетеродимера Fc типа по настоящему изобретению, содержит аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:85, аминокислотную последовательность аминокислоты 21-511 аминокислотной последовательности SEQ ID NO:87, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:88, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:89, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:90, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:91, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:92, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:93, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:94, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:95, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:96, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:86, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:149, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:150, аминокислотную последовательность аминокислот 20-511 аминокислотной последовательности SEQ ID NO:155, аминокислотную последовательность аминокислот 20-516 аминокислотной последовательности SEQ ID NO:156, или аминокислотную последовательность аминокислот 20-516 аминокислотной последовательности SEQ ID NO:157. Первый полипептид, включенный в еще одну предпочтительную биспецифическую молекулу taFv-гетеродимера Fc типа, содержит аминокислотную последовательность аминокислот 529-745 аминокислотной последовательности SEQ ID NO:85, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 86, 149, 150 или 155, или аминокислотную последовательность аминокислот 534-750 аминокислотной последовательности SEQ ID NO:156 или 157. Первый полипептид, включенный в еще более предпочтительную биспецифическую молекулу taFv-гетеродимера Fc типа, состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:85, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:87, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:88, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:89, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:90, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:91, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:92, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:93, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:94, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:95, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:96, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:86, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:149, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:150, или аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:155. Альтернативно, первый полипептид состоит из аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:156, или аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:157.
Второй полипептид, включенный в состав предпочтительной биспецифической молекулы taFv-гетеродимера Fc типа по настоящему изобретению, содержит шарнирную область и мутантный Fc, полученный из антитела человека. Второй полипептид, включенный в более предпочтительную биспецифическую молекулу TaFv-гетеродимера Fc типа, содержит аминокислотную последовательность аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84.
Из них, NYF-0016, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:85, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0022, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:87 связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0023, в которой первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:88, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0027, в котором первый полипептид, состоящий аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:89, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0035, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:90, связан со вторым полипептидом, состоящим из аминокислоты последовательность аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0044, в которой первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:91 связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0045, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:92, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0047, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:93, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0048, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:94, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0060, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:95, связан с вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0061, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 из аминокислотной последовательности SEQ ID NO:96, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0019, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:86, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, NYF-0014, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:149, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, и NYF-0082, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:150 связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 21-246 аминокислотной последовательности SEQ ID NO:84, могут быть приведены в качестве примера предпочтительных биспецифических молекул taFv-гетеродимера Fc типа по настоящему изобретению.
Дополнительные примеры предпочтительных биспецифических молекул taFv-гетеродимера Fc типа по настоящему изобретению включают NYZ-0038, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:155, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84, NYZ-0082, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:156, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84, и NYZ-0083, в котором первый полипептид, состоящий из аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:157, связан со вторым полипептидом, состоящим из аминокислотной последовательности из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84.
Из них NYF-0023, NYF-0047, NYF-0048, NYF-0060, NYF-0061, NYZ-0038, NYZ-0082 и NYZ-0083 демонстрируют превосходную биологическую активность, физические свойства и подобные и, таким образом, являются особенно предпочтительными.
Кроме того, taFv первого антитела может прикрепляться к одному Fc димера линкером или напрямую связываться с ним без линкера, и Fab первого антитела или второго антитела может прикрепляться к другому Fc линкером или напрямую связываться с ним без линкера. В такой биспецифической молекуле, Fab добавляется к сайту выше Fc области (ii) (сплошной) второго полипептида taFv-гетеродимера Fc типа. Такая биспецифическая молекула называется биспецифической молекулой taFv-Fab-гетеродимер Fc типа или taFv-Fab-гетеродимер Fc типа (фиг. 3).
В настоящем изобретении, например, taFv анти-HLA-A2/NY-ESO scFv, taFv анти-CD3 scFv и Fab HLA/NY-ESO могут быть использованы для включения в биспецифическую молекулу taFv-Fab-гетеродимера Fc типа.
Биспецифическая молекула taFv-Fab-гетеродимера Fc типа предпочтительно содержит (а) первый полипептид, содержащий scFv, который специфически связывается с HLA/NY-ESO человека, scFv, который специфически связывается с CD3, и Fc область (i) иммуноглобулина в указанном порядке от N конца к С концу, второй полипептид, состоящий из тяжелой цепи иммуноглобулина, включая Fc область (ii), и третий полипептид, состоящий из легкой цепи иммуноглобулина. Более предпочтительно, (b) второй полипептид связан с третьим полипептидом и (c) первый полипептид связан со вторым полипептидом в Fc области (i) и Fc области (ii). На фиг. 3(e) показан пример биспецифической молекулы taFv-Fab-гетеродимера Fc типа, на фиг. 3(f) показан первый полипептид, на фиг. 3(h) показан второй полипептид, и на фиг. 3 (i) показан третий полипептид. Как показано на фиг. 3(е), Fc область (ii) (закрашенная) второго полипептида, состоящая из тяжелой цепи иммуноглобулина, включающая Fc область (ii), связывается с Fc областью (i) первого полипептида, и легкая цепь иммуноглобулина дополнительно присоединяется ко второму полипептиду. Такая предпочтительная биспецифическая молекула TaFv-Fab-гетеродимера Fc типа может состоять из биспецифической молекулы taFv-гетеродимера Fc типа, содержащей taFv и Fc область иммуноглобулина, и Fab, связанные с ней. Примеры аминокислотных последовательностей, включенных в первый полипептид предпочтительной биспецифической молекулы taFv-Fab-гетеродимера Fc типа включают аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:85, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:87, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:88, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:89, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:90, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:91, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:92, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:93, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:94, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:95, аминокислотную последовательность аминокислот 21-511 аминокислотная последовательности SEQ ID NO:96, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:86, аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:149, и аминокислотную последовательность аминокислот 21-511 аминокислотной последовательности SEQ ID NO:150. Первый полипептид, включенный в более предпочтительную биспецифическую молекулу taFv-Fab-гетеродимера Fc типа состоит из аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:85, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:87, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:88, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:89, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:90, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:91, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:92, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:93, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:94, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:95, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:96, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:86, аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:149, или аминокислотной последовательности из аминокислот 21-745 аминокислотной последовательности SEQ ID NO:150.
Второй полипептид, включенный в состав предпочтительной биспецифической молекулы taFv-Fab-гетеродимера Fc типа по настоящему изобретению, содержит вариабельную область тяжелой цепи антитела человека или гуманизированного антитела, область CH1, шарнирную область и мутантный Fc. Второй полипептид, включенный в более предпочтительную биспецифическую молекулу taFv-Fab-гетеродимера Fc типа, содержит аминокислотную последовательность аминокислот 20-242 аминокислотной последовательности SEQ ID NO:99.
Третий полипептид, включенный в предпочтительную биспецифическую молекулу taFv-Fab-гетеродимера Fc типа по настоящему изобретению, содержит вариабельную область и константную область легкой цепи антитела человека или гуманизированного антитела. Третий полипептид, включенный в более предпочтительную биспецифическую молекулу taFv-Fab-гетеродимера Fc типа, содержит аминокислотную последовательность аминокислот 21-131 аминокислотной последовательности SEQ ID NO:100.
Из второго полипептида, включенного в такую предпочтительную биспецифическую молекулу taFv-Fab-гетеродимера Fc типа, вариабельная область, область CH1 и третий полипептид составляют Fab, где Fab предпочтительно представляет собой анти-HLA/NY-ESO антитело, такое как как Fab NYA-0001.
В настоящем изобретении, например, анти-HLA-A2/NY-ESO scFv, анти-CD3 scFv, HLA/NY-ESO Fab или анти-CD3 Fab могут быть включены в биспецифическую молекулу scFv-Fab-гетеродимера Fc типа.
Биспецифическая молекула scFv-Fab-гетеродимера Fc типа предпочтительно содержит (а) первый полипептид, содержащий scFv, который специфически связывается с HLA/NY-ESO человека, вариабельную область тяжелой цепи антитела и константную область CH1 антитела, которая специфически связывается с CD3, и Fc область (i) иммуноглобулина в указанном порядке от N конца к C концу, второй полипептид, содержащий шарнирную область иммуноглобулина и Fc область (ii), и третий полипептид, состоящий из легкой цепи антитела, содержащей вариабельную область и константную область. Более предпочтительно, (b) первый полипептид связан со вторым полипептидом через Fc область (i) и Fc область (ii), и первый полипептид связан с (легкой цепью антитела) третьим полипептидом через вариабельная область тяжелой цепи антитела и константную область CH1 первого полипептида. Fc области первого полипептида и второго полипептида могут быть областями дикого типа или могут содержать мутацию, образующую гетеродимер. На фиг. 6A (c) показан пример биспецифической молекулы scFv-Fab-гетеродимера Fc типа. На фиг. 6А(с) правая половина показывает первый полипептид и третий полипептид, и левая половина показывает второй полипептид. Как показано на фиг. 6А(с), Fc область (i) первого полипептида связана с Fc областью (ii) второго полипептида (сплошной), и первый полипептид связан с третьим полипептидом. На фиг. 6А(с) scFv, обозначенный положительным наклоном, представляет собой анти-HLA-A2/NY-ESO scFv, и каждое Fab, обозначенное пустым, клетчатым узором или горизонтальными линиями, представляет собой анти-CD3 Fab.
Примером аминокислотной последовательности, включенной в первый полипептид, включенный в предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа, является аминокислотная последовательность аминокислот 21-394 аминокислотной последовательности SEQ ID NO:160. Более предпочтительным ее примером является аминокислотная последовательность аминокислот 20-724 из аминокислотной последовательности SEQ ID NO:160.
Примером другой аминокислотной последовательности, включенной в первый полипептид, включенный в предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа, является аминокислотная последовательность аминокислот 21-389 аминокислотной последовательности SEQ ID NO:197. Более предпочтительным ее примером является аминокислотная последовательность аминокислот 20-719 из аминокислотной последовательности SEQ ID NO:197.
Примером другой последовательности, включенной в первый полипептид, входящий в состав предпочтительной биспецифической молекулы scFv-Fab-гетеродимера Fc типа, является аминокислотная последовательность аминокислот 21-389 аминокислотной последовательности SEQ ID NO:198. Более предпочтительным ее примером является аминокислотная последовательность аминокислот 20-719 из аминокислотной последовательности SEQ ID NO:198.
Второй полипептид, включенный в предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера типа Fc по настоящему изобретению, содержит шарнирную область и мутантный Fc, полученный из антитела человека. Второй полипептид, включенный в более предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа, содержит аминокислотную последовательность аминокислот 20-246 из аминокислотной последовательности SEQ ID NO:84.
Третий полипептид, включенный в предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа по настоящему изобретению, содержит легкую цепь, полученную из антитела человека. Третий полипептид, включенный в более предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа, содержит, например, аминокислотную последовательность аминокислот 21-127 аминокислотной последовательности SEQ ID NO:161. Еще более предпочтительным ее примером является аминокислотная последовательность аминокислот 21-233 из аминокислотной последовательности SEQ ID NO:161.
Из них NYZ-1010, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 20-724 аминокислотной последовательности SEQ ID NO:160, второй полипептид состоит из аминокислотной последовательности из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84, и третий полипептид, состоящий из аминокислотной последовательности из аминокислот 21-233 аминокислотной последовательности SEQ ID NO:161, связаны друг с другом, может быть приведен в качестве примера предпочтительной биспецифической молекулы scFv-Fab-гетеродимера Fc типа по настоящему изобретению.
Кроме того, NYZ-1007, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 20-719 аминокислотной последовательности SEQ ID NO:197, второй полипептид состоит из аминокислотной последовательности из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84, и третий полипептид состоит из аминокислотной последовательности из аминокислот 21-233 аминокислотной последовательности SEQ ID NO:161, связаны друг с другом, может быть приведен в качестве примера предпочтительной биспецифической молекулы scFv-Fab-гетеродимера Fc типа по настоящему изобретению.
Кроме того, NYZ-1017, в котором первый полипептид состоит из аминокислотной последовательности из аминокислот 20-719 аминокислотной последовательности SEQ ID NO:198, второй полипептид состоит из аминокислотной последовательности из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84, и третий полипептид состоит из аминокислотной последовательности из аминокислот 21-233 аминокислотной последовательности SEQ ID NO:161, связаны друг с другом, может быть приведен в качестве примера предпочтительной биспецифической молекулы scFv-Fab-гетеродимера Fc типа по настоящему изобретению.
По меньшей мере 1, 2 или 3 пептида, включенные в биспецифическую молекулу по настоящему изобретению, могут представлять собой «делеционный мутант», описанный выше. В частности, 1 или 2 (или более) аминокислот на его карбоксильном конце (в частности, на карбоксильном конце, полученном из тяжелой цепи антитела) могут быть мутированы (также могут быть удалены). Например, карбоксильный конец аминокислотной последовательности первого полипептида, включенного в NYZ-1010, который представляет собой предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа по настоящему изобретению, может представлять собой любой из Lys в положении 724 SEQ ID NO:160, Gly в положении 723 в результате делеции аминокислоты, или смесь первых полипептидов, включающих Lys и Gly на карбоксильном конце. Кроме того, карбоксильный конец аминокислотной последовательности второго полипептида, включенного в предпочтительную биспецифическую молекулу scFv-Fab-гетеродимера Fc типа по настоящему изобретению, может представлять собой любой из Lys в положении 246 SEQ ID NO:84, Gly в положении 245 в результате делеции аминокислоты, или смеси вторых полипептидов, включая Lys и Gly на карбоксильном конце.
scFv и Fab, включенные в биспецифическую молекулу по настоящему изобретению, предпочтительно происходят из гуманизированного антитела или антитела человека, и Fc предпочтительно происходит из антитела человека.
В вариабельной области биспецифической молекулы по настоящему изобретению, вариабельная область тяжелой цепи и вариабельная область легкой цепи могут быть присоединены в указанном порядке от амино конца антитела. Альтернативно, вариабельная область легкой цепи и вариабельная область тяжелой цепи могут быть присоединены в указанном порядке. Линкер может необязательно присутствовать между двумя вариабельными областями. Остаток глицина может необязательно присутствовать на амино конце амино концевой вариабельной области. В тандемной биспецифической молекуле типа scFv линкер, метка FLAG и/или метка His могут быть необязательно присоединены к карбоксильному концу вариабельной области карбоксильного конца. В предпочтительном варианте осуществления, например, вариабельная область тяжелой цепи, первый линкер, вариабельная область легкой цепи, второй линкер, метка FLAG и метка His присоединяются в указанном порядке от амино конца.
Примеры линкеров включают одноцепочечный полипептид, одноцепочечный олигопептид и синтетические продукты, такие как PEG, нуклеотид, сахарная цепь и соединения. Другие известные линкеры также могут быть использованы без особых ограничений, если они могут присоединять 2 полипептида друг к другу.
Пептидный линкер может содержать, например, от 5 до 30 аминокислот. Когда биспецифическая молекула содержит множество линкеров, все пептидные линкеры могут иметь одинаковую длину или могут использоваться пептидные линкеры разной длины.
Примером пептидного линкера является повторение (Gly⋅Gly⋅Gly⋅Gly⋅Ser) (SEQ ID NO:161). К ним могут быть добавлены от одного до нескольких аминокислотных остатков, отличных от Gly и Ser.
Среди конструкций (форматов) мультиспецифического антитела и, в частности, биспецифического антитела по настоящему изобретению, как описано выше, taFv-гетеродимер Fc типа, taFv-Fab-гетеродимер Fc типа и scFv-Fab-гетеродимер Fc типа являются предпочтительными, и taFv-гетеродимер Fc типа является более предпочтительным. Формат, в котором анти-HLA/NY-ESO scFv и анти-CD3 scFv расположены в таком порядке от N конца к С концу (taFv-гетеродимер Fc типа), является более предпочтительным, чем формат, в котором анти-CD3 scFv и анти-HLA/NY-ESO scFv расположены в таком порядке (taFv (инвертированный)-гетеродимер Fc типа) (например, пример 11). Другим примером более предпочтительного формата является scFv-Fab-гетеродимер Fc типа.
Настоящее изобретение также охватывает молекулу, которая содержит аминокислотную последовательность, кодируемую нуклеотидной последовательностью, включенной в полинуклеотид, который гибридизуется в жестких условиях с комплементарной цепью полинуклеотида, содержащего нуклеотидную последовательность, кодирующую аминокислотную последовательность, включенную в молекулу настоящего изобретения, и который связывается с HLA/NY-ESO и, кроме того, связывается с CD3.
Настоящее изобретение также охватывает молекулу, содержащую аминокислотную последовательность, имеющую, по меньшей мере, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичность с аминокислотной последовательностью, включенной в молекулу по настоящему изобретению, и которая связывается с HLA/NY-ESO, и также связывается с CD3.
Антитело по настоящему изобретению, его связывающий фрагмент и полиспецифическое антитело, содержащее его, обладают превосходной биологической активностью, физико-химическими свойствами (далее именуемыми «физическими свойствами»), безопасностью, фармакокинетикой и другими свойствами. (а) Примеры биологической активности или ее показателей включают антигенсвязывающую активность, цитотоксичность in vitro и противоопухолевую активность in vivo. Например, константа диссоциации (значение KD) для HLA/NY-ESO составляет 100 нМ или меньше, или 50 нМ или меньше, предпочтительно, 20 нМ или меньше, или 10 нМ или меньше, и более предпочтительно, 5 нМ или меньше. Величина EC50 цитотоксичности, проявляемой при использовании мононуклеарных клеток периферической крови человека в качестве эффекторных клеток по отношению к клеткам U266B1 и/или NCI-H1703 человека, эндогенно экспрессирующим NY-ESO, составляет 20 нМ или меньше, предпочтительно, 10 нМ или меньше, и более предпочтительно, 5 нМ или меньше (цитотоксичность in vitro можно анализировать и определять в соответствии со способом, описанным в примере 8, хотя способ этим не ограничивается). Например, 0,1 мл суспензии клеток плоскоклеточного рака легкого человека линии NCI-H1703 (6×107 клеток/мл) вводят подкожно мышам линии NOG, 0,2 мл суспензии мононуклеарных клеток периферической крови человека (3,75×107 клеток/мл) вводят внутривенно через 4 дня, и введение антител начинают через 14 дней и проводят один раз в неделю в 3 случаях. После этого измеряют объем опухоли. Ингибирующая активность в отношении роста опухоли по сравнению с контрольной группой, которой вводят растворитель, составляет 50% или выше, предпочтительно, 75% или выше, и более предпочтительно, 90% или выше (противоопухолевую активность in vivo можно анализировать и определять в соответствии со способом, описанным в примере 9, хотя способ этим не ограничивается). (b) Примеси, содержащиеся в биофармацевтических продуктах, связаны с безопасностью лекарственного средства. Таким образом, необходимо установить соответствующий стандарт и контролировать количество примесей во время производства и во время хранения. В частности, HMWS (агрегат) является основной примесью и связан с иммуногенным риском или снижением эффективности лекарственного средства. Таким образом, HMWS следует более строго контролировать. В целях контроля примесей следует также проводить оценку стабильности (независимо от того, увеличилось ли количество примесей) по прошествии времени во время и после производства, а также во время производства. Поскольку период действия фармацевтического продукта определяется на основании результатов теста на долговременную стабильность, более длительный период действия может быть определен для антитела, проявляющего стабильность во времени. Таким образом, долгосрочная стабильность может быть использована в качестве индикатора для выбора предпочтительного антитела для биофармацевтического продукта. Примеры физико-химических свойств в настоящем изобретении включают кислотостойкость (для ингибирования образования HMWS) и стабильность в растворе (для ингибирования образования HMWS). Примером другого показателя является высокий выход продукта культуры рекомбинантных клеток, полученного путем введения гена, кодирующего аминокислотную последовательность антитела по настоящему изобретению, его связывающего фрагмента или молекулы, содержащей его, в подходящую клетку-хозяина для их продуцирования, такую как клетка Expi293F. Предпочтительное антитело по настоящему изобретению, его антигенсвязывающий фрагмент и содержащее его мультиспецифическое антитело с такими физико-химическими свойствами являются предпочтительными по следующим причинам. То есть раствор, содержащий антитело, его связывающий фрагмент или мультиспецифическое антитело, можно подвергать воздействию кислых условий, и таким образом можно проводить или облегчать процессы его получения, такие как протеин А или ионообменная хроматография и инактивация вируса. Кроме того, производство HMWS можно контролировать на низких уровнях даже в форме раствора антитела, его связывающего фрагмента или мультиспецифического антитела и, таким образом, его производство, приготовление лекарственного средства, распространение или хранение фармацевтических продуктов, содержащих его, может быть проведено и облегчено. Кроме того, фармацевтические продукты, содержащие их, могут быть эффективно произведены. Что касается кислотостойкости, то, например, содержание HMWS, определенное нагреванием при pH 3,5 при комнатной температуре в течение 1 часа и последующим измерением HMWS с помощью эксклюзионной хроматографии, составляет 5% или меньше, предпочтительно, 2% или меньше, и более предпочтительно 1% или меньше (содержание HMWS может быть измерено и определено для оценки кислотостойкости в соответствии со способом, описанным в примере 19-1, хотя способ этим не ограничивается). Что касается стабильности раствора, то, например, анализируемое вещество растворяют до концентрации 25 мг/мл в растворе, содержащем 25 мМ гистидина и 5% сорбита при pH 6,0, полученную смесь хранят при 25°C в течение 6 дней, а затем HMWS измеряют эксклюзионной хроматографией. Определенное таким образом содержание ВМС составляет 20% или меньше, предпочтительно, 10% или меньше (содержание ВМС может быть измерено и определено для оценки стабильности раствора в соответствии со способом, описанным в примере 19-2, хотя способ этим не ограничивается). Эффективность производства или выход можно измерить и определить в соответствии со способом, описанным в примерах 20 и 21, хотя способ этим не ограничивается. (c) Примеры безопасности и ее показателей включают свойства распознавания антигена и наблюдения во время введения. Например, представляющее интерес антитело, которое распознает множество аминокислот на пептиде NY-ESO дикого типа, но не связывается с гомологичным пептидом, содержащим аминокислотную последовательность, аналогичную, но не идентичную аминокислотной последовательности пептида NY-ESO дикого типа, приведет к низкому риску побочных эффектов, вызванных эффектами вне мишени. Кроме того, можно предсказать, что такое антитело будет демонстрировать низкую иммуногенность при скрининге иммуногенности на основе ISPRI web (EpiVax, Inc) и демонстрировать низкий риск побочных эффектов, таких как продуцирование цитокинов. Кроме того, мышам Balb/c вводят NYF-0023, NYF-0045, NYF-0047, NYF-0048, NYF-0060, NYF-0061, NYZ-0082 или NYZ-1010, включенные в биспецифическое антитело по настоящему изобретению. В результате не наблюдается значительных проблем с периодом полужизни в крови, и также не наблюдается потеря массы тела или другие явные проблемы с токсичностью. В результате однократного введения NYZ-0082 или NYZ-1010 яванским макакам не наблюдается существенных проблем с периодом полужизни в крови, и также не наблюдаются изменения в результате введения в отношении общего состояния, массы тела, количества потребления корма, температуры тела и уровня цитокинов в плазме. (d) Примеры кинетики или ее показателей включают период полужизни в крови. В настоящем изобретении не наблюдается значительных проблем с периодом полужизни в крови в результате введения нескольких биспецифических антител мышам Balb/c или яванским макакам. Антитело по настоящему изобретению, его связывающий фрагмент и молекулы, обладающие такой превосходной биологической активностью, физико-химическими свойствами, безопасностью и кинетикой, предпочтительно могут быть интегрированы в фармацевтическую композицию. Предпочтительные примеры антитела по настоящему изобретению или его антигенсвязывающего фрагмента, обладающего антигенсвязывающей активностью, описанной в (а), и различными свойствами, описанными в (b), включают, но не ограничены ими, NYA-1143, NYA-1163 и NYA-3061. Их более предпочтительные примеры включают, но не ограничены ими, NYA-2047, NYA-2061, NYA-2143 и NYA-3061. Предпочтительные примеры мультиспецифического антитела по настоящему изобретению, обладающего свойствами (а)-(d), включают, но не ограничены ими, NYF-0016, NYF-0019, NYF-0022, NYF-0023, NYF-0027, NYF-0035, NYF-0044, NYF-0045, NYF-0047, NYF-0048, NYF-00058, NYF-0060, NYF-0061, NYZ-0038, NYZ-0082, NYZ-0088 и NYZ-1010. Более предпочтительные их примеры включают, но не ограничены ими, NYF-0061, NYZ-0038, NYZ-0082, NYZ-0088, NYZ-1007, NYZ-1010 и NYZ-1017.
В настоящем изобретении «сайт», с которой связывается антитело; т.е. «сайт», который распознается антителом, представляет собой частичный пептид или частичную структуру более высокого порядка антигена, с которым связывается антитело или который распознается антителом. В настоящем изобретении, такой сайт также называют эпитопом или сайтом связывания антитела. Примеры сайтов, с которыми связывается анти-HLA/NY-ESO антитело по настоящему изобретению, или сайтов, которые распознаются им на HLA/NY-ESO, включают множество аминокислот и частичных структур более высокого порядка в HLA/NY-ESO пептиде.
«Антитело или его связывающий фрагмент, который связывается с тем же сайтом», что и антитело по настоящему изобретению, или его связывающий фрагмент также входят в объем настоящего изобретения. «Антитело, которое связывается с тем же сайтом», что и данное антитело, представляет собой другое антитело, которое связывается с сайтом на молекуле антигена, распознаваемым таким указанным антителом. Если второе антитело связывается с частичным пептидом или частичной конформацией молекулы антигена, с которой связывается первое антитело, можно оценить связывание первого и второго антител с одним и тем же сайтом. Когда первое антитело по настоящему изобретению обладает антигенсвязывающей активностью, описанной в пункте (а) выше, второе антитело, которое связывается с тем же сайтом на HLA/NY-ESO, с высокой вероятностью будет обладать аналогичной активностью, и, таким образом, такое второе антитело включено в объем настоящего изобретения. Антитело, которое связывается с HLA/NY-ESO конкурентно с первым антителом по настоящему изобретению, обладающим антигенсвязывающей активностью, описанной в (а), входит в объем настоящего изобретения. Антитело, которое связывается с сайтом на HLA/NY-ESO, распознаваемым моноклональным антителом по настоящему изобретению, антитело, которое связывается с HLA/NY-ESO конкурентно с моноклональным антителом по настоящему изобретению, и его связывающие фрагменты предпочтительно имеют, по меньшей мере, одну из цитотоксичности in vitro и противоопухолевой активности in vivo, описанных в (а), и свойств, описанных в (b)-(d), более предпочтительно, 3 или более из них, и оптимально, все из них.
Сайт связывания антитела можно определить способом, хорошо известным специалисту в данной области техники, таким как способ иммуноанализа. Например, ряд пептидов получают путем удаления аминокислотной последовательности антигена с С или N конца, соответственно, исследуют реактивность антитела к нему, приблизительно определяют сайт узнавания, синтезируют более короткую пептидную последовательность и определяют реакционную способность антитела к нему. Таким образом, можно определить сайт связывания. Альтернативно, конкретный сайт или область аминокислотной последовательности антигена или пептидного фрагмента антигена удаляют или заменяют другой аминокислотной последовательностью, или в такой сайт или область вводят мутацию. Изучая реактивность антитела к такому пептиду, можно определить сайт связывания. Пептидный фрагмент антигена может быть получен, например, генетической рекомбинацией или пептидным синтезом.
Когда антитело связывается или распознает частичную структуру антигена более высокого порядка, такой антигенсвязывающий сайт антитела можно определить путем идентификации аминокислотного остатка на антигене, соседнем с антителом, с помощью рентгеноструктурного анализа. Например, антитело или его фрагмент и антиген или его фрагмент связывают друг с другом, кристаллизуют и подвергают структурному анализу. Таким образом, можно идентифицировать аминокислотный остаток на антигене, находящийся на расстоянии взаимодействия с антителом. Расстояние взаимодействия составляет 8 Å или менее, предпочтительно, 6 Å или менее, и более предпочтительно, 4 Å или менее. Один или несколько аминокислотных остатков, находящихся на таком расстоянии взаимодействия с антителом, могут составлять антигенсвязывающий сайт антитела (эпитоп). Когда имеется два или несколько таких аминокислотных остатка, такие аминокислотные остатки могут не быть соседними друг с другом в одномерной последовательности.
Анти-HLA/NY-ESO антитело по настоящему изобретению или его связывающий фрагмент специфически распознает множество аминокислот в аминокислотной последовательности HLA/NY-ESO. Антитело или его связывающий фрагмент, которое распознает множество аминокислот, антитело или его связывающий фрагмент, которое связывается с HLA/NY-ESO конкурентно с антителом по настоящему изобретению или его связывающим фрагментом, и антитело или его связывающий фрагмент, который имеет расстояние взаимодействия с таким множеством аминокислот, входит в объем настоящего изобретения. Мультиспецифическое антитело, содержащее такое антитело или его связывающий фрагмент, также входит в объем настоящего изобретения.
6. Фармацевтическая композиция
Настоящее изобретение охватывает противораковый агент, содержащий, в качестве активного ингредиента, анти-HLA/NY-ESO антитело по настоящему изобретению или мультиспецифическую молекулу, которая связывается с HLA/NY-ESO.
Противораковый агент по настоящему изобретению может быть использован для лечения одного или двух или нескольких типов видов рака, выбранных из карциномы, саркомы, лимфомы, лейкоза, миеломы, герминомы, опухоли головного мозга, карциноида, нейробластомы, ретинобластомы и нефробластомы. Конкретные примеры карциномы включают рак почки, меланому, плоскоклеточный рак, базальноклеточный рак, рак конъюнктивы, рак полости рта, рак гортани, рак глотки, рак щитовидной железы, рак легкого (немелкоклеточный рак легкого (аденокарциному, эпидермоидный рак, крупноклеточный рак и мелкоклеточный рак легкого), рак молочной железы, рак пищевода, рак желудка, рак двенадцатиперстной кишки, рак тонкой кишки, рак толстой кишки, рак прямой кишки, рак аппендикса, рак анального канала, рак печени, рак желчного пузыря, рак желчных протоков, рак поджелудочной железы, рак надпочечников, рак мочевого пузыря, рак предстательной железы, рак матки и рак влагалища. Конкретные примеры саркомы включают липосаркому, ангиосаркому, хондросаркому, рабдомиосаркому, саркому Юинга, остеосаркому, недифференцированную плеоморфную саркому, миксофибросаркому, злокачественную периферическую неврилеммому, ретроперитонеальную саркому, синовиосаркому, саркому матки, гастроинтестинальную стромальную опухоль, лейомиосаркому и эпителиоидную саркому. Конкретные примеры лимфомы включают В-клеточную лимфому, Т/NK-клеточную лимфому и лимфому Ходжкина. Конкретные примеры лейкоза включают миелогенный лейкоз, лимфатический лейкоз, миелопролиферативное заболевание и миелодиспластический синдром. Конкретные примеры миеломы включают множественную миелому. Конкретные примеры герминомы включают рак яичек и рак яичников. Конкретные примеры опухоли головного мозга включают нейроглиому и менингиому.
Противораковый агент по настоящему изобретению может содержать анти-HLA/NY-ESO антитело или мультиспецифическую молекулу, которая связывается с HLA/NY-ESO в количестве, эффективном для лечения, а также фармацевтически приемлемые носители, разбавители, солюбилизаторы, эмульгаторы, консерванты, добавки и подобные. «Фармацевтически приемлемые носители» и подобные могут быть подходящим образом выбраны из широкого диапазона в соответствии с типом заболевания-мишени и дозированной формой лекарственного средства. Способ введения противоракового агента по настоящему изобретению может быть выбран подходящим образом. Например, противораковый агент можно вводить инъекцией, и можно применять местную инъекцию, внутрибрюшинную инъекцию, селективную внутривенную инфузию, внутривенную инъекцию, подкожную инъекцию, перфузат органов и подобные. Кроме того, раствор для инъекций может быть составлен с использованием носителя, включающего раствор соли, раствор глюкозы или смесь соленой воды и раствора глюкозы, различные типы буферных растворов и подобные. Кроме того, можно приготовить порошок и смешать его с жидким носителем для приготовления раствора для инъекций перед применением.
Другие способы введения могут быть подходящим образом выбраны наряду с разработкой состава. Например, пероральные растворы, порошки, пилюли, капсулы, таблетки и подобные могут применяться для перорального введения. Что касается пероральных растворов, жидкие пероральные препараты, такие как суспензии и сиропы, могут быть получены с использованием воды, сахаридов, таких как сахароза, сорбит и фруктоза, гликолей, таких как PEG, масел, таких как кунжутное масло и соевое масло, консервантов, таких как алкилпарагидроксибензоаты, ароматизаторов, таких как как аромат клубники и мяты перечной, и подобных. Порошки, пилюли, капсулы и таблетки могут быть приготовлены с использованием эксципиентов, таких как лактоза, глюкоза, сахароза и маннит, разрыхлителей, таких как крахмал и альгинат натрия, смазывающих веществ, таких как стеарат магния и тальк, связующих веществ, таких как поливиниловый спирт, гидроксипропилцеллюлоза, и желатин, поверхностно-активных веществ, таких как сложные эфиры жирных кислот, пластификаторов, таких как глицерин, и подобных. Таблетки и капсулы являются предпочтительными стандартными дозированными формами для композиции по настоящему изобретению, поскольку их легко вводить. Твердые производственные носители используют для производства таблеток и капсул.
Эффективная доза анти-HLA/NY-ESO антитела или мультиспецифической молекулы, которая связывается с HLA/NY-ESO, используемая для лечения, может быть изменена в соответствии с характеристиками симптомов, подлежащих лечению, а также возрастом и состоянием пациента, и может быть окончательно изменена лечащим врачом. Например, одна доза составляет от 0,0001 мг до 100 мг на кг массы тела. Заданную дозу можно вводить один раз каждые 1-180 дней, или доза может быть разделена на две дозы, три дозы, четыре дозы или более доз в день с соответствующими интервалами.
Настоящее изобретение также охватывает полинуклеотид, содержащий аминокислотную последовательность, включенную в анти-HLA/NY-ESO антитело по настоящему изобретению, или мультиспецифическую молекулу, связывающуюся с HLA/NY-ESO, вектор, содержащий такой полинуклеотид, клетку, содержащую такой полинуклеотид или вектор, и фармацевтическую композицию, содержащую в качестве активного ингредиента любой из полинуклеотида, вектора и клетки. Такая фармацевтическая композиция предпочтительно представляет собой противораковый агент.
Кроме того, фармацевтическую композицию по настоящему изобретению можно использовать в сочетании с другими терапевтическими агентами или терапевтическими методами. Примеры других терапевтических агентов или терапевтических методов включают, но не ограничены ими, химиотерапевтические агенты, лучевую терапию и биофармацевтические продукты. Фармацевтическую композицию по настоящему изобретению и другие терапевтические агенты можно вводить одновременно или по разным схемам в виде одного препарата или двух или нескольких разных препаратов, содержащих фармацевтическую композицию по настоящему изобретению. Введение фармацевтической композиции можно проводить в комбинации с другими терапевтическими методами.
Примеры
Далее настоящее изобретение описано более подробно со ссылкой на примеры, хотя настоящее изобретение не ограничено этими примерами.
В следующих примерах, процедуры генной инженерии проводят в соответствии со способом, описанным в Molecular Cloning (Sambrook, J., Fritsch, E. F. and Maniatis, T., Cold Spring Harbor Laboratory Press, 1989), и способами, описанными в других используемых экспериментальных протоколах, применяемых специалистом в данной области, если не указано иное. Когда нужно использовать коммерчески доступный реагент или набор, применяют процедуры в соответствии с инструкциями такого коммерческого продукта. Синтез праймеров, необходимых для синтеза гена или конструирования векторов, по необходимости, передают на аутсорсинг (Fasmac Co., Ltd., Thermo Fisher Scientific и Eurofins Genomics).
(Пример 1) Получение анти-HLA/NY-ESO-антитела, полученного из фаговой библиотеки антител человека
1)-1 Получение антигенного белка HLA/NY-ESO
Тельца включения, полученные из клеток E. coli (BL21 (DE3), Agilent Technologies), экспрессирующих HLA-A*0201 (GenBank: ASA47534.1) усекают (добавляют последовательность распознавания биотинлигазы: SEQ ID NO:33), и β2-микроглобулин (UniProtKB-P61769: SEQ ID NO:34) и пептид NY-ESO: SLLMWITQC (SEQ ID NO:1 из перечня последовательностей) подвергают рефолдингу при высоком коэффициенте разведения, и затем получают пептидный комплекс HLA-A*0201/β2-микроглобин/NY-ESO (далее обозначенный как «HLA/NY-ESO») с использованием колонок для гель-фильтрации (Superdex 200 10/300, GE Healthcare).
В качестве антигена отрицательного контроля получают комплекс HLA-A*0201/β2-микроглобин/пептид MAGEC-1 (далее именуемый «HLA/MC1») путем рефолдинга с использованием пептида MAGEC-1: ILFGISLREV (SEQ ID NO:2 из Перечня последовательностей). Далее, полученные HLA/NY-ESO и HLA/MC1 биотинилируют биотинлигазой E. coli, и затем фракционируют с использованием колонок для гель-фильтрации (Superdex 200 10/300, GE Healthcare) для получения биотинилированных белков.
1)-2 Выделение scFv, способного связываться с HLA/NY-ESO
scFv, связывающийся с HLA/NY-ESO, выделяют из фаговой библиотеки антитела человека. Вначале фаги добавляют к биотинилированным HLA/MC1-иммобилизованным Dynabeads Streptavidin M-280 (Thermo Fisher Scientific) и собирают несвязанные фаги. Затем к биотинилированным HLA/NY-ESO-иммобилизованным Dynabeads Streptavidin M-280Ag добавляют фаги, и не связавшиеся фаги удаляют путем промывки с использованием магнитной подставки (DynaMag-2, Thermo Fisher Scientific).
После этого E.coli (XL-1 Blue, Agilent Technologies) инфицируют фагами, связанными с HLA/NY-ESO, и фаги собирают и амплифицируют. После проведения процедуры пэннинга в общей сложности 3 раза, фаги переносят из поликлональной фагмиды в вектор экспрессии E. coli, содержащий метку FLAG и метку His, добавленную к карбоксильному концу scFv, E. coli трансформируют, scFv экспрессируют в присутствии IPTG (изопропил-β-D-тиогалактопиранозида) (Sigma-Aldrich), и экспрессированный scFv подвергают скринингу с помощью ELISA.
1)-3 Скрининг scFv, связанного с HLA/NY-ESO, с помощью ELISA
NeutrAvidin (Life Technologies), разведенный до 1 мкг/мл в PBS (0,01 М фосфатно-солевом буфере, содержащем 0,138 М хлорида натрия и 0,0027 М хлорида калия (pH 7,4), Sigma-Aldrich), добавляют по 50 мкл каждого в 384-луночный планшет Maxi-sorp (Black, Nunc), и планшет выдерживают при 4°C в течение ночи для иммобилизации. Планшет промывают 3 раза PBS (буфер ELISA), содержащим 0,05% Tween-20 (BioRad), добавляют биотинилированный HLA/NY-ESO, разведенный до 1 мкг/мл в PBS и использованный в примере 1)-2, и полученную смесь перемешивают при комнатной температуре в течение 1 часа. Планшет промывают 3 раза буфером ELISA, блокируют Blocker Casein (Thermo Fisher Scientific) и затем промывают 3 раза буфером ELISA. После этого добавляют культуральный раствор, экспрессирующей scFv E. coli и оставляют реакцию протекать при комнатной температуре на 2 часа. После того, как полученную смесь промывают 3 раза буфером для ELISA, добавляют 50 мкл меченого пероксидазой хрена (HRP) анти-FLAG антитела (Sigma-Aldrich), разведенного в 5000 раз буфером для ELISA, и реакцию продолжают при температуре комнатной температуре в течение 1 часа. После того, как полученный продукт промывают 5 раз буфером ELISA, добавляют хемилюминесцентный субстрат SuperSignal Pico ELISA (Thermo Fisher Scientific), хемилюминесценцию анализируют через 10 минут с использованием планшетного ридера (Envision 2104 Multilabel Reader, Perkin Elmer) и селектируют положительные клоны в HLA/NY-ESO-связывающем ELISA.
1)-4 Определение нуклеотидной последовательности и аминокислотной последовательности ELISA-положительного клона NY-R119
Из числа ELISA-положительных клонов, полученных в 1)-3, NY-R119 выбирают как scFv, демонстрирующий высокую аффинность связывания с HLA/NY-ESO и превосходную специфичность распознавания. Нуклеотидные последовательности вариабельных областей тяжелой цепи и легкой цепи NY-R119 анализируют способом терминатора красителя (BigDye®, Terminator v3.1, Thermo Fisher Scientific). Последовательности праймеров, использованных для анализа последовательности, представлены ниже.
Праймер A: 5'-CTCTTCGTATTACGCCAGCTGGCGA-3' (SEQ ID NO:3 из перечня последовательностей (фиг. 10))
Праймер B: 5'-ATAACAATTTCACACAGGAAACAGCTATGA-3' (SEQ ID NO:4 из перечня последовательностей (фиг. 11))
SEQ ID NO:5 (фиг. 12) показывает определенную нуклеотидную последовательность кДНК, кодирующую вариабельную область тяжелой цепи NY-R119, и SEQ ID NO:6 (фиг. 13) показывает определенную ее аминокислотную последовательность.
SEQ ID NO:7 (фиг. 14) показывает определенную нуклеотидную последовательность кДНК, кодирующую вариабельную область легкой цепи NY-R119, и SEQ ID NO:8 (фиг. 15) показывает определенную ее аминокислотную последовательность.
Последовательности CDR NY-R119 в соответствии с определением CDR, предоставленным IMGT, представлены следующим образом: CDRH1: SEQ ID NO:54 (фиг. 61); CDRH2: SEQ ID NO:55 (фиг. 61); CDRH3: SEQ ID NO:56 (фиг. 61); CDRL1: SEQ ID NO:57 (фиг. 61); CDRL2: SEQ ID NO:58 (фиг. 61); и CDRL3: SEQ ID NO:59 (фиг. 61).
1)-5 Получение NYA-0001
1)-5-1 Конструирование вектора экспрессии NYA-0001
Для приготовления различных образцов для оценки конструируют вектор экспрессии NY-R119 для клеток млекопитающих. NY-R119, экспрессированный в культивируемых клетках млекопитающих, обозначен как «NYA-0001». Аминокислотные последовательности, составляющие область scFv, вариабельные области тяжелой цепи и легкой цепи, и CDRH1-3 и CDRL1-3 NY-R119, идентичны последовательности NYA-0001. Фрагмент ДНК, кодирующий NYA-0001, встраивают в вектор экспрессии для клеток млекопитающих, содержащий pcDNA3.3 (Thermo Fisher Scientific) в качестве скелета, с использованием набора для клонирования In-Fusion HD (CLONTECH) для конструирования вектора экспрессии NYA-0001.
Нуклеотидную последовательность сконструированного вектора экспрессии NYA-0001 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYA-0001 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:69 из перечня последовательностей (фиг. 70). Кроме того, на основе нуклеотидной последовательности обнаруживают, что аминокислотная последовательность всего NYA-0001, закодированного таким образом, представляет собой аминокислотную последовательность, показанную в SEQ ID NO:70 (фиг. 71). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-0001, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
1)-5-2 Экспрессия и очистка NYA-0001
Клетки Expi293F (Thermo Fisher Scientific) субкультивируют в соответствии с инструкциями. Культуральный раствор клеток Expi293F в логарифмической фазе роста разводят до 2,5×106 клеток/мл в среде для экспрессии Expi293 (Thermo Fisher Scientific) и используют для получения NYA-0001. Вектор экспрессии NYA-0001 (0,3 мг) и 0,9 мг полиэтиленимина (Polyscience #24765) добавляют к 20 мл среды Opti-Pro SFM (Thermo Fisher Scientific). Смесь осторожно перемешивают, оставляют на пять минут и затем добавляют к клеткам Expi293F. Культуральный супернатант, полученный встряхиванием культуры в инкубаторе при 37°С в присутствии 8% СО2 при 135 об/мин в течение 6 дней, фильтруют через 0,2 мкм фильтр (Millipore). Таким образом, получают культуральный супернатант NYA-0001. Очистку проводят элюированием и концентрированием с использованием Ni Sepharose excel (GE Healthcare), и затем фильтрованием через колонку для гель-фильтрации (Superdex 200 Increase, GE Healthcare), уравновешенную 25 мМ гистидином, 300 мМ NaCl и 5% сорбитом, pH 6,0. Образцы очищенного белка подвергают аналитической эксклюзионной хроматографии (SEC), определяют степень очистки и концентрацию, и затем образцы подвергают различным видам оценки.
(Пример 2) Получение мутанта NYA-0001
2)-1 Получение мутанта NYA-0001
Фаговую библиотеку конструируют с помощью способа, в котором ген NYA-0001 используют в качестве матрицы, и мутацию вводят с помощью ПЦР (т. е. библиотеки с ошибками), или способом, в котором олигомер синтезируют путем случайной мутации из 20 типов аминокислот для каждого из всех остатков CDR (т.е. библиотеки на основе олигонуклеотидов), селектируют клоны с высокой аффинностью связывания, NYA-0060, NYA-0068 и NYA-0082 получают как мутанты с высокой связывающей способностью затем определяют аффинность и их нуклеотидные последовательности.
SEQ ID NO:9 (фиг. 16) показывает нуклеотидную последовательность кДНК, кодирующую вариабельную область тяжелой цепи NYA-0060, и SEQ ID NO:10 (фиг. 17) показывает ее аминокислотную последовательность.
SEQ ID NO:11 (фиг. 18) показывает нуклеотидную последовательность кДНК, кодирующую вариабельную область легкой цепи NYA-0060, и SEQ ID NO:12 (фиг. 19) показывает ее аминокислотную последовательность.
SEQ ID NO:13 (фиг. 20) показывает нуклеотидную последовательность кДНК, кодирующую вариабельную область тяжелой цепи NYA-0068, и SEQ ID NO:14 (фиг. 21) показывает ее аминокислотную последовательность.
SEQ ID NO:15 (фиг. 22) показывает нуклеотидную последовательность кДНК, кодирующую вариабельную область легкой цепи NYA-0068, и SEQ ID NO:16 (фиг. 23) показывает ее аминокислотную последовательность.
SEQ ID NO:17 (фиг. 24) показывает нуклеотидную последовательность кДНК, кодирующую вариабельную область тяжелой цепи NYA-0082, и SEQ ID NO:18 (фиг. 25) показывает ее аминокислотную последовательность.
SEQ ID NO:19 (фиг. 26) показывает нуклеотидную последовательность кДНК, кодирующую вариабельную область легкой цепи NYA-0082, и SEQ ID NO:20 (фиг. 27) показывает ее аминокислотную последовательность.
2)-2 Получение мутантов с высокой аффинностью связывания с использованием сайтов мутаций и комбинаций, идентифицированных в NYA-0060, NYA-0068 и NYA-0082.
Фрагменты ДНК, кодирующие NYA-1163 и NYA-2023 с сайтами мутаций NYA-0060 и NYA-0068, встраивают в вектор экспрессии для клеток млекопитающих, содержащий pcDNA3.3 (Thermo Fisher Scientific) в качестве скелета, с использованием набора для клонирования In-Fusion HD (CLONTECH) для конструирования векторов экспрессии scFv для культуры клеток млекопитающих.
В соответствии с сайтами мутаций и их комбинаций, идентифицированных в NYA-0060, NYA-0068 и NYA-0082, конструируют NYA-2027, NYA-1143 и NYA-2143. Фрагменты ДНК, кодирующие scFv-мишень, встраивают в вектор экспрессии NYA-0001, сконструированный в 1)-5, посредством сайт-направленного мутагенеза, или вектор экспрессии для клеток млекопитающих, содержащий pcDNA3.3 (Thermo Fisher Scientific) в качестве скелета, с использованием набора для клонирования In-Fusion HD (CLONTECH) для конструирования векторов экспрессии scFv для культуры клеток млекопитающих.
Нуклеотидные последовательности сконструированных векторов экспрессии scFv повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYA-1163 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:21 из перечня последовательностей (фиг. 28).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2023 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:22 из перечня последовательностей (фиг. 29).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2027 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:23 из перечня последовательностей (фиг. 30).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-1143 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:24 из перечня последовательностей (фиг. 31).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2143 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:25 из перечня последовательностей (фиг. 32).
Кроме того, на основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности кодируемых ими полноразмерных NYA-1163, NYA-2023, NYA-2027, NYA-1143 и NYA-2143.
Аминокислотная последовательность полноразмерного NYA-1163 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:26 из перечня последовательностей (фиг. 33). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-1163, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2023 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:27 из перечня последовательностей (фиг. 34). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2023, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2027 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:28 из перечня последовательностей (фиг. 35). В такой аминокислотной последовательности из аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2027, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-1143 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:26 из перечня последовательностей (фиг. 36). В такой аминокислотной последовательности из аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-1143, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2143 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:30 из перечня последовательностей (фиг. 37). В такой аминокислотной последовательности из аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2143, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Все последовательности CDR NYA-1163 идентичны последовательностям CDR NYA-0001. Последовательности CDR NYA-1143, NYA-2143 и NYA-2023 идентичны друг другу, и эти последовательности представлены следующим образом: CDRH1: SEQ ID NO:54 (фиг. 61); CDRH2: SEQ ID NO:55 (фиг. 61); CDRH3: SEQ ID NO:56 (фиг. 61); CDRL1: SEQ ID NO:60 (фиг. 62); CDRL2: SEQ ID NO:58 (фиг. 61); и CDRL3: SEQ ID NO:59 (фиг. 61). Последовательности CDR NYA-2027 представлены следующим образом: CDRH1: SEQ ID NO:54 (фиг. 61); CDRH2: SEQ ID NO:55 (фиг. 61); CDRH3: SEQ ID NO:56 (фиг. 61); CDRL1: SEQ ID NO:57 (фиг. 61); CDRL2: SEQ ID NO:58 (фиг. 61); и CDRL3: SEQ ID NO:61 (фиг. 63).
Кроме того, NYA-1154, содержащий сайты связывания и мутации, наблюдаемые во время скрининга клонов с высокой аффинностью связывания в комбинации, конструируют и вводят в вектор экспрессии NYA-0001 посредством сайт-направленного мутагенеза для конструирования вектора экспрессии NYA-1154, содержащего pcDNA3.3 (Thermo Fisher Scientific) в качестве скелета. Нуклеотидные последовательности сконструированных векторов экспрессии scFv повторно анализируют, и обнаруживают, что они представляют собой нуклеотидную последовательность, показанную в SEQ ID NO:31 (фиг. 38). На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотную последовательность кодируемого ими полноразмерного NYA-1154 (SEQ ID NO:32 (фиг. 39)). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-1154, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Последовательности CDR NYA-1154 представлены следующим образом: CDRH1: SEQ ID NO:54 (фиг. 61); CDRH2: SEQ ID NO:55 (фиг. 61); CDRH3: SEQ ID NO:62 (фиг. 64); CDRL1: SEQ ID NO:57 (фиг. 61); CDRL2: SEQ ID NO:58 (фиг. 61); и CDRL3: SEQ ID NO:63 (фиг. 64).
NYA-1163, NYA-2023, NYA-2027, NYA-1143, NYA-2143 и NYA-1154 экспрессируют таким же образом, как в 1)-5-2, для очистки scFv конструкций-мишеней. Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, и образцы подвергают различным анализам.
2)-3 Получение мутанта NYA-1143
2)-3-1 Получение NYA-2035
NYA-2035 конструируют как мутант NYA-1143 и вводят в вектор экспрессии NYA-1143 для культуры клеток млекопитающих посредством сайт-направленного мутагенеза для создания вектора экспрессии NYA-2035. Нуклеотидную последовательность сконструированного вектора экспрессии scFv повторно анализируют, и обнаруживают, что она представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:35 (фиг. 42). На основе приведенных выше нуклеотидных последовательностей идентифицируют нуклеотидная последовательность кодируемого ими полноразмерного NYA-2025 (SEQ ID NO:36 (фиг. 43)). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2035, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3. Последовательности CDR NYA-2035 в соответствии с определением CDR, предоставленным IMGT, представлены следующим образом: CDRH1: SEQ ID NO:54 (фиг. 61); CDRH2: SEQ ID NO:55 (фиг. 61); CDRH3: SEQ ID NO:56 (фиг. 61); CDRL1: SEQ ID NO:64 (фиг. 65); CDRL2: SEQ ID NO:58 (фиг. 61); и CDRL3: SEQ ID NO:59 (фиг. 61).
2)-3-2 Получение трансплантата CDR NYA-1143 CDR
Для улучшения физических свойств конструируют CDR-мутант NYA-1143. Каркасные последовательности VH и VL NYA-1143 сравнивают с каркасными последовательностями консенсусной последовательности подгруппы человека или последовательностью зародышевой линии, определенной в KABAT et al. (Sequences of Proteins of Immunological Interest, 5th Ed., Public Health Service National Institutes of Health, Bethesda, MD., 1991). В результате, консенсусная последовательность между последовательностью зародышевой линии человека IGHV3_30*15 и подгруппой 3 γ цепи человека выбирают для VH, и последовательность зародышевой линии человека IGLV1-44*01 выбирают для VL в качестве акцепторов, обладающих высокой идентичностью в каркасных областях. Затем аминокислотные остатки в каркасной области акцептора выравнивают с аминокислотными остатками NYA-1143 для идентификации различных аминокислотных остатков.
Затем трехмерную модель NYA-1143 используют для выбора остатков каркаса для переноса на акцептор в соответствии с критериями, предоставленными Queen et al. (Proc. Natl. Acad. Sci. U.S.A., 86, 10029-10033, 1989). В соответствии с методикой, описанной выше, CDR прививают мутантные аминокислотные последовательности VH областей NYA-1143; т.е. конструируют NYA-1143-VH01, NYA-1143-VH02 и NYA-1143-VH03. Такие аминокислотные последовательности представлены в SEQ ID NO:37 (фиг. 44), SEQ ID NO:38 (фиг. 45) и SEQ ID NO:39 (фиг. 46). В качестве мутантной аминокислотной последовательности CDR-трансплантата VL-области NYA-1143, конструируют NYA-1143-VL01. Такая аминокислотная последовательность показана в SEQ ID NO:40 (фиг. 47).
Конструируют различные конструкции scFv с использованием комбинации последовательностей VH и VL. scFv, полученный в результате замены области VH NYA-1143 с аминокислотной последовательностью NYA-1143-VH01, обозначают как NYA-2044. scFv, полученный в результате замены области VL NYA-2044 аминокислотной последовательностью NYA-1143-VL01, обозначают как NYA-2045.
scFv, полученный в результате замены области VH NYA-1143 аминокислотной последовательностью NYA-1143-VH02, обозначают как NYA-2047. scFv, полученный в результате замены области VL NYA-2047 аминокислотной последовательностью NYA-1143-VL01, обозначают как NYA-2048.
scFv, полученный в результате замены области VH NYA-1143 аминокислотной последовательностью NYA-1143-VH03, обозначают как NYA-2060. scFv, полученный в результате замены области VL NYA-2060 аминокислотной последовательностью NYA-1143-VL01, обозначают как NYA-2061.
Фрагменты ДНК различных сконструированных мутантов трансплантата CDR NYA-1143 полностью синтезируют (Fasmac Co., Ltd.) и связывают друг с другом с использованием набора для клонирования In-Fusion HD (CLONTECH) для конструирования вектора экспрессии scFv для клеток млекопитающих, содержащего pcDNA3.3 (Thermo Fisher Scientific) в качестве скелета.
Нуклеотидные последовательности сконструированных векторов экспрессии scFv повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2044 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:41 из перечня последовательностей (фиг. 48).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2045 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:42 из перечня последовательностей (фиг. 49).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2047 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:43 из перечня последовательностей (фиг. 50).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2048 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:44 из перечня последовательностей (фиг. 51).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2060 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:45 из перечня последовательностей (фиг. 52).
Обнаруживают, что нуклеотидная последовательность полноразмерного NYA-2061 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:46 из перечня последовательностей (фиг. 53).
На основе приведенных выше нуклеотидных последовательностей, идентифицируют аминокислотные последовательности кодируемых ими полноразмерных NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060 и NYA-2061. Последовательности CDR NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060 и NYA-2061 идентичны таковым NYA-1143.
Аминокислотная последовательность полноразмерного NYA-2044 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:47 из перечня последовательностей (фиг. 54). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2044, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2045 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:48 из перечня последовательностей (фиг. 55). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2045, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2047 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:50 из перечня последовательностей (фиг. 57). В такой аминокислотной последовательности из аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2047, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2048 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:51 из перечня последовательностей (фиг. 58). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2048, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2060 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:52 из перечня последовательностей (фиг. 59). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2060, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
Аминокислотная последовательность полноразмерного NYA-2061 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:53 из перечня последовательностей (фиг. 60). В такой аминокислотной последовательности из аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-2061, и аминокислоты 267-292 составляют метку Flag-His. Кроме того, аминокислоты 46-53 составляют CDRH1, аминокислоты 71-78 составляют CDRH2, и аминокислоты 117-129 составляют CDRH3. Кроме того, аминокислоты 181-188 составляют CDRL1, аминокислоты 206-208 составляют CDRL2, и аминокислоты 245-256 составляют CDRL3.
2)-3-3 Экспрессия и очистка мутантов NYA-1143
NYA-2035, NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060 и NYA-2061, полученные в 2)-3-1 и 2)-3-2, экспрессируют таким же образом, как в 1)-5-2 для очистки scFv конструкций-мишеней. Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, а затем образцы подвергают различным анализам.
(Пример 3) Получение scFv эталонного анти-HLA/NY-ESO антитела
Конструируют конструкции scFv антител 3M4E5 и T1 с высокой аффинностью связывания с HLA/NY-ESO (WO2010/106431); scFv 3M4E5 обозначают как NYC-0003, и scFv T1 обозначают как NYC-0004.
Фрагменты ДНК NYC-0003 и NYC-0004 полностью синтезируют (Thermo Fisher Scientific), и вектор экспрессии scFv для клеток млекопитающих, содержащий pcDNA3.3 (Thermo Fisher Scientific) в качестве основы, конструируют с использованием набора для клонирования In-Fusion HD (CLONTECH).
Нуклеотидные последовательности сконструированных векторов экспрессии scFv повторно анализируют, и обнаруживают, что нуклеотидные последовательности полноразмерных NYC-0003 и NYC-0004 представляют собой нуклеотидные последовательности, показанные в SEQ ID NO:65 из перечня последовательностей (фиг. 66) и его SEQ ID NO:66 (фиг. 67). На основании нуклеотидных последовательностей обнаруживают, что аминокислотная последовательность кодируемого ими полноразмерного NYC-0003 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:67 (фиг. 68), и обнаруживают, что аминокислотная последовательность полноразмерного NYC-0004 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:68 (фиг. 69).
NYC-0003 и NYC-0004 экспрессируют таким же образом, как в 1)-5-2, для очистки scFv конструкций-мишеней. Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, и затем образцы подвергают различным анализам.
(Пример 4) Оценка аффинности связывания с HLA/NY-ESO с использованием Biacore
С использованием Biacore T200, анти-HLA/NY-ESO scFv захватывают в качестве лиганда иммобилизованным анти-His антителом, и антиген анализируют в качестве анализируемого вещества. В качестве антигена используют HLA/NY-ESO, полученный в 1)-1. Анти-His антитело (набор His Capture, GE Healthcare) иммобилизуют на сенсорном чипе CM5 (GE Healthcare) в соответствии с инструкциями набора. Исследуемые конструкции scFv анти-HLA/NY-ESO, разведенные до 0,5 мкг/мл в HBS-EP+ (GE Healthcare), контактируют с ним со скоростью 10 мкл/мин в течение 60 секунд для иммобилизации. После этого образцы добавляют к анализируемым веществам HLA/NY-ESO, разведенным до различных уровней с помощью HBS-EP+, при скорости потока 30 мкл/мин в течение 120 секунд и анализируют диссоциацию в течение 600 секунд. Результаты расчета, полученные с помощью такого анализа кинетики одиночного цикла, KD, показаны в таблице 1. Обнаруживают, что по сравнению с исходным антителом NYA-0001 все его мутанты демонстрируют более высокую аффинность связывания с HLA/NY-ESO. Кроме того, обнаруживают, что по сравнению с NYC-0004, NYA-1143, NYA-2023, NYA-2143, NYA-2044, NYA-2045, NYA-2060 и NYA-2061 демонстрируют более высокую аффинность связывания и, в частности, NYA-1143, NYA-2044, NYA-2045 и NYA-2143 демонстрируют KD 1 нМ или меньше.
[Таблица 1]
(Пример 5) Анализ распознаваемой аминокислоты в пептиде NY-ESO анти-HLA/NY-ESO scFv
Концентрацию слитых клеток лимфобластов человека Т2 (ATCC) доводят до адекватного уровня в среде AIM-V (Thermo Fisher Scientific), содержащей 20% FBS, раствор пептида NY-ESO (SEQ ID NO:1), точечные мутантные пептиды NY-ESO 1F, 2M, 3A, 4A, 5A, 6L, 7F, 8A и 9A (SEQ ID NO:121 (фиг. 122), SEQ ID NO:122 (фиг. 123), SEQ ID NO:123 (фиг. 124), SEQ ID NO:124 (фиг. 125), SEQ ID NO:125 (фиг. 126), SEQ ID NO:126 (фиг. 127), SEQ ID NO:127 (фиг. 128), SEQ ID NO:128 (фиг. 129) и SEQ ID NO:129 (фиг. 130)), или пептид gp100 (SEQ ID NO:130 (фиг. 131)) (Sigma Genosys), растворенный до 5 мМ в ДМСО добавляют до конечной концентрации 50 мкМ, или добавляют ДМСО в количестве 1/100, и полученную смесь инкубируют при 37°С в течение 4 часов. Полученную смесь дважды промывают в среде AIM-V, содержащей 20% FBS, доводят ее концентрацию до адекватного уровня с помощью PBS, содержащего 5% FBS, добавляют набор LIVE/DEAD Fixable Dead Cell Stain Kit (Thermo Fisher Scientific), и полученный продукт выдерживают при 4°C в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, отмытые клетки делят на две группы, клетки в PBS, содержащем 5% FBS, высевают на 96-луночный микропланшет с U-образным дном в количестве 105 клеток/лунку, и планшет подвергают центрифугированию с последующим удалением супернатанта. scFv анти-HLA/NY-ESO, разведенный до 100 нМ PBS, содержащим 5% FBS, добавляют по 25 мкл/лунку к одной из разделенных клеток, и полученную смесь выдерживают при 4°C в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, добавляют Penta-His Alexa Fluor488 (QIAGEN), разведенный в PBS, содержащем 5% FBS, по 25 мкл/лунку, и полученную смесь выдерживают при 4°C в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, добавляют Alexa Fluor488 против IgG мыши (Thermo Fisher Scientific), разведенный в PBS, содержащем 5% FBS, по 25 мкл/лунку, и полученную смесь выдерживают при 4°С. С в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, иммобилизуют Mildform 10 N (FUJIFILM Wako Pure Chemical Corporation) в течение ночи и ресуспендируют в PBS, содержащем 5% FBS. Для стандартизации количества комплекса HLA/пептид, добавляют HLA-A2-антитело BB7.2-Alexa Fluor 488, разведенное до 10 мкг/мл в PBS, содержащем 5% FBS, или IgG2b-Alexa Fluor 488 мыши в количестве 25 мкл/лунку добавляют к другой группе клеток, и полученную смесь выдерживают при 4°С в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, иммобилизуют Mildform 10 N (FUJIFILM Wako Pure Chemical Corporation) в течение ночи и ресуспендируют в PBS, содержащем 5% FBS. Суспензии клеток подвергают обнаружению с использованием проточного цитометра (CantoII, Becton Dickinson). Анализ данных проводят с использованием Flowjo (Treestar) и измеряют среднюю геометрическую интенсивность флуоресценции (gMFI) Alexa Fluor 488 во фракции Т2-клеток, из которых удалены мертвые клетки. Стандартизированную gMFI, указывающую аффинность связывания каждого scFv, стандартизированного по количеству комплекса HLA/пептид на клетках T2, определяют в соответствии со следующим уравнением.
Стандартизированная gMFI=(A/B)×((C/D)/(E/F))
A/B=относительная gMFI
A: gMFI Т2-клеток с добавлением ДМСО или пептида в присутствии антитела
B: gMFI Т2-клеток с добавлением ДМСО или пептида
(C/D)/(E/F)=скорректированное количество комплекса HLA/пептид T2-клеток, дополненных ДМСО или пептидом.
C: gMFI Т2-клеток с добавлением ДМСО или пептида в присутствии антитела HLA-A2.
D: gMFI Т2-клеток с добавлением ДМСО или пептида в присутствии антитела IgG2b мыши или пептида
E: gMFI Т2-клеток с добавлением ДМСО, содержащего антитело HLA-A2.
F: gMFI Т2-клеток с добавлением ДМСО, содержащего антитело IgG2b мыши.
Как показано на фиг. 1, анти-HLA/NY-ESO scFv: NYA-0001, 1143, 2044, 2045, 2047, 2048, 2060 и 2061 демонстрируют аффинность связывания с Т2-клетками, дополненными пептидами, содержащими точечные мутации, введенные в аминокислоты 1, 4, 5 и 7 уменьшились наполовину или меньше по сравнению с пептидом NY-ESO дикого типа. Это указывает на то, что такой scFv распознает аминокислоты 1, 4, 5 и 7 пептида NY-ESO. Это также указывает на то, что NYA-1154 распознает аминокислоты 1 и 5, NYA-1163 распознает аминокислоты 1, 3, 4, 5, 6 и 7, NYA-2023, 2027 и 2035 распознают аминокислоты 1, 4 и 5, и NYA-2143 распознает аминокислоты 1, 5 и 7. Это также указывает на то, что NYC-0003 и 0004 распознают аминокислоты 4 и 5.
(Пример 6) Оценка антигенсвязывающей специфичности анти-HLA/NY-ESO scFv
Для поиска человеческого пептида, имеющего аминокислотную последовательность, аналогичную, но не идентичную аминокислотной последовательности пептида NY-ESO: SLLMWITQC (SEQ ID NO:1), с которым может связываться антитело (далее такой пептид обозначается как как «гомологичный пептид») среди протеомов человека (Swiss-Prot), проводят поиск 9-mer пептида, содержащего аминокислоты 1, 4 и 5, которые согласуются с пептидами, распознаваемыми антителом. Искомый 9-mer пептид анализируют с использованием NetMHCPan2.8 в отношении аффинности связывания с HLA-A0201, и прогнозируют, что его IC50 составляет 500 нМ или меньше. 9-mer пептид, показанный на фиг. 2А, выбирают в качестве гомологичного пептида для оценки специфичности связывания анти-HLA/NY-ESO scFv. Концентрацию Т2-клеток доводят до адекватного уровня в среде AIM-V (Thermo Fisher Scientific), содержащей 20% FBS, раствор пептида NY-ESO (SEQ ID NO:1), гомологичные пептиды DOLPP1, IL20RB, PRKD2, CD163 и P2RY8 (SEQ ID NO:131 (фиг. 132), SEQ ID NO:132 (фиг. 133), SEQ ID NO:133 (фиг. 134), SEQ ID NO:134 (фиг. 135) и SEQ ID NO:135 (фиг. 136)), или пептид gp100 (SEQ ID NO:130 (фиг. 131)) (Sigma Genosys), растворенный до 5 мМ в ДМСО, добавляют до конечной концентрации 50 мкМ, или ДМСО добавляют в количестве 1/100, и аффинность связывания антител оценивают таким же образом, как в примере 5. Стандартизированную gMFI, указывающую аффинность связывания каждого scFv, стандартизированного с количеством HLA/пептида комплекса на Т2-клетках определяют в соответствии со следующим уравнением.
Стандартизированная gMFI=(A/B)×((C/D)/(E/F))
A/B=относительная gMFI
A: gMFI Т2-клеток с добавлением ДМСО или пептида в присутствии антитела
B: gMFI Т2-клеток с добавлением ДМСО или пептида
(C/D)/(E/F)=скорректированное количество комплекса HLA/пептид Т2-клеток, дополненных ДМСО или пептидом.
C: gMFI Т2-клеток с добавлением ДМСО, содержащего антитело или пептид HLA-A2.
D: gMFI Т2-клеток с добавлением ДМСО или пептида в присутствии антитела IgG2b мыши.
E: gMFI Т2-клеток с добавлением ДМСО, содержащего антитело HLA-A2.
F: gMFI Т2-клеток с добавлением ДМСО, содержащего антитело IgG2b мыши.
Как показано на фиг. 2В, анти-HLA/NY-ESO scFv: NYA-0001, 1143, 1163, 2023, 2027, 2035, 2044, 2045, 2047, 2048, 2060, 2061 и 2143 не связываются с Т2-клетками, дополненными любыми гомологичными пептидами. Это указывает на то, что такой scFv обладает высокой специфичностью. Напротив, обнаруживают, что NYA-1154, NYC-0003 и 0004 связываются с Т2-клетками, дополненными некоторыми гомологичными пептидами.
(Пример 7) Получение Fc конъюгированной анти-HLA/NY-ESO-анти-CD3 биспецифической молекулы
7)-1 Получение Fc-конъюгированного вектора экспрессии биспецифической молекулы анти-HLA/NY-ESO-анти-CD3
7)-1-1 Получение вектора экспрессии биспецифической молекулы TaFv-гетеродимера Fc типа
Чтобы оценить биспецифические молекулы анти-HLA/NY-ESO-анти-CD3 типа taFv-гетеродимера Fc, конструируют векторы экспрессии для различных молекул. Используемые анти-HLA/NY-ESO антитела: NYA-1143, NYA-2143, NYA-1163, NYA-2023, NYA-2027, NYA-2035, NYA-2044, NYA-2045, NYA-2047, NYA-2048, NYA-2060 и NYA-2061. В качестве анти-CD3-антитела используют гуманизированное анти-CD3 scFv; то есть C3E-7085 (WO 2018/117237). В качестве гетеродимерной последовательности Fc используют последовательность Fc, содержащую введенную в нее мутацию для снижения эффекторных функций и образования гетерополимера (WO 2014/190441).
Синтезируют фрагмент ДНК, кодирующий Fc (HC1 или HC2), содержащий введенную в него мутацию для снижения эффекторных функций и образования гетерополимера (Fasmac Co., Ltd.), вектор экспрессии для клеток млекопитающих, содержащий pcDNA3.3 (Thermo Fisher Scientific) в качестве скелета получают с использованием набора для клонирования In-Fusion HD (CLONTECH), и полученный вектор обозначают как «p_HC1».
Получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-1143, лигированный с C3E-7085 с помощью линкера GGGGS, и обозначенный как «p_NYF-0016-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2143, лигированный с C3E-7085 с помощью линкера GGGGS, получают посредством сайт-направленного мутагенеза в p_NYF-0016-HC2 и обозначают как «p_NYF-0019-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-1163, лигированный с C3E-7085 с помощью линкера GGGGS, получают путем замены нуклеотидной последовательности, кодирующей NYA-1143 p_NYF-0016-HC2 с фрагментом ДНК, кодирующим NYA-1163, с использованием набора для клонирования In-Fusion HD (CLONTECH) и обозначают как «p_NYF-0022-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2023, лигированный с C3E-7085 с помощью линкера GGGGS, получают посредством сайт-направленного мутагенеза в p_NYF-0016-HC2 и обозначают как «p_NYF-0023-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2027, лигированный с C3E-7085 с помощью линкера GGGGS, получают посредством сайт-направленного мутагенеза вектора экспрессии для клеток млекопитающих, содержащего фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-0001, лигированный с C3E-7085 линкером GGGGS, и обозначают как «p_NYF-0027-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2035, лигированный с C3E-7085 с помощью линкера GGGGS, получают посредством сайт-направленного мутагенеза в p_NYF-0016-HC2, и обозначают как «p_NYF-0035-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2044, лигированный с C3E-7085 с помощью линкера GGGGS, получают путем замены нуклеотидной последовательности, кодирующей NYA-1143 p_NYF-0016-HC2 с фрагментом ДНК, кодирующим NYA-2044, с использованием набора для клонирования In-Fusion HD (CLONTECH), и обозначают как «p_NYF-0044-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2045, лигированный с C3E-7085 с помощью линкера GGGGS, получают путем замены нуклеотидной последовательности, кодирующей NYA-1143 p_NYF-0016-HC2 с фрагментом ДНК, кодирующим NYA-2045, с использованием набора для клонирования In-Fusion HD (CLONTECH), и обозначают как «p_NYF-0045-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2047, лигированный с C3E-7085 с помощью линкера GGGGS, получают путем замены нуклеотидной последовательности, кодирующей NYA-1143 p_NYF-0016-HC2 с фрагментом ДНК, кодирующим NYA-2047, с использованием набора для клонирования In-Fusion HD (CLONTECH), и обозначают как «p_NYF-0047-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2048, лигированный с C3E-7085 с помощью линкера GGGGS, получают путем замены нуклеотидной последовательности, кодирующей NYA-1143 p_NYF-0016-HC2 с фрагментом ДНК, кодирующим NYA-2048, с использованием набора для клонирования In-Fusion HD (CLONTECH), и обозначают как «p_NYF-0048-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2060, лигированный с C3E-7085 с помощью линкера GGGGS, получают посредством сайт-направленного мутагенеза в p_NYF-0044-HC2, и обозначают как «p_NYF-0060-HC2».
Вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий HC2, интегрированный в карбоксильный конец taFv, содержащий NYA-2061, лигированный с C3E-7085 с помощью линкера GGGGS, получают посредством сайт-направленного мутагенеза в p_NYF-0045-HC2 и обозначают как «p_NYF-0061-HC2».
Нуклеотидную последовательность p_HC1 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного HC1 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:71 из перечня последовательностей (фиг. 72).
Нуклеотидную последовательность p_NYF-0016-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0016-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:72 из перечня последовательностей (фиг. 73).
Нуклеотидную последовательность p_NYF-0019-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0019-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:73 из перечня последовательностей (фиг. 74).
Нуклеотидную последовательность p_NYF-0022-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0022-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:74 из перечня последовательностей (фиг. 75).
Нуклеотидную последовательность p_NYF-0023-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0023-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:75 из перечня последовательностей (фиг. 76).
Нуклеотидную последовательность p_NYF-0027-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0027-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:76 из перечня последовательностей (фиг. 77).
Нуклеотидную последовательность p_NYF-0035-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0035-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:77 из перечня последовательностей (фиг. 78).
Нуклеотидную последовательность p_NYF-0044-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0044-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:78 из перечня последовательностей (фиг. 79).
Нуклеотидную последовательность p_NYF-0045-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0045-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:79 из перечня последовательностей (фиг. 80).
Нуклеотидную последовательность p_NYF-0047-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0047-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:80 из перечня последовательностей (фиг. 81).
Нуклеотидную последовательность p_NYF-0048-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0048-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:81 из перечня последовательностей (фиг. 82).
Нуклеотидную последовательность p_NYF-0060-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0060-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:82 из перечня последовательностей (фиг. 83).
Нуклеотидную последовательность p_NYF-0061-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYF-0061-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:83 из перечня последовательностей (фиг. 84).
На основе приведенных выше нуклеотидных последовательностей, идентифицируют аминокислотные последовательности полноразмерных HC1, NYF-0016-HC2, NYF-0019-HC2, NYF-0022-HC2, NYF-0023-HC2, NYF-0027-HC2, NYF-0035-HC2, NYF-0044-HC2, NYF-0045-HC2, NYF-0047-HC2, NYF-0048-HC2, NYF-0060-HC2 и NYF-0061-HC2, закодированные таким образом.
Аминокислотная последовательность полноразмерного HC1 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:84 из перечня последовательностей (фиг. 85). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, и аминокислоты 20-246 составляют НС.
Аминокислотная последовательность полноразмерного NYF-0016-HC2 показана в SEQ ID NO:85 из перечня последовательностей (фиг. 86). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-1143-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0019-HC2 показана в SEQ ID NO:86 из перечня последовательностей (фиг. 87). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2143-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0022-HC2 показана в SEQ ID NO:87 из перечня последовательностей (фиг. 88). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-1163-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0023-HC2 показана в SEQ ID NO:88 из перечня последовательностей (фиг. 89). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2023-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0027-HC2 показана в SEQ ID NO:89 из перечня последовательностей (фиг. 90). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2027-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0035-HC2 показана в SEQ ID NO:90 из перечня последовательностей (фиг. 91). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2035-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0044-HC2 показана в SEQ ID NO:91 из перечня последовательностей (фиг. 92). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2044-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0045-HC2 показана в SEQ ID NO:92 из перечня последовательностей (фиг. 93). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2045-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0047-HC2 показана в SEQ ID NO:93 из перечня последовательностей (фиг. 94). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2047-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0048-HC2 показана в SEQ ID NO:94 из перечня последовательностей (фиг. 95). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2048-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0060-HC2 показана в SEQ ID NO:95 из перечня последовательностей (фиг. 96). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2060-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYF-0061-HC2 показана в SEQ ID NO:96 из перечня последовательностей (фиг. 97). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2061-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
7)-1-2 Получение вектора экспрессии биспецифической молекулы taFv-Fab-гетеродимера Fc типа
Конструируют вектор экспрессии биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-Fab-гетеродимера Fc типа. В качестве анти-HLA/NY-ESO антитела используют NYA-0001. В качестве анти-CD3-антитела используют гуманизированное анти-CD3 scFv; то есть C3E-7085 (WO 2018/117237). В качестве гетеродимерной последовательности Fc используют последовательность Fc, содержащую введенную в нее мутацию для снижения эффекторных функций и образования гетерополимера (WO 2014/190441).
Получают вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие вариабельную область тяжелой цепи NYA-0001, область CH1, полученную из IgG человека, и область, содержащую мутацию, которая снижает эффекторные функции и кодирует интегрированную в нее делецию HC1-k, и обозначают как «p_NYA-0001-Fab-HC1-k делецию». Кроме того, получают вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие вариабельную область легкой цепи NYA-0001, и интегрированную в нее область CL, полученную из IgG человека, и обозначают как «p_NYA-0001-LC».
Нуклеотидную последовательность делеции p_NYA-0001-Fab-HC1-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции NYA-0001-Fab-HC1-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:97 (фиг. 98) Перечня последовательностей.
Нуклеотидную последовательность p_NYA-0001-LC повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYA-0001-LC представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:98 (фиг. 99) из перечня последовательностей.
На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности кодируемой ими полноразмерной делеции NYA-0001-Fab-HC1-k и NYA-0001-LC.
Аминокислотная последовательность полноразмерной делеции NYA-0001-Fab-HC1-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:99 из перечня последовательностей (фиг. 100). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 20-139 составляют вариабельную область, и аминокислоты 140-468 составляют константную область. Кроме того, аминокислоты 45-52 составляют CDRH1 (SEQ ID NO:54 (фиг. 61)), аминокислоты 70-77 составляют CDRH2 (SEQ ID NO:55 (фиг. 61)), и аминокислоты 116-128 составляют CDRH3 (SEQ ID NO:56 (фиг. 61)).
Аминокислотная последовательность полноразмерного NYA-0001-LC показана в SEQ ID NO:100 из перечня последовательностей (фиг. 101). В такой аминокислотной последовательности, аминокислоты 1-20 составляют сигнальную последовательность, аминокислоты 21-131 составляют вариабельную область, и аминокислоты 132-237 составляют константную область. Кроме того, аминокислоты 46-53 составляют CDRL1 (SEQ ID NO:57 (фиг. 61)), аминокислоты 71-73 составляют CDRL2 (SEQ ID NO:58 (фиг. 61)), и аминокислоты 110-121 составляют CDRL3. (SEQ ID NO:59 (фиг. 61)).
7)-2 Экспрессия Fc-конъюгированной анти-HLA/NY-ESO-анти-CD3-биспецифической молекулы
7)-2-1 Экспрессия биспецифической молекулы taFv-гетеродимера Fc типа
Клетки Expi293F (Thermo Fisher Scientific) субкультивируют в соответствии с инструкциями. Культуральный раствор клеток Expi293F в логарифмической фазе роста разводят до 2,5×106 клеток/мл в среде Expi293 Expression (Thermo Fisher Scientific) и используют для получения различных биспецифических молекул. Смесь (0,3 мг), включающую вектор p_NYF-0016-HC2 и p_HC1 в соотношении 1:1,5 и 0,9 мг полиэтиленимина (Polyscience #24765), добавляют к 20 мл среды Opti-Pro SFM (Thermo Fisher Scientific). Смесь осторожно перемешивают, оставляют на пять минут и затем добавляют к клеткам Expi293F. Культуральный супернатант, полученный встряхиванием культуры в инкубаторе при 37°С в присутствии 8% СО2 при 135 об/мин в течение 6 дней, фильтруют через 0,2 мкм фильтр (Millipore). Таким образом, получают культуральный супернатант биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0016). Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0016, показаны в SEQ ID NO:85 из перечня последовательностей (фиг. 86) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Таким же образом получают культуральный супернатант биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (NYF-0019) с использованием p_NYF-0019-HC2 и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0019, показаны в SEQ ID NO:86 из перечня последовательностей (фиг. 87) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (NYF-0022) готовят с использованием p_NYF-0022-HC2 и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0022, показаны в SEQ ID NO:87 из перечня последовательностей (фиг. 88) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (NYF-0023) получают с использованием p_NYF-0023-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYF-0023, показаны в SEQ ID NO:88 из перечня последовательностей (фиг. 89) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0027) получают с использованием p_NYF-0027-HC2 и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0027, показаны в SEQ ID NO:89 из перечня последовательностей (фиг. 90) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0035) получают с использованием p_NYF-0035-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYF-0035, показаны в SEQ ID NO:90 из перечня последовательностей (фиг. 91) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0044) получают с использованием p_NYF-0044-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYF-0044, показаны в SEQ ID NO:91 из перечня последовательностей (фиг. 92) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (NYF-0045) получают с использованием p_NYF-0045-HC2 и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0045, показаны в SEQ ID NO:92 из перечня последовательностей (фиг. 93) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0047) получают с использованием p_NYF-0047-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYF-0047, показаны в SEQ ID NO:93 из перечня последовательностей (фиг. 94) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0048) получают с использованием p_NYF-0048-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYF-0048, показаны в SEQ ID NO:94 из перечня последовательностей (фиг. 95) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0060) получают с использованием p_NYF-0060-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYF-0060, показаны в SEQ ID NO:95 из перечня последовательностей (фиг. 96) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYF-0061) получают с использованием p_NYF-0061-HC2 и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0061, показаны в SEQ ID NO:96 из перечня последовательностей (фиг. 97) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
7)-2-2 Экспрессия биспецифических молекул taFv-Fab-гетеродимера Fc типа
Таким же образом, как в 7)-2-1, культуральный супернатант биспецифической молекулы taFv-Fab-гетеродимера Fc типа (NYF-0058) получают с использованием векторной смеси, содержащей p_NYF-0023-HC2, p_NYA-0001-Fab-HC1-k делеции и p_NYA-0001-LC в соотношении 1:1:1,5. Аминокислотные последовательности, полученные путем экспрессии векторов, составляющих NYF-0058, представлены аминокислотами 20-745 в SEQ ID NO:88 из перечня последовательностей (фиг. 89), аминокислотами 20-468 в SEQ ID NO:99 из перечня последовательностей (фиг. 100) и аминокислоты 21-237 SEQ ID NO:100 из перечня последовательностей (фиг. 101).
7)-3 Очистка Fc-конъюгированной биспецифической молекулы анти-HLA/NY-ESO-анти-CD3
Различные биспецифические молекулы очищают из культуральных супернатантов, полученных в 7)-2, посредством 2 стадий аффинной хроматографии с белком А и гель-фильтрационной хроматографии.
Культуральный супернатант наносят на колонку MabSelectSuRe, уравновешенную PBS при pH 7,4 (GE Healthcare Bioscience, также называемую просто «GE Healthcare»), чтобы дать возможность биспецифическим молекулам-мишеням адсорбироваться на ней. После удаления не адсорбированных компонентов с помощью PBS, адсорбированные компоненты элюируют ацетатным буфером (рН 3,5). Элюированную фракцию нейтрализуют с помощью Tris буфера (pH 9,5), концентрируют и затем наносят на колонку для гель-фильтрации Superdex 200 10/300 (GE Healthcare Bioscience), уравновешенную 25 мМ гистидином, 300 мМ NaCl, 5% сорбитором при рН 5,5 заранее. Из пиковой фракции, полученной с помощью гель-фильтрационной хроматографии, собирают фракции, эквивалентные гетеродимеру-мишени, и образование биспецифической молекулы-мишени анти-HLA/NY-ESO-анти-CD3 подтверждают с помощью электрофореза на SDS-полиакриламиде (SDS-PAGE). Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, и затем образцы подвергают различным видам оценки.
(Пример 8) Оценка цитотоксичности Fc-конъюгированной биспецифической молекулы анти-HLA-A2/NY-ESO-анти-CD3
8)-1 Получение клеток-мишеней
Эндогенные экспрессирующие NY-ESO клетки человека (U266B1 и NCI-H1703) и эндогенные не экспрессирующие NY-ESO клетки человека (AGS и CFPAC-1) доводят до 1×106 клеток/мл в среде RPMI 1640, содержащей 10% FBS (FUJIFILM Wako Pure Chemical Corporation), 100 мкл радионуклида хрома-51 (PerkinElmer) добавляют на 1 мл каждой клеточной суспензии, и полученные продукты культивируют при 37°C в присутствии 5% CO2 в течение 2 часов. Клетки промывают 2 раза в среде RPMI 1640, содержащей 10% FBS, ресуспендируют до 1×105 клеток/мл в среде RPMI 1640, содержащей 10% FBS, и затем используют в качестве клеток-мишеней.
8)-2 Получение эффекторных клеток
Коммерчески доступные замороженные РВМС человека (Cellular Technology Limited) размораживают при 37°C, переносят в раствор, содержащий среду RPMI 1640, содержащую 10% FBS с добавлением реагента Anti-aggregate Wash (Cellular Technology Limited), промывают 2 раза и доводят до 1×106 клеток/мл в среде RPMI 1640, содержащей 10% FBS, для получения эффекторных клеток.
8)-3 Анализ цитотоксичности
Клетки-мишени, полученные в 8)-1, высевают на 96-луночный микропланшет с U-образным дном при 50 мкл/лунку. Туда добавляют различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3, конъюгированные с Fc, полученные в примере 7, доведенные до различных концентраций в количестве 50 мкл/лунку, туда добавляют эффекторные клетки, полученные в примере 8)-2 при 100 мкл/лунку, центрифугирование проводят при комнатной температуре и 1000 об/мин в течение 1 минуты, и затем инкубируют при 37°С в присутствии 5% СО2 в течение 20-24 часов. Супернатант (50 мкл) собирают на LumaPlate (PerkinElmer), сушат при 50°C в течение примерно 2 часов, и затем анализируют с использованием планшетного ридера (TopCount, PerkinElmer). Тест проводят трижды, и скорость лизиса клеток определяют в соответствии со следующим уравнением.
Скорость лизиса клеток (%)=(A - B) / (C - B) × 100
A: количество лунок для проб
B: Среднее фоновое значение (лунки без добавления антител) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Остальные процедуры такие же, как и для лунок с образцами.
C: среднее значение максимального высвобождения (лунки, в которых клетки-мишени лизируют поверхностно-активным веществом) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Добавляют поверхностно-активное вещество в количестве 100 мкл, фракцию 50 мкл переносят в LumaPlate, как и в случае лунок для образцов, после чего проводят анализы.
Как показано на фиг. 4A - фиг. 4F, различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 демонстрируют цитотоксичность в отношении эндогенных клеток, экспрессирующих NY-ESO человека (U266B1 и NCI-H1703). Результаты расчета EC50, полученные с использованием уравнения четырехпараметрической логистической кривой аналитического программного обеспечения (Sigmaplot, версия 12.0) для клеток U266B1, показаны в таблице 2, и результаты для клеток NCI-H1703 показаны в таблице 3. «Неприменимо (NA)» указывает на невозможность подгонки кривой, поскольку коэффициент корреляции (R) не рассчитан. Как показано на фиг. 4G-4L, напротив, цитотоксичность не наблюдается на эндогенных клетках, не экспрессирующих NY-ESO человека (AGS и CFPAC-1).
[Таблица 2]
[Таблица 3]
(Пример 9) Оценка in vivo активности Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3 в РВМС-трансфицированных моделях человека
Линии клеток плоскоклеточного рака легкого человека NCI-H1703 (ATCC) доводят до 6×107 клеток/мл в PBS, содержащем 50% Matrigel (Corning), и 0,1 мл этого препарата вводят подкожно мышам NOG (самки в возрасте 6-7 недель) (0 день). На 4 день РВМС человека доводят до 3,75×107 клеток/мл в PBS, и 0,2 мл этого соединения вводят внутривенно. Приблизительно через 1 неделю после этого (с 6 по 7 день) начинают измерение большого диаметра (мм) и малого диаметра (мм) опухоли, измерение проводят с помощью электронного цифрового штангенциркуля по прошествии времени, и расчетной объем опухоли рассчитывают в соответствии с приведенным ниже уравнением.
Оценочный объем опухоли (мм3)=средний предполагаемый объем опухоли среди отдельных индивидуумов
Оценочный объем опухоли у человека=большой диаметр × [малый диаметр]2/2
На 14 день мышей делят на группы, каждая из которых состоит из 5 или 6 мышей в зависимости от объема опухоли, и внутривенно вводят различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 (1 мг/кг, 1,5 мг/кг NYF-0058 для сравнения при той же молекулярной массе). Введение проводят на 14, 21 и 28 день. Противоопухолевые эффекты наблюдают в группах лечения, которым вводят различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 (фиг. 5A - фиг. 5С). Ингибирование роста опухоли (%) с 31 дня по 32 день рассчитывают в соответствии со следующим уравнением и отображают в таблице 4.
Ингибирование роста опухоли (%)=100 - (оценочный объем опухоли в группе лечения/оценочный объем опухоли в контрольной группе × 100)
[Таблица 4]
(Пример 10) Получение различных Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3
10)-1 Получение различных векторов экспрессии Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3.
10)-1-1 Получение вектора экспрессии биспецифической молекулы гибридного типа
Для оценки биспецифических молекул анти-HLA/NY-ESO-анти-CD3 гибридного типа конструируют векторы экспрессии для различных молекул. В качестве анти-HLA/NY-ESO антитела используют NYA-1143. В качестве анти-CD3-антитела используют гуманизированное анти-CD3 scFv; т.е. используют C3E-7085 (PCT/JP2017/046006). В качестве гетеродимерной последовательности Fc используют последовательность Fc, содержащую введенную в нее мутацию для снижения эффекторных функций и образования гетерополимера (WO 2014/190441).
Получают вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие вариабельную область тяжелой цепи NYA-1143, область CH1, полученную из IgG человека, и область Fc, в которую введена мутация для снижения эффекторных функций и образования гетерополимера, и обозначают как «p_NYA-1143-Fab-HC1-k делеция». Кроме того, получают вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие вариабельную область легкой цепи NYA-1143, и область CL, полученную из IgG человека, и обозначают как «p_NYA-1143-LC». Кроме того, получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий область Fc, содержащую мутацию, снижающую эффекторные функции и образующую гетерополимер, введенный в карбоксильный конец гуманизированного анти-CD3 scFv (C3E-7085), и обозначают как «p_C3E-7085-HC2-k делеция».
Нуклеотидную последовательность делеции p_NYA-1143-Fab-HC1-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции NYA-1143-Fab-HC1-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:101 из перечня последовательностей (фиг. 102).
Нуклеотидную последовательность p_NYA-1143-LC повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной NYA-1143-LC представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:102 из перечня последовательностей (фиг. 103).
Нуклеотидную последовательность делеции p_C3E-7085-HC2-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции C3E-7085-HC2-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:103 из перечня последовательностей (фиг. 104).
На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности кодируемых ими полноразмерных делеций NYA-1143-Fab-HC1-k, NYA-1143-LC и C3E-7085-HC2-k.
Аминокислотная последовательность полноразмерной делеции NYA-1143-Fab-HC1-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:104 из перечня последовательностей (фиг. 105). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-139 составляют вариабельную область, и аминокислоты 140-468 составляют константную область. Кроме того, аминокислоты 45-52 составляют CDRH1 (SEQ ID NO:54 (фиг. 61)), аминокислоты 70-77 составляют CDRH2 (SEQ ID NO:55 (фиг. 61)), аминокислоты 116-128 составляют CDRH3 ( SEQ ID NO:56 (фиг. 61)).
Аминокислотная последовательность полноразмерного NYA-1143-LC представляет собой аминокислотную последовательность, показанную в SEQ ID NO:105 из перечня последовательностей (фиг. 106). В такой аминокислотной последовательности, аминокислоты 1-20 составляют сигнальную последовательность, аминокислоты 21-131 составляют вариабельную область, и аминокислоты 132-237 составляют константную область. Кроме того, аминокислоты 46-53 составляют CDRL1 (SEQ ID NO:60 (фиг. 62)), аминокислоты 71-73 составляют CDRL2 (SEQ ID NO:58 (фиг. 61)), и аминокислоты 110-121 составляют CDRL3 (SEQ ID NO:59 (фиг. 61)).
Аминокислотная последовательность полноразмерной делеции C3E-7085-HC2-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:106 из перечня последовательностей (фиг. 107). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-260 составляют C3E-7085, аминокислоты 261-262 составляют линкер, и аминокислоты 263-493 составляют Fc, содержащий мутацию, введенную в него для снижения эффекторных функций и образования гетеродимера.
10)-1-2 Получение вектора экспрессии биспецифической молекулы двойного типа
Для оценки биспецифических молекул двойного типа анти-HLA/NY-ESO-анти-CD3 конструируют вектор экспрессии, описанный меньше. Получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий Fc область, содержащую мутацию, снижающую эффекторные функции и образующую гетерополимер, интегрированный в карбоксильный конец NYA-1143, и обозначают как «p_NYA-1143-HC1-k делеция».
Нуклеотидную последовательность делеции p_NYA-1143-HC1-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции NYA-1143-HC1-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:107 из перечня последовательностей (фиг. 108). На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотную последовательность кодированной полноразмерной NYA-1143-HC1-k делеции. Аминокислотная последовательность полноразмерной NYA-1143-HC1-k делеции представляет собой аминокислотную последовательность, показанную в SEQ ID NO:108 (фиг. 109) из перечня последовательностей. В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-266 составляют NYA-1143, аминокислоты 267-268 составляют линкер, аминокислоты 269-499 составляют Fc, содержащую мутацию, введенную в него для снижения эффекторных функций и образования гетеродимера.
10)-1-3 Получение вектора экспрессии биспецифической молекулы scFv-Fab-гетеродимера Fc типа
Для оценки биспецифических молекул анти-HLA/NY-ESO-анти-CD3 scFv-Fab-гетеродимера Fc типа конструируют векторы экспрессии для различных молекул. В качестве анти-HLA/NY-ESO антитела используют NYA-1154, и в качестве анти-CD3 антитела используют C3E-7085. В качестве гетеродимерной последовательности Fc, используют последовательность Fc, содержащую введенную в нее мутацию для снижения эффекторных функций и образования гетерополимера.
Вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие вариабельную область тяжелой цепи NYA-1154, область CH1, полученную из IgG человека, и область Fc, содержащую мутацию, снижающую эффекторные функции, и образующую гетерополимер, интегрированную в карбоксильный конец C3E-7085, получают и обозначают как «p_C3E-7085-NYA-1154-Fab-HC2-k делеция». Кроме того, получают вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие вариабельную область легкой цепи NYA-1154, и интегрированную в нее область CL, полученную из IgG человека, и обозначают как «p_NYA-1154-LC». Кроме того, получают вектор экспрессии для клеток млекопитающих, содержащий интегрированный в него фрагмент ДНК, кодирующий делецию HC1-k, который обозначают как «p_OAA-HC1-k делеция».
Нуклеотидную последовательность p_C3E-7085-NYA-1154-Fab-HC2-k делеции повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции C3E-7085-NYA-1154-Fab-HC2-k представляет собой нуклеотид последовательность, показанной в SEQ ID NO:109 из перечня последовательностей (фиг. 110).
Нуклеотидную последовательность p_NYA-1154-LC повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной NYA-1154-LC представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:110 (фиг. 111) из перечня последовательностей.
Нуклеотидную последовательность p_OAA-HC1-k делеции повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции OAA-HC1-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:111 из перечня последовательностей (фиг. 112).
На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности кодируемых ими полноразмерных делеций C3E-7085-NYA-1154-Fab-HC2-k, NYA-1154-LC и OAA-HC1-k.
Аминокислотная последовательность полноразмерной делеции C3E-7085-NYA-1154-Fab-HC2-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:112 из перечня последовательностей (фиг. 113). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-260 составляют C3E-7085, аминокислоты 261-385 составляют линкер, и аминокислоты 266-385 составляют вариабельную область тяжелой цепи NYA-1154. Кроме того, аминокислоты 386-714 составляют делецию HC2-k.
Аминокислотная последовательность полноразмерного NYA-1154-LC представляет собой аминокислотную последовательность, показанную в SEQ ID NO:113 из перечня последовательностей (фиг. 114). В такой аминокислотной последовательности, аминокислоты 1-20 составляют сигнальную последовательность, аминокислоты 21-131 составляют вариабельную область, и аминокислоты 132-237 составляют константную область. Кроме того, аминокислоты 46-53 составляют CDRL1 (SEQ ID NO:57 (фиг. 61)), аминокислоты 71-73 составляют CDRL2 (SEQ ID NO:58 (фиг. 61)), и аминокислоты 110-121 составляют CDRL3. (SEQ ID NO:63 (фиг. 64)).
Аминокислотная последовательность полноразмерной делеции OAA-HC1-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:114 из перечня последовательностей (фиг. 115). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, и аминокислоты 20-245 составляют делецию OAA-HC1-k.
10)-1-4 Получение векторов экспрессии биспецифических молекул TaFv-гетеродимера Fc типа для оценки различных форматов
Чтобы оценить биспецифические молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа для оценки различных форматов, конструируют векторы экспрессии для различных молекул. В качестве анти-HLA/NY-ESO антител используют NYA-1143 и NYA-1154. В качестве анти-CD3-антитела используют гуманизированное анти-CD3 scFv; т.е. используют C3E-7085. В качестве гетеродимерной последовательности Fc, используют последовательность Fc, содержащую введенную в нее мутацию для снижения эффекторных функций и образования гетерополимера.
Получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий делецию HC2-k, интегрированный в карбоксильный конец taFv, содержащий NYA-1154, лигированный с C3E-7085 с помощью линкера GGGGS, и обозначают как «p_NYF-0010-HC2-k делеция».
Получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий делецию HC2-k, интегрированный в карбоксильный конец taFv, содержащий C3E-7085, лигированный с NYA-1154 с помощью линкера GGGGS, и обозначают как «p_NYF-0004-HC2-k делеция».
Получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий делецию HC2-k, интегрированный в карбоксильный конец taFv, содержащий NYA-1143, лигированный с C3E-7085 с помощью линкера GGGGS, и обозначают как «p_NYF-0011-HC2-k делеция».
Нуклеотидную последовательность делеции p_NYF-0010-HC2-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции NYF-0010-HC2-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:115 из перечня последовательностей (фиг. 116).
Нуклеотидную последовательность делеции p_NYF-0004-HC2-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции NYF-0004-HC2-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:116 из перечня последовательностей (фиг. 117).
Нуклеотидную последовательность делеции p_NYF-0011-HC2-k повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерной делеции NYF-0011-HC2-k представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:117 из перечня последовательностей (фиг. 118).
На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности полноразмерной делеции NYF-0010-HC2-k, делеции NYF-0004-HC2-k и делеции NYF-0011-HC2-k, кодируемые ими.
Аминокислотная последовательность полноразмерной делеции NYF-0010-HC2-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:118 (фиг. 119) из перечня последовательностей. В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-1154-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-744 составляют HC2-k делецию.
Аминокислотная последовательность полноразмерной делеции NYF-0004-HC2-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:119 (фиг. 120) из перечня последовательностей. В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют C3E-7085-NYA-1154 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-744 составляют HC2-k делецию.
Аминокислотная последовательность полноразмерной делеции NYF-0011-HC2-k представляет собой аминокислотную последовательность, показанную в SEQ ID NO:120 из перечня последовательностей (фиг. 121). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-1143-C3E-7085 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2-k делецию.
10)-2 Экспрессия Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3 различных форматов
10)-2-1 Экспрессия биспецифических молекул гибридного типа
Таким же образом, как в 7)-2-1, культуральный супернатант биспецифической молекулы гибридного типа (NYG-3143) получают с использованием векторной смеси, содержащей делецию p_NYA-1143-Fab-HC1-k, делецию p_C3E-7085- HC2-k и p_NYA-1143-LC в соотношении 1:1:1,5. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYG-3143, показаны в SEQ ID NO:104 из перечня последовательностей (фиг. 105), SEQ ID NO:105 из перечня последовательностей (фиг. 106) и SEQ ID NO:106 из перечня последовательностей (фиг. 107).
10)-2-2 Экспрессия биспецифических молекул двойного типа
Таким же образом, как в 7)-2-1, культуральный супернатант биспецифической молекулы двойного типа (NYG-2143) получают с использованием векторной смеси, содержащей делецию p_NYA-1143-HC1-k и делецию p_C3E-7085-HC2-k в соотношении 2:1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYG-2143, показаны в SEQ ID NO:108 из перечня последовательностей (фиг. 109) и в SEQ ID NO:106 из перечня последовательностей (фиг. 107).
10)-2-3 Экспрессия биспецифических молекул scFv-Fab-гетеродимера Fc типа
Таким же образом, как в 7)-2-1, супернатант культуры биспецифической молекулы scFv-Fab-гетеродимер Fc типа (scFv-Fab-Fc типа) (NYF-0003) получают с использованием векторной смеси, содержащей p_C3E-7085-NYA-1154-Fab-HC2-k делецию, p_OAA-HC1-k делецию и p_NYA-1154-LC в соотношении 1:1:1,5. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0003, показаны в SEQ ID NO:112 из перечня последовательностей (фиг. 113), SEQ ID NO:113 из перечня последовательностей (фиг. 114) и SEQ ID NO:114 из перечня последовательностей (фиг. 115).
10)-2-4 Экспрессия биспецифических молекул taFv-гетеродимера Fc типа для оценки различных форматов
Таким же образом, как и в 7)-2-1, культуральный супернатант биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (taFv-Fc типа) (NYF-0010) получают с использованием p_NYF-0010-HC2-k делеции и p_OAA-HC1-k делеции. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0010, показаны в SEQ ID NO:118 из перечня последовательностей (фиг. 119) и в SEQ ID NO:114 из перечня последовательностей (фиг. 115).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 (NYF-0004) taFv (инвертированного)-гетеродимера Fc типа (taFv (инвертированного)-Fc типа) анти-HLA/NY-ESO-анти-CD3 (NYF-0004) получают с использованием p_NYF-0004-HC2-k делеции и p_OAA-HC1-k делеции. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0004, показаны в SEQ ID NO:119 из перечня последовательностей (фиг. 120) и SEQ ID NO:114 из перечня последовательностей (фиг. 115).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-Fc типа (NYF-0011) получают с использованием p_NYF-0011-HC2-k делеции и p_OAA-HC1-k делеции. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYF-0011, показаны в SEQ ID NO:120 из перечня последовательностей (фиг. 121) и в SEQ ID NO:114 из перечня последовательностей (фиг. 115).
10)-3 Очистка Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3 различных форматов
Различные биспецифические молекулы очищают из культуральных супернатантов, полученных в 10)-2-1 и 10)-2-2, посредством 2-стадийной аффинной хроматографии с белком А и хроматографии на керамическом гидроксиапатите.
Культуральный супернатант наносят на колонку MabSelectSuRe, уравновешенную PBS при pH 7,4 (GE Healthcare Bioscience), чтобы дать возможность биспецифическим молекулам-мишеням адсорбироваться на ней. После удаления не адсорбированных компонентов с помощью PBS, адсорбированные компоненты элюируют ацетатным буфером (рН 3,6). Элюированную фракцию нейтрализуют с помощью Tris буфера (pH 9,5) и буфер заменяют 25 мМ гистидином, 150 мМ NaCl, 5% сорбитором при pH 5,5. Раствор фракции-мишени, разведенный в 5 раз буфером, содержащим 10 мМ фосфата калия и 50 мМ MES (pH 6,5), наносят на керамическую гидроксиапатитовую колонку (Bio-Scale CHT Type-1 Hydroxyapatite Column, BioRad Japan), уравновешенную 10 мМ фосфата калия и 50 мМ MES (pH 6,5). Элюирование в линейном градиенте концентрации проводят с использованием хлорида натрия и собирают фракции, эквивалентные гетеродимеру-мишени. Фракции предварительно наносят на колонку для гель-фильтрации Superdex 200 10/300 (GE Healthcare Bioscience), уравновешенную 25 мМ гистидином, 300 мМ NaCl, 5% сорбитором при рН 6,0. Из пиковой фракции, полученной с помощью гель-фильтрационной хроматографии, собирают фракции, эквивалентные гетеродимеру-мишени, и буфер заменяют 25 мМ гистидином, 300 мМ NaCl, 5% сорбитором при рН 5,5. Образование биспецифической молекулы-мишени анти-HLA/NY-ESO-анти-CD3 подтверждают с помощью электрофореза в SDS-полиакриламиде (SDS-PAGE). Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, и затем образцы подвергают различным видам оценки.
Различные биспецифические молекулы очищают из культуральных супернатантов, полученных в 10)-2-3 и 10)-2-4, посредством 2-стадийной аффинной хроматографии с белком А и гель-фильтрационной хроматографии таким же образом, как в 7)-3. Образование биспецифической молекулы-мишени анти-HLA/NY-ESO-анти-CD3 подтверждают с помощью электрофореза в SDS-полиакриламиде (SDS-PAGE). Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, и затем образцы подвергают различным видам оценки.
(Пример 11) Оценка in vitro активности Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3 различных форматов
11)-1 Оценка in vitro активности биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3 гибридного типа, двойного типа и taFv-Fc типа
11)-1-1 Получение клеток-мишеней
U266B1, полученные так же, как в примере 8)-1, используют в качестве клеток-мишеней.
11)-1-2 Получение эффекторных клеток
Коммерчески доступные замороженные РВМС человека (Cellular Technology Limited), полученные по методике примера 8)-2, используют в качестве эффекторных клеток.
11)-1-3 Анализ цитотоксичности
Клетки-мишени, полученные в примере 11)-1-1, высевают на 96-луночный микропланшет с U-образным дном при 50 мкл/лунку. Туда добавляют биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 различных форматов, полученные в примере 10 и доведенные до различных уровней концентрации, в количестве 50 мкл/лунку, эффекторные клетки, полученные в примере 11)-1-2, добавляют по 100 мкл/лунку, центрифугируют при комнатной температуре и 1000 об/мин в течение 1 минуты, и затем инкубируют при 37°С в присутствии 5% СО2 в течение 20-24 часов. Супернатант (50 мкл) собирают на LumaPlate (PerkinElmer), сушат при 50°C в течение примерно 2 часов, и затем анализируют с использованием планшет-ридера (TopCount, PerkinElmer). Тест проводят трижды, и скорость лизиса клеток определяют в соответствии со следующим уравнением.
Скорость лизиса клеток (%)=(A - B) / (C - B) × 100
A: количество лунок для проб
B: Среднее фоновое значение (лунки без добавления антител) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Остальные процедуры такие же, как и для лунок с образцами.
C: среднее значение максимального высвобождения (лунки, в которых клетки-мишени лизируют поверхностно-активным веществом) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Добавляют поверхностно-активное вещество в количестве 100 мкл, фракцию 50 мкл переносят в LumaPlate, как и в случае лунок для образцов, после чего проводят анализы. Как показано на фиг. 7А, тип taFv-Fc демонстрирует цитотоксичность. Результаты расчетов EC50, полученные с использованием уравнения четырехпараметрической логистической кривой аналитического программного обеспечения (Sigmaplot, версия 12.0), показаны в таблице 5. «Не применимо (NA)» означает, что невозможно выполнить подбор кривой, поскольку коэффициент корреляции (R) не рассчитывался. Ни гибридный, ни двойной тип не демонстрируют цитотоксичность.
[Таблица 5]
11)-2 Оценка in vitro активности биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3 scFv-Fab-Fc типа, taFv-Fc типа и taFv (инвертированный)-Fc типа
11)-2-1 Получение клеток-мишеней
U266B1, полученные так же, как в примере 8)-1, используют в качестве клеток-мишеней.
11)-2-2 Получение эффекторных клеток
Коммерчески доступные замороженные РВМС человека (Cellular Technology Limited), полученные по методике примера 8)-2, используют в качестве эффекторных клеток.
11)-2-3 Анализ цитотоксичности
Клетки-мишени, полученные в примере 11)-2-1, высевают на 96-луночный микропланшет с U-образным дном при 50 мкл/лунку. Туда добавляют биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 различных форматов, полученные в примере 10 и доведенные до различных уровней концентрации, в количестве 50 мкл/лунку, эффекторные клетки, полученные в примере 11)-1-2, добавляют по 100 мкл/лунку, центрифугируют при комнатной температуре и 1000 об/мин в течение 1 минуты, и затем инкубируют при 37°С в присутствии 5% СО2 в течение 20-24 часов. Супернатант (50 мкл) собирают на LumaPlate (PerkinElmer), сушат при 50°C в течение примерно 2 часов, и затем анализируют с использованием планшет-ридера (TopCount, PerkinElmer). Тест проводят трижды, и скорость лизиса клеток определяют в соответствии со следующим уравнением.
Скорость лизиса клеток (%)=(A - B) / (C - B) × 100
A: количество лунок для проб
B: Среднее фоновое значение (лунки без добавления антител) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Остальные процедуры такие же, как и для лунок с образцами.
C: среднее значение максимального высвобождения (лунки, в которых клетки-мишени лизируют поверхностно-активным веществом) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Добавляют поверхностно-активное вещество в количестве 100 мкл, фракцию 50 мкл переносят в LumaPlate, как и в случае лунок для образцов, после чего проводят анализы. Как показано на фиг. 7В, тип taFv-Fc проявляет цитотоксичность. Результаты расчетов EC50, полученные с использованием уравнения четырехпараметрической логистической кривой аналитического программного обеспечения (Sigmaplot, версия 12.0), показаны в таблице 6. «Не применимо (NA)» означает, что невозможно выполнить подбор кривой, поскольку коэффициент корреляции (R) не рассчитывался. По сравнению с taFv-гетеродимером Fc типа, scFv-Fab-гетеродимер Fc типа и taFv (инвертированный)-гетеродимер Fc типа демонстрируют более низкую активность.
[Таблица 6]
(Пример 12) Получение различных Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3 посредством мутагенеза и модификации формата NYF-0061
12)-1 Получение различных Fc-конъюгированных векторов экспрессии биспецифических молекул анти-HLA/NY-ESO-анти-CD3 посредством мутагенеза и модификации формата NYF-0061
Для улучшения физических свойств конструируют мутанты NYF-0061 и получают векторы, составляющие молекулы, описанные ниже.
Мутацию вводят в последовательность C3E-7085 вектора p_NYF-0061-HC2, содержащую нуклеотидную последовательность, кодирующую аминокислотную последовательность NYF-0061, и полученный результат обозначают как «p_NYZ-0038-HC2».
Мутацию вводят в последовательность NYA-2061 и последовательность C3E-7085 вектора p_NYF-0061-HC2, содержащую нуклеотидную последовательность, кодирующую аминокислотную последовательность NYF-0061, и полученные результаты обозначают как «p_NYZ-0082-HC2» и «p_NYZ-0083-HC2», соответственно.
Нуклеотидную последовательность p_NYZ-0038-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYZ-0038-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:152 из перечня последовательностей (фиг. 148).
Нуклеотидную последовательность p_NYZ-0082-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYZ-0082-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:153 из перечня последовательностей (фиг. 149).
Нуклеотидную последовательность p_NYZ-0083-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYZ-0083-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:154 из перечня последовательностей (фиг. 150).
На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности кодируемых ими полноразмерных NYZ-0038-HC2, NYZ-0082-HC2 и NYZ-0083-HC2.
Аминокислотная последовательность полноразмерного NYZ-0038-HC2 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:155 из перечня последовательностей (фиг. 151). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-511 составляют NYA-2061-C3E-7096 taFv, аминокислоты 512-513 составляют линкер, и аминокислоты 514-745 составляют HC2.
Аминокислотная последовательность полноразмерного NYZ-0082-HC2 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:156 из перечня последовательностей (фиг. 152). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-516 составляют NYA-3061-C3E-7096 taFv, аминокислоты 517-518 составляют линкер, и аминокислоты 519-750 составляют HC2.
Аминокислотная последовательность полноразмерного NYZ-0083-HC2 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:157 из перечня последовательностей (фиг. 153). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-516 составляют NYA-3061-C3E-7097 taFv, аминокислоты 517-518 составляют линкер, и аминокислоты 519-750 составляют HC2.
Для изучения модификации формата NYF-0061 в scFv-Fab-гетеродимере Fc типа конструируют векторы, составляющие молекулы, описанные ниже.
Вектор экспрессии для клеток млекопитающих, содержащий фрагменты ДНК, кодирующие аминокислотную последовательность, включенную в полипептид, содержащий (i) вариабельную область тяжелой цепи C3E-7085, (ii) CH1 область, полученную из IgG человека, и (iii) Fc область содержащую мутацию для снижения эффекторных функций и образования гетерополимера, интегрированные в указанном порядке в карбоксильный конец NYA-3061, получают и обозначают как «p_NYZ-1010-HC2». Кроме того, получают вектор экспрессии для клеток млекопитающих, содержащий фрагмент ДНК, кодирующий аминокислотную последовательность, включенную в полипептид, содержащий CL область, полученную из IgG человека, добавленную к карбоксильному концу вариабельной области легкой цепи C3E-7085, и обозначают как «p_C3E-7085-LC».
Нуклеотидную последовательность p_NYZ-1010-HC2 повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного NYZ-1010-HC2 представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:158 из перечня последовательностей (фиг. 154).
Нуклеотидную последовательность p_C3E-7085-LC повторно анализируют, и обнаруживают, что нуклеотидная последовательность полноразмерного C3E-7085-LC представляет собой нуклеотидную последовательность, показанную в SEQ ID NO:159 из перечня последовательностей (фиг. 155).
На основе приведенных выше нуклеотидных последовательностей идентифицируют аминокислотные последовательности кодируемых ими полноразмерных NYZ-1010-HC2 и C3E-7085-LC.
Аминокислотная последовательность полноразмерного NYZ-1010-HC2 представляет собой аминокислотную последовательность, показанную в SEQ ID NO:160 из перечня последовательностей (фиг. 156). В такой аминокислотной последовательности, аминокислоты 1-19 составляют сигнальную последовательность, аминокислоты 21-271 составляют NYA-3061, аминокислоты 272-276 составляют линкер, и аминокислоты 277-394 составляют вариабельную область тяжелой цепи C3E-7085. Кроме того, аминокислоты 395-724 составляют константную область.
Аминокислотная последовательность полноразмерного C3E-7085-LC представляет собой аминокислотную последовательность, показанную в SEQ ID NO:161 из перечня последовательностей (фиг. 157). В такой аминокислотной последовательности, аминокислоты 1-20 составляют сигнальную последовательность, аминокислоты 21-127 составляют вариабельную область, и аминокислоты 128-233 составляют константную область. Кроме того, аминокислоты 46-53 составляют CDRL1 (SEQ ID NO:144 (фиг. 142)), аминокислоты 71-73 составляют CDRL2 (SEQ ID NO:145 (фиг. 142)), и аминокислоты 110-117 составляют CDRL3 (SEQ ID NO:146 (фиг. 142)).
12)-2 Экспрессия и очистка различных Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3 посредством мутагенеза и модификации формата или NYF-0061
Таким же образом, как и в 7)-2-1, культуральный супернатант биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (NYZ-0038) получают с использованием p_NYZ-0038-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYZ-0038, показаны в SEQ ID NO:155 из перечня последовательностей (фиг. 151) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера Fc типа (NYZ-0082) получают с использованием p_NYZ-0082-HC2 и p_HC1. Аминокислотные последовательности, полученные с помощью экспрессии векторов, составляющих NYZ-0082, показаны в SEQ ID NO:156 из перечня последовательностей (фиг. 152) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 taFv-гетеродимера типа Fc (NYZ-0083) получают с использованием p_NYZ-0083-HC2 и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYZ-0083, показаны в SEQ ID NO:157 из перечня последовательностей (фиг. 153) и в SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Супернатант культуры биспецифической молекулы анти-HLA/NY-ESO-анти-CD3 scFv-Fab-гетеродимера Fc типа (NYZ-1010) получают с использованием p_NYZ-1010-HC2, p_C3E-7085-LC и p_HC1. Аминокислотные последовательности, полученные посредством экспрессии векторов, составляющих NYZ-1010, представлены в SEQ ID NO:160 из перечня последовательностей (фиг. 156), SEQ ID NO:161 из перечня последовательностей (фиг. 157) и SEQ ID NO:84 из перечня последовательностей (фиг. 85).
Полученные выше культуральные супернатанты очищают таким же образом, как в 7)-3. Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, а затем образцы подвергают различным видам оценки.
(Пример 13) Оценка аффинности связывания с HLA/NY-ESO с использованием Biacore
С использованием Biacore T200, различные Fc-конъюгированные биспецифические молекулы анти-HLA/NY-ESO-анти-CD3 захватывают в качестве лигандов к иммобилизованному антителу против IgG человека (Fc), и антиген анализируют в качестве анализируемого вещества. В качестве антигена используют HLA/NY-ESO, полученный в 1)-1. Антитело против IgG (Fc) человека (набор для захвата антител человека, GE Healthcare) иммобилизуют на сенсорном чипе CM5 (GE Healthcare) в соответствии с инструкциями к набору. Fc-конъюгированные биспецифические молекулы анти-HLA/NY-ESO-анти-CD3, разведенные до 0,5 мкг/мл в HBS-EP+ (GE Healthcare) для оценки, приводят в контакт с ним при 10 мкл/мин в течение 60 секунд для иммобилизации. После этого образцы добавляют к анализируемым веществам HLA/NY-ESO, разведенным до различных уровней с помощью HBS-EP+, при скорости потока 30 мкл/мин в течение 120 секунд и анализируют диссоциацию в течение 600 секунд. Результаты расчетов, полученные с помощью такого анализа кинетики одиночного цикла, KD, показаны в таблице 7. NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 сохраняют аффинность связывания, эквивалентную аффинности связывания NYF-0061.
[Таблица 7]
(Пример 14) Получение слитых Т2-клеток лимфобластов человека с нокаутом CD3e
Для нокаута CD3e в геномной последовательности слитых Т2-клеток лимфобластов человека (ATCC) методом CRISPR, плазмиду экспрессии Cas9 (GE Healthcare) и плазмиду экспрессии енРНК вводят с помощью электропорации (LONZA). После этого проводят клонирование клеток путем ограниченного разведения. Клетки, в которых делеция фрагмента гена-мишени из введенных клеток и экспрессия гена CD3e не наблюдается в результате анализа генома и ОТ-ПЦР анализа общей РНК, соответственно, подвергают последующим экспериментам.
(Пример 15) Анализ распознавания аминокислоты в пептиде NY-ESO Fc-конъюгированной биспецифической молекулы анти-HLA-A2/NY-ESO-анти-CD3
Концентрацию Т2-клеток с нокаутом CD3e, полученных в примере 14, доводят до адекватного уровня в среде AIM-V (Thermo Fisher Scientific), содержащей 20% FBS, пептид NY-ESO (SEQ ID NO:1), пептиды NY-ESO с точечными мутациями 1F, 2M, 3A, 4A, 5A, 6L, 7F, 8A и 9A (SEQ ID NO:121 (фиг. 122), 122 (фиг. 123), 123 (фиг. 124), 124 (фиг. 125), 125 (фиг. 126), 126 (фиг. 127), 127 (фиг. 128), 128 (фиг. 129) и 129 (фиг. 130)) и пептид gp100 (SEQ ID NO:130 (фиг. 131)) (Sigma Genosys) добавляют так же, как в примере 5, клетки, окрашенные с использованием набора LIVE/DEAD Fixable Dead Cell Stain Kit (Thermo Fisher Scientific), делят на две группы, клетки в PBS, содержащем 5% FBS, высевают на 96-луночный микропланшет с U-образным дном в количестве 105 клеток/лунку, и планшет подвергают центрифугированию с последующим удалением надосадочной жидкости. Различные Fc-конъюгированные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3, полученные в примере 12 и разведенные до 100 нМ с помощью PBS, содержащего 5% FBS, добавляют по 25 мкл/лунку к одной из двух групп, и полученный продукт выдерживают при 4°С в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, добавляют PE-конъюгированный F(ab')2 фрагмент против IgG человека (Jackson Immuno Research Laboratories), разведенный PBS, содержащим 5% FBS, по 25 мкл/лунку, и полученный продукт выдерживают при 4°С в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, иммобилизуют Mildform 10 N (FUJIFILM Wako Pure Chemical Corporation) в течение ночи и ресуспендируют в PBS, содержащем 5% FBS. Для стандартизации по количеству комплекса HLA/пептид добавляют HLA-A2-антитело BB7.2-Alexa Fluor 488, разведенное до 10 мкг/мл в PBS, содержащем 5% FBS, или IgG2b-Alexa Fluor 488 мыши (BioRAD) при 25 мкл/лунку, и полученную смесь выдерживают при 4°С в течение 30 минут. Клетки промывают 2 раза в PBS, содержащем 5% FBS, иммобилизуют Mildform 10 N (FUJIFILM Wako Pure Chemical Corporation) в течение ночи и ресуспендируют в PBS, содержащем 5% FBS. Суспензии клеток подвергают обнаружению с использованием проточного цитометра (CantoII, Becton Dickinson). Анализ данных проводят с использованием Flowjo (Treestar) и измеряют среднюю геометрическую интенсивность флуоресценции (gMFI) PE или Alexa Fluor 488 в Т2-клетках с нокаутом CD3e, из которых удаляют мертвые клетки. Стандартизированный gMFI, показывающий аффинность связывания каждой Fc-конъюгированной биспецифической молекулы анти-HLA-A2/NY-ESO-анти-CD3, стандартизированной по количеству комплекса HLA/пептид на Т2-клетках с нокаутом CD3e, определяют в соответствии со следующим уравнением.
Стандартизированная gMFI=(A/B)×((C/D)/(E/F))
A/B=относительная gMFI
A: gMFI PE в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом в присутствии антитела
B: gMFI PE в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом.
(C/D)/(E/F)=скорректированное количество комплекса HLA/пептид в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом.
C: gMFI Alexa488 в Т2-клетках с нокаутом по CD3e, дополненных ДМСО или пептидом в присутствии антитела к HLA-A2.
D: gMFI Alexa488 в Т2-клетках с нокаутом по CD3e, дополненных ДМСО или пептидом в присутствии антитела IgG2b мыши.
E: gMFI Alexa488 в Т2-клетках с нокаутом CD3e, дополненных ДМСО, содержащим антитело HLA-A2.
F: gMFI Alexa488 в Т2-клетках с нокаутом CD3e, дополненных ДМСО, содержащим антитело IgG2b мыши.
Как показано на фиг. 158А, Fc-конъюгированные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3; т.е. NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 демонстрируют аффинность связывания с Т2-клетками с нокаутом CD3e, дополненными пептидами, содержащими точечные мутации, введенные в аминокислоты 1, 4, 5 и 7, которые имели была снижены до четверти аффинности связывания для пептида NY-ESO дикого типа или меньше. Это указывает на то, что такие конструкции хорошо распознают аминокислоты 1, 4, 5 и 7 пептида NY-ESO.
(Пример 16) Оценка антигенсвязывающей специфичности Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3
Таким же образом, как и в примере 6), 9-mer пептид, показанный на фиг. 2А, проявляет идентичность последовательности пептида NY-ESO: SLLMWITQC (SEQ ID NO:1) в аминокислотах 1, 4 и 5, которые, как показано, хорошо распознаются четырьмя типами антител, выбирают в качестве гомологичного пептида и оценивают специфичность связывания с ним. Концентрацию Т2-клеток с нокаутом CD3e доводят до адекватного уровня в среде AIM-V (Thermo Fisher Scientific), содержащей 20% FBS, раствор пептида NY-ESO (SEQ ID NO:1), гомологичные пептиды DOLPP1, IL20RB, PRKD2, CD163 и P2RY8 (SEQ ID NO:131 (фиг. 132), SEQ ID NO:132 (фиг. 133), SEQ ID NO:133 (фиг. 134), SEQ ID NO:134 (фиг. 135) и SEQ ID NO:135 (фиг. 136)), или пептид gp100 (SEQ ID NO:130 (фиг. 131)) (Sigma Genosys), растворенный до 5 мМ в ДМСО, добавляют для получения конечной концентрации 50 мкМ или ДМСО в количестве 1/100, и аффинность связывания антител оценивают таким же образом, как в примере 15. Стандартизированную gMFI, указывающую аффинность связывания каждой Fc-конъюгированной биспецифической молекулы анти-HLA-A2/NY-ESO-анти-CD3, стандартизированной по количеству комплекса HLA/пептид на Т2-клетках с нокаутом CD3e, определяют в соответствии со следующим уравнением.
Стандартизированная gMFI=(A/B)×((C/D)/(E/F))
A: gMFI PE в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом в присутствии антитела
B: gMFI PE в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом.
(C/D)/(E/F)=скорректированное количество комплекса HLA/пептид в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом.
C: gMFI Alexa488 в Т2-клетках с нокаутом по CD3e, дополненных ДМСО или пептидом в присутствии HLA-A2 антитела.
D: gMFI Alexa488 в Т2-клетках с нокаутом CD3e, дополненных ДМСО или пептидом, содержащим антитело IgG2b мыши.
E: gMFI Alexa488 в Т2-клетках с нокаутом CD3e, дополненных ДМСО, содержащим HLA-A2 антитело.
F: gMFI Alexa488 в Т2-клетках с нокаутом CD3e, дополненных ДМСО, содержащим антитело IgG2b мыши.
Как показано на фиг. 158В, ни одна из Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3; т.е. NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 не связывается с клетками, дополненными гомологичными пептидами, и эти молекулы демонстрируют очень высокую специфичность.
(Пример 17) Оценка цитотоксичности Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3
17)-1 Получение клеток-мишеней
Таким же образом, как в примере 8)-1, суспензии эндогенных клеток, экспрессирующих NY-ESO человека (U266B1 и NCI-H1703), и клеток, не экспрессирующих эндогенный NY-ESO человека (AGS и CFPAC-1), получают и используют в качестве клеток-мишеней.
17)-2 Получение эффекторных клеток
Таким же образом, как в примере 8)-2, получают суспензию коммерчески доступных замороженных РВМС человека (Cellular Technology Limited) и используют в качестве эффекторных клеток.
17)-3 Анализ цитотоксичности
Клетки-мишени, полученные в примере 17)-1, высевают на 96-луночный микропланшет с U-образным дном при 50 мкл/лунку. Туда добавляют различные Fc-конъюгированные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3, полученные в примере 12 и доведенные до различных уровней концентрации, в количестве 50 мкл на лунку, туда добавляют эффекторные клетки, полученные в примере 17)-2, по 100 мкл/лунку, центрифугируют при комнатной температуре и 1000 об/мин в течение 1 минуты, и затем инкубируют при 37°С в присутствии 5% СО2 в течение 20-24 часов. Супернатант (50 мкл) собирают на LumaPlate (PerkinElmer), сушат при 50°C в течение примерно 2 часов, и затем анализируют с использованием планшетного ридера (TopCount, PerkinElmer). Тест проводят трижды, и скорость лизиса клеток определяют в соответствии со следующим уравнением.
Скорость лизиса клеток (%)=(A - B)/(C - B) × 100
A: количество лунок для проб
B: Среднее фоновое значение (лунки без добавления антител) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Остальные процедуры такие же, как и для лунок с образцами.
C: среднее значение максимального высвобождения (лунки, в которых клетки-мишени лизируют поверхностно-активным веществом) (n=3).
Когда добавляют антитело, добавляют 50 мкл среды для анализа. Добавляют поверхностно-активное вещество в количестве 100 мкл, фракцию 50 мкл переносят в LumaPlate, как и в случае лунок для образцов, после чего проводят анализы.
Как показано на фиг. 159A - фиг. 159D, различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 демонстрируют цитотоксичность в отношении эндогенных клеток, экспрессирующих NY-ESO человека (U266B1 и NCI-H1703). Результаты расчетов EC50, полученные с использованием уравнения четырехпараметрической логистической кривой аналитического программного обеспечения (Sigmaplot, версия 12.0) для клеток U266B1, показаны в таблице 8, и результаты для клеток NCI-H1703 показаны в таблице 9. Как показано на фиг. 159Е - фиг. 159Н, наоборот, цитотоксичность не наблюдается на эндогенных клетках, не экспрессирующих NY-ESO человека (AGS и CFPAC-1).
[Таблица 8]
[Таблица 9]
(Пример 18) Оценка in vivo активности Fc-конъюгированных биспецифических молекул анти-HLA-A2/NY-ESO-анти-CD3 в РВМС-трансфицированных моделях человека
Линии клеток плоскоклеточного рака легкого человека NCI-H1703 (ATCC) доводят до 6×107 клеток/мл в PBS, содержащем 50% Matrigel (Corning), и 0,1 мл этого раствора вводят подкожно мышам NOG (самки, в возрасте 6-7 недель) (0 день). На 4 день, РВМС человека доводят до 3,75×107-5×107 клеток/мл в PBS и 0,2 мл этого соединения вводят внутривенно. Приблизительно через 1 неделю после этого (6 день - 7 день) начинают измерение большого диаметра (мм) и малого диаметра (мм) опухоли, измерение проводят с помощью электронного цифрового штангенциркуля по прошествии времени, и оценочный объем опухоли рассчитывают в соответствии с приведенным ниже уравнением.
Оценочный объем опухоли (мм3)=средний оценочный объем опухоли среди индивидуумов.
Оценочный объем опухоли у человека=большой диаметр × [малый диаметр]2/2
На 14 день мышей делят на группы, каждая из которых состоит из 5 или 6 мышей в зависимости от объема опухоли, и внутривенно вводят различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 (NYZ-1010 в дозе 1 мг/кг, NYZ-1010 в дозе 1,2 мг/кг для сравнения при одинаковой молекулярной массе). Введение проводят на 14 и 21 день. Противоопухолевые эффекты наблюдают в группах лечения, которым вводят различные биспецифические молекулы анти-HLA-A2/NY-ESO-анти-CD3 (фиг. 160A - фиг. 160D). Ингибирование роста опухоли (%) с 28 дня по 29 день рассчитывают в соответствии со следующим уравнением, и оно показано в таблице 10.
Ингибирование роста опухоли (%)=100 - (оценочный объем опухоли в группе лечения/оценочный объем опухоли в контрольной группе × 100)
[Таблица 10]
(Пример 19) Сравнение физико-химических свойств различных Fc-конъюгированных биспецифических молекул анти-HLA/NY-ESO-анти-CD3
19)-1 Оценка обработки кислотой
NYF-0016 и различные трансплантированные CDR-мутанты NYA-1143 (NYF-0044, NYF-0045, NYF-0047, NYF-0048, NYF-0060 и NYF-0061), полученные в 7)-2-1, концентрируют центрифугированием с использованием Amicon (Millipore), буфер заменяют 25 мМ ацетатом натрия, 5% сорбитом (pH 5,5) и доводят концентрацию до 15 мг/мл (NYF-0016 при 10 мг/мл). Затем образцы подвергают диализу с использованием Xpress Micro Dialyzer (Scienova) против 100 мМ ацетата натрия (рН 3,5) или 25 мМ ацетата натрия, 5% сорбита (рН 5,5) в качестве контроля, образцы собирают и выдерживают при комнатной температуре в течение 1 часа. Затем уровни pH образцов доводят до 5,0 с использованием 500 мМ Tris-HCl (pH 9,0). Образцы анализируют с помощью эксклюзионной хроматографии (SEC) с использованием ACQUITY UPLC BEH200 SEC 1,7 мкм 4,6*150 мм (Waters). В качестве подвижной фазы используют 0,2 М Ki/200 мМ KCl/pH 7,0, и анализ проводят при скорости потока 0,2 мл/мин (длина волны обнаружения: 280 нм). Пиковое содержание (%) полимеров, содержащихся в образцах, анализируют и рассчитывают способом процента площади. Результаты представлены на фиг. 162. В результате обработки кислотой, содержание полимера в NYF-0016 увеличивается до 96%. Напротив, в различных привитых мутантах NYA-1143 CDR количество образующегося полимера при кислотной обработке уменьшается. В частности, в NYF-0047, NYF-0048, NYF-0060 и NYF-0061 количество образующегося полимера при обработке кислотой снижается до 1% или меньше. Это свидетельствует о значительном улучшении кислотостойкости.
19)-2 Оценка стабильности раствора
NYF-0061, полученный в 7)-2-1, и NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010, полученный в 12)-2, концентрируют центрифугированием с использованием Amicon Ultra-4 (Millipore), буфер заменяют 25 мМ гистидином, 5% сорбитом (рН 6,0), туда добавляют 25 мМ гистидина и 5% сорбит (рН 6,0), и концентрацию образца доводят до 25 мг/мл для приготовления оценочных образцов. Сначала, высокомолекулярные соединения (HMWS) каждого оценочного образца определяют способом процента площади с помощью эксклюзионной хроматографии с использованием AdvanceBio SEC 300A 2,7 мкм 4,6×300 мм (Agilent). В качестве подвижной фазы используют 0,2 М Ki/200 мМ KCl/pH 7,0, и анализ проводят при скорости потока 0,2 мл/мин (длина волны обнаружения: 280 нм). Испытание стабильности раствора каждого образца проводят путем хранения при 25°C в течение 6 дней (NYF-0061: 7 дней), эксклюзионной хроматографией в условиях, описанных выше, и вычислением HMWS (%) каждого образца способом процента площади. Результаты показаны в таблице 11. HMWS (%) NYF-0061 увеличивается приблизительно до 10,6% по мере истечения времени хранения. Напротив, увеличение HMWS (%) NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 составляет приблизительно 2% или меньше. Это показывает, что стабильность растворов NYZ-0038, NYZ-0082, NYZ-0083 и NYZ-1010 выше, чем у NYF-0061.
[Таблица 11]
(Пример 20) Получение анти-HLA/NY-ESO scFv
20)-1 Конструирование вектора экспрессии анти-HLA/NY-ESO scFv
Конструируют scFv конструкции антител, которые могут иметь высокую аффинность связывания с HLA-A2/NY-ESO-1; т.е., mAb24955N, mAb24956N, mAb28075P, mAb28105P, mAb28113P и mAb29822P2 (WO2021/003357), и scFv mAb24955N обозначают как NYC-0005, scFv mAb24956N обозначают как NYC-0006, scFv mAb28075P обозначают как NYC-0007, scFv mAb28105P обозначают как NYC-0008, scFv mAb28113P обозначают как NYC-0009 и scFv mAb29822P2 обозначают как NYC-0010.
Кроме того, конструируют векторы экспрессии scFv для NYC-0005, NYC-0006, NYC-0007, NYC-0008, NYC-0009 и NYC-0010, каждый из которых содержит pcDNA3.4 (ThermoFisher Scientific) в качестве скелета в клетках млекопитающих. Для того чтобы сравнить уровни экспрессии одних и тех же векторных скелетов и форматов в клетках млекопитающих, кроме того, конструируют векторы экспрессии scFv для NYA-2047, NYA-2061 и NYA-3061 в клетках млекопитающих.
Нуклеотидные последовательности сконструированных векторов экспрессии scFv повторно анализируют, и нуклеотидные последовательности полноразмерных NYA-2047, NYA-2061, NYA-3061, NYC-0005, NYC-0006, NYC-0007, NYC-0008, NYC-0009 и NYC-0010 оказались нуклеотидными последовательностями, показанными в SEQ ID NO:43 (фиг. 50), SEQ ID NO:46 (фиг. 53), SEQ ID NO:163 (фиг. 164), SEQ ID NO:165 (фиг. 166), SEQ ID NO:167 (фиг. 168), SEQ ID NO:169 (фиг. 170), SEQ ID NO:171 (фиг. 172), SEQ ID NO:173 (фиг. 174) и SEQ ID NO:175 (фиг. 176) из перечня последовательностей. На основе нуклеотидных последовательностей, указанных выше, аминокислотные последовательности, кодируемые ими, являются следующими: аминокислотная последовательность полноразмерного NYA-2047 показана в SEQ ID NO:50 (фиг. 57); аминокислотная последовательность полноразмерного NYA-2061 показана в SEQ ID NO:53 (фиг. 60); аминокислотная последовательность полноразмерного NYA-3061 показана в SEQ ID NO:164 (фиг. 165); аминокислотная последовательность полноразмерного NYC-0005 показана в SEQ ID NO:166 (фиг. 167); аминокислотная последовательность полноразмерного NYC-0006 показана в SEQ ID NO:168 (фиг. 169); аминокислотная последовательность полноразмерного NYC-0007 показана в SEQ ID NO:170 (фиг. 171); аминокислотная последовательность полноразмерного NYC-0008 показана в SEQ ID NO:172 (фиг. 173); аминокислотная последовательность полноразмерного NYC-0009 показана в SEQ ID NO:174 (фиг. 175); и аминокислотная последовательность полноразмерного NYC-0010 показана в SEQ ID NO:176 (фиг. 177).
20)-2 Экспрессия и очистка анти-HLA/NY-ESO scFv
Культивирование и трансфекцию гена в клетках Expi293F (ThermoFisher Scientific) проводят таким же образом, как описано в 1)-5-2. Для оценки количества продуцирования, клетки подвергают встряхиванию культуры в инкубаторе при 37°С в присутствии 8% СО2 при 135 об/мин в течение 4 дней после введения гена, культуральный супернатант фильтруют через 0,2 мкм фильтр (ThermoFisher Scientific), и получают культуральный супернатант scFv анти-HLA/NY-ESO. Очистку проводят с использованием Ni Sepharose excel (Cytiva), элюат подвергают аналитической эксклюзионной хроматографии (SEC) и определяют концентрацию. В таблице 12 показаны объемы продуцирования. Количество продуцирования NYA-2047, NYA-2061 и NYA-3061, определенное с использованием Ni Sepharose excel, относительно 1 л культурального супернатанта в очищенном элюате, выше, чем у NYC-0005, NYC-0006, NYC-0007, NYC-0008, NYC-0009 и NYC-0010.
[Таблица 12]
Элюат каждого scFv, полученного с использованием Ni Sepharose excel, концентрируют и очищают через колонку для гель-фильтрации (Superdex 200 Increase, Cytiva), уравновешенную 25 мМ гистидином, 300 мМ NaCl, 5% сорбитором (pH 5,5). Образцы очищенного белка подвергают аналитической SEC, определяют степень очистки и концентрацию, и затем образцы подвергают различным видам оценки.
(Пример 21) Экспрессия и очистка scFv-гетеродимера-Fc анти-HLA/NY-ESO
21)-1 Конструирование вектора экспрессии scFv-гетеродимер-Fc анти-HLA/NY-ESO
Для сравнения продуктивности, полученной в результате различных форматов, вектор экспрессии конструируют путем превращения формата scFv анти-HLA/NY-ESO в слитый тип Fc. В качестве гетеродимерных Fc последовательностей (далее обозначенных как HC-h и HC-k), используют те, о которых сообщается в Nat. Biotechnol., Jul 1998, 16 (7), 677-81.
Конструируют конструкции антител scFv-гетеродимер-Fc, которые могут обладать высокой аффинностью связывания с HLA-A2/NY-ESO-1; т.е. mAb24955N, mAb24956N, mAb28075P, mAb28105P, mAb28113P и mAb29822P2 (WO 2021/003357), и scFv-гетеродимер-Fc mAb24955N обозначают как NYC-0011, scFv-гетеродимер-Fc mAb24956N обозначают как NYC-0012, scFv-гетеродимер-Fc mAb28075P обозначают как NYC-0013, scFv-гетеродимер-Fc mAb28105P обозначают как NYC-0014, scFv-гетеродимер-Fc mAb28113P обозначают как NYC-0015 и scFv-гетеродимер-Fc mAb29822P2 обозначают как NYC-0016. Также, scFv-гетеродимер-Fc NYA-2047 обозначают как NYD-2047, scFv-гетеродимер-Fc NYA-2061 обозначают как NYD-2061 и scFv-гетеродимер-Fc NYA-3061 обозначают как NYD-3061.
Конструируют векторы экспрессии для HC-h, NYC-0011-HC-k, NYC-0012-HC-k, NYC-0013-HC-k, NYC-0014-HC-k, NYC-0015-HC-k и NYC- 0016-HC-k, каждый из которых содержит pcDNA3.3 или pcDNA3.4 (ThermoFisher Scientific) в качестве скелета в клетках млекопитающих. Кроме того, векторы экспрессии NYD-2047-HC-k, NYD-2061-HC-k и NYD-3061-HC-k в клетках млекопитающих конструируют для сравнения уровней экспрессии в клетках млекопитающих с одинаковыми скелетами и форматами векторов.
Нуклеотидные последовательности сконструированных векторов экспрессии scFv-гетеродимера-Fc, и нуклеотидные последовательности полноразмерных HC-h, NYD-2047-HC-k, NYD-2061-HC-k, NYD-3061-HC-k, NYC-0011-HC-k, NYC-0012-HC-k, NYC-0013-HC-k, NYC-0014-HC-k, NYC-0015-HC-k и NYC-0016-HC-k представляют собой нуклеотидные последовательности, показанные в SEQ ID NO:177 (фиг. 178), SEQ ID NO:179 (фиг. 180), SEQ ID NO:181 (фиг. 182), SEQ ID NO:183 (фиг. 184), SEQ ID NO:185 (фиг. 186), SEQ ID NO:187 (фиг. 188), SEQ ID NO:189 (фиг. 190), SEQ ID NO:191 (фиг. 192), SEQ ID NO:193 (фиг. 194) и SEQ ID NO:195 (фиг. 196) из перечня последовательностей. На основе нуклеотидных последовательностей, указанных выше, кодируемые ими полноразмерные аминокислотные последовательности являются следующими: аминокислотная последовательность всего HC-h показана в SEQ ID NO:178 (фиг. 179); аминокислотная последовательность полноразмерного NYD-2047-HC-k показана в SEQ ID NO:180 (фиг. 181); аминокислотная последовательность полноразмерного NYD-2061-HC-k показана в SEQ ID NO:182 (фиг. 183); аминокислотная последовательность полноразмерного NYD-3061-HC-k показана в SEQ ID NO:184 (фиг. 185); аминокислотная последовательность полноразмерного NYC-0011-HC-k показана в SEQ ID NO:186 (фиг. 187); аминокислотная последовательность полноразмерного NYC-0012-HC-k показана в SEQ ID NO:188 (фиг. 189); аминокислотная последовательность полноразмерного NYC-0013-HC-k показана в SEQ ID NO:190 (фиг. 191); аминокислотная последовательность полноразмерного NYC-0014-HC-k показана в SEQ ID NO:192 (фиг. 193); аминокислотная последовательность полноразмерного NYC-0015-HC-k показана в SEQ ID NO:194 (фиг. 195); и аминокислотная последовательность полноразмерного NYC-0016-HC-k показана в SEQ ID NO:196 (фиг. 197).
21)-2 Экспрессия и очистка scFv-гетеродимера-Fc анти-HLA/NY-ESO
Культивирование и введение гена в клетки Expi293F (ThermoFisher Scientific) проводят таким же образом, как описано в 1)-5-2. Плазмиды, использованные для получения клонов, показаны в таблице 13 ниже.
[Таблица 13]
Для оценки количества продуцирования, клетки подвергают встряхиванию культуры в инкубаторе при 37°С в присутствии 8% СО2 при 135 об/мин в течение 4 дней после введения гена, культуральный супернатант фильтруют через 0,2 мкм фильтр (ThermoFisher Scientific), и получают культуральный супернатант scFv-гетеродимера-Fc анти-HLA/NY-ESO. Для очистки, культуральный супернатант наносят на смолу MabSelectSuRe (Cytiva), уравновешенную PBS (рН 7,4), чтобы позволить scFv-гетеродимеру-Fc-мишени анти-HLA/NY-ESO адсорбироваться на нем. После того как не адсорбированные компоненты удаляют с помощью PBS, адсорбированные компоненты элюируют с помощью ацетатного буфера, элюат нейтрализуют с помощью Tris буфера и элюат подвергают аналитической эксклюзионной хроматографии (SEC) для определения чистоты и концентрации. В таблице 14 показано количество продуцирования относительно 1 литра культурального супернатанта. Количество продуцирования NYA-2047, NYA-2061 и NYA-3061, определенное с использованием смолы MabSelectSuRe, на 1 л культурального супернатанта в очищенном элюате выше, чем у NYC-0011, NYC-0012, NYC-0013, NYC-0014, NYC-0015 и NYC-0016.
[Таблица 14]
Элюат каждого scFv-гетеродимера-Fc, полученного с использованием смолы MabSelectSuRe, концентрируют и очищают на колонке для гель-фильтрации (Superdex 200 Increase, Cytiva), уравновешенной 25 мМ гистидином, 300 мМ NaCl, 5% сорбитором (pH 5,5). Образцы очищенного белка подвергают аналитической эксклюзионной хроматографии (SEC), и в качестве образцов используют NYD-2047, NYD-2061, NYD-3061, NYC-0011, NYC-0013 и NYC-0015 с достаточной степенью чистоты и количествами, для оценки стабильности раствора. Мишень не была обнаружена в NYC-0016 в течение времени элюирования, установленного SEC. Таким образом, было определено, что мишень не экспрессируется в супернатанте культуры.
(Пример 22) Оценка стабильности раствора scFv-гетеродимера-Fc анти-HLA/NY-ESO
NYD-2047, NYD-2061, NYD-3061, NYC-0011, NYC-0013 и NYC-0015, полученные в примере 21, концентрируют центрифугированием с использованием Amicon Ultra-4 (Millipore), буфер заменяют PBS, туда добавляют PBS, и концентрацию образца доводят до 5 мг/мл для приготовления образцов для оценки. Вначале HMWS (%) каждого оцениваемого образца определяют способом процента площади с помощью эксклюзионной хроматографии с использованием AdvanceBio SEC 300A 2,7 мкм 4,6×150 мм (Agilent). В качестве подвижной фазы используют 0,2 М Ki/200 мМ KCl/pH 7,0, и анализ проводят при скорости потока 0,2 мл/мин (длина волны обнаружения: 280 нм). Испытание на ускоренное старение каждого образца проводят путем хранения при 40°C в течение 1 дня и 7 дней, эксклюзионной хроматографии в условиях, описанных выше, и расчета HMWS (%) каждого образца способом процента площади. На фиг. 163 показаны результаты, демонстрирующие изменения, происходящие в каждом образце при хранении при 40°C с течением времени.
HMWS (%) NYD-2047, NYD-2061 и NYD-3061 увеличивается до 8,7%, 8,1% и 4,4%, соответственно, по истечении времени хранения. Наоборот, HMWS (%) NYC-0011, NYC-0013 и NYC-0015 значительно увеличивается; 71,8%, 42,4% и 97,3%, соответственно, по истечении времени хранения.
Результаты показывают, что NYD-2047, NYD-2061 и NYD-3061 превосходят NYC-0011, NYC-0013 и NYC-0015 с точки зрения стабильности раствора. Кроме того, HMWS (%) NYD-3061 является самым низким среди оценочных образцов. То есть NYD-3061 является превосходным с точки зрения стабильности раствора.
(Пример 23) Оценка аффинности связывания с HLA/NY-ESO с использованием Biacore
NYC-0005, NYC-0007 и NYC-0008, эквивалентные scFv, составляющим анти-HLA/NY-ESO NYC-0011, NYC-0013 и NYC-0015, используют для оценки аффинности связывания с HLA/NY-ESO.
С использованием Biacore T200, анти-HLA/NY-ESO scFv захватывают в качестве лиганда к иммобилизованному анти-His антителу, и антиген анализируют в качестве анализируемого вещества. В качестве антигена используют HLA/NY-ESO, полученный в 1)-1. Анти-His антитело (набор His Capture, Cytiva) иммобилизуют на сенсорном чипе CM5 (Cytiva) в соответствии с инструкциями набора. Исследуемые конструкции scFv анти-HLA/NY-ESO, разведенные до 0,5 мкг/мл в HBS-EP+ (Cytiva), приводят в контакт с ним при 10 мкл/мин в течение 60 секунд для иммобилизации. После этого образцы добавляют к анализируемым веществам HLA/NY-ESO, разведенным до различных уровней с помощью HBS-EP+, при скорости потока 30 мкл/мин в течение 120 секунд, и анализируют диссоциацию в течение 600 секунд. Результаты расчетов, полученные с помощью такого анализа кинетики одного цикла, KD, показаны в таблице 15. Подтверждают, что NYC-0005, NYC-0007 и NYC-0008 будут связываться с HLA/NY-ESO в условиях, в которых аффинность связывания NYA-2047, используемого в качестве положительного контроля, с HLA/NY-ESO стабильно подтверждена.
[Таблица 15]
Промышленная применимость
Биспецифическое антитело по настоящему изобретению можно использовать в качестве терапевтического или профилактического агента против рака и подобных.
Все публикации, патенты и заявки на патенты, цитируемые в настоящем документе, полностью включены в настоящий документ посредством ссылки.
Свободный текст из перечня последовательностей
SEQ ID NO 1: аминокислотная последовательность пептида в NY-ESO (фиг. 8).
SEQ ID NO 2: аминокислотная последовательность пептида в MAGEC-1 (фиг. 9).
SEQ ID NO 3: праймер 1 для анализа последовательности scFv (фиг. 10).
SEQ ID NO 4: праймер 2 для анализа последовательности scFv (фиг. 11).
SEQ ID NO 5: нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0001 (фиг. 12).
SEQ ID NO 6: аминокислотная последовательность вариабельной области тяжелой цепи NYA-0001 (фиг. 13).
SEQ ID NO 7: нуклеотидная последовательность вариабельной области легкой цепи NYA-0001 (фиг. 14).
SEQ ID NO:8: аминокислотная последовательность вариабельной области легкой цепи NYA-0001 (фиг. 15).
SEQ ID NO 9: нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0060 (фиг. 16).
SEQ ID NO 10: аминокислотная последовательность вариабельной области тяжелой цепи NYA-0060 (фиг. 17).
SEQ ID NO 11: нуклеотидная последовательность вариабельной области легкой цепи NYA-0060 (фиг. 18).
SEQ ID NO 12: аминокислотная последовательность вариабельной области легкой цепи NYA-0060 (фиг. 19).
SEQ ID NO 13: нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0068 (фиг. 20).
SEQ ID NO 14: аминокислотная последовательность вариабельной области тяжелой цепи NYA-0068 (фиг. 21).
SEQ ID NO 15: нуклеотидная последовательность вариабельной области легкой цепи NYA-0068 (фиг. 22).
SEQ ID NO 16: аминокислотная последовательность вариабельной области легкой цепи NYA-0068 (фиг. 23).
SEQ ID NO 17: нуклеотидная последовательность вариабельной области тяжелой цепи NYA-0082 (фиг. 24).
SEQ ID NO 18: аминокислотная последовательность вариабельной области тяжелой цепи NYA-0082 (фиг. 25).
SEQ ID NO 19: нуклеотидная последовательность вариабельной области легкой цепи NYA-0082 (фиг. 26).
SEQ ID NO 20: аминокислотная последовательность вариабельной области легкой цепи NYA-0082 (фиг. 27).
SEQ ID NO 21: нуклеотидная последовательность аддукта метки NYA-1163 (фиг. 28).
SEQ ID NO 22: нуклеотидная последовательность аддукта метки NYA-2023 (фиг. 29).
SEQ ID NO 23: нуклеотидная последовательность аддукта метки NYA-2027 (фиг. 30).
SEQ ID NO 24: нуклеотидная последовательность аддукта метки NYA-1143 (фиг. 31).
SEQ ID NO 25: нуклеотидная последовательность аддукта метки NYA-2143 (фиг. 32).
SEQ ID NO 26: аминокислотная последовательность аддукта метки NYA-1163 (фиг. 33); NYA-1163: аминокислоты 21-266.
SEQ ID NO 27: аминокислотная последовательность аддукта метки NYA-2023 (фиг. 34); NYA-2023: аминокислоты 21-266.
SEQ ID NO 28: аминокислотная последовательность аддукта метки NYA-2027 (фиг. 35); NYA-2027: аминокислоты 21-266.
SEQ ID NO 29: аминокислотная последовательность аддукта метки NYA-1143 (фиг. 36); NYA-1143: аминокислоты 21-266.
SEQ ID NO 30: аминокислотная последовательность аддукта метки NYA-2143 (фиг. 37); NYA-2143: аминокислоты 21-266.
SEQ ID NO 31: нуклеотидная последовательность аддукта метки NYA-1154 (фиг. 38).
SEQ ID NO 32: аминокислотная последовательность аддукта метки NYA-1154 (фиг. 39); NYA-1154: аминокислоты 21-266.
SEQ ID NO 33: усеченная аминокислотная последовательность HLA-A*0201 (GenBank: ASA47534.1) (фиг. 40).
SEQ ID NO 34: аминокислотная последовательность β2-микроглобина (фиг. 41).
SEQ ID NO 35: нуклеотидная последовательность аддукта метки NYA-2035 (фиг. 42).
SEQ ID NO 36: аминокислотная последовательность аддукта метки NYA-2035 (фиг. 43); NYA-2035: аминокислоты 21-266.
SEQ ID NO 37: аминокислотная последовательность NYA-1143-VH01 (фиг. 44).
SEQ ID NO 38: аминокислотная последовательность NYA-1143-VH02 (фиг. 45).
SEQ ID NO 39: аминокислотная последовательность NYA-1143-VH03 (фиг. 46).
SEQ ID NO 40: аминокислотная последовательность NYA-1143-VL01 (фиг. 47).
SEQ ID NO 41: нуклеотидная последовательность аддукта метки NYA-2044 (фиг. 48).
SEQ ID NO 42: нуклеотидная последовательность аддукта метки NYA-2045 (фиг. 49).
SEQ ID NO 43: нуклеотидная последовательность аддукта метки NYA-2047 (фиг. 50).
SEQ ID NO 44: нуклеотидная последовательность аддукта метки NYA-2048 (фиг. 51).
SEQ ID NO 45: нуклеотидная последовательность аддукта метки NYA-2060 (фиг. 52).
SEQ ID NO 46: нуклеотидная последовательность аддукта метки NYA-2061 (фиг. 53).
SEQ ID NO 47: аминокислотная последовательность аддукта метки NYA-2044 (фиг. 54); NYA-2044: аминокислоты 21-266.
SEQ ID NO 48: аминокислотная последовательность аддукта метки NYA-2045 (фиг. 55); NYA-2045: аминокислоты 21-266.
SEQ ID NO 49: аминокислотная последовательность NYA-0082 (фиг. 56).
SEQ ID NO 50: аминокислотная последовательность аддукта метки NYA-2047 (фиг. 57); NYA-2047: аминокислоты 21-266.
SEQ ID NO 51: аминокислотная последовательность аддукта метки NYA-2048 (фиг. 58); NYA-2048: аминокислоты 21-266.
SEQ ID NO 52: аминокислотная последовательность аддукта метки NYA-2060 (фиг. 59); NYA-2060: аминокислоты 21-266.
SEQ ID NO 53: аминокислотная последовательность аддукта метки NYA-2061 (фиг. 60); NYA-2061: аминокислоты 21-266.
SEQ ID NO 54: CDRH1 тяжелой цепи NYA-0001 (фиг. 61).
SEQ ID NO 55: CDRH2 тяжелой цепи NYA-0001 (фиг. 61).
SEQ ID NO 56: CDRH3 тяжелой цепи NYA-0001 (фиг. 61).
SEQ ID NO 57: CDRL1 легкой цепи NYA-0001 (фиг. 61).
SEQ ID NO 58: CDRL2 легкой цепи NYA-0001 (фиг. 61).
SEQ ID NO 59: CDRL3 легкой цепи NYA-0001 (фиг. 61).
SEQ ID NO 60: аминокислотная последовательность CDRL1 NYA-2023 (фиг. 62).
SEQ ID NO 61: аминокислотная последовательность CDRL3 NYA-2027 (фиг. 63).
SEQ ID NO 62: CDRH3 тяжелой цепи NYA-1154 (фиг. 64).
SEQ ID NO 63: CDRL3 легкой цепи NYA-1154 (фиг. 64).
SEQ ID NO 64: аминокислотная последовательность CDRL1 NYA-0035 (фиг. 65).
SEQ ID NO 65: нуклеотидная последовательность аддукта метки NYC-0003 (фиг. 66).
SEQ ID NO 66: нуклеотидная последовательность аддукта метки NYC-0004 (фиг. 67).
SEQ ID NO 67: аминокислотная последовательность аддукта метки NYC-0003 (фиг. 68); NYC-0003: аминокислоты 21-263.
SEQ ID NO 68: аминокислотная последовательность аддукта метки NYC-0004 (фиг. 69); NYC-0004: аминокислоты 21-263.
SEQ ID NO 69: нуклеотидная последовательность аддукта метки NYA-0001 (фиг. 70).
SEQ ID NO 70: аминокислотная последовательность аддукта метки NYA-0001 (фиг. 71); NYA-0001: аминокислоты 21-266.
SEQ ID NO 71: нуклеотидная последовательность HC1 (фиг. 72).
SEQ ID NO 72: нуклеотидная последовательность NYF-0016-HC2 (фиг. 73).
SEQ ID NO 73: нуклеотидная последовательность NYF-0019-HC2 (фиг. 74).
SEQ ID NO 74: нуклеотидная последовательность NYF-0022-HC2 (фиг. 75).
SEQ ID NO 75: нуклеотидная последовательность NYF-0023-HC2 (фиг. 76).
SEQ ID NO 76: нуклеотидная последовательность NYF-0027-HC2 (фиг. 77).
SEQ ID NO 77: нуклеотидная последовательность NYF-0035-HC2 (фиг. 78).
SEQ ID NO 78: нуклеотидная последовательность NYF-0044-HC2 (фиг. 79).
SEQ ID NO 79: нуклеотидная последовательность NYF-0045-HC2 (фиг. 80).
SEQ ID NO 80: нуклеотидная последовательность NYF-0047-HC2 (фиг. 81).
SEQ ID NO 81: нуклеотидная последовательность NYF-0048-HC2 (фиг. 82).
SEQ ID NO 82: нуклеотидная последовательность NYF-0060-HC2 (фиг. 83).
SEQ ID NO 83: нуклеотидная последовательность NYF-0061-HC2 (фиг. 84).
SEQ ID NO 84: аминокислотная последовательность HC1 (фиг. 85).
SEQ ID NO 85: аминокислотная последовательность NYF-0016-HC2 (фиг. 86); NYA-1143: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 86: аминокислотная последовательность NYF-0019-HC2 (фиг. 87); NYA-2143: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 87: аминокислотная последовательность NYF-0022-HC2 (фиг. 88); NYA-1163: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 88: аминокислотная последовательность NYF-0023-HC2 (фиг. 89); NYA-2023: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 89: аминокислотная последовательность NYF-0027-HC2 (фиг. 90); NYA-2027: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 90: аминокислотная последовательность NYF-0035-HC2 (фиг. 91); NYA-2035: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 91: аминокислотная последовательность NYF-0044-HC2 (фиг. 92); NYA-2044: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 92: аминокислотная последовательность NYF-0045-HC2 (фиг. 93); NYA-2045: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 93: аминокислотная последовательность NYF-0047-HC2 (фиг. 94); NYA-2047: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 94: аминокислотная последовательность NYF-0048-HC2 (фиг. 95); NYA-2048: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 95: аминокислотная последовательность NYF-0060-HC2 (фиг. 96); NYA-2060: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 96: аминокислотная последовательность NYF-0061-HC2 (фиг. 97); NYA-2061: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 97: нуклеотидная последовательность делеции NYA-0001-Fab-HC1-k (фиг. 98).
SEQ ID NO 98: нуклеотидная последовательность NYA-0001-LC (фиг. 99).
SEQ ID NO 99: аминокислотная последовательность делеции NYA-0001-Fab-HC1-k (фиг. 100); вариабельная область тяжелой цепи NYA-0001: аминокислоты 20-139
SEQ ID NO 100: аминокислотная последовательность NYA-0001-LC (фиг. 101); вариабельная область легкой цепи NYA-0001: аминокислоты 21-131
SEQ ID NO 101: нуклеотидная последовательность делеции NYA-1143-Fab-HC1-k (фиг. 102).
SEQ ID NO 102: нуклеотидная последовательность NYA-1143-LC (фиг. 103).
SEQ ID NO 103: нуклеотидная последовательность C3E-7085-HC2-k с делецией C (фиг. 104).
SEQ ID NO 104: аминокислотная последовательность делеции NYA-1143-Fab-HC1-k (фиг. 105); вариабельная область тяжелой цепи NYA-1143: аминокислоты 20-139
SEQ ID NO 105: аминокислотная последовательность NYA-1143-LC (фиг. 106); вариабельная область легкой цепи NYA-1143: аминокислоты 21-131
SEQ ID NO 106: аминокислотная последовательность делеции C3E-7085-HC2-k (фиг. 107); C3E-7085: аминокислоты 21-260.
SEQ ID NO 107: нуклеотидная последовательность делеции NYA-1143-HC1-k (фиг. 108).
SEQ ID NO 108: аминокислотная последовательность делеции NYA-1143-HC1-k (фиг. 109); NYA-1143: аминокислоты 21-266.
SEQ ID NO 109: нуклеотидная последовательность делеции C3E-7085-NYA-1154-Fab-HC2-k (фиг. 110).
SEQ ID NO 110: нуклеотидная последовательность NYA-1154-LC (фиг. 111).
SEQ ID NO 111: нуклеотидная последовательность делеции OAA-HC1-k (фиг. 112).
SEQ ID NO 112: аминокислотная последовательность делеции C3E-7085-NYA-1154-Fab-HC2-k (фиг. 113); C3E-7085: аминокислоты 21-260; вариабельная область тяжелой цепи NYA-1154: аминокислоты 266-285
SEQ ID NO 113: аминокислотная последовательность NYA-1154-LC (фиг. 114); вариабельная область легкой цепи NYA-1154: аминокислоты 21-131
SEQ ID NO 114: аминокислотная последовательность делеции OAA-HC1-k (фиг. 115).
SEQ ID NO 115: нуклеотидная последовательность делеции NYF-0010-HC2-k (фиг. 116).
SEQ ID NO 116: нуклеотидная последовательность делеции NYF-0004-HC2-k (фиг. 117).
SEQ ID NO 117: нуклеотидная последовательность делеции NYF-0011-HC2-k (фиг. 118).
SEQ ID NO 118: аминокислотная последовательность делеции NYF-0010-HC2-k (фиг. 119); NYA-1154: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 119: аминокислотная последовательность делеции NYF-0004-HC2-k (фиг. 120); C3E-7085: аминокислоты 21-260; и NYA-1154: аминокислоты 272-511.
SEQ ID NO 120: аминокислотная последовательность делеции NYF-0011-HC2-k (фиг. 121); NYA-1143: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 121: аминокислотная последовательность точечного мутанта NY-ESO пептида 1F (фиг. 122).
SEQ ID NO 122: аминокислотная последовательность точечного мутантного пептида NY-ESO 2M (фиг. 123).
SEQ ID NO 123: аминокислотная последовательность точечного мутантного пептида 3A NY-ESO (фиг. 124).
SEQ ID NO 124: аминокислотная последовательность точечного мутантного пептида 4A NY-ESO (фиг. 125).
SEQ ID NO 125: аминокислотная последовательность точечного мутантного пептида 5A NY-ESO (фиг. 126).
SEQ ID NO 126: аминокислотная последовательность точечного мутанта NY-ESO пептида 6L (фиг. 127).
SEQ ID NO 127: аминокислотная последовательность точечного мутанта NY-ESO пептида 7F (фиг. 128).
SEQ ID NO 128: аминокислотная последовательность точечного мутанта NY-ESO пептида 8A (фиг. 129).
SEQ ID NO 129: аминокислотная последовательность точечного мутантного пептида 9A NY-ESO (фиг. 130).
SEQ ID NO 130: аминокислотная последовательность пептида gp100 (фиг. 131).
SEQ ID NO 131: аминокислотная последовательность гомологичного пептида DOLPP1 (фиг. 132).
SEQ ID NO 132: аминокислотная последовательность гомологичного пептида IL20RB (фиг. 133).
SEQ ID NO 133: аминокислотная последовательность гомологичного пептида PRKD2 (фиг. 134).
SEQ ID NO 134: аминокислотная последовательность гомологичного пептида CD163 (фиг. 135).
SEQ ID NO 135: аминокислотная последовательность гомологичного пептида P2RY8 (фиг. 136).
SEQ ID NO 136: аминокислотная последовательность C3E-7034 (фиг. 137).
SEQ ID NO 137: аминокислотная последовательность C3E-7036 (фиг. 138).
SEQ ID NO 138: аминокислотная последовательность C3E-7085 (фиг. 139).
SEQ ID NO 139: аминокислотная последовательность C3E-7088 (фиг. 140).
SEQ ID NO 140: аминокислотная последовательность C3E-7093 (фиг. 141).
SEQ ID NO 141: CDRH1 тяжелой цепи C3E-7085 (фиг. 142).
SEQ ID NO 142: CDRH2 тяжелой цепи C3E-7085 (фиг. 142).
SEQ ID NO 143: CDRH3 тяжелой цепи C3E-7085 (фиг. 142).
SEQ ID NO 144: CDRL1 легкой цепи C3E-7085 (фиг. 142).
SEQ ID NO 145: CDRL2 легкой цепи C3E-7085 (фиг. 142).
SEQ ID NO 146: CDRL3 легкой цепи C3E-7085 (фиг. 142).
SEQ ID NO 147: аминокислотная последовательность C3E-7078 (фиг. 143).
SEQ ID NO 148: нуклеотидная последовательность NYF-0014-HC2 (фиг. 144).
SEQ ID NO 149: аминокислотная последовательность NYF-0014-HC2 (фиг. 145); NYA-0001: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 150: аминокислотная последовательность NYF-0082-HC2 (фиг. 146); NYA-0082: аминокислоты 21-266; и C3E-7085: аминокислоты 272-511.
SEQ ID NO 151: аминокислотная последовательность CD3 ε человека (фиг. 147).
SEQ ID NO 152: нуклеотидная последовательность полноразмерного NYZ-0038-HC2 (фиг. 148).
SEQ ID NO 153: нуклеотидная последовательность полноразмерного NYZ-0082-HC2 (фиг. 149).
SEQ ID NO 154: нуклеотидная последовательность полноразмерного NYZ-0083-HC2 (фиг. 150).
SEQ ID NO 155: аминокислотная последовательность полноразмерного NYZ-0038-HC2 (фиг. 151).
SEQ ID NO 156: аминокислотная последовательность полноразмерного NYZ-0082-HC2 (фиг. 152).
SEQ ID NO 157: аминокислотная последовательность полноразмерного NYZ-0083-HC2 (фиг. 153).
SEQ ID NO 158: нуклеотидная последовательность полноразмерного NYZ-1010-HC2 (фиг. 154).
SEQ ID NO 159: нуклеотидная последовательность полноразмерного C3E-7085-LC (фиг. 155).
SEQ ID NO 160: аминокислотная последовательность полноразмерного NYZ-1010-HC2 (фиг. 156).
SEQ ID NO 161: аминокислотная последовательность полноразмерного C3E-7085-LC (фиг. 157).
SEQ ID NO 162: аминокислотная последовательность пептидного линкера (фиг. 161).
SEQ ID NO 163: нуклеотидная последовательность полноразмерного NYA-3061 (фиг. 164).
SEQ ID NO 164: аминокислотная последовательность полноразмерного NYA-3061 (фиг. 165).
SEQ ID NO 165: нуклеотидная последовательность полноразмерного NYC-0005 (фиг. 166).
SEQ ID NO 166: аминокислотная последовательность полноразмерного NYC-0005 (фиг. 167).
SEQ ID NO 167: нуклеотидная последовательность полноразмерного NYC-0006 (фиг. 168).
SEQ ID NO 168: аминокислотная последовательность полноразмерного NYC-0006 (фиг. 169).
SEQ ID NO 169: нуклеотидная последовательность полноразмерного NYC-0007 (фиг. 170).
SEQ ID NO 170: аминокислотная последовательность полноразмерного NYC-0007 (фиг. 171).
SEQ ID NO 171: нуклеотидная последовательность полноразмерного NYC-0008 (фиг. 172).
SEQ ID NO 172: аминокислотная последовательность полноразмерного NYC-0008 (фиг. 173).
SEQ ID NO 173: нуклеотидная последовательность полноразмерного NYC-0009 (фиг. 174).
SEQ ID NO 174: аминокислотная последовательность полноразмерного NYC-0009 (фиг. 175).
SEQ ID NO 175: нуклеотидная последовательность полноразмерного NYC-0010 (фиг. 176).
SEQ ID NO 176: аминокислотная последовательность полноразмерного NYC-0010 (фиг. 177).
SEQ ID NO 177: нуклеотидная последовательность полноразмерного HC-h (фиг. 178).
SEQ ID NO 178: аминокислотная последовательность полноразмерного HC-h (фиг. 179).
SEQ ID NO 179: нуклеотидная последовательность полноразмерного NYD-2047-HC-k (фиг. 180).
SEQ ID NO 180: аминокислотная последовательность полноразмерного NYD-2047-HC-k (фиг. 181).
SEQ ID NO 181: нуклеотидная последовательность полноразмерного NYD-2061-HC-k (фиг. 182).
SEQ ID NO 182: аминокислотная последовательность полноразмерного NYD-2061-HC-k (фиг. 183).
SEQ ID NO 183: нуклеотидная последовательность полноразмерного NYD-3061-HC-k (фиг. 184).
SEQ ID NO 184: аминокислотная последовательность полноразмерного NYD-3061-HC-k (фиг. 185).
SEQ ID NO 185: нуклеотидная последовательность полноразмерного NYC-0011-HC-k (фиг. 186).
SEQ ID NO 186: аминокислотная последовательность полноразмерного NYC-0011-HC-k (фиг. 187).
SEQ ID NO 187: нуклеотидная последовательность полноразмерного NYC-0012-HC-k (фиг. 188).
SEQ ID NO 188: аминокислотная последовательность полноразмерного NYC-0012-HC-k (фиг. 189).
SEQ ID NO 189: нуклеотидная последовательность полноразмерного NYC-0013-HC-k (фиг. 190).
SEQ ID NO 190: аминокислотная последовательность полноразмерного NYC-0013-HC-k (фиг. 191).
SEQ ID NO 191: нуклеотидная последовательность полноразмерного NYC-0014-HC-k (фиг. 192).
SEQ ID NO 192: аминокислотная последовательность полноразмерного NYC-0014-HC-k (фиг. 193).
SEQ ID NO 193: нуклеотидная последовательность полноразмерного NYC-0015-HC-k (фиг. 194).
SEQ ID NO 194: аминокислотная последовательность полноразмерного NYC-0015-HC-k (фиг. 195).
SEQ ID NO 195: нуклеотидная последовательность полноразмерного NYC-0016-HC-k (фиг. 196).
SEQ ID NO 196: аминокислотная последовательность полноразмерного NYC-0016-HC-k (фиг. 197).
SEQ ID NO 197: аминокислотная последовательность полноразмерного NYZ-1007-HC (фиг. 198).
SEQ ID NO 198: аминокислотная последовательность полноразмерного NYZ-1017-HC (фиг. 199).
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ДАЙИТИ САНКИО КОМПАНИ, ЛИМИТЕД
МИЕ ЮНИВЕРСИТИ
<120> БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА
<130> PH-8759-PCT
<150> JP 2020-061476
<151> 2020-03-30
<160> 198
<170> PatentIn version 3.5
<210> 1
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Пептидная последовательность NY-ESO
<400> 1
Ser Leu Leu Met Trp Ile Thr Gln Cys
1 5
<210> 2
<211> 10
<212> БЕЛОК
<213> Искусственная
<220>
<223> Пептидная последовательность MAGEC-1
<400> 2
Ile Leu Phe Gly Ile Ser Leu Arg Glu Val
1 5 10
<210> 3
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер для анализа последовательности scFv
<400> 3
ctcttcgcta ttacgccagc tggcga 26
<210> 4
<211> 30
<212> ДНК
<213> Искусственная
<220>
<223> Праймер для анализа последовательности scFv
<400> 4
ataacaattt cacacaggaa acagctatga 30
<210> 5
<211> 360
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области тяжелой цепи
NYA-0001
<400> 5
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 60
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 120
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 180
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 240
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 300
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 360
<210> 6
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области тяжелой цепи
NYA-0001
<400> 6
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn Met Val Tyr
65 70 75 80
Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 7
<211> 333
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области легкой цепи
NYA-0001
<400> 7
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcagctc 120
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 240
actggggacg aggccgatta ttactgcgga acatgggata gcagcctgag tgccccttgg 300
gtgttcggcg gagggaccaa ggtcaccgtc cta 333
<210> 8
<211> 111
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области легкой цепи
NYA-0001
<400> 8
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 9
<211> 360
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области тяжелой цепи
NYA-0060
<400> 9
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 60
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 120
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 180
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 240
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 300
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 360
<210> 10
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области тяжелой цепи
NYA-0060
<400> 10
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn Met Val Tyr
65 70 75 80
Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 11
<211> 333
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области легкой цепи
NYA-0060
<400> 11
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcagctc 120
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 240
actggggacg aggccgatta ttactgcgga acatgggata gcagcctgag tgccccttgg 300
gtgttcggcg gagggaccaa ggtcaccgtc cta 333
<210> 12
<211> 111
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области легкой цепи
NYA-0060
<400> 12
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 13
<211> 360
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области тяжелой цепи
NYA-0068
<400> 13
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 60
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 120
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 180
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 240
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 300
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 360
<210> 14
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области тяжелой цепи
NYA-0068
<400> 14
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn Met Val Tyr
65 70 75 80
Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 15
<211> 333
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области легкой цепи
NYA-0068
<400> 15
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcagctc caacattggg aattggtatg tatcctggta ccagcagctc 120
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 240
actggggacg aggccgatta ttactgcgga acatgggata gcagcctgag tgccccttgg 300
gtgttcggcg gagggaccaa ggtcaccgtc cta 333
<210> 16
<211> 111
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области легкой цепи
NYA-0068
<400> 16
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 17
<211> 360
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области тяжелой цепи
NYA-0082
<400> 17
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 60
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 120
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 180
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 240
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 300
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 360
<210> 18
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области тяжелой цепи
NYA-0082
<400> 18
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn Met Val Tyr
65 70 75 80
Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 19
<211> 333
<212> ДНК
<213> Искусственная
<220>
<223> Нуклеотидная последовательность вариабельной области легкой цепи
NYA-0082
<400> 19
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 60
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcggctc 120
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 180
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 240
actggggacg aggccgatta ttactgcgga tcatgggata gcagcctgag tgccccttgg 300
gtgttcggcg gagggaccaa ggtcaccgtc cta 333
<210> 20
<211> 111
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность вариабельной области легкой цепи
NYA-0082
<400> 20
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Ser Trp Asp Ser Ser Leu
85 90 95
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 21
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1163
<400> 21
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 22
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2023
<400> 22
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 23
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2027
<400> 23
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggatcatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 24
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1143
<400> 24
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 25
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2143
<400> 25
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc ggctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 26
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1163
<400> 26
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 27
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2023
<400> 27
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 28
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2027
<400> 28
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Ser Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 29
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1143
<400> 29
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 30
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2143
<400> 30
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 31
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1154
<400> 31
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tgcactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggccgttt atcactgtgc gagagggagt 360
agtaatcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc ggctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattactact gcggagcatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 32
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1154
<400> 32
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Asn His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Ala Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 33
<211> 290
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность усеченной формы
HLA-A*0201(UniProtKB-P01892)
<400> 33
Met Ser His Ser Met Arg Tyr Phe Phe Thr Ser Val Ser Arg Pro Gly
1 5 10 15
Arg Gly Glu Pro Arg Phe Ile Ala Val Gly Tyr Val Asp Asp Thr Gln
20 25 30
Phe Val Arg Phe Asp Ser Asp Ala Ala Ser Gln Arg Met Glu Pro Arg
35 40 45
Ala Pro Trp Ile Glu Gln Glu Gly Pro Glu Tyr Trp Asp Gly Glu Thr
50 55 60
Arg Lys Val Lys Ala His Ser Gln Thr His Arg Val Asp Leu Gly Thr
65 70 75 80
Leu Arg Gly Tyr Tyr Asn Gln Ser Glu Ala Gly Ser His Thr Val Gln
85 90 95
Arg Met Tyr Gly Cys Asp Val Gly Ser Asp Trp Arg Phe Leu Arg Gly
100 105 110
Tyr His Gln Tyr Ala Tyr Asp Gly Lys Asp Tyr Ile Ala Leu Lys Glu
115 120 125
Asp Leu Arg Ser Trp Thr Ala Ala Asp Met Ala Ala Gln Thr Thr Lys
130 135 140
His Lys Trp Glu Ala Ala His Val Ala Glu Gln Leu Arg Ala Tyr Leu
145 150 155 160
Glu Gly Thr Cys Val Glu Trp Leu Arg Arg Tyr Leu Glu Asn Gly Lys
165 170 175
Glu Thr Leu Gln Arg Thr Asp Ala Pro Lys Thr His Met Thr His His
180 185 190
Ala Val Ser Asp His Glu Ala Thr Leu Arg Cys Trp Ala Leu Ser Phe
195 200 205
Tyr Pro Ala Glu Ile Thr Leu Thr Trp Gln Arg Asp Gly Glu Asp Gln
210 215 220
Thr Gln Asp Thr Glu Leu Val Glu Thr Arg Pro Ala Gly Asp Gly Thr
225 230 235 240
Phe Gln Lys Trp Ala Ala Val Val Val Pro Ser Gly Gln Glu Gln Arg
245 250 255
Tyr Thr Cys His Val Gln His Glu Gly Leu Pro Lys Pro Leu Thr Leu
260 265 270
Arg Trp Glu Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp
275 280 285
His Glu
290
<210> 34
<211> 100
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность бета 2-микроглобулина
<400> 34
Met Ile Gln Arg Thr Pro Lys Ile Gln Val Tyr Ser Arg His Pro Ala
1 5 10 15
Glu Asn Gly Lys Ser Asn Phe Leu Asn Cys Tyr Val Ser Gly Phe His
20 25 30
Pro Ser Asp Ile Glu Val Asp Leu Leu Lys Asn Gly Glu Arg Ile Glu
35 40 45
Lys Val Glu His Ser Asp Leu Ser Phe Ser Lys Asp Trp Ser Phe Tyr
50 55 60
Leu Leu Tyr Tyr Thr Glu Phe Thr Pro Thr Glu Lys Asp Glu Tyr Ala
65 70 75 80
Cys Arg Val Asn His Val Thr Leu Ser Gln Pro Lys Ile Val Lys Trp
85 90 95
Asp Arg Asp Met
100
<210> 35
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2035
<400> 35
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gaaggtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 36
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2035
<400> 36
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Lys Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 37
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-1143-VH01
<400> 37
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 38
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-1143-VH02
<400> 38
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 39
<211> 120
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-1143-VH03
<400> 39
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 40
<211> 111
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-1143-VL01
<400> 40
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 41
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2044
<400> 41
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgactcgcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 42
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2045
<400> 42
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgactcgcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 43
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2047
<400> 43
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
caagtacagc tggtggaatc cggtggaggc gtggtccagc cgggacgcag cttgagactg 120
tcctgcgctg catctggctt ttccatacga tcctacgata tgcactgggt tcgccaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattac 240
gcggattcag tgaaggggcg gttcacaatt tcacgggaca attcaaagaa taccttgtat 300
ctccagatgt cttcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 44
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2048
<400> 44
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
caagttcaac tcgtggaatc tggtggaggg gtggtacaac caggccggtc acttagactg 120
tcctgcgctg cgagtggatt ttccatcaga tcttacgaca tgcactgggt tcgccaagca 180
cccggaaagg gtttggaatg ggtggctacg atctcctacg atggatccca gaagtattac 240
gccgacagcg tcaagggccg atttacaata tcacgggaca attcaaagaa taccttgtat 300
ctccagatgt cttcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 45
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2060
<400> 45
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 46
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-2061
<400> 46
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 47
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2044
<400> 47
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 48
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2045
<400> 48
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 49
<211> 246
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-0082
<400> 49
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile Arg Ser Tyr
20 25 30
Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn Met Val Tyr
65 70 75 80
Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val Tyr His Cys
85 90 95
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr Gln Pro Pro Ser
130 135 140
Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser Cys Ser Gly Ser
145 150 155 160
Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr Gln Arg Leu Pro
165 170 175
Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn Lys Arg Pro Ser
180 185 190
Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Thr
195 200 205
Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala Asp Tyr Tyr Cys
210 215 220
Gly Ser Trp Asp Ser Ser Leu Ser Ala Pro Trp Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu
245
<210> 50
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2047
<400> 50
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 51
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2048
<400> 51
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 52
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2060
<400> 52
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 53
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-2061
<400> 53
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 54
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0001-CDRH1
<400> 54
Gly Phe Ser Ile Arg Ser Tyr Asp
1 5
<210> 55
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0001-CDRH2
<400> 55
Ile Ser Tyr Asp Gly Ser Gln Lys
1 5
<210> 56
<211> 13
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0001-CDRH3
<400> 56
Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile
1 5 10
<210> 57
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0001-CDRL1
<400> 57
Ser Ser Asn Ile Gly Asn Asn Tyr
1 5
<210> 58
<211> 3
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0001-CDRL2
<400> 58
Asp Asn Asn
1
<210> 59
<211> 12
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0001-CDRL3
<400> 59
Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
1 5 10
<210> 60
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-2023-CDRL1
<400> 60
Ser Ser Asn Ile Gly Asn Trp Tyr
1 5
<210> 61
<211> 12
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-2027-CDRL3
<400> 61
Gly Ser Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
1 5 10
<210> 62
<211> 13
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-1154-CDRH3
<400> 62
Ala Arg Gly Ser Ser Asn His Tyr Glu Ala Phe Asp Ile
1 5 10
<210> 63
<211> 12
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-1154-CDRL3
<400> 63
Gly Ala Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
1 5 10
<210> 64
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность NYA-0035-CDRL1
<400> 64
Ser Ser Asn Ile Gly Asn Trp Lys
1 5
<210> 65
<211> 867
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0003
<400> 65
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggc 60
gaagtccagc tgttggagtc aggtggcgga ttggtgcaac ctggcgggtc actgaggctg 120
agttgtgcag ctagcggctt cacattctct acgtatcaga tgagctgggt gagacaggct 180
ccaggaaagg gtctggaatg ggtcagcggg attgtgtcta gcggtggctc cactgcctat 240
gccgatagcg taaaaggccg ctttacgatc tctcgggaca actctaagaa cacactctat 300
ctgcagatga attcccttag agccgaagat accgccgtgt actactgtgc tggggaactg 360
ctcccgtatt acggtatgga cgtttggggc caaggcacca ctgtcacagt gagttccggt 420
ggaggcgggt caggcggagg cggtagtgga ggtggaggat cacaaagcga gctgacacag 480
cctaggtccg tatccggaag tccagggcag agcgtcacca tcagctgcac tggcacctct 540
cgagatgtcg gcggatacaa ctacgtgtct tggtatcagc agcatcccgg caaagcgccc 600
aaactcatca tacacgacgt gattgagcgg tccagtgggg ttcccgatcg tttcagcggg 660
tcaaagtccg gaaataccgc aagcctgacc atttctgggc ttcaggcaga ggatgaggct 720
gactactact gctggtcctt tgccgggagc tattacgtgt ttgggacagg gactgacgtg 780
actgttctgg gcgcggccgc aggtgcaggt ggtgattaca aagatgatga cgataaaggt 840
gcagcggcgc atcaccatca tcaccac 867
<210> 66
<211> 867
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0004
<400> 66
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggc 60
gaagtccagc tgttggagtc aggtggcgga ttggtgcaac ctggcgggtc actgaggctg 120
agttgtgcag ctagcggctt cacattctct acgtatcaga tgagctgggt gagacaggct 180
ccaggaaagg gtctggaatg ggtcagcggg attgtgtcta gcggtggctc cactgcctat 240
gccgatagcg taaaaggccg ctttacgatc tctcgggaca actctaagaa cacactctat 300
ctgcagatga attcccttag agccgaagat accgccgtgt actactgtgc tggggaactg 360
ctcccgtatt acggtatgga cgtttggggc caaggcacca ctgtcacagt gagttccggt 420
ggaggcgggt caggcggagg cggtagtgga ggtggaggat cacaaagcga gctgacacag 480
cctaggtccg tatccggaag tccagggcag agcgtcacca tcagctgcac tggcaccgag 540
cgagatgtcg gcggatacaa ctacgtgtct tggtatcagc agcatcccgg caaagcgccc 600
aaactcatca tacacgacgt gattgagcgg tccagtgggg ttcccgatcg tttcagcggg 660
tcaaagtccg gaaataccgc aagcctgacc atttctgggc ttcaggcaga ggatgaggct 720
gactactact gctggtcctt tgccgggggc tattacgtgt ttgggacagg gactgacgtg 780
actgttctgg gcgcggccgc aggtgcaggt ggtgattaca aagatgatga cgataaaggt 840
gcagcggcgc atcaccatca tcaccac 867
<210> 67
<211> 289
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0003
<400> 67
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Thr Tyr Gln Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Gly Ile Val Ser Ser Gly Gly Ser Thr Ala Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Gly Glu Leu Leu Pro Tyr Tyr Gly Met Asp Val
115 120 125
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Glu Leu Thr Gln
145 150 155 160
Pro Arg Ser Val Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys
165 170 175
Thr Gly Thr Ser Arg Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln His Pro Gly Lys Ala Pro Lys Leu Ile Ile His Asp Val Ile
195 200 205
Glu Arg Ser Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Trp Ser Phe Ala Gly Ser Tyr Tyr Val Phe Gly Thr
245 250 255
Gly Thr Asp Val Thr Val Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp
260 265 270
Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His His His His
275 280 285
His
<210> 68
<211> 289
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0004
<400> 68
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Thr Tyr Gln Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Gly Ile Val Ser Ser Gly Gly Ser Thr Ala Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Gly Glu Leu Leu Pro Tyr Tyr Gly Met Asp Val
115 120 125
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Glu Leu Thr Gln
145 150 155 160
Pro Arg Ser Val Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys
165 170 175
Thr Gly Thr Glu Arg Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln His Pro Gly Lys Ala Pro Lys Leu Ile Ile His Asp Val Ile
195 200 205
Glu Arg Ser Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Trp Ser Phe Ala Gly Gly Tyr Tyr Val Phe Gly Thr
245 250 255
Gly Thr Asp Val Thr Val Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp
260 265 270
Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His His His His
275 280 285
His
<210> 69
<211> 876
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-0001
<400> 69
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg cgcggccgca ggtgcaggtg gtgattacaa agatgatgac 840
gataaaggtg cagcggcgca tcaccatcat caccac 876
<210> 70
<211> 292
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-0001
<400> 70
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Ala Ala Ala Gly Ala
260 265 270
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala Ala His His
275 280 285
His His His His
290
<210> 71
<211> 738
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность HC1
<400> 71
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgac 60
aaaactcaca catgcccacc ctgcccagca cctgaagccg cagggggacc ctcagtcttc 120
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 180
gtggtggtgt cagtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 240
gtggaggtgc ataatgccaa gacaaagccc cgggaggagc agtacaacag cacgtaccgg 300
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 360
aaggtctcca acaaagccct cccagccccc atcgagaaaa ccatctccaa agccaaaggc 420
cagccccggg aaccacaggt gtacgtgctg cccccatccc gggacgagct gaccaagaac 480
caggtcagcc tgctgtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 540
gagagcaatg gccagcccga gaacaactac ctgacctggc ctcccgtgct ggactccgac 600
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcagggcaac 660
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacaccca gaagagcctc 720
tccctgtctc ccggcaag 738
<210> 72
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0016-HC2
<400> 72
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 73
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0019-HC2
<400> 73
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc ggctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 74
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0022-HC2
<400> 74
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 75
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0023-HC2
<400> 75
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 76
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0027-HC2
<400> 76
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggatcatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 77
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0035-HC2
<400> 77
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gaaggtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 78
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0044-HC2
<400> 78
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgactcgcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 79
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0045-HC2
<400> 79
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgactcgcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 80
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0047-HC2
<400> 80
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
caagtacagc tggtggaatc cggtggaggc gtggtccagc cgggacgcag cttgagactg 120
tcctgcgctg catctggctt ttccatacga tcctacgata tgcactgggt tcgccaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattac 240
gcggattcag tgaaggggcg gttcacaatt tcacgggaca attcaaagaa taccttgtat 300
ctccagatgt cttcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 81
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0048-HC2
<400> 81
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
caagttcaac tcgtggaatc tggtggaggg gtggtacaac caggccggtc acttagactg 120
tcctgcgctg cgagtggatt ttccatcaga tcttacgaca tgcactgggt tcgccaagca 180
cccggaaagg gtttggaatg ggtggctacg atctcctacg atggatccca gaagtattac 240
gccgacagcg tcaagggccg atttacaata tcacgggaca attcaaagaa taccttgtat 300
ctccagatgt cttcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 82
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0060-HC2
<400> 82
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 83
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0061-HC2
<400> 83
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 84
<211> 246
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность HC1
<400> 84
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
20 25 30
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
35 40 45
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ser
50 55 60
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
65 70 75 80
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
85 90 95
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
100 105 110
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
115 120 125
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
130 135 140
Pro Gln Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
145 150 155 160
Gln Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
165 170 175
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr
180 185 190
Trp Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
195 200 205
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
210 215 220
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
225 230 235 240
Ser Leu Ser Pro Gly Lys
245
<210> 85
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0016-HC
<400> 85
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 86
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0019-HC2
<400> 86
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 87
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0022-HC2
<400> 87
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 88
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0023-HC2
<400> 88
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 89
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0027-HC2
<400> 89
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Ser Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 90
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0035-HC2
<400> 90
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Lys Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 91
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0044-HC2
<400> 91
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 92
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0045-HC2
<400> 92
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 93
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0047-HC2
<400> 93
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 94
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0048-HC2
<400> 94
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 95
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0060-HC2
<400> 95
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 96
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0061-HC2
<400> 96
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 97
<211> 1404
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-0001-Fab-HC1-k
делеция
<400> 97
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgag 60
gtgcagctgg tggagtctgg gggaggcgtg gtccagcctg ggaggtccct gacaccctcc 120
tgttcagcgt ctggattctc catccgtagt tatgatatac actgggtccg ccaggctcca 180
ggcaaggggc tagagtgggt ggccactata tcatatgatg gaagtcagaa gtacttcgca 240
gactccgtga agggccgatt taccatcttc agagacgaat cggagaacat ggtgtatctg 300
caaatgagcg gcctgagagt tgaggacacg gctgtttatc actgtgcgag agggagtagt 360
ggtcattatg aggcttttga tatctggggc caagggacaa tggtcaccgt ctcttcagcc 420
tccaccaagg gcccaagcgt cttccccctg gcaccctcct ccaagagcac ctctggcggc 480
acagccgccc tgggctgcct ggtcaaggac tacttccccg aacccgtgac cgtgagctgg 540
aactcaggcg ccctgaccag cggcgtgcac accttccccg ctgtcctgca gtcctcagga 600
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 660
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagagagt tgagcccaaa 720
tcttgtgaca aaactcacac atgcccaccc tgcccagcac ctgaagccgc agggggaccc 780
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 840
gtcacatgcg tggtggtgtc agtgagccac gaagaccctg aggtcaagtt caactggtac 900
gtggacggcg tggaggtgca taatgccaag acaaagcccc gggaggagca gtacaacagc 960
acgtaccggg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 1020
tacaagtgca aggtctccaa caaagccctc ccagccccca tcgagaaaac catctccaaa 1080
gccaaaggcc agccccggga accacaggtg tacgtgctgc ccccatcccg ggacgagctg 1140
accaagaacc aggtcagcct gctgtgcctg gtcaaaggct tctatcccag cgacatcgcc 1200
gtggagtggg agagcaatgg ccagcccgag aacaactacc tgacctggcc tcccgtgctg 1260
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1320
cagggcaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacccag 1380
aagagcctct ccctgtctcc cggc 1404
<210> 98
<211> 711
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-0001-LC
<400> 98
atggtgctgc agacccaggt gttcatctcc ctgctgctgt ggatctccgg cgcgtacggc 60
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 120
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcagctc 180
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 240
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 300
actggggacg aggccgatta ttactgcgga acatgggata gcagcctgag tgccccttgg 360
gtgttcggcg gagggaccaa ggtcaccgtc ctaggccagc ctaaggctgc ccctagcgtg 420
accctgttcc ctccttccag cgaggagctt caagctaaca aggccaccct ggtgtgtctt 480
atctctgact tctaccctgg cgctgtgacc gtggcctgga aggctgacag ctcccctgtg 540
aaggccggag tggagaccac cacacctagc aagcagtcta acaacaagta cgctgccagc 600
tcctacctga gccttacccc tgagcagtgg aagtctcaca gaagctactc ctgtcaagtg 660
acccacgagg gcagcaccgt ggagaagacc gtggctccta ccgagtgttc c 711
<210> 99
<211> 468
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-0001-Fab-HC1-k
делеция
<400> 99
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln
20 25 30
Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile
35 40 45
Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn
85 90 95
Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val
100 105 110
Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile
115 120 125
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala
245 250 255
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ser Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly
465
<210> 100
<211> 237
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-0001-LC
<400> 100
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala
20 25 30
Ala Pro Gly Gln Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn
35 40 45
Ile Gly Asn Asn Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala
50 55 60
Pro Lys Leu Leu Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile
85 90 95
Thr Gly Leu Gln Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp
100 105 110
Asp Ser Ser Leu Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val
115 120 125
Thr Val Leu Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro
130 135 140
Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu
145 150 155 160
Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp
165 170 175
Ser Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln
180 185 190
Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu
195 200 205
Gln Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly
210 215 220
Ser Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
225 230 235
<210> 101
<211> 1404
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1143-Fab-HC1-k
делеция
<400> 101
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgag 60
gtgcagctgg tggagtctgg gggaggcgtg gtccagcctg ggaggtccct gacaccctcc 120
tgttcagcgt ctggattctc catccgtagc tatgatatac actgggtccg cctggctcca 180
ggcaaggggc tagagtgggt ggccactata tcatatgatg gaagtcagaa gtacttcgca 240
gactccgtga agggccgatt taccatcttc agagacgaat cggagaacat ggtgtatctg 300
caaatgagcg gcctgagagt tgaggacacg gctgtttatc actgtgcgag agggagtagt 360
ggtcattatg aggcttttga tatctggggc caagggacaa tggtcaccgt ctcttcagcc 420
tccaccaagg gcccaagcgt cttccccctg gcaccctcct ccaagagcac ctctggcggc 480
acagccgccc tgggctgcct ggtcaaggac tacttccccg aacccgtgac cgtgagctgg 540
aactcaggcg ccctgaccag cggcgtgcac accttccccg ctgtcctgca gtcctcagga 600
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 660
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagagagt tgagcccaaa 720
tcttgtgaca aaactcacac atgcccaccc tgcccagcac ctgaagccgc agggggaccc 780
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 840
gtcacatgcg tggtggtgtc agtgagccac gaagaccctg aggtcaagtt caactggtac 900
gtggacggcg tggaggtgca taatgccaag acaaagcccc gggaggagca gtacaacagc 960
acgtaccggg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 1020
tacaagtgca aggtctccaa caaagccctc ccagccccca tcgagaaaac catctccaaa 1080
gccaaaggcc agccccggga accacaggtg tacgtgctgc ccccatcccg ggacgagctg 1140
accaagaacc aggtcagcct gctgtgcctg gtcaaaggct tctatcccag cgacatcgcc 1200
gtggagtggg agagcaatgg ccagcccgag aacaactacc tgacctggcc tcccgtgctg 1260
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1320
cagggcaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacccag 1380
aagagcctct ccctgtctcc cggc 1404
<210> 102
<211> 711
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1143-LC
<400> 102
atggtgctgc agacccaggt gttcatctcc ctgctgctgt ggatctccgg cgcgtacggc 60
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 120
tcctgctctg gaagcagctc caacattggg aattggtatg tatcctggta ccagcagctc 180
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 240
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 300
actggggacg aggccgatta ttactgcgga acatgggata gcagcctgag tgccccttgg 360
gtgttcggcg gagggaccaa ggtcaccgtc ctaggccagc ctaaggctgc ccctagcgtg 420
accctgttcc ctccttccag cgaggagctt caagctaaca aggccaccct ggtgtgtctt 480
atctctgact tctaccctgg cgctgtgacc gtggcctgga aggctgacag ctcccctgtg 540
aaggccggag tggagaccac cacacctagc aagcagtcta acaacaagta cgctgccagc 600
tcctacctga gccttacccc tgagcagtgg aagtctcaca gaagctactc ctgtcaagtg 660
acccacgagg gcagcaccgt ggagaagacc gtggctccta ccgagtgttc c 711
<210> 103
<211> 1476
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность C3E-7085-HC2-k делеция
<400> 103
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaagtgcagc tggtggaatc cggggggggc ctggtgcagc ctggggggag cctgagactg 120
agttgtgccg cctctggggt gacatttaac tactatggca tgtcttggat ccgccaggca 180
cctggaaagg gcctggagtg ggtggccagc atcactaggt ccggcgggcg aatctactat 240
cccgacagcg tcaagggcag gttcacaatt tcccgcgaga acacacagaa aactctgtac 300
ctgcagatga atagcctgag agccgaagat acagctgtgt actattgcac tctggacggc 360
agggatgggt gggtcgccta ttgggggcag ggaaccctgg tgacagtcag ctccggagga 420
ggaggatctg gcggaggagg cagtggggga ggcgggtcaa actttatgct cactcagccg 480
tcctctgttt ctggcgtacc tggccaacgg gtgaccatta gctgtacggg taataccggg 540
aatatcgggt ctaactacgt gaactggtat cagcagcttc cagggacagc tcccaagttg 600
ctgatctatc gcgacgacaa aagaccctca ggggtccctg accgatttag tggcagcaaa 660
agcggtactt ccgcttccct ggcgataacc ggctttcagg ccgaagatga ggcagactac 720
tattgccagt catattccag cggcttcatc ttcggaggcg gaactaagct gacagtgttg 780
gcagccgagc ccaaatcttc tgacaaaact cacacatgcc caccctgccc agcacctgaa 840
gccgcagggg gaccctcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 900
tcccggaccc ctgaggtcac atgcgtggtg gtgtcagtga gccacgaaga ccctgaggtc 960
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccccgggag 1020
gagcagtaca acagcacgta ccgggtggtc agcgtcctca ccgtcctgca ccaggactgg 1080
ctgaatggca aggagtacaa gtgcaaggtc tccaacaaag ccctcccagc ccccatcgag 1140
aaaaccatct ccaaagccaa aggccagccc cgggaaccac aggtgtacgt gtacccccca 1200
tcccgggacg agctgaccaa gaaccaggtc agcctgacct gcctggtcaa aggcttctat 1260
cccagcgaca tcgccgtgga gtgggagagc aatggccagc ccgagaacaa ctacaagacc 1320
acccctcccg tgctggactc cgacggctcc ttcgccctcg tgagcaagct caccgtggac 1380
aagagcaggt ggcagcaggg caacgtcttc tcatgctccg tgatgcatga ggctctgcac 1440
aaccactaca cccagaagag cctctccctg tctccc 1476
<210> 104
<211> 468
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1143-Fab-HC1-k
делеция
<400> 104
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln
20 25 30
Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser Ile
35 40 45
Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe Ala
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu Asn
85 90 95
Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala Val
100 105 110
Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp Ile
115 120 125
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
210 215 220
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys
225 230 235 240
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala
245 250 255
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
260 265 270
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ser Val
275 280 285
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
290 295 300
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
305 310 315 320
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
325 330 335
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
340 345 350
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
355 360 365
Gln Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
370 375 380
Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
385 390 395 400
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp
405 410 415
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
420 425 430
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
435 440 445
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
450 455 460
Leu Ser Pro Gly
465
<210> 105
<211> 237
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1143-LC
<400> 105
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala
20 25 30
Ala Pro Gly Gln Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn
35 40 45
Ile Gly Asn Trp Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala
50 55 60
Pro Lys Leu Leu Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile
85 90 95
Thr Gly Leu Gln Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp
100 105 110
Asp Ser Ser Leu Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val
115 120 125
Thr Val Leu Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro
130 135 140
Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu
145 150 155 160
Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp
165 170 175
Ser Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln
180 185 190
Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu
195 200 205
Gln Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly
210 215 220
Ser Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
225 230 235
<210> 106
<211> 493
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность C3E-7085-HC2-k делеция
<400> 106
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr
35 40 45
Phe Asn Tyr Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr
65 70 75 80
Pro Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln
85 90 95
Lys Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp
115 120 125
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro
145 150 155 160
Ser Ser Val Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr
165 170 175
Gly Asn Thr Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln
180 185 190
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg
195 200 205
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
210 215 220
Ala Ser Leu Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr
225 230 235 240
Tyr Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys
245 250 255
Leu Thr Val Leu Ala Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr
260 265 270
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
275 280 285
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
290 295 300
Glu Val Thr Cys Val Val Val Ser Val Ser His Glu Asp Pro Glu Val
305 310 315 320
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
325 330 335
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
340 345 350
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
355 360 365
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
370 375 380
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro
385 390 395 400
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
405 410 415
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
420 425 430
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
435 440 445
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
450 455 460
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
465 470 475 480
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
485 490
<210> 107
<211> 1497
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1143-HC1-k делеция
<400> 107
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagc agccgagccc aaatcttctg acaaaactca cacatgccca 840
ccctgcccag cacctgaagc cgcaggggga ccctcagtct tcctcttccc cccaaaaccc 900
aaggacaccc tcatgatctc ccggacccct gaggtcacat gcgtggtggt gtcagtgagc 960
cacgaagacc ctgaggtcaa gttcaactgg tacgtggacg gcgtggaggt gcataatgcc 1020
aagacaaagc cccgggagga gcagtacaac agcacgtacc gggtggtcag cgtcctcacc 1080
gtcctgcacc aggactggct gaatggcaag gagtacaagt gcaaggtctc caacaaagcc 1140
ctcccagccc ccatcgagaa aaccatctcc aaagccaaag gccagccccg ggaaccacag 1200
gtgtacgtgc tgcccccatc ccgggacgag ctgaccaaga accaggtcag cctgctgtgc 1260
ctggtcaaag gcttctatcc cagcgacatc gccgtggagt gggagagcaa tggccagccc 1320
gagaacaact acctgacctg gcctcccgtg ctggactccg acggctcctt cttcctctac 1380
agcaagctca ccgtggacaa gagcaggtgg cagcagggca acgtcttctc atgctccgtg 1440
atgcatgagg ctctgcacaa ccactacacc cagaagagcc tctccctgtc tcccggc 1497
<210> 108
<211> 499
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1143-HC1-k делеция
<400> 108
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Ala Ala Glu Pro Lys Ser
260 265 270
Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
275 280 285
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ser Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr Trp Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly
<210> 109
<211> 2142
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность C3E-7085-NYA-1154-Fab-HC2-k
делеция
<400> 109
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaagtgcagc tggtggaatc cggggggggc ctggtgcagc ctggggggag cctgagactg 120
agttgtgccg cctctggggt gacatttaac tactatggca tgtcttggat ccgccaggca 180
cctggaaagg gcctggagtg ggtggccagc atcactaggt ccggcgggcg aatctactat 240
cccgacagcg tcaagggcag gttcacaatt tcccgcgaga acacacagaa aactctgtac 300
ctgcagatga atagcctgag agccgaagat acagctgtgt actattgcac tctggacggc 360
agggatgggt gggtcgccta ttgggggcag ggaaccctgg tgacagtcag ctccggagga 420
ggaggatctg gcggaggagg cagtggggga ggcgggtcaa actttatgct cactcagccg 480
tcctctgttt ctggcgtacc tggccaacgg gtgaccatta gctgtacggg taataccggg 540
aatatcgggt ctaactacgt gaactggtat cagcagcttc cagggacagc tcccaagttg 600
ctgatctatc gcgacgacaa aagaccctca ggggtccctg accgatttag tggcagcaaa 660
agcggtactt ccgcttccct ggcgataacc ggctttcagg ccgaagatga ggcagactac 720
tattgccagt catattccag cggcttcatc ttcggaggcg gaactaagct gacagtgttg 780
gggggaggcg gttcagaggt gcagctggtg gagtctgggg gaggcgtggt ccagcctggg 840
aggtccctga caccctcctg ttcagcgtct ggattctcca tccgtagtta tgatatgcac 900
tgggtccgcc aggctccagg caaggggcta gagtgggtgg ccactatatc atatgatgga 960
agtcagaagt acttcgcaga ctccgtgaag ggccgattta ccatcttcag agacgaatcg 1020
gagaacatgg tgtatctgca aatgagcggc ctgagagttg aggacacggc cgtttatcac 1080
tgtgcgagag ggagtagtaa tcattatgag gcttttgata tctggggcca agggacaatg 1140
gtcaccgtct cttcagcctc caccaagggc ccaagcgtct tccccctggc accctcctcc 1200
aagagcacct ctggcggcac agccgccctg ggctgcctgg tcaaggacta cttccccgaa 1260
cccgtgaccg tgagctggaa ctcaggcgcc ctgaccagcg gcgtgcacac cttccccgct 1320
gtcctgcagt cctcaggact ctactccctc agcagcgtgg tgaccgtgcc ctccagcagc 1380
ttgggcaccc agacctacat ctgcaacgtg aatcacaagc ccagcaacac caaggtggac 1440
aagagagttg agcccaaatc ttgtgacaaa actcacacat gcccaccctg cccagcacct 1500
gaagccgcag ggggaccctc agtcttcctc ttccccccaa aacccaagga caccctcatg 1560
atctcccgga cccctgaggt cacatgcgtg gtggtgtcag tgagccacga agaccctgag 1620
gtcaagttca actggtacgt ggacggcgtg gaggtgcata atgccaagac aaagccccgg 1680
gaggagcagt acaacagcac gtaccgggtg gtcagcgtcc tcaccgtcct gcaccaggac 1740
tggctgaatg gcaaggagta caagtgcaag gtctccaaca aagccctccc agcccccatc 1800
gagaaaacca tctccaaagc caaaggccag ccccgggaac cacaggtgta cgtgtacccc 1860
ccatcccggg acgagctgac caagaaccag gtcagcctga cctgcctggt caaaggcttc 1920
tatcccagcg acatcgccgt ggagtgggag agcaatggcc agcccgagaa caactacaag 1980
accacccctc ccgtgctgga ctccgacggc tccttcgccc tcgtgagcaa gctcaccgtg 2040
gacaagagca ggtggcagca gggcaacgtc ttctcatgct ccgtgatgca tgaggctctg 2100
cacaaccact acacccagaa gagcctctcc ctgtctcccg gc 2142
<210> 110
<211> 711
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-1154-LC
<400> 110
atggtgctgc agacccaggt gttcatctcc ctgctgctgt ggatctccgg cgcgtacggc 60
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 120
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcggctc 180
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 240
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 300
actggggacg aggccgatta ctactgcgga gcatgggata gcagcctgag tgccccttgg 360
gtgttcggcg gagggaccaa ggtcaccgtc ctaggccagc ctaaggctgc ccctagcgtg 420
accctgttcc ctccttccag cgaggagctt caagctaaca aggccaccct ggtgtgtctt 480
atctctgact tctaccctgg cgctgtgacc gtggcctgga aggctgacag ctcccctgtg 540
aaggccggag tggagaccac cacacctagc aagcagtcta acaacaagta cgctgccagc 600
tcctacctga gccttacccc tgagcagtgg aagtctcaca gaagctactc ctgtcaagtg 660
acccacgagg gcagcaccgt ggagaagacc gtggctccta ccgagtgttc c 711
<210> 111
<211> 735
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность OAA-HC1-k делеция
<400> 111
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgac 60
aaaactcaca catgcccacc ctgcccagca cctgaagccg cagggggacc ctcagtcttc 120
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 180
gtggtggtgt cagtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 240
gtggaggtgc ataatgccaa gacaaagccc cgggaggagc agtacaacag cacgtaccgg 300
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 360
aaggtctcca acaaagccct cccagccccc atcgagaaaa ccatctccaa agccaaaggc 420
cagccccggg aaccacaggt gtacgtgctg cccccatccc gggacgagct gaccaagaac 480
caggtcagcc tgctgtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 540
gagagcaatg gccagcccga gaacaactac ctgacctggc ctcccgtgct ggactccgac 600
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcagggcaac 660
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacaccca gaagagcctc 720
tccctgtctc ccggc 735
<210> 112
<211> 714
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность C3E-7085-NYA-1154-Fab-
HC2-k делеция
<400> 112
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr
35 40 45
Phe Asn Tyr Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr
65 70 75 80
Pro Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln
85 90 95
Lys Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp
115 120 125
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro
145 150 155 160
Ser Ser Val Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr
165 170 175
Gly Asn Thr Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln
180 185 190
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg
195 200 205
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
210 215 220
Ala Ser Leu Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr
225 230 235 240
Tyr Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys
245 250 255
Leu Thr Val Leu Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
260 265 270
Gly Gly Gly Val Val Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser
275 280 285
Ala Ser Gly Phe Ser Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln
290 295 300
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly
305 310 315 320
Ser Gln Lys Tyr Phe Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe
325 330 335
Arg Asp Glu Ser Glu Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg
340 345 350
Val Glu Asp Thr Ala Val Tyr His Cys Ala Arg Gly Ser Ser Asn His
355 360 365
Tyr Glu Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser
370 375 380
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
385 390 395 400
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
405 410 415
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
420 425 430
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
435 440 445
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
450 455 460
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
465 470 475 480
Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
485 490 495
Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro
500 505 510
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
515 520 525
Cys Val Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
530 535 540
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
545 550 555 560
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
565 570 575
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
580 585 590
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
595 600 605
Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp
610 615 620
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
625 630 635 640
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
645 650 655
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
660 665 670
Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
675 680 685
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
690 695 700
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
705 710
<210> 113
<211> 237
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-1154-LC
<400> 113
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala
20 25 30
Ala Pro Gly Gln Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn
35 40 45
Ile Gly Asn Asn Tyr Val Ser Trp Tyr Gln Arg Leu Pro Gly Thr Ala
50 55 60
Pro Lys Leu Leu Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile
85 90 95
Thr Gly Leu Gln Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Ala Trp
100 105 110
Asp Ser Ser Leu Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val
115 120 125
Thr Val Leu Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro
130 135 140
Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu
145 150 155 160
Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp
165 170 175
Ser Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln
180 185 190
Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu
195 200 205
Gln Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly
210 215 220
Ser Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
225 230 235
<210> 114
<211> 245
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность OAA-HC1-k делеция
<400> 114
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
20 25 30
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
35 40 45
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ser
50 55 60
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
65 70 75 80
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
85 90 95
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
100 105 110
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
115 120 125
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
130 135 140
Pro Gln Val Tyr Val Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
145 150 155 160
Gln Val Ser Leu Leu Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
165 170 175
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Leu Thr
180 185 190
Trp Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
195 200 205
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
210 215 220
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
225 230 235 240
Ser Leu Ser Pro Gly
245
<210> 115
<211> 2232
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0010-HC2-k делеция
<400> 115
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tgcactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggccgttt atcactgtgc gagagggagt 360
agtaatcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc ggctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattactact gcggagcatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gc 2232
<210> 116
<211> 2232
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0004-HC2-k делеция
<400> 116
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaagtgcagc tggtggaatc cggggggggc ctggtgcagc ctggggggag cctgagactg 120
agttgtgccg cctctggggt gacatttaac tactatggca tgtcttggat ccgccaggca 180
cctggaaagg gcctggagtg ggtggccagc atcactaggt ccggcgggcg aatctactat 240
cccgacagcg tcaagggcag gttcacaatt tcccgcgaga acacacagaa aactctgtac 300
ctgcagatga atagcctgag agccgaagat acagctgtgt actattgcac tctggacggc 360
agggatgggt gggtcgccta ttgggggcag ggaaccctgg tgacagtcag ctccggagga 420
ggaggatctg gcggaggagg cagtggggga ggcgggtcaa actttatgct cactcagccg 480
tcctctgttt ctggcgtacc tggccaacgg gtgaccatta gctgtacggg taataccggg 540
aatatcgggt ctaactacgt gaactggtat cagcagcttc cagggacagc tcccaagttg 600
ctgatctatc gcgacgacaa aagaccctca ggggtccctg accgatttag tggcagcaaa 660
agcggtactt ccgcttccct ggcgataacc ggctttcagg ccgaagatga ggcagactac 720
tattgccagt catattccag cggcttcatc ttcggaggcg gaactaagct gacagtgttg 780
gggggaggcg gttcagaggt gcagctggtg gagtctgggg gaggcgtggt ccagcctggg 840
aggtccctga caccctcctg ttcagcgtct ggattctcca tccgtagtta tgatatgcac 900
tgggtccgcc aggctccagg caaggggcta gagtgggtgg ccactatatc atatgatgga 960
agtcagaagt acttcgcaga ctccgtgaag ggccgattta ccatcttcag agacgaatcg 1020
gagaacatgg tgtatctgca aatgagcggc ctgagagttg aggacacggc cgtttatcac 1080
tgtgcgagag ggagtagtaa tcattatgag gcttttgata tctggggcca agggacaatg 1140
gtcaccgtct cttcaggtgg aggcggttca ggcggaggtg gcagcggcgg tggcgggagt 1200
cagtctgtgt tgacgcagcc gccctcagtg tctgcggccc caggacagaa ggtcaccatc 1260
tcctgctctg gaagcagctc caacattggg aataattatg tatcctggta ccagcggctc 1320
ccaggaacag cccccaaact cctcatttat gacaataata agcgaccctc agggattcct 1380
gaccgattct ctggctccaa gtctggcacg tcagccaccc tgggcatcac cggactccag 1440
actggggacg aggccgatta ctactgcgga gcatgggata gcagcctgag tgccccttgg 1500
gtgttcggcg gagggaccaa ggtcaccgtc ctagcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gc 2232
<210> 117
<211> 2232
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0011-HC2-k делеция
<400> 117
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agctatgata tacactgggt ccgcctggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaattg gtatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gc 2232
<210> 118
<211> 744
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0010-HC2-k делеция
<400> 118
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Asn His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Ala Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly
740
<210> 119
<211> 744
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0004-HC2-k делеция
<400> 119
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr
35 40 45
Phe Asn Tyr Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr
65 70 75 80
Pro Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln
85 90 95
Lys Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp
115 120 125
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro
145 150 155 160
Ser Ser Val Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr
165 170 175
Gly Asn Thr Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln
180 185 190
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg
195 200 205
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
210 215 220
Ala Ser Leu Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr
225 230 235 240
Tyr Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys
245 250 255
Leu Thr Val Leu Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
260 265 270
Gly Gly Gly Val Val Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser
275 280 285
Ala Ser Gly Phe Ser Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln
290 295 300
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly
305 310 315 320
Ser Gln Lys Tyr Phe Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe
325 330 335
Arg Asp Glu Ser Glu Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg
340 345 350
Val Glu Asp Thr Ala Val Tyr His Cys Ala Arg Gly Ser Ser Asn His
355 360 365
Tyr Glu Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser
370 375 380
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
385 390 395 400
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
405 410 415
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
420 425 430
Tyr Val Ser Trp Tyr Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu
435 440 445
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
450 455 460
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
465 470 475 480
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Ala Trp Asp Ser Ser Leu
485 490 495
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly
740
<210> 120
<211> 744
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0011-HC2-k делеция
<400> 120
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Leu Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly
740
<210> 121
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 1F
<400> 121
Phe Leu Leu Met Trp Ile Thr Gln Cys
1 5
<210> 122
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 2M
<400> 122
Ser Met Leu Met Trp Ile Thr Gln Cys
1 5
<210> 123
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 3A
<400> 123
Ser Leu Ala Met Trp Ile Thr Gln Cys
1 5
<210> 124
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 4A
<400> 124
Ser Leu Leu Ala Trp Ile Thr Gln Cys
1 5
<210> 125
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 5A
<400> 125
Ser Leu Leu Met Ala Ile Thr Gln Cys
1 5
<210> 126
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 6L
<400> 126
Ser Leu Leu Met Trp Leu Thr Gln Cys
1 5
<210> 127
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 7F
<400> 127
Ser Leu Leu Met Trp Ile Phe Gln Cys
1 5
<210> 128
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 8A
<400> 128
Ser Leu Leu Met Trp Ile Thr Ala Cys
1 5
<210> 129
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность точечно мутированного NY-ESO пептида 9A
<400> 129
Ser Leu Leu Met Trp Ile Thr Gln Ala
1 5
<210> 130
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность gp100 пептида
<400> 130
Ile Met Asp Gln Val Pro Phe Ser Val
1 5
<210> 131
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность гомологичного пептида DOLPP1
<400> 131
Ser Gln Phe Met Trp Phe Phe Ser Val
1 5
<210> 132
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность гомологичного пептида IL20RB
<400> 132
Ser Leu Phe Met Trp Phe Phe Tyr Ala
1 5
<210> 133
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность гомологичного пептида PRKD2
<400> 133
Ser Leu Asp Met Trp Ser Val Gly Val
1 5
<210> 134
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность гомологичного пептида CD163
<400> 134
Ser Ile Pro Met Trp Val Asp Asn Val
1 5
<210> 135
<211> 9
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность гомологичного пептида P2RY8
<400> 135
Ser Val Ala Met Trp Ala Val Phe Leu
1 5
<210> 136
<211> 269
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность C3E-7034
<400> 136
Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr
20 25 30
Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Ser Ile Thr Asn Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser
50 55 60
Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro His Ser Val
130 135 140
Ser Glu Ser Pro Gly Lys Thr Val Thr Ile Ser Cys Lys Arg Asn Thr
145 150 155 160
Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln His Glu Gly
165 170 175
Ser Ser Pro Thr Thr Ile Ile Tyr Arg Asp Asp Lys Arg Pro Asp Gly
180 185 190
Val Ser Asp Arg Phe Ser Gly Ser Ile Asp Arg Ser Ser Lys Ser Ala
195 200 205
Ser Leu Thr Ile Ser Asn Leu Lys Thr Glu Asp Glu Ala Asp Tyr Phe
210 215 220
Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu
225 230 235 240
Thr Val Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp
245 250 255
Asp Asp Lys Gly Ala Ala Ala His His His His His His
260 265
<210> 137
<211> 267
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность C3E-7036
<400> 137
Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr
20 25 30
Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Ser Ile Thr Asn Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser
50 55 60
Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val
130 135 140
Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr
145 150 155 160
Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly
165 170 175
Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu
195 200 205
Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln
210 215 220
Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val
225 230 235 240
Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp
245 250 255
Lys Gly Ala Ala Ala His His His His His His
260 265
<210> 138
<211> 267
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность C3E-7085
<400> 138
Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr
20 25 30
Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser
50 55 60
Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val
130 135 140
Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr
145 150 155 160
Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly
165 170 175
Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu
195 200 205
Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln
210 215 220
Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val
225 230 235 240
Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp
245 250 255
Lys Gly Ala Ala Ala His His His His His His
260 265
<210> 139
<211> 269
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность C3E-7088
<400> 139
Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr
20 25 30
Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser
50 55 60
Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro His Ser Val
130 135 140
Ser Glu Ser Pro Gly Lys Thr Val Thr Ile Ser Cys Lys Arg Asn Thr
145 150 155 160
Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln His Glu Gly
165 170 175
Ser Ser Pro Thr Thr Ile Ile Tyr Arg Asn Asp Lys Arg Pro Asp Gly
180 185 190
Val Ser Asp Arg Phe Ser Gly Ser Ile Asp Arg Ser Ser Lys Ser Ala
195 200 205
Ser Leu Thr Ile Ser Asn Leu Lys Thr Glu Asp Glu Ala Asp Tyr Phe
210 215 220
Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu
225 230 235 240
Thr Val Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp
245 250 255
Asp Asp Lys Gly Ala Ala Ala His His His His His His
260 265
<210> 140
<211> 269
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность C3E-7093
<400> 140
Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr
20 25 30
Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Ser Ile Thr Ser Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser
50 55 60
Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro His Ser Val
130 135 140
Ser Glu Ser Pro Gly Lys Thr Val Thr Ile Ser Cys Lys Arg Asn Thr
145 150 155 160
Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln His Glu Gly
165 170 175
Ser Ser Pro Thr Thr Ile Ile Tyr Arg Asn Asp Lys Arg Pro Asp Gly
180 185 190
Val Ser Asp Arg Phe Ser Gly Ser Ile Asp Arg Ser Ser Lys Ser Ala
195 200 205
Ser Leu Thr Ile Ser Asn Leu Lys Thr Glu Asp Glu Ala Asp Tyr Phe
210 215 220
Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu
225 230 235 240
Thr Val Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp
245 250 255
Asp Asp Lys Gly Ala Ala Ala His His His His His His
260 265
<210> 141
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность CDR1 тяжелой цепи C3E-7085
<400> 141
Gly Val Thr Phe Asn Tyr Tyr Gly
1 5
<210> 142
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность CDR2 тяжелой цепи C3E-7085
<400> 142
Ile Thr Arg Ser Gly Gly Arg Ile
1 5
<210> 143
<211> 11
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность CDR3 тяжелой цепи C3E-7085
<400> 143
Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr
1 5 10
<210> 144
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность CDR1 легкой цепи C3E-7085
<400> 144
Thr Gly Asn Ile Gly Ser Asn Tyr
1 5
<210> 145
<211> 3
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность CDR2 легкой цепи C3E-7085
<400> 145
Arg Asp Asp
1
<210> 146
<211> 8
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность CDR3 легкой цепи C3E-7085
<400> 146
Gln Ser Tyr Ser Ser Gly Phe Ile
1 5
<210> 147
<211> 269
<212> БЕЛОК
<213> Искусственная
<220>
<223> Аминокислотная последовательность C3E-7078
<400> 147
Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
1 5 10 15
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr
20 25 30
Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
35 40 45
Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser
50 55 60
Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu
65 70 75 80
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro His Ser Val
130 135 140
Ser Glu Ser Pro Gly Lys Thr Val Thr Ile Ser Cys Lys Arg Asn Thr
145 150 155 160
Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln His Glu Gly
165 170 175
Ser Ser Pro Thr Thr Ile Ile Tyr Arg Asp Asp Lys Arg Pro Asp Gly
180 185 190
Val Ser Asp Arg Phe Ser Gly Ser Ile Asp Arg Ser Ser Lys Ser Ala
195 200 205
Ser Leu Thr Ile Ser Asn Leu Lys Thr Glu Asp Glu Ala Asp Tyr Phe
210 215 220
Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu
225 230 235 240
Thr Val Leu Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp
245 250 255
Asp Asp Lys Gly Ala Ala Ala His His His His His His
260 265
<210> 148
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYF-0014-HC2
<400> 148
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgga 60
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgacaccc 120
tcctgttcag cgtctggatt ctccatccgt agttatgata tacactgggt ccgccaggct 180
ccaggcaagg ggctagagtg ggtggccact atatcatatg atggaagtca gaagtacttc 240
gcagactccg tgaagggccg atttaccatc ttcagagacg aatcggagaa catggtgtat 300
ctgcaaatga gcggcctgag agttgaggac acggctgttt atcactgtgc gagagggagt 360
agtggtcatt atgaggcttt tgatatctgg ggccaaggga caatggtcac cgtctcttca 420
ggtggaggcg gttcaggcgg aggtggcagc ggcggtggcg ggagtcagtc tgtgttgacg 480
cagccgccct cagtgtctgc ggccccagga cagaaggtca ccatctcctg ctctggaagc 540
agctccaaca ttgggaataa ttatgtatcc tggtaccagc agctcccagg aacagccccc 600
aaactcctca tttatgacaa taataagcga ccctcaggga ttcctgaccg attctctggc 660
tccaagtctg gcacgtcagc caccctgggc atcaccggac tccagactgg ggacgaggcc 720
gattattact gcggaacatg ggatagcagc ctgagtgccc cttgggtgtt cggcggaggg 780
accaaggtca ccgtcctagg gggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
cagggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac agctcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 149
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0014-HC2
<400> 149
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 150
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYF-0082-HC2
<400> 150
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Thr Pro Ser Cys Ser Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Phe
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Phe Arg Asp Glu Ser Glu
85 90 95
Asn Met Val Tyr Leu Gln Met Ser Gly Leu Arg Val Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr
180 185 190
Gln Arg Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Ser Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 151
<211> 207
<212> БЕЛОК
<213> Homo sapiens
<400> 151
Met Gln Ser Gly Thr His Trp Arg Val Leu Gly Leu Cys Leu Leu Ser
1 5 10 15
Val Gly Val Trp Gly Gln Asp Gly Asn Glu Glu Met Gly Gly Ile Thr
20 25 30
Gln Thr Pro Tyr Lys Val Ser Ile Ser Gly Thr Thr Val Ile Leu Thr
35 40 45
Cys Pro Gln Tyr Pro Gly Ser Glu Ile Leu Trp Gln His Asn Asp Lys
50 55 60
Asn Ile Gly Gly Asp Glu Asp Asp Lys Asn Ile Gly Ser Asp Glu Asp
65 70 75 80
His Leu Ser Leu Lys Glu Phe Ser Glu Leu Glu Gln Ser Gly Tyr Tyr
85 90 95
Val Cys Tyr Pro Arg Gly Ser Lys Pro Glu Asp Ala Asn Phe Tyr Leu
100 105 110
Tyr Leu Arg Ala Arg Val Cys Glu Asn Cys Met Glu Met Asp Val Met
115 120 125
Ser Val Ala Thr Ile Val Ile Val Asp Ile Cys Ile Thr Gly Gly Leu
130 135 140
Leu Leu Leu Val Tyr Tyr Trp Ser Lys Asn Arg Lys Ala Lys Ala Lys
145 150 155 160
Pro Val Thr Arg Gly Ala Gly Ala Gly Gly Arg Gln Arg Gly Gln Asn
165 170 175
Lys Glu Arg Pro Pro Pro Val Pro Asn Pro Asp Tyr Glu Pro Ile Arg
180 185 190
Lys Gly Gln Arg Asp Leu Tyr Ser Gly Leu Asn Gln Arg Arg Ile
195 200 205
<210> 152
<211> 2235
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYZ-0038-HC2
<400> 152
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg aggaggcggt tcagaagtgc agctggtgga atccgggggg 840
ggcctggtgc agcctggggg gagcctgaga ctgagttgtg ccgcctctgg ggtgacattt 900
aactactatg gcatgtcttg gatccgccag gcacctggaa agggcctgga gtgggtggcc 960
agcatcacta ggtccggcgg gcgaatctac tatcccgaca gcgtcaaggg caggttcaca 1020
atttcccgcg agaacacaca gaaaactctg tacctgcaga tgaatagcct gagagccgaa 1080
gatacagctg tgtactattg cactctggac ggcagggatg ggtgggtcgc ctattggggg 1140
tgtggaaccc tggtgacagt cagctccgga ggaggaggat ctggcggagg aggcagtggg 1200
ggaggcgggt caaactttat gctcactcag ccgtcctctg tttctggcgt acctggccaa 1260
cgggtgacca ttagctgtac gggtaatacc gggaatatcg ggtctaacta cgtgaactgg 1320
tatcagcagc ttccagggac atgtcccaag ttgctgatct atcgcgacga caaaagaccc 1380
tcaggggtcc ctgaccgatt tagtggcagc aaaagcggta cttccgcttc cctggcgata 1440
accggctttc aggccgaaga tgaggcagac tactattgcc agtcatattc cagcggcttc 1500
atcttcggag gcggaactaa gctgacagtg ttggcagccg agcccaaatc ttctgacaaa 1560
actcacacat gcccaccctg cccagcacct gaagccgcag ggggaccctc agtcttcctc 1620
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 1680
gtggtgtcag tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 1740
gaggtgcata atgccaagac aaagccccgg gaggagcagt acaacagcac gtaccgggtg 1800
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 1860
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaaggccag 1920
ccccgggaac cacaggtgta cgtgtacccc ccatcccggg acgagctgac caagaaccag 1980
gtcagcctga cctgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 2040
agcaatggcc agcccgagaa caactacaag accacccctc ccgtgctgga ctccgacggc 2100
tccttcgccc tcgtgagcaa gctcaccgtg gacaagagca ggtggcagca gggcaacgtc 2160
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacccagaa gagcctctcc 2220
ctgtctcccg gcaag 2235
<210> 153
<211> 2250
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYZ-0082-HC2
<400> 153
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtggagg aggtggatct 480
caatccgttc tgactcagcc accctccgcc tctggcaccc cgggccaacg ggtcacaata 540
tcatgttctg gctcttcaag caacatcgga aactggtacg tgagctggta ccagcagctc 600
cccggcacgg cgcccaagct tctgatatat gacaacaaca aacggcccag tggagttcct 660
gacagattca gtgggtctaa aagtggtaca agcgctagcc tggccataag tggtctgcag 720
agtgaagatg aggcggacta ttattgcgga acctgggact ccagcctgag cgctccctgg 780
gttttcggcg gaggtactaa gctgaccgtc ttgggaggag gcggttcaga agtgcagctg 840
gtggaatccg gggggggcct ggtgcagcct ggggggagcc tgagactgag ttgtgccgcc 900
tctggggtga catttaacta ctatggcatg tcttggatcc gccaggcacc tggaaagggc 960
ctggagtggg tggccagcat cactaggtcc ggcgggcgaa tctactatcc cgacagcgtc 1020
aagggcaggt tcacaatttc ccgcgagaac acacagaaaa ctctgtacct gcagatgaat 1080
agcctgagag ccgaagatac agctgtgtac tattgcactc tggacggcag ggatgggtgg 1140
gtcgcctatt gggggtgtgg aaccctggtg acagtcagct ccggaggagg aggatctggc 1200
ggaggaggca gtgggggagg cgggtcaaac tttatgctca ctcagccgtc ctctgtttct 1260
ggcgtacctg gccaacgggt gaccattagc tgtacgggta ataccgggaa tatcgggtct 1320
aactacgtga actggtatca gcagcttcca gggacatgtc ccaagttgct gatctatcgc 1380
gacgacaaaa gaccctcagg ggtccctgac cgatttagtg gcagcaaaag cggtacttcc 1440
gcttccctgg cgataaccgg ctttcaggcc gaagatgagg cagactacta ttgccagtca 1500
tattccagcg gcttcatctt cggaggcgga actaagctga cagtgttggc agccgagccc 1560
aaatcttctg acaaaactca cacatgccca ccctgcccag cacctgaagc cgcaggggga 1620
ccctcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 1680
gaggtcacat gcgtggtggt gtcagtgagc cacgaagacc ctgaggtcaa gttcaactgg 1740
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cccgggagga gcagtacaac 1800
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 1860
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1920
aaagccaaag gccagccccg ggaaccacag gtgtacgtgt accccccatc ccgggacgag 1980
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 2040
gccgtggagt gggagagcaa tggccagccc gagaacaact acaagaccac ccctcccgtg 2100
ctggactccg acggctcctt cgccctcgtg agcaagctca ccgtggacaa gagcaggtgg 2160
cagcagggca acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacc 2220
cagaagagcc tctccctgtc tcccggcaag 2250
<210> 154
<211> 2250
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYZ-0083-HC2
<400> 154
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtggagg aggtggatct 480
caatccgttc tgactcagcc accctccgcc tctggcaccc cgggccaacg ggtcacaata 540
tcatgttctg gctcttcaag caacatcgga aactggtacg tgagctggta ccagcagctc 600
cccggcacgg cgcccaagct tctgatatat gacaacaaca aacggcccag tggagttcct 660
gacagattca gtgggtctaa aagtggtaca agcgctagcc tggccataag tggtctgcag 720
agtgaagatg aggcggacta ttattgcgga acctgggact ccagcctgag cgctccctgg 780
gttttcggcg gaggtactaa gctgaccgtc ttgggaggag gcggttcaga agtgcagctg 840
gtggaatccg gggggggcct ggtgcagcct ggggggagcc tgagactgag ttgtgccgcc 900
tctggggtga catttaacta ctatggcatg tcttggatcc gccaggcacc tggaaagtgt 960
ctggagtggg tggccagcat cactaggtcc ggcgggcgaa tctactatcc cgacagcgtc 1020
aagggcaggt tcacaatttc ccgcgagaac acacagaaaa ctctgtacct gcagatgaat 1080
agcctgagag ccgaagatac agctgtgtac tattgcactc tggacggcag ggatgggtgg 1140
gtcgcctatt gggggcaggg aaccctggtg acagtcagct ccggaggagg aggatctggc 1200
ggaggaggca gtgggggagg cgggtcaaac tttatgctca ctcagccgtc ctctgtttct 1260
ggcgtacctg gccaacgggt gaccattagc tgtacgggta ataccgggaa tatcgggtct 1320
aactacgtga actggtatca gcagcttcca gggacagctc ccaagttgct gatctatcgc 1380
gacgacaaaa gaccctcagg ggtccctgac cgatttagtg gcagcaaaag cggtacttcc 1440
gcttccctgg cgataaccgg ctttcaggcc gaagatgagg cagactacta ttgccagtca 1500
tattccagcg gcttcatctt cggatgtgga actaagctga cagtgttggc agccgagccc 1560
aaatcttctg acaaaactca cacatgccca ccctgcccag cacctgaagc cgcaggggga 1620
ccctcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 1680
gaggtcacat gcgtggtggt gtcagtgagc cacgaagacc ctgaggtcaa gttcaactgg 1740
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cccgggagga gcagtacaac 1800
agcacgtacc gggtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 1860
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1920
aaagccaaag gccagccccg ggaaccacag gtgtacgtgt accccccatc ccgggacgag 1980
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 2040
gccgtggagt gggagagcaa tggccagccc gagaacaact acaagaccac ccctcccgtg 2100
ctggactccg acggctcctt cgccctcgtg agcaagctca ccgtggacaa gagcaggtgg 2160
cagcagggca acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacc 2220
cagaagagcc tctccctgtc tcccggcaag 2250
<210> 155
<211> 745
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYZ-0038-HC2
<400> 155
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Cys Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
405 410 415
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
420 425 430
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Cys
435 440 445
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
450 455 460
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
465 470 475 480
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
485 490 495
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Ala
500 505 510
Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
515 520 525
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
530 535 540
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
545 550 555 560
Val Val Ser Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
565 570 575
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
580 585 590
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
595 600 605
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
610 615 620
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
625 630 635 640
Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu
645 650 655
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
660 665 670
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
675 680 685
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu
690 695 700
Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
705 710 715 720
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
725 730 735
Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745
<210> 156
<211> 750
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYZ-0082-HC2
<400> 156
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
165 170 175
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
180 185 190
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
225 230 235 240
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
245 250 255
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
260 265 270
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
275 280 285
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr
290 295 300
Phe Asn Tyr Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly
305 310 315 320
Leu Glu Trp Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr
325 330 335
Pro Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln
340 345 350
Lys Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
355 360 365
Val Tyr Tyr Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp
370 375 380
Gly Cys Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro
405 410 415
Ser Ser Val Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr
420 425 430
Gly Asn Thr Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln
435 440 445
Leu Pro Gly Thr Cys Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg
450 455 460
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
465 470 475 480
Ala Ser Leu Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr
485 490 495
Tyr Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys
500 505 510
Leu Thr Val Leu Ala Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr
515 520 525
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
530 535 540
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
545 550 555 560
Glu Val Thr Cys Val Val Val Ser Val Ser His Glu Asp Pro Glu Val
565 570 575
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
580 585 590
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
595 600 605
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
610 615 620
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
625 630 635 640
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro
645 650 655
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
660 665 670
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
675 680 685
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
690 695 700
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
705 710 715 720
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
725 730 735
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745 750
<210> 157
<211> 750
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYZ-0083-HC2
<400> 157
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
165 170 175
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
180 185 190
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
225 230 235 240
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
245 250 255
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
260 265 270
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
275 280 285
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr
290 295 300
Phe Asn Tyr Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Cys
305 310 315 320
Leu Glu Trp Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr
325 330 335
Pro Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln
340 345 350
Lys Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
355 360 365
Val Tyr Tyr Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp
370 375 380
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly
385 390 395 400
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro
405 410 415
Ser Ser Val Ser Gly Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr
420 425 430
Gly Asn Thr Gly Asn Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln
435 440 445
Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg
450 455 460
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
465 470 475 480
Ala Ser Leu Ala Ile Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr
485 490 495
Tyr Cys Gln Ser Tyr Ser Ser Gly Phe Ile Phe Gly Cys Gly Thr Lys
500 505 510
Leu Thr Val Leu Ala Ala Glu Pro Lys Ser Ser Asp Lys Thr His Thr
515 520 525
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
530 535 540
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
545 550 555 560
Glu Val Thr Cys Val Val Val Ser Val Ser His Glu Asp Pro Glu Val
565 570 575
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
580 585 590
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
595 600 605
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
610 615 620
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
625 630 635 640
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro Pro
645 650 655
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
660 665 670
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
675 680 685
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
690 695 700
Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
705 710 715 720
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
725 730 735
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
740 745 750
<210> 158
<211> 2172
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYZ-1010-HC2
<400> 158
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtggagg aggtggatct 480
caatccgttc tgactcagcc accctccgcc tctggcaccc cgggccaacg ggtcacaata 540
tcatgttctg gctcttcaag caacatcgga aactggtacg tgagctggta ccagcagctc 600
cccggcacgg cgcccaagct tctgatatat gacaacaaca aacggcccag tggagttcct 660
gacagattca gtgggtctaa aagtggtaca agcgctagcc tggccataag tggtctgcag 720
agtgaagatg aggcggacta ttattgcgga acctgggact ccagcctgag cgctccctgg 780
gttttcggcg gaggtactaa gctgaccgtc ttgggaggag gcggttcaga agtgcagctg 840
gtggaatccg gggggggcct ggtgcagcct ggggggagcc tgagactgag ttgtgccgcc 900
tctggggtga catttaacta ctatggcatg tcttggatcc gccaggcacc tggaaagggc 960
ctggagtggg tggccagcat cactaggtcc ggcgggcgaa tctactatcc cgacagcgtc 1020
aagggcaggt tcacaatttc ccgcgagaac acacagaaaa ctctgtacct gcagatgaat 1080
agcctgagag ccgaagatac agctgtgtac tattgcactc tggacggcag ggatgggtgg 1140
gtcgcctatt ggggtcaggg aaccctggtg acagtcagct ccgcctccac caagggccca 1200
agcgtcttcc ccctggcacc ctcctccaag agcacctctg gcggcacagc cgccctgggc 1260
tgcctggtca aggactactt ccccgaaccc gtgaccgtga gctggaactc aggcgccctg 1320
accagcggcg tgcacacctt ccccgctgtc ctgcagtcct caggactcta ctccctcagc 1380
agcgtggtga ccgtgccctc cagcagcttg ggcacccaga cctacatctg caacgtgaat 1440
cacaagccca gcaacaccaa ggtggacaag agagttgagc ccaagtcttg cgacaaaact 1500
cacacatgcc caccctgccc agcacctgaa gccgcagggg gaccctcagt cttcctcttc 1560
cccccaaaac ccaaggacac cctcatgatc tcccggaccc ctgaggtcac atgcgtggtg 1620
gtgtcagtga gccacgaaga ccctgaggtc aagttcaact ggtacgtgga cggcgtggag 1680
gtgcataatg ccaagacaaa gccccgggag gagcagtaca acagcacgta ccgggtggtc 1740
agcgtcctca ctgtcctgca ccaggactgg ctgaatggca aggagtacaa gtgcaaggtc 1800
tccaacaaag ccctcccagc ccccatcgag aaaaccatct ccaaagccaa aggccagccc 1860
cgggaaccac aggtgtacgt gtacccccca tcccgggacg agctgaccaa gaaccaggtc 1920
agcctgacct gcctggtcaa aggcttctat cccagcgaca tcgccgtgga gtgggagagc 1980
aatggccagc ccgagaacaa ctacaagacc acccctcccg tgctggactc cgacggctcc 2040
ttcgccctcg tgagcaagct caccgtggac aagagcaggt ggcagcaggg caacgtcttc 2100
tcatgctccg tgatgcatga ggctctgcac aaccactaca cccagaagag cctctccctg 2160
tctcccggca ag 2172
<210> 159
<211> 699
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность C3E-7085-LC
<400> 159
atggtgctgc agacccaggt gttcatctcc ctgctgctgt ggatctccgg cgcgtacggc 60
aactttatgc tcactcagcc gtcctctgtt tctggcgtac ctggccaacg ggtgaccatt 120
agctgtacgg gtaataccgg gaatatcggg tctaactacg tgaactggta tcagcagctt 180
ccagggacag ctcccaagtt gctgatctat cgcgacgaca aaagaccctc aggggtccct 240
gaccgattta gtggcagcaa aagcggtact tccgcttccc tggcgataac cggctttcag 300
gccgaagatg aggcagacta ctattgccag tcatattcca gcggcttcat cttcggaggc 360
ggaactaagc tgacagtgct gggccagcct aaggctgccc ctagcgtgac cctgttccct 420
ccttccagcg aggagcttca agctaacaag gccaccctgg tgtgtcttat ctctgacttc 480
taccctggcg ctgtgaccgt ggcctggaag gctgacagct cccctgtgaa ggccggagtg 540
gagaccacca cacctagcaa gcagtctaac aacaagtacg ctgccagctc ctacctgagc 600
cttacccctg agcagtggaa gtctcacaga agctactcct gtcaagtgac ccacgagggc 660
agcaccgtgg agaagaccgt ggctcctacc gagtgttcc 699
<210> 160
<211> 724
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYZ-1010-HC2
<400> 160
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
165 170 175
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
180 185 190
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
225 230 235 240
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
245 250 255
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
260 265 270
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
275 280 285
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr
290 295 300
Phe Asn Tyr Tyr Gly Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly
305 310 315 320
Leu Glu Trp Val Ala Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr
325 330 335
Pro Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln
340 345 350
Lys Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
355 360 365
Val Tyr Tyr Cys Thr Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp
370 375 380
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
385 390 395 400
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
405 410 415
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
420 425 430
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
435 440 445
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
450 455 460
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
465 470 475 480
His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser
485 490 495
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
500 505 510
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
515 520 525
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ser Val Ser
530 535 540
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
545 550 555 560
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
565 570 575
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
580 585 590
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
595 600 605
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
610 615 620
Val Tyr Val Tyr Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
625 630 635 640
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
645 650 655
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
660 665 670
Pro Val Leu Asp Ser Asp Gly Ser Phe Ala Leu Val Ser Lys Leu Thr
675 680 685
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
690 695 700
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
705 710 715 720
Ser Pro Gly Lys
<210> 161
<211> 233
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность C3E-7085-LC
<400> 161
Met Val Leu Gln Thr Gln Val Phe Ile Ser Leu Leu Leu Trp Ile Ser
1 5 10 15
Gly Ala Tyr Gly Asn Phe Met Leu Thr Gln Pro Ser Ser Val Ser Gly
20 25 30
Val Pro Gly Gln Arg Val Thr Ile Ser Cys Thr Gly Asn Thr Gly Asn
35 40 45
Ile Gly Ser Asn Tyr Val Asn Trp Tyr Gln Gln Leu Pro Gly Thr Ala
50 55 60
Pro Lys Leu Leu Ile Tyr Arg Asp Asp Lys Arg Pro Ser Gly Val Pro
65 70 75 80
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile
85 90 95
Thr Gly Phe Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr
100 105 110
Ser Ser Gly Phe Ile Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
115 120 125
Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu
130 135 140
Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe
145 150 155 160
Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val
165 170 175
Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys
180 185 190
Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser
195 200 205
His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu
210 215 220
Lys Thr Val Ala Pro Thr Glu Cys Ser
225 230
<210> 162
<211> 5
<212> БЕЛОК
<213> Искусственная
<220>
<223> Peptide linker
<400> 162
Gly Gly Gly Gly Ser
1 5
<210> 163
<211> 891
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYA-3061
<400> 163
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtggagg aggtggatct 480
caatccgttc tgactcagcc accctccgcc tctggcaccc cgggccaacg ggtcacaata 540
tcatgttctg gctcttcaag caacatcgga aactggtacg tgagctggta ccagcagctc 600
cccggcacgg cgcccaagct tctgatatat gacaacaaca aacggcccag tggagttcct 660
gacagattca gtgggtctaa aagtggtaca agcgctagcc tggccataag tggtctgcag 720
agtgaagatg aggcggacta ttattgcgga acctgggact ccagcctgag cgctccctgg 780
gttttcggcg gaggtactaa gctgaccgtc ttgggcgcgg ccgcaggtgc aggtggtgat 840
tacaaagatg atgacgataa aggtgcagcg gcgcatcacc atcatcacca c 891
<210> 164
<211> 297
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYA-3061
<400> 164
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
165 170 175
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
180 185 190
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
225 230 235 240
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
245 250 255
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
260 265 270
Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly
275 280 285
Ala Ala Ala His His His His His His
290 295
<210> 165
<211> 903
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0005
<400> 165
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gttgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
agctgtgtgg ccagcggctt caccttcgac gactacgcca tgtactgggt ccgacaggtg 180
ccaggcaagg acctggaatg ggtgtccctg atttctggcg acggcgacat cacctactac 240
gtggactctg tgaagggcag attcaccgtg tccagagaca acaacaagaa cagcctgtac 300
ctgcagatga agtccctgcg cgtggaagat acagccctgt actactgcgc caaggacatg 360
atctactacg cctcttggag cggctacggc agcagcgatt actactacta tgtgatggac 420
gtgtggggcc agggcaccac cgttacagtt tctagcggag gcggaggaag tggcggcgga 480
ggatctggcg gtggtggttc tgatatccag atgacacaga gccccagcag cctgtctgcc 540
tctgtgggag acagagtgac catcacctgt agagccagcc agagcatcag cagctacctg 600
aactggtatc agcagaagcc cggcaaggcc cctaaactgc tgatctatgc cgcctccagt 660
ctgcagagcg gagtgcctag cagattttct ggcagcggct ccggcaccga tttcaccctg 720
accatatcta gcctgcagcc tgaggacttc gccacctact attgccagca gagctacagc 780
acccctccta tcacctttgg ccagggaacc agactggaaa tcaaaggcgc tgctgcaggc 840
gctggcggcg actacaaaga cgatgatgat aagggcgctg ccgctcacca ccaccatcac 900
cat 903
<210> 166
<211> 301
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0005
<400> 166
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met Tyr Trp Val Arg Gln Val Pro Gly Lys Asp
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Asp Ile Thr Tyr Tyr
65 70 75 80
Val Asp Ser Val Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Asn Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Lys Ser Leu Arg Val Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Ile Tyr Tyr Ala Ser Trp Ser Gly
115 120 125
Tyr Gly Ser Ser Asp Tyr Tyr Tyr Tyr Val Met Asp Val Trp Gly Gln
130 135 140
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser
165 170 175
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
180 185 190
Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
195 200 205
Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
210 215 220
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
225 230 235 240
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
245 250 255
Gln Ser Tyr Ser Thr Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu
260 265 270
Glu Ile Lys Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp
275 280 285
Asp Asp Lys Gly Ala Ala Ala His His His His His His
290 295 300
<210> 167
<211> 903
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0006
<400> 167
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gttgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
tcttgtgccg ccagcggctt caccttcgac gactacgcca tgtactgggt ccgacaggcc 180
cctggcaaag gccttgaatg ggtgtccctg atcagcggag atggcggcag catgtactac 240
gccgatagcg tgaagggcag attcaccatc agccgggaca acagcaagaa cagcctgtac 300
ctgcagatga actccctgcg gaccgaggac acagccctgt actactgcgc caaggatatg 360
atcttctacg ccttttggag cggctacggc agcagcgatt actactacta cgtgatggac 420
gtgtggggcc agggcaccac agtgacagtt tattctggcg gcggaggatc tggcggaggt 480
ggaagcggag gcggtggatc tgatatccag atgacacaga gccccagcag cctgtctgcc 540
tctgtgggag acagagtgac catcacctgt cgggccagcc agagaatcag cacctacctg 600
aactggtatc agcagaagcc cggcaaggcc cctaagctgc tgatctatgc tgcctccagt 660
ctgcagagcg gcgtgccaag cagattttct ggcagcggct ctggcaccga cttcaccctg 720
accatatcta gcctgcagcc tgaggacttc gccacctact actgccagca gagctacagc 780
acccctccta tcacctttgg ccagggaacc agactggaaa tcaaaggcgc tgctgcaggc 840
gcaggcggcg actacaaaga cgatgatgat aagggcgctg ccgctcacca ccaccatcac 900
cat 903
<210> 168
<211> 301
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0006
<400> 168
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Gly Ser Met Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Ile Phe Tyr Ala Phe Trp Ser Gly
115 120 125
Tyr Gly Ser Ser Asp Tyr Tyr Tyr Tyr Val Met Asp Val Trp Gly Gln
130 135 140
Gly Thr Thr Val Thr Val Tyr Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser
165 170 175
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
180 185 190
Ser Gln Arg Ile Ser Thr Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
195 200 205
Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
210 215 220
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
225 230 235 240
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
245 250 255
Gln Ser Tyr Ser Thr Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu
260 265 270
Glu Ile Lys Gly Ala Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp
275 280 285
Asp Asp Lys Gly Ala Ala Ala His His His His His His
290 295 300
<210> 169
<211> 888
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0007
<400> 169
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gttgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
tcttgtgccg ccagcggctt caccttcgac gattatgcca tgcactgggt ccgacaggcc 180
cctggcaaag gacttgaatg ggtgtccctg atcagcggcg acggcgacaa tacctactac 240
gccgatagcg tgaagggcag attcaccatc agccgggaca acaacaagaa cagcctgtac 300
ctgcagatga actccctgcg gaccgaggac accgccttct actactgtgc caaagagctg 360
atcttcggca aggtgctgca cgacttttac tactacgtga tggacgtgtg gggccagggc 420
accacagtga cagtttctag cggaggcgga ggaagtggcg gcggaggatc tggcggtggt 480
ggttctgata tccagatgac acagagcccc agcagcctgt ctgcctctgt gggagacaga 540
gtgaccatca cctgtagagc cagccagagc atcagcagct acctgaactg gtatcagcag 600
aagcccggca aggcccctaa actgctgatc tatgccgcct ccagtctgca gagcggagtg 660
cctagcagat tttctggcag cggctccggc accgatttca ccctgaccat atctagcctg 720
cagcctgagg acttcgccac ctactactgc cagcagagct acagcacccc tcctatcacc 780
tttggccagg gaaccagact ggaaatcaaa ggcgctgctg caggcgcagg cggcgactac 840
aaagacgatg atgataaggg cgctgccgct caccaccacc atcaccat 888
<210> 170
<211> 296
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0007
<400> 170
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Asp Asn Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Asn Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Glu Asp Thr Ala
100 105 110
Phe Tyr Tyr Cys Ala Lys Glu Leu Ile Phe Gly Lys Val Leu His Asp
115 120 125
Phe Tyr Tyr Tyr Val Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly Ala
260 265 270
Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala
275 280 285
Ala Ala His His His His His His
290 295
<210> 171
<211> 888
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0008
<400> 171
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gttgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
agctgtgaag cctccggctt catcttcgac gactacgcca tgcactgggt ccgacaggca 180
cctggcaaag gccttgaatg ggtgtccctg atctctggcg acggcgacat catctactac 240
gccgactctg tgaagggcag attcaccatc agccgggaca acagcaagaa cagcctgtac 300
ctgcagatga actccctgat catcgaggac acagccctgt actactgcgc caaggattgg 360
gtgttcggcg tcgtgatgac ccactactgg tacttcggcc tggatgtgtg gggccaggga 420
accacagtga cagtttctag cggaggcgga ggaagtggcg gcggaggatc tggcggtggt 480
ggttctgata tccagatgac acagagcccc agcagcctgt ctgcctctga aggcgacaga 540
gtgaccatca cctgtagagc cagccagagc atcagcacct acctgaactg gtatcagcag 600
aagcccggca aggcccctaa gctgctgatc tatggtgcct ccagtctgca gagcggcgtg 660
ccaagcagat tttctggcag cggctctggc accgacttca ccctgaccat atctagcctg 720
cagcctgagg acttcgccac ctactactgc cagcagtcct acagcacccc tcctatcaca 780
tttggccagg gcaccaaggt ggaaatcaaa ggcgctgctg caggcgctgg cggcgactac 840
aaagacgatg atgataaggg cgctgccgct caccaccacc atcaccat 888
<210> 172
<211> 296
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0008
<400> 172
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe Ile
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Asp Ile Ile Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Ile Ile Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Trp Val Phe Gly Val Val Met Thr His
115 120 125
Tyr Trp Tyr Phe Gly Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Glu Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Thr Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Pro Ile Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Ala
260 265 270
Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala
275 280 285
Ala Ala His His His His His His
290 295
<210> 173
<211> 885
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0009
<400> 173
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gttgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
tcttgtgccg ccagcggctt caccttcgac gattatgcca tgcactgggt ccgacaggcc 180
cctggcaaag gacttgaatg ggtgtccctg atctctggcg gaggcggcgg aacctactac 240
agcgattctg tgaagggcag attcaccatc agccgggaca ccagcaagga cagcctgtac 300
ctgcagatga acagcctgcg gaccgaggac acagccctgt actactgcgc caaggacatg 360
gtgttcggcg tggtcacccc ttactactac ttcgccctgg atgtgtgggg ccagggcaca 420
acagtgacag tctcttctgg cggcggagga agcggaggcg gaggatccgg tggtggtgga 480
tctgacatcc agatgacaca gagccccagc agcctgtctg cctctgtggg agacagagtg 540
accatcacct gtagagccag ccagagcatc aacagctacc tgaactggta tcagcagaag 600
cccggcaagg cccctaagct gctgatctat gctgcctcca gtctgcagag cggcgtgcca 660
agcagatttt ctggcagcgg ctctggcacc gacttcaccc tgaccatatc tagcctgcag 720
cctgaggact tcgccaccta ctactgccag cagagctaca gcgcccctcc tatcacattt 780
ggccagggaa ccagactgga aatcaaaggc gctgctgcag gcgcaggcgg cgactacaaa 840
gacgatgatg ataagggcgc tgccgctcac caccatcacc atcat 885
<210> 174
<211> 295
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0009
<400> 174
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Gly Gly Gly Gly Thr Tyr Tyr
65 70 75 80
Ser Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys
85 90 95
Asp Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Val Phe Gly Val Val Thr Pro Tyr
115 120 125
Tyr Tyr Phe Ala Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
130 135 140
Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Asn Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Ala Pro
245 250 255
Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly Ala Ala
260 265 270
Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala
275 280 285
Ala His His His His His His
290 295
<210> 175
<211> 888
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0010
<400> 175
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gttgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
agctgtgctg tgtccggctt caccttcgac gactacgcca tgcactgggt ccgacaggca 180
cctggcaaag gccttgaatg ggtgtccctg atcagcggag atggcggcag cacacactac 240
gccgattctg tgaagggcag attcaccatc agccgggaca acagcaagaa cagcctgtac 300
ctgcagatga actccctgcg gacaggcgac acagccctgt actactgcgc caaggacatg 360
atcttcgccg tggtcatcac cgactaccac tactacggca tggacgtgtg gggccaggga 420
accacagtga cagtttctag cggaggcgga ggaagtggcg gcggaggatc tggcggtggt 480
ggttctgata tccagatgac acagagcccc agcagcctgt ctgcctctgt gggagacaga 540
gtgaccatca cctgtagagc cagccagagc atcagcagct acctgaactg gtatcagcag 600
aagcccggca aggcccctaa actgctgatc tatgccgcct ccagtctgca gagcggagtg 660
cctagcagat tttctggcag cggctccggc accgatttca ccctgaccat atctagcctg 720
cagcctgagg acttcgccac ctactactgc cagcagagct acagcacccc tcctatcacc 780
tttggccagg gcaccagact ggaaatcaaa ggcgctgctg caggcgctgg cggcgactac 840
aaagacgatg atgataaggg cgctgccgct caccaccacc atcaccat 888
<210> 176
<211> 296
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0010
<400> 176
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Gly Ser Thr His Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Gly Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Ile Phe Ala Val Val Ile Thr Asp
115 120 125
Tyr His Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly Ala
260 265 270
Ala Ala Gly Ala Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala
275 280 285
Ala Ala His His His His His His
290 295
<210> 177
<211> 738
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность HC-h
<400> 177
atgaaacacc tgtggttctt cctcctgctg gtggcagctc ccagatgggt gctgagcgac 60
aaaactcaca catgcccacc ctgcccagca cctgaagccg ccggcggacc ctcagtcttc 120
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 180
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 240
gtggaggtgc ataatgccaa gaccaagcct agagaggaac agtacaacag cacctaccgg 300
gtggtgtccg tgctgacagt gctgcaccag gactggctga acggcaaaga gtacaagtgc 360
aaggtgtcca acaaggccct gcctgccccc atcgagaaaa ccatcagcaa ggccaagggc 420
cagccccgcg aacctcaagt gtgcaccctg ccaccctccc gggatgagct gaccaagaac 480
caggtgtccc tgagctgtgc cgtgaagggc ttctacccct ccgatatcgc cgtggaatgg 540
gagagcaacg gccagcccga gaacaactac aagaccaccc cccctgtgct ggacagcgac 600
ggctcattct tcctggtgtc caagctgacc gtggacaagt ccagatggca gcagggcaac 660
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacaccca gaagagcctc 720
tccctgtctc ccggcaaa 738
<210> 178
<211> 246
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность HC-h
<400> 178
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
20 25 30
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
35 40 45
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
50 55 60
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
65 70 75 80
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
85 90 95
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
100 105 110
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
115 120 125
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
130 135 140
Pro Gln Val Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
145 150 155 160
Gln Val Ser Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile
165 170 175
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
180 185 190
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys
195 200 205
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
210 215 220
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
225 230 235 240
Ser Leu Ser Pro Gly Lys
245
<210> 179
<211> 1500
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYD-2047-HC-k
<400> 179
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggt 60
caagtacagc tggtggaatc cggtggaggc gtggtccagc cgggacgcag cttgagactg 120
tcctgcgctg catctggctt ttccatacga tcctacgata tgcactgggt tcgccaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattac 240
gcggattcag tgaaggggcg gttcacaatt tcacgggaca attcaaagaa taccttgtat 300
ctccagatgt cttcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgtgagtgc agcgcctgga cagaaggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggaa tccctgacag attcagtggg 660
tctaaaagtg gtacaagcgc taccctgggt atcaccggat tgcagaccgg agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaaggtta ccgtcttggg cggagaaccc aagagcgcag acaagaccca cacctgtcct 840
ccatgtcctg ctccagaagc tgcaggcggc ccttccgtgt ttctgttccc tccaaagcct 900
aaggacaccc tgatgatcag ccggacacct gaagtgacct gcgtggtggt ggatgtgtcc 960
cacgaggatc ccgaagtgaa gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 1020
aagaccaagc ctagagagga acagtacaac agcacctaca gagtggtgtc tgtgctgaca 1080
gtgctgcacc aggactggct gaacggcaaa gagtacaagt gcaaggtgtc caacaaggcc 1140
ctgcctgctc ctatcgagaa aaccatcagc aaggccaagg gccagcctag ggaaccccag 1200
gtttacaccc tgcctccatg cagggatgag ctgaccaaga accaggtgtc cctgtggtgc 1260
ctggttaagg gcttctaccc ctccgatatc gccgtggaat gggagagcaa tggccagcca 1320
gagaacaact acaagacaac ccctcctgtg ctggactccg acggctcatt cttcctgtac 1380
agcaagctga ccgtggacaa gagcagatgg cagcagggca acgtgttcag ctgcagcgtg 1440
atgcacgagg ccctgcacaa ccactacaca cagaagtccc tgtctctgag ccccggcaaa 1500
<210> 180
<211> 500
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYD-2047-HC-k
<400> 180
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Glu Pro Lys Ser
260 265 270
Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
275 280 285
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Lys
500
<210> 181
<211> 1500
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYD-2061-HC-k
<400> 181
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtcaatc cgttctgact 480
cagccaccct ccgcctctgg caccccgggc caacgggtca caatatcatg ttctggctct 540
tcaagcaaca tcggaaactg gtacgtgagc tggtaccagc agctccccgg cacggcgccc 600
aagcttctga tatatgacaa caacaaacgg cccagtggag ttcctgacag attcagtggg 660
tctaaaagtg gtacaagcgc tagcctggcc ataagtggtc tgcagagtga agatgaggcg 720
gactattatt gcggaacctg ggactccagc ctgagcgctc cctgggtttt cggcggaggt 780
actaagctga ccgtcttggg cggagaaccc aagagcgcag acaagaccca cacctgtcct 840
ccatgtcctg ctccagaagc tgcaggcggc ccttccgtgt ttctgttccc tccaaagcct 900
aaggacaccc tgatgatcag ccggacacct gaagtgacct gcgtggtggt ggatgtgtcc 960
cacgaggatc ccgaagtgaa gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 1020
aagaccaagc ctagagagga acagtacaac agcacctaca gagtggtgtc tgtgctgaca 1080
gtgctgcacc aggactggct gaacggcaaa gagtacaagt gcaaggtgtc caacaaggcc 1140
ctgcctgctc ctatcgagaa aaccatcagc aaggccaagg gccagcctag ggaaccccag 1200
gtttacaccc tgcctccatg cagggatgag ctgaccaaga accaggtgtc cctgtggtgc 1260
ctggttaagg gcttctaccc ctccgatatc gccgtggaat gggagagcaa tggccagcca 1320
gagaacaact acaagacaac ccctcctgtg ctggactccg acggctcatt cttcctgtac 1380
agcaagctga ccgtggacaa gagcagatgg cagcagggca acgtgttcag ctgcagcgtg 1440
atgcacgagg ccctgcacaa ccactacaca cagaagtccc tgtctctgag ccccggcaaa 1500
<210> 182
<211> 500
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYD-2061-HC-k
<400> 182
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Glu Pro Lys Ser
260 265 270
Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
275 280 285
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Lys
500
<210> 183
<211> 1515
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYD-3061-HC-k
<400> 183
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggt 60
gaggtacagc tggtggaatc cggtggaggc cttgtgcaac cgggaggctc cctgcgactc 120
tcttgcgccg catctggctt ttccatacga tcctacgata ttcattgggt ccgacaagcc 180
cctggcaaag gtcttgaatg ggttgccact atctcctacg acgggtctca gaaatattat 240
gcggattcag tgaaggggcg gttcacaatt tcacgagacg agtcaaagaa tacactgtac 300
ctccaaatga attcactgag agccgaggat actgcagtct atcattgtgc aagagggtcc 360
tcaggccact acgaggcctt tgatatatgg ggccaaggca ccttggtaac cgttagtagc 420
ggaggtggag gaagcggagg cggcggttcc ggaggaggtg gaagtggagg aggtggatct 480
caatccgttc tgactcagcc accctccgcc tctggcaccc cgggccaacg ggtcacaata 540
tcatgttctg gctcttcaag caacatcgga aactggtacg tgagctggta ccagcagctc 600
cccggcacgg cgcccaagct tctgatatat gacaacaaca aacggcccag tggagttcct 660
gacagattca gtgggtctaa aagtggtaca agcgctagcc tggccataag tggtctgcag 720
agtgaagatg aggcggacta ttattgcgga acctgggact ccagcctgag cgctccctgg 780
gttttcggcg gaggtactaa gctgaccgtc ttgggcggag aacccaagag cgcagacaag 840
acccacacct gtcctccatg tcctgctcca gaagctgcag gcggcccttc cgtgtttctg 900
ttccctccaa agcctaagga caccctgatg atcagccgga cacctgaagt gacctgcgtg 960
gtggtggatg tgtcccacga ggatcccgaa gtgaagttca attggtacgt ggacggcgtg 1020
gaagtgcaca acgccaagac caagcctaga gaggaacagt acaacagcac ctacagagtg 1080
gtgtctgtgc tgacagtgct gcaccaggac tggctgaacg gcaaagagta caagtgcaag 1140
gtgtccaaca aggccctgcc tgctcctatc gagaaaacca tcagcaaggc caagggccag 1200
cctagggaac cccaggttta caccctgcct ccatgcaggg atgagctgac caagaaccag 1260
gtgtccctgt ggtgcctggt taagggcttc tacccctccg atatcgccgt ggaatgggag 1320
agcaatggcc agccagagaa caactacaag acaacccctc ctgtgctgga ctccgacggc 1380
tcattcttcc tgtacagcaa gctgaccgtg gacaagagca gatggcagca gggcaacgtg 1440
ttcagctgca gcgtgatgca cgaggccctg cacaaccact acacacagaa gtccctgtct 1500
ctgagccccg gcaaa 1515
<210> 184
<211> 505
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYD-3061-HC-k
<400> 184
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln
165 170 175
Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp
180 185 190
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Asp Asn Asn Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln
225 230 235 240
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
245 250 255
Ser Ala Pro Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
260 265 270
Gly Glu Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro
275 280 285
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
290 295 300
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
305 310 315 320
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
325 330 335
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
340 345 350
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
355 360 365
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
370 375 380
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
385 390 395 400
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu
405 410 415
Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro
420 425 430
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
435 440 445
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
450 455 460
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
465 470 475 480
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
485 490 495
Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505
<210> 185
<211> 1527
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0011-HC-k
<400> 185
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
agctgtgtgg ccagcggctt caccttcgac gactacgcca tgtactgggt ccgacaggtg 180
ccaggcaagg acctggaatg ggtgtccctg atttctggcg acggcgacat cacctactac 240
gtggactctg tgaagggcag attcaccgtg tccagagaca acaacaagaa cagcctgtac 300
ctgcagatga agtccctgcg cgtggaagat acagccctgt actactgcgc caaggacatg 360
atctactacg cctcttggag cggctacggc agcagcgatt actactacta tgtgatggac 420
gtgtggggcc agggcaccac cgttacagtt tctagcggag gcggaggaag tggcggcgga 480
ggatctggcg gtggtggttc tgatatccag atgacacaga gccccagcag cctgtctgcc 540
tctgtgggag acagagtgac catcacctgt agagccagcc agagcatcag cagctacctg 600
aactggtatc agcagaagcc cggcaaggcc cctaaactgc tgatctatgc cgcctccagt 660
ctgcagagcg gagtgcctag cagattttct ggcagcggct ccggcaccga tttcaccctg 720
accatatcta gcctgcagcc tgaggacttc gccacctact attgccagca gagctacagc 780
acccctccta tcacctttgg ccagggaacc agactggaaa tcaaaggcgg agaacccaag 840
agcgcagaca agacccacac ctgtcctcca tgtcctgctc cagaagctgc aggcggccct 900
tccgtgtttc tgttccctcc aaagcctaag gacaccctga tgatcagccg gacacctgaa 960
gtgacctgcg tggtggtgga tgtgtcccac gaggatcccg aagtgaagtt caattggtac 1020
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gtacaacagc 1080
acctacagag tggtgtctgt gctgacagtg ctgcaccagg actggctgaa cggcaaagag 1140
tacaagtgca aggtgtccaa caaggccctg cctgctccta tcgagaaaac catcagcaag 1200
gccaagggcc agcctaggga accccaggtt tacaccctgc ctccatgcag ggatgagctg 1260
accaagaacc aggtgtccct gtggtgcctg gttaagggct tctacccctc cgatatcgcc 1320
gtggaatggg agagcaatgg ccagccagag aacaactaca agacaacccc tcctgtgctg 1380
gactccgacg gctcattctt cctgtacagc aagctgaccg tggacaagag cagatggcag 1440
cagggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacacag 1500
aagtccctgt ctctgagccc cggcaaa 1527
<210> 186
<211> 509
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0011-HC-k
<400> 186
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Val Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met Tyr Trp Val Arg Gln Val Pro Gly Lys Asp
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Asp Ile Thr Tyr Tyr
65 70 75 80
Val Asp Ser Val Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Asn Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Lys Ser Leu Arg Val Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Ile Tyr Tyr Ala Ser Trp Ser Gly
115 120 125
Tyr Gly Ser Ser Asp Tyr Tyr Tyr Tyr Val Met Asp Val Trp Gly Gln
130 135 140
Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser
165 170 175
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
180 185 190
Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
195 200 205
Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
210 215 220
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
225 230 235 240
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
245 250 255
Gln Ser Tyr Ser Thr Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu
260 265 270
Glu Ile Lys Gly Gly Glu Pro Lys Ser Ala Asp Lys Thr His Thr Cys
275 280 285
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
290 295 300
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
305 310 315 320
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
325 330 335
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
340 345 350
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
355 360 365
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
370 375 380
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
385 390 395 400
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys
405 410 415
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys
420 425 430
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
435 440 445
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
450 455 460
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
465 470 475 480
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
485 490 495
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505
<210> 187
<211> 1527
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0012-HC-k
<400> 187
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
tcttgtgccg ccagcggctt caccttcgac gactacgcca tgtactgggt ccgacaggcc 180
cctggcaaag gccttgaatg ggtgtccctg atcagcggag atggcggcag catgtactac 240
gccgatagcg tgaagggcag attcaccatc agccgggaca acagcaagaa cagcctgtac 300
ctgcagatga actccctgcg gaccgaggac acagccctgt actactgcgc caaggatatg 360
atcttctacg ccttttggag cggctacggc agcagcgatt actactacta cgtgatggac 420
gtgtggggcc agggcaccac agtgacagtt tattctggcg gcggaggatc tggcggaggt 480
ggaagcggag gcggtggatc tgatatccag atgacacaga gccccagcag cctgtctgcc 540
tctgtgggag acagagtgac catcacctgt cgggccagcc agagaatcag cacctacctg 600
aactggtatc agcagaagcc cggcaaggcc cctaagctgc tgatctatgc tgcctccagt 660
ctgcagagcg gcgtgccaag cagattttct ggcagcggct ctggcaccga cttcaccctg 720
accatatcta gcctgcagcc tgaggacttc gccacctact actgccagca gagctacagc 780
acccctccta tcacctttgg ccagggaacc agactggaaa tcaaaggcgg agaacccaag 840
agcgcagaca agacccacac ctgtcctcca tgtcctgctc cagaagctgc aggcggccct 900
tccgtgtttc tgttccctcc aaagcctaag gacaccctga tgatcagccg gacacctgaa 960
gtgacctgcg tggtggtgga tgtgtcccac gaggatcccg aagtgaagtt caattggtac 1020
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gtacaacagc 1080
acctacagag tggtgtctgt gctgacagtg ctgcaccagg actggctgaa cggcaaagag 1140
tacaagtgca aggtgtccaa caaggccctg cctgctccta tcgagaaaac catcagcaag 1200
gccaagggcc agcctaggga accccaggtt tacaccctgc ctccatgcag ggatgagctg 1260
accaagaacc aggtgtccct gtggtgcctg gttaagggct tctacccctc cgatatcgcc 1320
gtggaatggg agagcaatgg ccagccagag aacaactaca agacaacccc tcctgtgctg 1380
gactccgacg gctcattctt cctgtacagc aagctgaccg tggacaagag cagatggcag 1440
cagggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacacag 1500
aagtccctgt ctctgagccc cggcaaa 1527
<210> 188
<211> 509
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0012-HC-k
<400> 188
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Gly Ser Met Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Ile Phe Tyr Ala Phe Trp Ser Gly
115 120 125
Tyr Gly Ser Ser Asp Tyr Tyr Tyr Tyr Val Met Asp Val Trp Gly Gln
130 135 140
Gly Thr Thr Val Thr Val Tyr Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser
165 170 175
Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala
180 185 190
Ser Gln Arg Ile Ser Thr Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
195 200 205
Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly
210 215 220
Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
225 230 235 240
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln
245 250 255
Gln Ser Tyr Ser Thr Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu
260 265 270
Glu Ile Lys Gly Gly Glu Pro Lys Ser Ala Asp Lys Thr His Thr Cys
275 280 285
Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu
290 295 300
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
305 310 315 320
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
325 330 335
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
340 345 350
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
355 360 365
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
370 375 380
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
385 390 395 400
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys
405 410 415
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys
420 425 430
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
435 440 445
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
450 455 460
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
465 470 475 480
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
485 490 495
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
500 505
<210> 189
<211> 1512
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0013-HC-k
<400> 189
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
tcttgtgccg ccagcggctt caccttcgac gattatgcca tgcactgggt ccgacaggcc 180
cctggcaaag gacttgaatg ggtgtccctg atcagcggcg acggcgacaa tacctactac 240
gccgatagcg tgaagggcag attcaccatc agccgggaca acaacaagaa cagcctgtac 300
ctgcagatga actccctgcg gaccgaggac accgccttct actactgtgc caaagagctg 360
atcttcggca aggtgctgca cgacttttac tactacgtga tggacgtgtg gggccagggc 420
accacagtga cagtttctag cggaggcgga ggaagtggcg gcggaggatc tggcggtggt 480
ggttctgata tccagatgac acagagcccc agcagcctgt ctgcctctgt gggagacaga 540
gtgaccatca cctgtagagc cagccagagc atcagcagct acctgaactg gtatcagcag 600
aagcccggca aggcccctaa actgctgatc tatgccgcct ccagtctgca gagcggagtg 660
cctagcagat tttctggcag cggctccggc accgatttca ccctgaccat atctagcctg 720
cagcctgagg acttcgccac ctactactgc cagcagagct acagcacccc tcctatcacc 780
tttggccagg gaaccagact ggaaatcaaa ggcggagaac ccaagagcgc agacaagacc 840
cacacctgtc ctccatgtcc tgctccagaa gctgcaggcg gcccttccgt gtttctgttc 900
cctccaaagc ctaaggacac cctgatgatc agccggacac ctgaagtgac ctgcgtggtg 960
gtggatgtgt cccacgagga tcccgaagtg aagttcaatt ggtacgtgga cggcgtggaa 1020
gtgcacaacg ccaagaccaa gcctagagag gaacagtaca acagcaccta cagagtggtg 1080
tctgtgctga cagtgctgca ccaggactgg ctgaacggca aagagtacaa gtgcaaggtg 1140
tccaacaagg ccctgcctgc tcctatcgag aaaaccatca gcaaggccaa gggccagcct 1200
agggaacccc aggtttacac cctgcctcca tgcagggatg agctgaccaa gaaccaggtg 1260
tccctgtggt gcctggttaa gggcttctac ccctccgata tcgccgtgga atgggagagc 1320
aatggccagc cagagaacaa ctacaagaca acccctcctg tgctggactc cgacggctca 1380
ttcttcctgt acagcaagct gaccgtggac aagagcagat ggcagcaggg caacgtgttc 1440
agctgcagcg tgatgcacga ggccctgcac aaccactaca cacagaagtc cctgtctctg 1500
agccccggca aa 1512
<210> 190
<211> 504
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0013-HC-k
<400> 190
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Asp Asn Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Asn Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Glu Asp Thr Ala
100 105 110
Phe Tyr Tyr Cys Ala Lys Glu Leu Ile Phe Gly Lys Val Leu His Asp
115 120 125
Phe Tyr Tyr Tyr Val Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly Gly
260 265 270
Glu Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
275 280 285
Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
290 295 300
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
305 310 315 320
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
325 330 335
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
340 345 350
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
355 360 365
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
370 375 380
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
385 390 395 400
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr
405 410 415
Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser
420 425 430
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
435 440 445
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
450 455 460
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
465 470 475 480
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
485 490 495
Ser Leu Ser Leu Ser Pro Gly Lys
500
<210> 191
<211> 1512
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0014-HC-k
<400> 191
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
agctgtgaag cctccggctt catcttcgac gactacgcca tgcactgggt ccgacaggca 180
cctggcaaag gccttgaatg ggtgtccctg atctctggcg acggcgacat catctactac 240
gccgactctg tgaagggcag attcaccatc agccgggaca acagcaagaa cagcctgtac 300
ctgcagatga actccctgat catcgaggac acagccctgt actactgcgc caaggattgg 360
gtgttcggcg tcgtgatgac ccactactgg tacttcggcc tggatgtgtg gggccaggga 420
accacagtga cagtttctag cggaggcgga ggaagtggcg gcggaggatc tggcggtggt 480
ggttctgata tccagatgac acagagcccc agcagcctgt ctgcctctga aggcgacaga 540
gtgaccatca cctgtagagc cagccagagc atcagcacct acctgaactg gtatcagcag 600
aagcccggca aggcccctaa gctgctgatc tatggtgcct ccagtctgca gagcggcgtg 660
ccaagcagat tttctggcag cggctctggc accgacttca ccctgaccat atctagcctg 720
cagcctgagg acttcgccac ctactactgc cagcagtcct acagcacccc tcctatcaca 780
tttggccagg gcaccaaggt ggaaatcaaa ggcggagaac ccaagagcgc agacaagacc 840
cacacctgtc ctccatgtcc tgctccagaa gctgcaggcg gcccttccgt gtttctgttc 900
cctccaaagc ctaaggacac cctgatgatc agccggacac ctgaagtgac ctgcgtggtg 960
gtggatgtgt cccacgagga tcccgaagtg aagttcaatt ggtacgtgga cggcgtggaa 1020
gtgcacaacg ccaagaccaa gcctagagag gaacagtaca acagcaccta cagagtggtg 1080
tctgtgctga cagtgctgca ccaggactgg ctgaacggca aagagtacaa gtgcaaggtg 1140
tccaacaagg ccctgcctgc tcctatcgag aaaaccatca gcaaggccaa gggccagcct 1200
agggaacccc aggtttacac cctgcctcca tgcagggatg agctgaccaa gaaccaggtg 1260
tccctgtggt gcctggttaa gggcttctac ccctccgata tcgccgtgga atgggagagc 1320
aatggccagc cagagaacaa ctacaagaca acccctcctg tgctggactc cgacggctca 1380
ttcttcctgt acagcaagct gaccgtggac aagagcagat ggcagcaggg caacgtgttc 1440
agctgcagcg tgatgcacga ggccctgcac aaccactaca cacagaagtc cctgtctctg 1500
agccccggca aa 1512
<210> 192
<211> 504
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0014-HC-k
<400> 192
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe Ile
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Asp Ile Ile Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Ile Ile Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Trp Val Phe Gly Val Val Met Thr His
115 120 125
Tyr Trp Tyr Phe Gly Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Glu Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Thr Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Gly Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Pro Ile Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly
260 265 270
Glu Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
275 280 285
Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
290 295 300
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
305 310 315 320
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
325 330 335
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
340 345 350
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
355 360 365
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
370 375 380
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
385 390 395 400
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr
405 410 415
Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser
420 425 430
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
435 440 445
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
450 455 460
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
465 470 475 480
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
485 490 495
Ser Leu Ser Leu Ser Pro Gly Lys
500
<210> 193
<211> 1509
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0015-HC-k
<400> 193
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
tcttgtgccg ccagcggctt caccttcgac gattatgcca tgcactgggt ccgacaggcc 180
cctggcaaag gacttgaatg ggtgtccctg atctctggcg gaggcggcgg aacctactac 240
agcgattctg tgaagggcag attcaccatc agccgggaca ccagcaagga cagcctgtac 300
ctgcagatga acagcctgcg gaccgaggac acagccctgt actactgcgc caaggacatg 360
gtgttcggcg tggtcacccc ttactactac ttcgccctgg atgtgtgggg ccagggcaca 420
acagtgacag tctcttctgg cggcggagga agcggaggcg gaggatccgg tggtggtgga 480
tctgacatcc agatgacaca gagccccagc agcctgtctg cctctgtggg agacagagtg 540
accatcacct gtagagccag ccagagcatc aacagctacc tgaactggta tcagcagaag 600
cccggcaagg cccctaagct gctgatctat gctgcctcca gtctgcagag cggcgtgcca 660
agcagatttt ctggcagcgg ctctggcacc gacttcaccc tgaccatatc tagcctgcag 720
cctgaggact tcgccaccta ctactgccag cagagctaca gcgcccctcc tatcacattt 780
ggccagggaa ccagactgga aatcaaaggc ggagaaccca agagcgcaga caagacccac 840
acctgtcctc catgtcctgc tccagaagct gcaggcggcc cttccgtgtt tctgttccct 900
ccaaagccta aggacaccct gatgatcagc cggacacctg aagtgacctg cgtggtggtg 960
gatgtgtccc acgaggatcc cgaagtgaag ttcaattggt acgtggacgg cgtggaagtg 1020
cacaacgcca agaccaagcc tagagaggaa cagtacaaca gcacctacag agtggtgtct 1080
gtgctgacag tgctgcacca ggactggctg aacggcaaag agtacaagtg caaggtgtcc 1140
aacaaggccc tgcctgctcc tatcgagaaa accatcagca aggccaaggg ccagcctagg 1200
gaaccccagg tttacaccct gcctccatgc agggatgagc tgaccaagaa ccaggtgtcc 1260
ctgtggtgcc tggttaaggg cttctacccc tccgatatcg ccgtggaatg ggagagcaat 1320
ggccagccag agaacaacta caagacaacc cctcctgtgc tggactccga cggctcattc 1380
ttcctgtaca gcaagctgac cgtggacaag agcagatggc agcagggcaa cgtgttcagc 1440
tgcagcgtga tgcacgaggc cctgcacaac cactacacac agaagtccct gtctctgagc 1500
cccggcaaa 1509
<210> 194
<211> 503
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0015-HC-k
<400> 194
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Gly Gly Gly Gly Thr Tyr Tyr
65 70 75 80
Ser Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys
85 90 95
Asp Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Glu Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Val Phe Gly Val Val Thr Pro Tyr
115 120 125
Tyr Tyr Phe Ala Leu Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
130 135 140
Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Asn Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Ala Pro
245 250 255
Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly Gly Glu
260 265 270
Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
370 375 380
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys
405 410 415
Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
Leu Ser Leu Ser Pro Gly Lys
500
<210> 195
<211> 1512
<212> ДНК
<213> Искусственная
<220>
<223> Полноразмерная нуклеотидная последовательность NYC-0016-HC-k
<400> 195
atgaagcacc tgtggttctt tctgctgctg gtggccgctc ctagatgggt gctgtctggc 60
gaagtgcagc tggtggaatc tggtggcgga gttgttcagc ctggcggctc tctgagactg 120
agctgtgctg tgtccggctt caccttcgac gactacgcca tgcactgggt ccgacaggca 180
cctggcaaag gccttgaatg ggtgtccctg atcagcggag atggcggcag cacacactac 240
gccgattctg tgaagggcag attcaccatc agccgggaca acagcaagaa cagcctgtac 300
ctgcagatga actccctgcg gacaggcgac acagccctgt actactgcgc caaggacatg 360
atcttcgccg tggtcatcac cgactaccac tactacggca tggacgtgtg gggccaggga 420
accacagtga cagtttctag cggaggcgga ggaagtggcg gcggaggatc tggcggtggt 480
ggttctgata tccagatgac acagagcccc agcagcctgt ctgcctctgt gggagacaga 540
gtgaccatca cctgtagagc cagccagagc atcagcagct acctgaactg gtatcagcag 600
aagcccggca aggcccctaa actgctgatc tatgccgcct ccagtctgca gagcggagtg 660
cctagcagat tttctggcag cggctccggc accgatttca ccctgaccat atctagcctg 720
cagcctgagg acttcgccac ctactactgc cagcagagct acagcacccc tcctatcacc 780
tttggccagg gcaccagact ggaaatcaaa ggcggagaac ccaagagcgc agacaagacc 840
cacacctgtc ctccatgtcc tgctccagaa gctgcaggcg gcccttccgt gtttctgttc 900
cctccaaagc ctaaggacac cctgatgatc agccggacac ctgaagtgac ctgcgtggtg 960
gtggatgtgt cccacgagga tcccgaagtg aagttcaatt ggtacgtgga cggcgtggaa 1020
gtgcacaacg ccaagaccaa gcctagagag gaacagtaca acagcaccta cagagtggtg 1080
tctgtgctga cagtgctgca ccaggactgg ctgaacggca aagagtacaa gtgcaaggtg 1140
tccaacaagg ccctgcctgc tcctatcgag aaaaccatca gcaaggccaa gggccagcct 1200
agggaacccc aggtttacac cctgcctcca tgcagggatg agctgaccaa gaaccaggtg 1260
tccctgtggt gcctggttaa gggcttctac ccctccgata tcgccgtgga atgggagagc 1320
aatggccagc cagagaacaa ctacaagaca acccctcctg tgctggactc cgacggctca 1380
ttcttcctgt acagcaagct gaccgtggac aagagcagat ggcagcaggg caacgtgttc 1440
agctgcagcg tgatgcacga ggccctgcac aaccactaca cacagaagtc cctgtctctg 1500
agccccggca aa 1512
<210> 196
<211> 504
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYC-0016-HC-k
<400> 196
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Thr
35 40 45
Phe Asp Asp Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Leu Ile Ser Gly Asp Gly Gly Ser Thr His Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Thr Gly Asp Thr Ala
100 105 110
Leu Tyr Tyr Cys Ala Lys Asp Met Ile Phe Ala Val Val Ile Thr Asp
115 120 125
Tyr His Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly Gly
260 265 270
Glu Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
275 280 285
Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
290 295 300
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
305 310 315 320
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
325 330 335
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
340 345 350
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
355 360 365
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
370 375 380
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
385 390 395 400
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr
405 410 415
Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser
420 425 430
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
435 440 445
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
450 455 460
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
465 470 475 480
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
485 490 495
Ser Leu Ser Leu Ser Pro Gly Lys
500
<210> 197
<211> 719
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYZ-1007-HC2
<400> 197
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Glu Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Ser Leu Ala Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
385 390 395 400
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
405 410 415
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
420 425 430
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
435 440 445
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
450 455 460
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
465 470 475 480
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
485 490 495
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
500 505 510
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
515 520 525
Pro Glu Val Thr Cys Val Val Val Ser Val Ser His Glu Asp Pro Glu
530 535 540
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
545 550 555 560
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
565 570 575
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
580 585 590
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
595 600 605
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro
610 615 620
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
625 630 635 640
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
645 650 655
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
660 665 670
Asp Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
675 680 685
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
690 695 700
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
705 710 715
<210> 198
<211> 719
<212> БЕЛОК
<213> Искусственная
<220>
<223> Полноразмерная аминокислотная последовательность NYZ-1017-HC2
<400> 198
Met Lys His Leu Trp Phe Phe Leu Leu Leu Val Ala Ala Pro Arg Trp
1 5 10 15
Val Leu Ser Gly Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val
20 25 30
Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser
35 40 45
Ile Arg Ser Tyr Asp Met His Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ala Thr Ile Ser Tyr Asp Gly Ser Gln Lys Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr His Cys Ala Arg Gly Ser Ser Gly His Tyr Glu Ala Phe Asp
115 120 125
Ile Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Val Leu Thr
145 150 155 160
Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser
165 170 175
Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Trp Tyr Val Ser Trp Tyr
180 185 190
Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Asp Asn Asn
195 200 205
Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
210 215 220
Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala
225 230 235 240
Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Pro Trp Val
245 250 255
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser Glu
260 265 270
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
275 280 285
Leu Arg Leu Ser Cys Ala Ala Ser Gly Val Thr Phe Asn Tyr Tyr Gly
290 295 300
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
305 310 315 320
Ser Ile Thr Arg Ser Gly Gly Arg Ile Tyr Tyr Pro Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Arg Glu Asn Thr Gln Lys Thr Leu Tyr Leu
340 345 350
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Thr
355 360 365
Leu Asp Gly Arg Asp Gly Trp Val Ala Tyr Trp Gly Gln Gly Thr Leu
370 375 380
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
385 390 395 400
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
405 410 415
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
420 425 430
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
435 440 445
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
450 455 460
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
465 470 475 480
Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr His
485 490 495
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
500 505 510
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
515 520 525
Pro Glu Val Thr Cys Val Val Val Ser Val Ser His Glu Asp Pro Glu
530 535 540
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
545 550 555 560
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
565 570 575
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
580 585 590
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
595 600 605
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Val Tyr Pro
610 615 620
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
625 630 635 640
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
645 650 655
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
660 665 670
Asp Gly Ser Phe Ala Leu Val Ser Lys Leu Thr Val Asp Lys Ser Arg
675 680 685
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
690 695 700
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
705 710 715
230
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| АНТАГОНИСТЫ NGF ДЛЯ МЕДИЦИНСКОГО ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2829812C2 |
| АНТИТЕЛА К РЕЦЕПТОРУ IL4 ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2837141C2 |
| ВАРИАНТЫ IgG-FC ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2018 |
|
RU2814952C2 |
| ВАРИАНТ Fc И ЕГО ПОЛУЧЕНИЕ | 2021 |
|
RU2828852C1 |
| Слитые молекулы, происходящие от Cholix-токсина, для пероральной доставки биологически активных нагрузок | 2015 |
|
RU2723178C2 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2837821C1 |
| ТРИСПЕЦИФИЧЕСКИЕ И/ИЛИ ТРИВАЛЕНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ПРИМЕНЕНИЕМ КРОССОВЕРНОГО ФОРМАТА С ДВОЙНЫМ ВАРИАБЕЛЬНЫМ ДОМЕНОМ (CODV) ДЛЯ ЛЕЧЕНИЯ ИНФЕКЦИИ, ВЫЗВАННОЙ HIV | 2020 |
|
RU2820164C2 |
| КОНСТРУКЦИИ СЛИТОГО БЕЛКА ДЛЯ ЗАБОЛЕВАНИЯ, СВЯЗАННОГО С КОМПЛЕМЕНТОМ | 2019 |
|
RU2824402C2 |
| ПОЛИВАЛЕТНЫЕ И ПОЛИСПЕЦИФИЧНЫЕ GITR-СВЯЗЫВАЮЩИЕ СЛИТЫЕ БЕЛКИ | 2016 |
|
RU2753439C2 |
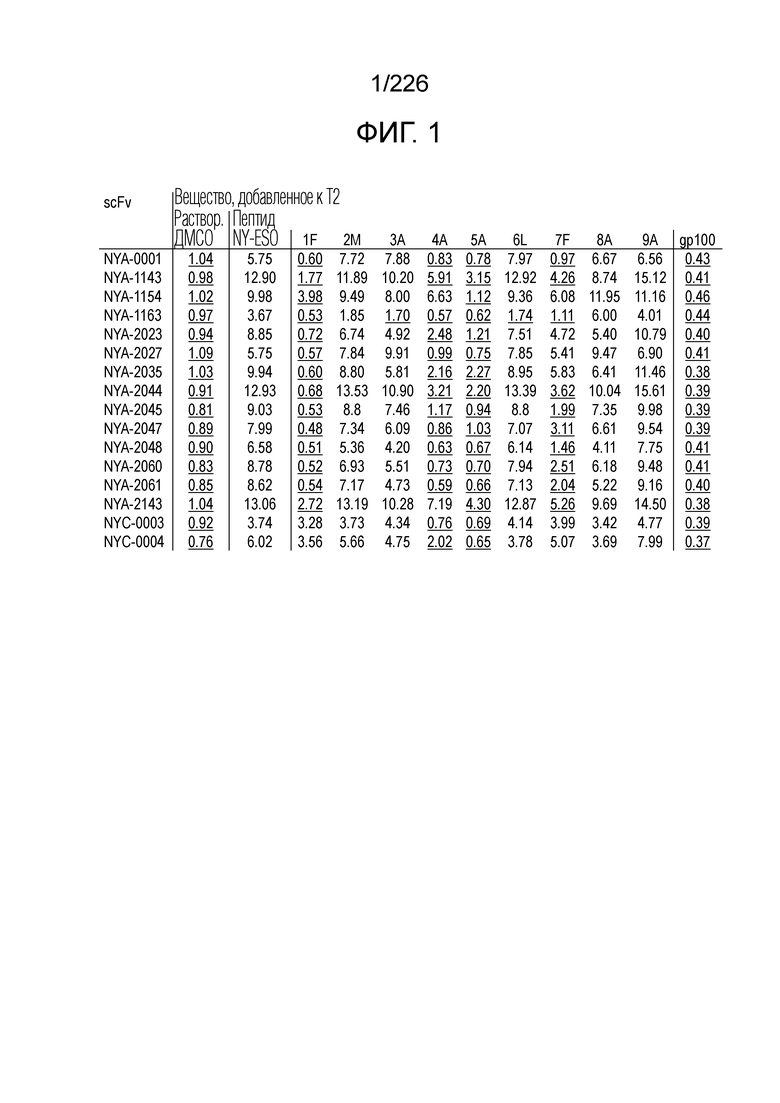
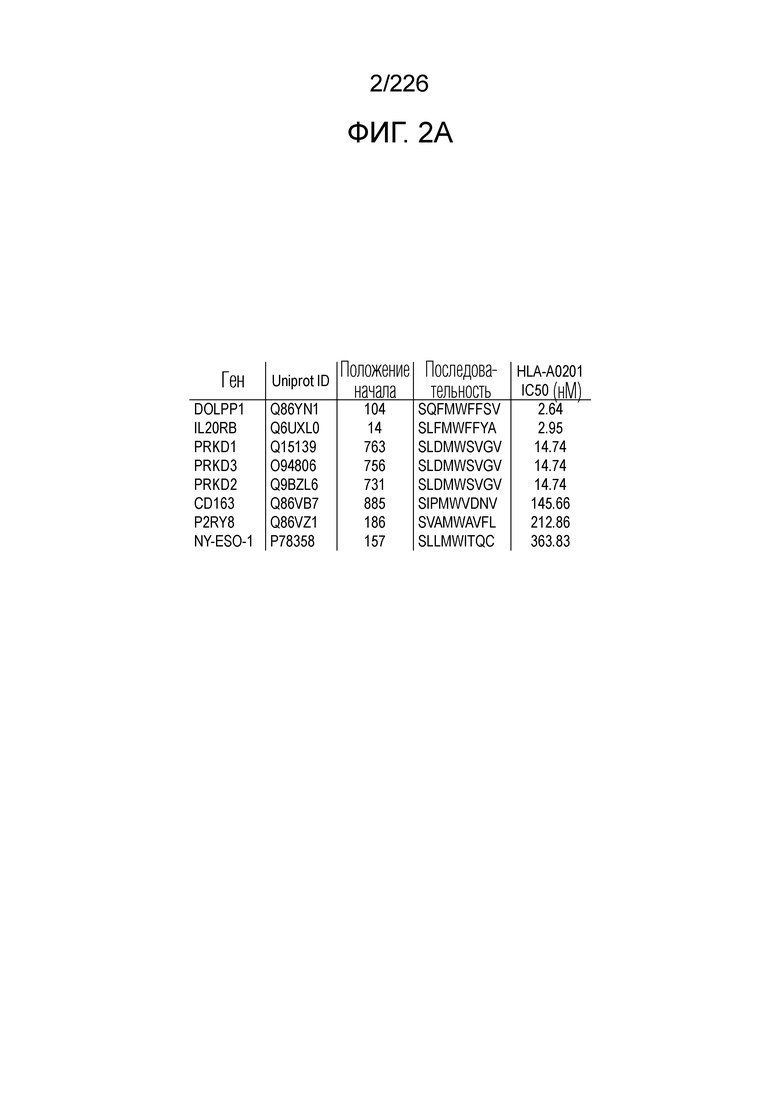
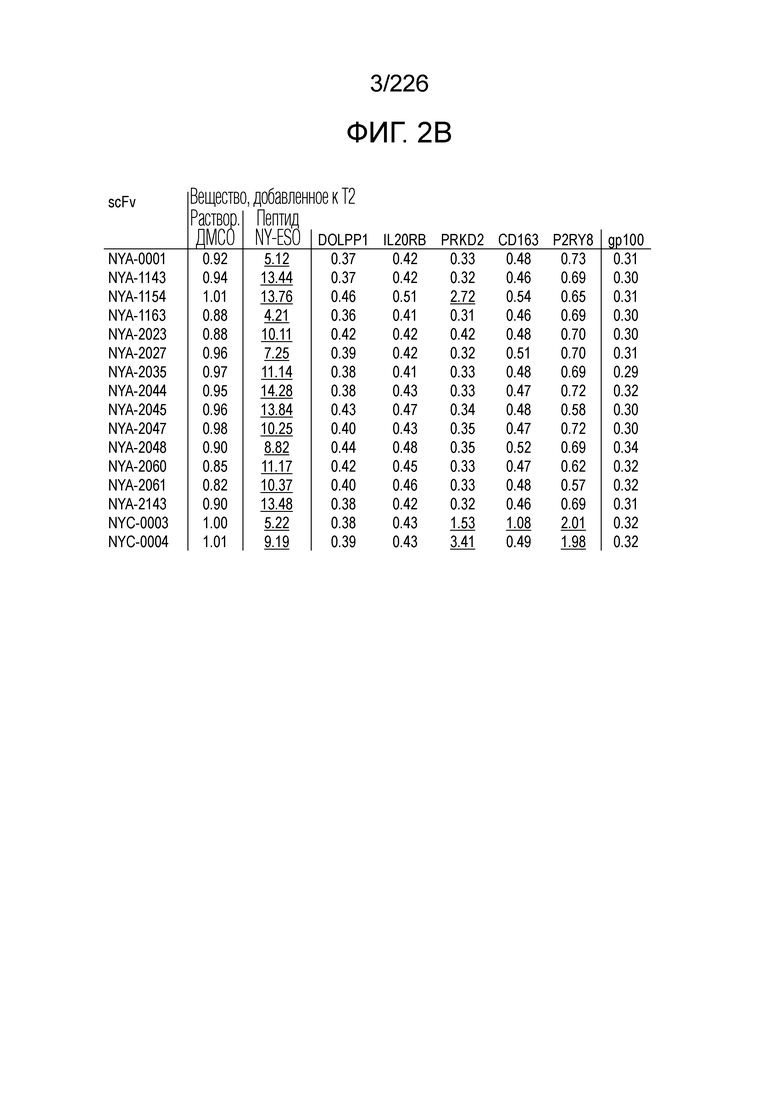
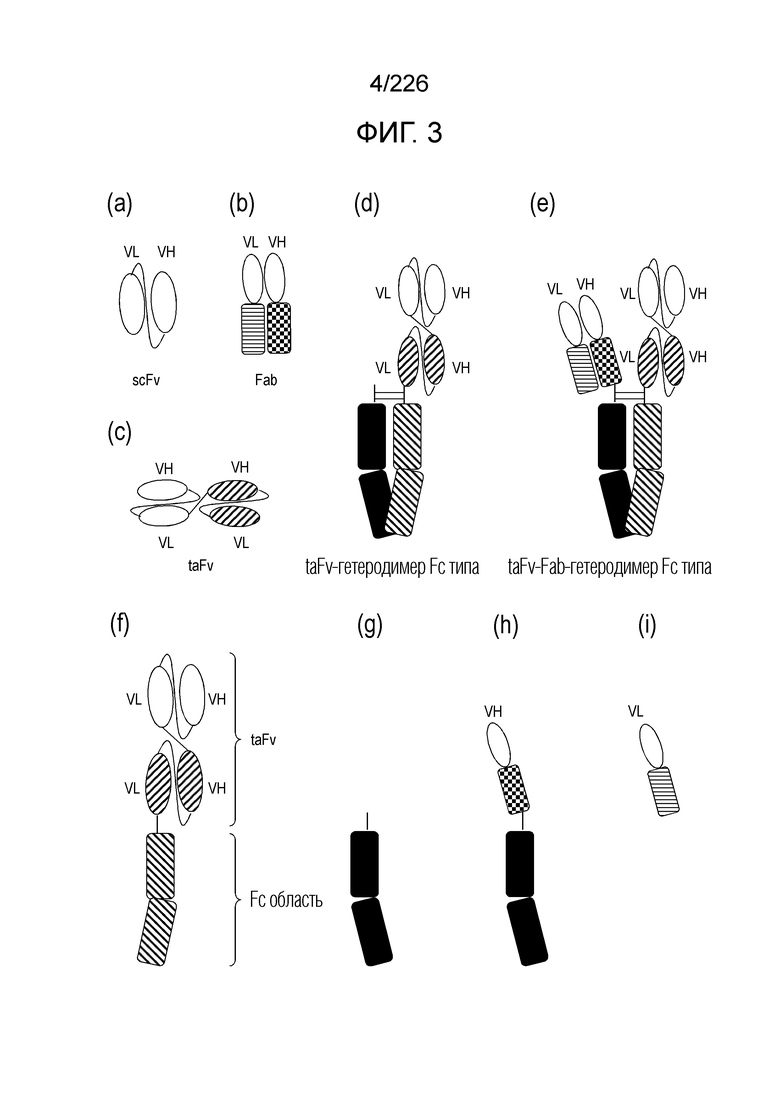
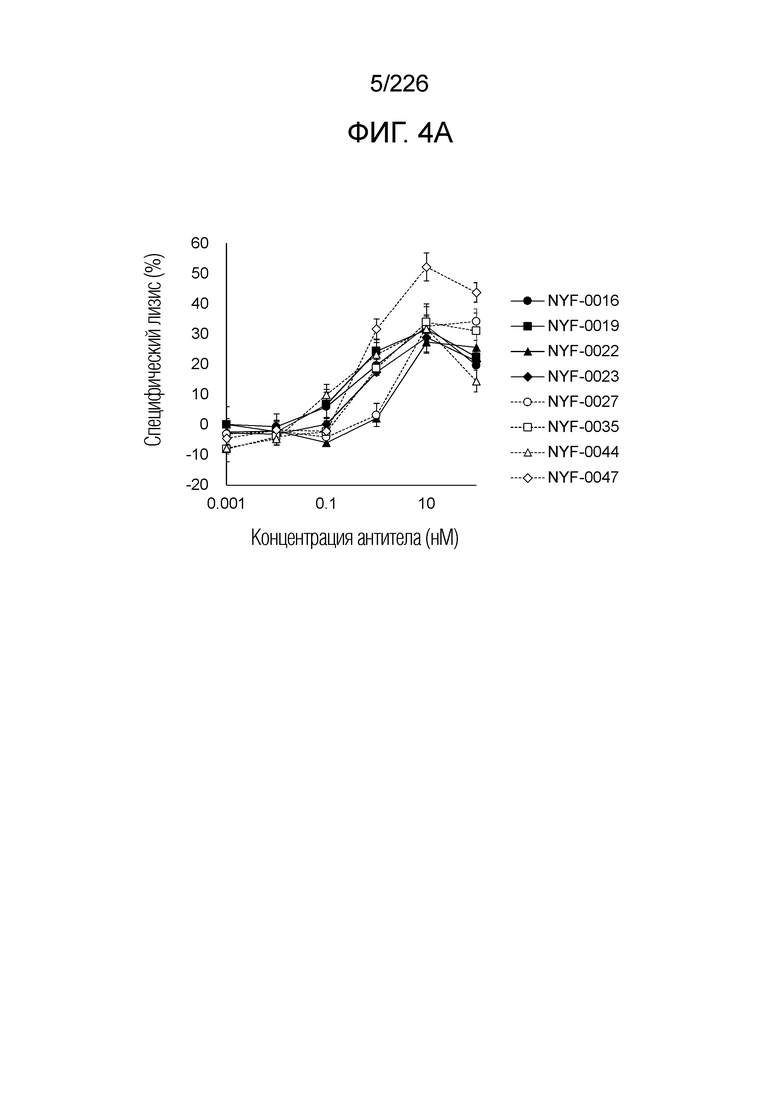
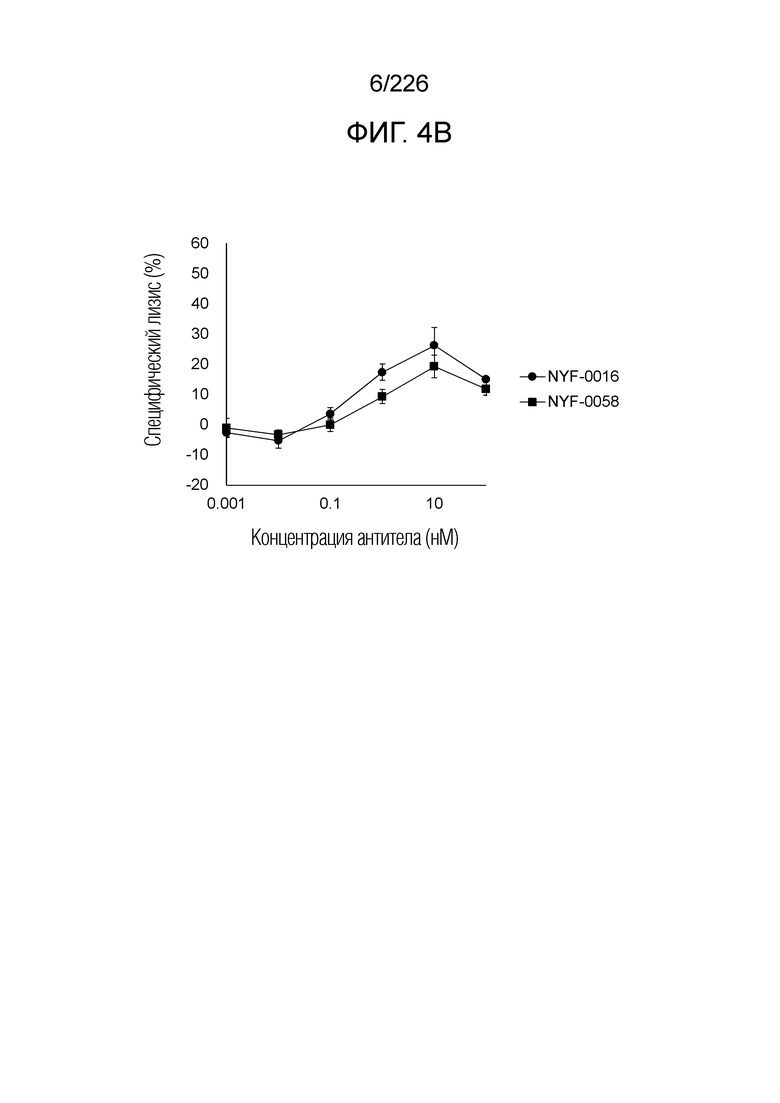
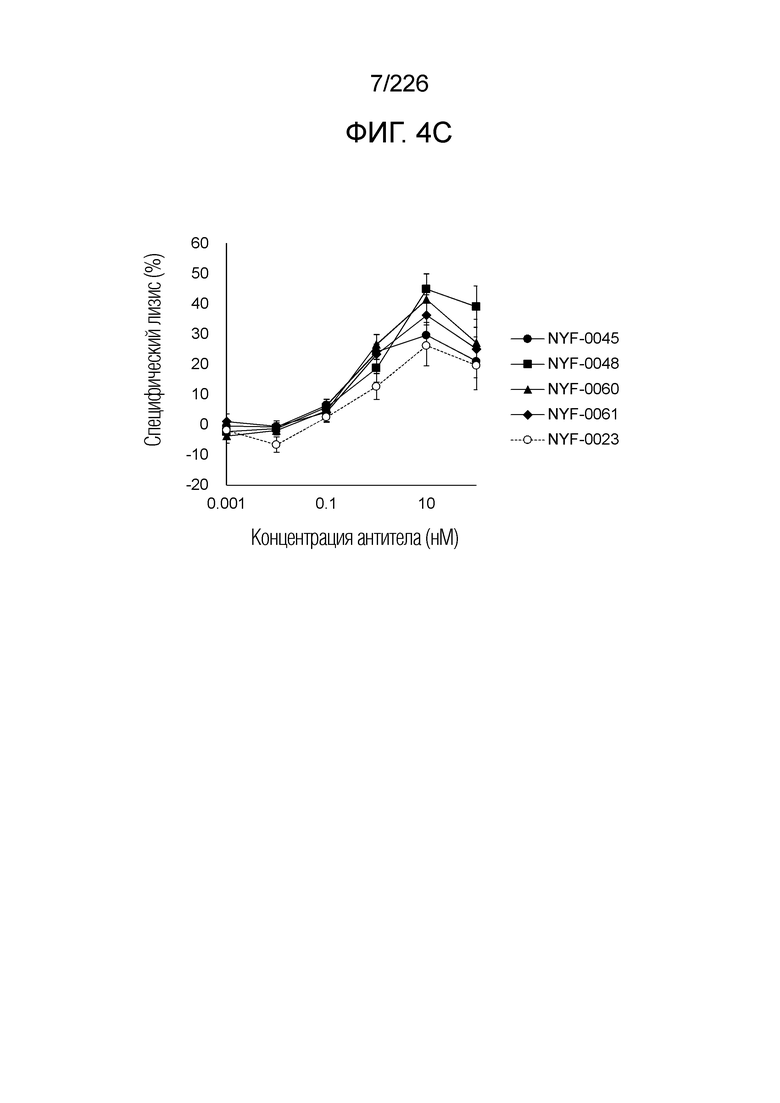
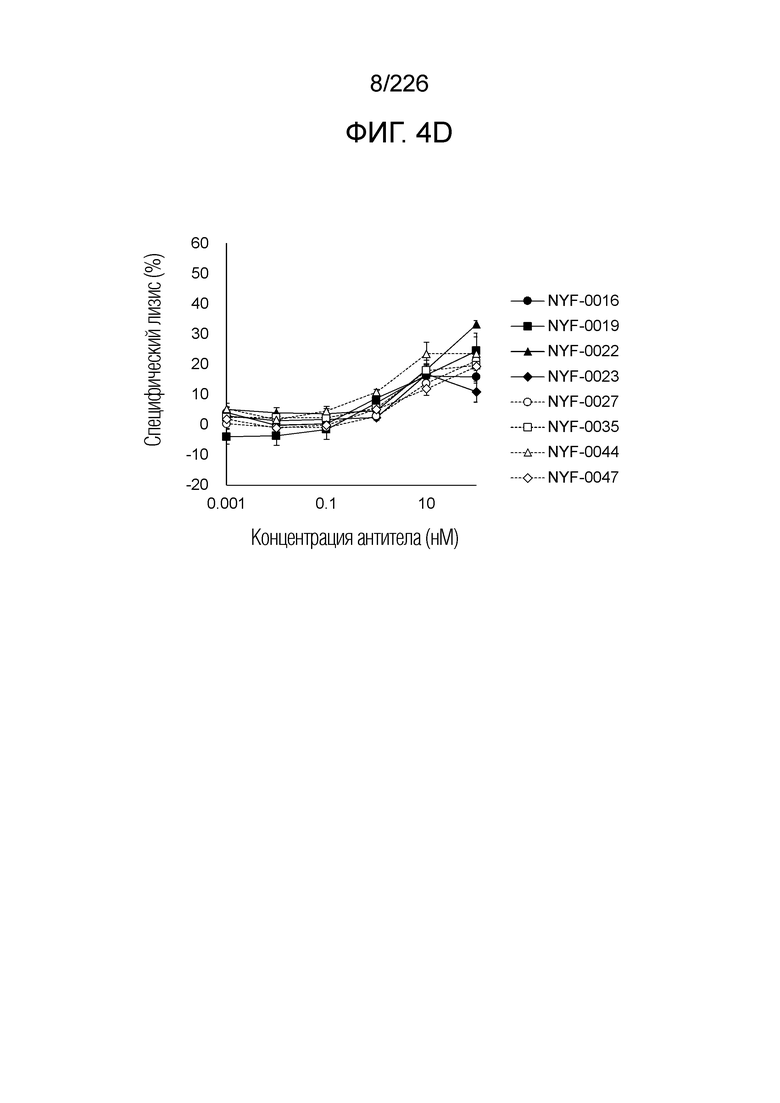
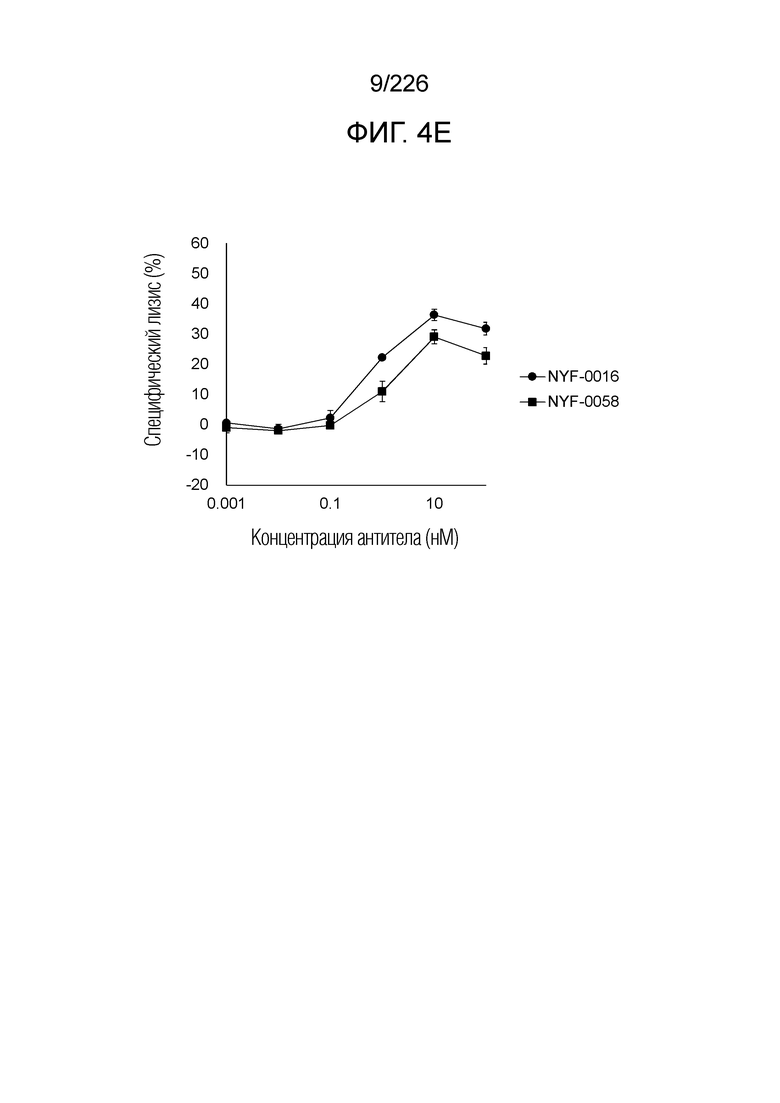
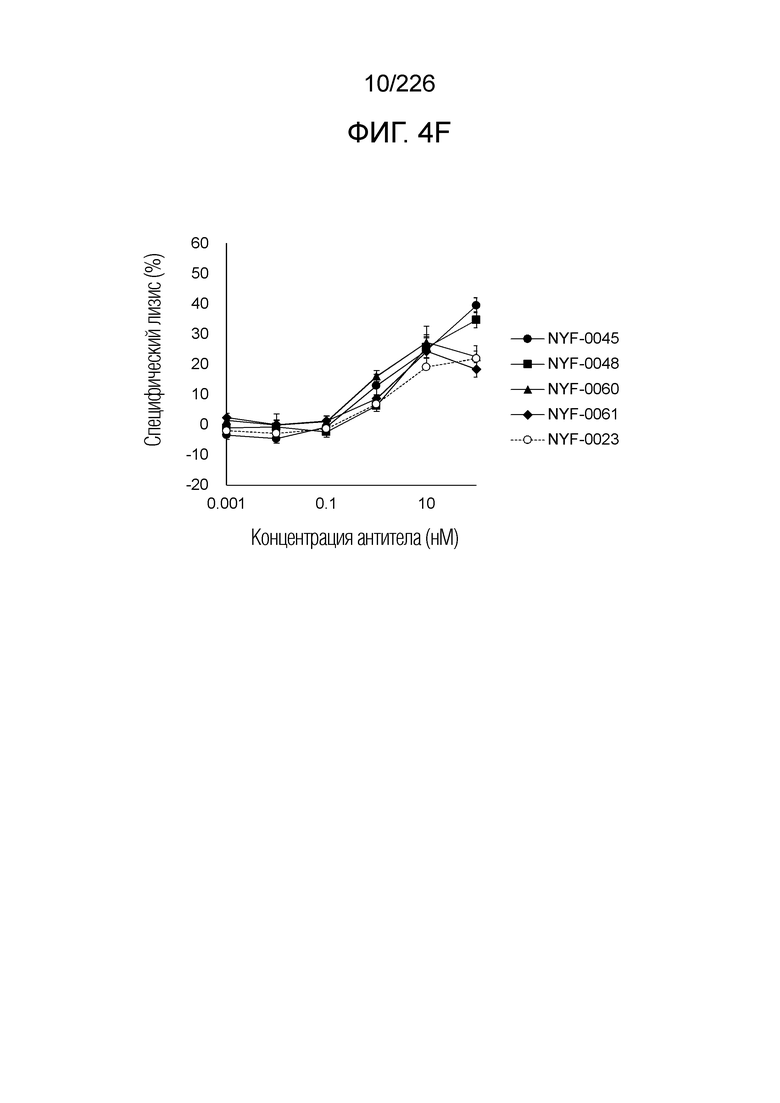
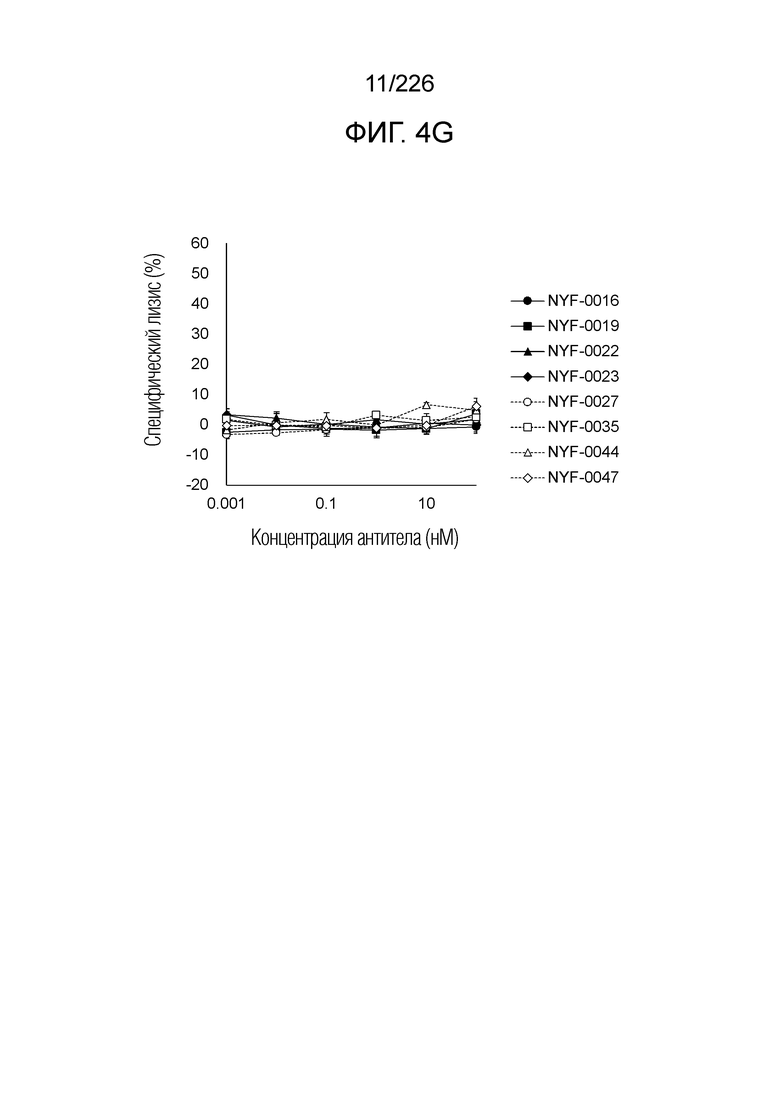
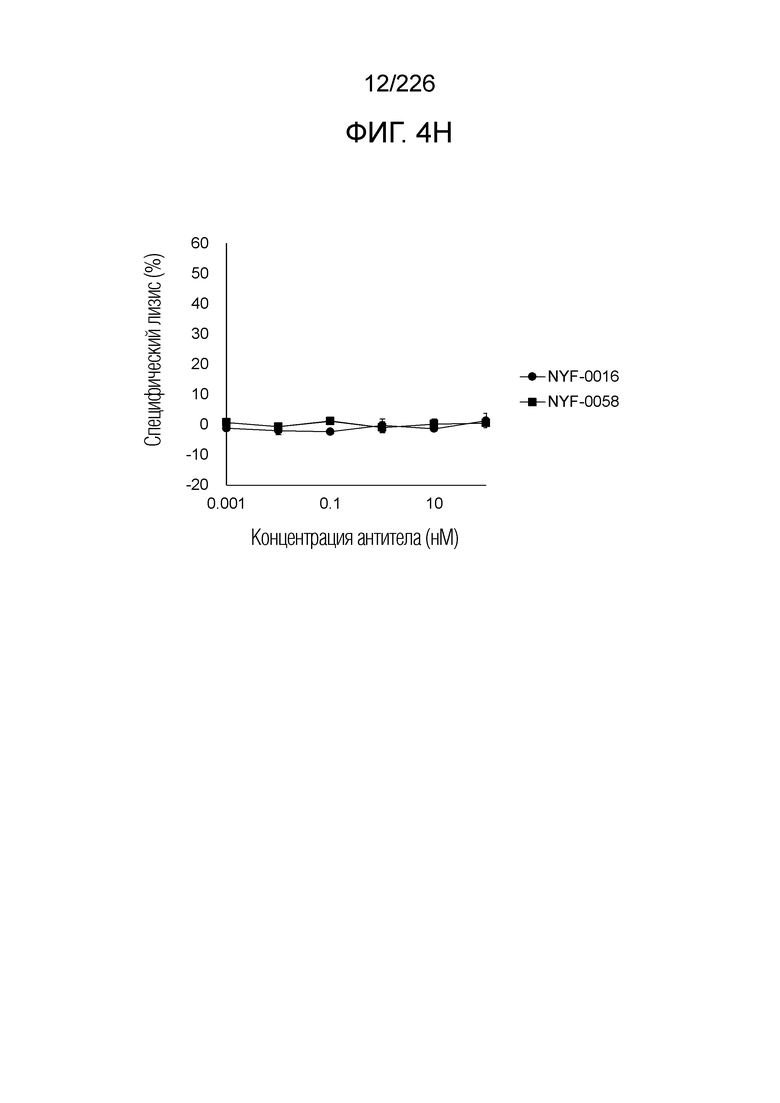
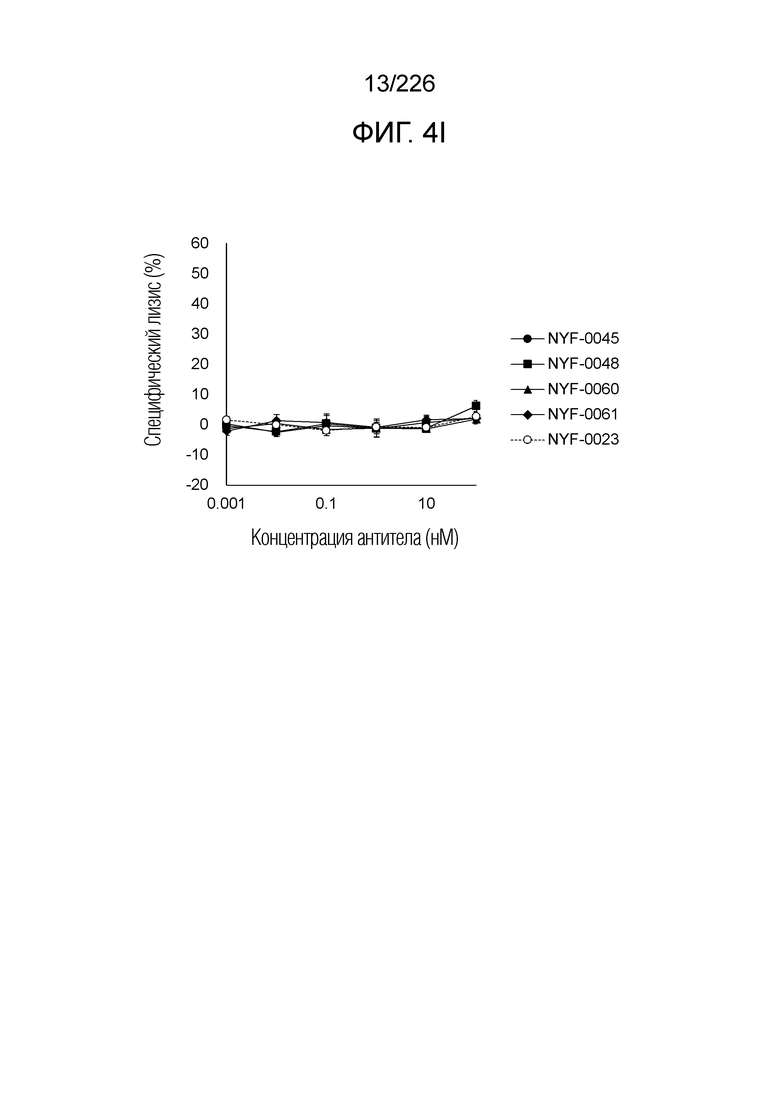
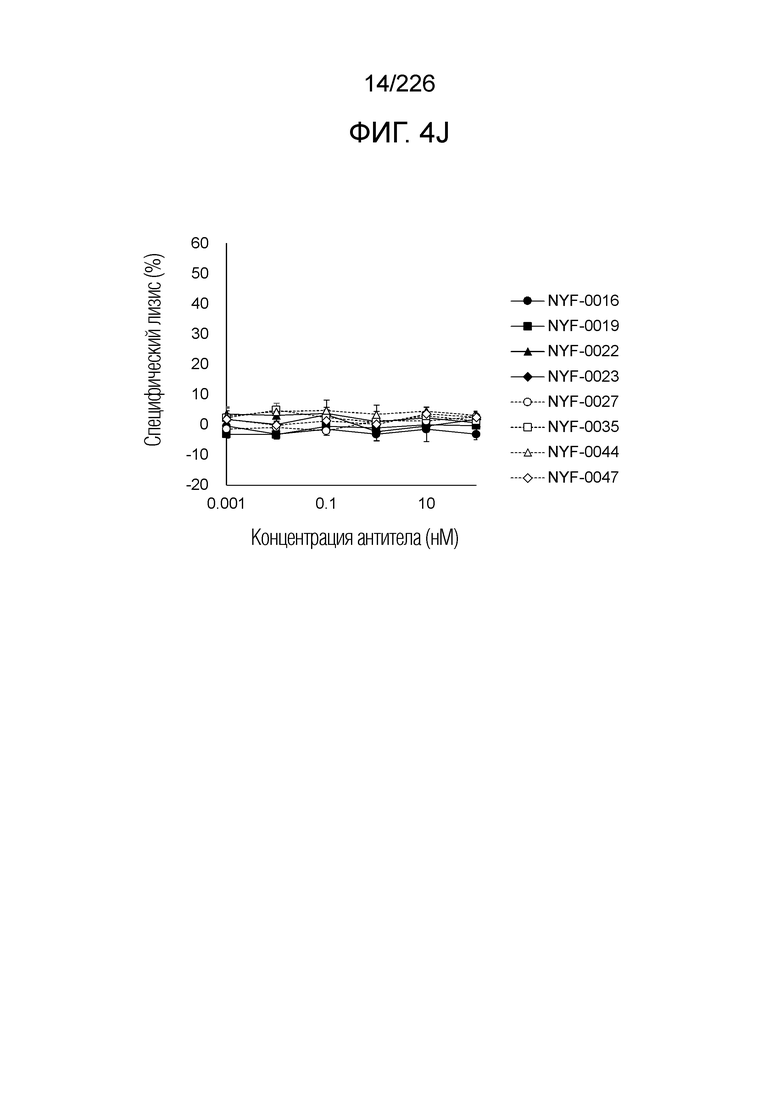
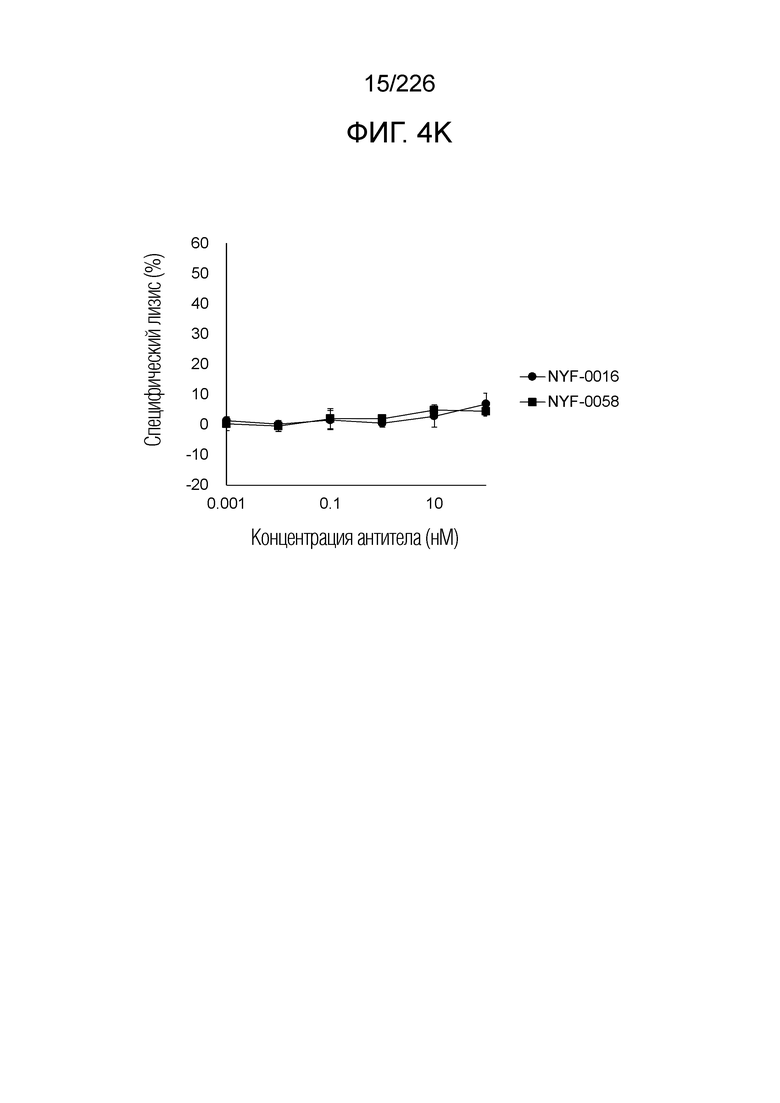
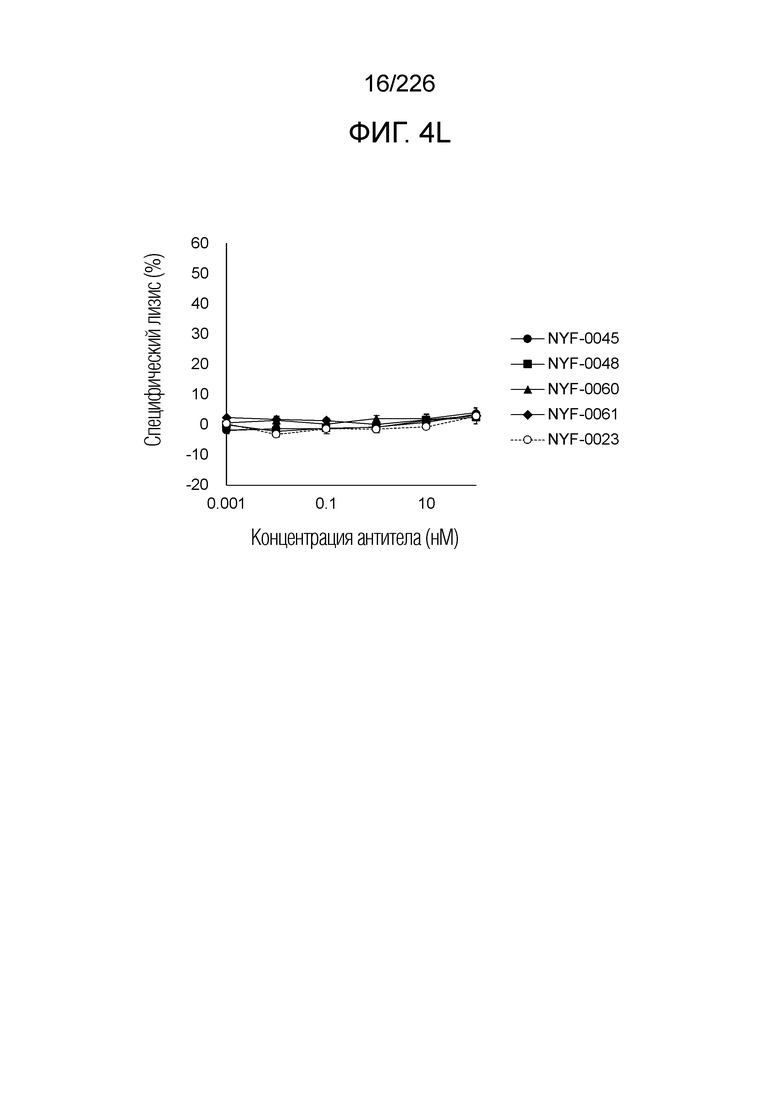
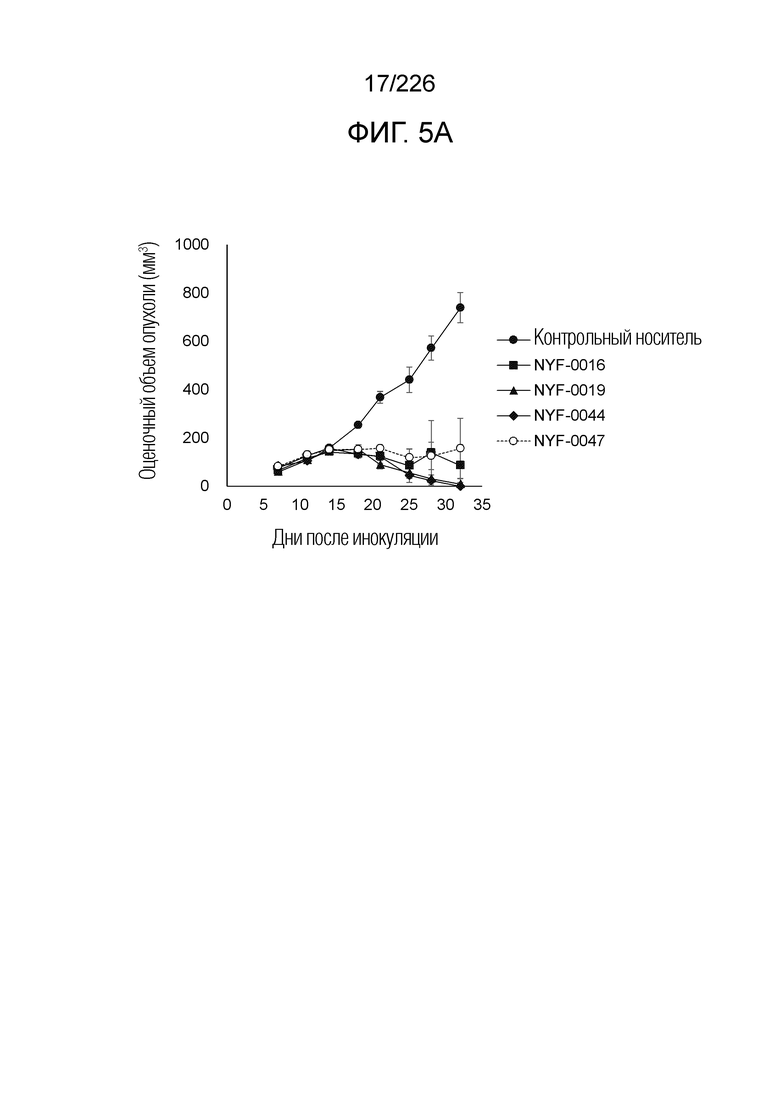
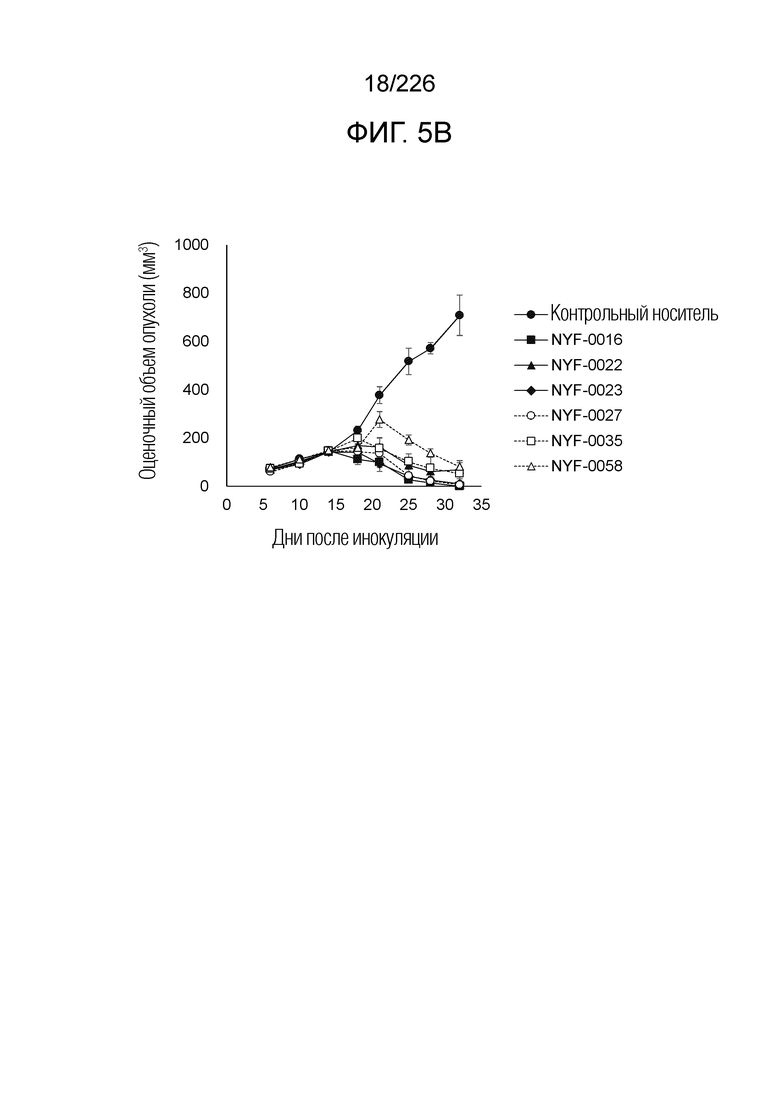
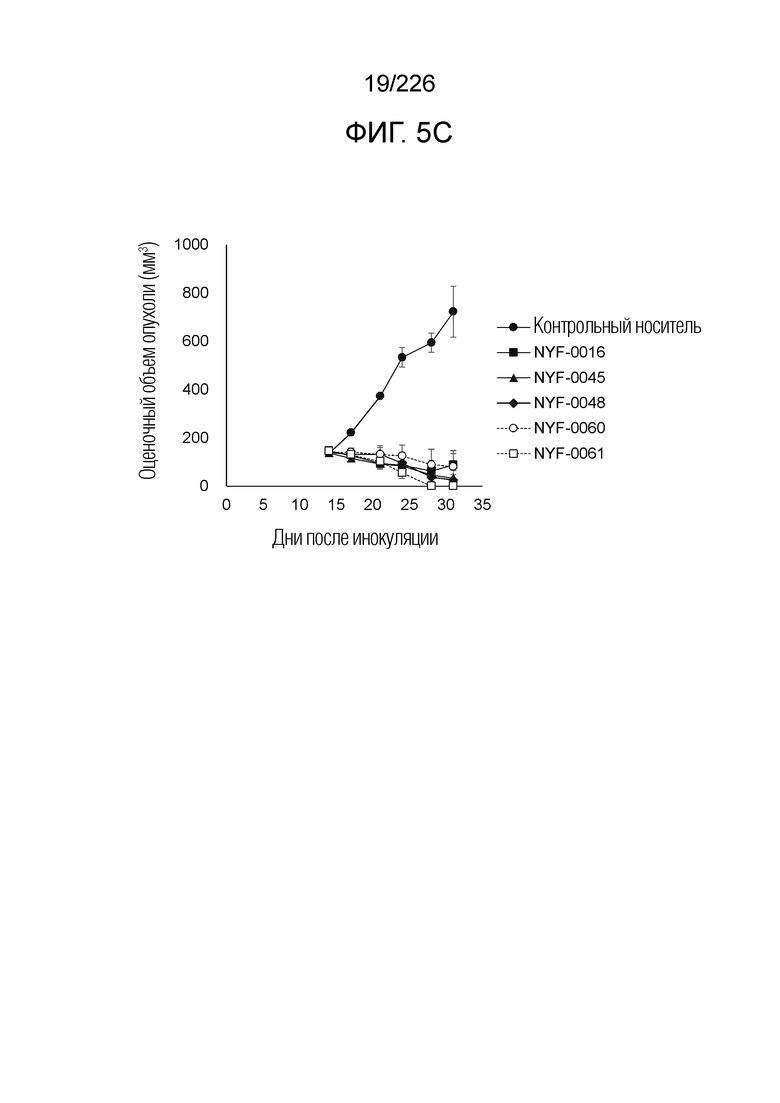
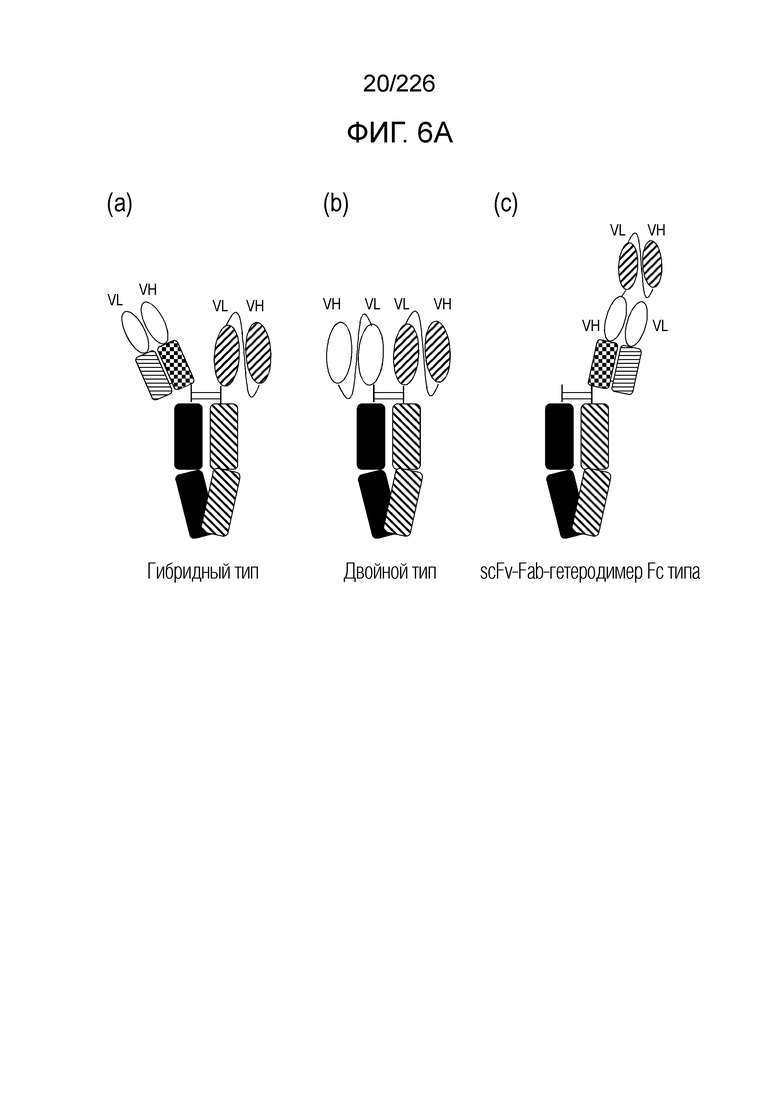
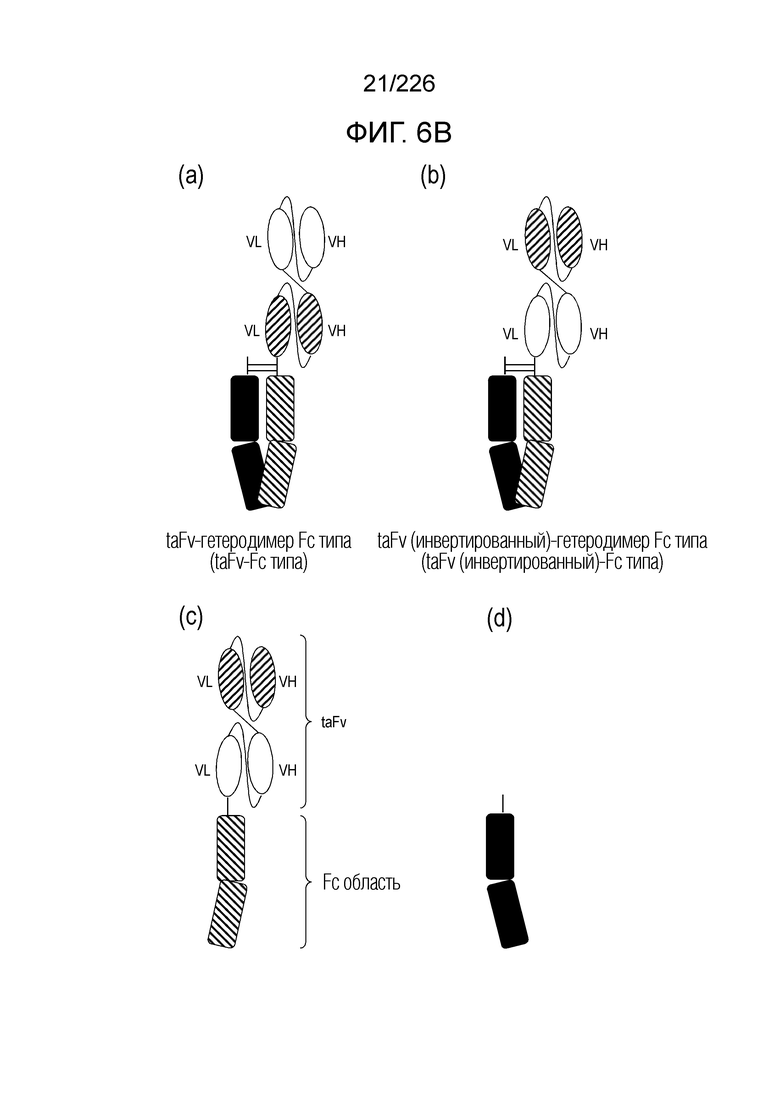
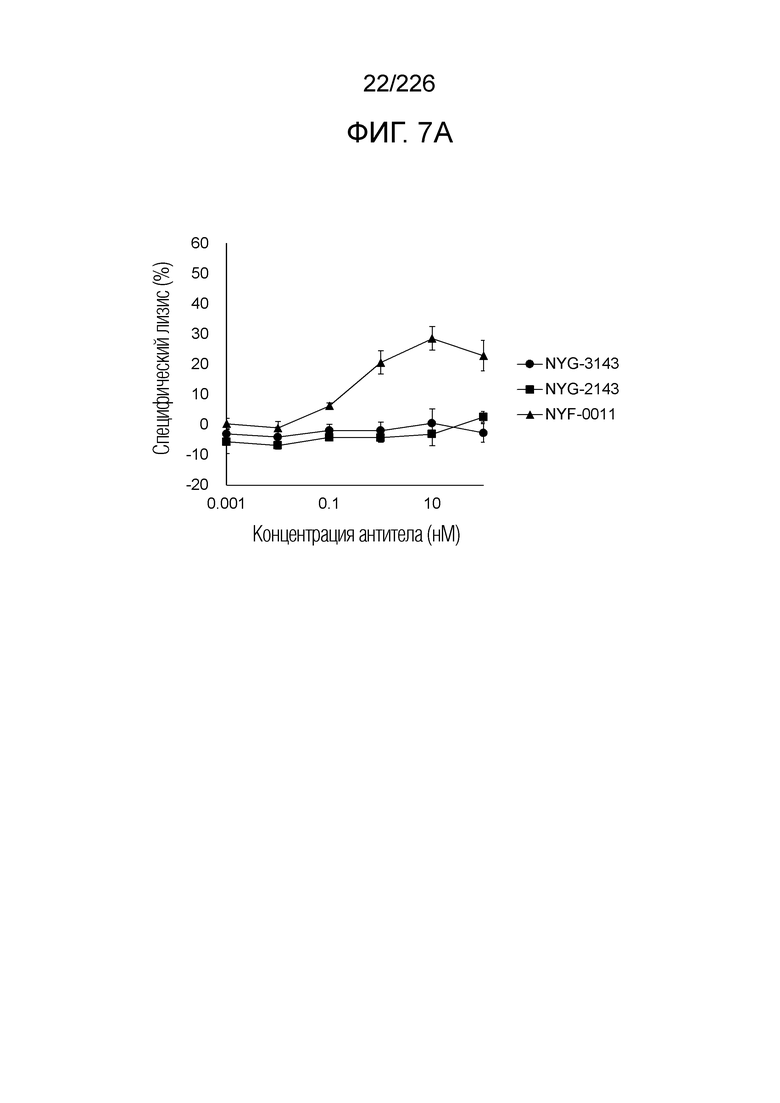
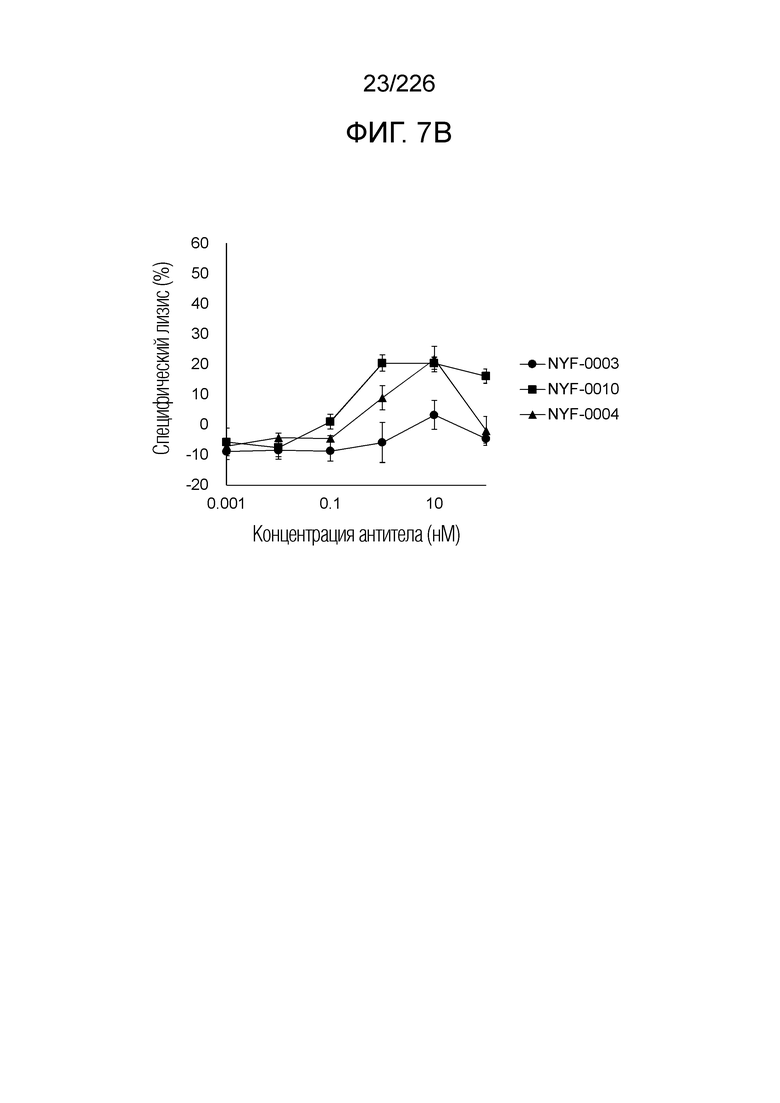























































































































































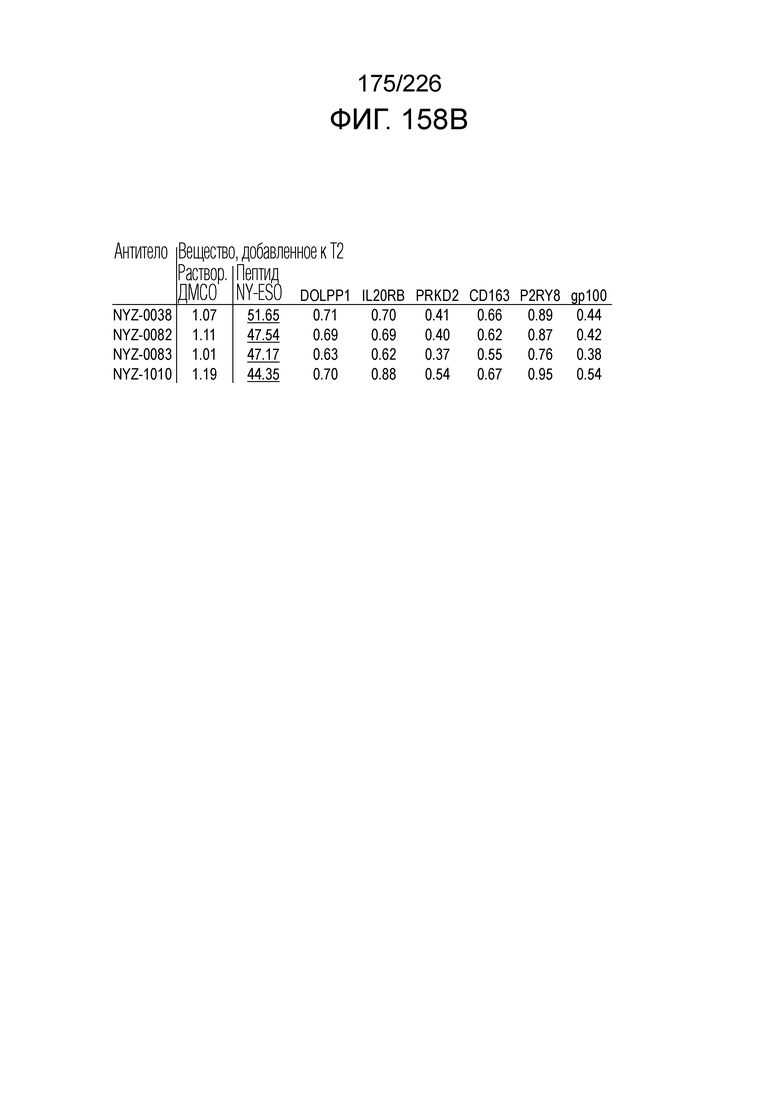

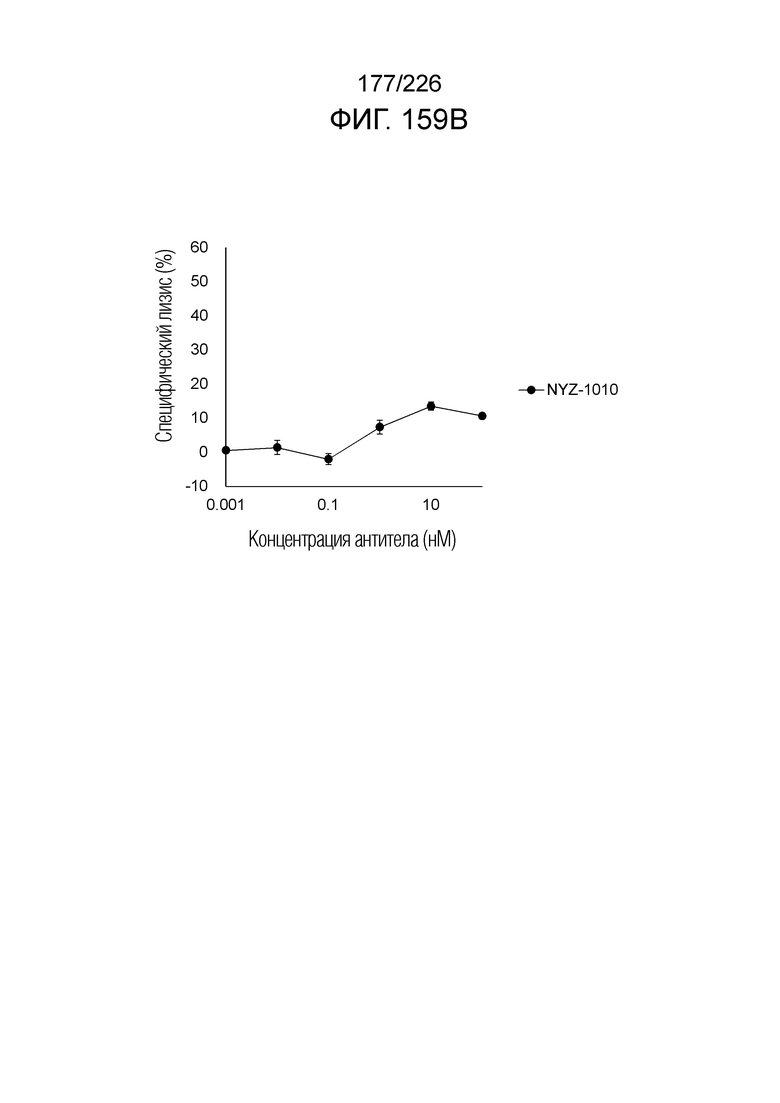
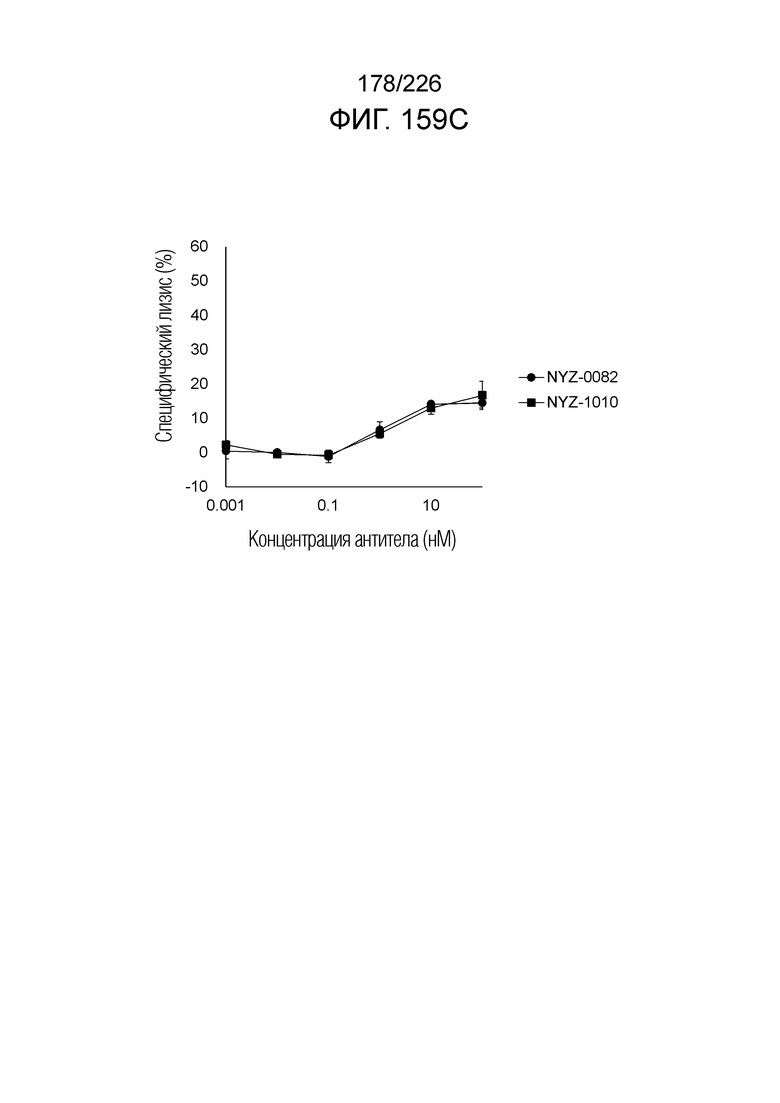
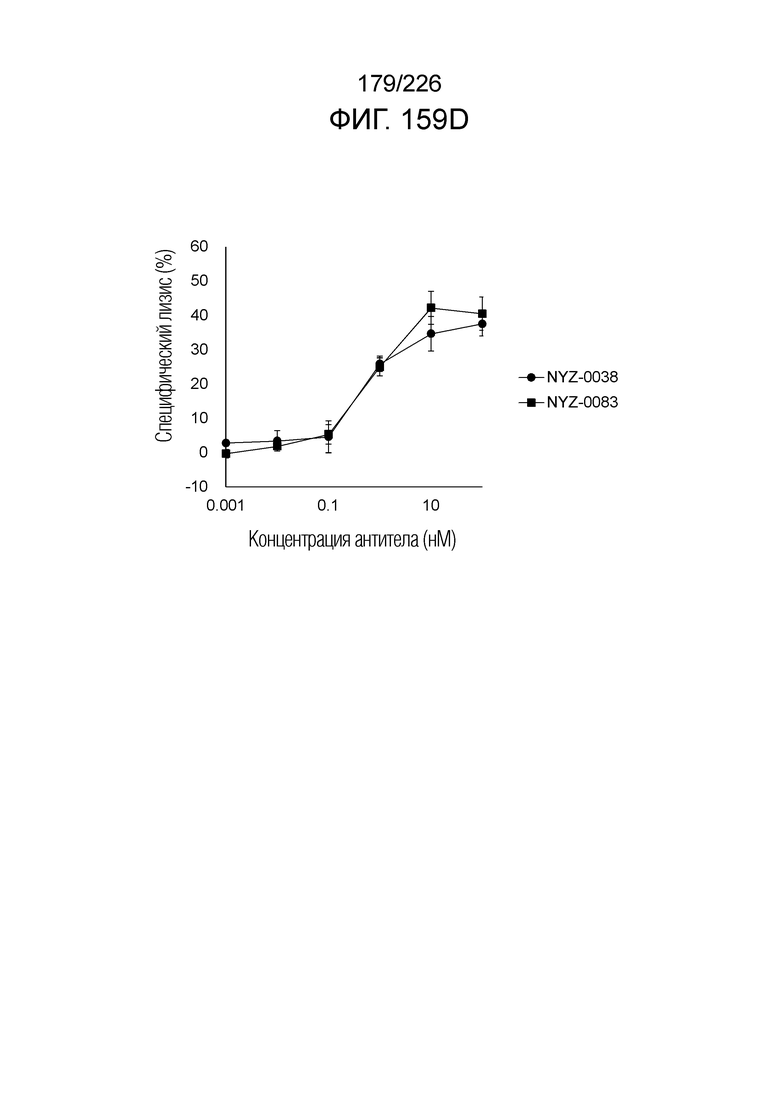
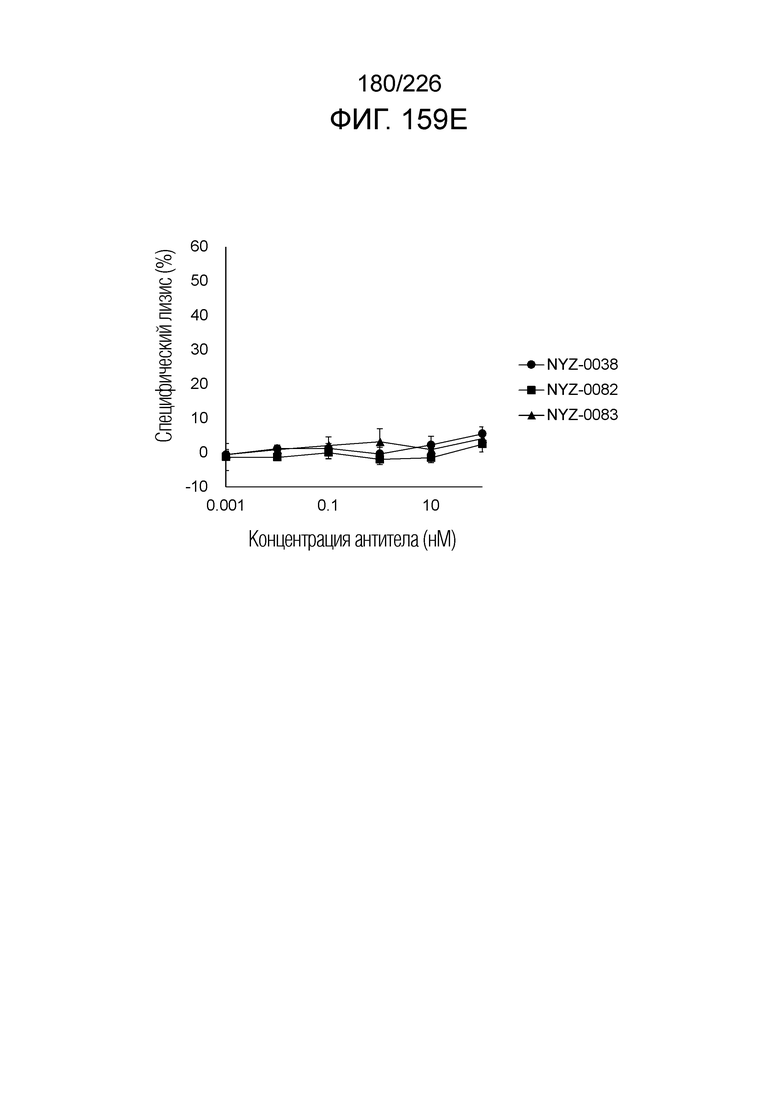
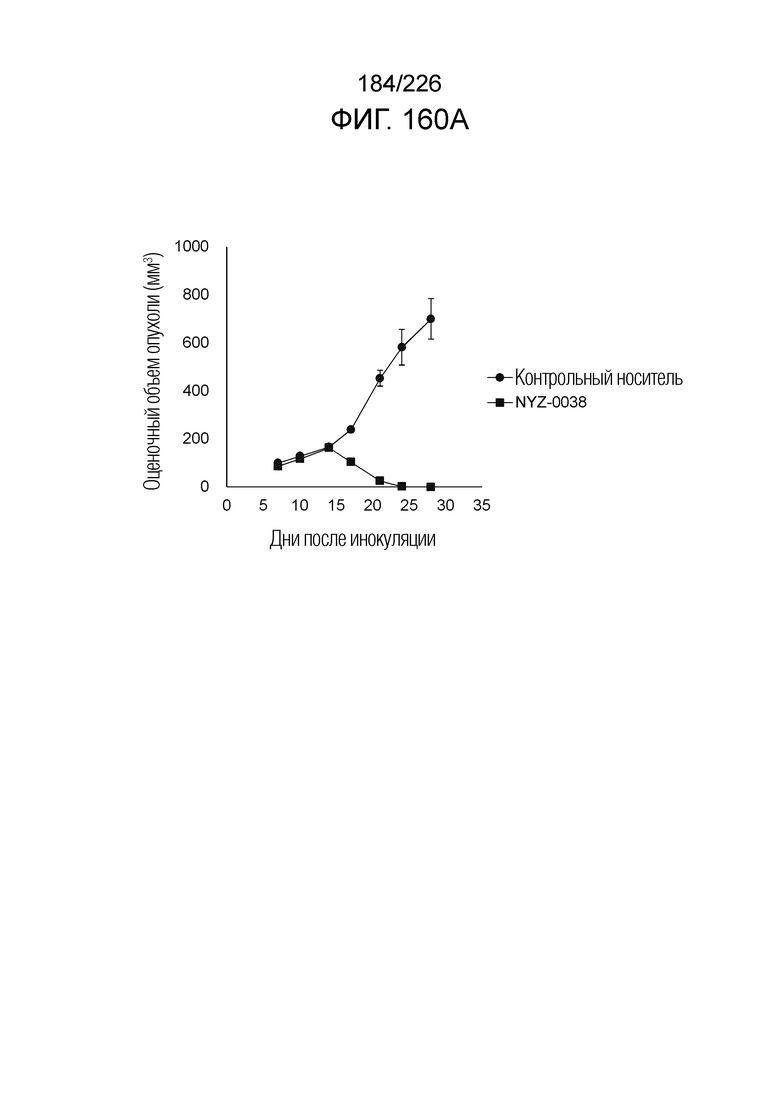
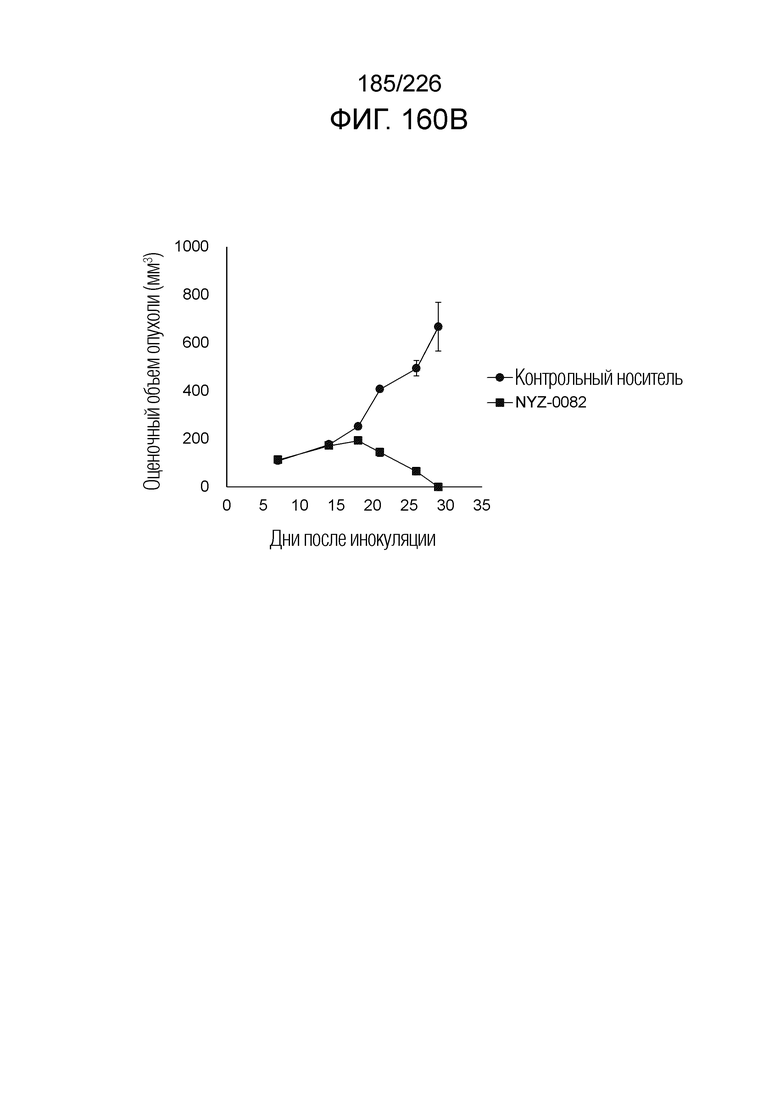
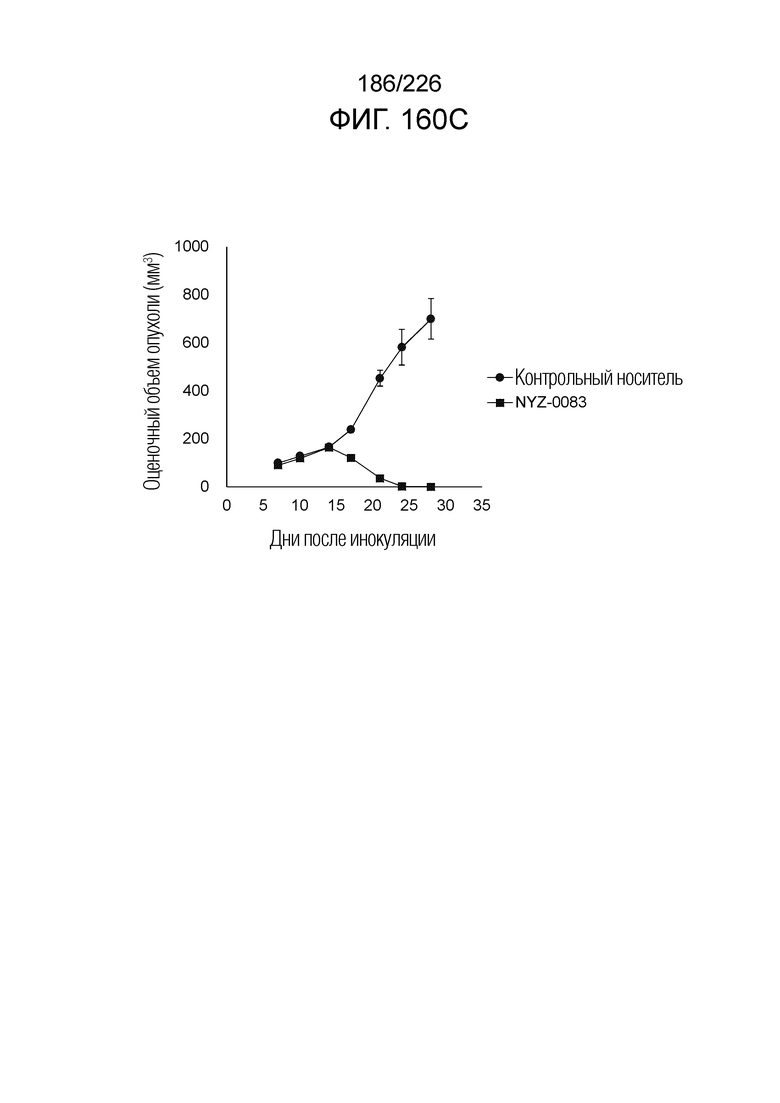
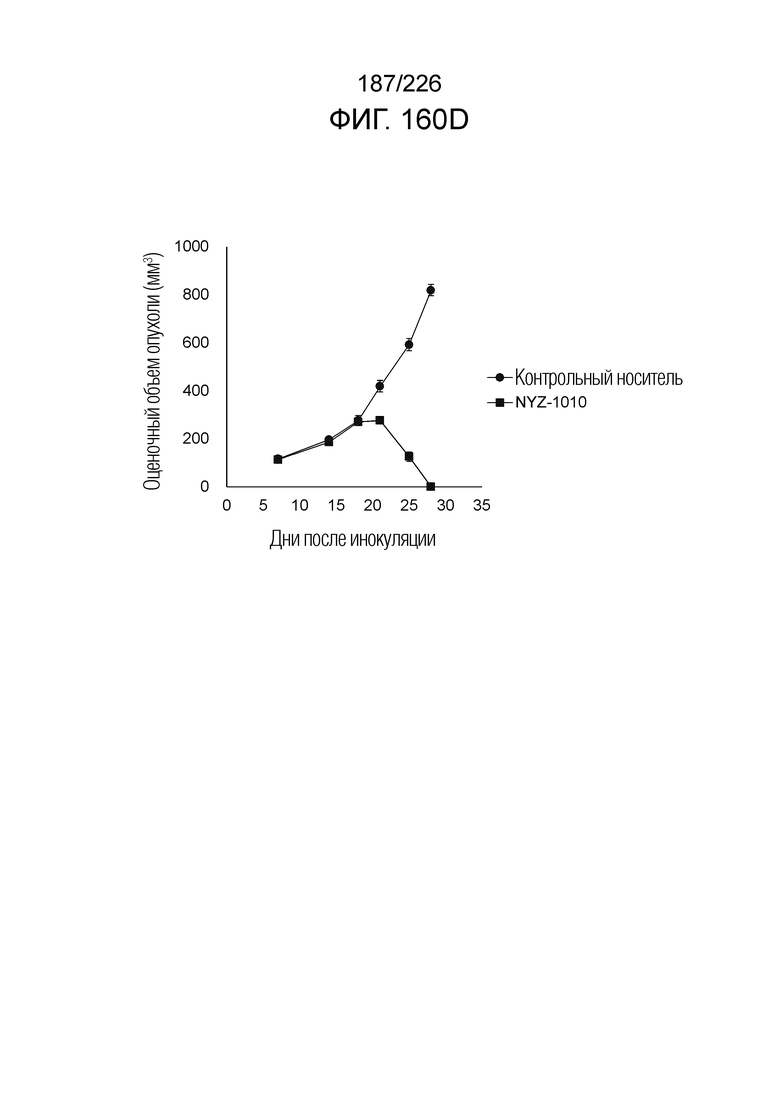

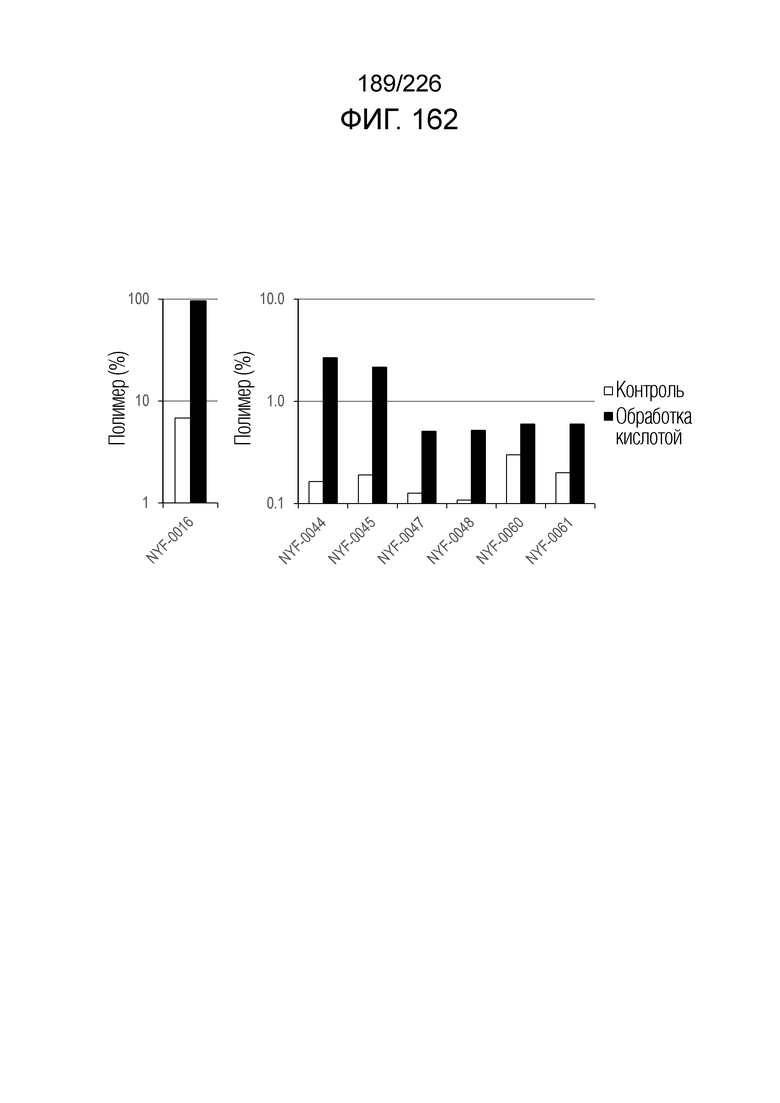




































Изобретение относится к области биотехнологии, в частности к антителу, которое специфически связывается с HLA/NY-ESO человека, или его связывающему фрагменту, к биспецифическому антителу, фармацевтической композиции, его содержащим, а также к способам получения вышеуказанных антител. Также раскрыты полинуклеотид, кодирующий вышеуказанное антитело или его фрагмент, полинуклеотид, кодирующий вышеуказанное биспецифическое антитело, а также клетка и вектор, содержащие вышеуказанные полинуклеотиды. Изобретение эффективно для профилактики или лечения рака с клетками, экспрессирующими HLA-A2/NY-ESO. 12 н. и 18 з.п. ф-лы, 199 ил., 15 табл., 23 пр.
1. Антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент, содержащее:
CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:54,
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:55,
CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:56,
CDRL1 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:57, в которой аминокислота 7 представляет собой W,
CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:58, и
CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:59.
2. Антитело, или его связывающий фрагмент, по п.1, содержащее
вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:53, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 156-266 аминокислотной последовательности SEQ ID NO:53, или
вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 21-140 аминокислотной последовательности SEQ ID NO:156, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 161-271 аминокислотной последовательности SEQ ID NO:156.
3. Антитело, или его связывающий фрагмент, по п.1 или 2, которое представляет собой scFv;
scFv состоит из аминокислотной последовательности из аминокислот 21-266 аминокислотной последовательности SEQ ID NO:53, или
scFv состоит из аминокислотной последовательности из аминокислот 21-271 аминокислотной последовательности SEQ ID NO:156.
4. Полинуклеотид, кодирующий антитело или его связывающий фрагмент по любому из пп.1-3.
5. Вектор экспрессии, содержащий полинуклеотид по п.4.
6. Клетка-хозяин для получения антитела или его связывающего фрагмента по любому из пп.1-3,
где клетка содержит полинуклеотид по п.4 или вектор по п.5.
7. Способ получения антитела, которое специфически связывается с HLA/NY-ESO человека, или его связывающего фрагмента, по п.1, включающий:
(i) стадию культивирования клетки-хозяина по п.6; и
(ii) стадию очистки антитела или его связывающего фрагмента из культурального продукта, полученного на стадии (i).
8. Антитело, или его связывающий фрагмент, по п.1, которое получено способом по п.7.
9. Фармацевтическая композиция для профилактики или лечения рака с клетками, экспрессирующими HLA-A2/NY-ESO, содержащая в качестве активного ингредиента антитело, или его связывающий фрагмент, по любому из пп.1-3 и 8, полинуклеотид по п.4, вектор по п.5 или клетку по п.6.
10. Биспецифическое антитело, которое
специфически связывается с HLA/NY-ESO человека и содержит антитело, или его связывающий фрагмент, по любому из пп.1-3 и 8, и
специфически связывается с CD3 и содержит антитело, которое специфически связывается с CD3, или его связывающий фрагмент, где антитело, которое специфически связывается с CD3, или его связывающий фрагмент, содержит:
(CCH1) CDRH1 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:141;
(CCH2) CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:142, или
CDRH2 тяжелой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:142, в которой аминокислота 3 представляет собой N или S;
(CCH3) CDRH3 тяжелой цепи, состоящую из аминокислотной последовательности SEQ ID NO:143;
(CCL1) CDRL1 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:144;
(CCL2) CDRL2 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:145, или
CDRL2 легкой цепи, состоящую из аминокислотной последовательности, полученной из аминокислотной последовательности SEQ ID NO:145, в которой аминокислота 2 представляет собой N; и
(CCL3) CDRL3 легкой цепи, состоящую из аминокислотной последовательности SEQ ID NO:146.
11. Биспецифическое антитело по п.10, где антитело, которое специфически связывается с CD3, или его связывающий фрагмент, содержит:
(CH1CL1) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:136, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:136;
(CH2CL2) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:137, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:137;
(CH3CL3) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:147, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:147;
(CH4CL4) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:138, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-241 аминокислотной последовательности SEQ ID NO:138;
(CH5CL5) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:139, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:139;
(CH6CL6) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 2-119 аминокислотной последовательности SEQ ID NO:140, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 135-243 аминокислотной последовательности SEQ ID NO:140;
(CH7CL7) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 272-389 аминокислотной последовательности SEQ ID NO:155, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 405-511 аминокислотной последовательности SEQ ID NO:155;
(CH8CL8) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:156, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:156; или
(CH9CL9) вариабельную область тяжелой цепи, состоящую из аминокислотной последовательности из аминокислот 277-394 аминокислотной последовательности SEQ ID NO:157, и
вариабельную область легкой цепи, состоящую из аминокислотной последовательности из аминокислот 410-516 аминокислотной последовательности SEQ ID NO:157.
12. Биспецифическое антитело по п.11, где антитело, которое специфически связывается с CD3, или его связывающий фрагмент, представляет собой scFv;
предпочтительно, где scFv содержит:
(CS1) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:136;
(CS2) scFv, состоящий из аминокислотной последовательности из аминокислот 2-241 аминокислотной последовательности SEQ ID NO:137;
(CS3) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:147;
(CS4) scFv, состоящий из аминокислотной последовательности из аминокислот 2-241 аминокислотной последовательности SEQ ID NO:138;
(CS5) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:139;
(CS6) scFv, состоящий из аминокислотной последовательности из аминокислот 2-243 аминокислотной последовательности SEQ ID NO:140;
(CS7) scFv, состоящий из аминокислотной последовательности из аминокислот 272-511 из аминокислотной последовательности SEQ ID NO:155;
(CS8) scFv, состоящий из аминокислотной последовательности из аминокислот 277-516 из аминокислотной последовательности SEQ ID NO:156; или
(CS9) scFv, состоящий из аминокислотной последовательности из аминокислот 277-516 аминокислотной последовательности SEQ ID NO:157.
13. Биспецифическое антитело по п.11 или 12, содержащее первый полипептид,
где первый полипептид содержит антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент, антитело, которое специфически связывается с CD3, или его антигенсвязывающий фрагмент; и Fc область (i) в порядке от N конца к С концу;
где антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент, представляет собой scFc,
где антитело, которое специфически связывается с CD3, или его связывающий фрагмент, представляет собой scFc; и
второй полипептид, содержащий Fc область (ii),
где первый полипептид связан со вторым полипептидом в Fc области (i) и Fc области (ii).
14. Биспецифическое антитело по п.13, где scFv, который специфически связывается с HLA/NY-ESO человека, содержит
аминокислотную последовательность, состоящую из аминокислотной последовательности из аминокислот 21-266 SEQ ID NO:53, или
аминокислотную последовательность, состоящую из аминокислотной последовательности из аминокислот 21-271 SEQ ID NO:156.
15. Биспецифическое антитело по п.13 или 14, которое содержит аминокислотную последовательность, выбранную из группы, состоящей из
аминокислотной последовательности из аминокислот 21-511 аминокислотной последовательности SEQ ID NO:96,
аминокислотной последовательности из аминокислот 21-516 аминокислотной последовательности SEQ ID NO:156, и
аминокислотной последовательности из аминокислот 21-516 аминокислотной последовательности SEQ ID NO:157.
16. Биспецифическое антитело по любому из пп.13-15, где первый полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из
аминокислотной последовательности из аминокислот 529-745 аминокислотной последовательности SEQ ID NO:96,
аминокислотной последовательности из аминокислот 534-750 аминокислотной последовательности SEQ ID NO:156, и
аминокислотной последовательности из аминокислот 534-750 аминокислотной последовательности SEQ ID NO:157; или
аминокислотной последовательности из аминокислот 20-745 аминокислотной последовательности SEQ ID NO:96,
аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:156, и
аминокислотной последовательности из аминокислот 20-750 аминокислотной последовательности SEQ ID NO:157.
17. Биспецифическое антитело по любому из пп.13-16, где второй полипептид содержит аминокислотную последовательность из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84.
18. Биспецифическое антитело по п.11 или 12, содержащее
первый полипептид,
где первый полипептид содержит антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающий фрагмент, антитело, которое специфически связывается с CD3, или его связывающий фрагмент, и Fc область (i) в порядке от N конца к С концу,
где антитело, которое специфически связывается с CD3, или его связывающийся фрагмент, содержит вариабельную область тяжелой цепи и константную область CH1,
второй полипептид и третий полипептид,
где антитело, которое специфически связывается с HLA/NY-ESO человека, или его связывающийся фрагмент, представляет собой scFv, и
антитело, которое специфически связывается с CD3, или его связывающийся фрагмент, представляет собой scFv,
второй полипептид содержит тяжелую цепь иммуноглобулина, содержащую Fc-область (ii), и
третий полипептид содержит легкую цепь иммуноглобулина; и
второй полипептид связан с третьим полипептидом, и
первый полипептид связан со вторым полипептидом в Fc области,
второй полипептид содержит шарнирную область иммуноглобулина и Fc область (ii) иммуноглобулина; и
третий полипептид содержит легкую цепь антитела, состоящую из вариабельной области и константной области,
где первый полипептид связан со вторым полипептидом в Fc области (i) и Fc области (ii), и первый полипептид связан с третьим полипептидом в вариабельной области тяжелой цепи и константной области CH1 антитела.
19. Биспецифическое антитело по п.17, где антитело, которое специфически связывается с CD3, представляет собой Fab.
20. Биспецифическое антитело по п.18 или 19, где первый полипептид содержит
аминокислотную последовательность из аминокислот 21-394 аминокислотной последовательности SEQ ID NO:160,
аминокислотную последовательность из аминокислот 20-724 аминокислотной последовательности SEQ ID NO:160; или
аминокислотную последовательность из аминокислот 20-719 аминокислотной последовательности SEQ ID NO:197.
21. Биспецифическое антитело по любому из пп.18-20,
где второй полипептид содержит аминокислотную последовательность из аминокислот 20-246 аминокислотной последовательности SEQ ID NO:84 и/или
где третий полипептид содержит аминокислотную последовательность из аминокислот 21-127 аминокислотной последовательности SEQ ID NO:161;
предпочтительно, где второй полипептид содержит аминокислотную последовательность из аминокислот 20-246 аминокислотной последовательности SEQ ID NO: 84, и
где третий полипептид содержит аминокислотную последовательность из аминокислот 21-233 аминокислотной последовательности SEQ ID NO: 161,
более предпочтительно, где первый полипептид содержит аминокислотную последовательность из аминокислот 20-724 аминокислотной последовательности SEQ ID NO:160; или аминокислотную последовательность из аминокислот 20-719 аминокислотной последовательности SEQ ID NO:197; и
где второй полипептид содержит аминокислотную последовательность аминокислот 20-246 из аминокислотной последовательности SEQ ID NO:84, и
где третий полипептид содержит аминокислотную последовательность из аминокислот 21-233 аминокислотной последовательности SEQ ID NO:161.
22. Биспецифическое антитело по любому из пп.10-21, где с карбоксильного конца аминокислотной последовательности по меньшей мере одного полипептида, включенного в молекулу, удалены 1 или 2 аминокислоты.
23. Молекула нуклеиновой кислоты, кодирующая биспецифическое антитело по любому из пп.10-22.
24. Вектор экспрессии, содержащий молекулу нуклеиновой кислоты по п.23.
25. Клетка-хозяин для получения биспецифического антитела по любому из пп.10-21, где клетка содержит вектор по п.24.
26. Способ получения биспецифического антитела, которое специфически связывается с HLA/NY-ESO человека и CD3 человека, по п.10, включающий:
(i) стадию культивирования клетки-хозяина по п.25; и
(ii) стадию очистки антитела, или его связывающего фрагмента, из культурального продукта, полученного на стадии (i).
27. Биспецифическое антитело, которое специфически связывается с HLA/NY-ESO человека и CD3 человека, по п.10, которое получено способом по п.26.
28. Фармацевтическая композиция для профилактики или лечения рака с клетками, экспрессирующими HLA-A2/NY-ESO, содержащая в качестве активного ингредиента биспецифическое антитело по любому из пп.10-22 и 27, полинуклеотид по п.23, вектор по п.24 или клетку-хозяин по п.25.
29. Фармацевтическая композиция по п.9 или 28 для применения в профилактике или лечении рака,
предпочтительно, где рак представляет собой один или несколько видов рака, выбранных из группы, состоящей из рака почки, меланомы, плоскоклеточного рака, базальноклеточного рака, рака конъюнктивы, рака полости рта, рака гортани, рака глотки, рака щитовидной железы, рака легкого, рака молочной железы, рака пищевода, рака желудка, рака двенадцатиперстной кишки, рака тонкой кишки, рака толстой кишки, рака прямой кишки, рака аппендикса, рака анального канала, рака печени, рака желчного пузыря, рака желчных протоков, рака поджелудочной железы, рака надпочечников, рака мочевого пузыря, рака предстательной железы, рака матки, рака влагалища, липосаркомы, ангиосаркомы, хондросаркомы, рабдомиосаркомы, саркомы Юинга, остеосаркомы, недифференцированной плеоморфной саркомы, миксофибросаркомы, злокачественной опухоли оболочек периферических нервов, забрюшинной саркомы, синовиальной саркомы, саркомы матки, стромальной опухоли желудочно-кишечного тракта, лейомиосаркомы, эпителиоидной саркомы, В-клеточной лимфомы, Т/NK-клеточной лимфомы, лимфомы Ходжкина, миелогенного лейкоза, лимфатического лейкоза, миелопролиферативного заболевания, миелодиспластического синдрома, множественной миеломы, карциномы яичек и рака яичников;
более предпочтительно, где рак легкого представляет собой немелкоклеточный рак легкого и мелкоклеточный рак легкого;
еще более предпочтительно, где немелкоклеточный рак легких представляет собой аденокарциному, эпидермоидный рак или крупноклеточный рак;
и, еще более предпочтительно, где композиция используется в комбинации с другим средством.
30. Биспецифическое антитело по любому из пп.10-22 и 27 для применения в профилактике или лечении рака,
предпочтительно, где рак представляет собой один или несколько видов рака, выбранных из группы, состоящей из рака почки, меланомы, плоскоклеточного рака, базальноклеточного рака, рака конъюнктивы, рака полости рта, рака гортани, рака глотки, рака щитовидной железы, рака легкого, рака молочной железы, рака пищевода, рака желудка, рака двенадцатиперстной кишки, рака тонкой кишки, рака толстой кишки, рака прямой кишки, рака аппендикса, рака анального канала, рака печени, рака желчного пузыря, рака желчных протоков, рака поджелудочной железы, рака надпочечников, рака мочевого пузыря, рака предстательной железы, рака матки, рака влагалища, липосаркомы, ангиосаркомы, хондросаркомы, рабдомиосаркомы, саркомы Юинга, остеосаркомы, недифференцированной плеоморфной саркомы, миксофибросаркомы, злокачественной опухоли оболочек периферических нервов, забрюшинной саркомы, синовиальной саркомы, саркомы матки, стромальной опухоли желудочно-кишечного тракта, лейомиосаркомы, эпителиоидной саркомы, В-клеточной лимфомы, Т/NK-клеточной лимфомы, лимфомы Ходжкина, миелогенного лейкоза, лимфатического лейкоза, миелопролиферативного заболевания, миелодиспластического синдрома, множественной миеломы, карциномы яичек и рака яичников.
более предпочтительно, где рак легкого представляет собой немелкоклеточный рак легкого и мелкоклеточный рак легкого;
еще более предпочтительно, где немелкоклеточный рак легких представляет собой аденокарциному, эпидермоидный рак или крупноклеточный рак;
и, еще более предпочтительно, где биспецифическое антитело используется в комбинации с другим средством.
| MASAKI MARUTA, MD, Development of T-Cell Therapy By Exploiting Modified Antibodies Specific for A2/NY-ESO-1 for Refractory Myeloma, Blood blood, 2017, 130 (Suppl_1):1913 | |||
| WO 2016210365 A2, 29.12.2016 | |||
| WO 2010106431 A2, 23.09.2010 | |||
| GUILLAUME STEWART-JONES et al., Rational development of high-affinity T-cell receptor-like antibodies, Proc Natl Acad |
Авторы
Даты
2025-05-22—Публикация
2021-03-29—Подача