ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
Настоящая заявка испрашивает приоритет на основании заявки на патент США с регистрационным номером 62/940509, поданной 26 ноября 2019 года, содержание которой включено в данный документ посредством ссылки во всей своей полноте.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Настоящая заявка содержит перечень последовательностей, который был подан в электронном виде в формате ASCII и настоящим включен посредством ссылки во всей своей полноте. Указанная копия в формате ASCII, созданная 24 ноября 2020 года, имеет название N2067-7166WO_SL.txt, и ее размер составляет 598194 байта.
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится в общем к иммунным эффекторным клеткам (например, T-клеткам или NK-клеткам), сконструированным таким образом, что они экспрессируют химерный антигенный рецептор (CAR), а также к композициям на их основе и путям их применения.
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
Терапия на основе адоптивного переноса клеток (ACT) с использованием Т-клеток, в особенности T-клеток, трансдуцированных с помощью химерных антигенных рецепторов (CAR), показала перспективность в нескольких исследованиях в отношении гематологического рака. Существует потребность в способах и процессах для улучшения получения терапевтического продукта на основе клеток, экспрессирующих CAR, повышения качества продукта и доведения до максимума терапевтической эффективности продукта.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
В одном аспекте в настоящем изобретении описана клетка, например, иммунная клетка, например, T-клетка или NK-клетка, содержащая первый антигенсвязывающий домен и второй антигенсвязывающий домен. В некоторых вариантах осуществления первый антигенсвязывающий домен представляет собой домен, связывающий BCMA. В некоторых вариантах осуществления домен, связывающий BCMA, содержит последовательность, связывающую BCMA, раскрытую в данном документе, например, последовательность CDR, VH, VL или scFv, раскрытую в таблицах 3-15, 19, 20, 22, 26 и 31. В некоторых вариантах осуществления второй антигенсвязывающий домен представляет собой домен, связывающий CD19. В некоторых вариантах осуществления домен, связывающий CD19, содержит последовательность, связывающую CD19, раскрытую в данном документе, например, последовательность CDR, VH, VL или scFv, раскрытую в таблицах 2, 19, 22 и 31.
В некоторых вариантах осуществления в настоящем изобретении предусмотрена клетка, содержащая (a) первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит вариабельную область тяжелой цепи (VH), содержащую определяющую комплементарность область 1 тяжелой цепи (CDR1 HC), определяющую комплементарность область 2 тяжелой цепи (CDR2 HC) и определяющую комплементарность область 3 тяжелой цепи (CDR3 HC), и вариабельную область легкой цепи (VL), содержащую определяющую комплементарность область 1 легкой цепи (CDR1 LC), определяющую комплементарность область 2 легкой цепи (CDR2 LC) и определяющую комплементарность область 3 легкой цепи (CDR3 LC), где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под (i) SEQ ID NO: 86, 130, 88, 95, 131 и 132 соответственно; (ii) SEQ ID NO: 44, 45, 84, 54, 55 и 56 соответственно или (iii) SEQ ID NO: 179, 180, 181, 147, 182 и 183 соответственно; и (b) второй антигенсвязывающий домен. В некоторых вариантах осуществления первый антигенсвязывающий домен и второй антигенсвязывающий домен расположены в двух химерных антигенных рецепторах (CAR). В некоторых вариантах осуществления первый антигенсвязывающий домен и второй антигенсвязывающий домен расположены в одном CAR.
В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 86, 130, 88, 95, 131 и 132 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 86, 109, 88, 95, 114 и 115 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 86, 109, 88, 95, 114 и 97 соответственно. В некоторых вариантах осуществления VH содержит аминокислотную последовательность под SEQ ID NO: 93 или 112 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VH кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 260, 94 или 113 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VL содержит аминокислотную последовательность под SEQ ID NO: 102, 118 или 124 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VL кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 261, 103, 119 или 125 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 93 и 102 соответственно. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 112 и 118 соответственно. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 112 и 124 соответственно. В некоторых вариантах осуществления первый антигенсвязывающий домен содержит одноцепочечный вариабельный фрагмент (scFv), содержащий аминокислотную последовательность под SEQ ID NO: 105, 120 или 126 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый антигенсвязывающий домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 253, 106, 121 или 127 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый антигенсвязывающий домен расположен в первом CAR. В некоторых вариантах осуществления первый CAR содержит аминокислотную последовательность под SEQ ID NO: 107, 226, 122 или 128 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 259, 258, 108, 123 или 129 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 84, 54, 55 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 46, 54, 55 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 68, 54, 55 и 56 соответственно. В некоторых вариантах осуществления VH содержит аминокислотную последовательность под SEQ ID NO: 78, 52 или 70 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VH кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 79, 53 или 71 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VL содержит аминокислотную последовательность под SEQ ID NO: 61 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VL кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 62 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 78 и 61 соответственно. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 52 и 61 соответственно. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 70 и 61 соответственно. В некоторых вариантах осуществления первый антигенсвязывающий домен содержит одноцепочечный вариабельный фрагмент (scFv), содержащий аминокислотную последовательность под SEQ ID NO: 80, 64 или 72 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый антигенсвязывающий домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 81, 65 или 73 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый антигенсвязывающий домен расположен в первом CAR. В некоторых вариантах осуществления первый CAR содержит аминокислотную последовательность под SEQ ID NO: 224, 82, 66 или 74 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 83, 67 или 75 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 179, 180, 181, 147, 182 и 183 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 137, 138, 139, 147, 148 и 149 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 160, 161, 162, 147, 170 и 171 соответственно. В некоторых вариантах осуществления VH содержит аминокислотную последовательность под SEQ ID NO: 145 или 168 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VH кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 146 или 169 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VL содержит аминокислотную последовательность под SEQ ID NO: 154 или 173 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VL кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 155 или 174 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 145 и 154 соответственно. В некоторых вариантах осуществления VH и VL содержат аминокислотные последовательности под SEQ ID NO: 168 и 173 соответственно. В некоторых вариантах осуществления первый антигенсвязывающий домен содержит одноцепочечный вариабельный фрагмент (scFv), содержащий аминокислотную последовательность под SEQ ID NO: 156 или 175 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый антигенсвязывающий домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 157 или 176 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый антигенсвязывающий домен расположен в первом CAR. В некоторых вариантах осуществления первый CAR содержит аминокислотную последовательность под SEQ ID NO: 158 или 177 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 159 или 178 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления в данном документе предусмотрена клетка, содержащая (a) первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит (i) VH, содержащую CDR1 HC, CDR2 HC и CDR3 HC последовательности, связывающей BCMA, представленные в таблице 20 или 26, и VL, содержащую CDR1 LC, CDR2 LC и CDR3 LC последовательности, связывающей BCMA, представленные в таблице 20 или 26, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243; (ii) VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 239 и 242 соответственно, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243; или (iii) scFv, содержащий аминокислотную последовательность под SEQ ID NO: 200; и (b) второй антигенсвязывающий домен. В некоторых вариантах осуществления первый антигенсвязывающий домен и второй антигенсвязывающий домен расположены в двух химерных антигенных рецепторах (CAR). В некоторых вариантах осуществления первый антигенсвязывающий домен и второй антигенсвязывающий домен расположены в одном CAR. В некоторых вариантах осуществления второй антигенсвязывающий домен связывается с антигеном, выбранным из CD19, CD5, CD10, CD20, CD21, CD22, CD23, CD24, CD25, CD27, CD30, CD34, CD37, CD38, CD40, CD53, CD69, CD72, CD73, CD74, CD75, CD77, CD79a, CD79b, CD80, CD81, CD82, CD83, CD84, CD85, CD86, CD123, CD135, CD138, CD179, CD269, Flt3, ROR1, FcRn5, FcRn2, CS-1, CXCR4, 5, 7, IL-7/3R, IL7/4/3R или IL4R, где В-клеточный антиген необязательно выбран из CD19, CD20, CD22, FcRn5, FcRn2, CS-1, CD138, CD123, CD33, CD34, CLL-1, рецептора фолиевой кислоты бета или FLT3. В некоторых вариантах осуществления второй антигенсвязывающий домен связывается с CD19. В некоторых вариантах осуществления второй антигенсвязывающий домен связывается с антигеном, выбранным из EGFRvIII, мезотелина, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC.
В некоторых вариантах осуществления второй антигенсвязывающий домен связывается с CD19. В некоторых вариантах осуществления второй антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и/или CDR3 LC последовательности, связывающей CD19, представленные в таблице 19 или таблице 22, например, CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC, содержащие аминокислотные последовательности под SEQ ID NO: 295 и 245-249 соответственно. В некоторых вариантах осуществления второй антигенсвязывающий домен содержит VH и/или VL последовательности, связывающей CD19, представленные в таблице 19 или таблице 22, например, VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними. В некоторых вариантах осуществления второй антигенсвязывающий домен содержит scFv последовательности, связывающей CD19, представленный в таблице 19 или таблице 22, например, scFv, содержащий аминокислотную последовательность под SEQ ID NO: 211 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления второй антигенсвязывающий домен расположен во втором CAR, где CAR включает в себя CAR с последовательностью, связывающей CD19, представленный в таблице 19 или таблице 22, например, CAR, содержащий аминокислотную последовательность под SEQ ID NO: 225 или 229 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления первый антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR LC, содержащие аминокислотные последовательности под (a) SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно; (b) SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно или (c) SEQ ID NO: 44, 45, 46, 54, 55 и 56 соответственно. В некоторых вариантах осуществления второй антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC, содержащие аминокислотные последовательности под SEQ ID NO: 295 и 245-249 соответственно. В некоторых вариантах осуществления первый антигенсвязывающий домен содержит VH и VL, содержащие аминокислотные последовательности под (a) SEQ ID NO: 93 и 102 соответственно; (b) SEQ ID NO: 78 и 61 соответственно или (c) SEQ ID NO: 52 и 61 соответственно. В некоторых вариантах осуществления второй антигенсвязывающий домен содержит VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно. В некоторых вариантах осуществления первый антигенсвязывающий домен содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 105, 80 или 64. В некоторых вариантах осуществления второй антигенсвязывающий домен содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 211. В некоторых вариантах осуществления первый антигенсвязывающий домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 253, 106, 81 или 65. В некоторых вариантах осуществления второй антигенсвязывающий домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 212.
В некоторых вариантах осуществления первый антигенсвязывающий домен расположен в первом CAR, а второй антигенсвязывающий домен расположен во втором CAR. В некоторых вариантах осуществления первый CAR дополнительно содержит первый трансмембранный домен и первый внутриклеточный сигнальный домен. В некоторых вариантах осуществления второй CAR дополнительно содержит второй трансмембранный домен и второй внутриклеточный сигнальный домен.
В некоторых вариантах осуществления первый CAR кодируется первой последовательностью нуклеиновой кислоты, а второй CAR кодируется второй последовательностью нуклеиновой кислоты, где первая и вторая последовательности нуклеиновой кислоты расположены в отдельных молекулах нуклеиновой кислоты.
В некоторых вариантах осуществления первый CAR кодируется первой последовательностью нуклеиновой кислоты, а второй CAR кодируется второй последовательностью нуклеиновой кислоты, где первая и вторая последовательности нуклеиновой кислоты расположены в одной молекуле нуклеиновой кислоты. В некоторых вариантах осуществления отдельная молекула нуклеиновой кислоты имеет следующую конфигурацию в ориентации 5'-3': последовательность нуклеиновой кислоты, кодирующая первый антигенсвязывающий домен - последовательность нуклеиновой кислоты, кодирующая первый трансмембранный домен - последовательность нуклеиновой кислоты, кодирующая первый внутриклеточный сигнальный домен - последовательность нуклеиновой кислоты, кодирующая линкер - последовательность нуклеиновой кислоты, кодирующая второй антигенсвязывающий домен - последовательность нуклеиновой кислоты, кодирующая второй трансмембранный домен - последовательность нуклеиновой кислоты, кодирующая второй внутриклеточный сигнальный домен. В некоторых вариантах осуществления отдельная молекула нуклеиновой кислоты имеет следующую конфигурацию в ориентации 5'-3': последовательность нуклеиновой кислоты, кодирующая второй антигенсвязывающий домен - последовательность нуклеиновой кислоты, кодирующая второй трансмембранный домен - последовательность нуклеиновой кислоты, кодирующая второй внутриклеточный сигнальный домен - последовательность нуклеиновой кислоты, кодирующая линкер - последовательность нуклеиновой кислоты, кодирующая первый антигенсвязывающий домен - последовательность нуклеиновой кислоты, кодирующая первый трансмембранный домен - последовательность нуклеиновой кислоты, кодирующая первый внутриклеточный сигнальный домен. В некоторых вариантах осуществления линкер содержит сайт саморасщепления. В некоторых вариантах осуществления линкер содержит сайт P2A, сайт Т2A, сайт Е2A или сайт F2A. В некоторых вариантах осуществления линкер содержит сайт P2A. В некоторых вариантах осуществления линкер кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 209 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления линкер содержит аминокислотную последовательность под SEQ ID NO: 208 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления отдельная молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты под SEQ ID NO: 215, 217, 219, 221 или 223 или последовательность нуклеиновой кислоты, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления отдельная молекула нуклеиновой кислоты кодирует аминокислотную последовательность под SEQ ID NO: 214, 216, 218, 220 или 222 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления первый антигенсвязывающий домен и второй антигенсвязывающий домен расположены в одном CAR, где CAR дополнительно содержит трансмембранный домен и внутриклеточный сигнальный домен. В некоторых вариантах осуществления первый антигенсвязывающий домен содержит первую VH (VH1) и первую VL (VL1), а второй антигенсвязывающий домен содержит вторую VH (VH2) и вторую VL (VL2). В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH2 - необязательно линкер 1 ("L1") - VL1 - необязательно линкер 2 ("L2") - VH1 - необязательно линкер 3 ("L3") - VL2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VL1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1. В некоторых вариантах осуществления VH1 и VL1 содержат аминокислотные последовательности под SEQ ID NO: 93 и 102 соответственно (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления VH1 и VL1 содержат аминокислотные последовательности под SEQ ID NO: 333 и 334 соответственно (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления VH1 и VL1 содержат аминокислотные последовательности под SEQ ID NO: 78 и 61 соответственно (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления VH1 и VL1 содержат аминокислотные последовательности под SEQ ID NO: 335 и 336 соответственно (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления VH2 и VL2 содержат аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления VH2 и VL2 содержат аминокислотные последовательности под SEQ ID NO: 331 и 332 соответственно (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления L1 или L3 содержит аминокислотную последовательность под SEQ ID NO: 5 (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления L2 содержит аминокислотную последовательность под SEQ ID NO: 63 (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления CAR содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 321-330, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней. В некоторых вариантах осуществления CAR содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 339-348, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней.
В некоторых вариантах осуществления CAR кодируется молекулой нуклеиновой кислоты, имеющей следующую конфигурацию в ориентации 5'-3': последовательность нуклеиновой кислоты, кодирующая первый антигенсвязывающий домен - необязательно последовательность нуклеиновой кислоты, кодирующая линкер - последовательность нуклеиновой кислоты, кодирующая второй антигенсвязывающий домен - последовательность нуклеиновой кислоты, кодирующая трансмембранный домен - последовательность нуклеиновой кислоты, кодирующая внутриклеточный сигнальный домен. В некоторых вариантах осуществления CAR кодируется молекулой нуклеиновой кислоты, имеющей следующую конфигурацию в ориентации 5'-3': последовательность нуклеиновой кислоты, кодирующая второй антигенсвязывающий домен - необязательно последовательность нуклеиновой кислоты, кодирующая линкер - последовательность нуклеиновой кислоты, кодирующая первый антигенсвязывающий домен - последовательность нуклеиновой кислоты, кодирующая трансмембранный домен - последовательность нуклеиновой кислоты, кодирующая внутриклеточный сигнальный домен.
В некоторых вариантах осуществления CAR имеет следующую конфигурацию в ориентации от N- до C-конца: первый антигенсвязывающий домен - необязательно линкер - второй антигенсвязывающий домен - трансмембранный домен - внутриклеточный сигнальный домен. В некоторых вариантах осуществления CAR имеет следующую конфигурацию в ориентации от N- до C-конца: второй антигенсвязывающий домен - необязательно линкер - первый антигенсвязывающий домен - трансмембранный домен - внутриклеточный сигнальный домен.
В некоторых вариантах осуществления первый антигенсвязывающий домен или второй антигенсвязывающий домен содержит VH и VL. В некоторых вариантах осуществления VH и VL соединены линкером. В некоторых вариантах осуществления линкер содержит аминокислотную последовательность под SEQ ID NO: 5, 63, 104 или 243 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления трансмембранный домен, первый трансмембранный домен или второй трансмембранный домен включает в себя трансмембранный домен белка, выбранного из альфа-, бета- или дзета-цепи T-клеточного рецептора, CD28, CD3-эпсилон, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137 или CD154. В некоторых вариантах осуществления трансмембранный домен, первый трансмембранный домен или второй трансмембранный домен содержит аминокислотную последовательность под SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления трансмембранный домен, первый трансмембранный домен или второй трансмембранный домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 17 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления первый антигенсвязывающий домен или второй антигенсвязывающий домен соединены с трансмембранным доменом, первым трансмембранным доменом или вторым трансмембранным доменом с помощью шарнирной области (например, первой или второй шарнирной области). В некоторых вариантах осуществления шарнирная область содержит аминокислотную последовательность под SEQ ID NO: 2, 3 или 4 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления шарнирная область кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 13, 14 или 15 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления шарнирная область и трансмембранный домен содержат аминокислотную последовательность под SEQ ID NO: 202 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления шарнирная область и трансмембранный домен кодируются последовательностью нуклеиновой кислоты под SEQ ID NO: 203 или 213 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления внутриклеточный сигнальный домен, первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит первичный сигнальный домен (например, первый или второй первичный сигнальный домен). В некоторых вариантах осуществления первичный сигнальный домен предусматривает функциональный сигнальный домен, полученный из CD3-дзета, TCR-дзета, FcR-гамма, FcR-бета, CD3-гамма, CD3-дельта, CD3-эпсилон, CD5, CD22, CD79a, CD79b, CD278 (ICOS), FcεRI, DAP10, DAP12 или CD66d. В некоторых вариантах осуществления первичный сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 9 или 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первичный сигнальный домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 20, 21 или 205 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления внутриклеточный сигнальный домен, первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит костимулирующий сигнальный домен (например, первый или второй костимулирующий сигнальный домен). В некоторых вариантах осуществления костимулирующий сигнальный домен предусматривает функциональный сигнальный домен, полученный из молекулы MHC I класса, белкового рецептора TNF, иммуноглобулиноподобного белка, рецептора цитокина, интегрина, сигнальной молекулы активации лимфоцитов (белка SLAM), активирующего рецептора NK-клеток, BTLA, лиганда Toll-подобного рецептора, OX40, CD2, CD7, CD27, CD28, CD30, CD40, CDS, ICAM-1, 4-1BB (CD137), B7-H3, ICOS (CD278), GITR, BAFFR, LIGHT, HVEM (LIGHTR), KIRDS2, SLAMF7, NKp80 (KLRF1), NKp44, NKp30, NKp46, CD19, CD4, CD8-альфа, CD8-бета, IL2R-бета, IL2R-гамма, IL7R-альфа, ITGA4, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, ITGB7, NKG2D, NKG2C, TNFR2, TRANCE/RANKL, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (Tactile), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), CD69, SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR, LAT, GADS, SLP-76, PAG/Cbp, CD19a, CD28-OX40, CD28-4-1BB или лиганда, который специфично связывается с CD83. В некоторых вариантах осуществления костимулирующий сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления костимулирующий сигнальный домен кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 18 или 204 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления внутриклеточный сигнальный домен, первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит функциональный сигнальный домен, полученный из 4-1BB, и функциональный сигнальный домен, полученный из CD3-дзета. В некоторых вариантах осуществления внутриклеточный сигнальный домен, первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 7 (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней) и аминокислотную последовательность под SEQ ID NO: 9 или 10 (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления внутриклеточный сигнальный домен, первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 7 и аминокислотную последовательность под SEQ ID NO: 9 или 10.
В некоторых вариантах осуществления CAR, первый CAR или второй CAR дополнительно содержат лидерную последовательность (например, первую или вторую лидерную последовательность). В некоторых вариантах осуществления лидерная последовательность предусматривает аминокислотную последовательность под SEQ ID NO: 1 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления лидерная последовательность кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 199 или 210 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%). В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%). В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%). В некоторых вариантах осуществления первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%). В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%). В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%). В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность предусматривают одинаковые аминокислотные последовательности (например, первая лидерная последовательность и вторая лидерная последовательность предусматривают аминокислотную последовательность под SEQ ID NO: 1 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность предусматривают разные аминокислотные последовательности. В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область содержат одинаковые аминокислотные последовательности (например, первая шарнирная область и вторая шарнирная область содержат аминокислотную последовательность под SEQ ID NO: 2 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область содержат разные аминокислотные последовательности. В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен содержат одинаковые аминокислотные последовательности (например, первый трансмембранный домен и второй трансмембранный домен содержат аминокислотную последовательность под SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен содержат разные аминокислотные последовательности. В некоторых вариантах осуществления первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен содержат одинаковые аминокислотные последовательности. В некоторых вариантах осуществления первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен содержат разные аминокислотные последовательности. В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен содержат одинаковые аминокислотные последовательности (например, первый первичный сигнальный домен и второй первичный сигнальный домен содержат аминокислотную последовательность под SEQ ID NO: 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен содержат разные аминокислотные последовательности. В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат одинаковые аминокислотные последовательности (например, первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат аминокислотную последовательность под SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат разные аминокислотные последовательности (например, первый и второй костимулирующие сигнальные домены содержат последовательность костимулирующего домена 4-1BB и последовательность костимулирующего домена CD28 соответственно или содержат последовательность костимулирующего домена CD28 и последовательность костимулирующего домена 4-1BB соответственно). В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 199 и 210 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 210 и 199 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 337 и 13 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 13 и 337 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 338 и 17 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 17 и 338 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 204 и 18 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются последовательностями нуклеиновой кислоты под SEQ ID NO: 18 и 204 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 205 и 21 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними). В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 21 и 205 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними).
В некоторых вариантах осуществления CAR, первый CAR или второй CAR кодируется молекулой нуклеиновой кислоты, содержащей посттранскрипционный регуляторный элемент вируса гепатита лесных сурков (WPRE).
В некоторых вариантах осуществления в данном документе предусмотрена молекула нуклеиновой кислоты, содержащая (a) первую последовательность нуклеиновой кислоты, кодирующую первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит вариабельную область тяжелой цепи (VH), содержащую определяющую комплементарность область 1 тяжелой цепи (CDR1 HC), определяющую комплементарность область 2 тяжелой цепи (CDR2 HC) и определяющую комплементарность область 3 тяжелой цепи (CDR3 HC), и вариабельную область легкой цепи (VL), содержащую определяющую комплементарность область 1 легкой цепи (CDR1 LC), определяющую комплементарность область 2 легкой цепи (CDR2 LC) и определяющую комплементарность область 3 легкой цепи (CDR3 LC), где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под (i) SEQ ID NO: 86, 130, 88, 95, 131 и 132 соответственно; (ii) SEQ ID NO: 44, 45, 84, 54, 55 и 56 соответственно или (iii) SEQ ID NO: 179, 180, 181, 147, 182 и 183 соответственно; и (b) вторую последовательность нуклеиновой кислоты, кодирующую второй антигенсвязывающий домен.
В некоторых вариантах осуществления выделенная молекула нуклеиновой кислоты предусматривает первую молекулу нуклеиновой кислоты и вторую молекулу нуклеиновой кислоты, которые являются отдельными молекулами нуклеиновой кислоты, и где первая последовательность нуклеиновой кислоты расположена в первой молекуле нуклеиновой кислоты, а вторая последовательность нуклеиновой кислоты расположена во второй молекуле нуклеиновой кислоты.
В некоторых вариантах осуществления в данном документе предусмотрена молекула нуклеиновой кислоты, содержащая (a) первую последовательность нуклеиновой кислоты, кодирующую первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит (i) VH, содержащую CDR1 HC, CDR2 HC и CDR3 HC последовательности, связывающей BCMA, представленные в таблице 20 или 26, и VL, содержащую CDR1 LC, CDR2 LC и CDR3 LC последовательности, связывающей BCMA, представленные в таблице 20 или 26, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243; (ii) VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 239 и 242 соответственно, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243; или (iii) scFv, содержащий аминокислотную последовательность под SEQ ID NO: 200; и (b) вторую последовательность нуклеиновой кислоты, кодирующую второй антигенсвязывающий домен.
В некоторых вариантах осуществления в данном документе предусмотрена молекула нуклеиновой кислоты, содержащая первую последовательность нуклеиновой кислоты, кодирующую первый CAR, и вторую последовательность нуклеиновой кислоты, кодирующую второй CAR, где первый CAR содержит первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, первый трансмембранный домен и первый внутриклеточный сигнальный домен, и где второй CAR содержит второй антигенсвязывающий домен, который представляет собой домен, связывающий CD19, второй трансмембранный домен и второй внутриклеточный сигнальный домен, где (i) первый антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR LC, содержащие аминокислотные последовательности под SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно, и второй антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR LC, содержащие аминокислотные последовательности под SEQ ID NO: 295 и 245-249 соответственно; (ii) первый антигенсвязывающий домен содержит VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 93 и 102 соответственно, и второй антигенсвязывающий домен содержит VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно; (iii) первый антигенсвязывающий домен содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 105, и второй антигенсвязывающий домен содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 211; (iv) первый CAR содержит аминокислотную последовательность под SEQ ID NO: 107 или 226, и второй CAR содержит аминокислотную последовательность под SEQ ID NO: 225 или 229; или (v) выделенная молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты под SEQ ID NO: 271.
В некоторых вариантах осуществления в данном документе предусмотрена молекула нуклеиновой кислоты, содержащая первую последовательность нуклеиновой кислоты, кодирующую первый CAR, и вторую последовательность нуклеиновой кислоты, кодирующую второй CAR, где первый CAR содержит первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, первый трансмембранный домен и первый внутриклеточный сигнальный домен, и где второй CAR содержит второй антигенсвязывающий домен, который представляет собой домен, связывающий CD19, второй трансмембранный домен и второй внутриклеточный сигнальный домен, где (i) первый антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR LC, содержащие аминокислотные последовательности под SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно, второй антигенсвязывающий домен содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC, содержащие аминокислотные последовательности под SEQ ID NO: 295 и 245-249 соответственно; (ii) первый антигенсвязывающий домен содержит VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 78 и 61 соответственно, и второй антигенсвязывающий домен содержит VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно; (iii) первый антигенсвязывающий домен содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 80, и второй антигенсвязывающий домен содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 211; (iv) первый CAR содержит аминокислотную последовательность под SEQ ID NO: 82 или 224, и второй CAR содержит аминокислотную последовательность под SEQ ID NO: 225 или 229; или (v) выделенная молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты под SEQ ID NO: 215.
В некоторых вариантах осуществления в данном документе предусмотрена молекула полипептида, кодируемая молекулой нуклеиновой кислоты, раскрытой в данном документе.
В некоторых вариантах осуществления в данном документе предусмотрен CAR, где CAR содержит (a) первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит вариабельную область тяжелой цепи (VH), содержащую определяющую комплементарность область 1 тяжелой цепи (CDR1 HC), определяющую комплементарность область 2 тяжелой цепи (CDR2 HC) и определяющую комплементарность область 3 тяжелой цепи (CDR3 HC), и вариабельную область легкой цепи (VL), содержащую определяющую комплементарность область 1 легкой цепи (CDR1 LC), определяющую комплементарность область 2 легкой цепи (CDR2 LC) и определяющую комплементарность область 3 легкой цепи (CDR3 LC), где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под (i) SEQ ID NO: 86, 130, 88, 95, 131 и 132 соответственно; (ii) SEQ ID NO: 44, 45, 84, 54, 55 и 56 соответственно или (iii) SEQ ID NO: 179, 180, 181, 147, 182 и 183 соответственно; и (b) второй антигенсвязывающий домен.
В некоторых вариантах осуществления в данном документе предусмотрен CAR, где CAR содержит (a) первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит (i) VH, содержащую CDR1 HC, CDR2 HC и CDR3 HC последовательности, связывающей BCMA, представленные в таблице 20 или 26, и VL, содержащую CDR1 LC, CDR2 LC и CDR3 LC последовательности, связывающей BCMA, представленные в таблице 20 или 26, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243; (ii) VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 239 и 242 соответственно, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243; или (iii) scFv, содержащий аминокислотную последовательность под SEQ ID NO: 200; и (b) второй антигенсвязывающий домен.
В некоторых вариантах осуществления в данном документе предусмотрен вектор, содержащий молекулу нуклеиновой кислоты, раскрытую в данном документе, или молекулу нуклеиновой кислоты, кодирующую CAR, раскрытый в данном документе. В некоторых вариантах осуществления вектор выбран из ДНК-вектора, РНК-вектора, плазмиды, лентивирусного вектора, аденовирусного вектора или ретровирусного вектора. В некоторых вариантах осуществления вектор содержит промотор EF-1, содержащий последовательность нуклеиновой кислоты под SEQ ID NO: 11.
В некоторых вариантах осуществления в данном документе предусмотрена клетка, содержащая молекулу нуклеиновой кислоты, раскрытую в данном документе, молекулу нуклеиновой кислоты, кодирующую CAR, раскрытый в данном документе, полипептид, раскрытый в данном документе, CAR, раскрытый в данном документе, или вектор, раскрытый в данном документе. В некоторых вариантах осуществления клетка представляет собой T-клетку или NK-клетку.
В некоторых вариантах осуществления в данном документе раскрыт способ получения клетки, включающий трансдукцию клетки вектором, раскрытым в данном документе, где клетка необязательно представляет собой T-клетку или NK-клетку. В некоторых вариантах осуществления в данном документе раскрыт способ получения РНК-сконструированной клетки, включающий введение РНК, транскрибированной in vitro, или синтетической РНК в клетку, где РНК содержит молекулу нуклеиновой кислоты, раскрытую в данном документе, молекулу нуклеиновой кислоты, кодирующую CAR, раскрытый в данном документе. В некоторых вариантах осуществления клетка представляет собой T-клетку или NK-клетку.
В некоторых вариантах осуществления в данном документе раскрыт способ получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR), при этом способ включает (i) приведение популяции клеток (например, T-клеток, например, T-клеток, выделенных из замороженного или свежего продукта лейкафереза) в контакт (например, связывание) со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток; (ii) приведение популяции клеток (например, T-клеток) в контакт с молекулой нуклеиновой кислоты, раскрытой в данном документе, или молекулой нуклеиновой кислоты, кодирующей CAR, раскрытый в данном документе, за счет чего обеспечивается получение популяции клеток (например, T-клеток), содержащих молекулу нуклеиновой кислоты, и (iii) сбор популяции клеток (например, T-клеток) для хранения (например, повторного составления популяции клеток в среде для криоконсервации) или введения, где:
(a) стадию (ii) проводят вместе со стадией (i) или не позже чем через 20 часов после начала стадии (i), например, не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i), например, не позже чем через 18 часов после начала стадии (i), и
стадию (iii) проводят не позже чем через 30 (например, 26) часов после начала стадии (i), например, не позже чем через 22, 23, 24, 25, 26, 27, 28, 29 или 30 часов после начала стадии (i), например, не позже чем через 24 часа после начала стадии (i),
(b) стадию (ii) проводят вместе со стадией (i) или не позже чем через 20 часов после начала стадии (i), например, не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i), например, не позже чем через 18 часов после начала стадии (i), и
стадию (iii) проводят не позже чем через 30 часов после начала стадии (ii), например, не позже чем через 22, 23, 24, 25, 26, 27, 28, 29 или 30 часов после начала стадии (ii), или
(c) популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i),
где молекула нуклеиновой кислоты на стадии (ii) необязательно находится в вирусном векторе, где молекула нуклеиновой кислоты на стадии (ii) необязательно представляет собой молекулу РНК в вирусном векторе, где стадия (ii) необязательно включает трансдукцию популяции клеток (например, T-клеток) с помощью вирусного вектора, содержащего молекулу нуклеиновой кислоты, кодирующую CAR.
В некоторых вариантах осуществления в данном документе раскрыт способ получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR), при этом способ включает (1) приведение популяции клеток (например, T-клеток, например, T-клеток, выделенных из замороженного продукта лейкафереза) в контакт с цитокином, выбранным из IL-2, IL-7, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-21, IL-6 (например, IL-6/sIL-6Ra) или их комбинации, (2) приведение популяции клеток (например, T-клеток) в контакт с молекулой нуклеиновой кислоты, раскрытой в данном документе, или молекулой нуклеиновой кислоты, кодирующей CAR, раскрытый в данном документе, за счет чего обеспечивается получение популяции клеток (например, T-клеток), содержащих молекулу нуклеиновой кислоты, и (3) сбор популяции клеток (например, T-клеток) для хранения (например, повторного составления популяции клеток в среде для криоконсервации) или введения, где:
(a) стадию (2) проводят вместе со стадией (1) или не позже чем через 5 часов после начала стадии (1), например, не позже чем через 1, 2, 3, 4 или 5 часов после начала стадии (1), и
стадию (3) проводят не позже чем через 26 часов после начала стадии (1), например, не позже чем через 22, 23 или 24 часа после начала стадии (1), например, не позже чем через 24 часа после начала стадии (1), или
(b) популяция клеток из стадии (3) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1),
где молекула нуклеиновой кислоты на стадии (2) необязательно находится в вирусном векторе, где молекула нуклеиновой кислоты на стадии (ii) необязательно представляет собой молекулу РНК в вирусном векторе, где стадия (ii) необязательно включает трансдукцию популяции клеток (например, T-клеток) с помощью вирусного вектора, содержащего молекулу нуклеиновой кислоты, кодирующую CAR.
В некоторых вариантах осуществления в данном документе раскрыта популяция клеток, сконструированных таким образом, что они экспрессируют CAR ("популяция клеток, экспрессирующих CAR"), при этом указанная популяция предусматривает: (a) приблизительно такую же процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (b) изменение количества необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, находящееся в пределах от приблизительно 5% до приблизительно 10%, например, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (c) увеличенную процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, например, увеличенную в по меньшей мере 1,2, 1,4, 1,6, 1,8, 2,0, 2,2, 2,4, 2,6, 2,8 или 3 раза, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (d) приблизительно такую же процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (e) изменение количества центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, находящееся в пределах от приблизительно 5% до приблизительно 10%, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (f) уменьшенную процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, например, уменьшенную на по меньшей мере 20, 25, 30, 35, 40, 45 или 50%, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (g) приблизительно такую же процентную долю стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (h) изменение количества стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, находящееся в пределах от приблизительно 5% до приблизительно 10%, по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; или (i) увеличенную процентную долю стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR. В некоторых вариантах осуществления популяция содержит клетку, раскрытую в данном документе. В некоторых вариантах осуществления популяция содержит клетку, содержащую двойной CAR или CAR в формате диатела, раскрытые в данном документе. В некоторых вариантах осуществления популяция содержит клетку, содержащую (a) первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA, где домен, связывающий BCMA, содержит вариабельную область тяжелой цепи (VH), содержащую определяющую комплементарность область 1 тяжелой цепи (CDR1 HC), определяющую комплементарность область 2 тяжелой цепи (CDR2 HC) и определяющую комплементарность область 3 тяжелой цепи (CDR3 HC), и вариабельную область легкой цепи (VL), содержащую определяющую комплементарность область 1 легкой цепи (CDR1 LC), определяющую комплементарность область 2 легкой цепи (CDR2 LC) и определяющую комплементарность область 3 легкой цепи (CDR3 LC), где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под (i) SEQ ID NO: 86, 130, 88, 95, 131 и 132 соответственно; (ii) SEQ ID NO: 44, 45, 84, 54, 55 и 56 соответственно или (iii) SEQ ID NO: 179, 180, 181, 147, 182 и 183 соответственно; и (b) второй антигенсвязывающий домен.
В некоторых вариантах осуществления в данном документе раскрыта фармацевтическая композиция, содержащая клетку, раскрытую в данном документе, или популяцию клеток, раскрытую в данном документе, и фармацевтически приемлемый носитель.
В некоторых вариантах осуществления популяцию клеток получают посредством способа, раскрытого в данном документе. В некоторых вариантах осуществления популяция содержит:
(a) первую популяцию клеток, содержащих CAR для BCMA, но не CAR для CD19;
(b) вторую популяцию клеток, содержащих CAR для CD19, но не CAR для BCMA; и
(c) третью популяцию клеток, содержащих как CAR для BCMA, так и CAR для CD19.
В некоторых вариантах осуществления:
(i) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях;
(ii) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях; и/или
(iii) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в популяции.
В некоторых вариантах осуществления популяция дополнительно содержит четвертую популяцию клеток, которые не содержат CAR.
В некоторых вариантах осуществления:
(i) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях;
(ii) общее количество жизнеспособных клеток во второй популяции меньше или равняется от приблизительно 45% до приблизительно 50% (например, приблизительно 47%); от приблизительно 50 до приблизительно 55% (например, приблизительно 53%); от приблизительно 60% до приблизительно 65% (например, приблизительно 63%) или от приблизительно 80 до приблизительно 85% (например, приблизительно 82%) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях.
В некоторых вариантах осуществления в данном документе раскрыт способ обеспечения противоопухолевого иммунитета у субъекта, включающий введение субъекту эффективного количества клетки, раскрытой в данном документе, популяции клеток, раскрытых в данном документе, или фармацевтической композиции, раскрытой в данном документе. В некоторых вариантах осуществления в данном документе раскрыт способ лечения субъекта, у которого имеется заболевание, ассоциированное с экспрессией BCMA, включающий введение субъекту эффективного количества клетки, раскрытой в данном документе, популяции клеток, раскрытой в данном документе, или фармацевтической композиции, раскрытой в данном документе. В некоторых вариантах осуществления заболевание, ассоциированное с экспрессией BCMA, представляет собой (i) рак, или злокачественное новообразование, или предраковое состояние, выбранное из одного или нескольких из миелодисплазии, миелодиспластического синдрома или предлейкоза, или (ii) не связанное с раком показание, ассоциированное с экспрессией BCMA. В некоторых вариантах осуществления заболевание представляет собой гематологический рак или солидный рак. В некоторых вариантах осуществления заболевание выбрано из острого лейкоза, B-клеточного острого лимфоидного лейкоза ("BALL"), T-клеточного острого лимфоидного лейкоза ("TALL"), острого лимфоидного лейкоза (ALL), хронического миелогенного лейкоза (CML), хронического лимфоцитарного лейкоза (CLL), B-клеточного пролимфоцитарного лейкоза, новообразования из бластных плазмоцитоидных дендритных клеток, лимфомы Беркитта, диффузной крупноклеточной B-клеточной лимфомы, фолликулярной лимфомы, волосатоклеточного лейкоза, мелкоклеточной или крупноклеточной фолликулярной лимфомы, злокачественных лимфопролиферативных состояний, MALT-лимфомы, мантийноклеточной лимфомы, лимфомы из клеток маргинальной зоны, множественной миеломы, миелодисплазии и миелодиспластического синдрома, неходжкинской лимфомы, плазмобластной лимфомы, новообразования из плазмоцитоидных дендритных клеток, макроглобулинемии Вальденстрема, рака предстательной железы (например, кастрационнорезистентного или резистентного к терапии рака предстательной железы или метастатического рака предстательной железы), рака поджелудочной железы, рака легкого, нарушения пролиферации плазматических клеток (например, бессимптомной миеломы ("тлеющей" множественной миеломы или вялотекущей миеломы), моноклональной гаммапатии неустановленной этиологии (MGUS), макроглобулинемии Вальденстрема, плазмоцитомы (например, плазмоклеточной дискразии, солитарной миеломы, солитарной плазмоцитомы, экстрамедуллярной плазмоцитомы и множественной плазмоцитомы), системного амилоидоза в форме амилоидоза легких цепей или POEMS-синдрома (также известного как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром)) или их комбинации. В некоторых вариантах осуществления заболевание представляет собой множественную миелому.
В некоторых вариантах осуществления популяцию клеток или фармацевтическую композицию вводят субъекту в дозе от приблизительно 1×106 до приблизительно 1×108 (например, от приблизительно 2×106 до приблизительно 5×107, от приблизительно 5×106 до приблизительно 2×107, от приблизительно 1×106 до приблизительно 1×107, от приблизительно 1×107 до приблизительно 1×108, от приблизительно 1×106 до приблизительно 3×106, от приблизительно 2×106 до приблизительно 4×106, от приблизительно 3×106 до приблизительно 5×106, от приблизительно 4×106 до приблизительно 6×106, от приблизительно 5×106 до приблизительно 7×106, от приблизительно 6×106 до приблизительно 8×106, от приблизительно 7×106 до приблизительно 9×106, от приблизительно 8×106 до приблизительно 1×107, от приблизительно 9×106 до приблизительно 2×107, от приблизительно 1×107 до приблизительно 3×107, от приблизительно 2×107 до приблизительно 4×107, от приблизительно 3×107 до приблизительно 5×107, от приблизительно 4×107 до приблизительно 6×107, от приблизительно 5×107 до приблизительно 7×107, от приблизительно 6×107 до приблизительно 8×107, от приблизительно 7×107 до приблизительно 9×107, от приблизительно 8×107 до приблизительно 1×108, приблизительно 1×106, приблизительно 2×106, приблизительно 3×106, приблизительно 4×106, приблизительно 5×106, приблизительно 6×106, приблизительно 7×106, приблизительно 8×106, приблизительно 9×106, приблизительно 1×107, приблизительно 2×107, приблизительно 3×107, приблизительно 4×107, приблизительно 5×107, приблизительно 6×107, приблизительно 7×107, приблизительно 8×107, приблизительно 9×107 или приблизительно 1×108) CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток). В некоторых вариантах осуществления популяцию клеток или фармацевтическую композицию вводят субъекту в дозе от приблизительно 5×106 до приблизительно 2×107 CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток).
В некоторых вариантах осуществления популяцию клеток или фармацевтическую композицию вводят субъекту в одной или нескольких (например, 2, 3, 4 или больше) дозах. В некоторых вариантах осуществления популяцию клеток или фармацевтическую композицию вводят субъекту в двух дозах. В некоторых вариантах осуществления одна или несколько доз включают первую дозу и вторую дозу, где количество CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) в первой дозе больше количества, равняется количеству или меньше количества CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) во второй дозе.
В некоторых вариантах осуществления одна или несколько доз включают первую дозу и вторую дозу, где:
(a) первая доза содержит от приблизительно 1×106 до приблизительно 1×107 (например, от приблизительно 2×106 до приблизительно 8×106, от приблизительно 4×106 до приблизительно 6×106, от приблизительно 1×106 до приблизительно 5×106, от приблизительно 5×106 до приблизительно 1×107, от приблизительно 1×106 до приблизительно 3×106, от приблизительно 2×106 до приблизительно 4×106, от приблизительно 3×106 до приблизительно 5×106, от приблизительно 4×106 до приблизительно 6×106, от приблизительно 5×106 до приблизительно 7×106, от приблизительно 6×106 до приблизительно 8×106, от приблизительно 7×106 до приблизительно 9×106, от приблизительно 8×106 до приблизительно 1×107, приблизительно 1×106, приблизительно 2×106, приблизительно 3×106, приблизительно 4×106, приблизительно 5×106, приблизительно 6×106, приблизительно 7×106, приблизительно 8×106, приблизительно 9×106 или приблизительно 1×107) жизнеспособных CAR-положительных клеток (например, BCMA-CAR+ Т-клеток);
(b) вторая доза содержит от приблизительно 1×107 до приблизительно 1×108 (например, от приблизительно 2×107 до приблизительно 8×107, от приблизительно 4×107 до приблизительно 6×107, от приблизительно 1×107 до приблизительно 5×107, от приблизительно 5×107 до приблизительно 1×108, от приблизительно 1×107 до приблизительно 3×107, от приблизительно 2×107 до приблизительно 4×107, от приблизительно 3×107 до приблизительно 5×107, от приблизительно 4×107 до приблизительно 6×107, от приблизительно 5×107 до приблизительно 7×107, от приблизительно 6×107 до приблизительно 8×107, от приблизительно 7×107 до приблизительно 9×107, от приблизительно 8×107 до приблизительно 1×108, приблизительно 1×107, приблизительно 2×107, приблизительно 3×107, приблизительно 4×107, приблизительно 5×107, приблизительно 6×107, приблизительно 7×107, приблизительно 8×107, приблизительно 9×107 или приблизительно 1×108) CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток);
(c) количество CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) в первой дозе составляет не более чем 1/X, где X равняется 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90 или 100, от количества CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) во второй дозе; и/или
(d) количество CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) в первой дозе составляет от приблизительно 1% до 100% (например, от приблизительно 10% до приблизительно 90%, от приблизительно 20% до приблизительно 80%, от приблизительно 30% до приблизительно 70%, от приблизительно 40% до приблизительно 60%, от приблизительно 10% до приблизительно 50%, от приблизительно 50% до приблизительно 90%, от приблизительно 10% до приблизительно 30%, от приблизительно 20% до приблизительно 40%, от приблизительно 30% до приблизительно 50%, от приблизительно 50% до приблизительно 70%, от приблизительно 60% до приблизительно 80% или от приблизительно 70% до приблизительно 90%) от количества CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) во второй дозе.
В некоторых вариантах осуществления первая доза содержит приблизительно 5×106 жизнеспособных CAR-положительных клеток (например, BCMA-CAR+ Т-клеток). В некоторых вариантах осуществления вторая доза содержит приблизительно 1×107 или приблизительно 2×107 жизнеспособных CAR-положительных клеток (например, BCMA-CAR+ Т-клеток).
В некоторых вариантах осуществления способ дополнительно включает введение субъекту второго терапевтического средства. В некоторых вариантах осуществления второе терапевтическое средство выбрано из (i) ингибитора PD-1, где ингибитор PD-1 необязательно выбран из группы, состоящей из PDR001, ниволумаба, пембролизумаба, пидилизумаба, MEDI0680, REGN2810, TSR-042, PF-06801591 и AMP-224; (ii) ингибитора PD-L1, где ингибитор PD-L1 необязательно выбран из группы, состоящей из FAZ053, атезолизумаба, авелумаба, дурвалумаба и BMS-936559; (iii) ингибитора LAG-3, где ингибитор LAG-3 необязательно выбран из группы, состоящей из LAG525, BMS-986016, TSR-033, MK-4280 и REGN3767; (iv) ингибитора TIM-3, где ингибитор TIM-3 необязательно выбран из группы, состоящей из MBG453, TSR-022 и LY3321367; (v) ингибитора CTLA-4, где ингибитор CTLA-4 необязательно представляет собой ипилимумаб или тремелимумаб; (vi) полипептида интерлейкина-15 (IL-15), полипептида альфа-субъединицы рецептора интерлейкина-15 (IL-15Ra) или комбинации как полипептида IL-15, так и полипептида IL-15Ra, например, hetIL-15; (vii) полипептида интерлейкина-12 (IL-12); или (viii) ингибитора mTOR, где ингибитор mTOR необязательно представляет собой RAD001 или рапамицин.
В некоторых вариантах осуществления в данном документе предусмотрена клетка, содержащая (a) первый CAR, содержащий первый антигенсвязывающий домен, который связывается с первым антигеном, первый трансмембранный домен и первый внутриклеточный сигнальный домен (например, первый первичный сигнальный домен и/или первый костимулирующий сигнальный домен), где первый CAR необязательно дополнительно содержит первую лидерную последовательность и/или первую шарнирную область; и (b) второй CAR, содержащий второй антигенсвязывающий домен, который связывается со вторым антигеном, второй трансмембранный домен и второй внутриклеточный сигнальный домен (например, второй первичный сигнальный домен и/или второй костимулирующий сигнальный домен), где второй CAR необязательно дополнительно содержит вторую лидерную последовательность и/или вторую шарнирную область, где (i) первая лидерная последовательность и вторая лидерная последовательность кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первая и вторая лидерные последовательности необязательно содержат одинаковые аминокислотные последовательности; (ii) первая шарнирная область и вторая шарнирная область кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первая и вторая шарнирные области необязательно содержат одинаковые аминокислотные последовательности; (iii) первый трансмембранный домен и второй трансмембранный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первый и второй трансмембранные домены необязательно содержат одинаковые аминокислотные последовательности; и/или (iv) первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где необязательно первый первичный сигнальный домен и второй первичный сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%) и/или первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%).
В некоторых вариантах осуществления в данном документе предусмотрена молекула нуклеиновой кислоты, содержащая (a) первую последовательность нуклеиновой кислоты, кодирующую первый CAR, где первый CAR содержит первый антигенсвязывающий домен, который связывается с первым антигеном, первый трансмембранный домен и первый внутриклеточный сигнальный домен (например, первый первичный сигнальный домен и/или первый костимулирующий сигнальный домен), где первый CAR необязательно дополнительно содержит первую лидерную последовательность и/или первую шарнирную область; и (b) вторую последовательность нуклеиновой кислоты, кодирующую второй CAR, где второй CAR содержит второй антигенсвязывающий домен, который связывается со вторым антигеном, второй трансмембранный домен и второй внутриклеточный сигнальный домен (например, второй первичный сигнальный домен и/или второй костимулирующий сигнальный домен), где второй CAR необязательно дополнительно содержит вторую лидерную последовательность и/или вторую шарнирную область, где (i) первая лидерная последовательность и вторая лидерная последовательность кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первая и вторая лидерные последовательности необязательно содержат одинаковые аминокислотные последовательности; (ii) первая шарнирная область и вторая шарнирная область кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первая и вторая шарнирные области необязательно содержат одинаковые аминокислотные последовательности; (iii) первый трансмембранный домен и второй трансмембранный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первый и второй трансмембранные домены необязательно содержат одинаковые аминокислотные последовательности; и/или (iv) первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен кодируются разными последовательностями нуклеиновой кислоты (например, отличающимися на по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 85%, 90%, 95%, или 100%), где первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен необязательно содержат одинаковые аминокислотные последовательности.
В некоторых вариантах осуществления первая и вторая лидерные последовательности содержат одинаковые аминокислотные последовательности. Не ограничиваясь какой-либо теорией, полагают, что такая молекула нуклеиновой кислоты демонстрирует меньшую рекомбинацию, чем в остальном сходная молекула нуклеиновой кислоты, в которой первая лидерная последовательность и вторая лидерная последовательность кодируются одинаковыми последовательностями нуклеиновой кислоты.
В некоторых вариантах осуществления первая и вторая шарнирные области содержат одинаковые аминокислотные последовательности. Не ограничиваясь какой-либо теорией, полагают, что такая молекула нуклеиновой кислоты демонстрирует меньшую рекомбинацию, чем в остальном сходная молекула нуклеиновой кислоты, в которой первая шарнирная область и вторая шарнирная область кодируются одинаковыми последовательностями нуклеиновой кислоты.
В некоторых вариантах осуществления первый и второй трансмембранные домены содержат одинаковые аминокислотные последовательности. Не ограничиваясь какой-либо теорией, полагают, что такая молекула нуклеиновой кислоты демонстрирует меньшую рекомбинацию, чем в остальном сходная молекула нуклеиновой кислоты, в которой первый трансмембранный домен и второй трансмембранный домен кодируются одинаковыми последовательностями нуклеиновой кислоты.
В некоторых вариантах осуществления первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен содержат одинаковые аминокислотные последовательности. Не ограничиваясь какой-либо теорией, полагают, что такая молекула нуклеиновой кислоты демонстрирует меньшую рекомбинацию, чем в остальном сходная молекула нуклеиновой кислоты, в которой первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен кодируются одинаковыми последовательностями нуклеиновой кислоты.
В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен содержат одинаковые аминокислотные последовательности. Не ограничиваясь какой-либо теорией, полагают, что такая молекула нуклеиновой кислоты демонстрирует меньшую рекомбинацию, чем в остальном сходная молекула нуклеиновой кислоты, в которой первый первичный сигнальный домен и второй первичный сигнальный домен кодируются одинаковыми последовательностями нуклеиновой кислоты.
В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен содержат разные аминокислотные последовательности.
В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат одинаковые аминокислотные последовательности. Не ограничиваясь какой-либо теорией, полагают, что такая молекула нуклеиновой кислоты демонстрирует меньшую рекомбинацию, чем в остальном сходная молекула нуклеиновой кислоты, в которой первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются одинаковыми последовательностями нуклеиновой кислоты.
В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат разные аминокислотные последовательности (например, первый и второй костимулирующие сигнальные домены содержат последовательность костимулирующего домена 4-1BB и последовательность костимулирующего домена CD28 соответственно или содержат последовательность костимулирующего домена CD28 и последовательность костимулирующего домена 4-1BB соответственно).
В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность содержат аминокислотную последовательность под SEQ ID NO: 1 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первая лидерная последовательность и вторая лидерная последовательность кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 199 и 210 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними) или SEQ ID NO: 210 и 199 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними).
В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область содержат аминокислотную последовательность под SEQ ID NO: 2 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первая шарнирная область и вторая шарнирная область кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 337 и 13 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними) или SEQ ID NO: 13 и 337 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними).
В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен содержат аминокислотную последовательность под SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый трансмембранный домен и второй трансмембранный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 338 и 17 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними) или SEQ ID NO: 17 и 338 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними).
В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат аминокислотную последовательность под SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 204 и 18 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними) или SEQ ID NO: 18 и 204 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними).
В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен содержат аминокислотную последовательность под SEQ ID NO: 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления первый первичный сигнальный домен и второй первичный сигнальный домен кодируются последовательностями нуклеиновой кислоты, содержащими SEQ ID NO: 205 и 21 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними) или SEQ ID NO: 21 и 205 соответственно (или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними).
В некоторых вариантах осуществления первый и второй антигены являются разными. В некоторых вариантах осуществления первый или второй антиген выбран из BCMA, CD19, CD5, CD10, CD20, CD21, CD22, CD23, CD24, CD25, CD27, CD30, CD34, CD37, CD38, CD40, CD53, CD69, CD72, CD73, CD74, CD75, CD77, CD79a, CD79b, CD80, CD81, CD82, CD83, CD84, CD85, CD86, CD123, CD135, CD138, CD179, CD269, Flt3, ROR1, FcRn5, FcRn2, CS-1, CXCR4, 5, 7, IL-7/3R, IL7/4/3R или IL4R, где В-клеточный антиген необязательно выбран из CD19, CD20, CD22, FcRn5, FcRn2, CS-1, CD138, CD123, CD33, CD34, CLL-1, рецептора фолиевой кислоты бета, FLT3, EGFRvIII, мезотелина, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC. В некоторых вариантах осуществления первый или второй антигенсвязывающий домен содержит CDR, VH, VL или scFv, раскрытые в данном документе, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними.
В некоторых вариантах осуществления в данном документе предусмотрен CAR, содержащий первую VH (VH1), первую VL (VL1), вторую VH (VH2), вторую VL (VL2), трансмембранный домен и внутриклеточный сигнальный домен, где VH1 и VL1 связываются с первым антигеном, а VH2 и VL2 связываются со вторым антигеном. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно линкер 1 ("L1") - VH2 - необязательно линкер 2 ("L2") - VL2 - необязательно линкер 3 ("L3") - VL1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VL2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - линкер 1 ("L1") - VH2 - линкер 2 ("L2") - VL2 - линкер 3 ("L3") - VL1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - L1 - VL2 - L2 - VH2 - L3 - VL1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL1 - L1 - VH2 - L2 - VL2 - L3 - VH1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL1 - L1 - VL2 - L2 - VH2 - L3 - VH1. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH2 - L1 - VH1 - L2 - VL1 - L3 - VL2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH2 - L1 - VL1 - L2 - VH1 - L3 - VL2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL2 - L1 - VH1 - L2 - VL1 - L3 - VH2. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VL2 - L1 - VL1 - L2 - VH1 - L3 - VH2. В некоторых вариантах осуществления L1 или L3 содержит аминокислотную последовательность под SEQ ID NO: 5 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления L2 содержит аминокислотную последовательность под SEQ ID NO: 63 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления CAR имеет следующую конфигурацию от N-конца до C-конца: (i) VH1 - необязательно линкер 1 ("L1") - VH2 - необязательно линкер 2 ("L2") - VL2 - необязательно линкер 3 ("L3") - VL1 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; (ii) VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; (iii) VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; (iv) VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; (v) VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; (vi) VH2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VL2 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; (vii) VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен; или (viii) VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2 - необязательно шарнирная область - трансмембранный домен - внутриклеточный сигнальный домен. В некоторых вариантах осуществления первый и второй антигены являются разными. В некоторых вариантах осуществления первый или второй антиген выбран из BCMA, CD19, CD5, CD10, CD20, CD21, CD22, CD23, CD24, CD25, CD27, CD30, CD34, CD37, CD38, CD40, CD53, CD69, CD72, CD73, CD74, CD75, CD77, CD79a, CD79b, CD80, CD81, CD82, CD83, CD84, CD85, CD86, CD123, CD135, CD138, CD179, CD269, Flt3, ROR1, FcRn5, FcRn2, CS-1, CXCR4, 5, 7, IL-7/3R, IL7/4/3R или IL4R, где В-клеточный антиген необязательно выбран из CD19, CD20, CD22, FcRn5, FcRn2, CS-1, CD138, CD123, CD33, CD34, CLL-1, рецептора фолиевой кислоты бета, FLT3, EGFRvIII, мезотелина, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC. В некоторых вариантах осуществления VH1, VL1, VH2 или VL2 содержат последовательность CDR, VH или VL, раскрытую в данном документе, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления шарнирная область, трансмембранный домен или внутриклеточный сигнальный домен (например, первичный сигнальный домен и/или костимулирующий сигнальный домен) содержат последовательность шарнирной области, последовательность трансмембранного домена или последовательность внутриклеточного сигнального домена (например, последовательность первичного сигнального домена и/или последовательность костимулирующего сигнального домена), раскрытую в данном документе, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления в данном документе предусмотрена молекула нуклеиновой кислоты, кодирующая CAR в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления в данном документе предусмотрен вектор, содержащий молекулу нуклеиновой кислоты, кодирующую CAR в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления в данном документе предусмотрена клетка, содержащая CAR, раскрытый в данном документе, молекулу нуклеиновой кислоты, кодирующую CAR в формате диатела, раскрытый в данном документе, или вектор, содержащий молекулу нуклеиновой кислоты, кодирующую CAR в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления в данном документе предусмотрена фармацевтическая композиция, которая содержит клетку, содержащую CAR в формате диатела, раскрытый в данном документе, и фармацевтически приемлемый носитель. В некоторых вариантах осуществления в данном документе раскрыт способ получения клетки, содержащей CAR в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления в данном документе раскрыт способ лечения субъекта, например, субъекта с раком, с применением клетки, содержащей CAR в формате диатела, раскрытый в данном документе.
В некоторых вариантах осуществления настоящее изобретение относится к способам получения иммунных эффекторных клеток (например, T-клеток или NK-клеток), сконструированных таким образом, что они экспрессируют CAR, и композиций, полученных с применением таких способов. Способы, раскрытые в данном документе (например, способ ARM или способ с использованием цитокинов, раскрытый в данном документе), можно применять для получения клеток, экспрессирующих двойные CAR или CAR в формате диатела, раскрытые в данном документе. Также раскрыты способы применения таких композиций для лечения заболевания, например, рака, у субъекта.
В некоторых вариантах осуществления в настоящем изобретении описан способ получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR), при этом способ включает: (i) приведение популяции клеток (например, T-клеток, например, T-клеток, выделенных из замороженного или свежего продукта лейкафереза) в контакт (например, связывание) со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток; (ii) приведение популяции клеток (например, T-клеток) в контакт с молекулой нуклеиновой кислоты (например, молекулой ДНК или РНК), кодирующей CAR, за счет чего обеспечивается получение популяции клеток (например, T-клеток), содержащих молекулу нуклеиновой кислоты, и (iii) сбор популяции клеток (например, T-клеток) для хранения (например, повторного составления популяции клеток в среде для криоконсервации) или введения, где: (a) стадию (ii) проводят вместе со стадией (i) или не позже чем через 20 часов после начала стадии (i), например, не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i), например, не позже чем через 18 часов после начала стадии (i), и стадию (iii) проводят не позже чем через 26 часов после начала стадии (i), например, не позже чем через 22, 23, 24 или 25 часов после начала стадии (i), например, не позже чем через 24 часа после начала стадии (i); (b) стадию (ii) проводят вместе со стадией (i) или не позже чем через 20 часов после начала стадии (i), например, не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i), например, не позже чем через 18 часов после начала стадии (i), и стадию (iii) проводят не позже чем через 30 часов после начала стадии (ii), например, не позже чем через 22, 23, 24, 25, 26, 27, 28, 29 или 30 часов после начала стадии (ii); или (c) популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i). В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) представляет собой молекулу ДНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) представляет собой молекулу РНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) находится в вирусном векторе, например, в вирусном векторе, выбранном из лентивирусного вектора, аденовирусного вектора или ретровирусного вектора. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) находится в невирусном векторе. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) находится в плазмиде. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) не находится в каком-либо векторе. В некоторых вариантах осуществления стадия (ii) включает трансдукцию популяции клеток (например, T-клеток) c помощью вирусного вектора, содержащего молекулу нуклеиновой кислоты, кодирующую CAR. В некоторых вариантах осуществления стадию (ii) проводят вместе со стадией (i). В некоторых вариантах осуществления стадию (ii) проводят не позже чем через 20 часов после начала стадии (i). В некоторых вариантах осуществления стадию (ii) проводят не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i). В некоторых вариантах осуществления стадию (ii) проводят не позже чем через 18 часов после начала стадии (i). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 26 часов после начала стадии (i). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 22, 23, 24 или 25 часов после начала стадии (i). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 24 часа после начала стадии (i). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 30 часов после начала стадии (ii). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 22, 23, 24, 25, 26, 27, 28, 29 или 30 часов после начала стадии (ii). В некоторых вариантах осуществления молекула нуклеиновой кислоты, кодирующая CAR, представляет собой молекулу нуклеиновой кислоты, раскрытую в данном документе. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит первую последовательность нуклеиновой кислоты, кодирующую первый CAR, и вторую последовательность нуклеиновой кислоты, кодирующую второй CAR. В некоторых вариантах осуществления первая и вторая последовательности нуклеиновой кислоты расположены в одной молекуле нуклеиновой кислоты, например, где первая последовательность нуклеиновой кислоты и вторая последовательность нуклеиновой кислоты разделены третьей последовательностью нуклеиновой кислоты, кодирующей сайт саморасщепления (например, сайт P2A, сайт Т2A, сайт Е2A или сайт F2A). В некоторых вариантах осуществления первая и вторая последовательности нуклеиновой кислоты расположены в отдельных молекулах нуклеиновой кислоты. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую CAR, где CAR содержит первую VH (VH1), первую VL (VL1), вторую VH (VH2), вторую VL (VL2), трансмембранный домен и внутриклеточный сигнальный домен, где VH1 и VL1 связываются с первым антигеном, а VH2 и VL2 связываются со вторым антигеном, где VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно линкер 1 ("L1") - VH2 - необязательно линкер 2 ("L2") - VL2 - необязательно линкер 3 ("L3") - VL1, VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1, VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1, VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1, VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2, VH2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VL2, VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2 или VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2.
В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся. В некоторых вариантах осуществления популяция клеток из стадии (iii) является размножившейся с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, или 40%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i). В некоторых вариантах осуществления популяция клеток из стадии (iii) является размножившейся с увеличением численности на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i).
В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит первую последовательность нуклеиновой кислоты, кодирующую первый CAR, и вторую последовательность нуклеиновой кислоты, кодирующую второй CAR, где первая и вторая последовательности нуклеиновой кислоты расположены в отдельных молекулах нуклеиновой кислоты.
В некоторых вариантах осуществления первая и вторая молекулы нуклеиновой кислоты находятся в отдельных вирусных векторах, и при этом стадия (ii) включает трансдукцию популяции клеток (например, Т-клеток) с помощью первого вирусного вектора, содержащего молекулу нуклеиновой кислоты, кодирующую первый CAR, и второго вирусного вектора, содержащего вторую молекулу нуклеиновой кислоты, кодирующую второй CAR.
В некоторых вариантах осуществления первый CAR содержит домен, связывающий BCMA (например, CAR для BCMA), а второй CAR содержит домен, связывающий CD19 (например, CAR для CD19).
В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с первым вирусным вектором при множественности заражения (MOI), которая больше, равняется или меньше MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором. В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с первым вирусным вектором при множественности заражения (MOI), которая больше MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором.
В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с первым вирусным вектором при первой MOI и со вторым вирусным вектором при второй MOI, так что полученная в результате популяция клеток содержит первую популяцию клеток, которые содержат CAR для BCMA, но не CAR для CD19, вторую популяцию клеток, которые содержат CAR для CD19, но не CAR для BCMA, и третью популяцию клеток, которые содержат как CAR для BCMA, так и CAR для CD19, где:
(a) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(b) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(c) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в полученной в результате популяции, например, при определении посредством способа, описанного в примере 10;
(d) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10; или
(e) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток во второй популяции, например, при определении посредством способа, описанного в примере 10. В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт со вторым вирусным вектором при MOI, например, MOI, которая достаточно ниже, чем MOI, при которой популяцию клеток приводят в контакт с первым вирусным вектором, так что в полученной в результате популяции клеток:
(a) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(b) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000%, 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(c) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в полученной в результате популяции, например, при определении посредством способа, описанного в примере 10;
(d) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10; или
(e) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000%, 5000, 10000% или больше) от общего количества жизнеспособных клеток во второй популяции, например, при определении посредством способа, описанного в примере 10.
В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с первым вирусным вектором при первой MOI и популяцию клеток приводят в контакт со вторым вирусным вектором при второй MOI, так что полученная в результате популяция клеток содержит:
(a) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(b) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(c) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в полученной в результате популяции, например, при определении посредством способа, описанного в примере 10;
(d) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10; или
(e) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000%, 5000, 10000% или больше) от общего количества жизнеспособных клеток во второй популяции, например, при определении посредством способа, описанного в примере 10.
В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с:
(a) первым вирусным вектором при MOI, составляющей от приблизительно 1 до приблизительно 10 (например, от приблизительно 2 до приблизительно 9, от приблизительно 3 до приблизительно 8, от приблизительно 4 до приблизительно 7, от приблизительно 5 до приблизительно 6, от приблизительно 1 до приблизительно 8, от приблизительно 1 до приблизительно 6, от приблизительно 1 до приблизительно 4, от приблизительно 8 до приблизительно 10, от приблизительно 6 до приблизительно 10, от приблизительно 4 до приблизительно 10, от приблизительно 1 до приблизительно 3, от приблизительно 2 до приблизительно 4, от приблизительно 3 до приблизительно 5, от приблизительно 4 до приблизительно 6, от приблизительно 5 до приблизительно 7, от приблизительно 6 до приблизительно 8, от приблизительно 7 до приблизительно до приблизительно 9, от приблизительно 8 до приблизительно 10, от приблизительно 2,5 до приблизительно 5, приблизительно 1, приблизительно 2, приблизительно 3, приблизительно 4, приблизительно 5, приблизительно 6, приблизительно 7, приблизительно 8, приблизительно 9 или приблизительно 10);
(b) вторым вирусным вектором при MOI, составляющей от приблизительно 0,1 до приблизительно 5 (например, от приблизительно 0,2 до приблизительно 4, от приблизительно 0,3 до приблизительно 3, от приблизительно 0,4 до приблизительно 2, от приблизительно 0,5 до приблизительно 1, от приблизительно 0,6 до приблизительно 0,9, от приблизительно 0,7 до приблизительно 0,8, от приблизительно 0,1 до приблизительно 4, от приблизительно 0,1 до приблизительно 3, от приблизительно 0,1 до приблизительно 2, от приблизительно 0,1 до приблизительно 1, от приблизительно 0,1 до приблизительно 0,5, от приблизительно 4 до приблизительно 5, от приблизительно 3 до приблизительно 5, от приблизительно 2 до приблизительно 5, от приблизительно 1 до приблизительно 5, от приблизительно 0,5 до приблизительно 5, от приблизительно 0,2 до приблизительно 5, от приблизительно 0,1 до приблизительно 0,5, от приблизительно 0,2 до приблизительно 1, от приблизительно 0,5 до приблизительно 2, от приблизительно 1 до приблизительно 3, от приблизительно 2 до приблизительно 4, от приблизительно 3 до приблизительно 5, от приблизительно 0,5 до приблизительно 1, приблизительно 0,1, приблизительно 0,2, приблизительно 0,3, приблизительно 0,4, приблизительно 0,5, приблизительно 0,6, приблизительно 0,7, приблизительно 0,8, приблизительно 0,9, приблизительно 1, приблизительно 2, приблизительно 3, приблизительно 4 или приблизительно 5);
(c) первым вирусным вектором при MOI, составляющей по меньшей мере приблизительно 10% (например, по меньшей мере приблизительно 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором, или в по меньшей мере приблизительно 1 раз (например, в по меньшей мере приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 35, 40, 50, 60, 70, 80, 90, или 100 раз, например, от приблизительно 2 до приблизительно 50 раз, от приблизительно 3 до 20 раз, от приблизительно 5 до приблизительно 15 раз или от приблизительно 8 до приблизительно 10 раз) выше нее; и/или
(d) вторым вирусным вектором при MOI, которая составляет не более чем 1/X, где X равняется 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 15, 20, 30, 40, 50, 60, 70. 80, 90, или 100, от MOI, при которой популяцию клеток приводят в контакт с первым вирусным вектором.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с первым вирусным вектором при MOI, составляющей от приблизительно 2,5 до приблизительно 5. В некоторых вариантах осуществления популяцию клеток приводят в контакт со вторым вирусным вектором при MOI, составляющей от приблизительно 0,5 до приблизительно 1,0. В некоторых вариантах осуществления MOI для первого вирусного вектора в от приблизительно 8 до приблизительно 10 раз выше, чем MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором. В некоторых вариантах осуществления MOI для второго вирусного вектора составляет не более чем 1/X, где X равняется 6, 8, 10 или 12, от MOI, при которой популяцию клеток приводят в контакт с первым вирусным вектором.
В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с:
(a) первым вирусным вектором при MOI, составляющей от приблизительно 4 до приблизительно 5 (например, приблизительно 4,75); и/или
(b) вторым вирусным вектором при MOI, составляющей от приблизительно 0,2 до приблизительно 1 (например, приблизительно 0,5).
В некоторых вариантах осуществления на стадии (ii) популяция клеток содержит от приблизительно 1×108 до приблизительно 5×109 (например, от приблизительно 2×108 до приблизительно 2×109 или от приблизительно 4×108 до приблизительно 1×109) всех жизнеспособных клеток. В некоторых вариантах осуществления клетки суспендированы в культуре при концентрации, составляющей от приблизительно 1×106 до приблизительно 1 ×107 (например, от приблизительно 2×106 до приблизительно 5 ×106 или от приблизительно 3×106 до приблизительно 4 ×106) жизнеспособных клеток/мл.
В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, представляет собой средство, которое стимулирует CD3. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой средство, которое стимулирует CD28, ICOS, CD27, HVEM, LIGHT, CD40, 4-1BB, OX40, DR3, GITR, CD30, TIM1, CD2, CD226 или любую их комбинацию. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой средство, которое стимулирует CD28. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, выбрано из антитела (например, однодоменного антитела (например, антитела из одного вариабельного домена тяжелой цепи), пептитела, Fab-фрагмента или scFv), малой молекулы или лиганда (например, существующего в природе, рекомбинантного или химерного лиганда). В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, выбрано из антитела (например, однодоменного антитела (например, антитела из одного вариабельного домена тяжелой цепи), пептитела, Fab-фрагмента или scFv), малой молекулы или лиганда (например, существующего в природе, рекомбинантного или химерного лиганда). В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, не содержит гранулу. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, не содержит гранулу. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, содержит антитело к CD3. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, содержит антитело к CD28. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, содержит антитело к CD3, ковалентно присоединенное к коллоидной полимерной наноматрице. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, содержит антитело к CD28, ковалентно присоединенное к коллоидной полимерной наноматрице. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, и средство, которое стимулирует костимулирующую молекулу, содержат TransAct™ для T-клеток.
В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, не содержит гидрогель. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, не содержит гидрогель. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, не содержит альгинат. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, не содержит альгинат.
В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, содержит гидрогель. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, содержит гидрогель. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, содержит альгинат. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, содержит альгинат. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, или средство, которое стимулирует костимулирующую молекулу, содержат MagCloudz™ от Quad Technologies.
В некоторых вариантах осуществления стадия (i) обеспечивает увеличение процентной доли клеток, экспрессирующих CAR, в популяции клеток из стадии (iii), например, популяция клеток из стадии (iii) демонстрирует более высокую процентную долю клеток, экспрессирующих CAR (например, на по меньшей мере 10, 20, 30, 40, 50 или 60% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа без стадии (i).
В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (iii) является такой же, как процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (iii) отличается на не более чем 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12% от процентной доли необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (iii) отличается на не более чем 5 или 10% от процентной доли необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (i).
В некоторых вариантах осуществления популяция клеток из стадии (iii) демонстрирует более высокую процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ Т-клеток (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления популяция клеток из стадии (iii) демонстрирует более высокую процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ Т-клеток (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных Т-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (iii) является такой же, как процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (iii) отличается на не более чем 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12% от процентной доли центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (iii) отличается на не более чем 5 или 10% от процентной доли центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (i).
В некоторых вариантах осуществления популяция клеток из стадии (iii) демонстрирует более низкую процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% ниже), по сравнению с клетками, полученными посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления популяция клеток из стадии (iii) демонстрирует более низкую процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% ниже), по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления процентная доля стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в популяции клеток из стадии (iii) является увеличенной по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля стволовых T-клеток памяти, экспрессирующих CAR, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, экспрессирующих CAR, в популяции клеток из стадии (iii) является увеличенной по сравнению с процентной долей стволовых T-клеток памяти, экспрессирующих CAR, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, экспрессирующих CAR, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в популяции клеток из стадии (iii) является более высокой, чем процентная доля стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, среди клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления процентная доля стволовых T-клеток памяти, экспрессирующих CAR, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, экспрессирующих CAR, в популяции клеток из стадии (iii) является более высокой, чем процентная доля стволовых T-клеток памяти, экспрессирующих CAR, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, экспрессирующих CAR, среди клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления процентная доля стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в популяции клеток из стадии (iii) является более высокой, чем процентная доля стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, среди клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней. В некоторых вариантах осуществления процентная доля стволовых T-клеток памяти, экспрессирующих CAR, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, экспрессирующих CAR, в популяции клеток из стадии (iii) является более высокой, чем процентная доля стволовых T-клеток памяти, экспрессирующих CAR, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, экспрессирующих CAR, среди клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для популяции клеток из стадии (iii) является приблизительно таким же, как медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для популяции клеток в начале стадии (i), или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 25, 50, 75, 100 или 125%. В некоторых вариантах осуществления медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 100, 150, 200, 250 или 300% ниже), чем медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 100, 150, 200, 250 или 300% ниже), чем медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней. В некоторых вариантах осуществления медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для популяции клеток из стадии (iii) является приблизительно таким же, как медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для популяции клеток в начале стадии (i), или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 25, 50, 100, 150 или 200%. В некоторых вариантах осуществления медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 50, 100, 125, 150 или 175% ниже), чем медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 50, 100, 125, 150 или 175% ниже), чем медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней. В некоторых вариантах осуществления медианный балл набора генов (подавляемые в состоянии стволовости) для популяции клеток из стадии (iii) является приблизительно таким же, как медианный балл набора генов (подавляемые в состоянии стволовости) для популяции клеток в начале стадии (i), или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 25, 50, 100, 150, 200 или 250%. В некоторых вариантах осуществления медианный балл набора генов (подавляемые в состоянии стволовости) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 50, 100 или 125% ниже), чем медианный балл набора генов (подавляемые в состоянии стволовости) для клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления медианный балл набора генов (подавляемые в состоянии стволовости) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 50, 100 или 125% ниже), чем медианный балл набора генов (подавляемые в состоянии стволовости) для клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней. В некоторых вариантах осуществления медианный балл набора генов (активируемые при гипоксии) для популяции клеток из стадии (iii) является приблизительно таким же, как медианный балл набора генов (активируемые при гипоксии) для популяции клеток в начале стадии (i), или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 125, 150, 175 или 200%. В некоторых вариантах осуществления медианный балл набора генов (активируемые при гипоксии) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 40, 50, 60, 70 или 80% ниже), чем медианный балл набора генов (активируемые при гипоксии) для клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления медианный балл набора генов (активируемые при гипоксии) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 40, 50, 60, 70 или 80% ниже), чем медианный балл набора генов (активируемые при гипоксии) для клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней. В некоторых вариантах осуществления медианный балл набора генов (активируемые при аутофагии) для популяции клеток из стадии (iii) является приблизительно таким же, как медианный балл набора генов (активируемые при аутофагии) для популяции клеток в начале стадии (i), или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 180, 190, 200 или 210%. В некоторых вариантах осуществления медианный балл набора генов (активируемые при аутофагии) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере приблизительно 20, 30 или 40% ниже), чем медианный балл набора генов (активируемые при аутофагии) для клеток, полученных посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления медианный балл набора генов (активируемые при аутофагии) для популяции клеток из стадии (iii) является более низким (например, на по меньшей мере 20, 30 или 40% ниже), чем медианный балл набора генов (активируемые при аутофагии) для клеток, полученных посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления популяция клеток из стадии (iii) после инкубирования с клеткой, экспрессирующей антиген, распознаваемый CAR, секретирует IL-2 на более высоком уровне (например, в по меньшей мере 2, 4, 6, 8, 10, 12 или 14 раз более высоком), чем клетки, полученные посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i), или клетки, полученные посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления популяция клеток из стадии (iii) после введения in vivo персистирует дольше или размножается на более высоком уровне (например, на по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, или 90% более высоком) по сравнению с клетками, полученными посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i). В некоторых вариантах осуществления популяция клеток из стадии (iii) после введения in vivo персистирует дольше или размножается на более высоком уровне (например, на по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, или 90% более высоком) по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления популяция клеток из стадии (iii) после введения in vivo демонстрирует более сильную противоопухолевую активность (например, более сильную противоопухолевую активность при низкой дозе, например, дозе, составляющей не более 0,15×106, 0,2×106, 0,25×106 или 0,3×106 жизнеспособных клеток, экспрессирующих CAR), чем клетки, полученные посредством в остальном аналогичного способа, в котором стадию (iii) проводят через более чем 26 часов после начала стадии (i), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (i), или клетки, полученные посредством в остальном аналогичного способа, который дополнительно включает после стадии (ii) и перед стадией (iii) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i). В некоторых вариантах осуществления популяция клеток из стадии (iii) является уменьшенной по сравнению с количеством живых клеток в популяции клеток в начале стадии (i), например, согласно оценке по количеству живых клеток. В некоторых вариантах осуществления популяция клеток из стадии (iii) является размножившейся с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i). В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся или является размножившейся за менее чем 0,5, 1, 1,5 или 2 часа, например, менее чем 1 или 1,5 часа, по сравнению с популяцией клеток в начале стадии (i).
В некоторых вариантах осуществления стадии (i) и (ii) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-6 (например, IL-6/sIL-6Ra), ингибитор LSD1 или ингибитор MALT1. В некоторых вариантах осуществления стадии (i) и (ii) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-7, IL-21 или их комбинацию. В некоторых вариантах осуществления стадии (i) и (ii) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-21, IL-7, IL-6 (например, IL-6/sIL-6Ra), ингибитор LSD1, ингибитор MALT1 или их комбинацию. В некоторых вариантах осуществления стадию (i) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-6 (например, IL-6/sIL-6Ra), ингибитор LSD1 или ингибитор MALT1. В некоторых вариантах осуществления стадию (ii) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-6 (например, IL-6/sIL-6Ra), ингибитор LSD1 или ингибитор MALT1. В некоторых вариантах осуществления стадию (i) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-7, IL-21 или их комбинацию. В некоторых вариантах осуществления стадию (ii) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-7, IL-21 или их комбинацию. В некоторых вариантах осуществления стадию (i) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-21, IL-7, IL-6 (например, IL-6/sIL-6Ra), ингибитор LSD1, ингибитор MALT1 или их комбинацию. В некоторых вариантах осуществления стадию (ii) проводят в средах для клеток (например, бессывороточных средах), содержащих IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-21, IL-7, IL-6 (например, IL-6/sIL-6Ra), ингибитор LSD1, ингибитор MALT1 или их комбинацию. В некоторых вариантах осуществления среда для клеток представляет собой бессывороточную среду, содержащую заменитель сыворотки. В некоторых вариантах осуществления заменитель сыворотки представляет собой заменитель сыворотки для иммунных клеток (ICSR) CTS™.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (iv) получение свежего продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как свежий продукт, представляющий собой цельную кровь, свежий продукт, представляющий собой костный мозг, или свежий продукт, полученный в результате биопсии или удаления опухоли или органа (например, свежий продукт, полученный в результате тимэктомии)), от организации, например, лаборатории, больницы или учреждения здравоохранения.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (v) выделение популяции клеток (например, T-клеток, например, CD8+ и/или CD4+ T-клеток), приводимых в контакт на стадии (i), из свежего продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как свежий продукт, представляющий собой цельную кровь, свежий продукт, представляющий собой костный мозг, или свежий продукт, полученный в результате биопсии или удаления опухоли или органа (например, свежий продукт, полученный в результате тимэктомии)). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 35 часов после начала стадии (v), например, не позже чем через 27, 28, 29, 30, 31, 32, 33, 34 или 35 часов после начала стадии (v), например, не позже чем через 30 часов после начала стадии (v). В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в конце стадии (v).
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) получение криоконсервированных T-клеток, выделенных из продукта лейкафереза (или альтернативного источника гемопоэтической ткани, как, например, криоконсервированных T-клеток, выделенных из цельной крови, костного мозга или продукта, полученного в результате биопсии или удаления опухоли или органа (например, тимэктомии)), от организации, например, лаборатории, больницы или учреждения здравоохранения.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (iv) получение криоконсервированного продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как криоконсервированный продукт, представляющий собой цельную кровь, криоконсервированный продукт, представляющий собой костный мозг, или криоконсервированный продукт, полученный в результате биопсии или удаления опухоли или органа (например, криоконсервированный продукт, полученный в результате тимэктомии)), от организации, например, лаборатории, больницы или учреждения здравоохранения.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (v) выделение популяции клеток (например, T-клеток, например, CD8+ и/или CD4+ T-клеток), приводимых в контакт на стадии (i), из криоконсервированного продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как криоконсервированный продукт, представляющий собой цельную кровь, криоконсервированный продукт, представляющий собой костный мозг, или криоконсервированный продукт, полученный в результате биопсии или удаления опухоли или органа (например, криоконсервированный продукт, полученный в результате тимэктомии)). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 35 часов после начала стадии (v), например, не позже чем через 27, 28, 29, 30, 31, 32, 33, 34 или 35 часов после начала стадии (v), например, не позже чем через 30 часов после начала стадии (v). В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в конце стадии (v).
В некоторых вариантах осуществления клетки из стадии (iii) культивируют в течение от приблизительно двух до приблизительно четырех дней, например, приблизительно трех дней (например, приблизительно 72 часов после сбора), перед измерением уровня экспрессии CAR в их части (например, перед измерением процентной доли жизнеспособных клеток, экспрессирующих CAR, в их части, например, перед измерением процентной доли жизнеспособных клеток, экспрессирующих CAR для BCMA, в их части). В некоторых вариантах осуществления измерение экспрессии CAR осуществляют через приблизительно 4 дня (например, 96 часов) после стадии (ii). В некоторых вариантах осуществления уровень экспрессии CAR измеряют посредством проточной цитометрии.
В некоторых вариантах осуществления в настоящем изобретении описан способ получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR), при этом способ включает (1) приведение популяции клеток (например, T-клеток, например, T-клеток, выделенных из замороженного продукта лейкафереза) в контакт с цитокином, выбранным из IL-2, IL-7, IL-15, IL-21, IL-6 или их комбинации, (2) приведение популяции клеток (например, T-клеток) в контакт с молекулой нуклеиновой кислоты (например, молекулой ДНК или РНК), кодирующей CAR, за счет чего обеспечивается получение популяции клеток (например, T-клеток), содержащих молекулу нуклеиновой кислоты, и (3) сбор популяции клеток (например, T-клеток) для хранения (например, повторного составления популяции клеток в среде для криоконсервации) или введения, где (a) стадию (2) проводят вместе со стадией (1) или не позже чем через 5 часов после начала стадии (1), например, не позже чем через 1, 2, 3, 4 или 5 часов после начала стадии (1), и стадию (3) проводят не позже чем через 26 часов после начала стадии (1), например, не позже чем через 22, 23, 24 или 25 часов после начала стадии (1), например, не позже чем через 24 часа после начала стадии (1), или (b) популяция клеток из стадии (3) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) представляет собой молекулу ДНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) представляет собой молекулу РНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) находится в вирусном векторе, например, в вирусном векторе, выбранном из лентивирусного вектора, аденовирусного вектора или ретровирусного вектора. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) находится в невирусном векторе. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) находится в плазмиде. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) не находится в каком-либо векторе. В некоторых вариантах осуществления стадия (2) включает трансдукцию популяции клеток (например, T-клеток) c помощью вирусного вектора, содержащего молекулу нуклеиновой кислоты, кодирующую CAR. В некоторых вариантах осуществления молекула нуклеиновой кислоты, кодирующая CAR, представляет собой молекулу нуклеиновой кислоты, раскрытую в данном документе. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит первую последовательность нуклеиновой кислоты, кодирующую первый CAR, и вторую последовательность нуклеиновой кислоты, кодирующую второй CAR. В некоторых вариантах осуществления первая и вторая последовательности нуклеиновой кислоты расположены в одной молекуле нуклеиновой кислоты, например, где первая последовательность нуклеиновой кислоты и вторая последовательность нуклеиновой кислоты разделены третьей последовательностью нуклеиновой кислоты, кодирующей сайт саморасщепления (например, сайт P2A, сайт Т2A, сайт Е2A или сайт F2A). В некоторых вариантах осуществления первая и вторая последовательности нуклеиновой кислоты расположены в отдельных молекулах нуклеиновой кислоты. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую CAR, где CAR содержит первую VH (VH1), первую VL (VL1), вторую VH (VH2), вторую VL (VL2), трансмембранный домен и внутриклеточный сигнальный домен, где VH1 и VL1 связываются с первым антигеном, а VH2 и VL2 связываются со вторым антигеном, где VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно линкер 1 ("L1") - VH2 - необязательно линкер 2 ("L2") - VL2 - необязательно линкер 3 ("L3") - VL1, VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1, VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1, VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1, VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2, VH2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VL2, VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2 или VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2.
В некоторых вариантах осуществления стадию (2) проводят вместе со стадией (1). В некоторых вариантах осуществления стадию (2) проводят не позже чем через 5 часов после начала стадии (1). В некоторых вариантах осуществления стадию (2) проводят не позже чем через 1, 2, 3, 4 или 5 часов после начала стадии (1). В некоторых вариантах осуществления стадию (3) проводят не позже чем через 26 часов после начала стадии (1). В некоторых вариантах осуществления стадию (3) проводят не позже чем через 22, 23, 24 или 25 часов после начала стадии (1). В некоторых вариантах осуществления стадию (3) проводят не позже чем через 24 часа после начала стадии (1).
В некоторых вариантах осуществления популяция клеток из стадии (3) не является размножившейся, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) является размножившейся с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, или 40%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) является размножившейся с увеличением численности на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1).
В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-2. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-7. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-21. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-2 и IL-7. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-2 и IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-2 и IL-21. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-2 и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-7 и IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-7 и IL-21. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-7 и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) и IL-21. В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-21 и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления стадия (1) включает приведение популяции клеток (например, T-клеток) в контакт с IL-7, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) и IL-21.
В некоторых вариантах осуществления популяция клеток из стадии (3) демонстрирует более высокую процентную долю необученных клеток среди клеток, экспрессирующих CAR (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает приведение популяции клеток в контакт, например, с антителом к CD3.
В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (3) является такой же, как процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (3) отличается на не более чем 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12% от процентной доли необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (3) отличается на не более чем 5 или 10% от процентной доли необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (3) является увеличенной по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (3) увеличена на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20% по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток из стадии (3) увеличена на по меньшей мере 10 или 20% по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале стадии (1).
В некоторых вариантах осуществления популяция клеток из стадии (3) демонстрирует более высокую процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ Т-клеток (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, в котором стадию (3) проводят через более чем 26 часов после начала стадии (1), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) демонстрирует более высокую процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает после стадии (2) и перед стадией (3) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных Т-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (3) является такой же, как процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (3) отличается на не более чем 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12% от процентной доли центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (3) отличается на не более чем 5 или 10% от процентной доли центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (i). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (3) является уменьшенной по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (3) уменьшена на по меньшей мере 10 или 20% по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (1). В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток из стадии (3) уменьшена на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20% по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале стадии (1).
В некоторых вариантах осуществления популяция клеток из стадии (3) демонстрирует более низкую процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% ниже), по сравнению с клетками, полученными посредством в остальном аналогичного способа, в котором стадию (3) проводят через более чем 26 часов после начала стадии (1), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) демонстрирует более низкую процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти (например, на по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35 или 40% ниже), по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает после стадии (2) и перед стадией (3) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления популяция клеток из стадии (3) после введения in vivo персистирует дольше или размножается на более высоком уровне (например, на по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, или 90% более высоком) по сравнению с клетками, полученными посредством в остальном аналогичного способа, в котором стадию (3) проводят через более чем 26 часов после начала стадии (1), например, через более чем 5, 6, 7, 8, 9, 10, 11 или 12 дней после начала стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) после введения in vivo персистирует дольше или размножается на более высоком уровне (например, на по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, или 90% более высоком) по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает после стадии (2) и перед стадией (3) размножение популяции клеток (например, T-клеток) in vitro в течение более чем 3 дней, например, в течение 5, 6, 7, 8 или 9 дней.
В некоторых вариантах осуществления популяция клеток из стадии (3) не является размножившейся, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) является размножившейся с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, или 40%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления популяция клеток из стадии (3) является размножившейся с увеличением численности на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления количество живых клеток в популяции клеток из стадии (3) является уменьшенным по сравнению с количеством живых клеток в популяции клеток в начале стадии (1), например, согласно оценке по количеству живых клеток.
В некоторых вариантах осуществления популяция клеток из стадии (3) не является размножившейся по сравнению с популяцией клеток в начале стадии (1), например, согласно оценке по количеству живых клеток. В некоторых вариантах осуществления популяция клеток из стадии (3) является размножившейся за менее чем 0,5, 1, 1,5 или 2 часа, например, менее чем 1 или 1,5 часа, по сравнению с популяцией клеток в начале стадии (1).
В некоторых вариантах осуществления популяцию клеток не приводят в контакт in vitro со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, или, в случае приведения в контакт, продолжительность стадии приведения в контакт составляет менее 2 часов, например, не более 1 или 1,5 часа. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, представляет собой средство, которое стимулирует CD3 (например, антитело к CD3). В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой средство, которое стимулирует CD28, ICOS, CD27, HVEM, LIGHT, CD40, 4-1BB, OX40, DR3, GITR, CD30, TIM1, CD2, CD226 или любую их комбинацию. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой средство, которое стимулирует CD28. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, или средство, которое стимулирует костимулирующую молекулу, выбрано из антитела (например, однодоменного антитела (например, антитела из одного вариабельного домена тяжелой цепи), пептитела, Fab-фрагмента или scFv), малой молекулы или лиганда (например, существующего в природе, рекомбинантного или химерного лиганда).
В некоторых вариантах осуществления стадии (1) и/или (2) проводят в средах для клеток, содержащих не более 5, 4, 3, 2, 1 или 0% сыворотки. В некоторых вариантах осуществления стадии (1) и/или (2) проводят в средах для клеток, содержащих не более 2% сыворотки. В некоторых вариантах осуществления стадии (1) и/или (2) проводят в средах для клеток, содержащих приблизительно 2% сыворотки. В некоторых вариантах осуществления стадии (1) и/или (2) проводят в средах для клеток, содержащих ингибитор LSD1 или ингибитор MALT1. В некоторых вариантах осуществления стадию (1) проводят в средах для клеток, содержащих не более 5, 4, 3, 2, 1 или 0% сыворотки. В некоторых вариантах осуществления стадию (1) проводят в средах для клеток, содержащих не более 2% сыворотки. В некоторых вариантах осуществления стадию (1) проводят в средах для клеток, содержащих приблизительно 2% сыворотки. В некоторых вариантах осуществления стадию (2) проводят в средах для клеток, содержащих не более 5, 4, 3, 2, 1 или 0% сыворотки. В некоторых вариантах осуществления стадию (2) проводят в средах для клеток, содержащих не более 2% сыворотки. В некоторых вариантах осуществления стадию (2) проводят в средах для клеток, содержащих приблизительно 2% сыворотки. В некоторых вариантах осуществления стадию (1) проводят в средах для клеток, содержащих ингибитор LSD1 или ингибитор MALT1. В некоторых вариантах осуществления стадию (2) проводят в средах для клеток, содержащих ингибитор LSD1 или ингибитор MALT1.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (iv) получение свежего продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как свежий продукт, представляющий собой цельную кровь, свежий продукт, представляющий собой костный мозг, или свежий продукт, полученный в результате биопсии или удаления опухоли или органа (например, свежий продукт, полученный в результате тимэктомии)), от организации, например, лаборатории, больницы или учреждения здравоохранения.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (v) выделение популяции клеток (например, T-клеток, например, CD8+ и/или CD4+ T-клеток), приводимых в контакт на стадии (i), из свежего продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как свежий продукт, представляющий собой цельную кровь, свежий продукт, представляющий собой костный мозг, или свежий продукт, полученный в результате биопсии или удаления опухоли или органа (например, свежий продукт, полученный в результате тимэктомии)). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 35 часов после начала стадии (v), например, не позже чем через 27, 28, 29, 30, 31, 32, 33, 34 или 35 часов после начала стадии (v), например, не позже чем через 30 часов после начала стадии (v). В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в конце стадии (v).
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) получение криоконсервированных T-клеток, выделенных из продукта лейкафереза (или альтернативного источника гемопоэтической ткани, как, например, криоконсервированных T-клеток, выделенных из цельной крови, костного мозга или продукта, полученного в результате биопсии или удаления опухоли или органа (например, тимэктомии)), от организации, например, лаборатории, больницы или учреждения здравоохранения.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (iv) получение криоконсервированного продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как криоконсервированный продукт, представляющий собой цельную кровь, криоконсервированный продукт, представляющий собой костный мозг, или криоконсервированный продукт, полученный в результате биопсии или удаления опухоли или органа (например, криоконсервированный продукт, полученный в результате тимэктомии)), от организации, например, лаборатории, больницы или учреждения здравоохранения.
В некоторых вариантах осуществления вышеуказанные способы дополнительно включают перед стадией (i) (v) выделение популяции клеток (например, T-клеток, например, CD8+ и/или CD4+ T-клеток), приводимых в контакт на стадии (i), из криоконсервированного продукта лейкафереза (или альтернативного источника гемопоэтической ткани, такого как криоконсервированный продукт, представляющий собой цельную кровь, криоконсервированный продукт, представляющий собой костный мозг, или криоконсервированный продукт, полученный в результате биопсии или удаления опухоли или органа (например, криоконсервированный продукт, полученный в результате тимэктомии)). В некоторых вариантах осуществления стадию (iii) проводят не позже чем через 35 часов после начала стадии (v), например, не позже чем через 27, 28, 29, 30, 31, 32, 33, 34 или 35 часов после начала стадии (v), например, не позже чем через 30 часов после начала стадии (v). В некоторых вариантах осуществления популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в конце стадии (v).
В некоторых вариантах осуществления популяция клеток в начале стадии (i) или стадии (1) была обогащена клетками, экспрессирующими IL6R (например, клетками, которые являются положительными по IL6Rα и/или IL6Rβ). В некоторых вариантах осуществления популяция клеток в начале стадии (i) или стадии (1) содержит не менее 40, 45, 50, 55, 60, 65 или 70% клеток, экспрессирующих IL6R (например, клеток, которые являются положительными по IL6Rα и/или IL6Rβ).
В некоторых вариантах осуществления стадии (i) и (ii) или стадии (1) и (2) проводят в средах для клеток, содержащих IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления IL-15 увеличивает способность популяции клеток к размножению, например, спустя 10, 15, 20 или 25 дней. В некоторых вариантах осуществления IL-15 увеличивает процентную долю клеток, экспрессирующих IL6Rβ, в популяции клеток.
В некоторых вариантах осуществления вышеуказанных способов способы осуществляют в закрытой системе. В некоторых вариантах осуществления все из отделения, активации, трансдукции, инкубирования и промывания Т-клеток проводят в закрытой системе. В некоторых вариантах осуществления вышеуказанных способов способы осуществляют в отдельных устройствах. В некоторых вариантах осуществления отделение, активацию, трансдукцию, инкубирование и промывание Т-клеток проводят в отдельных устройствах.
В некоторых вариантах осуществления вышеуказанных способов способы дополнительно включают добавление вспомогательного вещества или реагента, усиливающего трансдукцию, в среду для культивирования клеток для повышения эффективности трансдукции. В некоторых вариантах осуществления вспомогательное вещество или реагент, усиливающий трансдукцию, содержит катионный полимер. В некоторых вариантах осуществления вспомогательное вещество или реагент, усиливающий трансдукцию, выбраны из LentiBOOST™ (Sirion Biotech), вектофусина-1, F108, бромида гексадиметрина (полибрена), PEA, Pluronic F68, Pluronic F127, Synperonic или LentiTrans™. В некоторых вариантах осуществления вспомогательное вещество представляет собой LentiBOOST™ (Sirion Biotech).
В некоторых вариантах осуществления вышеуказанных способов трансдукция популяции клеток (например, T-клеток) c помощью вирусного вектора включает воздействие на популяцию клеток и вирусный вектор центробежной силы в условиях, при которых повышается эффективность трансдукции. В одном варианте осуществления клетки трансдуцируют путем спинокуляции.
В некоторых вариантах осуществления вышеуказанных способов клетки (например, T-клетки) активируют и трансдуцируют в колбе для культивирования клеток, содержащей газопроницаемую мембрану в основании, которая предоставляет возможность работы с большими объемами сред без существенного нарушения газообмена. В некоторых вариантах осуществления рост клеток достигается путем предоставления доступа, например, по существу непрерывного доступа, к питательным веществам посредством конвекции.
В некоторых вариантах осуществления вышеуказанных способов CAR содержит антигенсвязывающий домен, трансмембранный домен и внутриклеточный сигнальный домен.
В некоторых вариантах осуществления антигенсвязывающий домен связывается с антигеном, выбранным из CD19, CD20, CD22, BCMA, мезотелина, EGFRvIII, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC. В некоторых вариантах осуществления антигенсвязывающий домен содержит последовательность CDR, VH, VL, scFv или CAR, раскрытую в данном документе. В некоторых вариантах осуществления антигенсвязывающий домен содержит VH и VL, где VH и VL соединены линкером, где линкер необязательно содержит аминокислотную последовательность под SEQ ID NO: 63 или 104.
В некоторых вариантах осуществления трансмембранный домен включает в себя трансмембранный домен белка, выбранного из альфа-, бета- или дзета-цепи T-клеточного рецептора, CD28, CD3-эпсилон, CD45, CD4, CD5, CD8, CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137 и CD154. В некоторых вариантах осуществления трансмембранный домен включает в себя трансмембранный домен CD8. В некоторых вариантах осуществления трансмембранный домен содержит аминокислотную последовательность под SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую трансмембранный домен, где последовательность нуклеиновой кислоты включает в себя последовательность нуклеиновой кислоты под SEQ ID NO: 17 или последовательность нуклеиновой кислоты, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления антигенсвязывающий домен соединен с трансмембранным доменом с помощью шарнирной области. В некоторых вариантах осуществления шарнирная область содержит аминокислотную последовательность под SEQ ID NO: 2, 3 или 4 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую шарнирную область, где последовательность нуклеиновой кислоты включает в себя последовательность нуклеиновой кислоты под SEQ ID NO: 13, 14 или 15 или последовательность нуклеиновой кислоты, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит первичный сигнальный домен. В некоторых вариантах осуществления первичный сигнальный домен предусматривает функциональный сигнальный домен, полученный из CD3-дзета, TCR-дзета, FcR-гамма, FcR-бета, CD3-гамма, CD3-дельта, CD3-эпсилон, CD5, CD22, CD79a, CD79b, CD278 (ICOS), FcεRI, DAP10, DAP12 или CD66d. В некоторых вариантах осуществления первичный сигнальный домен предусматривает функциональный сигнальный домен, полученный из CD3-дзета. В некоторых вариантах осуществления первичный сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 9 или 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую первичный сигнальный домен, где последовательность нуклеиновой кислоты включает в себя последовательность нуклеиновой кислоты под SEQ ID NO: 20 или 21 или последовательность нуклеиновой кислоты, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит костимулирующий сигнальный домен. В некоторых вариантах осуществления костимулирующий сигнальный домен предусматривает функциональный сигнальный домен, полученный из молекулы MHC I класса, белкового рецептора TNF, иммуноглобулиноподобного белка, рецептора цитокина, интегрина, сигнальной молекулы активации лимфоцитов (белка SLAM), активирующего рецептора NK-клеток, BTLA, лиганда Toll-подобного рецептора, OX40, CD2, CD7, CD27, CD28, CD30, CD40, CDS, ICAM-1, 4-1BB (CD137), B7-H3, ICOS (CD278), GITR, BAFFR, LIGHT, HVEM (LIGHTR), KIRDS2, SLAMF7, NKp80 (KLRF1), NKp44, NKp30, NKp46, CD19, CD4, CD8-альфа, CD8-бета, IL2R-бета, IL2R-гамма, IL7R-альфа, ITGA4, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, ITGB7, NKG2D, NKG2C, TNFR2, TRANCE/RANKL, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (Tactile), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), CD69, SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR, LAT, GADS, SLP-76, PAG/Cbp, CD19a, CD28-OX40, CD28-4-1BB или лиганда, который специфично связывается с CD83. В некоторых вариантах осуществления костимулирующий сигнальный домен предусматривает функциональный сигнальный домен, полученный из 4-1BB. В некоторых вариантах осуществления костимулирующий сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую костимулирующий сигнальный домен, где последовательность нуклеиновой кислоты включает в себя последовательность нуклеиновой кислоты под SEQ ID NO: 18 или последовательность нуклеиновой кислоты, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
В некоторых вариантах осуществления внутриклеточный сигнальный домен предусматривает функциональный сигнальный домен, полученный из 4-1BB, и функциональный сигнальный домен, полученный из CD3-дзета. В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 7 (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней) и аминокислотную последовательность под SEQ ID NO: 9 или 10 (или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит аминокислотную последовательность под SEQ ID NO: 7 и аминокислотную последовательность под SEQ ID NO: 9 или 10.
В некоторых вариантах осуществления CAR дополнительно содержит лидерную последовательность, предусматривающую аминокислотную последовательность под SEQ ID NO: 1.
В некоторых вариантах осуществления в настоящем изобретении описана популяция клеток, экспрессирующих CAR (например, аутологичных или аллогенных T-клеток или NK-клеток, экспрессирующих CAR), полученных посредством любого из вышеуказанных способов или любого другого способа, раскрытого в данном документе. В некоторых вариантах осуществления в данном документе раскрыта фармацевтическая композиция, содержащая популяцию клеток, экспрессирующих CAR, раскрытую в данном документе, и фармацевтически приемлемый носитель.
В некоторых вариантах осуществления популяция содержит:
(a) первую популяцию клеток, содержащих CAR для BCMA, но не CAR для CD19;
(b) вторую популяцию клеток, содержащих CAR для CD19, но не CAR для BCMA; и
(c) третью популяцию клеток, содержащих как CAR для BCMA, так и CAR для CD19.
В некоторых вариантах осуществления:
(i) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях;
(ii) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях; и/или
(iii) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в популяции.
В некоторых вариантах осуществления популяция дополнительно содержит четвертую популяцию клеток, которые не содержат CAR.
В некоторых вариантах осуществления:
(i) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях;
(ii) общее количество жизнеспособных клеток во второй популяции меньше или равняется от приблизительно 45% до приблизительно 50% (например, приблизительно 47%); от приблизительно 50 до приблизительно 55% (например, приблизительно 53%); от приблизительно 60% до приблизительно 65% (например, приблизительно 63%) или от приблизительно 80 до приблизительно 85% (например, приблизительно 82%) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях.
В некоторых вариантах осуществления в конечном продукте на основе клеток с CAR, изготовленном с использованием способов, описанных в данном документе, общее количество гранул (например, гранул с антителами к CD4, гранул с антителами к CD8 и/или гранул TransAct) составляет не более 0,04, 0,05, 0,06, 0,07, 0,08, 0,09, 0,1, 0,2, 0,3, 0,4 или 0,5% от общего количества гранул, добавляемых в ходе способа изготовления.
В некоторых вариантах осуществления в настоящем изобретении описана популяция клеток, экспрессирующих CAR (например, аутологичных или аллогенных T-клеток или NK-клеток, экспрессирующих CAR), предусматривающая одну или несколько из следующих характеристик: (a) приблизительно такую же процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (b) изменение количества необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, находящееся в пределах от приблизительно 5% до приблизительно 10%, например, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (c) увеличенную процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, например, увеличенную в по меньшей мере 1,2, 1,4, 1,6, 1,8, 2,0, 2,2, 2,4, 2,6, 2,8 или 3 раза, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RO- CCR7+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (d) приблизительно такую же процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (e) изменение количества центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, находящееся в пределах от приблизительно 5% до приблизительно 10%, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (f) уменьшенную процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, например, уменьшенную на по меньшей мере 20, 25, 30, 35, 40, 45 или 50%, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CCR7+ CD45RO+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (g) приблизительно такую же процентную долю стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; (h) изменение количества стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, находящееся в пределах от приблизительно 5% до приблизительно 10%, по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR; или (i) увеличенную процентную долю стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, по сравнению с процентной долей стволовых T-клеток памяти, например, CD45RA+ CD95+ рецептор IL-2 β+ CCR7+ CD62L+ T-клеток, в той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR.
В некоторых вариантах осуществления в настоящем изобретении описана популяция клеток, экспрессирующих CAR (например, аутологичных или аллогенных T-клеток или NK-клеток, экспрессирующих CAR), где (a) медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для популяции клеток является приблизительно таким же, как медианный балл набора генов (активируемые в TEM относительно подавляемых в TSCM) для той же популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR, или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 25, 50, 75, 100 или 125%; (b) медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для популяции клеток является приблизительно таким же, как медианный балл набора генов (активируемые в Treg относительно подавляемых в Teff) для популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR, или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 25, 50, 100, 150 или 200%; (c) медианный балл набора генов (подавляемые в состоянии стволовости) для популяции клеток является приблизительно таким же, как медианный балл набора генов (подавляемые в состоянии стволовости) для популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR, или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 25, 50, 100, 150, 200 или 250%; (d) медианный балл набора генов (активируемые при гипоксии) для популяции клеток является приблизительно таким же, как медианный балл набора генов (активируемые при гипоксии) для популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR, или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 125, 150, 175 или 200%; или (e) медианный балл набора генов (активируемые при аутофагии) для популяции клеток является приблизительно таким же, как медианный балл набора генов (активируемые при аутофагии) для популяции клеток до их конструирования таким образом, чтобы они экспрессировали CAR, или отличается от него на не более чем (например, увеличен на не более чем) приблизительно 180, 190, 200 или 210%.
В некоторых вариантах осуществления в настоящем изобретении описан способ усиления иммунного ответа у субъекта, включающий введение субъекту популяции клеток, экспрессирующих CAR, раскрытой в данном документе, или фармацевтической композиции, раскрытой в данном документе, за счет чего обеспечивается усиление иммунного ответа у субъекта.
В некоторых вариантах осуществления в данном документе раскрыт способ лечения рака у субъекта, включающий введение субъекту популяции клеток, экспрессирующих CAR, раскрытой в данном документе, или фармацевтической композиции, раскрытой в данном документе, за счет чего обеспечивается лечение рака у субъекта. В некоторых вариантах осуществления рак представляет собой солидный рак, например, выбранный из одного или нескольких из мезотелиомы, злокачественной мезотелиомы плевры, немелкоклеточного рака легкого, мелкоклеточного рака легкого, плоскоклеточного рака легкого, крупноклеточного рака легкого, рака поджелудочной железы, протоковой аденокарциномы поджелудочной железы, аденокарциномы пищевода, рака молочной железы, глиобластомы, рака яичника, колоректального рака, рака предстательной железы, рака шейки матки, рака кожи, меланомы, рака почки, рака печени, рака головного мозга, тимомы, саркомы, карциномы, рака матки, рака почки, рака желудочно-кишечного тракта, уротелиального рака, рака глотки, рака головы и шеи, рака прямой кишки, рака пищевода или рака мочевого пузыря или их метастазов. В некоторых вариантах осуществления рак представляет собой гемобластоз, например, выбранный из хронического лимфоцитарного лейкоза (CLL), мантийноклеточной лимфомы (MCL), множественной миеломы, острого лимфоидного лейкоза (ALL), лимфомы Ходжкина, B-клеточного острого лимфоидного лейкоза (BALL), T-клеточного острого лимфоидного лейкоза (TALL), мелкоклеточного лимфоцитарного лейкоза (SLL), B-клеточного пролимфоцитарного лейкоза, новообразования из бластных плазмоцитоидных дендритных клеток, лимфомы Беркитта, диффузной крупноклеточной В-клеточной лимфомы (DLBCL), DLBCL, ассоциированной с хроническим воспалением, хронического миелоидного лейкоза, миелопролиферативных новообразований, фолликулярной лимфомы, педиатрической фолликулярной лимфомы, волосатоклеточного лейкоза, мелкоклеточной или крупноклеточной фолликулярной лимфомы, злокачественных лимфопролиферативных состояний, MALT-лимфомы (экстранодальной лимфомы из клеток маргинальной зоны из лимфоидной ткани слизистых оболочек), лимфомы из клеток маргинальной зоны, миелодисплазии, миелодиспластического синдрома, неходжкинской лимфомы, плазмобластной лимфомы, новообразования из плазмоцитоидных дендритных клеток, макроглобулинемии Вальденстрема, лимфомы из клеток маргинальной зоны селезенки, лимфомы/лейкоза селезенки, диффузной мелкоклеточной B-клеточной лимфомы красной пульпы селезенки, варианта волосатоклеточного лейкоза, лимфоплазмоцитарной лимфомы, болезни тяжелых цепей, плазмоклеточной миеломы, солитарной плазмоцитомы кости, внекостной плазмоцитомы, нодулярной лимфомы из клеток маргинальной зоны, педиатрической нодулярной лимфомы из клеток маргинальной зоны, первичной кожной лимфомы из клеток центра фолликула, лимфоматоидного гранулематоза, первичной медиастинальной (тимической) крупноклеточной B-клеточной лимфомы, внутрисосудистой крупноклеточной B-клеточной лимфомы, ALK+ крупноклеточной B-клеточной лимфомы, крупноклеточной B-клеточной лимфомы, возникающей при HHV8-ассоциированной многоочаговой болезни Кастлемана, первичной выпотной лимфомы, B-клеточной лимфомы, острого миелоидного лейкоза (AML) или неклассифицируемой лимфомы.
В некоторых вариантах осуществления способ дополнительно включает введение субъекту второго терапевтического средства. В некоторых вариантах осуществления второе терапевтическое средство представляет собой противораковое терапевтическое средство, например, средство химиотерапии, лучевой терапии или иммунорегуляторной терапии. В некоторых вариантах осуществления второе терапевтическое средство представляет собой IL-15 (например, hetIL-15 (IL15/sIL-15Ra)).
В некоторых вариантах осуществления в данном документе предусмотрена выделенная клетка или популяция клеток, полученная посредством способа, описанного в данном документе, предусматривающая одну или несколько клеток, содержащих:
(a) первую молекулу нуклеиновой кислоты, кодирующую первый CAR, который содержит домен, связывающий BCMA, первый трансмембранный домен и первый внутриклеточный сигнальный домен, где домен, связывающий BCMA, содержит вариабельную область тяжелой цепи (VH), содержащую определяющую комплементарность область 1 тяжелой цепи (CDR1 HC), определяющую комплементарность область 2 тяжелой цепи (CDR2 HC) и определяющую комплементарность область 3 тяжелой цепи (CDR3 HC), и вариабельную область легкой цепи (VL), содержащую определяющую комплементарность область 1 легкой цепи (CDR1 LC), определяющую комплементарность область 2 легкой цепи (CDR2 LC) и определяющую комплементарность область 3 легкой цепи (CDR3 LC), где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно; и
(b) вторую молекулу нуклеиновой кислоты, кодирующую второй CAR, который содержит домен, связывающий CD19, второй трансмембранный домен и второй внутриклеточный сигнальный домен, где домен, связывающий CD19, содержит VH, содержащую CDR1 HC, CDR2 HC и CDR3 HC, и VL, содержащую CDR1 LC, CDR2 LC и CDR3 LC, где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 295, 304 и 297-300 соответственно.
В некоторых вариантах осуществления в данном документе предусмотрена выделенная клетка, содержащая:
(a) первую молекулу нуклеиновой кислоты, кодирующую первый CAR, который содержит домен, связывающий BCMA, первый трансмембранный домен и первый внутриклеточный сигнальный домен, где домен, связывающий BCMA, содержит вариабельную область тяжелой цепи (VH), содержащую определяющую комплементарность область 1 тяжелой цепи (CDR1 HC), определяющую комплементарность область 2 тяжелой цепи (CDR2 HC) и определяющую комплементарность область 3 тяжелой цепи (CDR3 HC), и вариабельную область легкой цепи (VL), содержащую определяющую комплементарность область 1 легкой цепи (CDR1 LC), определяющую комплементарность область 2 легкой цепи (CDR2 LC) и определяющую комплементарность область 3 легкой цепи (CDR3 LC), где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно; и
(b) вторую молекулу нуклеиновой кислоты, кодирующую второй CAR, который содержит домен, связывающий CD19, второй трансмембранный домен и второй внутриклеточный сигнальный домен, где домен, связывающий CD19, содержит VH, содержащую CDR1 HC, CDR2 HC и CDR3 HC, и VL, содержащую CDR1 LC, CDR2 LC и CDR3 LC, где CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 295, 304 и 297-300 соответственно.
В некоторых вариантах осуществления VH и VL домена, связывающего BCMA, содержат аминокислотные последовательности под SEQ ID NO: 93 и 102 соответственно. В некоторых вариантах осуществления VH и VL домена, связывающего CD19, содержат аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно. В некоторых вариантах осуществления VH и VL домена, связывающего BCMA, содержат аминокислотные последовательности под SEQ ID NO: 93 и 102 соответственно, а VH и VL домена, связывающего CD19, содержат аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно. В некоторых вариантах осуществления домен, связывающий BCMA, содержит аминокислотную последовательность под SEQ ID NO: 105. В некоторых вариантах осуществления домен, связывающий CD19, содержит аминокислотную последовательность под SEQ ID NO: 293. В некоторых вариантах осуществления домен, связывающий BCMA, содержит аминокислотную последовательность под SEQ ID NO: 105, а домен, связывающий CD19, содержит аминокислотную последовательность под SEQ ID NO: 293. В некоторых вариантах осуществления первый CAR содержит аминокислотную последовательность под SEQ ID NO: 107. В некоторых вариантах осуществления второй CAR содержит аминокислотную последовательность под SEQ ID NO: 225. В некоторых вариантах осуществления первый CAR содержит аминокислотную последовательность под SEQ ID NO: 107; и второй CAR содержит аминокислотную последовательность под SEQ ID NO: 225. В некоторых вариантах осуществления первый CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 259, 258 или 416. В некоторых вариантах осуществления второй CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 417, 355, 356 или 354. В некоторых вариантах осуществления первый CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 259, 258 или 416, и второй CAR кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 417, 355, 356 или 354.
В некоторых вариантах осуществления в данном документе предусмотрена фармацевтическая композиция, содержащая клетку или популяцию клеток, описанные в данном документе.
В некоторых вариантах осуществления в данном документе предусмотрен способ обеспечения противоопухолевого иммунитета у субъекта или лечения субъекта, у которого имеется заболевание, ассоциированное с экспрессией BCMA, включающий введение субъекту эффективного количества клетки или популяции клеток или фармацевтической композиции, описанных в данном документе.
В некоторых вариантах осуществления заболевание, ассоциированное с экспрессией BCMA, представляет собой гематологический рак или солидный рак, например, гематологический рак или солидный рак, описанный в данном документе.
В некоторых вариантах осуществления заболевание выбрано из острого лейкоза, B-клеточного острого лимфоидного лейкоза ("BALL"), T-клеточного острого лимфоидного лейкоза ("TALL"), острого лимфоидного лейкоза (ALL), хронического миелогенного лейкоза (CML), хронического лимфоцитарного лейкоза (CLL), B-клеточного пролимфоцитарного лейкоза, новообразования из бластных плазмоцитоидных дендритных клеток, лимфомы Беркитта, диффузной крупноклеточной B-клеточной лимфомы, фолликулярной лимфомы, волосатоклеточного лейкоза, мелкоклеточной или крупноклеточной фолликулярной лимфомы, злокачественных лимфопролиферативных состояний, MALT-лимфомы, мантийноклеточной лимфомы, лимфомы из клеток маргинальной зоны, множественной миеломы, миелодисплазии и миелодиспластического синдрома, неходжкинской лимфомы, плазмобластной лимфомы, новообразования из плазмоцитоидных дендритных клеток, макроглобулинемии Вальденстрема, рака предстательной железы (например, кастрационнорезистентного или резистентного к терапии рака предстательной железы или метастатического рака предстательной железы), рака поджелудочной железы, рака легкого, нарушения пролиферации плазматических клеток (например, бессимптомной миеломы ("тлеющей" множественной миеломы или вялотекущей миеломы), моноклональной гаммапатии неустановленной этиологии (MGUS), макроглобулинемии Вальденстрема, плазмоцитомы (например, плазмоклеточной дискразии, солитарной миеломы, солитарной плазмоцитомы, экстрамедуллярной плазмоцитомы и множественной плазмоцитомы), системного амилоидоза в форме амилоидоза легких цепей или POEMS-синдрома (также известного как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром)) или их комбинации.
В некоторых вариантах осуществления заболевание представляет собой множественную миелому.
Хотя при осуществлении на практике или тестировании настоящего изобретения можно применять способы и материалы, аналогичные или эквивалентные описанным в данном документе, ниже описываются подходящие способы и материалы. Все публикации, патентные заявки, патенты и другие литературные источники (например, идентификационные номера последовательностей в базах данных), упоминаемые в данном документе, включены посредством ссылки во всей своей полноте. Например, все последовательности из GenBank, Unigene и Entrez, упоминаемые в данном документе, например, в любой таблице в данном документе, включены посредством ссылки. Если для одного гена или белка представлены ссылки на несколько номеров доступа последовательностей, охватываются все варианты последовательности.
Кроме того, материалы, способы и примеры являются исключительно иллюстративными и не предполагаются как ограничивающие. Заголовки, подзаголовки или пронумерованные или обозначенные буквами элементы, например, (a) (b) (i) и т. д., представлены исключительно для удобства прочтения. Использование заголовков или пронумерованных или обозначенных буквами элементов в данном документе не требует того, чтобы стадии или элементы проводились в алфавитном порядке, или того, чтобы стадии или элементы обязательно были отделены друг от друга. Другие признаки, цели и преимущества настоящего изобретения будут очевидны из описания и графических материалов, а также из формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Фиг. 1A-1H. Анализ в репортерных клетках Jurkat с NFAT-люциферазой (JNL) с помощью автоматической системы применяли для тестирования функции CAR для BCMA. Клоны CAR оценивали в анализе в репортерных клетках JNL в отношении антигензависимой активности. Клетки JNL, содержащие указанные клоны CAR, или нетрансдуцированные клетки JNL (UTD) совместно культивировали со средой в отдельности (фиг. 1G и 1H) или с линиями клеток-мишеней (KMS11 в качестве BCMA-положительной линии клеток (фиг. 1A и 1C) и NALM6 в качестве BCMA-отрицательной линии клеток (фиг. 1E и 1F)) при различных соотношениях, и активность люциферазы измеряли в виде интенсивности люминесценции. Клоны считались активными, если интенсивность люминесценции в 2 раза превышала уровень в клетках UTD в присутствии клеток, экспрессирующих антиген. Регистрируемый показатель люминесценции является прямой мерой стимуляции CAR. Фиг. 1B и 1D представляют собой графики, на которых показан уровень экспрессии CAR для ВСМА на клетках JNL, выявленный с помощью проточной цитометрии с использованием рекомбинантного (r)BCMA_Fc-AF647 человека. Платформа 1x или 2x указывала на 40000 клеток H293 или 80000 клеток H293, высеянных для продуцирования вируса.
Фиг. 2. Уровень экспрессии CAR для BCMA на первичных Т-клетках человека. Клетки окрашивали реагентом rBCMA_Fc-AF647 человека и анализировали с помощью проточной цитометрии. Процентная доля CAR+ клеток и MFI показаны на графике для дня 5 и дня 9 культивирования клеток. Данные обобщены в таблице 27, которая включает титр вируса, достигнутый для соответствующих CAR.
Фиг. 3A-3C. Способность Т-клеток, экспрессирующих указанные CAR, опосредовать лизис клеток и продуцирование цитокинов оценивали в отношении линии клеток-мишеней KMS11, экспрессирующих люциферазу светлячков (KMS11-luc). Фиг. 3A. CAR-T-клетки совместно культивировали с клетками-мишенями KMS11-luc при указанных соотношениях E:T. % уничтожения клеток определяли по разнице в сигнале люциферазы между клетками-мишенями без эффекторных Т-клеток (контроль) и с эффекторными Т-клетками (эксперимент), выраженной в виде процента от контроля. UTD представляет нетрансдуцированные Т-клетки. Фиг. 3B. Фоновый уровень уничтожения наблюдался для BCMA-отрицательной линии NALM6. Фиг. 3C. Уровень IFNγ измеряли с помощью MSD в надосадочных жидкостях, собранных через 24 часа из этих систем совместного культивирования при соотношении Е:Т 2,5. Все данные выражены в виде среднего значения +/- стандартное отклонение.
Фиг. 4A-4C. Экспрессия CAR в Т-клетках, трансдуцированных при MOI=5 (титр вируса определен по первому CAR, экспрессируемому в клетках SupT1). На фиг. 4A представлена таблица, в которой обобщены % CAR19, % CAR для BCMA, % дважды положительных, % только CAR19 и % только CAR для BCMA для различных конструкций. На фиг. 4B представлен ряд графиков проточной цитометрии, на которых показано окрашивание клеток для выявления поверхностной экспрессии CAR для BCMA (ось x) и поверхностной экспрессии CAR для CD19 (ось y). На фиг. 4C представлена пара столбчатых диаграмм, на которых показана MFI для CAR для BCMA (верхняя панель) и MFI для CAR для CD19 (нижняя панель).
Фиг. 5A-5C. Анализ уничтожения in vitro с использованием CAR-T-клеток в день 8. На фиг. 5A-5C представлен ряд графиков, на которых показан % уничтожения BCMA-положительных клеток KMS11, CD19-положительных клеток Nalm6 или BCMA/CD19-отрицательных клеток соответственно при указанных соотношениях E:T.
Фиг. 6A-6D. Анализ продуцирования цитокинов in vitro с использованием CAR-T-клеток в день 8. На фиг. 6A-6D представлен ряд столбчатых диаграмм, на которых показано продуцирование IFN-гамма CAR-T-клетками при совместном культивировании с BCMA-положительными клетками KMS11 или CD19-положительными клетками Nalm6.
Фиг. 7A-7C. Индивидуальная экспрессия CAR в клетках, изготовленных с использованием способа ARM. На фиг. 7A-7B представлены гистограммы, на которых показан паттерн экспрессии как CAR для BCMA, так и CAR для CD19 через 24 часа или 72 часа после трансдукции первичных Т-клеток человека, изготовленных с использованием способа ARM. В исследованиях использовали MOI, составляющую 1, исходя из титра в SupT1, определенного по экспрессии вышерасположенного CAR. На каждой из фиг. 7A и 7B левая часть представляет собой панель гистограмм, на которых показано окрашивание с использованием rBCMA-Fc, а правая часть представляет собой панель гистограмм, на которых показано окрашивание с использованием антиидиотипического антитела, связывающегося с CAR для CD19. Конструкции № 244 ("c244") и № 245 ("c245") представляют собой моно-CAR для CD19 и моно-CAR для BCMA соответственно. На фиг. 7C представлена панель графиков проточной цитометрии, на которых показан паттерн экспрессии CAR для BCMA и CD19 через 72 часа после трансдукции первичных Т-клеток человека с использованием MOI, составляющей 1, исходя из титра для вышерасположенного CAR.
Фиг. 8A-8C. Противоопухолевая активность in vivo конструкции № 236 ("c236") и конструкции № 238 ("c238") с использованием трех мышиных моделей: модели диссеминированной множественной миеломы из KMS-11 (BCMA+ CD19-), экспрессирующей репортерный ген люциферазы (KMS11-Luc) (фиг. 8A), ксенотрансплантатной мышиной модели из Nalm6-Luc (CD19+BCMA-) (фиг. 8B) и смешанной модели из 95% клеток KMS-luc и 5% клеток NALM6-Luc (фиг. 8C). Опухолевая нагрузка выражена в виде люминесценции всего тела (фотонов/с) и изображена в виде среднего значения опухолевой нагрузки+SEM. В день 7 или 8 после инокуляции опухоли мышей обрабатывали с помощью c236 и c238 в обозначенных дозах BCMA-CAR+ или CD19-CAR+ Т-клеток (приблизительное количество жизнеспособных CAR+ Т-клеток), как показано в таблице 30. Среда-носитель (PBS) и нетрансдуцированные Т-клетки (UTD) служили в качестве отрицательных контролей. Также в качестве контролей использовали моно-CAR для BCMA PI61 и моно-CAR для CD19 CTL119.
Фиг. 9A-9C. Потеря массы тела, индуцированная реакцией "трансплантат против хозяина". У всех мышей индивидуально отслеживали потерю массы тела, что является регистрируемым показателем X-GvHD, путем измерения массы тела с течением времени. Массу тела (BWT) наносили на график в виде % изменения относительно исходного уровня.
Фиг. 10A-10C. Размножение CD3+ Т-клеток периферической крови in vivo анализировали с помощью проточной цитометрии в течение вплоть до 4 недель после инфузии.
Фиг. 11A-11C. Размножение CAR+ Т-клеток in vivo (процентную долю BCMA-CAR+) анализировали с помощью проточной цитометрии в течение вплоть до 4 недель после инфузии.
Фиг. 12A-12C. Размножение CAR+ Т-клеток in vivo (показатели количества дважды CAR+) анализировали с помощью проточной цитометрии в течение вплоть до 4 недель после инфузии.
Фиг. 13A-13C. Кинетические показатели IFNγ в плазме крови in vivo. Уровни IFNγ в плазме крови во всех трех мышиных моделях при обработке с помощью c236 и c238, а также моно-CAR в качестве контролей в соответствующих дозах CAR-T наносили на графики. У мышей отбирали кровь, и уровень цитокинов в плазме крови измеряли посредством MSD-анализа.
Фиг. 14A и 14B. Эффективность in vivo и размножение клеток у клеток, полученных с использованием 236 и c238, в ксенотрансплантатной мышиной модели множественной миеломы. Фиг. 14A. Мышам NSG инъецировали линию клеток множественной миеломы KMS11, которые экспрессировали репортерный ген люциферазы. Опухолевая нагрузка выражена в виде люминесценции всего тела (фотонов/с) и изображена в виде среднего значения опухолевой нагрузки+SEM. В день 8 после инокуляции опухоли мышей обрабатывали с помощью c236 и c238 в дозе 9e4 дважды BCMA/CD19-CAR+ T-клеток (приблизительное количество жизнеспособных CAR+ Т-клеток). Среда-носитель (PBS) и нетрансдуцированные Т-клетки (UTD) служили в качестве отрицательных контролей. Фиг. 14B. Размножение CAR+ Т-клеток периферической крови анализировали с помощью проточной цитометрии в течение вплоть до 4 недель после инфузии. Размножение дважды BCMA/CD19-CAR+ T-клеток наблюдалось в группах Rx CAR-T c236 и c238.
Фиг. 15A и 15B. Экспрессия CAR в клетках, изготовленных с использованием способа ARM. Графики проточной цитометрии, на которых показана экспрессия CAR для BCMA и CD19 в дважды положительных клетках через 96 часов (фиг. 15A) и 7 дней (фиг. 15B) после добавления вируса к первичным Т-клеткам человека, изготовленным с использованием способа ARM. В исследованиях использовали MOI, составляющую 2, исходя из титра в SupT1, определенного по экспрессии CAR в дважды положительных клетках (положительных по клонам PI61 или R1G5 и CTL119), выявленной с помощью антиидиотипического антитела, связывающегося с CAR для CD19, и рекомбинантного BCMA_Fc (AF647), связывающегося с PI61 или R1G5. Моно-BCMA-CAR-T PI61 и R1G5, а также моно-CD19-CAR-T CTL119 служили в качестве контролей.
Фиг. 16. Экспрессия CAR в день 7 в способе ТМ с использованием MOI, составляющей 5. Графики проточной цитометрии, на которых показана экспрессия CAR для BCMA и CD19 в дважды положительных клетках через 7 дней после добавления вируса к первичным Т-клеткам человека, изготовленным с использованием способа TM. В исследованиях использовали MOI, составляющую 5, исходя из титра в SupT1, определенного по экспрессии CAR в дважды положительных клетках (положительных по клонам PI61 или R1G5 и CTL119), выявленной с помощью антиидиотипического антитела, связывающегося с CAR для CD19, и рекомбинантного BCMA_Fc (AF647), связывающегося с PI61 или R1G5.
Фиг. 17A и 17B. Специфичное уничтожение in vitro опухолевых клеток, экспрессирующих BCMA или CD19, Т-клетками, сконструированными с использованием конструкций CAR для BCMA и CAR для CD19 в формате диатела. Способность Т-клеток, экспрессирующих клоны PI61/CTL119, опосредовать лизис клеток оценивали в отношении линии клеток-мишеней KMS11-Luc или NALM6-Luc. CAR-Т-клетки совместно культивировали с BCMA+ клетками-мишенями KMS-11-luc или BCMA- клетками-мишенями NALM6-Luc при указанных соотношениях E:T в течение 20 часов, и в качестве косвенного показателя лизиса клеток-мишеней измеряли % уничтожения клеток, определяемый по разнице в сигнале люциферазы между клетками-мишенями без эффекторных Т-клеток (контроль) и с эффекторными Т-клетками (эксперимент), выраженной в виде процента от контроля. UTD представляет нетрансдуцированные Т-клетки. Моноварианты PI61 или CTL119 служили в качестве контролей.
Фиг. 18A и 18B. Продуцирование цитокинов Т-клетками, сконструированными с использованием конструкций CAR для BCMA и CAR для CD19 в формате диатела, в ответ на опухолевые клетки, экспрессирующие BCMA или CD19. Уровни IFNγ (фиг. 18A) и IL-2 (фиг. 18B) измеряли с помощью MSD в надосадочных жидкостях совместной культуры из анализа уничтожения при соотношении 1,25:1.
Фиг. 19. Процентные доли дважды CAR-положительной популяции, положительной по CAR для BCMA популяции и положительной по CAR для CD19 популяции в день 4.
Фиг. 20. Графики проточной цитометрии, на которых показано окрашивание клеток с помощью rBCMA-Fc и антиидиотипического антитела, которое связывается с CAR для CD19.
Фиг. 21. Процентные доли общих CAR-положительных популяций в день 4 и день 7 в указанных условиях.
Фиг. 22. Показатели количества клеток (левая панель) и процентная доля живых клеток (правая панель) в дни 0, 1, 3 и 7 в указанных условиях.
Фиг. 23A, 23B и 23C. Данные секвенирования РНК одиночных клеток для исходных клеток (фиг. 23A), клеток дня 1 (фиг. 23B) и клеток дня 9 (фиг. 23C). Графики "n генов" демонстрируют количество экспрессируемых генов на клетку. Графики "n UMI" демонстрируют количество уникальных молекулярных идентификаторов (UMI) на клетку.
Фиг. 24A, 24B, 24C и 24D. Графики, построенные методом нелинейного снижения размерности и визуализации многомерных переменных (TSNE), на которых сравниваются исходные клетки (фиг. 24A), клетки дня 1 (фиг. 24B) и клетки дня 9 (фиг. 24C) в отношении сигнатуры пролиферации, которую определяли на основании экспрессии генов CCNB1, CCND1, CCNE1, PLK1 и MKI67. Каждая точка представляет собой клетку в рассматриваемом образце. Клетки, показанные светло-серым цветом, не экспрессируют гены, отвечающие за пролиферацию, тогда как клетки, заштрихованные темным цветом, экспрессируют один или несколько генов, отвечающих за пролиферацию. Фиг. 24D представляет собой скрипичный график, на котором показано распределение баллов набора генов для набора генов, состоящего из генов, которые характеризуют покоящееся состояние T-клеток, относительно таковых, которые характеризуют активированное состояние T-клеток, для клеток дня 1, клеток дня 9 и исходных клеток. На фиг. 24D более высокий балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) указывает на увеличение выраженности фенотипа покоящихся Т-клеток, тогда как более низкий балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) указывает на увеличение выраженности фенотипа активированных T-клеток. Исходные клетки в целом были в большинстве своем в покоящемся состоянии по сравнению с клетками дня 9 и дня 1. Клетки дня 1 демонстрируют наиболее высокий балл набора генов, отвечающих за активацию.
Фиг. 25A, 25B, 25C, 25D и 25E. Анализ наборов генов для исходных клеток, клеток дня 1 и клеток дня 9. На фиг. 25A более высокий балл набора генов для набора генов "активируемые в TEM относительно подавляемых в TSCM" указывает на увеличение выраженности фенотипа эффекторных Т-клеток памяти (TEM) в рассматриваемом образце, тогда как более низкий балл набора генов указывает на увеличение выраженности фенотипа стволовых T-клеток памяти (TSCM). На фиг. 25B более высокий балл набора генов для набора генов "активируемые в Treg относительно подавляемых в Teff" указывает на увеличение выраженности фенотипа регуляторных Т-клеток (Treg), тогда как более низкий балл набора генов указывает на увеличение выраженности фенотипа эффектoрных Т-клеток (Teff). На фиг. 25C более низкий балл набора генов для набора генов "подавляемые в состоянии стволовости" указывает на увеличение выраженности фенотипа стволовости. На фиг. 25D более высокий балл набора генов для набора генов "активируемые при гипоксии" указывает на увеличение выраженности фенотипа, характерного для гипоксии. На фиг. 25E более высокий балл набора генов для набора генов "активируемые при аутофагии" указывает на увеличение выраженности фенотипа, характерного для аутофагии. Клетки дня 1 выглядели похожими на исходные клетки в отношении сигнатуры памяти, подобия стволовым клеткам и дифференцировки. Клетки дня 9 в то же время демонстрируют более высокую степень выраженности метаболического стресса.
Фиг. 26A, 26B и 26C. Анализ генов в кластерах исходных клеток. Фиг. 26A-26C представляют собой скрипичные графики, на которых показаны баллы наборов генов из анализа наборов генов в четырех кластерах исходных клеток. Каждая точка, лежащая на скрипичных графиках на фиг. 26A-26C, представляет балл набора генов в клетке. На фиг. 26A более высокий балл набора генов для набора генов "активируемые в Treg относительно подавляемых в Teff" указывает на увеличение выраженности фенотипа Treg-клеток, тогда как более низкий балл набора генов для набора генов "активируемые в Treg относительно подавляемых в Teff" указывает на увеличение выраженности фенотипа Teff-клеток. На фиг. 26B более высокий балл набора генов для набора генов "постепенно активируемые при дифференцировке в клетки памяти" указывает на увеличение выраженности фенотипа поздних Т-клеток памяти, тогда как более низкий балл набора генов для набора генов "постепенно активируемые при дифференцировке в клетки памяти" указывает на увеличение выраженности фенотипа ранних Т-клеток памяти. На фиг. 26C более высокий балл набора генов для набора генов "активируемые в TEM относительно подавляемых в TN" указывает на увеличение выраженности фенотипа эффекторных Т-клеток памяти, тогда как более низкий балл набора генов для набора генов "активируемые в TEM относительно подавляемых в TN" указывает на увеличение выраженности фенотипа необученных Т-клеток памяти. Показано, что клетки в кластере 3 находились в состоянии поздних, более дифференцированных Т-клеток памяти по сравнению с клетками в кластере 1 и кластере 2, которые находились в состоянии ранних, менее дифференцированных Т-клеток памяти. Кластер 0, по-видимому, соответствует промежуточному состоянию Т-клеток. Взятые вместе, эти данные демонстрируют, что среди исходных клеток имеется значительный уровень гетерогенности.
Фиг. 27A, 27B и 27C. Секвенирование TCR и измерение разнообразия клонотипов. Клетки дня 9 характеризовались более плосковершинным распределением частот клонотипов (более высоким разнообразием).
Фиг. 28A и 28B. Анализы методом проточной цитометрии для определения экспрессии CAR в дни 4 и 7 после трансдукции. Анализы методом проточной цитометрии для CAR-T-клеток, полученных путем котрансдукции клеток с помощью векторов, кодирующих CAR для BCMA и CD19, при разных комбинациях MOI с использованием способа ARM в 24-луночном планшете. Фиг. 28A. Графики проточной цитометрии, на которых показана экспрессия моно-CAR для BCMA, моно-CAR для CD19 и CAR в дважды+ клетках в дни 4 и 7 после трансдукции в условиях четырех разных MOI в дополнение к контролям (UTD и одиночный вектор). Фиг. 28B. Количественное оценивание субпопуляций в CAR+ популяциях, включающих общую популяцию BCMA-CAR+ Т-клеток, общую популяцию CD19-CAR+ Т-клеток, а также общую популяцию CAR+ Т-клеток (сумму двух групп моно-CAR+ Т-клеток и дважды CAR+ Т-клеток) при каждом условии, как описано на фиг. 28A. Приведенные данные являются иллюстративными для Т-клеток от трех доноров с согласующимися результатами. Процентные доли CAR+ клеток гейтированы по популяции живых CD3+ T-клеток.
Фиг. 29. Анализы методом проточной цитометрии для определения экспрессии CAR в день 4 после трансдукции. Анализы методом проточной цитометрии конечных продуктов в виде коктейля CAR-T с двойным нацеливанием, моно-BCMA-CAR-T и моно-CD19-CAR-T в отношении экспрессии CAR в день 4 после трансдукции. Небольшие аликвоты каждого продукта, собранные через 24 часа, повторно культивировали в течение трех дней перед окрашиванием в ходе проточной цитометрии.
Фиг. 30A, 30B, 30C, 30D и 30E. Эффективность in vivo CAR-T с двойной специфичностью по сравнению с моно-BCMA-CAR-T и CD19-CAR-T в ксенотрансплантатных моделях. Мышам NSG инъецировали линии клеток, экспрессирующие репортерный ген люциферазы (KMS-11, или Nalm-6, или смесь обоих с 5% Nalm-6-luc). Опухолевая нагрузка выражена в виде люминесценции всего тела (фотонов/с) и изображена в виде среднего значения опухолевой нагрузки+SEM. В день 7 или 8 после инокуляции опухоли мышей обрабатывали с помощью коктейля CAR-T с двойным нацеливанием, BCMA-CAR-T или CD19-CAR-T в соответствующих дозах (приблизительное количество жизнеспособных CAR+ Т-клеток). Среда-носитель (PBS) и нетрансдуцированные Т-клетки (UTD) служили в качестве отрицательных контролей. N=5 мышей для всех групп. BCMA-CAR-T и CD19-CAR-T служили в качестве соответствующих контролей при использовании наивысшего уровня дозы. Все эксперименты завершали в день 23 после введения CAR-T.
Фиг. 31. На фиг. 31 представлена столбчатая диаграмма, на которой показан % моно-CD19-CAR+ клеток, % моно-BCMA-CAR+ клеток и % дважды BCMA/CD19-CAR+ клеток в день 4 после трансдукции (день 3 после сбора).
Фиг. 32. Определение характеристик субпопуляций T-клеток. На фиг. 32 представлен график, на котором показан % CD4+ Т-клеток, CD8+ Т-клеток, необученных Т-клеток (Tn), центральных Т-клеток памяти (Tcm), эффекторных Т-клеток памяти (Tem) и эффекторных Т-клеток памяти, реэкспрессирующих CD45RA (TEMRA), в исходном материале, материале после обогащения и в материале после сбора в день 1.
Фиг. 33A и 33B. Кинетические показатели IFNγ в плазме крови мышей, обработанных клеточным продуктом на основе CAR-T с двойной специфичностью к BCMA/CD19, BCMA-CAR-T и CD19-CAR-T. Животных обрабатывали с помощью PBS, UTD, клеточного продукта на основе CAR-T с двойной специфичностью к BCMA/CD19, BCMA-CAR-T или CD19-CAR-T в соответствующих дозах CAR-T. У мышей отбирали кровь, и уровень цитокинов в плазме крови измеряли посредством MSD-анализа.
ПОДРОБНОЕ ОПИСАНИЕ
Определения
Если не указано иное, то все технические и научные термины, используемые в данном документе, имеют то же значение, которое обычно понятно специалисту средней квалификации в области техники, к которой относится настоящее изобретение.
Форма единственного числа относится к одному или нескольким (т. e. по меньшей мере одному) грамматическим объектам настоящей заявки. В качестве примера, "элемент" означает один элемент или несколько элементов.
Подразумевается, что термин "приблизительно" в отношении измеряемой величины, такой как количество, временной промежуток и т. п., охватывает отклонения на ± 20%, или в некоторых случаях на ± 10%, или в некоторых случаях на ± 5%, или в некоторых случаях на ± 1%, или в некоторых случаях на ± 0,1% от указанного значения, поскольку такие отклонения являются допустимыми для осуществления раскрытых способов.
Композиции и способы по настоящему изобретению охватывают полипептиды и нуклеиновые кислоты, имеющие указанные последовательности или последовательности, по существу идентичные им или сходные с ними, например, последовательности, идентичные указанной последовательности на по меньшей мере 85%, 90% или 95% или больше. Применительно к аминокислотной последовательности термин "по существу идентичная" используется в данном документе для обозначения первой аминокислотной последовательности, которая содержит достаточное или минимальное количество аминокислотных остатков, которые i) идентичны аминокислотным остаткам во второй аминокислотной последовательности после их выравнивания или ii) являются их консервативными заменами, так что первая и вторая аминокислотные последовательности могут иметь общий структурный домен и/или характеризоваться общей функциональной активностью, например, аминокислотные последовательности, которые содержат общий структурный домен, характеризуются по меньшей мере приблизительно 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичностью с эталонной последовательностью, например, последовательностью, представленной в данном документе.
Применительно к нуклеотидной последовательности термин "по существу идентичная" используется в данном документе для обозначения первой последовательности нуклеиновой кислоты, которая содержит достаточное или минимальное количество нуклеотидов, которые идентичны нуклеотидам во второй последовательности нуклеиновой кислоты после их выравнивания, так что первая и вторая нуклеотидные последовательности кодируют полипептиды, характеризующиеся общей функциональной активностью, или кодируют общий структурный домен полипептидов или отвечают за общую функциональную активность полипептидов, например, нуклеотидные последовательности характеризуются по меньшей мере приблизительно 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичностью с эталонной последовательностью, например, последовательностью, представленной в данном документе.
Термин "вариант" относится к полипептиду, который имеет аминокислотную последовательность, по существу идентичную эталонной аминокислотной последовательности, или кодируется по существу идентичной нуклеотидной последовательностью. В некоторых вариантах осуществления вариант представляет собой функциональный вариант.
Термин "функциональный вариант" относится к полипептиду, который имеет аминокислотную последовательность, по существу идентичную эталонной аминокислотной последовательности, или кодируется по существу идентичной нуклеотидной последовательностью и способен характеризоваться одним или несколькими видами активности эталонной аминокислотной последовательности.
Термин "цитокин" (например, IL-2, IL-7, IL-15, IL-21 или IL-6) включает полноразмерный встречающийся в природе цитокин, его фрагмент или вариант, например, функциональный вариант (в том числе его фрагменты и функциональные варианты, характеризующиеся по меньшей мере 10%, 30%, 50% или 80% активности, например, иммуномодулирующей активности, встречающегося в природе цитокина). В некоторых вариантах осуществления цитокин имеет аминокислотную последовательность, которая по существу идентична (например, идентична на по меньшей мере приблизительно 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%) встречающемуся в природе цитокину или кодируется нуклеотидной последовательностью, которая по существу идентична (например, идентична на по меньшей мере приблизительно 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%) встречающейся в природе нуклеотидной последовательности, кодирующей цитокин. В некоторых вариантах осуществления, как понятно из контекста, цитокин дополнительно содержит рецепторный домен, например, домен, представляющий собой рецептор цитокина (например, IL-15/IL-15R).
Используемый в данном документе термин "ВСМА" относится к антигену созревания В-клеток. BCMA (также известный как TNFRSF17, BCM или CD269) является представителем семейства рецепторов фактора некроза опухоли (TNFR) и преимущественно экспрессируется на терминально дифференцированных B-клетках, например, B-клетках памяти и плазматических клетках. Его лиганд называют B-клеточным активатором семейства TNF (BAFF) и лигандом, индуцирующим пролиферацию (APRIL). BCMA вовлечен в опосредование выживания плазматических клеток для поддержания долгосрочного гуморального иммунитета. Ген BCMA кодируется в хромосоме 16, продуцируя первичный мРНК-транскрипт длиной 994 нуклеотида (№ доступа в NCBI NM_001192.2), который кодирует белок из 184 аминокислот (NP_001183.2). Был описан второй антисмысловой транскрипт, происходящий из локуса BCMA, который может играть роль в регуляции экспрессии BCMA. (Laabi Y. et al., Nucleic Acids Res., 1994, 22:1147-1154). Были описаны дополнительные варианты транскрипта с неизвестным значением (Smirnova AS et al. Mol Immunol., 2008, 45(4):1179-1183). Была идентифицирована вторая изоформа, также известная как TV4 (идентификатор UniProt Q02223-2). Как используется в данном документе, "BCMA" включает белки, содержащие мутации, например, точечные мутации, фрагменты, вставки, делеции и сплайс-варианты полноразмерного BCMA дикого типа.
Фраза "заболевание, ассоциированное с экспрессией BCMA" включает без ограничения заболевание, ассоциированное с клеткой, которая экспрессирует BCMA (например, BCMA дикого типа или мутантный BCMA), или состояние, ассоциированное с клеткой, которая экспрессирует BCMA (например, BCMA дикого типа или мутантный BCMA), в том числе, например, пролиферативные заболевания, такие как рак или злокачественное новообразование, или предраковое состояние, такое как миелодисплазия, миелодиспластический синдром или предлейкоз; или показание, не связанное с раком, ассоциированное с клеткой, которая экспрессирует BCMA (например, BCMA дикого типа или мутантный BCMA). Во избежание неоднозначности, заболевание, ассоциированное с экспрессией ВСМА, может включать состояние, ассоциированное с клеткой, которая в настоящее время не экспрессирует ВСМА, например, из-за того, что экспрессия ВСМА была подавлена, например, вследствие лечения молекулой, нацеливающейся на ВСМА, например, ингибитором ВСМА, описанным в данном документе, но которая в свое время экспрессировала ВСМА. В одном аспекте рак, ассоциированный с экспрессией ВСМА (например, ВСМА дикого типа или мутантного ВСМА), представляет собой гематологический рак. В одном аспекте гематологический рак представляет собой лейкоз или лимфому. В одном аспекте рак, ассоциированный с экспрессией ВСМА (например, ВСМА дикого типа или мутантного ВСМА), представляет собой злокачественное новообразование из дифференцированных плазматических В-клеток. В одном аспекте рак, ассоциированный с экспрессией BCMA (например, BCMA дикого типа или мутантного BCMA), включает формы рака и злокачественные новообразования, в том числе без ограничения, например, одну или несколько форм острого лейкоза, в том числе без ограничения, например, B-клеточный острый лимфоидный лейкоз ("BALL"), Т-клеточный острый лимфоидный лейкоз ("TALL"), острый лимфоидный лейкоз (ALL); одну или несколько форм хронического лейкоза, в том числе без ограничения, например, хронический миелогенный лейкоз (CML), хронический лимфоидный лейкоз (CLL). Дополнительные формы рака или гематологические состояния, ассоциированные с экспрессией BMCA (например, BCMA дикого типа или мутантного BCMA), включают без ограничения, например, В-клеточный пролимфоцитарный лейкоз, новообразование из бластных плазмоцитоидных дендритных клеток, лимфому Беркитта, диффузную крупноклеточную B-клеточную лимфому, фолликулярную лимфому, волосатоклеточный лейкоз, мелкоклеточную или крупноклеточную фолликулярную лимфому, злокачественные лимфопролиферативные состояния, MALT-лимфому, мантийноклеточную лимфому, лимфому из клеток маргинальной зоны, множественную миелому, миелодисплазию и миелодиспластический синдром, неходжкинскую лимфому, плазмобластную лимфому, новообразование из плазмоцитоидных дендритных клеток, макроглобулинемию Вальденстрема и "предлейкоз", который представляет собой разнообразную группу гематологических состояний, объединенных неэффективным образованием (или дисплазией) миелоидных клеток крови, и т. п. В некоторых вариантах осуществления рак представляет собой множественную миелому, лимфому Ходжкина, неходжкинскую лимфому или глиобластому. В ряде вариантов осуществления заболевание, ассоциированное с экспрессией ВСМА, включает нарушение пролиферации плазматических клеток, например, бессимптомную миелому ("тлеющую" множественную миелому или вялотекущую миелому), моноклональную гаммапатию неустановленной этиологии (MGUS), макроглобулинемию Вальденстрема, формы плазмоцитомы (например, дискразию плазматических клеток, солитарную миелому, солитарную плазмоцитому, экстрамедуллярную плазмоцитому и множественную плазмоцитому), системный амилоидоз в форме амилоидоза легких цепей и POEMS-синдром (также известный как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром). Дополнительные заболевания, ассоциированные с экспрессией BCMA (например, BCMA дикого типа или мутантного BCMA), включают без ограничения, например, атипичные и/или неклассические формы рака, злокачественные новообразования, предраковые состояния или пролиферативные заболевания, ассоциированные с экспрессией BCMA (например, BCMA дикого типа или мутантного BCMA), например, рак, описанный в данном документе, например, рак предстательной железы (например, кастрационнорезистентный или резистентный к терапии рак предстательной железы или метастатический рак предстательной железы), рак поджелудочной железы или рак легкого.
Состояния, не связанные с раком, которые ассоциированы с ВСМА (например, ВСМА дикого типа или мутантным ВСМА), включают вирусные инфекции; например, вызванные HIV, грибковые инфекции, например, вызванные C. neoformans; аутоиммунное заболевание; например, ревматоидный артрит, системную красную волчанку (SLE или волчанку), пузырчатку обыкновенную и синдром Шегрена; воспалительное заболевание кишечника, неспецифический язвенный колит; аллоспецифический иммунный ответ, связанный с трансплантацией, нарушения, связанные с иммунитетом слизистых оболочек; и нежелательные иммунные ответы на биопрепараты (например, фактор VIII), при которых важен гуморальный иммунитет. В ряде вариантов осуществления показание, не связанное с раком, ассоциированное с экспрессией BCMA, включает без ограничения, например, аутоиммунное заболевание (например, волчанку), воспалительные нарушения (аллергию и астму) и трансплантацию. В некоторых вариантах осуществления клетка, экспрессирующая опухолевый антиген, экспрессирует или в какое-либо время экспрессировала мРНК, кодирующую опухолевый антиген. В одном варианте осуществления клетка, экспрессирующая опухолевый антиген, продуцирует опухолевый антигенный белок (например, дикого типа или мутантный), и при этом опухолевый антигенный белок может присутствовать на нормальных уровнях или сниженных уровнях. В одном варианте осуществления клетка, экспрессирующая опухолевый антиген, в один момент времени продуцировала опухолевый антигенный белок на выявляемых уровнях, а затем по существу не продуцировала опухолевый антигенный белок на выявляемых уровнях.
Термин "химерный антигенный рецептор" или в качестве альтернативы "CAR" или "молекула CAR" относится к рекомбинантной полипептидной конструкции, содержащей по меньшей мере внеклеточный антигенсвязывающий домен, трансмембранный домен и цитоплазматический сигнальный домен (также называемый в данном документе "внутриклеточным сигнальным доменом"), содержащий функциональный сигнальный домен, полученный из стимулирующей молекулы, определенной ниже. В некоторых вариантах осуществления домены в полипептидной конструкции CAR находятся в одной и той же полипептидной цепи, например, образуют химерный слитый белок. В некоторых вариантах осуществления домены в полипептидной конструкции CAR не являются смежными друг с другом, например, находятся в разных полипептидных цепях, например, как представлено в RCAR, описанном в данном документе.
В некоторых вариантах осуществления цитоплазматический сигнальный домен содержит первичный сигнальный домен (например, первичный сигнальный домен CD3-дзета). В некоторых вариантах осуществления цитоплазматический сигнальный домен дополнительно содержит один или несколько функциональных сигнальных доменов, полученных из по меньшей мере одной костимулирующей молекулы, определенной ниже. В некоторых вариантах осуществления костимулирующая молекула выбрана из 4-1BB (т. е. CD137), CD27, ICOS и/или CD28. В некоторых вариантах осуществления CAR содержит химерный слитый белок, содержащий внеклеточный антигенраспознающий домен, трансмембранный домен и внутриклеточный сигнальный домен, содержащий функциональный сигнальный домен, полученный из стимулирующей молекулы. В некоторых вариантах осуществления CAR содержит химерный слитый белок, содержащий внеклеточный антигенраспознающий домен, трансмембранный домен и внутриклеточный сигнальный домен, содержащий функциональный сигнальный домен, полученный из костимулирующей молекулы, и функциональный сигнальный домен, полученный из стимулирующей молекулы. В некоторых вариантах осуществления CAR содержит химерный слитый белок, содержащий внеклеточный антигенраспознающий домен, трансмембранный домен и внутриклеточный сигнальный домен, содержащий два функциональных сигнальных домена, полученных из одной или нескольких костимулирующих молекул, и функциональный сигнальный домен, полученный из стимулирующей молекулы. В некоторых вариантах осуществления CAR содержит химерный слитый белок, содержащий внеклеточный антигенраспознающий домен, трансмембранный домен и внутриклеточный сигнальный домен, содержащий по меньшей мере два функциональных сигнальных домена, полученных из одной или нескольких костимулирующих молекул, и функциональный сигнальный домен, полученный из стимулирующей молекулы. В некоторых вариантах осуществления CAR содержит необязательную лидерную последовательность на амино-конце (N-конце) слитого белка CAR. В некоторых вариантах осуществления CAR дополнительно содержит лидерную последовательность на N-конце внеклеточного антигенраспознающего домена, где лидерная последовательность необязательно отщепляется от антигенраспознающего домена (например, scFv) в ходе клеточного процессинга и локализации CAR в клеточной мембране.
CAR, содержащий антигенсвязывающий домен (например, scFv, однодоменное антитело или TCR (например, связывающий домен альфа-цепи TCR или связывающий домен бета-цепи TCR)), который нацеливается на конкретный опухолевый маркер X, где X может быть опухолевым маркером, описанным в данном документе, также называется XCAR. Например, CAR, который содержит антигенсвязывающий домен, нацеливающийся на BCMA, называется CAR для BCMA. CAR может экспрессироваться в любой клетке, например, иммунной эффекторной клетке, описанной в данном документе (например, T-клетке или NK-клетке).
Термин "сигнальный домен" относится к функциональной части белка, которая действует путем передачи информации внутри клетки для регуляции клеточной активности посредством определенных сигнальных путей, образуя вторичные мессенджеры или функционируя в качестве эффекторов путем ответа на такие мессенджеры.
Используемый в данном документе термин "антитело" относится к последовательности белка или полипептида, полученной из молекулы иммуноглобулина, который специфично связывается с антигеном. Антитела могут являться поликлональными или моноклональными, многоцепочечными или одноцепочечными или интактными иммуноглобулинами и могут быть получены из природных источников или из рекомбинантных источников. Антитела могут являться тетрамерными молекулами иммуноглобулинов.
Термин "фрагмент антитела" относится к по меньшей мере одной части интактного антитела или его рекомбинантных вариантов и относится к антигенсвязывающему домену, например, антигенраспознающей вариабельной области интактного антитела, достаточной для обеспечения распознавания и специфичного связывания фрагмента антитела с мишенью, такой как антиген. Примеры фрагментов антител включают без ограничения Fab-, Fab'-, F(ab')2- и Fv-фрагменты, scFv-фрагменты антител, линейные антитела, однодоменные антитела, такие как sdAb (VL либо VH), VHH-домены верблюдовых и полиспецифические молекулы, образованные из фрагментов антител, такие как бивалентный фрагмент, содержащий два или более, например, два, Fab-фрагмента, соединенных дисульфидным мостиком в шарнирной области, или две или более, например, две, связанные выделенные CDR или другие эпитопсвязывающие фрагменты антитела. Фрагмент антитела также может быть включен в состав однодоменных антител, максиантител, миниантител, нанотел, интрател, диател, триател, тетрател, v-NAR и бис-scFv (см., например, Hollinger and Hudson, Nature Biotechnology 23:1126-1136, 2005). Фрагменты антитела также могут быть пересажены на каркасные структуры на основе полипептидов, таких как фибронектин III типа (Fn3) (см. патент США № 6703199, в котором описаны миниантитела на основе полипептида фибронектина).
Термин "scFv" относится к слитому белку, содержащему по меньшей мере один фрагмент антитела, содержащий вариабельную область легкой цепи, и по меньшей мере один фрагмент антитела, содержащий вариабельную область тяжелой цепи, где вариабельные области легкой и тяжелой цепи присоединены друг к другу в смежном положении посредством короткого гибкого полипептидного линкера и способны экспрессироваться в виде одноцепочечного полипептида, и где scFv сохраняет специфичность интактного антитела, из которого он получен. Если не указано иное, то scFv, как используется в данном документе, может иметь вариабельные VL- и VH-области, расположенные в любом порядке, например, относительно N-конца и С-конца полипептида scFv может содержать VL-линкер-VH или может содержать VH-линкер-VL. В некоторых вариантах осуществления scFv может содержать структуру NH2-VL-линкер-VH-COOH или NH2-VH-линкер-VL-COOH.
Термины "область, определяющая комплементарность" или "CDR", используемые в данном документе, относятся к последовательностям из аминокислот в вариабельных областях антитела, которые придают антигенную специфичность и аффинность связывания. Например, как правило, в каждой вариабельной области тяжелой цепи находятся три CDR (HCDR1, HCDR2 и HCDR3), и в каждой вариабельной области легкой цепи находятся три CDR (LCDR1, LCDR2 и LCDR3). Точные границы аминокислотной последовательности указанной CDR могут быть определены с использованием любой из ряда хорошо известных схем, в том числе описанных в Kabat et al. (1991), "Sequences of Proteins of Immunological Interest", 5th Ed. Public Health Service, National Institutes of Health, Bethesda, MD (схема нумерации "по Kabat"), Al-Lazikani et al., (1997) JMB 273,927-948 (схема нумерации "по Chothia") или их комбинации. Согласно комбинированной схеме нумерации по Kabat и Chothia в некоторых вариантах осуществления CDR соответствуют аминокислотным остаткам, которые являются частью CDR по Kabat, CDR по Chothia или как тех, так и других.
Часть композиции на основе CAR по настоящему изобретению, предусматривающая соответствующее антитело или фрагмент антитела, может существовать в различных формах, например, в которых антигенсвязывающий домен экспрессируется в виде части полипептидной цепи, в том числе, например, в виде фрагмента однодоменного антитела (sdAb), одноцепочечного антитела (scFv) или, например, человеческого или гуманизированного антитела (Harlow et al., 1999, в: Using Antibodies: A Laboratory Manual, Cold Spring Harbor Laboratory Press, NY; Harlow et al., 1989, в: Antibodies: A Laboratory Manual, Cold Spring Harbor, New York; Houston et al., 1988, Proc. Natl. Acad. Sci. USA 85:5879-5883; Bird et al., 1988, Science 242:423-426). В некоторых вариантах осуществления антигенсвязывающий домен в композиции на основе CAR по настоящему изобретению содержит фрагмент антитела. В некоторых вариантах осуществления CAR содержит фрагмент антитела, который содержит scFv.
Используемый в данном документе термин "связывающий домен" или "молекула антитела" (также называемые в данном документе связывающим доменом для мишени) относится к белку, например, к цепи иммуноглобулина или ее фрагменту, содержащему по меньшей мере одну последовательность вариабельного домена иммуноглобулина. Термин "связывающий домен" или "молекула антитела" охватывает антитела и фрагменты антител. В некоторых вариантах осуществления молекула антитела представляет собой молекулу полиспецифического антитела, например, она содержит множество последовательностей вариабельных доменов иммуноглобулина, причем первая последовательность вариабельного домена иммуноглобулина из множества характеризуется специфичностью связывания с первым эпитопом, а вторая последовательность вариабельного домена иммуноглобулина из множества характеризуется специфичностью связывания со вторым эпитопом. В некоторых вариантах осуществления молекула полиспецифического антитела представляет собой молекулу биспецифического антитела. Биспецифическое антитело характеризуется специфичностью в отношении не более чем двух антигенов. Молекула биспецифического антитела характеризуется наличием первой последовательности вариабельного домена иммуноглобулина, которая характеризуется специфичностью связывания с первым эпитопом, и второй последовательности вариабельного домена иммуноглобулина, которая характеризуется специфичностью связывания со вторым эпитопом.
Термины "биспецифическое антитело" и "биспецифические антитела" относятся к молекулам, в которых объединены антигенсвязывающие участки двух антител в пределах одной молекулы. Таким образом, биспецифическое антитело способно связывать два разных антигена одновременно или последовательно. Способы получения биспецифических антител хорошо известны из уровня техники. Различные форматы объединения двух антител также известны из уровня техники. Формы биспецифических антител по настоящему изобретению включают без ограничения диатело, одноцепочечное диатело, продукт димеризации Fab (Fab-Fab), Fab-scFv и тандемное антитело, как известно специалистам в данной области.
Термин "тяжелая цепь антитела" относится к большей из полипептидных цепей двух типов, присутствующих в молекулах антител в их встречающихся в природе конформациях, которая обычно определяет класс, к которому принадлежит антитело.
Термин "легкая цепь антитела" относится к меньшей из полипептидных цепей двух типов, присутствующих в молекулах антител в их встречающихся в природе конформациях. Легкие каппа- (κ-) и лямбда- (λ-) цепи относятся к двум основным изотипам легких цепей антител.
Термин "рекомбинантное антитело" относится к антителу, образованному с помощью технологии рекомбинантных ДНК, такому как, например, антитело, экспрессируемое в бактериофаговой или дрожжевой системе экспрессии. Термин также следует истолковывать как означающий антитело, которое было образовано в результате синтеза молекулы ДНК, кодирующей антитело, при этом данная молекула ДНК экспрессирует белковое антитело или аминокислотную последовательность, определяющую антитело, где последовательность ДНК или аминокислотная последовательность были получены с помощью технологии рекомбинантных ДНК или аминокислотных последовательностей, доступной и хорошо известной из уровня техники.
Термин "антиген" или "Ag" относится к молекуле, которая вызывает иммунный ответ. Такой иммунный ответ может предусматривать продуцирование антител либо активацию специфических иммунокомпетентных клеток или как то, так и другое. Специалисту в данной области будет понятно, что любая макромолекула, в том числе практически все белки или пептиды, может служить в качестве антигена. Кроме того, антигены могут быть получены из рекомбинантной или геномной ДНК. Специалисту в данной области будет понятно, что любая ДНК, которая содержит нуклеотидную последовательность или частичную нуклеотидную последовательность, кодирующую белок, который вызывает иммунный ответ, тем самым кодирует "антиген", как этот термин используется в данном документе. Кроме того, специалисту в данной области будет понятно, что антиген не должен кодироваться исключительно полноразмерной нуклеотидной последовательностью гена. Очевидно, что настоящее изобретение включает без ограничения применение частичных нуклеотидных последовательностей более одного гена, и что эти нуклеотидные последовательности составлены в различных комбинациях для того, чтобы кодировать полипептиды, которые вызывают требуемый иммунный ответ. Более того, специалисту в данной области будет понятно, что антиген вообще не обязательно должен кодироваться "геном". Очевидно, что антиген может быть образован в результате синтеза, или может быть получен из биологического образца, или может представлять собой макромолекулу, отличную от полипептида. Такой биологический образец может включать без ограничения образец ткани, образец опухоли, клетку или жидкость с другими биологическими компонентами.
Термины "противоопухолевый эффект" и "противораковый эффект" используются взаимозаменяемо и относятся к биологическому эффекту, который может проявляться различным образом, в том числе без ограничения, например, в виде уменьшения объема опухоли или объема ракового образования, уменьшения количества опухолевых клеток или раковых клеток, уменьшения количества метастазов, увеличения ожидаемой продолжительности жизни, уменьшения уровня пролиферации опухолевых клеток или пролиферации раковых клеток, уменьшения выживаемости опухолевых клеток или выживаемости раковых клеток или ослабления различных физиологических симптомов, ассоциированных с раковым состоянием. "Противоопухолевый эффект" или "противораковый эффект" также может проявляться в виде способности пептидов, полинуклеотидов, клеток и антител по настоящему изобретению в первую очередь предупреждать появление опухоли или рака.
Термин "аутологичный" относится к любому материалу, полученному от того же индивидуума, которому он позднее должен быть повторно введен.
Термин "аллогенный" относится к любому материалу, полученному от другого животного того же вида, что и индивидуум, которому данный материал вводят. Говорят, что два или более индивидуума являются аллогенными по отношению друг к другу, если их гены в одном или нескольких локусах не являются идентичными. В некоторых вариантах осуществления аллогенный материал от индивидуумов одного и того же вида может характеризоваться степенью генетической разнородности, достаточной для их антигенного взаимодействия.
Термин "ксеногенный" относится к трансплантату, полученному от животного другого вида.
Термин "аферез", используемый в данном документе, относится к признанному в данной области экстракорпоральному способу, при котором кровь донора или пациента отбирают у донора или пациента и пропускают через аппарат, который отделяет выбранные определенные составляющие компоненты, а остальную часть возвращает в кровоток донору или пациенту, например, посредством ретрансфузии. Таким образом, применительно к "образцу, полученному путем афереза" подразумевают образец, полученный с использованием афереза.
Термин "рак" относится к заболеванию, которое характеризуется быстрым и неконтролируемым ростом аберрантных клеток. Раковые клетки могут распространяться локально или через кровоток и лимфатическую систему в другие части организма. Примеры различных форм рака описаны в данном документе и включают без ограничения рак молочной железы, рак предстательной железы, рак яичника, рак шейки матки, рак кожи, рак поджелудочной железы, колоректальный рак, рак почки, рак печени, рак головного мозга, лимфому, лейкоз, рак легкого и т. п. В некоторых вариантах осуществления формы рака, лечение которых осуществляют посредством способов, описанных в данном документе, включают множественную миелому, лимфому Ходжкина или неходжкинскую лимфому.
Термины "опухоль" и "рак" используются в данном документе взаимозаменяемо, например, оба термина охватывают солидные опухоли и опухоли жидких тканей, например, диффузные или циркулирующие. Используемый в данном документе термин "рак" или "опухоль" включает предзлокачественные, а также злокачественные формы рака и опухоли.
Используемый в данном документе термин "полученный из" указывает на взаимосвязь между первой и второй молекулами. Это обычно относится к структурному сходству между первой молекулой и второй молекулой и не имеет дополнительного значения или не включает ограничение способа или источника в отношении первой молекулы, которая получена из второй молекулы. Например, в случае с внутриклеточным сигнальным доменом, который получен из молекулы CD3-дзета, внутриклеточный сигнальный домен сохраняет структуру CD3-дзета в достаточной мере для того, чтобы обладать необходимой функцией, а именно способностью образовывать сигнал в подходящих условиях. Это не имеет дополнительного значения или не включает ограничение в отношении конкретного способа получения внутриклеточного сигнального домена, например, это не означает, что для обеспечения наличия внутриклеточного сигнального домена нужно начинать с последовательности CD3-дзета и удалять нежелательную последовательность или вводить мутации для получения внутриклеточного сигнального домена.
Термин "консервативные модификации последовательности" относится к аминокислотным модификациям, которые не оказывают значительного влияния на характеристики связывания антитела или фрагмента антитела, содержащего аминокислотную последовательность, или не изменяют их в значительной степени. Такие консервативные модификации включают аминокислотные замены, добавления и делеции. Модификации могут быть введены в антитело или фрагмент антитела по настоящему изобретению с помощью стандартных методик, известных из уровня техники, таких как сайт-направленный мутагенез и ПЦР-опосредованный мутагенез. Консервативные замены представляют собой замены, при которых аминокислотный остаток заменяется аминокислотным остатком, имеющим сходную боковую цепь. Семейства аминокислотных остатков, имеющих сходные боковые цепи, были определены в уровне техники. Эти семейства включают аминокислоты с основными боковыми цепями (например, лизин, аргинин, гистидин), кислыми боковыми цепями (например, аспарагиновая кислота, глутаминовая кислота), незаряженными полярными боковыми цепями (например, глицин, аспарагин, глутамин, серин, треонин, тирозин, цистеин, триптофан), неполярными боковыми цепями (например, аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин), бета-разветвленными боковыми цепями (например, треонин, валин, изолейцин) и ароматическими боковыми цепями (например, тирозин, фенилаланин, триптофан, гистидин). Таким образом, один или несколько аминокислотных остатков в CAR по настоящему изобретению могут быть заменены другими аминокислотными остатками из того же семейства боковых цепей, и измененный CAR можно протестировать с использованием функциональных анализов, описанных в данном документе.
Термин "стимуляция" применительно к стимуляции стимулирующей и/или костимулирующей молекулой относится к ответу, например, первичному или вторичному ответу, индуцированному путем связывания стимулирующей молекулы (например, комплекса TCR/CD3) и/или костимулирующей молекулы (например, CD28 или 4-1BB) с ее когнатным лигандом, которое опосредует событие передачи сигнала, такое как без ограничения передача сигнала через комплекс TCR/CD3. Стимуляция может опосредовать изменение экспрессии определенных молекул и/или реорганизацию структур цитоскелета и т. п.
Термин "стимулирующая молекула" относится к молекуле, экспрессируемой Т-клеткой, которая обеспечивает первичную(первичные) цитоплазматическую(цитоплазматические) сигнальную(сигнальные) последовательность(последовательности), регулирующую(регулирующие) первичную активацию комплекса TCR стимулирующим образом в по меньшей мере некоторых аспектах сигнального пути T-клетки. В некоторых вариантах осуществления ITAM-содержащий домен в CAR воспроизводит передачу сигнала от первичного TCR независимо от эндогенных комплексов TCR. В некоторых вариантах осуществления первичный сигнал инициируется, например, путем связывания комплекса TCR/CD3 с молекулой МНС, нагруженной пептидом, и это приводит к опосредованию ответа Т-клеток, в том числе без ограничения пролиферации, активации, дифференцировки и т. п. Первичная цитоплазматическая сигнальная последовательность (также называемая "первичным сигнальным доменом"), которая действует стимулирующим образом, может содержать сигнальный мотив, известный как иммунорецепторный тирозиновый активирующий мотив или ITAM. Примеры ITAM-содержащей первичной цитоплазматической сигнальной последовательности, которая является особенно применимой в настоящем изобретении, включают без ограничения последовательности, полученные из TCR-дзета, FcR-гамма, FcR-бета, CD3-гамма, CD3-дельта, CD3-эпсилон, CD5, CD22, CD79a, CD79b, CD278 (также известного как "ICOS"), FcεRI и CD66d, DAP10 и DAP12. В конкретном CAR по настоящему изобретению внутриклеточный сигнальный домен в любом одном или нескольких CAR по настоящему изобретению содержит внутриклеточную сигнальную последовательность, например, первичную сигнальную последовательность CD3-дзета. Термин "антигенпрезентирующая клетка" или "APC" относится к клетке иммунной системы, такой как вспомогательная клетка (например, B-клетка, дендритная клетка и т. п.), которая представляет чужеродный антиген в комплексе с молекулами главного комплекса гистосовместимости (MHC) на своей поверхности. Т-клетки могут распознавать эти комплексы с помощью своих T-клеточных рецепторов (TCR). АРС процессируют антигены и презентируют их Т-клеткам.
Используемый в данном документе термин "внутриклеточный сигнальный домен" относится к внутриклеточной части молекулы. В ряде вариантов осуществления внутриклеточный сигнальный домен передает сигнал эффекторной функции и направляет клетку на выполнение специализированной функции. Хотя может использоваться весь внутриклеточный сигнальный домен, во многих случаях отсутствует необходимость использовать всю цепь. В том случае, если используют усеченную часть внутриклеточного сигнального домена, такую усеченную часть можно использовать вместо интактной цепи, при условии, что она передает сигнал эффекторной функции. Таким образом, подразумевается, что термин "внутриклеточный сигнальный домен" включает любую усеченную часть внутриклеточного сигнального домена, достаточную для передачи сигнала эффекторной функции.
Внутриклеточный сигнальный домен образует сигнал, который способствует осуществлению иммунной эффекторной функции клеткой, содержащей CAR, например, CAR-T-клеткой. Примеры иммунной эффекторной функции, например, у CAR-T-клетки, включают цитолитическую активность и хелперную активность, в том числе секрецию цитокинов.
В некоторых вариантах осуществления внутриклеточный сигнальный домен может содержать первичный внутриклеточный сигнальный домен. Иллюстративные первичные внутриклеточные сигнальные домены включают домены, полученные из молекул, отвечающих за первичную стимуляцию или антигензависимую стимуляцию. В некоторых вариантах осуществления внутриклеточный сигнальный домен может содержать костимулирующий внутриклеточный домен. Иллюстративные костимулирующие внутриклеточные сигнальные домены включают домены, полученные из молекул, отвечающих за костимулирующие сигналы или антигеннезависимую стимуляцию. Например, в случае с CAR-T первичный внутриклеточный сигнальный домен может содержать цитоплазматическую последовательность Т-клеточного рецептора, и костимулирующий внутриклеточный сигнальный домен может содержать цитоплазматическую последовательность корецептора или костимулирующей молекулы.
Первичный внутриклеточный сигнальный домен может содержать сигнальный мотив, который известен как иммунорецепторный тирозиновый активирующий мотив или ITAM. Примеры ITAM-содержащих первичных цитоплазматических сигнальных последовательностей включают без ограничения последовательности, полученные из CD3-дзета, FcR-гамма, FcR-бета, CD3-гамма, CD3-дельта, CD3-эпсилон, CD5, CD22, CD79a, CD79b, CD278 (также известного как "ICOS"), FcεRI, CD66d, DAP10 и DAP12.
Термин "дзета" или, в качестве альтернативы, "дзета-цепь", "CD3-дзета" или "TCR-дзета" относится к CD247. Под номером доступа Swiss-Prot P20963 представлены иллюстративные аминокислотные последовательности CD3-дзета человека. "Стимулирующий домен дзета" или, в качестве альтернативы, "стимулирующий домен CD3-дзета" или "стимулирующий домен TCR-дзета" относится к стимулирующему домену CD3-дзета или его варианту (например, молекуле, имеющей мутации, например, точечные мутации, фрагменты, вставки или делеции). В некоторых вариантах осуществления цитоплазматический домен дзета содержит остатки 52-164 из последовательности под номером доступа в GenBank BAG36664.1 или ее варианта (например, молекулы, имеющей мутации, например, точечные мутации, фрагменты, вставки или делеции). В некоторых вариантах осуществления "стимулирующий домен дзета" или "стимулирующий домен CD3-дзета" представляет собой последовательность, представленную под SEQ ID NO: 9 или 10, или ее вариант (например, молекулу, имеющую мутации, например, точечные мутации, фрагменты, вставки или делеции).
Термин "костимулирующая молекула" относится к когнатному партнеру по связыванию на Т-клетке, который специфично связывается с костимулирующим лигандом, за счет чего происходит опосредование костимулируемого ответа Т-клетки, такого как, без ограничения, пролиферация. Костимулирующие молекулы представляют собой молекулы клеточной поверхности, отличные от антигенных рецепторов или их лигандов, которые необходимы для эффективного иммунного ответа. Костимулирующие молекулы включают без ограничения молекулу MHC I класса, белковые рецепторы TNF, иммуноглобулиноподобные белки, рецепторы цитокинов, интегрины, сигнальные молекулы активации лимфоцитов (белки SLAM), активирующие рецепторы NK-клеток, BTLA, лиганд Toll-подобного рецептора, OX40, CD2, CD7, CD27, CD28, CD30, CD40, CDS, ICAM-1, LFA-1 (CD11a/CD18), 4-1BB (CD137), B7-H3, CDS, ICAM-1, ICOS (CD278), GITR, BAFFR, LIGHT, HVEM (LIGHTR), KIRDS2, SLAMF7, NKp80 (KLRF1), NKp44, NKp30, NKp46, CD19, CD4, CD8-альфа, CD8-бета, IL2R-бета, IL2R-гамма, IL7R-альфа, ITGA4, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, LFA-1, ITGB7, NKG2D, NKG2C, TNFR2, TRANCE/RANKL, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (Tactile), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), CD69, SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR, LAT, GADS, SLP-76, PAG/Cbp, CD19a, CD28-OX40, CD28-4-1BB и лиганд, который специфично связывается с CD83.
Костимулирующий внутриклеточный сигнальный домен относится к внутриклеточной части костимулирующей молекулы.
Внутриклеточный сигнальный домен может содержать всю внутриклеточную часть или весь нативный внутриклеточный сигнальный домен молекулы, из которой он получен, или их функциональный фрагмент.
Термин "4-1BB" относится к CD137 или представителю 9 суперсемейства рецепторов фактора некроза опухоли. Под номером доступа Swiss-Prot P20963 представлены иллюстративные аминокислотные последовательности 4-1BB человека. "Кoстимулирующий домен 4-1BB" относится к костимулирующему домену 4-1BB или его варианту (например, молекуле, имеющей мутации, например, точечные мутации, фрагменты, вставки или делеции). В некоторых вариантах осуществления "костимулирующий домен 4-1BB" представляет собой последовательность, представленную под SEQ ID NO: 7, или ее вариант (например, молекулу, имеющую мутации, например, точечные мутации, фрагменты, вставки или делеции).
Используемый в данном документе термин "иммунная эффекторная клетка" относится к клетке, которая вовлечена в иммунный ответ, например, способствуя осуществлению иммунного эффекторного ответа. Примеры иммунных эффекторных клеток включают Т-клетки, например, альфа/бета-Т-клетки и гамма/дельта-Т-клетки, В-клетки, естественные клетки-киллеры (NK), естественные T-клетки-киллеры (NKT), тучные клетки и фагоциты миелоидного происхождения.
Используемый в данном документе термин "иммунная эффекторная функция или иммунный эффекторный ответ" относится к функции или ответу, например, иммунной эффекторной клетки, которые усиливают иммунную атаку на клетку-мишень или способствуют ей. Например, иммунная эффекторная функция или ответ относятся к свойству T- или NK-клетки, которое способствует уничтожению или ингибированию роста или пролиферации клетки-мишени. В случае с Т-клеткой первичная стимуляция и костимуляция являются примерами иммунных эффекторных функции или ответа.
Термин "эффекторная функция" относится к специализированной функции клетки. Например, эффекторная функция Т-клетки может представлять собой цитолитическую активность или хелперную активность, в том числе секрецию цитокинов.
Термин "кодирование" относится к внутренне присущему свойству специфических последовательностей нуклеотидов в полинуклеотиде, таком как ген, кДНК или мРНК, служить в качестве матриц для синтеза в ходе биологических процессов других полимеров и макромолекул, имеющих определенную последовательность нуклеотидов (т. е. рРНК, тРНК и мРНК) либо определенную последовательность аминокислот и биологические свойства, обусловленные ими. Таким образом, ген, кДНК или РНК кодирует белок, если в результате транскрипции и трансляции мРНК, соответствующей этому гену, в клетке или другой биологической системе продуцируется белок. Как кодирующая нить, нуклеотидная последовательность которой идентична последовательности мРНК и обычно представлена в перечнях последовательностей, так и некодирующая нить, используемая в качестве матрицы для транскрипции гена или кДНК, могут обозначаться как кодирующие белок или другой продукт данного гена или кДНК.
Если не указано иное, то "нуклеотидная последовательность, кодирующая аминокислотную последовательность" включает все нуклеотидные последовательности, которые являются вырожденными вариантами друг друга и которые кодируют одну и ту же аминокислотную последовательность. Фраза "нуклеотидная последовательность, которая кодирует белок или РНК" может также включать интроны в той степени, в которой нуклеотидная последовательность, кодирующая белок, может в каком-либо варианте содержать интрон(интроны).
Термины "эффективное количество" или "терапевтически эффективное количество" используются в данном документе взаимозаменяемо и относятся к количеству соединения, состава, материала или композиции, описанных в данном документе, эффективному для достижения конкретного биологического результата.
Термин "эндогенный" относится к любому материалу, полученному из организма, клетки, ткани или системы или продуцируемому внутри них.
Термин "экзогенный" относится к любому материалу, введенному в организм, клетку, ткань или систему или продуцируемому вне их.
Термин "экспрессия" относится к транскрипции и/или трансляции конкретной нуклеотидной последовательности. В некоторых вариантах осуществления экспрессия включает трансляцию мРНК, введенной в клетку.
Термин "вектор для переноса" относится к композиции, которая содержит выделенную нуклеиновую кислоту и которую можно применять для доставки выделенной нуклеиновой кислоты внутрь клетки. Из уровня техники известны многочисленные векторы, в том числе без ограничения линейные полинуклеотиды, полинуклеотиды, ассоциированные с ионными или амфифильными соединениями, плазмиды и вирусы. Таким образом, термин "вектор для переноса" включает автономно реплицирующуюся плазмиду или вирус. Данный термин также следует толковать таким образом, что он дополнительно включает неплазмидные и невирусные соединения, которые содействуют переносу нуклеиновой кислоты в клетки, такие как, например, полилизиновое соединение, липосома и т. п. Примеры вирусных векторов для переноса включают без ограничения аденовирусные векторы, векторы на основе аденоассоциированного вируса, ретровирусные векторы, лентивирусные векторы и т. п.
Термин "экспрессионный вектор" относится к вектору, который содержит рекомбинантный полинуклеотид, содержащий последовательности, контролирующие экспрессию, функционально связанные с нуклеотидной последовательностью, которая должна экспрессироваться. Экспрессионный вектор содержит достаточное количество цис-действующих элементов для экспрессии; а другие элементы для экспрессии могут предоставляться клеткой-хозяином или в системе экспрессии in vitro. Экспрессионные векторы включают все векторы, известные из уровня техники, в том числе космиды, плазмиды (например, "голые" или содержащиеся в липосомах) и вирусы (например, лентивирусы, ретровирусы, аденовирусы и аденоассоциированные вирусы), в состав которых включен рекомбинантный полинуклеотид.
Термин "лентивирус" относится к роду из семейства Retroviridae. Лентивирусы являются уникальными среди ретровирусов в том, что способны инфицировать неделящиеся клетки; они могут доставлять значительное количество генетической информации в ДНК клетки-хозяина, поэтому они в качестве векторов являются одним из наиболее эффективных способов доставки генов. Все из HIV, SIV и FIV являются примерами лентивирусов.
Термин "лентивирусный вектор" относится к вектору, полученному из по меньшей мере части генома лентивируса, в том числе, в частности, к самоинактивирующемуся лентивирусному вектору, представленному в Milone et al., Mol. Ther. 17(8): 1453-1464 (2009). Другие примеры лентивирусных векторов, которые можно использовать в клинической практике, включают без ограничения, например, технологию доставки генов LENTIVECTOR® от Oxford BioMedica, векторную систему LENTIMAX™ от Lentigen и т. п. Неклинические типы лентивирусных векторов также являются доступными и должны быть известны специалисту в данной области.
Термин "гомологичный" или "идентичность" относится к идентичности последовательностей субъединиц у двух полимерных молекул, например, двух молекул нуклеиновой кислоты, таких как две молекулы ДНК или две молекулы РНК, или двух молекул полипептидов. Если положение субъединицы в обеих из двух молекул занимает одна и та же мономерная субъединица, например, если положение в каждой из двух молекул ДНК занято аденином, то они являются гомологичными или идентичными по этому положению. Гомология между двумя последовательностями находится в прямой зависимости от количества совпадающих или гомологичных положений; например, если половина (например, пять положений в полимере длиной десять субъединиц) положений в двух последовательностях является гомологичной, то эти две последовательности являются гомологичными на 50%; если 90% положений (например, 9 из 10) являются совпадающими или гомологичными, то две последовательности являются гомологичными на 90%.
"Гуманизированные" формы антител, отличных от человеческих (например, мышиных), представляют собой химерные иммуноглобулины, цепи иммуноглобулинов или их фрагменты (такие как Fv, Fab, Fab', F(ab')2 или другие антигенсвязывающие подпоследовательности антител), которые содержат минимальную последовательность, полученную из иммуноглобулина, отличного от человеческого. В большинстве случаев гуманизированные антитела и фрагменты данных антител представляют собой иммуноглобулины человека (реципиентное антитело или фрагмент антитела), в которых остатки из определяющей комплементарность области (CDR) реципиента заменены остатками из CDR вида, отличного от человека (донорного антитела), такого как мышь, крыса или кролик, обладающими требуемой специфичностью, аффинностью и емкостью. В некоторых случаях остатки каркасной области (FR) Fv иммуноглобулина человека заменены соответствующими остатками, отличными от человеческих. Кроме того, гуманизированное антитело/фрагмент антитела может содержать остатки, не обнаруживаемые ни в реципиентном антителе, ни в импортированных последовательностях CDR или каркасных областей. С помощью этих модификаций можно дополнительно улучшить и оптимизировать функциональные характеристики антитела или фрагмента антитела. Как правило, гуманизированное антитело или фрагмент этого антитела будет содержать по существу все из по меньшей мере одного и, как правило, двух, вариабельных доменов, в которых все или по существу все CDR-области соответствуют областям иммуноглобулина, отличного от человеческого, и все FR-области или их значительная часть представляют собой области с последовательностью иммуноглобулина человека. Гуманизированное антитело или фрагмент антитела также может содержать по меньшей мере часть константной области (Fc) иммуноглобулина, как правило, иммуноглобулина человека. Дополнительные подробности см. в Jones et al., Nature, 321: 522-525, 1986; Reichmann et al., Nature, 332: 323-329, 1988; Presta, Curr. Op. Struct. Biol., 2: 593-596, 1992.
"Полностью человеческий" относится к иммуноглобулину, такому как антитело или фрагмент антитела, где целая молекула имеет человеческое происхождение или состоит из аминокислотной последовательности, идентичной человеческой форме антитела или иммуноглобулина.
Термин "выделенный" означает измененный относительно природного состояния или извлеченный из него. Например, нуклеиновая кислота или пептид, в природных условиях присутствующие в живом животном, не являются "выделенными", однако те же нуклеиновая кислота или пептид, частично или полностью отделенные от материалов, сопутствующих им в их природном состоянии, являются "выделенными". Выделенные нуклеиновая кислота или белок могут находиться в по существу очищенной форме или могут находиться в ненативной среде, такой как, например, клетка-хозяин.
В контексте настоящего изобретения используются следующие сокращения для общераспространенных оснований нуклеиновых кислот. "А" относится к аденозину, "С" относится к цитозину, "G" относится к гуанозину, "Т" относится к тимидину, и "U" относится к уридину.
Термин "функционально связанный" или "транскрипционный контроль" относится к функциональной связи между регуляторной последовательностью и гетерологичной последовательностью нуклеиновой кислоты, приводящей к экспрессии последней. Например, первая последовательность нуклеиновой кислоты функционально связана со второй последовательностью нуклеиновой кислоты в тех случаях, когда первая последовательность нуклеиновой кислоты размещена в функциональной взаимосвязи со второй последовательностью нуклеиновой кислоты. Например, промотор функционально связан с кодирующей последовательностью, если промотор влияет на транскрипцию или экспрессию кодирующей последовательности. Функционально связанные последовательности ДНК могут быть смежными друг с другом и, например, если необходимо соединить две области, кодирующие белок, находятся в одной и той же рамке считывания.
Термин "парентеральное" введение иммуногенной композиции включает, например, методики подкожной (s.c.), внутривенной (i.v.), внутримышечной (i.m.) или внутригрудинной инъекции, внутриопухолевого введения или инфузии.
Термин "нуклеиновая кислота", "молекула нуклеиновой кислоты", "полинуклеотид" или "молекула полинуклеотида" относится к дезоксирибонуклеиновым кислотам (ДНК) или рибонуклеиновым кислотам (РНК) и их полимерам в одно- или двухнитевой форме. Если специально не ограничено, то данный термин охватывает нуклеиновые кислоты, содержащие известные аналоги природных нуклеотидов, которые обладают свойствами связывания, сходными со свойствами эталонной нуклеиновой кислоты, и метаболизируются посредством механизма, сходного с механизмом для встречающихся в природе нуклеотидов. В некоторых вариантах осуществления "нуклеиновая кислота", "молекула нуклеиновой кислоты", "полинуклеотид" или "молекула полинуклеотида" предусматривает производное или аналог нуклеотида/нуклеозида. Если не указано иное, конкретная последовательность нуклеиновой кислоты также в неявной форме охватывает ее варианты с консервативными модификациями (например, с заменами вырожденными кодонами, например, консервативными заменами), аллели, ортологи, SNP и комплементарные последовательности, а также последовательность, указанную в явной форме. В частности, замены вырожденными кодонами, например, консервативные замены, можно осуществлять посредством образования последовательностей, в которых в третьем положении одного или нескольких выбранных (или всех) кодонов произведена замена любым из канонических оснований и/или остатками дезоксиинозина (Batzer et al., Nucleic Acid Res. 19:5081 (1991); Ohtsuka et al., J. Biol. Chem. 260:2605-2608 (1985); и Rossolini et al., Mol. Cell. Probes 8:91-98 (1994)).
Термины "пептид", "полипептид" и "белок" используются взаимозаменяемо и относятся к соединению, состоящему из аминокислотных остатков, ковалентно связанных пептидными связями. Белок или пептид должен содержать по меньшей мере две аминокислоты, и не установлено ограничение на максимальное количество аминокислот, которые может содержать последовательность белка или пептида. Полипептиды включают любой пептид или белок, содержащий две или более аминокислоты, соединенные друг с другом пептидными связями. Используемый в данном документе термин относится как к коротким цепям, которые также обычно называются в уровне техники, например, пептидами, олигопептидами и олигомерами, так и к более длинным цепям, которые обычно называются в уровне техники белками, которых существует множество типов. "Полипептиды" включают, например, среди прочих, биологически активные фрагменты, по существу гомологичные полипептиды, олигопептиды, гомодимеры, гетеродимеры, варианты полипептидов, модифицированные полипептиды, производные, аналоги, слитые белки. Полипептид включает природный пептид, рекомбинантный пептид или их комбинацию.
Термин "промотор" относится к последовательности ДНК, распознаваемой синтетическим аппаратом клетки или введенным синтетическим аппаратом, которая необходима для инициации специфической транскрипции полинуклеотидной последовательности.
Термин "промоторная/регуляторная последовательность" относится к последовательности нуклеиновой кислоты, которая необходима для экспрессии продукта гена, функционально связанного с промоторной/регуляторной последовательностью. В некоторых случаях эта последовательность может являться коровой промоторной последовательностью, а в других случаях эта последовательность может также содержать энхансерную последовательность и другие регуляторные элементы, которые необходимы для экспрессии продукта гена. Например, промоторная/регуляторная последовательность может представлять собой последовательность, которая обеспечивает экспрессию продукта гена тканеспецифическим образом.
Термин "конститутивный промотор" относится к нуклеотидной последовательности, которая, находясь в функциональной связи с полинуклеотидом, который кодирует или определяет продукт гена, обуславливает продуцирование продукта гена в клетке при большинстве или всех физиологических условиях клетки.
Термин "индуцируемый промотор" относится к нуклеотидной последовательности, которая, находясь в функциональной связи с полинуклеотидом, который кодирует или определяет продукт гена, обуславливает продуцирование продукта гена в клетке по существу только в случае присутствия в клетке индуктора, который соответствует данному промотору.
Термин "тканеспецифический промотор" относится к нуклеотидной последовательности, которая, находясь в функциональной связи с полинуклеотидом, который кодирует или определяет ген, обуславливает продуцирование продукта гена в клетке по существу только в том случае, если клетка является клеткой из того типа ткани, который соответствует данному промотору.
Термины "антиген, ассоциированный с раком", "опухолевый антиген", "антиген, связанный с гиперпролиферативным нарушением" и "антиген, ассоциированный с гиперпролиферативным нарушением" взаимозаменяемо относятся к антигенам, которые являются общими для специфических гиперпролиферативных нарушений. В некоторых вариантах осуществления такие термины относятся к молекуле (как правило, белку, углеводу или липиду), которая экспрессируется на поверхности раковой клетки полностью либо в виде фрагмента (например, молекула MHC/пептид) и которая применима для предпочтительного нацеливания фармакологического средства на раковую клетку. В некоторых вариантах осуществления опухолевый антиген представляет собой маркер, экспрессируемый как нормальными клетками, так и раковыми клетками, например, маркер линии дифференцировки, например, CD19 на В-клетках. В некоторых вариантах осуществления опухолевый антиген представляет собой молекулу клеточной поверхности, которая в раковой клетке сверхэкспрессируется по сравнению с нормальной клеткой, например, характеризуется 1-кратной сверхэкспрессией, 2-кратной сверхэкспрессией, 3-кратной или большей сверхэкспрессией по сравнению с нормальной клеткой. В некоторых вариантах осуществления опухолевый антиген представляет собой молекулу клеточной поверхности, которая синтезируется в раковой клетке ненадлежащим образом, например, молекулу, которая содержит делеции, добавления или мутации по сравнению с молекулой, экспрессируемой на нормальной клетке. В некоторых вариантах осуществления опухолевый антиген будет экспрессироваться исключительно на клеточной поверхности раковой клетки, полностью или в виде фрагмента (например, молекула MHC/пептид), и не будет синтезироваться или экспрессироваться на поверхности нормальной клетки. В некоторых вариантах осуществления антигены, связанные с гиперпролиферативным нарушением, по настоящему изобретению получены из раковых образований, включающих без ограничения первичную или метастатическую меланому, тимому, лимфому, саркому, рак легкого, рак печени, неходжкинскую лимфому, лимфому Ходжкина, формы лейкоза, рак матки, рак шейки матки, рак мочевого пузыря, рак почки и формы аденокарциномы, такие как рак молочной железы, рак предстательной железы (например, кастрационнорезистентный или резистентный к терапии рак предстательной железы или метастатический рак предстательной железы), рак яичника, рак поджелудочной железы и т. п., или нарушение пролиферации плазматических клеток, например, бессимптомную миелому ("тлеющую" множественную миелому или вялотекущую миелому), моноклональную гаммапатию неустановленной этиологии (MGUS), макроглобулинемию Вальденстрема, формы плазмоцитомы (например, плазмоклеточную дискразию, солитарную миелому, солитарную плазмоцитому, экстрамедуллярную плазмоцитому и множественную плазмоцитому), системный амилоидоз в форме амилоидоза легких цепей и POEMS-синдром (также известный как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром). В некоторых вариантах осуществления CAR по настоящему изобретению включают CAR, содержащие антигенсвязывающий домен (например, антитело или фрагмент антитела), который связывается с пептидом, презентируемым молекулой MHC. Обычно пептиды, происходящие из эндогенных белков, заполняют карманы молекул главного комплекса гистосовместимости (MHC) I класса и распознаются T-клеточными рецепторами (TCR) на CD8+ T-лимфоцитах. Комплексы молекул MHC I класса конститутивно экспрессируются всеми ядросодержащими клетками. При раке комплексы вирусоспецифический и/или опухолеспецифический пептид/молекула MHC представляют собой уникальный класс мишеней клеточной поверхности для иммунотерапии. Были описаны TCR-подобные антитела, нацеливающиеся на пептиды, происходящие из вирусных или опухолевых антигенов, в окружении человеческого лейкоцитарного антигена (HLA)-A1 или HLA-A2 (см., например, Sastry et al., J Virol. 2011 85(5):1935-1942; Sergeeva et al., Blood, 2011 117(16):4262-4272; Verma et al., J Immunol 2010 184(4):2156-2165; Willemsen et al., Gene Ther 2001 8(21) :1601-1608; Dao et al., Sci Transl Med 2013 5(176) :176ra33 ; Tassev et al., Cancer Gene Ther 2012 19(2):84-100). Например, TCR-подобное антитело можно идентифицировать при скрининге библиотеки, такой как фаг-дисплейная библиотека scFv человека.
Термины "антиген, обеспечивающий поддержание опухоли" или "антиген, обеспечивающий поддержание рака" взаимозаменяемо относятся к молекуле (как правило, белку, углеводу или липиду), которая экспрессируется на поверхности клетки, которая сама по себе не является раковой, но обеспечивает поддержание раковых клеток, например, путем способствования их росту или выживанию, например, устойчивости к иммунным клеткам. Иллюстративные клетки данного типа включают стромальные клетки и супрессорные клетки миелоидного происхождения (MDSC). Антиген, обеспечивающий поддержание опухоли, сам по себе не обязательно играет роль в поддержании опухолевых клеток, при условии, что антиген присутствует в клетке, которая обеспечивает поддержание раковых клеток.
Термин "гибкий полипептидный линкер" или "линкер", используемый применительно к scFv, относится к пептидному линкеру, который состоит из аминокислот, таких как глициновые и/или сериновые остатки, используемые в отдельности или в комбинации, для связывания вариабельной области тяжелой цепи и вариабельной области легкой цепи друг с другом. В некоторых вариантах осуществления гибкий полипептидный линкер представляет собой линкер Gly/Ser и содержит аминокислотную последовательность (Gly-Gly-Gly-Ser)n, где n представляет собой положительное целое число, которое равняется или больше 1 (SEQ ID NO: 41). Например, n=1, n=2, n=3, n=4, n=5 и n=6, n=7, n=8, n=9 и n=10. В некоторых вариантах осуществления гибкие полипептидные линкеры включают без ограничения (Gly4Ser)4 (SEQ ID NO: 27) или (Gly4 Ser)3 (SEQ ID NO: 28). В некоторых вариантах осуществления линкеры содержат множественные повторы (Gly2Ser) (GlySer) или (Gly3Ser) (SEQ ID NO: 29). Также в объем настоящего изобретения включены линкеры, описанные в WO2012/138475, включенной в данный документ посредством ссылки.
Как используется в данном документе, 5'-кэп (также называемый РНК-кэпом, 7-метилгуанозиновым РНК-кэпом или РНК-кэпом m7G) представляет собой модифицированный гуаниновый нуклеотид, который добавляется к "переднему", или 5'-концу, эукариотической матричной РНК вскоре после начала транскрипции. 5'-кэп состоит из концевой группы, которая связана с первым транскрибированным нуклеотидом. Его присутствие является критически важным для распознавания рибосомой и защиты от РНКаз. Добавление кэпа сопряжено с транскрипцией и происходит совместно с транскрипцией, так что каждое из этих событий влияет на другое. Вскоре после начала транскрипции 5'-конец синтезируемой мРНК связывается с кэп-синтезирующим комплексом, ассоциированным с РНК-полимеразой. Этот ферментативный комплекс катализирует химические реакции, которые необходимы для кэпирования мРНК. Синтез протекает в виде многоступенчатой биохимической реакции. Кэпирующий фрагмент можно модифицировать для модулирования функциональных свойств мРНК, таких как ее стабильность или эффективность трансляции.
Как используется в данном документе, "РНК, транскрибированная in vitro" относится к РНК, предпочтительно мРНК, которая была синтезирована in vitro. В некоторых вариантах осуществления РНК представляет собой мРНК. Обычно РНК, транскрибированная in vitro, образуется из вектора транскрипции in vitro. Вектор транскрипции in vitro содержит матрицу, используемую для образования РНК, транскрибированной in vitro.
Как используется в данном документе, "поли(А)" представляет собой последовательный ряд аденозиновых остатков, присоединенных путем полиаденилирования к мРНК. В некоторых вариантах осуществления конструкции для транзиентной экспрессии длина поли(А) составляет от 50 до 5000 (SEQ ID NO: 30). В некоторых вариантах осуществления длина поли(А) составляет более 64. В некоторых вариантах осуществления длина поли(А) составляет более 100. В некоторых вариантах осуществления длина поли(А) составляет более 300. В некоторых вариантах осуществления длина поли(А) составляет более 400. Последовательности поли(A) можно модифицировать с помощью химических средств или ферментов для модулирования функциональных свойств мРНК, таких как локализация, стабильность или эффективность трансляции.
Как используется в данном документе, "полиаденилирование" относится к ковалентной связи полиаденилилового фрагмента или его модифицированного варианта с молекулой матричной РНК. У эукариотических организмов большинство молекул матричной РНК (мРНК) являются полиаденилированными на 3'-конце. 3'-концевой поли(А)-хвост представляет собой длинную последовательность адениновых нуклеотидов (часто несколько сотен), добавленных к пре-мРНК под действием фермента полиаденилатполимеразы. У высших эукариот поли(А)-хвост добавляется к транскриптам, которые содержат определенную последовательность - сигнал полиаденилирования. Поли(А)-хвост и связанный с ним белок содействуют защите мРНК от разрушения экзонуклеазами. Полиаденилирование также важно для терминации транскрипции, экспорта мРНК из ядра и трансляции. Полиаденилирование происходит в ядре сразу после транскрипции ДНК в РНК, но дополнительно может также происходить позднее в цитоплазме. После терминации транскрипции цепь мРНК расщепляется под действием эндонуклеазного комплекса, ассоциированного с РНК-полимеразой. Сайт расщепления обычно характеризуется наличием последовательности оснований AAUAAA вблизи сайта расщепления. После того, как мРНК была расщеплена, к свободному 3'-концу в сайте расщепления добавляются аденозиновые остатки.
Как используется в данном документе, "транзиентный" относится к экспрессии неинтегрированного трансгена в течение периода нескольких часов, дней или недель, где период времени экспрессии является меньшим, чем период времени для экспрессии гена в случае, если он интегрирован в геном или содержится в стабильном плазмидном репликоне в клетке-хозяине.
Используемые в данном документе термины "лечить", "лечение" и "осуществление лечения" относятся к снижению или ослаблению прогрессирования, тяжести и/или продолжительности пролиферативного нарушения или ослаблению одного или нескольких симптомов (предпочтительно одного или нескольких различимых симптомов) пролиферативного нарушения в результате введения одного или нескольких средств терапии (например, одного или нескольких терапевтических средств, таких как CAR по настоящему изобретению). В конкретных вариантах осуществления термины "лечить", "лечение" и "осуществление лечения" относятся к ослаблению по меньшей мере одного измеряемого физического параметра пролиферативного нарушения, такого как рост опухоли, не обязательно различимого для пациента. В других вариантах осуществления термины "лечить", "лечение" и "осуществление лечения" относятся к ингибированию прогрессирования пролиферативного нарушения либо на физическом уровне, например, посредством стабилизации различимого симптома, либо на физиологическом уровне, например, посредством стабилизации физического параметра, либо как на одном, так и на другом уровнях. В других вариантах осуществления термины "лечить", "лечение" и "осуществление лечения" относятся к снижению или стабилизации размера опухоли или количества раковых клеток.
Термин "путь передачи сигнала" относится к биохимической взаимосвязи между различными молекулами-переносчиками сигналов, которые играют роль в передаче сигнала от одной части клетки к другой части клетки. Фраза "рецептор клеточной поверхности" включает молекулы и комплексы молекул, способные принимать сигнал и передавать сигнал через мембрану клетки.
Термин "субъект" предполагает включение живых организмов, у которых можно вызвать иммунный ответ (например, млекопитающих, человека).
Термин "по существу очищенная клетка" относится к клетке, которая по сути свободна от других типов клеток. "По существу очищенная клетка" также относится к клетке, которая была отделена от других типов клеток, с которыми она обычно ассоциирована в своем встречающемся в природе состоянии. В некоторых случаях популяция по существу очищенных клеток относится к однородной популяции клеток. В других случаях данный термин просто относится к клетке, которая была отделена от клеток, с которыми она в природных условиях ассоциирована в своем природном состоянии. В некоторых вариантах осуществления клетки культивируют in vitro. В некоторых вариантах осуществления клетки не культивируют in vitro.
Используемый в данном документе термин "терапевтический" означает лечение. Терапевтический эффект получают путем ослабления, подавления, ремиссии или устранения болезненного состояния.
Используемый в данном документе термин "профилактика" означает предупреждение или профилактическое лечение заболевания или болезненного состояния.
Термины "трансфицированный", или "трансформированный", или "трансдуцированный" относятся к процессу, в ходе которого экзогенную нуклеиновую кислоту переносят или вводят в клетку-хозяина. "Трансфицированная", или "трансформированная", или "трансдуцированная" клетка представляет собой клетку, которая была подвергнута трансфекции, трансформации или трансдукции с помощью экзогенной нуклеиновой кислоты. Такая клетка включает первичную клетку субъекта и ее потомство.
Термин "специфично связывается" относится к антителу или лиганду, которые распознают своего когнатного белкового партнера по связыванию (например, стимулирующую и/или костимулирующую молекулу, присутствующую на Т-клетке), присутствующего в образце, и связываются с ним, но при этом это антитело или лиганд по существу не распознают или не связывают другие молекулы в образце.
Как используется в данном документе, "регулируемый химерный антигенный рецептор (RCAR)" относится к набору полипептидов, как правило, в простейших вариантах осуществления двух, которые, находясь в иммунной эффекторной клетке, наделяют клетку специфичностью в отношении клетки-мишени, как правило, раковой клетки, и образованием внутриклеточного сигнала. В некоторых вариантах осуществления RCAR содержит по меньшей мере внеклеточный антигенсвязывающий домен, трансмембранный домен и цитоплазматический сигнальный домен (также называемый в данном документе "внутриклеточным сигнальным доменом"), содержащий функциональный сигнальный домен, полученный из стимулирующей молекулы и/или костимулирующей молекулы, как определено в данном документе применительно к молекуле CAR. В некоторых вариантах осуществления полипептиды из набора в RCAR не являются смежными друг с другом, например, находятся в разных полипептидных цепях. В некоторых вариантах осуществления RCAR содержит димеризационный переключатель, который при наличии димеризационной молекулы может связывать полипептиды друг с другом, например, может связывать антигенсвязывающий домен с внутриклеточным сигнальным доменом. В некоторых вариантах осуществления RCAR экспрессируется в клетке (например, иммунной эффекторной клетке), описанной в данном документе, например, в клетке, экспрессирующей RCAR (также называемой в данном документе "RCARX-клеткой"). В некоторых вариантах осуществления RCARX-клетка представляет собой T-клетку и называется RCAR-T-клеткой. В некоторых вариантах осуществления RCARX-клетка представляет собой NK-клетку и называется RCAR-N-клеткой. RCAR может наделять клетку, экспрессирующую RCAR, специфичностью в отношении клетки-мишени, как правило, раковой клетки, и регулируемым образованием внутриклеточного сигнала или пролиферацией, что может оптимизировать иммуноэффекторное свойство клетки, экспрессирующей RCAR. В ряде вариантов осуществления клетка с RCAR по меньшей мере частично зависит от антигенсвязывающего домена в обеспечении специфичности в отношении клетки-мишени, которая содержит антиген, связываемый антигенсвязывающим доменом.
Используемый в данном документе термин "мембранный якорь" или "домен прикрепления к мембране" относится к полипептиду или фрагменту, например, к миристоильной группе, достаточному для заякоривания внеклеточного или внутриклеточного домена на плазматической мембране.
Используемый в данном документе термин "переключающий домен", например, в отношении RCAR, относится к объекту, обычно полипептидному объекту, который в присутствии димеризующей молекулы ассоциирует с другим переключающим доменом. Ассоциация приводит к функциональному связыванию первого объекта, связанного, например, слитого, с первым переключающим доменом, и второго объекта, связанного, например, слитого, со вторым переключающим доменом. Первый и второй переключающие домены в совокупности называются димеризационным переключателем. В ряде вариантов осуществления первый и второй переключающие домены являются одинаковыми, например, они являются полипептидами, имеющими одинаковую первичную аминокислотную последовательность, и в совокупности называются гомодимеризационным переключателем. В ряде вариантов осуществления первый и второй переключающие домены отличаются друг от друга, например, они являются полипептидами, имеющими разные первичные аминокислотные последовательности, и в совокупности называются гетеродимеризационным переключателем. В ряде вариантов осуществления переключатель является внутриклеточным. В ряде вариантов осуществления переключатель является внеклеточным. В ряде вариантов осуществления переключающий домен представляет собой полипептидный объект, например, объект на основе FKBP или FRB, а димеризующая молекула представляет собой малую молекулу, например, рапалог. В ряде вариантов осуществления переключающий домен представляет собой полипептидный объект, например, scFv, который связывает пептид myc, а димеризующая молекула представляет собой полипептид, его фрагмент или мультимер полипептида, например, лиганд myc или мультимеры лиганда myc, которые связываются с одним или несколькими scFv для myc. В ряде вариантов осуществления переключающий домен представляет собой полипептидный объект, например, рецептор для myc, а димеризующая молекула представляет собой антитело или его фрагменты, например, антитело к myc.
Используемый в данном документе термин "димеризующая молекула", например, в отношении RCAR, относится к молекуле, которая способствует ассоциации первого переключающего домена со вторым переключающим доменом. В ряде вариантов осуществления димеризующая молекула не встречается у субъекта в природе или не встречается в концентрациях, которые могут привести к значительной степени димеризации. В ряде вариантов осуществления димеризующая молекула представляет собой малую молекулу, например, рапамицин или рапалог, например, RAD001.
Термин "низкая доза, усиливающая иммунный ответ" при использовании в отношении ингибитора mTOR, например, аллостерического ингибитора mTOR, например, RAD001 или рапамицина, или каталитического ингибитора mTOR относится к дозе ингибитора mTOR, которая обеспечивает частичное, но не полное, ингибирование активности mTOR, например, согласно измерению по ингибированию активности киназы P70-S6. Способы оценивания активности mTOR, например, по ингибированию киназы P70-S6, обсуждаются в данном документе. Доза является недостаточной, чтобы вызвать полное подавление иммунитета, но достаточной для усиления иммунного ответа. В некоторых вариантах осуществления низкая доза, усиливающая иммунный ответ, ингибитора mTOR приводит к уменьшению количества PD-1-положительных Т-клеток и/или увеличению количества PD-1-отрицательных Т-клеток или увеличению соотношения PD-1-отрицательные Т-клетки/PD-1-положительные Т-клетки. В некоторых вариантах осуществления низкая доза, усиливающая иммунный ответ, ингибитора mTOR приводит к увеличению количества необученных Т-клеток. В некоторых вариантах осуществления низкая доза, усиливающая иммунный ответ, ингибитора mTOR приводит к одному или нескольким из следующего:
повышению экспрессии одного или нескольких из следующих маркеров: CD62Lвысокий, CD127высокий, CD27+ и BCL2, например, на Т-клетках памяти, например, на предшественниках Т-клеток памяти;
снижению экспрессии KLRG1, например, на Т-клетках памяти, например, на предшественниках Т-клеток памяти; и
увеличению количества предшественников Т-клеток памяти, например, клеток с любой из следующих характеристик или их комбинацией: повышенным уровнем CD62Lвысокий, повышенным уровнем CD127высокий, повышенным уровнем CD27+, сниженным уровнем KLRG1 и повышенным уровнем BCL2;
где любое из изменений, описанных выше, происходит, например, по меньшей мере транзиентно, например, по сравнению с субъектом, не получающим лечение.
Как используется в данном документе, "рефрактерный" относится к заболеванию, например, раку, которое не отвечает на лечение. В ряде вариантов осуществления рефрактерный рак может быть устойчивым к лечению до или в начале лечения. В других вариантах осуществления рефрактерный рак может стать устойчивым в ходе лечения. Рефрактерный рак также называют устойчивым раком.
Как используется в данном документе, "рецидивирующий" или "рецидив" относится к возвращению или повторному появлению заболевания (например, рака) или признаков и симптомов заболевания, такого как рак, после периода улучшения или восприимчивости, например, после предшествующего лечения с помощью терапии, например, противораковой терапии. Начальный период восприимчивости может предусматривать падение уровня раковых клеток ниже определенного порога, например, ниже 20%, 1%, 10%, 5%, 4%, 3%, 2% или 1%. Повторное появление может предусматривать возрастание уровня раковых клеток выше определенного порога, например, выше 20%, 1%, 10%, 5%, 4%, 3%, 2% или 1%. Например, применительно к B-ALL повторное появление может предусматривать, например, повторное появление бластных клеток в крови, костном мозге (> 5%) или любом экстрамедуллярном участке после достижения полного ответа. Полный ответ в данном контексте может предусматривать < 5% бластных клеток в BM. В более широком смысле в некоторых вариантах осуществления ответ (например, полный ответ или частичный ответ) может предусматривать отсутствие выявляемого MRD (минимального остаточного заболевания). В некоторых вариантах осуществления начальный период восприимчивости продолжается в течение по меньшей мере 1, 2, 3, 4, 5 или 6 дней; по меньшей мере 1, 2, 3 или 4 недель; по меньшей мере 1, 2, 3, 4, 6, 8, 10 или 12 месяцев или по меньшей мере 1, 2, 3, 4 или 5 лет.
Диапазоны: во всем настоящем раскрытии различные варианты осуществления настоящего изобретения могут быть представлены в формате диапазонов. Следует понимать, что описание в формате диапазонов служит лишь для удобства и краткости и не должно рассматриваться как жесткое ограничение объема настоящего изобретения. Соответственно, описание диапазона следует рассматривать как конкретно раскрывающее все возможные поддиапазоны, а также отдельные числовые значения в пределах этого диапазона. Например, описание такого диапазона, как от 1 до 6, следует рассматривать как конкретно раскрывающее такие поддиапазоны, как от 1 до 3, от 1 до 4, от 1 до 5, от 2 до 4, от 2 до 6, от 3 до 6 и т. д., а также отдельные числа в пределах этого диапазона, например, 1, 2, 2,7, 3, 4, 5, 5,3 и 6. В качестве другого примера, такой диапазон, как 95-99% идентичность, включает что-либо с 95%, 96%, 97%, 98% или 99% идентичностью и включает такие поддиапазоны, как 96-99%, 96-98%, 96-97%, 97-99%, 97-98% и 98-99% идентичность. Это применимо независимо от ширины диапазона.
Используемый в данном документе термин "система редактирования генов" относится к системе, например, одной или нескольким молекулам, которые управляют изменением, например, делецией, и осуществляют его в одной или нескольких нуклеиновых кислотах в сайте геномной ДНК, на которую нацеливается указанная система, или рядом с ним. Системы редактирования генов известны из уровня техники и более подробно описаны ниже.
Как используется в данном документе, вводимый "в комбинации" означает, что два (или более) различных средства для лечения доставляют субъекту в период, когда субъект страдает нарушением, например, два или более средства для лечения доставляют после того, как у субъекта было диагностировано нарушение, и до того, как нарушение было излечено или устранено или лечение было прекращено по другим причинам. В некоторых вариантах осуществления доставка одного средства для лечения по-прежнему происходит, когда начинается доставка второго, так что в отношении введения имеет место перекрывание. Это иногда упоминается в данном документе как "одновременная" или "параллельная" доставка. В других вариантах осуществления доставка одного средства для лечения заканчивается до начала доставки другого средства для лечения. В некоторых вариантах осуществления в любом из случаев лечение является более эффективным благодаря комбинированному введению. Например, второе средство для лечения является более эффективным, например, эквивалентный эффект наблюдается при меньшем количестве второго средства для лечения, или второе средство для лечения снижает интенсивность симптомов в большей степени, чем наблюдалось бы при введении второго средства для лечения в отсутствие первого средства для лечения, или аналогичная ситуация наблюдается с первым средством для лечения. В некоторых вариантах осуществления доставка является такой, при которой снижение интенсивности симптома или другого параметра, связанного с нарушением, является большим, чем наблюдалось бы при доставке одного средства для лечения в отсутствие другого. Эффект двух средств для лечения может быть частично аддитивным, полностью аддитивным или превышающим аддитивный. Доставка может быть такой, что эффект от первого доставленного средства для лечения все еще поддается выявлению при доставке второго.
Термины "истощение" или "истощать", используемые взаимозаменяемо в данном документе, относятся к уменьшению или снижению уровня или количества клетки, белка или макромолекулы в образце после осуществления процесса, например, стадии отбора, например, отрицательного отбора. Истощение может быть полным или частичным истощением по клетке, белку или макромолекуле. В некоторых вариантах осуществления истощение представляет собой по меньшей мере 1%, 2%, 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, или 99% уменьшение или снижение уровня или количества клетки, белка или макромолекулы по сравнению с уровнем или количеством клетки, белка или макромолекулы в образце перед осуществлением процесса.
Как используется в данном документе, "необученная Т-клетка" относится к Т-клетке, которая является неопытной в отношении антигена. В некоторых вариантах осуществления Т-клетка, которая является неопытной в отношении антигена, встречалась со своим когнатным антигеном в тимусе, но не на периферии. В некоторых вариантах осуществления необученные Т-клетки являются предшественниками клеток памяти. В некоторых вариантах осуществления необученные Т-клетки экспрессируют как CD45RA, так и CCR7, но не экспрессируют CD45RO. В некоторых вариантах осуществления необученные Т-клетки могут характеризоваться экспрессией CD62L, CD27, CCR7, CD45RA, CD28 и CD127 и отсутствием CD95 или изоформы CD45RO. В некоторых вариантах осуществления необученные Т-клетки экспрессируют CD62L, рецептор IL-7 α, рецептор IL-6 и CD132, но не экспрессируют CD25, CD44, CD69 или CD45RO. В некоторых вариантах осуществления необученные Т-клетки экспрессируют CD45RA, CCR7 и CD62L и не экспрессируют CD95 или рецептор IL-2 β. В некоторых вариантах осуществления уровни экспрессии маркеров на поверхности оценивают с помощью проточной цитометрии.
Термин "центральные T-клетки памяти" относится к субпопуляции T-клеток, которые у людей являются CD45RO-положительными и экспрессируют CCR7. В некоторых вариантах осуществления центральные T-клетки памяти экспрессируют CD95. В некоторых вариантах осуществления центральные T-клетки памяти экспрессируют IL-2R, IL-7R и/или IL-15R. В некоторых вариантах осуществления центральные T-клетки памяти экспрессируют CD45RO, CD95, рецептор IL-2 β, CCR7 и CD62L. В некоторых вариантах осуществления уровни экспрессии маркеров на поверхности оценивают с помощью проточной цитометрии.
Термин "стволовые T-клетки памяти", "T-клетки памяти, представляющие собой стволовые клетки", "T-клетки памяти, подобные стволовым клеткам", "стволовые T-клетки, являющиеся клетками памяти", "стволовые клетки, являющиеся Т-клетками памяти", "клетки памяти, представляющие собой стволовые Т-клетки" или "TSCM-клетки" относится к субпопуляции T-клеток памяти со способностью, подобной таковой у стволовых клеток, например, способностью к самообновлению и/или с мультипотентным потенциалом преобразования в субпопуляции T-клеток памяти и/или эффектoрных Т-клеток. В некоторых вариантах осуществления стволовые T-клетки памяти экспрессируют CD45RA, CD95, рецептор IL-2 β, CCR7 и CD62L. В некоторых вариантах осуществления уровни экспрессии маркеров на поверхности оценивают с помощью проточной цитометрии. В некоторых вариантах осуществления иллюстративные стволовые T-клетки памяти раскрыты в Gattinoni et al., Nat Med. 2017 January 06; 23(1): 18-27, включенной в данный документ посредством ссылки во всей своей полноте.
Для ясности, если не указано иное, классификация клетки или популяции клеток в качестве "не экспрессирующих", или характеризующихся "отсутствием" определенного маркера, или являющихся "отрицательными в отношении" определенного маркера не обязательно может означать абсолютное отсутствие маркера. Специалист в данной области может без труда сравнить клетку с положительным и/или отрицательным контролем и/или установить предварительно определенное пороговое значение и классифицировать клетку или популяцию клеток в качестве не экспрессирующих или являющихся отрицательными в отношении маркера, если клетка характеризуется уровнем экспрессии ниже предварительно определенного порогового значения или популяция клеток характеризуется общим уровнем экспрессии ниже предварительно определенного порогового значения, с помощью традиционных способов выявления, например, с помощью проточной цитометрии, например, как описано в примерах в данном документе.
Используемый в данном документе термин "балл набора генов (активируемые в TEM относительно подавляемых в TSCM)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип эффектoрной Т-клетки памяти (TEM) по сравнению с фенотипом T-клетки памяти, представляющей собой стволовую клетку (TSCM). Более высокий балл набора генов (активируемые в TEM относительно подавляемых в TSCM) указывает на увеличение выраженности фенотипа TEM, тогда как более низкий балл набора генов (активируемые в TEM относительно подавляемых в TSCM) указывает на увеличение выраженности фенотипа TSCM. В некоторых вариантах осуществления балл набора генов (активируемые в TEM относительно подавляемых в TSCM) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в TEM-клетках и/или подавляются в TSCM-клетках, например, одного или нескольких генов, выбранных из группы, состоящей из MXRA7, CLIC1, NAT13, TBC1D2B, GLCCI1, DUSP10, APOBEC3D, CACNB3, ANXA2P2, TPRG1, EOMES, MATK, ARHGAP10, ADAM8, MAN1A1, SLFN12L, SH2D2A, EIF2C4, CD58, MYO1F, RAB27B, ERN1, NPC1, NBEAL2, APOBEC3G, SYTL2, SLC4A4, PIK3AP1, PTGDR, MAF, PLEKHA5, ADRB2, PLXND1, GNAO1, THBS1, PPP2R2B, CYTH3, KLRF1, FLJ16686, AUTS2, PTPRM, GNLY и GFPT2. В некоторых вариантах осуществления балл набора генов (активируемые в TEM относительно подавляемых в TSCM) определяют для каждой клетки с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), что, например, проиллюстрировано в примере 7 в отношении фиг. 25A. В некоторых вариантах осуществления балл набора генов (активируемые в TEM относительно подавляемых в TSCM) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (активируемые в Treg относительно подавляемых в Teff)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип регуляторной Т-клетки (Treg) по сравнению с фенотипом эффекторной Т-клетки (Teff). Более высокий балл набора генов (активируемые в Treg относительно подавляемых в Teff) указывает на увеличение выраженности фенотипа Treg, тогда как более низкий балл набора генов (активируемые в Treg относительно подавляемых в Teff) указывает на увеличение выраженности фенотипа Teff. В некоторых вариантах осуществления балл набора генов (активируемые в Treg относительно подавляемых в Teff) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в Treg-клетках и/или подавляются в Teff-клетках, например, одного или нескольких генов, выбранных из группы, состоящей из C12orf75, SELPLG, SWAP70, RGS1, PRR11, SPATS2L, SPATS2L, TSHR, C14orf145, CASP8, SYT11, ACTN4, ANXA5, GLRX, HLA-DMB, PMCH, RAB11FIP1, IL32, FAM160B1, SHMT2, FRMD4B, CCR3, TNFRSF13B, NTNG2, CLDND1, BARD1, FCER1G, TYMS, ATP1B1, GJB6, FGL2, TK1, SLC2A8, CDKN2A, SKAP2, GPR55, CDCA7, S100A4, GDPD5, PMAIP1, ACOT9, CEP55, SGMS1, ADPRH, AKAP2, HDAC9, IKZF4, CARD17, VAV3, OBFC2A, ITGB1, CIITA, SETD7, HLA-DMA, CCR10, KIAA0101, SLC14A1, PTTG3P, DUSP10, FAM164A, PYHIN1, MYO1F, SLC1A4, MYBL2, PTTG1, RRM2, TP53INP1, CCR5, ST8SIA6, TOX, BFSP2, ITPRIPL1, NCAPH, HLA-DPB2, SYT4, NINJ2, FAM46C, CCR4, GBP5, C15orf53, LMCD1, MKI67, NUSAP1, PDE4A, E2F2, CD58, ARHGEF12, LOC100188949, FAS, HLA-DPB1, SELP, WEE1, HLA-DPA1, FCRL1, ICA1, CNTNAP1, OAS1, METTL7A, CCR6, HLA-DRB4, ANXA2P3, STAM, HLA-DQB2, LGALS1, ANXA2, PI16, DUSP4, LAYN, ANXA2P2, PTPLA, ANXA2P1, ZNF365, LAIR2, LOC541471, RASGRP4, BCAS1, UTS2, MIAT, PRDM1, SEMA3G, FAM129A, HPGD, NCF4, LGALS3, CEACAM4, JAKMIP1, TIGIT, HLA-DRA, IKZF2, HLA-DRB1, FANK1, RTKN2, TRIB1, FCRL3 и FOXP3. В некоторых вариантах осуществления балл набора генов (активируемые в Treg относительно подавляемых в Teff) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), что, например, проиллюстрировано в примере 7 в отношении фиг. 25B. В некоторых вариантах осуществления балл набора генов (активируемые в Treg относительно подавляемых в Teff) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (подавляемые в состоянии стволовости)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип стволовости. Более низкий балл набора генов (подавляемые в состоянии стволовости) указывает на увеличение выраженности фенотипа стволовости. В некоторых вариантах осуществления балл набора генов (подавляемые в состоянии стволовости) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в дифференцирующейся стволовой клетке, относительно генов, которые подавляются в гемопоэтической стволовой клетке, например, одного или нескольких генов, выбранных из группы, состоящей из ACE, BATF, CDK6, CHD2, ERCC2, HOXB4, MEOX1, SFRP1, SP7, SRF, TAL1 и XRCC5. В некоторых вариантах осуществления балл набора генов (подавляемые в состоянии стволовости) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), как, например, проиллюстрировано в примере 7 в отношении фиг. 25C. В некоторых вариантах осуществления балл набора генов (подавляемые в состоянии стволовости) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (активируемые при гипоксии)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип, характерный для гипоксии. Более высокий балл набора генов (активируемые при гипоксии) указывает на увеличение выраженности фенотипа, характерного для гипоксии. В некоторых вариантах осуществления балл набора генов (активируемые при гипоксии) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в клетках, подвергающихся гипоксии, например, одного или нескольких генов, выбранных из группы, состоящей из ABCB1, ACAT1, ADM, ADORA2B, AK2, AK3, ALDH1A1, ALDH1A3, ALDOA, ALDOC, ANGPT2, ANGPTL4, ANXA1, ANXA2, ANXA5, ARHGAP5, ARSE, ART1, BACE2, BATF3, BCL2L1, BCL2L2, BHLHE40, BHLHE41, BIK, BIRC2, BNIP3, BNIP3L, BPI, BTG1, C11orf2, C7orf68, CA12, CA9, CALD1, CCNG2, CCT6A, CD99, CDK1, CDKN1A, CDKN1B, CITED2, CLK1, CNOT7, COL4A5, COL5A1, COL5A2, COL5A3, CP, CTSD, CXCR4, D4S234E, DDIT3, DDIT4, 1-Dec, DKC1, DR1, EDN1, EDN2, EFNA1, EGF, EGR1, EIF4A3, ELF3, ELL2, ENG, ENO1, ENO3, ENPEP, EPO, ERRFI1, ETS1, F3, FABP5, FGF3, FKBP4, FLT1, FN1, FOS, FTL, GAPDH, GBE1, GLRX, GPI, GPRC5A, HAP1, HBP1, HDAC1, HDAC9, HERC3, HERPUD1, HGF, HIF1A, HK1, HK2, HLA-DQB1, HMOX1, HMOX2, HSPA5, HSPD1, HSPH1, HYOU1, ICAM1, ID2, IFI27, IGF2, IGFBP1, IGFBP2, IGFBP3, IGFBP5, IL6, IL8, INSIG1, IRF6, ITGA5, JUN, KDR, KRT14, KRT18, KRT19, LDHA, LDHB, LEP, LGALS1, LONP1, LOX, LRP1, MAP4, MET, MIF, MMP13, MMP2, MMP7, MPI, MT1L, MTL3P, MUC1, MXI1, NDRG1, NFIL3, NFKB1, NFKB2, NOS1, NOS2, NOS2P1, NOS2P2, NOS3, NR3C1, NR4A1, NT5E, ODC1, P4HA1, P4HA2, PAICS, PDGFB, PDK3, PFKFB1, PFKFB3, PFKFB4, PFKL, PGAM1, PGF, PGK1, PGK2, PGM1, PIM1, PIM2, PKM2, PLAU, PLAUR, PLIN2, PLOD2, PNN, PNP, POLM, PPARA, PPAT, PROK1, PSMA3, PSMD9, PTGS1, PTGS2, QSOX1, RBPJ, RELA, RIOK3, RNASEL, RPL36A, RRP9, SAT1, SERPINB2, SERPINE1, SGSM2, SIAH2, SIN3A, SIRPA, SLC16A1, SLC16A2, SLC20A1, SLC2A1, SLC2A3, SLC3A2, SLC6A10P, SLC6A16, SLC6A6, SLC6A8, SORL1, SPP1, SRSF6, SSSCA1, STC2, STRA13, SYT7, TBPL1, TCEAL1, TEK, TF, TFF3, TFRC, TGFA, TGFB1, TGFB3, TGFBI, TGM2, TH, THBS1, THBS2, TIMM17A, TNFAIP3, TP53, TPBG, TPD52, TPI1, TXN, TXNIP, UMPS, VEGFA, VEGFB, VEGFC, VIM, VPS11 и XRCC6. В некоторых вариантах осуществления балл набора генов (активируемые при гипоксии) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), как, например, проиллюстрировано в примере 7 в отношении фиг. 25D. В некоторых вариантах осуществления балл набора генов (активируемые при гипоксии) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (активируемые при аутофагии)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип, характерный для аутофагии. Более высокий балл набора генов (активируемые при аутофагии) указывает на увеличение выраженности фенотипа, характерного для аутофагии. В некоторых вариантах осуществления балл набора генов (активируемые при аутофагии) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в клетках, подвергающихся аутофагии, например, одного или нескольких генов, выбранных из группы, состоящей из ABL1, ACBD5, ACIN1, ACTRT1, ADAMTS7, AKR1E2, ALKBH5, ALPK1, AMBRA1, ANXA5, ANXA7, ARSB, ASB2, ATG10, ATG12, ATG13, ATG14, ATG16L1, ATG16L2, ATG2A, ATG2B, ATG3, ATG4A, ATG4B, ATG4C, ATG4D, ATG5, ATG7, ATG9A, ATG9B, ATP13A2, ATP1B1, ATPAF1-AS1, ATPIF1, BECN1, BECN1P1, BLOC1S1, BMP2KL, BNIP1, BNIP3, BOC, C11orf2, C11orf41, C12orf44, C12orf5, C14orf133, C1orf210, C5, C6orf106, C7orf59, C7orf68, C8orf59, C9orf72, CA7, CALCB, CALCOCO2, CAPS, CCDC36, CD163L1, CD93, CDC37, CDKN2A, CHAF1B, CHMP2A, CHMP2B, CHMP3, CHMP4A, CHMP4B, CHMP4C, CHMP6, CHST3, CISD2, CLDN7, CLEC16A, CLN3, CLVS1, COX8A, CPA3, CRNKL1, CSPG5, CTSA, CTSB, CTSD, CXCR7, DAP, DKKL1, DNAAF2, DPF3, DRAM1, DRAM2, DYNLL1, DYNLL2, DZANK1, EI24, EIF2S1, EPG5, EPM2A, FABP1, FAM125A, FAM131B, FAM134B, FAM13B, FAM176A, FAM176B, FAM48A, FANCC, FANCF, FANCL, FBXO7, FCGR3B, FGF14, FGF7, FGFBP1, FIS1, FNBP1L, FOXO1, FUNDC1, FUNDC2, FXR2, GABARAP, GABARAPL1, GABARAPL2, GABARAPL3, GABRA5, GDF5, GMIP, HAP1, HAPLN1, HBXIP, HCAR1, HDAC6, HGS, HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3H, HIST1H3I, HIST1H3J, HK2, HMGB1, HPR, HSF2BP, HSP90AA1, HSPA8, IFI16, IPPK, IRGM, IST1, ITGB4, ITPKC, KCNK3, KCNQ1, KIAA0226, KIAA1324, KRCC1, KRT15, KRT73, LAMP1, LAMP2, LAMTOR1, LAMTOR2, LAMTOR3, LARP1B, LENG9, LGALS8, LIX1, LIX1L, LMCD1, LRRK2, LRSAM1, LSM4, MAP1A, MAP1LC3A, MAP1LC3B, MAP1LC3B2, MAP1LC3C, MAP1S, MAP2K1, MAP3K12, MARK2, MBD5, MDH1, MEX3C, MFN1, MFN2, MLST8, MRPS10, MRPS2, MSTN, MTERFD1, MTMR14, MTMR3, MTOR, MTSS1, MYH11, MYLK, MYOM1, NBR1, NDUFB9, NEFM, NHLRC1, NME2, NPC1, NR2C2, NRBF2, NTHL1, NUP93, OBSCN, OPTN, P2RX5, PACS2, PARK2, PARK7, PDK1, PDK4, PEX13, PEX3, PFKP, PGK2, PHF23, PHYHIP, PI4K2A, PIK3C3, PIK3CA, PIK3CB, PIK3R4, PINK1, PLEKHM1, PLOD2, PNPO, PPARGC1A, PPY, PRKAA1, PRKAA2, PRKAB1, PRKAB2, PRKAG1, PRKAG2, PRKAG3, PRKD2, PRKG1, PSEN1, PTPN22, RAB12, RAB1A, RAB1B, RAB23, RAB24, RAB33B, RAB39, RAB7A, RB1CC1, RBM18, REEP2, REP15, RFWD3, RGS19, RHEB, RIMS3, RNF185, RNF41, RPS27A, RPTOR, RRAGA, RRAGB, RRAGC, RRAGD, S100A8, S100A9, SCN1A, SERPINB10, SESN2, SFRP4, SH3GLB1, SIRT2, SLC1A3, SLC1A4, SLC22A3, SLC25A19, SLC35B3, SLC35C1, SLC37A4, SLC6A1, SLCO1A2, SMURF1, SNAP29, SNAPIN, SNF8, SNRPB, SNRPB2, SNRPD1, SNRPF, SNTG1, SNX14, SPATA18, SQSTM1, SRPX, STAM, STAM2, STAT2, STBD1, STK11, STK32A, STOM, STX12, STX17, SUPT3H, TBC1D17, TBC1D25, TBC1D5, TCIRG1, TEAD4, TECPR1, TECPR2, TFEB, TM9SF1, TMBIM6, TMEM203, TMEM208, TMEM39A, TMEM39B, TMEM59, TMEM74, TMEM93, TNIK, TOLLIP, TOMM20, TOMM22, TOMM40, TOMM5, TOMM6, TOMM7, TOMM70A, TP53INP1, TP53INP2, TRAPPC8, TREM1, TRIM17, TRIM5, TSG101, TXLNA, UBA52, UBB, UBC, UBQLN1, UBQLN2, UBQLN4, ULK1, ULK2, ULK3, USP10, USP13, USP30, UVRAG, VAMP7, VAMP8, VDAC1, VMP1, VPS11, VPS16, VPS18, VPS25, VPS28, VPS33A, VPS33B, VPS36, VPS37A, VPS37B, VPS37C, VPS37D, VPS39, VPS41, VPS4A, VPS4B, VTA1, VTI1A, VTI1B, WDFY3, WDR45, WDR45L, WIPI1, WIPI2, XBP1, YIPF1, ZCCHC17, ZFYVE1, ZKSCAN3, ZNF189, ZNF593 и ZNF681. В некоторых вариантах осуществления балл набора генов (активируемые при аутофагии) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), как, например, проиллюстрировано в примере 7 в отношении фиг. 25E. В некоторых вариантах осуществления балл набора генов (активируемые при аутофагии) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип покоящейся Т-клетки по сравнению с фенотипом активированной Т-клетки. Более высокий балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) указывает на увеличение выраженности фенотипа покоящихся Т-клеток, тогда как более низкий балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) указывает на увеличение выраженности фенотипа активированных T-клеток. В некоторых вариантах осуществления балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в покоящихся T-клетках и/или подавляются в активированных T-клетках, например, одного или нескольких генов, выбранных из группы, состоящей из ABCA7, ABCF3, ACAP2, AMT, ANKH, ATF7IP2, ATG14, ATP1A1, ATXN7, ATXN7L3B, BCL7A, BEX4, BSDC1, BTG1, BTG2, BTN3A1, C11orf21, C19orf22, C21orf2, CAMK2G, CARS2, CCNL2, CD248, CD5, CD55, CEP164, CHKB, CLK1, CLK4, CTSL1, DBP, DCUN1D2, DENND1C, DGKD, DLG1, DUSP1, EAPP, ECE1, ECHDC2, ERBB2IP, FAM117A, FAM134B, FAM134C, FAM169A, FAM190B, FAU, FLJ10038, FOXJ2, FOXJ3, FOXL1, FOXO1, FXYD5, FYB, HLA-E, HSPA1L, HYAL2, ICAM2, IFIT5, IFITM1, IKBKB, IQSEC1, IRS4, KIAA0664L3, KIAA0748, KLF3, KLF9, KRT18, LEF1, LINC00342, LIPA, LIPT1, LLGL2, LMBR1L, LPAR2, LTBP3, LYPD3, LZTFL1, MANBA, MAP2K6, MAP3K1, MARCH8, MAU2, MGEA5, MMP8, MPO, MSL1, MSL3, MYH3, MYLIP, NAGPA, NDST2, NISCH, NKTR, NLRP1, NOSIP, NPIP, NUMA1, PAIP2B, PAPD7, PBXIP1, PCIF1, PI4KA, PLCL2, PLEKHA1, PLEKHF2, PNISR, PPFIBP2, PRKCA, PRKCZ, PRKD3, PRMT2, PTP4A3, PXN, RASA2, RASA3, RASGRP2, RBM38, REPIN1, RNF38, RNF44, ROR1, RPL30, RPL32, RPLP1, RPS20, RPS24, RPS27, RPS6, RPS9, RXRA, RYK, SCAND2, SEMA4C, SETD1B, SETD6, SETX, SF3B1, SH2B1, SLC2A4RG, SLC35E2B, SLC46A3, SMAGP, SMARCE1, SMPD1, SNPH, SP140L, SPATA6, SPG7, SREK1IP1, SRSF5, STAT5B, SVIL, SYF2, SYNJ2BP, TAF1C, TBC1D4, TCF20, TECTA, TES, TMEM127, TMEM159, TMEM30B, TMEM66, TMEM8B, TP53TG1, TPCN1, TRIM22, TRIM44, TSC1, TSC22D1, TSC22D3, TSPYL2, TTC9, TTN, UBE2G2, USP33, USP34, VAMP1, VILL, VIPR1, VPS13C, ZBED5, ZBTB25, ZBTB40, ZC3H3, ZFP161, ZFP36L1, ZFP36L2, ZHX2, ZMYM5, ZNF136, ZNF148, ZNF318, ZNF350, ZNF512B, ZNF609, ZNF652, ZNF83, ZNF862 и ZNF91. В некоторых вариантах осуществления балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), как, например, проиллюстрировано в примере 7 в отношении фиг. 24D. В некоторых вариантах осуществления балл набора генов (активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (постепенно активируемые при дифференцировке в клетки памяти)" для клетки относится к баллу, который отражает стадию дифференцировки в клетки памяти. Более высокий балл набора генов (постепенно активируемые при дифференцировке в клетки памяти) указывает на увеличение выраженности фенотипа поздних Т-клеток памяти, тогда как более низкий балл набора генов (постепенно активируемые при дифференцировке в клетки памяти) указывает на увеличение выраженности фенотипа ранних Т-клеток памяти. В некоторых вариантах осуществления балл набора генов (активируемые при аутофагии) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в ходе дифференцировки в клетки памяти, например, одного или нескольких генов, выбранных из группы, состоящей из MTCH2, RAB6C, KIAA0195, SETD2, C2orf24, NRD1, GNA13, COPA, SELT, TNIP1, CBFA2T2, LRP10, PRKCI, BRE, ANKS1A, PNPLA6, ARL6IP1, WDFY1, MAPK1, GPR153, SHKBP1, MAP1LC3B2, PIP4K2A, HCN3, GTPBP1, TLN1, C4orf34, KIF3B, TCIRG1, PPP3CA, ATG4D, TYMP, TRAF6, C17orf76, WIPF1, FAM108A1, MYL6, NRM, SPCS2, GGT3P, GALK1, CLIP4, ARL4C, YWHAQ, LPCAT4, ATG2A, IDS, TBC1D5, DMPK, ST6GALNAC6, REEP5, ABHD6, KIAA0247, EMB, TSEN54, SPIRE2, PIWIL4, ZSCAN22, ICAM1, CHD9, LPIN2, SETD8, ZC3H12A, ULBP3, IL15RA, HLA-DQB2, LCP1, CHP, RUNX3, TMEM43, REEP4, MEF2D, ABL1, TMEM39A, PCBP4, PLCD1, CHST12, RASGRP1, C1orf58, C11orf63, C6orf129, FHOD1, DKFZp434F142, PIK3CG, ITPR3, BTG3, C4orf50, CNNM3, IFI16, AK1, CDK2AP1, REL, BCL2L1, MVD, TTC39C, PLEKHA2, FKBP11, EML4, FANCA, CDCA4, FUCA2, MFSD10, TBCD, CAPN2, IQGAP1, CHST11, PIK3R1, MYO5A, KIR2DL3, DLG3, MXD4, RALGDS, S1PR5, WSB2, CCR3, TIPARP, SP140, CD151, SOX13, KRTAP5-2, NF1, PEA15, PARP8, RNF166, UEVLD, LIMK1, CACNB1, TMX4, SLC6A6, LBA1, SV2A, LLGL2, IRF1, PPP2R5C, CD99, RAPGEF1, PPP4R1, OSBPL7, FOXP4, SLA2, TBC1D2B, ST7, JAZF1, GGA2, PI4K2A, CD68, LPGAT1, STX11, ZAK, FAM160B1, RORA, C8orf80, APOBEC3F, TGFBI, DNAJC1, GPR114, LRP8, CD69, CMIP, NAT13, TGFB1, FLJ00049, ANTXR2, NR4A3, IL12RB1, NTNG2, RDX, MLLT4, GPRIN3, ADCY9, CD300A, SCD5, ABI3, PTPN22, LGALS1, SYTL3, BMPR1A, TBK1, PMAIP1, RASGEF1A, GCNT1, GABARAPL1, STOM, CALHM2, ABCA2, PPP1R16B, SYNE2, PAM, C12orf75, CLCF1, MXRA7, APOBEC3C, CLSTN3, ACOT9, HIP1, LAG3, TNFAIP3, DCBLD1, KLF6, CACNB3, RNF19A, RAB27A, FADS3, DLG5, APOBEC3D, TNFRSF1B, ACTN4, TBKBP1, ATXN1, ARAP2, ARHGEF12, FAM53B, MAN1A1, FAM38A, PLXNC1, GRLF1, SRGN, HLA-DRB5, B4GALT5, WIPI1, PTPRJ, SLFN11, DUSP2, ANXA5, AHNAK, NEO1, CLIC1, EIF2C4, MAP3K5, IL2RB, PLEKHG1, MYO6, GTDC1, EDARADD, GALM, TARP, ADAM8, MSC, HNRPLL, SYT11, ATP2B4, NHSL2, MATK, ARHGAP18, SLFN12L, SPATS2L, RAB27B, PIK3R3, TP53INP1, MBOAT1, GYG1, KATNAL1, FAM46C, ZC3HAV1L, ANXA2P2, CTNNA1, NPC1, C3AR1, CRIM1, SH2D2A, ERN1, YPEL1, TBX21, SLC1A4, FASLG, PHACTR2, GALNT3, ADRB2, PIK3AP1, TLR3, PLEKHA5, DUSP10, GNAO1, PTGDR, FRMD4B, ANXA2, EOMES, CADM1, MAF, TPRG1, NBEAL2, PPP2R2B, PELO, SLC4A4, KLRF1, FOSL2, RGS2, TGFBR3, PRF1, MYO1F, GAB3, C17orf66, MICAL2, CYTH3, TOX, HLA-DRA, SYNE1, WEE1, PYHIN1, F2R, PLD1, THBS1, CD58, FAS, NETO2, CXCR6, ST6GALNAC2, DUSP4, AUTS2, C1orf21, KLRG1, TNIP3, GZMA, PRR5L, PRDM1, ST8SIA6, PLXND1, PTPRM, GFPT2, MYBL1, SLAMF7, FLJ16686, GNLY, ZEB2, CST7, IL18RAP, CCL5, KLRD1 и KLRB1. В некоторых вариантах осуществления балл набора генов (постепенно активируемые при дифференцировке в клетки памяти) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), как, например, проиллюстрировано в примере 7 в отношении фиг. 26B. В некоторых вариантах осуществления балл набора генов (постепенно активируемые при дифференцировке в клетки памяти) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Используемый в данном документе термин "балл набора генов (активируемые в TEM относительно подавляемых в TN)" для клетки относится к баллу, который отражает степень, в которой клетка демонстрирует фенотип эффекторной Т-клетки памяти (TEM) по сравнению с фенотипом необученной Т-клетки (TN). Более высокий балл набора генов (активируемые в TEM относительно подавляемых в TN) указывает на увеличение выраженности фенотипа TEM, тогда как более низкий балл набора генов (активируемые в TEM относительно подавляемых в TN) указывает на увеличение выраженности фенотипа TN. В некоторых вариантах осуществления балл набора генов (активируемые в TEM относительно подавляемых в TN) определяют путем измерения экспрессии одного или нескольких генов, которые активируются в TEM-клетках и/или подавляются в TN-клетках, например, одного или нескольких генов, выбранных из группы, состоящей из MYO5A, MXD4, STK3, S1PR5, GLCCI1, CCR3, SOX13, KRTAP5-2, PEA15, PARP8, RNF166, UEVLD, LIMK1, SLC6A6, SV2A, KPNA2, OSBPL7, ST7, GGA2, PI4K2A, CD68, ZAK, RORA, TGFBI, DNAJC1, JOSD1, ZFYVE28, LRP8, OSBPL3, CMIP, NAT13, TGFB1, ANTXR2, NR4A3, RDX, ADCY9, CHN1, CD300A, SCD5, PTPN22, LGALS1, RASGEF1A, GCNT1, GLUL, ABCA2, CLDND1, PAM, CLCF1, MXRA7, CLSTN3, ACOT9, METRNL, BMPR1A, LRIG1, APOBEC3G, CACNB3, RNF19A, RAB27A, FADS3, ACTN4, TBKBP1, FAM53B, MAN1A1, FAM38A, GRLF1, B4GALT5, WIPI1, DUSP2, ANXA5, AHNAK, CLIC1, MAP3K5, ST8SIA1, TARP, ADAM8, MATK, SLFN12L, PIK3R3, FAM46C, ANXA2P2, CTNNA1, NPC1, SH2D2A, ERN1, YPEL1, TBX21, STOM, PHACTR2, GBP5, ADRB2, PIK3AP1, DUSP10, PTGDR, EOMES, MAF, TPRG1, NBEAL2, NCAPH, SLC4A4, FOSL2, RGS2, TGFBR3, MYO1F, C17orf66, CYTH3, WEE1, PYHIN1, F2R, THBS1, CD58, AUTS2, FAM129A, TNIP3, GZMA, PRR5L, PRDM1, PLXND1, PTPRM, GFPT2, MYBL1, SLAMF7, ZEB2, CST7, CCL5, GZMK и KLRB1. В некоторых вариантах осуществления балл набора генов (активируемые в TEM относительно подавляемых в TN) определяют с помощью секвенирования РНК, например, секвенирования РНК одиночных клеток (scRNA-seq), как, например, проиллюстрировано в примере 7 в отношении фиг. 26C. В некоторых вариантах осуществления балл набора генов (активируемые в TEM относительно подавляемых в TN) рассчитывают с использованием среднего логарифмически нормализованного значения экспрессии гена для всех генов в наборе генов.
Применительно к значениям балла набора генов (например, медианным значениям балла набора генов), если положительный балл набора генов уменьшить на 100%, получим значение 0. Если отрицательный балл набора генов увеличить на 100%, получим значение 0. Например, на фиг. 25A медианный балл набора генов для образца в день 1 составляет -0,084; медианный балл набора генов для образца в день 9 составляет 0,035; и медианный балл набора генов для исходного образца составляет -0,1. На фиг. 25A увеличение медианного балла набора генов для исходного образца на 100% приводит к получению значения балла набора генов, составляющего 0; и увеличение медианного балла набора генов для исходного образца на 200% приводит к получению значения балла набора генов, составляющего 0,1. На фиг. 25A уменьшение медианного балла набора генов для образца в день 9 на 100% приводит к получению значения балла набора генов, составляющего 0; и уменьшение медианного балла набора генов для образца в день 9 на 200% приводит к получению значения балла набора генов, составляющего -0,035.
Используемый в данном документе термин "гранула" относится к дискретной частице с твердой поверхностью, размер которой находится в пределах от примерно 0,1 мкм до нескольких миллиметров в диаметре. Гранулы могут быть сферическими (например, микросферы) или иметь неправильную форму. Гранулы могут содержать разнообразные материалы, включающие без ограничения парамагнитные материалы, керамику, пластик, стекло, полистирол, метилстирол, акриловые полимеры, титан, латекс, Sepharose™, целлюлозу, нейлон и т. п. В некоторых вариантах осуществления гранулы представляют собой относительно однородные сферические суперпарамагнитные полистирольные гранулы диаметром приблизительно 4,5 мкм, например, покрытые смесью антител к CD3 (например, CD3-эпсилон) и CD28, например, ковалентно связанные с ней. В некоторых вариантах осуществления гранулы представляют собой Dynabeads®. В некоторых вариантах осуществления как антитела к CD3, так и антитела к CD28, связаны с одной и той же гранулой, имитируя стимуляцию T-клетки антигенпрезентирующими клетками. Свойство Dynabeads® и применение Dynabeads® для выделения и размножения клеток хорошо известны из уровня техники, например, см., Neurauter et al., Cell isolation and expansion using Dynabeads, Adv Biochem Eng Biotechnol. 2007;106:41-73, включенную в данный документ посредством ссылки во всей своей полноте.
Используемый в данном документе термин "наноматрица" относится к наноструктуре, содержащей матрицу из подвижных полимерных цепей. Размер наноматрицы составляет от 1 до 500 нм, например, от 10 до 200 нм. В некоторых вариантах осуществления матрица из подвижных полимерных цепей присоединена к одному или нескольким агонистам, которые передают сигналы активации к T-клеткам, например, агонистам, представляющим собой антитела к CD3 и/или антитела к CD28. В некоторых вариантах осуществления наноматрица предусматривает коллоидную полимерную наноматрицу, присоединенную, например, ковалентно присоединенную, к агонисту одной или нескольких стимулирующих молекул и/или агонисту одной или нескольких костимулирующих молекул. В некоторых вариантах осуществления агонист одной или нескольких стимулирующих молекул представляет собой агонист CD3 (например, агонистическое антитело к CD3). В некоторых вариантах осуществления агонист одной или нескольких костимулирующих молекул представляет собой агонист CD28 (например, агонистическое антитело к CD28). В некоторых вариантах осуществления наноматрица характеризуется отсутствием твердой поверхности, например, в качестве точки присоединения агонистов, таких как антитела к CD3 и/или антитела к CD28. В некоторых вариантах осуществления наноматрица представляет собой наноматрицу, раскрытую в WO2014/048920A1 или предоставляемую в наборе MACS® GMP TransAct™ для T-клеток от Miltenyi Biotec GmbH, включенных в данный документ посредством ссылки во всей своей полноте. MACS® GMP TransAct™ для T-клеток состоит из коллоидной полимерной наноматрицы, ковалентно присоединенной к гуманизированным рекомбинантным агонистическим антителам к CD3 и CD28 человека.
Ниже более подробно описаны различные варианты осуществления композиций и способов, представленных в данном документе. Дополнительные определения изложены по всему тексту настоящего описания.
Описание
В данном документе предусмотрены композиции и способы применения для лечения заболевания, такого как рак, с помощью клеток, экспрессирующих один или несколько химерных антигенных рецепторов (CAR). В некоторых вариантах осуществления в настоящем изобретении предусмотрена клетка (например, иммунная эффекторная клетка, например, T-клетка или NK-клетка), сконструированная таким образом, что она экспрессирует один или несколько CAR, где CAR-T-клетка ("CAR-T") или CAR-NK-клетка демонстрирует противоопухолевое свойство.
В некоторых вариантах осуществления клетка экспрессирует по меньшей мере два CAR. В некоторых вариантах осуществления клетка экспрессирует первый CAR, который связывается с первым антигеном, и второй CAR, который связывается со вторым антигеном. В некоторых вариантах осуществления первый антиген и второй антиген являются разными. В некоторых вариантах осуществления первый антиген представляет собой BCMA. В некоторых вариантах осуществления первый CAR представляет собой CAR для BCMA, содержащий последовательность CDR, VH, VL, scFv или CAR, раскрытую в данном документе, например, последовательность, раскрытую в таблицах 3-15, 19, 20, 22 и 26, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней, например, CAR для BCMA, раскрытый в данном документе. В некоторых вариантах осуществления второй антиген представляет собой CD19. В некоторых вариантах осуществления второй антиген представляет собой CAR для CD19, содержащий последовательность CDR, VH, VL, scFv или CAR, раскрытую в данном документе, например, последовательность, раскрытую в таблицах 2, 19 и 22, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней, например, CAR для CD19, раскрытый в данном документе. В некоторых вариантах осуществления первый CAR и второй CAR экспрессируются последовательностями нуклеиновой кислоты, расположенными в одной молекуле нуклеиновой кислоты. В некоторых вариантах осуществления последовательность нуклеиновой кислоты, кодирующая первый CAR, и последовательность нуклеиновой кислоты, кодирующая второй CAR, разделены последовательностью нуклеиновой кислоты, кодирующей сайт саморасщепления, например, сайт P2A, сайт Т2A, сайт Е2A или сайт F2A. В некоторых вариантах осуществления клетка представляет собой клетку, экспрессирующую двойные CAR, раскрытые в данном документе. В некоторых вариантах осуществления первый CAR и второй CAR экспрессируются последовательностями нуклеиновой кислоты, расположенными в отдельных молекулах нуклеиновой кислоты. В некоторых вариантах осуществления клетка сконструирована с использованием системы котрансдукции, раскрытой в данном документе.
В некоторых вариантах осуществления клетка экспрессирует CAR, который связывается с первым антигеном и вторым антигеном. В некоторых вариантах осуществления CAR представляет собой CAR в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления CAR содержит связывающий домен, который содержит первую VH (VH1), первую VL (VL1), вторую VH (VH2) и вторую VL (VL2). В некоторых вариантах осуществления VH1 и VL1 связываются с первым антигеном, а VH2 и VL2 связываются со вторым антигеном. В некоторых вариантах осуществления VH1, VL1, VH2 и VL2 расположены в следующей конфигурации от N-конца до C-конца: VH1 - необязательно линкер 1 ("L1") - VH2 - необязательно линкер 2 ("L2") - VL2 - необязательно линкер 3 ("L3") - VL1, VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1, VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1, VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1, VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2, VH2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VL2, VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2 или VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2.
В некоторых вариантах осуществления в CAR по настоящему изобретению антигенсвязывающий домен объединен с внутриклеточной сигнальной молекулой. Например, в некоторых вариантах осуществления внутриклеточная сигнальная молекула содержит без ограничения дзета-цепь CD3, сигнальные модули 4-1BB и CD28 и их комбинации.
Кроме того, в настоящем изобретении предусмотрены композиции на основе CAR и их применение в лекарственных препаратах или способах лечения, среди прочих заболеваний, рака, или любого злокачественного новообразования, или аутоиммунных заболеваний.
Химерный антигенный рецептор (CAR)
В настоящем изобретении предусмотрены иммунные эффекторные клетки (например, T-клетки, NK-клетки), сконструированные таким образом, что они содержат один или несколько CAR, которые направляют иммунные эффекторные клетки на раковое образование. Это достигается с помощью антигенсвязывающего домена в CAR, который является специфичным в отношении антигена, ассоциированного с раком. Существует два класса антигенов, ассоциированных с раком (опухолевых антигенов), на которые могут нацеливаться CAR, описанные в данном документе: (1) антигены, ассоциированные с раком, которые экспрессируются на поверхности раковых клеток; и (2) антигены, ассоциированные с раком, которые сами по себе являются внутриклеточными, однако фрагменты (пептиды) такого антигена презентируются на поверхности раковых клеток с помощью MHC (главного комплекса гистосовместимости).
Соответственно, иммунная эффекторная клетка, например, полученная посредством способа, описанного в данном документе, может быть сконструирована таким образом, чтобы она содержала CAR, нацеливающийся на один из следующих антигенов, ассоциированных с раком (опухолевых антигенов): CD19, CD123, CD22, CD30, CD171, CS-1, CLL-1, CD33, EGFRvIII, GD2, GD3, BCMA, Ag Tn, PSMA, ROR1, FLT3, FAP, TAG72, CD38, CD44v6, CEA, EPCAM, B7H3, KIT, IL-13Ra2, мезотелин, IL-11Ra, PSCA, VEGFR2, антиген Y системы Льюис, CD24, PDGFR-бета, PRSS21, SSEA-4, CD20, рецептор фолиевой кислоты альфа, ERBB2 (Her2/neu), MUC1, EGFR, NCAM, простазу, PAP, ELF2M, эфрин B2, рецептор IGF-I, CAIX, LMP2, gp100, bcr-abl, тирозиназу, EphA2, фукозил-GM1, sLe, GM3, TGS5, HMWMAA, o-ацетил-GD2, рецептор фолиевой кислоты бета, TEM1/CD248, TEM7R, CLDN6, TSHR, GPRC5D, CXORF61, CD97, CD179a, ALK, полисиаловую кислоту, PLAC1, GloboH, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, GPR20, LY6K, OR51E2, TARP, WT1, NY-ESO-1, LAGE-1a, легумаин, E6 и E7 HPV, MAGE-A1, MAGE A1, ETV6-AML, белок спермы 17, XAGE1, Tie 2, MAD-CT-1, MAD-CT-2, Fos-родственный антиген 1, p53, мутантный p53, простеин, сурвивин и теломеразу, PCTA-1/галектин 8, MelanA/MART1, мутантный Ras, hTERT, антигены саркомы с точечными разрывами при транслокации, ML-IAP, ERG (продукт слитого гена TMPRSS2-ETS), NA17, PAX3, андрогеновый рецептор, циклин B1, MYCN, RhoC, TRP-2, CYP1B1, BORIS, SART3, PAX5, OY-TES1, LCK, AKAP-4, SSX2, RAGE-1, обратную транскриптазу теломеразы человека, RU1, RU2, кишечную карбоксилэстеразу и мутантный вариант hsp70-2.
Последовательности из неограничивающих примеров различных компонентов, которые могут быть частью молекулы CAR, описанной в данном документе, перечислены в таблице 1, где "а. к." обозначает аминокислоты, а "н. к." обозначает нуклеиновые кислоты, которые кодируют соответствующий пептид.
(н. к.)
(с мутацией Q/K)
(с мутацией Q/K)
(эталонная последовательность NM_000734.3 в NCBI)
(эталонная последовательность NM_000734.3 в NCBI)
Эта последовательность может охватывать 50-5000 адениновых оснований.
Эта последовательность может охватывать 50-5000 тиминовых оснований.
Эта последовательность может охватывать 100-5000 адениновых оснований.
Эта последовательность может охватывать 100-400 адениновых оснований.
Эта последовательность может охватывать 50-2000 адениновых оснований.
(ECD PD1 подчеркнут)
(ECD PD1 подчеркнут)
В некоторых вариантах осуществления антигенсвязывающий домен содержит внеклеточный домен или его противолигандсвязывающий фрагмент из молекулы, которая связывает противолиганд на поверхности клетки-мишени.
Иммунные эффекторные клетки могут содержать рекомбинантную ДНК-конструкцию, содержащую последовательности, кодирующие CAR, где CAR содержит антигенсвязывающий домен (например, антитело или фрагмент антитела, TCR или фрагмент TCR), который специфично связывается с опухолевым антигеном, например, опухолевым антигеном, описанным в данном документе, и внутриклеточный сигнальный домен. Внутриклеточный сигнальный домен может содержать костимулирующий сигнальный домен и/или первичный сигнальный домен, например, дзета-цепь. Как описано в другом месте, способы, описанные в данном документе, могут включать трансдукцию клетки, например, из популяции, истощенной по регуляторным T-клеткам, с помощью нуклеиновой кислоты, кодирующей CAR, например, CAR, описанный в данном документе.
В некоторых вариантах осуществления CAR содержит scFv-домен, где перед scFv может располагаться необязательная лидерная последовательность, такая как представленная под SEQ ID NO: 1, и за ней может располагаться необязательная последовательность шарнирной области, такая как представленная под SEQ ID NO: 2, или SEQ ID NO: 36, или SEQ ID NO: 38, трансмембранная область, такая как представленная под SEQ ID NO: 6, внутриклеточный сигнальный домен, который содержит SEQ ID NO: 7 или SEQ ID NO: 16, и последовательность CD3-дзета, которая содержит SEQ ID NO: 9 или SEQ ID NO: 10, например, где домены являются смежными и находятся в одной и той же рамке считывания, образуя единый слитый белок.
В некоторых вариантах осуществления иллюстративные конструкции CAR содержат необязательную лидерную последовательность (например, лидерную последовательность, описанную в данном документе), внеклеточный антигенсвязывающий домен (например, антигенсвязывающий домен, описанный в данном документе), шарнирную область (например, шарнирную область, описанную в данном документе), трансмембранный домен (например, трансмембранный домен, описанный в данном документе) и внутриклеточный стимулирующий домен (например, внутриклеточный стимулирующий домен, описанный в данном документе). В некоторых вариантах осуществления иллюстративная конструкция CAR содержит необязательную лидерную последовательность (например, лидерную последовательность, описанную в данном документе), внеклеточный антигенсвязывающий домен (например, антигенсвязывающий домен, описанный в данном документе), шарнирную область (например, шарнирную область, описанную в данном документе), трансмембранный домен (например, трансмембранный домен, описанный в данном документе), внутриклеточный костимулирующий сигнальный домен (например, костимулирующий сигнальный домен, описанный в данном документе) и/или внутриклеточный первичный сигнальный домен (например, первичный сигнальный домен, описанный в данном документе).
Иллюстративная лидерная последовательность представлена под SEQ ID NO: 1. Следующие иллюстративные лидерные последовательности включают представленные под SEQ ID NO: 351 или кодируются представленными под SEQ ID NO: 352 или 353. Иллюстративная последовательность шарнирной области/спейсера представлена под SEQ ID NO: 2, или SEQ ID NO: 36, или SEQ ID NO: 38. Иллюстративная последовательность трансмембранного домена представлена под SEQ ID NO: 6. Иллюстративная последовательность внутриклеточного сигнального домена белка 4-1BB представлена под SEQ ID NO: 7. Иллюстративная последовательность внутриклеточного сигнального домена CD27 представлена под SEQ ID NO:16. Иллюстративная последовательность домена CD3-дзета представлена под SEQ ID NO: 9 или SEQ ID NO:10.
В некоторых вариантах осуществления иммунная эффектoрная клетка содержит рекомбинантную конструкцию нуклеиновой кислоты, содержащую молекулу нуклеиновой кислоты, кодирующую CAR, где молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты, кодирующую антигенсвязывающий домен, где последовательность является смежной с последовательностью нуклеиновой кислоты, кодирующей внутриклеточный сигнальный домен, и находится с ней в одной и той же рамке считывания. Иллюстративный внутриклеточный сигнальный домен, который может использоваться в CAR, включает без ограничения один или несколько внутриклеточных сигнальных доменов, например, CD3-дзета, CD28, CD27, 4-1BB и т. п. В некоторых случаях CAR может содержать любую комбинацию из CD3-дзета, CD28, 4-1BB и т. п.
Последовательности нуклеиновой кислоты, кодирующие требуемые молекулы, можно получить с помощью рекомбинантных способов, известных из уровня техники, как, например, путем скрининга библиотек из клеток, экспрессирующих молекулу нуклеиновой кислоты, путем получения молекулы нуклеиновой кислоты из вектора, о котором известно, что он ее содержит, или путем непосредственного выделения из клеток и тканей, содержащих ее, с помощью стандартных методик. В качестве альтернативы нуклеиновая кислота, представляющая интерес, может быть получена путем синтеза, а не клонирования.
Нуклеиновые кислоты, кодирующие CAR, можно вводить в иммунные эффекторные клетки с помощью, например, ретровирусных или лентивирусных векторных конструкций.
Нуклеиновые кислоты, кодирующие CAR, также можно вводить в иммунную эффекторную клетку с помощью, например, РНК-конструкции, которая может быть непосредственно введена в клетку путем трансфекции. Способ получения мРНК для применения в трансфекции включает обеспечение транскрипции in vitro (IVT) матрицы с использованием специально разработанных праймеров с последующим добавлением поли(А) с получением конструкции, содержащей последовательности 3'- и 5'-нетранслируемых областей ("UTR") (например, 3'- и/или 5'-UTR, описанных в данном документе), 5'-кэп (например, 5'-кэп, описанный в данном документе) и/или участок внутренней посадки рибосомы (IRES) (например, IRES, описанный в данном документе), нуклеиновую кислоту, которая должна экспрессироваться, и поли(A)-хвост, как правило, длиной 50-2000 оснований (например, описанный в примерах, например, под SEQ ID NO: 35). С помощью РНК, полученной таким образом, можно эффективно трансфицировать различные типы клеток. В некоторых вариантах осуществления матрица содержит последовательности для CAR. В некоторых вариантах осуществления вектор на основе РНК, кодирующей CAR, вводят путем трансдукции в клетку, например, T-клетку, с помощью электропорации.
Антигенсвязывающий домен
В некоторых вариантах осуществления множество иммунных эффекторных клеток, например, популяция, истощенная по регуляторным T-клеткам, содержит нуклеиновую кислоту, кодирующую CAR, которая содержит специфичный в отношении мишени связывающий элемент, иначе называемый антигенсвязывающим доменом. Выбор связывающего элемента зависит от типа и количества лигандов, которые определяют поверхность клетки-мишени. Например, антигенсвязывающий домен может быть выбран таким образом, чтобы он распознавал лиганд, который выступает в качестве маркера клеточной поверхности на клетках-мишенях, ассоциированных с конкретным болезненным состоянием. Таким образом, примеры маркеров клеточной поверхности, которые могут выступать в качестве лигандов для антигенсвязывающего домена в CAR, описанного в данном документе, включают маркеры, ассоциированные с вирусными, бактериальными и паразитарными инфекциями, аутоиммунным заболеванием и раковыми клетками.
В некоторых вариантах осуществления часть CAR, содержащая антигенсвязывающий домен, содержит антигенсвязывающий домен, который нацеливается на опухолевый антиген, например, на опухолевый антиген, описанный в данном документе.
Антигенсвязывающий домен может представлять собой любой домен, который связывается с антигеном, в том числе без ограничения моноклональное антитело, поликлональное антитело, рекомбинантное антитело, антитело человека, гуманизированное антитело и их функциональный фрагмент, в том числе без ограничения однодоменное антитело, такое как вариабельный домен тяжелой цепи (VH), вариабельный домен легкой цепи (VL) и вариабельный домен (VHH) нанотела верблюдовых, а также альтернативную каркасную структуру, которая, как известно из уровня техники, функционирует в качестве антигенсвязывающего домена, такую как рекомбинантный фибронектиновый домен, T-клеточный рецептор (TCR) или его фрагмент, например, одноцепочечный TCR, и т. п. В некоторых случаях целесообразно, чтобы антигенсвязывающий домен был получен из того же вида, в организме которого в конечном итоге будет применяться CAR. Например, для применения у людей может быть целесообразно, чтобы антигенсвязывающий домен CAR содержал человеческие или гуманизированные остатки в антигенсвязывающем домене антитела или фрагмента антитела.
CAR для CD19
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для CD19 (например, клетку, экспрессирующую CAR, который связывается с CD19 человека).
В некоторых вариантах осуществления антигенсвязывающий домен CAR для CD19 характеризуется такой же или сходной специфичностью связывания по сравнению с scFv-фрагментом FMC63, описанным в Nicholson et al. Mol. Immun. 34 (16-17): 1157-1165 (1997). В некоторых вариантах осуществления антигенсвязывающий домен CAR для CD19 включает в себя scFv-фрагмент, описанный в Nicholson et al. Mol. Immun. 34 (16-17): 1157-1165 (1997).
В некоторых вариантах осуществления CAR для CD19 содержит антигенсвязывающий домен (например, гуманизированный антигенсвязывающий домен) согласно таблице 3 из WO2014/153270, включенной в данный документ посредством ссылки. В WO2014/153270 также описаны способы анализа связывания и эффективности различных конструкций CAR.
В некоторых вариантах осуществления исходная последовательность scFv мыши представляет собой конструкцию CAR19, представленную в публикации согласно PCT WO2012/079000 (включенной в данный документ посредством ссылки). В некоторых вариантах осуществления домен, связывающий CD19, представляет собой scFv, описанный в WO2012/079000.
В некоторых вариантах осуществления молекула CAR содержит последовательность слитого полипептида, представленную под SEQ ID NO: 12 в публикации согласно PCT WO2012/079000, в которой представлен scFv-фрагмент мышиного происхождения, который специфично связывается с CD19 человека.
В некоторых вариантах осуществления CAR для CD19 содержит аминокислотную последовательность, представленную под SEQ ID NO: 12 в публикации согласно PCT WO2012/079000.
В некоторых вариантах осуществления аминокислотная последовательность представляет собой
Diqmtqttsslsaslgdrvtiscrasqdiskylnwyqqkpdgtvklliyhtsrlhsgvpsrfsgsgsgtdysltisnleqediatyfcqqgntlpytfgggtkleitggggsggggsggggsevklqesgpglvapsqslsvtctvsgvslpdygvswirqpprkglewlgviwgsettyynsalksrltiikdnsksqvflkmnslqtddtaiyycakhyyyggsyamdywgqgtsvtvsstttpaprpptpaptiasqplslrpeacrpaaggavhtrgldfacdiyiwaplagtcgvlllslvitlyckrgrkkllyifkqpfmrpvqttqeedgcscrfpeeeeggcelrvkfsrsadapaykqgqnqlynelnlgrreeydvldkrrgrdpemggkprrknpqeglynelqkdkmaeayseigmkgerrrgkghdglyqglstatkdtydalhmqalppr (SEQ ID NO: 292), или последовательность, по существу гомологичную ей.
В некоторых вариантах осуществления CAR для CD19 имеет обозначение согласно USAN тисагенлеклейсел-T. В ряде вариантов осуществления CTL019 получают посредством генной модификации T-клеток, опосредованной стабильной вставкой путем трансдукции с помощью самоинактивирующегося лентивирусного (LV) вектора, дефектного по репликации, содержащего трансген CTL019 под контролем промотора гена EF-1-альфа. CTL019 может представлять собой смесь положительных и отрицательных по трансгену Т-клеток, которые доставляются субъекту, исходя из процентной доли Т-клеток, положительных по трансгену.
В других вариантах осуществления CAR для CD19 содержит антигенсвязывающий домен (например, гуманизированный антигенсвязывающий домен) согласно таблице 3 из WO2014/153270, включенной в данный документ посредством ссылки.
Гуманизация антитела к CD19 мыши необходима для клинических условий, когда специфические для мышей остатки могут индуцировать ответ с образованием антител человека к антигенам мыши (HAMA) у пациентов, которые получают лечение с помощью CART19, т. е. лечение с помощью T-клеток, трансдуцированных конструкцией CAR19. Получение, определение характеристик и эффективность гуманизированных последовательностей CAR для CD19 описаны в международной заявке WO2014/153270, включенной в данный документ посредством ссылки во всей своей полноте, включая примеры 1-5 (стр. 115-159).
В некоторых вариантах осуществления молекула CAR представляет собой гуманизированный CAR для CD19, содержащий следующую аминокислотную последовательность: eivmtqspatlslspgeratlscrasqdiskylnwyqqkpgqaprlliyhtsrlhsgiparfsgsgsgtdytltisslqpedfavyfcqqgntlpytfgqgtkleikggggsggggsggggsqvqlqesgpglvkpsetlsltctvsgvslpdygvswirqppgkglewigviwgsettyyqsslksrvtiskdnsknqvslklssvtaadtavyycakhyyyggsyamdywgqgtlvtvss (SEQ ID NO: 293)
В некоторых вариантах осуществления молекула CAR представляет собой гуманизированный CAR для CD19, содержащий следующую аминокислотную последовательность:
eivmtqspatlslspgeratlscrasqdiskylnwyqqkpgqaprlliyhtsrlhsgiparfsgsgsgtdytltisslqpedfavyfcqqgntlpytfgqgtkleikggggsggggsggggsqvqlqesgpglvkpsetlsltctvsgvslpdygvswirqppgkglewigviwgsettyyqsslksrvtiskdnsknqvslklssvtaadtavyycakhyyyggsyamdywgqgtlvtvsstttpaprpptpaptiasqplslrpeacrpaaggavhtrgldfacdiyiwaplagtcgvlllslvitlyckrgrkkllyifkqpfmrpvqttqeedgcscrfpeeeeggcelrvkfsrsadapaykqgqnqlynelnlgrreeydvldkrrgrdpemggkprrknpqeglynelqkdkmaeayseigmkgerrrgkghdglyqglstatkdtydalhmqalppr (sEQ ID NO: 294)
В некоторых вариантах осуществления молекула CAR представляет собой гуманизированный CAR для CD19, содержащий следующую аминокислотную последовательность:
MALPVTALLLPLALLLHAARPEIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 349)
В некоторых вариантах осуществления молекула CAR представляет собой гуманизированный CAR для CD19, содержащий следующую аминокислотную последовательность:
EIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 350)
В соответствии с настоящим изобретением можно применять любой известный CAR для CD19, например, антигенсвязывающий домен для CD19 из любого известного CAR для CD19. Например, CAR для CD19 LG-740 описан в патенте США № 8399645; патенте США № 7446190; Xu et al., Leuk Lymphoma. 2013 54(2):255-260(2012); Cruz et al., Blood 122(17):2965-2973 (2013); Brentjens et al., Blood, 118(18):4817-4828 (2011); Kochenderfer et al., Blood 116(20):4099-102 (2010); Kochenderfer et al., Blood 122 (25):4129-39 (2013) и 16th Annu Meet Am Soc Gen Cell Ther (ASGCT) (May 15-18, Salt Lake City) 2013, Abst 10.
Иллюстративные CAR для CD19 включают CAR для CD19, описанный в данном документе, или CAR для CD19, описанный в Xu et al. Blood 123.24(2014):3750-9; Kochenderfer et al. Blood 122.25(2013):4129-39, Cruz et al. Blood 122.17(2013):2965-73, NCT00586391, NCT01087294, NCT02456350, NCT00840853, NCT02659943, NCT02650999, NCT02640209, NCT01747486, NCT02546739, NCT02656147, NCT02772198, NCT00709033, NCT02081937, NCT00924326, NCT02735083, NCT02794246, NCT02746952, NCT01593696, NCT02134262, NCT01853631, NCT02443831, NCT02277522, NCT02348216, NCT02614066, NCT02030834, NCT02624258, NCT02625480, NCT02030847, NCT02644655, NCT02349698, NCT02813837, NCT02050347, NCT01683279, NCT02529813, NCT02537977, NCT02799550, NCT02672501, NCT02819583, NCT02028455, NCT01840566, NCT01318317, NCT01864889, NCT02706405, NCT01475058, NCT01430390, NCT02146924, NCT02051257, NCT02431988, NCT01815749, NCT02153580, NCT01865617, NCT02208362, NCT02685670, NCT02535364, NCT02631044, NCT02728882, NCT02735291, NCT01860937, NCT02822326, NCT02737085, NCT02465983, NCT02132624, NCT02782351, NCT01493453, NCT02652910, NCT02247609, NCT01029366, NCT01626495, NCT02721407, NCT01044069, NCT00422383, NCT01680991, NCT02794961 или NCT02456207, каждый из которых включен в данный документ посредством ссылки во всей своей полноте.
В некоторых вариантах осуществления CAR для CD19 содержат последовательность, например, CDR, VH, VL, scFv или полную последовательность CAR, раскрытую в таблице 2, или последовательность, характеризующуюся по меньшей мере 80%, 85%, 90%, 95% или 99% идентичностью с ней.
полная аминокислотная последовательность; сигнальный пептид подчеркнут
аминокислотная последовательность; сигнальный пептид отсутствует
CAR для BCMA
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для BCMA (например, клетку, экспрессирующую CAR, который связывается с BCMA человека). Иллюстративные CAR для BCMA могут включать последовательности, раскрытые в таблице 1 или 16 из WO2016/014565, включенной в данный документ посредством ссылки. Конструкция CAR для BCMA может содержать необязательную лидерную последовательность; необязательный шарнирный домен, например, шарнирный домен CD8; трансмембранный домен, например, трансмембранный домен CD8; внутриклеточный домен, например, внутриклеточный домен 4-1BB; и функциональный сигнальный домен, например, домен CD3-дзета. В определенных вариантах осуществления домены являются смежными и находятся в одной и той же рамке считывания, образуя единый слитый белок. В других вариантах осуществления домены находятся в отдельных полипептидах, как, например, в молекуле RCAR, описанной в данном документе.
В некоторых вариантах осуществления молекула CAR для BCMA содержит одну или несколько CDR, VH, VL, scFv или полноразмерных последовательностей BCMA-1, BCMA-2, BCMA-3, BCMA-4, BCMA-5, BCMA-6, BCMA-7, BCMA-8, BCMA-9, BCMA-10, BCMA-11, BCMA-12, BCMA-13, BCMA-14, BCMA-15, 149362, 149363, 149364, 149365, 149366, 149367, 149368, 149369, BCMA_EBB-C1978-A4, BCMA_EBB-C1978-G1, BCMA_EBB-C1979-C1, BCMA_EBB-C1978-C7, BCMA_EBB-C1978-D10, BCMA_EBB-C1979-C12, BCMA_EBB-C1980-G4, BCMA_EBB-C1980-D2, BCMA_EBB-C1978-A10, BCMA_EBB-C1978-D4, BCMA_EBB-C1980-A2, BCMA_EBB-C1981-C3, BCMA_EBB-C1978-G4, A7D12.2, C11D5.3, C12A3.2 или C13F12.1, раскрытых в WO2016/014565, или последовательность, по существу (например, на 95-99%) идентичную им.
Дополнительные иллюстративные последовательности, нацеливающиеся на BCMA, которые можно использовать в конструкциях CAR для BCMA, раскрыты в WO 2017/021450, WO 2017/011804, WO 2017/025038, WO 2016/090327, WO 2016/130598, WO 2016/210293, WO 2016/090320, WO 2016/014789, WO 2016/094304, WO 2016/154055, WO 2015/166073, WO 2015/188119, WO 2015/158671, US 9243058, US 8920776, US 9273141, US 7083785, US 9034324, US 2007/0049735, US 2015/0284467, US 2015/0051266, US 2015/0344844, US 2016/0131655, US 2016/0297884, US 2016/0297885, US 2017/0051308, US 2017/0051252, US 2017/0051252, WO 2016/020332, WO 2016/087531, WO 2016/079177, WO 2015/172800, WO 2017/008169, US 9340621, US 2013/0273055, US 2016/0176973, US 2015/0368351, US 2017/0051068, US 2016/0368988 и US 2015/0232557, включенных в данный документ посредством ссылки во всей своей полноте. В некоторых вариантах осуществления дополнительные иллюстративные конструкции CAR для BCMA получают с использованием последовательностей VH и VL из публикации согласно PCT WO2012/0163805 (содержание которой включено в данный документ посредством ссылки во всей своей полноте).
В некоторых вариантах осуществления CAR для BCMA содержат последовательность, например, CDR, VH, VL, scFv или полную последовательность CAR, раскрытую в таблицах 3-15, или последовательность, характеризующуюся по меньшей мере 80%, 85%, 90%, 95% или 99% идентичностью с ней. В одном аспекте антигенсвязывающий домен содержит антитело человека или фрагмент антитела человека. В некоторых вариантах осуществления человеческий домен, связывающий BCMA, содержит одну или несколько (например, все три) из CDR1 LC, CDR2 LC и CDR3 LC человеческого домена, связывающего BCMA, описанного в данном документе (например, в таблицах 3-15), и/или одну или несколько (например, все три) из CDR1 HC, CDR2 HC и CDR3 HC человеческого домена, связывающего BCMA, описанного в данном документе (например, в таблицах 3-15). В некоторых вариантах осуществления человеческий домен, связывающий BCMA, содержит VL человека, описанную в данном документе (например, в таблицах 3, 7, 11, 11a и 12), и/или VH человека, описанную в данном документе (например, в таблицах 3, 7, 11, 11a и 12). В некоторых вариантах осуществления домен, связывающий BCMA, представляет собой scFv, содержащий VL и VH с аминокислотной последовательностью из таблиц 3, 7, 11, 11a и 12. В некоторых вариантах осуществления домен, связывающий BCMA (например, scFv), содержит: VL, содержащую аминокислотную последовательность, имеющую по меньшей мере одну, две или три модификации (например, замены, например, консервативные замены), но не более 30, 20 или 10 модификаций (например, замен, например, консервативных замен) аминокислотной последовательности, представленной в таблицах 3, 7, 11, 11a и 12, или последовательность с 95-99% идентичностью с аминокислотной последовательностью из таблиц 3, 7, 11, 11a и 12; и/или VH, содержащую аминокислотную последовательность, имеющую по меньшей мере одну, две или три модификации (например, замены, например, консервативные замены), но не более 30, 20 или 10 модификаций (например, замен, например, консервативных замен) аминокислотной последовательности, представленной в таблицах 3, 7, 11, 11a и 12, или последовательность с 95-99% идентичностью с аминокислотной последовательностью из таблиц 3, 7, 11, 11a и 12.
описание
описание
описание
В некоторых вариантах осуществления человеческий домен, связывающий BCMA, содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC.
В определенных вариантах осуществления молекула CAR, описанная в данном документе, или домен, связывающий BCMA, описанный в данном документе, содержат:
(1) одну, две или три CDR легкой цепи (LC), выбранные из
(i) CDR1 LC под SEQ ID NO: 54, CDR2 LC под SEQ ID NO: 55 и CDR3 LC под SEQ ID NO: 56; и/или
(2) одну, две или три CDR тяжелой цепи (HC) из любой из следующих:
(i) CDR1 HC под SEQ ID NO: 44, CDR2 HC под SEQ ID NO: 45 и CDR3 HC под SEQ ID NO: 84; (ii) CDR1 HC под SEQ ID NO: 44, CDR2 HC под SEQ ID NO: 45 и CDR3 HC под SEQ ID NO: 46; (iii) CDR1 HC под SEQ ID NO: 44, CDR2 HC под SEQ ID NO: 45 и CDR3 HC под SEQ ID NO: 68 или (iv) CDR1 HC под SEQ ID NO: 44, CDR2 HC под SEQ ID NO: 45 и CDR3 HC под SEQ ID NO: 76.
В определенных вариантах осуществления молекула CAR, описанная в данном документе, или домен, связывающий BCMA, описанный в данном документе, содержат:
(1) одну, две или три CDR легкой цепи (LC) из любой из следующих:
(i) CDR1 LC под SEQ ID NO: 95, CDR2 LC под SEQ ID NO: 131 и CDR3 LC под SEQ ID NO: 132; (ii) CDR1 LC под SEQ ID NO: 95, CDR2 LC под SEQ ID NO: 96 и CDR3 LC под SEQ ID NO: 97; (iii) CDR1 LC под SEQ ID NO: 95, CDR2 LC под SEQ ID NO: 114 и CDR3 LC под SEQ ID NO: 115 или (iv) CDR1 LC под SEQ ID NO: 95, CDR2 LC под SEQ ID NO: 114 и CDR3 LC под SEQ ID NO: 97; и/или
(2) одну, две или три CDR тяжелой цепи (HC) из любой из следующих:
(i) CDR1 HC под SEQ ID NO: 86, CDR2 HC под SEQ ID NO: 130 и CDR3 HC под SEQ ID NO: 88; (ii) CDR1 HC под SEQ ID NO: 86, CDR2 HC под SEQ ID NO: 87 и CDR3 HC под SEQ ID NO: 88 или (iii) CDR1 HC под SEQ ID NO: 86, CDR2 HC под SEQ ID NO: 109 и CDR3 HC под SEQ ID NO: 88.
В определенных вариантах осуществления молекула CAR, описанная в данном документе, или домен, связывающий BCMA, описанный в данном документе, содержат:
(1) одну, две или три CDR легкой цепи (LC) из любой из следующих:
(i) CDR1 LC под SEQ ID NO: 147, CDR2 LC под SEQ ID NO: 182 и CDR3 LC под SEQ ID NO: 183; (ii) CDR1 LC под SEQ ID NO: 147, CDR2 LC под SEQ ID NO: 148 и CDR3 LC под SEQ ID NO: 149 или (iii) CDR1 LC под SEQ ID NO: 147, CDR2 LC под SEQ ID NO: 170 и CDR3 LC под SEQ ID NO: 171; и/или
(2) одну, две или три CDR тяжелой цепи (HC) из любой из следующих:
(i) CDR1 HC под SEQ ID NO: 179, CDR2 HC под SEQ ID NO: 180 и CDR3 HC под SEQ ID NO: 181; (ii) CDR1 HC под SEQ ID NO: 137, CDR2 HC под SEQ ID NO: 138 и CDR3 HC под SEQ ID NO: 139 или (iii) CDR1 HC под SEQ ID NO: 160, CDR2 HC под SEQ ID NO: 161 и CDR3 HC под SEQ ID NO: 162.
В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 84, 54, 55 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 46, 54, 55 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 68, 54, 55 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно.
В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 47, 48, 84, 57, 58 и 59 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 47, 48, 46, 57, 58 и 59 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 47, 48, 68, 57, 58 и 59 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 47, 48, 76, 57, 58 и 59 соответственно.
В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 49, 50, 85, 60, 58 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 49, 50, 51, 60, 58 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 49, 50, 69, 60, 58 и 56 соответственно. В некоторых вариантах осуществления CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC содержат аминокислотные последовательности под SEQ ID NO: 49, 50, 77, 60, 58 и 56 соответственно.
В некоторых вариантах осуществления человеческий домен, связывающий BCMA, содержит scFv, содержащий VH (например, VH, описанную в данном документе) и VL (например, VL, описанную в данном документе). В некоторых вариантах осуществления VH присоединена к VL посредством линкера, например, линкера, описанного в данном документе, например, линкера, описанного в таблице 1. В некоторых вариантах осуществления человеческий домен, связывающий BCMA, содержит линкер (Gly4-Ser)n, где n равняется 1, 2, 3, 4, 5 или 6, предпочтительно 3 или 4 (SEQ ID NO: 26). Вариабельная область легкой цепи и вариабельная область тяжелой цепи scFv могут быть представлены, например, в любой из следующих ориентаций: вариабельная область легкой цепи-линкер-вариабельная область тяжелой цепи или вариабельная область тяжелой цепи-линкер-вариабельная область легкой цепи.
В некоторых вариантах осуществления домен, связывающий BCMA, представляет собой фрагмент, например, одноцепочечный вариабельный фрагмент (scFv). В некоторых вариантах осуществления домен, связывающий BCMA, представляет собой Fv, Fab (Fab')2 или бифункциональное (например, биспецифическое) гибридное антитело (например, Lanzavecchia et al., Eur. J. Immunol. 17, 105 (1987)). В некоторых вариантах осуществления антитела и их фрагменты по настоящему изобретению связываются c белком BCMA с аффинностью дикого типа или усиленной аффинностью.
В некоторых случаях scFv можно получить в соответствии со способом, известным из уровня техники (см., например, Bird et al., (1988) Science 242:423-426 и Huston et al., (1988) Proc. Natl. Acad. Sci. USA 85:5879-5883). Молекулы scFv можно получить путем связывания VH- и VL-областей друг с другом с помощью гибких полипептидных линкеров. Молекулы scFv содержат линкер (например, линкер Ser-Gly) с оптимизированными длиной и/или аминокислотным составом. Длина линкера может сильно влиять на укладку и взаимодействие вариабельных областей scFv. В действительности, если используют короткий полипептидный линкер (например, длиной 5-10 аминокислот), то внутрицепочечное сворачивание предотвращается. Межцепочечное сворачивание также необходимо для объединения двух вариабельных областей вместе с образованием функционального эпитопсвязывающего участка. Примеры ориентации и размера линкеров см., например, в Hollinger et al. 1993 Proc Natl Acad. Sci. U.S.A. 90:6444-6448, публикациях заявок на патент США №№ 2005/0100543, 2005/0175606, 2007/0014794 и публикациях согласно PCT №№ WO2006/020258 и WO2007/024715, включенных в данный документ посредством ссылки.
ScFv может содержать линкер из по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, или больше аминокислотных остатков между его VL- и VH-областями. Линкерная последовательность может содержать любую встречающуюся в природе аминокислоту. В некоторых вариантах осуществления линкерная последовательность содержит аминокислоты глицин и серин. В некоторых вариантах осуществления линкерная последовательность содержит группы глициновых и сериновых повторов, таких как (Gly4Ser)n, где n представляет собой положительное целое число, которое равняется или больше 1 (SEQ ID NO: 25). В некоторых вариантах осуществления линкер может представлять собой (Gly4Ser)4 (SEQ ID NO: 27) или (Gly4Ser)3(SEQ ID NO: 28). При изменении длины линкера может сохраняться или усиливаться активность, что приводит к улучшению эффективности в исследованиях активности.
CAR для CD20
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для CD20 (например, клетку, экспрессирующую CAR, который связывается с CD20 человека). В некоторых вариантах осуществления клетка, экспрессирующая CAR для CD20, содержит антигенсвязывающий домен согласно WO2016164731 и WO2018067992, включенным в данный документ посредством ссылки. Иллюстративные последовательности, связывающие CD20, или последовательности CAR для CD20 раскрыты, например, в таблицах 1-5 из WO2018067992. В некоторых вариантах осуществления CAR для CD20 содержит CDR, вариабельную область, scFv или полноразмерную последовательность CAR для CD20, раскрытую в WO2018067992 или WO2016164731.
CAR для CD22
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для CD22 (например, клетку, экспрессирующую CAR, который связывается с CD22 человека). В некоторых вариантах осуществления клетка, экспрессирующая CAR для CD22, содержит антигенсвязывающий домен согласно WO2016164731 и WO2018067992, включенным в данный документ посредством ссылки. Иллюстративные последовательности, связывающие CD22, или последовательности CAR для CD22 раскрыты в, например, таблицах 6A, 6B, 7A, 7B, 7C, 8A, 8B, 9A, 9B, 10A и 10B согласно WO2016164731 и таблицах 6-10 согласно WO2018067992. В некоторых вариантах осуществления последовательность CAR для CD22 содержит CDR, вариабельную область, scFv или полноразмерную последовательность CAR для CD22, раскрытую в WO2018067992 или WO2016164731.
В ряде вариантов осуществления молекула CAR содержит антигенсвязывающий домен, который связывается с CD22 (CAR для CD22). В некоторых вариантах осуществления антигенсвязывающий домен нацеливается на CD22 человека. В некоторых вариантах осуществления антигенсвязывающий домен содержит последовательность одноцепочечного Fv, описанного в данном документе.
Последовательности CAR для CD22 человека представлены ниже. В некоторых вариантах осуществления CAR для CD22 человека представляет собой CAR22-65.
Последовательность scFv CAR для CD22 человека
EVQLQQSGPGLVKPSQTLSLTCAISGDSMLSNSDTWNWIRQSPSRGLEWLGRTYHRSTWYDDYASSVRGRVSINVDTSKNQYSLQLNAVTPEDTGVYYCARVRLQDGNSWSDAFDVWGQGTMVTVSSGGGGSGGGGSGGGGSQSALTQPASASGSPGQSVTISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGVSNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSSSTLYVFGTGTQLTVL (SEQ ID NO: 285)
Вариабельная область тяжелой цепи CAR для CD22 человека
EVQLQQSGPGLVKPSQTLSLTCAISGDSMLSNSDTWNWIRQSPSRGLEWLGRTYHRSTWYDDYASSVRGRVSINVDTSKNQYSLQLNAVTPEDTGVYYCARVRLQDGNSWSDAFDVWGQGTMVTVSS (SEQ ID NO 286)
Вариабельная область легкой цепи CAR для CD22 человека
QSALTQPASASGSPGQSVTISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGVSNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSSSTLYVFGTGTQLTVL (SEQ ID NO 287)
В некоторых вариантах осуществления антигенсвязывающий домен содержит CDR1 HC, CDR2 HC и CDR3 HC с любыми аминокислотными последовательностями тяжелой цепи связывающего домена, перечисленными в таблице 16. В ряде вариантов осуществления антигенсвязывающий домен дополнительно содержит CDR1 LC, CDR2 LC и CDR3 LC. В ряде вариантов осуществления антигенсвязывающий домен содержит аминокислотные последовательности CDR1 LC, CDR2 LC и CDR3 LC, перечисленные в таблице 17.
В некоторых вариантах осуществления антигенсвязывающий домен содержит одну, две или все из CDR1 LC, CDR2 LC и CDR3 LC с любыми аминокислотными последовательностями легкой цепи связывающего домена, перечисленными в таблице 17, и одну, две или все из CDR1 HC, CDR2 HC и CDR3 HC с любыми аминокислотными последовательностями тяжелой цепи связывающего домена, перечисленными в таблице 16.
В некоторых вариантах осуществления CDR определены в соответствии со схемой нумерации по Kabat, схемой нумерации по Chothia или их комбинацией.
Порядок, в котором VL- и VH-домены представлены в scFv, может варьироваться (т. е. в ориентации VL-VH или VH-VL), и при этом любое количество из одной, двух, трех или четырех копий субъединиц "G4S" (SEQ ID NO: 25), где каждая субъединица содержит последовательность GGGGS (SEQ ID NO: 25) (например, (G4S)3 (SEQ ID NO: 28) или (G4S)4 (SEQ ID NO: 27)), может соединять вариабельные домены для создания целого scFv-домена. В качестве альтернативы конструкция CAR может содержать, например, линкер, содержащий последовательность GSTSGSGKPGSGEGSTKG (SEQ ID NO: 43). В качестве альтернативы конструкция CAR может содержать, например, линкер содержащий последовательность LAEAAAK (SEQ ID NO: 308). В одном варианте осуществления конструкция CAR не содержит линкер между VL- и VH-доменами.
Все эти клоны содержали изменение остатка Q/K в сигнальном домене костимулирующего домена, полученного из дзета-цепи CD3.
CAR для EGFR
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для EGFR (например, клетку, экспрессирующую CAR, который связывается с EGFR человека). В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для EGFRvIII (например, клетку, экспрессирующую CAR, который связывается с EGFRvIII человека). Иллюстративные CAR для EGFRvIII могут содержать последовательности, раскрытые в WO2014/130657, например, в таблице 2 из WO2014/130657, включенной в данный документ посредством ссылки.
Иллюстративные последовательности, связывающие EGFRvIII, или последовательности CAR для EGFR могут содержать CDR, вариабельную область, scFv или полноразмерную последовательность CAR из CAR для EGFR, раскрытого в WO2014/130657.
CAR для мезотелина
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, представляет собой клетку, экспрессирующую CAR для мезотелина (например, клетку, экспрессирующую CAR, который связывается с мезотелином человека). Иллюстративные CAR для мезотелина могут содержать последовательности, раскрытые в WO2015090230 и WO2017112741, например, в таблицах 2, 3, 4 и 5 из WO2017112741, включенных в данный документ посредством ссылки.
Другие иллюстративные CAR
В других вариантах осуществления клетки, экспрессирующие CAR, могут специфично связываться с CD123, например, могут содержать молекулу CAR (например, любую из CAR1-CAR8) или антигенсвязывающий домен согласно таблицам 1-2 из WO 2014/130635, включенной в данный документ посредством ссылки. Аминокислотные последовательности молекул и антигенсвязывающих доменов CAR для CD123 (например, содержащих одну, две, три CDR VH и одну, две, три CDR VL согласно Kabat или Chothia) и нуклеотидные последовательности, кодирующие их, указаны в WO 2014/130635. В других вариантах осуществления клетки, экспрессирующие CAR, могут специфично связываться с CD123, например, могут содержать молекулу CAR (например, любую из молекул от CAR123-1 до CAR123-4 и от hzCAR123-1 до hzCAR123-32) или ее антигенсвязывающий домен согласно таблицам 2, 6 и 9 из WO2016/028896, включенной в данный документ посредством ссылки. Аминокислотные последовательности молекул и антигенсвязывающих доменов CAR для CD123 (например, содержащих одну, две, три CDR VH и одну, две, три CDR VL согласно Kabat или Chothia) и нуклеотидные последовательности, кодирующие их, указаны в WO2016/028896.
В некоторых вариантах осуществления молекула CAR предусматривает CAR для CLL1, описанный в данном документе, например, CAR для CLL1, описанный в US2016/0051651A1, включенной в данный документ посредством ссылки. В ряде вариантов осуществления CAR для CLL1 содержит аминокислотную последовательность или имеет нуклеотидную последовательность, показанные в US2016/0051651A1, включенной в данный документ посредством ссылки. В других вариантах осуществления клетки, экспрессирующие CAR, могут специфично связываться с CLL-1, например, могут содержать молекулу CAR или антигенсвязывающий домен согласно таблице 2 из WO2016/014535, включенной в данный документ посредством ссылки. Аминокислотные последовательности молекул и антигенсвязывающих доменов CAR для CLL-1 (например, содержащих одну, две, три CDR VH и одну, две, три CDR VL согласно Kabat или Chothia) и нуклеотидные последовательности, кодирующие их, указаны в WO2016/014535.
В некоторых вариантах осуществления молекула CAR предусматривает CAR для CD33, описанный в данном документе, например, CAR для CD33, описанный в US2016/0096892A1, включенной в данный документ посредством ссылки. В ряде вариантов осуществления CAR для CD33 содержит аминокислотную последовательность или имеет нуклеотидную последовательность, показанные в US2016/0096892A1, включенной в данный документ посредством ссылки. В других вариантах осуществления клетки, экспрессирующие CAR, могут специфично связываться с CD33, например, могут содержать молекулу CAR (например, любую из молекул от CAR33-1 до CAR-33-9) или антигенсвязывающий домен согласно таблице 2 или 9 из WO2016/014576, включенной в данный документ посредством ссылки. Аминокислотные последовательности молекул и антигенсвязывающих доменов CAR для CD33 (например, содержащих одну, две, три CDR VH и одну, две, три CDR VL согласно Kabat или Chothia) и нуклеотидные последовательности, кодирующие их, указаны в WO2016/014576.
В некоторых вариантах осуществления антигенсвязывающий домен содержит одну, две, три (например, все три) CDR тяжелой цепи - CDR1 HC, CDR2 HC и CDR3 HC - из антитела, описанного в данном документе (например, антитела, описанного в WO2015/142675, US-2015-0283178-A1, US-2016-0046724-A1, US2014/0322212A1, US2016/0068601A1, US2016/0051651A1, US2016/0096892A1, US2014/0322275A1 или WO2015/090230, включенных в данный документ посредством ссылки), и/или одну, две, три (например, все три) CDR легкой цепи - CDR1 LC, CDR2 LC и CDR3 LC - из антитела, описанного в данном документе (например, антитела, описанного в WO2015/142675, US-2015-0283178-A1, US-2016-0046724-A1, US2014/0322212A1, US2016/0068601A1, US2016/0051651A1, US2016/0096892A1, US2014/0322275A1 или WO2015/090230, включенных в данный документ посредством ссылки). В некоторых вариантах осуществления антигенсвязывающий домен содержит вариабельную область тяжелой цепи и/или вариабельную область легкой цепи антитела, указанного выше.
В ряде вариантов осуществления антигенсвязывающий домен представляет собой антигенсвязывающий домен, описанный в WO2015/142675, US-2015-0283178-A1, US-2016-0046724-A1, US2014/0322212A1, US2016/0068601A1, US2016/0051651A1, US2016/0096892A1, US2014/0322275A1 или WO2015/090230, включенных в данный документ посредством ссылки.
В ряде вариантов осуществления антигенсвязывающий домен нацеливается на BCMA и описан в US-2016-0046724-A1. В ряде вариантов осуществления антигенсвязывающий домен нацеливается на CD19 и описан в US-2015-0283178-A1. В ряде вариантов осуществления антигенсвязывающий домен нацеливается на CD123 и описан в US2014/0322212A1, US2016/0068601A1. В ряде вариантов осуществления антигенсвязывающий домен нацеливается на CLL1 и описан в US2016/0051651A1. В ряде вариантов осуществления антигенсвязывающий домен нацеливается на CD33 и описан в US2016/0096892A1.
Иллюстративные антигены-мишени, на которые можно нацеливаться с помощью клеток, экспрессирующих CAR, включают без ограничения, среди прочих, CD19, CD123, EGFRvIII, CD33, мезотелин, BCMA и GFR-альфа 4, описанные, например, в WO2014/153270, WO 2014/130635, WO2016/028896, WO 2014/130657, WO2016/014576, WO 2015/090230, WO2016/014565, WO2016/014535 и WO2016/025880, каждая из которых включена в данный документ посредством ссылки во всей своей полноте.
В других вариантах осуществления клетки, экспрессирующие CAR, могут специфично связываться с GFR-альфа 4, например, могут содержать молекулу CAR или антигенсвязывающий домен согласно таблице 2 из WO2016/025880, включенной в данный документ посредством ссылки. Аминокислотные последовательности молекул и антигенсвязывающих доменов CAR для GFR-альфа 4 (например, содержащих одну, две, три CDR VH и одну, две, три CDR VL согласно Kabat или Chothia) и нуклеотидные последовательности, кодирующие их, указаны в WO2016/025880.
В некоторых вариантах осуществления антигенсвязывающий домен любой из молекул CAR, описанных в данном документе (например, для любого из CD19, CD123, EGFRvIII, CD33, мезотелина, BCMA и GFR-альфа 4), содержит одну, две, три (например, все три) CDR тяжелой цепи - CDR1 HC, CDR2 HC и CDR3 HC - из антитела, указанного выше, и/или одну, две, три (например, все три) CDR легкой цепи - CDR1 LC, CDR2 LC и CDR3 LC - из антигенсвязывающего домена, указанного выше. В некоторых вариантах осуществления антигенсвязывающий домен содержит вариабельную область тяжелой цепи и/или вариабельную область легкой цепи антитела, указанного или описанного выше.
В некоторых вариантах осуществления антигенсвязывающий домен содержит одну, две, три (например, все три) CDR тяжелой цепи - CDR1 HC, CDR2 HC и CDR3 HC - из антитела, указанного выше, и/или одну, две, три (например, все три) CDR легкой цепи - CDR1 LC, CDR2 LC и CDR3 LC - из антитела, указанного выше. В некоторых вариантах осуществления антигенсвязывающий домен содержит вариабельную область тяжелой цепи и/или вариабельную область легкой цепи антитела, указанного или описанного выше.
В некоторых вариантах осуществления опухолевый антиген представляет собой опухолевый антиген, описанный в международной заявке WO2015/142675, поданной 13 марта 2015 года, включенной в данный документ посредством ссылки во всей своей полноте. В некоторых вариантах осуществления опухолевый антиген выбран из одного или нескольких из CD19; CD123; CD22; CD30; CD171; CS-1 (также называемого CD2 субпопуляции 1, CRACC, SLAMF7, CD319 и 19A24); молекулы 1, подобной лектинам С-типа (CLL-1 или CLECL1); CD33; варианта III рецептора эпидермального фактора роста (EGFRvIII); ганглиозида G2 (GD2); ганглиозида GD3 (aNeu5Ac(2-8)aNeu5Ac(2-3)bDGalp(1-4)bDGlcp(1-1)Cer); антигена созревания В-клеток (BCMA), являющегося представителем семейства рецепторов TNF; Tn-антигена ((Ag Tn) или (GalNAcα-Ser/Thr)); простатического специфического мембранного антигена (PSMA); орфанного рецептора 1, подобного рецепторной тирозинкиназе (ROR1); Fms-подобной тирозинкиназы 3 (FLT3); опухолеассоциированного гликопротеина 72 (TAG72); CD38; CD44v6; раково-эмбрионального антигена (СЕА); молекулы адгезии эпителиальных клеток (ЕРСАМ); В7Н3 (CD276); KIT (CD117); альфа-2-субъединицы рецептора интерлейкина-13 (IL-13Ra2 или CD213A2); мезотелина; альфа-субъединицы рецептора интерлейкина 11 (IL-11Ra); антигена стволовых клеток предстательной железы (PSCA); сериновой протеазы 21 (тестизина или PRSS21); рецептора 2 фактора роста эндотелия сосудов (VEGFR2); антигена системы Льюис (Y); CD24; рецептора фактора роста тромбоцитов бета-типа (PDGFR-бета); стадиеспецифического эмбрионального антигена 4 (SSEA-4); CD20; рецептора фолиевой кислоты альфа; рецепторной тирозинпротеинкиназы ERBB2 (Her2/neu); муцина 1, ассоциированного с клеточной поверхностью (MUC1); рецептора эпидермального фактора роста (EGFR); молекулы адгезии нервных клеток (NCAM); простазы; простатической кислой фосфатазы (РАР); мутантного фактора элонгации 2 (ELF2M); эфрина В2; белка активации фибробластов альфа (FAP); рецептора инсулиноподобного фактора роста 1 (рецептора IGF-I), карбоангидразы IX (CAIX); субъединицы бета-типа 9 протеасомы (просомы, мультикаталитической протеазы) (LMP2); гликопротеина 100 (gp100); онкогенного слитого белка, состоящего из кластерного региона точечных разрывов (BCR) и гомолога 1 онкогена вируса лейкоза мышей Абельсона (Abl) (bcr-abl); тирозиназы; рецептора 2 эфрина А-типа (EphA2); фукозил-GM1; молекулы адгезии, представляющей собой сиалилированный антиген системы Льюис (sLe); ганглиозида GM3 (aNeu5Ac(2-3)bDGalp(1-4)bDGlcp(1-1)Cer); трансглутаминазы 5 (TGS5); высокомолекулярного антигена, ассоциированного с меланомой (HMWMAA); ганглиозида о-ацетил-GD2 (OAcGD2); рецептора фолиевой кислоты бета; опухолевого эндотелиального маркера 1 (TEM1/CD248); белка, родственного опухолевому эндотелиальному маркеру 7 (TEM7R); клаудина 6 (CLDN6); рецептора тиреотропного гормона (TSHR); представителя D группы 5 класса С рецепторов, сопряженных с G-белками (GPRC5D); белка, кодируемого открытой рамкой считывания 61 X-хромосомы (CXORF61); CD97; CD179a; киназы анапластической лимфомы (ALK); полисиаловой кислоты; плацентоспецифического белка 1 (PLAC1); гексасахаридной части гликоцерамида globoH (GloboH); дифференцировочного антигена молочной железы (NY-BR-1); уроплакина 2 (UPK2); клеточного рецептора 1 вируса гепатита A (HAVCR1); бета-3-адренорецептора (ADRB3); паннексина 3 (PANX3); рецептора 20, сопряженного с G-белком (GPR20); антигена локуса K9 комплекса лимфоцитарных антигенов 6 (LY6K); обонятельного рецептора 51Е2 (OR51E2); белка, кодируемого альтернативной рамкой считывания гена TCR-гамма (TARP); белка опухоли Вильмса (WT1); раково-тестикулярного антигена 1 (NY-ESO-1); раково-тестикулярного антигена 2 (LAGE-1a); антигена 1, ассоциированного с меланомой (MAGE-A1); белка, кодируемого транслокационным вариантом 6 гена ETS, локализованным в р-плече 12 хромосомы (ETV6-AML); белка спермы 17 (SPA17); представителя 1А семейства X-антигенов (XAGE1); ангиопоэтин-связывающего рецептора клеточной поверхности 2 (TIE-2); раково-тестикулярного антигена меланомы 1 (MAD-CT-1); раково-тестикулярного антигена меланомы 2 (MAD-CT-2); Fos-родственного антигена 1; опухолевого белка р53 (р53); мутантного р53; простеина; сурвивина; теломеразы; опухолевого антигена 1 карциномы предстательной железы (РСТА-1 или галектина 8), антигена меланомы 1, распознаваемого Т-клетками (MelanA или MART1); мутантного антигена саркомы крыс (Ras); обратной транскриптазы теломеразы человека (hTERT); антигена саркомы с точечными разрывами при транслокации; меланомного ингибитора апоптоза (ML-IAP); ERG (продукта слитого гена трансмембранной сериновой протеазы 2 (TMPRSS2) и ETS); N-ацетилглюкозаминилтрансферазы V (NA17); белка с парным box-доменом Рах-3 (РАХЗ); андрогенового рецептора; циклина В1; нейробластомного гомолога онкогена вируса миелоцитоматоза птиц v-myc (MYCN); представителя С семейства гомологов Ras (RhoC); белка 2, родственного тирозиназе (TRP-2); цитохрома Р450 1В1 (CYP1B1); белка, подобного CCCTC-связывающему фактору (белку с "цинковыми пальцами") (BORIS или "брата регулятора импринтированных сайтов"), антигена плоскоклеточной карциномы 3, распознаваемого Т-клетками (SART3); белка с парным box-доменом Рах-5 (РАХ5); проакрозин-связывающего белка sp32 (OY-TES1); лимфоцитоспецифической протеинтирозинкиназы (LCK); якорного белка 4 киназы A (AKAP-4); антигена 2 синовиальной саркомы с точечным разрывом в X-хромосоме (SSX2); рецептора конечных продуктов гликирования (RAGE-1); почечного убиквитарного белка 1 (RU1); почечного убиквитарного белка 2 (RU2); легумаина; Е6 вируса папилломы человека (Е6 HPV); Е7 вируса папилломы человека (Е7 HPV); кишечной карбоксилэстеразы; мутантного белка теплового шока 70-2 (мутантного варианта hsp70-2); CD79a; CD79b; CD72; иммуноглобулиноподобного рецептора 1, ассоциированного с лейкоцитами (LAIR1); рецептора Fc-фрагмента IgA (FCAR или CD89); представителя 2 подсемейства А лейкоцитарных иммуноглобулиноподобных рецепторов (LILRA2); представителя f семейства белков, подобных молекулам CD300 (CD300LF); представителя А семейства 12 лектиновых доменов С-типа (CLEC12A); антигена 2 стромальных клеток костного мозга (BST2); белка 2, подобного муциноподобному рецептору гормона, содержащего EGF-подобный модуль (EMR2); лимфоцитарного антигена 75 (LY75); глипикана-3 (GPC3); антигена 5, подобного Fc-рецептору (FCRL5); и полипептида 1, подобного иммуноглобулину лямбда (IGLL1).
В некоторых вариантах осуществления антигенсвязывающий домен содержит одну, две, три (например, все три) CDR тяжелой цепи - CDR1 HC, CDR2 HC и CDR3 HC - из антитела, указанного выше, и/или одну, две, три (например, все три) CDR легкой цепи - CDR1 LC, CDR2 LC и CDR3 LC - из антитела, указанного выше. В некоторых вариантах осуществления антигенсвязывающий домен содержит вариабельную область тяжелой цепи и/или вариабельную область легкой цепи антитела, указанного или описанного выше.
В некоторых вариантах осуществления домен, связывающий опухолевый антиген, представляет собой фрагмент, например, одноцепочечный вариабельный фрагмент (scFv). В некоторых вариантах осуществления связывающий домен для антигена, ассоциированного с раком, описанного в данном документе, представляет собой Fv, Fab (Fab')2 или бифункциональное (например, биспецифическое) гибридное антитело (например, Lanzavecchia et al., Eur. J. Immunol. 17, 105 (1987)). В некоторых вариантах осуществления антитела и их фрагменты по настоящему изобретению связываются с белком, представляющим собой антиген, ассоциированный с раком, описанный в данном документе, с аффинностью дикого типа или усиленной аффинностью.
В некоторых случаях scFv можно получить согласно способу, известному из уровня техники (см., например, Bird et al. (1988) Science 242:423-426 и Huston et al. (1988) Proc. Natl. Acad. Sci. USA 85:5879-5883). Молекулы scFv можно получить путем связывания VH- и VL-областей друг с другом с помощью гибких полипептидных линкеров. Молекулы scFv содержат линкер (например, линкер Ser-Gly) с оптимизированными длиной и/или аминокислотным составом. Длина линкера может сильно влиять на укладку и взаимодействие вариабельных областей scFv. В действительности, если используют короткий полипептидный линкер (например, длиной 5-10 аминокислот), то внутрицепочечное сворачивание предотвращается. Межцепочечное сворачивание также необходимо для объединения двух вариабельных областей вместе с образованием функционального эпитопсвязывающего участка. Примеры ориентации и размера линкеров см., например, в Hollinger et al. 1993 Proc Natl Acad. Sci. U.S.A. 90:6444-6448, публикациях заявок на патент США №№ 2005/0100543, 2005/0175606, 2007/0014794 и публикациях согласно PCT №№ WO2006/020258 и WO2007/024715, которые включены в данный документ посредством ссылки.
ScFv может содержать линкер из по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, или больше аминокислотных остатков между его VL- и VH-областями. Линкерная последовательность может содержать любую встречающуюся в природе аминокислоту. В некоторых вариантах осуществления линкерная последовательность содержит аминокислоты глицин и серин. В некоторых вариантах осуществления линкерная последовательность содержит группы глициновых и сериновых повторов, таких как (Gly4Ser)n, где n представляет собой положительное целое число, которое равняется или больше 1 (SEQ ID NO: 25). В некоторых вариантах осуществления линкер может представлять собой (Gly4Ser)4 (SEQ ID NO: 27) или (Gly4Ser)3(SEQ ID NO: 28). При изменении длины линкера может сохраняться или усиливаться активность, что приводит к улучшению эффективности в исследованиях активности.
В некоторых вариантах осуществления антигенсвязывающий домен представляет собой T-клеточный рецептор ("TCR") или его фрагмент, например, одноцепочечный TCR (scTCR). Способы получения таких TCR известны из уровня техники. См., например, Willemsen RA et al, Gene Therapy 7: 1369-1377 (2000); Zhang T et al, Cancer Gene Ther 11: 487-496 (2004); Aggen et al, Gene Ther. 19(4):365-74 (2012) (литературные источники включены в данный документ во всей своей полноте). Например, может быть сконструирован scTCR, который содержит гены Vα и Vβ из клона Т-клеток, связанные с помощью линкера (например, гибкого пептида). Данный подход является весьма применимым для мишени, ассоциированной с раком, которая сама по себе является внутриклеточной, однако фрагмент такого антигена (пептида) презентируется на поверхности раковых клеток с помощью MHC.
Трансмембранный домен
Что касается трансмембранного домена, в различных вариантах осуществления CAR можно разработать таким образом, чтобы он содержал трансмембранный домен, присоединенный к внеклеточному домену CAR. Трансмембранный домен может содержать одну или несколько дополнительных аминокислот, прилегающих к трансмембранной области, например, одну или несколько аминокислот, относящихся к внеклеточной области белка, из которого получен трансмембранный домен (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 и вплоть до 15 аминокислот внеклеточной области), и/или одну или несколько дополнительных аминокислот, относящихся к внутриклеточной области белка, из которого получен трансмембранный домен (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 и вплоть до 15 аминокислот внутриклеточной области). В некоторых вариантах осуществления трансмембранный домен представляет собой домен, ассоциированный с одним из остальных используемых доменов CAR. В некоторых случаях трансмембранный домен можно выбрать или модифицировать с помощью аминокислотной замены, чтобы избежать связывания таких доменов с трансмембранными доменами тех же или других поверхностных мембранных белков, например, чтобы свести к минимуму взаимодействия с другими элементами рецепторного комплекса. В некоторых вариантах осуществления трансмембранный домен способен к гомодимеризации с другим CAR на поверхности клетки, экспрессирующей CAR, например, CAR-T-клетки. В некоторых вариантах осуществления аминокислотную последовательность трансмембранного домена можно модифицировать или подвергнуть замене таким образом, чтобы свести к минимуму взаимодействия со связывающими доменами нативного партнера по связыванию, присутствующего в той же клетке, экспрессирующей CAR, например, CAR-T.
Трансмембранный домен может быть получен из природного либо из рекомбинантного источника. Если источник является природным, то домен может быть получен из любого мембраносвязанного или трансмембранного белка. В некоторых вариантах осуществления трансмембранный домен способен передавать сигнал внутриклеточному(внутриклеточным) домену(доменам) всякий раз, когда CAR связался с мишенью. Трансмембранный домен, особенно применимый в настоящем изобретении, может содержать по меньшей мере трансмембранную(трансмембранные) область(области), например, альфа-, бета- или дзета-цепи Т-клеточного рецептора, CD28, CD3-эпсилон, CD45, CD4, CD5, CD8 (например, CD8-альфа, CD8-бета), CD9, CD16, CD22, CD33, CD37, CD64, CD80, CD86, CD134, CD137, CD154. В некоторых вариантах осуществления трансмембранный домен может содержать по меньшей мере трансмембранную(трансмембранные) область(области) костимулирующей молекулы, например, молекулы MHC I класса, белковых рецепторов TNF, иммуноглобулиноподобных белков, рецепторов цитокинов, интегринов, сигнальных молекул активации лимфоцитов (белков SLAM), активирующих рецепторов NK-клеток, BTLA, лиганда Toll-подобного рецептора, OX40, CD2, CD7, CD27, CD28, CD30, CD40, CDS, ICAM-1, LFA-1 (CD11a/CD18), 4-1BB (CD137), B7-H3, CDS, ICAM-1, ICOS (CD278), GITR, BAFFR, LIGHT, HVEM (LIGHTR), KIRDS2, SLAMF7, NKp80 (KLRF1), NKp44, NKp30, NKp46, CD19, CD4, CD8-альфа, CD8-бета, IL2R-бета, IL2R-гамма, IL7R-альфа, ITGA4, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, LFA-1, ITGB7, NKG2D, NKG2C, TNFR2, TRANCE/RANKL, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (Tactile), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), CD69, SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR, LAT, GADS, SLP-76, PAG/Cbp, CD19a и лиганда, который специфично связывается с CD83.
В некоторых случаях трансмембранный домен может быть присоединен к внеклеточной области CAR, например, антигенсвязывающему домену CAR, с помощью шарнирной области, например, шарнирной области из белка человека. Например, в некоторых вариантах осуществления шарнирная область может представлять собой шарнирную область Ig (иммуноглобулина) человека, например, шарнирную область IgG4 или шарнирную область CD8a. В некоторых вариантах осуществления шарнирная область или спейсер содержат аминокислотную последовательность под SEQ ID NO: 2 (например, состоят из нее). В некоторых вариантах осуществления трансмембранный домен содержит трансмембранный домен под SEQ ID NO: 6 (например, состоит из него).
В некоторых вариантах осуществления шарнирная область или спейсер включают шарнирную область IgG4. Например, в некоторых вариантах осуществления шарнирная область или спейсер включают шарнирную область под SEQ ID NO: 3. В некоторых вариантах осуществления шарнирная область или спейсер включают шарнирную область, кодируемую нуклеотидной последовательностью под SEQ ID NO: 14.
В некоторых вариантах осуществления шарнирная область или спейсер включают шарнирную область IgD. Например, в некоторых вариантах осуществления шарнирная область или спейсер включают шарнирную область с аминокислотной последовательностью под SEQ ID NO: 4. В некоторых вариантах осуществления шарнирная область или спейсер включают шарнирную область, кодируемую нуклеотидной последовательностью под SEQ ID NO: 15.
В некоторых вариантах осуществления трансмембранный домен может быть рекомбинантным, и в этом случае он будет содержать главным образом гидрофобные остатки, такие как лейцин и валин. В некоторых вариантах осуществления на каждом конце рекомбинантного трансмембранного домена может находиться триплет из фенилаланина, триптофана и валина.
Короткий олиго- или полипептидный линкер длиной от 2 до 10 аминокислот необязательно может образовывать связь между трансмембранным доменом и цитоплазматической областью CAR. Глицин-сериновый дублет является особенно подходящим линкером. Например, в некоторых вариантах осуществления линкер содержит аминокислотную последовательность под SEQ ID NO: 5. В некоторых вариантах осуществления линкер кодируется нуклеотидной последовательностью под SEQ ID NO: 16.
В некоторых вариантах осуществления шарнирная область или спейсер включают в себя шарнирную область KIR2DS2.
Цитоплазматический домен
Цитоплазматические домен или область CAR по настоящему изобретению содержат внутриклеточный сигнальный домен. Внутриклеточный сигнальный домен обычно отвечает за активацию по меньшей мере одной из нормальных эффекторных функций иммунной клетки, в которую был введен CAR.
Примеры внутриклеточных сигнальных доменов для применения в CAR по настоящему изобретению включают цитоплазматические последовательности Т-клеточного рецептора (TCR) и корецепторов, которые действуют совместно с инициированием передачи сигнала после вступления в контакт с антигенным рецептором, а также любое производное или вариант этих последовательностей и любую рекомбинантную последовательность, которая обладает такими же функциональными способностями.
Известно, что сигналы, образуемые при участии только TCR, являются недостаточными для полной активации Т-клетки, и что также необходим вторичный и/или костимулирующий сигнал. Таким образом, можно сказать, что активация Т-клеток опосредуется двумя различными классами цитоплазматических сигнальных последовательностей: теми, которые инициируют антигензависимую первичную активацию посредством TCR (первичные внутриклеточные сигнальные домены), и теми, которые действуют антигеннезависимым образом, передавая вторичный или костимулирующий сигнал (вторичный цитоплазматический домен, например, костимулирующий домен).
Первичный сигнальный домен регулирует первичную активацию комплекса TCR стимулирующим образом либо ингибирующим образом. Первичные внутриклеточные сигнальные домены, которые действуют стимулирующим образом, могут содержать сигнальные мотивы, известные как иммунорецепторные тирозиновые активирующие мотивы или ITAM.
Примеры ITAM-содержащих первичных внутриклеточных сигнальных доменов, которые являются особенно применимыми в настоящем изобретении, включают домены из TCR-дзета, FcR-гамма, FcR-бета, CD3-гамма, CD3-дельта, CD3-эпсилон, CD5, CD22, CD79a, CD79b, CD278 (также известного как "ICOS"), FcεRI, DAP10, DAP12 и CD66d. В некоторых вариантах осуществления CAR по настоящему изобретению содержит внутриклеточный сигнальный домен, например, первичный сигнальный домен CD3-дзета.
В некоторых вариантах осуществления первичный сигнальный домен содержит модифицированный домен ITAM, например, мутантный домен ITAM, который обладает измененной (например, повышенной или пониженной) активностью по сравнению с нативным доменом ITAM. В некоторых вариантах осуществления первичный сигнальный домен содержит модифицированный ITAM-содержащий первичный внутриклеточный сигнальный домен, например, оптимизированный и/или усеченный ITAM-содержащий первичный внутриклеточный сигнальный домен. В некоторых вариантах осуществления первичный сигнальный домен содержит один, два, три, четыре или больше мотивов ITAM.
Дополнительные примеры молекул, содержащих первичный внутриклеточный сигнальный домен, являющихся особенно применимыми в настоящем изобретении, включают молекулы DAP10, DAP12 и CD32.
Внутриклеточный сигнальный домен CAR может содержать первичный сигнальный домен, например, сигнальный домен CD3-дзета, в отдельности или в комбинации с любым(любыми) другим(другими) требуемым(требуемыми) внутриклеточным(внутриклеточными) сигнальным(сигнальными) доменом(доменами), применимым(применимыми) в качестве составной части CAR по настоящему изобретению. Например, внутриклеточный сигнальный домен CAR может содержать первичный сигнальный домен, например, часть дзета-цепи CD3, и костимулирующий сигнальный домен. Костимулирующий сигнальный домен относится к части CAR, содержащей внутриклеточный домен костимулирующей молекулы. Костимулирующая молекула представляет собой молекулу клеточной поверхности, отличную от антигенного рецептора или его лигандов, которая необходима для эффективного ответа лимфоцитов на антиген. Примеры такой молекулы включают молекулу MHC I класса, белковые рецепторы TNF, иммуноглобулиноподобные белки, рецепторы цитокинов, интегрины, сигнальные молекулы активации лимфоцитов (белки SLAM), активирующие рецепторы NK-клеток, BTLA, лиганд Toll-подобного рецептора, OX40, CD2, CD7, CD27, CD28, CD30, CD40, CDS, ICAM-1, LFA-1 (CD11a/CD18), 4-1BB (CD137), B7-H3, CDS, ICAM-1, ICOS (CD278), GITR, BAFFR, LIGHT, HVEM (LIGHTR), KIRDS2, SLAMF7, NKp80 (KLRF1), NKp44, NKp30, NKp46, CD19, CD4, CD8-альфа, CD8-бета, IL2R-бета, IL2R-гамма, IL7R-альфа, ITGA4, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, LFA-1, ITGB7, NKG2D, NKG2C, TNFR2, TRANCE/RANKL, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (Tactile), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), CD69, SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR, LAT, GADS, SLP-76, PAG/Cbp, CD19a и лиганд, который специфично связывается с CD83, и т. п. Например, было продемонстрировано, что костимуляция с помощью CD27 усиливает размножение, эффекторную функцию и выживание CAR-T-клеток человека in vitro и усиливает персистенцию и противоопухолевую активность T-клеток человека in vivo (Song et al. Blood. 2012; 119(3):696-706). Внутриклеточные сигнальные последовательности в цитоплазматической части CAR по настоящему изобретению могут быть связаны друг с другом в случайном или установленном порядке. Короткий олиго- или полипептидный линкер, например, длиной от 2 до 10 аминокислот (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислот) необязательно может образовывать связь между внутриклеточными сигнальными последовательностями. В некоторых вариантах осуществления в качестве подходящего линкера может применяться глицин-сериновый дублет. В некоторых вариантах осуществления в качестве подходящего линкера может применяться одна аминокислота, например, аланин, глицин.
В некоторых вариантах осуществления внутриклеточный сигнальный домен разработан таким образом, что он содержит два или более, например, 2, 3, 4, 5 или более, костимулирующих сигнальных доменов. В некоторых вариантах осуществления два или более, например, 2, 3, 4, 5 или более, костимулирующих сигнальных доменов разделены линкерной молекулой, например, линкерной молекулой, описанной в данном документе. В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит два костимулирующих сигнальных домена. В некоторых вариантах осуществления линкерная молекула представляет собой глициновый остаток. В некоторых вариантах осуществления линкер представляет собой аланиновый остаток.
В некоторых вариантах осуществления внутриклеточный сигнальный домен разработан таким образом, что он содержит сигнальный домен CD3-дзета и сигнальный домен CD28. В некоторых вариантах осуществления внутриклеточный сигнальный домен разработан таким образом, что он содержит сигнальный домен CD3-дзета и сигнальный домен 4-1BB. В некоторых вариантах осуществления сигнальный домен 4-1BB представляет собой сигнальный домен под SEQ ID NO: 7. В некоторых вариантах осуществления сигнальный домен CD3-дзета представляет собой сигнальный домен под SEQ ID NO: 9 (мутантный CD3-дзета) или SEQ ID NO: 10 (CD3-дзета человека дикого типа).
В некоторых вариантах осуществления внутриклеточный сигнальный домен разработан таким образом, что он содержит сигнальный домен CD3-дзета и сигнальный домен CD27. В некоторых вариантах осуществления сигнальный домен CD27 содержит аминокислотную последовательность под SEQ ID NO: 8. В некоторых вариантах осуществления сигнальный домен CD27 кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 19.
В некоторых вариантах осуществления внутриклеточный домен разработан таким образом, что он содержит сигнальный домен CD3-дзета и сигнальный домен CD28. В некоторых вариантах осуществления сигнальный домен CD28 содержит аминокислотную последовательность под SEQ ID NO: 36. В некоторых вариантах осуществления сигнальный домен CD28 кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 37.
В некоторых вариантах осуществления внутриклеточный домен разработан таким образом, что он содержит сигнальный домен CD3-дзета и сигнальный домен ICOS. В некоторых вариантах осуществления сигнальный домен ICOS содержит аминокислотную последовательность под SEQ ID NO: 38. В некоторых вариантах осуществления сигнальный домен ICOS кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 39.
Конфигурации CAR
Двойные CAR
В одном варианте осуществления иммунная клетка (например, T-клетка или NK-клетка) экспрессирует два CAR, например, первый CAR, который связывается с первым антигеном, и второй CAR, который связывается со вторым антигеном. В одном варианте осуществления первый антиген и второй антиген являются разными. В одном варианте осуществления первый или второй антиген выбран из антигена, экспрессируемого на B-клетках, антигена, экспрессируемого на клетках острого миелоидного лейкоза, или антигена на клетках солидной опухоли. В одном варианте осуществления первый или второй антиген выбран из CD10, CD19, CD20, CD22, CD34, CD123, BCMA, FLT-3, ROR1, CD79b, CD179b, CD79a, CD34, CLL-1, рецептора фолиевой кислоты бета, FLT3, EGFRvIII, мезотелина, EGFRvIII, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC.
В одном варианте осуществления первый антиген представляет собой CD19. В одном варианте осуществления второй антиген не представляет собой CD19. В одном варианте осуществления второй антиген представляет собой антиген, раскрытый в данном документе, который не представляет собой CD19.
В одном варианте осуществления первый антиген представляет собой BCMA. В одном варианте осуществления второй антиген не представляет собой BCMA. В одном варианте осуществления второй антиген представляет собой антиген, раскрытый в данном документе, который не представляет собой BCMA. В одном варианте осуществления второй антиген выбран из антигена, экспрессируемого на B-клетках, антигена, экспрессируемого на клетках острого миелоидного лейкоза, или антигена на клетках солидной опухоли. В одном варианте осуществления второй антиген выбран из CD10, CD19, CD20, CD22, CD34, CD123, FLT-3, ROR1, CD79b, CD179b, CD79a, CD34, CLL-1, рецептора фолиевой кислоты бета, FLT3, EGFRvIII, мезотелина, EGFRvIII, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC. В одном варианте осуществления первый антиген представляет собой BCMA, а второй антиген представляет собой CD19.
В одном варианте осуществления первый CAR кодируется первой последовательностью нуклеиновой кислоты. В одном варианте осуществления второй CAR кодируется второй последовательностью нуклеиновой кислоты. В одном варианте осуществления первая и вторая последовательности нуклеиновой кислоты расположены в одной молекуле нуклеиновой кислоты. В одном варианте осуществления первая и вторая последовательности нуклеиновой кислоты расположены в отдельных молекулах нуклеиновой кислоты. В одном варианте осуществления молекула нуклеиновой кислоты или молекулы нуклеиновой кислоты представляют собой молекулы ДНК или РНК. В ряде вариантов осуществления первая и вторая последовательности нуклеиновой кислоты находятся в одинаковой ориентации, например, транскрипция первой и второй последовательностей нуклеиновой кислоты происходит в одном и том же направлении. В ряде вариантов осуществления первая и вторая последовательности нуклеиновой кислоты находятся в разных ориентациях. В ряде вариантов осуществления один промотор контролирует экспрессию первой и второй последовательностей нуклеиновой кислоты. В ряде вариантов осуществления нуклеиновая кислота, кодирующая сайт расщепления протеазами (такой как сайт расщепления T2A, P2A, E2A или F2A), находится между первой и второй последовательностями нуклеиновой кислоты. В ряде вариантов осуществления сайт расщепления протеазами размещен таким образом, что клетка может экспрессировать слитый белок, содержащий первый CAR и второй CAR, и слитый белок впоследствии подвергается процессингу посредством протеолитического расщепления с образованием двух пептидов. В некоторых вариантах осуществления первая последовательность нуклеиновой кислоты расположена выше второй последовательности нуклеиновой кислоты, или вторая последовательность нуклеиновой кислоты расположена выше первой последовательности нуклеиновой кислоты. В ряде вариантов осуществления первый промотор контролирует экспрессию первой последовательности нуклеиновой кислоты, а второй промотор контролирует экспрессию второй последовательности нуклеиновой кислоты. В ряде вариантов осуществления молекула нуклеиновой кислоты представляет собой плазмиду. В ряде вариантов осуществления молекула нуклеиновой кислоты содержит вирусный элемент упаковки. В ряде вариантов осуществления иммунная клетка может содержать протеазу (например, эндогенную или экзогенную протеазу), которая расщепляет сайт расщепления T2A, P2A, E2A или F2A.
В одном варианте осуществления первый CAR содержит первый антигенсвязывающий домен, а второй CAR содержит второй антигенсвязывающий домен. В одном варианте осуществления первый или второй антигенсвязывающий домен содержит CDR, VH, VL или scFv, раскрытые в данном документе, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними. В одном варианте осуществления первый или второй антигенсвязывающий домен содержит CDR, VH, VL или scFv антигенсвязывающего домена для BCMA, раскрытого в данном документе (например, аминокислотную последовательность, раскрытую в таблицах 3-15, 19, 20, 22 и 26, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней). В одном варианте осуществления первый или второй антигенсвязывающий домен содержит CDR, VH, VL или scFv антигенсвязывающего домена для CD19, раскрытого в данном документе (например, аминокислотную последовательность, раскрытую в таблицах 2, 19 и 22, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней).
В одном варианте осуществления первый антиген представляет собой BCMA, а второй антиген представляет собой CD19. В одном варианте осуществления иммунная клетка (например, T-клетка или NK-клетка) экспрессирует CAR для BCMA, например, CAR для BCMA, описанный в данном документе, и CAR для CD19, например, CAR для CD19, описанный в данном документе. В одном варианте осуществления иммунная клетка (например, T-клетка или NK-клетка) содержит первую последовательность нуклеиновой кислоты, кодирующую CAR для BCMA, например, CAR для BCMA, описанный в данном документе, и вторую последовательность нуклеиновой кислоты, кодирующую CAR для CD19, например, CAR для CD19, описанный в данном документе. В таблице 19 показаны иллюстративные аминокислотные последовательности и последовательности нуклеиновой кислоты конструкций двойных CAR.
(в плече CAR для BCMA)
(в плече CAR для BCMA)
(в плече CAR для BCMA)
(в плече CAR для BCMA)
(в плече CAR для BCMA)
(в плече CAR для BCMA)
(в плече CAR для CD19)
(в плече CAR для CD19)
(в плече CAR для CD19)
(в плече CAR для CD19)
(в плече CAR для CD19)
(в плече CAR для CD19)
R1G5-P2A-duCD19.1
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYAMSWVRQAPGKGLEWVSAISGSGGSTYYADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARREWWGESWLFDYWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSYSTPLTFGQGTKVEIKTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPG (SEQ ID NO: 224)
Плечо CAR для CD19:
EIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 225)
PI61-P2A-duCD19.1
QVQLQESGGGVVQPGRSLRLSCAASGFTFSSYGMHWVRQAPGKGLEWVAVISYDGSNKYYADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCGGSGYALHDDYYGLDVWGQGTLVTVSSGGGGSGGGGSGGGGSQSALTQPASVSGSPGQSITISCTGTSSDVGGYNYVSWYQQHPGKAPKLMIYDVSNRPSGVSNRFSGSKSGNTASLTISGLQAEDEADYYCSSYTSSSTLYVFGSGTKVTVLTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPG (SEQ ID NO: 226)
Плечо CAR для CD19:
EIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 225)
R1B6-P2A-duCD19.1
EVQLLESGGGLVQPGGSLRLSCAASGFTFSSYAMSWVRQAPGKGLEWVSAISGSGGSTYYADSVKGRFTISRDNSKNTLYLQMNSLRAEDTAVYYCARREWVPYDVSWYFDYWGQGTLVTVSSGGGGSGGGGSGGGGSGGGGSDIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSYSTPLTFGQGTKVEIKTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPG (SEQ ID NO: 227)
Плечо CAR для CD19:
EIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 225)
duBCMA.4-P2A-duCD19.1
EVQLVESGGGLVQPGGSLRLSCAVSGFALSNHGMSWVRRAPGKGLEWVSGIVYSGSTYYAASVKGRFTISRDNSRNTLYLQMNSLRPEDTAIYYCSAHGGESDVWGQGTTVTVSSASGGGGSGGGGSGGGGSDIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSYSTPYTFGQGTKVEIKTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPG (SEQ ID NO: 228)
Плечо CAR для CD19:
EIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 225)
duCD19.1-P2A-duBCMA.4
EIVMTQSPATLSLSPGERATLSCRASQDISKYLNWYQQKPGQAPRLLIYHTSRLHSGIPARFSGSGSGTDYTLTISSLQPEDFAVYFCQQGNTLPYTFGQGTKLEIKGGGGSGGGGSGGGGSQVQLQESGPGLVKPSETLSLTCTVSGVSLPDYGVSWIRQPPGKGLEWIGVIWGSETTYYQSSLKSRVTISKDNSKNQVSLKLSSVTAADTAVYYCAKHYYYGGSYAMDYWGQGTLVTVSSTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPG (SEQ ID NO: 229)
Плечо CAR для BCMA:
EVQLVESGGGLVQPGGSLRLSCAVSGFALSNHGMSWVRRAPGKGLEWVSGIVYSGSTYYAASVKGRFTISRDNSRNTLYLQMNSLRPEDTAIYYCSAHGGESDVWGQGTTVTVSSASGGGGSGGGGSGGGGSDIQLTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQKPGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQSYSTPYTFGQGTKVEIKTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 230)
описание
В некоторых вариантах осуществления в данном документе раскрыта выделенная молекула нуклеиновой кислоты, содержащая первую последовательность нуклеиновой кислоты, кодирующую первый полипептид CAR, и вторую последовательность нуклеиновой кислоты, кодирующую второй полипептид CAR, где первый полипептид CAR содержит первый антигенсвязывающий домен, который представляет собой домен, связывающий BCMA (например, человеческий домен, связывающий BCMA), первый трансмембранный домен и первый внутриклеточный сигнальный домен, и где второй полипептид CAR содержит второй антигенсвязывающий домен, который представляет собой домен, связывающий CD19, второй трансмембранный домен и второй внутриклеточный сигнальный домен. В некоторых вариантах осуществления первый полипептид CAR содержит VH, содержащую CDR1 HC, CDR2 HC и CDR3 HC последовательности, связывающей BCMA, представленные в таблице 20 или 26, и VL, содержащую CDR1 LC, CDR2 LC и CDR3 LC последовательности, связывающей BCMA, представленные в таблице 20 или 26, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243. В определенном варианте осуществления первый полипептид CAR содержит VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 239 и 242 соответственно, где VH и VL соединены линкером, содержащим аминокислотную последовательность под SEQ ID NO: 243. В определенном варианте осуществления первый полипептид CAR содержит scFv, содержащий аминокислотную последовательность под SEQ ID NO: 200. В определенном варианте осуществления первый полипептид CAR содержит аминокислотную последовательность под SEQ ID NO: 230 или 228. В некоторых вариантах осуществления второй полипептид CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и/или CDR3 LC последовательности, связывающей CD19, представленные в таблице 19 или таблице 22 (например, CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC, содержащие аминокислотные последовательности под SEQ ID NO: 295 и 245-249 соответственно). В некоторых вариантах осуществления второй полипептид CAR содержит VH и/или VL последовательности, связывающей CD19, представленные в таблице 19 или таблице 22 (например, VH и VL, содержащие аминокислотные последовательности под SEQ ID NO: 250 и 251 соответственно), или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ними. В некоторых вариантах осуществления второй полипептид CAR содержит scFv последовательности, связывающей CD19, представленный в таблице 19 или таблице 22 (например, scFv, содержащий аминокислотную последовательность под SEQ ID NO: 211), или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ним. В некоторых вариантах осуществления второй полипептид CAR включает в себя полипептид CAR с последовательностью, связывающей CD19, представленный в таблице 19 или таблице 22 (например, полипептид CAR, содержащий аминокислотную последовательность под SEQ ID NO: 225 или 229), или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ним. В некоторых вариантах осуществления молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты под SEQ ID NO: 221 или 223. В некоторых вариантах осуществления молекула нуклеиновой кислоты кодирует аминокислотную последовательность под SEQ ID NO: 220 или 222 с сигнальным пептидом или без сигнального пептида под SEQ ID NO: 1.
Полиспецифические CAR
В одном варианте осуществления CAR по настоящему изобретению представляет собой полиспецифический CAR. В одном варианте осуществления полиспецифический CAR представляет собой биспецифический CAR. В одном варианте осуществления биспецифический CAR содержит антигенсвязывающий домен, который представляет собой молекулу биспецифического антитела. Биспецифическое антитело характеризуется специфичностью в отношении не более чем двух антигенов. Молекула биспецифического антитела характеризуется наличием первой последовательности вариабельного домена иммуноглобулина, которая характеризуется специфичностью связывания с первым эпитопом, и второй последовательности вариабельного домена иммуноглобулина, которая характеризуется специфичностью связывания со вторым эпитопом. В одном варианте осуществления первый и второй эпитопы находятся на одном и том же антигене, например, на одном и том же белке (или субъединице мультимерного белка). В одном варианте осуществления первый и второй эпитопы перекрываются. В одном варианте осуществления первый и второй эпитопы не перекрываются. В одном варианте осуществления первый и второй эпитопы находятся на разных антигенах, например, на разных белках (или на разных субъединицах мультимерного белка). В одном варианте осуществления молекула биспецифического антитела содержит последовательность вариабельного домена тяжелой цепи и последовательность вариабельного домена легкой цепи, которые характеризуются специфичностью связывания с первым эпитопом, и последовательность вариабельного домена тяжелой цепи и последовательность вариабельного домена легкой цепи, которые характеризуются специфичностью связывания со вторым эпитопом. В одном варианте осуществления молекула биспецифического антитела содержит полуантитело, характеризующееся специфичностью связывания с первым эпитопом, и полуантитело, характеризующееся специфичностью связывания со вторым эпитопом. В одном варианте осуществления молекула биспецифического антитела содержит полуантитело или его фрагмент, характеризующиеся специфичностью связывания с первым эпитопом, и полуантитело или его фрагмент, характеризующиеся специфичностью связывания со вторым эпитопом. В одном варианте осуществления молекула биспецифического антитела содержит scFv или его фрагмент, характеризующиеся специфичностью связывания с первым эпитопом, и scFv или его фрагмент, характеризующиеся специфичностью связывания со вторым эпитопом.
В некоторых вариантах осуществления CAR по настоящему изобретению содержит антигенсвязывающий домен, который представляет собой молекулу полиспецифического (например, биспецифического или триспецифического) антитела. Протоколы получения молекул биспецифических или гетеродимерных антител известны из уровня техники, включая без ограничения, например, подход "выступ во впадину", как описано, например, в US 5731168; спаривание Fc с использованием направленного электростатического взаимодействия, как описано, например, в WO 09/089004, WO 06/106905 и WO 2010/129304; образование гетеродимеров путем конструирования доменов с обменом нитей (SEED), как описано, например, в WO 07/110205; обмен Fab-фрагментами, как описано, например, в WO 08/119353, WO 2011/131746 и WO 2013/060867; двойные конъюгаты антител, например, получаемые путем сшивания антител с образованием биспецифической структуры с помощью гетеробифункционального реагента, содержащего реакционноспособную аминогруппу и реакционноспособную сульфгидрильную группу, как описано, например, в US 4433059; детерминанты биспецифических антител, получаемые путем рекомбинации полуантител (пары тяжелая-легкая цепь или Fab) из разных антител посредством цикла восстановления и окисления дисульфидных связей между двумя тяжелыми цепями, как описано, например, в US 4444878; трифункциональные антитела, например, три Fab'-фрагмента, сшитые с помощью реакционноспособных сульфгидрильных групп, как описано, например, в US5273743; биосинтетические связывающие белки, например, пары scFv, сшитых на С-концевых хвостах, предпочтительно посредством химической сшивки с помощью дисульфидных связей или реакционноспособных аминогрупп, как описано, например, в US5534254; бифункциональные антитела, например, Fab-фрагменты с различной специфичностью связывания, димеризованные с помощью лейциновых "застежек" (например, c-fos и c-jun), заменивших константный домен, как описано, например, в US5582996; биспецифические и олигоспецифические моно- и олиговалентные рецепторы, например, VH-CH1-области двух антител (два Fab-фрагмента), связанные полипептидным спейсером между CH1-областью одного антитела и VH-областью другого антитела, обычно с ассоциированными легкими цепями, как описано, например, в US5591828; биспецифические конъюгаты ДНК-антитело, например, получаемые путем сшивки антител или Fab-фрагментов двухнитевым фрагментом ДНК, как описано, например, в US5635602; биспецифические слитые белки, например, экспрессионную конструкцию, содержащую два scFv с гидрофильным спиральным пептидным линкером между ними и полную константную область, как описано, например, в US5637481; поливалентные и полиспецифические связывающие белки, например, димеры полипептидов, имеющие первый домен со связывающей областью вариабельной области тяжелой цепи Ig и второй домен со связывающей областью вариабельной области легкой цепи Ig, как правило, называемые диателами (также охватываются структуры более высокого порядка для создания биспецифических, триспецифических или тетраспецифических молекул, как описано, например, в US5837242; конструкции миниантител со связанными VL- и VH-цепями, дополнительно соединенными пептидными спейсерами с шарнирной областью и СН3-областью антитела, которые могут быть димеризованы с образованием биспецифических/поливалентных молекул, как описано, например, в US5837821; VH- и VL-домены, связанные коротким пептидным линкером (например, из 5 или 10 аминокислот) или вовсе не имеющие линкера, в любой ориентации, которые могут образовывать димеры с образованием биспецифических диател; тримеры и тетрамеры, как описано, например, в US5844094; нить из VH-доменов (или VL-доменов у представителей семейства), соединенных пептидными связями со сшиваемыми группами на С-конце, дополнительно ассоциированных с VL-доменами с образованием последовательно соединенных Fv (или scFv), как описано, например, в US5864019; и одноцепочечные связывающие полипептиды как с VH-, так и с VL-доменами, связанными пептидным линкером, объединенные в поливалентные структуры посредством нековалентной или химической сшивки с образованием, например, гомобивалентных, гетеробивалентных, тривалентных и тетравалентных структур с использованием формата как типа scFv, так и типа диатела, как описано, например, в US5869620. Дополнительные иллюстративные полиспецифические и биспецифические молекулы и способы их получения можно найти, например, в US 5910573, US 5932448, US 5959083, US 5989830, US 6005079, US 6239259, US 6294353, US 6333396, US 6476198, US 6511663, US 6670453, US 6743896, US 6809185, US 6833441, US 7129330, US 7183076, US 7521056, US 7527787, US 7534866, US 7612181, US 2002004587 A1, US 2002076406 A1, US 2002103345 A1, US 2003207346 A1, US 2003211078 A1, US 2004219643 A1, US 2004220388 A1, US 2004242847 A1, US 2005003403 A1, US 2005004352 A1, US 2005069552 A1, US 2005079170 A1, US 2005100543 A1, US 2005136049 A1, US 2005136051 A1, US 2005163782 A1, US 2005266425 A1, US 2006083747 A1, US 2006120960 A1, US 2006204493 A1, US 2006263367 A1, US 2007004909 A1, US 2007087381 A1, US 2007128150 A1, US 2007141049 A1, US 2007154901 A1, US 2007274985 A1, US 2008050370 A1, US 2008069820 A1, US 2008152645 A1, US 2008171855 A1, US 2008241884 A1, US 2008254512 A1, US 2008260738 A1, US 2009130106 A1, US 2009148905 A1, US 2009155275 A1, US 2009162359 A1, US 2009162360 A1, US 2009175851 A1, US 2009175867 A1, US 2009232811 A1, US 2009234105 A1, US 2009263392 A1, US 2009274649 A1, EP 346087 A2, WO 0006605 A2, WO 02072635 A2, WO 04081051 A1, WO 06020258 A2, WO 2007044887 A2, WO 2007095338 A2, WO 2007137760 A2, WO 2008119353 A1, WO 2009021754 A2, WO 2009068630 A1, WO 9103493 A1, WO 9323537 A1, WO 9409131 A1, WO 9412625 A2, WO 9509917 A1, WO 9637621 A2, WO 9964460 A1. Содержание вышеупомянутых заявок включено в данный документ посредством ссылки во всей своей полноте.
В каждом антителе или фрагменте антитела (например, scFv) в молекуле биспецифического антитела VH может находиться выше или ниже VL. В некоторых вариантах осуществления вышерасположенные антитело или фрагмент антитела (например, scFv) имеют такую организацию, при которой их VH (VH1) расположена выше их VL (VL1), а нижерасположенные антитело или фрагмент антитела (например, scFv) имеют такую организацию, при которой их VL (VL2) расположена выше их VH (VH2), так что молекула биспецифического антитела в целом имеет организацию VH1-VL1-VL2-VH2. В других вариантах осуществления вышерасположенные антитело или фрагмент антитела (например, scFv) имеют такую организацию, при которой их VL (VL1) расположена выше их VH (VH1), а нижерасположенные антитело или фрагмент антитела (например, scFv) имеют такую организацию, при которой их VH (VH2) расположена выше их VL (VL2), так что молекула биспецифического антитела в целом имеет организацию VL1-VH1-VH2-VL2. Линкер необязательно располагается между двумя антителами или фрагментами антител (например, scFv), например, между VL1 и VL2, если конструкция имеет организацию VH1-VL1-VL2-VH2, или между VH1 и VH2, если конструкция имеет организацию VL1-VH1-VH2-VL2. Линкер может представлять собой линкер, описанный в данном документе, например, линкер (Gly4-Ser)n, где n равняется 1, 2, 3, 4, 5 или 6, предпочтительно 4 (SEQ ID NO: 26). Обычно линкер между двумя scFv должен иметь достаточную длину для предотвращения неправильного спаривания между доменами двух scFv. Линкер необязательно располагается между VL и VH первого scFv. Линкер необязательно располагается между VL и VH второго scFv. В конструкциях, которые имеют несколько линкеров, любые два или более линкера могут быть одинаковыми или разными. Соответственно, в некоторых вариантах осуществления биспецифический CAR содержит VL, VH и необязательно один или несколько линкеров в виде организации, описанной в данном документе.
В одном аспекте молекула биспецифического антитела характеризуется наличием первой последовательности вариабельного домена иммуноглобулина, например, scFv, которая характеризуется специфичностью связывания с BCMA, например, содержит scFv, описанный в данном документе, или содержит CDR легкой цепи и/или CDR тяжелой цепи из scFv для BCMA, описанного в данном документе, и второй последовательности вариабельного домена иммуноглобулина, которая характеризуется специфичностью связывания со вторым эпитопом на другом антигене. В одном аспекте вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с антигеном, экспрессируемым на клетках AML, например, антигеном, отличным от BCMA. Например, вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с CD123. В качестве другого примера вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с CLL-1. В качестве другого примера вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с CD34. В качестве другого примера вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с FLT3. Например, вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с рецептором фолиевой кислоты бета. В некоторых аспектах вторая последовательность вариабельного домена иммуноглобулина характеризуется специфичностью связывания с антигеном, экспрессируемым на B-клетках, например, CD10, CD19, CD20, CD22, CD34, CD123, FLT-3, ROR1, CD79b, CD179b или CD79a.
CAR в формате диатела
В некоторых вариантах осуществления CAR по настоящему изобретению представляет собой биспецифический CAR. В некоторых вариантах осуществления CAR по настоящему изобретению представляет собой CAR в формате диатела. В некоторых вариантах осуществления CAR в формате диатела содержит антигенсвязывающий домен, который связывается с первым антигеном и вторым антигеном. В некоторых вариантах осуществления антигенсвязывающий домен содержит VH1, VL1, VH2 и VL2, где VH1 и VL1 связываются с первым антигеном, а VH2 и VL2 связываются со вторым антигеном. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH1 - необязательно линкер 1 ("L1") - VH2 - необязательно линкер 2 ("L2") - VL2 - необязательно линкер 3 ("L3") - VL1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VL1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL1 - необязательно L1 - VH2 - необязательно L2 - VL2 - необязательно L3 - VH1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL1 - необязательно L1 - VL2 - необязательно L2 - VH2 - необязательно L3 - VH1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VL2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VL2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL2 - необязательно L1 - VH1 - необязательно L2 - VL1 - необязательно L3 - VH2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL2 - необязательно L1 - VL1 - необязательно L2 - VH1 - необязательно L3 - VH2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH1 - линкер 1 ("L1") - VH2 - линкер 2 ("L2") - VL2 - линкер 3 ("L3") - VL1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH1 - L1 - VL2 - L2 - VH2 - L3 - VL1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL1 - L1 - VH2 - L2 - VL2 - L3 - VH1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL1 - L1 - VL2 - L2 - VH2 - L3 - VH1 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH2 - L1 - VH1 - L2 - VL1 - L3 - VL2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH2 - L1 - VL1 - L2 - VH1 - L3 - VL2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL2 - L1 - VH1 - L2 - VL1 - L3 - VH2 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL2 - L1 - VL1 - L2 - VH1 - L3 - VH2 от N-конца до C-конца. В некоторых вариантах осуществления вариабельные области слиты с помощью линкера, содержащего аминокислотную последовательность GGGGSGGGGS (SEQ ID NO: 5). В некоторых вариантах осуществления вариабельные области слиты с помощью линкера, содержащего аминокислотную последовательность GGGGSGGGGSGGGGSGGGGS (SEQ ID NO: 63). В некоторых вариантах осуществления L1 содержит аминокислотную последовательность под SEQ ID NO: 5. В некоторых вариантах осуществления L2 содержит аминокислотную последовательность под SEQ ID NO: 63. В некоторых вариантах осуществления L3 содержит аминокислотную последовательность под SEQ ID NO: 5. В некоторых вариантах осуществления VH1, VL1, VH2 или VL2 содержат последовательность CDR, VH или VL, раскрытую в данном документе, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней. В некоторых вариантах осуществления диатело, раскрытое в данном документе, содержит сконструированный дисульфидный мостик, например, для стабилизации диатела и/или для содействия правильному спариванию VH и VL. В некоторых вариантах осуществления сконструированный дисульфидный мостик находится между вариабельной областью, которая расположена наиболее проксимально относительно шарнирной области (например, областью VH или VL, которая расположена наиболее проксимально относительно шарнирной области), и ее соответствующим партнером по спариванию (например, соответствующей VL или соответствующей VH).
В некоторых вариантах осуществления первый антиген и второй антиген являются разными. В некоторых вариантах осуществления первый или второй антиген выбран из антигена, экспрессируемого на B-клетках, антигена, экспрессируемого на клетках острого миелоидного лейкоза, или антигена на клетках солидной опухоли. В некоторых вариантах осуществления первый или второй антиген выбран из CD10, CD19, CD20, CD22, CD34, CD123, BCMA, FLT-3, ROR1, CD79b, CD179b, CD79a, CD34, CLL-1, рецептора фолиевой кислоты бета, FLT3, EGFRvIII, мезотелина, EGFRvIII, GD2, Tn-антигена, sTn-антигена, Tn-O-гликопептидов, sTn-O-гликопептидов, PSMA, CD97, TAG72, CD44v6, CEA, EPCAM, KIT, IL-13Ra2, легумана, GD3, CD171, IL-11Ra, PSCA, MAD-CT-1, MAD-CT-2, VEGFR2, антигена Y системы Льюис, CD24, PDGFR-бета, SSEA-4, рецептора фолиевой кислоты альфа, ERBB (например, ERBB2), Her2/neu, MUC1, EGFR, NCAM, эфрина B2, CAIX, LMP2, sLe, HMWMAA, o-ацетил-GD2, рецептора фолиевой кислоты бета, TEM1/CD248, TEM7R, FAP, легумаина, E6 или E7 HPV, ML-IAP, CLDN6, TSHR, GPRC5D, ALK, полисиаловой кислоты, Fos-родственного антигена, эластазы нейтрофилов, TRP-2, CYP1B1, белка спермы 17, бета-субъединицы хорионического гонадотропина человека, AFP, тиреоглобулина, PLAC1, globoH, RAGE1, MN-CA IX, обратной транскриптазы теломеразы человека, кишечной карбоксилэстеразы, мутантного варианта hsp 70-2, NA-17, NY-BR-1, UPK2, HAVCR1, ADRB3, PANX3, NY-ESO-1, GPR20, Ly6k, OR51E2, TARP, GFRα4 или пептида любого из этих антигенов, презентированного на MHC.
В некоторых вариантах осуществления первый антиген представляет собой BCMA, а второй антиген представляет собой CD19. В некоторых вариантах осуществления CAR содержит антигенсвязывающий домен, который связывается с BCMA и CD19. В некоторых вариантах осуществления антигенсвязывающий домен содержит VH1 и VL1, которые связываются с BCMA ("VH для BCMA" и "VL для BCMA"), а также VH2 и VL2, которые связываются с CD19 ("VH для CD19" и "VL для CD19"). В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для BCMA - необязательно линкер 1 ("L1") - VH для CD19 - необязательно линкер 2 ("L2") - VL для CD19 - необязательно линкер 3 ("L3") - VL для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для BCMA - необязательно L1 - VL для CD19 - необязательно L2 - VH для CD19 - необязательно L3 - VL для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для BCMA - необязательно L1 - VH для CD19 - необязательно L2 - VL для CD19 - необязательно L3 - VH для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для BCMA - необязательно L1 - VL для CD19 - необязательно L2 - VH для CD19 - необязательно L3 - VH для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для CD19 - необязательно L1 - VH для BCMA - необязательно L2 - VL для BCMA - необязательно L3 - VL для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для CD19 - необязательно L1 - VL для BCMA - необязательно L2 - VH для BCMA - необязательно L3 - VL для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для CD19 - необязательно L1 - VH для BCMA - необязательно L2 - VL для BCMA - необязательно L3 - VH для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для CD19 - необязательно L1 - VL для BCMA - необязательно L2 - VH для BCMA - необязательно L3 - VH для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для BCMA - линкер 1 ("L1") - VH для CD19 - линкер 2 ("L2") - VL для CD19 - линкер 3 ("L3") - VL для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для BCMA - L1 - VL для CD19 - L2 - VH для CD19 - L3 - VL для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для BCMA - L1 - VH для CD19 - L2 - VL для CD19 - L3 - VH для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для BCMA - L1 - VL для CD19 - L2 - VH для CD19 - L3 - VH для BCMA от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для CD19 - L1 - VH для BCMA - L2 - VL для BCMA - L3 - VL для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VH для CD19 - L1 - VL для BCMA - L2 - VH для BCMA - L3 - VL для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для CD19 - L1 - VH для BCMA - L2 - VL для BCMA - L3 - VH для CD19 от N-конца до C-конца. В некоторых вариантах осуществления антигенсвязывающий домен имеет организацию VL для CD19 - L1 - VL для BCMA - L2 - VH для BCMA - L3 - VH для CD19 от N-конца до C-конца. В некоторых вариантах осуществления вариабельные области слиты с помощью линкера, содержащего аминокислотную последовательность под SEQ ID NO: 5 или 63 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней. В некоторых вариантах осуществления L1 содержит аминокислотную последовательность под SEQ ID NO: 5. В некоторых вариантах осуществления L2 содержит аминокислотную последовательность под SEQ ID NO: 63. В некоторых вариантах осуществления L3 содержит аминокислотную последовательность под SEQ ID NO: 5.
В некоторых вариантах осуществления VH для BCMA содержит последовательности CDR или VH, раскрытые в данном документе, например, последовательности CDR или VH, раскрытые в таблицах 3-15, 19, 20, 22, 26 и 31, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ними. В некоторых вариантах осуществления VL для BCMA содержит последовательности CDR или VL, раскрытые в данном документе, например, последовательности CDR или VL, раскрытые в таблицах 3-15, 19, 20, 22, 26 и 31, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ними. В некоторых вариантах осуществления VH для CD19 содержит последовательности CDR или VH, раскрытые в данном документе, например, последовательности CDR или VH, раскрытые в таблицах 2, 19, 22 и 31, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ними. В некоторых вариантах осуществления VL для CD19 содержит последовательности CDR или VL, раскрытые в данном документе, например, последовательности CDR или VL, раскрытые в таблицах 2, 19, 22 и 31, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ними.
В некоторых вариантах осуществления CAR, например, CAR в формате диатела, дополнительно содержит шарнирную область, трансмембранный домен и/или внутриклеточный сигнальный домен. В некоторых вариантах осуществления шарнирная область включает в себя шарнирную область CD8. В некоторых вариантах осуществления шарнирная область содержит последовательность шарнирной области, раскрытую в данном документе, например, аминокислотную последовательность под SEQ ID NO: 2 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней. В некоторых вариантах осуществления трансмембранный домен включает в себя трансмембранный домен CD8. В некоторых вариантах осуществления трансмембранный домен содержит последовательность трансмембранного домена, раскрытую в данном документе, например, аминокислотную последовательность под SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней. В некоторых вариантах осуществления внутриклеточный сигнальный домен включает в себя внутриклеточный домен 4-1BB. В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит последовательность костимулирующего сигнального домена, раскрытую в данном документе, например, аминокислотную последовательность под SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней. В некоторых вариантах осуществления внутриклеточный сигнальный домен включает в себя внутриклеточный домен CD3. В некоторых вариантах осуществления внутриклеточный сигнальный домен содержит последовательность первичного сигнального домена, раскрытую в данном документе, например, аминокислотную последовательность под SEQ ID NO: 9 или 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней.
Иллюстративные последовательности диател раскрыты в таблице 31. В некоторых вариантах осуществления CAR содержит антигенсвязывающий домен, содержащий аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 321-330, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней. В некоторых вариантах осуществления CAR содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 339-348, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 80%, 85%, 90%, 95% или 99% идентичностью с ней.
Химерный TCR
В одном аспекте антитела и фрагменты антител по настоящему изобретению могут быть пересажены на один или несколько константных доменов цепи Т-клеточного рецептора ("TCR"), например, альфа-цепи TCR или бета-цепи TCR, с созданием химерного TCR. Не ограничиваясь какой-либо теорией, полагают, что химерные TCR будут передавать сигналы через комплекс TCR при связывании антигена. Например, scFv, раскрытый в данном документе, можно пересадить на константный домен, например, на по меньшей мере часть внеклеточного константного домена, трансмембранного домена и цитоплазматического домена, цепи TCR, например, альфа-цепи TCR и/или бета-цепи TCR. В качестве другого примера, фрагмент антитела, например, VL-домен, описанный в данном документе, можно пересадить на константный домен альфа-цепи TCR, и фрагмент антитела, например, VH-домен, описанный в данном документе, можно пересадить на константный домен бета-цепи TCR (или в качестве альтернативы VL-домен можно пересадить на константный домен бета-цепи TCR, а VH-домен можно пересадить на альфа-цепь TCR). В качестве другого примера CDR антитела или фрагмента антитела, например, CDR антитела или фрагмента антитела, описанного в данном документе, можно пересадить на альфа- и/или бета-цепь TCR для создания химерного TCR. Например, раскрытые в данном документе LCDR можно пересадить на вариабельный домен альфа-цепи TCR, а раскрытые в данном документе HCDR можно пересадить на вариабельный домен бета-цепи TCR или наоборот. Такие химерные TCR можно получить посредством способов, известных из уровня техники (например, Willemsen RA et al, Gene Therapy 2000; 7: 1369-1377; Zhang T et al, Cancer Gene Ther 2004; 11: 487-496; Aggen et al, Gene Ther. 2012 Apr;19(4):365-74).
Дополнительные варианты осуществления
В одном аспекте описанная в данном документе клетка, экспрессирующая CAR, может дополнительно содержать второй CAR, например, второй CAR, который содержит другой антигенсвязывающий домен, например, для той же мишени (BCMA) или другой мишени (например, CD19, CD20 или CS-1 или других мишеней множественной миеломы, например, легкой каппа-цепи, CD138, антигена Y системы Льюис или CD38 (Garfall et al., Discovery Medicine, 2014, 17(91):37-46)). В одном варианте осуществления клетка, экспрессирующая CAR, содержит первый CAR, который нацеливается на первый антиген и содержит внутриклеточный сигнальный домен, имеющий костимулирующий сигнальный домен, а не первичный сигнальный домен, и второй CAR, который нацеливается на второй, другой, антиген и содержит внутриклеточный сигнальный домен, имеющий первичный сигнальный домен, а не костимулирующий сигнальный домен. Без ограничения какой-либо теорией, размещение костимулирующего сигнального домена, например, 4-1BB, CD28, CD27 или OX-40, в первом CAR и первичного сигнального домена, например, CD3-дзета, во втором CAR может ограничивать активность CAR клетками, в которых экспрессируются обе мишени. В одном варианте осуществления клетка, экспрессирующая CAR, содержит первый CAR для BCMA, который содержит домен, связывающий BCMA, трансмембранный домен и костимулирующий домен, и второй CAR, который нацеливается на антиген, отличный от BCMA (например, антиген, экспрессируемый на клетках лейкоза или лимфомы, например, CD19, CD20, CS-1, легкую каппа-цепь, CD139, антиген Y системы Льюиса или CD38), и содержит антигенсвязывающий домен, трансмембранный домен и первичный сигнальный домен. В другом варианте осуществления клетка, экспрессирующая CAR, содержит первый CAR для BCMA, который содержит домен, связывающий BCMA, трансмембранный домен и первичный сигнальный домен, и второй CAR, который нацеливается на антиген, отличный от BCMA (например, антиген, экспрессируемый на клетках лейкоза или лимфомы, например, CD19, CD20, CS-1, легкую каппа-цепь, CD139, антиген Y системы Льюис или CD38), и содержит антигенсвязывающий домен для антигена, трансмембранный домен и костимулирующий сигнальный домен. В одном варианте осуществления клетка, экспрессирующая CAR, содержит CAR для BCMA, описанный в данном документе, и CAR, который нацеливается на CD19 (CAR для CD19).
В одном варианте осуществления клетка, экспрессирующая CAR, содержит CAR для BCMA, описанный в данном документе, и ингибирующий CAR. В одном варианте осуществления ингибирующий CAR содержит антигенсвязывающий домен, который связывает антиген, обнаруживаемый на нормальных клетках, но не на раковых клетках. В одном варианте осуществления ингибирующий CAR содержит антигенсвязывающий домен, трансмембранный домен и внутриклеточный домен ингибирующей молекулы. Например, внутриклеточный домен ингибирующего CAR может представлять собой внутриклеточный домен PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулы MHC I класса, молекулы MHC II класса, GAL9, аденозина и TGFR-бета.
В одном варианте осуществления в случае, если клетка, экспрессирующая CAR содержит два или более различных CAR, то антигенсвязывающие домены различных CAR могут быть такими, что антигенсвязывающие домены не взаимодействуют друг с другом. Например, клетка, экспрессирующая первый и второй CAR, может иметь антигенсвязывающий домен первого CAR, например, в качестве фрагмента, например, scFv, который не ассоциирует с антигенсвязывающим доменом второго CAR, например, антигенсвязывающий домен второго CAR представляет собой VHH.
В некоторых вариантах осуществления антигенсвязывающий домен содержит однодоменные антигенсвязывающие (SDAB) молекулы, в том числе молекулы, в которых определяющие комплементарность области являются частью однодоменного полипептида. Примеры включают без ограничения вариабельные домены тяжелых цепей, связывающие молекулы, в природных условиях лишенные легких цепей, отдельные домены, полученные из традиционных 4-цепочечных антител, сконструированные домены и однодоменные каркасные структуры, отличные от полученных из антител. Молекулы SDAB могут являться любыми молекулами, известными из уровня техники, или любыми однодоменными молекулами, которые будут известны в будущем. Молекулы SDAB могут быть получены из любых видов, в том числе без ограничения из мыши, человека, верблюда, ламы, миноги, рыбы, акулы, козы, кролика и быка. Этот термин также охватывает встречающиеся в природе молекулы однодоменных антител из видов, отличных от Camelidae и акул.
В одном аспекте молекула SDAB может быть получена из вариабельной области иммуноглобулина, обнаруживаемого у рыб, как, например, та, которая получена из изотипа иммуноглобулина, известного как новый антигенный рецептор (NAR), обнаруживаемого в сыворотке крови акулы. Способы получения однодоменных молекул, получаемых из вариабельной области NAR ("IgNAR"), описаны в WO 03/014161 и Streltsov (2005) Protein Sci. 14:2901-2909.
Согласно другому аспекту молекула SDAB представляет собой встречающуюся в природе однодоменную антигенсвязывающую молекулу, известную как тяжелая цепь, лишенная легких цепей. Такие однодоменные молекулы раскрыты, например, в WO 9404678 и Hamers-Casterman, C. et al. (1993) Nature 363:446-448. В целях ясности этот вариабельный домен, полученный из молекулы тяжелой цепи, в природных условиях лишенной легкой цепи, именуется в данном документе как VHH или нанотело, чтобы отличать его от традиционного VH четырехцепочечных иммуноглобулинов. Такая молекула VHH может быть получена из видов Camelidae, например, из верблюда, ламы, дромадера, альпаки и гуанако. Другие виды, помимо Camelidae, могут продуцировать молекулы тяжелых цепей, в природных условиях лишенные легкой цепи; такие VHH входят в объем настоящего изобретения.
Молекулы SDAB могут быть рекомбинантными, с пересаженными CDR, гуманизированными, камелизированными, деиммунизированными и/или образованными in vitro (например, отобранными с помощью фагового дисплея).
Также было обнаружено, что в клетках, имеющих множество химерных рецепторов, встроенных в мембрану, содержащих антигенсвязывающий домен, взаимодействия между антигенсвязывающими доменами рецепторов могут быть нежелательными, например, поскольку они ингибируют способность одного или нескольких антигенсвязывающих доменов связываться с их когнатными антигенами. Соответственно, в данном документе раскрыты клетки, имеющие первый и второй не встречающиеся в природе химерные рецепторы, встроенные в мембрану, содержащие антигенсвязывающие домены, для которых сведены к минимуму такие взаимодействия. Также в данном документе раскрыты нуклеиновые кислоты, кодирующие первый и второй не встречающиеся в природе химерные рецепторы, встроенные в мембрану, содержащие антигенсвязывающие домены, для которых сведены к минимуму такие взаимодействия, а также способы получения и применения таких клеток и нуклеиновых кислот. В одном варианте осуществления антигенсвязывающий домен одного из указанного первого и указанного второго не встречающихся в природе химерных рецепторов, встроенных в мембрану, содержит scFv, а другой содержит одиночный VH-домен, например, одиночный VH-домен верблюдовых, акулы или миноги или одиночный VH-домен, полученный из последовательности человека или мыши.
В некоторых вариантах осуществления в заявляемом изобретении предусмотрены первый и второй CAR, где антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR не содержит вариабельный домен легкой цепи и вариабельный домен тяжелой цепи. В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR представляет собой scFv, а другой не представляет собой scFv. В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR содержит одиночный VH-домен, например, одиночный VH-домен верблюдовых, акулы или миноги или одиночный VH-домен, полученный из последовательности человека или мыши. В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR содержит нанотело. В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR содержит VHH-домен верблюдовых.
В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR содержит scFv, а другой содержит одиночный VH-домен, например, одиночный VH-домен верблюдовых, акулы или миноги или одиночный VH-домен, полученный из последовательности человека или мыши. В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR содержит scFv, а другой содержит нанотело. В некоторых вариантах осуществления антигенсвязывающий домен одного из указанного первого CAR и указанного второго CAR содержит scFv, а другой содержит VHH-домен верблюдовых.
В некоторых вариантах осуществления связывание антигенсвязывающего домена указанного первого CAR, при наличии его на поверхности клетки, со своим когнатным антигеном существенно не снижается благодаря наличию указанного второго CAR. В некоторых вариантах осуществления связывание антигенсвязывающего домена указанного первого CAR со своим когнатным антигеном в присутствии указанного второго CAR составляет 85%, 90%, 95%, 96%, 97%, 98% или 99% от связывания антигенсвязывающего домена указанного первого CAR со своим когнатным антигеном в отсутствие указанного второго CAR.
В некоторых вариантах осуществления антигенсвязывающие домены указанного первого CAR и указанного второго CAR при наличии их на поверхности клетки ассоциируют друг с другом в меньшей степени, чем в случае, если бы они оба являлись антигенсвязывающими доменами scFv. В некоторых вариантах осуществления антигенсвязывающие домены указанного первого CAR и указанного второго CAR ассоциируют друг с другом в на 85%, 90%, 95%, 96%, 97%, 98% или 99% меньшей степени, чем в случае, если бы они оба являлись антигенсвязывающими доменами scFv.
В другом аспекте описанная в данном документе клетка, экспрессирующая CAR, может дополнительно экспрессировать другое средство, например, средство, которое усиливает активность клетки, экспрессирующей CAR. Например, в одном варианте осуществления средство может представлять собой средство, которое ингибирует ингибирующую молекулу, например, средство, описанное в данном документе. Ингибирующие молекулы, например, PD1, в некоторых вариантах осуществления могут снижать способность клетки, экспрессирующей CAR, осуществлять иммунный эффекторный ответ. Примеры ингибирующих молекул включают PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулу MHC I класса, молекулу MHC II класса, GAL9, аденозин и TGFR-бета. В одном варианте осуществления средство, которое ингибирует ингибирующую молекулу, содержит первый полипептид, например, ингибирующую молекулу, ассоциированный со вторым полипептидом, который обеспечивает положительный сигнал для клетки, например, с внутриклеточным сигнальным доменом, описанным в данном документе. В одном варианте осуществления средство содержит первый полипептид, например, из ингибирующей молекулы, такой как PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекула MHC I класса, молекула MHC II класса, GAL9, аденозин и TGFR-бета или фрагмент любого из них (например, по меньшей мере часть внеклеточного домена любого из них), и второй полипептид, который представляет собой внутриклеточный сигнальный домен, описанный в данном документе (например, содержащий костимулирующий домен (например, 4-1BB, CD27, ICOS или CD28, например, описанный в данном документе) и/или первичный сигнальный домен (например, сигнальный домен CD3-дзета, описанный в данном документе). В одном варианте осуществления средство содержит первый полипептид из PD1 или его фрагмента (например, по меньшей мере часть внеклеточного домена PD1) и второй полипептид, представляющий собой внутриклеточный сигнальный домен, описанный в данном документе (например, сигнальный домен CD28, описанный в данном документе, и/или сигнальный домен CD3-дзета, описанный в данном документе). В ряде вариантов осуществления описанная в данном документе клетка, экспрессирующая CAR, содержит переключающий костимулирующий рецептор, например, описанный в WO 2013/019615, которая включена в данный документ посредством ссылки во всей своей полноте. PD1 является ингибирующим представителем семейства рецепторов CD28, которое также включает CD28, CTLA-4, ICOS и BTLA. PD-1 экспрессируется на активированных B-клетках, T-клетках и миелоидных клетках (Agata et al. 1996 Int. Immunol 8:765-75). Было показано, что два лиганда PD1 - PD-L1 и PD-L2 - подавляют активацию Т-клеток при связывании с PD1 (Freeman et al. 2000 J Exp Med 192:1027-34; Latchman et al. 2001 Nat Immunol 2:261-8; Carter et al. 2002 Eur J Immunol 32:634-43). PD-L1 является широко распространенным при различных формах рака у человека (Dong et al. 2003 J Mol Med 81:281-7; Blank et al. 2005 Cancer Immunol. Immunother 54:307-314; Konishi et al. 2004 Clin Cancer Res 10:5094). Иммуносупрессию можно устранить путем ингибирования локального взаимодействия PD1 с PD-L1.
В одном варианте осуществления средство, содержащее внеклеточный домен (ECD) ингибирующей молекулы, например, белка 1 запрограммированной гибели клеток (PD1), может быть слито с трансмембранным доменом и внутриклеточными сигнальными доменами, такими как 4-1BB и CD3-дзета (также называется в данном документе CAR на основе PD1). В одном варианте осуществления CAR на основе PD1 при применении в комбинациях с CAR для BCMA, описанным в данном документе, улучшает персистенцию клетки, экспрессирующей CAR, например, T-клетки или NK-клетки. В одном варианте осуществления CAR представляет собой CAR на основе PD1, содержащий внеклеточный домен PD1, указанный подчеркиванием в SEQ ID NO: 24. В одном варианте осуществления CAR на основе PD1 содержит аминокислотную последовательность под SEQ ID NO: 24.
В одном варианте осуществления CAR на основе PD1 содержит аминокислотную последовательность, представленную ниже (SEQ ID NO: 22).
В одном варианте осуществления средство содержит последовательность нуклеиновой кислоты, кодирующую CAR на основе PD1, например, CAR на основе PD1, описанный в данном документе. В одном варианте осуществления последовательность нуклеиновой кислоты для CAR на основе PD1 представлена под SEQ ID NO: 23, при этом ECD PD1 подчеркнут.
В другом аспекте в настоящем изобретении предусмотрена популяция клеток, экспрессирующих CAR, например, CAR-T-клеток или NK-клеток, экспрессирующих CAR. В некоторых вариантах осуществления популяция клеток, экспрессирующих CAR, содержит смесь клеток, экспрессирующих различные CAR. Например, в одном варианте осуществления популяция клеток, экспрессирующих CAR (например, CAR-T-клеток или NK-клеток, экспрессирующих CAR), может содержать первую клетку, экспрессирующую CAR, имеющий домен, связывающий BCMA, описанный в данном документе, и вторую клетку, экспрессирующую CAR, имеющий другой домен, связывающий BCMA, например, домен, связывающий BCMA, описанный в данном документе, который отличается от домена, связывающего BCMA, в CAR, экспрессируемом первой клеткой. В качестве другого примера, популяция клеток, экспрессирующих CAR, может содержать первую клетку, экспрессирующую CAR, который содержит домен, связывающий BCMA, например, описанный в данном документе, и вторую клетку, экспрессирующую CAR, который содержит антигенсвязывающий домен для мишени, отличной от BCMA (например, CD19, CD20, CS-1, легкой каппа-цепи, CD139, антигена Y системы Льюис или CD38). В одном варианте осуществления популяция клеток, экспрессирующих CAR, содержит первую клетку, экспрессирующую CAR, содержащий домен, связывающий BCMA, например, описанный в данном документе, и вторую клетку, экспрессирующую CAR, содержащий антигенсвязывающий домен, нацеливающийся на CD19 (CAR для CD19). В одном варианте осуществления популяция клеток, экспрессирующих CAR, содержит, например, первую клетку, экспрессирующую CAR, который содержит первичный внутриклеточный сигнальный домен, и вторую клетку, экспрессирующую CAR, который содержит вторичный сигнальный домен.
В другом аспекте в настоящем изобретении предусмотрена популяция клеток, где по меньшей мере одна клетка в популяции экспрессирует CAR, имеющий домен, связывающий BCMA, описанный в данном документе, и вторая клетка экспрессирует другое средство, например, средство, которое усиливает активность клетки, экспрессирующей CAR. Например, в одном варианте осуществления средство может представлять собой средство, которое ингибирует ингибирующую молекулу. Ингибирующие молекулы, например, могут в некоторых вариантах осуществления снижать способность клетки, экспрессирующей CAR, осуществлять иммунный эффекторный ответ. Примеры ингибирующих молекул включают PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулу MHC I класса, молекулу MHC II класса, GAL9, аденозин и TGFR-бета. В одном варианте осуществления средство, которое ингибирует ингибирующую молекулу, содержит первый полипептид, например, ингибирующую молекулу, ассоциированный со вторым полипептидом, который обеспечивает положительный сигнал для клетки, например, с внутриклеточным сигнальным доменом, описанным в данном документе. В одном варианте осуществления средство содержит первый полипептид, например, из ингибирующей молекулы, такой как PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекула MHC I класса, молекула MHC II класса, GAL9, аденозин и TGFR-бета или фрагмент любого из них (например, по меньшей мере часть внеклеточного домена любого из них), и второй полипептид, который представляет собой внутриклеточный сигнальный домен, описанный в данном документе (например, содержащий костимулирующий домен (например, 4-1BB, CD27, ICOS или CD28, например, описанный в данном документе) и/или первичный сигнальный домен (например, сигнальный домен CD3-дзета, описанный в данном документе). В одном варианте осуществления средство содержит первый полипептид из PD1 или его фрагмента (например, по меньшей мере часть внеклеточного домена PD1) и второй полипептид, представляющий собой внутриклеточный сигнальный домен, описанный в данном документе (например, сигнальный домен CD28, описанный в данном документе, и/или сигнальный домен CD3-дзета, описанный в данном документе).
В одном аспекте в настоящем изобретении предусмотрены способы, включающие введение популяции клеток, экспрессирующих CAR (например, CAR-T-клеток или NK-клеток, экспрессирующих CAR), например, смеси клеток, экспрессирующих различные CAR, в комбинации с другим средством, например, ингибитором киназы, таким как ингибитор киназы, описанный в данном документе. В другом аспекте в настоящем изобретении предусмотрены способы, включающие введение популяции клеток, где по меньшей мере одна клетка в популяции экспрессирует CAR, имеющий антигенсвязывающий домен для антигена, ассоциированного с раком, описанного в данном документе, и вторую клетку, экспрессирующую другое средство, например, средство, которое усиливает активность клетки, экспрессирующей CAR, в комбинации с другим средством, например, ингибитором киназы, таким как ингибитор киназы, описанный в данном документе.
CAR, содержащие рецепторы естественных клеток-киллеров (NKR)
В одном варианте осуществления молекула CAR, описанная в данном документе, содержит один или несколько компонентов рецептора естественных клеток-киллеров (NKR), образуя тем самым NKR-CAR. Компонент NKR может представлять собой трансмембранный домен, шарнирный домен или цитоплазматический домен любого из следующих рецепторов естественных клеток-киллеров: иммуноглобулиноподобного рецептора клеток-киллеров (KIR), например, KIR2DL1, KIR2DL2/L3, KIR2DL4, KIR2DL5A, KIR2DL5B, KIR2DS1, KIR2DS2, KIR2DS3, KIR2DS4, DIR2DS5, KIR3DL1/S1, KIR3DL2, KIR3DL3, KIR2DP1 и KIR3DP1; рецептора естественной цитотоксичности (NCR), например, NKp30, NKp44, NKp46; семейства рецепторов иммунных клеток, являющихся сигнальными молекулами активации лимфоцитов (SLAM), например, CD48, CD229, 2B4, CD84, NTB-A, CRACC, BLAME и CD2F-10; Fc-рецептора (FcR), например, CD16 и CD64; и рецепторов Ly49, например, LY49A, LY49C. Молекулы NKR-CAR, описанные в данном документе, могут взаимодействовать с адапторной молекулой или внутриклеточным сигнальным доменом, например, DAP12. Иллюстративные конфигурации и последовательности молекул CAR, содержащих компоненты NKR, описаны в международной публикации № WO2014/145252, содержание которой включено в данный документ посредством ссылки.
Каркасные структуры, отличные от антител
В ряде вариантов осуществления антигенсвязывающий домен содержит каркасную структуру, отличную от антитела, например, фибронектин, анкирин, доменное антитело, липокалин, малое модульное иммунофармацевтическое средство, максиантитело, белок A или аффилин. Каркасная структура, отличная от антитела, обладает способностью связываться с антигеном-мишенью на клетке. В ряде вариантов осуществления антигенсвязывающий домен представляет собой полипептид или его фрагмент из встречающегося в природе белка, экспрессируемого на клетке. В некоторых вариантах осуществления антигенсвязывающий домен содержит каркасную структуру, отличную от антитела. Можно использовать большое разнообразие каркасных структур, отличных от антител, при условии, что полученный в результате полипептид содержит по меньшей мере одну связывающую область, которая специфично связывается с антигеном-мишенью на клетке-мишени.
Каркасные структуры, отличные от антител, включают фибронектин (Novartis, Массачусетс), анкирин (Molecular Partners AG, Цюрих, Швейцария), доменные антитела (Domantis, Ltd., Кембридж, Массачусетс и Ablynx NV, Звейнарде, Бельгия), липокалин (Pieris Proteolab AG, Фрайзинг, Германия), малые модульные иммунофармацевтические средства (Trubion Pharmaceuticals Inc., Сиэтл, Вашингтон), максиантитела (Avidia, Inc., Маунтин-Вью, Калифорния), белок A (Affibody AG, Швеция) и аффилин (гамма-кристаллин или убиквитин) (Scil Proteins GmbH, Галле, Германия).
Стратегии регуляции химерных антигенных рецепторов
Существует множество путей, которыми можно регулировать формы активности CAR. В некоторых вариантах осуществления регулируемый CAR (RCAR), в котором можно контролировать активность CAR, является желательным для оптимизации безопасности и эффективности CAR-терапии. Например, индуцирование апоптоза с помощью, например, каспазы, слитой с димеризационным доменом (см., например, Di et al., N Engl. J. Med. 2011 Nov. 3; 365(18):1673-1683), можно применять в качестве защитного переключателя в CAR-терапии по настоящему изобретению. В другом примере клетки, экспрессирующие CAR, могут также экспрессировать молекулу индуцируемой каспазы-9 (iCaspase-9), которая при введении димеризующего лекарственного средства (например, римидуцида (также называемого AP1903 (Bellicum Pharmaceuticals) или AP20187 (Ariad)) приводит к активации каспазы-9 и апоптозу клеток. Молекула iCaspase-9 содержит связывающий домен для химического индуктора димеризации (CID), который опосредует димеризацию в присутствии CID. Это приводит к индуцируемому и селективному истощению по клеткам, экспрессирующим CAR. В некоторых случаях молекула iCaspase-9 кодируется молекулой нуклеиновой кислоты, отдельной от вектора(векторов), кодирующего(кодирующих) CAR. В некоторых случаях молекула iCaspase-9 кодируется той же молекулой нуклеиновой кислоты, что и вектор, кодирующий CAR. iCaspase-9 может обеспечивать защитное переключение для избежания возможной токсичности клеток, экспрессирующих CAR. См., например, Song et al. Cancer Gene Ther. 2008; 15(10):667-75; Clinical Trial Id. № NCT02107963; и Di Stasi et al. N. Engl. J. Med. 2011; 365:1673-83.
Альтернативные стратегии регуляции средства CAR-терапии по настоящему изобретению включают использование малых молекул или антител, которые деактивируют или выключают активность CAR, например, путем удаления клеток, экспрессирующих CAR, например, путем индукции антителозависимой клеточноопосредованной цитотоксичности (ADCC). Например, клетки, экспрессирующие CAR, описанные в данном документе, могут также экспрессировать антиген, который распознается молекулами, способными индуцировать гибель клеток, например, ADCC или комплемент-индуцированную гибель клеток. Например, клетки, экспрессирующие CAR, описанные в данном документе, также могут экспрессировать рецептор, на который могут нацеливаться антитело или фрагмент антитела. Примеры таких рецепторов включают EpCAM, VEGFR, интегрины (например, интегрины ανβ3, α4, αΙ¾β3, α4β7, α5β1, ανβ3, αν), представителей суперсемейства рецепторов TNF (например, TRAIL-R1 , TRAIL-R2), рецептор PDGF, рецептор интерферона, рецептор фолиевой кислоты, GPNMB, ICAM-1, HLA-DR, CEA, CA-125, MUC1, TAG-72, рецептор IL-6, 5T4, GD2, GD3, CD2, CD3, CD4, CD5, CD1 1, CD1 1 a/LFA-1, CD15, CD18/ITGB2, CD19, CD20, CD22, CD23/рецептор IgE, CD25, CD28, CD30, CD33, CD38, CD40, CD41, CD44, CD51, CD52, CD62L, CD74, CD80, CD125, CD147/базиджин, CD152/CTLA-4, CD154/CD40L, CD195/CCR5, CD319/SLAMF7 и EGFR и их усеченные варианты (например, варианты, в которых сохраняются один или несколько внеклеточных эпитопов, но отсутствуют одна или несколько областей в цитоплазматическом домене). Например, клетки, экспрессирующие CAR, описанные в данном документе, также могут экспрессировать усеченный рецептор эпидермального фактора роста (EGFR), у которого отсутствует способность к передаче сигнала, но сохраняется эпитоп, распознаваемый молекулами, способными индуцировать ADCC, например, цетуксимабом (ERBITUX®), так что введение цетуксимаба индуцирует ADCC и последующее истощение популяции клеток, экспрессирующих CAR (см., например, WO2011/056894 и Jonnalagadda et al., Gene Ther. 2013; 20(8)853-860). Другая стратегия предусматривает обеспечение экспрессии высококомпактного маркерного/"суицидального" гена, в котором объединены эпитопы-мишени обоих из антигенов CD32 и CD20 в клетках, экспрессирующих CAR, описанных в данном документе, которые связываются с ритуксимабом, что приводит к избирательному истощению популяции клеток, экспрессирующих CAR, например, посредством ADCC (см., например, Philip et al., Blood. 2014; 124(8)1277-1287). Другие способы истощения популяции клеток, экспрессирующих CAR, описанных в данном документе, включают введение CAMPATH® - моноклонального антитела к CD52, которое избирательно связывается со зрелыми лимфоцитами, например, клетками, экспрессирующими CAR, и направляет их на разрушение, например, посредством индукции ADCC. В других вариантах осуществления на клетки, экспрессирующие CAR, можно осуществлять избирательное нацеливание с помощью лиганда CAR, например, антиидиотипического антитела. В некоторых вариантах осуществления антиидиотипическое антитело может обуславливать активность эффекторных клеток, например, формы активности ADCC или ADC, за счет чего обеспечивается снижение количества клеток, экспрессирующих CAR. В других вариантах осуществления лиганд CAR, например, антиидиотипическое антитело, может быть связан со средством, которое индуцирует уничтожение клеток, например, токсином, за счет чего обеспечивается снижение количества клеток, экспрессирующих CAR. В качестве альтернативы молекулы CAR сами по себе могут быть представлены в такой конфигурации, при которой активность можно регулировать, например, путем включения или выключения, как описано ниже.
В некоторых вариантах осуществления RCAR содержит набор полипептидов, как правило, в простейших вариантах осуществления два, в которых составляющие стандартного CAR, описанного в данном документе, например, антигенсвязывающий домен и внутриклеточный сигнальный домен, разделены на отдельные полипептиды или компоненты. В некоторых вариантах осуществления набор полипептидов содержит димеризационный переключатель, который при наличии димеризующей молекулы может связывать полипептиды друг с другом, например, может связывать антигенсвязывающий домен с внутриклеточным сигнальным доменом. Дополнительное описание и иллюстративные конфигурации таких регулируемых CAR предусмотрены в данном документе и в международной публикации № WO 2015/090229, включенной в данный документ посредством ссылки во всей своей полноте.
В одном варианте осуществления RCAR содержит два полипептида или компонента: 1) внутриклеточный сигнальный компонент, содержащий внутриклеточный сигнальный домен, например, первичный внутриклеточный сигнальный домен, описанный в данном документе, и первый переключающий домен; 2) антигенсвязывающий компонент, содержащий антигенсвязывающий домен, например, который нацеливается на опухолевый антиген, описанный в данном документе, и второй переключающий домен. RCAR необязательно содержит трансмембранный домен, описанный в данном документе. В одном варианте осуществления трансмембранный домен может быть расположен во внутриклеточном сигнальном компоненте, в антигенсвязывающем компоненте или в них обоих. (Если не указано иное, то при описании в данном документе компонентов или элементов RCAR порядок может быть таким, как предусмотрено, но также включены и другие порядки. Другими словами, в одном варианте осуществления порядок является таким, как изложено в тексте, но в других вариантах осуществления порядок может быть другим. Например, порядок элементов с одной стороны от трансмембранной области может отличаться от примера, например, размещение переключающего домена относительно внутриклеточного сигнального домена может быть другим, например, обратным).
В одном варианте осуществления первый и второй переключающий домены могут образовывать внутриклеточный или внеклеточный димеризационный переключатель. В одном варианте осуществления димеризационный переключатель может представлять собой гомодимеризационный переключатель, например, если первый и второй переключающие домены являются одинаковыми, или гетеродимеризационный переключатель, например, если первый и второй переключающие домены отличаются друг от друга.
В ряде вариантов осуществления RCAR может содержать "мультипереключатель". Мультипереключатель может содержать гетеродимеризационные переключающие домены или гомодимеризационные переключающие домены. Мультипереключатель содержит множество, например, 2, 3, 4, 5, 6, 7, 8, 9 или 10, переключающих доменов отдельно в первом компоненте, например, антигенсвязывающем компоненте, и втором компоненте, например, внутриклеточном сигнальном компоненте. В одном варианте осуществления первый компонент может содержать множество первых переключающих доменов, например, переключающих доменов на основе FKBP, и второй компонент может содержать множество вторых переключающих доменов, например, переключающих доменов на основе FRB. В одном варианте осуществления первый компонент может содержать первый и второй переключающий домен, например, переключающий домен на основе FKBP и переключающий домен на основе FRB, и второй компонент может содержать первый и второй переключающий домен, например, переключающий домен на основе FKBP и переключающий домен на основе FRB.
В одном варианте осуществления внутриклеточный сигнальный компонент содержит один или несколько внутриклеточных сигнальных доменов, например, первичный внутриклеточный сигнальный домен и один или несколько костимулирующих сигнальных доменов.
В одном варианте осуществления антигенсвязывающий компонент может содержать один или несколько внутриклеточных сигнальных доменов, например, один или несколько костимулирующих сигнальных доменов. В одном варианте осуществления антигенсвязывающий компонент содержит множество, например, 2 или 3, костимулирующих сигнальных доменов, описанных в данном документе, например, выбранных из 4-1BB, CD28, CD27, ICOS и OX40, и в ряде вариантов осуществления первичный внутриклеточный сигнальный домен отсутствует. В одном варианте осуществления антигенсвязывающий компонент содержит следующие костимулирующие сигнальные домены в направлении от внеклеточного к внутриклеточному пространству: 4-1BB-CD27; 4-1BB-CD27; CD27-4-1BB; 4-1BB-CD28; CD28-4-1BB; OX40-CD28; CD28-OX40; CD28-4-1BB или 4-1BB-CD28. В таких вариантах осуществления внутриклеточный связывающий компонент содержит домен CD3-дзета. В одном таком варианте осуществления RCAR содержит (1) антигенсвязывающий компонент, содержащий антигенсвязывающий домен, трансмембранный домен и два костимулирующих домена, а также первый переключающий домен; и (2) внутриклеточный сигнальный домен, содержащий трансмембранный домен или домен прикрепления к мембране и по меньшей мере один первичный внутриклеточный сигнальный домен и второй переключающий домен.
В одном варианте осуществления представлены RCAR, где антигенсвязывающий компонент не прикреплен к поверхности CAR-клетки. Это дает возможность клетке, имеющей внутриклеточный сигнальный компонент, успешно соединяться с одним или несколькими антигенсвязывающими доменами без трансформации клетки последовательностью, которая кодирует антигенсвязывающий компонент. В таких вариантах осуществления RCAR содержит 1) внутриклеточный сигнальный компонент, содержащий первый переключающий домен, трансмембранный домен, внутриклеточный сигнальный домен, например, первичный внутриклеточный сигнальный домен, и первый переключающий домен; и 2) антигенсвязывающий компонент, содержащий антигенсвязывающий домен и второй переключающий домен, где антигенсвязывающий компонент не содержит трансмембранный домен или домен прикрепления к мембране и необязательно не содержит внутриклеточный сигнальный домен. В некоторых вариантах осуществления RCAR может дополнительно содержать 3) второй антигенсвязывающий компонент, содержащий второй антигенсвязывающий домен, например, второй антигенсвязывающий домен, который связывает антиген, отличный от того, который связывается антигенсвязывающим доменом; и второй переключающий домен.
Также в данном документе предусмотрены RCAR, где антигенсвязывающий компонент обладает способностью к биспецифической активации и нацеливанию. В таком варианте осуществления антигенсвязывающий компонент может содержать множество, например, 2, 3, 4 или 5, антигенсвязывающих доменов, например, scFv, где каждый антигенсвязывающий домен связывается с антигеном-мишенью, например, с разными антигенами или одним тем же антигеном, например, с одним и тем же или разными эпитопами на одном и том же антигене. В одном варианте осуществления множество антигенсвязывающих доменов находятся в тандеме, и между каждыми двумя следующими друг за другом антигенсвязывающими доменами необязательно располагается линкер или шарнирная область. Подходящие линкеры и шарнирные области описаны в данном документе.
В одном варианте осуществления представлены RCAR, имеющие конфигурацию, которая обеспечивает переключение пролиферации. В таком варианте осуществления RCAR содержит 1) внутриклеточный сигнальный компонент, содержащий необязательно трансмембранный домен или домен прикрепления к мембране; один или несколько костимулирующих сигнальных доменов, например, выбранных из 4-1BB, CD28, CD27, ICOS и OX40, и переключающий домен; и 2) антигенсвязывающий компонент, содержащий антигенсвязывающий домен, трансмембранный домен и первичный внутриклеточный сигнальный домен, например, домен CD3-дзета, где антигенсвязывающий компонент не содержит переключающий домен или не содержит переключающий домен, который димеризуется с переключающим доменом во внутриклеточном сигнальном компоненте. В одном варианте осуществления антигенсвязывающий компонент не содержит костимулирующий сигнальный домен. В одном варианте осуществления внутриклеточный сигнальный компонент содержит переключающий домен из гомодимеризационного переключателя. В одном варианте осуществления внутриклеточный сигнальный компонент содержит первый переключающий домен из гетеродимеризационного переключателя, и RCAR содержит второй внутриклеточный сигнальный компонент, который содержит второй переключающий домен из гетеродимеризационного переключателя. В таких вариантах осуществления второй внутриклеточный сигнальный компонент содержит те же внутриклеточные сигнальные домены, что и внутриклеточный сигнальный компонент. В одном варианте осуществления димеризационный переключатель является внутриклеточным. В одном варианте осуществления димеризационный переключатель является внеклеточным.
В любой из конфигураций RCAR, описанных в данном документе, первый и второй переключающие домены содержат переключатель на основе FKBP-FRB, как описано в данном документе.
Также в данном документе предусмотрены клетки, содержащие RCAR, описанный в данном документе. Любая клетка, сконструированная таким образом, что она экспрессирует RCAR, может применяться в качестве RCARX-клетки. В одном варианте осуществления RCARX-клетка представляет собой T-клетку и называется RCAR-T-клеткой. В одном варианте осуществления RCARX-клетка представляет собой NK-клетку и называется RCAR-N-клеткой.
Также в данном документе предусмотрены нуклеиновые кислоты и векторы, содержащие последовательности, кодирующие RCAR. Последовательности, кодирующие различные составляющие RCAR, могут быть расположены в одной и той же молекуле нуклеиновой кислоты, например, в одних и тех же плазмиде или векторе, например, в вирусном векторе, например, лентивирусном векторе. В одном варианте осуществления (i) последовательность, кодирующая антигенсвязывающий компонент, и (ii) последовательность, кодирующая внутриклеточный сигнальный компонент, могут находиться в одной и той же нуклеиновой кислоте, например, векторе. Продуцирование соответствующих белков может быть достигнуто, например, путем применения отдельных промоторов или путем применения продукта бицистронной транскрипции (что может приводить к продуцированию двух белков путем расщепления одного продукта трансляции или путем трансляции двух отдельных белковых продуктов). В одном варианте осуществления последовательность, кодирующая расщепляемый пептид, например, последовательность P2A или F2А, располагается между (i) и (ii). В одном варианте осуществления последовательность, кодирующая IRES, например, IRES EMCV или EV71, располагается между (i) и (ii). В этих вариантах осуществления (i) и (ii) транскрибируются в виде одной РНК. В одном варианте осуществления первый промотор функционально связан с (i), и второй промотор функционально связан с (ii), так что (i) и (ii) транскрибируются в виде отдельных мРНК.
В качестве альтернативы последовательности, кодирующие различные составляющие RCAR, могут быть расположены в разных молекулах нуклеиновой кислоты, например, в разных плазмидах или векторах, например, в вирусных векторах, например, в лентивирусных векторах. Например, (i) последовательность, кодирующая антигенсвязывающий компонент, может находиться в первой нуклеиновой кислоте, например, в первом векторе, и (ii) последовательность, кодирующая внутриклеточный сигнальный компонент, может находиться во второй нуклеиновой кислоте, например, во втором векторе.
Димеризационные переключатели
Димеризационные переключатели могут быть нековалентными или ковалентными. В нековалентном димеризационном переключателе димеризующая молекула способствует нековалентному взаимодействию между переключающими доменами. В ковалентном димеризационном переключателе димеризующая молекула способствует ковалентному взаимодействию между переключающими доменами.
В одном варианте осуществления RCAR содержит димеризационный переключатель на основе FKBP/FRAP или FKBP/FRB. FKBP12 (FKBP или FK506-связывающий белок) является широко распространенным цитоплазматическим белком, который служит в качестве исходной внутриклеточной мишени для рапамицина - природного продукта, представляющего собой иммуносупрессивное лекарственное средство. Рапамицин связывается с FKBP и с большим гомологом PI3K, представляющим собой FRAP (RAFT, mTOR). FRB является частью FRAP из 93 аминокислот, которая является достаточной для связывания комплекса FKBP-рапамицин (Chen, J., Zheng, X. F., Brown, E. J. & Schreiber, S. L. (1995) Identification of an 11-kDa FKBP12-rapamycin-binding domain within the 289-kDa FKBP12-rapamycin-associated protein and characterization of a critical serine residue. Proc Natl Acad Sci U S A 92: 4947-51).
В ряде вариантов осуществления для переключателя на основе FKBP/FRAP, например, на основе FKBP/FRB, может применяться димеризующая молекула, например, рапамицин или аналог рапамицина.
Иллюстративная аминокислотная последовательность FKBP является следующей:
D V P D Y A S L G G P S S P K K K R K V S R G V Q V E T I S P G D G R T F P K R G Q T C V V H Y T G M L E D G K K F D S S R D R N K P F K F M L G K Q E V I R G W E E G V A Q M S V G Q R A K L T I S P D Y A Y G A T G H P G I I P P H A T L V F D V E L L K L E T S Y (SEQ ID NO: 275)
В ряде вариантов осуществления переключающий домен на основе FKBP может содержать фрагмент FKBP, обладающий способностью связываться с FRB или его фрагментом или аналогом в присутствии рапамицина или рапалога. В одном варианте осуществления переключающий домен на основе FKBP содержит следующую аминокислотную последовательность:
V Q V E T I S P G D G R T F P K R G Q T C V V H Y T G M L E D G K K F D S S R D R N K P F K F M L G K Q E V I R G W E E G V A Q M S V G Q R A K L T I S P D Y A Y G A T G H P G I I P P H A T L V F D V E L L K L E T S (SEQ ID NO: 276)
Аминокислотная последовательность FRB является следующей:
ILWHEMWHEG LEEASRLYFG ERNVKGMFEV LEPLHAMMER GPQTLKETSF NQAYGRDLME AQEWCRKYMK SGNVKDLTQA WDLYYHVFRR ISK (SEQ ID NO: 277)
Используемый в данном документе термин "переключатель на основе FKBP/FRAP, например, на основе FKBP/FRB" относится к димеризационному переключателю, содержащему первый переключающий домен, который содержит фрагмент FKBP или его аналог, обладающий способностью связываться с FRB или его фрагментом или аналогом в присутствии рапамицина или рапалога, например, RAD001, и характеризующийся по меньшей мере 70, 75, 80, 85, 90, 95, 96, 97, 98 или 99% идентичностью с последовательностью FKBP под SEQ ID NO: 275 или 276 или отличающийся не более чем 30, 25, 20, 15, 10, 5, 4, 3, 2 или 1 аминокислотным остатком от нее; и второй переключающий домен, который содержит фрагмент FKBP или его аналог, обладающий способностью связываться с FRB или его фрагментом или аналогом в присутствии рапамицина или рапалога и характеризующийся по меньшей мере 70, 75, 80, 85, 90, 95, 96, 97, 98 или 99% идентичностью с последовательностью FRB под SEQ ID NO: 277 или отличающийся не более чем 30, 25, 20, 15, 10, 5, 4, 3, 2 или 1 аминокислотным остатком от нее. В одном варианте осуществления RCAR, описанный в данном документе, содержит один переключающий домен, содержащий аминокислотные остатки, раскрытые под SEQ ID NO: 275 (или SEQ ID NO: 276), и один переключающий домен, содержащий аминокислотные остатки, раскрытые под SEQ ID NO: 277.
В ряде вариантов осуществления димеризационный переключатель на основе FKBP/FRB содержит модифицированный переключающий домен на основе FRB, который демонстрирует измененное, например, улучшенное, образование комплекса между переключающим доменом на основе FRB, например, модифицированным переключающим доменом на основе FRB, переключающим доменом на основе FKBP и димеризующей молекулой, например, рапамицином или рапалогом, например, RAD001. В одном варианте осуществления модифицированный переключающий домен на основе FRB содержит одну или несколько мутаций, например, 2, 3, 4, 5, 6, 7, 8, 9, 10 или больше, выбранных из мутаций в аминокислотных положениях L2031, E2032, S2035, R2036, F2039, G2040, T2098, W2101, D2102, Y2105 и F2108, где аминокислота дикого типа в результате мутации заменяется любой другой встречающейся в природе аминокислотой. В одном варианте осуществления мутантный FRB содержит мутацию в E2032, где E2032 в результате мутации заменяется фенилаланином (E2032F), метионином (E2032M), аргинином (E2032R), валином (E2032V), тирозином (E2032Y), изолейцином (E2032I), например, в SEQ ID NO: 278, или лейцином (E2032L), например, в SEQ ID NO: 279. В одном варианте осуществления мутантный FRB содержит мутацию в T2098, где T2098 в результате мутации заменяется фенилаланином (T2098F) или лейцином (T2098L), например, в SEQ ID NO: 280. В одном варианте осуществления мутантный FRB содержит мутацию в E2032 и в T2098, где E2032 в результате мутации заменяется любой аминокислотой, и где T2098 в результате мутации заменяется любой аминокислотой, например, в SEQ ID NO: 281. В одном варианте осуществления мутантный FRB содержит мутацию E2032I и T2098L, например, в SEQ ID NO: 282. В одном варианте осуществления мутантный FRB содержит мутацию E2032L и T2098L, например, в SEQ ID NO: 283.
E2032L, T2098L
Другие подходящие димеризационные переключатели включают димеризационный переключатель на основе GyrB-GyrB, димеризационный переключатель на основе гиббереллина, димеризационный переключатель на основе метки/связывающего средства и димеризационный переключатель на основе галогеновой метки/SNAP-метки. В соответствии с указаниями, представленными в данном документе, такие переключатели и подходящие димеризующие молекулы будут очевидны специалисту средней квалификации.
Димеризующая молекула
Ассоциации между переключающими доменами способствует димеризующая молекула. В присутствии димеризующей молекулы взаимодействие или ассоциация между переключающими доменами обеспечивает передачу сигнала между полипептидом, ассоциированным, например, слитым, с первым переключающим доменом, и полипептидом, ассоциированным, например, слитым, со вторым переключающим доменом. В присутствии неограничивающих уровней димеризующей молекулы передача сигнала усиливается в 1,1, 1,2, 1,3, 1,4, 1,5, 1,6, 1,7, 1,8, 1,9, 2, 5, 10, 50, 100 раз, например, при измерении в системе, описанной в данном документе.
Рапамицин и аналоги рапамицина (иногда называемые рапалогами), например, RAD001, могут применяться в качестве димеризующих молекул в димеризационном переключателе на основе FKBP/FRB, описанном в данном документе. В одном варианте осуществления димеризующая молекула может быть выбрана из рапамицина (сиролимуса), RAD001 (эверолимуса), зотаролимуса, темсиролимуса, AP-23573 (ридафоролимуса), биолимуса и AP21967. Дополнительные аналоги рапамицина, подходящие для применения с димеризационными переключателями на основе FKBP/FRB, далее описываются в разделе под названием "Виды комбинированной терапии" или в подразделе под названием "Комбинация с низкой дозой, усиливающей иммунный ответ, ингибитора mTOR".
Разделенный CAR
В некоторых вариантах осуществления в клетке, экспрессирующей CAR, используется разделенный CAR. Подход с разделенным CAR более подробно описан в публикациях WO2014/055442 и WO2014/055657, включенных в данный документ посредством ссылки. Вкратце, система разделенного CAR содержит клетку, экспрессирующую первый CAR, имеющий первый антигенсвязывающий домен и костимулирующий домен (например, 4-1BB), и эта клетка также экспрессирует второй CAR, имеющий второй антигенсвязывающий домен и внутриклеточный сигнальный домен (например, CD3-дзета). Когда клетка встречается с первым антигеном, то активируется костимулирующий домен, и клетка пролиферирует. Когда клетка встречается со вторым антигеном, то активируется внутриклеточный сигнальный домен, и запускается активность уничтожения клеток. Таким образом, клетка, экспрессирующая CAR, полностью активируется только в присутствии обоих антигенов. В ряде вариантов осуществления первый антигенсвязывающий домен распознает BCMA, например, содержит антигенсвязывающий домен, описанный в данном документе, и второй антигенсвязывающий домен распознает антиген, экспрессируемый на клетках острого миелоидного лейкоза, например, CD123, CLL-1, CD34, FLT3 или рецептор фолиевой кислоты бета. В ряде вариантов осуществления первый антигенсвязывающий домен распознает BCMA, например, содержит антигенсвязывающий домен, описанный в данном документе, и второй антигенсвязывающий домен распознает антиген, экспрессируемый на B-клетках, например, CD10, CD19, CD20, CD22, CD34, CD123, FLT-3, ROR1, CD79b, CD179b или CD79a.
Совместная экспрессия CAR с другими молекулами или средствами
Совместная экспрессия второго CAR
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, может дополнительно содержать второй CAR, например, второй CAR, который содержит другой антигенсвязывающий домен, например, для той же мишени (например, CD19) или другой мишени (например, для мишени, отличной от CD19, например, мишени, описанной в данном документе). В некоторых вариантах осуществления клетка, экспрессирующая CAR, содержит первый CAR, который нацеливается на первый антиген и содержит внутриклеточный сигнальный домен, имеющий костимулирующий сигнальный домен, но не первичный сигнальный домен, и второй CAR, который нацеливается на второй, другой, антиген и содержит внутриклеточный сигнальный домен, имеющий первичный сигнальный домен, но не костимулирующий сигнальный домен. Размещение костимулирующего сигнального домена, например, 4-1BB, CD28, CD27, OX-40 или ICOS, в первом CAR и первичного сигнального домена, например, CD3-дзета, во втором CAR может ограничивать активность CAR клетками, в которых экспрессируются обе мишени. В некоторых вариантах осуществления клетка, экспрессирующая CAR, содержит первый CAR, который содержит антигенсвязывающий домен, трансмембранный домен и костимулирующий домен, и второй CAR, который нацеливается на другой антиген и содержит антигенсвязывающий домен, трансмембранный домен и первичный сигнальный домен. В некоторых вариантах осуществления клетка, экспрессирующая CAR, содержит первый CAR, который содержит антигенсвязывающий домен, трансмембранный домен и первичный сигнальный домен, и второй CAR, который нацеливается на другой антиген и содержит антигенсвязывающий домен для антигена, трансмембранный домен и костимулирующий сигнальный домен.
В некоторых вариантах осуществления клетка, экспрессирующая CAR, содержит XCAR, описанный в данном документе, и ингибирующий CAR. В некоторых вариантах осуществления ингибирующий CAR содержит антигенсвязывающий домен, который связывается с антигеном, обнаруживаемым на нормальных клетках, но не на раковых клетках, например, на нормальных клетках, которые также экспрессируют X. В некоторых вариантах осуществления ингибирующий CAR содержит антигенсвязывающий домен, трансмембранный домен и внутриклеточный домен ингибирующей молекулы. Например, внутриклеточный домен ингибирующего CAR может представлять собой внутриклеточный домен PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулы MHC I класса, молекулы MHC II класса, GAL9, аденозина и TGF (например, TGF-бета).
В некоторых вариантах осуществления в случае, если клетка, экспрессирующая CAR, содержит два или более различных CAR, то антигенсвязывающие домены различных CAR могут быть такими, что антигенсвязывающие домены не взаимодействуют друг с другом. Например, клетка, экспрессирующая первый и второй CAR, может иметь антигенсвязывающий домен первого CAR, например, в качестве фрагмента, например, scFv, который не ассоциирует с антигенсвязывающим доменом второго CAR, например, антигенсвязывающий домен второго CAR представляет собой VHH.
В некоторых вариантах осуществления антигенсвязывающий домен содержит однодоменные антигенсвязывающие (SDAB) молекулы, в том числе молекулы, в которых определяющие комплементарность области являются частью однодоменного полипептида. Примеры включают без ограничения вариабельные домены тяжелых цепей, связывающие молекулы, в природных условиях лишенные легких цепей, отдельные домены, полученные из традиционных 4-цепочечных антител, сконструированные домены и однодоменные каркасные структуры, отличные от полученных из антител. Молекулы SDAB могут являться любыми молекулами, известными из уровня техники, или любыми однодоменными молекулами, которые будут известны в будущем. Молекулы SDAB могут быть получены из любых видов, в том числе без ограничения из мыши, человека, верблюда, ламы, миноги, рыбы, акулы, козы, кролика и быка. Этот термин также охватывает встречающиеся в природе молекулы однодоменных антител из видов, отличных от Camelidae и акул.
В некоторых вариантах осуществления молекула SDAB может быть получена из вариабельной области иммуноглобулина, обнаруживаемого у рыб, как, например, та, которая получена из изотипа иммуноглобулина, известного как новый антигенный рецептор (NAR), обнаруживаемого в сыворотке крови акулы. Способы получения однодоменных молекул, получаемых из вариабельной области NAR ("IgNAR"), описаны в WO 03/014161 и Streltsov (2005) Protein Sci. 14:2901-2909.
В некоторых вариантах осуществления молекула SDAB представляет собой встречающуюся в природе однодоменную антигенсвязывающую молекулу, известную как тяжелая цепь, лишенная легких цепей. Такие однодоменные молекулы раскрыты, например, в WO 9404678 и Hamers-Casterman, C. et al. (1993) Nature 363:446-448. В целях ясности этот вариабельный домен, полученный из молекулы тяжелой цепи, в природных условиях лишенной легкой цепи, именуется в данном документе как VHH или нанотело, чтобы отличать его от традиционного VH четырехцепочечных иммуноглобулинов. Такая молекула VHH может быть получена из видов Camelidae, например, из верблюда, ламы, дромадера, альпаки и гуанако. Другие виды, помимо Camelidae, могут продуцировать молекулы тяжелых цепей, в природных условиях лишенные легкой цепи; такие VHH входят в объем настоящего изобретения.
Молекулы SDAB могут быть рекомбинантными, с пересаженными CDR, гуманизированными, камелизированными, деиммунизированными и/или образованными in vitro (например, отобранными с помощью фагового дисплея).
Также было обнаружено, что в клетках, имеющих множество химерных рецепторов, встроенных в мембрану, содержащих антигенсвязывающий домен, взаимодействия между антигенсвязывающими доменами рецепторов могут быть нежелательными, например, поскольку они ингибируют способность одного или нескольких антигенсвязывающих доменов связываться с их когнатными антигенами. Соответственно, в данном документе раскрыты клетки, имеющие первый и второй не встречающиеся в природе химерные рецепторы, встроенные в мембрану, содержащие антигенсвязывающие домены, для которых сведены к минимуму такие взаимодействия. Также в данном документе раскрыты нуклеиновые кислоты, кодирующие первый и второй не встречающиеся в природе химерные рецепторы, встроенные в мембрану, содержащие антигенсвязывающие домены, для которых сведены к минимуму такие взаимодействия, а также способы получения и применения таких клеток и нуклеиновых кислот. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго не встречающихся в природе химерных рецепторов, встроенных в мембрану, содержит scFv, а другой содержит одиночный VH-домен, например, одиночный VH-домен верблюдовых, акулы или миноги или одиночный VH-домен, полученный из последовательности человека или мыши.
В некоторых вариантах осуществления композиция, описанная в данном документе, содержит первый и второй CAR, где антигенсвязывающий домен одного из первого и второго CAR не содержит вариабельный домен легкой цепи и вариабельный домен тяжелой цепи. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR представляет собой scFv, а другой не представляет собой scFv. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR содержит одиночный VH-домен, например, одиночный VH-домен верблюдовых, акулы или миноги или одиночный VH-домен, полученный из последовательности человека или мыши. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR содержит нанотело. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR содержит VHH-домен верблюдовых.
В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR содержит scFv, а другой содержит одиночный VH-домен, например, одиночный VH-домен верблюдовых, акулы или миноги или одиночный VH-домен, полученный из последовательности человека или мыши. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR содержит scFv, а другой содержит нанотело. В некоторых вариантах осуществления антигенсвязывающий домен одного из первого и второго CAR содержит scFv, а другой содержит VHH-домен верблюдовых.
В некоторых вариантах осуществления связывание антигенсвязывающего домена первого CAR, при наличии его на поверхности клетки, со своим когнатным антигеном существенно не снижается благодаря наличию второго CAR. В некоторых вариантах осуществления связывание антигенсвязывающего домена первого CAR со своим когнатным антигеном в присутствии второго CAR составляет по меньшей мере 85%, 90%, 95%, 96%, 97%, 98% или 99%, например, 85%, 90%, 95%, 96%, 97%, 98% или 99%, от связывания антигенсвязывающего домена первого CAR со своим когнатным антигеном в отсутствие второго CAR.
В некоторых вариантах осуществления антигенсвязывающие домены первого и второго CAR при наличии их на поверхности клетки ассоциируют друг с другом в меньшей степени, чем в случае, если бы они оба являлись антигенсвязывающими scFv-доменами. В некоторых вариантах осуществления антигенсвязывающие домены первого и второго CAR ассоциируют друг с другом в степени, на по меньшей мере 85%, 90%, 95%, 96%, 97%, 98% или 99% меньшей, например, на 85%, 90%, 95%, 96%, 97%, 98% или 99% меньшей, чем в случае, если бы они оба являлись антигенсвязывающими scFv-доменами.
Совместная экспрессия средства, усиливающего активность CAR
В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, может дополнительно экспрессировать другое средство, например, средство, усиливающее активность или приспособленность клетки, экспрессирующей CAR.
Например, в некоторых вариантах осуществления данное средство может представлять собой средство, ингибирующее молекулу, которая модулирует или регулирует, например, ингибирует, Т-клеточную функцию. В некоторых вариантах осуществления молекула, которая модулирует или регулирует Т-клеточную функцию, представляет собой ингибирующую молекулу. Ингибирующие молекулы, например, PD1, в некоторых вариантах осуществления могут снижать способность клеток, экспрессирующих CAR, осуществлять иммунный эффекторный ответ. Примеры ингибирующих молекул включают PD1, PD-L1, CTLA4, TIM3, LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулу MHC I класса, молекулу MHC II класса, GAL9, аденозин или TGF-бета.
В ряде вариантов осуществления средство, например, ингибирующую нуклеиновую кислоту, например, dsRNA, например, siRNA или shRNA; или, например, ингибирующие белок или систему, например, короткие палиндромные повторы, регулярно расположенные группами (CRISPR), эффекторную нуклеазу, подобную активаторам транскрипции (TALEN), или эндонуклеазу с "цинковыми пальцами" (ZFN), например, описанные в данном документе, можно применять для ингибирования экспрессии молекулы, которая модулирует или регулирует, например, ингибирует, Т-клеточную функцию в клетке, экспрессирующей CAR. В некоторых вариантах осуществления средство представляет собой shRNA, например, shRNA, описанную в данном документе. В некоторых вариантах осуществления средство, которое модулирует или регулирует, например, ингибирует, Т-клеточную функцию, ингибируется в клетке, экспрессирующей CAR. Например, молекула dsRNA, ингибирующая экспрессию молекулы, которая модулирует или регулирует, например, ингибирует, Т-клеточную функцию, связана с нуклеиновой кислотой, которая кодирует компонент, например, все компоненты, CAR.
В некоторых вариантах осуществления средство, которое ингибирует ингибирующую молекулу, содержит первый полипептид, например, ингибирующую молекулу, ассоциированный со вторым полипептидом, который обеспечивает положительный сигнал для клетки, например, внутриклеточный сигнальный домен, описанный в данном документе. В некоторых вариантах осуществления средство содержит первый полипептид, например, из ингибирующей молекулы, такой как PD1, PD-L1, CTLA4, TIM3, LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекула MHC I класса, молекула MHC II класса, GAL9, аденозин или TGF-бета или фрагмент любого из них (например, по меньшей мере часть внеклеточного домена любого из них), и второй полипептид, который представляет собой внутриклеточный сигнальный домен, описанный в данном документе (например, содержащий костимулирующий домен (например, 4-1BB, CD27 или CD28, например, описанные в данном документе) и/или первичный сигнальный домен (например, сигнальный домен CD3-дзета, описанный в данном документе). В некоторых вариантах осуществления средство содержит первый полипептид из PD1 или его фрагмента (например, по меньшей мере часть внеклеточного домена PD1) и второй полипептид, представляющий собой внутриклеточный сигнальный домен, описанный в данном документе (например, сигнальный домен CD28, описанный в данном документе, и/или сигнальный домен CD3-дзета, описанный в данном документе). PD1 является ингибирующим представителем семейства рецепторов CD28, которое также включает CD28, CTLA-4, ICOS и BTLA. PD-1 экспрессируется на активированных B-клетках, T-клетках и миелоидных клетках (Agata et al. 1996 Int. Immunol 8:765-75). Было показано, что два лиганда PD1 - PD-L1 и PD-L2 - подавляют активацию Т-клеток при связывании с PD1 (Freeman et al. 2000 J Exp Med 192:1027-34; Latchman et al. 2001 Nat Immunol 2:261-8; Carter et al. 2002 Eur J Immunol 32:634-43). PD-L1 является широко распространенным при различных формах рака у человека (Dong et al. 2003 J Mol Med 81:281-7; Blank et al. 2005 Cancer Immunol. Immunother 54:307-314; Konishi et al. 2004 Clin Cancer Res 10:5094). Иммуносупрессию можно устранить путем ингибирования локального взаимодействия PD1 с PD-L1.
В некоторых вариантах осуществления средство, содержащее внеклеточный домен (ECD) ингибирующей молекулы, например, белка 1 запрограммированной гибели клеток (PD1), может быть слито с трансмембранным доменом и внутриклеточными сигнальными доменами, такими как 4-1BB и CD3-дзета (также называется в данном документе CAR на основе PD1). В некоторых вариантах осуществления CAR на основе PD1 при применении в комбинациях с XCAR, описанным в данном документе, улучшает персистенцию Т-клетки. В некоторых вариантах осуществления CAR представляет собой CAR на основе PD1, содержащий внеклеточный домен PD1, указанный подчеркиванием в SEQ ID NO: 24. В некоторых вариантах осуществления CAR на основе PD1 содержит аминокислотную последовательность под SEQ ID NO: 24.
В некоторых вариантах осуществления CAR на основе PD1 содержит аминокислотную последовательность под SEQ ID NO: 22.
В некоторых вариантах осуществления средство содержит последовательность нуклеиновой кислоты, кодирующую CAR на основе PD1, например, CAR на основе PD1, описанный в данном документе. В некоторых вариантах осуществления последовательность нуклеиновой кислоты для CAR на основе PD1 представлена под SEQ ID NO: 23, при этом ECD PD1 подчеркнут.
В другом примере в некоторых вариантах осуществления средство, которое усиливает активность клетки, экспрессирующей CAR, может представлять собой костимулирующую молекулу или лиганд костимулирующей молекулы. Примеры костимулирующих молекул включают молекулу MHC I класса, BTLA и лиганд Toll-подобного рецептора, а также OX40, CD27, CD28, CDS, ICAM-1, LFA-1 (CD11a/CD18), ICOS (CD278) и 4-1BB (CD137). Дополнительные примеры таких костимулирующих молекул включают CDS, ICAM-1, GITR, BAFFR, HVEM (LIGHTR), SLAMF7, NKp80 (KLRF1), NKp44, NKp30, NKp46, CD160, CD19, CD4, CD8-альфа, CD8-бета, IL2R-бета, IL2R-гамма, IL7R-альфа, ITGA4, VLA1, CD49a, ITGA4, IA4, CD49D, ITGA6, VLA-6, CD49f, ITGAD, CD11d, ITGAE, CD103, ITGAL, CD11a, LFA-1, ITGAM, CD11b, ITGAX, CD11c, ITGB1, CD29, ITGB2, CD18, LFA-1, ITGB7, NKG2D, NKG2C, TNFR2, TRANCE/RANKL, DNAM1 (CD226), SLAMF4 (CD244, 2B4), CD84, CD96 (Tactile), CEACAM1, CRTAM, Ly9 (CD229), CD160 (BY55), PSGL1, CD100 (SEMA4D), CD69, SLAMF6 (NTB-A, Ly108), SLAM (SLAMF1, CD150, IPO-3), BLAME (SLAMF8), SELPLG (CD162), LTBR, LAT, GADS, SLP-76, PAG/Cbp, CD19a и лиганд, который специфично связывается с CD83, например, описанные в данном документе. Примеры лигандов костимулирующих молекул включают CD80, CD86, CD40L, ICOSL, CD70, OX40L, 4-1BBL, GITRL и LIGHT. В ряде вариантов осуществления лиганд костимулирующей молекулы является лигандом костимулирующей молекулы, отличной от костимулирующей молекулы, домен которой используется в CAR. В ряде вариантов осуществления лиганд костимулирующей молекулы является лигандом той же костимулирующей молекулы, что и костимулирующая молекула, домен которой используется в CAR. В некоторых вариантах осуществления лиганд костимулирующей молекулы представляет собой 4-1BBL. В одном варианте осуществления лиганд костимулирующей молекулы представляет собой CD80 или CD86. В некоторых вариантах осуществления лиганд костимулирующей молекулы представляет собой CD70. В ряде вариантов осуществления описанная в данном документе иммунная эффекторная клетка, экспрессирующая CAR, может быть дополнительно сконструирована таким образом, чтобы она экспрессировала одну или несколько дополнительных костимулирующих молекул или лигандов костимулирующих молекул.
Совместная экспрессия CAR с рецептором хемокина
В ряде вариантов осуществления описанная в данном документе клетка, экспрессирующая CAR, например, клетка, экспрессирующая CAR для CD19, дополнительно содержит молекулу рецептора хемокина. Трансгенная экспрессия рецепторов хемокинов CCR2b или CXCR2 в Т-клетках усиливает их миграцию к солидным опухолям, секретирующим CCL2 или CXCL1, в том числе к меланоме и нейробластоме (Craddock et al., J Immunother. 2010 Oct; 33(8):780-8 и Kershaw et al., Hum Gene Ther. 2002 Nov 1; 13(16):1971-80). Таким образом, не ограничиваясь какой-либо теорией, полагают, что рецепторы хемокинов, экспрессируемые в клетках, экспрессирующих CAR, которые распознают хемокины, секретируемые опухолями, например, солидными опухолями, могут улучшать "хоминг" клетки, экспрессирующей CAR, в опухоль, содействовать инфильтрации клетки, экспрессирующей CAR, в опухоль и усиливать противоопухолевую эффективность клетки, экспрессирующей CAR. Молекула рецептора хемокина может содержать встречающийся в природе или рекомбинантный рецептор хемокина или его хемокинсвязывающий фрагмент. Молекула рецептора хемокина, подходящая для экспрессии в клетке, экспрессирующей CAR (например, CAR-Tx), описанной в данном документе, включает рецептор хемокина CXC (например, CXCR1, CXCR2, CXCR3, CXCR4, CXCR5, CXCR6 или CXCR7), рецептор хемокина CC (например, CCR1, CCR2, CCR3, CCR4, CCR5, CCR6, CCR7, CCR8, CCR9, CCR10 или CCR11), рецептор хемокина CX3C (например, CX3CR1), рецептор хемокина XC (например, XCR1) или их хемокинсвязывающий фрагмент. В некоторых вариантах осуществления молекулу рецептора хемокина, которая должна экспрессироваться вместе с CAR, описанным в данном документе, выбирают с учетом хемокина(хемокинов), секретируемых опухолью. В некоторых вариантах осуществления описанная в данном документе клетка, экспрессирующая CAR, дополнительно содержит, например, экспрессирует, рецептор CCR2b или рецептор CXCR2. В некоторых вариантах осуществления CAR, описанный в данном документе, и молекула рецептора хемокина находятся в одном и том же векторе или находятся в двух разных векторах. В ряде вариантов осуществления, в которых CAR, описанный в данном документе, и молекула рецептора хемокина находятся в одном и том же векторе, CAR и молекула рецептора хемокина находятся под контролем двух разных промоторов или находятся под контролем одного и того же промотора.
Конструкции нуклеиновых кислот, кодирующие CAR
В настоящем изобретении также предусмотрена иммунная эффекторная клетка, например, полученная посредством способа, описанного в данном документе, которая содержит молекулу нуклеиновой кислоты, кодирующую одну или несколько конструкций CAR, описанных в данном документе. В некоторых вариантах осуществления молекула нуклеиновой кислоты представлена в виде транскрипта, представляющего собой матричную РНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты представлена в виде ДНК-конструкции.
Молекулы нуклеиновой кислоты, описанные в данном документе, могут представлять собой молекулу ДНК, молекулу РНК или их комбинацию. В некоторых вариантах осуществления молекула нуклеиновой кислоты представляет собой мРНК, кодирующую полипептид CAR, описанный в данном документе. В других вариантах осуществления молекула нуклеиновой кислоты представляет собой вектор, который содержит любую из вышеуказанных молекул нуклеиновой кислоты.
В некоторых вариантах осуществления антигенсвязывающий домен CAR по настоящему изобретению (например, scFv) кодируется молекулой нуклеиновой кислоты, последовательность которой была кодон-оптимизирована для экспрессии в клетке млекопитающего. В некоторых вариантах осуществления целая конструкция CAR по настоящему изобретению кодируется молекулой нуклеиновой кислоты, целая последовательность которой была кодон-оптимизирована для экспрессии в клетке млекопитающего. Оптимизация кодонов относится к обнаружению того, что частота встречаемости синонимичных кодонов (т. е. кодонов, кодирующих одну и ту же аминокислоту) в кодирующей ДНК смещена у разных видов. Такая вырожденность кодонов обеспечивает возможность кодирования идентичного полипептида различными нуклеотидными последовательностями. Из уровня техники известны различные способы оптимизации кодонов, и они включают, например, способы, раскрытые по меньшей мере в патентах США №№ 5786464 и 6114148.
Соответственно, в некоторых вариантах осуществления иммунная эффекторная клетка, например, полученная посредством способа, описанного в данном документе, содержит молекулу нуклеиновой кислоты, кодирующую химерный антигенный рецептор (CAR), где CAR содержит антигенсвязывающий домен, который связывается с опухолевым антигеном, описанным в данном документе, трансмембранный домен (например, трансмембранный домен, описанный в данном документе) и внутриклеточный сигнальный домен (например, внутриклеточный сигнальный домен, описанный в данном документе), содержащий стимулирующий домен, например, костимулирующий сигнальный домен (например, костимулирующий сигнальный домен, описанный в данном документе), и/или первичный сигнальный домен (например, первичный сигнальный домен, описанный в данном документе, например, дзета-цепь, описанную в данном документе).
В настоящем изобретении также предусмотрены векторы, в которые вставлена молекула нуклеиновой кислоты, кодирующая CAR, например, молекула нуклеиновой кислоты, описанная в данном документе. Векторы, полученные из ретровирусов, таких как лентивирус, являются подходящими инструментами для достижения долгосрочного переноса генов, поскольку они обеспечивают долгосрочную стабильную интеграцию трансгена и его размножение в дочерних клетках. Лентивирусные векторы обладают дополнительным преимуществом по сравнению с векторами, полученными из онкоретровирусов, таких как вирусы лейкоза мышей, заключающимся в том, что ими можно трансдуцировать непролиферирующие клетки, такие как гепатоциты. Они также обладают дополнительным преимуществом низкой иммуногенности. Ретровирусный вектор также может представлять собой, например, гамма-ретровирусный вектор. Гамма-ретровирусный вектор может содержать, например, промотор, сигнал упаковки (ψ), сайт связывания праймера (PBS), один или несколько (например, два) длинных концевых повторов (LTR) и трансген, представляющий интерес, например, ген, кодирующий CAR. В гамма-ретровирусном векторе могут отсутствовать структурные гены вирусов, такие как gag, pol и env. Иллюстративные гамма-ретровирусные векторы включают вирус лейкоза мышей (MLV), вирус некроза селезенки (SFFV) и вирус миелопролиферативной саркомы (MPSV), а также векторы, полученные из них. Другие гамма-ретровирусные векторы описаны, например, в Tobias Maetzig et al., "Gammaretroviral Vectors: Biology, Technology and Application" Viruses. 2011 Jun; 3(6): 677-713.
В некоторых вариантах осуществления вектор, содержащий нуклеиновую кислоту, кодирующую требуемый CAR, представляет собой аденовирусный вектор (A5/35). В некоторых вариантах осуществления экспрессии нуклеиновых кислот, кодирующих CAR, можно достичь с помощью транспозонов, таких как "спящая красавица", CRISPR, CAS9 и нуклеазы с "цинковыми пальцами". См. ниже June et al. 2009, Nature Reviews Immunology 9.10: 704-716, включенную в данный документ посредством ссылки.
Вкратце, экспрессия природных или синтетических нуклеиновых кислот, кодирующих CAR, как правило, достигается путем образования функциональной связи нуклеиновой кислоты, кодирующей полипептид CAR или его части, с промотором и включения конструкции в состав экспрессионного вектора. Векторы могут подходить для репликации и интеграции у эукариот. Типичные клонирующие векторы содержат терминаторы транскрипции и трансляции, последовательности инициации и промоторы, применимые для регуляции экспрессии необходимой последовательности нуклеиновой кислоты.
Нуклеиновую кислоту можно клонировать в векторы множества типов. Например, нуклеиновую кислоту можно клонировать в вектор, включающий без ограничения плазмиду, фагмиду, производное фага, вирус животных и космиду. Векторы, представляющие особый интерес, включают экспрессионные векторы, репликационные векторы, векторы для образования зондов и векторы для секвенирования.
Кроме того, экспрессионный вектор может предоставляться клетке в форме вирусного вектора. Технология вирусных векторов хорошо известна из уровня техники и описана, например, в Sambrook et al., 2012, MOLECULAR CLONING: A LABORATORY MANUAL, тома 1-4, Cold Spring Harbor Press, Нью-Йорк, и в других руководствах по вирусологии и молекулярной биологии. Вирусы, применимые в качестве векторов, включают без ограничения ретровирусы, аденовирусы, аденоассоциированные вирусы, герпесвирусы и лентивирусы. Как правило, подходящий вектор содержит точку начала репликации, функционирующую в по меньшей мере одном организме, промоторную последовательность, подходящие сайты для эндонуклеаз рестрикции и один или несколько селектируемых маркеров (например, из WO 01/96584; WO 01/29058 и патента США № 6326193).
Для переноса генов в клетки млекопитающих был разработан целый ряд систем на основе вирусов. Например, ретровирусы обеспечивают удобную платформу для систем доставки генов. Выбранный ген можно вставить в вектор и упаковать в ретровирусные частицы с помощью методик, известных из уровня техники. Затем рекомбинантный вирус можно выделить и доставить в клетки субъекта in vivo либо ex vivo. Из уровня техники известен целый ряд ретровирусных систем. В некоторых вариантах осуществления применяются аденовирусные векторы. Из уровня техники известен целый ряд аденовирусных векторов. В некоторых вариантах осуществления применяются лентивирусные векторы.
Дополнительные промоторные элементы, например, энхансеры, регулируют частоту инициации транскрипции. Как правило, они расположены в области на 30-110 п. о. выше сайта начала транскрипции, хотя было показано, что ряд промоторов также содержит функциональные элементы ниже сайта начала транскрипции. Расстояние между промоторными элементами часто является гибким, так что промоторная функция сохраняется при инверсии элементов или их перемещении друг относительно друга. В промоторе гена тимидинкиназы (tk) расстояние между промоторными элементами можно увеличить до 50 п. о., прежде чем активность начнет снижаться. В зависимости от промотора оказывается, что отдельные элементы могут функционировать для активации транскрипции совместно либо независимо. Иллюстративные промоторы включают промоторы генов IE CMV, EF-1α, убиквитина С или фосфоглицераткиназы (PGK).
Примером промотора, который способен обеспечивать экспрессию молекулы нуклеиновой кислоты, кодирующей CAR, в Т-клетке млекопитающего, является промотор EF1a. Нативный промотор EF1a управляет экспрессией альфа-субъединицы комплекса фактора элонгации-1, который отвечает за ферментативную доставку аминоацил-тРНК к рибосоме. Промотор EF1a широко использовался в экспрессионных плазмидах для млекопитающих, и было показано, что он эффективно управляет экспрессией CAR с молекул нуклеиновой кислоты, клонированных в лентивирусный вектор. См., например, Milone et al., Mol. Ther. 17(8): 1453-1464 (2009). В некоторых вариантах осуществления промотор EF1a содержит последовательность, представленную в примерах.
Другим примером промотора является последовательность немедленно-раннего промотора цитомегаловируса (CMV). Эта промоторная последовательность является последовательностью сильного конститутивного промотора, способного управлять экспрессией любой полинуклеотидной последовательности, функционально связанной с ней, на высоких уровнях. Однако можно также использовать последовательности других конститутивных промоторов, в том числе без ограничения раннего промотора вируса обезьян 40 (SV40), промотора вируса опухоли молочной железы мышей (MMTV), промотора длинного концевого повтора (LTR) вируса иммунодефицита человека (HIV), промотора MoMuLV, промотора вируса лейкоза птиц, немедленно-раннего промотора вируса Эпштейна-Барр, промотора вируса саркомы Рауса, а также промоторы генов человека, такие как, без ограничения, промотор гена актина, промотор гена миозина, промотор гена фактора элонгации-1α, промотор гена гемоглобина и промотор гена креатинкиназы. Кроме того, настоящее изобретение не должно ограничиваться использованием конститутивных промоторов. Индуцируемые промоторы также рассматриваются как часть настоящего изобретения. Использование индуцируемого промотора обеспечивает наличие молекулярного переключателя, способного включать экспрессию полинуклеотидной последовательности, с которой он функционально связан, если такая экспрессия требуется, или отключать экспрессию, если экспрессия не требуется. Примеры индуцируемых промоторов включают без ограничения металлотионеин-индуцируемый промотор, глюкокортикоид-индуцируемый промотор, прогестерон-индуцируемый промотор и тетрациклин-индуцируемый промотор.
Другим примером промотора является промотор гена фосфоглицераткиназы (PGK). В ряде вариантов осуществления может быть необходим усеченный промотор гена PGK (например, промотор гена PGK с одной или несколькими, например, 1, 2, 5, 10, 100, 200, 300 или 400, делециями нуклеотидов по сравнению с последовательностью промотора гена PGK дикого типа).
Нуклеотидные последовательности иллюстративных промоторов гена PGK представлены ниже.
Промотор гена PGK WT:
ACCCCTCTCTCCAGCCACTAAGCCAGTTGCTCCCTCGGCTGACGGCTGCACGCGAGGCCTCCGAACGTCTTACGCCTTGTGGCGCGCCCGTCCTTGTCCCGGGTGTGATGGCGGGGTGTGGGGCGGAGGGCGTGGCGGGGAAGGGCCGGCGACGAGAGCCGCGCGGGACGACTCGTCGGCGATAACCGGTGTCGGGTAGCGCCAGCCGCGCGACGGTAACGAGGGACCGCGACAGGCAGACGCTCCCATGATCACTCTGCACGCCGAAGGCAAATAGTGCAGGCCGTGCGGCGCTTGGCGTTCCTTGGAAGGGCTGAATCCCCGCCTCGTCCTTCGCAGCGGCCCCCCGGGTGTTCCCATCGCCGCTTCTAGGCCCACTGCGACGCTTGCCTGCACTTCTTACACGCTCTGGGTCCCAGCCGCGGCGACGCAAAGGGCCTTGGTGCGGGTCTCGTCGGCGCAGGGACGCGTTTGGGTCCCGACGGAACCTTTTCCGCGTTGGGGTTGGGGCACCATAAGCT (SEQ ID NO: 190)
Иллюстративные усеченные промоторы гена PGK:
PGK100:
ACCCCTCTCTCCAGCCACTAAGCCAGTTGCTCCCTCGGCTGACGGCTGCACGCGAGGCCTCCGAACGTCTTACGCCTTGTGGCGCGCCCGTCCTTGTCCCGGGTGTGATGGCGGGGTG (SEQ ID NO: 198)
PGK200:
ACCCCTCTCTCCAGCCACTAAGCCAGTTGCTCCCTCGGCTGACGGCTGCACGCGAGGCCTCCGAACGTCTTACGCCTTGTGGCGCGCCCGTCCTTGTCCCGGGTGTGATGGCGGGGTGTGGGGCGGAGGGCGTGGCGGGGAAGGGCCGGCGACGAGAGCCGCGCGGGACGACTCGTCGGCGATAACCGGTGTCGGGTAGCGCCAGCCGCGCGACGGTAACG (SEQ ID NO: 191)
PGK300:
ACCCCTCTCTCCAGCCACTAAGCCAGTTGCTCCCTCGGCTGACGGCTGCACGCGAGGCCTCCGAACGTCTTACGCCTTGTGGCGCGCCCGTCCTTGTCCCGGGTGTGATGGCGGGGTGTGGGGCGGAGGGCGTGGCGGGGAAGGGCCGGCGACGAGAGCCGCGCGGGACGACTCGTCGGCGATAACCGGTGTCGGGTAGCGCCAGCCGCGCGACGGTAACGAGGGACCGCGACAGGCAGACGCTCCCATGATCACTCTGCACGCCGAAGGCAAATAGTGCAGGCCGTGCGGCGCTTGGCGTTCCTTGGAAGGGCTGAATCCCCG (SEQ ID NO: 192)
PGK400:
ACCCCTCTCTCCAGCCACTAAGCCAGTTGCTCCCTCGGCTGACGGCTGCACGCGAGGCCTCCGAACGTCTTACGCCTTGTGGCGCGCCCGTCCTTGTCCCGGGTGTGATGGCGGGGTGTGGGGCGGAGGGCGTGGCGGGGAAGGGCCGGCGACGAGAGCCGCGCGGGACGACTCGTCGGCGATAACCGGTGTCGGGTAGCGCCAGCCGCGCGACGGTAACGAGGGACCGCGACAGGCAGACGCTCCCATGATCACTCTGCACGCCGAAGGCAAATAGTGCAGGCCGTGCGGCGCTTGGCGTTCCTTGGAAGGGCTGAATCCCCGCCTCGTCCTTCGCAGCGGCCCCCCGGGTGTTCCCATCGCCGCTTCTAGGCCCACTGCGACGCTTGCCTGCACTTCTTACACGCTCTGGGTCCCAGCCG (SEQ ID NO: 193)
Вектор также может содержать, например, сигнальную последовательность, содействующую секреции, сигнал полиаденилирования и терминатор транскрипции (например, из гена бычьего гормона роста (BGH)), элемент, обеспечивающий возможность эписомальной репликации и репликации у прокариот (например, точку начала репликации SV40 и ColE1 или другие, известные из уровня техники), и/или элементы для обеспечения возможности осуществления отбора (например, ген устойчивости к ампициллину и/или маркер устойчивости к зеоцину).
Для оценки экспрессии полипептида CAR или его частей экспрессионный вектор, который должен быть введен в клетку, также может содержать селектируемый маркерный ген или репортерный ген либо их оба для облегчения идентификации и отбора экспрессирующих клеток из популяции клеток, в отношении которых стремились провести трансфекцию или инфицирование посредством вирусных векторов. В некоторых вариантах осуществления селектируемый маркер может переноситься на отдельном фрагменте ДНК и использоваться в процедуре котрансфекции. Как селектируемые маркеры, так и репортерные гены могут быть фланкированы соответствующими регуляторными последовательностями для обеспечения экспрессии в клетках-хозяевах. Применимые селектируемые маркеры включают, например, гены устойчивости к антибиотикам, такие как neo и т. п.
Репортерные гены используют для идентификации потенциально трансфицированных клеток и для оценивания функциональных свойств регуляторных последовательностей. Как правило, репортерный ген представляет собой ген, который отсутствует или не экспрессируется в организме- или ткани-реципиенте и который кодирует полипептид, экспрессия которого проявляется в некотором легко выявляемом свойстве, например, ферментативной активности. Экспрессию репортерного гена анализируют в подходящее время после введения ДНК в клетки-реципиенты. Подходящие репортерные гены могут включать гены, кодирующие люциферазу, бета-галактозидазу, хлорамфениколацетилтрансферазу, секретируемую щелочную фосфатазу, или ген зеленого флуоресцентного белка (например, Ui-Tei et al., 2000 FEBS Letters 479: 79-82). Подходящие системы экспрессии хорошо известны и могут быть получены с помощью известных методик или приобретены коммерческим путем. Как правило, конструкцию с минимальной 5'-фланкирующей областью, демонстрирующую наивысший уровень экспрессии репортерного гена, идентифицируют как промотор. Такие промоторные области могут быть связаны с репортерным геном и использоваться для оценивания средств в отношении способности к модулированию транскрипции, управляемой промотором.
В ряде вариантов осуществления вектор может содержать две или более последовательности нуклеиновой кислоты, кодирующие CAR, например, CAR, описанный в данном документе, например, CAR для CD19, и второй CAR, например, ингибирующий CAR или CAR, который специфично связывается с антигеном, отличным от CD19. В таких вариантах осуществления две или более последовательности нуклеиновой кислоты, кодирующие CAR, кодируются одной молекулой нуклеиновой кислоты в одной и той же рамке считывания и в виде одной полипептидной цепи. В некоторых вариантах осуществления два или более CAR могут, например, быть разделены одним или несколькими сайтами расщепления пептидов (например, сайтом саморасщепления или субстратом для внутриклеточной протеазы). Примеры сайтов расщепления пептидов включают сайты T2A, P2A, E2A или F2A.
Способы введения генов в клетку и обеспечения их экспрессии в ней известны из уровня техники. Что касается экспрессионного вектора, то вектор можно легко ввести в клетку-хозяина, например, клетку млекопитающего, бактерии, дрожжей или насекомого, любым способом, например, способом, известным из уровня техники. Например, экспрессионный вектор можно перенести в клетку-хозяина физическим, химическим или биологическим способом.
Физические способы введения полинуклеотида в клетку-хозяина включают осаждение фосфатом кальция, липофекцию, бомбардировку частицами, микроинъекцию, электропорацию и т. п. Способы получения клеток, содержащих векторы и/или экзогенные нуклеиновые кислоты, хорошо известны из уровня техники. См., например, Sambrook et al., 2012, MOLECULAR CLONING: A LABORATORY MANUAL, тома 1-4, Cold Spring Harbor Press, Нью-Йорк. Подходящим способом введения полинуклеотида в клетку-хозяина является трансфекция с использованием фосфата кальция.
Биологические способы введения полинуклеотида, представляющего интерес, в клетку-хозяина включают использование векторов на основе ДНК и РНК. Вирусные векторы, и особенно ретровирусные векторы, стали наиболее широко применяемым способом вставки генов в клетки млекопитающего, например, человека. Другие вирусные векторы могут быть получены из лентивируса, поксвирусов, вируса простого герпеса I типа, аденовирусов и аденоассоциированных вирусов и т. п. См., например, патенты США №№ 5350674 и 5585362.
Химические средства для введения полинуклеотида в клетку-хозяина включают коллоидные дисперсные системы, такие как макромолекулярные комплексы, нанокапсулы, микросферы, гранулы и липидные системы, в том числе эмульсии типа "масло в воде", мицеллы, смешанные мицеллы и липосомы. Иллюстративной коллоидной системой для применения в качестве средства для доставки in vitro и in vivo является липосома (например, искусственная мембранная везикула). Из уровня техники доступны другие способы целенаправленной доставки нуклеиновых кислот, такие как доставка полинуклеотидов с помощью нацеливающихся наночастиц или другой подходящей системы доставки субмикронного размера.
В случае использования системы доставки, отличной от вирусной, иллюстративным средством для доставки является липосома. Для введения нуклеиновых кислот в клетку-хозяина (in vitro, ex vivo или in vivo) предполагается применение липидных составов. В некоторых вариантах осуществления нуклеиновая кислота может быть ассоциирована с липидом. Нуклеиновая кислота, ассоциированная с липидом, может быть инкапсулированной в водной внутренней части липосомы, помещенной в липидный бислой липосомы, присоединенной к липосоме посредством связывающей молекулы, которая ассоциирована как с липосомой, так и с олигонуклеотидом, захваченной в липосому, образовывать комплекс с липосомой, быть диспергированной в растворе, содержащем липид, смешанной с липидом, объединенной с липидом, содержаться в виде суспензии в липиде, содержаться в мицелле или образовывать комплекс с ней или иным образом быть ассоциированной с липидом. Композиции для ассоциации на основе липидов, липидов/ДНК или липидов/экспрессионного вектора не ограничиваются какой-либо конкретной структурой в растворе. Например, они могут присутствовать в бислойной структуре, в виде мицелл или в "сжатой" структуре. Они также могут быть просто помещены в раствор, при этом по возможности образуют агрегаты, которые не являются однородными по размеру или форме. Липиды представляют собой жирные вещества, которые могут являться встречающимися в природе или синтетическими липидами. Например, липиды включают жировые капли, которые встречаются в природе в цитоплазме, а также класс соединений, который содержит длинноцепочечные алифатические углеводороды и их производные, такие как жирные кислоты, спирты, амины, аминоспирты и альдегиды.
Подходящие для применения липиды можно получить из коммерческих источников. Например, димиристилфосфатидилхолин ("DMPC") можно получить от Sigma, Сент-Луис, Миссури; дицетилфосфат ("DCP") можно получить от K & K Laboratories (Плейнвью, Нью-Йорк); холестерин ("Choi") можно получить от Calbiochem-Behring; димиристилфосфатидилглицерин ("DMPG") и другие липиды можно получить от Avanti Polar Lipids, Inc. (Бирмингем, Алабама). Исходные растворы липидов в хлороформе или хлороформе/метаноле можно хранить при температуре приблизительно -20°C. Хлороформ используется в качестве единственного растворителя, поскольку он испаряется легче, чем метанол. "Липосома" является общим термином, охватывающим различные одно- и мультиламеллярные липидные носители, образованные путем формирования замкнутых липидных бислоев или агрегатов. Липосомы можно охарактеризовать как имеющие везикулярные структуры с фосфолипидной бислойной мембраной и внутренней водной средой. Мультиламеллярные липосомы имеют несколько липидных слоев, разделенных водной средой. Они образуются самопроизвольно при суспендировании фосфолипидов в избытке водного раствора. Липидные компоненты подвергаются самореорганизации перед образованием замкнутых структур и захватывают воду и растворенные вещества между липидными бислоями (Ghosh et al., 1991, Glycobiology 5: 505-10). Однако также охватываются композиции, которые в растворе имеют структуры, отличные от нормальной везикулярной структуры. Например, липиды могут принимать вид мицеллярной структуры или просто существовать в виде неоднородных агрегатов липидных молекул. Также предполагаются комплексы липофектамин-нуклеиновая кислота.
Чтобы подтвердить присутствие последовательности рекомбинантной нуклеиновой кислоты в клетке-хозяине, можно проводить различные анализы, независимо от способа, применяемого для введения экзогенных нуклеиновых кислот в клетку-хозяина, или иного воздействия ингибитора по настоящему изобретению на клетку. Такие анализы включают, например, "молекулярно-биологические" анализы, хорошо известные специалистам в данной области, такие как саузерн- и нозерн-блоттинг, ОТ-ПЦР и ПЦР; "биохимические" анализы, такие как выявление присутствия или отсутствия конкретного пептида, например, с помощью иммунологических способов (ELISA и вестерн-блоттинга) или с помощью описанных в данном документе анализов, для идентификации средств, находящихся в пределах объема настоящего изобретения.
Трансфекция РНК
В данном документе раскрыты способы получения РНК, транскрибированной in vitro, кодирующей CAR. РНК, кодирующая CAR, и способы ее применения описаны, например, в абзацах 553-570 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Иммунная эффектoрная клетка может содержать CAR, кодируемый матричной РНК (мРНК). В некоторых вариантах осуществления мРНК, кодирующую CAR, описанную в данном документе, вводят в иммунную эффекторную клетку, например, полученную посредством способа, описанного в данном документе, для получения клетки, экспрессирующей CAR.
В некоторых вариантах осуществления РНК, транскрибированную in vitro, кодирующую CAR, можно вводить в клетку в форме транзиентной трансфекции. РНК получают путем транскрипции in vitro с использованием матрицы, полученной посредством полимеразной цепной реакции (ПЦР). ДНК, представляющая интерес, из любого источника может быть непосредственно преобразована с помощью ПЦР в матрицу для синтеза мРНК in vitro с использованием соответствующих праймеров и РНК-полимеразы. Источником ДНК может быть, например, геномная ДНК, плазмидная ДНК, фаговая ДНК, кДНК, синтетическая последовательность ДНК или любой другой подходящий источник ДНК. Требуемая матрица для транскрипции in vitro представляет собой CAR, описанный в данном документе. Например, матрица для РНК, кодирующей CAR, содержит внеклеточную область, содержащую одноцепочечный вариабельный домен антитела к опухолеассоциированному антигену, описанный в данном документе; шарнирную область (например, шарнирную область, описанную в данном документе), трансмембранный домен (например, трансмембранный домен, описанный в данном документе, такой как трансмембранный домен CD8a) и цитоплазматическую область, которая содержит внутриклеточный сигнальный домен, например, внутриклеточный сигнальный домен, описанный в данном документе, например, содержащий сигнальный домен CD3-дзета и сигнальный домен 4-1BB.
В некоторых вариантах осуществления ДНК, подлежащая применению в ПЦР, содержит открытую рамку считывания. ДНК может быть получена из встречающейся в природе последовательности ДНК из генома организма. В некоторых вариантах осуществления нуклеиновая кислота может содержать некоторые или все из 5'- и/или 3'-нетранслируемых областей (UTR). Нуклеиновая кислота может содержать экзоны и интроны. В некоторых вариантах осуществления ДНК, подлежащая применению в ПЦР, представляет собой последовательность нуклеиновой кислоты человека. В некоторых вариантах осуществления ДНК, подлежащая применению в ПЦР, представляет собой последовательность нуклеиновой кислоты человека, содержащую 5'- и 3'-UTR. В качестве альтернативы ДНК может представлять собой искусственную последовательность ДНК, которая обычно не экспрессируется у встречающегося в природе организма. Иллюстративная искусственная последовательность ДНК представляет собой последовательность, которая содержит части генов, лигированные друг с другом с образованием открытой рамки считывания, которая кодирует слитый белок. Части ДНК, лигированные друг с другом, могут быть получены из одного организма или из более чем одного организма.
ПЦР применяют для получения матрицы, предназначенной для транскрипции in vitro мРНК, которая применяется для трансфекции. Способы осуществления ПЦР хорошо известны из уровня техники. Праймеры для применения в ПЦР разрабатывают таким образом, чтобы они имели области, по существу комплементарные областям ДНК, подлежащей применению в качестве матрицы для ПЦР. Используемый в данном документе термин "по существу комплементарные" относится к последовательностям нуклеотидов, где большинство или все основания в последовательности праймера являются комплементарными, или одно или несколько оснований являются некомплементарными или несовпадающими. По существу комплементарные последовательности способны отжигаться или гибридизироваться с предполагаемой ДНК-мишенью в условиях отжига, используемых для ПЦР. Праймеры можно разрабатывать таким образом, чтобы они были по существу комплементарными любой части ДНК-матрицы. Например, праймеры можно разрабатывать таким образом, чтобы обеспечить амплификацию части нуклеиновой кислоты, которая обычно транскрибируется в клетках (открытой рамки считывания), включающей 5'- и 3'-UTR. Праймеры также можно разрабатывать таким образом, чтобы обеспечить амплификацию части нуклеиновой кислоты, которая кодирует конкретный домен, представляющий интерес. В некоторых вариантах осуществления праймеры разработаны таким образом, что они обеспечивают амплификацию кодирующей области кДНК человека, в том числе 5'- и 3'-UTR целиком или их частей. Праймеры, применимые для ПЦР, могут быть получены посредством способов синтеза, которые хорошо известны из уровня техники. "Прямые праймеры" представляют собой праймеры, которые содержат область из нуклеотидов, по существу комплементарных нуклеотидам в ДНК-матрице, которые расположены выше последовательности ДНК, подлежащей амплификации. "Вышерасположенный" используется в данном документе для обозначения местоположения в 5'-направлении от последовательности ДНК, подлежащей амплификации, относительно кодирующей нити. "Обратные праймеры" представляют собой праймеры, которые содержат области из нуклеотидов, по существу комплементарных нуклеотидам в двухнитевой ДНК-матрице, которые расположены ниже последовательности ДНК, подлежащей амплификации. "Нижерасположенный" используется в данном документе для обозначения местоположения в 3'-направлении от последовательности ДНК, подлежащей амплификации, относительно кодирующей нити.
В способах, раскрытых в данном документе, может применяться любая ДНК-полимераза, применимая для ПЦР. Реагенты и полимераза являются коммерчески доступными из ряда источников.
Также можно использовать химические структуры, которые могут способствовать стабильности и/или эффективности трансляции. РНК в ряде вариантов осуществления содержит 5'- и 3'-UTR. В некоторых вариантах осуществления длина 5'-UTR составляет от одного до 3000 нуклеотидов. Длину последовательностей 5'- и 3'-UTR, которые подлежат добавлению к кодирующей области, можно изменять с помощью различных способов, включая без ограничения разработку праймеров для ПЦР, которые отжигаются с различными областями UTR. Используя данный подход, специалист средней квалификации в данной области может модифицировать длину 5'- и 3'-UTR, необходимую для достижения оптимальной эффективности трансляции после трансфекции транскрибированной РНК.
5'- и 3'-UTR могут представлять собой встречающиеся в природе эндогенные 5'- и 3'-UTR для нуклеиновой кислоты, представляющей интерес. В качестве альтернативы последовательности UTR, которые не являются эндогенными для нуклеиновой кислоты, представляющей интерес, могут быть добавлены путем включения последовательностей UTR в состав прямого и обратного праймеров или с помощью любых других модификаций матрицы. Использование последовательностей UTR, которые не являются эндогенными для нуклеиновой кислоты, представляющей интерес, может быть применимым для модификации стабильности и/или эффективности трансляции РНК. Например, известно, что AU-богатые элементы в последовательностях 3'-UTR могут уменьшать стабильность мРНК. Следовательно, 3'-UTR могут быть выбраны или разработаны таким образом, чтобы они увеличивали стабильность транскрибированной РНК, исходя из свойств UTR, которые хорошо известны из уровня техники.
В некоторых вариантах осуществления 5'-UTR может содержать последовательность Козак из эндогенной нуклеиновой кислоты В качестве альтернативы, если 5'-UTR, которая не является эндогенной для нуклеиновой кислоты, представляющей интерес, добавляют с помощью ПЦР, как описано выше, консенсусная последовательность Козак может быть видоизменена путем добавления последовательности 5'-UTR. Последовательности Козак могут увеличивать эффективность трансляции некоторых РНК-транскриптов, но, по-видимому, они не являются необходимыми для обеспечения эффективной трансляции всех РНК. Требование наличия последовательностей Козак для многих мРНК известно из уровня техники. В других вариантах осуществления 5'-UTR может представлять собой 5'-UTR из РНК-содержащего вируса, РНК-геном которого является стабильным в клетках. В других вариантах осуществления можно использовать различные аналоги нуклеотидов в 3'- или 5'-UTR, чтобы воспрепятствовать разрушению мРНК под действием экзонуклеаз.
Для обеспечения синтеза РНК с ДНК-матрицы без необходимости клонирования гена промотор транскрипции должен быть присоединен к ДНК-матрице выше последовательности, подлежащей транскрипции. Если последовательность, которая функционирует в качестве промотора для РНК-полимеразы, добавляют к 5'-концу прямого праймера, то промотор для РНК-полимеразы включается в состав продукта ПЦР выше открытой рамки считывания, которая подлежит транскрипции. В некоторых вариантах осуществления промотор представляет собой промотор для полимеразы Т7, как описано в другом разделе в данном документе. Другие применимые промоторы включают без ограничения промоторы для РНК-полимераз Т3 и SP6. Консенсусные нуклеотидные последовательности для промоторов T7, T3 и SP6 известны из уровня техники.
В некоторых вариантах осуществления мРНК содержит как кэп на 5'-конце, так и 3'-поли(А)-хвост, которые определяют связывание с рибосомой, инициацию трансляции и стабильность мРНК в клетке. На кольцевой ДНК-матрице, например, плазмидной ДНК, РНК-полимераза продуцирует длинный конкатемерный продукт, который не подходит для экспрессии в эукариотических клетках. Транскрипция плазмидной ДНК, линеаризованной на конце 3'-UTR, приводит к образованию мРНК нормального размера, которая не является эффективной для трансфекции у эукариот даже в случае ее полиаденилирования после транскрипции.
На линейной ДНК-матрице РНК-полимераза фага Т7 может удлинять 3'-конец транскрипта за пределы последнего основания матрицы (Schenborn and Mierendorf, Nuc Acids Res., 13:6223-36 (1985); Nacheva and Berzal-Herranz, Eur. J. Biochem., 270:1485-65 (2003).
Традиционный способ интеграции отрезков поли(А)/(Т) в ДНК-матрицу представляет собой молекулярное клонирование. Однако последовательность поли(А)/(Т), интегрированная в плазмидную ДНК, может обуславливать нестабильность плазмиды, вследствие чего плазмидные ДНК-матрицы, полученные из бактериальных клеток, зачастую сильно засорены делециями и другими аберрациями. Это приводит к тому, что процедуры клонирования являются не только трудоемкими и времязатратными, но зачастую и ненадежными. Поэтому существует большая потребность в способе, который позволяет конструировать ДНК-матрицы с отрезками поли(А)/(Т) на 3'-конце без клонирования.
Сегмент поли(A)/(T) в транскрипционной ДНК-матрице можно получить в ходе ПЦР с применением обратного праймера, содержащего поли(T)-хвост, такой как хвост из 100 T (SEQ ID NO: 31) (размер может составлять 50-5000 T (SEQ ID NO: 32)) или после ПЦР с помощью любого другого способа, в том числе без ограничения лигирования или рекомбинации ДНК in vitro. Поли(А)-хвосты также обеспечивают стабильность РНК и уменьшают их разрушение. Как правило, длина поли(А)-хвоста положительно коррелирует со стабильностью транскрибированной РНК. В некоторых вариантах осуществления длина поли(А)-хвоста составляет от 100 до 5000 аденозиновых остатков (например, SEQ ID NO: 33).
Поли(А)-хвосты РНК могут быть дополнительно удлинены после транскрипции in vitro путем использования поли(А)-полимеразы, такой как поли(А)-полимераза Е. coli (E-PAP). В некоторых вариантах осуществления увеличение длины поли(А)-хвоста от 100 нуклеотидов до 300-400 нуклеотидов (SEQ ID NO: 34) дает в результате приблизительно двукратное увеличение эффективности трансляции РНК. Кроме того, присоединение различных химических групп к 3'-концу может увеличивать стабильность мРНК. Такое присоединение может включать присоединение модифицированных/искусственных нуклеотидов, аптамеров и других соединений. Например, в состав поли(А)-хвоста с помощью поли(А)-полимеразы могут быть включены аналоги ATP. Аналоги ATP могут дополнительно увеличивать стабильность РНК.
5'-кэп-структуры также обеспечивают стабильность молекул РНК. В некоторых вариантах осуществления РНК, полученные посредством способов, раскрытых в данном документе, содержат 5'-кэп. Наличие 5'-кэпа обеспечивают с помощью методик, известных из уровня техники и описанных в данном документе (Cougot, et al., Trends in Biochem. Sci., 29:436-444 (2001); Stepinski, et al., RNA, 7:1468-95 (2001); Elango, et al., Biochim. Biophys. Res. Commun., 330:958-966 (2005)).
РНК, полученные посредством способов, раскрытых в данном документе, также могут содержать последовательность участка внутренней посадки рибосомы (IRES). Последовательность IRES может быть любой вирусной, хромосомной или искусственно разработанной последовательностью, которая инициирует кэп-независимое связывание рибосомы с мРНК и содействует инициации трансляции. Могут быть включены любые растворенные вещества, подходящие для электропорации клеток, которые могут включать факторы, содействующие проникновению в клетку и жизнеспособности клеток, такие как сахара, пептиды, липиды, белки, антиоксиданты и поверхностно-активные вещества.
РНК можно вводить в клетки-мишени с помощью любого из целого ряда различных способов, например, коммерчески доступных способов, которые включают без ограничения электропорацию (Amaxa Nucleofector-II (Amaxa Biosystems, Кельн, Германия)), ECM 830 (BTX) (Harvard Instruments, Бостон, Массачусетс) или Gene Pulser II (BioRad, Денвер, Колорадо), Multiporator (Eppendorf, Гамбург, Германия)), трансфекцию, опосредованную катионными липосомами, с применением липофекции, инкапсуляцию в полимеры, трансфекцию, опосредованную пептидами, или биобаллистические системы доставки частиц, такие как "генные пушки" (см., например, Nishikawa, et al. Hum Gene Ther., 12(8):861-70 (2001).
Невирусные способы доставки
В некоторых вариантах осуществления можно применять невирусные способы для доставки нуклеиновой кислоты, кодирующей CAR, описанный в данном документе, в клетку, или ткань, или организм субъекта.
В некоторых вариантах осуществления невирусный способ включает применение транспозона (также называемого мобильным генетическим элементом). В некоторых вариантах осуществления транспозон представляет собой фрагмент ДНК, который может самостоятельно вставляться в определенное местоположение в геноме, например, фрагмент ДНК, который способен к саморепликации и вставке своей копии в геном, или фрагмент ДНК, который с помощью сплайсинга может быть вырезан из более длинной нуклеиновой кислоты и вставлен в другое место в геноме. Например, транспозон содержит последовательность ДНК, состоящую из инвертированных повторов, фланкирующих гены для транспозиции.
Иллюстративные способы доставки нуклеиновой кислоты с применением транспозона включают транспозонную систему "спящая красавица" (SBTS) и транспозонную систему piggyBac™ (PB). См., например, Aronovich et al. Hum. Mol. Genet. 20.R1(2011):R14-20; Singh et al. Cancer Res. 15(2008):2961-2971; Huang et al. Mol. Ther. 16(2008):580-589; Grabundzija et al. Mol. Ther. 18(2010):1200-1209; Kebriaei et al. Blood. 122.21(2013):166; Williams. Molecular Therapy 16.9(2008):1515-16; Bell et al. Nat. Protoc. 2.12(2007):3153-65; и Ding et al. Cell. 122.3(2005):473-83, все из которых включены в данный документ посредством ссылки.
SBTS содержит два компонента: 1) транспозон, содержащий трансген, и 2) источник фермента транспозазы. Транспозаза может обеспечивать перенос транспозона из плазмиды-носителя (или другой донорной ДНК) в целевую ДНК, такую как хромосома/геном клетки-хозяина. Например, транспозаза связывается с плазмидой-носителем/донорной ДНК, вырезает транспозон (в том числе трансген(трансгены)) из плазмиды и вставляет его в геном клетки-хозяина. См., например, Aronovich et al. выше.
Иллюстративные транспозоны включают транспозон на основе pT2. См., например, Grabundzija et al. Nucleic Acids Res. 41.3(2013):1829-47 и Singh et al. Cancer Res. 68.8(2008): 2961-2971, все из которых включены в данный документ посредством ссылки. Иллюстративные транспозазы включают транспозазу типа Tc1/mariner, например, транспозазу SB10 или транспозазу SB11 (гиперактивную транспозазу, которая может экспрессироваться, например, под контролем промотора цитомегаловируса). См., например, Aronovich et al.; Kebriaei et al. и Grabundzija et al., все из которых включены в данный документ посредством ссылки.
Применение SBTS обеспечивает эффективную интеграцию и экспрессию трансгена, например, нуклеиновой кислоты, кодирующей CAR, описанный в данном документе. В данном документе представлены способы получения клетки, например, T-клетки или NK-клетки, которая стабильно экспрессирует CAR, описанный в данном документе, например, с применением транспозонной системы, такой как SBTS.
В соответствии со способами, описанными в данном документе, в некоторых вариантах осуществления одна или несколько нуклеиновых кислот, например, плазмид, содержащих компоненты SBTS, доставляются в клетку (например, T- или NK-клетку). Например, нуклеиновая(нуклеиновые) кислота(кислоты) доставляются с помощью стандартных способов доставки нуклеиновых кислот (например, плазмидной ДНК), например, с помощью описанных в данном документе способов, например, электропорации, трансфекции или липофекции. В некоторых вариантах осуществления нуклеиновая кислота содержит транспозон, содержащий трансген, например, нуклеиновую кислоту, кодирующую CAR, описанный в данном документе. В некоторых вариантах осуществления нуклеиновая кислота содержит транспозон, содержащий трансген (например, нуклеиновую кислоту, кодирующую CAR, описанный в данном документе), а также последовательность нуклеиновой кислоты, кодирующую фермент транспозазу. В других вариантах осуществления представлена система с двумя нуклеиновыми кислотами, например, система с двумя плазмидами, например, где первая плазмида содержит транспозон, содержащий трансген, а вторая плазмида содержит последовательность нуклеиновой кислоты, кодирующую фермент транспозазу. Например, первая и вторая нуклеиновые кислоты совместно доставляются в клетку-хозяина.
В некоторых вариантах осуществления клетки, например, T- или NK-клетки, которые экспрессируют CAR, описанный в данном документе, получают с помощью комбинации вставки гена с помощью SBTS и генетического редактирования с помощью нуклеазы (например, нуклеаз с "цинковыми пальцами" (ZFN), эффекторных нуклеаз, подобных активаторам транскрипции (TALEN), системы CRISPR/Cas или сконструированных мегануклеаз, представляющих собой реконструированные хоминг-эндонуклеазы).
В некоторых вариантах осуществления применение невирусного способа доставки делает возможным перепрограммирование клеток, например, Т- или NK-клеток, и прямую инфузию клеток в организм субъекта. Преимущества невирусных векторов включают без ограничения простоту и относительно низкую стоимость получения достаточных количеств, необходимых для удовлетворения требованиям популяции пациентов, стабильность при хранении и отсутствие иммуногенности.
Способы изготовления/получения
В настоящем изобретении также предусмотрены способы получения клетки, раскрытой в данном документе, например, способы конструирования T-клетки или NK-клетки таким образом, чтобы она экспрессировала молекулу нуклеиновой кислоты, кодирующую одну или несколько конструкций CAR, описанных в данном документе. В некоторых вариантах осуществления способы изготовления, раскрытые в данном документе, применяют для изготовления клетки, содержащей молекулу нуклеиновой кислоты, кодирующую два CAR, раскрытых в данном документе (например, CAR для BCMA и CAR для CD19, раскрытые в данном документе). В некоторых вариантах осуществления способы изготовления, раскрытые в данном документе, применяют для изготовления клетки, содержащей молекулу нуклеиновой кислоты, кодирующую CAR в формате диатела, раскрытый в данном документе, например, CAR для BCMA/CD19 в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления способы изготовления, раскрытые в данном документе, применяют для изготовления клетки, содержащей две молекулы нуклеиновой кислоты, каждая из которых кодирует CAR, раскрытый в данном документе (например, одну молекулу нуклеиновой кислоты, кодирующую CAR для BCMA, и одну молекулу нуклеиновой кислоты, кодирующую CAR для CD19). В некоторых вариантах осуществления в данном документе предусмотрена популяция клеток (например, иммунных эффектoрных клеток, например, T-клеток или NK-клеток), полученных посредством любого из способов изготовления, описанных в данном документе.
Способ с использованием активации
В некоторых вариантах осуществления посредством способов, раскрытых в данном документе, можно изготавливать иммунные эффектoрные клетки, сконструированные таким образом, что они экспрессируют один или несколько CAR, за менее чем 24 часа. Не ограничиваясь какой-либо теорией, полагают, что способы, предусмотренные в данном документе, обеспечивают сохранение недифференцированного фенотипа T-клеток, таких как необученные Т-клетки, в ходе способа изготовления. Такие клетки, экспрессирующие CAR, с недифференцированным фенотипом могут персистировать дольше и/или размножаться лучше in vivo после инфузии. В некоторых вариантах осуществления CAR-T-клетки, полученные посредством способов изготовления, предусмотренных в данном документе, содержат более высокую процентную долю T-клеток памяти, представляющих собой стволовые клетки, по сравнению с CAR-T-клетками, полученными посредством способа традиционного изготовления, например, согласно измерению с помощью scRNA-seq (например, согласно измерению посредством способов, описанных в примере 7 в отношении фиг. 25A). В некоторых вариантах осуществления CAR-T-клетки, полученные посредством способов изготовления, предусмотренных в данном документе, содержат более высокую процентную долю эффекторных T-клеток по сравнению с CAR-T-клетками, полученными посредством способа традиционного изготовления, например, согласно измерению с помощью scRNA-seq (например, согласно измерению посредством способов, описанных в примере 7 в отношении фиг. 25B). В некоторых вариантах осуществления CAR-T-клетки, полученные посредством способов изготовления, предусмотренных в данном документе, лучше сохраняют стволовость T-клеток по сравнению с CAR-T-клетками, полученными посредством способа традиционного изготовления, например, согласно измерению с помощью scRNA-seq (например, согласно измерению посредством способов, описанных в примере 7 в отношении фиг. 25C). В некоторых вариантах осуществления CAR-T-клетки, полученные посредством способов изготовления, предусмотренных в данном документе, демонстрируют более низкий уровень гипоксии по сравнению с CAR-T-клетками, полученными посредством способа традиционного изготовления, например, согласно измерению с помощью scRNA-seq (например, согласно измерению посредством способов, описанных в примере 7 в отношении фиг. 25D). В некоторых вариантах осуществления CAR-T-клетки, полученные посредством способов изготовления, предусмотренных в данном документе, демонстрируют более низкий уровень аутофагии по сравнению с CAR-T-клетками, полученными посредством способа традиционного изготовления, например, согласно измерению с помощью scRNA-seq (например, согласно измерению посредством способов, описанных в примере 7 в отношении фиг. 25E). В некоторых вариантах осуществления иммунные эффекторные клетки конструируют таким образом, чтобы они содержали молекулу нуклеиновой кислоты, кодирующую два CAR, раскрытых в данном документе (например, CAR для BCMA и CAR для CD19, раскрытые в данном документе). В некоторых вариантах осуществления иммунные эффекторные клетки конструируют таким образом, чтобы они содержали молекулу нуклеиновой кислоты, кодирующую CAR в формате диатела, раскрытый в данном документе, например, CAR для BCMA/CD19 в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления иммунные эффекторные клетки конструируют таким образом, чтобы они содержали две молекулы нуклеиновой кислоты, каждая из которых кодирует CAR, раскрытый в данном документе (например, одну молекулу нуклеиновой кислоты, кодирующую CAR для BCMA, и одну молекулу нуклеиновой кислоты, кодирующую CAR для CD19).
В некоторых вариантах осуществления способы, раскрытые в данном документе, не предусматривают применение гранулы, такой как Dynabeads® (например, Dynabeads® с антителами к CD3/CD28), и не предусматривают стадию удаления гранул. В некоторых вариантах осуществления CAR-T-клетки, изготовленные посредством способов, раскрытых в данном документе, можно вводить субъекту при минимальном размножении ex vivo, например, в течение менее 1 дня, менее 12 часов, менее 8 часов, менее 6 часов, менее 4 часов, менее 3 часов, менее 2 часов, менее 1 часа или без размножения ex vivo. Соответственно, способы, описанные в данном документе, предусматривают способ быстрого изготовления для получения улучшенных продуктов на основе клеток, экспрессирующих CAR, для применения в лечении заболевания у субъекта.
В некоторых вариантах осуществления в настоящем изобретении предусмотрены способы получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR) (например, один или несколько CAR, например, два CAR), включающие (i) приведение популяции клеток (например, T-клеток, например, T-клеток, выделенных из замороженного или свежего продукта лейкафереза) в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток; (ii) приведение популяции клеток (например, T-клеток) в контакт с молекулой(молекулами) нуклеиновой кислоты (например, молекулой ДНК или РНК), кодирующими CAR, за счет чего обеспечивается получение популяции клеток (например, T-клеток), содержащих молекулу нуклеиновой кислоты, и (iii) сбор популяции клеток (например, T-клеток) для хранения (например, повторного составления популяции клеток в среде для криоконсервации) или введения, где (a) стадию (ii) проводят вместе со стадией (i) или не позже чем через 20 часов после начала стадии (i), например, не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i), например, не позже чем через 18 часов после начала стадии (i), и стадию (iii) проводят не позже чем через 26 часов после начала стадии (i), например, не позже чем через 22, 23 или 24 часа после начала стадии (i), например, не позже чем через 24 часа после начала стадии (i); (b) стадию (ii) проводят вместе со стадией (i) или не позже чем через 20 часов после начала стадии (i), например, не позже чем через 12, 13, 14, 15, 16, 17 или 18 часов после начала стадии (i), например, не позже чем через 18 часов после начала стадии (i), и стадию (iii) проводят не позже чем через 30 часов после начала стадии (ii), например, не позже чем через 22, 23, 24, 25, 26, 27, 28, 29 или 30 часов после начала стадии (ii); или (c) популяция клеток из стадии (iii) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (i). В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) представляет собой молекулу ДНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) представляет собой молекулу РНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) находится в вирусном векторе, например, в вирусном векторе, выбранном из лентивирусного вектора, аденовирусного вектора или ретровирусного вектора. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) находится в невирусном векторе. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) находится в плазмиде. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (ii) не находится в каком-либо векторе. В некоторых вариантах осуществления стадия (ii) включает трансдукцию популяции клеток (например, T-клеток) c помощью вирусного(вирусных) вектора(векторов), содержащих молекулу нуклеиновой кислоты, кодирующую CAR.
В некоторых вариантах осуществления популяцию клеток (например, T-клеток) собирают из образца, полученного путем афереза (например, образца, полученного путем лейкафереза) у субъекта.
В некоторых вариантах осуществления образец, полученный путем афереза (например, образец, полученный путем лейкафереза), собирают у субъекта и транспортируют в виде замороженного образца (например, криоконсервированного образца) в учреждение, осуществляющее изготовление клеток. Затем замороженный образец, полученный путем афереза, размораживают, и T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) отбирают из образца, полученного путем афереза, например, с помощью устройства для сортировки клеток (например, устройства CliniMACS® Prodigy®). Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) затем высевают для изготовления CAR-T посредством способа с использованием активации, описанного в данном документе. В некоторых вариантах осуществления отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) подвергают одному или нескольким циклам замораживания-размораживания перед высеванием для изготовления CAR-T.
В некоторых вариантах осуществления образец, полученный путем афереза (например, образец, полученный путем лейкафереза), собирают у субъекта и транспортируют в виде свежего продукта (например, незамороженного продукта) в учреждение, осуществляющее изготовление клеток. T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) отбирают из образца, полученного путем афереза, например, с помощью устройства для сортировки клеток (например, устройства CliniMACS® Prodigy®). Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) затем высевают для изготовления CAR-T посредством способа с использованием активации, описанного в данном документе. В некоторых вариантах осуществления отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) подвергают одному или нескольким циклам замораживания-размораживания перед высеванием для изготовления CAR-T.
В некоторых вариантах осуществления у субъекта собирают образец, полученный путем афереза (например, образец, полученный путем лейкафереза). T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) отбирают из образца, полученного путем афереза, например, с помощью устройства для сортировки клеток (например, устройства CliniMACS® Prodigy®). Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) затем транспортируют в виде замороженного образца (например, криоконсервированного образца) в учреждение, осуществляющее изготовление клеток. Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) впоследствии размораживают и высевают для изготовления CAR-T посредством способа с использованием активации, описанного в данном документе.
В некоторых вариантах осуществления клетки (например, T-клетки) приводят в контакт с антителами к CD3 и антителами к CD28 в течение, например, 12 часов с последующей трансдукцией с помощью вектора (например, лентивирусного вектора) (например, одного или нескольких векторов), кодирующего CAR (например, один или несколько CAR). Через 24 часа после начала культивирования клетки промывают и составляют для хранения или введения.
Не ограничиваясь какой-либо теорией, полагают, что кратковременная стимуляция CD3 и CD28 может способствовать эффективной трансдукции самообновляющихся T-клеток. По сравнению с традиционными подходами к изготовлению CAR-T способ с использованием активации, предусмотренный в данном документе, не предусматривает продолжительного размножения ex vivo. Подобно способу с использованием цитокинов, способ с использованием активации, предусмотренный в данном документе, также обеспечивает сохранение недифференцированных T-клеток в ходе изготовления CAR-T.
В некоторых вариантах осуществления популяцию клеток приводят в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток.
В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, представляет собой средство, которое стимулирует CD3. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой средство, которое стимулирует CD28, ICOS, CD27, HVEM, LIGHT, CD40, 4-1BB, OX40, DR3, GITR, CD30, TIM1, CD2, CD226 или любую их комбинацию. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой средство, которое стимулирует CD28. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, выбрано из антитела (например, однодоменного антитела (например, антитела из одного вариабельного домена тяжелой цепи), пептитела, Fab-фрагмента или scFv), малой молекулы или лиганда (например, существующего в природе, рекомбинантного или химерного лиганда). В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, представляет собой антитело. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, представляет собой антитело к CD3. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, выбрано из антитела (например, однодоменного антитела (например, антитела из одного вариабельного домена тяжелой цепи), пептитела, Fab-фрагмента или scFv), малой молекулы или лиганда (например, существующего в природе, рекомбинантного или химерного лиганда). В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой антитело. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, представляет собой антитело к CD28. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, или средство, которое стимулирует костимулирующую молекулу, не содержит гранулу. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, содержит антитело к CD3, ковалентно присоединенное к коллоидной полимерной наноматрице. В некоторых вариантах осуществления средство, которое стимулирует костимулирующую молекулу, содержит антитело к CD28, ковалентно присоединенное к коллоидной полимерной наноматрице. В некоторых вариантах осуществления средство, которое стимулирует комплекс CD3/TCR, и средство, которое стимулирует костимулирующую молекулу, содержат TransAct™ для T-клеток.
В некоторых вариантах осуществления матрица содержит полимерный, например, биоразлагаемый или биосовместимый, инертный материал, например, который является нетоксичным для клеток, или состоит из такого материала. В некоторых вариантах осуществления матрица состоит из гидрофильных полимерных цепей, которые достигают максимальной подвижности в водном растворе благодаря гидратации цепей. В некоторых вариантах осуществления подвижная матрица может состоять из композиций на основе коллагена, очищенных белков, очищенных пептидов, полисахаридов, гликозаминогликанов или внеклеточного матрикса. Полисахарид может включать, например, простые эфиры целлюлозы, крахмал, аравийскую камедь, агарoзу, декстран, хитозан, гиалуроновую кислоту, пектины, ксантан, гуаровую камедь или альгинат. Другие полимеры могут включать сложные полиэфиры, простые полиэфиры, полиакрилаты, полиакриламиды, полиамины, полиэтиленимины, полимеры поликватерниумы, полифосфазены, поливиниловые спирты, поливинилацетаты, поливинилпирролидоны, блок-сополимеры или полиуретаны. В некоторых вариантах осуществления подвижная матрица представляет собой полимер декстран.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с молекулой нуклеиновой кислоты (например, одной или несколькими молекулами нуклеиновой кислоты), кодирующей CAR (например, один или несколько CAR). В некоторых вариантах осуществления популяцию клеток трансдуцируют с помощью молекулы ДНК (например, одной или нескольких молекул ДНК), кодирующей CAR (например, один или несколько CAR).
В некоторых вариантах осуществления в случае котрансдукции двумя молекулами нуклеиновой кислоты (например, лентивирусными векторами), каждая из которых кодирует CAR, раскрытый в данном документе (например, одной молекулой нуклеиновой кислоты, кодирующей CAR для BCMA, и одной молекулой нуклеиновой кислоты, кодирующей CAR для CD19, раскрытые в данном документе), каждый из векторов, содержащих молекулы нуклеиновой кислоты, кодирующие CAR, можно добавлять в реакционную смесь (например, содержащую популяцию клеток) при различной множественности заражения (MOI).
Не ограничиваясь какой-либо теорией, полагают, что в некоторых вариантах осуществления с использование различных MOI для векторов, содержащих молекулы нуклеиновой кислоты, которые кодируют различные молекулы CAR, может влиять на конечный состав клеточной популяции. Например, в случае котрансдукции лентивирусным вектором, кодирующим CAR для BCMA, и лентивирусным вектором, кодирующим CAR для CD19, можно использовать различные MOI для доведения до максимума процента моно-BCMA-CAR-Т-клеток и CAR-Т-клеток с двойной специфичностью к BCMA/CD19 с получением при этом меньшего количества моно-CD19-CAR-T-клеток и нетрансдуцированных клеток.
В некоторых вариантах осуществления в случае котрансдукции лентивирусным вектором, кодирующим CAR для BCMA, и лентивирусным вектором, кодирующим CAR для CD19, популяцию клеток приводят в контакт с первым вирусным вектором при множественности заражения (MOI), которая больше, равняется или меньше MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором. В некоторых вариантах осуществления популяцию клеток приводят в контакт с первым вирусным вектором при множественности заражения (MOI), которая больше MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с первым вирусным вектором при первой MOI и со вторым вирусным вектором при второй MOI, так что полученная в результате популяция клеток содержит первую популяцию клеток, которые содержат CAR для BCMA, но не CAR для CD19, вторую популяцию клеток, которые содержат CAR для CD19, но не CAR для BCMA, и третью популяцию клеток, которые содержат как CAR для BCMA, так и CAR для CD19, где:
(a) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(b) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(c) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в полученной в результате популяции, например, при определении посредством способа, описанного в примере 10;
(d) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10; или
(e) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток во второй популяции, например, при определении посредством способа, описанного в примере 10.
В некоторых вариантах осуществления популяцию клеток приводят в контакт со вторым вирусным вектором при MOI, например, MOI, которая достаточно ниже, чем MOI, при которой популяцию клеток приводят в контакт с первым вирусным вектором, так что в полученной в результате популяции клеток:
(a) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(b) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000%, 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(c) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в полученной в результате популяции, например, при определении посредством способа, описанного в примере 10;
(d) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10; или
(e) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000%, 5000, 10000% или больше) от общего количества жизнеспособных клеток во второй популяции, например, при определении посредством способа, описанного в примере 10.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с первым вирусным вектором при первой MOI и популяцию клеток приводят в контакт со вторым вирусным вектором при второй MOI, так что полученная в результате популяция клеток содержит:
(a) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(b) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях, например, при определении посредством способа, описанного в примере 10;
(c) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в полученной в результате популяции, например, при определении посредством способа, описанного в примере 10;
(d) общее количество жизнеспособных клеток во второй популяции меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях, например, при определении посредством способа, описанного в примере 10; или
(e) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000%, 5000, 10000% или больше) от общего количества жизнеспособных клеток во второй популяции, например, при определении посредством способа, описанного в примере 10.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с
(a) первым вирусным вектором при MOI, составляющей от приблизительно 1 до приблизительно 10 (например, от приблизительно 2 до приблизительно 9, от приблизительно 3 до приблизительно 8, от приблизительно 4 до приблизительно 7, от приблизительно 5 до приблизительно 6, от приблизительно 1 до приблизительно 8, от приблизительно 1 до приблизительно 6, от приблизительно 1 до приблизительно 4, от приблизительно 8 до приблизительно 10, от приблизительно 6 до приблизительно 10, от приблизительно 4 до приблизительно 10, от приблизительно 1 до приблизительно 3, от приблизительно 2 до приблизительно 4, от приблизительно 3 до приблизительно 5, от приблизительно 4 до приблизительно 6, от приблизительно 5 до приблизительно 7, от приблизительно 6 до приблизительно 8, от приблизительно 7 до приблизительно до приблизительно 9, от приблизительно 8 до приблизительно 10, от приблизительно 2,5 до приблизительно 5, приблизительно 1, приблизительно 2, приблизительно 3, приблизительно 4, приблизительно 5, приблизительно 6, приблизительно 7, приблизительно 8, приблизительно 9 или приблизительно 10);
(b) вторым вирусным вектором при MOI, составляющей от приблизительно 0,1 до приблизительно 5 (например, от приблизительно 0,2 до приблизительно 4, от приблизительно 0,3 до приблизительно 3, от приблизительно 0,4 до приблизительно 2, от приблизительно 0,5 до приблизительно 1, от приблизительно 0,6 до приблизительно 0,9, от приблизительно 0,7 до приблизительно 0,8, от приблизительно 0,1 до приблизительно 4, от приблизительно 0,1 до приблизительно 3, от приблизительно 0,1 до приблизительно 2, от приблизительно 0,1 до приблизительно 1, от приблизительно 0,1 до приблизительно 0,5, от приблизительно 4 до приблизительно 5, от приблизительно 3 до приблизительно 5, от приблизительно 2 до приблизительно 5, от приблизительно 1 до приблизительно 5, от приблизительно 0,5 до приблизительно 5, от приблизительно 0,2 до приблизительно 5, от приблизительно 0,1 до приблизительно 0,5, от приблизительно 0,2 до приблизительно 1, от приблизительно 0,5 до приблизительно 2, от приблизительно 1 до приблизительно 3, от приблизительно 2 до приблизительно 4, от приблизительно 3 до приблизительно 5, от приблизительно 0,5 до приблизительно 1, приблизительно 0,1, приблизительно 0,2, приблизительно 0,3, приблизительно 0,4, приблизительно 0,5, приблизительно 0,6, приблизительно 0,7, приблизительно 0,8, приблизительно 0,9, приблизительно 1, приблизительно 2, приблизительно 3, приблизительно 4 или приблизительно 5);
(c) первым вирусным вектором при MOI, составляющей по меньшей мере приблизительно 10% (например, по меньшей мере приблизительно 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором, или в по меньшей мере приблизительно 1 раз (например, в по меньшей мере приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 35, 40, 50, 60, 70, 80, 90, или 100 раз, например, от приблизительно 2 до приблизительно 50 раз, от приблизительно 3 до 20 раз, от приблизительно 5 до приблизительно 15 раз или от приблизительно 8 до приблизительно 10 раз) выше нее; и/или
(d) вторым вирусным вектором при MOI, которая составляет не более чем 1/X, где X равняется 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 15, 20, 30, 40, 50, 60, 70. 80, 90, или 100, от MOI, при которой популяцию клеток приводят в контакт с первым вирусным вектором.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с первым вирусным вектором при MOI, составляющей от приблизительно 2,5 до приблизительно 5. В некоторых вариантах осуществления популяцию клеток приводят в контакт со вторым вирусным вектором при MOI, составляющей от приблизительно 0,5 до приблизительно 1,0. В некоторых вариантах осуществления MOI для первого вирусного вектора в от приблизительно 8 до приблизительно 10 раз выше, чем MOI, при которой популяцию клеток приводят в контакт со вторым вирусным вектором. В некоторых вариантах осуществления MOI для второго вирусного вектора составляет не более чем 1/X, где X равняется 6, 8, 10 или 12, от MOI, при которой популяцию клеток приводят в контакт с первым вирусным вектором.
В некоторых вариантах осуществления на стадии (ii) популяцию клеток приводят в контакт с:
(a) первым вирусным вектором при MOI, составляющей от приблизительно 4 до приблизительно 5 (например, приблизительно 4,75); и/или
(b) вторым вирусным вектором при MOI, составляющей от приблизительно 0,2 до приблизительно 1 (например, приблизительно 0,5).
В некоторых вариантах осуществления на стадии (ii) популяция клеток содержит от приблизительно 1×108 до приблизительно 5×109 (например, от приблизительно 2×108 до приблизительно 2×109 или от приблизительно 4×108 до приблизительно 1×109) всех жизнеспособных клеток. В некоторых вариантах осуществления клетки суспендированы в культуре при концентрации, составляющей от приблизительно 1×106 до приблизительно 1 ×107 (например, от приблизительно 2×106 до приблизительно 5 ×106 или от приблизительно 3×106 до приблизительно 4 ×106) жизнеспособных клеток/мл.
Точную MOI, используемую для каждого вектора, можно корректировать или определять на основании ряда факторов, включающих без ограничения свойства партии вирусного вектора, характеристики клеток, подлежащих трансдукции, и эффективность трансдукции. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит одновременно с приведением популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 19, 18, 17, 16, 15, 14, 13, 12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 2, 1, или 0,5 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 20 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 19 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 18 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 17 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 16 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 15 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 14 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 14 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 13 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 12 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 11 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 10 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 9 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 8 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 7 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 6 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 5 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 4 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 3 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 2 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 1 час после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой(молекулами) нуклеиновой кислоты, кодирующей(кодирующими) CAR, происходит не позже чем через 30 минут после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше.
В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения.
В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 72, 60, 48, 36, 32, 31, 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 19, или 18 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 26 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 25 часов после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 24 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 23 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 22 часа после начала приведения популяции клеток в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше.
В некоторых вариантах осуществления популяцию клеток не размножают ex vivo.
В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, или 60%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 5%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 15%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 20%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 25%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 30%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 35%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 40%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт со средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток, описанными выше.
В некоторых вариантах осуществления популяцию клеток размножают в течение не более чем 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 6, 7, 8, 9, 10, 11, 12, 16, 20, 24, 36, или 48 часов, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше.
В некоторых вариантах осуществления способ с использованием активации осуществляют в бессывороточных средах для клеток. В некоторых вариантах осуществления способ с использованием активации осуществляют в средах для клеток, содержащих один или несколько цитокинов, выбранных из IL-2, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) или IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления hetIL-15 содержит аминокислотную последовательность NWVNVISDLKKIEDLIQSMHIDATLYTESDVHPSCKVTAMKCFLLELQVISLESGDASIHDTVENLIILANNSLSSNGNVTESGCKECEELEEKNIKEFLQSFVHIVQMFINTSITCPPPMSVEHADIWVKSYSLYSRERYICNSGFKRKAGTSSLTECVLNKATNVAHWTTPSLKCIRDPALVHQRPAPPSTVTTAGVTPQPESLSPSGKEPAASSPSSNNTAATTAAIVPGSQLMPSKSPSTGTTEISSHESSHGTPSQTTAKNWELTASASHQPPGVYPQG (SEQ ID NO: 309). В некоторых вариантах осуществления hetIL-15 содержит аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 70, 75, 80, 85, 90, 95 или 99% идентичностью с SEQ ID NO: 309. В некоторых вариантах осуществления способ с использованием активации осуществляют в средах для клеток, содержащих ингибитор LSD1. В некоторых вариантах осуществления способ с использованием активации осуществляют в средах для клеток, содержащих ингибитор MALT1. В некоторых вариантах осуществления бессывороточные среды для клеток содержат заменитель сыворотки. В некоторых вариантах осуществления заменитель сыворотки представляет собой заменитель сыворотки для иммунных клеток (ICSR) CTS™. В некоторых вариантах осуществления уровень ICSR может составлять, например, вплоть до 5%, например, приблизительно 1%, 2%, 3%, 4% или 5%. Не ограничиваясь какой-либо теорией, полагают, что применение сред для клеток, например, сред для быстрого изготовления, показанных в таблице 21 или таблице 25, содержащих ICSR, например, 2% ICSR, может обеспечивать улучшение жизнеспособности клеток в ходе способа изготовления, описанного в данном документе.
В некоторых вариантах осуществления в настоящем изобретении предусмотрены способы получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR), включающие (a) получение образца, полученного путем афереза (например, свежего или криоконсервированного образца, полученного путем лейкафереза), собранного у субъекта; (b) отбор T-клеток из образца, полученного путем афереза (например, с помощью отрицательного отбора, положительного отбора или отбора без гранул); (c) высевание выделенных T-клеток при, например, от 1×106 до 1×07 клеток/мл; (d) приведение T-клеток в контакт со средством, которое стимулирует T-клетки, например, средством, которое стимулирует комплекс CD3/TCR, и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток (например, приведение T-клеток в контакт с антителом к CD3 и/или антителом к CD28, например, приведение T-клеток в контакт с TransAct); (e) приведение T-клеток в контакт с молекулой(молекулами) нуклеиновой кислоты (например, молекулой ДНК или РНК), кодирующей(кодирующими) CAR (например, приведение T-клеток в контакт с вирусом, содержащим молекулу(молекулы) нуклеиновой кислоты, кодирующую(кодирующие) CAR), в течение, например, 6-48 часов, например, 20-28 часов; и (f) промывание и сбор T-клеток для хранения (например, повторного составления T-клеток в среде для криоконсервации) или введения. В некоторых вариантах осуществления стадию (f) проводят не позже чем через 30 часов после начала стадии (d) или (e), например, не позже чем через 22, 23, 24, 25, 26, 27, 28, 29 или 30 часов после начала стадии (d) или (e).
В некоторых вариантах осуществления в данном документе предусмотрена популяция клеток (например, иммунных эффекторных клеток, например, Т-клеток или NK-клеток), полученных посредством любого из способов изготовления, описанных в данном документе (например, способа с использованием активации, описанного в данном документе).
В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) (1) является такой же, (2) отличается, например, на не более чем 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, или 15%, или (3) является увеличенной, например, на по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, или 25%, по сравнению с процентной долей необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ клеток, в популяции клеток в начале способа изготовления (например, в начале способа с использованием цитокинов или способа с использованием активации, описанных в данном документе). В некоторых вариантах осуществления популяция клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) демонстрирует более высокую процентную долю необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток (например, на по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, или 50% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, продолжительность которого составляет, например, более 26 часов (например, продолжительность которого составляет более 5, 6, 7, 8, 9, 10, 11 или 12 дней) или который предусматривает размножение популяции клеток in vitro в течение, например, более чем 3 дней (например, размножение популяции клеток in vitro в течение 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, или 15 дней).
В некоторых вариантах осуществления процентная доля необученных клеток, например, необученных Т-клеток, например, CD45RA+ CD45RO- CCR7+ T-клеток, в популяции клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) составляет не менее 20, 25, 30, 35, 40, 45, 50, 55 или 60%.
В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) (1) является такой же, (2) отличается, например, на не более чем 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, или 15%, или (3) является уменьшенной, например, на по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, или 25%, по сравнению с процентной долей центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти, в популяции клеток в начале способа изготовления (например, в начале способа с использованием цитокинов или способа с использованием активации, описанных в данном документе). В некоторых вариантах осуществления популяция клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) демонстрирует более низкую процентную долю центральных клеток памяти, например, центральных T-клеток памяти, например, CD95+ центральных Т-клеток памяти (например, на по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, или 50% ниже), по сравнению с клетками, полученными посредством в остальном аналогичного способа, продолжительность которого составляет, например, более 26 часов (например, продолжительность которого составляет более 5, 6, 7, 8, 9, 10, 11 или 12 дней) или который предусматривает размножение популяции клеток in vitro в течение, например, более чем 3 дней (например, размножение популяции клеток in vitro в течение 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, или 15 дней).
В некоторых вариантах осуществления процентная доля центральных клеток памяти, например, центральных T-клеток памяти, например, центральных CD95+ Т-клеток памяти, в популяции клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) составляет не более 40, 45, 50, 55, 60, 65, 70, 75 или 80%.
В некоторых вариантах осуществления популяция клеток в конце способа изготовления (например, в конце способа с использованием цитокинов или способа с использованием активации, описанных в данном документе) после введения in vivo персистирует дольше или размножается на более высоком уровне (например, на по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, или 90% более высоком) по сравнению с клетками, полученными посредством в остальном аналогичного способа, продолжительность которого составляет, например, более 26 часов (например, продолжительность которого составляет более 5, 6, 7, 8, 9, 10, 11 или 12 дней) или который предусматривает размножение популяции клеток in vitro в течение, например, более чем 3 дней (например, размножение популяции клеток in vitro в течение 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, или 15 дней).
В некоторых вариантах осуществления популяция клеток была обогащена клетками, экспрессирующими IL6R (например, клетками, которые являются положительными по IL6Rα и/или IL6Rβ), до начала способа изготовления (например, до начала способа с использованием цитокинов или способа с использованием активации, описанных в данном документе). В некоторых вариантах осуществления популяция клеток содержит, например, не менее 30, 35, 40, 45, 50, 55, 60, 65, 70, 75 или 80% клеток, экспрессирующих IL6R (например, клеток, которые являются положительными по IL6Rα и/или IL6Rβ), в начале способа изготовления (например, в начале способа с использованием цитокинов или способа с использованием активации, описанных в данном документе).
Способ с использованием цитокинов
В некоторых вариантах осуществления в настоящем изобретении предусмотрены способы получения популяции клеток (например, T-клеток), которые экспрессируют химерный антигенный рецептор (CAR) (например, один или несколько CAR, например, два CAR), включающие (1) приведение популяции клеток в контакт с цитокином, выбранным из IL-2, IL-7, IL-15, IL-21, IL-6 или их комбинации, (2) приведение популяции клеток (например, T-клеток) в контакт с молекулой(молекулами) нуклеиновой кислоты (например, молекулой ДНК или РНК), кодирующей(кодирующими) CAR, за счет чего обеспечивается получение популяции клеток (например, T-клеток), содержащих молекулу нуклеиновой кислоты, и (3) сбор популяции клеток (например, T-клеток) для хранения (например, повторного составления популяции клеток в среде для криоконсервации) или введения, где (a) стадию (2) проводят вместе со стадией (1) или не позже чем через 5 часов после начала стадии (1), например, не позже чем через 1, 2, 3, 4 или 5 часов после начала стадии (1), и стадию (3) проводят не позже чем через 26 часов после начала стадии (1), например, не позже чем через 22, 23 или 24 часа после начала стадии (1), например, не позже чем через 24 часа после начала стадии (1), или (b) популяция клеток из стадии (3) не является размножившейся или является размножившейся с увеличением численности на не более чем 5, 10, 15, 20, 25, 30, 35 или 40%, например, на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток в начале стадии (1). В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) представляет собой молекулу ДНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) представляет собой молекулу РНК. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) находится в вирусном векторе, например, в вирусном векторе, выбранном из лентивирусного вектора, аденовирусного вектора или ретровирусного вектора. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) находится в невирусном векторе. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) находится в плазмиде. В некоторых вариантах осуществления молекула нуклеиновой кислоты на стадии (2) не находится в каком-либо векторе. В некоторых вариантах осуществления стадия (2) включает трансдукцию популяции клеток (например, T-клеток) c помощью вирусного вектора, содержащего молекулу(молекулы) нуклеиновой кислоты, кодирующую(кодирующие) CAR. В некоторых вариантах осуществления клетки конструируют таким образом, чтобы они содержали молекулу нуклеиновой кислоты, кодирующую два CAR, раскрытых в данном документе (например, CAR для BCMA и CAR для CD19, раскрытые в данном документе). В некоторых вариантах осуществления клетки конструируют таким образом, чтобы они содержали молекулу нуклеиновой кислоты, кодирующую CAR в формате диатела, раскрытый в данном документе, например, CAR для BCMA/CD19 в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления клетки конструируют таким образом, чтобы они содержали две молекулы нуклеиновой кислоты, каждая из которых кодирует CAR, раскрытый в данном документе (например, одну молекулу нуклеиновой кислоты, кодирующую CAR для BCMA, и одну молекулу нуклеиновой кислоты, кодирующую CAR для CD19).
В некоторых вариантах осуществления популяцию клеток (например, T-клеток) собирают из образца, полученного путем афереза (например, образца, полученного путем лейкафереза) у субъекта.
В некоторых вариантах осуществления образец, полученный путем афереза (например, образец, полученный путем лейкафереза), собирают у субъекта и транспортируют в виде замороженного образца (например, криоконсервированного образца) в учреждение, осуществляющее изготовление клеток. Затем замороженный образец, полученный путем афереза, размораживают, и T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) отбирают из образца, полученного путем афереза, например, с помощью устройства для сортировки клеток (например, устройства CliniMACS® Prodigy®). Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) затем высевают для изготовления CAR-T посредством способа с использованием цитокинов, описанного в данном документе. В некоторых вариантах осуществления в конце способа с использованием цитокинов CAR-T-клетки криоконсервируют и впоследствии размораживают и вводят субъекту. В некоторых вариантах осуществления отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) подвергают одному или нескольким циклам замораживания-размораживания перед высеванием для изготовления CAR-T.
В некоторых вариантах осуществления образец, полученный путем афереза (например, образец, полученный путем лейкафереза), собирают у субъекта и транспортируют в виде свежего продукта (например, незамороженного продукта) в учреждение, осуществляющее изготовление клеток. T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) отбирают из образца, полученного путем афереза, например, с помощью устройства для сортировки клеток (например, устройства CliniMACS® Prodigy®). Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) затем высевают для изготовления CAR-T посредством способа с использованием цитокинов, описанного в данном документе. В некоторых вариантах осуществления отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) подвергают одному или нескольким циклам замораживания-размораживания перед высеванием для изготовления CAR-T.
В некоторых вариантах осуществления у субъекта собирают образец, полученный путем афереза (например, образец, полученный путем лейкафереза). T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) отбирают из образца, полученного путем афереза, например, с помощью устройства для сортировки клеток (например, устройства CliniMACS® Prodigy®). Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) затем транспортируют в виде замороженного образца (например, криоконсервированного образца) в учреждение, осуществляющее изготовление клеток. Отобранные T-клетки (например, CD4+ T-клетки и/или CD8+ T-клетки) впоследствии размораживают и высевают для изготовления CAR-T посредством способа с использованием цитокинов, описанного в данном документе.
В некоторых вариантах осуществления после высевания клеток (например, T-клеток) к клеткам добавляют один или несколько цитокинов (например, один или несколько цитокинов, выбранных из IL-2, IL-7, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-21 или IL-6 (например, IL-6/sIL-6R)), а также вектор (например, лентивирусный вектор) (например, один или несколько векторов), кодирующий CAR (например, один или несколько CAR). После инкубирования в течение 20-24 часов клетки промывают и составляют для хранения или введения.
В отличие от традиционных подходов к изготовлению CAR-T, способ с использованием цитокинов, предусмотренный в данном документе, не предусматривает стимуляцию CD3 и/или CD28 или размножение Т-клеток ex vivo. T-клетки, которые приводятся в контакт с антителами к CD3 и антителами к CD28 и которые интенсивно размножаются ex vivo, склонны демонстрировать дифференцировку в направлении фенотипа центральных клеток памяти. Не ограничиваясь какой-либо теорией, полагают, что способ с использованием цитокинов, предусмотренный в данном документе, обеспечивает сохранение или увеличение выраженности недифференцированного фенотипа T-клеток в ходе изготовления CAR-T, обеспечивая при этом получение продукта на основе CAR-T, который может дольше персистировать после инфузии субъекту.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с одним или несколькими цитокинами (например, одним или несколькими цитокинами, выбранными из IL-2, IL-7, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)), IL-21 или IL-6 (например, IL-6/sIL-6Ra).
В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-2. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-7. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-21. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-2 и IL-7. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-2 и IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-2 и IL-21. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-2 и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-7 и IL-15 (например, hetIL-15 (IL15/sIL-15Ra)). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-7 и IL-21. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-7 и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) и IL-21. В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-21 и IL-6 (например, IL-6/sIL-6Ra). В некоторых вариантах осуществления популяцию клеток приводят в контакт с IL-7, IL-15 (например, hetIL-15 (IL15/sIL-15Ra)) и IL-21. В некоторых вариантах осуществления популяцию клеток дополнительно приводят в контакт с ингибитором LSD1. В некоторых вариантах осуществления популяцию клеток дополнительно приводят в контакт с ингибитором MALT1.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 210, 220, 230, 240, 250, 260, 270, 280, 290 или 300 ед/мл IL-2. В некоторых вариантах осуществления популяцию клеток приводят в контакт с 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 нг/мл IL-7. В некоторых вариантах осуществления популяцию клеток приводят в контакт с 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 нг/мл IL-15.
В некоторых вариантах осуществления популяцию клеток приводят в контакт с молекулой нуклеиновой кислоты (например, одной или несколькими молекулами нуклеиновой кислоты), кодирующей CAR (например, один или несколько CAR). В некоторых вариантах осуществления популяцию клеток трансдуцируют с помощью молекулы ДНК (например, одной или нескольких молекул ДНК), кодирующей CAR (например, один или несколько CAR).
В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит одновременно с приведением популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 5,5, 6, 6,5, 7, 7,5, 8, 8,5, 9, 9,5 или 10 часов после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 5 часов после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 4 часа после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 3 часа после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 2 часа после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления приведение популяции клеток в контакт с молекулой нуклеиновой кислоты, кодирующей CAR, происходит не позже чем через 1 час после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше.
В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения.
В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 72, 60, 48, 36, 32, 31, 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 19, или 18 часов после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 26 часов после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 25 часов после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 24 часа после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 23 часа после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток собирают для хранения или введения не позже чем через 22 часа после начала приведения популяции клеток в контакт с одним или несколькими цитокинами, описанными выше.
В некоторых вариантах осуществления популяцию клеток не размножают ex vivo.
В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, или 60%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 5%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 10%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 15%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 20%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 25%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 30%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 35%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше. В некоторых вариантах осуществления популяцию клеток размножают с увеличением численности на не более чем 40%, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше.
В некоторых вариантах осуществления популяцию клеток размножают в течение не более чем 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 6, 7, 8, 9, 10, 11, 12, 16, 20, 24, 36, или 48 часов, например, согласно оценке по количеству живых клеток, по сравнению с популяцией клеток до ее приведения в контакт с одним или несколькими цитокинами, описанными выше.
В некоторых вариантах осуществления популяцию клеток не приводят в контакт in vitro со средством, которое стимулирует комплекс CD3/TCR (например, антителом к CD3), и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток (например, антителом к CD28), или, в случае приведения в контакт, продолжительность стадии приведения в контакт составляет менее 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5 или 5 часов.
В некоторых вариантах осуществления популяцию клеток приводят в контакт in vitro со средством, которое стимулирует комплекс CD3/TCR (например, антителом к CD3), и/или средством, которое стимулирует костимулирующую молекулу на поверхности клеток (например, антителом к CD28), в течение 20, 21, 22, 23, 24, 25, 26, 27 или 28 часов.
В некоторых вариантах осуществления популяция клеток, изготовленная посредством способа с использованием цитокинов, предусмотренного в данном документе, демонстрирует более высокую процентную долю необученных клеток среди клеток, экспрессирующих CAR (например, на по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 35, 40, 45, 50, 55, или 60% выше), по сравнению с клетками, полученными посредством в остальном аналогичного способа, который дополнительно включает приведение популяции клеток в контакт, например, со средством, которое связывает комплекс CD3/TCR (например, антителом к CD3), и/или средством, которое связывает костимулирующую молекулу на поверхности клеток (например, антителом к CD28).
В некоторых вариантах осуществления способ с использованием цитокинов в данном документе осуществляют в средах для клеток, содержащих не более 0, 0,5, 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 5,5, 6, 6,5, 7, 7,5 или 8% сыворотки. В некоторых вариантах осуществления способ с использованием цитокинов, предусмотренный в данном документе, осуществляют в средах для клеток, содержащих ингибитор LSD1, ингибитор MALT1 или их комбинацию.
Дополнительные иллюстративные способы изготовления
В некоторых вариантах осуществления клетки, например, Т-клетки или NK клетки, активируют, например, с использованием Dynabeads®, покрытых антителами к CD3/антителами к CD28, приводят в контакт с одной или несколькими молекулами нуклеиновой кислоты, кодирующими CAR (например, один или несколько CAR), а затем размножают in vitro в течение, например, 7, 8, 9, 10 или 11 дней. В некоторых вариантах осуществления клетки, например, Т-клетки или NK-клетки, отбирают из свежего или криоконсервированного образца, полученного путем лейкафереза, например, с помощью положительного или отрицательного отбора. В некоторых вариантах осуществления клетки приводят в контакт с молекулой нуклеиновой кислоты (например, одной или несколькими молекулами нуклеиновой кислоты), кодирующей CAR (например, один или несколько CAR). В некоторых вариантах осуществления клетки приводят в контакт с молекулой нуклеиновой кислоты, кодирующей два CAR, раскрытых в данном документе (например, CAR для BCMA и CAR для CD19). В некоторых вариантах осуществления клетки приводят в контакт с двумя молекулами нуклеиновой кислоты, одна из которых экспрессирует первый CAR (например, CAR для BCMA), а другая экспрессирует второй CAR (например, CAR для CD19). В некоторых вариантах осуществления клетки приводят в контакт с молекулой нуклеиновой кислоты, кодирующей CAR в формате диатела (например, CAR для BCMA/CD19 в формате диатела, раскрытый в данном документе).
Элютриация
В некоторых вариантах осуществления способы, описанные в данном документе, включают способ элютриации, с помощью которого удаляют нежелательные клетки, например, моноциты и бластные клетки, что в результате приводит к улучшенному обогащению требуемыми иммунными эффектoрными клетками, подходящими для экспрессии CAR. В одном варианте осуществления способ элютриации, описанный в данном документе, оптимизирован для обогащения требуемыми иммунными эффектoрными клетками, подходящими для экспрессии CAR, из ранее замороженного образца, например, размороженного образца. В некоторых вариантах осуществления способ элютриации, описанный в данном документе, обеспечивает получение клеток с улучшенной чистотой по сравнению с получением клеток, собранных согласно протоколам элютриации, известным из уровня техники. В некоторых вариантах осуществления способ элютриации, описанный в данном документе, включает использование оптимизированной вязкости исходного образца, например, образца клеток, например, размороженного образца клеток, путем разбавления определенными изотоническими растворами (например, PBS), а также использование оптимизированной комбинации скоростей потока и объема собираемого материала для каждой фракции, собираемой с помощью устройства для элютриации. Иллюстративные способы элютриации, которые можно применять в настоящем изобретении, описаны на страницах 48-51 в WO 2017/117112, которая включена в данный документ посредством ссылки во всей своей полноте.
Центрифугирование в градиенте плотности
Изготовление продукта для адоптивной клеточной терапии требует обработки требуемых клеток, например, иммунных эффекторных клеток, для отделения от сложной смеси клеток крови и элементов крови, присутствующих в исходных материалах периферической крови, полученных путем афереза. Образцы лимфоцитов периферической крови были успешно выделены с помощью центрифугирования в градиенте плотности в растворе Ficoll. Однако Ficoll не является предпочтительным реагентом для выделения клеток для терапевтического применения, поскольку Ficoll не пригоден для клинического применения. Кроме того, Ficoll содержит гликоль, который обладает токсичным потенциалом для клеток. Кроме того, при центрифугировании в градиенте плотности Ficoll размороженных продуктов афереза после криоконсервации получают неоптимальный продукт на основе T-клеток, например, как описано в примерах в данном документе. Например, в препаратах клеток, выделенных посредством центрифугирования в градиенте плотности в растворе Ficoll, наблюдалась утрата Т-клеток в конечном продукте с относительным увеличением количества клеток, отличных от T-клеток, особенно нежелательных B-клеток, бластных клеток и моноцитов.
Не ограничиваясь какой-либо теорией, полагают, что иммунные эффектoрные клетки, например, T-клетки, обезвоживаются во время криоконсервации, становясь более плотными, чем свежие клетки. Не ограничиваясь какой-либо теорией, также полагают, что иммунные эффектoрные клетки, например, T-клетки, остаются более плотными дольше, чем другие клетки крови, и, таким образом, легче утрачиваются при разделении в градиенте плотности Ficoll по сравнению с другими клетками. Соответственно, не ограничиваясь какой-либо теорией, полагают, что среда с плотностью, превышающей плотность Ficoll, будет обеспечивать улучшенное выделение требуемых иммунных эффектoрных клеток по сравнению с Ficoll или другими средами с такой же плотностью, как у Ficoll, например, 1,077 г/мл.
В некоторых вариантах осуществления способ центрифугирования в градиенте плотности, описанный в данном документе, включает применение среды с градиентом плотности, содержащей йодиксанол. В некоторых вариантах осуществления среда с градиентом плотности содержит приблизительно 60% йодиксанола в воде.
В некоторых вариантах осуществления способ центрифугирования в градиенте плотности, описанный в данном документе, включает применение среды с градиентом плотности, характеризующейся плотностью, превышающей плотность Ficoll. В некоторых вариантах осуществления способ центрифугирования в градиенте плотности, описанный в данном документе, включает применение среды с градиентом плотности, характеризующейся плотностью более 1,077 г/мл, например, более 1,077 г/мл, более 1,1 г/мл, более 1,15 г/мл, более 1,2 г/мл, более 1,25 г/мл, более 1,3 г/мл, более 1,31 г/мл. В некоторых вариантах осуществления среда с градиентом плотности характеризуется плотностью, составляющей приблизительно 1,32 г/мл.
Дополнительные варианты осуществления центрифугирования в градиенте плотности описаны на страницах 51-53 в WO 2017/117112, которая включена в данный документ посредством ссылки во всей своей полноте.
Обогащение посредством отбора
В данном документе предусмотрены способы отбора конкретных клеток для обеспечения улучшения обогащения требуемыми иммунными эффектoрными клетками, подходящими для экспрессии CAR. В некоторых вариантах осуществления отбор включает положительный отбор, например, отбор по требуемым иммунным эффектoрным клеткам. В некоторых вариантах осуществления отбор включает отрицательный отбор, например, отбор по нежелательным клеткам, например, удаление нежелательных клеток. В ряде вариантов осуществления способы положительного или отрицательного отбора, описанные в данном документе, проводят в условиях потока, например, с помощью проточного устройства, например, проточного устройства, описанного в данном документе. Иллюстративные способы положительного и отрицательного отбора описаны на страницах 53-57 в WO 2017/117112, которая включена в данный документ посредством ссылки во всей своей полноте. Способы отбора можно проводить в условиях потока, например, с помощью проточного устройства, также называемого системой для обработки клеток, для дополнительного обогащения препарата клеток требуемыми иммунными эффектoрными клетками, например, T-клетками, подходящими для экспрессии CAR. Иллюстративные проточные устройства описаны на страницах 57-70 в WO 2017/117112, которая включена в данный документ посредством ссылки во всей своей полноте. Иллюстративные способы разделения клеток и удаления гранул описаны на страницах 70-78 в WO 2017/117112, которая включена в данный документ посредством ссылки во всей своей полноте.
Процедуры отбора не ограничиваются теми, которые описаны на страницах 57-70 в WO 2017/117112. Можно применять отрицательный отбор Т-клеток путем удаления нежелательных клеток с помощью гранул с антителами к CD19, CD14 и CD26 от Miltenyi в комбинации с колоночной технологией (CliniMACS® Plus или CliniMACS® Prodigy®) или положительный отбор Т-клеток с помощью комбинации гранул с антителами к CD4 и CD8 от Miltenyi и колоночной технологии (CliniMACS® Plus или CliniMACS® Prodigy®). В качестве альтернативы можно применять бесколоночную технологию с гранулами с высвобождаемыми антителами к CD3 (GE Healthcare).
Кроме того, можно также применять технологии без использования гранул, такие как устройства ThermoGenesis серии X.
Пути клинического применения
Все способы в данном документе можно осуществлять согласно клиническим стандартам надлежащей производственной практики (cGMP).
Способы можно применять для очистки, обогащения, сбора, промывания, концентрирования клеток или для замены сред для клеток, в частности в ходе сбора необработанных исходных материалов (в частности клеток) в начале способа изготовления, а также в ходе способа изготовления для отбора или размножения клеток для клеточной терапии.
Клетки могут включать любое множество клеток. Клетки могут относиться к одному и тому же типу клеток или представлять собой смесь типов клеток. Кроме того, клетки могут быть получены от одного донора, такого как аутологичный донор или один аллогенный донор, для клеточной терапии. Клетки можно получать от пациентов с помощью, например, лейкафереза или афереза. Клетки могут включать T-клетки, например, могут включать популяцию, которая содержит более 50% T-клеток, более 60% T-клеток, более 70% T-клеток, более 80% T-клеток или 90% T-клеток.
Способы отбора могут быть особенно применимыми для отбора клеток перед культивированием и размножением. Например, можно применять парамагнитные частицы, покрытые антителами к CD3 и/или к CD28, для отбора T-клеток для размножения или для введения нуклеиновой кислоты, кодирующей химерный антигенный рецептор (CAR) или другой белок. Такой способ используют для получения T-клеток CTL019 для лечения острого лимфобластного лейкоза (ALL).
Способы и модули для удаления гранул, раскрытые в данном документе, могут быть особенно применимыми при изготовлении клеток для клеточной терапии, например, при очистке клеток до или после культивирования и размножения. Например, парамагнитные частицы, покрытые антителами к CD3 и/или к CD28, можно применять для селективного размножения T-клеток, например, тех T-клеток, которые являются или будут модифицированными посредством введения нуклеиновой кислоты, кодирующей химерный антигенный рецептор (CAR) или другой белок, таким образом, чтобы CAR экспрессировался T-клетками. В ходе изготовления таких T-клеток способы или модули для удаления гранул можно применять для отделения T-клеток от парамагнитных частиц. Такой способ или модуль для удаления гранул применяют для получения, например, T-клеток CTL019 для лечения острого лимфобластного лейкоза (ALL).
В одном таком способе, проиллюстрированном в данном документе в качестве примера, клетки, например, T-клетки, собирают у донора (например, пациента, подлежащего лечению с помощью продукта на основе аутологичных Т-клеток с химерным антигенным рецептором) посредством афереза (например, лейкафереза). Собранные клетки затем можно необязательно очищать, например, с помощью стадии элютриации или с помощью положительного или отрицательного отбора клеток-мишеней (например, T-клеток). Парамагнитные частицы, например, парамагнитные частицы, покрытые антителами к CD3/антителами к CD28, можно затем добавлять к популяции клеток для размножения T-клеток. Способ также может включать стадию трансдукции, где в клетку вводят нуклеиновую кислоту, кодирующую один или несколько требуемых белков, например, CAR, например, CAR, который нацеливается на CD19. Нуклеиновая кислота может быть введена в лентивирусный вектор. Клетки, например, клетки, трансдуцированные лентивирусным вектором, затем можно размножать в течение периода нескольких дней, например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или больше дней, например, в присутствии подходящей среды. После размножения можно применять способы/модули для удаления гранул, раскрытые в данном документе, для отделения требуемых T-клеток от парамагнитных частиц. Способ может включать одну или несколько стадий удаления гранул согласно способам по настоящему изобретению. Клетки, от которых были удалены гранулы, затем можно составлять для введения пациенту. Примеры CAR-T-клеток и их изготовление дополнительно описаны, например, в WO2012/079000, которая включена в данный документ посредством ссылки во всей своей полноте. Системы и способы по настоящему изобретению можно применять для любых способов разделения клеток/очистки клеток/удаления гранул от клеток, описанных в WO2012/079000 или связанных с ней заявках. Дополнительные способы изготовления CAR-T описаны, например, в WO2016109410 и WO2017117112, которые включены в данный документ посредством ссылки во всей своей полноте.
Системы и способы, описанные в данном документе, могут аналогичным образом обеспечивать благоприятный эффект для других продуктов для клеточной терапии, обеспечивая уменьшение потерь требуемых клеток, уменьшение степени повреждения клеток и более надежное удаление магнитных и любых непарамагнитных частиц от клеток с меньшим воздействием химических средств или его отсутствием по сравнению с традиционными системами и способами.
Хотя только иллюстративные варианты осуществления настоящего изобретения конкретно описаны выше, будет понятно, что возможны модификации и видоизменения таких примеров без отступления от сущности и подразумеваемого объема настоящего изобретения. Например, магнитные модули и системы, содержащие их, можно организовывать и применять в разнообразных конфигурациях в дополнение к тем, что описаны. Кроме этого, также можно использовать немагнитные модули. Кроме того, системы и способы могут включать дополнительные компоненты и стадии, которые не описаны конкретно в данном документе. Например, способы могут включать первичную обработку, где жидкость сначала вносят в компонент для удаления пузырей и уменьшения противодействия движению суспензии клеток или буфера. Кроме того, варианты осуществления могут включать только часть систем, описанных в данном документе, для применения со способами, описанными в данном документе. Например, варианты осуществления могут относиться к одноразовым модулям, рукавам и т. д., применимым внутри неодноразового оборудования с образованием целостной системы, способной отделять клетки или удалять гранулы от клеток с получением продукта на основе клеток.
Дополнительные способы и процессы изготовления, которые можно применять в комбинации с настоящим изобретением, были описаны в уровне техники. Например, на страницах 86-91 в WO 2017/117112 описаны улучшенные стадии промывания и улучшенный способ изготовления.
Источники иммунных эффекторных клеток
В этом разделе предусмотрены дополнительные способы или стадии для получения исходного образца, содержащего требуемые иммунные эффекторные клетки, выделения и обработки требуемых иммунных эффекторных клеток, например, T-клеток, и удаления нежелательных материалов, например, нежелательных клеток. Дополнительные способы или стадии, описанные в этом разделе, можно применять в комбинации с любым из элютриации, центрифугирования в градиенте плотности, отбора в условиях потока или улучшенной стадии промывания, описанных в предыдущих разделах.
Источник клеток, например, T-клеток или естественных клеток-киллеров (NK), можно получить от субъекта. Примеры субъектов включают людей, обезьян, шимпанзе, собак, кошек, мышей, крыс и их трансгенные формы. Т-клетки можно получить из ряда источников, в том числе из мононуклеарных клеток периферической крови, костного мозга, ткани лимфатических узлов, пуповинной крови, ткани тимуса, ткани из очага инфекции, асцита, плеврального выпота, ткани селезенки и опухолей.
В некоторых вариантах осуществления настоящего изобретения иммунные эффекторные клетки, например, Т-клетки, можно получать из единицы крови, собранной у субъекта, с помощью любого количества методик, известных специалисту в данной области, и любого из способов, раскрытых в данном документе, при любой комбинации их стадий. В некоторых вариантах осуществления клетки из циркулирующей крови организма индивидуума получают путем афереза. Продукт афереза обычно содержит лимфоциты, в том числе Т-клетки, моноциты, гранулоциты, В-клетки, другие ядросодержащие лейкоциты, эритроциты и тромбоциты. В некоторых вариантах осуществления клетки, собранные путем афереза, можно промыть, чтобы удалить фракцию плазмы крови и необязательно поместить клетки в соответствующий буфер или среду для последующих стадий обработки. В некоторых вариантах осуществления клетки промывают фосфатно-солевым буферным раствором (PBS). В некоторых вариантах осуществления в растворе для промывания отсутствует кальций и может отсутствовать магний или могут отсутствовать многие, если не все, двухвалентные катионы. В некоторых вариантах осуществления клетки промывают с использованием улучшенной стадии промывания, описанной в данном документе.
Начальные стадии активации в отсутствие кальция могут приводить к усиленной активации. Как будет очевидно для специалистов средней квалификации в данной области, стадию промывания можно осуществлять посредством способов, известных специалистам в данной области, как, например, с помощью полуавтоматической "проточной" центрифуги (например, устройства для обработки клеток Cobe 2991, CytoMate™ от Baxter или Cell Saver 5 от Haemonetics), Cell Saver Elite от Haemonetics (Sepax или Sefia от GE Healthcare) или устройства, в котором используется технология фильтрации на вращающейся мембране (LOVO от Fresenius Kabi), в соответствии с инструкциями производителя. После промывания клетки можно ресуспендировать в различных биосовместимых буферах, таких как, например, PBS, не содержащий Ca и не содержащий Mg, PlasmaLyte A, PBS-EDTA, дополненный человеческим сывороточным альбумином (HSA), или другой солевой раствор с буфером или без него. В качестве альтернативы нежелательные компоненты из образца, полученного путем афереза, можно удалить, и клетки можно ресуспендировать непосредственно в культуральной среде.
В некоторых вариантах осуществления требуемые иммунные эффекторные клетки, например, Т-клетки, выделяют из лимфоцитов периферической крови путем лизиса эритроцитов и истощения по моноцитам, например, посредством центрифугирования в градиенте PERCOLL™ или посредством противоточного элютриационного центрифугирования.
Способы, описанные в данном документе, могут включать, например, отбор конкретной субпопуляции иммунных эффекторных клеток, например, Т-клеток, которая представляет собой популяцию клеток, истощенную по регуляторным Т-клеткам, например, с истощением по CD25+ клеткам или истощением по CD25высокий клеткам, с помощью, например, методики отрицательного отбора, например, описанной в данном документе. В некоторых вариантах осуществления популяция клеток, истощенная по регуляторным T-клеткам, содержит менее 30%, 25%, 20%, 15%, 10%, 5%, 4%, 3%, 2%, 1% CD25+ клеток или CD25высокий клеток.
В некоторых вариантах осуществления регуляторные T-клетки, например, CD25+ T-клетки или CD25высокий Т-клетки, удаляют из популяции с помощью антитела к CD25 или его фрагмента или CD25-связывающего лиганда, например, IL-2. В некоторых вариантах осуществления антитело к CD25 или его фрагмент или CD25-связывающий лиганд конъюгированы с субстратом, например, гранулой, или иным образом покрывают субстрат, например, гранулу. В некоторых вариантах осуществления антитело к CD25 или его фрагмент конъюгированы с субстратом, описанным в данном документе.
В некоторых вариантах осуществления регуляторные T-клетки, например, CD25+ T-клетки или CD25высокий Т-клетки, удаляют из популяции с помощью реагента для истощения по CD25 от Miltenyi™. В некоторых вариантах осуществления соотношение клеток и реагента для истощения по CD25 составляет 1e7 клеток на 20 мкл, или 1e7 клеток на 15 мкл, или 1e7 клеток на 10 мкл, или 1e7 клеток на 5 мкл, или 1e7 клеток на 2,5 мкл, или 1e7 клеток на 1,25 мкл. В некоторых вариантах осуществления, например, для регуляторных Т-клеток используют более 500 миллионов клеток/мл. В некоторых вариантах осуществления используют концентрацию клеток, составляющую 600, 700, 800 или 900 миллионов клеток/мл.
В некоторых вариантах осуществления популяция иммунных эффекторных клеток, подлежащая истощению, содержит приблизительно 6×109 CD25+ T-клеток. В некоторых вариантах осуществления популяция иммунных эффекторных клеток, подлежащая истощению, содержит от приблизительно 1×109 до 1×1010 CD25+ Т-клеток, включая любое целочисленное значение в этом промежутке. В некоторых вариантах осуществления полученная в результате популяция, истощенная по регуляторным T-клеткам, содержит 2×109 регуляторных T-клеток, например, CD25+ клеток или CD25высокий клеток, или меньше (например, 1×109, 5×108, 1×108, 5×107, 1×107 или меньше регуляторных T-клеток).
В некоторых вариантах осуществления регуляторные T-клетки, например, CD25+ клетки или CD25высокий клетки, удаляют из популяции с помощью системы CliniMACS с набором трубок для истощения, таким как, например, трубки 162-01. В некоторых вариантах осуществления система CliniMACS работает с установленными параметрами истощения, например, такими как DEPLETION2.1.
Не ограничиваясь конкретной теорией, полагают, что при уменьшении уровня отрицательных регуляторов иммунных клеток (например, уменьшении количества нежелательных иммунных клеток, например, Treg-клеток) у субъекта до афереза или в ходе изготовления продукта на основе клеток, экспрессирующих CAR, значительно снижается риск рецидива у субъекта. Например, способы истощения по Treg-клеткам известны из уровня техники. Способы уменьшения количества Treg-клеток включают без ограничения применение циклофосфамида, антитела к GITR (антитела к GITR, описанного в данном документе), реагента для истощения по CD25 и их комбинаций.
В некоторых вариантах осуществления способы изготовления включают снижение количества (например, истощение популяции) Treg-клеток перед изготовлением клетки, экспрессирующей CAR. Например, способы изготовления включают приведение образца, например, образца, полученного путем афереза, в контакт с антителом к GITR и/или антителом к CD25 (или его фрагментом или CD25-связывающим лигандом), например, для истощения по Treg-клеткам перед изготовлением продукта на основе клеток (например, T-клеток, NK-клеток), экспрессирующих CAR.
Не ограничиваясь конкретной теорией, полагают, что при уменьшении уровня отрицательных регуляторов иммунных клеток (например, уменьшении количества нежелательных иммунных клеток, например, Treg-клеток) у субъекта до афереза или в ходе изготовления продукта на основе клеток, экспрессирующих CAR, может снижаться риск рецидива у субъекта. В некоторых вариантах осуществления субъект получает предварительное лечение с помощью одного или нескольких средств терапии, которые обеспечивают снижение количества Treg-клеток, перед сбором клеток для изготовления продукта на основе клеток, экспрессирующих CAR, за счет чего обеспечивается снижение риска рецидива у субъекта при лечении с помощью клеток, экспрессирующих CAR. В некоторых вариантах осуществления способы уменьшения количества Treg-клеток включают без ограничения введение субъекту одного или нескольких из циклофосфамида, антитела к GITR, реагента для истощения по CD25 или их комбинации. В некоторых вариантах осуществления способы уменьшения количества Treg-клеток включают без ограничения введение субъекту одного или нескольких из циклофосфамида, антитела к GITR, реагента для истощения по CD25 или их комбинации. Введение одного или нескольких из циклофосфамида, антитела к GITR, реагента для истощения по CD25 или их комбинации может происходить до, во время или после инфузии продукта на основе клеток, экспрессирующих CAR. Введение одного или нескольких из циклофосфамида, антитела к GITR, реагента для истощения по CD25 или их комбинации может происходить до, во время или после инфузии продукта на основе клеток, экспрессирующих CAR.
В некоторых вариантах осуществления способы изготовления включают снижение количества (например, истощение популяции) Treg-клеток перед изготовлением клетки, экспрессирующей CAR. Например, способы изготовления включают приведение образца, например, образца, полученного путем афереза, в контакт с антителом к GITR и/или антителом к CD25 (или его фрагментом или CD25-связывающим лигандом), например, для истощения по Treg-клеткам перед изготовлением продукта на основе клеток (например, T-клеток, NK-клеток), экспрессирующих CAR.
В некоторых вариантах осуществления субъект получает предварительное лечение с помощью циклофосфамида перед сбором клеток для изготовления продукта на основе клеток, экспрессирующих CAR, за счет чего обеспечивается снижение риска рецидива у субъекта при лечении с помощью клеток, экспрессирующих CAR (например, лечении с помощью CTL019). В некоторых вариантах осуществления субъект получает предварительное лечение с помощью антитела к GITR перед сбором клеток для изготовления продукта на основе клеток (например, T-клеток или NK-клеток), экспрессирующих CAR, за счет чего обеспечивается снижение риска рецидива у субъекта при лечении с помощью клеток, экспрессирующих CAR.
В некоторых вариантах осуществления способ изготовления клеток, экспрессирующих CAR (например, T-клеток, NK-клеток), модифицируют для обеспечения истощения по Treg-клеткам перед изготовлением продукта на основе клеток (например, T-клеток, NK-клеток), экспрессирующих CAR (например, продукта CTL019). В некоторых вариантах осуществления истощение по CD25 применяют для истощения по Treg-клеткам перед изготовлением продукта на основе клеток (например, T-клеток или NK-клеток), экспрессирующих CAR (например, продукта CTL019).
В некоторых вариантах осуществления популяция клеток, которая должна быть удалена, не представляет собой ни регуляторные Т-клетки, ни опухолевые клетки, а представляет собой клетки, которые в иных отношениях отрицательно влияют на размножение и/или функцию CAR-T-клеток, например, клетки, экспрессирующие CD14, CD11b, CD33, CD15 или другие маркеры, экспрессируемые потенциально иммуносупрессорными клетками. В некоторых вариантах осуществления предполагается, что такие клетки удаляют одновременно с регуляторными Т-клетками и/или опухолевыми клетками, или после указанного истощения, или в другом порядке.
Способы, описанные в данном документе, могут включать более одной стадии отбора, например, более одной стадии истощения. Обогащение популяции Т-клеток путем отрицательного отбора можно осуществлять, например, с помощью комбинации антител, направленных на поверхностные маркеры, уникальные для клеток, подвергаемых отрицательному отбору. Один из способов представляет собой сортировку и/или отбор клеток с помощью отрицательной магнитной иммуноадгезии или проточной цитометрии, в которых используется коктейль моноклональных антител, направленных на маркеры клеточной поверхности, присутствующие на клетках, подвергаемых отрицательному отбору. Например, для обогащения CD4+ клетками путем отрицательного отбора коктейль моноклональных антител может содержать антитела к CD14, CD20, CD11b, CD16, HLA-DR и CD8.
Способы, описанные в данном документе, могут дополнительно включать удаление из популяции клеток, экспрессирующих опухолевый антиген, например, опухолевый антиген, который не включает CD25, например, CD19, CD30, CD38, CD123, CD20, CD14 или CD11b, за счет чего обеспечивается получение популяции, истощенной по регуляторным Т-клеткам, например, истощенной по CD25+ клеткам или истощенной по CD25высокий клеткам и истощенной по клеткам с опухолевым антигеном, которая подходит для экспрессии CAR, например, CAR, описанного в данном документе. В некоторых вариантах осуществления клетки, экспрессирующие опухолевый антиген, удаляют одновременно с регуляторными Т-клетками, например, CD25+ клетками или CD25высокий клетками. Например, антитело к CD25 или его фрагмент и антитело к опухолевому антигену или его фрагмент могут быть присоединены к одному и тому же субстрату, например, грануле, который можно использовать для удаления клеток, или антитело к CD25 или его фрагмент или антитело к опухолевому антигену или его фрагмент могут быть присоединены к отдельным гранулам, смесь которых можно использовать для удаления клеток. В других вариантах осуществления удаление регуляторных Т-клеток, например, CD25+ клеток или CD25высокий клеток, и удаление клеток, экспрессирующих опухолевый антиген, является последовательным и может происходить, например, в любом порядке.
Также предусмотрены способы, которые включают удаление из популяции клеток, экспрессирующих ингибитор контрольной точки иммунного ответа, например, ингибитор контрольной точки иммунного ответа, описанный в данном документе, например, одного или нескольких из PD1+ клеток, LAG3+ клеток и TIM3+ клеток, за счет чего обеспечивается получение популяции, истощенной по регуляторным Т-клеткам, например, истощенной по CD25+ клеткам и истощенной по клеткам с ингибитором контрольной точки иммунного ответа, например, истощенной по PD1+, LAG3+ и/или TIM3+ клеткам. Иллюстративные ингибиторы контрольных точек иммунного ответа включают PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулу MHC I класса, молекулу MHC II класса, GAL9, аденозин и TGF (например, TGF-бета), например, как описано в данном документе. В некоторых вариантах осуществления клетки, экспрессирующие ингибитор контрольной точки иммунного ответа, удаляют одновременно с регуляторными Т-клетками, например, CD25+ клетками или CD25высокий клетками. Например, антитело к CD25 или его фрагмент и антитело к ингибитору контрольной точки иммунного ответа или его фрагмент могут быть присоединены к одной и той же грануле, которую можно использовать для удаления клеток, или антитело к CD25 или его фрагмент и антитело к ингибитору контрольной точки иммунного ответа или его фрагмент могут быть присоединены к отдельным гранулам, смесь которых можно использовать для удаления клеток. В других вариантах осуществления удаление регуляторных Т-клеток, например, CD25+ клеток или CD25высокий клеток, и удаление клеток, экспрессирующих ингибитор контрольной точки иммунного ответа, является последовательным и может происходить, например, в любом порядке.
Способы, описанные в данном документе, могут включать стадию положительного отбора. Например, Т-клетки можно выделять путем инкубирования с гранулами, конъюгированными с антителами к CD3/антителами к CD28 (например, 3×28), такими как Dynabeads® M-450 CD3/CD28 T, в течение периода времени, достаточного для положительного отбора требуемых Т-клеток. В некоторых вариантах осуществления период времени составляет приблизительно 30 минут. В некоторых вариантах осуществления период времени находится в диапазоне от 30 минут до 36 часов или больше и включает все целочисленные значения в этом промежутке. В некоторых вариантах осуществления период времени составляет по меньшей мере 1, 2, 3, 4, 5 или 6 часов. В некоторых вариантах осуществления период времени составляет от 10 до 24 часов, например, 24 часа. Более длительное время инкубирования можно использовать для выделения Т-клеток в любой ситуации, при которой Т-клеток немного по сравнению с другими типами клеток, как, например, при выделении лимфоцитов, инфильтрирующих опухоль (TIL), из опухолевой ткани или у индивидуумов с ослабленным иммунитетом. Кроме того, при использовании более длительного времени инкубирования может увеличиваться эффективность захвата CD8+ Т-клеток. Таким образом, просто сокращая или удлиняя время, в течение которого Т-клетки имеют возможность связываться с гранулами с антителами к CD3/CD28, и/или увеличивая или уменьшая соотношение гранул и Т-клеток (как дополнительно описано в данном документе), можно предпочтительно осуществлять положительный или отрицательный отбор субпопуляций Т-клеток в начале культивирования или в другие моменты времени в ходе процесса. Кроме того, путем увеличения или уменьшения соотношения антител к CD3 и/или антител к CD28 на гранулах или другой поверхности можно предпочтительно осуществлять положительный или отрицательный отбор субпопуляций Т-клеток в начале культивирования или в другие необходимые моменты времени.
В некоторых вариантах осуществления можно выбрать популяцию Т-клеток, которые экспрессируют один или несколько из IFNγ, TNFα, IL-17A, IL-2, IL-3, IL-4, GM-CSF, IL-10, IL-13, гранзима В и перфорина или других соответствующих молекул, например, других цитокинов. Способы скрининга в отношении клеточной экспрессии можно определить, например, посредством способов, описанных в публикации согласно PCT № WO 2013/126712.
Для выделения необходимой популяции клеток путем положительного или отрицательного отбора можно варьировать концентрацию клеток и поверхность (например, частицы, такие как гранулы). В определенных аспектах может быть необходимым значительно уменьшить объем, в котором гранулы и клетки смешиваются друг с другом (например, увеличить концентрацию клеток), чтобы обеспечить максимальный контакт клеток и гранул. Например, в некоторых вариантах осуществления используют концентрацию, составляющую 10 миллиардов клеток/мл, 9 миллиардов/мл, 8 миллиардов/мл, 7 миллиардов/мл, 6 миллиардов/мл или 5 миллиардов/мл. В некоторых вариантах осуществления используют концентрацию, составляющую 1 миллиард клеток/мл. В некоторых вариантах осуществления используют концентрацию клеток, составляющую от 75, 80, 85, 90, 95 или 100 миллионов клеток/мл. В некоторых вариантах осуществления можно использовать концентрации, составляющие 125 или 150 миллионов клеток/мл.
Использование высоких концентраций может приводить к увеличению выхода клеток, активации клеток и размножения клеток. Кроме того, использование высоких концентраций клеток позволяет более эффективно захватывать клетки, которые могут характеризоваться слабой экспрессией антигенов-мишеней, представляющих интерес, такие как CD28-отрицательные Т-клетки, или клетки из образцов, в которых присутствует много опухолевых клеток (например, лейкозной крови, опухолевой ткани и т. д.). Такие популяции клеток могут иметь терапевтическую ценность, и может потребоваться их получение. Например, использование высокой концентрации клеток позволяет проводить более эффективный отбор CD8+ Т-клеток, которые обычно характеризуются более слабой экспрессией CD28.
В некоторых вариантах осуществления может потребоваться использование более низких концентраций клеток. Благодаря значительному разбавлению смеси Т-клеток и поверхности (например, частиц, таких как гранулы) взаимодействие между частицами и клетками сводится к минимуму. При этом отбираются клетки, которые экспрессируют необходимые антигены, подлежащие связыванию с частицами, в высоких количествах. Например, CD4+ T-клетки экспрессируют CD28 на более высоких уровнях и захватываются в слабых концентрациях более эффективно, чем CD8+ T-клетки. В некоторых вариантах осуществления используемая концентрация клеток составляет 5×106/мл. В некоторых вариантах осуществления используемая концентрация может составлять от приблизительно 1×105/мл до 1×106/мл, включая любое целочисленное значение в этом промежутке.
В некоторых вариантах осуществления клетки можно инкубировать на ротаторе в течение различных промежутков времени с различными скоростями при 2-10°C либо при комнатной температуре.
В некоторых вариантах осуществления множество иммунных эффекторных клеток в популяции не экспрессируют диацилглицеринкиназу (DGK), например, характеризуются дефицитом DGK. В некоторых вариантах осуществления множество иммунных эффекторных клеток в популяции не экспрессируют Ikaros, например, характеризуются дефицитом Ikaros. В некоторых вариантах осуществления множество иммунных эффекторных клеток в популяции не экспрессируют DGK и Ikaros, например, характеризуются дефицитом как DGK, так и Ikaros.
Т-клетки для стимуляции также можно замораживать после стадии промывания. Не ограничиваясь какой-либо теорией, полагают, что стадия замораживания и последующего размораживания обеспечивает получение более однородного продукта за счет удаления гранулоцитов и в некоторой степени моноцитов из популяции клеток. После стадии промывания, на которой удаляют плазму крови и тромбоциты, клетки можно суспендировать в замораживающем растворе. Хотя многие замораживающие растворы и параметры для замораживания известны из уровня техники и будут применимыми в данном случае, один способ предусматривает использование PBS, содержащего 20% DMSO и 8% человеческий сывороточный альбумин, или культуральных сред, содержащих 10% декстран 40 в 5% декстрозе, 20% человеческий сывороточный альбумин и 7,5% DMSO или 31,25% PlasmaLyte-А, 31,25% раствор с 5% декстрозой в 0,45% NaCl, 10% декстран 40 в 5% декстрозе, 20% человеческий сывороточный альбумин и 7,5% DMSO, или других подходящих сред для замораживания клеток, содержащих, например, Hespan и PlasmaLyte A, и затем клетки замораживают до -80°C со скоростью 1° в минуту и хранят в паровой фазе в резервуаре для хранения жидкого азота. Можно применять другие способы контролируемого замораживания, а также неконтролируемого замораживания немедленно при -20°C или в жидком азоте.
В некоторых вариантах осуществления криоконсервированные клетки размораживают и промывают, как описано в данном документе, и оставляют на один час при комнатной температуре перед активацией посредством способов по настоящему изобретению.
В контексте настоящего изобретения также предусмотрен сбор образцов крови или продукта афереза у субъекта в период времени до того, как могут понадобиться размножившиеся клетки, описанные в данном документе. Ввиду этого источник клеток, подлежащих размножению, можно собирать в любой необходимый момент времени, и необходимые клетки, такие как Т-клетки, можно выделять и замораживать для последующего применения в терапии с использованием иммунных эффекторных клеток для любых заболеваний или состояний, при которых будет благоприятной терапия с использованием иммунных эффекторных клеток, таких как описанные в данном документе. В некоторых вариантах осуществления образец крови или образец, полученный путем афереза, берут у в целом здорового субъекта. В некоторых вариантах осуществления образец крови или образец, полученный путем афереза, берут у в целом здорового субъекта, для которого существует риск развития заболевания, но у которого заболевание еще не развилось, и клетки, представляющие интерес, выделяют и замораживают для последующего применения. В некоторых вариантах осуществления Т-клетки можно размножать, замораживать и использовать позднее. В некоторых вариантах осуществления образцы собирают у пациента вскоре после диагностирования конкретного заболевания, описанного в данном документе, однако до проведения каких-либо видов лечения. В некоторых вариантах осуществления клетки выделяют из образца крови или продукта афереза у субъекта до осуществления какого-либо количества соответствующих способов лечения, в том числе без ограничения лечения такими средствами, как натализумаб, эфализумаб, противовирусные средства, химиотерапия, лучевая терапия, иммуносупрессивные средства, такие как циклоспорин, азатиоприн, метотрексат, микофенолат и FK506, антитела или другие иммунодеструктивные средства, такие как CAMPATH, антитела к CD3, цитоксан, флударабин, циклоспорин, FK506, рапамицин, микофеноловая кислота, стероиды, FR901228 и облучение.
В некоторых вариантах осуществления настоящего изобретения Т-клетки получают от пациента непосредственно после лечения, при котором у субъекта остаются функциональные Т-клетки. В данном отношении наблюдалось, что после определенных видов лечения рака, в частности видов лечения лекарственными средствами, которые повреждают иммунную систему, вскоре после лечения в течение периода, когда пациенты обычно восстанавливаются после лечения, качество полученных Т-клеток может быть оптимальным или улучшенным в отношении их способности размножаться ex vivo. Аналогично, после манипуляций ex vivo с применением способов, описанных в данном документе, эти клетки могут находиться в состоянии, предпочтительном для улучшенного приживления и размножения in vivo. Таким образом, в контексте настоящего изобретения рассматривается сбор клеток крови, в том числе Т-клеток, дендритных клеток или других клеток гемопоэтической линии дифференцировки, во время этой фазы восстановления. Кроме того, в некоторых вариантах осуществления можно использовать мобилизацию (например, мобилизацию с помощью GM-CSF) и режимы кондиционирования для создания у субъекта состояния, благоприятного для репопуляции, рециркуляции, регенерации и/или размножения определенных типов клеток, особенно в течение определенного временного окна после терапии. Иллюстративные типы клеток включают Т-клетки, В-клетки, дендритные клетки и другие клетки иммунной системы.
В некоторых вариантах осуществления иммунные эффекторные клетки, экспрессирующие молекулу CAR, например, молекулу CAR, описанную в данном документе, получают от субъекта, который получил низкую дозу, усиливающую иммунный ответ, ингибитора mTOR. В некоторых вариантах осуществления популяцию иммунных эффекторных клеток, например, Т-клеток, подлежащих конструированию таким образом, чтобы они экспрессировали CAR, собирают спустя достаточное количество времени или после достаточного введения низкой дозы, усиливающей иммунный ответ, ингибитора mTOR, так чтобы уровень PD1-отрицательных иммунных эффекторных клеток, например, Т-клеток, или соотношение PD1-отрицательных иммунных эффекторных клеток, например, Т-клеток, и PD1-положительных иммунных эффекторных клеток, например, Т-клеток, у субъекта или в материале, собранном у субъекта, по меньшей мере транзиентно были повышены.
В других вариантах осуществления популяция иммунных эффекторных клеток, например, Т-клеток, которые были сконструированы или будут сконструированы таким образом, чтобы они экспрессировали CAR, может быть обработана ex vivo путем приведения ее в контакт с таким количеством ингибитора mTOR, которое обеспечивает увеличение количества PD1-отрицательных иммунных эффекторных клеток, например, Т-клеток, или обеспечивает увеличение соотношения PD1-отрицательных иммунных эффекторных клеток, например, Т-клеток, и PD1-положительных иммунных эффекторных клеток, например, Т-клеток.
Принято считать, что в способах применения можно использовать условия для культуральных сред, включающие человеческую сыворотку крови AB в концентрации 5% или меньше, например, 2%, и использовать известные условия и составы культуральных сред, например, описанные в Smith et al., "Ex vivo expansion of human T cells for adoptive immunotherapy using the novel Xeno-free CTS™ Immune Cell Serum Replacement" Clinical & Translational Immunology (2015) 4, e31; doi:10.1038/cti.2014.31.
В некоторых вариантах осуществления в способах применения можно использовать условия среды, включающие по меньшей мере приблизительно 0,1%, 0,5%, 1,0%, 1,5%, 2%, 2,5%, 3%, 3,5%, 4%, 4,5%, 5%, 6%, 7%, 8%, 9% или 10% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 0,5%-5%, приблизительно 0,5%-4,5%, приблизительно 0,5%-4%, приблизительно 0,5%-3,5%, приблизительно 0,5%-3%, приблизительно 0,5%-2,5%, приблизительно 0,5%-2%, приблизительно 0,5%-1,5%, приблизительно 0,5%-1,0%, приблизительно 1,0%-5%, приблизительно 1,5%-5%, приблизительно 2%-5%, приблизительно 2,5%-5%, приблизительно 3%-5%, приблизительно 3,5%-5%, приблизительно 4%-5% или приблизительно 4,5%-5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 0,5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 0,5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 1% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 1,5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 2% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 2,5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 3% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 3,5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 4% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 4,5% сыворотки. В некоторых вариантах осуществления среда содержит приблизительно 5% сыворотки. В некоторых вариантах осуществления сыворотка предусматривает человеческую сыворотку, например, человеческую сыворотку AB. В некоторых вариантах осуществления сыворотка представляет собой человеческую сыворотку, которой предоставили возможность естественным образом свернуться после сбора, например, сыворотку после коагуляции (OTC). В некоторых вариантах осуществления сыворотка представляет собой человеческую сыворотку, полученную из плазмы крови. Сыворотку, полученную из плазмы крови, можно получить путем удаления фибрина из объединенной человеческой плазмы крови, собранной в присутствии антикоагулянта, например, цитрата натрия.
В некоторых вариантах осуществления в способах применения можно использовать условия для культуральных сред, включающие бессывороточную среду. В некоторых вариантах осуществления бессывороточная среда представляет собой OpTmizer™ CTS™ (LifeTech), ImmunoCult™ XF (Stemcell Technologies), CellGro™ (CellGenix), TexMacs™ (Miltenyi), Stemline™ (Sigma), Xvivo15™ (Lonza), PrimeXV® (Irvine Scientific) или StemXVivo® (R&D Systems). Бессывороточную среду можно дополнять имитатором сыворотки, таким как ICSR (заменитель сыворотки для иммунных клеток) от LifeTech. Уровень имитатора сыворотки (например, ICSR) может составлять, например, вплоть до 5%, например, приблизительно 1%, 2%, 3%, 4% или 5%. В некоторых вариантах осуществления бессывороточную среду можно дополнять сывороткой, например, человеческой сывороткой, например, человеческой сывороткой AB. В некоторых вариантах осуществления сыворотка представляет собой человеческую сыворотку, которой предоставили возможность естественным образом свернуться после сбора, например, сыворотку после коагуляции (OTC). В некоторых вариантах осуществления сыворотка представляет собой человеческую сыворотку, полученную из плазмы крови. Сыворотку, полученную из плазмы крови, можно получить путем удаления фибрина из объединенной человеческой плазмы крови, собранной в присутствии антикоагулянта, например, цитрата натрия.
В некоторых вариантах осуществления Т-клетки в популяции характеризуются дефицитом диацилглицеринкиназы (DGK). Клетки с дефицитом DGK включают клетки, которые не экспрессируют DGK в виде РНК или белка или характеризуются сниженной или ингибированной активностью DGK. Клетки с дефицитом DGK можно получить с помощью генетических подходов, например, путем введения средств для РНК-интерференции, например, siRNA, shRNA, miRNA, для снижения или предотвращения экспрессии DGK. В качестве альтернативы клетки с дефицитом DGK можно получить путем обработки ингибиторами DGK, описанными в данном документе.
В некоторых вариантах осуществления Т-клетки в популяции характеризуются дефицитом Ikaros. Клетки с дефицитом Ikaros включают клетки, которые не экспрессируют Ikaros в виде РНК или белка или характеризуются сниженной или ингибированной активностью Ikaros; клетки с дефицитом Ikaros можно получить с помощью генетических подходов, например, путем введения средств для РНК-интерференции, например, siRNA, shRNA, miRNA, для снижения или предотвращения экспрессии Ikaros. В качестве альтернативы клетки с дефицитом Ikaros можно получить путем обработки ингибиторами Ikaros, например, леналидомидом.
В ряде вариантов осуществления популяция Т-клеток характеризуется дефицитом DGK и дефицитом Ikaros, например, в ней не происходит экспрессия DGK и Ikaros или она характеризуется сниженной или ингибированной активностью DGK и Ikaros. Такие клетки с дефицитом DGK и Ikaros можно получить любым из способов, описанных в данном документе.
В некоторых вариантах осуществления NK-клетки получают от субъекта. В некоторых вариантах осуществления NK-клетки представляют собой линию NK-клеток, например, линию клеток NK-92 (Conkwest).
Аллогенные клетки, экспрессирующие CAR
В ряде вариантов осуществления, описанных в данном документе, иммунная эффекторная клетка может представлять собой аллогенную иммунную эффекторную клетку, например, Т-клетку или NK-клетку. Например, клетка может представлять собой аллогенную Т-клетку, например, аллогенную Т-клетку, в которой отсутствует экспрессия функционального Т-клеточного рецептора (TCR) и/или человеческого лейкоцитарного антигена (HLA), например, HLA I класса и/или HLA II класса.
T-клетку, в которой отсутствует функциональный TCR, можно, например, сконструировать таким образом, чтобы она вообще не экспрессировала функциональный TCR на своей поверхности, сконструировать таким образом, чтобы она не экспрессировала одну или несколько субъединиц, которые образуют функциональный TCR (например, сконструировать таким образом, чтобы она не экспрессировала (или демонстрировала сниженную экспрессию) TCR-альфа, TCR-бета, TCR-гамма, TCR-дельта, TCR-эпсилон и/или TCR-дзета), или сконструировать таким образом, чтобы она продуцировала очень небольшое количество функционального TCR на своей поверхности. В качестве альтернативы Т-клетка может экспрессировать существенно нарушенный TCR, например, посредством экспрессии мутантных или усеченных форм одной или нескольких субъединиц TCR. Термин "существенно нарушенный TCR" означает, что этот TCR не будет вызывать нежелательную иммунную реакцию у хозяина.
Т-клетку, описанную в данном документе, можно, например, сконструировать таким образом, чтобы она не экспрессировала функциональный HLA на своей поверхности. Например, Т-клетку, описанную в данном документе, можно сконструировать таким образом, чтобы экспрессия HLA, например, HLA I класса и/или HLA II класса, на поверхности клетки была подавлена. В некоторых вариантах осуществления подавление экспрессии HLA можно осуществлять путем снижения или устранения экспрессии бета-2-микроглобулина (B2M).
В некоторых вариантах осуществления в Т-клетке может отсутствовать функциональный TCR и функциональный HLA, например, HLA I класса и/или HLA II класса.
Модифицированные Т-клетки, в которых отсутствует экспрессия функционального TCR и/или HLA, можно получить любым подходящим способом, в том числе путем нокаута или нокдауна одной или нескольких субъединиц TCR или HLA. Например, можно предусмотреть нокдаун TCR и/или HLA в Т-клетке с помощью siRNA, shRNA, коротких палиндромных повторов, регулярно расположенных группами (CRISPR), эффекторной нуклеазы, подобной активаторам транскрипции (TALEN), или эндонуклеазы с "цинковыми пальцами" (ZFN).
В некоторых вариантах осуществления аллогенная клетка может представлять собой клетку, в которой не экспрессируется или экспрессируется на низких уровнях ингибирующая молекула, например, благодаря любому способу, описанному в данном документе. Например, клетка может представлять собой клетку, в которой не экспрессируется или экспрессируется на низких уровнях ингибирующая молекула, например, молекула, которая может уменьшать способность клетки, экспрессирующей CAR, осуществлять иммунный эффекторный ответ. Примеры ингибирующих молекул включают PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулу MHC I класса, молекулу MHC II класса, GAL9, аденозин и TGF (например, TGF-бета). Ингибирование ингибирующей молекулы, например, путем ингибирования на уровне ДНК, РНК или белка, может оптимизировать функциональные характеристики клетки, экспрессирующей CAR. В ряде вариантов осуществления можно применять ингибирующую нуклеиновую кислоту, например, ингибирующую нуклеиновую кислоту, например, dsRNA, например, siRNA или shRNA, короткие палиндромные повторы, регулярно расположенные группами (CRISPR), эффекторную нуклеазу, подобную активаторам транскрипции (TALEN), или эндонуклеазу с "цинковыми пальцами" (ZFN), например, описанные в данном документе.
siRNA и shRNA для ингибирования TCR или HLA
В некоторых вариантах осуществления экспрессию TCR и/или экспрессию HLA можно ингибировать с помощью siRNA или shRNA, нацеливающихся на нуклеиновую кислоту, кодирующую TCR и/или HLA, и/или ингибирующую молекулу, описанную в данном документе (например, PD1, PD-L1, PD- L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулу MHC I класса, молекулу MHC II класса, GAL9, аденозин и TGF-бета), в клетке, например, Т-клетке.
Системы экспрессии для siRNA и shRNA и иллюстративные shRNA описаны, например, в абзацах 649 и 650 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена посредством ссылки во всей своей полноте.
CRISPR для ингибирования TCR или HLA
Как используется в данном документе, "CRISPR", или "CRISPR для TCR и/или HLA", или "CRISPR для ингибирования TCR и/или HLA" относится к набору коротких палиндромных повторов, регулярно расположенных группами, или к системе, содержащей такой набор повторов. Как используется в данном документе, "Cas" относится к CRISPR-ассоциированному белку. Система "CRISPR/Cas" относится к системе, полученной из CRISPR и Cas, которую можно применять для сайленсинга или осуществления мутации гена TCR и/или HLA и/или ингибирующей молекулы, описанной в данном документе (например, PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулы MHC I класса, молекулы MHC II класса, GAL9, аденозина и TGF-бета), в клетке, например, Т-клетке.
Система CRISPR/Cas и пути ее применения описаны, например, в абзацах 651-658 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена посредством ссылки во всей своей полноте.
TALEN для ингибирования TCR и/или HLA
"TALEN", или "TALEN для HLA и/или TCR", или "TALEN для ингибирования HLA и/или TCR" относится к эффекторной нуклеазе, подобной активаторам транскрипции, представляющей собой искусственную нуклеазу, которую можно применять для редактирования гена HLA и/или TCR и/или ингибирующей молекулы, описанной в данном документе (например, PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулы MHC I класса, молекулы MHC II класса, GAL9, аденозина и TGF-бета), в клетке, например, T-клетке.
TALEN и пути их применения описаны, например, в абзацах 659-665 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена посредством ссылки во всей своей полноте.
Нуклеаза с "цинковыми пальцами" для ингибирования HLA и/или TCR
"ZFN", или "нуклеаза с "цинковыми пальцами", или "ZFN для HLA и/или TCR", или "ZFN для ингибирования HLA и/или TCR" относится к нуклеазе с "цинковыми пальцами", представляющей собой искусственную нуклеазу, которую можно применять для редактирования гена HLA и/или TCR и/или ингибирующей молекулы, описанной в данном документе (например, PD1, PD-L1, PD-L2, CTLA4, TIM3, CEACAM (например, CEACAM-1, CEACAM-3 и/или CEACAM-5), LAG3, VISTA, BTLA, TIGIT, LAIR1, CD160, 2B4, CD80, CD86, B7-H3 (CD276), B7-H4 (VTCN1), HVEM (TNFRSF14 или CD270), KIR, A2aR, молекулы MHC I класса, молекулы MHC II класса, GAL9, аденозина и TGF-бета), в клетке, например, T-клетке.
ZFN и пути их применения описаны, например, в абзацах 666-671 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена посредством ссылки во всей своей полноте.
Экспрессия теломеразы
Теломеры играют важнейшую роль в персистенции соматических клеток, и их длину поддерживает теломераза (TERT). Длина теломер в клетках CLL может быть очень короткой (Roth et al., "Significantly shorter telomeres in T-cells of patients with ZAP-70+/CD38 chronic lymphocytic leukaemia" British Journal of Haematology, 143, 383-386., August 28 2008) и может быть еще короче в изготовленных клетках, экспрессирующих CAR, например, клетках CART19, что ограничивает их потенциал размножения после адоптивного переноса пациенту. Экспрессия теломеразы может "спасти" клетки, экспрессирующие CAR, от репликативного истощения.
Не ограничиваясь какой-либо конкретной теорией, полагают, что в некоторых вариантах осуществления для терапевтической Т-клетки характерна кратковременная персистенция в организме пациента ввиду укороченных теломер в Т-клетке; соответственно, трансфекция с использованием гена теломеразы может приводить к удлинению теломер в Т-клетке и улучшению персистенции Т-клетки в организме пациента. См. Carl June, "Adoptive T cell therapy for cancer in the clinic", Journal of Clinical Investigation, 117:1466-1476 (2007). Таким образом, в некоторых вариантах осуществления иммунная эффекторная клетка, например, Т-клетка, характеризуется эктопической экспрессией субъединицы теломеразы, например, каталитической субъединицы теломеразы, например, TERT, например, hTERT. В некоторых вариантах осуществления в настоящем изобретении предусмотрен способ получения клетки, экспрессирующей CAR, включающий приведение клетки в контакт с нуклеиновой кислотой, кодирующей субъединицу теломеразы, например, каталитическую субъединицу теломеразы, например, TERT, например, hTERT. Клетку можно приводить в контакт с нуклеиновой кислотой перед приведением в контакт с конструкцией, кодирующей CAR, одновременно с этим или после этого.
Экспрессия теломеразы может быть стабильной (например, нуклеиновая кислота может интегрироваться в геном клетки) или транзиентной (например, нуклеиновая кислота не интегрируется, и уровень экспрессии снижается спустя некоторый период времени, например, через несколько дней). Стабильная экспрессия может быть достигнута путем трансфекции или трансдукции клетки с помощью ДНК, кодирующей субъединицу теломеразы и селектируемый маркер, и отбора в отношении стабильных интегрантов. В качестве альтернативы или в комбинации, стабильная экспрессия может быть достигнута посредством сайт-специфической рекомбинации, например, с помощью системы Cre/Lox или FLP/FRT.
Транзиентная экспрессия может предусматривать трансфекцию или трансдукцию с помощью нуклеиновой кислоты, например, ДНК или РНК, такой как мРНК. В некоторых вариантах осуществления транзиентная трансфекция мРНК позволяет избежать генетической нестабильности, иногда связанной со стабильной трансфекцией TERT. Транзиентная экспрессия экзогенной теломеразной активности описана, например, в международной заявке WO2014/130909, которая включена в данный документ посредством ссылки во всей своей полноте. В ряде вариантов осуществления трансфекцию субъединицы теломеразы с использованием мРНК проводят в соответствии с платформой Therapeutics™ на основе матричных РНК, коммерциализированной Moderna Therapeutics. Например, способ может представлять собой способ, описанный в патенте США № 8710200, 8822663, 8680069, 8754062, 8664194 или 8680069.
В некоторых вариантах осуществления hTERT имеет аминокислотную последовательность с ID белка в GenBank AAC51724.1 (Meyerson et al., "hEST2, the Putative Human Telomerase Catalytic Subunit Gene, Is Up-Regulated in Tumor Cells and during Immortalization" Cell, том 90, выпуск 4, 22 августа 1997 г., страницы 785-795): MPRAPRCRAVRSLLRSHYREVLPLATFVRRLGPQGWRLVQRGDPAAFRALVAQCLVCVPWDARPPPAAPSFRQVSCLKELVARVLQRLCERGAKNVLAFGFALLDGARGGPPEAFTTSVRSYLPNTVTDALRGSGAWGLLLRRVGDDVLVHLLARCALFVLVAPSCAYQVCGPPLYQLGAATQARPPPHASGPRRRLGCERAWNHSVREAGVPLGLPAPGARRRGGSASRSLPLPKRPRRGAAPEPERTPVGQGSWAHPGRTRGPSDRGFCVVSPARPAEEATSLEGALSGTRHSHPSVGRQHHAGPPSTSRPPRPWDTPCPPVYAETKHFLYSSGDKEQLRPSFLLSSLRPSLTGARRLVETIFLGSRPWMPGTPRRLPRLPQRYWQMRPLFLELLGNHAQCPYGVLLKTHCPLRAAVTPAAGVCAREKPQGSVAAPEEEDTDPRRLVQLLRQHSSPWQVYGFVRACLRRLVPPGLWGSRHNERRFLRNTKKFISLGKHAKLSLQELTWKMSVRGCAWLRRSPGVGCVPAAEHRLREEILAKFLHWLMSVYVVELLRSFFYVTETTFQKNRLFFYRKSVWSKLQSIGIRQHLKRVQLRELSEAEVRQHREARPALLTSRLRFIPKPDGLRPIVNMDYVVGARTFRREKRAERLTSRVKALFSVLNYERARRPGLLGASVLGLDDIHRAWRTFVLRVRAQDPPPELYFVKVDVTGAYDTIPQDRLTEVIASIIKPQNTYCVRRYAVVQKAAHGHVRKAFKSHVSTLTDLQPYMRQFVAHLQETSPLRDAVVIEQSSSLNEASSGLFDVFLRFMCHHAVRIRGKSYVQCQGIPQGSILSTLLCSLCYGDMENKLFAGIRRDGLLLRLVDDFLLVTPHLTHAKTFLRTLVRGVPEYGCVVNLRKTVVNFPVEDEALGGTAFVQMPAHGLFPWCGLLLDTRTLEVQSDYSSYARTSIRASLTFNRGFKAGRNMRRKLFGVLRLKCHSLFLDLQVNSLQTVCTNIYKILLLQAYRFHACVLQLPFHQQVWKNPTFFLRVISDTASLCYSILKAKNAGMSLGAKGAAGPLPSEAVQWLCHQAFLLKLTRHRVTYVPLLGSLRTAQTQLSRKLPGTTLTALEAAANPALPSDFKTILD (SEQ ID NO: 284)
В некоторых вариантах осуществления hTERT имеет последовательность, характеризующуюся по меньшей мере 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% идентичностью с последовательностью под SEQ ID NO: 284. В некоторых вариантах осуществления hТРЕТ имеет последовательность под SEQ ID NO: 284. В некоторых вариантах осуществления hTERT содержит делецию (например, размером не более 5, 10, 15, 20 или 30 аминокислот) на N-конце, С-конце или на них обоих. В некоторых вариантах осуществления hTERT содержит трансгенную аминокислотную последовательность (например, размером не более 5, 10, 15, 20 или 30 аминокислот) на N-конце, C-конце или на них обоих.
В некоторых вариантах осуществления hTERT кодируется последовательностью нуклеиновой кислоты под № доступа в GenBank AF018167 (Meyerson et al., "hEST2, the Putative Human Telomerase Catalytic Subunit Gene, Is Up-Regulated in Tumor Cells and during Immortalization" Cell, том 90, выпуск 4, 22 августа 1997 г., страницы 785-795).
Активация и размножение иммунных эффекторных клеток (например, T-клеток)
Иммунные эффекторные клетки, такие как Т-клетки, полученные или обогащенные посредством способов, описанных в данном документе, можно активировать и размножать, как правило, посредством способов, описанных, например, в патентах США №№ 6352694; 6534055; 6905680; 6692964; 5858358; 6887466; 6905681; 7144575; 7067318; 7172869; 7232566; 7175843; 5883223; 6905874; 6797514; 6867041 и публикации заявки на патент США № 20060121005.
Как правило, популяцию иммунных эффекторных клеток можно размножать путем приведения их в контакт с поверхностью, к которой присоединены средство, стимулирующее передачу сигнала, ассоциированного с комплексом CD3/TCR, и лиганд, стимулирующий костимулирующую молекулу на поверхности Т-клеток. В частности, популяции Т-клеток можно стимулировать, как описано в данном документе, как, например, путем приведения в контакт с антителом к CD3 или его антигенсвязывающим фрагментом или антителом к CD2, иммобилизованным на поверхности, или путем приведения в контакт с активатором протеинкиназы С (например, бриостатином) в сочетании с ионофором кальция. Для костимуляции вспомогательной молекулы на поверхности Т-клеток применяют лиганд, который связывает вспомогательную молекулу. Например, популяцию Т-клеток можно приводить в контакт с антителом к CD3 и антителом к CD28 в условиях, подходящих для стимуляции пролиферации Т-клеток. Для стимуляции пролиферации CD4+ T-клеток либо CD8+ T-клеток можно применять антитело к CD3 и антитело к CD28. Примеры антитела к CD28 включают 9.3, B-T3, XR-CD28 (Diaclone, Безансон, Франция), и их можно применять, равно как и другие способы, общеизвестные из уровня техники (Berg et al., Transplant Proc. 30(8):3975-3977, 1998; Haanen et al., J. Exp. Med. 190(9):1319-1328, 1999; Garland et al., J. Immunol Meth. 227(1-2):53-63, 1999).
В некоторых вариантах осуществления первичный стимулирующий сигнал и костимулирующий сигнал для Т-клетки можно обеспечивать с помощью различных протоколов. Например, средства, обеспечивающие каждый сигнал, могут находиться в растворе или быть связаны с поверхностью. В случае, если они связаны с поверхностью, средства могут быть связаны с одной и той же поверхностью (т. е. в "цис"-расположении) или с отдельными поверхностями (т. е. в "транс"-расположении). В качестве альтернативы одно средство может быть связано с поверхностью, а другое средство может находиться в растворе. В некоторых вариантах осуществления средство, обеспечивающее костимулирующий сигнал, связано с клеточной поверхностью, а средство, обеспечивающее сигнал первичной активации, находится в растворе или связано с поверхностью. В некоторых вариантах осуществления оба средства могут находиться в растворе. В некоторых вариантах осуществления средства могут находиться в растворимой форме, а затем быть сшиты с поверхностью, такой как клетка, экспрессирующая Fc-рецепторы или антитело или другое связывающее средство, которое будет связываться с данными средствами. В этом отношении см., например, публикации заявок на патент США №№ 20040101519 и 20060034810 в том, что касается искусственных антигенпрезентирующих клеток (aAPC), которые предполагаются для применения в активации и размножении Т-клеток в настоящем изобретении.
В некоторых вариантах осуществления два средства иммобилизованы на гранулах: либо на одной и той же грануле, т. е. в "цис"-положении, либо на отдельных гранулах, т. е. в "транс"-положении. В качестве примера, средство, обеспечивающее сигнал первичной активации, представляет собой антитело к CD3 или его антигенсвязывающий фрагмент, а средство, обеспечивающее костимулирующий сигнал, представляет собой антитело к CD28 или его антигенсвязывающий фрагмент; и оба средства совместно иммобилизованы на одной и той же грануле в эквивалентных молекулярных количествах. В некоторых вариантах осуществления для размножения CD4+ T-клеток и роста T-клеток каждое антитело, связанное с гранулами, применяют в соотношении 1:1. В некоторых вариантах осуществления настоящего изобретения используют такое соотношение антител к CD3:CD28, связанных с гранулами, при котором наблюдается увеличение интенсивности размножения Т-клеток по сравнению с размножением, наблюдаемым при использовании соотношения 1:1. В некоторых вариантах осуществления наблюдается увеличение в от приблизительно 1 до приблизительно 3 раз по сравнению с размножением, наблюдаемым при использовании соотношения 1:1. В некоторых вариантах осуществления соотношение антител к CD3:CD28, связанных с гранулами, находится в диапазоне от 100:1 до 1:100 и включает все целочисленные значения в этом промежутке. В некоторых вариантах осуществления с частицами связано больше антител к CD28, чем антител к CD3, т. е. соотношение антител к CD3:CD28 составляет меньше единицы. В некоторых вариантах осуществления соотношение антитела к CD28 и антитела к CD3, связанных с гранулами, составляет более чем 2:1. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 1:100. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 1:75. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 1:50. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 1:30. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 1:10. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 1:3. В некоторых вариантах осуществления антитела к CD3 и CD28, связанные с гранулами, применяют в соотношении 3:1.
Для стимуляции Т-клеток или других клеток-мишеней можно использовать соотношения частиц и клеток, составляющие от 1:500 до 500:1, с включением любых целочисленных значений в этом промежутке. Как будет очевидно для специалистов средней квалификации в данной области, соотношение частиц и клеток может зависеть от размера частиц относительно клетки-мишени. Например, гранулы небольшого размера могут связывать лишь немного клеток, тогда как более крупные гранулы могут связывать много клеток. В некоторых вариантах осуществления соотношение клеток и частиц находится в диапазоне от 1:100 до 100:1 и включает любые целочисленные значения в этом промежутке, и в некоторых вариантах осуществления соотношение составляет от 1:9 до 9:1 и включает любые целочисленные значения в этом промежутке, и его также можно использовать для стимуляции Т-клеток. Соотношение частиц, связанных с антителами к CD3 и антителами к CD28, и Т-клеток, которое приводит к стимуляции Т-клеток, может варьироваться, как отмечено выше, однако определенные подходящие значения включают 1:100, 1:50, 1:40, 1:30, 1:20, 1:10, 1:9, 1:8, 1:7, 1:6, 1:5, 1:4, 1:3, 1:2, 1:1, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1, 10:1, и 15:1 , при этом одно подходящее соотношение составляет по меньшей мере 1:1 для частиц на Т-клетку. В некоторых вариантах осуществления используют соотношение частиц и клеток, составляющее 1:1 или меньше. В некоторых вариантах осуществления подходящее соотношение частицы:клетки составляет 1:5. В некоторых вариантах осуществления соотношение частиц и клеток можно варьировать в зависимости от дня стимуляции. Например, в некоторых вариантах осуществления соотношение частиц и клеток составляет от 1:1 до 10:1 в первый день, и после этого к клеткам ежедневно или раз в два дня добавляют дополнительные частицы в течение периода вплоть до 10 дней при конечных соотношениях от 1:1 до 1:10 (в расчете на количество клеток в день добавления). В некоторых вариантах осуществления соотношение частиц и клеток составляет 1:1 в первый день стимуляции и доводится до 1:5 в третий и пятый дни стимуляции. В некоторых вариантах осуществления частицы добавляют каждый день или раз в два дня до конечного соотношения 1:1 в первый день и 1:5 в третий и пятый дни стимуляции. В некоторых вариантах осуществления соотношение частиц и клеток составляет 2:1 в первый день стимуляции и доводится до 1:10 в третий и пятый дни стимуляции. В некоторых вариантах осуществления частицы добавляют каждый день или раз в два дня до конечного соотношения 1:1 в первый день и 1:10 в третий и пятый дни стимуляции. Специалисту в данной области будет понятно, что для использования в настоящем изобретении может подходить множество других соотношений. В частности, соотношения будут варьироваться в зависимости от размера частиц, а также от размера и типа клеток. В некоторых вариантах осуществления наиболее типичные соотношения для использования составляют примерно 1:1, 2:1 и 3:1 в первый день.
В некоторых вариантах осуществления клетки, такие как Т-клетки, объединяют с гранулами, покрытыми средством, впоследствии гранулы и клетки разделяют, а затем клетки культивируют. В некоторых вариантах осуществления гранулы, покрытые средством, и клетки не разделяют перед культивированием, а культивируют вместе. В некоторых вариантах осуществления гранулы и клетки вначале концентрируют путем приложения силы, такой как сила магнитного поля, что приводит к увеличению лигирования маркеров клеточной поверхности, за счет чего обеспечивается индукция стимуляции клеток.
В качестве примера, белки клеточной поверхности можно лигировать, обеспечивая возможность контакта парамагнитных гранул, к которым присоединены антитела к CD3 и антитела к CD28 (гранул 3 × 28), и Т-клеток. В некоторых вариантах осуществления клетки (например, от 104 до 109 T-клеток) и гранулы (например, парамагнитные гранулы DYNABEADS® M-450 CD3/CD28 T в соотношении 1:1) объединяют в буфере, например, PBS (без двухвалентных катионов, таких как кальций и магний). Специалистам средней квалификации в данной области также будет очевидно, какую концентрацию клеток можно использовать. Например, клетка-мишень может очень редко встречаться в образце и составлять только 0,01% образца, или весь образец (т. е. 100%) может состоять из клетки-мишени, представляющей интерес. Соответственно, любое количество клеток находится в рамках контекста настоящего изобретения. В некоторых вариантах осуществления может потребоваться значительно уменьшить объем, в котором частицы и клетки смешивают друг с другом (т. е. увеличить концентрацию клеток), чтобы обеспечить максимальный контакт клеток и частиц. Например, в некоторых вариантах осуществления используют концентрацию, составляющую приблизительно 10 миллиардов клеток/мл, 9 миллиардов/мл, 8 миллиардов/мл, 7 миллиардов/мл, 6 миллиардов/мл, 5 миллиардов/мл или 2 миллиарда клеток/мл. В некоторых вариантах осуществления используют более 100 миллионов клеток/мл. В некоторых вариантах осуществления используют концентрацию клеток, составляющую 10, 15, 20, 25, 30, 35, 40, 45 или 50 миллионов клеток/мл. В некоторых вариантах осуществления используют концентрацию клеток, составляющую от 75, 80, 85, 90, 95 или 100 миллионов клеток/мл. В некоторых вариантах осуществления можно использовать концентрации, составляющие 125 или 150 миллионов клеток/мл. Использование высоких концентраций может приводить к увеличению выхода клеток, активации клеток и размножения клеток. Кроме того, использование высоких концентраций клеток позволяет более эффективно захватывать клетки, которые могут характеризоваться слабой экспрессией антигенов-мишеней, представляющих интерес, такие как CD28-отрицательные Т-клетки. Такие популяции клеток могут иметь терапевтическую ценность, и в некоторых вариантах осуществления может требоваться их получение. Например, использование высокой концентрации клеток позволяет проводить более эффективный отбор CD8+ Т-клеток, которые обычно характеризуются более слабой экспрессией CD28.
В некоторых вариантах осуществления клетки, трансдуцированные нуклеиновой кислотой, кодирующей CAR, например, CAR, описанный в данном документе, например, CAR для CD19, описанный в данном документе, размножают, например, посредством способа, описанного в данном документе. В некоторых вариантах осуществления клетки размножают в культуре в течение периода времени, который составляет от нескольких часов (например, приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 18, 21 час) до приблизительно 14 дней (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14 дней). В некоторых вариантах осуществления клетки размножают в течение периода от 4 до 9 дней. В некоторых вариантах осуществления клетки размножают в течение периода, который составляет 8 дней или меньше, например, 7, 6 или 5 дней. В некоторых вариантах осуществления клетки размножают в культуре в течение 5 дней, и полученные клетки являются более активными, чем те же самые клетки, размножаемые в культуре в течение 9 дней при тех же условиях культивирования. Активность можно определить, например, по различным Т-клеточным функциям, например, пролиферации, уничтожению клеток-мишеней, продуцированию цитокинов, активации, миграции, поверхностной экспрессии CAR, уровню CAR, определенному с помощью количественной ПЦР, или их комбинаций. В некоторых вариантах осуществления клетки, например, клетки с CAR для CD19, описанные в данном документе, размножаемые в течение 5 дней, демонстрируют по меньшей мере одно-, двух-, трех- или четырехкратное увеличение интенсивности удвоения клеток при стимуляции антигеном по сравнению с теми же клетками, размножаемыми в культуре в течение 9 дней при тех же условиях культивирования. В некоторых вариантах осуществления клетки, например, клетки, экспрессирующие CAR для CD19, описанные в данном документе, размножают в культуре в течение 5 дней, и полученные клетки демонстрируют более высокие уровни продуцирования провоспалительных цитокинов, например, уровни IFNγ и/или GM-CSF, по сравнению с теми же клетками, размножаемыми в культуре в течение 9 дней при тех же условиях культивирования. В некоторых вариантах осуществления клетки, например, клетки с CAR для CD19, описанные в данном документе, размножаемые в течение 5 дней, демонстрируют по меньшей мере одно-, двух-, трех-, четырех-, пяти-, десятикратное или большее повышение уровней продуцирования провоспалительных цитокинов в пг/мл, например, уровней IFNγ и/или GM-CSF, по сравнению с теми же клетками, размножаемыми в культуре в течение 9 дней при тех же условиях культивирования.
Также могут потребоваться несколько циклов стимуляции, вследствие чего время культивирования Т-клеток может составлять 60 дней или больше. Условия, подходящие для культивирования Т-клеток, включают подходящие среды (например, минимальную питательную среду, α-MEM, среду RPMI 1640, AIM-V, DMEM, F-12 или X-vivo 15 (Lonza), X-Vivo 20, OpTmizer и IMDM), которые могут содержать факторы, необходимые для пролиферации и жизнеспособности, в том числе сыворотку (например, фетальную бычью или человеческую сыворотку), интерлейкин-2 (IL-2), инсулин, IFNγ, IL-4, IL-7, GM-CSF, IL-10, IL-12, IL-15, TGFβ и TNF-α или любые другие добавки для роста клеток, известные специалисту в данной области. Другие добавки для роста клеток включают без ограничения поверхностно-активное вещество, плазманат и восстановители, такие как N-ацетилцистеин и 2-меркаптоэтанол. Среды могут включать без ограничения RPMI 1640, AIM-V, DMEM, MEM, α-MEM, F-12, X-Vivo 15 и X-Vivo 20, OpTmizer и IMDM с добавлением аминокислот, пирувата натрия и витаминов, бессывороточные либо с добавлением соответствующего количества сыворотки (или плазмы крови), или определенного набора гормонов и/или количества цитокина(цитокинов), достаточных для роста и размножения Т-клеток. Антибиотики, например, пенициллин и стрептомицин, включают только в экспериментальные культуры, но не в культуры клеток, которые предназначены для инфузии субъекту. Клетки-мишени содержатся в условиях, необходимых для поддержания роста, например, при соответствующей температуре (например, 37°C) и атмосфере (например, воздух с 5% CO2).
В некоторых вариантах осуществления клетки размножают в подходящей среде (например, среде, описанной в данном документе), содержащей один или несколько интерлейкинов, которые приводят к по меньшей мере 200-кратному (например, к 200-кратному, 250-кратному, 300-кратному, 350-кратному) увеличению количества клеток в течение 14-дневного периода размножения, например, согласно измерению посредством способа, описанного в данном документе, такого как проточная цитометрия. В некоторых вариантах осуществления клетки размножают в присутствии IL-15 и/или IL-7 (например, IL-15 и IL-7).
В ряде вариантов осуществления способы, описанные в данном документе, например, способы изготовления клеток, экспрессирующих CAR, включают удаление регуляторных T-клеток, например, CD25+ T-клеток или CD25высокий Т-клеток, из популяции клеток, например, с помощью антитела к CD25 или его фрагмента или CD25-связывающего лиганда IL-2. Способы удаления регуляторных Т-клеток, например, CD25+ Т-клеток или CD25высокий Т-клеток, из популяции клеток описаны в данном документе. В ряде вариантов осуществления способы, например, способы изготовления, дополнительно включают приведение популяции клеток (например, популяции клеток, которая была истощена по регуляторным T-клеткам, таким как CD25+ T-клетки или CD25высокий Т-клетки; или популяции клеток, которая ранее была приведена в контакт с антителом к CD25, его фрагментом или CD25-связывающим лигандом) в контакт с IL-15 и/или IL-7. Например, популяцию клеток (например, популяцию, которая ранее была приведена в контакт с антителом к CD25, его фрагментом или CD25-связывающим лигандом) размножают в присутствии IL-15 и/или IL-7.
В некоторых вариантах осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей полипептид интерлейкин-15 (IL-15), полипептид альфа-субъединицу рецептора интерлейкина-15 (IL-15Ra) или комбинацию как полипептида IL-15, так и полипептида IL-15Ra, например, hetIL-15, в ходе изготовления клетки, экспрессирующей CAR, например, ex vivo. В ряде вариантов осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей полипептид IL-15, в ходе изготовления клетки, экспрессирующей CAR, например, ex vivo. В ряде вариантов осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей комбинацию как полипептида IL-15, так и полипептида IL-15Ra, в ходе изготовления клетки, экспрессирующей CAR, например, ex vivo. В ряде вариантов осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей hetIL-15, в ходе изготовления клетки, экспрессирующей CAR, например, ex vivo.
В некоторых вариантах осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей hetIL-15, в ходе размножения ex vivo. В некоторых вариантах осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей полипептид IL-15, в ходе размножения ex vivo. В некоторых вариантах осуществления описанную в данном документе клетку, экспрессирующую CAR, приводят в контакт с композицией, содержащей как полипептид IL-15, так и полипептид IL-15Ra, в ходе размножения ex vivo. В некоторых вариантах осуществления приведение в контакт приводит к выживанию и пролиферации субпопуляции лимфоцитов, например, CD8+ T-клеток.
Т-клетки, которые подвергались стимуляции в течение разных периодов времени, могут демонстрировать разные характеристики. Например, в типичных препаратах крови или продуктах афереза мононуклеарных клеток периферической крови популяция Т-клеток-хелперов (TH, CD4+) превосходит по числу популяцию цитотоксических Т-клеток или Т-клеток-супрессоров (TC, CD8+). Размножение Т-клеток ex vivo путем стимуляции рецепторов CD3 и CD28 приводит к получению популяции Т-клеток, которая до приблизительно дней 8-9 состоит главным образом из ТН-клеток, тогда как после приблизительно дней 8-9 популяция Т-клеток содержит все более многочисленную популяцию TC-клеток. Соответственно, в зависимости от цели лечения может быть преимущественной инфузия субъекту популяции Т-клеток, содержащей главным образом ТН-клетки. Аналогично, если была выделена антигенспецифическая субпопуляция TC-клеток, то может быть целесообразным размножение этой субпопуляции в большей степени.
Кроме того, в ходе процесса размножения клеток в дополнение к маркерам CD4 и CD8 в значительной степени, но по большей части воспроизводимо, варьируются уровни других фенотипических маркеров. Таким образом, подобная воспроизводимость дает возможность адаптировать продукт на основе активированных Т-клеток для конкретных целей.
После конструирования CAR, описанного в данном документе, можно применять различные анализы для оценивания активности молекулы, такой как, без ограничения, способность Т-клеток размножаться после стимуляции антигеном, устойчивое размножение Т-клеток при отсутствии повторной стимуляции и формы противораковой активности, в соответствующих моделях in vitro и животных моделях. Анализы для оценивания эффектов CAR по настоящему изобретению описаны более подробно ниже.
Вестерн-блот-анализ экспрессии CAR в первичных T-клетках можно применять для выявления присутствия мономеров и димеров, например, как описано в абзаце 695 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Размножение CAR+ Т-клеток in vitro после стимуляции антигеном можно измерить с помощью проточной цитометрии. Например, смесь CD4+ и CD8+ Т-клеток стимулируют с помощью aAPC с αCD3/αCD28, а затем трансдуцируют с помощью лентивирусных векторов, экспрессирующих GFP под контролем промоторов, подлежащих анализу. Иллюстративные промоторы включают промоторы генов IE CMV, EF-1α, убиквитина С или фосфоглицераткиназы (PGK). Флуоресценцию GFP оценивают с помощью проточной цитометрии в день 6 культивирования в субпопуляциях CD4+ и/или CD8+ T-клеток. См., например, Milone et al., Molecular Therapy 17(8): 1453-1464 (2009). В качестве альтернативы смесь CD4+ и CD8+ Т-клеток стимулируют с помощью магнитных гранул, покрытых αCD3/αCD28, в день 0 и трансдуцируют с помощью CAR в день 1 с применением бицистронного лентивирусного вектора, экспрессирующего CAR вместе с eGFP с использованием последовательности 2A для "проскока" рибосомы. Культуры повторно стимулируют клетками K562, положительными+ в отношении антигена, ассоциированного с раком, описанного в данном документе (K562, экспрессирующими антиген, ассоциированный с раком, описанный в данном документе), клетками K562 дикого типа (K562 дикого типа) либо клетками K562, экспрессирующими hCD32 и 4-1BBL в присутствии антитела к CD3 и антитела к CD28 (K562-BBL-3/28). Экзогенный IL-2 добавляют в культуры раз в два дня при 100 МЕ/мл. GFP+ Т-клетки подсчитывают с помощью проточной цитометрии с применением подсчета по гранулам. См., например, Milone et al., Molecular Therapy 17(8): 1453-1464 (2009).
Также можно измерять уровень устойчивости размножения CAR+ T-клеток при отсутствии повторной стимуляции. См., например, Milone et al., Molecular Therapy 17(8): 1453-1464 (2009). Вкратце, измеряют средний объем Т-клеток (фл) в день 8 культивирования с использованием устройства для подсчета частиц Multisizer III или более поздней версии от Coulter, Cellometer Vision от Nexcelom или Scepter от Millipore или других устройств для подсчета клеток после стимуляции с помощью магнитных гранул, покрытых αCD3/αCD28, в день 0 и трансдукции с помощью указанного CAR в день 1.
Животные модели также можно использовать для измерения активности клеток, экспрессирующих CAR, например, как описано в абзаце 698 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Дозозависимый ответ на лечение с помощью CAR можно оценивать, например, как описано в абзаце 699 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Оценка пролиферации клеток и продуцирования цитокинов была описана ранее, как описано в абзаце 700 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Цитотоксичность можно оценить с помощью стандартного анализа высвобождения 51Cr, например, как описано в абзаце 701 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте. Также можно использовать альтернативные способы без применения радиоактивных изотопов.
Цитотоксичность можно также оценить посредством измерения изменений электрического импеданса адгезивных клеток, например, с помощью анализатора клеток в реальном времени (RTCA) xCELLigence. В некоторых вариантах осуществления цитотоксичность измеряют в несколько моментов времени.
Для оценки специфической миграции и пролиферации CAR в моделях на животных, несущих опухоль, можно применять технологии визуализации, например, как описано в абзаце 702 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Также можно применять другие анализы, включая анализы, описанные в данном документе в разделе "Примеры", а также анализы, которые известны из уровня техники, для оценивания CAR, описанных в данном документе.
В качестве альтернативы или в комбинации со способами, раскрытыми в данном документе, раскрыты способы и композиции для одного или нескольких из выявления и/или количественного определения клеток, экспрессирующих CAR (например, in vitro или in vivo (например, клинического мониторинга)); размножения и/или активации иммунных клеток и/или CAR-специфического отбора, которые предусматривают применение лиганда CAR. В некоторых вариантах осуществления лиганд CAR представляет собой антитело, которое связывается с молекулой CAR, например, связывается с внеклеточным антигенсвязывающим доменом CAR (например, антитело, которое связывается с антигенсвязывающим доменом, например, антиидиотипическое антитело; или антитело, которое связывается с константной областью внеклеточного связывающего домена). В других вариантах осуществления лиганд CAR представляет собой молекулу антигена CAR (например, молекулу антигена CAR, описанную в данном документе).
В некоторых вариантах осуществления раскрыт способ выявления и/или количественного определения клеток, экспрессирующих CAR. Например, лиганд CAR можно применять для выявления и/или количественного определения клеток, экспрессирующих CAR, in vitro или in vivo (например, для клинического мониторинга клеток, экспрессирующих CAR, у пациента или введения доз пациенту). Способ включает:
получение лиганда CAR (необязательно меченого лиганда CAR, например, лиганда CAR, который содержит маркер, гранулу, радиоактивную или флуоресцентную метку);
получение клетки, экспрессирующей CAR (например, получение образца, содержащего клетки, экспрессирующие CAR, такого как изготовленный образец или клинический образец);
приведение клетки, экспрессирующей CAR, в контакт с лигандом CAR в условиях, при которых происходит связывание, за счет чего обеспечивается выявление уровня (например, количества) присутствующих клеток, экспрессирующих CAR. Связывание клетки, экспрессирующей CAR, с лигандом CAR можно выявлять с помощью стандартных методик, таких как FACS, ELISA и т. п.
В некоторых вариантах осуществления раскрыт способ размножения и/или активации клеток (например, иммунных эффектoрных клеток). Способ включает:
получение клетки, экспрессирующей CAR (например, первой клетки, экспрессирующей CAR, или клетки, транзиентно экспрессирующей CAR);
приведение указанной клетки, экспрессирующей CAR, в контакт с лигандом CAR (например, лигандом CAR, описанным в данном документе) в условиях, при которых происходит размножение и/или пролиферация иммунных клеток, за счет чего обеспечивается получение популяции активированных и/или размноженных клеток.
В определенных вариантах осуществления лиганд CAR присутствует на субстрате (например, является иммобилизованным на субстрате, например, не встречающемся в природе субстрате, или присоединенным к нему). В некоторых вариантах осуществления субстрат представляет собой неклеточный субстрат. Неклеточный субстрат может представлять собой твердую подложку, выбранную из, например, планшета (например, титрационного микропланшета), мембраны (например, нитроцеллюлозной мембраны), матрицы, чипа или гранулы. В ряде вариантов осуществления лиганд CAR присутствует в субстрате (например, на поверхности субстрата). Лиганд CAR может быть иммобилизован на субстрате, присоединен к нему или ковалентно или нековалентно ассоциирован (например, сшит) с ним. В некоторых вариантах осуществления лиганд CAR присоединен (например, ковалентно присоединен) к грануле. В вышеуказанных вариантах осуществления популяцию иммунных клеток можно размножать in vitro или ex vivo. Способ может дополнительно включать культивирование популяции иммунных клеток в присутствии лиганда молекулы CAR, например, посредством любого из способов, описанных в данном документе.
В других вариантах осуществления способ размножения и/или активации клеток дополнительно включает добавление второй стимулирующей молекулы, например, CD28. Например, лиганд CAR и вторая стимулирующая молекула могут быть иммобилизованы на субстрате, например, на одной или нескольких гранулах, за счет чего обеспечивается увеличение интенсивности размножения и/или активации клеток.
В некоторых вариантах осуществления предусмотрен способ отбора клетки, экспрессирующей CAR, или обогащения такой клеткой. Способ включает приведение клетки, экспрессирующей CAR, в контакт с лигандом CAR, описанным в данном документе; и отбор клетки на основании связывания с лигандом CAR.
В еще нескольких других вариантах осуществления представлен способ истощения популяции, снижения количества и/или уничтожения клеток, экспрессирующих CAR. Способ включает приведение клетки, экспрессирующей CAR, в контакт с лигандом CAR, описанным в данном документе; и нацеливание на клетку на основании связывания с лигандом CAR, за счет чего обеспечивается снижение количества и/или уничтожение клеток, экспрессирующих CAR. В некоторых вариантах осуществления лиганд CAR связан с токсичным средством (например, токсином или лекарственным средством, разрушающим клетки). В некоторых вариантах осуществления антиидиотипическое антитело может обуславливать активность эффекторных клеток, например, формы активности ADCC или ADC.
Иллюстративные антитела к CAR, которые можно применять в способах, раскрытых в данном документе, описаны, например, в WO 2014/190273 и в Jena et al.,"Chimeric Antigen Receptor (CAR)-Specific Monoclonal Antibody to Detect CD19-Specific T cells in Clinical Trials", PLOS March 2013 8:3 e57838, содержание которых включено посредством ссылки.
В некоторых вариантах осуществления композиции и способы в данном документе оптимизированы для конкретной субпопуляции Т-клеток, например, как описано в заявке на патент США с регистрационным № PCT/US2015/043219, поданной 31 июля 2015 года, содержание которой включено в данный документ посредством ссылки во всей своей полноте. В некоторых вариантах осуществления оптимизированные субпопуляции T-клеток проявляют повышенную персистенцию по сравнению с контрольной T-клеткой, например, T-клеткой другого типа (например, CD8+ или CD4+), экспрессирующей такую же конструкцию.
В некоторых вариантах осуществления CD4+ T-клетка содержит CAR, описанный в данном документе, при этом CAR содержит внутриклеточный сигнальный домен, подходящий для CD4+ T-клетки (например, оптимизированный для, например, приведения к ее повышенной персистенции), например, домен ICOS. В некоторых вариантах осуществления CD8+ T-клетка содержит CAR, описанный в данном документе, при этом CAR содержит внутриклеточный сигнальный домен, подходящий для CD8+ T-клетки (например, оптимизированный для, например, приведения к ее повышенной персистенции), например, домен 4-1BB, домен CD28 или другой костимулирующий домен, отличный от домена ICOS. В некоторых вариантах осуществления CAR, описанный в данном документе, содержит антигенсвязывающий домен, описанный в данном документе, например, представляет собой CAR, содержащий антигенсвязывающий домен.
В некоторых вариантах осуществления в данном документе описан способ лечения субъекта, например, субъекта, у которого имеется рак. Способ включает введение указанному субъекту эффективного количества:
1) CD4+ T-клетки, содержащей CAR (CARCD4+), который содержит:
антигенсвязывающий домен, например, антигенсвязывающий домен, описанный в данном документе;
трансмембранный домен и
внутриклеточный сигнальный домен, например, первый костимулирующий домен, например, домен ICOS; и
2) CD8+ T-клетки, содержащей CAR (CARCD8+), который содержит:
антигенсвязывающий домен, например, антигенсвязывающий домен, описанный в данном документе;
трансмембранный домен и
внутриклеточный сигнальный домен, например, второй костимулирующий домен, например, домен 4-1BB, домен CD28 или другой костимулирующий домен, отличный от домена ICOS;
где CARCD4+ и CARCD8+ отличаются друг от друга.
Способ необязательно дополнительно включает введение:
3) второй CD8+ T-клетки, содержащей CAR (второй CARCD8+), который содержит:
антигенсвязывающий домен, например, антигенсвязывающий домен, описанный в данном документе;
трансмембранный домен и
внутриклеточный сигнальный домен, где второй CARCD8+ содержит внутриклеточный сигнальный домен, например, костимулирующий сигнальный домен, отсутствующий в CARCD8+, и необязательно не содержит сигнального домена ICOS.
Способы доставки с помощью биополимеров
В некоторых вариантах осуществления одну или несколько клеток, экспрессирующих CAR, раскрытых в данном документе, можно вводить или доставлять субъекту посредством биополимерного каркаса, например, биополимерного имплантата. Биополимерные каркасы могут поддерживать или усиливать доставку, размножение и/или распространение клеток, экспрессирующих CAR, описанных в данном документе. Биополимерный каркас содержит биосовместимый (например, который по существу не индуцирует воспалительный или иммунный ответ) и/или биоразлагаемый полимер, который может быть встречающимся в природе или синтетическим. Иллюстративные биополимеры описаны, например, в абзацах 1004-1006 международной заявки WO2015/142675, поданной 13 марта 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте.
Фармацевтические композиции и средства для лечения
В некоторых вариантах осуществления в настоящем изобретении предусмотрен способ лечения пациента, включающий введение клеток, экспрессирующих CAR, полученных согласно описанному в данном документе, необязательно в комбинации с одним или несколькими другими видами терапии. В некоторых вариантах осуществления в настоящем изобретении предусмотрен способ лечения пациента, включающий введение реакционной смеси, содержащей клетки, экспрессирующие CAR, описанные в данном документе, необязательно в комбинации с одним или несколькими другими видами терапии. В некоторых вариантах осуществления в настоящем изобретении предусмотрен способ транспортировки или приема реакционной смеси, содержащей клетки, экспрессирующие CAR, описанные в данном документе. В некоторых вариантах осуществления в настоящем изобретении предусмотрен способ лечения пациента, включающий прием клетки, экспрессирующей CAR, которая была получена согласно описанному в данном документе, и дополнительно включающий введение клетки, экспрессирующей CAR, пациенту, необязательно в комбинации с одним или несколькими другими видами терапии. В некоторых вариантах осуществления в настоящем изобретении предусмотрен способ лечения пациента, включающий получение клетки, экспрессирующей CAR, описанной в данном документе, и дополнительно включающий введение клетки, экспрессирующей CAR, пациенту, необязательно в комбинации с одним или несколькими другими видами терапии. Другим видом терапии может быть, например, противораковая терапия, такая как химиотерапия.
В некоторых вариантах осуществления клетки, экспрессирующие CAR, описанные в данном документе, вводят субъекту в комбинации с молекулой, которая обеспечивает уменьшение популяции Treg-клеток. Способы, которые обеспечивают уменьшение количества (например, приводят к истощению) Treg-клеток, известны из уровня техники и включают, например, истощение по CD25, введение циклофосфамида, модулирование функции GITR. Не ограничиваясь какой-либо теорией, полагают, что снижение количества Treg-клеток у субъекта до афереза или до введения клетки, экспрессирующей CAR, описанной в данном документе, приводит к снижению количества нежелательных иммунных клеток (например, Treg) в микроокружении опухоли и снижению риска рецидива у субъекта.
В некоторых вариантах осуществления средство терапии, описанное в данном документе, например, клетку, экспрессирующую CAR, вводят субъекту в комбинации с молекулой, которая нацеливается на GITR и/или модулирует функции GITR, такой как антагонист GITR и/или антитело к GITR, которые обеспечивают истощение по регуляторным Т-клеткам (Treg). В ряде вариантов осуществления клетки, экспрессирующие CAR, описанные в данном документе, вводят субъекту в комбинации с циклофосфамидом. В некоторых вариантах осуществления молекулы, связывающие GITR, и/или молекулы, модулирующие функции GITR (например, агонист GITR и/или антитела к GITR, обеспечивающие истощение по Treg), вводят до введения клетки, экспрессирующей CAR. Например, в некоторых вариантах осуществления агонист GITR можно вводить до афереза клеток. В ряде вариантов осуществления циклофосфамид вводят субъекту до введения (например, до инфузии или повторной инфузии) клетки, экспрессирующей CAR, или до афереза клеток. В ряде вариантов осуществления циклофосфамид и антитело к GITR вводят субъекту до введения (например, инфузии или повторной инфузии) клетки, экспрессирующей CAR, или до афереза клеток. В некоторых вариантах осуществления у субъекта имеется рак (например, солидный рак или гематологический рак, такой как ALL или CLL). В некоторых вариантах осуществления у субъекта имеется CLL. В ряде вариантов осуществления у субъекта имеется ALL. В ряде вариантов осуществления у субъекта имеется солидный рак, например, солидный рак, описанный в данном документе. Иллюстративные агонисты GITR включают, например, слитые белки на основе GITR и антитела к GITR (например, бивалентные антитела к GITR), такие как, например, слитый белок на основе GITR, описанный в патенте США № 6111090, европейском патенте № 090505B1, патенте США № 8586023, публикациях согласно PCT №№ WO 2010/003118 и 2011/090754, или антитело к GITR, описанное, например, в патенте США № 7025962, европейском патенте № 1947183B1, патенте США № 7812135, патенте США № 8388967, патенте США № 8591886, европейском патенте № EP 1866339, публикации согласно PCT № WO 2011/028683, публикации согласно PCT № WO 2013/039954, публикации согласно PCT № WO2005/007190, публикации согласно PCT № WO 2007/133822, публикации согласно PCT № WO2005/055808, публикации согласно PCT № WO 99/40196, публикации согласно PCT № WO 2001/03720, публикации согласно PCT № WO99/20758, публикации согласно PCT № WO2006/083289, публикации согласно PCT № WO 2005/115451, патенте США № 7618632 и публикации согласно PCT № WO 2011/051726.
В некоторых вариантах осуществления описанную в данном документе клетку, экспрессирующую CAR, вводят субъекту в комбинации с агонистом GITR, например, агонистом GITR, описанным в данном документе. В некоторых вариантах осуществления агонист GITR вводят до введения клетки, экспрессирующей CAR. Например, в некоторых вариантах осуществления агонист GITR можно вводить до афереза клеток. В некоторых вариантах осуществления у субъекта имеется CLL.
Способы, описанные в данном документе, могут дополнительно включать составление клетки, экспрессирующей CAR, в виде фармацевтической композиции. Фармацевтические композиции могут содержать клетку, экспрессирующую CAR, например, множество клеток, экспрессирующих CAR, описанных в данном документе, в комбинации с одним или несколькими фармацевтически или физиологически приемлемыми носителями, разбавителями или наполнителями. Такие композиции могут содержать буферы, такие как нейтральный буферный солевой раствор, фосфатно-солевой буферный раствор и т. п.; углеводы, такие как глюкоза, манноза, сахароза или декстраны, маннит; белки; полипептиды или аминокислоты, такие как глицин; антиоксиданты; хелатообразующие средства, такие как EDTA или глутатион; адъюванты (например, гидроксид алюминия) и консерванты. Композиции можно составлять, например, для внутривенного введения.
В некоторых вариантах осуществления фармацевтическая композиция по существу не содержит загрязнителя, например, выбранного из группы, состоящей из эндотоксина, микоплазмы, репликационно-компетентного лентивируса (RCL), p24, нуклеиновой кислоты VSV-G, gag HIV, остаточных количеств гранул, покрытых антителами к CD3/антителами к CD28, антител мыши, объединенной человеческой сыворотки, бычьего сывороточного альбумина, бычьей сыворотки, компонентов культуральных сред, клеточных или плазмидных компонентов для упаковки векторов, бактерии и гриба, например, в ней отсутствуют его поддающиеся выявлению уровни. В некоторых вариантах осуществления бактерия представляет собой по меньшей мере бактерию, выбранную из группы, состоящей из Alcaligenes faecalis, Candida albicans, Escherichia coli, Haemophilus influenzae, Neisseria meningitidis, Pseudomonas aeruginosa, Staphylococcus aureus, Streptococcus pneumoniae и Streptococcus pyogenes группы A.
В случае, если указано "иммунологически эффективное количество", "эффективное противораковое количество", "количество, эффективное для ингибирования рака" или "терапевтическое количество", точное количество композиций, подлежащих введению, может быть определено врачом с учетом индивидуальных различий в возрасте, массе тела, размере опухоли, степени инфицирования или метастазирования и состояния пациента (субъекта). В целом, можно отметить, что фармацевтическую композицию, содержащую иммунные эффекторные клетки (например, T-клетки, NK-клетки), описанные в данном документе, можно вводить в дозе от 104 до 109 клеток/кг массы тела, в некоторых случаях от 105 до 106 клеток/кг массы тела, включая все целочисленные значения в пределах этих диапазонов. Композиции на основе Т-клеток можно также вводить несколько раз в этих дозах. Клетки можно вводить с помощью методик инфузии, которые являются общеизвестными в иммунотерапии (см., например, Rosenberg et al., New Eng. J. of Med. 319:1676, 1988).
Иллюстративные фармацевтические композиции на основе BCMA/CD19-CAR-T
В некоторых вариантах осуществления композицию на основе CAR-T-клеток с двойной специфичностью к BCMA/CD19 получают путем котрансдукции с помощью двух уникальных векторов. Соответственно, в некоторых вариантах осуществления композиция на основе клеток содержит гетерогенную популяцию клеток. В некоторых вариантах осуществления композиция на основе гетерогенных клеток включает нетрансдуцированные Т-клетки, моно-BCMA-специфичные CAR-Т-клетки, моно-CD19-специфичные CAR-Т-клетки и CAR-Т-клетки с двойной специфичностью, экспрессирующие как молекулы BCMA-специфичного CAR, так и молекулы CD19-специфичного CAR. Эти различные популяции клеток могут демонстрировать различные формы активности в контексте лечения заболевания у субъекта.
Не ограничиваясь какой-либо теорией, полагают, что в контексте лечения множественной миеломы, например, активация BCMA-специфичных CAR-T-клеток может быть основным фактором противоопухолевого ответа у пациентов, тогда как CD19-специфичная активность может играть роль в устранении менее распространенных CD19-положительных опухолевых клеток.
В некоторых вариантах осуществления композицию на основе клеток можно оценивать для определения относительных процентных долей четырех различных клеточных популяций, например, для отбора композиции на основе клеток, содержащей более высокую процентную долю клеток с BCMA-специфичным CAR (например, клеток с моно-BCMA-специфичным CAR и клеток с двойным CAR для BCMA/CD19), чем клеток с моно-CD19-специфичным CAR.
В некоторых вариантах осуществления композиция на основе клеток содержит:
(a) первую популяцию клеток, содержащих CAR для BCMA, но не CAR для CD19;
(b) вторую популяцию клеток, содержащих CAR для CD19, но не CAR для BCMA; и
(c) третью популяцию клеток, содержащих как CAR для BCMA, так и CAR для CD19.
В некоторых вариантах осуществления:
(i) общее количество жизнеспособных клеток в объединенных второй и третьей популяциях меньше или равняется приблизительно 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от общего количества жизнеспособных клеток в объединенных первой и третьей популяциях;
(ii) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 90% (например, больше или равняется приблизительно 100%, 125%, 150%, 175%, 200%, 250%, 300%, 400%, 500%, 750%, 1000%, 2000% , 5000, 10000% или больше) от общего количества жизнеспособных клеток в объединенных второй и третьей популяциях; и/или
(iii) общее количество жизнеспособных клеток в объединенных первой и третьей популяциях больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%) от общего количества жизнеспособных клеток в популяции.
В некоторых вариантах осуществления композиция на основе клеток дополнительно содержит четвертую популяцию клеток, которые не содержат CAR. В некоторых вариантах осуществления композиция на основе клеток содержит популяцию клеток с моно-CD19-специфичным CAR, численность которой меньше или равняется 110% (например, меньше или равняется приблизительно 105%, 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5%, 1% или меньше) от количества клеток с BCMA-специфичным CAR (например, общего количества клеток с моно-BCMA-специфичным CAR и клеток с двойным CAR для BCMA/CD19). В некоторых вариантах осуществления композиция на основе клеток содержит популяцию моно-CD19-CAR+ клеток, численность которой составляет от приблизительно 45% до приблизительно 50% (например, приблизительно 47%) от количества BCMA-специфичных CAR-Т-клеток. В некоторых вариантах осуществления композиция на основе клеток содержит популяцию моно-CD19-CAR+ клеток, численность которой составляет от приблизительно 60% до приблизительно 65% (например, приблизительно 63%) от количества BCMA-специфичных CAR-Т-клеток. В некоторых вариантах осуществления композиция на основе клеток содержит популяцию моно-CD19-CAR+ клеток, численность которой составляет от приблизительно 50% до приблизительно 55% (например, приблизительно 53%) от количества BCMA-специфичных CAR-Т-клеток. В некоторых вариантах осуществления композиция на основе клеток содержит популяцию моно-CD19-CAR+ клеток, численность которой составляет приблизительно 82% от количества BCMA-специфичных CAR-Т-клеток.
В некоторых вариантах осуществления композиция на основе клеток можно оценивать для определения процентной доли CAR-положительных жизнеспособных клеток для обеспечения достаточного введения дозы. Соответственно, в некоторых вариантах осуществления композиция на основе клеток содержит популяцию клеток с BCMA-специфичным CAR (например, общее количество клеток с моно-BCMA-специфичным CAR и клеток с двойным CAR для BCMA/CD19), численность которой больше или равняется приблизительно 5% (например, больше или равняется приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 99% или 100%) от общего количества жизнеспособных клеток в композиции на основе клеток.
В некоторых вариантах осуществления клетки с BCMA-специфичным CAR представляют собой BCMA-специфичные CAR-Т-клетки. В некоторых вариантах осуществления клетки с моно-BCMA-специфичным CAR представляют собой моно-BCMA-специфичные CAR-Т-клетки. В некоторых вариантах осуществления клетки с CD19-специфичным CAR представляют собой CD19-специфичные CAR-Т-клетки. В некоторых вариантах осуществления клетки с моно-CD19-специфичным CAR представляют собой моно-CD19-специфичные CAR-Т-клетки. В некоторых вариантах осуществления клетки с двойным CAR для BCMA/CD19 представляют собой CAR-Т-клетки с двойной специфичностью к BCMA/CD19.
Введение доз
В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет приблизительно 1×106, 1,1×106, 2×106, 3,6×106, 5×106, 1×107, 1,8×107, 2×107, 5×107, 1×108, 2×108 или 5×108 клеток/кг. В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет по меньшей мере приблизительно 1×106, 1,1×106, 2×106, 3,6×106, 5×106, 1×107, 1,8×107, 2×107, 5×107, 1×108, 2×108 или 5×108 клеток/кг. В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет вплоть до приблизительно 1×106, 1,1×106, 2×106, 3,6×106, 5×106, 1×107, 1,8×107, 2×107, 5×107, 1×108, 2×108 или 5×108 клеток/кг. В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет приблизительно 1,1×106-1,8×107 клеток/кг. В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет приблизительно 1×107, 2×107, 5×107, 1×108, 2×108, 5×108, 1×109, 2×109 или 5×109 клеток. В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет по меньшей мере приблизительно 1×107, 2×107, 5×107, 1×108, 2×108, 5×108, 1×109, 2×109 или 5×109 клеток. В некоторых вариантах осуществления доза клеток с CAR (например, клеток с CAR для CD19) составляет вплоть до приблизительно 1×107, 2×107, 5×107, 1×108, 2×108, 5×108, 1×109, 2×109 или 5×109 клеток.
В некоторых вариантах осуществления субъекту может потребоваться введение активированных иммунных эффекторных клеток (например, T-клеток, NK-клеток), и тогда последовательно повторно берут кровь (или осуществляют аферез), активируют иммунные эффекторные клетки (например, T-клетки, NK-клетки) из нее и проводят повторную инфузию этих активированных и размноженных иммунных эффекторных клеток (например, T-клеток, NK-клеток) пациенту. Этот процесс можно осуществлять несколько раз каждые несколько недель. В некоторых вариантах осуществления иммунные эффекторные клетки (например, T-клетки, NK-клетки) можно активировать из отобранных образцов крови объемом от 10 куб. см до 400 куб. см. В некоторых вариантах осуществления иммунные эффекторные клетки (например, T-клетки, NK-клетки) активируют из отобранных образцов крови объемом 20 куб. см, 30 куб. см, 40 куб. см, 50 куб. см, 60 куб. см, 70 куб. см, 80 куб. см, 90 куб. см или 100 куб. см.
Введение рассматриваемых композиций можно осуществлять любым удобным способом. Композиции, описанные в данном документе, можно вводить пациенту трансартериально, подкожно, внутрикожно, интратуморально, интранодально, интрамедуллярно, внутримышечно, с помощью внутривенной (i.v.) инъекции или внутрибрюшинно, например, с помощью внутрикожной или подкожной инъекции. Композиции на основе иммунных эффекторных клеток (например, T-клеток, NK-клеток) можно инъецировать непосредственно в опухоль, лимфатический узел или очаг инфекции.
Истощение популяции Т-клеток
В некоторых вариантах осуществления способы, раскрытые в данном документе, дополнительно включают введение средства, обеспечивающего истощение популяции Т-клеток, после лечения с помощью клетки (например, иммунной эффектoрной клетки, описанной в данном документе), за счет чего обеспечивается снижение количества (например, истощение популяции) клеток, экспрессирующих CAR (например, клеток, экспрессирующих CAR для CD19). Такие средства, обеспечивающие истощение популяции Т-клеток, можно применять для эффективного истощения популяции клеток, экспрессирующих CAR (например, клеток, экспрессирующих CAR для CD19), для уменьшения токсичности. В некоторых вариантах осуществления клетки, экспрессирующие CAR, были изготовлены в соответствии со способом, описанным в данном документе, например, подвергнуты анализу (например, до или после трансфекции или трансдукции) в соответствии со способом, описанным в данном документе.
В некоторых вариантах осуществления средство, обеспечивающее истощение популяции Т-клеток, вводят через одну, две, три, четыре или пять недель после введения клетки, например, популяции иммунных эффекторных клеток, описанной в данном документе.
В некоторых вариантах осуществления средство, обеспечивающее истощение популяции Т-клеток, представляет собой средство, которое обеспечивает истощение популяции Т-клеток, экспрессирующих CAR, например, посредством индукции антителозависимой клеточноопосредованной цитотоксичности(ADCC) и/или комплемент-индуцированной гибели клеток. Например, клетки, экспрессирующие CAR, описанные в данном документе, могут также экспрессировать антиген (например, антиген-мишень), который распознается молекулами, способными индуцировать гибель клеток, например, ADCC или комплемент-индуцированную гибель клеток. Например, клетки, экспрессирующие CAR, описанные в данном документе, также могут экспрессировать белок-мишень (например, рецептор), на который могут нацеливаться антитело или фрагмент антитела. Примеры таких белков-мишеней включают без ограничения EpCAM, VEGFR, интегрины (например, интегрины ανβ3, α4, αΙ3/4β3, α4β7, α5β1, ανβ3, αν), представителей суперсемейства рецепторов TNF (например, TRAIL-R1, TRAIL-R2), рецептор PDGF, рецептор интерферона, рецептор фолиевой кислоты, GPNMB, ICAM-1, HLA-DR, CEA, CA-125, MUC1, TAG-72, рецептор IL-6, 5T4, GD2, GD3, CD2, CD3, CD4, CD5, CD11, CD11a/LFA-1, CD15, CD18/ITGB2, CD19, CD20, CD22, CD23/рецептор IgE, CD25, CD28, CD30, CD33, CD38, CD40, CD41, CD44, CD51, CD52, CD62L, CD74, CD80, CD125, CD147/базиджин, CD152/CTLA-4, CD154/CD40L, CD195/CCR5, CD319/SLAMF7 и EGFR и их усеченные варианты (например, варианты, в которых сохраняются один или несколько внеклеточных эпитопов, но отсутствуют одной или нескольких областей в цитоплазматическом домене).
В некоторых вариантах осуществления клетка, экспрессирующая CAR, совместно экспрессирует CAR и белок-мишень, например, естественным образом экспрессирует белок-мишень или сконструирована таким образом, что она экспрессирует белок-мишень. Например, клетка, например, популяция иммунных эффекторных клеток, может содержать нуклеиновую кислоту (например, вектор), содержащую нуклеиновую кислоту CAR (например, нуклеиновую кислоту CAR, описанную в данном документе) и нуклеиновую кислоту, кодирующую белок-мишень.
В некоторых вариантах осуществления средство, обеспечивающее истощение популяции Т-клеток, представляет собой ингибитор CD52, например, молекулу антитела к CD52, например, алемтузумаб.
В других вариантах осуществления клетка, например, популяция иммунных эффекторных клеток, экспрессирует молекулу CAR, описанную в данном документе (например, CAR для CD19), и белок-мишень, распознаваемый средством, обеспечивающим истощение популяции Т-клеток. В некоторых вариантах осуществления белок-мишень представляет собой CD20. В ряде вариантов осуществления, в которых белок-мишень представляет собой CD20, средство, обеспечивающее истощение популяции Т-клеток, представляет собой антитело к CD20, например, ритуксимаб.
В дополнительных вариантах осуществления любого из вышеупомянутых способов способы дополнительно включают трансплантацию клетки, например, гемопоэтической стволовой клетки, или костного мозга в организм млекопитающего.
В некоторых вариантах осуществления настоящего изобретения описан способ подготовки млекопитающего перед трансплантацией клеток. Способ включает введение млекопитающему эффективного количества клетки, содержащей нуклеиновую кислоту или полипептид CAR, например, нуклеиновую кислоту или полипептид CAR для CD19. В некоторых вариантах осуществления трансплантация клеток представляет собой трансплантацию стволовых клеток, например, трансплантацию гемопоэтических стволовых клеток, или трансплантацию костного мозга. В других вариантах осуществления подготовка субъекта перед трансплантацией клеток включает снижение у субъекта количества клеток, экспрессирующих мишень, например, нормальных клеток, экспрессирующих CD19, или раковых клеток, экспрессирующих CD19.
Режим введения доз
В некоторых вариантах осуществления доза жизнеспособных клеток, экспрессирующих CAR (например, жизнеспособных клеток, экспрессирующих CAR для CD19, BCMA, CD20 или CD22), или фармацевтической композиции, содержащей указанные клетки содержит от приблизительно 1×106 до приблизительно 1×108 (например, от приблизительно 2×106 до приблизительно 5×107, от приблизительно 5×106 до приблизительно 2×107, от приблизительно 1×106 до приблизительно 1×107, от приблизительно 1×107 до приблизительно 1×108, от приблизительно 1×106 до приблизительно 3×106, от приблизительно 2×106 до приблизительно 4×106, от приблизительно 3×106 до приблизительно 5×106, от приблизительно 4×106 до приблизительно 6×106, от приблизительно 5×106 до приблизительно 7×106, от приблизительно 6×106 до приблизительно 8×106, от приблизительно 7×106 до приблизительно 9×106, от приблизительно 8×106 до приблизительно 1×107, от приблизительно 9×106 до приблизительно 2×107, от приблизительно 1×107 до приблизительно 3×107, от приблизительно 2×107 до приблизительно 4×107, от приблизительно 3×107 до приблизительно 5×107, от приблизительно 4×107 до приблизительно 6×107, от приблизительно 5×107 до приблизительно 7×107, от приблизительно 6×107 до приблизительно 8×107, от приблизительно 7×107 до приблизительно 9×107, от приблизительно 8×107 до приблизительно 1×108, приблизительно 1×106, приблизительно 2×106, приблизительно 3×106, приблизительно 4×106, приблизительно 5×106, приблизительно 6×106, приблизительно 7×106, приблизительно 8×106, приблизительно 9×106, приблизительно 1×107, приблизительно 2×107, приблизительно 3×107, приблизительно 4×107, приблизительно 5×107, приблизительно 6×107, приблизительно 7×107, приблизительно 8×107, приблизительно 9×107 или приблизительно 1×108) CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток). В некоторых вариантах осуществления доза жизнеспособных клеток, экспрессирующих CAR (например, жизнеспособных клеток, экспрессирующих CAR для CD19, BCMA, CD20 или CD22), составляет от приблизительно 0,5×106 жизнеспособных клеток, экспрессирующих CAR, до приблизительно 1,25×109 жизнеспособных клеток, экспрессирующих CAR (например, от 0,5×106 жизнеспособных клеток, экспрессирующих CAR, до 1,25×109 жизнеспособных клеток, экспрессирующих CAR). В некоторых вариантах осуществления доза жизнеспособных клеток, экспрессирующих CAR (например, жизнеспособных клеток, экспрессирующих CAR для CD19, BCMA, CD20 или CD22), составляет приблизительно 1×106, приблизительно 2,5×106, приблизительно 5×106, приблизительно 1,25×107, приблизительно 2,5×107, приблизительно 5×107, приблизительно 5,75×107 или приблизительно 8×107 жизнеспособных клеток, экспрессирующих CAR.
В некоторых вариантах осуществления дозу рассчитывают на основании количества жизнеспособных BCMA-CAR+ T-клеток (например, клеток, положительных по одному CAR для BCMA, и клеток, положительных по двум CAR для BCMA+/CD19+), измеренном с помощью проточной цитометрии в день 4 (через 96 часов) после трансдукции (день 3 (через 72 часа) после сбора), как описано в данном документе. В некоторых вариантах осуществления доза жизнеспособных клеток, экспрессирующих CAR (например, клеточного продукта на основе CAR-T с двойной специфичностью к BCMA/CD19), содержит от приблизительно 5×106 до приблизительно 2×107 CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток).
В некоторых вариантах осуществления дозу жизнеспособных клеток, экспрессирующих CAR (например, жизнеспособных клеток, экспрессирующих CAR для CD19, BCMA, CD20 или CD22), или фармацевтической композиции, содержащей указанные клетки, вводят субъекту в одной или нескольких (например, 2, 3, 4 или больше) дозах. В некоторых вариантах осуществления клетки или фармацевтическую композицию вводят субъекту в двух дозах. В некоторых вариантах осуществления одна или несколько доз включают первую дозу и вторую дозу, где количество CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) в первой дозе больше количества, равняется количеству или меньше количества CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) во второй дозе.
В некоторых вариантах осуществления одна или несколько доз включают первую дозу и вторую дозу, где:
(a) первая доза содержит от приблизительно 1×106 до приблизительно 1×107 (например, от приблизительно 2×106 до приблизительно 8×106, от приблизительно 4×106 до приблизительно 6×106, от приблизительно 1×106 до приблизительно 5×106, от приблизительно 5×106 до приблизительно 1×107, от приблизительно 1×106 до приблизительно 3×106, от приблизительно 2×106 до приблизительно 4×106, от приблизительно 3×106 до приблизительно 5×106, от приблизительно 4×106 до приблизительно 6×106, от приблизительно 5×106 до приблизительно 7×106, от приблизительно 6×106 до приблизительно 8×106, от приблизительно 7×106 до приблизительно 9×106, от приблизительно 8×106 до приблизительно 1×107, приблизительно 1×106, приблизительно 2×106, приблизительно 3×106, приблизительно 4×106, приблизительно 5×106, приблизительно 6×106, приблизительно 7×106, приблизительно 8×106, приблизительно 9×106 или приблизительно 1×107) жизнеспособных CAR-положительных клеток (например, BCMA-CAR+ Т-клеток);
(b) вторая доза содержит от приблизительно 1×107 до приблизительно 1×108 (например, от приблизительно 2×107 до приблизительно 8×107, от приблизительно 4×107 до приблизительно 6×107, от приблизительно 1×107 до приблизительно 5×107, от приблизительно 5×107 до приблизительно 1×108, от приблизительно 1×107 до приблизительно 3×107, от приблизительно 2×107 до приблизительно 4×107, от приблизительно 3×107 до приблизительно 5×107, от приблизительно 4×107 до приблизительно 6×107, от приблизительно 5×107 до приблизительно 7×107, от приблизительно 6×107 до приблизительно 8×107, от приблизительно 7×107 до приблизительно 9×107, от приблизительно 8×107 до приблизительно 1×108, приблизительно 1×107, приблизительно 2×107, приблизительно 3×107, приблизительно 4×107, приблизительно 5×107, приблизительно 6×107, приблизительно 7×107, приблизительно 8×107, приблизительно 9×107 или приблизительно 1×108) CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток);
(c) количество CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) в первой дозе составляет не более чем 1/X, где X равняется 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 30, 40, 50, 60, 70, 80, 90 или 100, от количества CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) во второй дозе; и/или
(d) количество CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) в первой дозе составляет от приблизительно 1% до 100% (например, от приблизительно 10% до приблизительно 90%, от приблизительно 20% до приблизительно 80%, от приблизительно 30% до приблизительно 70%, от приблизительно 40% до приблизительно 60%, от приблизительно 10% до приблизительно 50%, от приблизительно 50% до приблизительно 90%, от приблизительно 10% до приблизительно 30%, от приблизительно 20% до приблизительно 40%, от приблизительно 30% до приблизительно 50%, от приблизительно 50% до приблизительно 70%, от приблизительно 60% до приблизительно 80% или от приблизительно 70% до приблизительно 90%) от количества CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток) во второй дозе.
В некоторых вариантах осуществления первая доза содержит приблизительно 5×106 жизнеспособных CAR-положительных клеток (например, BCMA-CAR+ Т-клеток). В некоторых вариантах осуществления вторая доза содержит приблизительно 1×107 или приблизительно 2×107 жизнеспособных CAR-положительных клеток (например, BCMA-CAR+ Т-клеток).
В некоторых вариантах осуществления доза CAR-положительных клеток может быть увеличена относительно начальной дозы при последующем введении. Например, пациент может получать начальную дозу, составляющую от приблизительно 1×106 до приблизительно 1×107 (например, приблизительно 5×106) жизнеспособных CAR-положительных клеток, и может получать вторую дозу, составляющую от приблизительно 1×107 до приблизительно 1×108 (например, приблизительно 1×107 или приблизительно 2×107) CAR-положительных жизнеспособных клеток (например, BCMA-CAR+ Т-клеток).
Отбор пациентов
В некоторых вариантах осуществления любого из способов лечения субъекта или композиции для применения, раскрытых в данном документе, у субъекта имеется рак, например, гематологический рак. В некоторых вариантах осуществления рак выбран из лимфоцитарного лейкоза (CLL), мантийноклеточной лимфомы (MCL), множественной миеломы, острого лимфоидного лейкоза (ALL), лимфомы Ходжкина, B-клеточного острого лимфоидного лейкоза (BALL), T-клеточного острого лимфоидного лейкоза (TALL), мелкоклеточного лимфоцитарного лейкоза (SLL), B-клеточного пролимфоцитарного лейкоза, новообразования из бластных плазмоцитоидных дендритных клеток, лимфомы Беркитта, диффузной крупноклеточной В-клеточной лимфомы (DLBCL), DLBCL, ассоциированной с хроническим воспалением, хронического миелоидного лейкоза, миелопролиферативных новообразований, фолликулярной лимфомы, педиатрической фолликулярной лимфомы, волосатоклеточного лейкоза, мелкоклеточной или крупноклеточной фолликулярной лимфомы, злокачественных лимфопролиферативных состояний, MALT-лимфомы (экстранодальной лимфомы из клеток маргинальной зоны из лимфоидной ткани слизистых оболочек), лимфомы из клеток маргинальной зоны, миелодисплазии, миелодиспластического синдрома, неходжкинской лимфомы, плазмобластной лимфомы, новообразования из плазмоцитоидных дендритных клеток, макроглобулинемии Вальденстрема, лимфомы из клеток маргинальной зоны селезенки, лимфомы/лейкоза селезенки, диффузной мелкоклеточной B-клеточной лимфомы красной пульпы селезенки, варианта волосатоклеточного лейкоза, лимфоплазмоцитарной лимфомы, болезни тяжелых цепей, плазмоклеточной миеломы, солитарной плазмоцитомы кости, внекостной плазмоцитомы, нодулярной лимфомы из клеток маргинальной зоны, педиатрической нодулярной лимфомы из клеток маргинальной зоны, первичной кожной лимфомы из клеток центра фолликула, лимфоматоидного гранулематоза, первичной медиастинальной (тимической) крупноклеточной B-клеточной лимфомы, внутрисосудистой крупноклеточной B-клеточной лимфомы, ALK+ крупноклеточной B-клеточной лимфомы, крупноклеточной B-клеточной лимфомы, возникающей при HHV8-ассоциированной многоочаговой болезни Кастлемана, первичной выпотной лимфомы, B-клеточной лимфомы, острого миелоидного лейкоза (AML) или неклассифицируемой лимфомы. В некоторых вариантах осуществления рак представляет собой рецидивирующий и/или рефрактерный рак.
В некоторых вариантах осуществления любого из способов лечения субъекта или композиции для применения, раскрытых в данном документе, у субъекта имеется CLL или SLL В некоторых вариантах осуществления субъекту, у которого имеется CLL или SLL, ранее вводили средство терапии на основе ингибитора BTK, например, ибрутиниб, в течение по меньшей мере 1-12 месяцев, например, 6 месяцев. В некоторых вариантах осуществления средство терапии на основе ингибитора BTK, например, средство терапии ибрутиниб, представляет собой средство терапии второй линии. В некоторых вариантах осуществления у субъекта наблюдался частичный ответ или наблюдалась стабилизация заболевания в ответ на средство терапии на основе ингибитора BTK. В некоторых вариантах осуществления у субъекта не наблюдался ответ на средство терапии на основе ингибитора BTK. В некоторых вариантах осуществления у субъекта развилась устойчивость, например, развились мутации, придающие устойчивость к ибрутинибу. В некоторых вариантах осуществления мутации, придающие устойчивость к ибрутинибу, включают мутацию в гене, кодирующем BTK, и/или гене, кодирующем PLCg2. В некоторых вариантах осуществления субъект представляет собой взрослую особь, например, в возрасте по меньшей мере 18 лет.
В некоторых вариантах осуществления любого из способов лечения субъекта или композиции для применения, раскрытых в данном документе, у субъекта имеется DLBCL, например, рецидивирующая и/или рефрактерная DLBCL. В некоторых вариантах осуществления субъекту, у которого имеется DLBCL, например, рецидивирующая и/или рефрактерная DLBCL, ранее вводили по меньшей мере 2 линии средств химиотерапии, например, средство терапии на основе антитела к CD20 и/или средство химиотерапии на основе антрациклина. В некоторых вариантах осуществления субъект ранее получал средство терапии на основе стволовых клеток, например, средство терапии на основе аутологичных стволовых клеток, и у него не наблюдался ответ на указанное средство терапии на основе стволовых клеток. В некоторых вариантах осуществления субъект не соответствует критериям для применения средства терапии на основе стволовых клеток, например, средства терапии на основе аутологичных стволовых клеток. В некоторых вариантах осуществления субъект представляет собой взрослую особь, например, в возрасте по меньшей мере 18 лет.
Терапевтическое применение
Заболевания и/или нарушения, ассоциированные с ВСМА
В одном аспекте в настоящем изобретении предусмотрены способы лечения заболевания, ассоциированного с экспрессией ВСМА. В одном аспекте в настоящем изобретении предусмотрены способы лечения заболевания, где часть опухоли является отрицательной по BCMA, а часть опухоли является положительной по BCMA. Например, CAR по настоящему изобретению является применимым для лечения субъектов, которые прошли лечение заболевания, ассоциированного с повышенной экспрессией BCMA, где у субъекта, прошедшего лечение при повышенных уровнях BCMA, проявляется заболевание, ассоциированное с повышенными уровнями BCMA. В ряде вариантов осуществления CAR по настоящему изобретению является применимым для лечения субъектов, которые прошли лечение заболевания, ассоциированного с экспрессией BCMA, где у субъекта, прошедшего лечение, связанное с экспрессией BCMA, проявляется заболевание, ассоциированное с экспрессией BCMA.
В одном варианте осуществления в настоящем изобретении предусмотрены способы лечения заболевания, при котором BCMA экспрессируется как на нормальных клетках, так и на раковых клетках, но на нормальных клетках экспрессируется при более низких уровнях. В одном варианте осуществления способ дополнительно включает отбор CAR по настоящему изобретению, который связывается с такой аффинностью, которая позволяет CAR для ВСМА связывать и уничтожать раковые клетки, экспрессирующие ВСМА, но при этом уничтожать менее 30%, 25%, 20%, 15%, 10%, 5% или меньше нормальных клеток, экспрессирующих ВСМА, например, согласно определению с помощью анализа, описанного в данном документе. Например, можно применять анализ уничтожения, такой как проточная цитометрия с использованием CTL с Cr51. В одном варианте осуществления CAR для ВСМА имеет антигенсвязывающий домен, который характеризуется аффинностью связывания с KD в отношении антигена-мишени, составляющей от 10-4 М до 10-8 М, например, от 10-5 М до 10-7 М, например, 10-6 М или 10-7 М. В одном варианте осуществления антигенсвязывающий домен для ВСМА характеризуется аффинностью связывания, в по меньшей мере пять раз, 10 раз, 20 раз, 30 раз, 50 раз, 100 раз или 1000 раз меньшей, чем у эталонного антитела, например, антитела, описанного в данном документе.
В одном аспекте настоящее изобретение относится к вектору, содержащему CAR для ВСМА, функционально связанный с промотором для экспрессии в иммунных эффекторных клетках млекопитающего, например, T-клетках или NK-клетках. В одном аспекте в настоящем изобретении предусмотрена рекомбинантная иммунная эффектoрная клетка, например, T-клетка или NK-клетка, экспрессирующая CAR для BCMA, для применения в лечении опухолей, экспрессирующих BCMA, где рекомбинантная иммунная эффектoрная клетка (например, T-клетка или NK-клетка), экспрессирующая CAR для BCMA, называется клеткой, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клеткой или NK-клеткой, экспрессирующей CAR для BCMA). В одном аспекте клетка, экспрессирующая CAR для BCMA (например, BCMA-CAR-Т-клетка или NK-клетка, экспрессирующая CAR для BCMA), по настоящему изобретению способна вступать в контакт с опухолевой клеткой с по меньшей мере одним CAR для BCMA по настоящему изобретению, экспрессируемым на ее поверхности, так что клетка, экспрессирующая CAR для BCMA (например, BCMA-CAR-Т-клетка или NK-клетка, экспрессирующая CAR для BCMA), нацеливается на опухолевую клетку, и рост опухоли ингибируется.
В одном аспекте настоящее изобретение относится к способу ингибирования роста опухолевой клетки, экспрессирующей BCMA, включающий приведение опухолевой клетки в контакт с клеткой, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клеткой или NK-клеткой, экспрессирующей CAR для BCMA), по настоящему изобретению, так что клетка, экспрессирующая CAR для BCMA (например, BCMA-CAR-T-клетка или NK-клетка, экспрессирующая CAR для BCMA), активируется в ответ на антиген и нацеливается на раковую клетку, при этом рост опухоли ингибируется.
В одном аспекте настоящее изобретение относится к способу лечения рака у субъекта. Способ включает введение субъекту клетки, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клетки или NK-клетки, экспрессирующей CAR для BCMA), по настоящему изобретению, так что у субъекта осуществляется лечение рака. Примером рака, поддающегося лечению с помощью клетки, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клетки или NK-клетки, экспрессирующей CAR для BCMA) по настоящему изобретению, является рак, ассоциированный с экспрессией BCMA.
Настоящее изобретение включает тип клеточной терапии, где иммунные эффекторные клетки (например, T-клетки или NK-клетки) подвергнуты генетической модификации таким образом, что они экспрессируют химерный антигенный рецептор (CAR), и при этом клетку, экспрессирующую CAR для BCMA (например, BCMA-CAR-T-клетку или NK-клетку, экспрессирующую CAR для BCMA), вводят посредством инфузии нуждающемуся в этом реципиенту. Введенная посредством инфузии клетка способна уничтожать опухолевые клетки у реципиента. В отличие от терапевтических средств на основе антител, CAR-модифицированные клетки, например, Т-клетки или NK-клетки, способны реплицироваться in vivo, что приводит в результате к долгосрочной персистенции, которая может обеспечивать устойчивый контроль опухоли. В различных аспектах клетки (например, T-клетки, NK-клетки), введенные пациенту, или их потомство персистируют у пациента в течение по меньшей мере четырех месяцев, пяти месяцев, шести месяцев, семи месяцев, восьми месяцев, девяти месяцев, десяти месяцев, одиннадцати месяцев, двенадцати месяцев, тринадцати месяцев, четырнадцати месяцев, пятнадцати месяцев, шестнадцати месяцев, семнадцати месяцев, восемнадцати месяцев, девятнадцати месяцев, двадцати месяцев, двадцати одного месяца, двадцати двух месяцев, двадцати трех месяцев, двух лет, трех лет, четырех лет или пяти лет после введения пациенту клетки (например, T-клетки или NK-клетки).
Настоящее изобретение также включает тип клеточной терапии, при которой иммунные эффекторные клетки (например, Т-клетки или NK-клетки) модифицированы, например, с помощью РНК, транскрибированной in vitro, таким образом, что они транзиентно экспрессируют химерный антигенный рецептор (CAR), и иммунную эффекторную клетку (например, Т-клетку или NK-клетку) вводят посредством инфузии реципиенту, нуждающемуся в этом. Введенная посредством инфузии клетка способна уничтожать опухолевые клетки у реципиента. Таким образом, в различных аспектах иммунные эффекторные клетки (например, Т-клетки или NK-клетки), вводимые пациенту, присутствуют в течение менее одного месяца, например, в течение трех недель, двух недель, одной недели, после введения пациенту иммунной эффекторной клетки (например, Т-клетки или NK-клетки).
Не ограничиваясь какой-либо теорией, полагают, что противоопухолевый иммунный ответ, вызываемый CAR-модифицированными иммунными эффекторными клетками (например, T-клетками или NK-клетками), может быть активным или пассивным иммунным ответом или в качестве альтернативы может быть обусловлен прямым или непрямым иммунным ответом. В одном аспекте иммунные эффекторные клетки (например, Т-клетки или NK-клетки), трансдуцированные с помощью CAR, демонстрируют секрецию специфических провоспалительных цитокинов и сильную цитолитическую активность в ответ на опухолевые клетки человека, экспрессирующие ВСМА, противодействуют ингибированию со стороны растворимого ВСМА, опосредуют неспецифический цитолиз и опосредуют регрессию развившейся опухоли у человека. Например, опухолевые клетки, не содержащие антигена, в гетерогенной области опухоли, экспрессирующей ВСМА, могут быть восприимчивы к непрямому разрушению ВСМА-перенаправленными иммунными эффекторными клетками (например, Т-клетками или NK-клетками), которые ранее вступали в реакцию против соседних раковых клеток, положительных по антигену.
В одном аспекте полностью человеческие CAR-модифицированные иммунные эффекторные клетки (например, T-клетки или NK-клетки) по настоящему изобретению могут представлять собой тип вакцины для иммунизации ex vivo и/или терапии in vivo у млекопитающего. В одном аспекте млекопитающее является человеком.
Что касается иммунизации ex vivo, то перед введением клетки млекопитающему происходит по меньшей мере одно из следующих событий in vitro: i) размножение клеток, ii) введение нуклеиновой кислоты, кодирующей CAR, в клетки или iii) криоконсервация клеток.
Процедуры ex vivo хорошо известны из уровня техники и более подробно обсуждаются ниже. Вкратце, клетки выделяют у млекопитающего (например, человека) и генетически модифицируют (т. е. трансдуцируют или трансфицируют in vitro) с помощью вектора, экспрессирующего CAR, раскрытый в данном документе. CAR-модифицированную клетку можно вводить реципиенту-млекопитающему для обеспечения терапевтически благоприятного эффекта. Реципиентом-млекопитающим может быть человек, и CAR-модифицированная клетка может быть аутологичной по отношению к реципиенту. В качестве альтернативы клетки могут быть аллогенными, сингенными или ксеногенными по отношению к реципиенту.
Процедуру размножения ex vivo гемопоэтических стволовых клеток и клеток-предшественников, описанную в патенте США № 5199942, включенном в данный документ посредством ссылки, можно применять по отношению к клеткам по настоящему изобретению. Другие подходящие способы известны из уровня техники, поэтому настоящее изобретение не ограничивается каким-либо конкретным способом размножения клеток ex vivo. Вкратце, культивирование и размножение Т-клеток ex vivo включает (1) сбор CD34+ гемопоэтических стволовых клеток и клеток-предшественников млекопитающего из собранного материала периферической крови или эксплантатов костного мозга и (2) размножение таких клеток ex vivo. В дополнение к клеточным факторам роста, описанным в патенте США № 5199942, для культивирования и размножения клеток можно применять другие факторы, такие как flt3-L, IL-1, IL-3 и лиганд c-kit.
В дополнение к применению клеточной вакцины с целью иммунизации ex vivo, в настоящем изобретении также предусмотрены композиции и способы для иммунизации in vivo для того, чтобы вызвать иммунный ответ, направленный на антиген у пациента.
Как правило, клетки, активированные и размноженные так, как это описано в данном документе, можно использовать в лечении и предупреждении заболеваний, возникающих у индивидуумов с ослабленным иммунитетом. Например, CAR-модифицированные иммунные эффекторные клетки (например, Т-клетки или NK-клетки) по настоящему изобретению применяют в лечении заболеваний, нарушений и состояний, ассоциированных с экспрессией ВСМА. В некоторых аспектах клетки по настоящему изобретению применяют в лечении пациентов с риском развития заболеваний, нарушений и состояний, ассоциированных с экспрессией ВСМА. Таким образом, в настоящем изобретении предусмотрены способы лечения или предупреждения заболеваний, нарушений и состояний, ассоциированных с экспрессией ВСМА, включающие введение субъекту, нуждающемуся в этом, терапевтически эффективного количества CAR-модифицированных иммунных эффекторных клеток (например, Т-клеток или NK-клеток) по настоящему изобретению.
В одном аспекте клетки, экспрессирующие CAR (например, CAR-T-клетки или NK-клетки, экспрессирующие CAR), по настоящему изобретению можно применять для лечения пролиферативного заболевания, такого как рак или злокачественное новообразование, или предракового состояния, такого как миелодисплазия, миелодиспластический синдром или предлейкоз. В одном аспекте рак представляет собой гематологический рак. Состояния, связанные с гематологическими формами рака, представляют собой типы рака, такие как лейкоз и злокачественные лимфопролиферативные состояния, при которых поражается кровь, костный мозг и лимфатическая система. В одном аспекте гематологический рак представляет собой лейкоз или гематологическое заболевание. Примером заболевания или нарушения, ассоциированного с BCMA, является множественная миелома (также известная как MM) (см. Claudio et al., Blood. 2002, 100(6):2175-86; и Novak et al., Blood. 2004, 103(2):689-94). Множественная миелома, также известная как плазмоклеточная миелома или болезнь Калера, представляет собой рак, характеризующийся накоплением аномальных или злокачественных плазматических B-клеток в костном мозге. Часто раковые клетки внедряются в смежную кость, разрушая скелетные структуры, что приводит к боли в костях и переломам. Большинство случаев миеломы также характеризуются продуцированием парапротеина (также известного как M-белки или миеломные белки), который представляет собой аномальный иммуноглобулин, продуцируемый в избытке вследствие клональной пролиферации злокачественных плазматических клеток. Уровни парапротеина в сыворотке крови, составляющие более 30 г/л, являются диагностическим критерием множественной миеломы в соответствии с диагностическими критериями Международной группы по изучению множественной миеломы (IMWG) (см. Kyle et al. (2009), Leukemia. 23:3-9). Другие симптомы или признаки множественной миеломы включают снижение функции почек или почечную недостаточность, очаги поражения костей, анемию, гиперкальциемию и неврологические симптомы.
Критерии для проведения различий между множественной миеломой и другими нарушениями пролиферации плазматических клеток были установлены Международной группой по изучению множественной миеломы (см. Kyle et al. (2009), Leukemia. 23:3-9). Должны удовлетворяться все три следующих критерия:
- клональные плазматические клетки костного мозга ≥ 10%;
- наличие моноклонального белка в сыворотке крови и/или моче (за исключением пациентов с истинной несекретирующей множественной миеломой);
- свидетельство поражения органов-мишеней, обусловленного лежащим в его основе нарушением пролиферации плазматических клеток, а именно:
o гиперкальциемия: уровень кальция в сыворотке крови ≥ 11,5 мг/100 мл,
o почечная недостаточность: уровень креатинина в сыворотке крови > 1,73 ммоль/л,
o анемия: нормохромная, нормоцитарная с величиной уровня гемоглобина, которая на > 2 г/100 мл ниже нижней границы нормы, или с величиной уровня гемоглобина < 10 г/100 мл,
o очаги поражения костей: очаги остеолитического поражения, тяжелая остеопения или патологические переломы.
Другие нарушения пролиферации плазматических клеток, которые можно лечить с помощью композиций и способов, описанных в данном документе, включают без ограничения бессимптомную миелому ("тлеющую" множественную миелому или вялотекущую миелому), моноклональную гаммапатию неустановленной этиологии (MGUS), макроглобулинемию Вальденстрема, формы плазмоцитомы (например, плазмоклеточную дискразию, солитарную миелому, солитарную плазмоцитому, экстрамедуллярную плазмоцитому и множественную плазмоцитому), системный амилоидоз в форме амилоидоза легких цепей и POEMS-синдром (также известный как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром).
Для стадирования множественной миеломы используют две системы стадирования: Международную систему стадирования (ISS)(см. Greipp et al. (2005), J. Clin. Oncol. 23 (15):3412-3420) и систему стадирования по Дьюри-Сэлмону (DSS) (см. Durie et al. (1975), Cancer 36 (3): 842-854).
Третья система стадирования множественной миеломы называется пересмотренной Международной системой стадирования (R-ISS) (см. Рalumbo A, Avet-Loiseau H, Oliva S, et al. Journal of clinical oncology: official journal of the American Society of Clinical Oncology 2015;33:2863-9, включенную в данный документ посредством ссылки во всей своей полноте). Стадия I R-ISS включает стадию I ISS (уровень сывороточного β2-микроглобулина < 3,5 мг/л и уровень сывороточного альбумина ≥ 3,5 г/дл), отсутствие CA, соответствующих высокому риску [del(17p), и/или t(4;14), и/или t(14;16)], и уровень LDH в пределах нормы (меньше верхней границы нормального диапазона). Стадия III R-ISS включает стадию III ISS (уровень сывороточного β2-микроглобулина > 5,5 мг/л) и CA, соответствующие высокому риску, или высокий уровень LDH. Стадия II R-ISS включает все остальные возможные комбинации.
Ответы у пациентов можно определить согласно критериям IMWG 2016, раскрытым в Kumar S, Paiva B, Anderson KC, et al. International Myeloma Working Group consensus criteria for response and minimal residual disease assessment in multiple myeloma. The Lancet Oncology;17(8):e328-e346, включенной в данный документ посредством ссылки во всей своей полноте.
Стандартное лечение множественной миеломы и ассоциированных заболеваний включает химиотерапию, трансплантацию стволовых клеток (аутологичную или аллогенную), лучевую терапию и другие виды лекарственной терапии. Лекарственные средства, часто применяемые для лечения миеломы, включают алкилирующие средства (например, бендамустин, циклофосфамид и мелфалан), ингибиторы протеасом (например, бортезомиб), кортикостероиды (например, дексаметазон и преднизон) и иммуномодуляторы (например, талидомид и леналидомид или Revlimid®) или любую их комбинацию. Лекарственные средства из группы бисфосфонатов также часто вводят в комбинации со стандартными средствами лечения MM для предупреждения потери костной ткани. Пациенты старше 65-70 лет являются маловероятными кандидатами на трансплантацию стволовых клеток. В некоторых случаях возможными вариантами лечения для пациентов младше 60 лет с субоптимальным ответом на первую трансплантацию являются двойные трансплантации аутологичных стволовых клеток. Композиции и способы по настоящему изобретению можно вводить в комбинации с любым из назначаемых в настоящее время средств лечения множественной миеломы.
Другим примером заболевания или нарушения, ассоциированного с BCMA, является лимфома Ходжкина и неходжкинская лимфома (см. Chiu et al., Blood. 2007, 109(2):729-39; He et al., J Immunol. 2004, 172(5):3268-79).
Лимфома Ходжкина (HL), также известная как болезнь Ходжкина, представляет собой рак лимфатической системы, который происходит из белых кровяных телец или лимфоцитов. Аномальные клетки, которые образуют лимфому, называются клетками Рид-Штернберга. При лимфоме Ходжкина происходит распространение рака из одной группы лимфатических узлов в другую. Лимфому Ходжкина можно подразделить на четыре патологических подтипа на основании морфологических характеристик клеток Рид-Штернберга и клеточного состава вокруг клеток Рид-Штернберга (что определяют посредством биопсии лимфатических узлов): HL с нодулярным склерозом, смешанно-клеточный подтип, богатая лимфоцитами или с лимфоидным преобладанием, с лимфоидным истощением. Иногда лимфома Ходжкина также может представлять собой нодулярную лимфому Ходжкина с лимфоидным преобладанием или может быть неуточненной. Симптомы и признаки лимфомы Ходжкина включают безболезненное увеличение лимфатических узлов в области шеи, подмышечных впадин или паха, лихорадку, ночную потливость, потерю веса, утомляемость, кожный зуд или боль в животе.
Неходжкинская лимфома (NHL) представляет собой группу разнообразных форм рака крови, которые включают любую разновидность лимфомы, отличную от лимфомы Ходжкина. Подтипы неходжкинской лимфомы классифицируют в основном по морфологическим характеристикам клеток, хромосомным аберрациям и поверхностным маркерам. Подтипы NHL (или NHL-ассоциированные формы рака) включают В-клеточные лимфомы, такие как, без ограничения, лимфома Беркитта, хронический лимфоцитарный В-клеточный лейкоз (B-CLL), пролимфоцитарный B-клеточный лейкоз (B-PLL), хронический лимфоцитарный лейкоз (CLL), диффузная крупноклеточная В-клеточная лимфома (DLBCL) (например, внутрисосудистая крупноклеточная B-клеточная лимфома и первичная медиастинальная B-клеточная лимфома), фолликулярная лимфома (например, лимфома из клеток центра фолликула, фолликулярная мелкоклеточная лимфома с расщепленными ядрами), волосатоклеточный лейкоз, B-клеточная лимфома высокой степени злокачественности (подобная лимфоме Беркитта), лимфоплазмоцитарная лимфома (макроглобулинемия Вальденстрема), мантийноклеточная лимфома, формы B-клеточной лимфомы из клеток маргинальной зоны (например, экстранодальная B-клеточная лимфома из клеток маргинальной зоны или лимфома из лимфоидной ткани слизистых оболочек (MALT), нодулярная В-клеточная лимфома из клеток маргинальной зоны и В-клеточная лимфома из клеток маргинальной зоны селезенки), плазмоцитома/миелома, B-лимфобластный лейкоз/лимфома из клеток-предшественников (PB-LBL/L), первичная лимфома центральной нервной системы (CNS), первичная внутриглазная лимфома, мелкоклеточная лимфоцитарная лимфома (SLL); и T-клеточные лимфомы, такие как, без ограничения, анапластическая крупноклеточная лимфома (ALCL), T-клеточная лимфома/лейкоз взрослых (например, "тлеющая", хроническая, острая и лимфоматозная), лимфангиома, ангиоиммунобластная Т-клеточная лимфома, формы T-клеточной лимфомы кожи (например, фунгоидный микоз, синдром Сезари и т. д.), внеузловая лимфома из естественных киллеров/T-клеток (назального типа), Т-клеточная лимфома кишечника энтеропатического типа, лейкоз из больших гранулярных лимфоцитов, T-лимфобластная лимфома/лейкоз из клеток-предшественников (T-LBL/L), T-клеточный хронический лимфоцитарный лейкоз/пролимфоцитарный лейкоз (T-CLL/PLL) и неуточненная периферическая T-клеточная лимфома. Симптомы и признаки лимфомы Ходжкина включают безболезненное увеличение лимфатических узлов в области шеи, подмышечных впадин или паха, лихорадку, ночную потливость, потерю веса, утомляемость, кожный зуд, боль в животе, кашель или боль в груди.
Стадирование лимфомы Ходжкина и неходжкинской лимфомы является одинаковым и относится к степени распространения раковых клеток в организме. На стадии I клетки лимфомы присутствуют в одной группе лимфатических узлов. На стадии II клетки лимфомы присутствуют в по меньшей мере двух группах лимфатических узлов, но обе группы расположены с одной стороны диафрагмы или в одной части ткани или органа, а лимфатические узлы рядом с этим органом с той же стороны диафрагмы. На стадии III клетки лимфомы присутствуют в лимфатических узлах с обеих сторон диафрагмы или в одной части ткани или органа рядом с этими группами лимфатических узлов или в селезенке. На стадии IV клетки лимфомы обнаруживаются в нескольких частях по меньшей мере одного органа или ткани, или клетки лимфомы присутствуют в органе и в лимфатических узлах с другой стороны диафрагмы. В дополнение к обозначению стадий римскими цифрами стадии также могут быть описаны буквами A, B, E и S, где A обозначает пациентов без симптомов, B обозначает пациентов с симптомами, E обозначает пациентов, у которых лимфома обнаружена в тканях вне лимфатической системы, а S обозначает пациентов, у которых лимфома обнаружена в селезенке.
Лимфому Ходжкина обычно лечат с помощью лучевой терапии, химиотерапии или трансплантации гемопоэтических стволовых клеток. Наиболее распространенным средством терапии неходжкинской лимфомы является режим R-CHOP, который состоит из четырех различных химиотерапевтических препаратов (циклофосфамид, доксорубицин, винкристин и преднизолон) и ритуксимаба (Rituxan®). Другие средства терапии, обычно применяемые для лечения NHL, включают другие химиотерапевтические средства, лучевую терапию, трансплантацию стволовых клеток (аутологичную или аллогенную трансплантацию костного мозга) или биологическую терапию, такую как иммунотерапия. Другие примеры биологических терапевтических средств включают без ограничения ритуксимаб (Rituxan®), тозитумомаб (Bexxar®), эпратузумаб (LymphoCide®) и алемтузумаб (MabCampath®). Композиции и способы по настоящему изобретению можно вводить в комбинации с любым из назначаемых в настоящее время средств лечения лимфомы Ходжкина или неходжкинской лимфомы.
Также было установлено, что с экспрессией BCMA ассоциирована макроглобулинемия Вальденстрема (WM), также известная как лимфоплазмоцитарная лимфома (LPL) (см. Elsawa et al., Blood. 2006, 107(7):2882-8). Ранее считалось, что макроглобулинемия Вальденстрема является родственной по отношению к множественной миеломе, но совсем недавно ее классифицировали как подтип неходжкинской лимфомы. WM характеризуется неконтролируемой пролиферацией В-клеточных лимфоцитов, что приводит к анемии и продуцированию избыточных количеств парапротеина или иммуноглобулина М (IgM), который сгущает кровь и приводит к синдрому повышенной вязкости крови. Другие симптомы или признаки WM включают лихорадку, ночную потливость, утомляемость, анемию, потерю веса, лимфаденопатию или спленомегалию, нечеткость зрения, головокружение, носовые кровотечения, кровоточивость десен, нетипичные кровоподтеки, нарушение функции почек или почечную недостаточность, амилоидоз или периферическую нейропатию.
Стандартное лечение WM заключается в химиотерапии, в частности ритуксимабом (Rituxan®). Другие химиотерапевтические лекарственные средства, такие как хлорамбуцил (Leukeran®), циклофосфамид (Neosar®), флударабин (Fludara®), кладрибин (Leustatin®), винкристин и/или талидомид, можно применять в комбинации. Кортикостероиды, такие как преднизон, также можно вводить в комбинации с химиотерапией. Плазмаферез или замещение плазмы обычно применяют в ходе всего лечения пациента для облегчения некоторых симптомов посредством удаления парапротеина из крови. В некоторых случаях трансплантация стволовых клеток является вариантом лечения для некоторых пациентов.
Другим примером заболевания или нарушения, ассоциированного с BCMA, является рак головного мозга. В частности, было установлено, что с экспрессией ВСМА ассоциирована астроцитома или глиобластома (см. Deshayes et al, Oncogene. 2004, 23(17):3005-12, Pelekanou et al., PLoS One. 2013, 8(12):e83250). Астроцитомы представляют собой опухоли, которые возникают из астроцитов, представляющих собой тип глиальных клеток в головном мозге. Глиобластома (также известная как мультиформная глиобластома или GBM) является наиболее злокачественной формой астроцитомы и считается наиболее поздней стадией рака головного мозга (стадией IV). Существуют два варианта глиобластомы: гигантоклеточная глиобластома и глиосаркома. Другие формы астроцитомы включают ювенильную пилоцитарную астроцитому (JPA), фибриллярную астроцитому, плеоморфную ксантоастроцитому (PXA), дисэмбриопластическую нейроэпителиальную опухоль (DNET) и анапластическую астроцитому (AA).
Симптомы или признаки, ассоциированные с глиобластомой или астроцитомой, включают повышенное давление в головном мозге, головные боли, эпилептические припадки, потерю памяти, изменения в поведении, нарушение движения или потерю чувствительности на одной стороне тела, речевую дисфункцию, когнитивные нарушения, нарушения зрения, тошноту, рвоту и слабость в руках или ногах.
Хирургическое удаление опухоли (или резекция) является стандартным лечением для удаления как можно большей части глиомы без повреждения или с минимальным повреждением окружающей нормальной ткани головного мозга. Лучевая терапия и/или химиотерапия часто применяются после хирургического вмешательства для подавления и замедления рецидивирующего заболевания, обусловленного любыми оставшимися раковыми клетками или сопутствующими очагами поражения. Лучевая терапия включает лучевую терапию всего головного мозга (традиционную дистанционную лучевую терапию), целенаправленную трехмерную конформную лучевую терапию и применение целенаправленно воздействующих радионуклидов. Химиотерапевтические средства, обычно применяемые для лечения глиобластомы, включают темозоломид, гефитиниб или эрлотиниб и цисплатин. Ингибиторы ангиогенеза, такие как бевацизумаб (Avastin®), также обычно применяются в комбинации с химиотерапией и/или лучевой терапией.
Поддерживающее лечение также часто применяют для облегчения неврологических симптомов и улучшения неврологической функции и используют в комбинации с любым из средств терапии рака, описанных в данном документе. Основные поддерживающие средства включают противосудорожные средства и кортикостероиды. Таким образом, композиции и способы по настоящему изобретению можно применять в комбинации с любыми видами стандартного или поддерживающего лечения с целью лечения глиобластомы или астроцитомы.
Заболевания и нарушения, не связанные с раком, но ассоциированные с экспрессией ВСМА, также можно лечить с помощью композиций и способов, раскрытых в данном документе. Примеры заболеваний и нарушений, не связанных с раком, ассоциированных с экспрессией BCMA, включают без ограничения вирусные инфекции, например, вызванные HIV; грибковые инфекции, например, вызванные C. neoformans; синдром раздраженного кишечника; неспецифический язвенный колит и нарушения, связанные с иммунитетом слизистых оболочек.
CAR-модифицированные иммунные эффекторные клетки (например, T-клетки или NK-клетки) по настоящему изобретению можно вводить отдельно либо в фармацевтической композиции в комбинации с разбавителями и/или другими компонентами, такими как IL-2 или другие цитокины, или популяциями клеток.
В настоящем изобретении предусмотрены композиции и способы для лечения рака. В одном аспекте рак представляет собой гематологический рак, в том числе без ограничения гематологический рак, представляющий собой лейкоз или лимфому. В одном аспекте клетки, экспрессирующие CAR (например, CAR-T-клетки или NK-клетки, экспрессирующие CAR), по настоящему изобретению можно применять для лечения форм рака и злокачественных новообразований, таких как, без ограничения, например, формы острого лейкоза, в том числе без ограничения, например, B-клеточный острый лимфоидный лейкоз ("BALL"), T-клеточный острый лимфоидный лейкоз ("TALL"), острый лимфоидный лейкоз (ALL); одна или несколько форм хронического лейкоза, в том числе без ограничения, например, хронический миелогенный лейкоз (CML), хронический лимфоцитарный лейкоз (CLL); дополнительные формы гематологического рака или гематологические состояния, в том числе без ограничения, например, B-клеточный пролимфоцитарный лейкоз, новообразование из бластных плазмоцитоидных дендритных клеток, лимфома Беркитта, диффузная крупноклеточная В-клеточная лимфома, фолликулярная лимфома, волосатоклеточный лейкоз, мелкоклеточная или крупноклеточная фолликулярная лимфома, злокачественные лимфопролиферативные состояния, MALT-лимфома, мантийноклеточная лимфома, лимфома из клеток маргинальной зоны, множественная миелома, миелодисплазия и миелодиспластический синдром, неходжкинская лимфома, плазмобластная лимфома, новообразование из плазмоцитоидных дендритных клеток, макроглобулинемия Вальденстрема и "предлейкоз", который представляет собой разнообразную группу гематологических состояний, объединенных неэффективным образованием (или дисплазией) миелоидных клеток крови, и т. п. Кроме того, заболевание, ассоциированное с экспрессией ВСМА, включает без ограничения, например, атипичные и/или неклассические формы рака, злокачественные новообразования, предраковые состояния или пролиферативные заболевания, характеризующиеся экспрессией ВСМА.
В ряде вариантов осуществления композицию, описанную в данном документе, можно применять для лечения заболевания, включающего без ограничения нарушение пролиферации плазматических клеток, например, бессимптомную миелому ("тлеющую" множественную миелому или вялотекущую миелому), моноклональную гаммапатию неустановленной этиологии (MGUS), макроглобулинемию Вальденстрема, формы плазмоцитомы (например, дискразию плазматических клеток, солитарную миелому, солитарную плазмоцитому, экстрамедуллярную плазмоцитому и множественную плазмоцитому), системный амилоидоз в форме амилоидоза легких цепей и POEMS-синдром (также известный как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром).
В ряде вариантов осуществления композицию, описанную в данном документе, можно применять для лечения заболевания, включающего без ограничения рак, например, рак, описанный в данном документе, например, рак предстательной железы (например, кастрационнорезистентный или резистентный к терапии рак предстательной железы или метастатический рак предстательной железы), рак поджелудочной железы или рак легкого.
В настоящем изобретении также предусмотрены способы ингибирования пролиферации или уменьшения популяции клеток, экспрессирующих BCMA, при этом способы включают приведение популяции клеток, содержащей клетку, экспрессирующую BCMA, в контакт с клеткой, экспрессирующей CAR для BCMA (например, ВСМА-CAR-Т-клеткой или NK-клеткой, экспрессирующей CAR для BCMA), по настоящему изобретению, которая связывается с клеткой, экспрессирующей BCMA. В конкретном аспекте в настоящем изобретении предусмотрены способы ингибирования пролиферации или уменьшения популяции раковых клеток, экспрессирующих BCMA, при этом способы включают приведение популяции раковых клеток, экспрессирующих BCMA, в контакт с клеткой, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клеткой или NK-клеткой, экспрессирующей CAR для BCMA), по настоящему изобретению, которая связывается с клеткой, экспрессирующей BCMA. В одном аспекте в настоящем изобретении представлены способы ингибирования пролиферации или уменьшения популяции раковых клеток, экспрессирующих BCMA, при этом способы включают приведение популяции раковых клеток, экспрессирующих BCMA, в контакт с клеткой, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клеткой или NK-клеткой, экспрессирующей CAR для BCMA), по настоящему изобретению, которая связывается с клеткой, экспрессирующей BCMA. В некоторых аспектах клетка, экспрессирующая CAR для BCMA (например, BCMA-CAR-T-клетка или NK-клетка, экспрессирующая CAR для BCMA), по настоящему изобретению обеспечивает снижение уровня, числа, количества или процентной доли клеток и/или раковых клеток на по меньшей мере 25%, по меньшей мере 30%, по меньшей мере 40%, по меньшей мере 50%, по меньшей мере 65%, по меньшей мере 75%, по меньшей мере 85%, по меньшей мере 95% или по меньшей мере 99% у субъекта с миелоидным лейкозом или другой формой рака, ассоциированного с клетками, экспрессирующими BCMA, или в соответствующей животной модели по сравнению с отрицательным контролем. В одном аспекте субъект является человеком.
В настоящем изобретении также предусмотрены способы предупреждения, лечения и/или контроля заболевания, ассоциированного с клетками, экспрессирующими BCMA (например, гематологического рака или атипичного рака, характеризующегося экспрессией BCMA), при этом способы включают введение субъекту, нуждающемуся в этом, клетки, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клетки или NK-клетки, экспрессирующей CAR для BCMA), по настоящему изобретению, которая связывается с клеткой, экспрессирующей BCMA. В одном аспекте субъект является человеком. Неограничивающие примеры нарушений, ассоциированных с клетками, экспрессирующими ВСМА, включают вирусные или грибковые инфекции и нарушения, связанные с иммунитетом слизистых оболочек.
В настоящем изобретении также предусмотрены способы предупреждения, лечения и/или контроля заболевания, ассоциированного с клетками, экспрессирующими BCMA, при этом способы включают введение субъекту, нуждающемуся в этом, клетки, экспрессирующей CAR для BCMA, (например, BCMA-CAR-T-клетки или NK-клетки, экспрессирующей CAR для BCMA), по настоящему изобретению, которая связывается с клеткой, экспрессирующей BCMA. В одном аспекте субъект является человеком.
В настоящем изобретении предусмотрены способы предупреждения рецидива рака, ассоциированного с клетками, экспрессирующими BCMA, при этом способы включают введение субъекту, нуждающемуся в этом, клетки, экспрессирующей CAR для BCMA, (например, BCMA-CAR-T-клетки или NK-клетки, экспрессирующей CAR для BCMA), по настоящему изобретению, которая связывается с клеткой, экспрессирующей BCMA. В одном аспекте способы включают введение субъекту, нуждающемуся в этом, эффективного количества клетки, экспрессирующей CAR для BCMA (например, BCMA-CAR-T-клетки или NK-клетки, экспрессирующей CAR для BCMA), описанной в данном документе, которая связывается с клеткой, экспрессирующей BCMA, в комбинации с эффективным количеством другого средства терапии.
Виды комбинированной терапии
Описанную в данном документе клетку, экспрессирующую CAR, можно применять в комбинации с другими известными средствами и видами терапии. Описанную в данном документе клетку, экспрессирующую CAR, и по меньшей мере одно дополнительное терапевтическое средство можно вводить одновременно в одной и той же или отдельных композициях или последовательно. В случае последовательного введения описанную в данном документе клетку, экспрессирующую CAR, можно вводить первой, а дополнительное средство можно вводить вторым, или порядок введения может быть обратным. Средство CAR-терапии и/или другие терапевтические средства, процедуры или способы можно применять в периоды активного проявления нарушения или в период ремиссии или менее активного проявления заболевания. Средство CAR-терапии можно вводить перед другим средством лечения, параллельно со средством лечения, после средства лечения или во время ремиссии нарушения. При введении в комбинации средство CAR-терапии и дополнительное средство (например, второе или третье средство) или все из них можно вводить в количестве или дозе, которые являются более высокими, более низкими или такими же по сравнению с количеством или дозой каждого средства, применяемого отдельно, например, в качестве монотерапии. В некоторых вариантах осуществления вводимые количество или доза средства CAR-терапии, дополнительного средства (например, второго или третьего средства) или всех из них являются более низкими (например, на по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40% или по меньшей мере 50%), чем количество или доза каждого средства, применяемого отдельно, например, в качестве монотерапии. В других вариантах осуществления количество или доза средства CAR-терапии, дополнительного средства (например, второго или третьего средства) или всех из них, которые приводят к требуемому эффекту (например, лечению рака), являются более низкими (например, на по меньшей мере 20%, по меньшей мере 30%, по меньшей мере 40% или по меньшей мере 50% ниже), чем количество или доза каждого средства, применяемого отдельно, например, в качестве монотерапии, необходимые для достижения того же терапевтического эффекта. В следующих аспектах описанная в данном документе клетка, экспрессирующая CAR, может применяться в режиме лечения в комбинации с хирургическим вмешательством, химиотерапией, лучевой терапией, иммуносупрессивными средствами. Иллюстративные средства и виды терапии, которые можно применять в комбинации с клеткой, экспрессирующей CAR, описанной в данном документе, раскрыты на страницах 266-313 в WO2016164731, которая включена в данный документ посредством ссылки во всей своей полноте.
Биомаркеры для оценивания эффективности CAR
В некоторых вариантах осуществления в данном документе раскрыт способ оценивания или мониторинга эффективности средства терапии на основе клеток, экспрессирующих CAR (например, средства терапии на основе CAR для CD19 или BCMA), у субъекта (например, субъекта, у которого имеется рак, например, гематологический рак). Способ включает сбор данных о величине эффективности средства CAR-терапии, где указанная величина является показателем эффективности или пригодности средства терапии на основе клеток, экспрессирующих CAR.
В ряде вариантов осуществления величина эффективности средства CAR-терапии у субъекта, у которого имеется CLL или SLL, включает показатель одного, двух, трех или всех из следующих параметров:
(i) мутации в гене, кодирующем BTK, в образце (например, образце, полученном путем афереза, или образце изготовленного продукта на основе клеток, экспрессирующих CAR);
(ii) мутации в гене, кодирующем PLCg2, в образце (например, образце, полученном путем афереза, или образце изготовленного продукта на основе клеток, экспрессирующих CAR);
(iii) минимального остаточного заболевания, например, оцененного по уровню и/или активности CD8, CD4, CD3, CD5, CD19, CD20, CD22, CD43, CD79b, CD27, CD45RO, CD45RA, CCR7, CD95, LAG3, PD-1, TIM-3 и/или CD81; или оцененного с помощью глубокого секвенирования иммуноглобулинов; в образце (например, образце, полученном путем афереза, или образце опухоли от субъекта); или
(iv) уровня или активности одного, двух, трех, четырех, пяти, шести, семи, восьми, девяти, десяти или всех из цитокинов, выбранных из IFN-g, IL-2, IL-4, IL-6, IL-8, IL-10, IL-15, TNF-a, IP-10, MCP1, MIP1a, в образце, например, образце, полученном путем афереза, от субъекта.
В ряде вариантов осуществления величина эффективности средства CAR-терапии у субъекта, у которого имеется DLBCL, например, рецидивирующая и/или рефрактерная DLBCL, включает показатель одного или обоих из следующих параметров:
(i) минимального остаточного заболевания, например, оцененного по уровню и/или активности CD8, CD4, CAR19, CD3, CD27, CD45RO, CD45RA, CCR7, CD95, LAG3, PD-1 и/или TIM-3; или оцененного с помощью глубокого секвенирования иммуноглобулинов; в образце (например, образце, полученном путем афереза, или образце опухоли от субъекта); или
(ii) уровня или активности одного, двух, трех, четырех, пяти, шести, семи, восьми, девяти, десяти или всех из цитокинов, выбранных из IFN-g, IL-2, IL-4, IL-6, IL-8, IL-10, IL-15, TNF-a, IP-10, MCP1, MIP1a, в образце (например, образце, полученном путем афереза, от субъекта).
В других вариантах осуществления величина эффективности средства CAR-терапии дополнительно включает показатель одного, двух, трех, четырех, пяти, шести или более (всех) из следующих параметров:
(i) уровня или активности одного, двух, трех или более (например, всех) из покоящихся TEFF-клеток, покоящихся TREG-клеток, более молодых T-клеток (например, необученных Т-клеток (например, необученных CD4 или CD8 T-клеток, необученных гамма/дельта-T-клеток)), или стволовых T-клеток памяти (например, стволовых CD4 или CD8 T-клеток памяти или стволовых гамма/дельта-T-клеток памяти), или ранних T-клеток памяти или их комбинации в образце (например, образце, полученном путем афереза, или образце изготовленного продукта на основе клеток, экспрессирующих CAR);
(ii) уровня или активности одного, двух, трех или более (например, всех) из активированных TEFF-клеток, активированных TREG-клеток, более старых T-клеток (например, более старых CD4 или CD8 клеток) или поздних T-клеток памяти или их комбинации в образце (например, образце, полученном путем афереза, или образце изготовленного продукта на основе клеток, экспрессирующих CAR);
(iii) уровня или активности маркера истощения иммунных клеток, например, одного, двух или более ингибиторов контрольных точек иммунного ответа (например, PD-1, PD-L1, TIM-3, TIGIT и/или LAG-3), в образце (например, образце, полученном путем афереза, или образце изготовленного продукта на основе клеток, экспрессирующих CAR). В некоторых вариантах осуществления иммунная клетка имеет фенотип истощения, например, совместно экспрессирует по меньшей мере два маркера истощения, например, совместно экспрессирует PD-1 и TIM-3. В других вариантах осуществления иммунная клетка имеет фенотип истощения, например, совместно экспрессирует по меньшей мере два маркера истощения, например, совместно экспрессирует PD-1 и LAG-3;
(iv) уровня или активности CD27 и/или CD45RO- (например, CD27+ CD45RO-) иммунных эффектoрных клеток, например, в популяции CD4+ или CD8+ Т-клеток, в образце (например, образце, полученном путем афереза, или образце изготовленного продукта на основе клеток, экспрессирующих CAR);
(v) уровня или активности одного, двух, трех, четырех, пяти, шести, семи, восьми, девяти, десяти, одиннадцати или всех из биомаркеров, выбранных из CCL20, IL-17a, IL-6, PD-1, PD-L1, LAG-3, TIM-3, CD57, CD27, CD122, CD62L, KLRG1;
(vi) уровня или активности цитокинов (например, качества репертуара цитокинов) в образце продукта на основе клеток, экспрессирующих CAR, например, образце продукта на основе клеток, экспрессирующих CLL-1; или
(vii) эффективности трансдукции клетки, экспрессирующей CAR, в образце изготовленного продукта на основе клеток, экспрессирующих CAR.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, средство терапии на основе клеток, экспрессирующих CAR, содержит множество (например, популяцию) иммунных эффекторных клеток, экспрессирующих CAR, например, множество (например, популяцию) T-клеток или NK-клеток или их комбинацию. В некоторых вариантах осуществления средство терапии на основе клеток, экспрессирующих CAR, представляет собой средство терапии на основе CAR для CD19.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, показатель одного или нескольких параметров, раскрытых в данном документе, получают из образца, полученного путем афереза, взятого у субъекта. Образец, полученный путем афереза, можно оценить перед инфузией или повторной инфузией.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, показатель одного или нескольких параметров, раскрытых в данном документе, получают из образца опухоли, взятого у субъекта.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, показатель одного или нескольких параметров, раскрытых в данном документе, получают из образца изготовленного продукта на основе клеток, экспрессирующих CAR, например, образца продукта на основе клеток, экспрессирующих CAR для CD19. Изготовленный продукт на основе клеток, экспрессирующих CAR, можно оценить перед инфузией или повторной инфузией.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, оценивание субъекта проводят перед получением, во время или после получения средства терапии на основе клеток, экспрессирующих CAR.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, по показателю одного или нескольких параметров, раскрытых в данном документе, оценивается профиль одного или нескольких из экспрессии гена, проточной цитометрии или экспрессии белка.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, способ дополнительно включает идентификацию субъекта как пациента, отвечающего на лечение, пациента, не отвечающего на лечение, пациента с рецидивом или пациента без рецидива на основании показателя одного или нескольких из параметров, раскрытых в данном документе.
В некоторых вариантах осуществления любого из способов раскрытых в данном документе, пациент, отвечающий на лечение, например, пациент с полным ответом на лечение, имеет или идентифицирован как имеющий более высокую, например, статистически значимо более высокую процентную долю CD8+ T-клеток по сравнению с эталонным значением, например, процентной долей CD8+ T-клеток у пациента, не отвечающего на лечение.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, пациент, отвечающий на лечение, например, пациент с полным ответом на лечение, имеет или идентифицирован как имеющий более высокую процентную долю CD27+ CD45RO- иммунных эффекторных клеток, например, в популяции CD8+, по сравнению с эталонным значением, например, количеством CD27+ CD45RO- иммунных эффекторных клеток у пациента, не отвечающего на лечение.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, пациент, отвечающий на лечение, например, пациент с полным ответом на лечение или пациент с частичным ответом на лечение, имеет или идентифицирован как имеющий более высокую, например, статистически значимо более высокую, процентную долю CD4+ T-клеток по сравнению с эталонным значением, например, процентной долей CD4+ T-клеток у пациента, не отвечающего на лечение.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, пациент, отвечающий на лечение, например, пациент с полным ответом на лечение, имеет или идентифицирован как имеющий более высокую процентную долю одного, двух, трех или более (например, всех) из покоящихся TEFF-клеток, покоящихся TREG-клеток, более молодых T-клеток или ранних T-клеток памяти или их комбинации по сравнению с эталонным значением, например, количеством покоящихся TEFF-клеток, покоящихся TREG-клеток, более молодых T-клеток или ранних T-клеток памяти у пациента, не отвечающего на лечение.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, пациент, не отвечающий на лечение, имеет или идентифицирован как имеющий более высокую процентную долю одного, двух, трех или более (например, всех) из активированных TEFF-клеток, активированных TREG-клеток, более старых T-клеток (например, более старых CD4 или CD8 клеток) или поздних T-клеток памяти или их комбинации по сравнению с эталонным значением, например, количеством активированных TEFF-клеток, активированных TREG-клеток, более старых T-клеток (например, более старых CD4 или CD8 клеток) или поздних T-клеток памяти у пациента, отвечающего на лечение.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, пациент, не отвечающий на лечение, имеет или идентифицирован как имеющий более высокий процентный уровень маркера истощения иммунных клеток, например, одного, двух или более ингибиторов контрольных точек иммунного ответа (например, PD-1, PD-L1, TIM-3, TIGIT и/или LAG-3). В некоторых вариантах осуществления пациент, не отвечающий на лечение, имеет или идентифицирован как имеющий более высокую процентную долю иммунных эффекторных клеток, экспрессирующих PD-1, PD-L1 или LAG-3 (например, CD4+ T-клеток и/или CD8+ T-клеток) (например, CD4+ клеток и/или CD8+ T-клеток, экспрессирующих CAR), по сравнению с процентной долей иммунных эффекторных клеток, экспрессирующих PD-1 или LAG-3, у пациента, отвечающего на лечение.
В некоторых вариантах осуществления пациент, не отвечающий на лечение, имеет или идентифицирован как имеющий более высокую процентную долю иммунных клеток, имеющих фенотип истощения, например, иммунных клеток, которые совместно экспрессируют по меньшей мере два маркера истощения, например, совместно экспрессируют PD-1, PD-L1 и/или TIM-3. В других вариантах осуществления пациент, не отвечающий на лечение, имеет или идентифицирован как имеющий более высокую процентную долю иммунных клеток, имеющих фенотип истощения, например, иммунных клеток, которые совместно экспрессируют по меньшей мере два маркера истощения, например, совместно экспрессируют PD-1 и LAG-3.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, пациент, не отвечающий на лечение, имеет или идентифицирован как имеющий более высокую процентную долю PD-1/PD-L1+/LAG-3+ клеток в популяции клеток, экспрессирующих CAR (например, популяции CLL-1-CAR+ клеток), по сравнению с пациентом, отвечающим на лечение (например, пациентом с полным ответом на лечение) с помощью средства терапии на основе клеток, экспрессирующих CAR.
В некоторых вариантах осуществления любого из способов, раскрытых в данном документе, у пациента, отвечающего на лечение (например, пациента с полным или частичным ответом на лечение), имеются один, два, три или больше (или все) из следующих профилей:
(i) имеется большее количество CD27+ иммунных эффекторных клеток по сравнению с эталонным значением, например, количеством CD27+ иммунных эффектoрных клеток у пациента, не отвечающего на лечение;
(ii) имеется большее количество CD8+ T-клеток по сравнению с эталонным значением, например, количеством CD8+ T-клеток у пациента, не отвечающего на лечение;
(iii) имеется меньшее количество иммунных клеток, экспрессирующих один или несколько ингибиторов контрольных точек иммунного ответа, например, ингибиторов контрольных точек иммунного ответа, выбранных из PD-1, PD-L1, LAG-3, TIM-3 или KLRG-1 или их комбинации, по сравнению с эталонным значением, например, количеством клеток, экспрессирующих один или несколько ингибиторов контрольных точек иммунного ответа, у пациента, не отвечающего на лечение; или
(iv) имеется большее количество одного, двух, трех, четырех или более (всех) из покоящихся TEFF-клеток, покоящихся TREG-клеток, необученных CD4 клеток, нестимулированных клеток памяти или ранних T-клеток памяти или их комбинации по сравнению с эталонным значением, например, количеством покоящихся TEFF-клеток, покоящихся TREG-клеток, необученных CD4 клеток, нестимулированных клеток памяти или ранних T-клеток памяти у пациента, не отвечающего на лечение.
В ряде вариантов осуществления субъект, который является пациентом, отвечающим на лечение, пациентом, не отвечающим на лечение, пациентом с рецидивом или пациентом без рецидива, идентифицированным посредством способов, изложенных в данном документе, может быть подвергнут дополнительной оценке в соответствии с клиническими критериями. Например, пациент с полным ответом на лечение является субъектом или идентифицирован как субъект, у которого имеется заболевание, например, рак, у которого проявляется полный ответ на лечение, например, полная ремиссия. Полный ответ на лечение можно идентифицировать, например, с помощью NCCN Guidelines® или руководства, утвержденного на международном семинаре по хроническому лимфоцитарному лейкозу (iwCLL) 2018 года, как раскрыто в Hallek M et al., Blood (2018) 131:2745-2760 "iwCLL guidelines for diagnosis, indications for treatment, response assessment, and supportive management of CLL", полное содержание которого включено в данный документ посредством ссылки во всей своей полноте. Пациент с частичным ответом на лечение является субъектом или идентифицирован как субъект, у которого имеется заболевание, например, рак, у которого проявляется частичный ответ на лечение, например, частичная ремиссия. Частичный ответ можно идентифицировать, например, с помощью критериев NCCN Guidelines® или iwCLL 2018 года, как описано в данном документе. Пациент, не отвечающий на лечение, является субъектом или идентифицирован как субъект, у которого имеется заболевание, например, рак, у которого не проявляется ответ на лечение, например, у пациента наблюдается стабилизация заболевания или прогрессирование заболевания. Пациента, не отвечающего на лечение, можно идентифицировать, например, с помощью критериев NCCN Guidelines® или iwCLL 2018 года, как описано в данном документе.
В качестве альтернативы или в комбинации со способами, раскрытыми в данном документе, с учетом указанной величины осуществляется одно, два, три, четыре или более из следующего:
введения, например, пациенту, отвечающему на лечение, или пациенту без рецидива средства терапии на основе клеток, экспрессирующих CAR;
введения измененных доз средства терапии на основе клеток, экспрессирующих CAR;
изменения схемы или периода введения средства терапии на основе клеток, экспрессирующих CAR;
введения, например, пациенту, не отвечающему на лечение, или пациенту с частичным ответом на лечение дополнительного средства в комбинации со средством терапии на основе клеток, экспрессирующих CAR, например, ингибитора контрольной точки иммунного ответа, например, ингибитора контрольной точки иммунного ответа, описанного в данном документе;
введения пациенту, не отвечающему на лечение, или пациенту с частичным ответом на лечение средства терапии, которое обеспечивает увеличение количества более молодых T-клеток у субъекта, перед проведением лечения с помощью средства терапии на основе клеток, экспрессирующих CAR;
модификации способа изготовления средства терапии на основе клеток, экспрессирующих CAR, например, обогащения более молодыми T-клетками перед введением нуклеиновой кислоты, кодирующей CAR, или увеличения эффективности трансдукции, например, у субъекта, идентифицированного как пациент, не отвечающий на лечение, или пациент с частичным ответом на лечение;
введения альтернативного средства терапии, например, пациенту, не отвечающему на лечение, или пациенту с частичным ответом на лечение, или пациенту с рецидивом; или,
в случае, если субъект является пациентом, не отвечающим на лечение, или пациентом с рецидивом или идентифицирован в таковом качестве, уменьшения популяции TREG-клеток и/или уровня экспрессии генной сигнатуры TREG, например, посредством одного или нескольких из истощения по CD25, введения циклофосфамида, антитела к GITR или их комбинации.
ПРИМЕРЫ
Настоящее изобретение дополнительно подробно описано посредством ссылки на следующие экспериментальные примеры. Эти примеры приведены лишь в целях иллюстрации и не предполагаются как ограничивающие, если не указано иное. Таким образом, настоящее изобретение никоим образом не следует истолковывать как ограниченное следующими примерами, а, наоборот, следует истолковывать как охватывающее все без исключения видоизменения, которые становятся очевидными как следствие идеи, приведенной в данном документе.
Пример 1. Определение характеристик CAR для BCMA человека in vitro
Набор полностью человеческих одноцепочечных вариабельных фрагментов (scFv) клонировали в лентивирусные векторы экспрессии CAR с CD3-дзета-цепью и стимулирующими молекулами 4-1BB: R1B6, R1F2, R1G5, PI61, B61-10, B61-02, Hy03 и Hy52. Первоначально конструкции подвергали скринингу с помощью автоматизированного анализа в репортерных клетках с последующим отбором оптимальных клонов на основании экспрессии на первичных Т-клетках, а также количества и качества ответов с участием эффекторных Т-клеток ("BCMA-CAR-T" или "BCMA-CAR-T-клеток") в ответ на мишени, экспрессирующие BCMA ("BCMA+" или "BCMA-положительные"). Ответы с участием эффекторных T-клеток включают без ограничения размножение, пролиферацию, удвоение, продуцирование цитокинов и уничтожение клеток-мишеней или цитолитическую активность (дегрануляцию) у клеток.
Получение лентивирусного вектора, кодирующего CAR для BCMA
Все вышеуказанные лентивирусные векторы для переноса, кодирующие scFv, использовали для получения геномного материала, упакованного в псевдотипированные с VSVg лентивирусные частицы. ДНК лентивирусного вектора для переноса, кодирующую CAR, смешивали с тремя упаковочными компонентами VSVg, gag/pol и rev в комбинации с реагентом липофектамином для трансфекции клеток Lenti-X 293T (Clontech) с последующей заменой среды спустя 12-18 ч. Через 30 часов после замены среды среду собирали, фильтровали и хранили при -80°C.
JNL с CAR для BCMA и скрининговый анализ в репортерных клетках JNL с помощью автоматической системы
Для анализа в репортерных клетках лентивирус, кодирующий CAR для BCMA, получали в клетках HEK293 при двух различных показателях плотности клеток (40000 клеток (1x H293) или 80000 клеток (2x H293)) в автоматизированном мелкомасштабном режиме в 96-луночных планшетах, при этом содержащую вирус надосадочную жидкость собирали через 48 часов после трансфекции и использовали в свежем виде без замораживания для трансдукции линии репортерных Т-клеток Jurkat. Линия репортерных клеток Jurkat с NFAT-люциферазой (JNL) получена на основе линии острого Т-клеточного лейкоза Jurkat. Линию модифицировали таким образом, чтобы она экспрессировала люциферазу под контролем элемента ответа на ядерный фактор активированных Т-клеток (NFAT). Для трансдукции с помощью CAR для BCMA 10000 клеток JNL/лунка 96-луночного планшета трансдуцировали 50 мкл свежей содержащей вирус надосадочной жидкости, отфильтрованной через фильтр на 45 мкм. Планшеты культивировали в течение 5 дней перед совместным культивированием с клетками-мишенями.
Для оценивания функциональной способности CAR для BCMA активировать клетки JNL их совместно культивировали с раковыми клетками-мишенями при различных соотношениях эффекторных клеток и клеток-мишеней (соотношениях E:T) для регистрации их активации путем количественного определения экспрессии люциферазы. Оценивали CAR R1B6, R1F2, R1G5, PI61, B61-10, B61-02, Hy03 и Hy52 на основе scFv. Клетки JNL с CAR для CD19 использовали в качестве контроля, специфичного для мишени, и среда в отдельности без клеток-мишеней служила в качестве отрицательного контроля.
Вышеупомянутые пятидневные трансдуцированные клетки JNL с CAR совместно культивировали с BCMA-положительной линией клеток множественной миеломы (MM) KMS11 или NALM6 - линией клеток острого лимфоцитарного лейкоза, служившей в качестве BCMA-отрицательного контроля. Оставшиеся CAR-Т-клетки JNL оценивали в отношении экспрессии CAR для BCMA с помощью проточной цитометрии. Совместные культуры подготавливали в 384-луночных планшетах при соотношениях эффекторов и мишеней (E:T), составляющих 4:1, 2:1, 1:1 и 0,5:1, и инкубировали в течение 24 часов, после чего количественно определяли экспрессию люциферазы по активированным CAR-Т-клеткам JNL с помощью системы анализа люциферазы Bright-Glo™ (Promega, Мэдисон, Висконсин). Количество света, испускаемого из каждой лунки (люминесценция), было прямым регистрируемым показателем активации JNL соответствующим CAR. Клетки JNL считались активированными, если уровень люминесценции равнялся уровню в клетках UTD или более чем в два раза превышал его. BCMA+ линия клеток KMS11 приводила к активации клеток JNL, экспрессирующих R1B6, R1F2, R1G5, PI61, B61-10, B61-02, Hy03 и Hy52 (фиг. 1A и 1C). Ни один из CAR для ВСМА не продемонстрировал активации BCMA-отрицательной линией NALM6 (фиг. 1E и 1F). Среда в отдельности без клеток-мишеней не активировала никакие из тестируемых JNL, трансдуцированных с помощью CAR (фиг. 1G и 1H). Анализы методом FACS продемонстрировали, что экспрессия CAR для BCMA в трансдуцированных JNL обнаруживалась в разной степени; % CAR обычно положительно коррелирует с активацией JNL клетками KMS11 для наиболее активных CAR-T JNL (фиг. 1B и 1D).
Получение BCMA-CAR-T-клеток
Следующие 8 CAR были выбраны для анализа экспрессии, стабильности и эффективности CAR в первичных Т-клетках: R1B6, R1F2, R1G5, PI61, B61-10, B61-02, Hy03 и Hy52. BCMA-CAR-T-клетки получали, начиная с крови здоровых подвергнутых аферезу доноров, Т-клетки которых (CD4+ и CD8+ лимфоциты) были получены путем отрицательного отбора в отношении CD3+ Т-клеток. Эти клетки активировали путем добавления гранул с антителами к CD3/CD28 (Dynabeads® Human T-Expander CD3/CD28, Thermo Fisher Scientific) при соотношении 1:3 (T-клеток и гранул) в среде для T-клеток (RPMI-1640, 10% инактивированной нагреванием эмбриональной телячьей сыворотки (FCS), 2 мM L-глутамина, 1x пенициллина/стрептомицина, 100 мкM заменимых аминокислот, 1 мM пирувата натрия, 10 мM HEPES и 55 мкM 2-меркаптоэтанола). T-клетки культивировали при 0,5×106 Т-клеток в 1 мл среды на лунку 24-луночного планшета при 37°C, 5% CO2. Через 24 часа, когда Т-клетки проходили бластообразование, Т-клетки трансдуцировали с помощью вируса, кодирующего CAR для BCMA, при множественности заражения (MOI), составляющей 5. Т-клетки начинали делиться с логарифмическим паттерном роста, который отслеживали путем измерения количества клеток на мл, и Т-клетки разбавляли свежей средой каждые два дня, от них удаляли гранулы, и их собирали для дальнейших анализов в день 9. Аликвоты Т-клеток окрашивали для измерения экспрессии CAR с помощью проточной цитометрии в дни 5 и 9 на FACS Fortessa (BD). Все BCMA-CAR-T-клетки были получены в условиях изготовления исследовательского уровня (т. е. не клинического уровня).
Поверхностную экспрессию CAR для BCMA и ее стабильность оценивали путем измерения % CAR и MFI (средней интенсивности флуоресценции) в день 5 и день 9 с использованием анализов методом проточной цитометрии клеток, окрашенных с помощью rBCMA_Fc-AF647 (фиг. 2 и таблица 27). Экспрессия CAR для BCMA в конечном продукте в день 9 отличается для разных конструкций и находится в диапазоне от 18% до 42,4%, а MFI - от 672 до 5238. Конструкции из полученных из PALLAS клонов R1F2, R1B6 и R1G5 и гибридомного клона Hy03 демонстрировали потерю CAR от ~ 30% до 50% с дня 5 по день 9, тогда как PI61, B61-10 и -02, а также Hy52 были относительно стабильными с точки зрения процентного значения экспрессии CAR, хотя все конструкции CAR демонстрировали уменьшение MFI с дня 5 по день 9, что, вероятно, было обусловлено меньшим размером Т-клеток на их стадии покоя в день 9. Подсчет клеток в культурах CAR-Т-клеток указывал на отсутствие поддающегося выявлению отрицательного эффекта CAR для BCMA, несущего scFv человека, в отношении способности клеток нормально размножаться по сравнению с нетрансдуцированными Т-клетками ("UTD").
Оценивание функциональных свойств T-клеток, перенаправленных с помощью CAR для BCMA
Для оценивания функциональных возможностей BCMA-CAR-T-клеток подготавливали совместные культуры с BCMA-положительными и BCMA-отрицательными линиями раковых клеток. CAR-T-клетки размораживали, подсчитывали и культивировали совместно с клетками-мишенями для регистрации их способностей к уничтожению и секреции цитокинов. Тестировали клоны CAR для BCMA R1B6, R1F2, R1G5, B61-02, B61-10, PI61, Hy03 и Hy52. Нетрансдуцированные Т-клетки (UTD) использовали в качестве контрольных ненацеливающихся Т-клеток.
Уничтожение с помощью CAR-T-клеток осуществляли путем совместного культивирования CAR-T-клеток с клетками-мишенями KMS11-Luc и NALM6-Luc при различных соотношениях E:T в течение 20 часов. Популяции CAR-Т-клеток нормализовали по эквивалентным процентным долям CAR-положительных клеток перед посевом. Цитокин IFNγ измеряли в надосадочных жидкостях 20-часовых совместных культур CAR-T-клеток с клетками-мишенями при соотношении эффекторов и мишеней, составляющем 2,5:1, используя Meso Scale Discovery (MSD; Гейтерсберг, Мэриленд), и результаты для каждого цитокина рассчитывали в пг/мл с использованием известных стандартов. Все анализы выполняли в двух повторностях из одного источника донорских клеток. Данные об уничтожении показывают, что все клоны CAR для BCMA эффективно уничтожают раковые клетки KMS11 (фиг. 3A). Контрольные клетки-мишени NALM6 не уничтожались ни одним из этих BCMA-специфичных CAR (фиг. 3B). Также тестировали способность этих CAR продуцировать IFNγ при культивировании с KMS11 (фиг. 3C). CAR для BCMA R1F2, R1G5 и PI61 приводили к продуцированию наиболее высоких количеств IFNγ. Уровни цитокинов, продуцируемых BCMA-CAR-T после воздействия контрольных клеток NALM6, были низкими (фиг. 3C), что указывает на отсутствие неспецифической активации с помощью CAR для BCMA.
Выводы
Новые scFv, связывающие BCMA, тестировали в качестве составной части CAR-Т-клеток. Восемь CAR анализировали в анализе в репортерных клетках JNL, а также в первичных Т-клетках: R1B6, R1F2, R1G5, B61-02, B61-10, PI61, Hy03 и Hy52. Все восемь типов CAR-T-клеток демонстрировали уничтожение, специфичное для мишени. Т-клетки, экспрессирующие R1F2, R1G5 или PI61, продуцировали наиболее высокие количества IFNγ в присутствии клеток-мишеней. В целом, перенос CAR для ВСМА в первичные Т-клетки индуцировал реактивность CAR в отношении ВСМА, но не индуцировал нецелевую функцию.
Пример 2. Экспрессия двойных CAR и активность in vitro CAR-T с двойной специфичностью к BCMA/CD19
Ряд бицистронных конструкций, содержащих две полные цепи CAR (химерного антигенного рецептора), одна из которых направлена на BCMA, а другая - на CD19, конструировали в лентивирусном векторе (таблица 28). Экспрессия CAR управляется промотором EF1-альфа. Такие CAR содержат ряд человеческих одноцепочечных вариабельных фрагментов (scFv), нацеливающихся на BCMA (duBCMA.4, PI61, R1G5 и R1B6). Во всех конструкциях использовался один и тот же сконструированный гуманизированный scFv, нацеливающийся на CD19. На N-конце каждого scFv сигнальный пептид, полученный из CD8-альфа, нацеливает CAR на секреторный путь. Предполагают, что такой сигнальный пептид будет отщепляться котрансляционно и поэтому будет отсутствовать в зрелой форме CAR, представляемой на поверхности клетки. На С-конце каждого scFv находится шарнирная область и трансмембранный домен CD8-альфа, слитый с внутриклеточным доменом 4-1BB, за которым следует внутриклеточный домен CD3-дзета. Между двумя CAR, точнее, между последней аминокислотой первого домена CD3-дзета и сигнальным пептидом последующего CAR, сконструирован линкер (GSG (SEQ ID NO: 206), за которым следует саморасщепляющийся пептид 2A из тешовируса-1 2A свиней (т. е. последовательность P2A). Можно использовать и другие линкеры и/или саморасщепляющиеся пептиды. Такая разработка позволяет экспрессировать два независимых CAR из одного мРНК-транскрипта. Последовательности ДНК, кодирующие области, перекрывающиеся между двумя CAR (сигнальный пептид, шарнирную область, трансмембранный домен, 4-1BB и CD3-дзета), отличаются друг от друга, чтобы свести к минимуму потенциальную рекомбинацию.
Конструкции использовали для получения векторного материала, который использовали для инфицирования первичных Т-клеток человека. Экспрессию CAR оценивали с помощью проточной цитометрии. Также измеряли количество и качество ответов с участием эффекторных Т-клеток ("CAR-T-клеток с двойной специфичностью к BCMA/CD19" или "Т-клеток") в ответ на экспрессирующие BCMA ("BCMA+" или "BCMA-положительные") и CD19+ опухоли-мишени. Ответы с участием эффекторных T-клеток включают без ограничения размножение, пролиферацию, удвоение, продуцирование цитокинов и уничтожение клеток-мишеней или цитолитическую активность (дегрануляцию) у клеток.
Получение лентивируса и определение титра
Пять конструкций, кодирующих двойные CAR для BCMA/CD19, описанные выше, использовали для получения геномного материала, упакованного в псевдотипированные с VSVg лентивирусные частицы. Две конструкции использовали в качестве контроля: одну, кодирующую моно-CAR, направленный против BCMA (duBCMA.4), и другую, кодирующую моно-CAR, направленный против CD19. Все семь конструкций конструировали в одном и том же каркасе плазмиды. Каждую из этих ДНК смешивали с тремя упаковочными компонентами VSVg, gag/pol и rev в комбинации с реагентом липофектамином для трансфекции клеток Lenti-X 293T (Clontech) с последующей заменой среды спустя 12-18 ч. Через 30 часов после замены среды среду собирали, фильтровали и хранили при -80°C.
Титр лентивируса определяли путем оценивания поверхностной экспрессии двойных CAR для BCMA/CD19 на трансдуцированных клетках Sup-T1 с использованием рекомбинантного белка BCMA человека с Fc-меткой, конъюгированного с Alexa-647 (BCMA-Fc), и антиидиотипического антитела к duCD19.1 (PE). Клетки Sup-T1 трансдуцировали с помощью содержащих вирус надосадочных жидкостей в 3-кратном серийном разведении с начальным разведением 1:3. Процентную долю клеток, экспрессирующих CAR (CAR+ клеток), оценивали спустя четыре дня. Титр вируса рассчитывали с использованием популяции, положительной по вышерасположенному CAR либо дважды положительной по CAR, согласно следующей формуле:
(% CAR+) x (количество высеянных клеток Sup-T1) x (разведение) / (количество вируса (мл)).
Титр вируса рассчитывали по центральной точке наибольшего разведения в линейном диапазоне, дающем от 5 до 25% CAR+ клеток (таблица 29).
Получение BCMA/CD19-CAR-Т-клеток с использованием традиционного 10-дневного способа получения
Получали CAR-Т-клетки с двойной специфичностью к BCMA/CD19, или моно-BCMA-, или моно-CD19-CAR-Т-клетки с использованием первичных Т-клеток человека (CD4+ и CD8+ лимфоцитов), полученных путем отрицательного отбора или положительного отбора с помощью Prodigy при обработке крови от здоровых подвергнутых аферезу доноров. Перед трансдукцией Т-клетки активировали с помощью гранул с антителами к CD3/CD28 (Dynabeads® Human T-Expander CD3/CD28, Thermo Fisher Scientific) при соотношении 1:3 (T-клеток и гранул) в среде для T-клеток (RPMI-1640, 10% инактивированной нагреванием эмбриональной телячьей сыворотки (FCS), 2 мM L-глутамина, 1x пенициллина/стрептомицина, 100 мкM заменимых аминокислот, 1 мM пирувата натрия, 10 мM HEPES и 55 мкM 2-меркаптоэтанола). Клетки культивировали при плотности 0,5×106/мл среды на лунку в 24-луночном планшете при 37°C, 5% CO2. Через 24 часа, когда Т-клетки проходили бластообразование, их трансдуцировали с помощью вирусов при множественности заражения (MOI), составляющей 5, исходя из титра для вышерасположенного CAR. Т-клетки начинали делиться с логарифмическим паттерном роста, который отслеживали путем измерения количества клеток на мл, и Т-клетки разбавляли свежей средой каждые два дня, от них удаляли гранулы, и их собирали для дальнейших анализов в день 8 или позднее в зависимости от размера клеток. Аликвоты Т-клеток окрашивали для измерения экспрессии CAR с помощью проточной цитометрии в дни 7 или 8 на FACS Fortessa (BD).
Поверхностную экспрессию CAR для BCMA и CAR для CD19 оценивали путем измерения % CAR и MFI (средней интенсивности флуоресценции) в день 8 с использованием анализов методом проточной цитометрии клеток, окрашенных с помощью rBCMA_Fc-AF647 и антиидиотипического антитела к duCD19.1 (PE) (фиг. 4A-4C). Три вектора, кодирующие двойные CAR, содержащие R1G5, R1B6 или PI61, приводили к сходной процентной доле клеток, дважды положительных по CAR (~ 10%), при этом конструкция 236 (R1G5/duCD19.1) демонстрировала наиболее высокие уровни MFI для CAR для BMCA и CD19 (фиг. 4A). Моно-duCD19.1 (конструкция 244) демонстрирует наиболее высокую MFI для CAR19 среди различных конструкций (фиг. 4B).
Оценивание функциональных свойств продукта на основе Т-клеток, перенаправленных с помощью двойного CAR для BCMA/CD19, в D8 in vitro и in vivo
Для оценивания противоопухолевой эффективности каждого из CAR в CAR-T-клетках с двойной специфичностью к BCMA/CART подготавливали совместные культуры с BCMA-положительными (KMS11), CD19-положительными (NALM6) и BCMA/CD19-отрицательными линиями раковых клеток (CD19KO; полученная из NALM6). CAR-T-клетки размораживали, подсчитывали и культивировали совместно с клетками-мишенями для регистрации их способностей к уничтожению и секреции цитокинов. Нетрансдуцированные Т-клетки (UTD) использовали в качестве контрольных ненацеливающихся Т-клеток.
Анализ цитотоксичности in vitro выполняли путем совместного культивирования CAR-T-клеток с клетками-мишенями, экспрессирующими люциферазу, при различных соотношениях E:T в течение 20 часов. Популяции CAR-T-клеток нормализовали по эквивалентным процентным долям CAR-положительных клеток перед посевом в соответствии с CAR, направленным на соответствующую опухоль (т. е. проводили нормализацию на основании CAR для BCMA, когда CAR-T культивировали совместно с клетками KMS11; и нормализацию на основании CAR19, когда CAR-T культивировали совместно с NALM6). Цитокин IFNγ измеряли в надосадочных жидкостях 20-часовых совместных культур, соответствующих целевому соотношению 1,25:1 или 2,5:1 (CAR-T-клетки:клетки-мишени), с использованием Meso Scale Discovery (MSD; Гейтерсберг, Мэриленд). Результаты рассчитывали в пг/мл с использованием известных стандартов. Все анализы выполняли в двух повторностях из одного источника донорских клеток. Данные об уничтожении показывают, что все двойные клоны CAR были эффективными в отношении BCMA-положительных клеток KMS11 и CD19-положительных клеток Nalm6 (фиг. 5A и 5B соответственно). В отношении BCMA/CD19-отрицательных опухолевых клеток наблюдался только фоновый уровень уничтожения (фиг. 5C). Способность этих клеток с двойными CAR продуцировать IFNγ при совместном культивировании с KMS11 или Nalm6 была сходной при оценке с использованием двух разных соотношений E:T (фиг. 6A-6D).
Противоопухолевую активность этих CAR-T в D8 in vivo анализировали с использованием смешанной ксенотрансплантатной модели опухоли (5% Nalm6 и 95% KMS11) у мышей NSG.
Получение и измерение CAR-T с двойной специфичностью к BCMA/CD19 дня 1 с использованием способа быстрого изготовления с активацией (ARM)
В некоторых вариантах осуществления этот способ ARM начинали с замороженного или свежего продукта лейкафереза. После получения образца для подсчета и QC продукт соединяли с устройством для сортировки клеток (например, установленным набором устройства CliniMACS Prodigy) и запускали программу. Клетки промывали и инкубировали с микрогранулами, которые связываются с требуемыми поверхностными маркерами, такими как CD4 и CD8. Меченные гранулами клетки отбирали путем пропускания клеток через магнитную колонку. Выделенные клетки снова промывали, и среды для клеток заменяли буфером для разделения. Очищенные Т-клетки затем переносили в культуру либо криоконсервировали для последующего применения. Чистота выделенных T-клеток проходила стадию QC посредством оценки с помощью проточной цитометрии. Криоконсервированные клетки размораживали, промывали в предварительно нагретых средах для клеток и ресуспендировали в средах для клеток. Свежие клетки добавляли непосредственно в культуру. Клетки высевали в мембранные биореакторы при 0,4-1,2e6 клеток/см2 мембраны, добавляли активирующий реагент, такой как гранулы/полимеры, содержащие антитела к CD3/CD28, наночастицы или коллоидные наносистемы, и среды для клеток добавляли в конечном объеме 0,25-2 мл/см2 мембраны. Для исследования кинетических показателей экспрессии CAR in vitro использовали 24-луночный GRex. Использовали GRex 100, колбу или CentriCult для тестирования масштабируемости этого способа изготовления и для тестирования противоопухолевой эффективности in vivo. Во время посева клетки подвергали трансдукции с помощью лентивирусного вектора, кодирующего двойной CAR для BCMA/CD19, при множественности заражения (MOI), составляющей 1 или 2. MOI определяли на основании титра вируса, полученного в клетках SupT1, исходя из титра для CAR, сконструированного выше последовательности 2A. Через 24 часа клетки промывали для удаления ненужных реагентов перед окрашиванием для измерения уровня экспрессии CAR с помощью проточной цитометрии и повторно составляли в среде для криоконсервации в виде "продукта на основе CAR-T дня 1" для исследования in vivo. Во всех случаях аликвоту клеток собирали через 72 часа после трансдукции для измерения кинетических показателей экспрессии CAR in vitro. Ответы, обеспечиваемые CAR-T дня 1, включают без ограничения цитолитическую активность и размножение in vivo.
На фиг. 7A-7B показан паттерн экспрессии CAR как для BCMA, так и для CD19 через 24 часа и 72 часа после трансдукции первичных Т-клеток человека, изготовленных с использованием способа ARM, при использовании MOI, составляющей 1, исходя из титра в SupT1, определяемого по экспрессии только вышерасположенного CAR. Через двадцать четыре часа после трансдукции при анализе методом проточной цитометрии наблюдалось, что вся популяция живых CD3+ T-клеток смещалась вправо в разной степени (фиг. 7A). Этот паттерн экспрессии отличался от типичных гистограмм проточной цитометрии клеток, трансдуцированных таким образом, чтобы они экспрессировали CAR, где CAR-положительная популяция четко отделяется от отрицательной популяции. Эти данные позволяют предположить, что "псевдотрансдукция или транзиентная экспрессия" может быть выявлена с помощью реагента для окрашивания в проточной цитометрии rBCMA_Fc. Ранее сообщалось, что лентивирусная псевдотрансдукция наблюдалась от момента добавления вектора и до 24 часов в CD34+ клетках и вплоть до 72 часов в клетках 293 (Haas DL, et al. Mol Ther. 2000. 291: 71-80). Дефектный по интегразе лентивирусный вектор вызывал транзиентную экспрессию eGFP в течение вплоть до 10 дней в CD34+клетках и в течение вплоть до 14 дней в клетках 293. Хотя наблюдаемая экспрессия CAR в день 1 после трансдукции Т-клеток может потенциально объясняться псевдотрансдукцией, в день 3, однако, появлялись две четко выраженные популяции, одна из которых была положительной по rBCMA_Fc, а другая была положительной по антиидиотипическому антителу к duCD19.1 (фиг. 7B и фиг. 7C). Более того, 20-30% клеток были моно-BCMA-CAR-положительными (CD19-CAR-отрицательными) среди клеток, сконструированных с использованием двойных конструкций c235, c236, c237 и c238 (фиг. 7C). В целом, эти результаты демонстрируют, что оба CAR хорошо экспрессировались в системе CAR-T с двойной специфичностью при использовании R1G5, R1B6 и PI61 в комбинации с duCD19.1. 72 часа после вирусной трансдукции могут служить в качестве суррогатного момента времени для измерения экспрессии CAR для определения стратегии введения доз in vivo.
Оценивание функциональных свойств in vivo BCMA-CAR-T дня 1, обработанных с помощью ARM
CAR-T дня 1, полученные с помощью системы с использованием CentriCult или колбы, изучали в отношении их противоопухолевой активности in vivo с использованием трех мышиных моделей: модели диссеминированной множественной миеломы из KMS-11-luc (BCMA+ CD19-), ксенотрансплантатной мышиной модели из Nalm6-Luc (CD19+ BCMA-) и смешанной модели из 95% клеток KMS-luc и 5% клеток NALM6-Luc. Данное исследование in vivo имело три цели: (1) продемонстрировать эффективность плеч как для BCMA, так и для CD19 в CAR-T с двойной специфичностью; (2) сравнить смешанную модель (содержащую BCMA+ опухолевые клетки и CD19+ опухолевые клетки) с моделью множественной миеломы из KMS-11-luc (BCMA+ CD19-), чтобы понять потенциальную активацию CAR-T с двойной специфичностью посредством мишени CD19; и (3) протестировать CAR-T с двойной специфичностью в модели только из Nalm6 (CD19+ BCMA-), чтобы изучить активность плеча для CD19. Измерения методом BLI проводили дважды в неделю. Отбирали периферическую кровь в дни 6, 13, 20 и 27 для анализа методом проточной цитометрии. Плазму крови собирали в вышеуказанные дни, а также в день 2 для анализа цитокинов. План исследования и информация о дозах обобщены в таблице 30.
CAR-T с двойной специфичностью обеспечивали очистку от смешанных опухолей (BCMA+ CD19+) при дозе 5e4 и ингибировали, но не полностью устраняли, опухоли при дозе 1e4 на протяжении всего исследования (фиг. 8C). Ни PI61, ни CTL119 не могли контролировать смешанную опухоль (фиг. 8C). Обе двойные c236 и c238 демонстрировали сходную или более высокую эффективность по сравнению с их моноаналогами в моделях KMS11 и Nalm6 (фиг. 8A и 8B). В ходе исследования масса тела увеличивалась во всех трех моделях (фиг. 9A-9C). В конце исследования для KMS11 и NALM6 наблюдалось небольшое снижение, возможно, вследствие GVHD.
Размножение Т-клеток происходило с дня 6 по день 20, а затем их численность выравнивалась с дня 20 по день 27 (фиг. 10A-10C). Размножение было связано с дозой во всех трех моделях (фиг. 11A-11C). Модель KMS11 демонстрировала в 3-4 раза более интенсивное размножение по сравнению со смешанной моделью при более высоких дозах (фиг. 10A и 10C). Двойные c236 и c238 демонстрировали более интенсивное размножение, чем моно-CAR-T (фиг. 10A-10C).
Общая процентная доля BCMA-CAR+ достигала пикового значения в день 13, а затем начинала уменьшаться до дня 27 включительно (фиг. 11A-11C). В смешанной модели группы 5e4 демонстрировали более низкие процентные доли BCMA-CAR+ по сравнению с группами 1e4 после дня 13 (фиг. 11C). Процентная доля дважды CAR+ была связана с притоком общей популяции CD3+ клеток, что могло быть возможным признаком GVHD. Размножение дважды BCMA/CD19-CAR+ T-клеток наблюдалось в группах Rx CAR-T c236 и c238 во всех трех моделях (фиг. 12A-12C).
Индукция IFNγ зависела от дозы во всех моделях (фиг. 13A-13C). Двойные конструкции при обеих дозах приводили к продуцированию в 3-4 раза большего количества IFNγ на пиковом уровне в смешанной модели, чем в модели с KMS11 в отдельности (фиг. 13A и 13C). Пик индукции наблюдался в течение 13 дней в большинстве групп во всех моделях (пики в день 20 и день 27 по всей вероятности были обусловлены GVHD) (фиг. 13A-13C).
В отдельном исследовании CAR-T дня 1, полученные с помощью GRex 100 и 6-луночной системы GRex, изучали в отношении их противоопухолевой активности in vivo с использованием модели диссеминированной множественной миеломы из KMS-11-luc. Репортер люцифераза позволяет проводить мониторинг бремени заболевания посредством количественной биолюминесцентной визуализации (BLI). Вкратце, CAR-T дня 1, изготовленные согласно описанному выше, вводили мышам, несущим опухоль. Еженедельно отбирали образцы крови для измерения размножения CAR-T в периферической крови и анализировали с помощью проточной цитометрии. Т-клетки, сконструированные с использованием конструкции № 236 и конструкции № 238, демонстрировали сильную противоопухолевую активность (фиг. 14A) и хорошее размножение CAR-T (фиг. 14B) in vivo в модели KMS11 (BCMA+ CD19-).
Пример 3. Определение характеристик CAR-T в формате диатела
В данном примере описано определение характеристик CAR JL1 - JL10 в формате диатела. JL1 - JL8 представляют собой конструкции диател PI61/CTL119, а JL9 - JL10 представляют собой конструкции диател R1G5/CTL119. Информация о последовательностях JL1 - JL10 раскрыта в таблице 31.
Получение и измерение CAR-T в формате диател к BCMA/CD19 дня 1 с использованием способа быстрого изготовления с активацией (ARM)
В некоторых вариантах осуществления этот способ ARM начинали с замороженного или свежего продукта лейкафереза. После получения образца для подсчета и QC продукт соединяли с устройством для сортировки клеток (например, установленным набором устройства CliniMACS Prodigy) и запускали программу. Клетки промывали и инкубировали с микрогранулами, которые связываются с требуемыми поверхностными маркерами, такими как CD4 и CD8. Меченные гранулами клетки отбирали путем пропускания клеток через магнитную колонку. Выделенные клетки снова промывали, и среды для клеток заменяли буфером для разделения. Очищенные Т-клетки затем переносили в культуру либо криоконсервировали для последующего применения. Чистота выделенных T-клеток проходила стадию QC посредством оценки с помощью проточной цитометрии. Криоконсервированные клетки размораживали, промывали в предварительно нагретых средах для клеток и ресуспендировали в средах для клеток. Свежие клетки добавляли непосредственно в культуру. Аликвоты замороженной общей популяции выделенных Т-клеток размораживали на водяной бане при 37°C, помещали в Optimizer CM (среду Gibco Optimizer с добавлением+100 ед/мл IL2 человека) и центрифугировали в течение 5 минут при 1500 оборотах в минуту. Клетки подсчитывали и высевали в 24-луночный планшет при 3e6/мл, 1 мл/лунка. В каждую лунку добавляли TransAct при 1/100 (10 мкл/лунка).
Для исследования кинетических показателей экспрессии CAR in vitro в способе ARM использовали 24-луночный планшет. Во время посева клетки подвергали трансдукции с помощью лентивирусного вектора, кодирующего CAR для BCMA/CD19 в формате диатела, при множественности заражения (MOI), составляющей 2. MOI определяли на основании титра вируса, полученного в клетках SupT1, исходя из экспрессии CAR в дважды положительных клетках. После 24 часов в культуре клетки собирали и трижды промывали в PBS+1% HSA. Затем клетки подсчитывали и повторно высевали в 24-луночный планшет в конечном количестве 1e6/мл. Через 72 часа после повторного посева клетки собирали, подсчитывали, и из каждого образца отбирали аликвоту 5e5 клеток для анализа методом проточной цитометрии. Эту процедуру повторяли спустя 72 часа для момента времени, соответствующего дню седьмому.
На фиг. 15A и 15B показан паттерн экспрессии CAR как для BCMA, так и для CD19 через 96 часов (фиг. 15A) или 7 дней (фиг. 15B) после добавления вируса, кодирующего диатело, в первичные Т-клетки человека, изготовленные с использованием способа ARM, при использовании MOI, составляющей 2. В целом, эти результаты демонстрируют, что оба CAR хорошо экспрессировались в системе CAR-T в формате диатела при использовании R1G5 или PI61 в комбинации с duCD19.1 в день 4 (фиг. 15A) или день 7 (фиг. 15B) после добавления вируса. JL1 - JL4, JL9 и JL10 демонстрировали линейную экспрессию CAR в дважды положительных клетках, тогда как JL5 - JL8 демонстрировали небольшое смещение в сторону CD19+ популяции, что может быть обусловлено различиями в связывании CAR с соответствующими реагентами для выявления. Здесь показаны данные одного из двух доноров.
Получение CAR-T-клеток с использованием способа традиционного изготовления
Способ традиционного изготовления (TM) представляет собой способ, при котором Т-клетки размножают ex vivo в течение 8-9 дней после активации и трансдукции перед сбором. Обработанные в Prodigy Т-клетки ресуспендировали в теплой полной среде для Т-клеток RPMI (RPMI, 10% инактивированной нагреванием FBS, 2 мМ L-глутамина, 100 ед/мл пенициллина/стрептомицина, 1x NEAA, 1 мМ пирувата натрия, 10 мМ HEPES и 55 мкМ β-меркаптоэтанола) и высевали в 24-луночные планшеты при 0,5e6 клеток/мл на лунку. T-клетки инкубировали в течение ночи при 37°C с гранулами Human T-Expander CD3/CD28 при соотношении гранул и клеток, составляющем 3:1.
В день 1 добавляли лентивирусы при MOI, составляющей 5, исходя из титра в SUP-T1. Вирус не добавляли в нетрансдуцированный контроль (UTD). T-клетки инкубировали в течение ночи при 37°C с последующим добавлением 1 мл полной среды для Т-клеток на лунку, после чего их инкубировали в течение ночи при 37°C. В течение оставшихся шести дней размножения культуры T-клетки переносили в колбы для тканевых культур и разбавляли полной средой для Т-клеток каждые два дня, добиваясь концентрации 0,5e6 клеток/мл. Типичные коэффициенты разведения находились в диапазоне от 1:2 до 1:4 на протяжении фазы размножения.
В день 7 от T-клеток удаляли гранулы, и их собирали и криоконсервировали в замораживающей среде CryoStor CS10, замораживали при -80°C в контейнерах для замораживания клеток CoolCell (BioCision) и переносили в LN2 на следующий день. Небольшие аликвоты T-клеток окрашивали для выявления экспрессии CAR. Одноцветные контроли были включены для компенсации. Образцы измеряли на проточном цитометре (BD LSRFortessa), и данные анализировали с помощью программного обеспечения FlowJo.
На фиг. 16 показана экспрессия CAR в день 7 в способе ТМ с использованием MOI, составляющей 5. Продукты TM демонстрировали паттерн экспрессии, сходный с таковым у продуктов ARM (фиг. 15A, 15B и 16).
Анализ уничтожения in vitro
Потенциал уничтожения Т-клеток, сконструированных с различными конструкциями диател, в ответ на клетки-мишени, экспрессирующие BCMA или CD19, оценивали путем инкубирования CAR-T-клеток с клетками-мишенями при соотношениях E:T с 2-кратными разведениями, начиная с 20:1. Количество клеток-мишеней фиксировали при 2,5×105 клеток/лунка, и клетки культивировали в 96-луночных плоскодонных планшетах. Эффекторные клетки представляли собой CAR-T-клетки, полученные посредством традиционного изготовления путем трансдукции Т-клеток с помощью диател. Клетки-мишени включают BCMA-положительные клетки KMS11-luc или BCMA-отрицательные клетки NALM6-luc. Для данного анализа % трансдукции CAR-T-клеток нормализовали путем добавления UTD к BCMA-CAR-T. Это позволяло проводить сравнение одного и того же количества CAR-T и одного и того же общего количества Т-клеток в каждом образце.
Потерю сигнала люциферазы в результате уничтожения клеток измеряли с использованием субстрата Bright-Glo через 16 часов после высевания клеток, а специфический лизис рассчитывали по следующей формуле:
специфичный лизис (%)=100 - (люминесценция образца/средняя максимальная люминесценция) * 100.
На фиг. 17A и 17B продемонстрирована способность различных диател PI61/CTL119 эффективно уничтожать специфические линии клеток-мишеней NALM-6 (CD19+, BCMA-) (фиг. 17A) или KMS-11 (CD19-, BCMA+) (фиг. 17B). Данные позволяют предположить, что клоны JL6, JL5, JL3 и JL8 в равной степени уничтожали оба типа клеток-мишеней.
Анализ секреции цитокинов
Собирали надосадочные жидкости из совместных культур (соотношение 1,25:1), использованных в вышеприведенном анализе уничтожения, после 20 часов инкубирования для использования в V-плексном анализе MSD с IFNγ и IL-2 человека. Наблюдалась разная величина специфичной для мишени индукции IFNγ или IL-2 клетками, трансдуцированными диателом, в ответ на стимуляцию клетками KMS11 или NALM6 (фиг. 18A и 18B). Клетки UTD не демонстрировали какую-либо неспецифичную секрецию IFNγ. в ответ на любые клетки-мишени (фиг. 18А). Сообразно результатам анализа уничтожения клоны JL6, JL5, JL3 и JL8 продуцировали больше цитокинов в ответ на клетки-мишени (фиг. 18A и 18B).
Пример 4. Котрансдукция CAR для BCMA и CAR для CD19
Аликвоты замороженной общей популяции выделенных Т-клеток размораживали на водяной бане при 37°C, помещали в Optimizer CM (среду Gibco Optimizer с добавлением+100 ед/мл IL2 человека) и центрифугировали в течение 5 минут при 1500 оборотах в минуту. Клетки подсчитывали и высевали в 24-луночный планшет при 3e6/мл, 1 мл/лунка. В каждую лунку добавляли TransAct при 1/100 (10 мкл/лунка). Добавляли вирус при разных значениях множественности заражения (MOI), исходя из титра в SupT1 для PI61 либо титра, определенного с помощью qPCR, для CTL119. PI61 использовали при MOI, составляющей 2 или 1, и задействовали в паре с тремя разными MOI для CTL119: 1, 0,5 и 0,25. Добавляли моно-CAR при MOI, составляющей 1 или 2 для PI61 и 1 для CTL119, а также высевали контроль UTD. После 24 часов в культуре клетки собирали и трижды промывали в PBS+1% HSA. Затем клетки подсчитывали и повторно высевали в 24-луночный планшет в конечном количестве 1e6/мл.
Через 72 часа после повторного посева клетки собирали, подсчитывали, и из каждого образца отбирали аликвоту 5e5 клеток для анализа методом проточной цитометрии. Клетки окрашивали с помощью LIVE/DEAD Aqua (BV510) в течение 15 минут в 100 мкл/лунка и затем дважды промывали. Затем добавляли антитело к MM (таблица 32) в количестве 50 мкл/лунка в течение 25 минут при 4°C. Клетки промывали еще два раза, а затем фиксировали в течение 15 минут в 1,6% PFA в PBS, 100 мкл/лунка. После фиксации клетки промывали, как описано ранее, и ресуспендировали в конечном объеме 150 мкл/образец в буфере для проточной цитометрии. Для каждого образца получали 5e4 клеток в гейте по живым CD3-положительным клеткам на BD LSRFortessa (BD Biosciences, Сан-Хосе, Калифорния), и данные анализировали с использованием программного обеспечения FlowJo v.10 (Эшленд, Орегон). Эту процедуру повторяли спустя 72 часа для момента времени, соответствующего дню седьмому.
DEAD
В день четвертый после добавления вируса анализ методом проточной цитометрии показал значительно более высокие процентные доли моно-BCMA-CAR+ популяции по сравнению с CAR19-положительной популяцией и дважды CAR-положительной популяцией в условиях MOI, составляющей 2, в случае CAR для BCMA (фиг. 19 и 20). Эти результаты были согласующимися для обоих доноров. При добавлении PI61 при MOI, составляющей 1, наблюдалось хорошее титрование всех популяций (фиг. 19 и 20). CAR19+ популяция, а также дважды CAR+ популяция уменьшались по мере уменьшения MOI в случае CAR для CD19 с 1 до 0,25 (фиг. 19). BCMA-CAR+ популяция увеличивалась при добавлении меньшего количества вируса, кодирующего CTL119 (фиг. 19). Процентные доли общих CAR+ популяций коррелировали с общей MOI, добавленной в каждую лунку (фиг. 21). Процентные доли общих популяций с CAR для BCMA и с CAR19 также коррелировали. Все тенденции были стабильными с дня четвертого по день седьмой (фиг. 21). Жизнеспособность и скорости размножения не зависели от общей добавленной MOI (фиг. 22). Здесь показаны данные одного из четырех доноров.
Пример 5. Описание способа быстрого изготовления с активацией (ARM)
В некоторых вариантах осуществления CAR-T-клетки изготавливают с использованием непрерывного способа быстрого изготовления с активацией (ARM) в течение примерно 2 дней, что будет потенциально позволять получить большее количество менее дифференцированных Т-клеток (необученных T-клеток и TSCM-клеток (стволовых T-клеток центральной памяти)), подлежащих возвращению пациенту для размножения клеток in vivo. Короткий период времени изготовления позволяет T-клеткам с профилем ранней дифференцировки пролиферировать в организме до их требуемого конечного дифференцированного состояния, а не в сосуде для культивирования ex vivo.
В некоторых вариантах осуществления CAR-T-клетки изготавливают с использованием криоконсервированного исходного материала, полученного путем лейкафереза, например, материала немобилизованной аутологичной периферической крови, полученного путем лейкафереза (LKPK). Криоконсервированный исходный материал подвергают стадиям обработки для обогащения Т-клетками в первый день получения (день 0) с помощью иммуномагнитной системы, содержащей антитела к CD4/CD8. Положительную фракцию затем высевают в сосуд для культивирования GRex, активируют с помощью системы, содержащей антитела к CD3/CD28 (TransAct), и в тот же день подвергают трансдукции с помощью лентивирусного вектора (LV), кодирующего CAR. На следующий день, спустя 20-28 часов трансдукции, T-клетки собирают, промывают четыре раза, составляют в замораживающей среде и затем замораживают с помощью программируемого криозамораживателя (CRF). От начала способа в день 0 до начала сбора на следующий день клетки культивируют в течение 20-28 часов с целевым временем культивирования, составляющем 24 часа после высевания в день 0.
Среды для дня 0 получали в соответствии с таблицей 21.
Криоконсервированный материал, полученный путем лейкафереза, размораживают. Размороженные клетки разбавляют с помощью буфера для способа быстрого изготовления (таблица 21) и промывают на устройстве CliniMACS® Prodigy®. Т-клетки отбирают с помощью микрогранул CliniMACS® с антителами к CD4 и CD8. После завершения программы отбора Т-клеток (примерно от 3 ч. 40 мин. до 4 ч. 40 мин.) мешок для повторного применения, содержащий клетки, суспендированные в среде для способа быстрого изготовления (таблица 21), переносят в пакет для переноса. Отбирают образец для оценки жизнеспособности и количества клеток. Данные о количестве и жизнеспособности клеток из положительной фракции в мешке используют для определения концентрации клеток при высевании в сосуд для культивирования для активации и трансдукции вектором.
После положительного отбора T-клеток с помощью микрогранул CliniMACS® (с антителами к CD4 и CD8) клетки высевают в сосуд для культивирования G-Rex. После высевания клеток затем добавляют реагент для активации (TransAct) в сосуд для культивирования. Клетки затем подвергают трансдукции с помощью лентивирусного вектора, кодирующего CAR, при целевой MOI, составляющей 1,0 (0,8-1,2). После добавления вектора сосуд для культивирования транспортируют в инкубатор, где его инкубируют в течение целевого времени, составляющего 24 часа (рабочий диапазон 20-28 часов), при номинальной температуре 37°C (рабочий диапазон 36-38°C) при номинальных 5% CO2 (рабочий диапазон 4,5-5,5%). После инкубирования клетки промывают с помощью раствора для промывания собранного материала (таблица 21) четыре раза для удаления любого неинтегрировашегося вектора и остаточных вирусных частиц, а также любых других примесей, связанных со способом. Затем клетки элюируют, и отбирают образец для тестирования для подсчета количества и жизнеспособности клеток, и результаты используют для определения объема, требуемого для ресуспендирования клеток, для конечного составления с помощью CryoStor® CS10. Клетки затем центрифугируют с удалением раствора для промывания собранного материала, и приступают к их криоконсервации.
В некоторых вариантах осуществления CAR, экспрессируемый в CAR-T-клетках, связывается с CD19. В некоторых вариантах осуществления IL-2, применяемый в средах для способа быстрого изготовления (RM) (таблица 21), может быть заменен на IL-15, hetIL-15 (IL-15/sIL-15Ra), IL-6 или IL-6/sIL-6Ra.
В некоторых вариантах осуществления CAR, экспрессируемый в CAR-T-клетках, связывается с BCMA. В некоторых вариантах осуществления IL-2, применяемый в средах для способа быстрого изготовления (RM) (таблица 21), может быть заменен на IL-15, hetIL-15 (IL-15/sIL-15Ra), IL-6 или IL-6/sIL-6Ra.
В некоторых вариантах осуществления CAR-Т-клетки экспрессируют двойные CAR, раскрытые в данном документе, например, двойные CAR для BCMA/CD19, раскрытые в данном документе. В некоторых вариантах осуществления CAR-Т-клетки экспрессируют CAR в формате диатела, раскрытый в данном документе, например, CAR для BCMA/CD19 в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления CAR-Т-клетки конструируют таким образом, чтобы они экспрессировали CAR для BCMA и CAR для CD19, с помощью котрансдукции, как раскрыто в данном документе.
Пример 6. Изготовление CAR-T-клеток с использованием способа быстрого изготовления с активацией (ARM)
Способ ARM для получения CAR-T-клеток начинается с получения сред, как указано в таблице 25.
Криоконсервированный продукт лейкафереза используют в качестве исходного материала и обрабатывают для обогащения Т-клетками. Документация по аферезу при ее доступности используется для определения процентной доли Т-клеток. При отсутствии данных о процентной доле Т-клеток в документации по аферезу тестирование в сигнальном флаконе проводят на поступающих криоконсервированных продуктах лейкафереза с получением целевой процентной доли Т-клеток для афереза. Результаты для процентной доли Т-клеток определяют то, как много мешков размораживается в день 0 способа ARM.
Криоконсервированный продукт лейкафереза размораживают, промывают и затем подвергают отбору Т-клеток и обогащению Т-клетками с использованием технологии на основе микрогранул CliniMACS®. Жизнеспособные ядросодержащие клетки (VNC) активируют с помощью TransAct (Miltenyi) и подвергают трансдукции с помощью лентивирусного вектора, кодирующего CAR. Жизнеспособные клетки, отобранные с помощью микрогранул Miltenyi, высевают на CentriCult в Prodigy®, который представляет собой инкубационную камеру без увлажнения. Находящиеся в культуре клетки суспендируют в среде для способа быстрого изготовления, которая представляет собой среду на основе OpTmizer™ CTS™, которая содержит добавку CTS™ (ThermoFisher), GlutaMAX, IL-2 и 2% заменитель сыворотки для иммунных клеток вместе с ее компонентами, способствующими активации и трансдукции Т-клеток. Лентивирусную трансдукцию проводят один раз в день высевания после добавления TransAct к разбавленным клеткам в культуральную среду. Лентивирусный вектор будут размораживать непосредственно перед применением в день высевания в течение вплоть до 30 минут при комнатной температуре.
От начала способа в день 0 до начала промывания и сбора культуры CAR-T-клетки культивируют в течение 20-28 часов после высевания. После культивирования суспензию клеток подвергают двум промываниям культивируемого материала и одному промыванию собранного материала в камере CentriCult (Miltenyi Biotech).
После промывания собранного материала на CliniMACS® Prodigy® в день 1 из суспензии клеток отбирают образцы для определения количества и жизнеспособности жизнеспособных клеток. Суспензию клеток затем переносят в центрифугу для осаждения вручную. Надосадочную жидкость удаляют, и клеточный осадок ресуспендируют в CS10 (BioLife Solution), что приводит к составлению продукта при конечной концентрации DMSO, составляющей ~ 10,0%. Количество жизнеспособных клеток составляют в конце сбора для введения дозы. Дозы затем распределяют в отдельные мешки для криоконсервации, и отбор аналитических образцов осуществляют в криофлаконы.
Криоконсервированные продукты хранятся в контролируемых резервуарах для хранения с LN2 в безопасной зоне с ограниченным доступом до окончательного выпуска и транспортировки.
В некоторых вариантах осуществления CAR-Т-клетки экспрессируют двойные CAR, раскрытые в данном документе, например, двойные CAR для BCMA/CD19, раскрытые в данном документе. В некоторых вариантах осуществления CAR-Т-клетки экспрессируют CAR в формате диатела, раскрытый в данном документе, например, CAR для BCMA/CD19 в формате диатела, раскрытый в данном документе. В некоторых вариантах осуществления CAR-Т-клетки конструируют таким образом, чтобы они экспрессировали CAR для BCMA и CAR для CD19, с помощью котрансдукции, как раскрыто в данном документе.
Пример 7. Изготовление CAR-Т-клеток, экспрессирующих CAR для BCMA и CAR для CD19
Способ быстрого изготовления CAR-Т-клеток начинается с получения сред, как указано в таблице 33.
Добавка CTS
ICSR
GlutaMAX
Восстановленный IL-2
HSA
HSA
Криоконсервированный продукт лейкафереза размораживают. Размороженные клетки разбавляют с помощью буфера для способа быстрого изготовления (таблица 33) и промывают на устройстве CliniMACS® Prodigy®. Т-клетки отбирают с помощью микрогранул CliniMACS® с антителами к CD4 и CD8. После завершения программы отбора Т-клеток (примерно от 3 ч. 40 мин. до 4 ч. 40 мин.) мешок для повторного применения будет содержать клетки, суспендированные в буфере для способа быстрого изготовления (таблица 33). Отбирают образец для оценки жизнеспособности и количества клеток. Данные о количестве и жизнеспособности клеток из положительной фракции в мешке используют для определения концентрации клеток при высевании в сосуд для культивирования для активации и трансдукции вектором.
После положительного отбора Т-клеток с помощью микрогранул CliniMACS® (с антителами к CD4 и CD8) клетки высевают в сосуд для культивирования CentriCult в Prodigy® при целевом количестве 4,0×108-1,0×109 всех жизнеспособных клеток при целевой концентрации приблизительно 4,0×106 жизнеспособных клеток/мл. После высевания клеток затем добавляют реагент для активации (TransAct) в сосуд для культивирования.
Клетки затем подвергают трансдукции с помощью лентивирусного вектора, кодирующего CAR для BCMA, и лентивирусного вектора, кодирующего CAR для CD19. Объем вектора, который должен использоваться для трансдукции Т-клеток после положительного отбора, рассчитывают, исходя из целевой множественности заражения (MOI), составляющей 4,75 для лентивирусного вектора, кодирующего CAR для BCMA, и целевой MOI, составляющей 0,5 для лентивирусного вектора, кодирующего CAR для CD19.
После инкубирования в течение целевого 24-часового периода (рабочий диапазон 20-28 часов) при температуре 37°C при номинальных 5% CO2 клетки обрабатывают для промывания собранного материала.
После инкубирования клетки промывают с помощью раствора для промывания собранного материала (таблица 33) три раза для удаления любого неинтегрировашегося вектора и остаточных вирусных частиц, а также любых других примесей, связанных со способом. Затем клетки элюируют, и отбирают образец для тестирования для подсчета количества и жизнеспособности клеток, и результаты используют для определения объема, требуемого для ресуспендирования клеток, для конечного составления с помощью CryoStor® CS10. Клетки затем центрифугируют с удалением раствора для промывания собранного материала, и приступают к их криоконсервации.
Пример 8. Анализ генных сигнатур CAR-T-клеток, изготовленных с использованием способа ARM
Способы
Секвенирование РНК одиночных клеток
Библиотеки для секвенирования РНК одиночных клеток создавали с помощью устройства 10X Genomics Chromium Controller и дополнительных наборов для конструирования библиотек.
Криоконсервированные клетки размораживали, подсчитывали и сортировали с помощью проточной цитометрии (если это необходимо для исследуемого вопроса) до загрузки в устройство 10X Genomics. Отдельные клетки загружали в капли, и осуществляли штрихкодирование РНК в отдельных каплях с помощью гранулы GemCode. Штрихкодированные РНК высвобождали из капель и преобразовывали в библиотеку секвенирования полного транскриптома, совместимую с Illumina.
Созданные библиотеки секвенировали на устройстве Illumina HiSeq и анализировали с применением программного конвейера для анализа 10X Genomics и программного обеспечения Loupe Cell Browser.
Иммунологическое профилирование одиночных клеток
Созданные с помощью 10X Genomics библиотеки полного транскриптома одиночных клеток использовали в качестве опорного материала для осуществления профилирования и анализа репертуара иммунных клеток. Последовательности Т-клеточных рецепторов подвергали ПЦР-амплификации из 5'-библиотек секвенирования одиночных клеток Chromium и анализировали на устройстве для секвенирования Illumina.
Программный конвейер для анализа
Данные секвенирования РНК одиночных клеток обрабатывали с помощью программного конвейера для анализа Cell Ranger, начиная с файлов FASTQ. Подробное описание программного конвейера для анализа Cell Ranger можно найти на support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger. Общий программный конвейер включает выравнивание, фильтрацию, подсчет штрихкодов и подсчет UMI. Клеточные штрихкоды применяли для образования матриц на основе генов и штрихкодов, определения кластеров и осуществления анализа экспрессии генов. Данные о количественных показателях экспрессии генов нормализовали с помощью пакета Seurat Bioconductor. Клетки, которые имели менее 200 экспрессируемых генов, отбрасывали из анализа. Гены, которые экспрессировались только в 2 клетках или меньше, отбрасывали из анализа. Оставшиеся данные нормализовали с помощью способа логарифмической нормализации Seurat с использованием коэффициента пересчета, составляющего 10000. Данные масштабировали посредством регрессии по количеству выявленных молекул на клетку. Балл набора генов (балл для набора генов) рассчитывали, используя среднее логарифмически нормализованное значение экспрессии гена для всех генов в наборе генов. Каждое значение экспрессии гена нормализуют с помощью Z-оценки так, что среднее значение экспрессии гена среди образцов составляет 0, и стандартное отклонение составляет 1. Балл набора генов затем рассчитывают как среднее значение для нормализованных значений экспрессии гена в наборе генов. Иллюстративный расчет балла набора генов описан ниже.
Для данного примера расчета балла набора генов нормализованные значения экспрессии гена в двух (2) образцах для шести (6) генов представлены в таблице 23. Для целей данного иллюстративного расчета набор генов состоит из генов 1-4. Следовательно, оба из образцов 1 и 2 имеют балл набора генов, составляющий 0.
Набор генов "активируемые в TEM относительно подавляемых в TSCM" включает следующие гены: MXRA7, CLIC1, NAT13, TBC1D2B, GLCCI1, DUSP10, APOBEC3D, CACNB3, ANXA2P2, TPRG1, EOMES, MATK, ARHGAP10, ADAM8, MAN1A1, SLFN12L, SH2D2A, EIF2C4, CD58, MYO1F, RAB27B, ERN1, NPC1, NBEAL2, APOBEC3G, SYTL2, SLC4A4, PIK3AP1, PTGDR, MAF, PLEKHA5, ADRB2, PLXND1, GNAO1, THBS1, PPP2R2B, CYTH3, KLRF1, FLJ16686, AUTS2, PTPRM, GNLY и GFPT2.
Набор генов "активируемые в Treg относительно подавляемых в Teff" включает следующие гены: C12orf75, SELPLG, SWAP70, RGS1, PRR11, SPATS2L, SPATS2L, TSHR, C14orf145, CASP8, SYT11, ACTN4, ANXA5, GLRX, HLA-DMB, PMCH, RAB11FIP1, IL32, FAM160B1, SHMT2, FRMD4B, CCR3, TNFRSF13B, NTNG2, CLDND1, BARD1, FCER1G, TYMS, ATP1B1, GJB6, FGL2, TK1, SLC2A8, CDKN2A, SKAP2, GPR55, CDCA7, S100A4, GDPD5, PMAIP1, ACOT9, CEP55, SGMS1, ADPRH, AKAP2, HDAC9, IKZF4, CARD17, VAV3, OBFC2A, ITGB1, CIITA, SETD7, HLA-DMA, CCR10, KIAA0101, SLC14A1, PTTG3P, DUSP10, FAM164A, PYHIN1, MYO1F, SLC1A4, MYBL2, PTTG1, RRM2, TP53INP1, CCR5, ST8SIA6, TOX, BFSP2, ITPRIPL1, NCAPH, HLA-DPB2, SYT4, NINJ2, FAM46C, CCR4, GBP5, C15orf53, LMCD1, MKI67, NUSAP1, PDE4A, E2F2, CD58, ARHGEF12, LOC100188949, FAS, HLA-DPB1, SELP, WEE1, HLA-DPA1, FCRL1, ICA1, CNTNAP1, OAS1, METTL7A, CCR6, HLA-DRB4, ANXA2P3, STAM, HLA-DQB2, LGALS1, ANXA2, PI16, DUSP4, LAYN, ANXA2P2, PTPLA, ANXA2P1, ZNF365, LAIR2, LOC541471, RASGRP4, BCAS1, UTS2, MIAT, PRDM1, SEMA3G, FAM129A, HPGD, NCF4, LGALS3, CEACAM4, JAKMIP1, TIGIT, HLA-DRA, IKZF2, HLA-DRB1, FANK1, RTKN2, TRIB1, FCRL3 и FOXP3.
Набор генов "подавляемые в состоянии стволовости" включает следующие гены: ACE, BATF, CDK6, CHD2, ERCC2, HOXB4, MEOX1, SFRP1, SP7, SRF, TAL1 и XRCC5.
Набор генов "активируемые при гипоксии" включает следующие гены: ABCB1, ACAT1, ADM, ADORA2B, AK2, AK3, ALDH1A1, ALDH1A3, ALDOA, ALDOC, ANGPT2, ANGPTL4, ANXA1, ANXA2, ANXA5, ARHGAP5, ARSE, ART1, BACE2, BATF3, BCL2L1, BCL2L2, BHLHE40, BHLHE41, BIK, BIRC2, BNIP3, BNIP3L, BPI, BTG1, C11orf2, C7orf68, CA12, CA9, CALD1, CCNG2, CCT6A, CD99, CDK1, CDKN1A, CDKN1B, CITED2, CLK1, CNOT7, COL4A5, COL5A1, COL5A2, COL5A3, CP, CTSD, CXCR4, D4S234E, DDIT3, DDIT4, 1-Dec, DKC1, DR1, EDN1, EDN2, EFNA1, EGF, EGR1, EIF4A3, ELF3, ELL2, ENG, ENO1, ENO3, ENPEP, EPO, ERRFI1, ETS1, F3, FABP5, FGF3, FKBP4, FLT1, FN1, FOS, FTL, GAPDH, GBE1, GLRX, GPI, GPRC5A, HAP1, HBP1, HDAC1, HDAC9, HERC3, HERPUD1, HGF, HIF1A, HK1, HK2, HLA-DQB1, HMOX1, HMOX2, HSPA5, HSPD1, HSPH1, HYOU1, ICAM1, ID2, IFI27, IGF2, IGFBP1, IGFBP2, IGFBP3, IGFBP5, IL6, IL8, INSIG1, IRF6, ITGA5, JUN, KDR, KRT14, KRT18, KRT19, LDHA, LDHB, LEP, LGALS1, LONP1, LOX, LRP1, MAP4, MET, MIF, MMP13, MMP2, MMP7, MPI, MT1L, MTL3P, MUC1, MXI1, NDRG1, NFIL3, NFKB1, NFKB2, NOS1, NOS2, NOS2P1, NOS2P2, NOS3, NR3C1, NR4A1, NT5E, ODC1, P4HA1, P4HA2, PAICS, PDGFB, PDK3, PFKFB1, PFKFB3, PFKFB4, PFKL, PGAM1, PGF, PGK1, PGK2, PGM1, PIM1, PIM2, PKM2, PLAU, PLAUR, PLIN2, PLOD2, PNN, PNP, POLM, PPARA, PPAT, PROK1, PSMA3, PSMD9, PTGS1, PTGS2, QSOX1, RBPJ, RELA, RIOK3, RNASEL, RPL36A, RRP9, SAT1, SERPINB2, SERPINE1, SGSM2, SIAH2, SIN3A, SIRPA, SLC16A1, SLC16A2, SLC20A1, SLC2A1, SLC2A3, SLC3A2, SLC6A10P, SLC6A16, SLC6A6, SLC6A8, SORL1, SPP1, SRSF6, SSSCA1, STC2, STRA13, SYT7, TBPL1, TCEAL1, TEK, TF, TFF3, TFRC, TGFA, TGFB1, TGFB3, TGFBI, TGM2, TH, THBS1, THBS2, TIMM17A, TNFAIP3, TP53, TPBG, TPD52, TPI1, TXN, TXNIP, UMPS, VEGFA, VEGFB, VEGFC, VIM, VPS11 и XRCC6.
Набор генов "активируемые при аутофагии" включает следующие гены: ABL1, ACBD5, ACIN1, ACTRT1, ADAMTS7, AKR1E2, ALKBH5, ALPK1, AMBRA1, ANXA5, ANXA7, ARSB, ASB2, ATG10, ATG12, ATG13, ATG14, ATG16L1, ATG16L2, ATG2A, ATG2B, ATG3, ATG4A, ATG4B, ATG4C, ATG4D, ATG5, ATG7, ATG9A, ATG9B, ATP13A2, ATP1B1, ATPAF1-AS1, ATPIF1, BECN1, BECN1P1, BLOC1S1, BMP2KL, BNIP1, BNIP3, BOC, C11orf2, C11orf41, C12orf44, C12orf5, C14orf133, C1orf210, C5, C6orf106, C7orf59, C7orf68, C8orf59, C9orf72, CA7, CALCB, CALCOCO2, CAPS, CCDC36, CD163L1, CD93, CDC37, CDKN2A, CHAF1B, CHMP2A, CHMP2B, CHMP3, CHMP4A, CHMP4B, CHMP4C, CHMP6, CHST3, CISD2, CLDN7, CLEC16A, CLN3, CLVS1, COX8A, CPA3, CRNKL1, CSPG5, CTSA, CTSB, CTSD, CXCR7, DAP, DKKL1, DNAAF2, DPF3, DRAM1, DRAM2, DYNLL1, DYNLL2, DZANK1, EI24, EIF2S1, EPG5, EPM2A, FABP1, FAM125A, FAM131B, FAM134B, FAM13B, FAM176A, FAM176B, FAM48A, FANCC, FANCF, FANCL, FBXO7, FCGR3B, FGF14, FGF7, FGFBP1, FIS1, FNBP1L, FOXO1, FUNDC1, FUNDC2, FXR2, GABARAP, GABARAPL1, GABARAPL2, GABARAPL3, GABRA5, GDF5, GMIP, HAP1, HAPLN1, HBXIP, HCAR1, HDAC6, HGS, HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3H, HIST1H3I, HIST1H3J, HK2, HMGB1, HPR, HSF2BP, HSP90AA1, HSPA8, IFI16, IPPK, IRGM, IST1, ITGB4, ITPKC, KCNK3, KCNQ1, KIAA0226, KIAA1324, KRCC1, KRT15, KRT73, LAMP1, LAMP2, LAMTOR1, LAMTOR2, LAMTOR3, LARP1B, LENG9, LGALS8, LIX1, LIX1L, LMCD1, LRRK2, LRSAM1, LSM4, MAP1A, MAP1LC3A, MAP1LC3B, MAP1LC3B2, MAP1LC3C, MAP1S, MAP2K1, MAP3K12, MARK2, MBD5, MDH1, MEX3C, MFN1, MFN2, MLST8, MRPS10, MRPS2, MSTN, MTERFD1, MTMR14, MTMR3, MTOR, MTSS1, MYH11, MYLK, MYOM1, NBR1, NDUFB9, NEFM, NHLRC1, NME2, NPC1, NR2C2, NRBF2, NTHL1, NUP93, OBSCN, OPTN, P2RX5, PACS2, PARK2, PARK7, PDK1, PDK4, PEX13, PEX3, PFKP, PGK2, PHF23, PHYHIP, PI4K2A, PIK3C3, PIK3CA, PIK3CB, PIK3R4, PINK1, PLEKHM1, PLOD2, PNPO, PPARGC1A, PPY, PRKAA1, PRKAA2, PRKAB1, PRKAB2, PRKAG1, PRKAG2, PRKAG3, PRKD2, PRKG1, PSEN1, PTPN22, RAB12, RAB1A, RAB1B, RAB23, RAB24, RAB33B, RAB39, RAB7A, RB1CC1, RBM18, REEP2, REP15, RFWD3, RGS19, RHEB, RIMS3, RNF185, RNF41, RPS27A, RPTOR, RRAGA, RRAGB, RRAGC, RRAGD, S100A8, S100A9, SCN1A, SERPINB10, SESN2, SFRP4, SH3GLB1, SIRT2, SLC1A3, SLC1A4, SLC22A3, SLC25A19, SLC35B3, SLC35C1, SLC37A4, SLC6A1, SLCO1A2, SMURF1, SNAP29, SNAPIN, SNF8, SNRPB, SNRPB2, SNRPD1, SNRPF, SNTG1, SNX14, SPATA18, SQSTM1, SRPX, STAM, STAM2, STAT2, STBD1, STK11, STK32A, STOM, STX12, STX17, SUPT3H, TBC1D17, TBC1D25, TBC1D5, TCIRG1, TEAD4, TECPR1, TECPR2, TFEB, TM9SF1, TMBIM6, TMEM203, TMEM208, TMEM39A, TMEM39B, TMEM59, TMEM74, TMEM93, TNIK, TOLLIP, TOMM20, TOMM22, TOMM40, TOMM5, TOMM6, TOMM7, TOMM70A, TP53INP1, TP53INP2, TRAPPC8, TREM1, TRIM17, TRIM5, TSG101, TXLNA, UBA52, UBB, UBC, UBQLN1, UBQLN2, UBQLN4, ULK1, ULK2, ULK3, USP10, USP13, USP30, UVRAG, VAMP7, VAMP8, VDAC1, VMP1, VPS11, VPS16, VPS18, VPS25, VPS28, VPS33A, VPS33B, VPS36, VPS37A, VPS37B, VPS37C, VPS37D, VPS39, VPS41, VPS4A, VPS4B, VTA1, VTI1A, VTI1B, WDFY3, WDR45, WDR45L, WIPI1, WIPI2, XBP1, YIPF1, ZCCHC17, ZFYVE1, ZKSCAN3, ZNF189, ZNF593 и ZNF681.
Набор генов "активируемые в покоящемся состоянии относительно подавляемых в активированном состоянии" включает следующие гены: ABCA7, ABCF3, ACAP2, AMT, ANKH, ATF7IP2, ATG14, ATP1A1, ATXN7, ATXN7L3B, BCL7A, BEX4, BSDC1, BTG1, BTG2, BTN3A1, C11orf21, C19orf22, C21orf2, CAMK2G, CARS2, CCNL2, CD248, CD5, CD55, CEP164, CHKB, CLK1, CLK4, CTSL1, DBP, DCUN1D2, DENND1C, DGKD, DLG1, DUSP1, EAPP, ECE1, ECHDC2, ERBB2IP, FAM117A, FAM134B, FAM134C, FAM169A, FAM190B, FAU, FLJ10038, FOXJ2, FOXJ3, FOXL1, FOXO1, FXYD5, FYB, HLA-E, HSPA1L, HYAL2, ICAM2, IFIT5, IFITM1, IKBKB, IQSEC1, IRS4, KIAA0664L3, KIAA0748, KLF3, KLF9, KRT18, LEF1, LINC00342, LIPA, LIPT1, LLGL2, LMBR1L, LPAR2, LTBP3, LYPD3, LZTFL1, MANBA, MAP2K6, MAP3K1, MARCH8, MAU2, MGEA5, MMP8, MPO, MSL1, MSL3, MYH3, MYLIP, NAGPA, NDST2, NISCH, NKTR, NLRP1, NOSIP, NPIP, NUMA1, PAIP2B, PAPD7, PBXIP1, PCIF1, PI4KA, PLCL2, PLEKHA1, PLEKHF2, PNISR, PPFIBP2, PRKCA, PRKCZ, PRKD3, PRMT2, PTP4A3, PXN, RASA2, RASA3, RASGRP2, RBM38, REPIN1, RNF38, RNF44, ROR1, RPL30, RPL32, RPLP1, RPS20, RPS24, RPS27, RPS6, RPS9, RXRA, RYK, SCAND2, SEMA4C, SETD1B, SETD6, SETX, SF3B1, SH2B1, SLC2A4RG, SLC35E2B, SLC46A3, SMAGP, SMARCE1, SMPD1, SNPH, SP140L, SPATA6, SPG7, SREK1IP1, SRSF5, STAT5B, SVIL, SYF2, SYNJ2BP, TAF1C, TBC1D4, TCF20, TECTA, TES, TMEM127, TMEM159, TMEM30B, TMEM66, TMEM8B, TP53TG1, TPCN1, TRIM22, TRIM44, TSC1, TSC22D1, TSC22D3, TSPYL2, TTC9, TTN, UBE2G2, USP33, USP34, VAMP1, VILL, VIPR1, VPS13C, ZBED5, ZBTB25, ZBTB40, ZC3H3, ZFP161, ZFP36L1, ZFP36L2, ZHX2, ZMYM5, ZNF136, ZNF148, ZNF318, ZNF350, ZNF512B, ZNF609, ZNF652, ZNF83, ZNF862 и ZNF91.
Набор генов "постепенно активируемые при дифференцировке в клетки памяти" включает следующие гены: MTCH2, RAB6C, KIAA0195, SETD2, C2orf24, NRD1, GNA13, COPA, SELT, TNIP1, CBFA2T2, LRP10, PRKCI, BRE, ANKS1A, PNPLA6, ARL6IP1, WDFY1, MAPK1, GPR153, SHKBP1, MAP1LC3B2, PIP4K2A, HCN3, GTPBP1, TLN1, C4orf34, KIF3B, TCIRG1, PPP3CA, ATG4D, TYMP, TRAF6, C17orf76, WIPF1, FAM108A1, MYL6, NRM, SPCS2, GGT3P, GALK1, CLIP4, ARL4C, YWHAQ, LPCAT4, ATG2A, IDS, TBC1D5, DMPK, ST6GALNAC6, REEP5, ABHD6, KIAA0247, EMB, TSEN54, SPIRE2, PIWIL4, ZSCAN22, ICAM1, CHD9, LPIN2, SETD8, ZC3H12A, ULBP3, IL15RA, HLA-DQB2, LCP1, CHP, RUNX3, TMEM43, REEP4, MEF2D, ABL1, TMEM39A, PCBP4, PLCD1, CHST12, RASGRP1, C1orf58, C11orf63, C6orf129, FHOD1, DKFZp434F142, PIK3CG, ITPR3, BTG3, C4orf50, CNNM3, IFI16, AK1, CDK2AP1, REL, BCL2L1, MVD, TTC39C, PLEKHA2, FKBP11, EML4, FANCA, CDCA4, FUCA2, MFSD10, TBCD, CAPN2, IQGAP1, CHST11, PIK3R1, MYO5A, KIR2DL3, DLG3, MXD4, RALGDS, S1PR5, WSB2, CCR3, TIPARP, SP140, CD151, SOX13, KRTAP5-2, NF1, PEA15, PARP8, RNF166, UEVLD, LIMK1, CACNB1, TMX4, SLC6A6, LBA1, SV2A, LLGL2, IRF1, PPP2R5C, CD99, RAPGEF1, PPP4R1, OSBPL7, FOXP4, SLA2, TBC1D2B, ST7, JAZF1, GGA2, PI4K2A, CD68, LPGAT1, STX11, ZAK, FAM160B1, RORA, C8orf80, APOBEC3F, TGFBI, DNAJC1, GPR114, LRP8, CD69, CMIP, NAT13, TGFB1, FLJ00049, ANTXR2, NR4A3, IL12RB1, NTNG2, RDX, MLLT4, GPRIN3, ADCY9, CD300A, SCD5, ABI3, PTPN22, LGALS1, SYTL3, BMPR1A, TBK1, PMAIP1, RASGEF1A, GCNT1, GABARAPL1, STOM, CALHM2, ABCA2, PPP1R16B, SYNE2, PAM, C12orf75, CLCF1, MXRA7, APOBEC3C, CLSTN3, ACOT9, HIP1, LAG3, TNFAIP3, DCBLD1, KLF6, CACNB3, RNF19A, RAB27A, FADS3, DLG5, APOBEC3D, TNFRSF1B, ACTN4, TBKBP1, ATXN1, ARAP2, ARHGEF12, FAM53B, MAN1A1, FAM38A, PLXNC1, GRLF1, SRGN, HLA-DRB5, B4GALT5, WIPI1, PTPRJ, SLFN11, DUSP2, ANXA5, AHNAK, NEO1, CLIC1, EIF2C4, MAP3K5, IL2RB, PLEKHG1, MYO6, GTDC1, EDARADD, GALM, TARP, ADAM8, MSC, HNRPLL, SYT11, ATP2B4, NHSL2, MATK, ARHGAP18, SLFN12L, SPATS2L, RAB27B, PIK3R3, TP53INP1, MBOAT1, GYG1, KATNAL1, FAM46C, ZC3HAV1L, ANXA2P2, CTNNA1, NPC1, C3AR1, CRIM1, SH2D2A, ERN1, YPEL1, TBX21, SLC1A4, FASLG, PHACTR2, GALNT3, ADRB2, PIK3AP1, TLR3, PLEKHA5, DUSP10, GNAO1, PTGDR, FRMD4B, ANXA2, EOMES, CADM1, MAF, TPRG1, NBEAL2, PPP2R2B, PELO, SLC4A4, KLRF1, FOSL2, RGS2, TGFBR3, PRF1, MYO1F, GAB3, C17orf66, MICAL2, CYTH3, TOX, HLA-DRA, SYNE1, WEE1, PYHIN1, F2R, PLD1, THBS1, CD58, FAS, NETO2, CXCR6, ST6GALNAC2, DUSP4, AUTS2, C1orf21, KLRG1, TNIP3, GZMA, PRR5L, PRDM1, ST8SIA6, PLXND1, PTPRM, GFPT2, MYBL1, SLAMF7, FLJ16686, GNLY, ZEB2, CST7, IL18RAP, CCL5, KLRD1 и KLRB1.
Набор генов "активируемые в TEM относительно подавляемых в TN" включает следующие гены: MYO5A, MXD4, STK3, S1PR5, GLCCI1, CCR3, SOX13, KRTAP5-2, PEA15, PARP8, RNF166, UEVLD, LIMK1, SLC6A6, SV2A, KPNA2, OSBPL7, ST7, GGA2, PI4K2A, CD68, ZAK, RORA, TGFBI, DNAJC1, JOSD1, ZFYVE28, LRP8, OSBPL3, CMIP, NAT13, TGFB1, ANTXR2, NR4A3, RDX, ADCY9, CHN1, CD300A, SCD5, PTPN22, LGALS1, RASGEF1A, GCNT1, GLUL, ABCA2, CLDND1, PAM, CLCF1, MXRA7, CLSTN3, ACOT9, METRNL, BMPR1A, LRIG1, APOBEC3G, CACNB3, RNF19A, RAB27A, FADS3, ACTN4, TBKBP1, FAM53B, MAN1A1, FAM38A, GRLF1, B4GALT5, WIPI1, DUSP2, ANXA5, AHNAK, CLIC1, MAP3K5, ST8SIA1, TARP, ADAM8, MATK, SLFN12L, PIK3R3, FAM46C, ANXA2P2, CTNNA1, NPC1, SH2D2A, ERN1, YPEL1, TBX21, STOM, PHACTR2, GBP5, ADRB2, PIK3AP1, DUSP10, PTGDR, EOMES, MAF, TPRG1, NBEAL2, NCAPH, SLC4A4, FOSL2, RGS2, TGFBR3, MYO1F, C17orf66, CYTH3, WEE1, PYHIN1, F2R, THBS1, CD58, AUTS2, FAM129A, TNIP3, GZMA, PRR5L, PRDM1, PLXND1, PTPRM, GFPT2, MYBL1, SLAMF7, ZEB2, CST7, CCL5, GZMK и KLRB1.
Другие наборы генов, описывающие аналогичные процессы и/или характеристики, могут также использоваться для определения характеристик фенотипов клеток, описанных выше.
Использовали Cell Ranger VDJ для получения последовательностей и аннотаций VDJ одиночных клеток для каждой 5'-библиотеки одиночной клетки. Программное обеспечение Loupe Cell Browser и пакеты Bioconductor использовали для анализа и визуализации данных.
Результаты
Целью данного примера является сравнение состояния Т-клеток между очищенными T-клетками, которые выступали в качестве исходных клеток, CAR-T-клетками, изготовленными с использованием способа ARM (обозначенными как клетки "дня 1"), и CAR-T-клетками, изготовленными с использованием способа TM (обозначенными как клетки "дня 9"), с помощью секвенирования РНК одиночных клеток (scRNA-seq). Кроме того, проводили секвенирование TCR одиночных клеток (scTCR-seq) для исследования клональности и отслеживания дифференцировки клеток от исходного материала до материала после изготовления.
Как показано на фиг. 23A-23C, исходные клетки характеризовались наименьшей экспрессией генов и UMI, что позволяет предположить, что такие клетки не были транскрипционно активными и находились в покоящемся состоянии. Клетки дня 1 и дня 9 экспрессировали больше генов, при этом клетки дня 9 были наиболее транскрипционно активными. Аналогичные результаты показаны на фиг. 24A-24D. Исходные клетки не экспрессировали гены, отвечающие за пролиферацию (фиг. 24A и 24D).
Дополнительные данные анализа набора генов показаны на фиг. 25A-25E. Разные популяции клеток сравнивали с использованием медианных баллов наборов генов. Клетки дня 1 и исходные клетки находились в более молодом состоянии, более подобном стволовым клеткам памяти (фиг. 25A-25C). На фиг. 25A значения медианного балла набора генов (активируемые в TEM относительно подавляемых в TSCM) для клеток дня 1, клеток дня 9 и исходных клеток составляют -0,084, 0,035 и -0,1 соответственно. На фиг. 25B значения медианного балла набора генов (активируемые в Treg относительно подавляемых в Teff) для клеток дня 1, клеток дня 9 и исходных клеток составляют -0,082, 0,087 и -0,071 соответственно. На фиг. 25C значения медианного балла набора генов (подавляемые в состоянии стволовости) для клеток дня 1, клеток дня 9 и исходных клеток составляют -0,062, 0,14 и -0,081 соответственно.
Кроме того, клетки дня 1 находились в более оптимальном метаболическом состоянии по сравнению с клетками дня 9 (фиг. 25D и 25E). На фиг. 25D значения медианного балла набора генов (активируемые при гипоксии) для клеток дня 1, клеток дня 9 и исходных клеток составляют 0,019, 0,11 и -0,096 соответственно. На фиг. 25E значения медианного балла набора генов (активируемые при аутофагии) для клеток дня 1, клеток дня 9 и исходных клеток составляют 0,066, 0,11 и -0,09 соответственно.
Исходные клетки включали четыре кластера, выделенные на основании экспрессии генов. Кластер 0 характеризуется высокой экспрессией LMNA, S100A4 и т. д. Кластер 1 характеризуется высокой экспрессией RP913, PRKCQ-AS1 и т. д. Кластер 2 характеризуется высокой экспрессией PR11-291B21.2, CD8B и т. д. Кластер 3 характеризуется высокой экспрессией NKG7, GZMH, CCL5, CST7, GNLY, FGFBP2, GZMA, CCL4, CTSW, CD8A и т. д. На графике, построенном методом нелинейного снижения размерности и визуализации многомерных переменных (TSNE), для исходных клеток кластер 3 отделялся от других клеток, и кластер 1 и кластер 2 было тяжело отличить.
В соответствии с анализом набора генов, показанным на фиг. 26A-26C, кластер 0 и кластер 3 были обогащены фенотипом регуляторных T-клеток по сравнению с кластером 1 и кластером 2, которые были обогащены фенотипом эффектoрных T-клеток. В кластере 3 преобладали поздние T-клетки памяти/эффектoрные T-клетки памяти (TEM), в кластере 1 и кластере 2 - ранние Т-клетки памяти и необученные Т-клетки, и кластер 0 находится посередине. Большинство исходных клеток находились в состоянии необученных и ранних Т-клеток памяти. Не ограничиваясь какой-либо теорией, полагают, что такие клетки могут проявлять себя лучше всего в ходе процедуры изготовления.
Меньшая транскрипционная гетерогенность наблюдалась у клеток дня 1 и клеток дня 9 (данные не показаны).
Как и исходная популяция, клетки дня 1 демонстрировали более крупный кластер ранних клеток памяти и менее крупный кластер поздних клеток памяти на графике TSNE. Это было сходно с тем, что наблюдалось с кластером 3 исходных клеток. В отличие от этого, клетки дня 9 не демонстрировали отдельных кластеров ранних клеток памяти на графике TSNE. Это означает, что ко дню 9 клетки стали более однородными.
Секвенировали TCR, и измеряли разнообразие клонотипов. В целом, три профиля клонотипов были очень плоскими - большинство клонов встречались только один раз (фиг. 27A-27C и таблица 24). Энтропия Шеннона в таблице 24 является мерой плосковершинности распределения. Преобладающими клонами в исходных клетках являлись поздние клетки памяти. График для клеток дня 1 выглядел сходно с графиком для исходных клеток, но начинал выравниваться. Ко дню 9 график для преобладающих клонов существенно выравнивался, и распределение было намного более плосковершинным. Показатель разнообразия был наиболее высоким в день 9, поскольку для клеток дня 9 наблюдалось намного более равномерное и плосковершинное распределение, чем для исходных клеток или клеток дня 1.
Сводка результатов
Наблюдались значительные отличия в состоянии Т-клеток между продуктами дня 1 и дня 9. Клетки дня 1 были намного более подобными исходным клеткам и характеризовались обогащением сигнатурами стволовости, что указывало на более эффективный продукт.
Пример 9. Котрансдукция CAR для BCMA и CAR для CD19, оценивание MOI и исследования эффективности
В данном примере описано определение характеристик CAR-Т-клеток, полученных путем котрансдукции Т-клеток с помощью лентивирусного вектора, кодирующего CAR для BCMA, и лентивирусного вектора, кодирующего CAR для CD19. CAR для BCMA содержит аминокислотную последовательность под SEQ ID NO: 107, а CAR для CD19 содержит аминокислотную последовательность под SEQ ID NO: 225.
Получение клеток и трансдукция CAR
Четыре разные комбинации MOI для CAR для BCMA и CD19 при трансдукции Т-клеток тестировали в формате 24-луночного планшета. Лентивирусный вектор, кодирующий CAR для BCMA, и лентивирусный вектор, кодирующий CAR для CD19, использовали при MOI, составляющей 5 и 1 соответственно (5/1 на фиг. 28A и 28B); 5 и 0,5 соответственно (5/0,5 на фиг. 28A и 28B); 2,5 и 1 соответственно (2,5/1 на фиг. 28A и 28B) или 2,5 и 0,5 соответственно (2,5/0,5 на фиг. 28A и 28B). Т-клетки, очищенные на Prodigy, высевали при 3e6/мл, 1 мл/лунка в 24-луночные планшеты, и добавляли векторы, кодирующие CAR как для BCMA, так и для CD19 при разных MOI, как указано выше. После высевания добавляли полимерную наноматрицу TransAct (Miltenyi Biotec), конъюгированную с агонистическими антителами к CD3 и к CD28. Клетки инкубировали в полной среде для Т-клеток OpTmizer, содержащей 100 МЕ/мл рекомбинантного IL-2 человека (Prometheus, Сан-Диего, Калифорния) и 2% ICRS (Life Technologies), при 37°C и 5% CO2 в течение 24 часов перед сбором.
Затем клетки трижды промывали 3-кратным объемом PBS+1% HAS. После культивирования и промывания собранного материала оценивали жизнеспособность и количество клеток, чтобы определить влияние титрования вектора на конечный продукт.
Затем клетки повторно культивировали при 1×106 VNC/мл в 24-луночном планшете и инкубировали в течение еще 6 дней. Анализ методом FACS для оценки экспрессии CAR для BCMA проводили в дни 4 и 7 после трансдукции (дни 3 и 6 после сбора).
Анализ экспрессии CAR
Образцы измеряли на проточном цитометре (BD LSRFortessa), и данные анализировали с помощью программного обеспечения FlowJo. В способе ARM, поскольку CAR может не полностью интегрироваться и экспрессироваться ex vivo в течение 24 часов, 96 часов после добавления вируса могут служить в качестве суррогатного момента времени для определения стратегии введения доз in vitro и in vivo при стабильной экспрессии CAR. Ту же стратегию применяли для измерений коктейля CAR с двойным нацеливанием.
Анализы методом FACS указывали на то, что котрансдукция Т-клеток с помощью CAR для BCMA и CD19 приводила к образованию трех разных CAR+ субпопуляций, и их относительные доли варьировались при тестировании четырех разных комбинаций MOI (фиг. 28A), что указывает на возможность успешного получения продукта на основе коктейля CAR-T путем котрансдукции CAR для BCMA и CD19. При MOI, составляющей 5/0,5, доля моно-BCMA-CAR+ (CD19-CAR-отрицательных) была наиболее высокой и составляла 20,7%, за ней следовала доля дважды CAR+, составляющая 16,2%, и моно-CD19-CAR+, составляющая 10,5%. Общая доля BCMA-CAR+, рассчитанная как % дважды CAR+ и моно-BCMA-CAR+, составляла 36,9%, а общая доля CD19-CAR+ составляла 26,7% (фиг. 28A и 28B). Оба из соотношений MOI, составляющих 2,5/0,5 и 2,5/1, приводили к намного более высокому % моно-CD19-CAR+, чем % дважды+ и % моно-BCMA-CAR+. Более того, % дважды CAR+ положительно коррелирует с показателем общего использования MOI (фиг. 28B). Общий % CAR+ является сходным между MOI, составляющей 5/1, и MOI, составляющей 5/0,5. Наблюдалось, что экспрессия CAR в каждой популяции была относительно стабильной с дня 4 по день 7 после трансдукции, в том числе в Т-клетках, трансдуцированных с помощью CAR для BCMA или CD19 (фиг. 28A и 28B). Данные были согласующимися для трех доноров. В некоторых вариантах осуществления использовали MOI, составляющую 4~5 для CAR для BCMA, и MOI, составляющую 0,5 для CAR для CD19, для котрансдукции с целью получения коктейля CAR-T с двойным нацеливанием.
Кроме того, эксперименты в крупномасштабном объеме проводили полностью на CliniMACS Prodigy от обогащения Т-клеток, посева Т-клеток, активации, трансдукции и культивирования до промывания собранного материала в CentriCult перед составлением и криоконсервацией. Использовали MOI, составляющую 4/0,5 или 4,75/0,5, для CAR для BCMA/CD19. В данном исследовании использовали плотность высевания 3e6/мл в общем объеме 250 мл для получения общего количества 7,5e8 клеток. Клетки собирали через 24 часа, промывали 3 раза с помощью PBS+1% HAS и криоконсервировали для последующего применения. Оставшиеся Т-клетки собирали для получения соответствующих контрольных групп UTD, BCMA-CAR-T и CD19-CAR-T. Отбирали аликвоту и повторно культивировали при 1e6/мл в 24-луночном планшете для анализов методом проточной цитометрии с целью оценки экспрессии BCMA-CAR в день 4 после трансдукции.
На фиг. 29 показано, что коктейль CAR-T с двойным нацеливанием содержал 11,8% моно-BCMA-CAR-Т-клеток, 4,31% CAR-Т-клеток с двойной специфичностью и ~ 9% моно-CD19-CAR-Т-клеток в день 4 после трансдукции. В данном исследовании общий % BCMA-CAR+, рассчитанный как % дважды CAR+ плюс % моно-BCMA-CAR+, составлял 16%; а общий % CD19-CAR+, рассчитанный как % дважды CAR+ плюс % моно-CD19-CAR+, составлял 13,4%.
Активность коктейля CAR-T с двойным нацеливанием in vivo анализировали в трех ксенотрансплантатных мышиных моделях: 1) модели множественной миеломы из KMS-11-luc (эта модель, экспрессирующая BCMA, мечена репортерной люциферазной конструкцией, что позволяет отслеживать заболевание системно в костном мозге посредством биолюминесцентной визуализации); 2) смешанной модели опухоли, созданной путем смешивания 95% клеток миеломы KMS-11-luc с 5% CD19+ опухолевых клеток (например, Nalm-6-luc) для имитации гетерогенности у пациентов с ММ; и 3) модели Nalm-6-luc для оценивания специфичности нацеливания на CD19 и дополнительного размножения популяции дважды положительных CAR-T в коктейле CAR-T с двойным нацеливанием. BCMA-CAR-T и CD19-CAR-T служили в качестве контролей для их соответствующих моделей. Для расчета дозы CAR-T-клеток для моделей 1 и 2 измеряли общий % BCMA-CAR+ в день 4 после трансдукции, тогда как для модели 3 измеряли общий % CD19-CAR+ в день 4 после трансдукции. Доза UTD отражала наиболее высокую дозу общей популяции Т-клеток в соответствующем способе.
Кривые регрессии опухоли для всех групп в трех моделях показаны на фиг. 30A-E. В модели KMS-11-luc как коктейль CAR-T с двойным нацеливанием, так и BCMA-CAR-T индуцировали регрессию опухоли дозозависимым образом, как показано на фиг. 30A. При дозе 1e4 как коктейль CAR-T с двойным нацеливанием, так и BCMA-CAR-T демонстрировали отсроченное ингибирование опухоли со сходной скоростью, и оба они смогли обеспечить очистку от опухоли в конце исследования. Коктейль CAR-T с двойным нацеливанием продемонстрировал более эффективную очистку от опухоли, чем BCMA-CAR-T, при дозе 5e4, при этом коктейль CAR-T с двойным нацеливанием обеспечивал очистку от опухоли ко дню 14 после инфузии CAR-T, тогда как BCMA-CAR-T демонстрировал эффект, отсроченный на по меньшей мере одну неделю. В повторном исследовании с использованием остальных клеточных продуктов из той же партии, охватывающем более широкие диапазоны доз (1e4, 5e4, 1,5e5) (фиг. 30B), зависимость от дозы подтверждали при регрессии опухоли с помощью коктейля CAR-T с двойным нацеливанием. Кривые регрессии для коктейля CAR-T с двойным нацеливанием при 1,5e5, 5e4 и для BCMA-CAR-T при 1,5e5 накладываются друг на друга. BCMA-CAR-T при 5e4 смог устранить опухоль ко дню 14 после инфузии CAR-T у этой когорты мышей, несмотря на несколько более медленную регрессию опухоли, чем в случае с коктейлем CAR-T с двойным нацеливанием при той же дозе в течение 14 дней после инфузии CAR-T. При дозе 1е4 коктейль CAR-T с двойным нацеливанием демонстрировал лучшую эффективность, чем BCMA-CAR-T. Вкратце, с использованием модели KMS-11-luc было продемонстрировано, что коктейль CAR-T с двойным нацеливанием был способен специфично нацеливаться на BCMA, демонстрировал такую же активность, как BCMA-CAR-T, при уничтожении BCMA+ опухолевых клеток множественной миеломы и был даже более активным, чем BCMA-CAR-T, при более низких дозах.
Далее эффективность коктейля CAR-T с двойным нацеливанием оценивали при наличии как BCMA, так и CD19 в "смешанной" модели, в которой клетки KMS-11-luc, смешанные с 5% клеток Nalm-6-luc, имплантировали мышам. В смешанной модели опухоли, как показано на фиг. 30C, только коктейль CAR-T с двойным нацеливанием при 5e4 и 1e4 демонстрировал частичное ингибирование опухоли дозозависимым образом, тогда как ни BCMA-CAR-T, ни CD19-CAR-T не показали никакого эффекта при дозе 5e4. В повторном исследовании, охватывающем группу с более высокой дозой, как показано на фиг. 30D, было продемонстрировано, что коктейль CAR-T с двойным нацеливанием при 1,5e5 был способен устранить смешанные опухоли ко дню 14 после инфузии CAR-T, и подавление опухоли было дозозависимым. В отличие от этого, BCMA-CAR-T и CD19-CAR-T при 1,5e5 демонстрировали лишь частичное ингибирование опухоли в этой смешанной модели при наличии как BCMA, так и CD19.
Наконец, модель Nalm-6-luc использовали для того, чтобы показать, может ли коктейль CAR-T с двойным нацеливанием специфично нацеливаться на CD19+ опухоль. Как показано на фиг. 30E, коктейль CAR-T с двойным нацеливанием также был способен устранять опухоли Nalm-6-luc дозозависимым образом, что было сравнимым с моно-CD19-CAR-T при более высокой дозе и было лучше, чем для моно-CD19-CAR-T при более низкой дозе.
Пример 10. Оценивание MOI для изготовления клеточного продукта на основе BCMA/CD19-CAR-T
Исходя из титра, определенного с помощью qPCR, проводили титрование вируса, кодирующего CAR для BCMA, для определения оптимального соотношения векторов, кодирующих CAR для BCMA/CAR для CD19. Вкратце, Т-клетки размораживали и ресуспендировали при плотности 3×106 VNC/мл или 6×106 VNC/мл. Для каждой тестируемой MOI 1 мл суспензии клеток высевали в 24-луночный планшет, подвергали трансдукции в момент времени 0 и инкубировали в течение 20-24 часов. Затем 1 мл каждой культуры вручную промывали с помощью 3×2 мл PBS+1% HSA. После культивирования и промывания собранного материала оценивали жизнеспособность и количество клеток, чтобы определить влияние титрования вектора на конечный продукт. Затем собранные клетки помещали в культуру при 1×106 VNC/мл и измеряли экспрессию CAR (для BCMA и для CD19) через 72 часа и 144 часа после сбора (в день 3 и день 6 после сбора (PH) (в день 4 и день 7 после трансдукции), таблица 34 и фиг. 31).
CAR для BCMA/CAR для CD19
День 3 PH День 6 PH
День 3 PH
День 6 PH
В некоторых вариантах осуществления общее количество жизнеспособных BCMA-CAR+ Т-клеток, измеренное с помощью проточной цитометрии, в день 4 после трансдукции (или в день 3 после сбора) используют для расчетов, связанных с дозой. В некоторых вариантах осуществления Т-клетки высевают при плотности, составляющей приблизительно 4e6 VNC/мл, в CentriCult. В некоторых вариантах осуществления вектор, кодирующий CAR для BCMA, используют при MOI, составляющей 4,75, а вектор, кодирующий CAR для CD19, используют при MOI, составляющей 0,5.
В отдельном исследовании BCMA/CD19-CAR-Т-клетки изготавливали с использованием описанного выше подхода быстрого изготовления с котрансдукцией в большом масштабе в устройстве с автоматизированной закрытой системой CliniMACS Prodigy. Вкратце, процесс начинается с отбора Т-клеток из криоконсервированной лейкоцитарной массы. Т-клетки подвергают положительному отбору с помощью микрогранул с антителами к CD4 и CD8. После отбора Т-клетки элюируют в мешок для повторного применения, и образец отбирают для оценки концентрации, жизнеспособности и чистоты клеток. Затем клетки активируют и подвергают трансдукции с помощью лентивирусного вектора, кодирующего CAR для BCMA, и лентивирусного вектора, кодирующего CAR для CD19. Как показано на фиг. 32, субпопуляции Т-клеток в выходном материале из Prodigy не отличались от исходного материала для афереза.
Пример 11. Дополнительное определение характеристик Т-клеток, сконструированных таким образом, что они экспрессируют CAR для BCMA и CAR для CD19
В данном примере описано дополнительное определение характеристик клеточного продукта на основе BCMA/CD19-CAR-T, который получали, как описано в примере 9. Этот клеточный продукт содержит три разные CAR+ популяции: моно-BCMA-CAR-T-клетки, моно-CD19-CAR-T-клетки и дважды положительные BCMA/CD19-CAR-T-клетки. Кроме того, этот клеточный продукт также содержит популяцию нетрансдуцированных Т-клеток.
IFNγ в плазме крови мышей, обработанных с помощью клеточного продукта на основе CAR-T с двойной специфичностью к BCMA/CD19
IFNγ является отличительным признаком активации CAR-T-клеток в ответ на связывание с мишенью. Кинетические показатели IFNγ в плазме крови анализировали в исследованиях in vivo, описанных в примере 9. Как показано на фиг. 33A и 33B, все группы, обработанные с помощью CAR-T, демонстрировали низкие уровни циркулирующего IFNγ (10-50 пг/мл) в день 2, а затем продолжающееся увеличение секреции IFNγ.
Фармакокинетические показатели и метаболизм в неклинических исследованиях
Размножение клеточного продукта на основе CAR-T с двойной специфичностью к BCMA/CD19 в периферической крови (содержащего моно-BCMA-, моно-CD19- и дважды положительные BCMA/CD19-CAR-T-клетки) анализировали с помощью проточной цитометрии в течение вплоть до 3 недель после инфузии и сравнивали с эталонным BCMA-CAR-T в модели KMS-11-luc. Во всех группах обработки с помощью CAR-T наблюдалось размножение как CD3+ Т-клеток, так и CAR+ Т-клеток. Для клеточного продукта на основе CAR-T с двойной специфичностью к BCMA/CD19 в популяции дважды+ или моно-CAR+ T-клеток наблюдалось дозозависимое размножение клеток. На основании данных из двух исследований клеточный продукт на основе CAR-T с двойной специфичностью к BCMA/CD19 демонстрировал несколько более интенсивное размножение CAR+ Т-клеток, нацеливающихся на BCMA, и общей популяции CAR+ Т-клеток по сравнению с BCMA-CAR-T.
Антигензависимое размножение клеточного продукта на основе CAR-T с двойной специфичностью к BCMA/CD19 демонстрировали путем оценки размножения CAR+ Т-клеток в трех моделях (KMS-11, Nalm-6 и смешанной). В ксенотрансплантатной модели KMS-11, экспрессирующей BCMA, интенсивно размножались дважды положительные BCMA/CD19- и моно-BCMA-CAR+ Т-клетки, тогда как в ксенотрансплантатной модели Nalm-6, экспрессирующей CD19, интенсивно размножались дважды положительные BCMA/CD19- и моно-CD19-CAR+ Т-клетки. Популяция дважды положительных CAR-T-клеток была способна размножаться при активации антигеном BCMA либо CD19 в отдельности. Начальные скорости размножения дважды положительных BCMA/CD19- и моно-BCMA-CAR+ Т-клеток были сопоставимыми в модели KMS-11, равно как и скорости размножения дважды CAR-положительных и моно-CD19-CAR+ Т-клеток в модели Nalm-6.
Выводы
Клеточный продукт на основе CAR-T с двойной специфичностью к BCMA/CD19 представляет собой новый продукт на основе CAR-T-клеток с двойным нацеливанием на BCMA и на CD19, полученный с использованием способа быстрого изготовления, в котором сохраняется стволовость Т-клеток.
В трех ксенотрансплантатных мышиных моделях (KMS-11, Nalm-6 и смешанной) клеточный продукт на основе CAR-T с двойной специфичностью к BCMA/CD19 демонстрировал сильные фармакологические характеристики in vivo, контролируя рост опухоли, индуцируя размножение CAR-T и продуцирование цитокинов антигензависимым и дозозависимым образом.
Устранение опухоли в смешанной модели достигалось при использовании клеточного продукта на основе CAR-T с двойным нацеливанием на BCMA/CD19, при этом регрессия опухоли увеличивалась дозозависимым образом. Ни моно-BCMA-CAR-T, ни моно-CD19-CAR-T не демонстрировали регрессию опухоли в смешанной модели. Кроме того, клеточный продукт на основе CAR-T с двойной специфичностью к BCMA/CD19 демонстрировал обширное размножение CAR-T in vivo, тогда как дважды CAR-положительная популяция размножалась при активации с помощью BCMA либо CD19 в отдельности. В модели множественной миеломы из KMS-11-luc клеточный продукт на основе CAR-T с двойной специфичностью к BCMA/CD19 демонстрировал улучшение контроля роста опухоли при более высоких тестируемых уровнях дозы и лучший контроль роста опухоли при более низком тестируемом уровне дозы по сравнению с BCMA-CAR-T.
Эти результаты подтверждают, что клеточный продукт на основе CAR-T с двойной специфичностью к BCMA/CD19 представляет собой CAR-T с двойным нацеливанием, который может изменить клинические исходы путем решения проблемы потенциального вклада BCMA-/CD19+ стволовых клеток/клеток-предшественников в рецидив множественной миеломы, потенциально обеспечивая более глубокие и более продолжительные ответы, чем CAR-T, изготовленные традиционным способом или нацеливающиеся на один антиген BCMA.
Пример 12. Клиническое испытание фазы I BCMA-CAR-Т-клеток, изготовленных с использованием способа ARM
В данном примере описано открытое исследование фазы I для оценки безопасности и переносимости средства терапии на основе BCMA-CAR-T-клеток, изготовленного с использованием способа ARM, у взрослых пациентов с рецидивирующей и/или рефрактерной множественной миеломой.
Первичные показатели исхода включают частоту встречаемости и характер форм ограничивающей дозу токсичности (DLT) в течение первых 28 дней после введения BCMA-CAR-T-клеток, а также частоту встречаемости и тяжесть нежелательных явлений (AE) и серьезных нежелательных явлений (SAE), в том числе изменений лабораторных показателей, ECG и показателей жизненно важных функций после введения BCMA-CAR-T-клеток.
Вторичные показатели исхода включают коэффициент успешности изготовления (определяемый как количество субъектов, получавших лечение в запланированной целевой дозе, деленное на общее количество субъектов, получавших лечение), ORR (долю субъектов с наилучшим общим ответом (BOR) sCR+CR+VGPR+PR в месяцы 3 и 6, определенную местным исследователем с использованием критериев IMWG (Kumar et al., Lancet Oncol. 2016 Aug;17(8): e328-e346, включенная в данный документ посредством ссылки во всей своей полноте)), CRR (долю субъектов с BOR sCR+CR в месяц 3, определенную местным исследователем с использованием критериев IMWG), DOR согласно оценке местного исследователя (время от достижения sCR+CR+VGPR+PR до рецидива или смерти в связи с ММ), выявляемые с помощью qPCR концентрации трансгена CAR-T с течением времени в периферической крови и костном мозге, а также сводную информацию об уже имеющейся и индуцированной лечением иммуногенности (клеточной и гуморальной) средства терапии на основе BCMA-CAR-T-клеток.
Критерии включения являются следующими:
субъекты с MM, у которых проявляется рецидив и/или рефрактерность к по меньшей мере 2 предшествующим режимам лечения, включая IMiD (например, леналидомид или помалидомид), ингибитор протеасом (например, бортезомиб, карфилзомиб) и одобренное антитело к CD38 (например, даратумумаб), при наличии таковых и у которых имеется документально подтвержденное свидетельство прогрессирования заболевания (согласно критериям IMWG);
поддающееся измерению заболевание, определенное протоколом;
показатель общего состояния по шкале ECOG, составляющий 0 или 1 на момент скрининга;
надлежащие гематологические показатели и
наличие материала, полученного путем лейкафереза из немобилизованных клеток, принятого для изготовления.
Критерии исключения являются следующими:
предшествующее введение генетически модифицированного клеточного продукта, в том числе предшествующего средства терапии на основе BCMA-CAR-T. Пациенты, получавшие ранее биспецифические антитела или конъюгаты антитело-лекарственное средство (ADC), направленные на BCMA, не исключаются;
аутологичная HSCT в течение 6 недель до включения в исследование или любая предшествующая аллогенная трансплантация гемопоэтических стволовых клеток (HSCT) в анамнезе;
химиотерапия или любая сопутствующая противораковая терапия (кроме предписанной протоколом химиотерапии с лимфодеплецией (LD)) в течение 2 недель до афереза;
лечение низкомолекулярными нацеленными антинеопластическими средствами в течение 2 недель до сбора продукта афереза или 5 периодов полужизни, в зависимости от того, какой срок является более коротким; и
получение антител или иммунотерапевтических средств (кроме даратумумаба) в течение 4 недель до сбора продукта афереза. Даратумумаб в течение 3 недель до сбора продукта афереза.
Пример 13. Оценивание BCMA/CD19-CAR-T в формате диатела
Эффективность элемента ответа на антиген CD19 в новых CAR-Т в формате одноцепочечного диатела оценивают с помощью модели NALM-6 с люциферазой. 1×106 клеток NALM-6-Luc имплантируют путем инъекции в боковую хвостовую вену в день -7 от введения дозы CAR-T. Измеряют массу тела, и дважды в неделю проводят биолюминесцентную визуализацию (BLI) in vivo для оценивания прогрессирования опухоли. Животных измеряют дважды в неделю, и после достижения опухолевой нагрузки, соответствующей фотонному потоку 3×106 (фотоны/секунда), животных рандомизировали в определенную группу (день -1). В день 0 CAR-Т в формате диатела извлекают из жидкого азота и размораживают для инъекции. 1×106 дважды CAR-положительных клеток инъецируют в боковую хвостовую вену. Экспериментальное оценивание проводят на протяжении нескольких недель, чтобы определить, какая конструкция обладает наилучшей функциональной эффективностью, путем оценивания уменьшения интенсивности в BLI с течением времени.
ЭКВИВАЛЕНТЫ
Раскрытия всех без исключения патентов, заявок на патент и публикаций, цитируемых в данном документе, настоящим включены в данный документ посредством ссылки во всей своей полноте. Несмотря на то, что настоящее изобретение было раскрыто со ссылкой на определенные варианты осуществления, очевидно, что дополнительные варианты осуществления и видоизменения настоящего изобретения могут быть разработаны другими специалистами в данной области без отступления от фактической сущности и объема настоящего изобретения. Предполагается, что прилагаемую формулу изобретения следует истолковывать как включающую все такие варианты осуществления и эквивалентные видоизменения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> NOVARTIS AG
<120> ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ПУТИ ИХ ПРИМЕНЕНИЯ
<130> N2067-7166WO
<140>
<141>
<150> 62/940,509
<151> 2019-11-26
<160> 417
<170> PatentIn версия 3.5
<210> 1
<211> 21
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности:
синтетический пептид"
<400> 1
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro
20
<210> 2
<211> 45
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 2
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 3
<211> 230
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 3
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys Met
225 230
<210> 4
<211> 282
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 4
Arg Trp Pro Glu Ser Pro Lys Ala Gln Ala Ser Ser Val Pro Thr Ala
1 5 10 15
Gln Pro Gln Ala Glu Gly Ser Leu Ala Lys Ala Thr Thr Ala Pro Ala
20 25 30
Thr Thr Arg Asn Thr Gly Arg Gly Gly Glu Glu Lys Lys Lys Glu Lys
35 40 45
Glu Lys Glu Glu Gln Glu Glu Arg Glu Thr Lys Thr Pro Glu Cys Pro
50 55 60
Ser His Thr Gln Pro Leu Gly Val Tyr Leu Leu Thr Pro Ala Val Gln
65 70 75 80
Asp Leu Trp Leu Arg Asp Lys Ala Thr Phe Thr Cys Phe Val Val Gly
85 90 95
Ser Asp Leu Lys Asp Ala His Leu Thr Trp Glu Val Ala Gly Lys Val
100 105 110
Pro Thr Gly Gly Val Glu Glu Gly Leu Leu Glu Arg His Ser Asn Gly
115 120 125
Ser Gln Ser Gln His Ser Arg Leu Thr Leu Pro Arg Ser Leu Trp Asn
130 135 140
Ala Gly Thr Ser Val Thr Cys Thr Leu Asn His Pro Ser Leu Pro Pro
145 150 155 160
Gln Arg Leu Met Ala Leu Arg Glu Pro Ala Ala Gln Ala Pro Val Lys
165 170 175
Leu Ser Leu Asn Leu Leu Ala Ser Ser Asp Pro Pro Glu Ala Ala Ser
180 185 190
Trp Leu Leu Cys Glu Val Ser Gly Phe Ser Pro Pro Asn Ile Leu Leu
195 200 205
Met Trp Leu Glu Asp Gln Arg Glu Val Asn Thr Ser Gly Phe Ala Pro
210 215 220
Ala Arg Pro Pro Pro Gln Pro Gly Ser Thr Thr Phe Trp Ala Trp Ser
225 230 235 240
Val Leu Arg Val Pro Ala Pro Pro Ser Pro Gln Pro Ala Thr Tyr Thr
245 250 255
Cys Val Val Ser His Glu Asp Ser Arg Thr Leu Leu Asn Ala Ser Arg
260 265 270
Ser Leu Glu Val Ser Tyr Val Thr Asp His
275 280
<210> 5
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 5
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 6
<211> 24
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 6
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr Cys
20
<210> 7
<211> 42
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 7
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 8
<211> 48
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 8
Gln Arg Arg Lys Tyr Arg Ser Asn Lys Gly Glu Ser Pro Val Glu Pro
1 5 10 15
Ala Glu Pro Cys Arg Tyr Ser Cys Pro Arg Glu Glu Glu Gly Ser Thr
20 25 30
Ile Pro Ile Gln Glu Asp Tyr Arg Lys Pro Glu Pro Ala Cys Ser Pro
35 40 45
<210> 9
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 9
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 10
<211> 112
<212> PRT
<213> Homo sapiens
<400> 10
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 11
<211> 1184
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 11
cgtgaggctc cggtgcccgt cagtgggcag agcgcacatc gcccacagtc cccgagaagt 60
tggggggagg ggtcggcaat tgaaccggtg cctagagaag gtggcgcggg gtaaactggg 120
aaagtgatgt cgtgtactgg ctccgccttt ttcccgaggg tgggggagaa ccgtatataa 180
gtgcagtagt cgccgtgaac gttctttttc gcaacgggtt tgccgccaga acacaggtaa 240
gtgccgtgtg tggttcccgc gggcctggcc tctttacggg ttatggccct tgcgtgcctt 300
gaattacttc cacctggctg cagtacgtga ttcttgatcc cgagcttcgg gttggaagtg 360
ggtgggagag ttcgaggcct tgcgcttaag gagccccttc gcctcgtgct tgagttgagg 420
cctggcctgg gcgctggggc cgccgcgtgc gaatctggtg gcaccttcgc gcctgtctcg 480
ctgctttcga taagtctcta gccatttaaa atttttgatg acctgctgcg acgctttttt 540
tctggcaaga tagtcttgta aatgcgggcc aagatctgca cactggtatt tcggtttttg 600
gggccgcggg cggcgacggg gcccgtgcgt cccagcgcac atgttcggcg aggcggggcc 660
tgcgagcgcg gccaccgaga atcggacggg ggtagtctca agctggccgg cctgctctgg 720
tgcctggcct cgcgccgccg tgtatcgccc cgccctgggc ggcaaggctg gcccggtcgg 780
caccagttgc gtgagcggaa agatggccgc ttcccggccc tgctgcaggg agctcaaaat 840
ggaggacgcg gcgctcggga gagcgggcgg gtgagtcacc cacacaaagg aaaagggcct 900
ttccgtcctc agccgtcgct tcatgtgact ccacggagta ccgggcgccg tccaggcacc 960
tcgattagtt ctcgagcttt tggagtacgt cgtctttagg ttggggggag gggttttatg 1020
cgatggagtt tccccacact gagtgggtgg agactgaagt taggccagct tggcacttga 1080
tgtaattctc cttggaattt gccctttttg agtttggatc ttggttcatt ctcaagcctc 1140
agacagtggt tcaaagtttt tttcttccat ttcaggtgtc gtga 1184
<210> 12
<211> 63
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 12
atggccctgc ctgtgacagc cctgctgctg cctctggctc tgctgctgca tgccgctaga 60
ccc 63
<210> 13
<211> 135
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 13
accacgacgc cagcgccgcg accaccaaca ccggcgccca ccatcgcgtc gcagcccctg 60
tccctgcgcc cagaggcgtg ccggccagcg gcggggggcg cagtgcacac gagggggctg 120
gacttcgcct gtgat 135
<210> 14
<211> 690
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 14
gagagcaagt acggccctcc ctgcccccct tgccctgccc ccgagttcct gggcggaccc 60
agcgtgttcc tgttcccccc caagcccaag gacaccctga tgatcagccg gacccccgag 120
gtgacctgtg tggtggtgga cgtgtcccag gaggaccccg aggtccagtt caactggtac 180
gtggacggcg tggaggtgca caacgccaag accaagcccc gggaggagca gttcaatagc 240
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg actggctgaa cggcaaggaa 300
tacaagtgta aggtgtccaa caagggcctg cccagcagca tcgagaaaac catcagcaag 360
gccaagggcc agcctcggga gccccaggtg tacaccctgc cccctagcca agaggagatg 420
accaagaacc aggtgtccct gacctgcctg gtgaagggct tctaccccag cgacatcgcc 480
gtggagtggg agagcaacgg ccagcccgag aacaactaca agaccacccc ccctgtgctg 540
gacagcgacg gcagcttctt cctgtacagc cggctgaccg tggacaagag ccggtggcag 600
gagggcaacg tctttagctg ctccgtgatg cacgaggccc tgcacaacca ctacacccag 660
aagagcctga gcctgtccct gggcaagatg 690
<210> 15
<211> 847
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 15
aggtggcccg aaagtcccaa ggcccaggca tctagtgttc ctactgcaca gccccaggca 60
gaaggcagcc tagccaaagc tactactgca cctgccacta cgcgcaatac tggccgtggc 120
ggggaggaga agaaaaagga gaaagagaaa gaagaacagg aagagaggga gaccaagacc 180
cctgaatgtc catcccatac ccagccgctg ggcgtctatc tcttgactcc cgcagtacag 240
gacttgtggc ttagagataa ggccaccttt acatgtttcg tcgtgggctc tgacctgaag 300
gatgcccatt tgacttggga ggttgccgga aaggtaccca cagggggggt tgaggaaggg 360
ttgctggagc gccattccaa tggctctcag agccagcact caagactcac ccttccgaga 420
tccctgtgga acgccgggac ctctgtcaca tgtactctaa atcatcctag cctgccccca 480
cagcgtctga tggcccttag agagccagcc gcccaggcac cagttaagct tagcctgaat 540
ctgctcgcca gtagtgatcc cccagaggcc gccagctggc tcttatgcga agtgtccggc 600
tttagcccgc ccaacatctt gctcatgtgg ctggaggacc agcgagaagt gaacaccagc 660
ggcttcgctc cagcccggcc cccaccccag ccgggttcta ccacattctg ggcctggagt 720
gtcttaaggg tcccagcacc acctagcccc cagccagcca catacacctg tgttgtgtcc 780
catgaagata gcaggaccct gctaaatgct tctaggagtc tggaggtttc ctacgtgact 840
gaccatt 847
<210> 16
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 16
ggtggcggag gttctggagg tggaggttcc 30
<210> 17
<211> 72
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 17
atctacatct gggcgccctt ggccgggact tgtggggtcc ttctcctgtc actggttatc 60
accctttact gc 72
<210> 18
<211> 126
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 18
aaacggggca gaaagaaact cctgtatata ttcaaacaac catttatgag accagtacaa 60
actactcaag aggaagatgg ctgtagctgc cgatttccag aagaagaaga aggaggatgt 120
gaactg 126
<210> 19
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 19
aggagtaaga ggagcaggct cctgcacagt gactacatga acatgactcc ccgccgcccc 60
gggcccaccc gcaagcatta ccagccctat gccccaccac gcgacttcgc agcctatcgc 120
tcc 123
<210> 20
<211> 336
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 20
agagtgaagt tcagcaggag cgcagacgcc cccgcgtaca agcagggcca gaaccagctc 60
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc 120
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 180
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 240
cggaggggca aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc 300
tacgacgccc ttcacatgca ggccctgccc cctcgc 336
<210> 21
<211> 336
<212> DNA
<213> Homo sapiens
<400> 21
agagtgaagt tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc 60
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc 120
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 180
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 240
cggaggggca aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc 300
tacgacgccc ttcacatgca ggccctgccc cctcgc 336
<210> 22
<211> 373
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 22
Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp Asn Pro Pro Thr
1 5 10 15
Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp Asn Ala Thr Phe
20 25 30
Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val Leu Asn Trp Tyr
35 40 45
Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala Ala Phe Pro Glu
50 55 60
Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg Val Thr Gln Leu
65 70 75 80
Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg Ala Arg Arg Asn
85 90 95
Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu Ala Pro Lys Ala
100 105 110
Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val Thr Glu Arg Arg
115 120 125
Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro Arg Pro Ala Gly
130 135 140
Gln Phe Gln Thr Leu Val Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr
145 150 155 160
Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala
165 170 175
Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe
180 185 190
Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val
195 200 205
Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys
210 215 220
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
225 230 235 240
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
245 250 255
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
260 265 270
Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
275 280 285
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
290 295 300
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
305 310 315 320
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
325 330 335
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
340 345 350
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
355 360 365
Ala Leu Pro Pro Arg
370
<210> 23
<211> 1182
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 23
atggccctcc ctgtcactgc cctgcttctc cccctcgcac tcctgctcca cgccgctaga 60
ccacccggat ggtttctgga ctctccggat cgcccgtgga atcccccaac cttctcaccg 120
gcactcttgg ttgtgactga gggcgataat gcgaccttca cgtgctcgtt ctccaacacc 180
tccgaatcat tcgtgctgaa ctggtaccgc atgagcccgt caaaccagac cgacaagctc 240
gccgcgtttc cggaagatcg gtcgcaaccg ggacaggatt gtcggttccg cgtgactcaa 300
ctgccgaatg gcagagactt ccacatgagc gtggtccgcg ctaggcgaaa cgactccggg 360
acctacctgt gcggagccat ctcgctggcg cctaaggccc aaatcaaaga gagcttgagg 420
gccgaactga gagtgaccga gcgcagagct gaggtgccaa ctgcacatcc atccccatcg 480
cctcggcctg cggggcagtt tcagaccctg gtcacgacca ctccggcgcc gcgcccaccg 540
actccggccc caactatcgc gagccagccc ctgtcgctga ggccggaagc atgccgccct 600
gccgccggag gtgctgtgca tacccgggga ttggacttcg catgcgacat ctacatttgg 660
gctcctctcg ccggaacttg tggcgtgctc cttctgtccc tggtcatcac cctgtactgc 720
aagcggggtc ggaaaaagct tctgtacatt ttcaagcagc ccttcatgag gcccgtgcaa 780
accacccagg aggaggacgg ttgctcctgc cggttccccg aagaggaaga aggaggttgc 840
gagctgcgcg tgaagttctc ccggagcgcc gacgcccccg cctataagca gggccagaac 900
cagctgtaca acgaactgaa cctgggacgg cgggaagagt acgatgtgct ggacaagcgg 960
cgcggccggg accccgaaat gggcgggaag cctagaagaa agaaccctca ggaaggcctg 1020
tataacgagc tgcagaagga caagatggcc gaggcctact ccgaaattgg gatgaaggga 1080
gagcggcgga ggggaaaggg gcacgacggc ctgtaccaag gactgtccac cgccaccaag 1140
gacacatacg atgccctgca catgcaggcc cttccccctc gc 1182
<210> 24
<211> 394
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 24
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro
20 25 30
Trp Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly
35 40 45
Asp Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe
50 55 60
Val Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu
65 70 75 80
Ala Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe
85 90 95
Arg Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val
100 105 110
Arg Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser
115 120 125
Leu Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg
130 135 140
Val Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser
145 150 155 160
Pro Arg Pro Ala Gly Gln Phe Gln Thr Leu Val Thr Thr Thr Pro Ala
165 170 175
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
180 185 190
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
195 200 205
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
210 215 220
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
225 230 235 240
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
245 250 255
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
260 265 270
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
275 280 285
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
290 295 300
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
305 310 315 320
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
325 330 335
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
340 345 350
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
355 360 365
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
370 375 380
Ala Leu His Met Gln Ala Leu Pro Pro Arg
385 390
<210> 25
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 25
Gly Gly Gly Gly Ser
1 5
<210> 26
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> SITE
<222> (1)..(30)
<223> /note="This sequence may encompass 1-6 'Gly Gly Gly Gly Ser'
repeating units"
<220>
<221> source
<223> /note="See specification as filed for detailed description of
substitutions and preferred embodiments"
<400> 26
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
20 25 30
<210> 27
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 27
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 28
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 28
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 29
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 29
Gly Gly Gly Ser
1
<210> 30
<211> 5000
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> misc_feature
<222> (1)..(5000)
<223> /note="This sequence may encompass 50-5000 nucleotides"
<400> 30
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 60
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 180
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 300
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 360
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 420
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 480
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 540
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 600
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 660
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 720
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 780
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 840
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 900
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 960
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1020
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1080
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1140
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1200
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1260
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1320
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1380
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1440
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1500
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1560
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1620
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1680
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1740
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1800
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1860
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1920
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1980
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2040
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2100
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2160
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2220
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2280
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2340
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2400
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2460
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2520
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2580
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2640
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2700
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2760
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2820
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2880
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2940
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3000
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3060
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3180
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3300
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3360
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3420
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3480
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3540
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3600
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3660
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3720
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3780
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3840
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3900
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3960
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4020
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4080
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4140
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4200
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4260
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4320
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4380
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4440
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4500
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4560
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4620
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4680
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4740
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4800
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4860
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4920
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4980
aaaaaaaaaa aaaaaaaaaa 5000
<210> 31
<211> 100
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 31
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 60
tttttttttt tttttttttt tttttttttt tttttttttt 100
<210> 32
<211> 5000
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> misc_feature
<222> (1)..(5000)
<223> /note="This sequence may encompass 50-5000 nucleotides"
<400> 32
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 60
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 120
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 180
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 240
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 300
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 360
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 420
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 480
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 540
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 600
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 660
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 720
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 780
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 840
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 900
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 960
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1020
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1080
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1140
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1200
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1260
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1320
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1380
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1440
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1500
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1560
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1620
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1680
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1740
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1800
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1860
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1920
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 1980
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2040
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2100
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2160
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2220
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2280
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2340
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2400
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2460
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2520
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2580
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2640
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2700
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2760
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2820
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2880
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 2940
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3000
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3060
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3120
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3180
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3240
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3300
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3360
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3420
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3480
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3540
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3600
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3660
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3720
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3780
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3840
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3900
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 3960
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4020
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4080
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4140
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4200
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4260
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4320
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4380
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4440
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4500
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4560
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4620
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4680
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4740
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4800
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4860
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4920
tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt tttttttttt 4980
tttttttttt tttttttttt 5000
<210> 33
<211> 5000
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> misc_feature
<222> (1)..(5000)
<223> /note="This sequence may encompass 100-5000 nucleotides"
<400> 33
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 60
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 180
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 300
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 360
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 420
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 480
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 540
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 600
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 660
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 720
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 780
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 840
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 900
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 960
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1020
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1080
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1140
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1200
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1260
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1320
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1380
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1440
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1500
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1560
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1620
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1680
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1740
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1800
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1860
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1920
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1980
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2040
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2100
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2160
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2220
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2280
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2340
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2400
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2460
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2520
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2580
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2640
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2700
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2760
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2820
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2880
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 2940
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3000
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3060
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3180
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3300
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3360
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3420
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3480
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3540
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3600
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3660
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3720
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3780
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3840
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3900
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3960
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4020
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4080
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4140
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4200
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4260
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4320
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4380
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4440
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4500
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4560
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4620
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4680
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4740
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4800
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4860
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4920
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4980
aaaaaaaaaa aaaaaaaaaa 5000
<210> 34
<211> 400
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> misc_feature
<222> (1)..(400)
<223> /note="This sequence may encompass 100-400 nucleotides"
<400> 34
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 60
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 180
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 300
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 360
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 400
<210> 35
<211> 2000
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> misc_feature
<222> (1)..(2000)
<223> /note="This sequence may encompass 50-2000 nucleotides"
<400> 35
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 60
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 180
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 300
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 360
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 420
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 480
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 540
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 600
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 660
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 720
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 780
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 840
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 900
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 960
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1020
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1080
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1140
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1200
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1260
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1320
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1380
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1440
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1500
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1560
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1620
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1680
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1740
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1800
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1860
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1920
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1980
aaaaaaaaaa aaaaaaaaaa 2000
<210> 36
<211> 41
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 36
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 37
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 37
aggagtaaga ggagcaggct cctgcacagt gactacatga acatgactcc ccgccgcccc 60
gggcccaccc gcaagcatta ccagccctat gccccaccac gcgacttcgc agcctatcgc 120
tcc 123
<210> 38
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 38
Thr Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro Asn Gly Glu Tyr
1 5 10 15
Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser Arg Leu Thr Asp
20 25 30
Val Thr Leu
35
<210> 39
<211> 105
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 39
acaaaaaaga agtattcatc cagtgtgcac gaccctaacg gtgaatacat gttcatgaga 60
gcagtgaaca cagccaaaaa atccagactc acagatgtga cccta 105
<210> 40
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 40
ggtggcggag gttctggagg tgggggttcc 30
<210> 41
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 41
Gly Gly Gly Ser
1
<210> 42
<211> 40
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> SITE
<222> (1)..(40)
<223> /note="This sequence may encompass 1-10 'Gly Gly Gly Ser'
repeating units"
<400> 42
Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Ser
20 25 30
Gly Gly Gly Ser Gly Gly Gly Ser
35 40
<210> 43
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 43
Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr
1 5 10 15
Lys Gly
<210> 44
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 44
Ser Tyr Ala Met Ser
1 5
<210> 45
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 45
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 46
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 46
Arg Glu Trp Val Pro Tyr Asp Val Ser Trp Tyr Phe Asp Tyr
1 5 10
<210> 47
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 47
Gly Phe Thr Phe Ser Ser Tyr
1 5
<210> 48
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 48
Ser Gly Ser Gly Gly Ser
1 5
<210> 49
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 49
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 50
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 50
Ile Ser Gly Ser Gly Gly Ser Thr
1 5
<210> 51
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 51
Ala Arg Arg Glu Trp Val Pro Tyr Asp Val Ser Trp Tyr Phe Asp Tyr
1 5 10 15
<210> 52
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 52
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Val Pro Tyr Asp Val Ser Trp Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 53
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 53
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tgggtgccct acgatgtcag ctggtacttc gactactggg gacagggcac tctcgtgact 360
gtgtcctcc 369
<210> 54
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 54
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 55
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 55
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 56
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 56
Gln Gln Ser Tyr Ser Thr Pro Leu Thr
1 5
<210> 57
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 57
Ser Gln Ser Ile Ser Ser Tyr
1 5
<210> 58
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 58
Ala Ala Ser
1
<210> 59
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 59
Ser Tyr Ser Thr Pro Leu
1 5
<210> 60
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 60
Gln Ser Ile Ser Ser Tyr
1 5
<210> 61
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 61
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 62
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 62
gacattcaaa tgactcagtc cccgtcctcc ctctccgcct ccgtgggaga tcgcgtcacg 60
atcacgtgca gggccagcca gagcatctcc agctacctga actggtacca gcagaagcca 120
gggaaggcac cgaagctcct gatctacgcc gctagctcgc tgcagtccgg cgtcccttca 180
cggttctcgg gatcgggctc aggcaccgac ttcaccctga ccattagcag cctgcagccg 240
gaggacttcg cgacatacta ctgtcagcag tcatactcca cccctctgac cttcggccaa 300
gggaccaaag tggagatcaa g 321
<210> 63
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 63
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 64
<211> 250
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 64
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Val Pro Tyr Asp Val Ser Trp Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
245 250
<210> 65
<211> 750
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 65
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tgggtgccct acgatgtcag ctggtacttc gactactggg gacagggcac tctcgtgact 360
gtgtcctccg gtggtggtgg atcggggggt ggtggttcgg gcggaggagg atctggagga 420
ggagggtcgg acattcaaat gactcagtcc ccgtcctccc tctccgcctc cgtgggagat 480
cgcgtcacga tcacgtgcag ggccagccag agcatctcca gctacctgaa ctggtaccag 540
cagaagccag ggaaggcacc gaagctcctg atctacgccg ctagctcgct gcagtccggc 600
gtcccttcac ggttctcggg atcgggctca ggcaccgact tcaccctgac cattagcagc 660
ctgcagccgg aggacttcgc gacatactac tgtcagcagt catactccac ccctctgacc 720
ttcggccaag ggaccaaagt ggagatcaag 750
<210> 66
<211> 473
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 66
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Val Pro Tyr Asp Val Ser Trp Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro
245 250 255
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
260 265 270
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
275 280 285
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
290 295 300
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
305 310 315 320
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
325 330 335
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
340 345 350
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
355 360 365
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
370 375 380
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
385 390 395 400
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
405 410 415
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
420 425 430
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
435 440 445
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
450 455 460
Leu His Met Gln Ala Leu Pro Pro Arg
465 470
<210> 67
<211> 1419
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 67
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tgggtgccct acgatgtcag ctggtacttc gactactggg gacagggcac tctcgtgact 360
gtgtcctccg gtggtggtgg atcggggggt ggtggttcgg gcggaggagg atctggagga 420
ggagggtcgg acattcaaat gactcagtcc ccgtcctccc tctccgcctc cgtgggagat 480
cgcgtcacga tcacgtgcag ggccagccag agcatctcca gctacctgaa ctggtaccag 540
cagaagccag ggaaggcacc gaagctcctg atctacgccg ctagctcgct gcagtccggc 600
gtcccttcac ggttctcggg atcgggctca ggcaccgact tcaccctgac cattagcagc 660
ctgcagccgg aggacttcgc gacatactac tgtcagcagt catactccac ccctctgacc 720
ttcggccaag ggaccaaagt ggagatcaag accactaccc cagcaccgag gccacccacc 780
ccggctccta ccatcgcctc ccagcctctg tccctgcgtc cggaggcatg tagacccgca 840
gctggtgggg ccgtgcatac ccggggtctt gacttcgcct gcgatatcta catttgggcc 900
cctctggctg gtacttgcgg ggtcctgctg ctttcactcg tgatcactct ttactgtaag 960
cgcggtcgga agaagctgct gtacatcttt aagcaaccct tcatgaggcc tgtgcagact 1020
actcaagagg aggacggctg ttcatgccgg ttcccagagg aggaggaagg cggctgcgaa 1080
ctgcgcgtga aattcagccg cagcgcagat gctccagcct accagcaggg gcagaaccag 1140
ctctacaacg aactcaatct tggtcggaga gaggagtacg acgtgctgga caagcggaga 1200
ggacgggacc cagaaatggg cgggaagccg cgcagaaaga atccccaaga gggcctgtac 1260
aacgagctcc aaaaggataa gatggcagaa gcctatagcg agattggtat gaaaggggaa 1320
cgcagaagag gcaaaggcca cgacggactg taccagggac tcagcaccgc caccaaggac 1380
acctatgacg ctcttcacat gcaggccctg ccgcctcgg 1419
<210> 68
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 68
Arg Glu Trp Trp Tyr Asp Asp Trp Tyr Leu Asp Tyr
1 5 10
<210> 69
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 69
Ala Arg Arg Glu Trp Trp Tyr Asp Asp Trp Tyr Leu Asp Tyr
1 5 10
<210> 70
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 70
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Tyr Asp Asp Trp Tyr Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 71
<211> 363
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 71
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tggtggtacg acgattggta cctggactac tggggacagg gcactctcgt gactgtgtcc 360
tcc 363
<210> 72
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 72
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Tyr Asp Asp Trp Tyr Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Val Glu Ile Lys
245
<210> 73
<211> 744
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 73
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tggtggtacg acgattggta cctggactac tggggacagg gcactctcgt gactgtgtcc 360
tccggtggtg gtggatcggg gggtggtggt tcgggcggag gaggatctgg aggaggaggg 420
tcggacattc aaatgactca gtccccgtcc tccctctccg cctccgtggg agatcgcgtc 480
acgatcacgt gcagggccag ccagagcatc tccagctacc tgaactggta ccagcagaag 540
ccagggaagg caccgaagct cctgatctac gccgctagct cgctgcagtc cggcgtccct 600
tcacggttct cgggatcggg ctcaggcacc gacttcaccc tgaccattag cagcctgcag 660
ccggaggact tcgcgacata ctactgtcag cagtcatact ccacccctct gaccttcggc 720
caagggacca aagtggagat caag 744
<210> 74
<211> 471
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 74
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Tyr Asp Asp Trp Tyr Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro
245 250 255
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
260 265 270
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
275 280 285
Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
290 295 300
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
305 310 315 320
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
325 330 335
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
340 345 350
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
355 360 365
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
370 375 380
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
385 390 395 400
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
405 410 415
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
420 425 430
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
435 440 445
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
450 455 460
Met Gln Ala Leu Pro Pro Arg
465 470
<210> 75
<211> 1413
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 75
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tggtggtacg acgattggta cctggactac tggggacagg gcactctcgt gactgtgtcc 360
tccggtggtg gtggatcggg gggtggtggt tcgggcggag gaggatctgg aggaggaggg 420
tcggacattc aaatgactca gtccccgtcc tccctctccg cctccgtggg agatcgcgtc 480
acgatcacgt gcagggccag ccagagcatc tccagctacc tgaactggta ccagcagaag 540
ccagggaagg caccgaagct cctgatctac gccgctagct cgctgcagtc cggcgtccct 600
tcacggttct cgggatcggg ctcaggcacc gacttcaccc tgaccattag cagcctgcag 660
ccggaggact tcgcgacata ctactgtcag cagtcatact ccacccctct gaccttcggc 720
caagggacca aagtggagat caagaccact accccagcac cgaggccacc caccccggct 780
cctaccatcg cctcccagcc tctgtccctg cgtccggagg catgtagacc cgcagctggt 840
ggggccgtgc atacccgggg tcttgacttc gcctgcgata tctacatttg ggcccctctg 900
gctggtactt gcggggtcct gctgctttca ctcgtgatca ctctttactg taagcgcggt 960
cggaagaagc tgctgtacat ctttaagcaa cccttcatga ggcctgtgca gactactcaa 1020
gaggaggacg gctgttcatg ccggttccca gaggaggagg aaggcggctg cgaactgcgc 1080
gtgaaattca gccgcagcgc agatgctcca gcctaccagc aggggcagaa ccagctctac 1140
aacgaactca atcttggtcg gagagaggag tacgacgtgc tggacaagcg gagaggacgg 1200
gacccagaaa tgggcgggaa gccgcgcaga aagaatcccc aagagggcct gtacaacgag 1260
ctccaaaagg ataagatggc agaagcctat agcgagattg gtatgaaagg ggaacgcaga 1320
agaggcaaag gccacgacgg actgtaccag ggactcagca ccgccaccaa ggacacctat 1380
gacgctcttc acatgcaggc cctgccgcct cgg 1413
<210> 76
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 76
Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr
1 5 10
<210> 77
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 77
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr
1 5 10
<210> 78
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 78
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 79
<211> 363
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 79
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tggtggggag aaagctggct gttcgactac tggggacagg gcactctcgt gactgtgtcc 360
tcc 363
<210> 80
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 80
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Val Glu Ile Lys
245
<210> 81
<211> 744
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 81
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tggtggggag aaagctggct gttcgactac tggggacagg gcactctcgt gactgtgtcc 360
tccggtggtg gtggatcggg gggtggtggt tcgggcggag gaggatctgg aggaggaggg 420
tcggacattc aaatgactca gtccccgtcc tccctctccg cctccgtggg agatcgcgtc 480
acgatcacgt gcagggccag ccagagcatc tccagctacc tgaactggta ccagcagaag 540
ccagggaagg caccgaagct cctgatctac gccgctagct cgctgcagtc cggcgtccct 600
tcacggttct cgggatcggg ctcaggcacc gacttcaccc tgaccattag cagcctgcag 660
ccggaggact tcgcgacata ctactgtcag cagtcatact ccacccctct gaccttcggc 720
caagggacca aagtggagat caag 744
<210> 82
<211> 471
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 82
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro
245 250 255
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
260 265 270
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
275 280 285
Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
290 295 300
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
305 310 315 320
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
325 330 335
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
340 345 350
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
355 360 365
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
370 375 380
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
385 390 395 400
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
405 410 415
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
420 425 430
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
435 440 445
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
450 455 460
Met Gln Ala Leu Pro Pro Arg
465 470
<210> 83
<211> 1413
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 83
gaagtgcagt tgctggagtc aggcggagga ctggtgcagc ccggaggatc gcttcgcttg 60
agctgcgcag cctcaggctt taccttctcc tcctacgcca tgtcctgggt cagacaggct 120
cccgggaagg gactggaatg ggtgtccgcc attagcggtt ccggcggaag cacttactat 180
gccgactctg tgaagggccg cttcactatc tcccgggaca actccaagaa caccctgtat 240
ctccaaatga attccctgag ggccgaagat accgcggtgt actactgcgc tagacgggag 300
tggtggggag aaagctggct gttcgactac tggggacagg gcactctcgt gactgtgtcc 360
tccggtggtg gtggatcggg gggtggtggt tcgggcggag gaggatctgg aggaggaggg 420
tcggacattc aaatgactca gtccccgtcc tccctctccg cctccgtggg agatcgcgtc 480
acgatcacgt gcagggccag ccagagcatc tccagctacc tgaactggta ccagcagaag 540
ccagggaagg caccgaagct cctgatctac gccgctagct cgctgcagtc cggcgtccct 600
tcacggttct cgggatcggg ctcaggcacc gacttcaccc tgaccattag cagcctgcag 660
ccggaggact tcgcgacata ctactgtcag cagtcatact ccacccctct gaccttcggc 720
caagggacca aagtggagat caagaccact accccagcac cgaggccacc caccccggct 780
cctaccatcg cctcccagcc tctgtccctg cgtccggagg catgtagacc cgcagctggt 840
ggggccgtgc atacccgggg tcttgacttc gcctgcgata tctacatttg ggcccctctg 900
gctggtactt gcggggtcct gctgctttca ctcgtgatca ctctttactg taagcgcggt 960
cggaagaagc tgctgtacat ctttaagcaa cccttcatga ggcctgtgca gactactcaa 1020
gaggaggacg gctgttcatg ccggttccca gaggaggagg aaggcggctg cgaactgcgc 1080
gtgaaattca gccgcagcgc agatgctcca gcctaccagc aggggcagaa ccagctctac 1140
aacgaactca atcttggtcg gagagaggag tacgacgtgc tggacaagcg gagaggacgg 1200
gacccagaaa tgggcgggaa gccgcgcaga aagaatcccc aagagggcct gtacaacgag 1260
ctccaaaagg ataagatggc agaagcctat agcgagattg gtatgaaagg ggaacgcaga 1320
agaggcaaag gccacgacgg actgtaccag ggactcagca ccgccaccaa ggacacctat 1380
gacgctcttc acatgcaggc cctgccgcct cgg 1413
<210> 84
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace=" "
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace=" "
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="Y"
<220>
<221> VARIANT
<222> (7)..(7)
<223> /replace="Y" or "D"
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace="D" or "V"
<220>
<221> VARIANT
<222> (9)..(9)
<223> /replace="D"
<220>
<221> VARIANT
<222> (11)..(11)
<223> /replace="Y"
<220>
<221> VARIANT
<222> (12)..(12)
<223> /replace="L"
<220>
<221> SITE
<222> (1)..(14)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 84
Arg Glu Trp Val Pro Trp Gly Glu Ser Trp Leu Phe Asp Tyr
1 5 10
<210> 85
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace=" "
<220>
<221> VARIANT
<222> (7)..(7)
<223> /replace=" "
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace="Y"
<220>
<221> VARIANT
<222> (9)..(9)
<223> /replace="Y" or "D"
<220>
<221> VARIANT
<222> (10)..(10)
<223> /replace="D" or "V"
<220>
<221> VARIANT
<222> (11)..(11)
<223> /replace="D"
<220>
<221> VARIANT
<222> (13)..(13)
<223> /replace="Y"
<220>
<221> VARIANT
<222> (14)..(14)
<223> /replace="L"
<220>
<221> SITE
<222> (1)..(16)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 85
Ala Arg Arg Glu Trp Val Pro Trp Gly Glu Ser Trp Leu Phe Asp Tyr
1 5 10 15
<210> 86
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 86
Ser Tyr Gly Met His
1 5
<210> 87
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 87
Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 88
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 88
Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
1 5 10
<210> 89
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 89
Ser Tyr Asp Gly Ser Asn
1 5
<210> 90
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 90
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 91
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 91
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 92
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 92
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
1 5 10 15
<210> 93
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 93
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 94
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 94
caagtgcagc tgcaggaatc cggtggcgga gtcgtgcagc ctggaaggag cctgagactc 60
tcatgcgccg cgtcagggtt caccttttcc tcctacggga tgcattgggt cagacaggcc 120
cccggaaagg gactcgaatg ggtggctgtg atcagctacg acggctccaa caagtactac 180
gccgactccg tgaaaggccg gttcactatc tcccgggaca actccaagaa cacgctgtat 240
ctgcaaatga attcactgcg cgcggaggat accgctgtgt actactgcgg tggctccggt 300
tacgccctgc acgatgacta ttacggcctt gacgtctggg gccagggaac cctcgtgact 360
gtgtccagc 369
<210> 95
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 95
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 96
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 96
Asp Val Ser Asn Arg Pro Ser
1 5
<210> 97
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 97
Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr Val
1 5 10
<210> 98
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 98
Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr
1 5 10
<210> 99
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 99
Asp Val Ser
1
<210> 100
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 100
Tyr Thr Ser Ser Ser Thr Leu Tyr
1 5
<210> 101
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 101
Ser Ser Asp Val Gly Gly Tyr Asn Tyr
1 5
<210> 102
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 102
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 103
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 103
cagagcgcac tgactcagcc ggcatccgtg tccggtagcc ccggacagtc gattaccatc 60
tcctgtaccg gcacctcctc cgacgtggga gggtacaact acgtgtcgtg gtaccagcag 120
cacccaggaa aggcccctaa gttgatgatc tacgatgtgt caaaccgccc gtctggagtc 180
tccaaccggt tctccggctc caagtccggc aacaccgcca gcctgaccat tagcgggctg 240
caagccgagg atgaggccga ctactactgc tcgagctaca catcctcgag caccctctac 300
gtgttcggct cggggactaa ggtcaccgtg ctg 333
<210> 104
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 104
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 105
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 105
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln
130 135 140
Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile Ser Cys
145 150 155 160
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr
165 170 175
Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp Val Ser
180 185 190
Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys Ser Gly
195 200 205
Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala
210 215 220
Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr Val Phe
225 230 235 240
Gly Ser Gly Thr Lys Val Thr Val Leu
245
<210> 106
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 106
caagtgcagc tgcaggaatc cggtggcgga gtcgtgcagc ctggaaggag cctgagactc 60
tcatgcgccg cgtcagggtt caccttttcc tcctacggga tgcattgggt cagacaggcc 120
cccggaaagg gactcgaatg ggtggctgtg atcagctacg acggctccaa caagtactac 180
gccgactccg tgaaaggccg gttcactatc tcccgggaca actccaagaa cacgctgtat 240
ctgcaaatga attcactgcg cgcggaggat accgctgtgt actactgcgg tggctccggt 300
tacgccctgc acgatgacta ttacggcctt gacgtctggg gccagggaac cctcgtgact 360
gtgtccagcg gtggaggagg ttcgggcgga ggaggatcag gagggggtgg atcgcagagc 420
gcactgactc agccggcatc cgtgtccggt agccccggac agtcgattac catctcctgt 480
accggcacct cctccgacgt gggagggtac aactacgtgt cgtggtacca gcagcaccca 540
ggaaaggccc ctaagttgat gatctacgat gtgtcaaacc gcccgtctgg agtctccaac 600
cggttctccg gctccaagtc cggcaacacc gccagcctga ccattagcgg gctgcaagcc 660
gaggatgagg ccgactacta ctgctcgagc tacacatcct cgagcaccct ctacgtgttc 720
ggctcgggga ctaaggtcac cgtgctg 747
<210> 107
<211> 472
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 107
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln
130 135 140
Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile Ser Cys
145 150 155 160
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr
165 170 175
Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp Val Ser
180 185 190
Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys Ser Gly
195 200 205
Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala
210 215 220
Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr Val Phe
225 230 235 240
Gly Ser Gly Thr Lys Val Thr Val Leu Thr Thr Thr Pro Ala Pro Arg
245 250 255
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
260 265 270
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
275 280 285
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
290 295 300
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
305 310 315 320
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
325 330 335
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
340 345 350
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
355 360 365
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
370 375 380
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
385 390 395 400
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
405 410 415
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
420 425 430
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
435 440 445
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
450 455 460
His Met Gln Ala Leu Pro Pro Arg
465 470
<210> 108
<211> 1416
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 108
caagtgcagc tgcaggaatc cggtggcgga gtcgtgcagc ctggaaggag cctgagactc 60
tcatgcgccg cgtcagggtt caccttttcc tcctacggga tgcattgggt cagacaggcc 120
cccggaaagg gactcgaatg ggtggctgtg atcagctacg acggctccaa caagtactac 180
gccgactccg tgaaaggccg gttcactatc tcccgggaca actccaagaa cacgctgtat 240
ctgcaaatga attcactgcg cgcggaggat accgctgtgt actactgcgg tggctccggt 300
tacgccctgc acgatgacta ttacggcctt gacgtctggg gccagggaac cctcgtgact 360
gtgtccagcg gtggaggagg ttcgggcgga ggaggatcag gagggggtgg atcgcagagc 420
gcactgactc agccggcatc cgtgtccggt agccccggac agtcgattac catctcctgt 480
accggcacct cctccgacgt gggagggtac aactacgtgt cgtggtacca gcagcaccca 540
ggaaaggccc ctaagttgat gatctacgat gtgtcaaacc gcccgtctgg agtctccaac 600
cggttctccg gctccaagtc cggcaacacc gccagcctga ccattagcgg gctgcaagcc 660
gaggatgagg ccgactacta ctgctcgagc tacacatcct cgagcaccct ctacgtgttc 720
ggctcgggga ctaaggtcac cgtgctgacc actaccccag caccgaggcc acccaccccg 780
gctcctacca tcgcctccca gcctctgtcc ctgcgtccgg aggcatgtag acccgcagct 840
ggtggggccg tgcatacccg gggtcttgac ttcgcctgcg atatctacat ttgggcccct 900
ctggctggta cttgcggggt cctgctgctt tcactcgtga tcactcttta ctgtaagcgc 960
ggtcggaaga agctgctgta catctttaag caacccttca tgaggcctgt gcagactact 1020
caagaggagg acggctgttc atgccggttc ccagaggagg aggaaggcgg ctgcgaactg 1080
cgcgtgaaat tcagccgcag cgcagatgct ccagcctacc agcaggggca gaaccagctc 1140
tacaacgaac tcaatcttgg tcggagagag gagtacgacg tgctggacaa gcggagagga 1200
cgggacccag aaatgggcgg gaagccgcgc agaaagaatc cccaagaggg cctgtacaac 1260
gagctccaaa aggataagat ggcagaagcc tatagcgaga ttggtatgaa aggggaacgc 1320
agaagaggca aaggccacga cggactgtac cagggactca gcaccgccac caaggacacc 1380
tatgacgctc ttcacatgca ggccctgccg cctcgg 1416
<210> 109
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 109
Val Ile Ser Tyr Lys Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 110
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 110
Ser Tyr Lys Gly Ser Asn
1 5
<210> 111
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 111
Ile Ser Tyr Lys Gly Ser Asn Lys
1 5
<210> 112
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 112
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Lys Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 113
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 113
caagtgcagc ttgtcgaatc gggaggcgga gtggtgcagc ctggacgatc gctccggctc 60
tcatgtgccg cgagcggatt caccttctcg agctacggca tgcactgggt cagacaagcc 120
ccaggaaagg gcctggaatg ggtggctgtc atctcgtaca agggctcaaa caagtactac 180
gccgactccg tgaagggccg gttcaccatc tcccgcgata actccaagaa taccctctat 240
ctgcaaatga acagcctgag ggccgaggat actgcagtgt actactgcgg gggttcaggc 300
tacgcgctgc acgacgacta ctacggattg gacgtctggg gccaaggaac tcttgtgacc 360
gtgtcctct 369
<210> 114
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 114
Glu Val Ser Asn Arg Leu Arg
1 5
<210> 115
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 115
Ser Ser Tyr Thr Ser Ser Ser Ala Leu Tyr Val
1 5 10
<210> 116
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 116
Glu Val Ser
1
<210> 117
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 117
Tyr Thr Ser Ser Ser Ala Leu Tyr
1 5
<210> 118
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 118
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Leu Arg Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Ala Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 119
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 119
cagagcgcgc tgactcagcc tgcctccgtg agcggttcgc cgggacagtc cattaccatt 60
tcgtgcaccg ggacctcctc cgacgtggga ggctacaact acgtgtcctg gtaccagcag 120
catcccggaa aggccccgaa gctgatgatc tacgaagtgt cgaacagact gcggggagtc 180
tccaaccgct tttccgggtc caagtccggc aacaccgcca gcctgaccat cagcgggctc 240
caggcagaag atgaggctga ctattactgc tcctcctaca cgtcaagctc cgccctctac 300
gtgttcgggt ccgggaccaa agtcactgtg ctg 333
<210> 120
<211> 254
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 120
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Lys Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
145 150 155 160
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
165 170 175
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
180 185 190
Ile Tyr Glu Val Ser Asn Arg Leu Arg Gly Val Ser Asn Arg Phe Ser
195 200 205
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
210 215 220
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
225 230 235 240
Ala Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu
245 250
<210> 121
<211> 762
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 121
caagtgcagc ttgtcgaatc gggaggcgga gtggtgcagc ctggacgatc gctccggctc 60
tcatgtgccg cgagcggatt caccttctcg agctacggca tgcactgggt cagacaagcc 120
ccaggaaagg gcctggaatg ggtggctgtc atctcgtaca agggctcaaa caagtactac 180
gccgactccg tgaagggccg gttcaccatc tcccgcgata actccaagaa taccctctat 240
ctgcaaatga acagcctgag ggccgaggat actgcagtgt actactgcgg gggttcaggc 300
tacgcgctgc acgacgacta ctacggattg gacgtctggg gccaaggaac tcttgtgacc 360
gtgtcctctg gtggaggcgg atcagggggt ggcggatctg ggggtggtgg ttccggggga 420
ggaggatcgc agagcgcgct gactcagcct gcctccgtga gcggttcgcc gggacagtcc 480
attaccattt cgtgcaccgg gacctcctcc gacgtgggag gctacaacta cgtgtcctgg 540
taccagcagc atcccggaaa ggccccgaag ctgatgatct acgaagtgtc gaacagactg 600
cggggagtct ccaaccgctt ttccgggtcc aagtccggca acaccgccag cctgaccatc 660
agcgggctcc aggcagaaga tgaggctgac tattactgct cctcctacac gtcaagctcc 720
gccctctacg tgttcgggtc cgggaccaaa gtcactgtgc tg 762
<210> 122
<211> 477
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 122
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Lys Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
145 150 155 160
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
165 170 175
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
180 185 190
Ile Tyr Glu Val Ser Asn Arg Leu Arg Gly Val Ser Asn Arg Phe Ser
195 200 205
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
210 215 220
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
225 230 235 240
Ala Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Thr Thr
245 250 255
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
260 265 270
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
275 280 285
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala
290 295 300
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
305 310 315 320
Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
325 330 335
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
340 345 350
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
355 360 365
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
370 375 380
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
385 390 395 400
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
405 410 415
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
420 425 430
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
435 440 445
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
450 455 460
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
465 470 475
<210> 123
<211> 1431
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 123
caagtgcagc ttgtcgaatc gggaggcgga gtggtgcagc ctggacgatc gctccggctc 60
tcatgtgccg cgagcggatt caccttctcg agctacggca tgcactgggt cagacaagcc 120
ccaggaaagg gcctggaatg ggtggctgtc atctcgtaca agggctcaaa caagtactac 180
gccgactccg tgaagggccg gttcaccatc tcccgcgata actccaagaa taccctctat 240
ctgcaaatga acagcctgag ggccgaggat actgcagtgt actactgcgg gggttcaggc 300
tacgcgctgc acgacgacta ctacggattg gacgtctggg gccaaggaac tcttgtgacc 360
gtgtcctctg gtggaggcgg atcagggggt ggcggatctg ggggtggtgg ttccggggga 420
ggaggatcgc agagcgcgct gactcagcct gcctccgtga gcggttcgcc gggacagtcc 480
attaccattt cgtgcaccgg gacctcctcc gacgtgggag gctacaacta cgtgtcctgg 540
taccagcagc atcccggaaa ggccccgaag ctgatgatct acgaagtgtc gaacagactg 600
cggggagtct ccaaccgctt ttccgggtcc aagtccggca acaccgccag cctgaccatc 660
agcgggctcc aggcagaaga tgaggctgac tattactgct cctcctacac gtcaagctcc 720
gccctctacg tgttcgggtc cgggaccaaa gtcactgtgc tgaccactac cccagcaccg 780
aggccaccca ccccggctcc taccatcgcc tcccagcctc tgtccctgcg tccggaggca 840
tgtagacccg cagctggtgg ggccgtgcat acccggggtc ttgacttcgc ctgcgatatc 900
tacatttggg cccctctggc tggtacttgc ggggtcctgc tgctttcact cgtgatcact 960
ctttactgta agcgcggtcg gaagaagctg ctgtacatct ttaagcaacc cttcatgagg 1020
cctgtgcaga ctactcaaga ggaggacggc tgttcatgcc ggttcccaga ggaggaggaa 1080
ggcggctgcg aactgcgcgt gaaattcagc cgcagcgcag atgctccagc ctaccagcag 1140
gggcagaacc agctctacaa cgaactcaat cttggtcgga gagaggagta cgacgtgctg 1200
gacaagcgga gaggacggga cccagaaatg ggcgggaagc cgcgcagaaa gaatccccaa 1260
gagggcctgt acaacgagct ccaaaaggat aagatggcag aagcctatag cgagattggt 1320
atgaaagggg aacgcagaag aggcaaaggc cacgacggac tgtaccaggg actcagcacc 1380
gccaccaagg acacctatga cgctcttcac atgcaggccc tgccgcctcg g 1431
<210> 124
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 124
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Glu Val Ser Asn Arg Leu Arg Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 125
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 125
cagagcgcgc tgactcagcc tgcctccgtg agcggttcgc cgggacagtc cattaccatt 60
tcgtgcaccg ggacctcctc cgacgtggga ggctacaact acgtgtcctg gtaccagcag 120
catcccggaa aggccccgaa gctgatgatc tacgaagtgt cgaacagact gcggggagtc 180
tccaaccgct tttccgggtc caagtccggc aacaccgcca gcctgaccat cagcgggctc 240
caggcagaag atgaggctga ctattactgc tcctcctaca cgtcaagctc caccctctac 300
gtgttcgggt ccgggaccaa agtcactgtg ctg 333
<210> 126
<211> 254
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 126
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Lys Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
145 150 155 160
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
165 170 175
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
180 185 190
Ile Tyr Glu Val Ser Asn Arg Leu Arg Gly Val Ser Asn Arg Phe Ser
195 200 205
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
210 215 220
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
225 230 235 240
Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu
245 250
<210> 127
<211> 762
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 127
caagtgcagc ttgtcgaatc gggaggcgga gtggtgcagc ctggacgatc gctccggctc 60
tcatgtgccg cgagcggatt caccttctcg agctacggca tgcactgggt cagacaagcc 120
ccaggaaagg gcctggaatg ggtggctgtc atctcgtaca agggctcaaa caagtactac 180
gccgactccg tgaagggccg gttcaccatc tcccgcgata actccaagaa taccctctat 240
ctgcaaatga acagcctgag ggccgaggat actgcagtgt actactgcgg gggttcaggc 300
tacgcgctgc acgacgacta ctacggattg gacgtctggg gccaaggaac tcttgtgacc 360
gtgtcctctg gtggaggcgg atcagggggt ggcggatctg ggggtggtgg ttccggggga 420
ggaggatcgc agagcgcgct gactcagcct gcctccgtga gcggttcgcc gggacagtcc 480
attaccattt cgtgcaccgg gacctcctcc gacgtgggag gctacaacta cgtgtcctgg 540
taccagcagc atcccggaaa ggccccgaag ctgatgatct acgaagtgtc gaacagactg 600
cggggagtct ccaaccgctt ttccgggtcc aagtccggca acaccgccag cctgaccatc 660
agcgggctcc aggcagaaga tgaggctgac tattactgct cctcctacac gtcaagctcc 720
accctctacg tgttcgggtc cgggaccaaa gtcactgtgc tg 762
<210> 128
<211> 477
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 128
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Lys Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
145 150 155 160
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
165 170 175
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
180 185 190
Ile Tyr Glu Val Ser Asn Arg Leu Arg Gly Val Ser Asn Arg Phe Ser
195 200 205
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
210 215 220
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
225 230 235 240
Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Thr Thr
245 250 255
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
260 265 270
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
275 280 285
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala
290 295 300
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
305 310 315 320
Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
325 330 335
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
340 345 350
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
355 360 365
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
370 375 380
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
385 390 395 400
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
405 410 415
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
420 425 430
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
435 440 445
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
450 455 460
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
465 470 475
<210> 129
<211> 1431
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 129
caagtgcagc ttgtcgaatc gggaggcgga gtggtgcagc ctggacgatc gctccggctc 60
tcatgtgccg cgagcggatt caccttctcg agctacggca tgcactgggt cagacaagcc 120
ccaggaaagg gcctggaatg ggtggctgtc atctcgtaca agggctcaaa caagtactac 180
gccgactccg tgaagggccg gttcaccatc tcccgcgata actccaagaa taccctctat 240
ctgcaaatga acagcctgag ggccgaggat actgcagtgt actactgcgg gggttcaggc 300
tacgcgctgc acgacgacta ctacggattg gacgtctggg gccaaggaac tcttgtgacc 360
gtgtcctctg gtggaggcgg atcagggggt ggcggatctg ggggtggtgg ttccggggga 420
ggaggatcgc agagcgcgct gactcagcct gcctccgtga gcggttcgcc gggacagtcc 480
attaccattt cgtgcaccgg gacctcctcc gacgtgggag gctacaacta cgtgtcctgg 540
taccagcagc atcccggaaa ggccccgaag ctgatgatct acgaagtgtc gaacagactg 600
cggggagtct ccaaccgctt ttccgggtcc aagtccggca acaccgccag cctgaccatc 660
agcgggctcc aggcagaaga tgaggctgac tattactgct cctcctacac gtcaagctcc 720
accctctacg tgttcgggtc cgggaccaaa gtcactgtgc tgaccactac cccagcaccg 780
aggccaccca ccccggctcc taccatcgcc tcccagcctc tgtccctgcg tccggaggca 840
tgtagacccg cagctggtgg ggccgtgcat acccggggtc ttgacttcgc ctgcgatatc 900
tacatttggg cccctctggc tggtacttgc ggggtcctgc tgctttcact cgtgatcact 960
ctttactgta agcgcggtcg gaagaagctg ctgtacatct ttaagcaacc cttcatgagg 1020
cctgtgcaga ctactcaaga ggaggacggc tgttcatgcc ggttcccaga ggaggaggaa 1080
ggcggctgcg aactgcgcgt gaaattcagc cgcagcgcag atgctccagc ctaccagcag 1140
gggcagaacc agctctacaa cgaactcaat cttggtcgga gagaggagta cgacgtgctg 1200
gacaagcgga gaggacggga cccagaaatg ggcgggaagc cgcgcagaaa gaatccccaa 1260
gagggcctgt acaacgagct ccaaaaggat aagatggcag aagcctatag cgagattggt 1320
atgaaagggg aacgcagaag aggcaaaggc cacgacggac tgtaccaggg actcagcacc 1380
gccaccaagg acacctatga cgctcttcac atgcaggccc tgccgcctcg g 1431
<210> 130
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace="K"
<220>
<221> SITE
<222> (1)..(17)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 130
Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 131
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="E"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="L"
<220>
<221> VARIANT
<222> (7)..(7)
<223> /replace="R"
<220>
<221> SITE
<222> (1)..(7)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 131
Asp Val Ser Asn Arg Pro Ser
1 5
<210> 132
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace="A"
<220>
<221> SITE
<222> (1)..(11)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 132
Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr Val
1 5 10
<210> 133
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="K"
<220>
<221> SITE
<222> (1)..(6)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 133
Ser Tyr Asp Gly Ser Asn
1 5
<210> 134
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="E"
<220>
<221> SITE
<222> (1)..(3)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 134
Asp Val Ser
1
<210> 135
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="A"
<220>
<221> SITE
<222> (1)..(8)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 135
Tyr Thr Ser Ser Ser Thr Leu Tyr
1 5
<210> 136
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="K"
<220>
<221> SITE
<222> (1)..(8)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 136
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 137
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 137
Gly Phe Trp Met Ser
1 5
<210> 138
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 138
Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val Arg
1 5 10 15
Gly
<210> 139
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 139
Ala Leu Asp Tyr Tyr Gly Met Asp Val
1 5
<210> 140
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 140
Gly Phe Thr Phe Ser Gly Phe
1 5
<210> 141
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 141
Lys Gln Asp Gly Ser Glu
1 5
<210> 142
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 142
Gly Phe Thr Phe Ser Gly Phe Trp
1 5
<210> 143
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 143
Ile Lys Gln Asp Gly Ser Glu Lys
1 5
<210> 144
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 144
Ala Arg Ala Leu Asp Tyr Tyr Gly Met Asp Val
1 5 10
<210> 145
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 145
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly Phe
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Leu Asp Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 146
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 146
gaagtgcaac tggtggagag cggtggaggg cttgtccagc ccggaggatc gctgcggctg 60
tcctgtgctg cgtccgggtt caccttctcc ggcttctgga tgtcctgggt cagacaggca 120
ccgggaaagg gcctcgaatg ggtggccaac atcaagcagg atggctccga gaagtactac 180
gtcgactccg tgagaggccg cttcaccatc tcccgggaca acgccaagaa ctcgctgtac 240
ctccaaatga atagcctcag ggcggaagat actgctgtgt attactgcgc acgcgccctt 300
gactactacg gcatggacgt ctggggccaa gggaccactg tgaccgtgtc tagc 354
<210> 147
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 147
Arg Ser Ser Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr Leu
1 5 10 15
Asp
<210> 148
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 148
Thr Leu Ser Tyr Arg Ala Ser
1 5
<210> 149
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 149
Thr Gln Arg Leu Glu Phe Pro Ser Ile Thr
1 5 10
<210> 150
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 150
Ser Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 151
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 151
Thr Leu Ser
1
<210> 152
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 152
Arg Leu Glu Phe Pro Ser Ile
1 5
<210> 153
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 153
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 154
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 154
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Arg Leu Leu Ile Tyr Thr Leu Ser Tyr Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Leu Tyr Tyr Cys Thr Gln
85 90 95
Arg Leu Glu Phe Pro Ser Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu
100 105 110
Ile Lys
<210> 155
<211> 342
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 155
gatatcgtga tgacccagac tcccctgtcc ctgcctgtga ctcccggaga accagcctcc 60
atttcctgcc ggtcctccca gtccctgctg gacagcgacg acggcaacac ttacctggac 120
tggtacttgc agaagccggg ccaatcgcct cgcctgctga tctataccct gtcataccgg 180
gcctcaggag tgcctgaccg cttctcggga tcagggagcg ggaccgattt caccctgaaa 240
atttcccgag tggaagccga ggacgtcgga ctgtactact gcacccagcg cctcgaattc 300
ccgtcgatta cgtttggaca gggtacccgg cttgagatca ag 342
<210> 156
<211> 252
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 156
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly Phe
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Leu Asp Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln
130 135 140
Thr Pro Leu Ser Leu Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser
145 150 155 160
Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
165 170 175
Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Arg Leu Leu Ile
180 185 190
Tyr Thr Leu Ser Tyr Arg Ala Ser Gly Val Pro Asp Arg Phe Ser Gly
195 200 205
Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala
210 215 220
Glu Asp Val Gly Leu Tyr Tyr Cys Thr Gln Arg Leu Glu Phe Pro Ser
225 230 235 240
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
245 250
<210> 157
<211> 756
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 157
gaagtgcaac tggtggagag cggtggaggg cttgtccagc ccggaggatc gctgcggctg 60
tcctgtgctg cgtccgggtt caccttctcc ggcttctgga tgtcctgggt cagacaggca 120
ccgggaaagg gcctcgaatg ggtggccaac atcaagcagg atggctccga gaagtactac 180
gtcgactccg tgagaggccg cttcaccatc tcccgggaca acgccaagaa ctcgctgtac 240
ctccaaatga atagcctcag ggcggaagat actgctgtgt attactgcgc acgcgccctt 300
gactactacg gcatggacgt ctggggccaa gggaccactg tgaccgtgtc tagcggaggc 360
ggaggttcag ggggcggtgg atcaggcgga ggaggatcgg ggggtggtgg atcggatatc 420
gtgatgaccc agactcccct gtccctgcct gtgactcccg gagaaccagc ctccatttcc 480
tgccggtcct cccagtccct gctggacagc gacgacggca acacttacct ggactggtac 540
ttgcagaagc cgggccaatc gcctcgcctg ctgatctata ccctgtcata ccgggcctca 600
ggagtgcctg accgcttctc gggatcaggg agcgggaccg atttcaccct gaaaatttcc 660
cgagtggaag ccgaggacgt cggactgtac tactgcaccc agcgcctcga attcccgtcg 720
attacgtttg gacagggtac ccggcttgag atcaag 756
<210> 158
<211> 475
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 158
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly Phe
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Leu Asp Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln
130 135 140
Thr Pro Leu Ser Leu Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser
145 150 155 160
Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
165 170 175
Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Arg Leu Leu Ile
180 185 190
Tyr Thr Leu Ser Tyr Arg Ala Ser Gly Val Pro Asp Arg Phe Ser Gly
195 200 205
Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala
210 215 220
Glu Asp Val Gly Leu Tyr Tyr Cys Thr Gln Arg Leu Glu Phe Pro Ser
225 230 235 240
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Thr Thr Thr Pro
245 250 255
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
260 265 270
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
275 280 285
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
290 295 300
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
305 310 315 320
Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
325 330 335
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
340 345 350
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser
355 360 365
Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr
370 375 380
Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys
385 390 395 400
Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn
405 410 415
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
420 425 430
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
435 440 445
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
450 455 460
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
465 470 475
<210> 159
<211> 1425
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 159
gaagtgcaac tggtggagag cggtggaggg cttgtccagc ccggaggatc gctgcggctg 60
tcctgtgctg cgtccgggtt caccttctcc ggcttctgga tgtcctgggt cagacaggca 120
ccgggaaagg gcctcgaatg ggtggccaac atcaagcagg atggctccga gaagtactac 180
gtcgactccg tgagaggccg cttcaccatc tcccgggaca acgccaagaa ctcgctgtac 240
ctccaaatga atagcctcag ggcggaagat actgctgtgt attactgcgc acgcgccctt 300
gactactacg gcatggacgt ctggggccaa gggaccactg tgaccgtgtc tagcggaggc 360
ggaggttcag ggggcggtgg atcaggcgga ggaggatcgg ggggtggtgg atcggatatc 420
gtgatgaccc agactcccct gtccctgcct gtgactcccg gagaaccagc ctccatttcc 480
tgccggtcct cccagtccct gctggacagc gacgacggca acacttacct ggactggtac 540
ttgcagaagc cgggccaatc gcctcgcctg ctgatctata ccctgtcata ccgggcctca 600
ggagtgcctg accgcttctc gggatcaggg agcgggaccg atttcaccct gaaaatttcc 660
cgagtggaag ccgaggacgt cggactgtac tactgcaccc agcgcctcga attcccgtcg 720
attacgtttg gacagggtac ccggcttgag atcaagacca ctaccccagc accgaggcca 780
cccaccccgg ctcctaccat cgcctcccag cctctgtccc tgcgtccgga ggcatgtaga 840
cccgcagctg gtggggccgt gcatacccgg ggtcttgact tcgcctgcga tatctacatt 900
tgggcccctc tggctggtac ttgcggggtc ctgctgcttt cactcgtgat cactctttac 960
tgtaagcgcg gtcggaagaa gctgctgtac atctttaagc aacccttcat gaggcctgtg 1020
cagactactc aagaggagga cggctgttca tgccggttcc cagaggagga ggaaggcggc 1080
tgcgaactgc gcgtgaaatt cagccgcagc gcagatgctc cagcctacca gcaggggcag 1140
aaccagctct acaacgaact caatcttggt cggagagagg agtacgacgt gctggacaag 1200
cggagaggac gggacccaga aatgggcggg aagccgcgca gaaagaatcc ccaagagggc 1260
ctgtacaacg agctccaaaa ggataagatg gcagaagcct atagcgagat tggtatgaaa 1320
ggggaacgca gaagaggcaa aggccacgac ggactgtacc agggactcag caccgccacc 1380
aaggacacct atgacgctct tcacatgcag gccctgccgc ctcgg 1425
<210> 160
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 160
Ser Phe Arg Met Asn
1 5
<210> 161
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 161
Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 162
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 162
Trp Leu Ser Tyr Tyr Gly Met Asp Val
1 5
<210> 163
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 163
Gly Phe Thr Phe Ser Ser Phe
1 5
<210> 164
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 164
Ser Ser Ser Ser Ser Tyr
1 5
<210> 165
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 165
Gly Phe Thr Phe Ser Ser Phe Arg
1 5
<210> 166
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 166
Ile Ser Ser Ser Ser Ser Tyr Ile
1 5
<210> 167
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 167
Ala Arg Trp Leu Ser Tyr Tyr Gly Met Asp Val
1 5 10
<210> 168
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 168
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Arg Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Trp Leu Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 169
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 169
gaagtgcaac tggtggagag cggtggaggg cttgtcaagc ccggaggatc gctgcggctg 60
tcctgtgctg cgtccgggtt caccttctcc tcgttccgca tgaactgggt cagacaggca 120
ccgggaaagg gcctcgaatg ggtgtcctca atctcatcgt cctcgtccta catctactac 180
gccgactccg tgaaaggccg cttcaccatc tcccgggaca acgccaagaa ctcgctgtac 240
ctccaaatga atagcctcag ggcggaagat actgctgtgt attactgcgc acgctggctt 300
tcctactacg gcatggacgt ctggggccaa gggaccactg tgaccgtgtc tagc 354
<210> 170
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 170
Thr Leu Ser Phe Arg Ala Ser
1 5
<210> 171
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 171
Met Gln Arg Ile Gly Phe Pro Ile Thr
1 5
<210> 172
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 172
Arg Ile Gly Phe Pro Ile
1 5
<210> 173
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 173
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile Tyr Thr Leu Ser Phe Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Arg Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln
85 90 95
Arg Ile Gly Phe Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile
100 105 110
Lys
<210> 174
<211> 339
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 174
gatatcgtga tgacccagac tcccctgtcc ctgcctgtga ctcccggaga accagcctcc 60
atttcctgcc ggtcctccca gtccctgctg gacagcgacg acggcaacac ttacctggac 120
tggtacttgc agaagccggg ccaatcgcct cagctgctga tctataccct gtcattccgg 180
gcctcaggag tgcctgaccg cttctcggga tcagggagcg ggaccgattt caccctgaaa 240
attaggcgag tggaagccga ggacgtcgga gtgtactact gcatgcagcg catcggcttc 300
ccgattacgt ttggacaggg tacccggctt gagatcaag 339
<210> 175
<211> 251
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 175
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Arg Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Trp Leu Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln
130 135 140
Thr Pro Leu Ser Leu Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser
145 150 155 160
Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
165 170 175
Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile
180 185 190
Tyr Thr Leu Ser Phe Arg Ala Ser Gly Val Pro Asp Arg Phe Ser Gly
195 200 205
Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Arg Arg Val Glu Ala
210 215 220
Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Arg Ile Gly Phe Pro Ile
225 230 235 240
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
245 250
<210> 176
<211> 753
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 176
gaagtgcaac tggtggagag cggtggaggg cttgtcaagc ccggaggatc gctgcggctg 60
tcctgtgctg cgtccgggtt caccttctcc tcgttccgca tgaactgggt cagacaggca 120
ccgggaaagg gcctcgaatg ggtgtcctca atctcatcgt cctcgtccta catctactac 180
gccgactccg tgaaaggccg cttcaccatc tcccgggaca acgccaagaa ctcgctgtac 240
ctccaaatga atagcctcag ggcggaagat actgctgtgt attactgcgc acgctggctt 300
tcctactacg gcatggacgt ctggggccaa gggaccactg tgaccgtgtc tagcggaggc 360
ggaggttcag ggggcggtgg atcaggcgga ggaggatcgg ggggtggtgg atcggatatc 420
gtgatgaccc agactcccct gtccctgcct gtgactcccg gagaaccagc ctccatttcc 480
tgccggtcct cccagtccct gctggacagc gacgacggca acacttacct ggactggtac 540
ttgcagaagc cgggccaatc gcctcagctg ctgatctata ccctgtcatt ccgggcctca 600
ggagtgcctg accgcttctc gggatcaggg agcgggaccg atttcaccct gaaaattagg 660
cgagtggaag ccgaggacgt cggagtgtac tactgcatgc agcgcatcgg cttcccgatt 720
acgtttggac agggtacccg gcttgagatc aag 753
<210> 177
<211> 474
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 177
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Phe
20 25 30
Arg Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Ser Ser Tyr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Trp Leu Ser Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Thr Gln
130 135 140
Thr Pro Leu Ser Leu Pro Val Thr Pro Gly Glu Pro Ala Ser Ile Ser
145 150 155 160
Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
165 170 175
Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile
180 185 190
Tyr Thr Leu Ser Phe Arg Ala Ser Gly Val Pro Asp Arg Phe Ser Gly
195 200 205
Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Arg Arg Val Glu Ala
210 215 220
Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Arg Ile Gly Phe Pro Ile
225 230 235 240
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Thr Thr Thr Pro Ala
245 250 255
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
260 265 270
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
275 280 285
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
290 295 300
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
305 310 315 320
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
325 330 335
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
340 345 350
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
355 360 365
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
370 375 380
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
385 390 395 400
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
405 410 415
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
420 425 430
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
435 440 445
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
450 455 460
Ala Leu His Met Gln Ala Leu Pro Pro Arg
465 470
<210> 178
<211> 1422
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 178
gaagtgcaac tggtggagag cggtggaggg cttgtcaagc ccggaggatc gctgcggctg 60
tcctgtgctg cgtccgggtt caccttctcc tcgttccgca tgaactgggt cagacaggca 120
ccgggaaagg gcctcgaatg ggtgtcctca atctcatcgt cctcgtccta catctactac 180
gccgactccg tgaaaggccg cttcaccatc tcccgggaca acgccaagaa ctcgctgtac 240
ctccaaatga atagcctcag ggcggaagat actgctgtgt attactgcgc acgctggctt 300
tcctactacg gcatggacgt ctggggccaa gggaccactg tgaccgtgtc tagcggaggc 360
ggaggttcag ggggcggtgg atcaggcgga ggaggatcgg ggggtggtgg atcggatatc 420
gtgatgaccc agactcccct gtccctgcct gtgactcccg gagaaccagc ctccatttcc 480
tgccggtcct cccagtccct gctggacagc gacgacggca acacttacct ggactggtac 540
ttgcagaagc cgggccaatc gcctcagctg ctgatctata ccctgtcatt ccgggcctca 600
ggagtgcctg accgcttctc gggatcaggg agcgggaccg atttcaccct gaaaattagg 660
cgagtggaag ccgaggacgt cggagtgtac tactgcatgc agcgcatcgg cttcccgatt 720
acgtttggac agggtacccg gcttgagatc aagaccacta ccccagcacc gaggccaccc 780
accccggctc ctaccatcgc ctcccagcct ctgtccctgc gtccggaggc atgtagaccc 840
gcagctggtg gggccgtgca tacccggggt cttgacttcg cctgcgatat ctacatttgg 900
gcccctctgg ctggtacttg cggggtcctg ctgctttcac tcgtgatcac tctttactgt 960
aagcgcggtc ggaagaagct gctgtacatc tttaagcaac ccttcatgag gcctgtgcag 1020
actactcaag aggaggacgg ctgttcatgc cggttcccag aggaggagga aggcggctgc 1080
gaactgcgcg tgaaattcag ccgcagcgca gatgctccag cctaccagca ggggcagaac 1140
cagctctaca acgaactcaa tcttggtcgg agagaggagt acgacgtgct ggacaagcgg 1200
agaggacggg acccagaaat gggcgggaag ccgcgcagaa agaatcccca agagggcctg 1260
tacaacgagc tccaaaagga taagatggca gaagcctata gcgagattgg tatgaaaggg 1320
gaacgcagaa gaggcaaagg ccacgacgga ctgtaccagg gactcagcac cgccaccaag 1380
gacacctatg acgctcttca catgcaggcc ctgccgcctc gg 1422
<210> 179
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="S"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="R"
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace="N"
<220>
<221> SITE
<222> (1)..(5)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 179
Gly Phe Trp Met Ser
1 5
<210> 180
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="S"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="S"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="S"
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace="S"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="S"
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace="Y"
<220>
<221> VARIANT
<222> (9)..(9)
<223> /replace="I"
<220>
<221> VARIANT
<222> (12)..(12)
<223> /replace="A"
<220>
<221> VARIANT
<222> (16)..(16)
<223> /replace="K"
<220>
<221> SITE
<222> (1)..(17)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 180
Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val Arg
1 5 10 15
Gly
<210> 181
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="W"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="S"
<220>
<221> SITE
<222> (1)..(9)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 181
Ala Leu Asp Tyr Tyr Gly Met Asp Val
1 5
<210> 182
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="F"
<220>
<221> SITE
<222> (1)..(7)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 182
Thr Leu Ser Tyr Arg Ala Ser
1 5
<210> 183
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="M"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="I"
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace="G"
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace=" "
<220>
<221> SITE
<222> (1)..(10)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 183
Thr Gln Arg Leu Glu Phe Pro Ser Ile Thr
1 5 10
<210> 184
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="S"
<220>
<221> SITE
<222> (1)..(7)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 184
Gly Phe Thr Phe Ser Gly Phe
1 5
<210> 185
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (1)..(1)
<223> /replace="S"
<220>
<221> VARIANT
<222> (2)..(2)
<223> /replace="S"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> /replace="S"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="S"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="Y"
<220>
<221> SITE
<222> (1)..(6)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 185
Lys Gln Asp Gly Ser Glu
1 5
<210> 186
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (2)..(2)
<223> /replace="I"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="G"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace=" "
<220>
<221> SITE
<222> (1)..(7)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 186
Arg Leu Glu Phe Pro Ser Ile
1 5
<210> 187
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (6)..(6)
<223> /replace="S"
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace="R"
<220>
<221> SITE
<222> (1)..(8)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 187
Gly Phe Thr Phe Ser Gly Phe Trp
1 5
<210> 188
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (2)..(2)
<223> /replace="K"
<220>
<221> MOD_RES
<222> (3)..(3)
<223> /replace="S"
<220>
<221> VARIANT
<222> (4)..(4)
<223> /replace="S"
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace="S"
<220>
<221> VARIANT
<222> (7)..(7)
<223> /replace="Y"
<220>
<221> VARIANT
<222> (8)..(8)
<223> /replace="I"
<220>
<221> SITE
<222> (1)..(8)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 188
Ile Lys Gln Asp Gly Ser Glu Lys
1 5
<210> 189
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<220>
<221> VARIANT
<222> (3)..(3)
<223> /replace="W"
<220>
<221> VARIANT
<222> (5)..(5)
<223> /replace="S"
<220>
<221> SITE
<222> (1)..(11)
<223> /note="Variant residues given in the sequence have no
preference with respect to those in the annotations
for variant positions"
<400> 189
Ala Arg Ala Leu Asp Tyr Tyr Gly Met Asp Val
1 5 10
<210> 190
<211> 521
<212> DNA
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
PGK Promoter sequence"
<400> 190
acccctctct ccagccacta agccagttgc tccctcggct gacggctgca cgcgaggcct 60
ccgaacgtct tacgccttgt ggcgcgcccg tccttgtccc gggtgtgatg gcggggtgtg 120
gggcggaggg cgtggcgggg aagggccggc gacgagagcc gcgcgggacg actcgtcggc 180
gataaccggt gtcgggtagc gccagccgcg cgacggtaac gagggaccgc gacaggcaga 240
cgctcccatg atcactctgc acgccgaagg caaatagtgc aggccgtgcg gcgcttggcg 300
ttccttggaa gggctgaatc cccgcctcgt ccttcgcagc ggccccccgg gtgttcccat 360
cgccgcttct aggcccactg cgacgcttgc ctgcacttct tacacgctct gggtcccagc 420
cgcggcgacg caaagggcct tggtgcgggt ctcgtcggcg cagggacgcg tttgggtccc 480
gacggaacct tttccgcgtt ggggttgggg caccataagc t 521
<210> 191
<211> 221
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 191
acccctctct ccagccacta agccagttgc tccctcggct gacggctgca cgcgaggcct 60
ccgaacgtct tacgccttgt ggcgcgcccg tccttgtccc gggtgtgatg gcggggtgtg 120
gggcggaggg cgtggcgggg aagggccggc gacgagagcc gcgcgggacg actcgtcggc 180
gataaccggt gtcgggtagc gccagccgcg cgacggtaac g 221
<210> 192
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 192
acccctctct ccagccacta agccagttgc tccctcggct gacggctgca cgcgaggcct 60
ccgaacgtct tacgccttgt ggcgcgcccg tccttgtccc gggtgtgatg gcggggtgtg 120
gggcggaggg cgtggcgggg aagggccggc gacgagagcc gcgcgggacg actcgtcggc 180
gataaccggt gtcgggtagc gccagccgcg cgacggtaac gagggaccgc gacaggcaga 240
cgctcccatg atcactctgc acgccgaagg caaatagtgc aggccgtgcg gcgcttggcg 300
ttccttggaa gggctgaatc cccg 324
<210> 193
<211> 422
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 193
acccctctct ccagccacta agccagttgc tccctcggct gacggctgca cgcgaggcct 60
ccgaacgtct tacgccttgt ggcgcgcccg tccttgtccc gggtgtgatg gcggggtgtg 120
gggcggaggg cgtggcgggg aagggccggc gacgagagcc gcgcgggacg actcgtcggc 180
gataaccggt gtcgggtagc gccagccgcg cgacggtaac gagggaccgc gacaggcaga 240
cgctcccatg atcactctgc acgccgaagg caaatagtgc aggccgtgcg gcgcttggcg 300
ttccttggaa gggctgaatc cccgcctcgt ccttcgcagc ggccccccgg gtgttcccat 360
cgccgcttct aggcccactg cgacgcttgc ctgcacttct tacacgctct gggtcccagc 420
cg 422
<210> 194
<400> 194
000
<210> 195
<400> 195
000
<210> 196
<400> 196
000
<210> 197
<400> 197
000
<210> 198
<211> 118
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 198
acccctctct ccagccacta agccagttgc tccctcggct gacggctgca cgcgaggcct 60
ccgaacgtct tacgccttgt ggcgcgcccg tccttgtccc gggtgtgatg gcggggtg 118
<210> 199
<211> 63
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 199
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
ccc 63
<210> 200
<211> 239
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 200
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Ala Leu Ser Asn His
20 25 30
Gly Met Ser Trp Val Arg Arg Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr Ala Ala Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Arg Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ser
85 90 95
Ala His Gly Gly Glu Ser Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser Ala Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser
130 135 140
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
145 150 155 160
Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
165 170 175
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
180 185 190
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
195 200 205
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr
210 215 220
Ser Thr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
225 230 235
<210> 201
<211> 717
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 201
gaagtgcaat tggtggaatc agggggagga cttgtgcagc ctggaggatc gctgagactg 60
tcatgtgccg tgtccggctt tgccctgtcc aaccacggga tgtcctgggt ccgccgcgcg 120
cctggaaagg gcctcgaatg ggtgtcgggt attgtgtaca gcggtagcac ctactatgcc 180
gcatccgtga aggggagatt caccatcagc cgggacaact ccaggaacac tctgtacctc 240
caaatgaatt cgctgaggcc agaggacact gccatctact actgctccgc gcatggcgga 300
gagtccgacg tctggggaca ggggaccacc gtgaccgtgt ctagcgcgtc cggcggaggc 360
ggcagcgggg gtggtggttc agggggcggc ggatcggaca tccagctcac ccagtccccg 420
agctcgctgt ccgcctccgt gggagatcgg gtcaccatca cgtgccgcgc cagccagtcg 480
atttcctcct acctgaactg gtaccaacag aagcccggaa aagccccgaa gcttctcatc 540
tacgccgcct cgagcctgca gtcaggagtg ccctcacggt tctccggctc cggttccggt 600
actgatttca ccctgaccat ttcctccctg caaccggagg acttcgctac ttactactgc 660
cagcagtcgt actccacccc ctacactttc ggacaaggca ccaaggtcga aatcaag 717
<210> 202
<211> 69
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 202
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile
35 40 45
Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val
50 55 60
Ile Thr Leu Tyr Cys
65
<210> 203
<211> 207
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 203
accactaccc cagcaccgag gccacccacc ccggctccta ccatcgcctc ccagcctctg 60
tccctgcgtc cggaggcatg tagacccgca gctggtgggg ccgtgcatac ccggggtctt 120
gacttcgcct gcgatatcta catttgggcc cctctggctg gtacttgcgg ggtcctgctg 180
ctttcactcg tgatcactct ttactgt 207
<210> 204
<211> 126
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 204
aagcgcggtc ggaagaagct gctgtacatc tttaagcaac ccttcatgag gcctgtgcag 60
actactcaag aggaggacgg ctgttcatgc cggttcccag aggaggagga aggcggctgc 120
gaactg 126
<210> 205
<211> 336
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 205
cgcgtgaaat tcagccgcag cgcagatgct ccagcctacc agcaggggca gaaccagctc 60
tacaacgaac tcaatcttgg tcggagagag gagtacgacg tgctggacaa gcggagagga 120
cgggacccag aaatgggcgg gaagccgcgc agaaagaatc cccaagaggg cctgtacaac 180
gagctccaaa aggataagat ggcagaagcc tatagcgaga ttggtatgaa aggggaacgc 240
agaagaggca aaggccacga cggactgtac cagggactca gcaccgccac caaggacacc 300
tatgacgctc ttcacatgca ggccctgccg cctcgg 336
<210> 206
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 206
Gly Ser Gly
1
<210> 207
<211> 9
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 207
ggaagcgga 9
<210> 208
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 208
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
1 5 10 15
Pro Gly Pro
<210> 209
<211> 57
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 209
gctactaact tcagcctgct gaagcaggct ggagacgtgg aggagaaccc tggacct 57
<210> 210
<211> 63
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 210
atggccttac cagtgaccgc cttgctcctg ccgctggcct tgctgctcca cgccgccagg 60
ccg 63
<210> 211
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 211
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser
<210> 212
<211> 726
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 212
gaaattgtga tgacccagtc acccgccact cttagccttt cacccggtga gcgcgcaacc 60
ctgtcttgca gagcctccca agacatctca aaatacctta attggtatca acagaagccc 120
ggacaggctc ctcgccttct gatctaccac accagccggc tccattctgg aatccctgcc 180
aggttcagcg gtagcggatc tgggaccgac tacaccctca ctatcagctc actgcagcca 240
gaggacttcg ctgtctattt ctgtcagcaa gggaacaccc tgccctacac ctttggacag 300
ggcaccaagc tcgagattaa aggtggaggt ggcagcggag gaggtgggtc cggcggtgga 360
ggaagccagg tccaactcca agaaagcgga ccgggtcttg tgaagccatc agaaactctt 420
tcactgactt gtactgtgag cggagtgtct ctccccgatt acggggtgtc ttggatcaga 480
cagccaccgg ggaagggtct ggaatggatt ggagtgattt ggggctctga gactacttac 540
taccaatcat ccctcaagtc acgcgtcacc atctcaaagg acaactctaa gaatcaggtg 600
tcactgaaac tgtcatctgt gaccgcagcc gacaccgccg tgtactattg cgctaagcat 660
tactattatg gcgggagcta cgcaatggat tactggggac agggtactct ggtcaccgtg 720
tccagc 726
<210> 213
<211> 207
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 213
accacgacgc cagcgccgcg accaccaaca ccggcgccca ccatcgcgtc gcagcccctg 60
tccctgcgcc cagaggcgtg ccggccagcg gcggggggcg cagtgcacac gagggggctg 120
gacttcgcct gtgatatcta catctgggcg cccttggccg ggacttgtgg ggtccttctc 180
ctgtcactgg ttatcaccct ttactgc 207
<210> 214
<211> 1000
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 214
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
165 170 175
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
180 185 190
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
195 200 205
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr
245 250 255
Pro Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr Thr Thr
260 265 270
Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro
275 280 285
Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val
290 295 300
His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro
305 310 315 320
Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu
325 330 335
Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
340 345 350
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
355 360 365
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
370 375 380
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
385 390 395 400
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
405 410 415
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
420 425 430
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
435 440 445
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
450 455 460
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
465 470 475 480
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala
485 490 495
Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro
500 505 510
Gly Pro Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu
515 520 525
Leu Leu His Ala Ala Arg Pro Glu Ile Val Met Thr Gln Ser Pro Ala
530 535 540
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala
545 550 555 560
Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
565 570 575
Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly
580 585 590
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu
595 600 605
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln
610 615 620
Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
625 630 635 640
Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
645 650 655
Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser
660 665 670
Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp
675 680 685
Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
690 695 700
Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu
705 710 715 720
Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser
725 730 735
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
740 745 750
Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly
755 760 765
Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg
770 775 780
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
785 790 795 800
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
805 810 815
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
820 825 830
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
835 840 845
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
850 855 860
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
865 870 875 880
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
885 890 895
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
900 905 910
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
915 920 925
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
930 935 940
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
945 950 955 960
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
965 970 975
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
980 985 990
His Met Gln Ala Leu Pro Pro Arg
995 1000
<210> 215
<211> 3000
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 215
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
cccgaagtgc agttgctgga gtcaggcgga ggactggtgc agcccggagg atcgcttcgc 120
ttgagctgcg cagcctcagg ctttaccttc tcctcctacg ccatgtcctg ggtcagacag 180
gctcccggga agggactgga atgggtgtcc gccattagcg gttccggcgg aagcacttac 240
tatgccgact ctgtgaaggg ccgcttcact atctcccggg acaactccaa gaacaccctg 300
tatctccaaa tgaattccct gagggccgaa gataccgcgg tgtactactg cgctagacgg 360
gagtggtggg gagaaagctg gctgttcgac tactggggac agggcactct cgtgactgtg 420
tcctccggtg gtggtggatc ggggggtggt ggttcgggcg gaggaggatc tggaggagga 480
gggtcggaca ttcaaatgac tcagtccccg tcctccctct ccgcctccgt gggagatcgc 540
gtcacgatca cgtgcagggc cagccagagc atctccagct acctgaactg gtaccagcag 600
aagccaggga aggcaccgaa gctcctgatc tacgccgcta gctcgctgca gtccggcgtc 660
ccttcacggt tctcgggatc gggctcaggc accgacttca ccctgaccat tagcagcctg 720
cagccggagg acttcgcgac atactactgt cagcagtcat actccacccc tctgaccttc 780
ggccaaggga ccaaagtgga gatcaagacc actaccccag caccgaggcc acccaccccg 840
gctcctacca tcgcctccca gcctctgtcc ctgcgtccgg aggcatgtag acccgcagct 900
ggtggggccg tgcatacccg gggtcttgac ttcgcctgcg atatctacat ttgggcccct 960
ctggctggta cttgcggggt cctgctgctt tcactcgtga tcactcttta ctgtaagcgc 1020
ggtcggaaga agctgctgta catctttaag caacccttca tgaggcctgt gcagactact 1080
caagaggagg acggctgttc atgccggttc ccagaggagg aggaaggcgg ctgcgaactg 1140
cgcgtgaaat tcagccgcag cgcagatgct ccagcctacc agcaggggca gaaccagctc 1200
tacaacgaac tcaatcttgg tcggagagag gagtacgacg tgctggacaa gcggagagga 1260
cgggacccag aaatgggcgg gaagccgcgc agaaagaatc cccaagaggg cctgtacaac 1320
gagctccaaa aggataagat ggcagaagcc tatagcgaga ttggtatgaa aggggaacgc 1380
agaagaggca aaggccacga cggactgtac cagggactca gcaccgccac caaggacacc 1440
tatgacgctc ttcacatgca ggccctgccg cctcggggaa gcggagctac taacttcagc 1500
ctgctgaagc aggctggaga cgtggaggag aaccctggac ctatggcctt accagtgacc 1560
gccttgctcc tgccgctggc cttgctgctc cacgccgcca ggccggaaat tgtgatgacc 1620
cagtcacccg ccactcttag cctttcaccc ggtgagcgcg caaccctgtc ttgcagagcc 1680
tcccaagaca tctcaaaata ccttaattgg tatcaacaga agcccggaca ggctcctcgc 1740
cttctgatct accacaccag ccggctccat tctggaatcc ctgccaggtt cagcggtagc 1800
ggatctggga ccgactacac cctcactatc agctcactgc agccagagga cttcgctgtc 1860
tatttctgtc agcaagggaa caccctgccc tacacctttg gacagggcac caagctcgag 1920
attaaaggtg gaggtggcag cggaggaggt gggtccggcg gtggaggaag ccaggtccaa 1980
ctccaagaaa gcggaccggg tcttgtgaag ccatcagaaa ctctttcact gacttgtact 2040
gtgagcggag tgtctctccc cgattacggg gtgtcttgga tcagacagcc accggggaag 2100
ggtctggaat ggattggagt gatttggggc tctgagacta cttactacca atcatccctc 2160
aagtcacgcg tcaccatctc aaaggacaac tctaagaatc aggtgtcact gaaactgtca 2220
tctgtgaccg cagccgacac cgccgtgtac tattgcgcta agcattacta ttatggcggg 2280
agctacgcaa tggattactg gggacagggt actctggtca ccgtgtccag caccacgacg 2340
ccagcgccgc gaccaccaac accggcgccc accatcgcgt cgcagcccct gtccctgcgc 2400
ccagaggcgt gccggccagc ggcggggggc gcagtgcaca cgagggggct ggacttcgcc 2460
tgtgatatct acatctgggc gcccttggcc gggacttgtg gggtccttct cctgtcactg 2520
gttatcaccc tttactgcaa acggggcaga aagaaactcc tgtatatatt caaacaacca 2580
tttatgagac cagtacaaac tactcaagag gaagatggct gtagctgccg atttccagaa 2640
gaagaagaag gaggatgtga actgagagtg aagttcagca ggagcgcaga cgcccccgcg 2700
taccagcagg gccagaacca gctctataac gagctcaatc taggacgaag agaggagtac 2760
gatgttttgg acaagagacg tggccgggac cctgagatgg ggggaaagcc gagaaggaag 2820
aaccctcagg aaggcctgta caatgaactg cagaaagata agatggcgga ggcctacagt 2880
gagattggga tgaaaggcga gcgccggagg ggcaaggggc acgatggcct ttaccagggt 2940
ctcagtacag ccaccaagga cacctacgac gcccttcaca tgcaggccct gccccctcgc 3000
<210> 216
<211> 1001
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 216
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr
115 120 125
Tyr Gly Leu Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
145 150 155 160
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
165 170 175
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
180 185 190
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
195 200 205
Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
225 230 235 240
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
245 250 255
Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Thr Thr
260 265 270
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
275 280 285
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
290 295 300
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala
305 310 315 320
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
325 330 335
Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
340 345 350
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
355 360 365
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
370 375 380
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
385 390 395 400
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
405 410 415
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
420 425 430
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
435 440 445
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
450 455 460
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
465 470 475 480
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly
485 490 495
Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn
500 505 510
Pro Gly Pro Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala
515 520 525
Leu Leu Leu His Ala Ala Arg Pro Glu Ile Val Met Thr Gln Ser Pro
530 535 540
Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg
545 550 555 560
Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
565 570 575
Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser
580 585 590
Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr
595 600 605
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys
610 615 620
Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu
625 630 635 640
Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
645 650 655
Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro
660 665 670
Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro
675 680 685
Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
690 695 700
Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser
705 710 715 720
Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val
725 730 735
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
740 745 750
Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp
755 760 765
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro
770 775 780
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
785 790 795 800
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
805 810 815
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
820 825 830
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
835 840 845
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
850 855 860
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
865 870 875 880
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
885 890 895
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
900 905 910
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
915 920 925
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
930 935 940
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
945 950 955 960
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
965 970 975
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
980 985 990
Leu His Met Gln Ala Leu Pro Pro Arg
995 1000
<210> 217
<211> 3003
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 217
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
ccccaagtgc agctgcagga atccggtggc ggagtcgtgc agcctggaag gagcctgaga 120
ctctcatgcg ccgcgtcagg gttcaccttt tcctcctacg ggatgcattg ggtcagacag 180
gcccccggaa agggactcga atgggtggct gtgatcagct acgacggctc caacaagtac 240
tacgccgact ccgtgaaagg ccggttcact atctcccggg acaactccaa gaacacgctg 300
tatctgcaaa tgaattcact gcgcgcggag gataccgctg tgtactactg cggtggctcc 360
ggttacgccc tgcacgatga ctattacggc cttgacgtct ggggccaggg aaccctcgtg 420
actgtgtcca gcggtggagg aggttcgggc ggaggaggat caggaggggg tggatcgcag 480
agcgcactga ctcagccggc atccgtgtcc ggtagccccg gacagtcgat taccatctcc 540
tgtaccggca cctcctccga cgtgggaggg tacaactacg tgtcgtggta ccagcagcac 600
ccaggaaagg cccctaagtt gatgatctac gatgtgtcaa accgcccgtc tggagtctcc 660
aaccggttct ccggctccaa gtccggcaac accgccagcc tgaccattag cgggctgcaa 720
gccgaggatg aggccgacta ctactgctcg agctacacat cctcgagcac cctctacgtg 780
ttcggctcgg ggactaaggt caccgtgctg accactaccc cagcaccgag gccacccacc 840
ccggctccta ccatcgcctc ccagcctctg tccctgcgtc cggaggcatg tagacccgca 900
gctggtgggg ccgtgcatac ccggggtctt gacttcgcct gcgatatcta catttgggcc 960
cctctggctg gtacttgcgg ggtcctgctg ctttcactcg tgatcactct ttactgtaag 1020
cgcggtcgga agaagctgct gtacatcttt aagcaaccct tcatgaggcc tgtgcagact 1080
actcaagagg aggacggctg ttcatgccgg ttcccagagg aggaggaagg cggctgcgaa 1140
ctgcgcgtga aattcagccg cagcgcagat gctccagcct accagcaggg gcagaaccag 1200
ctctacaacg aactcaatct tggtcggaga gaggagtacg acgtgctgga caagcggaga 1260
ggacgggacc cagaaatggg cgggaagccg cgcagaaaga atccccaaga gggcctgtac 1320
aacgagctcc aaaaggataa gatggcagaa gcctatagcg agattggtat gaaaggggaa 1380
cgcagaagag gcaaaggcca cgacggactg taccagggac tcagcaccgc caccaaggac 1440
acctatgacg ctcttcacat gcaggccctg ccgcctcggg gaagcggagc tactaacttc 1500
agcctgctga agcaggctgg agacgtggag gagaaccctg gacctatggc cttaccagtg 1560
accgccttgc tcctgccgct ggccttgctg ctccacgccg ccaggccgga aattgtgatg 1620
acccagtcac ccgccactct tagcctttca cccggtgagc gcgcaaccct gtcttgcaga 1680
gcctcccaag acatctcaaa ataccttaat tggtatcaac agaagcccgg acaggctcct 1740
cgccttctga tctaccacac cagccggctc cattctggaa tccctgccag gttcagcggt 1800
agcggatctg ggaccgacta caccctcact atcagctcac tgcagccaga ggacttcgct 1860
gtctatttct gtcagcaagg gaacaccctg ccctacacct ttggacaggg caccaagctc 1920
gagattaaag gtggaggtgg cagcggagga ggtgggtccg gcggtggagg aagccaggtc 1980
caactccaag aaagcggacc gggtcttgtg aagccatcag aaactctttc actgacttgt 2040
actgtgagcg gagtgtctct ccccgattac ggggtgtctt ggatcagaca gccaccgggg 2100
aagggtctgg aatggattgg agtgatttgg ggctctgaga ctacttacta ccaatcatcc 2160
ctcaagtcac gcgtcaccat ctcaaaggac aactctaaga atcaggtgtc actgaaactg 2220
tcatctgtga ccgcagccga caccgccgtg tactattgcg ctaagcatta ctattatggc 2280
gggagctacg caatggatta ctggggacag ggtactctgg tcaccgtgtc cagcaccacg 2340
acgccagcgc cgcgaccacc aacaccggcg cccaccatcg cgtcgcagcc cctgtccctg 2400
cgcccagagg cgtgccggcc agcggcgggg ggcgcagtgc acacgagggg gctggacttc 2460
gcctgtgata tctacatctg ggcgcccttg gccgggactt gtggggtcct tctcctgtca 2520
ctggttatca ccctttactg caaacggggc agaaagaaac tcctgtatat attcaaacaa 2580
ccatttatga gaccagtaca aactactcaa gaggaagatg gctgtagctg ccgatttcca 2640
gaagaagaag aaggaggatg tgaactgaga gtgaagttca gcaggagcgc agacgccccc 2700
gcgtaccagc agggccagaa ccagctctat aacgagctca atctaggacg aagagaggag 2760
tacgatgttt tggacaagag acgtggccgg gaccctgaga tggggggaaa gccgagaagg 2820
aagaaccctc aggaaggcct gtacaatgaa ctgcagaaag ataagatggc ggaggcctac 2880
agtgagattg ggatgaaagg cgagcgccgg aggggcaagg ggcacgatgg cctttaccag 2940
ggtctcagta cagccaccaa ggacacctac gacgcccttc acatgcaggc cctgccccct 3000
cgc 3003
<210> 218
<211> 1002
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 218
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Arg Glu Trp Val Pro Tyr Asp Val Ser
115 120 125
Trp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
145 150 155 160
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
165 170 175
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
180 185 190
Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
195 200 205
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
210 215 220
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
225 230 235 240
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr
245 250 255
Ser Thr Pro Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr
260 265 270
Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser
275 280 285
Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly
290 295 300
Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp
305 310 315 320
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
325 330 335
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
340 345 350
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
355 360 365
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
370 375 380
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
385 390 395 400
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
405 410 415
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
420 425 430
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
435 440 445
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
450 455 460
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
465 470 475 480
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser
485 490 495
Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
500 505 510
Asn Pro Gly Pro Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu
515 520 525
Ala Leu Leu Leu His Ala Ala Arg Pro Glu Ile Val Met Thr Gln Ser
530 535 540
Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys
545 550 555 560
Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys
565 570 575
Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His
580 585 590
Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr
595 600 605
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe
610 615 620
Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys
625 630 635 640
Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
645 650 655
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
660 665 670
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
675 680 685
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu
690 695 700
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
705 710 715 720
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
725 730 735
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
740 745 750
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
755 760 765
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala
770 775 780
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
785 790 795 800
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
805 810 815
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
820 825 830
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
835 840 845
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
850 855 860
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
865 870 875 880
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
885 890 895
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
900 905 910
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
915 920 925
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
930 935 940
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
945 950 955 960
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
965 970 975
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
980 985 990
Ala Leu His Met Gln Ala Leu Pro Pro Arg
995 1000
<210> 219
<211> 3006
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 219
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
cccgaagtgc agttgctgga gtcaggcgga ggactggtgc agcccggagg atcgcttcgc 120
ttgagctgcg cagcctcagg ctttaccttc tcctcctacg ccatgtcctg ggtcagacag 180
gctcccggga agggactgga atgggtgtcc gccattagcg gttccggcgg aagcacttac 240
tatgccgact ctgtgaaggg ccgcttcact atctcccggg acaactccaa gaacaccctg 300
tatctccaaa tgaattccct gagggccgaa gataccgcgg tgtactactg cgctagacgg 360
gagtgggtgc cctacgatgt cagctggtac ttcgactact ggggacaggg cactctcgtg 420
actgtgtcct ccggtggtgg tggatcgggg ggtggtggtt cgggcggagg aggatctgga 480
ggaggagggt cggacattca aatgactcag tccccgtcct ccctctccgc ctccgtggga 540
gatcgcgtca cgatcacgtg cagggccagc cagagcatct ccagctacct gaactggtac 600
cagcagaagc cagggaaggc accgaagctc ctgatctacg ccgctagctc gctgcagtcc 660
ggcgtccctt cacggttctc gggatcgggc tcaggcaccg acttcaccct gaccattagc 720
agcctgcagc cggaggactt cgcgacatac tactgtcagc agtcatactc cacccctctg 780
accttcggcc aagggaccaa agtggagatc aagaccacta ccccagcacc gaggccaccc 840
accccggctc ctaccatcgc ctcccagcct ctgtccctgc gtccggaggc atgtagaccc 900
gcagctggtg gggccgtgca tacccggggt cttgacttcg cctgcgatat ctacatttgg 960
gcccctctgg ctggtacttg cggggtcctg ctgctttcac tcgtgatcac tctttactgt 1020
aagcgcggtc ggaagaagct gctgtacatc tttaagcaac ccttcatgag gcctgtgcag 1080
actactcaag aggaggacgg ctgttcatgc cggttcccag aggaggagga aggcggctgc 1140
gaactgcgcg tgaaattcag ccgcagcgca gatgctccag cctaccagca ggggcagaac 1200
cagctctaca acgaactcaa tcttggtcgg agagaggagt acgacgtgct ggacaagcgg 1260
agaggacggg acccagaaat gggcgggaag ccgcgcagaa agaatcccca agagggcctg 1320
tacaacgagc tccaaaagga taagatggca gaagcctata gcgagattgg tatgaaaggg 1380
gaacgcagaa gaggcaaagg ccacgacgga ctgtaccagg gactcagcac cgccaccaag 1440
gacacctatg acgctcttca catgcaggcc ctgccgcctc ggggaagcgg agctactaac 1500
ttcagcctgc tgaagcaggc tggagacgtg gaggagaacc ctggacctat ggccttacca 1560
gtgaccgcct tgctcctgcc gctggccttg ctgctccacg ccgccaggcc ggaaattgtg 1620
atgacccagt cacccgccac tcttagcctt tcacccggtg agcgcgcaac cctgtcttgc 1680
agagcctccc aagacatctc aaaatacctt aattggtatc aacagaagcc cggacaggct 1740
cctcgccttc tgatctacca caccagccgg ctccattctg gaatccctgc caggttcagc 1800
ggtagcggat ctgggaccga ctacaccctc actatcagct cactgcagcc agaggacttc 1860
gctgtctatt tctgtcagca agggaacacc ctgccctaca cctttggaca gggcaccaag 1920
ctcgagatta aaggtggagg tggcagcgga ggaggtgggt ccggcggtgg aggaagccag 1980
gtccaactcc aagaaagcgg accgggtctt gtgaagccat cagaaactct ttcactgact 2040
tgtactgtga gcggagtgtc tctccccgat tacggggtgt cttggatcag acagccaccg 2100
gggaagggtc tggaatggat tggagtgatt tggggctctg agactactta ctaccaatca 2160
tccctcaagt cacgcgtcac catctcaaag gacaactcta agaatcaggt gtcactgaaa 2220
ctgtcatctg tgaccgcagc cgacaccgcc gtgtactatt gcgctaagca ttactattat 2280
ggcgggagct acgcaatgga ttactgggga cagggtactc tggtcaccgt gtccagcacc 2340
acgacgccag cgccgcgacc accaacaccg gcgcccacca tcgcgtcgca gcccctgtcc 2400
ctgcgcccag aggcgtgccg gccagcggcg gggggcgcag tgcacacgag ggggctggac 2460
ttcgcctgtg atatctacat ctgggcgccc ttggccggga cttgtggggt ccttctcctg 2520
tcactggtta tcacccttta ctgcaaacgg ggcagaaaga aactcctgta tatattcaaa 2580
caaccattta tgagaccagt acaaactact caagaggaag atggctgtag ctgccgattt 2640
ccagaagaag aagaaggagg atgtgaactg agagtgaagt tcagcaggag cgcagacgcc 2700
cccgcgtacc agcagggcca gaaccagctc tataacgagc tcaatctagg acgaagagag 2760
gagtacgatg ttttggacaa gagacgtggc cgggaccctg agatgggggg aaagccgaga 2820
aggaagaacc ctcaggaagg cctgtacaat gaactgcaga aagataagat ggcggaggcc 2880
tacagtgaga ttgggatgaa aggcgagcgc cggaggggca aggggcacga tggcctttac 2940
cagggtctca gtacagccac caaggacacc tacgacgccc ttcacatgca ggccctgccc 3000
cctcgc 3006
<210> 220
<211> 991
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 220
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe
35 40 45
Ala Leu Ser Asn His Gly Met Ser Trp Val Arg Arg Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Ala Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Arg
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala
100 105 110
Ile Tyr Tyr Cys Ser Ala His Gly Gly Glu Ser Asp Val Trp Gly Gln
115 120 125
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Leu Thr Gln Ser
145 150 155 160
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
165 170 175
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys
180 185 190
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln
195 200 205
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
210 215 220
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
225 230 235 240
Cys Gln Gln Ser Tyr Ser Thr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
245 250 255
Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala
260 265 270
Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg
275 280 285
Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys
290 295 300
Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
305 310 315 320
Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu
325 330 335
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
340 345 350
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
355 360 365
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
370 375 380
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
385 390 395 400
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
405 410 415
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
420 425 430
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
435 440 445
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
450 455 460
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
465 470 475 480
Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala
485 490 495
Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Leu Pro Val Thr Ala
500 505 510
Leu Leu Leu Pro Leu Ala Leu Leu Leu His Ala Ala Arg Pro Glu Ile
515 520 525
Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg
530 535 540
Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn
545 550 555 560
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His
565 570 575
Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly
580 585 590
Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
595 600 605
Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe
610 615 620
Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
625 630 635 640
Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly
645 650 655
Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val
660 665 670
Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro
675 680 685
Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr
690 695 700
Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp
705 710 715 720
Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala
725 730 735
Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser
740 745 750
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
755 760 765
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
770 775 780
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
785 790 795 800
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile
805 810 815
Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val
820 825 830
Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
835 840 845
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
850 855 860
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
865 870 875 880
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
885 890 895
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
900 905 910
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
915 920 925
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
930 935 940
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
945 950 955 960
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
965 970 975
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
980 985 990
<210> 221
<211> 2973
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 221
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
cccgaagtgc aattggtgga atcaggggga ggacttgtgc agcctggagg atcgctgaga 120
ctgtcatgtg ccgtgtccgg ctttgccctg tccaaccacg ggatgtcctg ggtccgccgc 180
gcgcctggaa agggcctcga atgggtgtcg ggtattgtgt acagcggtag cacctactat 240
gccgcatccg tgaaggggag attcaccatc agccgggaca actccaggaa cactctgtac 300
ctccaaatga attcgctgag gccagaggac actgccatct actactgctc cgcgcatggc 360
ggagagtccg acgtctgggg acaggggacc accgtgaccg tgtctagcgc gtccggcgga 420
ggcggcagcg ggggtggtgg ttcagggggc ggcggatcgg acatccagct cacccagtcc 480
ccgagctcgc tgtccgcctc cgtgggagat cgggtcacca tcacgtgccg cgccagccag 540
tcgatttcct cctacctgaa ctggtaccaa cagaagcccg gaaaagcccc gaagcttctc 600
atctacgccg cctcgagcct gcagtcagga gtgccctcac ggttctccgg ctccggttcc 660
ggtactgatt tcaccctgac catttcctcc ctgcaaccgg aggacttcgc tacttactac 720
tgccagcagt cgtactccac cccctacact ttcggacaag gcaccaaggt cgaaatcaag 780
accactaccc cagcaccgag gccacccacc ccggctccta ccatcgcctc ccagcctctg 840
tccctgcgtc cggaggcatg tagacccgca gctggtgggg ccgtgcatac ccggggtctt 900
gacttcgcct gcgatatcta catttgggcc cctctggctg gtacttgcgg ggtcctgctg 960
ctttcactcg tgatcactct ttactgtaag cgcggtcgga agaagctgct gtacatcttt 1020
aagcaaccct tcatgaggcc tgtgcagact actcaagagg aggacggctg ttcatgccgg 1080
ttcccagagg aggaggaagg cggctgcgaa ctgcgcgtga aattcagccg cagcgcagat 1140
gctccagcct accagcaggg gcagaaccag ctctacaacg aactcaatct tggtcggaga 1200
gaggagtacg acgtgctgga caagcggaga ggacgggacc cagaaatggg cgggaagccg 1260
cgcagaaaga atccccaaga gggcctgtac aacgagctcc aaaaggataa gatggcagaa 1320
gcctatagcg agattggtat gaaaggggaa cgcagaagag gcaaaggcca cgacggactg 1380
taccagggac tcagcaccgc caccaaggac acctatgacg ctcttcacat gcaggccctg 1440
ccgcctcggg gaagcggagc tactaacttc agcctgctga agcaggctgg agacgtggag 1500
gagaaccctg gacctatggc cttaccagtg accgccttgc tcctgccgct ggccttgctg 1560
ctccacgccg ccaggccgga aattgtgatg acccagtcac ccgccactct tagcctttca 1620
cccggtgagc gcgcaaccct gtcttgcaga gcctcccaag acatctcaaa ataccttaat 1680
tggtatcaac agaagcccgg acaggctcct cgccttctga tctaccacac cagccggctc 1740
cattctggaa tccctgccag gttcagcggt agcggatctg ggaccgacta caccctcact 1800
atcagctcac tgcagccaga ggacttcgct gtctatttct gtcagcaagg gaacaccctg 1860
ccctacacct ttggacaggg caccaagctc gagattaaag gtggaggtgg cagcggagga 1920
ggtgggtccg gcggtggagg aagccaggtc caactccaag aaagcggacc gggtcttgtg 1980
aagccatcag aaactctttc actgacttgt actgtgagcg gagtgtctct ccccgattac 2040
ggggtgtctt ggatcagaca gccaccgggg aagggtctgg aatggattgg agtgatttgg 2100
ggctctgaga ctacttacta ccaatcatcc ctcaagtcac gcgtcaccat ctcaaaggac 2160
aactctaaga atcaggtgtc actgaaactg tcatctgtga ccgcagccga caccgccgtg 2220
tactattgcg ctaagcatta ctattatggc gggagctacg caatggatta ctggggacag 2280
ggtactctgg tcaccgtgtc cagcaccacg acgccagcgc cgcgaccacc aacaccggcg 2340
cccaccatcg cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc agcggcgggg 2400
ggcgcagtgc acacgagggg gctggacttc gcctgtgata tctacatctg ggcgcccttg 2460
gccgggactt gtggggtcct tctcctgtca ctggttatca ccctttactg caaacggggc 2520
agaaagaaac tcctgtatat attcaaacaa ccatttatga gaccagtaca aactactcaa 2580
gaggaagatg gctgtagctg ccgatttcca gaagaagaag aaggaggatg tgaactgaga 2640
gtgaagttca gcaggagcgc agacgccccc gcgtaccagc agggccagaa ccagctctat 2700
aacgagctca atctaggacg aagagaggag tacgatgttt tggacaagag acgtggccgg 2760
gaccctgaga tggggggaaa gccgagaagg aagaaccctc aggaaggcct gtacaatgaa 2820
ctgcagaaag ataagatggc ggaggcctac agtgagattg ggatgaaagg cgagcgccgg 2880
aggggcaagg ggcacgatgg cctttaccag ggtctcagta cagccaccaa ggacacctac 2940
gacgcccttc acatgcaggc cctgccccct cgc 2973
<210> 222
<211> 991
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 222
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu
20 25 30
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
35 40 45
Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala
50 55 60
Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile
85 90 95
Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly
100 105 110
Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
145 150 155 160
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
165 170 175
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
180 185 190
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
195 200 205
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
210 215 220
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
225 230 235 240
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
245 250 255
Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu
485 490 495
Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Ala Leu Pro
500 505 510
Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu His Ala Ala Arg
515 520 525
Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
530 535 540
Gly Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Ala Leu Ser Asn
545 550 555 560
His Gly Met Ser Trp Val Arg Arg Ala Pro Gly Lys Gly Leu Glu Trp
565 570 575
Val Ser Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr Ala Ala Ser Val
580 585 590
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Arg Asn Thr Leu Tyr
595 600 605
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
610 615 620
Ser Ala His Gly Gly Glu Ser Asp Val Trp Gly Gln Gly Thr Thr Val
625 630 635 640
Thr Val Ser Ser Ala Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
645 650 655
Gly Gly Gly Gly Ser Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu
660 665 670
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln
675 680 685
Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala
690 695 700
Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro
705 710 715 720
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
725 730 735
Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser
740 745 750
Tyr Ser Thr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
755 760 765
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
770 775 780
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
785 790 795 800
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile
805 810 815
Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val
820 825 830
Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
835 840 845
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
850 855 860
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
865 870 875 880
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
885 890 895
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
900 905 910
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
915 920 925
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
930 935 940
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
945 950 955 960
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
965 970 975
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
980 985 990
<210> 223
<211> 2973
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 223
atggccttac cagtgaccgc cttgctcctg ccgctggcct tgctgctcca cgccgccagg 60
ccggaaattg tgatgaccca gtcacccgcc actcttagcc tttcacccgg tgagcgcgca 120
accctgtctt gcagagcctc ccaagacatc tcaaaatacc ttaattggta tcaacagaag 180
cccggacagg ctcctcgcct tctgatctac cacaccagcc ggctccattc tggaatccct 240
gccaggttca gcggtagcgg atctgggacc gactacaccc tcactatcag ctcactgcag 300
ccagaggact tcgctgtcta tttctgtcag caagggaaca ccctgcccta cacctttgga 360
cagggcacca agctcgagat taaaggtgga ggtggcagcg gaggaggtgg gtccggcggt 420
ggaggaagcc aggtccaact ccaagaaagc ggaccgggtc ttgtgaagcc atcagaaact 480
ctttcactga cttgtactgt gagcggagtg tctctccccg attacggggt gtcttggatc 540
agacagccac cggggaaggg tctggaatgg attggagtga tttggggctc tgagactact 600
tactaccaat catccctcaa gtcacgcgtc accatctcaa aggacaactc taagaatcag 660
gtgtcactga aactgtcatc tgtgaccgca gccgacaccg ccgtgtacta ttgcgctaag 720
cattactatt atggcgggag ctacgcaatg gattactggg gacagggtac tctggtcacc 780
gtgtccagca ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 840
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 900
agggggctgg acttcgcctg tgatatctac atctgggcgc ccttggccgg gacttgtggg 960
gtccttctcc tgtcactggt tatcaccctt tactgcaaac ggggcagaaa gaaactcctg 1020
tatatattca aacaaccatt tatgagacca gtacaaacta ctcaagagga agatggctgt 1080
agctgccgat ttccagaaga agaagaagga ggatgtgaac tgagagtgaa gttcagcagg 1140
agcgcagacg cccccgcgta ccagcagggc cagaaccagc tctataacga gctcaatcta 1200
ggacgaagag aggagtacga tgttttggac aagagacgtg gccgggaccc tgagatgggg 1260
ggaaagccga gaaggaagaa ccctcaggaa ggcctgtaca atgaactgca gaaagataag 1320
atggcggagg cctacagtga gattgggatg aaaggcgagc gccggagggg caaggggcac 1380
gatggccttt accagggtct cagtacagcc accaaggaca cctacgacgc ccttcacatg 1440
caggccctgc cccctcgcgg aagcggagct actaacttca gcctgctgaa gcaggctgga 1500
gacgtggagg agaaccctgg acctatggcc ctccctgtca ccgccctgct gcttccgctg 1560
gctcttctgc tccacgccgc tcggcccgaa gtgcaattgg tggaatcagg gggaggactt 1620
gtgcagcctg gaggatcgct gagactgtca tgtgccgtgt ccggctttgc cctgtccaac 1680
cacgggatgt cctgggtccg ccgcgcgcct ggaaagggcc tcgaatgggt gtcgggtatt 1740
gtgtacagcg gtagcaccta ctatgccgca tccgtgaagg ggagattcac catcagccgg 1800
gacaactcca ggaacactct gtacctccaa atgaattcgc tgaggccaga ggacactgcc 1860
atctactact gctccgcgca tggcggagag tccgacgtct ggggacaggg gaccaccgtg 1920
accgtgtcta gcgcgtccgg cggaggcggc agcgggggtg gtggttcagg gggcggcgga 1980
tcggacatcc agctcaccca gtccccgagc tcgctgtccg cctccgtggg agatcgggtc 2040
accatcacgt gccgcgccag ccagtcgatt tcctcctacc tgaactggta ccaacagaag 2100
cccggaaaag ccccgaagct tctcatctac gccgcctcga gcctgcagtc aggagtgccc 2160
tcacggttct ccggctccgg ttccggtact gatttcaccc tgaccatttc ctccctgcaa 2220
ccggaggact tcgctactta ctactgccag cagtcgtact ccacccccta cactttcgga 2280
caaggcacca aggtcgaaat caagaccact accccagcac cgaggccacc caccccggct 2340
cctaccatcg cctcccagcc tctgtccctg cgtccggagg catgtagacc cgcagctggt 2400
ggggccgtgc atacccgggg tcttgacttc gcctgcgata tctacatttg ggcccctctg 2460
gctggtactt gcggggtcct gctgctttca ctcgtgatca ctctttactg taagcgcggt 2520
cggaagaagc tgctgtacat ctttaagcaa cccttcatga ggcctgtgca gactactcaa 2580
gaggaggacg gctgttcatg ccggttccca gaggaggagg aaggcggctg cgaactgcgc 2640
gtgaaattca gccgcagcgc agatgctcca gcctaccagc aggggcagaa ccagctctac 2700
aacgaactca atcttggtcg gagagaggag tacgacgtgc tggacaagcg gagaggacgg 2760
gacccagaaa tgggcgggaa gccgcgcaga aagaatcccc aagagggcct gtacaacgag 2820
ctccaaaagg ataagatggc agaagcctat agcgagattg gtatgaaagg ggaacgcaga 2880
agaggcaaag gccacgacgg actgtaccag ggactcagca ccgccaccaa ggacacctat 2940
gacgctcttc acatgcaggc cctgccgcct cgg 2973
<210> 224
<211> 492
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 224
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro
245 250 255
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
260 265 270
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
275 280 285
Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
290 295 300
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
305 310 315 320
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
325 330 335
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
340 345 350
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
355 360 365
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
370 375 380
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
385 390 395 400
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
405 410 415
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
420 425 430
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
435 440 445
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
450 455 460
Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu
465 470 475 480
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly
485 490
<210> 225
<211> 465
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 225
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
245 250 255
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
260 265 270
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
275 280 285
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
290 295 300
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
305 310 315 320
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
325 330 335
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
340 345 350
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
355 360 365
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
370 375 380
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
385 390 395 400
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
405 410 415
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
420 425 430
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
435 440 445
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
450 455 460
Arg
465
<210> 226
<211> 493
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 226
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln
130 135 140
Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile Ser Cys
145 150 155 160
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr
165 170 175
Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp Val Ser
180 185 190
Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys Ser Gly
195 200 205
Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp Glu Ala
210 215 220
Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr Val Phe
225 230 235 240
Gly Ser Gly Thr Lys Val Thr Val Leu Thr Thr Thr Pro Ala Pro Arg
245 250 255
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
260 265 270
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
275 280 285
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
290 295 300
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
305 310 315 320
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
325 330 335
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
340 345 350
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
355 360 365
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
370 375 380
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
385 390 395 400
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
405 410 415
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
420 425 430
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
435 440 445
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
450 455 460
His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser
465 470 475 480
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly
485 490
<210> 227
<211> 494
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 227
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Val Pro Tyr Asp Val Ser Trp Tyr Phe Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp
130 135 140
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
145 150 155 160
Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu
165 170 175
Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
180 185 190
Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
195 200 205
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
210 215 220
Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr
225 230 235 240
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro
245 250 255
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
260 265 270
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
275 280 285
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
290 295 300
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
305 310 315 320
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
325 330 335
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
340 345 350
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
355 360 365
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
370 375 380
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
385 390 395 400
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
405 410 415
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
420 425 430
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
435 440 445
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
450 455 460
Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe
465 470 475 480
Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly
485 490
<210> 228
<211> 483
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 228
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Ala Leu Ser Asn His
20 25 30
Gly Met Ser Trp Val Arg Arg Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr Ala Ala Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Arg Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ser
85 90 95
Ala His Gly Gly Glu Ser Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser Ala Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser
130 135 140
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
145 150 155 160
Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
165 170 175
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
180 185 190
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
195 200 205
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr
210 215 220
Ser Thr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr
225 230 235 240
Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser
245 250 255
Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly
260 265 270
Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp
275 280 285
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
290 295 300
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
305 310 315 320
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
325 330 335
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
340 345 350
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
355 360 365
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
370 375 380
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
385 390 395 400
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
405 410 415
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
420 425 430
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
435 440 445
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser
450 455 460
Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu
465 470 475 480
Asn Pro Gly
<210> 229
<211> 486
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 229
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
245 250 255
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
260 265 270
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
275 280 285
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
290 295 300
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
305 310 315 320
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
325 330 335
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
340 345 350
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
355 360 365
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
370 375 380
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
385 390 395 400
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
405 410 415
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
420 425 430
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
435 440 445
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
450 455 460
Arg Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp
465 470 475 480
Val Glu Glu Asn Pro Gly
485
<210> 230
<211> 462
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 230
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Ala Leu Ser Asn His
20 25 30
Gly Met Ser Trp Val Arg Arg Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr Ala Ala Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Arg Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ser
85 90 95
Ala His Gly Gly Glu Ser Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser Ala Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser
130 135 140
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
145 150 155 160
Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
165 170 175
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
180 185 190
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
195 200 205
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr
210 215 220
Ser Thr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr
225 230 235 240
Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser
245 250 255
Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly
260 265 270
Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp
275 280 285
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
290 295 300
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
305 310 315 320
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
325 330 335
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
340 345 350
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
355 360 365
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
370 375 380
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
385 390 395 400
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
405 410 415
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
420 425 430
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
435 440 445
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 231
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 231
Asn His Gly Met Ser
1 5
<210> 232
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 232
Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr Ala Ala Ser Val Lys Gly
1 5 10 15
<210> 233
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 233
His Gly Gly Glu Ser Asp Val
1 5
<210> 234
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 234
Gly Phe Ala Leu Ser Asn His
1 5
<210> 235
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 235
Val Tyr Ser Gly Ser
1 5
<210> 236
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 236
Gly Phe Ala Leu Ser Asn His Gly
1 5
<210> 237
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 237
Ile Val Tyr Ser Gly Ser Thr
1 5
<210> 238
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 238
Ser Ala His Gly Gly Glu Ser Asp Val
1 5
<210> 239
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 239
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Val Ser Gly Phe Ala Leu Ser Asn His
20 25 30
Gly Met Ser Trp Val Arg Arg Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Val Tyr Ser Gly Ser Thr Tyr Tyr Ala Ala Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Arg Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ser
85 90 95
Ala His Gly Gly Glu Ser Asp Val Trp Gly Gln Gly Thr Thr Val Thr
100 105 110
Val Ser Ser
115
<210> 240
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 240
Gln Gln Ser Tyr Ser Thr Pro Tyr Thr
1 5
<210> 241
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 241
Ser Tyr Ser Thr Pro Tyr
1 5
<210> 242
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 242
Asp Ile Gln Leu Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 243
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 243
Ala Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Ser
<210> 244
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 244
Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
1 5 10
<210> 245
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 245
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
1 5 10 15
<210> 246
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 246
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
1 5 10
<210> 247
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 247
Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn
1 5 10
<210> 248
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 248
His Thr Ser Arg Leu His Ser
1 5
<210> 249
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 249
Gln Gln Gly Asn Thr Leu Pro Tyr Thr
1 5
<210> 250
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 250
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 251
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 251
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 252
<211> 63
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 252
atggccctcc ctgtcaccgc tctgttgctg ccgcttgctc tgctgctcca cgcagcgcga 60
ccg 63
<210> 253
<211> 747
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 253
caggtacaat tgcaggagtc tggaggcggt gtggtgcaac ccggtcgcag cttgcgcctg 60
agttgtgctg cgtctggatt tacattttca tcttacggaa tgcattgggt acgccaggca 120
ccggggaaag gccttgaatg ggtggctgta atttcatacg atggttccaa caaatactat 180
gctgactcag tcaagggtcg atttacaatt agtcgggaca actccaagaa caccctttat 240
cttcaaatga attcccttag agcagaggat acggcggtct attactgtgg tggcagtggt 300
tatgcacttc atgatgatta ctatggcttg gatgtctggg ggcaagggac gcttgtaact 360
gtatcctctg gtggtggtgg tagtggtggg ggaggctccg gcggtggcgg ctctcaatct 420
gctctgactc aaccagcaag cgtatcaggg tcaccgggac agagtattac cataagttgc 480
acggggacct ctagcgatgt aggggggtat aattatgtat cttggtatca acaacacccc 540
gggaaagccc ctaaattgat gatctacgac gtgagcaatc gacctagtgg cgtatcaaat 600
cgcttctctg gtagcaagag tgggaatacg gcgtccctta ctattagcgg attgcaagca 660
gaagatgagg ccgattacta ctgcagctcc tatactagct cttctacatt gtacgtcttt 720
gggagcggaa caaaagtaac agtactc 747
<210> 254
<211> 207
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 254
acaacaacac ctgccccgag accgcctaca ccagccccga ctattgccag ccagcctctg 60
agcctcaggc ctgaggcctg taggcccgca gcgggcggcg cagttcatac acggggcttg 120
gatttcgctt gtgatattta tatttgggct cctttggcgg ggacatgtgg cgtgctgctt 180
ctgtcacttg ttattacact gtactgt 207
<210> 255
<211> 126
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 255
aaacgcgggc gaaaaaaatt gctgtatatt tttaagcagc catttatgag gcccgttcag 60
acgacgcagg aggaggacgg ttgctcttgc aggttcccag aagaggaaga agggggctgt 120
gaattg 126
<210> 256
<211> 336
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 256
cgggttaaat tttcaagatc cgcagacgct ccagcatacc aacagggaca aaaccaactc 60
tataacgagc tgaatcttgg aagaagggag gaatatgatg tgctggataa acggcgcggt 120
agagatccgg agatgggcgg aaaaccaagg cgaaaaaacc ctcaggaggg actctacaac 180
gaactgcaga aagacaaaat ggcggaggct tattccgaaa taggcatgaa gggcgagcgg 240
aggcgaggga aagggcacga cggactgtat caaggcctct caaccgcgac taaggatacg 300
tacgacgccc tgcacatgca ggccctgcct ccgaga 336
<210> 257
<211> 493
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 257
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr
115 120 125
Tyr Gly Leu Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
145 150 155 160
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
165 170 175
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
180 185 190
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
195 200 205
Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser
210 215 220
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
225 230 235 240
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
245 250 255
Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Thr Thr
260 265 270
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
275 280 285
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
290 295 300
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala
305 310 315 320
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
325 330 335
Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
340 345 350
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
355 360 365
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
370 375 380
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
385 390 395 400
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
405 410 415
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
420 425 430
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
435 440 445
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
450 455 460
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
465 470 475 480
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 258
<211> 1479
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 258
atggccctcc ctgtcaccgc tctgttgctg ccgcttgctc tgctgctcca cgcagcgcga 60
ccgcaggtac aattgcagga gtctggaggc ggtgtggtgc aacccggtcg cagcttgcgc 120
ctgagttgtg ctgcgtctgg atttacattt tcatcttacg gaatgcattg ggtacgccag 180
gcaccgggga aaggccttga atgggtggct gtaatttcat acgatggttc caacaaatac 240
tatgctgact cagtcaaggg tcgatttaca attagtcggg acaactccaa gaacaccctt 300
tatcttcaaa tgaattccct tagagcagag gatacggcgg tctattactg tggtggcagt 360
ggttatgcac ttcatgatga ttactatggc ttggatgtct gggggcaagg gacgcttgta 420
actgtatcct ctggtggtgg tggtagtggt gggggaggct ccggcggtgg cggctctcaa 480
tctgctctga ctcaaccagc aagcgtatca gggtcaccgg gacagagtat taccataagt 540
tgcacgggga cctctagcga tgtagggggg tataattatg tatcttggta tcaacaacac 600
cccgggaaag cccctaaatt gatgatctac gacgtgagca atcgacctag tggcgtatca 660
aatcgcttct ctggtagcaa gagtgggaat acggcgtccc ttactattag cggattgcaa 720
gcagaagatg aggccgatta ctactgcagc tcctatacta gctcttctac attgtacgtc 780
tttgggagcg gaacaaaagt aacagtactc acaacaacac ctgccccgag accgcctaca 840
ccagccccga ctattgccag ccagcctctg agcctcaggc ctgaggcctg taggcccgca 900
gcgggcggcg cagttcatac acggggcttg gatttcgctt gtgatattta tatttgggct 960
cctttggcgg ggacatgtgg cgtgctgctt ctgtcacttg ttattacact gtactgtaaa 1020
cgcgggcgaa aaaaattgct gtatattttt aagcagccat ttatgaggcc cgttcagacg 1080
acgcaggagg aggacggttg ctcttgcagg ttcccagaag aggaagaagg gggctgtgaa 1140
ttgcgggtta aattttcaag atccgcagac gctccagcat accaacaggg acaaaaccaa 1200
ctctataacg agctgaatct tggaagaagg gaggaatatg atgtgctgga taaacggcgc 1260
ggtagagatc cggagatggg cggaaaacca aggcgaaaaa accctcagga gggactctac 1320
aacgaactgc agaaagacaa aatggcggag gcttattccg aaataggcat gaagggcgag 1380
cggaggcgag ggaaagggca cgacggactg tatcaaggcc tctcaaccgc gactaaggat 1440
acgtacgacg ccctgcacat gcaggccctg cctccgaga 1479
<210> 259
<211> 1416
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 259
caggtacaat tgcaggagtc tggaggcggt gtggtgcaac ccggtcgcag cttgcgcctg 60
agttgtgctg cgtctggatt tacattttca tcttacggaa tgcattgggt acgccaggca 120
ccggggaaag gccttgaatg ggtggctgta atttcatacg atggttccaa caaatactat 180
gctgactcag tcaagggtcg atttacaatt agtcgggaca actccaagaa caccctttat 240
cttcaaatga attcccttag agcagaggat acggcggtct attactgtgg tggcagtggt 300
tatgcacttc atgatgatta ctatggcttg gatgtctggg ggcaagggac gcttgtaact 360
gtatcctctg gtggtggtgg tagtggtggg ggaggctccg gcggtggcgg ctctcaatct 420
gctctgactc aaccagcaag cgtatcaggg tcaccgggac agagtattac cataagttgc 480
acggggacct ctagcgatgt aggggggtat aattatgtat cttggtatca acaacacccc 540
gggaaagccc ctaaattgat gatctacgac gtgagcaatc gacctagtgg cgtatcaaat 600
cgcttctctg gtagcaagag tgggaatacg gcgtccctta ctattagcgg attgcaagca 660
gaagatgagg ccgattacta ctgcagctcc tatactagct cttctacatt gtacgtcttt 720
gggagcggaa caaaagtaac agtactcaca acaacacctg ccccgagacc gcctacacca 780
gccccgacta ttgccagcca gcctctgagc ctcaggcctg aggcctgtag gcccgcagcg 840
ggcggcgcag ttcatacacg gggcttggat ttcgcttgtg atatttatat ttgggctcct 900
ttggcgggga catgtggcgt gctgcttctg tcacttgtta ttacactgta ctgtaaacgc 960
gggcgaaaaa aattgctgta tatttttaag cagccattta tgaggcccgt tcagacgacg 1020
caggaggagg acggttgctc ttgcaggttc ccagaagagg aagaaggggg ctgtgaattg 1080
cgggttaaat tttcaagatc cgcagacgct ccagcatacc aacagggaca aaaccaactc 1140
tataacgagc tgaatcttgg aagaagggag gaatatgatg tgctggataa acggcgcggt 1200
agagatccgg agatgggcgg aaaaccaagg cgaaaaaacc ctcaggaggg actctacaac 1260
gaactgcaga aagacaaaat ggcggaggct tattccgaaa taggcatgaa gggcgagcgg 1320
aggcgaggga aagggcacga cggactgtat caaggcctct caaccgcgac taaggatacg 1380
tacgacgccc tgcacatgca ggccctgcct ccgaga 1416
<210> 260
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 260
caggtacaat tgcaggagtc tggaggcggt gtggtgcaac ccggtcgcag cttgcgcctg 60
agttgtgctg cgtctggatt tacattttca tcttacggaa tgcattgggt acgccaggca 120
ccggggaaag gccttgaatg ggtggctgta atttcatacg atggttccaa caaatactat 180
gctgactcag tcaagggtcg atttacaatt agtcgggaca actccaagaa caccctttat 240
cttcaaatga attcccttag agcagaggat acggcggtct attactgtgg tggcagtggt 300
tatgcacttc atgatgatta ctatggcttg gatgtctggg ggcaagggac gcttgtaact 360
gtatcctct 369
<210> 261
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 261
caatctgctc tgactcaacc agcaagcgta tcagggtcac cgggacagag tattaccata 60
agttgcacgg ggacctctag cgatgtaggg gggtataatt atgtatcttg gtatcaacaa 120
caccccggga aagcccctaa attgatgatc tacgacgtga gcaatcgacc tagtggcgta 180
tcaaatcgct tctctggtag caagagtggg aatacggcgt cccttactat tagcggattg 240
caagcagaag atgaggccga ttactactgc agctcctata ctagctcttc tacattgtac 300
gtctttggga gcggaacaaa agtaacagta ctc 333
<210> 262
<211> 345
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 262
gaagtgcaat tggtggaatc agggggagga cttgtgcagc ctggaggatc gctgagactg 60
tcatgtgccg tgtccggctt tgccctgtcc aaccacggga tgtcctgggt ccgccgcgcg 120
cctggaaagg gcctcgaatg ggtgtcgggt attgtgtaca gcggtagcac ctactatgcc 180
gcatccgtga aggggagatt caccatcagc cgggacaact ccaggaacac tctgtacctc 240
caaatgaatt cgctgaggcc agaggacact gccatctact actgctccgc gcatggcgga 300
gagtccgacg tctggggaca ggggaccacc gtgaccgtgt ctagc 345
<210> 263
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 263
gacatccagc tcacccagtc cccgagctcg ctgtccgcct ccgtgggaga tcgggtcacc 60
atcacgtgcc gcgccagcca gtcgatttcc tcctacctga actggtacca acagaagccc 120
ggaaaagccc cgaagcttct catctacgcc gcctcgagcc tgcagtcagg agtgccctca 180
cggttctccg gctccggttc cggtactgat ttcaccctga ccatttcctc cctgcaaccg 240
gaggacttcg ctacttacta ctgccagcag tcgtactcca ccccctacac tttcggacaa 300
ggcaccaagg tcgaaatcaa g 321
<210> 264
<400> 264
000
<210> 265
<400> 265
000
<210> 266
<400> 266
000
<210> 267
<400> 267
000
<210> 268
<400> 268
000
<210> 269
<400> 269
000
<210> 270
<400> 270
000
<210> 271
<400> 271
000
<210> 272
<400> 272
000
<210> 273
<400> 273
000
<210> 274
<400> 274
000
<210> 275
<211> 132
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 275
Asp Val Pro Asp Tyr Ala Ser Leu Gly Gly Pro Ser Ser Pro Lys Lys
1 5 10 15
Lys Arg Lys Val Ser Arg Gly Val Gln Val Glu Thr Ile Ser Pro Gly
20 25 30
Asp Gly Arg Thr Phe Pro Lys Arg Gly Gln Thr Cys Val Val His Tyr
35 40 45
Thr Gly Met Leu Glu Asp Gly Lys Lys Phe Asp Ser Ser Arg Asp Arg
50 55 60
Asn Lys Pro Phe Lys Phe Met Leu Gly Lys Gln Glu Val Ile Arg Gly
65 70 75 80
Trp Glu Glu Gly Val Ala Gln Met Ser Val Gly Gln Arg Ala Lys Leu
85 90 95
Thr Ile Ser Pro Asp Tyr Ala Tyr Gly Ala Thr Gly His Pro Gly Ile
100 105 110
Ile Pro Pro His Ala Thr Leu Val Phe Asp Val Glu Leu Leu Lys Leu
115 120 125
Glu Thr Ser Tyr
130
<210> 276
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 276
Val Gln Val Glu Thr Ile Ser Pro Gly Asp Gly Arg Thr Phe Pro Lys
1 5 10 15
Arg Gly Gln Thr Cys Val Val His Tyr Thr Gly Met Leu Glu Asp Gly
20 25 30
Lys Lys Phe Asp Ser Ser Arg Asp Arg Asn Lys Pro Phe Lys Phe Met
35 40 45
Leu Gly Lys Gln Glu Val Ile Arg Gly Trp Glu Glu Gly Val Ala Gln
50 55 60
Met Ser Val Gly Gln Arg Ala Lys Leu Thr Ile Ser Pro Asp Tyr Ala
65 70 75 80
Tyr Gly Ala Thr Gly His Pro Gly Ile Ile Pro Pro His Ala Thr Leu
85 90 95
Val Phe Asp Val Glu Leu Leu Lys Leu Glu Thr Ser
100 105
<210> 277
<211> 93
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 277
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Glu Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Thr Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys
85 90
<210> 278
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 278
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Ile Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Thr Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys Thr Ser
85 90 95
<210> 279
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 279
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Leu Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Thr Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys Thr Ser
85 90 95
<210> 280
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 280
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Glu Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Leu Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys Thr Ser
85 90 95
<210> 281
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<220>
<221> MOD_RES
<222> (12)..(12)
<223> Any amino acid
<220>
<221> MOD_RES
<222> (78)..(78)
<223> Any amino acid
<400> 281
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Xaa Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Xaa Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys Thr Ser
85 90 95
<210> 282
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 282
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Ile Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Leu Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys Thr Ser
85 90 95
<210> 283
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 283
Ile Leu Trp His Glu Met Trp His Glu Gly Leu Leu Glu Ala Ser Arg
1 5 10 15
Leu Tyr Phe Gly Glu Arg Asn Val Lys Gly Met Phe Glu Val Leu Glu
20 25 30
Pro Leu His Ala Met Met Glu Arg Gly Pro Gln Thr Leu Lys Glu Thr
35 40 45
Ser Phe Asn Gln Ala Tyr Gly Arg Asp Leu Met Glu Ala Gln Glu Trp
50 55 60
Cys Arg Lys Tyr Met Lys Ser Gly Asn Val Lys Asp Leu Leu Gln Ala
65 70 75 80
Trp Asp Leu Tyr Tyr His Val Phe Arg Arg Ile Ser Lys Thr Ser
85 90 95
<210> 284
<211> 1132
<212> PRT
<213> Homo sapiens
<400> 284
Met Pro Arg Ala Pro Arg Cys Arg Ala Val Arg Ser Leu Leu Arg Ser
1 5 10 15
His Tyr Arg Glu Val Leu Pro Leu Ala Thr Phe Val Arg Arg Leu Gly
20 25 30
Pro Gln Gly Trp Arg Leu Val Gln Arg Gly Asp Pro Ala Ala Phe Arg
35 40 45
Ala Leu Val Ala Gln Cys Leu Val Cys Val Pro Trp Asp Ala Arg Pro
50 55 60
Pro Pro Ala Ala Pro Ser Phe Arg Gln Val Ser Cys Leu Lys Glu Leu
65 70 75 80
Val Ala Arg Val Leu Gln Arg Leu Cys Glu Arg Gly Ala Lys Asn Val
85 90 95
Leu Ala Phe Gly Phe Ala Leu Leu Asp Gly Ala Arg Gly Gly Pro Pro
100 105 110
Glu Ala Phe Thr Thr Ser Val Arg Ser Tyr Leu Pro Asn Thr Val Thr
115 120 125
Asp Ala Leu Arg Gly Ser Gly Ala Trp Gly Leu Leu Leu Arg Arg Val
130 135 140
Gly Asp Asp Val Leu Val His Leu Leu Ala Arg Cys Ala Leu Phe Val
145 150 155 160
Leu Val Ala Pro Ser Cys Ala Tyr Gln Val Cys Gly Pro Pro Leu Tyr
165 170 175
Gln Leu Gly Ala Ala Thr Gln Ala Arg Pro Pro Pro His Ala Ser Gly
180 185 190
Pro Arg Arg Arg Leu Gly Cys Glu Arg Ala Trp Asn His Ser Val Arg
195 200 205
Glu Ala Gly Val Pro Leu Gly Leu Pro Ala Pro Gly Ala Arg Arg Arg
210 215 220
Gly Gly Ser Ala Ser Arg Ser Leu Pro Leu Pro Lys Arg Pro Arg Arg
225 230 235 240
Gly Ala Ala Pro Glu Pro Glu Arg Thr Pro Val Gly Gln Gly Ser Trp
245 250 255
Ala His Pro Gly Arg Thr Arg Gly Pro Ser Asp Arg Gly Phe Cys Val
260 265 270
Val Ser Pro Ala Arg Pro Ala Glu Glu Ala Thr Ser Leu Glu Gly Ala
275 280 285
Leu Ser Gly Thr Arg His Ser His Pro Ser Val Gly Arg Gln His His
290 295 300
Ala Gly Pro Pro Ser Thr Ser Arg Pro Pro Arg Pro Trp Asp Thr Pro
305 310 315 320
Cys Pro Pro Val Tyr Ala Glu Thr Lys His Phe Leu Tyr Ser Ser Gly
325 330 335
Asp Lys Glu Gln Leu Arg Pro Ser Phe Leu Leu Ser Ser Leu Arg Pro
340 345 350
Ser Leu Thr Gly Ala Arg Arg Leu Val Glu Thr Ile Phe Leu Gly Ser
355 360 365
Arg Pro Trp Met Pro Gly Thr Pro Arg Arg Leu Pro Arg Leu Pro Gln
370 375 380
Arg Tyr Trp Gln Met Arg Pro Leu Phe Leu Glu Leu Leu Gly Asn His
385 390 395 400
Ala Gln Cys Pro Tyr Gly Val Leu Leu Lys Thr His Cys Pro Leu Arg
405 410 415
Ala Ala Val Thr Pro Ala Ala Gly Val Cys Ala Arg Glu Lys Pro Gln
420 425 430
Gly Ser Val Ala Ala Pro Glu Glu Glu Asp Thr Asp Pro Arg Arg Leu
435 440 445
Val Gln Leu Leu Arg Gln His Ser Ser Pro Trp Gln Val Tyr Gly Phe
450 455 460
Val Arg Ala Cys Leu Arg Arg Leu Val Pro Pro Gly Leu Trp Gly Ser
465 470 475 480
Arg His Asn Glu Arg Arg Phe Leu Arg Asn Thr Lys Lys Phe Ile Ser
485 490 495
Leu Gly Lys His Ala Lys Leu Ser Leu Gln Glu Leu Thr Trp Lys Met
500 505 510
Ser Val Arg Gly Cys Ala Trp Leu Arg Arg Ser Pro Gly Val Gly Cys
515 520 525
Val Pro Ala Ala Glu His Arg Leu Arg Glu Glu Ile Leu Ala Lys Phe
530 535 540
Leu His Trp Leu Met Ser Val Tyr Val Val Glu Leu Leu Arg Ser Phe
545 550 555 560
Phe Tyr Val Thr Glu Thr Thr Phe Gln Lys Asn Arg Leu Phe Phe Tyr
565 570 575
Arg Lys Ser Val Trp Ser Lys Leu Gln Ser Ile Gly Ile Arg Gln His
580 585 590
Leu Lys Arg Val Gln Leu Arg Glu Leu Ser Glu Ala Glu Val Arg Gln
595 600 605
His Arg Glu Ala Arg Pro Ala Leu Leu Thr Ser Arg Leu Arg Phe Ile
610 615 620
Pro Lys Pro Asp Gly Leu Arg Pro Ile Val Asn Met Asp Tyr Val Val
625 630 635 640
Gly Ala Arg Thr Phe Arg Arg Glu Lys Arg Ala Glu Arg Leu Thr Ser
645 650 655
Arg Val Lys Ala Leu Phe Ser Val Leu Asn Tyr Glu Arg Ala Arg Arg
660 665 670
Pro Gly Leu Leu Gly Ala Ser Val Leu Gly Leu Asp Asp Ile His Arg
675 680 685
Ala Trp Arg Thr Phe Val Leu Arg Val Arg Ala Gln Asp Pro Pro Pro
690 695 700
Glu Leu Tyr Phe Val Lys Val Asp Val Thr Gly Ala Tyr Asp Thr Ile
705 710 715 720
Pro Gln Asp Arg Leu Thr Glu Val Ile Ala Ser Ile Ile Lys Pro Gln
725 730 735
Asn Thr Tyr Cys Val Arg Arg Tyr Ala Val Val Gln Lys Ala Ala His
740 745 750
Gly His Val Arg Lys Ala Phe Lys Ser His Val Ser Thr Leu Thr Asp
755 760 765
Leu Gln Pro Tyr Met Arg Gln Phe Val Ala His Leu Gln Glu Thr Ser
770 775 780
Pro Leu Arg Asp Ala Val Val Ile Glu Gln Ser Ser Ser Leu Asn Glu
785 790 795 800
Ala Ser Ser Gly Leu Phe Asp Val Phe Leu Arg Phe Met Cys His His
805 810 815
Ala Val Arg Ile Arg Gly Lys Ser Tyr Val Gln Cys Gln Gly Ile Pro
820 825 830
Gln Gly Ser Ile Leu Ser Thr Leu Leu Cys Ser Leu Cys Tyr Gly Asp
835 840 845
Met Glu Asn Lys Leu Phe Ala Gly Ile Arg Arg Asp Gly Leu Leu Leu
850 855 860
Arg Leu Val Asp Asp Phe Leu Leu Val Thr Pro His Leu Thr His Ala
865 870 875 880
Lys Thr Phe Leu Arg Thr Leu Val Arg Gly Val Pro Glu Tyr Gly Cys
885 890 895
Val Val Asn Leu Arg Lys Thr Val Val Asn Phe Pro Val Glu Asp Glu
900 905 910
Ala Leu Gly Gly Thr Ala Phe Val Gln Met Pro Ala His Gly Leu Phe
915 920 925
Pro Trp Cys Gly Leu Leu Leu Asp Thr Arg Thr Leu Glu Val Gln Ser
930 935 940
Asp Tyr Ser Ser Tyr Ala Arg Thr Ser Ile Arg Ala Ser Leu Thr Phe
945 950 955 960
Asn Arg Gly Phe Lys Ala Gly Arg Asn Met Arg Arg Lys Leu Phe Gly
965 970 975
Val Leu Arg Leu Lys Cys His Ser Leu Phe Leu Asp Leu Gln Val Asn
980 985 990
Ser Leu Gln Thr Val Cys Thr Asn Ile Tyr Lys Ile Leu Leu Leu Gln
995 1000 1005
Ala Tyr Arg Phe His Ala Cys Val Leu Gln Leu Pro Phe His Gln
1010 1015 1020
Gln Val Trp Lys Asn Pro Thr Phe Phe Leu Arg Val Ile Ser Asp
1025 1030 1035
Thr Ala Ser Leu Cys Tyr Ser Ile Leu Lys Ala Lys Asn Ala Gly
1040 1045 1050
Met Ser Leu Gly Ala Lys Gly Ala Ala Gly Pro Leu Pro Ser Glu
1055 1060 1065
Ala Val Gln Trp Leu Cys His Gln Ala Phe Leu Leu Lys Leu Thr
1070 1075 1080
Arg His Arg Val Thr Tyr Val Pro Leu Leu Gly Ser Leu Arg Thr
1085 1090 1095
Ala Gln Thr Gln Leu Ser Arg Lys Leu Pro Gly Thr Thr Leu Thr
1100 1105 1110
Ala Leu Glu Ala Ala Ala Asn Pro Ala Leu Pro Ser Asp Phe Lys
1115 1120 1125
Thr Ile Leu Asp
1130
<210> 285
<211> 253
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 285
Glu Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Met Leu Ser Asn
20 25 30
Ser Asp Thr Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Thr Tyr His Arg Ser Thr Trp Tyr Asp Asp Tyr Ala
50 55 60
Ser Ser Val Arg Gly Arg Val Ser Ile Asn Val Asp Thr Ser Lys Asn
65 70 75 80
Gln Tyr Ser Leu Gln Leu Asn Ala Val Thr Pro Glu Asp Thr Gly Val
85 90 95
Tyr Tyr Cys Ala Arg Val Arg Leu Gln Asp Gly Asn Ser Trp Ser Asp
100 105 110
Ala Phe Asp Val Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser
130 135 140
Ala Leu Thr Gln Pro Ala Ser Ala Ser Gly Ser Pro Gly Gln Ser Val
145 150 155 160
Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr
165 170 175
Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile
180 185 190
Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly
195 200 205
Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala
210 215 220
Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr
225 230 235 240
Leu Tyr Val Phe Gly Thr Gly Thr Gln Leu Thr Val Leu
245 250
<210> 286
<211> 127
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 286
Glu Val Gln Leu Gln Gln Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Ile Ser Gly Asp Ser Met Leu Ser Asn
20 25 30
Ser Asp Thr Trp Asn Trp Ile Arg Gln Ser Pro Ser Arg Gly Leu Glu
35 40 45
Trp Leu Gly Arg Thr Tyr His Arg Ser Thr Trp Tyr Asp Asp Tyr Ala
50 55 60
Ser Ser Val Arg Gly Arg Val Ser Ile Asn Val Asp Thr Ser Lys Asn
65 70 75 80
Gln Tyr Ser Leu Gln Leu Asn Ala Val Thr Pro Glu Asp Thr Gly Val
85 90 95
Tyr Tyr Cys Ala Arg Val Arg Leu Gln Asp Gly Asn Ser Trp Ser Asp
100 105 110
Ala Phe Asp Val Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120 125
<210> 287
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 287
Gln Ser Ala Leu Thr Gln Pro Ala Ser Ala Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Thr Gly Thr Gln Leu Thr Val Leu
100 105 110
<210> 288
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 288
Gly Asp Ser Met Leu Ser Asn Ser Asp Thr Trp Asn
1 5 10
<210> 289
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 289
Ser Asn Ser Asp Thr Trp Asn
1 5
<210> 290
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 290
Arg Thr Tyr His Arg Ser Thr Trp Tyr Asp Asp Tyr Ala Ser Ser Val
1 5 10 15
Arg Gly
<210> 291
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 291
Val Arg Leu Gln Asp Gly Asn Ser Trp Ser Asp Ala Phe Asp Val
1 5 10 15
<210> 292
<211> 465
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 292
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser Leu Ser Val Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser Arg Leu Thr Ile Ile
180 185 190
Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn Ser Leu Gln
195 200 205
Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val
225 230 235 240
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
245 250 255
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
260 265 270
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
275 280 285
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
290 295 300
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
305 310 315 320
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
325 330 335
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
340 345 350
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln
355 360 365
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
370 375 380
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
385 390 395 400
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
405 410 415
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
420 425 430
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
435 440 445
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
450 455 460
Arg
465
<210> 293
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 293
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser
<210> 294
<211> 465
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 294
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
245 250 255
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
260 265 270
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
275 280 285
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
290 295 300
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
305 310 315 320
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
325 330 335
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
340 345 350
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln
355 360 365
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
370 375 380
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
385 390 395 400
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
405 410 415
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
420 425 430
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
435 440 445
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
450 455 460
Arg
465
<210> 295
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 295
Asp Tyr Gly Val Ser
1 5
<210> 296
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 296
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser
1 5 10 15
<210> 297
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 297
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
1 5 10
<210> 298
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 298
Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn
1 5 10
<210> 299
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 299
His Thr Ser Arg Leu His Ser
1 5
<210> 300
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 300
Gln Gln Gly Asn Thr Leu Pro Tyr Thr
1 5
<210> 301
<211> 486
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 301
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu
20 25 30
Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln
35 40 45
Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr
50 55 60
Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val Pro
65 70 75 80
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile
85 90 95
Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly
100 105 110
Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
130 135 140
Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser
145 150 155 160
Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
165 170 175
Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly
180 185 190
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser
195 200 205
Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys
210 215 220
Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys
225 230 235 240
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
245 250 255
Thr Ser Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg
485
<210> 302
<211> 1458
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 302
atggccttac cagtgaccgc cttgctcctg ccgctggcct tgctgctcca cgccgccagg 60
ccggacatcc agatgacaca gactacatcc tccctgtctg cctctctggg agacagagtc 120
accatcagtt gcagggcaag tcaggacatt agtaaatatt taaattggta tcagcagaaa 180
ccagatggaa ctgttaaact cctgatctac catacatcaa gattacactc aggagtccca 240
tcaaggttca gtggcagtgg gtctggaaca gattattctc tcaccattag caacctggag 300
caagaagata ttgccactta cttttgccaa cagggtaata cgcttccgta cacgttcgga 360
ggggggacca agctggagat cacaggtggc ggtggctcgg gcggtggtgg gtcgggtggc 420
ggcggatctg aggtgaaact gcaggagtca ggacctggcc tggtggcgcc ctcacagagc 480
ctgtccgtca catgcactgt ctcaggggtc tcattacccg actatggtgt aagctggatt 540
cgccagcctc cacgaaaggg tctggagtgg ctgggagtaa tatggggtag tgaaaccaca 600
tactataatt cagctctcaa atccagactg accatcatca aggacaactc caagagccaa 660
gttttcttaa aaatgaacag tctgcaaact gatgacacag ccatttacta ctgtgccaaa 720
cattattact acggtggtag ctatgctatg gactactggg gccaaggaac ctcagtcacc 780
gtctcctcaa ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 840
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 900
agggggctgg acttcgcctg tgatatctac atctgggcgc ccttggccgg gacttgtggg 960
gtccttctcc tgtcactggt tatcaccctt tactgcaaac ggggcagaaa gaaactcctg 1020
tatatattca aacaaccatt tatgagacca gtacaaacta ctcaagagga agatggctgt 1080
agctgccgat ttccagaaga agaagaagga ggatgtgaac tgagagtgaa gttcagcagg 1140
agcgcagacg cccccgcgta caagcagggc cagaaccagc tctataacga gctcaatcta 1200
ggacgaagag aggagtacga tgttttggac aagagacgtg gccgggaccc tgagatgggg 1260
ggaaagccga gaaggaagaa ccctcaggaa ggcctgtaca atgaactgca gaaagataag 1320
atggcggagg cctacagtga gattgggatg aaaggcgagc gccggagggg caaggggcac 1380
gatggccttt accagggtct cagtacagcc accaaggaca cctacgacgc ccttcacatg 1440
caggccctgc cccctcgc 1458
<210> 303
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 303
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Lys Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser Leu Ser Val Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser Arg Leu Thr Ile Ile
180 185 190
Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn Ser Leu Gln
195 200 205
Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val
225 230 235 240
Ser Ser
<210> 304
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 304
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
1 5 10 15
<210> 305
<211> 726
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 305
gaaattgtga tgacccagtc acccgccact cttagccttt cacccggtga gcgcgcaacc 60
ctgtcttgca gagcctccca agacatctca aaatacctta attggtatca acagaagccc 120
ggacaggctc ctcgccttct gatctaccac accagccggc tccattctgg aatccctgcc 180
aggttcagcg gtagcggatc tgggaccgac tacaccctca ctatcagctc actgcagcca 240
gaggacttcg ctgtctattt ctgtcagcaa gggaacaccc tgccctacac ctttggacag 300
ggcaccaagc tcgagattaa aggtggaggt ggcagcggag gaggtgggtc cggcggtgga 360
ggaagccagg tccaactcca agaaagcgga ccgggtcttg tgaagccatc agaaactctt 420
tcactgactt gtactgtgag cggagtgtct ctccccgatt acggggtgtc ttggatcaga 480
cagccaccgg ggaagggtct ggaatggatt ggagtgattt ggggctctga gactacttac 540
taccaatcat ccctcaagtc acgcgtcacc atctcaaagg acaactctaa gaatcaggtg 600
tcactgaaac tgtcatctgt gaccgcagcc gacaccgccg tgtactattg cgctaagcat 660
tactattatg gcgggagcta cgcaatggat tactggggac agggtactct ggtcaccgtg 720
tccagc 726
<210> 306
<211> 486
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 306
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu
20 25 30
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
35 40 45
Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala
50 55 60
Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile
85 90 95
Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly
100 105 110
Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
145 150 155 160
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
165 170 175
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
180 185 190
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
195 200 205
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
210 215 220
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
225 230 235 240
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
245 250 255
Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg
485
<210> 307
<211> 1458
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 307
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
cccgaaattg tgatgaccca gtcacccgcc actcttagcc tttcacccgg tgagcgcgca 120
accctgtctt gcagagcctc ccaagacatc tcaaaatacc ttaattggta tcaacagaag 180
cccggacagg ctcctcgcct tctgatctac cacaccagcc ggctccattc tggaatccct 240
gccaggttca gcggtagcgg atctgggacc gactacaccc tcactatcag ctcactgcag 300
ccagaggact tcgctgtcta tttctgtcag caagggaaca ccctgcccta cacctttgga 360
cagggcacca agctcgagat taaaggtgga ggtggcagcg gaggaggtgg gtccggcggt 420
ggaggaagcc aggtccaact ccaagaaagc ggaccgggtc ttgtgaagcc atcagaaact 480
ctttcactga cttgtactgt gagcggagtg tctctccccg attacggggt gtcttggatc 540
agacagccac cggggaaggg tctggaatgg attggagtga tttggggctc tgagactact 600
tactaccaat catccctcaa gtcacgcgtc accatctcaa aggacaactc taagaatcag 660
gtgtcactga aactgtcatc tgtgaccgca gccgacaccg ccgtgtacta ttgcgctaag 720
cattactatt atggcgggag ctacgcaatg gattactggg gacagggtac tctggtcacc 780
gtgtccagca ccactacccc agcaccgagg ccacccaccc cggctcctac catcgcctcc 840
cagcctctgt ccctgcgtcc ggaggcatgt agacccgcag ctggtggggc cgtgcatacc 900
cggggtcttg acttcgcctg cgatatctac atttgggccc ctctggctgg tacttgcggg 960
gtcctgctgc tttcactcgt gatcactctt tactgtaagc gcggtcggaa gaagctgctg 1020
tacatcttta agcaaccctt catgaggcct gtgcagacta ctcaagagga ggacggctgt 1080
tcatgccggt tcccagagga ggaggaaggc ggctgcgaac tgcgcgtgaa attcagccgc 1140
agcgcagatg ctccagccta caagcagggg cagaaccagc tctacaacga actcaatctt 1200
ggtcggagag aggagtacga cgtgctggac aagcggagag gacgggaccc agaaatgggc 1260
gggaagccgc gcagaaagaa tccccaagag ggcctgtaca acgagctcca aaaggataag 1320
atggcagaag cctatagcga gattggtatg aaaggggaac gcagaagagg caaaggccac 1380
gacggactgt accagggact cagcaccgcc accaaggaca cctatgacgc tcttcacatg 1440
caggccctgc cgcctcgg 1458
<210> 308
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 308
Leu Ala Glu Ala Ala Ala Lys
1 5
<210> 309
<211> 284
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 309
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser Ile Thr Cys Pro Pro Pro Met Ser Val Glu His Ala Asp Ile
115 120 125
Trp Val Lys Ser Tyr Ser Leu Tyr Ser Arg Glu Arg Tyr Ile Cys Asn
130 135 140
Ser Gly Phe Lys Arg Lys Ala Gly Thr Ser Ser Leu Thr Glu Cys Val
145 150 155 160
Leu Asn Lys Ala Thr Asn Val Ala His Trp Thr Thr Pro Ser Leu Lys
165 170 175
Cys Ile Arg Asp Pro Ala Leu Val His Gln Arg Pro Ala Pro Pro Ser
180 185 190
Thr Val Thr Thr Ala Gly Val Thr Pro Gln Pro Glu Ser Leu Ser Pro
195 200 205
Ser Gly Lys Glu Pro Ala Ala Ser Ser Pro Ser Ser Asn Asn Thr Ala
210 215 220
Ala Thr Thr Ala Ala Ile Val Pro Gly Ser Gln Leu Met Pro Ser Lys
225 230 235 240
Ser Pro Ser Thr Gly Thr Thr Glu Ile Ser Ser His Glu Ser Ser His
245 250 255
Gly Thr Pro Ser Gln Thr Thr Ala Lys Asn Trp Glu Leu Thr Ala Ser
260 265 270
Ala Ser His Gln Pro Pro Gly Val Tyr Pro Gln Gly
275 280
<210> 310
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 310
Gly Val Ser Leu Pro Asp Tyr
1 5
<210> 311
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 311
Trp Gly Ser Glu Thr
1 5
<210> 312
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 312
Gly Val Ser Leu Pro Asp Tyr Gly
1 5
<210> 313
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 313
Ile Trp Gly Ser Glu Thr Thr
1 5
<210> 314
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 314
Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
1 5 10
<210> 315
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 315
caggtccaac tccaagaaag cggaccgggt cttgtgaagc catcagaaac tctttcactg 60
acttgtactg tgagcggagt gtctctcccc gattacgggg tgtcttggat cagacagcca 120
ccggggaagg gtctggaatg gattggagtg atttggggct ctgagactac ttactaccaa 180
tcatccctca agtcacgcgt caccatctca aaggacaact ctaagaatca ggtgtcactg 240
aaactgtcat ctgtgaccgc agccgacacc gccgtgtact attgcgctaa gcattactat 300
tatggcggga gctacgcaat ggattactgg ggacagggta ctctggtcac cgtgtccagc 360
<210> 316
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 316
Ser Gln Asp Ile Ser Lys Tyr
1 5
<210> 317
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 317
His Thr Ser
1
<210> 318
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 318
Gly Asn Thr Leu Pro Tyr
1 5
<210> 319
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 319
Gln Asp Ile Ser Lys Tyr
1 5
<210> 320
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 320
gaaattgtga tgacccagtc acccgccact cttagccttt cacccggtga gcgcgcaacc 60
ctgtcttgca gagcctccca agacatctca aaatacctta attggtatca acagaagccc 120
ggacaggctc ctcgccttct gatctaccac accagccggc tccattctgg aatccctgcc 180
aggttcagcg gtagcggatc tgggaccgac tacaccctca ctatcagctc actgcagcca 240
gaggacttcg ctgtctattt ctgtcagcaa gggaacaccc tgccctacac ctttggacag 300
ggcaccaagc tcgagattaa a 321
<210> 321
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 321
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gln Ser Ala
115 120 125
Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr
130 135 140
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
145 150 155 160
Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr
165 170 175
Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser
180 185 190
Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu
195 200 205
Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu
210 215 220
Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly
225 230 235 240
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
245 250 255
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
260 265 270
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
275 280 285
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
290 295 300
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
305 310 315 320
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
325 330 335
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
340 345 350
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
355 360 365
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
370 375 380
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
385 390 395 400
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
405 410 415
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
420 425 430
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
435 440 445
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
450 455 460
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
465 470 475 480
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
485 490
<210> 322
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 322
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
115 120 125
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
130 135 140
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
145 150 155 160
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
165 170 175
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
180 185 190
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
195 200 205
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
210 215 220
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
245 250 255
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
260 265 270
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
275 280 285
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
290 295 300
Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser
305 310 315 320
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
325 330 335
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
340 345 350
Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Gly
355 360 365
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
370 375 380
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
385 390 395 400
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu
405 410 415
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
420 425 430
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
435 440 445
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
450 455 460
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
485 490
<210> 323
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 323
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gln Val Gln
115 120 125
Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg Ser Leu Arg
130 135 140
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Gly Met His
145 150 155 160
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Val Ile
165 170 175
Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys Gly Arg
180 185 190
Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met
195 200 205
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Gly Gly Ser
210 215 220
Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
245 250 255
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu
260 265 270
Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile
275 280 285
Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
290 295 300
Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp
305 310 315 320
Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys
325 330 335
Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp
340 345 350
Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr
355 360 365
Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser
370 375 380
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
385 390 395 400
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
405 410 415
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
420 425 430
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
435 440 445
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
450 455 460
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
465 470 475 480
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
485 490
<210> 324
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 324
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
115 120 125
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
130 135 140
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
145 150 155 160
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
165 170 175
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
180 185 190
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
195 200 205
Ser Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly
210 215 220
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
225 230 235 240
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln
245 250 255
Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
260 265 270
Ser Ser Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
275 280 285
Glu Trp Val Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala
290 295 300
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
305 310 315 320
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
325 330 335
Tyr Tyr Cys Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly
340 345 350
Leu Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
355 360 365
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
370 375 380
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
385 390 395 400
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu
405 410 415
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
420 425 430
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
435 440 445
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
450 455 460
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
485 490
<210> 325
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 325
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
130 135 140
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
145 150 155 160
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
165 170 175
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
180 185 190
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
195 200 205
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
210 215 220
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
245 250 255
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
260 265 270
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
275 280 285
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
290 295 300
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
305 310 315 320
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
325 330 335
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
340 345 350
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
355 360 365
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu
370 375 380
Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile
385 390 395 400
Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
405 410 415
Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp
420 425 430
Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys
435 440 445
Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp
450 455 460
Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr
465 470 475 480
Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu
485 490
<210> 326
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 326
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu Gly
100 105 110
Gly Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val
115 120 125
Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser
130 135 140
Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly
145 150 155 160
Leu Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln
165 170 175
Ser Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn
180 185 190
Gln Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val
195 200 205
Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp
210 215 220
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
225 230 235 240
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
245 250 255
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
260 265 270
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
275 280 285
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
290 295 300
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
305 310 315 320
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
325 330 335
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
340 345 350
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
355 360 365
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
370 375 380
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
385 390 395 400
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
405 410 415
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
420 425 430
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
435 440 445
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
450 455 460
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
485 490
<210> 327
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 327
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
130 135 140
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
145 150 155 160
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
165 170 175
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
180 185 190
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
195 200 205
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
210 215 220
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
245 250 255
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met
260 265 270
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
275 280 285
Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr
290 295 300
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser
305 310 315 320
Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
325 330 335
Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
340 345 350
Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln
355 360 365
Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gln Ser Ala Leu
370 375 380
Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile
385 390 395 400
Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
405 410 415
Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp
420 425 430
Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys
435 440 445
Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp
450 455 460
Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr
465 470 475 480
Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu
485 490
<210> 328
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 328
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu Gly
100 105 110
Gly Gly Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser
115 120 125
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp
130 135 140
Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
145 150 155 160
Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala
165 170 175
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser
180 185 190
Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn
195 200 205
Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly
210 215 220
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
225 230 235 240
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
245 250 255
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
260 265 270
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu
275 280 285
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
290 295 300
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
305 310 315 320
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
325 330 335
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
340 345 350
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
355 360 365
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
370 375 380
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
385 390 395 400
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
405 410 415
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
420 425 430
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
435 440 445
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
450 455 460
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
485 490
<210> 329
<211> 485
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 329
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Asp Ile Gln
115 120 125
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
130 135 140
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
145 150 155 160
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
165 170 175
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
180 185 190
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
195 200 205
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
210 215 220
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
225 230 235 240
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu
245 250 255
Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
260 265 270
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp
275 280 285
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ser
290 295 300
Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe
305 310 315 320
Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn
325 330 335
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Arg Glu
340 345 350
Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly Gln Gly Thr Leu
355 360 365
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Glu Ile Val Met Thr Gln
370 375 380
Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser
385 390 395 400
Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln
405 410 415
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu
420 425 430
His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
435 440 445
Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr
450 455 460
Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Cys Gly Thr
465 470 475 480
Lys Leu Glu Ile Lys
485
<210> 330
<211> 485
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 330
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Glu Ile
115 120 125
Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg
130 135 140
Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn
145 150 155 160
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His
165 170 175
Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly
180 185 190
Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
195 200 205
Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe
210 215 220
Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
225 230 235 240
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
245 250 255
Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser
260 265 270
Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
275 280 285
Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile
290 295 300
Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val
305 310 315 320
Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser
325 330 335
Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr
340 345 350
Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
355 360 365
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
370 375 380
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
385 390 395 400
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
405 410 415
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
420 425 430
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
435 440 445
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
450 455 460
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Cys Gly Thr
465 470 475 480
Lys Val Glu Ile Lys
485
<210> 331
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 331
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 332
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 332
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 333
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 333
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 334
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 334
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 335
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 335
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 336
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 336
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu
85 90 95
Thr Phe Gly Cys Gly Thr Lys Val Glu Ile Lys
100 105
<210> 337
<211> 135
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 337
accactaccc cagcaccgag gccacccacc ccggctccta ccatcgcctc ccagcctctg 60
tccctgcgtc cggaggcatg tagacccgca gctggtgggg ccgtgcatac ccggggtctt 120
gacttcgcct gcgat 135
<210> 338
<211> 72
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 338
atctacattt gggcccctct ggctggtact tgcggggtcc tgctgctttc actcgtgatc 60
actctttact gt 72
<210> 339
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 339
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gln Ser Ala
115 120 125
Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr
130 135 140
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
145 150 155 160
Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr
165 170 175
Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser
180 185 190
Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu
195 200 205
Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu
210 215 220
Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly
225 230 235 240
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
245 250 255
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
260 265 270
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
275 280 285
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
290 295 300
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
305 310 315 320
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
325 330 335
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
340 345 350
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
355 360 365
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
370 375 380
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
385 390 395 400
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
405 410 415
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
420 425 430
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
435 440 445
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
450 455 460
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
465 470 475 480
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 340
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 340
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
115 120 125
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
130 135 140
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
145 150 155 160
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
165 170 175
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
180 185 190
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
195 200 205
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
210 215 220
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
245 250 255
Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser
260 265 270
Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn
275 280 285
Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met
290 295 300
Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser
305 310 315 320
Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln
325 330 335
Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser
340 345 350
Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Gly
355 360 365
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
370 375 380
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
385 390 395 400
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu
405 410 415
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
420 425 430
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
435 440 445
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
450 455 460
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 341
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 341
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gln Val Gln
115 120 125
Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg Ser Leu Arg
130 135 140
Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Gly Met His
145 150 155 160
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Val Ile
165 170 175
Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val Lys Gly Arg
180 185 190
Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met
195 200 205
Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Gly Gly Ser
210 215 220
Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
245 250 255
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu
260 265 270
Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile
275 280 285
Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
290 295 300
Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp
305 310 315 320
Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys
325 330 335
Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp
340 345 350
Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr
355 360 365
Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly Gly Gly Gly Ser
370 375 380
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
385 390 395 400
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
405 410 415
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
420 425 430
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
435 440 445
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
450 455 460
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
465 470 475 480
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 342
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 342
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
115 120 125
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
130 135 140
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
145 150 155 160
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
165 170 175
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
180 185 190
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
195 200 205
Ser Thr Leu Tyr Val Phe Gly Ser Gly Thr Lys Val Thr Val Leu Gly
210 215 220
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
225 230 235 240
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln
245 250 255
Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
260 265 270
Ser Ser Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
275 280 285
Glu Trp Val Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala
290 295 300
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
305 310 315 320
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
325 330 335
Tyr Tyr Cys Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly
340 345 350
Leu Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
355 360 365
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
370 375 380
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
385 390 395 400
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu
405 410 415
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
420 425 430
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
435 440 445
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
450 455 460
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 343
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 343
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
130 135 140
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
145 150 155 160
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
165 170 175
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
180 185 190
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
195 200 205
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
210 215 220
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
245 250 255
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
260 265 270
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
275 280 285
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
290 295 300
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
305 310 315 320
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
325 330 335
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
340 345 350
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
355 360 365
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu
370 375 380
Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile
385 390 395 400
Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
405 410 415
Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp
420 425 430
Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys
435 440 445
Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp
450 455 460
Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr
465 470 475 480
Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 344
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 344
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu Gly
100 105 110
Gly Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val
115 120 125
Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser
130 135 140
Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly
145 150 155 160
Leu Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln
165 170 175
Ser Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn
180 185 190
Gln Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val
195 200 205
Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp
210 215 220
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
225 230 235 240
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
245 250 255
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
260 265 270
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
275 280 285
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
290 295 300
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
305 310 315 320
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
325 330 335
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
340 345 350
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
355 360 365
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
370 375 380
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
385 390 395 400
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
405 410 415
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
420 425 430
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
435 440 445
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
450 455 460
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 345
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 345
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
115 120 125
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
130 135 140
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
145 150 155 160
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
165 170 175
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
180 185 190
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
195 200 205
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
210 215 220
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
245 250 255
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met
260 265 270
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
275 280 285
Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr
290 295 300
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser
305 310 315 320
Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
325 330 335
Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
340 345 350
Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln
355 360 365
Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gln Ser Ala Leu
370 375 380
Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln Ser Ile Thr Ile
385 390 395 400
Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
405 410 415
Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu Met Ile Tyr Asp
420 425 430
Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe Ser Gly Ser Lys
435 440 445
Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu Gln Ala Glu Asp
450 455 460
Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser Ser Thr Leu Tyr
465 470 475 480
Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 346
<211> 714
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 346
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Leu Tyr Val Phe Gly Cys Gly Thr Lys Val Thr Val Leu Gly
100 105 110
Gly Gly Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser
115 120 125
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp
130 135 140
Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
145 150 155 160
Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala
165 170 175
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser
180 185 190
Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn
195 200 205
Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly
210 215 220
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
225 230 235 240
Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys
245 250 255
Pro Ser Glu Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu
260 265 270
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu
275 280 285
Glu Trp Ile Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser
290 295 300
Ser Leu Lys Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln
305 310 315 320
Val Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr
325 330 335
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
340 345 350
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
355 360 365
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
370 375 380
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
385 390 395 400
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
405 410 415
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
420 425 430
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
435 440 445
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
450 455 460
Gly Gly Ser Gly Tyr Ala Leu His Asp Asp Tyr Tyr Gly Leu Asp Val
465 470 475 480
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala
485 490 495
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
500 505 510
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
515 520 525
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
530 535 540
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
545 550 555 560
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
565 570 575
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
580 585 590
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
595 600 605
Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn
610 615 620
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
625 630 635 640
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
645 650 655
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
660 665 670
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
675 680 685
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
690 695 700
Ala Leu His Met Gln Ala Leu Pro Pro Arg
705 710
<210> 347
<211> 708
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 347
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Cys Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Asp Ile Gln
115 120 125
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
130 135 140
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
145 150 155 160
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
165 170 175
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
180 185 190
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
195 200 205
Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly
210 215 220
Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly
225 230 235 240
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu
245 250 255
Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
260 265 270
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp
275 280 285
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ser
290 295 300
Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe
305 310 315 320
Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn
325 330 335
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Arg Glu
340 345 350
Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly Gln Gly Thr Leu
355 360 365
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Glu Ile Val Met Thr Gln
370 375 380
Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser
385 390 395 400
Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln
405 410 415
Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu
420 425 430
His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
435 440 445
Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr
450 455 460
Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Cys Gly Thr
465 470 475 480
Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
485 490 495
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
500 505 510
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
515 520 525
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
530 535 540
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
545 550 555 560
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
565 570 575
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
580 585 590
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
595 600 605
Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
610 615 620
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
625 630 635 640
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
645 650 655
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
660 665 670
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
675 680 685
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
690 695 700
Leu Pro Pro Arg
705
<210> 348
<211> 708
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 348
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Glu Trp Trp Gly Glu Ser Trp Leu Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Glu Ile
115 120 125
Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg
130 135 140
Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn
145 150 155 160
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His
165 170 175
Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly
180 185 190
Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp
195 200 205
Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe
210 215 220
Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly
225 230 235 240
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln
245 250 255
Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser
260 265 270
Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
275 280 285
Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile
290 295 300
Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val
305 310 315 320
Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser
325 330 335
Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr
340 345 350
Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu
355 360 365
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
370 375 380
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
385 390 395 400
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
405 410 415
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
420 425 430
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
435 440 445
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
450 455 460
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu Thr Phe Gly Cys Gly Thr
465 470 475 480
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
485 490 495
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
500 505 510
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
515 520 525
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
530 535 540
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
545 550 555 560
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
565 570 575
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
580 585 590
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
595 600 605
Tyr Lys Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
610 615 620
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
625 630 635 640
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
645 650 655
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
660 665 670
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
675 680 685
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
690 695 700
Leu Pro Pro Arg
705
<210> 349
<211> 486
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 349
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu
20 25 30
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
35 40 45
Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala
50 55 60
Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro
65 70 75 80
Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile
85 90 95
Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly
100 105 110
Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
145 150 155 160
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
165 170 175
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
180 185 190
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
195 200 205
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
210 215 220
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
225 230 235 240
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
245 250 255
Thr Leu Val Thr Val Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg
485
<210> 350
<211> 465
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 350
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
245 250 255
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
260 265 270
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
275 280 285
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
290 295 300
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
305 310 315 320
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
325 330 335
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
340 345 350
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
355 360 365
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
370 375 380
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
385 390 395 400
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
405 410 415
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
420 425 430
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
435 440 445
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
450 455 460
Arg
465
<210> 351
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
peptide"
<400> 351
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro
20
<210> 352
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 352
atgcttctcc tggtgacaag ccttctgctc tgtgagttac cacacccagc attcctcctg 60
atccca 66
<210> 353
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 353
atgctgctgc tggtgaccag cctgctgctg tgcgagctgc cccaccccgc ctttctgctg 60
atcccc 66
<210> 354
<211> 1461
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 354
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
cccgaaattg tgatgaccca gtcacccgcc actcttagcc tttcacccgg tgagcgcgca 120
accctgtctt gcagagcctc ccaagacatc tcaaaatacc ttaattggta tcaacagaag 180
cccggacagg ctcctcgcct tctgatctac cacaccagcc ggctccattc tggaatccct 240
gccaggttca gcggtagcgg atctgggacc gactacaccc tcactatcag ctcactgcag 300
ccagaggact tcgctgtcta tttctgtcag caagggaaca ccctgcccta cacctttgga 360
cagggcacca agctcgagat taaaggtgga ggtggcagcg gaggaggtgg gtccggcggt 420
ggaggaagcc aggtccaact ccaagaaagc ggaccgggtc ttgtgaagcc atcagaaact 480
ctttcactga cttgtactgt gagcggagtg tctctccccg attacggggt gtcttggatc 540
agacagccac cggggaaggg tctggaatgg attggagtga tttggggctc tgagactact 600
tactaccaat catccctcaa gtcacgcgtc accatctcaa aggacaactc taagaatcag 660
gtgtcactga aactgtcatc tgtgaccgca gccgacaccg ccgtgtacta ttgcgctaag 720
cattactatt atggcgggag ctacgcaatg gattactggg gacagggtac tctggtcacc 780
gtgtccagca ccactacccc agcaccgagg ccacccaccc cggctcctac catcgcctcc 840
cagcctctgt ccctgcgtcc ggaggcatgt agacccgcag ctggtggggc cgtgcatacc 900
cggggtcttg acttcgcctg cgatatctac atttgggccc ctctggctgg tacttgcggg 960
gtcctgctgc tttcactcgt gatcactctt tactgtaagc gcggtcggaa gaagctgctg 1020
tacatcttta agcaaccctt catgaggcct gtgcagacta ctcaagagga ggacggctgt 1080
tcatgccggt tcccagagga ggaggaaggc ggctgcgaac tgcgcgtgaa attcagccgc 1140
agcgcagatg ctccagccta ccagcagggg cagaaccagc tctacaacga actcaatctt 1200
ggtcggagag aggagtacga cgtgctggac aagcggagag gacgggaccc agaaatgggc 1260
gggaagccgc gcagaaagaa tccccaagag ggcctgtaca acgagctcca aaaggataag 1320
atggcagaag cctatagcga gattggtatg aaaggggaac gcagaagagg caaaggccac 1380
gacggactgt accagggact cagcaccgcc accaaggaca cctatgacgc tcttcacatg 1440
caggccctgc cgcctcggta a 1461
<210> 355
<211> 1458
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 355
atggccctcc ctgtcaccgc cctgctgctt ccgctggctc ttctgctcca cgccgctcgg 60
cccgaaattg tgatgaccca gtcacccgcc actcttagcc tttcacccgg tgagcgcgca 120
accctgtctt gcagagcctc ccaagacatc tcaaaatacc ttaattggta tcaacagaag 180
cccggacagg ctcctcgcct tctgatctac cacaccagcc ggctccattc tggaatccct 240
gccaggttca gcggtagcgg atctgggacc gactacaccc tcactatcag ctcactgcag 300
ccagaggact tcgctgtcta tttctgtcag caagggaaca ccctgcccta cacctttgga 360
cagggcacca agctcgagat taaaggtgga ggtggcagcg gaggaggtgg gtccggcggt 420
ggaggaagcc aggtccaact ccaagaaagc ggaccgggtc ttgtgaagcc atcagaaact 480
ctttcactga cttgtactgt gagcggagtg tctctccccg attacggggt gtcttggatc 540
agacagccac cggggaaggg tctggaatgg attggagtga tttggggctc tgagactact 600
tactaccaat catccctcaa gtcacgcgtc accatctcaa aggacaactc taagaatcag 660
gtgtcactga aactgtcatc tgtgaccgca gccgacaccg ccgtgtacta ttgcgctaag 720
cattactatt atggcgggag ctacgcaatg gattactggg gacagggtac tctggtcacc 780
gtgtccagca ccactacccc agcaccgagg ccacccaccc cggctcctac catcgcctcc 840
cagcctctgt ccctgcgtcc ggaggcatgt agacccgcag ctggtggggc cgtgcatacc 900
cggggtcttg acttcgcctg cgatatctac atttgggccc ctctggctgg tacttgcggg 960
gtcctgctgc tttcactcgt gatcactctt tactgtaagc gcggtcggaa gaagctgctg 1020
tacatcttta agcaaccctt catgaggcct gtgcagacta ctcaagagga ggacggctgt 1080
tcatgccggt tcccagagga ggaggaaggc ggctgcgaac tgcgcgtgaa attcagccgc 1140
agcgcagatg ctccagccta ccagcagggg cagaaccagc tctacaacga actcaatctt 1200
ggtcggagag aggagtacga cgtgctggac aagcggagag gacgggaccc agaaatgggc 1260
gggaagccgc gcagaaagaa tccccaagag ggcctgtaca acgagctcca aaaggataag 1320
atggcagaag cctatagcga gattggtatg aaaggggaac gcagaagagg caaaggccac 1380
gacggactgt accagggact cagcaccgcc accaaggaca cctatgacgc tcttcacatg 1440
caggccctgc cgcctcgg 1458
<210> 356
<211> 1398
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 356
gaaattgtga tgacccagtc acccgccact cttagccttt cacccggtga gcgcgcaacc 60
ctgtcttgca gagcctccca agacatctca aaatacctta attggtatca acagaagccc 120
ggacaggctc ctcgccttct gatctaccac accagccggc tccattctgg aatccctgcc 180
aggttcagcg gtagcggatc tgggaccgac tacaccctca ctatcagctc actgcagcca 240
gaggacttcg ctgtctattt ctgtcagcaa gggaacaccc tgccctacac ctttggacag 300
ggcaccaagc tcgagattaa aggtggaggt ggcagcggag gaggtgggtc cggcggtgga 360
ggaagccagg tccaactcca agaaagcgga ccgggtcttg tgaagccatc agaaactctt 420
tcactgactt gtactgtgag cggagtgtct ctccccgatt acggggtgtc ttggatcaga 480
cagccaccgg ggaagggtct ggaatggatt ggagtgattt ggggctctga gactacttac 540
taccaatcat ccctcaagtc acgcgtcacc atctcaaagg acaactctaa gaatcaggtg 600
tcactgaaac tgtcatctgt gaccgcagcc gacaccgccg tgtactattg cgctaagcat 660
tactattatg gcgggagcta cgcaatggat tactggggac agggtactct ggtcaccgtg 720
tccagcacca ctaccccagc accgaggcca cccaccccgg ctcctaccat cgcctcccag 780
cctctgtccc tgcgtccgga ggcatgtaga cccgcagctg gtggggccgt gcatacccgg 840
ggtcttgact tcgcctgcga tatctacatt tgggcccctc tggctggtac ttgcggggtc 900
ctgctgcttt cactcgtgat cactctttac tgtaagcgcg gtcggaagaa gctgctgtac 960
atctttaagc aacccttcat gaggcctgtg cagactactc aagaggagga cggctgttca 1020
tgccggttcc cagaggagga ggaaggcggc tgcgaactgc gcgtgaaatt cagccgcagc 1080
gcagatgctc cagcctacca gcaggggcag aaccagctct acaacgaact caatcttggt 1140
cggagagagg agtacgacgt gctggacaag cggagaggac gggacccaga aatgggcggg 1200
aagccgcgca gaaagaatcc ccaagagggc ctgtacaacg agctccaaaa ggataagatg 1260
gcagaagcct atagcgagat tggtatgaaa ggggaacgca gaagaggcaa aggccacgac 1320
ggactgtacc agggactcag caccgccacc aaggacacct atgacgctct tcacatgcag 1380
gccctgccgc ctcggtaa 1398
<210> 357
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 357
caggtccagc tgcaggaatc aggaccaggg ctggtgaaac ctagcgaaac tctgagtctg 60
acttgtaccg tctccggggt gtctctgcca gactacggcg tgagctggat cagacagccc 120
cctggcaagt gcctggagtg gatcggcgtg atctggggct ccgagaccac atactatcag 180
agctccctga agtctcgggt gaccatctcc aaggacaact ctaagaatca ggtgagcctg 240
aagctgtcta gcgtgaccgc cgccgataca gccgtgtact attgtgccaa gcactactat 300
tacggcggct cctatgccat ggattactgg ggccagggca ccctggtgac agtgtcctct 360
<210> 358
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 358
caggtccagc tgcaggaatc cggcccagga ctggtgaagc ctagcgagac cctgtccctg 60
acctgcacag tgagcggcgt gtccctgccc gattacggcg tgagctggat cagacagccc 120
cctggcaagt gtctggagtg gatcggcgtg atctggggct ctgagaccac atactatcag 180
tcctctctga agagcagggt gaccatctct aaggacaaca gcaagaatca ggtgtccctg 240
aagctgagct ccgtgaccgc agcagataca gccgtgtact attgcgccaa gcactactat 300
tacggcggct cctatgctat ggattattgg gggcagggca ctctggtcac tgtctcatca 360
<210> 359
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 359
caggtgcagc tgcaggaatc tggacccgga ctggtgaaac ctagtgaaac tctgtctctg 60
acttgtaccg tctcaggggt ctcactgcca gactacggcg tgtcctggat cagacagccc 120
cctggcaagt gcctggagtg gatcggcgtg atctggggct ctgagaccac atactatcag 180
agctccctga agagccgggt gaccatctcc aaggacaact ctaagaatca ggtgtccctg 240
aagctgtcta gcgtgaccgc cgccgataca gccgtgtact attgtgccaa gcactactat 300
tacggcggca gctatgccat ggattactgg ggccagggca ccctggtgac agtgtcctct 360
<210> 360
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 360
caggtccagc tgcaggaaag cggcccagga ctggtgaagc ctagcgagac cctgtccctg 60
acctgcacag tgagcggcgt gtccctgcct gattacggcg tgtcctggat cagacagccc 120
cctggcaagt gtctggagtg gatcggcgtg atctggggct ccgagaccac atactatcag 180
tcctctctga agtctagggt gacaatctct aaggacaaca gcaagaatca ggtgagcctg 240
aagctgagct ccgtgaccgc agcagataca gccgtgtact attgtgccaa gcactactat 300
tacggcggct cttatgctat ggattattgg gggcagggca ctctggtcac tgtctcaagc 360
<210> 361
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 361
caggtgcagc tgcaggagag cggcccagga ctggtgaagc cttccgagac actgtctctg 60
acctgtacag tgagcggcgt gtccctgccc gactacggcg tgtcctggat cagacagcca 120
cctggcaagg gactggagtg gatcggcgtg atctggggca gcgagaccac atactatcag 180
agctccctga agtccagggt gaccatcagc aaggacaact ccaagaatca ggtgagcctg 240
aagctgtcta gcgtgaccgc cgccgataca gccgtgtact attgcgccaa gcactactat 300
tacggcggct cctatgccat ggattactgg ggccagggca ccctggtcac agtgtcctct 360
<210> 362
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 362
caggtgcagc tgcaggagtc tggcccagga ctggtgaagc cttctgagac cctgagcctg 60
acctgcacag tgtccggcgt gtctctgccc gattacggcg tgtcctggat cagacagcca 120
cctggcaagg gactggagtg gatcggcgtg atctggggct ctgagaccac atactatcag 180
tctagcctga agagccgggt gacaatctcc aaggacaact ctaagaatca ggtgtccctg 240
aagctgtcct ctgtgaccgc cgccgataca gccgtgtact attgtgccaa gcactactat 300
tacggcggca gctatgccat ggactactgg ggccagggca ccctggtgac agtgagctcc 360
<210> 363
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 363
caggtgcagc tgcaggagtc tggcccagga ctggtgaagc cttctgagac cctgagcctg 60
acctgcacag tgagcggcgt gtccctgccc gattacggcg tgtcctggat cagacagcca 120
cctggcaagg gactggagtg gatcggcgtg atctggggca gcgagaccac atactatcag 180
tcctctctga agtccagggt gacaatctcc aaggacaact ctaagaatca ggtgagcctg 240
aagctgagct ccgtgaccgc agcagataca gccgtgtact attgcgccaa gcactactat 300
tacggcggct cctatgccat ggactactgg ggccagggca ccctggtcac agtgtctagc 360
<210> 364
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 364
caggtgcagc tgcaggagtc cggcccagga ctggtgaagc cttccgagac actgtctctg 60
acctgtacag tgtccggcgt gtctctgccc gactacggcg tgagctggat cagacagcca 120
cctggcaagg gactggagtg gatcggcgtg atctggggct ctgagaccac atactatcag 180
tcctctctga agagccgggt gaccatcagc aaggacaact ccaagaatca ggtgtccctg 240
aagctgagct ccgtgaccgc agcagataca gccgtgtact attgcgccaa gcactactat 300
tacggcggca gctatgccat ggattactgg ggccagggca ccctggtgac agtgtctagc 360
<210> 365
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 365
gagatcgtga tgacccagag cccagccaca ctgagcctgt ccccaggaga gagggccaca 60
ctgtcttgta gagccagcca ggatatctcc aagtatctga actggtacca gcagaagcct 120
ggacaggcac caaggctgct gatctaccac acctctagac tgcacagcgg catccctgcc 180
aggttttctg gcagcggctc cggcacagac tataccctga caatctctag cctgcagcca 240
gaggatttcg ccgtgtactt ttgtcagcag ggcaatactc tgccatacac ctttggatgc 300
ggaactaaac tggaaatcaa g 321
<210> 366
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 366
gaaattgtga tgacccagtc ccccgctact ctgtctctgt cccccggaga acgggctact 60
ctgtcttgtc gcgcttccca ggatattagc aagtacctga actggtatca gcagaagcca 120
ggacaggcac caaggctgct gatctaccac acctctcgcc tgcacagcgg catccctgca 180
cggttctctg gcagcggctc cggcacagac tacaccctga caatcagctc cctgcagcct 240
gaggatttcg ccgtgtactt ttgccagcag ggcaataccc tgccatatac atttggctgt 300
ggcaccaagc tggagatcaa g 321
<210> 367
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 367
gagatcgtga tgacccagtc cccagccaca ctgagcctgt ccccaggaga gagggccacc 60
ctgtcttgta gagccagcca ggatatctcc aagtatctga actggtacca gcagaagcct 120
ggacaggcac caaggctgct gatctaccac acctctagac tgcacagcgg catccctgcc 180
aggttttctg gcagcggctc cggcacagac tataccctga caatctctag cctgcagcca 240
gaggatttcg ccgtgtactt ttgtcagcag ggaaatactc tgccatacac ctttggatgc 300
ggaactaaac tggaaatcaa g 321
<210> 368
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 368
gagattgtga tgacccagtc ccccgccacc ctgagtctga gccccggaga acgagctacc 60
ctgagttgcc gagcttccca ggacatttcc aagtacctga actggtatca gcagaagcca 120
ggacaggcac caaggctgct gatctaccac acctctcgcc tgcacagcgg catcccagca 180
cggttctctg gcagcggctc cggcacagac tacaccctga caatcagctc cctgcagcct 240
gaggatttcg ccgtgtactt ttgccagcag ggcaataccc tgccatatac atttggctgt 300
ggcaccaagc tggagatcaa g 321
<210> 369
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 369
gagatcgtga tgacccagtc tccagccaca ctgtctctga gcccaggaga gagggccacc 60
ctgtcttgcc gcgccagcca ggatatctcc aagtatctga actggtacca gcagaagcca 120
ggacaggcac caaggctgct gatctaccac acctctcgcc tgcacagcgg catcccagca 180
cggttctccg gctctggcag cggcacagac tacaccctga caatctcctc tctgcagccc 240
gaggatttcg ccgtgtattt ttgccagcag ggcaataccc tgccttacac atttggccag 300
ggcaccaagc tggagatcaa g 321
<210> 370
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 370
gagatcgtga tgacccagag cccagccaca ctgagcctgt ccccaggaga gagggccacc 60
ctgagctgca gagcctccca ggatatctct aagtatctga actggtacca gcagaagcct 120
ggacaggcac caaggctgct gatctaccac accagcagac tgcactccgg catccctgca 180
aggttctctg gcagcggctc cggcacagac tacaccctga caatctctag cctgcagcct 240
gaggatttcg ccgtgtattt ttgtcagcag ggcaataccc tgccatacac atttggccag 300
ggcaccaagc tggagatcaa g 321
<210> 371
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 371
gagatcgtga tgacccagag cccagccaca ctgtctctga gcccaggaga gagggccacc 60
ctgagctgtc gcgcctccca ggatatctct aagtatctga actggtacca gcagaagcca 120
ggacaggcac caaggctgct gatctaccac accagccgcc tgcactccgg catcccagca 180
cggttctccg gctctggcag cggcacagac tacaccctga caatctcctc tctgcagccc 240
gaggatttcg ccgtgtattt ttgccagcag ggcaataccc tgccttacac atttggccag 300
ggcaccaagc tggagatcaa g 321
<210> 372
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 372
gagatcgtga tgacccagtc tccagccaca ctgagcctgt ccccaggaga gagggccacc 60
ctgtcttgca gagccagcca ggatatctcc aagtatctga actggtacca gcagaagcct 120
ggacaggcac caaggctgct gatctaccac acctctagac tgcacagcgg catcccagca 180
aggttctctg gcagcggctc cggcacagac tacaccctga caatctctag cctgcagcct 240
gaggatttcg ccgtgtattt ttgccagcag ggcaataccc tgccatacac atttggccag 300
ggcaccaagc tggagatcaa g 321
<210> 373
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 373
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Ser Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser
<210> 374
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 374
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Ser Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala
130 135 140
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala
145 150 155 160
Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly
180 185 190
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln
210 215 220
Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys
<210> 375
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 375
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala
130 135 140
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala
145 150 155 160
Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly
180 185 190
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln
210 215 220
Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys
<210> 376
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 376
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
115 120 125
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
130 135 140
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
145 150 155 160
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
165 170 175
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Ser Ser Ser Leu Lys Ser
180 185 190
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
195 200 205
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
210 215 220
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
225 230 235 240
Thr Leu Val Thr Val Ser Ser
245
<210> 377
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 377
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
115 120 125
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
130 135 140
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
145 150 155 160
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
165 170 175
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys Ser
180 185 190
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
195 200 205
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
210 215 220
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
225 230 235 240
Thr Leu Val Thr Val Ser Ser
245
<210> 378
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 378
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Ser Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met
130 135 140
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
145 150 155 160
Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser
180 185 190
Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
210 215 220
Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Leu Glu Ile Lys
245
<210> 379
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 379
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Gln Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met
130 135 140
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
145 150 155 160
Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser
180 185 190
Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
210 215 220
Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Leu Glu Ile Lys
245
<210> 380
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 380
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
115 120 125
Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr
130 135 140
Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly
145 150 155 160
Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly
165 170 175
Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ser Leu Lys Ser
180 185 190
Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys
195 200 205
Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys
210 215 220
His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly
225 230 235 240
Thr Leu Val Thr Val Ser Ser
245
<210> 381
<211> 247
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 381
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met
130 135 140
Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr
145 150 155 160
Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr
165 170 175
Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser
180 185 190
Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
195 200 205
Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala
210 215 220
Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln
225 230 235 240
Gly Thr Lys Leu Glu Ile Lys
245
<210> 382
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 382
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu
115 120 125
Ser Gly Pro Gly Leu Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys
130 135 140
Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser Trp Ile Arg
145 150 155 160
Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile Gly Val Ile Trp Gly Ser
165 170 175
Glu Thr Thr Tyr Tyr Asn Ser Ser Leu Lys Ser Arg Val Thr Ile Ser
180 185 190
Lys Asp Asn Ser Lys Asn Gln Val Ser Leu Lys Leu Ser Ser Val Thr
195 200 205
Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala Lys His Tyr Tyr Tyr Gly
210 215 220
Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
225 230 235 240
Ser Ser
<210> 383
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 383
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr
20 25 30
Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ser Leu Lys
50 55 60
Ser Arg Val Thr Ile Ser Lys Asp Asn Ser Lys Asn Gln Val Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala
130 135 140
Thr Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala
145 150 155 160
Ser Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly
180 185 190
Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu
195 200 205
Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Val Tyr Phe Cys Gln
210 215 220
Gln Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys
<210> 384
<211> 1467
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 384
atgcttctcc tggtgacaag ccttctgctc tgtgagttac cacacccagc attcctcctg 60
atcccagaca tccagatgac acagactaca tcctccctgt ctgcctctct gggagacaga 120
gtcaccatca gttgcagggc aagtcaggac attagtaaat atttaaattg gtatcagcag 180
aaaccagatg gaactgttaa actcctgatc taccatacat caagattaca ctcaggagtc 240
ccatcaaggt tcagtggcag tgggtctgga acagattatt ctctcaccat tagcaacctg 300
gagcaagaag atattgccac ttacttttgc caacagggta atacgcttcc gtacacgttc 360
ggagggggga ctaagttgga aataacaggc tccacctctg gatccggcaa gcccggatct 420
ggcgagggat ccaccaaggg cgaggtgaaa ctgcaggagt caggacctgg cctggtggcg 480
ccctcacaga gcctgtccgt cacatgcact gtctcagggg tctcattacc cgactatggt 540
gtaagctgga ttcgccagcc tccacgaaag ggtctggagt ggctgggagt aatatggggt 600
agtgaaacca catactataa ttcagctctc aaatccagac tgaccatcat caaggacaac 660
tccaagagcc aagttttctt aaaaatgaac agtctgcaaa ctgatgacac agccatttac 720
tactgtgcca aacattatta ctacggtggt agctatgcta tggactactg gggtcaagga 780
acctcagtca ccgtctcctc agcggccgca attgaagtta tgtatcctcc tccttaccta 840
gacaatgaga agagcaatgg aaccattatc catgtgaaag ggaaacacct ttgtccaagt 900
cccctatttc ccggaccttc taagcccttt tgggtgctgg tggtggttgg gggagtcctg 960
gcttgctata gcttgctagt aacagtggcc tttattattt tctgggtgag gagtaagagg 1020
agcaggctcc tgcacagtga ctacatgaac atgactcccc gccgccccgg gcccacccgc 1080
aagcattacc agccctatgc cccaccacgc gacttcgcag cctatcgctc cagagtgaag 1140
ttcagcagga gcgcagacgc ccccgcgtac cagcagggcc agaaccagct ctataacgag 1200
ctcaatctag gacgaagaga ggagtacgat gttttggaca agagacgtgg ccgggaccct 1260
gagatggggg gaaagccgag aaggaagaac cctcaggaag gcctgtacaa tgaactgcag 1320
aaagataaga tggcggaggc ctacagtgag attgggatga aaggcgagcg ccggaggggc 1380
aaggggcacg atggccttta ccagggtctc agtacagcca ccaaggacac ctacgacgcc 1440
cttcacatgc aggccctgcc ccctcgc 1467
<210> 385
<211> 489
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 385
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser
20 25 30
Leu Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser
35 40 45
Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly
50 55 60
Thr Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr
85 90 95
Ile Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln
100 105 110
Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile
115 120 125
Thr Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser
130 135 140
Thr Lys Gly Glu Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala
145 150 155 160
Pro Ser Gln Ser Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu
165 170 175
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu
180 185 190
Glu Trp Leu Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser
195 200 205
Ala Leu Lys Ser Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln
210 215 220
Val Phe Leu Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr
225 230 235 240
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
245 250 255
Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Ala Ala Ala Ile Glu
260 265 270
Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn Gly Thr
275 280 285
Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu Phe Pro
290 295 300
Gly Pro Ser Lys Pro Phe Trp Val Leu Val Val Val Gly Gly Val Leu
305 310 315 320
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
325 330 335
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
340 345 350
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
355 360 365
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser
370 375 380
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
385 390 395 400
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
405 410 415
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
420 425 430
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
435 440 445
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
450 455 460
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
465 470 475 480
Leu His Met Gln Ala Leu Pro Pro Arg
485
<210> 386
<211> 801
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 386
atgcttctcc tggtgacaag ccttctgctc tgtgagttac cacacccagc attcctcctg 60
atcccagaca tccagatgac acagactaca tcctccctgt ctgcctctct gggagacaga 120
gtcaccatca gttgcagggc aagtcaggac attagtaaat atttaaattg gtatcagcag 180
aaaccagatg gaactgttaa actcctgatc taccatacat caagattaca ctcaggagtc 240
ccatcaaggt tcagtggcag tgggtctgga acagattatt ctctcaccat tagcaacctg 300
gagcaagaag atattgccac ttacttttgc caacagggta atacgcttcc gtacacgttc 360
ggagggggga ctaagttgga aataacaggc tccacctctg gatccggcaa gcccggatct 420
ggcgagggat ccaccaaggg cgaggtgaaa ctgcaggagt caggacctgg cctggtggcg 480
ccctcacaga gcctgtccgt cacatgcact gtctcagggg tctcattacc cgactatggt 540
gtaagctgga ttcgccagcc tccacgaaag ggtctggagt ggctgggagt aatatggggt 600
agtgaaacca catactataa ttcagctctc aaatccagac tgaccatcat caaggacaac 660
tccaagagcc aagttttctt aaaaatgaac agtctgcaaa ctgatgacac agccatttac 720
tactgtgcca aacattatta ctacggtggt agctatgcta tggactactg gggtcaagga 780
acctcagtca ccgtctcctc a 801
<210> 387
<211> 266
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 387
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser
20 25 30
Leu Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser
35 40 45
Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly
50 55 60
Thr Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr
85 90 95
Ile Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln
100 105 110
Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile
115 120 125
Thr Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser
130 135 140
Thr Lys Gly Glu Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala
145 150 155 160
Pro Ser Gln Ser Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu
165 170 175
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu
180 185 190
Glu Trp Leu Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser
195 200 205
Ala Leu Lys Ser Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln
210 215 220
Val Phe Leu Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr
225 230 235 240
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
245 250 255
Trp Gly Gln Gly Thr Ser Val Thr Val Ser
260 265
<210> 388
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 388
gacatccaga tgacacagac tacatcctcc ctgtctgcct ctctgggaga cagagtcacc 60
atcagttgca gggcaagtca ggacattagt aaatatttaa attggtatca gcagaaacca 120
gatggaactg ttaaactcct gatctaccat acatcaagat tacactcagg agtcccatca 180
aggttcagtg gcagtgggtc tggaacagat tattctctca ccattagcaa cctggagcaa 240
gaagatattg ccacttactt ttgccaacag ggtaatacgc ttccgtacac gttcggaggg 300
gggactaagt tggaaataac aggctccacc tctggatccg gcaagcccgg atctggcgag 360
ggatccacca agggcgaggt gaaactgcag gagtcaggac ctggcctggt ggcgccctca 420
cagagcctgt ccgtcacatg cactgtctca ggggtctcat tacccgacta tggtgtaagc 480
tggattcgcc agcctccacg aaagggtctg gagtggctgg gagtaatatg gggtagtgaa 540
accacatact ataattcagc tctcaaatcc agactgacca tcatcaagga caactccaag 600
agccaagttt tcttaaaaat gaacagtctg caaactgatg acacagccat ttactactgt 660
gccaaacatt attactacgg tggtagctat gctatggact actggggtca aggaacctca 720
gtcaccgtct cctcagcggc cgcaattgaa gttatgtatc ctcctcctta cctagacaat 780
gagaagagca atggaaccat tatccatgtg aaagggaaac acctttgtcc aagtccccta 840
tttcccggac cttctaagcc cttttgggtg ctggtggtgg ttgggggagt cctggcttgc 900
tatagcttgc tagtaacagt ggcctttatt attttctggg tgaggagtaa gaggagcagg 960
ctcctgcaca gtgactacat gaacatgact ccccgccgcc ccgggcccac ccgcaagcat 1020
taccagccct atgccccacc acgcgacttc gcagcctatc gctccagagt gaagttcagc 1080
aggagcgcag acgcccccgc gtaccagcag ggccagaacc agctctataa cgagctcaat 1140
ctaggacgaa gagaggagta cgatgttttg gacaagagac gtggccggga ccctgagatg 1200
gggggaaagc cgagaaggaa gaaccctcag gaaggcctgt acaatgaact gcagaaagat 1260
aagatggcgg aggcctacag tgagattggg atgaaaggcg agcgccggag gggcaagggg 1320
cacgatggcc tttaccaggg tctcagtaca gccaccaagg acacctacga cgcccttcac 1380
atgcaggccc tgccccctcg c 1401
<210> 389
<211> 467
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 389
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr Gly Ser Thr Ser Gly
100 105 110
Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys Gly Glu Val Lys
115 120 125
Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser Leu Ser
130 135 140
Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
145 150 155 160
Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly Val Ile
165 170 175
Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser Arg Leu
180 185 190
Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn
195 200 205
Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys His Tyr
210 215 220
Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser
225 230 235 240
Val Thr Val Ser Ser Ala Ala Ala Ile Glu Val Met Tyr Pro Pro Pro
245 250 255
Tyr Leu Asp Asn Glu Lys Ser Asn Gly Thr Ile Ile His Val Lys Gly
260 265 270
Lys His Leu Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser Lys Pro Phe
275 280 285
Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu
290 295 300
Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg
305 310 315 320
Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro
325 330 335
Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala
340 345 350
Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
355 360 365
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
370 375 380
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
385 390 395 400
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
405 410 415
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
420 425 430
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
435 440 445
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
450 455 460
Pro Pro Arg
465
<210> 390
<211> 735
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 390
gacatccaga tgacacagac tacatcctcc ctgtctgcct ctctgggaga cagagtcacc 60
atcagttgca gggcaagtca ggacattagt aaatatttaa attggtatca gcagaaacca 120
gatggaactg ttaaactcct gatctaccat acatcaagat tacactcagg agtcccatca 180
aggttcagtg gcagtgggtc tggaacagat tattctctca ccattagcaa cctggagcaa 240
gaagatattg ccacttactt ttgccaacag ggtaatacgc ttccgtacac gttcggaggg 300
gggactaagt tggaaataac aggctccacc tctggatccg gcaagcccgg atctggcgag 360
ggatccacca agggcgaggt gaaactgcag gagtcaggac ctggcctggt ggcgccctca 420
cagagcctgt ccgtcacatg cactgtctca ggggtctcat tacccgacta tggtgtaagc 480
tggattcgcc agcctccacg aaagggtctg gagtggctgg gagtaatatg gggtagtgaa 540
accacatact ataattcagc tctcaaatcc agactgacca tcatcaagga caactccaag 600
agccaagttt tcttaaaaat gaacagtctg caaactgatg acacagccat ttactactgt 660
gccaaacatt attactacgg tggtagctat gctatggact actggggtca aggaacctca 720
gtcaccgtct cctca 735
<210> 391
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 391
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr Gly Ser Thr Ser Gly
100 105 110
Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys Gly Glu Val Lys
115 120 125
Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser Leu Ser
130 135 140
Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
145 150 155 160
Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly Val Ile
165 170 175
Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser Arg Leu
180 185 190
Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn
195 200 205
Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys His Tyr
210 215 220
Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser
225 230 235 240
Val Thr Val Ser Ser
245
<210> 392
<211> 1383
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 392
atgctgctgc tggtgaccag cctgctgctg tgcgagctgc cccaccccgc ctttctgctg 60
atccccgaca tccagatgac ccagaccacc tccagcctga gcgccagcct gggcgaccgg 120
gtgaccatca gctgccgggc cagccaggac atcagcaagt acctgaactg gtatcagcag 180
aagcccgacg gcaccgtcaa gctgctgatc taccacacca gccggctgca cagcggcgtg 240
cccagccggt ttagcggcag cggctccggc accgactaca gcctgaccat ctccaacctg 300
gaacaggaag atatcgccac ctacttttgc cagcagggca acacactgcc ctacaccttt 360
ggcggcggaa caaagctgga aatcaccggc agcacctccg gcagcggcaa gcctggcagc 420
ggcgagggca gcaccaaggg cgaggtgaag ctgcaggaaa gcggccctgg cctggtggcc 480
cccagccaga gcctgagcgt gacctgcacc gtgagcggcg tgagcctgcc cgactacggc 540
gtgagctgga tccggcagcc ccccaggaag ggcctggaat ggctgggcgt gatctggggc 600
agcgagacca cctactacaa cagcgccctg aagagccggc tgaccatcat caaggacaac 660
agcaagagcc aggtgttcct gaagatgaac agcctgcaga ccgacgacac cgccatctac 720
tactgcgcca agcactacta ctacggcggc agctacgcca tggactactg gggccagggc 780
accagcgtga ccgtgagcag cgaatctaag tacggaccgc cctgcccccc ttgccctatg 840
ttctgggtgc tggtggtggt cggaggcgtg ctggcctgct acagcctgct ggtcaccgtg 900
gccttcatca tcttttgggt gaaacggggc agaaagaaac tcctgtatat attcaaacaa 960
ccatttatga gaccagtaca aactactcaa gaggaagatg gctgtagctg ccgatttcca 1020
gaagaagaag aaggaggatg tgaactgcgg gtgaagttca gcagaagcgc cgacgcccct 1080
gcctaccagc agggccagaa tcagctgtac aacgagctga acctgggcag aagggaagag 1140
tacgacgtcc tggataagcg gagaggccgg gaccctgaga tgggcggcaa gcctcggcgg 1200
aagaaccccc aggaaggcct gtataacgaa ctgcagaaag acaagatggc cgaggcctac 1260
agcgagatcg gcatgaaggg cgagcggagg cggggcaagg gccacgacgg cctgtatcag 1320
ggcctgtcca ccgccaccaa ggatacctac gacgccctgc acatgcaggc cctgccccca 1380
agg 1383
<210> 393
<211> 461
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 393
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser
20 25 30
Leu Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser
35 40 45
Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly
50 55 60
Thr Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr
85 90 95
Ile Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln
100 105 110
Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile
115 120 125
Thr Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser
130 135 140
Thr Lys Gly Glu Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala
145 150 155 160
Pro Ser Gln Ser Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu
165 170 175
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu
180 185 190
Glu Trp Leu Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser
195 200 205
Ala Leu Lys Ser Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln
210 215 220
Val Phe Leu Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr
225 230 235 240
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
245 250 255
Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Glu Ser Lys Tyr Gly
260 265 270
Pro Pro Cys Pro Pro Cys Pro Met Phe Trp Val Leu Val Val Val Gly
275 280 285
Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile
290 295 300
Phe Trp Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
305 310 315 320
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
325 330 335
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
340 345 350
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
355 360 365
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
370 375 380
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
385 390 395 400
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
405 410 415
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
420 425 430
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
435 440 445
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 394
<211> 801
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 394
atgctgctgc tggtgaccag cctgctgctg tgcgagctgc cccaccccgc ctttctgctg 60
atccccgaca tccagatgac ccagaccacc tccagcctga gcgccagcct gggcgaccgg 120
gtgaccatca gctgccgggc cagccaggac atcagcaagt acctgaactg gtatcagcag 180
aagcccgacg gcaccgtcaa gctgctgatc taccacacca gccggctgca cagcggcgtg 240
cccagccggt ttagcggcag cggctccggc accgactaca gcctgaccat ctccaacctg 300
gaacaggaag atatcgccac ctacttttgc cagcagggca acacactgcc ctacaccttt 360
ggcggcggaa caaagctgga aatcaccggc agcacctccg gcagcggcaa gcctggcagc 420
ggcgagggca gcaccaaggg cgaggtgaag ctgcaggaaa gcggccctgg cctggtggcc 480
cccagccaga gcctgagcgt gacctgcacc gtgagcggcg tgagcctgcc cgactacggc 540
gtgagctgga tccggcagcc ccccaggaag ggcctggaat ggctgggcgt gatctggggc 600
agcgagacca cctactacaa cagcgccctg aagagccggc tgaccatcat caaggacaac 660
agcaagagcc aggtgttcct gaagatgaac agcctgcaga ccgacgacac cgccatctac 720
tactgcgcca agcactacta ctacggcggc agctacgcca tggactactg gggccagggc 780
accagcgtga ccgtgagcag c 801
<210> 395
<211> 267
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 395
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Asp Ile Gln Met Thr Gln Thr Thr Ser Ser
20 25 30
Leu Ser Ala Ser Leu Gly Asp Arg Val Thr Ile Ser Cys Arg Ala Ser
35 40 45
Gln Asp Ile Ser Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly
50 55 60
Thr Val Lys Leu Leu Ile Tyr His Thr Ser Arg Leu His Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr
85 90 95
Ile Ser Asn Leu Glu Gln Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln
100 105 110
Gly Asn Thr Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile
115 120 125
Thr Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser
130 135 140
Thr Lys Gly Glu Val Lys Leu Gln Glu Ser Gly Pro Gly Leu Val Ala
145 150 155 160
Pro Ser Gln Ser Leu Ser Val Thr Cys Thr Val Ser Gly Val Ser Leu
165 170 175
Pro Asp Tyr Gly Val Ser Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu
180 185 190
Glu Trp Leu Gly Val Ile Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser
195 200 205
Ala Leu Lys Ser Arg Leu Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln
210 215 220
Val Phe Leu Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr
225 230 235 240
Tyr Cys Ala Lys His Tyr Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr
245 250 255
Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
260 265
<210> 396
<211> 1317
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 396
gacatccaga tgacccagac cacctccagc ctgagcgcca gcctgggcga ccgggtgacc 60
atcagctgcc gggccagcca ggacatcagc aagtacctga actggtatca gcagaagccc 120
gacggcaccg tcaagctgct gatctaccac accagccggc tgcacagcgg cgtgcccagc 180
cggtttagcg gcagcggctc cggcaccgac tacagcctga ccatctccaa cctggaacag 240
gaagatatcg ccacctactt ttgccagcag ggcaacacac tgccctacac ctttggcggc 300
ggaacaaagc tggaaatcac cggcagcacc tccggcagcg gcaagcctgg cagcggcgag 360
ggcagcacca agggcgaggt gaagctgcag gaaagcggcc ctggcctggt ggcccccagc 420
cagagcctga gcgtgacctg caccgtgagc ggcgtgagcc tgcccgacta cggcgtgagc 480
tggatccggc agccccccag gaagggcctg gaatggctgg gcgtgatctg gggcagcgag 540
accacctact acaacagcgc cctgaagagc cggctgacca tcatcaagga caacagcaag 600
agccaggtgt tcctgaagat gaacagcctg cagaccgacg acaccgccat ctactactgc 660
gccaagcact actactacgg cggcagctac gccatggact actggggcca gggcaccagc 720
gtgaccgtga gcagcgaatc taagtacgga ccgccctgcc ccccttgccc tatgttctgg 780
gtgctggtgg tggtcggagg cgtgctggcc tgctacagcc tgctggtcac cgtggccttc 840
atcatctttt gggtgaaacg gggcagaaag aaactcctgt atatattcaa acaaccattt 900
atgagaccag tacaaactac tcaagaggaa gatggctgta gctgccgatt tccagaagaa 960
gaagaaggag gatgtgaact gcgggtgaag ttcagcagaa gcgccgacgc ccctgcctac 1020
cagcagggcc agaatcagct gtacaacgag ctgaacctgg gcagaaggga agagtacgac 1080
gtcctggata agcggagagg ccgggaccct gagatgggcg gcaagcctcg gcggaagaac 1140
ccccaggaag gcctgtataa cgaactgcag aaagacaaga tggccgaggc ctacagcgag 1200
atcggcatga agggcgagcg gaggcggggc aagggccacg acggcctgta tcagggcctg 1260
tccaccgcca ccaaggatac ctacgacgcc ctgcacatgc aggccctgcc cccaagg 1317
<210> 397
<211> 439
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 397
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr Gly Ser Thr Ser Gly
100 105 110
Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys Gly Glu Val Lys
115 120 125
Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser Leu Ser
130 135 140
Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
145 150 155 160
Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly Val Ile
165 170 175
Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser Arg Leu
180 185 190
Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn
195 200 205
Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys His Tyr
210 215 220
Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser
225 230 235 240
Val Thr Val Ser Ser Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys
245 250 255
Pro Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr
260 265 270
Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Lys Arg Gly
275 280 285
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
290 295 300
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
305 310 315 320
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
325 330 335
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
340 345 350
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
355 360 365
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
370 375 380
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
385 390 395 400
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
405 410 415
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
420 425 430
Met Gln Ala Leu Pro Pro Arg
435
<210> 398
<211> 735
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 398
gacatccaga tgacccagac cacctccagc ctgagcgcca gcctgggcga ccgggtgacc 60
atcagctgcc gggccagcca ggacatcagc aagtacctga actggtatca gcagaagccc 120
gacggcaccg tcaagctgct gatctaccac accagccggc tgcacagcgg cgtgcccagc 180
cggtttagcg gcagcggctc cggcaccgac tacagcctga ccatctccaa cctggaacag 240
gaagatatcg ccacctactt ttgccagcag ggcaacacac tgccctacac ctttggcggc 300
ggaacaaagc tggaaatcac cggcagcacc tccggcagcg gcaagcctgg cagcggcgag 360
ggcagcacca agggcgaggt gaagctgcag gaaagcggcc ctggcctggt ggcccccagc 420
cagagcctga gcgtgacctg caccgtgagc ggcgtgagcc tgcccgacta cggcgtgagc 480
tggatccggc agccccccag gaagggcctg gaatggctgg gcgtgatctg gggcagcgag 540
accacctact acaacagcgc cctgaagagc cggctgacca tcatcaagga caacagcaag 600
agccaggtgt tcctgaagat gaacagcctg cagaccgacg acaccgccat ctactactgc 660
gccaagcact actactacgg cggcagctac gccatggact actggggcca gggcaccagc 720
gtgaccgtga gcagc 735
<210> 399
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 399
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Lys Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr His Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Thr Gly Ser Thr Ser Gly
100 105 110
Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr Lys Gly Glu Val Lys
115 120 125
Leu Gln Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln Ser Leu Ser
130 135 140
Val Thr Cys Thr Val Ser Gly Val Ser Leu Pro Asp Tyr Gly Val Ser
145 150 155 160
Trp Ile Arg Gln Pro Pro Arg Lys Gly Leu Glu Trp Leu Gly Val Ile
165 170 175
Trp Gly Ser Glu Thr Thr Tyr Tyr Asn Ser Ala Leu Lys Ser Arg Leu
180 185 190
Thr Ile Ile Lys Asp Asn Ser Lys Ser Gln Val Phe Leu Lys Met Asn
195 200 205
Ser Leu Gln Thr Asp Asp Thr Ala Ile Tyr Tyr Cys Ala Lys His Tyr
210 215 220
Tyr Tyr Gly Gly Ser Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser
225 230 235 240
Val Thr Val Ser Ser
245
<210> 400
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 400
caggtgcagc tgcaggagtc cggcggcggc gtggtgcagc caggccggtc cctgagactg 60
tcttgtgccg ccagcggctt caccttttcc tcttatggca tgcactgggt gagacaggca 120
cctggcaagg gactggagtg ggtggccgtg atctcctacg acggctctaa caagtattac 180
gccgatagcg tgaagggcag gttcaccatc agccgcgaca actccaagaa tacactgtat 240
ctgcagatga atagcctgcg ggccgaggat accgccgtgt attactgcgg aggctccggc 300
tacgcactgc acgacgatta ttacggactg gacgtgtggg gacagggcac cctggtcaca 360
gtgagctcc 369
<210> 401
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 401
caggtgcagc tgcaggagtc tggcggagga gtggtgcagc caggccggtc cctgagactg 60
tcttgcgccg ccagcggctt cacattttct agctacggaa tgcactgggt gcgccaggca 120
cctggcaagg gactggagtg ggtggccgtg atctcctatg acggctctaa caagtactat 180
gccgattccg tgaagggcag gttcaccatc agccgcgaca actccaagaa tacactgtac 240
ctgcagatga attccctgcg ggccgaggat accgccgtgt actattgtgg cggctctggc 300
tatgccctgc acgacgatta ctatggactg gacgtgtggg gacagggcac cctggtgaca 360
gtgtcctct 369
<210> 402
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 402
caggtgcagc tgcaggagtc tggcggagga gtggtgcagc caggccggag cctgagactg 60
tcctgcgccg cctctggctt cacctttagc tcctatggca tgcactgggt gagacaggca 120
cctggcaagg gactggagtg ggtggccgtg atcagctacg acggctccaa caagtattac 180
gccgatagcg tgaagggcag gttcaccatc tctcgcgaca acagcaagaa tacactgtat 240
ctgcagatga attccctgcg ggccgaggat acagccgtgt attactgcgg aggcagcggc 300
tacgcactgc acgacgatta ttacggactg gacgtgtggg gacagggcac cctggtcaca 360
gtgtctagc 369
<210> 403
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 403
caggtgcagc tgcaggagag cggcggcggc gtggtgcagc ccggccggtc tctgagactg 60
agctgtgccg cctccggctt cacctttagc tcctacggaa tgcactgggt gcgccaggca 120
cctggcaagg gactggagtg ggtggccgtg atctcttatg acggcagcaa caagtactat 180
gccgatagcg tgaagggcag gttcaccatc tcccgcgaca actctaagaa tacactgtac 240
ctgcagatga atagcctgcg ggccgaggat accgccgtgt actattgcgg aggctccggc 300
tatgcactgc acgacgatta ctatggactg gacgtgtggg gacagggcac cctggtgaca 360
gtgtctagc 369
<210> 404
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 404
caggtccagc tgcaggagag tggggggggg gtcgtccagc ccggaagaag cctgagactg 60
tcatgtgccg catctgggtt tacttttagc tcctatggaa tgcactgggt gcgccaggca 120
cctggcaagt gcctggagtg ggtggccgtg atctcctacg acggctctaa caagtactat 180
gccgatagcg tgaagggccg gttcaccatc agcagagaca actccaagaa tacactgtat 240
ctgcagatga attctctgcg ggccgaggat accgccgtgt actattgtgg aggctccggc 300
tacgcactgc acgacgatta ctatggactg gacgtgtggg gacagggcac cctggtgaca 360
gtgtctagc 369
<210> 405
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 405
caggtccagc tgcaggaatc cggcggagga gtggtgcagc caggccggtc tctgagactg 60
agctgcgccg cctccggctt cacattttcc tcttatggca tgcactgggt gagacaggcc 120
cctggcaagt gtctggagtg ggtggccgtg atctcttacg acggcagcaa caagtattac 180
gccgatagcg tgaagggcag gttcaccatc tcccgcgaca actctaagaa tacactgtat 240
ctgcagatga attccctgcg ggccgaggat accgccgtgt attactgcgg cggctctggc 300
tacgccctgc acgacgacta ctatggactg gatgtctggg ggcagggcac actggtcact 360
gtctcttca 369
<210> 406
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 406
caggtccagc tgcaggaatc aggggggggg gtcgtccagc ccggaagaag tctgagactg 60
tcatgtgccg catcagggtt tacctttagc tcctatggaa tgcactgggt gcgccaggca 120
cctggcaagt gcctggagtg ggtggccgtg atctcctacg acggctctaa caagtactat 180
gccgatagcg tgaagggccg gttcaccatc agcagagaca actccaagaa tacactgtat 240
ctgcagatga attctctgcg ggccgaggat accgccgtgt actattgtgg aggctccggc 300
tacgcactgc acgacgatta ctatggactg gacgtgtggg gacagggcac cctggtgaca 360
gtgtctagc 369
<210> 407
<211> 369
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 407
caggtccagc tgcaggaatc cggcggagga gtggtgcagc caggccggtc tctgagactg 60
agctgcgccg cctccggctt caccttttcc tcttatggca tgcactgggt gagacaggcc 120
cctggcaagt gtctggagtg ggtggccgtg atctcttacg acggcagcaa caagtattac 180
gccgatagcg tgaagggcag gttcaccatc tcccgcgaca actctaagaa tacactgtat 240
ctgcagatga attccctgcg ggccgaggat acagccgtgt attactgtgg cggctctggc 300
tacgccctgc atgatgatta ttatggactg gatgtctggg ggcagggcac actggtcact 360
gtctcttcc 369
<210> 408
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 408
cagtctgccc tgacccagcc agcaagcgtg tccggctctc ctggccagag catcacaatc 60
tcctgcaccg gcacaagctc cgacgtggga ggctataact acgtgagctg gtatcagcag 120
cacccaggca aggcccccaa gctgatgatc tacgacgtga gcaacaggcc ttctggcgtg 180
agcaatcgct tcagcggctc caagtctggc aataccgcct ctctgacaat cagcggcctg 240
caggcagagg acgaggcaga ttattactgc tctagctata cctcctctag cacactgtac 300
gtgtttggca gcggcaccaa ggtgacagtg ctg 333
<210> 409
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 409
cagagcgccc tgacccagcc agcatccgtg tctggcagcc caggccagtc tatcacaatc 60
agctgcaccg gcacaagctc cgacgtggga ggctacaact atgtgagctg gtaccagcag 120
caccctggca aggccccaaa gctgatgatc tatgacgtga gcaaccggcc atccggcgtg 180
tctaatagat tctccggctc taagagcggc aataccgcct ccctgacaat ctctggcctg 240
caggcagagg acgaggcaga ttactattgt tctagctaca cctcctctag cacactgtac 300
gtgttcggca gcggcaccaa ggtgacagtg ctg 333
<210> 410
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 410
cagtctgccc tgacccagcc agcaagcgtg tccggctctc ctggccagtc catcacaatc 60
tcttgtaccg gcacatcctc tgacgtgggc ggctataact acgtgtcctg gtatcagcag 120
cacccaggca aggcccccaa gctgatgatc tacgatgtga gcaacaggcc ttctggcgtg 180
agcaatcgct tcagcggctc caagtctggc aataccgcca gcctgacaat ctccggcctg 240
caggcagagg acgaggcaga ttattactgc agctcctata cctctagctc cacactgtac 300
gtgtttggca gcggcaccaa ggtgacagtg ctg 333
<210> 411
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 411
cagagcgccc tgacccagcc agcatccgtg tctggcagcc caggccagtc catcacaatc 60
tcttgcaccg gcacatctag cgacgtgggc ggctacaact acgtgagctg gtaccagcag 120
caccctggca aggccccaaa gctgatgatc tatgatgtga gcaaccggcc ctccggcgtg 180
tctaatagat tctccggctc taagagcggc aataccgcca gcctgacaat ctccggcctg 240
caggcagagg acgaggcaga ttactattgc tcctcttaca ccagctcctc tacactgtac 300
gtgttcggct ccggcaccaa ggtgacagtg ctg 333
<210> 412
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 412
cagtctgccc tgacccagcc tgcaagcgtg tccggctctc caggccagtc tatcacaatc 60
agctgtaccg gcacaagctc cgacgtgggc ggctataact acgtgagctg gtatcagcag 120
caccctggca aggccccaaa gctgatgatc tacgacgtga gcaaccggcc ctctggcgtg 180
agcaatcggt tcagcggcag caagtctggc aataccgcct ccctgacaat ctctggcctg 240
caggcagagg acgaggcaga ttattactgt agcagttata cttcaagctc aaccctgtac 300
gtgtttggat gcggcactaa ggtcaccgtc ctg 333
<210> 413
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 413
cagtctgctc tgacccagcc cgcttccgtc tcagggtctc caggacagtc aattaccatt 60
agttgcacag gcacctcatc cgatgtgggc ggctataact acgtgtcctg gtatcagcag 120
cacccaggca aggcccccaa gctgatgatc tacgacgtga gcaacaggcc atctggcgtg 180
agcaatcgct tcagcggctc caagtctggc aataccgcca gcctgacaat ctccggcctg 240
caggcagagg acgaggcaga ttactattgc agctcctata cctctagctc cacactgtac 300
gtgtttggct gtggcaccaa ggtgacagtg ctg 333
<210> 414
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 414
cagtctgccc tgacccagcc tgcaagcgtg tccggctctc caggccagtc tatcacaatc 60
agctgtaccg gcacaagctc cgacgtgggc ggctataact acgtgagctg gtatcagcag 120
caccctggca aggccccaaa gctgatgatc tacgacgtga gcaaccggcc ctctggcgtg 180
agcaatcggt tcagcggcag caagtctggc aataccgcct ccctgacaat ctctggcctg 240
caggcagagg acgaggcaga ttattactgt agctcctaca cttcttcaag cacactgtat 300
gtctttggat gcggaactaa ggtcactgtc ctg 333
<210> 415
<211> 333
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 415
cagtctgctc tgacccagcc cgcttccgtc tcaggatctc caggacagtc tattacaatt 60
agttgcacag gaacctcttc cgatgtgggc ggctataact acgtgtcctg gtatcagcag 120
cacccaggca aggcccccaa gctgatgatc tacgacgtga gcaacaggcc ttctggcgtg 180
agcaatcgct tcagcggctc caagtctggc aataccgcca gcctgacaat ctccggcctg 240
caggcagagg acgaggcaga ttactattgc agctcctata cctctagctc cacactgtac 300
gtgtttggct gtggcaccaa ggtgacagtg ctg 333
<210> 416
<211> 1485
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 416
atggccctcc ctgtcaccgc tctgttgctg ccgcttgctc tgctgctcca cgcagcgcga 60
ccgcaggtac aattgcagga gtctggaggc ggtgtggtgc aacccggtcg cagcttgcgc 120
ctgagttgtg ctgcgtctgg atttacattt tcatcttacg gaatgcattg ggtacgccag 180
gcaccgggga aaggccttga atgggtggct gtaatttcat acgatggttc caacaaatac 240
tatgctgact cagtcaaggg tcgatttaca attagtcggg acaactccaa gaacaccctt 300
tatcttcaaa tgaattccct tagagcagag gatacggcgg tctattactg tggtggcagt 360
ggttatgcac ttcatgatga ttactatggc ttggatgtct gggggcaagg gacgcttgta 420
actgtatcct ctggtggtgg tggtagtggt gggggaggct ccggcggtgg cggctctcaa 480
tctgctctga ctcaaccagc aagcgtatca gggtcaccgg gacagagtat taccataagt 540
tgcacgggga cctctagcga tgtagggggg tataattatg tatcttggta tcaacaacac 600
cccgggaaag cccctaaatt gatgatctac gacgtgagca atcgacctag tggcgtatca 660
aatcgcttct ctggtagcaa gagtgggaat acggcgtccc ttactattag cggattgcaa 720
gcagaagatg aggccgatta ctactgcagc tcctatacta gctcttctac attgtacgtc 780
tttgggagcg gaacaaaagt aacagtactc acaacaacac ctgccccgag accgcctaca 840
ccagccccga ctattgccag ccagcctctg agcctcaggc ctgaggcctg taggcccgca 900
gcgggcggcg cagttcatac acggggcttg gatttcgctt gtgatattta tatttgggct 960
cctttggcgg ggacatgtgg cgtgctgctt ctgtcacttg ttattacact gtactgtaaa 1020
cgcgggcgaa aaaaattgct gtatattttt aagcagccat ttatgaggcc cgttcagacg 1080
acgcaggagg aggacggttg ctcttgcagg ttcccagaag aggaagaagg gggctgtgaa 1140
ttgcgggtta aattttcaag atccgcagac gctccagcat accaacaggg acaaaaccaa 1200
ctctataacg agctgaatct tggaagaagg gaggaatatg atgtgctgga taaacggcgc 1260
ggtagagatc cggagatggg cggaaaacca aggcgaaaaa accctcagga gggactctac 1320
aacgaactgc agaaagacaa aatggcggag gcttattccg aaataggcat gaagggcgag 1380
cggaggcgag ggaaagggca cgacggactg tatcaaggcc tctcaaccgc gactaaggat 1440
acgtacgacg ccctgcacat gcaggccctg cctccgagat gataa 1485
<210> 417
<211> 1395
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 417
gaaattgtga tgacccagtc acccgccact cttagccttt cacccggtga gcgcgcaacc 60
ctgtcttgca gagcctccca agacatctca aaatacctta attggtatca acagaagccc 120
ggacaggctc ctcgccttct gatctaccac accagccggc tccattctgg aatccctgcc 180
aggttcagcg gtagcggatc tgggaccgac tacaccctca ctatcagctc actgcagcca 240
gaggacttcg ctgtctattt ctgtcagcaa gggaacaccc tgccctacac ctttggacag 300
ggcaccaagc tcgagattaa aggtggaggt ggcagcggag gaggtgggtc cggcggtgga 360
ggaagccagg tccaactcca agaaagcgga ccgggtcttg tgaagccatc agaaactctt 420
tcactgactt gtactgtgag cggagtgtct ctccccgatt acggggtgtc ttggatcaga 480
cagccaccgg ggaagggtct ggaatggatt ggagtgattt ggggctctga gactacttac 540
taccaatcat ccctcaagtc acgcgtcacc atctcaaagg acaactctaa gaatcaggtg 600
tcactgaaac tgtcatctgt gaccgcagcc gacaccgccg tgtactattg cgctaagcat 660
tactattatg gcgggagcta cgcaatggat tactggggac agggtactct ggtcaccgtg 720
tccagcacca ctaccccagc accgaggcca cccaccccgg ctcctaccat cgcctcccag 780
cctctgtccc tgcgtccgga ggcatgtaga cccgcagctg gtggggccgt gcatacccgg 840
ggtcttgact tcgcctgcga tatctacatt tgggcccctc tggctggtac ttgcggggtc 900
ctgctgcttt cactcgtgat cactctttac tgtaagcgcg gtcggaagaa gctgctgtac 960
atctttaagc aacccttcat gaggcctgtg cagactactc aagaggagga cggctgttca 1020
tgccggttcc cagaggagga ggaaggcggc tgcgaactgc gcgtgaaatt cagccgcagc 1080
gcagatgctc cagcctacca gcaggggcag aaccagctct acaacgaact caatcttggt 1140
cggagagagg agtacgacgt gctggacaag cggagaggac gggacccaga aatgggcggg 1200
aagccgcgca gaaagaatcc ccaagagggc ctgtacaacg agctccaaaa ggataagatg 1260
gcagaagcct atagcgagat tggtatgaaa ggggaacgca gaagaggcaa aggccacgac 1320
ggactgtacc agggactcag caccgccacc aaggacacct atgacgctct tcacatgcag 1380
gccctgccgc ctcgg 1395
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ЛЕЧЕНИЕ ЗЛОКАЧЕСТВЕННОГО НОВООБРАЗОВАНИЯ С ИСПОЛЬЗОВАНИЕМ ГУМАНИЗИРОВАННОГО ХИМЕРНОГО АНТИГЕННОГО РЕЦЕПТОРА ПРОТИВ ВСМА | 2015 |
|
RU2751660C2 |
| СПОСОБЫ ПОЛУЧЕНИЯ КЛЕТОК, ЭКСПРЕССИРУЮЩИХ ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР | 2019 |
|
RU2822196C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ ДЛЯ BCMA И ПУТИ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2785658C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ ДЛЯ ЛЕЧЕНИЯ РАКА | 2017 |
|
RU2826270C2 |
| ВИДЫ КОМБИНИРОВАННОЙ ТЕРАПИИ С ИСПОЛЬЗОВАНИЕМ ХИМЕРНЫХ АНТИГЕННЫХ РЕЦЕПТОРОВ И ИНГИБИТОРОВ PD-1 | 2017 |
|
RU2809160C2 |
| ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР (CAR) ПРОТИВ CD123 ДЛЯ ИСПОЛЬЗОВАНИЯ В ЛЕЧЕНИИ ЗЛОКАЧЕСТВЕННЫХ ОПУХОЛЕЙ | 2015 |
|
RU2724999C2 |
| КОМПОЗИЦИИ И СПОСОБЫ СЕЛЕКТИВНОЙ ДЕГРАДАЦИИ БЕЛКА | 2018 |
|
RU2811700C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ИЗБИРАТЕЛЬНОЙ ЭКСПРЕССИИ БЕЛКА | 2017 |
|
RU2795467C2 |
| ЛЕЧЕНИЕ РАКА С ИСПОЛЬЗОВАНИЕМ ХИМЕРНОГО АНТИГЕНСПЕЦИФИЧЕСКОГО РЕЦЕПТОРА НА ОСНОВЕ ГУМАНИЗИРОВАННОГО АНТИТЕЛА ПРОТИВ EGFRvIII | 2014 |
|
RU2831418C2 |
| ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР | 2019 |
|
RU2810092C2 |
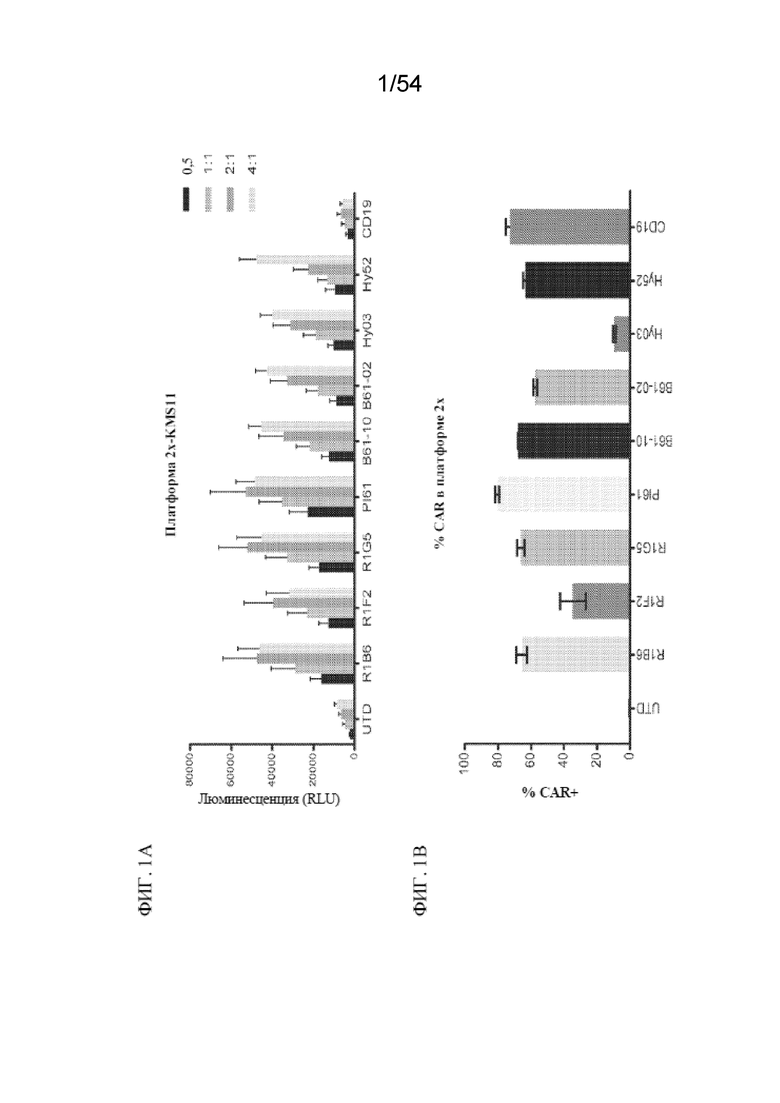
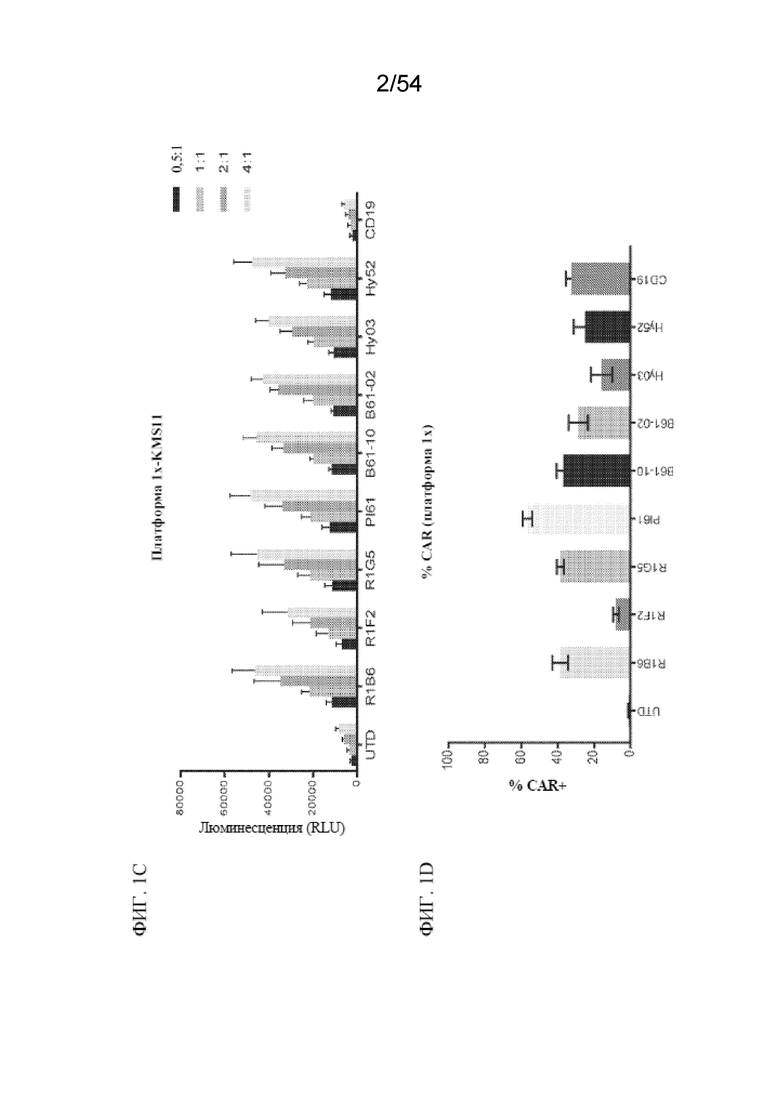


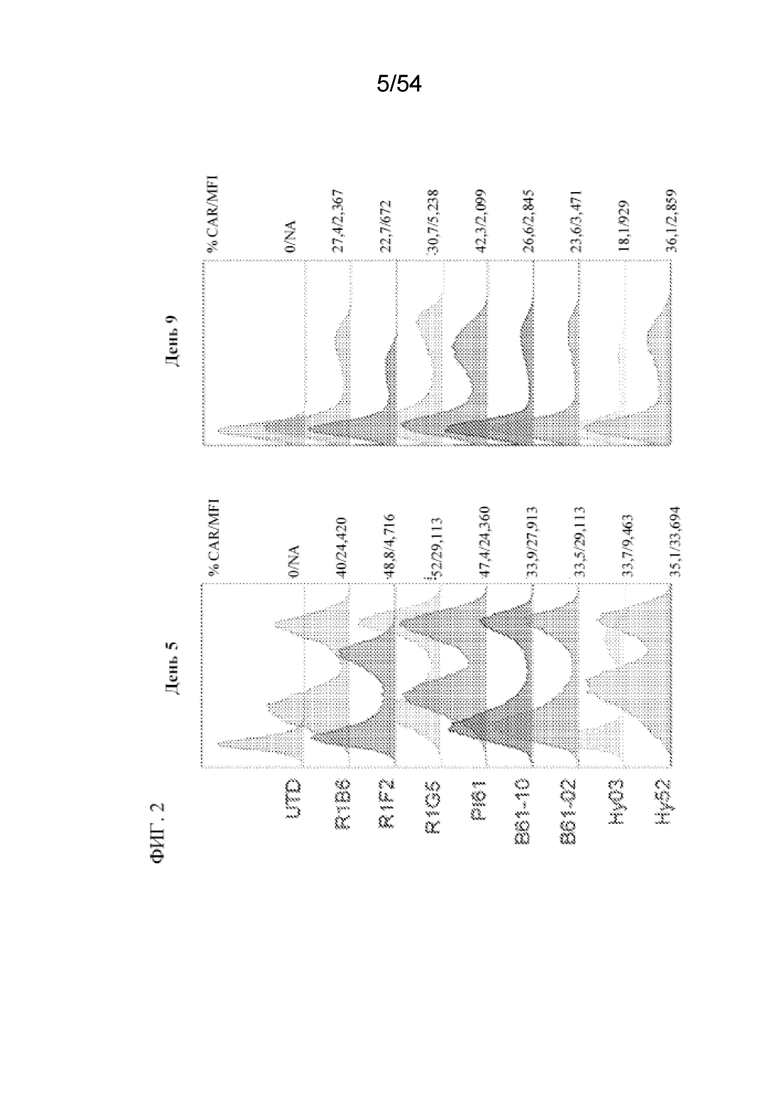
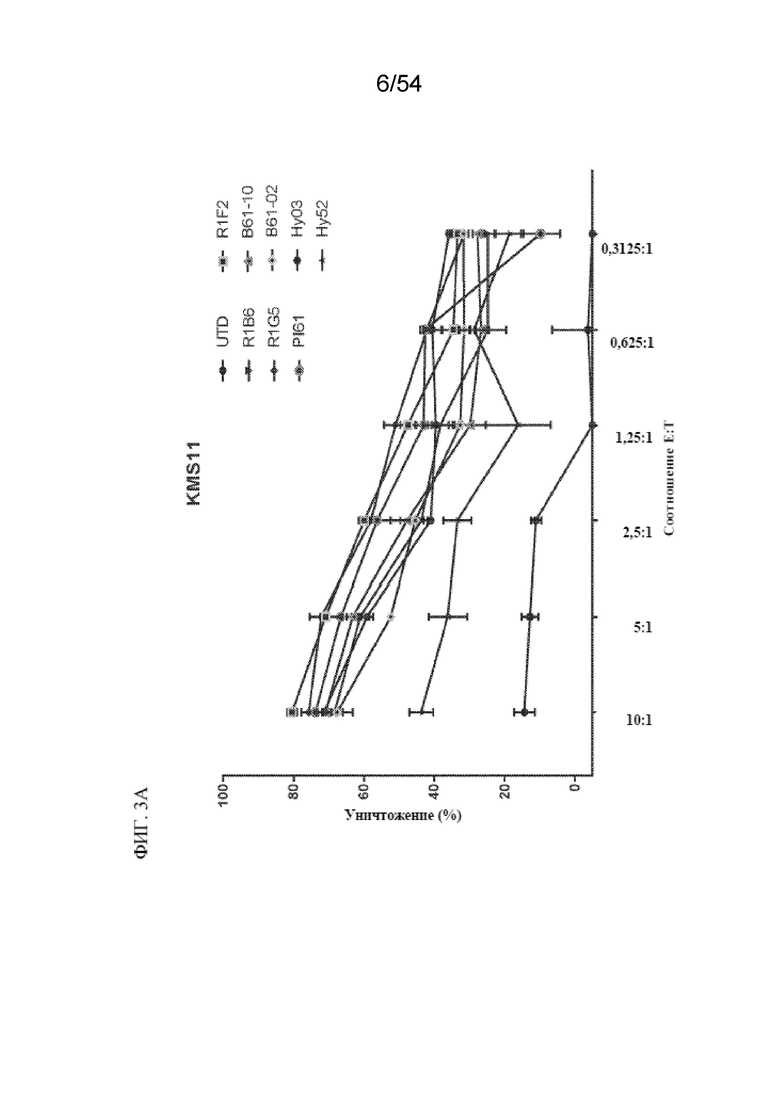
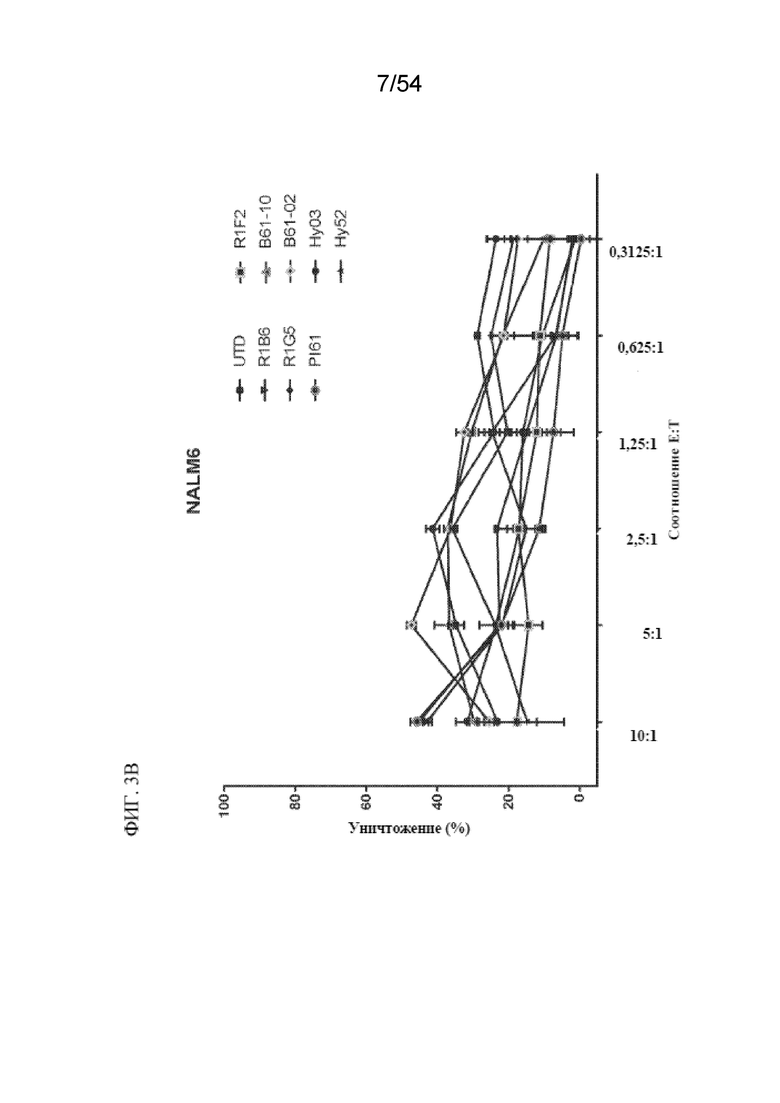

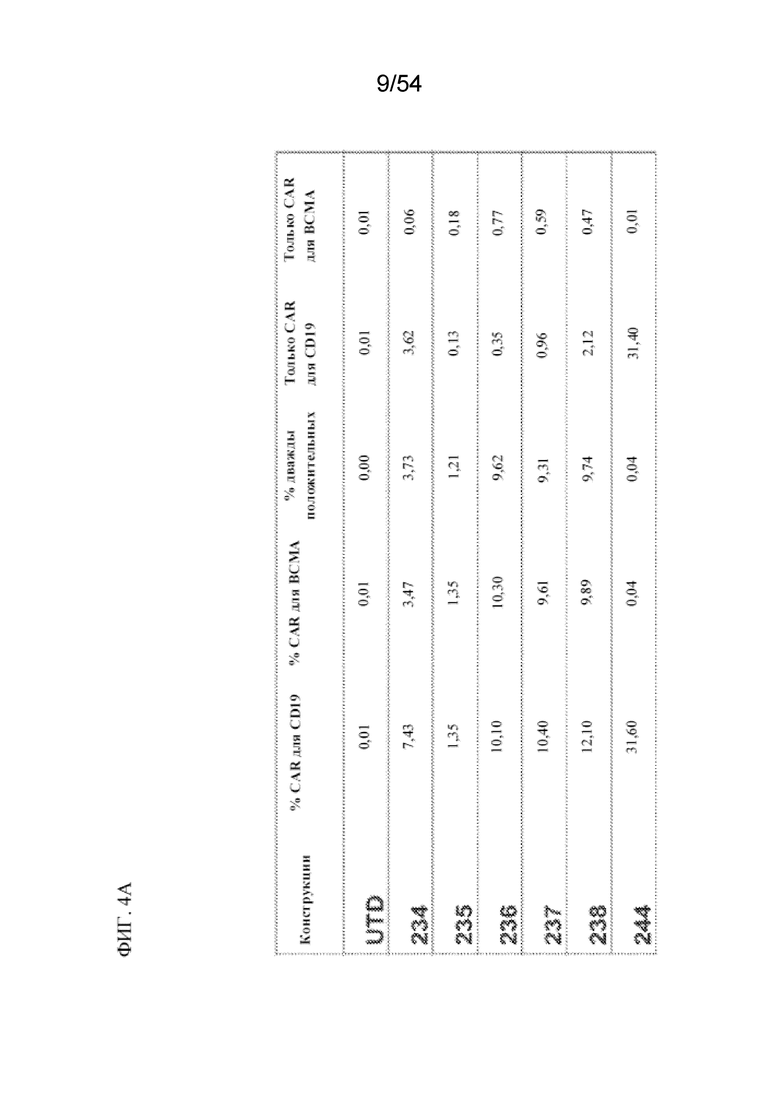
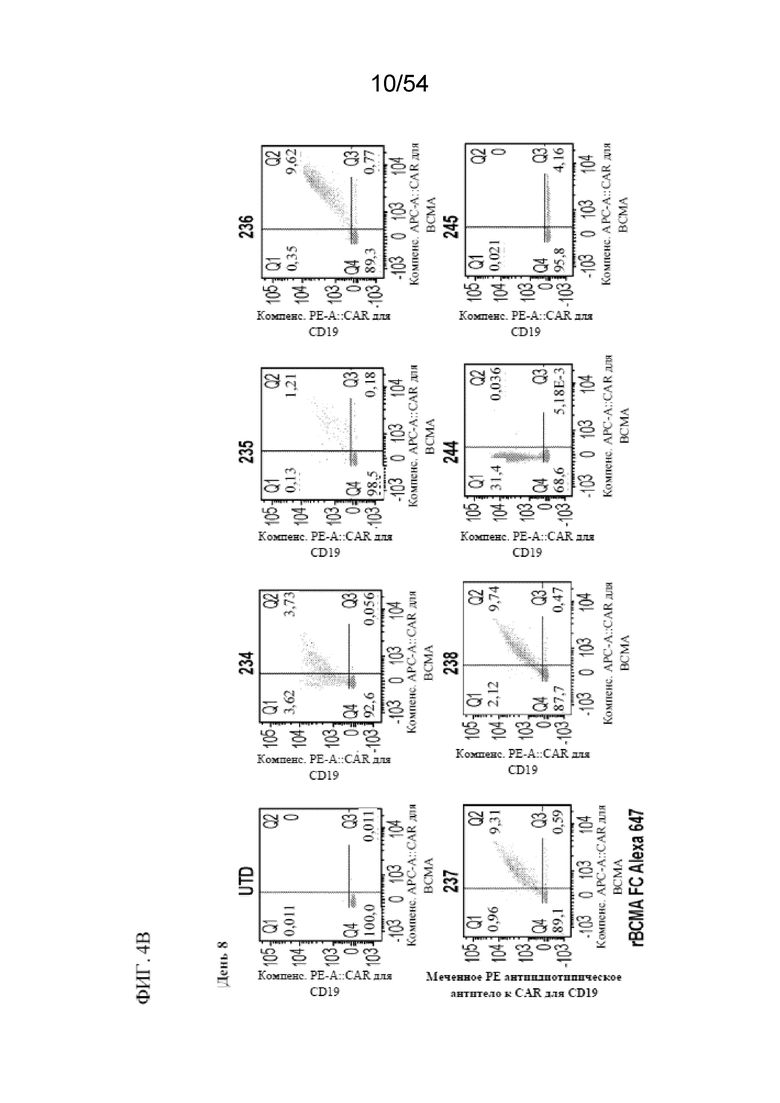
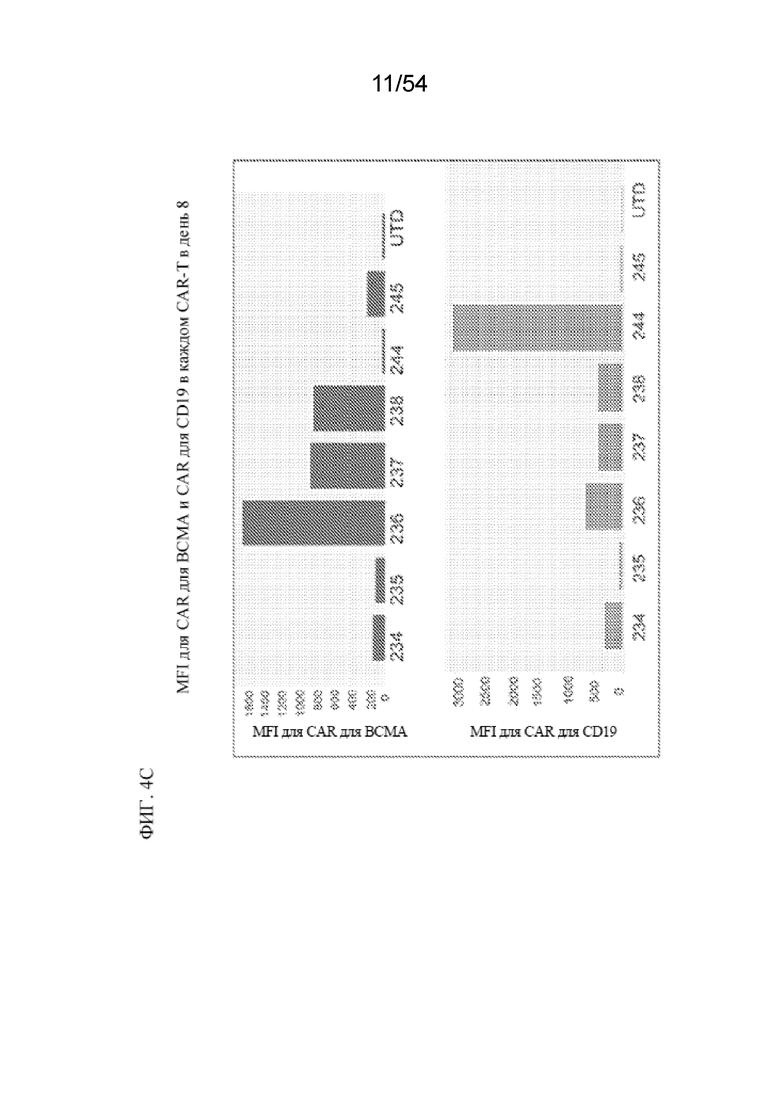
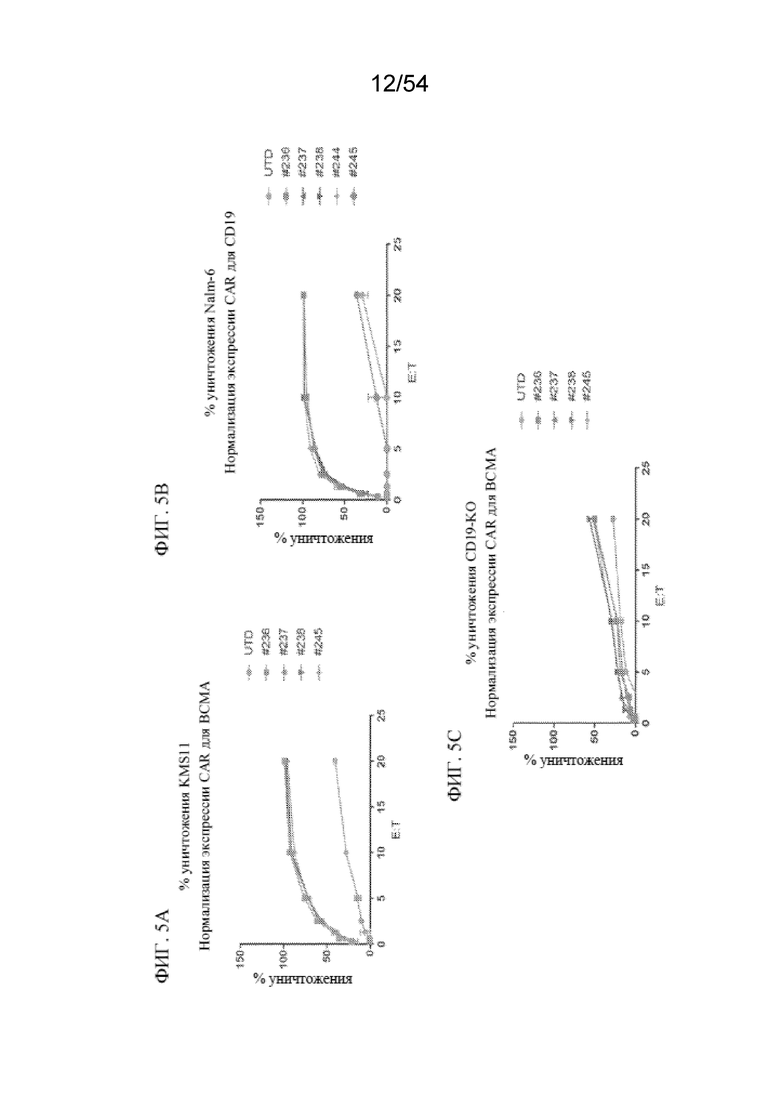
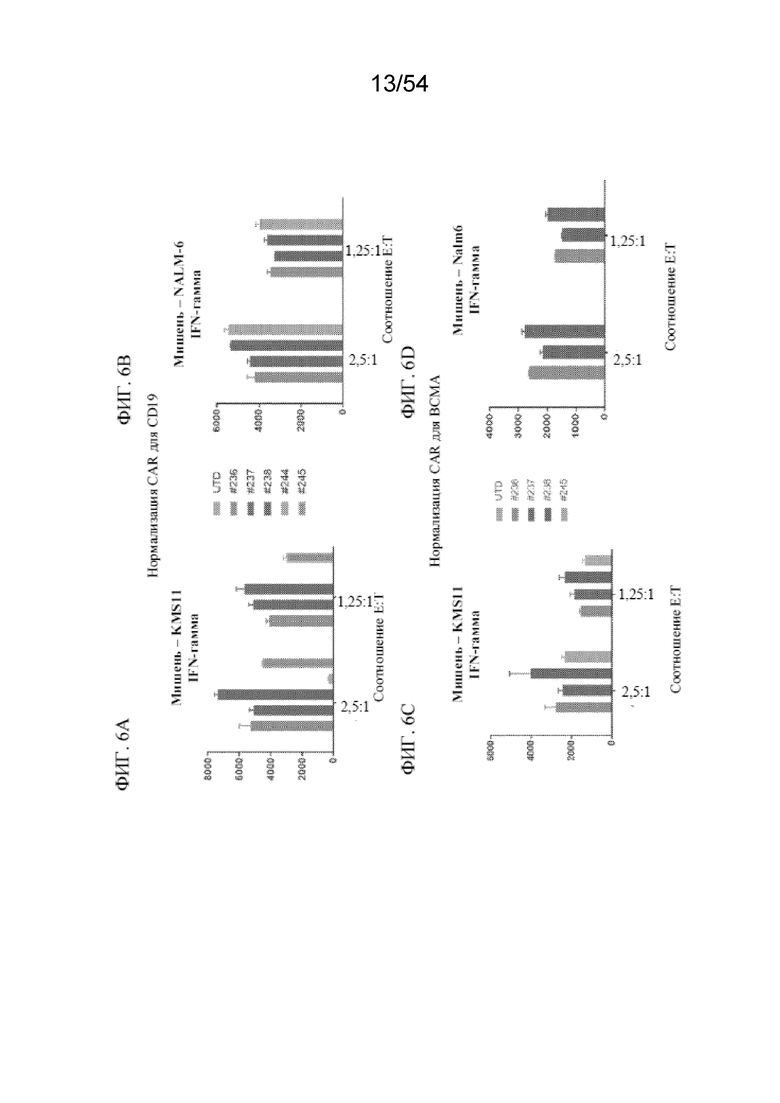
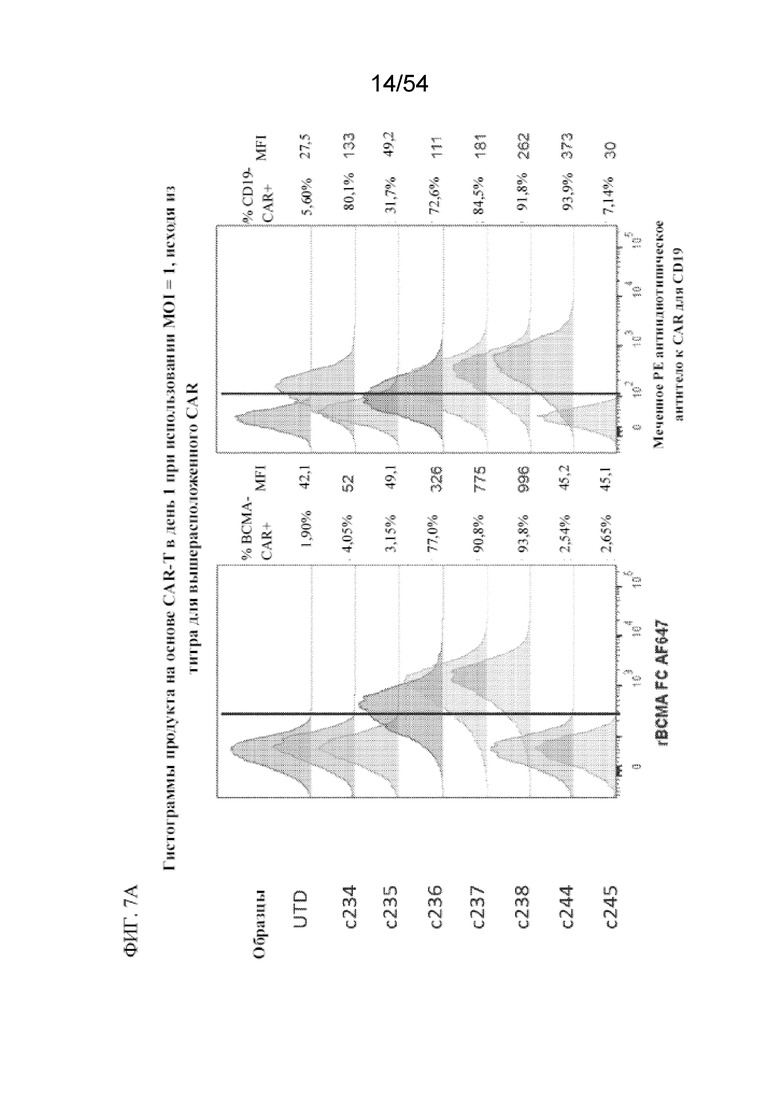
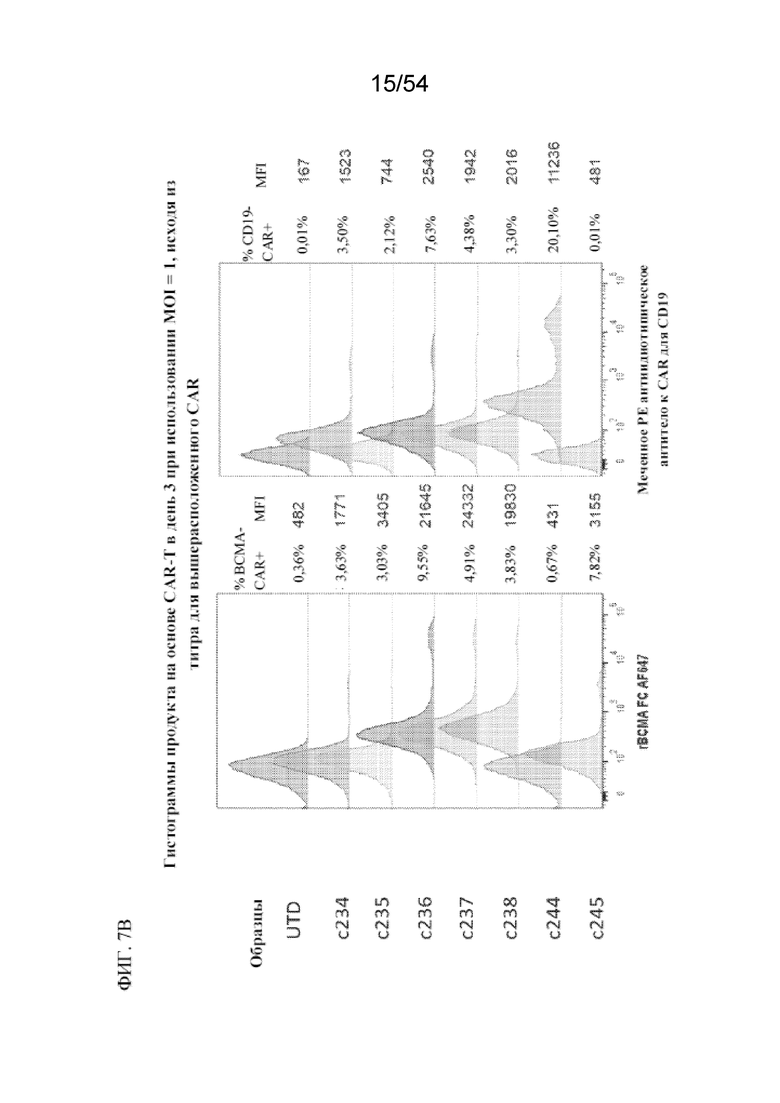
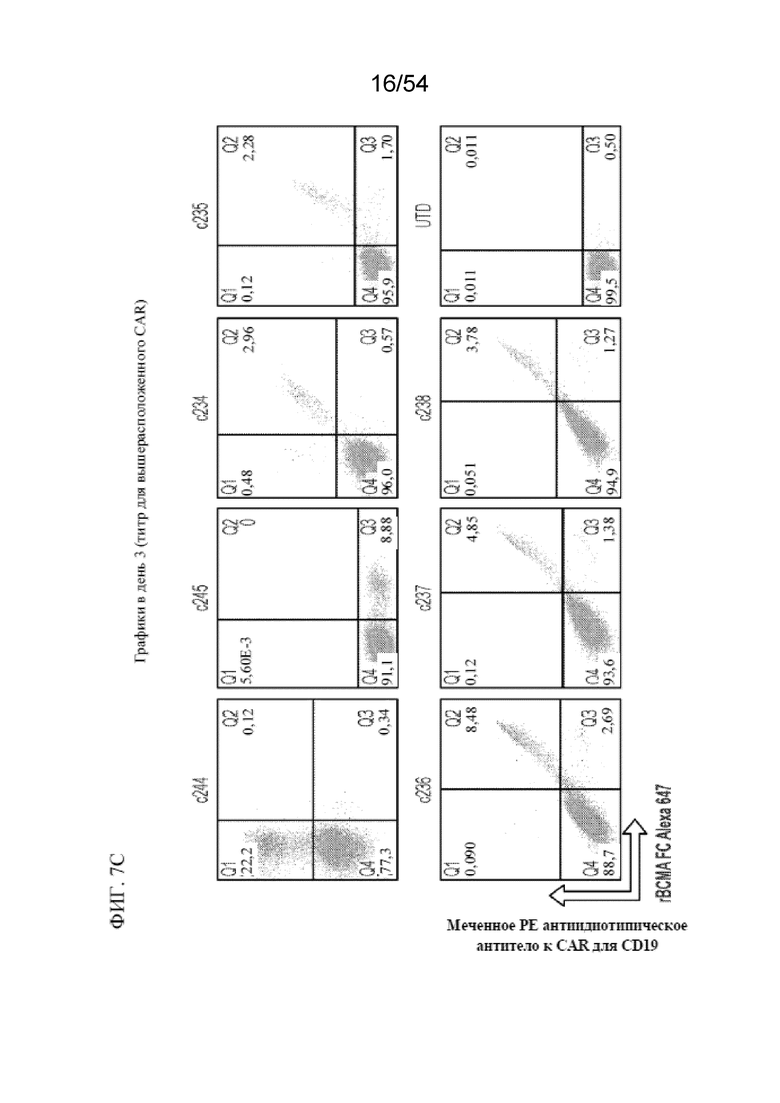
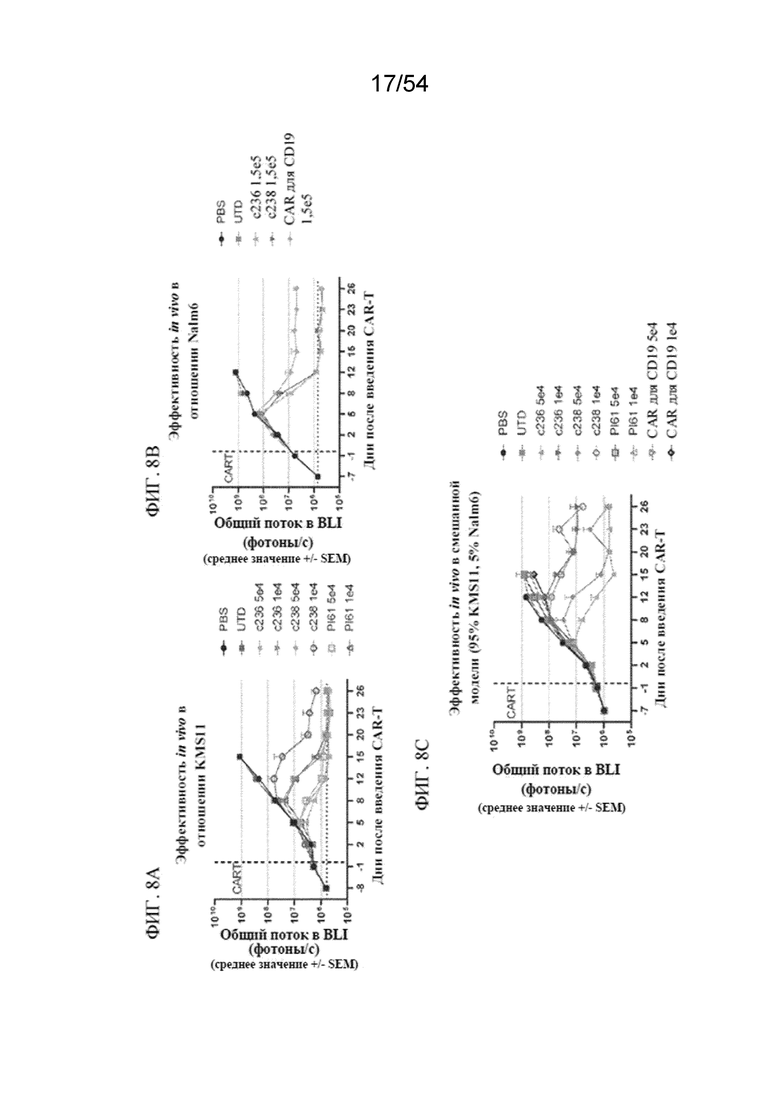
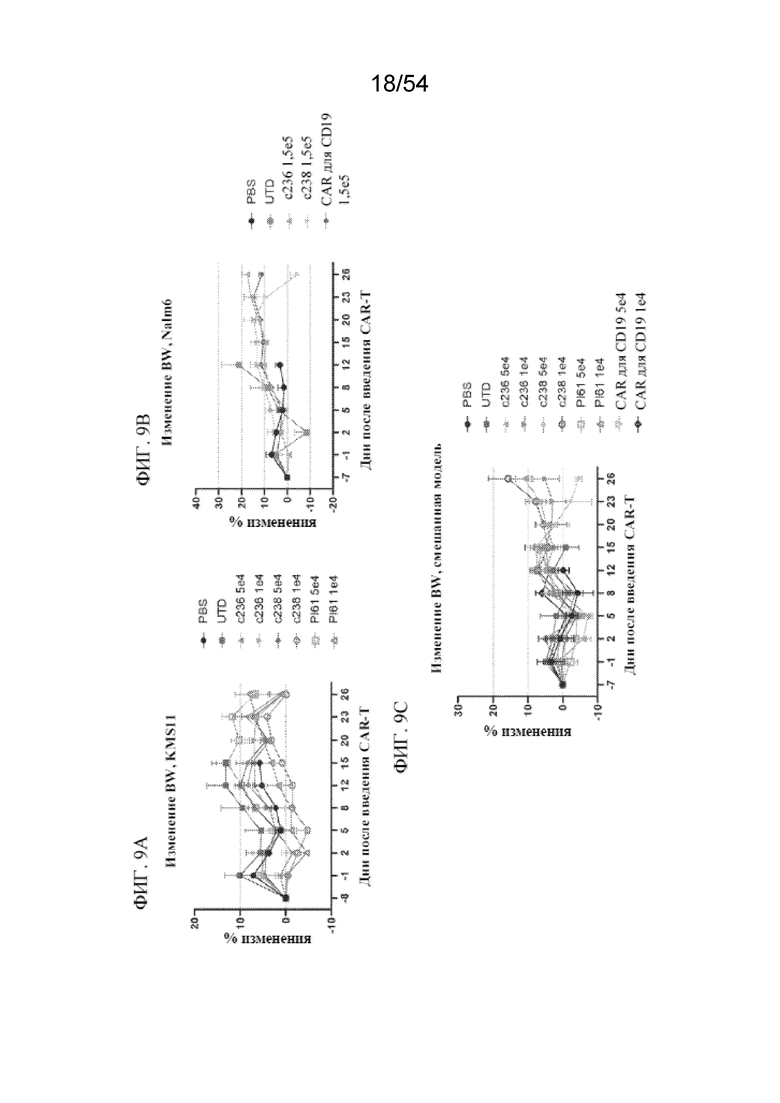

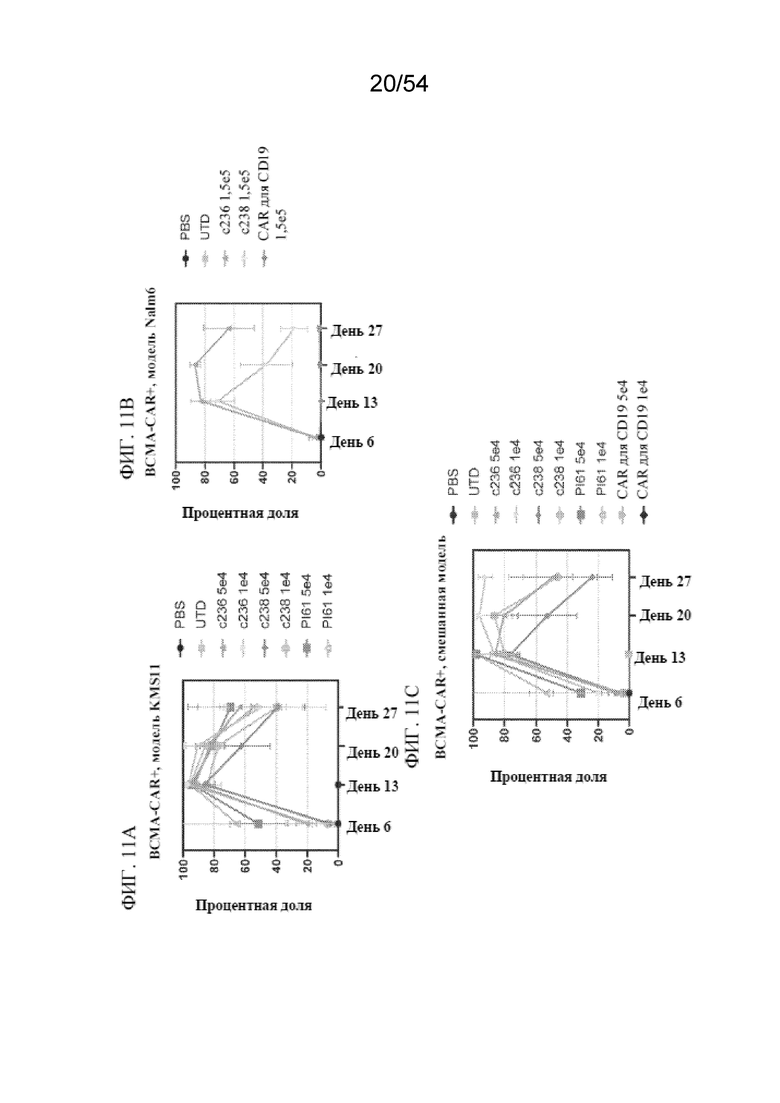
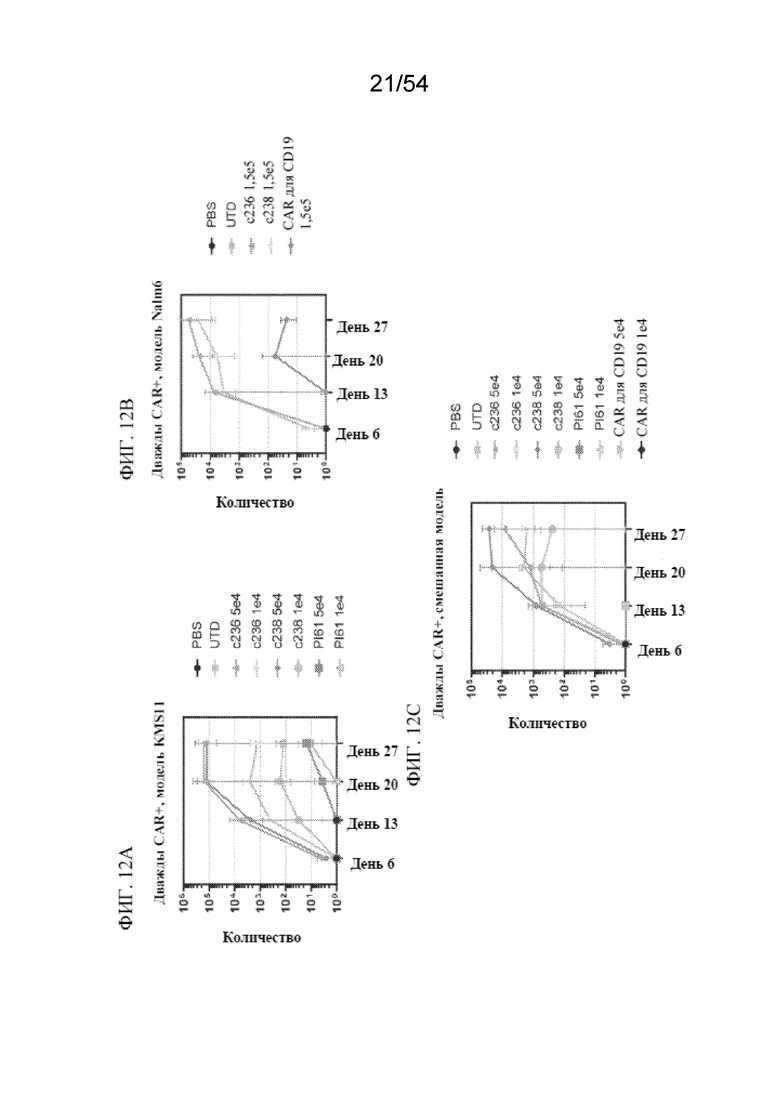

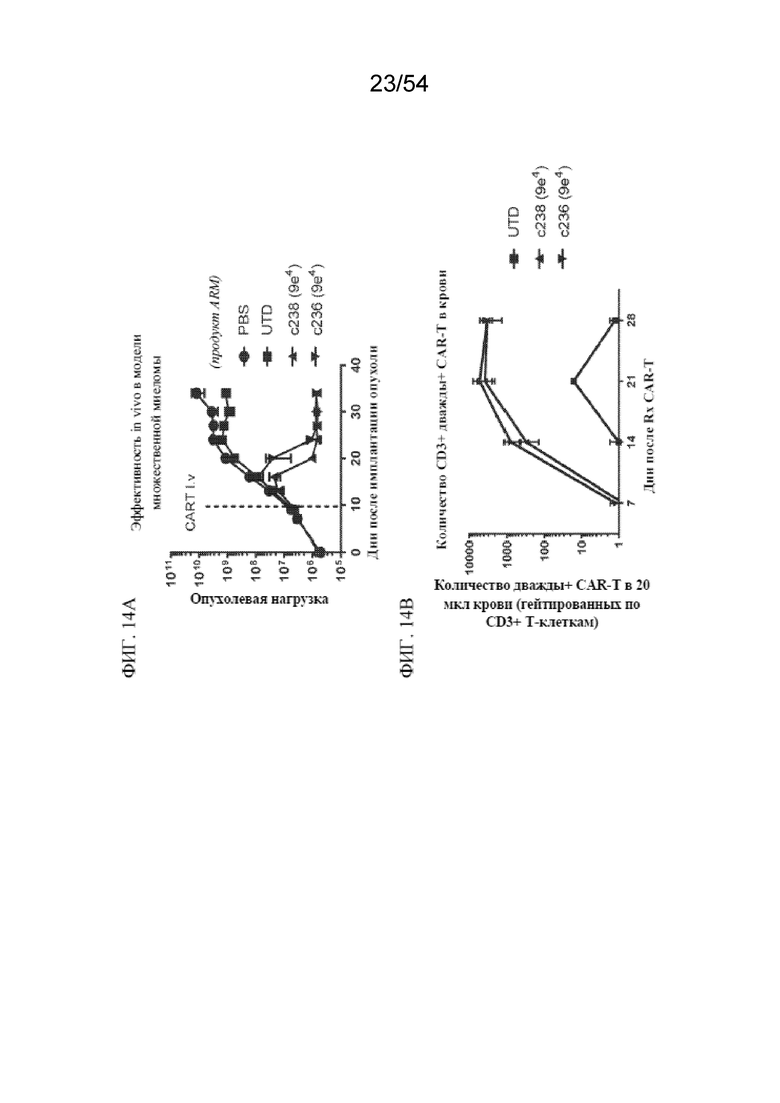
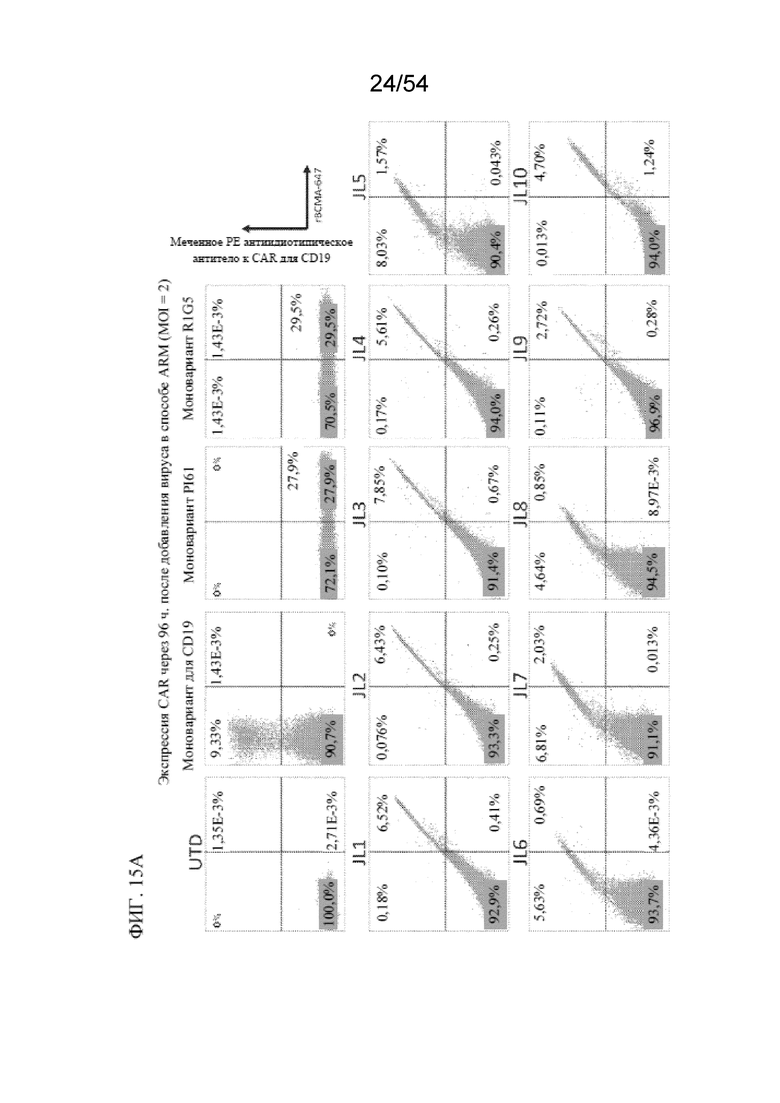

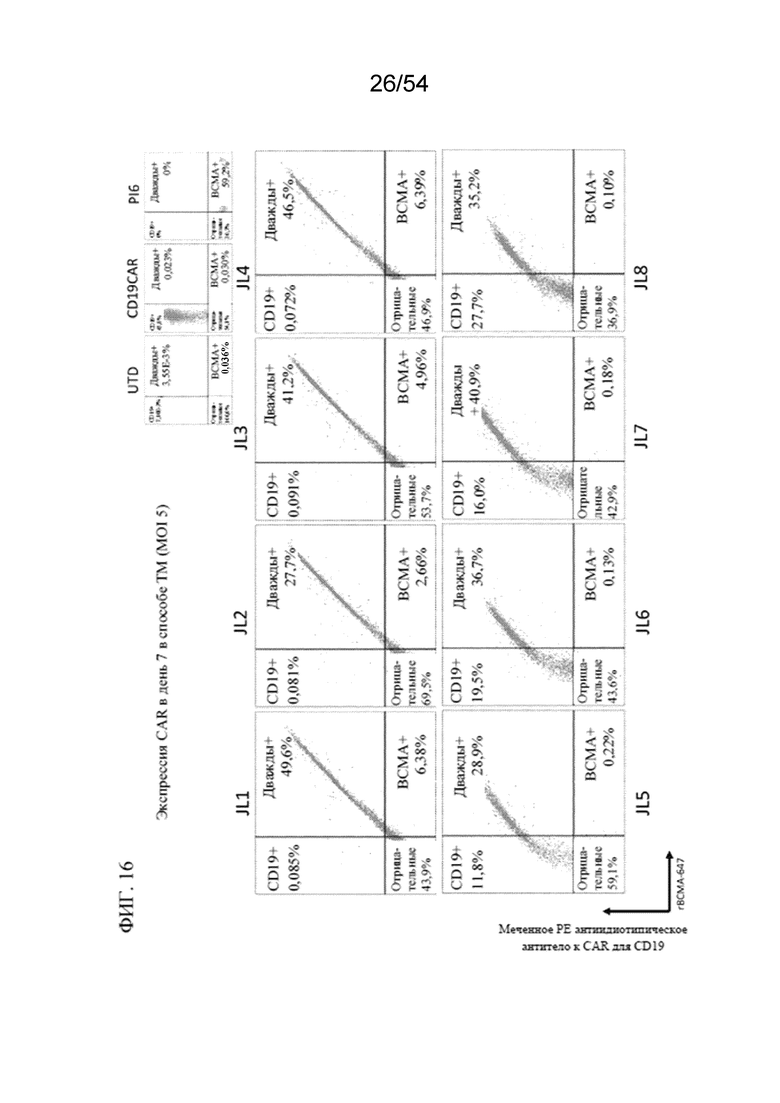
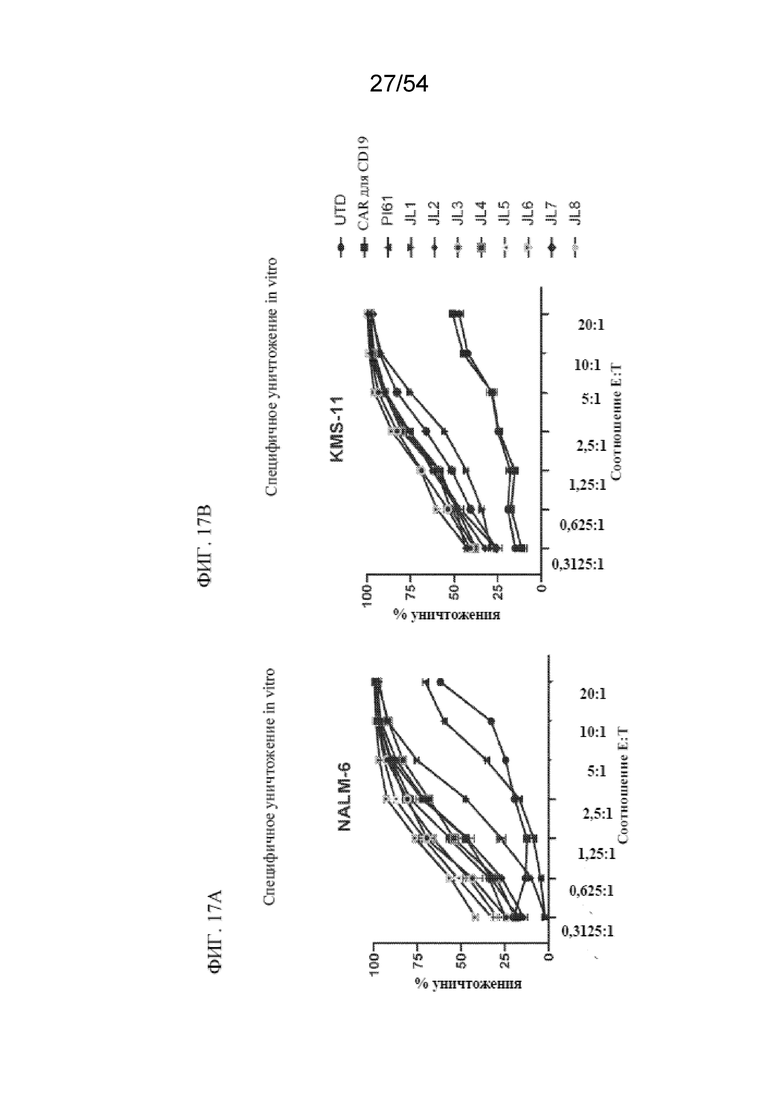
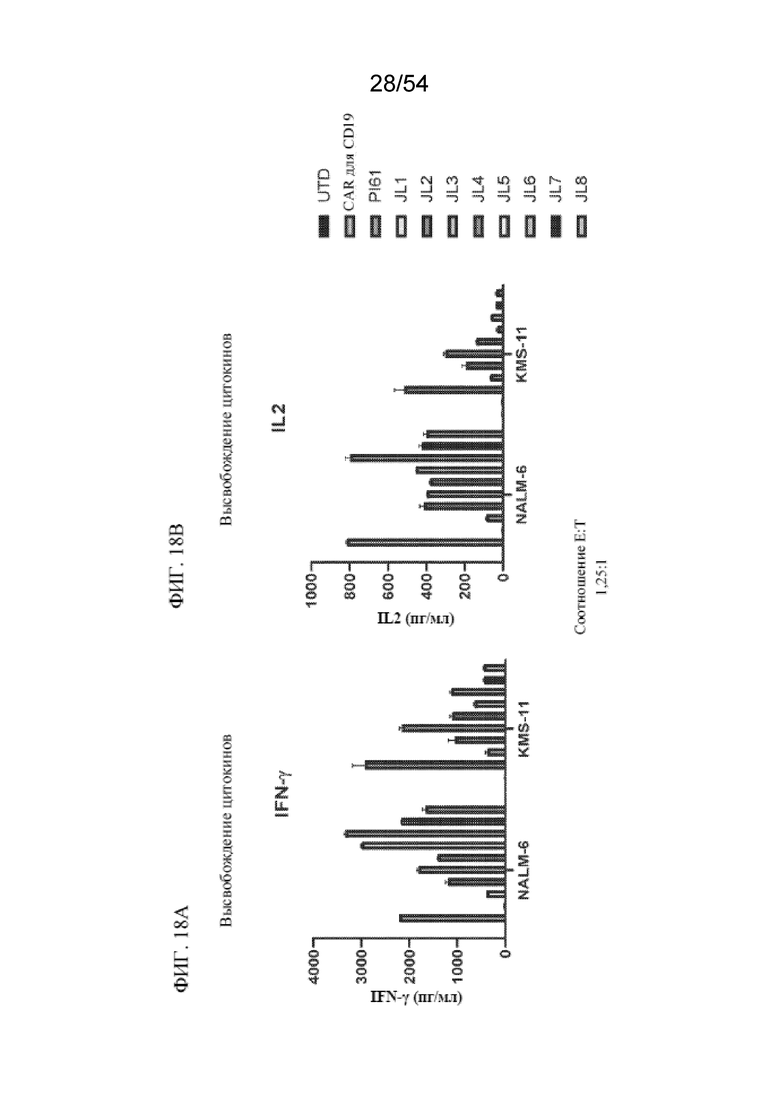
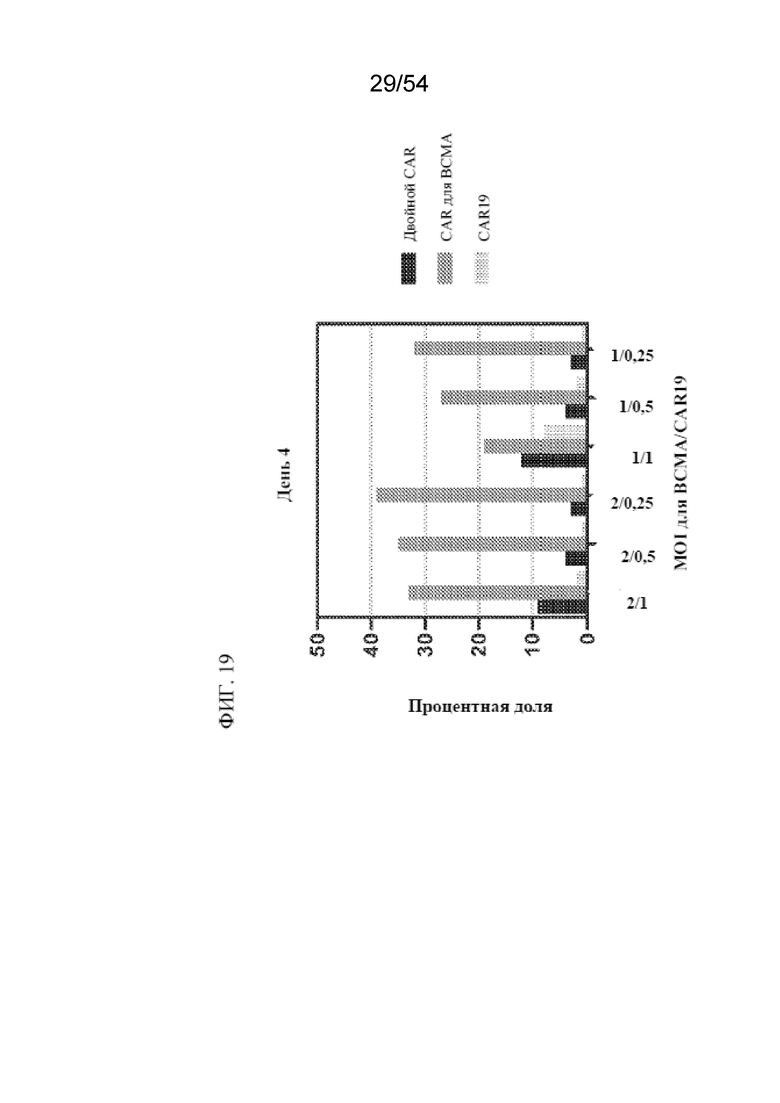

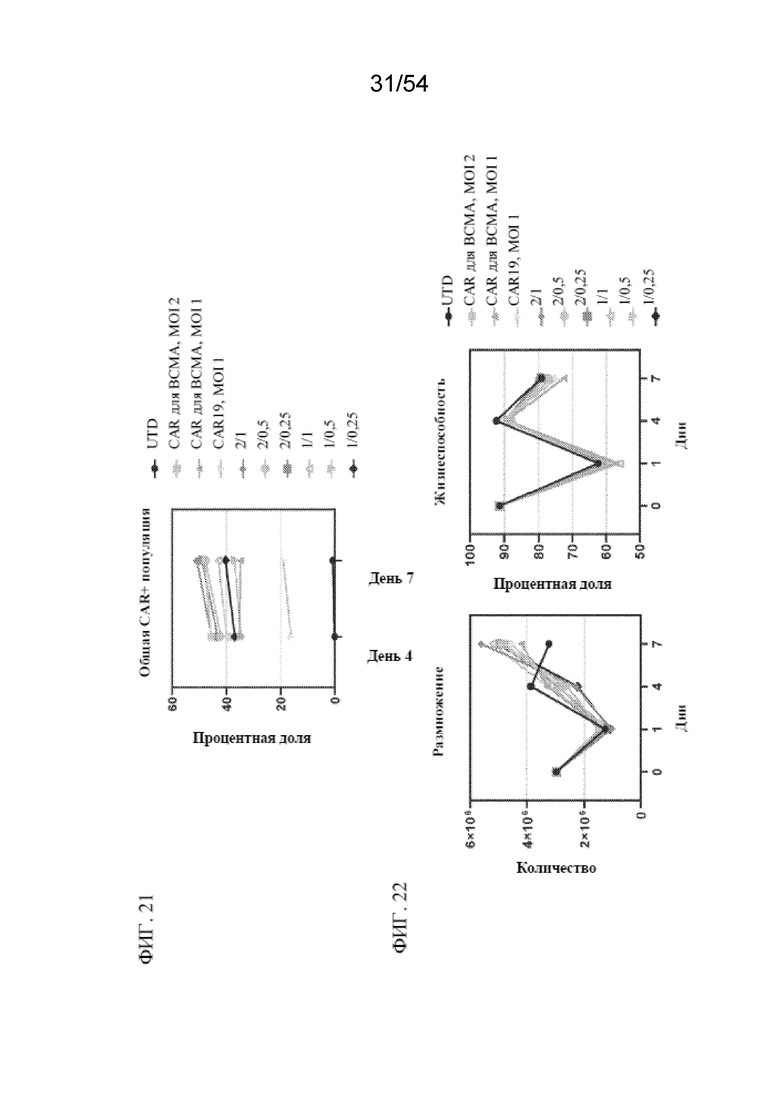
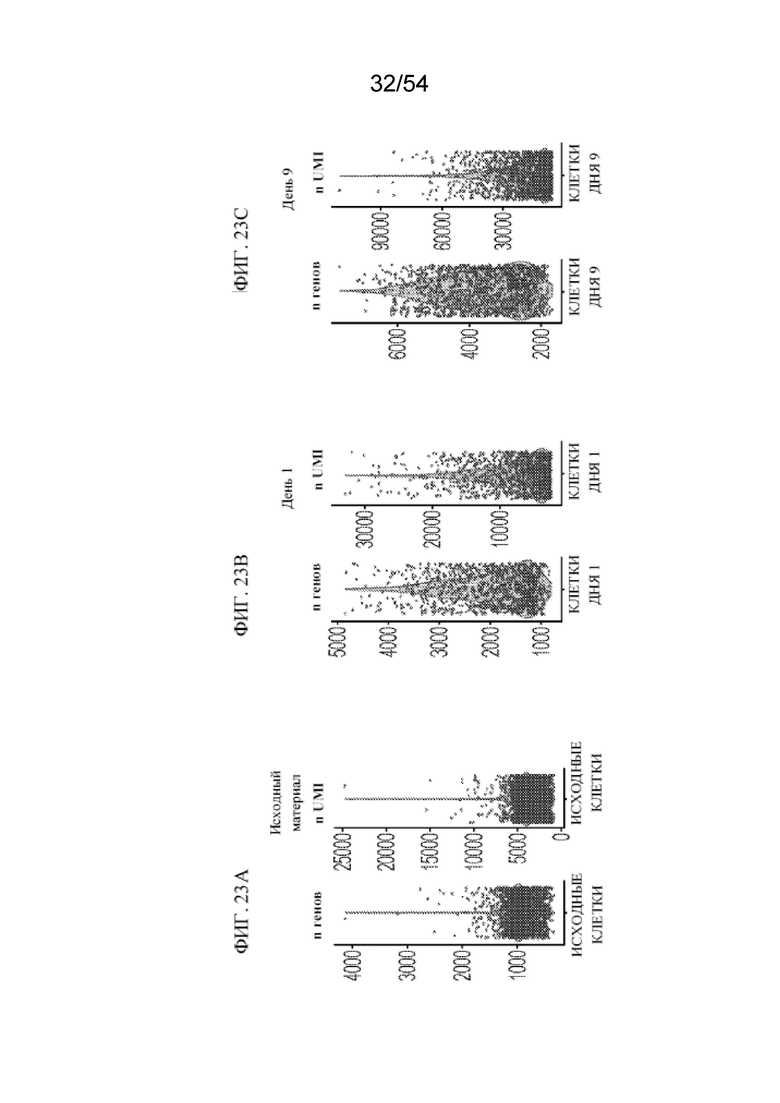

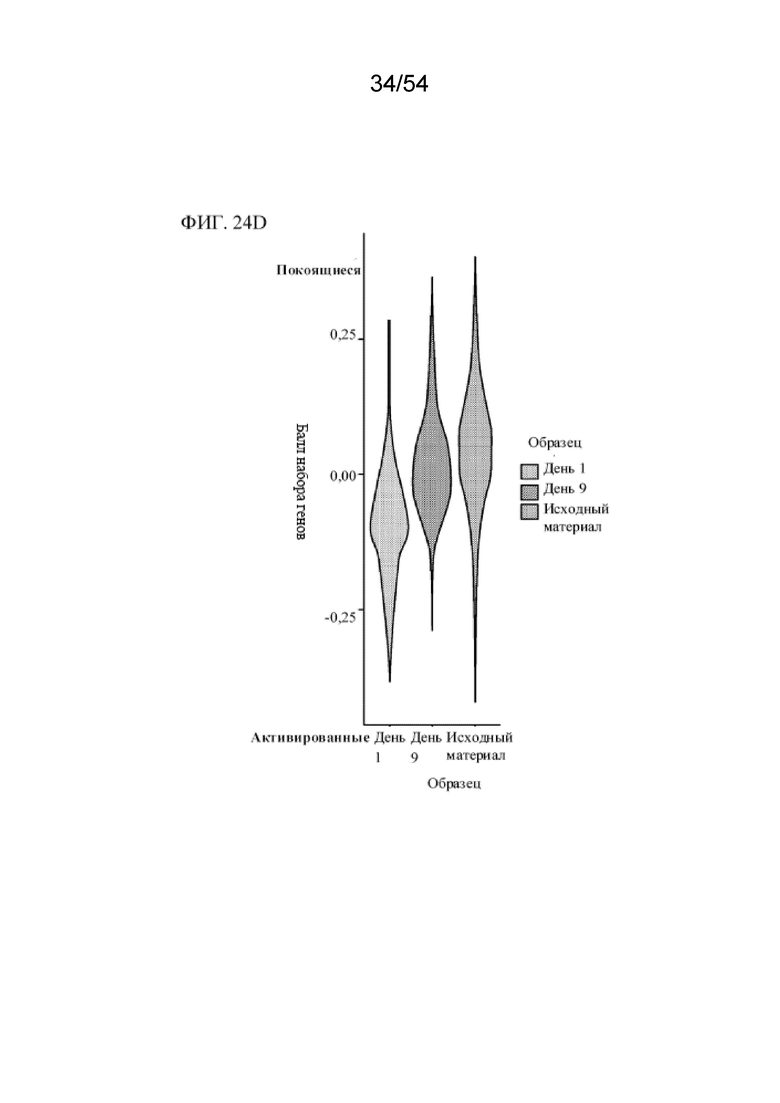
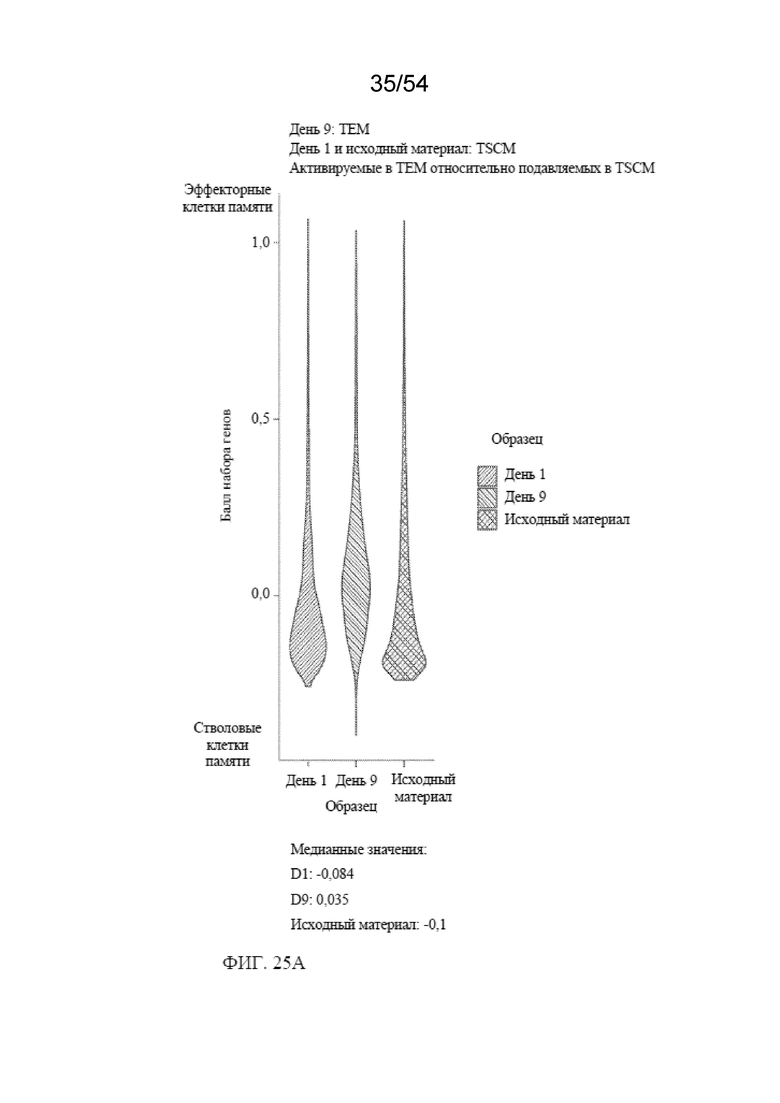
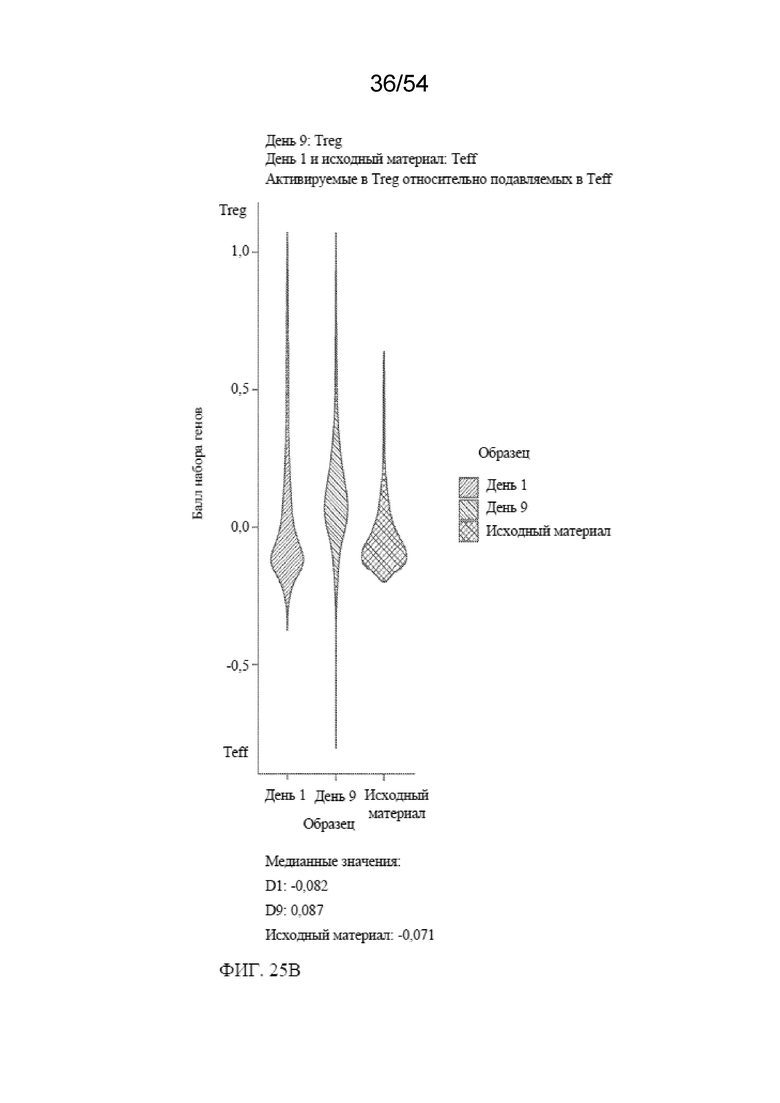
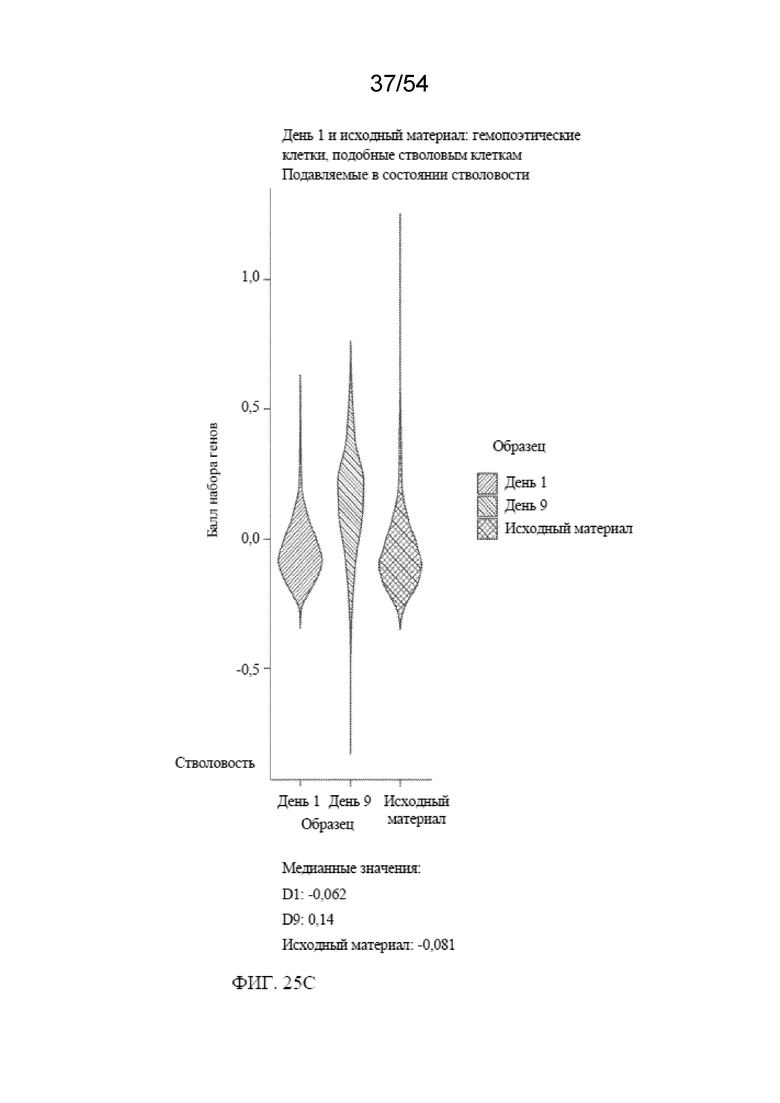
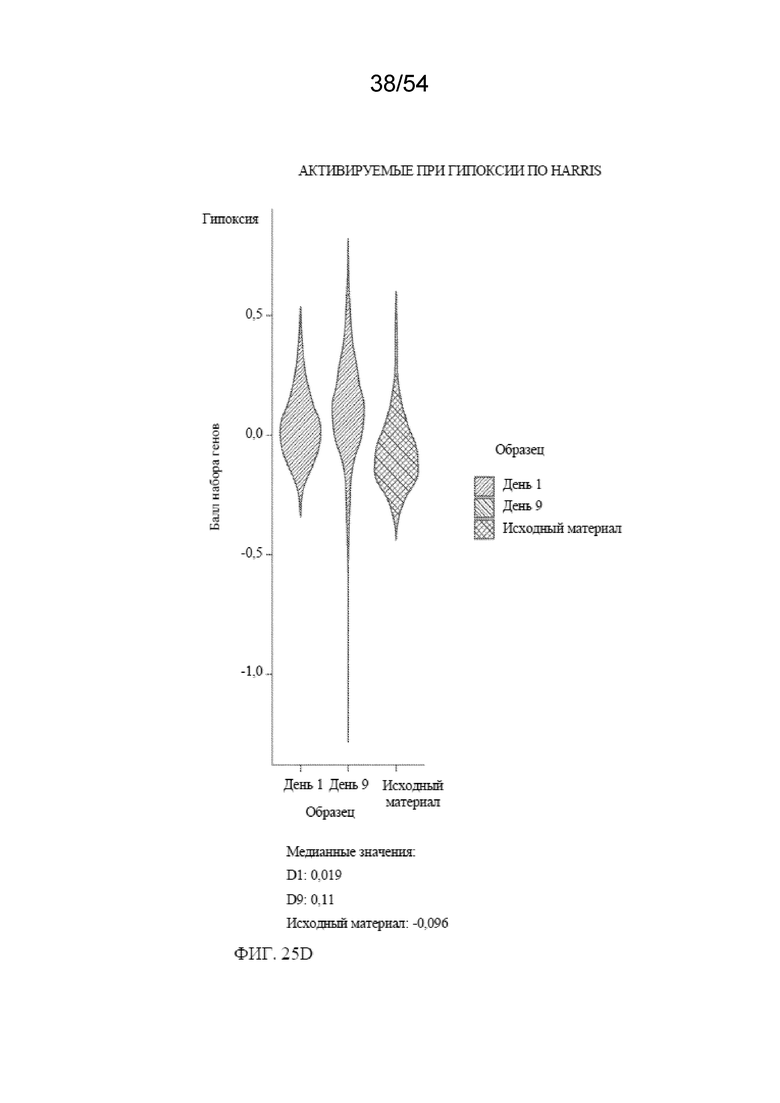
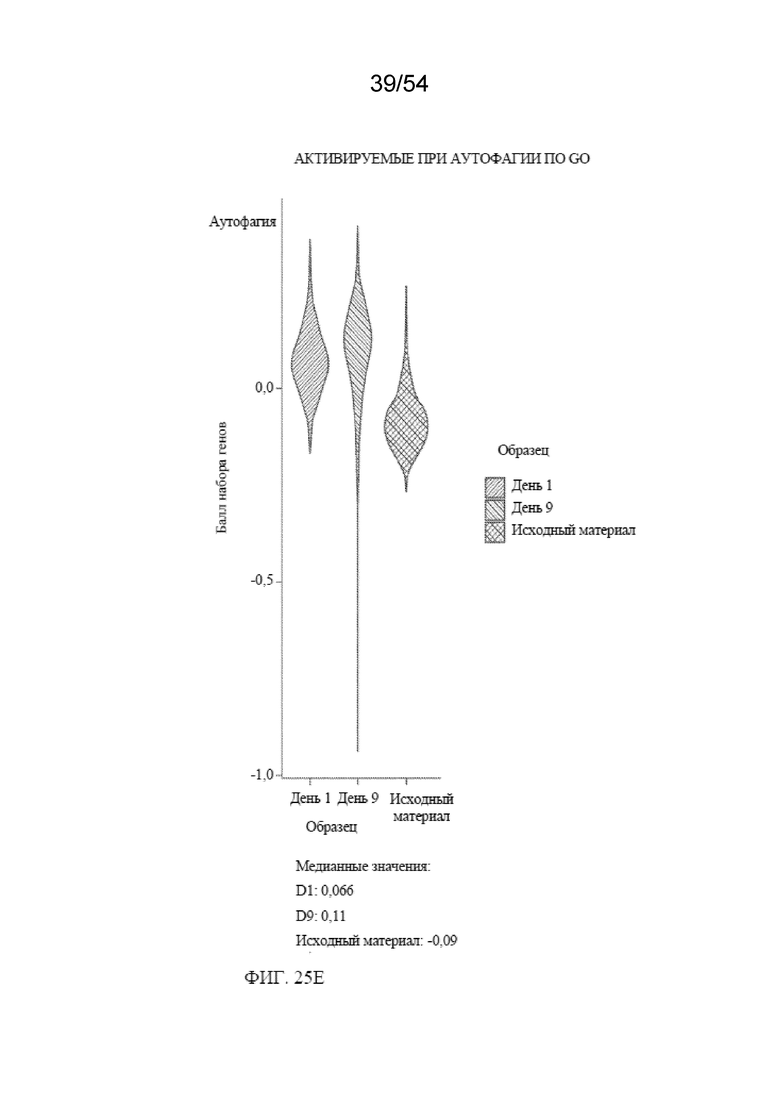
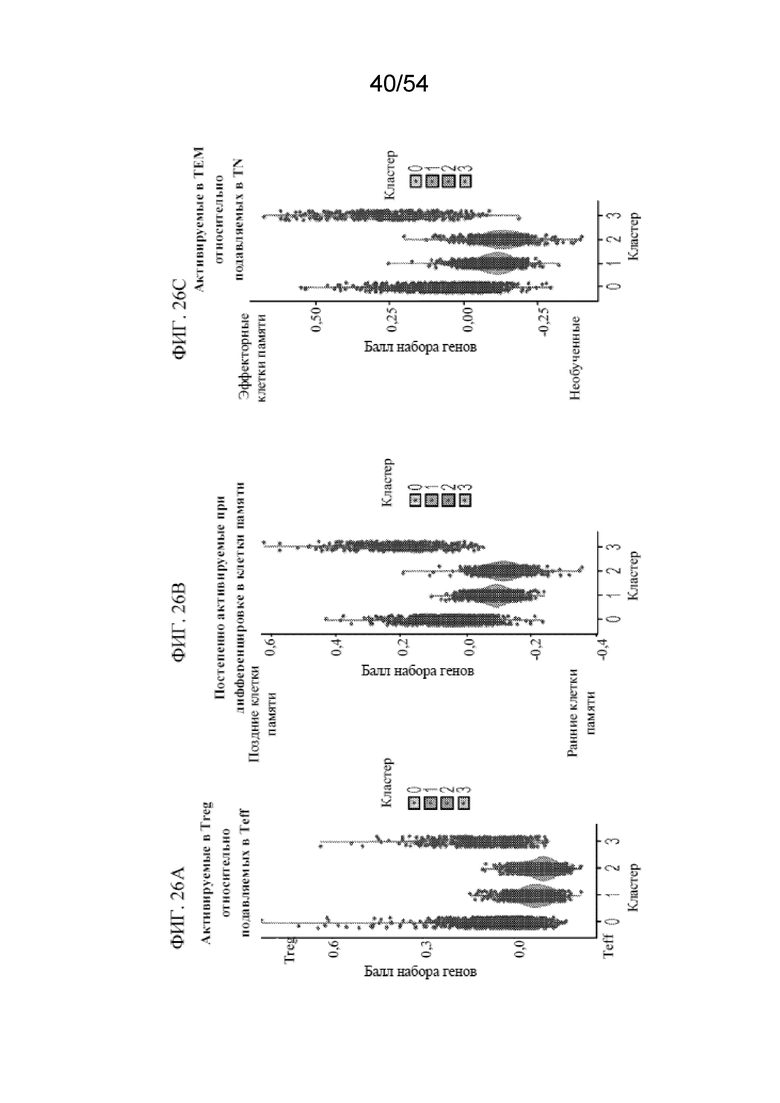
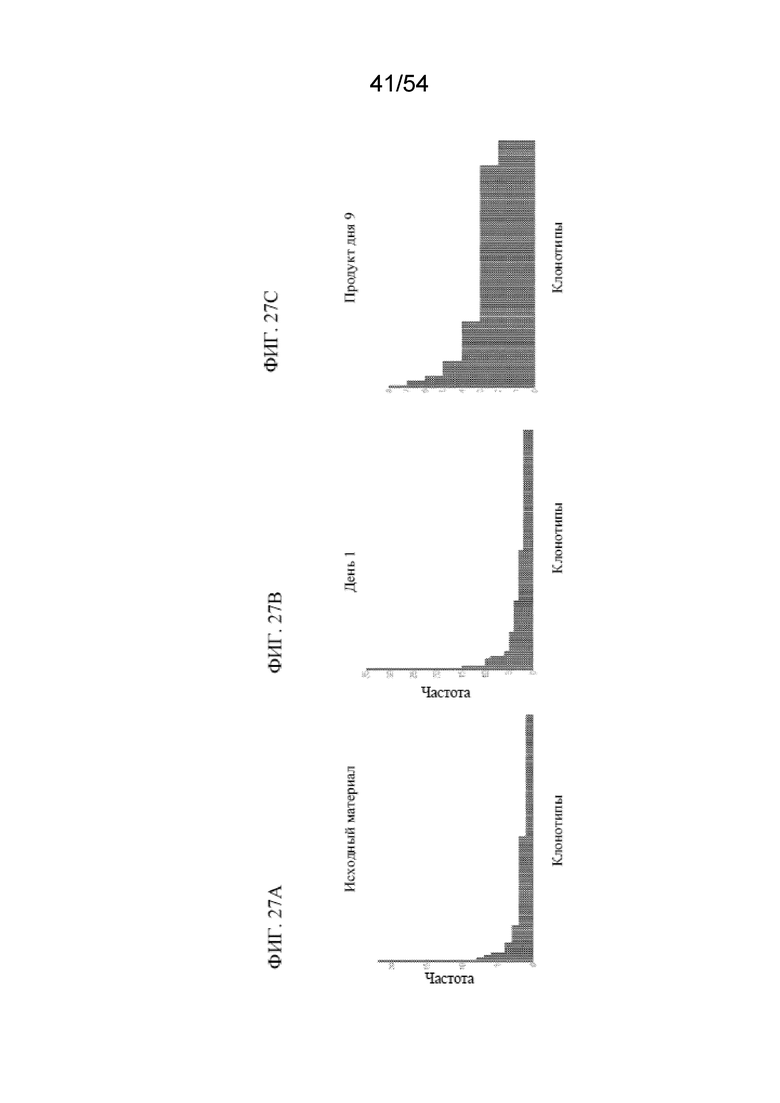
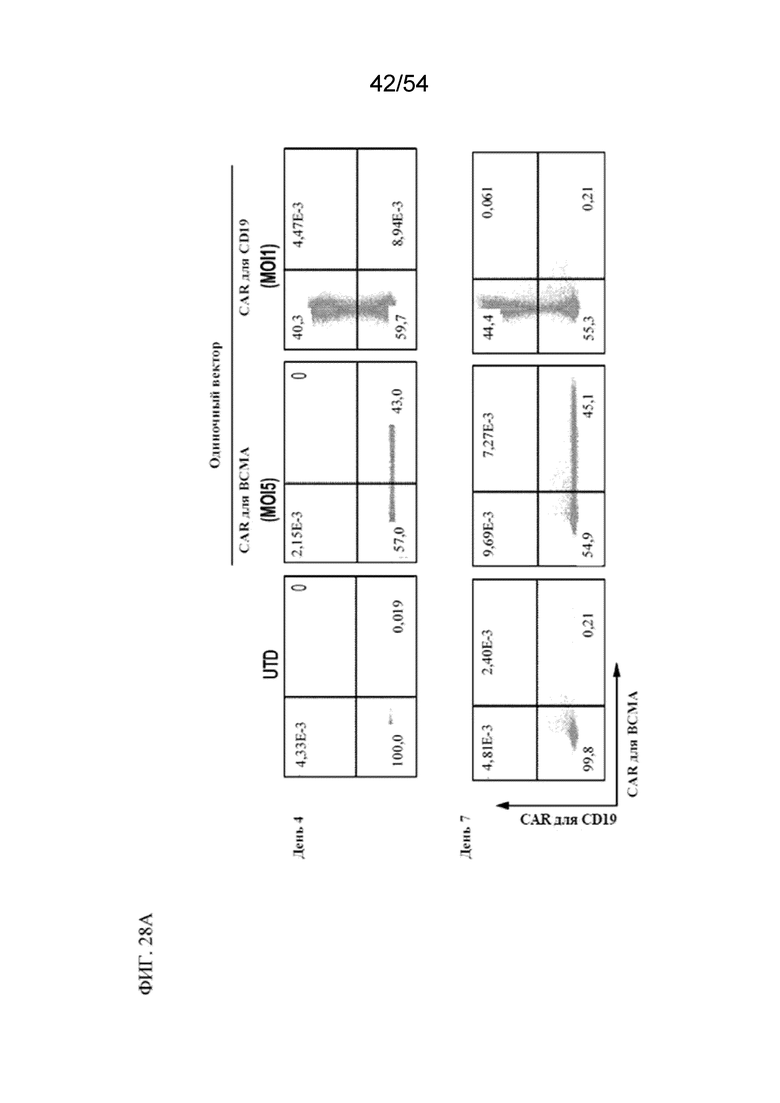

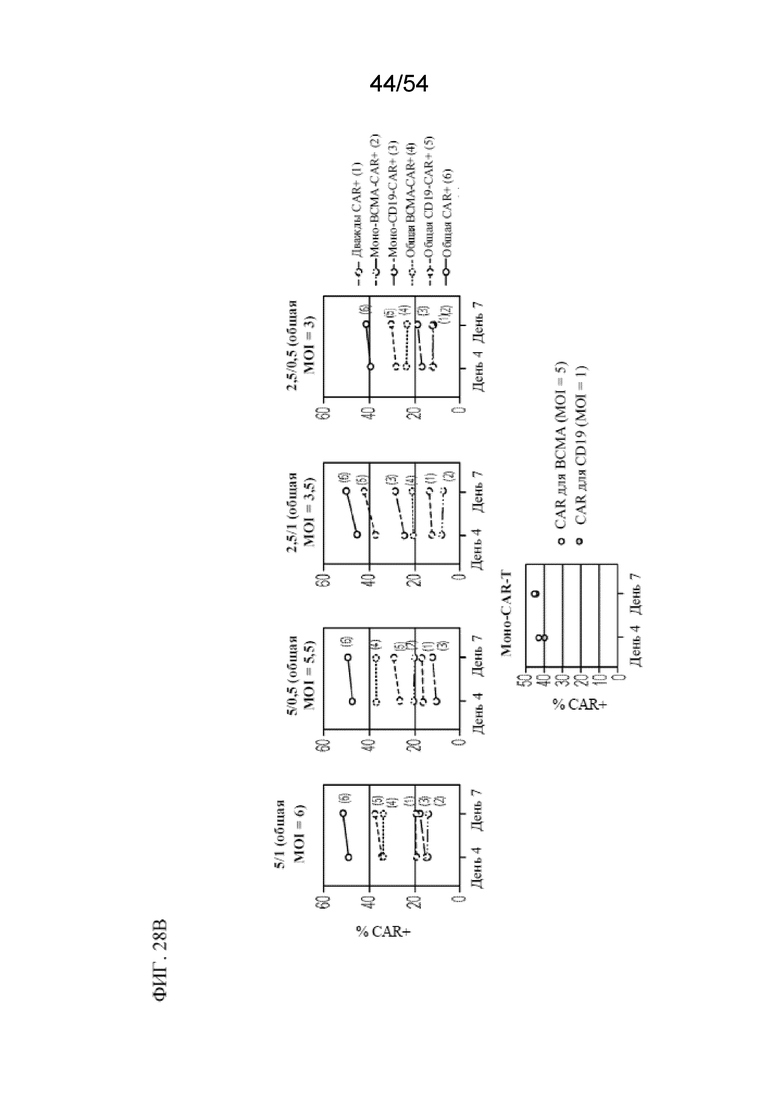
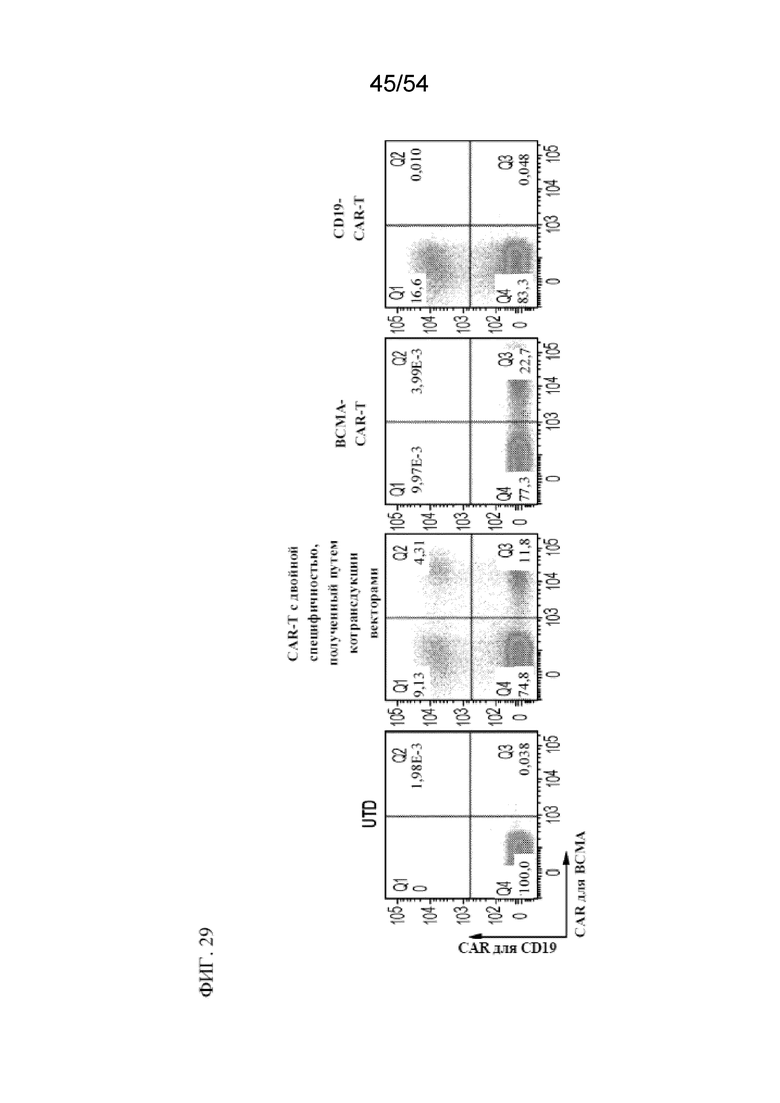
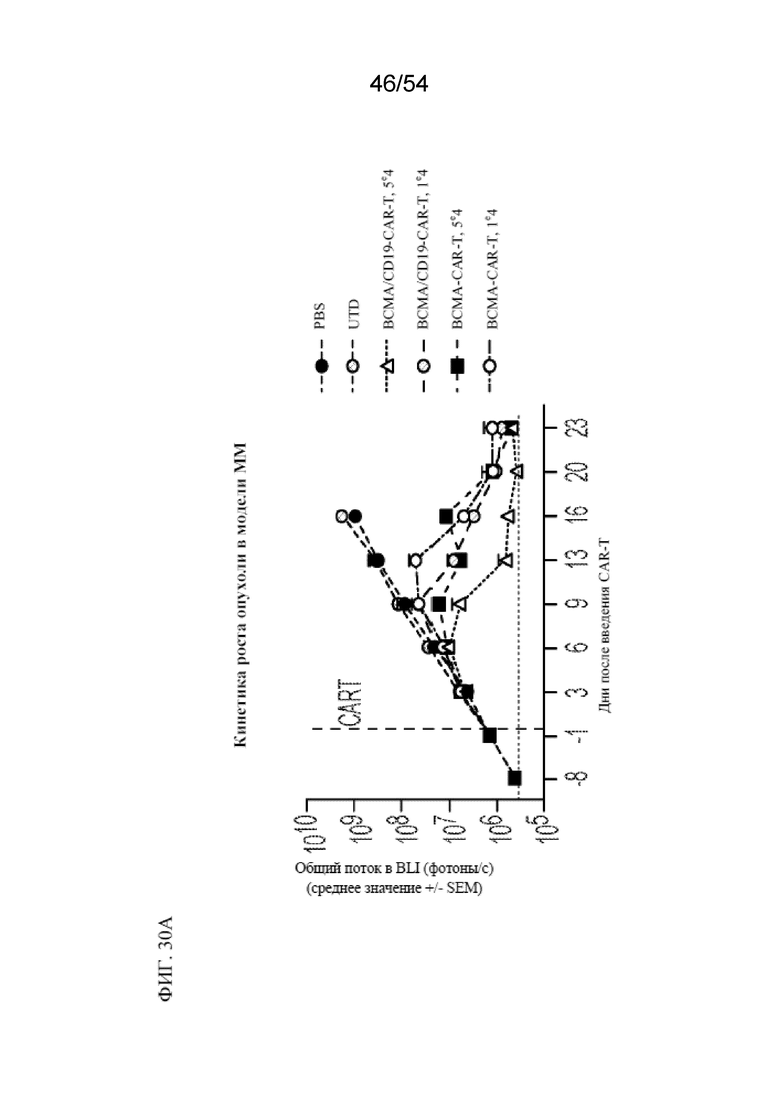
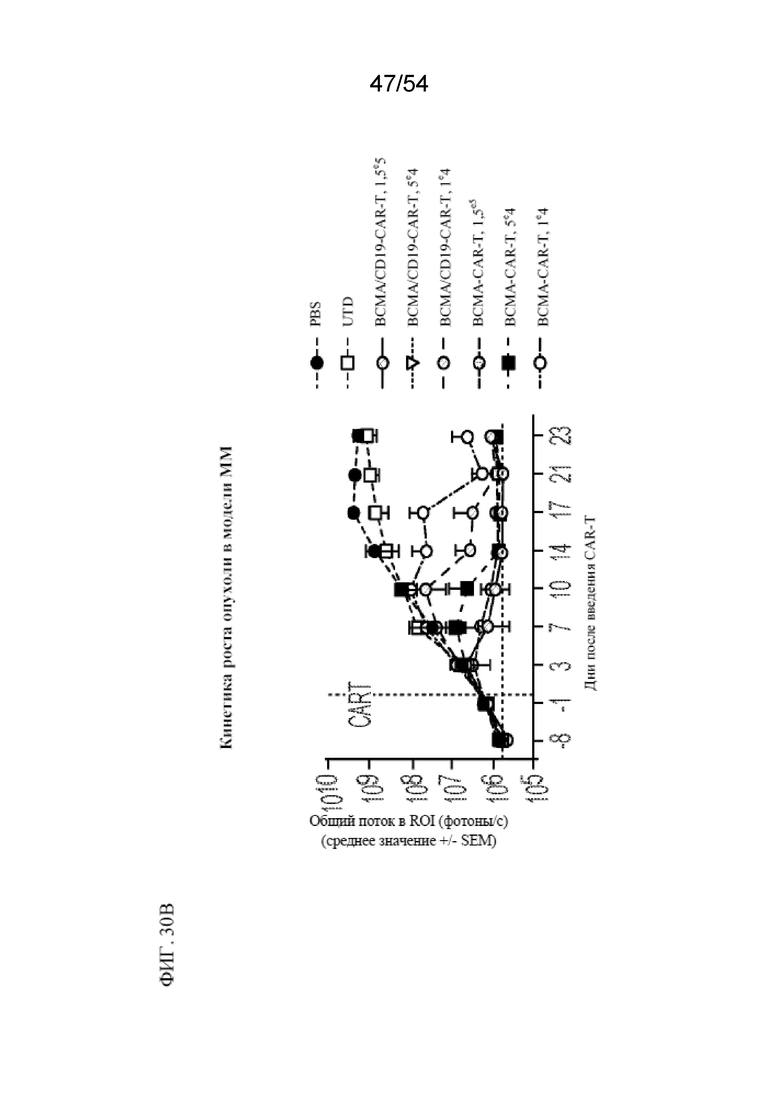
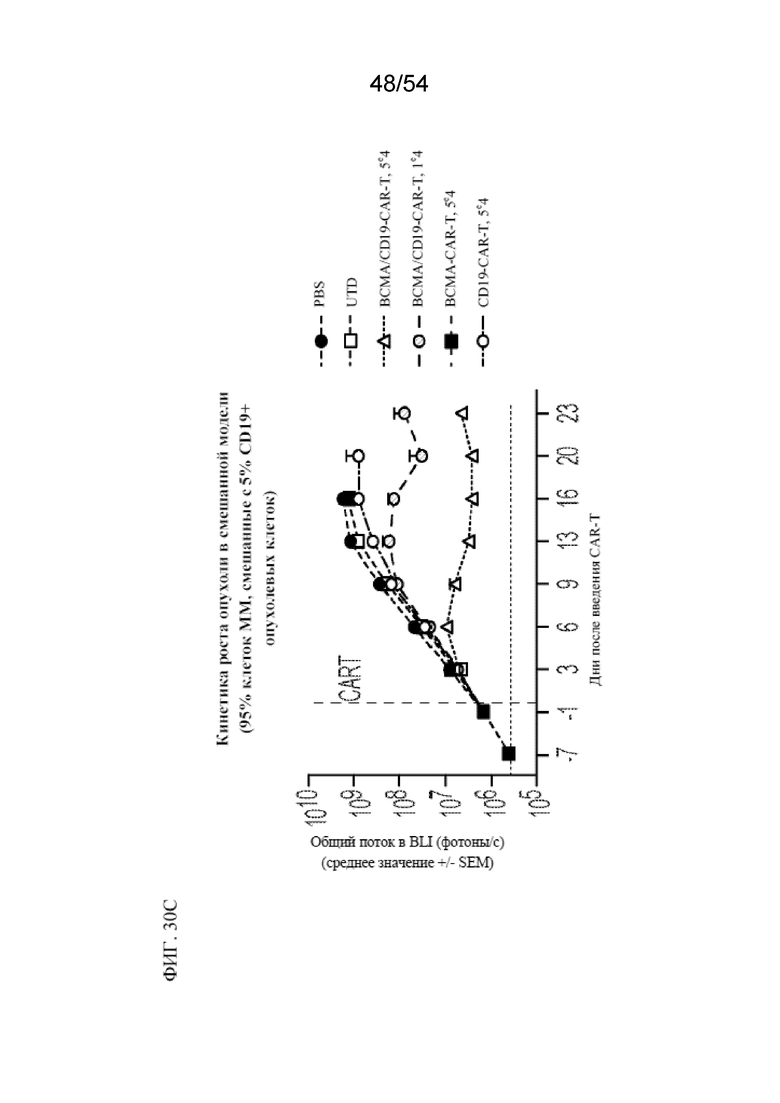
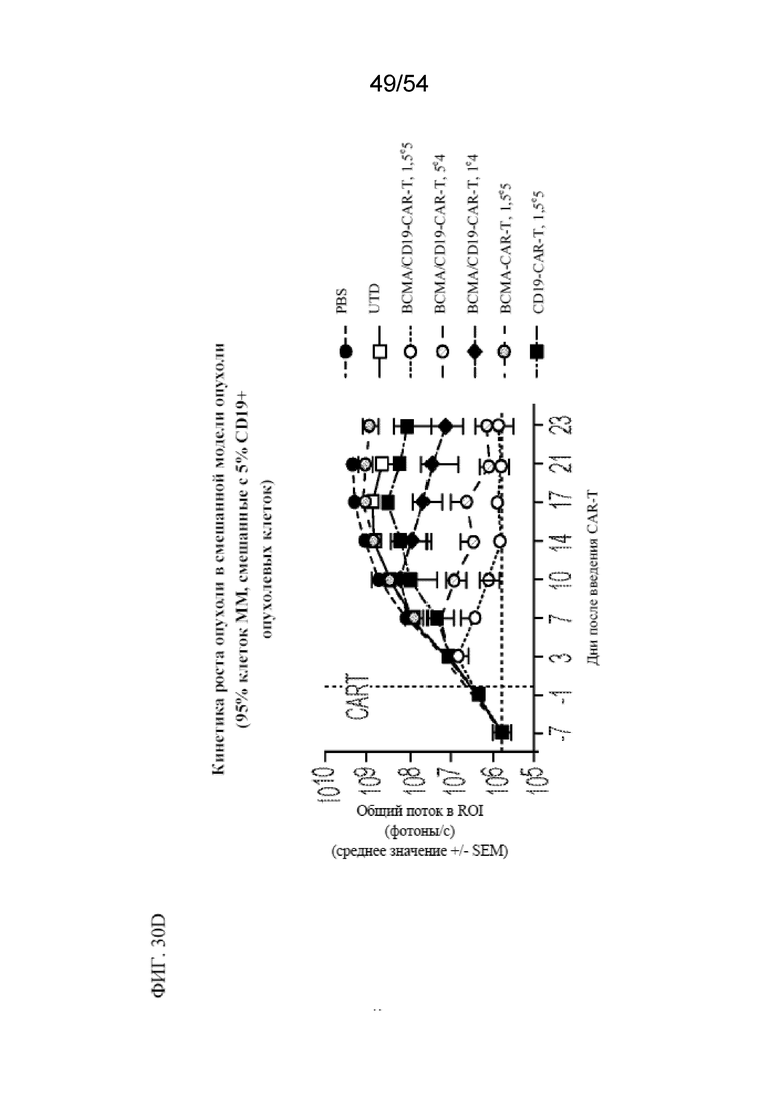
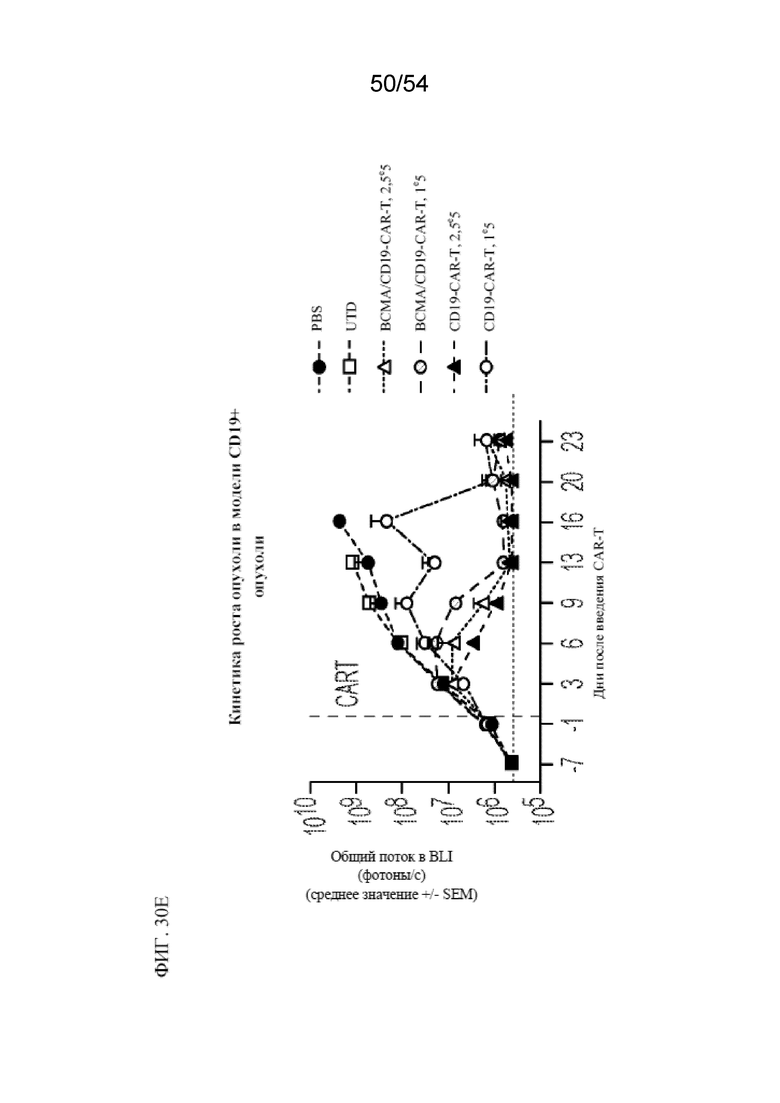
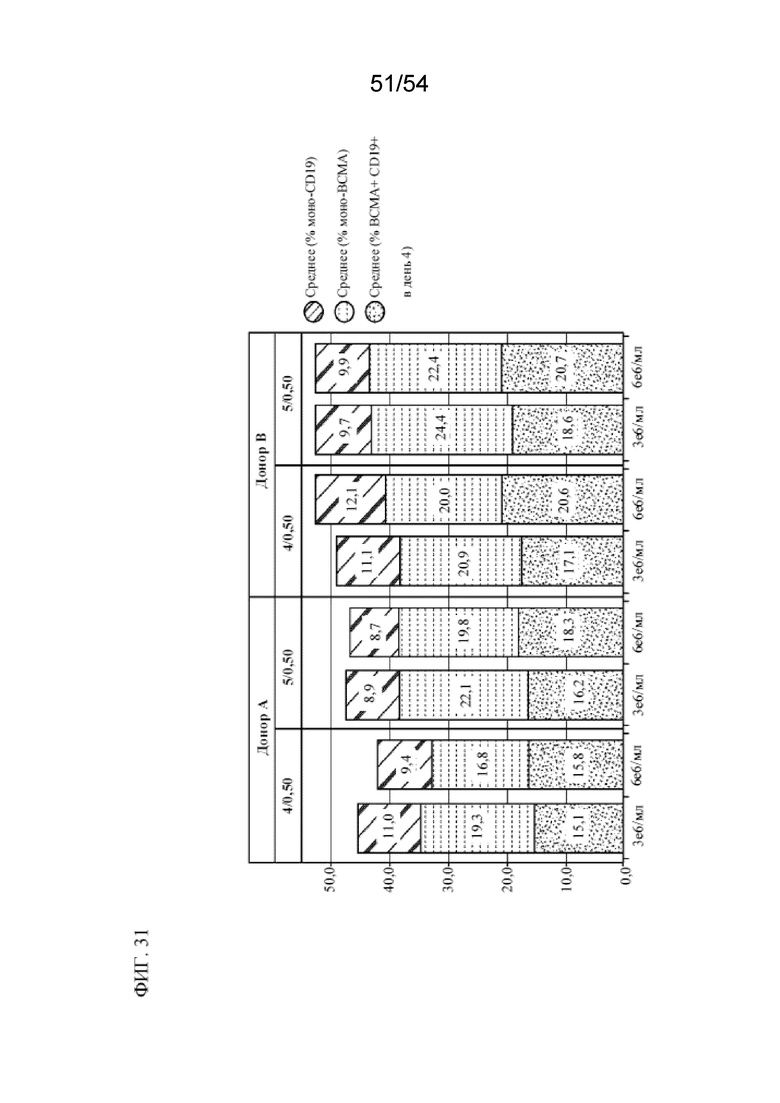
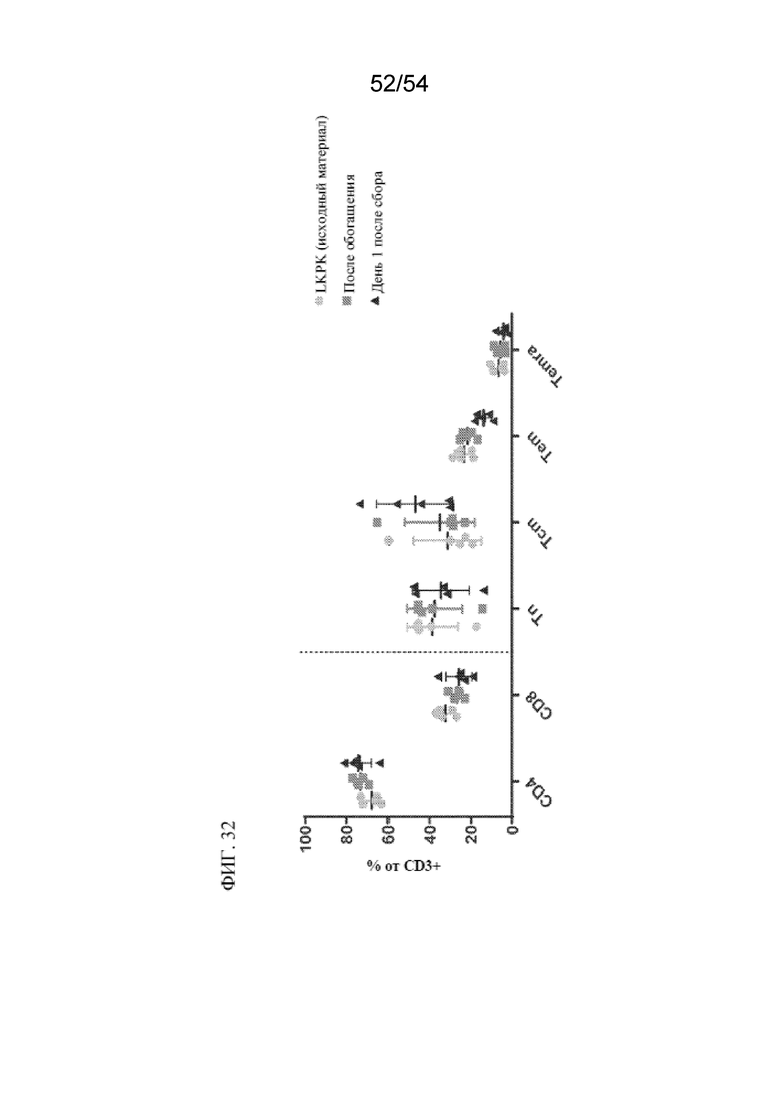

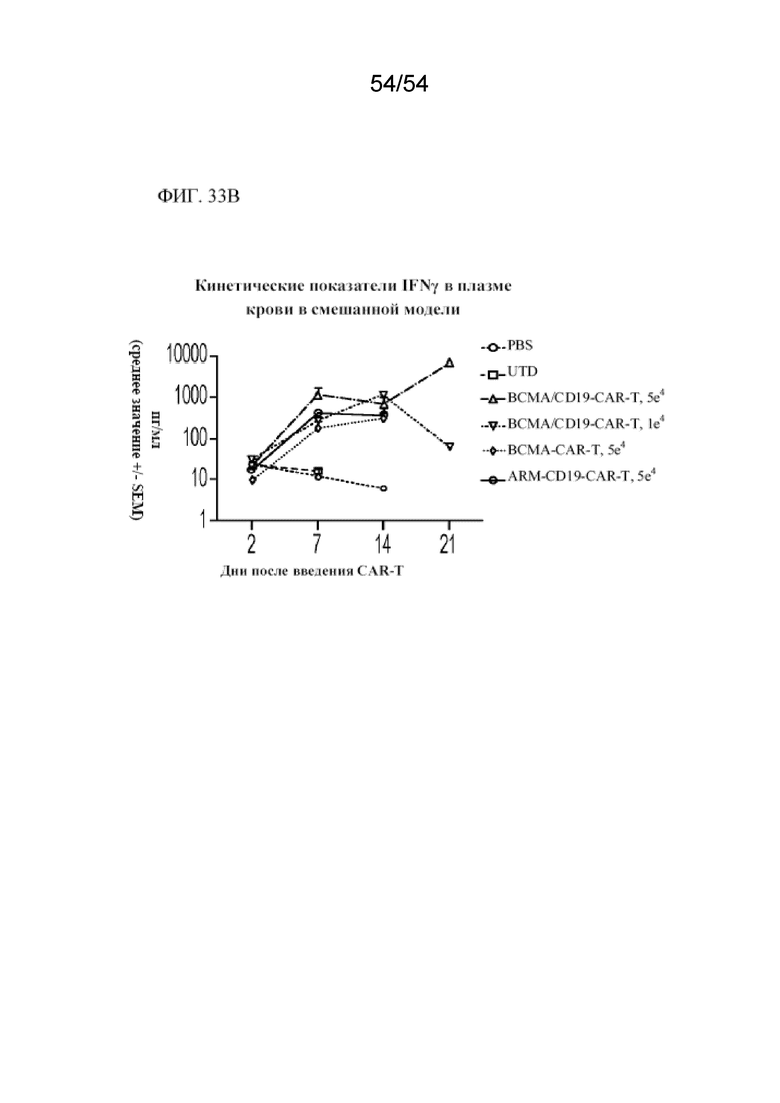
Изобретение относится к области биотехнологии, а именно к T-клетке или NK-клетке, которая включает молекулу CAR, которая содержит первый CAR, который связывается с BCMA (BCMA CAR), второй CAR, который связывается с CD19 (CD19 CAR), к композиции, ее содержащей, а также к выделенной молекуле нуклеиновой кислоты, кодирующей молекулу химерного антигенного рецептора (CAR), где указанная молекула нуклеиновой кислоты содержит первую последовательность нуклеиновой кислоты, кодирующую первый CAR, который связывается с BCMA (BCMA CAR), вторую последовательность нуклеиновой кислоты, кодирующую второй CAR, который связывается с CD19 (CD19 CAR), и выделенной молекуле CAR, связывающейся с BCMA и CD19. Также раскрыты вектор, клетка, содержащие вышеуказанную молекулу нуклеиновой кислоты, а также способ получения указанной клетки. Изобретение также относится к популяции T-клеток или NK-клеток для экспрессии молекулы CAR. Изобретение эффективно для обеспечения противоопухолевого иммунитета у субъекта. 12 н. и 10 з.п. ф-лы, 33 ил., 34 табл., 12 пр.
1. T-клетка или NK-клетка для экспрессии молекулы химерного антигенного рецептора (CAR), где клетка включает молекулу CAR, где указанная молекула CAR содержит:
(a) первый CAR, содержащий первый антигенсвязывающий домен, который связывается с BCMA (BCMA CAR), первый трансмембранный домен и первый внутриклеточный сигнальный домен, содержащий костимулирующий сигнальный домен и первый первичный сигнальный домен; и
(b) второй CAR, содержащий второй антигенсвязывающий домен, который связывается с CD19 (CD19 CAR), второй трансмембранный домен и второй внутриклеточный сигнальный домен, содержащий второй костимулирующий сигнальный домен и второй первичный сигнальный домен;
где:
(i) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно и второй CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(ii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 48, 76, 57, 58 и 59 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(iii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(iv) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 89, 88, 98, 99 и 100 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(v) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 44, 45, 46, 54, 55 и 56 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(vi) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 48, 46, 57, 58 и 59 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(vii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 231, 232, 233, 54, 55 и 240 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(viii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 234, 235, 233, 57, 58 и 241 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(ix) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 231, 232, 233, 54, 55 и 240 соответственно; или
(x) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 234, 235, 233, 57, 58 и 241 соответственно; и
где молекула CAR содержит аминокислотную последовательность SEQ ID NO: 214, 216, 218, 220, 222 или аминокислотную последовательность, характеризующуюся по меньшей мере 95%, 96%, 97%, 98% или 99% идентичностью с ней.
2. Клетка по п. 1, в которой первый CAR кодируется первой последовательностью нуклеиновой кислоты, а второй CAR кодируется второй последовательностью нуклеиновой кислоты, причем первая и вторая последовательности нуклеиновой кислоты расположены в одной молекуле нуклеиновой кислоты.
3. Клетка по п. 2, в которой:
(a) одна молекула нуклеиновой кислоты имеет следующую конфигурацию в ориентации 5'-3':
(i) последовательность нуклеиновой кислоты, кодирующая первый антигенсвязывающий домен, - последовательность нуклеиновой кислоты, кодирующая первый трансмембранный домен, - последовательность нуклеиновой кислоты, кодирующая первый внутриклеточный сигнальный домен, - последовательность нуклеиновой кислоты, кодирующая линкер, - последовательность нуклеиновой кислоты, кодирующая второй антигенсвязывающий домен, - последовательность нуклеиновой кислоты, кодирующая второй трансмембранный домен, - последовательность нуклеиновой кислоты, кодирующая второй внутриклеточный сигнальный домен; или
(ii) последовательность нуклеиновой кислоты, кодирующая второй антигенсвязывающий домен, - последовательность нуклеиновой кислоты, кодирующая второй трансмембранный домен, - последовательность нуклеиновой кислоты, кодирующая второй внутриклеточный сигнальный домен, - последовательность нуклеиновой кислоты, кодирующая линкер, - последовательность нуклеиновой кислоты, кодирующая первый антигенсвязывающий домен, - последовательность нуклеиновой кислоты, кодирующая первый трансмембранный домен, - последовательность нуклеиновой кислоты, кодирующая первый внутриклеточный сигнальный домен,
где линкер необязательно содержит сайт саморасщепления, где линкер необязательно содержит сайт P2A, сайт Т2A, сайт Е2A или сайт F2A, где линкер необязательно содержит сайт P2A, необязательно где:
(I) линкер кодируется последовательностью нуклеиновой кислоты под SEQ ID NO: 209 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней, или
(II) линкер содержит аминокислотную последовательность под SEQ ID NO: 208 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(b) одна молекула нуклеиновой кислоты содержит последовательность нуклеиновой кислоты под SEQ ID NO: 215, 217, 219, 221 или 223 или последовательность нуклеиновой кислоты, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(c) одна молекула нуклеиновой кислоты кодирует аминокислотную последовательность под SEQ ID NO: 214, 216, 218, 220 или 222 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
4. Клетка по п. 1, в которой первый антигенсвязывающий домен или второй антигенсвязывающий домен содержит VH и VL, где VH и VL соединены линкером, где линкер содержит аминокислотную последовательность SEQ ID NO: 5 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
5. Клетка по п. 2, в которой:
(i) первый трансмембранный домен или второй трансмембранный домен содержит аминокислотную последовательность SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(ii) первый трансмембранный домен или второй трансмембранный домен кодируется последовательностью нуклеиновой кислоты SEQ ID NO: 17 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(iii) первый антигенсвязывающий домен соединен с первым трансмембранным доменом с помощью первой шарнирной области и/или второй антигенный домен соединен с вторым трансмембранным доменом с помощью второй шарнирной области, необязательно где:
(a) шарнирная область содержит аминокислотную последовательность SEQ ID NO: 2 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(b) шарнирная область кодируется последовательностью нуклеиновой кислоты SEQ ID NO: 13 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(c) шарнирная область и трансмембранный домен содержат аминокислотную последовательность SEQ ID NO: 202 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(d) шарнирная область и трансмембранный домен кодируются последовательностью нуклеиновой кислоты SEQ ID NO: 203 или 213 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(iv) первичный сигнальный домен содержит функциональный сигнальный домен, полученный из CD3-дзета, где:
(a) первичный сигнальный домен содержит аминокислотную последовательность SEQ ID NO: 9 или 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(b) первичный сигнальный домен кодируется последовательностью нуклеиновой кислоты SEQ ID NO: 20, 21 или 205, последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(v) костимулирующий сигнальный домен содержит функциональный сигнальный домен, полученный из 4-1BB (CD137), где:
(a) костимулирующий сигнальный домен содержит аминокислотную последовательность SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(b) костимулирующий сигнальный домен кодируется последовательностью нуклеиновой кислоты SEQ ID NO: 18 или 204, последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(vi) первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит функциональный сигнальный домен, полученный из 4-1BB, и функциональный сигнальный домен, полученный из CD3-дзета, где:
(a) первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит аминокислотную последовательность SEQ ID NO: 7, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней и аминокислотную последовательность SEQ ID NO: 9 или 10, или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(b) первый внутриклеточный сигнальный домен или второй внутриклеточный сигнальный домен содержит аминокислотную последовательность SEQ ID NO: 7 и аминокислотную последовательность SEQ ID NO: 9 или 10; или
(vii) первый CAR дополнительно содержит первую лидерную последовательность или второй CAR дополнительно содержит вторую лидерную последовательность, где:
(a) первая или вторая лидерная последовательность содержит аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(b) первая или вторая лидерная последовательность кодируются последовательностью нуклеиновой кислоты SEQ ID NO: 199 или 210 или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней.
6. Клетка по п. 5, где:
(a)
(i) первая лидерная последовательность и вторая лидерная последовательность кодируются разными последовательностями нуклеиновых кислот;
(ii) первая шарнирная область и вторая шарнирная область кодируются разными последовательностями нуклеиновых кислот;
(iii) первый трансмембранный домен и второй трансмембранный домен кодируются разными последовательностями нуклеиновых кислот; и/или
(iv) первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен кодируются разными последовательностями нуклеиновых кислот;
(b)
(i) первая лидерная последовательность и вторая лидерная последовательность содержат одинаковую аминокислотную последовательность или содержат разные аминокислотные последовательности;
(ii) первая шарнирная область и вторая шарнирная область содержат одинаковую аминокислотную последовательность или содержат разные аминокислотные последовательности;
(iii) первый трансмембранный домен и второй трансмембранный домен содержат одинаковую аминокислотную последовательность или содержат разные аминокислотные последовательности; и/или
(iv) первый внутриклеточный сигнальный домен и второй внутриклеточный сигнальный домен содержат одинаковую аминокислотную последовательность или содержат разные аминокислотные последовательности, необязательно где:
(I) первый первичный сигнальный домен и второй первичный сигнальный домен кодируются разными последовательностями нуклеиновых кислот;
(II) первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются разными последовательностями нуклеиновых кислот;
(III) первая лидерная последовательность и вторая лидерная последовательность содержат аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(IV) первая шарнирная область и вторая шарнирная область содержат аминокислотную последовательность SEQ ID NO: 2 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(V) первый трансмембранный домен и второй трансмембранный домен содержат аминокислотную последовательность SEQ ID NO: 6 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(VI) первый первичный сигнальный домен и второй первичный сигнальный домен содержат аминокислотную последовательность SEQ ID NO: 10 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(VII) первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен содержат аминокислотную последовательность SEQ ID NO: 7 или аминокислотную последовательность, характеризующуюся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(c)
(i) первая лидерная последовательность и вторая лидерная последовательность кодируются последовательностями нуклеиновых кислот, включающими SEQ ID NO: 199 и 210 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней или SEQ ID NO: 210 и 199 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(ii) первая шарнирная область и вторая шарнирная область кодируются последовательностями нуклеиновых кислот, включающими SEQ ID NO: 337 и 13 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней или SEQ ID NO: 13 и 337 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(iii) первый трансмембранный домен и второй трансмембранный домен кодируются последовательностями нуклеиновых кислот, включающими SEQ ID NO: 338 и 17 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней или SEQ ID NO: 17 и 338 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней;
(iv) первый костимулирующий сигнальный домен и второй костимулирующий сигнальный домен кодируются последовательностями нуклеиновых кислот, включающими SEQ ID NO: 204 и 18 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней или SEQ ID NO: 18 и 204 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; и/или
(v) первый первичный сигнальный домен и второй первичный сигнальный домен кодируются последовательностями нуклеиновых кислот, включающими SEQ ID NO: 205 и 21 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней или SEQ ID NO: 21 и 205 соответственно, или последовательностью нуклеиновой кислоты, характеризующейся по меньшей мере приблизительно 85%, 90%, 95% или 99% идентичностью последовательности с ней; или
(d) первый CAR или второй CAR кодируется молекулой нуклеиновой кислоты, включающей посттранскрипционный регуляторный элемент вируса гепатита лесных сурков (WPRE).
7. Выделенная молекула нуклеиновой кислоты, кодирующая молекулу химерного антигенного рецептора (CAR), где указанная молекула нуклеиновой кислоты содержит:
(a) первую последовательность нуклеиновой кислоты, кодирующую первый CAR, содержащий первый антигенсвязывающий домен, который связывается с BCMA (BCMA CAR), первый трансмембранный домен и первый внутриклеточный сигнальный домен, содержащий костимулирующий сигнальный домен и первый первичный сигнальный домен; и
(b) вторую последовательность нуклеиновой кислоты, кодирующую второй CAR, содержащий второй антигенсвязывающий домен, который связывается с CD19 (CD19 CAR), второй трансмембранный домен и второй внутриклеточный сигнальный домен, содержащий второй костимулирующий сигнальный домен и второй первичный сигнальный домен;
где:
(i) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно и второй CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(ii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 48, 76, 57, 58 и 59 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(iii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(iv) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 89, 88, 98, 99 и 100 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(v) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 44, 45, 46, 54, 55 и 56 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(vi) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 48, 46, 57, 58 и 59 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(vii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 231, 232, 233, 54, 55 и 240 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(viii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 234, 235, 233, 57, 58 и 241 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(ix) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 231, 232, 233, 54, 55 и 240 соответственно; или
(x) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 234, 235, 233, 57, 58 и 241 соответственно; и
где кодируемая молекула CAR содержит аминокислотную последовательность SEQ ID NO: 214, 216, 218, 220, 222 или аминокислотную последовательность, характеризующуюся по меньшей мере 95%, 96%, 97%, 98% или 99% идентичностью с ней; и где первая последовательность нуклеиновой кислоты и вторая последовательность нуклеиновой кислоты расположены в одной молекуле нуклеиновой кислоты.
8. Выделенная молекула CAR, связывающаяся с BCMA и CD19, где молекула CAR содержит:
(a) первый CAR, содержащий первый антигенсвязывающий домен, который связывается с BCMA (BCMA CAR), первый трансмембранный домен и первый внутриклеточный сигнальный домен, содержащий костимулирующий сигнальный домен и первый первичный сигнальный домен; и
(b) второй CAR, содержащий второй антигенсвязывающий домен, который связывается с CD19 (CD19 CAR), второй трансмембранный домен и второй внутриклеточный сигнальный домен, содержащий второй костимулирующий сигнальный домен и второй первичный сигнальный домен;
где:
(i) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 44, 45, 76, 54, 55 и 56 соответственно и второй CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(ii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 48, 76, 57, 58 и 59 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(iii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 86, 87, 88, 95, 96 и 97 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(iv) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 89, 88, 98, 99 и 100 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(v) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 44, 45, 46, 54, 55 и 56 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(vi) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 47, 48, 46, 57, 58 и 59 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(vii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 231, 232, 233, 54, 55 и 240 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно;
(viii) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 234, 235, 233, 57, 58 и 241 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно;
(ix) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 295, 245, 246, 247, 248 и 249 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 231, 232, 233, 54, 55 и 240 соответственно; или
(x) первый CAR содержит CDR1 HC, CDR2 HC, CDR3 HC, CDR1 LC, CDR2 LC и CDR3 LC с SEQ ID NO: 310, 311, 246, 316, 317 и 318 соответственно и второй CAR, каждый, содержит HCDR1 HC, HCDR2 HC, HCDR3 HC, LCDR1 LC, LCDR2 LC и LCDR3 LC, как указано в одной из SEQ ID NO: 234, 235, 233, 57, 58 и 241 соответственно; и
где молекула CAR содержит аминокислотную последовательность SEQ ID NO: 214, 216, 218, 220, 222 или аминокислотную последовательность, характеризующуюся по меньшей мере 95%, 96%, 97%, 98% или 99% идентичностью с ней.
9. Вектор для экспрессии молекулы CAR, содержащий молекулу нуклеиновой кислоты по п. 7.
10. Вектор по п. 9, отличающийся тем, что вектор выбран из ДНК-вектора, РНК-вектора, плазмиды, лентивирусного вектора, аденовирусного вектора или ретровирусного вектора.
11. T-клетка или NK-клетка для экспрессии молекулы химерного антигенного рецептора (CAR), где клетка содержит молекулу нуклеиновой кислоты по п. 7.
12. Способ получения клетки, включающий трансдукцию T-клетки или NK-клетки с помощью вектора по п. 9.
13. Способ получения РНК-сконструированной клетки, включающий введение РНК, транскрибированной in vitro, или синтетической РНК в клетку, где РНК содержит молекулу нуклеиновой кислоты по п. 7, и где клетка представляет собой T-клетку или NK-клетку.
14. Фармацевтическая композиция для обеспечения противоопухолевого иммунитета у субъекта, где фармацевтическая композиция содержит T-клетку или NK-клетку по любому из пп. 1-6 или 11 и фармацевтически приемлемый носитель.
15. Применение эффективного количества популяции T-клеток или NK-клеток, содержащей молекулу нуклеиновой кислоты по п. 7, молекулу CAR по п. 8 или вектор по п. 9 или 10; T-клетку или NK-клетку по любому из пп. 1-6 или 11; или фармацевтическую композицию по п. 14 для обеспечения противоопухолевого иммунитета у субъекта.
16. Применение по п. 15, где у субъекта:
(i) рак, или злокачественное новообразование, или предраковое состояние, выбранное из одного или нескольких из миелодисплазии, миелодиспластического синдрома или предлейкоза,
(ii) гематологический рак или солидный рак;
(iii) острый лейкоз, B-клеточный острый лимфоидный лейкоз ("BALL"), T-клеточный острый лимфоидный лейкоз ("TALL"), острый лимфоидный лейкоз (ALL), хронический миелогенный лейкоз (CML), хронический лимфоцитарный лейкоз (CLL), B-клеточный пролимфоцитарный лейкоз, новообразование из бластных плазмоцитоидных дендритных клеток, лимфома Беркитта, диффузная крупноклеточная B-клеточная лимфома, фолликулярная лимфома, волосатоклеточный лейкоз, мелкоклеточная или крупноклеточная фолликулярная лимфома, злокачественные лимфопролиферативные состояния, MALT-лимфома, мантийноклеточная лимфома, лимфома из клеток маргинальной зоны, множественная миелома, миелодисплазия и миелодиспластический синдром, неходжкинская лимфома, плазмобластная лимфома, новообразование из плазмоцитоидных дендритных клеток, макроглобулинемия Вальденстрема, рак предстательной железы, кастрационнорезистентный или резистентный к терапии рак предстательной железы, метастатический рак предстательной железы, рак поджелудочной железы, рак легкого, нарушение пролиферации плазматических клеток, бессимптомная миелома, "тлеющая" множественная миелома, вялотекущая миелома, моноклональная гаммапатия неустановленной этиологии (MGUS), макроглобулинемия Вальденстрема, плазмоцитома, плазмоклеточная дискразия, солитарная миелома, солитарная плазмоцитома, экстрамедуллярная плазмоцитома, множественная плазмоцитома, системный амилоидоз в форме амилоидоза легких цепей или POEMS-синдром (также известный как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром)) или их комбинация; или
(iv) множественная миелома.
17. Применение по п. 15 или 16, где клетка или популяция T-клеток или NK-клеток составлена для введения в комбинации со вторым терапевтическим средством, необязательно где второе терапевтическое средство выбрано из:
(i) ингибитора PD-1, где ингибитор PD-1 необязательно выбран из группы, состоящей из PDR001, ниволумаба, пембролизумаба, пидилизумаба, MEDI0680, REGN2810, TSR-042, PF-06801591 и AMP-224;
(ii) ингибитора PD-L1, где ингибитор PD-L1 необязательно выбран из группы, состоящей из FAZ053, атезолизумаба, авелумаба, дурвалумаба и BMS-936559;
(iii) ингибитора LAG-3, где ингибитор LAG-3 необязательно выбран из группы, состоящей из LAG525, BMS-986016, TSR-033, MK-4280 и REGN3767;
(iv) ингибитора TIM-3, где ингибитор TIM-3 необязательно выбран из группы, состоящей из MBG453, TSR-022 и LY3321367;
(v) ингибитора CTLA-4, где ингибитор CTLA-4 необязательно представляет собой ипилимумаб или тремелимумаб;
(vi) полипептида интерлейкина-15 (IL-15), полипептида альфа-субъединицы рецептора интерлейкина-15 (IL-15Ra) или комбинации как полипептида IL-15, так и полипептида IL-15Ra, например hetIL-15;
(vii) полипептида интерлейкина-12 (IL-12); или
(viii) ингибитора mTOR, где ингибитор mTOR необязательно представляет собой RAD001 или рапамицин.
18. Популяция T-клеток или NK-клеток для экспрессии молекулы CAR, где популяция включает:
первую популяцию клеток, экспрессирующую BCMA CAR, но не CD19 CAR;
вторую популяцию клеток, экспрессирующую CD19 CAR, но не BCMA CAR; и
третью популяцию клеток, экспрессирующую как BCMA CAR, так и CD19 CAR;
где клетки первой, второй и третьей популяции содержат молекулу нуклеиновой кислоты по п. 7.
19. Популяция T-клеток или NK-клеток для экспрессии молекулы CAR, включающая молекулу нуклеиновой кислоты по п. 7, где первая последовательность нуклеиновой кислоты и вторая последовательность нуклеиновой кислоты разделены третьей последовательностью нуклеиновой кислоты, кодирующей сайт саморасщепления.
20. Способ обеспечения противоопухолевого иммунитета у субъекта, включающий введение субъекту эффективного количества популяции T-клеток или NK-клеток, содержащей молекулу нуклеиновой кислоты по п. 7, молекулу CAR по п. 8 или вектор по п. 9 или 10; клетки по любому из пп. 1-6 или 11; или фармацевтической композиции по п. 14.
21. Способ по п. 20, в котором у субъекта:
(i) рак, или злокачественное новообразование, или предраковое состояние, выбранное из одного или нескольких из миелодисплазии, миелодиспластического синдрома или предлейкоза,
(ii) гематологический рак или солидный рак;
(iii) острый лейкоз, B-клеточный острый лимфоидный лейкоз ("BALL"), T-клеточный острый лимфоидный лейкоз ("TALL"), острый лимфоидный лейкоз (ALL), хронический миелогенный лейкоз (CML), хронический лимфоцитарный лейкоз (CLL), B-клеточный пролимфоцитарный лейкоз, новообразование из бластных плазмоцитоидных дендритных клеток, лимфома Беркитта, диффузная крупноклеточная B-клеточная лимфома, фолликулярная лимфома, волосатоклеточный лейкоз, мелкоклеточная или крупноклеточная фолликулярная лимфома, злокачественные лимфопролиферативные состояния, MALT-лимфома, мантийноклеточная лимфома, лимфома из клеток маргинальной зоны, множественная миелома, миелодисплазия и миелодиспластический синдром, неходжкинская лимфома, плазмобластная лимфома, новообразование из плазмоцитоидных дендритных клеток, макроглобулинемия Вальденстрема, рак предстательной железы, кастрационнорезистентный или резистентный к терапии рак предстательной железы, метастатический рак предстательной железы, рак поджелудочной железы, рак легкого, нарушение пролиферации плазматических клеток, бессимптомная миелома, "тлеющая" множественная миелома, вялотекущая миелома, моноклональная гаммапатия неустановленной этиологии (MGUS), макроглобулинемия Вальденстрема, плазмоцитома, плазмоклеточная дискразия, солитарная миелома, солитарная плазмоцитома, экстрамедуллярная плазмоцитома, множественная плазмоцитома, системный амилоидоз в форме амилоидоза легких цепей или POEMS-синдром (также известный как синдром Кроу-Фукаса, болезнь Такатсуки и PEP-синдром)) или их комбинация; или
(iv) множественная миелома.
22. Способ по п. 20 или 21, в котором популяция T-клеток или NK-клеток составлена для введения в комбинации с вторым терапевтическим средством, необязательно где второе терапевтическое средство выбрано из:
(i) ингибитора PD-1, где ингибитор PD-1 необязательно выбран из группы, состоящей из PDR001, ниволумаба, пембролизумаба, пидилизумаба, MEDI0680, REGN2810, TSR-042, PF-06801591 и AMP-224;
(ii) ингибитора PD-L1, где ингибитор PD-L1 необязательно выбран из группы, состоящей из FAZ053, атезолизумаба, авелумаба, дурвалумаба и BMS-936559;
(iii) ингибитора LAG-3, где ингибитор LAG-3 необязательно выбран из группы, состоящей из LAG525, BMS-986016, TSR-033, MK-4280 и REGN3767;
(iv) ингибитора TIM-3, где ингибитор TIM-3 необязательно выбран из группы, состоящей из MBG453, TSR-022 и LY3321367;
(v) ингибитора CTLA-4, где ингибитор CTLA-4 необязательно представляет собой ипилимумаб или тремелимумаб;
(vi) полипептида интерлейкина-15 (IL-15), полипептида альфа-субъединицы рецептора интерлейкина-15 (IL-15Ra) или комбинации как полипептида IL-15, так и полипептида IL-15Ra, например hetIL-15;
(vii) полипептида интерлейкина-12 (IL-12); или
(viii) ингибитора mTOR, где ингибитор mTOR необязательно представляет собой RAD001 или рапамицин.
| WO 2018237022 A1, 20.06.2018 | |||
| ZHILING YAN et al., A combination of humanised anti-CD19 and anti-BCMA CAR T cells in patients with relapsed or refractory multiple myeloma: a single-arm, phase 2 trial, The Lancet Haematology, Published online August 1, 2019, v | |||
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Колосниковая решетка для генераторов | 1918 |
|
SU521A1 |
| WO 2019089798 A1, 09.05.2019 | |||
| Химерные антигенные рецепторы, нацеленные на антиген созревания B-клеток | 2016 |
|
RU2706582C2 |
Авторы
Даты
2025-06-04—Публикация
2020-11-25—Подача