Область изобретения
Настоящее изобретение относится к способам идентификации мишеней для антибиотиков в бактерии, к способам идентификации антибиотиков и к способам получения антибиотиков и фармацевтических композиций, содержащих антибиотики.
Уровень техники
Существует насущная необходимость разработки новых антибиотиков, чтобы противостоять появлению новых патогенов и резистентности к существующим противомикробным препаратам. Важной задачей является идентификация мишеней для антибиотиков-кандидатов, поскольку такая информация может открыть доступ к большому количеству семейств функционально родственных новых лекарственных препаратов. Например, открытие пенициллин-связывающих белков как мишени пенициллина привело к разработке большого семейства антибиотиков, включая несколько поколений цефалоспоринов, пенициллинов и карбапенемов (см. Schmid (2006) Nature Biotechnology 24(4): 419-420).
Недавно было описано транспозон-направленное секвенирование сайтов встраивания (TraDIS - см. Langridge et al. (2009) Genome Research 19: 2308-2316), и его применили для идентификации: (а) жизненно важных генов; (б) генов, способствующих росту (но не жизненно важных); (в) генов, неблагоприятных для роста при определенных условиях; и (г) генов, участвующих в придании устойчивости к определенным условиям («нише-специфичные» жизненно важные гены). Похожие технологии были описаны, например, у Gawronski et al. (2009) PNAS 106: 16422-16427; Goodman et al. (2009) Cell Host Microbe 6: 279-289; van Opijnen et al. (2009) Nat. Methods 6: 767-772 and Gallagher et al. (2011) mBio 2(1):e00315-10, и в настоящее время такие технологии в совокупности назвали технологией «Tn-seq».
Однако важным классом мишеней для воздействия антибиотиков являются продукты генов, участвующие в клеточных процессах, важных для жизнеспособности при применяемых условиях роста. Такие мишени нельзя идентифицировать способом Tn-seq (включая TraDIS), поскольку вставки транспозонов в жизненно важные гены (включая гены, выступающие в роли мишеней воздействии антибиотиков) не представлены в значительной степени в общем числе мутантов. Таким образом, различий в распределении транспозонов после роста общей совокупности мутантов в присутствии или в отсутствии антибиотиков (или с разным их количеством), не возникает, вследствие чего Tn-seq не позволяет провести различие между жизненно важным геном и жизненно важным геном, выступающим в роли мишени воздействия антибиотиков.
Следовательно, существует потребность в высокопроизводительных подходах функционального скрининга мишеней действия антибиотиков, которые позволили бы выявлять жизненно важные гены, служащие мишенями для антибиотиков.
Сущность изобретения
Согласно одному аспекту настоящего изобретения предложен способ идентификации жизненно важных генов, которые служат мишенями для антибиотиков у бактерий, способ, включающий этапы:
(а) создания совокупности мутантных бактерий путем транспозонного мутагенеза с применением активирующего транспозона (TnA), причем указанный TnA содержит промотор, такой, что при встраивании транспозона в ДНК бактерии увеличивается транскрипция гена поблизости от сайта встраивания;
(б) выращивания бактерий из указанной совокупности мутантов в присутствии разных количеств указанного антибиотика с получением двух или более культур для тестирования; и
(в) сравнения распределения вставок TnA между культурами для тестирования с целью идентификации предполагаемого жизненно важного гена, служащего мишенью для указанного антибиотика у указанной бактерии.
Указанный способ также может включать этапы: создания устойчивого к антибиотикам мутанта указанной бактерии при помощи способа, включающего этап отбора в ходе роста в присутствии указанного антибиотика для получения клона устойчивого к антибиотикам мутанта (мутант AbR); трансформации мутанта AbR: (i) одним или более жизненно важными генами указанной бактерии; и (ii) транспозоном, который при встраивании инактивирует бактериальную ДНК, с получением совокупности транспозонных мутантов, которые являются меродиплоидными по указанному одному или более жизненно важным генам; выращивания бактерий из указанной меродиплоидной совокупности в присутствии разных количеств указанного антибиотика с получением двух или более культур для тестирования; и сравнения распределения вставок транспозонов между культурами для тестирования с целью идентификации предполагаемого жизненно важного гена, служащего мишенью для указанного антибиотика у указанной бактерии.
Согласно другому аспекту предложен способ идентификации антибиотика, включающий идентификацию жизненно важного гена, который служит мишенью для указанного антибиотика согласно способу, предложенному в настоящем изобретении.
Согласно дополнительному аспекту предложен способ получения антибиотика, включающий идентификацию антибиотика при помощи способа, включающего идентификацию жизненно важного гена, служащего мишенью для указанного антибиотика согласно способу, предложенному в настоящем изобретении. Такой способ может необязательно включать этап синтеза указанного антибиотика, а также необязательно может включать смешивание синтезированного антибиотика с фармацевтически приемлемым вспомогательным веществом с получением фармацевтической композиции.
Применение активирующего транспозона гарантирует, что встраивание транспозона в жизненно важные гены представлено в исходной совокупности мутантов, поскольку встраивание транспозонов может теперь приводить к активации генов, а не к инсерционной инактивации. Таким образом, можно изучать влияние присутствия антибиотика во время последующего культивирования совокупности мутантов на распределение транспозонов (и идентифицировать гены-мишени, определяемые при этом).
Другие аспекты и предпочтительные варианты реализации настоящего изобретения охарактеризованы и описаны в других пунктах формулы изобретения ниже.
Подробное описание изобретения
Все публикации, патенты, заявки на патенты и другие материалы, упоминаемые в настоящей заявке, включены в нее посредством ссылок на их полные версии для любых целей, как если бы каждая отдельная публикация, патент или заявка на патент были специально и индивидуально указаны и включены посредством ссылки, и их содержание было цитировано целиком.
Определения и общие предпочтения
Подразумевается, что следующие термины, применяемые в настоящей заявке, за исключением случаев, когда указано другое, имеют следующие значения наряду с любыми более широкими (или узкими) значениями указанных терминов, которые могут применяться в данной области техники:
Если контекст не требует иного, применение в настоящей заявке единственного числа следует понимать так, что оно включает множественное число, и наоборот. Неопределенные артикли единственного числа применительно к какой-либо сущности следует понимать так, что они относятся к одной или более таким сущностям. Сами по себе неопределенные артикли единственного числа, термин «один или более» и «по меньшей мере один» в настоящей заявке применяются взаимозаменяемо.
В настоящей заявке термин «включает» или его разновидности, такие как «содержит» или «содержащий» следует понимать так, что они указывают на включение любого из перечисленных целых чисел (например, особенность, элемент, характеристика, свойство, этап способа или ограничение) или группы целых чисел (например, особенностей, элементов, характеристик, свойств, этапов способа или ограничений), но не на исключение любого другого целого числа или группы целых чисел. Таким образом, в настоящей заявке термин «включающий» является охватывающим или неисчерпывающим, и не исключает дополнительных, не перечисленных целых чисел и этапов способов.
Термин «ген» представляет собой термин, описывающий единицу наследования, включающую последовательность ДНК, которая занимает специфическое место в хромосоме или плазмиде и определяет конкретные характеристики организма. Ген может определять характеристику организма за счет того, что он обусловливает полипептидную цепь, которая образует белок или часть белка (структурный ген); или может кодировать молекулу РНК; или регулировать работу других генов, или же угнетать такую работу; или влиять на фенотип посредством какого-либо другого, еще не идентифицированного, механизма.
Термин «геномная ДНК» представляет собой термин в области техники, применяемый в настоящей заявке для описания хромосомной ДНК в отличие от внехромосомной плазмидной ДНК.
Термин «геном» представляет собой термин в области техники, который описывает весь набор генетического материала в организме и, таким образом, включает хромосомную, плазмидную, профаговую и любую другую ДНК.
Термин «грамположительная бактерия» представляет собой термин в области техники, описывающий конкретный класс бактерий, которые объединены в одну группу на основании определенных характеристик окрашивания клеточной стенки.
Термин «грамположительная бактерия с низким G+С» представляет собой термин в области техники, описывающий конкретный подкласс родственных с эволюционной точки зрения бактерий в пределах класса грамположительных бактерий на основе состава оснований в ДНК. Данный подкласс включает виды Streptococcus, виды Staphylococcus, виды Listeria, виды Bacillus, виды Clostridium, виды Enterococcus и виды Lactobacillus).
Термин «грамположительная бактерия с высоким G+С» представляет собой термин в области техники, описывающий конкретный подкласс родственных с эволюционной точки зрения бактерий в пределах класса грамположительных бактерий на основе состава оснований в ДНК. Данный подкласс включает актиномицеты (актинобактерии), включая виды Actinomyces, виды Arthrobacter, виды Corynebacterium. виды Frankia, виды Micrococcus, виды Micromonospora, виды Mycobacterium, виды Nocardia, виды Propionibacterium и виды Streptomyces.
Термин «грамотрицательная бактерия» представляет собой термин в области техники, описывающий конкретный класс бактерий, которые объединены в одну группу на основании определенных характеристик окрашивания клеточной стенки. Примеры грамотрицательных бактерий включают Klebsiella, Acinetobacter, Escherichia, Pseudomonas, Enterobacter и Neisseria.
В настоящей заявке термин «жизненно важной ген» представляет собой термин в области техники, описывающий конкретный класс генов, продукты которых необходимы для жизнеспособности, либо при всех условиях, либо при применяемых условиях роста. Важным подклассом жизненно важных генов являются гены, кодирующие продукты (например, белки, пептиды и регуляторные полинуклеотиды), которые вносят вклад в метаболические процессы, необходимы для жизнеспособности при важных условиях роста (например, и в случае патогенных бактерий, при условиях, которые преобладают при инфекции или размножении в организме хозяина).
Антибиотики и мишени воздействия антибиотиков
Антибиотик, применяемый для получения тестируемых культур согласно настоящему изобретению, является новым исследуемым антибиотиком (антибактериальным химиотерапевтических агентом), механизм действия (и, следовательно, биологические мишени) которого не известен. Во многих областях применения антибиотик выбирают из комбинаторных библиотек, библиотек природных продуктов, описанных химических объектов, пептидов, миметиков пептидов и олигонуклеотидов. Мишень воздействия антибиотика, идентифицируемая согласно настоящему изобретению, является жизненно важным геном/продуктом жизненно важного гена, и, следовательно, может участвовать в одном или более из следующих биологических процессов в клетке бактерии-хозяина:
(а) деление клеток;
(б) репликация ДНК (включая полимеризацию и суперспирализацию);
(в) транскрипция (включая праймирование, элонгацию и терминацию);
(г) трансляция (включая рибосомальные компоненты, инициацию, элонгацию и высвобождение);
(д) пути биосинтеза (включая пептидогликаны й жирные кислоты);
(е) сборка клеточной стенки; и/или
(ж) целостность клетки бактерий.
Бактерии для применения при реализации способов согласно изобретению
Способы согласно настоящему изобретению можно применять для идентификации мишеней для действия антибиотиков в любой бактерии. Таким образом, способы согласно настоящему изобретению находят применение при идентификации мишеней действия антибиотиков в: (а) грамположительных, грамотрицательных и/или грамвариабельных бактериях; (б) спорообразующих бактериях; (в) неспорообразующих бактериях; (г) нитчатых бактериях; (д) внутриклеточных бактериях; (е) облигатных анаэробах; (ж) факультативных анаэробах; (з) микроаэрофильных бактериях и/или (и) оппортунистических бактериальных патогенах.
Согласно определенным вариантам реализации способ согласно настоящему изобретению применяют для идентификации мишени действия антибиотиков в бактериях следующих родов: Acinetobacter (например, A. baumannii); Aeromonas (например, А. hydrophila); Bacillus (например, В. anthracis); Bacteroides (например, В. fragilis); Bordetella (например, В. pertussis); Borrelia (например, В. burgdorferi); Brucella (например, В. abortus, В. canis, В. melitensis и 6. suis); Burkholderia (например, комплекс S. cepacia); Campylobacter (например, С. jejuni); Chlamydia (например, С trachomatis, С suis и С muridarum); Chlamydophila (например, (например, С. pneumoniae, С pecorum, С. psittaci, С. abortus, С. felis и С. caviae); Citrobacter (например, С. freundii); Closthdium (например, С. botulinum, С difficile, С perfringens и С. tetani); Corynebacterium (например, С. diphteriae и С glutamicum); Enterobacter (например, Ε. cloacae и Ε. aerogenes); Enterococcus (например, Ε. faecalis и L faecium); Escherichia (например, Ε. coli); Flavobacterium; Francisella (например, F. tularensis); Fusobacterium (например, F. necrophorum); Haemophilus (например, Н. somnus, H. influenzae и H. parainfluenzae); Helicobacter (например, H. pylori); Klebsiella (например, К. oxytoca и К. pneumoniae), Legionella (например, L pneumophila); Leptospira (например, L interrogans); Listeha (например, L. monocytogenes); Moraxella (например, M. catarrhalis); Morganella (например, M. morganii); Mycobacterium (например, M.leprae и M. tuberculosis); Mycoplasma (например, M. pneumoniae); Neisseria (например, N. gonorrhoeae и N. meningitidis); Pasteurella (например, P. multocida); Peptostreptococcus; Prevotella; Proteus (например, P. mirabilis и P. vulgaris), Pseudomonas (например, P. aeruginosa); Rickettsia (например, R rickettsii); Salmonella (например, serotypes. Typhi и Typhimurium); Serratia (например, S. marcesens); Shigella (например, S. flexnaria, S. dysenteriae и S. sonnei); Staphylococcus (например, S. aureus, S. haemolyticus, S. intermedius, S. epidermidis и S. saprophytics); Stenotrophomonas (например, S. maltophila); Streptococcus (например, S. agalactiae, S. mutans, S. pneumoniae и S. pyogenes); Treponema (например, Т. pallidum); Vibrio (например, V. cholerae) и Yersinia (например, Y. pestis).
Способы согласно настоящему изобретению можно применять для идентификации мишени для антибиотиков у бактерий с множественной лекарственной устойчивостью, включая. без ограничений устойчивые к пенициллину, устойчивые к метициллину, устойчивые к хинолонам, устойчивые к макролидам и/или устойчивые к ванкомицину штаммы бактерий, включая, например, Streptococcus pneumoniae, устойчивый к пенициллину, метициллину, макролидам, ванкомицину и/или к хинолонам; Staphylococcus aureus, устойчивый к пенициллину, метициллину, макролидам, ванкомицину и/или к хинолонам; Streptococcus pyogenes, устойчивый к пенициллину, метициллину, макролидам, ванкомицину и/или к хинолонам, и энтерококки, устойчивые к пенициллину, метициллину, макролидам, ванкомицину и/или к хинолонам.
Таким образом, способы согласно настоящему изобретению можно применять для идентификации мишени для антибиотика у устойчивого к метициллину Staphylococcus aureus (MRSA), например, выбранного из C-MSRA1, C-MRSA2, C-MRSA3, C-MSRA4, бельгийского MRSA, швейцарского MRSA и любого из штаммов EMRSA.
Соединения согласно настоящему изобретению можно применять для идентификации мишени для действия антибиотиков как у грамположительных бактерий с высоким G+C, так и у грамположительных бактерий с низким G+C.
Способы согласно настоящему изобретению находят конкретное применение при идентификации мишени для действия антибиотиков у бактерий, выбранных из Klebsiella pneumoniae, Acinetobacter baumanii, Escherichia coli (включая ST131), Enterococcus faecalis, Enterococcus faecium, Pseudomonas aeruginosa, Enterobacter cloacae, Enterobacter aerogenes и Neisseria gonorrhoeae.
Особенно предпочтительны способы идентификации мишеней для действия антибиотиков у Klebsiella pneumoniae, Acinetobacter baumanii или Escherichia coli.
Совокупности мутантов
Способы согласно настоящему изобретению подразумевают создание совокупности мутантных бактерий при помощи транспозонного мутагенеза. Размер совокупности мутантов влияет на разрешение способа: по мере увеличения размера совокупности, будет представлено все больше разных генов с встраиванием TnA (и таким образом эффективно анализироваться). По мере уменьшения размера совокупности разрешение способа снижается, гены анализируются менее эффективно и все больше генов вообще не анализируется.
В идеале, совокупность мутантов, создаваемая при реализации способов согласно настоящему изобретению является всеобъемлющей, в том смысле, что представлены вставки в каждый ген. Количество мутантов с встраиванием TnA (т.е. размер совокупности мутантов), которое требуется для достижения этого, зависит от разных факторов, включая (а) размер генома бактерии; (б) средний размер генов; и (в) наличие любого предпочтения для сайтов встраивания TnA.
Что касается последнего фактора, некоторые области геномов бактерий соответствуют низкой частоте вставок (особенно GC-богатые области). Таким образом, предпочтительны частота встраивания и размер совокупности, которые достаточно велики для того, чтобы гарантировать встраивание в области, плохо поддающиеся встраиванию.
Как правило, для получения всеобъемлющей совокупности/библиотеки, требуется минимальный уровень встраивания один транспозон на 25 пар оснований, который обычно предусматривает минимальный размер совокупности для бактерии с размером генома 4-7 Мб - от 0,5×105 до 1×105, например, 5×105, предпочтительно по меньшей мере приблизительно 1×106 мутантов. Во многих случаях размер 1×106 мутантов позволяет проводить идентификацию ~300000 разных сайтов встраивания и соответствует встраиванию 1 транспозона на каждые 13-23 п.о. (или приблизительно 40-70 разных сайтов встраивания на один ген).
Однако способы согласно настоящему изобретению не обязательно требуют всеобъемлющей совокупности мутантов (в том смысле, которые оговаривался выше) для извлечения полезной информации, такой как информация для идентификации мишеней действия антибиотиков. Как раз размеры совокупности меньше идеальной всеобъемлющей совокупности можно применять при условии, что снижение разрешения (и сопутствующая невозможность анализа определенных генов) может быть приемлемой. Это может быть справедливо, например, когда способ разработан так, чтобы он выполнялся итерационно до тех пор, пока мишень не будет идентифицирована; в таких вариантах реализации эффективный размер совокупности растет с каждой итерацией указанного способа.
Транспозонный мутагенез
Транспозоны, которые иногда называют мобильными генетическими элементами, представляют собой полинуклеотиды, способные к встраиванию своих копий в другие полинуклеотиды. Термин «транспозон» хорошо известен специалистам в данной области техники и включает классы транспозонов, которые можно различать по организации последовательностей, например, короткие инвертированные повторы на каждом-конце; прямо повторяемые длинные концевые повторы (LTR) на концах; и последовательности полиА на 3'-концах РНК-транскриптов, у которых 5'-конец часто процессирован.
Транспосомы представляют собой комплексы транспозаза/транспозон, в которых указанный транспозон не кодирует указанную транспозазу. Таким образом, будучи встроенным, транспозон становится стабильным. Для обеспечения стабильности совокупности мутантов предпочтительно, чтобы указанный транспозон не кодировал указанную транспозазу и был представлен в форме транспосомы (т.е. в виде комплекса с ферментом транспозазой), как описано ниже.
В настоящей заявке термин «активирующий транспозон» (здесь и далее обозначается сокращенно «TnA») относится к транспозону, который содержит промотор, такой, что встраивание транспозона усиливает транскрипцию гена в сайте встраивания или поблизости от него. Примеры таких транспозонов описаны у Troeschel et at. (2010) Methods Mol Biol. 668:117-39 и Kim et al. (2008) Curr Microbiol. 57(4): 391-394.
Активирующий транспозон/транспосому можно встраивать в геном бактерии (включая хромосомную ДНК и/или ДНК плазмид) при помощи любого из широкого спектра стандартных способов, которые хорошо известны специалистам в данной области техники. Например, транспосомы ТпА можно вводить посредством электропорации (или любого другого подходящего способа трансформации).
Предпочтительно, чтобы способ трансформации позволял получить от 1×103 до 5×103 трансформантов/нг ДНК, и такая производительность трансформации обычно достигается при электропорации.
В качестве альтернативы, транспозонный мутагенез с применением TnA можно проводить in vitro, и рекомбинантные молекулы трансформировать/трансфецировать в клетки бактерий. Согласно таким вариантам реализации транспосомы можно получить в соответствии со стандартным протоколом путем смешивания коммерческого фермента транспозазы с транспозонным фрагментом ДНК. Образующиеся транспосомы затем смешивают с плазмидной ДНК рассматриваемой плазмиды, чтобы могла произойти транспозиция, затем указанную ДНК вводят в штамм бактерии-хозяина при помощи электротрансформации, чтобы создать совокупность плазмид транспозонных мутантов.
При вариантах реализации, в которых мутагенез проводят in vitro, возможно смешивать транспосомы с геномной ДНК in vitro и затем вводить подвергнутую мутагенезу ДНК (необязательно, после фрагментации и/или образования кольцевой формы) в штамм бактерии-хозяина (например, посредством электропорации), вследствие чего аппарат эндогенной рекомбинации включает ее в геном. Такой способ может быть особенно полезен в случае бактерии, которая от природы компетентна (например, виды Acinetobacter) и/или может включать ДНК посредством событий рекомбинации гомологичного кроссовера (например, двойного кроссовера).
Активирующие транспозоны для применения при реализации способов согласно настоящему изобретению
При реализации способов согласно настоящему изобретению можно применять любой активирующий транспозон. Подходящие транспозоны включают транспозоны на основе Tn3 и Тп3-подобных транспозонов (II класса), включая γδ (Tn1000), Tn507, Tn2507, Tn27, Tn977 и их производные. Также подходят Tn10, Tn5, TnphoA, Tn903, бактериофаг мю и родственные транспонируемые бактериофаги. Также разнообразные подходящие транспозоны можно получить из коммерческих источников, включая, например, транспозон ΕΖ-Τn5™<R6Κγοri/ΚΑΝ-2>.
Предпочтительны транспозоны, которые несут в себе гены устойчивости к антибиотикам (которые могут быть полезны при идентификации мутантов, несущих транспозон), включая Tn5, Tn10 и TnphoA. Например, Τn10 несет ген устойчивости к тетрациклину, который встроен между его IS-элементами, а Tn5 несет гены, кодирующие полипептиды, придающие устойчивость к канамицину, стрептомицину и блеомицину. Другие подходящие гены устойчивости включают гены, содержащие хлорамфеникол-ацетилтрансферазу (придающую устойчивость к хлорамфениколу). Конечно, возможно получать новые транспозоны путем встраивания разных комбинаций генов устойчивости к антибиотикам между IS-элементами или путем встраивания комбинаций генов устойчивости к антибиотикам между мозаичными концами транспозонов (предпочтительно), или путем изменения полинуклеотидной последовательности транспозона, например путем создания замены избыточных оснований или замены оснований другого типа, которые не влияют на транспозицию или характеристики устойчивости к антибиотикам, присущие транспозону, в кодирующей области гена устойчивости к антибиотику или еще где-либо в транспозоне. Такие транспозоны включены в область настоящего изобретения.
Согласно многим вариантам реализации для создания совокупности мутантов применяют единственный транспозон. Однако, как объяснялось выше, количество мутантов с встраиванием Τn (т.е. размер совокупности мутантов), которое нужно для достижения всеобъемлющей совокупности или библиотеки, зависит, в том числе, и от смещения сайта встраивания Τn. Таким образом, в тех случаях, когда возникает смещение сайта встраивания транспозона, можно применять два или более разных транспозона, чтобы уменьшить или исключить смещение сайта встраивания. Например, можно применять комбинацию из двух разных транспозонов на основе Tn5 и Tn70.
Промоторы, применяемые для активирующих транспозонов
Природа промоторов, присутствующих в TnA, зависит от природы указанного транспозона и от конечной бактерии-хозяина. Как правило, выбирают эффективный, ориентированный на внешнюю работу промотор, который запускает транскрипцию на высоком уровне ДНК, расположенной в сайте встраивания или поблизости от него. Промотор может включать: (а) рамку Прибноу (элемент -10); (б) элемент -35 и/или (в) UP-элемент.
Например, промотор lac можно применять с транспозоном ΕΖ-Τn5™<R6Κγοri/ΚΑΝ-2>, и такие конструкции применимы для анализа, например, видов Escherichia coli, Enterobacter и других членов семейства Enterobacteriaceae, таких как виды Klebsiella. Другие подходящие промоторы включают: rplJ (белок большой субъединицы рибосомы; промотор средней силы); tac (искусственный гибрид lacltrp; сильный промотор) и rrnB (промотор гена рибосомальной РНК; очень сильный промотор). Последовательности последних из перечисленных промоторов показаны ниже:
Определение распределения вставок TnA
Распределение вставок транспозонов предпочтительно определяют посредством секвенирования бактериальной ДНК, примыкающей или расположенной рядом с (5' и/или 3') с сайтом встраивания TnA (например, посредством секвенирования ДНК, которая содержит участок соединения TnA-геномная ДНК). Обычно секвенируют бактериальную ДНК, фланкирующую или примыкающую с одним или более концами TnA. Длина секвенируемой соседней ДНК не должна быть избыточной, и предпочтительно должна быть короткой (например, менее 200 пар оснований).
Для определения распределения вставок TnA можно применять разные способы с применением секвенирования ДНК: такие способы недавно получили названия процедур Tn-seq (van Opijnen et al. (2009) Nat. Methods 6: 767-772). Например, процедуры Tn-seq включают аффинную очистку амплифицированных участков соединения (Gawronski et al. (2009) PNAS 106: 16422-16427); сшивку адаптеров в геномные последовательности, расположенные дистально на конце транспозона, с применением специализированного сайта рестрикции (Goodman et al. (2009) Cell Host Microbe 6: 279-289; van Opijnen ef al. (2009) Nat. Methods 6: 767-772); селективную амплификацию (Langridge ef al. (2009) Genome Research 19: 2308-2316) и получение колец одноцепочечной ДНК, несущих участки соединения Τn, которые служат матрицами для амплификации и секвенирования после исключения геномной ДНК путем расщепления экзонуклеазами (Gallagher ef al. (2011) mBio 2(1):e00315-10).
Можно применять любые подходящие высокопроизводительные технологии, и существует множество коммерческих платформ для секвенирования, которые подходят для применения при реализации способов согласно настоящему изобретению. Особенно для применения при реализации способов согласно настоящему изобретению подходят платформы секвенирования на основе SBS (секвенирования путем синтеза): например, система Illumina™ генерирует миллионы относительно коротких считываний последовательностей (54, 75 или 100 по) и является особенно предпочтительной.
Другие подходящие технологии включают способы, основанные на обратимых красителях-теминаторах. При реализации таких способов молекулы ДНК сначала присоединяют к праймерам на предметном стекле и амплифицируют так, чтобы образовались локальные клональные колонии (мостовая амплификация). Добавляют четыре типа ddNTP, а нуклеотиды, не включившиеся в цепь, отмывают. В отличие от пиросеквенирования, указанную ДНК можно за один раз увеличить только на один нуклеотид. Камера создает изображения нуклеотидов с флуоресцентной меткой, затем краситель вместе с 3'-концевым блокатором отщепляют от ДНК химическим путем, и становится возможен следующий цикл.
Другие системы, способные к считыванию коротких последовательностей, включают SOLiD™ и технологии ионного Торрента (обе от компании «Applied Biosystems™»). Технология SOLiD™ основана на секвенировании путем сшивания. При реализации данного способа совокупность всех возможных олигонуклеотидов фиксированной длины метят в зависимости от секвенированного положения. Олигонуклеотиды отжигают и сшивают; предпочтительно сшивание при помощи ДНК-лигазы для соответствующих друг другу последовательностей приводит к получению сигнала, информативного в отношении нуклеотида в таком положении. Перед секвенированием ДНК амплифицируют при помощи эмульсионной ПЦР (полимеразной цепной реакции). На предметном стекле откладывается полученная в результате гранула, причем каждая гранула содержит копии только одной и той же молекулы ДНК. Результатом является секвенирование таких количеств и длин последовательностей, которые сопоставимы с секвенированием по технологии Illumina.
Компания «Ion Torrent Systems Inc.» разработала систему на основе применения стандартной химии секвенирования, но с применением новой системы детекции, основанной на полупроводниках. Данный способ секвенирования основан на детекции ионов водорода, которые выделяются во время полимеризации ДНК, в противоположность оптическим способам, применяемым в других системах секвенирования. Микролунку, содержащую нить ДНК-матрицы, которую требуется секвенировать, заливают нуклеотидом одного типа. Если введенный нуклеотид комплементарен лидирующему нуклеотиду матрицы, он включается в растущую комплементарную цепь. Это приводит к высвобождению иона водорода, который инициирует гиперчувствительный ионный сенсор, который показывает, что реакция прошла. Если в последовательности матрицы присутствует гомополимерные повторы, в одном цикле будут включено несколько нуклеотидов. Это приведет к выделению соответствующего числа ионов водорода, и будет получен пропорционально высокий электронный сигнал.
Функциональная оценка предполагаемых жизненно важных генов
Предполагаемые жизненно важные гены, идентифицированные путем сравнения распределения вставок TnA между исследуемыми культурами, также можно охарактеризовать при помощи разных технологий, которые позволяют прямо или косвенно оценить их функцию. Таким путем можно однозначно приписать определенную жизненно важную функцию указанному предполагаемому жизненно важному гену.
Подходящие технологии включают способы биоинформатики, в которых последовательность (полную или частичную) предполагаемого жизненно важного гена применяют для запросов в базах данных последовательностей, содержащих информацию, полученную при анализе бактерий и/или других видов, чтобы идентифицировать гены (например, ортологичные гены у другого вида), для которых жизненно важная биохимическая функция (функции) уже была определена и/или для которого уже было показано, что он является жизненно важным геном.
Подходящие бионформационные программы хорошо известны специалистам в данной области техники и включают программу «Средство поиска основного локального выравнивания (BLAST)» (Altschul et al. (1990) J. Mol. Biol. 215: 403-410 uAltschul et al. (1997) Nucl. Acids Res. 25: 3389-3402). Подходящие базы данных включают, например, EMBL, GENBANK, TIGR, EBI, SWISS-PROT и tremble.
В качестве альтернативы, или как дополнение, последовательность (полную или частичную) предполагаемого жизненно важного гена применяют для запросов в базах данных последовательностей, содержащих информацию, касающуюся идентичности жизненно важных генов, которые ранее были сконструированы при помощи традиционных способов Tn-seq, описанных на предыдущем уровне техники (например, как описано у Gawronski et al. (2009) PNAS 106: 16422-16427; Goodman er al. (2009) Cell Host Microbe 6: 279-289; van Opijnen et al. (2009) Nat. Methods 6: 767-772; Langridge et al. (2009) Genome Research 19: 2308-2316; Gallagher et al. (2011) mBio 2(1): e00315-10) и/или технологий, описанных в WO 01/07651 (содержание которой включено в настоящую заявку посредством ссылки).
В качестве альтернативы, или как дополнение, значимость можно приписать путем исключения возможности того, что предполагаемый жизненно важный ген выступает в роли гена устойчивости к антибиотикам. Например, последовательность (полную или частичную) предполагаемого жизненно важного гена применяют для запросов в базах данных последовательностей, содержащих информацию о последовательностях генов, ранее идентифицированных в качестве генов устойчивости к антибиотикам при помощи способов Tn-seq, описанных например у Gawronski er al. (2009) PNAS 106: 16422-16427; Goodman et al. (2009) Cell Host Microbe 6: 279-289; Langridge et al. (2009) Genome Research 19: 2308-2316 or Gallagher er al. (2011) mBio 2(1):e00315-10. При помощи таких способов гены устойчивости к антибиотикам можно идентифицировать как класс нише-специфичных/условно жизненно важных генов.
Несмотря на присутствие промотора во встроенной последовательности, многие вставки TnA будут нарушать функцию гена/ДНК и создадут возможность идентификации незаменимых/важных областей ДНК, как при применении стандартной технологии Tn-seq (включая TraDIS). Однако некоторые транспозоны позиционируют должным образом относительно значимых областей ДНК, посредством чего транскрипция таких специфических областей, запускаемая встроенным промотором, значительно усиливается по сравнению с эндогенной транскрипцией. Если выращивать совокупность мутантов при возрастающих концентрациях антибиотика и повторять секвенирование, можно увидеть изменения в количестве считываний, которые указывают не только на то, какая область ДНК вносит вклад в выживание в присутствии антибиотика, но и на относительный ее вклад. Более высокий уровень транскрипции мишени воздействия антибиотиков (запускаемой встроенным при помощи транспозона промотором) будет благоприятен для выживания бактерии в присутствии антибиотика и будет связываться с сайтом встраивания с областью ДНК за счет их пространственной близости.
Для идентификации мишени (мишеней) воздействия специфических антибиотиков положение встроенного промотора можно оценить в отношении его вклада в повышенную транскрипцию соответствующих нижележащих последовательностей ДНК. Понятный с математической/технической точки зрения биоинформационный компонент данной технологии позволяет устанавливать вклад последовательности встроенного промотора в транскрипцию гена - предполагаемой мишени воздействия антибиотиков. Например, транскрипции продукта конкретного гена - мишени воздействия антибиотиков, может быть достаточно, чтобы придать бактерии устойчивость к антибиотику, а биоинформационный анализ может дать объяснение. Например, транскрипт конкретного гена может все еще кодировать достаточно информации, чтобы была возможна транскрипция укороченного, но все еще функционально значимого белка. Биоинформатика позволяет рассмотреть эффекты транскрипционного считывания генов, расположенных в последовательности до гена, примыкающего с встроенным транспозоном, где нет охарактеризованной последовательности терминации транскрипции РНК.
Например, транспозон/промотор, расположенный в последовательности после генов А, В и С, приводит к появлению полицистронного транскрипта всех трех генов (А-С), расположенный в последовательности после гена В - полицистронный транскрипт генов В и С, а расположенный в последовательности после гена С - только ген С, и если считывание в присутствии антибиотика для первых двух транспозонов было высоким, а для третьего низким, то мишенью воздействия антибиотика может быть ген В.
Вспомогательные способы анализа
Способы согласно настоящему изобретению находят конкретное применение в тех случаях, когда мишень воздействия антибиотиков экспрессируется в избытке на таком уровне, когда она вносит вклад и играет значительную роль в кинетике связывания с антибиотиком при том уровне, который применяют для деконволюции мишени. Следовательно, указанные способы идеально подходят для тех мишеней воздействия антибиотиков, с которыми антибиотики связываются и меняют функцию мономерной макромолекулы. Однако в тех случаях, когда мишень воздействия антибиотиков доступна только в тройном комплексе, когда дополнительная мишень из-за гиперэкспрессии может не функционировать как эффективная фракция, и тем самым изменяет действие антибиотика, чувствительность способа может быть снижена.
Таким образом, в некоторых обстоятельствах способы согласно настоящему изобретению можно применять в сочетании с другими, дополняющими их, технологиями для идентификации жизненно важных, условно жизненно важных, не жизненно важных и/или жизненно важных генов, служащих мишенями воздействия антибиотиков, как описано ниже:
(а) Дополнительное секвенирование
Способ согласно настоящему изобретению может также необязательно включать этап:
(а) создания устойчивого к антибиотику мутанта указанной бактерии посредством способа, включающего этап отбора в ходе роста в присутствии указанного антибиотика с получением клона мутантов, устойчивых к антибиотику (мутант AbR);
(б) трансформации указанного мутанта AbR: (i) одним или более жизненно важными генами указанной бактерии; и (ii) транспозоном, который при встраивании инактивирует бактериальную ДНК, с получением совокупности транспозонных мутантов, которые меродиплоидны по указанному одному или более жизненно важным генам;
(в) выращивания бактерий из меродиплоидной совокупности в присутствии разных количеств указанного антибиотика с получением двух или более культур для тестирования; и
(г) сравнения распределения вставок транспозонов между анализируемыми культурами с целью идентификации предполагаемого жизненно важного гена, служащего мишенью воздействия указанного антибиотика у указанной бактерии.
Применение совокупностей транспозонных мутантов, полученных из мутантов, устойчивых к антибиотикам, которые меродиплоидны по одному или более жизненно важным генам, гарантирует, что встраивание транспозонов в жизненно важные гены (включая гены, служащие мишенями воздействия антибиотиков) представлены в исходной совокупности мутантов, поскольку встраивание транспозонов в ген-мишень воздействия антибиотиков приводит к образованию жизнеспособных генотипов при неселективных условиях (когда копия жизненно важного гена дикого типа дополняет активированную вследствие встраивания мутантную копию), но не при селективных условиях (когда копия жизненно важного гена дикого типа не дополняет активированную вследствие встраивания мутантную копию).
Таким образом, можно легко определить различия в распределении транспозонов после выращивания указанной совокупности меродиплоидных мутантов с антибиотиком или без него, и это создает возможность для идентификации жизненно важных генов, служащих мишенями действия антибиотиков.
На необязательных последующих этапах, описанных выше:
- меродиплоидная совокупность может содержать по меньшей мере 0,5×105 мутантов, например, по меньшей мере 1×105 мутантов;
- меродиплоидная совокупность может содержать по меньшей мере 5×105 мутантов;
- меродиплоидная совокупность может содержать по меньшей мере 1×106 мутантов;
- меродиплоидная совокупность может содержать от 0,5×106 до 2×106 мутантов;
- меродиплоидная совокупность может содержать приблизительно 1×106 мутантов;
- трансформация с применением транспозона на этапе (б) может приводить к проценту встраивания по меньшей мере один транспозон на 50 пар оснований бактериальной ДНК, по меньшей мере один транспозон на 30 пар оснований бактериальной ДНК, по меньшей мере один транспозон на 25 пар оснований бактериальной ДНК, по меньшей мере один транспозон на 15 пар оснований бактериальной ДНК или по меньшей мере один транспозон на 10 пар оснований бактериальной ДНК;
- бактериальная ДНК в соответствии с этапом (б) может быть хромосомной ДНК, плазмидной ДНК или смесью хромосомной и плазмидной ДНК, и может включать весь геном бактерии;
- указанный мутант AbR может быть получен при помощи способа, который включает мутагенез указанной бактерии перед отбором в ходе роста в присутствии указанного антибиотика;
- указанным этапом мутагенеза может быть: (а) химический мутагенез; и/или (б) радиационный мутагенез;
- указанной бактерией может быть грамположительная бактерия;
- указанная бактерия может быть выбрана из: Enterococcus faecalis, Enterococcus faecium и Neisseria gonorrhoeae;
- указанной бактерией может быть грамотрицательная бактерия;
- указанная бактерия может быть выбрана из: Klebsiella pneumoniae, Acinetobacter baumanii, Escherichia coli, штаммов Ε. coli ST131, Pseudomonas aeruginosa, Enterobacter cloacae, Enterobacter aerogenes и Neisseria gonorrhoeae;
- указанная бактерия может быть выращена из меродиплоидной совокупности на этапе (в) путем посева на питательную среду от 107 до 109, например приблизительно 108 КОЕ из указанной меродиплоидной совокупности;
- на этапе (в) могут быть получены по меньшей мере две анализируемые культуры, одна из которых выращена в отсутствии антибиотика, а другая - в присутствии антибиотика (например, в концентрации от приблизительно 1 до приблизительно 4×MIC (минимальная ингибирующая концентрация));
- указанное распределение встраивания транспозонов между анализируемыми культурами можно сравнивать путем секвенирования ДНК, примыкающей или расположенной поблизости от указанного сайта встраивания транспозона;
- секвенирование ДНК, примыкающей или расположенной поблизости от указанного сайта встраивания транспозона, может включать селективную амплификацию участков соединения транспозон-бактериальная ДНК;
- указанное секвенирование может включать биохимический способ секвенирования путем синтеза (SBS);
- можно секвенировать приблизительно 25, 50, 75, 100 или более 100 пар оснований, примыкающих или расположенных поблизости от указанного сайта встраивания;
- секвенированная ДНК может быть расположена с 5' и/или 3'-стороны от указанного сайта встраивания;
- указанный способ также может включать этап приписывания определенной жизненно важной функции указанному предполагаемому жизненно важному гену путем сравнения последовательностей с одним или более жизненно важными генами указанной бактерии, например, путем транспозон-направленного секвенирования сайтов встраивания;
- указанный способ также может включать этап приписывания определенной жизненно важной функции указанному предполагаемому жизненно важному гену путем определения того, является ли он геном устойчивости к антибиотику, например, посредством способа, включающего идентификацию гена устойчивости к антибиотику путем транспозон-направленного секвенирования сайтов встраивания с применением транспозона, который при встраивании оказывает инактивирующее действие;
- один или более жизненно важных генов в соответствии с этапом (б) может быть представлен в составе вектора экспрессии, интегрирующегося вектора экспрессии, который встраивается в хромосому бактерии после трансформации; в составе вектора экспрессии с одной копией или малым числом копий, который после трансформации стабильно сохраняется вне хромосом;
- указанным вектором экспрессии может быть комбинация плазмид, содержащая фрагменты нативной хромосомы указанной бактерии или искусственной бактериальной хромосомы (ВАС);
- один или более жизненно важных генов в соответствии с этапом (б) может быть представлен в составе линейной ДНК (например, фрагменты геномной ДНК указанной бактерии);
- один или более жизненно важных генов в соответствии с этапом (б) может содержать по меньшей мере 10, по меньшей мере 20, по меньшей мере 50, по меньшей мере 100, по меньшей мере 150, по меньшей мере 200, по меньшей мере 250, по меньшей мере 300 жизненно важных генов указанной бактерии;
- один или более жизненно важных генов в соответствии с этапом (б) может быть получен при помощи способа, включающего идентификацию одного или более жизненно важных генов указанной бактерии путем транспозон-направленного секвенирования сайтов встраивания;
- один или более жизненно важных генов в соответствии с этапом (б) может быть выбран из генов, участвующих в делении клеток; репликации ДНК (например, полимеризации или суперспирализации); и/или транскрипции (например, праймировании, элонгации и/или терминации); и/или трансляции (например, гены, кодирующие рибосомальные компоненты, гены, участвующие в инициации, элонгации и/или высвобождении); и/или в путях биосинтеза (например, в обмене пептидогликанов и/или жирных кислот);
- на этапе (б) указанный мутант AbR может быть трансформирован одновременно одним или более жизненно важными генами указанной бактерии и транспозоном;
- на этапе (б) указанный мутант AbR может быть трансформирован указанным транспозоном, а затем одним или более жизненно важными генами указанной бактерии; и/или
- на этапе (б) указанный мутант AbR может быть трансформирован сначала одним или более жизненно важными генами указанной бактерии, а затем указанным транспозоном.
Когда мутанта AbR трансформируют одновременно одним или более жизненно важными генами указанной бактерии и указанным транспозоном или трансформируют сначала одним или более жизненно важными генами указанной бактерии, а затем указанным транспозоном, может возникать нежелательное встраивание транспозона во введенные жизненно важные гены, которое может снижать эффективность формирования меродиплоидов, и может осложнять анализ данных, полученных на основании таких библиотек.
Таких проблем удается избежать, когда указанного мутанта AbR сначала трансформируют транспозоном, а затем одним или более жизненно важными генами указанной бактерии. Однако в зависимости от эффективности процесса, применяемого для введения жизненно важного гена (генов), для такой стратегии может потребоваться значительно больше экспериментов по трансформации, чтобы получить библиотеку достаточного размера.
Существует альтернативное решение, которое основано на феномене иммунитета к транспозиции. При реализации данного способа нежелательную транспозицию во введенные жизненно важные гены удается исключить (или уменьшить) путем включения мозаичных концов последовательностей с мозаикой транспозонов во внехромосомную ДНК (обычно плазмидную или ВАС), содержащие жизненно важные гены, применяемые для создания меродиплоидов. Такую стратегию можно применять в сочетании с транспозонами на основе транспозонов II класса (Tn3-подобных), например, Tn3 или близкими ему формами, которые описаны ниже.
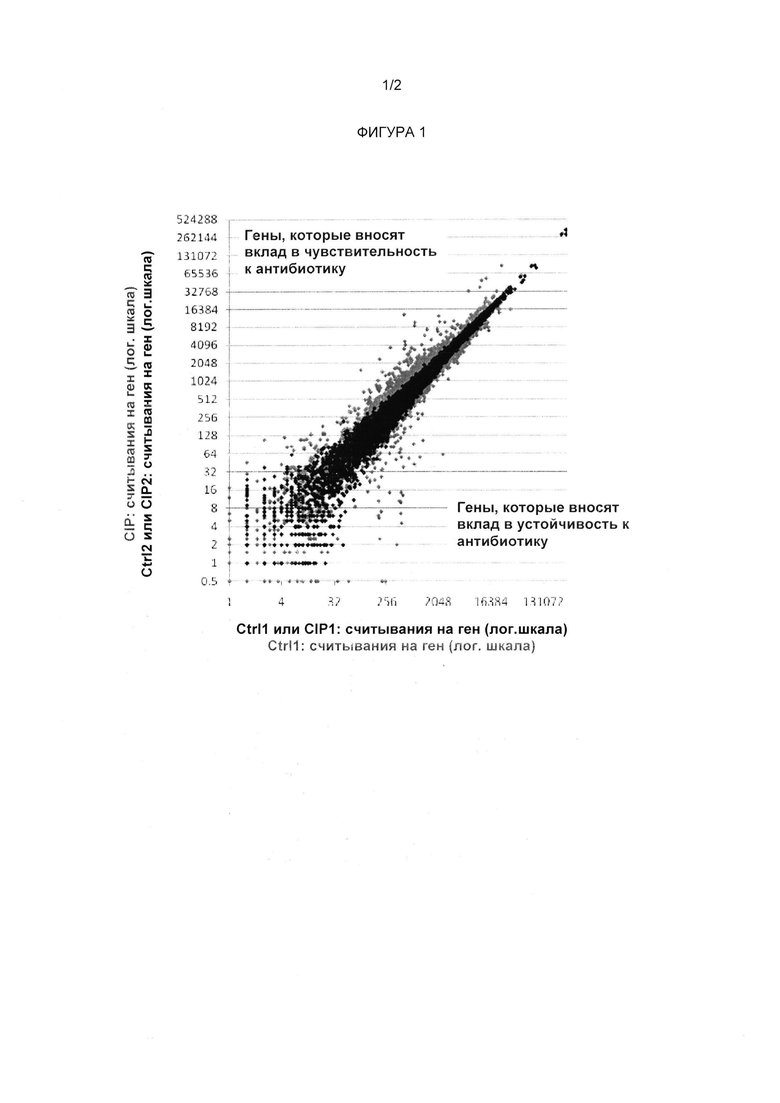
Системы транспозонов, подходящие для применения при реализации способов, основанных на феномене иммунитета к транспозиции (описанных выше), включают транспозоны на основе Tn3 или близкие ему формы. Например, плазмида для доставки транспозона pAMICS2 (см. Фигуру 2) содержит целую основанную на Tn3 транспозон-генерирующую систему (включая гены, кодирующие ферменты резолвазы и транспозазы, TnpR и TnpA) и точку начала репликации (or/R6K), которая активна только тогда, когда она дополнена геном pir, что препятствует размножению бактерии-реципиента после транспозиции. Также указанная плазмида содержит точку мобилизации mobRP4, которая создает возможность переноса из подходящего штамма донора путем конъюгации, который является разрешающим сигналом для репликации плазмиды (например, содержит ген pir). Самопроизвольное воспроизведение плазмиды можно выявить по наличию гена устойчивости к тобрамицину (аасА4). Другие подходящие транспозоны включают транспозоны на основе Tn3 и Тп3-подобных транспозонов (II класса), включая Tn1000), Tn507, Tn2507, Tn27, Tn977 и их производные. Также подходят Tn10, Tn5, TnphoA, Tn903, бактериофаг мю и родственные им транспонируемые бактериофаги. Также разнообразные подходящие транспозоны можно получить из коммерческих источников, включая, например, транспозон EZ-Tn5™<R6Kγori/KAN-2>.
Предпочтительны транспозоны, которые несут в себе гены устойчивости к антибиотикам (которые могут быть полезны при идентификации мутантов, несущих транспозон), включая Tn5, Tn10 и TnphoA. Например, Tn10 несет ген устойчивости к тетрациклину, который встроен между его IS-элементами, а Tn5 несет гены, кодирующие полипептиды, придающие устойчивость к канамицину, стрептомицину и блеомицину. Другие подходящие гены устойчивости включают гены, содержащие хлорамфеникол-ацетилтрансферазу (придающую устойчивость к хлорамфениколу).
Конечно, возможно создавать новые транспозоны путем встраивания разных комбинаций генов устойчивости к антибиотикам между IS-элементами или путем встраивания комбинаций генов устойчивости к антибиотикам между мозаичными концами транспозонов (предпочтительно), или путем изменения полинуклеотидной последовательности транспозона, например, путем создания замены избыточных оснований или замены оснований другого типа, которые не влияют на транспозицию или характеристики устойчивости к антибиотикам, присущие транспозону, в кодирующей области гена устойчивости к антибиотику или еще где-либо в транспозоне. Такие транспозоны включены в область настоящего изобретения.
Согласно многим вариантам реализации для создания совокупности мутантов применяют один транспозон. Однако, как объяснялось выше, количество мутантов с вставками Τn (т.е. размер совокупности мутантов), который требуется для получения всеобъемлющей совокупности или библиотеки, зависит, помимо прочего, от смещения сайта встраивания. Таким образом, в тех случаях, когда возникает смещение сайта встраивания транспозона, можно применять два или более разных транспозонов, чтобы уменьшить или исключить смещение сайта встраивания. Например, можно применять комбинацию из двух разных транспозонов на основе Tn5 и Tn70.
Таким образом, способ согласно настоящему изобретению может дополнительно включать этапы идентификации гена (например, жизненно важного гена), который служит мишенью воздействия антибиотиков в бактерии, способ, включающий этапы:
(а) трансформации бактерии при помощи внехромосомного элемента (например, рлазмиднрго или ВАС), содержащего: (i) один или более жизненно важных генов указанной бактерии; и (ii) одну или более последовательность транспозонных повторов, с получением совокупности бактерий, которые являются меродиплоидными по указанному одному или более жизненно важным генам; и
(б) трансформации меродиплоидов в соответствии с этапом (а) плазмидой для доставки транспозона, содержащей: (i) ген, кодирующий транспозазу и резолвазу; и (ii) сайты распознавания транспозазы обратных повторов;
причем указанная одна или более последовательность транспозонных повторов для указанного внехромосомного элемента в соответствии с этапом (а) придает свойство иммунитета к транспозиции в отношении транспозона, доставляемого плазмидой в соответствии с этапом (б).
Согласно данному аспекту настоящего изобретения система доставки транспозона предпочтительно создана на основе Tn3, например, содержит гены Tn3 tnpA и tnpR. Предпочтительны плазмиды для доставки транспозонов, которые также содержат один или более генов устойчивости к антибиотикам.
(б) Tn-seq
Способ согласно настоящему изобретению может дополнительно включать необязательный этап анализа на основе Tn-seq, который описан, например, у Gawronski er al. (2009) PNAS 106: 16422-16427; Goodman et al. (2009) Cell Host Microbe 6: 279-289; Langridge et al. (2009) Genome Research 19: 2308-2316 или Gallagher et al. (2011) mBio 2(1):e00315-10. При применении в сочетании с анализом на основе Tn-seq настоящее изобретение позволяет дополнительно идентифицировать: (а) жизненно важные гены; (б) гены, благоприятствующие росту (но не жизненно важные); (в) гены, неблагоприятные для роста при конкретных условиях; и (г) гены, участвующие в придании устойчивости к определенным условиям («нише-специфичные» жизненно важные гены), наряду с жизненно важными генами, которые служат мишенями воздействия антибиотиков.
Примеры
В данном разделе настоящее изобретения описано применительно к специфическим примерам. Они выступают исключительно в качестве примеров и только с целью иллюстрирования; предполагается, что они никоим образом не должны ограничивать область заявленной монополии или описанного изобретения. Указанные примеры представляют собой наилучший предполагаемый в настоящее время вариант реализации изобретения.
Получение бактерий для электропорации
Бактерии выращивают на бульоне 2×ΤΥ, пока оптическая плотность OD6oo не достигнет 0,3-0,5. Затем клетки собирают и промывают три раза в 10% глицерине объема исходной культуры), повторно суспендируют в 10% глицерине (1/1000 объема исходной культуры) и хранят при -80°С.
Получение транспосом
ДНК транспозона (производное EZ-Tn5™<R6Kγori/KAN-2>, содержащее внутренний промотор lac) амплифицировали с применением олигонуклеотидов 5'-CTGTCTCTTATACACATCTCCCT и 5'-CTGTCTCTTATACACATCTCTTC и с участием ДНК-полимеразы II Pfu Ultra Fusion («Stratagene»). В качестве альтернативы внутренний промотор lac можно заменить на промотор tac (как было описано выше). Полученный ампликон затем фосфорилировали с применением полинуклеотид киназы фага Т4 («New England Biolabs»). Затем двести нанограмм указанной ДНК инкубировали с транспозазой EZ-Tn5™ («Epicenter Biotechnologies») при 37°С в течение 1 часа, затем хранили при -20°С при концентрации ДНК 20 нг/мкл.
Создание совокупности бактерий-мутантов
Шестьдесят микролитров клеток (ранее хранимых при -80°С) смешивали с 0,2 мкл (4 нг) 10 транспосом и 1 мкл (20 г) комплементарной плазмиды, содержащей жизненно важные гены, и проводили электротрансформацию в кювете с расстоянием между электродами 2 мм с применением Bio-Rad GenePulser II, установленным на 2,4 кВ, 25 мкФ и 200 Ом. Клетки повторно суспендировали в 1 мл среды SOC («Invitrogen») и инкубировали при 37°С в течение 2 часов, затем распределяли на бактериологической питательной среде с L-агаром с добавлением канамицина в соответствующей концентрации. Применяемая концентрация канамицина зависит от штамма, и ее определяют эмпирически.
После инкубации в течение ночи при 37°С оценивают количество колоний путем подсчета доли колоний, а на основании нее консервативным способом оценивают общее количество колоний во всех чашках. Устойчивые к канамицину колонии отбирают путем повторного суспендирования в стерилизованной деионизированной воде при помощи микробиологического распределителя. Затем повторно суспендированные клетки, полученные в ходе нескольких процедур электропорации, объединяют и создают смеси библиотек мутантов, которые по оценкам содержат свыше 1 миллиона мутантов.
Идентификация генов-мишеней воздействия антибиотиков
Подготавливают восемь культур, состоящих из 100 мл бульонной питательной среды, шесть из которых содержат добавки анализируемого антибиотика в концентрациях 0,5, 1 и 2×MIC (каждая концентрация в двойном экземпляре). Также в это же время в указанную среду добавляют какой-либо необходимый индуктор промотора для обеспечения активной транскрипции, направленной на хромосомную ДНК, с последовательности транспозона.
В предположении, что совокупность транспозонных мутантов составляет 1 миллион, для засевания каждой культуры применяют 108-109 КОЕ из указанной совокупности. Культуры выращивают до наступления стационарной фазы и клетки отбирают с целью экстракции геномной ДНК. Также готовят свежие культуры и засевают их 108-109 КОЕ из первичных культур. Их выращивают до наступления стационарной фазы и клетки отбирают с целью экстракции геномной ДНК.
Геномную ДНК секвенируют с применением платформы Illumina™, включая модификацию TraDIS, и получают считывание последовательностей, инициируемое с сайтов встраивания транспозонов. Затем считывание последовательностей вносят в карту последовательностей генома бактерии и сравнивают с аннотацией генома, определяя число считываний последовательностей, которые картированы для каждого гена для 8 указанных культур (6 анализируемых и 2 контрольных). Сравнение комплектов контрольных данных друг с другом и комплектов анализируемых данных друг с другом позволяет оценить степень разброса между экспериментами.
Сравнение контрольных данных с комплектами анализируемых данных позволяет оценить воспроизводимость экспериментов и выявить ген (гены), на которые направлен антибиотик. Считывание последовательностей системой Illumina™ с вставки транспозона в пределах жизненно важного гена - мишени воздействия антибиотиков увеличивается в клетках, выращенных в присутствии антибиотика в тех случаях, где промотор вызвал повышение транскрипции специфического гена. Кроме того, относительное количество считываний с гена (генов) - мишени повышается при повышении концентрации применяемого антибиотика.
Исключение генов устойчивости к антибиотикам
Для идентификации генов устойчивости к антибиотикам, которые не являются жизненно важными для роста в нормальных условиях, но которые придают устойчивость к антибиотикам (т.е. класс «нише-специфичных» жизненно важных генов, обсуждаемых у Langridge et al. (2009)) можно применять традиционное транспозон-направленное секвенирование сайтов встраивания TraDIS - см. Langridge er al. (2009) Genome Research 19: 2308-2316). Это позволяет исключить гены устойчивости к антибиотикам из числа кандидатов на мишени воздействия антибиотиков, как описано ниже.
Для рассматриваемой бактерии определяют MIC антибиотика, который требуется анализировать. Подготавливают четыре культуры, состоящие из 100 мл бульонной питательной среды, две из которых содержат добавки анализируемого антибиотика в концентрациях 0,5-0,75×MIC (т.е. чуть ниже MIC). В предположении, что совокупность транспозонных мутантов составляет 1 миллион, для засевания каждой культуры применяют 108-109 КОЕ из указанной совокупности. Культуры выращивают до наступления стационарной фазы, и клетки отбирают с целью экстракции геномной ДНК. Также готовят свежие культуры, и засевают их 108-109 КОЕ из первичных культур. Их выращивают до наступления стационарной фазы, и клетки отбирают с целью экстракции геномной ДНК. Геномную ДНК секвенируют с применением платформы Illumina™, включая модификацию TraDIS, и получают считывание последовательностей, инициируемое с сайтов встраивания транспозонов. Затем считывание последовательностей вносят в карту последовательностей генома бактерии и сравнивают с аннотацией генома, определяя число считываний последовательностей, которые картированы для каждого гена для 4 указанных культур (2 анализируемых и 2 контрольных).
Сравнение комплектов контрольных данных друг с другом и комплектов анализируемых данных друг с другом позволяет оценить степень разброса между экспериментами. Сравнение контрольных данных с комплектами анализируемых данных позволяет оценить воспроизводимость экспериментов и выявить гены, которые вносят вклад в устойчивость.
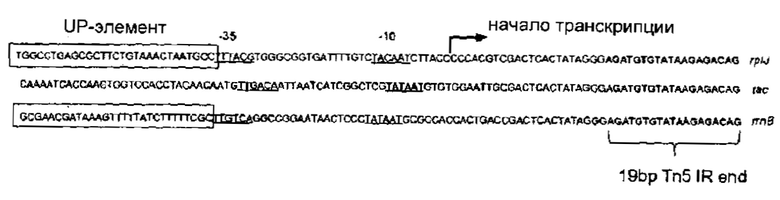
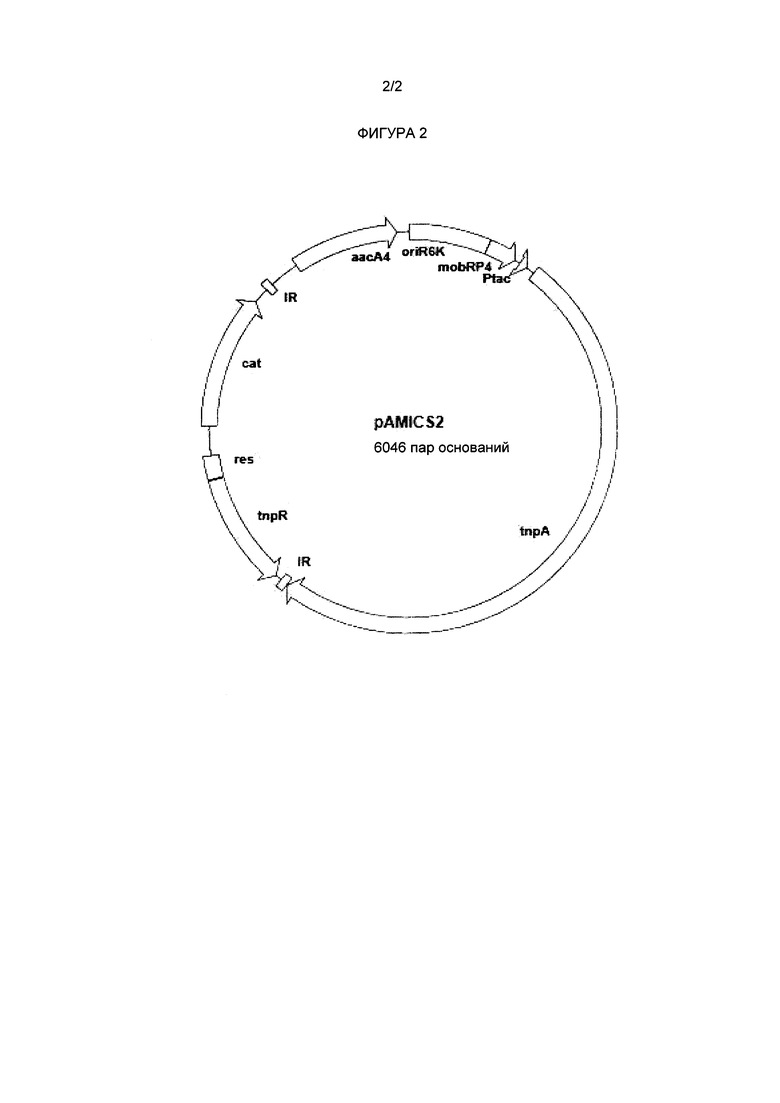
На Фигуре 1 показаны результаты поискового исследования, направленного на идентификацию генов, которые вносят вклад в устойчивость Salmonella Typhi к ципрофлоксацину. На графике представлены данные для каждого нежизненно важного гена в геноме указанной бактерии. Библиотеку мутантов с встроенными транспозонами культивировали при 4 вариантах условий: 2 контрольные культуры (без антибиотика) и 2 культуры с неоптимальной концентрацией ципрофлоксацина. Каждая точка соответствует гену, и каждый ген отложен на графике 3 раза (ctrl1 и ctrl2; CIP1 и CIP2 = черный, и указана степень разброса между экспериментами; среднее значение CIΡ и среднее значение Ctrl = серый; серые точки показывают, что график выше кластера черных точек соответствует генам, данные для которых демонстрируют значительные различия). На Фигуре 1 серые точки ниже диагонального кластера соответствуют генам, которые вносят вклад в устойчивость. Чем дальше от кластера черных точек расположены серые точки, тем более значимые данные. Гены, которые, как известно, вносят вклад в устойчивость к ципрофлоксацину у сальмонеллы, находятся в данной области графика, как и гены, для которых вклад в устойчивость ранее был неизвестен. Серые точки выше кластера черных точек соответствуют генам, которые вносят вклад в чувствительность. Опять же, гены, которые, как известно, вносят вклад в чувствительность, находятся в данной области графика, и эти данные позволяют идентифицировать гены, для которых вклад в чувствительность ранее был неизвестен. В целом данные достаточно наглядные и, следовательно, не требуют статистического анализа.
Эквиваленты
В приведенном выше описании подробно изложены предпочтительные варианты реализации настоящего изобретения. Ожидается, что на практике, при рассмотрении данного описания специалистами в данной области техники возникнет целый ряд модификаций и вариантов. Подразумевается, что такие модификации и варианты должны быть включены в пункты формулы изобретения, прилагаемой ниже.


Предложен способ идентификации предполагаемого жизненно важного гена, необходимого для жизнеспособности в условиях роста в присутствии антибиотика, который кодирует мишень для антибиотика в бактерии. Способ включает создание совокупности мутантных бактерий путем транспозонного мутагенеза с применением двух или более разных активирующих транспозонов (TnAS), причем каждый TnA содержит промотор, такой, что встраивание транспозона в ДНК бактерии приводит к нарушению функции или увеличению транскрипции гена в сайте встраивания или поблизости от него позиционно-зависимым образом. При этом транспозонный мутагенез приводит к уровню встраивания, равному по меньшей мере одному транспозону на 10 пар оснований бактериальной ДНК. Осуществляют выращивание бактерий из указанной мутантной совокупности в присутствии антибиотика в концентрации приблизительно 0,5, приблизительно 1 и приблизительно 2×MIC с получением по меньшей мере трех культур для тестирования. Сравнивают распределение вставок TnA между культурами для тестирования и контроля с целью идентификации сайтов встраивания TnA, расположенных таким образом, что транскрипция жизненно важного гена увеличивается, приводя к гиперэкспрессии продукта жизненно важного гена до уровня, при котором он подавляет действие указанного антибиотика и таким образом изменяет эффект антибиотика в отношении указанной бактерии, тем самым обеспечивая идентификацию предполагаемого жизненно важного гена, необходимого для жизнеспособности в условиях роста, применяемых на этапе выращивания бактерий из мутантной совокупности, и кодирующего мишень для указанного антибиотика у указанной бактерии. 9 з.п. ф-лы, 2 ил. 
1. Способ идентификации предполагаемого жизненно важного гена, необходимого для жизнеспособности в условиях роста в присутствии антибиотика, который кодирует мишень для антибиотика в бактерии, указанный способ включает этапы:
(а) создания совокупности мутантных бактерий путем транспозонного мутагенеза с применением двух или более разных активирующих транспозонов (TnAS), причем каждый TnA содержит промотор, такой, что встраивание транспозона в ДНК бактерии приводит к нарушению функции или увеличению транскрипции гена в сайте встраивания или поблизости от него позиционно-зависимым образом, и при этом транспозонный мутагенез приводит к уровню встраивания, равному по меньшей мере одному транспозону на 10 пар оснований бактериальной ДНК;
(б) выращивания бактерий из указанной мутантной совокупности в присутствии антибиотика в концентрации приблизительно 0,5, приблизительно 1 и приблизительно 2×MIC с получением по меньшей мере трех культур для тестирования; и
(в) сравнения распределения вставок TnA между культурами для тестирования и контроля с целью идентификации сайтов встраивания TnA, расположенных таким образом, что транскрипция жизненно важного гена увеличивается, приводя к гиперэкспрессии продукта жизненно важного гена до уровня, при котором он подавляет действие указанного антибиотика и таким образом изменяет эффект антибиотика в отношении указанной бактерии, тем самым обеспечивая идентификацию предполагаемого жизненно важного гена, необходимого для жизнеспособности в условиях роста, применяемых на этапе (б), и кодирующего мишень для указанного антибиотика у указанной бактерии.
2. Способ по п.1, отличающийся тем, что указанная совокупность мутантных бактерий содержит по меньшей мере 0,5×105 мутантов, например: (а) по меньшей мере 1×105 мутантов; (б) по меньшей мере 5×105 мутантов; (в) по меньшей мере 1×106 мутантов; (г) от 0,5×106 до 2×106 мутантов; или (д) приблизительно 1×106 мутантов.
3. Способ по п.1 или 2, отличающийся тем, что бактериальная ДНК на этапе (а) является: (а) хромосомной (геномной) ДНК; или (б) плазмидной ДНК или смесью хромосомной (геномной) и плазмидной ДНК.
4. Способ по п.1, отличающийся тем, что транспозонный мутагенез в соответствии с этапом (а) происходит in vivo или in vitro.
5. Способ по п.1, отличающийся тем, что указанная бактерия представляет собой: (а) грамположительную бактерию, необязательно выбранную из Enterococcus faecalis, Enterococcus faecium и Neisseria gonorrhoeae; или (б) грамотрицательную бактерию, необязательно выбранную из: Klebsiella pneumoniae, Acinetobacter baumanii, Escherichia coli, штаммов E. coli ST131, Pseudomonas aeruginosa, Enterobacter cloacae, Enterobacter aerogenes и Neisseria gonorrhoeae.
6. Способ по п.1, отличающийся тем, что бактерии выращивают из совокупности мутантов на этапе (б) путем посева на питательную среду от 107 до 109, например, приблизительно 108 КОЕ из указанной совокупности мутантов.
7. Способ по п.1, отличающийся тем, что указанное распределение встраивания TnA между анализируемыми культурами сравнивают путем секвенирования ДНК, примыкающей или расположенной поблизости от указанного сайта встраивания TnA.
8. Способ по п.7, отличающийся тем, что секвенирование ДНК, примыкающей или расположенной поблизости от указанного сайта встраивания TnA включает:
(а) селективную амплификацию участков соединения транспозон-бактериальная ДНК; и/или
(б) биохимический способ секвенирования путем синтеза (SBS), и, необязательно, при этом: (а) секвенируют приблизительно 25, 50, 75, 100 или более 100 пар оснований ДНК, примыкающих или расположенных поблизости от указанного сайта встраивания TnA; и/или (б) секвенированная ДНК расположена с 5’и/или 3’-стороны от указанного сайта встраивания TnA.
9. Способ по п.1, дополнительно включающий этап приписывания определенной жизненно важной функции указанному предполагаемому жизненно важному гену: (а) путем сравнения последовательностей с одним или более жизненно важными генами указанной бактерии, необязательно, при этом указанные жизненно важные гены идентифицируют путем транспозон-направленного секвенирования сайтов встраивания с применением транспозона, который при встраивании оказывает инактивирующее действие; или (б) путем определения того, что он не является геном устойчивости к антибиотику, необязательно, при этом указанный этап определения включает идентификацию гена устойчивости к антибиотику путем транспозон-направленного секвенирования сайтов встраивания с применением транспозона, который при встраивании оказывает инактивирующее действие.
10. Способ по п.1, дополнительно включающий этапы: создания устойчивого к антибиотику мутанта указанной бактерии посредством способа, включающего этап отбора в ходе роста в присутствии указанного антибиотика с получением клона мутантов, устойчивых к антибиотику (мутант AbR); трансформации указанного мутанта AbR: (i) одним или более жизненно важными генами указанной бактерии; и (ii) транспозоном, который при встраивании инактивирует бактериальную ДНК, с получением совокупности транспозонных мутантов, которые являются меродиплоидными по указанному одному или более жизненно важным генам; выращивания бактерий из указанной меродиплоидной совокупности в присутствии разных количеств указанного антибиотика с получением двух или более культур для тестирования; и сравнения распределения вставок транспозонов между культурами для тестирования с целью идентификации предполагаемого жизненно важного гена, кодирующего мишень для указанного антибиотика у указанной бактерии.
Авторы
Даты
2017-09-11—Публикация
2012-05-03—Подача