Перекрестная ссылка на родственные заявки
По настоящей заявке испрашивается приоритет в соответствии с 35 U.S.C. § 119(e) предварительной патентной заявки США № 61/759276, озаглавленной: METHODS, SYSTEMS, AND SOFTWARE FOR IDENTIFYING BIO-MOLECULES WITH INTERACTING COMPONENTS, поданной 31 января 2013 года, и предварительной патентной заявки США № 61/799377, озаглавленной: METHODS, SYSTEMS, AND SOFTWARE FOR IDENTIFYING BIO-MOLECULES USING MODELS OF MULTIPLICATIVE FORM, поданной 15 марта 2013 года, которые включены в настоящий документ посредством ссылки во всей полноте для всех целей.
Уровень техники
Настоящее раскрытие относится к областям молекулярной биологии, молекулярной эволюции, биоинформатики и цифровых систем. Более конкретно, настоящее раскрытие относится к способам предсказания посредством вычислений активности биомолекулы и/или управления направленной эволюцией. Также предлагаются системы, включая цифровые системы, и системное программное обеспечение для осуществления данных способов. Способы настоящего раскрытия применимы в оптимизации белков для промышленного и терапевтического применения.
Дизайн белков в течение долгого времени считается сложной задачей хотя бы из-за комбинаторного взрыва возможных молекул, которые образуют пространство доступных для поиска последовательностей. Пространство последовательностей белков огромно, и его невозможно исчерпывающе исследовать с помощью способов, известных в настоящее время в данной области техники. Из-за этой сложности для дизайна лучших белков использовалось много приближенных способов; главным среди них является способ направленной эволюции. В настоящее время в направленной эволюции белков преобладают различные форматы высокопроизводительного скрининга и рекомбинации, часто выполняемые итерационно.
Параллельно были предложены различные вычислительные методы для изучения пространства последовательность-активность. Хотя каждый вычислительный метод имеет преимущества в некоторых контекстах, новые способы эффективного поиска пространства последовательностей, для того чтобы идентифицировать функциональные белки, были бы весьма желательны.
Сущность изобретения
Настоящее изобретение предлагает способы идентификации биомолекул с желаемыми свойствами или наиболее подходящих для получения таких свойств из библиотек сложных биомолекул или наборов таких библиотек. Более конкретно, некоторые варианты осуществления настоящего изобретения предлагают способы построения моделей последовательность-активность, которые включают произведение множества мультипликативных членов, и применения данных моделей для управления направленной эволюцией.
Форма модели
Некоторые варианты осуществления, раскрытые в настоящем документе, предлагают способы построения моделей последовательность-активность. Каждая из моделей включает произведение множества мультипликативных членов. По меньшей мере некоторые из мультипликативных членов представляют собой мультипликативные члены, не характеризующие взаимодействие, каждый из которых содержит коэффициент, представляющий вклад определенной аминокислоты или нуклеотида в представляющую интерес активность. Определенная аминокислота или нуклеотид имеет конкретный тип остатка в конкретном положении в белковой или нуклеиновокислотной последовательности. Каждый из мультипликативных членов, не характеризующих взаимодействие, также включает независимую переменную (как правило, только одну независимую переменную), которая может принимать форму фиктивной переменной. Независимая переменная представляет присутствие или отсутствие определенной аминокислоты или нуклеотида конкретного типа в конкретном местоположении последовательности. Данные мультипликативные члены называются членами, не характеризующими взаимодействие, поскольку каждый из них представляет вклад одного остатка в одном положении в белковой или нуклеиновокислотной последовательности. Независимые переменные в членах, не характеризующих взаимодействие, не представляют два или более взаимодействующих остатка. Кроме того, каждая из моделей последовательность-активность в некоторых вариантах осуществления включает зависимую переменную, представляющую активность белкового варианта, когда модель описывает зависимость между активностью белкового варианта и произведением множества мультипликативных членов.
В некоторых вариантах осуществления настоящего изобретения модели последовательность-активность также включают члены, характеризующие взаимодействие, каждый из которых содержит коэффициент взаимодействия, представляющий вклад в активность определенной комбинации (i) первой аминокислоты или нуклеотида в первом положении в белковой последовательности, и (ii) второй аминокислоты или нуклеотида во втором положении в белковой последовательности. Вклад, представляемый коэффициентом взаимодействия, является независимым и отличным от вклада каждого из первого остатка и второго остатка в изоляции.
В некоторых вариантах осуществления модели объединяют члены, характеризующие взаимодействие, посредством умножения. В некоторых вариантах осуществления модели объединяют члены, характеризующие взаимодействие, посредством сложения. В некоторых вариантах осуществления модели объединяют члены, характеризующие взаимодействие, с мультипликативными членами, не характеризующими взаимодействие, посредством умножения или сложения. В некоторых вариантах осуществления модели являются чисто мультипликативными, объединяющими члены, не характеризующие и характеризующие взаимодействие, в одном произведении. В других вариантах осуществления модели включают по меньшей мере одно произведение множественных членов, объединенное с одним или более другими членами посредством сложения.
В некоторых вариантах осуществления настоящего изобретения модели последовательность-активность имеют аддитивную форму, содержащую сумму одного или более членов, не характеризующих взаимодействие, и по меньшей мере одного члена, характеризующего взаимодействие. В контексте аддитивных моделей член, не характеризующий взаимодействие, также называется линейным членом, тогда как член, характеризующий взаимодействие, также называется нелинейным членом или членом перекрестного произведения. Каждый из членов, не характеризующих взаимодействие, представляет присутствие определенного остатка конкретного типа в конкретном положении в последовательности в обучающем наборе библиотеки белковых вариантов. По меньшей мере один член, характеризующий взаимодействие, представляет собой член перекрестного произведения, содержащий произведение одной переменной, представляющей присутствие одного взаимодействующего остатка, и другой переменной, представляющей присутствие другого взаимодействующего остатка.
В некоторых вариантах осуществления мультипликативные члены, не характеризующие взаимодействие, и/или члены, характеризующие взаимодействие, имеют форму (коэффициент × независимая переменная). В других вариантах осуществления члены принимают форму (1 + коэффициент × независимая переменная). Специалист в данной области техники может применить другие выражения для членов, сохраняя мультипликативные свойства модели. В некоторых вариантах осуществления коэффициенты представлены в таблице соответствия.
В некоторых вариантах осуществления вместо использования аминокислотных последовательностей способы используют для генерации моделей и предсказания активности нуклеотидные последовательности. Вариации в группах нуклеотидов, например кодонов, влияют на активность пептидов, кодируемых нуклеотидными последовательностями. В некоторых вариантах осуществления модель может предлагать смещение для кодонов, которые предпочтительно экспрессируются (по сравнению с другими кодонами, кодирующими ту же самую аминокислоту), в зависимости от хозяина, используемого для того, чтобы экспрессировать пептид.
В некоторых вариантах осуществления предлагаются способы направленной эволюции. Хотя направленная эволюция может применяться в отношении белков или нуклеиновых кислот, которые кодируют белки, в некоторых случаях направленная эволюция применяется в отношении биологических молекул помимо белков. В таких вариантах осуществления модели последовательность-активность могут применяться для того, чтобы характеризовать зависимости между активностью и последовательностью различных биологических молекул. Например, последовательность может представлять собой последовательность всего генома, целую хромосому, сегмент хромосомы, коллекцию последовательностей генов взаимодействующих генов, ген, нуклеиновокислотную последовательность, белок, полисахарид и так далее. В одном или более вариантах осуществления субъединицы последовательности представляют собой хромосомы, сегменты хромосом, гаплотипы, гены, нуклеотиды, кодоны, мутации, аминокислоты, углеводы (моно-, ди-, три- или олигомерные), липид и так далее.
В некоторых вариантах осуществления способы направленной эволюции биологических молекул могут быть охарактеризованы следующим образом:
(a) получение данных о последовательности и активности для множества биологических молекул, причем каждая биологическая молекула содержит последовательность, содержащую субъединицы различных типов и положений в последовательности;
(b) построение модели последовательность-активность по полученным данным, причем модель последовательность-активность предсказывает активность как функцию типа субъединиц и положения субъединиц в последовательности, модель последовательность-активность содержит произведение множества мультипликативных членов, не характеризующих взаимодействие, причем каждый из мультипликативных членов, не характеризующих взаимодействие, содержит (1) фиктивную переменную, представляющую присутствие/отсутствие определенной субъединицы конкретного типа в конкретном положении в последовательности, и (2) коэффициент, представляющий вклад определенной субъединицы в активность; и
(c) использование модели последовательность-активность, для того чтобы идентифицировать одну или более субъединиц конкретных типов в конкретных положениях для вариации для воздействия на желаемую активность биологических молекул.
Построение и уточнение модели
В одном или более воплощениях в соответствии с вышеуказанными вариантами осуществления обучающий набор для генерации модели последовательность-активность получают из множества вариантов белков, которое может предлагаться в виде библиотеки белков. Библиотека белков может включать белки из различных источников. В одном примере члены включают природные белки, такие как кодируемые членами одного семейства генов. В другом примере последовательности включают белки, полученные посредством использования основанного на рекомбинации механизма генерации разнообразия. Например, для данной цели в отношении нуклеиновых кислот, кодирующих все или некоторые из одного или более природных родительских белков, могут быть осуществлены рекомбинация, опосредованная фрагментацией ДНК, рекомбинация, опосредованная синтетическими олигонуклеотидами, или их комбинация. В еще одном примере члены получают посредством реализации протокола плана эксперимента (DOE), для того чтобы идентифицировать систематически варьируемые последовательности.
В некоторых вариантах осуществления данные о последовательности и активности для каждого из множества вариантов белков получают посредством (i) анализирования каждого из множества вариантов белков, для того чтобы определить его активность, и (ii) секвенирования каждого из множества вариантов белков, для того чтобы определить его последовательность. Полученные данные для каждой последовательности также называются наблюдением. Вместе наблюдения содержат обучающий набор.
Некоторые варианты осуществления предлагают методы выбора членов и коэффициентов для модели, которые наилучшим образом описывают активность последовательности. Следует отметить, что часто существует гораздо больше возможных членов, характеризующих парное или более высокого порядка взаимодействие, чем существует реальных взаимодействий между остатками. Следовательно, для того чтобы избежать переобучения, обычно рассматривают только ограниченное количество членов, характеризующих взаимодействие, и используемые члены должны отражать взаимодействия, которые влияют на активность.
Некоторые варианты осуществления предлагают способы генерации модели последовательность-активность посредством осуществления пошагового добавления, удаления или умножения членов, характеризующих взаимодействие и/или не характеризующих взаимодействие.
Генетические алгоритмы относятся к методам, которые можно использовать для генерации моделей, имеющих форму произведений членов, характеризующих взаимодействие или не характеризующих взаимодействие, как описано выше. Регрессионные методы и генетические алгоритмы относятся к методам, которые можно использовать для генерации моделей, имеющих форму сумм членов, характеризующих взаимодействие или не характеризующих взаимодействие, как описано выше.
Один аспект настоящего раскрытия предлагает пошаговый способ получения модели последовательность-активность, которая может помогать при идентифицировании биологических молекул для воздействия на желаемую активность, причем данный способ включает: (a) получение данных о последовательности и активности для множества биологических молекул; (b) получение основной модели по данным о последовательности и активности, причем основная модель предсказывает активность как функцию присутствия или отсутствия субъединиц последовательности; (c) получение по меньшей мере одной новой модели посредством добавления, удаления или умножения по меньшей мере одного нового члена, характеризующего взаимодействие, к/из основной модели, где новый член, характеризующий взаимодействие, представляет взаимодействие между двумя или более взаимодействующими субъединицами; (d) определение способности по меньшей мере одной новой модели предсказывать активность как функцию присутствия или отсутствия субъединиц; и (e) определение того, добавлять/удалять ли новый член, характеризующий взаимодействие, к/из основной модели, на основании способности по меньшей мере одной новой модели предсказывать активность, определенную в (d), и со смещением в сторону от добавления нового члена, характеризующего взаимодействие. Затем полученную модель можно использовать в различных применениях, как например в направленной эволюции библиотек белков, для того чтобы идентифицировать белки с желаемой биологической активностью и свойствами.
Некоторые варианты осуществления предлагают способы использования генетического алгоритма для выбора одного или более членов моделей последовательность-активность. Другие варианты осуществления предлагают способы использования генетического алгоритма для того, чтобы корректировать значения коэффициентов для подгонки моделей к полученным данным.
В одном или более вариантах осуществления модель, включающую члены, характеризующие взаимодействие, подгоняют к наблюдаемым данным, используя методы байесовской регрессии, причем для определения апостериорных распределений вероятностей модели используют априорное знание.
В одном или более вариантах осуществления создают две или более новых модели, каждая из которых включает по меньшей мере один отличающийся член, характеризующий взаимодействие. В таких вариантах осуществления способ дополнительно содержит получение ансамблевой модели, основанной на двух или более новых моделях. Ансамблевая модель включает члены, характеризующие взаимодействие, из двух или более новых моделей. Ансамблевая модель взвешивает члены, характеризующие взаимодействие, в соответствии со способностями двух или более новых моделей предсказывать представляющую интерес активность.
Модель последовательность-активность может быть получена из обучающего набора с помощью множества различных методов. В определенных вариантах осуществления модель представляет собой регрессионную модель, такую как модель частных наименьших квадратов, модель байесовской регрессии или модель регрессии на главные компоненты. В другом варианте осуществления модель представляет собой нейронную сеть.
Если явно не указано иное, или если они иным образом несовместимы по своей природе, способы уточнения моделей и различных форм моделей, описанные в настоящем документе, совместимы друг с другом. Их можно применять в различных комбинациях для генерации желаемых моделей последовательность-активность. В некоторых вариантах осуществления настоящего изобретения генерируемые модели можно применять для управления направленной эволюцией.
Использование модели для управления направленной эволюцией
В определенных вариантах осуществления варианты белков с желаемой активностью идентифицируют посредством направленной эволюции. Некоторые варианты осуществления предлагают способы управления направленной эволюцией вариантов белков с использованием генерируемых моделей последовательность-активность. Различные модели последовательность-активность, полученные и уточненные в соответствии со способами, описанными выше, подходят для управления направленной эволюцией белков или биологических молекул. В качестве части данного процесса способы могут идентифицировать последовательности, которые следует использовать для генерации новой библиотеки белковых вариантов. Такие последовательности включают вариации в определенных остатках, идентифицированных выше, или представляют собой предшественники, используемые для последующего введения таких вариаций. Последовательности могут быть модифицированы с помощью мутагенеза или основанного на рекомбинации механизма генерации разнообразия для генерации новой библиотеки вариантов белков. Любая или обе из форм генерации разнообразия могут образовывать часть процедуры направленной эволюции. Новую библиотеку можно также использовать при разработке новой модели последовательность-активность. Новую библиотеку белковых вариантов анализируют для определения воздействия на определенную активность, такую как стабильность, каталитическая активность, терапевтическая активность, устойчивость к патогену или токсину, токсичность и так далее.
В некоторых вариантах осуществления получение олигонуклеотидов или нуклеиновокислотных последовательностей осуществляют посредством синтезирования олигонуклеотидов или нуклеиновокислотных последовательностей с помощью синтезатора нуклеиновых кислот. Некоторые варианты осуществления настоящего изобретения включают осуществление цикла направленной эволюции с использованием полученных олигонуклеотидов или белковой последовательности в качестве строительных элементов для направленной эволюции. Различные варианты осуществления настоящего изобретения применяют рекомбинацию и/или мутагенез к этим строительным элементам для генерации разнообразия.
В качестве одного примера, некоторые варианты осуществления применяют к олигонуклеотидам методы рекомбинации. В данных вариантах осуществления способы включают выбор одной или более мутаций для цикла направленной эволюции посредством оценивания коэффициентов членов модели последовательность-активность. Выбранные мутации представляют определенные аминокислоты или нуклеотиды конкретных типов в конкретных положениях на основании их вкладов в активность белков, предсказанных моделями. В некоторых вариантах осуществления выбор мутаций включает определение одного или более коэффициентов, которые, как определено, больше, чем другие коэффициенты (или иным образом указывают на сильное воздействие на активность), и выбор определенных аминокислот или нуклеотидов в определенных положениях, представляемых одним или более коэффициентами, определенными таким образом. В некоторых вариантах осуществления после выбора мутаций в соответствии с моделями последовательность-активность способы включают получение множества олигонуклеотидов, содержащих или кодирующих одну или более мутаций, и осуществление цикла направленной эволюции с использованием олигонуклеотидов, полученных таким образом. В некоторых вариантах осуществления методы направленной эволюции включают комбинирование и/или рекомбинирование олигонуклеотидов. Может быть осуществлена реакция перетасовки с использованием олигонуклеотидов.
Другие варианты осуществления применяют методы рекомбинации к белковым последовательностям. В некоторых вариантах осуществления способы включают идентифицирование новой белковой или новой нуклеиновокислотной последовательности и получение и анализирование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью. В некоторых вариантах осуществления способы дополнительно включают использование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью, в качестве исходной точки для дальнейшей направленной эволюции. В некоторых вариантах осуществления процесс направленной эволюции включает фрагментирование и рекомбинирование белковой последовательности, о которой модель предсказывает, что она имеет желаемый уровень активности.
В некоторых вариантах осуществления способы идентифицируют и/или получают новую белковую или новую нуклеиновокислотную последовательность на основании отдельных мутаций, о которых модель предсказывает, что они важны. Данные способы включают: выбор одной или более мутаций посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность; идентифицирование новой белковой или новой нуклеиновокислотной последовательности, содержащей одну или более мутаций, выбранных выше, и получение и анализирование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью.
В других вариантах осуществления способы идентифицируют и/или получают новую белковую или новую нуклеиновокислотную последовательность на основании предсказанной активности всей последовательности, а не отдельных мутаций. В некоторых из данных вариантов осуществления способы включают использование множественных белковых последовательностей или множественных аминокислотных последовательностей в модели последовательность-активность и определение значений активности, предсказанных моделью последовательность-активность для каждой из множественных белковых последовательностей или нуклеиновокислотных последовательностей. Способы дополнительно включают выбор новой белковой последовательности или новой нуклеиновокислотной последовательности из множественных белковых последовательностей или множественных аминокислотных последовательностей, использованных выше, посредством оценивания значений активности, предсказанных моделью последовательность-активность для данных множественных последовательностей. Способы также включают получение и анализирование белка, имеющего новую белковую последовательность, или белка, кодируемого новой нуклеиновокислотной последовательностью.
Некоторые варианты осуществления включают выбор одного или более положений в белковой последовательности или нуклеиновокислотной последовательности и проведение насыщающего мутагенеза в одном или более положениях, идентифицированных таким образом. В некоторых вариантах осуществления положения выбирают посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность. Соответственно, в некоторых вариантах осуществления цикл направленной эволюции включает осуществление насыщающего мутагенеза в отношении белковой последовательности в положениях, выбранных с использованием моделей последовательность-активность. В некоторых вариантах осуществления, включающих модели, содержащие один или более членов, характеризующих взаимодействие, способы включают применение мутагенеза одновременно в отношении двух или более взаимодействующих остатков.
В некоторых вариантах осуществления способ включает выбор одного или более членов новой библиотеки белковых вариантов для получения. Один или более из них могут быть затем синтезированы и/или экспрессированы в системе экспрессии. В конкретном варианте осуществления способ продолжается следующим образом: (i) предоставление системы экспрессии, с которой может быть экспрессирован выбранный член новой библиотеки белковых вариантов; и (ii) экспрессия выбранного члена новой библиотеки белковых вариантов.
Некоторые варианты осуществления используют модель последовательность-активность, для того чтобы идентифицировать основную или референсную последовательность, в которой идентифицируют остатки для фиксации или вариации. В некоторых случаях референсная последовательность представляет собой последовательность, о которой модель предсказывает, что она имеет наивысшее значение (или одно из наивысших значений) желаемой активности. В другом случае референсная последовательность является членом исходной библиотеки белковых вариантов. Из референсной последовательности способ может выбирать подпоследовательности для осуществления вариаций. Дополнительно или альтернативно модель последовательность-активность ранжирует положения остатков (или конкретных остатков в некоторых положениях) в порядке воздействия на желаемую активность.
Другой аспект настоящего раскрытия относится к приспособлению и компьютерным программным продуктам, включая машиночитаемые среды, на которых предоставляются программные инструкции и/или конфигурации данных для воплощения способов и программных систем, описанных выше. Часто программные инструкции предоставляются в виде кода для осуществления операций некоторого способа. Данные, если они используются для воплощения признаков настоящего раскрытия, могут предоставляться в виде структур данных, таблиц баз данных, объектов данных или других соответствующих конфигураций указанной информации. Все способы или системы, описанные в настоящем документе, могут быть полностью или частично представлены в виде таких программных инструкций и/или данных, предоставляемых на любых подходящих машиночитаемых средах.
Эти и другие признаки более подробно описаны ниже в подробном описании и в сочетании со следующими фигурами.
Краткое описание чертежей
Фигура 1A представляет собой блок-схему, изображающую общий процесс генерации мультипликативной модели последовательность-активность.
Фигура 1B представляет собой блок-схему, изображающую генетический алгоритм для подгонки мультипликативной модели последовательность-активность к наблюдаемым данным в соответствии с вариантом осуществления настоящего изобретения.
Фигура 2 представляет собой блок-схему, изображающую процесс направленной эволюции для генерации одного или более поколений библиотек белковых вариантов, в котором операции используют мультипликативную модель последовательность-активность, такую как одна из полученных на фигуре 1, для управления генерацией библиотек белковых вариантов. Генерируемый вариант библиотеки может предоставлять данные о последовательности и активности для получения одной или более новых моделей последовательность-активность.
Фигуры 3A-H представляют собой диаграммы, показывающие примеры, в которых сравниваются прогностические возможности линейных и нелинейных аддитивных моделей.
Фигуры 3I-J представляют собой диаграммы, показывающие примеры в который сравниваются прогностические возможности некоторых мультипликативных и аддитивных моделей.
Фигура 4A-4B иллюстрирует блок-схемы процессов воплощения способы пошагового добавления и удаления для получения модели последовательность-активность. Фигура 4A иллюстрирует конкретный пример способа пошагового добавления для получения модели; и фигура 4B иллюстрирует конкретный пример способа пошагового удаления для получения модели.
Фигура 5 иллюстрирует блок-схему процесса, реализующего байесовскую регрессию в направленной эволюции вариантов последовательностей в соответствии с вариантом осуществления.
Фигура 6 иллюстрирует блок-схему процесса, реализующего регрессию ансамбля в направленной эволюции вариантов последовательностей в соответствии с вариантом осуществления.
Фигура 7 представляет собой блок-схему, изображающую бутстреп-способ p-значений для генерации библиотек белковых вариантов в соответствии с вариантом осуществления.
Фигура 8 представляет собой схему примера цифрового устройства.
Фигуры 9A-C представляют собой диаграммы, которые показывают, соответственно, предсказательную силу линейной аддитивной модели, мультипликативной модели без взаимодействия и мультипликативной модели с взаимодействием.
Подробное описание
I. Определения
Если в настоящем документе не указано иное, все технические и научные термины, используемые в настоящем документе, имеют значение, обычно понимаемое средним специалистом в данной области техники. Различные научные словари, которые содержат термины, включенные в настоящий документ, хорошо известны и доступны специалистам в данной области техники. Все способы и материалы, подобные или эквивалентные описанным в настоящем документе, находят применение при осуществлении вариантов осуществления, раскрытых в настоящем документе, на практике.
Термины, определенные непосредственно ниже, становятся лучше понятны посредством ссылки на описание изобретения в целом. Определения служат только для целей описания конкретных вариантов осуществления и помощи в понимании сложных концепций, описанных в данном описании изобретения. Они не предназначены для ограничения полного объема настоящего раскрытия. В частности, следует понимать, что настоящее раскрытие не ограничено конкретными описанными последовательностями, композициями, алгоритмами, системами, методологией, протоколами и реагентами, поскольку они могут варьироваться в зависимости от контекста, в котором они используются специалистами в данной области техники.
Как используется в данном описании изобретения и прилагаемой формуле изобретения, формы единственного числа включают ссылки на множественное число, если содержание и контекст явно не требует иного. Таким образом, например, ссылка на "устройство" включает комбинацию двух или более таких устройств, и тому подобное.
Если не указано иное, союз "или" предназначен для использования в его правильном смысле как булев логический оператор, охватывающий как альтернативный выбор признаков (A или B, где выбор A является взаимоисключающим с B), так и выбор признаков в сочетании (A или B, где выбраны как A, так и B). В некоторых местах в тексте термин "и/или" используется для той же цели, что не должно быть истолковано как означающее, что "или" используется со ссылкой на взаимоисключающие альтернативы.
"Биомолекула" или "биологическая молекула" относится к молекуле, которая обычно встречается в биологическом организме. В некоторых вариантах осуществления биологические молекулы содержат полимерные биологические макромолекулы, содержащие множественные субъединицы (то есть "биополимеры"). Типичные биомолекулы включают, но без ограничения, молекулы, которые разделяют некоторые структурные особенности с природными полимерами, такие как РНК (образованные из нуклеотидных субъединиц), ДНК (образованные из нуклеотидных субъединиц) и пептиды или полипептиды (образованные из аминокислотных субъединиц), включая, например, РНК, аналоги РНК, ДНК, аналоги ДНК, полипептиды, аналоги полипептидов, пептидные нуклеиновые кислоты (ПНК), комбинации РНК и ДНК (например, химерапласты) или тому подобное. Не предполагается, что биомолекулы ограничены какой-либо конкретной молекулой, поскольку любая подходящая биологическая молекула находит применение в настоящем изобретении, включая, но без ограничения, например, липиды, углеводы или другие органические молекулы, которые создаются одной или более генетически кодируемыми молекулами (например, одним или более ферментами или ферментативными путями), или тому подобное.
Тeрмины "полинуклеотид" и "нуклеиновая кислота" относятся к дезоксирибонуклеотидам или рибонуклеотидам и их полимерам (например, олигонуклеотидам, полинуклеотидам и так далее) или в одно-, или в двухцепочечной форме. Данные термины включают, но без ограничения, одно-, двух- или трехцепочечную ДНК, геномную ДНК, кДНК, РНК, гибрид ДНК-РНК, полимеры, содержащие пуриновые и пиримидиновые основания, и/или другие природные, химически или биохимически модифицированные, неприродные или дериватизированные нуклеотидные основания. Ниже приведены неограничивающие примеры полинуклеотидов: гены, фрагменты генов, хромосомные фрагменты, EST, экзоны, интроны, мРНК, тРНК, рРНК, рибозимы, кДНК, рекомбинантные полинуклеотиды, разветвленные полинуклеотиды, плазмиды, векторы, выделенная ДНК любой последовательности, выделенная РНК любой последовательности, нуклеиновокислотные зонды и праймеры. В некоторых вариантах осуществления полинуклеотиды содержат модифицированные нуклеотиды, такие как метилированные нуклеотиды и аналоги нуклеотидов, урацил, другие сахара и линкерные группы, такие как фторрибоза и тиоат, и нуклеотидные разветвления. В некоторых альтернативных вариантах осуществления последовательность нуклеотидов прерывается ненуклеотидными компонентами.
Если специально не ограничено, данный термин охватывает нуклеиновые кислоты, содержащие известные аналоги природных нуклеотидов, которые обладают связывающими свойствами, аналогичными референсной нуклеиновой кислоте, и метаболизируются аналогичным природным нуклеотидам образом. Если не указано иное, конкретная нуклеиновокислотная последовательность также неявно охватывает ее консервативно модифицированные варианты (например, замены вырожденных кодонов) и комплементарные последовательности, а также явно указанную последовательность. В частности, замены вырожденных кодонов могут быть получены посредством генерации последовательностей, в которых третье положение одного или более выбранных (или всех) кодонов заменено остатками смешанных оснований и/или дезоксиинозина (Batzer et al., (1991), Nucleic Acid Res. 19: 5081; Ohtsuka et al., (1985), J. Biol. Chem. 260: 2605-2608; и Rossolini et al., (1994), Mol. Cell. Probes 8: 91-98). Термин «нуклеиновая кислота» используется взаимозаменяемо с, например, терминами «олигонуклеотид, полинуклеотид, кДНК и мРНК».
Термины "белок", "полипептид" и "пептид" используются взаимозаменяемо для обозначения полимера из по меньшей мере двух аминокислот, ковалентно связанных амидной связью, независимо от длины или посттрансляционной модификации (например, гликозилирования, фосфорилирования, липидирования, миристилирования, убиквитинирования и так далее). В некоторых случаях полимер имеет по меньшей мере приблизительно 30 аминокислотных остатков, а обычно по меньшей мере приблизительно 50 аминокислотных остатков. Более типично, они содержат по меньшей мере приблизительно 100 аминокислотных остатков. Данные термины включают композиции, обычно рассматриваемые как фрагменты белков или пептидов полной длины. К данному определению относятся D- и L-аминокислоты и смеси D- и L-аминокислот. Полипептиды, описанные в настоящем документе, не ограничены генетически кодируемыми аминокислотами. Более того, в дополнение к генетически кодируемым аминокислотам полипептиды, описанные в настоящем документе, могут быть составлены, или полностью, или частично, из природных и/или синтетических некодируемых аминокислот. В некоторых вариантах осуществления полипептид представляет собой часть предкового или родительского полипептида полной длины, содержащего присоединения, или делеции (например, пропуски), или замены аминокислот по сравнению с аминокислотной последовательностью родительского полипептида полной длины, при этом сохраняя функциональную активность (например, каталитическую активность).
Как используется в настоящем документе, термин "целлюлаза" относится к категории ферментов, способных к гидролизу целлюлозы (β-1,4-глюкановых или β-D-глюкозидных связей) до более коротких целлюлозных цепей, олигосахаридов, целлобиозы и/или глюкозы. В некоторых вариантах осуществления термин "целлюлаза" охватывает бета-глюкозидазы, эндоглюканазы, целлобиогидролазы, целлобиозодегидрогеназы, эндоксиланазы, бета-ксилозидазы, арабинофуранозидазы, альфа-глюкуронидазы, ацетилксиланэстеразы, ферулоилэстеразы и/или альфа-глюкуронилэстеразы. В некоторых вариантах осуществления термин "целлюлаза" охватывает гидролизующие гемицеллюлозу ферменты, включая, но без ограничения, эндоксиланазы, бета-ксилозидазы, арабинофуранозидазы, альфа-глюкуронидазы, ацетилксиланэстеразу, ферулоилэстеразу и альфа-глюкуронилэстеразу. "Производящая целлюлазу грибная клетка" представляет собой грибную клетку, которая экспрессирует и секретирует по меньшей мере один гидролизующий целлюлозу фермент. В некоторых вариантах осуществления производящие целлюлазу грибные клетки экспрессируют и секретируют смесь гидролизующих целлюлозу ферментов. "Целлюлолитический", "гидролизующий целлюлозу", "разрушающий целлюлозу" и аналогичные термины относятся к ферментам, таким как эндоглюканазы и целлобиогидролазы (последние также называют "экзоглюканазами"), которые действуют синергически, расщепляя целлюлозу до растворимых ди- или олигосахаридов, таких как целлобиоза, которые затем далее гидролизуются до глюкозы с помощью бета-глюкозидазы. В некоторых вариантах осуществления целлюлаза представляет собой рекомбинантную целлюлазу, выбранную из β-глюкозидаз (BGL), целлобиогидролаз типа 1 (CBH1), целлобиогидролаз типа 2 (CBH2), гликозидгидролаз 61 (GH61) и/или эндоглюканаз (EG). В некоторых вариантах осуществления целлюлаза представляет собой рекомбинантную целлюлазу из Myceliophthora, выбранную из β-глюкозидаз (BGL), целлобиогидролаз типа 1 (CBH1), целлобиогидролаз типа 2 (CBH2), гликозидгидролаз 61 (GH61) и/или эндоглюканаз (EG). В некоторых дополнительных вариантах осуществления целлюлаза представляет собой рекомбинантную целлюлазу, выбранную из EG1b, EG2, EG3, EG4, EG5, EG6, CBH1a, CBH1b, CBH2a, CBH2b, GH61a и/или BGL.
Термин "последовательность" используется в настоящем документе для обозначения порядка и идентичности любой биологической последовательности, включая, но без ограничения весь геном, целую хромосому, сегмент хромосомы, коллекцию последовательностей генов взаимодействующих генов, ген, нуклеиновокислотную последовательность, белок, полисахарид и так далее. В некоторых контекстах последовательность обозначает порядок и идентичность аминокислотных остатков в белке (то есть белковую последовательность или цепочку символов белка) или порядок и идентичность нуклеотидов в нуклеиновой кислоте (то есть нуклеиновокислотную последовательность или цепочку символов нуклеиновой кислоты). Последовательность может быть представлена цепочкой символов. "Нуклеиновокислотная последовательность" обозначает порядок и идентичность нуклеотидов, образующих нуклеиновую кислоту. "Белковая последовательность" обозначает порядок и идентичность аминокислот, образующих белок или пептид.
"Кодон" относится к конкретной последовательности трех последовательных нуклеотидов, которая представляет собой часть генетического кода, и которая определяет конкретную аминокислоту в белке или начинает или заканчивает синтез белка.
"Нативная последовательность" или "последовательность дикого типа" относится к полинуклеотиду или полипептиду, выделенному из природного источника. "Нативная последовательность" включает рекомбинантные формы нативного полипептида или полинуклеотида, которые имеют последовательность, идентичную нативной форме.
Термин "ген" широко используется для обозначения любого сегмента ДНК или другой нуклеиновой кислоты, связанного с биологической функцией. Таким образом, гены включают кодирующие последовательности и, необязательно, регуляторные последовательности, требуемые для их экспрессии. Гены также, необязательно, включают неэкспрессируемые нуклеиновокислотные сегменты, которые, например, образуют последовательности распознавания для других белков. Гены могут быть получены из множества источников, включая клонирование из представляющего интерес источника или синтезирование по известной или предсказанной информации о последовательности, и могут включать последовательности, разработанные так, чтобы они имели желаемые параметры.
"Мотив" относится к структуре субъединиц в биологических молекулах или среди биологических молекул. Например, термин "мотив" можно использовать в отношении структуры субъединиц некодируемой биологической молекулы или структуры субъединиц кодируемого представления биологической молекулы.
Термин "хромосома" используется в отношении организованной структуры ДНК и соответствующего белка, обнаруживаемой в клетках, содержащей один фрагмент спиральной ДНК, включающий множество генов, регуляторных элементов и других нуклеотидных последовательностей. Данный термин также используется в отношении последовательности ДНК данной структуры.
В контексте генетического алгоритма, термин "хромосома" используется в качестве альтернативного названия для отдельной модели (или набора параметров модели) в популяции моделей. Он используется таким образом, поскольку модель из родительского поколения передает свои параметры (или гены) моделям дочернего поколения, что похоже на то, как родительская хромосома передает свои гены дочерней хромосоме.
"Скрининг" относится к процессу, в котором определяют одно или более свойств одной или более биомолекул. Например, типичные процессы скрининга включают те, в которых определяют одно или более свойств одного или более членов одной или более библиотек.
"Система экспрессии" представляет собой систему для экспрессии белка или пептида, кодируемого геном или другой нуклеиновой кислотой.
"Клетка-хозяин" или "рекомбинантная клетка-хозяин" относится к клетке, которая содержит по меньшей мере одну рекомбинантную молекулу нуклеиновой кислоты. Таким образом, например, в некоторых вариантах осуществления рекомбинантные клетки-хозяева экспрессируют гены, которые не обнаруживаются в нативной (то есть нерекомбинантной) форме клетки.
"Направленная эволюция", "управляемая эволюция" или "искусственная эволюция" относится к in vitro или in vivo процессам искусственного изменения одной или более последовательностей биомолекул (или цепочки символов, представляющей такую последовательность) с помощью искусственного отбора, мутации, рекомбинации или другой манипуляции. В некоторых вариантах осуществления направленная эволюция происходит в репродуктивной популяции, в которой имеют место (1) различия индивидуумов, причем некоторые различия являются (2) наследуемыми, из которых некоторые различия (3) отличаются по приспособленности. Репродуктивный успех определяют по результату отбора по предварительно определенному свойству, такому как полезное свойство. Репродуктивная популяция может представлять собой, например, физическую популяцию или виртуальную популяцию в компьютерной системе.
В определенных вариантах осуществления способы направленной эволюции генерируют библиотеки белковых вариантов посредством рекомбинирования генов, кодирующих варианты родительской библиотеки белковых вариантов. Способы могут использовать олигонуклеотиды, содержащие последовательности или подпоследовательности для кодирования белков родительской библиотеки вариантов. Некоторые из олигонуклеотидов родительской библиотеки вариантов могут быть близкородственными, отличающимися только по выбору кодонов для изменяющихся аминокислот, выбранных для варьирования посредством рекомбинации с другими вариантами. Могут быть осуществлены один или более циклов способа, до тех пор, пока не будут достигнуты желаемые результаты. Если используют множественные циклы, то каждый включает этап скрининга, для того чтобы определить, какие из имеющих приемлемую эффективность вариантов следует использовать в следующем цикле рекомбинации.
В некоторых вариантах осуществления способы направленной эволюции генерируют варианты белков посредством сайт-направленного мутагенеза в определенных местоположениях, идентифицированных с помощью моделей последовательность-активность. Некоторые варианты осуществления используют насыщающий мутагенез, в котором предпринимает попытка генерации всех возможных (или настолько близко к этому, насколько возможно) мутаций в конкретном сайте или узкой области гена.
"Перетасовка" и "перетасовка генов" относятся к способам направленной эволюции, вносящим разнообразие с помощью рекомбинирования коллекции фрагментов родительских полинуклеотидов посредством ряда циклов удлинения цепи. В определенных вариантах осуществления один или более из циклов удлинения цепи являются самопраймирующимися; то есть осуществляемыми без добавления праймеров, отличных от самих фрагментов. Каждый цикл включает отжиг одноцепочечных фрагментов посредством гибридизации, последующую элонгацию отожженных фрагментов посредством удлинения цепи и денатурацию. Во время перетасовки растущая нить нуклеиновой кислоты, как правило, подвергается воздействию множественных различных партнеров по отжигу в процессе, иногда называемом "переключение матрицы". Как используется в настоящем документе, "переключение матрицы" относится к возможности замены одного нуклеиновокислотного домена из одной нуклеиновой кислоты вторым доменом из второй нуклеиновой кислоты (то есть первая и вторая нуклеиновые кислоты служат в качестве матриц в процедуре перетасовки).
Переключение матрицы часто производит химерные последовательности, что является результатом введения кроссинговеров между фрагментами различного происхождения. Кроссинговеры создают посредством рекомбинаций с переключением матрицы во время множественных циклов отжига, удлинения и денатурации. Таким образом, перетасовка, как правило, приводит к получению вариантных полинуклеотидных последовательностей. В некоторых вариантах осуществления вариантные последовательности образуют "библиотеку" вариантов. В некоторых вариантах осуществления данных библиотек варианты содержат сегменты последовательностей из двух или более родительских полинуклеотидов.
Когда используют два или более родительских полинуклеотида, отдельные родительские полинуклеотиды являются достаточно гомологичными для того, чтобы фрагменты от различных родителей гибридизировались в условиях отжига, используемых в циклах перетасовки. В некоторых вариантах осуществления перетасовка делает возможной рекомбинацию родительских полинуклеотидов, имеющих относительно ограниченную гомологию. Часто отдельные родительские полинуклеотиды имеют отличающиеся и/или уникальные домены и/или другие представляющие интерес характеристики последовательностей. Когда используются родительские полинуклеотиды, имеющие отличающиеся характеристики последовательностей, перетасовка может производить сильно различающиеся вариантные полинуклеотиды.
В данной области техники известны различные методы перетасовки. См., например, патенты США №№ 6917882, 7776598, 8029988, 7024312 и 7795030, которые включены в настоящий документ посредством ссылки во всей их полноте.
"Сплайсинг генов посредством удлинения перекрывания" или "SOEing генов" представляет собой основанный на ПЦР способ рекомбинирования последовательностей ДНК, не опирающегося на сайты рестрикции, и непосредственной генерации мутировавших фрагментов ДНК in vitro. Посредством модифицирования последовательностей, введенных с 5'-концов праймеров, можно сделать так, что любая пара продуктов полимеразной цепной реакции будет иметь общую последовательность с одного конца. В условиях полимеразной цепной реакции общая последовательность позволяет нитям из двух различных фрагментов быть комплементарными друг другу и гибридизироваться друг на друге, образуя новую последовательность, содержащую два фрагмента на каждом конце, соединенные перекрыванием общей последовательности. Удлинение данного перекрывания ДНК-полимеразой дает рекомбинантную молекулу.
"Мутагенез" представляет собой процесс введения мутации в стандартную или референсную последовательность, такую как родительская нуклеиновая кислота или родительский полипептид.
Одним примером полезного метода введения мутаций является сайт-направленный мутагенез, хотя находит применение любой подходящий способ. Таким образом, альтернативно или дополнительно, мутанты могут быть получены посредством синтеза генов, насыщающего случайного мутагенеза, полусинтетических комбинаторных библиотек остатков, направленной эволюции, рекурсивной рекомбинации последовательностей ("RSR") (см., например, заявку на патент США № 2006/0223143, включена посредством ссылки в настоящий документ во всей полноте), перетасовки генов, ПЦР сниженной точности и/или любого другого подходящего способа.
Один пример подходящей процедуры насыщающего мутагенеза описан в опубликованной патентной заявке США № 20100093560, которая включена в настоящий документ посредством ссылки во всей полноте.
"Фрагмент" представляет собой любую часть последовательности нуклеотидов или аминокислот. Фрагменты могут быть получены с помощью любого подходящего способа, известного в данной области техники, включая, но без ограничения расщепление полипептидной или полинуклеотидной последовательности. В некоторых вариантах осуществления фрагменты получают с использованием нуклеаз, которые расщепляют полинуклеотиды. В некоторых дополнительных вариантах осуществления фрагменты генерируют с использованием методов химического и/или биологического синтеза. В некоторых вариантах осуществления фрагменты содержат подпоследовательности по меньшей мере одной родительской последовательности, генерируемые с использованием частичной элонгации цепи комплементарной нуклеиновой кислоты (кислот).
"Родительский полипептид", "родительский полинуклеотид", "родительская нуклеиновая кислота" и "родитель" обычно используются для обозначения полипептида дикого типа, полинуклеотида дикого типа или варианта, используемого в качестве исходной точки в процедуре генерации разнообразия, такой как направленная эволюция. В некоторых вариантах осуществления сам родитель получают посредством перетасовки или другой процедуры генерации разнообразия. В некоторых вариантах осуществления мутанты, используемые в направленной эволюции, непосредственно связаны с родительским полипептидом. В некоторых вариантах осуществления родительский полипептид стабилен под воздействием экстремальных условий температуры, pH и/или растворителя и может служить в качестве основы для генерации вариантов для перетасовки. В некоторых вариантах осуществления родительский полипептид не стабилен в отношении экстремальных условий температуры, pH и/или растворителя, и родительский полипептид подвергается эволюции для создания устойчивых вариантов.
"Родительская нуклеиновая кислота" кодирует родительский полипептид.
"Мутант", "вариант" и "вариантная последовательность", как используется в настоящем документе, относятся к биологической последовательности, которая отличается в некоторых отношениях от стандартной или референсной последовательности. Отличие может быть названо "мутацией". В некоторых вариантах осуществления мутант представляет собой аминокислотную (то есть полипептид) или полинуклеотидную последовательность, которая была изменена с помощью по меньшей мере одной замены, вставки, кроссинговера, делеции и/или другой генетической операции. Для целей настоящего раскрытия мутанты и варианты не ограничены конкретным способом, посредством которого он были сгенерированы. В некоторых вариантах осуществления мутант или вариантная последовательность имеет повышенные, пониженные или по существу такие же активности или свойства по сравнению с родительской последовательностью. В некоторых вариантах осуществления вариантный полипептид содержит один или более аминокислотных остатков, которые подверглись мутации, по сравнению с аминокислотной последовательностью полипептида дикого типа (например, родительского полипептида). В некоторых вариантах осуществления один или более аминокислотных остатков полипептида остаются постоянными, являются инвариантными или являются не мутировавшими по сравнению с родительским полипептидом в вариантных полипептидах, составляющих множество. В некоторых вариантах осуществления родительский полипептид используется в качестве основы для генерации вариантов с улучшенной стабильностью, активностью или другим свойством.
"Библиотека" или "популяция" относится к коллекции из по меньшей мере двух различных молекул, цепочек символов и/или моделей, таких как нуклеиновокислотные последовательности (например, гены, олигонуклеотиды и так далее), или продуктов их экспрессии (например, ферментов или других белков). Библиотека или популяция обычно включает ряд различных молекул. Например, библиотека или популяция, как правило, включает по меньшей мере приблизительно 10 различных молекул. Большие библиотеки, как правило, включают по меньшей мере приблизительно 100 различных молекул, более типично по меньшей мере приблизительно 1000 различных молекул. Для некоторых применений библиотека включает по меньшей мере приблизительно 10000 или больше различных молекул. В определенных вариантах осуществления библиотека содержит ряд вариантных или химерных нуклеиновых кислот или белков, получаемых посредством процедуры направленной эволюции.
Две нуклеиновые кислоты являются "рекомбинированными", когда последовательности каждой из двух нуклеиновых кислот объединены в нуклеиновой кислоте-потомке. Две последовательности являются "непосредственно" рекомбинированными, когда обе нуклеиновые кислоты являются субстратами для рекомбинации.
"Выбор" относится к процессу, в котором одну или более биомолекул идентифицируют как имеющих одно или более свойств, представляющих интерес. Таким образом, например, можно осуществлять скрининг библиотеки для определения одного или более свойств одного или более членов библиотеки. Если один или более членов библиотеки идентифицированы как обладающие представляющим интерес свойством, их выбирают. Выбор может включать выделение члена библиотеки, но это не обязательно. Кроме того, выбор и скрининг могут быть и часто являются одновременными.
"Зависимая переменная" представляет выходные данные или результат или проверяется, не является ли она результатом. "Независимые переменные" представляют входные данные или причины или проверяются, не являются ли они причиной. Может быть исследовано, изменяется ли и насколько зависимая переменная, когда изменяются независимые переменные.
В простой стохастической линейной модели
yi=a+bxi+ei
где член yi представляет собой i-е значение зависимой переменной, и xi представляет собой i-е значение независимой переменной. Член ei известен как "ошибка" и содержит изменчивость зависимой переменной, которая не объясняется независимой переменной.
Независимая переменная также известна как "предикторная переменная", "регрессор", "управляемая переменная", "регулируемая переменная", "объясняющая переменная" или "входная переменная".
"Аддитивная модель" представляет собой модель, описывающую зависимость зависимой переменной y как функции множественных независимых переменных xi, причем данная модель объединяет вклады независимых переменных в зависимую переменную посредством сложения множественных членов, причем каждый член включает выражение независимой переменной. Выражение независимой переменной отражает вклад независимой переменной в зависимую переменную.
В некоторых вариантах осуществления используют математическую модель для описания зависимости между одной или более независимыми переменными (IV) и зависимой переменной (DV). Данная модель может быть записана как DV = алгебраическое выражение (IV). "Алгебраическое выражение" может включать переменные, коэффициенты, постоянные и символы операций, такие как знаки плюс и минус. 4x2+3xy+7y+5 представляет собой алгебраическое выражение с двумя переменными.
В аддитивной модели члены представляют собой элементы, разделенные знаками плюс или минус. Вышеприведенный пример принимает форму аддитивной модели. Он имеет четыре члена, 4x2, 3xy, 7y и 5. Члены могут состоять из переменных и коэффициентов (4x2, 3xy и 7y) или постоянных (5). В алгебраических выражениях переменные могут принимать различные значения, для того чтобы представлять изменяющиеся условия системы. Например, переменная может представлять собой непрерывную переменную, представляющую скорость движущегося автомобиля, или дискретную переменную с множественными не непрерывными значениями, представляющую типы аминокислот. Переменная может представлять собой переменную со значением бита, представляющую присутствие или отсутствие объекта, например присутствие или отсутствие остатка конкретного типа в конкретном положении. В алгебраическом выражении выше переменными являются x и y.
В некоторых вариантах осуществления "члены" выражения могут представлять собой элементы выражения, которые разделены другими знаками. Например, мультипликативная модель имеет члены, соединенные умножением, как подробнее объяснено ниже.
"Коэффициент" относится к скалярному значению, умноженному на зависимую переменную или выражение, содержащее зависимую переменную. В вышеприведенном примере, "коэффициенты" представляют собой числовую часть членов в алгебраическом выражении. В 4x2+3xy+7y+5 коэффициентом первого члена является 4. Коэффициентом второго члена является 3, а коэффициентом третьего члена является 7. Если член состоит только из переменных, его коэффициент равен 1.
"Постоянные" представляют собой члены в алгебраическом выражении, которые содержат только числа, то есть они представляют собой члены без переменных. В выражении 4x2+3xy+7y+5 постоянным членом является "5".
"Линейный член" представляет собой член со степенью, равной 1, или одну переменную, возведенную в степень, равную 1. В вышеприведенном примере член 7y представляет собой линейный член, поскольку его степень равна 1 (y1 или просто y). Напротив, член 4x2 представляет собой квадратичный член, поскольку x имеет степень, равную 2, и 3xy представляет собой квадратичный член с двумя переменными, поскольку x и y оба имеют степень, равную 1, что ведет к произведению степени 2.
Аддитивная модель может включать линейные и/или нелинейные члены. "Линейный член" относится к члену аддитивной модели, содержащему произведение одной независимой переменной и соответствующего коэффициента, причем величина модели изменяется линейно, когда изменяется независимая переменная. Термин "линейная модель" или "линейная аддитивная модель" относится к любой аддитивной модели, все члены которой представляют собой линейные члены. Она предполагает, что отсутствуют взаимодействия между независимыми переменными (то есть каждая независимая переменная вносит свой вклад независимо от общей приспособленности белка). Простая стохастическая линейная модель, описанная выше, является примером линейной аддитивной модели.
В контексте аддитивных моделей, если не указано иное, "нелинейный член", "член перекрестного произведения" и "член, характеризующий взаимодействие" используются взаимозаменяемо и относятся к члену модели, содержащему выражение, включающее произведение двух или более независимых переменных. В некоторых вариантах осуществления данное выражение может представлять собой просто постоянный коэффициент, умноженный на произведение: ci,j × xixj. В контексте аддитивных моделей термин "нелинейная модель" или "нелинейная аддитивная модель" относится к любой аддитивной модели, включающей по меньшей мере один нелинейный член в вышеуказанном значении. Однако в некоторых местах в тексте "нелинейный член" используется в более широком смысле, причем данный член содержит различные формы единственной независимой переменной, включая, но без ограничения, степенную функцию или экспоненциальную функцию независимой переменной.
"Мультипликативная модель" представляет собой модель, описывающую зависимость зависимой переменной y как функции множественных независимых переменных xi, причем данная модель объединяет вклады независимых переменных в зависимую переменную посредством умножения множественных членов, каждый из которых содержит выражение независимой переменной. Выражение независимой переменной отражает вклад независимой переменной в зависимую переменную.
Следует отметить, что "линейный член" и "линейная модель" не применимы к мультипликативной модели. Поэтому мультипликативная модель описывают только как учитывающую взаимодействие или не учитывающую взаимодействие, а не как линейную или нелинейную. Мультипликативная модель может включать члены, не характеризующие взаимодействие и/или характеризующие взаимодействие. "Член, не характеризующий взаимодействие" в мультипликативной модели относится к члену модели, содержащему выражение единственной независимой переменной.
Аналогично аддитивным моделям, "член, характеризующий взаимодействие" в мультипликативной модели относится к члену модели, содержащему выражение, включающее произведение двух или более независимых переменных. В некоторых вариантах осуществления данное выражение может представлять собой просто постоянный коэффициент, умноженный на произведение: ci,j × xixj. В других вариантах осуществления выражение может представлять собой (1+ci,j × xixj).
"Взаимодействующие субъединицы" относится к двум или более субъединицам последовательности, которые оказывают синергические воздействия на моделируемую активность последовательности, причем синергические воздействия отделяются и отличаются от отдельных воздействий субъединиц на моделируемую активность.
"Произведение" относится к результату умножения двух или более переменных или членов.
"Коэффициент" относится к скалярному значению, умноженному на зависимую переменную или выражение, содержащее зависимую переменную.
"Ортогональная/ортогональность" относится к независимой переменной, которая является некоррелированной с другими независимыми переменными в модели или другой зависимости.
Термин "модель последовательность-активность" относится к любым математическим моделям, которые описывают зависимость между активностями, характеристиками или свойствами биологических молекул с одной стороны и различными биологическими последовательностями с другой стороны.
Термин "кодированная цепочка символов" относится к представлению биологической молекулы, которое сохраняет информацию о последовательности/структуре в отношении данной молекулы. В некоторых вариантах осуществления кодированная цепочка символов содержит информацию о мутациях последовательности в библиотеке вариантов. Кодированные цепочки символов биомолекул наряду с информацией об активности для биомолекул могут быть использованы в качестве обучающего набора для модели последовательность-активность. Не относящиеся к последовательности свойства биомолекул могут быть сохранены или иным образом связаны с кодированными цепочками символов для биомолекул.
"Референсная последовательность" представляет собой последовательность, из которой осуществляют вариацию последовательности. В некоторых случаях "референсная последовательность" используется для определения вариаций. Такая последовательность может представлять собой последовательность, о которой модель предсказывает, что она имеет наивысшее значение (или одно из наивысших значений) желаемой активности. В другом случае референсная последовательность может представлять собой последовательность члена исходной библиотеки белковых вариантов. В определенных вариантах осуществления референсная последовательность представляет собой последовательность родительского белка или нуклеиновой кислоты.
"Обучающий набор" относится к набору данных или наблюдений о последовательности и активности, к которым подогнаны и на которых основаны одна или более моделей. Например, для модели последовательность-активность белка обучающий набор содержит последовательности остатков для исходной или улучшенной библиотеки белковых вариантов. Как правило, эти данные включают полную или частичную информацию о последовательности остатков вместе со значением активности для каждого белка в библиотеке. В некоторых случаях множественные типы активностей (например, данные о константе скорости и данные о термической стабильности) представлены в обучающем наборе вместе. Активность иногда представляет собой полезное свойство.
Термин "наблюдение" представляет собой информацию о белке или другом биологическом объекте, который может быть использован в обучающем наборе для генерации модели, такой как модель последовательность-активность. Термин "наблюдение" может относиться к любым секвенированным и проанализированным биологическим молекулам, включая варианты белков. В определенных вариантах осуществления каждое наблюдение представляет собой значение активности и соответствующую последовательность для варианта в библиотеке. Обычно, чем больше наблюдений используется для создания модели последовательность-активность, тем лучше предсказательная сила данной модели последовательность-активность.
Как используется в настоящем документе, термин "полезное свойство" предназначен для обозначения фенотипического или другого идентифицируемого признака, который придает некоторую выгоду белку или композиции веществ или процессу, связанному с данным белком. Примеры полезных свойств включают увеличение или уменьшение у вариантного белка по сравнению с родительским белком каталитических свойств, свойств связывания, стабильности при воздействии экстремальных температур, pH и так далее, чувствительности к стимулам, ингибирования и тому подобного. Другие полезные свойства могут включать изменение профиля при реакции на определенный стимул. Дополнительные примеры полезных свойств приведены ниже. Значения полезных свойств могут быть использованы в качестве значений активности в наблюдениях, используемых в обучающем наборе для модели последовательность-активность.
"Секвенирование нового поколения" или "высокопроизводительное секвенирование" представляют собой методы секвенирования, которые параллелизуют процесс секвенирования, производя тысячи или миллионы последовательностей за один раз. Примеры подходящих способов секвенирования нового поколения включают, но без ограничения, одномолекулярное секвенирование в реальном времени (например, Pacific Biosciences, Menlo Park, California), ионное полупроводниковое секвенирование (например, Ion Torrent, South San Francisco, California), пиросеквенирование (например, 454, Branford, Connecticut), секвенирование лигированием (например, секвенирование SOLid, Life Technologies, Carlsbad, California), секвенирование посредством синтеза и обратимого терминирования (например, Illumina, San Diego, California), технологии визуализации нуклеиновой кислоты, такие как просвечивающая электронная микроскопия, и тому подобное. Дополнительные описания примеров этих методов даны в подробном описании настоящего раскрытия.
"Предсказательная сила" относится к способности модели корректно предсказывать значения зависимой переменной для данных в различных условиях. Например, предсказательная сила модели последовательность-активность относится к способности модели предсказывать активность по информации о последовательности.
"Перекрестная проверка" относится к способу тестирования обобщаемости способности модели для предсказания представляющего интерес значения (то есть значения зависимой переменной). Способ получает модель с использованием одного набора данных и тестирует ошибку модели с использованием другого набора данных. Первый набор данных рассматривают как обучающий набор, а второй набор данных представляет собой контрольный набор.
"Систематическая дисперсия" относится к различным дескрипторам элемента или набора элементов, изменяемым в различных комбинациях.
"Систематически варьируемые данные" относится к данным, получаемым, происходящим или следующим из различных дескрипторов элемента или набора элементов, изменяемых в различных комбинациях. Множество различных дескрипторов может быть изменено в одно и то же время, но в различных комбинациях. Например, данные об активности, собранные от полипептидов, в которых изменялись комбинации аминокислот, представляют собой систематически варьируемые данные.
Термин "систематически варьируемые последовательности" относится к набору последовательностей, в которых каждый остаток рассматривают во множественных контекстах. В принципе, уровень систематической вариации может быть количественно охарактеризован с помощью степени, в которой последовательности ортогональны друг другу (то есть максимально отличаются по сравнению со средним).
Термин "переключение" относится к введению множественных типов аминокислотных остатков в конкретное положение в последовательностях вариантов белков в оптимизированной библиотеке.
Термины "регрессия" и "регрессионный анализ" относятся к методам, используемым для того, чтобы понять, какие из независимых переменных связаны с зависимой переменной, и для того, чтобы исследовать формы данных зависимостей. В ограниченных условиях регрессионный анализ можно использовать для получения вывода о причинно-следственных зависимостях между независимыми и зависимыми переменными. Он представляет собой статистический метод определения зависимостей между переменными. Он включает множество методов моделирования и анализа множественных переменных при фокусировании на зависимости между зависимой переменной и одной или более независимыми переменными. Более конкретно, регрессионный анализ помогает понять, как типичное значение зависимой переменной изменяется, когда варьируется любая из независимых переменных, в то время как другие независимые переменные остаются фиксированными. Регрессионные методы можно использовать для генерации моделей последовательность-активность из обучающих наборов, содержащих множественные наблюдения, которые могут содержать информацию о последовательности и активности.
Частные наименьшие квадраты или PLS представляет собой семейство способов, которое находит линейную регрессионную модель посредством проецирования предсказанных переменных (например, значений активности) и наблюдаемых переменных (например, последовательностей) на новое пространство. PLS также известен как проекция на латентные структуры. Данные как X (независимые переменные), так и Y (зависимые переменные) проецируют на новые пространства. PLS используют для поиска фундаментальных связей между двумя матрицами (X и Y). Подход латентных переменных используют для моделирования ковариационных структур в пространствах X и Y. Модель PLS пытается найти многомерное направление в пространстве X, которое раскрывает направление максимальной многомерной дисперсии в пространстве Y. Регрессия PLS является особенно подходящей, когда матрица предикторов имеет больше переменных, чем наблюдения, и когда имеет место мультиколлинеарность среди значений X.
"Дескриптор" относится к чему-либо, что служит для описания или идентификации элемента. Например, знаки в цепочке символов могут быть дескрипторами аминокислот в полипептиде, представляемом данной цепочкой символов.
В регрессионной модели зависимая переменная связана с независимыми переменными посредством суммы членов. Каждый член включает произведение независимой переменной и соответствующего коэффициента регрессии. В случае чисто линейной регрессионной модели коэффициенты регрессии задаются посредством β в выражении следующей формы:
yi = β1xi1 +… + βpxip + εi = xiTβ + εi
где yi представляет собой зависимую переменную, xi представляют собой независимые переменные, εi представляет собой переменную ошибки, и T обозначает транспонирование, то есть скалярное произведение векторов xi и β.
"Регрессия на главные компоненты" (PCR) относится к регрессионному анализу, который использует анализ главных компонент при определении коэффициентов регрессии. При PCR вместо регрессии зависимой переменной непосредственно на независимые переменные используют главные компоненты независимых переменных. PCR, как правило, использует в регрессии только поднабор главных компонент.
"Анализ главных компонент" (PCA) относится к математической процедуре, которая использует ортогональное преобразование для превращения набора наблюдений возможно коррелированных переменных в набор значений линейно некоррелированных переменных, называемых главными компонентами. Число главных компонент меньше или равно числу исходных переменных. Данное преобразование определяют таким образом, чтобы первая главная компонента имела наибольшую возможную дисперсию (то есть учитывал настолько большую изменчивость в данных, насколько возможно), а каждая следующая компонента в свою очередь имела наивысшую возможную дисперсию при том ограничении, чтобы она была ортогональна предшествующим компонентам (то есть некоррелированна с ними).
"Нейронная сеть" представляет собой модель, содержащую взаимосвязанную группу элементов обработки или "нейронов", которые обрабатывают информацию, используя коннекционистский подход к вычислениям. Нейронные сети используют для моделирования сложных зависимостей между входными и выходными данными или для поиска шаблонов в данных. Большинство нейронных сетей обрабатывают данные нелинейным распределенным параллельным образом. В большинстве случаев нейронная сеть представляет собой адаптивную систему, которая меняет свою структуру во время фазы обучения. Функции скорее осуществляются коллективно и параллельно посредством обрабатывающих элементов, чем имеет место четкое разграничение подзадач, на которые назначаются различные узлы.
Обычно нейронная сеть включает сеть простых обрабатывающих элементов, которые демонстрируют сложное общее поведение, определяемое соединениями между обрабатывающими элементами и параметрами элементов. Нейронные сети используют с алгоритмами, разработанными для изменения силы соединений в сети для получения желаемого потока сигналов. Данная сила изменяется во время тренировки или обучения.
"Случайный лес" относится к такой комбинации предикторов классификационных деревьев, что каждое дерево зависит от значений случайного вектора, выбранных независимо и с одним и тем же распределением для всех деревьев в лесу. Случайный лес представляет собой обучающий ансамбль, состоящий из бэггинга неусеченных обучающих деревьев принятия решений с рандомизированным выбором признаков при каждом разветвлении дерева принятия решений. Случайный лес выращивает большое количество классификационных деревьев, каждое из которых голосует за наиболее популярный класс. Затем случайный лес классифицирует переменную посредством принятия определенного голосованием наиболее популярного класса из всех предикторов деревьев в лесу.
"Априорное распределение вероятностей" или "априорная вероятность" неопределенной величины p представляет собой распределение вероятностей, которое выражает неопределенность в p до того, как были учтены представляющие интерес данные (например, обучающий набор белковых последовательностей). Неизвестная величина может представлять собой параметр, коэффициент, переменную, латентную переменную или тому подобное (например, коэффициент в модели множественной регрессии).
"Апостериорное распределение вероятностей" или "апостериорная вероятность" неопределенной величины p представляет собой распределение вероятностей, которое выражает неопределенность в p после того, как были учтены представляющие интерес данные.
Термин "байесовская линейная регрессия" относится к подходу к линейной регрессии, в котором статистический анализ осуществляется в контексте байесовского вывода. Априорное предположение в отношении линейной регрессионной модели, включающее функцию априорного распределения вероятностей параметра модели, объединяют с функцией правдоподобия данных в соответствии с теоремой Байеса для получения апостериорного распределения вероятностей параметров.
"Переобучение" относится к состоянию, которое имеет место, когда статистическая модель описывает случайную ошибку или шум вместо основной зависимости. Переобучение обычно происходит, когда модель излишне сложна, как, например, имеет слишком много параметров относительно количества наблюдений. Модель, которая подверглась переобучению, обычно имеет низкую предсказательную эффективность, поскольку она может завышать слабые флуктуации в данных.
Термин "основная модель" используют в отношении модели последовательность-активность, предлагаемой в начале процесса улучшения модели.
Термин "обновленная модель" используют в отношении модели последовательность-активность, которая получена прямо или косвенно из основной модели, которая имеет улучшенную предсказательную силу по сравнению с основной моделью и/или другой моделью, из которой она была получена.
"Функция правдоподобия" или "правдоподобие" модели представляет собой функцию параметров статистической модели. Правдоподобие набора значений параметров при некоторых заданных наблюдаемых результатах равно вероятности этих наблюдаемых результатов при этих заданных значениях параметров, то есть L(θ|x)=P(x|θ).
"Моделирование по методу Монте-Карло" представляет собой моделирование, которое основано на большом количестве случайных выборок для получения числовых результатов, которые моделируют реальное явление. Например, взятие большого количества псевдослучайных равномерно распределенных переменных из интервала (01] и сопоставление значений, меньших или равных 0,50, орлам, а больших 0,50 решкам представляет собой моделирование по методу Монте-Карло поведения многократно подбрасываемой монеты.
"Алгоритм Метрополиса" или "алгоритм Метрополиса-Гастингса" представляет собой способ Монте-Карло с цепями Маркова (MCMC) для получения последовательности случайных выборок из распределения вероятностей, для которого непосредственная выборка затруднена. Данная последовательность выборок может быть использована для аппроксимации распределения (то есть для генерации гистограммы) или для вычисления интеграла (такого как ожидаемое значение). Алгоритм Метрополиса-Гастингса и другие алгоритмы MCMC обычно используют для семплирования из многомерного распределения, особенно при большом числе измерений. Целью алгоритма Метрополиса-Гастингса является асимптотическая генерация состояний x в соответствии с желаемым распределением P(x), и для ее достижения он использует стохастический процесс. Идея алгоритма заключается в подборе таких условий стохастического процесса, что он асимптотически сходится к уникальному распределению P(x).
"Цепь Маркова" представляет собой последовательность случайных переменных X1, X2, X3… с марковским свойством. Другими словами, если задано состояние настоящего, состояния будущего и прошлого независимы. Формально
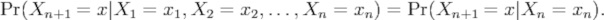
Возможные значения Xi образуют счетный набор S, называемый пространством состояний цепи. Система "цепей Маркова" представляет собой математическую систему, которая подвергается переходам из одного состояния в другое между конечным или счетным числом возможных состояний. Это является случайным процессом, обычно характеризуемым как отсутствие памяти: следующее состояние зависит только от текущего состояния, а не от последовательности событий, которые ему предшествуют.
"Информационный критерий Акаике" (AIC) представляет собой меру относительного качества подгонки статистической модели, и его часто используют в качестве критерия выбора моделей среди конечного набора моделей. AIC основывается на концепции информационной энтропии, по сути предлагая относительную меру информации, теряемой, когда заданную модель используют для описания реальности. Можно сказать, для описания компромисса между смещением и дисперсией при построении модели или, грубо говоря, между точностью и сложностью модели. AIC может быть вычислен как: AIC=-2logeL+2k, где L представляет собой максимальное правдоподобие функции, а k представляет собой количество свободных параметров модели, подлежащих определению.
"Байесовский информационный критерий" представляет собой критерий выбора моделей среди конечного набора моделей и тесно связан с AIC. BIC может быть вычислен как: BIC= -2logeL+kloge(n), где n представляет собой число наблюдений данных. Когда число наблюдений растет, BIC часто штрафует излишнее число свободных параметров более строго, чем AIC.
"Генетический алгоритм" представляет собой процесс, который имитирует эволюционные процессы. Генетические алгоритмы (GA) используются в самых различных областях для решения проблем, которые не в полной мере охарактеризованы или слишком сложны, чтобы их можно было полностью охарактеризовать, но для которых доступна определенная аналитическая оценка, то есть GA используется для решения проблем, которые могут быть оценены с помощью некоторой поддающейся количественному определению меры относительного значения решения (или по меньшей мере относительного значения одного потенциального решения по сравнению с другим). В контексте настоящего раскрытия генетический алгоритм представляет собой процесс выбора цепочек символов или манипулирования ими на компьютере, как правило, когда цепочка символов соответствует одной или более биологическим молекулам (например, нуклеиновым кислотам, белкам или тому подобным).
Термин "генетическая операция" (или "GO") относится к биологическим и/или вычислительным генетическим операциям, причем все изменения в любой популяции любого типа цепочек символов (и, следовательно, в любых физических свойствах физических объектов, кодируемых такими цепочками) могут быть описаны как результат случайного и/или предварительно определенного применения конечного набора логических алгебраических функций. Примеры GO включают, но без ограничения, умножение, кроссинговер, рекомбинацию, мутацию, лигирование, фрагментацию и так далее.
"Ансамблевая модель" представляет собой модель, члены которой включают все члены группы моделей, причем коэффициенты членов ансамблевой модели основаны на взвешенных коэффициентах соответствующих членов отдельных моделей в группе. Взвешивание коэффициентов основано на предсказательной силе и/или приспособленности отдельных моделей.
II. Обзор процесса поиска улучшенных вариантов белков
В подходе управляемой эволюции к исследованию белковых последовательностей используют модели последовательность-активность для управления генерацией вариантов белков. Один аспект настоящего раскрытия предлагает различные способы получения моделей последовательность-активность, которые основаны на библиотеках белков и могут быть использованы для поиска новых и улучшенных библиотек белков. Настоящий раздел, во-первых, предлагает обзор процесса поиска новых и улучшенных белков, и затем предлагает дополнительные подробности в отношении вопросов, связанных с выбором исходной библиотеки, построением модели последовательность-активность и использованием модели для управления исследованием новых белков.
Настоящее раскрытие предлагает иллюстративные примеры с использованием последовательностей аминокислотных остатков и активности белков, но следует понимать, что подход, описанный в настоящем документе, может быть также реализован для других биологических последовательностей и видов активности. Например, в различных вариантах осуществления последовательность может представлять собой весь геном, целую хромосому, сегмент хромосомы, коллекцию последовательностей генов взаимодействующих генов, ген, нуклеиновокислотную последовательность, белок, полисахарид и так далее. В одном или более вариантах осуществления субъединицы последовательностей могут представлять собой хромосомы, сегменты хромосом, гаплотипы, гены, нуклеотиды, кодоны, мутации, аминокислоты, моно-, ди-, три- или олигомерные углеводы и так далее.
Как правило, в начале определенного цикла направленной эволюции последовательностей получают обучающий набор секвенированных и проанализированных вариантов белков. Данный цикл направленной эволюции производит ряд вариантных белков, которые отличаются по одной или более мутациям от родительского пептида или пептидов, использованных в начале цикла направленной эволюции. Вариантные пептиды, полученные во время цикла направленной эволюции, анализируют на активность. Те пептиды, которые имеют желаемую активность и/или улучшенную активность по сравнению с родительским пептидом(ами), выбирают для использования в по меньшей мере одном последующем цикле направленной эволюции.
Секвенированные и проанализированные варианты белков можно также использовать для получения модели последовательность-активность. Как правило, их используют в модели последовательность-активность, если они фактически секвенированы. Каждый из секвенированных и проанализированных вариантов белков называется "наблюдением". Обычно, чем больше наблюдений используют для создания модели последовательность-активность, тем лучше предсказательная сила данной модели последовательность-активность.
До появления технологий массового параллельного секвенирования нового поколения было трудно экономно секвенировать больше, чем 10-30 вариантных пептидов, получаемых в любом цикле направленной эволюции. В настоящее время при применении секвенирования нового поколения можно секвенировать гораздо больше вариантных белков, получаемых в цикле направленной эволюции. Следовательно, для получения моделей последовательность-активность можно использовать гораздо большую совокупность данных обучающего набора. Модели последовательность-активность можно в настоящее время генерировать с использованием обучающего набора, который включает не только пептиды с наивысшей эффективностью из цикла, но также некоторые пептиды, который могут не представлять интерес для последующих циклов направленной эволюции, но информация о последовательности и активности которых может быть применена для получения более робастной модели последовательность-активность.
В некоторых вариантах осуществления обычно является желательным получать модели последовательность-активность, обладающие хорошей способностью предсказывать активность произвольной последовательности. Предсказательная сила может быть охарактеризована точностью предсказания, а также постоянством, с которым модель точно предсказывает активность. Кроме того, модель может быть охарактеризована по своей способности точно предсказывать активность в широком диапазоне пространства последовательностей. Например, предсказательная сила может быть охарактеризована посредством остатков между вычисленными и фактическими значениями активности для заданного тестового и/или контрольного набора пептидов. Модель с более высокой обобщенной предсказательной силой имеет тенденцию к тому, чтобы давать меньшие и более постоянные остатки в различных наборах контрольных данных. Модель, которая переобучена на тестовом наборе данных, имеет тенденцию к тому, чтобы давать большие и менее постоянные остатки для контрольных данных, как показано с помощью примера ниже. Один аспект настоящего раскрытия предлагает способ эффективного поиска модели с высокой предсказательной силой по различным наборам данных.
Модели последовательность-активность, как описано в настоящем документе, можно использовать для помощи в идентифицировании одного или более родительских "генов" в исходной библиотеке вариантов, которые должны быть подвергнуты направленной эволюции. После осуществления цикла эволюции определяют новую библиотеку вариантов, предоставляя новый набор наблюдений, который затем может быть возвращен в качестве данных для получения новой или уточненной модели последовательность-активность. Данный процесс чередования получения модели последовательность-активность на основании новых наблюдений и проведения направленной эволюции на основании модели последовательность-активность может образовывать итерационный цикл моделирования-исследования, который можно повторять до тех пор, пока не будут получены желаемые белки и библиотеки.
Благодаря циклу обратной связи между моделями последовательность-активность и библиотеками вариантов в исследовании белков с улучшенной активностью лучшие модели и лучшие библиотеки вариантов зависят друг от друга. Поэтому узкие места и улучшения в областях моделирования и/или секвенирования могут влиять на обе области. В некоторых вариантах осуществления настоящего изобретения улучшения эффективности моделирования благодаря лучшим методам моделирования обеспечивает лучшие модели для управления исследованием последовательности. В некоторых вариантах осуществления технологии секвенирования нового поколения используются для улучшения скорости секвенирования in vitro, а также для предоставления данных перекрестной проверки для улучшения вычислительных моделей in silico.
В некоторых вариантах осуществления настоящего изобретения полезные модели последовательность-активность требуют робастных методов математического моделирования и большого количества "наблюдений". Эти наблюдения представляют собой данные, предоставляемые в обучающем наборе для модели. А именно, каждое наблюдение представляет собой значение активности и соответствующую последовательность для варианта в библиотеке. Исторически, секвенирование представляло собой лимитирующий этап в разработке больших обучающих наборов и, соответственно, все более робастных моделей последовательность-активность. В способах, обычно используемых в настоящее время, генерируются библиотеки вариантов, содержащие, возможно, сотни вариантов. Однако только малая часть этих вариантов фактически секвенирована. В типичном цикле направленной эволюции только приблизительно 10-30 вариантов с наивысшей активностью фактически секвенированы. В идеальном случае секвенирована гораздо большая доля вариантов в библиотеке, включая некоторые варианты с относительно низкой активностью. Инструменты для секвенирования нового поколения обладают значительно улучшенной скоростью секвенирования, что делает возможным включение в обучающий набор вариантов с низкой активностью и высокой активностью. В некоторых вариантах осуществления включение вариантов, обладающих диапазоном уровней активности, приводит к получению моделей, которые лучше работают и/или имеют лучшую предсказательную активность в более широком диапазоне пространства последовательности и активности.
Некоторые не учитывающие взаимодействие модели последовательность-активность, упомянутые в настоящем документе, включают отдельные остатки в качестве независимых переменных для предсказания любой представляющей интерес активности. Не учитывающие взаимодействие модели последовательность-активность не включают члены для учета взаимодействий между двумя или более остатками. Если взаимодействие между двумя из остатков оказывает синергическое воздействие на активность, не учитывающая взаимодействие, или линейная модель может предоставлять искусственно завышенное значение коэффициентов, связанных с двумя взаимодействующими остатками. В результате тот, кто работает с моделью, может ошибочно заключить, что просто благодаря осуществлению замены остатка, предлагаемой относительно высоким значением коэффициента, активность полученного пептида станет выше, чем ожидалось. Это объясняется тем, что исследователь не понимает из использования не учитывающей взаимодействие или линейной модели, что повышенная активность, связанная с заменой остатка, главным образом является результатом взаимодействия данной замены с другой заменой. Если исследователь поймет важность данного взаимодействия, то он или она сможет произвести обе замены одновременно и получить повышение активности, предполагаемое учитывающей взаимодействие моделью.
Если два остатка взаимодействуют так, что активность подавляется нелинейным образом, учитывающая взаимодействие модель приписывает коэффициентам, соответствующим данным остаткам, более низкие значения, чем те, которые соответствуют рассмотрению остатков как полностью изолированных друг от друга. Другими словами, осуществление одной из замен взаимодействующих остатков, но не другой приведет к тому, что активность будет больше, чем предполагалось не учитывающей взаимодействие или линейной моделью.
Поскольку не учитывающая взаимодействие модель может быть неадекватной, когда взаимодействия остаток-остаток оказывают сильное воздействие на активность, для точного предсказания активности часто необходимы учитывающие взаимодействие модели с членами, характеризующими взаимодействие, учитывающими взаимодействия между остатками. Однако, модели, которые используют члены, характеризующие взаимодействие, представляют вычислительные и эмпирические проблемы. Прежде всего, при разработке/использовании модели следует принимать во внимание очень много потенциальных членов, характеризующих взаимодействие, что требует значительного объема вычислений. Гораздо более существенное ограничение заключается в потенциальном числе наблюдений, необходимых для получения модели со значительным количеством членов, характеризующих взаимодействие остаток-остаток. Кроме того, может иметь место тенденция метода создания модели к переобучению данных при заданном конкретном числе доступных наблюдений. Для решения данной проблемы важным фактором в разработке многих моделей является осторожный выбор и ограничение членов, характеризующих взаимодействие, предусмотренных в модели последовательность-активность.
Фигура 1 представляет блок-схему, показывающую одно воплощение способа получения модели последовательность-активность. Как изображено, способ 100 начинается в блоке 103 с предоставления данных о последовательности и активности для вариантных генов или белков ("наблюдения"). Данные о последовательности могут быть взяты из, например, обучающего набора, содержащего последовательности остатков для исходной или улучшенной библиотеки белковых вариантов. Как правило, эти данные включают полную или частичную информацию о последовательности остатков вместе со значением активности для каждого белка в библиотеке. В некоторых случаях множественные типы активности (например, данные о константе скорости и данные о термической стабильности) предлагаются в обучающем наборе вместе. Могут также быть рассмотрены другие источники данных, что определяется желаемыми результатами. Некоторые подходящие источники данных включают, но без ограничения, литературные ссылки, которые описывают информацию о конкретных пептидах, имеющих отношение к разрабатываемой модели последовательность-активность. Дополнительные источники информации включают, но без ограничения, предшествующие или другие циклы направленной эволюции в том же проекте. Более того, предполагается, что информация, получаемая из предыдущих циклов направленной эволюции (с помощью любого подходящего способа, включая, но без ограничения, предлагаемые в настоящем документе), найдет применение в разработке получаемых позже библиотек, вариантов и так далее.
Во многих вариантах осуществления отдельные члены библиотеки белковых вариантов представляют широкий диапазон последовательностей и активности. Это облегчает генерацию модели последовательность-активность, которая применима в широкой области пространства последовательностей. Методы генерации таких различных библиотек включают, но без ограничения, систематическую вариацию белковых последовательностей и методы направленной эволюции, как описано в настоящем документе. Однако в некоторых альтернативных вариантах осуществления желательно генерировать модели из последовательностей генов конкретного семейства генов (например, конкретной киназы, обнаруживаемой во множественных видах или организмах). Поскольку многие остатки будут идентичны во всех членах семейств, модель описывает только те остатки, которые варьируются. Таким образом, в некоторых вариантах осуществления статистические модели на основании таких относительно небольших по сравнению с набором всех возможных вариантов обучающих наборов являются применимыми в локальном смысле. А именно, модели являются применимыми только для данных наблюдений данных вариантов. В некоторых вариантах осуществления целью не является обнаружение глобальной функции приспособленности, как понимается в некоторых моделях, это выходит за пределы возможностей и/или потребностей рассматриваемой модельной системы (систем).
Данные об активности могут быть получены с помощью любого подходящего средства, известного в данной области техники, включая, но без ограничения, способы анализа и/или скрининга, выполненные таким образом, чтобы измерять величины вида/видов активности, представляющих интерес. Такие методы хорошо известны и не являются неотъемлемой частью настоящего изобретения. Принципы разработки соответствующих способов анализа или скрининга широко известны и понятны в данной области техники. Методы получения белковых последовательностей также хорошо известны и не являются ключевыми для настоящего изобретения. Как упомянуто, могут быть использованы технологии секвенирования нового поколения. В некоторых вариантах осуществления представляющая интерес активность может представлять собой стабильность белка (например, термическую стабильность). Однако многие важные варианты осуществления рассматривают другие активности, такие как каталитическая активность, устойчивость к патогенам и/или токсинам, терапевтическая активность, токсичность и тому подобное. Более того, не предполагается, что настоящее изобретение ограничено каким-либо конкретным способом(ами) анализа/скрининг и/или способом(ами) секвенирования, поскольку любой подходящий способ, известный в данной области техники, находит применение в настоящем изобретении.
После того, как обучающий набор данных быт сгенерирован или получен, способ использует его для генерации основной модели последовательность-активность, которая предсказывает активность как функцию информации о последовательности. См. блок 105. Данная модель представляет собой выражение, алгоритм или другой инструмент, который предсказывает относительную активность конкретного белка, когда получает информацию о последовательности для данного белка. Другими словами, информация о белковой последовательности представляет собой вход, а предсказание активности представляет собой выход.
В некоторых вариантах осуществления каждая из моделей включает произведение множественных мультипликативных членов, причем каждый из мультипликативных членов отражает вклад в активность определенного остатка конкретного типа в конкретном местоположении последовательности. В других вариантах осуществления каждая модель включает сумму множественных членов, причем каждый из мультипликативных членов отражает вклад в активность определенного остатка конкретного типа в конкретном местоположении последовательности.
В некоторых вариантах осуществления основная модель не включает никаких членов, характеризующих взаимодействие. В таких случаях основная модель может быть описана как линейная или не учитывающая взаимодействие модель. В других вариантах осуществления основная модель включает все доступные члены, характеризующие взаимодействие, в каком случае основная модель может быть описана как нелинейная или учитывающая взаимодействие модель.
Для многих вариантов осуществления основная модель может ранжировать вклад различных остатков в активность. Способы генерации таких моделей, все из которых попадают в категорию машинного обучения (например, регрессия частных наименьших квадратов (PLS), регрессия на главные компоненты (PCR) и множественная линейная регрессия (MLR), байесовская линейная регрессия) рассмотрены ниже наряду с форматом независимых переменных (информация о последовательности), форматом зависимой переменной(ых) (активность) и самой формой модели (например, линейное выражение первого порядка, или произведение множественных членов, или гибрид как мультипликативной, так и аддитивной комбинации членов).
После генерации основной модели последовательность-активность способ уточняет модель посредством корректировки значений коэффициентов в членах моделей для минимизации остаточной ошибки между предсказанием модели и наблюдаемыми данными. См. блок 107. Такой тип коррекции также называется подгонкой модели. Могут быть использованы различные способы подгонки моделей, известные в данной области техники. Например, для того чтобы корректировать значения коэффициентов, можно использовать генетический алгоритм. Для аддитивных моделей можно использовать для подгонки модели различные методы регрессии.
В некоторых вариантах осуществления настоящего изобретения способ также уточняет модель посредством выбора подходящих членов для включения в модель или исключения из нее таким образом, чтобы минимизировать остаточные ошибки и/или улучшить предсказательную силу модели. См. блок 107. Поскольку все члены рассматриваемых моделей выбраны из одной и той же совокупности членов, данный процесс уточнения также известен как выбор модели среди вложенных моделей. Некоторые варианты осуществления настоящего изобретения используют для выбора подходящих членов генетический алгоритм. Дополнительно или альтернативно, некоторые варианты осуществления настоящего изобретения итерационно добавляют или удаляют члены, характеризующие взаимодействие, из совокупности доступных членов, характеризующих взаимодействие, к или из основной модели и оценивают получаемые новые модели в отношении улучшения по сравнению с основной моделью для получения итоговой модели. Когда основная модель включает все доступные члены, характеризующие взаимодействие, способ удаляет такие члены пошаговым образом. Когда основная модель не включает членов, характеризующие взаимодействие, способ добавляет такие члены пошаговым образом. Как корректировка значений коэффициентов модели, так и выбор членов модели для улучшения предсказательной силы моделей известны как методы оптимизации моделей.
В процессе выбора модели некоторые варианты осуществления настоящего изобретения предлагают способы, которые принимают во внимание не только дисперсию, которую учитывает модель при заданном наборе данных, но также способность модели к предсказанию новых данных. В некоторых вариантах осуществления данный подход к выбору модели штрафует модели, имеющие больше коэффициентов/параметров, чем эквивалентные модели, имеющие меньше коэффициентов/параметров, для избежания переобучения модели на заданном наборе данных. Примеры способов выбора включают, но без ограничения, информационный критерий Акаике (AIC) и байесовский информационный критерий (BIC) и их вариации.
В ряду вложенных моделей, как в регрессионных моделях с прогрессивно большим количеством членов, характеризующих взаимодействие (и соответствующих коэффициентов), чем в основной модели, более сложные модели обеспечивают столь же хорошие или лучшие подгонки, чем более простые, даже если дополнительные коэффициенты являются ложными, поскольку более сложная модель имеет дополнительные степени свободы. Определенные варианты осуществления настоящего раскрытия используют способы выбора модели, которые штрафуют более сложные модели таким образом, чтобы выигрыш в качестве подгонки был больше, чем смещение из-за стоимости ложных параметров.
Примеры алгоритмов для генерации моделей последовательность-активность в соответствии с операциями в блоках 105 и 107 представлены ниже. Такие методы включают, но без ограничения, генетический алгоритм и пошаговые методы со смещением от включения дополнительных членов, характеризующих взаимодействие, в модель. Однако не предполагается, что настоящее раскрытие ограничено данными конкретными примерами.
В одном аспекте настоящее раскрытие предлагает способы проведения направленной эволюции, причем данный способ включает: (a) получение данных о последовательности и активности для каждого из множества вариантов белков; (b) генерацию модели последовательность-активность по данным о последовательности и активности для каждого из множества вариантов белков, причем модель последовательность-активность содержит: (1) произведение множественных членов, причем каждый из по меньшей мере некоторых из этих членов содержит коэффициент, представляющий вклад в активность определенной аминокислоты или нуклеотида в определенном положении в белковой или нуклеиновокислотной последовательности, и (2) зависимую переменную, представляющую активность вариантов белков; и (c) использование модели для управления циклом направленной эволюции.
В некоторых вариантах осуществления использование модели для управления циклом направленной эволюции включает выбор одной или более мутаций для цикла направленной эволюции посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность. Данные способы также включают получение множества олигонуклеотидов, содержащих или кодирующих одну или более мутаций, выбранных выше; и осуществление цикла направленной эволюции с использованием олигонуклеотидов, полученных выше.
В других вариантах осуществления способы также включают идентифицирование новой белковой или новой нуклеиновокислотной последовательности, содержащей одну или более мутаций, выбранных выше, и получение и анализирование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью. В некоторых вариантах осуществления данный способ дополнительно включает использование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью, в качестве исходной точки для дальнейшей направленной эволюции.
В альтернативных вариантах осуществления использование модели для управления циклом направленной эволюции включает идентифицирование полной белковой последовательности на основании предсказания моделью активности всей последовательности, а не вклада отдельной определенной субъединицы в активность последовательности. Данные способы включают использование множественных белковых последовательностей или множественных аминокислотных последовательностей в модели последовательность-активность и определение значений активности, предсказанных моделью последовательность-активность для каждой из множественных белковых последовательностей или нуклеиновокислотных последовательностей. Данные способы также включают выбор новой белковой последовательности или новой нуклеиновокислотной последовательности из множественных белковых последовательностей или множественных аминокислотных последовательностей, использованных выше, посредством оценивания значений активности, предсказанных моделью последовательность-активность для данных множественных последовательностей. Данные способы также включают получение и анализирование белка, имеющего новую белковую последовательность, или белка, кодируемого новой нуклеиновокислотной последовательностью.
В других вариантах осуществления способы применяют методы насыщающего мутагенеза. Данные способы включают выбор одного или более положений в белковой последовательности или нуклеиновокислотных последовательностях посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов конкретных типов в конкретных положениях в последовательности, которые вносят вклад в активность. Данные способы затем осуществляют насыщающий мутагенез в одном или более идентифицированных положениях.
В другом аспекте настоящее раскрытие предлагает способы получения модели последовательность-активность, которая может помогать при идентифицировании биологических молекул для воздействия на желаемую активность. В некоторых вариантах осуществления данный способ включает: (a) получение данных о последовательности и активности для множества биологических молекул; (b) получение основной модели по данным о последовательности и активности, причем основная модель предсказывает активность как функцию присутствия или отсутствия субъединиц последовательности; (c) получение по меньшей мере одной новой модели посредством добавления/удаления по меньшей мере одного нового члена, характеризующего взаимодействие, к/из основной модели, причем новый член, характеризующий взаимодействие, представляет взаимодействие между двумя или более взаимодействующими субъединицами; (d) определение способности по меньшей мере одной новой модели предсказывать активность как функцию присутствия или отсутствия субъединиц; и (e) определение того, добавлять/удалять ли новый член, характеризующий взаимодействие, к/из основной модели, на основании способности по меньшей мере одной новой модели предсказывать активность, определенную в (d), и со смещением в сторону от добавления нового члена, характеризующего взаимодействие. Затем полученную модель можно использовать в различных применениях, как, например, в направленной эволюции библиотек белков, для того чтобы идентифицировать белки с желаемой биологической активностью и свойствами.
В некоторых вариантах осуществления, в которых способ определяет, что к основной модели для получения обновленной модели должен быть добавлен новый член, характеризующий взаимодействие, способ также включает: (f) повторение (c) с использованием обновленной модели вместо основной модели и добавление/удаление члена, характеризующего взаимодействие, отличного от добавленного/удаленного в (c); и (g) повторение (d) и (e) с использованием обновленной модели вместо основной модели. В некоторых вариантах осуществления данный способ дополнительно включает: (h) повторение (f) и (g) с использованием дополнительно обновленной модели.
После выбора наблюдений для обучающего набора и выбора математического метода для получения модели последовательность-активность создается основная модель. Основная модель, как правило, генерируется без учета ее предсказательной способности. Ее просто получают в соответствии с определенной процедурой получения основной модели из доступных наблюдений (то есть набора наблюдений), как описано в настоящем документе. Как указано выше, модели последовательностей могут описывать различные последовательности, при этом в некоторых вариантах осуществления модели описывают белки. В последнем случае основная модель представляет собой просто не учитывающую взаимодействие/линейную модель с одним членом для каждой из мутаций, присутствующей в коллекции пептидов, используемой для создания обучающего набора. В данных вариантах осуществления основная модель не включает никаких членов, представляющих взаимодействия между остатками в пептидах. В некоторых вариантах осуществления основная модель не включает отдельного члена для всех без исключения мутаций, присутствующих в наборе наблюдений.
В альтернативных подходах основная модель включает не только члены, описывающие каждую мутацию в изоляции, но дополнительно включает члены для всех потенциально взаимодействующих остатков. В крайнем случае в основной модели используют все возможные взаимодействия между известными мутациями. В нее включают член для всех без исключения парных взаимодействий между мутациями, а также члены для всех без исключения возможных взаимодействий трех остатков, а также все возможные взаимодействия четырех остатков и так далее. Некоторые варианты осуществления включают только парные взаимодействия или парные взаимодействия и тройные взаимодействия. Тройные взаимодействия представляют собой воздействующее на активность взаимодействие между тремя отличающимися субъединицами.
В одном или более вариантах осуществления настоящего раскрытия, которые в качестве основной модели используют простую не учитывающую взаимодействие или линейную модель, последующие действия по улучшению модели включают добавление новых членов, представляющих отличающиеся взаимодействия. В альтернативных вариантах осуществления, в которых основная модель включает все члены, не характеризующие и характеризующие взаимодействие, последующие действия по улучшению модели включают селективное удаление некоторых из членов, характеризующих взаимодействие.
В одном или более вариантах осуществления настоящего изобретения процесс улучшения основной модели включает итерационное добавление или удаление членов, характеризующих взаимодействие, из основной модели с определением того, достаточно ли улучшает получаемая модель качество модели. При каждой итерации предсказательную силу текущей модели определяют и сравнивают с другой моделью, например с основной моделью или с обновленной моделью.
В вариантах осуществления, в которых мера предсказательной силы уже учитывает способность модели к обобщению на другие наборы данных, сама эта мера может определять, следует ли выбрать модель-кандидат. Например, мера, такая как AIC или BIC, учитывает как правдоподобие (или остаточную ошибку) модели, так и число параметров. "Функция правдоподобия" или "правдоподобие" модели представляет собой функцию параметров статистической модели. Правдоподобие набора значений параметров при некоторых заданных наблюдаемых результатах равно вероятности этих наблюдаемых результатов при этих заданных значениях параметров, то есть L(θ|x)=P(x|θ). Пример вычисления правдоподобия модели описан в нижеследующем разделе. Меры, такие как AIC и BIC, смещены в сторону от модели, имеющей больше параметров, если модель, имеющая больше параметров, получает ту же величину дисперсии данных, что и модель, имеющая меньше параметров.
Если мера предсказательной силы использует только остаточную ошибку, должна быть рассмотрена величина улучшения остаточной ошибки, для того чтобы определить, включать или нет изменение, связанное с текущей итерацией, в текущую наилучшую обновленную модель. Это может быть выполнено посредством сравнения величины улучшения с порогом. Если величина меньше порога, рассматриваемое изменение в текущей итерации не принимают. Если, напротив, величина улучшения превышает порог, то рассматриваемое изменение включают в обновленную модель, и обновленная модель служит в дальнейшем в качестве новой наилучшей модели для оставшихся итераций.
В определенных вариантах осуществления каждая итерация рассматривает добавление или удаление одного члена, характеризующего взаимодействие, из текущей наилучшей рассматриваемой модели. В случае, когда основная модель содержит только члены, не характеризующие взаимодействие, может быть рассмотрена совокупность всех доступных членов, характеризующих взаимодействие. Каждый из этих членов, характеризующих взаимодействие, рассматривается последовательно, до тех пор, пока процесс не будет завершен, и будет получена итоговая наилучшая модель.
В некоторых случаях после определения того, что процесс эффективно сошелся, и дальнейшее улучшение маловероятно, процесс генерации моделей останавливают до того, как будут рассмотрены все доступные в совокупности члены, характеризующие взаимодействие.
Фигура 2 иллюстрирует то, как модель может быть использована для итерационного управления созданием новых библиотек белковых вариантов для цели исследования пространства белковых последовательностей и активности, в процессе (см. 200). В одном примере после генерации итоговой модели итоговую модель используют для выбора мутаций множественных положений остатков (например, положения 35) или конкретных типов остатков (например, глутамина в положении 35), о которых предсказано, что они влияют на активность. В некоторых вариантах осуществления определение основано на коэффициентах членов. См. блок 207. Кроме того, для идентифицирования таких положений можно использовать модель для "ранжирования" положений остатков или типов остатков на основании их вкладов в активность, например значений их коэффициентов. Эта функция является желательной. Например, модель может предсказывать, что глутамин в положении 35 оказывает наиболее выраженное положительное воздействие на активность; фенилаланин в положении 208 оказывает второе наиболее выраженное положительное воздействие на активность; и так далее. В одном конкретном подходе, описанном ниже, для ранжирования важности конкретных остатков используют коэффициенты регрессии PLS или PCR. В другом конкретном подходе используют матрицу нагрузок PLS для ранжирования важности положений конкретных остатков.
Остатки выбирают с использованием любого из ряда различных протоколов выбора, некоторые из которых описаны ниже. В одном иллюстративном примере конкретные остатки, о которых предсказано, что они оказывают наибольшее полезное воздействие на активность, сохраняют (то есть не варьируют). Некоторое количество других остатков, о которых предсказано, что они оказывают меньшее воздействие, тем не менее выбирают для вариации. В другом иллюстративном примере положения остатков, которые, как обнаружено, оказывают наибольшее воздействие на активность, выбирают для вариации, но только если обнаружено, что они варьируют в высокоэффективных членах обучающего набора. Например, если модель предсказывает, что положение 197 остатка оказывают наибольшее воздействие на активность, но все или большинство белков с высокой активностью имеют лейцин в этом положении, положение 197 не будет выбрано для вариации при данном подходе. Другими словами, все или большинство белков в библиотеке следующего поколения будут иметь лейцин в положении 197. Однако, если некоторые "хорошие" белки имеют в этом положении валин, а другие имеют лейцин, то способ выберет варьировать аминокислоту в этом положении. В некоторых случаях будет обнаружено, что комбинация двух или более взаимодействующих остатков оказывает наибольшее воздействие на активность. Следовательно, в некоторых стратегиях эти остатки будут варьироваться совместно. Примером совместного варьирования является одновременное применение насыщающего мутагенеза к этим остаткам.
После того, как способ идентифицировал мутации, способ осуществляет получение олигонуклеотидов, содержащих или кодирующих идентифицированные мутации, как указано в блоке 209 (фигура 2). Для получения олигонуклеотидов можно использовать различные способы. В некоторых вариантах осуществления олигонуклеотиды получают с помощью синтезатора последовательностей.
После идентификации остатков для вариации способ генерирует новую библиотеку вариантов, имеющую указанные вариации остатков. См. блок 211 (фигура 2). Для данной цели доступны различные методы. В одном примере осуществляют основанный на рекомбинации механизм генерации разнообразия in vitro или in vivo для генерации новой библиотеки вариантов. Такие процедуры могут использовать олигонуклеотиды, содержащие последовательности или подпоследовательности для кодирования белков родительской библиотеки вариантов. Некоторые из олигонуклеотидов будут близкородственными, отличающимися только по выбору кодонов для изменяющихся аминокислот, выбранных для вариации в 209. Могут быть осуществлены один или более циклов основанного на рекомбинации механизма генерации разнообразия. Если используют множественные циклы, то каждый включает этап скрининга, для того чтобы определить, какие из вариантов имеют приемлемую эффективность для использования в следующем цикле рекомбинации. Это представляет собой форму направленной эволюции. При этом не предполагается, что настоящее изобретение ограничено каким-либо конкретным способом основанного на рекомбинации способа генерации разнообразия, поскольку любой подходящий способ/метод находит применение в настоящем изобретении. В некоторых вариантах осуществления, например, настоящее изобретение осуществляет насыщающий мутагенез в отдельных местоположениях в последовательности или комбинациях местоположений в последовательности, которые взаимодействуют, как указывают значения коэффициентов членов, не характеризующих или характеризующих взаимодействие, моделей последовательность-активность.
В дополнительном иллюстративном примере выбирают "референсную" белковую последовательность, и остатки, выбранные в 207 фигуры 2, "переключают", для того чтобы идентифицировать отдельные члены библиотеки вариантов. Новые белки, идентифицированные таким образом, синтезируют подходящим методом для генерации новой библиотеки. В одном примере референсная последовательность может представлять собой наиболее эффективный член обучающего набора или "наилучшую" последовательность, предсказанную моделью PLS или PCR.
В другом подходе модель последовательность-активность используется в виде "функции приспособленности" в генетическом алгоритме для исследования пространства последовательностей. После одного или более циклов генетического алгоритма (причем каждый цикл использует функцию приспособленности для выбора одной или более возможный последовательностей для генетической операции) определяется библиотека следующего поколения для использования, описанного в данной блок-схеме. Данная стратегия может в прямом смысле слова рассматриваться как направленная эволюция in silico. В идеальном случае, при наличии точной прецизионной глобальной или локальной функции приспособленности, можно выполнить всю эволюцию in silico и синтезировать единственный наилучший вариант для использования в конечном коммерческом или научном применении. Хотя этого, вероятно, невозможно достичь в большинстве случаев, такой взгляд на способ дает понимание целей и подхода применения методов машинного обучения для направленной эволюции.
В другом иллюстративном примере остатки для вариации в цикле направленной эволюции выбирают в единственной родительской последовательности. Родитель может быть идентифицирован с использованием результатов модели из предшествующего цикла направленной эволюции или с использованием данных, которые идентифицируют член библиотеки, имеющий наилучшую эффективность при анализе. Олигонуклеотиды для следующего цикла направленной эволюции могут быть определены так, чтобы они включали участки остова выбранного родителя с одной или более мутациями, предсказанными алгоритмически из модели последовательность-активность для текущего цикла. Данные олигонуклеотиды могут быть получены с использованием любого подходящего средства, включая, но без ограничения, синтетические способы.
После того, как получена новая библиотека, ее подвергают скринингу на активность, как указано в блоке 213 (фигура 2). В идеальном случае новая библиотека предоставляет один или более членов с лучшей активностью, чем наблюдалась в прошлой библиотеке. Однако даже без такого преимущества новая библиотека может предоставлять полезную информацию. Ее члены могут быть использованы для генерации улучшенных моделей, которые учитывают эффекты новых вариантов и благодаря этому более точно предсказывают активность в более широких областях пространства последовательностей. Кроме того, библиотека может представлять проход в пространстве последовательностей из локального максимума в направлении глобального максимума (например, в активности).
В зависимости от цели способа 200 (фигура 2) в некоторых вариантах осуществления желательно генерировать ряд новых библиотек белковых вариантов, причем каждая предоставляет новые члены обучающего набора. Затем обновленный обучающий набор используют для генерации улучшенной модели. Для того чтобы получить улучшенную модель, способ 200 показан с операцией принятия решения, как показано в блоке 215, которая определяет, следует ли получать еще одну библиотеку белковых вариантов. Можно использовать различные критерии для принятия данного решения. Примеры критериев принятия решений включают, но без ограничения, количество библиотек белковых вариантов, сгенерированных на данный момент, активность наилучших белков из текущей библиотеки, желаемую величину активности и уровень улучшения, наблюдаемого в последних новых библиотеках.
В том случае, если способ используют для того, чтобы продолжить с новой библиотекой, способ возвращается на операцию блока 100 (фигура 2), где генерируется новая модель последовательность-активность из данных о последовательности и активности, полученных для текущей библиотеки белковых вариантов. Другими словами, данные о последовательности и активности для текущей библиотеки белковых вариантов служат в качестве части обучающего набора для новой модели (или они могут служить в качестве всего обучающего набора). В дальнейшем выполняют операции, показанные в блоках 207, 209, 211, 213 и 215 (фигура 2), как описано выше, но с новой моделью.
Когда определяют, что достигнута конечная точка способа, цикл, проиллюстрированный на фигуре 2, заканчивается и новые библиотеки не генерируются. В этой точке способ или просто заканчивается, или, в некоторых вариантах осуществления, одну или более последовательностей из одной или более из библиотек выбирают для разработки и/или производства. См. блок 217.
III. Генерация наблюдений
Библиотеки белковых вариантов представляют собой группы из множественных белков, имеющих один или более остатков, которые варьируются в библиотеке от члена к члену. Эти библиотеки могут быть сгенерированы с использованием способов, описанных в настоящем документе, и/или любых подходящих средств, известных в данной области техники. Эти библиотеки находят применение в предоставлении данных для обучающих наборов, используемых для генерации моделей последовательность-активность в соответствии с различными вариантами осуществления настоящего изобретения. Количество белков, включенное в библиотеку белковых вариантов, часто зависит от применения и затрат по их генерации. Не предполагается, что настоящее изобретение ограничено каким-либо конкретным количеством белков в библиотеках белков, используемых в способах настоящего изобретения. Кроме того, не предполагается, что настоящее изобретение ограничено любой конкретной библиотекой или библиотеками белковых вариантов.
В одном примере библиотеку белковых вариантов генерируют из одного или более природных белков, которые могут быть закодированы одним семейством генов. Могут быть использованы другие исходные точки, включая, но без ограничения, рекомбинанты известных белков или новые синтетические белки. Из этих начальных или исходных белков библиотека может быть сгенерирована с помощью различных методов. В одном случае библиотеку генерируют с помощью опосредованной фрагментацией ДНК рекомбинации, как описано в документах Stemmer (1994), Proceedings of the National Academy of Sciences, USA, 10747-10751 и WO 95/22625 (которые включены в настоящий документ посредством ссылки), опосредованной синтетическими олигонуклеотидами рекомбинации, как описано в документах Ness et al. (2002), Nature Biotechnology 20:1251-1255 и WO 00/42561 (которые включены в настоящий документ посредством ссылки), или нуклеиновых кислот, кодирующих часть или все из одного или более родительских белков. Также можно использовать комбинации данных способов (например, рекомбинацию фрагментов ДНК и синтетических олигонуклеотидов), а также другие основанные на рекомбинации способы, описанные, например, в документах WO 97/20078 и WO 98/27230, которые включены в настоящий документ посредством ссылки. Любые подходящие способы, используемые для генерации библиотек белковых вариантов, находят применение в настоящем изобретении. Более того, не предполагается, что настоящее изобретение ограничено каким-либо конкретным способом получения библиотек вариантов.
В некоторых вариантах осуществления единственная "исходная" последовательность (которая может представлять собой "предковую" последовательность) может использоваться для целей определения группы мутаций, используемой в процессе моделирования. В некоторых вариантах осуществления по меньшей мере одна из исходных последовательностей представляет собой последовательность дикого типа.
В определенных вариантах осуществления мутации (a) определены в литературе как воздействующие на субстратную специфичность, селективность, стабильность или другое полезное свойство, и/или (b) о них предсказано посредством вычислений, что они улучшают конфигурации укладки белка (например, упаковку внутренних остатков белка), связывание лигандов, взаимодействия субъединиц, перетасовку семейств между множественными различными гомологами и так далее. Альтернативно, мутации могут быть физически введены в исходную последовательность, а продукты экспрессии подвергнуты скринингу на полезные свойства. Сайт-направленный мутагенез является одним примером полезного метода для введения мутаций, хотя находит применение любой подходящий способ. Таким образом, альтернативно или дополнительно, мутанты могут быть получены посредством синтеза генов, насыщающего случайного мутагенеза, полусинтетических комбинаторных библиотек остатков, направленной эволюции, рекурсивной рекомбинации последовательностей ("RSR") (см., например, заявку на патент США № 2006/0223143, включена посредством ссылки в настоящий документ во всей полноте), перетасовки генов, ПЦР сниженной точности и/или любого другого подходящего способа. Один пример подходящей процедуры насыщающего мутагенеза описан в опубликованной патентной заявке США № 20100093560, которая включена в настоящий документ посредством ссылки во всей полноте.
Исходная последовательность не обязательно должна быть идентична аминокислотной последовательности белка дикого типа. Однако в некоторых вариантах осуществления исходная последовательность представляет собой последовательность белка дикого типа. В некоторых вариантах осуществления исходная последовательность включает мутации, не присутствующие в белке дикого типа. В некоторых вариантах осуществления исходная последовательность представляет собой консенсусную последовательность, получаемую из группы белков, имеющих общее свойство, например, семейства белков.
Неограничивающий репрезентативный список семейств или классов ферментов, которые могут служить в качестве источников родительских последовательностей, включает, но без ограничения, следующие: оксидоредуктазы (E.C.1); трансферазы (E.C.2); гидролазы (E.C.3); лиазы (E.C.4); изомеразы (E.C.5) и лигазы (E.C.6). Более определенные, но неограничивающие подгруппы оксидоредуктаз включают дегидрогеназы (например, алкогольдегидрогеназы (карбонилредуктазы), ксилулозоредуктазы, альдегидредуктазы, фарнезолдегидрогеназу, лактатдегидрогеназы, арабинозодегидрогеназы, глюкозодегидрогеназа, фруктозодегидрогеназы, ксилозоредуктазы и сукцинатдегидрогеназы), оксидазы (например, глюкозооксидазы, гексозооксидазы, галактозооксидазы и лакказы), моноаминоксидазы, липоксигеназы, пероксидазы, альдегиддегидрогеназы, редуктазы, длинноцепочечный ацил-[ацилпереносящий белок]редуктазы, ацил-КоА-дегидрогеназы, ен-редуктазы, синтазы (например, глутаматсинтазы), нитратредуктазы, моно- и диоксигеназы и каталазы. Более определенные, но неограничивающие подгруппы трансфераз включают метил-, амидино- и карбоксилтрансферазы, транскетолазы, трансальдолазы, ацилтрансферазы, гликозилтрансферазы, трнасаминазы, трансглутаминазы и полимеразы. Более определенные, но неограничивающие подгруппы гидролаз включают гидролазы сложных эфиров, пептидазы, гликозилазы, амилазы, целлюлазы, гемицеллюлазы, ксиланазы, хитиназы, глюкозидазы, глюканазы, глюкоамилазы, ацилазы, галактозидазы, пуллуланазы, фитазы, лактазы, арабинозидазы, нуклеозидазы, нитрилазы, фосфатазы, липазы, фосфолипазы, протеазы, АТФазы и дегалогеназы. Более определенные, но неограничивающие подгруппы лиаз включают декарбоксилазы, альдолазы, гидратазы, дегидратазы (например, карбоангидразы), синтазы (например, изопрен-, пинен- и фарнезенсинтазы), пектиназы (например, пектинлиазы) и галогенгидриндегидрогеназы. Более определенные, но неограничивающие подгруппы изомераз включают рацемазы, эпимеразы, изомеразы (например, ксилозо-, арабинозо-, рибозо-, глюкозо-, галактозо- и маннозоизомеразы), таутомеразы и мутазы (например, переносящие ацилмутазы, фосфомутазы и аминомутазы. Более определенные, но неограничивающие подгруппы лигаз включают синтазы сложных эфиров. Другие семейства или классы ферментов, которые можно использовать в качестве источников родительских последовательностей, включают трансаминазы, протеазы, киназы и синтазы. Данный список, при том, что он иллюстрирует некоторые конкретные аспекты возможных ферментов настоящего раскрытия, не рассматривается как исчерпывающий и не отображает ограничения или не очерчивает объем настоящего раскрытия.
В некоторых случаях ферменты-кандидаты, полезные в способах, описанных в настоящем документе, способны к катализу энантиоселективной реакции, такой как, например, энантиоселективная реакция восстановления. Такие ферменты можно использовать для получения промежуточных продуктов, полезных, например, при синтезе фармацевтических соединений.
В некоторых вариантах осуществления ферменты-кандидаты выбирают из эндоксиланаз (EC 3.2.1.8); β-ксилозидаз (EC 3.2.1.37); альфа-L-арабинофуранозидаз (EC 3.2.1.55); альфа-глюкуронидаз (EC 3.2.1.139); ацетилксиланэстераз (EC 3.1.1.72); ферулоилэстераз (EC 3.1.1.73); кумароилэстераз (EC 3.1.1.73); альфа-галактозидаз (EC 3.2.1.22); бета-галактозидаз (EC 3.2.1.23); бета-маннаназ (EC 3.2.1.78); бета-маннозидаз (EC 3.2.1.25); эндополигалактуроназ (EC 3.2.1.15); пектинметилэстераз (EC 3.1.1.11); эндогалактаназ (EC 3.2.1.89); пектинацетилэстераз (EC 3.1.1.6); эндопектинлиаз (EC 4.2.2.10); пектатлиаз (EC 4.2.2.2); альфа-рамнозидаз (EC 3.2.1.40); экзополи-альфа-галактуронозидаз (EC 3.2.1.82); 1,4-альфа-галактуронозидаз (EC 3.2.1.67); экзополигалактуронатлиаз (EC 4.2.2.9); рамногалактуронанэндолиаз (EC 4.2.2.B3); рамногалактуронанацетилэстераз (EC 3.2.1.B11); рамногалактуронангалактуроногидролаз (EC 3.2.1.B11); эндоарабинаназ (EC 3.2.1.99); лакказ (EC 1.10.3.2); марганец-зависимых пероксидаз (EC 1.10.3.2); амилаз (EC 3.2.1.1), глюкоамилаз (EC 3.2.1.3), протеаз, липаз и лигнинпероксидаз (EC 1.11.1.14). Любые комбинации из одного, двух, трех, четырех, пяти или более чем пяти ферментов находят применение в композициях настоящего изобретения.
В одном или более вариантах осуществления настоящего изобретения единственную исходную последовательность модифицируют различным образом для генерации библиотеки. В некоторых вариантах осуществления библиотеку генерируют посредством систематического варьирования отдельных остатков исходной последовательности. В одном иллюстративном примере для того, чтобы идентифицировать систематически варьируемые последовательности, используют методику плана эксперимента (DOE). В другом примере используют процедуру "влажной лаборатории", такую как опосредованная олигонуклеотидами рекомбинация для введения определенного уровня систематической вариации. Не предполагается, что настоящее изобретение ограничено каким-либо конкретным способом генерации систематически варьируемых последовательностей, поскольку применение находит любой подходящий способ.
Как используется в настоящем документе, термин "систематически варьируемые последовательности" относится к набору последовательностей, в котором каждый остаток рассматривают во множественных контекстах. В принципе, уровень систематической вариации может быть количественно охарактеризован с помощью степени, в которой последовательности ортогональны друг другу (то есть максимально отличаются по сравнению со средним). В некоторых вариантах осуществления способ не зависит от наличия максимально ортогональных последовательностей. Однако качество модели улучшается в непосредственной связи с ортогональностью исследуемого пространства последовательностей. В простом иллюстративном примере пептидную последовательность систематически варьируют с помощью идентифицирования двух положений остатков, каждое из которых может содержать одну из двух различных аминокислот. Максимально разнообразная библиотека включает все четыре возможные последовательности. Такая максимальная систематическая вариация возрастает экспоненциально с количеством варьируемых положений; например на 2N, когда наличествуют 2 варианта в каждом из N положений остатков. Средним специалистам в данной области техники будет легко понять, что максимальная систематическая вариация, однако, не является необходимой. Систематическая вариация обеспечивает механизм для идентификации относительно небольшого набора последовательностей для исследования, который обеспечивает хорошее семплирование пространства последовательностей.
Варианты белков, содержащие систематически варьируемые последовательности, могут быть получены рядом способов с использованием методов, которые хорошо известны средним специалистам в данной области техники. Как указано, подходящие способы включают, но без ограничения, основанные на рекомбинации способы, которые генерируют варианты на основании одной или более "родительских" полинуклеотидных последовательностей. Полинуклеотидные последовательности могут быть рекомбинированы с использованием множества методов, включая, например, расщепление полинуклеотидов, подлежащих рекомбинированнию, ДНКазой с последующей повторной сборкой нуклеиновых кислот посредством лигирования и/или ПЦР. Данные способы включают, но без ограничения, описанные, например, в документе Stemmer (1994), Proceedings of the National Academy of Sciences USA, 91: 10747-10751, патенте США № 5605793 "Methods for In Vitro Recombination," патенте США № 5811238 "Methods for Generating Polynucleotides having Desired Characteristics by Iterative Selection and Recombination", патенте США № 5830721 "DNA Mutagenesis by Random Fragmentation and Reassembly", патенте США № 5834252 "End Complementary Polymerase Reaction", патенте США № 5837458 "Methods and Compositions for Cellular and Metabolic Engineering", WO 98/42832 "Recombination of Polynucleotide Sequences Using Random or Defined Primers", WO 98/27230 "Methods and Compositions for Polypeptide Engineering", WO 99/29902 "Method for Creating Polynucleotide and Polypeptide Sequences" и тому подобных, все из которых включены в настоящий документ посредством ссылки.
Также особенно хорошо для генерации библиотек белковых вариантов с систематической вариацией подходят способы синтетической рекомбинации. В способах синтетической рекомбинации синтезируют множество олигонуклеотидов, которые вместе кодируют множество генов, подлежащих рекомбинированию. В некоторых вариантах осуществления олигонуклеотиды вместе кодируют последовательности, получаемые из гомологичных родительских генов. Например, гомологичные гены, представляющие интерес, выравнивают с использованием программы выравнивания последовательностей, такой как BLAST (см., например, документ Atschul, et al., Journal of Molecular Biology, 215: 403-410 (1990)). Отмечают нуклеотиды, соответствующие аминокислотным вариациям между гомологами. Данные вариации, необязательно, дополнительно ограничивают поднабором всех возможных вариаций на основании ковариационного анализа родительских последовательностей, функциональной информации относительно родительских последовательностей, выбора консервативных или не консервативных изменений между родительскими последовательностями или других подходящих критериев. Вариации, необязательно, дополнительно увеличивают для кодирования дополнительного разнообразия аминокислот в положениях, идентифицированных посредством, например, ковариационного анализа родительских последовательностей, функциональной информации относительно родительских последовательностей, выбора консервативных или не консервативных изменений между родительскими последовательностями или очевидной толерантности положения для вариации. Результатом является вырожденная последовательность генов, кодирующая консенсусную аминокислотную последовательность, получаемую из родительских последовательностей генов, с вырожденными нуклеотидами в положениях, кодирующих аминокислотные вариации. Разрабатывают олигонуклеотиды, которые содержат нуклеотиды, требуемые для создания разнообразия, имеющего место в вырожденном гене. Подробности в отношении таких подходов можно найти, например, в документах Ness et al. (2002), Nature Biotechnology, 20: 1251-1255, WO 00/42561 "Oligonucleotide Mediated Nucleic Acid Recombination", WO 00/42560 "Methods for Making Character Strings, Polynucleotides and Polypeptides having Desired Characteristics", WO 01/75767 "In Silico Cross-Over Site Selection" и WO 01/64864 "Single-Stranded Nucleic Acid Template-Mediated Recombination and Nucleic Acid Fragment Isolation", каждый из которых включен в настоящий документ посредством ссылки. Идентифицированные полинуклеотидные вариантные последовательности могут быть транскрибированы и транслированы или in vitro, или in vivo для создания набора или библиотеки последовательностей вариантов белков.
Набор систематически варьируемых последовательностей может также быть разработан a priori с использованием способов плана эксперимента (DOE) для определения последовательностей в наборе данных. Описание способов DOE можно найти в документе Diamond, W.J. (2001), Practical Experiment Designs: for Engineers and Scientists, John Wiley & Sons, и в документах "Practical Experimental Design for Engineers and Scientists", William J. Drummond (1981), Van Nostrand Reinhold Co New York, "Statistics for experimenters", George E.P. Box, William G. Hunter and J. Stuart Hunter (1978), John Wiley and Sons, New York, или, например, во Всемирной паутине по адресу itl.nist.gov/div898/handbook/. Существует несколько доступных вычислительных пакетов для осуществления соответствующей математики, включая Statistics Toolbox (MATLAB®), JMP®, STATISTICA® и STAT-EASE® DESIGN EXPERT®. Результатом является систематически варьируемый и ортогонально распределенный набор данных последовательностей, который подходит для построения модели последовательность-активность настоящего изобретения. Основанные на DOE наборы данных также могут быть легко сгенерированы с использованием или планов Плакетта-Бермана, или дробных факторных планов, как известно в данной области техники. Diamond, W.J. (2001).
В инженерных и химических науках дробные факторные планы используют для определения меньшего количества экспериментов по сравнению с полными факторными планами. В данных способах фактор варьируют (то есть "переключают") между двумя или более уровнями. Используют методы оптимизации для обеспечения того, что выбранные эксперименты являются максимально информативными при учете дисперсии пространства факторов. Подходы с таким же планом (например, дробный факторный D-оптимальный план) могут применяться в белковой инженерии для построения меньшего количества последовательностей, в которых заданное число положений переключают между двумя или более остатками. В некоторых вариантах осуществления данный набор последовательностей обеспечивает оптимальное описание систематической дисперсии, присутствующей в рассматриваемом пространстве белковых последовательностей.
Иллюстративный пример подхода DOE, примененного к белковой инженерии, включает следующие операции:
1) Идентифицирование положений для переключения на основании принципов, описанных в настоящем документе (например, присутствие в родительских последовательностях, уровень консервации и так далее).
2) Создание эксперимента DOE с использованием одного из общедоступных пакетов статистического программного обеспечения посредством определения числа факторов (то есть варьируемых положений), числа уровней (то есть вариантов в каждом положении) и числа проводимых экспериментов для получения матрицы выхода. Информационное содержание матрицы выхода (как правило, состоящей из единиц и нулей, которые представляют варианты выбора остатков в каждом положении) зависит непосредственно от числа проводимых экспериментов (как правило, чем больше, тем лучше).
3) Использование матрицы выхода для построения выравнивания белков, которое перекодирует единицы и нули обратно в конкретные варианты выбора остатков в каждом положении.
4) Синтез генов, кодирующих белки, представленные в выравнивании белков.
5) Тестирование белков, кодируемых синтезированными генами, в соответствующем анализе(ах).
6) Построение модели на основании протестированных генов/белков.
7) Следование этапам, описанным в настоящем документе, для того чтобы идентифицировать важные положения, и для того чтобы построить одну или более последующих библиотек с улучшенной приспособленностью.
В иллюстративном примере исследуют белок, в котором следует определить функционально наилучшие аминокислотные остатки в 20 положениях (например, когда существует 2 возможные аминокислоты, доступные в каждом положении). В данном примере подходящим будет факторный план с разрешением IV. План с разрешением IV определяется как план, который способен к выявлению воздействий всех отдельных переменных без двухфакторных воздействий с их перекрыванием. Затем план определяет набор из 40 конкретных аминокислотных последовательностей, покрывающих общее разнообразие, составляющее 220 (~1 миллион) возможных последовательностей. Затем эти последовательности генерируют с использованием любого стандартного протокола синтеза генов, и определяют функцию и приспособленность данных клонов.
Альтернатива вышеуказанным подходам заключается в использовании некоторых или всех доступных последовательностей (например, базы данных GENBANK® и других открытых источников) для получения библиотеки белковых вариантов. Данный подход обеспечивает индикацию представляющих интерес областей пространства последовательностей.
IV. Способы секвенирования
Исторически секвенирование было лимитирующим этапом при разработке больших обучающих наборов и, следовательно, все более робастных моделей последовательность-активность. Высокая стоимость и длительное время, требуемое для вариантов последовательностей, ограничивало количество наблюдений несколькими десятками вариантов. Инструменты секвенирования нового поколения значительно снизили стоимость и повысили скорость и объем секвенирования, сделав возможным включение в обучающий набор вариантов как с низкой, так и с высокой активностью.
Инструменты секвенирования нового поколения могут недорого секвенировать большие количества пар оснований (например, по меньшей мере приблизительно 1000000000 пар оснований) за один проход. Эта возможность может быть использована при секвенировании вариантных белков, которые, как правило, имеют длину только несколько тысяч пар оснований, за один проход. Часто инструменты секвенирования нового поколения оптимизированы для секвенирования за один проход отдельных больших геномов (например, человеческого генома), а не множества небольших последовательностей. Для реализации потенциала инструментов секвенирования нового поколения для секвенирования множества наблюдений параллельно следует однозначно идентифицировать происхождение каждого из наблюдений, подвергаемых секвенированию за один проход. В некоторых вариантах осуществления используют последовательности со штриховым кодом на всех без исключения фрагментах, подаваемых в секвенатор нового поколения за один проход. В одном примере штриховые коды однозначно идентифицируют конкретную лунку на конкретном планшете (например, 96-луночных планшетах). В некоторых из данных вариантов осуществления каждая лунка каждого планшета содержит один уникальный вариант. Посредством штрихового кодирования каждого варианта или, более конкретно, каждого фрагмента каждого варианта, можно секвенировать и идентифицировать за один проход последовательности генов множественных различных вариантов. В данном способе все считываемые фрагменты, имеющие один и тот же штриховой код, идентифицируют и обрабатывают вместе с помощью алгоритма, определяющего длину последовательностей для вариантов.
В некоторых вариантах осуществления ДНК из клеток варианта в заданной лунке экстрагируют и затем фрагментируют. Затем на фрагменты наносят штриховой код, для того чтобы идентифицировать по меньшей мере лунку, а иногда лунку и планшет, соответствующие данному варианту. Затем получаемые фрагменты сортируют по размеру для получения последовательностей подходящей для секвенатора нового поколения длины. В одном иллюстративном примере длины прочтения составляют приблизительно 200 пар оснований. В некоторых вариантах осуществления штриховой код планшета не наносят до тех пор, пока сначала не объединят фрагменты ДНК из различных лунок планшета. Затем на объединенную ДНК наносят штриховой код, для того чтобы идентифицировать планшет. В некоторых вариантах осуществления каждый фрагмент, независимо от того, из какой лунки он был получен, имеет один и тот же штриховой код планшета. Однако в некоторых альтернативных вариантах осуществления фрагменты имеют различные штриховые коды. Кроме того, штриховые коды лунки и планшета могут быть нанесены для того, чтобы идентифицировать ДНК, экстрагированную из данной лунки.
В одном или более вариантах осуществления данные о последовательности могут быть получены с использованием способов объемного секвенирования включая, например, секвенирование по Сэнгеру или секвенирование по Максаму-Гилберту, которые считаются способами секвенирования первого поколения. Секвенирование по Сэнгеру, которое включает использование меченых дидезокси терминаторов цепи, хорошо известно в данной области техники; см., например, Sanger et al., Proceedings of the National Academy of Sciences of the United States of America 74, 5463-5467 (1997). Cеквенирование по Максаму-Гилберту, которое включает осуществление множественных реакций частичной химической деградации в отношении фракций образца нуклеиновой кислоты с последующими детектированием и анализом фрагментов для получения заключения о последовательности, также хорошо известно в данной области техники; см., например, Maxam et al., Proceedings of the National Academy of Sciences of the United States of America 74, 560-564 (1977). Другим способом объемного секвенирования является секвенирование гибридизацией, при которой последовательность образца получают на основании свойств ее гибридизации с множеством последовательностей, например, на микроматице или генном чипе; см., например, Drmanac, et al., Nature Biotechnology 16, 54-58 (1998).
В одном или более вариантах осуществления данные о последовательности получают с использованием способов секвенирования нового поколения. Секвенирование нового поколения также называется "высокопроизводительным секвенированием". Данные методы параллелизуют процесс секвенирования, производя тысячи или миллионы последовательностей за один раз. Примеры подходящих способов секвенирования нового поколения включают, но без ограничения, одномолекулярное секвенирование в реальном времени (например, Pacific Biosciences, Menlo Park, California), ионное полупроводниковое секвенирование (например, Ion Torrent, South San Francisco, California), пиросеквенирование (например, 454, Branford, Connecticut), секвенирование лигированием (например, секвенирование SOLid, Life Technologies, Carlsbad, California), секвенирование посредством синтеза и обратимого терминирования (например, Illumina, San Diego, California), технологии визуализации нуклеиновой кислоты, такие как просвечивающая электронная микроскопия, и тому подобное.
Вообще говоря, способы секвенирования нового поколения используют, как правило, этап клонирования in vitro для амплификации отдельных молекул ДНК. Эмульсионная ПЦР (emPCR) выделяет отдельные молекулы ДНК вместе с покрытыми праймером гранулами в водных каплях в масляной фазе. ПЦР производит копии молекулы ДНК, которые связываются с праймерами на грануле, за чем следует иммобилизация для последующего секвенирования. EmPCR используют в способах Marguilis et al. (коммерциализирован 454 Life Sciences, Branford, CT), Shendure и Porreca et al. (также известен как "полони-секвенирование") и секвенирования SOLiD, (Applied Biosystems Inc., Foster City, CA). См. M. Margulies, et al. (2005), "Genome sequencing in microfabricated high-density picolitre reactors" Nature 437: 376-380; J. Shendure, et al. (2005), "Accurate Multiplex Polony Sequencing of an Evolved Bacterial Genome" Science 309 (5741): 1728-1732. Клональная амплификация in vitro может также быть осуществлена с помощью "мостиковой ПЦР", когда фрагменты амплифицируют после того, как праймеры прикрепятся к твердой поверхности. Braslavsky et al. разработали одномолекулярный способ (коммерциализирован Helicos Biosciences Corp., Cambridge, MA), который опускает этот этап амплификации, непосредственно фиксируя молекулы ДНК на поверхности. I. Braslavsky, et al. (2003), "Sequence information can be obtained from single DNA molecules", Proceedings of the National Academy of Sciences of the United States of America 100: 3960-3964.
Молекулы ДНК, которые физически связаны с поверхностью, могут быть секвенированы параллельно. При "секвенировании посредством синтеза" комплементарную нить строят на основании последовательности матричной нити с использованием ДНК-полимеразы, как, например, при электрофоретическом секвенировании с терминированием красителем. Способы с обратимым терминированием (коммерциализированы Illumina, Inc., San Diego, CA и Helicos Biosciences Corp., Cambridge, MA) используют обратимые варианты красителей-терминаторов, добавляя один нуклеотид за один раз, и детектируют флуоресценцию в каждом положении в реальном времени посредством повторяющегося удаления блокирующей группы, для того чтобы обеспечить возможность полимеризации другого нуклеотида. "Пиросеквенирование" также использует полимеризацию ДНК, добавляя один нуклеотид за один раз и детектируя и количественно анализируя число нуклеотидов, добавленных в данное местоположение, с помощью света, испускаемого при высвобождении прикрепленных пирофосфатов (коммерциализировано 454 Life Sciences, Branford, CT). См. M. Ronaghi, et al. (1996), "Real-time DNA sequencing using detection of pyrophosphate release", Analytical Biochemistry 242: 84-89.
Конкретные примеры способов секвенирования нового поколения описаны более подробно ниже. Одно или более воплощений настоящего изобретения могут использовать один или более из следующих способов секвенирования без отхода от принципов настоящего изобретения.
Одномолекулярное секвенирование в реальном времени (также известное как SMRT) представляет собой метод параллелизованного одномолекулярного секвенирования ДНК посредством синтеза, разработанный Pacific Biosciences. Одномолекулярное секвенирование в реальном времени использует волновод нулевой моды (ZMW). Единственный фермент ДНК-полимераза закреплен в нижней части ZMW с одной молекулой ДНК в качестве матрицы. ZMW представляет собой структуру, которая создает освещенный объем наблюдения, который достаточно мал для того, чтобы наблюдать только один нуклеотид ДНК (также известный как основание), вставляемый ДНК-полимеразой. Каждое из четырех оснований ДНК прикрепляют к одному из четырех различных флуоресцентных красителей. Когда нуклеотид вставлен ДНК-полимеразой, флуоресцентная метка отщепляется и диффундирует из области наблюдения ZMW, где ее флуоресценцию больше не наблюдают. Детектор детектирует флуоресцентный сигнал от вставки нуклеотида, и определение основания осуществляется по соответствующей флуоресценции красителя.
Другим применимым методом одномолекулярного секвенирования является метод одномолекулярного секвенирования Helicos True (tSMS) (например, как описано в Harris T.D. et al., Science 320: 106-109 [2008]). В методе tSMS образец ДНК расщепляют на нити приблизительно по 100-200 нуклеотидов, и добавляют последовательность поли-A к 3'-концу каждой нити ДНК. Каждую нить помечают посредством добавления флуоресцентно меченого нуклеотида аденозина. Затем нити ДНК гибридизуют к проточной кювете, которая содержит миллионы сайтов захвата олиго-T, которые иммобилизованы на поверхности проточной кюветы. В определенных вариантах осуществления матрицы могут иметь плотность приблизительно 100 миллионов матриц/см2. Затем проточную кювету загружают в аппарат, например секвенатор HeliScope™, и лазер освещает поверхность проточной кюветы, показывая положение каждой матрицы. Камера CCD может картировать положение матриц на поверхности проточной кюветы. Затем флуоресцентную метку матриц отщепляют и смывают. Реакция секвенирования начинается с внесения ДНК-полимеразы и флуоресцентно меченого нуклеотида. Нуклеиновая кислота олиго-T служит в качестве праймера. Полимераза вставляет меченые нуклеотиды в праймер матрично-управляемым образом. Полимеразу и невставленные нуклеотиды удаляют. Матрицы, на которых произошло управляемое включение флуоресцентно меченого нуклеотида, отличают с помощью визуализации поверхности проточной кюветы. После визуализации этап отщепления удаляет флуоресцентные метки, и процесс повторяют с другими флуоресцентно мечеными нуклеотидами до тех пор, пока не будет достигнута желаемая длина прочтения. Информацию о последовательности получают на каждом этапе добавления нуклеотидов. Секвенирование всего генома с помощью технологий одномолекулярного секвенирования исключает или в основном устраняет основанную на ПЦР амплификацию при получении библиотек секвенирования, и данные способы делают возможным непосредственное измерение образца, а не измерение копий этого образца.
Ионное полупроводниковое секвенирование представляет собой способ секвенирования ДНК на основании детектирования ионов водорода, которые высвобождаются во время полимеризации ДНК. Оно является способом "секвенирования посредством синтеза", во время которого комплементарная нить строится на основании последовательности матричной нити. Микролунку, содержащую матричную нить ДНК для секвенирования, наполняют дезоксирибонуклеотидтрифосфатом (дНТФ) одного вида. Если введенный дНТФ является комплементарным к нуклеотиду ведущей матрицы, он включается в растущую комплементарную цепь. Это вызывает высвобождение иона водорода, который вызывает срабатывание ионного датчика ISFET, что указывает на то, что реакция произошла. Если в матричной последовательности присутствуют гомополимерные повторы, множественные молекулы дНТФ будут присоединены в одном цикле. Это приводит к соответствующему количеству высвобождаемого водорода и пропорционально более высокому электрическому сигналу. Эта технологии отличается от других технологий секвенирования тем, что не используются модифицированные нуклеотиды и оптика. Ионное полупроводниковое секвенирование может также называться секвенированием Ion Torrent, pH-опосредованным секвенированием, кремниевым секвенированием или полупроводниковым секвенированием.
При пиросеквенировании ион пирофосфата, высвобождаемый реакцией полимеризации, реагирует с аденозин-5′-фосфосульфатом с помощью АТФ-сульфурилазы для получения АТФ; затем АТФ запускает превращение люциферина в оксилюциферин плюс свет с помощью люциферазы. Поскольку флуоресценция является временной, в данном способе нет необходимости в отдельном этапе для удаления флуоресценции. За один раз добавляют один тип дезоксирибонуклеотидтрифосфата (дНТФ), и информацию о последовательности получают в соответствии с тем, какой из дНТФ генерирует значительный сигнал в реакционном сайте. Коммерчески доступный аппарат Roche GS FLX получает последовательность с использованием данного способа. Данный метод и его применения подробно рассмотрены, например, в документах Ronaghi et al., Analytical Biochemistry 242, 84-89 (1996), и Margulies et al., Nature 437, 376-380 (2005) (исправления в Nature 441, 120 (2006)). Коммерчески доступной технологией пиросеквенирования является секвенирование 454 (Roche) (например, как описано в документе Margulies, M. et al. Nature 437:376-380 [2005]).
При секвенировании лигированием используют фермент лигазу для соединения частично двухцепочечного олигонуклеотида с липким концом с секвенируемой нуклеиновой кислотой, которая имеет липкий конец; для того чтобы произошло лигирование, липкие концы должны быть комплементарны. Основания в липком конце частично двухцепочечного олигонуклеотида могут быть идентифицированы по флуорофору, конъюгированному с частично двухцепочечным олигонуклеотидом, и/или по второму олигонуклеотиду, который гибридизуется с другой частью частично двухцепочечного олигонуклеотида. После получения данных флуоресценции лигированный комплекс расщепляется выше сайта лигирования, например, с помощью фермента рестрикции типа IIs, например Bbvl, который режет в сайте на фиксированном расстоянии от своего сайта распознавания (который включен в частично двухцепочечный олигонуклеотид). Данная реакция расщепления обнажает новый липкий конец непосредственно выше предыдущего липкого конца, и процесс повторяется. Данный метод и его применения подробно рассмотрены, например, в документе Brenner et al., Nature Biotechnology 18, 630-634 (2000). В некоторых вариантах осуществления секвенирование лигированием адаптируют к способам настоящего изобретения посредством получения продукта амплификации по типу катящегося кольца кольцевой молекулы нуклеиновой кислоты и использования продукта амплификации по типу катящегося кольца в качестве матрицы для секвенирования лигированием.
Коммерчески доступным примером технологии секвенирования лигированием является технология SOLiD™ (Applied Biosystems). При секвенировании лигированием SOLiD™ геномную ДНК нарезают на фрагменты, и прикрепляют к 5' и 3'-концам фрагментов адаптеры для генерации библиотеки фрагментов. Альтернативно, могут быть введены внутренние адаптеры посредством лигирования адаптеров к 5' и 3'-концам фрагментов, закольцовывания фрагментов, расщепления закольцованного фрагмента для генерации внутреннего адаптера и прикрепления адаптеров к 5' и 3'-концам получаемых фрагментов для генерации библиотеки спаренных концов. Затем получают клональные популяции гранул в микрореакторах, содержащих гранулы, праймеры, матрицу и компоненты ПЦР. После ПЦР матрицы денатурируют, и обогащают гранулы для отделения гранул с удлиненными матрицами. Матрицы на выбранных гранулах подвергают 3'-модификации, которая делает возможным связывание со стеклянной пластиной. Последовательность может быть определена посредством последовательной гибридизации и лигирования частично случайных олигонуклеотидов с определенным центральным основанием (или парой оснований), которые идентифицированы определенным флуорофором. После регистрации цвета лигированный олигонуклеотид отщепляют и удаляют, и затем процесс повторяют.
При секвенировании с обратимыми терминаторами аналог нуклеотида, меченный флуоресцентным красителем, который представляет собой обратимый терминатор цепи благодаря присутствию блокирующей группы, вводят в реакцию удлинения на одно основание. Идентичность основания определяют по флуорофору; другими словами, каждое основание образует пару с различными флуорофорами. После получения данных о флуоресценции/последовательности флуорофор и блокирующую группу химически удаляют, и цикл повторяют для получения информации о следующем основании в последовательности. Аппарат Illumina GA работает в соответствии с данным способом. Данный метод и его применения подробно рассмотрены, например, в документах Ruparel et al., Proceedings of the National Academy of Sciences of the United States of America 102, 5932-5937 (2005), и Harris et al., Science 320, 106-109 (2008).
Коммерчески доступным примером способа секвенирования с обратимыми терминаторами является секвенирование посредством синтеза Illumina и основанное на обратимых терминаторах секвенирование (например, как описано в документе Bentley et al., Nature 6:53-59 [2009]). Технология секвенирования Illumina основана на прикреплении фрагментированной геномной ДНК к плоской оптически прозрачной поверхности, с которой связаны олигонуклеотидные якоря. Матричная ДНК подвергается репарации концов для генерации 5'-фосфорилированных тупых концов, и полимеразную активность фрагмента Кленова используют для добавления одного основания A с 3'-конца тупых фосфорилированных фрагментов ДНК. Данное добавление подготавливает фрагменты ДНК для лигирования к олигонуклеотидным адаптерам, которые имеют липкий конец из одного основания T на своем 3'-конце для повышения эффективности лигирования. Адаптерные олигонуклеотиды комплементарны якорям проточной кюветы. В условиях предельных разведений модифицированную адаптером одноцепочечную матричную ДНК добавляют в проточную кювету и иммобилизуют посредством гибридизации с якорями. Прикрепленные фрагменты ДНК удлиняют и подвергают мостиковой амплификации для создания проточной кюветы для секвенирования сверхвысокой плотности с сотнями миллионов кластеров, причем каждый содержит ~1000 копий одной и той же матрицы. Матрицы секвенируют с использованием робастной технологии четырехцветного секвенирования ДНК посредством синтеза, которая использует обратимые терминаторы с удаляемыми флуоресцентными красителями. Высокочувствительного детектирования флуоресценции достигают, используя возбуждение лазером и оптику с полным внутренним отражением. Короткие считываемые последовательности приблизительно по 20-40 п.о., например 36 п.о., выравнивают по референсному геному с маскировкой повторов, и идентифицируют уникальное картирование этих коротких считываемых последовательностей на референсном геноме с использованием специально разработанного конвейерного программного обеспечения для анализа данных. Также можно использовать референсные геномы без маскировки повторов. Используются ли референсные геномы с маскировкой повторов или без маскировки повторов, подсчитывают только прочтения, которые однозначно картируются на референсный геном. После завершения первого прочтения матрицы могут быть регенерированы in situ, для того чтобы позволить второе прочтение с противоположного конца фрагментов. Таким образом, можно использовать секвенирование или неспаренных концов, или спаренных концов фрагментов ДНК. Осуществляют частичное секвенирование фрагментов ДНК, присутствующих в образце, и подсчитывают метки последовательностей, содержащих прочтения предварительно определенной длины, например 36 п.о., картируемые на известный референсный геном.
При нанопоровом секвенировании одноцепочечную молекулу нуклеиновой кислоты продевают через пору, например используя электрофоретическую движущую силу, и делают заключение в отношении последовательности посредством анализа данных, полученных, когда одноцепочечная молекула нуклеиновой кислоты проходит через пору. Данные могут представлять собой данные об ионном токе, причем каждое основание изменяет ток, например, посредством частичной блокировки тока, проходящего через пору, в различной различимой степени.
В другом иллюстративном, но неограничивающем варианте осуществления способы, описанные в настоящем документе, содержат получение информации о последовательности с использованием просвечивающей электронной микроскопии (TEM). Данный способ включает использование визуализации посредством просвечивающего электронного микроскопа с одноатомным разрешением имеющей высокий молекулярный вес (150 п.о. или более) ДНК, селективно меченой тяжелыми атомами-маркерами, и расположение данных молекул на сверхтонких пленках в сверхплотных (3 нм от нити до нити) параллельных компоновках с постоянным расстоянием от основания до основания. Электронный микроскоп используют для визуализации молекул на пленках для определения положения тяжелых атомов-маркеров и для извлечения информации о последовательности оснований из ДНК. Данный способ далее описан в патентной публикации PCT WO 2009/046445.
В другом иллюстративном, но неограничивающем варианте осуществления способы, описанные в настоящем документе, содержат получение информации о последовательности с использованием секвенирования третьего поколения. При секвенировании третьего поколения пластина с алюминиевым покрытием с множеством маленьких (~50 нм) отверстий используется в качестве волновода нулевой моды (см., например, Levene et al., Science 299, 682-686 (2003)). Алюминиевая поверхность защищена от прикрепления ДНК-полимеразы с помощью полифосфонатной химии, например, поливинилфосфонатной химии (см., например, Korlach et al., Proceedings of the National Academy of Sciences of the United States of America 105, 1176-1181 (2008)). Это приводит к предпочтительному прикреплению молекул ДНК-полимеразы к обнаженному оксиду кремния в отверстиях алюминиевого покрытия. Такая конфигурация позволяет использовать явления нераспространяющихся волн для снижения фона флуоресценции, что позволяет использовать более высокие концентрации флуоресцентно меченных дНТФ. Флуорофор прикрепляют к терминальному фосфату дНТФ, так что после присоединения дНТФ испускается флуоресценция, но флуорофор не остается прикреплен к только что присоединенному нуклеотиду, что означает, что комплекс немедленно готов к другому циклу присоединения. Посредством данного способа можно детектировать включение дНТФ в отдельные комплексы праймер-матрица, присутствующие в отверстиях алюминиевого покрытия. См., например, Eid et al., Science 323, 133-138 (2009).
V. Генерация модели последовательность-активность
Как указано выше, модель последовательность-активность, используемая с вариантами осуществления настоящего документа, связывает информацию о белковой последовательности с активностью белка. Информация о белковой последовательности, используемая моделью, может принимать множество форм. В некоторых вариантах осуществления она представляет собой полную последовательность аминокислотных остатков в белке. Однако, в некоторых вариантах осуществления полная аминокислотная последовательность не является необходимой. Например, в некоторых вариантах осуществления достаточно предоставить только те остатки, которые подлежат варьированию в конкретной научно-исследовательской работе. В некоторых вариантах осуществления, включающих стадии последних исследований, многие остатки зафиксированы, и только ограниченные области пространства последовательностей остаются для исследования. В некоторых из таких ситуаций удобно предоставлять модели последовательность-активность, которые требуют в качестве входных данных идентификацию только остатков в областях белка, в которых продолжается исследование. В некоторых дополнительных вариантах осуществления модели не требуют, чтобы были известны точные идентичности остатков в положениях. В некоторых таких вариантах осуществления определяют одно или более физических или химических свойств, которые характеризуют аминокислоту в конкретном положении остатка. В одном иллюстративном примере модель требует спецификации положений остатков по размерам, гидрофобности, кислотности и так далее. Кроме того, в некоторых моделях используют комбинации таких свойств. Более того, не предполагается, что настоящее изобретение ограничено каким-либо конкретным подходом, поскольку модели находят применение в различных конфигурациях информации о последовательности, информации об активности и/или других физических свойств (например, гидрофобности и так далее).
Таким образом, форма модели последовательность-активность может широко варьироваться до тех пор, пока она обеспечивает по желанию инструмент для корректной аппроксимации относительной активности белков на основании информации о последовательности. В некоторых вариантах осуществления модели обычно рассматривают активность в качестве зависимой переменной, а значения последовательности/остатков в качестве независимых переменных. Примеры математической/логической формы моделей включают аддитивные, мультипликативные, линейные и нелинейные математические выражения различных порядков, нейронные сети, классификационные и регрессионные деревья/графики, подходы кластеризации, рекурсивное секционирование, машины опорных векторов и тому подобное.
Некоторые варианты осуществления моделей включают произведение множества мультипликативных членов. По меньшей мере некоторые из мультипликативных членов представляют собой мультипликативные члены, не характеризующие взаимодействие, каждый из которых содержит коэффициент, представляющий вклад определенной аминокислоты или нуклеотида в представляющую интерес активность. Определенная аминокислота или нуклеотид имеет конкретный тип остатка в конкретном положении в белковой или нуклеиновокислотной последовательности. Каждый из мультипликативных членов, не характеризующих взаимодействие, также включает одну независимую переменную, или фиктивную переменную, которая представляет присутствие или отсутствие определенной аминокислоты или нуклеотида. Кроме того, каждая из моделей последовательность-активность в некоторых вариантах осуществления настоящего изобретения содержит зависимую переменную, представляющую активность белкового варианта.
В некоторых вариантах осуществления настоящего изобретения, модели последовательность-активность также включают члены, характеризующие взаимодействие, каждый из которых содержит коэффициент взаимодействия, представляющий вклад в активность определенной комбинации (i) первой аминокислоты или нуклеотида в первом положении в белковой последовательности, и (ii) второй аминокислоты или нуклеотида во втором положении в белковой последовательности.
В некоторых вариантах осуществления настоящего изобретения модели объединяют члены, характеризующие взаимодействие, посредством умножения или сложения. В некоторых вариантах осуществления модели объединяют члены, характеризующие взаимодействие, с мультипликативными членами, не характеризующими взаимодействие, посредством умножения или сложения. В некоторых вариантах осуществления являются чисто мультипликативными, объединяющими члены, не характеризующие и характеризующие взаимодействие, в одном произведении. В других вариантах осуществления модели включают по меньшей мере одно произведение множественных членов, объединенное с одним или более другими членами посредством сложения.
В некоторых альтернативных вариантах осуществления настоящего изобретения модели последовательность-активность имеют аддитивную форму, содержащую сумму одного или более членов, не характеризующих взаимодействие, и по меньшей мере одного члена, характеризующего взаимодействие. По меньшей мере один член, характеризующий взаимодействие, представляет собой член перекрестного произведения, содержащий произведение одной переменной, представляющей присутствие одного взаимодействующего остатка, и другой переменной, представляющей присутствие другого взаимодействующего остатка.
В некоторых вариантах осуществления модели разрабатывают по обучающему набору информации об активности в зависимости от последовательности для получения математической/логической зависимости между активностью и последовательностью. Данную зависимость, как правило, проверяют перед использованием для предсказания активности новых последовательностей или воздействия остатков на представляющую интерес активность.
Различные методы генерации моделей доступны и находят применение в настоящем изобретении. В некоторых вариантах осуществления данные методы включают оптимизацию моделей или минимизацию ошибок моделей. Конкретные примеры включают частные наименьшие квадраты, регрессию ансамбля, случайный лес, различные другие методы регрессии, а также методы нейронных сетей, рекурсивное секционирование, методы машин опорных векторов, CART (классификационные и регрессионные деревья) и/или тому подобное. Обычно метод должен давать модель, которая может отличать остатки, которые оказывают значительное воздействие на активность, от тех, которые не оказывают. В некоторых вариантах осуществления модели также ранжируют отдельные остатки или положения остатков на основании их воздействия на активность. Не предполагается, что настоящее изобретение ограничено каким-либо конкретным методом генерации модели, поскольку любой подходящий способ, известный в данной области техники, находит применение в настоящем изобретении.
В некоторых вариантах осуществления, включающих аддитивные модели, модели генерируют посредством регрессионного метода, который определяет ковариацию независимых и зависимых переменных в обучающем наборе. Различные регрессионные методы известны и широко используются. Примеры включают множественную линейную регрессию (MLR), регрессию на главные компоненты (PCR) и регрессию частных наименьших квадратов (PLS). В некоторых вариантах осуществления модели генерируют с использованием методов, которые включают множественные составляющие, включая, но без ограничения, регрессию ансамбля и случайный лес. Эти и любые другие подходящие способы находят применение в настоящем изобретении. Не предполагается, что настоящее изобретение ограничено каким-либо конкретным методом.
MLR является наиболее простым из этих методов. Ее используют для простого решения системы уравнений с коэффициентами для членов обучающего набора. Каждое уравнение относится к активности члена обучающего набора (то есть зависимых переменных) при присутствии или отсутствии конкретного остатка в конкретном положении (то есть независимых переменных). В зависимости от количества вариантов остатков в обучающем наборе число данных уравнений может быть достаточно большим.
Как и MLR, PLS и PCR генерируют модели из уравнений, связывающих активность последовательности со значениями остатков. Однако, данные методы осуществляют это другим образом. Сначала они осуществляют преобразование координат для уменьшения числа независимых переменных. Затем они выполняют регрессию в отношении преобразованных переменных. В MLR имеет место потенциально очень большое число независимых переменных: две или больше для каждого положения остатков, которые варьируют в обучающем наборе. При условии, что представляющие интерес белки и пептиды часто достаточно велики, и обучающий набор может предоставлять множество различных последовательностей, число независимых переменных может быстро стать очень большим. Посредством уменьшения числа переменных для того, чтобы сфокусироваться на тех, которые обеспечивают наибольшую вариацию в наборе данных, PLS и PCR обычно требуют меньше образцов и упрощают этапы, участвующие в генерации моделей.
PCR аналогична регрессии PLS в том, что фактическую регрессию осуществляют в отношении относительно небольшого числа латентных переменных, полученных посредством преобразования координат необработанных независимых переменных (то есть значений остатков). Отличие между PLS и PCR заключается в том, что латентные переменные в PCR конструируют посредством максимизации ковариации между независимыми переменными (то есть значениями остатков). В регрессии PLS латентные переменные конструируют таким образом, чтобы максимизировать ковариацию между независимыми переменными и зависимыми переменными (то есть значениями активности). Регрессия частных наименьших квадратов описана в документе Hand D.J. et al. (2001), Principles of Data Mining (Adaptive Computation and Machine Learning), Boston, MA, MIT Press, и в документе Geladi et al. (1986), "Partial Least-Squares Regression: a Tutorial", Analytica Chimica Acta, 198: 1-17. Обе эти ссылки включены в настоящий документ посредством ссылки для всех целей.
В PCR и PLS непосредственным результатом регрессионного анализа является выражение для активности, которая является функцией взвешенных латентных переменных. Это выражение может быть преобразовано в выражение для активности как функции исходных независимых переменных посредством осуществления преобразования координат, которое превращает латентные переменные обратно в исходные независимые переменные.
По существу как PCR, так и PLS сначала уменьшают размерность информации, содержащейся в обучающем наборе, и затем проводят регрессионный анализ на преобразованном наборе данных, который был преобразован для получения новых независимых переменных, но сохраняет исходные значения зависимых переменных. Преобразованные варианты наборов данных могут приводить всего лишь к относительно небольшому числу выражений для осуществления регрессионного анализа. В протоколах, в которых не осуществляют никакого уменьшения размерности, нужно учитывать каждый отдельный остаток, для которого может иметь место вариация. Это может представлять собой очень большой набор коэффициентов (например, 2N коэффициентов для двойных взаимодействий, где N представляет собой число положений остатков, которые могут варьироваться в обучающем наборе). В типичном анализе главных компонент используют только 3, 4, 5, 6 главных компонент.
Способность методов машинного обучения к подгонке обучающих данных часто называют "подгонкой модели", и в регрессионных методах, таких как MLR, PCR и PLS, подгонку модели, как правило, измеряют по сумме квадратов разностей между измеренными и предсказанными значениями. Для заданного обучающего набора оптимальная подгонка модели будет достигнута с использованием MLR, причем PCR и PLS часто имеют худшую подгонку модели (выше сумма квадратов ошибок между измерениями и предсказаниями). Однако главное преимущество использования регрессионных методов с латентными переменными, таких как PCR и PLS, заключается в предсказательной способности таких моделей. Получение подгонки модели с очень малой суммой квадратов ошибок никаким образом не гарантирует, что модель будет способна точно предсказывать новые образцы, отсутствующие в обучающем наборе, - фактически, часто имеет место противоположный случай, особенно когда существует много переменных, и только несколько наблюдений (то есть образцов). Таким образом регрессионные методы с латентными переменными (например, PCR, PLS), при том что они часто имеют худшие подгонки моделей на обучающих данных, обычно являются более робастными и способны к более точному предсказанию новых образцов за пределами обучающего набора.
Другим классом инструментов, которые можно использовать для генерации моделей в соответствии с настоящим раскрытием, являются машины опорных векторов (SVM). Эти математические инструменты принимают обучающие наборы последовательностей, которые классифицированы по двум или более группам на основании активности, в качестве входных данных. Машины опорных векторов работают посредством различного взвешивания различных членов обучающего набора в зависимости от того, насколько они близки к поверхности гиперплоскости, разделяющей "активные" и "неактивные" члены обучающего набора. Данный метод требует, чтобы исследователь вначале принял решение, какой из членов обучающего набора помещать в "активную" группу, и какой из членов обучающего набора помещать в "неактивную" группу. В некоторых вариантах осуществления это осуществляется посредством выбора соответствующего числового значения для уровня активности, который служит в качестве границы между "активными" и "неактивными" членами обучающего набора. Из этой классификации машина опорных векторов генерирует вектор W, который может предоставлять значения коэффициентов для отдельных независимых переменных, определяющих последовательности членов активной и неактивной групп в обучающем наборе. Данные коэффициенты можно использовать для "ранжирования" отдельных остатков, как описано в другом месте настоящего документа. Данный метод используют для того, чтобы идентифицировать гиперплоскость, которая максимизирует расстояние между ближайшими членами обучающего набора на противоположных сторонах этой плоскости. В другом варианте осуществления проводят регрессионное моделирование на опорных векторах. В данном случае зависимая переменная представляет собой вектор непрерывных значений активности. Регрессионная модель на опорных векторах генерирует вектор коэффициентов W, который можно использовать для ранжирования отдельных остатков.
SVM используют для проверки больших наборов данных во многих исследованиях, и они находят широкое применение с микроматрицами ДНК. Их потенциальные сильные стороны включают способность точно дискриминировать (посредством взвешивания) факторы, которые отделяют образцы друг от друга. До той степени, в которой SVM может точно выделять остатки, которые вносят вклад в функционирование, она может представлять собой особенно полезный инструмент для ранжирования остатков. SVM описаны в документе S. Gunn (1998), "Support Vector Machines for Classification and Regression", Technical Report, Faculty of Engineering and Applied Science, Department of Electronics and Computer Science, University of Southampton, который включен в настоящий документ посредством ссылки для всех целей.
В некоторых вариантах осуществления настоящего изобретения другим классом инструментов, который можно использовать для генерации моделей, является классификация и регрессия на основании ансамбля классификационных деревьев с использованием случайных входных данных, примером которой является случайный лес. См. Breiman (2001). Random Forests. Machine Learning 45 (1): 5-32. Случайные леса представляют собой комбинацию предикторов деревьев, такую, что каждое дерево зависит от значений случайного вектора, выбранных независимо и с одним и тем же распределением для всех деревьев в лесу. Случайный лес представляет собой обучающий ансамбль, состоящий из бэггинга неусеченных обучающих деревьев принятия решений с рандомизированным выбором признаков при каждом разветвлении дерева принятия решений. Ошибка обобщения для леса стремится к пределу, когда количество деревьев в лесу становится большим.
Случайные леса могут быть сконструированы следующим образом:
1) Если число случаев в обучающем наборе составляет N, выбирают N случаев случайным образом, но с возвращением из исходных данных. Данная выборка будет представлять собой обучающий набор для растущего дерева.
2) Если присутствуют M входных независимых переменных, определяют такое число m<<M, что в каждом узле дерева случайным образом выбирают m переменных из M, и используют для расщепления этого узла наилучшее расщепление на этих m. Значение m поддерживается постоянным во время роста леса.
3) В некоторых воплощениях каждое дерево растет насколько возможно большим. Усечение отсутствует.
4) Затем генерируют большое количество деревьев k=1, …, K (обычно K>=100).
5) После генерации большого количества деревьев все они голосуют для классифицирования представляющих интерес переменных. Например, каждое из них может вносить вклад в итоговое предсказание активности или вклада конкретных мутаций.
6) Затем случайный лес классифицирует × (например, последовательность мутаций или другую независимую переменную) посредством принятия определенного голосованием наиболее популярного класса из всех предикторов деревьев в лесу.
Частота ошибок леса зависит от корреляции между любыми двумя деревьями в лесу. Повышение корреляции повышает частоту ошибок леса. Частота ошибок леса зависит от мощности каждого отдельного дерева в лесу. Дерево с низкой частотой ошибок является сильным классификатором. Повышение мощности отдельных деревьев снижает частоту ошибок леса. Уменьшение m снижает как корреляцию, так и мощность. Ее увеличение повышает оба показателя. Где-то посередине расположен "оптимальный" диапазон m - обычно достаточно широкий.
Методы случайного леса можно использовать в регрессионных моделях для категориальных переменных, а также непрерывных переменных. В некоторых вариантах осуществления настоящего изобретения модели на основании случайного леса имеют предсказательную силу, сравнимую с моделями на основании SVM и нейронных сетей, но имеют тенденцию к более высокой вычислительной эффективности, поскольку, помимо прочего, перекрестная проверка встроена в процесс моделирования, и отсутствует необходимость в отдельном процессе перекрестной проверки.
A. Мультипликативные модели
Некоторые варианты осуществления настоящего изобретения предлагают способы построения моделей последовательность-активность, которые включают произведение множества мультипликативных членов, и использования данных моделей для управления направленной эволюцией. В некоторых вариантах осуществления модель последовательность-активность предсказывает активность варианта белка как функцию присутствия или отсутствия двух или более субъединиц последовательности. В некоторых вариантах осуществления субъединицы представляют собой аминокислоты, образующие вариант белка. В некоторых вариантах осуществления субъединицы представляют собой нуклеиновые кислоты или кодоны, которые кодируют варианты белков.
В некоторых вариантах осуществления по меньшей мере некоторые из мультипликативных членов модели представляют собой мультипликативные члены, не характеризующие взаимодействие, каждый из которых содержит коэффициент, представляющий вклад определенной аминокислоты или нуклеотида в представляющую интерес активность. Определенная аминокислота или нуклеотид имеет конкретный тип остатка в конкретном положении в белковой или нуклеиновокислотной последовательности. Каждый из мультипликативных членов, не характеризующих взаимодействие, также включает одну независимую переменную, или фиктивную переменную, которая представляет присутствие или отсутствие определенной аминокислоты или нуклеотида конкретного типа в конкретном местоположении последовательности. В различных вариантах осуществления фиктивная переменная может быть реализована в виде значений битов, таких как 1 для присутствия и 1/коэффициент для отсутствия определенного остатка. В некоторых вариантах осуществления вместо аминокислот или нуклеиновых кислот моделируют кодоны.
В некоторых вариантах осуществления настоящего изобретения модели последовательность-активность также включают члены, характеризующие взаимодействие, каждый из которых содержит коэффициент взаимодействия, представляющий вклад в активность определенной комбинации (i) первой аминокислоты или нуклеотида в первом положении в белковой последовательности, и (ii) второй аминокислоты или нуклеотида во втором положении в белковой последовательности.
Как объяснено выше, совокупность членов, характеризующих взаимодействие, обычно велика. Включение большого числа членов, характеризующих взаимодействие, отрицательно влияет на эффективность направленной эволюции. Дополнительные члены также имеют тенденцию вызывать переобучение данных, тем самым снижая предсказательную силу моделей. Мультипликативные модели могут быть способны уменьшать число членов, характеризующих взаимодействие, и улучшать предсказательной силу модели, поскольку множественные члены модели, не характеризующие взаимодействие, образуют произведение, что позволяет независимым переменным множественных членов, не характеризующих взаимодействие, вносить вклад в зависимую переменную мультипликативным образом. Без связи с какими-либо теориями, постулируемыми в настоящем документе, мультипликативные модели могут улучшать предсказательную силу моделей и/или эффективность направленной эволюции.
Вырождение кодона имеет место, когда два или более кодона кодируют одну и ту же аминокислоту. В некоторых вариантах осуществления вырождение кодона можно, необязательно, не учитывать посредством представления двух или более вырожденных кодонов с помощью одной и той же независимой переменной. Однако, вырождение кодона может также в некоторых вариантах осуществления сохраняться в модели. Например, модель может связывать две различные нуклеиновые кислоты с синонимичными мутациями с одной и той активностью белка. Причины того, что мультипликативная модель может адекватно отражать зависимость между активностью и информацией о белковой последовательности, можно продемонстрировать посредством использования уравнений Аррениуса для описания активности белка как функции различных мутаций в вариантном белке. Исходное уравнение Аррениуса
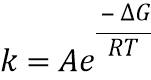
описывает константу скорости химический реакция k как функцию энергии активации ΔG и температуры реакции T (в кельвинах), причем A представляет собой предэкспоненциальный множитель (эмпирическую постоянную), и R представляет собой универсальную газовую постоянную, равную 8,314 Дж/моль⋅K. Уравнение Аррениуса может быть записано в виде:
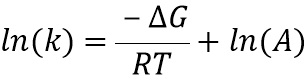
показывающем, что константа скорости реакции положительно коррелирует с температурой T и отрицательно коррелирует с энергией активации. Другими словами, более высокая температура реакции приводит к более быстрой реакции, а более высокая энергия активации приводит к более медленной реакции.
В принципе, разумно провести параллель между скоростью реакции химической реакции и активностью белка и между энергией активации и вкладом мутации в активность белка. На основании этого параллелизма уравнение Аррениуса можно использовать для описания активности белка k как функции воздействия мутации ΔG. Если остаток остова белка оказывает воздействие, равное ΔG0, мутация 1 оказывает воздействие, равное ΔG1, и мутация 2 воздействие, равное ΔG2, объединенное воздействие мутаций 1 и 2 может быть выражено в виде:
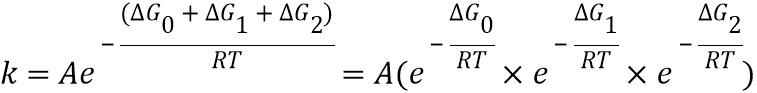
Как видно из данного уравнения, воздействия двух мутаций могут быть объединены в произведение в мультипликативной форме. В некоторых вариантах осуществления настоящего изобретения мультипликативные модели могут быть представлены в виде произведения различных членов. Каждый член представляет вклад в активность конкретной комбинаций положения в последовательности и конкретного остатка или нуклеотида/кодона в этом положении. Каждый из этих членов включает коэффициент и независимую переменную. Независимая переменная может принимать одно из двух значений: одно значение, когда рассматриваемая мутация присутствует, и другое значение, когда рассматриваемая мутация отсутствует.
В некоторых вариантах осуществления каждый член в модели последовательность-активность может представлять собой простое произведение его коэффициента и соответствующей независимой переменной. В некоторых вариантах осуществления настоящего изобретения, когда нуклеотид или остаток, соответствующий члену, присутствует, значение независимой переменной устанавливают равным 1. Когда независимая переменная не присутствует, значение переменной составляет единицу, деленную на значение коэффициента для этого члена, или 1/Cn. Для членов, характеризующих взаимодействие, коэффициент предоставляется для вклада взаимодействия в активность. Соответствующий коэффициент имеет значение, равное 1, когда все взаимодействующие члены (m и n) присутствуют, и значение, равное 1/Cm,n, когда они не присутствуют.
В другой форме модели в каждом члене независимой переменной дается значение, равное единице, когда рассматриваемый остаток или нуклеотид присутствует в данном положении, и нулю, когда он не присутствует в данном положении. В данной форме модели члену дается значение, равное единице плюс коэффициент, умноженный на независимую переменную: (1+Cnxn). Члены, характеризующие взаимодействие, рассматриваются аналогично первому случаю: (1+Cm,n xmxn). В одном варианте осуществления, модель может принимать такую форму:
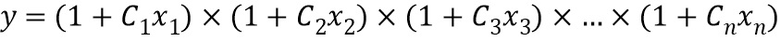
Члены, характеризующие взаимодействие, могут присутствовать или не присутствовать в модели. Возможен вариант, что в силу самой своей природы произведение учитывает взаимодействия между взаимодействующими остатками. В том случае, когда мультипликативная модель изначально не учитывает полный вклад таких взаимодействий, в мультипликативную модель в виде членов могут быть включены члены, характеризующие взаимодействие. В таком случае один или более членов, характеризующих взаимодействие, могут быть включены посредством включения коэффициентов, представляющих присутствие двух или более мутаций, и фиктивных переменных, представляющих две мутации:
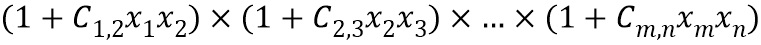
В некоторых вариантах осуществления члены, характеризующие взаимодействие, перемножают с членами, не характеризующими взаимодействие. В других вариантах осуществления члены, характеризующие взаимодействие, могут быть объединены посредством суммирования с членами, не характеризующими взаимодействие. В вышеуказанных вариантах осуществления члены, характеризующие взаимодействие, объединяются между собой посредством умножения. В альтернативных вариантах осуществления члены, характеризующие взаимодействие, могут быть объединены между собой посредством сложения.
Таблица I предлагает пример, который включает остов и варианты белка, имеющие 4 представляющих интерес положения: 10, 166, 175 и 340. Остатками остова являются Ala в 10, Phe в 166, Gly в 175, и Phe в 340. Активность белков измеряют как y для остова и yn для вариантов.
Иллюстративные данные об активности последовательности для учитывающих взаимодействие моделей*
Применяя мультипликативную модель в следующей форме к данному примеру
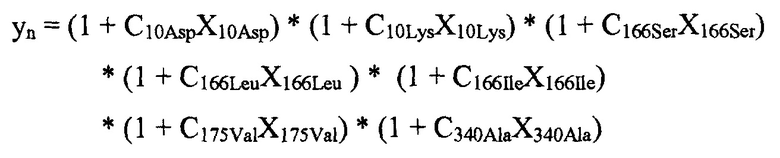
можно получить предсказываемую моделью активность варианта 2 следующим образом:
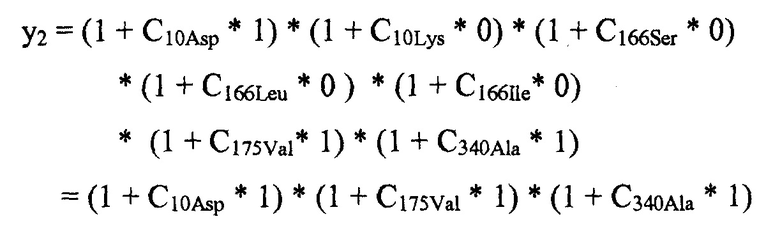
Хотя уравнение модели может содержать все аминокислоты варьируемых положениях, так что:
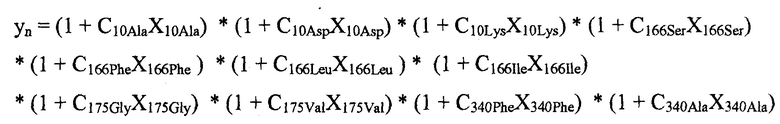
Члены в остовах часто выбрасывают из уравнения. Такой подход приемлем, поскольку часто способы подгоняют модель с использованием кратного улучшения вариантов (FIOP), так что коэффициенты аминокислот остова составляют 0 (так что y остова = 1).
В некоторых альтернативных вариантах осуществления другой подход заключается в использовании таблицы соответствия, в которой предусмотрены значения коэффициентов для всех без исключения комбинаций положения и соответствующих мутаций. Для предсказания активности для вариантной последовательности (коллекции мутаций) алгоритм просто идентифицирует те мутации, которые присутствуют в варианте, и берет соответствующие значения коэффициентов для этих мутаций из таблицы соответствия. Выбранные коэффициенты затем умножают друг на друга для получения предсказываемых значений активности, которые представляют собой зависимую переменную.
Фигура 3I-J показывает пример, в котором мультипликативная модель подгоняет наблюдаемые данные лучше, чем аддитивная модель, обеспечивая при некоторых условиях более высокую предсказательную силу. Наблюдаемые данные содержат варианты белков, имеющие мутации, которые имеют тенденцию к оказанию серьезного вредного воздействия на активность белка. Фигура 3I изображает предсказанные не учитывающей взаимодействие аддитивной моделью значения активности против наблюдаемых значений активности обучающего набора вариантов белков. Очевидно, что предсказания аддитивной модели страдают значительным занижением для наблюдений, которые имеют высокие значения активности. Напротив, предсказанные мультипликативной моделью значения активности лучше соответствуют наблюдаемым значениям активности, как показано на фигуре 3J, со значительно меньшими ошибками, чем аддитивная модель.
B. Линейные аддитивные модели
В некоторых вариантах осуществления линейная модель используется в качестве "основной" модели в пошаговом процессе генерации нелинейной модели. В общем линейная регрессионная модель активности в зависимости от последовательности имеет следующую форму:
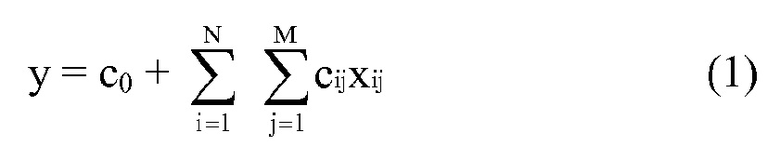
В данном линейном выражении y представляет собой предсказанную реакцию, тогда как cij и xij представляют собой коэффициент регрессии и значение бита или фиктивную переменную, используемую для представления выбора остатка, соответственно в положении i в последовательности. Существует N положений остатков в последовательностях библиотеки белковых вариантов, и каждое из них может быть занято одним или более остатками. В любом заданном положении может присутствовать j = от 1 до M отдельных типов остатков. Данная модель предполагает линейную (аддитивную) зависимость между остатками в каждом положении. Развернутый вариант уравнения 1 имеет следующий вид:
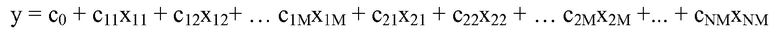
Как указано, данные в форме информации об активности и последовательности получают из исходной библиотеки белковых вариантов и используют для определения коэффициентов регрессии модели. Фиктивные переменные сначала определяют из выравнивания белковых вариантных последовательностей. Аминокислотные положения остатков идентифицируют из белковых вариантных последовательностей, в которых аминокислотные остатки в этих положениях различаются между последовательностями. Информация об аминокислотных остатках в некоторых или всех из этих положений варьируемых остатков может быть включена в модель последовательность-активность.
Таблица I содержит информацию о последовательности в форме положения варьируемых остатков и типы остатков для 10 иллюстративных вариантных белков вместе со значениями активности, соответствующими каждому вариантному белку. Они являются репрезентативными членами большего набора, который требуется для генерации достаточного количества уравнений для нахождения всех коэффициентов. Таким образом, например, для иллюстративных белковых вариантных последовательностей в таблице II положения 10, 166, 175 и 340 представляют собой положения вариабельных остатков, а все другие положения, то есть те, которые не указанные в таблице, содержат остатки, которые идентичны между вариантами 1-10.
В данном примере 10 вариантов могу включать или не включать последовательность остова дикого типа. В некоторых вариантах осуществления модель, разрабатываемая для учета данных всех вариантов, включая последовательность остова дикого типа, может создавать проблему совершенной мультиколлинеарности или ловушки фиктивных переменных. Эта проблема может решаться различными методами. Некоторые варианты осуществления могут исключать данные остова дикого типа из разработки модели. Некоторые варианты осуществления могут отбрасывать коэффициенты, представляющие остов дикого типа. Некоторые варианты осуществления могут использовать такие методы, как регрессия PLS, для рассмотрения мультиколлинеарности.
Иллюстративные данные о последовательности и активности
Таким образом, на основании уравнения 1, регрессионная модель может быть получена из систематически варьируемой библиотеки в таблице II, то есть:
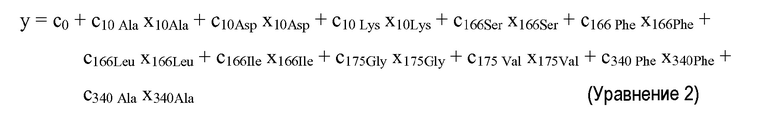
Значения битов (фиктивные переменные x) могут быть представлены как или 1, или 0, что отражает присутствие или отсутствие означенного аминокислотного остатка, или, альтернативно, как 1 или -1, или как некоторое другое суррогатное представление. Например, если использовать обозначение 1 или 0, x10Ala будет равно "1" для варианта 1 и "0" для варианта 2. Если использовать обозначение 1 или -1, x10Ala будет равно "1" для варианта 1 и "-1" для варианта 2. Коэффициенты регрессии могут, таким образом, быть получены из уравнений регрессии на основании информации о последовательности и активности для всех вариантов в библиотеке. Примеры таких уравнений для вариантов 1-10 (при использовании для x обозначения 1 или 0) имеют вид:



Полную систему уравнений можно легко решить с использованием любого подходящего регрессионного метода (например, PCR, PLS или MLR) для определения значения для коэффициентов регрессии, соответствующих каждому остатку и положению, представляющим интерес. В данном примере относительная величина коэффициента регрессии коррелирует с относительной величиной вклада данного конкретного остатка в конкретном положении в активность. Коэффициенты регрессии могут затем быть ранжированы или иным образом категоризированы для определения того, какие из остатков с наибольшей вероятностью должны успешно вносить вклад в желаемую активность. Таблица III предлагает иллюстративные значения коэффициентов регрессии, соответствующие систематически варьируемой библиотеке, приведенной в качестве примера в таблице II:
Иллюстративный ранговый порядок коэффициентов регрессии
Упорядоченный по рангу список коэффициентов регрессии можно использовать для конструирования новой библиотеки вариантов белков, которая оптимизирована в отношении желаемой активности (то есть улучшенной приспособленности). Это может быть осуществлено различными способами. В одном варианте осуществления это осуществляют посредством сохранения аминокислотных остатков, имеющих коэффициенты с наивысшими наблюдаемыми значениями. Они представляют собой остатки, на которые регрессионная модель указывает, как на вносящие наибольший вклад в желаемую активность. Если для идентифицирования остатков используют отрицательные дескрипторы (например, 1 для лейцина и -1 для глицина), становится необходимым ранжировать положения остатков на основании абсолютного значения коэффициента. Следует отметить, что в таких ситуациях имеет место, как правило, только один коэффициент для каждого остатка. Абсолютное значение величины коэффициента дает ранжирование соответствующих положений остатков. Затем становится необходимым рассмотреть знаки отдельных остатков, для того чтобы решить, является ли каждый из них вредным или полезным с точки зрения желаемой активности.
C. Нелинейные аддитивные модели
Нелинейное моделирование используют для учета взаимодействий остаток-остаток, которые вносят вклад в активность в белках. Данную проблему описывает N-K ландшафт. Параметр N относится к числу варьируемых остатков в коллекции соответствующих полипептидных последовательностей. Параметр K представляет взаимодействие между отдельными остатками в любом из данных полипептидов. Взаимодействие обычно является результатом тесной физической близости между различными остатками в первичной, вторичной или третичной структуре полипептида. Взаимодействие может быть обусловлено непосредственными взаимодействиями, косвенными взаимодействиями, физико-химическими взаимодействиями, взаимодействиями из-за промежуточных продуктов фолдинга, эффектами трансляции и тому подобным. См. Kauffman, S. and Levin, S. (1987), "Towards a general theory of adaptive walks on rugged landscapes", Journal of Theoretical Biology 128 (1), 11-45.
Параметр K определяют так, что для значения K=1 каждый варьируемый остаток (например, имеют место 20 остатков) взаимодействует ровно с одним другим остатком в своей последовательности. В случае, когда все остатки физически и химически изолированы от воздействий всех других остатков, значение K равно нулю. Очевидно, в зависимости от структуры полипептида K может принимать широкий диапазон различных значений. Если точно определена структура рассматриваемого полипептида, можно оценить значение K. Часто, однако, это не имеет места.
Строго линейная аддитивная модель активности полипептида (как описано выше) может быть улучшена посредством включения одного или более нелинейных членов, характеризующих взаимодействие, представляющих конкретные взаимодействия между 2 или более остатками. В контексте формы модели, представленной выше, эти члены описывают как "перекрестные произведения", содержащие две или более фиктивных переменных, представляющих два или более конкретных остатка (каждый соответствует конкретному положению в последовательности), которые взаимодействуют, оказывая значительное положительное или отрицательное воздействие на активность. Например, член перекрестного произведения может иметь форму cabxaxb, где xa представляет собой фиктивную переменную, представляющую присутствие конкретного остатка в конкретном положении в последовательности, а переменная xb представляет присутствие конкретного остатка в другом положении (которое взаимодействует с первым положением) в полипептидной последовательности. Подробный пример формы модели показан ниже.
Присутствие всех остатков, представленных в члене перекрестного произведения (то есть каждого из двух или более конкретных типов остатков в конкретно определенных положениях), воздействует на общую активность полипептида. Данное воздействие может проявляться многими способами. Например, каждый из отдельных взаимодействующих остатков, когда он один присутствует в полипептиде, может оказывать отрицательное воздействие на активность, но когда они присутствуют в полипептиде, общий эффект является положительным. В других случаях верным может оказаться обратное. Кроме того, может иметь место получаемый синергический эффект, при котором каждый из отдельных остатков в отдельности оказывает относительно ограниченное воздействие на активность, но когда все они присутствуют, эффект на активность больше, чем кумулятивные эффекты всех отдельных остатков.
В некоторых вариантах осуществления нелинейные модели включают член перекрестного произведения для всех возможных комбинаций взаимодействующих варьируемых остатков в последовательности. Однако это не представляет физическую реальность, поскольку только поднабор варьируемых остатков фактически взаимодействуют друг с другом. Кроме того, это будет приводить к "переобучению" с получением модели, которая предоставляет ложные результаты, которые являются проявлениями конкретных полипептидов, используемых для создания модели, и не представляет реальные взаимодействия в полипептиде. Правильное число членов перекрестного произведения для модели, которая представляет физическую реальность и избегает переобучения, определяется значением K. Например, если K=1, число членов перекрестного произведения, характеризующих взаимодействие, равно N.
При конструировании нелинейной модели в некоторых вариантах осуществления важно идентифицировать те члены перекрестного произведения, характеризующие взаимодействие, представляющие истинные структурные взаимодействия, которые оказывают значительное воздействие на активность. Это может быть осуществлено различными путями, включая, но без ограничения, прямое добавление, при котором члены-кандидаты перекрестного произведения добавляют к исходной чисто линейной модели по одному за один раз, до тех пор, пока добавление данных членов не перестанет быть статистически значимым, и обратное удаление, при котором все возможные члены перекрестного произведения предлагаются в исходной модели и удаляются по одному за один раз. Иллюстративные примеры, представленные ниже, включают применение методов пошагового добавления и удаления для того, чтобы идентифицировать полезные нелинейные члены.
В некоторых вариантах осуществления подход к генерации нелинейной модели, содержащей такие члены, характеризующие взаимодействие, является таким же, как подход, описанный выше для генерации линейной модели. Другими словами, используют обучающий набор для "подгонки" данных к модели. При этом к модели добавляют один или более нелинейных членов, предпочтительно членов перекрестного произведения, рассмотренных выше. Кроме того, получаемую нелинейную модель, как и линейные модели, описанные выше, можно использовать для ранжирования важности различных остатков для общей активности полипептида. Можно использовать различные методы, для того чтобы идентифицировать наилучшую комбинацию варьируемых остатков, предсказываемую нелинейным уравнением. Подходы к ранжированию остатков описаны ниже. В некоторых вариантах осуществления используются очень большие количества возможных членов перекрестного произведения для варьируемых остатков, даже при ограничении взаимодействиями, вызываемыми только двумя остатками. Если имеет место большее количество взаимодействий, число рассматриваемых потенциальных взаимодействий для нелинейной модели растет экспоненциальным образом. Если модель включает возможность взаимодействий, которые включают три или более остатка, число потенциальных членов растет даже более быстро.
В простом иллюстративном примере, в котором имеют место 20 варьируемых остатков, и K=1 (этим предполагается, что каждый варьируемый остаток взаимодействует с одним другим варьируемым остатком), в модели должно быть 20 членов, характеризующих взаимодействие (перекрестных произведений). Если имеет место любое меньшее число членов, характеризующих взаимодействие, модель не будет полностью описывать взаимодействия (хотя некоторые из взаимодействий могут не оказывать значительное воздействие на активность). Напротив, если имеет место любое большее число членов, характеризующих взаимодействие, модель может переобучить набор данных. В данном примере имеет место N*(N-1)/2 или 190 возможных пар взаимодействий. Нахождение комбинации из 20 уникальных пар, которые описывают 20 взаимодействий в последовательности, представляет собой значительную вычислительную проблему, поскольку существует приблизительно 5,48×1026 возможных комбинаций.
Можно использовать многочисленные методы, для того чтобы идентифицировать релевантные члены перекрестного произведения. В зависимости от величины проблемы и доступной вычислительной мощности можно исследовать все возможные комбинации и посредством этого идентифицировать одну модель, которая наилучшим образом соответствует данным. Однако часто проблема является требовательной к вычислениям. Таким образом, в некоторых вариантах осуществления используется эффективный алгоритм поиска или аппроксимация. Как указано в настоящем документе, одним подходящим методом поиска является пошаговый метод. Однако не предполагается, что настоящее изобретение ограничено каким-либо конкретным способом идентификации релевантных членов перекрестного произведения.
Ниже в таблице IV представлен иллюстративный пример, для того чтобы показать значение включения нелинейных членов перекрестного произведения в модель, предсказывающую активность по информации о последовательности. Данный пример представляет собой нелинейную модель, в которой предполагается, что существует только два варианта остатков в каждом варьируемом положении в последовательности. В данном примере белковую последовательность переводят в закодированную последовательность посредством использования фиктивных переменных, которые соответствуют выбору A или выбору B, используя +1 и -1, соответственно. Модель является иммунной к произвольному выбору того, какое из числовых значений используется для соответствия выбору каждого остатка. Варьируемые положения, показанные в первой строке таблицы IV, не указывают фактические положения в последовательности белковой последовательности. Вместо этого они представляют собой произвольные метки, представляющие все 10 гипотетических положений в белковой последовательности, которые можно варьировать в зависимости от одного из двух вариантов, показанных во второй и третьей строках таблицы IV выбора остатка A и выбора остатка B.
Пример кодирования остатков в положениях, каждое из которых имеет два варианта
При такой схеме кодирования линейная модель, используемая для связывания белковых последовательностей с активностью, может быть записана следующим образом:
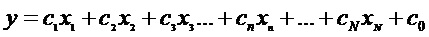
где y представляет собой реакцию (активность), cn коэффициент регрессии для выбора остатка в положении n, x фиктивную переменную, кодирующую выбор остатка (+1/-1) в положении n, и c0 среднее значение реакции. Данная форма модели предполагает, что отсутствуют взаимодействия между варьируемыми остатками (то есть выбор каждого остатка независимо вносит вклад в общую приспособленность белка).
Нелинейная модель включает некоторое количество (еще не определенных) членов перекрестного произведения для учета взаимодействий между остатками:
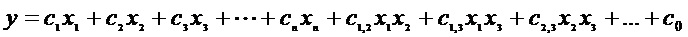
где переменные являются теми же, что и в уравнении (3), но теперь присутствуют нелинейные члены, например, c12 представляет собой коэффициент регрессии для взаимодействия между варьируемыми положениями 1 и 2.
Для того чтобы определить эффективность линейной и нелинейной моделей, использовали синтетический источник данных, известный как NK-ландшафт (Kauffman and Levin, 1987). Как упомянуто выше, N представляет собой число варьируемых положений в моделируемом белке, а K представляет собой эпистатическое парное взаимодействие между остатками. Кроме того, генерировали in silico синтетический набор данных.
Этот набор данных использовали для генерации исходного обучающего набора с S=40 синтетическими образцами, с N=20 варьируемыми положениями и K=1 (повторимся, что для K=1 каждое варьируемое положение функционально связано с одним другим варьируемым положением). При генерации рандомизированных белков каждое варьируемое положение имело равную вероятность получения фиктивной переменной +1 или -1. Взаимодействия остаток-остаток (представляемые перекрестными произведениями) и фактические активности были известны для каждого члена синтетического обучающего набора. Другие V=100 образцов генерировали для использования при проверке. Взаимодействия остаток-остаток и активности также были известны для каждого члена контрольного набора.
Обучающие наборы использовали для конструирования как линейных, так и нелинейных моделей. Некоторые нелинейные модели генерировали с выбором членов перекрестного произведения, а другие нелинейные модели генерировали без выбора таких членов. Модели для фигуры 3A-F генерировали с использованием способа моделирования генетического алгоритма, тогда как модели для фигуры G-H генерировали с использованием пошагового способа моделирования. Хотя количественное преимущество моделей, содержащих как линейные, так и нелинейные члены, относительно моделей, содержащих только линейные члены, отличается между способами генетического алгоритма и пошагового моделирования, результаты показывают обобщаемое преимущество моделей с нелинейными членами вне зависимости от способов моделирования. Более того, не предполагается, что настоящее изобретение ограничено какими-либо конкретными способами, поскольку любые подходящие способы моделирования находят применение в настоящем изобретении.
Для размера обучающего набора S=40, описанного выше, линейная модель оказалась способна к довольно хорошей корреляции измеренных и предсказанных значений, но продемонстрировала более слабую корреляцию при проверке данными, отсутствовавшими в обучающем наборе (см. фигуру 3A). Как показано, темные точки данных представляют наблюдаемую активность 40 обучающих точек данных против предсказаний, сделанных линейной моделью. Светлые точки данных представляют предсказания, сделанные той же моделью, сконструированной из 40 обучающих образцов и используемой для предсказания контрольных образцов V, ни один из которых не присутствовал в исходном обучающем наборе. Контрольный набор обеспечивает хорошую меру истинной предсказательной способности модели, в противоположность обучающему набору, который может испытывать проблемы из-за переобучения модели, особенно в нелинейных случаях, описанных ниже.
Этот результат для обучающего набора S=40, описанного выше, примечателен, учитывая, что линейную модель использовали для моделирования нелинейного ландшафта приспособленности. В данном случае линейная модель может, в лучшем случае, зафиксировать средний вклад в приспособленность для выбора заданного остатка. Если задано достаточное число средних вкладов, учитываемых в комбинации, линейная модель грубо предсказывает фактическую измеренную реакцию. Результаты проверки для линейной модели были несколько лучше, когда размер обучения был увеличен до S=100 (см. фигуру 3B). Тенденция относительно простых моделей к недообучению данных известна как "смещение".
Когда нелинейную модель обучали с использованием только S=40 образцов, корреляция с членами обучающего набора была отличной (см. фигуру 3C). К сожалению, в данном иллюстративном примере модель обеспечивала ограниченную предсказательную силу за пределами обучающего набора, о чем свидетельствует ее ограниченная корреляция с измеренными значениями в контрольном наборе. Данная нелинейная модель с множеством потенциальных переменных (возможно 210) и ограниченными обучающими данными для облегчения идентификации соответствующих членов перекрестного произведения оказалась способна по существу лишь запомнить набор данных, на котором ее обучали. Данная тенденция моделей с высокой сложностью к переобучению данных известна как "дисперсия". Компромисс смещение-дисперсия представляет фундаментальную проблему в машинном обучении, и почти всегда требуются некоторые формы проверки для решения связанных с ним вопросов при работе с новыми или не охарактеризованными проблемами машинного обучения.
Тем не менее, когда нелинейную модель обучали с использованием большего обучающего набора (S=100), как показано на фигуре 3D, нелинейная модель работала чрезвычайно хорошо как для обучающего предсказания, так и, что важнее, контрольного предсказания. Контрольные предсказания были достаточно точными для того, чтобы большинство точек данных оказались закрыты темными кружками, используемыми для нанесения обучающего набора.
Для сравнения, фигуры 3E и 3F показывают эффективность нелинейных моделей, полученных без тщательного выбора членов перекрестного произведения. В отличие от моделей на фигурах 3C и 3D был выбран каждый возможный член перекрестного произведения (то есть 190 членов перекрестного произведения для N=20). Как показано на данных фигурах, способность к предсказанию активности контрольного набора оказалась относительно плохой по сравнению с нелинейными моделями, генерируемыми с тщательным выбором членов перекрестного произведения. Эта плохая способность к предсказанию контрольных данных является проявлением переобучения.
Фигуры 3G и 3H соответственно показывают предсказательную силу, указанную по остаткам, линейной модели и пошаговой нелинейной модели для данных, смоделированных in silico. Пошаговую нелинейную модель реализовали, как в целом описано выше и, более конкретно, ниже.
Для тестирования этих моделей создали моделированные данные. Был создан генератор случайных чисел R на основании нормального распределения со средним MN и стандартным отклонением SD. Затем был определен набор из 10 мутаций. Они были названы M1, M2…M10 (данная схема наименования является произвольной). Данный этап моделирует создание разнообразия.
Каждая мутация представляла замену аминокислоты в заданном положении в белковой последовательности, и каждое положение является независимым от других положений. Каждая вышеуказанная мутация имеет случайное значение активности A, присваиваемое на основании R (MN=0, SD=0,2). Шесть вышеуказанных мутаций выбрали и попарно объединили в три пары P. Данные пары представляли эпистатические взаимодействия между мутациями.
Значение активности AP присваивали каждой паре P на основании R (MN=0, SD=0,2). Сконструировали библиотеку L из 50 вариантов, в которой каждый вариант содержал случайное количество мутаций M, определенное выше - случайное количество мутаций определяли по округленному абсолютному значению R (MN=4, S=0,25). Данный этап моделирует конструирование библиотеки и секвенирование.
Активность каждого варианта в L вычисляли, сначала добавляя к 1,0 (определенная активность дикого типа, последовательность без мутаций) значение активности от каждой парной мутации PA (если присутствовали обе мутации), а затем добавляя значения оставшихся одиночных мутаций (A). Шум анализа моделировали посредством добавления к итоговому значению для каждого варианта случайного значения из R (MN=0, SD=0,005). Данный этап моделирует скрининг вариантов.
Линейную модель LM конструировали на основании данных от последнего этапа. Эта модель содержала десять независимых переменных/коэффициентов, причем каждый представлял одну мутацию M. Затем линейную модель подгоняли с использованием обычной регрессии наименьших квадратов и данных, полученных выше.
Для выбора модели MM на основании данных, полученных выше, использовали способ пошагового добавления, причем основной моделью являлась LM, используя AIC в качестве критерия выбора и выбирая только те модели, которые содержат коэффициенты, представляющие одиночные мутации и попарные взаимодействия. Для дополнительных подробностей о способе выбора модели см. описание выбора модели ниже. Наилучшую модель, выбранную с помощью AIC, подгоняли с использованием обычной регрессии наименьших квадратов.
Для определения предсказательной способности линейной модели и нелинейной модели процедуры, описанные выше, повторяли 20 раз. Предсказание моделей изображали на диаграмме против моделируемых данных, причем фигура 3G показывает линейную модель, а фигура 3H показывает пошаговую нелинейную модель. Модели использовали для предсказания значений одиночных мутаций, описанных выше. Это предсказание осуществляли посредством использования моделей для предсказания варианта, содержащего только одну представляющую интерес мутацию, и удаления 1,0 (дикого типа). Как очевидно из фигур 3G и 3H, нелинейная модель более точно предсказывает значения, причем она имеет более линейный тренд и меньшие остатки.
D. Выбор модели
После генерации основной модели последовательность-активность способ уточняет модель посредством корректировки значений коэффициентов в членах моделей для минимизации остаточной ошибки между предсказанием модели и наблюдаемыми данными. См. блок 107. Такой тип коррекции также называется подгонкой модели. Могут быть использованы различные способы подгонки моделей, известные в данной области техники. Например, для того чтобы корректировать значения коэффициентов, можно использовать генетический алгоритм. Для аддитивных моделей можно использовать для подгонки модели различные методы регрессии.
В некоторых вариантах осуществления настоящего изобретения способ также уточняет модель посредством выбора подходящих членов для включения в модель или исключения из нее таким образом, чтобы минимизировать остаточные ошибки и/или улучшить предсказательную силу модели. См. блок 107. Поскольку все члены рассматриваемых моделей выбраны из одной и той же совокупности членов, данный процесс уточнения также известен как выбор модели среди вложенных моделей. Некоторые варианты осуществления настоящего изобретения используют для выбора подходящих членов генетический алгоритм. Дополнительно или альтернативно, некоторые варианты осуществления настоящего изобретения итерационно добавляют или удаляют члены, характеризующие взаимодействие, из совокупности доступных членов, характеризующих взаимодействие, к или из основной модели и оценивают получаемые новые модели в отношении улучшения по сравнению с основной моделью для получения итоговой модели.
Как корректировка значений коэффициентов модели, так и выбор членов модели для улучшения предсказательной силы моделей известны как методы оптимизации моделей. Примеры алгоритмов для генерации моделей последовательность-активность в соответствии с операциями в блоках 105 и 107 представлены ниже. Такие методы включают, но без ограничения, генетический алгоритм и пошаговые методы со смещением от включения дополнительных членов, характеризующих взаимодействие, в модель. Однако не предполагается, что настоящее раскрытие ограничено данными конкретными примерами.
Генетический алгоритм
Некоторые варианты осуществления настоящего изобретения предлагают способы использования генетического алгоритма для выбора одного или более членов моделей последовательность-активность. Другие варианты осуществления предлагают способы использования генетического алгоритма для того, чтобы корректировать значения коэффициентов для подгонки моделей к полученным данным.
В генетическом алгоритме определяют соответствующую функцию приспособленности и соответствующую процедуру скрещивания. Функция приспособленности дает критерий для определения того, какие из моделей (комбинаций членов перекрестного произведения) являются "наиболее приспособленными" или имеют наивысшую предсказательную силу (то есть, вероятно, обеспечивают наилучшие результаты). В некоторых вариантах осуществления алгоритм предоставляет механизм поиска в пространствах параметров для нахождения оптимальных значений параметров (то есть коэффициентов для моделей последовательность-активность). В данных вариантах осуществления каждый из индивидуумов или хромосом популяции включает гены, представляющие все тестируемые коэффициенты, и для гена выбирают значения в определенных диапазонах коэффициентов. Например, хромосома может содержать ген, представляющий коэффициент для Gly в положении 131, имеющий значение, равное 0,4.
В некоторых вариантах осуществления генетический алгоритм можно также использовать для выбора подходящих членов для моделей. Один пример такого алгоритма может быть аналогичен предыдущему примеру, за исключением того, что все индивидуумы/хромосомы включают все гены, представляющие все приемлемые параметры (коэффициенты), и значениям генов разрешено принимать значение, равное 0. Если коэффициент для члена стремится к 0 среди наиболее приспособленных индивидуумов в конце алгоритма, то член исключают из модели. В ином случае этот член сохраняют.
В других вариантах осуществления процедура скрещивания обеспечивает механизм для введения новых комбинаций членов, характеризующих взаимодействие, из успешных "родительских" моделей предыдущего поколения.
Один пример генетического алгоритма для подгонки модели к данным посредством корректировки значений коэффициентов и, необязательно, выбора подходящих членов для максимизации предсказательной силы модели. Данный пример описан со ссылкой на фигуру 1B. Данный алгоритм начинается с первого поколения, содержащего множественные возможные модели, причем все они содержат переменные значения коэффициентов, причем некоторые модели лучше выполняют работу по представлению физической реальности, чем другие. См. блок 131. Первое и каждое последующее поколение представлено в виде числа моделей (также называемых индивидуумами или хромосомами) в "популяции". Каждая модель/хромосома включает гены, представляющие значения коэффициентов всех членов, тестируемых в данном поколении. Генетический алгоритм продолжается в направлении схождения посредством прохождения через последовательные поколения моделей, каждое из которых характеризуется различными наборами значений для коэффициентов членов модели последовательность-активность.
Для обучающего набора полипептидов (имеющих известные последовательности и соответствующие активности) вычисляют приспособленность каждой модели в поколении. См. блоки 133, 135, 141 и 143 на фигуре 2. В некоторых вариантах осуществления приспособленность измеряют по среднеквадратичной ошибке. В других вариантах осуществления приспособленность измеряют по правдоподобию. В дополнительных вариантах осуществления приспособленность измеряют посредством AIC или BIC.
После оценки каждой комбинации членов в конкретном поколении по ее предсказательной силе (то есть ответ решения 143 отрицателен) генетический алгоритм проверяют на сходимость или другие критерии (такие как фиксированное число поколений) для определения того, следует ли продолжать процесс для следующего поколения. См. блок 145. В случае, если генетический алгоритм еще не удовлетворяет критерию остановки, модели текущего поколения ранжируют. Те, которые наилучшим образом выполняют работу по предсказанию активности, могут быть сохранены и использованы в следующем поколении. См. блок 147. Например, можно использовать уровень элитизма, равный 10%. Другими словами, лучшие 10% моделей (как определено с использованием функции приспособленности и измерено с помощью, например, среднеквадратичной ошибки или AIC) выделяют для того, чтобы они стали членами следующего поколения. Остальные 90% членов в следующем поколении получают посредством скрещивания "родителей" из предыдущего поколения. См. блоки 149, 151 и 153.
Как указано, "родители" являются моделями, выбранными из предыдущего поколения. См. блок 149. Обычно выбор является взвешенным в пользу более приспособленных членов предыдущего поколения, хотя в их выборе может иметь место случайный компонент. Например, родительские модели могут быть выбраны с использованием линейного взвешивания (например, модель, которая работает в 1,2 раза лучше, чем другая модель, имеет на 20% большую вероятность быть выбранной) или геометрического взвешивания (то есть для того, чтобы получить вероятность выбора, предсказательные различия моделей возводят в степень). В некоторых вариантах осуществления родителей выбирают просто посредством выбора двух или более наиболее эффективных моделей из ранжирования моделей в предыдущем поколении, а другие модели не выбирают. В данных вариантах осуществления все выбранные модели из предыдущего поколения скрещивают. В других вариантах осуществления некоторые модели из предыдущего поколения выбирают для включения в модель следующего поколения без скрещивания, а другие менее эффективные модели из предыдущего поколения случайным образом выбирают в качестве родителей. Эти родители могу быть скрещены друг с другом и/или с наиболее эффективными моделями, выбранными для включения, как они есть в следующее поколение.
После выбора набора родительских моделей пары таких моделей скрещивают (блок 151) для получения дочерних моделей посредством предоставления некоторых генов (значений коэффициентов) от одного родителя, а других значений коэффициентов от другого родителя. В одном подходе коэффициенты двух родителей выравнивают, и каждое значение рассматривают последовательно для определения того, следует ли ребенку принимать член от родителя A или от родителя B. В одном воплощении процесс скрещивания начинается с родителя A и случайным образом определяет, должно ли происходить событие "кроссинговера" в первом встретившемся члене. Если это так, член берется от родителя B. Если нет, член берется от родителя A. Следующий член по порядку рассматривается в отношении кроссинговера, и так далее. Члены продолжают поступать от родителя, давшего предыдущий рассмотренный член, до тех пор, пока не произойдет событие кроссинговера. В этот момент следующий член дается другим родителем, и все последующие члены даются этим родителем до тех пор, пока не произойдет другое событие кроссинговера. Для обеспечения того, чтобы один и тот же член не был выбран в два различных местоположения в дочерней модели, могут быть использованы различные методы, например метод частично соответствующего кроссинговера. В некоторых вариантах осуществления вместо использования значений коэффициентов генов от любого из родителей для дочерней хромосомы может быть принято среднее значений гена.
В некоторых вариантах осуществления настоящего изобретения генетический алгоритм также использует один или более механизмов мутаций для генерации дополнительного разнообразия моделей (блок 152), что помогает в исследовании областей пространства параметров, которые не покрываются никакими существующими генами в родительском поколении. С другой стороны, механизмы мутаций влияют на сходимость, так что чем выше скорость мутаций или больше диапазон мутаций, тем больше времени потребуется для сходимости (если она вообще будет иметь место). В некоторых вариантах осуществления мутацию реализовывают с помощью случайного выбора хромосомы/модели и случайного выбора коэффициента упомянутой хромосомы, который затем случайным образом изменяется. В некоторых вариантах осуществления случайным образом измененные значения коэффициентов выводят из случайного равномерного распределения с определенной широтой. В других вариантах осуществления случайным образом измененные значения коэффициентов выводят из случайного нормального распределения с определенной широтой.
После рассмотрения каждого члена, характеризующего взаимодействие, определяют дочернюю "модель" для следующего поколения. Затем выбирают два других родителя для получения другой дочерней модели и так далее. В конце концов, после такого выбора всего поколения (блок 153) следующее поколение готово для оценивания, и тогда управление способом возвращается к блоку 133, где оценивают члены следующего поколения, как описано выше.
Способ продолжается поколение за поколением до тех пор, пока не будет выполнен критерий остановки (то есть решение блока 145 станет положительным). В этот момент по меньшей мере одну из высоко ранжированных моделей выбирают из текущего поколения в качестве общей наилучшей модели. См. блок 155. Сходимость может быть проверена множеством обычных методов. Как правило, они включают определение того, что эффективность наилучшей модели из ряда последовательных поколений заметно не изменяется. Примеры критериев принятия решений или сходимости включают, но без ограничения, число поколений, сгенерированных на данный момент, активность наилучших белков из текущей библиотеки, желаемую величину активности и уровень улучшения, наблюдаемого в последнем поколении моделей.
Далее представлен пример, для того чтобы продемонстрировать применение генетического алгоритма для уточнения модели посредством корректировки значений коэффициента модели. Для каждой модели коэффициенты выбирают для каждой возможной мутации (то есть для каждого члена модели). Значения коэффициентов в начальной популяции случайным образом выбирают в пределах некоторых максимальных и минимальных границ. Затем каждую модель используют для предсказания активности по каждой вариантной последовательности в обучающем наборе вариантов. Для заданной модели все вычисленные значения активности сравнивают с наблюдаемыми значениями активности и генерируют остаток среднеквадратичной ошибки. Данную процедуру проводят для каждой из случайным образом сгенерированных моделей в первом поколении. Те модели, которые имеют наименьшую остаточную ошибку, выбирают для включения в следующее поколение.
Хромосомы генетического алгоритма для последовательности из таблицы I
Для данного примера генетический алгоритм оценивает приспособленность каждой хромосомы с использованием уравнения модели для вычисления ожидаемой активности для отдельной хромосомы.
Для хромосомы/модели 2 ожидаемая активность y=(1+1,15)* * (1-0,001)* * (1+1,0). Данную ожидаемую активность модели сравнивают с наблюдаемой активностью последовательности, и приспособленность индивидуума вычисляют по среднеквадратичной ошибке (MSE).
Затем генетический алгоритм выбирает 20% лучших моделей, которые имеют наименьшую MSE в текущем поколении. Как правило, следующее поколение включает скрещивание выбранных моделей из предыдущего поколения. Скрещивание может просто включать выбор некоторых коэффициентов от одной "родительской" модели и остальных коэффициентов от другого "родителя". Выбор членов может быть проведен как "кроссинговер" или другая генетическая операция.
Затем все модели следующего поколения (получаемые скрещиванием) используют для предсказания активности в обучающем наборе вариантов. Предсказанные активности сравнивают с наблюдаемыми значениями активности и генерируют остаточные ошибки. Модели второго поколения, имеющие наилучшую активность, выбирают для следующего поколения скрещивания и отбора. Данный процесс продолжается до тех пор, пока эффективность моделей сходится. Например, генетический алгоритм выполняется для 50-100 поколений. В этот момент по меньшей мере одну из высоко ранжированных моделей выбирают из текущего поколения в качестве общей наилучшей модели. Наиболее приспособленная модель определяет значения коэффициентов для модели последовательность-активность.
Пошаговый выбор
В некоторых вариантах осуществления для получения моделей с членами, характеризующими взаимодействие, используют способы пошагового добавления или удаления. Посредством реализации операции, показанной в блоке 107 фигуры 1, итоговая модель с высокой предсказательной силой, включающая члены, характеризующие взаимодействие, обеспечивается с помощью пошагового добавления или удаления членов, характеризующих взаимодействие, из основной модели.
Фигура 4A предлагает блок-схему реализации операции блока 107 фигуры 1 посредством добавления членов, характеризующих взаимодействие, к основной модели и оценивания новой модели для создания итоговой наилучшей модели.
В данном примере основная модель последовательности не включает члены, характеризующие взаимодействие. Способ вначале принимает в качестве текущей модели последовательности и наилучшей модели последовательности основную модель последовательности, блок 409. Способ определяет совокупность членов, характеризующих взаимодействие, для вариантов последовательностей. Данные члены, характеризующие взаимодействие, могут включать любое число парных или более высокого порядка взаимодействий двух или более аминокислотных остатков. См. блок 411. Хотя блок 409 проиллюстрирован как имеющий место до блока 411, порядок этих двух этапов не важен. В некоторых вариантах осуществления совокупность членов, характеризующих взаимодействие, включает факторные комбинации всех представляющих интерес аминокислотных остатков. В некоторых дополнительных вариантах осуществления включены по меньшей мере все члены, характеризующие парное взаимодействие. В некоторых дополнительных вариантах осуществления включены парные и тройные члены, характеризующие взаимодействие.
После создания основной модели способ выбирает из совокупности член, характеризующий взаимодействие, который еще не был протестирован. Затем способ создает новую модель последовательности посредством добавления выбранного члена, характеризующего взаимодействие, к текущей модели последовательности. См. блок 413. Затем способ оценивает предсказательную силу новой модели последовательности с использованием способа выбора модели, имеющего смещение в сторону от включения дополнительных членов, характеризующих взаимодействие. См. блок 415. Способ определяет, больше ли предсказательная сила новой модели последовательности, чем наилучшей модели последовательности, или нет. См. блок принятия решения 417. В качестве примере, способ может применять метод, использующий определение "правдоподобия" (например, AIC) в качестве критерия выбора модели. В таких случаях только модель, имеющая значение AIC, меньшее, чем тестированная ранее модель, рассматривается как имеющая более высокую предсказательную силу.
В некоторых вариантах осуществления способ выбора смещен в сторону от моделей с большим количеством параметров. Примеры таких способов выбора включают, но без ограничения, информационный критерий Акаике (AIC) и байесовский информационный критерий (BIC) и их варианты. Например, AIC может быть вычислен как:
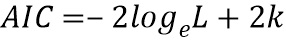
где L представляет собой правдоподобие модели при заданном наборе данных, а k представляет собой число свободных параметров в модели.
В некоторых вариантах осуществления правдоподобие модели при заданном наборе данных может быть вычислено различными способами, включая, но без ограничения, способ максимального правдоподобия. Например, для бинарной зависимой переменной, где активность или присутствует, или отсутствует для одного наблюдения, правдоподобие модели может быть вычислено как:
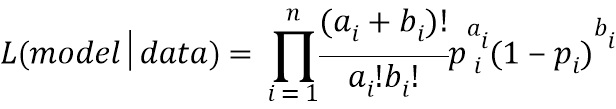
где n представляет собой полное число точек данных в наборе данных; ai и bi представляют собой число наблюдаемых испытаний, содержащих i-е условие; p представляют собой вероятность того, что зависимая переменная будет наблюдаться, как предсказано моделью.
В некоторых вариантах осуществления, включающих ряд вложенных моделей, как в регрессионных моделях с прогрессивно большим количеством членов, характеризующих взаимодействие (и соответствующих коэффициентов), чем в основной модели, более сложные модели обеспечивают столь же хорошие или лучшие подгонки, чем более простые, даже если дополнительные коэффициенты являются ложными, поскольку более сложная модель имеет дополнительные степени свободы. В некоторых вариантах осуществления AIC штрафует более сложную модель таким образом, чтобы выигрыш в качестве подгонки был больше, чем смещение из-за стоимости ложных параметров. При выборе модели меньшее значение AIC указывает лучшую модель.
В примере, показанном на фигуре 4A, если предсказательная сила новой модели последовательности больше, чем наилучшей модели последовательности, то способ устанавливает новую модель в качестве наилучшей модели. См. блок 419. Затем способ проверяет, остались ли какие-либо дополнительные члены, характеризующие взаимодействие, в совокупности, которые не были протестированы. См. блок принятия решения 421. Если это так, способ возвращается назад к блоку 413, что образует внутренний цикл, для тестирования всех доступных членов, характеризующих взаимодействие, доступных в совокупности, учитывающей взаимодействие. Посредством итераций внутреннего цикла может быть найден и добавлен в модель один наилучший член, характеризующий взаимодействие.
После того как все члены, характеризующие взаимодействие, были протестированы и внутренний цикл закончился, идентифицируют наилучшую модель, имеющую один дополнительный член, характеризующий взаимодействие, при условии, что модель, имеющая большую предсказательную силу, чем предыдущая наилучшая модель, действительно существует. См. блок принятия решения 423. В таких вариантах осуществления способ принимает текущую модель в качестве наилучшей модели и исключает члены наилучшей модели, характеризующие взаимодействие, из доступной совокупности членов, характеризующих взаимодействие. См. блок 425. Затем, способ возвращается назад к блоку 413. Данный внешний цикл осуществляет поиск следующего наилучшего члена, характеризующего взаимодействие, который может улучшить предсказательную силу модели. Если такой член, характеризующий взаимодействие, обнаружен, поиск следующего наилучшего члена, характеризующего взаимодействие, продолжается во внешнем цикле до тех пор, пока не перестанут быть идентифицированы новые модели, имеющие предсказательную силу, большую, чем у предыдущей наилучшей модели последовательности.
Когда больше не могут быть обнаружены члены, характеризующие взаимодействие, улучшающие модель, способ принимает наилучшую модель в качестве итоговой модели. См. блок 427. Поиск наилучшей модели при заданных данных о последовательности и активности заканчивается. Затем модель используют для предсказания активности новых последовательностей. Такие предсказания могут управлять выбором последовательностей для дополнительного варьирования и тестирования.
В определенных вариантах осуществления каждый из доступных членов, характеризующих взаимодействие, в совокупности членов, характеризующих взаимодействие, рассматривают как оказывающий потенциально равное воздействие на качество или предсказательную силу модели. Другими словами, при воплощении, каждый из доступных в совокупности членов, характеризующих взаимодействие, имеет равную вероятность быть выбранным для рассмотрения во время конкретной итерации. В некоторых вариантах осуществления доступные члены, характеризующие взаимодействие, выбирают случайным образом или в некотором произвольном порядке. В некоторых других вариантах осуществления члены, характеризующие взаимодействие, смещают или взвешивают таким образом, что некоторые члены имеют большую вероятность быть выбранными во время заданной итерации для рассмотрения, чем другие. Смещение или взвешивание могут, в определенных вариантах осуществления, быть применены на основании физической или теоретической информации о взаимодействиях. Например, может быть известно, что мутации в двух определенных областях белка являются, вероятно, физически близкими друг к другу и в связи с этим взаимодействуют. Члены, характеризующие взаимодействие, относящиеся к остаткам в этих двух общих областях могут быть смещены для выбора во время итерационного процесса уточнения модели.
Псевдокод, иллюстрирующий способы, аналогичные способу с фигуры 4A, имеет следующий вид:


Элемент 1 добавляет тестируемый член, характеризующий взаимодействие, в регрессионную модель.
Элемент 2 представляет сравнение моделей, один или более из информационного критерия Акаике (AIC), байесовского информационного критерия (BIC), перекрестной проверки (средней ошибки), ANOVA или вклада коэффициентов.
Элемент 3 предусмотрен для того, чтобы избежать повторных тестов членов, характеризующих взаимодействие.
Фигура 4B предлагает блок-схему, показывающую вариант осуществления операции, показанной в блоке 107 фигуры 1. В данном процессе члены, характеризующие взаимодействие, удаляют из основной модели, которая включает все возможные члены, характеризующие взаимодействие, из совокупности таких членов, для создания итоговой наилучшей модели.
В данном варианте осуществления основная модель последовательности включает все члены, характеризующие взаимодействие, в пределах определенной совокупности. Способ вначале принимает, что текущая модель последовательности и наилучшая модель последовательности равны основной модели последовательности в начале процесса, блок 439. Данный вариант осуществления аналогичен последней модели, описанной выше, в том, что полная совокупность членов, характеризующих взаимодействие, может включать любое число парных или более высокого порядка взаимодействий двух или более аминокислотных остатков. В некоторых вариантах осуществления совокупность членов, характеризующих взаимодействие, включает факторные комбинации всех аминокислотных остатков, которые представляют интерес.
После создания основной модели способ выбирает член, характеризующий взаимодействие, который еще не был протестирован, из совокупности членов, уже включенных в основную модель. Затем способ создает новую модель последовательности посредством удаления выбранного члена, характеризующего взаимодействие, из текущей модели последовательности. См. блок 441. Затем способ оценивает предсказательную силу новой модели последовательности, используя способ выбора модели, имеющий смещение в сторону от дополнительных членов, характеризующих взаимодействие. См. блок 443. Способ оценивает, больше ли предсказательная сила новой модели последовательности, чем наилучшей модели последовательности, или нет. См. операцию принятия решения, показанную в блоке 445. В некоторых вариантах осуществления в качестве критерия выбора модель используется AIC, так что модель, имеющая значение AIC, меньшее, чем у ранее протестированной модели, рассматривается как имеющая более высокую предсказательную силу.
В данном иллюстративном примере, если предсказательная сила новой модели последовательности больше, чем у наилучшей модели последовательности, то способ принимает новую модель в качестве наилучшей модели. См. блок 447. Затем способ проверяет, остались ли в совокупности какие-либо дополнительные члены, характеризующие взаимодействие, которые не были протестированы (то есть удалены из текущей модели последовательности). См. блок принятия решения 449. Если существуют какие-либо не протестированные члены, способ возвращается к блоку 441, что образует внутренний цикл, для тестирования всех доступных членов, характеризующих взаимодействие, доступных в совокупности, учитывающей взаимодействие. Посредством итераций внутреннего цикла идентифицируют один член, характеризующий взаимодействие. Исключение его из модели улучшает модель в наиболее значительной степени (и наиболее существенно снижает AIC, если используется AIC для измерения предсказательной силы модели).
После того как все члены, характеризующие взаимодействие, были протестированы и внутренний цикл закончился, идентифицируют наилучшую модель, имеющую на один член, характеризующий взаимодействие, меньше, при условии, что модель, имеющая большую предсказательную силу, чем предыдущая наилучшая модель, действительно существует. См. блок принятия решения 451. В данном случае способ принимает текущую модель в качестве наилучшей модели. См. блок 453. Затем, способ возвращается назад к блоку 441. Данный внешний цикл осуществляет поиск следующего члена, характеризующего взаимодействие, который может улучшить предсказательную силу модели наиболее существенно. Если такой член, характеризующий взаимодействие, обнаружен, поиск следующего члена, характеризующего взаимодействие, подлежащего удалению, продолжается во внешнем цикле до тех пор, пока не перестанут быть идентифицированы новые модели, имеющие предсказательную силу, большую, чем у предыдущей наилучшей модели последовательности.
Когда внутренний цикл завершен и больше не могут быть обнаружены члены, характеризующие взаимодействие, подлежащие удалению для улучшения модели (то есть ответ операции принятия решения, показанной в блоке 451, отрицателен), способ принимает последнюю наилучшую модель в качестве итоговой модели. См. блок 455. Поиск наилучшей модели при заданных данных о последовательности и активности заканчивается.
E. Альтернативные варианты моделирования
Множественные дополнительные варианты вышеуказанного подхода лежат в пределах объема настоящего раскрытия. Более того, не предполагается, что настоящее изобретение ограничено какой-либо конкретной моделью, поскольку любая подходящая модель находит применение в настоящем изобретении. В качестве одного иллюстративного примера, переменные xij представляют собой представления физических или химических свойств аминокислот - а не точные идентичности самих аминокислот (лейцин по сравнению с валином по сравнению с пролином…). Примеры таких свойств включают липофильность, размеры и электронные свойства (например, формальный заряд, площадь поверхности Ван-дер-Ваальса, связанная с частичным зарядом, и так далее). Для реализации данного подхода значения xij, представляющие аминокислотные остатки, могут быть представлены с помощью своих свойств или главных компонент, сконструированных из данных свойств. Не предполагается, что настоящее изобретение ограничено каким-либо конкретным свойством аминокислот, пептидов и/или полипептидов, поскольку любое подходящее свойство находит применение в способах настоящего изобретения.
В некоторых дополнительных вариантах осуществления переменные xij представляют нуклеотиды, а не аминокислотные остатки. В данных вариантах осуществления цель заключается в идентификации нуклеиновокислотных последовательностей, которые кодируют белки для библиотеки белковых вариантов. Посредством использования нуклеотидов, а не аминокислот, можно по желанию оптимизировать параметры, отличные от активности (например, специфической активности). Например, экспрессия белка в определенном хозяине или векторе может быть функцией нуклеотидной последовательности. Две различные нуклеотидные последовательности могут кодировать белок, имеющий одну и ту же аминокислотную последовательность, но одна из нуклеотидных последовательностей может приводить к получению больших количеств белка, и/или белок более активен. Посредством использования нуклеотидных последовательностей, а не аминокислотных последовательностей способы, описанные в настоящем документе, можно использовать для оптимизации штаммов микроорганизмов, которые проявляют улучшенные свойства экспрессии генов и/или улучшенные свойства (например, специфическую активность, стабильность и так далее).
В некоторых вариантах осуществления нуклеотидная последовательность представлена в виде последовательности кодонов. В некоторых вариантах осуществления модели используют кодоны в качестве неделимых единиц нуклеотидной последовательности, так что предсказываемые активности являются функцией различных кодонов, присутствующих в нуклеотидной последовательности. Каждый кодон вместе с его положением во всей нуклеотидной последовательности служит в качестве независимой переменной для генерации модели последовательность-активность. Следует отметить, что в некоторых случаях различные кодоны для данной аминокислоты экспрессируются по-разному в данном организме. В некоторых вариантах осуществления каждый организм имеет предпочтительный кодон или распределение частоты кодонов для заданной аминокислоты. Посредством использования кодонов в качестве независимых переменных вариант осуществления учитывает данные предпочтения. Таким образом, вариант осуществления можно использовать для генерации библиотеки вариантов экспрессии (например, когда "активность" включает уровень экспрессии генов конкретного организма-хозяина).
В некоторых вариантах осуществления способы включают следующие операции: (a) получение данных, характеризующих обучающий набор библиотеки белковых вариантов; (b) разработку учитывающей взаимодействие модели последовательность-активность, которая предсказывает активность как функцию типов нуклеотидов и соответствующих положений в нуклеотидной последовательности, на основании данных, полученных в (a); (c) использование модели последовательность-активность для ранжирования положений в нуклеотидной последовательности и/или типов нуклеотидов в конкретных положениях в нуклеотидной последовательности в порядке воздействия на желаемую активность; и (d) использование ранжирования, для того чтобы идентифицировать один или более нуклеотидов в нуклеотидной последовательности, которые подлежат варьированию или фиксации для того, чтобы улучшить желаемую активность. Как указано, в некоторых вариантах осуществления нуклеотиды, подлежащие варьированию, кодируют конкретные аминокислоты.
В некоторых других вариантах осуществления способы включают использование различных методов для ранжирования или иной характеризации остатков по их важности в отношении некоторого свойства. Как описано выше для линейных или не учитывающих взаимодействие моделей, для ранжирования остатков использовали величины коэффициентов регрессии. Остатки, имеющие коэффициенты с большими величинами (например, 166 Ile), рассматривали как высоко ранжированные остатки. Эту характеризацию использовали для принятия решения, варьировать ли конкретный остаток при генерации новой оптимизированной библиотеки вариантов белков или нет. Для учитывающих взаимодействие моделей анализ чувствительности оказался более сложным, как описано в настоящем документе.
PLS и другие методы предоставляют дополнительную информацию помимо величины коэффициентов регрессии, которую можно использовать для ранжирования конкретных остатков или положений остатков. Методы, такие как PLS и анализ главных компонент (PCA) или PCR, предоставляют информацию в форме главных компонент или латентных векторов. Они представляют направления или векторы максимальной вариации в многомерных наборах данных, таких как пространство белковая последовательность-активность, используемых с вариантами осуществления настоящего изобретения, раскрытыми в настоящем документе. Эти латентные векторы являются функциями различных измерений последовательности; то есть отдельных остатков или положений остатков, которые содержат белковые последовательности, содержащие библиотеку вариантов, используемую для конструирования обучающего набора. Латентные векторы, следовательно, содержат сумму вкладов от каждого из положений остатков в обучающий набор. Некоторые положения вносят больший вклад в направление вектора. Это проявляется в относительно больших "нагрузках", то есть коэффициентах, используемых для описания вектора. В качестве простого иллюстративного примера, обучающий набор может состоять из трипептидов. В данном примере первый латентный вектор содержит вклады от всех трех остатков.
Вектор 1=a1(положение остатка 1)+a2(положение остатка 2)+a3(положение остатка 3)
Коэффициенты a1, a2 и a3 представляют собой нагрузки. Поскольку они отражают важность соответствующих положений остатков для вариации в наборе данных, их можно использовать для ранжирования важности отдельных положений остатков для целей "переключения" решений, как описано выше. Нагрузки, такие как коэффициенты регрессии, можно использовать для ранжирования остатков в каждом переключаемом положении. Различные параметры описывают важность данных нагрузок. Некоторые варианты осуществления используют такие способы, как важность переменной в проекции (VIP), для использования матрицы нагрузок. Эта матрица нагрузок состоит из нагрузок для множественных латентных векторов, взятых из обучающего набора. В способах важности переменной для проекции PLS важность переменной (например, положения остатка) вычисляется посредством расчета VIP. Для заданной размерности PLS, a, (VIN)ak2 равно квадрату веса PLS (wak)2 переменной, умноженному на процентную объяснимую изменчивость в y (зависимая переменная, например некоторая функция) по этой размерности PLS. (VIN)ak2 суммируют по всем размерностям (компонентам) PLS. Затем VIP вычисляют посредством деления суммы на общую процентную изменчивость в y, объяснимую посредством модели PLS, и умножения на число переменных в модели. Переменные с VIP больше, чем 1 являются наиболее релевантными для корреляции с некоторой функцией (y) и, следовательно, имеют наивысшие ранги для целей принятия решений о переключении.
Во многих вариантах осуществления настоящее изобретение использует способы общей линейной регрессии для того, чтобы идентифицировать эффекты мутаций в комбинаторной библиотеке на представляющие интерес последовательность-активность. Альтернативные варианты и методы моделирования, например байесовская регрессия, регрессия ансамбля, бутстреппинг, можно использовать в комбинации со способами, отмеченными выше, или вместо них. Более того, не предполагается, что настоящее изобретение ограничено какими-либо конкретными вариантами и/или методами моделирования, поскольку любой подходящий способ(ы) находит применение в настоящем изобретении.
Байесовская линейная регрессия
В некоторых вариантах осуществления настоящего изобретения находит применение байесовская линейная регрессия. Данный способ представляет собой подход к линейной регрессии, в котором статистический анализ осуществляется в контексте байесовского вывода. Когда регрессионная модель имеет ошибки, которые имеют нормальное распределение, и если предполагается определенная форма априорного распределения, апостериорные распределения вероятностей параметров модели можно определить с использованием методов байесовского вывода.
Решение линейной регрессионной модели с помощью обычных наименьших квадратов оценивает вектор коэффициентов и ошибку модели на основании функции правдоподобия данных с использованием аналитического вычислительного способа, такого как псевдоинверсия Мура-Пенроуза. Она представляет собой частотный подход, который предполагает, что имеется достаточно наблюдений данных для представления связи последовательности с активностью для всех последовательностей. Однако фактические наблюдения выборки почти никогда не бывают достаточными для представления всех членов популяции. Это становится особенной проблемой, когда размер выборки (или обучающего набора) ограничен. В байесовском подходе данные выборки дополняют дополнительной информацией в форме априорного распределения вероятностей. Априорное предположение в отношении параметров объединяют с функцией правдоподобия данных в соответствии с теоремой Байеса для получения апостериорного предположения в отношении параметров. Априорное предположение может принимать различные функциональные формы в зависимости от домена и информации, которая доступна a priori.
Например, в некоторых вариантах осуществления байесовская регрессия может использовать априорную информацию для взвешивания коэффициентов до подгонки модели. В некоторых вариантах осуществления для взвешивания линейных коэффициентов можно использовать данные о последовательности/активности, взятые из предыдущего цикла направленной эволюции, например цикла, выполненного с использованием родительского или референсного остова, и по меньшей мере некоторые из мутаций, используемых в предыдущих циклах. Кроме того, для взвешивания коэффициентов взаимодействия можно использовать предсказания эпистатической зависимости между двумя или более мутациями. Одним из основных преимуществ данного подхода является включение априорной информации в непосредственные предсказания модели.
Одним иллюстративным примером источника априорной информации является модель с независимыми и характеризующими взаимодействие членами для каждой из множественных мутаций в референсном остове. В некоторых вариантах осуществления данные получают из коллекции вариантов, которая содержит одну мутацию на вариант.
Дополнительные примеры априорной информации, которые находят применение в настоящем изобретении, включают, но без ограничения, интуитивную или физическую информацию о роли некоторых мутаций или типов мутаций. Независимо от источника, априорная информация служит в качестве заранее определенного представления о зависимости между последовательностью и активностью.
В некоторых вариантах осуществления для оценки параметров модели байесовская линейная регрессия использует моделирование по методу Монте-Карло, такое как семплирование по Гиббсу или алгоритмы Метрополиса, для подгонки модели при заданных данных. Семплирование по Гиббсу представляет собой алгоритм Монте-Карло с цепями Маркова для получения последовательности наблюдений, которые приблизительно относятся к указанному многомерному распределению вероятностей (то есть к совместному распределению вероятностей двух или более случайных переменных), когда непосредственная выборка затруднена.
Фигура 5 представляет собой блок-схему, иллюстрирующую использование байесовской регрессии в управляемой эволюции библиотек вариантов. Каждый цикл эволюции последовательности включает мутации на основании последовательностей из предыдущего цикла, который может управляться посредством знания, такого как модель последовательность-активность. В цикле n эволюции, как например, в блоке 501, имеет место одна мутация на вариант. Следующий или n+1 цикл эволюции представляет собой текущий цикл, как показано в блоке 503. Имеется по меньшей мере одна новая мутация для каждого варианта, что составляет две или более мутации на вариант. В данном цикле в данном иллюстративном примере реализована байесовская регрессия.
Варианты последовательностей цикла n+1 предоставляют обучающий набор данных для новых моделей. Новые модели могут содержать основную модель, которая включает только члены, не характеризующие взаимодействие, для отдельных остатков, или полную модель, которая содержит все возможные члены/коэффициенты, характеризующие взаимодействие, как указано в блоке 507. Новые модели могут также содержать модель, выбранную с помощью различных методов, включая методы пошагового добавления или удаления, рассмотренные выше, см. блок 505. Модель может, альтернативно, быть выбрана с использованием генетического алгоритма или методов бутстрепа, как рассмотрено ниже. Все эти модели основаны на текущих/новых данных из данных обучающего набора цикла n+1. К этим моделям может быть применен метод байесовского вывода, так что модель будет основана как на функции вероятности текущих данных, так и на распределении априорной информации. Априорная информация может быть получена из данных предыдущего цикла вариантов последовательностей, как в цикле n, показанном в блоке 501. Информация также может быть получена из данных о последовательности-активности из любого предыдущего цикла эволюции или другом априорном интуитивном знании, как указано в блоке 513. Модель байесовской регрессии, показанной в блоке 509, предсказывает активность на основании информации, предоставляемой текущими данными, и априорной информации, см. блок 511. Хотя фигура 5 иллюстрирует применение метода байесовской регрессии к только циклу n+1, он может быть применен на различных стадиях. Также не предполагается, что настоящее изобретение ограничено конкретными этапами, предложенными на фигуре 5, поскольку любые подходящие способы находят применение в настоящем изобретении.
Регрессия ансамбля
В некоторых вариантах осуществления настоящее изобретение использует для получения модели последовательность-активность метод регрессии ансамбля. Модель регрессии ансамбля основана на нескольких регрессионных моделях. Предсказание каждой модели взвешивают на основании определенного информационного критерия (IC), и предсказанием ансамбля является взвешенная сумма предсказаний всех моделей, которые он содержит. В некоторых вариантах осуществления разработка модели начинается с основной модели, содержащей все из членов, характеризующих взаимодействие. Последующие модели конструируют посредством добавления коэффициентов взаимодействия в некоторых или всех возможных комбинациях. В некоторых вариантах осуществления коэффициенты взаимодействия добавляют в пошаговом процессе. Каждую модель подгоняют к данным и генерируют IC. Вес для каждой модели основан на IC, который может представлять собой сам IC или его преобразованный вариант, например логарифмическое значение, обратное значение и так далее. Предсказания для наблюдения могут быть сделаны посредством генерации предсказания каждой модели в ансамбле и определения ансамблевого предсказания посредством получения взвешенного среднего предсказания из каждой модели. Полный ансамбль содержит все возможные модели, но может быть сокращен с удалением моделей с низкой эффективностью посредством установления порога или на количество моделей, которое он содержит, или на IC.
Составляющие ансамбль модели могут быть получены с использованием различных методов. Например, в некоторых вариантах осуществления используют генетический алгоритм для создания моделей-составных частей. Данные о последовательности/активности используют для получения множества регрессионных моделей, каждая из которых имеет свой собственный набор коэффициентов. Наилучшие модели выбирают в соответствии с критерием приспособленности (например, AIC или BIC). Эти модели "скрещивают" для получения новых гибридных моделей, которые затем подвергают эволюции на приспособленность и выбирают соответствующим образом. В некоторых вариантах осуществления данный процесс повторяют множественными циклами "вычислительной эволюции" для получения ансамбля наилучших моделей. Альтернативно, в некоторых вариантах осуществления составные части ансамбля создают посредством пошаговой регрессии, как описано выше, и выбирают наилучшие n моделей для формирования ансамбля.
Фигура 6 предлагает блок-схему процесса, который реализует регрессию ансамбля в направленной эволюции вариантов последовательностей в соответствии с вариантом осуществления настоящего изобретения. В данном варианте осуществления метод регрессии ансамбля может быть применен на любой стадии множественных циклов эволюции последовательности. Например, в цикле n варианты последовательностей, показанные в блоке 601, предоставляют обучающий набор данных для различных моделей для формирования совокупности моделей, как показано в блоке 603. Модели в совокупности моделей могут представлять собой модели, генерируемые посредством генетического алгоритма и/или пошагового выбора. В других вариантах осуществления совокупность моделей содержит модели с n-кратной перекрестной проверкой и/или бутстреппингом. В некоторых вариантах осуществления для вхождения в совокупность выбирают только модели с наивысшей предсказательной силой на основании различных критериев выбора модели, таких как AIC или BIC.
Альтернативно или дополнительно, в некоторых вариантах осуществления модели, которые не были отобраны с помощью выбора модели, также входят в совокупность моделей. В одном варианте осуществления все модели со всеми членами, не характеризующими и характеризующими взаимодействие, входят в совокупность моделей. Для большого количества остатков и гораздо большего количества факторных взаимодействий между остатками данный вариант осуществления может быть очень трудоемким вычислительно. В некоторых альтернативных вариантах осуществления только модели, содержащие члены, не характеризующие взаимодействие, и члены, характеризующие парное взаимодействие, входят в совокупность моделей. Независимо от способа включения в совокупность моделей, ансамблевая модель включает все члены своих составных частей. Совокупность моделей может содержать любое число моделей, включая, но без ограничения байесовские модели, в каковом случае априорная информация может быть включена в ансамбль.
В некоторых вариантах осуществления ансамбль предсказывает активность последовательности на основании взвешенного среднего коэффициентов каждой модели в совокупности, причем веса определяют по предсказательной силе соответствующих моделей, как показано в блоке 605.
В некоторых вариантах осуществления регрессия ансамбля использует следующую последовательность действий: (1) предоставление пустого ансамбля; (2) выбор размера группы n, составляющего 1 или более; (3) категоризацию точек данных по группам размера n, где точки данных группируют без возвращения; и (4) получение ансамблевой модели для предсказания индивидуальных коэффициентов и коэффициентов взаимодействия. В некоторых вариантах осуществления этап (4) получения ансамблевой модели дополнительно содержит: a) удаление точек данных из каждой группы, причем остающиеся данные образуют обучающий набор, а удаленные данные образуют контрольный набор; b) получение модели посредством подгонки обучающего набора с использованием пошаговой регрессии; c) тестирование модели с использованием контрольного набора, что обеспечивает индикацию предсказательной способности модели; d) добавление модели в совокупность моделей, которые используют для генерации ансамблевой модели, как описано выше.
Бутстреп-подход
В настоящем изобретении находят применение другие методы характеризации предсказательной силы рассматриваемой модели в данной итерации. В некоторых вариантах осуществления эти методы включают перекрестную проверку или бутстреп-методы. В некоторых вариантах осуществления перекрестная проверка использует набор наблюдений, используемый для генерации модели, но отбрасывает некоторые из наблюдений для определения мощности модели. В некоторых вариантах осуществления бутстреп-метод включает использование набора образцов, которые тестируют с возвращением. В некоторых вариантах осуществления модели, генерируемые перекрестной проверкой или бутстреппингом, могут быть объединены в ансамблевую модель, как описано выше.
В некоторых дополнительных вариантах осуществления способы ранжируют остатки не просто по величинам предсказываемых ими вкладов в активность, но также по достоверности предсказываемых ими вкладов. В некоторых случаях исследователь имеет дело с обобщаемостью модели с одного набора данных на другой набор. Другими словами, исследователь хочет знать, являются ли значения коэффициентов или главные компоненты ложными или нет. Методы перекрестной проверки и бутстреппинга обеспечивают меры для указания доверительного уровня того, что модели являются обобщаемыми на различные данные.
В некоторых вариантах осуществления используют более статистически строгий подход, в котором ранжирование основано на комбинации величины и распределения. В некоторых из данных вариантов осуществления коэффициенты как с высокими величинами, так и с тесными распределениями дают наивысшее ранжирование. В некоторых случаях один коэффициент с более низкой величиной, чем другой может получить более высокое ранжирование в силу того, что он имеет меньшую вариацию. Таким образом, некоторые варианты осуществления ранжируют аминокислотные остатки или нуклеотиды на основании как величины, так и стандартного отклонения или дисперсии. Для осуществления этого можно использовать различные методы. Более того, не предполагается, что настоящее изобретение ограничено каким-либо конкретным методом ранжирования. Один вариант осуществления, использующий бутстреп-подход p-значений, описан ниже.
Иллюстративный пример способа, который использует бутстреп-способ, изображен на фигуре 7. Как показано на фигуре 7, способ 725 начинается в блоке 727, где предоставляется исходный набор данных S. В некоторых вариантах осуществления он представляет собой обучающий набор, как описано выше. Например, в некоторых вариантах осуществления его генерируют посредством систематического варьирования отдельных остатков исходной последовательности любым образом (например, как описано выше). В случае, проиллюстрированном способом 725, набор данных S содержит M различных точек данных (информацию об активности и последовательности, полученную от аминокислотных или нуклеотидных последовательностей) для использования в анализе.
Из набора данных S создают различные бутстреп-наборы B. Каждый из этих наборов получают посредством семплирования с возвращением из набора S для создания нового набора из M членов - все взяты из исходного набора S. См. блок 729. Условие "с возвращением" производит вариации в отношении исходного набора S. Новый бутстреп-набор, B, будет иногда содержать повторы образцов из S. В некоторых случаях в бутстреп-наборе B также будут отсутствовать некоторые образцы, исходно содержащиеся в S.
В качестве иллюстративного примера предлагается набор S из 100 последовательностей. Бутстреп-набор B создают посредством случайного выбора 100 членов последовательности из 100 последовательностей в исходном наборе S. Каждый бутстреп-набор B, используемый в способе, содержит 100 последовательностей. Таким образом, возможно, что некоторые последовательности будут выбраны больше, чем один раз, а другие не будут выбраны совсем. Затем, используя бутстреп-набор B, получаемый из набора S из 100 последовательностей, способ строит модель. См. блок 731. Модель можно строить, как описано выше, используя PLS, PCR, SVM, пошаговую регрессию и так далее. Более того, предполагается, что любой подходящий способ найдет применение в построении модели. Данная модель предоставляет коэффициенты или другие признаки ранжирования для остатков или нуклеотидов, обнаруженных в различных образцах из набора B. Как показано в блоке 733, эти коэффициенты или другие признаки фиксируют для последующего использования.
Затем в блоке 735 принятия решения способ определяет, следует ли создавать другой бутстреп-набор. Если да, способ возвращается к блоку 729, где создается новый бутстреп-набор B, как описано выше. Если нет, способ переходит к блоку 737, рассмотренному ниже. Решение в блоке 735 зависит от того, как много различных наборов значений коэффициентов следует использовать при определении распределений этих значений. Количество наборов B должно быть достаточным для генерации точной статистики. В некоторых вариантах осуществления получают и анализируют от 100 до 1000 бутстреп-наборов. Их представляет приблизительно от 100 до 1000 проходов через блоки 729, 731 и 733 способа 725. Однако не предполагается, что настоящее изобретение ограничено каким-либо конкретным количеством бутстреп-наборов, поскольку любое количество, подходящее для желаемого анализа, находит применение.
После получения и анализа достаточного количества бутстреп-наборов B решение 735 становится отрицательным. Как указано, затем способ переходит к блоку 737. Здесь вычисляются среднее и стандартное отклонение коэффициента (или другого индикатора, генерируемого моделью) для каждого остатка или нуклеотида (включая кодоны) с использованием значений коэффициентов (например, значений 100-1000, одно из каждого бутстреп-набора). Из данной информации способ может вычислить t-статистику и определить доверительный интервал того, что измеренное значение отличается от нуля. Из t-статистики он вычисляет p-значение для доверительного интервала. В данном иллюстративном случае чем меньше p-значение, тем больше достоверность того, что измеренный коэффициент регрессии отличается от нуля.
Следует отметить, что p-значение является всего лишь одним из многих различных типов характеризации, который может учитывать статистическую вариацию в коэффициенте или другом индикаторе важности остатка. Примеры включают, но без ограничения, вычисление 95-процентных доверительных интервалов для коэффициентов регрессии и исключение любого рассматриваемого коэффициента регрессии, для которого 95-процентный доверительный интервал пересекает линию нуля. По существу, в некоторых вариантах осуществления находит применение любая характеризация, которая учитывает стандартное отклонение, дисперсию или другую статистически релевантную меру распределения данных. В некоторых вариантах осуществления данный этап характеризации также учитывает величину коэффициентов.
В некоторых вариантах осуществления получается большое стандартное отклонение. Данное большое стандартное отклонение может быть обусловлено различными причинами, включая, но без ограничения, плохие измерения в наборе данных и/или ограниченное представление конкретного остатка или нуклеотида в исходном наборе данных. В этом последнем случае некоторые бутстреп-наборы не будут содержать вхождений конкретного остатка или нуклеотида. В таких случаях значение коэффициента для этого остатка будет равно нулю. Другие бутстреп-наборы будут содержать по меньшей мере некоторое количество вхождений этого остатка или нуклеотида и дадут ненулевое значение соответствующего коэффициента. Но наборы, дающие нулевое значение будут причиной того, что стандартное отклонение коэффициента станет относительно большим. Это уменьшает достоверность значения коэффициента и приводит к более низкому рангу. Но этого следует ожидать при условии, что имеется относительно мало данных о рассматриваемом остатке или нуклеотиде.
Затем в блоке 739 способ ранжирует коэффициенты регрессии (или другие индикаторы) от самых низких (наилучших) p-значений до самых высоких (худших) p-значений. Данное ранжирование сильно коррелирует с абсолютным значением самих коэффициентов регрессии по причине того, что чем больше абсолютное значение, тем больше стандартные отклонения удалены от нуля. Таким образом, для заданного стандартного отклонения p-значение становится меньше, если коэффициент регрессии становится больше. Тем не менее абсолютное ранжирование не всегда будет совпадать со способами как p-значений, так и чистой величины, особенно когда относительно мало точек данных доступно для начала в наборе S.
Наконец, как показано в блоке 741, способ фиксирует и переключает некоторые остатки на основании ранжирований, наблюдаемых в операции блока 739. Это по существу то же использование ранжирований, что и описанное выше для других вариантов осуществления. В одном подходе способ фиксирует наилучшие остатки (в данном случае с наименьшими p-значениями) и переключает другие (с наивысшими p-значениями).
Было показано, что данный способ 725 хорошо работает in silico. Кроме того, в некоторых вариантах осуществления подход ранжирования по p-значениям естественным образом работает с единственными или встречающимися несколько раз остатками: p-значения обычно будут выше (хуже), поскольку в бутстреп-процессе эти остатки, которые не часто появляются в исходном наборе данных, будут с меньшей вероятностью выбираться случайным образом. Даже если их коэффициенты велики, их изменчивость (измеренная в стандартных отклонениях) будет также достаточно высока. В некоторых вариантах осуществления это является желаемым результатом, поскольку те остатки, которые плохо представлены (то есть или наблюдаются не с достаточной частотой, или имеют более низкие коэффициенты регрессии) могут быть хорошими кандидатами для переключения в следующем цикле дизайна библиотеки.
VI. Генерация оптимизированной библиотеки белковых вариантов посредством модифицирования предсказанных моделью последовательностей
Одной из целей настоящего изобретения является генерация оптимизированной библиотеки белковых вариантов с помощью направленной эволюции. Некоторые варианты осуществления настоящего изобретения предлагают способы управления направленной эволюцией вариантов белков с использованием генерируемых моделей последовательность-активность. Различные модели последовательность-активность, полученные и уточненные в соответствии со способами, описанными выше, подходят для управления направленной эволюцией белков или биологических молекул. В качестве части данного процесса способы могут идентифицировать последовательности, которые следует использовать для генерации новой библиотеки белковых вариантов. Такие последовательности включают вариации в определенных остатках, идентифицированных выше, или представляют собой предшественники, используемые для последующего введения таких вариаций. Последовательности могут быть модифицированы с помощью мутагенеза или основанного на рекомбинации механизма генерации разнообразия для генерации новой библиотеки вариантов белков. Новую библиотеку можно также использовать при разработке новой модели последовательность-активность.
В некоторых вариантах осуществления получение олигонуклеотидов или нуклеиновокислотных последовательностей осуществляют посредством синтезирования олигонуклеотидов или нуклеиновокислотных последовательностей с помощью синтезатора нуклеиновых кислот. Некоторые варианты осуществления настоящего изобретения включают осуществление цикла направленной эволюции с использованием полученных олигонуклеотидов или белковой последовательности в качестве строительных элементов для направленной эволюции. Различные варианты осуществления настоящего изобретения применяют рекомбинацию и/или мутагенез к этим строительным элементам для генерации разнообразия.
В качестве одного конкретного примера, некоторые варианты осуществления применяют к олигонуклеотидам методы рекомбинации. В данных вариантах осуществления способы включают выбор одной или более мутаций для цикла направленной эволюции посредством оценивания коэффициентов членов модели последовательность-активность. Мутации выбирают из комбинаций определенных аминокислот или нуклеотидов конкретных типов в конкретных положениях на основании их вкладов в активность белков, предсказанных моделями. В некоторых вариантах осуществления выбор мутаций включает идентифицирование одного или более коэффициентов, которые, как определено, больше, чем другие коэффициенты, и выбор определенной аминокислоты или нуклеотида в определенном положении, представляемых одним или более коэффициентами, идентифицированными таким образом. В некоторых вариантах осуществления после выбора мутаций в соответствии с моделями последовательность-активность способы включают получение множества олигонуклеотидов, содержащих или кодирующих одну или более мутаций, и осуществление цикла направленной эволюции с использованием полученных олигонуклеотидов. В некоторых вариантах осуществления методы направленной эволюции включают комбинирование и/или рекомбинирование олигонуклеотидов.
Другие варианты осуществления настоящего изобретения применяют методы рекомбинации к белковым последовательностям. В некоторых вариантах осуществления способы включают идентифицирование новой белковой или новой нуклеиновокислотной последовательности и получение и анализирование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью. В некоторых вариантах осуществления способы дополнительно включают использование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью, в качестве исходной точки для дальнейшей направленной эволюции. В некоторых вариантах осуществления процесс направленной эволюции включает фрагментирование и рекомбинирование белковой последовательности, о которой модель предсказывает, что она имеет желаемый уровень активности.
В некоторых вариантах осуществления способы идентифицируют и/или получают новую белковую или новую нуклеиновокислотную последовательность на основании отдельных мутаций, о которых модель предсказывает, что они важны. Данные способы включают: выбор одной или более мутаций посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность; идентифицирование новой белковой или новой нуклеиновокислотной последовательности, содержащей одну или более мутаций, выбранных выше, и получение и анализирование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью.
В других вариантах осуществления способы идентифицируют и/или получают новую белковую или новую нуклеиновокислотную последовательность на основании предсказанной активности всей последовательности, а не отдельных мутаций. В некоторых из данных вариантов осуществления способы включают использование множественных белковых последовательностей или множественных аминокислотных последовательностей в модели последовательность-активность и определение значений активности, предсказанных моделью последовательность-активность для каждой из множественных белковых последовательностей или нуклеиновокислотных последовательностей. Способы дополнительно включают выбор новой белковой последовательности или новой нуклеиновокислотной последовательности из множественных белковых последовательностей или множественных аминокислотных последовательностей, использованных выше, посредством оценивания значений активности, предсказанных моделью последовательность-активность для данных множественных последовательностей. Способы также включают получение и анализирование белка, имеющего новую белковую последовательность, или белка, кодируемого новой нуклеиновокислотной последовательностью.
В некоторых вариантах осуществления вместо простого синтезирования одного белка с наилучшим предсказанием генерируют комбинаторную библиотеку белков на основании анализа чувствительности наилучших изменений выбора остатков в каждом местоположении в белке. В данном варианте осуществления чем более чувствителен заданный выбор остатка для предсказанного белка, тем больше будет предсказанное изменение приспособленности. В некоторых вариантах осуществления эти чувствительности изменяются от самых высоких до самых низких, и показатели чувствительности используют для создания комбинаторных библиотек белков в последующих циклах (то есть посредством включения этих остатков на основании чувствительности). В одном варианте осуществления, в котором используют не учитывающую взаимодействие модель, чувствительность определяют посредством простого учета величины коэффициента, соответствующего члену заданного остатка в модели. Однако, это не возможно для учитывающих взаимодействие моделей. Вместо этого в вариантах осуществления, использующих учитывающие взаимодействие модели, чувствительность остатков определяют посредством использования модели для расчета изменений активности, когда один остаток варьируют в "наилучшей" предсказанной последовательности.
Некоторые варианты осуществления настоящего изобретения включают выбор одного или более положений в белковой последовательности или нуклеиновокислотной последовательности и проведение насыщающего мутагенеза в одном или более положениях, идентифицированных таким образом. В некоторых вариантах осуществления положения выбирают посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность. Соответственно, в некоторых вариантах осуществления цикл направленной эволюции включает осуществление насыщающего мутагенеза в отношении белковой последовательности в положениях, выбранных с использованием моделей последовательность-активность. В некоторых вариантах осуществления, включающих модели, содержащие один или более членов, характеризующих взаимодействие, способы включают применение мутагенеза одновременно в отношении двух или более взаимодействующих остатков.
В некоторых вариантах осуществления остатки учитывают в порядке, в котором они ранжированы. В некоторых вариантах осуществления для каждого рассматриваемого остатка способ определяет, "переключать" ли этот остаток. Термин "переключение" относится к введению множественных типов аминокислотных остатков в конкретное положение в последовательностях вариантов белков в оптимизированной библиотеке. Например, серин может оказаться в положении 166 в одном белковом варианте, тогда как в другом белковом варианте в той же библиотеке в положении 166 может оказаться фенилаланин. Аминокислотные остатки, которые не варьируются между белковыми вариантными последовательностями в обучающем наборе, как правило, остаются фиксированными в оптимизированной библиотеке. Однако это не всегда имеет место, поскольку может иметь место вариация в оптимизированных библиотеках.
В некоторых вариантах осуществления оптимизированную библиотеку белковых вариантов разрабатывают так, чтобы все идентифицированные остатки с "высоко" ранжированными коэффициентами регрессии были зафиксированы, а остальные остатки с более низко ранжированными коэффициентами регрессии были переключены. Обоснованием для этого варианта осуществления является то, что следует проводить поиск в локальном пространстве, окружающем "наилучший" предсказанный белок. Следует отметить, что "остов" исходной точки, в который вводят переключения, может представлять собой наилучший белок, предсказанный моделью, и/или уже проверенный "наилучший" белок из подвергаемой скринингу библиотеки. Более того, не предполагается, что остов исходной точки ограничен каким-либо конкретным белком.
В альтернативном варианте осуществления по меньшей мере один или более, но не все идентифицированные остатки с высоко ранжированными коэффициентами регрессии фиксируют в оптимизированной библиотеке, а другие переключают. Данный подход рекомендован в некоторых вариантах осуществления, если есть желание не изменять кардинально контекст других аминокислотных остатков посредством внесения слишком большого числа изменений за один раз. В этом случае также исходная точка для переключения может представлять собой наилучший набор остатков, как предсказано моделью, наилучший проверенный белок из существующей библиотеки или "средний" клон, который хорошо смоделирован. В последнем случае может быть желательно переключать остатки, о которых предсказано, что они имеют более высокую важность, поскольку нужно исследовать большее пространство при поиске холмов активности, ранее исключенных из семплирования. Данный тип библиотеки, как правило, является более релевантным в ранних циклах получения библиотек, поскольку он генерирует более уточненную картину для последующих циклов. Также не предполагается, что остов исходной точки ограничен каким-либо конкретным белком.
Некоторые альтернативы вышеуказанным вариантам осуществления включают различные процедуры для использования важности остатков (ранжирования) при определении того, какие из остатков переключать. В одном таком альтернативном варианте осуществления более высокоранжированные положения остатков более агрессивно используют для переключения. Информация, необходимая для данного подхода, включает последовательность наилучшего белка из обучающего набора, наилучшую предсказанную с помощью PLS или PCR последовательность и ранжирование остатков по модели PLS или PCR. "Наилучший" белок представляет собой проверенный во влажной лаборатории "наилучший" клон в наборе данных (то есть клон с наивысшей измеренной функцией, который все еще хорошо смоделирован в том смысле, что он оказывается относительно близок к предсказанному значению при перекрестной проверке). Способ сравнивает каждый остаток из данного белка с соответствующим остатком из "наилучшей предсказанной" последовательности, имеющей наивысшее значение желаемой активности. Если остаток с наивысшей нагрузкой или коэффициентом регрессии не присутствует в "наилучшем" клоне, способ включает данное положение в качестве положения переключения для последующей библиотеки. Если остаток присутствует в наилучшем клоне, способ не рассматривает данное положение как положение переключения, и он переходит к следующему по порядку положению. Данный процесс повторяют для различных остатков, переходя чрез последовательно уменьшающиеся значения нагрузок до тех пор, пока не будет сгенерирована библиотека достаточного размера.
В некоторых вариантах осуществления количество остатков коэффициентов регрессии для сохранения и количество остатков коэффициентов регрессии для переключения варьируют. Определение того, какие из остатков переключать, и какие оставлять, основано на различных факторах, включая, но без ограничения, желаемый размер библиотеки, величину различия между коэффициентами регрессии и степень, до которой предполагается существование нелинейности. Сохранение остатков с малыми (нейтральными) коэффициентами может раскрывать важные нелинейности в последующих циклах эволюции. В некоторых вариантах осуществления оптимизированные библиотеки белковых вариантов содержат приблизительно 2N вариантов белков, где N представляет число положений, которые переключают между двумя остатками. Иначе говоря, разнообразие, добавляемое каждым дополнительным переключением, удваивает размер библиотеки таким образом, что 10 положений переключения дают ~1000 клонов (1024), 13 положений ~10000 клонов (8192), и 20 положений ~1000000 клонов (1048576). Подходящий размер библиотеки зависит от таких факторов, как стоимость скрининга, неровность ландшафта, предпочтительное процентное семплирование пространства и так далее. В некоторых случаях было обнаружено, что относительно большое число измененных остатков производит библиотеку, в которой чрезмерно большой процент клонов являются нефункциональными. Поэтому в некоторых вариантах осуществления число остатков для переключения лежит в диапазоне от приблизительно 2 до приблизительно 30; то есть размер библиотеки лежит в диапазоне между приблизительно 4 и 230~109 клонов.
Кроме того, предусмотрено, что различные стратегии библиотек последующих циклов используют одновременно, причем некоторые стратегии являются более агрессивными (фиксируют больше "полезных" остатков), а другие стратегии являются более консервативными (фиксируют меньше "полезных" остатков с целью более тщательного исследования пространства).
В некоторых вариантах осуществления группы, или остатки, или "мотивы", которые встречаются в большинстве природных или иным образом успешных пептидов, идентифицируют и/или сохраняют, поскольку они могут быть важны для функциональности белка (например, активности, стабильности и так далее). Например, может быть обнаружено, что Ile в варьируемом положении 3 всегда связан с Val в варьируемом положении 11 в природных пептидах. Следовательно, в одном варианте осуществления требуется сохранение таких групп при любой стратегии переключения. Другими словами, единственными допускаемыми переключениями являются те, которые сохраняют определенную группировку в основном белке, или те, которые генерируют другую группировку, которая также обнаружена в активных белках. В последнем случае необходимо переключать два или более остатка.
В некоторых дополнительных вариантах осуществления проверенный во влажной лаборатории "наилучший" (или один из более наилучших) белок в текущей оптимизированной библиотеке (то есть белок с наивысшей или одной из более наивысших измеренный функций, который все еще хорошо смоделирован, то есть оказывается относительно близок к предсказанному значению при перекрестной проверке) служит в качестве остова, в который вносят различные изменения. В другом подходе проверенный во влажной лаборатории "наилучший" (или один из нескольких наилучших) белок в текущей библиотеке, который может не быть хорошо смоделирован, служит в качестве остова, в который вносят различные изменения. В некоторых других подходах последовательность, о которой модель последовательность-активность предсказывает, что она имеет наивысшее значение (или одно из наивысших значений) желаемой активности, служит в качестве остова. В данных подходах набор данных для библиотеки "следующего поколения" (и, возможно, соответствующую модель) получают посредством изменения остатков в одном или более из наилучших белков. В одном варианте осуществления эти изменения содержат систематическую вариацию остатков в остове. В некоторых случаях изменения содержат различный мутагенез, рекомбинацию и/или методы выбора подпоследовательности. Все это может быть осуществлено in vitro, in vivo и/или in silico. Более того, не предполагается, что настоящее изобретение ограничено каким-либо конкретным форматом, поскольку любой подходящий формат находит применение.
В некоторых вариантах осуществления при том, что оптимальная последовательность, предсказываемая не учитывающей взаимодействие моделью, может быть идентифицирована посредством проверки, как описано выше, это не верно для учитывающих взаимодействие моделей. Некоторые остатки появляются как в не характеризующих, так и в характеризующих взаимодействие членах, и их общее воздействие на активность в контексте множества возможных комбинаций других остатков может быть проблематичным. Таким образом, как и в случае выбора членов, характеризующих взаимодействие, для учитывающей взаимодействие модели оптимальная последовательность, предсказываемая учитывающей взаимодействие моделью, может быть идентифицирована посредством тестирования всех возможных последовательностей с помощью модели (в предположении достаточных вычислительных ресурсов) или посредством использования алгоритма поиска, такого как пошаговый алгоритм.
В некоторых вариантах осуществления информацию, содержащуюся в подвергавшихся эволюции с помощью компьютера белках, идентифицированных, как описано выше, используют для синтезирования новых белков и их тестирования в физических анализах. Точное представление in silico фактической определенной во влажной лаборатории функции приспособленности позволяет исследователям уменьшить количество циклов эволюции и/или количество вариантов, которые нужно подвергать скринингу в лаборатории. В некоторых вариантах осуществления оптимизированные библиотеки белковых вариантов генерируют с использованием способов рекомбинации, описанных в настоящем документе, или, альтернативно, способов синтеза генов с последующей экспрессией in vivo или in vitro. В некоторых вариантах осуществления после скрининга оптимизированных библиотек белковых вариантов на желаемую активность их секвенируют. Как указано выше при рассмотрении фигур 1 и 2, информацию об активности и последовательности из оптимизированной библиотеки белковых вариантов можно использовать для генерации другой модели последовательность-активность, на основании которой может быть разработана следующая оптимизированная библиотека с использованием способов, описанных в настоящем документе. В одном варианте осуществления все белки из данной новой библиотеки используют в качестве части набора данных.
VII. Цифровое приспособление и системы
Как должно быть очевидно, варианты осуществления, описанные в настоящем документе, используют процессы, работающие под управлением инструкций и/или данных, сохраненных в одной или более компьютерных системах или передаваемых через них. Варианты осуществления, раскрытые в настоящем документе, также относятся к приспособлению для осуществления данных операций. В некоторых вариантах осуществления приспособление специально разработано и/или сконструировано для требуемых целей, или оно может представлять собой компьютер общего назначения, селективно активированный или переконфигурированный с помощью компьютерной программы и/или структуры данных, сохраненной в компьютере. Способы, предлагаемые настоящим изобретением, не связаны по своей сути с каким-либо конкретным компьютером или другим конкретным приспособлением. В частности, различные машины общего применения находят применение с программами, написанными в соответствии с идеями настоящего документа. Однако в некоторых вариантах осуществления конструируют специализированное приспособление для выполнения операций требуемого способа. Один вариант осуществления специальной структуры для множества данных машин описан ниже.
Кроме того, определенные варианты осуществления настоящего изобретения относятся к читаемым компьютером средам или компьютерным программным продуктам, которые включают программные инструкции и/или данные (включая структуры данных) для осуществления различных реализуемых компьютером операций. Примеры читаемых компьютером сред включают, но без ограничения, магнитные среды, такие как жесткие диски, гибкие диски, магнитную ленту; оптические среды, такие как устройства CD-ROM и голографические устройства; магнитно-оптические среды; полупроводниковые устройства памяти; и аппаратные устройства, которые специально сконфигурированы для хранения и выполнения программных инструкций, такие как устройства памяти, доступной только для чтения (ROM), и память с произвольным доступом (RAM), интегральные схемы специального назначения (ASIC) и программируемые логические устройства (PLD). Данные и программные инструкции могут также быть воплощены на несущей волне или другой транспортной среде (например, оптических линиях, электрических линиях и/или радиоволнах). Более того, не предполагается, что настоящее изобретение ограничено какими-либо конкретными читаемыми компьютером средами или какими-либо другими компьютерными программными продуктами, которые включают инструкции и/или данные для осуществления реализуемых компьютером операций.
Примеры программных инструкций включают, но без ограничения, низкоуровневый код, такой как производимый компилятором, и файлы, содержащие высокоуровневый код, который может быть выполнен компьютером с использованием интерпретатора. Кроме того, программные инструкции включают, но без ограничения, машинный код, исходный код и любой другой код, который прямо или косвенно управляет операциями вычислительной машины в соответствии с настоящим изобретением. Код может определять ввод, вывод, вычисления, условные конструкции, переходы, итерационные циклы и так далее.
В одном иллюстративном примере код, реализующий способы, раскрытые в настоящем документе, реализован в фиксированных средах или передаваемых программных компонентах, содержащих логические инструкции и/или данные, которые при загрузке в соответствующим образом сконфигурированное вычислительное устройство вынуждают это устройство выполнять моделируемую генетическую операцию (GO) над одной или более цепочками символов. Фигура 8 показывает пример цифрового устройства 800, которое представляет собой логическое приспособление, которое может считывать инструкции из среды 817, сетевого порта 819, клавиатуры 809 пользовательского ввода, пользовательского ввода 811 или других средств ввода. Приспособление 800 может в дальнейшем использовать эти инструкции для управления статистическими операциями в пространстве данных, например для конструирования одного или более наборов данных (например, для определения множества репрезентативных членов пространства данных). Одним типом логического приспособления, которое может реализовывать раскрытые варианты осуществления, является компьютерная система, как в компьютерной системе 800, содержащей CPU 807, необязательную клавиатуру 809 устройств пользовательского ввода и указывающее устройство 811 GUI, а также периферийные компоненты, такие как приводы 815 дисков и монитор 805 (который отображает модифицированные GO цепочки символов и обеспечивает упрощенный выбор поднаборов таких цепочек символов пользователем). Фиксированные среды 817, необязательно, используются для программирования всей системы и могут включать, например, оптические или магнитные среды дискового типа или другие электронные элементы запоминающих устройств. Коммуникационный порт 819 может использоваться для программирования системы и может представлять любой тип коммуникационного соединения.
В некоторых вариантах осуществления настоящее раскрытие предлагает компьютерную систему, содержащую: один или более процессоров; системную память; и одну или более читаемых компьютером сред для хранения, несущих сохраненные на них выполнимые компьютером инструкции, которые, когда выполняются одним или более процессорами, вынуждают компьютерную систему осуществлять способ проведения направленной эволюции биологических молекул. Способ включает: (a) получение данных о последовательности и активности для множества биологических молекул, причем каждая биологическая молекула содержит последовательность, содержащую субъединицы различных типов и положений в последовательности; (b) построение модели последовательность-активность по полученным данным; и (c) использование модели последовательность-активность, для того чтобы идентифицировать одну или более субъединиц конкретных типов в конкретных положениях для вариации для воздействия на желаемую активность биологических молекул. В некоторых вариантах осуществления модель последовательность-активность включает произведение множества основных мультипликативных членов, причем каждый из основных мультипликативных членов содержит (1) фиктивную переменную, представляющую присутствие/отсутствие определенной субъединицы конкретного типа в конкретном положении в последовательности, и (2) коэффициент, представляющий вклад определенной субъединицы в активность.
Определенные варианты осуществления могут также быть воплощены в системе цепей интегральной схемы специального назначения (ASIC) или программируемого логического устройства (PLD). В таком случае варианты осуществления реализуют в читаемом компьютером дескрипторном языке, который можно использовать для создания ASIC или PLD. Некоторые варианты осуществления настоящего изобретения реализованы в системе цепей или логических процессорах множества других цифровых приспособлений, таких как КПК, портативные компьютерные системы, дисплеи, оборудование для редактирования изображений и так далее.
В некоторых вариантах осуществления настоящее изобретение относится к компьютерному программному продукту, содержащему одну или более читаемых компьютером сред для хранения, несущих сохраненные на них выполнимые компьютером инструкции, которые, когда выполняются одним или более процессорами компьютерной системы, вынуждают компьютерную систему осуществлять способ идентификации биологических молекул для воздействия на желаемую активность. Такой способ может представлять собой любой способ, описанный в настоящем документе, такой как охватываемые фигурами и псевдокодом. В некоторых вариантах осуществления способ получает данные о последовательности и активности для множества биологических молекул и получает основную модель и улучшенную модель по данным о последовательности и активности. В некоторых вариантах осуществления модель предсказывает активность как функцию присутствия или отсутствия субъединиц последовательности.
В некоторых вариантах осуществления настоящего изобретения способ, воплощенный посредством компьютерного программного продукта, получает по меньшей мере одну новую модель посредством добавления/удаления по меньшей мере одного нового члена, характеризующего взаимодействие, к/из основной модели, причем новый член, характеризующий взаимодействие, представляет взаимодействие между двумя или более взаимодействующими субъединицами. В некоторых вариантах осуществления способ определяет способность по меньшей мере одной новой модели предсказывать активность как функцию присутствия или отсутствия субъединиц. Способ также определяет, добавлять/удалять ли новый член, характеризующий взаимодействие, к/из основной модели, на основании способности по меньшей мере одной новой модели предсказывать активность, как определено выше и со смещением в сторону от включения дополнительных членов, характеризующих взаимодействие.
VIII. Примеры
Следующие примеры демонстрируют способ направленной эволюции, воплощающий различные варианты осуществления настоящего изобретения. Способ включает создание библиотеки белковых вариантов, генерацию различных форм моделей последовательность-активность на основании вариантов библиотеки и проведение направленной эволюции белков для достижения желаемого уровня представляющей интерес активности белка.
В начале конструировали комбинаторную библиотеку белковых вариантов, причем варианты белков имели 11 мутаций, используя технологию автоматического параллельного сплайсинга посредством удлинения перекрывания (SOEing) или APS. Библиотека включает мутации случайным образом в количестве приблизительно 3 мутации на вариант. 11 мутаций вариантов белков показаны в первых 11 строках таблицы VI.
Значения коэффициентов для линейной аддитивной модели, не учитывающей взаимодействие мультипликативной и учитывающей взаимодействие мультипликативной моделей
Затем способ получал данные о последовательности/активности для библиотеки. Сконструированные варианты секвенировали с использованием способа секвенирования нового поколения с возможностью штрихового кодирования. Исследование по секвенированию включало 6 96-луночных планшетов, причем каждый планшет содержал 6 лунок положительного контроля с остовом и 6 лунок отрицательного контроля. Исследование также измеряло приспособленность или желаемую активность белков. Приспособленность фиксировали в виде отношения к положительному контролю как меру кратного улучшения по сравнению с положительным контролем (FIOP).
Данные о последовательности/активности, полученные выше, использовали для построения матрицы данных, представляющей присутствие/отсутствие мутаций. Каждая строка матрицы данных представляла вариант (последовательность), в общей сложности nrows (179). Первый столбец матрицы содержал значения FIOP, представляющие активность вариантов. Все остальные столбцы содержали значения, представляющие присутствие/отсутствие мутации.
Полученную матрицу данных подгоняли с использованием не учитывающей взаимодействие аддитивной основной модели следующей формы:
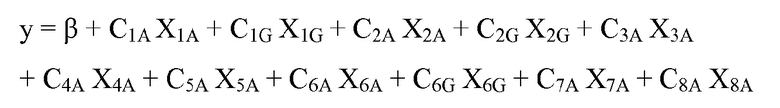
где y представляет собой активность, предсказываемую моделью, и β представляет собой "свободный член" линейного уравнения.
Применяли бутстреп-метод, использующий случайную выборку с возвращением, для подгонки модели к полученным данным с использованием следующей процедуры:
(a) Строки данных в матрице данных выбирали с возвращением nrows (179) раз.
(b) Выбранные данные подгоняли с использованием обычной регрессии наименьших квадратов.
(c) Этапы (a) и (b) повторяли 500 раз.
(d) Усредняли коэффициенты из каждого (c) для получения значений коэффициентов итоговой модели.
(e) Использовали модель, полученную из (d), для предсказания значения FIOP данных последовательностей в матрице данных. Наблюдаемые и предсказанные значения вместе наносили на диаграмму на фигуре 9A для каждого наблюдения.
Значения коэффициентов данной не учитывающей взаимодействие аддитивной модели показаны во втором столбце таблицы VI. Как можно видеть на фигуре 9A, не учитывающая взаимодействие аддитивная модель переоценивает уровни активности для многих наблюдений, особенно для тех, которые имеют низкие наблюдаемые уровни активности.
Второй пример использовал мультипликативную модель для подгонки тех же данных. Модель имеет следующую форму:
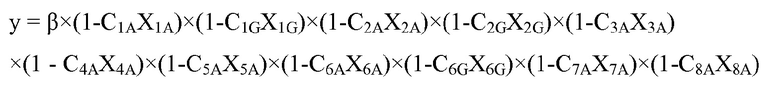
Альтернативно, модель может принимать следующую форму, если значения коэффициентов установлены так, чтобы эффективно включать постоянную 1 в вычисление значения y. Например, если коэффициенты для вышеуказанной модели установлены так, чтобы перекрывать диапазон от -0,9 до 1,5, то коэффициенты могут быть установлены так, чтобы перекрывать диапазон от 0,1 до 2,5 для следующей альтернативной модели для достижения аналогичного выхода модели:
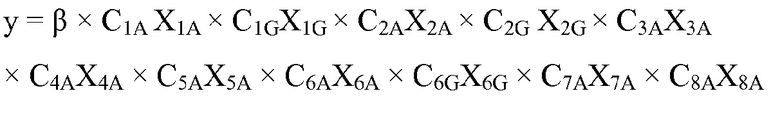
Модель уточняли посредством корректировки значений коэффициентов с использованием методов как бутстреппинга, так и генетического алгоритма. Подгонку модели осуществляли посредством минимизации среднеквадратичной ошибки предсказанной активности (или приспособленности) относительно наблюдения следующим образом:
(a) Выбирали данные о последовательности/активности строк с возвращением nrows раз для получения данных бутстреппинга.
(b) Подгоняли модель к выбранным данным с использованием генетического алгоритма, который ограничивал значения коэффициентов между -0,9 и 1,5. Генетический алгоритм имел размер популяции 200, и его выполняли для 100 поколений.
(c) Повторяли этапы (a) и (b) 500 раз.
(d) Усредняли 10 лучших хромосом/индивидуумов из каждого (c) для получения коэффициентов итоговой модели.
(e) Для каждой строки матрицы данных использовали (d) для предсказания значения FIOP. Наблюдаемые и предсказанные значения вместе наносили на диаграмму на фигуре 9B для каждой строки данных (наблюдения). Значения коэффициентов данной мультипликативной основной модели показаны в третьем столбце таблицы VI.
Как можно видеть на фигуре 9B, данная не учитывающая взаимодействие мультипликативная модель делает предсказания, которые лучше соответствуют наблюдаемым значениям активности библиотеки вариантов, так что она не имеет значительных постоянных завышенных оценок для вариантов белков, имеющих низкие наблюдаемые уровни активности. Однако остаточные ошибки все еще относительно велики, причем точки данных рассредоточены от диагональной линии диаграммы.
Следующий пример демонстрирует мультипликативную основную модель, уточненную с использованием пошагового способа для идентификации соответствующих членов, характеризующих взаимодействие, образующих учитывающую взаимодействие мультипликативную модель. Данная процедура запускала уточнение мультипликативной модели относительно мультипликативной основной модели, приведенной в предыдущем примере, со следующими этапами.
(a) Основную модель, полученную выше, принимали в качестве наилучшей модели.
(b) Все возможные коэффициенты парного взаимодействия помещали в совокупность коэффициентов. (1А*2А, 1А*3А, 1А*4А, …, 6G*8A, 7A*8A)
(c) Каждый коэффициент добавляли к наилучшей модели, которую подгоняли с использованием генетического алгоритма, имеющего те же параметры, что и описанные в предыдущем примере.
(d) Получали приспособленность каждой новой модели из (c) с использованием AIC.
(e) Модель с самым низким AIC (то есть наиболее приспособленную модель) из (d) принимали в качестве наилучшей модели текущего цикла.
(f) Если модель из (e) была лучше, чем наилучшая модель, в качестве наилучшей модели принимали эту модель, а коэффициент в этой модели удаляли из совокупности коэффициентов, и алгоритм переходил к (c) - иначе говоря, отсутствовали новые модели, и алгоритм завершался.
(g) Новую модель из (f) подгоняли с использованием способа бутстреппинга/подгонки, приведенного в предыдущем примере.
Значения коэффициента для этой учитывающей взаимодействие мультипликативной модели показаны в четвертом столбце таблицы VI, где последние 4 коэффициента относятся к членам, характеризующим взаимодействие. Как можно видеть в таблице во всех трех столбцах значений коэффициентов, относительные величины коэффициентов в модели сохраняли схожую структуру, что указывает на корректную сходимость процедур оптимизации для всех моделей.
Фигура 9C показывает в виде диаграммы активность вариантов белков, предсказанную учитывающей взаимодействие мультипликативной моделью, против наблюдаемых уровней активности вариантов белков. В этом случае учитывающая взаимодействие мультипликативная модель наилучшим образом предсказывает активность наблюдений среди трех моделей. Остаточные ошибки являются постоянно малыми во всем диапазоне уровней активности, причем точки данных распределены рядом с диагональной линией.
Следующие примеры показывают, как учитывающую взаимодействие мультипликативную модель, полученную выше, использовали для управления направленной эволюцией вариантов белков для достижения желаемой активности белка.
Один пример использует мутации, которые уже существуют в текущей библиотеке. В данном примере активность всех возможных комбинаций мутаций была предсказана с использованием учитывающей взаимодействие мультипликативной модели, полученной выше. Затем конструировали последовательности для всех возможных вариантов in silico, и их активности (FIOP) вычисляли с использованием учитывающей взаимодействие мультипликативной модели. Предсказанные FIOP были упорядочены от самых высоких до самых низких и приведены в таблице VII.
Лучшие 5 предсказаний показаны ниже вместе с их предсказанными значениями и наблюдаемыми значениями (если доступны). Ненаблюдаемые варианты синтезировали и повторно тестировали и сравнивали с наблюдаемыми вариантами. Наиболее эффективный вариант при повторном тестировании переносят для создания нового остова для эволюции.
Наиболее приспособленные последовательности, предсказанные учитывающей взаимодействие мультипликативной моделью
В данном примере направленной эволюции вредные мутации и комбинация мутаций были зафиксированы и были исключены из ближайших циклов эволюции. Кроме того, полезное разнообразие, идентифицированное, но не включенное в остов следующего цикла, рекомбинировали (если доступно) с ранее идентифицированным полезным разнообразием комбинаторным образом.
Другой пример направленной эволюции генерировал новое разнообразие с использованием механизмов разнообразия на основе мутагенеза. По пространству последовательностей осуществляли поиск посредством генерации разнообразия с помощью насыщающего мутагенеза комбинаторным образом в положениях, которые были идентифицированы учитывающей взаимодействие мультипликативной моделью как производящие значительные эффекты взаимодействия на представляющую интерес активность. Этими положениями являются те, которые имеют высокие значения коэффициентов взаимодействия: в данном случае положения 2, 3, 4 и 7. Насыщающий мутагенез осуществляли на этих положениях одновременно. Получаемые библиотеки подвергали скринингу на активность относительно остова, что помогает идентифицировать наиболее эффективные варианты.
В то время как вышеприведенное было описано достаточно подробно для целей ясности и понимания, специалисту в данной области техники после ознакомления с настоящим раскрытием будет понятно, что могут быть осуществлены различные изменения в форме и подробностях без отхода от фактического объема настоящего раскрытия. Например, все способы и приспособления, описанные выше, можно использовать в различных комбинациях. Все публикации, патенты, патентные заявки или другие документы, процитированные в данной заявке, включены посредством ссылки во всей полноте для всех целей в той же степени, как если бы все отдельные публикации, патенты, патентные заявки или другие документы были по отдельности указаны как включенные посредством ссылки для всех целей.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБЫ, СИСТЕМЫ И ПРОГРАММНОЕ ОБЕСПЕЧЕНИЕ ДЛЯ ИДЕНТИФИКАЦИИ БИОМОЛЕКУЛ СО ВЗАИМОДЕЙСТВУЮЩИМИ КОМПОНЕНТАМИ | 2014 |
|
RU2695146C2 |
| ОСНОВАННОЕ НА СТРУКТУРЕ ПРОГНОЗНОЕ МОДЕЛИРОВАНИЕ | 2014 |
|
RU2694321C2 |
| СПОСОБЫ ОБУЧЕНИЯ ГЛУБОКИХ СВЕРТОЧНЫХ НЕЙРОННЫХ СЕТЕЙ НА ОСНОВЕ ГЛУБОКОГО ОБУЧЕНИЯ | 2018 |
|
RU2767337C2 |
| ИДЕНТИФИКАЦИЯ, ПРОИЗВОДСТВО И ПРИМЕНЕНИЕ НЕОАНТИГЕНОВ | 2016 |
|
RU2729116C2 |
| ПРИМЕНЕНИЕ АДАПТИРОВАННЫХ РЕКОМБИНАЗ ДЛЯ ЛЕЧЕНИЯ РЕТРОВИРУСНЫХ ИНФЕКЦИЙ | 2008 |
|
RU2501860C2 |
| КОМПОЗИЦИИ И СПОСОБЫ С УЧАСТИЕМ НУКЛЕИНОВЫХ КИСЛОТ, НАЦЕЛЕННЫХ НА НУКЛЕИНОВЫЕ КИСЛОТЫ | 2014 |
|
RU2662932C2 |
| Компьютерно-реализуемый интегральный способ для оценки качества результатов таргетного секвенирования | 2018 |
|
RU2717809C1 |
| ПРЕДСКАЗАНИЕ ИММУНОГЕННОСТИ Т-КЛЕТОЧНЫХ ЭПИТОПОВ | 2014 |
|
RU2724370C2 |
| СПОСОБЫ УЛУЧШЕНИЯ ЭФФЕКТИВНОСТИ БЕЛКОВ | 2008 |
|
RU2569106C2 |
| ОБНАРУЖЕНИЕ МУТАЦИЙ И ПЛОИДНОСТИ В ХРОМОСОМНЫХ СЕГМЕНТАХ | 2015 |
|
RU2717641C2 |
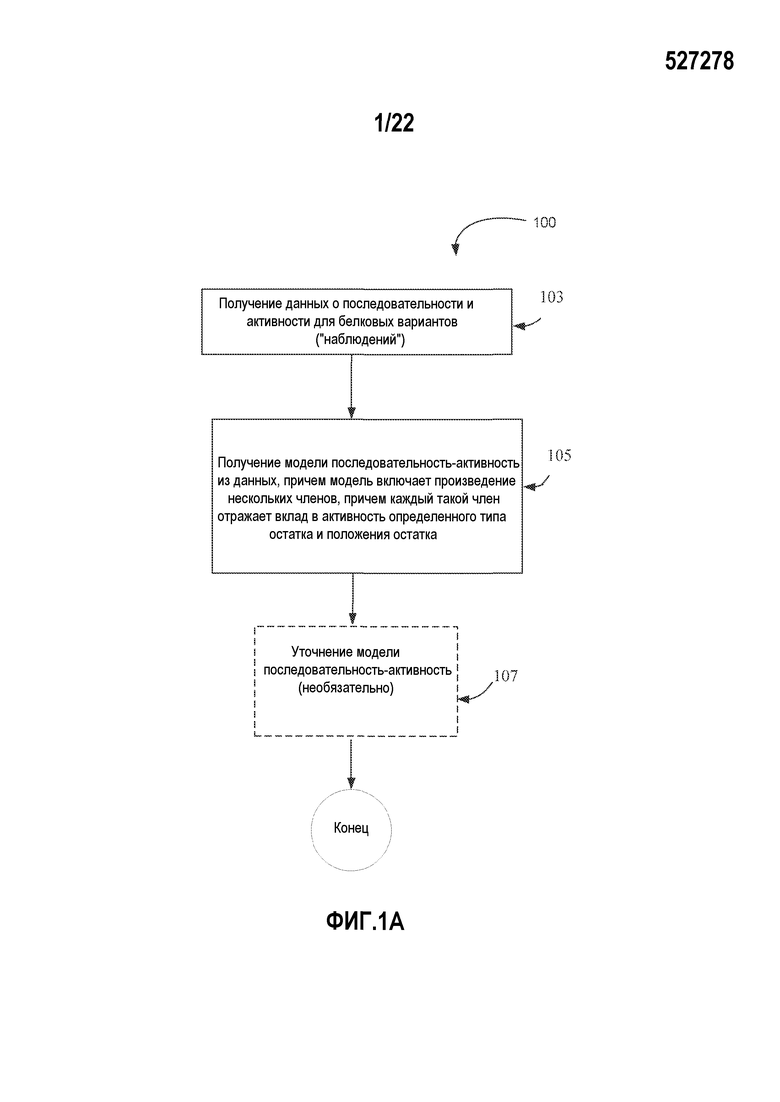
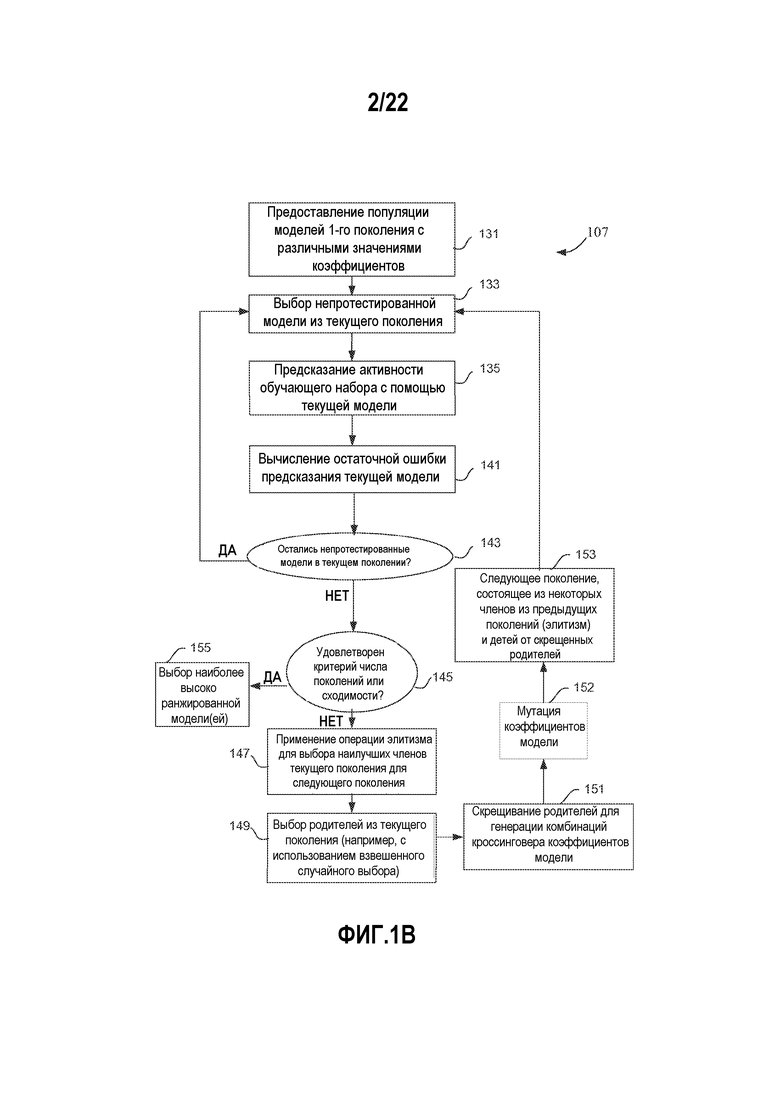
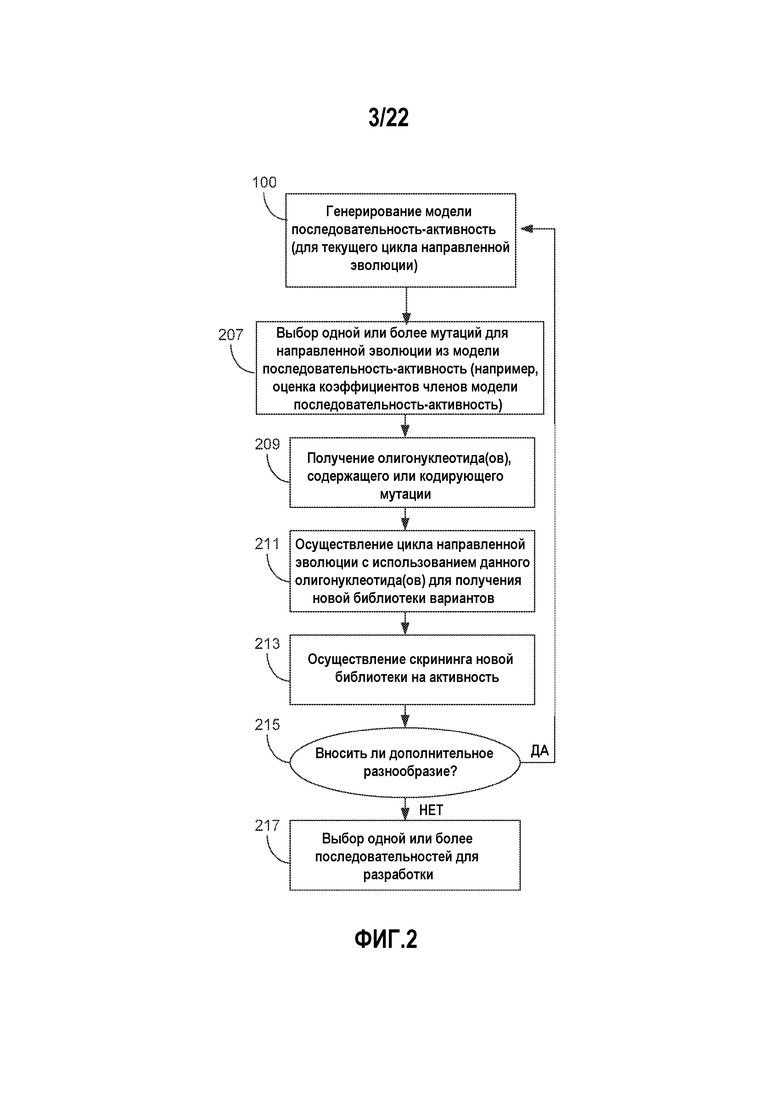
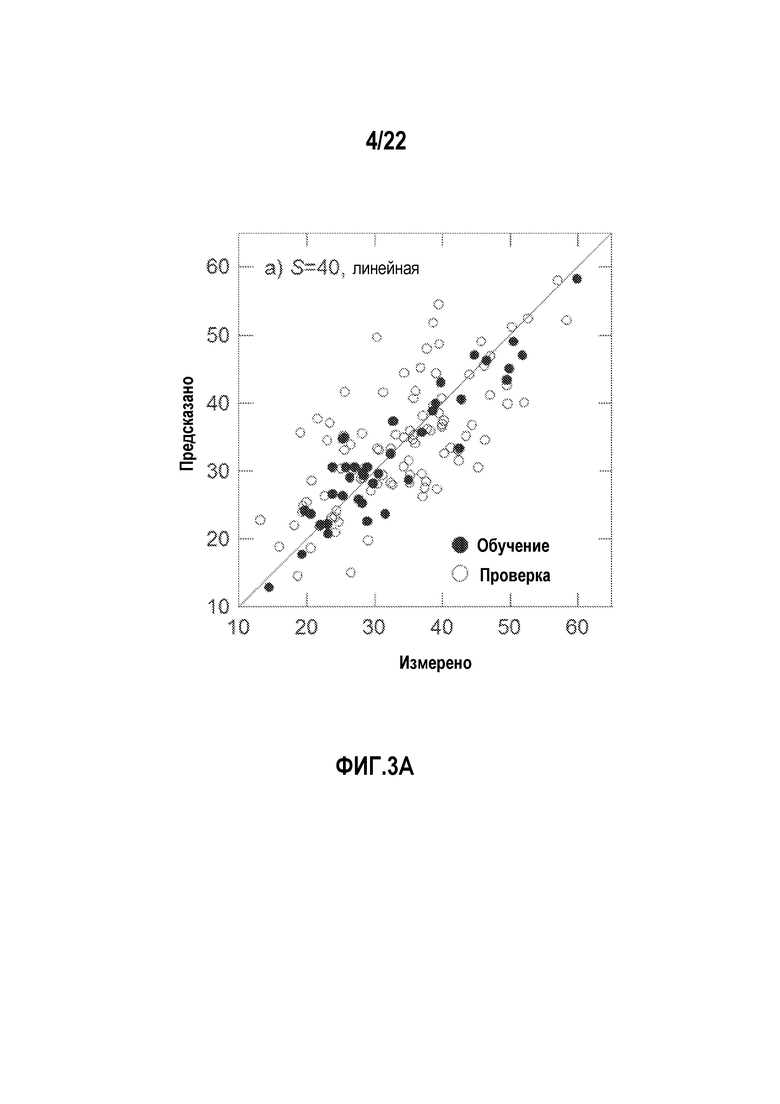
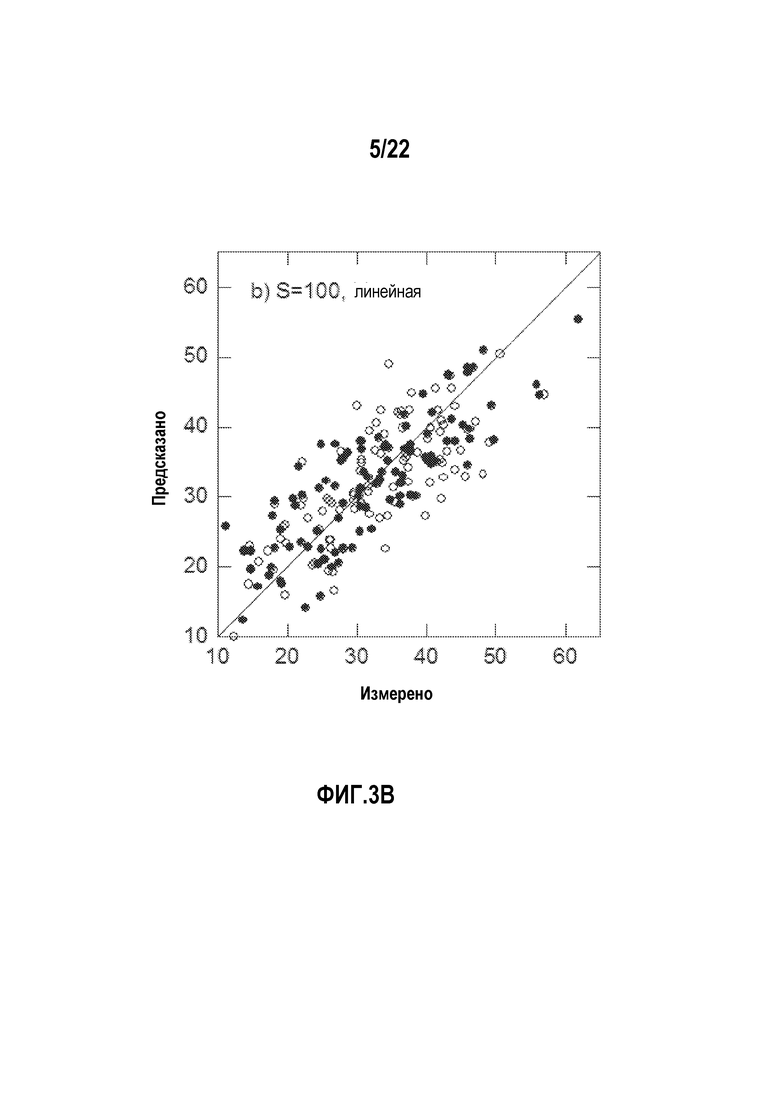
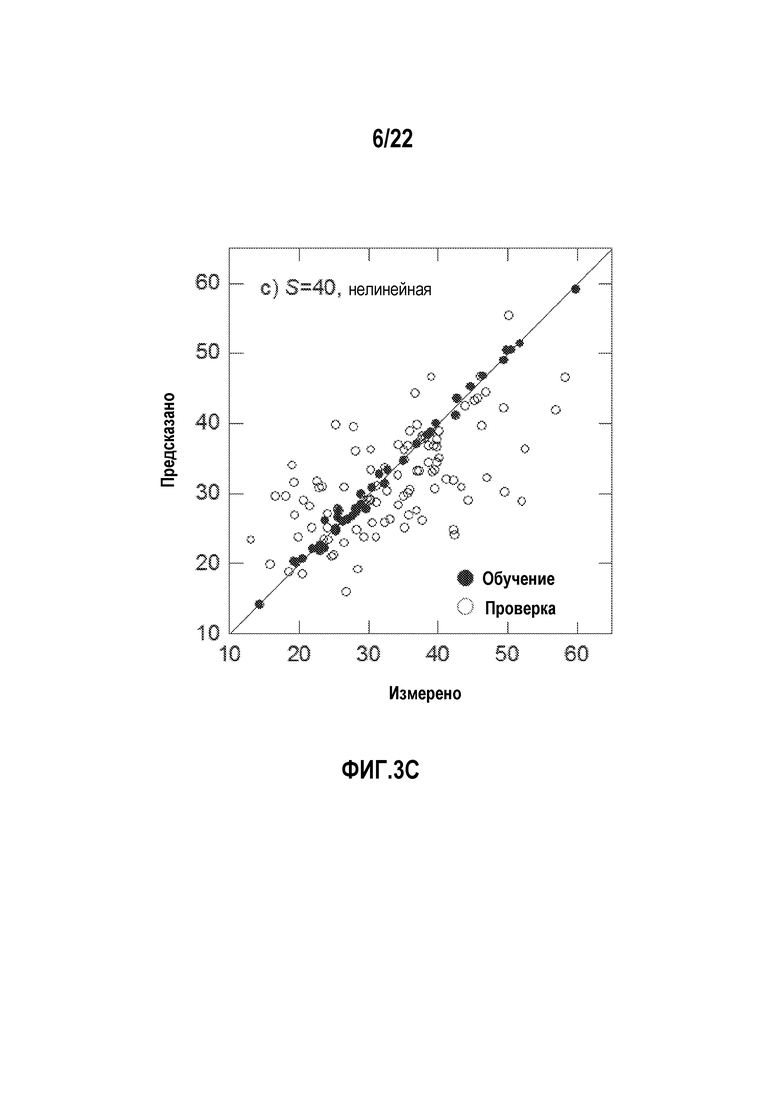
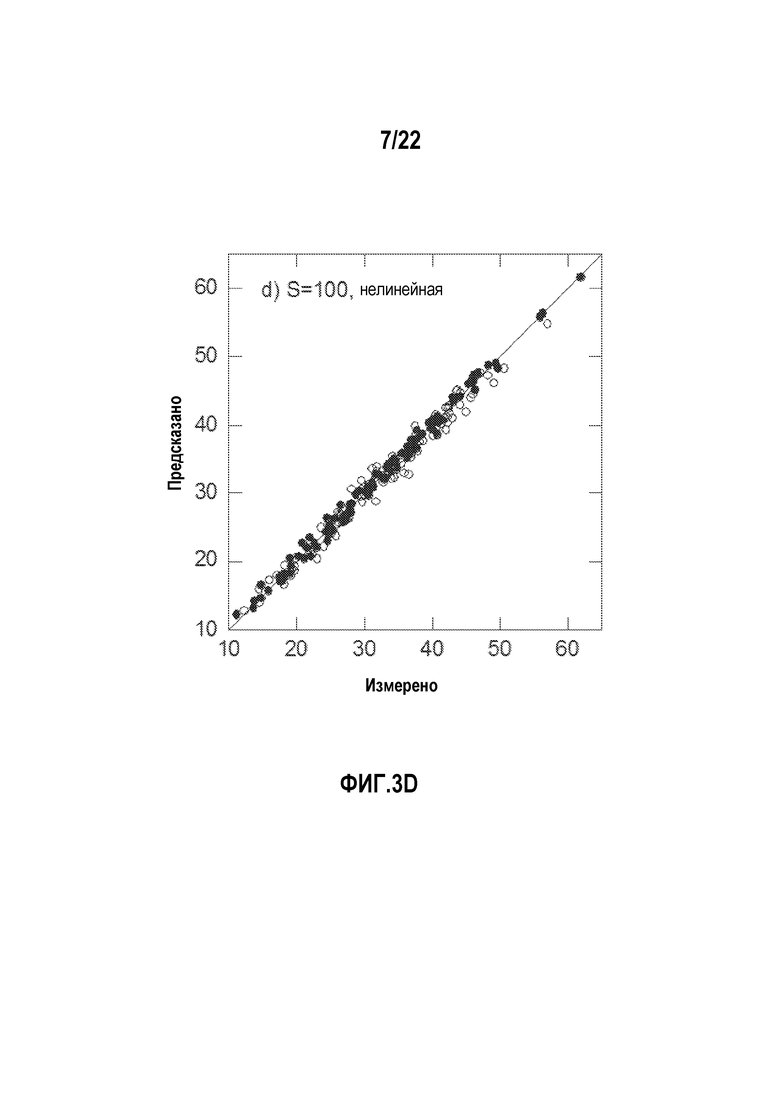
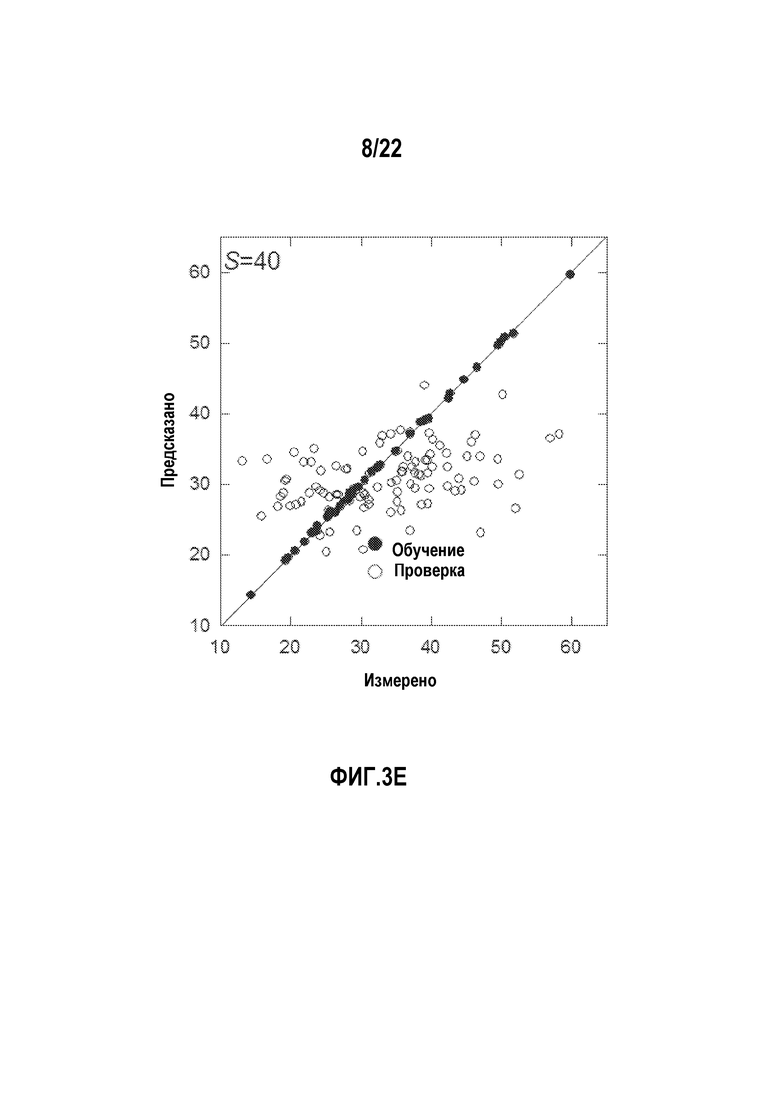
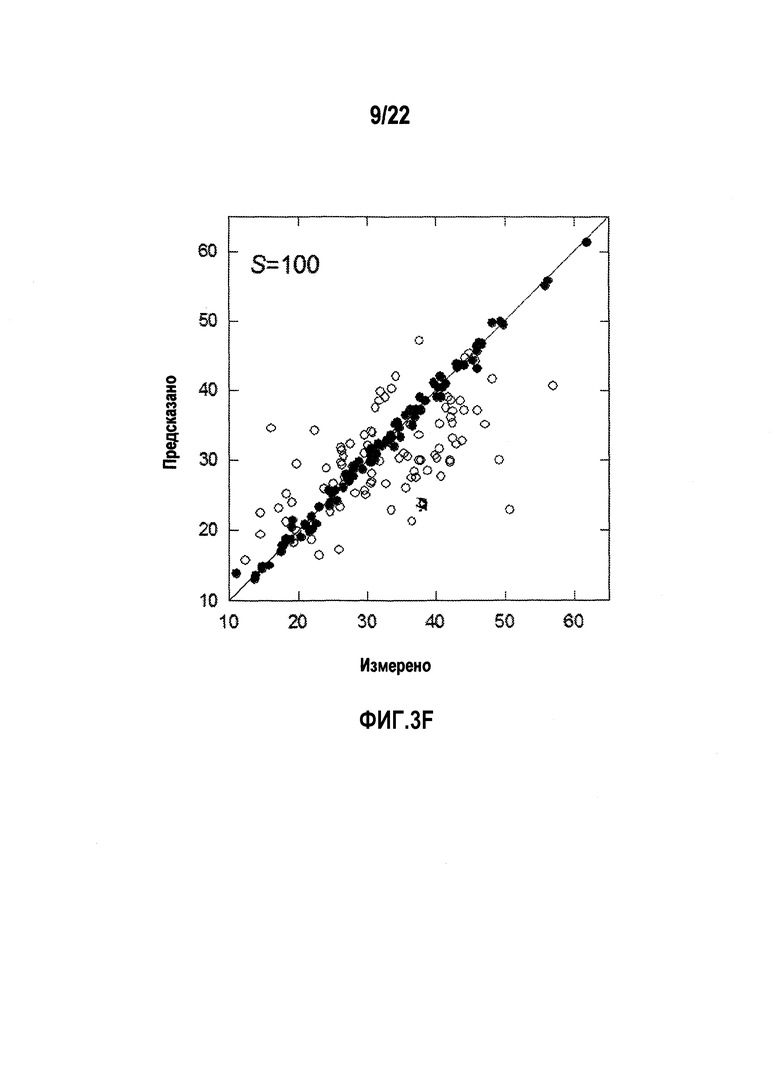
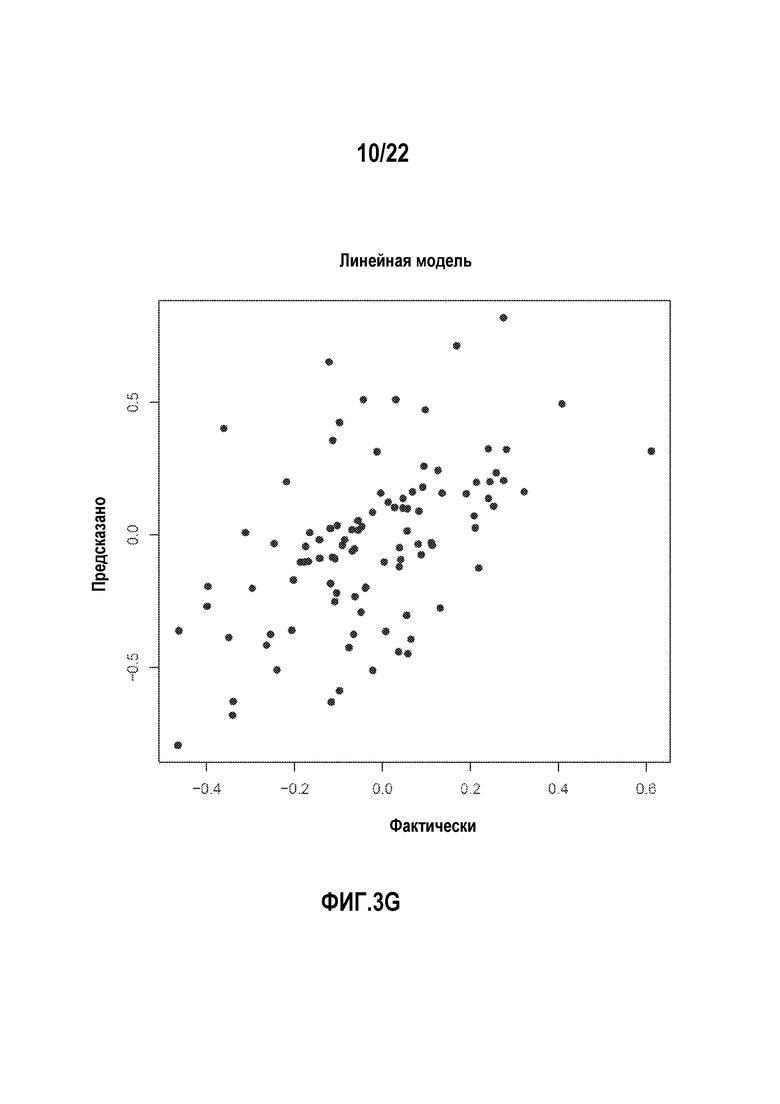
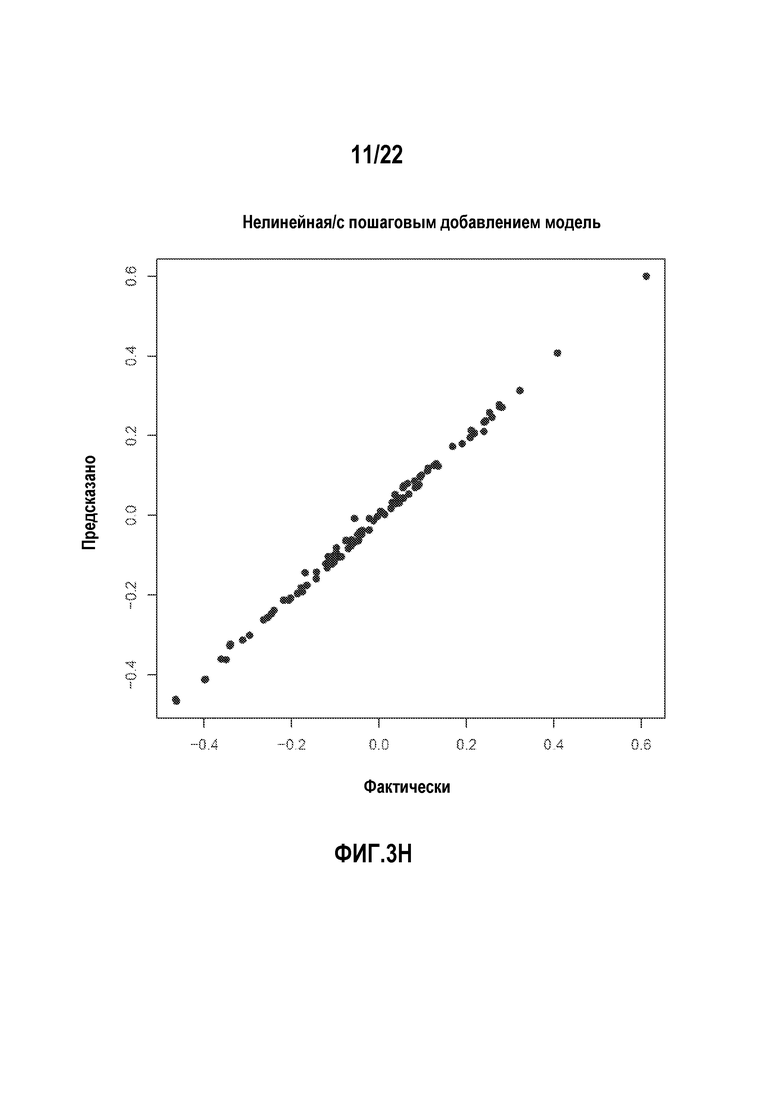
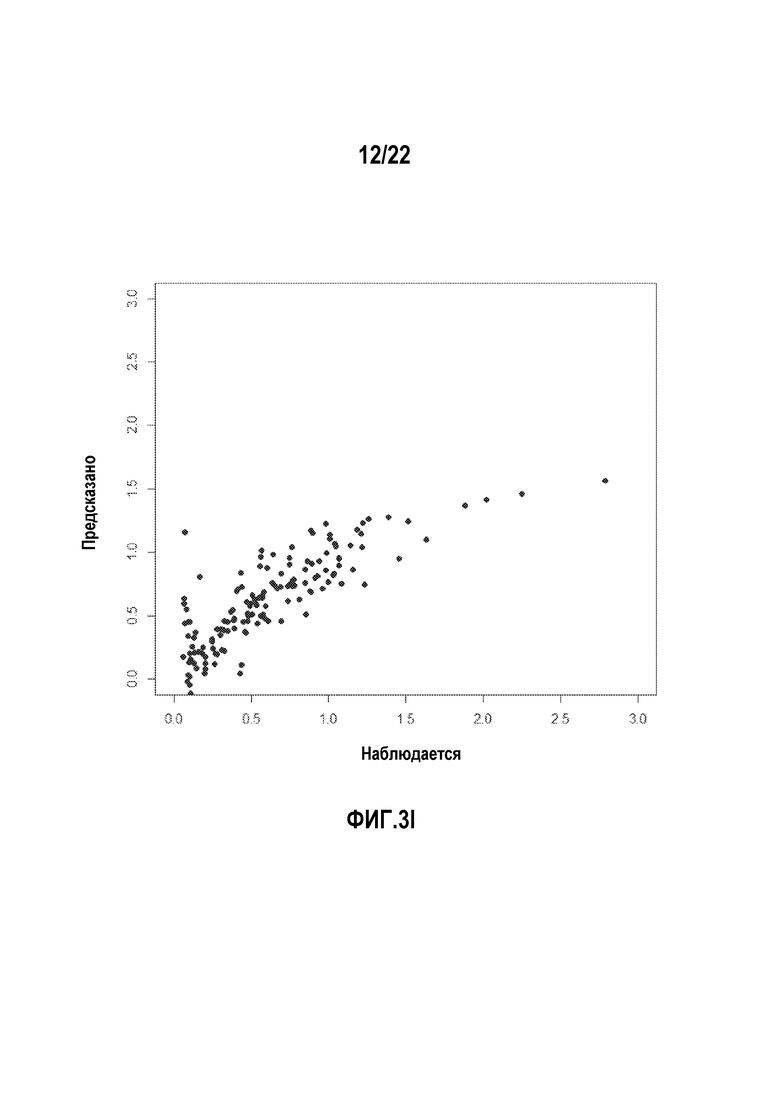
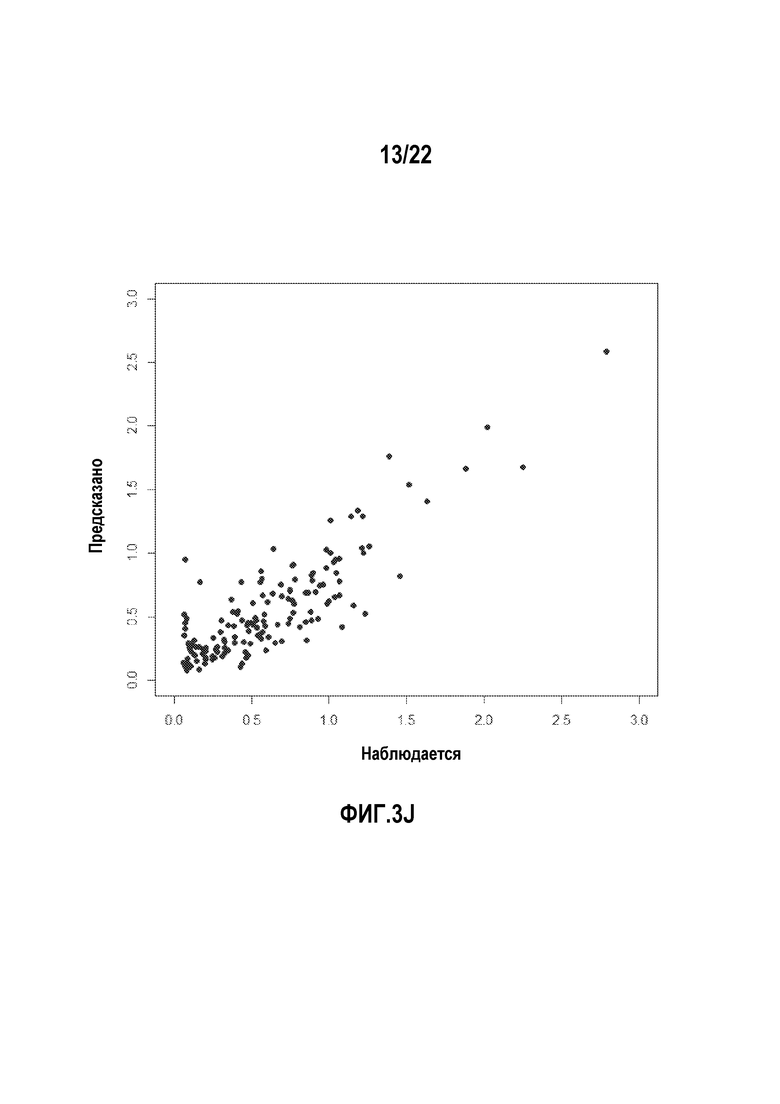
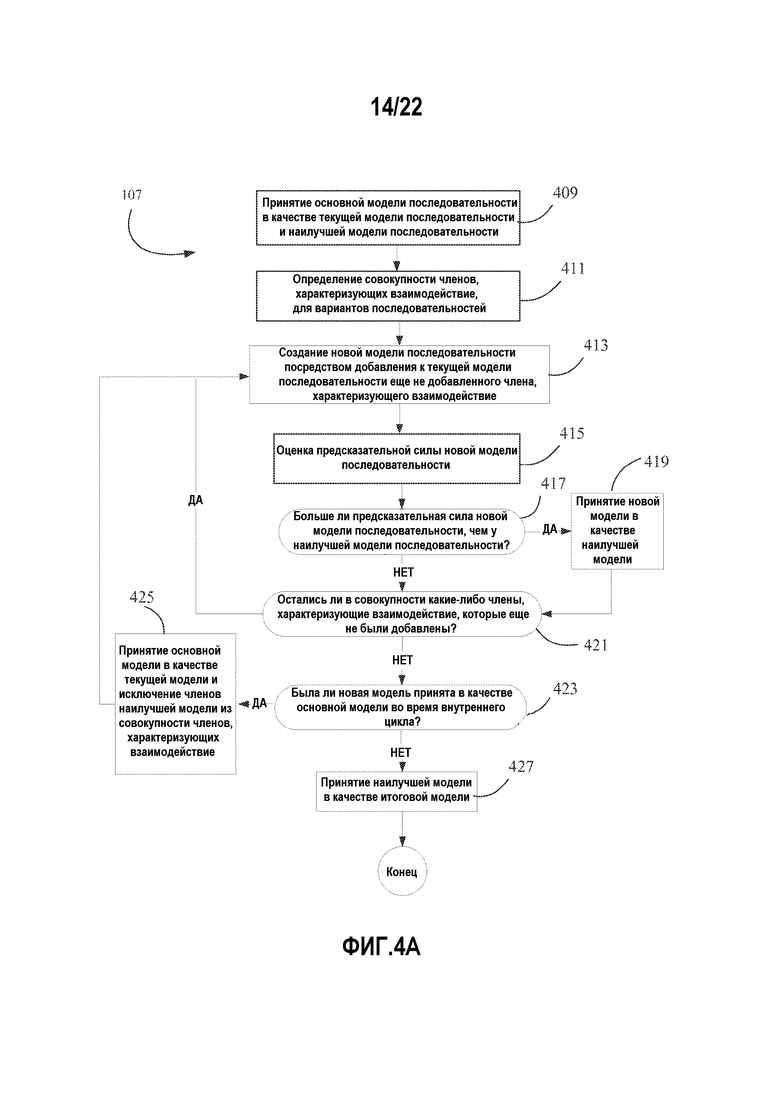
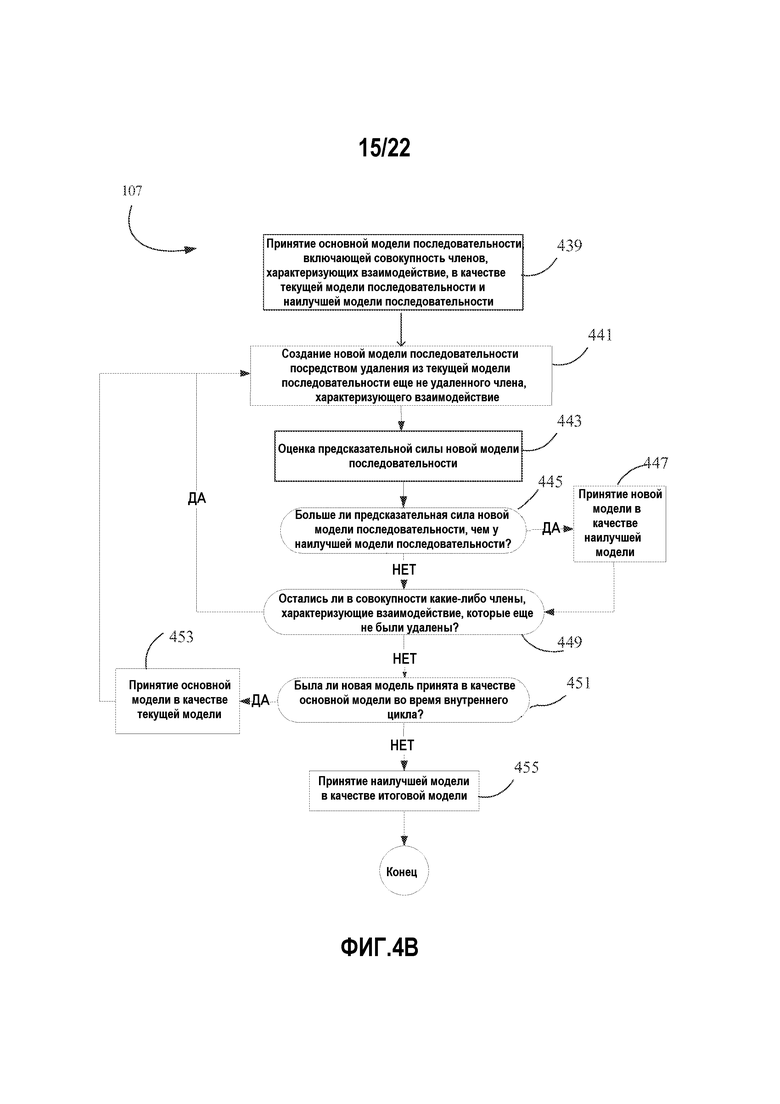
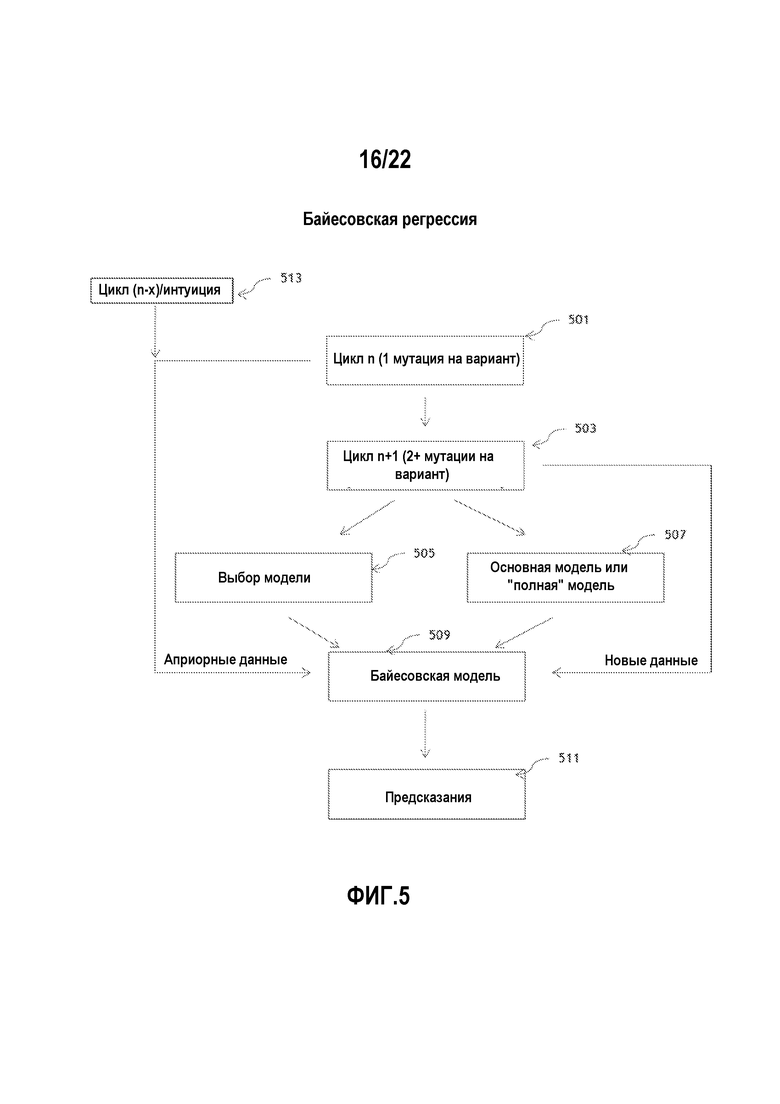
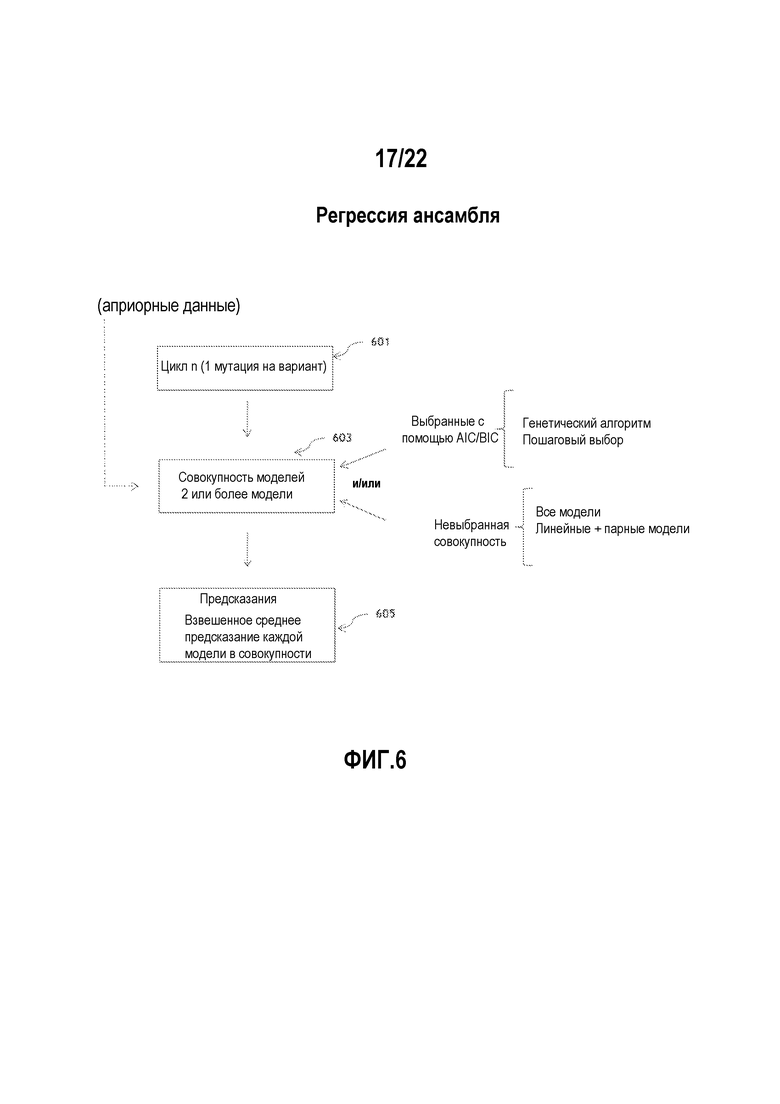
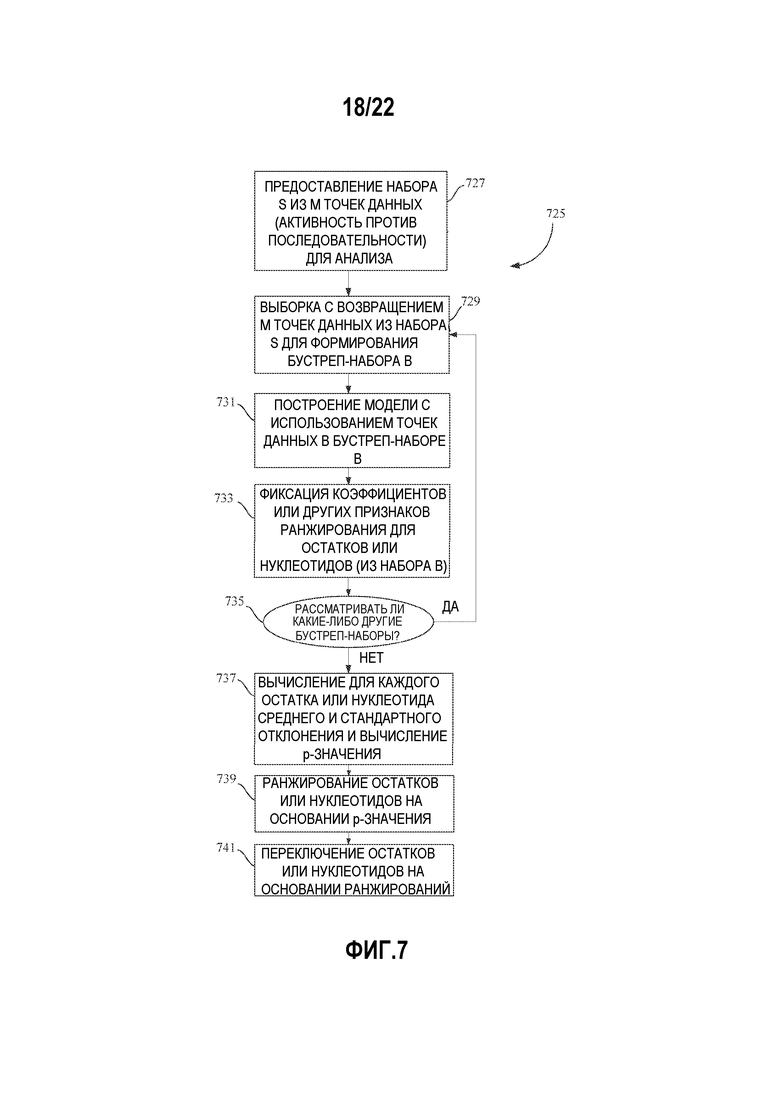

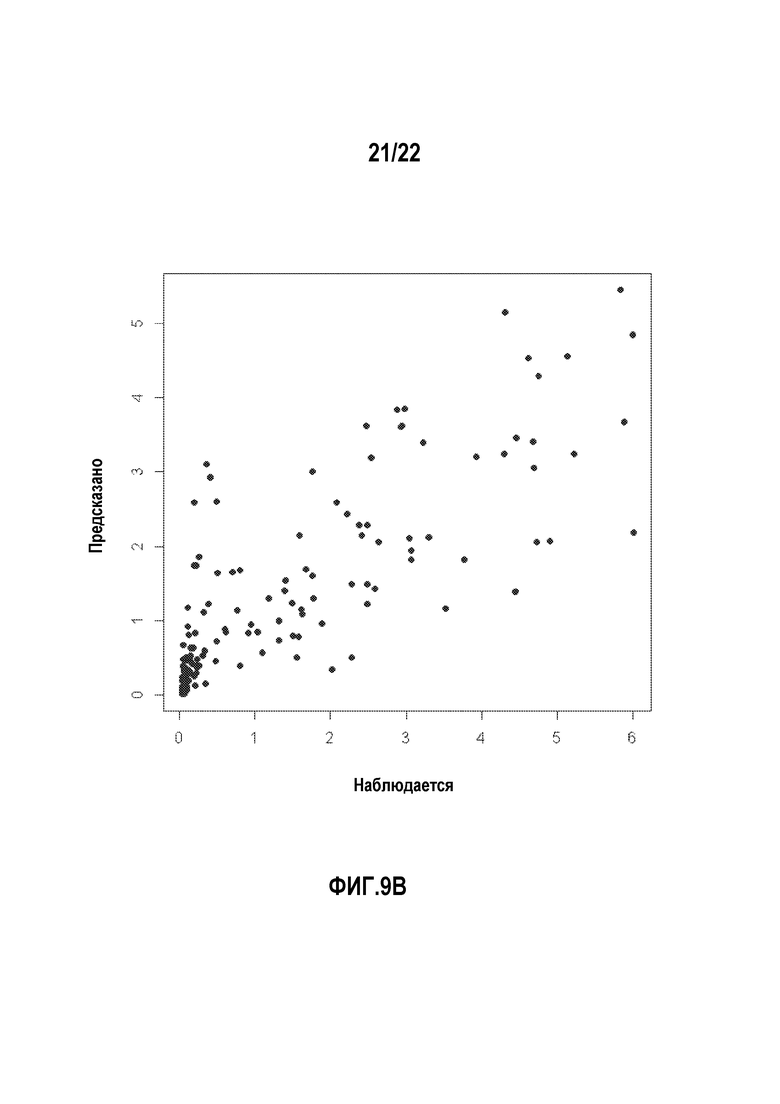
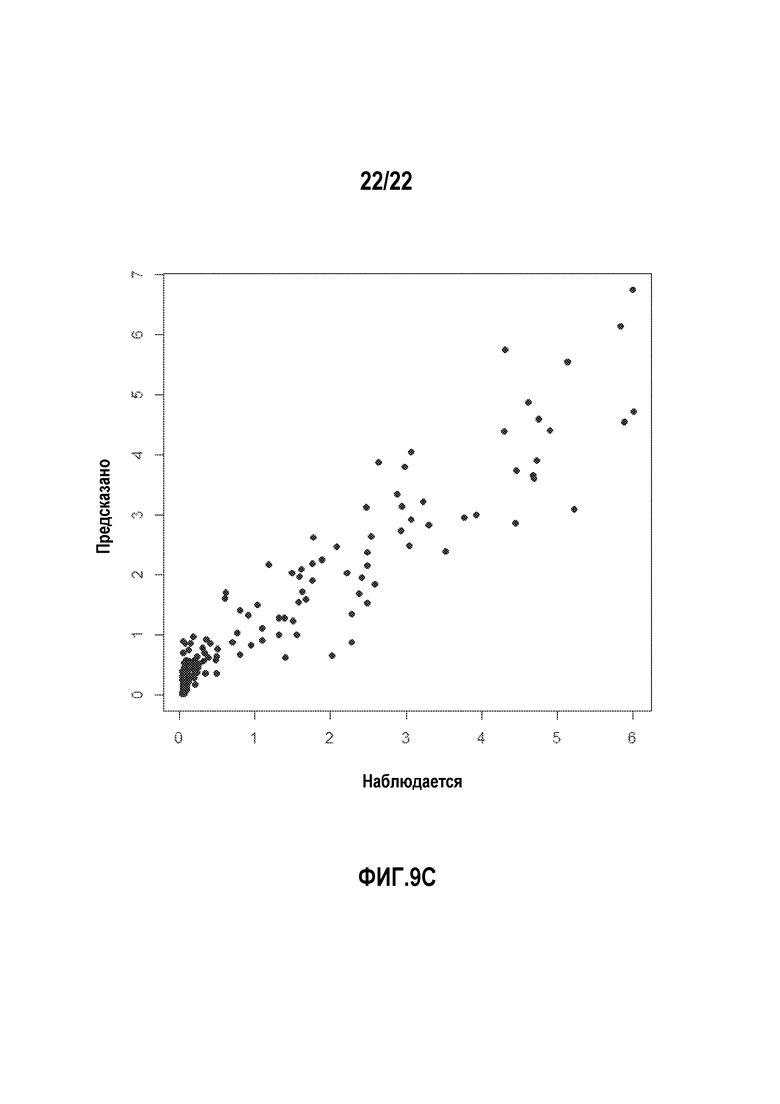
Группа изобретений относится к медицине и биотехнологии, и может быть использована для проведения направленной эволюции белков. Для этого проводят (a) получение данных о последовательности и данных об активности для каждого из множества вариантов белков; (b) генерацию мультипликативной модели последовательность-активность по данным о последовательности и данным об активности, где модель последовательность-активность связывает аминокислоты варианта белка или нуклеотидов, кодирующих вариант белка, с активностью варианта белка, и мультипликативная модель последовательность-активность содержит: произведение множественных невзаимодействующих мультипликативных членов, причем каждый невзаимодействующий мультипликативный член содержит произведение независимой переменной и коэффициента, где: (i) независимая переменная представляет определенную аминокислоту варианта белка или определенный нуклеотид, кодирующий вариант белка, и (ii) коэффициент представляет вклад в активность варианта бека только определенной аминокислотой или определенным нуклеотидом (c) использование модели последовательность-активность для осуществления цикла направленной эволюции одного или нескольких вариантов белка. Также предложена компьютерная система для проведения направленной эволюции белков. Группа изобретений обеспечивает идентификацию биомолекул с желаемыми свойствами или наиболее подходящих для получения таких свойств из библиотек сложных биомолекул или наборов таких библиотек. 6 н. и 26 з.п. ф-лы, 9 ил., 7 табл.
1. Способ проведения направленной эволюции белков, причем данный способ включает:
(a) получение данных о последовательности и данных об активности для каждого из множества вариантов белков;
(b) генерацию мультипликативной модели последовательность-активность по данным о последовательности и данным об активности, где
Мультипликативная модель последовательность-активность связывает аминокислоты варианта белка или нуклеотидов, кодирующих вариант белка, с активностью варианта белка, и
мультипликативная модель последовательность-активность содержит:
произведение множественных невзаимодействующих мультипликативных членов, причем каждый невзаимодействующий мультипликативный член содержит произведение независимой переменной и коэффициента, где: (i) независимая переменная представляет определенную аминокислоту варианта белка или определенный нуклеотид, кодирующий вариант белка, и (ii) коэффициент представляет вклад в активность варианта белка только определенной аминокислотой или определенным нуклеотидом;
(c) использование мультипликативной модели последовательность-активность для осуществления цикла направленной эволюции одного или нескольких вариантов белка.
2. Способ по п. 1, где использование модели последовательность-активность для осуществления цикла направленной эволюции одного или нескольких вариантов белка включает:
выбор одной или нескольких мутаций для цикла направленной эволюции путем оценки коэффициентов модели последовательность-активность для идентификации одной или нескольких аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность;
получение множества олигонуклеотидов, содержащих или кодирующих одну или несколько мутаций; и
проведение цикла направленной эволюции с использованием множества олигонуклеотидов.
3. Способ по п. 2, в котором получение множества олигонуклеотидов, содержащих или кодирующих мутации, выбранные в (с), содержит синтезирование олигонуклеотидов с помощью синтезатора нуклеиновых кислот.
4. Способ по п. 1 или 2, в котором осуществление цикла направленной эволюции содержит фрагментирование и рекомбинирование белка, о которой модель предсказывает, что она имеет желаемый уровень активности.
5. Способ по п. 1 или 2, в котором осуществление цикла направленной эволюции содержит осуществление насыщающего мутагенеза в отношении белка, о которой модель предсказывает, что она имеет желаемый уровень активности.
6. Способ проведения направленной эволюции белков, причем данный способ включает:
(a) получение данных о последовательности и активности для каждого из множества вариантов белков;
(b) генерацию мультипликативной модели последовательность-активность по данным о последовательности и активности для каждого из множества вариантов белков, причем мультипликативная модель последовательность-активность содержит:
произведение множественных невзаимодействующих мультипликативных членов, причем каждый невзаимодействующий мультипликативный член содержит произведение независимой переменной и коэффициента, где коэффициент представляет вклад в активность определенной аминокислоты или нуклеотида в определенном положении в белковой последовательности или нуклеиновокислотной последовательности, кодирующей белковую последовательность; и
независимая переменная представляет активность вариантов белков;
(c) выбор одной или более мутаций посредством оценивания коэффициентов членов модели последовательность-активность для идентификации одной или более из определенных аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность;
(d) идентифицирование новой белковой или новой нуклеиновокислотной последовательности, содержащей одну или более мутаций, выбранных в (с); и
(e) получение и анализирование нового белка или белка, кодируемого новой нуклеиновокислотной последовательностью.
7. Способ по п. 6, дополнительно содержащий использование новой белковой или новой нуклеиновокислотной последовательности в качестве исходной точки для дальнейшей направленной эволюции.
8. Способ по п. 6, дополнительно включающий проведение насыщающего мутагенеза в положениях одной или более мутаций.
9. Способ проведения направленной эволюции белков, причем данный способ включает:
(a) получение данных о последовательности и активности для каждого из множества вариантов белков;
(b) генерацию мультипликативной модели последовательность-активность по данным о последовательности и активности для каждого из множества вариантов белков, причем мультипликативная модель последовательность-активность содержит:
произведение множественных невзаимодействующих мультипликативных членов, причем каждый невзаимодействующий мультипликативный член содержит произведение независимой переменной и коэффициента, где коэффициент представляет вклад в активность определенной аминокислоты или нуклеотида в определенном положении в белковой последовательности или нуклеиновокислотной последовательности, кодирующей белковую последовательность; и
независимая переменная представляет активность вариантов белков;
(c) выбор одного или более положений в белковой последовательности или нуклеиновокислотной последовательности посредством оценивания коэффициентов модели последовательность-активность для идентификации одной или более аминокислот или нуклеотидов в определенных положениях, которые вносят вклад в активность; и
(d) проведение насыщающего мутагенеза в выбранных одном или более положениях.
10. Способ проведения направленной эволюции белков, причем данный способ включает:
(a) получение данных о последовательности и активности для каждого из множества вариантов белков;
(b) генерацию мультипликативной модели последовательность-активность по данным о последовательности и активности для каждого из множества вариантов белков, причем мультипликативная модель последовательность-активность содержит:
произведение множественных невзаимодействующих мультипликативных членов, причем каждый невзаимодействующий мультипликативный член содержит произведение независимой переменной и коэффициента, где коэффициент представляет вклад в активность определенной аминокислоты или нуклеотида в определенном положении в белковой последовательности или нуклеиновокислотной последовательности, кодирующей белковую последовательность; и
независимая переменная представляет активность вариантов белков;
(c) использование множественных белковых последовательностей или множественных аминокислотных последовательностей в модели последовательность-активность и определение значений активности, предсказанных моделью последовательность-активность для каждой из множественных белковых последовательностей или нуклеиновокислотных последовательностей;
(d) выбор новой белковой последовательности или новой нуклеиновокислотной последовательности из множественных белковых последовательностей или множественных аминокислотных последовательностей посредством оценивания значений активности, предсказанных моделью последовательность-активность для данных множественных белковых или аминокислотных последовательностей; и
(e) получение и анализирование белка, имеющего новую белковую последовательность, или белка, кодируемого новой нуклеиновокислотной последовательностью.
11. Способ по п. 10, в котором получение белка, имеющего новую белковую последовательность, или белка, кодируемого новой нуклеиновокислотной последовательностью, содержит синтезирование нового белка или новой нуклеиновокислотной последовательности.
12. Способ по любому из пп. 1, 6, 9 и 10, в котором каждый невзаимодействующий мультипликативный член имеет вид (коэффициент × независимая переменная).
13. Способ по любому из пп. 1, 6, 9 и 10, в котором каждый невзаимодействующий мультипликативный член представлен в форме (1 + коэффициент × независимая переменная).
14. Способ по любому из пп. 1, 6, 9 и 10, в котором модель последовательность-активность содержит зависимость между зависимой переменной, представляющей активность, и произведением множественных членов.
15. Способ по любому из пп. 1, 6, 9 и 10, в котором осуществление цикла направленной эволюции содержит перетасовку множества олигонуклеотидов, содержащих или кодирующих мутации, выбранные с использованием модели последовательность-активность.
16. Способ по любому из пп. 1, 6, 9 и 10, в котором осуществление цикла направленной эволюции содержит формирование библиотеки белковых вариантов.
17. Способ по п. 16, дополнительно содержащий анализирование активности и секвенирование членов библиотеки белковых вариантов.
18. Способ по п. 17, дополнительно содержащий использование информации об активности и последовательности для библиотеки белковых вариантов для генерации и использования новой модели последовательность-активность посредством осуществления операций (b)-(с) в отношении информации об активности и последовательности для библиотеки белковых вариантов.
19. Способ по любому из пп. 1, 6, 9 и 10, в котором по меньшей мере один из множественных членов модели последовательность-активность содержит коэффициент взаимодействия, представляющий вклад в активность определенной комбинацией из (i) первой аминокислоты или нуклеотида в первом положении в последовательности и (ii) второй аминокислоты или нуклеотида во втором положении в последовательности, и в котором коэффициент взаимодействия представляет вклад в активность упомянутой определенной комбинацией.
20. Способ по п. 19, в котором генерация модели последовательность-активность содержит осуществление пошагового добавления или удаления членов, содержащих коэффициенты взаимодействия.
21. Способ по п. 19, в котором генерация модели последовательность-активность содержит использование генетического алгоритма для выбора одного или более членов, содержащих коэффициенты взаимодействия.
22. Способ по любому из пп. 1, 6, 9 и 10, в котором генерация модели последовательность-активность содержит использование генетического алгоритма для уточнения значений коэффициентов модели последовательность-активность.
23. Способ по п. 21, в котором генерация модели последовательность-активность содержит:
(i) получение поколения моделей, причем каждое содержит произведение множественных членов;
(ii) использование каждой из моделей в поколении моделей для предсказания активности по меньшей мере некоторых из множества вариантов белков с использованием последовательностей вариантов белков;
(iii) выбор одной или более моделей из поколения моделей на основании способности моделей точно предсказывать активность множества вариантов белков;
(iv) модифицирование выбранных одной или более моделей для получения следующего поколения моделей; и
(v) многократное повторение (ii)-(iv) со следующим поколением моделей для генерации модели последовательность-активность (b).
24. Способ по п. 22, в котором генерация модели последовательность-активность содержит:
(i) получение поколения моделей, причем каждое содержит произведение множественных членов;
(ii) использование каждой из моделей в поколении моделей для предсказания активности по меньшей мере некоторых из множества вариантов белков с использованием последовательностей вариантов белков;
(iii) выбор одной или более моделей из поколения моделей на основании способности моделей точно предсказывать активность множества вариантов белков;
(iv) модифицирование выбранных одной или более моделей для получения следующего поколения моделей; и
(v) многократное повторение (ii)-(iv) со следующим поколением моделей для генерации модели последовательность-активность (b).
25. Способ по любому из пп. 1, 6, 9 и 10, в котором генерация модели последовательность-активность содержит использование априорной информации для определения апостериорных распределений вероятностей модели.
26. Способ по любому из пп. 1, 6, 9 и 10, дополнительно содержащий генерацию две или более модели последовательность-активность, причем каждая имеет форму, приведенную в (b).
27. Способ по п. 26, дополнительно содержащий генерацию ансамблевой модели, включающей члены из двух или более моделей последовательность-активность, причем данные члены ансамблевой модели взвешены по способности данных двух или более моделей предсказывать активность.
28. Способ по п. 26, дополнительно содержащий использование ансамблевой модели для выбора мутаций для цикла направленной эволюции.
29. Способ проведения направленной эволюции белков, причем данный способ включает:
(a) получение данных о последовательности и данных об активности для множества биологических молекул, причем каждая биологическая молекула содержит последовательность, содержащую субъединицы различных типов и положений в последовательности;
(b) построение мультипликативной модели последовательность-активность по полученным данным, причем мультипликативная модель последовательность-активность связывает активность биологической молекулы и субъединицы в биологической молекуле,
мультипликативная модель последовательность-активность содержит произведение множества основных невзаимодействующих мультипликативных членов, причем каждый из основных невзаимодействующих мультипликативных членов содержит произведение фиктивной независимой переменной и коэффициента, где (1) фиктивная независимая переменная представляет присутствие/отсутствие определенной субъединицы конкретного типа в конкретном положении биологической молекулы, и (2) коэффициент представляет вклад только определенной субъединицы в активность биологической молекулы; и
(с) использование мультипликативной модели последовательность-активность, для того чтобы идентифицировать одну или более субъединиц конкретных типов в конкретных положениях для вариации для воздействия на активность биологических молекул.
30. Способ по п. 29, в котором последовательность представляет собой весь геном, целую хромосому, сегмент хромосомы, коллекцию последовательностей генов взаимодействующих генов, ген, белок или полисахарид или любую их комбинацию.
31. Способ по п. 29, в котором субъединица представляет собой хромосому, сегмент хромосомы, гаплотип, ген, кодон, мутацию, нуклеотид, аминокислоту, моносахарид, липид или любую их комбинацию.
32. Компьютерная система для выполнения способа направленной эволюции белков по любому из пп. 1-31, содержащая:
один или более процессоров;
системную память; и
одну или более читаемых компьютером сред для хранения, несущих сохраненные на них выполнимые компьютером инструкции, которые, когда выполняются одним или более процессорами, вынуждают компьютерную систему осуществлять способ проведения направленной эволюции белков, причем данный способ включает:
(a) получение данных о последовательности и данных об активности для множества белков, причем каждый белок содержит последовательность, содержащую субъединицы различных типов и положений в последовательности;
(b) построение мультипликативной модели последовательность-активность по полученным данным, причем
мультипликативная модель последовательность-активность связывает активность биологической молекулы и субъединицы в биологической молекуле,
мультипликативная модель последовательность-активность содержит произведение множества основных невзаимодействующих мультипликативных членов, причем каждый из основных невзаимодействующих мультипликативных членов содержит произведение фиктивной определенной переменной и коэффициента, где (1) фиктивная определенная переменная представляет присутствие/отсутствие определенной субъединицы конкретного типа в конкретном положении биологической молекулы, и (2) коэффициент представляет вклад только определенной субъединицы в активность биологической молекулы; и
(с) использование мультипликативной модели последовательность-активность, для того чтобы идентифицировать одну или более субъединиц конкретных типов в конкретных положениях для вариации для воздействия на активность белков .
| US2011257023 A1, 20.01.2011 | |||
| US6171820 B1, 09.01.2001 | |||
| WO2006002267 A1, 05.01.2006 | |||
| RICHARD FOX | |||
| Крутильный аппарат | 1922 |
|
SU234A1 |
Авторы
Даты
2018-08-21—Публикация
2014-01-29—Подача