Область техники, к которой относится изобретение
Изобретение относится к пептидам, способным реактивировать мутантные белки р53, а также к их применению в терапии.
ПРЕДПОСЫЛКИ СОЗДАНИЯ ИЗОБРЕТЕНИЯ
Рак является главной причиной смерти в развитых странах, и поскольку средний возраст населения продолжает расти, то растет и число диагностированных случаев, что влечет за собой экономические последствия. Рак не является одним заболеванием, это группа более чем 200 заболеваний, характеризующихся неконтролируемым ростом и распространением аномальных клеток. Рак является в высокой степени гетерогенным заболеванием со значительными молекулярными различиями в экспрессии и распределении маркеров поверхности опухолевых клеток даже среди больных с раком одного и того же типа и степени. Кроме того, клеточные мутации, как правило, накапливаются по мере того, как рак прогрессирует, приводя к дальнейшему повышению гетерогенности опухоли. Большинство опухолевых клеток демонстрируют геномную нестабильность при повышенной экспрессии онкогенов и инактивации генов-супрессоров опухолей.
Ген р53 считается наиболее важным геном-супрессором опухолей, который действует в качестве главного барьера для прогрессирования рака. Белок р53 реагирует на различные типы клеточного стресса и запускает блокировку клеточного цикла, апоптоз или старение (Levine, J.A., p53, the cellular gatekeeper for growth and division. Cell, 1997. 88: p. 323-331). Это достигается за счет транскрипционной трансактивации специфических генов-мишеней, несущих ДНК-связывающие мотивы р53. Общепризнанно, что метаболический путь р53 нарушается почти во всех злокачественных опухолях человека. Мутация р53 рассматривается как важный шаг в процессе злокачественной трансформации и более чем в 50% случаев злокачественные опухоли несут мутации в своих генах р53. Большинство из этих мутаций являются точечными миссенс-мутациями, которые нацелены на кóровый ДНК-связывающий домен (DBD) р53, тем самым отменяя специфическое ДНК-связывание р53 с его целевым сайтом. Эти мутации предотвращают р53-зависимую транскрипцию и, следовательно, р53-опосредованную супрессию опухоли. Исключительно высокая частота мутаций р53 в человеческих злокачественных опухолях различных типов делает р53 уникальным среди генов, участвующих в развитии опухоли, что делает мутированный р53 (Mut-p53) привлекательной мишенью для новых методов лечения рака.
Структурные исследования показали, что полученные из опухоли миссенс-мутации в ДНК-связывающем домене р53 дают общий эффект: дестабилизация сворачивания DBD при физиологической температуре. (Joerger, A.C., M.D. Allen, and A.R. Fersht, Crystal structure of a superstable mutant of human p53 core domain. Insights into the mechanism of rescuing oncogenic mutations. J Biol Chem, 2004. 279(2): p. 1291-6). Эта дестабилизация может быть обратимой, так как некоторые мутанты могут реверсировать в конформацию дикого типа и связываться с ДНК при пониженных температурах. Таким образом, большинство мутаций р53 дестабилизируют сворачивание белка р53, вызывая частичную денатурацию при физиологической температуре.
Мутантные белки р53 накапливаются на высоких уровнях в опухолевых клетках, в основном из-за их неспособности повышающе регулировать экспрессию собственного деструктора Mdm2 белка p53. Кроме того, многие активирующие р53 стресс-сигналы (например, гипоксия, геномная нестабильность и экспрессия онкогенов) конститутивно индуцируются в раковых клетках. Таким образом, реактивация Mut-p53, как предполагается, проявляет значительные противоопухолевые эффекты. Кроме того, было показано на мышиной модели, что восстановление функций р53 хорошо переносится в нормальных тканях и не вызывает никаких видимых токсических эффектов (Ventura, A., et al., Restoration of p53 function leads to tumour regression in vivo. Nature, 2007. 445(7128): p. 661-5).
Белок р53 эволюционировал и стал динамичным и конформационно нестабильным. Отсутствие жесткой структуры белка р53 может привести к ряду конформеров р53, отображая различную активность в зависимости от типа стресса и клеточного контекста. В упрощенной модели p53 может принимать либо дикий тип, активную конформацию, либо мутантную, неправильно сложенную, неактивную конформацию. Два конформационных состояния р53 могут различаться двумя специфическими моноклональными антителами. PAb240 и PAb1620 (Wang, P.L., F. Sait, and G. Winter, The 'wild type' conformation of p53: epitope mapping using hybrid proteins. Oncogene, 2001. 20(18): p. 2318-24). Антитело PAb240 связывается с остатками 212-217 в DBD р53. Этот участок недоступен для антитела (Ab) в конформации дикого типа (WT). Однако в денатурированным или мутантном р53 он открыт (Vojtesek, B., et al., Conformational changes in p53 analyzed using new antibodies to the core DNA binding domain of the protein. Oncogene, 1995. 10(2): p. 389-93). Антитело PAb1620 распознает конформационный, нелинейный эпитоп в DBD, состоящий из двух отдельных участков p53 и включающий остатки R156, L206, R209 и N210 (Cook, A. and J. Milner, Evidence for allosteric variants of wild-type p53, a tumor suppressor protein. Br J Cancer, 1990. 61(4): p. 548-52). В конформации дикого типа белок складывается таким образом, что удерживает петли в непосредственной близости друг к другу (Ravera, M.W., et al., Identification of an allosteric binding site on the transcription factor p53 using a phage-displayed peptide library. Oncogene, 1998. 16(15): p. 1993-9), образуя полный эпитоп, распознаваемый антителом PAb1620. Когда белок р53 свернут неправильно (под действием мутации, температуры, денатурации или тому подобного), эти две петли отодвигаются дальше, эпитоп разрушается и вследствие этого мутантная конформация является PAb1620-отрицательной. Было показано, что р53 является конформационно гибким белком. Тем не менее, дефект складывания таких мутантов не является необратимым: некоторые мутанты р53 сохраняют остаточную ДНК-связывающую способность, мутанты, которые не связываются с ДНК при 37°С, могут связываться при значениях температуры ниже физиологической (32°C или 25°C) и активировать транскрипцию от р53-чувствительного промотора при 26°C. Кроме того, было показано, что выделенные ДНК-связывающие домены мутантных белков R245S, R282W и V143A и другие обладают остаточной (30-60%) ДНК-связывающей активностью при 20°С.
Структурные исследования показывают, что степень неправильного сворачивания отличается среди мутантов; однако нет определенного альтернативного складывания, а скорее частичная денатурация. Это говорит о том, что способ «малой молекулы» для реверсирования действия мутации р53 на сворачивание может быть применим к широкому диапазону мутантных форм. Другим важным прогнозом структурных исследований является то, что лиганд, который связывается с правильно сложенной фракцией белка, по предположению, смещает равновесие в сторону нативного складывания согласно закону действия масс.
р53 был впервые идентифицирован как клеточный белок, взаимодействующий с большим Т-антигеном (LT) SV40. Интерфейсная область между LT и р53 большая: в общей сложности 23 остатка LT и 19 остатков р53 либо спрятаны в этом интерфейсе, либо, оказывается, непосредственно участвуют во взаимодействиях между этими двумя молекулами. Остатки взаимодействия p53/ДНК являются смежными и перекрываются с интерфейсом p53/LT. Связывание LT с этими остатками р53 может эффективно защитить всю ДНК-связывающую поверхность р53, включая три наиболее часто мутированных остатков р53 в раковой опухоли: R273, R248 и G245. Это препятствует трансактивации р53-зависимых промоторов. Поскольку интерфейс р53/LT включает в себя несколько различных участков и петель p53, белок р53 должен быть сложен правильно, чтобы аминокислоты совмещались в правильном положении и правильной ориентации для сформирования контекста связывания с LT. Таким образом, связывание р53 с LT может служить маркером конформационного состояния p53.
В области конформации р53 было предпринято несколько коррекционных подходов. Доказательство принципа в отношении стабилизирующих конформацию пептидов было представлено автором Фридлером и коллегами (Friedler, A., et al., A peptide that binds and stabilizes p53 core domain: chaperone strategy for rescue of oncogenic mutants. Proc. Natl. Acad. Sci. USA, 2002. 99(2): p. 937-42). На основе кристаллической структуры комплекса между ДНК-связывающим доменом p53 и ASPP был разработан пептид с девятью остатками, CDB3 (Samuels-Lev, Y., et al., ASPP proteins specifically stimulate the apoptotic function of p53. Mol. Cell, 2001. 8(4): p. 781-94). Было показано, что этот пептид связывается с Mut-P53 и действует как шаперон, сдвигающий равновесие в сторону WT конформации, на что указывает повышенная реакционная способность к PAb1620. Однако биологические эффекты CDB3 (Issaeva, N., et al., Rescue of mutants of the tumor suppressor p53 in cancer cells by a designed peptide. Proc. Natl. Acad. Sci. USA, 2003. 100(23): p. 13303-7) только частичны, поскольку конформация комплекса Mut-p53/CDB3 находится в промежуточном состоянии между диким типом (WT) и мутантом.
Соединения малой молекулы, нацеленные на Mut-p53, были идентифицированы с помощью анализов либо на основе белка, либо на основе клеток (Peng, Y., et al., Rescue of mutant p53 transcription function by ellipticine. Oncogene, 2003. 22(29): p. 4478-87). С. 4478-87). CP-31398 было идентифицировано путем скрининга молекул, которые защищают выделенный ДНК-связывающий домен p53 от термической денатурации, что подтверждается сохранением реактивности PAb1620 при нагревании белка (Foster, B.A., et al., Pharmacological rescue of mutant p53 conformation and function. Science, 1999. 286(5449): p. 2507-10). Механизм действия СР-31398 остается неясным. ЯМР-исследования не обнаружили никакого связывания CP-31398 с DBD р53 (Rippin, T.M., et al., Characterization of the p53-rescue drug CP-31398 in vitro and in living cells. Oncogene, 2002. 21(14): p. 2119-29). CP-31398 влияет на экспрессию генов и индуцирует гибель клеток как р53-зависимым обазом, так и независимым образом. Таким образом, оказывается, что CP-3138 имеет и другие клеточные мишени, помимо р53, что может объяснить его клеточную токсичность.
Две другие малые молекулы, которые восстанавливают функцию р53 в живых раковых клетках, PRIMA-1 и MIRA-1, были обнаружены с помощью анализов скрининга на основе клеток. PRIMA-1 и MIRA-1 имеют сходные профили активности (Bykov, V.J., et al., Reactivation of mutant p53 and induction of apoptosis in human tumor cells by maleimide analogs. J Biol Chem, 2005. 280(34): p. 30384-91), но структурно не родственные. До настоящего времени прямое связывание с Мut-p53 не было продемонстрировано. По-видимому, механизм может задействовать JNK путь.
В области обнаружения и разработки препарата против рака могут использоваться две разные, а иногда дополняющие друг друга стратегии. Рациональная разработка, в которой используют биологические, математические или вычислительные инструменты для разработки молекул для определенной цели, был использован в случае с CDB3. Однако, поскольку взаимодействия между различными белками и их окружением сложные, это чрезвычайно трудно и часто приводит к получению молекул с ограниченным биологическим воздействием. Второй стратегией является высокопроизводительный скрининг библиотек молекул с выделением соединений с лучшими признаками. При таком скрининге могут использоваться либо химические библиотеки, библиотеки малых молекул, либо пептидные библиотеки. Большинство лекарственных препаратов, доступных на сегодняшний день, представляют собой малые молекулы благодаря их способности проникать через клеточные мембраны. Химические библиотеки обычно состоят из 104-105 различных соединений; скрининг такой библиотеки требует функциональной оценки отдельных молекул, что делает его непрактичным для небольшой лаборатории, так как для этого требуются большие инвестиции в робототехнику и/или рабочую силу. Библиотеки пептидного дисплея значительно больше. Селекция пептидов основана на связывании пептидов (и следовательно фага), с иммобилизованной мишенью, элюировании и амплификации, а затем идентификации путем секвенирования.
В процедуре фагового дисплея обогащение фагов, которые представляют пептид, достигается с помощью аффинной селекции в фаговой библиотеке на иммобилизованной мишени. В этом процессе «пэннинга» связывающиеся фаги захватываются, тогда как несвязывающиеся фаги смываются. На следующем этапе связанные фаги элюируются и амплифицируются путем реинфекции клеток E.coli. Амплифицированная популяция фагов может, в свою очередь, подвергаться следующему раунду пэннинга. Отбор из библиотек фагового дисплея представляет собой циклический процесс селективного обогащения и амплификации. После нескольких раундов отбора фаги разбавляют таким образом, что позволяет выделить отдельные фаговые клоны. Отдельные клоны затем собирают, культивируют в E-coli, ДНК фага извлекают и затем отправляют на секвенирование. Разработанные за последнее время технологии секвенирования следующего поколения значительно повышают эффективность фагового дисплея, что позволяет выполнять анализ всей отобранной совокупности пептидов при меньшем числе раундов отбора.
Фаговый дисплей дает несколько важных преимуществ по сравнению с другими методами скрининга; основным преимуществом фагового дисплея является разнообразие последовательностей, которые могут быть представлены, позволяя находить молекулы с очень высокой аффинностью и биологическим эффектом. После обнаружения консенсусной пептидной последовательности, она может быть дополнительно улучшена либо за счет технологий направленной эволюции, либо за счет рационального конструирования.
Тем не менее, остается неудовлетворенной потребность в данной области в агентах, которые могут реактивировать мутантные белки р53 эффективно и специфически. Такие специфические и эффективные агенты могут быть также использованы в качестве эффективного средства для лечения различных состояний, в которых р53 мутирует, в частности, путем восстановления нативного складывания и активности мутантных белков р53.
Краткое изложение сущности изобретения
В настоящем изобретении предлагаются высокоактивные пептиды и модифицированные пептидные агенты, которые могут эффективно реактивировать конформационные мутанты р53, в идеале путем изменения конформации и/или активности мутантных белков р53, чтобы были похожи на таковые функционального белка р53 дикого типа. Таким образом, в настоящем изобретении предлагаются пептиды и их применение в лечении состояний, связанных с мутантным р53, когда активация присутствующих еще конформационно дефектных белков р53 может быть полезна.
Настоящее изобретение основано на неожиданной идентификации высокоактивных пептидов и агентов на основе пептидов, которые могут эффективно реактивировать конформационные мутанты р53, более эффективно, чем ранее известные пептиды, идентифицированные для этого применения. Таким образом, в настоящем изобретении предлагается, в одном аспекте, рекомбинантный или синтетический пептид, состоящий из аминокислотной последовательности, представленной любой из SEQ ID NO: 321-286.
В настоящем изобретении также предлагается, в другом аспекте, рекомбинантный или синтетический пептид, содержащий аминокислотную последовательность, представленную любой из SEQ ID NO: 321-286, в котором указанный пептид по меньшей мере частично реактивирует мутантный белок р53.
В настоящем изобретении, кроме того, в еще одном аспекте предлагается рекомбинантный или синтетический пептид, содержащий консенсусный мотив аминокислотной последовательности, представленной любой из SEQ ID NO: 314, 268, 282, 340, 376, 298, 377, 378, 253, 20, 379, 302, 275, 380, 273, 381, 280 или 382, в котором указанный пептид по меньшей мере частично реактивирует мутантный белок р53.
В некоторых вариантах осуществления пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO: 321, SEQ ID NO: 314, SEQ ID NO: 313, SEQ ID NO: 310 или SEQ ID NO: 307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO: 321-302. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO: 321-312. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO: 321-316. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO: 321, SEQ ID NO: 314, SEQ ID NO: 313, SEQ ID NO: 310 или SEQ ID NO: 307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид включает аминокислотную последовательность, представленную любой из SEQ ID NO:321-302. В некоторых вариантах осуществления описанный выше пептид включает аминокислотную последовательность, представленную любой из SEQ ID NO: 321-312. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO: 321-316. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления пептид конъюгирован по меньшей мере с одним фрагментом жирной кислоты. В некоторых вариантах осуществления настоящего изобретения жирную кислоту выбирают из группы, состоящей из миристиновой кислоты, лауриновой кислоты, пальмитиновой кислоты и стеариновой кислоты. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления настоящего изобретения жирная кислота представляет собой миристоильную жирную кислоту.
В некоторых вариантах осуществления пептид по меньшей мере частично изменяет конформацию указанного мутантного белка р53 на конформацию белка р53 дикого типа (WT).
В некоторых вариантах осуществления пептид по меньшей мере частично изменяет конформацию указанного мутантного белка р53 таким образом, что указанный мутантный белок р53 распознается моноклональным антителом, направленным против белка p53 дикого типа. В некоторых вариантах осуществления моноклональное антитело представляет собой Ab1620.
В некоторых вариантах осуществления пептид по меньшей мере частично восстанавливает активность указанного мутантного белка р53 до активности белка р53 дикого типа.
В некоторых вариантах осуществления активность представляет собой уменьшение жизнеспособности клеток, экспрессирующих указанный мутантный белок р53. В некоторых вариантах осуществления активность представляет собой содействие апоптозу клеток, экспрессирующих указанный мутантный белок р53. В некоторых вариантах осуществления активность представляет собой активирование проапоптотических генов клеток, экспрессирующих указанный мутантный белок р53. В некоторых вариантах осуществления проапоптотические гены выбирают из группы, состоящей из CD95, Bax, DR4, DR5, PUMA, NOXA, Bid, 53AIP1 и PERP. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления активность представляет собой связывание с консенсусным ДНК-связывающим элементом р53 в клетках, экспрессирующих указанный мутантный белок p53. В некоторых вариантах осуществления консенсусный ДНК-связывающий элемент содержит аминокислотную последовательность, представленную SEQ ID NO: 339.
В некоторых вариантах осуществления связывание вызывает по меньшей мере частичную активацию эндогенного гена-мишени р53. В некоторых вариантах осуществления эндогенный ген-мишень выбирают из группы, состоящей из p21, MDM2 и PUMA. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления мутантный белок р53 имеет другую конформацию, чем белок р53 дикого типа. В некоторых вариантах осуществления мутантный белок р53 является, по меньшей мере, частично неактивным по сравнению с белком р53 дикого типа.
В некоторых вариантах осуществления мутантный белок р53 не распознается моноклональным антителом, направленным против белка p53 дикого типа. В некоторых вариантах осуществления мутантный белок р53 при связывании с указанным пептидом распознается моноклональным антителом, направленным против белка p53 дикого типа. В некоторых вариантах осуществления моноклональное антитело представляет собой Ab1620.
В некоторых вариантах осуществления мутантный белок р53 включает мутацию, выбранную из группы, состоящей из R175H, V143A, R249S, R273H, R280K, P309S, P151S, P151H, C176S, C176F, H179L, Q192R, R213Q, Y220C, Y220D, R245S, R282W, D281G, S241F, C242R, R248Q, R248W, D281G, R273C и V274F. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления пептид содержит консенсусный мотив, представленный SEQ ID NO: 314. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO:321, SEQ ID NO:314, SEQ ID NO:313, SEQ ID NO:310 или SEQ ID NO:307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO:321, SEQ ID NO:314, SEQ ID NO:313, SEQ ID NO:310 или SEQ ID NO:307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NOs:268, 282, 340, 376, 298, 377, 378, 253, 20, 379, 302, 275, 380, 273, 381, 280 или 382. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NOs:379, 302, 275, 380, 273, 381, 280 или 382. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NOs: SEQ ID NOs:302, 275, 380, 273, 381, 280 или 382. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В настоящем изобретении также предлагается, в другом аспекте, вектор экспрессии, способный экспрессировать пептиды, описанные выше.
В настоящем изобретении, кроме того, предлагается в еще одном аспекте фармацевтическая композиция, содержащая описанные выше пептиды.
В настоящем изобретении, кроме того, предлагается в еще одном аспекте фармацевтическая композиция, содержащая описанный выше вектор экспрессии.
В одном аспекте описанные выше фармацевтические композиции предназначены для применения в лечении заболевания, нарушения или состояния, ассоциированого с мутантным белком р53.
В некоторых вариантах осуществления настоящего изобретения заболевание представляет собой рак. В некоторых вариантах осуществления рак выбирают из группы, состоящей из рака молочной железы, рака толстой кишки и рака легких. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления клетки рака экспрессируют мутантный белок р53.
В настоящем изобретении также предлагается в другом аспекте способ лечения заболевания, нарушения или состояния, ассоциированного с мутантным белком р53, включающий этап введения терапевтически эффективного количества фармацевтических композиций, описанных выше, субъекту, нуждающемуся в этом, тем самым обеспечивая лечение указанного заболевания, нарушения или состояния.
В настоящем изобретении, кроме того, предлагается в еще одном аспекте набор, содержащий описанные выше фармацевтические композиции.
В одном аспекте описанный выше набор предназначен для применения в лечении заболевания, нарушения или состояния, ассоциированного с мутантным белком р53.
Другие цели, отличительные признаки и преимущества настоящего изобретения станут очевидными из последующего описания и рисунков.
КРАТКОЕ ОПИСАНИЕ РИСУНКОВ
На Фигуре 1А показана блок-схема этапов способа скрининга, который предусматривает отбор партнеров связывания (как например, пептидов) непрямым образом по их действию на конформацию или структуру молекулы-мишени.
На Фигуре 1В представлен схематический чертеж, поясняющий способ идентификации, скрининга и отбора реактивирующих мутантный р53 пептидов. Способ включает чередующиеся различные стратегии отбора, с возрастающей строгостью, для скрининга и идентификации реактивирующих мутантный р53 пептидов, с использованием способа фагового дисплея. Стратегия А (слева): отбор в соответствии с конформацией: отбор пептидов, экспрессированных и представленных фагом, которые могут связываться с мутантным белком p53 (например, R175H Mut-р53). Белок Mut-р53 связан со специфическим антителом p53 (например, PAb1620), который иммобилизован на подложке, тем самым обеспечивается отбор связанного фага. Стратегия B (справа): отбор в соответствии с функцией: отбор пептидов, экспрессированных и представленных фагом, которые могут реактивировать Mut-p53 (например, R175H Mut-р53), в результате чего активация определяется по способности белка Mut-р53 связываться с его консенсусным ДНК-связывающим элементом. ДНК-связывающий элемент (например, р53-RE дикого типа) иммобилизован на подложке. Mut-p53 не может связываться с р53-RE дикого типа, пока он по меньшей мере частично не будет реактивирован реактивирующим пептидом, связанным с ним. Способ может дополнительно включать секвенирование (например, глубокое секвенирование) идентифицированных пептидов для определения их последовательностей и, по желанию, идентификации консенсусной последовательности для реактивирующих пептидов.
Фигура 2 представляет собой диаграмму с вестерн-блоттинг анализа в экспериментах по иммунопреципитации (IP), в которых гранулы агарозы, ковалентно сшитые с антителами (PAb1620 или PAb240) или белками (ASPP2 или Bcl2), были инкубированы с белком р53 дикого типа, мутантным р53 R175H или мутантным р53 V143A (каждый получен из клеток sf9, трансфектированных бакуловирусом, экспрессирующих соответствующий белок) в течение 3 часов при 4ºC. Полученный иммунопреципитат, а также супернатант (sup) были подвергнуты экспериментам по вестерн-блоттингу при использовании анти-p53 (αp53) антитела, конъюгированного с пероксидазой хрена (HRP), с определением уровня белка p53 в каждом образце.
Фигура 3 представляет собой диаграмму вестерн-блоттинг анализа в экспериментах по IP, в которых гранулы, которые были ковалентно сшиты с антителами PAb1620 или PAb240, были инкубированы с р53 дикого типа или мутантным р53 R175H в течение 3 часов при 4°С с различными растворами (буфер A-I и IP). Полученный иммунопреципитат, а также супернатант (sup) были подвергнуты экспериментам по вестерн-блоттингу при использовании анти-p53 (αp53) антитела, конъюгированного с пероксидазой хрена, с определением уровня белка p53 в каждом образце. Раствор А - 50 мМ Трис; раствор В - Трис, 150 мМ NaCl, раствор С - Трис, NaCl, 0,5% Тритон; раствор D - Трис, 0,5% глицин; раствор E - 40мМ Na4O7P2; раствор F - 400 мМ гуанидин-HCl; раствор G - 800 мМ гуанидин-HCl; раствор Н - 1М мочевина; раствор I - 3M мочевина; IP - буфер IP.
Фигура 4 представляет собой последовательность олигонуклеотида, используемого в качестве связующего элемента для белков р53. Олигонуклеотид (SEQ ID NO:61) содержит метку 5' биотин, затем следует сайт узнавания HindIII (подчеркнуто), затем следует сайт узнавания EcoRI (подчеркнуто), затем следует консенсусный элемент связывания р53 (подчеркнуто, сайт связывания р53 состоит из двух полусайтов, каждый полусайт связывается с димером p53 и вместе с ним этот сайт образует комплекс ДНК и тетрамер p53), затем следуют две копии элемента узнавания р53 промотора р21 (подчеркнуто). Для экспериментов по связыванию этот олигонуклеотид отжигали с комплементарным олигонуклеотидом с образованием двухцепочечного (ds) олигонуклеотида.
Фигура 5 представляет собой пиктограммы вестерн-блоттинг анализа в IP-экспериментах, в которых гранулы, ковалентно сшитые с антителом PAb1620, были инкубированы с очищенным мутантным р53 R175H в присутствии фага, полученного путем отбора методом фагового дисплея либо с Mut-р53 R175H полной длины (175), либо с рекомбинантным Mut-р53 R249S (249 DBD), причем предшествующий этап предварительной очистки выполняли путем инкубации фагового пула с гранулами PAb1620. Неотобранные фаги (NS) использовали в качестве контроля. Инкубацию проводили в течение 3 часов при 4°С. Связанный р53 в иммунопреципитате проанализировали с помощью вестерн-блоттинга при использовании антитела против р53 (αp53). Неотобранные фаги (NS) использовали в качестве контроля. «In» обозначает 10% от исходного материала по IP, который был загружен непосредственно на гель. Иммунопреципитацию с PAb-421 использовали в качестве положительного контроля и в качестве стандарта для иммунопреципитированного p53, так как это антитело связывается с эпитопом р53 на С-конце независимо от конформации белка р53.
Фигура 6 представляет собой пиктограммы вестерн-блоттинг анализа в экспериментах по IP, в которых покрытые стрептавидином гранулы, связанные либо с p53-RE-ДНК, либо с контрольными RE-ДНК олигонуклеотидами, меченными биотином, были инкубированы с очищенным WT-p53-DBD или мутантным p53-R249S-DBD в присутствии фага, полученного путем отбора методом фагового дисплея, с Mut-p53 R175H (175), клоном 27 (одиночный клон, выделенный из отбора 175, SEQ ID NO:328); пулы #69 и #94, отобранные с р53 дикого типа и Mut-p53 R175H с использованием комбинаций большого Т-антигена SV-40 (T-ag) и PAb1620 в чередующихся раундах отбора. Неотобранные фаги (NS) использовали в качестве контроля. Инкубацию проводили в течение 3 часов при 4°С. Связанный р53 визуализировали с помощью вестерн-блоттинг анализа при использовании антитела против р53 (αp53).
Фигура 7 представляет собой схематическую иллюстрацию нескольких консенсусных пептидных мотивов, идентифицированных, как описано в настоящем документе.
На Фигурах 8А и 8В представлены гистограммы, демонстрирующие репрезентативные эксперименты ELISA по определению действия тестируемых пептидов на изменения конформации Мut-p53 в клетках H1299, стабильно сверхэкспрессирующих Mut-p53 (R175H p53), как определено с помощью иммуноанализа. Для измерения конформационного действия пептидов на Mut-p53 титрационный микропланшет покрывали либо PAb240, PAb 1620Ю, либо pAb421 (в качестве положительного контроля и стандарта для общего белка р53, так как используемое антитело распознает и конформации дикого типа, и мутантные конформации), в течение ночи инкубировали, промыли, блокировали и клеточные экстракты (с или без пептидов) добавляли в течение еще 2 часов. После удаления экстрактов планшеты промыли и инкубировали с αp53-HRP- конъюгированным антителом Ab для определения уровней р53. Выполнили анализ ТМВ (подложка HRP) и определили оптическую плотность при 450 нм. р53 дикого типа служил в качестве положительного контроля для реактивности с PAb1620, а Mut-p53 служил в качестве отрицательного контроля. Результаты представлены как соотношение поглощения между образцами PAb1620 или PAb240 и контрольным образцом pAb241. Клетки MCF7 и H1299-Mut-р53 (ts) A135V (TS) использовали в качестве положительных контролей для конформации р53 дикого типа (соотношение 1620/240 равно или превышает 5:1).
Фигура 9 представляет собой гистограмму, демонстрирующую репрезентативные эксперименты ELISA по определению действия тестируемых пептидов на активность связывания Mut-р53 с ДНК в клетках H1299, стабильно сверхэкспрессирующих Mut-P53 (R175H p53). Коммерческий набор связывания р53/ДНК (R&D) использовали в соответствии с инструкциями производителя. Вкратце, 96-луночные планшеты покрывали в течение ночи анти-p53 антителом. Клеточные экстракты, содержащие р53, подвергались реакции с олигонуклеотидом, который содержит консенсусный сайт связывания р53, меченный биотином, в присутствии или в отсутствие (NT) тестовых пептидов. Предполагается, что р53 дикого типа связывается с этим сайтом связывания с ДНК, а также с антителами, покрывающими тестовые лунки планшета. Избыток p53 и олиго вымываются и стрептавидин-HRP используется для определения количества олигонуклеотидов в лунке, которое пропорционально ДНК, связанной белком р53. Был проведен анализ ТМВ для определения уровней HRP (450нм). Результаты представлены в виде относительного поглощения (при 450 нм) (по оси Y) каждого тестируемого образца. Клетки MCF7 и H1299-Mut-p53 (ts) A135V служат в качестве положительных контролей для p53 дикого типа.
Фигура 10 представляет собой гистограмму, изображающую репрезентативные эксперименты ELISA по определению связывания тестируемых пептидов с рекомбинантным р53 дикого типа и Mut-p53. Коммерческий набор связывания пептид-белок (TAKARA) использовали в соответствии с инструкциями изготовителя. Вкратце, 96-луночные планшеты покрывали пептидами в течение 2 часов. Растворимые пептиды добавляли в соответствующие лунки в качестве конкурентного контроля для подтверждения специфичности связывания пептида с p53 (+comp). В другие лунки добавляли р53-RE ДНК олиго (+ДНК) для исследования, влияет ли он на связывание пептидов с p53. После удаления рекомбинантного белка планшеты промывали и инкубировали с αp53-HRP- конъюгированным антителом для количественного определения р53. Наконец, выполнили анализ ТМВ (подложка HRP) и определили поглощение при 450 нм. Результаты представлены в виде относительного поглощения при 450 нм (по оси Y) каждого тестируемого образца. Следующие моноклональные антитела αp53 служили в качестве внутренних контролей: PAb1801; PAb1620 и PAb240.
Фигура 11 представляет собой гистограмму, демонстрирующую связывание Mut-p53 с промоутерами репрезентативных генов-мишеней р53 в живых клетках. Клетки рака молочной железы ВТ-549, эндогенно экспрессирующие мутантный p53R249S обрабатывали в течение 5 часов смесью 3-х pCAP - 250, 308 и 325. Клетки, обработанные смесью контрольных пептидов (инертные пептиды) служили в качестве отрицательного контроля. Клетки фиксировали 1% формальдегидом, собрали и ДНК расщепили с помощью ультразвука. Сшитую с p53 ДНК подвергли иммунопреципитации с использованием поликлонального анти-p53 антитела (H47). ДНК очистили и связывание с р53-чувствительнымиыми элементами промоторами генов PUMA, p21, CD95 и MDM2 определяли количественно с помощью количественного анализа ПЦР (qPCR). Результаты были нормализованы до исходных образцов, которые представляют общие уровни ДНК. В качестве отрицательного контроля, экстракты подвергали иммунопреципитации с гранулами без антитела (гранулы). Геномный сайт, не содержащий никакого p53-связывающего элемента, служил в качестве отрицательного контроля (черный).
Фигура 12 представляет собой диаграмму, иллюстрирующую относительную активность люциферазы (cLuc/gLuc), согласно измерениям в различных тестируемых образцах. Транзиентная трансфекция клеток H1299 р53-/- осуществлялась с помощью плазмид, экспрессирующих WT p53, R175H p53, R249S p53 или пустой вектор в качестве контроля, вместе с ТК-RGC-luc, где экспрессия люциферазы находится под контролем тандемного массива множественных p53-чувствительных элементов. Через 24 часа после трансфекции клетки обрабатывали тест-пептидами. Через 48 часов после трансфекции взяли образец культуральной среды для измерения биолюминесценции.
Фигуры 13А и 13В представляют собой гистограммы, иллюстрирующие действие различных тестируемых пептидов на жизнеспособность клеток, экспрессирующих Mut-p53, согласно определению с помощью кристаллвиолет-анализа. Фибробласты WI-38, экспрессирующие эндогенный р53 дикого типа, были инфицированы ретровирусом, экспрессирующим либо кшРНК мыши Noxa (WI38-m-NOXA-i) в качестве неспецифического контроля, либо мутанта R175H р53 для стабильной сверхэкспрессии мутантного р53 (WI38-175). Клетки (WI38-m-NOXA-i или WI38-175) высевали по 3000 клеток на лунку в 96-луночные планшеты. К клеткам были добавлены тестируемые пептиды. Различные концентрации этопозида (цитотоксический лекарственный препарат, 4'-диметил-эпиподофиллотоксин 9-[4,6-O-(R)-этилиден-бета-D-глюкопиранозид], 4'- (дигидроген фосфат) использовали в качестве положительного контроля для некроза клеток и в качестве стандартной эталонной кривой для оценки действия тестируемых пептидов. Через 48 часов после обработки клетки промывали с помощью PBS для исключения мертвых клеток и дебриса, а клетки, которые остались прикрепленными к планшету, окрашивали кристаллвиолетом в течение 30 минут. Кристаллвиолет удалили и клетки промывали 4 раза раствором PBS для удаления оставшегося кристаллвиолета. Затем окрашенные клетки растворили в 10% уксусной кислоте, планшеты взяли для измерения оптической плотности при 595 нм (оптимальное значение для кристаллвиолета). В гистограммах на Фигурах 13А и 13В представлены показания оптической плотности при 595 нм, которые отражают количество клеток в планшете после обработки, нормализованных до необработанных (NT) образцов.
Фигура 14 представляет собой гистограмму, иллюстрирующую действие тестируемых пептидов на активацию Mut-p53 путем измерения трансактивации генов-мишеней р53, согласно определению с помощью qRT-PCR. Клетки H1299 являются р53-нулевыми и широко используются для исследования р53. Были использованы клетки Н1299, стабильно трансфицированные Mut-p53 (ts) A135V. Клетки поместили в 12-луночные чашки, указанные пептиды добавили непосредственно в среду в концентрации 5 μг/мл, и клетки затем либо переместили на 32°C, либо вернули на 37°C. Через 18 часов клетки собрали, а затем извлекли РНК, выполнили синтез кДНК и ПЦР-анализ в режиме реального времени. Исследовали уровень экспрессии 3-х репрезентативных генов-мишеней р53, р21, PUMA и Mdm2. Гистограммы, показанные на Фигуре 14, иллюстрируют относительную кратную индукцию транскрипции тестируемых генов в различных образцах по сравнению с их уровнем транскрипции в необработанных клетках.
Фигуры 15А и 15В представляют собой гистограммы, иллюстрирующие действие различных указанных пептидов на жизнеспособность клеток рака молочной железы, экспрессирующих различные изоформы Mut-p53, как определено путем анализа с кристаллвиолетом. Фигура 15А: клетки MDA-MB-231, экспрессирующие Mut-p53 с мутацией в позиции 280 домена DBD. Фигура 15B: клетки SKBR3, экспрессирующие Mut-p53 с мутацией в позиции 175 в пределах DBD. На гистограммах Фигур 15А и 15B ппредставляют для каждого тестируемого пептида показания оптической плотности при 595 нм, отражающие количество клеток в планшете после обработки, нормализованных до необработанных (NT) образцов.
Фигура 16 представляет собой гистограмму, иллюстрирующую действие указанных пептидов на активацию Mut-p53 путем измерения трансактивации генов-мишеней р53, согласно определению с помощью QRT-PCR. Были использованы клетки SKBR3 ShCon и клетки SKBR3 Shp53, инактивированные для экспрессии p53. Клетки высевали в 12-луночные чашки и указанные пептиды добавляли непосредственно в среду в концентрации 5 μг/мл. Через 18 часов клетки собрали, после этого выполнили анализ qRT-PCR. Оценили уровень экспрессии p21, PUMA и Mdm2. На Фигуре 16 иллюстрируется относительная кратная индукция транскрипции тестируемых генов в различных образцах по сравнению с их уровнем транскрипции в необработанных клетках. Параллельно измеряли мРНК GAPDH в качестве контроля.
На Фигурах 17А, 17В, 17С и 17D иллюстрируются репрезентативные эксперименты, выполненные на клетках карциномы яичников ES2, экспрессирующих Mut-p53, мутированный в позиции 241 в пределах DBD. По существу, клетки высевали в 6-см чашки, и указанные пептиды добавляли непосредственно в среду в концентрации 12 μг/мл в указанные моменты времени. Клетки собрали и выполнили анализ на апоптоз (Фигуры 17А и 17В) при использовании аннексин-V окрашивающего набора (Roche, REF 11 988 549 001). Нефиксированное клетки окрашивали как анти-аннексин FITC- конъюгированными антителами для обнаружения апоптотических клеток, так и PI (пропидиум йодид) для окрашивания мертвых клеток в соответствии с инструкциями изготовителя. Окрашенные клетки проанализировали с помощью проточной цитометрии. В общей сложности для каждого образца насчитали 10000 клеток, которые разделили на четыре субпопуляции по интенсивности окрашивания; клетки, отрицательные как по IP, так и по аннексину (-PI, - Annexin), называются живыми; клетки, отрицательные по PI и положительные по аннексину (-PI + Annexin), проходят через ранние стадии апоптоза; клетки, положительные по IP и аннексину (+ PI, + Annexin), являются мертвыми клетками, которые претерпели процесс апоптоза; а клетки, положительные по PI и отрицательные по аннексину (+ PI, -Annexin), считаются мертвыми клетками, которые погибли под действием неапоптотического процесса, как например некроз.
На Фигурах 18А, 18В и 18С иллюстрируется эффект in vivo указанных пептидов в мышиной модели ксенотрансплантата. Клетки MDA-MB-231, экспрессирующие эндогенный мутантный р53 и стабильно экспрессирующие люциферазу, вводили в левое бедро голых мышей CD1/голых мышей. Когда опухоли достигали видимого размера, с помощью системы IVIS200 измеряли биолюминесценцию (указывающую число раковых клеток). Мышей затем обрабатывали с помощью внутриопухолевой инъекции три раза в неделю со смесью 3-х контрольных пептидов, которые не демонстрировали фенотип in vitro (pCAP 76, 77 и 12; 2 мг каждого пептида). Через 35 дней после начала обработки эксперимент был завершен. На Фигуре 18А показан график в логарифмическом масштабе, демонстрирующий показания по люциферазе в каждой опухоли как функции времени после начала обработки (инъекция пептида). На Фигуре 18B показаны изображения прямой передачи мышей (7-10) в начале обработки. На Фигуре 18С показаны изображения прямой передачи обработанных мышей (7-9) на день 35, когда эксперимент был завершен. Мышь 10 пришлось умертвить через 28 дней из-за большого размера опухоли.
На Фигурах 19А, 19В и 19С иллюстрируется эффект in vivo указанных пептидов в мышиной модели ксенотрансплантата. Клетки MDA-MB-231, экспрессирующие эндогенный мутантный р53 и стабильно экспрессирующие люциферазу, вводили в левое бедро голых мышей CD1/голых мышей. Когда опухоли достигали видимого размера, с помощью системы IVIS200 измеряли биолюминесценцию (указывающую число раковых клеток). Мышей затем обрабатывали путем внутриопухолевой инъекции три раза в неделю со смесью 3-х тестовых пептидов, которые продемонстрировали способность реактивировать мутантный р53 (pCAP 159, 155 и 174; 2 мг каждого пептида). Через 35 дней после начала обработки эксперимент был завершен. На Фигуре 19А показан график в логарифмическом масштабе, демонстрирующий показания по люциферазе в каждой опухоли как функции времени после начала обработки (инъекция пептида). На Фигуре 19B показаны изображения прямой передачи мышей 1-6 в начале обработки. На Фигуре 19C показаны изображения прямой передачи обработанных мышей 1-6 на день 35, когда эксперимент был завершен. Две из опухолей (мышь 1 и мышь 4) показали частичный ответ на обработку согласно измерению с уменьшением на 50% и 65%, соответственно, сигнала люциферазы спустя 35 дней. Мыши 2 и 5 показали полный ответ при достижении показаний биолюминесценции, которые были такие же низкие или близки к пороговым уровням фона системы IVIS (5×106 фотонов) даже через 21 день обработки. После прекращения обработки спустя 35 дней мыши под номерами 2 и 5 были живы, их мониторинг проводили в течение еще 21 дня; нового появления опухолей не было обнаружено ни визуально, ни с помощью прямого изображения.
На Фигурах 20А, 20В, 20С и 20D иллюстрируется эффект in vivo указанных пептидов в мышиной модели ксенотрансплантата. Клетки MDA-MB-231, экспрессирующие мутантный эндогенный р53 и стабильно экспрессирующие люциферазу, вводили в левое бедро голых мышей CD1/голых мышей. Когда опухоли достигали видимого размера, с помощью системы IVIS200 измеряли биолюминесценцию (указывающую число раковых клеток). Мышей затем обрабатывали внутриопухолевой инъекцией три раза в неделю, либо смесью 3-х контрольных пептидов, которые не демонстрировали фенотип in vitro (pCAP 76, 77 и 12; 2 мкг каждого пептида), либо смесью из 3-х тестовых пептидов, которые демонстрировали способность реактивировать мутантный р53 (pCAP 159, 155 и 174; 2 μг каждого пептида). На Фигурах 20А и 20В показан график в логарифмическом масштабе, демонстрирующий средние значения показаний по люциферазе в опухолях как функции времени, до (вплоть до дня 18) и после начала обработки (инъекция пептида). На Фигурах 20С и 20D показаны изображения прямой передачи мышей в начале обработки (день 18, слева) и 12 дней обработки (день 30, справа). 40% мышей показали полный ответ, достигая показаний биолюминесценции, которые такие же низкие или близки к пороговым уровням фона системы IVIS (5×106 фотонов).
Фигуры 21А, 21В, 21С, 21D и 21Е иллюстрируют эффект in vivo указанных пептидов в мышиной модели ксенотрансплантата. Клетки SW-480 рака толстой кишки, экспрессирующие эндогенный мутантный р53 и стабильно экспрессирующие люциферазу, вводили в левое бедро голых мышей CD1/голых мышей. Когда опухоли достигали видимого размера, с помощью системы IVIS200 измеряли биолюминесценцию (указывающую число раковых клеток). Мышей затем обрабатывали путем внутриопухолевой инъекции три раза в неделю либо смесью из 3-х контрольных пептидов, которые не демонстрировали фенотип in vitro (pCAP 76, 77 и 12; 2 μг каждого пептида), либо смесью из 3-х тестовых пептидов, которые демонстрировали способность реактивировать мутантный р53 (pCAP 250, 308 и 325; 2 μг каждого пептида). На Фигурах 21А, 21В и 21С показан график в логарифмическом масштабе, демонстрирующий средние значения показаний по люциферазе в опухолях как функции времени, до (вплоть до дня 0) и после начала обработки (инъекция пептида). На Фигурах 21D и 21Е показаны коробчатые диаграммы объема опухоли и массы опухоли, соответственно. Как видно на Фигурах 21D и 21Е, опухоли, извлеченные из мышей, которым вводили либо пептидную смесь, либо один пептид pCAP-325, значительно меньше по размеру и массе по сравнению с опухолями, извлеченными из мышей, обработанных контрольными пептидами (р-величина <0,05).
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В настоящем изобретении предлагаются высокоактивные пептиды и модифицированные пептидные агенты, которые могут эффективно реактивировать конформационные мутанты р53, в идеале путем изменения конформации и/или активности мутантного белка р53, чтобы были похожи на таковые функционального белка р53 дикого типа. Таким образом, в настоящем изобретении предлагаются пептиды и их применение в лечении связанных с мутантом р53 состояний, когда активация присутствующих еще конформационно дефектных белков р53 может быть полезна.
Настоящее изобретение основано на неожиданной идентификации высокоактивных пептидов и агентов на основе пептидов, которые могут эффективно реактивировать конформационные мутанты р53, более эффективно, чем ранее известные пептиды, идентифицированные для такого применения.
В настоящем изобретении предлагаются агенты, способные по меньшей мере частично повышать противораковое и/или проапоптотическое действие мутантных белков р53, а также их применение в лечении любого заболевания или состояния, вызванного конформационно-аберрантным белком р53 или коррелированным с ним. Не привязываясь к какому-либо механизму или теории, авторы полагают, что конформационные изменения мутантных белков р53 при связывании с агентами, предлагаемыми в настоящем изобретении, приближает их к 3D-конформации белка р53 дикого типа, и, таким образом, по меньшей мере частично восстанавливает по меньшей мере часть функций белка р53 дикого типа до мутантных белков р53.
Более конкретно, в настоящем изобретении предлагается, в одном аспекте, рекомбинантный или синтетический пептид, состоящий из аминокислотной последовательности, представленной любой из SEQ ID NO: 321-286.
В настоящем изобретении также предлагается, в другом аспекте, рекомбинантный или синтетический пептид, содержащий аминокислотную последовательность, представленную любой из SEQ ID NO: 321-286, в котором пептид по меньшей мере частично реактивирует мутантный белок р53.
В настоящем изобретении, кроме того, в еще одном аспекте предлагается рекомбинантный или синтетический пептид, содержащий консенсусный мотив аминокислотной последовательности, представленной любой из SEQ ID NOs:314, 268, 282, 340, 376, 298, 377, 378, 253, 20, 379, 302, 275, 380, 273, 381, 280 или 382, в котором пептид по меньшей мере частично реактивирует мутантный белок р53.
В некоторых вариантах осуществления пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO: 321, SEQ ID NO: 314, SEQ ID NO: 313, SEQ ID NO: 310 или SEQ ID NO:307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO:321-302. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO:321-312. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO:321-316. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO:321, SEQ ID NO:314, SEQ ID NO:313, SEQ ID NO:310 или SEQ ID NO:307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO:321-302. В некоторых вариантах осуществления описанный выше пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO: 321-312. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления описанный выше пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO:321-316. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления пептид конъюгирован по меньшей мере с одним фрагментом жирной кислоты. В некоторых вариантах осуществления жирную кислоту выбирают из группы, состоящей из миристиновой кислоты, лауриновой кислоты, пальмитиновой кислоты и стеариновой кислоты. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления жирная кислота представляет собой миристоильную жирную кислоту.
В некоторых вариантах осуществления пептид по меньшей мере частично изменяет конформацию мутантного белка р53 на конформацию белка р53 дикого типа (WT).
Известные в данной области антитела представляют собой антитела, которые специфически распознают только белки р53 дикого типа. Такие антитела являются весьма полезными при определении, имеет ли определенный белок р53, либо дикого типа, либо мутантный, конформацию функционального белка р53 дикого типа. Таким образом, в некоторых вариантах осуществления пептид по меньшей мере частично изменяет конформацию мутантного белка р53 таким образом, что мутантный белок р53 распознается моноклональным антителом, направленным исключительно против белка р53 дикого типа или против белка р53, имеющего конформацию белка р53 дикого типа. В некоторых вариантах осуществления моноклональное антитело представляет собой Ab1620.
Следует понимать, что поскольку р53 экспрессируется с обоих аллелей, общее содержание внутриклеточного р53 может быть либо дикого типа (wt/wt), смесью р53 дикого типа (wt) и мутантного (wt/mut), или только мутантным р53 (когда оба аллеля мутированы (mut/mut), либо один аллель ликвидируется (mut/-)). При раке ситуация часто такая: wt/ mut, mut/mut или mut/-. Поскольку р53 действует как тетрамер, мутантные белки р53 могут отменять активность белков р53дикого типа, которые могут находиться в клетках рака. Таким образом, пептиды, предлагаемые в настоящем изобретении, являются особенно полезными при лечении раковых заболеваний, в которых повышение уровня белков р53 дикого типа не продуктивно.
В некоторых вариантах осуществления пептид по меньшей мере частично восстанавливает активность мутантного белка р53, по меньшей мере, до одной из функций белка р53 дикого типа.
В некоторых вариантах осуществления активность представляет собой уменьшение жизнеспособности клеток, экспрессирующих мутантный белок р53. В некоторых вариантах осуществления активность представляет собой содействие апоптозу клеток, экспрессирующих мутантный белок р53. В некоторых вариантах осуществления активность представляет собой активирование проапоптотических генов клеток, экспрессирующих указанный мутантный белок р53. В некоторых вариантах осуществления проапоптотические гены выбирают из группы, состоящей из CD95, Bax, DR4, DR5, PUMA, NOXA, Bid, 53AIP1 и PERP. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления активность представляет собой связывание с консенсусным ДНК-связывающим элементом р53 в клетках, экспрессирующих мутантный белок p53. В некоторых вариантах осуществления консенсусный ДНК-связывающий элемент содержит аминокислотную последовательность или состоит из аминокислотной последовательности, представленной SEQ ID NO: 339.
В некоторых вариантах осуществления связывание вызывает по меньшей мере частичную активацию эндогенного гена-мишени р53. В некоторых вариантах осуществления эндогенный ген-мишень выбирают из группы, состоящей из p21, MDM2 и PUMA. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления мутантный белок р53 имеет другую конформацию, чем белок р53 дикого типа. В некоторых вариантах осуществления мутантный белок р53 является по меньшей мере частично неактивным по сравнению с белком р53 дикого типа.
В некоторых вариантах осуществления мутантный белок р53 не распознается моноклональным антителом, направленным против белка p53 дикого типа. В некоторых вариантах осуществления мутантный белок р53 после связывания с пептидом распознается моноклональным антителом, направленным против белка p53 дикого типа. В некоторых вариантах осуществления моноклональное антитело представляет собой Ab1620.
В некоторых вариантах осуществления мутантный белок р53 содержит мутацию, выбранную из группы, состоящей из R175H, V143A, R249S, R273H, R280K, P309S, P151S, P151H, C176S, C176F, H179L, Q192R, R213Q, Y220C, Y220D, R245S, R282W, D281G, S241F, C242R, R248Q, R248W, D281G, R273C и V274F. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления пептид содержит консенсусный мотив, представленный SEQ ID NO:314. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NO: 321, SEQ ID NO: 314, SEQ ID NO: 313, SEQ ID NO: 310 или SEQ ID NO:307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид состоит из аминокислотной последовательности, представленной любой из SEQ ID NO:321, SEQ ID NO:314, SEQ ID NO:313, SEQ ID NO:310 или SEQ ID NO:307. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NOs:268, 282, 340, 376, 298, 377, 378, 253, 20, 379, 302, 275, 380, 273, 381, 280 или 382. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NOs:379, 302, 275, 380, 273, 381, 280 или 382. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления пептид содержит аминокислотную последовательность, представленную любой из SEQ ID NOs:302, 275, 380, 273, 381, 280 или 382. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В настоящем изобретении, кроме того, в другом аспекте предлагается вектор экспрессии, способный экспрессировать описанные выше пептиды.
В настоящем изобретении, кроме того, предлагается, в другом аспекте, фармацевтическая композиция, содержащая описанные выше пептиды.
В настоящем изобретении, кроме того, в еще одном аспекте, предлагается фармацевтическая композиция, содержащая описанный выше вектор экспрессии.
В одном аспекте, описанные выше фармацевтические композиции предназначены для применения в лечении заболевания, нарушения или состояния, ассоциированного с мутантным белком р53.
В некоторых вариантах осуществления заболевание представляет собой рак. В некоторых вариантах осуществления рак выбирают из группы, состоящей из рака молочной железы, рака толстой кишки и рака легких. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления раковые клетки экспрессируют мутантный белок р53.
В настоящем изобретении, кроме того, в другом аспекте предлагается способ лечения заболевания, нарушения или состояния, ассоциированного с мутантным белком р53, включающий этап введения терапевтически эффективного количества описанных выше фармацевтических композиций субъекту, нуждающемуся в этом, тем самым обеспечивая лечение заболевания, нарушения или состояния.
В настоящем изобретении, кроме того, в еще одном аспекте, предлагается набор, содержащий описанные выше фармацевтические композиции.
В одном аспекте описанный выше набор предназначен для применения в лечении заболевания, нарушения или состояния, ассоциированного с мутантным белком р53.
Определения
Для облегчения понимания настоящего изобретения ниже определен ряд терминов и фраз. Следует понимать, что эти термины и фразы предназначены для описания, но не ограничения, так что терминология или фразеология настоящего описания должна интерпретироваться квалифицированным специалистом в свете идей и руководства, представленных в настоящем документе, в сочетании со знанием обычного специалиста в данной области техники.
Термин «рекомбинантный или синтетический пептид», используемый в данном описании, относится к пептиду, который получают стандартными биотехнологическими способами, известными в данной области техники, как например экспрессия в бактериях или твердофазный пептидный синтез (SPPS).
Термин «способный по меньшей мере частично реактивировать мутантный белок р53» или «по меньшей мере частично реактивирует мутантный белок р53», используемый взаимозаменяемо в данном описании, относится к пептиду, в котором после связывания пептида с мутантным белком р53 мутантный белок р53 приобретает или увеличивает активность, аналогичную соответствующей активности белка р53 дикого типа.
Термин «консенсусный мотив», используемый в данном описании, относится к аминокислотной последовательности, по меньшей мере, из трех аминокислот, которые были обнаружены в нескольких пептидах, предлагаемых в настоящем изобретении.
Термин «фрагмент жирной кислоты», используемый в данном описании, относится к части жирной кислоты, которая обладает определенным набором химических и фармакологических характеристик, сходных соответствующей исходной полной молекуле жирной кислоты. Термин также относится к любым молекулярным частицам и/или молекулярному фрагменту, содержащим ацильный компонент жирной (карбоновой) кислоты.
Повышающий проницаемость фрагмент в соответствии с настоящим изобретением предпочтительно ковалентно соединен с пептидной последовательностью через прямую связь или через линкер, с образованием пептидного конъюгата. Повышающий проницаемость фрагмент может соединяться с любой позицией в пептидном фрагменте, напрямую или через спейсер, предпочтительно с амино-концом пептида. Согласно некоторым вариантам осуществления повышающий проницаемость фрагмент представляет собой жирную кислоту.
Любой фрагмент, известный в данной области техники как активно или пассивно способствующий повышению проницаемости соединения в клетки, может быть использован для конъюгации с ядром пептида в соответствии с настоящим изобретением. Неограничивающие примеры включают: гидрофобные фрагменты, такие как жирные кислоты, стероиды и массивные ароматические или алифатические соединения; фрагменты, которые могут иметь рецепторы или носители клеточных мембран, такие как стероиды, витамины и сахара, природные и неприродные аминокислоты и транспортные пептиды. Согласно некоторым вариантам осуществления гидрофобный фрагмент представляет собой липидный фрагмент или аминокислотный фрагмент.
Термин «проницаемость», используемый в данном описании, относится к способности агента или вещества проникать или диффундировать через барьер, мембрану или слой кожи. «Проницаемость клетки» или «проникающий в клетку» фрагмент относится к любой молекуле, известной в данной области, которая способна облегчить или улучшить проникновение молекул через мембраны. Неограничивающие примеры включают: гидрофобные фрагменты, такие как липиды, жирные кислоты, стероиды и массивные ароматические или алифатические соединения; фрагменты, которые могут иметь рецепторы или носители клеточной мембраны, такие как стероиды, витамины и сахара, природные и неприродные аминокислоты, транспортные пептиды, наночастицы и липосомы.
Гидрофобный фрагмент в соответствии с настоящим изобретением может предпочтительно содержать липидный фрагмент молекулы или аминокислотный фрагмент. В соответствии с конкретным вариантом осуществления гидрофобный фрагмент выбирают из группы в составе: фосфолипиды, стероиды, сфингозины, керамиды, октил-глицин, 2-циклогексилаланин, бензоилфенилаланин, пропионил (С3); бутаноил (С4); пентаноил (С5); капроил (С6); гептаноил (C7); каприлоил (C8); нонаноил (С9); каприл (С10); ундеканоил (С11); лаурил (С12); тридеканоил (C13); миристоил (С14); пентадеканоил (C15); пальмитоил (С16); фтаноил ((СН3)4); гептадеканоил (С17); стеароил (С18); нонадеканоил (C19); арахидоил (С20); генэйкозаноил (C21); бегеноил (C22); труцизаноил (C23); и лигноцероил (C24); в котором указанный гидрофобный фрагмент прикреплен к указанному химерному полипептиду с амидными связями, сульфидрилами, аминами, спиртами, фенольными группами или связями углерод-углерод.
Другие примеры липидных фрагментов, которые могут быть использованы в соответствии с настоящим изобретением: липофектамин, Transfectace, трансфектам, цитофектин, DMRIE, DLRIE, GAP-DLRIE, DOTAP, DOPE, DMEAP, DODMP, ДОФХ, DDAB, DOSPA, EDLPC, EDMPC, DPH , TMADPH, СТАВ, лизил-PE, DC-Cho, -аланил холестерин; DCGS, DPPES, DCPE, DMAP, DMPE, DOGS, DOHME, DPEPC, Pluronic, твин, BRIJ, плазмалоген, фосфатидилэтаноламин, фосфатидилхолин, глицерин-3-этил фосфатидилхолин, диметил аммоний пропан, триметил аммоний пропан, диэтиламмоний пропан, триэтиламмоний пропан, диметилдиоктадециламмоний бромид, сфинголипид, сфингомиелин, лизолипид, гликолипид, сульфатид, гликосфинголипид, холестерин, холестериновый эфир, холестериновая соль, растительное масло, N-сукцинилдиолеоил фосфатидилэтаноламин, 1,2-диолеоил-sn-глицерин, 1,3-дипальмитоил- 2-сукцинилглицерин, 1,2-дипальмитоил-sn-3- сукцинилглицерин, 1-гексадецил-2-пальмитоилглицерофосфатидилэтаноламин, пальмитоилгомоцистеин, N,N'-бис(додециламинокарбонилметилен) -N,N'-бис((-N,N,N-триметиламмоний этиламинокарбонилметилен) этилендиамин тетрайодид, N,N"-бис (гексадециламинокарбонилметилен)-N,N',N"трис ((-N,N,N-триметиламмоний-этиламинокарбонилметилендиэтилентри амин гексайодид, N,N'-бис (додециламинокарбонилметилен) -N,N"-бис ((- N,N,N-триметиламмоний этиламинокарбонилметилен) циклогексилен-1,4-диамин тетрайодид; 1,7,7-тетра - ((- N,N,N,N-тетраметиламмоний этиламино-карбонилметилен) -3- гексадециламинокарбонил-метилен-1,3, 7-триаазагептан гептайодид, N, N, N', N'-тетра ((- N,N,N-триметиламмоний-этиламинокарбонилметилен) -N'- (1,2-диолеоилглицеро -3-фосфоэтаноламино карбонилметилен) диэтилентриамин тетрайодид; диолеоилфосфатидилэтаноламин, жирная кислота, лизолипид, фосфатидилхолин, фосфатидилэтаноламин, фосфатидилсерин, фосфатидилглицерин, фосфатидилинозитол, сфинголипид, гликолипид, глюколипид, сульфатид, гликосфинголипид, фосфатидная кислота, пальмитиновая кислота, стеариновая кислота, арахидоновая кислота, олеиновая кислота, липид, несущий полимер, липид, несущий сульфированный сахарид, холестерин, токоферол гемисукцинат, липид с жирной кислотой с простой эфирной связью, липид с жирной кислотой со сложной эфирной связью, полимеризованный липид, диацетил фосфат, стеариламин, кардиолипин, фосфолипид с жирной кислотой с 6-8 атомами углерода в длину, фосфолипид с асимметричными ацильными цепями, 6- (5-холестен-3b-илокси) -1-тио-b-D-галактопиранозид, дигалактозилдиглицерид, 6- (5-холестен-3b-илокси) гексил-6-амино-6-дезокси -1-тио- b-D -галактопиранозид, 6- (5-холестен-3b-илокси) гексил-6-амино-6-дезоксил -1-тио-a-D-маннопиранозид, 12 - (((7'-диэтиламино-кумарин -3-ил) карбонил) метиламино) - стеариновая кислота; N- [12 - (((7'-диэтиламинокумарин -3-ил) карбонил) метил-амино) октадеканоил] -2-амино пальмитиновая кислота; холестерил) 4'-триметил-аммонио) бутаноат; N-сукцинилдиолеоил-фосфатидилэтаноламин; 1,2-диолеоил-sn-глицерин; 1,2-дипальмитоил-sn-3-сукцинил-глицерин; 1,3-дипальмитоил-2- сукцинилглицерин, 1-гексадецил-2-пальмитоилглицеро-фосфоэтаноламин и пальмитоилгомоцистеин.
Термин «клетки, экспрессирующие мутантный белок p53», используемый в данном описании, относится к клеткам, которые экспрессируют по меньшей мере с одного аллеля мутантный белок р53. В некоторых вариантах осуществления термин «клетки, экспрессирующие мутантный белок р53» является взаимозаменяемым с термином «раковые клетки».
Термин «про-апоптотические гены» относится к гену, или большому числу генов, участвующих в апоптозе, либо напрямую (как например, некоторые каспазы), либо опосредованно (например, как часть каскада сигнальной трансдукции).
Термин «фармацевтическая композиция», используемый в данном описании, относится к любой композиции, содержащей по меньшей мере один фармацевтически активный ингредиент.
Термин «ассоциированный с мутантным белком р53», используемый в данном описании, относится к любому заболеванию, нарушению или состоянию, которое вызывается мутантным белком р53 или связано с наличием мутантного белка р53 в клетке или органе.
Следует понимать, что поскольку р53 экспрессируется с обоих аллелей, общее содержание внутриклеточного р53 может быть либо дикого типа (wt/wt), смесью р53 дикого типа (wt) и мутантного (wt/mut), либо только мутантным р53 (если мутируют оба аллеля (mut/mut) или один аллель ликвидируется (mut/-)). При раке ситуация часто такая: wt/mut, mut/mut или mut/-. Поскольку р53 действует как тетрамер, мутантные белки р53 могут отменять активность белков р53дикого типа, которые действительно присутствуют в клетках рака. Таким образом, пептиды, предлагаемые в настоящем изобретении, являются особенно полезными при лечении раковых опухолей, в которых повышение уровня белков р53 дикого типа не продуктивно.
Термин «терапевтически эффективное количество», используемый в данном описании, относится к количеству композиции, содержащей пептид в соответствии с настоящим изобретением, которое является достаточным, чтобы уменьшить, снизить и/или ингибировать заболевание, нарушение или состояние у индивидуума.
Используемый в данном описании термин р53 относится к белку р53, который может иметь конформацию р53 дикого типа, мутированного р53 или промежуточную конформацию между р53 дикого типа и мутированным р53.
Используемые в данном описании термины «р53 дикого типа», «wt р53" и «WT р53» могут использоваться взаимозаменяемо и относятся к белку р53 дикого типа, имеющего конформацию белка р53 дикого типа, и, следовательно, активность белка p53 дикого типа. В некоторых вариантах осуществления р53 дикого типа может быть идентифицирован с помощью специфического моноклонального антитела.
Используемые в настоящем описании термины «мутантный р53», «Mut-p53», «мутированный p53» и «мутант р53» могут использоваться взаимозаменяемо и относятся к мутированному белку р53, не способному эффективно функционировать в клетке-мишени. В некоторых вариантах осуществления Mut-p53 не может связываться со своим целевым сайтом. В некоторых вариантах осуществления Mut-р53 мутирует на участке ДНК-связывающего домена (DBD). В некоторых вариантах осуществления Mut-р53 неправильно сворачивается в неактивную конформацию. В некоторых иллюстративных вариантах осуществления Mut-р53 представляет собой чувствительный к температуре (ts) mut р53 R249S (R249S р53), мутантный р53 полной длины горячей точки Mut-р53 R175H (R175H р53) или любой другой белок Mut-р53. В некоторых вариантах осуществления Mut-p53 идентифицируется с помощью специфического моноклонального антитела, способного распознавать неправильно сложенную конформацию р53 (индуцированную мутацией белка р53). В некоторых вариантах осуществления Mut-р53 идентифицируется с помощью специфического моноклонального антитела.
Фраза «пептид реактивирует мутантный белок p53», используемый в данном описании, относится к пептиду, который при его взаимодействии с мутантным белком р53, мутантный белок р53 увеличивает по меньшей мере одну из его активностей, в которой активность - это активность белка р53 дикого типа. Например, при его взаимодействии с пептидом, предлагаемым в настоящем изобретении, мутантный белок р53 может увеличить, напрямую или опосредованно, экспрессию про-апоптотических белков, таких как каспазы, в раковых клетках, аналогично тому, что бы делал в подобной ситуации белок р53 дикого типа.
Как указано в настоящем описании, термины «реактивирующий пептид», «реактивирующий Mut-р53 пептид» могут использоваться взаимозаменяемо и относятся к пептидному агенту, способному по меньшей мере частично возвращать активность белку Mut-p53. В некоторых вариантах осуществления реактивирующий агент может реактивировать Mut-p53 путем воздействия на конформацию Mut-p53, принимая конформацию, которая больше похожа на нативную или идентична нативной, WT р53. В некоторых вариантах осуществления реактивирующий агент может реактивировать Mut-p53 с восстановлением связывания Mut-p53 с сайтом связывания WT р53 в ДНК-мишени. В некоторых вариантах осуществления реактивирующий агент может восстанавливать биохимические свойства Mut-p53. В некоторых вариантах осуществления реактивирующий агент может индуцировать белок Mut-p53 к проявлению p53-селективного ингибирования раковых клеток. В некоторых вариантах осуществления реактивирующий агент может реактивировать Mut-p53 с приобретением структурных свойств, биохимических свойств, физиологических свойств и/или функциональных свойств, аналогичных или идентичных белку р53 дикого типа. В некоторых вариантах осуществления реактивирующий агент представляет собой пептид. В некоторых вариантах осуществления реактивирующий агент представляет собой пептид, имеющий 3-25 аминокислот в длину. В некоторых вариантах осуществления реактивирующий агент представляет собой пептид, имеющий 5-20 аминокислот в длину. В некоторых вариантах осуществления реактивирующий агент представляет собой пептид, имеющий 6-15 аминокислот в длину. В некоторых вариантах осуществления реактивирующий агент представляет собой пептид, имеющий 7 или 12 аминокислот в длину.
Термин «конформация» по отношению к белку относится к конструктивному построению (складыванию) белка в пространстве.
Термины «глубокое секвенирование» и «секвенирование следующего поколения» могут использоваться взаимозаменяемо и относятся к способу усиленного секвенирования, обеспечивающего быстрое параллельное секвенирование множества нуклеиновокислотных последовательностей.
Способ «фагового дисплея» включает скрининг библиотеки фагов, каждый из которых экспрессирует и представляет специфическую экзогенную молекулу, как например пептид. Обогащение фагов, которые экспрессируют и представляют специфический пептид, достигается с помощью аффинной селекции из фаговой библиотеки на иммобилизованной мишени. В этом «пэннинг»-процессе связывающиеся фаги (т.е. фаги, экспрессирующие и представляющие пептид, который может связываться с иммобилизованной мишенью) захватываются, тогда как несвязывающиеся фаги (т.е. фаги, не экспрессирующие и не представляющие пептид, который может связываться с иммобилизованной мишенью) вымываются. Следующий этап способа может включать элюцию и амплификацию связанных фагов путем реинфицирования клеток E.coli идентифицированными фагами. В некоторых вариантах осуществления фаговая библиотека может быть исходной библиотекой или коммерчески доступной библиотекой фагового дисплея.
Термины «полипептид» и «пептид» используются в данном описании взаимозаменяемо для обозначения полимера аминокислотных остатков. Термины применимы к аминокислотным полимерам, в которых один или несколько аминокислотных остатков представляют собой искусственный химический аналог соответствующей природной аминокислоты, а также к встречающимся в природе аминокислотным полимерам.
Термины «нуклеиновая кислота», «полинуклеотид», «олигонуклеотид» или «олиго» относится к одноцепочечному или двухцепочечному полимеру, состоящему из нуклеотидов ДНК (дезоксирибонуклеиновая кислота), нуклеотидов РНК (рибонуклеиновая кислота) или комбинации обоих типов, а также могут включать природные нуклеотиды, химически модифицированные нуклеотиды и синтетические нуклеотиды.
«Аминокислота» относится к любой из 20 природных аминокислот, аминокислот, которые были химически модифицированы (смотри ниже), или синтетических аминокислот.
«Консервативная замена» относится к замене аминокислоты в одном классе аминокислотой того же класса, где класс определяется общими физико-химическими свойствами боковой цепи аминокислот и высокой частотой замещений в гомологичных белках, обнаруженных в природе, согласно определению, например, с помощью стандартной частотно-обменной матрицы Дайхоффа или матрицы BLOSUM. Шесть общих классов боковых цепей аминокислот разделены на категории и включают: Класс I (Cys); Класс II (Ser, Thr, Pro, Ala, Gly ); Класс III (Asn, Asp, Gin, Glu); Класс IV (His, Arg, Lys); Класс V (Ile, Leu, Val, Met);. и Класс VI (Phe, Tyr, Trp). Например, замещение Asp на другой остаток класса III, как например Asn, Gin или Glu, является консервативным замещением.
«Неконсервативное замещение» относится к замещению аминокислоты в одном классе аминокислотой другого класса; например, замещение Ala, остаток Класса II, остатком Класса III, как например Asp, Asn, Glu или Gin.
Термин «химически модифицированный» относится к аминокислоте, которая модифицирована либо с помощью естественных процессов, либо с помощью методов химической модификации, которые хорошо известны в данной области техники. Среди многочисленных известных модификаций типичные, но не исключительные примеры включают: ацетилирование, ацилирование, аминирование, АДФ-рибозилирование, гликозилирование, образование гликозамингликанов, формирование GPI-якоря, ковалентное присоединение липида или липидного производного, метилирование, миристоилирование, пэгилирование, пренилирование, фосфорилирование, убиквитинирование или любой подобный процесс.
Как указано в данном описании, термин «лечение заболевания» или «лечение состояния» относится к введению композиции, которая включает по меньшей мере один агент, эффективный для улучшения ассоциированных с заболеванием симптомов, для уменьшения тяжести или излечения заболевания или для предупреждения возникновения заболевания у субъекта. Введение может включать любой способ введения. В некоторых вариантах осуществления заболевание представляет собой заболевание, которое вызвано или связано с наличием мутантного р53 в клетке, ткани, органе, организме и тому подобное. В некоторых вариантах осуществления заболевание представляет собой рак. В некоторых вариантах осуществления рак выбирают из группы, состоящей из рака молочной железы, рака толстой кишки и рака легких. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения. В некоторых вариантах осуществления субъектом является млекопитающее, как например человек. В некоторых вариантах осуществления субъектом является млекопитающее животное. В некоторых вариантах осуществления субъектом является не млекопитающее животное.
Термин «экспрессия», используемый в данном описании, относится к получению желаемой молекулы конечного продукта в клетке-мишени. Молекула конечного продукта может включать, например, молекулы РНК; пептид или белок; и тому подобное; или их комбинации.
Термин «конструкция», используемый в данном описании, относится к искусственно собранной или выделенной молекуле нуклеиновой кислоты, которая может представлять собой одну или несколько нуклеиновокислотных последовательностей, причем нуклеиновокислотные последовательности могут содержать кодирующие последовательности (то есть, последовательность, которая кодирует конечный продукт), регуляторные последовательности, некодирующие последовательности или любую их комбинацию. Термин «конструкция» включает в себя, например, вектор, но не следует его рассматривать как ограничивающийся этим.
«Вектор экспрессии» относится к векторам, которые обладают способностью инкорпорировать и экспрессировать гетерологичные фрагменты нуклеиновых кислот (таких как, например, ДНК), в чужой клетке. Другими словами, вектор экспрессии содержит нуклеиновокислотные последовательности/фрагменты (как например, ДНК, мРНК, тРНК, рРНК), способные к транскрипции. Известно и/или коммерчески доступно много прокариотических и эукариотических векторов экспрессии. Отбор соответствующих векторов экспрессии находится в пределах знаний специалистов в данной области техники.
Термины «в обратном направлении (Upstream)» и «в прямом направлении (Downstream)», используемые в данном описании, касаются относительной позиции в нуклеотидной последовательности, как например, ДНК-последовательности или РНК-последовательности. Как хорошо известно, нуклеотидная последовательность имеет 5' конец и 3' конец, называемые так в отношении углеродов на сахарном (дезоксирибоза или рибоза) кольце нуклеотидной цепи. Следовательно, по отношению к позиции на нуклеотидной последовательности термин «в прямом направлении» относится к участку в направлении к 3' концу последовательности. Термин «в обратном направлении» относится к участку в направлении к 5' концу нити.
Используемые в настоящем описании термины «внедрение», «трансфекция» или «транфектирование» и «инфекция» или «инфицирование» могут использоваться взаимозаменяемо и относятся к переносу молекул, как например, нуклеиновые кислоты, полинуклеотидные молекулы, векторы и тому подобное, в клетку-мишень (клетки-мишени) и, более конкретно, внутрь ограниченного мембраной пространства клетки-мишени (клеток-мишеней). Молекулы могут «внедряться» в клетку-мишень (клетки-мишени) с помощью любых средств, известных специалистам в данной области, например, как предлагают в Sambrook et al. Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, New York (2001), содержание которого включено в настоящее описание посредством ссылки. Средства «внедрения» молекул в клетку включают, например, но не ограничиваются этим: температурный шок, кальций- фосфатная трансфекция, PEI трансфекция, электропорация, липофекция, агент (агенты) трансфекции, вирус-опосредованный перенос и тому подобное, или их комбинации. Трансфекция клетки может выполняться на любом типе клетки, любого происхождения.
Используемый в настоящем описании термин «экзогенный ген» относится к гену (или любой ее части), который внедряется извне в клетку. В некоторых вариантах осуществления экзогенный ген встраивается в виде полинуклеотида (например, ДНК, РНК и т.п.). В некоторых вариантах осуществления экзогенный ген способен экспрессироваться в клетке. В некоторых вариантах осуществления экзогенный ген сверхэкспрессируется внутри клетки.
Используемый в данном описании термин «примерно» относительно численного значения, указанному в настоящем описании, следует понимать как указанное значение +/- 10%.
В некоторых вариантах осуществления реактивирующий пептид может реактивировать Mut-p53 с получением структурных свойств, биохимических свойств, физиологических свойств и/или функциональных свойств, аналогичных или идентичных белку р53 дикого типа.
В соответствии с некоторыми вариантами осуществления предлагаются реактивирующие Mut-p53 пептиды, в которых пептиды имеют длину примерно 3-25 аминокислот. В некоторых вариантах осуществления реактивирующие Mut-p53 пептиды имеют длину примерно 4-15 аминокислот. В некоторых вариантах осуществления реактивирующие Mut-p53 пептиды имеют длину примерно 7-12 аминокислот. В некоторых вариантах осуществления реактивирующие Mut-p53 пептиды имеют длину 7 аминокислот. В некоторых вариантах осуществления реактивирующие Mut-p53 пептиды имеют длину 12 аминокислот. Каждая возможность представляет отдельный вариант осуществления настоящего изобретения.
В некоторых вариантах осуществления предлагается реактивирующий Mut-p53 пептид, имеющий аминокислотную последовательность, как обозначено с помощью любой из пептидных последовательностей в Таблицах 6, 7 или 8 ниже.
Согласно некоторым вариантам осуществления реактивирующий Mut-p53 пептид может влиять на Mut-p53 таким образом, что он может трансактивировать ген-репортер (как например люцифераза), имеющий элемент, связывающий р53 дикого типа, в его промотор. В некоторых вариантах осуществления трансактивация гена-репортера может выполняться in vitro (например, в тест-пробирке или лунке) или in-vivo в клетке, укрывающей конструкцию гена-репортера.
Согласно некоторым вариантам осуществления реактивирующий Mut-p53 пептид может связываться с ДНК-связывающим доменом (DBD) мутантного р53. В некоторых вариантах осуществления мутантный р53 укрывает мутацию в своем ДНК-связывающем домене (DBD).
В некоторых вариантах осуществления рак представляет собой адренокортикальную карциному, рак анального канала, рак мочевого пузыря, опухоль мозга, глиому ствола мозга, опухоль головного мозга, астроцитому мозжечка, астроцитому головного мозга, эпендимому, медуллобластому, супратенториальные примитивные нейроэктодермальные шишковидные опухоли, гипоталамическую глиому, рак молочной железы, карциноидную опухоль, карциному, рак шейки матки, рак толстой кишки, эндометриальный рак, рак пищевода, внепеченочный рак желчных протоков, опухоли семейства Юинга (pnet), внепеченочную опухоль зародышевых клеток, рак глаза, внутриглазную меланому, рак желчного пузыря, рак желудка, опухоль зародышевых клеток, внегонадную, гестационную трофобластическую опухоль, рак головы и шеи, гипофарингеальный рак, карциному островковых клеток, рак гортани, лейкоз, острый лимфобластный лейкоз, рак ротовой полости, рак печени, рак легкого, мелкоклеточную лимфому, связанную со СПИДом лимфому, лимфому центральной нервной система (первичную), кожную Т-клеточную лимфому, заболевание Ходжкина, неходжкинское заболевание, злокачественную мезотелиому, меланому, Меркель-клеточную карциному, метастатическую плоскоклеточную карциному, множественную миелому, неоплазмы плазматических клеток, грибовидный микоз, миелодиспластический синдром, миелопролиферативные заболевания, носоглоточный рак, нейробластому, ротоглоточный рак, остеосаркому, эпителиальный овариальный рак, овариальный рак зародышевых клеток, овариальную опухоль с низким злокачественным потенциалом, рак поджелудочной железы, экзокринный рак поджелудочной железы, карциному островковых клеток, рак околоносовой пазухи и носовой полости, паратиреоидный рак, рак полового члена, рак феохромоцитома, рак гипофиза, неоплазма плазматических клеток, рак предстательной железы, рабдомиосаркому, рак прямой кишки, рак клеток почечного эпителия, рак слюнной железы, синдром Сезари, рак кожи, кожную Т-клеточную лимфому, рак кожи, саркому Капоши, рак кожи, меланому, мелкоклеточный рак кишечника, саркому мягких тканей, саркому мягких тканей, рак яичка, тимому, злокачественный рак щитовидной железы, рак мочеиспускательного канала, рак матки, саркому, редкий рак у детей, рак влагалища, рак вульвы или опухоль Вильмса.
В некоторых вариантах осуществления рак представляет собой несолидную опухоль, как например рак крови. В другом варианте осуществления несолидная опухоль или рак крови представляет собой лейкоз или лимфому. В другом варианте осуществления несолидная опухоль или рак крови представляет собой острый лимфобластный лейкоз (ALL). В другом варианте осуществления несолидная опухоль или рак крови представляет собой острый миелобластный лейкоз (AML). В другом варианте осуществления несолидная опухоль или рак крови представляет собой хронический лимфоцитарный лейкоз (CLL). В другом варианте осуществления несолидная опухоль или рак крови представляет собой малую лимфоцитарную лимфому (SLL). В другом варианте осуществления несолидная опухоль или рак крови представляет собой хронический миелобластный лейкоз (CML). В другом варианте осуществления несолидная опухоль или рак крови представляет собой острый моноцитарный лейкоз (AMOL). В другом варианте осуществления несолидная опухоль или рак крови представляет собой ходжкинские лимфомы (любая из четырех подтипов). В другом варианте осуществления несолидная опухоль или рак крови представляет собой неходжкинские лимфомы (любая из подтипов). В другом варианте осуществления несолидная опухоль или рак крови представляет собой миелоидный лейкоз.
Для использования в способах по изобретению реактивирующие пептиды могут быть приготовлены обычным способом при использовании одного или нескольких фармацевтически приемлемых носителей, стабилизаторов или эксципиентов (наполнителей) с образованием фармацевтической композиции, известной в данной области, в частности в отношении белковых активных веществ. Носитель (-и) являются «приемлемыми» в смысле совместимости с другими ингредиентами композиции и безвредны для их реципиента. Подходящие носители, как правило, включают физиологический солевой раствор или этанол полиолы, такие как глицерин или пропиленгликоль.
Реактивирующие пептиды могут быть приготовлены в виде нейтральных или солевых форм. Фармацевтически приемлемые соли включают кислотно-аддитивные соли (образованные со свободными аминогруппами) и которые образованы с неорганическими кислотами, как например соляная кислота или фосфорная кислота, или такими органическими кислотами, как например уксусная, щавелевая, винная и малеиновая кислоты. Соли, образованные со свободными карбоксильными группами, также могут быть получены из неорганических оснований, как например натрий, калий, аммоний, кальций или гидроокиси железа, и из органических оснований, как изопропиламин, триметиламин, 2-метиламино этанол, гистидин и прокаин.
Композиции могут быть надлежащим образом составлены для внутривенного, внутримышечного, подкожного или внутрибрюшинного введения и обычно содержат стерильные водные растворы реактивирующих пептидов, которые предпочтительно изотоничные крови реципиента. Такие препараты, как правило, получают путем растворения твердого активного ингредиента в воде, содержащей физиологически совместимые вещества, как например хлорид натрия, глицин и тому подобное, и имеющей забуференное значение рН, совместимое с физиологическими условиями для получения водного раствора, с обеспечением стерильности указанного раствора. Они могут быть приготовлены в виде однодозовых или многодозовых контейнеров, например, запаянные ампулы или флаконы.
Композиции могут включать стабилизатор, такой как, например, полиэтиленгликоль, белки, сахариды (например, трегалоза), аминокислоты, неорганические кислоты и их смеси. Стабилизаторы используются в водных растворах при соответствующей концентрации и рН. Значение рН водного раствора регулируется в диапазоне 5,0-9,0, предпочтительно в диапазоне 6-8. При составлении реактивирующих пептидов может использоваться анти-адсорбционный агент. Другие подходящие эксципиенты могут включать, как правило, антиоксидант, как например аскорбиновая кислота.
Композиции могут быть составлены в виде препаратов с контролируемым высвобождением, которое может быть достигнуто за счет использования полимера для образования комплекса или поглощения белков. Подходящие полимеры для препаратов с контролируемым высвобождением включают, например, сложный полиэфир, полиаминокислоты, поливинилпирролидон, этиленвинилацетат и метилцеллюлозуа. Другим возможным способом контролируемого высвобождения является инкорпорация реактивирующих пептидов в частицы полимерного материала, как например сложные полиэфиры, полиаминокислоты, гидрогели, поли (молочная кислота) или сополимеры этилена и винилацетата. В качестве альтернативы, вместо инкорпорации этих агентов в полимерные частицы эти материалы можно помещать в микрокапсулы, приготовленные, например, с помощью методов коацервации или путем межфазной полимеризации, например, гидроксиэтилцеллюлозные или желатиновые микрокапсулы и поли(метилметакрилатные) микрокапсулы, соответственно, или в коллоидные системы доставки лекарственного средства, например, липосомы, альбуминовые микросферы, микроэмульсии, наночастицы и нанокапсулы или в макроэмульсии.
В некоторых вариантах осуществления реактивирующие пептиды по изобретению могут быть составлены в виде пероральных, или для приема внутрь, композиций, а в некоторых вариантах осуществления они включают жидкие растворы, эмульсии, суспензии и тому подобное. В некоторых вариантах осуществления фармацевтически приемлемые носители, пригодные для приготовления таких композиций, хорошо известны в данной области техники. В некоторых вариантах осуществления жидкие пероральные композиции содержат от примерно 0,001% до примерно 0,9% реактивирующих пептидов или в другом варианте осуществления от примерно 0,01% до примерно 10%.
В некоторых вариантах осуществления композиции для применения в способах по настоящему изобретению включают растворы или эмульсии, которые в некоторых вариантах осуществления представляют собой водные растворы или эмульсии, содержащие безопасное и эффективное количество реактивирующего пептида и, по желанию, другие соединения, предназначенные для местного интраназального введения.
В некоторых вариантах осуществления инъекционные растворы по изобретению приготовлены в виде водных растворов. В одном варианте осуществления инъекционные растворы по изобретению приготовлены в физиологически совместимых буферах, как например раствор Хэнкса, раствор Рингера или физиологический солевой буфер. В некоторых вариантах осуществления для чресслизистого введения в рецептуре используются пенетранты, подходящие для проникания через барьер. Такие пенетранты, как правило, известны в данной области техники.
В одном варианте осуществления препараты, описанные здесь, составлены для парентерального введения, напр., путем болюсной инъекции или непрерывной инфузии. В некоторых вариантах осуществления составы для инъекций представлены в стандартной лекарственной форме, напр., в ампулах или в многодозовых контейнерах с добавлением консерванта, по желанию. В некоторых вариантах осуществления композиции представляют собой суспензии, растворы или эмульсии в масляных или водных наполнителях и содержат вспомогатеьные агенты, как например суспендирующие, стабилизирующие и/или диспергирующие агенты.
Реактивирующие пептиды по изобретению могут быть введены любым подходящим способом введения, выбранным из перорального, местного, чрескожного или парентерального введения. Согласно некоторым вариантам осуществления способ введения является способом местного применения, выбранным из следующих способов: дермальный, вагинальный, ректальный, путем ингаляции, интраназальный, офтальмологический, аурикулярный и буккальный. Согласно некоторым вариантам осуществления способ введения осуществляется с помощью парентеральной инъекции. В различных вариантах осуществления этап введения выполняется парентеральным путем, выбранным из группы, состоящей из способов: внутривенного, внутримышечного, подкожного, внутрикожного, внутрибрюшинного, внутриартериального, внутримозгового, интрацеребровентрикулярного, внутрикостного и интратекального. Например, реактивирующие пептиды могут вводиться системно, например, парентеральным способом, таким как внутрибрюшинный (i.p.), внутривенный (i.v.), подкожный или внутримышечный пути введения. Реактивирующие пептиды по изобретению и/или, по желанию, любой дополнительный агент могут вводиться системно, например, путем интраназального введения. Реактивирующие пептиды по изобретению и/или, по желанию, любой дополнительный агент могут вводиться системно, например, путем перорального введения, с использованием специфических композиций или составов, способных обеспечивать пероральную биодоступность к белкам. Реактивирующие пептиды по изобретению и/или, по желанию, любой дополнительный агент могут вводиться местно.
Реактивирующие пептиды могут вводиться в диапазоне от примерно 0,1 до примерно 20 мг/кг веса субъекта, обычно от примерно 0,5 до примерно 10 мг/кг и часто от примерно 1 до примерно 5 мг/кг. В некоторых случаях может оказаться предпочтительным введение большой ударной дозы с последующими периодическими (напр., раз в неделю) поддерживающими дозами в течение всего периода лечения. Реактивирующие пептиды также могут доставляться с помощью систем доставки с медленным высвобождением, насосов и других известных систем доставки для непрерывного вливания. Режимы дозирования могут варьироваться для обеспечения желаемых уровней циркулирующих конкретных реактивирующих пептидов на основе их фармакокинетики. Таким образом, дозы рассчитываются таким образом, чтобы поддерживался желаемый уровень циркулирующего терапевтического агента.
Как правило, эффективная доза определяется активностью реактивирующих пептидов и состояния субъекта, а также массой тела или площадью поверхности субъекта, подлежащего лечению. Размер дозы и режим дозирования также определяется наличием, природой и степенью любых неблагоприятных побочных эффектов, которые сопровождают введение реактивирующих пептидов конкретному субъекту.
В некоторых вариантах осуществления предлагается набор для лечения или предупреждения связанного с р53 состояния. В некоторых вариантах осуществления набор содержит контейнер (например, флакон), содержащий реактивирующий Mut-p53 пептид в подходящем буфере, и инструкции по применению реактивирующего пептида.
Следующие примеры представлены для более полной иллюстрации некоторых вариантов осуществления настоящего изобретения. Они, однако, никоим образом не должны рассматриваться как ограничивающие широкий объем изобретения. Специалист в данной области может легко разработать много вариаций и модификаций принципов, описанных в данном документе, не отступая от объема настоящего изобретения.
ПРИМЕРЫ
Материалы и способы
Очистка рекомбинантных белков полной длины (FL) от sf9 клеток: мутант p53 R249S, мутант р53 R175H и WT р53:
2×107клеток sf9 в лог-фазе выращивали в девяти 175- см2 колбах, содержащих 25 мл среды, и инкубировали в течение ночи при 27oС. В каждую колбу добавили бакуловирусы с рекомбинантным p53 и инкубировали в течение 72 часов. Клетки выскоблили из колб и центрифугировали при темпетаруре 4°С (3200g в течение 5 мин), среду удалили и клеточный осадок дважды промыли охлажденным на льду изотоническим буфером (10 мМ Na2HPO4, рН 7,2, 130 мМ NaCl, 1 мМ DTPA - диэтилентриаминпентауксусная кислота). Для лизиса клетки ресуспендировали в 50 мл буфера А (20 мМ Трис-HCl, рН 8,0, 12% сахароза, 2 мМ EGTA, 2 мМ PMSF, 5 мМ DTT) с 0,2% тритон Х-100 при мягком переворачивании. Ядра, центрифугированные при 5600G в течение 8 мин, и супернатант удалили. Ядра лизировали добавлением 20 мл буфера В (20 мМ Трис-HCl, рН 8,0, 12% сахароза, 2 мМ EGTA, 2 мМ PMSF, 10 мМ DTT + ингибиторы протеазы) с 0,5М NaCl, энергично встряхивали и инкубировали в течение 20 мин на льду. Ядерный лизат перенесли в трубки ультрацентрифуги и центрифугировали при 100000g в течение 60 минут при 4°С. Супернатант удалили и разбавили буфером B до конечной концентрации 0,04 М NaCl, затем центрифугировали при 20000g в течение 5 мин при 4°С. Ядерный лизат загрузили на 5-мл ионообменную колонку Hitrap Q FF (быстропоточную) (Amersham Pharmacia), предварительно промытую 50 мл буфера А. Затем колонку промыли буферами с содержанием более высоких концентраций соли для элюции белка. Например, в случае мутантного р53 R249S, белок элюировался из ионообменной колонки при ~150 мМ NaCl. Белок далее очищали с помощью гель-фильтрационной хроматографии с использованием препаративной колонки Superdex 75 (Amersham Pharmacia Biotech), предварительно уравновешенной 20 мМ цитрата натрия, рН 6,1, 150 мМ NaCl, 10 μМ ZnCl2, и 10 мМ DTT. Фракции, содержащие очищенный белок, объединили и концентрировали до 6 - 7 мг/мл, подвергли аликвотированию и хранили при -80OC. Фракции, полученные после каждого этапа очистки, проанализировали на дот-блоттинге на присутствие мутантного p53, а затем на SDS-PAGE с помощью окрашивания Кумасси синим для проверки чистоты фракций.
Сэндвич-ELISA
96-луночные планшеты покрыли 3-мя различными антителами (1 тип антитела (Ab) в каждой лунке): PAb421 распознает обе конформации р53 и связывается с С-концевым эпитопом; PAb240 распознает мутантную конформацию р53, связывается с эпитопом в пределах ядерного домена (аминокислоты 212-217) (Stephen, C.W. and D.P. Lane, Mutant conformation of p53. Precise epitope mapping using a filamentous phage epitope library. J. Mol. Biol., 1992. 225(3): p. 577-83), который доступен для Ab, когда белок частично денатурирован (например, когда DBD мутирован); и PAb1620, который распознает конформацию белка р53дикого типа, связывается с эпитопом в пределах ядерного домена (аа 156, 206-210), образованного когда складывание происходит в конформации дикого типа (Wang, P.L., F. Sait, and G. Winter, The 'wild type' conformation of p53: epitope mapping using hybrid proteins. Oncogene, 2001. 20(18): p. 2318-24).
Лунки инкубировали в течение ночи (ON) с 100 μл Ab (5 μг/мл) при комнатной температуре (RT). Жидкость слили, а лунки промыли 3 раза фосфатно-солевым буфером (PBS), по 200 μл на каждую промывку. Далее была выполнена блокировка с помощью 200 μл 5% бычьего сывороточного альбумина (BSA), разбавленного в PBS в каждой лунке в течение 1,5 часов при комнатной температуре (RT). Блокирующий буфер отбросили, а затем 3 раза промыли в PBS, как описано выше. Образцы мутантных белков р53 и белков р53 дикого типа (100 μл, 10 μг/мл) вместе с контрольными пептидами pCAP-710 (LPNPPER, SEQ ID NO:340) и pCAP-1220 (FRSFAIPLVVPF, SEQ ID NO:368) (5 μг/мл, Sigma Aldrich) или с тестовыми пептидами 1-153 (5 μг/мл) инкубировали в течение 1,5 часов вместе, а затем добавили в лунки. Образцы вращали и инкубировали в течение 1 часа при комнатной температуре. Образцы отбросили, после этого выполнили 4 промывки, как описано выше, с использованием трифосфат-солевого буферного раствора (TPBS). Далее в лунки добавили конъюгированные пероксидазой хрена (HRP) р53-антитела с стрептавидином (10 μг/мл HAF1355 (R & D)) и инкубировали при комнатной температуре в течение 1 часа. После того, как планшет промыли 3 раза в TPBS, добавили раствор субстрата TMB (50 μл в каждую лунку, Thermo, (Cat. No. ES001-1L-K)) и инкубировали при 37°С в течение 20 мин. Реакцию остановили с помощью 2 М серной кислоты (50 μл). Поглощение измеряли при 450 нм с помощью спектрофотометра. Концентрацию белка определяли путем деления оптической плотности каждого образца на поглощение образцов Ab 421.
Анализ на связывание с ДНК
Для этих экспериментов использовали коммерческий набор связывания p53/ДНК «R&D» (Cat-DYC1355-5 Lot-1273366FA) в соответствии с руководящими указаниями производителя. Вкратце, 96-луночные планшеты покрываются анти-p53 антителом в течение ночи. Клеточные экстракты, содержащие р53, подвергаются реакции с олигонуклеотидом, который содержит консенсусный сайт связывания р53 (входящий в набор), меченный биотином, в присутствии или в отсутствие (NT) тестовых пептидов. Предполагается, что белок р53 дикого типа связывается с этим ДНК-связывающим сайтом, а также с антителами, покрывающими тестовые лунки планшета. Избыток p53 и олигонуклеотидов смыли и использовали стрептавидин-HRP для определения количества олигонуклеотидов в лунке, которое пропорционально ДНК, связанной белком р53. Для определения уровней HRP (ES001-1L-K) был проведен TMB анализ (450нм).
Анализ с помощью кристаллвиолета
Клетки культивировали в 96-луночных планшетах при 2500-4000 клеток/лунку в 0,1 мл и инкубировали в течение ночи при 37°С, чтобы они прикрепились к планшету. Добавили серийные разведения различных пептидов (0,5 μг/мл) в 0,1 мл аликвот и планшеты инкубировали в течение еще 48 часов при 37°С. Затем среду удалили и лизис клеток определяли путем окрашивания клеток кристаллвиолетом (0,5%) в смеси метанол/вода (1:4, об/об), 50 μл на каждую лунку, в течение 10 мин, затем 3 раза промывали PBS. После этого в каждую лунку добавляли 10% уксусную кислоту (50 μл) и встряхивали в течение 10 мин. Затем провели автоматическое считывание планшета при 595 нм.
Иммунофлуоресценция
Клетки культивировали на покровных стеклах в течение ночи, а затем обработали пептидами с использованием X-fect трансфекции. После 2-х часового восстановления клетки фиксировали 4% параформальдегидом в течение 30 мин при комнатной температуре, а затем 3 раза промыли (PBS). Образцы пермеабилизовали с помощью 0,1% Тритона (1% BSA в PBS) в течение 10 мин при комнатной температуре с последующим блокированием (3 промывки, 0,5% BSA в PBS), 5 мин на каждую промывку. Клетки затем исследовали с помощью мышиных анти-p53 (DO-1) антител, разведенных 1:500, в течение 1,5 часов с последующим блокированием (3 промывки, 0,5% BSA в PBS), 5 мин на каждую промывку. Затем клетки исследовали с помощью козьих анти-мышиных антител Су3, разведенных 1: 600, и DAPI, разведенных 1: 1000, в течение 45 мин. Образцы были подготовлены с помощью Elvanol.
Анализ с использованием люциферазы
Построение люциферазных конструкций
Олигонуклеотид (RGC-W), который имеет последовательность TCGAGTTGCCTGGACTTGCCTGGCCTTGCCTTTTC-3' (SEQ ID NO:362), и олигонуклеотидный мутантный олигонуклеотид RGC (RGC-M), который имеет последовательность 5'-TCGAGTTTAATGGACTTTAATGGCCTTTAATTTTC-3' (SEQ ID NO:363), получены от Kern et al. (Kern, S.E., et al., Identification of p53 as a sequence-specific DNA-binding protein. Science, 1991. 252(5013): p. 1708-11)) и служат в качестве консенсусных сайтов связывания для р53 дикого типа.
Эти мотивы были клонированы в сайты KPN и Eco53IK в pCLuc Mini-ТК 2 вектор. (NEB, Cat No. N0324S). Люциферазную конструкцию использовали для оценки транскрипционной активации р53 в тестовых клетках.
Анализ ChIP
Вкратце, клоны были сшиты формальдегидом (1% конечная концентрация) при комнатной температуре в течение 10 минут. Формальдегид нейтрализовали с помощью 2,5М глицина (конечная концентрация 0,25 М) в течение 5 минут. Клетки промывали последовательно 1 мл охлажденного на льду PBS, буфером I (0,25% Тритон Х-100, 10 мМ EDTA, 0,5 мМ EGTA, 10 мМ HEPES, рН 6,5) и буфером II (200 мМ NaCl, 1 мМ EDTA, 0,5 мМ EGTA, 10 мМ HEPES, рН 6,5) и собирали путем соскабливания. Затем клетки ресуспендировали в 0,3 мл буфера для лизиса (1% SDS, 10 мМ EDTA, 50 мМ Трис-HCl, рН 8,1, 1X коктейль ингибиторов протеазы (Roche Molecular Biochemicals, Indianapolis, IN) и обработали ультразвуком 10 раз (20 сек 'вкл', затем 40 сек 'выкл') при максимальных установочных параметрах (Bioruptor, Diagenode, NY) с последующим центрифугированием в течение 10 минут на льду с получением фрагментов 200-500 п.о. [bp]. Супернатанты собрали и разбавили в 10 раз в буфере для разведения ChIP (1% Тритон Х-100, 2 мМ EDTA, 150 мМ NaCl, 20 мМ Трис-HCl, рН 8,1) с последующей иммунологической очисткой с помощью 40 μл предварительно блокированной протеин-A-сефарозы (Santa Cruz Biotech) с 2 μг деградированной в результате гидродинамического сдвига ДНК спермы лосося и преиммуной сывороткой (1 μг сыворотки кролика) с 10 μл 100 мг/мл BSA в течение 2 часов при 4°С. Образец сохраняли для подготовки исходного образца.
Провели иммунопреципитацию в течение ночи при 4°C со специфическими антителами, полученными от. После иммунопреципитации добавили 40 μл протеин-А-сефарозы (предварительно блокированной с помощью ДНК спермы лосося) и дополнительно инкубировали в течение еще 1 часа. Преципитаты промывали последовательно в течение 10 минут каждый в TSE I (0,1% SDS, 1% Тритон Х-100, 2 мМ EDTA, 20 мМ Трис-HCl, рН 8,1, 150 мМ NaCl), TSE II (0,1% SDS, 1% Тритон Х-100, 2 мМ EDTA, 20 мМ Трис-HCl, рН 8,1, 500 мМ NaCl) и буфере III (0,25 М LiCl, 1% NP-40, 1% дезоксихолат, 1 мМ EDTA, 10 мМ Трис-HCl, рН 8,1). Преципитаты затем трижды промывали буфером ТЕ и дважды экстрагировали с помощью 1% SDS, 0,1 М NaHCO3. Элюаты объединили и нагревали при 65°С в течение минимум 6 часов в течение ночи, чтобы реверсировать формальдегидную сшивку. ДНК-фрагменты очистили с помощью набора QIAquick Spin Kit (Qiagen, CA). Реакции иммунопреципитации проводили в трех повторностях с использованием гранул только в качестве неспецифического контроля. Количественный анализ активных и репрессивных гистоновых меток в продуктах ChIP из клонов оценивали с помощью количественной ПЦР в режиме реального времени. Для нормализации эффективности иммунопреципитации (IP) выполнили нормализацию IP хроматина с использованием специфических праймеров для участка промотора некдина и 5' участка (который соответствует репрессивному участку хроматина).
Анализы клеточной культуры и люциферазного репортера
p53-нулевые клетки H1299 культивировали в течение ночи и затем трансфицировали люциферазными конструкциями при использовании агента для трансфекции MaxFect (Mediatech) в соответствии с протоколом производителя. Перед трансфекцией клеточную среду заменили на OPTI-MEM.
Клетки обработали различными пептидами через 24 часа после трансфекции. Еще через 24 часа среду для выращивания собрали на 96 черные планшеты: 40 μл для Cluc анализа и 20 μл для Gluc анализа. Анализ проводили с использованием микропланшета Turner BioSystems Modulus. Значение рассчитали по Cluc/gluc/NT (необработанные клетки).
RT-PCR
РНК получили с использованием набора Macherey-Nagel NucleoSpin РНК II на клеточном остатке согласно протоколу производителя. Аликвоты по 0,4-1 μг подвергли обратной транскрипции с помощью Bio-RT 2000 (Bio-Lab) и случайных гексамерных праймеров. Была проведена количественная полимеразная цепная реакция в режиме реального времени (QRT-PCR) на приборе ABI 7300 (Applied Biosystems) с использованием PerfeCTa SYBR Green FastMix ROX (Quanta). Использованные праймеры для RT-PCR представлены в Таблице 1 (последовательности праймеров представлены от 5' до 3 ').
Библиотека фагового дисплея
Использованная библиотека фагового дисплея представляла собой коммерчески доступные фаговые библиотеки, разработанные New England Biolabs (NEB). Одна библиотека состоит из линейных гепта-пептидов (PhD-7), другая библиотека из линейных додека-пептидов (PhD-12) (CAT NOs.: PhD-7, E8100S; PhD-12, E8110S). Рандомизированные пептидные последовательности в обеих библиотеках экспрессируются на N-конце минорного оболочечного белка pIII, что приводит к валентности 5 копий отображаемого пептида на вирион. Все библиотеки содержат короткую линкерную последовательность между отображаемым пептидом и pIII.
Глубокое секвенирование
Перед секвенированием была проведена ПЦР-реакция с праймерами, фланкирующими вставленные библиотеки Прямой-5'- NNNNNNNNCATGGAAAGATAGTG (SEQ ID NO:364) и Обратный-5'- NNNNNNNNCCTAAAACGATTTGTG (SEQ ID NO:365), первые 8 оснований каждого праймера рандомизированные и были инкорпорированы в виде смеси всех четырех оснований. Была применена рандомизация первых оснований, так как аппарат для секвенирования Solexa неспособен секвенировать повторяющиеся последовательности в течение первых нескольких циклов. Реакция ПЦР дала ДНК в требуемом количестве 5μг и требуемой длины (примерно 120 bp), которая включает фланкирующие праймеры и библиотеку клонированных пептидов для глубокого секвенирования по методу Solexa.
Пример 1: Калибровка экспериментальных условий
Выбор источника белка p53
При выборе источника белка для селекции методом фагового дисплея учитывается ряд соображений; рекомендуется использовать очищенные белки, поскольку взаимодействие фаговых клонов с различными белками в растворе может порождать неспецифические ложные положительные пептиды. В следующих экспериментах использовали человеческий белок р53 полной длины, очищенный от клеток SF9 (смотри выше) (Accession No. CG3336). Следовательно, была использована система экспрессии р53 в клеточной линии насекомых SF9, инфицированная бакуловирусом (как подробно описано выше). Основным преимуществом р53, экспрессированного в этой системе, является то, что он уже содержит посттрансляционные модификации.
Конформация бакуловирус-экспрессированных белков p53 дикого типа и Mut-p53
Первые эксперименты с Baculo-p53 проводили с использованием ядерных экстрактов лизатов клеток Sf9, экспрессирующих либо WT р53, мутантный р53 полной длины горячей точки (R175H), либо чувствительный к температуре (ts) мутантный р53 (V143A). Клетки SF9 были инфицированы вирусами, несущими любой из трех экспрессирующих векторов. Через 48 часов после инфицирования клетки собрали, ядра извлекли, а экстракты подвергли иммунопреципитации при помощи: PAb1620, PAb240, ASPP2 (также имеющие название (Р53-ВР2)) и/или Bcl2 в течение 3-х часов при 4°С. Иммунопреципитированный p53 детектировали с помощью вестерн-блоттинга, используя αp53-HRP Ab (Cat No. HAF1355 (R & D). Результаты этого эксперимента с использованием IP-вестерн блоттинга показаны на Фигуре 2. Как видно, и чувствительный к температуре (ts) мутантный р53 V143A (4ºС), и р53 дикого типа хорошо связываются с антителом PAb 1620, но не с PAb240. С другой стороны, мутантный р53 R175H проявляет более сильное связывание с PAb240, чем с PAb1620. Это говорит о том, что экспрессированный бакуловирусом мутантный р53 R175H принимает конформацию, которая является промежуточной между мутантным р53 и р53 дикого типа. Bcl2 не проявляет связывания ни с одной из форм р53, тогда как ASPP2 (P53-BP2) связывается со всеми формами р53 с приблизительно одинаковой аффинностью. Следовательно, можно сделать заключение, что ASPP2 и Bcl2 не могут использоваться в качестве маркеров конформации р53 в этих экспериментальных условиях.
Калибровка условий раствора
Для снижения относительно высокого остаточного связывания мутантного р53 R175H с PAb1620 и повышения связывания WT р53 с этим антителом была выполнена тонкая настройка условий анализа. Результаты показаны на Фигуре 3, на которой представлено пятно очищенного мутантного р53 (R175H) и WT р53, извлеченное из ядер клеток Sf9, инфицированных соответствующим бакуловирусом (как описано выше). Очищенный р53 растворили в предусмотренных буферах (A-Трис-50 мМ; В-Трис, NaCl 150 мМ; С-Трис, NaCl, Тритон 0,5%; D-Трис, Глицин 0,5%; Е-Na4O7P2 40мМ; F- GndCl 400мМ; G-GndCl 800мМ, H-Мочевина 1М; I-Мочевина 3М; буфер IP-IP), а затем подвергли иммунопреципитации при помощи PAb1620 и PAb240 в течение 3-х часов при 4°С и вестерн-блоттингу с использованием αp53-HRP-Ab. Как видно, раствор (А) содержит только 50 мМ Трис. В этом растворе связывание мутантного р53 R175H с PAb1620 составляет только примерно 5% по сравнению со связыванием с PAb240. Добавление либо 150 мМ NaCl, (В), 150 мМ NaCl + 0,5% Тритон (C), либо 0,5% глицина (D) усилило связывание мутантного R175H с PAb1620. 3M мочевины (I) уменьшили связывание мутанта р53 R175H с PAb1620, вероятно за счет вызванной денатурация белка. Более низкая концентрация мочевины, 1 М (Н), увеличила связывание мутантного р53 R175H (R175H р53) с PAb1620. 40 мМ Na4O7P2 (Е) уменьшили связывание R175H р53 PAb1620 до самого низкого уровня. И, наконец, в IP-буфере R175H p53 остался PAb1620-отрицательным; однако в этом буфере WT р53 показал сильное связывание с PAB 240 и уменьшенное связывание с PAb1620, что говорит о том, что IP-буфер вызывает умеренное неправильное сворачивание формы дикого типа. Следовательно, для дальнейших экспериментов используется буфер, содержащий только Трис.
Пример 2: Исходный скрининг библиотеки фагового дисплея и селекция по реактивирующим Мut-p53 пептидам
Сначала выполнили скриннинг методом фагового дисплея, при использовании белка R175H p53, одной phd-12 фаговой библиотеки (NEB, Cat. No. E8110S) и селекцию с антителом PAb1620. 200 нг белка р53 R175H подвергли реакции с 1011 фага в течение 1 часа, обеспечивая связывание представленных пептидов фага с Mut-p53 (R175H). Затем дополнительно в течение 1 часа добавляли гранулы, сшитые с PAb1620, для иммунопреципитации всего комплекса. Эту пэннинг-процедуру повторяли в течение трех раундов, увеличивая строгость отбора после каждого раунда путем уменьшения количества инкубированного Mut-p53: 1-й раунд - 200 нг, 2-й раунд - 100 нг и 3-ий раунд - 50 нг. Фаги элюировали с помощью очищенного ДНК-связывающего домена (DBD) p53 дикого типа при концентрации 2 μг/мл (DBD р53 (остатки 94-293) был субклонирован в pET-27b (Novagen)). Плазмиду трансформировали в штамм E.coli BL21 (DE3). Продуцирование белка проводили по методике, описанной для DBD мышиного р53 (Suad, O., et al., Structural basis of restoring sequence-specific DNA binding and transactivation to mutant p53 by suppressor mutations. J Mol. Biol., 2009. 385(1): p. 249-65). После каждого раунда отбора проводили титрование элюированного фага для оценки количества отобранных фагов (Таблица 2). Элюированные фаги амплифицировали путем инфицирования E-Coli с получением примерно 1013 фага для отбора в следующем раунде. Со второго раунда пэннинга проводили эксперимент контрольного пэннинга только с PAb1620 (без инкубации с Mut-p53); этот титр свидетельствует о специфичности пэннинга.
Как видно из Таблицы 2, в первом раунде отбора было получено 100 инфекционных фаговых частиц/μл и типичные значения обогащения между раундами отбора, что приводит к более высокому обогащению в первых двух раундах, а затем достигается плато в третьем и четвертом раунде пэннинга. Однако, число фагов, элюированных в реакциях специфического пэннинга отбора, а также в реакциях неспецифического контрольного пэннинга с PAb1620 было одинаковое. Такое обогащение предполагает, что фаг может связываться непосредственно с PAb1620, а не за счет взаимодействия с мишенью р53 R175H.
Для того чтобы снизить фон (неспецифическое связывание), были введены дополнительные этапы предварительной очистки и увеличено время предварительной очистки; однако доля фонового связывания остается высокой. Таким образом, чтобы снизить фоновое связывание, в процессе фагового дисплея были реализованы чередующиеся этапы отбора. С этой целью были выполнены различные стратегии отбора в каждом раунде отбора с попыткой минимизировать общие неспецифические элементы в экспериментальной системе (и, следовательно, снизить связывание с этими неспецифическими элементами).
Поскольку предполагается, что предпосылкой конформационного изменения р53 является связывание пептида с p53, был введен дополнительный этап отбора по связыванию р53 дикого типа между отборами с PAb1620. Было сделано предположение что, поскольку PAb1620 не будет присутствовать во втором раунде пэннинга, связывание фага непосредственно с ним будет исключено. К тому же, так как предварительным условием любого функционального пептида является связывание с р53, предполагается, что пептиды, предпочтительно связывающиеся с формой дикого типа, стабилизируют эту конформацию. Первый и третий раунды пэннинга были такие же, как в предыдущем эксперименте. Во втором раунде отбора, однако, был выполнен отбор по связыванию с фагом в отношении WT-p53 (His-меченный), и комплекс p53/фаг подвергли иммунопреципитации с использованием никелевых гранул (которые связываются с His-меткой). Титр элюированного фага оценивали после каждого раунда отбора. Как показано в Таблице 3, при элюции фагов было достигнуто 10-кратное обогащение, когда второй раунд сравнили с первым. Хотя это можно было считать немного ниже по стандартам метода фагового дисплея, причиной такого относительно низкого обогащения, возможно, является использование различных стратегий отбора в каждом раунде пэннинга, увеличение специфичности, но с другой стороны, уменьшение общего выхода отобранного фага. Обогащение со второго раунда отбора к третьему было на уровне 100- кратного, что указывает на заметное увеличение фагового обогащения по сравнению с предыдущим показателем 10. Это заметное увеличение связано с повторным отбором с PAb1620. Важно отметить, что количество фагов после третьего раунда составило примерно 105, в то время как с контрольным PAb1620 это количество составляло 4×103. Таким образом, неспецифический контроль (т.е. фон) составляет только примерно 5% от общих отобранных фагов.
Пример 3: Способ скрининга и идентификации реактивирующих Mut-p53 пептидов
Для скрининга, идентификации и выделения специфических пептидов, реактивирующих р53, был разработан и осуществлен способ, в котором используется комбинация отличающихся и взаимодополняющих стратегий отбора.
В данном примере были объединены три стратегии отбора. Первая стратегия отбора основана на реактивности с PAb1620, как описано выше. Вторая стратегия отбора основана на связывании WT р53 с его мотивом последовательности консенсусной ДНК: р53-чувствительный элемент (р53-RE). Связывание p53 с его консенсусной ДНК in-vitro было широко продемонстрировано [Joerger, A.C., M.D. Allen, and A.R. Fersht, Crystal structure of a superstable mutant of human p53 core domain. Insights into the mechanism of rescuing oncogenic mutations. J Biol Chem, 2004. 279(2): p. 1291-6]. Соответственно, для получения дцДНК (после отжига) были разработаны два комплементарных олигонуклеотида. Эти олигонуклеотиды содержат две тандемные копии консенсусных последовательностей p53-RE: одна консенсусная последовательность представляет собой идеальный консенсусный сайт связывания, полученный в результате экспериментов по связыванию (AGACATGCCCAGACATGTCC (SEQ ID NO:339)), а вторая последовательность представляет собой сайт связывания ДНК р53, полученный из промотора p21 (GAACATGTCCCAACATGTTG (SEQ ID NO:340)), который расположен в прямом направлении по отношению к первой консенсусной последовательности (Фигура 4). Кроме того, дополнительно были введены два сайта фермента рестрикции (HindIII (AAGCTT (SEQ ID NO:341)) и EcoRI (GAATTC (SEQ ID NO:342)), которые обеспечивают этап более специфической элюции после отбора. Одна нить олигонуклеотида также была мечена биотином, чтобы обеспечить иммунопреципитацию комплекса ДНК/p53/фаг при помощи покрытых стрептавидином гранул. На Фигуре 4 показана схематичная последовательность р53-RE олигонуклеотида и его элементы последовательности. Последовательность верхней нити олигонуклеотида:
Биотин-5'-CTGCTGAAGCTTCGAATTCCTAGACATGCCCAGACATGTCCTACTGCTGCTGCTGCTGCTG CTGCGAACATGTCCCAACATGTTGCTGCTGCTGCTGCTG-3' (SEQ ID NO:361).
В процедуре отбора, выполняемой с использованием стратегии ДНК-связывания (как описано ниже), 0,5-3 пмоль биотин-р53-RE олигонуклеотида подвергли реакции с 200 нг очищенного р53 дикого типа в течение 1 часа для обеспечения связывания. 1010 фагов либо из PhD-7, либо из PhD-12 фаговых библиотек затем вводили в течение еще одного часа. После этого добавили покрытые стрептавидином агарозные гранулы в течение 30 минут. Затем выполнили 5-12 этапов промывки с последующей элюцией, выполненной путем добавления либо рестрикционных ферментов, либо избытка небиотинилированной ДНК в течение 30 минут. Эти меры предосторожности позволяли сократить отбор фагового связывания с ДНК, биотин и стрептавидин.
Третья стратегия отбора основана на большом Т (LT) антигене SV40. Связывание между р53 и SV40 LT считается очень сильным. Таким образом, р53 должен складываться правильно, чтобы сформировалась платформа эпитопа связывания с SV40 LT. Для этой цели клетки Sf9 инфицировали бакуловирусом, кодирующим SV40 LT. Клетки лизировали и SV40 LT выделили с использованием гранул белка-А, сшитых с PAb 419 (антитело, SV40 LT-специфичное, (Abcam-ab 1684)). Гранулы промывали несколько раз, а затем использовали для отборов методом фагового дисплея. Процедура пэннинга для SV40 LT- связывания была аналогична стратегии на основе конформации, за исключением того, что для отбора вместо гранул PAb 1620 использовали гранулы PAb 419-SV40 LT.
Комбинация всех трех стратегий отбора в чередующихся раундах дает наилучшие результаты, поскольку каждый цикл постепенно увеличивает процент фагов, который укрывают желаемые специфические пептиды, при одновременном снижении неспецифического фона. Схематическое изображение способа идентификации и отбора иллюстрируется на Фигурах 1А и 1В.
Скрининг фагового дисплея проводили параллельно с пептидными библиотеками PhD-7 и PhD-12 фагов. На каждом этапе выполнялись чередующиеся циклы отбора фага с использованием другой иммобилизованной платформы (PAb1620, p53-RE DNA или SV40 LT). В Таблице 4 показаны различные маршруты отбора, принятые для создания обогащенной фаговой библиотеки, и представлены значения титра после каждого раунда отбора. При использовании таких различных комбинаций платформ отбора (напр., PAb1620 с последующей консенсусной ДНК р53, за которой снова следует PAb1620, или SV40 LT с последующим PAb1620, за которым следует SV40 LT), а также 2-х различных фаговых библиотек был получен список подбиблиотек, которые можно было бы затем сравнивать после секвенирования. После 3-х циклов отбора получили более 60 различных пулов (подбиблиотек), содержащих высокую долю Mut-p53-реактивирующего фага (Таблица 4).
Пример 5: Отобранные фаговые пулы индуцируют связывание Mut-р53 с PAb1620
Для того, чтобы определить, может ли метод отбора фагового дисплея, как представлено выше, обогащаться фагом, который реактивирует Mut-p53, исследовали способность фаговых пулов, полученных после 3 циклов отбора, индуцировать связывание либо белка Mut-p53 R175H полной длины (BD Pharmingen, Cat. No. 556439), либо рекомбинантного белка ДНК-связывающего домена р53 R249S (249 DBD) с PAb1620. Чтобы уменьшить нежелательный эффект загрязнения фага, который демонстрирует прямое связывание с PAb1620, ввели этап предварительной очистки, в силу чего фаговый пул сначала инкубировали только с PAb1620, перед добавлением в реакцию испытания. Гранулы, ковалентно сшитые с PAb1620, инкубировали с очищенным мутантным р53 R175H в присутствии фага, полученного путем отбора в фаговом дисплее либо с Mut-p53 R175H (175), либо Mut-p53 R249S (249), без выполнения или с выполнением этапа предварительной очистки путем инкубации фагового пула с гранулами PAb1620. Неотобранные фаги (ns) использовали в качестве контроля. Инкубацию проводили в течение 3 часов при 4°С. Связанный р53 визуализировали с помощью вестерн-блот-анализа с использованием антитела против р53. Как видно по результатам, представленным на Фигуре 5, некоторые из отобранных фаговых пулов действительно индуцировали связывание Mut-p53 с PAb1620, по сравнению с отсутствием фага или неотобранным исходным фагом (ns).
Пример 6: Отобранные фаговые пулы индуцируют связывание Mut-p53 с консенсусной ДНК p53
Для дальнейшей проверки, могут ли отобранные фаговые пулы способствовать связыванию Mut-р53 с связывающим элементом консенсусной ДНК белка р53, меченные биотином олигонуклеотиды, соответствующие p53-чувствительный элемент-консенсусной (p53-RE) биотин-AGACATGCCCAGACATGTC CTTATAGACATGCCCAGACATGTCC (SEQ ID NO:366), или контрольные олигонуклеотиды, мутированные в ключевые остатки, важные для связывания р53 (Con-RE биотин-AGAaATGCCCAGA aATGTCCTTATAGAaATGCCCAGAaATGTCC (SEQ ID NO:367), иммобилизовали путем реакции этих олигонуклеотидов с покрытыми стрептавидином гранулами. Гранулы р53-RE или Con-RE инкубировали либо с DBD белка р53 дикого типа, либо с DBD мутанта 249 вместе с фаговыми пулами, полученными после 3 циклов отбора. Покрытые стрептавидином гранулы, связанные либо с олигонуклеотидами p53-RE-ДНК, либо с олигонуклеотидами Con-RE-ДНК, меченными биотином, инкубировали с DBD очищенного WT р53 или с DBD мутантного р53 R249S в присутствии фага, полученного путем отбора методом фагового дисплея с Mut-p53 R175H (175), клон 27 (LPNPPER, SEQ ID NO: 328) (единственный клон, выделенный из отбора R175H), пулы #69 и #94, отобранные с р53 дикого типа и Mut-p53 R175H с использованием комбинаций T-AG и PAb1620 в чередующихся раундах отбора. Неотобранные фаги (NS) использовали в качестве контроля. Инкубация проводилась в течение 3-х часов при 4°С. Связанный р53 визуализировали с помощью вестерн-блот-анализа. Как видно по результатам, представленным на Фигуре 6, DBD р53 дикого типа связывался с p53-RE лучше, чем с Con-RE, как и следовало ожидать. DBD 249 не связывался с р53-RE в соответствии с его известной потерей способности последовательность-специфического связывания с ДНК. Важно отметить, что отобранные фаговые пулы были способны индуцировать связывание Mut-p53 с p53-RE, демонстрируя, что они действительно способны реактивировать и восстанавливать утраченную функцию Mut-p53.
Пример 7: Глубокое секвенирование отобранных фаговых пулов
Было выполнено секвенирование следующего поколения, которое значительно повышает эффективность фагового дисплея, позволяя извлекать и анализировать весь комплект отобранных пептидов с меньшим числом циклов отбора. Для глубокого секвенирования отобрали восемь фаговых пулов с помощью критериев повышенного обогащения между раундами отбора и функциональной активности. Перед секвенированием провели ПЦР-реакцию с праймерами, фланкирующими вставленные библиотеки: прямой -5'-NNNNNNNNCATGGAAAGATAGTG (SEQ ID NO:364), и обратный - 5'- NNNNNNNNCCTAAAACGATTTGTG (SEQ ID NO:365); первые 8 оснований каждого праймера рандомизированные и были инкорпорированы в виде смеси из всех четырех оснований. С целью повышения эффективности и точности секвенирования была введена рандомизация первых оснований. В результате ПЦР-реакции получили ДНК в необходимом количестве 5 μг и нужной длины (примерно 120 bp), которая включает фланкирующие праймеры и библиотеку клонированных пептидов для глубокого секвенирования по методу Solexa.
В результате глубокого секвенирования получили базу данных из 36 миллионов прочтений. 95% последовательностей содержали последовательности праймера, используемые в ПЦР при извлечении библиотек. Далее был выполнен предварительный биоинформатический анализ данных. Этот анализ включал удаление последовательностей, которые не содержат исходные праймеры, удаление последовательностей, которые не находятся в правильной рамке считывания, разделение базы данных на исходные библиотеки из 12 аминокислот и 7 аминокислот в соответствии с длиной вставки, и наконец, подсчет уникальных последовательностей и их сортировку по количеству появлений в базе данных. Было установлено, что большинство последовательностей появились только один раз или два раза в базе данных, вероятно, соответственно фоновому фагу. 12 прочтений были определены как порог отсечки, ниже которого обогащение последовательностей рассматривалось как незначительное. Последовательности ДНК в базе данных затем были преобразованы в аминокислотные последовательности.
В качестве внутреннего контроля качества сравнивали последовательности и их обилие как процент от общей библиотеки между двумя нитями, которые были секвенированы с противоположных направлений и, следовательно, содержали другой праймер на 5'-конце. Сравнение показало, что последовательности и их обилие были одинаковыми между двумя нитями, указывая, что полученная база данных последовательностей достоверна.
В Таблице 5 показан перечень пептидных последовательностей, полученных из базы данных глубокого секвенирования 5'-нитей. Эта база данных содержит в итоге 107 последовательностей после фильтрации несоответствующих последовательностей. Затем выполнили подсчет по порогу отсечки и трансляцию. В колонке (#Прочтения) показано, сколько раз последовательность повторяется в описываемой базе данных и, следовательно, это число соответствует обогащению данной специфической последовательности. Поскольку биоинформатический анализ проводился на ДНК-последовательностях, а отдельные пептиды могут быть кодированы несколькими различными ДНК-последовательностями в результате вырождения генетического кода, существует довольно много пептидов, которые появляются в таблице несколько раз. Если определенный пептид кодируется различными ДНК-последовательностями, это означает, что он был выбран независимо в различных фаговых клонах.
Альтернативно, число ДНК-последовательностей, кодирующих один и тот же пептид, может быть результатом ошибок секвенирования: однако, в этом случае следовало ожидать, что результатом такой ошибки будет случайное основание и, следовательно, не будет обогащено большим числом прочтений. Таким образом, ДНК- последовательности, которые находились ниже 30 прочтений в #Повторы, были исключены. В колонке (#Повторы) представлено число ДНК-последовательностей, кодирующих одну и ту же пептидную последовательность, и, следовательно, является еще одним свидетельством специфичности и эффективности отбора.
Как видно из Таблицы 5, последовательности могут быть разделены на их две библиотеки происхождения. Пептидная последовательность изображена в средней колонке и последовательности сортируются в порядке убывания в соответствии с количеством прочтений, что соответствует обогащению в каждой библиотеке. Было установлено, что библиотека 12aa доминирует за счет одной последовательности - KPPDRLWHYTQP (SEQ ID NO:322), что составляет почти 20% от общего числа последовательностей. Библиотека 7aa более многообразна и содержит намного больше последовательностей, но с более низкими значениями обогащения.
В Таблице 5 представлен анализ базы данных глубокого секвенирования - последовательности разделены на их две библиотеки происхождения, пептидная последовательность изображена в средней колонке и последовательности сортируются в порядке убывания в соответствии с числом прочтений, что соответствует обогащению в каждой библиотеке. В колонке (#Повторы) показано число ДНК-последовательностей, кодирующих одну и ту же пептидную последовательность.
Пример 8: Биоинформатический анализ мотива базы данных глубокого секвенирования
Далее был проведен более широкий биоинформатический анализ с целью идентификации консенсусных мотивов. Такие мотивы можно объяснить несколькими способами. Во-первых, сравнением пептидных последовательностей, идентифицированных в библиотеках 12aa и 7aa. Появление общих мотивов в обеих библиотеках будет поддерживать силу такого мотива, так как он был четко отобран в двух полностью независимых экспериментах. Во-вторых, обилие определенной аминокислоты в конкретной позиции и сходство с другими аминокислотами в той же позиции мотива могут служить свидетельством значимости такой аминокислоты в этой конкретной позиции. В-третьих, позиция мотива может иметь решающее значение для его функции: короткий мотив может смещаться вдоль более длинной пептидной последовательности при изменчивости других аминокислотных последовательностей и расстояние от свободного N-конца пептида может говорить о значимости для его активности. Был разработан следующий алгоритм проверки аминокислотной последовательности в растущем окне длины пептида:
1. Балльная оценка каждого пептида, объединение числа различных нуклеотидных последовательностей, которые преобразуются в тот же пептид при появлении каждого такого типа нуклеотидной последовательности;
2. кластеризация различных пептидов, балльная оценка сходства последовательностей между различными пептидами; и
3. идентификация групп родственных пептидных последовательностей и извлечение консенсуса из них.
Пептиды-кандидаты представляли собой пептиды с лучшими появлениями ≥ 0,2%: 40 из библиотеки 7aa и 32 из библиотеки 12aa. Они могут быть сгруппированы в 40 групп по их сходству согласно Blastp и появлению короткой аминокислоты (аа мотив). Большинство групп включало единственный пептид, но 9 групп включали 2-13 пептидов, и 6 из этих групп включали как пептиды из 7aa, так и пептиды из 12aa.
Группы были преобразованы в множественные выравнивания блока, причем % появлений представляет вес последовательности. Блоки использовали для запроса файлов с последовательностями пептидных групп 7aa и 12aa, и лучшие результаты были вновь преобразованы в блоки таким же образом. В некоторых блоках, но не во всех, результаты из двух библиотек были похожи друг на друга.
Результат глубокого секвенирования (т.е. создание базы данных из миллионов пептидных последовательностей по сравнению с сотнями последовательностей с помощью обычного секвенирования) позволил провести гораздо более детальный и всесторонний анализ консенсусных мотивов. В целом, идентифицировали примерно 130 мотивов существенно обогащенных последовательностей; большинство из этих пептидных мотивов представлено несколькими ДНК-последовательностями и 16 из этих мотивов распределяются между обеими библиотеками 7aa и 12aa. На Фигуре 7 показано несколько таких мотивов. Некоторые из мотивов получены в результате объединения перекрывающихся последовательностей и, следовательно, они длиннее, чем исходные пептидные библиотеки.
Пример 9: Синтез пептидов
Из полученного перечня идентифицированных пептидных мотивов, как описано выше, 128 пептидов были синтезированы химическим путем при помощи PEPTIDE 2.0 при грубой чистоте при использовании преимущества 96-луночного формата. Этот полусинтез с высокой пропускной способностью обеспечил относительно низкую стоимость каждого пептида. В Таблице 6 ниже перечислены синтезированные пептиды. Этот перечень также включает несколько пептидов, полученных из белков, о которых известно из литературы, что они взаимодействуют с р53. В перечень также входят 10 пептидов, синтезированных в двух версиях, без С-концевого присоединения полиаргинина и с С-концевым присоединением полиаргинина. Как сообщалось, присоединение полиаргинина обеспечивает пересечение пептидами клеточных мембран. Это позволяет оценить и способность С-концевого присоединения полиаргинина для обеспечения доставки пептида в клетки, и возможность влияния на нее активности этих специфических пептидов in-vivo. Полиаргинин может включать 0-10 остатков аргенина и обозначается как R0-10.
Между химически синтезированными пептидамив и пептидами, которые были отобраны из фаговых библиотек, могут существовать различия. В частности, отобранные пептиды были представлены в контексте фага как слитые белки с покрытым pIII фагом белком. Таким образом, этот переход к синтетическому пептиду не тривиальный, и известно, что в некоторых случаях, как сообщалось, пептиды активны, когда присутствуют на фаге, утратившем свою активность, когда такая же последовательность синтезируется как свободный пептид.
Пример 10: Функциональный скрининг основных тестовых пептидов
Использовали несколько альтернативных и взаимодополняющих способов скрининга основных пептидов-кандидатов по конформационным и функциональным действиям на Mut-p53. Поскольку нет никакой информации относительно проникновения каждого тестового пептида через клеточные мембраны, для оценки сначала выполнили анализы на основе in-vitro: ELISA для оценки конформации р53 и последовательность- специфического ДНК-связывающего белка р53. Затем в живых клетках исследовали активность пептидов с помощью анализов на жизнеспособность, транскрипционную активность p53 на люциферазном гене-репортере, а также исследовали гены-мишени р53 in-vivo. Комбинация этих анализов (все выполнены в 96-луночном формате) позволила идентифицировать и проверить действие пептидов на различную активность p53 и их способность придавать такую способность белкам Mut -p53.
Скрининг пептидов по действию на конформацию p53
Первая стратегия скрининга основывалась на ELISA. Версию сэндвич-ELISA использовали для изучения действия основных тестовых пептидов на конформацию р53. Для измерения конформационного действия пептидов на Mut-p53 титрационный микропланшет покрыли PAb240, PAb1620 или pAb421 (в качестве положительного контроля), а затем исследовали реакционную способность р53 по отношению к этим антителам. Белок р53 дикого типа служил в качестве положительного контроля для реакционной способности с PAb1620, а Mut-p53 служил в качестве отрицательного контроля. Для исследования действия тестируемого пептида его добавили к раствору, содержащему Mut-P53, и проверили изменение реакционной способности по отношению к любому Ab. Если после добавления пептида наблюдалось увеличение реакционной способности Mut-p53 по отношению к PAb1620 и снижение реакционной способности по отношению к PAb240, это указывало на то, что тестируемый пептид активизировал конформацию дикого типа Mut-p53. Были проведены несколько экспериментов ELISA с использованием различных клеточных экстрактов. Результаты представлены на Фигуре 8, которые показывают репрезентативный эксперимент, выполненный на экстракте клеток H1299, стабильно сверхэкспрессирующих Mut-p53, (R175H p53). Экстракты были приготовлены при концентрации 750 нг/μл в стандартном буфере иммунопреципитации при физиологическом рН и солевых концентрациях и с добавлением 3% BSA для блокировки, а затем подвергли реакции с различными пептидами при концентрации 50 нг/мл в течение 2-х часов. Планшеты покрыли различными антителами (Ab) в течение ночи, промыли, блокировали и добавляли клеточные экстракты (с пептидами или без пептидов) в течение еще 2-х часов. После удаления экстрактов планшеты промыли и инкубировали с антителами, конъюгированными αp53- HRP, для выявления уровней р53. И, наконец, провели ТМВ (субстрат HRP) анализ, оптическую плотность определяли при 450 нм (как описано выше). Клетки MCF7 и H1299-MUT-р53 (ts) A135V (Zhang, W., et al., A temperature-sensitive mutant of human p53. Embo J, 1994. 13(11): p. 2535-44) использовали в качестве положительных контролей для конформации р53 дикого типа (соотношение 1620/240 равно или превышает 5:1). Экстракты p53 H1299-R175H, хотя проявляют более значительную конформацию мутантного р53, по-прежнему сохраняют реакционную способность к PAb1620 (соотношение 1620/240 составляет 1:2) PAb1620 или PAb240 по сравнению с PAb421. Тем не менее, при рассмотрении итогов анализа, ссылка делается на расчетное соотношение PAb1620/PAb240, которое лучше отражает степень конформационного изменения. Чтобы проверить, является ли это фоновым связыванием с антителом или фактической конформацией складывания дикого типа, индуцировали повышение уровней денатурации путем нагрева экстрактов в течение различных временных интервалов, контролируя их реакционную способность к PAb1620 и PAb240. Как видно на Фигурах 8A и 8B, повышенная тепловая обработка индуцировала повышение реакционной способности с PAb240 и снижение реакционной способности с PAb1620, что указывает на то, что р53 R175H в этих экстрактах оставался частично в конформации дикого типа при таких экспериментальных условиях. Следует отметить, что после инкубации с некоторыми из тестируемых пептидов была обнаружена повышенная реакционная способность Mut-p53 R175H по отношению к PAb1620 и сниженная реакционная способность к PAb240. Это происходило, например, с пептидами 24, 36, 47, 60, 68 (Таблица 6), что указывает на то, что эти пептиды вызывают конформационное изменение в мутантном белке p53.
Скрининг пептидов по действию на связывание Mut-p53 с p53-RE ДНК.
Для измерения действия тестируемых пептидов на связывание Mut-p53 с ДНК в качестве анализа с высокой пропускной способностью для количественного определения активации р53 использовали коммерческий набор ELISA (R&D Systems DYC1355-5, Lot-1273366FA). В этом наборе используется формат 96-луночного планшета. Набор использовали в соответствии с инструкциями производителя. Лунки покрывали анти-p53 антителом в течение ночи. Клеточные экстракты, содержащие р53, подвергали реакции с биотин-меченным олигонуклеотидом, содержащим консенсусный сайт связывания р53 (входит в комплект). р53 дикого типа, как предполагалось, связывается с этим олиго, а также с антителом, покрывающим лунки. Избыток P53 и олиго смыли в промывочном буфере (0,05% Твин 20 в PBS, рН 7,2 - 7,4; R&D Systems, Catalog#WA126). Затем добавили стрептавидин-HRP (R & D Systems, компонент 890803, входит в комплект) в течение 15-45 мин, чтобы определить количество олиго в лунке, которое пропорционально ДНК, связанной белком р53. Если добавление пептида к экстрактам Mut-p53 увеличивает показания ELISA по сравнению с фоном, этот пептид считается функционально эффективным и может быть отобран для дальнейшего анализа. На Фигуре 9 показан репрезентативный эксперимент: аналогично конформационному анализу ELISA клеточные экстракты инкубировали с биотин-p53-RE либо в присутствии, либо в отсутствие (NT) тестовых пептидов. Как и в случае конформационного скрининга, клетки MCF7 и H1299- Mut-p53 (ts) A135V служат в качестве положительных контролей для WT р53. Экстракты добавили в лунки, покрытые αp53-Ab, и после нескольких этапов промывки добавляли в течение 1 часа стрептавидин-HRP, а затем планшеты промыли снова и выполнили ТМВ (субстрат для HRP) анализ. Как видно на Фигуре 9, экстракты H1299-R175H p53 продемонстрировали некоторое фоновое связывание с р53-RE олиго, которое уменьшилось еще за счет немеченного конкурирующего олиго. Положительные контроли показали 3-4-кратное усиление сигнала по сравнению с фоном. Оказалось, что некоторые пептиды повышают связывание экстрактов H1299-R175H р53 с p53-RE ДНК, например: 68, 75, 83, 93, 97.
Связывание пептидов с р53 дикого типа и мутантным р53.
Для измерения связывания пептидов с Mut-p53 и WT p53 использовали коммерческий набор ELISA от «TAKARA» (MK100 Lot AK401) в качестве анализа с высокой пропускной способностью для количественного определения связывания различных пептидов с белками или антителами. Набор использовали в соответствии с инструкциями производителя. Лунки засевали пептидами путем проведения химической реакции, прикрепляя С-конец пептида к планшету.
Рекомбинантный WT р53 или Mut-р53 R175H при концентрации 10 нг/мл растворили в PBS и блокирующем буфере и затем добавили в покрытые пептидами планшеты в течение 2-х часов. Растворимые пептиды добавляли в соответствующие лунки в качестве конкурентного контроля, показывающего специфичность связывания пептида с p53 (+ comp), а p53-RE ДНК олиго добавляли в другие лунки (+ДНК) с целью исследования его воздействия на связывание пептидов с р53. После удаления рекомбинантного белка планшеты промыли и инкубировали с αp53-HRP-конъюгированным Ab для количественного определения уровней p53. И наконец, провели ТМВ (субстрат HRP) анализ и определили оптическую плотность при 450 нм. На Фигуре 10 показан репрезентативный эксперимент, выполненный с соответствующими пептидами и антителами. Как видно, к лункам присоединены моноклональные антитела αp53, которые служат в качестве внутренних контролей этого анализа; как предполагалось, PAb1801 связывается с обеими формами р53; PAb1620 специфичен к WT р53, а PAb240 более реактивный с мутантной формой. Лунки (заблокированные) не были покрыты пептидами, и контрольной пептидной последовательностью является pep76. Как можно видеть, большинство пептидов, изображенных на фигуре, связываются с более высокой аффинностью с рекомбинантным р53 дикого типа по сравнению с мутантным р53.
Действие pCAP на связывание Mut-p53 с его чувствительными элементами в живых клетках.
Затем провели исследование способности р53 также связываться с хроматином его генов-мишеней. С помощью анализа иммунопреципитации хроматина (ChIP) проверили, могут ли pCAP восстановить способность связывания ДНК Mut-p53 с p53-чувствительными элементами (p53-RE). Карциному молочной железы BT-549, эндогенно экспрессирующую мутантные p53R249S, обрабатывали в течение 5 часов смесью 3-х pCAP; 250, 308 и 325. Клетки, обработанные смесью контрольных пептидов, служили в качестве отрицательного контроля. Затем клетки фиксировали и ДНК расщепили путем обработки ультразвуком. Сшитую с p53 ДНК подвергли иммунопреципитации при использовании поликлонального анти-p53 антитела. ДНК очистили и затем определили количество р53-чувствительных элементов различных генов-мишеней р53 с помощью различных праймеров в реакции qPCR. Результаты были нормализованы до полной исходной ДНК. В качестве отрицательного контроля экстракты подвергли иммунопреципитации с гранулами без антитела (гранулы). Как видно на Фигуре 11, связывание хроматина с контрольными гранулами было на базальном уровне 0,005% от исходной ДНК. Смесь pCAP не увеличила связывание р53 с контрольной последовательностью неспецифической геномной ДНК, но связывание р53 с чувствительными элементами в генах PUMA, р21 и CD95 увеличилось в 2,34, 9,78 и 4,54 раза, соответственно, при помощи pCAP по сравнению с контрольными пептидами.
Скрининг пептидов по действию на транскрипционную активность р53
В качестве дополнительной стратегии скрининга, используемой для идентификации реактивирующих пептидов, стратегия была выполнена in vivo и на основе анализа гена-репортера. Она измеряет транскрипционную активность p53 путем количественного определения активности гена-репортера, находящегося под контролем промотора, содержащего 17 повторов консенсусного сайта связывания р53 (RGC). Люциферазный анализ проводится на живых клетках и, следовательно, отражает действие тестовых пептидов на функцию Mut-p53 в контексте интактных клеток. Был использован промотор на основе RGC, клонированный в обратном направлении от секретируемого люциферазного репортера (TK-RGC-luc) (New England Biolabs (CAT. NO. N0324S)), поскольку он не требует лизиса клеток и позволяет применять 96- луночный формат.
На Фигуре 12 показаны репрезентативные эксперименты по люциферазному анализу, которые были выполнены для оценки способности пептидов возвращать транскрипционную активность мутантному р53. Для скрининга in vivo на основе люциферазы использовали клетки Н1299. Переходная трансфекция этих p53-/- клеток проводилась с векторами, экспрессирующими p53 дикого типа, p53 R175H, p53 R249S или с пустым вектором в качестве контроля (Suad, O., et al., Structural basis of restoring sequence-specific DNA binding and transactivation to mutant p53 by suppressor mutations. J. Mol. Biol., 2009. 385(1): p. 249-65). Клетки также были котрансфицированы с ТК-RGC-luc (CAT. No. NEB, N0324S). Через 24 часа после трансфекции клетки обработали тестовыми пептидами. Через 48 часов после трансфекции взяли образец культуральной среды для измерения биолюминесценции. Как можно видеть на Фигуре 12, в необработанных образцах трансфекция WT р53, (положительный контроль), индуцировала транскрипцию с ТК-RGC-luc в 20-30 раз больше по сравнению с TK-RGC-luc отдельно. При исследовании обработанных образцов пептидов видно, что пептиды не оказали существенного действия на активность р53 дикого типа; это обнадеживающий результат, так как пептиды, значительно увеличивающие активность WT р53, как предполагается, оказывают токсическое воздействие на нормальные клетки. Два из тестируемых пептидов, а именно pCAP-68 и pCAP-75, индуцируют транскрипцию из ТК-RGC-Luc в присутствии R175H р53 и R249S р53.
Скрининг пептидов по действию на жизнеспособность клеток, экспрессирующих мутантный р53
Важным показателем активности реактивирующих пептидов является их действие in-vivo на раковые клетки, которые экспрессируют Mut-p53. В частности, требуются реактивирующие пептиды, которые могут вызвать специфически Mut-p53-зависимую гибель раковых клеток при минимальных токсических действиях на нормальные клетки. Для определения действия различных тестовых пептидов на Mut-p53-зависимую гибель использовали анализ на жизнеспособность на основе кристаллвиолета, в котором кристаллвиолет используется для окрашивания клеток, которые прилипают к планшету и, следовательно, количество красителя пропорционально числу клеток. Анализ с использованием кристаллвиолета простой, быстрый, надежный, недорогой и для него не требуется сложная подготовка образцов.
Клетки высевали в 96-луночные планшеты с точно определенной плотностью, что давало возможность клеткам расти в течение 48 часов, не достигая конфлюэнтности. Пептиды добавили через 6 часов. Различные концентрации этопозида (цитотоксический препарат) использовали в качестве положительного контроля для гибели клеток и в качестве стандартной эталонной кривой для оценки действия тестируемых пептидов. Через 48 часов после обработки клетки промыли с помощью PBS, чтобы исключить мертвые клетки и дебрис, а клетки, которые остались прикрепленными к планшету, окрашивали кристаллвиолетом в течение 30 минут. Кристаллвиолет удалили и клетки промыли PBS 4 раза, чтобы устранить остатки кристаллвиолета. Затем окрашенные клетки растворили в 10% уксусной кислоте и планшеты взяли для измерения оптической плотности при 595 нМ (специфичной для кристаллвиолета).
На Фигурах 13А и 13В иллюстрируется репрезентативный эксперимент скрининга, выполненного на 128 синтезированных пептидах. В этом эксперименте использовали фибробласты WI-38. Эти клетки экспрессируют эндогенный WT p53 и они были дополнительно инфицированы вирусом, экспрессирующим либо кшРНК [shRNA] мыши Noxa, как неспецифический контроль, либо мутант р53 R175H для стабильной сверхэкспрессии мутантного р53. Обе эти сублинии (mNoxa или p53 R175H) высевали по 3000 клеток на лунку и обрабатывали, как описано выше. Показания оптической плотности (595 нм) отражает количество клеток на планшете после обработки, нормализованное до необработанных образцов, которые считаются 100% жизнеспособными. Как видно, хотя клетки WI-38 относительно устойчивы к уничтожению, возрастающие концентрации этопозида служат хорошим положительным контролем для гибели клеток и остановки роста, причем самая высокая концентрация сокращает число клеток на 50% через 48 часов.
Некоторые из испытуемых пептидов действительно вызвали значительное снижение числа клеток; это снижение зависело от мутантного р53, так как оно было гораздо более заметным в клетках, экспрессирующих R175H р53, по сравнению с контрольными клетками mNOXA-i. Эти пептиды включают, например, pCAP-36, pCAP- 46, pCAP-47, pCAP-60, pCAP-97. С другой стороны, было обнаружено, что некоторые пептиды оказывают токсическое действие на обе клеточные сублинии; одним из примеров является pCAP-68. Подобный анализ провели на нескольких различных MUT-p53-экспрессирующих клеточных линиях рака человека, результаты для различных пептидов приведены в Таблице 7.
Пример 11: Гомология основных пептидов с последовательностями известных белков, связывающихся с р53
После выполнения функционального скрининга пептидных мотивов, предсказанных фаговым дисплеем, были идентифицированы 20 пептидов, которые проявили функциональное воздействие на мутантный р53 в различных анализах и клеточных линиях. Далее было исследовано сходство этих пептидов с последовательностями человеческих белков в целом и с белками, известными как взаимодействующие с р53, в частности, поскольку большое сходство с белками, взаимодействующими с р53, может служить свидетельством биологической значимости конкретного мотива и может подтвердить предположение о том, что пептиды, отобранные в искусственных условиях in-vitro, действительно могут взаимодействовать с р53. Кроме того, структура белка и окружающая последовательность могут быть полезны при разработке улучшенных пептидов, которые основаны как на сеоекции, так и на рациональной конструкции. Для установления сходства между пептидными последовательностями и известными человеческими белками был использован алгоритм BLAST (Basic Local Alignment Search Tool). Пептидные мотивы были введены как последовательности запроса против базы данных последовательностей, содержащих последовательности человеческого белка. Программа BLAST находит подпоследовательности в базе данных, которые похожи на подпоследовательности в запросе. Основная идея BLAST состоит в том, что имеются часто встречающиеся пары сегментов с высоким оценочным баллом (HSP), содержащиеся в статистически значимом выравнивании. BLAST выполняет поиск выравниваний последовательности с высоким оценочным баллом между последовательностью запроса и последовательностями в базе данных при использовании эвристического подхода, который аппроксимирует алгоритм Смита-Ватермана. На основании сходств между пептидными мотивами и известными человеческими белками и структурными данными этих белков был создан перечень новых пептидных последовательностей (показано ниже в Таблице 8). В этом перечне аминокислоты, подобные пептидным мотивам, фланкированы другими аминокислотами, полученными из белковой последовательности, любая фланкирующая мотив, или из структурных элементов, физически близко расположенных к гомологичному мотиву, в соответствии с 3-мерными кристаллографическими данными.
Было выявлено более 70 различных белков с различной степенью сходства с отобранными пептидными мотивами. Ранее было доказано, что многие из этих белков физически взаимодействуют с р53, в то время как о других сообщалось, что они вовлечены в сигнальный путь р53, либо в обратном направлении, либо в прямом направлении от р53. Было установлено, что несколько мотивов имеют очень высокую степень гомологии с известными взаимодействующими белками р53; например, pCAP-97 (WNHHHSTPHPAH, SEQ ID NO:10) имеет 100% гомологию с RAD9A (при р-значении 10-8), которое, как было доказано, взаимодействует с и активирует p53; pCAP-60 (SFILFIRRGRLG, SEQ ID NO:302) и pCAP-63 (HNHHHSQHTPQH, SEQ ID NO:226) имеют 90% гомологию с последовательностью белка GAS2 (KILFIRLMHNKH, SEQ ID NO:369), в которой эти мотивы разделены двумя аминокислотами (аминокислоты, подобные пептидным мотивам, выделены жирным шрифтом).
Для скрининга основных пептидов-кандидатов по конформационным и функциональным действиям на Mut-p53 использовали несколько альтернативных и взаимодополняющих спососбов. Для повышенного проникновения пептида через клеточные мембраны каждый пептид содержит 3-6 остатков аргинина либо как часть его последовательности, либо добавленных на N-конце или С-конце. 40 пептидов также были конъюгированы с миристоильной жирной кислотой (myr) для усиления слияния с клеточными мембранами, которые потенциально были способны улучшить доставку в клетки. Сначала для оценки были выполнены анализы in-vitro, как например ELISA для оценки конформации р53 и последовательность-специфического ДНК-связывания белка р53. После этого с помощью анализов на жизнеспособность в живых клетках исследовали активность пептидов, транскрипционную активность р53 на люциферазном гене- репортере, а также исследование генов-мишеней р53 in-vivo. Комбинация этих анализов (все выполнены в 96-луночном формате) позволила идентифицировать и проверить действие пептидов на различную активность p53 и их способность придавать такую способность белкам Mut-p53. Как видно из Таблицы 8, было установлено, что 12 пептидов имеют общий балл активности выше 30; все эти 12 пептидов показали свою эффективность во многих различных анализах, включая конформацию p53 и последовательность-специфическое ДНК-связывание, снижение жизнеспособности экспрессирующих Mut-p53 клеток и активацию генов-мишеней р53. Некоторые из этих основных пептидов, которые имеют кóровый мотив, полученный из фагового дисплея с добавленными последовательностями известных белков (pCAP 201-326), показали значительно более сильное действие по сравнению с пептидами, полученными только из одного фагового дисплея (pCAP 1-180), в то время как другие были сравнимы с pCAP 1-180.
После тщательного изучения пептидных последовательностей, которые показали наиболее значительное действие в комбинации анализов, было установлено, что основные пептиды могут быть классифицированы по нескольким основным группам в соответствии с их консенсусными мотивами. Консенсусные мотивы состоят, по меньшей мере, из 3-х последовательных аминокислот, которые гипотетически образуют последовательный или конформационный сайт связывания для мутантов р53. Было установлено, что эти консенсусные мотивы представляют собой HSTPHP, FPGHTIH, IRGRIIR, LPNPPER, SFILFIR, HANLHHT, YPTQGHL, WNHHHSTPHP, TLYLPHWHRH, YRRLLIGMMW, IRILMFLIGCG, SFILFIRRGRLG, LRCLLLLIGRVG, SWQALALYAAGW, IRILMFLIGCGR, glrgrriflifs, HSSHHHPVHSWN, LRCLLLLIGRVGRKKRRQ (SEQ ID NOs:314, 268, 282, 340, 376, 298, 377, 378, 253, 20, 379, 302, 275, 380, 273, 381, 280 и 382, соответственно).
Действие тестовых пептидов на гены-мишени р53
Белок р53 дикого типа работает в основном как фактор транскрипции. При активации различными формами стресса он накапливается, связывается со своими чувствительными элементами во многих генах-мишенях и трансактивирует их транскрипцию. Белки, которые являются продуктами этих генов-мишеней, выполняют свои функции; трансактивация р21, например, приводит к остановке роста, в то время как трансактивация PUMA привела бы к апоптозу. Поэтому одним из наиболее важных показателей функциональной активации р53 является индукция его различных генов-мишеней. Проверка действия различных тестовых пептидов на гены-мишени р53, следовательно, проводилась in-vivo.
Для функционального скрининга in vivo использовали несколько экспериментальных систем. Одна система основана на клетках H1299, которые являются р53-нулевыми и широко используются для исследования р53. Были использованы клетки Н1299, стабильно трансфицированные Mut-p53 (ts) A135V. Эта форма р53 является чувствительным к температуре мутантом, который имеет мутантную конформацию при 37°C и конформацию дикого типа при 32°C. На Фигуре 14 показан репрезентативный эксперимент. По существу, клетки высевали в 12-луночные чашки, указанные пептиды добавляли непосредственно в среду при концентрации 5 μг/мл, и затем клетки либо перемещали на 32°C, либо возвращали на 37°C. Через 18 часов клетки собирали, а затем извлекали РНК, проводили синтез кДНК и ПЦР-анализ в режиме реального времени. Был исследован уровень экспрессии 3-х представителей генов-мишеней р53: p21, PUMA и Mdm2. Экспрессия генов в H1299-ts при 37°C рассматривается как фоновый уровень и все результаты нормализуются к нему, а также к гену «домашнего хозяйства» GAPDH. Экспрессия генов в H1299-ts при 32°C представляет конформацию WT р53 и, следовательно, служит в качестве положительного контроля. Как можно видеть, сдвиг температуры к 32°C значительно увеличил экспрессию всех 3-х генов-мишеней.
Как видно на Фигуре 14, отрицательный контрольный пептид pCAP-76 не вызвал индукцию мишеней р53. Несколько тестируемых пептидов действительно вызвали значительное увеличение экспрессии p21, PUMA и Mdm2. Это имело место для pCAP-130, pCAP-135, pCAP-142, pCAP-144 и pCAP-148. Эти пептиды индуцировали транскрипцию генов-мишеней 2-4-кратно по сравнению с 9-11-кратным со стороны положительного контрольного, аутентичного р53 дикого типа. Тот факт, что обработка пептидами индуцировала все три гена, но не оказала никакого действия на экспрессию этих генов в контрольной клетке H1299 (р53-/-), говорит о том, что эта индукция зависит от р53.
Поскольку доставка пептидов является основным препятствием для их использования в качестве терапевтических агентов, для преодоления этого препятствия были предприняты различные подходы. Во-первых, на основании тестируемых основных последовательностей были изучены и синтезированы короткие мотивы пептидной последовательности (до 6 аминокислот), поскольку эти малые пептиды могли пересекать клеточные мембраны путем диффузии. Второй подход касался синтеза тестируемых пептидов с полиаргининовым C-концевым фрагментом («хвостом») для облегчения их активного поглощения основанными на эндоцитозе механизмами.
Добавление полиаргининового хвоста к пептидам резко увеличивает растворимость пептидов с высоким содержанием гидрофобных аминокислот. В некоторых случаях это также значительно увеличивает активность пептидов как in-vitro, так и in-vivo; pCAP-25, например, был не растворим в ДМСО при концентрации 10 мг/мл и не показал никакого действия на активность р53 ни при испытании на изменение конформации, ни на жизнеспособность. В то время как pCAP-68, который имеет такую же аминокислотную последовательность, при добавлении хвоста 9R вызвал значительный сдвиг в конформации Mut-p53 по отношению к PAb1620, а также массовую гибель клеток. Основные пептиды дополнительно подвергли тщательной проверке действий на жизнеспособность клеток Mut-p53- специфическим образом.
Были проведены эксперименты с использованием различных линий раковых клеток, эндогенно экспрессирующих различные изоформы мутанта р53. Фигуры 15А и 15В иллюстрируют два репрезентативных эксперимента, проведенных на клетках рака молочной железы MDA-MB-231 (Фигура 15А) и SKBR3 (Фигура 15В), экспрессирующих Mut-p53 с мутациями в позициях 280 или 175, соответственно, в пределах ДНК-связывающего домена (DBD). Для изучения специфичности пептидов к Мut-p53 в качестве используемого контроля выступали такие клетки с нокдауном Mut-p53 (shp53). Как видно на Фигурах 15А и 15В, многие из тестируемых пептидов показали снижение жизнеспособности клеток Mut-p53-специфическим образом, со значительными показаниями 30%-80% по отношению к 100% жизнеспособности, которую показали необработанные Mut-p53-экспрессирующие клетки. Некоторые пептиды показывают некоторую степень токсического действия на жизнеспособность клеток в целом, как это видно в клетках shp53. Например, pCAP-155 продемонстрировали сокращение на 30% - 40% жизнеспособности в двух сублиниях инфицированных клеток shp53. Кроме того, также видно, что некоторые пептиды показывают специфическое уменьшение количества клеток в конкретных типах клеток по сравнению с минимальной активностью в других. pCAP-146, например, вызвали значительное уменьшение клеток MDA-MB-231 shCon, но не показали почти никакого специфического действия на клетки SKBR3 shCon.
Тестируемые пептиды дополнительно испытали на их действие на экспрессию гена-мишени p53 в клетках, экспрессирующих эндогенный R175H p53. Результаты показаны на Фигуре 16, где представлена гистограмма репрезентативного эксперимента, выполненного на клетках SKBR3 ShCon и клетках SKBR3 Shp53, инактивированных для экспрессии p53. По существу, эти клетки высевали в 12-луночные чашки; указанные пептиды добавляли непосредственно в среду при концентрации 5 μг/мл. Через 18 часов клетки собрали, затем провели анализ qRT-PCR. Оценили уровень экспрессии p21, PUMA и Mdm2. Экспрессия этих генов в необработанных клетках считается фоновой и все результаты были нормализованы к ней, а также к GAPDH. Как видно, некоторые из основных пептидов продемонстрировали значительную трансактивацию генов-мишеней р53. Это действие было опосредовано через Mut-p53, так как он не наблюдался в клетках SKBR3 shp53. pCAP-155, pCAP-144 и pCAP-148 показали одни из самых высоких уровней трансактивации.
Действие тестовых пептидов на апоптоз и корреляцию активации генов-мишеней р53
Фигуры 17А и 17В иллюстрируют репрезентативные эксперименты, проведенные на клетках карциномы яичников ES2 (Фигура 17A-D), экспрессирующих Mut-p53 с мутацией в позициях 241 в пределах DBD. Вкратце, клетки поместили в 6-см чашки и указанные пептиды добавляли непосредственно в среду при концентрации 12 μг/мл в указанные моменты времени. Клетки собрали и 60% клеток взяли для аннексин-PI анализа на апоптоз, а 40% для экстракции РНК, синтеза кДНК и ПЦР-анализа в режиме реального времени. Апоптоз оценивали с использованием комплекта для аннексин-V окрашивания (Roche, REF 11 988 549 001). Нефиксированные клетки окрашивали как с помощью анти-аннексин FITC конъюгированных антител для обнаружения апоптотических клеток, так и с помощью PI (пропидиум-йодид) для окрашивания мертвых клеток, проницаемых для соединения, в соответствии с инструкциями изготовителя. Окрашенные клетки затем проанализировали с помощью проточной цитометрии. Всего для каждого образца насчитывалось 10000 клеток. Их разделили на четыре субпопуляции по интенсивности окрашивания: клетки, отрицательные и по PI, и по аннексину (-PI, -аннексин), называются живыми; клетки, отрицательные по PI и положительные по аннексину (-PI +аннексин), проходят ранние стадии апоптоза; клетки, положительные по PI и аннексину (+ PI, + аннексин), являются мертвыми клетками, которые прошли процесс апоптоза; и клетки, положительные по PI и отрицательные по аннексину (+ PI, -аннексин) считаются мертвыми клетками, которые умерли не апоптотической смертью, как например некроз. Как видно на Фигуре 17А, 17В, необработанные клетки (время 0 час) в основном (на 94%) отрицательны и по PI, и по аннексину, а это означает, что клетки жизнеспособные и здоровые. Обработка с помощью pCAP 242 и 250 приводит к быстрому увеличению апоптотических клеток с последующей гибелью клеток, а через 5 часов после обработки 12% клеток являются положительными по аннексину и примерно 7% погибает. Через 16 и 24 часа после обработки с помощью pCAP 250 апоптотическая популяция увеличивается до примерно 27%, а мертвых клеток накапливается до 29% через 16 часов и до 36% через 24 часа. Эта тенденция также справедлива и для pCAP 242, хотя его действие ослабленное и более замедленное. Действие пептидов на жизнеспособность клеток сопровождается существенной трансактивацией генов-мишеней р53, как видно на Фигурах 17С и 17D, где демонстрируется экспрессия 4-х репрезентативных мишеней. Как видно, все гены активируются после обработки пептидами, и экспрессия мРНК p21 и PUMA увеличивается с течением времени до 10 раз и до 6 раз после обработки с помощью pCAP 250 и pCAP 242, соответственно. Экспрессия CD95 и Btg-2 повышается до 6 раз по сравнению с необработанными клетками.
Пример 12: Испытания in-vivo (доклинические) реактивирующих Мut-p53 пептидов
Эксперименты in-vivo (доклинические) проводились на моделях двух типов: модели человеческих ксенотрансплантатов у голых мышей и Mut-p53 «нокин» мышей. В каждой модели определяется действие внутриопухолевой инъекции тестируемых пептидов на рост опухоли и выживаемость животных.
В доклинической модели ксенотрансплантата опухолевые клетки трансфицированы люциферазным вектором экспрессии, позволяющим осуществлять мониторинг опухоли путем прямого изображения.
В модели Mut-p53 «нокин» мыши используется условная «нокин» мышь с специфическим к легким Mut-p53 (Kim, C.F., et al., Mouse models of human non-small-cell lung cancer: raising the bar. Cold Spring Harb. Symp. Quant. Biol., 2005. 70: p. 241-50. Olive, K.P., et al., Mutant p53 gain of function in two mouse models of Li-Fraumeni syndrome. Cell, 2004. 119(6): p. 847-60). Эта модель дает сложных условных «нокин» мышей с мутациями в K-ras в сочетании с одним из трех аллелей p53: R273H, R175H, или p53-нулевой аллель. Инфицирование с помощью AdenoCre вызывает рекомбинацию условных аллелей и, как было показано, продуцирует K-ras-индуцированные аденокарциномы легкого уже через 6 недель после зарождения опухоли. В этой модели подробно обобщаются некоторые аспекты продвинутой легочной аденокарциномы человека, и она обеспечивает экспрессию двух различных мутантов (175 и 273) от промотора эндогенного р53, на физиологических уровнях, с правильным пространственным и временным профилем. Эта модель дает возможность продемонстрировать характеристики тестируемых реактивирующих пептидов, in vivo, по отношению к нескольким критическим; безопасное пренебрежимо малое действие на нормальную ткань мыши или неинфицированных мышей; эффективное уменьшение размера и числа опухолей у обработанных мышей по сравнению с контрольными; и специфичность к уменьшению опухоли у экспрессирующих Mut-p53 мышей по сравнению с нокаутными р53-мышами. Кроме того, чтобы оценить минимальные активные концентрации и максимальную переносимую дозу, выполняются эксперименты с повышением дозы с положительными контрольными пептидами.
Доклинические испытания на модели ксенотрансплантата
Клетки MDA-MB-231, эндогенно экспрессирующие p53 R280K, были инфицированы люциферазным вектором экспрессии, в качестве неспецифического контроля выступал либо shp53 для нокдауна p53, либо кшРНК мыши NOXA (shmNOXA). Клетки MDA-MB-231 являются высокоонкогенными, образуя агрессивные, быстро растущие опухоли, а также метастатическими в организме человека. В общей сложности инфицировали 10 мышей. Каждой мыши подкожно вводили в правый бок 2*106 клеток MDA-MB-231, экспрессирующих shp53, и 2*106 клеток MDA-MB-231, экспрессирующих shmNOXA, с левой стороны. Опухоли оставляли расти в течение 14 дней до достижения видимого размера. Рост контролировали прямым изображением с помощью системы IVIS200. В этой системе люциферазная биолюминесценция пропорциональна числу раковых клеток. Результаты представлены на Фигурах 18А-18С и на Фигурах 19А-19С. Через 14 дней после инъекции клеток 4 мыши (мыши 7-10) были отнесены к контрольной группе (Фигуры 18А-18С), а 6 мышей (мыши 1-6) были отнесены к группе обработки (Фигуры 19А-19С). Контрольная обработка состояла из смеси 3-х контрольных пептидов (pCAP 76, 77 и 12), которые не показали никакого действия (фенотип) на p53 in vitro. Мышам из группы обработки ввели смесь из 3 пептидов (pCAP 174, 155 и 159), которые показали самое лучшее фенотипическое действие in vitro на р53. pCAP-159 (SEQ ID NO: 312) имеет сходную последовательность с pCAP-60 (SEQ ID NO:302) с добавлением остатков аргинина; пептид состоит из D-аминокислот и синтезируется в обратном порядке (pCAP -159: rrrrrrrrglrgrriflifs (SEQ ID NO: 312)) по сравнению с pCAP-60: SFILFIRRGRLG, (SEQ ID NO: 302) (строчные буквы обозначают D-аминокислоты) по «ретро-инверсо» стратегии. Пептиды вводили непосредственно в опухоль (внутриопухолевая инъекция) три раза в неделю в объеме 40 μл на каждую опухоль и в концентрации 50 μг/мл для каждого пептида в смеси. Поэтому в общей сложности каждой мыши каждый раз вводили 6 μг смеси либо контрольных пептидов, либо пептидов для обработки. Мышей наблюдали в течение в общей сложности 5 недель от начала пептидной обработки. Биолюминесценцию измеряли раз в 7 дней. Как видно на Фигуре 18А, опухоли shmNOXA, экспрессирующие эндогенный Mut-p53, показали 6-15-кратное (логарифмическая шкала) увеличение интенсивности люциферазы в течение периода действия эксперимента при обработке смесью контрольных пептидов. Мышь 10 пришлось умертвить после 28 дней обработки, так как опухоли достигли предельно большого размера. На Фигуре 19А показан анализ мышей, обработанных параллельно смесью из 3-х Mut-p53-активирующих пептидов. Как видно на Фигуре 19А, ни одна из опухолей не показала значительного увеличения числа раковых клеток в течение 35-дневного периода эксперимента. Две из опухолей (мышь-1 и мышь-4) показали частичный ответ на обработку, выраженный в виде уменьшения на 50% - 65%, соответственно, биолюминесценции. Мыши номер 2 и 5 показали полный ответ, с люциферазными показаниями, которые были такими же или близкими к пороговому уровню обнаружения фона системы IVIS (5*106 фотонов) даже после 21 дня обработки. Введение пептидов было прекращено через 35 дней, и мышей номер 2 и 5 оставили без какой-либо дальнейшей обработки и наблюдали в течение еще 21 дней. Никакого повторного возникновения опухоли не было обнаружено у этих мышей ни визуально, ни с помощью прямого изображения.
Доклиническое испытание #2
Клетки MDA-MB-231, эндогенно экспрессирующие p53 R280K, инфицировали люциферазным вектором экспрессии и 15 мышам ввели подкожно 1×106 клеток MDA-MB-231-Luc на обоих бедрах. Опухоли оставили расти в течение 10 дней до достижения видимого размера и далее с этого момента времени рост опухоли контролировали с помощью живого изображения. Результаты представлены на Фигурах 20А-20С. Через 18 дней после инъекции клеток 6 мышей были отнесены к контрольной группе и 9 мышей были отнесены к группе обработки. Как и раньше, контрольная обработка включала смесь из 3-х контрольных пептидов (pCAP 76, 77 и 12). Мышам из группы обработки вводили смесь из 3-х пептидов (pCAP 174, 155 и 159). Пептиды вводили непосредственно в опухоль (внутриопухолевая инъекция) три раза в неделю в объеме 40 μл на каждую опухоль и в концентрации 50 μг/мл для каждого пептида в смеси. Как показано на Фигуре 20, перед обработкой и в контрольной группе, и в группе обработки наблюдалось одинаковое состояние; примерно 2-3-кратное (логарифмическая шкала) увеличение интенсивности люциферазы (день 10-18). На Фигуре 20А представлен анализ мышей, обработанных параллельно смесью из 3-х контрольных пептидов: как видно, контрольная обработка оказывает лишь очень сьабое действие на рост опухоли, снижая темп роста по сравнению с периодом до обработки. Тем не менее, как видно на Фигуре 20В, обработка смесью из трех р53- реактивирующих pCAP вызвала значительное уменьшение люминесценции опухолей MDA-MB-231. После однократной инъекции pCAP смеси средняя люминесценция была снижена на 70% и 7 из 18 опухолей показали полную регрессию при показаниях живых изображений, близких к порогу обнаружения фона (данные не показаны). Как показано на Фигуре 20В, через 12 дней после начала обработки (4 инъекции) средняя люминесценция опухоли уменьшилась на 93%, и 11 из 18 опухолей показали полный ответ. Только одна из 18 опухолей либо не показала никакого ответа, либо показала слабый ответ. Эта опухоль была относительно большой до начала обработки, поэтому возможно, что доза pCAP была недостаточной, чтобы вызвать существенный ответ.
Доклинические испытания # 3 - клетки карциномы толстой кишки SW-480
После наблюдения весьма значительного результата в эксперименте с MDA-MB-231 дополнительные исследования были направлены на расширение наблюдения и исследование клеток различного происхождения, содержащие другую точечную мутацию p53. Линия клеток карциномы толстой кишки SW-480 содержит две эндогенные мутации р53: R273H и P309S. Клетки SW480 были стабильно инфицированы люциферазным геном-репортером и 106 клеток подкожно ввели голым мышам. Эксперимент охватывал 15 мышей, которых наугад разделили в ходе эксперимента на 3 группы: контрольная группа, получившая коктейль из 3-х pCAP, которые ранее показали себя как неэффективные; группа, получившая коктейль из 3-х эффективных pCAP (250, 308, 325), и, наконец, группа, получившая один пептид, pCAP-325. Эксперимент с SW-480 продолжался 42 дня с момента имплантации клеток. Временная линия относится к первому дню обработки, который отмечается как день 0. На Фигуре 21 показан рост опухоли с течением времени во всех трех группах согласно измерению с помощью живого изображения в IVIS. Как видно, с течением времени контрольные опухоли показывают среднее увеличение размера опухоли в 2,75 раза (как вытекает из изменения логарифма значения интенсивности люминесценции от 9,24 в день 0 до 9,68 в день 35, представленные на Фигуре 21А). Опухоли в группе обработки смесью показывают уменьшение, эквивалентное 96,7% уменьшению размера опухоли (как выведено из изменения логарифма значения интенсивности люминесценции от 9,13 в день 0 до 7,65 в день 35, представленные на Фигуре 21В). Аналогично этому, опухоли в pCAP 325- группе показали среднее кратное изменение 0,043, которое эквивалентно 95,6% уменьшению размера опухоли (как выведено из изменения логарифма значения интенсивности люминесценции от 8,97 в день 0 до 7,61 в день 35, представленное на Фигуре 21С).
Резюме доклинических экспериментов
К настоящему времени уже проведено 4 доклинических эксперимента с использованием модели ксенотрансплантата экспрессирующих Mut-p53 клеток, трансфицированных люциферазным вектором экспрессии, что позволяет осуществлять мониторинг опухоли путем живого изображения. Два эксперимента были проведены с тройными негативными клетками рака молочной железы MDA-MB-231 (p53 R280K), в одном эксперименте использовались клетки карциномы рака толстой кишки SW-480 (p53 R273H) и еще в одном эксперименте использовались клетки рака молочной железы SKBR3 (p53 R175H). В каждом эксперименте клетки из соответствующей линии клеток вводили подкожно и оставляли для образования хорошо сформировавшихся опухолей, видных как невооруженным глазом, так и с помощью живого изображения (обычно 2-3 недели). Затем была введена схема обработки, состоявшая из внутриопухолевой инъекции либо эффективных основных пептидов, либо контрольных пептидов (не проявляющих активности in-vitro) раз в три дня в течение периода вплоть до 42 дней.
Во всех доклинических экспериментах мыши, обработанные основными пептидами, показали очень значительное уменьшение всех параметров опухолей (в разных экспериментах проценты отличаются); средний сигнал люминесценции (81% -99% согласно измерению с помощью IVIS), масса опухоли и объем (72% -93%, измеренные после извлечения опухоли). Опухоли у мышей, получавших контрольные пептиды с другой стороны, продолжали расти, хотя и с меньшей скоростью по сравнению с темпом роста до обработки. Почти все опухоли, обработанные основными пептидами, отреагировали на обработку, а 35% -70% обработанных опухолей показали полный ответ при регрессии опухолей ниже уровней порога обнаружения. Шесть из мышей, показывающих полный ответ, оставались живыми в течение двух месяцев после завершения эксперимента (без обработки) и рецидива опухолей не было обнаружено.
Проверка токсичности пептидов in-vivo
В общей сложности для проверки токсичности пептидной смеси использовали 6 мышей: по две мыши на каждую пептидную концентрацию. Используемая в этом эксперименте пептидная смесь была такой же, как описано выше (Фигуры 19А-19С) (pCAP 174, 155 и 159). Мышам в течение трех недель вводили внутрибрюшинно три раза в неделю пептидную смесь, приготовленную в концентрации 100 μг/мл. Две мыши получили объем 40 μл, совпадающий с общим количеством, которое получили мыши в доклиническом испытании. Две мыши получили 120 μл, а оставшиеся две мыши 400 μл. Учитывая, что средний вес мыши составляет 20 г, эти количества представляют концентрации 0,6, 1,8 и 6 мг/кг, соответственно. После инъекции мышей проверяли каждый день. Никаких видимых изменений не было обнаружено ни у одной из мышей. Кроме того, ткани, окружающие опухоли мышей, использованных в доклиническом эксперименте (Фигуры 18А - 18С и Фигуры 19А-19С), были исследованы после умерщвления мышей на признаки некроза или воспаления. Однако ткани, окружающие опухоль, оказались нормальными во всех случаях, что указывает на отсутствие значительного токсического действия обработки пептидами pCAP.
В Таблице 10 суммируется активность пептидов, тестируемых в настоящем изобретении.
Таблица 1.
Таблица 2. Отбор по p53 R175H.
Таблица 3. Чередующийся отбор по Mut-p53 и p53 дикого типа.
Таблица 4
Таблица 5
Таблица 6
Таблица 7.
*AS - балл оценки активности.
Таблица 8
*AS - Балл оценки активности.
Таблица 9
p53R280H
Таблица 10
Предшествующее описание конкретных вариантов осуществления настолько полно раскрывает общий характер изобретения, что другие специалисты могут, применяя современные знания, легко модифицировать и/или адаптировать к различным применениям такие конкретные варианты осуществления без чрезмерного экспериментирования и без отступления от общей концепции, и следовательно, такие адаптации и модификации должны и предназначены быть понятными в рамках смысла и диапазона эквивалентов раскрытых вариантов осуществления. Следует понимать, что фразеология или терминология, используемая в настоящем документе, применяется для целей описания, а не ограничения. Средства, материалы и этапы для осуществления различных раскрытых функций, могут принимать различные альтернативные формы, не выходящие за рамки изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ЙЕДА РЕСЕАРЧ ЭНД ДЕВЕЛОПМЕНТ КО. ЛТД.
<120> ПЕПТИДЫ, СПОСОБНЫЕ РЕАКТИВИРОВАТЬ МУТАНТЫ Р53
<130> YEDA0137PCT
<150> 61/862977
<151> 2013-08-07
<160> 382
<170> PatentIn версия 3.5
<210> 1
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (12)..(12)
<223> X = 0-10 остатков Arg
<400> 1
Ile Arg Ile Leu Met Phe Leu Ile Gly Cys Gly Xaa
1 5 10
<210> 2
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (19)..(19)
<223> X = 0-10 остатков Arg
<400> 2
Leu Arg Cys Leu Leu Leu Leu Ile Gly Arg Val Gly Arg Lys Lys Arg
1 5 10 15
Arg Gln Xaa
<210> 3
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (8)..(8)
<223> X = 0-10 остатков Arg
<400> 3
Tyr Pro Thr Gln Gly His Leu Xaa
1 5
<210> 4
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (13)..(13)
<223> X = 0-10 остатков Arg
<400> 4
Ser Trp Gln Ala Leu Ala Leu Tyr Ala Ala Gly Trp Xaa
1 5 10
<210> 5
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (11)..(11)
<223> X = 0-10 остатков Arg
<400> 5
Thr Leu Tyr Leu Pro His Trp His Arg His Xaa
1 5 10
<210> 6
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (7)..(7)
<223> X = 0-10 остатков Arg
<400> 6
Ile Arg Gly Arg Ile Ile Xaa
1 5
<210> 7
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (13)..(13)
<223> X = 0-10 остатков Arg
<400> 7
Ser Phe Ile Leu Phe Ile Arg Arg Gly Arg Leu Gly Xaa
1 5 10
<210> 8
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (13)..(13)
<223> X = 0-10 остатков Arg
<400> 8
His Ser Ser His His His Pro Val His Ser Trp Asn Xaa
1 5 10
<210> 9
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (8)..(8)
<223> X = 0-10 остатков Arg
<400> 9
His Ala Asn Leu His His Thr Xaa
1 5
<210> 10
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (11)..(11)
<223> X = 0-10 остатков Arg
<400> 10
Trp Asn His His His Ser Thr Pro His Pro Xaa
1 5 10
<210> 11
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (7)..(7)
<223> X = 0-10 остатков Arg
<400> 11
His Ser Thr Pro His Pro Xaa
1 5
<210> 12
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (5)..(5)
<223> X = 0-10 остатков Arg
<400> 12
Ser Ile Leu Thr Xaa
1 5
<210> 13
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (11)..(11)
<223> X = 0-10 остатков Arg
<400> 13
Tyr Arg Arg Leu Leu Ile Gly Met Met Trp Xaa
1 5 10
<210> 14
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (11)..(11)
<223> X = 0-10 остатков Arg
<400> 14
Tyr Arg Arg Leu Leu Ile Gly Met Met Trp Xaa
1 5 10
<210> 15
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (8)..(8)
<223> X = 0-10 остатков Arg
<400> 15
Phe Pro Gly His Thr Ile His Xaa
1 5
<210> 16
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Остатки Arg
<222> (1)..(1)
<223> X = 0-10 остатков Arg
<220>
<221> Пептид, образованный D-аминокислотами
<222> (1)..(13)
<400> 16
Xaa Gly Leu Arg Gly Arg Arg Ile Phe Leu Ile Phe Ser
1 5 10
<210> 17
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 17
Leu Thr Phe Glu His Tyr Trp Ala Gln Leu Thr Ser
1 5 10
<210> 18
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 18
Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly Gly
1 5 10
<210> 19
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 19
Asn Pro Asn Thr Tyr Val Pro His Trp Met Arg Gln
1 5 10
<210> 20
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 20
Tyr Arg Arg Leu Leu Ile Gly Met Met Trp
1 5 10
<210> 21
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 21
Asp Glu Phe His Ser Phe Tyr Thr Ala Arg Gln Thr Gly
1 5 10
<210> 22
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 22
Lys Pro Asp Ser Pro Arg Val
1 5
<210> 23
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 23
Pro Pro Tyr Ser Gln Phe Leu Gln Trp Tyr Leu Ser
1 5 10
<210> 24
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 24
Ser Glu Phe Pro Arg Ser Trp Asp Met Glu Thr Asn
1 5 10
<210> 25
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 25
His Asp Thr His Asn Ala His Val Gly
1 5
<210> 26
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 26
Trp Ser Glu Tyr Asp Ile Pro Thr Pro Gln Ile Pro Pro
1 5 10
<210> 27
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 27
Ser Ile Leu Thr Leu Ser Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
<210> 28
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 28
Ser Cys Arg Cys Arg Leu Arg Gly Asp Arg Gly Asp Arg
1 5 10
<210> 29
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 29
Gly Gly Gly Gly Gly Gly Gly Gly Gly Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
<210> 30
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 30
Ser Glu Tyr Leu Cys Ser Ser Leu Asp Ala Ala Gly
1 5 10
<210> 31
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 31
Gly Glu Ser Phe Val Gln His Val Phe Arg Gln Asn
1 5 10
<210> 32
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 32
Ser Val His His His His Arg Met His Leu Val Ala
1 5 10
<210> 33
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 33
Gly Arg Arg Arg Phe Cys Met
1 5
<210> 34
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 34
Lys Leu Thr Ile His His His
1 5
<210> 35
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 35
Phe Gly Ser His His Glu Leu
1 5
<210> 36
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 36
Gly Thr Val Asp His His Ala
1 5
<210> 37
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 37
Asp Arg Leu Ser Val Phe Leu Phe Ile Met
1 5 10
<210> 38
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 38
Ala Ile Ser His His Thr Arg
1 5
<210> 39
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 39
Lys His His Pro Phe Asp His Arg Leu Gly Asn Gln
1 5 10
<210> 40
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 40
His Ser Ala His His Thr Met
1 5
<210> 41
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 41
Glu Leu Gly Leu His Arg His
1 5
<210> 42
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 42
Arg Arg Leu Arg Ile Cys Val
1 5
<210> 43
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 43
Val Pro His Ile His Glu Phe Thr Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
<210> 44
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 44
Pro Leu Thr Leu Ile
1 5
<210> 45
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 45
Ser Leu Leu Ile Gly
1 5
<210> 46
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 46
Lys Pro Pro Glu Arg
1 5
<210> 47
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 47
Cys Arg Ile Ile Arg
1 5
<210> 48
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 48
Ser Phe Ile Leu Ile
1 5
<210> 49
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 49
Pro His His His Ser
1 5
<210> 50
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 50
Glu Phe His Ser
1
<210> 51
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 51
Arg Leu Arg Arg Leu
1 5
<210> 52
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 52
Asp Ser Pro Arg
1
<210> 53
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 53
His Pro Trp Thr His
1 5
<210> 54
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 54
His Phe Ser His His
1 5
<210> 55
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 55
Arg Arg Val Ile
1
<210> 56
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 56
Ile Leu Val Ile
1
<210> 57
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 57
Arg Arg Ser Arg Ser Asn Glu Asp Val Glu Asp Lys Thr Glu Asp Glu
1 5 10 15
<210> 58
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 58
Arg Arg Ile Arg Ser Gly Gly Lys Asp His Ala Trp Thr Pro Leu His
1 5 10 15
Glu Asn His
<210> 59
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 59
His Thr Pro His Pro Pro Val Ala Arg Thr Ser Pro Leu Gln Thr Pro
1 5 10 15
Arg Arg
<210> 60
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 60
Pro Asp Ser Glu Pro Pro Arg Met Glu Leu Arg Arg Arg
1 5 10
<210> 61
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 61
Arg Arg Asp Thr Phe Asp Ile Arg Ile Leu Met Ala Phe
1 5 10
<210> 62
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 62
Arg Arg Glu Val Thr Glu Leu His His Thr His Glu Asp Arg Arg
1 5 10 15
<210> 63
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 63
Ser Pro Trp Thr His Glu Arg Arg Cys Arg Gln Arg
1 5 10
<210> 64
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 64
Arg Ser Arg Ser Ser His Leu Arg Asp His Glu Arg Thr His Thr
1 5 10 15
<210> 65
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 65
Arg Arg Arg Ser Thr Asn Thr Phe Leu Gly Glu Asp Phe Asp Gln
1 5 10 15
<210> 66
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 66
Leu Ile Gly Leu Ser Thr Ser Pro Arg Pro Arg Ile Ile Arg
1 5 10
<210> 67
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 67
Glu Ile Tyr Gly Glu Ser Gly Lys Thr Asp Glu His Ala Leu Asp Thr
1 5 10 15
Glu Tyr Arg Arg
20
<210> 68
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 68
Arg Arg Val Ile Leu Arg Ser Tyr Asp Gly Gly His Ser Thr Pro His
1 5 10 15
Pro Asp
<210> 69
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 69
Thr Gly Lys Thr Phe Val Lys Arg His Leu Thr Glu Phe Glu Lys Lys
1 5 10 15
Tyr Arg
<210> 70
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 70
Asn His Phe Asp Tyr Asp Thr Ile Glu Leu Asp Thr Ala Gly Glu Tyr
1 5 10 15
Ser Arg Arg Arg
20
<210> 71
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 71
Asp Pro Glu Pro Pro Arg Tyr Leu Pro Pro Pro Pro Glu Arg Arg
1 5 10 15
<210> 72
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 72
Arg Arg Thr Phe Ile Arg His Arg Ile Asp Ser Thr Glu Val Ile Tyr
1 5 10 15
Gln Asp Glu Asp
20
<210> 73
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 73
Glu Ser Lys Thr Gly His Lys Ser Glu Glu Gln Arg Leu Arg Arg Tyr
1 5 10 15
Arg
<210> 74
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 74
Tyr Asp Asp Glu His Asn His His Pro His His Ser Thr His Arg Arg
1 5 10 15
Arg
<210> 75
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 75
Arg Arg Arg Arg Glu Val His Thr Ile His Gln His Gly Ile Val His
1 5 10 15
Ser Asp
<210> 76
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 76
Asp Glu Pro Leu Pro Pro Pro Glu Arg Arg Arg
1 5 10
<210> 77
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 77
Ser Pro His Pro Pro Tyr
1 5
<210> 78
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 78
Ser Pro His Pro Pro Tyr Ser Pro His Pro Pro Tyr Ser Pro His Pro
1 5 10 15
Pro Tyr Pro
<210> 79
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 79
Arg Arg Pro His Asn Leu His His Asp
1 5
<210> 80
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 80
Leu Arg Asp Pro His Pro Pro Glu Arg Arg Ile Arg
1 5 10
<210> 81
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 81
Arg Arg Pro Ala Asp Gln Ile Ser Tyr Leu His Pro Pro Glu Arg
1 5 10 15
<210> 82
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 82
Asp Leu Gln Tyr Asp Phe Pro Arg Ile Arg Arg
1 5 10
<210> 83
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 83
Tyr Asp Glu Leu Tyr Gln Lys Glu Asp Pro His Arg Arg Arg
1 5 10
<210> 84
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 84
Phe Lys Pro Glu Arg Phe Pro Gln Asn Asp Arg Arg Arg
1 5 10
<210> 85
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 85
Arg Pro Ala Asp Arg Ile Arg Arg
1 5
<210> 86
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 86
His Asp Phe Asp Pro Arg Tyr Arg Asp Arg Arg
1 5 10
<210> 87
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 87
Arg Ile Arg Arg Asp Pro Asp Ser Pro Leu Pro His Pro Glu
1 5 10
<210> 88
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 88
Xaa Arg Arg Ile Arg Ile Leu Met Phe Leu Ile Gly Cys Gly Arg Val
1 5 10 15
<210> 89
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 89
His Pro His Val Ile Leu Pro Arg Ile Arg Ile Arg Ile Arg
1 5 10
<210> 90
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 90
Glu Ile His Thr Ile His Leu Leu Pro Glu Arg Arg
1 5 10
<210> 91
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 91
Glu Pro Ser His Pro Arg Ser Arg Tyr Pro Arg Thr Phe
1 5 10
<210> 92
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 92
Arg Asn Ile Ile Ile Arg Asp Phe Ile His Phe Ser His Ile Asp Arg
1 5 10 15
<210> 93
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (10)..(10)
<223> X = МИРИСТАТ
<400> 93
Arg Arg Ile Arg Asp Pro Gln Ile Lys Xaa Leu Glu Ile His Phe Ser
1 5 10 15
His Ile Asp
<210> 94
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 94
Xaa Asp Leu His Thr Ile His Ile Pro Arg Asp Arg Arg
1 5 10
<210> 95
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 95
Ser His Asp Phe Pro His Arg Glu Pro Arg Pro Glu Arg Arg
1 5 10
<210> 96
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 96
Ser Tyr Arg His Tyr Ser Asp His Trp Glu Asp Arg Arg Arg
1 5 10
<210> 97
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 97
Val Trp Val His Asp Ser Cys His Ala Asn Leu Gln Asn Tyr Arg Asn
1 5 10 15
Tyr Leu Leu Pro
20
<210> 98
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 98
Glu His Asp Phe Glu Val Arg Gly Asp Val Val Asn Gly Arg Asn His
1 5 10 15
Gln Gly Pro Lys
20
<210> 99
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 99
Leu Glu Val Ile Tyr Met Ile
1 5
<210> 100
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 100
Trp Thr Leu Ser Asn Tyr Leu
1 5
<210> 101
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 101
Asp Ser Leu His Ser Thr Tyr
1 5
<210> 102
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 102
Trp His His Arg Gln Gln Ile Pro Arg Pro Leu Glu
1 5 10
<210> 103
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 103
Ala Pro Ser Ile Phe Thr Pro His Ala Trp Arg Gln
1 5 10
<210> 104
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 104
Thr His Phe Ser His His Leu Lys Gly Gly Gly Arg Arg Gln Arg Arg
1 5 10 15
Arg Pro
<210> 105
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 105
Leu His Ser Lys Thr Leu Val Leu Gly Gly Gly Arg Arg Arg Arg Gly
1 5 10 15
Asp Arg
<210> 106
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 106
Trp Thr Leu Ser Asn Tyr Leu Gly Gly Arg Lys Lys Arg Arg Gln Arg
1 5 10 15
Arg Arg Arg
<210> 107
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 107
Val Arg Cys Ile Phe Arg Gly Ile Trp Val Arg Leu
1 5 10
<210> 108
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 108
His Ser Ser Gly His Asn Phe Val Leu Val Arg Gln
1 5 10
<210> 109
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 109
Leu Phe Ile Leu Val Phe Arg
1 5
<210> 110
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 110
Thr Thr Ser His His Pro Lys
1 5
<210> 111
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 111
Val Met Val Leu Phe Arg Ile Leu Arg Gly Ser Met
1 5 10
<210> 112
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 112
Ser Ile Leu Thr
1
<210> 113
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 113
Arg Arg Arg Glu Ser Glu Gln Arg Ser Ile Ser Leu His His His Ser
1 5 10 15
Thr
<210> 114
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 114
Xaa His Phe Asn His Tyr Thr Phe Glu Ser Thr Cys Arg Arg Arg Arg
1 5 10 15
Cys
<210> 115
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 115
His Ser Thr Pro His Pro Pro Gln Pro Pro Glu Arg Arg Arg
1 5 10
<210> 116
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 116
Arg Arg Lys Ser Glu Pro His Ser Leu Ser Gly Gly Tyr Gln Thr Gly
1 5 10 15
Ala Asp
<210> 117
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 117
His Arg Thr Gly His Tyr Thr Arg Cys Arg Gln Arg Cys Arg Ser Arg
1 5 10 15
Ser His Asn Arg His
20
<210> 118
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 118
Arg Arg Cys Arg Ser Ile Leu Pro Leu Leu Leu Leu Ser Arg
1 5 10
<210> 119
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 119
Arg Thr Leu His Gly Arg Arg Val Ile Leu His Glu Gly Gly His Ser
1 5 10 15
Ile Ser Asp Leu Lys
20
<210> 120
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 120
His His Arg Leu Ser Tyr Phe Ile Val Arg Arg His Ser Thr His Ala
1 5 10 15
Ser Arg
<210> 121
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 121
Arg Arg Ile Arg Ile Asp Pro Gln His Asp
1 5 10
<210> 122
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 122
Ile Leu Gln Pro Asp Phe Leu Ile Arg Pro Glu
1 5 10
<210> 123
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 123
His Asp Pro Arg Ile Ile Arg Ile Arg
1 5
<210> 124
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 124
Ser Pro Tyr Pro Ile Arg Thr
1 5
<210> 125
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 125
Ile Leu Val Ile Ile Gln Arg Ile Met
1 5
<210> 126
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 126
Ile Arg Phe Ile Leu Ile Arg
1 5
<210> 127
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 127
Ser Ser Val His His Arg Gly
1 5
<210> 128
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 128
Leu Arg Arg Gln Leu Gln Leu
1 5
<210> 129
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 129
His Thr Thr Ala His Thr His
1 5
<210> 130
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 130
His Pro His Asn His Thr Val His Asn Val Val Tyr
1 5 10
<210> 131
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 131
Asp His Ser Lys Phe Val Pro Leu Phe Val Arg Gln
1 5 10
<210> 132
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 132
Ser Ile Arg Thr Leu Gly Arg Phe Leu Ile Ile Arg Val
1 5 10
<210> 133
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 133
Gly Leu Cys Arg Ile Ile Leu
1 5
<210> 134
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 134
Ser Pro Pro Ile Arg His His
1 5
<210> 135
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 135
His Pro Thr His Pro Ile Arg Leu Arg Asp Asn Leu Thr Arg
1 5 10
<210> 136
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Миристат
<222> (1)..(1)
<223> X = миристат
<400> 136
Xaa Arg Glu Glu Glu Thr Ile Leu Ile Ile Arg Arg Arg
1 5 10
<210> 137
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 137
His Thr Ile His Ser Ile Ser Asp Phe Pro Glu Pro Pro Asp Arg Arg
1 5 10 15
Arg Arg
<210> 138
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 138
Asp Glu Asp Ala Ala His Ser Thr Gly His Pro His Asn Ser Gln His
1 5 10 15
Arg Arg Arg Arg
20
<210> 139
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 139
Thr Glu Gln His His Tyr Ile Pro His Arg Arg Arg
1 5 10
<210> 140
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 140
Arg Leu Arg Arg Val Ile Leu Arg Ser Tyr His Glu
1 5 10
<210> 141
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 141
Glu Glu Pro Asp Arg Gln Pro Ser Gly Lys Arg Gly Gly Arg Lys Arg
1 5 10 15
Arg Ser Arg
<210> 142
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 142
Arg Asp Phe His Thr Ile His Pro Ser Ile Ser Arg Arg
1 5 10
<210> 143
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 143
Arg Arg Val Asp Ile His Asp Gly Gln Arg Arg
1 5 10
<210> 144
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 144
Asp Gln Pro Tyr Pro His Arg Arg Ile Arg
1 5 10
<210> 145
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 145
Xaa Arg Asp Phe Ile Leu Phe Ile Arg Arg Leu Gly Arg Arg
1 5 10
<210> 146
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 146
Leu Asp Leu Tyr His Pro Arg Glu Arg Arg
1 5 10
<210> 147
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 147
Arg Arg Ile Arg Asp Pro Leu Gly Asn Glu His Glu
1 5 10
<210> 148
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 148
Ile Val Glu Phe Arg Ile Arg Arg
1 5
<210> 149
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 149
Arg Arg Pro Arg Ile Pro Asp Tyr Ile Leu
1 5 10
<210> 150
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 150
Arg Ser Thr Pro His Ile His Glu Phe Ile Arg Arg
1 5 10
<210> 151
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 151
Ser His Asp Phe Tyr Pro His Trp Met Arg Glu Arg Ile Arg
1 5 10
<210> 152
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 152
His Phe Ser His His Leu Lys
1 5
<210> 153
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 153
Thr Ser Pro Leu Gln Ser Leu Lys
1 5
<210> 154
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 154
Ala Ile Leu Thr Leu Ile Leu Arg Arg Val Ile Trp Pro
1 5 10
<210> 155
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 155
Leu Arg Phe Ile Asp Tyr Pro
1 5
<210> 156
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 156
Gly Pro Ile Lys His His Leu Gln His His
1 5 10
<210> 157
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 157
Leu Thr Leu Ser
1
<210> 158
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 158
Arg Tyr Glu Glu Asn Asn Gly Val Asn Pro Pro Val Gln Val Phe Glu
1 5 10 15
Ser Arg Thr Arg
20
<210> 159
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 159
Arg Glu Gly Phe Tyr Gly Pro Trp His Glu Gln Arg Arg Arg
1 5 10
<210> 160
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 160
Arg Arg Asp Ile Ile Arg His Asn Ala His Ser
1 5 10
<210> 161
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 161
His Asp Phe His Asp Tyr Leu Glu Arg Arg
1 5 10
<210> 162
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 162
Ile Arg Glu Phe Asp Pro Arg Arg Ile Arg
1 5 10
<210> 163
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 163
Arg Leu Arg Cys Leu Leu Leu Leu Ile Gly Arg Val Gly Arg Arg
1 5 10 15
<210> 164
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 164
Leu Gly Ile Asp Glu Asp Glu Glu Thr Glu Thr Ala Pro Glu
1 5 10
<210> 165
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 165
Ser Leu Leu Ile Gly Phe Gly Ile Ile Arg Ser Arg
1 5 10
<210> 166
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 166
Val His Glu Val Thr His His Trp Leu
1 5
<210> 167
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 167
Ala Thr Pro Phe His Gln Thr
1 5
<210> 168
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 168
Ser Ile Leu Pro Leu Phe Leu Ile Arg Arg Ser Gly
1 5 10
<210> 169
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 169
Ser Cys Arg Cys Arg Leu Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
<210> 170
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 170
Ser Arg Ile Val Leu Gly Trp
1 5
<210> 171
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 171
Ser Asn Ile His His Gln Val
1 5
<210> 172
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 172
Leu Thr Leu Met Arg Leu Arg Ile Ile Gly
1 5 10
<210> 173
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 173
His Ser Tyr Ser Pro Tyr Tyr Thr Phe Arg Gln His
1 5 10
<210> 174
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 174
Phe Ile Leu Ile Arg
1 5
<210> 175
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 175
Arg Cys Arg Asn Arg Lys Lys Glu Lys Thr Glu Cys Leu Gln Lys Glu
1 5 10 15
Ser Glu Lys
<210> 176
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 176
Arg Arg Ile Lys Met Ile Arg Thr Ser Glu Ser Phe Ile Gln His Ile
1 5 10 15
Val Ser
<210> 177
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 177
Arg Arg Val Ser Glu Leu Gln Arg Asn Lys His Gly Arg Lys His Glu
1 5 10 15
Leu
<210> 178
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 178
Arg Arg Arg Leu Asp Asp Glu Asp Val Gln Thr Pro Thr Pro Ser Glu
1 5 10 15
Tyr Gln Asn
<210> 179
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 179
Arg Arg Arg Gln Pro Leu Pro Ser Ala Pro Glu Asn Glu Glu
1 5 10
<210> 180
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 180
Ser Pro Leu Gln Thr Pro Ala Ala Pro Gly Ala Ala Ala Gly Pro Ala
1 5 10 15
Leu Ser Pro Val
20
<210> 181
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 181
Ser His Gln Val His Thr His His Asn Asn
1 5 10
<210> 182
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 182
Lys Leu Gln Val Pro Ile Lys
1 5
<210> 183
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 183
Ile Arg Gly Arg Ile Ile Arg Arg Lys Lys Arg Arg Gln Arg Arg Arg
1 5 10 15
Arg Gly Asp Arg
20
<210> 184
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 184
Gln Ile Pro His Arg Ser Ser Thr Ala Leu Gln Leu
1 5 10
<210> 185
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 185
Ser Tyr Gln Thr Met Gln Pro
1 5
<210> 186
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 186
Thr Asp Ser His Ser His His Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 187
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 187
Ile Pro Met Asn Phe Thr Ser His Ser Leu Arg Gln Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg Arg
20
<210> 188
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 188
Tyr Trp Ser Ala Pro Gln Pro Ala Thr Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg
20
<210> 189
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 189
Ser Thr Thr His Pro His Pro Gly Thr Ser Ala Pro Glu Pro Ala Thr
1 5 10 15
Arg Arg Arg
<210> 190
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 190
Asp Asp Ser Asp Asn Arg Ile Ile Arg Tyr Arg Arg
1 5 10
<210> 191
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 191
Thr Ser Pro His Pro Ser Leu Pro Arg His Ile Tyr Pro Arg Arg
1 5 10 15
<210> 192
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 192
Arg Arg Ile Thr Glu Ile Arg Gly Arg Thr Gly Lys Thr Thr Leu Thr
1 5 10 15
Tyr Ile Glu Asp
20
<210> 193
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 193
Xaa Asp Glu Arg Thr Gly Lys Thr Arg Arg Tyr Ile Asp Thr Arg Asp
1 5 10 15
Ile Arg Arg
<210> 194
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 194
Xaa Met Thr Tyr Ser Asp Met Pro Arg Arg Ile Ile Thr Asp Glu Asp
1 5 10 15
Arg Arg Arg
<210> 195
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 195
Arg Arg Tyr Asp Thr Val Ile Asp Asp Ile Glu Tyr Arg Arg
1 5 10
<210> 196
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 196
Arg Asp Thr Ile Glu Arg Pro Glu Ile Arg Arg
1 5 10
<210> 197
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 197
Xaa Arg Tyr Arg Arg Leu Ile Leu Glu Ile Trp Arg Arg
1 5 10
<210> 198
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 198
Xaa Arg His Asp Thr His Asn Ala His Ile Arg Arg
1 5 10
<210> 199
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 199
Thr His Asp Phe Asp Arg Leu Leu Arg Ile Arg Arg Arg
1 5 10
<210> 200
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 200
Arg His Asn His Ile Arg Pro Asp Asn Gln
1 5 10
<210> 201
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 201
Arg Tyr Lys Glu Pro Arg Ile Thr Pro Arg Glu
1 5 10
<210> 202
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 202
Leu Arg Ile Glu Pro Ile Arg Ile Arg
1 5
<210> 203
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 203
Xaa Arg Leu Ile Arg Ile Arg Ile Leu Met
1 5 10
<210> 204
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 204
Arg Pro Glu Phe His Ser Phe His Pro Ile Tyr Glu Arg Arg
1 5 10
<210> 205
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 205
Ser Thr Thr His Ile His Ala
1 5
<210> 206
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 206
Phe Pro His Leu Val Ser Ser Leu Thr Thr
1 5 10
<210> 207
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 207
Gly Leu His Leu Phe Thr Thr Asp Arg Gln Gly Trp
1 5 10
<210> 208
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 208
Asn His Pro Trp Gln Phe Pro Asn Arg Trp Thr Arg Arg Arg Arg Arg
1 5 10 15
Arg
<210> 209
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 209
His Ser Ser His His His Pro Val His Ser Trp Asn Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 210
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 210
Xaa Asp Ile His Thr Ile His Leu Pro Asp Thr His Arg Arg
1 5 10
<210> 211
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 211
Val Ala Glu Phe Ala Gln Ser Ile Gln Ser Arg Ile Val Glu Trp Lys
1 5 10 15
Glu Arg Leu Asp
20
<210> 212
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 212
Thr Arg Ile Leu Cys Ile Val Met Met
1 5
<210> 213
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 213
Phe Leu Leu Pro Glu Pro Asp Glu Asn Thr Arg Trp
1 5 10
<210> 214
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 214
Leu Met Ser Asn Ala Gln Tyr
1 5
<210> 215
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 215
Ser Ile Leu Thr Leu Ser Cys Arg Cys Arg Leu Arg Leu Trp Arg
1 5 10 15
<210> 216
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 216
His Gln Ile His Arg Asn His Thr Tyr
1 5
<210> 217
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 217
Leu Ile Arg Arg Cys Ser Leu Gln Arg
1 5
<210> 218
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 218
Gly Ala Met His Leu Pro Trp His Met Gly Thr Arg Arg Arg Arg Arg
1 5 10 15
Arg
<210> 219
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 219
Asp Glu Asp Ala Lys Phe Arg Ile Arg Ile Leu Met Arg Arg
1 5 10
<210> 220
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 220
Asn His Ile Thr Asn Gly Gly Glu Glu Asp Ser Asp Cys Ser Ser Arg
1 5 10 15
Arg Arg Arg Leu
20
<210> 221
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 221
Xaa His Ser Ser His His His Pro Thr Val Gln His Arg Arg
1 5 10
<210> 222
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 222
Arg Asp Phe Glu Arg Thr Ile Val Asp Ile
1 5 10
<210> 223
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 223
Xaa Arg Arg Arg Glu Ile Leu His Pro Glu Phe Arg Ile Leu Tyr Glu
1 5 10 15
<210> 224
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 224
His His Phe Ser His His Trp Lys Thr
1 5
<210> 225
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 225
Phe Leu Ile Arg Arg Ser Gly
1 5
<210> 226
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 226
His Asn His His His Ser Gln His Thr Pro Gln His
1 5 10
<210> 227
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 227
His Leu His Lys His His Tyr Lys Asp Ser Arg Met
1 5 10
<210> 228
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 228
His Arg Thr Gln Ser Thr Leu Ile Leu Phe Ile Arg Arg Gly Arg Glu
1 5 10 15
Thr
<210> 229
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 229
Leu His Phe Ser His Ile Asp Arg Arg
1 5
<210> 230
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 230
Tyr Glu Leu Pro His His Ala Tyr Pro Ala
1 5 10
<210> 231
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 231
Ser Leu Leu Ile Gly Phe Gly Ile Ile Arg Ser Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 232
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 232
His Thr Asp Ser His Pro His His His His Pro His Arg Arg Arg Arg
1 5 10 15
Arg
<210> 233
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 233
Ala Thr Gln His His Tyr Ile Lys Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 234
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 234
Phe Arg Ser Phe Ala Ile Pro Leu Val Val Pro Phe Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 235
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 235
Tyr Pro Thr Gln Gly His Leu Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 236
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 236
His Ala Asn Leu His His Thr Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 237
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 237
Tyr Arg Arg Leu Leu Ile Gly Met Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg
20
<210> 238
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 238
Ser His Tyr His Thr Pro Gln Asn Pro Pro Ser Thr Arg Arg Arg
1 5 10 15
<210> 239
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 239
Arg Ser Tyr Ser Lys Leu Leu Cys Leu Leu Glu Arg Leu Arg Ile Ser
1 5 10 15
Pro
<210> 240
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 240
Phe Trp Thr Gln Ser Ile Lys Glu Arg Lys Met Leu Asn Glu His Asp
1 5 10 15
Phe Glu Val Arg
20
<210> 241
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 241
Thr His Phe Ser His His Leu Lys His
1 5
<210> 242
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 242
Ser Cys Arg Cys Arg Leu Arg
1 5
<210> 243
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 243
Met His Pro Pro Asp Trp Tyr His His Thr Pro Lys Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 244
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 244
His Thr Ile His Val His Tyr Pro Gly Asn Arg Gln Pro Asn Pro Pro
1 5 10 15
Leu Ile Leu Gln Arg
20
<210> 245
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 245
Thr Pro Ser Tyr Gly His Thr Pro Ser His His Arg Arg Arg
1 5 10
<210> 246
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 246
Xaa Ile Arg Gly Arg Ile Arg Ile Ile Arg Arg Ile Arg
1 5 10
<210> 247
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 247
His His Pro Trp Thr His His Gln Arg Trp Ser
1 5 10
<210> 248
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 248
Ile Pro Met Asn Phe Thr Ser His Ser Leu Arg Gln
1 5 10
<210> 249
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 249
Ser Asn His His His Arg His His Thr Asn Thr His
1 5 10
<210> 250
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 250
Glu Val Thr Phe Arg His Ser Val Val Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg
20
<210> 251
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 251
Phe Pro Gly His Thr Ile His Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 252
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 252
Ser Ile Leu Thr Leu Ser Arg Ile Val Leu Gly Trp Trp
1 5 10
<210> 253
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 253
Thr Leu Tyr Leu Pro His Trp His Arg His
1 5 10
<210> 254
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 254
Ser Ile Leu Thr Leu Arg Leu Arg Arg Leu Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 255
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 255
Thr Leu Tyr Leu Pro His Trp His Arg His Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg
20
<210> 256
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 256
Thr Asp Ser His Ser His His
1 5
<210> 257
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 257
Glu Trp Lys Glu Arg Leu Asp Lys Glu Phe Ser Leu Ser Val Tyr Gln
1 5 10 15
Lys Met Lys Phe
20
<210> 258
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 258
Thr Ile His Pro Ser Ile Ser
1 5
<210> 259
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 259
Ser Ile Leu Thr Leu Arg Leu Arg Arg Leu Arg Arg
1 5 10
<210> 260
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 260
Val Pro His Ile His Glu Phe Thr
1 5
<210> 261
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 261
Thr Ile Ile His Arg Glu Asp Glu Asp Glu Ile Glu Trp
1 5 10
<210> 262
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 262
Lys Asp Leu Pro Phe Tyr Ser His Leu Ser Arg Gln
1 5 10
<210> 263
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 263
Thr His Phe Ser His His Leu Lys His Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 264
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 264
Ala Thr Gln His His Tyr Ile Lys
1 5
<210> 265
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 265
Ile Ile Arg Gly Asn Phe Leu Ile Gly Gly Arg Leu
1 5 10
<210> 266
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 266
Leu Pro Asn Pro Pro Glu Arg His His Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg
20
<210> 267
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 267
Ser Phe Ile Leu Phe Ile Arg Arg Gly Arg Leu Gly Arg Gly Asp Arg
1 5 10 15
<210> 268
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 268
Phe Pro Gly His Thr Ile His
1 5
<210> 269
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 269
Cys Ile Leu Arg Leu Trp Trp
1 5
<210> 270
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 270
Arg Arg Arg Ser His Ser Gln Glu Asn Val Asp Gln Asp Thr Asp Glu
1 5 10 15
<210> 271
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 271
Met Ser Thr Glu Ser Asn Met Pro Arg Leu Ile Gln Asn Asp Asp Arg
1 5 10 15
Arg Arg
<210> 272
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 272
Leu Leu Arg Leu Gly Leu Ile
1 5
<210> 273
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 273
Ile Arg Ile Leu Met Phe Leu Ile Gly Cys Gly Arg
1 5 10
<210> 274
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 274
Leu His Ser Lys Thr Leu Val Leu
1 5
<210> 275
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 275
Leu Arg Cys Leu Leu Leu Leu Ile Gly Arg Val Gly
1 5 10
<210> 276
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 276
Phe Leu Ile Gly Pro Asp Arg Leu Ile Arg Ser Arg
1 5 10
<210> 277
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 277
Leu Pro Asn Pro Pro Glu Arg His His
1 5
<210> 278
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 278
His Thr Asp Ser His Pro His His His His Pro His
1 5 10
<210> 279
<211> 22
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> Флуоресцеин изотиоцианат (ФИТЦ)
<222> (1)..(1)
<223> X = Флуоресцеин изотиоцианат (ФИТЦ)
<400> 279
Xaa Ser Phe Ile Leu Phe Ile Arg Arg Gly Arg Leu Gly Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg Arg Arg
20
<210> 280
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 280
His Ser Ser His His His Pro Val His Ser Trp Asn
1 5 10
<210> 281
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 281
Xaa Arg Thr Leu Ile Gly Ile Ile Arg Ser His His Leu Thr Leu Ile
1 5 10 15
Arg Arg
<210> 282
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 282
Ile Arg Gly Arg Ile Ile Arg
1 5
<210> 283
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 283
Ile Ile Arg Gly Asn Phe Leu Ile Gly Gly Arg Leu Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg Arg
20
<210> 284
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 284
Ile Arg Ile Leu Met
1 5
<210> 285
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 285
Gly Ala Met His Leu Pro Trp His Met Gly Thr Leu
1 5 10
<210> 286
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 286
Lys Arg Gly Gly Arg Lys Arg Arg Gly Gly Gly His Arg Leu Ser Tyr
1 5 10 15
Phe Ile Arg Arg
20
<210> 287
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 287
Asn His Pro Trp Gln Phe Pro Asn Arg Trp Thr Val
1 5 10
<210> 288
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 288
Met His Pro Pro Asp Trp Tyr His His Thr Pro Lys His
1 5 10
<210> 289
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 289
Ser Trp Gln Ala Leu Ala Leu Tyr Ala Ala Gly Trp Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 290
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 290
His Asn Ala His
1
<210> 291
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 291
Asp Glu Phe Glu Arg Tyr Arg Arg Phe Ser Thr Ser Arg Arg Arg
1 5 10 15
<210> 292
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 292
Glu Val Thr Phe Arg His Ser Val Val
1 5
<210> 293
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 293
Thr Arg Ile Leu Cys Ile Val Arg Lys Lys Arg Arg Gln Arg Arg Arg
1 5 10 15
Arg Gly Asp Arg
20
<210> 294
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 294
Ser Ile Leu Thr Leu Ser Arg Gly Arg Lys Lys Arg Arg Gln Arg Arg
1 5 10 15
Arg Arg
<210> 295
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 295
Cys Ile Leu Arg Leu Trp Trp Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg
<210> 296
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 296
Ala Ser Trp Gln Ala Leu Ala Leu Tyr Ala Ala Gly Trp
1 5 10
<210> 297
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 297
Xaa Pro Arg Val Leu Pro Ser Pro His Thr Ile His Pro Ser Gln Tyr
1 5 10 15
Pro
<210> 298
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 298
His Ala Asn Leu His His Thr
1 5
<210> 299
<211> 22
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 299
Ser Phe Ile Leu Phe Ile Arg Arg Gly Arg Leu Gly Arg Lys Lys Arg
1 5 10 15
Arg Gln Arg Arg Arg Pro
20
<210> 300
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 300
Tyr Pro Thr Gln Gly His Leu Arg
1 5
<210> 301
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 301
Tyr Arg Arg Leu Leu Ile Gly Met Met Trp Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg Arg
20
<210> 302
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 302
Ser Phe Ile Leu Phe Ile Arg Arg Gly Arg Leu Gly
1 5 10
<210> 303
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 303
Ile Arg Ile Leu Met Phe Leu Ile Gly Cys Gly Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg
<210> 304
<211> 14
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 304
Xaa Arg Arg Ile Cys Arg Phe Ile Arg Ile Cys Arg Val Arg
1 5 10
<210> 305
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 305
Ile Arg Gly Arg Ile Ile Arg Arg Arg Arg Arg Arg Arg Arg Arg Arg
1 5 10 15
<210> 306
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 306
Arg Arg Arg His Asp Ser Cys His Asn Gln Leu Gln Asn Tyr Asp His
1 5 10 15
Ser Thr Glu
<210> 307
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 307
Trp Asn His His His Ser Thr Pro His Pro Arg Arg Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg
20
<210> 308
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 308
Xaa Arg Arg Pro Val Ala Pro Asp Leu Arg His Thr Ile His Ile Pro
1 5 10 15
Pro Glu Arg
<210> 309
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 309
Arg Arg Asp Ile His Thr Ile His Pro Phe Tyr Gln
1 5 10
<210> 310
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 310
Trp Asn His His His Ser Thr Pro His Pro Ala His
1 5 10
<210> 311
<211> 21
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 311
Ser Phe Ile Leu Phe Ile Arg Arg Gly Arg Leu Gly Arg Arg Arg Arg
1 5 10 15
Arg Arg Arg Arg Arg
20
<210> 312
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> D-аминокислота
<222> (1)..(20)
<400> 312
Arg Arg Arg Arg Arg Arg Arg Arg Gly Leu Arg Gly Arg Arg Ile Phe
1 5 10 15
Leu Ile Phe Ser
20
<210> 313
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 313
Xaa Arg Arg His Asn Ala His His Ser Thr Pro His Pro Asp Asp Arg
1 5 10 15
<210> 314
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 314
His Ser Thr Pro His Pro
1 5
<210> 315
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 315
Leu Arg Cys Leu Leu Leu Leu Ile Gly Arg Val Gly Arg Lys Lys Arg
1 5 10 15
Arg Gln Arg Arg
20
<210> 316
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 316
Xaa Arg Arg Lys His Asn Lys His Arg Pro Glu Pro Asp Ser Asp Glu
1 5 10 15
Arg
<210> 317
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 317
Xaa Arg Arg Ile Arg Asp Pro Arg Ile Leu Leu Leu His Phe Asp
1 5 10 15
<210> 318
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 318
Arg Lys Arg Gly Lys Ser Tyr Ala Phe Phe Val Pro Pro Ser Glu Ser
1 5 10 15
Lys Glu Arg Trp
20
<210> 319
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 319
Xaa Arg Arg Lys Ile Leu Phe Ile Arg Leu Met His Asn Lys His
1 5 10 15
<210> 320
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 320
Xaa Arg Arg Leu Ile Val Arg Ile Leu Lys Leu Pro Asn Pro Pro Glu
1 5 10 15
Arg
<210> 321
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> МИРИСТАТ
<222> (1)..(1)
<223> X = МИРИСТАТ
<400> 321
Xaa Arg Arg His Ser Thr Pro His Pro Asp
1 5 10
<210> 322
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 322
Lys Pro Pro Asp Arg Leu Trp His Tyr Thr Gln Pro
1 5 10
<210> 323
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 323
Ala Thr Leu Pro Phe Val Thr Asp Arg Gln Gly Trp
1 5 10
<210> 324
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 324
Phe Tyr Ser His Ser Thr Ser Pro Ala Pro Ala Lys
1 5 10
<210> 325
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 325
Cys Tyr Ser His Ser Tyr Pro Thr Gln Gly His Leu
1 5 10
<210> 326
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 326
Glu Phe His Ser Phe Tyr Thr Ala Arg Gln Thr Gly
1 5 10
<210> 327
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 327
Ser Asp Gly Phe Val Pro His Phe Lys Arg Gln His
1 5 10
<210> 328
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 328
Leu Pro Asn Pro Pro Glu Arg
1 5
<210> 329
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 329
Leu His Ser Lys Thr Leu Val
1 5
<210> 330
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 330
His Val His Thr His Gln
1 5
<210> 331
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 331
Ser Ser Ser Leu Gly Thr His
1 5
<210> 332
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 332
His Glu Val Thr His His Trp
1 5
<210> 333
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 333
Ser Ala Pro Gln Pro Ala Thr
1 5
<210> 334
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 334
Thr Pro Pro Leu Thr Leu Ile
1 5
<210> 335
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 335
His Pro Trp Thr His His
1 5
<210> 336
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 336
Ser Ala Ala Ser Asp Leu Arg
1 5
<210> 337
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 337
Ser Pro Leu Gln Ser Leu Lys
1 5
<210> 338
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 338
Arg Pro Thr Gln Val Leu His
1 5
<210> 339
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 339
agacatgccc agacatgtcc 20
<210> 340
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 340
gaacatgtcc caacatgttg 20
<210> 341
<211> 6
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 341
aagctt 6
<210> 342
<211> 6
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 342
gaattc 6
<210> 343
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 343
cccaagcaat ggatgatttg a 21
<210> 344
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 344
ggcattctgg gagcttcatc t 21
<210> 345
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 345
ggcagaccag catgacagat t 21
<210> 346
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 346
gcggattagg gcttcctctt 20
<210> 347
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 347
gacctcaacg cacagtacga g 21
<210> 348
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 348
aggagtccca tgatgagatt gt 22
<210> 349
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 349
aggcaaatgt gcaataccaa ca 22
<210> 350
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 350
ggttacagca ccatcagtag gtacag 26
<210> 351
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 351
cggcagagaa ttccacgtga t 21
<210> 352
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 352
atctcttcgc cagctccaac a 21
<210> 353
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 353
gcagagctgg aagtcgagtg t 21
<210> 354
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 354
aagtttctgc cggaagttca g 21
<210> 355
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 355
actgtgaccc ttgcaccaaa t 21
<210> 356
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 356
gccaccccaa gttagatctg g 21
<210> 357
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 357
aggcactcac agagcactac aaac 24
<210> 358
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 358
gcccttggac ggcttttc 18
<210> 359
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 359
acccactcct ccacctttga 20
<210> 360
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 360
ctgttgctgt agccaaattc gt 22
<210> 361
<211> 100
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 361
ctgctgaagc ttcgaattcc tagacatgcc cagacatgtc ctactgctgc tgctgctgct 60
gctgcgaaca tgtcccaaca tgttgctgct gctgctgctg 100
<210> 362
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 362
tcgagttgcc tggacttgcc tggccttgcc ttttc 35
<210> 363
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 363
tcgagtttaa tggactttaa tggcctttaa ttttc 35
<210> 364
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<220>
<221> misc_feature
<222> (1)..(8)
<223> n is a, c, g, or t
<400> 364
nnnnnnnnca tggaaagata gtg 23
<210> 365
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<220>
<221> misc_feature
<222> (1)..(8)
<223> n is a, c, g, or t
<400> 365
nnnnnnnncc taaaacgatt tgtg 24
<210> 366
<211> 44
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 366
agacatgccc agacatgtcc ttatagacat gcccagacat gtcc 44
<210> 367
<211> 44
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Олигонуклеотид
<400> 367
agaaatgccc agaaatgtcc ttatagaaat gcccagaaat gtcc 44
<210> 368
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 368
Phe Arg Ser Phe Ala Ile Pro Leu Val Val Pro Phe
1 5 10
<210> 369
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 369
Lys Ile Leu Phe Ile Arg Leu Met His Asn Lys His
1 5 10
<210> 370
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 370
His His His Pro
1
<210> 371
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 371
His Thr Ile His
1
<210> 372
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 372
His Asn Lys His
1
<210> 373
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 373
Leu Leu Leu Ile Gly
1 5
<210> 374
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 374
Ile Leu Phe Ile Arg
1 5
<210> 375
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 375
Ile Arg Gly Arg Ile Ile
1 5
<210> 376
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 376
Ser Phe Ile Leu Phe Ile Arg
1 5
<210> 377
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 377
Tyr Pro Thr Gln Gly His Leu
1 5
<210> 378
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 378
Trp Asn His His His Ser Thr Pro His Pro
1 5 10
<210> 379
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 379
Ile Arg Ile Leu Met Phe Leu Ile Gly Cys Gly
1 5 10
<210> 380
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 380
Ser Trp Gln Ala Leu Ala Leu Tyr Ala Ala Gly Trp
1 5 10
<210> 381
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<220>
<221> D-amino_acids
<222> (1)..(12)
<400> 381
Gly Leu Arg Gly Arg Arg Ile Phe Leu Ile Phe Ser
1 5 10
<210> 382
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептид
<400> 382
Leu Arg Cys Leu Leu Leu Leu Ile Gly Arg Val Gly Arg Lys Lys Arg
1 5 10 15
Arg Gln
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ПЕПТИДЫ И ИХ ПРИМЕНЕНИЕ В ЛЕЧЕНИИ ЗАБОЛЕВАНИЙ, НАРУШЕНИЙ ИЛИ СОСТОЯНИЙ, АССОЦИИРОВАННЫХ С МУТАНТНЫМ р53 | 2017 |
|
RU2762089C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| МОДИФИЦИРОВАННЫЕ МИТОХОНДРИИ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2811467C2 |
| НЕОАНТИГЕНЫ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2805196C2 |
| АНТИТЕЛА, КОТОРЫЕ СВЯЗЫВАЮТ ОПУХОЛЕВУЮ ТКАНЬ, ДЛЯ ДИАГНОСТИКИ И ЛЕЧЕНИЯ | 2020 |
|
RU2806915C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА АНТИГЕН СОЗРЕВАНИЯ В-КЛЕТОК, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2829097C2 |
| БЕЛОК, СВЯЗЫВАЮЩИЙСЯ С NKG2D, CD16 И С ОПУХОЛЕСПЕЦИФИЧЕСКИМ АНТИГЕНОМ | 2018 |
|
RU2826991C2 |
| АНТИТЕЛА, СПЕЦИФИЧЕСКИЕ В ОТНОШЕНИИ CD70, И ПУТИ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2801093C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА ПОДОБНЫЙ FC-РЕЦЕПТОРУ БЕЛОК 5, И ИХ ПРИМЕНЕНИЕ | 2015 |
|
RU2779747C2 |
| НАПРАВЛЕННОЕ ИНГИБИРОВАНИЕ TGF БЕТА | 2015 |
|
RU2752424C2 |
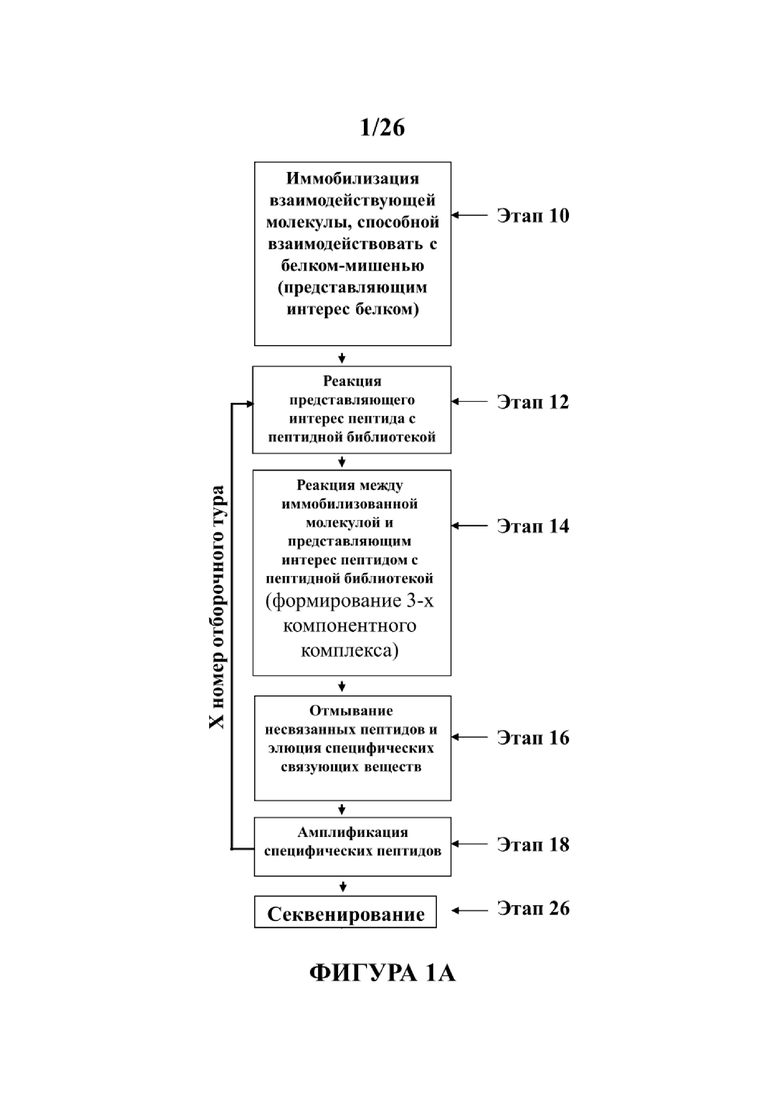


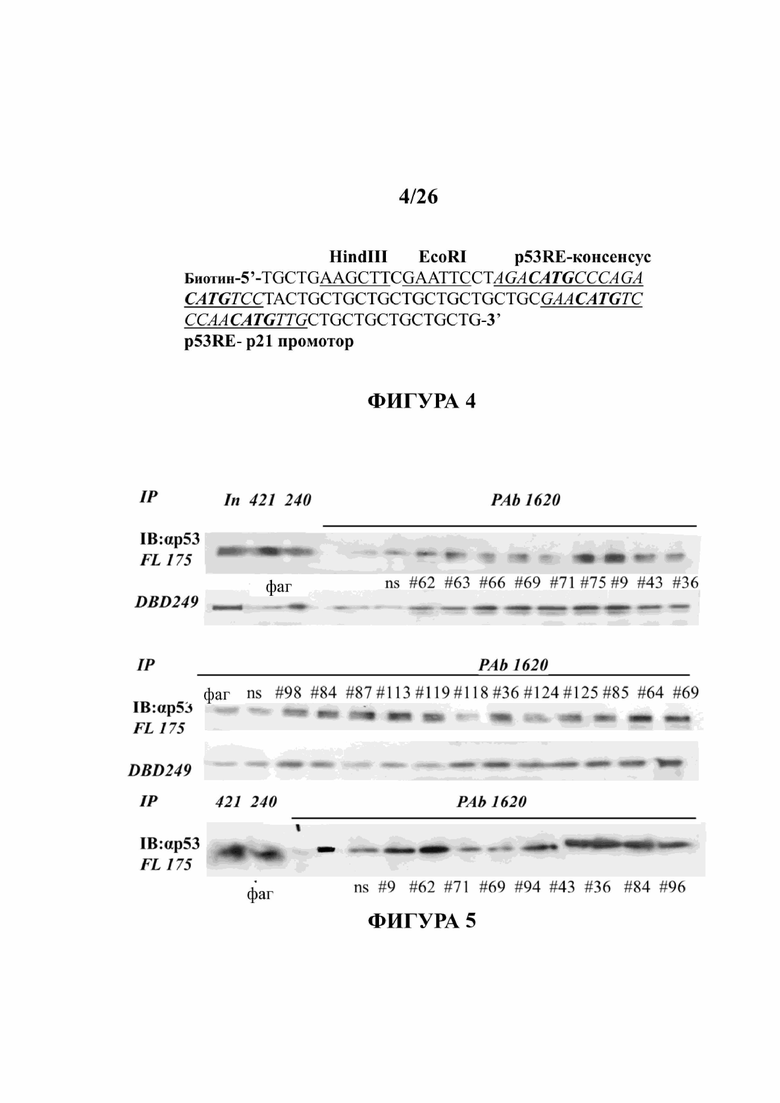

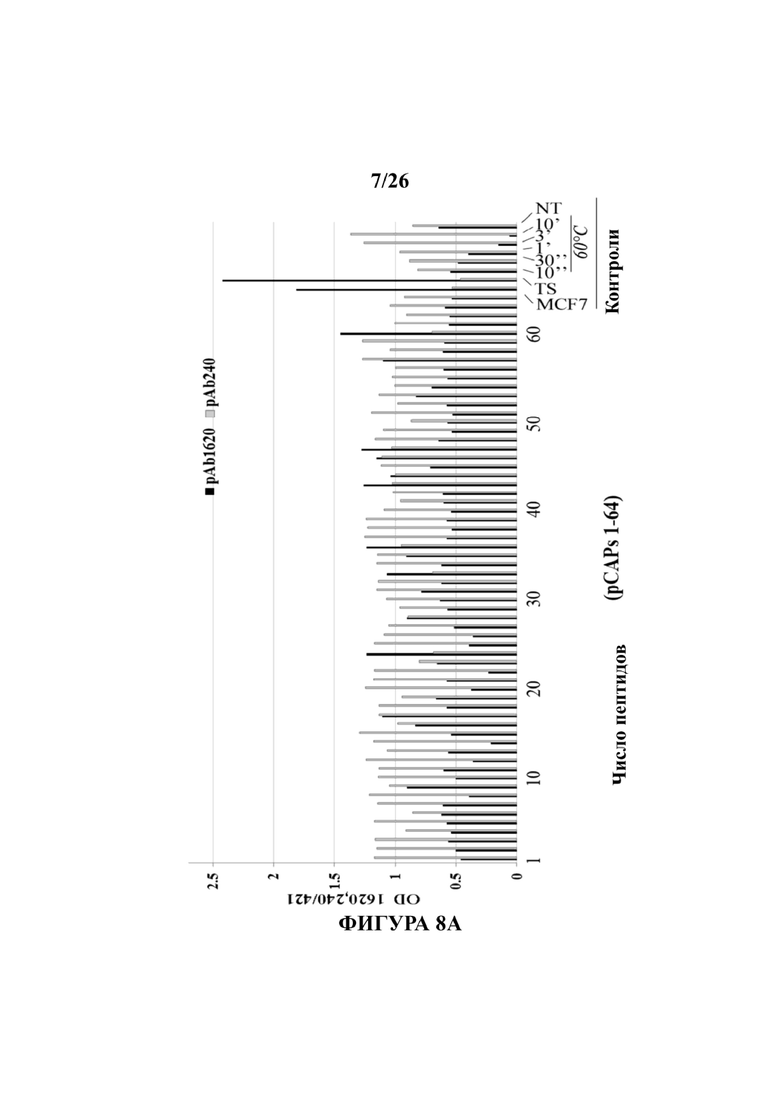
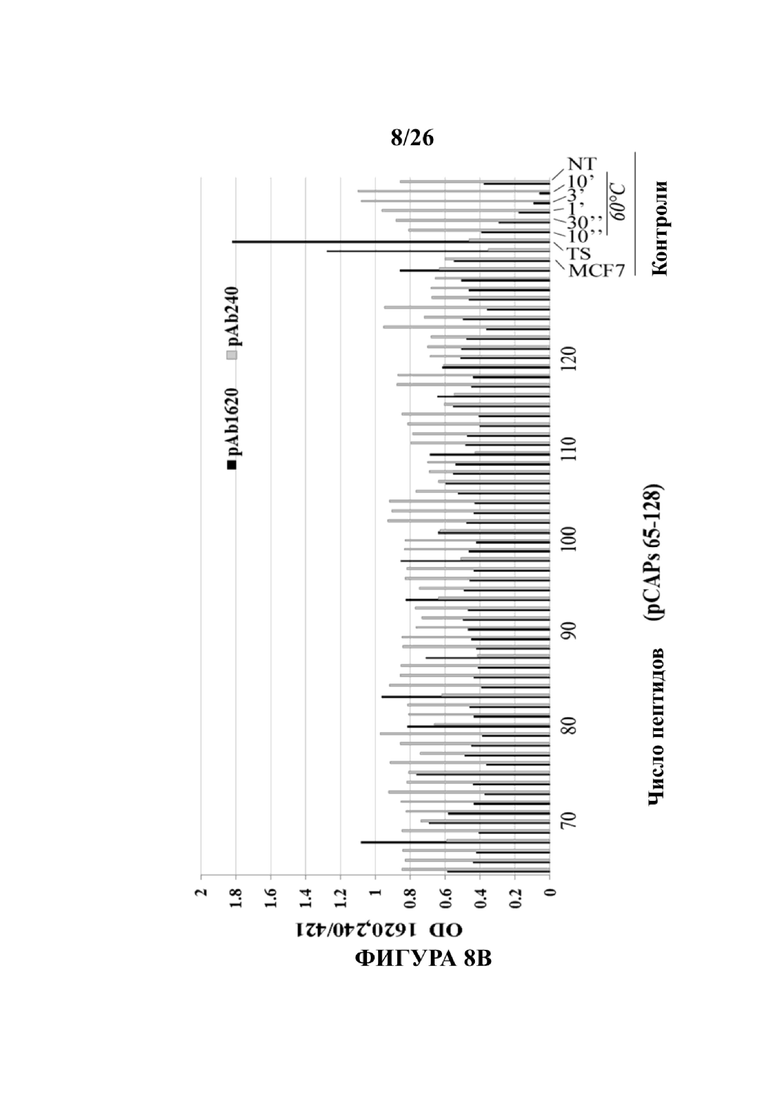
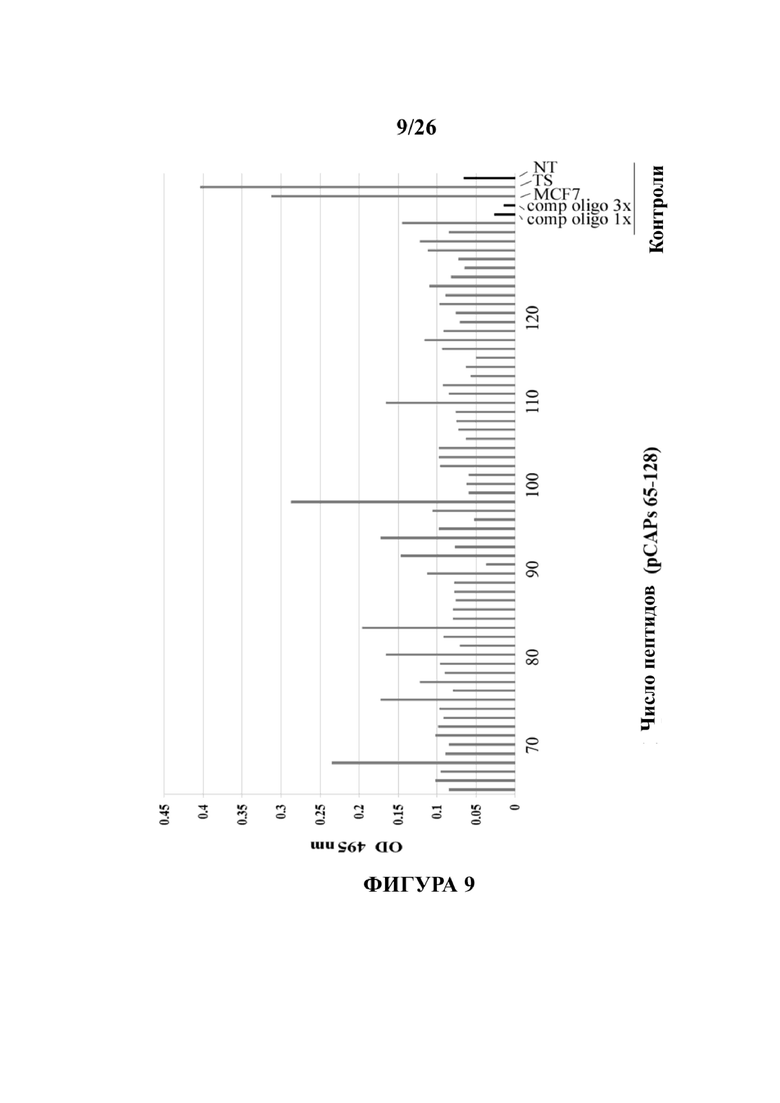
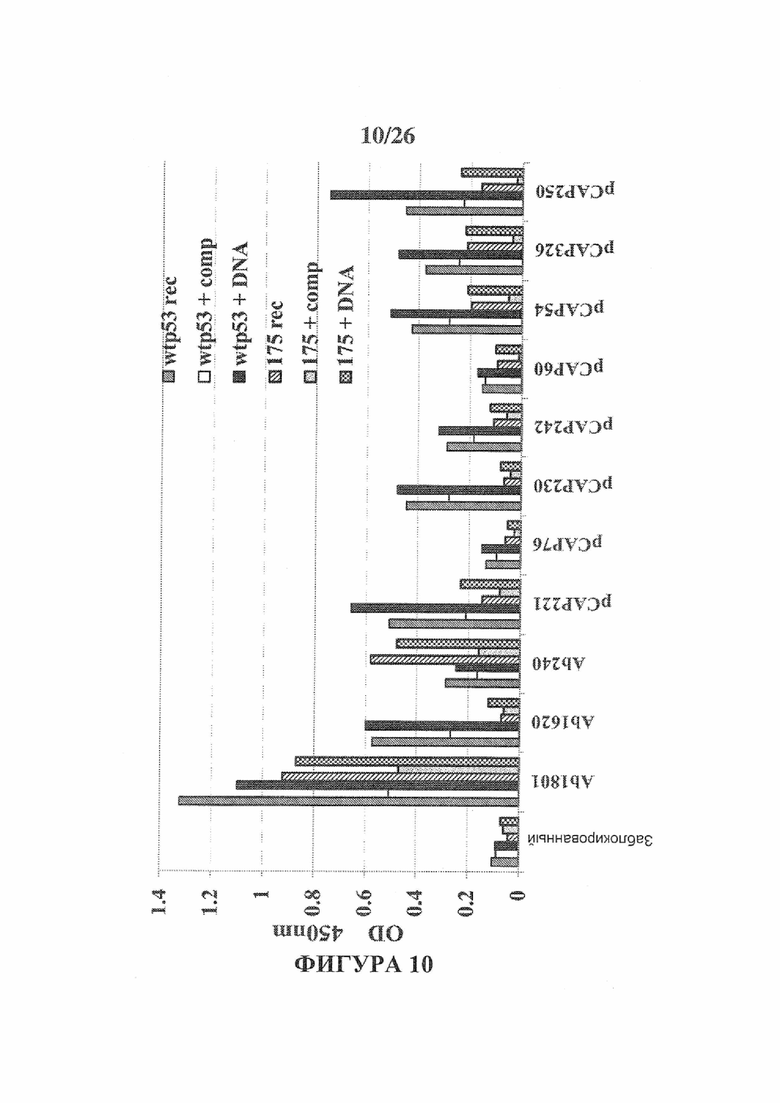
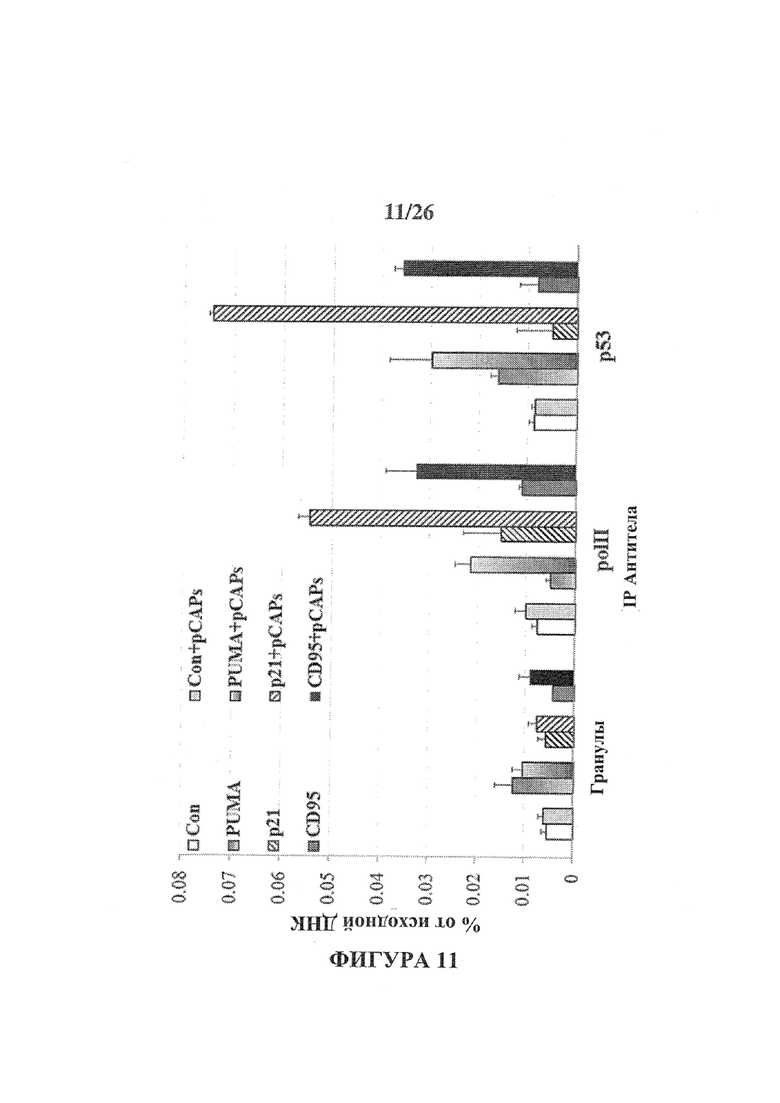

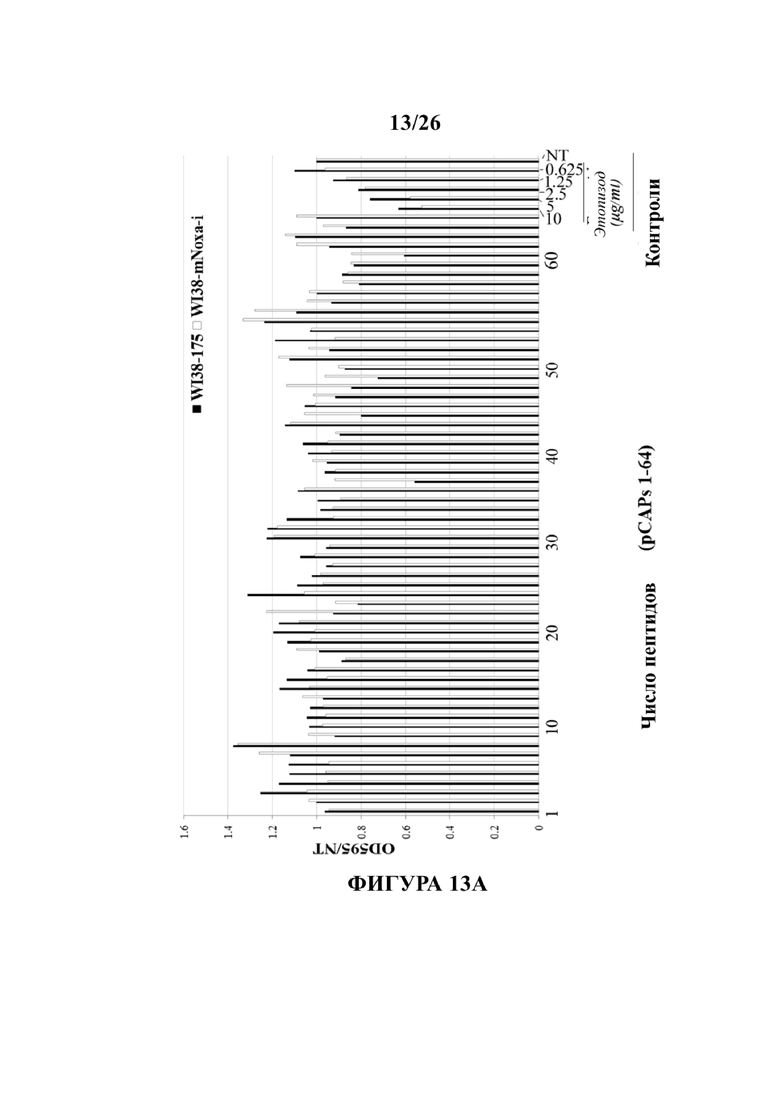
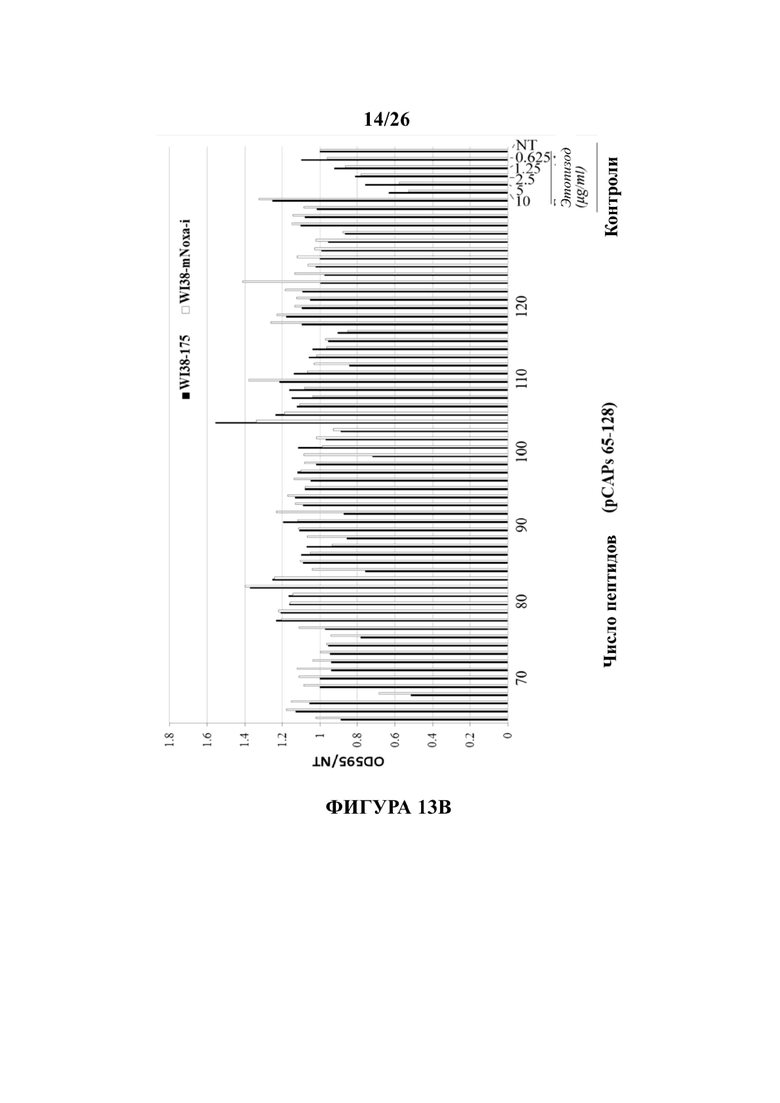
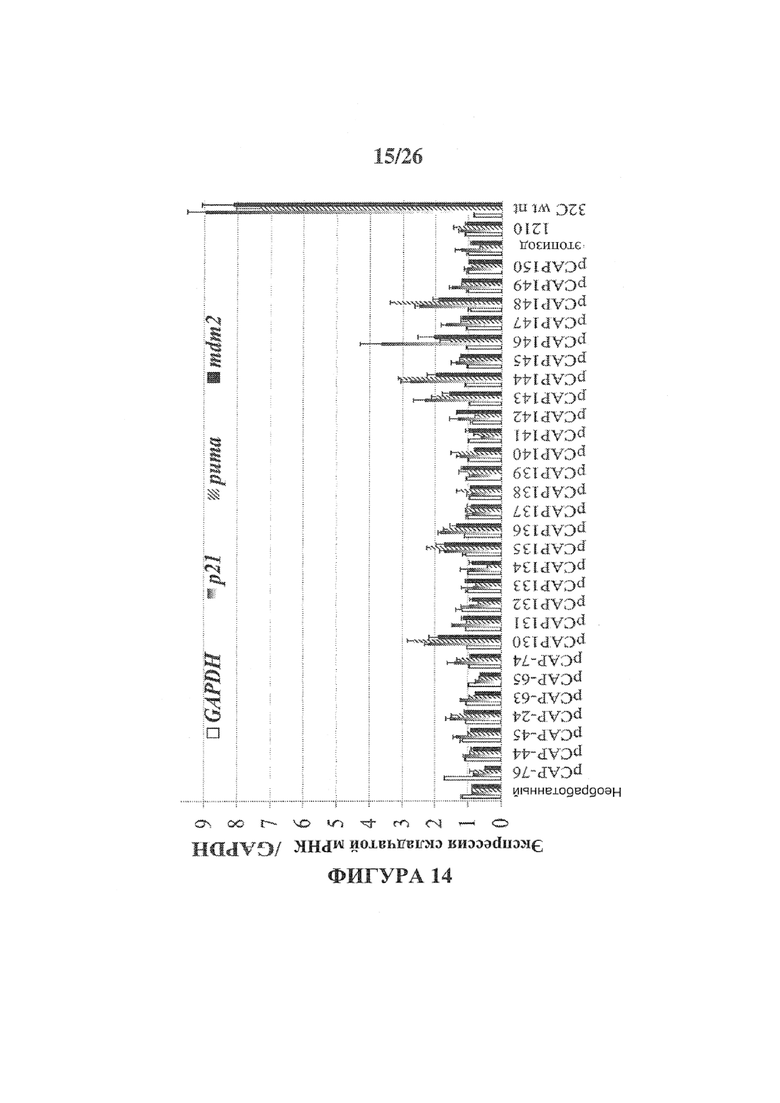

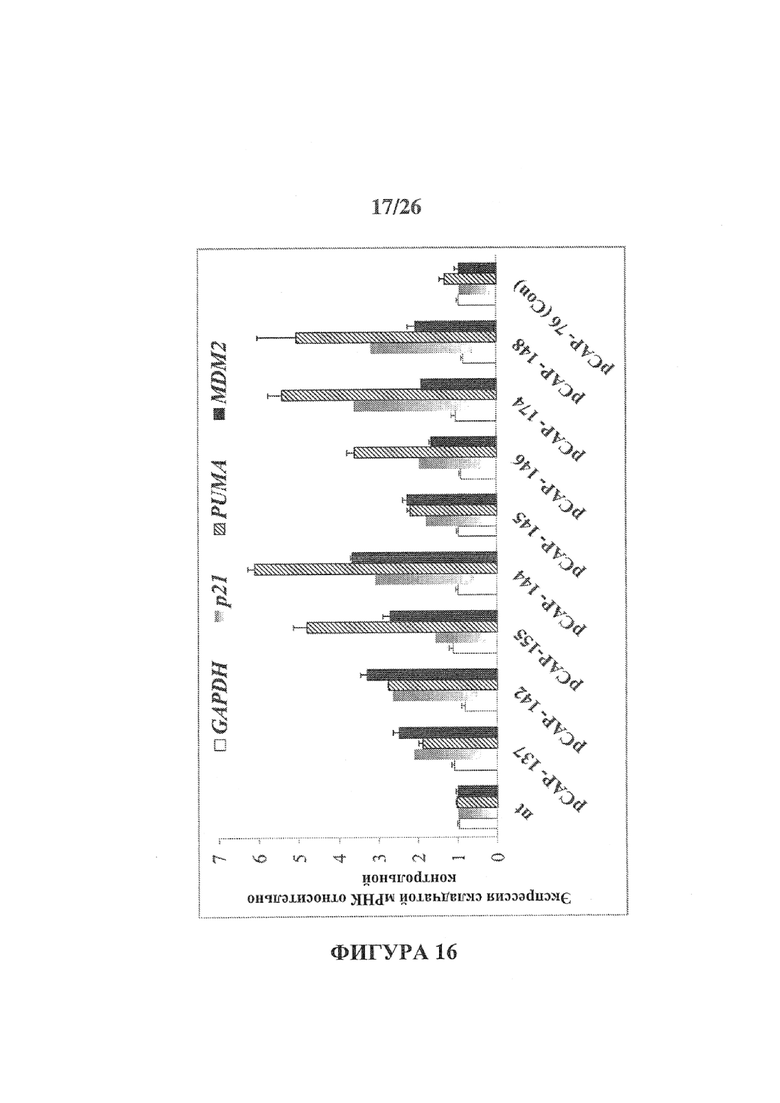
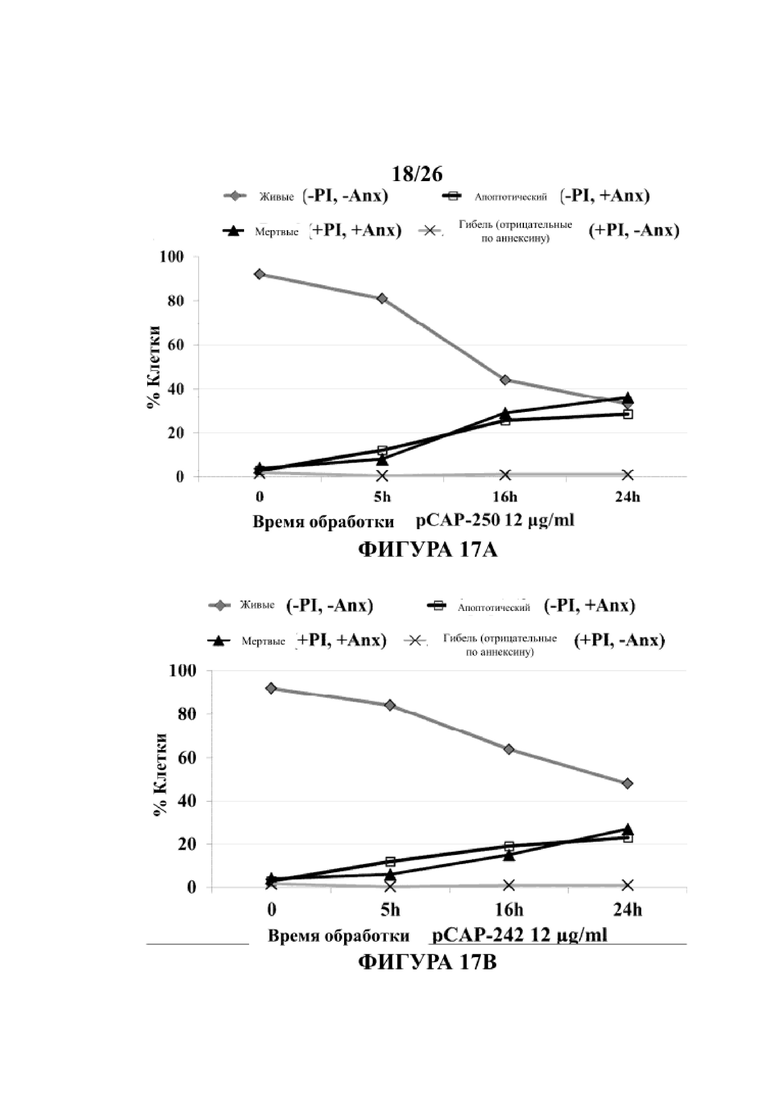

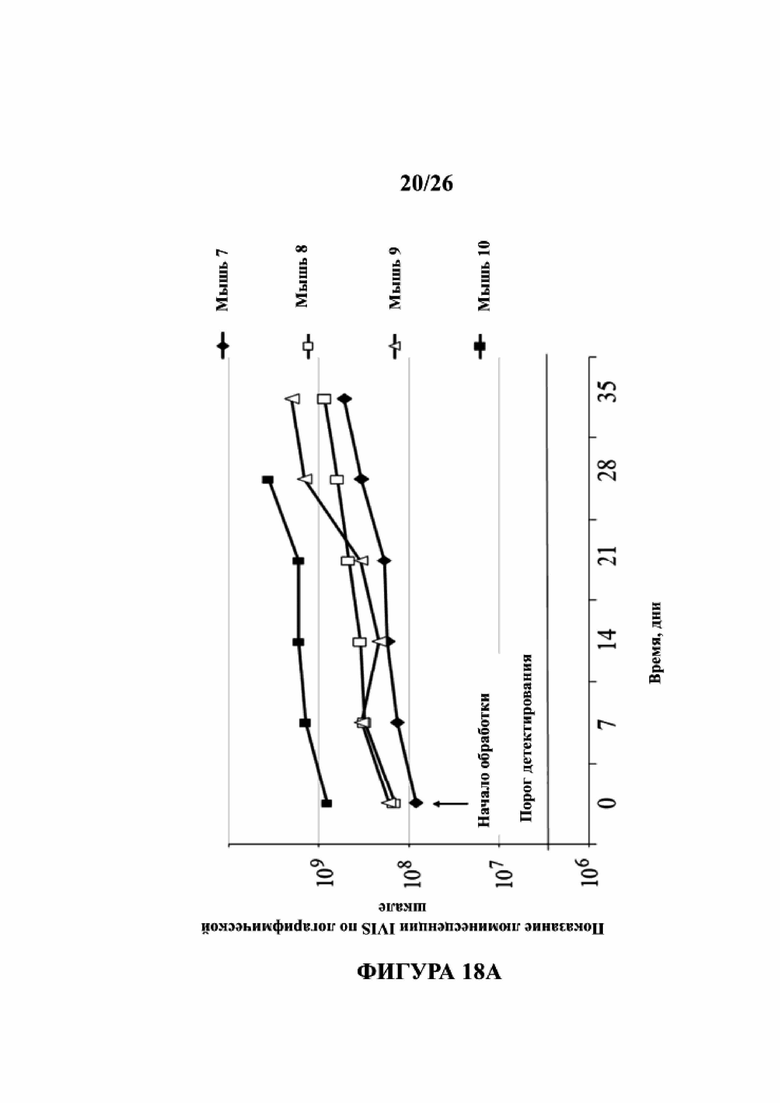
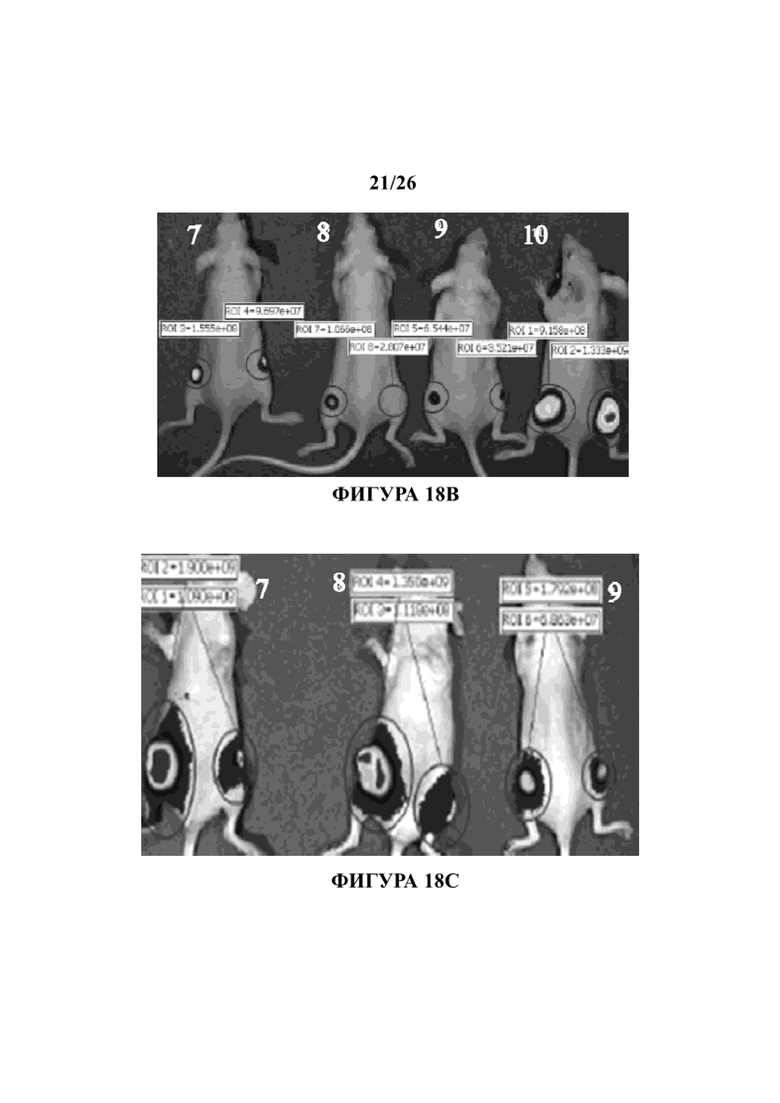
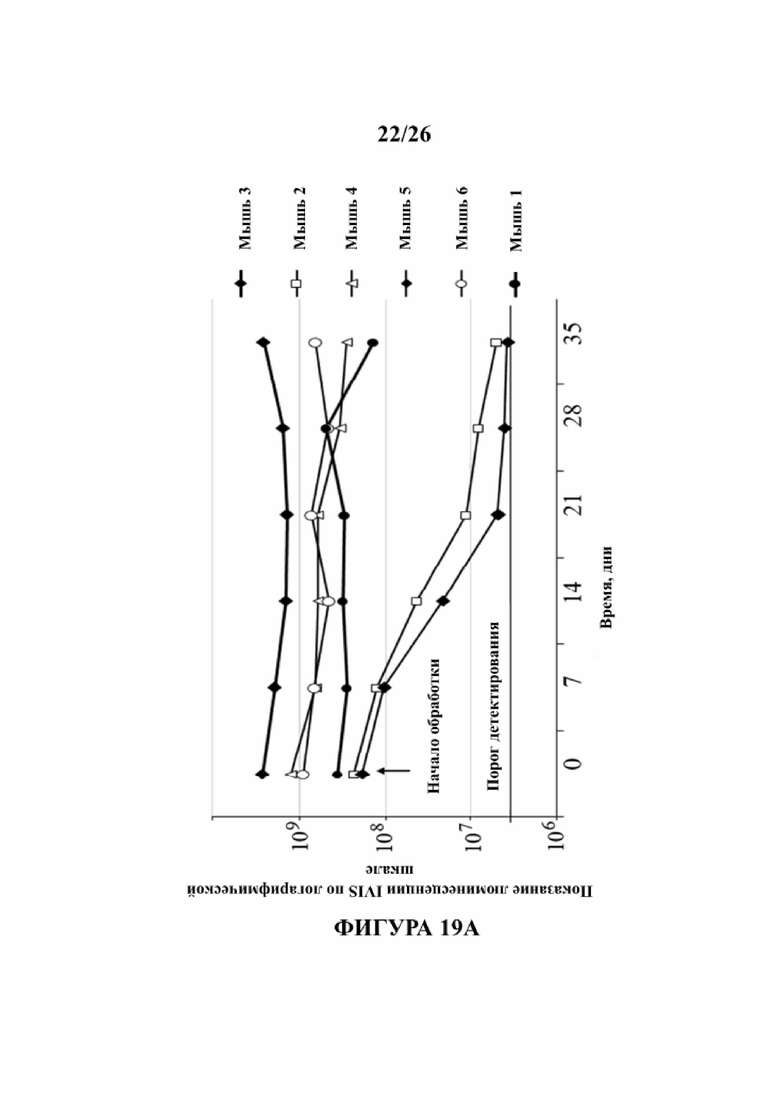
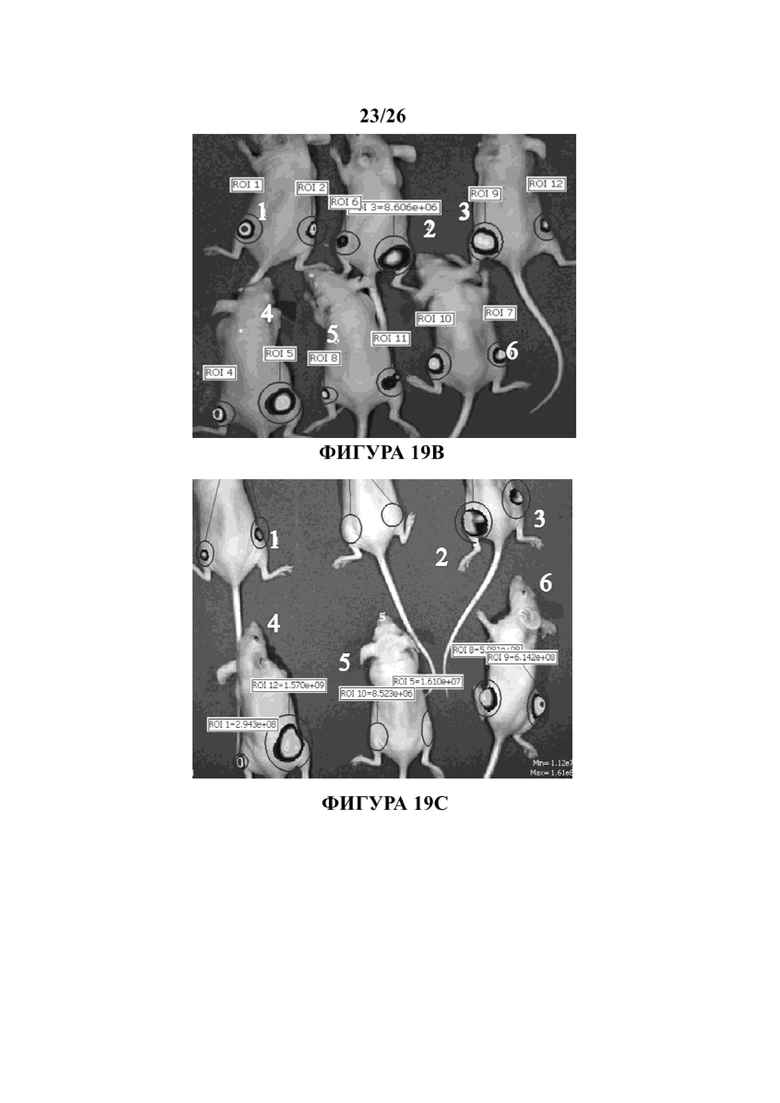

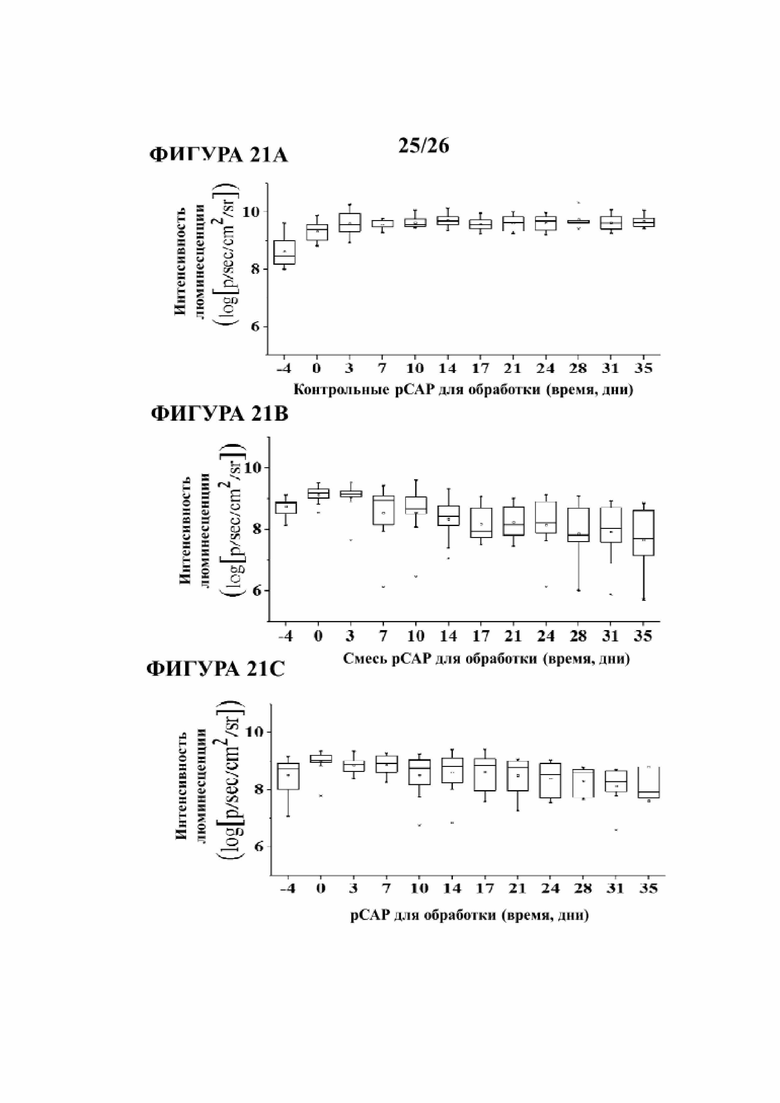
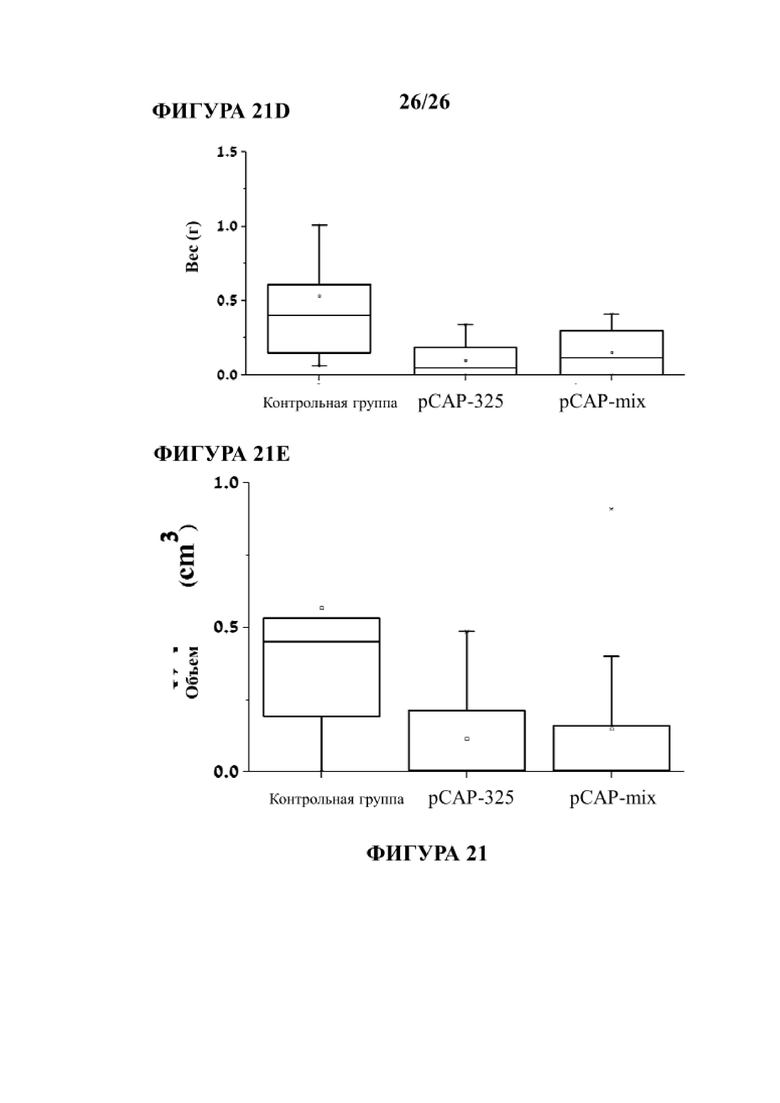
Изобретение относится к пептидам, которые могут реактивировать эффективно и специфически мутанты р53. В частности, предлагаются пептиды, реактивирующие мутантный р53, которые могут восстанавливать нативное складывание р53 дикого типа и, следовательно, активность супрессоров опухоли по отношению к мутантному белку p53. Такие пептиды могут быть пригодны для лечения различных состояний и заболеваний, в которых белок р53 мутирован. 2 н. и 9 з.п. ф-лы, 21 ил., 10 табл., 12 пр.
1. Пептид, содержащий аминокислотную последовательность, представленную любой одной из SEQ ID NO:
172, 208, 209, 216, 226, 227, 244, 247, 262, 267, 274, 281, 283, 297, 316, 318,319, 320,
отличающийся тем, что указанный пептид по меньшей мере частично реактивирует мутантный белок p53; и
отличающийся тем, что указанный пептид имеет длину вплоть до 30 аминокислот.
2. Пептид по п. 1, конъюгированный по меньшей мере с одной группой, улучшающей проницаемость клетки, выбранной из группы, состоящей из аминокислотной группы и жирнокислотной группы.
3. Пептид по п. 1, где указанный пептид:
(i) по меньшей мере частично изменяет конформацию указанного мутантного белка p53 на конформацию белка p53 дикого типа (WT),
(ii) указанный пептид по меньшей мере частично изменяет конформацию указанного мутантного белка p53 таким образом, что указанный мутантный белок p53 распознается моноклональным антителом, направленным против белка p53 дикого типа,
(iii) указанный мутантный белок p53 не распознается моноклональным антителом, направленным против белка p53 дикого типа.
4. Пептид по п. 1, отличающийся тем, что указанный пептид по меньшей мере частично восстанавливает активность указанного мутантного белка p53 до активности белка p53 дикого типа.
5. Пептид по п. 4, отличающийся тем, что указанная активность заключается в снижении жизнеспособности клеток, экспрессирующих указанный мутантный белок p53;
указанная активность представляет собой промотирование апоптоза клеток, экспрессирующих указанный мутантный белок p53; или
указанная активность представляет собой связывание с консенсусным ДНК-связывающим элементом p53 в клетках, экспрессирующих указанный мутантный белок p53.
6. Пептид по п. 5, отличающийся тем, что указанное связывание приводит по меньшей мере к частичной активации эндогенного гена-мишени p53.
7. Пептид по п. 1, отличающийся тем, что указанный мутантный белок p53 имеет иную конформацию, чем белок p53 дикого типа.
8. Пептид по п. 1, отличающийся тем, что указанный мутантный белок p53 является по меньшей мере частично неактивным по сравнению с белком p53 дикого типа.
9. Пептид по п. 1, отличающийся тем, что указанный мутантный белок p53 содержит мутацию, выбранную из группы, состоящей из R175H, V143A, R249S, R273H, R280K, P309S, P151S, P151H, C176S, C176F, H179L, Q192R, R213Q, Y220C, Y220D, R245S, R282W, D281G, S241F, C242R, R248Q, R248W, D281G, R273C и V274F.
10. Применение пептида последовательности SEQ ID NO: 320 для лечения заболевания, нарушения или состояния, связанного с мутантным белком p53.
11. Применение по п. 10, отличающееся тем, что указанное заболевание, нарушение или состояние, связанное с мутантным белком p53, представляет собой рак.
| US 20030224379 A1, 04.12.2003 | |||
| WO 2013036208 A1, 14.03.2013 | |||
| WO 2013040142 A2, 21.03.2013 | |||
| СИНТЕЗ ГАММА-ЛИНОЛЕНОВОЙ КИСЛОТЫ ПОД ДЕЙСТВИЕМ ДЕЛЬТА-6-ДЕСАТУРАЗЫ | 1995 |
|
RU2181772C2 |
| Поршневой компрессор | 1980 |
|
SU989136A1 |
| СИНТЕТИЧЕСКИЙ МУТАНТНЫЙ ГЕН RB (ВАРИАНТЫ), СИНТЕТИЧЕСКИЙ МУТАНТНЫЙ ГЕН P53, ПЛАЗМИДА (ВАРИАНТЫ), МУТАНТНЫЙ БЕЛОК (ВАРИАНТЫ), СПОСОБ ИНГИБИРОВАНИЯ КЛЕТОЧНОЙ ПРОЛИФЕРАЦИИ (ВАРИАНТЫ), СПОСОБ ИНДУЦИРОВАНИЯ АПОПТОЗА | 1995 |
|
RU2192431C2 |
Авторы
Даты
2020-05-19—Публикация
2014-08-07—Подача