УРОВЕНЬ ТЕХНИКИ
Рекомбиназы могут использоваться в рекомбиназо-опосредованной амплификации нуклеиновых кислот. Так, например, рекомбиназы могут способствовать специфическому взаимодействию олигонуклеотидов с ДНК-мишенью, позволяя репликацию ДНК с помощью полимеразы. Поэтому, сохраняется потребность в модифицированных рекомбиназах с улучшенными свойствами.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Данная заявка подается вместе со списком последовательностей в электронном формате. Список последовательностей предоставляется в виде файла под названием IP1264.TXT, созданного 26-го сентября 2014 года и имеющего размер 98 кб. Информация в электронном виде в списке последовательностей полностью включена в данное описание путем ссылки.
КРАТКОЕ ИЗЛОЖЕНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
В настоящем документе представлены рекомбиназы для улучшения рекомбиназо-опосредованной амплификации нуклеиновых кислот. Авторы настоящего изобретения неожиданно идентифицировали некоторые измененные рекомбиназы, которые имеют существенно улучшенные характеристики при посеве нуклеиновых кислот на разграфленную поверхность проточной ячейки. В некоторых вариантах осуществления предложены измененные рекомбиназы, улучшающие посев ПЦР-свободной библиотеки, например, ПЦР-библиотеки, имеющей одноцепочечные адаптерные области, на разграфленную поверхность проточной ячейки для улучшенной кластерной амплификации.
В некоторых вариантах осуществления рекомбиназа представляет собой рекомбинантную UvsX и включает аминокислотную заместительную мутацию в положении, функционально эквивалентном Pro256 в аминокислотной последовательности RB49 UvsX. Аминокислотная последовательность RB49 UvsX дикого типа представлена в SEQ ID NO: 1. В некоторых вариантах осуществления рекомбинантная UvsX включает аминокислотную последовательность, которая содержит аминокислоту, которая по меньшей мере на 60%, 70%, 80%, 90%, 95%, 99% идентична SEQ ID NO: 1, и включает в себя аминокислотную заместительную мутацию в положении, функционально эквивалентном Pro256 в аминокислотной последовательности RB49 UvsX. В некоторых вариантах осуществления заместительная мутация включает мутацию на заряженный остаток. В некоторых вариантах осуществления заместительная мутация включает мутацию на основный остаток. В некоторых вариантах осуществления заместительная мутация включает мутацию, гомологичную Pro256Lys в аминокислотной последовательности RB49 UvsX.
В некоторых вариантах осуществления, в дополнение к вышеуказанным мутациям рекомбинантная UvsX может дополнительно включать заместительные мутации в положениях, функционально эквивалентных His63 в аминокислотной последовательности RB49 UvsX. Например, в некоторых вариантах осуществления рекомбинантная UvsX содержит заместительную мутацию, гомологичную His63Ser в аминокислотной последовательности RB49 UvsX.
В некоторых вариантах осуществления, в дополнение к любой из вышеуказанных мутаций рекомбинантная UvsX может дополнительно включать мутацию, выбранную из группы, состоящей из: добавления одного или нескольких остатков глутаминовой кислоты на С-конце; добавления одного или нескольких остатков аспарагиновой кислоты на С-конце; и их комбинации.
В некоторых вариантах осуществления рекомбинантную UvsX получают из миовирусного фага, выбранного из группы, состоящей из: Т4, Т6, Rb69, Aeh1, KVP40, фага Acinetobacter 133, фага Aeromonas 65, цианофага Р-SSM2, цианофага PSSM4, цианофага S-PM2, Rb32, фага Vibrio nt-1, Rb16, Rb43 и Rb49.
В некоторых вариантах осуществления рекомбинантную UvsX получают из миовирусного фага, выбранного из группы, состоящей из: T2, Rb14, фага Aeromonas 25, phi-1, фага 31, фага 44RR2.8t, фага RB3 и фага LZ2.
Также, в настоящем изобретении представлена рекомбинантная UvsX, включающая аминокислотную последовательность по любой одной из SEQ ID NO: 2 и 22-35. В некоторых вариантах осуществления рекомбинантная UvsX включает аминокислотную последовательность, которая содержит аминокислоту, которая по меньшей мере на 60%, 70%, 80%, 90%, 95%, 99% идентична любой одной из SEQ ID NO: 2 и 22-35, и которая включает в себя аминокислотную заместительную мутацию в положении, функционально эквивалентном Pro256 в аминокислотной последовательности RB49 UvsX.
Также, в настоящем изобретении представлена рекомбинантная UvsX, включающая заместительную мутацию в полуконсервативном домене, содержащем аминокислотную последовательность по любой из SEQ ID NO: 3-5, где заместительная мутация включает мутацию, выбранную из замены в положении 7 на любой остаток, кроме Phe, Pro, Asp, Glu или Asn. В некоторых вариантах осуществления рекомбинантная UvsX включает аминокислоту, которая по меньшей мере на 60%, 70%, 80%, 90%, 95%, 99% идентична рекомбиназе, которая включает полуконсервативный домен, содержащий аминокислотную последовательность по любой из SEQ ID NO: 3-5, и где рекомбинантная UvsX включает заместительную мутацию, выбранную из замены в положении 7 на любой остаток, кроме Phe, Pro, Asp, Glu или Asn. В некоторых вариантах осуществления мутация включает мутацию на заряженный остаток. В некоторых вариантах осуществления мутация включает мутацию на основный остаток. В некоторых вариантах осуществления мутация включает замену в положении 7 на Lys.
Также, в настоящем изобретении представлена рекомбинантная UvsX, включающая заместительную мутацию в полуконсервативном домене, включающем аминокислотную последовательность по любой из SEQ ID NO: 6-7, где заместительная мутация включает мутацию, выбранную из замены в положении 12 на любой остаток, кроме Phe, Pro, Asp, Glu или Asn. В некоторых вариантах осуществления рекомбинантная UvsX включает аминокислоту, которая по меньшей мере на 60%, 70%, 80%, 90%, 95%, 99% идентична рекомбиназе, которая включает в себя полуконсервативный домен, содержащий аминокислотную последовательность по любой из SEQ ID NO: 6-7, и где рекомбинантная UvsX включает заместительную мутацию, выбранную из замены в положении 12 на любой остаток, кроме Phe, Pro, Asp, Glu или Asn. В некоторых вариантах осуществления мутация включает мутацию на заряженный остаток. В некоторых вариантах осуществления мутация включает мутацию на основный остаток. В некоторых вариантах осуществления мутация включает замену в положении 12 на Lys.
В некоторых вариантах осуществления, в дополнение к вышеуказанным мутациям рекомбинантная UvsX может дополнительно включать заместительные мутации в положениях, функционально эквивалентных His63 в аминокислотной последовательности RB49 UvsX. Например, в некоторых вариантах осуществления рекомбинантная UvsX включает заместительную мутацию, гомологичную His63Ser в аминокислотной последовательности RB49 UvsX.
В некоторых вариантах осуществления, в дополнение к любой из вышеуказанных мутаций, рекомбинантная UvsX может дополнительно включать мутацию, выбранную из группы, состоящей из: добавления одного или нескольких остатков глутаминовой кислоты на С-конец; добавления одного или нескольких остатков аспарагиновой кислоты на С-конец; и их комбинации.
В некоторых вариантах осуществления рекомбинантную UvsX получают из миовирусного фага, выбранного из группы, состоящей из: Т4, Т6, Rb69, Aeh1, KVP40, фага Acinetobacter 133, фага Aeromonas 65, цианофага Р-SSM2, цианофага PSSM4, цианофага S-PM2, Rb32, фага Vibrio nt-1, Rb16, Rb43 и Rb49.
В некоторых вариантах осуществления рекомбинантную UvsX получают из миовирусного фага, выбранного из группы, состоящей из: T2, Rb14, фага Aeromonas 25, phi-1, фага 31, фага 44RR2.8t, фага RB3 и фага LZ2.
Также, в настоящем изобретении представлена молекула нуклеиновой кислоты, кодирующая рекомбинантную UvsX по любому из вышеуказанных вариантов осуществления. Также, в настоящем изобретении представлен экспрессионный вектор, включающий молекулу нуклеиновой кислоты, описанную выше. Также, в настоящем изобретении представлена клетка-хозяин, содержащая вектор, описанный выше.
Также, в настоящем изобретении представлен способ рекомбиназной полимеразной амплификации молекулы нуклеиновой кислоты-мишени, включающий следующие стадии: (а) контактирования рекомбинантной UvsX по любому из вышеуказанных вариантов осуществления с первым и вторым нуклеотидными праймерами с образованием первого и второго нуклеопротеинового праймера, где указанный нуклеотидный праймер содержит одноцепочечную область на своем 3'-конце; (b) контактирования первого и второго нуклеопротеиновых праймеров с указанной молекулой нуклеиновой кислоты-мишени, таким образом, образуя первую двухцепочечную структуру на первом участке указанной первой цепи и образуя вторую двухцепочечную структуру на втором участке указанной второй цепи таким образом, что 3'-концы указанного первого нуклеотидного праймера и указанного второго нуклеотидного праймера направлены друг на друга на одной двухцепочечной нуклеотидной матричной молекуле; (с) удлинения 3'-концов указанных первого и второго нуклеотидных праймеров с одной или несколькими полимеразами и dNTP, чтобы синтезировать первую и вторую двухцепочечную нуклеиновые кислоты, и первую и вторую вытесненные цепи нуклеиновой кислоты; и (d) продолжения реакции путем повторения стадий (b) и (с) до достижения желаемой степени амплификации.
В некоторых вариантах осуществления процесса, молекула нуклеиновой кислоты-мишени включает двухцепочечную нуклеиновую кислоту. В некоторых вариантах осуществления молекула нуклеиновой кислоты-мишени включает одноцепочечную нуклеиновую кислоту. Например, в некоторых вариантах осуществления нуклеиновая кислота-мишень включает одноцепочечную адаптерную область. В некоторых вариантах осуществления процесс осуществляют в присутствии белка-загрузчика рекомбиназы. Так, например, белок-загрузчик рекомбиназы может быть выбран из группы, состоящей из T4 UvsY, E. coli recO, E. coli recR и их комбинации. В некоторых вариантах осуществления процесс осуществляют в присутствии стабилизирующего одноцепочечные нуклеиновые кислоты агента, выбранного из группы, состоящей из gp32, SSB-белка E. coli, gp32-белка фага T4 и их производных. В некоторых вариантах осуществления процесс осуществляют в присутствии концентрирующего агента, выбранного из группы, включающей полиэтиленгликоль, полиэтиленоксид, полистирол, фиколл, декстран, ПВП и альбумин, так что концентрирующий агент стимулирует амплификацию.
В некоторых вариантах осуществления процесс осуществляют на массиве амплификационных сайтов. В некоторых вариантах осуществления изобретения каждый амплификационный сайт содержит множество амплификационных праймеров для амплификации нуклеиновой кислоты-мишени. В некоторых вариантах осуществления массив амплификационных сайтов содержит массив признаков на поверхности. Например, признаки могут быть несмежными и могут быть отделены друг от друга промежуточными областями поверхности, на которых отсутствуют амплификационные праймеры. В некоторых вариантах осуществления изобретения массив амплификационных сайтов содержит шарики в растворе или шарики на поверхности. В некоторых вариантах осуществления изобретения массив амплификационных сайтов содержит эмульсию. В некоторых вариантах осуществления процесс происходит изотермически.
Также, в настоящем изобретении представлен набор для проведения рекомбиназной полимеразной реакции. В некоторых вариантах осуществления набор может включать в себя рекомбинантную UvsX по любому из вышеуказанных вариантов осуществления и одно или несколько из следующего: связывающего одноцепочечную ДНК белка; ДНК-полимеразы; dNTP или смеси dNTP и ddNTP; концентрирующего агента; буфера; восстанавливающего агента; АТР или аналога АТР; белка-загрузчика рекомбиназы; первого праймера и, необязательно, второго праймера.
Подробности одного или нескольких вариантов осуществления изложены в прилагаемых чертежах и в нижеследующем описании. Другие признаки, цели и преимущества будут очевидны из описания и чертежей, а также из формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
Фиг.1 представляет собой схематическое изображение выравнивания аминокислотных последовательностей UvsX из Enterobacteria phage T4 (T4) (SEQ ID NO: 8), Enterobacteria phage T6 (T6) (SEQ ID NO: 9), Acinetobacter phage 133 (Phage133) (SEQ ID NO: 10), Enterobacteria phage RB69 (Rb69) (SEQ ID NO: 11), Aeromonas phage Aeh1 (Aeh1) (SEQ ID NO: 12), Aeromonas phage 65 (Ae65) (SEQ ID NO: 13), Vibrio phage KVP40 (Kvp40) (SEQ ID NO: 14), Enterobacteria phage RB43 (Rb43) (SEQ ID NO: 15), Prochlorococcus phage P-SSM2 (PSSM2) (SEQ ID NO: 16) и Prochlorococcus phage P-SSM4 (PSSM4) (SEQ ID NO: 17), которые также изложены в материалах, включенных в US 2009/0029421. Остатки, которые позиционно и/или функционально эквивалентны Pro256 в аминокислотной последовательности RB49 UvsX, выделены и обозначены треугольным символом.
Фиг.2 представляет собой схематическое изображение выравнивания аминокислотных последовательностей UvsX из Enterobacteria phage RB49 (RB49) (SEQ ID NO: 1) и Enterobacteria phage T4 (T4) (SEQ ID NO: 8). Остатки, которые позиционно и/или функционально эквивалентны Pro256 в аминокислотной последовательности RB49 UvsX, выделены и обозначены треугольным символом.
На фиг.3А показан снимок с экрана изображения кластеров ПЦР-свободной библиотеки, высеянной на разграфленную поверхность проточной ячейки с использованием состава T4 UvsX.
На фиг.3В показан снимок экрана изображения кластеров ПЦР-свободной библиотеки, высеянной на разграфленную поверхность проточной ячейки с использованием жидкого состава, который включает в себя рекомбиназу RB49 P256K.
На фиг.4А показан снимок с экрана изображения кластеров одноцепочечной (оцДНК) ПЦР-свободной библиотеки, высеянной на разграфленную поверхность проточной ячейки с использованием состава Т4 UvsX.
На фиг.4В показан снимок с экрана изображения кластеров одноцепочечной ПЦР-свободной библиотеки, высеянной на разграфленную поверхность проточной ячейки с использованием жидкого состава, который включает рекомбиназу RB49 P256K.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В настоящем документе представлены рекомбиназы для улучшения рекомбиназо-опосредованной амплификации нуклеиновых кислот. Авторы настоящего изобретения неожиданно идентифицировали некоторые измененные рекомбиназы, которые имеют существенно улучшенные характеристики при посеве нуклеиновых кислот на разграфленную поверхность проточной ячейки.
Как более подробно описано ниже, авторы настоящего изобретения неожиданно обнаружили, что одна или большее число мутаций по одному или нескольким остаткам рекомбиназы приводят к значительному улучшению при посеве ДНК-библиотеки, такой как, например, ПЦР-библиотека, имеющая одноцепочечные адаптерные области, на разграфленную поверхность проточной ячейки, обеспечивая улучшенную кластерную амплификацию.
В некоторых вариантах осуществления заместительная мутация включает мутацию на остаток, имеющий заряженную боковую цепь. Например, в некоторых вариантах осуществления заряженный аминокислотный остаток представляет собой положительно заряженный аминокислотный остаток. Термин «положительно заряженная аминокислота» относится к гидрофильной аминокислоте с значением рКа для боковой цепи выше 7, а именно, к основной аминокислоте. Основные аминокислоты, как правило, имеют положительно заряженные боковые цепи при физиологическом рН за счет ассоциации с ионом гидроксония. Встречающиеся в природе (генетически кодируемые) основные аминокислоты включают лизин (Lys, K), аргинин (Arg, R) и гистидин (His, H), в то время как неприродные (генетически некодируемые или нестандартные) основные аминокислоты включают, например, орнитин, 2,3-диаминопропионовую кислоту, 2,4-диаминомасляную кислоту, 2,5,6-триаминогексановую кислоту, 2-амино-4-гуанидинобутановую кислоту и гомоаргинин. Термин «отрицательно заряженная аминокислота» относится к природной или неприродной аминокислоте, независимо от хиральности, содержащей в дополнение к C-концевой карбоксильной группе по меньшей мере одну дополнительную отрицательно заряженную группу, такую как карбоксил, фосфат, фосфонат, сульфонат и т.п.
Также, в настоящем изобретении представлена рекомбинантная UvsX, содержащая заместительную мутацию в полуконсервативном домене рекомбинантной UvsX. Используемый в данном описании термин «полуконсервативный домен» относится к участку рекомбинантной UvsX, который является полностью консервативным или по меньшей мере частично консервативным среди различных биологических видов. Было неожиданно обнаружено, что мутация одного или нескольких остатков в полуконсервативном домене влияет на активность рекомбиназы, особенно, в присутствии одноцепочечной матричной нуклеиновой кислоты, что приводит к улучшению посева и/или амплификации в рекомбиназо-опосредованных реакциях амплификации. Эти мутированные рекомбиназы имеют улучшенные показатели по посеву ПЦР-свободных библиотек, таких как ПЦР-библиотеки, имеющие одноцепочечные адаптерные области, на разграфленную поверхность проточной ячейки, что приводит к улучшению кластерной амплификации, как описано в разделе примеров ниже.
В некоторых вариантах осуществления полуконсервативный домен содержит аминокислоты, имеющие последовательность, представленную в любой из SEQ ID NO: 3-7. SEQ ID NO: 3-7 соответствуют остаткам в полуконсервативной области среди различных биологических видов. Последовательность SEQ ID NO: 3 соответствует остаткам 251-258 в аминокислотной последовательности Т4 UvsX, которая представлена в данном описании как SEQ ID NO: 8. Выравнивание, показывающее консервативность последовательности у различных видов в полуконсервативной области, приведено на фиг.1 и 2. Последовательности UvsX, приведенные на фиг.1, были получены из последовательностей с номерами доступа в базе данных Genbank: NP_049656 (T4), YP_004300647 (фаг 133); NP_861734 (RB69); NP_943894.1 (Aeh1); YP_004300858 (Ae65); NP_899256 (KVP40); YP_239013 (RB43); YP_214417 (P-SSM2); YP_214708 (P-SSM4); и из публикации патента США № 2009/0029421 (T6). Фиг.2 представляет собой схематическое изображение выравнивания аминокислотных последовательностей UvsX из Enterobacteria phage RB49 (RB49) (SEQ ID NO: 1) и Enterobacteria phage T4 (T4) (SEQ ID NO: 8). Остатки, которые позиционно и/или функционально эквивалентны Pro256 в аминокислотной последовательности RB49 UvsX, выделены и обозначены треугольным символом. Последовательности UvsX, приведенные на фиг.2, были получены из последовательностей с номерами доступа в базе данных Genbank: NP_891595 (RB49) и NP_049656 (T4).
Было неожиданно обнаружено, что мутации в одном или нескольких остатках в полуконсервативной области повышают активность рекомбиназы, особенно, в присутствии одноцепочечной матричной нуклеиновой кислоты, что приводит к усилению посева и/или амплификации в рекомбиназо-опосредованных реакциях амплификации. Эти мутированные рекомбиназы имели улучшенные показатели в посеве ПЦР-свободных библиотек, таких как ПЦР-библиотеки, имеющие одноцепочечные адаптерные области, на разграфленную поверхность проточной ячейки, приводя к улучшению кластерной амплификации, как описано в разделе примеров ниже. Например, в некоторых вариантах рекомбинантной UvsX, представленных в настоящем описании, заместительная мутация включает мутацию в положении 7 любой из SEQ ID NO: 3-5 на любой остаток, кроме Phe, Pro, Asp, Glu или Asn. В некоторых вариантах осуществления рекомбинантная UvsX содержит мутацию на Lys в положении 7 любой из SEQ ID NO: 3-5. В некоторых вариантах рекомбинантной UvsX, представленных в настоящем описании, заместительная мутация включает мутацию в положении 12 любой из SEQ ID NO: 6-7 на любой остаток, кроме Phe, Pro, Asp, Glu или Asn. В некоторых вариантах осуществления рекомбинантная UvsX содержит мутацию на Lys в положении в 12 любой из SEQ ID NO: 6-7.
В некоторых вариантах осуществления рекомбиназа представляет собой белок UvsX. Любая фаговая рекомбиназа может быть использована в вариантах осуществления изобретения, представленных в настоящем документе, включая, например, фаговые рекомбиназы, такие как UvsX или UvsX-подобная рекомбиназа, полученные из миовирусных фагов, таких как, например, Т4, Т6, Rb69, Aeh1, KVP40, фаг Acinetobacter 133, фаг Aeromonas 65, цианофаг Р-SSM2, цианофаг PSSM4, цианофаг S-PM2, Rb32, фаг Vibrio nt-1, Rb16, Rb43 и Rb49. В некоторых вариантах осуществления рекомбиназой является UvsX или UvsX-подобная рекомбиназа, полученная из миовирусного фага, такого как, например, Т2, Rb14, фаг Aeromonas 25, phi-1, фаг 31, фаг 44RR2.8t, фаг Rb3 и фаг LZ2. Специалисту в данной области техники будет очевидно, что и другие рекомбиназы могут быть использованы в вариантах осуществления, представленных в данном документе. Подходящие рекомбиназы могут быть идентифицированы по гомологии с UvsX с использованием любого из рядя способов, известных в данной области техники, таких как, например, выравнивание с помощью BLAST, что описано более подробно ниже.
Под «функционально эквивалентной» подразумевается, что контрольная рекомбиназа в случае исследований с использованием полностью другой рекомбиназы будет содержать аминокислотную замену, которую считают возникшей в другой рекомбиназе в аминокислотной позиции, которая имеет ту же функциональную роль в ферменте. В качестве примера, мутация в положении 257 фенилаланина на лизин (F257K) в T4 UvsX будет функционально эквивалентна замене в положении 256 пролина на лизин (P256K) в RB49 UvsX.
В общем, функционально эквивалентные заместительные мутации в двух или нескольких различных рекомбиназах находятся в гомологичных аминокислотных позициях аминокислотных последовательностей рекомбиназ. Следовательно, используемый в данном описании термин «функционально эквивалентная» также включает в себя мутацию, которые являются «позиционно эквивалентной» или «гомологичной» данной мутации, независимо от того, известна ли конкретная функция мутируемой аминокислоты. Можно определить позиционно эквивалентные или гомологичные аминокислотные остатки в аминокислотных последовательностях двух или нескольких различных рекомбиназ на основе выравнивания последовательностей и/или молекулярного моделирования. Пример выравнивания последовательностей для идентификации позиционно эквивалентных и/или функционально эквивалентных остатков приведен на фиг.1, на которой представлено выравнивание аминокислотных последовательностей UvsX из Enterobacteria phage T4 (T4) (SEQ ID NO: 8), Enterobacteria phage T6 (T6) (SEQ ID NO: 9), Acinetobacter phage 133 (фиг) (SEQ ID NO: 10), Enterobacteria phage RB69 (Rb69) (SEQ ID NO: 11), Aeromonas phage Aeh1 (Aeh1) (SEQ ID NO: 12), Aeromonas phage 65 (Ae65) (SEQ ID NO: 13), Vibrio phage KVP40 (Kvp40) (SEQ ID NO: 14), Enterobacteria phage RB43 (Rb43) (SEQ ID NO: 15), Prochlorococcus phage P-SSM2 (PSSM2) (SEQ ID NO: 16) и Prochlorococcus phage P-SSM4 (PSSM4) (SEQ ID NO: 17), которые также изложены в материалах, включенных в US 2009/0029421. Последовательности UvsX, показанные на фиг.1, были получены из последовательностей с номерами доступа в базе данных Genbank: NP_049656 (T4), YP_004300647 (фиг 133); NP_861734 (RB69); NP_943894.1 (Aeh1); YP_004300858 (Ae65); NP_899256 (KVP40); YP_239013 (RB43); YP_214417 (P-SSM2); YP_214708 (P-SSM4); и из публикации патента США № 2009/0029421 (T6).
Фиг.2 представляет собой схематическое изображение выравнивания аминокислотных последовательностей UvsX из Enterobacteria phage RB49 (RB49) (SEQ ID NO: 1) и Enterobacteria phage T4 (T4) (SEQ ID NO: 8). Остатки, которые позиционно и/или функционально эквивалентны Pro256 в аминокислотной последовательности RB49 UvsX, выделены и обозначены треугольником. Последовательности UvsX, приведенные на фиг.2, были получены из последовательностей с номерами доступа в базе данных Genbank: NP_891595 (RB49) и NP_049656 (T4).
Позиционно эквивалентный и/или функционально эквивалентный остаток может быть определен для одной или нескольких из любого числа других последовательностей UvsX путем выравнивания этих последовательностей с эталонной последовательностью, такой как Т4 и RB49. В качестве неограничивающего примера, последовательности UvsX из Synechococcus phage S-PM2, Enterobacteria phage RB32, Vibrio phage nt-1, Enterobacteria phage RB16, представленные в SEQ ID NO: 18-21 и полученные из последовательностей с номерами доступа в базы данных Genbank YP_195169.1; YP_802982.1; YP_008125207.1; YP_003858336.1, могут быть выровнены с эталонной последовательностью UvsX, такой как, например, Т4 UvsX (SEQ ID NO: 8) и RB49 UvsX (SEQ ID NO: 1), и могут быть идентифицированы позиционно эквивалентные и/или функционально эквивалентные остатки. В качестве примера, остатки, показанные в приведенной ниже таблице, идентифицированы как позиционно эквивалентные и/или функционально эквивалентные Pro256 в аминокислотной последовательности RB49 UvsX. Специалисту в данной области техники будет очевидно, что позиционно эквивалентные и/или функционально эквивалентные позиции для других белков UvsX может быть определены с использованием такого же подхода.
Рекомбинантные белки UvsX, описанные выше, могут содержать дополнительные заместительные мутации, которые известны, как улучшающие один или несколько из аспектов активности, стабильности или любого другого желательного свойства рекомбиназы. Например, в некоторых вариантах осуществления в дополнение к любой из вышеуказанных мутаций рекомбинантная UvsX может дополнительно включать заместительные мутации в положениях, функционально эквивалентных His63 в аминокислотной последовательности RB49 UvsX, как известно в данной области техники и проиллюстрировано в патентной публикации США 2009/0029421, которая включена путем ссылки в полном объеме. Например, в некоторых вариантах осуществления рекомбинантная UvsX содержит заместительную мутацию, гомологичную His63Ser в аминокислотной последовательности RB49 UvsX.
В некоторых вариантах осуществления, в дополнение к любой из вышеуказанных мутаций рекомбинантная UvsX может включать дополнительные заместительные, делеционные и/или добавочные мутации по сравнению с рекомбиназой дикого типа. Могут быть осуществлены любые из множества заместительных мутаций в одном или нескольких положениях, известных в данной области техники и проиллюстрированых материалами, включенными в 2009/0029421. Например, в некоторых вариантах осуществления, в дополнение к вышеуказанным мутациям рекомбинантная UvsX может дополнительно содержать мутацию, выбранную из группы, состоящей из: добавления одного или нескольких остатков глутаминовой кислоты на С-конце; добавления одного или нескольких остатков аспарагиновой кислоты на С-конце; и их комбинации.
Мутагенез рекомбиназ
в настоящем изобретении, необязательно, используются различные типы мутагенеза, например, для модификации рекомбиназ, чтобы получить варианты, например, в соответствии с моделями рекомбиназ и прогнозируемыми моделями, или с использованием подходов случайного или полуслучайного мутагенеза. В общем, для получения мутантов рекомбиназ может быть использована любая доступная процедура мутагенеза. Такие процедуры мутагенеза, необязательно, включают отбор мутантных нуклеиновых кислот и полипептидов по одному или нескольким представляющим интерес типам активности (например, улучшенному посеву и/или амплификации на твердой подложке). Процедуры, которые могут быть использованы, включают, но не ограничиваются ими: сайт-направленный точечный мутагенез, случайный точечный мутагенез, in vitro или in vivo гомологичную рекомбинацию (перетасовку ДНК и комбинаторную перекрывающуюся ПЦР), мутагенез с помощью урацил-содержащих матриц, олигонуклеотид-направленный мутагенез, мутагенез с фосфоротиоат-модифицированной ДНК, мутагенез с использованием дуплексной ДНК с разрывом, репарацию точечных несовпадений, мутагенез с использованием дефицитных по репарации штаммов-хозяев, рестрикционный отбор и рестрикционную очистку, делеционный мутагенез, мутагенез путем синтеза всего гена, вырожденную ПЦР, репарацию двухцепочечного разрыва и многие другие, известные специалистам в данной области. Исходной рекомбиназой для мутагенеза может быть любая из тех, которые указаны в настоящем документе, включая доступные мутанты рекомбиназ, такие как те, которые описаны, например, в патентной заявке США 2009/0029421, которая включена путем ссылки в полном объеме.
Необязательно, при проведении мутагенеза можно руководствоваться известной информацией о природной молекуле рекомбиназы или известной измененной или мутантной рекомбиназы (например, используя существующую мутантную рекомбиназу, указанную в предшествующих ссылках), например, иноформацией о последовательности, сравнении последовательностей, физических свойствах, кристаллической структуре и/или т.п., как описано выше. Однако в другом классе вариантов осуществления, модификации могут быть по существу случайными (например, в классической или «семейственной» перетасовке ДНК, см., например, Crameri et al. (1998) "DNA shuffling of a family of genes from diverse species accelerates directed evolution" Nature 391:288-291).
Дополнительную информацию о форматах мутаций можно найти в: Sambrook et al., Molecular Cloning--A Laboratory Manual (3rd Ed.), Vol. 1-3, Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y., 2000 ("Sambrook"); Current Protocols in Molecular Biology, F. M. Ausubel et al., eds., Current Protocols, совместное издание Greene Publishing Associates, Inc. и John Wiley & Sons, Inc. (дополненное в 2011) ("Ausubel")) и PCR Protocols A Guide to Methods and Applications (Innis et al. eds) Academic Press Inc. San Diego, Calif. (1990) ("Innis"). Следующие публикации и ссылки, приведенные в них, предоставляют дополнительную информацию о форматах мутаций: Arnold, Protein engineering for unusual environments, Current Opinion in Biotechnology 4:450-455 (1993); Bass et al., Mutant Trp repressors with new DNA-binding specificities, Science 242:240-245 (1988); Bordo and Argos (1991) Suggestions for "Safe" Residue Substitutions in Site-directed Mutagenesis 217:721-729; Botstein & Shortle, Strategies and applications of in vitro mutagenesis, Science 229:1193-1201 (1985); Carter et al., Improved oligonucleotide site-directed mutagenesis using M13 vectors, Nucl. Acids Res. 13: 4431-4443 (1985); Carter, Site-directed mutagenesis, Biochem. J. 237:1-7 (1986); Carter, Improved oligonucleotide-directed mutagenesis using M13 vectors, Methods in Enzymol. 154: 382-403 (1987); Dale et al., Oligonucleotide-directed random mutagenesis using the phosphorothioate method, Methods Mol. Biol. 57:369-374 (1996); Eghtedarzadeh & Henikoff, Use of oligonucleotides to generate large deletions, Nucl. Acids Res. 14: 5115 (1986); Fritz et al., Oligonucleotide-directed construction of mutations: a gapped duplex DNA procedure without enzymatic reactions in vitro, Nucl. Acids Res. 16: 6987-6999 (1988); Grundstrom et al., Oligonucleotide-directed mutagenesis by microscale `shot-gun` gene synthesis, Nucl. Acids Res. 13: 3305-3316 (1985); Hayes (2002) Combining Computational and Experimental Screening for rapid Optimization of Protein Properties PNAS 99(25) 15926-15931; Kunkel, The efficiency of oligonucleotide directed mutagenesis, in Nucleic Acids & Molecular Biology (Eckstein, F. and Lilley, D. M. J. eds., Springer Verlag, Berlin)) (1987); Kunkel, Rapid and efficient site-specific mutagenesis without phenotypic selection, Proc. Natl. Acad. Sci. USA 82:488-492 (1985); Kunkel et al., Rapid and efficient site-specific mutagenesis without phenotypic selection, Methods in Enzymol. 154, 367-382 (1987); Kramer et al., The gapped duplex DNA approach to oligonucleotide-directed mutation construction, Nucl. Acids Res. 12: 9441-9456 (1984); Kramer & Fritz Oligonucleotide-directed construction of mutations via gapped duplex DNA, Methods in Enzymol. 154:350-367 (1987); Kramer et al., Point Mismatch Repair, Cell 38:879-887 (1984); Kramer et al., Improved enzymatic in vitro reactions in the gapped duplex DNA approach to oligonucleotide-directed construction of mutations, Nucl. Acids Res. 16: 7207 (1988); Ling et al., Approaches to DNA mutagenesis: an overview, Anal Biochem. 254(2): 157-178 (1997); Lorimer and Pastan Nucleic Acids Res. 23, 3067-8 (1995); Mandecki, Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis, Proc. Natl. Acad. Sci. USA, 83:7177-7181(1986); Nakamaye & Eckstein, Inhibition of restriction endonuclease Nci I cleavage by phosphorothioate groups and its application to oligonucleotide-directed mutagenesis, Nucl. Acids Res. 14: 9679-9698 (1986); Nambiar et al., Total synthesis and cloning of a gene coding for the ribonuclease S protein, Science 223: 1299-1301(1984); Sakamar and Khorana, Total synthesis and expression of a gene for the a-subunit of bovine rod outer segment guanine nucleotide-binding protein (transducin), Nucl. Acids Res. 14: 6361-6372 (1988); Sayers et al., Y-T Exonucleases in phosphorothioate-based oligonucleotide-directed mutagenesis, Nucl. Acids Res. 16:791-802 (1988); Sayers et al., Strand specific cleavage of phosphorothioate-containing DNA by reaction with restriction endonucleases in the presence of ethidium bromide, (1988) Nucl. Acids Res. 16: 803-814; Sieber, et al., Nature Biotechnology, 19:456-460 (2001); Smith, In vitro mutagenesis, Ann. Rev. Genet. 19:423-462 (1985); Methods in Enzymol. 100: 468-500 (1983); Methods in Enzymol. 154: 329-350 (1987); Stemmer, Nature 370, 389-91(1994); Taylor et al., The use of phosphorothioate-modified DNA in restriction enzyme reactions to prepare nicked DNA, Nucl. Acids Res. 13: 8749-8764 (1985); Taylor et al., The rapid generation of oligonucleotide-directed mutations at high frequency using phosphorothioate-modified DNA, Nucl. Acids Res. 13: 8765-8787 (1985); Wells et al., Importance of hydrogen-bond formation in stabilizing the transition state of subtilisin, Phil. Trans. R. Soc. Lond. A 317: 415-423 (1986); Wells et al., Cassette mutagenesis: an efficient method for generation of multiple mutations at defined sites, Gene 34:315-323 (1985); Zoller & Smith, Oligonucleotide-directed mutagenesis using M 13-derived vectors: an efficient and general procedure for the production of point mutations in any DNA fragment, Nucleic Acids Res. 10:6487-6500 (1982); Zoller & Smith, Oligonucleotide-directed mutagenesis of DNA fragments cloned into M13 vectors, Methods in Enzymol. 100:468-500 (1983); Zoller & Smith, Oligonucleotide-directed mutagenesis: a simple method using two oligonucleotide primers and a single-stranded DNA template, Methods in Enzymol. 154:329-350 (1987); Clackson et al. (1991) "Making antibody fragments using phage display libraries" Nature 352:624-628; Gibbs et al. (2001) "Degenerate oligonucleotide gene shuffling (DOGS): a method for enhancing the frequency of recombination with family shuffling" Gene 271:13-20; и Hiraga and Arnold (2003) "General method for sequence-independent site-directed chimeragenesis: J. Mol. Biol. 330:287-296. Дополнительные сведения о многих из указанных выше способов можно найти в Methods Enzymology, том 154, где также описаны полезные методы контроля для устранения проблем при различных способах мутагенеза.
Получение и выделение рекомбинантной рекомбиназы
В общем, нуклеиновые кислоты, кодирующие рекомбиназу, представленную в настоящем описании, могут быть получены путем клонирования, рекомбинации, in vitro синтеза, in vitro амплификации и/или другими доступными способами. Множество рекомбинантных способов может быть использовано для экспрессии экспрессионного вектора, который кодирует рекомбиназу, представленную в данном описании. Способы получения рекомбинантных нуклеиновых кислот, экспрессии и выделения экспрессированных продуктов хорошо известны и описаны в данной области. Ряд иллюстративных мутаций и комбинаций мутаций, а также стратегии создания желаемых мутаций, описаны в данном документе.
Дополнительные полезные ссылки на способы проведения мутагенеза, рекомбинантных и in vitro способов манипуляций с нуклеиновыми кислотами (включая клонирование, экспрессию, ПЦР и т.п.) включают Berger and Kimmel, Guide to Molecular Cloning Techniques, Methods in Enzymology volume 152 Academic Press, Inc., San Diego, Calif. (Berger); Kaufman et al. (2003) Handbook of Molecular and Cellular Methods in Biology and Medicine Second Edition Ceske (ed) CRC Press (Kaufman); и The Nucleic Acid Protocols Handbook Ralph Rapley (ed) (2000) Cold Spring Harbor, Humana Press Inc (Rapley); Chen et al. (ed) PCR Cloning Protocols, Second Edition (Methods in Molecular Biology, volume 192) Humana Press; и Viljoen et al. (2005)Molecular Diagnostic PCR Handbook Springer, ISBN 1402034032.
Кроме того, в продаже доступно множество наборов для очистки плазмид или других соответствующих нуклеиновых кислот из клеток (см., например, EasyPrep™, FlexiPrep™, оба от Pharmacia Biotech; StrataClean™ от Stratagene; и QIAprep™ от Qiagen). С любой выделенной и/или очищенной нуклеиновой кислотой могут быть проведены дополнительные манипуляции, чтобы получить другие нуклеиновые кислоты, используемые для трансфекции клеток, включенные в соответствующие векторы, для заражения организмов для экспрессии и/или других целей. Типичные клонирующие векторы содержат транскрипционные и трансляционные терминаторы, последовательность инициации трансляции и транскрипции и промоторы, подходящие для регуляции экспрессии конкретной нуклеиновой кислоты-мишени. Векторы, необязательно, включают универсальные кассеты экспрессии, содержащие по меньшей мере одну независимую терминаторную последовательность, последовательности, разрешающие репликацию кассеты в эукариотах или прокариот, или них обоих (например, челночные векторы), и селективные маркеры для обоих прокариотических и эукариотических систем. Векторы подходят для репликации и интеграции в прокариотах, эукариотах или в них обоих.
Другие полезные ссылки, например, для выделения клеток и культур (например, для последующего выделения нуклеиновых кислот) включают Freshney (1994) Culture of Animal Cells, a Manual of Basic Technique, third edition, Wiley-Liss, New York и приведенные там ссылки; Payne et al. (1992) Plant Cell and Tissue Culture in Liquid Systems John Wiley & Sons, Inc. New York, N.Y.; Gamborg and Phillips (eds) (1995) Plant Cell, Tissue and Organ Culture; Fundamental Methods Springer Lab Manual, Springer-Verlag (Berlin Heidelberg New York) и Atlas and Parks (eds) The Handbook of Microbiological Media (1993) CRC Press, Boca Raton, Fla.
Нуклеиновые кислоты, кодирующие рекомбинантные рекомбиназы из раскрытых в настоящем документе, также являются признаком вариантов осуществления, представленных в настоящем документе. Одна аминокислота может кодироваться несколькими кодонами, и некоторые системы трансляции (например, прокариотические или эукариотические клетки) часто демонстрируют избирательность в отношении кодонов, например, разные организмы часто предпочитают один из нескольких синонимичных кодонов, которые кодируют одну и ту же аминокислоту. Как таковые, нуклеиновые кислоты по настоящему изобретению, необязательно, являются «кодон-оптимизированными», что означает, что нуклеиновые кислоты синтезируют таким образом, чтобы они включали кодоны, которые являются предпочтительными для конкретной системы трансляции, используемой для экспрессии рекомбиназы. Например, если желательно получить экспрессию рекомбиназы в бактериальной клетке (или даже в конкретном бактериальном штамме), для эффективной экспрессии рекомбиназы нуклеиновая кислота может быть синтезирована таким образом, чтобы она включала кодоны, наиболее часто встречающиеся в геноме этой бактериальной клетки. Аналогичная стратегия может быть использована, когда желательно получить экспрессию рекомбиназы в эукариотической клетке, например, нуклеиновая кислота может включать в себя кодоны, предпочитаемые этой эукариотической клеткой.
Множество способов выделения и обнаружения белка известны и могут быть использованы для выделения рекомбиназы, например, из культур рекомбинантных клеток, экспрессирующих рекомбинантные рекомбиназы, представленные в настоящем документе. В данной области техники хорошо известны множество способов выделения и обнаружения белка, включая, например, те, которые приведены в R. Scopes, Protein Purification, Springer-Verlag, N.Y. (1982); Deutscher, Methods in Enzymology Vol. 182: Guide to Protein Purification, Academic Press, Inc. N.Y. (1990); Sandana (1997) Bioseparation of Proteins, Academic Press, Inc.; Bollag et al. (1996) Protein Methods, 2nd Edition Wiley-Liss, NY; Walker (1996) The Protein Protocols Handbook Humana Press, NJ, Harris and Angal (1990) Protein Purification Applications: A Practical Approach IRL Press at Oxford, Oxford, England; Harris and Angal Protein Purification Methods: A Practical Approach IRL Press at Oxford, Oxford, England; Scopes (1993) Protein Purification: Principles and Practice 3rd Edition Springer Verlag, NY; Janson and Ryden (1998) Protein Purification: Principles, High Resolution Methods and Applications, Second Edition Wiley-VCH, NY; и Walker (1998) Protein Protocols on CD-ROM Humana Press, NJ; и приведенные в них ссылки. Дополнительные подробности, касающиеся способов очистки и обнаружения белков, можно найти в Satinder Ahuja ed., Handbook of Bioseparations, Academic Press (2000).
Способы применения
Измененные рекомбиназы, представленные в настоящем документе, могут быть использованы в процедуре рекомбиназо-опосредованной амплификации, такой как метод рекомбиназной полимеразной амплификации (RPA). Вкратце, RPA может инициироваться путем контактирования нуклеиновой кислоты-мишени с рекомбиназой и одноцепочечным нуклеотидным праймером, специфичным к молекуле нуклеиновой кислоты-мишени. Гибридизованный праймер может быть удлинен с помощью полимеразы, такой как полимераза, способная к вытеснению цепи в присутствии смеси dNTP, с образованием двухцепочечной молекулы нуклеиновой кислоты-мишени и вытесненной цепи молекулы нуклеиновой кислоты. Дальнейшая амплификация может происходить путем рекомбиназо-опосредованного привлечения праймеров к вытесненной цепи молекулы нуклеиновой кислоты и удлинения праймера, с образованием двухцепочечной молекулы нуклеиновой кислоты. Процесс RPA может модулироваться путем объединения описанных выше компонентов, например, факторов-загрузчиков рекомбиназы, специфичных вытесняющих полимераз и надежной системы регенерации энергии. Примеры способов, систем и компонентов RPA, которые могут быть легко адаптированы для использования с рекомбинантными белками UvsX по настоящему изобретению, описаны, например, в патентах США №№ 8071308, 7399590, 7485428, 7270981, 8030000, 7666598, 7763427, 8017399, 8062850 и 7435561, каждый из которых включен в данное описание путем ссылки.
В некоторых вариантах осуществления, изотермическая амплификация может быть осуществлена с использованием динамической исключающей амплификации (КЕА), также называемой как исключающей амплификацией (ExAmp). Библиотека нуклеиновых кислот по настоящему изобретению может быть изготовлена с использованием способа, в котором используется динамическое исключение. Динамическое исключение может осуществляться, когда процесс происходит при достаточно быстрой скорости, чтобы эффективно исключить возникновение другого события или процесса. Если взять, например, создание массива нуклеиновых кислот, где сайты массива случайным образом «засеваются» нуклеиновыми кислотами-мишенями из раствора, и копии нуклеиновой кислоты-мишени образуются в процессе амплификации, заполняя каждый из засеянных участков до его емкости. В соответствии со способами динамического исключения по настоящему изобретению, процессы посева и амплификации могут протекать одновременно в условиях, когда скорость амплификации превышает скорость посева. Таким образом, относительно высокая скорость, с которой копии синтезируются на сайте, который был «засеян» с помощью первой нуклеиновой кислоты-мишени, будет эффективно препятствовать («исключать») посев второй нуклеиновой кислоты на этом сайте амплификации. Способы динамической исключающей амплификации могут быть выполнены, как описано подробно в раскрытии публикации заявки на патент США № 2013/0338042, которая включена в настоящее описание путем ссылки во всей полноте.
В некоторых вариантах осуществления амплифицируемая нуклеиновая кислота-мишень является полностью двухцепочечной. В некоторых вариантах осуществления амплифицируемая нуклеиновая кислота-мишень содержит область двухцепочечной нуклеиновой кислоты, а также включает область, имеющую одноцепочечную нуклеиновую кислоту. В некоторых вариантах осуществления нуклеиновая кислота-мишень содержит один или несколько раздвоенных адаптеров с областью около 5, 10, 15, 20, 25, 30, 35, 40 или более чем примерно 40 оснований одноцепочечной последовательности на каждом конце фрагментов библиотеки. Конструкция и использование раздвоенных адаптеров описаны более подробно в описании патентов США №№ 7742463 и 8563748, каждый из которых включен в данное описание путем ссылки во всей полноте.
В процессе динамического исключения может использоваться относительно низкая скорость синтеза первой копии нуклеиновой кислоты-мишени по сравнению с относительно высокой скоростью синтеза последующих копий нуклеиновой кислоты-мишени или первой копии. В примере предшествующего параграфа, динамическое исключение возникает вследствие относительно низкой скорости посева нуклеиновой кислоты-мишени (например, относительно медленных диффузии или переноса) по сравнению с относительно высокой скоростью, при которой происходит амплификация, чтобы заполнить участок (сайт) копиями посеянной нуклеиновой кислоты. В другом иллюстративном варианте осуществления динамическое исключение может возникать вследствие задержки в образовании первой копии нуклеиновой кислоты-мишени, которая была посеяна на сайте (например, отложенная или медленная активация) по сравнению с относительно высокой скоростью, с которой синтезируются последующие копии, заполняющие сайт. В этом примере, на индивидуальном сайте могут быть посеяны несколько различных нуклеиновых кислот-мишеней (например, несколько нуклеиновых кислот-мишеней могут присутствовать на каждом сайте до амплификации). Однако образование первой копии для любой данной нуклеиновой кислоты-мишени может активироваться случайным образом, так что средняя скорость образования первой копии является относительно медленной по сравнению со скоростью, с которой синтезируются последующие копии. В этом случае, несмотря на то, что на отдельном сайте, возможно, были посеяны несколько различных нуклеиновых кислот-мишеней, динамическое исключение позволит амплифицироваться только одной из этих нуклеиновых кислот-мишеней. Более конкретно, после того как первая нуклеиновая кислота-мишень активируется для амплификации, сайт будет быстро заполнен до конца ее копиями, тем самым предотвращая синтез копий второй нуклеиновой кислоты-мишени на этом сайте.
Амплификационный реагент может включать дополнительные компоненты, которые облегчают образование ампликона, а в некоторых случаях, увеличивают скорость образования ампликона. Рекомбиназа, такая как, например, UvsX, может облегчить образование ампликона, позволяя повторное внедрение/удлинение. Более конкретно, рекомбиназа может облегчить внедрение нуклеиновой кислоты-мишени с помощью полимеразы и удлинение праймера полимеразой с использованием нуклеиновой кислоты-мишени в качестве матрицы для формирования ампликона. Этот процесс может быть повторен в виде цепной реакции, где ампликоны, полученные из каждого раунда внедрения/удлинения, служат в качестве матриц в последующем раунде. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку не требуется цикл денатурации (например, путем нагрева или химической денатурации). Таким образом, рекомбиназо-опосредованную амплификацию можно проводить изотермически. В общем случае, для проведения амплификации желательно включать АТР или другие нуклеотиды (или в некоторых случаях - их негидролизуемые аналоги) в реагент для рекомбиназо-опосредованной амплификации. Смесь рекомбиназы и связывающего одноцепочечные нуклеиновые кислоты (SSB) белка является особенно полезной, поскольку SSB может дополнительно способствовать амплификации. Типичные составы для рекомбиназо-опосредованной амплификации включают в себя те, которые продаются в качестве наборов TwistAmp от TwistDx (Cambridge, UK). Полезные компоненты реагента для рекомбиназо-опосредованной амплификации и условия реакции приведены в US 5223414 и US 7399590, каждый из которых включен в данное описание путем ссылки.
Сравнение, идентичность и гомология последовательностей
Термины «идентичный» или «процент идентичности» в контексте двух или нескольких последовательностей нуклеиновых кислот или полипептидов относятся к двум или нескольким последовательностям или подпоследовательностям, которые являются одинаковыми или имеют определенный процент одинаковых аминокислотных остатков или нуклеотидов при сравнении и выравнивании для максимального соответствия, который измеряется с использованием одного из алгоритмов сравнения последовательностей, описанных ниже (или других алгоритмов, доступных для специалистов), или путем визуальной проверки.
Фраза «по существу идентичны» в контексте двух нуклеиновых кислот или полипептидов (например, ДНК, кодирующих рекомбиназу, или аминокислотных последовательностей рекомбиназ) относится к двум или нескольким последовательностям или подпоследовательностям, которые имеют по меньшей мере примерно 60%, примерно 80%, примерно 90-95%, примерно 98%, примерно 99% или больший процент идентичности нуклеотидных или аминокислотных остатков при сравнении и выравнивании для максимального соответствия, который измеряется с использованием алгоритма сравнения последовательностей или путем визуальной проверки. Такие «по существу идентичные» последовательности, как правило, считаются «гомологичными» без ссылки на фактического предшественника. Предпочтительно, чтобы «значительная идентичность» присутствовала на протяжении области последовательностей, которая имеет по меньшей мере около 50 остатков в длину, более предпочтительно, на протяжении области по меньшей мере около 100 остатков в длину и, наиболее предпочтительно, чтобы последовательности были по существу идентичными на протяжении по меньшей мере примерно 150 остатков или по всей длине двух сравниваемых последовательностей.
Белки и/или белковые последовательности являются «гомологичными», когда они получены в природе или искусственно из общего родительского белка или белковой последовательности. Аналогичным образом, нуклеиновые кислоты и/или последовательности нуклеиновых кислот являются гомологичными, когда они получены в природе или искусственно из общей родительской нуклеиновой кислоты или последовательности нуклеиновой кислоты. Гомология, как правило, основана на сходстве последовательностей у двух или нескольких нуклеиновых кислот или белков (или их последовательностей). Точный процент сходства между последовательностями, который соответствует установлению гомологии, варьирует в зависимости от рассматриваемых нуклеиновой кислоты и белка, но всего лишь 25% сходства последовательностей на протяжении 50, 100, 150 или большего числа остатков обычно используется для установления гомологии. Более высокий уровень сходства последовательностей, например, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% или 99% или более, также может использоваться для установления гомологии. Способы определения сходства последовательностей в процентах (например, BLASTP и BLASTN с использованием параметров по умолчанию) описаны в данном документе и повсеместно доступны.
Для сравнения последовательностей и определения гомологии, как правило, одна последовательность выступает в качестве эталонной последовательности, с которой сравниваются тестируемые последовательности. При использовании алгоритма сравнения последовательностей, тестируемую и эталонную последовательности вводят в компьютер, в случае необходимости указывают координаты подпоследовательностей и указывают параметры для программы сравнения последовательностей. С использованием алгоритма сравнения последовательностей затем вычисляют процент идентичности последовательностей для тестируемой последовательности (тестируемых последовательностей) относительно эталонной последовательности на основании указанных параметров программы.
Оптимальное выравнивание последовательностей для сравнения можно проводить, например, с помощью алгоритма локальной гомологии Smith & Waterman, Adv. Appl. Math. 2:482 (1981), с помощью алгоритма выравнивания по гомологии Needleman & Wunsch, J. Mol. Biol. 48: 443 (1970), с помощью метода поиска сходства Pearson & Lipman, Proc. Nat'l. Acad. Sci. USA 85: 2444 (1988), с помощью компьютерных реализаций этих алгоритмов (GAP, BESTFIT, FASTA и TFASTA в Wisconsin Genetics Software Package, Genetics Computer Group, 575 Science Dr., Madison, WI) или путем визуальной проверки (см., в общем Current Protocols in Molecular Biology, Ausubel et al., eds., Current Protocols, совместное издание Greene Publishing Associates, Inc. и John Wiley & Sons, Inc., дополненное в 2004).
Одним примером алгоритма, который подходит для определения процента идентичности последовательностей и сходства последовательностей, является алгоритм BLAST, который описан в Altschul et al., J. Mol. Biol. 215: 403-410 (1990). Программное обеспечение для осуществления BLAST-анализа является общедоступным через Национальный центр биотехнологической информации. Этот алгоритм включает первичную идентификацию пар последовательностей с высокими баллами (HSP) путем идентификации в запрашиваемой последовательности коротких слов с длиной W, которые или совпадают, или удовлетворяют некоторому положительно-оцениваемому пороговому баллу Т при выравнивании со словом той же длины в последовательности из базы данных. Т называется пороговым баллом соседних слов (Altschul et al., см.выше). Эти начальные совпадения соседних слов действуют в качестве затравки для инициации поиска, целью которого является идентификация более длинных HSP, содержащих их. Совпадения слов затем расширяют в двух направлениях по каждой последовательности до тех пор, пока кумулятивная оценка выравнивания может увеличиваться. Кумулятивная оценка вычисляется с использованием следующих параметров для нуклеотидных последовательностей: М (наградные баллы для пары совпадающих остатков; всегда >0) и N (штрафные баллы за несовпадающие остатки; всегда <0). В случае аминокислотных последовательностей для вычисления кумулятивной оценки используется матрица баллов. Удлинение совпадений слов в каждом направлении прекращают, когда: кумулятивная оценка выравнивания падает на величину X от своего максимального достигнутого значения; кумулятивная оценка стремится к нулю или становится отрицательной вследствие накопления одного или нескольких выравниваний остатков, дающих отрицательные баллы; или достигнут конец какой-либо последовательности. Параметры W, Т и Х в алгоритме BLAST определяют чувствительность и скорость выравнивания. В программе BLASTN (для нуклеотидных последовательностей) используют в качестве значений по умолчанию: длину слова (W), равную 11, ожидание (Е), равное 10, порог 100, M=5, N=-4 и сравнение обеих цепей. Для аминокислотных последовательностей в программе BLASTP используют в качестве значений по умолчанию: длину слова (W), равную 3, ожидание (Е), равное 10 и матрицу баллов BLOSUM62 (см. Henikoff & Henikoff (1989), Proc. Natl. Acad. Sci. USA 89: 10915).
В дополнении к вычислению процента идентичности последовательностей, алгоритм BLAST также выполняет статистический анализ сходства между двумя последовательностями (см., например, Karlin & Altschul, Proc. Nat'l. Acad. Sci. USA 90: 5873-5787 (1993)). Одной мерой сходства, обеспечиваемой алгоритмом BLAST, является наименьшая суммарная вероятность (P (N)), указывающая вероятность, с которой совпадение между двумя нуклеотидными или аминокислотными последовательностями будет происходить случайно. Например, нуклеиновая кислота считается похожей на эталонную последовательность, если наименьшая суммарная вероятность при сравнении тестируемой нуклеиновой кислоты с эталонной нуклеиновой кислотой составляет менее примерно 0,1, более предпочтительно, менее примерно 0,01 и, наиболее предпочтительно, менее примерно 0,001.
Нуклеиновые кислоты, кодирующие измененные рекомбиназы
Дополнительно в настоящем описании представлены молекулы нуклеиновых кислот, кодирующих измененные рекомбиназы, представленные в настоящем документе. Согласно базовым принципам молекулярной биологии для любой данной измененной рекомбиназы, которая представляет собой мутантный вариант рекомбиназы, для которой известна аминокислотная последовательность, а также, предпочтительно, нуклеотидная последовательность дикого типа, кодирующая эту рекомбиназу, можно получить кодирующую мутант нуклеотидную последовательность. Например, учитывая, что известна нуклеотидная последовательность дикого типа, кодирующая рекомбиназу RB49 UvsX, с использованием стандартного генетического кода можно вывести нуклеотидную последовательность, кодирующую любой данный мутантный вариант RB49 UvsX, имеющий одну или несколько аминокислотных замен. Аналогичным образом, нуклеотидные последовательности могут быть легко получены для мутантных версий других рекомбиназ, таких как, например, T4,, Rb69, Aeh1, KVP40, фаг Acinetobacter 133, фаго Aeromonas phage 65, цианофаг P-SSM2, цианофаг PSSM4, цианофаг S-PM2, Rb32, фаг Vibrio nt-1, Rb16, Rb43, T2, Rb14, фаг Aeromonas 25, phi-1, фаг 31, фаг 44RR2.8t, фаг Rb3 и фаг LZ2 и т.д. Молекулы нуклеиновых кислот, содержащие требуемую нуклеотидную последовательность, могут затем быть сконструированы с использованием стандартных методов молекулярной биологии, известных в данной области техники.
В соответствии с вариантами осуществления, представленными в настоящем описании, указанная нуклеиновая кислота включает не только идентичную нуклеиновую кислоту, но также и любые незначительные вариации оснований, включая, в частности, замены в случаях, которые приводят к синонимичному кодону (другому кодону, кодирующему такой же аминокислотный остаток) вследствие вырожденности кода в консервативных аминокислотных заменах. Термин «последовательность нуклеиновой кислоты» также включает в себя последовательность, комплементарную любой одноцепочечной последовательности, приведенной в отношении вариаций оснований.
Молекулы нуклеиновых кислот, описанные в данном документе, могут также, предпочтительно, быть включены в подходящий экспрессионный вектор для экспрессии кодируемых ими рекомбиназ в подходящем хозяине. Включение клонированной ДНК в подходящий экспрессионный вектор для последующей трансформации указанной клетки и последующий отбор трансформированных клеток хорошо известны специалистам в данной области техники и приведены в руководстве Sambrook et al. (1989), Molecular cloning: A Laboratory Manual, Cold Spring Harbor Laboratory, которое включено путем ссылки во всей полноте.
Такой экспрессионный вектор включает вектор, имеющий нуклеиновую кислоту в соответствии с вариантами осуществления, представленными в настоящем описании, функционально связанную с регуляторными последовательностями, такими как промоторные области, которые способны осуществлять экспрессию указанных фрагментов ДНК. Термин «функционально связанная» относится к такому совместному расположению, при котором описанные компоненты являются взаимосвязанными, что позволяет им функционировать заданным образом. Такие векторы могут быть трансформированы в подходящую клетку-хозяина, чтобы обеспечить экспрессию белка в соответствии с вариантами осуществления, представленными в данном документе.
Молекула нуклеиновой кислоты может кодировать зрелый белок или белок, имеющий пропоследовательность, включая последовательность, кодирующую лидерную последовательность на пребелке, которая затем отщепляется клеткой-хозяином с образованием зрелого белка. Векторы могут представлять собой, например, плазмидные, вирусные или фаговые векторы, снабженные точкой инициации репликации и, необязательно, промотором для экспрессии указанного нуклеотида и, необязательно, регулятором промотора. Векторы могут содержать один или несколько селективных маркеров, таких как, например, ген устойчивости к антибиотику.
Регуляторные элементы, необходимые для экспрессии, включают промоторные последовательности для связывания РНК-полимеразы и для обеспечения соответствующего уровня инициации транскрипции, а также последовательности инициации трансляции для связывания с рибосомой. Например, бактериальный экспрессионный вектор может включать в себя промотор, такой как lac-промотор и для инициации трансляции - последовательность Шайна-Дальгарно и старт-кодон AUG. Аналогичным образом, эукариотической экспрессионный вектор может включать гетерологичный или гомологичный промотор для РНК-полимеразы II, расположенный ниже сигнал полиаденилирования, старт-кодон AUG и терминирующий кодон для отсоединения рибосомы. Такие векторы могут быть получены из коммерческих источников или могут быть собраны из описанных последовательностей способами, хорошо известными в данной области техники.
Транскрипция ДНК, кодирующей рекомбиназу, высшими эукариотами может быть оптимизирована путем включения в вектор энхансерной последовательности. Энхансеры представляют собой цис-действующие элементы ДНК, которые действуют на промотор, повышая уровень транскрипции. Векторы также обычно включают точку инициации репликации в дополнении к селективным маркерам.
ПРИМЕР 1
В этом примере приведены способы посева ПЦР-свободной библиотеки на разграфленную поверхность проточной ячейки для улучшения кластерной амплификации. В одном варианте осуществления в способе по изобретению используют состав для посева, который включает UvsX, содержащую мутации, указанные выше, например, мутанта RB49 UvsX, содержащего Pro256Lys (приведенного в настоящем описании как SEQ ID NO: 2, указывается в настоящем описании как «RB49 P256K»). Было неожиданно обнаружено, что рекомбиназо-опосредованная амплификация с его использованием существенно улучшает посев ПЦР-библиотек с одноцепочечными адаптерными областями на разграфленную поверхность проточной ячейки. В другом варианте осуществления, в способе по изобретению используют состав для посева, который включает относительно высокую концентрацию ДНК-полимеразы (например, полимеразы eBsu) в комбинации с рекомбиназой RB49 P256K.
Для того чтобы оценить эффективность состава RB49 P256K в посеве ПЦР-свободной библиотеки на разграфленную поверхность проточной ячейки, ПЦР-свободная библиотека была создана с помощью набора для приготовления образцов TruSeq® DNA PCR-free (Illumina, Inc.). ПЦР-свободные библиотеки, созданные с использованием набора для получения библиотек TruSeq®, имеют раздвоенные адаптеры с областью около 40 оснований одноцепочечной последовательности на каждом конце фрагментов библиотеки.
На фиг.3А показан снимок с экрана 100 изображения кластеров ПЦР-свободной библиотеки, посеянной на разграфленную проточную ячейку с использованием стандартного состава, содержащего рекомбиназу Т4 UvsX (приведенную в настоящем описании как SEQ ID NO: 8, указанную в настоящем описании как «T4 UvsX»). На фиг.3В показан снимок с экрана 150 изображения кластеров ПЦР-свободной библиотеки, посеянной на разграфленную проточную ячейку с использованием жидкого состава, который включает рекомбиназу RB49 P256K. В этом примере библиотеку смешивали с составом Т4 UvsX или составом RB49 P256K до конечной концентрации 100 пМ, которым промывали проточную ячейку и инкубировали на cBot при 38°C. После инкубации в течение 1 часа температуру понижали до 20°С, и проточную ячейку промывали буфером для промывки НТ2 (Illumina). Кластеры окрашивали SYBR® Green (Life Technologies), разведенным 1:5000 в 0,1 М Tris/0,1 М аскорбате натрия и получали изображения на флуоресцентном микроскопе. В отношении фиг.3А плотность кластеров, полученная посевом ПЦР-свободной библиотеки на разграфленную проточную ячейку со стандартным составом (например, Т4 UvsX), является относительно невысокой. В отношении фиг.3В плотность кластеров, полученная посевом ПЦР-свободной библиотеки на разграфленную проточную ячейку с использованием состава, который включает рекомбиназу RB49 P256K, существенно увеличивается.
На фиг.4А показан снимок с экрана 200 изображения кластеров одноцепочечной (оцДНК) ПЦР-свободной библиотеки, посеянной на разграфленную проточную ячейку с использованием стандартного состава Т4 UvsX. На фиг.4В показан снимок с экрана 250 изображения кластеров одноцепочечной (оцДНК) ПЦР-свободной библиотеки, посеянной на разграфленную проточную ячейку с использованием жидкого состава, который включает рекомбиназу RB49 P256K. В этом примере двухцепочечную ПЦР-свободную библиотеку денатурировали с помощью NaOH, а затем высевали на разграфленную проточную ячейку в концентрации 50 мкМ. Как показано на фиг.4A, плотность кластеров, полученная путем посева оцДНК ПЦР-свободной библиотеки ДНК на разграфленную проточную ячейку со стандартным составом (например, Т4 UvsX), является относительно невысокой. Как показано на фиг.4B, плотность кластеров, полученная путем посева оцДНК ПЦР-свободной библиотеки ДНК на разграфленную проточную ячейку с использованием состава, который включает рекомбиназу RB49 P256K, существенно увеличивается.
ПРИМЕР 2
Улучшенная амплификации с использованием мутантов RB49 P256
В этом примере описано сравнение амплификационной производительности между рекомбиназами с описанной в данном документе мутацией P256K и без нее. В этом примере «контрольная» рекомбиназа RB49 UvsX (приведена в SEQ ID NO: 1) дополнительно содержит мутацию H63S. Мутанта P256K получали путем дополнительного мутагенеза контрольного фермента, чтобы он нес остаток Lys в положении 256, что представлено в данном документе в SEQ ID NO: 2).
Кластеризацию ПЦР-свободной библиотеки на разграфленную проточную ячейку выполняли на cBot, как описано выше в примере 1, с использованием либо контроля, либо мутанта P256K. Секвенирование затем выполняли на инструменте HiSeq (Illumina, Inc.), и результаты секвенирования анализировали, чтобы определить «отвечаемость» ряда областей, которые, как правило, слабо представлены в пуле данных от предшествующих секвенирований.
«Отвечаемость» является мерой доли участков, в которых корректно идентифицируется однонуклеотидный полиморфизм (SNP). В идеале это значение равно 1 (для 100% участков), означая, что на 100% участков в пределах области определенного типа (т.е. с высоким содержанием GC, т.д.) SNP идентифицируются корректно. «Покрытие» является мерой доли участков, которые имеют покрытие >n, где n обычно 30х (то есть, стандарт покрытия для генома человека). Фосмидные промоторы представляют собой набор из промоторов 100 генов, которые были идентифицированы как слабо представлены в данных предыдущих секвенирований. Промоторы были клонированы в фосмидные векторы. Область с высоким содержанием GC может быть определена как область с по меньшей мере 100 п.о., где содержание GC равно или выше 75% (N50 (G+C≥0,75) 100 N50). Область с очень высоким содержанием GC может быть определена как область с по меньшей мере 100 п.о., где содержание GC равно или выше 85% (N50 (G+C≥0,85) 100 N50). Область с низким содержанием GC может быть определена как область с по меньшей мере 100 п.о., где содержание GC равно или меньше 40% (N50 (G+C≥0,40) 100 N50). Область с высоким содержанием АТ может быть определена как область с по меньшей мере 100 п.о., где содержание АТ равно или выше 75% (N50 (A+T≥0,75) 100 N50), с размером области сниженным приблизительно до 50 т.п.о. Область с очень высоким содержанием АТ может быть определена как область с по меньшей мере 100 п.о., где содержание АТ равно или выше 85% (N50 (A+T≥0,85) 100 N50), с размером области сниженным приблизительно до 50 т.п.о. Область динуклеотидных АТ-повторов может быть определена как область, которая включает в себя длинные отрезки ATAT-повторов.
Сравнение данных по отвечаемости для контрольной рекомбиназы относительно P256K-мутантов показывает, что мутант P256K обеспечивает неожиданное и значительное улучшение в отвечаемости одной или нескольких из фосмидных промоторных областей, областей с высоким содержанием GC, с очень высоким содержанием GC, с низким содержанием GC, с высоким содержанием AT, с очень высоким содержанием AT и AT-динуклеотидными повторами по сравнению с контролем.
ПРИМЕР 3
Улучшение амплификации с использованием мутантов, имеющих мутации, гомологичные P256
Сравнение производительности, описанное выше в примере 2, повторяли для других рекомбиназ. В этом примере «контрольные» рекомбиназы создавали путем модификации рекомбиназы дикого типа, чтобы они содержали мутации, гомологичные H63S в RB49, как указано в колонке «контроль» в приведенной ниже таблице. Мутанты «P256K-гомологичные» создавали путем дальнейшего изменения контрольных ферментов, чтобы они несли мутации, гомологичные P256K в RB49, как указано в колонке «P256K-гомолог» в приведенной ниже таблице.
Например, для T6 UvsX контроль создают путем модификации Т6 UvsX дикого типа (SEQ ID NO: 9), чтобы она несла мутацию H66S. P256K-гомолог дополнительно модифицируют, чтобы фермент имел обе мутации, H66S и F259K.
F257K
F259K
P257K
P258K
P269K
D266K
P267K
P259K
Q261K
E264K
E264K
F259K
P267K
P259K
Кластеризацию ПЦР-свободной библиотеки на разграфленной проточной ячейке выполняли на cBot, как описано выше в примере 1, с использованием либо контроля, либо мутанта P256K. Секвенирование затем выполняли на инструменте HiSeq (Illumina, Inc.), и результаты секвенирования анализировали, как описано выше в примере 2, чтобы определить «отвечаемость» у набора областей, которые, как правило, слабо представлены в пуле данных от предшествующих секвенирований.
Сравнение данных по отвечаемости для контрольной рекомбиназы относительно P256K-гомологичных мутантов показывает, что P256K-гомологичный мутант обеспечивает неожиданное и значительное улучшение в отвечаемости одной или нескольких из фосмидных промоторных областей, областей с высоким содержанием GC, с очень высоким содержанием GC, с низким содержанием GC, с высоким содержанием AT, с очень высоким содержанием AT и AT-динуклеотидными повторами по сравнению с контролем.
В данной заявке приведены ссылки на различные публикации, патенты и/или заявки на патент. Раскрытие этих публикаций во всей их полноте включено в данное описание путем ссылки в данной заявке.
Предполагается, что термин «содержащий» в данном описании является открытым, включая не только перечисленные элементы, но дополнительно охватывая любые дополнительные элементы.
В данном документе описан ряд вариантов осуществления изобретения. Тем не менее, следует понимать, что могут быть осуществлены различные его модификации. Поэтому, в объем приведенной ниже формулы изобретения входят и другие варианты осуществления изобретения.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ILLUMINA, INC.
KELLINGER, MATTHEW
BOUTELL, JONATHAN
<120> МУТАНТЫ РЕКОМБИНАЗЫ
<130> IP-1264-PRV
<160> 35
<170> PatentIn version 3.5
<210> 1
<211> 356
<212> Белок
<213> Фаг Enterobacteria RB49
<400> 1
Met Ser Val Leu Glu Lys Leu Lys Lys Asn Ser Thr Leu Lys Thr Thr
1 5 10 15
Ala Val Leu Ser Lys Ser Ser Phe Phe Asn Glu Lys Thr Asn Thr Arg
20 25 30
Thr Lys Ile Pro Met Leu Asn Ile Ala Phe Ser Gly Asp Leu Lys Lys
35 40 45
Gly Phe Gln Ser Gly Leu Ile Phe Phe Ala Gly Pro Ser Lys His Phe
50 55 60
Lys Ser Asn Met Gly Leu Thr Cys Val Ser Ala Tyr Met Lys Gln Asn
65 70 75 80
Pro Asp Ala Ala Cys Leu Phe Phe Asp Ser Glu Phe Gly Ile Thr Ser
85 90 95
Ala Tyr Leu Glu Ser Met Gly Val Asp Pro Asp Arg Val Val His Val
100 105 110
Pro Ile Lys Asn Ile Glu Glu Leu Lys Phe Glu Ile Met Asn Gln Leu
115 120 125
Glu Gln Ile Thr Arg Glu Asp Lys Val Ile Ile Phe Ile Asp Ser Ile
130 135 140
Gly Asn Leu Ala Ser Lys Lys Glu Val Glu Asp Ala Ile Asn Glu Lys
145 150 155 160
Ser Ala Gln Asp Met Thr Arg Ala Lys Ala Leu Lys Gly Leu Phe Arg
165 170 175
Met Val Thr Pro Tyr Leu Thr Met Asn Asp Ile Pro Cys Ile Ala Ile
180 185 190
Asn His Thr Tyr Glu Thr Gln Glu Met Phe Ser Lys Thr Val Met Ser
195 200 205
Gly Gly Thr Gly Ala Met Tyr Ser Ala Asn Glu Val Phe Ile Ile Gly
210 215 220
Arg Arg Gln Gln Lys Glu Gly Thr Glu Ile Thr Gly Tyr Asp Phe Ile
225 230 235 240
Leu Asn Ala Glu Lys Ser Arg Thr Val Lys Glu Lys Ser Lys Phe Pro
245 250 255
Ile Ser Val Thr Phe Ser Gly Gly Ile Asp Pro Tyr Ser Gly Leu Leu
260 265 270
Glu Leu Ala Val Glu Leu Gly Trp Val Val Lys Pro Ser Asn Gly Trp
275 280 285
Tyr Ser Arg Ser Ile Leu Asn Thr Glu Thr Gly Glu Met Glu Thr Glu
290 295 300
Glu Arg Lys Phe Arg Ala Lys Glu Thr Asn Ser Ile Glu Phe Trp Lys
305 310 315 320
Pro Leu Leu Thr Asn Asp Lys Phe Asn Glu Ala Ile Asn Asp His Tyr
325 330 335
Lys Leu Gly Gln Val Ile Ser Asp Glu Ala Val Asp Lys Glu Ile Glu
340 345 350
Asp Met Leu Ala
355
<210> 2
<211> 356
<212> Белок
<213> Фаг Enterobacteria RB49
<400> 2
Met Ser Val Leu Glu Lys Leu Lys Lys Asn Ser Thr Leu Lys Thr Thr
1 5 10 15
Ala Val Leu Ser Lys Ser Ser Phe Phe Asn Glu Lys Thr Asn Thr Arg
20 25 30
Thr Lys Ile Pro Met Leu Asn Ile Ala Phe Ser Gly Asp Leu Lys Lys
35 40 45
Gly Phe Gln Ser Gly Leu Ile Phe Phe Ala Gly Pro Ser Lys Ser Phe
50 55 60
Lys Ser Asn Met Gly Leu Thr Cys Val Ser Ala Tyr Met Lys Gln Asn
65 70 75 80
Pro Asp Ala Ala Cys Leu Phe Phe Asp Ser Glu Phe Gly Ile Thr Ser
85 90 95
Ala Tyr Leu Glu Ser Met Gly Val Asp Pro Asp Arg Val Val His Val
100 105 110
Pro Ile Lys Asn Ile Glu Glu Leu Lys Phe Glu Ile Met Asn Gln Leu
115 120 125
Glu Gln Ile Thr Arg Glu Asp Lys Val Ile Ile Phe Ile Asp Ser Ile
130 135 140
Gly Asn Leu Ala Ser Lys Lys Glu Val Glu Asp Ala Ile Asn Glu Lys
145 150 155 160
Ser Ala Gln Asp Met Thr Arg Ala Lys Ala Leu Lys Gly Leu Phe Arg
165 170 175
Met Val Thr Pro Tyr Leu Thr Met Asn Asp Ile Pro Cys Ile Ala Ile
180 185 190
Asn His Thr Tyr Glu Thr Gln Glu Met Phe Ser Lys Thr Val Met Ser
195 200 205
Gly Gly Thr Gly Ala Met Tyr Ser Ala Asn Glu Val Phe Ile Ile Gly
210 215 220
Arg Arg Gln Gln Lys Glu Gly Thr Glu Ile Thr Gly Tyr Asp Phe Ile
225 230 235 240
Leu Asn Ala Glu Lys Ser Arg Thr Val Lys Glu Lys Ser Lys Phe Lys
245 250 255
Ile Ser Val Thr Phe Ser Gly Gly Ile Asp Pro Tyr Ser Gly Leu Leu
260 265 270
Glu Leu Ala Val Glu Leu Gly Trp Val Val Lys Pro Ser Asn Gly Trp
275 280 285
Tyr Ser Arg Ser Ile Leu Asn Thr Glu Thr Gly Glu Met Glu Thr Glu
290 295 300
Glu Arg Lys Phe Arg Ala Lys Glu Thr Asn Ser Ile Glu Phe Trp Lys
305 310 315 320
Pro Leu Leu Thr Asn Asp Lys Phe Asn Glu Ala Ile Asn Asp His Tyr
325 330 335
Lys Leu Gly Gln Val Ile Ser Asp Glu Ala Val Asp Lys Glu Ile Glu
340 345 350
Asp Met Leu Ala
355
<210> 3
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полуконсервативный домен
<400> 3
Lys Glu Lys Ser Lys Phe Phe Ile
1 5
<210> 4
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полуконсервативный домен
<400> 4
Lys Glu Lys Ser Lys Phe Pro Ile
1 5
<210> 5
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полуконсервативный домен
<220>
<221> ВАРИАНТ
<222> (5)..(5)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (7)..(7)
<223> X = любая AA
<400> 5
Lys Glu Lys Ser Xaa Phe Xaa Ile
1 5
<210> 6
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полуконсервативный домен
<220>
<221> ВАРИАНТ
<222> (4)..(4)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (5)..(5)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (8)..(8)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (9)..(9)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (10)..(10)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (11)..(11)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (12)..(12)
<223> X = любая AA
<220>
<221> ВАРИАНТ
<222> (13)..(13)
<223> X = любая AA
<400> 6
Lys Ser Arg Xaa Xaa Lys Glu Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 7
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полуконсервативный домен
<400> 7
Lys Ser Arg Thr Val Lys Glu Lys Ser Lys Phe Phe Ile
1 5 10
<210> 8
<211> 391
<212> Белок
<213> Фаг Enterobacteria T4
<400> 8
Met Ser Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser Lys Leu
1 5 10 15
Thr Ala Glu Leu Thr Ala Ser Lys Phe Phe Asn Glu Lys Asp Val Val
20 25 30
Arg Thr Lys Ile Pro Met Met Asn Ile Ala Leu Ser Gly Glu Ile Thr
35 40 45
Gly Gly Met Gln Ser Gly Leu Leu Ile Leu Ala Gly Pro Ser Lys Ser
50 55 60
Phe Lys Ser Asn Phe Gly Leu Thr Met Val Ser Ser Tyr Met Arg Gln
65 70 75 80
Tyr Pro Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Phe Gly Ile Thr
85 90 95
Pro Ala Tyr Leu Arg Ser Met Gly Val Asp Pro Glu Arg Val Ile His
100 105 110
Thr Pro Val Gln Ser Leu Glu Gln Leu Arg Ile Asp Met Val Asn Gln
115 120 125
Leu Asp Ala Ile Glu Arg Gly Glu Lys Val Val Val Phe Ile Asp Ser
130 135 140
Leu Gly Asn Leu Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu Asn Glu
145 150 155 160
Lys Val Val Ser Asp Met Thr Arg Ala Lys Thr Met Lys Ser Leu Phe
165 170 175
Arg Ile Val Thr Pro Tyr Phe Ser Thr Lys Asn Ile Pro Cys Ile Ala
180 185 190
Ile Asn His Thr Tyr Glu Thr Gln Glu Met Phe Ser Lys Thr Val Met
195 200 205
Gly Gly Gly Thr Gly Pro Met Tyr Ser Ala Asp Thr Val Phe Ile Ile
210 215 220
Gly Lys Arg Gln Ile Lys Asp Gly Ser Asp Leu Gln Gly Tyr Gln Phe
225 230 235 240
Val Leu Asn Val Glu Lys Ser Arg Thr Val Lys Glu Lys Ser Lys Phe
245 250 255
Phe Ile Asp Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser Gly Leu
260 265 270
Leu Asp Met Ala Leu Glu Leu Gly Phe Val Val Lys Pro Lys Asn Gly
275 280 285
Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met Ile Arg
290 295 300
Glu Glu Lys Ser Trp Arg Ala Lys Asp Thr Asn Cys Thr Thr Phe Trp
305 310 315 320
Gly Pro Leu Phe Lys His Gln Pro Phe Arg Asp Ala Ile Lys Arg Ala
325 330 335
Tyr Gln Leu Gly Ala Ile Asp Ser Asn Glu Ile Val Glu Ala Glu Val
340 345 350
Asp Glu Leu Ile Asn Ser Lys Val Glu Lys Phe Lys Ser Pro Glu Ser
355 360 365
Lys Ser Lys Ser Ala Ala Asp Leu Glu Thr Asp Leu Glu Gln Leu Ser
370 375 380
Asp Met Glu Glu Phe Asn Glu
385 390
<210> 9
<211> 393
<212> Белок
<213> Фаг Enterobacteria T6
<400> 9
Met Ser Ile Ala Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser
1 5 10 15
Lys Met Thr Ala Glu Leu Thr Thr Ser Lys Phe Phe Asn Glu Lys Asp
20 25 30
Val Ile Arg Thr Lys Ile Pro Met Leu Asn Ile Ala Ile Ser Gly Ala
35 40 45
Ile Asp Gly Gly Met Gln Ser Gly Leu Thr Ile Phe Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Met Ser Leu Thr Met Val Ala Ala Tyr Leu
65 70 75 80
Asn Lys Tyr Pro Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Phe Gly
85 90 95
Ile Thr Pro Ala Tyr Leu Arg Ser Met Gly Val Asp Pro Glu Arg Val
100 105 110
Ile His Thr Pro Ile Gln Ser Val Glu Gln Leu Lys Ile Asp Met Val
115 120 125
Asn Gln Leu Glu Ala Ile Glu Arg Gly Glu Lys Val Ile Val Phe Ile
130 135 140
Asp Ser Ile Gly Asn Met Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu
145 150 155 160
Asn Glu Lys Ser Val Ala Asp Met Thr Arg Ala Lys Ser Leu Lys Ser
165 170 175
Leu Phe Arg Ile Val Thr Pro Tyr Phe Ser Ile Lys Asn Ile Pro Cys
180 185 190
Val Ala Val Asn His Thr Ile Glu Thr Ile Glu Met Phe Ser Lys Thr
195 200 205
Val Met Thr Gly Gly Thr Gly Val Met Tyr Ser Ala Asp Thr Val Phe
210 215 220
Ile Ile Gly Lys Arg Gln Ile Lys Asp Gly Ser Asp Leu Gln Gly Tyr
225 230 235 240
Gln Phe Val Leu Asn Val Glu Lys Ser Arg Thr Val Lys Glu Lys Ser
245 250 255
Lys Phe Phe Ile Asp Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser
260 265 270
Gly Leu Leu Asp Met Ala Leu Glu Leu Gly Phe Val Val Lys Pro Lys
275 280 285
Asn Gly Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met
290 295 300
Ile Arg Glu Glu Lys Ser Trp Arg Ala Lys Asp Thr Asn Cys Thr Thr
305 310 315 320
Phe Trp Gly Pro Leu Phe Lys His Gln Pro Phe Arg Asp Ala Ile Lys
325 330 335
Arg Ala Tyr Gln Leu Gly Ala Ile Asp Ser Asn Glu Ile Val Glu Ala
340 345 350
Glu Val Asp Glu Leu Ile Asn Ser Lys Val Glu Lys Phe Lys Ser Pro
355 360 365
Glu Ser Lys Ser Lys Ser Ala Ala Asp Leu Glu Thr Asp Leu Glu Gln
370 375 380
Leu Ser Asp Met Glu Glu Phe Asn Glu
385 390
<210> 10
<211> 387
<212> Белок
<213> Фаг Acinetobacter 133
<400> 10
Met Ser Ser Leu Lys Glu Arg Leu Ile Lys Ala Ser Thr Ser Lys Met
1 5 10 15
Thr Ala Glu Leu Thr Lys Ser Lys Phe Phe Asn Asp Lys Thr Val Val
20 25 30
Arg Thr Arg Ile Pro Met Leu Asn Ile Ala Ile Ser Gly Ala Leu Asn
35 40 45
Gly Gly Met Gln Ser Gly Leu Thr Ile Phe Ala Gly Pro Ser Lys His
50 55 60
Phe Lys Ser Asn Met Gly Leu Thr Met Val Ala Ala Tyr Met Lys Ala
65 70 75 80
Phe Pro Asp Ala Val Cys Met Phe Tyr Asp Ser Glu Phe Gly Ile Thr
85 90 95
Pro Ala Tyr Leu Lys Ala Met Gly Val Asp Pro Asp Arg Val Ile His
100 105 110
Thr Pro Val Gln Ser Val Glu Gln Leu Lys Ile Asp Met Thr Asn Gln
115 120 125
Leu Glu Glu Val Lys Arg Gly Glu Lys Val Ile Val Phe Ile Asp Ser
130 135 140
Ile Gly Asn Leu Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu Asn Glu
145 150 155 160
Lys Thr Thr Ala Asp Met Thr Arg Ala Lys Ala Leu Lys Ser Leu Phe
165 170 175
Arg Ile Val Thr Pro Tyr Phe Ser Ile Lys Asp Ile Pro Cys Val Ala
180 185 190
Val Asn His Thr Leu Gln Thr Leu Glu Met Phe Ser Lys Glu Val Met
195 200 205
Thr Gly Gly Thr Gly Val Met Tyr Ser Ala Asp Thr Val Phe Phe Ile
210 215 220
Gly Lys Arg Gln Val Lys Asp Gly Thr Glu Leu Ala Gly Tyr Glu Phe
225 230 235 240
Ile Leu Lys Ala Glu Lys Ser Arg Met Val Lys Glu Lys Ser Val Phe
245 250 255
Pro Ile Thr Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser Gly Leu
260 265 270
Leu Glu Met Ala Thr Asp Leu Gly Phe Val Val Lys Pro Lys Val Gly
275 280 285
Trp Tyr Lys Arg Ala Met Met Val Asp Gly Val Met Gln His Glu Glu
290 295 300
Lys Ser Trp Arg Ala Lys Asp Thr Asp Ser Ile Asp Phe Trp Gly Pro
305 310 315 320
Leu Phe Lys His Asp Glu Phe Arg Lys Ala Ile Glu Thr Arg Tyr Gln
325 330 335
Leu Gly Ser Ile Glu Ser Asp Ala Glu Val Asp Ala Glu Val Asp Ala
340 345 350
Leu Ile Gly Ser Lys Thr Thr Ala Lys Ile Ser Gly Val Asn Phe Gly
355 360 365
Pro Ala Glu Ser Ala Ala Asp Lys Glu Gln Gln Leu Glu Asp Phe Val
370 375 380
Asp Glu Asp
385
<210> 11
<211> 390
<212> Белок
<213> Фаг Enterobacteria RB69
<400> 11
Met Ser Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser Lys Met
1 5 10 15
Thr Ala Asp Leu Thr Lys Ser Lys Leu Phe Asn Asn Arg Asp Glu Val
20 25 30
Pro Thr Arg Ile Pro Met Leu Asn Ile Ala Leu Gly Gly Ala Leu Asn
35 40 45
Ala Gly Leu Gln Ser Gly Leu Thr Ile Phe Ala Ala Pro Ser Lys His
50 55 60
Phe Lys Thr Leu Phe Gly Leu Thr Met Val Ala Ala Tyr Met Lys Lys
65 70 75 80
Tyr Lys Asp Ala Ile Cys Leu Phe Tyr Asp Ser Glu Phe Gly Ala Ser
85 90 95
Glu Ser Tyr Phe Arg Ser Met Gly Val Asp Leu Asp Arg Val Val His
100 105 110
Thr Pro Ile Gln Ser Val Glu Gln Leu Lys Val Asp Met Thr Asn Gln
115 120 125
Leu Asp Ala Ile Glu Arg Gly Asp Lys Val Ile Ile Phe Ile Asp Ser
130 135 140
Ile Gly Asn Thr Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu Asn Glu
145 150 155 160
Lys Val Val Gly Asp Met Ser Arg Ala Lys Ala Leu Lys Ser Leu Phe
165 170 175
Arg Ile Val Thr Pro Tyr Leu Thr Ile Lys Asp Ile Pro Cys Val Ala
180 185 190
Ile Asn His Thr Ala Met Glu Ile Gly Gly Leu Tyr Pro Lys Glu Ile
195 200 205
Met Gly Gly Gly Thr Gly Ile Leu Tyr Ser Ala Asn Thr Val Phe Phe
210 215 220
Ile Ser Lys Arg Gln Val Lys Glu Gly Thr Glu Leu Thr Gly Tyr Asp
225 230 235 240
Phe Thr Leu Lys Ala Glu Lys Ser Arg Thr Val Lys Glu Lys Ser Thr
245 250 255
Phe Pro Ile Thr Val Asn Phe Asp Gly Gly Ile Asp Pro Phe Ser Gly
260 265 270
Leu Leu Glu Met Ala Thr Glu Ile Gly Phe Val Val Lys Pro Lys Ala
275 280 285
Gly Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met Ile
290 295 300
Arg Glu Glu Lys Ser Trp Arg Ala Lys Ala Thr Asp Cys Val Glu Phe
305 310 315 320
Trp Gly Pro Leu Phe Lys His Lys Pro Phe Arg Asp Ala Ile Glu Thr
325 330 335
Lys Tyr Lys Leu Gly Ala Ile Ser Ser Ile Lys Glu Val Asp Asp Ala
340 345 350
Val Asn Asp Leu Ile Asn Cys Lys Ala Thr Thr Lys Val Pro Val Lys
355 360 365
Thr Ser Asp Ala Pro Ser Ala Ala Asp Ile Glu Asn Asp Leu Asp Glu
370 375 380
Met Glu Asp Phe Asp Glu
385 390
<210> 12
<211> 411
<212> Белок
<213> Фаг Aeromonas Aeh1
<400> 12
Met Ala Lys Gly Ile Lys Thr Ala Lys Thr Gly Asn Leu Gly Ser Leu
1 5 10 15
Met Ser Lys Leu Ala Gly Thr Ser Ser Asn Lys Met Ser Ser Val Leu
20 25 30
Ala Asp Ser Lys Phe Phe Asn Asp Lys Asp Cys Val Arg Thr Arg Val
35 40 45
Pro Leu Leu Asn Leu Ala Met Ser Gly Glu Leu Asp Gly Gly Leu Thr
50 55 60
Pro Gly Leu Thr Val Leu Ala Gly Pro Ser Lys His Phe Lys Ser Asn
65 70 75 80
Leu Ser Leu Val Phe Val Ala Ala Tyr Leu Arg Lys Tyr Pro Asp Ala
85 90 95
Val Cys Ile Phe Phe Asp Asn Glu Phe Gly Ser Thr Pro Gly Tyr Phe
100 105 110
Glu Ser Gln Gly Val Asp Ile Ser Arg Val Ile His Cys Pro Phe Lys
115 120 125
Asn Ile Glu Glu Leu Lys Phe Asp Ile Val Lys Lys Leu Glu Ala Ile
130 135 140
Glu Arg Gly Asp Arg Val Ile Val Phe Val Asp Ser Ile Gly Asn Ala
145 150 155 160
Ala Ser Lys Lys Glu Ile Asp Asp Ala Ile Asp Glu Lys Ser Val Ser
165 170 175
Asp Met Thr Arg Ala Lys Gln Ile Lys Ser Leu Thr Arg Met Met Thr
180 185 190
Pro Tyr Leu Thr Val Asn Asp Ile Pro Ala Ile Met Val Ala His Thr
195 200 205
Tyr Asp Thr Gln Glu Met Tyr Ser Lys Lys Val Val Ser Gly Gly Thr
210 215 220
Gly Ile Thr Tyr Ser Ser Asp Thr Val Ile Ile Ile Gly Arg Gln Gln
225 230 235 240
Glu Lys Asp Gly Lys Glu Leu Leu Gly Tyr Asn Phe Val Leu Asn Met
245 250 255
Glu Lys Ser Arg Phe Val Lys Glu Gln Ser Lys Leu Pro Leu Glu Val
260 265 270
Thr Phe Gln Gly Gly Ile Asn Thr Tyr Ser Gly Met Leu Asp Ile Ala
275 280 285
Leu Glu Val Gly Phe Val Val Lys Pro Ser Asn Gly Trp Phe Ser Arg
290 295 300
Ala Phe Leu Asp Glu Glu Thr Gly Glu Leu Val Glu Glu Asp Arg Lys
305 310 315 320
Trp Arg Arg Ala Asp Thr Asn Cys Leu Glu Phe Trp Lys Pro Met Phe
325 330 335
Ala His Gln Pro Phe Lys Thr Ala Cys Ser Asp Met Phe Lys Leu Lys
340 345 350
Ser Val Ala Val Lys Asp Glu Val Phe Asp Glu Val Asp Glu Leu Phe
355 360 365
Ser Gly Glu Ala Glu Met Pro Val Asn Met Gly Arg Lys Leu Asp Thr
370 375 380
Ala Asp Gln Glu Glu Ile Asp Gln Leu Glu Glu Val Asp Val Glu Gly
385 390 395 400
Ser Asp Ser Asp Glu Leu Phe Ala Asn Leu Asp
405 410
<210> 13
<211> 368
<212> Белок
<213> Фаг Aeromonas 65
<400> 13
Met Ala Lys Lys Ala Lys Val Val Asn Ser Gly Asp Leu Leu Glu Arg
1 5 10 15
Leu Asn Gly Thr Ser Ser Asn Lys Met Ser Ala Met Leu Ala Glu Ser
20 25 30
Ile Phe Phe Asn Glu Lys Asp Thr Ile Arg Thr Arg Val Pro Ile Ile
35 40 45
Asn Leu Met Met Ser Gly Arg Leu Asp Gly Gly Ile Thr Pro Gly Leu
50 55 60
Thr Cys Ile Ala Gly Pro Ser Lys His Phe Lys Ser Asn Leu Ser Leu
65 70 75 80
Val Met Val Ser Ala Tyr Leu Arg Lys Tyr Pro Lys Ala Val Cys Leu
85 90 95
Phe Phe Asp Asn Glu Phe Gly Ser Thr Pro Asp Tyr Phe Thr Ser Gln
100 105 110
Gly Val Asp Ile Ser Arg Val Val His Cys Pro Phe Ile Asp Val Glu
115 120 125
Glu Leu Lys Phe Asp Ile Val Lys Lys Leu Glu Ser Ile Thr Arg Gly
130 135 140
Asp Lys Val Ile Ile Tyr Ile Asp Ser Ile Gly Asn Val Ala Ser Lys
145 150 155 160
Lys Glu Leu Gln Asp Ala Lys Asp Glu Lys Ser Ala Gln Asp Met Thr
165 170 175
Arg Ala Lys Gln Ile Lys Ser Leu Phe Arg Met Val Thr Pro Tyr Leu
180 185 190
Thr Val Leu Asp Ile Pro Cys Ile Ala Val Asn His Thr Tyr Glu Thr
195 200 205
Gln Glu Met Phe Ser Lys Thr Val Met Ser Gly Gly Thr Gly Pro Met
210 215 220
Tyr Ser Ala Asp Thr Val Ile Ile Leu Gly Lys Gln Gln Asp Lys Asp
225 230 235 240
Gly Lys Glu Leu Leu Gly Tyr Asn Phe Val Met Asn Ala Glu Lys Ser
245 250 255
Arg Ala Ile Lys Glu Lys Ser Lys Leu Asp Leu Met Val Ser Phe Glu
260 265 270
Gly Gly Ile Asn Thr Tyr Ser Gly Leu Leu Lys Ile Ala Gln Glu Leu
275 280 285
Gly Phe Val Thr Lys Pro Gln Asn Ala Arg Tyr Gln Arg Asn Phe Leu
290 295 300
Asp Leu Glu Pro Gly Glu Met Val Ile Pro Glu Asp Glu Lys Lys Trp
305 310 315 320
Thr Glu Glu Glu Ser Asp Ser Leu Glu Phe Trp Lys Pro Met Phe Ser
325 330 335
His Lys Pro Phe Met Asp Ala Val Ser Asn Ala Tyr Lys Leu Lys Ala
340 345 350
Val Glu Val Ser Gln Glu Val Phe Asp Glu Val Asp Gln Leu Phe Gly
355 360 365
<210> 14
<211> 366
<212> Белок
<213> Фаг Vibrio KVP40
<400> 14
Met Ser Asp Leu Met Lys Ser Leu Lys Lys Ser Ser Thr Ser Gly Tyr
1 5 10 15
Ala Gln Val Leu Ser Glu Ser Gln Phe Met Phe Asp Lys Asp His Thr
20 25 30
Arg Thr Tyr Val Pro Ala Ile Asn Ile Ala Phe Ser Gly Glu Val Asp
35 40 45
Gly Gly Leu Thr Ser Gly Leu Thr Val Leu Ala Gly Pro Ser Lys His
50 55 60
Phe Lys Ser Asn Leu Gly Leu Val Gly Val Ala Ala Tyr Leu Lys Lys
65 70 75 80
Tyr Pro Asp Ala Val Cys Val Phe Ile Asp Thr Glu Phe Gly Ile Thr
85 90 95
Pro Ser Tyr Leu Arg Ser Gln Gly Val Asp Pro Asp Arg Val Leu His
100 105 110
Ile Gln Cys Glu Ser Val Glu Arg Met Lys Phe Glu Met Ala Asn Gln
115 120 125
Leu Lys Asp Leu Ala Glu Arg Lys Arg Ala Lys Lys Ala Gly Glu Glu
130 135 140
Pro Asp Arg Val Ile Phe Phe Ile Asp Ser Val Gly Asn Val Ala Ser
145 150 155 160
Ala Lys Glu Ile Asp Asp Ala Gln Asn Glu Lys Ser Val Ala Asp Met
165 170 175
Ser Arg Ala Lys Gln Leu Lys Ser Leu Phe Arg Ile Ile Thr Pro Tyr
180 185 190
Phe Thr Met Leu Asp Ile Pro Cys Ile Ala Ile Asn His Thr Tyr Gln
195 200 205
Thr Gln Glu Ile Tyr Ser Lys Thr Val Met Ser Gly Gly Thr Gly Ile
210 215 220
Met Tyr Ser Ala Asp Thr Val Ile Ile Leu Gly Lys Gln Gln Glu Lys
225 230 235 240
Asp Gly Lys Asp Ile Ile Gly Tyr His Phe Ile Met Asn Ile Glu Lys
245 250 255
Ser Arg Phe Val Lys Glu Lys Met Lys Val Pro Leu Thr Val Thr Tyr
260 265 270
Glu Asn Gly Ile Asp Pro Phe Ser Gly Leu Leu Asp Ile Ala Leu Gln
275 280 285
Thr Gly His Val Val Lys Pro Ser Asn Gly Trp Tyr Gln Arg Ala Thr
290 295 300
Val Asp Glu Glu Thr Gly Glu Met Ile Val Glu Glu Lys Lys Tyr Arg
305 310 315 320
Ala Lys Glu Thr Gln Thr Ile Ser Phe Trp Lys Asp Ile Ile Asn Ser
325 330 335
Pro Thr Phe Lys Glu Gly Val Lys Arg Ile Tyr Cys Leu Gly Gln Leu
340 345 350
Asp Glu Ser Glu Leu Phe Gly Glu Val Asp Ser Leu Phe Asp
355 360 365
<210> 15
<211> 386
<212> Белок
<213> Фаг Enterobacteria RB43
<400> 15
Met Ser Asn Lys Ala Leu Leu Lys Lys Leu Ile Lys Asn Ser Asn Ser
1 5 10 15
Gln Ser Ala Ala Ile Leu Ser Glu Ser Asp Val Phe Asn Asn Ile Thr
20 25 30
Lys Thr Arg Thr Arg Val Pro Ile Leu Asn Leu Ala Leu Ser Gly Ala
35 40 45
Phe Asp Gly Gly Leu Thr Ser Gly Leu Thr Leu Phe Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Leu Gly Leu Val Thr Val Ser Ala Tyr Leu
65 70 75 80
Lys Ala Asn Glu Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Lys Gly
85 90 95
Val Thr Lys Ser Tyr Leu Lys Ser Met Gly Val Asp Pro Asp Arg Val
100 105 110
Val Tyr Thr Arg Ile Thr Thr Val Glu Gln Leu Arg Asn Asp Val Val
115 120 125
Ser Gln Leu Asp Ala Leu Glu Arg Gly Asp Lys Val Ile Ile Phe Val
130 135 140
Asp Ser Val Gly Asn Thr Ala Ser Lys Lys Glu Leu Ala Asp Ala Leu
145 150 155 160
Ser Asp Asn Asp Lys Gln Asp Met Thr Arg Ala Lys Ala Leu Lys Gly
165 170 175
Met Phe Arg Met Val Thr Pro Tyr Leu Ala Asp Leu Asp Ile Pro Met
180 185 190
Val Cys Ile Cys His Thr Tyr Asp Thr Gln Glu Met Tyr Ser Lys Lys
195 200 205
Val Ile Ser Gly Gly Thr Gly Leu Met Tyr Ser Ala Asp Thr Ala Ile
210 215 220
Ile Leu Gly Lys Gln Gln Val Lys Glu Gly Thr Glu Val Val Gly Tyr
225 230 235 240
Asp Phe Ile Met Asn Ile Glu Lys Ser Arg Phe Val Lys Glu Lys Ser
245 250 255
Lys Phe Pro Leu His Val Thr Tyr Glu Gly Gly Ile Ser Met Tyr Ser
260 265 270
Gly Leu Leu Asp Leu Ala Met Glu Met Asn Phe Val Gln Thr Pro Thr
275 280 285
Lys Gly Trp Arg Gly Arg Ala Phe Leu Asn Thr Glu Thr Gly Glu Leu
290 295 300
Glu Leu Glu Glu Lys Lys Trp Arg Glu Ser Glu Thr Asn Ser Ile Glu
305 310 315 320
Phe Trp Arg Pro Leu Phe Thr His Gln Pro Phe Leu Asp Ala Ile Gln
325 330 335
Asp Lys Tyr Arg Ile Pro Asp Lys Glu Ile Thr Asp Gly Ala Ala Leu
340 345 350
Glu Asp Leu Tyr Ser Thr Asp Glu Pro Glu Ser Asn Lys Ile Asp Leu
355 360 365
Asp Asp Asp Ile Pro Asp Asp Ile Gly Ile Asp Gln Asp Glu Glu Pro
370 375 380
Ile Met
385
<210> 16
<211> 335
<212> Белок
<213> Фаг Prochlorococcus P-SSM2
<400> 16
Met Asp Phe Leu Lys Glu Ile Val Lys Glu Ile Gly Asp Glu Tyr Thr
1 5 10 15
Gln Val Ala Ala Asp Ile Gln Glu Asn Glu Arg Phe Ile Asp Thr Gly
20 25 30
Ser Tyr Ile Phe Asn Gly Leu Val Ser Gly Ser Ile Phe Gly Gly Val
35 40 45
Ser Ser Ser Arg Ile Thr Ala Ile Ala Gly Glu Ser Ser Thr Gly Lys
50 55 60
Thr Tyr Phe Ser Leu Ala Val Val Lys Asn Phe Leu Asp Asn Asn Pro
65 70 75 80
Asp Gly Tyr Cys Leu Tyr Phe Asp Thr Glu Ala Ala Val Asn Lys Gly
85 90 95
Leu Leu Glu Ser Arg Gly Ile Asp Met Asn Arg Leu Val Val Val Asn
100 105 110
Val Val Thr Ile Glu Glu Phe Arg Ser Lys Ala Leu Arg Ala Val Asp
115 120 125
Ile Tyr Leu Lys Thr Ser Glu Glu Glu Arg Lys Pro Cys Met Phe Val
130 135 140
Leu Asp Ser Leu Gly Met Leu Ser Thr Glu Lys Glu Ile Arg Asp Ala
145 150 155 160
Leu Asp Asp Lys Gln Val Arg Asp Met Thr Lys Ser Gln Leu Val Lys
165 170 175
Gly Ala Phe Arg Met Leu Thr Leu Lys Leu Gly Gln Ala Asn Ile Pro
180 185 190
Leu Ile Val Thr Asn His Thr Tyr Asp Val Ile Gly Ser Tyr Val Pro
195 200 205
Thr Lys Glu Met Gly Gly Gly Ser Gly Leu Lys Tyr Ala Ala Ser Thr
210 215 220
Ile Ile Tyr Leu Ser Lys Lys Lys Glu Lys Asp Gln Lys Glu Val Ile
225 230 235 240
Gly Asn Leu Ile Lys Ala Lys Thr His Lys Ser Arg Leu Ser Lys Glu
245 250 255
Asn Lys Glu Val Gln Ile Arg Leu Tyr Tyr Asp Glu Arg Gly Leu Asp
260 265 270
Arg Tyr Tyr Gly Leu Leu Glu Leu Gly Glu Ile Gly Gly Met Trp Lys
275 280 285
Asn Val Ala Gly Arg Tyr Glu Met Asn Gly Lys Lys Ile Tyr Ala Lys
290 295 300
Glu Ile Leu Lys Asn Pro Thr Glu Tyr Phe Thr Asp Asp Ile Met Glu
305 310 315 320
Gln Leu Asp Asn Ile Ala Lys Glu His Phe Ser Tyr Gly Thr Asn
325 330 335
<210> 17
<211> 336
<212> Белок
<213> Фаг Prochlorococcus P-SSM4
<400> 17
Met Asn Phe Leu Lys Asp Ile Ala Lys Glu Ile Gly Asn Asp Tyr Ala
1 5 10 15
Ser Leu Val Ser Glu Gly Val Ser Ala Gly Asp Thr Ala Gly Phe Ile
20 25 30
Asp Thr Gly Ser Tyr Ile Phe Asn Ala Leu Leu Ser Gly Ser Ile Tyr
35 40 45
Gly Gly Ile Pro Asn Asn Lys Ile Thr Ala Ile Ala Gly Glu Thr Ser
50 55 60
Thr Gly Lys Thr Phe Phe Cys Leu Gly Met Val Gln His Phe Leu Glu
65 70 75 80
Ser Asn Pro Asp Ala Gly Val Ile Tyr Phe Glu Ser Glu Ser Ala Ile
85 90 95
Ser Lys Gln Met Ile Glu Asp Arg Gly Ile Asp Ser Asn Arg Met Leu
100 105 110
Leu Val Pro Val Thr Thr Val Gln Glu Phe Arg Leu Gln Ala Ile Lys
115 120 125
Ile Leu Asp Lys Tyr Asn Glu Gln Thr Ala Glu Glu Arg Lys Pro Leu
130 135 140
Met Phe Val Leu Asp Ser Leu Gly Met Leu Ser Thr Ser Lys Glu Val
145 150 155 160
Glu Asp Ser Glu Ala Gly Lys Glu Thr Arg Asp Met Thr Arg Ala Gln
165 170 175
Val Val Lys Ser Ile Phe Arg Val Leu Thr Leu Lys Leu Gly Lys Ala
180 185 190
Asn Val Pro Leu Ile Val Thr Asn His Thr Tyr Asp Val Val Gly Ala
195 200 205
Tyr Ile Pro Thr Lys Glu Met Gly Gly Gly Ser Gly Leu Lys Tyr Ala
210 215 220
Ala Ser Thr Ile Val Tyr Leu Ser Lys Lys Lys Glu Lys Asn Gly Lys
225 230 235 240
Glu Val Val Gly Asn Ile Ile Lys Cys Lys Thr Ala Lys Ser Arg Leu
245 250 255
Thr Lys Glu Asn Ser Asp Val Glu Thr Arg Leu Tyr Tyr Asp Arg Gly
260 265 270
Leu Asp Arg Tyr Tyr Gly Leu Leu Glu Leu Gly Glu Lys His Gly Val
275 280 285
Phe Ser Arg Lys Gly Asn Arg Val Val Val Gly Asp Ser Ser Val Tyr
290 295 300
Pro Ser Ala Ile Leu Ala Asp Pro Asp Lys Tyr Phe Thr Glu Glu Leu
305 310 315 320
Met Glu Lys Leu Asp Glu Ala Ala Ala Lys Glu Phe Arg Tyr Gly Asn
325 330 335
<210> 18
<211> 343
<212> Белок
<213> Фаг Synechococcus S-PM2
<400> 18
Met Ser Phe Leu Asp Ser Val Ile Lys Asp Ser Lys Asn Glu Tyr Ala
1 5 10 15
Ala Phe Ala Ser Glu Gly Val Ala Ala Gly Asp Val Glu Ser Phe Val
20 25 30
Asp Thr Gly Ser Tyr Ile Phe Asn Ala Leu Val Ser Gly Ser Ile Phe
35 40 45
Gly Gly Ile Pro Ser Asn Lys Ile Thr Ala Leu Ala Gly Glu Ser Gly
50 55 60
Thr Gly Lys Thr Phe Phe Cys Leu Ser Val Val Arg Asn Phe Leu Asn
65 70 75 80
Thr Asp Pro Asp Ala Gly Val Ile Tyr Phe Glu Thr Glu Ser Ala Ile
85 90 95
Ser Lys Gln Met Ile Glu Ser Arg Gly Ile Asp Ser Thr Arg Met Ile
100 105 110
Ile Phe Pro Val Asp Thr Ile Glu Asp Phe Arg Thr Gln Ala Val Arg
115 120 125
Ile Ile Asp Lys Tyr Met Glu Gln Asn Lys Ser Glu Arg Lys Pro Leu
130 135 140
Met Phe Val Leu Asp Ser Leu Gly Met Leu Ala Thr Lys Lys Glu Val
145 150 155 160
Glu Asp Ala Ser Asn Asp Lys Gln Val Arg Asp Met Thr Lys Ala Gln
165 170 175
Ile Val Lys Ser Ala Phe Arg Ile Leu Thr Leu Lys Met Gly Lys Ala
180 185 190
Asn Ile Pro Met Leu Val Thr Asn His Thr Tyr Asp Val Val Gly Ser
195 200 205
Tyr Val Pro Thr Lys Glu Met Gly Gly Gly Ser Gly Leu Lys Tyr Ser
210 215 220
Ala Ser Thr Ile Val Tyr Leu Gly Lys Lys Lys Glu Lys Asp Gly Thr
225 230 235 240
Asp Leu Val Gly Asn Ile Ile Lys Cys Glu Ala Lys Lys Ser Arg Leu
245 250 255
Thr Arg Glu Gly Ser Lys Val Glu Thr Arg Leu Phe Phe Asp Gln Arg
260 265 270
Gly Leu Glu Arg Tyr Tyr Gly Met Leu Glu Leu Gly Glu Arg Ala Gly
275 280 285
Leu Trp Lys Asn Thr Ala Gly Arg Tyr Glu Ile Asn Gly Lys Lys Val
290 295 300
Tyr Gly Lys Gln Ile Leu Ala Asn Pro Asp Glu Phe Phe Thr Glu Glu
305 310 315 320
Ile Leu Gln Glu Leu Asp Lys Gln Ala Gln Arg Glu Phe Leu Tyr Gly
325 330 335
Ala Ser Asp Asp Gly Glu Asp
340
<210> 19
<211> 393
<212> Белок
<213> Фаг Enterobacteria RB32
<400> 19
Met Ser Ile Ala Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser
1 5 10 15
Lys Met Thr Ala Glu Leu Thr Thr Ser Lys Phe Phe Asn Glu Lys Asp
20 25 30
Val Ile Arg Thr Lys Ile Pro Met Leu Asn Ile Ala Ile Ser Gly Ala
35 40 45
Ile Asp Gly Gly Met Gln Ser Gly Leu Thr Ile Phe Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Met Ser Leu Thr Met Val Ala Ala Tyr Leu
65 70 75 80
Asn Lys Tyr Pro Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Phe Gly
85 90 95
Ile Thr Pro Ala Tyr Leu Arg Ser Met Gly Val Asp Pro Glu Arg Val
100 105 110
Ile His Thr Pro Ile Gln Ser Val Glu Gln Leu Lys Ile Asp Met Val
115 120 125
Asn Gln Leu Glu Ala Ile Glu Arg Gly Glu Lys Val Ile Val Phe Ile
130 135 140
Asp Ser Ile Gly Asn Met Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu
145 150 155 160
Asn Glu Lys Ser Val Ala Asp Met Thr Arg Ala Lys Ser Leu Lys Ser
165 170 175
Leu Phe Arg Ile Val Thr Pro Tyr Phe Ser Ile Lys Asn Ile Pro Cys
180 185 190
Val Ala Val Asn His Thr Ile Glu Thr Ile Glu Met Phe Ser Lys Thr
195 200 205
Val Met Thr Gly Gly Thr Gly Val Met Tyr Ser Ala Asp Thr Val Phe
210 215 220
Ile Ile Gly Lys Arg Gln Ile Lys Asp Gly Ser Asp Leu Gln Gly Tyr
225 230 235 240
Gln Phe Val Leu Asn Val Glu Lys Ser Arg Thr Val Lys Glu Lys Ser
245 250 255
Lys Phe Phe Ile Asp Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser
260 265 270
Gly Leu Leu Asp Met Ala Leu Glu Leu Gly Phe Val Val Lys Pro Lys
275 280 285
Asn Gly Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met
290 295 300
Ile Arg Glu Glu Lys Ser Trp Arg Ala Lys Asp Thr Asn Cys Thr Thr
305 310 315 320
Phe Trp Gly Pro Leu Phe Lys His Gln Pro Phe Arg Asp Ala Ile Lys
325 330 335
Arg Ala Tyr Gln Leu Gly Ala Ile Asp Ser Asn Glu Ile Val Glu Ala
340 345 350
Glu Val Asp Glu Leu Ile Asn Ser Lys Val Glu Lys Phe Lys Ser Pro
355 360 365
Glu Ser Lys Ser Lys Ser Ala Ala Asp Leu Glu Thr Asp Leu Glu Gln
370 375 380
Leu Ser Asp Met Glu Glu Phe Asn Glu
385 390
<210> 20
<211> 366
<212> Белок
<213> Фаг Vibrio nt-1
<400> 20
Met Ser Asp Leu Leu Lys Ser Leu Lys Lys Ser Ser Thr Ser Gly Tyr
1 5 10 15
Ala His Val Leu Ser Glu Ser Gln Phe Met Phe Glu Lys Asp His Thr
20 25 30
Arg Thr Tyr Val Pro Ala Ile Asn Ile Ala Phe Ser Gly Glu Val Asp
35 40 45
Gly Gly Leu Thr Ser Gly Leu Thr Val Leu Ala Gly Pro Ser Lys His
50 55 60
Phe Lys Ser Asn Leu Gly Leu Val Gly Val Ala Ala Tyr Leu Lys Lys
65 70 75 80
Tyr Pro Glu Ala Ile Cys Val Phe Ile Asp Thr Glu Phe Gly Ile Thr
85 90 95
Pro Ser Tyr Leu Lys Ser Gln Gly Val Asp Pro Glu Arg Val Leu His
100 105 110
Ile Gln Cys Glu Ser Val Glu Arg Met Lys Phe Glu Met Ala Asn Gln
115 120 125
Leu Lys Asp Leu Ala Glu Arg Lys Arg Ala Lys Lys Ala Gly Glu Glu
130 135 140
Pro Asp Arg Val Val Phe Phe Ile Asp Ser Val Gly Asn Val Ala Ser
145 150 155 160
Ala Lys Glu Ile Asp Asp Ala Gln Asn Glu Lys Ser Val Ala Asp Met
165 170 175
Ser Arg Ala Lys Gln Leu Lys Ser Leu Phe Arg Ile Ile Thr Pro Tyr
180 185 190
Phe Thr Met Leu Asp Ile Pro Cys Ile Ala Ile Asn His Thr Tyr Gln
195 200 205
Thr Gln Glu Met Tyr Ser Lys Thr Val Met Ser Gly Gly Thr Gly Ile
210 215 220
Met Tyr Ser Ala Asp Thr Val Ile Ile Leu Gly Lys Gln Gln Glu Lys
225 230 235 240
Asp Gly Lys Glu Ile Ile Gly Tyr His Phe Ile Met Asn Ile Glu Lys
245 250 255
Ser Arg Phe Val Lys Glu Lys Met Lys Val Pro Leu Thr Val Thr Tyr
260 265 270
Glu His Gly Ile Asp Gln Phe Ser Gly Leu Leu Asp Ile Ala Leu Gln
275 280 285
Thr Gly His Val Val Lys Pro Ser Asn Gly Trp Tyr Gln Arg Ala Phe
290 295 300
Ile Asp Glu Glu Thr Gly Glu Ile Glu Ile Glu Glu Lys Lys Tyr Arg
305 310 315 320
Ala Lys Glu Thr Gln Thr Leu Ser Phe Trp Lys Glu Ile Ile Asn Ser
325 330 335
Pro Thr Phe Lys Thr Gly Val Lys Arg Leu Tyr Cys Leu Gly Gln Leu
340 345 350
Asp Glu Ser Glu Leu Leu Asp Glu Val Asp Ser Leu Phe Asp
355 360 365
<210> 21
<211> 381
<212> Белок
<213> Фаг Enterobacteria RB16
<400> 21
Met Ser Asn Lys Ala Leu Leu Lys Lys Leu Ile Lys Asn Ser Asn Ser
1 5 10 15
Gln Ser Ala Ser Ile Leu Ser Glu Ser Asp Val Phe Asn Asn Ile Thr
20 25 30
Lys Thr Arg Thr Arg Val Pro Ile Leu Asn Leu Val Leu Ser Gly Ala
35 40 45
Phe Asp Gly Gly Leu Thr Ser Gly Leu Thr Leu Ile Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Leu Gly Leu Val Ala Val Ala Ala Tyr Leu
65 70 75 80
Lys Ala Asn Glu Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Lys Gly
85 90 95
Val Thr Lys Ser Tyr Leu Lys Ser Met Gly Val Asp Pro Asp Arg Val
100 105 110
Val Tyr Thr Arg Ile Thr Thr Val Glu Gln Leu Arg Asn Asp Val Val
115 120 125
Ser Gln Leu Asp Ala Leu Glu Arg Gly Asp Lys Val Ile Ile Phe Val
130 135 140
Asp Ser Val Gly Asn Thr Ala Ser Lys Lys Glu Leu Lys Asp Ala Leu
145 150 155 160
Glu Asp Asn Asp Lys Gln Asp Met Thr Arg Ala Lys Ala Leu Lys Gly
165 170 175
Met Phe Arg Met Val Thr Pro Tyr Leu Ala Asp Ile Asp Ile Pro Met
180 185 190
Val Cys Ile Cys His Thr Tyr Asp Thr Gln Glu Met Tyr Ser Lys Lys
195 200 205
Val Ile Ser Gly Gly Thr Gly Leu Met Tyr Ser Ala Asp Thr Ala Ile
210 215 220
Ile Leu Gly Lys Gln Gln Val Lys Glu Gly Thr Glu Val Val Gly Tyr
225 230 235 240
Asp Phe Ile Met Asn Val Glu Lys Ser Arg Phe Val Lys Glu Lys Ser
245 250 255
Lys Phe Pro Leu His Val Thr Tyr Glu Gly Gly Ile Ser Met Phe Ser
260 265 270
Gly Leu Leu Asp Leu Ala Met Glu Met Asn Phe Val Gln Thr Pro Thr
275 280 285
Lys Gly Trp Arg Gly Arg Ala Phe Leu Asn Thr Glu Thr Gly Glu Leu
290 295 300
Glu Leu Glu Glu Lys Lys Trp Arg Glu Ala Glu Thr Asn Cys Ile Glu
305 310 315 320
Phe Trp Lys Pro Leu Phe Lys His Gln Pro Phe Ile Asp Ala Ile Gln
325 330 335
Asp Lys Tyr Arg Ile Pro Asp Lys Glu Ile Thr Asp Gly Ala Ala Leu
340 345 350
Glu Asp Leu Tyr Ser Asp Asp Val Val Glu Ser Asn Lys Val Asp Phe
355 360 365
Asp Asp Asp Ile Pro Asp Asp Val Asp Leu Met Glu Glu
370 375 380
<210> 22
<211> 391
<212> Белок
<213> Фаг Enterobacteria T4
<400> 22
Met Ser Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser Lys Leu
1 5 10 15
Thr Ala Glu Leu Thr Ala Ser Lys Phe Phe Asn Glu Lys Asp Val Val
20 25 30
Arg Thr Lys Ile Pro Met Met Asn Ile Ala Leu Ser Gly Glu Ile Thr
35 40 45
Gly Gly Met Gln Ser Gly Leu Leu Ile Leu Ala Gly Pro Ser Lys Ser
50 55 60
Phe Lys Ser Asn Phe Gly Leu Thr Met Val Ser Ser Tyr Met Arg Gln
65 70 75 80
Tyr Pro Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Phe Gly Ile Thr
85 90 95
Pro Ala Tyr Leu Arg Ser Met Gly Val Asp Pro Glu Arg Val Ile His
100 105 110
Thr Pro Val Gln Ser Leu Glu Gln Leu Arg Ile Asp Met Val Asn Gln
115 120 125
Leu Asp Ala Ile Glu Arg Gly Glu Lys Val Val Val Phe Ile Asp Ser
130 135 140
Leu Gly Asn Leu Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu Asn Glu
145 150 155 160
Lys Val Val Ser Asp Met Thr Arg Ala Lys Thr Met Lys Ser Leu Phe
165 170 175
Arg Ile Val Thr Pro Tyr Phe Ser Thr Lys Asn Ile Pro Cys Ile Ala
180 185 190
Ile Asn His Thr Tyr Glu Thr Gln Glu Met Phe Ser Lys Thr Val Met
195 200 205
Gly Gly Gly Thr Gly Pro Met Tyr Ser Ala Asp Thr Val Phe Ile Ile
210 215 220
Gly Lys Arg Gln Ile Lys Asp Gly Ser Asp Leu Gln Gly Tyr Gln Phe
225 230 235 240
Val Leu Asn Val Glu Lys Ser Arg Thr Val Lys Glu Lys Ser Lys Phe
245 250 255
Lys Ile Asp Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser Gly Leu
260 265 270
Leu Asp Met Ala Leu Glu Leu Gly Phe Val Val Lys Pro Lys Asn Gly
275 280 285
Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met Ile Arg
290 295 300
Glu Glu Lys Ser Trp Arg Ala Lys Asp Thr Asn Cys Thr Thr Phe Trp
305 310 315 320
Gly Pro Leu Phe Lys His Gln Pro Phe Arg Asp Ala Ile Lys Arg Ala
325 330 335
Tyr Gln Leu Gly Ala Ile Asp Ser Asn Glu Ile Val Glu Ala Glu Val
340 345 350
Asp Glu Leu Ile Asn Ser Lys Val Glu Lys Phe Lys Ser Pro Glu Ser
355 360 365
Lys Ser Lys Ser Ala Ala Asp Leu Glu Thr Asp Leu Glu Gln Leu Ser
370 375 380
Asp Met Glu Glu Phe Asn Glu
385 390
<210> 23
<211> 393
<212> Белок
<213> Фаг Enterobacteria T6
<400> 23
Met Ser Ile Ala Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser
1 5 10 15
Lys Met Thr Ala Glu Leu Thr Thr Ser Lys Phe Phe Asn Glu Lys Asp
20 25 30
Val Ile Arg Thr Lys Ile Pro Met Leu Asn Ile Ala Ile Ser Gly Ala
35 40 45
Ile Asp Gly Gly Met Gln Ser Gly Leu Thr Ile Phe Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Met Ser Leu Thr Met Val Ala Ala Tyr Leu
65 70 75 80
Asn Lys Tyr Pro Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Phe Gly
85 90 95
Ile Thr Pro Ala Tyr Leu Arg Ser Met Gly Val Asp Pro Glu Arg Val
100 105 110
Ile His Thr Pro Ile Gln Ser Val Glu Gln Leu Lys Ile Asp Met Val
115 120 125
Asn Gln Leu Glu Ala Ile Glu Arg Gly Glu Lys Val Ile Val Phe Ile
130 135 140
Asp Ser Ile Gly Asn Met Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu
145 150 155 160
Asn Glu Lys Ser Val Ala Asp Met Thr Arg Ala Lys Ser Leu Lys Ser
165 170 175
Leu Phe Arg Ile Val Thr Pro Tyr Phe Ser Ile Lys Asn Ile Pro Cys
180 185 190
Val Ala Val Asn His Thr Ile Glu Thr Ile Glu Met Phe Ser Lys Thr
195 200 205
Val Met Thr Gly Gly Thr Gly Val Met Tyr Ser Ala Asp Thr Val Phe
210 215 220
Ile Ile Gly Lys Arg Gln Ile Lys Asp Gly Ser Asp Leu Gln Gly Tyr
225 230 235 240
Gln Phe Val Leu Asn Val Glu Lys Ser Arg Thr Val Lys Glu Lys Ser
245 250 255
Lys Phe Lys Ile Asp Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser
260 265 270
Gly Leu Leu Asp Met Ala Leu Glu Leu Gly Phe Val Val Lys Pro Lys
275 280 285
Asn Gly Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met
290 295 300
Ile Arg Glu Glu Lys Ser Trp Arg Ala Lys Asp Thr Asn Cys Thr Thr
305 310 315 320
Phe Trp Gly Pro Leu Phe Lys His Gln Pro Phe Arg Asp Ala Ile Lys
325 330 335
Arg Ala Tyr Gln Leu Gly Ala Ile Asp Ser Asn Glu Ile Val Glu Ala
340 345 350
Glu Val Asp Glu Leu Ile Asn Ser Lys Val Glu Lys Phe Lys Ser Pro
355 360 365
Glu Ser Lys Ser Lys Ser Ala Ala Asp Leu Glu Thr Asp Leu Glu Gln
370 375 380
Leu Ser Asp Met Glu Glu Phe Asn Glu
385 390
<210> 24
<211> 387
<212> Белок
<213> Фаг Acinetobacter 133
<400> 24
Met Ser Ser Leu Lys Glu Arg Leu Ile Lys Ala Ser Thr Ser Lys Met
1 5 10 15
Thr Ala Glu Leu Thr Lys Ser Lys Phe Phe Asn Asp Lys Thr Val Val
20 25 30
Arg Thr Arg Ile Pro Met Leu Asn Ile Ala Ile Ser Gly Ala Leu Asn
35 40 45
Gly Gly Met Gln Ser Gly Leu Thr Ile Phe Ala Gly Pro Ser Lys His
50 55 60
Phe Lys Ser Asn Met Gly Leu Thr Met Val Ala Ala Tyr Met Lys Ala
65 70 75 80
Phe Pro Asp Ala Val Cys Met Phe Tyr Asp Ser Glu Phe Gly Ile Thr
85 90 95
Pro Ala Tyr Leu Lys Ala Met Gly Val Asp Pro Asp Arg Val Ile His
100 105 110
Thr Pro Val Gln Ser Val Glu Gln Leu Lys Ile Asp Met Thr Asn Gln
115 120 125
Leu Glu Glu Val Lys Arg Gly Glu Lys Val Ile Val Phe Ile Asp Ser
130 135 140
Ile Gly Asn Leu Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu Asn Glu
145 150 155 160
Lys Thr Thr Ala Asp Met Thr Arg Ala Lys Ala Leu Lys Ser Leu Phe
165 170 175
Arg Ile Val Thr Pro Tyr Phe Ser Ile Lys Asp Ile Pro Cys Val Ala
180 185 190
Val Asn His Thr Leu Gln Thr Leu Glu Met Phe Ser Lys Glu Val Met
195 200 205
Thr Gly Gly Thr Gly Val Met Tyr Ser Ala Asp Thr Val Phe Phe Ile
210 215 220
Gly Lys Arg Gln Val Lys Asp Gly Thr Glu Leu Ala Gly Tyr Glu Phe
225 230 235 240
Ile Leu Lys Ala Glu Lys Ser Arg Met Val Lys Glu Lys Ser Val Phe
245 250 255
Lys Ile Thr Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser Gly Leu
260 265 270
Leu Glu Met Ala Thr Asp Leu Gly Phe Val Val Lys Pro Lys Val Gly
275 280 285
Trp Tyr Lys Arg Ala Met Met Val Asp Gly Val Met Gln His Glu Glu
290 295 300
Lys Ser Trp Arg Ala Lys Asp Thr Asp Ser Ile Asp Phe Trp Gly Pro
305 310 315 320
Leu Phe Lys His Asp Glu Phe Arg Lys Ala Ile Glu Thr Arg Tyr Gln
325 330 335
Leu Gly Ser Ile Glu Ser Asp Ala Glu Val Asp Ala Glu Val Asp Ala
340 345 350
Leu Ile Gly Ser Lys Thr Thr Ala Lys Ile Ser Gly Val Asn Phe Gly
355 360 365
Pro Ala Glu Ser Ala Ala Asp Lys Glu Gln Gln Leu Glu Asp Phe Val
370 375 380
Asp Glu Asp
385
<210> 25
<211> 390
<212> Белок
<213> Фаг Enterobacteria RB69
<400> 25
Met Ser Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser Lys Met
1 5 10 15
Thr Ala Asp Leu Thr Lys Ser Lys Leu Phe Asn Asn Arg Asp Glu Val
20 25 30
Pro Thr Arg Ile Pro Met Leu Asn Ile Ala Leu Gly Gly Ala Leu Asn
35 40 45
Ala Gly Leu Gln Ser Gly Leu Thr Ile Phe Ala Ala Pro Ser Lys His
50 55 60
Phe Lys Thr Leu Phe Gly Leu Thr Met Val Ala Ala Tyr Met Lys Lys
65 70 75 80
Tyr Lys Asp Ala Ile Cys Leu Phe Tyr Asp Ser Glu Phe Gly Ala Ser
85 90 95
Glu Ser Tyr Phe Arg Ser Met Gly Val Asp Leu Asp Arg Val Val His
100 105 110
Thr Pro Ile Gln Ser Val Glu Gln Leu Lys Val Asp Met Thr Asn Gln
115 120 125
Leu Asp Ala Ile Glu Arg Gly Asp Lys Val Ile Ile Phe Ile Asp Ser
130 135 140
Ile Gly Asn Thr Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu Asn Glu
145 150 155 160
Lys Val Val Gly Asp Met Ser Arg Ala Lys Ala Leu Lys Ser Leu Phe
165 170 175
Arg Ile Val Thr Pro Tyr Leu Thr Ile Lys Asp Ile Pro Cys Val Ala
180 185 190
Ile Asn His Thr Ala Met Glu Ile Gly Gly Leu Tyr Pro Lys Glu Ile
195 200 205
Met Gly Gly Gly Thr Gly Ile Leu Tyr Ser Ala Asn Thr Val Phe Phe
210 215 220
Ile Ser Lys Arg Gln Val Lys Glu Gly Thr Glu Leu Thr Gly Tyr Asp
225 230 235 240
Phe Thr Leu Lys Ala Glu Lys Ser Arg Thr Val Lys Glu Lys Ser Thr
245 250 255
Phe Lys Ile Thr Val Asn Phe Asp Gly Gly Ile Asp Pro Phe Ser Gly
260 265 270
Leu Leu Glu Met Ala Thr Glu Ile Gly Phe Val Val Lys Pro Lys Ala
275 280 285
Gly Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met Ile
290 295 300
Arg Glu Glu Lys Ser Trp Arg Ala Lys Ala Thr Asp Cys Val Glu Phe
305 310 315 320
Trp Gly Pro Leu Phe Lys His Lys Pro Phe Arg Asp Ala Ile Glu Thr
325 330 335
Lys Tyr Lys Leu Gly Ala Ile Ser Ser Ile Lys Glu Val Asp Asp Ala
340 345 350
Val Asn Asp Leu Ile Asn Cys Lys Ala Thr Thr Lys Val Pro Val Lys
355 360 365
Thr Ser Asp Ala Pro Ser Ala Ala Asp Ile Glu Asn Asp Leu Asp Glu
370 375 380
Met Glu Asp Phe Asp Glu
385 390
<210> 26
<211> 411
<212> Белок
<213> Фаг Aeromonas Aeh1
<400> 26
Met Ala Lys Gly Ile Lys Thr Ala Lys Thr Gly Asn Leu Gly Ser Leu
1 5 10 15
Met Ser Lys Leu Ala Gly Thr Ser Ser Asn Lys Met Ser Ser Val Leu
20 25 30
Ala Asp Ser Lys Phe Phe Asn Asp Lys Asp Cys Val Arg Thr Arg Val
35 40 45
Pro Leu Leu Asn Leu Ala Met Ser Gly Glu Leu Asp Gly Gly Leu Thr
50 55 60
Pro Gly Leu Thr Val Leu Ala Gly Pro Ser Lys His Phe Lys Ser Asn
65 70 75 80
Leu Ser Leu Val Phe Val Ala Ala Tyr Leu Arg Lys Tyr Pro Asp Ala
85 90 95
Val Cys Ile Phe Phe Asp Asn Glu Phe Gly Ser Thr Pro Gly Tyr Phe
100 105 110
Glu Ser Gln Gly Val Asp Ile Ser Arg Val Ile His Cys Pro Phe Lys
115 120 125
Asn Ile Glu Glu Leu Lys Phe Asp Ile Val Lys Lys Leu Glu Ala Ile
130 135 140
Glu Arg Gly Asp Arg Val Ile Val Phe Val Asp Ser Ile Gly Asn Ala
145 150 155 160
Ala Ser Lys Lys Glu Ile Asp Asp Ala Ile Asp Glu Lys Ser Val Ser
165 170 175
Asp Met Thr Arg Ala Lys Gln Ile Lys Ser Leu Thr Arg Met Met Thr
180 185 190
Pro Tyr Leu Thr Val Asn Asp Ile Pro Ala Ile Met Val Ala His Thr
195 200 205
Tyr Asp Thr Gln Glu Met Tyr Ser Lys Lys Val Val Ser Gly Gly Thr
210 215 220
Gly Ile Thr Tyr Ser Ser Asp Thr Val Ile Ile Ile Gly Arg Gln Gln
225 230 235 240
Glu Lys Asp Gly Lys Glu Leu Leu Gly Tyr Asn Phe Val Leu Asn Met
245 250 255
Glu Lys Ser Arg Phe Val Lys Glu Gln Ser Lys Leu Lys Leu Glu Val
260 265 270
Thr Phe Gln Gly Gly Ile Asn Thr Tyr Ser Gly Met Leu Asp Ile Ala
275 280 285
Leu Glu Val Gly Phe Val Val Lys Pro Ser Asn Gly Trp Phe Ser Arg
290 295 300
Ala Phe Leu Asp Glu Glu Thr Gly Glu Leu Val Glu Glu Asp Arg Lys
305 310 315 320
Trp Arg Arg Ala Asp Thr Asn Cys Leu Glu Phe Trp Lys Pro Met Phe
325 330 335
Ala His Gln Pro Phe Lys Thr Ala Cys Ser Asp Met Phe Lys Leu Lys
340 345 350
Ser Val Ala Val Lys Asp Glu Val Phe Asp Glu Val Asp Glu Leu Phe
355 360 365
Ser Gly Glu Ala Glu Met Pro Val Asn Met Gly Arg Lys Leu Asp Thr
370 375 380
Ala Asp Gln Glu Glu Ile Asp Gln Leu Glu Glu Val Asp Val Glu Gly
385 390 395 400
Ser Asp Ser Asp Glu Leu Phe Ala Asn Leu Asp
405 410
<210> 27
<211> 368
<212> Белок
<213> Фаг Aeromonas 65
<400> 27
Met Ala Lys Lys Ala Lys Val Val Asn Ser Gly Asp Leu Leu Glu Arg
1 5 10 15
Leu Asn Gly Thr Ser Ser Asn Lys Met Ser Ala Met Leu Ala Glu Ser
20 25 30
Ile Phe Phe Asn Glu Lys Asp Thr Ile Arg Thr Arg Val Pro Ile Ile
35 40 45
Asn Leu Met Met Ser Gly Arg Leu Asp Gly Gly Ile Thr Pro Gly Leu
50 55 60
Thr Cys Ile Ala Gly Pro Ser Lys His Phe Lys Ser Asn Leu Ser Leu
65 70 75 80
Val Met Val Ser Ala Tyr Leu Arg Lys Tyr Pro Lys Ala Val Cys Leu
85 90 95
Phe Phe Asp Asn Glu Phe Gly Ser Thr Pro Asp Tyr Phe Thr Ser Gln
100 105 110
Gly Val Asp Ile Ser Arg Val Val His Cys Pro Phe Ile Asp Val Glu
115 120 125
Glu Leu Lys Phe Asp Ile Val Lys Lys Leu Glu Ser Ile Thr Arg Gly
130 135 140
Asp Lys Val Ile Ile Tyr Ile Asp Ser Ile Gly Asn Val Ala Ser Lys
145 150 155 160
Lys Glu Leu Gln Asp Ala Lys Asp Glu Lys Ser Ala Gln Asp Met Thr
165 170 175
Arg Ala Lys Gln Ile Lys Ser Leu Phe Arg Met Val Thr Pro Tyr Leu
180 185 190
Thr Val Leu Asp Ile Pro Cys Ile Ala Val Asn His Thr Tyr Glu Thr
195 200 205
Gln Glu Met Phe Ser Lys Thr Val Met Ser Gly Gly Thr Gly Pro Met
210 215 220
Tyr Ser Ala Asp Thr Val Ile Ile Leu Gly Lys Gln Gln Asp Lys Asp
225 230 235 240
Gly Lys Glu Leu Leu Gly Tyr Asn Phe Val Met Asn Ala Glu Lys Ser
245 250 255
Arg Ala Ile Lys Glu Lys Ser Lys Leu Lys Leu Met Val Ser Phe Glu
260 265 270
Gly Gly Ile Asn Thr Tyr Ser Gly Leu Leu Lys Ile Ala Gln Glu Leu
275 280 285
Gly Phe Val Thr Lys Pro Gln Asn Ala Arg Tyr Gln Arg Asn Phe Leu
290 295 300
Asp Leu Glu Pro Gly Glu Met Val Ile Pro Glu Asp Glu Lys Lys Trp
305 310 315 320
Thr Glu Glu Glu Ser Asp Ser Leu Glu Phe Trp Lys Pro Met Phe Ser
325 330 335
His Lys Pro Phe Met Asp Ala Val Ser Asn Ala Tyr Lys Leu Lys Ala
340 345 350
Val Glu Val Ser Gln Glu Val Phe Asp Glu Val Asp Gln Leu Phe Gly
355 360 365
<210> 28
<211> 366
<212> Белок
<213> Фаг Vibrio KVP40
<400> 28
Met Ser Asp Leu Met Lys Ser Leu Lys Lys Ser Ser Thr Ser Gly Tyr
1 5 10 15
Ala Gln Val Leu Ser Glu Ser Gln Phe Met Phe Asp Lys Asp His Thr
20 25 30
Arg Thr Tyr Val Pro Ala Ile Asn Ile Ala Phe Ser Gly Glu Val Asp
35 40 45
Gly Gly Leu Thr Ser Gly Leu Thr Val Leu Ala Gly Pro Ser Lys His
50 55 60
Phe Lys Ser Asn Leu Gly Leu Val Gly Val Ala Ala Tyr Leu Lys Lys
65 70 75 80
Tyr Pro Asp Ala Val Cys Val Phe Ile Asp Thr Glu Phe Gly Ile Thr
85 90 95
Pro Ser Tyr Leu Arg Ser Gln Gly Val Asp Pro Asp Arg Val Leu His
100 105 110
Ile Gln Cys Glu Ser Val Glu Arg Met Lys Phe Glu Met Ala Asn Gln
115 120 125
Leu Lys Asp Leu Ala Glu Arg Lys Arg Ala Lys Lys Ala Gly Glu Glu
130 135 140
Pro Asp Arg Val Ile Phe Phe Ile Asp Ser Val Gly Asn Val Ala Ser
145 150 155 160
Ala Lys Glu Ile Asp Asp Ala Gln Asn Glu Lys Ser Val Ala Asp Met
165 170 175
Ser Arg Ala Lys Gln Leu Lys Ser Leu Phe Arg Ile Ile Thr Pro Tyr
180 185 190
Phe Thr Met Leu Asp Ile Pro Cys Ile Ala Ile Asn His Thr Tyr Gln
195 200 205
Thr Gln Glu Ile Tyr Ser Lys Thr Val Met Ser Gly Gly Thr Gly Ile
210 215 220
Met Tyr Ser Ala Asp Thr Val Ile Ile Leu Gly Lys Gln Gln Glu Lys
225 230 235 240
Asp Gly Lys Asp Ile Ile Gly Tyr His Phe Ile Met Asn Ile Glu Lys
245 250 255
Ser Arg Phe Val Lys Glu Lys Met Lys Val Lys Leu Thr Val Thr Tyr
260 265 270
Glu Asn Gly Ile Asp Pro Phe Ser Gly Leu Leu Asp Ile Ala Leu Gln
275 280 285
Thr Gly His Val Val Lys Pro Ser Asn Gly Trp Tyr Gln Arg Ala Thr
290 295 300
Val Asp Glu Glu Thr Gly Glu Met Ile Val Glu Glu Lys Lys Tyr Arg
305 310 315 320
Ala Lys Glu Thr Gln Thr Ile Ser Phe Trp Lys Asp Ile Ile Asn Ser
325 330 335
Pro Thr Phe Lys Glu Gly Val Lys Arg Ile Tyr Cys Leu Gly Gln Leu
340 345 350
Asp Glu Ser Glu Leu Phe Gly Glu Val Asp Ser Leu Phe Asp
355 360 365
<210> 29
<211> 386
<212> Белок
<213> Фаг Enterobacteria RB43
<400> 29
Met Ser Asn Lys Ala Leu Leu Lys Lys Leu Ile Lys Asn Ser Asn Ser
1 5 10 15
Gln Ser Ala Ala Ile Leu Ser Glu Ser Asp Val Phe Asn Asn Ile Thr
20 25 30
Lys Thr Arg Thr Arg Val Pro Ile Leu Asn Leu Ala Leu Ser Gly Ala
35 40 45
Phe Asp Gly Gly Leu Thr Ser Gly Leu Thr Leu Phe Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Leu Gly Leu Val Thr Val Ser Ala Tyr Leu
65 70 75 80
Lys Ala Asn Glu Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Lys Gly
85 90 95
Val Thr Lys Ser Tyr Leu Lys Ser Met Gly Val Asp Pro Asp Arg Val
100 105 110
Val Tyr Thr Arg Ile Thr Thr Val Glu Gln Leu Arg Asn Asp Val Val
115 120 125
Ser Gln Leu Asp Ala Leu Glu Arg Gly Asp Lys Val Ile Ile Phe Val
130 135 140
Asp Ser Val Gly Asn Thr Ala Ser Lys Lys Glu Leu Ala Asp Ala Leu
145 150 155 160
Ser Asp Asn Asp Lys Gln Asp Met Thr Arg Ala Lys Ala Leu Lys Gly
165 170 175
Met Phe Arg Met Val Thr Pro Tyr Leu Ala Asp Leu Asp Ile Pro Met
180 185 190
Val Cys Ile Cys His Thr Tyr Asp Thr Gln Glu Met Tyr Ser Lys Lys
195 200 205
Val Ile Ser Gly Gly Thr Gly Leu Met Tyr Ser Ala Asp Thr Ala Ile
210 215 220
Ile Leu Gly Lys Gln Gln Val Lys Glu Gly Thr Glu Val Val Gly Tyr
225 230 235 240
Asp Phe Ile Met Asn Ile Glu Lys Ser Arg Phe Val Lys Glu Lys Ser
245 250 255
Lys Phe Lys Leu His Val Thr Tyr Glu Gly Gly Ile Ser Met Tyr Ser
260 265 270
Gly Leu Leu Asp Leu Ala Met Glu Met Asn Phe Val Gln Thr Pro Thr
275 280 285
Lys Gly Trp Arg Gly Arg Ala Phe Leu Asn Thr Glu Thr Gly Glu Leu
290 295 300
Glu Leu Glu Glu Lys Lys Trp Arg Glu Ser Glu Thr Asn Ser Ile Glu
305 310 315 320
Phe Trp Arg Pro Leu Phe Thr His Gln Pro Phe Leu Asp Ala Ile Gln
325 330 335
Asp Lys Tyr Arg Ile Pro Asp Lys Glu Ile Thr Asp Gly Ala Ala Leu
340 345 350
Glu Asp Leu Tyr Ser Thr Asp Glu Pro Glu Ser Asn Lys Ile Asp Leu
355 360 365
Asp Asp Asp Ile Pro Asp Asp Ile Gly Ile Asp Gln Asp Glu Glu Pro
370 375 380
Ile Met
385
<210> 30
<211> 335
<212> Белок
<213> Фаг Prochlorococcus P-SSM2
<400> 30
Met Asp Phe Leu Lys Glu Ile Val Lys Glu Ile Gly Asp Glu Tyr Thr
1 5 10 15
Gln Val Ala Ala Asp Ile Gln Glu Asn Glu Arg Phe Ile Asp Thr Gly
20 25 30
Ser Tyr Ile Phe Asn Gly Leu Val Ser Gly Ser Ile Phe Gly Gly Val
35 40 45
Ser Ser Ser Arg Ile Thr Ala Ile Ala Gly Glu Ser Ser Thr Gly Lys
50 55 60
Thr Tyr Phe Ser Leu Ala Val Val Lys Asn Phe Leu Asp Asn Asn Pro
65 70 75 80
Asp Gly Tyr Cys Leu Tyr Phe Asp Thr Glu Ala Ala Val Asn Lys Gly
85 90 95
Leu Leu Glu Ser Arg Gly Ile Asp Met Asn Arg Leu Val Val Val Asn
100 105 110
Val Val Thr Ile Glu Glu Phe Arg Ser Lys Ala Leu Arg Ala Val Asp
115 120 125
Ile Tyr Leu Lys Thr Ser Glu Glu Glu Arg Lys Pro Cys Met Phe Val
130 135 140
Leu Asp Ser Leu Gly Met Leu Ser Thr Glu Lys Glu Ile Arg Asp Ala
145 150 155 160
Leu Asp Asp Lys Gln Val Arg Asp Met Thr Lys Ser Gln Leu Val Lys
165 170 175
Gly Ala Phe Arg Met Leu Thr Leu Lys Leu Gly Gln Ala Asn Ile Pro
180 185 190
Leu Ile Val Thr Asn His Thr Tyr Asp Val Ile Gly Ser Tyr Val Pro
195 200 205
Thr Lys Glu Met Gly Gly Gly Ser Gly Leu Lys Tyr Ala Ala Ser Thr
210 215 220
Ile Ile Tyr Leu Ser Lys Lys Lys Glu Lys Asp Gln Lys Glu Val Ile
225 230 235 240
Gly Asn Leu Ile Lys Ala Lys Thr His Lys Ser Arg Leu Ser Lys Glu
245 250 255
Asn Lys Glu Val Lys Ile Arg Leu Tyr Tyr Asp Glu Arg Gly Leu Asp
260 265 270
Arg Tyr Tyr Gly Leu Leu Glu Leu Gly Glu Ile Gly Gly Met Trp Lys
275 280 285
Asn Val Ala Gly Arg Tyr Glu Met Asn Gly Lys Lys Ile Tyr Ala Lys
290 295 300
Glu Ile Leu Lys Asn Pro Thr Glu Tyr Phe Thr Asp Asp Ile Met Glu
305 310 315 320
Gln Leu Asp Asn Ile Ala Lys Glu His Phe Ser Tyr Gly Thr Asn
325 330 335
<210> 31
<211> 336
<212> Белок
<213> Фаг Prochlorococcus P-SSM4
<400> 31
Met Asn Phe Leu Lys Asp Ile Ala Lys Glu Ile Gly Asn Asp Tyr Ala
1 5 10 15
Ser Leu Val Ser Glu Gly Val Ser Ala Gly Asp Thr Ala Gly Phe Ile
20 25 30
Asp Thr Gly Ser Tyr Ile Phe Asn Ala Leu Leu Ser Gly Ser Ile Tyr
35 40 45
Gly Gly Ile Pro Asn Asn Lys Ile Thr Ala Ile Ala Gly Glu Thr Ser
50 55 60
Thr Gly Lys Thr Phe Phe Cys Leu Gly Met Val Gln His Phe Leu Glu
65 70 75 80
Ser Asn Pro Asp Ala Gly Val Ile Tyr Phe Glu Ser Glu Ser Ala Ile
85 90 95
Ser Lys Gln Met Ile Glu Asp Arg Gly Ile Asp Ser Asn Arg Met Leu
100 105 110
Leu Val Pro Val Thr Thr Val Gln Glu Phe Arg Leu Gln Ala Ile Lys
115 120 125
Ile Leu Asp Lys Tyr Asn Glu Gln Thr Ala Glu Glu Arg Lys Pro Leu
130 135 140
Met Phe Val Leu Asp Ser Leu Gly Met Leu Ser Thr Ser Lys Glu Val
145 150 155 160
Glu Asp Ser Glu Ala Gly Lys Glu Thr Arg Asp Met Thr Arg Ala Gln
165 170 175
Val Val Lys Ser Ile Phe Arg Val Leu Thr Leu Lys Leu Gly Lys Ala
180 185 190
Asn Val Pro Leu Ile Val Thr Asn His Thr Tyr Asp Val Val Gly Ala
195 200 205
Tyr Ile Pro Thr Lys Glu Met Gly Gly Gly Ser Gly Leu Lys Tyr Ala
210 215 220
Ala Ser Thr Ile Val Tyr Leu Ser Lys Lys Lys Glu Lys Asn Gly Lys
225 230 235 240
Glu Val Val Gly Asn Ile Ile Lys Cys Lys Thr Ala Lys Ser Arg Leu
245 250 255
Thr Lys Glu Asn Ser Asp Val Lys Thr Arg Leu Tyr Tyr Asp Arg Gly
260 265 270
Leu Asp Arg Tyr Tyr Gly Leu Leu Glu Leu Gly Glu Lys His Gly Val
275 280 285
Phe Ser Arg Lys Gly Asn Arg Val Val Val Gly Asp Ser Ser Val Tyr
290 295 300
Pro Ser Ala Ile Leu Ala Asp Pro Asp Lys Tyr Phe Thr Glu Glu Leu
305 310 315 320
Met Glu Lys Leu Asp Glu Ala Ala Ala Lys Glu Phe Arg Tyr Gly Asn
325 330 335
<210> 32
<211> 343
<212> Белок
<213> Фаг Synechococcus S-PM2
<400> 32
Met Ser Phe Leu Asp Ser Val Ile Lys Asp Ser Lys Asn Glu Tyr Ala
1 5 10 15
Ala Phe Ala Ser Glu Gly Val Ala Ala Gly Asp Val Glu Ser Phe Val
20 25 30
Asp Thr Gly Ser Tyr Ile Phe Asn Ala Leu Val Ser Gly Ser Ile Phe
35 40 45
Gly Gly Ile Pro Ser Asn Lys Ile Thr Ala Leu Ala Gly Glu Ser Gly
50 55 60
Thr Gly Lys Thr Phe Phe Cys Leu Ser Val Val Arg Asn Phe Leu Asn
65 70 75 80
Thr Asp Pro Asp Ala Gly Val Ile Tyr Phe Glu Thr Glu Ser Ala Ile
85 90 95
Ser Lys Gln Met Ile Glu Ser Arg Gly Ile Asp Ser Thr Arg Met Ile
100 105 110
Ile Phe Pro Val Asp Thr Ile Glu Asp Phe Arg Thr Gln Ala Val Arg
115 120 125
Ile Ile Asp Lys Tyr Met Glu Gln Asn Lys Ser Glu Arg Lys Pro Leu
130 135 140
Met Phe Val Leu Asp Ser Leu Gly Met Leu Ala Thr Lys Lys Glu Val
145 150 155 160
Glu Asp Ala Ser Asn Asp Lys Gln Val Arg Asp Met Thr Lys Ala Gln
165 170 175
Ile Val Lys Ser Ala Phe Arg Ile Leu Thr Leu Lys Met Gly Lys Ala
180 185 190
Asn Ile Pro Met Leu Val Thr Asn His Thr Tyr Asp Val Val Gly Ser
195 200 205
Tyr Val Pro Thr Lys Glu Met Gly Gly Gly Ser Gly Leu Lys Tyr Ser
210 215 220
Ala Ser Thr Ile Val Tyr Leu Gly Lys Lys Lys Glu Lys Asp Gly Thr
225 230 235 240
Asp Leu Val Gly Asn Ile Ile Lys Cys Glu Ala Lys Lys Ser Arg Leu
245 250 255
Thr Arg Glu Gly Ser Lys Val Lys Thr Arg Leu Phe Phe Asp Gln Arg
260 265 270
Gly Leu Glu Arg Tyr Tyr Gly Met Leu Glu Leu Gly Glu Arg Ala Gly
275 280 285
Leu Trp Lys Asn Thr Ala Gly Arg Tyr Glu Ile Asn Gly Lys Lys Val
290 295 300
Tyr Gly Lys Gln Ile Leu Ala Asn Pro Asp Glu Phe Phe Thr Glu Glu
305 310 315 320
Ile Leu Gln Glu Leu Asp Lys Gln Ala Gln Arg Glu Phe Leu Tyr Gly
325 330 335
Ala Ser Asp Asp Gly Glu Asp
340
<210> 33
<211> 393
<212> Белок
<213> Фаг Enterobacteria RB32
<400> 33
Met Ser Ile Ala Asp Leu Lys Ser Arg Leu Ile Lys Ala Ser Thr Ser
1 5 10 15
Lys Met Thr Ala Glu Leu Thr Thr Ser Lys Phe Phe Asn Glu Lys Asp
20 25 30
Val Ile Arg Thr Lys Ile Pro Met Leu Asn Ile Ala Ile Ser Gly Ala
35 40 45
Ile Asp Gly Gly Met Gln Ser Gly Leu Thr Ile Phe Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Met Ser Leu Thr Met Val Ala Ala Tyr Leu
65 70 75 80
Asn Lys Tyr Pro Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Phe Gly
85 90 95
Ile Thr Pro Ala Tyr Leu Arg Ser Met Gly Val Asp Pro Glu Arg Val
100 105 110
Ile His Thr Pro Ile Gln Ser Val Glu Gln Leu Lys Ile Asp Met Val
115 120 125
Asn Gln Leu Glu Ala Ile Glu Arg Gly Glu Lys Val Ile Val Phe Ile
130 135 140
Asp Ser Ile Gly Asn Met Ala Ser Lys Lys Glu Thr Glu Asp Ala Leu
145 150 155 160
Asn Glu Lys Ser Val Ala Asp Met Thr Arg Ala Lys Ser Leu Lys Ser
165 170 175
Leu Phe Arg Ile Val Thr Pro Tyr Phe Ser Ile Lys Asn Ile Pro Cys
180 185 190
Val Ala Val Asn His Thr Ile Glu Thr Ile Glu Met Phe Ser Lys Thr
195 200 205
Val Met Thr Gly Gly Thr Gly Val Met Tyr Ser Ala Asp Thr Val Phe
210 215 220
Ile Ile Gly Lys Arg Gln Ile Lys Asp Gly Ser Asp Leu Gln Gly Tyr
225 230 235 240
Gln Phe Val Leu Asn Val Glu Lys Ser Arg Thr Val Lys Glu Lys Ser
245 250 255
Lys Phe Lys Ile Asp Val Lys Phe Asp Gly Gly Ile Asp Pro Tyr Ser
260 265 270
Gly Leu Leu Asp Met Ala Leu Glu Leu Gly Phe Val Val Lys Pro Lys
275 280 285
Asn Gly Trp Tyr Ala Arg Glu Phe Leu Asp Glu Glu Thr Gly Glu Met
290 295 300
Ile Arg Glu Glu Lys Ser Trp Arg Ala Lys Asp Thr Asn Cys Thr Thr
305 310 315 320
Phe Trp Gly Pro Leu Phe Lys His Gln Pro Phe Arg Asp Ala Ile Lys
325 330 335
Arg Ala Tyr Gln Leu Gly Ala Ile Asp Ser Asn Glu Ile Val Glu Ala
340 345 350
Glu Val Asp Glu Leu Ile Asn Ser Lys Val Glu Lys Phe Lys Ser Pro
355 360 365
Glu Ser Lys Ser Lys Ser Ala Ala Asp Leu Glu Thr Asp Leu Glu Gln
370 375 380
Leu Ser Asp Met Glu Glu Phe Asn Glu
385 390
<210> 34
<211> 366
<212> Белок
<213> Фаг Vibrio nt-1
<400> 34
Met Ser Asp Leu Leu Lys Ser Leu Lys Lys Ser Ser Thr Ser Gly Tyr
1 5 10 15
Ala His Val Leu Ser Glu Ser Gln Phe Met Phe Glu Lys Asp His Thr
20 25 30
Arg Thr Tyr Val Pro Ala Ile Asn Ile Ala Phe Ser Gly Glu Val Asp
35 40 45
Gly Gly Leu Thr Ser Gly Leu Thr Val Leu Ala Gly Pro Ser Lys His
50 55 60
Phe Lys Ser Asn Leu Gly Leu Val Gly Val Ala Ala Tyr Leu Lys Lys
65 70 75 80
Tyr Pro Glu Ala Ile Cys Val Phe Ile Asp Thr Glu Phe Gly Ile Thr
85 90 95
Pro Ser Tyr Leu Lys Ser Gln Gly Val Asp Pro Glu Arg Val Leu His
100 105 110
Ile Gln Cys Glu Ser Val Glu Arg Met Lys Phe Glu Met Ala Asn Gln
115 120 125
Leu Lys Asp Leu Ala Glu Arg Lys Arg Ala Lys Lys Ala Gly Glu Glu
130 135 140
Pro Asp Arg Val Val Phe Phe Ile Asp Ser Val Gly Asn Val Ala Ser
145 150 155 160
Ala Lys Glu Ile Asp Asp Ala Gln Asn Glu Lys Ser Val Ala Asp Met
165 170 175
Ser Arg Ala Lys Gln Leu Lys Ser Leu Phe Arg Ile Ile Thr Pro Tyr
180 185 190
Phe Thr Met Leu Asp Ile Pro Cys Ile Ala Ile Asn His Thr Tyr Gln
195 200 205
Thr Gln Glu Met Tyr Ser Lys Thr Val Met Ser Gly Gly Thr Gly Ile
210 215 220
Met Tyr Ser Ala Asp Thr Val Ile Ile Leu Gly Lys Gln Gln Glu Lys
225 230 235 240
Asp Gly Lys Glu Ile Ile Gly Tyr His Phe Ile Met Asn Ile Glu Lys
245 250 255
Ser Arg Phe Val Lys Glu Lys Met Lys Val Lys Leu Thr Val Thr Tyr
260 265 270
Glu His Gly Ile Asp Gln Phe Ser Gly Leu Leu Asp Ile Ala Leu Gln
275 280 285
Thr Gly His Val Val Lys Pro Ser Asn Gly Trp Tyr Gln Arg Ala Phe
290 295 300
Ile Asp Glu Glu Thr Gly Glu Ile Glu Ile Glu Glu Lys Lys Tyr Arg
305 310 315 320
Ala Lys Glu Thr Gln Thr Leu Ser Phe Trp Lys Glu Ile Ile Asn Ser
325 330 335
Pro Thr Phe Lys Thr Gly Val Lys Arg Leu Tyr Cys Leu Gly Gln Leu
340 345 350
Asp Glu Ser Glu Leu Leu Asp Glu Val Asp Ser Leu Phe Asp
355 360 365
<210> 35
<211> 381
<212> Белок
<213> Фаг Enterobacteria RB16
<400> 35
Met Ser Asn Lys Ala Leu Leu Lys Lys Leu Ile Lys Asn Ser Asn Ser
1 5 10 15
Gln Ser Ala Ser Ile Leu Ser Glu Ser Asp Val Phe Asn Asn Ile Thr
20 25 30
Lys Thr Arg Thr Arg Val Pro Ile Leu Asn Leu Val Leu Ser Gly Ala
35 40 45
Phe Asp Gly Gly Leu Thr Ser Gly Leu Thr Leu Ile Ala Gly Pro Ser
50 55 60
Lys His Phe Lys Ser Asn Leu Gly Leu Val Ala Val Ala Ala Tyr Leu
65 70 75 80
Lys Ala Asn Glu Asp Ala Val Cys Leu Phe Tyr Asp Ser Glu Lys Gly
85 90 95
Val Thr Lys Ser Tyr Leu Lys Ser Met Gly Val Asp Pro Asp Arg Val
100 105 110
Val Tyr Thr Arg Ile Thr Thr Val Glu Gln Leu Arg Asn Asp Val Val
115 120 125
Ser Gln Leu Asp Ala Leu Glu Arg Gly Asp Lys Val Ile Ile Phe Val
130 135 140
Asp Ser Val Gly Asn Thr Ala Ser Lys Lys Glu Leu Lys Asp Ala Leu
145 150 155 160
Glu Asp Asn Asp Lys Gln Asp Met Thr Arg Ala Lys Ala Leu Lys Gly
165 170 175
Met Phe Arg Met Val Thr Pro Tyr Leu Ala Asp Ile Asp Ile Pro Met
180 185 190
Val Cys Ile Cys His Thr Tyr Asp Thr Gln Glu Met Tyr Ser Lys Lys
195 200 205
Val Ile Ser Gly Gly Thr Gly Leu Met Tyr Ser Ala Asp Thr Ala Ile
210 215 220
Ile Leu Gly Lys Gln Gln Val Lys Glu Gly Thr Glu Val Val Gly Tyr
225 230 235 240
Asp Phe Ile Met Asn Val Glu Lys Ser Arg Phe Val Lys Glu Lys Ser
245 250 255
Lys Phe Lys Leu His Val Thr Tyr Glu Gly Gly Ile Ser Met Phe Ser
260 265 270
Gly Leu Leu Asp Leu Ala Met Glu Met Asn Phe Val Gln Thr Pro Thr
275 280 285
Lys Gly Trp Arg Gly Arg Ala Phe Leu Asn Thr Glu Thr Gly Glu Leu
290 295 300
Glu Leu Glu Glu Lys Lys Trp Arg Glu Ala Glu Thr Asn Cys Ile Glu
305 310 315 320
Phe Trp Lys Pro Leu Phe Lys His Gln Pro Phe Ile Asp Ala Ile Gln
325 330 335
Asp Lys Tyr Arg Ile Pro Asp Lys Glu Ile Thr Asp Gly Ala Ala Leu
340 345 350
Glu Asp Leu Tyr Ser Asp Asp Val Val Glu Ser Asn Lys Val Asp Phe
355 360 365
Asp Asp Asp Ile Pro Asp Asp Val Asp Leu Met Glu Glu
370 375 380
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Конструкции для направленного редактирования генов и способы с их применением | 2020 |
|
RU2832109C2 |
| Способ получения модифицированного капсида аденоассоциированного вируса | 2021 |
|
RU2831732C1 |
| СПОСОБ ПРОДУЦИРОВАНИЯ АРЕНАВИРУСА, А ТАКЖЕ МУТАНТОВ АРЕНАВИРУСА С ПРОТИВООПУХОЛЕВЫМИ СВОЙСТВАМИ | 2019 |
|
RU2812046C2 |
| СПЕЦИФИЧНЫЕ К MUC16 ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2795198C2 |
| ВАРИАНТЫ FC-ОБЛАСТИ С МОДИФИЦИРОВАННОЙ СПОСОБНОСТЬЮ СВЯЗЫВАТЬСЯ С FCRN И С СОХРАНЕННОЙ СПОСОБНОСТЬЮ СВЯЗЫВАТЬСЯ С БЕЛКОМ А | 2015 |
|
RU2727639C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| ВАРИАНТЫ FC-ОБЛАСТИ С МОДИФИЦИРОВАННЫМИ СПОСОБНОСТЯМИ СВЯЗЫВАТЬСЯ С FCRN | 2015 |
|
RU2730592C2 |
| Выделенная нуклеиновая кислота, которая кодирует слитый белок на основе FVIII-BDD и гетерологичного сигнального пептида, и ее применение | 2022 |
|
RU2818229C2 |
| Выделенный модифицированный белок VPI капсида аденоассоциированного вируса 9 серотипа (AAV9), капсид и вектор на его основе | 2021 |
|
RU2825667C2 |
| СНИЖЕНИЕ ПРЕВРАЩЕНИЯ НИКОТИНА В НОРНИКОТИН В РАСТЕНИЯХ | 2015 |
|
RU2733837C2 |




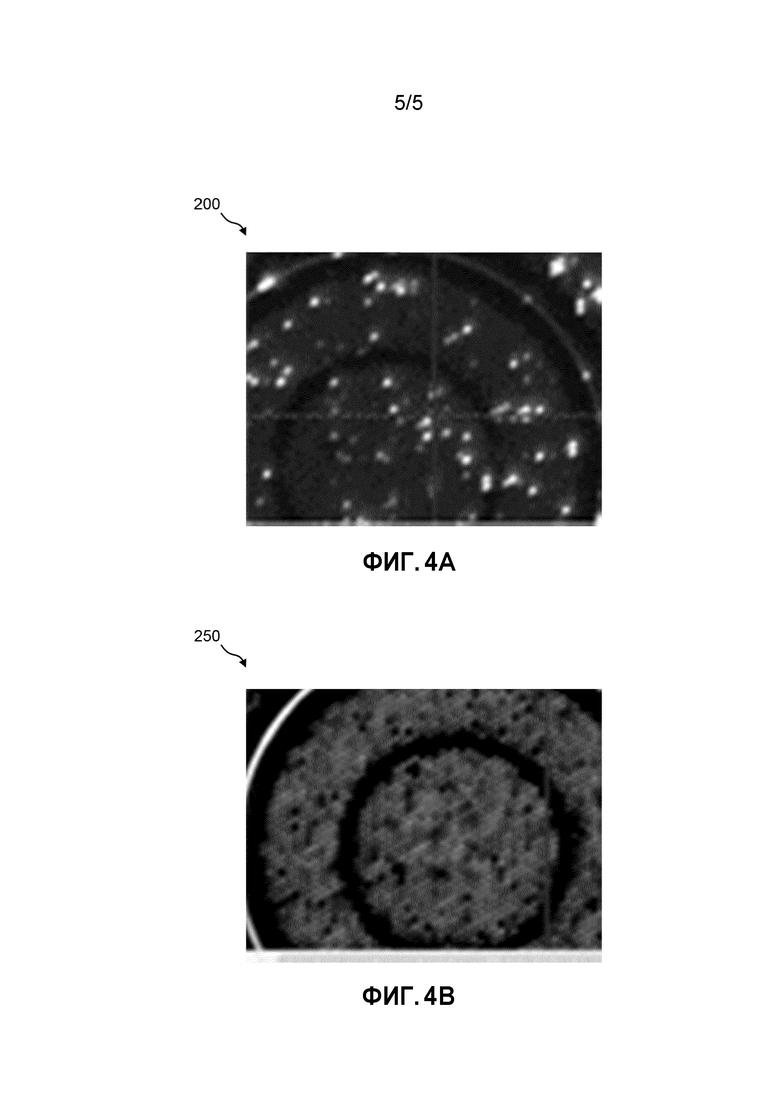
Группа изобретений относится к вариантам рекомбинантной рекомбиназы UvsX, молекуле нуклеиновой кислоты, кодирующей рекомбинантную рекомбиназу UvsX, способу рекомбиназной полимеразной амплификации для амплификации мишеневой молекулы нуклеиновой кислоты с использованием указанной рекомбиназы, а также набору, ее содержащему. Предложена рекомбинантная рекомбиназа UvsX, содержащая аминокислотную последовательность, которая по меньшей мере на 90%, 95% или 99% идентична последовательности SEQ ID NO:1, где рекомбинантная рекомбиназа UvsX содержит замену на лизиновый остаток в положении, функционально эквивалентном положению 256 в последовательности SEQ ID NO:1 UvsX RB49.Также предложена рекомбинантная рекомбиназа UvsX, содержащая аминокислотную последовательность, которая по меньшей мере на 90%, 95% или 99% идентична последовательности SEQ ID NO:1, где рекомбинантная рекомбиназа UvsX содержит замену на лизиновый остаток в положении, функционально эквивалентном положению 256 в последовательности SEQ ID NO:1 UvsX RB49, и замену на сериновый остаток в положении, функционально эквивалентном His63 в аминокислотной последовательности RB49 UvsX. Также предложены молекула нуклеиновой кислоты, кодирующая рекомбинантную рекомбиназу UvsX, способ рекомбиназной полимеразной амплификации для амплификации мишеневой молекулы нуклеиновой кислоты с использованием указанной рекомбиназы, а также набор для осуществления рекомбиназной полимеразной реакции, содержащий указанную рекомбиназу. Группа изобретений обеспечивает улучшение рекомбиназо-опосредованной амплификации нуклеиновых кислот. 5 н. и 17 з.п. ф-лы, 7 ил., 3 табл., 3 пр.
1. Рекомбинантная рекомбиназа UvsX, содержащая аминокислотную последовательность, которая по меньшей мере на 90%, 95% или 99% идентична последовательности SEQ ID NO:1,
где рекомбинантная рекомбиназа UvsX содержит замену на лизиновый остаток в положении, функционально эквивалентном положению 256 в последовательности SEQ ID NO:1 UvsX RB49.
2. Рекомбинантная рекомбиназа UvsX по п.1, содержащая аминокислотную последовательность SEQ ID NO:2.
3. Рекомбинантная рекомбиназа UvsX по п.1, содержащая замещающую мутацию в полуконсервативном домене, содержащем аминокислотную последовательность любой последовательности SEQ ID NO:3-5, где замещающая мутация содержит мутацию в положении 7 последовательности SEQ ID NO:3-5 на остаток лизина.
4. Рекомбинантная рекомбиназа UvsX по п.1, содержащая замещающую мутацию в полуконсервативном домене, содержащем аминокислотную последовательность любой последовательности SEQ ID NO:6, 7, где замещающая мутация содержит мутацию в положении 12 последовательностей SEQ ID NO:6, 7 на остаток лизина.
5. Рекомбинантная рекомбиназа UvsX, содержащая аминокислотную последовательность, которая по меньшей мере на 90%, 95% или 99% идентична последовательности SEQ ID NO:1,
где рекомбинантная рекомбиназа UvsX содержит замену на лизиновый остаток в положении, функционально эквивалентном положению 256 в последовательности SEQ ID NO:1 UvsX RB49, и замену на сериновый остаток в положении, функционально эквивалентном His63 в аминокислотной последовательности RB49 UvsX.
6. Рекомбинантная рекомбиназа UvsX по п.5, содержащая аминокислотную последовательность SEQ ID NO:2.
7. Рекомбинантная рекомбиназа UvsX по п.5, содержащая замещающую мутацию в полуконсервативном домене, содержащем аминокислотную последовательность любой последовательности SEQ ID NO:3-5, где замещающая мутация содержит мутацию в положении 7 последовательности SEQ ID NO:3-5 на остаток лизина.
8. Рекомбинантная рекомбиназа UvsX по п.5, содержащая замещающую мутацию в полуконсервативном домене, содержащем аминокислотную последовательность любой последовательности SEQ ID NO:6, 7, где замещающая мутация содержит мутацию в положении 12 последовательностей SEQ ID NO:6, 7 на остаток лизина.
9. Рекомбинантная рекомбиназа UvsX по любому из предыдущих пунктов, которая содержит мутацию, выбранную из группы, состоящей из:
добавления одного или нескольких остатков глутаминовой кислоты на C-конце;
добавления одного или нескольких остатков аспарагиновой кислоты на С-конце; и
их сочетание.
10. Молекула нуклеиновой кислоты, кодирующая рекомбинантную рекомбиназу UvsX по любому из предыдущих пунктов.
11. Способ рекомбиназной полимеразной амплификации для амплификации мишеневой молекулы нуклеиновой кислоты, включающий стадии:
(a) приведение рекомбинантной рекомбиназы UvsX по любому из пп.1-9 в контакт с первым и вторым праймерами на основе нуклеиновой кислоты с образованием первого и второго нуклеопротеиновых праймеров, где указанный праймер на основе нуклеиновой кислоты содержит одноцепочечную область на своем 3' конце;
(b) приведение первого и второго нуклеопротеиновых праймеров в контакт с указанной мишеневой молекулой нуклеиновой кислоты с образованием первой двухцепочечной структуры на первом участке указанной первой цепи и с образованием второй двухцепочечной структуры на втором участке указанной второй цепи, при этом 3'-концы указанного первого нуклеотидного праймера и указанного второго нуклеотидного праймера направлены друг на друга в одной и той же двухцепочечной нуклеотидной матричной молекуле;
(с) удлинения 3'-конца указанного первого и второго нуклеотидного праймера с помощью одной или нескольких полимераз и dNTP с получением первой и второй двухцепочечной нуклеиновой кислоты и первой и второй нуклеиновой кислоты с заменой цепей; и
(d) повторение стадий (b) и (с) при дальнейшем осуществлении реакции до достижения желаемой степени амплификации.
12. Способ по п.11, где указанная мишеневая молекула нуклеиновой кислоты содержит двухцепочечную нуклеиновую кислоту и/или одноцепочечную область адаптера.
13. Способ по п.11, который проводят в присутствии белка-загрузчика рекомбиназы.
14. Способ по п.13, где белок-загрузчик рекомбиназы выбран из группы, состоящей из Т4 UvsY, E.coli recO, E.coli recR или их комбинации.
15. Способ по п.11, который проводят в присутствии одноцепочечного стабилизирующего агента, выбранного из группы, состоящей из gp32, белка E.coli SSB, белка gp32 T4.
16. Способ по п.11, который проводят в присутствии концентрирующего агента, выбранного из группы, состоящей из полиэтиленгликоля, полиэтиленоксида, полистирола, фиколла, декстрана, PVP и альбумина, так что концентрирующий агент стимулирует амплификацию.
17. Способ по п.11, который проводят на массиве амплификационных сайтов.
18. Способ по п.17, где каждый амплификационный сайт содержит множество амплификационных праймеров для амплификации мишеневой нуклеиновой кислоты.
19. Способ по п.11, где массив амплификационных сайтов содержит массив признаков на поверхности.
20. Способ по п.19, где признаки являются несмежными и отделены друг от друга промежуточными областями поверхности, на которых отсутствуют амплификационные праймеры.
21. Способ по п.11, в котором процесс проходит изотермически.
22. Набор для осуществления рекомбиназной полимеразной реакции, включающий:
рекомбинантную рекобминазу UvsX по любому из пп.1-9 и одно или несколько из следующего:
связывающего одноцепочечную ДНК белка; ДНК-полимеразы;
dNTP или смеси dNTP и ddNTP; концентрирующего агента; буфера;
восстанавливающего агента; АТР или аналога АТР;
белка-загрузчика рекомбиназы;
первого праймера и
второго праймера.
| WO 2008035205 A2, 27.03.2008 | |||
| WO 2010141940 A1, 09.12.2010 | |||
| PIEPENBURG O | |||
| ET AL | |||
| DNA detection using recombination proteins | |||
| PLoS Biol | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| PAKULA AA ET.AL., Genetic analysis of protein stability and function, Anna | |||
| Rev | |||
| Genet, 1989, No.23, pp.289-310 (см | |||
| Держатель для поленьев при винтовом колуне | 1920 |
|
SU305A1 |
| СИНГЕР М., БЕРГ П | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Пер | |||
| с | |||
Авторы
Даты
2020-05-25—Публикация
2015-09-29—Подача