ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к не встречающимся в природе микроорганизмам, которые способны продуцировать N-ацетилнейраминовую кислоту, к способам получения N-ацетилнейраминовой кислоты посредством ферментации с использованием таких не встречающихся в природе микроорганизмов, к применению N-ацетилнейраминовой кислоты, получаемой посредством ферментации, а также к продуктам, содержащим N-ацетилнейраминовую кислоту, полученную таким образом.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Сиаловые кислоты (Sia) представляют собой семейство отрицательно заряженных моносахаридов, имеющих остов из девяти атомов углерода. В природе было обнаружено более 50 форм этих α-кетокислот. Как оказалось, наиболее широко распространенной сиаловой кислотой является N-ацетилнейраминовая кислота (обозначаемая NANA, NeuNAc, Neu5Ac).
Сиаловые кислоты присутствуют в виде терминальных сахаридов в гликанах, содержащихся в гликоконъюгатах (гликопротеины и гликолипиды) на поверхности клеток позвоночных и высших беспозвоночных. Сиаловые кислоты являются компонентами липополисахаридов и капсульных полисахаридов патогенных бактерий, примеры которых включают: Escherichia coli K1, Haemophilus influenzae, Haemophilus ducreyi, Pateurella multocida, Neisseria gonorrhoeae, Neisseria meningitidis, Campylobacter jejuni и Streptococcus agalactiae.
Сиаловые кислоты играют важную роль во множестве физиологических и патофизиологических процессов, включая развитие нервной системы эмбриона, метастазирование, регулирование иммунных ответов и инфицирование бактериями или вирусами. Сиаловые кислоты представляют собой необходимый компонент ганглиозидов мозга и цепочек полисиаловых кислот, которые модифицируют нейрональные молекулы клеточной адгезии (НМКА), способствующие взаимодействию клеток друг с другом, разрастанию нейронов, модификации конфигурации синаптических связей и формированию памяти. Питание поросят пищей, обогащенной сиаловыми кислотами, повышает концентрацию сиаловых кислот в мозгу и экспрессию двух генов, связанных с обучением. Соответственно, такое питание также усиливает способности к обучению и улучшает память.
Из-за быстрого роста мозга и развития иммунной системы для младенцев, в частности, недоношенных младенцев, на соответствующей стадии развития требуются питательные вещества, включающие сиаловые кислоты. Высокие концентрации сиаловых кислот, в частности, N-ацетилнейраминовой кислоты, содержатся в женском грудном молоке (приблизительно 0,5 г⋅л-1). Напротив, детские питательные смеси содержат низкие или даже незначительные количества N-ацетилнейраминовой кислоты.
Таким образом, необходимо получить сиаловые кислоты, в частности, Neu5Ac, приемлемого качества и в количестве, достаточном для восполняющего введения в детские питательные смеси и другие питательные композиции. Для достижения этой цели уже предпринимались многочисленные попытки.
В документе ЕР 1484406 А1 рассмотрен способ получения N-ацетилнейраминовой кислоты с использованием микроорганизма, который способен продуцировать Neu5Ac, но лишь ограниченно может разлагать или вовсе не может разлагать Neu5Ac в сравнении со штаммом дикого типа, в результате чего Neu5Ac накапливается в культуральной среде и может быть из нее выделена. Для того, чтобы получение Neu5Ac стало возможным, микроорганизм должен обладать высокой активностью синтазы N-ацетилнейраминовой кислоты и/или высокой активностью N-ацетилглюкозамин-2-эпимеразы. В частности, был проведен случайный мутагенез клеток Е. Coli, и клеточную линию, которая преимущественно росла в среде, содержащей глюкозу, но ограниченно или совсем не разрасталась в среде, содержащей N-ацетилнейраминовую кислоту, трансформировали экспрессионной плазмидой, кодирующей синтазу N-ацетилнейраминовой кислоты и N-ацетилглюкозамин-2-эпимеразу. По завершении периода культивирования, клетки собирали в гранулы центрифугированием, хранили при -20°С в виде так называемых "мокрых клеток" и при необходимости использовали после размораживания. Для получения N-ацетилнейраминовой кислоты использовали реакционную смесь (30 мл), включающую 90 г⋅л-1 N-ацетилглюкозамина, 50 г⋅л-1 глюкозы, 10 мл⋅л-1 ксилола и 200 г⋅л-1 указанных мокрых клеток, пермеабилизированных в присутствии 4 г⋅л-1 моющего средства. По завершении реакции in vitro, образование Neu5Ac оценивали способом ВЭЖХ (высокоэффективной жидкостной хроматографии).
В документе WO 94/29476 А1 раскрыт in vitro способ получения N-ацетил-D-нейраминовой кислоты из N-ацетил-D-глюкозамина (NAG, GlcNAc). В процессе получения NAG превращается в N-ацетил-D-маннозамин (NAM, ManNAc) в результате эпимеризации, катализируемой основанием. Затем NAM реагирует с пируватом с образованием Neu5Ac в результате реакции, катализируемой Neu5Ac-альдолазой. Neu5Ac-альдолазу получали из рекомбинантных клеток Е. coli, экспрессирующих Neu5Ac-альдолазу. Фермент альдолаза был иммобилизован на смеси гранул Eupergit-C® с неочищенным экстрактом рекомбинантных клеток Е. coli. Превращение NAM в Neu5Ac инициировали добавлением к смеси NAM и пирувата гранул, содержащих названный иммобилизованный фермент. По окончании реакции Neu5Ac выделяли из реакционной смеси.
В качестве альтернативы приведенному выше способу, в документе ЕР 0578825 А1 раскрыт способ получения in vitro N-ацетилнейраминовой кислоты, включающий обработку смеси N-ацетилглюкозамина и пировиноградной кислоты лиазой N-ацетилнейраминовой кислоты в щелочных условиях.
В патенте US 7579175 раскрыт способ получения N-ацетилнейраминовой кислоты с использованием пермеабилизированных микроорганизмов. Способ включает получение смеси, содержащей: (i) культуру микроорганизма, в котором имеется активность альдолазы N-ацетилнейраминовой кислоты или активность синтетазы N-ацетилнейраминовой кислоты, или обработанный материал этой культуры, (ii) культуру микроорганизма, который может продуцировать пировиноградную кислоту, или обработанный материал этой культуры или культуру микроорганизма, который может продуцировать фосфоенолпировиноградную кислоту, или обработанный материал этой культуры, (iii) N-ацетилманнозамин и (iv) источник энергии, необходимой для образования пировиноградной кислоты или фосфоенолпировиноградной кислоты. Смесь готовят в водной среде, содержащей хелатирующий агент или поверхностно-активное вещество, способствующее образованию и накоплению N-ацетилнейраминовой кислоты в водной среде, после чего извлекают N-ацетилнейраминовую кислоту из водной среды.
Рассмотренные выше способы имеют следующие недостатки: (i) синтез возможен лишь в малом масштабе и (ii) для сдвига равновесия реакции в сторону Neu5Ac требуется избыток пирувата. Кроме того, необходимые для проведения указанных реакций материалы N-ацетилглюкозамин, N-ацетилманнозамин и фосфоенолпируват имеют высокую стоимость.
В опубликованной международной патентной заявке WO 2008/040717 А2 раскрыт способ получения сиаловой кислоты, включающий культивирование микроорганизма в среде, причем микроорганизм содержит гетерологичные гены, кодирующие синтазу сиаловой кислоты (NeuB) и эпимеразу UDP-GlcNAc (NeuC), причем указанный микроорганизм не имеет гена, кодирующего синтазу CMP-Neu5Ac (NeuA), или любые гены, кодирующие синтазу CMP-Neu5Ac (NeuA), были инактивированы или удалены, и эндогенные гены, кодирующие альдолазу сиаловой кислоты (NanA), транспортер сиаловой кислоты (NanT) и, необязательно, ManNAc-киназу (NanK), были удалены или были инактивированы. Neu5Ac отделяли от культуральной жидкости над осадком (2 литра) осаждением посредством добавления ледяной уксусной кислоты.
В опубликованной международной патентной заявке WO 2008/097366 А2 рассмотрены клетки Е. coli с искусственно измененным метаболизмом, продуцирующие сиаловую кислоту. В этих клетках инактивированы гены nanT (транспортера сиаловой кислоты) и nanA (альдолазы сиаловой кислоты), и в указанные клетки nanT- nanA- у Е. coli введены гены neuC и neuB, способствующие биосинтезу сиаловой кислоты в организмах Neisseria meningitidis группы В, где они свехэкспрессируются при помощи экспрессионных плазмид. Кроме того, наряду с neuB и neuC свехэкспрессируется ген синтазы глюкозамина Е. coli (glmS).
В опубликованной международной патентной заявке WO 2012/083329 А1 раскрыты способы и агенты для получения Neu5Ac в клетках грибков рода Trichoderma, которые конститутивно экспрессируют N-ацетилглюкозамин-2-эпимеразу и синтазу N-ацетилнейраминовой кислоты. Клетки Trichoderma культивировали в присутствии GlcNAc, и мицелий анализировали способом ВЭЖХ-МС на наличие Neu5Ac.
В китайской патентной заявке CN 106929461 А раскрыт способ получения N-ацетилнейраминовой кислоты в клетках Bacillus subtilis, которые экспрессируют гены, кодирующие глюкозамин-фруктозо-6-фосфат-трансаминазу, глюкозамин-6-фосфат-N-ацетилтрансферазу, N-ацетилглюкозаминизомеразу и синтазу N-ацетилнейраминовой кислоты. Кроме того, в этих клетках отсутствует ген ptsG, который кодирует глюкозоспецифичный компонент системы фосфотрансферазы EIICBA. При культивировании этих клеток в глюкозосодержащей среде выход Neu5Ac составлял 0,66 г⋅л-1.
Zhu D. и соавт. (Zhu, D. et al. (2017) Biotechnol. Lett. 39: 227-234) сообщали, что использование многокопийного вектора совместной экспрессии для сверхэкспрессии генов, связанных с синтезом PEP (сокр. от phosphoenolpyruvate, т.е. фосфоенолпирувата), pck и ppsA, в культуре Е. coli повышает продукцию Neu5Ac.
Таким образом, задача настоящего изобретения состоит в предоставлении микробных организмов, которые способны более эффективно продуцировать сиаловую кислоту в промышленном масштабе и с использованием недорого источника углерода в качестве единственного источника углерода.
Поставленная задача может быть решена посредством предоставления не встречающегося в природе микроорганизма, у которого имеется путь синтеза сиаловой кислоты, который включает по меньшей мере один гетерологичный фермент, причем в микроорганизме блокирован естественный катаболический путь сиаловой кислоты, улучшена доступность фосфоенолпировиноградной кислоты для биосинтеза Neu5Ac, и микроорганизм способен использовать единственный недорогой источник экзогенного углерода, присутствующий в ферментативном бульоне и не использовать систему фосфоенолпировиноградная кислота : фосфотрансфераза в качестве источника экзогенного углерода.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Первый аспект изобретения относится к не встречающемуся в природе микроорганизму для получения Neu5Ac, причем у не встречающегося в природе микроорганизма имеется путь синтеза сиаловой кислоты, включающий по меньшей мере один гетерологичный фермент, при этом в микроорганизме блокирован природный катаболический путь сиаловой кислоты, блокирована по меньшей мере одна система фосфотрансферазы для импорта сахарида, который не используется в качестве источника углерода в процессе ферментативного продуцирования Neu5Ac, и при этом указанный не встречающийся в природе микроорганизм может использовать источник экзогенного углерода, присутствующий в ферментативном бульоне, не используя систему фосфотрансферазы для доступа к указанному источнику экзогенного углерода.
Второй аспект относится к применению не встречающихся в природе микроорганизмов согласно первому аспекту для продуцирования Neu5Ac.
Третий аспект относится к способу получения Neu5Ac посредством ферментации с использованием не встречающегося в природе микроорганизма согласно первому аспекту.
Четвертый аспект относится к Neu5Ac, полученной способом согласно второму аспекту.
Пятый аспект относится к применению Neu5Ac согласно четвертому аспекту для изготовления питательной композиции.
Шестой аспект относится к питательной композиции, включающей Neu5Ac, которая была получена способом согласно третьему аспекту.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
На Фиг. 1 представлена схема метаболических путей синтеза Neu5Ac.
На Фиг. 2 представлена схема дополнительных и/или альтернативных метаболических путей синтеза Neu5Ac.
На Фиг. 3 представлена гистограмма, на которой показаны уровни продукции Neu5Ac различными не встречающимися в природе штаммами Е. coli.
На Фиг. 4 представлена гистограмма, на которой показаны уровни продукции Neu5Ac различными не встречающимися в природе штаммами Е. coli.
СВЕДЕНИЯ, ПОДТВЕРЖДАЮЩИЕ ВОЗМОЖНОСТЬ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Первый аспект изобретения относится к не встречающемуся в природе микроорганизму, который способен продуцировать Neu5Ac. У не встречающегося в природе микроорганизма имеется путь биосинтеза сиаловой кислоты, включающий по меньшей мере один гетерологичный фермент, который экспрессируется из гетерологичной нуклеотидной последовательности, что позволяет микроорганизму вырабатывать сиаловую кислоту. Естественный катаболический путь сиаловой кислоты в таких микроорганизмах заблокирован. Также заблокирована по меньшей мере одна система фосфоенолпируват : сахар-фосфотрансфераза. Не встречающийся в природе микроорганизм может использовать предоставляемый извне источник углерода в качестве единственного источника углерода, то есть для доступа к этому источнику углерода ему не требуется система фосфоенолпируват : сахар-фосфотрансфераза.
Употребляемый в настоящей работе термин "не встречающийся в природе микроорганизм" относится к микроорганизму, который был генетически модифицирован с возможностью введения по меньшей мере одной гетерологичной нуклеотидной последовательности и/или модификации природной нуклеотидной последовательности, имеющейся в микроорганизме, т.е. внесение изменений, замен, вставок или делеций в эту последовательность.
Употребляемый в настоящей работе термин "гетерологичный" относится к соединению, полипептиду, белку, ферменту, молекуле нуклеиновой кислоты или нуклеотидной последовательности - как части молекулы нуклеиновой кислоты - которые не могут содержаться в организме-хозяине, встречающемся в природе. "Гетерологичная нуклеотидная последовательность" может представлять собой ген или фрагмент гена. Термин "гетерологичная экспрессия" относится к экспрессии гетерологичного гена или фрагмента гена, которые не могут экспрессироваться в организме-хозяине, встречающемся в природе. Экспрессия гетерологичного гена приводит к образованию гетерологичного полипептида, белка или фермента в организме-хозяине.
Не встречающийся в природе микроорганизм может продуцировать (вырабатывать, синтезировать) Neu5Ac. Употребляемый в настоящей работе термин "продуцировать" относится к получению Neu5Ac посредством микробной ферментации. Под "микробной ферментацией" понимают обычно крупномасштабный промышленный способ, в котором целевой продукт, например, Neu5Ac, получают культивированием микроорганизма в ферментативном бульоне, содержащем питательные вещества, в котором микроорганизм может превращать одни соединения в другие соединения. Термины "крупномасштабный" и "промышленный" указывают на то, что получение микробной ферментацией может происходить в объеме ферментативного бульона, который превышает 100 л, 500 л, 1000 л, 5000 л, 10000 л, 50000 л, 100000 л или даже 200000 л.
Употребляемый в настоящей работе термин "который может продуцировать (вырабатывать, синтезировать)" или "способный продуцировать" относится к способности микроорганизма вырабатывать Neu5Ac, при условии, что организм культивируют в среде или бульоне в условиях, которые позволяют микроорганизму синтезировать Neu5Ac.
Путь биосинтеза сиаловой кислоты
Не встречающийся в природе микроорганизм представляет собой микроорганизм, подходящий для получения Neu5Ac. Таким образом, не встречающийся в природе микроорганизм способен вырабатывать Neu5Ac. Не встречающийся в природе микроорганизм представляет собой микроорганизм, измененный способами генетической инженерии, что позволяет микроорганизму реализовывать путь биосинтеза сиаловой кислоты.
В одном из примеров осуществления путь биосинтеза сиаловой кислоты в не встречающемся в природе микроорганизме включает по меньшей мере один гетерологичный фермент, выбранный из группы, состоящей из глутамин-фруктозо-6-фосфатаминотрансферазы, глюкозамин-6-фосфат-N-ацетилтрансферазы, N-ацетилглюкозамин-2-эпимеразы, синтазы N-ацетилнейраминовой кислоты и сахар-фосфатазы суперсемейства дегидрогеназам галогенокислот-подобных фосфатаз, (англ. haloacid dehydrogenase, сокращенно HAD). Предпочтительно, не встречающийся в природе микроорганизм представляет собой микроорганизм, измененный способами генетической инженерии, в результате чего он содержит один или более генов, кодирующих названные ферменты. Следует понимать, что для выполнения пути биосинтеза сиаловой кислоты микроорганизм-хозяин, уже имеющий один или более генов, кодирующих названные ферменты, и экспрессирующий такие гены в степени, достаточной для продукции Neu5Ac, необязательно должен быть генетически модифицирован, но, тем не менее, может быть генетически модифицирован с целью изменения уровня экспрессии одного или более указанных генов для увеличения количества глутамин-фруктозо-6-фосфатаминотрансферазы, глюкозамин-6-фосфат-N-ацетилтрансферазы, N-ацетилглюкозамин-2-эпимеразы, синтазы N-ацетилнейраминовой кислоты и/или сахар-фосфатазы суперсемейства HAD-подобных фосфатаз с целью повышения выхода биосинтеза Neu5Ac в не встречающемся в природе микроорганизме.
Фермент глутамин-фруктозо-6-фосфатаминотрансфераза (ЕС 2.6.1.16) катализирует превращение фруктозо-6-фосфата в глюкозамин-6-фосфат с участием глутамина. Эту ферментативную реакцию обычно считают первым этапом гексозаминового пути биосинтеза. Глутамин-фруктозо-6-фосфатаминотрансфераза имеет следующие альтернативные наименования: D-фруктозо-6-фосфатамидотрансфераза, GFAT (сокр. от англ. glutamine:fructose-6-phosphate amido-transferase), глюкозамин-6-фосфатсинтаза, гексозофосфатаминотрансфераза и L-глутамин-D-фруктозо-6-фосфатамидотрансфераза.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит глутамин-фруктозо-6-фосфатаминотрансферазу (GlmS), предпочтительно гетерологичную глутамин-фруктозо-6-фосфатаминотрансферазу, более предпочтительно глутамин-фруктозо-6-фосфатаминотрансферазу, полученную из Е. coli, или функциональный вариант GlmS Е. coli. Наиболее предпочтительно, функциональный вариант представляет собой вариант GlmS Е. coli, который проявляет значительно более низкую чувствительность к ингибированию глюкозамин-6-фосфатом по сравнению с ферментом дикого типа, например, кодируемый мутантным геном glmS (glmS*54 или glmS* (см. SEQ ID NO: 6)).
Употребляемый в настоящей работе термин "функциональный вариант" относится к полипептидным вариантам означенного фермента, которые не теряют своей активности и имеют аминокислотную последовательность, которая по меньшей мере на 70%, предпочтительно по меньшей мере на 80%, предпочтительнее по меньшей мере на 90% и более предпочтительно по меньшей мере на 95% идентична аминокислотной последовательности исходного фермента. При этом следует учитывать возможность некоторой вариабельности данных о геномной последовательности, из которой получены указанные полипептиды, а также возможность того, что некоторые из аминокислот, присутствующих в этих полипептидах, могут быть замещены без существенного влияния на каталитическую активность фермента.
Термин "функциональные варианты" также включает полипептидные варианты соответствующих ферментов, которые представляют собой усеченные варианты фермента, не потерявшие в значительной степени каталитической активности. Таким образом, аминокислотная последовательность усеченных вариантов может отличаться от аминокислотных последовательностей соответствующего фермента тем, что в ней отсутствует один, два или более фрагментов, состоящих более чем из двух последовательно расположенных аминокислот. Усечение может находиться на амино-конце (N-конце), на карбоксильном конце (С-конце) и/или внутри аминокислотной последовательности соответствующего фермента.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую глутамин-фруктозо-6-фосфатаминотрансферазу. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая глутамин-фруктозо-6-фосфатаминотрансферазу, представляет собой гетерологичную нуклеотидную последовательность. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая глутамин-фруктозо-6-фосфатаминотрансферазу, кодирует глутамин-фруктозо-6-фосфатаминотрансферазу Е. coli или ее функциональный вариант. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую глутамин-фруктозо-6-фосфатаминотрансферазу или ее функциональный вариант, и/или содержит глутамин-фруктозо-6-фосфатаминотрансферазу или ее функциональный вариант.
Глутамин-фруктозо-6-фосфатаминотрансфераза Е. coli (UniProtKB - Р17169; SEQ ID NO: 11) кодируется геном glmS E. coli (SEQ ID NO: 10). В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую и экспрессирующую нуклеотидную последовательность, кодирующую GlmS Е. coli, или ее функциональный вариант, предпочтительно нуклеотидную последовательность, кодирующую GlmS* (SEQ ID NO: 12 и SEQ ID NO: 13).
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая GlmS Е. coli или один из функциональных вариантов GlmS Е. coli, идентична последовательности glmS Е. coli по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
Фермент глюкозамин-6-фосфат-N-ацетилтрансфераза (Gna1, ЕС 2.3.1.4) превращает глюкозамин-6-фосфат в N-ацетилглюкозамин-6-фосфат с участием ацетил-СоА. Эту ферментативную реакцию считают первым этапом субпути синтеза N-ацетил-альфа-D-глюкозамин-1-фосфата из альфа-D-глюкозамин-6-фосфата в организме Saccharomyces cerevisiae. Gna1 также называют фосфоглюкозамин-ацетилазой или фосфоглюкозамин-трансацетилазой.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм включает глюкозамин-6-фосфат-N-ацетилтрансферазу (Gna1), предпочтительно гетерологичную глюкозамин-6-фосфат-N-ацетилтрансферазу, более предпочтительно глюкозамин-6-фосфат-N-ацетилтрансферазу, полученную из S. cerevisiae (UniProtKB - Р43577, SEQ ID NO: 15), или функциональный вариант Gna1 S. cerevisiae.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, которая включает нуклеотидную последовательность, кодирующую глюкозамин-6-фосфат-N-ацетилтрансферазу. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая глюкозамин-6-фосфат-N-ацетилтрансферазу, представляет собой гетерологичную нуклеотидную последовательность. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая глюкозам ин-6-фосфат-N-ацетилтрансферазу, кодирует глюкозамин-6-фосфат-N-ацетилтрансферазу S. cerevisiae или ее функциональный фрагмент. Известно, однако, что глюкозамин-6-фосфат-N-ацетилтрансферазы, получаемые из них аминокислотные последовательности и нуклеотидные последовательности, кодирующие такие глюкозамин-6-фосфат-N-ацетилтрансферазы, содержатся во множестве различных биологических видов и также могут служить подходящими глюкозамин-6-фосфат-N-ацетилтрансферазами.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую глюкозамин-6-фосфат-N-ацетилтрансферазу или ее функциональный вариант, и/или включает глюкозамин-6-фосфат-N-ацетилтрансферазу или ее функциональный вариант.
Глюкозамин-6-фосфат-N-ацетилтрансфераза S. cerevisiae (UniProtKB - Р43577) кодируется геном Gna1 S. cerevisiae (SEQ ID NO: 14). В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм включает молекулу нуклеиновой кислоты, включающую и экспрессирующую нуклеотидную последовательность, кодирующую Gna1 S. cerevisiae или ее функциональный вариант, предпочтительно нуклеотидную последовательность, кодирующую Gna1 S. cerevisiae (SEQ ID NO: 14).
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая Gna1 S. cerevisiae или один из функциональных вариантов Gna1 S. cerevisiae, идентична последовательности Gna1 S. cerevisiae по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм экспрессирует сахар-фосфатазу суперсемейства HAD-подобных фосфатаз, которая катализирует превращение N-ацетилглюкозамин-6-фосфата (GlcNAc6P) в N-ацетилглюкозамин (GlcNAc). Суперсемейство HAD-подобных фосфатаз названо по бактериальному ферменту дегидрогеназе галогенокислот и включает фосфатазы. Подходящая фосфатаза суперсемейства HAD-подобных фосфатаз, катализирующая превращение GlcNAc6P в GlcNAc, может быть выбрана из группы, состоящей из фруктозо-1-фосфат-фосфатазы (YqaB, UniProtKB - Р77475) и альфа-D-глюкозо-1-фосфат-фосфатазы (YihX, UniProtKB - P0A8Y3). Предполагается, что ферменты YqaB Е. coli и YihX в Е. coli также воздействуют на GlcNAc6P (Lee, S.-W., Oh, M.-K. (2015) Metabolic Engineering 28: 143-150). В одном из примеров осуществления сахар-фосфатаза суперсемейства HAD-подобных фосфатаз, катализирующая превращение GlcNAc-6-фосфата в GlcNAc, представляет собой гетерологичный фермент не встречающегося в природе микроорганизма. В дополнительном и/или альтернативном примере осуществления сахар-фосфатаза суперсемейства HAD-подобных фосфатаз, катализирующая превращение GlcNAc6P в GlcNAc, выбрана из группы, состоящей из YqaB Е. coli, YihX Е. coli и их функциональных вариантов.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, которая включает нуклеотидную последовательность, кодирующую сахар-фосфатазу суперсемейства HAD-подобных фосфатаз, катализирующую превращение GlcNAc6P в GlcNAc. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая сахар-фосфатазу суперсемейства HAD-подобных фосфатаз, катализирующую превращение GlcNAc6P в GlcNAc, представляет собой гетерологичную нуклеотидную последовательность. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая сахар-фосфатазу суперсемейства HAD-подобных фосфатаз, катализирующую превращение GlcNAc6P в GlcNAc, кодирует фруктозо-1-фосфат-фосфатазу Е. coli или альфа-D-глюкозо-1-фосфат-фосфатазу Е. coli или функциональный фрагмент одного из этих двух ферментов.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую сахар-фосфатазу суперсемейства HAD-подобных фосфатаз, катализирующих превращение GlcNAc6P в GlcNAc, или функциональный фрагмент такой HAD фосфатазы, и/или включает сахар-фосфатазу суперсемейства HAD-подобных фосфатаз, катализирующую превращение GlcNAc6P в GlcNAc, или ее функциональный вариант.
Нуклеотидные последовательности, кодирующие подходящие сахар-фосфатазы суперсемейства HAD-подобных фосфатаз, катализирующие превращение GlcNAc6P в GlcNAc, могут быть выбраны из группы нуклеотидных последовательностей, кодирующих YqaB Е. coli, YihX Е. coli, и их функциональных вариантов.
YqaB Е. coli (SEQ ID NO: 16) и YihX E. coli (SEQ ID NO: 18) кодируются генами yqaB E. coli (SEQ ID NO: 17) и yihX E. coli (SEQ ID NO: 18), соответственно. Таким образом, в дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую YqaB Е. coli, YihX Е. coli или функциональный фрагмент одного из этих двух ферментов.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая YqaB Е. coli или ее функциональный вариант, идентична последовательности yqaB Е. coli по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая YihX Е. coli или ее функциональный вариант, идентична последовательности yihX Е. coli по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
N-ацетилглюкозамин-2-эпимераза (ЕС 5.1.3.8) представляет собой фермент, который катализирует превращение N-ацетилглюкозамина (GlcNAc) в N-ацетилманнозамин (ManNAc). Этот фермент действует на углеводы и их производные как рацемаза. Систематическое наименование класса указанных ферментов: N-ацил-D-глюкозамин-2-эпимераза. Этот фермент участвует в амино-сахарном метаболизме и нуклеотидно-сахарном метаболизме.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит N-ацетилглюкозамин-2-эпимеразу, предпочтительно гетерологичную N-ацетилглюкозамин-2-эпимеразу.
В дополнительном и/или альтернативном примере осуществления N-ацетилглюкозамин-2-эпимераза происходит от Anabena variabilis, Acaryochloris sp., Nostoc sp., Nostoc punctiforme, Bacteroides ovatus или Synechocystis sp. или представляет собой функциональный вариант такой эпимеразы. N-ацетилглюкозамин-2-эпимераза организма В. ovatus АТСС 8483 (UniProtKB - A7LVG6, SEQ ID NO: 21) кодируется геном BACOVA_01816 (SEQ ID NO: 20). N-ацетилглюкозамин-2-эпимераза (UniProtKB - P74124; SEQ ID NO: 23) организма Synechocystis sp. (штамм PCC 6803) также известна как ренин-связывающий белок и кодируется геном slr1975 (SEQ ID NO: 23).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую N-ацетилглюкозамин-2-эпимеразу или ее функциональный вариант. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая N-ацетилглюкозамин-2-эпимеразу, выбрана из группы, состоящей из нуклеотидных последовательностей, кодирующих N-ацетилглюкозамин-2-эпимеразу организмов Anabena variabilis, Acaryochloris sp., Nostoc sp., Nostoc punctiforme, Bacteroides ovatus или Synechocystis sp., и их функциональных вариантов. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая N-ацетилглюкозамин-2-эпимеразу, представляет собой гетерологичную нуклеотидную последовательность.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую N-ацетилглюкозамин-2-эпимеразу или ее функциональный вариант, и/или включает N-ацетилглюкозамин-2-эпимеразу или ее функциональный вариант.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая один из функциональных вариантов N-ацетилглюкозамин-2-эпимеразы, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична последовательности гена slr1975 Synechocystis sp.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм характеризуется активностью GlcNAc-6-фосфатэпимеразы и активностью ManNAc-6-фосфатфосфатазы.
GlcNAc-6-фосфатэпимераза (GlcNAc-6-phosphatase epimerase) превращает GlcNAc-6-фосфат в ManNAc-6-фосфат, в то время как ManNAc-6-фосфатфосфатаза дефосфорилирует ManNAc-6-фосфат с образованием ManNAc. Наличие активности GlcNAc-6-фосфатэпимеразы и активности ManNAc-6-фосфатфосфатазы дает дополнительный или альтернативный путь синтеза Neu5Ac, включающий превращение GlcNAc-6-фосфата в ManNAc, показанный на Фиг. 2.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он имеет ген, кодирующий GlcNAc-6-фосфатэпимеразу или ее функциональный вариант. Предпочтительно не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающую и экспрессирующую нуклеотидную последовательность, кодирующую GlcNAc-6-фосфатэпимеразу.
Предпочтительно GlcNAc-6-фосфатэпимераза получена из Enterobacter cloacae subsp. cloacae (SEQ ID NO: 25) или представляет собой ее функциональный вариант. Нуклеотидная последовательность, кодирующая GlcNAc-6-фосфатэпимеразу Е. cloacae subsp. cloacae, представляет собой кодирующую белок область гена nanE организма Enterobacter cloacae subsp. cloacae АТСС 13047 (SEQ ID NO: 24).
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая один из функциональных вариантов GlcNAc-6-фосфатэпимеразы, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична последовательности гена Е. cloacae subsp. cloacae nanE.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован таким образом, что он содержит ген, кодирующий ManNAc-6-фосфатфосфатазу или ее функциональный вариант.
Синтаза N-ацетилнейраминовой кислоты (ЕС 2.5.1.56) представляет собой фермент, который катализирует превращение N-ацетилманнозамина (ManNAc) в Neu5Ac с участием фосфоенолпирувата (PEP). Синтаза N-ацетилнейраминовой кислоты (NeuB) кодируется геном neuB.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит синтазу N-ацетилнейраминовой кислоты или ее функциональный вариант, предпочтительно гетерологичную синтазу N-ацетилнейраминовой кислоты. В дополнительном примере осуществления синтаза N-ацетилнейраминовой кислоты получена из Campylobacter jejuni SEQ ID NO: 29), Streptococcus agalactiae, Butyrivibrio proteoclasticus, Methanobrevibacter ruminatium, Acetobacterium woodii, Desulfobacula toluolica, Escherichia coli, Prevotella nigescens, Halorhabdus tiamatea, Desulfotignum phosphitoxidans или Candidatus Scalindua sp., Idomarina loihiensis, Fusobacterium nucleatum или Neisseria meningitidis.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую синтазу N-ацетилнейраминовой кислоты или ее функциональный вариант. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая синтазу N-ацетилнейраминовой кислоты, представляет собой гетерологичную нуклеотидную последовательность. В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая синтазу N-ацетилнейраминовой кислоты, выбрана из группы, состоящей из нуклеотидных последовательностей, кодирующих NeuB С. jejuni (SEQ ID NO: 28), NeuB S. agalactiae, NeuB B. proteoclasticus, NeuB M. ruminatium, NeuB A. woodii, NeuB D. toluolica, NeuB E. coli, NeuB P. nigescens, NeuB H. tiamatea, NeuB D. phosphitoxidans, NeuB Ca. scalindua sp., NeuB I. loihiensis, NeuB F. nucleatum, NeuB N. meningitidis и их функциональные варианты.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая синтазу N-ацетилнейраминовой кислоты или один из функциональных вариантов синтазы N-ацетилнейраминовой кислоты, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична одной из нуклеотидных последовательностей, кодирующих NeuB С. jejuni, NeuB S. agalactiae, NeuB В. proteoclasticus, NeuB M. ruminatium, NeuB A. woodii, NeuB D. toluolica, NeuB E. coli, NeuB P. nigescens, NeuB H. tiamatea, NeuB D. phosphitoxidans, NeuB Ca. Scalindua sp., NeuB I. loihiensis, NeuB F. nucleatum, NeuB N. Meningitidis.
Катаболический путь сиаловой кислоты
Не встречающийся в природе микроорганизм для получения Neu5Ac, не может использовать Neu5Ac. В дополнительном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован таким образом, что он не использует сиаловую кислоту. Таким образом, Neu5Ac, произведенная не встречающимся в природе микроорганизмом, ни разлагается естественными катаболическими путями, ни встраивается в липополисахариды и/или в полисиаловые кислоты. Напротив, не встречающийся в природе микроорганизм может вырабатывать Neu5Ac, которая выделяется в культуральную среду или ферментативный бульон.
Не встречающийся в природе микроорганизм может продуцировать Neu5Ac. Для того, чтобы он мог продуцировать Neu5Ac, природный катаболический путь сиаловой кислоты блокируют. Нарушение катаболического пути сиаловой кислоты в микроорганизме предотвращает дальнейший метаболизм сиаловой кислоты, синтезируемой этим микроорганизмом, и, таким образом, повышает выход сиаловой кислоты, которая может быть произведена не встречающимся в природе микроорганизмом.
В дополнительном и/или альтернативном примере осуществления природный катаболический путь сиаловой кислоты в микроорганизме блокирован способами генетической инженерии.
В дополнительном и/или альтернативном примере осуществления природный катаболический путь сиаловой кислоты был прерван за счет делеции или другой мутации одного или более генов, кодирующих ферменты, необходимые для катаболизма сиаловой кислоты. В результате прекращается продуцирование фермента (ферментов), необходимых для катаболизма сиаловой кислоты, или их вырабатываемое количество гораздо ниже, чем в нормальных условиях, например, в микроорганизме дикого типа. Например, из генома могут быть удалены один или более генов, кодирующих ферменты, необходимые для катаболизма сиаловой кислоты, в результате чего соответствующие ферменты не производятся. В альтернативном варианте замещения или мутации могут быть введены в регуляторные последовательности, контролирующие экспрессию генов, что приводит к невозможности транскрипции или трансляции гена. Такое нарушение транскрипции или трансляции включает постоянное нарушение транскрипции или трансляции, а также временное нарушение транскрипции или трансляции. Таким образом, возможно регулирование транскрипции или трансляции соответствующего гена посредством инициирования или подавления транскрипции или трансляции. Таким образом, в любой заданный момент времени в процессе культивирования микроорганизма может быть вызвана экспрессия соответствующего гена, предпочтительно посредством добавления соединения, вызывающего экспрессию соответствующего гена (индуктор, также называемый эвокатором) в культуральной среде. В другом примере осуществления экспрессия соответствующего гена может быть подавлена в любой заданный момент времени в процессе культивирования микроорганизма, предпочтительно посредством добавления соединения, которое подавляет экспрессию соответствующего гена (репрессор) в культуральной среде, или расходованием в культуральной среде любого соединения, действующего как индуктор. В другом примере нуклеотидная последовательность, кодирующая фермент, необходимый для катаболизма сиаловой кислоты, может быть изменена с целью устранения активности этого фермента. Это может быть выполнено за счет изменения нуклеотидной последовательности посредством замены смыслового кодона (кодирующего определенную аминокислоту) в первоначальной нуклеотидной последовательности терминирующим кодоном, что приводит к синтезу усеченного полипептида, который не имеет активности фермента, необходимого для катаболизма сиаловой кислоты, или посредством замены смыслового кодона другим кодоном, кодирующим другую аминокислоту, которая образует нефункциональный вариант фермента, необходимого для катаболизма сиаловой кислоты.
В дополнительном и/или альтернативном примере осуществления гены не встречающегося в природе микроорганизма, на которые направлено прерывающее или изменяющее воздействие с целью прекращения катаболизма сиаловой кислоты, кодируют один или более ферментов, выбранных из группы, состоящей из N-ацетилманнозаминкиназы, N-ацетилманнозамин-6-фосфатэпимеразы, альдолазы N-ацетилнейраминовой кислоты и пермеазы сиаловой кислоты.
N-ацетилманнозаминкиназа (ЕС 2.7.1.60) представляет собой фермент, который фосфорилирует N-ацетилманнозамин с образованием N-ацетилманнозамин-6-фосфата. N-ацетилманнозаминкиназа кодируется геном nanK. Нуклеотидная последовательность кодирующей белок области nanK Е. coli представлена последовательностью SEQ ID NO: 30.
N-ацетилманнозамин-6-фосфатэпимераза представляет собой фермент, который превращает N-ацетилманнозамин-6-фосфат (ManNAc-6-P) в N-ацетилглюкозамин-6-фосфат (GlcNAc-6-P). Эта ферментативная реакция представляет собой этап субпути, согласно которому из N-ацетилнейрамината синтезируется D-фруктозо-6-фосфат.N-ацетилманнозамин-6-фосфатэпимераза кодируется геном nanE. Нуклеотидная последовательность кодирующей белок области nanE Е. coli представлена последовательностью SEQ ID NO: 32.
Альдолаза N-ацетилнейраминовой кислоты, также называемая N-ацетилнейраминат-лиазой, катализирует обратимое альдольное расщепление N-ацетилнейраминовой кислоты с образованием пирувата и N-ацетилманнозамина (ManNAc). Альдолаза N-ацетилнейраминовой кислоты кодируется геном nanA. Нуклеотидная последовательность кодирующей белок области nanA Е. coli представлена последовательностью SEQ ID NO: 34.
Пермеаза сиаловой кислоты катализирует протон-зависимый транспорт сиаловой кислоты через клеточную мембрану. Пермеаза сиаловой кислоты может транспортировать N-ацетилнейраминовую кислоту. Варианты пермеазы сиаловой кислоты также могут транспортировать соответствующие сиаловые кислоты, N-гликолилнейраминовую кислоту (Neu5Gc) и 3-кето-3-дезокси-D-глицеро-D-галактонононовую кислоту (KDN). Несмотря на то, что известно, что in vitro пермеаза сиаловой кислоты функционирует как переносчик в обоих направлениях, in vivo она ответственна за клеточный импорт внеклеточной Neu5Ac. Пермеаза сиаловой кислоты кодируется геном nanT. Нуклеотидная последовательность кодирующей белок области nanT Е. coli представлена последовательностью SEQ ID NO: 36. Разрушение nanT предотвращает реимпорт Neu5Ac, которая уже была образована не встречающимся в природе микроорганизмом и выделена в культуральную среду.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм характеризуется пониженной или отсутствующей активностью по меньшей мере одного из ферментов, выбранных из группы, состоящей из N-ацетилманнозаминкиназы, N-ацетилманнозамин-6-фосфатэпимеразы, альдолазы N-ацетилнейраминовой кислоты и пермеазы сиаловой кислоты, по сравнению с микроорганизмом дикого типа. В дополнительном и/или альтернативном примере осуществления микроорганизм был генетически модифицирован, в результате чего микроорганизм характеризуется пониженной или отсутствующей активностью по меньшей мере одного из указанных ферментов. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм был генетически модифицирован, в результате чего в нем полностью отсутствует один или более генов, кодирующих N-ацетилманнозаминкиназу, N-ацетилманнозамин-6-фосфатэпимеразу, альдолазу N-ацетилнейраминовой кислоты и пермеазу сиаловой кислоты, нарушена экспрессия одного или более из этих генов или устранена активность одного или более соответствующих ферментов в результате введения мутаций в кодирующую белок область этого гена (генов), что приводит к тому, что полипептид, кодируемый измененной нуклеотидной последовательностью, не обладает ферментативной активностью фермента, кодируемого немодифицированной нуклеотидной последовательностью.
В дополнительном и/или альтернативном примере осуществления не встречающемся в природе микроорганизм не имеет ферментативной активности, обеспечиваемой по меньшей мере одним из следующих ферментов: N-ацетилглюкозамин-6-фосфатдезацетилазой и N-ацетилглюкозамин-6-фосфатдезаминазой.
В дополнительном и/или альтернативном примере осуществления в не встречающемся в природе микроорганизме инактивирован по меньшей мере один из следующих ферментов: N-ацетилглюкозамин-6-фосфатдезацетилаза и N-ацетилглюкозамин-6-фосфатдезаминаза.
N-ацетилглюкозамин-6-фосфатдезацетилаза (ЕС 3.5.1.25) представляет собой фермент, участвующий в первом этапе биосинтеза аминосахаронуклеотидов. Он катализирует гидролиз N-ацетильной группы N-ацетилглюкозамин-6-фосфата (GlcNAc-6-P), приводящий к образованию глюкозамино-6-фосфата и ацетата. N-ацетилглюкозамин-6-фосфатдезацетилаза кодируется геном nagA. Нуклеотидная последовательность области nagA Е. coli, кодирующей белок, представлена последовательностью SEQ ID NO: 38.
Глюкозамин-6-фосфатдезаминаза (ЕС 3.5.99.6) катализирует обратимую изомеризацию-дезаминирование глюкозамин-6-фосфата (GlcN6P), приводящую к образованию фруктозо-6-фосфата (Fru6P). Глюкозамин-6-фосфатдезаминаза кодируется геном nagB. Нуклеотидная последовательность области nagB Е. coli, кодирующей белок, представлена последовательностью SEQ ID NO: 40.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, что приводит к деактивации N-ацетилглюкозамин-6-фосфатдезацетилазы и/или глюкозамин-6-фосфатдезаминазы. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего в нем полностью удален один или более генов, кодирующих N-ацетилглюкозамин-6-фосфатдезацетилазу и глюкозамин-6-фосфатдезаминазу, нарушена экспрессия одного или более указанных генов или устранена активность одного или более соответствующих ферментов за счет введения мутаций в кодирующую белок область по меньшей мере одного указанных генов, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не обладает ферментативной активностью фермента, кодируемого неизмененной нуклеотидной последовательностью.
Система транспорта углевод-фосфотрансферазы
Для внутриклеточного продуцирования сиаловой кислоты требуется фосфоенолпируват (PEP). PEP представляет собой очень важное для метаболизма промежуточное соединение, поскольку он участвует в гликолизе и гликонеогенезе. Для повышения продуцирования Neu5Ac не встречающийся в природе микроорганизм был генетически модифицирован для обеспечения улучшенной доставки PEP для биосинтеза сиаловой кислоты. Для этого не встречающийся в природе микроорганизм генетически модифицировали таким образом, чтобы блокировать по меньшей мере одну РЕР-зависимую систему фосфотрансферазы - переносчик сахара (англ. phosphotransferase system, сокр. PTS), т.е. соответствующий ген удаляют или разрушают или нарушают экспрессию этого гена.
Подходящая для разрушения РЕР-зависимая система фосфотрансферазы - переносчик сахара представляет собой пермеазу GlcNAc, также называемую белок-Npi-фосфо-L-гистидин:N-ацетил-D-глюкозамин-Npi-фосфотрансферазой (ЕС 2.7.1.193), которая кодируется геном nagE. NagE (называемый ферментом II) представляет собой компонент РЕР-зависимой системы фосфотрансферазы - переносчика сахара. Эта система одновременно переносит субстрат из периплазмы или внеклеточного пространтсва в цитоплазму и фосфорилирует субстрат. Удаление или разрушение nagE или нарушение его экспрессии являются положительными факторами, поскольку они предотвращают импорт GlcNAc за счет PEP, который, в противном случае, снижал бы количество PEP, доступного для продукции сиаловой кислоты. Таким образом, удаление или разрушение гена nagE или нарушение его экспрессии повышает внутриклеточное количество PEP, которое может быть использовано не встречающимся в природе микроорганизмом для продуцирования Neu5Ac, что, таким образом, усиливает синтез Neu5Ac в не встречающемся в природе микроорганизме по сравнению с его синтезом в не встречающемся в природе микроорганизме, который может продуцировать сиаловую кислоту, но имеет интактный и функциональный ген nagE. Нуклеотидная последовательность области nagE Е. coli, кодирующей белок, представлена последовательностью SEQ ID NO: 42.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован с целью устранения активности белок-Npi-фосфо-L-гистидин:N-ацетил-D-глюкозамин-Npi-фосфотрансферазы.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для полного удаления гена nagE, для нарушения его экспрессии или для устранения активности фермента NagE посредством введения мутации в кодирующую белок область гена nagE, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не обладает ферментативной активностью фермента, кодируемого неизмененной нуклеотидной последовательностью.
Другой или дополнительной РЕР-зависимой системой фосфотрансферазы - переносчиком сахара для импорта углевода, подходящей для нарушения, является маннозопермеаза.
ManXYZ, комплекс фермента IIMan (маннозо-PTS-пермеаза, белок-Npi-фосфогистидин-D-маннозофосфотрансфераза) импортирует экзогенные гексозы (маннозу, глюкозу, глюкозамин, фруктозу, 2-дезоксиглюкозу, маннозамин, N-ацетилглюкозамин и т.д.) и высвобождает сложные фосфатные эфиры в цитоплазму клеток. Этот фермент также представляет собой компонент РЕР-зависимой системы фосфотрансферазы - переносчика сахара. В трех полипептидных цепочках ManXYZ содержит четыре домена: ManX=IIABMan, ManY=IICMan и ManZ=IIDMan. Они являются членами семейства маннозо-PTS-пермеаз, группы сплинтеров (англ. splinter), которые не гомологичны большинству других PTS-пермеаз. Нуклеотидные последовательности кодирующих белок областей manX, manY и manZ Е. coli представлены последовательностями SEQ ID NO: 44, SEQ ID NO: 46 и SEQ ID NO: 48, соответственно.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован с целью устранения активности белок-Npi-фосфо-L-гистидин:маннозо-Npi-фосфотрансферазы.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован с целью полного удаления одного или более генов, кодирующих ManX, ManY и ManZ, с целью нарушения экспрессии одного или более указанных генов или с целью устранения активности одного или более соответствующих ферментов посредством введения мутаций в кодирующую белок область (области) гена (генов), в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не обладает ферментативной активностью фермента, кодируемого неизмененной нуклеотидной последовательностью.
Другой или дополнительной РЕР-зависимой системой фосфотрансферазы - переносчиком сахара для импорта углевода, подходящей для нарушения, является транспортер глюкозы.
Глюкозоспецифичный PTS транспортер (PtsG/Crr) поглощает экзогенную глюкозу, высвобождая в цитоплазму сложный фосфатный эфир. Комплекс фермента IIGlc содержит два домена в одной полипептидной цепочке с порядком доменов IIC-IIB (PtsG), и он функционирует совместно с дополнительной полипептидной цепочкой, белком Crr или IIAGlc.
Делеция или разрушение ptsG и/или crr или нарушение его экспрессии являются предпочтительными факторами, поскольку они предотвращают импорт глюкозы за счет PEP, который, в противном случае, снижал бы количество PEP, доступного для продуцирования сиаловой кислоты. Таким образом, удаление или разрушение гена ptsG и/или гена crr или нарушение его экспрессии повышает внутриклеточное количество PEP, которое может быть использовано не встречающимся в природе микроорганизмом для синтеза Neu5Ac, что, таким образом, повышает выход Neu5Ac в не встречающемся в природе микроорганизме, который может продуцировать Neu5Ac, по сравнению с его выходом в не встречающемся в природе микроорганизме, который может продуцировать сиаловую кислоту, но имеет интактный и функциональный ген ptsG и/или crr.
Нуклеотидные последовательности кодирующих белок областей ptsG и crr Е. coli представлены последовательностями SEQ ID NO: 50 и SEQ ID NO: 52, соответственно.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован с целью устранения активности PtsG/Crr.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован с целью полного удаления гена ptsG и/или гена crr, с целью нарушения экспрессии гена ptsG и/или гена crr или с целью устранения активности PtsG/Crr посредством введения мутаций в кодирующую белок область гена ptsG и/или гена crr, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью (последовательностями) не обладает ферментативной активностью фермента (ферментов), кодируемого неизмененной нуклеотидной последовательностью.
Предоставление источника углерода
Для роста и пролиферации не встречающегося в природе микроорганизма и продукцирования Neu5Ac требуется источник углерода. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм может быть выращен на одном не дорогостоящем источнике углерода, таком как, например, глюкоза или сахароза. Единственный источник углерода предоставляет реагент для биосинтеза сиаловой кислоты в не встречающемся в природе микроорганизме. Таким образом, для получения Neu5Ac нет необходимости культивировать не встречающийся в природе микроорганизм в присутствии ManNAc, GlcNAc или глюкозамина (GlcN). Кроме того, для импорта единственного источника углерода в не встречающийся в природе микроорганизм не требуется наличия РЕР-зависимой систем фосфотрансферазы - переносчика сахара, и, таким образом, нет необходимости в использовании PEP для доступа к единственному источнику углерода.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован так, что он может потреблять сахарозу в качестве единственного источника углерода.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм имеет функциональную систему утилизации сахарозы. Функциональная система утилизации сахарозы обеспечивает импорт в клетку предоставляемой извне сахарозы и ее гидролиз, после чего полученные моносахариды глюкоза и фруктоза могут быть метаболизированы в не встречающемся в природе микроорганизме и направлены на синтез Neu5Ac.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм был генетически модифицирован для создания функциональной системы утилизации сахарозы. В дополнительном и/или альтернативном примере осуществления система утилизации сахарозы не встречающегося в природе микроорганизма включает систему переноса в одном направлении сахарозы и протона, фруктокиназу, инвертазу и репрессор сахарозного оперона.
Подходящей системой переноса сахарозы и протона в одном направлении является CscB, кодируемая геном cscB, например, CscB Е. coli (SEQ ID NO: 55), кодируемая геном cscB Е. coli (SEQ ID NO: 54).
Подходящей фруктокиназой (EC 2.7.1.4) является CscK, кодируемая геном cscK, например, CscK E. coli (SEQ ID NO: 57), кодируемая геном cscK E. coli (SEQ ID NO: 56).
Подходящей инвертазой (EC 3.2.1.26), которая гидролизует терминальные невосстанавливающие бета-D-фруктофуранозидные остатки в бета-D-фруктофуранозидах, является CscA, например, cscA Е. coli (SEQ ID NO: 59), кодируемая геном cscA Е. coli (SEQ ID NO: 58).
Подходящим репрессором сахарозного оперона является CscR, кодируемый геном cscR, например, CscR Е. coli (SEQ ID NO: 61), кодируемый геном cscR Е. coli (SEQ ID NO: 60).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит систему переноса сахарозы и протона в одном направлении, фруктокиназу, инвертазу и репрессор сахарозного оперона или функциональные варианты любого из указанных белков.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован,, в результате чего он содержит молекулу нуклеиновой кислоты, включающую нуклеотидные последовательности, кодирующие систему переноса сахарозы и протона в одном направлении, фруктокиназу, инвертазу и репрессор сахарозного оперона, для экспрессии системы переноса сахарозы и протона в одном направлении, фруктокиназы, инвертазы и репрессора сахарозного оперона. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован так, что в результате он экспрессирует гены cscB, cscK, cscA, предпочтительно гнеы cscB, cscK, cscA и cscR организма E. coli.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая функциональный вариант CscB, CscK, CscA или CscR, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична последовательности cscB, cscK, cscA или cscR организма Е. coli, соответственно.
Не встречающийся в природе микроорганизм, который может продуцировать Neu5Ac и имеет функциональную систему утилизации сахарозы, может быть подвергнут культивированию в присутствии сахарозы, которую применяют в качестве единственного источника углерода для метаболизма, а также биосинтеза Neu5Ac в микроорганизме. Сахароза имеет невысокую стоимость, и ее использование в качестве единственного источника углерода для получения Neu5Ac посредством ферментации более экономически выгодно, чем применение других предшественников сиаловой кислоты, например, GlcNAc.
Другой подходящей системой утилизации сахарида, которая позволяет выращивать не встречающийся в природе микроорганизм на единственном источнике углерода без использования РЕР-зависимой системы фосфотрансферазы - переносчика сахара, является LacY, кодируемая геном lacY лактозного оперона. LacY представляет собой β-галактозидпермеазу, которая импортирует лактозу через клеточные мембраны под действием протонного градиента того же направления. Внутриклеточная лактоза может быть гидролизована β-галактозидазой (LacZ), в результате чего внутри клетки образуются глюкоза и галактоза. Ген lacZ также является частью лактозного оперона.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм экспрессирует β-галактозидпермеазу и β-галактозидазу.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он экспрессирует β-галактозидпермеазу, предпочтительно пермеазу лактозы LacY Е. coli (SEQ ID NO: 63) или ее функциональный вариант, и β-галактозидазу, предпочтительно LacZ Е. coli (SEQ ID NO: 65) или ее функциональный вариант. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую β-галактозидпермеазу, предпочтительно нуклеотидную последовательность, кодирующую LacY Е. coli (SEQ ID NO: 62) или ее функциональный вариант, и/или нуклеотидную последовательность, кодирующую β-галактозидазу, предпочтительно нуклеотидную последовательность, кодирующую LacZ Е. coli (SEQ ID NO: 64) или ее функциональный вариант.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая LacY Е. coli или ее функциональный вариант, идентична последовательности lacY Е. coli по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
В дополнительном и/или альтернативном примере осуществления нуклеотидная последовательность, кодирующая LacZ Е. coli или ее функциональный вариант, идентична последовательности lacZ Е. coli по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99%.
Не встречающийся в природе микроорганизм, который может продуцировать Neu5Ac и который экспрессирует функциональную β-галактозидпермеазу и функциональную β-галактозидазу, может культивироваться на лактозе, которую применяют в качестве единственного источника углерода.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм экспрессирует симпортер глюкозы/Н+. Предпочтительно не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, включающей нуклеотидную последовательность, кодирующую и обеспечивающую экспрессию симпортера глюкозы/Н+ в не встречающемся в природе микроорганизме.
Подходящий симпортер глюкозы/Н+ выбран из группы, состоящей из симпортера глюкозы/Н Staphylococcus epidermis (UniProtKB - A0A0U5QDM9; SEQ ID NO: 67), симпортера глюкозы/Н+ Lactobacillus brevis (UniProtKB - A0A0C1PU75, SEQ ID NO: 69) и их функциональных вариантов.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он содержит молекулу нуклеиновой кислоты, кодирующей симпортер глюкозы/Н+ S. epidermis или симпортер глюкозы/Н+ L. brevis. Предпочтительно не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, выбранную из группы, состоящей из SEQ ID NO: 67 и SEQ ID NO: 69, и нуклеотидные последовательности по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентичные последовательностям SEQ ID NO: 67 или SEQ ID NO: 69.
He встречающийся в природе микроорганизм, который может продуцировать Neu5Ac и который экспрессирует либо симпортер глюкозы/Н+ S. epidermis, либо симпортер глюкозы/Н+ L. brevis, можно культивировать в присутствии глюкозы, используемой в качестве единственного источника углерода, и микроорганизм не нуждается в PEP для доступа к предоставляемой извне глюкозе.
Дополнительные генетические модификации
Не встречающийся в природе микроорганизм, который может продуцировать Neu5Ac, необязательно может иметь дополнительные особенности и может быть генетически модифицирован так, что он имеет дополнительные свойства,. Предполагается, что дополнительные свойства увеличивают продуктивность не встречающегося в природе микроорганизма, что приводит к повышению выходов Neu5Ac.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм синтезирует большее количество PEP, чем микроорганизм дикого типа. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он имеет улучшенный путь биосинтеза PEP. Предпочтительно не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он имеет повышенную активность фосфоенолпируватсинтазы, проявляющуюся, например, в сверхэкспрессии гена ppsA, кодирующего фосфоенолпируватсинтазу, и/или не встречающийся в природе микроорганизм содержит по меньшей мере одну дополнительную копию нуклеотидной последовательности, вызывающей экспрессию фосфоенолпируватсинтазы или ее функционального варианта. Сверхэкспрессия ppsA повышает внутриклеточный синтез PEP, в результате чего для получения сиаловой кислоты доступно большее количество PEP. Например, подходящей фосфоенолпируватсинтазой является PpsA Е. coli (SEQ ID NO: 71).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм содержит молекулу нуклеиновой кислоты, включающую нуклеотидную последовательность, кодирующую PpsA Е. coli или ее функциональный вариант. Эта нуклеотидная последовательность, кодирующая PpsA Е. coli или ее функциональный вариант по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична последовательности гена PpsA Е. coli (SEQ ID NO: 70).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для сверхэкспрессии фосфоенолпируваткарбоксикиназы. Подходящей фосфоенолпируваткарбоксикиназой является Pck Е. coli (SEQ ID NO: 73).
Фермент фосфоенолпируваткарбоксикиназа (ЕС 4.1.1.49) кодируется геном рек и катализирует следующую реакцию: оксалоацетат + АТФ → фосфоенолпируват + АДФ + CO2. Фосфоенолпируваткарбоксикиназа участвует в гликонеогенезе.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для сверхэкспрессии фосфоенолпируваткарбоксикиназы и/или так, что микроорганизм содержит по меньшей мере одну дополнительную нуклеотидную последовательность, позволяющую экспрессировать фосфоенолпируваткарбоксикиназу или ее функциональный вариант. Сверхэкспрессия фосфоенолпируваткарбоксикиназы повышает внутриклеточную концентрацию PEP, то есть PEP становится более доступен для синтеза сиаловой кислоты.
Нуклеотидная последовательность, кодирующая дополнительную нуклеотидную последовательность, кодирующую фосфоенолпируваткиназу или ее функциональный вариант, может быть последовательностью SEQ ID NO: 72 или нуклеотидной последовательностью, которая по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична последовательности гена рек Е. coli (SEQ ID NO: 72).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм не содержит функциональной фосфоенолпируваткарбоксилазы (ЕС 4.1.1.31). Фосфоенолпируваткарбоксилаза образует оксалоацетат, источник дикарбоновой кислоты, содержащей четыре атома углерода, для цикла трикарбоновых кислот. Фосфоенолпируваткарбоксилаза кодируется геном ррс. В организме Е. coli фосфоенолпируваткарбоксилаза (SEQ ID NO: 27) кодируется геном рерС (SEQ ID NO: 26).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для устранения активности PEP карбоксилазы.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для удаления гена ррс или гена рерС, нарушения его экспрессии или устранения активности PEP карбоксилазы за счет введения мутаций в кодирующую белок область гена ррс/рерС, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью не проявляет активности PEP карбоксилазы.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего активность пируваткиназы в организме снижена или отсутствует.
Фермент пируваткиназа образует аденозинтрифосфат (АТФ) из аденозиндифосфата (АДФ) и PEP. Образование АТФ из АДФ и PEP является последним этапом гликолиза, и в физиологических условиях этот этап необратим. Во многих энтеробактериях, включающих Е. coli, вырабатывается две изоморфных формы пируваткиназы, PykA (SEQ ID NO: 75) и PykF (SEQ ID NO: 77), которые для Е. coli идентичны на 37%.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для делеции одного или более генов, кодирующих пируваткиназу, предпочтительно гена pykA (SEQ ID NO: 74) и/или гена pykF (SEQ ID NO: 76), нарушения экспрессии одного или более указанных генов, кодирующих пируваткиназу, или устранения активности по меньшей мере одной пируваткиназы за счет введения одной или более мутаций в нуклеотидную последовательность области, кодирующей белок, одного или более указанных генов, кодирующих пируваткиназу, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не проявляет активности пируваткиназы.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм синтезирует большее количество глутамина по сравнению с микроорганизмом дикого типа. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего у микроорганизма имеется улучшенный путь биосинтеза глутамина.
Глутаминсинтетаза (GlnA) превращает глутамат в глутамин по следующей реакции: АТФ + L-глутамат + NH3 = АДФ + фосфат + L-глутамин. В организме Е. coli глутаминсинтетаза (SEQ ID NO: 79) кодируется геном glnA (SEQ ID NO: 78).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, что приводит к сверхэкспрессии глутаминсинтазы и/или к тому, что микроорганизм содержит по меньшей мере одну дополнительную нуклеотидную последовательность, обеспечивающую экспрессию глутаминсинтазы или ее функционального варианта. Сверхэкспрессия глутаминсинтазы повышает внутриклеточную концентрацию глутамина, что, в свою очередь, ускоряет внутриклеточное превращение фруктозо-6-фосфата (Frc-6P) в глюкозамин-6-фосфат (GlcN-6P). Предпочтительно нуклеотидная последовательность, кодирующая дополнительную нуклеотидную последовательность, кодирующую глутаминсинтазу или ее функциональный вариант, может быть последовательностью SEQ ID NO: 78 или нуклеотидной последовательностью, которая по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентична последовательности гена glnA Е. coli (SEQ ID NO: 78).
Моделирование метаболизма в Е. coli подтвердило, что усиленный синтез глутамина повышает продукцию Neu5Ac. Кроме того, транскриптомный анализ в штамме Е. coli, продуцирующем Neu5Ac (# NANA1), который не был генетически модифицирован для усиления синтеза глутамина, показал более высокую экспрессию глутаминсинтазы по сравнению с соответствующим штаммом Е. coli, который не был способен продуцировать Neu5Ac.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм не содержит функциональной глутаматсинтазы или отличается более низкой активностью глутаматсинтазы по сравнению с микроорганизмом дикого типа. Глутаматсинтаза Е. coli состоит из двух субъединиц, GltB (SEQ ID NO: 81) и GltD (SEQ ID NO: 83), и синтезирует глутамат из глутамина. GltB кодируется геном gltB (SEQ ID NO: 80), и GltD кодируется геном gltD (SEQ ID NO: 82).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для делеции гена gltB и/или гена gltD, нарушения экспрессии по меньшей мере одного из этих генов или снижения или устранения активности глутаматсинтазы за счет введения одной или более мутаций в кодирующую белок область гена gltB и/или гена gltD, в результате чего полипептид (полипептиды), кодируемый измененной нуклеотидной последовательностью (последовательностями), представляет собой нефункциональный вариант глутаматсинтазы.
В дополнительном и/или альтернативном примере осуществления в не встречающемся в природе микроорганизме активность глутаминазы не проявляется.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для делеции по меньшей мере одного из генов глутаминазы: asnB, ybaS и yneH, нарушения экспрессии по меньшей мере одного указанных генов или устранения активности глутаминазы за счет введения одной или более мутаций в кодирующую белок область генов asnB, ybaS и/или yneH таким образом, что полипептид, кодируемый одной из измененных нуклеотидных последовательностей, не проявляет активности глутаминазы.
AsnB представляет собой аспарагинсинтетазу, которая катализирует АТФ-зависимое превращение аспартата в аспарагин с использованием глутамина. Аспарагинсинтетаза AsnB Е. coli (SEQ ID NO: 85) кодируется геном asnB Е. coli (SEQ ID NO: 84).
YbaS, также называемая GlsA1 или Gls1, представляет собой глутаминазу 1, то есть глутаминазу с высокой селективностью к L-глутамину. YbaS превращает L-глутамин в L-глутамат. Глутаминаза YbaS Е. coli (SEQ ID NO: 87) кодируется геном ybaS Е. coli (SEQ ID NO: 86).
YneH, также называемая GlsA2, GlsB или глутаминазой 2, катализирует следующую реакцию: L-глутамин + H2O = L-глутамат + NH3. Глутаминаза YneH Е. coli (SEQ ID NO: 89) кодируется геном yneH Е. coli (SEQ ID NO: 88).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм отличается повышенной активностью глутаматдегидрогеназы по сравнению с микроорганизмом дикого типа. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для сверхэкспрессии глутаматдегидрогеназы, и/или так, что, что микроорганизмом содержит по меньшей мере одну дополнительную нуклеотидную последовательность, обеспечивающую экспрессию глутаматдегидрогеназы или ее функционального варианта.
Глутаматдегидрогеназа превращает глутамат в α-кетоглутарат. Сверхэкспрессия глутаматдегидрогеназы усиливает продукцию α-кетоглутарата, который, в свою очередь, может быть превращен в глутамат под действием глутаматсинтазы, например, глутаматсинтазы, кодируемой gltD Е. coli, или ее функционального варианта. Затем глутамат может быть превращен в глутамин под действием глутаминсинтетазы (GlnA) или ее функционального варианта.
В дополнительном и/или альтернативном примере осуществления дополнительная нуклеотидная последовательность, обеспечивающая экспрессию глутаматдегидрогеназы или ее функционального варианта, включает кодирующую белок область глутаматсинтазы Е. coli (SEQ ID NO: 91). Нуклеотидная последовательность, кодирующая глутаматсинтазу или ее функциональный вариант, может представлять собой последовательность SEQ ID NO: 90 или нуклеотидную последовательность, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности гена gltD Е. coli (SEQ ID NO: 90).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм не способен синтезировать липополисахариды (сокращенно ЛПС) и/или колановую кислоту. В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, в результате чего он не может синтезировать ЛПС и/или колановую кислоту.
В одном из примеров осуществления не встречающийся в природе микроорганизм генетически модифицирован для делеции гена wzxC, нарушения экспрессии гена wzxC или устранения активности фермента WzxC, посредством введения одной или более мутаций в кодирующую белок область гена, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не обладает ферментативной активностью WzxC. WzxC требуется для биосинтеза ЛПС и кодирует предполагаемый экспортный белок. Нуклеотидная последовательность wzxC Е. coli представлена последовательностью SEQ ID NO: 92, и полученная из нее аминокислотная представлена последовательностью SEQ ID NO: 93.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм не проявляет активности трансферазы уридин-дифосфатглюкозы (т.е. UDP-глюкозы):ундекапренилфосфат-глюкозо-1-фосфата.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для устранения активности трансферазы UDP-глюкозы:ундекапренилфосфат-глюкозо-1-фосфата, предпочтительно за счет делеции гена wcaJ или его функционального варианта, нарушения экспрессии гена wcaJ или его функционального варианта или устранения активности фермента WcaJ за счет введения мутаций в кодирующую белок область, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не обладает ферментативной активностью WcaJ. WcaJ кодирует трансферазу UDP-глюкозы:ундекапренилфосфат-глюкозо-1-фосфата. Трансфераза UDP-глюкозы:ундекапренилфосфат-глюкозо-1-фосфата представляет собой первый фермент биосинтеза колановой кислоты. Нуклеотидная последовательность wcaJ Е. coli представлена последовательностью SEQ ID NO: 94 и полученной из нее аминокислотной последовательностью SEQ ID NO: 95.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм может не содержать функциональной β-галактозидпермеазы (LacY) и/или функциональной β-галактозидазы (LacZ), при условии, что не встречающийся в природе микроорганизм можно культивировать на единственном источнике углерода, который не является лактозой, например, на сахарозе или глюкозе, применяемой в качестве единственного источника углерода.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, для делеции гена β-галактозидпермеазы (lacY) и/или гена β-галактозидазы (lacZ), нарушения экспрессии гена β-галактозидпермеазы и/или гена β-галактозидазы или изменения нуклеотидной последовательности области, кодирующей белок гена β-галактозидпермеазы и/или гена β-галактозидазы, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью (последовательностями), не обладает ферментативной активностью β-галактозидпермеазы и/или β-галактозидазы.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм не содержит функциональную YjhC. YjhC представляет собой оксидоредуктазу, кодируемую геном yjhC, или ее функциональный вариант.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для устранения активности оксидоредуктазы YjhC, предпочтительно за счет делеции гена yjhC, за счет нарушения экспрессии гена yjhC или за счет введения одной или более мутаций в кодирующую белок область гена yjhC, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не проявляет активности оксидоредуктазы YjhC.
Нуклеотидная последовательность yjhC Е. coli представлена последовательностью SEQ ID NO: 96 и полученной из нее аминокислотной последовательностью SEQ ID NO: 97.
В дополнительном и/или альтернативном примере осуществления в не встречающемся в природе микроорганизме не проявляется активность одного или более следующих ферментов: фукозоизомеразы, фукулокиназы и N-ацетилглутаминаминоацилазы. В одном из примеров осуществления не встречающийся в природе микроорганизм генетически модифицирован с целью устранения активности одного или более из перечисленных ферментов.
Фукозоизомераза превращает альдозо-L-фукозу в соответствующую кетозо-L-фукулозу. Фукозоизомераза представляет собой первый фермент субпути синтеза L-лактальдегида и глицеронфосфата (фосфата дигидроксиацетона) из L-фукозы. Фукозоизомераза Fucl Е. coli (SEQ ID NO: 99) кодируется геном fucI Е. coli (SEQ ID NO: 98).
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован для устранения активности фукозоизомеразы, предпочтительно посредством делеции гена fucI, посредством нарушения экспрессии гена fucI или посредством модификации кодирующей белок области гена fucI, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не проявляет активности фукозоизомеразы.
Фукулокиназа катализирует фосфорилирование фукозы. Фукулокиназа представляет собой второй фермент субпути синтеза L-лактальдегида и глицеронфосфата из L-фукозы. Фукулокиназа FucK Е. coli (SEQ ID NO: 101) кодируется геном fucK Е. coli (SEQ ID NO: 100). Фукулокиназа E. coli также может фосфорилировать, но с меньшей эффективностью, D-рибулозу, D-ксилулозу и D-фруктозу.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, для устранения активности фукозоизомеразы, предпочтительно посредством делеции гена fucK, посредством нарушения экспрессии гена fucK или посредством введения мутаций в кодирующую белок область гена fucK, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не проявляет активности фукозоизомеразы.
N-ацетилгалактозамин-6-фосфатдезацетилаза катализирует следующую реакцию: N-ацетил-D-галактозамин 6-фосфат + H2O → D-галактозамин 6-фосфат + ацетат. N-ацетилгалактозамин-6-фосфатдезацетилаза кодируется геном agaA. В отличие от штаммов С и ЕС3132 E. coli, штаммы K-12 не могут быть выращены на N-ацетилгалактозамине и D-галактозамине, поскольку они имеют делецию и, таким образом, не имеют активных систем PTS, специфичных для этих соединений. Таким образом, в штаммах K-12 AgaA не участвует в разрушении этих соединений. AgaA Е. coli (SEQ ID NO: 103) кодируется геном agaA Е. coli (SEQ ID NO: 102)..
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм генетически модифицирован, для устранения активности N-ацетилгалактозамин-6-фосфатдезацетилазы, предпочтительно посредством делеции гена agaA, посредством нарушения экспрессии гена agaA или посредством введения мутаций в кодирующую белок область гена agaA, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, не проявляет активности N-ацетилгалактозамин-6-фосфатдезацетилазы.
Не встречающийся в природе микроорганизм выбран из группы, состоящей из дрожжей, грибков и бактерий. Предпочтительно не встречающийся в природе микроорганизм представляет собой организм, общепризнанный безопасным (англ. generally recognized as safe, сокращенно GRAS), что установлено, например, Федеральным Управлением по лекарственным средствам США (англ. Federal Drug Administration, сокращенно FDA) или независимо определено квалифицированными экспертами, более предпочтительно прокариотический микроорганизм, наиболее предпочтительно бактериальный микроорганизм. Бактерии, подходящие для продукции Neu5Ac, могут быть выбраны из следующих видов: Bacillus, Lactobacillus, Lactococcus, Enterococcus, Bifidobacterium, Sporolactobacillus, Micromomospora, Micrococcus, Rhodococcus и Pseudomonas. Подходящие бактериальные виды включают: Bacillus subtilis, Bacillus licheniformis, Bacillus coagulans, Bacillus thermophilus, Bacillus laterosporus, Bacillus megaterium, Bacillus mycoides, Bacillus pumilus, Bacillus lentus, Bacillus cereus, Bacillus circulans, Bifidobacterium longum, Bifidobacterium infantis, Bifidobacterium bifidum, Citrobacter freundii, Clostridium cellulolyticum, Clostridium ljungdahlii, Clostridium autoethanogenum, Clostridium acetobutylicum, Corynebacterium glutamicum, Enterococcus faecium, Enterococcus thermophiles, Escherichia coli, Erwinia herbicola (Pantoea agglomerans), Lactobacillus acidophilus, Lactobacillus salivarius, Lactobacillus plantarum, Lactobacillus helveticus, Lactobacillus delbrueckii, Lactobacillus rhamnosus, Lactobacillus bulgaricus, Lactobacillus crispatus, Lactobacillus gasseri, Lactobacillus casei, Lactobacillus reuteri, Lactobacillus jensenii, Lactococcus lactis, Pantoea citrea, Pecto bacterium carotovorum, Proprionibacterium freudenreichii, Pseudomonas fluorescens, Pseudomonas aeruginosa, Streptococcus thermophiles и Xanthomonas campestris.
Второй аспект изобретения относится к применению не встречающегося в природе микроорганизма, рассмотренного выше в настоящей работе, для получения Neu5Ac. Не встречающийся в природе микроорганизм способен продуцировать Neu5Ac в промышленном масштабе. Термины "способный" и "может", употребляемые в настоящей работе для описания получения (продукции) Neu5Ac, относятся к способности не встречающегося в природе микроорганизма синтезировать Neu5Ac и секретировать Neu5Ac в ферментативный бульон, при условии, что не встречающийся в природе микроорганизм культивируют в условиях, которые позволяют образовываться Neu5Ac. Это включает способность не встречающегося в природе микроорганизма размножаться, достигая высоких плотностей клеток, и развиваться в крупных культуральных объемах, например, объемах, превышающих 1000 л, предпочтительно 10000 л, более предпочтительно 80000 л и наиболее предпочтительно 200000 л.
Третий аспект относится к способу получения Neu5Ac посредством микробной ферментации. Способ включает следующие этапы:
- предоставление не встречающегося в природе микроорганизма, который способен продуцировать Neu5Ac, предпочтительно не встречающегося в природе микроорганизма, рассмотренного выше в настоящей работе;
- культивирование не встречающегося в природе микроорганизма в ферментативном бульоне в условиях, пермессивных для продуцирования Neu5Ac указанным микроорганизмом; и
- необязательно выделение Neu5Ac из ферментативного бульона.
Ферментативный бульон содержит по меньшей мере один источник углерода для не встречающегося в природе микроорганизма. Этот источник углерода предпочтительно выбран из группы, состоящей из глюкозы, ксилозы, фруктозы, сахарозы, лактозы, глицерина, синтез-газа и их комбинаций.
В дополнительном и/или альтернативном примере осуществления не встречающийся в природе микроорганизм культивируют в отсутствие в ферментативном бульоне и/или без добавления в ферментативный бульон одного или более веществ, выбранных из группы, состоящей из пирувата, глюкозамина и N-ацетилглюкозамина.
Способ включает необязательный этап выделения Neu5Ac, синтезированной не встречающимся в природе микроорганизмом во время его культивирования в ферментативном бульоне. Neu5Ac может быть выделена из ферментативного бульона после удаления не встречающихся в природе микроорганизмов из ферментативного бульона, например, центрифугированием. Затем Neu5Ac может быть дополнительно очищена от осветленного таким образом ферментативного бульона с помощью подходящих методик, таких как микрофильтрация, ультрафильтрация, диафильтрация, хроматография с псевдодвижущимся слоем, электродиализ, обратный осмос, гель-фильтрация, анионообменная хроматография, катионообменная хроматография и подобных методик.
Способ подходит для крупномасштабного и экономически выгодного получения Neu5Ac посредством микробной ферментации.
Четвертый аспект относится к Neu5Ac, полученной микробной ферментацией, рассмотренной в настоящей работе.
Пятый аспект относится к применению Neu5Ac, полученной как раскрыто в настоящей работе, для изготовления питательной композиции.
Шестой аспект относится к питательной композиции, содержащий Neu5Ac, полученной способом согласно третьему аспекту.
В дополнительном и/или альтернативном примере осуществления питательная композиция дополнительно содержит по меньшей мере один олигосахарид грудного молока (англ. human milk oligosaccharide, сокращенно HMO), предпочтительно по меньшей мере один нейтральный НМО и/или по меньшей мере один кислотный НМО.
Нейтральный олигосахарид (олигосахариды) грудного молока может быть выбран из группы, состоящей из 2'-фукозиллактозы (2'-FL), 3-фукозиллактозы (3-FL), лакто-N-тетраозы (LNT), лакто-N-неотетраозы (LNnT) и лакто-N-фукопентаозы I (LNPFI).
Кислотный олигосахарид (олигосахариды) грудного молока может быть выбран из группы, состоящей из сиалилированных НМО, предпочтительно из группы, состоящей из 3'-сиалиллактозы (3-SL), 6'-сиалиллактозы (6-SL), сиалиллакто-N-тетраозы a (LST-a), сиалиллакто-N-тетраозы b (LST-b), сиалиллакто-N-тетраозы с (LST-c) и дисиалиллакто-N-тетраозы (DSLNT).
В дополнительном примере осуществления питательная композиция выбрана из группы, состоящей из лекарственных препаратов, детских питательных смесей и пищевых добавок.
Питательная композиция может быть предоставлена в жидкой форме или в твердой форме, примеры которой включают, без ограничений, порошки, гранулы, хлопья и крупинки.
В дополнительном и/или альтернативном примере осуществления питательная композиция также включает микроорганизмы, предпочтительно пробиотические микроорганизмы. Для приготовления питания для младенцев предпочтительные микроорганизмы получают из кишечной флоры здорового человека, или предпочтительными микроорганизмами являются микроорганизмы, которые обнаруживаются в кишечной флоре здорового человека. Предпочтительно микроорганизмы выбраны из следующих видов: Bifidobacterium, Lactobacillus, Enterococcus, Streptococcus, Staphylococcus, Peptostreptococcus, Leuconostoc, Clostridium, Eubacterium, Veilonella, Fusobacterium, Bacterioides, Prevotella, Escherichia, Propionibacterium или Saccharomyces, однако также могут быть применены другие микроорганизмы. В дополнительном и/или альтернативном примере осуществления микроорганизм выбран из группы, состоящей из Bifidobacterium breve, Bifidobacterium longum, Bifidobacterium lactis, Bifidobacterium animalis, Bifidobacterium bifidum, Bifidobacterium infantis, Bifidobacterium aldolescentis, Lactobacillus acidophilus, Lactobacillus plantarum, Lactobacillus salivarius, Lactobacillus casei, Lactobacillus gasseri, Lactobacillus reuteri, Lactobacillus rhamnosus, Lactobacillus plantarum, Lactobacillus salivarius, Lactococcus lactis, Lactobacillus paracasei, Lactobacillus bulgaricus, Lactobacillus helveticus, Lactobacillus fermentum, Leuconostoc mesenteroides; Escherichia coli, Enterococcus faecium и Streptococcus thermophilus (VSL#3).
Кроме комбинации Neu5Ac с живыми организмами, также может быть Neu5Ac применена ее комбинация с инактивированными (убитыми) культурами, которые иногда применяют в области техники, относящейся к пробиотикам (например, тиндализованные бактерии). Такие инактивированные культуры могут быть источником белков, пептидов, олигосахаридов, фрагментов клеточных мембран (стенок) и натуральных продуктов, которые вызывают кратковременное стимулирование иммунной системы.
Комбинация Neu5Ac и пробиотических микроорганизмов в питательной композиции особенно предпочтительна для создания или воссоздания подходящей кишечной флоры, которая поддерживает соответствующее здоровое состояние человека.
Более предпочтительной является комбинация сиаловой кислоты с одобренными пребиотиками, такими как галактоолигосахариды (сокращенно ГОС) и/или фруктоолигосахариды (сокращенно ФОС), включающие инулин.
Ниже настоящее изобретение раскрыто с помощью конкретных примеров осуществления и сопроводительных графических материалов; однако изобретение ограничено не приведенными примерами, а лишь объемом прилагаемых пунктов формулы изобретения. Кроме того, термины "первый", "второй" и подобные термины, употребляемые в описании и пунктах формулы изобретения, использованы для различия схожих элементов и не обязательно относятся к последовательности, как временной, так и пространственной, не определяют главенства или подобных факторов. Следует понимать, что при подходящих обстоятельствах используемые термины могут быть взаимозаменяемыми, и что примеры осуществления изобретения, рассмотренные в настоящей работе, могут быть воплощены в другой последовательности, отличной от рассмотренной или показанной в настоящей работе.
Следует отметить, что термин "включающий", употребляемый в пунктах формулы изобретения, не должен рассматриваться как ограниченный перечисленными после него значениями; напротив, он не исключает наличия других элементов или этапов. Таким образом, следует понимать, что этот термин определяет наличие указанных признаков, целых чисел, этапов или компонентов, но не исключает наличия или добавления одного или более других признаков, целых чисел, этапов или компонентов или их групп. Таким образом, объем определения "устройство, включающее средства А и В" не ограничен устройствами, состоящими только из компонентов А и В. Согласно настоящему изобретению, он означает, то единственными существенными компонентами устройства являются А и В.
Упоминание в настоящей работе "примера осуществления" означает, что конкретный признак, структура или характеристика, рассмотренная при раскрытии примера осуществления, включена в по меньшей мере один пример осуществления настоящего изобретения. Таким образом, упоминаемые в различных частях настоящей работы фразы "в примере осуществления" или "в одном из примеров осуществления" не обязательно относятся к одному и тому же примеру осуществления и могут относиться ко множеству и/или различным примерам осуществления. Кроме того, специалисту в данной области техники после прочтения настоящего описания должно быть понятно, что конкретные признаки, структуры или характеристики могут быть скомбинированы любым подходящим образом с получением одного или более примеров осуществления.
Аналогично, следует понимать, что при описании репрезентативных примеров осуществления изобретения различные признаки изобретения в некоторых случаях сгруппированы вместе в один пример осуществления, фигуру или их описание для упрощения понимания сущности изобретения и облегчения понимания одного или более различных аспектов изобретения. Однако такой способ раскрытия изобретения не должен рассматриваться как то, что заявляемое изобретение требует больше признаков, чем те признаки, которые ясно указаны в каждом пункте формулы изобретения. Напротив, как следует из приведенных пунктов формулы изобретения, аспекты изобретения определяются не всеми перечисленными признаками каждого из описанных выше примеров осуществления. Таким образом, пункты формулы изобретения, приведенные после подробного рассмотрения изобретения, полностью включены в подробное рассмотрение изобретения, и каждый пункт формулы изобретения должен рассматриваться как отдельный пример осуществления настоящего изобретения.
Кроме того, как должно быть понятно специалистам в данной области техники, в то время как некоторые примеры осуществления, рассмотренные в настоящей работе, включают некоторые, но все признаки, включенные в другие примеры осуществления, в объем изобретения включены комбинации признаков различных примеров осуществления, и они образуют другие примеры осуществления. Например, в приведенных ниже пунктах формулы изобретения любой из заявленных примеров осуществления может быть воплощен в любой комбинации.
Кроме того, некоторые из примеров осуществления рассмотрены в настоящей работе в виде способа или комбинации элементов способа, которые могут быть выполнены процессором компьютерной системы или другими средствами осуществления функций. Таким образом, процессор с инструкциями, необходимыми для выполнения способа или элемента способа, представляет собой средства осуществления способа или элемента способа. Так, рассмотренный в настоящей работе элемент примера осуществления установки является примером средств осуществления функции, выполняемой элементом для воплощения изобретения.
В описании и графических материалах, представленных в настоящей работе, показаны различные конкретные детали. Однако следует понимать, что примеры осуществления изобретения могут быть воплощены без использования этих конкретных деталей. В других примерах, для упрощения изложения и облегчения понимания, хорошо известные способы, структуры и методики подробно не рассмотрены.
Ниже изобретение раскрыто посредством подробного рассмотрения нескольких примеров осуществления изобретения. Специалисты в данной области техники могут создать другие примеры осуществления изобретения, не выходящие за пределы объема или технической сущности изобретения, которое ограничено лишь объемом прилагаемых пунктов формулы изобретения.
ОПИСАНИЕ ПРИМЕРОВ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Пример 1
Конструирование пути обмена веществ штамма BL21 (DE3) Е. coli, продуцирующего N-ацетилнейраминовую кислоту
Для конструирования пути обмена веществ проводили мутагенез и осуществляли делеции специфичных эндогенных генов и геномную интеграцию гетерологичных генов. Гены lacZ и araA были инактивированы мутагенезом с использованием ошибочных олигонуклеотидов, как рассмотрено в публикации Ellis с соавт. (Proc. Natl. Acad. Sci. USA 98: 6742-6746 (2001)).
Геномные делеции генерировали способом Datsenko и Wanner (Proc. Natl. Acad. Sci. USA 97:6640-6645 (2000)). Для предотвращения разрушения N-ацетилглюкозамина, из генома штамма BL21 (DE3) Е. coli удаляли следующие гены: N-ацетилглюкозамин-специфичного PTS фермента II (nagE), N-ацетилглюкозамин-6-фосфатдезацетилазы (nagA) и глюкозамин-6-фосфатдезаминазы (nagB). Также целиком удаляли катаболический генный кластер N-ацетилнейраминовой кислоты, кодирующий N-ацетилманнозаминкиназу (nanK), N-ацетилманнозамин-6-фосфатэпимеразу (nanE), альдолазу N-ацетилнейраминовой кислоты (nanA) и пермеазу сиаловой кислоты (nanT). Также были удалены гены manX, manY и manZ, кодирующие фосфоенолпируват-зависимую систему фосфотрансферазы, обеспечивающую импорт глюкозамина. Также были удалены гены wzxC-wcaJ. Ген wcaJ кодирует трансферазу UDP-глюкозы:ундекапренилфосфат-глюкозо-1-фосфата, катализирующую первый этап синтеза колановой кислоты (Stevenson с соавт., J. Bacteriol. 1996, 178:4885-4893). Кроме того, были удалены гены fucI, fucK и agaA, кодирующие L-фукозоизомеразу, L-фукулозокиназу и N-ацетилгалактозамин-6-фосфатдезацетилазу, соответственно.
Геномную интеграцию гетерологичных генов проводили, применяя транспозицию с участием либо транспозазы EZ-Tn5™ (Epicentre, USA), либо гиперактивного С9-мутанта транспозазы mariner Himar1 (Proc. Natl. Acad. Sci. 1999, USA 96:11428-11433). Для получения транспосом EZ-Tn5 проводили амплификацию целевого гена совместно с сайтом FRT, фланкированным маркером устойчивости к антибиотикам (в альтернативном варианте ген маркера устойчивости был фланкирован сайтами lox66-lox71). Полученный продукт ПЦР на обоих концах содержал сайты распознавания мозаичных концов 19-bp (Mosaic End recognition sites) для распознавания транспозазы EZ-Tn5. Для проведения интеграции под действием транспозазы Himar1 целевые экспрессионные конструкты (опероны) аналогично клонировали вместе с сайтом FRT/сайтом lox66-lox71, фланкированным маркерами устойчивости к антибиотикам, и внедряли в вектор pEcomar, который кодирует гиперактивный С9-мутант транспозазы mariner Himar1 под контролем индуцируемого арабинозой промотора ParaB. Все гены были кодон-оптимизированными для экспрессии в Е. coli и получены синтетическими способами в GenScript Corp.
Экспрессионный фрагмент <Ptet-lacY-FRT-aadA-FRT> (SEQ ID NO: 1) интегрировали с помощью транспозазы EZ-Tn5. После успешной интеграции гена импортера лактозы LacY из K12 TG1 Е. coli (GenBank: ABN72583), ген устойчивости был удален из устойчивых к стрептомицину клонов под действием рекомбиназы FLP, кодируемой на плазмиде рСР20 (Proc. Natl. Acad. Sci. 2000, USA 97:6640-6645). В геном также вводили генный кластер csc (SEQ ID NO: 2) из E. coli W (GenBank: CP002185.1), включающий гены сахарозопермеазы, фруктокиназы, сахарозогидролазы и транскрипционного репрессора (гены cscB, cscK, cscA, и cscR, соответственно), что позволяло выращивать штамм на сахарозе, применяемой в качестве единственного источника углерода. Этот кластер интегрировали в геном штамма Е. coli BL21 (DE3), применяя транспозицию с использованием плазмида pEcomar-cscABKR.
Полученный штамм дополнительно модифицировали для продукцирования Neu5Ac, применяя геномную интеграцию следующих экспрессионных кассет: <Ptet slr1975-gna1-lox66-aacC1-lox71> (SEQ ID NO: 3), <Ptet-neuB-lox66-kanR-lox71> (SEQ ID NO: 4) <Ptet-slr1975-Pt5-neuB-FRT-dhfr-FRT> (SEQ ID NO: 5), <Ptet-glmS*-gna1-lox66-aacC1-lox71> (SEQ ID NO: 6) и <Ptet-ppsA-lox66-aacC1-lox71> (SEQ ID NO: 7). 3a исключением экспрессионной кассеты dhfr последовательности SEQ ID NO 5, все гены маркеров устойчивости были поэтапно удалены из геномов (до проведения последующего раунда интеграции генов) посредством введения плазмида pKD-Cre (SEQ ID NO: 8), после чего проводили отбор на агаровых планшетах 2YT, содержащих 100 мкг⋅мл-1 ампициллина и 100 мМ L-арабинозы при 30°С. Устойчивые клоны затем перемещали на агаровые планшеты 2YT, не содержащие ни ампициллина, ни селективного антибиотика, применяемого для геномной интеграции. Планшеты инкубировали при 42°С для созревания клеточных плазмид. Для последующих экспериментов и модификаций отбирали клоны, которые были чувствительны к ампициллину и селективному антибиотику.
Ген slr1975 (GenBank: BAL35720) кодирует N-ацетилглюкозамин-2-эпимеразу Synechocystis sp. РСС6803. Ген gna1 (GenBank: NP_116637) кодирует глюкозамин-6-фосфатацетилтрансферазу Saccharomyces cerevisiae. Ген neuB (GenBank: AF305571) кодирует синтазу сиаловой кислоты Campylobacter jejuni. Ген glmS* представляет собой мутированную версию гена L-глутамин:D-фруктозо-6-фосфатаминотрансферазы Е. coli (Metab Eng. 2005 May; 7(3):201-14). Ген ppsA (GenBank: ACT43527) кодирует фосфоенолпируватсинтазу E. coli BL21 (DE3).
Для образования <Ptet-slr1975-gna1-lox66-aacC1-lox71> гены slr1975 и gna1 субклонируются в виде оперона позади конститутивного промотора Ptet, сплавляются с геном устойчивости к гентамицину (фланкированным сайтами lox66/lox71) и вводятся в вектор pEcomar сшиванием тупых концов. Полученную экспрессионную кассету интегрировали в геном с использованием вектора pEcomar-slr195-gna1-aacC1 и гиперактивного С9-мутанта транспозазы mariner Himar1 под контролем индуцируемого арабинозой промотора ParaB.
Для образования <Ptet-neuB-lox66-kanR-lox71> neuB клонируется позади конститутивного промотора Ptet и сплавляется с геном устойчивости к канамицину (фланкированным сайтами lox66/lox71). Полученную экспрессионную кассету интегрировали в геном под действием транспозазы EZ-Tn5. Для образования <Ptet-slr1975-Pt5-neuB-FRT-dhfr-FRT> гены slr1975 и neuB по отдельности субклонируются позади конститутивных промоторов Ptet и Pt5, соответственно, и сплавляются с геном устойчивости к триметоприму (фланкированным сайтами FRT). Полученную экспрессионную кассету интегрировали в геном под действием транспозазы EZ-Tn5.
Экспрессионную кассету <Ptet-glmS*-gna1-lox66-aacC1-lox71> получали клонированием glmS* и gna1 в виде оперона позади конститутивного промотора Ptet. Этот конструкт дополнительно сплавляли с геном устойчивости к гентамицину (фланкированным сайтами lox66/lox71). Полученную экспрессионную кассету интегрировали в геном под действием транспозазы EZ-Tn5.
Для образования <Ptet-ppsA-lox66-aacC1-lox71> ген ppsA клонируется позади конститутивного промотора Ptet и сплавляется с геном устойчивости к гентамицину (фланкированным сайтами lox66/lox71). Полученную экспрессионную кассету интегрировали в геном под действием транспозазы EZ-Tn5.
В итог суммарные модификации генома приводили к получению штамма Е. coli #NANA1, способного продуцировать Neu5Ac.
Пример 2
Получение N-ацетилнейраминовой кислоты способом периодической ферментации с подпиткой
Штамм #NANA1 Е. coli BL21 (DE3) культивировали при 30°С в ферментаторах емкостью 3 л (New Brunswick, Edison, USA), начиная с 1000 мл среды, содержащей минеральные соли в следующих концентрациях: 7 г⋅л-1 NH4H2PO4, 7 г⋅л-1 K2HPO4, 2 г⋅л-1 KOH, 0,3 г⋅л-1 лимонной кислоты, 2 г⋅л-1 MgSO4×7 H2O, 5 г⋅л-1 NH4Cl2 и 0,015 г⋅л-1 CaCl2×6⋅H2O, которую дополняли 1 мл⋅л-1 раствора микроэлементов (54,4 г⋅л-1 цитрата аммония-железа (III), 9,8 г⋅л-1 MnCl2×4⋅H2O, 1,6 г⋅л-1 CoCl2×6⋅H2O, 1 г⋅л-1 CuCl2×2⋅H2O, 1,9 г⋅л-1 Н3ВО3, 9 г⋅л-1 ZnSO4×7⋅H2O, 1,1 г⋅л-1 Na2MoO4×2⋅H2O, 1,5 г⋅л-1 Na2SeO3, 1,5 г⋅л-1 NiSO4×6⋅H2O), а также содержащей 2% (масс./об.) сахарозы, применяемой в качестве источника углерода, и антибиотик зеоцин (10 мкг⋅мл-1). Культивирование начинали с введения инокулята концентрацией 2,5% (об./об.) предварительно выращенной культуры, культивируемой в той же среде, содержащей сахарозу. Окончание порционной ферментации характеризовалось повышением концентрации растворенного кислорода. Подачу сахарозы проводили непосредственно после окончания порционной ферментации. Подаваемый 50% (масс./об.) раствор сахарозы дополняли 2 г⋅л-1 MgSO4×7⋅H2O, 0,015 г⋅л-1 CaCl2×6⋅H2O и 1 мл⋅л-1 раствора микроэлементов. Скорость подачи составляла от 9,0 до 11,0 мл⋅л-1 в пересчете на начальный объем. Скорость аэрации составляла 3 л⋅мин-1. Концентрацию растворенного кислорода поддерживали на уровне 20-30% насыщения, регулируя скорость перемешивания. Величину рН поддерживали на уровне 7,0 посредством добавления 25% раствора аммиака.
Для обнаружения Neu5Ac в культуральной жидкости над осадком применяли высокоэффективную жидкостную хроматографию (ВЭЖХ, Shimadzu). Оборудование включало детектор UV-VIS для обнаружения при длине волны λ=210 нм (SPD-10AVP, Shimadzu) и аналитическую колонку Rezex ROA-organic acid Н+ (300×7,8 мм) с подходящим защитным картриджем. Изократическое элюирование выполняли в 5 мМ H2SO4 при 50°С при скорости течения, составляющей 0,5 мл⋅мин-1. Культуральную жидкость над осадком центрифугировали, стерилизовали с помощью фильтра и нагревали до 95°С в течение 5 минут. После окончательного центрифугирования 5 мкл образца наносили на колонку. Концентрацию N-ацетилнейраминовой кислоты вычисляли из стандартной кривой, применяя коммерчески доступный стандарт (Carbosynth, Compton, UK). После инкубации в течение 88 часов окончательный титр Neu5Ac в культуральной жидкости над осадком составил 68,6 г⋅л-1.
Пример 3
Получение и культивирования организмов с одиночными нокаутными мутациями в штамме Е. coli #NANA1, которые обладают улучшенной способностью вырабатывать Neu5Ac
Штамм Е. coli BL21 (DE3) #NANA1 модифицировали дополнительно, получая мутантов с делециями, соответствующими удалению или разрушению генов gltB, yjhC и ррС. Штаммы #NANA1, #NANA1ΔgltB, #NANA1ΔyjhC и #NANA1Δppc культивировали на 96-луночных планшетах. Таким образом, одиночные колонии штаммов переносили с агаровых планшетов в микротитрационные планшеты, содержащие 200 мкл минимальной среды, рассмотренной в Примере 2, и инкубировали в течение ~20 часов при 30°С при энергичном встряхивании. Затем 50 мкл культурального бульона переносили в 96-луночные планшеты с глубокими лунками (2,0 мл), содержащие 400 мкл минимальной среды в каждой лунке.
После инкубации в течение дополнительных 48 часов культивирование прекращали, и количество N-ацетилнейраминовой кислоты в супернатанте определяли способом масс-спектрометрии в режиме мониторинга множественных реакций (англ. multiple reaction monitoring, сокращенно MRM) с помощью системы обнаружения ЖХ-трехквадрупольной масс-спектрометрии (англ. LC Triple-Quadrupole MS). Ионы-предшественники выбирали и анализировали в квадруполе 1, фрагментация происходила в ячейке столкновений с использованием аргона в качестве газа для соударений, и ионы-фрагменты выбирали в квадруполе 3. Образец N-ацетилнейраминовой кислоты объемом 1 мкл впрыскивали в устройство для проведения ВЭЖХ после разбавления культуральной жидкости над осадком 1:100 водой, подходящей для ЖХ/МС (жидкостной хроматографии/масс-спектрометрии). Образец разделяли на колонке для ВЭЖХ XBridge Amide (3,5 мкм, 2,1×50 мм (Waters, USA), снабженной защитным картриджем XBridge Amide (3,5 мкм, 2,1×10 мм) (Waters, USA), при 50°С в смеси ацетонитрил : H2O с добавлением 10 мМ ацетата аммония при скорости течения, составляющей 400 мкл⋅мин-1. Каждое разделение продолжалось 240 секунд. N-ацетилнейраминовую кислоту анализировали электрораспылительной ионизацией (англ. electrospray ionization, сокр. ESI) с MRM в режиме положительной ионизации. Масс-спектрометр работал при единичном разрешении. N-ацетилнейраминовая кислота образует ион с m/z 309,2 [М+Н]. Ион-предшественник N-ацетилнейраминовой кислоты дополнительно фрагментировался в ячейке столкновений на ионы-фрагменты с m/z 292,20, m/z 274,15 и m/z 121,15. Энергию столкновений, начальное смещение (Pre Bias) Q1 и Q3 оптимизировали для каждого анализируемого вещества индивидуально. Способ количественного определения был установлен в соответствии с коммерчески доступным стандартом (Carbosynth, Compton, UK).
На Фиг. 3 представлены относительные выходы Neu5Ac при культивировании штаммов. Для сравнения с материнским штаммом величину, полученную при культивировании штамма #NANA1, принимали за 100%, и выход N-ацетилнейраминовой кислоты в штаммах с одиночными нокаутными мутациями превосходил выходы материнского штамма на величину, составляющую от 20% до 25%.
Пример 4
Извлечение Neu5Ac из ферментативного бульона
По завершении ферментации биомассу отделяли от ферментационной среды ультрафильтрацией, после чего последовательно применяли фильтр с намоточным модулем (winding module) (порог пропускания 0,05 мкм) (мембранная методика CUT, Erkrath, Germany) и фильтр с перекрестным током (порог пропускания 150 кДа) (Microdyn-Nadir, Wiesbaden, Germany). Получали приблизительно 1 м3 ферментационной среды, не содержащей клеток, которая содержала более 19 г⋅л-1 сиаловой кислоты.
Жидкость, не содержащую клеток, затем подвергали деионизации с помощью ионообменной хроматографии. Сначала на сильной катионообменной смоле Lewatit® S 2568, находящейся в Н+-форме (Lanxess AG, Cologne, Germany), в объеме 200 л удаляли катионные загрязняющие вещества. Полученный раствор, имеющий рН, приблизительно составляющий 1,5, нейтрализовали до величины 7,0 добавлением NaOH. При выполнении второго этапа с помощью сильной анионообменной смолы Lewatit® S 6368 A (Lanxess AG, Cologne, Germany), находящейся в хлоридной форме, из раствора удаляли анионы и нежелательные окрашивающие вещества. Объем слоя в ионообменном устройстве составлял 200 л. Осадки, полученные при подкислении раствора, удаляли во втором этапе фильтрования с помощью фильтра с перекрестным током (порог пропускания 150 кДа) (Microdyn-Nadir GmbH, Wiesbaden, Germany). Для концентрирования сахара раствор подвергали нанофильтрации на устройстве Dow Filmtec® NF270-4040 (INAQUA Vertriebsgesellschaft mbH,  Germany) до получения приблизительно
Germany) до получения приблизительно 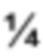 объема. Затем концентрированный раствор Neu5Ac вновь концентрировали на роторном испарителе до концентрации приблизительно 400 г⋅л-1 или более. Специфичную кристаллизацию продукта выполняли, используя 10-кратный избыток ледяной уксусной кислоты, при 5°С в течение периода времени от 12 до 60 часов. Твердую фракцию отфильтровывали, промывали этанолом и сушили при 40°С. Высушенный кристаллизованный продукт очищали дополнительно. Так, сухой продукт растворяли в 2 л H2O на кг и обрабатывали активированным углем (CAS-No: 7440-44-0, Carl Roth GmbH & Co. KG, Karlsruhe, Germany). После отделения угля осветленный раствор концентрировали испарением при 50°С до затвердевания. Твердый материал смешивали с 99% этанолом и инкубировали при 4°С в течение по меньшей мере 16 часов. Затем твердую фракцию отфильтровывали и сушили при 40°С. Получали белый кристаллический продукт, чистота которого составляла более 95% в соответствии с вычислениями по площади под хроматографической кривой, полученной способом ВЭЖХ с помощью колонки Rezex ROA-organic acid Н+ (Phenomenex, Aschaffenburg, Germany).
объема. Затем концентрированный раствор Neu5Ac вновь концентрировали на роторном испарителе до концентрации приблизительно 400 г⋅л-1 или более. Специфичную кристаллизацию продукта выполняли, используя 10-кратный избыток ледяной уксусной кислоты, при 5°С в течение периода времени от 12 до 60 часов. Твердую фракцию отфильтровывали, промывали этанолом и сушили при 40°С. Высушенный кристаллизованный продукт очищали дополнительно. Так, сухой продукт растворяли в 2 л H2O на кг и обрабатывали активированным углем (CAS-No: 7440-44-0, Carl Roth GmbH & Co. KG, Karlsruhe, Germany). После отделения угля осветленный раствор концентрировали испарением при 50°С до затвердевания. Твердый материал смешивали с 99% этанолом и инкубировали при 4°С в течение по меньшей мере 16 часов. Затем твердую фракцию отфильтровывали и сушили при 40°С. Получали белый кристаллический продукт, чистота которого составляла более 95% в соответствии с вычислениями по площади под хроматографической кривой, полученной способом ВЭЖХ с помощью колонки Rezex ROA-organic acid Н+ (Phenomenex, Aschaffenburg, Germany).
Пример 5
Альтернативный путь синтеза N-ацетилнейраминовой кислоты
Штамм Е. coli BL21 (DE3), рассмотренный в Примере 1 (ΔlacZ, ΔaraA, ΔnagABE, ΔnanATEK, ΔmanXYZ, ΔwcaJ, ΔfucIK, ΔagaA, lacY+, cscABKR+) дополнительно модифицировали интеграцией экспрессионной кассеты <Ptet-glmS*-gna1-lox66-aacC1-lox71>, получая штамм, способный синтезировать N-ацетилглюкозамин (штамм А). Штамм А модифицировали с целью получения штамма для синтеза N-ацетилнейраминовой кислоты. Для этого в геном штамма А по отдельности интегрировали экспрессионные конструкты <Ptet-slr1975-Pt5-neuB-FRT-dhfr-FRT> (SEQ ID NO: 5) или <PtetEcnanE-Pt5-neuB-FRT-dhfr-FRT> (SEQ ID NO: 9), получая штаммы В и С, соответственно. Ген EcnanE (GenBank:. YP_003614592) кодирует N-ацилглюкозамин-6-фосфат-2-эпимеразу Enterobacter cloacae subsp. cloacae АТСС 13047. Все экспрессионные кассеты интегрировали в геном под действием транспозазы EZ-Tn5.
Одиночные колонии этих штаммов переносили с агаровых планшетов на микротитрационные планшеты, содержащие 200 мкл минимальной среды, рассмотренной в Примере 2, и культивировали в течение ~20 часов при 30°С при энергичном встряхивании. Затем 50 мкл культурального бульона переносили в 96-луночные планшеты с глубокими лунками (2,0 мл), содержащие в каждой лунке 400 мкл минимальной среды. После инкубации в течение еще 48 часов культивирование прекращали и способом масс-спектрометрии определяли концентрацию N-ацетилнейраминовой кислоты в жидкости над осадком.
Синтезированную Neu5Ac обнаруживали только в культуральных жидкостях над осадками штаммов В и С. На Фиг. 4 показаны относительные выходы Neu5Ac, полученные при культивировании штаммов. Для сравнения вход Neu5Ac в штамме В был принят за 100%. Штамм С вырабатывал приблизительно 7,5% от выхода Neu5Ac в штамме В.
Пример 6
Состав детской питательной смеси, содержащей Neu5Ac
Детская питательная смесь:
Обезжиренное молоко
Растительные масла (пальмовое масло, рапсовое масло, подсолнечное масло)
Олигосахариды грудного молока
L-Фукоза
N-ацетилнейраминовая кислота
Порошкообразное обезжиренное молоко
Масло Mortierella alpine
Рыбий жир
Карбонат кальция
Хлорид калия
Витамин С
Хлорид натрия
Витамин Е
Ацетат железа
Сульфат цинка
Ниацин
Кальций-D-пантотенат
Сульфат меди
Витамин А
Витамин В1
Витамин В6
Сульфат магния
Йодат калия
Фолиевая кислота
Витамин K
Селенит натрия
Витамин D
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Jennewein Biotechnologie GmbH
<120> ФЕРМЕНТАТИВНЫЙ СИНТЕЗ N-АЦЕТИЛНЕЙРАМИНОВОЙ КИСЛОТЫ
<130> P 1705 WO
<150> EP 17196925.6
<151> 2017-10-17
<160> 103
<170> PatentIn version 3.5
<210> 1
<211> 2851
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Фрагмент экспрессии
<400> 1
tggccagatg attaattcct aatttttgtt gacactctat cattgataga gttattttac
60
cactccctat cagtgataga gaaaagtgaa atgaatagtt cgacaaaaat ctagaaataa
120
ttttgtttaa ctttaagaag gagatataca aatgtactat ttaaaaaaca caaacttttg
180
gatgttcggt ttattctttt tcttttactt ttttatcatg ggagcctact tcccgttttt
240
cccgatttgg ctacatgaca tcaaccatat cagcaaaagt gatacgggta ttatttttgc
300
cgctatttct ctgttctcgc tattattcca accgctgttt ggtctgcttt ctgacaaact
360
cgggctgcgc aaatacctgc tgtggattat taccggcatg ttagtgatgt ttgcgccgtt
420
ctttattttt atcttcgggc cactgttaca atacaacatt ttagtaggat cgattgttgg
480
tggtatttat ctaggctttt gttttaacgc cggtgcgcca gcagtagagg catttattga
540
gaaagtcagc cgtcgcagta atttcgaatt tggtcgcgcg cggatgtttg gctgtgttgg
600
ctgggcgctg tgtgcctcga ttgtcggcat catgttcacc atcaataatc agtttgtttt
660
ctggctgggc tctggctgtg cactcatcct cgccgtttta ctctttttcg ccaaaacgga
720
tgcgccctct tctgccacgg ttgccaatgc ggtaggtgcc aaccattcgg catttagcct
780
taagctggca ctggaactgt tcagacagcc aaaactgtgg tttttgtcac tgtatgttat
840
tggcgtttcc tgcacctacg atgtttttga ccaacagttt gctaatttct ttacttcgtt
900
ctttgctacc ggtgaacagg gtacgcgggt atttggctac gtaacgacaa tgggcgaatt
960
acttaacgcc tcgattatgt tctttgcgcc actgatcatt aatcgcatcg gtgggaaaaa
1020
cgccctgctg ctggctggca ctattatgtc tgtacgtatt attggctcat cgttcgccac
1080
ctcagcgctg gaagtggtta ttctgaaaac gctgcatatg tttgaagtac cgttcctgct
1140
ggtgggctgc tttaaatata ttaccagcca gtttgaagtg cgtttttcag cgacgattta
1200
tctggtctgt ttctgcttct ttaagcaact ggcgatgatt tttatgtctg tactggcggg
1260
caatatgtat gaaagcatcg gtttccaggg cgcttatctg gtgctgggtc tggtggcgct
1320
gggcttcacc ttaatttccg tgttcacgct tagcggcccc ggcccgcttt ccctgctgcg
1380
tcgtcaggtg aatgaagtcg ctgggagcta agcggccgcg tcgacacgca aaaaggccat
1440
ccgtcaggat ggccttctgc ttaatttgat gcctggcagt ttatggcggg cgtcctgccc
1500
gccaccctcc gggccgttgc ttcgcaacgt tcaaatccgc tcccggcgga tttgtcctac
1560
tcaggagagc gttcaccgac aaacaacaga taaaacgaaa ggcccagtct ttcgactgag
1620
cctttcgttt tatttgatgc ctggcagttc cctactctcg catggggaga ccccacacta
1680
ccatcatgta tgaatatcct ccttagttcc tattccgaag ttcctattct ctagaaagta
1740
taggaacttc ggcgcgtcct acctgtgaca cgcgtgccgc agtctcacgc ccggagcgta
1800
gcgaccgagt gagctagcta tttgtttatt tttctaaata cattcaaata tgtatccgct
1860
catgagacaa taaccctgat aaatgcttca ataatattga aaaaggaaga gtatgaggga
1920
agcggtgatc gccgaagtat cgactcaact atcagaggta gttggcgtca tcgagcgcca
1980
tctcgaaccg acgttgctgg ccgtacattt gtacggctcc gcagtggatg gcggcctgaa
2040
gccacacagt gatattgatt tgctggttac ggtgaccgta aggcttgatg aaacaacgcg
2100
gcgagctttg atcaacgacc ttttggaaac ttcggcttcc cctggagaga gcgagattct
2160
ccgcgctgta gaagtcacca ttgttgtgca cgacgacatc attccgtggc gttatccagc
2220
taagcgcgaa ctgcaatttg gagaatggca gcgcaatgac attcttgcag gtatcttcga
2280
gccagccacg atcgacattg atctggctat cttgctgaca aaagcaagag aacatagcgt
2340
tgccttggta ggtccagcgg cggaggaact ctttgatccg gttcctgaac aggatctatt
2400
tgaggcgcta aatgaaacct taacgctatg gaactcgccg cccgactggg ctggcgatga
2460
gcgaaatgta gtgcttacgt tgtcccgcat ttggtacagc gcagtaaccg gcaaaatcgc
2520
gccgaaggat gtcgctgccg actgggcaat ggagcgcctg ccggcccagt atcagcccgt
2580
catacttgaa gctagacagg cttatcttgg acaagaagaa gatcgcttgg cctcgcgcgc
2640
agatcagttg gaagaatttg tccactacgt gaaaggcgag atcaccaagg tagtcggcaa
2700
ataatgtcta acaattcgtt caagccgagg ggccgcaaga tccggccacg atgacccggt
2760
cgtcgggtac cggcagggcg gggcgtaagg cgcgccattt aaatgaagtt cctattccga
2820
agttcctatt ctctagaaag tataggaact t
2851
<210> 2
<211> 5226
<212> DNA
<213> Artificial Sequence
<220>
<223> генный кластер E. coli W содержащий гены сахарозопермеазы
(cscB),
фруктокиназы (cscK), сахарозогидролазы (cscA)
и транскрипционного репрессора(cscR)
<400> 2
acaggttggc tgataagtcc ccggtctggc agccgcgact gtaccagaac atgaatgagg
60
cgtttggatt aggcgattat tagcagggct aagcatttta ctattattat tttccggttg
120
agggatatag agctatcgac aacaaccgga aaaagtttac gtctatattg ctgaaggtac
180
aggcgtttcc ataactattt gctcgcgttt tttactcaag aagaaaatgc caaatagcaa
240
catcaggcag acaatacccg aaattgcgaa gaaaactgtc tggtagcctg cgtggtcaaa
300
gagtatccca gtcggcgttg aaagcagcac aatcccaagc gaactggcaa tttgaaaacc
360
aatcagaaag atcgtcgacg acaggcgctt atcaaagttt gccacgctgt atttgaagac
420
ggatatgaca caaagtggaa cctcaatggc atgtaacaac ttcactaatg aaataatcca
480
ggggttaacg aacagcgcgc aggaaaggat acgcaacgcc ataatcacaa ctccgataag
540
taatgcattt tttggcccta cccgattcac aaagaaagga ataatcgcca tgcacagcgc
600
ttcgagtacc acctggaatg agttgagata accatacagg cgcgttccta catcgtgtga
660
ttcgaataaa cctgaataaa agacaggaaa aagttgttga tcaaaaatgt tatagaaaga
720
ccacgtcccc acaataaata tgacgaaaac ccagaagttt cgatccttga aaactgcgat
780
aaaatcctct ttttttaccc ctcccgcatc tgccgctacg cactggtgat ccttatcttt
840
aaaacgcatg ttgatcatca taaatacagc gccaaatagc gagaccaacc agaagttgat
900
atggggactg atactaaaaa atatgccggc aaagaacgcg ccaatagcat agccaaaaga
960
tccccaggcg cgcgctgttc catattcgaa atgaaaattt cgcgccattt tttcggtgaa
1020
gctatcaagc aaaccgcatc ccgccagata ccccaagcca aaaaatagcg cccccagaat
1080
tagacctaca gaaaaattgc tttgcagtaa cggttcataa acgtaaatca taaacggtcc
1140
ggtcaagacc aggatgaaac tcatacacca gatgagcggt ttcttcagac cgagtttatc
1200
ctgaacgatg ccgtagaaca tcataaatag aatgctggta aactggttga ccgaataaag
1260
tgtacctaat tccgtccctg tcaaccctag atgtcctttc agccaaatag cgtataacga
1320
ccaccacagc gaccaggaaa taaaaaagag aaatgagtaa ctggatgcaa aacgatagta
1380
cgcatttctg aatggaatat tcagtgccat aattacctgc ctgtcgttaa aaaattcacg
1440
tcctatttag agataagagc gacttcgccg tttacttctc actattccag ttcttgtcga
1500
catggcagcg ctgtcattgc ccctttcgcc gttactgcaa gcgctccgca acgttgagcg
1560
agatcgataa ttcgtcgcat ttctctctca tctgtagata atcccgtaga ggacagacct
1620
gtgagtaacc cggcaacgaa cgcatctccc gcccccgtgc tatcgacaca attcacagac
1680
attccagcaa aatggtgaac ttgtcctcga taacagacca ccaccccttc tgcaccttta
1740
gtcaccaaca gcatggcgat ctcatactct tttgccaggg cgcatatatc ctgatcgttc
1800
tgtgtttttc cactgataag tcgccattct tcttccgaga gcttgacgac atccgccagt
1860
tgtagcgcct gccgcaaaca caagcggagc aaatgctcgt cttgccatag atcttcacga
1920
atattaggat cgaagctgac aaaacctccg gcatgccgga tcgccgtcat cgcagtaaat
1980
gcgctggtac gcgaaggctc ggcagacaac gcaattgaac agagatgtaa ccattcgcca
2040
tgtcgccagc agggcaagtc tgtcgtctct aaaaaaagat cggcactggg gcggaccata
2100
aacgtaaatg aacgttcccc ttgatcgttc agatcgacaa gcaccgtgga tgtccggtgc
2160
cattcatctt gcttcagata cgtgatatcg actccctcag ttagcagcgt tctttgcatt
2220
aacgcaccaa aaggatcatc ccccacccga cctataaacc cacttgttcc gcctaatctg
2280
gcgattccca ccgcaacgtt agctggcgcg ccgccaggac aaggcagtag gcgcccgtct
2340
gattctggca agagatctac gaccgcatcc cctaaaaccc atactttggc tgacattttt
2400
ttcccttaaa ttcatctgag ttacgcatag tgataaacct ctttttcgca aaatcgtcat
2460
ggatttacta aaacatgcat attcgatcac aaaacgtcat agttaacgtt aacatttgtg
2520
atattcatcg catttatgaa agtaagggac tttattttta taaaagttaa cgttaacaat
2580
tcaccaaatt tgcttaacca ggatgattaa aatgacgcaa tctcgattgc atgcggcgca
2640
aaacgcccta gcaaaacttc atgagcaccg gggtaacact ttctatcccc attttcacct
2700
cgcgcctcct gccgggtgga tgaacgatcc aaacggcctg atctggttta acgatcgtta
2760
tcacgcgttt tatcaacatc atccgatgag cgaacactgg gggccaatgc actggggaca
2820
tgccaccagc gacgatatga tccactggca gcatgagcct attgcgctag cgccaggaga
2880
cgataatgac aaagacgggt gtttttcagg tagtgctgtc gatgacaatg gtgtcctctc
2940
acttatctac accggacacg tctggctcga tggtgcaggt aatgacgatg caattcgcga
3000
agtacaatgt ctggctacca gtcgggatgg tattcatttc gagaaacagg gtgtgatcct
3060
cactccacca gaaggaatca tgcacttccg cgatcctaaa gtgtggcgtg aagccgacac
3120
atggtggatg gtagtcgggg cgaaagatcc aggcaacacg gggcagatcc tgctttatcg
3180
cggcagttcg ttgcgtgaat ggaccttcga tcgcgtactg gcccacgctg atgcgggtga
3240
aagctatatg tgggaatgtc cggacttttt cagccttggc gatcagcatt atctgatgtt
3300
ttccccgcag ggaatgaatg ccgagggata cagttaccga aatcgctttc aaagtggcgt
3360
aatacccgga atgtggtcgc caggacgact ttttgcacaa tccgggcatt ttactgaact
3420
tgataacggg catgactttt atgcaccaca aagcttttta gcgaaggatg gtcggcgtat
3480
tgttatcggc tggatggata tgtgggaatc gccaatgccc tcaaaacgtg aaggatgggc
3540
aggctgcatg acgctggcgc gcgagctatc agagagcaat ggcaaacttc tacaacgccc
3600
ggtacacgaa gctgagtcgt tacgccagca gcatcaatct gtctctcccc gcacaatcag
3660
caataaatat gttttgcagg aaaacgcgca agcagttgag attcagttgc agtgggcgct
3720
gaagaacagt gatgccgaac attacggatt acagctcggc actggaatgc ggctgtatat
3780
tgataaccaa tctgagcgac ttgttttgtg gcggtattac ccacacgaga atttagacgg
3840
ctaccgtagt attcccctcc cgcagcgtga cacgctcgcc ctaaggatat ttatcgatac
3900
atcatccgtg gaagtattta ttaacgacgg ggaagcggtg atgagtagtc gaatctatcc
3960
gcagccagaa gaacgggaac tgtcgcttta tgcctcccac ggagtggctg tgctgcaaca
4020
tggagcactc tggctactgg gttaacataa tatcaggtgg aacaacggat caacagcggg
4080
caagggatcc gcgtcactct tcccccttca cgaccttcaa taatatgcaa tgcagcttcc
4140
cgcccgataa tgtcatgtgg aagctgaatt gtggtcagcg gcggtaaaaa cagatgcccg
4200
acgccaacca gattatcaaa gcccattacg gcgacatcct gcgggattcg tacccccttc
4260
gccagaagaa cctgataagc cacaaaggct gcgcgatcgt taccacatat cagaacatca
4320
aaatctggtt tgcccggttt gaagtgggca ttgagtaaac ttgcgagatc ggtgtagtga
4380
tcatcacctg ttgccatgtg aaattgtttc acctcagcca gatctcgtcc agcatcacgc
4440
caggcctgct caaatccctg ccgacgatac cctgttgcca acgcactttc cggtagccag
4500
aagcataacg gttgacgata gcccgccgcg agcaaatgct gtgttgattc atattgtgca
4560
gtgtaatcat cagggatata actgggtaac gctgggtcat ccgccacaca gttcgccaat
4620
acaatatttt caccatacag agactcaggc agcgtgatat gtcgcagccc cattgtagta
4680
tagataatgc catccggacg gtgggcaagc agctgacgtg ccgcgcgggc agcgtcatct
4740
tcagaaaaaa tattgattaa aaaactattc cagccgaact cgctggcggt ttgctcaatg
4800
gcaagcagaa tatcaacaga gaaaggagtg gtagccgtgt cctgcgccag cacggcgaga
4860
gtcgacggct tacgtccttg agcgcgcatc ttacgggcgg aaagatcagg aacataattc
4920
agggtctgga ttgcctgcaa tacgcggtca cgcgttgcag gacgcacaga ttctgcatta
4980
tgcatcaccc gggagactgt catcatcgac actcccgcca ggcgtgcgac atcctttaat
5040
gaagccatac ccaagccgtt tgccgtaaaa cgggcactgt agcagaaaca gacgtcactg
5100
gcgagatcca acgccctatc acctgacaca gcaatacaat aaaaaataac aataattccc
5160
ggacaattgt ccccaattcc gcctctgttc tcgcattgta gaccggggac ttatcagcca
5220
acctgt
5226
<210> 3
<211> 3123
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Экспрессионная Кассета
<400> 3
ctgtctctta tacacatctc cggccagatg attaattcct aatttttgtt gacactctat
60
cattgataga gttattttac cactccctat cagtgataga gaaaagtgaa atgaatagtt
120
cgacaaaaat ctagaaataa ttttgtttaa ctttaagaag gagatataca aatgatcgct
180
caccgtcgtc aggaactggc tcaacagtat tatcaggctc tgcaccaaga tgtgctgccg
240
ttctgggaaa agtattcgct ggatcgtcaa ggcggtggct attttacctg cctggaccgc
300
aagggtcagg tttttgatac ggacaagttc atttggctgc aaaaccgtca agtgtggcaa
360
tttgcggttt tctacaatcg cctggaaccg aaaccgcagt ggctggaaat cgctcgtcat
420
ggtgcggatt ttctggcacg tcacggtcgt gatcaggacg gtaactggta tttcgccctg
480
gatcaggaag gcaaaccgct gcgccaaccg tacaatgtgt tttccgactg tttcgcggcg
540
atggcgttta gccagtatgc actggcttct ggtgctcaag aagcgaaggc cattgcactg
600
caagcgtata acaatgttct gcgtcgccag cataacccga aaggtcaata tgaaaagagt
660
tacccgggta cccgtccgct gaaatccctg gcagtgccga tgatcctggc taatctgacg
720
ctggaaatgg aatggctgct gccgccgacc acggtcgaag aagtgctggc ccagaccgtt
780
cgtgaagtca tgacggattt tctggacccg gaaattggcc tgatgcgcga agcagttacc
840
ccgacgggtg aatttgtcga ttcattcgaa ggccgcctgc tgaacccggg tcatggcatt
900
gaagcgatgt ggtttatgat ggatattgcc cagcgttcgg gtgaccgcca gctgcaagaa
960
caggctattg cggtggttct gaataccctg gaatatgcat gggatgaaga atttggtggc
1020
atcttttact tcctggaccg tcaaggtcac ccgccgcagc aactggaatg ggatcagaaa
1080
ctgtggtggg tccatctgga aaccctggtg gccctggcaa aaggtcacca ggcgacgggc
1140
caagaaaagt gctggcagtg gtttgaacgc gtgcatgatt atgcatggag ccactttgct
1200
gacccggaat atggtgaatg gttcggctac ctgaaccgtc gcggtgaagt gctgctgaat
1260
ctgaaaggtg gcaaatggaa gggctgcttc cacgttccgc gtgcgctgtg gctgtgtgcc
1320
gaaaccctgc aactgccggt ctcttaataa tcgaaggaga tacaacatga gcttacccga
1380
tggattttat ataaggcgaa tggaagaggg ggatttggaa caggtcactg agacgctaaa
1440
ggttttgacc accgtgggca ctattacccc cgaatccttc agcaaactca taaaatactg
1500
gaatgaagcc acagtatgga atgataacga agataaaaaa ataatgcaat ataaccccat
1560
ggtgattgtg gacaagcgca ccgagacggt tgccgctacg gggaatatca tcatcgaaag
1620
aaagatcatt catgaactgg ggctatgtgg ccacatcgag gacattgcag taaactccaa
1680
gtatcagggc caaggtttgg gcaagctctt gattgatcaa ttggtaacta tcggctttga
1740
ctacggttgt tataagatta ttttagattg cgatgagaaa aatgtcaaat tctatgaaaa
1800
atgtgggttt agcaacgcag gcgtggaaat gcaaattaga aaatagaata actagcataa
1860
acccccttgg ggcctctaaa cgggtcttga ggggtttttt gctgaaacca atttgcctgg
1920
cggcagtagc gcggtggtcc cacctgaccc catgccgaac tcagaagtga aacgccgtag
1980
cgccgatggt agtgtggggt ctccccatgc gagagtaggg aactgccagg catcaaataa
2040
aacgaaaggc tcagtcgaaa gactgggcct ttcgggatcc aggccggcct gttaagacgg
2100
ccagtgaatt cgagctcggt acctaccgtt cgtataatgt atgctatacg aagttatcga
2160
gctctagaga atgatcccct cattaggcca cacgttcaag tgcagcgcac accgtggaaa
2220
cggatgaagg cacgaaccca gttgacataa gcctgttcgg ttcgtaaact gtaatgcaag
2280
tagcgtatgc gctcacgcaa ctggtccaga accttgaccg aacgcagcgg tggtaacggc
2340
gcagtggcgg ttttcatggc ttgttatgac tgtttttttg tacagtctat gcctcgggca
2400
tccaagcagc aagcgcgtta cgccgtgggt cgatgtttga tgttatggag cagcaacgat
2460
gttacgcagc agcaacgatg ttacgcagca gggcagtcgc cctaaaacaa agttaggtgg
2520
ctcaagtatg ggcatcattc gcacatgtag gctcggccct gaccaagtca aatccatgcg
2580
ggctgctctt gatcttttcg gtcgtgagtt cggagacgta gccacctact cccaacatca
2640
gccggactcc gattacctcg ggaacttgct ccgtagtaag acattcatcg cgcttgctgc
2700
cttcgaccaa gaagcggttg ttggcgctct cgcggcttac gttctgccca ggtttgagca
2760
gccgcgtagt gagatctata tctatgatct cgcagtctcc ggcgagcacc ggaggcaggg
2820
cattgccacc gcgctcatca atctcctcaa gcatgaggcc aacgcgcttg gtgcttatgt
2880
gatctacgtg caagcagatt acggtgacga tcccgcagtg gctctctata caaagttggg
2940
catacgggaa gaagtgatgc actttgatat cgacccaagt accgccacct aacaattcgt
3000
tcaagccgag atcgtagaat ttcgacgacc tgcagccaag cataacttcg tataatgtat
3060
gctatacgaa cggtaggatc ctctagagtc gacctgcagg catgagatgt gtataagaga
3120
cag
3123
<210> 4
<211> 2965
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Экспрессионная Кассета
<400> 4
ctgtctctta tacacatctc cggccagatg attaattcct aatttttgtt gacactctat
60
cattgataga gttattttac cactccctat cagtgataga gaaaagtgaa atgaatagtt
120
cgacaaaaat ctagaaataa ttttgtttaa ctttaagaag gagatataca aatgaaagaa
180
atcaaaatcc agaacatcat catcagcgaa gaaaaagcgc cgctggttgt gccggaaatc
240
ggcattaacc ataatggtag tctggaactg gcaaaaatca tggtggatgc ggcctttagc
300
gccggtgcaa aaatcattaa acatcagacc cacattgtgg aagatgaaat gtctaaagca
360
gcgaaaaaag ttatcccggg caacgcgaaa atcagtatct acgaaatcat gcagaaatgc
420
gcgctggatt acaaagatga actggccctg aaagaatata ccgaaaaact gggtctggtg
480
tacctgtcta ccccgtttag tcgtgcgggt gcaaaccgtc tggaagatat gggtgttagt
540
gcgttcaaaa tcggcagcgg tgaatgtaac aattatccgc tgatcaaaca tattgccgca
600
tttaaaaaac cgatgattgt tagcaccggc atgaatagca tcgaatctat taaaccgacg
660
gtgaaaatcc tgctggataa cgaaattccg tttgttctga tgcataccac gaatctgtac
720
ccgaccccgc acaacctggt gcgtctgaat gccatgctgg aactgaaaaa agaattctct
780
tgcatggttg gtctgagtga tcacaccacg gataatctgg catgcctggg tgcagtggtt
840
ctgggtgcgt gtgtgctgga acgtcatttc accgatagca tgcaccgctc tggtccggat
900
attgtttgta gtatggatac gaaagcactg aaagaactga tcattcagag cgaacagatg
960
gcgatcattc gcggcaacaa tgaatctaaa aaagcggcca aacaggaaca ggtgaccatc
1020
gattttgcat tcgcgagtgt ggttagcatc aaagatatca aaaaaggcga agtgctgagc
1080
atggataata tttgggttaa acgtccgggt ctgggcggta tctctgcagc ggaatttgaa
1140
aacattctgg gcaaaaaagc actgcgcgat attgaaaatg atgcgcagct gtcttatgaa
1200
gatttcgcct aataaatcga tactagcata accccttggg gcctctaaac gcgtcgacac
1260
gcaaaaaggc catccgtcag gatggccttc tgcttaattt gatgcctggc agtttatggc
1320
gggcgtcctg cccgccaccc tccgggccgt tgcttcgcaa cgttcaaatc cgctcccggc
1380
ggatttgtcc tactcaggag agcgttcacc gacaaacaac agataaaacg aaaggcccag
1440
tctttcgact gagcctttcg ttttatttga tgcctggcag ttccctactc tcgcatgggg
1500
agaccccaca ctaccatccg gtatcgataa gcttgatggc gaaaggggga tgtgctgcaa
1560
ggcgattaag ttgggtaacg ccagggtttt cccagtcacg acgttgtaaa acgacggcca
1620
gtgaattcga gctcggtacc taccgttcgt ataatgtatg ctatacgaag ttatcgagct
1680
ctagagaatg atcccctccc tcacgctgcc gcaagcactc agggcgcaag ggctgctaaa
1740
ggaagcggaa cacgtagaaa gccagtccgc agaaacggtg ctgaccccgg atgaatgtca
1800
gctactgggc tatctggaca agggaaaacg caagcgcaaa gagaaagcag gtagcttgca
1860
gtgggcttac atggcgatag ctagactggg cggttttatg gacagcaagc gaaccggaat
1920
tgccagctgg ggcgccctct ggtaaggttg ggaagccctg caaagtaaac tggatggctt
1980
tcttgccgcc aaggatctga tggcgcaggg gatcaagatc tgatcaagag acaggatgag
2040
gatcgtttcg catgattgaa caagatggat tgcacgcagg ttctccggcc gcttgggtgg
2100
agaggctatt cggctatgac tgggcacaac agacaatcgg ctgctctgat gccgccgtgt
2160
tccggctgtc agcgcagggg cgcccggttc tttttgtcaa gaccgacctg tccggtgccc
2220
tgaatgaact gcaggacgag gcagcgcggc tatcgtggct ggccacgacg ggcgttcctt
2280
gcgcagctgt gctcgacgtt gtcactgaag cgggaaggga ctggctgcta ttgggcgaag
2340
tgccggggca ggatctcctg tcatctcacc ttgctcctgc cgagaaagta tccatcatgg
2400
ctgatgcaat gcggcggctg catacgcttg atccggctac ctgcccattc gaccaccaag
2460
cgaaacatcg catcgagcga gcacgtactc ggatggaagc cggtcttgtc gatcaggatg
2520
atctggacga agagcatcag gggctcgcgc cagccgaact gttcgccagg ctcaaggcgc
2580
gcatgcccga cggcgaggat ctcgtcgtga cccatggcga tgcctgcttg ccgaatatca
2640
tggtggaaaa tggccgcttt tctggattca tcgactgtgg ccggctgggt gtggcggacc
2700
gctatcagga catagcgttg gctacccgtg atattgctga agagcttggc ggcgaatggg
2760
ctgaccgctt cctcgtgctt tacggtatcg ccgctcccga ttcgcagcgc atcgccttct
2820
atcgccttct tgacgagttc ttctgagcgg gactctggga atttcgacga cctgcagcca
2880
agcataactt cgtataatgt atgctatacg aacggtagga tcctctagag tcgacctgca
2940
ggcatgagat gtgtataaga gacag
2965
<210> 5
<211> 3904
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Экспрессионная Кассета
<400> 5
ctgtctctta tacacatctc cggccagatg attaattcct aatttttgtt gacactctat
60
cattgataga gttattttac cactccctat cagtgataga gaaaagtgaa atgaatagtt
120
cgacaaaaat ctagaaataa ttttgtttaa ctttaagaag gagatataca aatgatcgct
180
caccgtcgtc aggaactggc tcaacagtat tatcaggctc tgcaccaaga tgtgctgccg
240
ttctgggaaa agtattcgct ggatcgtcaa ggcggtggct attttacctg cctggaccgc
300
aagggtcagg tttttgatac ggacaagttc atttggctgc aaaaccgtca agtgtggcaa
360
tttgcggttt tctacaatcg cctggaaccg aaaccgcagt ggctggaaat cgctcgtcat
420
ggtgcggatt ttctggcacg tcacggtcgt gatcaggacg gtaactggta tttcgccctg
480
gatcaggaag gcaaaccgct gcgccaaccg tacaatgtgt tttccgactg tttcgcggcg
540
atggcgttta gccagtatgc actggcttct ggtgctcaag aagcgaaggc cattgcactg
600
caagcgtata acaatgttct gcgtcgccag cataacccga aaggtcaata tgaaaagagt
660
tacccgggta cccgtccgct gaaatccctg gcagtgccga tgatcctggc taatctgacg
720
ctggaaatgg aatggctgct gccgccgacc acggtcgaag aagtgctggc ccagaccgtt
780
cgtgaagtca tgacggattt tctggacccg gaaattggcc tgatgcgcga agcagttacc
840
ccgacgggtg aatttgtcga ttcattcgaa ggccgcctgc tgaacccggg tcatggcatt
900
gaagcgatgt ggtttatgat ggatattgcc cagcgttcgg gtgaccgcca gctgcaagaa
960
caggctattg cggtggttct gaataccctg gaatatgcat gggatgaaga atttggtggc
1020
atcttttact tcctggaccg tcaaggtcac ccgccgcagc aactggaatg ggatcagaaa
1080
ctgtggtggg tccatctgga aaccctggtg gccctggcaa aaggtcacca ggcgacgggc
1140
caagaaaagt gctggcagtg gtttgaacgc gtgcatgatt atgcatggag ccactttgct
1200
gacccggaat atggtgaatg gttcggctac ctgaaccgtc gcggtgaagt gctgctgaat
1260
ctgaaaggtg gcaaatggaa gggctgcttc cacgttccgc gtgcgctgtg gctgtgtgcc
1320
gaaaccctgc aactgccggt ctcttaattt cgtcgacaca caggaaacat attaaaaatt
1380
aaaacctgca ggagtttaaa cgcggccgcg atatcgttgt aaaacgacgg ccagtgcaag
1440
aatcataaaa aatttatttg ctttcaggaa aatttttctg tataatagat tcataaattt
1500
gagagaggag tttttgtgag cggataacaa ttccccatct tagtatatta gttaagtata
1560
aatacacaag gagatataca tatgaaagaa atcaaaatcc agaacatcat catcagcgaa
1620
gaaaaagcgc cgctggttgt gccggaaatc ggcattaacc ataatggtag tctggaactg
1680
gcaaaaatca tggtggatgc ggcctttagc gccggtgcaa aaatcattaa acatcagacc
1740
cacattgtgg aagatgaaat gtctaaagca gcgaaaaaag ttatcccggg caacgcgaaa
1800
atcagtatct acgaaatcat gcagaaatgc gcgctggatt acaaagatga actggccctg
1860
aaagaatata ccgaaaaact gggtctggtg tacctgtcta ccccgtttag tcgtgcgggt
1920
gcaaaccgtc tggaagatat gggtgttagt gcgttcaaaa tcggcagcgg tgaatgtaac
1980
aattatccgc tgatcaaaca tattgccgca tttaaaaaac cgatgattgt tagcaccggc
2040
atgaatagca tcgaatctat taaaccgacg gtgaaaatcc tgctggataa cgaaattccg
2100
tttgttctga tgcataccac gaatctgtac ccgaccccgc acaacctggt gcgtctgaat
2160
gccatgctgg aactgaaaaa agaattctct tgcatggttg gtctgagtga tcacaccacg
2220
gataatctgg catgcctggg tgcagtggtt ctgggtgcgt gtgtgctgga acgtcatttc
2280
accgatagca tgcaccgctc tggtccggat attgtttgta gtatggatac gaaagcactg
2340
aaagaactga tcattcagag cgaacagatg gcgatcattc gcggcaacaa tgaatctaaa
2400
aaagcggcca aacaggaaca ggtgaccatc gattttgcat tcgcgagtgt ggttagcatc
2460
aaagatatca aaaaaggcga agtgctgagc atggataata tttgggttaa acgtccgggt
2520
ctgggcggta tctctgcagc ggaatttgaa aacattctgg gcaaaaaagc actgcgcgat
2580
attgaaaatg atgcgcagct gtcttatgaa gatttcgcct aaaataacta gcataacccc
2640
ttggggcctc taaacgggtc ttgaggggtt ttttgctgaa accaatttgc ctggcggcag
2700
tagcgcggtg gtcccacctg accccatgcc gaactcagaa gtgaaacgcc gtagcgccga
2760
tggtagtgtg gggtctcccc atgcgagagt agggaactgc caggcatcaa ataaaacgaa
2820
aggctcagtc gaaagactgg gcctttcggg atccaggccg gcctgttaac gaattaatct
2880
tccgcggcgg tatcgataag cttgatatcg aattccgaag ttcctattct ctagaaagta
2940
taggaacttc aggtctgaag aggagtttac gtccagccaa gctagcttgg ctgcaggtcg
3000
tcgaaattct accgggtagg ggaggcgctt ttcccaaggc agtctggagc atgcgcttta
3060
gcagccccgc tgggcacttg gcgctacaca agtggcctct ggcctcgcac acattccaca
3120
tccaccggta ggcgccaacc ggctccgttc tttggtggcc ccttcgcgcc accttctact
3180
cctcccctag tcaggaagtt cccccccgcc ccgcagctcg cgtcgtgcag gacgtgacaa
3240
atggaagtag cacgtctcac tagtctcgtg cagatggaca gcaccgctga gcaatggaag
3300
cgggtaggcc tttggggcag cggccaatag cagctttgct ccttcgcttt ctgggctcag
3360
gggcgggctc agggggcggg gcgggcgccc gaaggtcctc cggaggcccg gcattctgca
3420
cgcttcaaaa gcgcacgtct gccgcgctgt tctcctcttc ctcatctccg ggcctttcga
3480
cctgcagcct gttgacaatt aatcatcggc atagtatatc ggcatagtat aatacgacaa
3540
ggtgaggaac taaaccatgg gtcaaagtag cgatgaagcc aacgctcccg ttgcagggca
3600
gtttgcgctt cccctgagtg ccacctttgg cttaggggat cgcgtacgca agaaatctgg
3660
tgccgcttgg cagggtcaag tcgtcggttg gtattgcaca aaactcactc ctgaaggcta
3720
tgcggtcgag tccgaatccc acccaggctc agtgcaaatt tatcctgtgg ctgcacttga
3780
acgtgtggcc taatgagggg atcaattctc tagagctcgc tgatcagaag ttcctattct
3840
ctagaaagta taggaacttc gatggcgcct catccctgaa gccaaagatg tgtataagag
3900
acag
3904
<210> 6
<211> 3793
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Экспрессионная Кассета
<400> 6
ctgtctctta tacacatctc cggccagatg attaattcct aatttttgtt gacactctat
60
cattgataga gttattttac cactccctat cagtgataga gaaaagtgaa atgaatagtt
120
cgacaaaaat ctagaaataa ttttgtttgg cgtcgagaag gagatagaaa atgtgcggta
180
tcgttggtgc tatcgcacag cgtgatgtag cgaaaatcct cctggaaggt ctgcgtcgtc
240
tcgaataccg tggttacgac tctgccggtc tggcagtagt ggatgcagaa ggtcacatga
300
ctcgtctgcg tcgtctgggt aaagtgcaga tgctcgcgca ggcggcggaa gaacacccac
360
tccacggtgg tacgggtatc gcacacactc gttgggcaac ccacggtgaa ccgtctgagg
420
tcaacgcaca cccgcatgtt agcgagcaca tcgtagtcgt tcacaacggt atcatcgaga
480
accacgaacc actccgtgag gaactcaaag cccgtggtta caccttcgta agcgaaaccg
540
acacggaagt tatcgcccac ctcgttaact gggaactcaa acagggtggt actctgcgtg
600
aagcagttct gcgtgccatt ccacagctgc gtggtgcata cggtaccgtg atcatggact
660
ctcgtcatcc ggataccctg ctcgccgcac gttctggttc tccactcgtt atcggtctgg
720
gtatgggtga gaacttcatc gcctctgatc agctggccct gctcccagtt acccgtcgct
780
tcatcttcct ggaagagggt gacatcgccg aaatcacccg tcgttccgtt aacatcttcg
840
acaaaacggg tgcggaagtt aaacgtcagg acatcgagtc taacctgcag tatgacgctg
900
gtgacaaagg catctaccgt cactacatgc agaaagagat ctacgaacag ccgaacgcga
960
tcaaaaacac cctgaccggt cgtatctctc acggtcaggt tgacctgtct gagctgggtc
1020
caaacgcgga cgaactcctg tccaaagtcg agcacatcca gatcctggct tgtggtacct
1080
cttacaactc cggtatggtt tctcgttact ggttcgaatc tctggcaggt atcccatgcg
1140
acgttgaaat cgcctccgaa ttccgttatc gtaaatctgc ggtacgtcgt aactccctca
1200
tgatcaccct gtctcagtct ggtgaaaccg ctgatactct ggcaggtctg cgtctcagca
1260
aagaactggg ttacctgggt tctctggcca tctgcaacgt tccgggttct agcctggttc
1320
gtgagtctgt gctggctctg atgaccaacg cgggtacgga gatcggtgtt gcctctacca
1380
aagcgttcac tacccagctc actgtcctgc tgatgctggt tgccaaactg tctcgtctca
1440
aaggcctcga cgctagcatc gaacacgaca tcgtacacgg tctgcaggcc ctcccatctc
1500
gtatcgagca gatgctgccg caggacaaac gtatcgaagc actggcagaa gacttcagcg
1560
acaaacacca cgcgctgttt ctgggtcgtg gtgaccagta cccaattgcg ctggaaggtg
1620
ccctgaaact gaaagagatc agctacatcc atgcagaggc atacgcagcg ggtgagctga
1680
aacatggtcc actggccctg atcgacgcag atatgccggt tattgtggtt gctccgaaca
1740
acggcctgct ggagaaactg aaatccaaca tcgaggaagt acgtgcgcgt ggtggtcagc
1800
tgtacgtgtt tgctgaccag gacgcgggtt tcgtttccag cgacaacatg cacatcatcg
1860
aaatgccgca tgttgaagag gtaatcgcgc caatcttcta caccgtaccg ctgcagctgc
1920
tggcgtacca tgtagccctg atcaaaggta cggacgttga ccagccgcgt aacctggcga
1980
aatccgtgac cgtggaataa cgaaggagat agaaccatga gcttacccga tggattttat
2040
ataaggcgaa tggaagaggg ggatttggaa caggtcactg agacgctaaa ggttttgacc
2100
accgtgggca ctattacccc cgaatccttc agcaaactca taaaatactg gaatgaagcc
2160
acagtatgga atgataacga agataaaaaa ataatgcaat ataaccccat ggtgattgtg
2220
gacaagcgca ccgagacggt tgccgctacg gggaatatca tcatcgaaag aaagatcatt
2280
catgaactgg ggctatgtgg ccacatcgag gacattgcag taaactccaa gtatcagggc
2340
caaggtttgg gcaagctctt gattgatcaa ttggtaacta tcggctttga ctacggttgt
2400
tataagatta ttttagattg cgatgagaaa aatgtcaaat tctatgaaaa atgtgggttt
2460
agcaacgcag gcgtggaaat gcaaattaga aaatagcatc cgtatcggaa acactagcat
2520
aaccccttgg ggcctctaaa cgggtcttga ggggtttttt gctgaaacca atttgcctgg
2580
cggcagtagc gcggtggtcc cacctgaccc catgccgaac tcagaagtga aacgccgtag
2640
cgccgatggt agtgtggggt ctccccatgc gagagtaggg aactgccagg catcaaataa
2700
aacgaaaggc tcagtcgaaa gactgggcct ttcgcttcca caactttgta taataaagtt
2760
gtccccacgg ccagtgaatt cgagctcggt acctaccgtt cgtataatgt atgctatacg
2820
aagttatcga gctctagaga atgatcccct cattaggcca cacgttcaag tgcagcgcac
2880
accgtggaaa cggatgaagg cacgaaccca gttgacataa gcctgttcgg ttcgtaaact
2940
gtaatgcaag tagcgtatgc gctcacgcaa ctggtccaga accttgaccg aacgcagcgg
3000
tggtaacggc gcagtggcgg ttttcatggc ttgttatgac tgtttttttg tacagtctat
3060
gcctcgggca tccaagcagc aagcgcgtta cgccgtgggt cgatgtttga tgttatggag
3120
cagcaacgat gttacgcagc agcaacgatg ttacgcagca gggcagtcgc cctaaaacaa
3180
agttaggtgg ctcaagtatg ggcatcattc gcacatgtag gctcggccct gaccaagtca
3240
aatccatgcg ggctgctctt gatcttttcg gtcgtgagtt cggagacgta gccacctact
3300
cccaacatca gccggactcc gattacctcg ggaacttgct ccgtagtaag acattcatcg
3360
cgcttgctgc cttcgaccaa gaagcggttg ttggcgctct cgcggcttac gttctgccca
3420
ggtttgagca gccgcgtagt gagatctata tctatgatct cgcagtctcc ggcgagcacc
3480
ggaggcaggg cattgccacc gcgctcatca atctcctcaa gcatgaggcc aacgcgcttg
3540
gtgcttatgt gatctacgtg caagcagatt acggtgacga tcccgcagtg gctctctata
3600
caaagttggg catacgggaa gaagtgatgc actttgatat cgacccaagt accgccacct
3660
aacaattcgt tcaagccgag atcgtagaat ttcgacgacc tgcagccaag cataacttcg
3720
tataatgtat gctatacgaa cggtaggatc ctctagagtc gacctgcagg catgagatgt
3780
gtataagaga cag
3793
<210> 7
<211> 3847
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Экспрессионная Кассета
<400> 7
ctgtctctta tacacatctc cggccagatg attaattcct aatttttgtt gacactctat
60
cattgataga gttattttac cactccctat cagtgataga gaaaagtgaa atgaatagtt
120
cgacaaaaat ctagaaataa ttttgtttgg cgtcgagaag gagatagaac catgtccaac
180
aatggctcgt caccgctggt gctttggtat aaccaactcg gcatgaatga tgtagacagg
240
gttgggggca aaaatgcctc cctgggtgaa atgattacta acctttccgg aatgggtgtt
300
tccgttccga atggtttcgc cacaaccgcc gacgcgttta accagtttct ggaccaaagc
360
ggcgtaaacc agcgcattta tgaactgctg gataaaacgg atattgacga tgttactcag
420
cttgcgaaag cgggcgcgca aatccgccag tggattatcg acactccctt ccagcctgag
480
ctggaaaacg ccatcagcga agcctatgca cagctttctg ccgatgacga aaacgcctct
540
tttgcggtgc gctcctccgc caccgcagaa gatatgccgg acgcttcttt tgccggtcag
600
caggaaacct tcctcaacgt tcagggtttt gacgccgttc tcgtggcagt gaaacatgta
660
tttgcttctc tgtttaacga tcgcgccatc tcttatcgtg tgcaccaggg ttacgatcac
720
cgtggtgtgg cgctctccgc cggtgttcaa cggatggtgc gctctgacct cgcatcatct
780
ggcgtgatgt tctccattga taccgaatcc ggctttgacc aggtggtgtt tatcacttcc
840
gcatggggcc ttggtgagat ggtcgtgcag ggtgcggtta acccggatga gttttacgtg
900
cataaaccga cactggcggc gaatcgcccg gctatcgtgc gccgcaccat ggggtcgaaa
960
aaaatccgca tggtttacgc gccgacccag gagcacggca agcaggttaa aatcgaagac
1020
gtaccgcagg aacagcgtga catcttctcg ctgaccaacg aagaagtgca ggaactggca
1080
aaacaggccg tacaaattga gaaacactac ggtcgcccga tggatattga gtgggcgaaa
1140
gatggccaca ccggtaaact gttcattgtg caggcgcgtc cggaaaccgt gcgctcacgc
1200
ggtcaggtca tggagcgtta tacgctgcat tcacagggta agattatcgc cgaaggccgt
1260
gctatcggtc atcgcatcgg tgcgggtccg gtgaaagtca tccatgatat cagcgaaatg
1320
aaccgcatcg aacctggtga cgtgctggtc actgacatga ccgacccgga ctgggaaccg
1380
atcatgaaga aagcatctgc catcgtcacc aaccgtggcg gtcgtacctg tcacgcggcg
1440
atcatcgctc gtgaactggg cattccggcg gtagtgggct gtggtgatgc aacagaacgg
1500
atgaaagacg gtgagaacgt cactgtttct tgtgccgaag gtgataccgg ttacgtctat
1560
gcggagttgc tggaatttag cgtgaaaagc tccagcgtag aaacgatgcc ggatctgccg
1620
ttgaaagtga tgatgaacgt cggtaacccg gaccgagctt tcgacttcgc ctgtctgccg
1680
aacgaaggcg tgggacttgc gcgtctggaa tttatcatca accgtatgat tggcgtccac
1740
ccacgcgcac tgcttgagtt tgacgatcag gaaccgcagt tgcaaaacga aatccgcgag
1800
atgatgaaag gttttgattc tccgcgtgaa ttttacgttg gtcgtctgac tgaagggatc
1860
gcgacgctgg gtgccgcgtt ttatccgaag cgcgtcattg tccgtctctc tgattttaaa
1920
tcgaacgaat atgccaacct ggtcggtggt gagcgttacg agccagatga agagaacccg
1980
atgctcggct tccgtggcgc gggacgctat atttccgaca gcttccgcga ctgtttcgcg
2040
ctggagtgcg aagcagtgaa acgtgtgcgc aacgacatgg ggctgaccaa cgttgagatc
2100
atgatcccgt tcgtgcgaac cgtagatcag gcgaaagcgg tggttgagga actggcgcgt
2160
caggggctga aacgtggtga gaacgggctg aaaatcatca tgatgtgtga aatcccgtcc
2220
aacgccttgc tggccgagca gttcctcgaa tatttcgacg gcttctcaat tggctcaaac
2280
gacatgacgc agctggcgct cggtctggat cgtgactccg gcgtggtgtc tgaactgttc
2340
gatgagcgca acgatgcggt gaaagcactg ctgtcgatgg cgattcgtgc cgcgaagaaa
2400
cagggcaaat atgtcgggat ttgcggtcag ggtccgtccg accacgaaga ctttgccgca
2460
tggttgatgg aagaggggat cgatagcctg tctctgaacc cggacaccgt ggtgcaaacc
2520
tggttaagcc tggctgaact gaagaaataa catccgtatc ggaaacacta gcataacccc
2580
ttggggcctc taaacgggtc ttgaggggtt ttttgctgaa accaatttgc ctggcggcag
2640
tagcgcggtg gtcccacctg accccatgcc gaactcagaa gtgaaacgcc gtagcgccga
2700
tggtagtgtg gggtctcccc atgcgagagt agggaactgc caggcatcaa ataaaacgaa
2760
aggctcagtc gaaagactgg gcctttcgct tccacaactt tgtataataa agttgtcccc
2820
acggccagtg aattcgagct cggtacctac cgttcgtata atgtatgcta tacgaagtta
2880
tcgagctcta gagaatgatc ccctcattag gccacacgtt caagtgcagc gcacaccgtg
2940
gaaacggatg aaggcacgaa cccagttgac ataagcctgt tcggttcgta aactgtaatg
3000
caagtagcgt atgcgctcac gcaactggtc cagaaccttg accgaacgca gcggtggtaa
3060
cggcgcagtg gcggttttca tggcttgtta tgactgtttt tttgtacagt ctatgcctcg
3120
ggcatccaag cagcaagcgc gttacgccgt gggtcgatgt ttgatgttat ggagcagcaa
3180
cgatgttacg cagcagcaac gatgttacgc agcagggcag tcgccctaaa acaaagttag
3240
gtggctcaag tatgggcatc attcgcacat gtaggctcgg ccctgaccaa gtcaaatcca
3300
tgcgggctgc tcttgatctt ttcggtcgtg agttcggaga cgtagccacc tactcccaac
3360
atcagccgga ctccgattac ctcgggaact tgctccgtag taagacattc atcgcgcttg
3420
ctgccttcga ccaagaagcg gttgttggcg ctctcgcggc ttacgttctg cccaggtttg
3480
agcagccgcg tagtgagatc tatatctatg atctcgcagt ctccggcgag caccggaggc
3540
agggcattgc caccgcgctc atcaatctcc tcaagcatga ggccaacgcg cttggtgctt
3600
atgtgatcta cgtgcaagca gattacggtg acgatcccgc agtggctctc tatacaaagt
3660
tgggcatacg ggaagaagtg atgcactttg atatcgaccc aagtaccgcc acctaacaat
3720
tcgttcaagc cgagatcgta gaatttcgac gacctgcagc caagcataac ttcgtataat
3780
gtatgctata cgaacggtag gatcctctag agtcgacctg caggcatgag atgtgtataa
3840
gagacag
3847
<210> 8
<211> 5554
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Плазмида
<400> 8
catcgattta ttatgacaac ttgacggcta catcattcac tttttcttca caaccggcac
60
ggaactcgct cgggctggcc ccggtgcatt ttttaaatac ccgcgagaaa tagagttgat
120
cgtcaaaacc aacattgcga ccgacggtgg cgataggcat ccgggtggtg ctcaaaagca
180
gcttcgcctg gctgatacgt tggtcctcgc gccagcttaa gacgctaatc cctaactgct
240
ggcggaaaag atgtgacaga cgcgacggcg acaagcaaac atgctgtgcg acgctggcga
300
tatcaaaatt gctgtctgcc aggtgatcgc tgatgtactg acaagcctcg cgtacccgat
360
tatccatcgg tggatggagc gactcgttaa tcgcttccat gcgccgcagt aacaattgct
420
caagcagatt tatcgccagc agctccgaat agcgcccttc cccttgcccg gcgttaatga
480
tttgcccaaa caggtcgctg aaatgcggct ggtgcgcttc atccgggcga aagaaccccg
540
tattggcaaa tattgacggc cagttaagcc attcatgcca gtaggcgcgc ggacgaaagt
600
aaacccactg gtgataccat tcgcgagcct ccggatgacg accgtagtga tgaatctctc
660
ctggcgggaa cagcaaaata tcacccggtc ggcaaacaaa ttctcgtccc tgatttttca
720
ccaccccctg accgcgaatg gtgagattga gaatataacc tttcattccc agcggtcggt
780
cgataaaaaa atcgagataa ccgttggcct caatcggcgt taaacccgcc accagatggg
840
cattaaacga gtatcccggc agcaggggat cattttgcgc ttcagccata cttttcatac
900
tcccgccatt cagagaagaa accaattgtc catattgcat cagacattgc cgtcactgcg
960
tcttttactg gctcttctcg ctaaccaaac cggtaacccc gcttattaaa agcattctgt
1020
aacaaagcgg gaccaaagcc atgacaaaaa cgcgtaacaa aagtgtctat aatcacggca
1080
gaaaagtcca cattgattat ttgcacggcg tcacactttg ctatgccata gcatttttat
1140
ccataagatt agcggatcct acctgacgct ttttatcgca actctctact gtttctccat
1200
acccgttttt ttgggaattc gagctctaag gaggttataa aaaatgtcta atctgctgac
1260
ggtccaccaa aacctgccgg ctctgccggt cgatgctacc tctgatgaag ttcgcaaaaa
1320
cctgatggat atgtttcgtg atcgccaggc attcagcgaa catacctgga aaatgctgct
1380
gtccgtgtgc cgttcatggg cggcctggtg taaactgaac aatcgcaaat ggtttccggc
1440
ggaaccggaa gatgtccgtg actatctgct gtacctgcag gcccgcggtc tggcagttaa
1500
aacgatccag caacatctgg gccaactgaa tatgctgcac cgtcgctccg gtctgccgcg
1560
tccgagcgat tctaatgcgg tgtcactggt tatgcgtcgc attcgtaaag aaaacgtgga
1620
tgcaggcgaa cgcgctaaac aggcactggc ttttgaacgt accgatttcg accaagttcg
1680
ctcgctgatg gaaaacagcg atcgttgcca ggacatccgc aatctggcgt tcctgggtat
1740
tgcctataac accctgctgc gcattgcaga aatcgctcgt attcgcgtga aagatatcag
1800
ccgtacggac ggcggtcgca tgctgattca catcggccgt accaaaacgc tggtctctac
1860
cgcaggcgtg gaaaaagctc tgagtctggg tgtgacgaaa ctggttgaac gctggattag
1920
tgtctccggc gtggcggatg acccgaacaa ttacctgttt tgtcgtgttc gcaaaaatgg
1980
tgtcgcagct ccgtcagcca cctcgcagct gagcacgcgt gcactggaag gcatcttcga
2040
agctacccat cgcctgattt atggcgccaa agatgactcg ggtcaacgtt acctggcgtg
2100
gtctggtcac agtgcacgtg ttggtgccgc acgtgatatg gcccgtgccg gtgtttccat
2160
cccggaaatt atgcaggcag gcggttggac caacgttaat atcgtcatga actatattcg
2220
caatctggac tcggaaacgg gtgctatggt tcgcctgctg gaagacggtg actaatgagt
2280
gccggagttc atcgaaaaaa tggacgaggc actggctgaa attggttttg tatttgggga
2340
gcaatggcga tgacgcatcc tcacgataat atccgggtag gcgcaatcac tttcgtctac
2400
tccgttacaa agcgaggctg ggtatttccc ggcctttctg ttatccgaaa tccactgaaa
2460
gcacagcggc tggctgagga gataaataat aaacgagggg ctgtatgcac aaagcatctt
2520
ctgttgagtt aagaacgagt atcgagatgg cacatagcct tgctcaaatt ggaatcaggt
2580
ttgtgccaat accagtagaa acagacgaag aatccatggg tatggacagt tttccctttg
2640
atatgtaacg gtgaacagtt gttctacttt tgtttgttag tcttgatgct tcactgatag
2700
atacaagagc cataagaacc tcagatcctt ccgtatttag ccagtatgtt ctctagtgtg
2760
gttcgttgtt tttgcgtgag ccatgagaac gaaccattga gatcatactt actttgcatg
2820
tcactcaaaa attttgcctc aaaactggtg agctgaattt ttgcagttaa agcatcgtgt
2880
agtgtttttc ttagtccgtt acgtaggtag gaatctgatg taatggttgt tggtattttg
2940
tcaccattca tttttatctg gttgttctca agttcggtta cgagatccat ttgtctatct
3000
agttcaactt ggaaaatcaa cgtatcagtc gggcggcctc gcttatcaac caccaatttc
3060
atattgctgt aagtgtttaa atctttactt attggtttca aaacccattg gttaagcctt
3120
ttaaactcat ggtagttatt ttcaagcatt aacatgaact taaattcatc aaggctaatc
3180
tctatatttg ccttgtgagt tttcttttgt gttagttctt ttaataacca ctcataaatc
3240
ctcatagagt atttgttttc aaaagactta acatgttcca gattatattt tatgaatttt
3300
tttaactgga aaagataagg caatatctct tcactaaaaa ctaattctaa tttttcgctt
3360
gagaacttgg catagtttgt ccactggaaa atctcaaagc ctttaaccaa aggattcctg
3420
atttccacag ttctcgtcat cagctctctg gttgctttag ctaatacacc ataagcattt
3480
tccctactga tgttcatcat ctgagcgtat tggttataag tgaacgatac cgtccgttct
3540
ttccttgtag ggttttcaat cgtggggttg agtagtgcca cacagcataa aattagcttg
3600
gtttcatgct ccgttaagtc atagcgacta atcgctagtt catttgcttt gaaaacaact
3660
aattcagaca tacatctcaa ttggtctagg tgattttaat cactatacca attgagatgg
3720
gctagtcaat gataattact agtccttttc ctttgagttg tgggtatctg taaattctgc
3780
tagacctttg ctggaaaact tgtaaattct gctagaccct ctgtaaattc cgctagacct
3840
ttgtgtgttt tttttgttta tattcaagtg gttataattt atagaataaa gaaagaataa
3900
aaaaagataa aaagaataga tcccagccct gtgtataact cactacttta gtcagttccg
3960
cagtattaca aaaggatgtc gcaaacgctg tttgctcctc tacaaaacag accttaaaac
4020
cctaaaggct taagtagcac cctcgcaagc tcggttgcgg ccgcaatcgg gcaaatcgct
4080
gaatattcct tttgtctccg accatcaggc acctgagtcg ctgtcttttt cgtgacattc
4140
agttcgctgc gctcacggct ctggcagtga atgggggtaa atggcactac aggcgccttt
4200
tatggattca tgcaaggaaa ctacccataa tacaagaaaa gcccgtcacg ggcttctcag
4260
ggcgttttat ggcgggtctg ctatgtggtg ctatctgact ttttgctgtt cagcagttcc
4320
tgccctctga ttttccagtc tgaccacttc ggattatccc gtgacaggtc attcagactg
4380
gctaatgcac ccagtaaggc agcggtatca tcaacggggt ctgacgctca gtggaacgaa
4440
aactcacgtt aagggatttt ggtcatgaga ttatcaaaaa ggatcttcac ctagatcctt
4500
ttaaattaaa aatgaagttt taaatcaatc taaagtatat atgagtaaac ttggtctgac
4560
agttaccaat gcttaatcag tgaggcacct atctcagcga tctgtctatt tcgttcatcc
4620
atagttgcct gactccccgt cgtgtagata actacgatac gggagggctt accatctggc
4680
cccagtgctg caatgatacc gcgagaccca cgctcaccgg ctccagattt atcagcaata
4740
aaccagccag ccggaagggc cgagcgcaga agtggtcctg caactttatc cgcctccatc
4800
cagtctatta attgttgccg ggaagctaga gtaagtagtt cgccagttaa tagtttgcgc
4860
aacgttgttg ccattgctac aggcatcgtg gtgtcacgct cgtcgtttgg tatggcttca
4920
ttcagctccg gttcccaacg atcaaggcga gttacatgat cccccatgtt gtgcaaaaaa
4980
gcggttagct ccttcggtcc tccgatcgtt gtcagaagta agttggccgc agtgttatca
5040
ctcatggtta tggcagcact gcataattct cttactgtca tgccatccgt aagatgcttt
5100
tctgtgactg gtgagtactc aaccaagtca ttctgagaat agtgtatgcg gcgaccgagt
5160
tgctcttgcc cggcgtcaat acgggataat accgcgccac atagcagaac tttaaaagtg
5220
ctcatcattg gaaaacgttc ttcggggcga aaactctcaa ggatcttacc gctgttgaga
5280
tccagttcga tgtaacccac tcgtgcaccc aactgatctt cagcatcttt tactttcacc
5340
agcgtttctg ggtgagcaaa aacaggaagg caaaatgccg caaaaaaggg aataagggcg
5400
acacggaaat gttgaatact catactcttc ctttttcaat attattgaag catttatcag
5460
ggttattgtc tcatgagcgg atacatattt gaatgtattt agaaaaataa acaaataggg
5520
gttccgcgca catttccccg aaaagtgcca cctg
5554
<210> 9
<211> 3436
<212> ДНК
<213> Искусственная Последовательность
<220>
<223> Экспрессионная Кассета
<400> 9
ctgtctctta tacacatctc cggccagatg attaattcct aatttttgtt gacactctat
60
cattgataga gttattttac cactccctat cagtgataga gaaaagtgaa atgaatagtt
120
cgacaaaaat ctagaaataa ttttgtttaa ctttaagaag gagatataca aatgaagacc
180
gtgctggaca ccctgaaagg tcgcctggtt gtgagctgcc aagcgctgga aaatgagccg
240
ctgcatagcc cgtttattat gagccgtatg gcgctggcgg cgcgtcaggg tggtgcggcg
300
gcgatccgtg cgaacagcgt ggttgatatc gaggcgatta aggaacaagt taccctgccg
360
gtgatcggca tcattaagcg tgagtacccg gatagcgaag ttttcattac cgcgaccatg
420
aaagaggtgg acgaactgat gaccgtgagc ccggcgatca ttgcgctgga tgcgaccgac
480
cgtgcgcgtc cgggtggcga gagcctggcg atgctggtta cccgtatccg tacccgttat
540
ccgagcgtgc tgctgatggc ggatattgcg accgttgacg aagcggtgac cgcgcaggcg
600
ctgggtttcg attgcgttgg caccaccctg tacggttata ccgcgcagac cgtgggtcat
660
gcgctgccgg acgatgactg ccaatttctg aaagcggttc tggcggcggt taccgtgccg
720
gtggttgcgg aaggcaacgt ggacaccccg gaacgtgcgg cgcgttgcct ggcgctgggt
780
gcgcacatgg tggttgtggg tggcgcgatt acccgtccgc aacagattac cgaacgcttc
840
atggcggcga ttgatgcgca gagcaccgac cgtgcgtaat ttcgtcgaca cacaggaaac
900
atattaaaaa ttaaaacctg caggagttta aacgcggccg cgatatcgtt gtaaaacgac
960
ggccagtgca agaatcataa aaaatttatt tgctttcagg aaaatttttc tgtataatag
1020
attcataaat ttgagagagg agtttttgtg agcggataac aattccccat cttagtatat
1080
tagttaagta taaatacaca aggagatata catatgaaag aaatcaaaat ccagaacatc
1140
atcatcagcg aagaaaaagc gccgctggtt gtgccggaaa tcggcattaa ccataatggt
1200
agtctggaac tggcaaaaat catggtggat gcggccttta gcgccggtgc aaaaatcatt
1260
aaacatcaga cccacattgt ggaagatgaa atgtctaaag cagcgaaaaa agttatcccg
1320
ggcaacgcga aaatcagtat ctacgaaatc atgcagaaat gcgcgctgga ttacaaagat
1380
gaactggccc tgaaagaata taccgaaaaa ctgggtctgg tgtacctgtc taccccgttt
1440
agtcgtgcgg gtgcaaaccg tctggaagat atgggtgtta gtgcgttcaa aatcggcagc
1500
ggtgaatgta acaattatcc gctgatcaaa catattgccg catttaaaaa accgatgatt
1560
gttagcaccg gcatgaatag catcgaatct attaaaccga cggtgaaaat cctgctggat
1620
aacgaaattc cgtttgttct gatgcatacc acgaatctgt acccgacccc gcacaacctg
1680
gtgcgtctga atgccatgct ggaactgaaa aaagaattct cttgcatggt tggtctgagt
1740
gatcacacca cggataatct ggcatgcctg ggtgcagtgg ttctgggtgc gtgtgtgctg
1800
gaacgtcatt tcaccgatag catgcaccgc tctggtccgg atattgtttg tagtatggat
1860
acgaaagcac tgaaagaact gatcattcag agcgaacaga tggcgatcat tcgcggcaac
1920
aatgaatcta aaaaagcggc caaacaggaa caggtgacca tcgattttgc attcgcgagt
1980
gtggttagca tcaaagatat caaaaaaggc gaagtgctga gcatggataa tatttgggtt
2040
aaacgtccgg gtctgggcgg tatctctgca gcggaatttg aaaacattct gggcaaaaaa
2100
gcactgcgcg atattgaaaa tgatgcgcag ctgtcttatg aagatttcgc ctaaaataac
2160
tagcataacc ccttggggcc tctaaacggg tcttgagggg ttttttgctg aaaccaattt
2220
gcctggcggc agtagcgcgg tggtcccacc tgaccccatg ccgaactcag aagtgaaacg
2280
ccgtagcgcc gatggtagtg tggggtctcc ccatgcgaga gtagggaact gccaggcatc
2340
aaataaaacg aaaggctcag tcgaaagact gggcctttcg ggatccaggc cggcctgtta
2400
acgaattaat cttccgcggc ggtatcgata agcttgatat cgaattccga agttcctatt
2460
ctctagaaag tataggaact tcaggtctga agaggagttt acgtccagcc aagctagctt
2520
ggctgcaggt cgtcgaaatt ctaccgggta ggggaggcgc ttttcccaag gcagtctgga
2580
gcatgcgctt tagcagcccc gctgggcact tggcgctaca caagtggcct ctggcctcgc
2640
acacattcca catccaccgg taggcgccaa ccggctccgt tctttggtgg ccccttcgcg
2700
ccaccttcta ctcctcccct agtcaggaag ttcccccccg ccccgcagct cgcgtcgtgc
2760
aggacgtgac aaatggaagt agcacgtctc actagtctcg tgcagatgga cagcaccgct
2820
gagcaatgga agcgggtagg cctttggggc agcggccaat agcagctttg ctccttcgct
2880
ttctgggctc aggggcgggc tcagggggcg gggcgggcgc ccgaaggtcc tccggaggcc
2940
cggcattctg cacgcttcaa aagcgcacgt ctgccgcgct gttctcctct tcctcatctc
3000
cgggcctttc gacctgcagc ctgttgacaa ttaatcatcg gcatagtata tcggcatagt
3060
ataatacgac aaggtgagga actaaaccat gggtcaaagt agcgatgaag ccaacgctcc
3120
cgttgcaggg cagtttgcgc ttcccctgag tgccaccttt ggcttagggg atcgcgtacg
3180
caagaaatct ggtgccgctt ggcagggtca agtcgtcggt tggtattgca caaaactcac
3240
tcctgaaggc tatgcggtcg agtccgaatc ccacccaggc tcagtgcaaa tttatcctgt
3300
ggctgcactt gaacgtgtgg cctaatgagg ggatcaattc tctagagctc gctgatcaga
3360
agttcctatt ctctagaaag tataggaact tcgatggcgc ctcatccctg aagccaaaga
3420
tgtgtataag agacag
3436
<210> 10
<211> 1830
<212> ДНК
<213> Escherichia coli
<400> 10
atgtgtggaa ttgttggcgc gatcgcgcaa cgtgatgtag cagaaatcct tcttgaaggt
60
ttacgtcgtc tggaataccg cggatatgac tctgccggtc tggccgttgt tgatgcagaa
120
ggtcatatga cccgcctgcg tcgcctcggt aaagtccaga tgctggcaca ggcagcggaa
180
gaacatcctc tgcatggcgg cactggtatt gctcacactc gctgggcgac ccacggtgaa
240
ccttcagaag tgaatgcgca tccgcatgtt tctgaacaca ttgtggtggt gcataacggc
300
atcatcgaaa accatgaacc gctgcgtgaa gagctaaaag cgcgtggcta taccttcgtt
360
tctgaaaccg acaccgaagt gattgcccat ctggtgaact gggagctgaa acaaggcggg
420
actctgcgtg aggccgttct gcgtgctatc ccgcagctgc gtggtgcgta cggtacagtg
480
atcatggact cccgtcaccc ggataccctg ctggcggcac gttctggtag tccgctggtg
540
attggcctgg ggatgggcga aaactttatc gcttctgacc agctggcgct gttgccggtg
600
acccgtcgct ttatcttcct tgaagagggc gatattgcgg aaatcactcg ccgttcggta
660
aacatcttcg ataaaactgg cgcggaagta aaacgtcagg atatcgaatc caatctgcaa
720
tatgacgcgg gcgataaagg catttaccgt cactacatgc agaaagagat ctacgaacag
780
ccgaacgcga tcaaaaacac ccttaccgga cgcatcagcc acggtcaggt tgatttaagc
840
gagctgggac cgaacgccga cgaactgctg tcgaaggttg agcatattca gatcctcgcc
900
tgtggtactt cttataactc cggtatggtt tcccgctact ggtttgaatc gctagcaggt
960
attccgtgcg acgtcgaaat cgcctctgaa ttccgctatc gcaaatctgc cgtgcgtcgt
1020
aacagcctga tgatcacctt gtcacagtct ggcgaaaccg cggataccct ggctggcctg
1080
cgtctgtcga aagagctggg ttaccttggt tcactggcaa tctgtaacgt tccgggttct
1140
tctctggtgc gcgaatccga tctggcgcta atgaccaacg cgggtacaga aatcggcgtg
1200
gcatccacta aagcattcac cactcagtta actgtgctgt tgatgctggt ggcgaagctg
1260
tctcgcctga aaggtctgga tgcctccatt gaacatgaca tcgtgcatgg tctgcaggcg
1320
ctgccgagcc gtattgagca gatgctgtct caggacaaac gcattgaagc gctggcagaa
1380
gatttctctg acaaacatca cgcgctgttc ctgggccgtg gcgatcagta cccaatcgcg
1440
ctggaaggcg cattgaagtt gaaagagatc tcttacattc acgctgaagc ctacgctgct
1500
ggcgaactga aacacggtcc gctggcgcta attgatgccg atatgccggt tattgttgtt
1560
gcaccgaaca acgaattgct ggaaaaactg aaatccaaca ttgaagaagt tcgcgcgcgt
1620
ggcggtcagt tgtatgtctt cgccgatcag gatgcgggtt ttgtaagtag cgataacatg
1680
cacatcatcg agatgccgca tgtggaagag gtgattgcac cgatcttcta caccgttccg
1740
ctgcagctgc tggcttacca tgtcgcgctg atcaaaggca ccgacgttga ccagccgcgt
1800
aacctggcaa aatcggttac ggttgagtaa
1830
<210> 11
<211> 609
<212> ПРТ
<213> Escherichia coli
<400> 11
Met Cys Gly Ile Val Gly Ala Ile Ala Gln Arg Asp Val Ala Glu Ile
1 5 10 15
Leu Leu Glu Gly Leu Arg Arg Leu Glu Tyr Arg Gly Tyr Asp Ser Ala
20 25 30
Gly Leu Ala Val Val Asp Ala Glu Gly His Met Thr Arg Leu Arg Arg
35 40 45
Leu Gly Lys Val Gln Met Leu Ala Gln Ala Ala Glu Glu His Pro Leu
50 55 60
His Gly Gly Thr Gly Ile Ala His Thr Arg Trp Ala Thr His Gly Glu
65 70 75 80
Pro Ser Glu Val Asn Ala His Pro His Val Ser Glu His Ile Val Val
85 90 95
Val His Asn Gly Ile Ile Glu Asn His Glu Pro Leu Arg Glu Glu Leu
100 105 110
Lys Ala Arg Gly Tyr Thr Phe Val Ser Glu Thr Asp Thr Glu Val Ile
115 120 125
Ala His Leu Val Asn Trp Glu Leu Lys Gln Gly Gly Thr Leu Arg Glu
130 135 140
Ala Val Leu Arg Ala Ile Pro Gln Leu Arg Gly Ala Tyr Gly Thr Val
145 150 155 160
Ile Met Asp Ser Arg His Pro Asp Thr Leu Leu Ala Ala Arg Ser Gly
165 170 175
Ser Pro Leu Val Ile Gly Leu Gly Met Gly Glu Asn Phe Ile Ala Ser
180 185 190
Asp Gln Leu Ala Leu Leu Pro Val Thr Arg Arg Phe Ile Phe Leu Glu
195 200 205
Glu Gly Asp Ile Ala Glu Ile Thr Arg Arg Ser Val Asn Ile Phe Asp
210 215 220
Lys Thr Gly Ala Glu Val Lys Arg Gln Asp Ile Glu Ser Asn Leu Gln
225 230 235 240
Tyr Asp Ala Gly Asp Lys Gly Ile Tyr Arg His Tyr Met Gln Lys Glu
245 250 255
Ile Tyr Glu Gln Pro Asn Ala Ile Lys Asn Thr Leu Thr Gly Arg Ile
260 265 270
Ser His Gly Gln Val Asp Leu Ser Glu Leu Gly Pro Asn Ala Asp Glu
275 280 285
Leu Leu Ser Lys Val Glu His Ile Gln Ile Leu Ala Cys Gly Thr Ser
290 295 300
Tyr Asn Ser Gly Met Val Ser Arg Tyr Trp Phe Glu Ser Leu Ala Gly
305 310 315 320
Ile Pro Cys Asp Val Glu Ile Ala Ser Glu Phe Arg Tyr Arg Lys Ser
325 330 335
Ala Val Arg Arg Asn Ser Leu Met Ile Thr Leu Ser Gln Ser Gly Glu
340 345 350
Thr Ala Asp Thr Leu Ala Gly Leu Arg Leu Ser Lys Glu Leu Gly Tyr
355 360 365
Leu Gly Ser Leu Ala Ile Cys Asn Val Pro Gly Ser Ser Leu Val Arg
370 375 380
Glu Ser Asp Leu Ala Leu Met Thr Asn Ala Gly Thr Glu Ile Gly Val
385 390 395 400
Ala Ser Thr Lys Ala Phe Thr Thr Gln Leu Thr Val Leu Leu Met Leu
405 410 415
Val Ala Lys Leu Ser Arg Leu Lys Gly Leu Asp Ala Ser Ile Glu His
420 425 430
Asp Ile Val His Gly Leu Gln Ala Leu Pro Ser Arg Ile Glu Gln Met
435 440 445
Leu Ser Gln Asp Lys Arg Ile Glu Ala Leu Ala Glu Asp Phe Ser Asp
450 455 460
Lys His His Ala Leu Phe Leu Gly Arg Gly Asp Gln Tyr Pro Ile Ala
465 470 475 480
Leu Glu Gly Ala Leu Lys Leu Lys Glu Ile Ser Tyr Ile His Ala Glu
485 490 495
Ala Tyr Ala Ala Gly Glu Leu Lys His Gly Pro Leu Ala Leu Ile Asp
500 505 510
Ala Asp Met Pro Val Ile Val Val Ala Pro Asn Asn Glu Leu Leu Glu
515 520 525
Lys Leu Lys Ser Asn Ile Glu Glu Val Arg Ala Arg Gly Gly Gln Leu
530 535 540
Tyr Val Phe Ala Asp Gln Asp Ala Gly Phe Val Ser Ser Asp Asn Met
545 550 555 560
His Ile Ile Glu Met Pro His Val Glu Glu Val Ile Ala Pro Ile Phe
565 570 575
Tyr Thr Val Pro Leu Gln Leu Leu Ala Tyr His Val Ala Leu Ile Lys
580 585 590
Gly Thr Asp Val Asp Gln Pro Arg Asn Leu Ala Lys Ser Val Thr Val
595 600 605
Glu
<210> 12
<211> 1830
<212> ДНК
<213> Escherichia coli
<400> 12
atgtgcggta tcgttggtgc tatcgcacag cgtgatgtag cgaaaatcct cctggaaggt
60
ctgcgtcgtc tcgaataccg tggttacgac tctgccggtc tggcagtagt ggatgcagaa
120
ggtcacatga ctcgtctgcg tcgtctgggt aaagtgcaga tgctcgcgca ggcggcggaa
180
gaacacccac tccacggtgg tacgggtatc gcacacactc gttgggcaac ccacggtgaa
240
ccgtctgagg tcaacgcaca cccgcatgtt agcgagcaca tcgtagtcgt tcacaacggt
300
atcatcgaga accacgaacc actccgtgag gaactcaaag cccgtggtta caccttcgta
360
agcgaaaccg acacggaagt tatcgcccac ctcgttaact gggaactcaa acagggtggt
420
actctgcgtg aagcagttct gcgtgccatt ccacagctgc gtggtgcata cggtaccgtg
480
atcatggact ctcgtcatcc ggataccctg ctcgccgcac gttctggttc tccactcgtt
540
atcggtctgg gtatgggtga gaacttcatc gcctctgatc agctggccct gctcccagtt
600
acccgtcgct tcatcttcct ggaagagggt gacatcgccg aaatcacccg tcgttccgtt
660
aacatcttcg acaaaacggg tgcggaagtt aaacgtcagg acatcgagtc taacctgcag
720
tatgacgctg gtgacaaagg catctaccgt cactacatgc agaaagagat ctacgaacag
780
ccgaacgcga tcaaaaacac cctgaccggt cgtatctctc acggtcaggt tgacctgtct
840
gagctgggtc caaacgcgga cgaactcctg tccaaagtcg agcacatcca gatcctggct
900
tgtggtacct cttacaactc cggtatggtt tctcgttact ggttcgaatc tctggcaggt
960
atcccatgcg acgttgaaat cgcctccgaa ttccgttatc gtaaatctgc ggtacgtcgt
1020
aactccctca tgatcaccct gtctcagtct ggtgaaaccg ctgatactct ggcaggtctg
1080
cgtctcagca aagaactggg ttacctgggt tctctggcca tctgcaacgt tccgggttct
1140
agcctggttc gtgagtctgt gctggctctg atgaccaacg cgggtacgga gatcggtgtt
1200
gcctctacca aagcgttcac tacccagctc actgtcctgc tgatgctggt tgccaaactg
1260
tctcgtctca aaggcctcga cgctagcatc gaacacgaca tcgtacacgg tctgcaggcc
1320
ctcccatctc gtatcgagca gatgctgccg caggacaaac gtatcgaagc actggcagaa
1380
gacttcagcg acaaacacca cgcgctgttt ctgggtcgtg gtgaccagta cccaattgcg
1440
ctggaaggtg ccctgaaact gaaagagatc agctacatcc atgcagaggc atacgcagcg
1500
ggtgagctga aacatggtcc actggccctg atcgacgcag atatgccggt tattgtggtt
1560
gctccgaaca acggcctgct ggagaaactg aaatccaaca tcgaggaagt acgtgcgcgt
1620
ggtggtcagc tgtacgtgtt tgctgaccag gacgcgggtt tcgtttccag cgacaacatg
1680
cacatcatcg aaatgccgca tgttgaagag gtaatcgcgc caatcttcta caccgtaccg
1740
ctgcagctgc tggcgtacca tgtagccctg atcaaaggta cggacgttga ccagccgcgt
1800
aacctggcga aatccgtgac cgtggaataa
1830
<210> 13
<211> 609
<212> ПРТ
<213> Escherichia coli
<400> 13
Met Cys Gly Ile Val Gly Ala Ile Ala Gln Arg Asp Val Ala Lys Ile
1 5 10 15
Leu Leu Glu Gly Leu Arg Arg Leu Glu Tyr Arg Gly Tyr Asp Ser Ala
20 25 30
Gly Leu Ala Val Val Asp Ala Glu Gly His Met Thr Arg Leu Arg Arg
35 40 45
Leu Gly Lys Val Gln Met Leu Ala Gln Ala Ala Glu Glu His Pro Leu
50 55 60
His Gly Gly Thr Gly Ile Ala His Thr Arg Trp Ala Thr His Gly Glu
65 70 75 80
Pro Ser Glu Val Asn Ala His Pro His Val Ser Glu His Ile Val Val
85 90 95
Val His Asn Gly Ile Ile Glu Asn His Glu Pro Leu Arg Glu Glu Leu
100 105 110
Lys Ala Arg Gly Tyr Thr Phe Val Ser Glu Thr Asp Thr Glu Val Ile
115 120 125
Ala His Leu Val Asn Trp Glu Leu Lys Gln Gly Gly Thr Leu Arg Glu
130 135 140
Ala Val Leu Arg Ala Ile Pro Gln Leu Arg Gly Ala Tyr Gly Thr Val
145 150 155 160
Ile Met Asp Ser Arg His Pro Asp Thr Leu Leu Ala Ala Arg Ser Gly
165 170 175
Ser Pro Leu Val Ile Gly Leu Gly Met Gly Glu Asn Phe Ile Ala Ser
180 185 190
Asp Gln Leu Ala Leu Leu Pro Val Thr Arg Arg Phe Ile Phe Leu Glu
195 200 205
Glu Gly Asp Ile Ala Glu Ile Thr Arg Arg Ser Val Asn Ile Phe Asp
210 215 220
Lys Thr Gly Ala Glu Val Lys Arg Gln Asp Ile Glu Ser Asn Leu Gln
225 230 235 240
Tyr Asp Ala Gly Asp Lys Gly Ile Tyr Arg His Tyr Met Gln Lys Glu
245 250 255
Ile Tyr Glu Gln Pro Asn Ala Ile Lys Asn Thr Leu Thr Gly Arg Ile
260 265 270
Ser His Gly Gln Val Asp Leu Ser Glu Leu Gly Pro Asn Ala Asp Glu
275 280 285
Leu Leu Ser Lys Val Glu His Ile Gln Ile Leu Ala Cys Gly Thr Ser
290 295 300
Tyr Asn Ser Gly Met Val Ser Arg Tyr Trp Phe Glu Ser Leu Ala Gly
305 310 315 320
Ile Pro Cys Asp Val Glu Ile Ala Ser Glu Phe Arg Tyr Arg Lys Ser
325 330 335
Ala Val Arg Arg Asn Ser Leu Met Ile Thr Leu Ser Gln Ser Gly Glu
340 345 350
Thr Ala Asp Thr Leu Ala Gly Leu Arg Leu Ser Lys Glu Leu Gly Tyr
355 360 365
Leu Gly Ser Leu Ala Ile Cys Asn Val Pro Gly Ser Ser Leu Val Arg
370 375 380
Glu Ser Val Leu Ala Leu Met Thr Asn Ala Gly Thr Glu Ile Gly Val
385 390 395 400
Ala Ser Thr Lys Ala Phe Thr Thr Gln Leu Thr Val Leu Leu Met Leu
405 410 415
Val Ala Lys Leu Ser Arg Leu Lys Gly Leu Asp Ala Ser Ile Glu His
420 425 430
Asp Ile Val His Gly Leu Gln Ala Leu Pro Ser Arg Ile Glu Gln Met
435 440 445
Leu Pro Gln Asp Lys Arg Ile Glu Ala Leu Ala Glu Asp Phe Ser Asp
450 455 460
Lys His His Ala Leu Phe Leu Gly Arg Gly Asp Gln Tyr Pro Ile Ala
465 470 475 480
Leu Glu Gly Ala Leu Lys Leu Lys Glu Ile Ser Tyr Ile His Ala Glu
485 490 495
Ala Tyr Ala Ala Gly Glu Leu Lys His Gly Pro Leu Ala Leu Ile Asp
500 505 510
Ala Asp Met Pro Val Ile Val Val Ala Pro Asn Asn Gly Leu Leu Glu
515 520 525
Lys Leu Lys Ser Asn Ile Glu Glu Val Arg Ala Arg Gly Gly Gln Leu
530 535 540
Tyr Val Phe Ala Asp Gln Asp Ala Gly Phe Val Ser Ser Asp Asn Met
545 550 555 560
His Ile Ile Glu Met Pro His Val Glu Glu Val Ile Ala Pro Ile Phe
565 570 575
Tyr Thr Val Pro Leu Gln Leu Leu Ala Tyr His Val Ala Leu Ile Lys
580 585 590
Gly Thr Asp Val Asp Gln Pro Arg Asn Leu Ala Lys Ser Val Thr Val
595 600 605
Glu
<210> 14
<211> 480
<212> ДНК
<213> Saccharomyces cerevisiae
<400> 14
atgagcttac ccgatggatt ttatataagg cgaatggaag agggggattt ggaacaggtc
60
actgagacgc taaaggtttt gaccaccgtg ggcactatta cccccgaatc cttcagcaaa
120
ctcataaaat actggaatga agccacagta tggaatgata acgaagataa aaaaataatg
180
caatataacc ccatggtgat tgtggacaag cgcaccgaga cggttgccgc tacggggaat
240
atcatcatcg aaagaaagat cattcatgaa ctggggctat gtggccacat cgaggacatt
300
gcagtaaact ccaagtatca gggccaaggt ttgggcaagc tcttgattga tcaattggta
360
actatcggct ttgactacgg ttgttataag attattttag attgcgatga gaaaaatgtc
420
aaattctatg aaaaatgtgg gtttagcaac gcaggcgtgg aaatgcaaat tagaaaatag
480
<210> 15
<211> 159
<212> ПРТ
<213> Saccharomyces cerevisiae
<400> 15
Met Ser Leu Pro Asp Gly Phe Tyr Ile Arg Arg Met Glu Glu Gly Asp
1 5 10 15
Leu Glu Gln Val Thr Glu Thr Leu Lys Val Leu Thr Thr Val Gly Thr
20 25 30
Ile Thr Pro Glu Ser Phe Ser Lys Leu Ile Lys Tyr Trp Asn Glu Ala
35 40 45
Thr Val Trp Asn Asp Asn Glu Asp Lys Lys Ile Met Gln Tyr Asn Pro
50 55 60
Met Val Ile Val Asp Lys Arg Thr Glu Thr Val Ala Ala Thr Gly Asn
65 70 75 80
Ile Ile Ile Glu Arg Lys Ile Ile His Glu Leu Gly Leu Cys Gly His
85 90 95
Ile Glu Asp Ile Ala Val Asn Ser Lys Tyr Gln Gly Gln Gly Leu Gly
100 105 110
Lys Leu Leu Ile Asp Gln Leu Val Thr Ile Gly Phe Asp Tyr Gly Cys
115 120 125
Tyr Lys Ile Ile Leu Asp Cys Asp Glu Lys Asn Val Lys Phe Tyr Glu
130 135 140
Lys Cys Gly Phe Ser Asn Ala Gly Val Glu Met Gln Ile Arg Lys
145 150 155
<210> 16
<211> 567
<212> ДНК
<213> Escherichia coli
<400> 16
atgtacgagc gttatgcagg tttaattttt gatatggatg gcacaatcct ggatacggag
60
cctacgcacc gtaaagcgtg gcgcgaagta ttagggcact acggtcttca gtacgatatt
120
caggcgatga ttgcgcttaa tggatcgccc acctggcgta ttgctcaggc aattattgag
180
ctgaatcagg ccgatctcga cccgcatgcg ttagcgcgtg aaaaaacaga agcagtaaga
240
agtatgctgc tggatagcgt cgaaccgctt cctcttgttg atgtggtgaa aagttggcat
300
ggtcgtcgcc caatggctgt aggaacgggg agtgaaagcg ccatcgctga ggcattgctg
360
gcgcacctgg gattacgcca ttattttgac gccgtcgtcg ctgccgatca cgtcaaacac
420
cataaacccg cgccagacac atttttgttg tgcgcgcagc gtatgggcgt gcaaccgacg
480
cagtgtgtgg tctttgaaga tgccgatttc ggtattcagg cggcccgtgc agcaggcatg
540
gacgccgtgg atgttcgctt gctgtga
567
<210> 17
<211> 188
<212> ПРТ
<213> Escherichia coli
<400> 17
Met Tyr Glu Arg Tyr Ala Gly Leu Ile Phe Asp Met Asp Gly Thr Ile
1 5 10 15
Leu Asp Thr Glu Pro Thr His Arg Lys Ala Trp Arg Glu Val Leu Gly
20 25 30
His Tyr Gly Leu Gln Tyr Asp Ile Gln Ala Met Ile Ala Leu Asn Gly
35 40 45
Ser Pro Thr Trp Arg Ile Ala Gln Ala Ile Ile Glu Leu Asn Gln Ala
50 55 60
Asp Leu Asp Pro His Ala Leu Ala Arg Glu Lys Thr Glu Ala Val Arg
65 70 75 80
Ser Met Leu Leu Asp Ser Val Glu Pro Leu Pro Leu Val Asp Val Val
85 90 95
Lys Ser Trp His Gly Arg Arg Pro Met Ala Val Gly Thr Gly Ser Glu
100 105 110
Ser Ala Ile Ala Glu Ala Leu Leu Ala His Leu Gly Leu Arg His Tyr
115 120 125
Phe Asp Ala Val Val Ala Ala Asp His Val Lys His His Lys Pro Ala
130 135 140
Pro Asp Thr Phe Leu Leu Cys Ala Gln Arg Met Gly Val Gln Pro Thr
145 150 155 160
Gln Cys Val Val Phe Glu Asp Ala Asp Phe Gly Ile Gln Ala Ala Arg
165 170 175
Ala Ala Gly Met Asp Ala Val Asp Val Arg Leu Leu
180 185
<210> 18
<211> 600
<212> ДНК
<213> Escherichia coli
<400> 18
atgctctata tctttgattt aggtaatgtg attgtcgata tcgactttaa ccgtgtgctg
60
ggagcctgga gcgatttaac gcgtattccg ctggcatcgc ttaagaagag ttttcatatg
120
ggggaggcgt ttcatcagca tgagcgtggg gaaattagcg acgaagcgtt cgcagaggcg
180
ctgtgtcatg agatggctct accgctaagc tacgagcagt tctctcacgg ctggcaggcg
240
gtgtttgttg cgctgcgccc ggaagtgatc gccatcatgc ataaactgcg tgagcagggg
300
catcgcgtgg tggtgctttc caataccaac cgcctgcata ccaccttctg gccggaagaa
360
tacccggaaa ttcgtgatgc tgctgaccat atctatctgt cgcaagatct ggggatgcgc
420
aaacctgaag cacgaattta ccagcatgtt ttgcaggcgg aaggtttttc acccagcgat
480
acggtctttt tcgacgataa cgccgataat atagaaggag ccaatcagct gggcattacc
540
agtattctgg tgaaagataa aaccaccatc ccggactatt tcgcgaaggt gttatgctaa
600
<210> 19
<211> 199
<212> ПРТ
<213> Escherichia coli
<400> 19
Met Leu Tyr Ile Phe Asp Leu Gly Asn Val Ile Val Asp Ile Asp Phe
1 5 10 15
Asn Arg Val Leu Gly Ala Trp Ser Asp Leu Thr Arg Ile Pro Leu Ala
20 25 30
Ser Leu Lys Lys Ser Phe His Met Gly Glu Ala Phe His Gln His Glu
35 40 45
Arg Gly Glu Ile Ser Asp Glu Ala Phe Ala Glu Ala Leu Cys His Glu
50 55 60
Met Ala Leu Pro Leu Ser Tyr Glu Gln Phe Ser His Gly Trp Gln Ala
65 70 75 80
Val Phe Val Ala Leu Arg Pro Glu Val Ile Ala Ile Met His Lys Leu
85 90 95
Arg Glu Gln Gly His Arg Val Val Val Leu Ser Asn Thr Asn Arg Leu
100 105 110
His Thr Thr Phe Trp Pro Glu Glu Tyr Pro Glu Ile Arg Asp Ala Ala
115 120 125
Asp His Ile Tyr Leu Ser Gln Asp Leu Gly Met Arg Lys Pro Glu Ala
130 135 140
Arg Ile Tyr Gln His Val Leu Gln Ala Glu Gly Phe Ser Pro Ser Asp
145 150 155 160
Thr Val Phe Phe Asp Asp Asn Ala Asp Asn Ile Glu Gly Ala Asn Gln
165 170 175
Leu Gly Ile Thr Ser Ile Leu Val Lys Asp Lys Thr Thr Ile Pro Asp
180 185 190
Tyr Phe Ala Lys Val Leu Cys
195
<210> 20
<211> 1266
<212> ДНК
<213> Bacteroides ovatus
<400> 20
atggatagta agaataacat tggtcattca gcagacatct ctttaactgc tgaattaccc
60
ataccaatct ataatggaaa tacgattatg gatttcaaaa aactggcaag tctgtacaag
120
gatgagctcc tggacaacgt ccttcctttc tggcttgaac attcacaaga ccatgagtat
180
ggtggttact tcacctgtct ggaccgtgaa ggaaaagtat tcgatacgga taagtttatt
240
tggctgcaaa gtcgtgaggt atggatgttc tccatgcttt acaacaaagt ggagaaacgt
300
caggaatggc tagactgtgc cattcagggt ggcgaatttc taaaaaaata tggacatgac
360
ggcaattata actggtattt ttccctcgac cgttcgggta gaccattggt agaaccgtac
420
aatatattct cgtatacatt cgctaccatg gctttcggac agttgagcct tacaaccggt
480
aatcaggaat atgcggacat tgccaagaaa actttcgata taatcctttc caaagtggat
540
aatccgaaag ggagatggaa taagcttcat ccgggtaccc gtaatctgaa gaactttgcc
600
ttgccaatga tcctctgtaa cttggcactg gagatagagc atttattgga tgaaacgtat
660
ctgcgggaaa caatggatac ttgtatccat gaagtgatgg aagttttcta tcgtcctgaa
720
ctcggaggta tcattgttga aaacgtggac atagacggta atttggtcga ttgttttgaa
780
ggccgtcagg tgaccccggg acatgccatt gaagcgatgt ggtttatcat ggatctaggc
840
aagcgtctga atcgtccgga attgatagag aaagccaaag agactactct cacgatgctt
900
aattatggct gggacaagca atatggaggt atctactatt ttatggatcg taacggttgt
960
cctccccaac aattggagtg ggaccagaaa ctctggtggg tccatatcga aacgcttatt
1020
tccctgctga aaggctatca attgacggga gacaaaaaat gcttggaatg gtttgaaaag
1080
gtacatgact acacttggga gcatttcaag gataaagaat atcctgaatg gtatggctac
1140
ttgaaccgaa gaggcgaagt attgctacca ctcaaaggag gaaaatggaa aggatgcttc
1200
catgtgccaa gaggactgta tcagtgctgg aaaacattag aagaaataaa aaatatagta
1260
tcctaa
1266
<210> 21
<211> 421
<212> ПРТ
<213> Bacteroides ovatus
<400> 21
Met Asp Ser Lys Asn Asn Ile Gly His Ser Ala Asp Ile Ser Leu Thr
1 5 10 15
Ala Glu Leu Pro Ile Pro Ile Tyr Asn Gly Asn Thr Ile Met Asp Phe
20 25 30
Lys Lys Leu Ala Ser Leu Tyr Lys Asp Glu Leu Leu Asp Asn Val Leu
35 40 45
Pro Phe Trp Leu Glu His Ser Gln Asp His Glu Tyr Gly Gly Tyr Phe
50 55 60
Thr Cys Leu Asp Arg Glu Gly Lys Val Phe Asp Thr Asp Lys Phe Ile
65 70 75 80
Trp Leu Gln Ser Arg Glu Val Trp Met Phe Ser Met Leu Tyr Asn Lys
85 90 95
Val Glu Lys Arg Gln Glu Trp Leu Asp Cys Ala Ile Gln Gly Gly Glu
100 105 110
Phe Leu Lys Lys Tyr Gly His Asp Gly Asn Tyr Asn Trp Tyr Phe Ser
115 120 125
Leu Asp Arg Ser Gly Arg Pro Leu Val Glu Pro Tyr Asn Ile Phe Ser
130 135 140
Tyr Thr Phe Ala Thr Met Ala Phe Gly Gln Leu Ser Leu Thr Thr Gly
145 150 155 160
Asn Gln Glu Tyr Ala Asp Ile Ala Lys Lys Thr Phe Asp Ile Ile Leu
165 170 175
Ser Lys Val Asp Asn Pro Lys Gly Arg Trp Asn Lys Leu His Pro Gly
180 185 190
Thr Arg Asn Leu Lys Asn Phe Ala Leu Pro Met Ile Leu Cys Asn Leu
195 200 205
Ala Leu Glu Ile Glu His Leu Leu Asp Glu Thr Tyr Leu Arg Glu Thr
210 215 220
Met Asp Thr Cys Ile His Glu Val Met Glu Val Phe Tyr Arg Pro Glu
225 230 235 240
Leu Gly Gly Ile Ile Val Glu Asn Val Asp Ile Asp Gly Asn Leu Val
245 250 255
Asp Cys Phe Glu Gly Arg Gln Val Thr Pro Gly His Ala Ile Glu Ala
260 265 270
Met Trp Phe Ile Met Asp Leu Gly Lys Arg Leu Asn Arg Pro Glu Leu
275 280 285
Ile Glu Lys Ala Lys Glu Thr Thr Leu Thr Met Leu Asn Tyr Gly Trp
290 295 300
Asp Lys Gln Tyr Gly Gly Ile Tyr Tyr Phe Met Asp Arg Asn Gly Cys
305 310 315 320
Pro Pro Gln Gln Leu Glu Trp Asp Gln Lys Leu Trp Trp Val His Ile
325 330 335
Glu Thr Leu Ile Ser Leu Leu Lys Gly Tyr Gln Leu Thr Gly Asp Lys
340 345 350
Lys Cys Leu Glu Trp Phe Glu Lys Val His Asp Tyr Thr Trp Glu His
355 360 365
Phe Lys Asp Lys Glu Tyr Pro Glu Trp Tyr Gly Tyr Leu Asn Arg Arg
370 375 380
Gly Glu Val Leu Leu Pro Leu Lys Gly Gly Lys Trp Lys Gly Cys Phe
385 390 395 400
His Val Pro Arg Gly Leu Tyr Gln Cys Trp Lys Thr Leu Glu Glu Ile
405 410 415
Lys Asn Ile Val Ser
420
<210> 22
<211> 1176
<212> ДНК
<213> Synechocystis PCC6803
<400> 22
atgattgccc atcgccgtca ggagttagcc cagcaatatt accaggcttt acaccaggac
60
gtattgccct tttgggaaaa atattccctc gatcgccagg ggggcggtta ctttacctgc
120
ttagaccgta aaggccaggt ttttgacaca gataaattca tttggttaca aaaccgtcag
180
gtatggcagt ttgccgtttt ctacaaccgt ttggaaccaa aaccccaatg gttagaaatt
240
gcccgccatg gtgctgattt tttagctcgc cacggccgag atcaagacgg taattggtat
300
tttgctttgg atcaggaagg caaacccctg cgtcaaccct ataacgtttt ttccgattgc
360
ttcgccgcca tggcctttag tcaatatgcc ttagccagtg gggcgcagga agctaaagcc
420
attgccctgc aggcctacaa taacgtccta cgccgtcagc acaatcccaa aggtcaatac
480
gagaagtcct atccaggtac tagacccctc aaatccctgg cggtgccgat gattttagcc
540
aacctcaccc tggagatgga atggttatta ccgcctacta ccgtggaaga ggtgttggcc
600
caaaccgtca gagaagtgat gacggatttc ctcgacccag aaataggatt aatgcgggaa
660
gcggtgaccc ccacaggaga atttgttgat agttttgaag ggcggttgct caacccagga
720
cacggcattg aagccatgtg gttcatgatg gacattgccc aacgctccgg cgatcgccag
780
ttacaggagc aagccattgc agtggtgttg aacaccctgg aatatgcctg ggatgaagaa
840
tttggtggca tattttattt ccttgatcgc cagggccacc ctccccaaca actggaatgg
900
gaccaaaagc tctggtgggt acatttggaa accctggttg ccctagccaa gggccaccaa
960
gccactggcc aagaaaaatg ttggcaatgg tttgagcggg tccatgatta cgcctggagt
1020
catttcgccg atcctgagta tggggaatgg tttggctacc tgaatcgccg gggagaggtg
1080
ttactcaacc taaaaggggg gaaatggaaa gggtgcttcc acgtgccccg agctctgtgg
1140
ctctgtgcgg aaactctcca acttccggtt agttaa
1176
<210> 23
<211> 391
<212> ПРТ
<213> Synechocystis PCC6803
<400> 23
Met Ile Ala His Arg Arg Gln Glu Leu Ala Gln Gln Tyr Tyr Gln Ala
1 5 10 15
Leu His Gln Asp Val Leu Pro Phe Trp Glu Lys Tyr Ser Leu Asp Arg
20 25 30
Gln Gly Gly Gly Tyr Phe Thr Cys Leu Asp Arg Lys Gly Gln Val Phe
35 40 45
Asp Thr Asp Lys Phe Ile Trp Leu Gln Asn Arg Gln Val Trp Gln Phe
50 55 60
Ala Val Phe Tyr Asn Arg Leu Glu Pro Lys Pro Gln Trp Leu Glu Ile
65 70 75 80
Ala Arg His Gly Ala Asp Phe Leu Ala Arg His Gly Arg Asp Gln Asp
85 90 95
Gly Asn Trp Tyr Phe Ala Leu Asp Gln Glu Gly Lys Pro Leu Arg Gln
100 105 110
Pro Tyr Asn Val Phe Ser Asp Cys Phe Ala Ala Met Ala Phe Ser Gln
115 120 125
Tyr Ala Leu Ala Ser Gly Ala Gln Glu Ala Lys Ala Ile Ala Leu Gln
130 135 140
Ala Tyr Asn Asn Val Leu Arg Arg Gln His Asn Pro Lys Gly Gln Tyr
145 150 155 160
Glu Lys Ser Tyr Pro Gly Thr Arg Pro Leu Lys Ser Leu Ala Val Pro
165 170 175
Met Ile Leu Ala Asn Leu Thr Leu Glu Met Glu Trp Leu Leu Pro Pro
180 185 190
Thr Thr Val Glu Glu Val Leu Ala Gln Thr Val Arg Glu Val Met Thr
195 200 205
Asp Phe Leu Asp Pro Glu Ile Gly Leu Met Arg Glu Ala Val Thr Pro
210 215 220
Thr Gly Glu Phe Val Asp Ser Phe Glu Gly Arg Leu Leu Asn Pro Gly
225 230 235 240
His Gly Ile Glu Ala Met Trp Phe Met Met Asp Ile Ala Gln Arg Ser
245 250 255
Gly Asp Arg Gln Leu Gln Glu Gln Ala Ile Ala Val Val Leu Asn Thr
260 265 270
Leu Glu Tyr Ala Trp Asp Glu Glu Phe Gly Gly Ile Phe Tyr Phe Leu
275 280 285
Asp Arg Gln Gly His Pro Pro Gln Gln Leu Glu Trp Asp Gln Lys Leu
290 295 300
Trp Trp Val His Leu Glu Thr Leu Val Ala Leu Ala Lys Gly His Gln
305 310 315 320
Ala Thr Gly Gln Glu Lys Cys Trp Gln Trp Phe Glu Arg Val His Asp
325 330 335
Tyr Ala Trp Ser His Phe Ala Asp Pro Glu Tyr Gly Glu Trp Phe Gly
340 345 350
Tyr Leu Asn Arg Arg Gly Glu Val Leu Leu Asn Leu Lys Gly Gly Lys
355 360 365
Trp Lys Gly Cys Phe His Val Pro Arg Ala Leu Trp Leu Cys Ala Glu
370 375 380
Thr Leu Gln Leu Pro Val Ser
385 390
<210> 24
<211> 708
<212> ДНК
<213> Enterobacter cloacae
<400> 24
atgaaaactg tactggatac cctgaaggga agactggtcg tctcctgtca ggcgcttgag
60
aacgaaccgt tgcatagccc gtttattatg tcgcggatgg cgctggcggc gcgtcaggga
120
ggggctgcgg ccatccgtgc caacagcgtg gtggatattg aggcgatcaa agagcaggtt
180
acgctgccgg ttattggcat catcaagcgg gagtaccccg acagcgaggt gtttatcacc
240
gcaacgatga aagaggtgga tgaactgatg accgtctccc cggcgatcat tgcgcttgat
300
gcgaccgaca gggcgcggcc tggcggggaa tctctggcaa tgctggttac gcgcattcgt
360
acccgttatc cctcggtgct gcttatggct gatatagcca ctgttgatga ggccgtcacg
420
gcgcaggcgc tggggtttga ttgtgtcggg accacgcttt acggctacac cgcgcagacc
480
gtcggccacg ccttacccga tgatgactgt cagtttctga aagcggtact ggcagccgtc
540
acggtaccgg tggtggccga aggtaacgtg gacaccccgg aacgcgccgc cagatgtctg
600
gcgttggggg cgcatatggt ggtggtgggc ggggcaatca cccgcccgca gcagattacg
660
gaacgcttta tggcggcaat tgacgcgcaa agcaccgatc gagcatga
708
<210> 25
<211> 235
<212> ПРТ
<213> Enterobacter cloacae
<400> 25
Met Lys Thr Val Leu Asp Thr Leu Lys Gly Arg Leu Val Val Ser Cys
1 5 10 15
Gln Ala Leu Glu Asn Glu Pro Leu His Ser Pro Phe Ile Met Ser Arg
20 25 30
Met Ala Leu Ala Ala Arg Gln Gly Gly Ala Ala Ala Ile Arg Ala Asn
35 40 45
Ser Val Val Asp Ile Glu Ala Ile Lys Glu Gln Val Thr Leu Pro Val
50 55 60
Ile Gly Ile Ile Lys Arg Glu Tyr Pro Asp Ser Glu Val Phe Ile Thr
65 70 75 80
Ala Thr Met Lys Glu Val Asp Glu Leu Met Thr Val Ser Pro Ala Ile
85 90 95
Ile Ala Leu Asp Ala Thr Asp Arg Ala Arg Pro Gly Gly Glu Ser Leu
100 105 110
Ala Met Leu Val Thr Arg Ile Arg Thr Arg Tyr Pro Ser Val Leu Leu
115 120 125
Met Ala Asp Ile Ala Thr Val Asp Glu Ala Val Thr Ala Gln Ala Leu
130 135 140
Gly Phe Asp Cys Val Gly Thr Thr Leu Tyr Gly Tyr Thr Ala Gln Thr
145 150 155 160
Val Gly His Ala Leu Pro Asp Asp Asp Cys Gln Phe Leu Lys Ala Val
165 170 175
Leu Ala Ala Val Thr Val Pro Val Val Ala Glu Gly Asn Val Asp Thr
180 185 190
Pro Glu Arg Ala Ala Arg Cys Leu Ala Leu Gly Ala His Met Val Val
195 200 205
Val Gly Gly Ala Ile Thr Arg Pro Gln Gln Ile Thr Glu Arg Phe Met
210 215 220
Ala Ala Ile Asp Ala Gln Ser Thr Asp Arg Ala
225 230 235
<210> 26
<211> 2652
<212> ДНК
<213> Escherichia coli
<400> 26
atgaacgaac aatattccgc attgcgtagt aatgtcagta tgctcggcaa agtgctggga
60
gaaaccatca aggatgcgtt gggagaacac attcttgaac gcgtagaaac tatccgtaag
120
ttgtcgaaat cttcacgcgc tggcaatgat gctaaccgcc aggagttgct caccacctta
180
caaaatttgt cgaacgacga gctgctgccc gttgcgcgtg cgtttagtca gttcctgaac
240
ctggccaaca ccgccgagca ataccacagc atttcgccga aaggcgaagc tgccagcaac
300
ccggaagtga tcgcccgcac cctgcgtaaa ctgaaaaacc agccggaact gagcgaagac
360
accatcaaaa aagcagtgga atcgctgtcg ctggaactgg tcctcacggc tcacccaacc
420
gaaattaccc gtcgtacact gatccacaaa atggtggaag tgaacgcctg tttaaaacag
480
ctcgataaca aagatatcgc tgactacgaa cacaaccagc tgatgcgtcg cctgcgccag
540
ttgatcgccc agtcatggca taccgatgaa atccgtaagc tgcgtccaag cccggtagat
600
gaagccaaat ggggctttgc cgtagtggaa aacagcctgt ggcaaggcgt accaaattac
660
ctgcgcgaac tgaacgaaca actggaagag aacctcggct acaaactgcc cgtcgaattt
720
gttccggtcc gttttacttc gtggatgggc ggcgaccgcg acggcaaccc gaacgtcact
780
gccgatatca cccgccacgt cctgctactc agccgctgga aagccaccga tttgttcctg
840
aaagatattc aggtgctggt ttctgaactg tcgatggttg aagcgacccc tgaactgctg
900
gcgctggttg gcgaagaagg tgccgcagaa ccgtatcgct atctgatgaa aaacctgcgt
960
tctcgcctga tggcgacaca ggcatggctg gaagcgcgcc tgaaaggcga agaactgcca
1020
aaaccagaag gcctgctgac acaaaacgaa gaactgtggg aaccgctcta cgcttgctac
1080
cagtcacttc aggcgtgtgg catgggtatt atcgccaacg gcgatctgct cgacaccctg
1140
cgccgcgtga aatgtttcgg cgtaccgctg gtccgtattg atatccgtca ggagagcacg
1200
cgtcataccg aagcgctggg cgagctgacc cgctacctcg gtatcggcga ctacgaaagc
1260
tggtcagagg ccgacaaaca ggcgttcctg atccgcgaac tgaactccaa acgtccgctt
1320
ctgccgcgca actggcaacc aagcgccgaa acgcgcgaag tgctcgatac ctgccaggtg
1380
attgccgaag caccgcaagg ctccattgcc gcctacgtga tctcgatggc gaaaacgccg
1440
tccgacgtac tggctgtcca cctgctgctg aaagaagcgg gtatcgggtt tgcgatgccg
1500
gttgctccgc tgtttgaaac cctcgatgat ctgaacaacg ccaacgatgt catgacccag
1560
ctgctcaata ttgactggta tcgtggcctg attcagggca aacagatggt gatgattggc
1620
tattccgact cagcaaaaga tgcgggagtg atggcagctt cctgggcgca atatcaggca
1680
caggatgcat taatcaaaac ctgcgaaaaa gcgggtattg agctgacgtt gttccacggt
1740
cgcggcggtt ccattggtcg cggcggcgca cctgctcatg cggcgctgct gtcacaaccg
1800
ccaggaagcc tgaaaggcgg cctgcgcgta accgaacagg gcgagatgat ccgctttaaa
1860
tatggtctgc cagaaatcac cgtcagcagc ctgtcgcttt ataccggggc gattctggaa
1920
gccaacctgc tgccaccgcc ggagccgaaa gagagctggc gtcgcattat ggatgaactg
1980
tcagtcatct cctgcgatgt ctaccgcggc tacgtacgtg aaaacaaaga ttttgtgcct
2040
tacttccgct ccgctacgcc ggaacaagaa ctgggcaaac tgccgttggg ttcacgtccg
2100
gcgaaacgtc gcccaaccgg cggcgtcgag tcactacgcg ccattccgtg gatcttcgcc
2160
tggacgcaaa accgtctgat gctccccgcc tggctgggtg caggtacggc gctgcaaaaa
2220
gtggtcgaag acggcaaaca gagcgagctg gaggctatgt gccgcgattg gccattcttc
2280
tcgacgcgtc tcggcatgct ggagatggtc ttcgccaaag cagacctgtg gctggcggaa
2340
tactatgacc aacgcctggt agacaaagca ctgtggccgt taggtaaaga gttacgcaac
2400
ctgcaagaag aagacatcaa agtggtgctg gcgattgcca acgattccca tctgatggcc
2460
gatctgccgt ggattgcaga gtctattcag ctacggaata tttacaccga cccgctgaac
2520
gtattgcagg ccgagttgct gcaccgctcc cgccaggcag aaaaagaagg ccaggaaccg
2580
gatcctcgcg tcgaacaagc gttaatggtc actattgccg ggattgcggc aggtatgcgt
2640
aataccggct aa
2652
<210> 27
<211> 883
<212> ПРТ
<213> Escherichia coli
<400> 27
Met Asn Glu Gln Tyr Ser Ala Leu Arg Ser Asn Val Ser Met Leu Gly
1 5 10 15
Lys Val Leu Gly Glu Thr Ile Lys Asp Ala Leu Gly Glu His Ile Leu
20 25 30
Glu Arg Val Glu Thr Ile Arg Lys Leu Ser Lys Ser Ser Arg Ala Gly
35 40 45
Asn Asp Ala Asn Arg Gln Glu Leu Leu Thr Thr Leu Gln Asn Leu Ser
50 55 60
Asn Asp Glu Leu Leu Pro Val Ala Arg Ala Phe Ser Gln Phe Leu Asn
65 70 75 80
Leu Ala Asn Thr Ala Glu Gln Tyr His Ser Ile Ser Pro Lys Gly Glu
85 90 95
Ala Ala Ser Asn Pro Glu Val Ile Ala Arg Thr Leu Arg Lys Leu Lys
100 105 110
Asn Gln Pro Glu Leu Ser Glu Asp Thr Ile Lys Lys Ala Val Glu Ser
115 120 125
Leu Ser Leu Glu Leu Val Leu Thr Ala His Pro Thr Glu Ile Thr Arg
130 135 140
Arg Thr Leu Ile His Lys Met Val Glu Val Asn Ala Cys Leu Lys Gln
145 150 155 160
Leu Asp Asn Lys Asp Ile Ala Asp Tyr Glu His Asn Gln Leu Met Arg
165 170 175
Arg Leu Arg Gln Leu Ile Ala Gln Ser Trp His Thr Asp Glu Ile Arg
180 185 190
Lys Leu Arg Pro Ser Pro Val Asp Glu Ala Lys Trp Gly Phe Ala Val
195 200 205
Val Glu Asn Ser Leu Trp Gln Gly Val Pro Asn Tyr Leu Arg Glu Leu
210 215 220
Asn Glu Gln Leu Glu Glu Asn Leu Gly Tyr Lys Leu Pro Val Glu Phe
225 230 235 240
Val Pro Val Arg Phe Thr Ser Trp Met Gly Gly Asp Arg Asp Gly Asn
245 250 255
Pro Asn Val Thr Ala Asp Ile Thr Arg His Val Leu Leu Leu Ser Arg
260 265 270
Trp Lys Ala Thr Asp Leu Phe Leu Lys Asp Ile Gln Val Leu Val Ser
275 280 285
Glu Leu Ser Met Val Glu Ala Thr Pro Glu Leu Leu Ala Leu Val Gly
290 295 300
Glu Glu Gly Ala Ala Glu Pro Tyr Arg Tyr Leu Met Lys Asn Leu Arg
305 310 315 320
Ser Arg Leu Met Ala Thr Gln Ala Trp Leu Glu Ala Arg Leu Lys Gly
325 330 335
Glu Glu Leu Pro Lys Pro Glu Gly Leu Leu Thr Gln Asn Glu Glu Leu
340 345 350
Trp Glu Pro Leu Tyr Ala Cys Tyr Gln Ser Leu Gln Ala Cys Gly Met
355 360 365
Gly Ile Ile Ala Asn Gly Asp Leu Leu Asp Thr Leu Arg Arg Val Lys
370 375 380
Cys Phe Gly Val Pro Leu Val Arg Ile Asp Ile Arg Gln Glu Ser Thr
385 390 395 400
Arg His Thr Glu Ala Leu Gly Glu Leu Thr Arg Tyr Leu Gly Ile Gly
405 410 415
Asp Tyr Glu Ser Trp Ser Glu Ala Asp Lys Gln Ala Phe Leu Ile Arg
420 425 430
Glu Leu Asn Ser Lys Arg Pro Leu Leu Pro Arg Asn Trp Gln Pro Ser
435 440 445
Ala Glu Thr Arg Glu Val Leu Asp Thr Cys Gln Val Ile Ala Glu Ala
450 455 460
Pro Gln Gly Ser Ile Ala Ala Tyr Val Ile Ser Met Ala Lys Thr Pro
465 470 475 480
Ser Asp Val Leu Ala Val His Leu Leu Leu Lys Glu Ala Gly Ile Gly
485 490 495
Phe Ala Met Pro Val Ala Pro Leu Phe Glu Thr Leu Asp Asp Leu Asn
500 505 510
Asn Ala Asn Asp Val Met Thr Gln Leu Leu Asn Ile Asp Trp Tyr Arg
515 520 525
Gly Leu Ile Gln Gly Lys Gln Met Val Met Ile Gly Tyr Ser Asp Ser
530 535 540
Ala Lys Asp Ala Gly Val Met Ala Ala Ser Trp Ala Gln Tyr Gln Ala
545 550 555 560
Gln Asp Ala Leu Ile Lys Thr Cys Glu Lys Ala Gly Ile Glu Leu Thr
565 570 575
Leu Phe His Gly Arg Gly Gly Ser Ile Gly Arg Gly Gly Ala Pro Ala
580 585 590
His Ala Ala Leu Leu Ser Gln Pro Pro Gly Ser Leu Lys Gly Gly Leu
595 600 605
Arg Val Thr Glu Gln Gly Glu Met Ile Arg Phe Lys Tyr Gly Leu Pro
610 615 620
Glu Ile Thr Val Ser Ser Leu Ser Leu Tyr Thr Gly Ala Ile Leu Glu
625 630 635 640
Ala Asn Leu Leu Pro Pro Pro Glu Pro Lys Glu Ser Trp Arg Arg Ile
645 650 655
Met Asp Glu Leu Ser Val Ile Ser Cys Asp Val Tyr Arg Gly Tyr Val
660 665 670
Arg Glu Asn Lys Asp Phe Val Pro Tyr Phe Arg Ser Ala Thr Pro Glu
675 680 685
Gln Glu Leu Gly Lys Leu Pro Leu Gly Ser Arg Pro Ala Lys Arg Arg
690 695 700
Pro Thr Gly Gly Val Glu Ser Leu Arg Ala Ile Pro Trp Ile Phe Ala
705 710 715 720
Trp Thr Gln Asn Arg Leu Met Leu Pro Ala Trp Leu Gly Ala Gly Thr
725 730 735
Ala Leu Gln Lys Val Val Glu Asp Gly Lys Gln Ser Glu Leu Glu Ala
740 745 750
Met Cys Arg Asp Trp Pro Phe Phe Ser Thr Arg Leu Gly Met Leu Glu
755 760 765
Met Val Phe Ala Lys Ala Asp Leu Trp Leu Ala Glu Tyr Tyr Asp Gln
770 775 780
Arg Leu Val Asp Lys Ala Leu Trp Pro Leu Gly Lys Glu Leu Arg Asn
785 790 795 800
Leu Gln Glu Glu Asp Ile Lys Val Val Leu Ala Ile Ala Asn Asp Ser
805 810 815
His Leu Met Ala Asp Leu Pro Trp Ile Ala Glu Ser Ile Gln Leu Arg
820 825 830
Asn Ile Tyr Thr Asp Pro Leu Asn Val Leu Gln Ala Glu Leu Leu His
835 840 845
Arg Ser Arg Gln Ala Glu Lys Glu Gly Gln Glu Pro Asp Pro Arg Val
850 855 860
Glu Gln Ala Leu Met Val Thr Ile Ala Gly Ile Ala Ala Gly Met Arg
865 870 875 880
Asn Thr Gly
<210> 28
<211> 1041
<212> ДНК
<213> Campylobacter jejuni
<400> 28
atgaaagaaa taaaaataca aaatataatc ataagtgaag aaaaagcacc cttagtcgtg
60
cctgaaatag gcattaatca taatggcagt ttagaactag ctaaaattat ggtagatgca
120
gcctttagcg caggtgctaa gattataaag catcaaaccc acatcgttga agatgagatg
180
agtaaggccg ctaaaaaagt aattcctggt aatgcaaaaa taagcattta tgagattatg
240
caaaaatgtg ctttagatta taaagatgag ctagcactta aagaatacac agaaaaatta
300
ggtcttgttt atcttagcac acctttttct cgtgcaggtg caaaccgctt agaagatatg
360
ggagttagtg cttttaagat tggttcaggt gagtgtaata attatccgct tattaaacac
420
atagcagcct ttaaaaagcc tatgatagtt agcacaggaa tgaatagtat tgaaagtata
480
aaaccaactg taaaaatctt attagacaat gaaattccct ttgttttaat gcactcgacc
540
aatctttacc caaccccgca taatcttgta agattaaacg ctatgcttga attaaaaaaa
600
gaattttctt gcatggtagg cttaagcgac cacacaacag ataatcttgc gtgtttaggt
660
gcggttgcac ttggtgcttg tgtgcttgaa agacatttta ctgatagtat gcatagaagt
720
ggccctgata tagtttgttc tatggataca aaggctttaa aagagctaat tatccaaagt
780
gagcaaatgg ctataatgaa aggaaataat gaaagcaaaa aagcagctaa gcaagaacaa
840
gttacaattg attttgcctt tgcaagcgta gttagcatta aagatattaa aaaaggcgaa
900
gttttatcta tggacaatat ctgggttaaa agacctggac ttggtggaat tagtgcggct
960
gaatttgaaa atattttagg caaaaaagca ttaagagata tagaaaatga tactcagtta
1020
agctatgagg attttgcgtg a
1041
<210> 29
<211> 346
<212> ПРТ
<213> Campylobacter jejuni
<400> 29
Met Lys Glu Ile Lys Ile Gln Asn Ile Ile Ile Ser Glu Glu Lys Ala
1 5 10 15
Pro Leu Val Val Pro Glu Ile Gly Ile Asn His Asn Gly Ser Leu Glu
20 25 30
Leu Ala Lys Ile Met Val Asp Ala Ala Phe Ser Ala Gly Ala Lys Ile
35 40 45
Ile Lys His Gln Thr His Ile Val Glu Asp Glu Met Ser Lys Ala Ala
50 55 60
Lys Lys Val Ile Pro Gly Asn Ala Lys Ile Ser Ile Tyr Glu Ile Met
65 70 75 80
Gln Lys Cys Ala Leu Asp Tyr Lys Asp Glu Leu Ala Leu Lys Glu Tyr
85 90 95
Thr Glu Lys Leu Gly Leu Val Tyr Leu Ser Thr Pro Phe Ser Arg Ala
100 105 110
Gly Ala Asn Arg Leu Glu Asp Met Gly Val Ser Ala Phe Lys Ile Gly
115 120 125
Ser Gly Glu Cys Asn Asn Tyr Pro Leu Ile Lys His Ile Ala Ala Phe
130 135 140
Lys Lys Pro Met Ile Val Ser Thr Gly Met Asn Ser Ile Glu Ser Ile
145 150 155 160
Lys Pro Thr Val Lys Ile Leu Leu Asp Asn Glu Ile Pro Phe Val Leu
165 170 175
Met His Ser Thr Asn Leu Tyr Pro Thr Pro His Asn Leu Val Arg Leu
180 185 190
Asn Ala Met Leu Glu Leu Lys Lys Glu Phe Ser Cys Met Val Gly Leu
195 200 205
Ser Asp His Thr Thr Asp Asn Leu Ala Cys Leu Gly Ala Val Ala Leu
210 215 220
Gly Ala Cys Val Leu Glu Arg His Phe Thr Asp Ser Met His Arg Ser
225 230 235 240
Gly Pro Asp Ile Val Cys Ser Met Asp Thr Lys Ala Leu Lys Glu Leu
245 250 255
Ile Ile Gln Ser Glu Gln Met Ala Ile Met Lys Gly Asn Asn Glu Ser
260 265 270
Lys Lys Ala Ala Lys Gln Glu Gln Val Thr Ile Asp Phe Ala Phe Ala
275 280 285
Ser Val Val Ser Ile Lys Asp Ile Lys Lys Gly Glu Val Leu Ser Met
290 295 300
Asp Asn Ile Trp Val Lys Arg Pro Gly Leu Gly Gly Ile Ser Ala Ala
305 310 315 320
Glu Phe Glu Asn Ile Leu Gly Lys Lys Ala Leu Arg Asp Ile Glu Asn
325 330 335
Asp Thr Gln Leu Ser Tyr Glu Asp Phe Ala
340 345
<210> 30
<211> 876
<212> ДНК
<213> Escherichia coli
<400> 30
atgaccacac tggcgattga tatcggcggt actaaacttg ccgccgcgct gattggcgct
60
gacgggcaga tccgcgatcg tcgtgaactt cctacgccag ccagccagac accagaagcc
120
ttgcgtgatg ccttatccgc attagtctct ccgttgcaag ctcatgcgca gcgggttgcc
180
atcgcttcga ccgggataat ccgtgacggc agcttgctgg cgcttaatcc gcataatctt
240
ggtggattgc tacactttcc gttagtcaaa acgctggaac aacttaccaa tttgccgacc
300
attgccatta acgacgcgca ggccgcagca tgggcggagt ttcaggcgct ggatggcgat
360
ataaccgata tggtctttat caccgtttcc accggcgttg gcggcggtgt agtgagcggc
420
tgcaaactgc ttaccggccc tggcggtctg gcggggcata tcgggcatac gcttgccgat
480
ccacacggcc cagtctgcgg ctgtggacgc acaggttgcg tggaagcgat tgcttctggt
540
cgcggcattg cagcggcagc gcagggggag ttggctggcg cggatgcgaa aactattttc
600
acgcgcgccg ggcagggtga cgagcaggcg cagcagctga ttcaccgctc cgcacgtacg
660
cttgcaaggc tgatcgctga tattaaagcc acaactgatt gccagtgcgt ggtggtcggt
720
ggcagcgttg gtctggcaga agggtatctg gcgctggtgg aaacgtatct ggcgcaggag
780
ccagcggcat ttcatgttga tttactggcg gcgcattacc gccatgatgc aggtttactt
840
ggggctgcgc tgttggccca gggagaaaaa ttatga
876
<210> 31
<211> 291
<212> ПРТ
<213> Escherichia coli
<400> 31
Met Thr Thr Leu Ala Ile Asp Ile Gly Gly Thr Lys Leu Ala Ala Ala
1 5 10 15
Leu Ile Gly Ala Asp Gly Gln Ile Arg Asp Arg Arg Glu Leu Pro Thr
20 25 30
Pro Ala Ser Gln Thr Pro Glu Ala Leu Arg Asp Ala Leu Ser Ala Leu
35 40 45
Val Ser Pro Leu Gln Ala His Ala Gln Arg Val Ala Ile Ala Ser Thr
50 55 60
Gly Ile Ile Arg Asp Gly Ser Leu Leu Ala Leu Asn Pro His Asn Leu
65 70 75 80
Gly Gly Leu Leu His Phe Pro Leu Val Lys Thr Leu Glu Gln Leu Thr
85 90 95
Asn Leu Pro Thr Ile Ala Ile Asn Asp Ala Gln Ala Ala Ala Trp Ala
100 105 110
Glu Phe Gln Ala Leu Asp Gly Asp Ile Thr Asp Met Val Phe Ile Thr
115 120 125
Val Ser Thr Gly Val Gly Gly Gly Val Val Ser Gly Cys Lys Leu Leu
130 135 140
Thr Gly Pro Gly Gly Leu Ala Gly His Ile Gly His Thr Leu Ala Asp
145 150 155 160
Pro His Gly Pro Val Cys Gly Cys Gly Arg Thr Gly Cys Val Glu Ala
165 170 175
Ile Ala Ser Gly Arg Gly Ile Ala Ala Ala Ala Gln Gly Glu Leu Ala
180 185 190
Gly Ala Asp Ala Lys Thr Ile Phe Thr Arg Ala Gly Gln Gly Asp Glu
195 200 205
Gln Ala Gln Gln Leu Ile His Arg Ser Ala Arg Thr Leu Ala Arg Leu
210 215 220
Ile Ala Asp Ile Lys Ala Thr Thr Asp Cys Gln Cys Val Val Val Gly
225 230 235 240
Gly Ser Val Gly Leu Ala Glu Gly Tyr Leu Ala Leu Val Glu Thr Tyr
245 250 255
Leu Ala Gln Glu Pro Ala Ala Phe His Val Asp Leu Leu Ala Ala His
260 265 270
Tyr Arg His Asp Ala Gly Leu Leu Gly Ala Ala Leu Leu Ala Gln Gly
275 280 285
Glu Lys Leu
290
<210> 32
<211> 690
<212> ДНК
<213> Escherichia coli
<400> 32
atgtcgttac ttgcacaact ggatcaaaaa atcgctgcta acggtggcct gattgtctcc
60
tgccagccgg ttccggacag cccgctcgat aaacccgaaa tcgtcgccgc catggcatta
120
gcggcagaac aggcgggcgc ggttgccatt cgcattgaag gtgtggcaaa tctgcaagcc
180
acgcgtgcgg tggtgagcgt gccgattatt ggaattgtga aacgcgatct ggaggattct
240
ccggtacgca tcacggccta tattgaagat gttgatgcgc tggcgcaggc gggcgcggac
300
attatcgcca ttgacggcac cgaccgcccg cgtccggtgc ctgttgaaac gctgctggca
360
cgtattcacc atcacggttt actggcgatg accgactgct caacgccgga agacggcctg
420
gcatgccaaa agctgggagc cgaaattatt ggcactacgc tttctggcta taccacgcct
480
gaaacgccag aagagccgga tctggcgctg gtgaaaacgt tgagcgacgc cggatgtcgg
540
gtgattgccg aagggcgtta caacacgcct gctcaggcgg cggatgcgat gcgccacggc
600
gcgtgggcgg tgacggtcgg ttctgcaatc acgcgtcttg agcacatttg tcagtggtac
660
aacacagcga tgaaaaaggc ggtgctatga
690
<210> 33
<211> 229
<212> ПРТ
<213> Escherichia coli
<400> 33
Met Ser Leu Leu Ala Gln Leu Asp Gln Lys Ile Ala Ala Asn Gly Gly
1 5 10 15
Leu Ile Val Ser Cys Gln Pro Val Pro Asp Ser Pro Leu Asp Lys Pro
20 25 30
Glu Ile Val Ala Ala Met Ala Leu Ala Ala Glu Gln Ala Gly Ala Val
35 40 45
Ala Ile Arg Ile Glu Gly Val Ala Asn Leu Gln Ala Thr Arg Ala Val
50 55 60
Val Ser Val Pro Ile Ile Gly Ile Val Lys Arg Asp Leu Glu Asp Ser
65 70 75 80
Pro Val Arg Ile Thr Ala Tyr Ile Glu Asp Val Asp Ala Leu Ala Gln
85 90 95
Ala Gly Ala Asp Ile Ile Ala Ile Asp Gly Thr Asp Arg Pro Arg Pro
100 105 110
Val Pro Val Glu Thr Leu Leu Ala Arg Ile His His His Gly Leu Leu
115 120 125
Ala Met Thr Asp Cys Ser Thr Pro Glu Asp Gly Leu Ala Cys Gln Lys
130 135 140
Leu Gly Ala Glu Ile Ile Gly Thr Thr Leu Ser Gly Tyr Thr Thr Pro
145 150 155 160
Glu Thr Pro Glu Glu Pro Asp Leu Ala Leu Val Lys Thr Leu Ser Asp
165 170 175
Ala Gly Cys Arg Val Ile Ala Glu Gly Arg Tyr Asn Thr Pro Ala Gln
180 185 190
Ala Ala Asp Ala Met Arg His Gly Ala Trp Ala Val Thr Val Gly Ser
195 200 205
Ala Ile Thr Arg Leu Glu His Ile Cys Gln Trp Tyr Asn Thr Ala Met
210 215 220
Lys Lys Ala Val Leu
225
<210> 34
<211> 894
<212> ДНК
<213> Escherichia coli
<400> 34
atggcaacga atttacgtgg cgtaatggct gcactcctga ctccttttga ccaacaacaa
60
gcactggata aagcgagtct gcgtcgcctg gttcagttca atattcagca gggcatcgac
120
ggtttatacg tgggtggttc gaccggcgag gcctttgtac aaagcctttc cgagcgtgaa
180
caggtactgg aaatcgtcgc cgaagaggcg aaaggtaaga ttaaactcat cgcccacgtc
240
ggttgcgtca gcaccgccga aagccaacaa cttgcggcat cggctaaacg ttatggcttc
300
gatgccgtct ccgccgtcac gccgttctac tatcctttca gctttgaaga acactgcgat
360
cactatcggg caattattga ttcggcggat ggtttgccga tggtggtgta caacattcca
420
gccctgagtg gggtaaaact gaccctggat cagatcaaca cacttgttac attgcctggc
480
gtaggtgcgc tgaaacagac ctctggcgat ctctatcaga tggagcagat ccgtcgtgaa
540
catcctgatc ttgtgctcta taacggttac gacgaaatct tcgcctctgg tctgctggcg
600
ggcgctgatg gtggtatcgg cagtacctac aacatcatgg gctggcgcta tcaggggatc
660
gttaaggcgc tgaaagaagg cgatatccag accgcgcaga aactgcaaac tgaatgcaat
720
aaagtcattg atttactgat caaaacgggc gtattccgcg gcctgaaaac tgtcctccat
780
tatatggatg tcgtttctgt gccgctgtgc cgcaaaccgt ttggaccggt agatgaaaaa
840
tatctgccag aactgaaggc gctggcccag cagttgatgc aagagcgcgg gtga
894
<210> 35
<211> 297
<212> ПРТ
<213> Escherichia coli
<400> 35
Met Ala Thr Asn Leu Arg Gly Val Met Ala Ala Leu Leu Thr Pro Phe
1 5 10 15
Asp Gln Gln Gln Ala Leu Asp Lys Ala Ser Leu Arg Arg Leu Val Gln
20 25 30
Phe Asn Ile Gln Gln Gly Ile Asp Gly Leu Tyr Val Gly Gly Ser Thr
35 40 45
Gly Glu Ala Phe Val Gln Ser Leu Ser Glu Arg Glu Gln Val Leu Glu
50 55 60
Ile Val Ala Glu Glu Ala Lys Gly Lys Ile Lys Leu Ile Ala His Val
65 70 75 80
Gly Cys Val Ser Thr Ala Glu Ser Gln Gln Leu Ala Ala Ser Ala Lys
85 90 95
Arg Tyr Gly Phe Asp Ala Val Ser Ala Val Thr Pro Phe Tyr Tyr Pro
100 105 110
Phe Ser Phe Glu Glu His Cys Asp His Tyr Arg Ala Ile Ile Asp Ser
115 120 125
Ala Asp Gly Leu Pro Met Val Val Tyr Asn Ile Pro Ala Leu Ser Gly
130 135 140
Val Lys Leu Thr Leu Asp Gln Ile Asn Thr Leu Val Thr Leu Pro Gly
145 150 155 160
Val Gly Ala Leu Lys Gln Thr Ser Gly Asp Leu Tyr Gln Met Glu Gln
165 170 175
Ile Arg Arg Glu His Pro Asp Leu Val Leu Tyr Asn Gly Tyr Asp Glu
180 185 190
Ile Phe Ala Ser Gly Leu Leu Ala Gly Ala Asp Gly Gly Ile Gly Ser
195 200 205
Thr Tyr Asn Ile Met Gly Trp Arg Tyr Gln Gly Ile Val Lys Ala Leu
210 215 220
Lys Glu Gly Asp Ile Gln Thr Ala Gln Lys Leu Gln Thr Glu Cys Asn
225 230 235 240
Lys Val Ile Asp Leu Leu Ile Lys Thr Gly Val Phe Arg Gly Leu Lys
245 250 255
Thr Val Leu His Tyr Met Asp Val Val Ser Val Pro Leu Cys Arg Lys
260 265 270
Pro Phe Gly Pro Val Asp Glu Lys Tyr Leu Pro Glu Leu Lys Ala Leu
275 280 285
Ala Gln Gln Leu Met Gln Glu Arg Gly
290 295
<210> 36
<211> 1491
<212> ДНК
<213> Escherichia coli
<400> 36
atgagtacta caacccagaa tatcccgtgg tatcgccatc tcaaccgtgc acaatggcgc
60
gcattttccg ctgcctggtt gggatatctg cttgacggtt ttgatttcgt tttaatcgcc
120
ctggtactca ccgaagtaca aggtgaattc gggctgacga cggtgcaggc ggcaagtctg
180
atctctgcag cctttatctc tcgctggttc ggcggcctga tgctcggcgc tatgggtgac
240
cgctacgggc gtcgtctggc aatggtcacc agcatcgttc tcttctcggc cgggacgctg
300
gcctgcggct ttgcgccagg ctacatcacc atgtttatcg ctcgtctggt catcggcatg
360
gggatggcgg gtgaatacgg ttccagcgcc acctatgtca ttgaaagctg gccaaaacat
420
ctgcgtaaca aagccagtgg ttttttgatt tcaggcttct ctgtgggggc cgtcgttgcc
480
gctcaggtct atagcctggt ggttccggtc tggggctggc gtgcgctgtt ctttatcggc
540
attttgccaa tcatctttgc tctctggctg cgtaaaaaca tcccggaagc ggaagactgg
600
aaagagaaac acgcaggtaa agcaccagta cgcacaatgg tggatattct ctaccgtggt
660
gaacatcgca ttgccaatat cgtaatgaca ctggcggcgg ctactgcgct gtggttctgc
720
ttcgccggta acctgcaaaa tgccgcgatc gtcgctgttc ttgggctgtt atgcgccgca
780
atctttatca gctttatggt gcagagtgca ggcaaacgct ggccaacggg cgtaatgctg
840
atggtggtcg tgttgtttgc tttcctctac tcatggccga ttcaggcgct gctgccaacg
900
tatctgaaaa ccgatctggc ttataacccg catactgtag ccaatgtgct gttctttagt
960
ggctttggcg cggcggtggg atgctgcgta ggtggcttcc tcggtgactg gctgggaacc
1020
cgcaaagcgt acgtttgtag cctgctggcc tcgcagctgc tgattattcc ggtatttgcg
1080
attggcggcg caaacgtctg ggtgctcggt ctgttactgt tcttccagca aatgcttgga
1140
caagggatcg ccgggatctt accaaaactg attggcggtt atttcgatac cgaccagcgt
1200
gcagcgggcc tgggctttac ctacaacgtt ggcgcattgg gcggtgcact ggccccaatc
1260
atcggcgcgt tgatcgctca acgtctggat ctgggtactg cgctggcatc gctctcgttc
1320
agtctgacgt tcgtggtgat cctgctgatt gggctggata tgccttctcg cgttcagcgt
1380
tggttgcgcc cggaagcgtt gcgtactcat gacgctatcg acggtaaacc attcagcggt
1440
gccgtgccgt ttggcagcgc caaaaacgat ttagtcaaaa ccaaaagtta a
1491
<210> 37
<211> 496
<212> ПРТ
<213> Escherichia coli
<400> 37
Met Ser Thr Thr Thr Gln Asn Ile Pro Trp Tyr Arg His Leu Asn Arg
1 5 10 15
Ala Gln Trp Arg Ala Phe Ser Ala Ala Trp Leu Gly Tyr Leu Leu Asp
20 25 30
Gly Phe Asp Phe Val Leu Ile Ala Leu Val Leu Thr Glu Val Gln Gly
35 40 45
Glu Phe Gly Leu Thr Thr Val Gln Ala Ala Ser Leu Ile Ser Ala Ala
50 55 60
Phe Ile Ser Arg Trp Phe Gly Gly Leu Met Leu Gly Ala Met Gly Asp
65 70 75 80
Arg Tyr Gly Arg Arg Leu Ala Met Val Thr Ser Ile Val Leu Phe Ser
85 90 95
Ala Gly Thr Leu Ala Cys Gly Phe Ala Pro Gly Tyr Ile Thr Met Phe
100 105 110
Ile Ala Arg Leu Val Ile Gly Met Gly Met Ala Gly Glu Tyr Gly Ser
115 120 125
Ser Ala Thr Tyr Val Ile Glu Ser Trp Pro Lys His Leu Arg Asn Lys
130 135 140
Ala Ser Gly Phe Leu Ile Ser Gly Phe Ser Val Gly Ala Val Val Ala
145 150 155 160
Ala Gln Val Tyr Ser Leu Val Val Pro Val Trp Gly Trp Arg Ala Leu
165 170 175
Phe Phe Ile Gly Ile Leu Pro Ile Ile Phe Ala Leu Trp Leu Arg Lys
180 185 190
Asn Ile Pro Glu Ala Glu Asp Trp Lys Glu Lys His Ala Gly Lys Ala
195 200 205
Pro Val Arg Thr Met Val Asp Ile Leu Tyr Arg Gly Glu His Arg Ile
210 215 220
Ala Asn Ile Val Met Thr Leu Ala Ala Ala Thr Ala Leu Trp Phe Cys
225 230 235 240
Phe Ala Gly Asn Leu Gln Asn Ala Ala Ile Val Ala Val Leu Gly Leu
245 250 255
Leu Cys Ala Ala Ile Phe Ile Ser Phe Met Val Gln Ser Ala Gly Lys
260 265 270
Arg Trp Pro Thr Gly Val Met Leu Met Val Val Val Leu Phe Ala Phe
275 280 285
Leu Tyr Ser Trp Pro Ile Gln Ala Leu Leu Pro Thr Tyr Leu Lys Thr
290 295 300
Asp Leu Ala Tyr Asn Pro His Thr Val Ala Asn Val Leu Phe Phe Ser
305 310 315 320
Gly Phe Gly Ala Ala Val Gly Cys Cys Val Gly Gly Phe Leu Gly Asp
325 330 335
Trp Leu Gly Thr Arg Lys Ala Tyr Val Cys Ser Leu Leu Ala Ser Gln
340 345 350
Leu Leu Ile Ile Pro Val Phe Ala Ile Gly Gly Ala Asn Val Trp Val
355 360 365
Leu Gly Leu Leu Leu Phe Phe Gln Gln Met Leu Gly Gln Gly Ile Ala
370 375 380
Gly Ile Leu Pro Lys Leu Ile Gly Gly Tyr Phe Asp Thr Asp Gln Arg
385 390 395 400
Ala Ala Gly Leu Gly Phe Thr Tyr Asn Val Gly Ala Leu Gly Gly Ala
405 410 415
Leu Ala Pro Ile Ile Gly Ala Leu Ile Ala Gln Arg Leu Asp Leu Gly
420 425 430
Thr Ala Leu Ala Ser Leu Ser Phe Ser Leu Thr Phe Val Val Ile Leu
435 440 445
Leu Ile Gly Leu Asp Met Pro Ser Arg Val Gln Arg Trp Leu Arg Pro
450 455 460
Glu Ala Leu Arg Thr His Asp Ala Ile Asp Gly Lys Pro Phe Ser Gly
465 470 475 480
Ala Val Pro Phe Gly Ser Ala Lys Asn Asp Leu Val Lys Thr Lys Ser
485 490 495
<210> 38
<211> 1149
<212> ДНК
<213> Escherichia coli
<400> 38
atgtatgcat taacccaggg ccggatcttt accggccacg aatttcttga tgaccacgcg
60
gttgttatcg ctgatggcct gattaaaagc gtctgtccgg tagcggaact gccgccagag
120
atcgaacaac gttcactgaa cggggccatt ctctcccccg gttttatcga tgtgcagtta
180
aacggctgcg gcggcgtaca gtttaacgac accgctgaag cggtcagcgt ggaaacgctg
240
gaaatcatgc agaaagccaa tgagaaatca ggctgtacta actatctgcc gacgcttatc
300
accaccagcg atgagctgat gaaacagggc gtgcgcgtta tgcgcgagta cctggcaaaa
360
catccgaatc aggcgttagg tctgcatctg gaaggtccgt ggctgaatct ggtaaaaaaa
420
ggcacccata atccgaattt tgtgcgtaag cctgatgccg cgctggtcga tttcctgtgt
480
gaaaacgccg acgtcattac caaagtgacc ctggcaccgg aaatggttcc tgcggaagtc
540
atcagcaaac tggcaaatgc cgggattgtg gtttctgccg gtcactccaa cgcgacgttg
600
aaagaagcaa aagccggttt ccgcgcgggg attacctttg ccacccatct gtacaacgcg
660
atgccgtata ttaccggtcg tgaacctggc ctggcgggcg cgatcctcga cgaagctgac
720
atttattgcg gtattattgc tgatggcctg catgttgatt acgccaacat tcgcaacgct
780
aaacgtctga aaggcgacaa actgtgtctg gttactgacg ccaccgcgcc agcaggtgcc
840
aacattgaac agttcatttt tgcgggtaaa acaatatact accgtaacgg actttgtgtg
900
gatgagaacg gtacgttaag cggttcatcc ttaaccatga ttgaaggcgt gcgtaatctg
960
gtcgaacatt gcggtatcgc actggatgaa gtgctacgta tggcgacgct ctatccggcg
1020
cgtgcgattg gcgttgagaa acgtctcggc acactcgccg caggtaaagt agccaacctg
1080
actgcattca cacctgattt taaaatcacc aagaccatcg ttaacggtaa cgaggtcgta
1140
actcaataa
1149
<210> 39
<211> 382
<212> ПРТ
<213> Escherichia coli
<400> 39
Met Tyr Ala Leu Thr Gln Gly Arg Ile Phe Thr Gly His Glu Phe Leu
1 5 10 15
Asp Asp His Ala Val Val Ile Ala Asp Gly Leu Ile Lys Ser Val Cys
20 25 30
Pro Val Ala Glu Leu Pro Pro Glu Ile Glu Gln Arg Ser Leu Asn Gly
35 40 45
Ala Ile Leu Ser Pro Gly Phe Ile Asp Val Gln Leu Asn Gly Cys Gly
50 55 60
Gly Val Gln Phe Asn Asp Thr Ala Glu Ala Val Ser Val Glu Thr Leu
65 70 75 80
Glu Ile Met Gln Lys Ala Asn Glu Lys Ser Gly Cys Thr Asn Tyr Leu
85 90 95
Pro Thr Leu Ile Thr Thr Ser Asp Glu Leu Met Lys Gln Gly Val Arg
100 105 110
Val Met Arg Glu Tyr Leu Ala Lys His Pro Asn Gln Ala Leu Gly Leu
115 120 125
His Leu Glu Gly Pro Trp Leu Asn Leu Val Lys Lys Gly Thr His Asn
130 135 140
Pro Asn Phe Val Arg Lys Pro Asp Ala Ala Leu Val Asp Phe Leu Cys
145 150 155 160
Glu Asn Ala Asp Val Ile Thr Lys Val Thr Leu Ala Pro Glu Met Val
165 170 175
Pro Ala Glu Val Ile Ser Lys Leu Ala Asn Ala Gly Ile Val Val Ser
180 185 190
Ala Gly His Ser Asn Ala Thr Leu Lys Glu Ala Lys Ala Gly Phe Arg
195 200 205
Ala Gly Ile Thr Phe Ala Thr His Leu Tyr Asn Ala Met Pro Tyr Ile
210 215 220
Thr Gly Arg Glu Pro Gly Leu Ala Gly Ala Ile Leu Asp Glu Ala Asp
225 230 235 240
Ile Tyr Cys Gly Ile Ile Ala Asp Gly Leu His Val Asp Tyr Ala Asn
245 250 255
Ile Arg Asn Ala Lys Arg Leu Lys Gly Asp Lys Leu Cys Leu Val Thr
260 265 270
Asp Ala Thr Ala Pro Ala Gly Ala Asn Ile Glu Gln Phe Ile Phe Ala
275 280 285
Gly Lys Thr Ile Tyr Tyr Arg Asn Gly Leu Cys Val Asp Glu Asn Gly
290 295 300
Thr Leu Ser Gly Ser Ser Leu Thr Met Ile Glu Gly Val Arg Asn Leu
305 310 315 320
Val Glu His Cys Gly Ile Ala Leu Asp Glu Val Leu Arg Met Ala Thr
325 330 335
Leu Tyr Pro Ala Arg Ala Ile Gly Val Glu Lys Arg Leu Gly Thr Leu
340 345 350
Ala Ala Gly Lys Val Ala Asn Leu Thr Ala Phe Thr Pro Asp Phe Lys
355 360 365
Ile Thr Lys Thr Ile Val Asn Gly Asn Glu Val Val Thr Gln
370 375 380
<210> 40
<211> 801
<212> ДНК
<213> Escherichia coli
<400> 40
atgagactga tccccctgac taccgctgaa caggtcggca aatgggctgc tcgccatatc
60
gtcaatcgta tcaatgcgtt caaaccgact gccgatcgtc cgtttgtact gggcctgccg
120
actggcggca cgccgatgac cacctataaa gcgttagtcg aaatgcataa agcaggccag
180
gtcagcttta agcacgttgt caccttcaac atggacgaat atgtcggtct gccgaaagag
240
catccggaaa gctactacag ctttatgcac cgtaatttct tcgatcacgt tgatattcca
300
gcagaaaaca tcaaccttct caacggcaac gccccggata tcgacgccga gtgccgccag
360
tatgaagaaa aaatccgttc ttacggaaaa attcatctgt ttatgggcgg tgtaggtaac
420
gacggtcata ttgcatttaa cgaaccggcg tcttctctgg cttctcgtac tcgtatcaaa
480
accctgactc atgacactcg cgtcgcaaac tctcgtttct ttgataacga tgttaatcag
540
gtgccaaaat atgccctgac tgtcggtgtt ggtacactgc tggatgccga agaagtgatg
600
attctggtgc tgggtagcca gaaagcactg gcgctgcagg ccgccgttga aggttgcgtg
660
aaccatatgt ggaccatcag ctgtctgcaa ctgcatccga aagcgatcat ggtgtgcgat
720
gaaccttcca ccatggagct gaaagttaag actttaagat atttcaatga attagaagca
780
gaaaatatca aaggtctgta a
801
<210> 41
<211> 266
<212> ПРТ
<213> Escherichia coli
<400> 41
Met Arg Leu Ile Pro Leu Thr Thr Ala Glu Gln Val Gly Lys Trp Ala
1 5 10 15
Ala Arg His Ile Val Asn Arg Ile Asn Ala Phe Lys Pro Thr Ala Asp
20 25 30
Arg Pro Phe Val Leu Gly Leu Pro Thr Gly Gly Thr Pro Met Thr Thr
35 40 45
Tyr Lys Ala Leu Val Glu Met His Lys Ala Gly Gln Val Ser Phe Lys
50 55 60
His Val Val Thr Phe Asn Met Asp Glu Tyr Val Gly Leu Pro Lys Glu
65 70 75 80
His Pro Glu Ser Tyr Tyr Ser Phe Met His Arg Asn Phe Phe Asp His
85 90 95
Val Asp Ile Pro Ala Glu Asn Ile Asn Leu Leu Asn Gly Asn Ala Pro
100 105 110
Asp Ile Asp Ala Glu Cys Arg Gln Tyr Glu Glu Lys Ile Arg Ser Tyr
115 120 125
Gly Lys Ile His Leu Phe Met Gly Gly Val Gly Asn Asp Gly His Ile
130 135 140
Ala Phe Asn Glu Pro Ala Ser Ser Leu Ala Ser Arg Thr Arg Ile Lys
145 150 155 160
Thr Leu Thr His Asp Thr Arg Val Ala Asn Ser Arg Phe Phe Asp Asn
165 170 175
Asp Val Asn Gln Val Pro Lys Tyr Ala Leu Thr Val Gly Val Gly Thr
180 185 190
Leu Leu Asp Ala Glu Glu Val Met Ile Leu Val Leu Gly Ser Gln Lys
195 200 205
Ala Leu Ala Leu Gln Ala Ala Val Glu Gly Cys Val Asn His Met Trp
210 215 220
Thr Ile Ser Cys Leu Gln Leu His Pro Lys Ala Ile Met Val Cys Asp
225 230 235 240
Glu Pro Ser Thr Met Glu Leu Lys Val Lys Thr Leu Arg Tyr Phe Asn
245 250 255
Glu Leu Glu Ala Glu Asn Ile Lys Gly Leu
260 265
<210> 42
<211> 1947
<212> ДНК
<213> Escherichia coli
<400> 42
atgaatattt taggtttttt ccagcgactc ggtagggcgt tacagctccc tatcgcggtg
60
ctgccggtgg cggcactgtt gctgcgattc ggtcagccag atttacttaa cgttgcgttt
120
attgcccagg cgggcggtgc gatttttgat aacctcgcat taatcttcgc catcggtgtg
180
gcatccagct ggtcgaaaga cagcgctggt gcggcggcgc tggcgggtgc ggtaggttac
240
tttgtgttaa ccaaagcgat ggtgaccatc aacccagaaa ttaacatggg tgtactggcg
300
ggtatcatta ccggtctggt tggtggcgca gcctataacc gttggtccga tattaaactg
360
ccggacttcc tgagcttctt cggcggcaaa cgctttgtgc cgattgccac cggattcttc
420
tgcctggtgc tggcggccat ttttggttac gtctggccgc cggtacagca cgctatccat
480
gcaggcggcg agtggatcgt ttctgcgggc gcgctgggtt ccggtatctt tggtttcatc
540
aaccgtctgc tgatcccaac cggtctgcat caggtactga acaccatcgc ctggttccag
600
attggtgaat tcaccaacgc ggcgggtacg gttttccacg gtgacattaa ccgcttctat
660
gccggtgacg gcaccgcggg gatgttcatg tccggcttct tcccgatcat gatgttcggt
720
ctgccgggtg cggcgctggc gatgtacttc gcagcaccga aagagcgtcg tccgatggtt
780
ggcggtatgc tgctttctgt tgctgttact gcgttcctga ccggtgtgac tgagccgctg
840
gaattcctgt tcatgttcct tgctccgctg ctgtacctcc tgcacgcact gctgaccggt
900
atcagcctgt ttgtggcaac gctgctgggt atccacgcgg gcttctcttt ctctgcgggg
960
gctatcgact acgcgttgat gtataacctg ccggccgcca gccagaacgt ctggatgctg
1020
ctggtgatgg gcgttatctt cttcgctatc tacttcgtgg tgttcagttt ggttatccgc
1080
atgttcaacc tgaaaacgcc gggtcgtgaa gataaagaag acgagatcgt tactgaagaa
1140
gccaacagca acactgaaga aggtctgact caactggcaa ccaactatat tgctgcggtt
1200
ggcggcactg acaacctgaa agcgattgac gcctgtatca cccgtctgcg ccttacagtg
1260
gctgactctg cccgcgttaa cgatacgatg tgtaaacgtc tgggtgcttc tggggtagtg
1320
aaactgaaca aacagactat tcaggtgatt gttggcgcga aagcagaatc catcggcgat
1380
gcgatgaaga aagtcgttgc ccgtggtccg gtagccgctg cgtcagctga agcaactccg
1440
gcaactgccg cgcctgtagc aaaaccgcag gctgtaccaa acgcggtatc tatcgcggag
1500
ctggtatcgc cgattaccgg tgatgtcgtg gcactggatc aggttcctga cgaagcattc
1560
gccagcaaag cggtgggtga cggtgtggcg gtgaaaccga cagataaaat cgtcgtatca
1620
ccagccgcag ggacaatcgt gaaaatcttc aacaccaacc acgcgttctg cctggaaacc
1680
gaaaaaggcg cggagatcgt cgtccatatg ggtatcgaca ccgtagcgct ggaaggtaaa
1740
ggctttaaac gtctggtgga agagggtgcg caggtaagcg cagggcaacc gattctggaa
1800
atggatctgg attacctgaa cgctaacgcc cgctcgatga ttagcccggt ggtttgcagc
1860
aatatcgacg atttcagtgg cttgatcatt aaagctcagg gccatattgt ggcgggtcaa
1920
acaccgctgt atgaaatcaa aaagtaa
1947
<210> 43
<211> 648
<212> ПРТ
<213> Escherichia coli
<400> 43
Met Asn Ile Leu Gly Phe Phe Gln Arg Leu Gly Arg Ala Leu Gln Leu
1 5 10 15
Pro Ile Ala Val Leu Pro Val Ala Ala Leu Leu Leu Arg Phe Gly Gln
20 25 30
Pro Asp Leu Leu Asn Val Ala Phe Ile Ala Gln Ala Gly Gly Ala Ile
35 40 45
Phe Asp Asn Leu Ala Leu Ile Phe Ala Ile Gly Val Ala Ser Ser Trp
50 55 60
Ser Lys Asp Ser Ala Gly Ala Ala Ala Leu Ala Gly Ala Val Gly Tyr
65 70 75 80
Phe Val Leu Thr Lys Ala Met Val Thr Ile Asn Pro Glu Ile Asn Met
85 90 95
Gly Val Leu Ala Gly Ile Ile Thr Gly Leu Val Gly Gly Ala Ala Tyr
100 105 110
Asn Arg Trp Ser Asp Ile Lys Leu Pro Asp Phe Leu Ser Phe Phe Gly
115 120 125
Gly Lys Arg Phe Val Pro Ile Ala Thr Gly Phe Phe Cys Leu Val Leu
130 135 140
Ala Ala Ile Phe Gly Tyr Val Trp Pro Pro Val Gln His Ala Ile His
145 150 155 160
Ala Gly Gly Glu Trp Ile Val Ser Ala Gly Ala Leu Gly Ser Gly Ile
165 170 175
Phe Gly Phe Ile Asn Arg Leu Leu Ile Pro Thr Gly Leu His Gln Val
180 185 190
Leu Asn Thr Ile Ala Trp Phe Gln Ile Gly Glu Phe Thr Asn Ala Ala
195 200 205
Gly Thr Val Phe His Gly Asp Ile Asn Arg Phe Tyr Ala Gly Asp Gly
210 215 220
Thr Ala Gly Met Phe Met Ser Gly Phe Phe Pro Ile Met Met Phe Gly
225 230 235 240
Leu Pro Gly Ala Ala Leu Ala Met Tyr Phe Ala Ala Pro Lys Glu Arg
245 250 255
Arg Pro Met Val Gly Gly Met Leu Leu Ser Val Ala Val Thr Ala Phe
260 265 270
Leu Thr Gly Val Thr Glu Pro Leu Glu Phe Leu Phe Met Phe Leu Ala
275 280 285
Pro Leu Leu Tyr Leu Leu His Ala Leu Leu Thr Gly Ile Ser Leu Phe
290 295 300
Val Ala Thr Leu Leu Gly Ile His Ala Gly Phe Ser Phe Ser Ala Gly
305 310 315 320
Ala Ile Asp Tyr Ala Leu Met Tyr Asn Leu Pro Ala Ala Ser Gln Asn
325 330 335
Val Trp Met Leu Leu Val Met Gly Val Ile Phe Phe Ala Ile Tyr Phe
340 345 350
Val Val Phe Ser Leu Val Ile Arg Met Phe Asn Leu Lys Thr Pro Gly
355 360 365
Arg Glu Asp Lys Glu Asp Glu Ile Val Thr Glu Glu Ala Asn Ser Asn
370 375 380
Thr Glu Glu Gly Leu Thr Gln Leu Ala Thr Asn Tyr Ile Ala Ala Val
385 390 395 400
Gly Gly Thr Asp Asn Leu Lys Ala Ile Asp Ala Cys Ile Thr Arg Leu
405 410 415
Arg Leu Thr Val Ala Asp Ser Ala Arg Val Asn Asp Thr Met Cys Lys
420 425 430
Arg Leu Gly Ala Ser Gly Val Val Lys Leu Asn Lys Gln Thr Ile Gln
435 440 445
Val Ile Val Gly Ala Lys Ala Glu Ser Ile Gly Asp Ala Met Lys Lys
450 455 460
Val Val Ala Arg Gly Pro Val Ala Ala Ala Ser Ala Glu Ala Thr Pro
465 470 475 480
Ala Thr Ala Ala Pro Val Ala Lys Pro Gln Ala Val Pro Asn Ala Val
485 490 495
Ser Ile Ala Glu Leu Val Ser Pro Ile Thr Gly Asp Val Val Ala Leu
500 505 510
Asp Gln Val Pro Asp Glu Ala Phe Ala Ser Lys Ala Val Gly Asp Gly
515 520 525
Val Ala Val Lys Pro Thr Asp Lys Ile Val Val Ser Pro Ala Ala Gly
530 535 540
Thr Ile Val Lys Ile Phe Asn Thr Asn His Ala Phe Cys Leu Glu Thr
545 550 555 560
Glu Lys Gly Ala Glu Ile Val Val His Met Gly Ile Asp Thr Val Ala
565 570 575
Leu Glu Gly Lys Gly Phe Lys Arg Leu Val Glu Glu Gly Ala Gln Val
580 585 590
Ser Ala Gly Gln Pro Ile Leu Glu Met Asp Leu Asp Tyr Leu Asn Ala
595 600 605
Asn Ala Arg Ser Met Ile Ser Pro Val Val Cys Ser Asn Ile Asp Asp
610 615 620
Phe Ser Gly Leu Ile Ile Lys Ala Gln Gly His Ile Val Ala Gly Gln
625 630 635 640
Thr Pro Leu Tyr Glu Ile Lys Lys
645
<210> 44
<211> 972
<212> ДНК
<213> Escherichia coli
<400> 44
atgaccattg ctattgttat aggcacacat ggttgggctg cagagcagtt gcttaaaacg
60
gcagaaatgc tgttaggcga gcaggaaaac gtcggctgga tcgatttcgt tccaggtgaa
120
aatgccgaaa cgctgattga aaagtacaac gctcagttgg caaaactcga caccactaaa
180
ggcgtgctgt ttctcgttga tacatgggga ggcagcccgt tcaatgctgc cagccgcatt
240
gtcgtcgaca aagagcatta tgaagtcatt gcaggcgtta acattccaat gctcgtggaa
300
acgttaatgg cccgtgatga tgacccaagc tttgatgaac tggtggcact ggcagtagaa
360
acaggccgtg aaggcgtgaa agcactgaaa gccaaaccgg ttgaaaaagc cgcgccagca
420
cccgctgccg cagcaccaaa agcggctcca actccggcaa aaccaatggg gccaaacgac
480
tacatggtta ttggccttgc gcgtatcgac gaccgtctga ttcacggtca ggtcgccacc
540
cgctggacca aagaaaccaa tgtctcccgt attattgttg ttagtgatga agtggctgcg
600
gataccgttc gtaagacact gctcacccag gttgcacctc cgggcgtaac agcacacgta
660
gttgatgttg ccaaaatgat tcgcgtctac aacaacccga aatatgctgg cgaacgcgta
720
atgctgttat ttaccaaccc aacagatgta gagcgtctcg ttgaaggcgg cgtgaaaatc
780
acctctgtta acgtcggtgg tatggcattc cgtcagggta aaacccaggt gaataacgcg
840
gtttcggttg atgaaaaaga tatcgaggcg ttcaagaaac tgaatgcgcg cggtattgag
900
ctggaagtcc gtaaggtttc caccgatccg aaactgaaaa tgatggatct gatcagcaaa
960
atcgataagt aa
972
<210> 45
<211> 323
<212> ПРТ
<213> Escherichia coli
<400> 45
Met Thr Ile Ala Ile Val Ile Gly Thr His Gly Trp Ala Ala Glu Gln
1 5 10 15
Leu Leu Lys Thr Ala Glu Met Leu Leu Gly Glu Gln Glu Asn Val Gly
20 25 30
Trp Ile Asp Phe Val Pro Gly Glu Asn Ala Glu Thr Leu Ile Glu Lys
35 40 45
Tyr Asn Ala Gln Leu Ala Lys Leu Asp Thr Thr Lys Gly Val Leu Phe
50 55 60
Leu Val Asp Thr Trp Gly Gly Ser Pro Phe Asn Ala Ala Ser Arg Ile
65 70 75 80
Val Val Asp Lys Glu His Tyr Glu Val Ile Ala Gly Val Asn Ile Pro
85 90 95
Met Leu Val Glu Thr Leu Met Ala Arg Asp Asp Asp Pro Ser Phe Asp
100 105 110
Glu Leu Val Ala Leu Ala Val Glu Thr Gly Arg Glu Gly Val Lys Ala
115 120 125
Leu Lys Ala Lys Pro Val Glu Lys Ala Ala Pro Ala Pro Ala Ala Ala
130 135 140
Ala Pro Lys Ala Ala Pro Thr Pro Ala Lys Pro Met Gly Pro Asn Asp
145 150 155 160
Tyr Met Val Ile Gly Leu Ala Arg Ile Asp Asp Arg Leu Ile His Gly
165 170 175
Gln Val Ala Thr Arg Trp Thr Lys Glu Thr Asn Val Ser Arg Ile Ile
180 185 190
Val Val Ser Asp Glu Val Ala Ala Asp Thr Val Arg Lys Thr Leu Leu
195 200 205
Thr Gln Val Ala Pro Pro Gly Val Thr Ala His Val Val Asp Val Ala
210 215 220
Lys Met Ile Arg Val Tyr Asn Asn Pro Lys Tyr Ala Gly Glu Arg Val
225 230 235 240
Met Leu Leu Phe Thr Asn Pro Thr Asp Val Glu Arg Leu Val Glu Gly
245 250 255
Gly Val Lys Ile Thr Ser Val Asn Val Gly Gly Met Ala Phe Arg Gln
260 265 270
Gly Lys Thr Gln Val Asn Asn Ala Val Ser Val Asp Glu Lys Asp Ile
275 280 285
Glu Ala Phe Lys Lys Leu Asn Ala Arg Gly Ile Glu Leu Glu Val Arg
290 295 300
Lys Val Ser Thr Asp Pro Lys Leu Lys Met Met Asp Leu Ile Ser Lys
305 310 315 320
Ile Asp Lys
<210> 46
<211> 801
<212> ДНК
<213> Escherichia coli
<400> 46
atggagatta ccactcttca aattgtgctg gtatttatcg tagcctgtat cgcaggtatg
60
ggatcaatcc tcgatgaatt tcagtttcac cgtccgctaa tcgcgtgtac cctggtgggt
120
atcgttcttg gggatatgaa aaccggtatt attatcggtg gtacgctgga aatgatcgcg
180
ctgggctgga tgaacatcgg tgctgcagtt gcgcctgacg ccgctctggc ttctatcatt
240
tctaccattc tggttatcgc aggtcatcag agcattggtg caggtatcgc actggcaatc
300
cctctggccg ctgcgggcca ggtactgacc atcatcgttc gtactattac cgttgctttc
360
cagcacgctg cggataaggc tgctgataac ggcaacctga cagcgatttc ctggatccac
420
gtttcttctc tgttcctgca agcaatgcgt gtggctattc cggccgtcat cgttgcgctg
480
tctgttggta ccagcgaagt acagaacatg ctgaatgcga ttccggaagt ggtgaccaat
540
ggtctgaata tcgccggtgg catgatcgtg gtggttggtt atgcgatggt tatcaacatg
600
atgcgtgctg gctacctgat gccgttcttc tacctcggct tcgtaaccgc agcattcacc
660
aactttaacc tggttgctct gggtgtgatt ggtactgtta tggcagtgct ctacatccaa
720
cttagcccga aatacaaccg cgtagccggt gcgcctgctc aggcagctgg taacaacgat
780
ctcgataacg aactggacta a
801
<210> 47
<211> 266
<212> ПРТ
<213> Escherichia coli
<400> 47
Met Glu Ile Thr Thr Leu Gln Ile Val Leu Val Phe Ile Val Ala Cys
1 5 10 15
Ile Ala Gly Met Gly Ser Ile Leu Asp Glu Phe Gln Phe His Arg Pro
20 25 30
Leu Ile Ala Cys Thr Leu Val Gly Ile Val Leu Gly Asp Met Lys Thr
35 40 45
Gly Ile Ile Ile Gly Gly Thr Leu Glu Met Ile Ala Leu Gly Trp Met
50 55 60
Asn Ile Gly Ala Ala Val Ala Pro Asp Ala Ala Leu Ala Ser Ile Ile
65 70 75 80
Ser Thr Ile Leu Val Ile Ala Gly His Gln Ser Ile Gly Ala Gly Ile
85 90 95
Ala Leu Ala Ile Pro Leu Ala Ala Ala Gly Gln Val Leu Thr Ile Ile
100 105 110
Val Arg Thr Ile Thr Val Ala Phe Gln His Ala Ala Asp Lys Ala Ala
115 120 125
Asp Asn Gly Asn Leu Thr Ala Ile Ser Trp Ile His Val Ser Ser Leu
130 135 140
Phe Leu Gln Ala Met Arg Val Ala Ile Pro Ala Val Ile Val Ala Leu
145 150 155 160
Ser Val Gly Thr Ser Glu Val Gln Asn Met Leu Asn Ala Ile Pro Glu
165 170 175
Val Val Thr Asn Gly Leu Asn Ile Ala Gly Gly Met Ile Val Val Val
180 185 190
Gly Tyr Ala Met Val Ile Asn Met Met Arg Ala Gly Tyr Leu Met Pro
195 200 205
Phe Phe Tyr Leu Gly Phe Val Thr Ala Ala Phe Thr Asn Phe Asn Leu
210 215 220
Val Ala Leu Gly Val Ile Gly Thr Val Met Ala Val Leu Tyr Ile Gln
225 230 235 240
Leu Ser Pro Lys Tyr Asn Arg Val Ala Gly Ala Pro Ala Gln Ala Ala
245 250 255
Gly Asn Asn Asp Leu Asp Asn Glu Leu Asp
260 265
<210> 48
<211> 852
<212> ДНК
<213> Escherichia coli
<400> 48
atggttgata caactcaaac taccaccgag aaaaaactca ctcaaagtga tattcgtggc
60
gtcttcctgc gttctaacct cttccagggt tcatggaact tcgaacgtat gcaggcactg
120
ggtttctgct tctctatggt accggcaatt cgtcgcctct accctgagaa caacgaagct
180
cgtaaacaag ctattcgccg tcacctggag ttctttaaca cccagccgtt cgtggctgcg
240
ccgattctcg gcgtaaccct ggcgctggaa gaacagcgtg ctaatggcgc agagatcgac
300
gacggtgcta tcaacggtat caaagtcggt ttgatggggc cactggctgg tgtaggcgac
360
ccgatcttct ggggaaccgt acgtccggta tttgcagcac tgggtgccgg tatcgcgatg
420
agcggcagcc tgttaggtcc gctgctgttc ttcatcctgt ttaacctggt gcgtctggca
480
acccgttact acggcgtagc gtatggttac tccaaaggta tcgatatcgt taaagatatg
540
ggtggtggct tcctgcaaaa actgacggaa ggggcgtcta tcctcggcct gtttgtcatg
600
ggggcattgg ttaacaagtg gacacatgtc aacatcccgc tggttgtctc tcgcattact
660
gaccagacgg gcaaagaaca cgttactact gtccagacta ttctggacca gttaatgcca
720
ggcctggtac cactgctgct gacctttgct tgtatgtggc tactgcgcaa aaaagttaac
780
ccgctgtgga tcatcgttgg cttcttcgtc atcggtatcg ctggttacgc ttgcggcctg
840
ctgggactgt aa
852
<210> 49
<211> 283
<212> ПРТ
<213> Escherichia coli
<400> 49
Met Val Asp Thr Thr Gln Thr Thr Thr Glu Lys Lys Leu Thr Gln Ser
1 5 10 15
Asp Ile Arg Gly Val Phe Leu Arg Ser Asn Leu Phe Gln Gly Ser Trp
20 25 30
Asn Phe Glu Arg Met Gln Ala Leu Gly Phe Cys Phe Ser Met Val Pro
35 40 45
Ala Ile Arg Arg Leu Tyr Pro Glu Asn Asn Glu Ala Arg Lys Gln Ala
50 55 60
Ile Arg Arg His Leu Glu Phe Phe Asn Thr Gln Pro Phe Val Ala Ala
65 70 75 80
Pro Ile Leu Gly Val Thr Leu Ala Leu Glu Glu Gln Arg Ala Asn Gly
85 90 95
Ala Glu Ile Asp Asp Gly Ala Ile Asn Gly Ile Lys Val Gly Leu Met
100 105 110
Gly Pro Leu Ala Gly Val Gly Asp Pro Ile Phe Trp Gly Thr Val Arg
115 120 125
Pro Val Phe Ala Ala Leu Gly Ala Gly Ile Ala Met Ser Gly Ser Leu
130 135 140
Leu Gly Pro Leu Leu Phe Phe Ile Leu Phe Asn Leu Val Arg Leu Ala
145 150 155 160
Thr Arg Tyr Tyr Gly Val Ala Tyr Gly Tyr Ser Lys Gly Ile Asp Ile
165 170 175
Val Lys Asp Met Gly Gly Gly Phe Leu Gln Lys Leu Thr Glu Gly Ala
180 185 190
Ser Ile Leu Gly Leu Phe Val Met Gly Ala Leu Val Asn Lys Trp Thr
195 200 205
His Val Asn Ile Pro Leu Val Val Ser Arg Ile Thr Asp Gln Thr Gly
210 215 220
Lys Glu His Val Thr Thr Val Gln Thr Ile Leu Asp Gln Leu Met Pro
225 230 235 240
Gly Leu Val Pro Leu Leu Leu Thr Phe Ala Cys Met Trp Leu Leu Arg
245 250 255
Lys Lys Val Asn Pro Leu Trp Ile Ile Val Gly Phe Phe Val Ile Gly
260 265 270
Ile Ala Gly Tyr Ala Cys Gly Leu Leu Gly Leu
275 280
<210> 50
<211> 1434
<212> ДНК
<213> Escherichia coli
<400> 50
atgtttaaga atgcatttgc taacctgcaa aaggtcggta aatcgctgat gctgccggta
60
tccgtactgc ctatcgcagg tattctgctg ggcgtcggtt ccgcgaattt cagctggctg
120
cccgccgttg tatcgcatgt tatggcagaa gcaggcggtt ccgtctttgc aaacatgcca
180
ctgatttttg cgatcggtgt cgccctcggc tttaccaata acgatggcgt atccgcgctg
240
gccgcagttg ttgcctatgg catcatggtt aaaaccatgg ccgtggttgc gccactggta
300
ctgcatttac ctgctgaaga aatcgcctct aaacacctgg cggatactgg cgtactcgga
360
gggattatct ccggtgcgat cgcagcgtac atgtttaacc gtttctaccg tattaagctg
420
cctgagtatc ttggcttctt tgccggtaaa cgctttgtgc cgatcatttc tggcctggct
480
gccatcttta ctggcgttgt gctgtccttc atttggccgc cgattggttc tgcaatccag
540
accttctctc agtgggctgc ttaccagaac ccggtagttg cgtttggcat ttacggtttc
600
atcgaacgtt gcctggtacc gtttggtctg caccacatct ggaacgtacc tttccagatg
660
cagattggtg aatacaccaa cgcagcaggt caggttttcc acggcgacat tccgcgttat
720
atggcgggtg acccgactgc gggtaaactg tctggtggct tcctgttcaa aatgtacggt
780
ctgccagctg ccgcaattgc tatctggcac tctgctaaac cagaaaaccg cgcgaaagtg
840
ggcggtatta tgatctccgc ggcgctgacc tcgttcctga ccggtatcac cgagccgatc
900
gagttctcct tcatgttcgt tgcgccgatc ctgtacatca tccacgcgat tctggcaggc
960
ctggcattcc caatctgtat tcttctgggg atgcgtgacg gtacgtcgtt ctcgcacggt
1020
ctgatcgact tcatcgttct gtctggtaac agcagcaaac tgtggctgtt cccgatcgtc
1080
ggtatcggtt atgcgattgt ttactacacc atcttccgcg tgctgattaa agcactggat
1140
ctgaaaacgc cgggtcgtga agacgcgact gaagatgcaa aagcgacagg taccagcgaa
1200
atggcaccgg ctctggttgc tgcatttggt ggtaaagaaa acattactaa cctcgacgca
1260
tgtattaccc gtctgcgcgt cagcgttgct gatgtgtcta aagtggatca ggccggcctg
1320
aagaaactgg gcgcagcggg cgtagtggtt gctggttctg gtgttcaggc gattttcggt
1380
actaaatccg ataacctgaa aaccgagatg gatgagtaca tccgtaacca ctaa
1434
<210> 51
<211> 477
<212> ПРТ
<213> Escherichia coli
<400> 51
Met Phe Lys Asn Ala Phe Ala Asn Leu Gln Lys Val Gly Lys Ser Leu
1 5 10 15
Met Leu Pro Val Ser Val Leu Pro Ile Ala Gly Ile Leu Leu Gly Val
20 25 30
Gly Ser Ala Asn Phe Ser Trp Leu Pro Ala Val Val Ser His Val Met
35 40 45
Ala Glu Ala Gly Gly Ser Val Phe Ala Asn Met Pro Leu Ile Phe Ala
50 55 60
Ile Gly Val Ala Leu Gly Phe Thr Asn Asn Asp Gly Val Ser Ala Leu
65 70 75 80
Ala Ala Val Val Ala Tyr Gly Ile Met Val Lys Thr Met Ala Val Val
85 90 95
Ala Pro Leu Val Leu His Leu Pro Ala Glu Glu Ile Ala Ser Lys His
100 105 110
Leu Ala Asp Thr Gly Val Leu Gly Gly Ile Ile Ser Gly Ala Ile Ala
115 120 125
Ala Tyr Met Phe Asn Arg Phe Tyr Arg Ile Lys Leu Pro Glu Tyr Leu
130 135 140
Gly Phe Phe Ala Gly Lys Arg Phe Val Pro Ile Ile Ser Gly Leu Ala
145 150 155 160
Ala Ile Phe Thr Gly Val Val Leu Ser Phe Ile Trp Pro Pro Ile Gly
165 170 175
Ser Ala Ile Gln Thr Phe Ser Gln Trp Ala Ala Tyr Gln Asn Pro Val
180 185 190
Val Ala Phe Gly Ile Tyr Gly Phe Ile Glu Arg Cys Leu Val Pro Phe
195 200 205
Gly Leu His His Ile Trp Asn Val Pro Phe Gln Met Gln Ile Gly Glu
210 215 220
Tyr Thr Asn Ala Ala Gly Gln Val Phe His Gly Asp Ile Pro Arg Tyr
225 230 235 240
Met Ala Gly Asp Pro Thr Ala Gly Lys Leu Ser Gly Gly Phe Leu Phe
245 250 255
Lys Met Tyr Gly Leu Pro Ala Ala Ala Ile Ala Ile Trp His Ser Ala
260 265 270
Lys Pro Glu Asn Arg Ala Lys Val Gly Gly Ile Met Ile Ser Ala Ala
275 280 285
Leu Thr Ser Phe Leu Thr Gly Ile Thr Glu Pro Ile Glu Phe Ser Phe
290 295 300
Met Phe Val Ala Pro Ile Leu Tyr Ile Ile His Ala Ile Leu Ala Gly
305 310 315 320
Leu Ala Phe Pro Ile Cys Ile Leu Leu Gly Met Arg Asp Gly Thr Ser
325 330 335
Phe Ser His Gly Leu Ile Asp Phe Ile Val Leu Ser Gly Asn Ser Ser
340 345 350
Lys Leu Trp Leu Phe Pro Ile Val Gly Ile Gly Tyr Ala Ile Val Tyr
355 360 365
Tyr Thr Ile Phe Arg Val Leu Ile Lys Ala Leu Asp Leu Lys Thr Pro
370 375 380
Gly Arg Glu Asp Ala Thr Glu Asp Ala Lys Ala Thr Gly Thr Ser Glu
385 390 395 400
Met Ala Pro Ala Leu Val Ala Ala Phe Gly Gly Lys Glu Asn Ile Thr
405 410 415
Asn Leu Asp Ala Cys Ile Thr Arg Leu Arg Val Ser Val Ala Asp Val
420 425 430
Ser Lys Val Asp Gln Ala Gly Leu Lys Lys Leu Gly Ala Ala Gly Val
435 440 445
Val Val Ala Gly Ser Gly Val Gln Ala Ile Phe Gly Thr Lys Ser Asp
450 455 460
Asn Leu Lys Thr Glu Met Asp Glu Tyr Ile Arg Asn His
465 470 475
<210> 52
<211> 510
<212> ДНК
<213> Escherichia coli
<400> 52
atgggtttgt tcgataaact gaaatctctg gtttccgacg acaagaagga taccggaact
60
attgagatca ttgctccgct ctctggcgag atcgtcaata tcgaagacgt gccggatgtc
120
gtttttgcgg aaaaaatcgt tggtgatggt attgctatca aaccaacggg taacaaaatg
180
gtcgcgccag tagacggcac cattggtaaa atctttgaaa ccaaccacgc attctctatc
240
gaatctgata gcggcgttga actgttcgtc cacttcggta tcgacaccgt tgaactgaaa
300
ggcgaaggct tcaagcgtat tgctgaagaa ggtcagcgcg tgaaagttgg cgatactgtc
360
attgaatttg atctgccgct gctggaagag aaagccaagt ctaccctgac tccggttgtt
420
atctccaaca tggacgaaat caaagaactg atcaaactgt ccggtagcgt aaccgtgggt
480
gaaaccccgg ttatccgcat caagaagtaa
510
<210> 53
<211> 169
<212> ПРТ
<213> Escherichia coli
<400> 53
Met Gly Leu Phe Asp Lys Leu Lys Ser Leu Val Ser Asp Asp Lys Lys
1 5 10 15
Asp Thr Gly Thr Ile Glu Ile Ile Ala Pro Leu Ser Gly Glu Ile Val
20 25 30
Asn Ile Glu Asp Val Pro Asp Val Val Phe Ala Glu Lys Ile Val Gly
35 40 45
Asp Gly Ile Ala Ile Lys Pro Thr Gly Asn Lys Met Val Ala Pro Val
50 55 60
Asp Gly Thr Ile Gly Lys Ile Phe Glu Thr Asn His Ala Phe Ser Ile
65 70 75 80
Glu Ser Asp Ser Gly Val Glu Leu Phe Val His Phe Gly Ile Asp Thr
85 90 95
Val Glu Leu Lys Gly Glu Gly Phe Lys Arg Ile Ala Glu Glu Gly Gln
100 105 110
Arg Val Lys Val Gly Asp Thr Val Ile Glu Phe Asp Leu Pro Leu Leu
115 120 125
Glu Glu Lys Ala Lys Ser Thr Leu Thr Pro Val Val Ile Ser Asn Met
130 135 140
Asp Glu Ile Lys Glu Leu Ile Lys Leu Ser Gly Ser Val Thr Val Gly
145 150 155 160
Glu Thr Pro Val Ile Arg Ile Lys Lys
165
<210> 54
<211> 1248
<212> ДНК
<213> Escherichia coli
<400> 54
atggcactga atattccatt cagaaatgcg tactatcgtt ttgcatccag ttactcattt
60
ctctttttta tttcctggtc gctgtggtgg tcgttatacg ctatttggct gaaaggacat
120
ctaggattaa cagggacgga attaggtaca ctttattcgg tcaaccagtt taccagcatt
180
ctatttatga tgttctacgg catcgttcag gataaactcg gtctgaagaa accgctcatc
240
tggtgtatga gtttcattct ggtcttgacc ggaccgttta tgatttacgt ttatgaaccg
300
ttactgcaaa gcaatttttc tgtaggtcta attctggggg cgctcttttt tggcctgggg
360
tatctggcgg gatgcggttt gcttgacagc ttcaccgaaa aaatggcgcg aaattttcat
420
ttcgaatatg gaacagcgcg cgcctgggga tcttttggct atgctattgg cgcgttcttt
480
gccggtatat tttttagtat cagtccccat atcaacttct ggttggtctc gctatttggc
540
gctgtattta tgatgatcaa catgcgtttt aaagataagg atcaccagtg catagcggcg
600
gatgcgggag gggtaaaaaa agaggatttt atcgcagttt tcaaggatcg aaacttctgg
660
gttttcgtca tatttattgt ggggacgtgg tctttctata acatttttga tcaacaactc
720
tttcctgtct tttatgcagg tttattcgaa tcacacgatg taggaacgcg cctgtatggt
780
tatctcaact cattccaggt ggtactcgaa gcgctgtgca tggcgattat tcctttcttt
840
gtgaatcggg tagggccaaa aaatgcatta cttatcggtg ttgtgattat ggcgttgcgt
900
atcctttcct gcgcgttgtt cgttaacccc tggattattt cattagtgaa gctgttacat
960
gccattgagg ttccactttg tgtcatatcc gtcttcaaat acagcgtggc aaactttgat
1020
aagcgcctgt cgtcgacgat ctttctgatt ggttttcaaa ttgccagttc gcttgggatt
1080
gtgctgcttt caacgccgac tgggatactc tttgaccacg caggctacca gacagttttc
1140
ttcgcaattt cgggtattgt ctgcctgatg ttgctatttg gcattttctt cctgagtaaa
1200
aaacgcgagc aaatagttat ggaaacgcct gtaccttcag caatatag
1248
<210> 55
<211> 415
<212> ПРТ
<213> Escherichia coli
<400> 55
Met Ala Leu Asn Ile Pro Phe Arg Asn Ala Tyr Tyr Arg Phe Ala Ser
1 5 10 15
Ser Tyr Ser Phe Leu Phe Phe Ile Ser Trp Ser Leu Trp Trp Ser Leu
20 25 30
Tyr Ala Ile Trp Leu Lys Gly His Leu Gly Leu Thr Gly Thr Glu Leu
35 40 45
Gly Thr Leu Tyr Ser Val Asn Gln Phe Thr Ser Ile Leu Phe Met Met
50 55 60
Phe Tyr Gly Ile Val Gln Asp Lys Leu Gly Leu Lys Lys Pro Leu Ile
65 70 75 80
Trp Cys Met Ser Phe Ile Leu Val Leu Thr Gly Pro Phe Met Ile Tyr
85 90 95
Val Tyr Glu Pro Leu Leu Gln Ser Asn Phe Ser Val Gly Leu Ile Leu
100 105 110
Gly Ala Leu Phe Phe Gly Leu Gly Tyr Leu Ala Gly Cys Gly Leu Leu
115 120 125
Asp Ser Phe Thr Glu Lys Met Ala Arg Asn Phe His Phe Glu Tyr Gly
130 135 140
Thr Ala Arg Ala Trp Gly Ser Phe Gly Tyr Ala Ile Gly Ala Phe Phe
145 150 155 160
Ala Gly Ile Phe Phe Ser Ile Ser Pro His Ile Asn Phe Trp Leu Val
165 170 175
Ser Leu Phe Gly Ala Val Phe Met Met Ile Asn Met Arg Phe Lys Asp
180 185 190
Lys Asp His Gln Cys Ile Ala Ala Asp Ala Gly Gly Val Lys Lys Glu
195 200 205
Asp Phe Ile Ala Val Phe Lys Asp Arg Asn Phe Trp Val Phe Val Ile
210 215 220
Phe Ile Val Gly Thr Trp Ser Phe Tyr Asn Ile Phe Asp Gln Gln Leu
225 230 235 240
Phe Pro Val Phe Tyr Ala Gly Leu Phe Glu Ser His Asp Val Gly Thr
245 250 255
Arg Leu Tyr Gly Tyr Leu Asn Ser Phe Gln Val Val Leu Glu Ala Leu
260 265 270
Cys Met Ala Ile Ile Pro Phe Phe Val Asn Arg Val Gly Pro Lys Asn
275 280 285
Ala Leu Leu Ile Gly Val Val Ile Met Ala Leu Arg Ile Leu Ser Cys
290 295 300
Ala Leu Phe Val Asn Pro Trp Ile Ile Ser Leu Val Lys Leu Leu His
305 310 315 320
Ala Ile Glu Val Pro Leu Cys Val Ile Ser Val Phe Lys Tyr Ser Val
325 330 335
Ala Asn Phe Asp Lys Arg Leu Ser Ser Thr Ile Phe Leu Ile Gly Phe
340 345 350
Gln Ile Ala Ser Ser Leu Gly Ile Val Leu Leu Ser Thr Pro Thr Gly
355 360 365
Ile Leu Phe Asp His Ala Gly Tyr Gln Thr Val Phe Phe Ala Ile Ser
370 375 380
Gly Ile Val Cys Leu Met Leu Leu Phe Gly Ile Phe Phe Leu Ser Lys
385 390 395 400
Lys Arg Glu Gln Ile Val Met Glu Thr Pro Val Pro Ser Ala Ile
405 410 415
<210> 56
<211> 924
<212> ДНК
<213> Escherichia coli
<400> 56
atgtcagcca aagtatgggt tttaggggat gcggtcgtag atctcttgcc agaatcagac
60
gggcggctac tgccttgtcc tggcggcgcg ccagctaacg ttgcggtggg aatcgccaga
120
ttaggcggaa caagtgggtt tataggtcgg gtcggtgatg atccttttgg tgcgttaatg
180
caaagaacgc tgctaactga gggtgtcgat atcacgtatc tgaagcaaga tgaatggcac
240
cggacatcca cggtgcttgt cgatctgaac gatcaaggag aacgttcatt tacgtttatg
300
gtccgcccca gtgccgatct ttttttagag acgacagact tgccctgctg gcgacatggc
360
gaatggttac atctctgttc aattgcgttg tctgccgagc cttcgcgtac cagcgcattt
420
actgcgatga cggcgatccg gcatgccgga ggttttgtca gcttcgatcc caatattcgt
480
gaagatctat ggcaagacga gcatttgctc cgcttgtgtt tgcggcaggc gctacaactg
540
gcggatgtcg tcaagctctc ggaagaagaa tggcgactta tcagtggaaa aacacagaac
600
gatcgggata tatgcgccct ggcaaaagag tatgagatcg ccatgctgtt ggtgactaaa
660
ggtgcagaag gggtggtggt ctgttatcga ggacaagtcc accattttgc tggaatgtct
720
gtgaattgtg tcgatagcac tggggcggga gatgcgttcg ttgccgggtt actcacaggt
780
ctgtcctcta cgggattatc tacagatgag agagaaatgc gacgaattat cgatctcgct
840
caacgttgcg gagcgcttgc agtaacagcg aaaggggcaa tgacagcgct gccatgtcga
900
caagaactgg aaagtgagaa gtaa
924
<210> 57
<211> 307
<212> ПРТ
<213> Escherichia coli
<400> 57
Met Ser Ala Lys Val Trp Val Leu Gly Asp Ala Val Val Asp Leu Leu
1 5 10 15
Pro Glu Ser Asp Gly Arg Leu Leu Pro Cys Pro Gly Gly Ala Pro Ala
20 25 30
Asn Val Ala Val Gly Ile Ala Arg Leu Gly Gly Thr Ser Gly Phe Ile
35 40 45
Gly Arg Val Gly Asp Asp Pro Phe Gly Ala Leu Met Gln Arg Thr Leu
50 55 60
Leu Thr Glu Gly Val Asp Ile Thr Tyr Leu Lys Gln Asp Glu Trp His
65 70 75 80
Arg Thr Ser Thr Val Leu Val Asp Leu Asn Asp Gln Gly Glu Arg Ser
85 90 95
Phe Thr Phe Met Val Arg Pro Ser Ala Asp Leu Phe Leu Glu Thr Thr
100 105 110
Asp Leu Pro Cys Trp Arg His Gly Glu Trp Leu His Leu Cys Ser Ile
115 120 125
Ala Leu Ser Ala Glu Pro Ser Arg Thr Ser Ala Phe Thr Ala Met Thr
130 135 140
Ala Ile Arg His Ala Gly Gly Phe Val Ser Phe Asp Pro Asn Ile Arg
145 150 155 160
Glu Asp Leu Trp Gln Asp Glu His Leu Leu Arg Leu Cys Leu Arg Gln
165 170 175
Ala Leu Gln Leu Ala Asp Val Val Lys Leu Ser Glu Glu Glu Trp Arg
180 185 190
Leu Ile Ser Gly Lys Thr Gln Asn Asp Arg Asp Ile Cys Ala Leu Ala
195 200 205
Lys Glu Tyr Glu Ile Ala Met Leu Leu Val Thr Lys Gly Ala Glu Gly
210 215 220
Val Val Val Cys Tyr Arg Gly Gln Val His His Phe Ala Gly Met Ser
225 230 235 240
Val Asn Cys Val Asp Ser Thr Gly Ala Gly Asp Ala Phe Val Ala Gly
245 250 255
Leu Leu Thr Gly Leu Ser Ser Thr Gly Leu Ser Thr Asp Glu Arg Glu
260 265 270
Met Arg Arg Ile Ile Asp Leu Ala Gln Arg Cys Gly Ala Leu Ala Val
275 280 285
Thr Ala Lys Gly Ala Met Thr Ala Leu Pro Cys Arg Gln Glu Leu Glu
290 295 300
Ser Glu Lys
305
<210> 58
<211> 1434
<212> ДНК
<213> Escherichia coli
<400> 58
atgacgcaat ctcgattgca tgcggcgcaa aacgccctag caaaacttca tgagcaccgg
60
ggtaacactt tctatcccca ttttcacctc gcgcctcctg ccgggtggat gaacgatcca
120
aacggcctga tctggtttaa cgatcgttat cacgcgtttt atcaacatca tccgatgagc
180
gaacactggg ggccaatgca ctggggacat gccaccagcg acgatatgat ccactggcag
240
catgagccta ttgcgctagc gccaggagac gataatgaca aagacgggtg tttttcaggt
300
agtgctgtcg atgacaatgg tgtcctctca cttatctaca ccggacacgt ctggctcgat
360
ggtgcaggta atgacgatgc aattcgcgaa gtacaatgtc tggctaccag tcgggatggt
420
attcatttcg agaaacaggg tgtgatcctc actccaccag aaggaatcat gcacttccgc
480
gatcctaaag tgtggcgtga agccgacaca tggtggatgg tagtcggggc gaaagatcca
540
ggcaacacgg ggcagatcct gctttatcgc ggcagttcat tgcgtgaatg gaccttcgat
600
cgcgtactgg cccacgctga tgcgggtgaa agctatatgt gggaatgtcc ggactttttc
660
agccttggcg atcagcatta tctgatgttt tccccgcagg gaatgaatgc cgagggatac
720
agttaccgaa atcgctttca aagtggcgta atacccggaa tgtggtcgcc aggacgactt
780
tttgcacaat ccgggcattt tactgaactt gataacgggc atgactttta tgcaccacaa
840
agctttttag cgaaggatgg tcggcgtatt gttatcggat ggatggatat gtgggaatcg
900
ccaatgccct caaaacgtga aggctgggca ggctgcatga cgctggcgcg cgagctatca
960
gagagcaatg gcaaacttct acaacgcccg gttcacgaag ctgagtcgtt acgccagcag
1020
catcaatctg tctctccccg cacaatcagc aataaatatg ttttgcagga aaacgcgcaa
1080
gcagttgaga ttcagttgca gtgggcgctg aagaacagtg atgccgaaca ttacggatta
1140
cagctcggca ctggaatgcg gctgtatatt gataaccaat ctgagcgact tgttttgtgg
1200
cggtattacc cacacgagaa tttagacggc taccgtagta ttcccctccc gcagcgtgac
1260
acgctcgccc taaggatatt tatcgataca tcatccgtgg aagtatttat taacgacggg
1320
gaagcggtga tgagtagtcg aatctatccg cagccagaag aacgggaact gtcgctttat
1380
gcctcccacg gagtggctgt gctgcaacat ggagcactct ggctactggg ttaa
1434
<210> 59
<211> 480
<212> ПРТ
<213> Escherichia coli
<400> 59
Met Ile Lys Met Thr Gln Ser Arg Leu His Ala Ala Gln Asn Ala Leu
1 5 10 15
Ala Lys Leu His Glu His Arg Gly Asn Thr Phe Tyr Pro His Phe His
20 25 30
Leu Ala Pro Pro Ala Gly Trp Met Asn Asp Pro Asn Gly Leu Ile Trp
35 40 45
Phe Asn Asp Arg Tyr His Ala Phe Tyr Gln His His Pro Met Ser Glu
50 55 60
His Trp Gly Pro Met His Trp Gly His Ala Thr Ser Asp Asp Met Ile
65 70 75 80
His Trp Gln His Glu Pro Ile Ala Leu Ala Pro Gly Asp Asp Asn Asp
85 90 95
Lys Asp Gly Cys Phe Ser Gly Ser Ala Val Asp Asp Asn Gly Val Leu
100 105 110
Ser Leu Ile Tyr Thr Gly His Val Trp Leu Asp Gly Ala Gly Asn Asp
115 120 125
Asp Ala Ile Arg Glu Val Gln Cys Leu Ala Thr Ser Arg Asp Gly Ile
130 135 140
His Phe Glu Lys Gln Gly Val Ile Leu Thr Pro Pro Glu Gly Ile Met
145 150 155 160
His Phe Arg Asp Pro Lys Val Trp Arg Glu Ala Asp Thr Trp Trp Met
165 170 175
Val Val Gly Ala Lys Asp Pro Gly Asn Thr Gly Gln Ile Leu Leu Tyr
180 185 190
Arg Gly Ser Ser Leu Arg Glu Trp Thr Phe Asp Arg Val Leu Ala His
195 200 205
Ala Asp Ala Gly Glu Ser Tyr Met Trp Glu Cys Pro Asp Phe Phe Ser
210 215 220
Leu Gly Asp Gln His Tyr Leu Met Phe Ser Pro Gln Gly Met Asn Ala
225 230 235 240
Glu Gly Tyr Ser Tyr Arg Asn Arg Phe Gln Ser Gly Val Ile Pro Gly
245 250 255
Met Trp Ser Pro Gly Arg Leu Phe Ala Gln Ser Gly His Phe Thr Glu
260 265 270
Leu Asp Asn Gly His Asp Phe Tyr Ala Pro Gln Ser Phe Leu Ala Lys
275 280 285
Asp Gly Arg Arg Ile Val Ile Gly Trp Met Asp Met Trp Glu Ser Pro
290 295 300
Met Pro Ser Lys Arg Glu Gly Trp Ala Gly Cys Met Thr Leu Ala Arg
305 310 315 320
Glu Leu Ser Glu Ser Asn Gly Lys Leu Leu Gln Arg Pro Val His Glu
325 330 335
Ala Glu Ser Leu Arg Gln Gln His Gln Ser Val Ser Pro Arg Thr Ile
340 345 350
Ser Asn Lys Tyr Val Leu Gln Glu Asn Ala Gln Ala Val Glu Ile Gln
355 360 365
Leu Gln Trp Ala Leu Lys Asn Ser Asp Ala Glu His Tyr Gly Leu Gln
370 375 380
Leu Gly Thr Gly Met Arg Leu Tyr Ile Asp Asn Gln Ser Glu Arg Leu
385 390 395 400
Val Leu Trp Arg Tyr Tyr Pro His Glu Asn Leu Asp Gly Tyr Arg Ser
405 410 415
Ile Pro Leu Pro Gln Arg Asp Thr Leu Ala Leu Arg Ile Phe Ile Asp
420 425 430
Thr Ser Ser Val Glu Val Phe Ile Asn Asp Gly Glu Ala Val Met Ser
435 440 445
Ser Arg Ile Tyr Pro Gln Pro Glu Glu Arg Glu Leu Ser Leu Tyr Ala
450 455 460
Ser His Gly Val Ala Val Leu Gln His Gly Ala Leu Trp Leu Leu Gly
465 470 475 480
<210> 60
<211> 954
<212> ДНК
<213> Escherichia coli
<400> 60
atgatgacag tctcccgggt gatgcataat gcagaatctg tgcgtcctgc aacgcgtgac
60
cgcgtattgc aggcaatcca gaccctgaat tatgttcctg atctttccgc ccgtaagatg
120
cgcgctcaag gacgtaagcc gtcgactctc gccgtgctgg cgcaggacac ggctaccact
180
cctttctctg ttgatattct gcttgccatt gagcaaaccg ccagcgagtt cggctggaat
240
agttttttaa tcaatatttt ttctgaagat gacgctgccc gtgctgcacg tcagctgctt
300
gcccaccgtc cggatggcat tatctatact acaatggggc tgcgacatat cacgctgcct
360
gagtctctgt atggtgaaaa tattgtattg gcgaactgtg tggcggatga cccagcgtta
420
cccagttata tccctgatga ttacactgca caatatgaat caacacagca tttgctcgcg
480
gcgggctatc gtcaaccgtt atgcttctgg ctaccggaaa gtgcgttggc aacagggtat
540
cgtcggcagg gatttgagca ggcctggcgt gatgctggac gagatctggc tgaggtgaaa
600
caatttcaca tggcaacagg tgatgatcac tacaccgatc tcgcaagttt actcaatgcc
660
cacttcaaat cgggcaaacc agattttgat gttctgatat gtggtaacga tcgcgcagct
720
tttgtggctt atcaggttct tttggcgaag ggggtacgta tcccgcagga tgtcgccgta
780
atgggctttg ataatctggt tggcgtcggg catctgtttt taccgccgct gaccacaatt
840
cagcttccac atgacattat cgggcgggaa gctgcattgc atattattga aggtcgtgaa
900
gggggaagag tgacccggat cccttgcccg ctgttgatcc gttgttccac ctga
954
<210> 61
<211> 317
<212> ПРТ
<213> Escherichia coli
<400> 61
Met Met Thr Val Ser Arg Val Met His Asn Ala Glu Ser Val Arg Pro
1 5 10 15
Ala Thr Arg Asp Arg Val Leu Gln Ala Ile Gln Thr Leu Asn Tyr Val
20 25 30
Pro Asp Leu Ser Ala Arg Lys Met Arg Ala Gln Gly Arg Lys Pro Ser
35 40 45
Thr Leu Ala Val Leu Ala Gln Asp Thr Ala Thr Thr Pro Phe Ser Val
50 55 60
Asp Ile Leu Leu Ala Ile Glu Gln Thr Ala Ser Glu Phe Gly Trp Asn
65 70 75 80
Ser Phe Leu Ile Asn Ile Phe Ser Glu Asp Asp Ala Ala Arg Ala Ala
85 90 95
Arg Gln Leu Leu Ala His Arg Pro Asp Gly Ile Ile Tyr Thr Thr Met
100 105 110
Gly Leu Arg His Ile Thr Leu Pro Glu Ser Leu Tyr Gly Glu Asn Ile
115 120 125
Val Leu Ala Asn Cys Val Ala Asp Asp Pro Ala Leu Pro Ser Tyr Ile
130 135 140
Pro Asp Asp Tyr Thr Ala Gln Tyr Glu Ser Thr Gln His Leu Leu Ala
145 150 155 160
Ala Gly Tyr Arg Gln Pro Leu Cys Phe Trp Leu Pro Glu Ser Ala Leu
165 170 175
Ala Thr Gly Tyr Arg Arg Gln Gly Phe Glu Gln Ala Trp Arg Asp Ala
180 185 190
Gly Arg Asp Leu Ala Glu Val Lys Gln Phe His Met Ala Thr Gly Asp
195 200 205
Asp His Tyr Thr Asp Leu Ala Ser Leu Leu Asn Ala His Phe Lys Ser
210 215 220
Gly Lys Pro Asp Phe Asp Val Leu Ile Cys Gly Asn Asp Arg Ala Ala
225 230 235 240
Phe Val Ala Tyr Gln Val Leu Leu Ala Lys Gly Val Arg Ile Pro Gln
245 250 255
Asp Val Ala Val Met Gly Phe Asp Asn Leu Val Gly Val Gly His Leu
260 265 270
Phe Leu Pro Pro Leu Thr Thr Ile Gln Leu Pro His Asp Ile Ile Gly
275 280 285
Arg Glu Ala Ala Leu His Ile Ile Glu Gly Arg Glu Gly Gly Arg Val
290 295 300
Thr Arg Ile Pro Cys Pro Leu Leu Ile Arg Cys Ser Thr
305 310 315
<210> 62
<211> 1254
<212> ДНК
<213> Escherichia coli
<400> 62
atgtactatt taaaaaacac aaacttttgg atgttcggtt tattcttttt cttttacttt
60
tttatcatgg gagcctactt cccgtttttc ccgatttggc tacatgacat caaccatatc
120
agcaaaagtg atacgggtat tatttttgcc gctatttctc tgttctcgct attattccaa
180
ccgctgtttg gtctgctttc tgacaaactc gggctgcgca aatacctgct gtggattatt
240
accggcatgt tagtgatgtt tgcgccgttc tttattttta tcttcgggcc actgttacaa
300
tacaacattt tagtaggatc gattgttggt ggtatttatc taggcttttg ttttaacgcc
360
ggtgcgccag cagtagaggc atttattgag aaagtcagcc gtcgcagtaa tttcgaattt
420
ggtcgcgcgc ggatgtttgg ctgtgttggc tgggcgctgt gtgcctcgat tgtcggcatc
480
atgttcacca tcaataatca gtttgttttc tggctgggct ctggctgtgc actcatcctc
540
gccgttttac tctttttcgc caaaacggat gcgccctctt ctgccacggt tgccaatgcg
600
gtaggtgcca accattcggc atttagcctt aagctggcac tggaactgtt cagacagcca
660
aaactgtggt ttttgtcact gtatgttatt ggcgtttcct gcacctacga tgtttttgac
720
caacagtttg ctaatttctt tacttcgttc tttgctaccg gtgaacaggg tacgcgggta
780
tttggctacg taacgacaat gggcgaatta cttaacgcct cgattatgtt ctttgcgcca
840
ctgatcatta atcgcatcgg tgggaaaaac gccctgctgc tggctggcac tattatgtct
900
gtacgtatta ttggctcatc gttcgccacc tcagcgctgg aagtggttat tctgaaaacg
960
ctgcatatgt ttgaagtacc gttcctgctg gtgggctgct ttaaatatat taccagccag
1020
tttgaagtgc gtttttcagc gacgatttat ctggtctgtt tctgcttctt taagcaactg
1080
gcgatgattt ttatgtctgt actggcgggc aatatgtatg aaagcatcgg tttccagggc
1140
gcttatctgg tgctgggtct ggtggcgctg ggcttcacct taatttccgt gttcacgctt
1200
agcggccccg gcccgctttc cctgctgcgt cgtcaggtga atgaagtcgc ttaa
1254
<210> 63
<211> 417
<212> ПРТ
<213> Escherichia coli
<400> 63
Met Tyr Tyr Leu Lys Asn Thr Asn Phe Trp Met Phe Gly Leu Phe Phe
1 5 10 15
Phe Phe Tyr Phe Phe Ile Met Gly Ala Tyr Phe Pro Phe Phe Pro Ile
20 25 30
Trp Leu His Asp Ile Asn His Ile Ser Lys Ser Asp Thr Gly Ile Ile
35 40 45
Phe Ala Ala Ile Ser Leu Phe Ser Leu Leu Phe Gln Pro Leu Phe Gly
50 55 60
Leu Leu Ser Asp Lys Leu Gly Leu Arg Lys Tyr Leu Leu Trp Ile Ile
65 70 75 80
Thr Gly Met Leu Val Met Phe Ala Pro Phe Phe Ile Phe Ile Phe Gly
85 90 95
Pro Leu Leu Gln Tyr Asn Ile Leu Val Gly Ser Ile Val Gly Gly Ile
100 105 110
Tyr Leu Gly Phe Cys Phe Asn Ala Gly Ala Pro Ala Val Glu Ala Phe
115 120 125
Ile Glu Lys Val Ser Arg Arg Ser Asn Phe Glu Phe Gly Arg Ala Arg
130 135 140
Met Phe Gly Cys Val Gly Trp Ala Leu Cys Ala Ser Ile Val Gly Ile
145 150 155 160
Met Phe Thr Ile Asn Asn Gln Phe Val Phe Trp Leu Gly Ser Gly Cys
165 170 175
Ala Leu Ile Leu Ala Val Leu Leu Phe Phe Ala Lys Thr Asp Ala Pro
180 185 190
Ser Ser Ala Thr Val Ala Asn Ala Val Gly Ala Asn His Ser Ala Phe
195 200 205
Ser Leu Lys Leu Ala Leu Glu Leu Phe Arg Gln Pro Lys Leu Trp Phe
210 215 220
Leu Ser Leu Tyr Val Ile Gly Val Ser Cys Thr Tyr Asp Val Phe Asp
225 230 235 240
Gln Gln Phe Ala Asn Phe Phe Thr Ser Phe Phe Ala Thr Gly Glu Gln
245 250 255
Gly Thr Arg Val Phe Gly Tyr Val Thr Thr Met Gly Glu Leu Leu Asn
260 265 270
Ala Ser Ile Met Phe Phe Ala Pro Leu Ile Ile Asn Arg Ile Gly Gly
275 280 285
Lys Asn Ala Leu Leu Leu Ala Gly Thr Ile Met Ser Val Arg Ile Ile
290 295 300
Gly Ser Ser Phe Ala Thr Ser Ala Leu Glu Val Val Ile Leu Lys Thr
305 310 315 320
Leu His Met Phe Glu Val Pro Phe Leu Leu Val Gly Cys Phe Lys Tyr
325 330 335
Ile Thr Ser Gln Phe Glu Val Arg Phe Ser Ala Thr Ile Tyr Leu Val
340 345 350
Cys Phe Cys Phe Phe Lys Gln Leu Ala Met Ile Phe Met Ser Val Leu
355 360 365
Ala Gly Asn Met Tyr Glu Ser Ile Gly Phe Gln Gly Ala Tyr Leu Val
370 375 380
Leu Gly Leu Val Ala Leu Gly Phe Thr Leu Ile Ser Val Phe Thr Leu
385 390 395 400
Ser Gly Pro Gly Pro Leu Ser Leu Leu Arg Arg Gln Val Asn Glu Val
405 410 415
Ala
<210> 64
<211> 3075
<212> ДНК
<213> Escherichia coli
<400> 64
atgaccatga ttacggattc actggccgtc gttttacaac gtcgtgactg ggaaaaccct
60
ggcgttaccc aacttaatcg ccttgcagca catccccctt tcgccagctg gcgtaatagc
120
gaagaggccc gcaccgatcg cccttcccaa cagttgcgca gcctgaatgg cgaatggcgc
180
tttgcctggt ttccggcacc agaagcggtg ccggaaagct ggctggagtg cgatcttcct
240
gaggccgata ctgtcgtcgt cccctcaaac tggcagatgc acggttacga tgcgcccatc
300
tacaccaacg tgacctatcc cattacggtc aatccgccgt ttgttcccac ggagaatccg
360
acgggttgtt actcgctcac atttaatgtt gatgaaagct ggctacagga aggccagacg
420
cgaattattt ttgatggcgt taactcggcg tttcatctgt ggtgcaacgg gcgctgggtc
480
ggttacggcc aggacagtcg tttgccgtct gaatttgacc tgagcgcatt tttacgcgcc
540
ggagaaaacc gcctcgcggt gatggtgctg cgctggagtg acggcagtta tctggaagat
600
caggatatgt ggcggatgag cggcattttc cgtgacgtct cgttgctgca taaaccgact
660
acacaaatca gcgatttcca tgttgccact cgctttaatg atgatttcag ccgcgctgta
720
ctggaggctg aagttcagat gtgcggcgag ttgcgtgact acctacgggt aacagtttct
780
ttatggcagg gtgaaacgca ggtcgccagc ggcaccgcgc ctttcggcgg tgaaattatc
840
gatgagcgtg gtggttatgc cgatcgcgtc acactacgtc tgaacgtcga aaacccgaaa
900
ctgtggagcg ccgaaatccc gaatctctat cgtgcggtgg ttgaactgca caccgccgac
960
ggcacgctga ttgaagcaga agcctgcgat gtcggtttcc gcgaggtgcg gattgaaaat
1020
ggtctgctgc tgctgaacgg caagccgttg ctgattcgag gcgttaaccg tcacgagcat
1080
catcctctgc atggtcaggt catggatgag cagacgatgg tgcaggatat cctgctgatg
1140
aagcagaaca actttaacgc cgtgcgctgt tcgcattatc cgaaccatcc gctgtggtac
1200
acgctgtgcg accgctacgg cctgtatgtg gtggatgaag ccaatattga aacccacggc
1260
atggtgccaa tgaatcgtct gaccgatgat ccgcgctggc taccggcgat gagcgaacgc
1320
gtaacgcgaa tggtgcagcg cgatcgtaat cacccgagtg tgatcatctg gtcgctgggg
1380
aatgaatcag gccacggcgc taatcacgac gcgctgtatc gctggatcaa atctgtcgat
1440
ccttcccgcc cggtgcagta tgaaggcggc ggagccgaca ccacggccac cgatattatt
1500
tgcccgatgt acgcgcgcgt ggatgaagac cagcccttcc cggctgtgcc gaaatggtcc
1560
atcaaaaaat ggctttcgct acctggagag acgcgcccgc tgatcctttg cgaatacgcc
1620
cacgcgatgg gtaacagtct tggcggtttc gctaaatact ggcaggcgtt tcgtcagtat
1680
ccccgtttac agggcggctt cgtctgggac tgggtggatc agtcgctgat taaatatgat
1740
gaaaacggca acccgtggtc ggcttacggc ggtgattttg gcgatacgcc gaacgatcgc
1800
cagttctgta tgaacggtct ggtctttgcc gaccgcacgc cgcatccagc gctgacggaa
1860
gcaaaacacc agcagcagtt tttccagttc cgtttatccg ggcaaaccat cgaagtgacc
1920
agcgaatacc tgttccgtca tagcgataac gagctcctgc actggatggt ggcgctggat
1980
ggtaagccgc tggcaagcgg tgaagtgcct ctggatgtcg ctccacaagg taaacagttg
2040
attgaactgc ctgaactacc gcagccggag agcgccgggc aactctggct cacagtacgc
2100
gtagtgcaac cgaacgcgac cgcatggtca gaagccgggc acatcagcgc ctggcagcag
2160
tggcgtctgg cggaaaacct cagtgtgacg ctccccgccg cgtcccacgc catcccgcat
2220
ctgaccacca gcgaaatgga tttttgcatc gagctgggta ataagcgttg gcaatttaac
2280
cgccagtcag gctttctttc acagatgtgg attggcgata aaaaacaact gctgacgccg
2340
ctgcgcgatc agttcacccg tgcaccgctg gataacgaca ttggcgtaag tgaagcgacc
2400
cgcattgacc ctaacgcctg ggtcgaacgc tggaaggcgg cgggccatta ccaggccgaa
2460
gcagcgttgt tgcagtgcac ggcagataca cttgctgatg cggtgctgat tacgaccgct
2520
cacgcgtggc agcatcaggg gaaaacctta tttatcagcc ggaaaaccta ccggattgat
2580
ggtagtggtc aaatggcgat taccgttgat gttgaagtgg cgagcgatac accgcatccg
2640
gcgcggattg gcctgaactg ccagctggcg caggtagcag agcgggtaaa ctggctcgga
2700
ttagggccgc aagaaaacta tcccgaccgc cttactgccg cctgttttga ccgctgggat
2760
ctgccattgt cagacatgta taccccgtac gtcttcccga gcgaaaacgg tctgcgctgc
2820
gggacgcgcg aattgaatta tggcccacac cagtggcgcg gcgacttcca gttcaacatc
2880
agccgctaca gtcaacagca actgatggaa accagccatc gccatctgct gcacgcggaa
2940
gaaggcacat ggctgaatat cgacggtttc catatgggga ttggtggcga cgactcctgg
3000
agcccgtcag tatcggcgga attccagctg agcgccggtc gctaccatta ccagttggtc
3060
tggtgtcaaa aataa
3075
<210> 65
<211> 1024
<212> ПРТ
<213> Escherichia coli
<400> 65
Met Thr Met Ile Thr Asp Ser Leu Ala Val Val Leu Gln Arg Arg Asp
1 5 10 15
Trp Glu Asn Pro Gly Val Thr Gln Leu Asn Arg Leu Ala Ala His Pro
20 25 30
Pro Phe Ala Ser Trp Arg Asn Ser Glu Glu Ala Arg Thr Asp Arg Pro
35 40 45
Ser Gln Gln Leu Arg Ser Leu Asn Gly Glu Trp Arg Phe Ala Trp Phe
50 55 60
Pro Ala Pro Glu Ala Val Pro Glu Ser Trp Leu Glu Cys Asp Leu Pro
65 70 75 80
Glu Ala Asp Thr Val Val Val Pro Ser Asn Trp Gln Met His Gly Tyr
85 90 95
Asp Ala Pro Ile Tyr Thr Asn Val Thr Tyr Pro Ile Thr Val Asn Pro
100 105 110
Pro Phe Val Pro Thr Glu Asn Pro Thr Gly Cys Tyr Ser Leu Thr Phe
115 120 125
Asn Val Asp Glu Ser Trp Leu Gln Glu Gly Gln Thr Arg Ile Ile Phe
130 135 140
Asp Gly Val Asn Ser Ala Phe His Leu Trp Cys Asn Gly Arg Trp Val
145 150 155 160
Gly Tyr Gly Gln Asp Ser Arg Leu Pro Ser Glu Phe Asp Leu Ser Ala
165 170 175
Phe Leu Arg Ala Gly Glu Asn Arg Leu Ala Val Met Val Leu Arg Trp
180 185 190
Ser Asp Gly Ser Tyr Leu Glu Asp Gln Asp Met Trp Arg Met Ser Gly
195 200 205
Ile Phe Arg Asp Val Ser Leu Leu His Lys Pro Thr Thr Gln Ile Ser
210 215 220
Asp Phe His Val Ala Thr Arg Phe Asn Asp Asp Phe Ser Arg Ala Val
225 230 235 240
Leu Glu Ala Glu Val Gln Met Cys Gly Glu Leu Arg Asp Tyr Leu Arg
245 250 255
Val Thr Val Ser Leu Trp Gln Gly Glu Thr Gln Val Ala Ser Gly Thr
260 265 270
Ala Pro Phe Gly Gly Glu Ile Ile Asp Glu Arg Gly Gly Tyr Ala Asp
275 280 285
Arg Val Thr Leu Arg Leu Asn Val Glu Asn Pro Lys Leu Trp Ser Ala
290 295 300
Glu Ile Pro Asn Leu Tyr Arg Ala Val Val Glu Leu His Thr Ala Asp
305 310 315 320
Gly Thr Leu Ile Glu Ala Glu Ala Cys Asp Val Gly Phe Arg Glu Val
325 330 335
Arg Ile Glu Asn Gly Leu Leu Leu Leu Asn Gly Lys Pro Leu Leu Ile
340 345 350
Arg Gly Val Asn Arg His Glu His His Pro Leu His Gly Gln Val Met
355 360 365
Asp Glu Gln Thr Met Val Gln Asp Ile Leu Leu Met Lys Gln Asn Asn
370 375 380
Phe Asn Ala Val Arg Cys Ser His Tyr Pro Asn His Pro Leu Trp Tyr
385 390 395 400
Thr Leu Cys Asp Arg Tyr Gly Leu Tyr Val Val Asp Glu Ala Asn Ile
405 410 415
Glu Thr His Gly Met Val Pro Met Asn Arg Leu Thr Asp Asp Pro Arg
420 425 430
Trp Leu Pro Ala Met Ser Glu Arg Val Thr Arg Met Val Gln Arg Asp
435 440 445
Arg Asn His Pro Ser Val Ile Ile Trp Ser Leu Gly Asn Glu Ser Gly
450 455 460
His Gly Ala Asn His Asp Ala Leu Tyr Arg Trp Ile Lys Ser Val Asp
465 470 475 480
Pro Ser Arg Pro Val Gln Tyr Glu Gly Gly Gly Ala Asp Thr Thr Ala
485 490 495
Thr Asp Ile Ile Cys Pro Met Tyr Ala Arg Val Asp Glu Asp Gln Pro
500 505 510
Phe Pro Ala Val Pro Lys Trp Ser Ile Lys Lys Trp Leu Ser Leu Pro
515 520 525
Gly Glu Thr Arg Pro Leu Ile Leu Cys Glu Tyr Ala His Ala Met Gly
530 535 540
Asn Ser Leu Gly Gly Phe Ala Lys Tyr Trp Gln Ala Phe Arg Gln Tyr
545 550 555 560
Pro Arg Leu Gln Gly Gly Phe Val Trp Asp Trp Val Asp Gln Ser Leu
565 570 575
Ile Lys Tyr Asp Glu Asn Gly Asn Pro Trp Ser Ala Tyr Gly Gly Asp
580 585 590
Phe Gly Asp Thr Pro Asn Asp Arg Gln Phe Cys Met Asn Gly Leu Val
595 600 605
Phe Ala Asp Arg Thr Pro His Pro Ala Leu Thr Glu Ala Lys His Gln
610 615 620
Gln Gln Phe Phe Gln Phe Arg Leu Ser Gly Gln Thr Ile Glu Val Thr
625 630 635 640
Ser Glu Tyr Leu Phe Arg His Ser Asp Asn Glu Leu Leu His Trp Met
645 650 655
Val Ala Leu Asp Gly Lys Pro Leu Ala Ser Gly Glu Val Pro Leu Asp
660 665 670
Val Ala Pro Gln Gly Lys Gln Leu Ile Glu Leu Pro Glu Leu Pro Gln
675 680 685
Pro Glu Ser Ala Gly Gln Leu Trp Leu Thr Val Arg Val Val Gln Pro
690 695 700
Asn Ala Thr Ala Trp Ser Glu Ala Gly His Ile Ser Ala Trp Gln Gln
705 710 715 720
Trp Arg Leu Ala Glu Asn Leu Ser Val Thr Leu Pro Ala Ala Ser His
725 730 735
Ala Ile Pro His Leu Thr Thr Ser Glu Met Asp Phe Cys Ile Glu Leu
740 745 750
Gly Asn Lys Arg Trp Gln Phe Asn Arg Gln Ser Gly Phe Leu Ser Gln
755 760 765
Met Trp Ile Gly Asp Lys Lys Gln Leu Leu Thr Pro Leu Arg Asp Gln
770 775 780
Phe Thr Arg Ala Pro Leu Asp Asn Asp Ile Gly Val Ser Glu Ala Thr
785 790 795 800
Arg Ile Asp Pro Asn Ala Trp Val Glu Arg Trp Lys Ala Ala Gly His
805 810 815
Tyr Gln Ala Glu Ala Ala Leu Leu Gln Cys Thr Ala Asp Thr Leu Ala
820 825 830
Asp Ala Val Leu Ile Thr Thr Ala His Ala Trp Gln His Gln Gly Lys
835 840 845
Thr Leu Phe Ile Ser Arg Lys Thr Tyr Arg Ile Asp Gly Ser Gly Gln
850 855 860
Met Ala Ile Thr Val Asp Val Glu Val Ala Ser Asp Thr Pro His Pro
865 870 875 880
Ala Arg Ile Gly Leu Asn Cys Gln Leu Ala Gln Val Ala Glu Arg Val
885 890 895
Asn Trp Leu Gly Leu Gly Pro Gln Glu Asn Tyr Pro Asp Arg Leu Thr
900 905 910
Ala Ala Cys Phe Asp Arg Trp Asp Leu Pro Leu Ser Asp Met Tyr Thr
915 920 925
Pro Tyr Val Phe Pro Ser Glu Asn Gly Leu Arg Cys Gly Thr Arg Glu
930 935 940
Leu Asn Tyr Gly Pro His Gln Trp Arg Gly Asp Phe Gln Phe Asn Ile
945 950 955 960
Ser Arg Tyr Ser Gln Gln Gln Leu Met Glu Thr Ser His Arg His Leu
965 970 975
Leu His Ala Glu Glu Gly Thr Trp Leu Asn Ile Asp Gly Phe His Met
980 985 990
Gly Ile Gly Gly Asp Asp Ser Trp Ser Pro Ser Val Ser Ala Glu Phe
995 1000 1005
Gln Leu Ser Ala Gly Arg Tyr His Tyr Gln Leu Val Trp Cys Gln
1010 1015 1020
Lys
<210> 66
<211> 1119
<212> ДНК
<213> Staphylococcus epidermidis
<400> 66
atgactaaac tccatatctt ttacttttct ttaatgtact ttcttattgg catgatacac
60
acttttgtcg gttcatttaa tcaattctta aaaatagaac ttaatatgaa tcaatcagat
120
gtatcaaatc taattagtat tcagttcata acatttatga ttggagtatt ttattctacc
180
ttcttagtta ataaagacat aaagaatttt ttaaaaataa tacatttatt tattctatta
240
attactacta cttttattat atttgaacat tacttaataa tatatctgat tgtagctata
300
ttaggttttt gcgctggatt tattgaatca tctatcgcat catatatttt taatagtaag
360
tttgagtccg ctaaaacttt tggatatata gaatcatttt ttgcagtagg gtcatttttg
420
ctccctgtaa ttgtgaaagt gtttgaatac cattcagata caaaacacgc tattatcttt
480
atacttataa taaatataat cttattttta attatctatt cattagagtt tgaagtaagt
540
agcagcgata gaaataaaat acccatactg tcttttaata aaaagtcaat gctagttatg
600
attattttta catggtgttt cttttatatt agtatagaaa caaatttttc aaatttacta
660
ccatatatca acttagtttc tgaaaaatat agctatatta ctgtaagtat attttgggtt
720
ggaataatta taggaaggtt tttatatacg ctaatattga cgttaattag attcaggcta
780
gaatcattac tattaacata tacagtgaca tctttttttc tatatattat tttaatatat
840
ttgaatactc aagatgaagt caaattaata attttgtttt tacttactct attcttagca
900
cctatgttcc ctttaggtgt tagtatcatt aatcaacata gtagtaataa gaacttacta
960
actagtattt ttattgctgt agctggatgt ggtggtgcag ttggtgcagt aattataaaa
1020
tcagctttat acattcatat tcccgtacat ttatctattt tattaatatt aatgacttgc
1080
ttatttttaa gtactgtgat tttaaaaata aagctttaa
1119
<210> 67
<211> 372
<212> ПРТ
<213> Staphylococcus epidermidis
<400> 67
Met Thr Lys Leu His Ile Phe Tyr Phe Ser Leu Met Tyr Phe Leu Ile
1 5 10 15
Gly Met Ile His Thr Phe Val Gly Ser Phe Asn Gln Phe Leu Lys Ile
20 25 30
Glu Leu Asn Met Asn Gln Ser Asp Val Ser Asn Leu Ile Ser Ile Gln
35 40 45
Phe Ile Thr Phe Met Ile Gly Val Phe Tyr Ser Thr Phe Leu Val Asn
50 55 60
Lys Asp Ile Lys Asn Phe Leu Lys Ile Ile His Leu Phe Ile Leu Leu
65 70 75 80
Ile Thr Thr Thr Phe Ile Ile Phe Glu His Tyr Leu Ile Ile Tyr Leu
85 90 95
Ile Val Ala Ile Leu Gly Phe Cys Ala Gly Phe Ile Glu Ser Ser Ile
100 105 110
Ala Ser Tyr Ile Phe Asn Ser Lys Phe Glu Ser Ala Lys Thr Phe Gly
115 120 125
Tyr Ile Glu Ser Phe Phe Ala Val Gly Ser Phe Leu Leu Pro Val Ile
130 135 140
Val Lys Val Phe Glu Tyr His Ser Asp Thr Lys His Ala Ile Ile Phe
145 150 155 160
Ile Leu Ile Ile Asn Ile Ile Leu Phe Leu Ile Ile Tyr Ser Leu Glu
165 170 175
Phe Glu Val Ser Ser Ser Asp Arg Asn Lys Ile Pro Ile Leu Ser Phe
180 185 190
Asn Lys Lys Ser Met Leu Val Met Ile Ile Phe Thr Trp Cys Phe Phe
195 200 205
Tyr Ile Ser Ile Glu Thr Asn Phe Ser Asn Leu Leu Pro Tyr Ile Asn
210 215 220
Leu Val Ser Glu Lys Tyr Ser Tyr Ile Thr Val Ser Ile Phe Trp Val
225 230 235 240
Gly Ile Ile Ile Gly Arg Phe Leu Tyr Thr Leu Ile Leu Thr Leu Ile
245 250 255
Arg Phe Arg Leu Glu Ser Leu Leu Leu Thr Tyr Thr Val Thr Ser Phe
260 265 270
Phe Leu Tyr Ile Ile Leu Ile Tyr Leu Asn Thr Gln Asp Glu Val Lys
275 280 285
Leu Ile Ile Leu Phe Leu Leu Thr Leu Phe Leu Ala Pro Met Phe Pro
290 295 300
Leu Gly Val Ser Ile Ile Asn Gln His Ser Ser Asn Lys Asn Leu Leu
305 310 315 320
Thr Ser Ile Phe Ile Ala Val Ala Gly Cys Gly Gly Ala Val Gly Ala
325 330 335
Val Ile Ile Lys Ser Ala Leu Tyr Ile His Ile Pro Val His Leu Ser
340 345 350
Ile Leu Leu Ile Leu Met Thr Cys Leu Phe Leu Ser Thr Val Ile Leu
355 360 365
Lys Ile Lys Leu
370
<210> 68
<211> 1350
<212> ДНК
<213> Lactobacillus brevis
<400> 68
atgcaagcaa ctgaaaccaa gcacggctgg acccaattag cggacggcta cctcagcaaa
60
acgccattat ttcaatttat tttagtttca ttgatttttc cactgtgggg aactgcggca
120
agtttaaatg atattttgat tacgcagttc aagacagtct ttcaacttaa cgatgccgcg
180
acggcctttg ttcaaagtgc cttctatggt gggtatttct taattgccat tccggcatcc
240
ctgattatta agaagaacag ttataaattt gccatcatga ccgggttgat cttttatatc
300
atcgggtgtg ggctgttttt cccggcctca catctcgcaa cttacagtat gttcctggtg
360
gccatctttg ccattgccat tggtctgagc ttcttggaaa catcatgtga tacgtatagt
420
tcaatgctgg gaccgaagca acacgccacg atgcgcttga acttttccca gacattaatt
480
ccgttaggcg acatcatggg aattgtttta gggaagtact taatttttgg ttctgtaggt
540
aatttatctg aaaagatgag ccatatgcac ggcgcagcac gcattgctta cggcgaacag
600
atgttacaat tgacgttacg gccttacaaa tatatcttaa tcgtgttact cgtgatgctg
660
attatctttg ccgtaacgcc tatgccacgg gctaaggcga cgaaggaaat tggtggggaa
720
caacaagaag aacgtcctag tcttggggaa actctgaagt atctatcaca caacaagcac
780
tatattaaag gggtagtaac ccagttcttt tatgcgggtc tgcaaacaac cgtctggtcc
840
tttacgattc gtttggtatt aaacttgaac catcaaatta ccgacagcgg tgcatcaacc
900
tttatgattt atagttatgt ggcgtggttc gttggtaagc tggttgccaa tacctttatg
960
agtcgcttct caattacgaa ggtgctgacg tggtactcct tattggggac attagcatta
1020
gttgtgacct ttacggttcc gaatatgatt gcggtctacg cagccatctt aacgagtttc
1080
ttctttggtc cagaatggcc aacaatttat gcgcacacgt tggatgccgt tacggagaag
1140
aaatacactg aaacggctgg ggcaattatc gtgatggccc tgatcggtgg tgcagtcatt
1200
ccagccattc aaggcctggt ttctgatgcg accggttcaa tgcagttctc attcgttgta
1260
ccaatgctct gctacgcttt aattacaggg tactttttct tcgaacatcg ttttgagaaa
1320
gctcacccta acgaagttca agaacattaa
1350
<210> 69
<211> 449
<212> ПРТ
<213> Lactobacillus brevis
<400> 69
Met Gln Ala Thr Glu Thr Lys His Gly Trp Thr Gln Leu Ala Asp Gly
1 5 10 15
Tyr Leu Ser Lys Thr Pro Leu Phe Gln Phe Ile Leu Val Ser Leu Ile
20 25 30
Phe Pro Leu Trp Gly Thr Ala Ala Ser Leu Asn Asp Ile Leu Ile Thr
35 40 45
Gln Phe Lys Thr Val Phe Gln Leu Asn Asp Ala Ala Thr Ala Phe Val
50 55 60
Gln Ser Ala Phe Tyr Gly Gly Tyr Phe Leu Ile Ala Ile Pro Ala Ser
65 70 75 80
Leu Ile Ile Lys Lys Asn Ser Tyr Lys Phe Ala Ile Met Thr Gly Leu
85 90 95
Ile Phe Tyr Ile Ile Gly Cys Gly Leu Phe Phe Pro Ala Ser His Leu
100 105 110
Ala Thr Tyr Ser Met Phe Leu Val Ala Ile Phe Ala Ile Ala Ile Gly
115 120 125
Leu Ser Phe Leu Glu Thr Ser Cys Asp Thr Tyr Ser Ser Met Leu Gly
130 135 140
Pro Lys Gln His Ala Thr Met Arg Leu Asn Phe Ser Gln Thr Leu Ile
145 150 155 160
Pro Leu Gly Asp Ile Met Gly Ile Val Leu Gly Lys Tyr Leu Ile Phe
165 170 175
Gly Ser Val Gly Asn Leu Ser Glu Lys Met Ser His Met His Gly Ala
180 185 190
Ala Arg Ile Ala Tyr Gly Glu Gln Met Leu Gln Leu Thr Leu Arg Pro
195 200 205
Tyr Lys Tyr Ile Leu Ile Val Leu Leu Val Met Leu Ile Ile Phe Ala
210 215 220
Val Thr Pro Met Pro Arg Ala Lys Ala Thr Lys Glu Ile Gly Gly Glu
225 230 235 240
Gln Gln Glu Glu Arg Pro Ser Leu Gly Glu Thr Leu Lys Tyr Leu Ser
245 250 255
His Asn Lys His Tyr Ile Lys Gly Val Val Thr Gln Phe Phe Tyr Ala
260 265 270
Gly Leu Gln Thr Thr Val Trp Ser Phe Thr Ile Arg Leu Val Leu Asn
275 280 285
Leu Asn His Gln Ile Thr Asp Ser Gly Ala Ser Thr Phe Met Ile Tyr
290 295 300
Ser Tyr Val Ala Trp Phe Val Gly Lys Leu Val Ala Asn Thr Phe Met
305 310 315 320
Ser Arg Phe Ser Ile Thr Lys Val Leu Thr Trp Tyr Ser Leu Leu Gly
325 330 335
Thr Leu Ala Leu Val Val Thr Phe Thr Val Pro Asn Met Ile Ala Val
340 345 350
Tyr Ala Ala Ile Leu Thr Ser Phe Phe Phe Gly Pro Glu Trp Pro Thr
355 360 365
Ile Tyr Ala His Thr Leu Asp Ala Val Thr Glu Lys Lys Tyr Thr Glu
370 375 380
Thr Ala Gly Ala Ile Ile Val Met Ala Leu Ile Gly Gly Ala Val Ile
385 390 395 400
Pro Ala Ile Gln Gly Leu Val Ser Asp Ala Thr Gly Ser Met Gln Phe
405 410 415
Ser Phe Val Val Pro Met Leu Cys Tyr Ala Leu Ile Thr Gly Tyr Phe
420 425 430
Phe Phe Glu His Arg Phe Glu Lys Ala His Pro Asn Glu Val Gln Glu
435 440 445
His
<210> 70
<211> 2379
<212> ДНК
<213> Escherichia coli
<400> 70
atgtccaaca atggctcgtc accgctggtg ctttggtata accaactcgg catgaatgat
60
gtagacaggg ttgggggcaa aaatgcctcc ctgggtgaaa tgattactaa tctttccgga
120
atgggtgttt ccgttccgaa tggtttcgcc acaaccgccg acgcgtttaa ccagtttctg
180
gaccaaagcg gcgtaaacca gcgcatttat gaactgctgg ataaaacgga tattgacgat
240
gttactcagc ttgcgaaagc gggcgcgcaa atccgccagt ggattatcga cactcccttc
300
cagcctgagc tggaaaacgc catccgcgaa gcctatgcac agctttccgc cgatgacgaa
360
aacgcctctt ttgcggtgcg ctcctccgcc accgcagaag atatgccgga cgcttctttt
420
gccggtcagc aggaaacctt cctcaacgtt cagggttttg acgccgttct cgtggcagtg
480
aaacatgtat ttgcttctct gtttaacgat cgcgccatct cttatcgtgt gcaccagggt
540
tacgatcacc gtggtgtggc gctctccgcc ggtgttcaac ggatggtgcg ctctgacctc
600
gcatcatctg gcgtgatgtt ctccattgat accgaatccg gctttgacca ggtggtgttt
660
atcacttccg catggggcct tggtgagatg gtcgtgcagg gtgcggttaa cccggatgag
720
ttttacgtgc ataaaccgac actggcggcg aatcgcccgg ctatcgtgcg ccgcaccatg
780
gggtcgaaaa aaatccgcat ggtttacgcg ccgacccagg agcacggcaa gcaggttaaa
840
atcgaagacg taccgcagga acagcgtgac atcttctcgc tgaccaacga agaagtgcag
900
gaactggcaa aacaggccgt acaaattgag aaacactacg gtcgcccgat ggatattgag
960
tgggcgaaag atggccacac cggtaaactg ttcattgtgc aggcgcgtcc ggaaaccgtg
1020
cgctcacgcg gtcaggtcat ggagcgttat acgctgcatt cacagggtaa gattatcgcc
1080
gaaggccgtg ctatcggtca tcgcatcggt gcgggtccgg tgaaagtcat ccatgacatc
1140
agcgaaatga accgcatcga acctggcgac gtgctggtta ctgacatgac cgacccggac
1200
tgggaaccga tcatgaagaa agcatctgcc atcgtcacca accgtggcgg tcgtacctgt
1260
cacgcggcga tcatcgctcg tgaactgggc attccggcgg tagtgggctg tggagatgca
1320
acagaacgga tgaaagacgg tgagaacgtc actgtttctt gtgccgaagg tgataccggt
1380
tacgtctatg cggagttgct ggaatttagc gtgaaaagct ccagcgtaga aacgatgccg
1440
gatctgccgt tgaaagtgat gatgaacgtc ggtaacccgg accgtgcttt cgacttcgcc
1500
tgcctaccga acgaaggcgt gggccttgcg cgtctggaat ttatcatcaa ccgtatgatt
1560
ggcgtccacc cacgcgcact gcttgagttt gacgatcagg aaccgcagtt gcaaaacgaa
1620
atccgcgaga tgatgaaagg ttttgattct ccgcgtgaat tttacgttgg tcgtctgact
1680
gaagggatcg cgacgctggg tgccgcgttt tatccgaagc gcgtcattgt ccgtctctct
1740
gattttaaat cgaacgaata tgccaacctg gtcggtggtg agcgttacga gccagatgaa
1800
gagaacccga tgctcggctt ccgtggcgcg ggccgctatg tttccgacag cttccgcgac
1860
tgtttcgcgc tggagtgtga agcagtgaaa cgtgtgcgca acgacatggg actgaccaac
1920
gttgagatca tgatcccgtt cgtgcgtacc gtagatcagg cgaaagcggt ggttgaagaa
1980
ctggcgcgtc aggggctgaa acgtggcgag aacgggctga aaatcatcat gatgtgtgaa
2040
atcccgtcca acgccttgct ggccgagcag ttcctcgaat atttcgacgg cttctcaatt
2100
ggctcaaacg atatgacgca gctggcgctc ggtctggacc gtgactccgg cgtggtgtct
2160
gaattgttcg atgagcgcaa cgatgcggtg aaagcactgc tgtcgatggc tatccgtgcc
2220
gcgaagaaac agggcaaata tgtcgggatt tgcggtcagg gtccgtccga ccacgaagac
2280
tttgccgcat ggttgatgga agaggggatc gatagcctgt ctctgaaccc ggacaccgtg
2340
gtgcaaacct ggttaagcct ggctgaactg aagaaataa
2379
<210> 71
<211> 792
<212> ПРТ
<213> Escherichia coli
<400> 71
Met Ser Asn Asn Gly Ser Ser Pro Leu Val Leu Trp Tyr Asn Gln Leu
1 5 10 15
Gly Met Asn Asp Val Asp Arg Val Gly Gly Lys Asn Ala Ser Leu Gly
20 25 30
Glu Met Ile Thr Asn Leu Ser Gly Met Gly Val Ser Val Pro Asn Gly
35 40 45
Phe Ala Thr Thr Ala Asp Ala Phe Asn Gln Phe Leu Asp Gln Ser Gly
50 55 60
Val Asn Gln Arg Ile Tyr Glu Leu Leu Asp Lys Thr Asp Ile Asp Asp
65 70 75 80
Val Thr Gln Leu Ala Lys Ala Gly Ala Gln Ile Arg Gln Trp Ile Ile
85 90 95
Asp Thr Pro Phe Gln Pro Glu Leu Glu Asn Ala Ile Arg Glu Ala Tyr
100 105 110
Ala Gln Leu Ser Ala Asp Asp Glu Asn Ala Ser Phe Ala Val Arg Ser
115 120 125
Ser Ala Thr Ala Glu Asp Met Pro Asp Ala Ser Phe Ala Gly Gln Gln
130 135 140
Glu Thr Phe Leu Asn Val Gln Gly Phe Asp Ala Val Leu Val Ala Val
145 150 155 160
Lys His Val Phe Ala Ser Leu Phe Asn Asp Arg Ala Ile Ser Tyr Arg
165 170 175
Val His Gln Gly Tyr Asp His Arg Gly Val Ala Leu Ser Ala Gly Val
180 185 190
Gln Arg Met Val Arg Ser Asp Leu Ala Ser Ser Gly Val Met Phe Ser
195 200 205
Ile Asp Thr Glu Ser Gly Phe Asp Gln Val Val Phe Ile Thr Ser Ala
210 215 220
Trp Gly Leu Gly Glu Met Val Val Gln Gly Ala Val Asn Pro Asp Glu
225 230 235 240
Phe Tyr Val His Lys Pro Thr Leu Ala Ala Asn Arg Pro Ala Ile Val
245 250 255
Arg Arg Thr Met Gly Ser Lys Lys Ile Arg Met Val Tyr Ala Pro Thr
260 265 270
Gln Glu His Gly Lys Gln Val Lys Ile Glu Asp Val Pro Gln Glu Gln
275 280 285
Arg Asp Ile Phe Ser Leu Thr Asn Glu Glu Val Gln Glu Leu Ala Lys
290 295 300
Gln Ala Val Gln Ile Glu Lys His Tyr Gly Arg Pro Met Asp Ile Glu
305 310 315 320
Trp Ala Lys Asp Gly His Thr Gly Lys Leu Phe Ile Val Gln Ala Arg
325 330 335
Pro Glu Thr Val Arg Ser Arg Gly Gln Val Met Glu Arg Tyr Thr Leu
340 345 350
His Ser Gln Gly Lys Ile Ile Ala Glu Gly Arg Ala Ile Gly His Arg
355 360 365
Ile Gly Ala Gly Pro Val Lys Val Ile His Asp Ile Ser Glu Met Asn
370 375 380
Arg Ile Glu Pro Gly Asp Val Leu Val Thr Asp Met Thr Asp Pro Asp
385 390 395 400
Trp Glu Pro Ile Met Lys Lys Ala Ser Ala Ile Val Thr Asn Arg Gly
405 410 415
Gly Arg Thr Cys His Ala Ala Ile Ile Ala Arg Glu Leu Gly Ile Pro
420 425 430
Ala Val Val Gly Cys Gly Asp Ala Thr Glu Arg Met Lys Asp Gly Glu
435 440 445
Asn Val Thr Val Ser Cys Ala Glu Gly Asp Thr Gly Tyr Val Tyr Ala
450 455 460
Glu Leu Leu Glu Phe Ser Val Lys Ser Ser Ser Val Glu Thr Met Pro
465 470 475 480
Asp Leu Pro Leu Lys Val Met Met Asn Val Gly Asn Pro Asp Arg Ala
485 490 495
Phe Asp Phe Ala Cys Leu Pro Asn Glu Gly Val Gly Leu Ala Arg Leu
500 505 510
Glu Phe Ile Ile Asn Arg Met Ile Gly Val His Pro Arg Ala Leu Leu
515 520 525
Glu Phe Asp Asp Gln Glu Pro Gln Leu Gln Asn Glu Ile Arg Glu Met
530 535 540
Met Lys Gly Phe Asp Ser Pro Arg Glu Phe Tyr Val Gly Arg Leu Thr
545 550 555 560
Glu Gly Ile Ala Thr Leu Gly Ala Ala Phe Tyr Pro Lys Arg Val Ile
565 570 575
Val Arg Leu Ser Asp Phe Lys Ser Asn Glu Tyr Ala Asn Leu Val Gly
580 585 590
Gly Glu Arg Tyr Glu Pro Asp Glu Glu Asn Pro Met Leu Gly Phe Arg
595 600 605
Gly Ala Gly Arg Tyr Val Ser Asp Ser Phe Arg Asp Cys Phe Ala Leu
610 615 620
Glu Cys Glu Ala Val Lys Arg Val Arg Asn Asp Met Gly Leu Thr Asn
625 630 635 640
Val Glu Ile Met Ile Pro Phe Val Arg Thr Val Asp Gln Ala Lys Ala
645 650 655
Val Val Glu Glu Leu Ala Arg Gln Gly Leu Lys Arg Gly Glu Asn Gly
660 665 670
Leu Lys Ile Ile Met Met Cys Glu Ile Pro Ser Asn Ala Leu Leu Ala
675 680 685
Glu Gln Phe Leu Glu Tyr Phe Asp Gly Phe Ser Ile Gly Ser Asn Asp
690 695 700
Met Thr Gln Leu Ala Leu Gly Leu Asp Arg Asp Ser Gly Val Val Ser
705 710 715 720
Glu Leu Phe Asp Glu Arg Asn Asp Ala Val Lys Ala Leu Leu Ser Met
725 730 735
Ala Ile Arg Ala Ala Lys Lys Gln Gly Lys Tyr Val Gly Ile Cys Gly
740 745 750
Gln Gly Pro Ser Asp His Glu Asp Phe Ala Ala Trp Leu Met Glu Glu
755 760 765
Gly Ile Asp Ser Leu Ser Leu Asn Pro Asp Thr Val Val Gln Thr Trp
770 775 780
Leu Ser Leu Ala Glu Leu Lys Lys
785 790
<210> 72
<211> 1623
<212> ДНК
<213> Escherichia coli
<400> 72
atgcgcgtta acaatggttt gaccccgcaa gaactcgagg cttatggtat cagtgacgta
60
catgatatcg tttacaaccc aagctacgac ctgctgtatc aggaagagct cgatccgagc
120
ctgacaggtt atgagcgcgg ggtgttaact aatctgggtg ccgttgccgt cgataccggg
180
atcttcaccg gtcgttcacc aaaagataag tatatcgtcc gtgacgatac cactcgcgat
240
actttctggt gggcagacaa aggcaaaggt aagaacgaca acaaacctct ctctccggaa
300
acctggcagc atctgaaagg cctggtgacc aggcagcttt ccggcaaacg tctgttcgtt
360
gtcgacgctt tctgtggtgc gaacccggat actcgtcttt ccgtccgttt catcaccgaa
420
gtggcctggc aggcgcattt tgtcaaaaac atgtttattc gcccgagcga tgaagaactg
480
gcaggtttca aaccagactt tatcgttatg aacggcgcga agtgcactaa cccgcagtgg
540
aaagaacagg gtctcaactc cgaaaacttc gtggcgttta acctgaccga gcgcatgcag
600
ctgattggcg gcacctggta cggcggcgaa atgaagaaag ggatgttctc gatgatgaac
660
tacctgctgc cgctgaaagg tatcgcttct atgcactgct ccgccaacgt tggtgagaaa
720
ggcgatgttg cggtgttctt cggcctttcc ggcaccggta aaaccaccct ttccaccgac
780
ccgaaacgtc gcctgattgg cgatgacgaa cacggctggg acgatgacgg cgtgtttaac
840
ttcgaaggcg gctgctacgc aaaaactatc aagctgtcga aagaagcgga acctgaaatc
900
tacaacgcta tccgtcgtga tgcgttgctg gaaaacgtca ccgtgcgtga agatggcact
960
atcgactttg atgatggttc aaaaaccgag aacacccgcg tttcttatcc gatctatcac
1020
atcgataaca ttgttaagcc ggtttccaaa gcgggccacg cgactaaggt tatcttcctg
1080
actgctgatg ctttcggcgt gttgccgccg gtttctcgcc tgactgccga tcaaacccag
1140
tatcacttcc tctctggctt caccgccaaa ctggccggta ctgagcgtgg catcaccgaa
1200
ccgacgccaa ccttctccgc ttgcttcggc gcggcattcc tgtcgctgca cccgactcag
1260
tacgcagaag tgctggtgaa acgtatgcag gcggcgggcg cgcaggctta tctggttaac
1320
actggctgga acggcactgg caaacgtatc tcgattaaag atacccgcgc cattatcgac
1380
gccatcctca acggttcgct ggataatgca gaaaccttca ctctgccgat gtttaacctg
1440
gcgatcccaa ccgaactgcc gggcgtagac acgaagattc tcgatccgcg taacacctac
1500
gcttctccgg aacagtggca ggaaaaagcc gaaaccctgg cgaaactgtt tatcgacaac
1560
ttcgataaat acaccgacac ccctgcgggt gccgcgctgg tagcggctgg tccgaaactg
1620
taa
1623
<210> 73
<211> 540
<212> ПРТ
<213> Escherichia coli
<400> 73
Met Arg Val Asn Asn Gly Leu Thr Pro Gln Glu Leu Glu Ala Tyr Gly
1 5 10 15
Ile Ser Asp Val His Asp Ile Val Tyr Asn Pro Ser Tyr Asp Leu Leu
20 25 30
Tyr Gln Glu Glu Leu Asp Pro Ser Leu Thr Gly Tyr Glu Arg Gly Val
35 40 45
Leu Thr Asn Leu Gly Ala Val Ala Val Asp Thr Gly Ile Phe Thr Gly
50 55 60
Arg Ser Pro Lys Asp Lys Tyr Ile Val Arg Asp Asp Thr Thr Arg Asp
65 70 75 80
Thr Phe Trp Trp Ala Asp Lys Gly Lys Gly Lys Asn Asp Asn Lys Pro
85 90 95
Leu Ser Pro Glu Thr Trp Gln His Leu Lys Gly Leu Val Thr Arg Gln
100 105 110
Leu Ser Gly Lys Arg Leu Phe Val Val Asp Ala Phe Cys Gly Ala Asn
115 120 125
Pro Asp Thr Arg Leu Ser Val Arg Phe Ile Thr Glu Val Ala Trp Gln
130 135 140
Ala His Phe Val Lys Asn Met Phe Ile Arg Pro Ser Asp Glu Glu Leu
145 150 155 160
Ala Gly Phe Lys Pro Asp Phe Ile Val Met Asn Gly Ala Lys Cys Thr
165 170 175
Asn Pro Gln Trp Lys Glu Gln Gly Leu Asn Ser Glu Asn Phe Val Ala
180 185 190
Phe Asn Leu Thr Glu Arg Met Gln Leu Ile Gly Gly Thr Trp Tyr Gly
195 200 205
Gly Glu Met Lys Lys Gly Met Phe Ser Met Met Asn Tyr Leu Leu Pro
210 215 220
Leu Lys Gly Ile Ala Ser Met His Cys Ser Ala Asn Val Gly Glu Lys
225 230 235 240
Gly Asp Val Ala Val Phe Phe Gly Leu Ser Gly Thr Gly Lys Thr Thr
245 250 255
Leu Ser Thr Asp Pro Lys Arg Arg Leu Ile Gly Asp Asp Glu His Gly
260 265 270
Trp Asp Asp Asp Gly Val Phe Asn Phe Glu Gly Gly Cys Tyr Ala Lys
275 280 285
Thr Ile Lys Leu Ser Lys Glu Ala Glu Pro Glu Ile Tyr Asn Ala Ile
290 295 300
Arg Arg Asp Ala Leu Leu Glu Asn Val Thr Val Arg Glu Asp Gly Thr
305 310 315 320
Ile Asp Phe Asp Asp Gly Ser Lys Thr Glu Asn Thr Arg Val Ser Tyr
325 330 335
Pro Ile Tyr His Ile Asp Asn Ile Val Lys Pro Val Ser Lys Ala Gly
340 345 350
His Ala Thr Lys Val Ile Phe Leu Thr Ala Asp Ala Phe Gly Val Leu
355 360 365
Pro Pro Val Ser Arg Leu Thr Ala Asp Gln Thr Gln Tyr His Phe Leu
370 375 380
Ser Gly Phe Thr Ala Lys Leu Ala Gly Thr Glu Arg Gly Ile Thr Glu
385 390 395 400
Pro Thr Pro Thr Phe Ser Ala Cys Phe Gly Ala Ala Phe Leu Ser Leu
405 410 415
His Pro Thr Gln Tyr Ala Glu Val Leu Val Lys Arg Met Gln Ala Ala
420 425 430
Gly Ala Gln Ala Tyr Leu Val Asn Thr Gly Trp Asn Gly Thr Gly Lys
435 440 445
Arg Ile Ser Ile Lys Asp Thr Arg Ala Ile Ile Asp Ala Ile Leu Asn
450 455 460
Gly Ser Leu Asp Asn Ala Glu Thr Phe Thr Leu Pro Met Phe Asn Leu
465 470 475 480
Ala Ile Pro Thr Glu Leu Pro Gly Val Asp Thr Lys Ile Leu Asp Pro
485 490 495
Arg Asn Thr Tyr Ala Ser Pro Glu Gln Trp Gln Glu Lys Ala Glu Thr
500 505 510
Leu Ala Lys Leu Phe Ile Asp Asn Phe Asp Lys Tyr Thr Asp Thr Pro
515 520 525
Ala Gly Ala Ala Leu Val Ala Ala Gly Pro Lys Leu
530 535 540
<210> 74
<211> 1443
<212> ДНК
<213> Escherichia coli
<400> 74
atgtccagaa ggcttcgcag aacaaaaatc gttaccacgt taggcccagc aacagatcgc
60
gataataatc ttgaaaaagt tatcgcggcg ggtgccaacg ttgtacgtat gaacttttct
120
cacggctcgc ctgaagatca caaaatgcgc gcggataaag ttcgtgagat tgccgcaaaa
180
ctggggcgtc atgtggctat tctgggtgac ctccaggggc ccaaaatccg tgtatccacc
240
tttaaagaag gcaaagtttt cctcaatatt ggggataaat tcctgctcga cgccaacctg
300
ggtaaaggtg aaggcgacaa agaaaaagtc ggtatcgact acaaaggcct gcctgctgac
360
gtcgtgcctg gtgacatcct gctgctggac gatggtcgcg tccagttaaa agtactggaa
420
gttcagggca tgaaagtgtt caccgaagtc accgtcggtg gtcccctctc caacaataaa
480
ggtatcaaca aacttggcgg cggtttgtcg gctgaagcgc tgaccgaaaa agacaaagca
540
gacattaaga ctgcggcgtt gattggcgta gattacctgg ctgtctcctt cccacgctgt
600
ggcgaagatc tgaactatgc ccgtcgcctg gcacgcgatg caggatgtga tgcgaaaatt
660
gttgccaagg ttgaacgtgc ggaagccgtt tgcagccagg atgcaatgga tgacatcatc
720
ctcgcctctg acgtggtaat ggttgcacgt ggcgacctcg gtgtggaaat tggcgacccg
780
gaactggtcg gcattcagaa agcgttgatc cgtcgtgcgc gtcagctaaa ccgagcggta
840
atcacggcga cccagatgat ggagtcaatg attactaacc cgatgccgac gcgtgcagaa
900
gtcatggacg tagcaaacgc cgttctggat ggtactgacg ctgtgatgct gtctgcagaa
960
actgccgctg ggcagtatcc gtcagaaacc gttgcagcca tggcgcgcgt ttgcctgggt
1020
gcggaaaaaa tcccgagcat caacgtttct aaacaccgtc tggacgttca gttcgacaat
1080
gtggaagaag ctattgccat gtcagcaatg tacgcagcta accacctgaa aggcgttacg
1140
gcgatcatca ccatgaccga atcgggtcgt accgcgctga tgacctcccg tatcagctct
1200
ggtctgccaa ttttcgccat gtcgcgccat gaacgtacgc tgaacctgac tgctctctat
1260
cgtggcgtta cgccggtgca ctttgatagc gctaatgacg gcgtagcagc tgccagcgaa
1320
gcggttaatc tgctgcgcga taaaggttac ttgatgtctg gtgacctggt gattgtcacc
1380
cagggcgacg tgatgagtac cgtgggttct actaatacca cgcgtatttt aacggtagag
1440
taa
1443
<210> 75
<211> 480
<212> ПРТ
<213> Escherichia coli
<400> 75
Met Ser Arg Arg Leu Arg Arg Thr Lys Ile Val Thr Thr Leu Gly Pro
1 5 10 15
Ala Thr Asp Arg Asp Asn Asn Leu Glu Lys Val Ile Ala Ala Gly Ala
20 25 30
Asn Val Val Arg Met Asn Phe Ser His Gly Ser Pro Glu Asp His Lys
35 40 45
Met Arg Ala Asp Lys Val Arg Glu Ile Ala Ala Lys Leu Gly Arg His
50 55 60
Val Ala Ile Leu Gly Asp Leu Gln Gly Pro Lys Ile Arg Val Ser Thr
65 70 75 80
Phe Lys Glu Gly Lys Val Phe Leu Asn Ile Gly Asp Lys Phe Leu Leu
85 90 95
Asp Ala Asn Leu Gly Lys Gly Glu Gly Asp Lys Glu Lys Val Gly Ile
100 105 110
Asp Tyr Lys Gly Leu Pro Ala Asp Val Val Pro Gly Asp Ile Leu Leu
115 120 125
Leu Asp Asp Gly Arg Val Gln Leu Lys Val Leu Glu Val Gln Gly Met
130 135 140
Lys Val Phe Thr Glu Val Thr Val Gly Gly Pro Leu Ser Asn Asn Lys
145 150 155 160
Gly Ile Asn Lys Leu Gly Gly Gly Leu Ser Ala Glu Ala Leu Thr Glu
165 170 175
Lys Asp Lys Ala Asp Ile Lys Thr Ala Ala Leu Ile Gly Val Asp Tyr
180 185 190
Leu Ala Val Ser Phe Pro Arg Cys Gly Glu Asp Leu Asn Tyr Ala Arg
195 200 205
Arg Leu Ala Arg Asp Ala Gly Cys Asp Ala Lys Ile Val Ala Lys Val
210 215 220
Glu Arg Ala Glu Ala Val Cys Ser Gln Asp Ala Met Asp Asp Ile Ile
225 230 235 240
Leu Ala Ser Asp Val Val Met Val Ala Arg Gly Asp Leu Gly Val Glu
245 250 255
Ile Gly Asp Pro Glu Leu Val Gly Ile Gln Lys Ala Leu Ile Arg Arg
260 265 270
Ala Arg Gln Leu Asn Arg Ala Val Ile Thr Ala Thr Gln Met Met Glu
275 280 285
Ser Met Ile Thr Asn Pro Met Pro Thr Arg Ala Glu Val Met Asp Val
290 295 300
Ala Asn Ala Val Leu Asp Gly Thr Asp Ala Val Met Leu Ser Ala Glu
305 310 315 320
Thr Ala Ala Gly Gln Tyr Pro Ser Glu Thr Val Ala Ala Met Ala Arg
325 330 335
Val Cys Leu Gly Ala Glu Lys Ile Pro Ser Ile Asn Val Ser Lys His
340 345 350
Arg Leu Asp Val Gln Phe Asp Asn Val Glu Glu Ala Ile Ala Met Ser
355 360 365
Ala Met Tyr Ala Ala Asn His Leu Lys Gly Val Thr Ala Ile Ile Thr
370 375 380
Met Thr Glu Ser Gly Arg Thr Ala Leu Met Thr Ser Arg Ile Ser Ser
385 390 395 400
Gly Leu Pro Ile Phe Ala Met Ser Arg His Glu Arg Thr Leu Asn Leu
405 410 415
Thr Ala Leu Tyr Arg Gly Val Thr Pro Val His Phe Asp Ser Ala Asn
420 425 430
Asp Gly Val Ala Ala Ala Ser Glu Ala Val Asn Leu Leu Arg Asp Lys
435 440 445
Gly Tyr Leu Met Ser Gly Asp Leu Val Ile Val Thr Gln Gly Asp Val
450 455 460
Met Ser Thr Val Gly Ser Thr Asn Thr Thr Arg Ile Leu Thr Val Glu
465 470 475 480
<210> 76
<211> 1413
<212> ДНК
<213> Escherichia coli
<400> 76
atgaaaaaga ccaaaattgt ttgcaccatc ggaccgaaaa ccgaatctga agagatgtta
60
gctaaaatgc tggacgctgg catgaacgtt atgcgtctga acttctctca tggtgactat
120
gcagaacacg gtcagcgcat tcagaatctg cgcaacgtga tgagcaaaac tggtaaaacc
180
gccgctatcc tgcttgatac caaaggtccg gaaatccgca ccatgaaact ggaaggcggt
240
aacgacgttt ctctgaaagc tggtcagacc tttactttca ccactgataa atctgttatc
300
ggcaacagcg aaatggttgc ggtaacgtat gaaggtttca ctactgacct gtctgttggc
360
aacaccgtac tggttgacga tggtctgatc ggtatggaag ttaccgccat tgaaggtaac
420
aaagttatct gtaaagtgct gaacaacggt gacctgggcg aaaacaaagg tgtgaacctg
480
cctggcgttt ccattgctct gccagcactg gctgaaaaag acaaacagga cctgatcttt
540
ggttgcgaac aaggcgtaga ctttgttgct gcttccttta ttcgtaagcg ttctgacgtt
600
atcgaaatcc gtgagcacct gaaagcgcac ggcggcgaaa acatccacat catctccaaa
660
atcgaaaacc aggaaggcct caacaacttc gacgaaatcc tcgaagcctc tgacggcatc
720
atggttgcgc gtggcgacct gggtgtagaa atcccggtag aagaagttat cttcgcccag
780
aagatgatga tcgaaaaatg tatccgtgca cgtaaagtcg ttatcactgc gacccagatg
840
ctggattcca tgatcaaaaa cccacgcccg actcgcgcag aagccggtga cgttgcaaac
900
gccatcctcg acggtactga cgcagtgatg ctgtctggtg aatccgcaaa aggtaaatac
960
ccgctggaag cggtttctat catggcgacc atctgcgaac gtaccgaccg cgtgatgaac
1020
agccgtctcg agttcaacaa tgacaaccgt aaactgcgca ttaccgaagc ggtatgccgt
1080
ggtgccgttg aaactgctga aaaactggat gctccgctga tcgtggttgc tactcagggc
1140
ggtaaatctg ctcgcgcagt acgtaaatac ttcccggatg ccaccatcct ggcactgacc
1200
accaacgaaa aaacggctca tcagttggta ctgagcaaag gcgttgtgcc gcagcttgtt
1260
aaagagatca cttctactga tgatttctac cgtctgggta aagaactggc tctgcagagc
1320
ggtctggcac acaaaggtga cgttgtagtt atggtttctg gtgcactggt accgagcggc
1380
actactaaca ccgcatctgt tcacgtcctg taa
1413
<210> 77
<211> 470
<212> ПРТ
<213> Escherichia coli
<400> 77
Met Lys Lys Thr Lys Ile Val Cys Thr Ile Gly Pro Lys Thr Glu Ser
1 5 10 15
Glu Glu Met Leu Ala Lys Met Leu Asp Ala Gly Met Asn Val Met Arg
20 25 30
Leu Asn Phe Ser His Gly Asp Tyr Ala Glu His Gly Gln Arg Ile Gln
35 40 45
Asn Leu Arg Asn Val Met Ser Lys Thr Gly Lys Thr Ala Ala Ile Leu
50 55 60
Leu Asp Thr Lys Gly Pro Glu Ile Arg Thr Met Lys Leu Glu Gly Gly
65 70 75 80
Asn Asp Val Ser Leu Lys Ala Gly Gln Thr Phe Thr Phe Thr Thr Asp
85 90 95
Lys Ser Val Ile Gly Asn Ser Glu Met Val Ala Val Thr Tyr Glu Gly
100 105 110
Phe Thr Thr Asp Leu Ser Val Gly Asn Thr Val Leu Val Asp Asp Gly
115 120 125
Leu Ile Gly Met Glu Val Thr Ala Ile Glu Gly Asn Lys Val Ile Cys
130 135 140
Lys Val Leu Asn Asn Gly Asp Leu Gly Glu Asn Lys Gly Val Asn Leu
145 150 155 160
Pro Gly Val Ser Ile Ala Leu Pro Ala Leu Ala Glu Lys Asp Lys Gln
165 170 175
Asp Leu Ile Phe Gly Cys Glu Gln Gly Val Asp Phe Val Ala Ala Ser
180 185 190
Phe Ile Arg Lys Arg Ser Asp Val Ile Glu Ile Arg Glu His Leu Lys
195 200 205
Ala His Gly Gly Glu Asn Ile His Ile Ile Ser Lys Ile Glu Asn Gln
210 215 220
Glu Gly Leu Asn Asn Phe Asp Glu Ile Leu Glu Ala Ser Asp Gly Ile
225 230 235 240
Met Val Ala Arg Gly Asp Leu Gly Val Glu Ile Pro Val Glu Glu Val
245 250 255
Ile Phe Ala Gln Lys Met Met Ile Glu Lys Cys Ile Arg Ala Arg Lys
260 265 270
Val Val Ile Thr Ala Thr Gln Met Leu Asp Ser Met Ile Lys Asn Pro
275 280 285
Arg Pro Thr Arg Ala Glu Ala Gly Asp Val Ala Asn Ala Ile Leu Asp
290 295 300
Gly Thr Asp Ala Val Met Leu Ser Gly Glu Ser Ala Lys Gly Lys Tyr
305 310 315 320
Pro Leu Glu Ala Val Ser Ile Met Ala Thr Ile Cys Glu Arg Thr Asp
325 330 335
Arg Val Met Asn Ser Arg Leu Glu Phe Asn Asn Asp Asn Arg Lys Leu
340 345 350
Arg Ile Thr Glu Ala Val Cys Arg Gly Ala Val Glu Thr Ala Glu Lys
355 360 365
Leu Asp Ala Pro Leu Ile Val Val Ala Thr Gln Gly Gly Lys Ser Ala
370 375 380
Arg Ala Val Arg Lys Tyr Phe Pro Asp Ala Thr Ile Leu Ala Leu Thr
385 390 395 400
Thr Asn Glu Lys Thr Ala His Gln Leu Val Leu Ser Lys Gly Val Val
405 410 415
Pro Gln Leu Val Lys Glu Ile Thr Ser Thr Asp Asp Phe Tyr Arg Leu
420 425 430
Gly Lys Glu Leu Ala Leu Gln Ser Gly Leu Ala His Lys Gly Asp Val
435 440 445
Val Val Met Val Ser Gly Ala Leu Val Pro Ser Gly Thr Thr Asn Thr
450 455 460
Ala Ser Val His Val Leu
465 470
<210> 78
<211> 1410
<212> ДНК
<213> Escherichia coli
<400> 78
atgtccgctg aacacgtact gacgatgctg aacgagcacg aagtgaagtt tgttgatttg
60
cgcttcaccg atactaaagg taaagaacag cacgtcacta tccctgctca tcaggtgaat
120
gctgaattct tcgaagaagg caaaatgttt gacggctcct cgattggcgg ctggaaaggc
180
attaacgagt ccgacatggt gctgatgcca gacgcatcca ccgcagtgat tgacccgttc
240
ttcgccgact ccaccctgat tatccgttgc gacatccttg aacctggcac cctgcaaggc
300
tatgaccgtg acccgcgctc cattgcgaag cgcgccgaag attacctgcg ttccactggc
360
attgccgaca ccgtactgtt cgggccagaa cctgaattct tcctgttcga tgacatccgt
420
ttcggatcat ctatctccgg ttcccacgtt gctatcgacg atatcgaagg cgcatggaac
480
tcctccaccc aatacgaagg tggtaacaaa ggtcaccgtc cggcagtgaa aggcggttac
540
ttcccggttc caccggtaga ctcggctcag gatattcgtt ctgaaatgtg tctggtgatg
600
gaacagatgg gtctggtggt tgaagcccat caccacgaag tagcgactgc tggtcagaac
660
gaagtggcta cccgcttcaa taccatgacc aaaaaagctg acgaaattca gatctacaaa
720
tatgttgtgc acaacgtagc gcaccgcttc ggtaaaaccg cgacctttat gccaaaaccg
780
atgttcggtg ataacggctc cggtatgcac tgccacatgt ctctgtctaa aaacggcgtt
840
aacctgttcg caggcgacaa atacgcaggt ctgtctgagc aggcgctgta ctacattggc
900
ggcgtaatca aacacgctaa agcgattaac gccctggcaa acccgaccac caactcttat
960
aagcgtctgg tcccgggcta tgaagcaccg gtaatgctgg cttactctgc gcgtaaccgt
1020
tctgcgtcta tccgtattcc ggtggtttct tctccgaaag cacgtcgtat cgaagtacgt
1080
ttcccggatc cggcagctaa cccgtacctg tgctttgctg ccctgctgat ggccggtctt
1140
gatggtatca agaacaagat ccatccgggc gaagccatgg acaaaaacct gtatgacctg
1200
ccgccagaag aagcgaaaga gatcccacag gttgcaggct ctctggaaga agcactgaac
1260
gaactggatc tggaccgcga gttcctgaaa gccggtggcg tgttcactga cgaagcaatt
1320
gatgcgtaca tcgctctgcg tcgcgaagaa gatgaccgcg tgcgtatgac tccgcatccg
1380
gtagagtttg agctgtacta cagcgtctaa
1410
<210> 79
<211> 469
<212> ПРТ
<213> Escherichia coli
<400> 79
Met Ser Ala Glu His Val Leu Thr Met Leu Asn Glu His Glu Val Lys
1 5 10 15
Phe Val Asp Leu Arg Phe Thr Asp Thr Lys Gly Lys Glu Gln His Val
20 25 30
Thr Ile Pro Ala His Gln Val Asn Ala Glu Phe Phe Glu Glu Gly Lys
35 40 45
Met Phe Asp Gly Ser Ser Ile Gly Gly Trp Lys Gly Ile Asn Glu Ser
50 55 60
Asp Met Val Leu Met Pro Asp Ala Ser Thr Ala Val Ile Asp Pro Phe
65 70 75 80
Phe Ala Asp Ser Thr Leu Ile Ile Arg Cys Asp Ile Leu Glu Pro Gly
85 90 95
Thr Leu Gln Gly Tyr Asp Arg Asp Pro Arg Ser Ile Ala Lys Arg Ala
100 105 110
Glu Asp Tyr Leu Arg Ser Thr Gly Ile Ala Asp Thr Val Leu Phe Gly
115 120 125
Pro Glu Pro Glu Phe Phe Leu Phe Asp Asp Ile Arg Phe Gly Ser Ser
130 135 140
Ile Ser Gly Ser His Val Ala Ile Asp Asp Ile Glu Gly Ala Trp Asn
145 150 155 160
Ser Ser Thr Gln Tyr Glu Gly Gly Asn Lys Gly His Arg Pro Ala Val
165 170 175
Lys Gly Gly Tyr Phe Pro Val Pro Pro Val Asp Ser Ala Gln Asp Ile
180 185 190
Arg Ser Glu Met Cys Leu Val Met Glu Gln Met Gly Leu Val Val Glu
195 200 205
Ala His His His Glu Val Ala Thr Ala Gly Gln Asn Glu Val Ala Thr
210 215 220
Arg Phe Asn Thr Met Thr Lys Lys Ala Asp Glu Ile Gln Ile Tyr Lys
225 230 235 240
Tyr Val Val His Asn Val Ala His Arg Phe Gly Lys Thr Ala Thr Phe
245 250 255
Met Pro Lys Pro Met Phe Gly Asp Asn Gly Ser Gly Met His Cys His
260 265 270
Met Ser Leu Ser Lys Asn Gly Val Asn Leu Phe Ala Gly Asp Lys Tyr
275 280 285
Ala Gly Leu Ser Glu Gln Ala Leu Tyr Tyr Ile Gly Gly Val Ile Lys
290 295 300
His Ala Lys Ala Ile Asn Ala Leu Ala Asn Pro Thr Thr Asn Ser Tyr
305 310 315 320
Lys Arg Leu Val Pro Gly Tyr Glu Ala Pro Val Met Leu Ala Tyr Ser
325 330 335
Ala Arg Asn Arg Ser Ala Ser Ile Arg Ile Pro Val Val Ser Ser Pro
340 345 350
Lys Ala Arg Arg Ile Glu Val Arg Phe Pro Asp Pro Ala Ala Asn Pro
355 360 365
Tyr Leu Cys Phe Ala Ala Leu Leu Met Ala Gly Leu Asp Gly Ile Lys
370 375 380
Asn Lys Ile His Pro Gly Glu Ala Met Asp Lys Asn Leu Tyr Asp Leu
385 390 395 400
Pro Pro Glu Glu Ala Lys Glu Ile Pro Gln Val Ala Gly Ser Leu Glu
405 410 415
Glu Ala Leu Asn Glu Leu Asp Leu Asp Arg Glu Phe Leu Lys Ala Gly
420 425 430
Gly Val Phe Thr Asp Glu Ala Ile Asp Ala Tyr Ile Ala Leu Arg Arg
435 440 445
Glu Glu Asp Asp Arg Val Arg Met Thr Pro His Pro Val Glu Phe Glu
450 455 460
Leu Tyr Tyr Ser Val
465
<210> 80
<211> 4461
<212> ДНК
<213> Escherichia coli
<400> 80
atgttgtacg ataaatccct tgagagggat aactgtggtt tcggcctgat cgcccacata
60
gaaggcgaac ctagccacaa ggtagtgcgt actgcaatac acgcactggc ccgcatgcag
120
caccgtggcg cgattctcgc cgatggtaaa accggcgacg gttgcggctt gctgttacaa
180
aaaccggatc gcttttttcg catcgttgcg caggagcgcg gctggcgttt agcaaaaaac
240
tacgctgtcg ggatgctctt cctgaataaa gatcctgaac tcgccgctgc cgcacgccgc
300
atcgttgaag aagaactgca acgcgaaacc ttgtcgattg tgggctggcg tgatgtcccc
360
actaacgaag gcgtgctggg tgaaatcgcc ctctcctctc tgccacgcat tgagcaaatt
420
tttgtgaacg ccccggcagg ctggcgtcca cgcgatatgg agcgccgtct gtttatcgcc
480
cgccgccgca ttgaaaagcg tctcgaagcc gacaaagact tctacgtctg tagcctgtcg
540
aatctggtga acatctataa aggtctgtgt atgccgacgg atctgccgcg cttttatctg
600
gatcttgcgg acctgcgtct ggaatcggcc atttgcctgt tccaccagcg cttctccact
660
aacaccgtac cgcgctggcc gctggcgcaa ccgttccgct atctggcgca taacggtgaa
720
atcaacacca tcaccggtaa ccgccaatgg gcgcgtgcgc gtacttataa attccagaca
780
ccgcttatcc ctgacctgca cgacgccgca ccgttcgtca acgaaaccgg ctctgactcc
840
agttcgatgg ataacatgct ggaactgctg ctggcaggcg ggatggatat catccgcgcc
900
atgcgtctat tagtaccacc cgcctggcag aacaacccgg atatggaccc ggaactgcgt
960
gccttctttg actttaactc catgcatatg gagccgtggg atggcccggc gggcatcgtg
1020
atgtccgacg gtcgttttgc cgcctgtaac ctcgaccgta acggtctgcg tccggcgcgc
1080
tacgtcatca ccaaagataa gctcatcacc tgcgcctctg aagtcggtat ctgggattac
1140
cagcctgacg aagtggtcga aaaaggccgc gtcgggccag gcgaactgat ggttatcgac
1200
acccgcagtg ggcgtattct gcactcggca gaaaccgatg acgatctgaa aagccgccat
1260
ccatataaag agtggatgga gaaaaacgtc cgccgactgg taccgtttga agatctgccc
1320
gatgaagaag tgggtagccg cgaactggac gacgacacgc ttgccagcta ccagaaacag
1380
tttaactaca gcgcggaaga gctggactcc gtaattcgcg tactgggcga aaacggtcag
1440
gaagcggtcg gttcgatggg cgatgatacc ccattcgccg tgctctccag tcagccgcgc
1500
attatttacg actacttccg ccagcagttt gcccaggtga ctaacccgcc aatcgacccg
1560
ctgcgtgaag cgcatgttat gtcgctcgcc accagtatcg gtcgtgaaat gaacgtcttt
1620
tgcgaagcag agggccaggc gcaccgttta agctttaaat cgccgattct gctctactcc
1680
gatttcaaac agctcacgac gatgaaagag gagcactacc gcgcagatac gctggatatc
1740
acctttgacg tcactaaaac cacgctcgaa gcgacagtca aagagctgtg cgacaaagcc
1800
gaaaaaatgg tacgtagcgg caccgtgctg ctggtgctct ccgaccggaa tatcgctaaa
1860
gatcgcctgc cggttccagc cccgatggcg gttggcgcga tccagacccg tctggtcgat
1920
caaagcctgc gttgcgatgc caacatcatc gtcgaaaccg ccagcgcccg cgatccgcac
1980
cacttcgccg tgttgctggg cttcggcgcg acggctattt atccatacct tgcctatgaa
2040
acgctgggcc gcctggtaga cacccatgcg attgccaaag attatcgtac cgtgatgctc
2100
aactaccgta acggcatcaa caaaggcttg tacaaaatca tgtccaaaat gggcatctcc
2160
accatcgcct cttaccgctg ctcgaaactg tttgaagcgg tcggtctaca cgatgatgta
2220
gtgggcctgt gcttccaggg ggcggtcagc cgcattggtg gagcaagctt tgaagacttc
2280
cagcaggatc tgctgaatct gtcgaaacgt gcctggctgg cgcgtaagcc catcagccag
2340
ggcggtctgc tgaaatacgt ccacggcggc gaataccacg cctacaaccc ggacgtggtg
2400
cgcacgctgc aacaagcggt acaaagcggc gagtacagcg actatcagga atacgcgaag
2460
ctggttaatg agcgtccggc aaccacgctg cgcgatctgc tggcaattac gccgggtgaa
2520
aacgcggtca acattgctga tgttgaaccg gcaagcgaac tgtttaaacg ctttgatacc
2580
gccgcgatgt ctatcggcgc gttaagcccg gaagcccacg aggcgctggc ggaagcgatg
2640
aacagcatcg gcggtaattc gaactccggt gaaggcggcg aagacccggc gcgctacggc
2700
accaacaaag tgtcgcgcat caagcaggtg gcttccggtc gctttggggt tactccggcg
2760
tatctggtca atgccgacgt cattcagatt aaagtcgccc agggcgcgaa gccaggcgaa
2820
ggcggtcagt tgccgggtga taaagtcacg ccttacatcg ccaaactgcg ctattcggtg
2880
cccggagtga cgctgatctc cccgccgccg caccacgata tctactctat cgaggactta
2940
gcgcagctca ttttcgacct caagcaggtt aacccgaaag cgatgatctc cgtgaagctg
3000
gtttccgaac cgggagtagg caccatcgcg actggcgtgg caaaagctta tgcggacttg
3060
atcaccatcg caggctatga cggcggcacc ggcgcaagtc cgctttcatc ggtgaaatac
3120
gcaggctgtc cgtgggagct ggggcttgtt gaaacccagc aggcgctggt tgctaacggc
3180
ttgcgtcaca agatccgttt gcaggtcgat ggcggcctga aaacgggtgt cgatatcatc
3240
aaggcggcga ttctcggcgc agaaagcttc ggcttcggca ctggcccgat ggtggcgctc
3300
ggctgtaaat atctacgtat ttgccatctg aacaactgcg caacgggtgt agcaactcag
3360
gatgacaaac tgcgtaagaa ccactatcac ggcctgccat tcaaggtgac gaattacttt
3420
gagtttatcg cccgtgaaac ccgcgagctg atggcacagc ttggcgtaac acgtctggtg
3480
gatctgattg gtcgcaccga cctgctgaaa gagctggacg gtttcaccgc caaacagcag
3540
aagctggcgc tgtcgaagct gctggagact gccgaaccgc atccaggtaa ggcactctac
3600
tgcaccgaaa acaacccgcc gtttgataac ggcctgctga acgcgcagtt gctgcaacag
3660
gcgaaaccgt ttgtcgatga gcgccagagc aaaaccttct ggttcgatat tcgcaacacc
3720
gaccgttctg tcggcgcgtc gctttcaggc tatatcgccc agacgcacgg cgatcagggg
3780
ctggcagccg atcctatcaa agcgtacttc aacggcaccg caggccagag cttcggcgtg
3840
tggaacgcgg gcggcgtgga actgtacctg accggtgatg ccaacgacta tgtcggtaaa
3900
ggcatggcgg gcggcttaat cgccattcgt cctccggttg gttccgcctt ccgcagccat
3960
gaagcaagca ttatcggcaa cacctgcctg tatggcgcga ccggtggtcg tctgtatgcc
4020
gcaggccgcg cgggtgaacg tttcggcgtg cgtaactccg gtgctatcac cgtggtagaa
4080
ggcattggcg acaacggttg tgaatatatg acgggtggta tcgtctgcat tctgggtaaa
4140
accggcgtta acttcggtgc gggcatgacc ggcggtttcg cttacgttct cgatgaaagc
4200
ggcgatttcc gcaaacgcgt taacccggaa ctggtcgagg tcttaagcgt tgacgctctg
4260
gcgatccatg aagagcatct gcgtggtctt atcaccgagc atgtgcagca taccggctct
4320
cagcgcggtg aagagattct ggcgaactgg tcaaccttcg ccactaaatt tgcgctggtt
4380
aaaccgaagt ccagtgatgt aaaagcactg ctgggtcacc gtagtcgtag cgcagctgag
4440
ttgcgcgtgc aggcgcagta a
4461
<210> 81
<211> 1486
<212> ПРТ
<213> Escherichia coli
<400> 81
Met Leu Tyr Asp Lys Ser Leu Glu Arg Asp Asn Cys Gly Phe Gly Leu
1 5 10 15
Ile Ala His Ile Glu Gly Glu Pro Ser His Lys Val Val Arg Thr Ala
20 25 30
Ile His Ala Leu Ala Arg Met Gln His Arg Gly Ala Ile Leu Ala Asp
35 40 45
Gly Lys Thr Gly Asp Gly Cys Gly Leu Leu Leu Gln Lys Pro Asp Arg
50 55 60
Phe Phe Arg Ile Val Ala Gln Glu Arg Gly Trp Arg Leu Ala Lys Asn
65 70 75 80
Tyr Ala Val Gly Met Leu Phe Leu Asn Lys Asp Pro Glu Leu Ala Ala
85 90 95
Ala Ala Arg Arg Ile Val Glu Glu Glu Leu Gln Arg Glu Thr Leu Ser
100 105 110
Ile Val Gly Trp Arg Asp Val Pro Thr Asn Glu Gly Val Leu Gly Glu
115 120 125
Ile Ala Leu Ser Ser Leu Pro Arg Ile Glu Gln Ile Phe Val Asn Ala
130 135 140
Pro Ala Gly Trp Arg Pro Arg Asp Met Glu Arg Arg Leu Phe Ile Ala
145 150 155 160
Arg Arg Arg Ile Glu Lys Arg Leu Glu Ala Asp Lys Asp Phe Tyr Val
165 170 175
Cys Ser Leu Ser Asn Leu Val Asn Ile Tyr Lys Gly Leu Cys Met Pro
180 185 190
Thr Asp Leu Pro Arg Phe Tyr Leu Asp Leu Ala Asp Leu Arg Leu Glu
195 200 205
Ser Ala Ile Cys Leu Phe His Gln Arg Phe Ser Thr Asn Thr Val Pro
210 215 220
Arg Trp Pro Leu Ala Gln Pro Phe Arg Tyr Leu Ala His Asn Gly Glu
225 230 235 240
Ile Asn Thr Ile Thr Gly Asn Arg Gln Trp Ala Arg Ala Arg Thr Tyr
245 250 255
Lys Phe Gln Thr Pro Leu Ile Pro Asp Leu His Asp Ala Ala Pro Phe
260 265 270
Val Asn Glu Thr Gly Ser Asp Ser Ser Ser Met Asp Asn Met Leu Glu
275 280 285
Leu Leu Leu Ala Gly Gly Met Asp Ile Ile Arg Ala Met Arg Leu Leu
290 295 300
Val Pro Pro Ala Trp Gln Asn Asn Pro Asp Met Asp Pro Glu Leu Arg
305 310 315 320
Ala Phe Phe Asp Phe Asn Ser Met His Met Glu Pro Trp Asp Gly Pro
325 330 335
Ala Gly Ile Val Met Ser Asp Gly Arg Phe Ala Ala Cys Asn Leu Asp
340 345 350
Arg Asn Gly Leu Arg Pro Ala Arg Tyr Val Ile Thr Lys Asp Lys Leu
355 360 365
Ile Thr Cys Ala Ser Glu Val Gly Ile Trp Asp Tyr Gln Pro Asp Glu
370 375 380
Val Val Glu Lys Gly Arg Val Gly Pro Gly Glu Leu Met Val Ile Asp
385 390 395 400
Thr Arg Ser Gly Arg Ile Leu His Ser Ala Glu Thr Asp Asp Asp Leu
405 410 415
Lys Ser Arg His Pro Tyr Lys Glu Trp Met Glu Lys Asn Val Arg Arg
420 425 430
Leu Val Pro Phe Glu Asp Leu Pro Asp Glu Glu Val Gly Ser Arg Glu
435 440 445
Leu Asp Asp Asp Thr Leu Ala Ser Tyr Gln Lys Gln Phe Asn Tyr Ser
450 455 460
Ala Glu Glu Leu Asp Ser Val Ile Arg Val Leu Gly Glu Asn Gly Gln
465 470 475 480
Glu Ala Val Gly Ser Met Gly Asp Asp Thr Pro Phe Ala Val Leu Ser
485 490 495
Ser Gln Pro Arg Ile Ile Tyr Asp Tyr Phe Arg Gln Gln Phe Ala Gln
500 505 510
Val Thr Asn Pro Pro Ile Asp Pro Leu Arg Glu Ala His Val Met Ser
515 520 525
Leu Ala Thr Ser Ile Gly Arg Glu Met Asn Val Phe Cys Glu Ala Glu
530 535 540
Gly Gln Ala His Arg Leu Ser Phe Lys Ser Pro Ile Leu Leu Tyr Ser
545 550 555 560
Asp Phe Lys Gln Leu Thr Thr Met Lys Glu Glu His Tyr Arg Ala Asp
565 570 575
Thr Leu Asp Ile Thr Phe Asp Val Thr Lys Thr Thr Leu Glu Ala Thr
580 585 590
Val Lys Glu Leu Cys Asp Lys Ala Glu Lys Met Val Arg Ser Gly Thr
595 600 605
Val Leu Leu Val Leu Ser Asp Arg Asn Ile Ala Lys Asp Arg Leu Pro
610 615 620
Val Pro Ala Pro Met Ala Val Gly Ala Ile Gln Thr Arg Leu Val Asp
625 630 635 640
Gln Ser Leu Arg Cys Asp Ala Asn Ile Ile Val Glu Thr Ala Ser Ala
645 650 655
Arg Asp Pro His His Phe Ala Val Leu Leu Gly Phe Gly Ala Thr Ala
660 665 670
Ile Tyr Pro Tyr Leu Ala Tyr Glu Thr Leu Gly Arg Leu Val Asp Thr
675 680 685
His Ala Ile Ala Lys Asp Tyr Arg Thr Val Met Leu Asn Tyr Arg Asn
690 695 700
Gly Ile Asn Lys Gly Leu Tyr Lys Ile Met Ser Lys Met Gly Ile Ser
705 710 715 720
Thr Ile Ala Ser Tyr Arg Cys Ser Lys Leu Phe Glu Ala Val Gly Leu
725 730 735
His Asp Asp Val Val Gly Leu Cys Phe Gln Gly Ala Val Ser Arg Ile
740 745 750
Gly Gly Ala Ser Phe Glu Asp Phe Gln Gln Asp Leu Leu Asn Leu Ser
755 760 765
Lys Arg Ala Trp Leu Ala Arg Lys Pro Ile Ser Gln Gly Gly Leu Leu
770 775 780
Lys Tyr Val His Gly Gly Glu Tyr His Ala Tyr Asn Pro Asp Val Val
785 790 795 800
Arg Thr Leu Gln Gln Ala Val Gln Ser Gly Glu Tyr Ser Asp Tyr Gln
805 810 815
Glu Tyr Ala Lys Leu Val Asn Glu Arg Pro Ala Thr Thr Leu Arg Asp
820 825 830
Leu Leu Ala Ile Thr Pro Gly Glu Asn Ala Val Asn Ile Ala Asp Val
835 840 845
Glu Pro Ala Ser Glu Leu Phe Lys Arg Phe Asp Thr Ala Ala Met Ser
850 855 860
Ile Gly Ala Leu Ser Pro Glu Ala His Glu Ala Leu Ala Glu Ala Met
865 870 875 880
Asn Ser Ile Gly Gly Asn Ser Asn Ser Gly Glu Gly Gly Glu Asp Pro
885 890 895
Ala Arg Tyr Gly Thr Asn Lys Val Ser Arg Ile Lys Gln Val Ala Ser
900 905 910
Gly Arg Phe Gly Val Thr Pro Ala Tyr Leu Val Asn Ala Asp Val Ile
915 920 925
Gln Ile Lys Val Ala Gln Gly Ala Lys Pro Gly Glu Gly Gly Gln Leu
930 935 940
Pro Gly Asp Lys Val Thr Pro Tyr Ile Ala Lys Leu Arg Tyr Ser Val
945 950 955 960
Pro Gly Val Thr Leu Ile Ser Pro Pro Pro His His Asp Ile Tyr Ser
965 970 975
Ile Glu Asp Leu Ala Gln Leu Ile Phe Asp Leu Lys Gln Val Asn Pro
980 985 990
Lys Ala Met Ile Ser Val Lys Leu Val Ser Glu Pro Gly Val Gly Thr
995 1000 1005
Ile Ala Thr Gly Val Ala Lys Ala Tyr Ala Asp Leu Ile Thr Ile
1010 1015 1020
Ala Gly Tyr Asp Gly Gly Thr Gly Ala Ser Pro Leu Ser Ser Val
1025 1030 1035
Lys Tyr Ala Gly Cys Pro Trp Glu Leu Gly Leu Val Glu Thr Gln
1040 1045 1050
Gln Ala Leu Val Ala Asn Gly Leu Arg His Lys Ile Arg Leu Gln
1055 1060 1065
Val Asp Gly Gly Leu Lys Thr Gly Val Asp Ile Ile Lys Ala Ala
1070 1075 1080
Ile Leu Gly Ala Glu Ser Phe Gly Phe Gly Thr Gly Pro Met Val
1085 1090 1095
Ala Leu Gly Cys Lys Tyr Leu Arg Ile Cys His Leu Asn Asn Cys
1100 1105 1110
Ala Thr Gly Val Ala Thr Gln Asp Asp Lys Leu Arg Lys Asn His
1115 1120 1125
Tyr His Gly Leu Pro Phe Lys Val Thr Asn Tyr Phe Glu Phe Ile
1130 1135 1140
Ala Arg Glu Thr Arg Glu Leu Met Ala Gln Leu Gly Val Thr Arg
1145 1150 1155
Leu Val Asp Leu Ile Gly Arg Thr Asp Leu Leu Lys Glu Leu Asp
1160 1165 1170
Gly Phe Thr Ala Lys Gln Gln Lys Leu Ala Leu Ser Lys Leu Leu
1175 1180 1185
Glu Thr Ala Glu Pro His Pro Gly Lys Ala Leu Tyr Cys Thr Glu
1190 1195 1200
Asn Asn Pro Pro Phe Asp Asn Gly Leu Leu Asn Ala Gln Leu Leu
1205 1210 1215
Gln Gln Ala Lys Pro Phe Val Asp Glu Arg Gln Ser Lys Thr Phe
1220 1225 1230
Trp Phe Asp Ile Arg Asn Thr Asp Arg Ser Val Gly Ala Ser Leu
1235 1240 1245
Ser Gly Tyr Ile Ala Gln Thr His Gly Asp Gln Gly Leu Ala Ala
1250 1255 1260
Asp Pro Ile Lys Ala Tyr Phe Asn Gly Thr Ala Gly Gln Ser Phe
1265 1270 1275
Gly Val Trp Asn Ala Gly Gly Val Glu Leu Tyr Leu Thr Gly Asp
1280 1285 1290
Ala Asn Asp Tyr Val Gly Lys Gly Met Ala Gly Gly Leu Ile Ala
1295 1300 1305
Ile Arg Pro Pro Val Gly Ser Ala Phe Arg Ser His Glu Ala Ser
1310 1315 1320
Ile Ile Gly Asn Thr Cys Leu Tyr Gly Ala Thr Gly Gly Arg Leu
1325 1330 1335
Tyr Ala Ala Gly Arg Ala Gly Glu Arg Phe Gly Val Arg Asn Ser
1340 1345 1350
Gly Ala Ile Thr Val Val Glu Gly Ile Gly Asp Asn Gly Cys Glu
1355 1360 1365
Tyr Met Thr Gly Gly Ile Val Cys Ile Leu Gly Lys Thr Gly Val
1370 1375 1380
Asn Phe Gly Ala Gly Met Thr Gly Gly Phe Ala Tyr Val Leu Asp
1385 1390 1395
Glu Ser Gly Asp Phe Arg Lys Arg Val Asn Pro Glu Leu Val Glu
1400 1405 1410
Val Leu Ser Val Asp Ala Leu Ala Ile His Glu Glu His Leu Arg
1415 1420 1425
Gly Leu Ile Thr Glu His Val Gln His Thr Gly Ser Gln Arg Gly
1430 1435 1440
Glu Glu Ile Leu Ala Asn Trp Ser Thr Phe Ala Thr Lys Phe Ala
1445 1450 1455
Leu Val Lys Pro Lys Ser Ser Asp Val Lys Ala Leu Leu Gly His
1460 1465 1470
Arg Ser Arg Ser Ala Ala Glu Leu Arg Val Gln Ala Gln
1475 1480 1485
<210> 82
<211> 1419
<212> ДНК
<213> Escherichia coli
<400> 82
atgagtcaga atgtttatca atttatcgac ctgcagcgcg ttgatccgcc aaagaaaccg
60
ctgaagatcc gcaaaattga gtttgttgaa atttacgagc cgttttccga aggccaggcc
120
aaagcgcagg ctgaccgctg cctgtcgtgc ggcaacccat actgcgagtg gaaatgcccg
180
gtacacaact acatcccgaa ctggctgaag ctcgccaacg aggggcgtat ttttgaagcg
240
gcggaactgt cgcaccagac caacaccctg ccggaagttt gcggacgagt ctgcccgcaa
300
gaccgtctgt gcgaaggttc ctgcactctg aacgatgagt ttggcgcggt gaccatcggc
360
aacattgagc gctatatcaa cgataaagcg ttcgagatgg gctggcgtcc ggatatgtct
420
ggtgtgaaac agaccggtaa aaaagtggcg attatcggcg caggcccggc aggtctggcg
480
tgtgcggatg tcctgacgcg taacggcgta aaagccgttg tcttcgaccg tcatccagaa
540
attggcgggc tgctgacctt cggtattccg gccttcaagc tggaaaaaga ggtaatgacg
600
cgtcgccgtg aaatcttcac cggcatgggt attgaattca aactcaatac cgaagtgggc
660
cgcgacgtac agctggacga tctgctgagt gattacgatg ccgtgttcct tggcgtcggg
720
acttatcagt caatgcgcgg cgggctggaa aacgaagacg ccgatggcgt gtacgcagcg
780
ctgccgttcc tcatcgccaa caccaaacag ttaatgggct ttggtgaaac ccgcgacgaa
840
ccgttcgtca gcatggaagg caaacgcgtg gtggtccttg gcggtggcga cactgcgatg
900
gactgcgtgc gtacgtccgt gcgccaggga gcgaagcacg ttacctgtgc ctatcgtcgt
960
gatgaagaga acatgccggg ttcccgccgc gaagtgaaaa acgcgcggga agaaggcgta
1020
gagttcaaat tcaacgtcca gccgctgggt attgaagtga acggtaacgg caaagtcagc
1080
ggcgtaaaaa tggtgcgtac cgaaatgggc gaaccggacg ccaaaggccg tcgccgcgcg
1140
gagatcgttg caggttccga acatatcgtt ccggcagatg cggtgatcat ggcgtttggt
1200
ttccgtccac acaacatgga atggctggca aaacacagcg tcgagctgga ttcacaaggc
1260
cgcatcatcg ccccggaagg cagcgacaac gccttccaga ccagcaaccc gaaaatcttt
1320
gctggcggcg atatcgtccg tggttccgat ctggtggtga ccgctattgc cgaaggtcgt
1380
aaggcggcag acggtattat gaactggctg gaagtttaa
1419
<210> 83
<211> 472
<212> ПРТ
<213> Escherichia coli
<400> 83
Met Ser Gln Asn Val Tyr Gln Phe Ile Asp Leu Gln Arg Val Asp Pro
1 5 10 15
Pro Lys Lys Pro Leu Lys Ile Arg Lys Ile Glu Phe Val Glu Ile Tyr
20 25 30
Glu Pro Phe Ser Glu Gly Gln Ala Lys Ala Gln Ala Asp Arg Cys Leu
35 40 45
Ser Cys Gly Asn Pro Tyr Cys Glu Trp Lys Cys Pro Val His Asn Tyr
50 55 60
Ile Pro Asn Trp Leu Lys Leu Ala Asn Glu Gly Arg Ile Phe Glu Ala
65 70 75 80
Ala Glu Leu Ser His Gln Thr Asn Thr Leu Pro Glu Val Cys Gly Arg
85 90 95
Val Cys Pro Gln Asp Arg Leu Cys Glu Gly Ser Cys Thr Leu Asn Asp
100 105 110
Glu Phe Gly Ala Val Thr Ile Gly Asn Ile Glu Arg Tyr Ile Asn Asp
115 120 125
Lys Ala Phe Glu Met Gly Trp Arg Pro Asp Met Ser Gly Val Lys Gln
130 135 140
Thr Gly Lys Lys Val Ala Ile Ile Gly Ala Gly Pro Ala Gly Leu Ala
145 150 155 160
Cys Ala Asp Val Leu Thr Arg Asn Gly Val Lys Ala Val Val Phe Asp
165 170 175
Arg His Pro Glu Ile Gly Gly Leu Leu Thr Phe Gly Ile Pro Ala Phe
180 185 190
Lys Leu Glu Lys Glu Val Met Thr Arg Arg Arg Glu Ile Phe Thr Gly
195 200 205
Met Gly Ile Glu Phe Lys Leu Asn Thr Glu Val Gly Arg Asp Val Gln
210 215 220
Leu Asp Asp Leu Leu Ser Asp Tyr Asp Ala Val Phe Leu Gly Val Gly
225 230 235 240
Thr Tyr Gln Ser Met Arg Gly Gly Leu Glu Asn Glu Asp Ala Asp Gly
245 250 255
Val Tyr Ala Ala Leu Pro Phe Leu Ile Ala Asn Thr Lys Gln Leu Met
260 265 270
Gly Phe Gly Glu Thr Arg Asp Glu Pro Phe Val Ser Met Glu Gly Lys
275 280 285
Arg Val Val Val Leu Gly Gly Gly Asp Thr Ala Met Asp Cys Val Arg
290 295 300
Thr Ser Val Arg Gln Gly Ala Lys His Val Thr Cys Ala Tyr Arg Arg
305 310 315 320
Asp Glu Glu Asn Met Pro Gly Ser Arg Arg Glu Val Lys Asn Ala Arg
325 330 335
Glu Glu Gly Val Glu Phe Lys Phe Asn Val Gln Pro Leu Gly Ile Glu
340 345 350
Val Asn Gly Asn Gly Lys Val Ser Gly Val Lys Met Val Arg Thr Glu
355 360 365
Met Gly Glu Pro Asp Ala Lys Gly Arg Arg Arg Ala Glu Ile Val Ala
370 375 380
Gly Ser Glu His Ile Val Pro Ala Asp Ala Val Ile Met Ala Phe Gly
385 390 395 400
Phe Arg Pro His Asn Met Glu Trp Leu Ala Lys His Ser Val Glu Leu
405 410 415
Asp Ser Gln Gly Arg Ile Ile Ala Pro Glu Gly Ser Asp Asn Ala Phe
420 425 430
Gln Thr Ser Asn Pro Lys Ile Phe Ala Gly Gly Asp Ile Val Arg Gly
435 440 445
Ser Asp Leu Val Val Thr Ala Ile Ala Glu Gly Arg Lys Ala Ala Asp
450 455 460
Gly Ile Met Asn Trp Leu Glu Val
465 470
<210> 84
<211> 1665
<212> ДНК
<213> Escherichia coli
<400> 84
atgtgttcaa tttttggcgt attcgatatc aaaacagacg cagttgagct gcgtaagaaa
60
gccctcgagc tgtcacgcct gatgcgtcat cgtggcccgg actggtccgg tatttatgcc
120
agcgataacg ccattctcgc ccacgaacgt ctgtcaattg ttgacgttaa cgcgggggcg
180
caacctctct acaaccaaca aaaaacccac gtactggcgg taaacggtga aatctacaac
240
caccaggcat tgcgcgccga atatggcgat cgttaccagt tccagaccgg gtctgactgt
300
gaagtgatcc tcgcgctgta tcaggaaaaa gggccggaat ttctcgacga cttgcagggc
360
atgtttgcct ttgcactgta cgacagcgaa aaagatgcct acctgattgg tcgcgaccat
420
ctggggatca tcccactgta tatggggtat gacgaacacg gtcagctgta tgtggcctca
480
gaaatgaaag cgctggtgcc agtttgccgc acgattaaag agttcccggc ggggagctat
540
ttgtggagcc aggacggcga aatccgttct tactatcatc gcgactggtt cgactacgat
600
gcggtgaaag ataacgtgac cgacaaaaac gagctgcgtc aggcactgga agattcagtt
660
aaaagccatc tgatgtctga tgtgccttac ggtgtgctgc tttctggtgg tctggattcc
720
tcaattattt ccgctatcac caagaaatac gcagcccgtc gcgtggaaga tcaggaacgc
780
tctgaagcct ggtggccgca gttacactcc tttgctgtag gtctgccggg ttcaccggat
840
ctgaaagcag cccaggaagt ggcaaaccat ctgggcacgg tgcatcacga aattcacttc
900
actgtacagg aaggtctgga tgccatccgc gacgtgattt accacatcga aacttatgat
960
gtgaccacta ttcgcgcttc aacaccgatg tatttaatgt cgcgtaagat caaggcgatg
1020
ggcattaaaa tggtgctgtc cggtgaaggt tctgatgaag tgttcggcgg ttatctttac
1080
ttccacaaag caccgaatgc caaagaactg catgaagaga cggtgcgtaa actgctggcc
1140
ctgcatatgt atgactgcgc gcgtgccaac aaagcgatgt cagcctgggg cgtggaagca
1200
cgcgttccgt tcctcgacaa aaaattcctt gatgtggcga tgcgtattaa cccacaggat
1260
aaaatgtgcg gtaacggcaa aatggaaaaa cacatcctgc gtgaatgttt tgaagcgtat
1320
ctgcctgcaa gcgtggcctg gcggcagaaa gagcagttct ccgatggcgt cggttacagt
1380
tggatcgaca ccctgaaaga agtggctgcg cagcaggttt ctgatcagca actggaaact
1440
gcccgcttcc gcttcccgta caacacgcca acctctaaag aagcgtactt gtatcgggag
1500
atctttgaag aactattccc gcttccgagc gccgctgagt gcgtgccggg cggtccttcc
1560
gtcgcttgtt cttccgctaa agcgatcgaa tgggatgaag cgttcaagaa aatggacgat
1620
ccgtctggtc gcgcggttgg tgttcaccag tcggcgtata agtaa
1665
<210> 85
<211> 554
<212> ПРТ
<213> Escherichia coli
<400> 85
Met Cys Ser Ile Phe Gly Val Phe Asp Ile Lys Thr Asp Ala Val Glu
1 5 10 15
Leu Arg Lys Lys Ala Leu Glu Leu Ser Arg Leu Met Arg His Arg Gly
20 25 30
Pro Asp Trp Ser Gly Ile Tyr Ala Ser Asp Asn Ala Ile Leu Ala His
35 40 45
Glu Arg Leu Ser Ile Val Asp Val Asn Ala Gly Ala Gln Pro Leu Tyr
50 55 60
Asn Gln Gln Lys Thr His Val Leu Ala Val Asn Gly Glu Ile Tyr Asn
65 70 75 80
His Gln Ala Leu Arg Ala Glu Tyr Gly Asp Arg Tyr Gln Phe Gln Thr
85 90 95
Gly Ser Asp Cys Glu Val Ile Leu Ala Leu Tyr Gln Glu Lys Gly Pro
100 105 110
Glu Phe Leu Asp Asp Leu Gln Gly Met Phe Ala Phe Ala Leu Tyr Asp
115 120 125
Ser Glu Lys Asp Ala Tyr Leu Ile Gly Arg Asp His Leu Gly Ile Ile
130 135 140
Pro Leu Tyr Met Gly Tyr Asp Glu His Gly Gln Leu Tyr Val Ala Ser
145 150 155 160
Glu Met Lys Ala Leu Val Pro Val Cys Arg Thr Ile Lys Glu Phe Pro
165 170 175
Ala Gly Ser Tyr Leu Trp Ser Gln Asp Gly Glu Ile Arg Ser Tyr Tyr
180 185 190
His Arg Asp Trp Phe Asp Tyr Asp Ala Val Lys Asp Asn Val Thr Asp
195 200 205
Lys Asn Glu Leu Arg Gln Ala Leu Glu Asp Ser Val Lys Ser His Leu
210 215 220
Met Ser Asp Val Pro Tyr Gly Val Leu Leu Ser Gly Gly Leu Asp Ser
225 230 235 240
Ser Ile Ile Ser Ala Ile Thr Lys Lys Tyr Ala Ala Arg Arg Val Glu
245 250 255
Asp Gln Glu Arg Ser Glu Ala Trp Trp Pro Gln Leu His Ser Phe Ala
260 265 270
Val Gly Leu Pro Gly Ser Pro Asp Leu Lys Ala Ala Gln Glu Val Ala
275 280 285
Asn His Leu Gly Thr Val His His Glu Ile His Phe Thr Val Gln Glu
290 295 300
Gly Leu Asp Ala Ile Arg Asp Val Ile Tyr His Ile Glu Thr Tyr Asp
305 310 315 320
Val Thr Thr Ile Arg Ala Ser Thr Pro Met Tyr Leu Met Ser Arg Lys
325 330 335
Ile Lys Ala Met Gly Ile Lys Met Val Leu Ser Gly Glu Gly Ser Asp
340 345 350
Glu Val Phe Gly Gly Tyr Leu Tyr Phe His Lys Ala Pro Asn Ala Lys
355 360 365
Glu Leu His Glu Glu Thr Val Arg Lys Leu Leu Ala Leu His Met Tyr
370 375 380
Asp Cys Ala Arg Ala Asn Lys Ala Met Ser Ala Trp Gly Val Glu Ala
385 390 395 400
Arg Val Pro Phe Leu Asp Lys Lys Phe Leu Asp Val Ala Met Arg Ile
405 410 415
Asn Pro Gln Asp Lys Met Cys Gly Asn Gly Lys Met Glu Lys His Ile
420 425 430
Leu Arg Glu Cys Phe Glu Ala Tyr Leu Pro Ala Ser Val Ala Trp Arg
435 440 445
Gln Lys Glu Gln Phe Ser Asp Gly Val Gly Tyr Ser Trp Ile Asp Thr
450 455 460
Leu Lys Glu Val Ala Ala Gln Gln Val Ser Asp Gln Gln Leu Glu Thr
465 470 475 480
Ala Arg Phe Arg Phe Pro Tyr Asn Thr Pro Thr Ser Lys Glu Ala Tyr
485 490 495
Leu Tyr Arg Glu Ile Phe Glu Glu Leu Phe Pro Leu Pro Ser Ala Ala
500 505 510
Glu Cys Val Pro Gly Gly Pro Ser Val Ala Cys Ser Ser Ala Lys Ala
515 520 525
Ile Glu Trp Asp Glu Ala Phe Lys Lys Met Asp Asp Pro Ser Gly Arg
530 535 540
Ala Val Gly Val His Gln Ser Ala Tyr Lys
545 550
<210> 86
<211> 933
<212> ДНК
<213> Escherichia coli
<400> 86
atgttagatg caaacaaatt acagcaggca gtggatcagg cttacaccca atttcactca
60
cttaacggcg gacaaaatgc cgattacatt ccctttctgg cgaatgtacc aggtcaactg
120
gcggcagtgg ctatcgtgac ctgcgatggc aacgtctata gtgcgggtga cagtgattac
180
cgctttgcac tggaatccat ctcgaaagtc tgtacgttag cccttgcgtt agaagatgtc
240
ggcccgcagg cggtacagga caaaattggc gctgacccga ccggattgcc ctttaactca
300
gttatcgcct tagagttgca tggcggcaaa ccgctttcgc cactggtaaa tgctggcgct
360
attgccacca ccagcctgat taacgctgaa aatgttgaac aacgctggca gcgaatttta
420
catatccaac agcaactggc tggcgagcag gtagcgctct ctgacgaagt caaccagtcg
480
gaacaaacaa ccaacttcca taaccgggcc atagcctggc tgctgtactc cgccggatat
540
ctctattgtg atgcaatgga agcctgtgac gtgtataccc gtcagtgctc cacgctcctc
600
aatactattg aactggcaac gcttggcgcg acgctggcgg caggtggtgt gaatccgttg
660
acgcataaac gcgttcttca ggccgacaac gtgccgtaca ttctggccga aatgatgatg
720
gaagggctgt atggtcgctc cggtgactgg gcgtatcgtg ttggtttacc gggcaaaagc
780
ggtgtaggtg gcggtattct ggcggtcgtc cctggagtga tgggaattgc cgcgttctca
840
ccaccgctgg acgaagatgg caacagtgtt cgcggtcaaa aaatggtggc atcggtcgct
900
aagcaactcg gctataacgt gtttaagggc tga
933
<210> 87
<211> 310
<212> ПРТ
<213> Escherichia coli
<400> 87
Met Leu Asp Ala Asn Lys Leu Gln Gln Ala Val Asp Gln Ala Tyr Thr
1 5 10 15
Gln Phe His Ser Leu Asn Gly Gly Gln Asn Ala Asp Tyr Ile Pro Phe
20 25 30
Leu Ala Asn Val Pro Gly Gln Leu Ala Ala Val Ala Ile Val Thr Cys
35 40 45
Asp Gly Asn Val Tyr Ser Ala Gly Asp Ser Asp Tyr Arg Phe Ala Leu
50 55 60
Glu Ser Ile Ser Lys Val Cys Thr Leu Ala Leu Ala Leu Glu Asp Val
65 70 75 80
Gly Pro Gln Ala Val Gln Asp Lys Ile Gly Ala Asp Pro Thr Gly Leu
85 90 95
Pro Phe Asn Ser Val Ile Ala Leu Glu Leu His Gly Gly Lys Pro Leu
100 105 110
Ser Pro Leu Val Asn Ala Gly Ala Ile Ala Thr Thr Ser Leu Ile Asn
115 120 125
Ala Glu Asn Val Glu Gln Arg Trp Gln Arg Ile Leu His Ile Gln Gln
130 135 140
Gln Leu Ala Gly Glu Gln Val Ala Leu Ser Asp Glu Val Asn Gln Ser
145 150 155 160
Glu Gln Thr Thr Asn Phe His Asn Arg Ala Ile Ala Trp Leu Leu Tyr
165 170 175
Ser Ala Gly Tyr Leu Tyr Cys Asp Ala Met Glu Ala Cys Asp Val Tyr
180 185 190
Thr Arg Gln Cys Ser Thr Leu Leu Asn Thr Ile Glu Leu Ala Thr Leu
195 200 205
Gly Ala Thr Leu Ala Ala Gly Gly Val Asn Pro Leu Thr His Lys Arg
210 215 220
Val Leu Gln Ala Asp Asn Val Pro Tyr Ile Leu Ala Glu Met Met Met
225 230 235 240
Glu Gly Leu Tyr Gly Arg Ser Gly Asp Trp Ala Tyr Arg Val Gly Leu
245 250 255
Pro Gly Lys Ser Gly Val Gly Gly Gly Ile Leu Ala Val Val Pro Gly
260 265 270
Val Met Gly Ile Ala Ala Phe Ser Pro Pro Leu Asp Glu Asp Gly Asn
275 280 285
Ser Val Arg Gly Gln Lys Met Val Ala Ser Val Ala Lys Gln Leu Gly
290 295 300
Tyr Asn Val Phe Lys Gly
305 310
<210> 88
<211> 927
<212> ДНК
<213> Escherichia coli
<400> 88
atggcagtcg ccatggataa tgcaatttta gaaaacatct tgcggcaagt gcggccgctc
60
attggtcagg gtaaagtcgc ggattatatt ccggcgctgg ctacagtaga cggttcccga
120
ttggggattg ctatctgtac cgttgacgga cagctttttc aggccggaga cgcgcaagaa
180
cgtttttcca ttcagtctat ttccaaagtg ctgagtctcg ttgtcgccat gcgtcattac
240
tccgaagagg aaatctggca acgcgtcggc aaagatccgt ctggatcacc gttcaattcc
300
ttagtgcaac tggaaatgga gcagggtata ccgcgtaatc cgttcattaa tgccggtgcg
360
ctggtggtct gcgatatgtt gcaagggcga ttaagcgcac cacggcaacg tatgctggaa
420
gtcgtgcgcg gcttaagcgg tgtgtctgat atttcctacg atacggtggt agcgcgttcc
480
gaatttgaac attccgcgcg aaatgcggct atcgcctggc tgatgaagtc gtttggcaat
540
ttccatcatg acgtgacaac cgttctgcaa aactactttc attactgcgc tctgaaaatg
600
agctgtgtag agctggcccg gacgtttgtc tttctggcta atcaggggaa agctattcat
660
attgatgaac cagtggtgac gccaatgcag gcgcggcaaa ttaacgcgct gatggcgacc
720
agtggtatgt accagaacgc gggggagttt gcctggcggg tggggctacc ggcgaaatct
780
ggcgttggtg gcggtattgt ggcgattgtt ccgcatgaaa tggccatcgc tgtctggagt
840
ccggaactgg atgatgcagg taactcgctt gcgggtattg ccgttcttga acaattgacg
900
aaacagttag ggcgttcggt ttattaa
927
<210> 89
<211> 927
<212> ПРТ
<213> Escherichia coli
<400> 89
Gly Thr Gly Gly Cys Ala Gly Thr Cys Gly Cys Cys Ala Thr Gly Gly
1 5 10 15
Ala Thr Ala Ala Thr Gly Cys Ala Ala Thr Thr Thr Thr Ala Gly Ala
20 25 30
Ala Ala Ala Cys Ala Thr Cys Thr Thr Gly Cys Gly Gly Cys Ala Ala
35 40 45
Gly Thr Gly Cys Gly Gly Cys Cys Gly Cys Thr Cys Ala Thr Thr Gly
50 55 60
Gly Thr Cys Ala Gly Gly Gly Thr Ala Ala Ala Gly Thr Cys Gly Cys
65 70 75 80
Gly Gly Ala Thr Thr Ala Thr Ala Thr Thr Cys Cys Gly Gly Cys Gly
85 90 95
Cys Thr Gly Gly Cys Thr Ala Cys Ala Gly Thr Ala Gly Ala Cys Gly
100 105 110
Gly Thr Thr Cys Cys Cys Gly Ala Thr Thr Gly Gly Gly Gly Ala Thr
115 120 125
Thr Gly Cys Thr Ala Thr Cys Thr Gly Thr Ala Cys Cys Gly Thr Thr
130 135 140
Gly Ala Cys Gly Gly Ala Cys Ala Gly Cys Thr Thr Thr Thr Thr Cys
145 150 155 160
Ala Gly Gly Cys Cys Gly Gly Ala Gly Ala Cys Gly Cys Gly Cys Ala
165 170 175
Ala Gly Ala Ala Cys Gly Thr Thr Thr Thr Thr Cys Cys Ala Thr Thr
180 185 190
Cys Ala Gly Thr Cys Thr Ala Thr Thr Thr Cys Cys Ala Ala Ala Gly
195 200 205
Thr Gly Cys Thr Gly Ala Gly Thr Cys Thr Cys Gly Thr Thr Gly Thr
210 215 220
Cys Gly Cys Cys Ala Thr Gly Cys Gly Thr Cys Ala Thr Thr Ala Cys
225 230 235 240
Thr Cys Cys Gly Ala Ala Gly Ala Gly Gly Ala Ala Ala Thr Cys Thr
245 250 255
Gly Gly Cys Ala Ala Cys Gly Cys Gly Thr Cys Gly Gly Cys Ala Ala
260 265 270
Ala Gly Ala Thr Cys Cys Gly Thr Cys Thr Gly Gly Ala Thr Cys Ala
275 280 285
Cys Cys Gly Thr Thr Cys Ala Ala Thr Thr Cys Cys Thr Thr Ala Gly
290 295 300
Thr Gly Cys Ala Ala Cys Thr Gly Gly Ala Ala Ala Thr Gly Gly Ala
305 310 315 320
Gly Cys Ala Gly Gly Gly Thr Ala Thr Ala Cys Cys Gly Cys Gly Thr
325 330 335
Ala Ala Thr Cys Cys Gly Thr Thr Cys Ala Thr Thr Ala Ala Thr Gly
340 345 350
Cys Cys Gly Gly Thr Gly Cys Gly Cys Thr Gly Gly Thr Gly Gly Thr
355 360 365
Cys Thr Gly Cys Gly Ala Thr Ala Thr Gly Thr Thr Gly Cys Ala Ala
370 375 380
Gly Gly Gly Cys Gly Ala Thr Thr Ala Ala Gly Cys Gly Cys Ala Cys
385 390 395 400
Cys Ala Cys Gly Gly Cys Ala Ala Cys Gly Thr Ala Thr Gly Cys Thr
405 410 415
Gly Gly Ala Ala Gly Thr Cys Gly Thr Gly Cys Gly Cys Gly Gly Cys
420 425 430
Thr Thr Ala Ala Gly Cys Gly Gly Thr Gly Thr Gly Thr Cys Thr Gly
435 440 445
Ala Thr Ala Thr Thr Thr Cys Cys Thr Ala Cys Gly Ala Thr Ala Cys
450 455 460
Gly Gly Thr Gly Gly Thr Ala Gly Cys Gly Cys Gly Thr Thr Cys Cys
465 470 475 480
Gly Ala Ala Thr Thr Thr Gly Ala Ala Cys Ala Thr Thr Cys Cys Gly
485 490 495
Cys Gly Cys Gly Ala Ala Ala Thr Gly Cys Gly Gly Cys Thr Ala Thr
500 505 510
Cys Gly Cys Cys Thr Gly Gly Cys Thr Gly Ala Thr Gly Ala Ala Gly
515 520 525
Thr Cys Gly Thr Thr Thr Gly Gly Cys Ala Ala Thr Thr Thr Cys Cys
530 535 540
Ala Thr Cys Ala Thr Gly Ala Cys Gly Thr Gly Ala Cys Ala Ala Cys
545 550 555 560
Cys Gly Thr Thr Cys Thr Gly Cys Ala Ala Ala Ala Cys Thr Ala Cys
565 570 575
Thr Thr Thr Cys Ala Thr Thr Ala Cys Thr Gly Cys Gly Cys Thr Cys
580 585 590
Thr Gly Ala Ala Ala Ala Thr Gly Ala Gly Cys Thr Gly Thr Gly Thr
595 600 605
Ala Gly Ala Gly Cys Thr Gly Gly Cys Cys Cys Gly Gly Ala Cys Gly
610 615 620
Thr Thr Thr Gly Thr Cys Thr Thr Thr Cys Thr Gly Gly Cys Thr Ala
625 630 635 640
Ala Thr Cys Ala Gly Gly Gly Gly Ala Ala Ala Gly Cys Thr Ala Thr
645 650 655
Thr Cys Ala Thr Ala Thr Thr Gly Ala Thr Gly Ala Ala Cys Cys Ala
660 665 670
Gly Thr Gly Gly Thr Gly Ala Cys Gly Cys Cys Ala Ala Thr Gly Cys
675 680 685
Ala Gly Gly Cys Gly Cys Gly Gly Cys Ala Ala Ala Thr Thr Ala Ala
690 695 700
Cys Gly Cys Gly Cys Thr Gly Ala Thr Gly Gly Cys Gly Ala Cys Cys
705 710 715 720
Ala Gly Thr Gly Gly Thr Ala Thr Gly Thr Ala Cys Cys Ala Gly Ala
725 730 735
Ala Cys Gly Cys Gly Gly Gly Gly Gly Ala Gly Thr Thr Thr Gly Cys
740 745 750
Cys Thr Gly Gly Cys Gly Gly Gly Thr Gly Gly Gly Gly Cys Thr Ala
755 760 765
Cys Cys Gly Gly Cys Gly Ala Ala Ala Thr Cys Thr Gly Gly Cys Gly
770 775 780
Thr Thr Gly Gly Thr Gly Gly Cys Gly Gly Thr Ala Thr Thr Gly Thr
785 790 795 800
Gly Gly Cys Gly Ala Thr Thr Gly Thr Thr Cys Cys Gly Cys Ala Thr
805 810 815
Gly Ala Ala Ala Thr Gly Gly Cys Cys Ala Thr Cys Gly Cys Thr Gly
820 825 830
Thr Cys Thr Gly Gly Ala Gly Thr Cys Cys Gly Gly Ala Ala Cys Thr
835 840 845
Gly Gly Ala Thr Gly Ala Thr Gly Cys Ala Gly Gly Thr Ala Ala Cys
850 855 860
Thr Cys Gly Cys Thr Thr Gly Cys Gly Gly Gly Thr Ala Thr Thr Gly
865 870 875 880
Cys Cys Gly Thr Thr Cys Thr Thr Gly Ala Ala Cys Ala Ala Thr Thr
885 890 895
Gly Ala Cys Gly Ala Ala Ala Cys Ala Gly Thr Thr Ala Gly Gly Gly
900 905 910
Cys Gly Thr Thr Cys Gly Gly Thr Thr Thr Ala Thr Thr Ala Ala
915 920 925
<210> 90
<211> 1344
<212> ДНК
<213> Escherichia coli
<400> 90
atggatcaga catattctct ggagtcattc ctcaaccatg tccaaaagcg cgacccgaat
60
caaaccgagt tcgcgcaagc cgttcgtgaa gtaatgacca cactctggcc ttttcttgaa
120
caaaatccaa aatatcgcca gatgtcatta ctggagcgtc tggttgaacc ggagcgcgtg
180
atccagtttc gcgtggtatg ggttgatgat cgcaaccaga tacaggtcaa ccgtgcatgg
240
cgtgtgcagt tcagctctgc catcggcccg tacaaaggcg gtatgcgctt ccatccgtca
300
gttaaccttt ccattctcaa attcctcggc tttgaacaaa ccttcaaaaa tgccctgact
360
actctgccga tgggcggtgg taaaggcggc agcgatttcg atccgaaagg aaaaagcgaa
420
ggtgaagtga tgcgtttttg ccaggcgctg atgactgaac tgtatcgcca cctgggcgcg
480
gataccgacg ttccggcagg tgatatcggg gttggtggtc gtgaagtcgg ctttatggcg
540
gggatgatga aaaagctctc caacaatacc gcctgcgtct tcaccggtaa gggcctttca
600
tttggcggca gtcttattcg cccggaagct accggctacg gtctggttta tttcacagaa
660
gcaatgctaa aacgccacgg tatgggtttt gaagggatgc gcgtttccgt ttctggctcc
720
ggcaacgtcg cccagtacgc tatcgaaaaa gcgatggaat ttggtgctcg tgtgatcact
780
gcgtcagact ccagcggcac tgtagttgat gaaagcggat tcacgaaaga gaaactggca
840
cgtcttatcg aaatcaaagc cagccgcgat ggtcgagtgg cagattacgc caaagaattt
900
ggtctggtct atctcgaagg ccaacagccg tggtctctac cggttgatat cgccctgcct
960
tgcgccaccc agaatgaact ggatgttgac gccgcgcatc agcttatcgc taatggcgtt
1020
aaagccgtcg ccgaaggggc aaatatgccg accaccatcg aagcgactga actgttccag
1080
caggcaggcg tactatttgc accgggtaaa gcggctaatg ctggtggcgt cgctacatcg
1140
ggcctggaaa tggcacaaaa cgctgcgcgc ctgggctgga aagccgagaa agttgacgca
1200
cgtttgcatc acatcatgct ggatatccac catgcctgtg ttgagcatgg tggtgaaggt
1260
gagcaaacca actacgtgca gggcgcgaac attgccggtt ttgtgaaggt tgccgatgcg
1320
atgctggcgc agggtgtgat ttaa
1344
<210> 91
<211> 447
<212> ПРТ
<213> Escherichia coli
<400> 91
Met Asp Gln Thr Tyr Ser Leu Glu Ser Phe Leu Asn His Val Gln Lys
1 5 10 15
Arg Asp Pro Asn Gln Thr Glu Phe Ala Gln Ala Val Arg Glu Val Met
20 25 30
Thr Thr Leu Trp Pro Phe Leu Glu Gln Asn Pro Lys Tyr Arg Gln Met
35 40 45
Ser Leu Leu Glu Arg Leu Val Glu Pro Glu Arg Val Ile Gln Phe Arg
50 55 60
Val Val Trp Val Asp Asp Arg Asn Gln Ile Gln Val Asn Arg Ala Trp
65 70 75 80
Arg Val Gln Phe Ser Ser Ala Ile Gly Pro Tyr Lys Gly Gly Met Arg
85 90 95
Phe His Pro Ser Val Asn Leu Ser Ile Leu Lys Phe Leu Gly Phe Glu
100 105 110
Gln Thr Phe Lys Asn Ala Leu Thr Thr Leu Pro Met Gly Gly Gly Lys
115 120 125
Gly Gly Ser Asp Phe Asp Pro Lys Gly Lys Ser Glu Gly Glu Val Met
130 135 140
Arg Phe Cys Gln Ala Leu Met Thr Glu Leu Tyr Arg His Leu Gly Ala
145 150 155 160
Asp Thr Asp Val Pro Ala Gly Asp Ile Gly Val Gly Gly Arg Glu Val
165 170 175
Gly Phe Met Ala Gly Met Met Lys Lys Leu Ser Asn Asn Thr Ala Cys
180 185 190
Val Phe Thr Gly Lys Gly Leu Ser Phe Gly Gly Ser Leu Ile Arg Pro
195 200 205
Glu Ala Thr Gly Tyr Gly Leu Val Tyr Phe Thr Glu Ala Met Leu Lys
210 215 220
Arg His Gly Met Gly Phe Glu Gly Met Arg Val Ser Val Ser Gly Ser
225 230 235 240
Gly Asn Val Ala Gln Tyr Ala Ile Glu Lys Ala Met Glu Phe Gly Ala
245 250 255
Arg Val Ile Thr Ala Ser Asp Ser Ser Gly Thr Val Val Asp Glu Ser
260 265 270
Gly Phe Thr Lys Glu Lys Leu Ala Arg Leu Ile Glu Ile Lys Ala Ser
275 280 285
Arg Asp Gly Arg Val Ala Asp Tyr Ala Lys Glu Phe Gly Leu Val Tyr
290 295 300
Leu Glu Gly Gln Gln Pro Trp Ser Leu Pro Val Asp Ile Ala Leu Pro
305 310 315 320
Cys Ala Thr Gln Asn Glu Leu Asp Val Asp Ala Ala His Gln Leu Ile
325 330 335
Ala Asn Gly Val Lys Ala Val Ala Glu Gly Ala Asn Met Pro Thr Thr
340 345 350
Ile Glu Ala Thr Glu Leu Phe Gln Gln Ala Gly Val Leu Phe Ala Pro
355 360 365
Gly Lys Ala Ala Asn Ala Gly Gly Val Ala Thr Ser Gly Leu Glu Met
370 375 380
Ala Gln Asn Ala Ala Arg Leu Gly Trp Lys Ala Glu Lys Val Asp Ala
385 390 395 400
Arg Leu His His Ile Met Leu Asp Ile His His Ala Cys Val Glu His
405 410 415
Gly Gly Glu Gly Glu Gln Thr Asn Tyr Val Gln Gly Ala Asn Ile Ala
420 425 430
Gly Phe Val Lys Val Ala Asp Ala Met Leu Ala Gln Gly Val Ile
435 440 445
<210> 92
<211> 1479
<212> ДНК
<213> Escherichia coli
<400> 92
atgagcttac gtgaaaaaac catcagcggc gcgaagtggt cggcgattgc cacggtgatc
60
atcatcggcc tcgggctggt gcagatgacc gtgctggcgc ggattatcga caaccaccag
120
ttcggcctgc ttaccgtgtc gctggtgatt atcgcgctgg cagatacgct ttctgacttc
180
ggtatcgcta actcgattat tcagcgaaaa gaaatcagtc accttgaact caccacgttg
240
tactggctga acgtcgggct ggggatcgtg gtgtgcgtgg cggtgttttt gttgagtgat
300
ctcatcggcg acgtgctgaa taacccggac ctggcaccgt tgattaaaac attatcgctg
360
gcgtttgtgg taatccccca cgggcaacag ttccgcgcgt tgatgcaaaa agagctggag
420
ttcaacaaaa tcggcatgat cgaaaccagc gcggtgctgg cgggcttcac ttgtacggtg
480
gttagcgccc atttctggcc gctggcgatg accgcgatcc tcggttatct ggtcaatagt
540
gcggtgagaa cgctgctgtt tggctacttt ggccgcaaaa tttatcgccc cggtctgcat
600
ttctcgctgg cgtcggtggc accgaactta cgctttggtg cctggctgac ggcggacagc
660
atcatcaact atctcaatac caacctttca acgctcgtgc tggcgcgtat tctcggcgcg
720
ggcgtggcag ggggatacaa cctggcgtac aacgtggccg ttgtgccacc gatgaagctg
780
aacccaatca tcacccgcgt gttgtttccg gcattcgcca aaattcagga cgataccgaa
840
aagctgcgtg ttaacttcta caagctgctg tcggtagtgg ggattatcaa ctttccggcg
900
ctgctcgggc taatggtggt gtcgaataac tttgtaccgc tggtctttgg tgagaagtgg
960
aacagcatta ttccggtgct gcaattgctg tgtgtggtgg gtctgctgcg ctccgtaggt
1020
aacccgattg gttcgctgct gatggcgaaa gcgcgggtcg atatcagctt taaattcaac
1080
gtattcaaaa catttctgtt tattccggcg attgttatag gtgggcagat ggcgggcgcg
1140
atcggcgtca cgcttggctt cctgctggtg caaattatca acaccattct gagttacttc
1200
gtgatgatta aaccggttct tggttccagt tatcgccagt acatcctgag tttatggctg
1260
ccgttttatc tctcgctgcc gacgctggtg gtcagttatg cgctgggcat tgtgctgaaa
1320
gggcaactgg cgctggggat gctgctggcg gtgcaaatag ccacgggggt gctggcgttt
1380
gtggtgatga ttgtgctgtc gcgccatccg ctggtggtgg aagtgaagcg tcagttttgt
1440
cgcagcgaaa aaatgaaaat gcttttacgg gcggggtga
1479
<210> 93
<211> 492
<212> ПРТ
<213> Escherichia coli
<400> 93
Met Ser Leu Arg Glu Lys Thr Ile Ser Gly Ala Lys Trp Ser Ala Ile
1 5 10 15
Ala Thr Val Ile Ile Ile Gly Leu Gly Leu Val Gln Met Thr Val Leu
20 25 30
Ala Arg Ile Ile Asp Asn His Gln Phe Gly Leu Leu Thr Val Ser Leu
35 40 45
Val Ile Ile Ala Leu Ala Asp Thr Leu Ser Asp Phe Gly Ile Ala Asn
50 55 60
Ser Ile Ile Gln Arg Lys Glu Ile Ser His Leu Glu Leu Thr Thr Leu
65 70 75 80
Tyr Trp Leu Asn Val Gly Leu Gly Ile Val Val Cys Val Ala Val Phe
85 90 95
Leu Leu Ser Asp Leu Ile Gly Asp Val Leu Asn Asn Pro Asp Leu Ala
100 105 110
Pro Leu Ile Lys Thr Leu Ser Leu Ala Phe Val Val Ile Pro His Gly
115 120 125
Gln Gln Phe Arg Ala Leu Met Gln Lys Glu Leu Glu Phe Asn Lys Ile
130 135 140
Gly Met Ile Glu Thr Ser Ala Val Leu Ala Gly Phe Thr Cys Thr Val
145 150 155 160
Val Ser Ala His Phe Trp Pro Leu Ala Met Thr Ala Ile Leu Gly Tyr
165 170 175
Leu Val Asn Ser Ala Val Arg Thr Leu Leu Phe Gly Tyr Phe Gly Arg
180 185 190
Lys Ile Tyr Arg Pro Gly Leu His Phe Ser Leu Ala Ser Val Ala Pro
195 200 205
Asn Leu Arg Phe Gly Ala Trp Leu Thr Ala Asp Ser Ile Ile Asn Tyr
210 215 220
Leu Asn Thr Asn Leu Ser Thr Leu Val Leu Ala Arg Ile Leu Gly Ala
225 230 235 240
Gly Val Ala Gly Gly Tyr Asn Leu Ala Tyr Asn Val Ala Val Val Pro
245 250 255
Pro Met Lys Leu Asn Pro Ile Ile Thr Arg Val Leu Phe Pro Ala Phe
260 265 270
Ala Lys Ile Gln Asp Asp Thr Glu Lys Leu Arg Val Asn Phe Tyr Lys
275 280 285
Leu Leu Ser Val Val Gly Ile Ile Asn Phe Pro Ala Leu Leu Gly Leu
290 295 300
Met Val Val Ser Asn Asn Phe Val Pro Leu Val Phe Gly Glu Lys Trp
305 310 315 320
Asn Ser Ile Ile Pro Val Leu Gln Leu Leu Cys Val Val Gly Leu Leu
325 330 335
Arg Ser Val Gly Asn Pro Ile Gly Ser Leu Leu Met Ala Lys Ala Arg
340 345 350
Val Asp Ile Ser Phe Lys Phe Asn Val Phe Lys Thr Phe Leu Phe Ile
355 360 365
Pro Ala Ile Val Ile Gly Gly Gln Met Ala Gly Ala Ile Gly Val Thr
370 375 380
Leu Gly Phe Leu Leu Val Gln Ile Ile Asn Thr Ile Leu Ser Tyr Phe
385 390 395 400
Val Met Ile Lys Pro Val Leu Gly Ser Ser Tyr Arg Gln Tyr Ile Leu
405 410 415
Ser Leu Trp Leu Pro Phe Tyr Leu Ser Leu Pro Thr Leu Val Val Ser
420 425 430
Tyr Ala Leu Gly Ile Val Leu Lys Gly Gln Leu Ala Leu Gly Met Leu
435 440 445
Leu Ala Val Gln Ile Ala Thr Gly Val Leu Ala Phe Val Val Met Ile
450 455 460
Val Leu Ser Arg His Pro Leu Val Val Glu Val Lys Arg Gln Phe Cys
465 470 475 480
Arg Ser Glu Lys Met Lys Met Leu Leu Arg Ala Gly
485 490
<210> 94
<211> 1395
<212> ДНК
<213> Escherichia coli
<400> 94
atgacaaatc taaaaaagcg cgagcgagcg aaaaccaatg catcgttaat ctctatggtg
60
caacgctttt cagatatcac catcatgttt gccggactat ggctggtttg cgaagtcagc
120
ggactgtcat tcctctacat gcacctgttg gtggcgctga ttacgctggt ggtgttccag
180
atgctgggcg gcatcaccga tttttatcgc tcatggcgcg gtgttcgggc agcgacagaa
240
tttgccctgt tgctacaaaa ctggacctta agcgtgattt tcagcgccgg actggtggcg
300
ttcaacaatg atttcgacac gcaactgaaa atctggctgg cgtggtatgc gctgaccagc
360
atcggactgg tggtttgccg ttcgtgtatt cgcattgggg cgggctggct gcgtaatcat
420
ggctataaca agcgcatggt cgcggtggcg ggggatttag ccgccgggca aatgctgatg
480
gagagcttcc gtaaccagcc gtggttaggg tttgaagtgg tgggcgttta ccacgacccg
540
aaaccgggcg gcgtttctaa cgactgggcg ggtaacctgc aacagctggt cgaggacgcg
600
aaagcgggca agattcataa cgtctatatc gcgatgcaaa tgtgcgacgg cgcgcgagtg
660
aaaaaactgg tccatcaact ggcggacacc acctgttcgg tgctgctgat ccccgacgtc
720
tttaccttca acattctcca ttcacgcctc gaagagatga acggcgtacc ggtggtgccg
780
ctttacgaca cgccgctttc cggggttaac cgcctgctca aacgtgcgga agacattgtg
840
ctggcgacgc ttattctgct gctgatctcc ccggtgctgt gctgtattgc gctggcggtg
900
aaactcagtt caccagggcc ggttattttc cgccagactc gctacggcat ggatggcaag
960
ccgatcaaag tgtggaagtt ccgttccatg aaagtgatgg agaacgacaa agtggtgacc
1020
caggcgacgc agaacgatcc gcgcgtcacc aaagtgggga actttctgcg ccgtacctcg
1080
ctggatgaat tgccgcagtt tatcaatgtg ctgaccgggg ggatgtcgat tgtcggtcca
1140
cgtccgcacg cagtagcgca taacgaacag tatcgacagc tcattgaagg ctacatgctg
1200
cgccataagg tgaaaccggg cattaccggc tgggcgcaga ttaacggctg gcgcggcgaa
1260
accgacacgc tggagaaaat ggaaaaacgc gtcgagttcg accttgagta catccgcgaa
1320
tggagcgtct ggttcgatat caaaatcgtt ttcctgacgg tgttcaaagg tttcgttaac
1380
aaagcggcat attga
1395
<210> 95
<211> 464
<212> ПРТ
<213> Escherichia coli
<400> 95
Met Thr Asn Leu Lys Lys Arg Glu Arg Ala Lys Thr Asn Ala Ser Leu
1 5 10 15
Ile Ser Met Val Gln Arg Phe Ser Asp Ile Thr Ile Met Phe Ala Gly
20 25 30
Leu Trp Leu Val Cys Glu Val Ser Gly Leu Ser Phe Leu Tyr Met His
35 40 45
Leu Leu Val Ala Leu Ile Thr Leu Val Val Phe Gln Met Leu Gly Gly
50 55 60
Ile Thr Asp Phe Tyr Arg Ser Trp Arg Gly Val Arg Ala Ala Thr Glu
65 70 75 80
Phe Ala Leu Leu Leu Gln Asn Trp Thr Leu Ser Val Ile Phe Ser Ala
85 90 95
Gly Leu Val Ala Phe Asn Asn Asp Phe Asp Thr Gln Leu Lys Ile Trp
100 105 110
Leu Ala Trp Tyr Ala Leu Thr Ser Ile Gly Leu Val Val Cys Arg Ser
115 120 125
Cys Ile Arg Ile Gly Ala Gly Trp Leu Arg Asn His Gly Tyr Asn Lys
130 135 140
Arg Met Val Ala Val Ala Gly Asp Leu Ala Ala Gly Gln Met Leu Met
145 150 155 160
Glu Ser Phe Arg Asn Gln Pro Trp Leu Gly Phe Glu Val Val Gly Val
165 170 175
Tyr His Asp Pro Lys Pro Gly Gly Val Ser Asn Asp Trp Ala Gly Asn
180 185 190
Leu Gln Gln Leu Val Glu Asp Ala Lys Ala Gly Lys Ile His Asn Val
195 200 205
Tyr Ile Ala Met Gln Met Cys Asp Gly Ala Arg Val Lys Lys Leu Val
210 215 220
His Gln Leu Ala Asp Thr Thr Cys Ser Val Leu Leu Ile Pro Asp Val
225 230 235 240
Phe Thr Phe Asn Ile Leu His Ser Arg Leu Glu Glu Met Asn Gly Val
245 250 255
Pro Val Val Pro Leu Tyr Asp Thr Pro Leu Ser Gly Val Asn Arg Leu
260 265 270
Leu Lys Arg Ala Glu Asp Ile Val Leu Ala Thr Leu Ile Leu Leu Leu
275 280 285
Ile Ser Pro Val Leu Cys Cys Ile Ala Leu Ala Val Lys Leu Ser Ser
290 295 300
Pro Gly Pro Val Ile Phe Arg Gln Thr Arg Tyr Gly Met Asp Gly Lys
305 310 315 320
Pro Ile Lys Val Trp Lys Phe Arg Ser Met Lys Val Met Glu Asn Asp
325 330 335
Lys Val Val Thr Gln Ala Thr Gln Asn Asp Pro Arg Val Thr Lys Val
340 345 350
Gly Asn Phe Leu Arg Arg Thr Ser Leu Asp Glu Leu Pro Gln Phe Ile
355 360 365
Asn Val Leu Thr Gly Gly Met Ser Ile Val Gly Pro Arg Pro His Ala
370 375 380
Val Ala His Asn Glu Gln Tyr Arg Gln Leu Ile Glu Gly Tyr Met Leu
385 390 395 400
Arg His Lys Val Lys Pro Gly Ile Thr Gly Trp Ala Gln Ile Asn Gly
405 410 415
Trp Arg Gly Glu Thr Asp Thr Leu Glu Lys Met Glu Lys Arg Val Glu
420 425 430
Phe Asp Leu Glu Tyr Ile Arg Glu Trp Ser Val Trp Phe Asp Ile Lys
435 440 445
Ile Val Phe Leu Thr Val Phe Lys Gly Phe Val Asn Lys Ala Ala Tyr
450 455 460
<210> 96
<211> 1119
<212> ДНК
<213> Escherichia coli
<400> 96
atgattaatt atggcgttgt tggtgttgga tactttggcg ctgaattagc tcgttttatg
60
aatatgcatg ataatgcaaa aattacatgt gtatacgatc ctgaaaatgg agaaaatatt
120
gcccgtgaat tgcagtgtat caatatgtca agcttggatg ctttagtctc aagtaaatta
180
gtcgattgcg tgatcgtagc caccccaaat tatctgcata aagaaccagt aattaaagca
240
gcaaagaata agaagcatgt tttttgtgaa aaaccaattg cattaagtta tgaagattgt
300
gtggatatgg tcaaagcgtg taaagaagct ggtgtgacct ttatggccgg gcatattatg
360
aattttttca atggggttca atatgcacgg aagttaatta aagaaggtgt tatcggcgaa
420
atattatcat gtcatactaa gagaaatggc tgggaaaaca aacaagagag actttcctgg
480
aaaaagatga aagaacaatc tggtggacat ctatatcatc atatacatga gttagattgt
540
gttcagcatt tacttggaga aataccagag acggttacta tgattggtgg aaatttggcc
600
cattctggtc caggatttgg caatgaagat gatatgttat ttatgacctt ggaattcccg
660
tcaggaaaac tagcaacctt agagtggggg agtgcattta actggccgga acattatgtc
720
atcatcaatg gaactaaagg ctctattaaa attgatatgc aagaaacagc agggtcactt
780
aggattggcg gtcagacaaa gcattttttg gtccatgaaa cacaagaaga agatgatgat
840
cgtcggaaag gcaatatgac ctcagaaatg gatggcgcta tagcatatgg tcatccagga
900
aaaaaaacac cattatggct tgccagttta ataagaaagg agacgttatt cctccataat
960
atcctctgtg gtgcaaaacc tgaagaagat tatattgacc ttctcaatgg tgaggcggcc
1020
atgtcggcga ttgctactgc tgatgctgcc actctttcaa gatcgcagga caggaaagtg
1080
aaaatcagtg agatcattaa acatacatca gtaatgtaa
1119
<210> 97
<211> 372
<212> ПРТ
<213> Escherichia coli
<400> 97
Met Ile Asn Tyr Gly Val Val Gly Val Gly Tyr Phe Gly Ala Glu Leu
1 5 10 15
Ala Arg Phe Met Asn Met His Asp Asn Ala Lys Ile Thr Cys Val Tyr
20 25 30
Asp Pro Glu Asn Gly Glu Asn Ile Ala Arg Glu Leu Gln Cys Ile Asn
35 40 45
Met Ser Ser Leu Asp Ala Leu Val Ser Ser Lys Leu Val Asp Cys Val
50 55 60
Ile Val Ala Thr Pro Asn Tyr Leu His Lys Glu Pro Val Ile Lys Ala
65 70 75 80
Ala Lys Asn Lys Lys His Val Phe Cys Glu Lys Pro Ile Ala Leu Ser
85 90 95
Tyr Glu Asp Cys Val Asp Met Val Lys Ala Cys Lys Glu Ala Gly Val
100 105 110
Thr Phe Met Ala Gly His Ile Met Asn Phe Phe Asn Gly Val Gln Tyr
115 120 125
Ala Arg Lys Leu Ile Lys Glu Gly Val Ile Gly Glu Ile Leu Ser Cys
130 135 140
His Thr Lys Arg Asn Gly Trp Glu Asn Lys Gln Glu Arg Leu Ser Trp
145 150 155 160
Lys Lys Met Lys Glu Gln Ser Gly Gly His Leu Tyr His His Ile His
165 170 175
Glu Leu Asp Cys Val Gln His Leu Leu Gly Glu Ile Pro Glu Thr Val
180 185 190
Thr Met Ile Gly Gly Asn Leu Ala His Ser Gly Pro Gly Phe Gly Asn
195 200 205
Glu Asp Asp Met Leu Phe Met Thr Leu Glu Phe Pro Ser Gly Lys Leu
210 215 220
Ala Thr Leu Glu Trp Gly Ser Ala Phe Asn Trp Pro Glu His Tyr Val
225 230 235 240
Ile Ile Asn Gly Thr Lys Gly Ser Ile Lys Ile Asp Met Gln Glu Thr
245 250 255
Ala Gly Ser Leu Arg Ile Gly Gly Gln Thr Lys His Phe Leu Val His
260 265 270
Glu Thr Gln Glu Glu Asp Asp Asp Arg Arg Lys Gly Asn Met Thr Ser
275 280 285
Glu Met Asp Gly Ala Ile Ala Tyr Gly His Pro Gly Lys Lys Thr Pro
290 295 300
Leu Trp Leu Ala Ser Leu Ile Arg Lys Glu Thr Leu Phe Leu His Asn
305 310 315 320
Ile Leu Cys Gly Ala Lys Pro Glu Glu Asp Tyr Ile Asp Leu Leu Asn
325 330 335
Gly Glu Ala Ala Met Ser Ala Ile Ala Thr Ala Asp Ala Ala Thr Leu
340 345 350
Ser Arg Ser Gln Asp Arg Lys Val Lys Ile Ser Glu Ile Ile Lys His
355 360 365
Thr Ser Val Met
370
<210> 98
<211> 1776
<212> ДНК
<213> Escherichia coli
<400> 98
atgaaaaaaa tcagcttacc gaaaattggt atccgcccgg ttattgacgg tcgtcgcatg
60
ggtgttcgtg agtcgcttga agaacaaaca atgaatatgg cgaaagctac ggccgcactg
120
ctgaccgaga aactgcgcca tgcctgcgga gctgccgtcg agtgtgtcat ttccgatacc
180
tgtatcgcgg gtatggctga agccgctgct tgcgaagaaa aattcagcag tcagaatgta
240
ggcctcacca ttacggtaac gccttgctgg tgctatggca gtgaaaccat cgacatggat
300
ccaacccgcc cgaaggccat ttggggcttt aacggcactg aacgccccgg cgctgtttac
360
ctggcagcgg ctctggcagc tcacagccag aaaggcatcc cagcattctc catttacggt
420
catgacgttc aggatgccga tgacacatcg attcctgccg atgttgaaga aaaactgctg
480
cgctttgccc gcgccggttt ggccgtcgcc agcatgaaag gtaaaagcta tctgtcgctg
540
ggcggcgttt cgatgggtat cgccggttcc attgttgatc acaacttctt tgaatcctgg
600
ctgggaatga aagtccaggc ggtggatatg accgaactgc gtcgccgtat cgatcagaag
660
atttacgacg aagccgaatt ggaaatggca ctggcctggg ctgataaaaa cttccgctat
720
ggcgaagatg aaaataacaa acagtatcaa cgtaatgccg agcaaagccg cgcagttctg
780
cgcgaaagtt tactgatggc gatgtgtatc cgcgacatga tgcaaggcaa cagcaaactg
840
gccgatattg gtcgcgtgga agaatcactt ggctacaacg ccatcgctgc gggcttccag
900
gggcaacgtc actggaccga tcaatatccc aatggtgaca ccgccgaagc gatcctcaac
960
agttcatttg actggaatgg cgtgcgcgaa ccctttgtcg tggcgaccga aaacgacagt
1020
cttaacggcg tggcaatgct aatgggtcac cagctcaccg gcaccgctca ggtatttgcc
1080
gatgtgcgta cctactggtc accagaagca attgagcgtg taacggggca taaactggat
1140
ggactggcag aacacggcat catccatttg atcaactccg gttctgctgc gctggacggt
1200
tcctgtaaac aacgcgacag cgaaggtaac ccgacgatga agccacactg ggaaatctct
1260
cagcaagagg ctgacgcttg cctcgccgct accgaatggt gcccggcgat ccacgaatac
1320
ttccgtggcg gcggttactc ttcccgcttc cttaccgaag gcggcgtccc gttcaccatg
1380
actcgtgtca acatcatcaa aggcctggga ccggtactgc aaatcgcgga aggctggagc
1440
gtggaattgc cgaaggatgt gcatgacatc ctcaacaaac gcaccaactc aacctggcca
1500
accacctggt ttgcaccgcg cctcaccggt aaagggccgt ttacggatgt gtactcggta
1560
atggcgaact ggggcgctaa ccatggggtt ctgaccatcg gccacgttgg cgcagacttt
1620
atcactctcg cctccatgct gcgtatcccg gtatgtatgc acaacgttga agagaccaaa
1680
gtgtatcgtc cttctgcctg ggctgcgcac ggcatggata ttgaaggcca ggattaccgc
1740
gcttgccaga actacggtcc gttgtacaag cgttaa
1776
<210> 99
<211> 591
<212> ПРТ
<213> Escherichia coli
<400> 99
Met Lys Lys Ile Ser Leu Pro Lys Ile Gly Ile Arg Pro Val Ile Asp
1 5 10 15
Gly Arg Arg Met Gly Val Arg Glu Ser Leu Glu Glu Gln Thr Met Asn
20 25 30
Met Ala Lys Ala Thr Ala Ala Leu Leu Thr Glu Lys Leu Arg His Ala
35 40 45
Cys Gly Ala Ala Val Glu Cys Val Ile Ser Asp Thr Cys Ile Ala Gly
50 55 60
Met Ala Glu Ala Ala Ala Cys Glu Glu Lys Phe Ser Ser Gln Asn Val
65 70 75 80
Gly Leu Thr Ile Thr Val Thr Pro Cys Trp Cys Tyr Gly Ser Glu Thr
85 90 95
Ile Asp Met Asp Pro Thr Arg Pro Lys Ala Ile Trp Gly Phe Asn Gly
100 105 110
Thr Glu Arg Pro Gly Ala Val Tyr Leu Ala Ala Ala Leu Ala Ala His
115 120 125
Ser Gln Lys Gly Ile Pro Ala Phe Ser Ile Tyr Gly His Asp Val Gln
130 135 140
Asp Ala Asp Asp Thr Ser Ile Pro Ala Asp Val Glu Glu Lys Leu Leu
145 150 155 160
Arg Phe Ala Arg Ala Gly Leu Ala Val Ala Ser Met Lys Gly Lys Ser
165 170 175
Tyr Leu Ser Leu Gly Gly Val Ser Met Gly Ile Ala Gly Ser Ile Val
180 185 190
Asp His Asn Phe Phe Glu Ser Trp Leu Gly Met Lys Val Gln Ala Val
195 200 205
Asp Met Thr Glu Leu Arg Arg Arg Ile Asp Gln Lys Ile Tyr Asp Glu
210 215 220
Ala Glu Leu Glu Met Ala Leu Ala Trp Ala Asp Lys Asn Phe Arg Tyr
225 230 235 240
Gly Glu Asp Glu Asn Asn Lys Gln Tyr Gln Arg Asn Ala Glu Gln Ser
245 250 255
Arg Ala Val Leu Arg Glu Ser Leu Leu Met Ala Met Cys Ile Arg Asp
260 265 270
Met Met Gln Gly Asn Ser Lys Leu Ala Asp Ile Gly Arg Val Glu Glu
275 280 285
Ser Leu Gly Tyr Asn Ala Ile Ala Ala Gly Phe Gln Gly Gln Arg His
290 295 300
Trp Thr Asp Gln Tyr Pro Asn Gly Asp Thr Ala Glu Ala Ile Leu Asn
305 310 315 320
Ser Ser Phe Asp Trp Asn Gly Val Arg Glu Pro Phe Val Val Ala Thr
325 330 335
Glu Asn Asp Ser Leu Asn Gly Val Ala Met Leu Met Gly His Gln Leu
340 345 350
Thr Gly Thr Ala Gln Val Phe Ala Asp Val Arg Thr Tyr Trp Ser Pro
355 360 365
Glu Ala Ile Glu Arg Val Thr Gly His Lys Leu Asp Gly Leu Ala Glu
370 375 380
His Gly Ile Ile His Leu Ile Asn Ser Gly Ser Ala Ala Leu Asp Gly
385 390 395 400
Ser Cys Lys Gln Arg Asp Ser Glu Gly Asn Pro Thr Met Lys Pro His
405 410 415
Trp Glu Ile Ser Gln Gln Glu Ala Asp Ala Cys Leu Ala Ala Thr Glu
420 425 430
Trp Cys Pro Ala Ile His Glu Tyr Phe Arg Gly Gly Gly Tyr Ser Ser
435 440 445
Arg Phe Leu Thr Glu Gly Gly Val Pro Phe Thr Met Thr Arg Val Asn
450 455 460
Ile Ile Lys Gly Leu Gly Pro Val Leu Gln Ile Ala Glu Gly Trp Ser
465 470 475 480
Val Glu Leu Pro Lys Asp Val His Asp Ile Leu Asn Lys Arg Thr Asn
485 490 495
Ser Thr Trp Pro Thr Thr Trp Phe Ala Pro Arg Leu Thr Gly Lys Gly
500 505 510
Pro Phe Thr Asp Val Tyr Ser Val Met Ala Asn Trp Gly Ala Asn His
515 520 525
Gly Val Leu Thr Ile Gly His Val Gly Ala Asp Phe Ile Thr Leu Ala
530 535 540
Ser Met Leu Arg Ile Pro Val Cys Met His Asn Val Glu Glu Thr Lys
545 550 555 560
Val Tyr Arg Pro Ser Ala Trp Ala Ala His Gly Met Asp Ile Glu Gly
565 570 575
Gln Asp Tyr Arg Ala Cys Gln Asn Tyr Gly Pro Leu Tyr Lys Arg
580 585 590
<210> 100
<211> 1419
<212> ДНК
<213> Escherichia coli
<400> 100
atgaaacaag aagttatcct ggtactcgac tgtggcgcga ccaatgtcag ggccatcgcg
60
gttaatcggc agggcaaaat tgttgcccgc gcctcaacgc ctaatgccag cgatatcgcg
120
atggaaaaca acacctggca ccagtggtct ttagacgcca ttttgcaacg ctttgctgat
180
tgctgtcggc aaatcaatag tgaactgact gaatgccaca tccgcggtat cgccgtcacc
240
acctttggtg tggatggcgc tctggtagat aagcaaggca atctgctcta tccgattatt
300
agctggaaat gtccgcgaac agcagcggtt atggacaata ttgaacggtt aatctccgca
360
cagcggttgc aggctatttc tggcgtcgga gcctttagtt tcaatacgtt atataagttg
420
gtgtggttga aagaaaatca tccacaactg ctggaacgcg cgcacgcctg gctctttatt
480
tcgtcgctga ttaaccaccg tttaaccggc gaattcacta ctgatatcac gatggccgga
540
accagccaga tgctggatat ccagcaacgc gatttcagtc cgcaaatttt acaagccacc
600
ggtattccac gccgactctt ccctcgtctg gtggaagcgg gtgaacagat tggtacgcta
660
cagaacagcg ccgcagcaat gctcggctta cccgttggca taccggtgat ttccgcaggt
720
cacgataccc agttcgccct ttttggcgct ggtgctgaac aaaatgaacc cgtgctctct
780
tccggtacat gggaaatttt aatggttcgc agcgcccagg ttgatacttc gctgttaagt
840
cagtacgccg gttccacctg cgaactggat agccaggcag ggttgtataa cccaggtatg
900
caatggctgg catccggcgt gctggaatgg gtgagaaaac tgttctggac ggctgaaaca
960
ccctggcaaa tgttgattga agaagctcgt ctgatcgcgc ctggcgcgga tggcgtaaaa
1020
atgcagtgtg atttattgtc gtgtcagaac gctggctggc aaggagtgac gcttaatacc
1080
acgcgggggc atttctatcg cgcggcgctg gaagggttaa ctgcgcaatt acagcgcaat
1140
ctacagatgc tggaaaaaat cgggcacttt aaggcctctg aattattgtt agtcggtgga
1200
ggaagtcgca acacattgtg gaatcagatt aaagccaata tgcttgatat tccggtaaaa
1260
gttctcgacg acgccgaaac gaccgtcgca ggagctgcgc tgttcggttg gtatggcgta
1320
ggggaattta acagcccgga agaagcccgc gcacagattc attatcagta ccgttatttc
1380
tacccgcaaa ctgaacctga atttatagag gaagtgtga
1419
<210> 101
<211> 472
<212> ПРТ
<213> Escherichia coli
<400> 101
Met Lys Gln Glu Val Ile Leu Val Leu Asp Cys Gly Ala Thr Asn Val
1 5 10 15
Arg Ala Ile Ala Val Asn Arg Gln Gly Lys Ile Val Ala Arg Ala Ser
20 25 30
Thr Pro Asn Ala Ser Asp Ile Ala Met Glu Asn Asn Thr Trp His Gln
35 40 45
Trp Ser Leu Asp Ala Ile Leu Gln Arg Phe Ala Asp Cys Cys Arg Gln
50 55 60
Ile Asn Ser Glu Leu Thr Glu Cys His Ile Arg Gly Ile Ala Val Thr
65 70 75 80
Thr Phe Gly Val Asp Gly Ala Leu Val Asp Lys Gln Gly Asn Leu Leu
85 90 95
Tyr Pro Ile Ile Ser Trp Lys Cys Pro Arg Thr Ala Ala Val Met Asp
100 105 110
Asn Ile Glu Arg Leu Ile Ser Ala Gln Arg Leu Gln Ala Ile Ser Gly
115 120 125
Val Gly Ala Phe Ser Phe Asn Thr Leu Tyr Lys Leu Val Trp Leu Lys
130 135 140
Glu Asn His Pro Gln Leu Leu Glu Arg Ala His Ala Trp Leu Phe Ile
145 150 155 160
Ser Ser Leu Ile Asn His Arg Leu Thr Gly Glu Phe Thr Thr Asp Ile
165 170 175
Thr Met Ala Gly Thr Ser Gln Met Leu Asp Ile Gln Gln Arg Asp Phe
180 185 190
Ser Pro Gln Ile Leu Gln Ala Thr Gly Ile Pro Arg Arg Leu Phe Pro
195 200 205
Arg Leu Val Glu Ala Gly Glu Gln Ile Gly Thr Leu Gln Asn Ser Ala
210 215 220
Ala Ala Met Leu Gly Leu Pro Val Gly Ile Pro Val Ile Ser Ala Gly
225 230 235 240
His Asp Thr Gln Phe Ala Leu Phe Gly Ala Gly Ala Glu Gln Asn Glu
245 250 255
Pro Val Leu Ser Ser Gly Thr Trp Glu Ile Leu Met Val Arg Ser Ala
260 265 270
Gln Val Asp Thr Ser Leu Leu Ser Gln Tyr Ala Gly Ser Thr Cys Glu
275 280 285
Leu Asp Ser Gln Ala Gly Leu Tyr Asn Pro Gly Met Gln Trp Leu Ala
290 295 300
Ser Gly Val Leu Glu Trp Val Arg Lys Leu Phe Trp Thr Ala Glu Thr
305 310 315 320
Pro Trp Gln Met Leu Ile Glu Glu Ala Arg Leu Ile Ala Pro Gly Ala
325 330 335
Asp Gly Val Lys Met Gln Cys Asp Leu Leu Ser Cys Gln Asn Ala Gly
340 345 350
Trp Gln Gly Val Thr Leu Asn Thr Thr Arg Gly His Phe Tyr Arg Ala
355 360 365
Ala Leu Glu Gly Leu Thr Ala Gln Leu Gln Arg Asn Leu Gln Met Leu
370 375 380
Glu Lys Ile Gly His Phe Lys Ala Ser Glu Leu Leu Leu Val Gly Gly
385 390 395 400
Gly Ser Arg Asn Thr Leu Trp Asn Gln Ile Lys Ala Asn Met Leu Asp
405 410 415
Ile Pro Val Lys Val Leu Asp Asp Ala Glu Thr Thr Val Ala Gly Ala
420 425 430
Ala Leu Phe Gly Trp Tyr Gly Val Gly Glu Phe Asn Ser Pro Glu Glu
435 440 445
Ala Arg Ala Gln Ile His Tyr Gln Tyr Arg Tyr Phe Tyr Pro Gln Thr
450 455 460
Glu Pro Glu Phe Ile Glu Glu Val
465 470
<210> 102
<211> 492
<212> ДНК
<213> Escherichia coli
<400> 102
atgcattgct ataacgggat gacaggttta catcaccgcg aaccgggaat ggttggcgcg
60
ggattaacgg acaagcgcgc ctggctggaa ctgatagccg atggtcatca tgtgcatccg
120
gcggcaatgt cgctgtgttg ttgctgtgcg aaagagagaa tcgtactgat caccgacgcg
180
atgcaggcag ctgggatgcc ggatggtcgc tatacgttat gtggtgaaga agtgcagatg
240
cacggtggcg ttgtccgtac cgcgtctggt gggctggcgg gcagtacgct gtctgttgat
300
gcggcagtgc gcaatatggt cgagttgacg ggcgtaacgc tgcggaagcc atccatatgg
360
cgtcgctgca tccggcgcga atgctgggtg ttgatggtgt tctgggatcg cttaaaccgg
420
gcaaacgcgc cagagtcgtt gcgctggata gcgggctaca tgtgcaacaa atctggattc
480
agggtcaatt ag
492
<210> 103
<211> 167
<212> ПРТ
<213> Escherichia coli
<400> 103
Met His Cys Tyr Asn Gly Met Thr Gly Leu His His Arg Glu Pro Gly
1 5 10 15
Met Val Gly Ala Gly Leu Thr Asp Lys Arg Ala Trp Leu Glu Leu Ile
20 25 30
Ala Asp Gly His His Val His Pro Ala Ala Met Ser Leu Cys Cys Cys
35 40 45
Cys Ala Lys Glu Arg Ile Val Leu Ile Thr Asp Ala Met Gln Ala Ala
50 55 60
Gly Met Pro Asp Gly Arg Tyr Thr Leu Cys Gly Glu Glu Val Gln Met
65 70 75 80
His Gly Gly Val Val Arg Thr Ala Ser Gly Gly Leu Ala Gly Ser Thr
85 90 95
Leu Ser Val Asp Ala Ala Val Arg Asn Met Val Glu Leu Thr Gly Val
100 105 110
Thr Pro Ala Glu Ala Ile His Met Ala Ser Leu His Pro Ala Arg Met
115 120 125
Leu Gly Val Asp Gly Val Leu Gly Ser Leu Lys Pro Gly Lys Arg Ala
130 135 140
Ser Val Val Ala Leu Asp Ser Gly Leu His Val Gln Gln Ile Trp Ile
145 150 155 160
Gln Gly Gln Leu Ala Ser Phe
165
<---
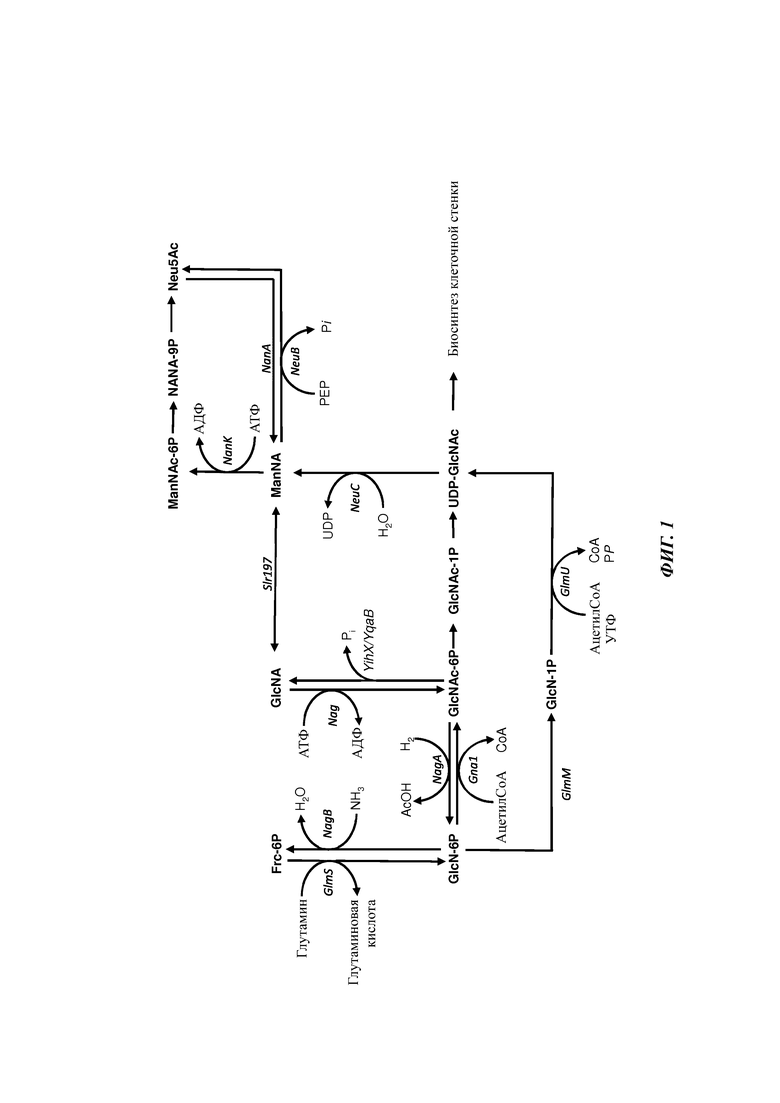
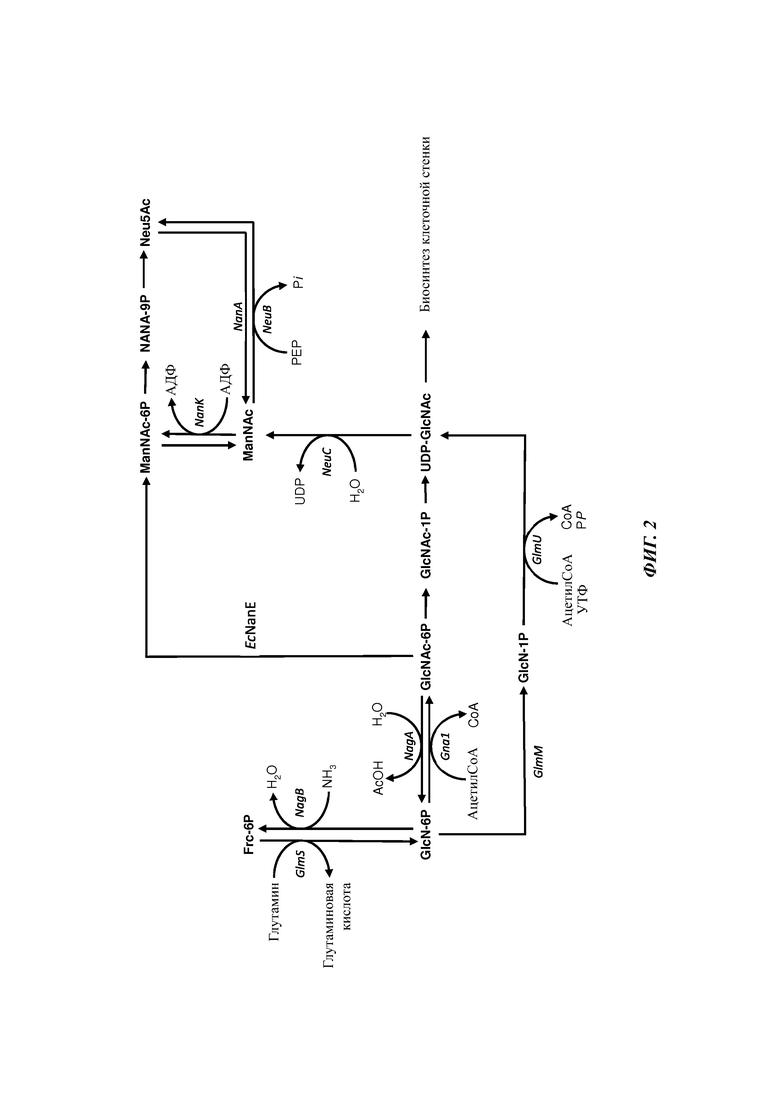
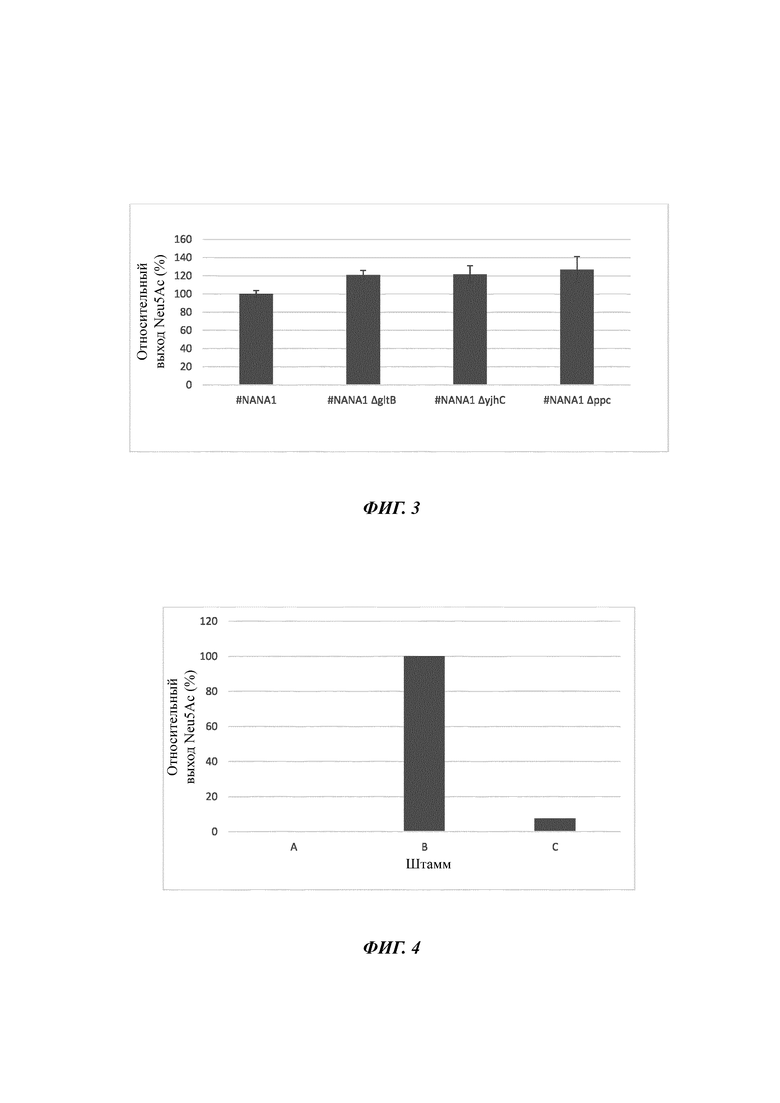
Изобретение относится к биотехнологии. Предложен не встречающийся в природе микроорганизм, синтезирующий ацетилнейраминовую кислоту (Neu5Ac). При этом у указанного микроорганизма имеется путь синтеза сиаловой кислоты, включающий по меньшей мере один гетерологичный фермент, при этом путь биосинтеза сиаловой кислоты включает глутамин-фруктозо-6-фосфатаминотрансферазу, глюкозамин-6-фосфат-N-ацетилтрансферазу, N-ацетилглюкозамин-2-эпимеразу, синтазу N-ацетилнейраминовой кислоты и сахар-фосфатазу суперсемейства HAD-подобных фосфатаз. Также в указанном микроорганизме блокированы природный катаболический путь сиаловой кислоты и система фосфоенолпируват:сахар-фосфотрансфераза для импорта сахарида, который не используется в качестве источника углерода в процессе ферментативного продуцирования Neu5Ac, и при этом микроорганизм может использовать источник экзогенного углерода, присутствующий в ферментационном бульоне, в качестве единственного источника углерода, не используя систему фосфоенолпируват:сахар-фосфотрансфераза для доступа к указанному источнику экзогенного углерода. Предложены также применение указанного микроорганизма для получения Neu5Ac и способ получения Neu5Ac с использованием указанного микроорганизма. Изобретение обеспечивает повышение продукции Neu5Ac. 3 н. и 8 з.п. ф-лы, 4 ил., 6 пр.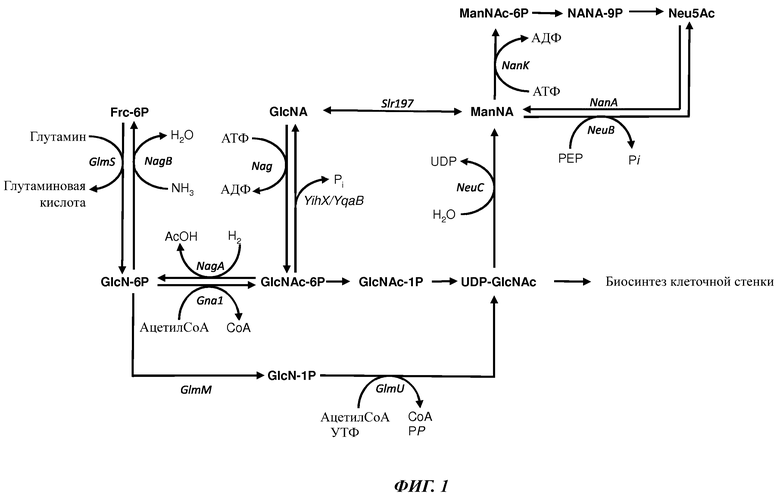
1. Не встречающийся в природе микроорганизм, синтезирующий ацетилнейраминовую кислоту (Neu5Ac), отличающийся тем, что у указанного не встречающегося в природе микроорганизма имеется путь синтеза сиаловой кислоты, включающий по меньшей мере один гетерологичный фермент, причем в микроорганизме природный катаболический путь сиаловой кислоты блокирован за счет того, что один или более генов, кодирующих фермент, который участвует в катаболическом пути сиаловой кислоты, удален из генома не встречающегося в природе микроорганизма за счет того, что нарушена экспрессия одного или более генов, кодирующих фермент, который участвует в катаболическом пути сиаловой кислоты, или за счет того, что нуклеотидная последовательность области, кодирующей белок, по меньшей мере одного гена, кодирующего фермент, который участвует в катаболическом пути сиаловой кислоты, изменена, в результате чего полипептид, кодируемый измененной нуклеотидной последовательностью, кодирующей белок, не обладает ферментативной активностью фермента, кодируемого не измененной нуклеотидной последовательностью, за счет делеции одного или более генов, выбранных из группы генов, кодирующих N-ацетилманнозаминкиназу, N-ацетилманнозамин-6-фосфатэпимеразу, альдолазу N-ацетилнейраминовой кислоты и пермеазу сиаловой кислоты, за счет нарушения экспрессии одного или более указанных генов или за счет изменения нуклеотидной последовательности области, кодирующей белок, по меньшей мере одного из указанных генов, в результате чего полипептид, кодируемый указанной измененной нуклеотидной последовательностью, не обладает ферментативной активностью фермента, кодируемого не измененной нуклеотидной последовательностью,
причем система фосфоенолпируват:сахар-фосфотрансфераза для импорта сахарида, который не используется в качестве источника углерода в процессе ферментативного продуцирования Neu5Ac, блокирована за счет того, что удалены гены manX, manY и manZ, и причем микроорганизм может использовать источник экзогенного углерода, присутствующий в ферментационном бульоне, в качестве единственного источника углерода, не используя систему фосфоенолпируват:сахар-фосфотрансфераза для доступа к указанному источнику экзогенного углерода,
при этом путь биосинтеза сиаловой кислоты включает следующие ферменты: глутамин-фруктозо-6-фосфатаминотрансферазу, глюкозамин-6-фосфат-N-ацетилтрансферазу, N-ацетилглюкозамин-2-эпимеразу, синтазу N-ацетилнейраминовой кислоты и сахар-фосфатазу суперсемейства HAD-подобных фосфатаз.
2. Не встречающийся в природе микроорганизм по п. 1, в котором по меньшей мере один из ферментов пути биосинтеза сиаловой кислоты представляет собой гетерологичный фермент.
3. Не встречающийся в природе микроорганизм по любому из пп. 1, 2, в котором устранены активности одного или более ферментов, выбранных из группы, состоящей из N-ацетилглюкозамин-6-фосфатдезацетилазы и N-ацетилглюкозамин-6-фосфатдезаминазы.
4. Не встречающийся в природе микроорганизм по любому из пп. 1-3, отличающийся тем, что указанный не встречающийся в природе микроорганизм генетически модифицирован для устранения активности N-ацетилглюкозамин-6-фосфатдезацетилазы и/или N-ацетилглюкозамин-6-фосфатдезаминазы за счет делеции одного или обоих генов, кодирующих указанные ферменты, за счет нарушения экспрессии одного или обоих указанных генов или за счет мутации в кодирующей белок области одного или обоих генов, в результате чего полипептид, кодируемый каждой из измененных нуклеотидных последовательностей, не обладает ферментативной активностью фермента, кодируемого не измененной нуклеотидной последовательностью.
5. Не встречающийся в природе микроорганизм по любому из пп. 1-4, отличающийся тем, что указанный не встречающийся в природе микроорганизм включает симпортер сахарида/H+, предпочтительно симпортер сахарида/H+, выбранный из группы, состоящей из симпортера сахарозы-протона, симпортера лактозы-протона и симпортера глюкозы-протона.
6. Не встречающийся в природе микроорганизм по любому из пп. 1-5, отличающийся тем, что указанный не встречающийся в природе микроорганизм обладает улучшенным биосинтезом PEP по сравнению с микроорганизмом дикого типа предпочтительно в результате сверхэкспрессии PEP-синтазы.
7. Не встречающийся в природе микроорганизм по любому из пп. 1-6, отличающийся тем, что не встречающийся в природе микроорганизм не содержит одного или более выбранных из группы, состоящей из функциональной PEP-карбоксилазы, функциональной глутаматсинтазы, функционального WzxC-белка, функциональной трансферазы UDP-глюкозы:ундекапренилфосфат-глюкозо-1-фосфата, функциональной β-галактозидпермеазы, функциональной β-галактозидазы, функционального YjhC-белка, функциональной фукозоизомеразы, функциональной фукулокиназы и функциональной N-ацетилглутаминаминоацилазы.
8. Не встречающийся в природе микроорганизм по любому из пп. 1-7, отличающийся улучшенным синтезом глутамина по сравнению с микроорганизмом дикого типа предпочтительно в результате сверхэкспрессии глутаминсинтазы.
9. Применение не встречающегося в природе микроорганизма по любому из пп. 1-8 для получения Neu5Ac.
10. Способ получения Neu5Ac посредством ферментации с использованием не встречающегося в природе микроорганизма, способного продуцировать Neu5Ac, где способ включает следующие этапы:
a) предоставление не встречающегося в природе микроорганизма по любому из пп. 1-8;
b) культивирование не встречающегося в природе микроорганизма в ферментационном бульоне в условиях, пермиссивных для продуцирования Neu5Ac не встречающимся в природе микроорганизмом; и
c) необязательно выделение Neu5Ac из ферментационного бульона.
11. Способ по п. 10, в котором ферментационный бульон содержит источник углерода для роста не встречающегося в природе микроорганизма, при этом указанный источник углерода предпочтительно выбран из группы, состоящей из глюкозы, ксилозы, сахарозы, фруктозы, лактозы, глицерина, синтез-газа и их комбинаций.
| ZHU, D | |||
| ET AL | |||
| Phosphoenolpyruvate-supply module in Escherichia coli improves N-acetyl-D-neuraminic acid biocatalysis | |||
| Машина для изготовления проволочных гвоздей | 1922 |
|
SU39A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Насос | 1917 |
|
SU13A1 |
| Найдено онлайн: | |||
Авторы
Даты
2023-12-18—Публикация
2018-10-17—Подача