Область техники, к которой относится изобретение
Лектинподобная молекула-1 С-типа (молекула 1 с доменом, подобным лектину С-типа) (CLL1) идентифицирована в качестве молекулы, для которой часто характерна сверхэкспрессия в основном на (миелоидных) стволовых клетках (LSC) при остром миелоидном лейкозе (AML) по сравнению со здоровыми гематопоэтическими стволовыми клетками или другими типами клеток. В настоящем изобретении предложены способы целенаправленного воздействия на CLL1-позитивные злокачественные клетки с помощью химерных антигенных рецепторов (анти-CLL1-CAR), которые представляют собой рекомбинантные химерные белки, обладающие способностью перенаправлять специфичность и реактивность иммунных клеток на выбранный мембранный антиген CLL1. Более конкретно, указанные анти-CLL1 CAR содержат внеклеточный лигандсвязывающий домен, содержащий scFV из некоторых специфичских моноклональных антител к CLL1. Сконструированная иммунные клетки, несущие указанные CAR, обеспечивают адаптивный иммунитет против CLL1-позитивных клеток в качестве компонента различных клеточных терапий, предназначенных для лечения рака, прежде всего гематологических видов рака, с более высокой эффективностью.
Предпосылки создания изобретения
Адаптивная иммунотерапия, в которой применяют перенос аутологичных антигенспецифических Т-клеток, созданных ex vivo, представляет собой перспективную стратегию для лечения вирусных инфекций и рака. Т-клетки, применяемые для адаптивной иммунотерапии, можно создавать либо путем размножения антигенспецифических Т-клеток, либо посредством перенаправления Т-клеток с помощью методов генетической инженерии (Park, Rosenberg и др., 2011). Перенос Т-клеток, специфических в отношении вирусных антигенов, является хорошо зарекомендовавшей себя процедурой, которую применяют для лечения ассоциированных с трансплантацией вирусных инфекций и редких связанных с вирусами злокачественных заболеваний. Продемонстрировано также, что выделение и перенос специфических в отношении опухоли Т-клеток приводит к успеху при лечении меланомы.
В Т-клетках были успешно созданы новые специфичности посредством генетического переноса трансгенных Т-клеточных рецепторов или химерных антигенных рецепторов (scCAR) (Jena, Dotti и др., 2010). scCAR представляют собой синтетические рецепторы, состоящие из «нацеливающего» фрагмента, ассоциированного с одним или несколькими сигнальными доменами в одной слитой молекуле. В целом, связывающий фрагмент scCAR состоит из антигенсвязывающего домена одноцепочечного антитела (scFv), который содержит фрагменты моноклонального антитела, представляющие собой вариабельные домены легкой и тяжелой цепи, соединенные гибким линкером. Успешно применяли также связывающие фрагменты на основе доменов рецептора или лиганда. Сигнальные домены для первого поколения scCAR получали из цитоплазматической области дзета-цепей CD3 или гамма-цепей Fc-рецептора. Было продемонстрировано, что первое поколение scCAR позволяло успешно перенаправлять Т-клеточную цитотоксичность, однако для них не удалось обеспечить пролонгированное размножение и противоопухолевую активность in vivo. Для повышения выживаемости и усиления пролиферации модифицированных с помощью scCAR Т-клеток осуществляли добавление сигнальных доменов из костимуляторных молекул, включая CD28, ОХ-40 (CD134) и 4-1 ВВ (CD137), по отдельности (второе поколение) или в комбинации (третье поколение). scCAR позволяли успешно перенаправлять Т-клетки на антигены, экспрессируемые на поверхности опухолевых клеток при различных злокачественных заболеваниях, включая лимфомы и солидные опухоли (Jena, Dotti и др., 2010)
Следует отметить, что индукционные терапии острого миелоидного лейкоза (AML) в основном не изменялись в течение практически 50 лет, и AML остается заболеванием с плохим прогнозом. AML представляет собой заболевание, характеризующееся быстрой пролиферацией незрелых миелоидных клеток в костном мозге, что приводит к нарушению гематопоэза. Хотя стандартная индукционная химиотерапия может приводить к полным ремиссиям, у многих пациентов, в конце концов, наступает рецидив и смерть в результате заболевания, что требует разработки новых терапевтических средств для лечения AML. Достигнутые в последние годы успехи в иммунофенотипировании AML-клеток позволили обнаружить несколько ассоциированные с AML антигенов клеточной поверхности, которые могут действовать в качестве мишеней для будущих терапий.
Среди прочего, CLL1 (лектинподобная молекула-1 С-типа), вероятно, может являться представляющим интерес опухолевым антигеном-мишенью, поскольку экспрессируется лейкозными бластными клетками у 85-92% проанализированных пациентов с диагностированным AML. Она является представителем семейства молекул группы V лектинподобного рецептора С-типа с молекулярной массой 75 кДа. Молекулы группы V имеют лектинподобный домен, который связывается с лигандами, не относящимися к сахарам. CLL1 представляет собой состоящий из 265 аминокислот трансмембранный гликопротеин типа II (база данных Uniprot: Q5QGZ9 для человеческого белка, кодируемого геном, имеющим код доступа (n°) 160364 в базе данных «Entrez Gene»), который содержит состоящий из 200 ак внеклеточный домен. CLL1 обозначают также в литературе и в базах данных как MICL, CLEC12 и KLRL1.
Bakker с соавторами (Bakker и др., 2004) продемонстрировали, что антиген CLL1 ассоциирован со стволовыми клетками AML. Подобно некоторым другим антигенам (таким как CD33), CLL1 представляет собой белок клеточной поверхности, который специфически экспрессируется на большинстве злокачественных лимфоидных стволовых клетках (AML LSC), но не экспрессируется на здоровых HSC (Van Rhenen и др., 2007). Поэтому CLL1 рассматривается в качестве диагностического маркера при AML (Larsen и др., 2012). Антитела к CLL-1 дают возможность как выявлять AML-специфические стволовые клетки и, вероятно, осуществлять направленный перенос к антигену, так и отличать злокачественные клетки от здоровых стволовых клеток, как при диагностировании, так и при определении ремиссии (van Rhenen и др., 2007). Однако ни одно из описанных к настоящему времени антител не прошло испытания в клинических условиях в качестве терапевтических антител.
Для лечения лимфом часто используются моноклональные антитела, но их применение при лейкозах является более ограниченным. Гемтузумабозогамицин (Mylotarg®) представляет собой моноклональное антитело с присоединенным к нему токсичным для клеток агентом. Ранее одобренный для лечения AML у престарелых пациентов этот препарат был отозван с рынка после проведения исследований, в которых была обнаружена некоторая токсичность, ассоциированная с этим препаратом (пресс-релиз от 10 декабря 2010 г. в PMLIVE «ASH: Pfizer eyes re-launch of Mylotarg»). В последние 10 лет побочные действия обнаружены у других моноклональных терапевтических антител (Klastersky J. «Adverse effects of the humanized antibodies used as cancer therapeutics» Current Opinion in Oncology. 18(4), 2006, сс. 316-320).
В публикации Zhang и др., 2011 описаны мицеллярные наночастицы, ковалентно сшитые с «нацеливающими» на CLL1 пептидами для направленного переноса лекарственного средства (даунорубицин); эти «нацеливающие наномицеллы» транспортируют груз в виде лекарственного средства к внутренней области клеток, экспрессирующих CLL1, и к LSC, выделенным из клинических образцов in vitro. Было установлено, что применение «нацеливающих» на CLL1 наномицелл является перспективным для направленного переноса лекарственных средств к лейкозным стволовым клеткам. Однако никакие терапевтические действия не связаны с самим «нацеливающим» на CLL1 пептидом.
В свете вышеизложенного при создании изобретения осуществлен поиск нового подхода к нацеливанию на CCL1 с использованием иммунных клеток, наделенных специфическими химерными антигенными рецепторами, на основе моноклональных антител к CLL1, которые перенаправляют специфичность иммунных клеток к CLL1-позитивным клеткам.
Для сконструированных иммунных клеток, полученных с помощью указанного подхода, доказана способность эффективно элиминировать CLL1-позитивные злокачественные клетки. В частности, они вероятно могут оказаться особенно ценными в контексте производства аллогеннных TCR-негативных сконструированных иммунных клеток, что позволяет снижать побочные действия, такие как GvHD.
Таким образом, настоящее изобретение открывает путь к лечению пациентов, пораженных состоянием, для которого характерен избыток CLL1-экспрессирующих клеток, с помощью адаптивной иммунотерапии. Более того, при создании настоящего изобретения сконструированы аллогенные иммунные клетки, которые можно применять в качестве «готовых (имеющихся в наличии)» аллогенных терапевтических продуктов. В качестве дополнительного преимущества изобретения CAR-позитивные сконструированные клетки можно получать так, чтобы они были совместимы (т.е. устойчивы) с химиотерапевтическими или истощающими иммунную систему режимами лечения, приводя к достижению синергетических действий между химиотерапией и иммунотерапией.
Краткое изложение сущности изобретения
При создании изобретения созданы CLL1-специфические одноцепочечные scCAR, обладающие различной структурой и содержащие различные scFV, полученные из различных специфических в отношении CLL1 антител.
В частности, при создании изобретения разработаны специфические в отношении CLL1 одноцепочечные CAR (scCAR), которые содержат VH- и VL-цепи, полученные из антител SC02-357, SC02-378, SC02-161, М26, М31, G4, М22, М29, М2, М5, G12, 21.26 и 1075.7 с различными структурами, и идентифицированы высокоспецифические и обладающие очень высокой селективностью конструкции scCAR, которые связываются с экспрессирующими CLL1 клетками и избирательно разрушают экспрессирующие CLL1 раковые клетки.
После неспецифической активации in vitro (например, с использованием покрытых CD3/CD28 гранул и рекомбинантного IL2) Т-клетки из доноров трансформировали полинуклеотидами, экспрессирующими указанные scCAR, используя вирусную трансдукцию. В некоторых случаях Т-клетки подвергали дополнительному конструированию для создания менее аллореактивных или неаллореактивных Т-клеток, более конкретно путем разрушения компонента TCR (αβ-цепи Т-клеточных рецепторов) для предупреждения реакции «трансплантат-против хозяина».
Т-клетки конструировали также с целью создания Т-клеток, устойчивых к противораковым лекарственным средствам, для их применения в комбинации с указанными классическими противораковыми лекарственными средствами.
Полученные сконструированные Т-клетки обладали разной степенью реактивности in vitro в отношении CLL1-позитивных клеток, что свидетельствует о том, что scCAR, предлагаемые в настоящем изобретении, принимали участие в антигензависимой активации, а также пролиферации Т-клеток, что делает возможным их применение для иммунотерапии.
Полученные сконструированные Т-клетки обладали реактивностью in vivo в отношении CLL1-позитивных клеток и значительно снижали количество раковых клеток in vivo.
Сконструированные Т-клетки, предлагаемые в изобретении, которые созданы для проявления реактивности in vivo в отношении CLL1-позитивных клеток, хорошо переносятся при их применении в сочетании с противораковыми лекарственными средствами. В конкретном варианте осуществления изобретения сконструированные Т-клетки, предлагаемые в изобретении, сохраняют эффективность даже после несколько введений, что делает их ценными для иммунотерапии в качестве первой обработки (индукции), в качестве консолидированного лечения, в качестве лечения в комбинации с классической противораковой химиотерапией.
В настоящей заявке подробно описаны полипептиды и полинуклеотидные последовательности, которые кодируют CAR, предлагаемые в настоящем изобретении.
Сконструированные иммунные клетки, предлагаемые в настоящем изобретении, наиболее пригодны для таких терапевтических применений, как лечение острого миелолейкоза (AML).
Краткое описание чертежей
На чертежах показано:
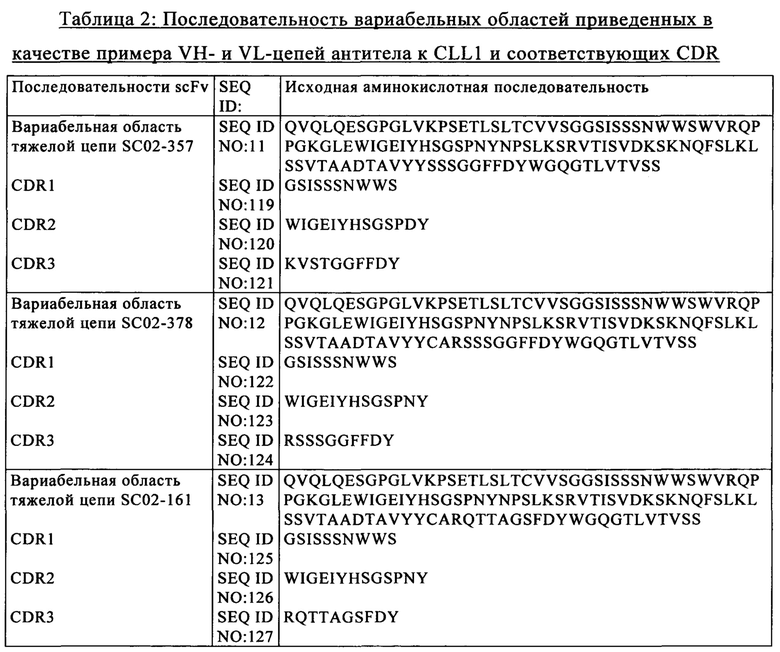
на фиг. 1 - схематическое изображение сконструированной иммунной клетки, предлагаемой в изобретении. Сконструированная иммунная клетка, указанная на этом чертеже, представляет собой Т-клетку, трансдуцированную ретровирусным вектором, кодирующим CLL1-scCAR. Указанную Т-клетку конструировали также с целью обеспечения улучшенного и более безопасного приживления трансплантата пациенту, что необязательно подпадает под объем настоящего изобретения. Ген Х может представлять собой, например, ген, экспрессирующий компонент TCR (TCRальфа или TCRбета), ген Y может представлять собой ген, с которым связана чувствительность Т-клетки к иммуносупрессорным лекарственным средствам типа средств, мишенью которых является CD52 (таких как Campath (Кэмпас) или HPRT (таких как 6-тиогуанин);
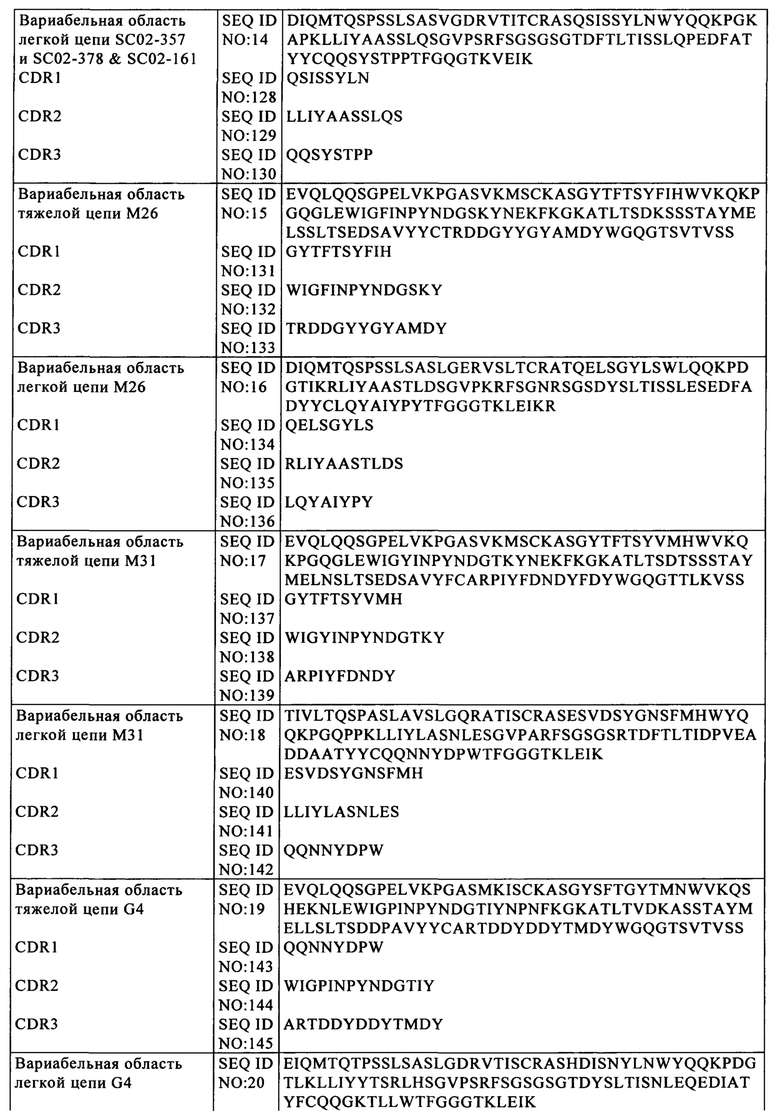
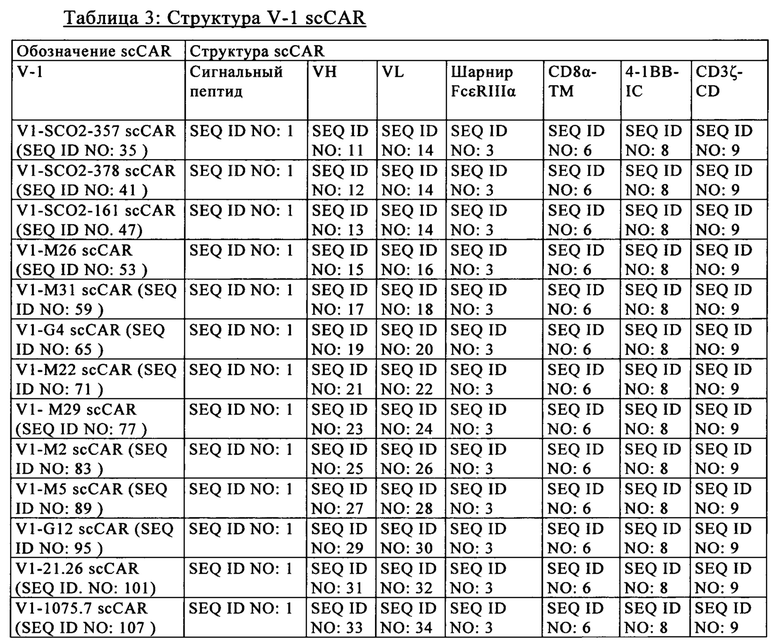
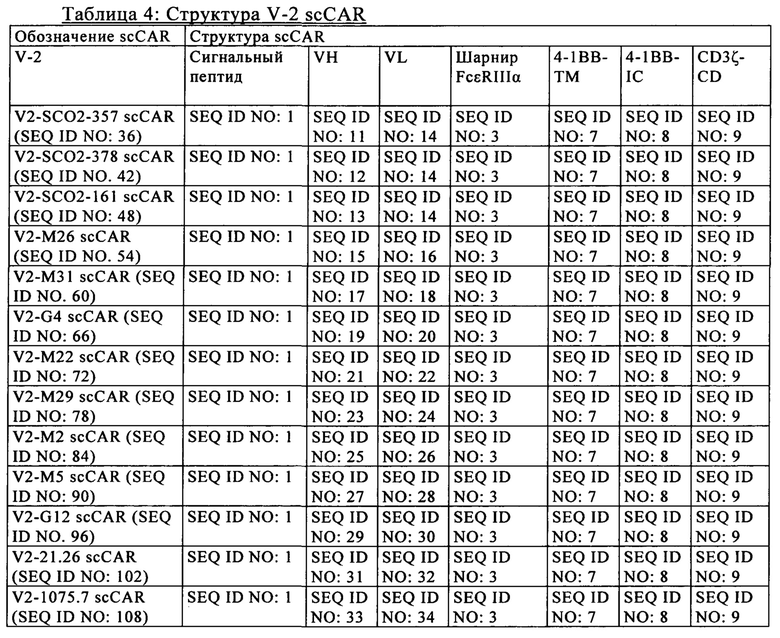
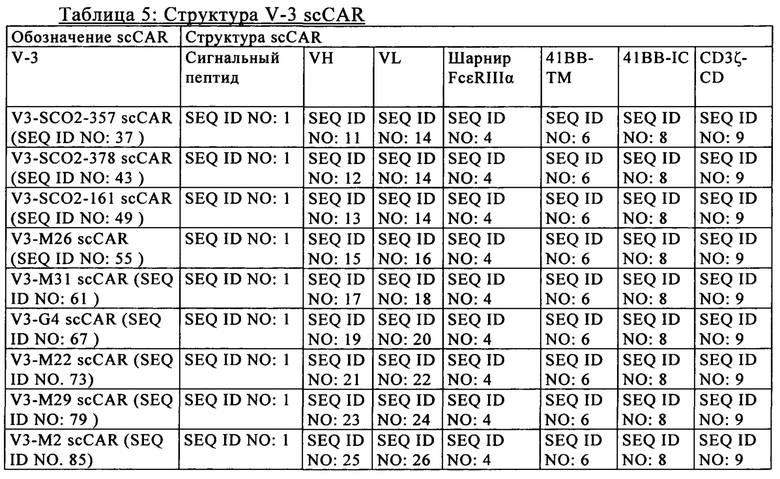
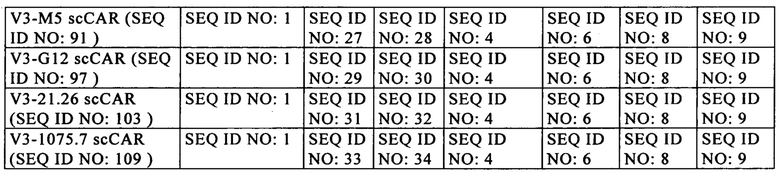
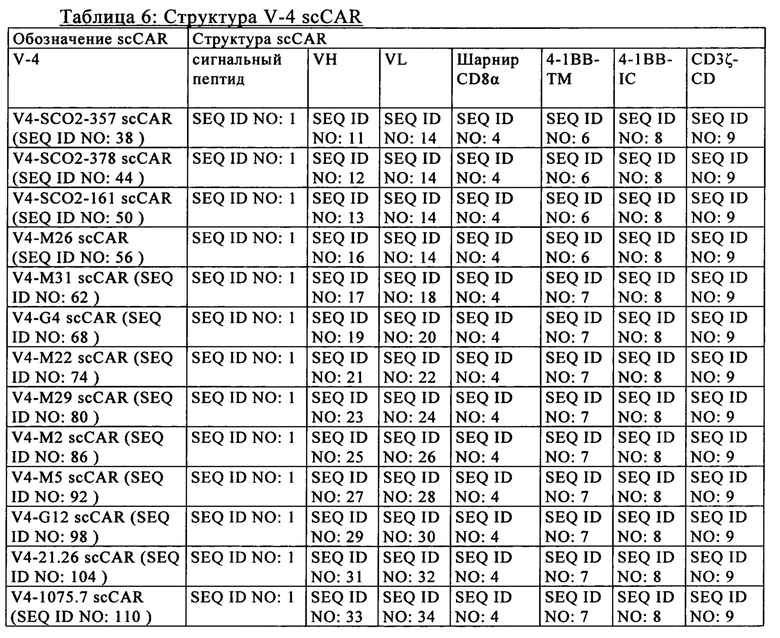
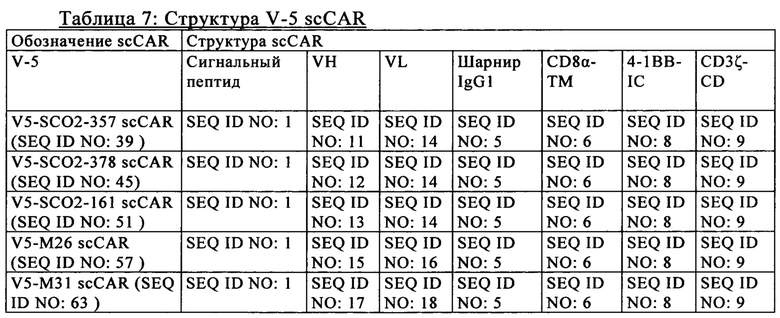
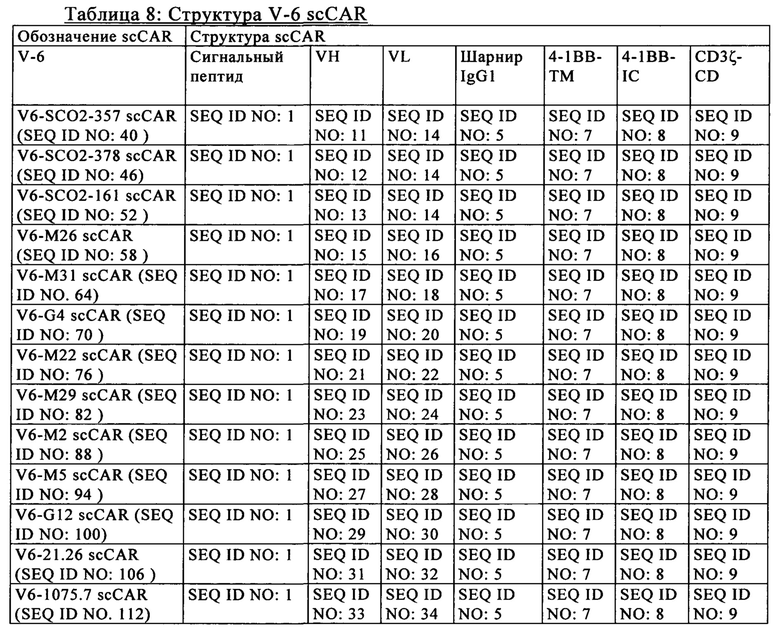
на фиг. 2 - схематическое изображение различной архитектуры scCAR (V1-V6), предлагаемого в изобретении (анти-CLL1 scCAR), с компонентами, которые представлены ниже в таблице 1;
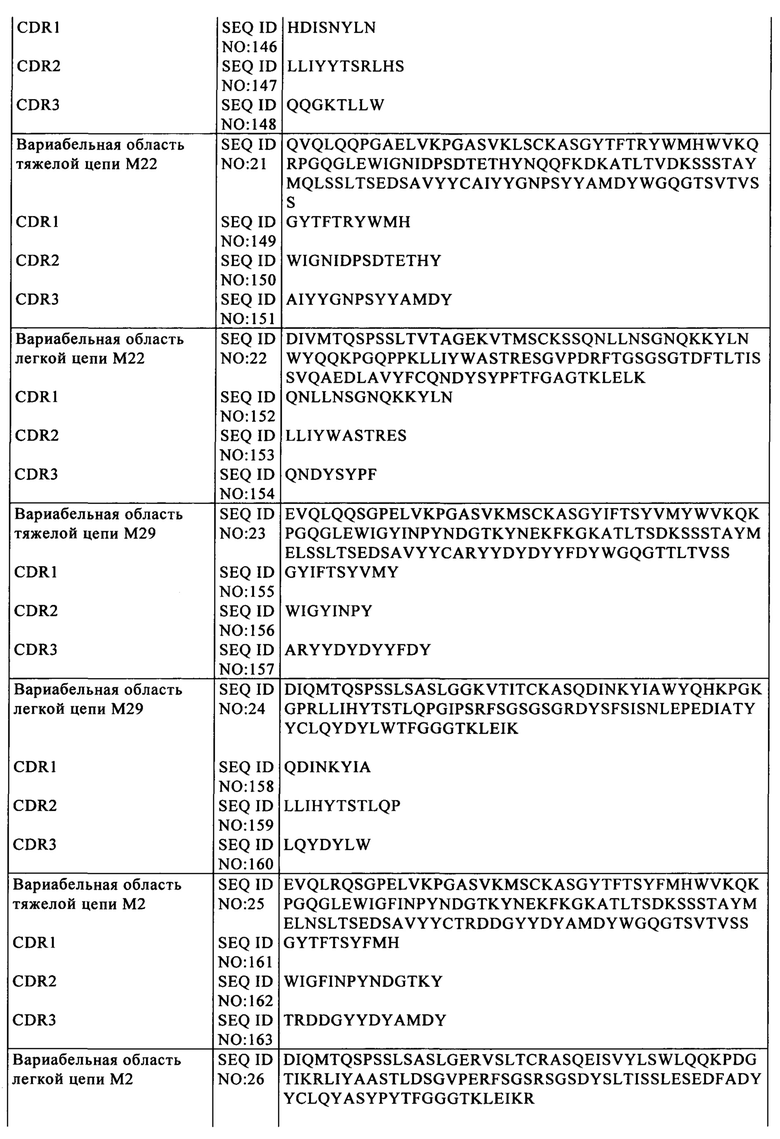
на фиг. 3А и фиг. 3Б - схематическое изображение приведенных в качестве примера CLL1-специфических CAR, предлагаемых в изобретения, которые включают различные эпитопы МАт, являющиеся метками для Т-клеточного истощения, прежде всего мимеотоп(ы) CD20, которые создают для уменьшения возможности возникновения побочных действий, связанных с инъекцией CAR-позитивных клеток;
(A) CLL1-специфический прототип CAR, предлагаемый в настоящем изобретении, не включающий метящую эпитоп последовательность для сортировки или истощения клеток: V1 и v2 обозначают либо VH-, либо VL-цепь соответственно антитела, которое связывается с CLL1, ТМ: трансмембранный домен, L: линкер, ТМ: трансмембранный домен (предпочтительно трансмембранный домен CD8α), 4-1BB: внутриклеточной костимуляторный домен, CD3 ITAM: активирующий домен.
(Б) Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, которые дополнительно включают по меньшей мере один эпитоп, встроенный во внеклеточный лигандсвязывающий домен CAR, где указанный эпитоп встроен между VH- и VL-цепями; к указанному эпитопу могут примыкать («окаймлять») различные линкеры;
(B): Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, в которых два эпитопа встроены во внеклеточный лигандсвязывающий домен CAR, один встроен между N-концом CAR и VH-цепью, к указанному эпитопу может примыкать по меньшей мере один или два линкера; второй эпитоп встроен между VH- и VL-цепями, к указанному 2-ому эпитопу могут также примыкать по меньшей мере один или два линкера. Архитектуры, проиллюстрированные в настоящем описании, различаются линкерами, применяемыми для «окаймления» 2-ого эпитопа.
(Г): Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, в которых два эпитопа встроены во внеклеточный лигандсвязывающий домен CAR, один встроен между VH- и VL-цепями; другой эпитоп встроен между VL-цепью и шарниром, к каждому из указанных эпитопов может примыкать также по меньшей мере один или два линкера. Архитектуры, проиллюстрированные в настоящем описании, различаются линкерами, применяемыми для «окаймления» 1-го эпитопа.
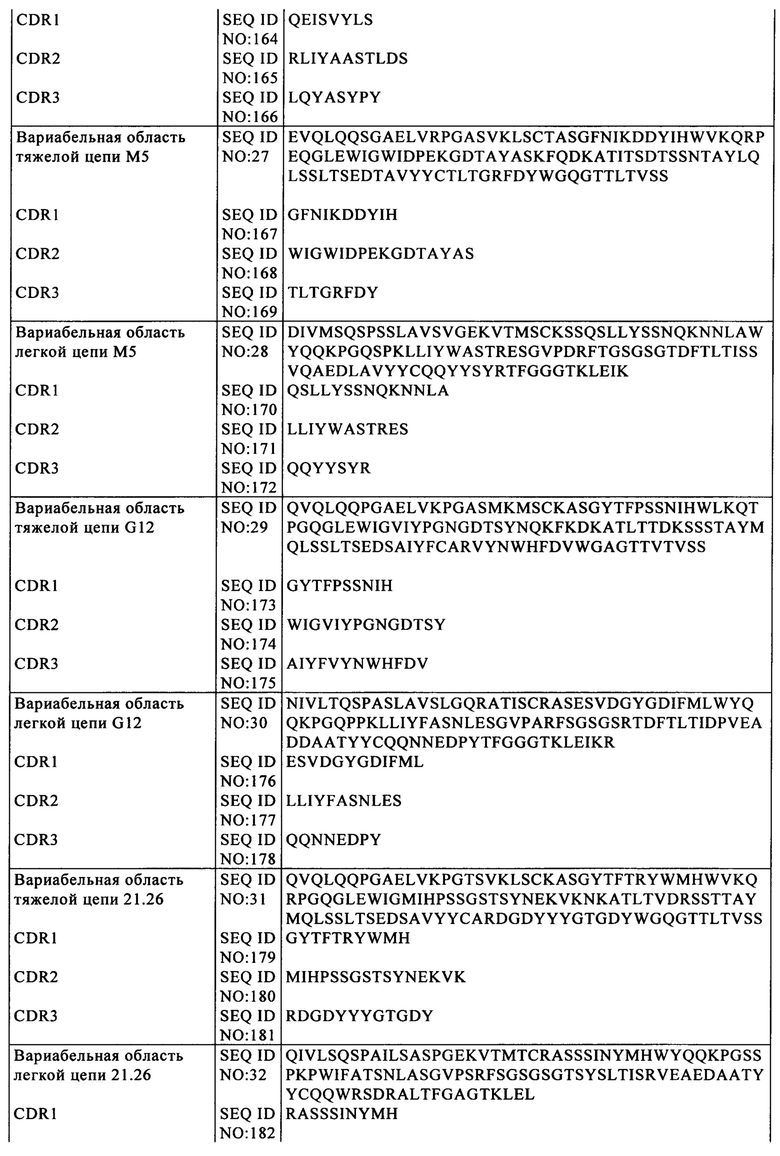
(Д): Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, в которых два эпитопа встроены во внеклеточный домен CAR, один встроен между N-концом CAR и VH-цепью, к указанному эпитопу примыкает по меньшей мере один или два линкера; второй эпитоп встроен между VL-цепью и шарниром, к указанному 2-ому эпитопу примыкают также указанные линкеры.
(Е): Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, в которых два эпитопа встроены во внеклеточный домен CAR, один встроен между N-концом CAR и VH-цепью, к указанному эпитопу примыкает по меньшей мере один или два линкера; второй эпитоп встроен между VH- и VL-цепями, к указанному эпитопу примыкают также указанные линкеры, и третий эпитоп встроен между VL-цепью и шарниром. Эти две архитектуры различаются линкерами, применяемыми для «окаймления» 2-ого эпитопа.
(Ж): Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, в которых по меньшей мере два эпитопа (предпочтительно эпитопы CD20) встроены во внеклеточный лигандсвязывающий домен между шарниром и VH- и VL-цепями анти-CLL1. В третьей приведенной в качестве примера архитектуре один эпитоп CD34 включен между двумя эпитопами CD20. Можно предусматривать дополнительные архитектуры, в которых CD34 заменяет любой другой из указанных ранее эпитопов CD20.
(З): Архитектуры CLL1-специфических CAR, предлагаемых в изобретении, в которых по меньшей мере два эпитопа встроены на конец внеклеточного лигандсвязывающего домена.




Подробное описание изобретения
Если специально не указано иное, то все технические и научные понятия, использованные в настоящем описании, имеют значение, которое является общепринятым для специалистов в области генной терапии, биохимии, генетики и молекулярной биологии.
Для осуществления на практике или тестирования настоящего изобретения можно применять все методы и материалы, сходные или эквивалентные тем, которые указаны в настоящем описании, при этом в настоящей заявке описаны приемлемые методы и материалы. Все публикации, заявки на патент, патенты и другие ссылки, упомянутые в настоящем описании, полностью включены в настоящее описание в качестве ссылки. В случае разночтения следует руководствоваться настоящим описанием, включая определения. Кроме того, материалы, методы и примеры представлены только для иллюстрации и не направлены на ограничение объема изобретения, если не указано иное.
При осуществлении на практике настоящего изобретения следует применять, если не указано иное, общепринятые методы клеточной биологии, культивирования клеток, молекулярной биологии, трансгенной биологии, микробиологии, рекомбинантной ДНК и иммунологии, известные специалисту в данной области. Такие методы подробно описаны в литературе, см., например, Frederick M. AUSUBEL, Current Protocols in Molecular Biology, изд-во Wiley and son Inc, Library of Congress, USA, 2000; Sambrook и др., Molecular Cloning: A Laboratory Manual, 3-е изд., изд-во Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, 2001; Oligonucleotide Synthesis, под ред. M.J. Gait, 1984; Mullis и др., US №4683195; Nucleic Acid Hybridization, под ред. В.D. Harries и S.J. Higgins, 1984; Transcription And Translation, под ред. В.D. Hames и S.J. Higgins, 1984; R.I. Freshney, Culture Of Animal Cells, изд-во Alan R. Liss, Inc., 1987; Immobilized Cells And Enzymes, изд-во IRL Press, 198; В. Perbal, A Practical Guide To Molecular Cloning, 1984; серия: Methods In ENZYMOLOGY, под. ред. J. Abelson и M. Simon, изд-во Academic Press, Inc., New York, прежде всего т. 154 и т. 155, под ред. Wu и др., и т. 185, «Gene Expression Technology», под ред. D. Goeddel; Gene Transfer Vectors For Mammalian Cells, под ред. J.H. Miller и M.P. Calos, 1987, изд-во Cold Spring Harbor Laboratory; Immunochemical Methods In Cell And Molecular Biology, под ред. Mayer и Walker, изд-во Academic Press, London, 1987; Handbook Of Experimental Immunology, тома I-IV, под ред. D.M. Weir и С.С. Blackwell, 1986; и Manipulating the Mouse Embryo, изд-во Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1986.
В настоящем изобретении предложены:
1. CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR), который содержит по меньшей мере:
- внеклеточный лигандсвязывающий домен анти-CLL1,
- трансмембранный домен и
- цитоплазматический сигнальный домен.
2. CLL1-специфический химерный антигенный рецептор по варианту осуществления изобретения 1, дополнительно содержащий костимуляторный домен.
3. CLL1-специфический химерный антигенный рецептор по варианту осуществления изобретения 1, дополнительно содержащий костимуляторный домен CD28 или 4-1 ВВ.
4. CLL1-специфический химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-3, в котором трансмембранный домен представляет собой трансмембранный домен CD8a.
5. CLL1-специфический химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-4, дополнительно содержащий шарнирную область.
6. CLL1-специфический химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-5, в котором указанный цитоплазматический сигнальный домен представляет собой активирующий Т-клетку домен.
7. CLL1-специфический химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-6, где указанный химерный антигенный рецептор экспрессируется в форме единичного полипептида.
8. CLL1-специфический химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-7, в котором указанный внеклеточный лигандсвязывающий домен происходит из моноклонального антитела к CLL1.
9. CLL1-специфический химерный антигенный рецептор по варианту осуществления изобретения 8, в котором внеклеточный лигандсвязывающий домен содержит CDR из VH- и VL-доменов моноклонального(ых) антитела(л) к CLL1.
10. CLL1-специфический химерный антигенный рецептор по варианту осуществления изобретения 9, в котором указанные CDR выбраны из SEQ ID NO: 109-190.
11. CLL1-специфический scCAR no одному из вариантов осуществления изобретения 1-10, который имеет одну из полипептидных структур, выбранных из V1, V3 и V5, которые проиллюстрированы на фиг. 2, указанная структура содержит внеклеточный лигандсвязывающий домен, который содержит VH и VL из моноклонального антитела к CLL1, шарнир, трансмембранный домен CD8α, цитоплазматический домен, включающий сигнальный домен СD3дзета и костимуляторный домен из 4-1 ВВ.
12. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-11, в котором указанная структура V1 содержит шарнир FcγRIIIα и трансмембранный домен CD8α.
13. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-11, в котором указанная структура V3 содержит шарнир CD8α и трансмембранный домен CD8α.
14. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-11, в котором указанная структура V5 содержит шарнир IgG1 и трансмембранный домен CD8α.
15. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-14, в котором указанные VH и VL идентичны по меньшей мере на 80% полипептидной последовательности, выбранной из SEQ ID NO: 11-34.
16. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-15, в котором костимуляторный домен из 4-1 ВВ идентичен по меньшей мере на 80% SEQ ID NO: 8.
17. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-16, в котором указанный сигнальный домен CD3дзeта идентичен по меньшей мере на 80% SEQ ID NO: 9.
18. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-12 или 15-17, в котором указанный шарнир FcγRIIIα идентичен по меньшей мере на 80% SEQ ID NO: 3.
19. CLL1-специфический scCAR no одному из вариантов осуществления изобретения 1-11, 13 или 15-17, в котором указанный шарнир CD8α идентичен по меньшей мере на 80% SEQ ID NO: 4.
20. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-11 или 14-19, в котором указанный шарнир IgG1 идентичен по меньшей мере на 80% SEQ ID NO: 5.
21. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-20, в котором указанный трансмембранный домен CD8α идентичен по меньшей мере на 80% SEQ ID NO: 6.
22. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-21, который дополнительно содержит другой внеклеточный лигандсвязывающий домен, не обладающий специфичностью в отношении CLL1.
23. CLL1-специфический scCAR, имеющий структуру V1, по одному из вариантов осуществления изобретения 1-12, 15-17 или 21-22, который содержит полипептидную последовательность, идентичную по меньшей мере на 80% SEQ ID NO: 35, 41, 47, 53, 59, 65, 71, 77, 83, 89, 95, 101 или 107.
24. CLL1-специфический scCAR, имеющий структуру V3, по одному из вариантов осуществления изобретения 1-11, 13, 15-17 или 21-22, который содержит полипептидную последовательность, идентичную по меньшей мере на 80% SEQ ID NO: 37, 43, 49, 55, 61, 67, 73, 79, 85, 91, 97, 103 или 109.
25. CLL1-специфический scCAR, имеющий структуру V5, по одному из вариантов осуществления изобретения 1-11, 14-19 или 21-22, который содержит полипептидную последовательность, идентичную по меньшей мере на 80% SEQ IDNO: 39, 45,51, 57,63,69, 75, 81, 87, 93, 99, 105 или 111.
26. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-25, дополнительно содержащий сигнальный пептид.
27. CLL1-специфический scCAR по варианту осуществления изобретения 26, в котором указанный сигнальный пептид имеет последовательность, идентичную по меньшей мере на 80% SEQ ID NO: 1 или SEQ ID NO: 2.
28. CLL1-специфический scCAR по одному из вариантов осуществления изобретения 1-27, в котором внеклеточный связывающий домен содержит 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 МАт-специфических эпитопов.
29. Полипептид по одному из вариантов осуществления изобретения 1-28, в котором внеклеточный связывающий домен содержит 1, 2, 3 или 4 МАт-специфических эпитопа.
30. Полипептид по одному из вариантов осуществления изобретения 1-29, в котором внеклеточный связывающий домен содержит 2, 3 или 4 МАт-специфических эпитопа.
31. Полипептид по одному из вариантов осуществления изобретения 1-30, в котором внеклеточный связывающий домен содержит следующую последовательность
V1-L1-V2-(L)х-эпитоп1-(L)x-;
V1-L1-V2-(L)х-эпитоп1-(L)x-эпитоп2-(L)x-;
V1-L1-V2-(L)х-эпитоп1-(L)x-эпитоп2-(L)х-эпитоп3-(L)x-;
(L)х-эпитоп1-(L)x-V1-L1-V2;
(L)х-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2;
эпитоп1-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-V1-L1-V2;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x-;
(L)x-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2-(L)x-эпитоп3-(L)x-;
(L)x-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x-;
V1-(L)x-эпитоп1-(L)x-V2;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x;
(L)x-эпитоп1-(L)x-V1-(L)x-эпитоп2-(L)x-V2; или
(L)х-эпитоп1-(L)x-V1-(L)х-эпитоп2-(L)х-V2-(L)х-эпитоп3-(L)x;
где
V1 обозначает VL и V2 обозначает VH или V1 обозначает VH и V2 обозначает VL;
L1 обозначает линкер, который можно применять для связи VH-цепи с VL-цепью;
L обозначает линкер, содержащий глициновые и сериновые остатки, и каждый из присутствующих во внеклеточном связывающем домене L может быть идентичен или отличаться от другого L, присутствующего в том же самом внеклеточном связывающем домене, и
х обозначает 0 или 1 и каждый из присутствующих х выбирают независимо от других; и
эпитоп 1, эпитоп 2 и эпитоп 3 представляют собой МАт-специфические эпитопы, и они могут быть идентичными или различными.
32. Полипептид по варианту осуществления изобретения 31, в котором внеклеточный связывающий домен содержит следующую последовательность V1-L1-V2-L-эпитоп; V1-L1-V2-L-эпитоп1-L; V1-L1-V2-L-эпитоп1-L-эпитоп2; V1-L1-V2-L-эпитоп1-L-эпитоп2-L; V1-L1-V2-L-эпитоп1-L-эпитоп2-L-эпитоп3; V1-L1-V2-L-эпитоп1-L-эпитоп2-L-эпитоп3-L; V1-L1-V2-эпитоп1; V1-L1-V2-эпитоп1-L; V1-L1-V2-эпитоп1-L-эпитоп2; V1-L1-V2-эпитоп1-L-эпитоп2-L; V1-L1-V2-эпитоп1-L-эпитоп2-L-эпитоп3; V1-L1-V2-эпитоп1-L-эпитоп2-L-эпитоп3-L; эпитоп1-V1-L1-V2; эпитоп1-L-V1-L1-V2; L-эпитоп1-V1-L1-V2; L-эпитоп1-L-V1-L1-V2; эпитоп1-L-эпитоп2-V1 -L1-V2; эпитоп1-L-эпитоп2-L-V1-L1-V2; L-эпитоп1-L-эпитоп2-V1-L1-V2; L-эпитоп1-L-эпитоп2-L-V1-L1-V2; эпитоп1-L-эпитоп2-L-эпитоп3-V1-L1-V2; эпитоп1-L-эпитоп2-L-эпитоп3-L-V1-L1-V2; L-эпитоп1-L-эпитоп2-L-эпитоп3-V1-L1-V2; L-эпитоп1-L-эпитоп2-L-эпитоп3-L-V1-L1-V2; V1-L-эпитоп1-L-V2; L-эпитоп1-L-V1-L-эпитоп2-L-V2; V1-L-эпитоп1-L-V2-L-эпитоп2-L; V1-L-эпитоп1-L-V2-L-эпитоп2-L-эпитоп3; V1-L-эпитоп1-L-V2-L-эпитоп2-эпитоп3; V1-L-эпитоп1-L-V2-L-эпитоп2-L-эпитоп3-эпитоп4; L-эпитоп1-L-V1-L-эпитоп2-L-V2-L-эпитоп3-L; эпитоп1-L-V1-L-эпитоп2-L-V2-L-эпитоп3-L; L-эпитоп1-L-V1-L-эпитоп2-L-V2-L-эпитоп3; L-эпитоп1-L-V1-L1-V2-L-эпитоп2-L; L-эпитоп1-L-V1-L1-V2-L-эпитоп2-L-эпитоп3; L-эпитоп1-L-V1-L1-V2-L-эпитоп2-эпитоп3 или эпитоп1-L-V1-L1 -V2-L-эпитоп2-L-эпитоп3-эпитоп4,
где
V1 обозначает VL и V2 обозначает VH или V1 обозначает VH и V2 обозначает VL;
L1 обозначает любой линкер, который можно применять для связи VH-цепи с VL-цепью;
L обозначает линкер, содержащий глициновые и сериновые остатки, и каждый из присутствующих во внеклеточном связывающем домене L может быть идентичен или отличаться от другого L в том же самом внеклеточном связывающем домене, и
эпитоп 1, эпитоп 2 и эпитоп 3 представляют собой МАт-специфические эпитопы, и они могут быть идентичными или различными.
33. Полипептид по варианту осуществления изобретения 31 или 32, в котором L1 обозначает линкер, содержащий глицин и/или серии.
34. Полипептид по варианту осуществления изобретения 33, в котором L1 обозначает линкер, содержащий аминокислотную последовательность (Gly-Gly-Gly-Ser)n или (Gly-Gly-Gly-Gly-Ser)n, где n обозначает 1, 2, 3, 4 или 5.
35. Полипептид по варианту осуществления изобретения 34, в котором L1 обозначает линкер, содержащий аминокислотную последовательность (Gly4Ser)4 или (Сly4Ser)3.
36. Полипептид по одному из вариантов осуществления изобретения 31-35, в котором L обозначает линкер, содержащий глицин и/или серин.
37. Полипептид по варианту осуществления изобретения 36, в котором L обозначает линкер, имеющий аминокислотную последовательность, выбранную из SGG, GGS, SGGS, SSGGS, GGGG, SGGGG, GGGGS, SGGGGS, GGGGGS, SGGGGGS, SGGGGG, GSGGGGS, GGGGGGGS, SGGGGGGG, SGGGGGGGS или SGGGGSGGGGS.
38. Полипептид по варианту осуществления изобретения 37, в котором L обозначает SGGGG, GGGGS или SGGGGS.
39. Полипептид по одному из варианту осуществления изобретения 31-38, в котором эпитоп 1, эпитоп 2, эпитоп 3 и эпитоп 4 независимо выбирают из МАт-специфических эпитопов, которые специфически распознаются ибритумомабом, тиоксетаном, муромонабом-CD3, тозитумомабом, абциксимабом, базиликсимабом, брентуксимабом ведотином, цетуксимабом, инфликсимабом, ритуксимабом, алемтузумабом, бевацизумабом, цертолизумаба пеголом, даклизумабом, экулизумабом, эфализумабом, гемтузумабом, натализумабом, омализумабом, паливизумабом, ранибизумабом, тоцилизумабом, трастузумабом, ведолизумабом, адалимумабом, белимумабом, канакинумабом, деносумабом, голимумабом, ипилимумабом, офатумумабом, панитумумабом, QBEND-10, алемтузумабом или устекинумабом.
40. Полипептид по одному из вариантов осуществления изобретения 31-38, в котором эпитоп 1, эпитоп 2, эпитоп 3 и эпитоп 4 независимо выбраны из МАт-специфических эпитопов, которые имеют аминокислотную последовательность SEQ ID NO: 113, SEQ ID NO: 191, SEQ ID NO: 192, SEQ ID NO: 193, SEQ ID NO: 194, SEQ ID NO: 195, SEQ ID NO: 195, SEQ ID NO: 196, EQ ID NO: 197, SEQ ID NO: 198 или SEQ ID NO: 199.
41. Полипептид по одному из вариантов осуществления изобретения 31-40, в котором эпитоп 1 представляет собой МАт-специфический эпитоп, имеющий аминокислотную последовательность SEQ ID NO: 113.
42. Полипептид по одному из вариантов осуществления изобретения 31-41, в котором эпитоп 2 представляет собой МАт-специфический эпитоп, имеющий аминокислотную последовательность SEQ ID NO: 113.
43. Полипептид по одному из вариантов осуществления изобретения 31-42, в котором эпитоп 3 представляет собой МАт-специфический эпитоп, имеющий аминокислотную последовательность SEQ ID NO: 113, SEQ ID NO: 198 или SEQ ID NO: 199.
44. Полипептид по одному из вариантов осуществления изобретения 31-43, в котором эпитоп 4 представляет собой МАт-специфический эпитоп, имеющий аминокислотную последовательность SEQ ID NO: 113.
45. Полинуклеотид, кодирующий химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-44.
46. Экспрессионный вектор, содержащий нуклеиновую кислоту по варианту осуществления изобретения 45.
47. Сконструированная лимфоидная иммунная клетка, экспрессирующая на поверхности клеточной мембраны анти-CLL1 CAR по одному из вариантов осуществления изобретения 1-44.
48. Сконструированная лимфоидная иммунная клетка по варианту осуществления изобретения 41, происходящая из воспалительных Т-лимфоцитов, цитотоксических Т-лимфоцитов, регуляторных Т-лимфоцитов или хелперных Т-лимфоцитов.
49. Сконструированная клетка по варианту осуществления изобретения 47 или 48, предназначенная для применения в терапии.
50. Сконструированная лимфоидная иммунная клетка по одному из вариантов осуществления изобретения 41-43, предназначенная для применения в терапии, где пациент представляет собой человека.
51. Сконструированная клетка по одному из вариантов осуществления изобретения 47-50, предназначенная для применения в терапии, где состояние представляет собой предзлокачественное или злокачественное связанное с раком состояние, отличающееся присутствием экспрессирующих CLL1 клеток.
52. Сконструированная клетка по одному из вариантов осуществления изобретения 47-51, предназначенная для применения в терапии, где состояние представляет собой состояние, отличающееся избыточным количеством экспрессирующих CLL1 клеток.
53. Сконструированная клетка по одному из вариантов осуществления изобретения 51-52, предназначенная для применения в терапии, где состояние представляет собой связанное с гематологическим раком состояние.
54. Сконструированная клетка по варианту осуществления изобретения 53, предназначенная для применения в терапии, где связанное с гематологическим раком состояние представляет собой лейкоз.
55. Сконструированная клетка по варианту осуществления изобретения 54, предназначенная для применения в терапии, где лейкоз представляет собой острый миелогенный лейкоз (AML).
56. Сконструированная клетка по одному из вариантов осуществления изобретения 47-55, где экспрессия TCR подавляется в указанной иммунной клетке.
57. Сконструированная клетка по одному из вариантов осуществления изобретения 47-55, где экспрессия по меньшей мере одного белка ГКГС, предпочтительно β2m или HLA, ингибируется или подавляется в указанной иммунной клетке.
58. Сконструированная клетка по одному из вариантов осуществления изобретения 47-57, где указанная клетка изменена в результате мутации для придания устойчивости по меньшей мере к одному иммуносупрессорному или химиотерапевтическому лекарственному средству.
59. Способ ослабления клетки гематологического рака, включающий приведение в контакт указанной клетки со сконструированной клеткой по одному из вариантов осуществления изобретения 47-58 в количестве, эффективном для ослабления указанной раковой клетки.
60. Способ конструирования иммунной клетки, включающий:
(а) получение иммунной клетки,
(б) экспрессию на поверхности указанной клетки по меньшей мере одного CLL1-специфического одноцепочечного химерного антигенного рецептора по одному из вариантов осуществления изобретения 1-44.
61. Способ конструированная иммунной клетки по варианту осуществления изобретения 60, включающий:
(а) получение иммунной клетки,
(б) интродукцию в указанную клетку по меньшей мере одного полинуклеотида, который кодирует указанный CLL1-специфический одноцепочечный химерный антигенный рецептор,
(в) экспрессию указанного полинуклеотида в указанной клетке.
62. Способ конструированная иммунной клетки по варианту осуществления изобретения 61, включающий:
(а) получение иммунной клетки,
(б) интродукцию в указанную клетку по меньшей мере одного полинуклеотида, который кодирует указанный CLL1-специфический одноцепочечный химерный антигенный рецептор,
(в) интродукцию по меньшей мере одного другого химерного антигенного рецептора, не специфического в отношении CLL1.
63. Способ лечения индивидуума, который нуждается в этом, включающий:
(а) получение иммунной клетки, экспрессирующей на поверхности анти-CLL1-специфический одноцепочечный химерный антигенный рецептор по одному из вариантов осуществления изобретения 1-44;
(б) введение указанных иммунных клеток указанному пациенту.
64. Способ по варианту осуществления изобретения 63, в котором иммунную клетку получают из донора.
65. Способ по варианту осуществления изобретения 63, в котором указанную иммунную клетку получают из самого пациента.
CLL1-специфические одноцепочечные химерные антигенные рецепторы
Настоящее изобретение относится к CLL1-специфическому химерному антигенному рецептору, содержащему внеклеточный лигандсвязывающий домен, который специфически направлен против одной части антигена CLL1, трансмембранный домен и домен трансдукции сигнала.
Под химерным антигенным рецептором (CAR) подразумеваются молекулы, в которых объединены внеклеточный связывающий домен, направленный против компонента, который присутствует на клетке-мишени, например, происходящая из антитела специфичность в отношении требуемого антигена (например, опухолевого антигена), с компонентом рецептора иммунной клетки, с образованием химерного белка, который может трансдуцировать активирующий или ингибирующий сигнал для иммунной активности клеток.
Более конкретно настоящее изобретение относится к CLL1-специфическому химерному антигенному рецептору (анти-CLL1 CAR), содержащему по меньшей мере:
- внеклеточный лигандсвязывающий домен анти-CLL1,
- трансмембранный домен и
- цитоплазматический сигнальный домен.
Предпочтительно CLL1-специфический химерный антигенный рецептор, предлагаемый в изобретении, содержит также костимуляторный домен, более предпочтительно костимуляторный домен из CD28 или 4-1ВВ, описанный, например, у Jena В., G. Dotti и др., 2010). Он может содержать также трансмембранный домен, который может представлять собой трансмембранный домен CD8α, а также необязательную шарнирную область.
Домен трансдукции сигнала или «цитоплазматический сигнальный домен» CAR, предлагаемого в настоящем изобретении, ответствен за внутриклеточную передачу сигнала после связывания внеклеточного лигандсвязывающего домена с мишенью, что приводит к активации или ингибированию иммунной клетки и иммунного ответа. Другими словами, домен трансдукции сигнала ответствен для активацию или инактивацию по меньшей мере одной из обычных эффекторных функций иммунной клетки, в которой экспрессируется CAR. Например, эффекторная функция Т-клетки может представлять собой цитолитическую активность или хелперную активность, включая секрецию цитокинов. Таким образом, понятие «цитоплазматический сигнальный домен» относится к части белка, которая трансдуцирует сигнал эффекторной сигнальной функции и принуждает клетку осуществлять специализированную функцию.
Цитоплазматический сигнальный домен, который предпочтительно имеет происхождение из человеческого белка, участвующего в пути(ях) трансдукции сигнала, в зависимости от природы сигнала определяет, представляет ли собой анти-CLL1 CAR позитивный CAR (PCAR), или негативный CAR (NCAR). Соответственно CAR представляет собой PCAR, когда сигнальный домен, такой как CD3дзета из человеческого TCR-рецептора, обладает способностью стимулировать клеточную иммунную активность иммунной клетки, когда внеклеточный лигандсвязывающий домен связывается с CLL1. И наоборот, анти-CLL1 CAR представляет собой NCAR или ингибирующий CAR (iCAR), когда сигнальный домен обладает способностью снижать клеточную иммунную активность, что имеет место в случае сигнальных доменов человеческих ингибирующих иммунную систему рецепторов CTLA-4 и PD-1 (Federov и др., Sci Transl Med. 5 (215), 11 декабря 2013 г., 215ra172). Предпочтительными примерами домена трансдукции сигнала для применения в анти-CLL1 CAR могут служить цитоплазматические последовательности Т-клеточного рецептора и корецепторов, совместное действие которых состоит в инициации трансдукции сигнала после контакта с рецептором антигена, а также любое производное или любой вариант указанных последовательностей и любой синтетической последовательности, которая имеет такую же функциональную способность. Домен трансдукции сигнала содержит два различных класса цитоплазматических сигнальных последовательностей, те, которые инициируют антигензависимую первичную активацию, и те, которые действуют независимым от антигена образом, обеспечивая вторичный или костимуляторный сигнал. Первичная цитоплазматическая сигнальная последовательность может содержать сигнальные мотивы, известные как мотивы активации иммунных рецепторов на основе тирозина, ITAM. ITAM представляют собой хорошо известные сигнальные мотивы, присутствующие во внутрицитоплазматическом «хвосте» различных рецепторов, которые служат в качестве сайтов связывания для класса syk/zap70 тирозинкиназ. Примерами ITAM, применяемых согласно изобретению, могут служить (но не ограничиваясь только ими) ITAM, происходящие из ТСRдзета, FcR-гамма, FcRбета, FcRэпсилон, CD3гамма, СD3дельта, СD3эпсилон, CD5, CD22, CD79a, CD79b и CD66d. В предпочтительном варианте осуществления изобретения домен трансдукции сигнала анти-CLL1 CAR может содержать происходящий из CD3дзета сигнальный домен, который имеет аминокислотную последовательность, идентичную по меньшей мере на 70%, предпочтительно по меньшей мере на 80%, более предпочтительно по меньшей мере на 90%, 95%, 97% или 99%, или 100% аминокислотной последовательности, выбранной из группы, включающей SEQ ID NO: 9.
Костимуляторная молекула представляет собой молекулу клеточной поверхности, отличную от антигенного рецептора или его лигандов, которая требуется для эффективного иммунного ответа. Понятие «костимуляторный лиганд» относится к молекуле на антигенпрезентирующей клетке, которая специфически связывается с когнатной костимуляторной молекулой на Т-клетке, создавая тем самым сигнал, который, в дополнение к первичному сигналу, создаваемому, например, при связывании комплекса TCR/CD3 с молекулой ГКГС, загруженной пептидом, опосредует Т-клеточный ответ, включая (но не ограничиваясь только ими) активацию пролиферации, дифференцировку и т.п. Костимуляторный лиганд может включать (но не ограничиваясь только ими) CD7, В7-1 (CD80), В7-2 (CD86), PD-L1, PD-L2, 4-1BBL, OX40L, индуцибельный костимуляторный лиганд (ICOS-L), молекулу межклеточной адгезии (ICAM), CD30L, CD40, CD70, CD83, HLA-G, MICA, M1CB, HVEM, рецептор лимфотоксина бета, 3/TR6, ILT3, ILT4, агонист или антитело, который/которое связывается с лигандом Толл-рецептора, и лиганд, который специфически связывается с В7-Н3. Под понятие «костимуляторный лиганд» подпадает, среди прочего, антитело, которое специфически связывается с костимуляторной молекулой, присутствующей на Т-клетке, такой как (но не ограничиваясь только ими) CD27, CD28, 4-1ВВ, OX40, CD30, CD40, PD-1, ICOS, антиген-1, ассоциированный с функцией лимфоцитов (LFA-1), CD2, CD7, LTGHT, NKG2C, В7-Н3, лиганд, специфически связывающийся с CD83. Понятие «костимуляторная молекула» относится к когнатному связывающему партнеру, присутствующему на Т-клетке, который специфически связывается с костимуляторным лигандом, опосредуя тем самым костимуляторный ответ клетки, такой как (но не ограничиваясь только им) пролиферация. Костимуляторные молекулы включают (но не ограничиваясь только ими) молекулу ГКГС класса I, BTLA и лиганд Толл-рецептора. Примерами костимуляторных молекул являются CD27, CD28, CD8, 4-1ВВ (CD137), OX40, CD30, CD40, PD-1, ICOS, антиген-1, ассоциированный с функцией лимфоцитов (LFA-1), CD2, CD7, LIGHT, NKG2C, В7-Н3 и лиганд, специфически связывающийся с CD83, и т.п.
В предпочтительном варианте осуществления изобретения костимуляторный домен анти-CLL1 CAR, предлагаемого в настоящем изобретении, содержит часть костимуляторной сигнальной молекулы, выбранной из группы, состоящей из фрагмента 4-1ВВ (GenBank: AAA53133.) и CD28 (NP_006130.1). В частности, домен трансдукции сигнала анти-CLL1 CAR, предлагаемого в настоящем изобретении, содержит аминокислотную последовательность, идентичную по меньшей мере на 70%, предпочтительно по меньшей мере на 80%, более предпочтительно по меньшей мере на 90%, 95%, 97% или 99% аминокислотной последовательности, выбранной из группы, включающей SEQ ID NO: 8.
Анти-CLL1 CAR, предлагаемый в настоящем изобретении, содержит также трансмембранный домен (ТМ). Отличительными особенностями соответствующих трансмембранных доменов являются способность экспрессироваться на поверхности клетки, согласно настоящему изобретению предпочтительно на поверхности иммунной клетки, прежде всего лимфоцитов или естественных клеток-киллеров (NK), и взаимодействовать друг с другом, направляя клеточный ответ иммунной клетки на заранее определенную клетку-мишень. Трансмембранный домен можно получать как из встречающегося в естественных условиях, так и из синтетического источника. Трансмембранный домен можно получать из любого связанного с мембраной или трансмембранного белка. Примерами трансмембранных полипептидов могут служить (но не ограничиваясь только ими) субъединица Т-клеточного рецептора, такая как α-, β-, γ- или ζ-субъединица, полипептид, образующий комплекс с CD3, р55 (α-цепь), р75 (β-цепь) или γ-цепь рецептора IL2, цепь субъединицы Fc-рецепторов, в частности, Fcγ-рецептора III, или CD-белки. Альтернативно этому, трансмембранный домен может быть синтетическим, и он может содержать преимущественно гидрофобные остатки, такие как лейцин и валин. В предпочтительном варианте осуществления изобретения указанный трансмембранный домен получают из альфа-цепи человеческого CD8 (например, NP_001139345.1). Трансмембранный домен может содержать также шарнирную область между указанным внеклеточным лигандсвязывающим доменом и указанным трансмембранным доменом.
В контексте настоящего описания понятие «шарнирная область, шарнир», как правило, обозначает любой олиго- или полипептид, функция которого заключается в сцеплении трансмембранного домена с внеклеточным лигандсвязывающим доменом. В частности, шарнирную область используют для обеспечения большей гибкости и доступности внеклеточного лигандсвязывающего домена. Шарнирная область может содержать вплоть до 300 аминокислот, предпочтительно от 10 до 100 аминокислот, и наиболее предпочтительно от 25 до 50 аминокислот.Шарнирную область можно получать из полных встречающихся в естественных условиях молекул, или их частей, таких как полная внеклеточная область CD8, CD4 или CD28, или ее часть, или полная константная область антитела или ее часть. Альтернативно этому, шарнирная область может представлять собой синтетическую последовательность, которая соответствует встречающейся в естественных условиях шарнирной последовательности, или может представлять собой полностью синтетическую шарнирную последовательность. В предпочтительном варианте осуществления изобретения указанная шарнирная область представляет собой часть альфа-цепи человеческого CD8, FcγRIIIα-рецептора или IgG1 соответственно, которые обозначены в настоящем описании как SEQ ID NO: 3, SEQ ID NO: 4 и SEQ ID NO: 5, или шарнирные полипептиды, последовательности которых идентичны предпочтительно по меньшей мере на 80%, более предпочтительно по меньшей мере на 90%, 95%, 97% или 99% последовательностям указанных полипептидов. Согласно одному из вариантов осуществления изобретения шарнир может представлять собой также шарнир человеческого Ig (иммуноглобулин), например, шарнир PD-1, шарнир IgG4.
Согласно предпочтительному варианту осуществления изобретения анти-CLL1 CAR, предлагаемый в изобретении, содержит трансмембранный домен, более предпочтительно выбранный из CD8α и 4-1ВВ, который идентичен полипептидам, имеющим SEQ ID NO: 6 или 7.
Анти-CLL1 CAR, предлагаемый в изобретении, как правило, дополнительно содержит трансмембранный домен (ТМ), более предпочтительно выбранный из CD8α и 4-1ВВ, и еще более предпочтительно идентичный полипептидам, имеющим SEQ ID NO: 6 или 7.
В предпочтительном варианте осуществления изобретения анти-CLL1 CAR, предлагаемый в изобретении, дополнительно содержит ТМ-домен из CD8α, имеющий SEQ ID NO: 6, или идентичный по меньшей мере на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% последовательности SEQ ID NO: 6.
В раковых клетках часто имеет место понижающая регуляция или мутация антигенов-мишеней, что приводит к появлению так называемых вариантов «потерянных антигенов» (антигенов, ускользающих от иммунологического надзора). Таким образом, для того, чтобы противостоять ускользанию опухоли от иммунологического надзора и придавать иммунной клетке большую специфичность в отношении мишени, CLL1-специфический анти-CLL1 CAR, предлагаемый в изобретении, может содержать другие внеклеточные лигандсвязывающие домены, предназначенные для одновременного связывания с другими элементами на мишени и усиления тем самым активации и функции иммунной клетки. В одном из вариантов осуществления изобретения внеклеточные лигандсвязывающие домены можно размещать тандемно на одном и том же трансмембранном полипептиде и их необязательно можно отделять друг от друга с помощью линкера. В другом варианте осуществления изобретения указанные другие внеклеточные лигандсвязывающие домены можно размещать на различных трансмембранных полипептидах, из которых состоит анти-CLL1 CAR. Другим вариантом осуществления настоящего изобретения является популяция анти-CLL1 CAR, каждый из которых содержит различные внеклеточные лигандсвязывающие домены. В частности, настоящее изобретение относится к способу создания иммунных клеток, включающему получение иммунной клетки и экспрессию на поверхности указанной клетки популяции анти-CLL1 CAR, каждый из которых содержит различные внеклеточные лигандсвязывающие домены. Другой конкретный вариант осуществления настоящего изобретения относится к способу создания иммунной клетки, включающему получение иммунной клетки и интродукцию в указанную клетку полинуклеотидов, кодирующих полипептиды, из которых состоит популяция анти-CLL1 CAR, каждый из которых содержит различные внеклеточные лигандсвязывающие домены. Под популяцией анти-CLL1 CAR подразумевают по меньшей мере два, три, четыре, пять, шесть или большее количество анти-CLL1 CAR, каждый из которых содержит различные внеклеточные лигандсвязывающие домены. Согласно настоящему изобретению различные внеклеточные лигандсвязывающие домены, предлагаемые в настоящем изобретении, предпочтительно могут связываться одновременно с различными элементами на мишени, усиливая тем самым активацию и функцию иммунной клетки. Настоящее изобретение относится также к выделенной иммунной клетке, которая содержит популяцию анти-CLL1 CAR, каждый из которых содержит различные внеклеточные лигандсвязывающие домены.
CLL1-специфические химерные антигенные рецепторы, предлагаемые в изобретении, могут иметь различные архитектуры, в которых они могут экспрессироваться, например, в форме одноцепочечного химерного белка (scCAR) или в форме нескольких полипептидов (несколько цепей), включающих по меньшей мере один указанный химерный белок. Такие включающие несколько цепей архитектуры CAR описаны в WO 2014/039523, включенной в настоящее описание в качестве ссылки, прежде всего на фиг. 2-4 и на страницах 14-21.
В целом, анти-CLL1 CAR содержит внеклеточное одноцепочечное антитело (scFv Fc), слитое с внутриклеточным сигнальным доменом дзета-цепи из комплекса Т-клеточного рецептора антигена (scFv Fc:ζ), который обладает способностью при экспрессии в Т-клетках перенаправлять распознавание антигена на основе специфичности моноклонального антитела.
В настоящем описании представлено несколько анти-CLL1 одноцепочечных CAR, направленных против антигена CLL1, которые, например, содержат аминокислотные последовательности SEQ ID NO: 35-112 (но не ограничиваясь только ими).
CLL1 CAR, предлагаемые в настоящем изобретении, могут представлять собой также ранее упомянутые «многоцепочечные CAR», это означает, что внеклеточный связывающий домен и сигнальные домены предпочтительно локализованы на различных полипептидных цепях, в то время как костимуляторные домены могут быть локализованы на этом же или на третьем полипептиде. Указанные многоцепочечные CAR можно получать из FcεRI (Ravetch и др., 1989) путем замены высокоаффинного IgE-связывающего домена альфа-цепи FcεRI на внеклеточный лигандсвязывающий домен, такой как scFv, в то время как N- и/или С-концевые хвосты бета и/или гамма цепей FcεRI сливают с доменами трансдукции сигнала и костимуляторными доменами соответственно. Внеклеточный лигандсвязывающий домен играет роль специфичности, перенаправляющей Т-клетку к клеткам-мишеням, а домен трансдукции сигнала активирует или снижает ответ иммунных клеток. Тот факт, что различные полипептиды, имеющие происхождение из альфа-, бета- и гамма-полипептидов FcεRI, представляют собой трансмембранные полипептиды, находящиеся в положении вблизи мембраны, делает архитектуру CAR более гибкой, повышая специфичность в отношении целевой молекулы и снижая фоновую активацию иммунных клеток, что описано в WO 2014/039523.
Внеклеточный лигандсвязывающий домен
Понятие «внеклеточный лигандсвязывающий домен» обозначает олиго- или полипептид, который обладает способностью связываться с лигандом. Предпочтительно домен должен обладать способностью взаимодействовать с молекулой клеточной поверхности. Например, можно выбирать внеклеточный лигандсвязывающий домен для распознавания лиганда, который функционирует в качестве маркера клеточной поверхности на клетках-мишенях, ассоциированных с конкретным болезненным состоянием. Они могут представлять собой, например, связывающие домены, полученные из лиганда, рецептора, человеческих или мышиных антител, или антигенраспознающие домены, полученные из верблюдов или хрящевых рыб.
В предпочтительном варианте осуществления изобретения указанный внеклеточный лигандсвязывающий домен содержит одноцепочечный фрагмент антитела (scFv), содержащий вариабельный фрагмент легкой (VL) и тяжелой (VH) цепи специфического в отношении антигена-мишени моноклонального антитела к CLL1, сцепленный с помощью гибкого линкера. Указанные VL и VH предпочтительно выбирают из антител, известных из литературы, таких как SCO02-357, SC02-378 и SC02-161, описанные в WO 2005/00894 (заявитель: фирма Crucell Holland BV); М26, М31, G4, М22, М29, М2, М5, G12, описанные в WO 2013/169625 (заявитель: фирма Cellerant Therapeutics); и 21.26, 1075.7, описанные в WO 2009/051974 (заявитель: фирма Nuvelo Inc.), и более конкретно содержащие CDR VH- и VL-доменов моноклональных антител к CLL1, выбранные из представленных ниже SEQ ID NO: 119-190.
Последовательности CDR VH-цепи моноклонального антитела к CLL1 можно выбирать из GSISSSNWWS (SEQ ID NO: 119), WIGEIYHSGSPDY (SEQ ID NO: 120), KVSTGGFFDY (SEQ ID NO: 121) и GSISSSNWWS (SEQ ID NO: 122), WIGEIYHSGSPNY (SEQ ID NO: 123), RSSSGGFFDY (SEQ ID NO: 124), и GSISSSNWWS (SEQ ID NO: 125), WIGEIYHSGSPNY (SEQ ID NO: 126), RQTTAGSFDY (SEQ ID NO: 127), и GYTFTSYFIH (SEQ ID NO: 131), WIGFINPYNDGSKY (SEQ ID NO: 132), TRDDGYYGYAMDY (SEQ ID NO: 133), и GYTFTSYVMH (SEQ ID NO: 137), WIGYINPYNDGTKY (SEQ ID NO: 138), ARPIYFDNDY (SEQ ID NO: 139), и QQNNYDPW (SEQ ID NO: 143), WIGPINPYNDGTI (SEQ ID NO: 144), ARTDDYDDYTMDY (SEQ ID NO: 145), и GYTFTRYWMH (SEQ ID NO: 149), WIGNIDPSDTETHY (SEQ ID NO: 150), AIYYGNPSYYAMDY (SEQ ID NO: 151), и GYIFTSYVMY (SEQ ID NO: 155), WIGYINPY (SEQ ID NO: 156), ARYYDYDYYFDY (SEQ ID NO: 157), и GYTFTSYFMH (SEQ ID NO: 161), WIGFINPYNDGTKY (SEQ ID NO: 162), TRDDGYYDYAMDY (SEQ ID NO: 163), и GFNIKDDYIH (SEQ ID NO: 167), WIGWIDPEKGDTAYA (SEQ ID NO: 168), TLTGRFDY (SEQ ID NO: 169), и GYTFPSSNIH (SEQ ID NO: 173), WIGVIYPGNGDTSY (SEQ ID NO: 174), AIYFVYNWHFDV (SEQ ID NO: 175) и GYTFTRYWMH (SEQ ID NO: 179), MIHPSSGSTSYNEKVK (SEQ ID NO: 180), RDGDYYYGTGDY (SEQ ID NO: 181), и GYSITSAYYWN (SEQ ID NO: 185), YISYDGRNNYNPSLKN (SEQ ID NO: 186) и AKEGDYDVGNYYAMDY (SEQ ID NO: 187).
Аналогично этому, последовательности CDR VL-цепи моноклонального антитела к CLL1 можно выбирать из: QSISSYLN (SEQ ID NO: 128), LLIYAASSLQS (SEQ ID NO: 129), QQSYSTPP (SEQ ID NO: 130) и QELSGYLS (SEQ ID NO: 134), RLIYAASTLDS (SEQ ID NO: 135), LQYAIYPY (SEQ ID NO: 136), и ESVDSYGNSFMH (SEQ ID NO: 140), LLIYLASNLES (SEQ ID NO: 141), QQNNYDPW (SEQ ID NO: 142), HDISNYLN (SEQ ID NO: 146), LLIYYTSRLHS (SEQ ID NO: 147), QQGKTLLW (SEQ ID NO: 148) и QNLLNSGNQKKYLN (SEQ ID NO: 152), LLIYWASTRES (SEQ ID NO: 153), QNDYSYPF (SEQ ID NO: 154), и QDINKYIA (SEQ ID NO: 158), LLIHYTSTLQP (SEQ ID NO: 159), LQYDYLW (SEQ ID NO: 160), и QEISVYLS (SEQ ID NO: 164), RLIYAASTLDS (SEQ ID NO: 165), LQYASYPY (SEQ ID NO: 166), и QSLLYSSNQKNNLA (SEQ ID NO: 170), LLIYWASTRES (SEQ ID NO: 171), QQYYSYR (SEQ ID NO: 172), и ESVDGYGDIFML (SEQ ID NO: 176), LLIYFASNLES (SEQ ID NO: 177), QQNNEDPY (SEQ ID NO: 178), и RASSSINYMH (SEQ ID NO: 182), PWIFATSNLAS (SEQ ID NO: 183), QQWRSDRALT (SEQ ID NO: 184), и RASSNVISSYVH (SEQ ID NO: 188), LWIYSTSNLAS (SEQ ID NO: 189), и QQYSGYPLT (SEQ ID NO: 190).
Внеклеточный домен и трансмембранный домен предпочтительно связаны друг с другом с помощью гибкого линкера, содержащего SEQ ID NO: 10. Другими словами, указанные анти-CLL1 CAR предпочтительно содержат внеклеточный лигандсвязывающий домен, который содержит полипептидную последовательность, идентичную по меньшей мере на 90%, 95% 97% или 99% аминокислотной последовательности, которая выбрана из группы, состоящей из SEQ ID NO: 11-SEQ ID NO: 34 (см. таблицу 2).
В контексте настоящего описания понятие «рекомбинантное антитело» означает антитело или фрагмент антитела, созданное/созданный с помощью технологии рекомбинантной ДНК, такое, например, как антитело или фрагмент антитела, экспрессируемое/экспрессируемый бактериофагом, системой экспрессии дрожжей или системой экспрессии клеток млекопитающих. Подразумевается также, что понятие относится к антителу или фрагменту антитела, созданному с помощью синтеза молекулы ДНК, которая кодирует антитело или фрагмент антитела, и эта молекула ДНК экспрессирует белок антитела или фрагмента антитела или аминокислотную последовательность, специфическую для антитела или фрагмента антитела, при этом ДНК или аминокислотную последовательность получали с использованием технологии создания рекомбинантной или синтетической ДНК или аминокислотной последовательности, которая доступна и хорошо известна в данной области.
В настоящем изобретении предложен CLL1-специфический химерный антигенный рецептор (анти-CLL1 scCAR), описанный выше, в котором указанный внеклеточный лигандсвязывающий домен содержит VH- и VL-цепи, которые являются гуманизированными.
В контексте настоящего описания понятие «гуманизированное антитело» означает, что полипептиды включают гуманизированную вариабельную область тяжелой цепи и гуманизированную вариабельную область легкой цепи. Например, полипептиды могут включать каркасные (FR) участки вариабельных областей тяжелой и легкой цепи человеческого антитела, практически сохраняя при этом антигенсвязывающую специфичность родительского моноклонального антитела. Гуманизированная вариабельная область тяжелой цепи и/или гуманизированная вариабельная область легкой цепи являются гуманизированными по меньшей мере примерно на 87%, гуманизированными по меньшей мере примерно на 90%, гуманизированными по меньшей мере примерно на 95%, гуманизированными по меньшей мере примерно на 98% или гуманизированными по меньшей мере примерно на 100% за исключением гипервариабельных участков (CDR). Молекулы антигенсвязывающих полипептидов могут происходить из донорских моноклональных антител (например, мышиных донорских моноклональных антител) и могут включать CDR из моноклональных антител (например, CDR из мышиных моноклональных антител).
В контексте настоящего описания понятие «моноклональное антитело» означает, что антитело получено с использованием выращенного в лаборатории клона клетки, либо гибридомы, либо трансформированного вирусом лимфоцита, такие антитела являются более многочисленными и однородными, чем встречающиеся в естественных условиях антитела, и обладают способностью специфически связываться с одним сайтом на антигене CLL1. Они представляют собой моноспецифические антитела, создающиеся идентичными иммунными клетками, которые все представляют собой клоны одной родительской клетки, в отличие от поликлональных антител, которые получают из нескольких различных иммунных клеток. Моноклональные антитела обладают одновалентной аффинностью, это означает, что они связываются с одним и тем же эпитопом. Современные методики, применяемые для гуманизации, описаны у Lefranc M.P. и др. (Lefranc, M.P., Ehrenmann F., Ginestoux С., Giudicelli V., Duroux P., «Use of IMGT (®) databases and tools for antibody engineering and humanization». Methods Mol Biol. 907, 2012, cc. 3-37). В этой публикации представлены данные о четырех выровненных последовательностях.
Гуманизированное антитело можно получать с помощью различных методов, известных в данной области, включая (но не ограничиваясь только ими) трансплантацию CDR (см., например, европейский патент ЕР 239400; международную публикацию WO 91/09967; и US №№5225539, 5530101 и 5585089, каждый из указанных документов полностью включен в настоящее описание в качестве ссылки), «облицовку» или «нанесение покрытия» (см, например, европейский патент ЕР 592106 и ЕР 519596; Padlan, Molecular Immunology, 28(4/5), 1991, cc. 489-498; Studnicka и др., Protein Engineering, 7(6), 1994, cc. 805-814; и Roguska и др., PNAS, 91, 1994, cc. 969-973, каждый из указанных документов полностью включен в настоящее описание в качестве ссылки), перестановку цепи (см., например, US №5565332, который полностью включен в настоящее описание в качестве ссылки) и методы, описанные, например, в публикации заявки на патент США US №2005/0042664, публикации заявки на патент США US 2005/0048617, патентах US №6407213, US №5766886, публикации международной заявки на патент WO 9317105, Tan и др., J. Immunol., 169, 2002, cc. 1119-1125, Caldas и др., Protein Eng., 13(5), 2000, cc. 353-360, Morea и др., Methods, 20(3), 2000, cc. 267-279, Васа и др., J. Biol. Chem., 272(16), 1997, cc. 10678-10684, Roguska и др., Protein Eng., 9(10), 1996, cc. 895-904, Couto и др., Cancer Res., 55 (приложение 23), 1995, cc. 5973-5977, Couto и др., Cancer Res., 55(8), 1995, cc. 1717-1722, Sandhu J.S., Gene, 150(2), 1994, cc. 409-410 и Pedersen и др., J. Mol. Biol., 235(3), 1994, cc. 959-973, каждый из указанных документов полностью включен в настоящее описание в качестве ссылки. Часто каркасные остатки в каркасных областях требуется заменять на соответствующие остатки из CDR донорского антитела для изменения, например, для улучшения, связывания с антигеном. Такие замены в каркасном участке идентифицируют с помощью методов, хорошо известных в данной области, например, путем моделирования взаимодействий остатков CDR и каркасного участка с целью идентификации остатков каркасного участка, важных для связывания с антигеном, и сравнения последовательностей для идентификации необычных остатков каркасного участка в определенных положениях (см., например. Queen и др., US №5585089; и Riechmann и др., Nature, 332, 1988, с. 323, указанные документы включены в настоящее описание в качестве ссылки).
Консервативные аминокислотные замены представляют собой замены, при которых аминокислотный остаток заменяют на аминокислотный остаток, который имеет сходную боковую цепь. Семейства аминокислотных остатков, имеющих сходные боковые цепи, известны в данной области. Эти семейства включают аминокислоты с 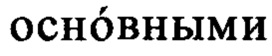 боковыми цепями (например, лизин, аргинин, гистидин), кислотными боковыми цепями (например, аспарагиновая кислота, глутаминовая кислота), незаряженными полярными боковыми цепями (например, глицин, аспарагин, глутамин, серии, треонин, тирозин, цистеин, триптофан), неполярными боковыми цепями (например, аланин, валин, лейцин, изолейцин, пролин, фениланин, метионин), бета-разветвленными боковыми цепями (например, треонин, валин, изолейцин) и ароматическими боковыми цепями (например, тирозин, фенилаланин, триптофан, гистидин). Таким образом, один или несколько аминокислотных остатков в анти-CLL1 CAR, предлагаемом в изобретении, можно заменять на другие аминокислотные остатки из того же семейства боковых цепей и измененный анти-CLL1 CAR можно тестировать в отношении способности связывать CLL1, используя функциональные анализы, представленные в настоящем описании.
боковыми цепями (например, лизин, аргинин, гистидин), кислотными боковыми цепями (например, аспарагиновая кислота, глутаминовая кислота), незаряженными полярными боковыми цепями (например, глицин, аспарагин, глутамин, серии, треонин, тирозин, цистеин, триптофан), неполярными боковыми цепями (например, аланин, валин, лейцин, изолейцин, пролин, фениланин, метионин), бета-разветвленными боковыми цепями (например, треонин, валин, изолейцин) и ароматическими боковыми цепями (например, тирозин, фенилаланин, триптофан, гистидин). Таким образом, один или несколько аминокислотных остатков в анти-CLL1 CAR, предлагаемом в изобретении, можно заменять на другие аминокислотные остатки из того же семейства боковых цепей и измененный анти-CLL1 CAR можно тестировать в отношении способности связывать CLL1, используя функциональные анализы, представленные в настоящем описании.
Анти-CLL1 CAR, предлагаемый в изобретении, необязательно включает домен суицида, предназначенный для истощения иммунных клеток, несущих CAR, в случае, который позднее может вызывать нежелательные действия in vivo. Указанный домен суицида можно получать, например, путем включения двух копий мимеотопа CD20, предпочтительно последовательность CPYSNPSLCS (SEQ ID NO: 113), в полипептидную последовательность CAR. Указанные две копии мимеотопа CD20 можно связывать друг с другом, а также с VL с помощью линкера. Их можно встраивать между анти-CLL1 scFv и шарниром (таким как CD8альфа) с помощью необязательного линкера. Мимеотопы CD20 можно связывать с помощью антител к CD20, таких как ритуксимаб (McLaughlin P. и др., 1998). Таким образом, анти-CLL1 CAR, предлагаемый в настоящем изобретении, может содержать VH- и VL-цепи, которые обладают способностью связываться с антигеном клеточной поверхности CLL1, необязательно гуманизированные, линкер L, домен суицида, шарнирную область или ее часть, трансмембранный домен, костимуляторный домен и стимуляторный домен.
В предпочтительном варианте осуществления настоящего изобретения описан анти-CLL1-специфический одноцепочечный химерный антигенный рецептор («анти-CLL1 scCAR» или «scCAR»), который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2 и в таблицах 3-8, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир, трансмембранный домен, цитоплазматический домен, включающий сигнальный домен и костимуляторный домен.
Более предпочтительным вариантом осуществления настоящего изобретения является описанный выше CLL1-специфический scCAR, в котором указанная структура V1, V3 или V5 содержит шарнир FcγRIIIα, СD8альфа или IgG1 и трансмембранный домен СD8альфа.
В другом более предпочтительном варианте осуществления изобретения указанный CLL1-специфический scCAR содержит костимуляторный домен из 4-1ВВ или CD28 или более предпочтительно костимуляторный домен из 4-1ВВ.
В настоящем изобретении предложен CLL1-специфический scCAR, описанный выше, в котором указанная структура V1, V3 или V5 содержит шарнир FcγRIIIα, CD8альфа или IgG1 и трансмембранный домен 4-1ВВ.
В настоящем изобретении предложен CLL1-специфический scCAR, описанный выше, в котором указанная структура V1, V3 или V5 содержит шарнир FcγRIIIα, СD8альфа или IgG1, цитоплазматический домен 4-1ВВ и трансмембранный домен СD8альфа.
Согласно предпочтительному варианту осуществления изобретения анти-CLL1 scCAR, предлагаемый в изобретении, имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир, трансмембранный домен, цитоплазматический домен, включающий сигнальный домен СОЗдзета и костимуляторный домен, где указанный сигнальный домен СОЗдзета предпочтительно имеет последовательность SEQ ID NO: 9.
Согласно другому предпочтительному варианту осуществления изобретения анти-CLL1 scCAR, предлагаемый в изобретении, имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир, трансмембранный домен, цитоплазматический домен, включающий сигнальный домен CD3дзета и костимуляторный домен из 4-1ВВ, где указанный костимуляторный домен из 4-1ВВ предпочтительно имеет последовательность SEQ ID NO: 8.
В настоящем изобретении описан анти-CLL1 scCAR, который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир FcγRIIIα, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзета и костимуляторный домен.
В настоящем изобретении описан анти-CLL1 scCAR, который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир CD8α, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта и костимуляторный домен.
В настоящем изобретении описан анти-CLL1 scCAR, который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир IgG1, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта и костимуляторный домен.
В настоящем изобретении описан анти-CLL1 scCAR, который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир FcγRIIIα, трансмембранный домен 4-1ВВ, предпочтительно имеющий SEQ ID NO: 7, цитоплазматический домен, включающий сигнальный домен CD3дзета и костимуляторный домен.
В настоящем изобретении описан анти-CLL1 scCAR, который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир CD8α, трансмембранный домен 4-1ВВ, предпочтительно имеющий SEQ ID NO: 7, цитоплазматический домен, включающий сигнальный домен CD3дзeта и костимуляторный домен.
В настоящем изобретении описан анти-CLL1 scCAR, который имеет одну из полипептидных структур, выбранную из V1-V6, которые проиллюстрированы на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир IgG1, трансмембранный домен 4-1ВВ, предпочтительно имеющий SEQ ID NO: 7, цитоплазматический домен, включающий сигнальный домен CD3дзeтa и костимуляторный домен.
Конкретным объектом настоящего изобретения является анти-CLL1-специфический scCAR, который имеет полипептидную структуру VI, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир FcγRIIIα, предпочтительно имеющий SEQ ID NO: 3, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8.
Более конкретно в настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V1, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир FcγRIIIα, предпочтительно имеющий SEQ ID NO: 3, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8, где указанная VH-цепь идентична по меньшей мере на 80% SEQ ID NO: 11-13, 15, 17, 19, 21, 23, 25, 27, 29, 31 или 33, и указанная VL идентична по меньшей мере на 80% SEQ ID NO: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32 или 34.
Другим конкретным объектом настоящего изобретения является анти-CLL1-специфический scCAR, который имеет полипептидную структуру V3, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир CD8α, предпочтительно имеющий SEQ ID NO: 4, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзета, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8.
Более конкретно в настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V3, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир CD8α, предпочтительно имеющий SEQ ID NO: 4, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1 ВВ, предпочтительно имеющий SEQ ID NO: 8, где указанная VH-цепь идентична по меньшей мере на 80% SEQ ID NO: 11-13, 15, 17, 19, 21, 23, 25, 27, 29, 31 или 33, и указанная VL-цепь идентична по меньшей мере на 80% SEQ ID NO: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32 или 34.
Другим конкретным объектом настоящего изобретения является анти-CLL1-специфический scCAR, который имеет полипептидную структуру V5, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир IgG1, предпочтительно имеющий SEQ ID NO: 5, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзета, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8.
Более конкретно в настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V5, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир IgG1, предпочтительно имеющий SEQ ID NO: 5, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8, где указанная VH-цепь идентична по меньшей мере на 80% SEQ ID NO: 11-13, 15, 17, 19, 21, 23, 25, 27, 29, 31 или 33, и указанная VL-цепь идентична по меньшей мере на 80% SEQ ID NO: 14, 16, 18, 20, 22, 24 ,26, 28, 30, 32 или 34.
В настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V1, проиллюстрированную на фиг. 2, где указанный полипептид идентичен по меньшей мере на 80% SEQ ID NO: 35, 41, 47, 53, 59, 65, 71, 77, 83, 89, 95, 101 или 107.
В частности, указанный анти-CLL1-специфический scCAR имеет полипептидную структуру V1, проиллюстрированную на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир FcγRIIIα, предпочтительно имеющий SEQ ID NO: 3, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзета, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный из домен 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8, где указанная VH-цепь идентична по меньшей мере на 80% SEQ ID NO: 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31 или 33, и указанная VL идентична по меньшей мере на 80% SEQ ID NO: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32 или 34, и где указанный полипептид идентичен по меньшей мере на 80% SEQ ID NO: 35, 41, 47, 53, 59, 65, 71, 77, 83, 89, 95, 101 или 107.
В настоящем изобретении описан анти-CLL1 -специфический scCAR, который имеет полипептидную структуру V3, проиллюстрированную на фиг. 2, где указанный полипептид идентичен по меньшей мере на 80% SEQ ID NO: 37, 43, 49, 55, 61, 67, 73, 79, 85, 91, 97, 103 или 109.
Более конкретно в настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V3, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир CD8α, предпочтительно имеющий SEQ ID NO: 4, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8, где указанная VH-цепь идентична по меньшей мере на 80% SEQ ID NO: 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31 или 33, и указанная VL-цепь идентична по меньшей мере на 80% SEQ ID NO: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32 или 34, и где указанный полипептид идентичен по меньшей мере на 80% SEQ ID NO: 37, 43, 49, 55, 61, 67, 73, 79, 85, 91, 97, 103 или 109.
В настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V5, проиллюстрированную на фиг. 2, где указанный полипептид идентичен по меньшей мере на 80% SEQ ID NO: 39, 45, 51, 57, 63, 69, 75, 81, 87, 93, 99, 105 или 111.
Более конкретно в настоящем изобретении описан анти-CLL1-специфический scCAR, который имеет полипептидную структуру V5, которая проиллюстрирована на фиг. 2, где указанная структура содержит внеклеточный лигансвязывающий домен, содержащий VH и VL из моноклонального антитела к CLL1, шарнир IgG1, предпочтительно имеющий SEQ ID NO: 5, трансмембранный домен CD8α, предпочтительно имеющий SEQ ID NO: 6, цитоплазматический домен, включающий сигнальный домен CD3дзeта, предпочтительно имеющий SEQ ID NO: 9, и костимуляторный домен из 4-1ВВ, предпочтительно имеющий SEQ ID NO: 8, где указанная VH-цепь идентична по меньшей мере на 80% SEQ ID NO: 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31 или 33, и указанная VL-цепь идентична по меньшей мере на 80% SEQ ID NO: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32 или 34, и где указанный полипептид идентичен по меньшей мере на 80% SEQ ID NO: 39, 45, 51, 57, 63, 69, 75, 81, 87, 93, 99, 105 или 111.
Более конкретно в настоящем изобретении описан CLL1-специфический одноцепочечный химерный антигенный рецептор (scCAR), который имеет полипептидную структуру V1, V3 или V5, проиллюстрированную на 2 и описанную выше, где указанная структура содержит VH внеклеточного лигандсвязывающего домена из моноклонального антитела к CLL1, содержащую следующие последовательности CDR: GSISSSNWWS (SEQ ID NO: 119), WIGEIYHSGSPDY (SEQ ID NO: 120), KVSTGGFFDY (SEQ ID NO: 121) и GSISSSNWWS (SEQ ID NO: 122), WIGEIYHSGSPNY (SEQ ID NO: 123), RSSSGGFFDY (SEQ ID NO: 124), и GSISSSNWWS (SEQ ID NO: 125), WIGEIYHSGSPNY (SEQ ID NO: 126), RQTTAGSFDY (SEQ ID NO: 127), и GYTFTSYFIH (SEQ ID NO: 131), WIGFINPYNDGSKY (SEQ ID NO: 132), TRDDGYYGYAMDY (SEQ ID NO: 133), и GYTFTSYVMH (SEQ ID NO: 137), WIGYINPYNDGTKY (SEQ ID NO: 138), ARPIYFDNDY (SEQ ID NO: 139), и QQNNYDPW (SEQ ID NO: 143), WIGPINPYNDGTI (SEQ ID NO: 144), ARTDDYDDYTMDY (SEQ ID NO: 145), и GYTFTRYWMH (SEQ ID NO: 149), WIGNIDPSDTETHY (SEQ ID NO: 150), AIYYGNPSYYAMDY (SEQ ID NO: 151), и GYIFTSYVMY (SEQ ID NO: 155), WIGYINPY (SEQ ID NO: 156), ARYYDYDYYFDY (SEQ ID NO: 157), и GYTFTSYFMH (SEQ ID NO: 161), WIGFINPYNDGTKY (SEQ ID NO: 162),TRDDGYYDYAMDY (SEQ ID NO: 163), и GFNIKDDYIH (SEQ ID NO: 167), WIGWIDPEKGDTAYA (SEQ ID NO: 168), TLTGRFDY (SEQ ID NO: 169), и GYTFPSSNIH (SEQ ID NO: 173), WIGVIYPGNGDTSY (SEQ ID NO: 174), AIYFVYNWHFDV (SEQ ID NO: 175), and GYTFTRYWMH (SEQ ID NO: 179), MIHPSSGSTSYNEKVK (SEQ ID NO: 180), RDGDYYYGTGDY (SEQ ID NO: 181), и GYSITSAYYWN (SEQ ID NO: 185), YISYDGRNNYNPSLKN (SEQ ID NO: 186), AKEGDYDVGNYYAMDY (SEQ ID NO: 187), и предпочтительно
VL внеклеточного лигандсвязывающего домена из моноклонального антитела к CLL1, содержащую следующие последовательности CDR: QSISSYLN (SEQ ID NO: 128), LLIYAASSLQS (SEQ ID NO: 129), QQSYSTPP (SEQ ID NO: 130) и QELSGYLS (SEQ ID NO: 134), RLIYAASTLDS (SEQ ID NO: 135), LQYAIYPY (SEQ ID NO: 136), и ESVDSYGNSFMH (SEQ ID NO: 140), LLIYLASNLES (SEQ ID NO: 141), QQNNYDPW (SEQ ID NO: 142), HDISNYLN (SEQ ID NO: 146), LLIYYTSRLHS (SEQ ID NO: 147), QQGKTLLW (SEQ ID NO: 148), и QNLLNSGNQKKYLN (SEQ ID NO: 152), LLIYWASTRES (SEQ ID NO: 153), QNDYSYPF (SEQ ID NO: 154), и QDINKYIA (SEQ ID NO: 158), LLIHYTSTLQP (SEQ ID NO: 159), LQYDYLW (SEQ ID NO: 160), и QEISVYLS (SEQ ID NO: 164), RLIYAASTLDS (SEQ ID NO: 165), LQYASYPY (SEQ ID NO: 166), и QSLLYSSNQKNNLA (SEQ ID NO: 170), LLIYWASTRES (SEQ ID NO: 171), QQYYSYR (SEQ ID NO: 172), и ESVDGYGDIFML (SEQ ID NO: 176), LLIYFASNLES (SEQ ID NO: 177), QQNNEDPY (SEQ ID NO: 178), и RASSSINYMH (SEQ ID NO: 182), PWIFATSNLAS (SEQ ID NO: 183), QQWRSDRALT (SEQ ID NO: 184), и RASSNVISSYVH (SEQ ID NO: 188), LWIYSTSNLAS (SEQ ID NO: 189), QQYSGYPLT (SEQ ID NO: 190),
и где указанная структура, как правило, содержит:
шарнир, трансмембранный домен и цитоплазматический домен, включающий сигнальный домен CD3дзета и костимуляторный домен из 4-1ВВ.
В настоящем изобретении описан CLL1-специфический одноцепочечный химерный антигенный рецептор (анти-CLL1 scCAR), указанный выше, в котором VH и VL указанного внеклеточного лигандсвязывающего домена гуманизированы.
В настоящем изобретении описан CLL1-специфический одноцепочечный химерный антигенный рецептор (scCAR), указанный выше, в котором VH указанного внеклеточного лигандсвязывающего домена из антитела к CLL1 содержит по меньшей мере одну из следующих последовательностей:
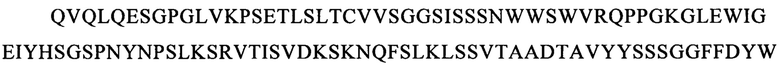
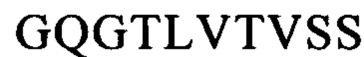 (соответствует SC02-357),
(соответствует SC02-357),
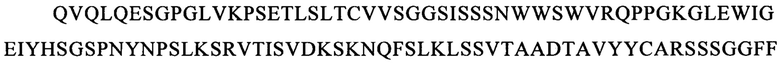
 (соответствует SC02-378),
(соответствует SC02-378),
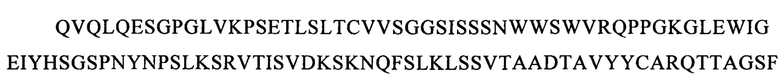
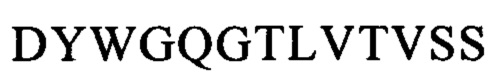 (соответствует SC02-161),
(соответствует SC02-161),

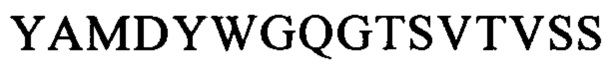 (соответствует М26),
(соответствует М26),
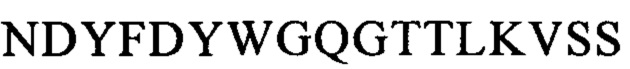 (соответствует М31),
(соответствует М31),
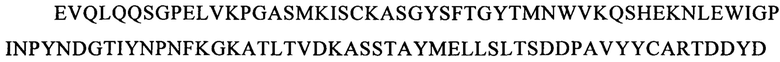
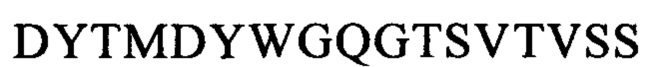 (соответствует G4),
(соответствует G4),

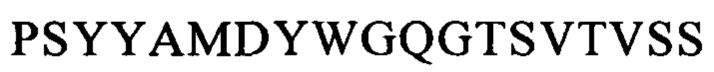 (соответствует М22),
(соответствует М22),
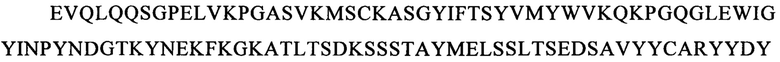
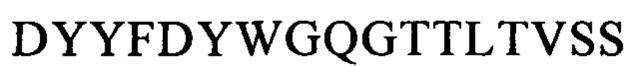 (соответствует М29),
(соответствует М29),
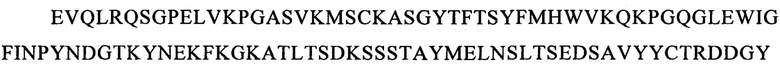
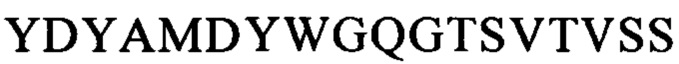 (соответствует М2),
(соответствует М2),

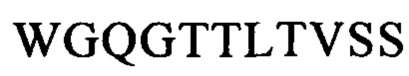 (соответствует М5),
(соответствует М5),
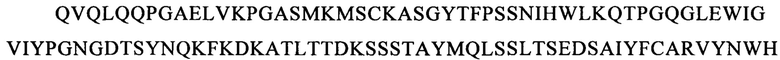
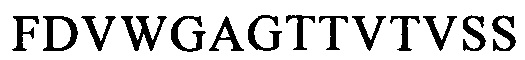 (соответствует G12),
(соответствует G12),

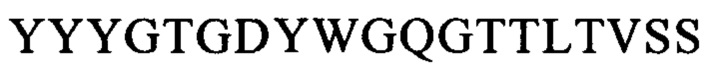 (соответствует 21.26),
(соответствует 21.26),
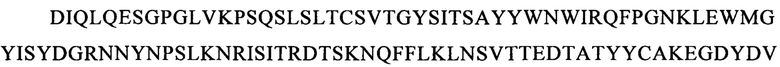
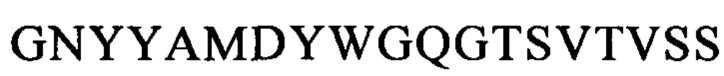 (соответствует 1075.7),
(соответствует 1075.7),
a VL из моноклонального антитела к CLL1 содержит по меньшей мере одну из следующих последовательностей:
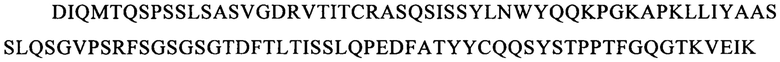 (соответствует SC02-357, SC02-378 и SC02161),
(соответствует SC02-357, SC02-378 и SC02161),
 (соответствует М26),
(соответствует М26),
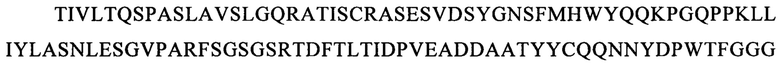
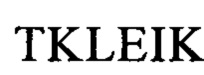 (соответствует М31),
(соответствует М31),
 (соответствует G4),
(соответствует G4),

 (соответствует М22),
(соответствует М22),
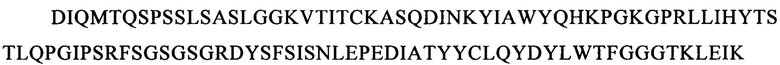 (соответствует М29),
(соответствует М29),
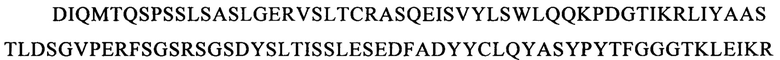 (соответствует М2),
(соответствует М2),
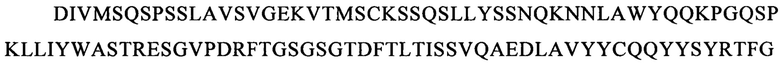
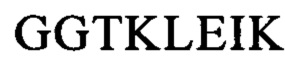 (соответствует М5),
(соответствует М5),
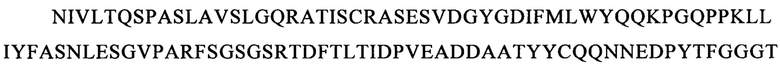
 (соответствует G12),
(соответствует G12),
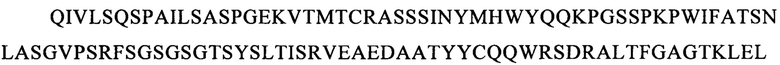 (соответствует 21.26),
(соответствует 21.26),
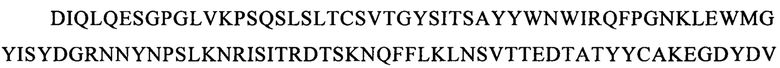
 (соответствует 1075.7)
(соответствует 1075.7)
В настоящем изобретении описаны также CLL1-специфические scCAR, указанные выше, которые дополнительно содержат другой внеклеточный лигандсвязывающий домен, не являющийся специфическим для CLL1, например, специфический в отношении антигена CD33, антигена CD44, антигена CD47, антигена CD123, антигена CD96 и Т-клеточного иммуноглобулина и домена 3 муцина (TIM-3).
В настоящем изобретении описаны также CLL1-специфические scCAR, указанные выше, которые дополнительно содержат сигнальный пептид, предпочтительно имеющий SEQ ID NO: 1 или SEQ ID NO: 2, помогающий полипептиду CAR достигать мембраны иммунной клетки.
В настоящем изобретении описаны также CLL1-специфические scCAR, указанные выше, в которых линкер, имеющий SEQ ID NO: 10, встроен между VH и VL.
Полинуклеотиды, векторы:
Настоящее изобретение относится также к полинуклеотидам и векторам, обеспечивающим гетерологичную экспрессию в клетках анти-CLL1 CAR, предлагаемого в изобретении, которые кодируют полипептидные последовательности, подробно описанные выше.
Полинуклеотиды могут быть включены в кассету экспрессии или экспрессионный вектор (например, в плазмиду, предназначенную для интродукции в бактериальную клетку-хозяина, или вирусный вектор, такой как бакуловирусный вектор, предназначенный для трансфекции клетки насекомого-хозяина, или плазмидный или вирусный вектор, такой как лентивирус, предназначенный для трансфекции клетки млекопитающего-хозяина).
В конкретном варианте осуществления изобретения можно включать различные нуклеотидные последовательности в один полинуклеотид или вектор, который содержит нуклеотидную последовательность, кодирующую рибосомальную skip-последовательность, такую как последовательность, кодирующую пептид 2А. Пептиды 2А, которые были идентифицированы в подгруппе афтовирусов группы пикорнавирусов, приводят к рибосомальному «перескоку» (skip) от одного кодона к следующему без образования пептидной связи между двумя аминокислотами, кодируемыми кодонами (см. Donnelly и Elliott, 2001; Atkins, Wills и др., 2007; Doronina, Wu и др., 2008). Под «кодоном» понимают три нуклеотида на мРНК (или на смысловой цепи молекулы ДНК), которые транслируются рибосомой в один аминокислотный остаток. Таким образом, можно синтезировать два полипептида из одной непрерывной рамки считывания в мРНК, если полипептиды разделены последовательностью олигопептида 2А, присутствующей в рамке считывания. Такие рибосомальные skip-механизмы хорошо известны в данной области и известно их применение в нескольких векторах для экспрессии нескольких белков, кодируемых одной матричной РНК.
Для направления трансмембранного полипептида в секреторный путь клетки-хозяина, в последовательность полинуклеотида или в последовательность вектора помещают секреторную сигнальную последовательность (известную также как лидерная последовательность, пре-про-последовательность или пре-последовательность). Секреторную сигнальную последовательность функционально связывают с трансмембранной нуклеотидной последовательностью, т.е. две последовательности соединяют в правильной рамке считывания и размещают так, чтобы направлять новый синтезированный полипептид в секреторный путь клетки-хозяина. Секреторные сигнальные последовательности, как правило, размещают в 5'-направлении относительно нуклеотидной последовательности, кодирующей представляющий интерес полипептид, хотя некоторые секреторные сигнальные последовательности можно размещать и в другом месте представляющей интерес нуклеотидной последовательности (см., например, Welch и др., US №5037743; Holland и др., US №5143830). В предпочтительном варианте осуществления изобретения сигнальный пептид содержит аминокислотные последовательности SEQ ID NO: 1 и 2 или идентичные по меньшей мере на 90%, 95%, 97% или 99% SEQ ID NO: 1 и/или 2.
Специалистам в данной области должно быть очевидно, что с учетом вырожденности генетического кода могут иметь место значительные вариации последовательности указанных полинуклеотидных молекул. Предпочтительно нуклеотидные последовательности, предлагаемые в настоящем изобретении, имеют оптимизированные кодоны для экспрессии в клетках млекопитающих, предпочтительно для экспрессии в человеческих клетках. Оптимизация кодонов относится к замене в представляющей интерес последовательности кодонов, встречающихся относительно редко в генах с высоким уровнем экспрессии, на кодоны, которые, как правило, часто встречаются в генах с высоким уровнем экспрессии в указанных видах, такие кодоны кодируют те же аминокислоты, что и подлежащие замене кодоны.
Методы доставки
В настоящем изобретении описаны различные средства, обеспечивающие экспрессию анти-CLL1 химерного антигенного рецептора (CAR), указанного в настоящем описании, в иммунных клетках.
Методы интродукции полинуклеотидной конструкции в клетки известны в данной области и включают, например (но не ограничиваясь только ими), методы стабильной трансформации, при осуществлении которых полинуклеотидную конструкцию, кодирующую указанный CAR, интегрируют в геном клетки, методы кратковременной трансформации, при осуществлении которых полинуклеотидную конструкцию не интегрируют в геном клетки, и опосредуемые вирусами методы.
Указанные полинуклеотиды можно интродуцировать в клетку, например, с использованием рекомбинантных вирусных векторов (например, ретровирусов, аденовирусов), липосом и т.п.Например, методы кратковременной трансформации включают микроинъекцию, электропорацию или бомбардировку частицами, слияние клеток. Указанные полинуклеотиды можно включать в векторы, более конкретно, в плазмиды или вирусы, с целью экспрессии в клетках. Указанный плазмидный вектор может содержать также селектируемый маркер, обеспечивающий идентификацию и/или отбор клеток, в которые встроен указанный вектор.
В один вектор можно включать различные трансгены. Указанный вектор может содержать нуклеотидную последовательность, кодирующую рибосомальную skip-последовательность, такую как последовательность, кодирующую пептид 2А. Пептиды 2А, которые были идентифицированы в подгруппе афтовирусов группы пикорнавирусов, приводят к рибосомальному «перескоку» (skip) от одного кодона к следующему без образования пептидной связи между двумя аминокислотами, кодируемыми кодонами (см. Donnelly и др., J. of General Virology 82, 2001, сс. 1013-1025; Donnelly и др., J. of Gen. Virology 78, 1997, сс. 13-21; Doronina и др., Mol. And. Cell. Biology 28(13), 2008, сс. 4227-4239; Atkins и др., RNA 13, 2007, сс. 803-810).
Под «кодоном» понимают три нуклеотида на мРНК (или на смысловой цепи молекулы ДНК), которые транслируются рибосомой в один аминокислотный остаток. Таким образом, можно синтезировать два полипептида из одной непрерывной рамки считывания в мРНК, если полипептиды разделены последовательностью олигопептида 2А, присутствующей в рамке считывания. Такие рибосомальные skip-механизмы хорошо известны в данной области и известно их применение в нескольких векторах для экспрессии нескольких белков, кодируемых одной матричной РНК.
В более предпочтительном варианте осуществления изобретения полинуклеотиды, которые кодируют полипептиды, предлагаемые в настоящем изобретении, могут представлять собой мРНК, которые интродуцируют непосредственно в клетки, например, путем электропорации. При создании изобретения были определены оптимальные условия для электропорации мРНК в Т-клетку. При создании изобретения применяли технологию cytoPulse, которая позволяет с помощью пульсирующих электрических полей кратковременно повышать проницаемость (пермеабилизовывать) живые клетки для доставки материала в клетки. Технология, основанная на применении системы создания волновых форм для электропорации PulseAgile® (фирма ВТХ Havard Apparatus, 84 October Hill Road, Holliston, MA 01746, США), гарантирует точный контроль продолжительности, интенсивности импульса, а также интервала между импульсами (US №6010613 и международная заявка РСТ WO 2004/083379). Все указанные параметры можно модифицировать для достижения наилучших условий для высокоэффективной трансфекции с минимальной смертностью. В целом, первые электрические импульсы высокой интенсивности приводят к образованию пор, а последующие электрические импульсы более низкой интенсивности позволяют полинуклеотиду проникать в клетку.
Различные методы, описанные выше, предполагают интродукцию scCAR в клетку. Например (но не ограничиваясь только этим), указанный scCAR можно интродуцировать в виде трансгенов, кодируемых одним плазмидным вектором. Указанный плазмидный вектор может содержать также селектируемый маркер, обеспечивающий идентификацию и/или отбор клеток, в которые встроен указанный вектор.
Полипептиды можно синтезировать in situ в клетке в результате интродукции в клетку полинуклеотидов, кодирующих указанные полипептиды. Альтернативно этому, указанные полипептиды можно получать вне клетки и затем интродуцировать в нее. Методы интродукции полинуклеотидных конструкций в клетки известны в данной области и их примерами являются (но не ограничиваясь только ими) методы стабильной трансформации, в которых полинуклеотидную конструкцию интегрируют в геном клетки, методы кратковременной трансформации, в которых полинуклеотидную конструкцию не интегрируют в геном клетки, и методы, основанные на использовании вирусов. Указанные полинуклеотиды можно интродуцировать в клетку, например, с использованием рекомбинантных вирусных векторов (например, ретровирусов, аденовирусов), липосом и т.п. Например, методы кратковременной трансформации включают микроинъекцию, электропорацию или бомбардировку частицами. Указанные полинуклеотиды можно включать в векторы, более конкретно, в плазмиды или вирусы, с целью экспрессии в клетках.
Активация и размножение Т-клеток
Как до, так и после осуществления генетической модификации Т-клеток, даже в том случае, если активация и пролиферация генетически модифицированных иммунных клеток, предлагаемых в настоящем изобретении, не зависит от механизмов связывания с антигеном, иммунные клетки, прежде всего Т-клетки, предлагаемые в настоящем изобретении, можно подвергать дополнительной активации и размножать с помощью методов, описанных, например, в US №№6352694; 6534055; 6905680; 6692964; 5858358; 6887466; 6905681;7144575;7067318; 7172869; 7232566; 7175843; 5883223; 6905874; 6797514; 6867041 и публикации заявки на патент США №2006/0121005. Т-клетки можно размножать in vitro или in vivo.
Как правило, Т-клетки, предлагаемые в изобретении, размножают путем приведения в контакт с агентом, который стимулирует комплекс CD3 TCR, и костимуляторной молекулой на поверхности Т-клеток с целью создания сигнала активации для Т-клетки. Например, для создания сигнала активации для Т-клетки можно применять химические соединения, такие как ионофор кальция А23187, форбол-12-миристат-13-ацетат (ФМА), или митогенные лектины, такие как фитогемагглютинин (ФГА).
Например (но не ограничиваясь только этим), популяции Т-клеток можно стимулировать in vitro путем приведения в контакт с антителом к CD3 или его антигенсвязывающим фрагментом, или антителом к CD2, иммобилизованным на поверхности, или путем приведения в контакт с активатором протеинкиназы С (например, бриостатином) в сочетании с ионофором кальция. Для костимуляции вспомогательной молекулы на поверхности Т-клеток используют лиганд, который связывается с вспомогательной молекулой. Например, популяцию Т-клеток можно приводить в контакт с антителом к CD3 и антителом к CD28 в условиях, обеспечивающих стимуляцию пролиферации Т-клеток. Условия, пригодные для культивирования Т-клеток, включают соответствующие среды (например, минимальную поддерживающую среду или среду RPMI 1640, или X-vivo 5, (фирма Lonza)), которые могут содержать факторы, необходимые для обеспечения пролиферации и жизнеспособности, включая сыворотку (например, фетальную бычью сыворотку или человеческую сыворотку), интерлейкин-2 (IL-2), инсулин, IFN-g, 1L-4, 1L-7, GM-CSF, -10, -2, 1L-15, TGFp и TNF, или любые другие добавки для роста клеток, известные специалисту в данной области. Другие добавки для роста клеток включают (но не ограничиваясь только ими) сурфактант, плазманат и восстановители, такие как N-ацетилцистеин и 2-меркаптоэтанол. Среды могут представлять собой RPMI 1640, A1M-V, DMEM, MEM, a-MEM, F-12, X-Vivo 1 и X-Vivo 20, Optimizer, содержащие в качестве добавок аминокислоты, пируват натрия и витамины, они могут быть бессывороточными или могут содержать в качестве добавок в определенном количестве сыворотку (или плазму) или определенный набор гормонов, и/или цитокин(ы) в количестве, достаточном для роста и размножения Т-клеток. Антибиотики, например, пенициллин и стрептомицин, включают только в экспериментальные культуры, но не в культуры клеток, которые предназначены для введения путем инфузии индивидууму. Клетки-мишени поддерживают в условиях, необходимых для поддержания роста, например, при соответствующей температуре (например, при 37°С) и атмосфере (например, в воздухе плюс 5% СО2). Клетки, которые подвергали стимуляции в течение различных промежутков времени, могут обладать различными характеристиками.
В другом конкретном варианте осуществления изобретения указанные клетки можно размножать путем совместного культивирования с тканью или клетками. Указанные клетки можно размножать также in vivo, например, в крови индивидуума после введения указанной клетки индивидууму.
Сконструированные иммунные клетки
Понятие «клетка» согласно настоящему изобретению относится, как правило, к клетке гематопоэтического происхождения, которая функционально участвует в инициации и/или реализации врожденного и/или адаптивного иммунного ответа. Клетка, предлагаемая в настоящем изобретении, предпочтительно представляет собой выделенную иммунную клетку и более предпочтительно Т-клетку, полученную из донора. Указанную иммунную клетку, предлагаемую в настоящем изобретении, можно получать также из стволовой клетки. Стволовые клетки могут представлять собой зрелые стволовые клетки, нечеловеческие эмбриональные стволовые клетки, более предпочтительно нечеловеческие стволовые клетки, стволовые клетки из пуповинной крови, клетки-предшественники, стволовые клетки костного мозга, индуцированные плюрипотентные стволовые клетки, тотипотентные стволовые клетки или гематопоэтические стволовые клетки. Репрезентативными человеческими клетками являются CD34+-клетки. Указанная выделенная клетка может представлять собой также дендритную клетку, дендритную киллерную клетку, тучную клетку, NK-клетку, В-клетку или Т-клетку, выбранную из группы, состоящей из воспалительных Т-лимфоцитов, цитотоксических Т-лимфоцитов, регуляторных Т-лимфоцитов или хелперных Т-лимфоцитов. В другом варианте осуществления изобретения указанная клетка может иметь происхождение из группы, состоящей из CD4+-Т-лимфоцитов и CD8+-Т-лимфоцитов. Перед осуществлением размножения и генетической модификации клеток, предлагаемых в изобретении, из организма индивидуума можно получать источник клеток с помощью различных методов, не ограничивающих объем изобретения. Клетки можно получать из многочисленных источников, включая (но не ограничиваясь только ими) мононуклеарные клетки периферической крови, костный мозг, ткань лимфатических узлов, пуповинную кровь, ткань тимуса, ткань из инфицированной области, асциты, плевральный выпот, ткань селезенки и опухоли. В некоторых вариантах осуществления настоящего изобретения можно применять любое количество Т-клеточных линий, доступных и известных специалистам в данной области.
В другом варианте осуществления изобретения указанную клетку можно получать из организма здорового донора, пациента, у которого диагностирован рак, или пациента, у которого диагностирована инфекция. В другом варианте осуществления изобретения указанная клетка представляет собой часть смешанной популяции клеток, обладающих различными фенотипическими характеристиками. Под объем настоящего изобретения подпадает также клеточная линия, полученная из Т-клетки, трансформированной согласно ранее описанному методу. Под объем настоящего изобретения подпадают модифицированные клетки, полученные с помощью ранее описанного метода, которые являются устойчивыми или чувствительными к иммуносупрессорной обработке.
Предпочтительными вариантами осуществления настоящего изобретения являются описанные выше Т-клетки или популяция Т-клеток, наделенных CLL1 CAR, которые не экспрессируют функциональный TCR и которые обладают реактивностью в отношении CLL1-позитивных клеток, предназначенные для их аллогенной трансплантации пациентам.
Более предпочтительным вариантом осуществления настоящего изобретения являются Т-клетки или популяция Т-клеток, наделенных CLL1 scCAR, и которые обладают реактивностью в отношении CLL1-позитивных клеток, которые не экспрессируют функциональный TCR и обладают устойчивостью к отобранному лекарственному средству, предназначенные для аллогенной трансплантации пациентам, которых лечат указанным отобранным лекарственным средством.
Настоящее изобретение относится к способу получения сконструированных иммунных клеток, предназначенных для иммунотерапии, который включает интродукцию ex vivo в указанные иммунные клетки полинуклеотидов или векторов, кодирующих CLL1 CAR, в соответствии с методами трансформации, описанными ранее в WO 2014/130635, WO 2013/176916, WO 2013/176915, которые включены в настоящее описание в качестве ссылки.
В предпочтительном варианте осуществления изобретения указанные полинуклеотиды интродуцируют в иммунные клетки с помощью ретровирусных векторов с целью стабильной интеграции в клеточный геном.
Способы конструирования иммунных клеток, наделенных CAR, предлагаемыми в изобретении
Настоящее изобретение относится также к получению иммунных клеток, наделенных анти-CLL1 CAR, которые являются менее аллореактивными или неаллореактивными, что позволяет применять их для аллогенных обработок (т.е. с пониженным риском индукции реакции трансплантат-против-хозяина и/или придает устойчивость к различным стандартным медицинским мероприятиям).
Как будет указано ниже в настоящем описании, указанные способы могут дополнительно включать стадию генетической модификации указанной иммунной клетки с использованием по меньшей мере одной эндонуклеазы.
- Понятие «эндонуклеаза» относится к ферменту дикого типа или его варианту, обладающему способностью катализировать гидролиз (расщепление) связей между нуклеиновыми кислотами в молекуле ДНК или РНК, предпочтительно в молекуле ДНК. Эндонуклеазы не расщепляют молекулу ДНК или РНК независимо от ее последовательности, но они распознают и расщепляют молекулу ДНК или РНК в специфических полинуклеотидных последовательностях, в дальнейшем обозначенных как «последовательности-мишени» или «сайты-мишени». Эндонуклеазы можно классифицировать как редко расщепляющие эндонуклеазы, для которых, как правило, полинуклеотидный сайт распознавания имеет длину более 12 пар оснований (bp), более предпочтительно 14-55 пар оснований.
Предпочтительно в способах, предлагаемых в настоящем изобретении, используют редко расщепляющие эндонуклеазы. Редко расщепляющие эндонуклеазы могут представлять собой, например, хоминг-эндонуклеазу (Paques и Duchateau, 2007), химерную нуклеазу с цинковыми пальцами (ZFN), образованную слиянием сконструированных доменов «цинковых пальцев» с каталитическим доменом рестриктазы, такой как FokI (Porteus и Carroll, 2005), TALE-нуклеазу, эндонуклеазу Cas9 из системы CRISPR (Gasiunas, Barrangou и др., 2012; Jinek, Chylinski и др., 2012; Cong, Ran и др., 2013; Mali, Yang и др., 2013) или химическую эндонуклеазу (Eisenschmidt, Lanio и др., 2005; Arimondo, Thomas и др., 2006). В химических эндонуклеазах химический или пептидный расщепляющий компонент конъюгируют либо с полимером нуклеиновой кислоты, либо с другой ДНК, распознающей специфическую последовательность-мишень, тем самым обеспечивая направление расщепляющей активности на специфическую последовательность. Химические эндонуклеазы включают также синтетические нуклеазы типа конъюгатов ортофенантролина, ДНК-расщепляющей молекулы и триплекс-образующих олигонуклеотидов (TFO), которые, как известно, связываются со специфическими последовательностями ДНК (Kalish и Glazer, 2005). Редко расщепляющие эндонуклеазы можно применять для инактивации генов в определенном локусе или для интеграции трансгенов с помощью гомологичной рекомбинации (HR), т.е. путем индукции двухцепочечных разрывов ДНК (DSB) в локусе и встраивания в этот локус экзогенной ДНК с помощью механизма генной репарации (Perrin, Buckle и др., 1993; Rouet, Smih и др., 1994; Choulika, Perrin и др., 1995; Pingoud и Silva, 2007).
- Под «TALE-нуклеазой» (TALEN) понимают слитый белок, который состоит из связывающего нуклеиновую кислоту домена, как правило, происходящего из подобного активатору транскрипции эффектора (TALE), и одного нуклеазного каталитического домена для расщепления последовательности нуклеиновой кислоты-мишени. Каталитический домен предпочтительно представляет собой нуклеазный домен и более предпочтительно домен, обладающий эндонуклеазной активностью, например, типа I-TevI, ColE7, NucA и Fok-I. В конкретном варианте осуществления изобретения домен TALE может быть слит с мегануклеазой, например, типа I-CreI и I-OnuI, или ее функциональным вариантом. В более предпочтительном варианте осуществления изобретения указанная нуклеаза представляет собой мономерную TALE-нуклеазу. Мономерная TALE-нуклеаза представляет собой TALE-нуклеазу, которая не требует димеризации для специфического распознавания и расщепления, например, в виде слияний сконструированных повторов TAL с каталитическим доменом I-TevI, описанным в WO 2012/138927. Подобные активатору транскрипции эффекторы (TALE) представляют собой белки из видов бактерий рода Xanthomonas, содержащие несколько повторяющихся последовательностей, где каждый повтор содержит di-остатки (двойные остатки) в положениях 12 и 13 (RVD), обладающие специфичностью в отношении каждого нуклеотидного основания последовательности нуклеиновой кислоты-мишени. Связывающие домены, обладающие сходными модульными свойствами связывания нуклеиновых кислот по типу основание-с-основанием («base-per-base») (MBBBD), могут быть получены также из новых модульных белков, недавно обнаруженных заявителями в различных видах бактерий. Преимущество новых модульных белков заключается в том, что они характеризуются большей вариабельностью последовательности, чем TAL-повторы. Предпочтительно RVD, ассоциированные с распознаванием различных нуклеотидов, представляют собой HD для распознавания С, NG для распознавания Т, NI для распознавания А, NN для распознавания G или A, NS для распознавания А, С, G или Т, HG для распознавания Т, IG для распознавания Т, NK для распознавания G, НА для распознавания С, ND для распознавания С, HI для распознавания С, HN для распознавания G, NA для распознавания G, SN для распознавания G или А и YG для распознавания Т, TL для распознавания А, VT для распознавания А или G и SW для распознавания А. В другом варианте осуществления изобретения имеющие решающее значение аминокислоты 12 и 13 можно изменять путем мутации на другие аминокислотные остатки для модуляции их специфичности в отношении нуклеотидов А, Т, С и G и, прежде всего, для повышения их специфичности. Ранее уже была описана TALE-нуклеаза, и ее применяли для стимуляции генного таргетинга и генных модификаций (Boch, Scholze и др., 2009; Moscou и Bogdanove, 2009; Christian, Cermak и др., 2010; Li, Huang и др., 2011). Сконструированные TAL-нуклеазы поступают в продажу под товарным знаком TALEN™ (фирма Cellectis, 8 rue de la Croix Jarry, 75013, Париж, Франция) и их можно заказывать у производителей, таких как фирма Life Technologies (Карсбад, шт. Калифорния, США).
Предпочтительные TALE-нуклеазы, которые распознают и расщепляют последовательность-мишень, описаны в РСТ/ЕР2014/075317. В частности, можно также интродуцировать дополнительный каталитический домен в клетку с указанной редко расщепляющей эндонуклеазой для усиления мутагенеза с целью повышения способности к инактивации генов-мишеней. Более конкретно, указанный дополнительный каталитический домен представляет собой фермент, процессирующий конец ДНК. Примерами ферментов, процессирующих конец ДНК, являются (но не ограничиваясь только ими) 5-3'-экзонуклеазы, 3-5'-экзонуклеазы, щелочные 5-3'-экзонуклеазы, 5'-флэп-эндонуклеазы, геликазы, фосфатаза, гидролазы и независимые от матриц ДНК-полимеразы. Примером указанного каталитического домена является (но не ограничиваясь только им) белковый домен или каталитически активное производное белкового домена, выбранного из группы, состоящей из hExoI (EXO1_HUMAN), ExoI дрожжей (EXO1_YEAST), ExoI E. coli, человеческой TREX2 (3'-репарационная экзонуклеаза), мышиной TREX1, человеческой TREX1, бычьей TREX1, крысиной TREX1, TdT (концевая дезоксинуклеотидил-трансфераза), человеческой ДНК2, ДНК2 дрожжей (DNA2_YEAST). В предпочтительном варианте осуществления изобретения указанный дополнительный каталитический домен обладает 3'-5'-экзонуклеазной активностью и в более предпочтительном варианте осуществления изобретения указанный дополнительный каталитический домен представляет собой каталитический домен TREX, более предпочтительно каталитический домен TREX2 (WO 2012/058458). В другом предпочтительном варианте осуществления изобретения указанный каталитический домен описывается одноцепочечным полипептидом TREX2. Указанный дополнительный каталитический домен можно сливать с содержащим нуклеазу слитым белком или химерным белком, предлагаемым в изобретении, необязательно с помощью пептидного линкера.
- Под «Cas9-эндонуклеазой» подразумевается любой созданный с помощью генной инженерии инструмент на основе РНК-направляемой нуклеазы Cas9 (Gasiunas, Barrangou и др., 2012; Jinek, Chylinski и др., 2012; Cong, Ran и др., 2013; Mali, Yang и др., 2013) из адаптивной иммунной системы CRISPR (короткие палиндромные повторы, регулярно расположенные группами (Clustered Regularly Interspaced Short palindromic Repeats)) типа II прокариот (см. обзор Sorek, Lawrence и др., 2013). CRISPR-ассоциированная (Cas) система впервые была обнаружена в бактериях, и ее функция состоит в защите от чужой вирусной или плазмидной ДНК, При осуществлении опосредуемой CRISPR генетической инженерии сначала проводят отбор последовательности-мишени, часто фланкированной короткой последовательностью мотива, который обозначают как примыкающий к протоспейсеру мотив (РАМ). После отбора последовательности-мишени конструируют специфическую crРНК (CRISPR-РНК), комплементарную указанной последовательности-мишени. Для систем CRISPR типа II требуется трансактивирующая crРНК (tracrPHК), спаренная с crРНК и связанная с внесенным белком Cas9. Cas9 функционирует в качестве молекулярного якоря, облегчающего спаривание оснований tracPHК с сРНК (Deltcheva, Chylinski и др., 2011). В этом тройном комплексе двойная структура tracrPHК:crРНК функционирует в качестве направляющей РНК, которая направляет эндонуклеазу Cas9 к когнатной последовательности-мишени. Распознавание мишени комплексом Cas9-tracrPHК:crPHК инициируется путем сканирования последовательности-мишени для обнаружения гомологии между последовательностью-мишенью и crРНК. Помимо комплементарности последовательности-мишени и crРНК, таргетинг ДНК требует присутствия короткого мотива, примыкающего к протоспейсеру (примыкающий к протоспейсеру мотив - РАМ). После спаривания двойной РНК и последовательности-мишени Cas9 интродуцирует «тупой» двухцепочечный разрыв на расстоянии 3 пар оснований в обратном направлении относительно мотива РАМ (Garneau, Dupuis и др., 2010). Применение Cas9 в иммунных клетках, прежде всего в Т-клетках, описано ранее в WO 2014191128.
Модификация Т-клетки путем инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR)
Согласно одному из объектов изобретения иммунную клетку можно делать менее аллогенной, например, путем инактивации по меньшей мере одного гена, экспрессирующего один или несколько компонентов Т-клеточного рецептора (TCR), согласно методу, описанному в WO 2013/176915. Указанную инактивацию можно объединять с инактивацией другого гена, такого как ген, кодирующий или регулирующий экспрессию белка HLA или β2m. Таким образом, существенно снижают риск возникновения реакции «трансплантат-против-хозяина» и отторжение трансплантата.
Методы получения менее аллогенных Т-клеток могут включать стадию инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR), в частности, генов ТСRальфа и/или ТСRбета.
Описанные в WO 2013/176915 методы получения экспрессирующей CAR иммунной клетки, пригодной для аллогенной трансплантации, путем инактивации одного или нескольких компонентов Т-клеточного рецептора (TCR), все включены в настоящее описание в качестве ссылки.
В настоящем изобретении предложена экспрессирующая анти-CLL1 CAR иммунная клетка, в которой инактивирован по меньшей мере один ген, экспрессирующий один или несколько компонентов Т-клеточного рецептора (TCR). Таким образом, настоящее изобретение относится к экспрессирующей анти-CLL1 CAR Т-клетке, в которой инавтивирован по меньшей мере один ген, экспрессирующий один или несколько компонентов Т-клеточного рецептора (TCR).
Под инактивированным геном TCR подразумевается, что представляющий интерес ген не экспрессируется в форме функционального белка. В конкретных вариантах осуществления изобретения способ генетической модификации основан на экспрессии в предназначенной для конструирования клетке редко расщепляющей эндонуклеазы, которая катализирует расщепление в требуемом гене, инактивируя тем самым указанный требуемый ген. Разрывы нуклеотидной цепи, вызываемые редко расщепляющей эндонуклеазой, как правило, репарируются с помощью различных механизмов гомологичной рекомбинации или соединения негомологичных концов (NHEJ). Однако NHEJ представляет собой несовершенный процесс репарации, который часто приводит к изменениям последовательности ДНК в сайте расщепления. Механизмы включают повторное соединение остатков двух концов ДНК посредством прямого повторного лигирования (Critchlow и Jackson, 1998) или посредством так называемого опосредуемого микрогомологией соединения концов (Betts, Brenchley и др., 2003; Ма, Kim и др., 2003). Репарация посредством соединения негомологичных концов (NHEJ) часто приводит к небольшим инсерциям или делециям и ее можно применять для создания специфических «выключений» генов. Указанная модификация может представлять собой замену, делецию или добавление по меньшей мере одного нуклеотида. Клетки, в которых произошел случай индуцированного расщеплением мутагенеза, т.е. случай мутагенеза, являющегося результатом NHEJ, можно идентифицировать и/или отбирать с помощью метода, хорошо известного в данной области. В конкретном варианте осуществления изобретения стадия инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR) в клетках каждого индивидуального образца, включает интродукцию в клетку редко расщепляющей эндонуклеазы, которая обладает способностью разрушать по меньшей мере один ген, кодирующий компонент Т-клеточного рецептора (TCR). В более конкретном варианте осуществления изобретения указанные клетки каждого индивидуального образца трансформируют нуклеиновой кислотой, кодирующей редко расщепляющую эндонуклеазу, которая обладает способностью разрушать по меньшей мере один ген, кодирующий компонент Т-клеточного рецептора (TCR), и указанную редко расщепляющую эндонуклеазу экспрессируют в указанных клетках.
В предпочтительном варианте осуществления изобретения указанный способ дополнительного конструирования иммунных клеток включает интродукцию в указанные Т-клетки полинуклеотидов, в частности мРНК, которые кодируют специфические редко расщепляющие эндонуклеазы, для избирательной инактивации вышеупомянутых генов путем расщепления ДНК. В более предпочтительном варианте осуществления изобретения указанные редко расщепляющие эндонуклеазы представляют собой TALE-нуклеазы или Cas9-эндонуклеазы. В настоящее время установлено, что TAL-нуклеазы обладают более высокой специфичностью и эффективностью расщепления по сравнению с другими типами редко расщепляющих эндонуклеаз, что обусловливает выбор этих эндонуклеаз для получения сконструированных иммунных клеток в промышленном масштабе с постоянным оборотом.
Согласно изобретению предполагается, что несущие анти-CLL1 CAR иммунные клетки с инактивированным одним или несколькими компонентами Т-клеточного рецептора (TCR), можно применять в качестве лекарственного средства.
Устойчивые к лекарственным средствам Т-клетки
Согласно другому объекту изобретения экспрессирующие анти-CLL1 CAR иммунные клетки, предлагаемые в изобретении, можно подвергать дополнительной генетической инженерии для повышения их устойчивости к иммуносупрессорным лекарственным средствам или химиотерапевтическим обработкам, которые применяют в качества стандартной медицинской помощи для лечения рака, ассоциированного с CLL1-позитивными злокачественными клетками, прежде всего AML.
Для уничтожения раковых клеток разработан ряд цитотоксических агентов (противораковых лекарственных средств), таких как антиметаболиты, алкилирующие агенты, антрациклины, ингибиторы ДНК-метилтрансферазы, соединения платины и веретенные яды. Однако применение этих агентов в сочетании с новыми терапиями, такими как иммунотерапии, является проблематичным. Например, химиотерапевтические агенты могут оказаться вредными при разработке сильных противоопухолевых иммунокомпетентных клеток из-за неспецифических профилей токсичности этих агентов. Основанные на применении малых молекул терапии, мишенью которых являются пути клеточной пролиферации, могут также мешать созданию противоопухолевого иммунитета. Если химиотерапевтические режимы, которые обладают кратковременной эффективностью, удается объединять с новыми терапиями на основе иммунокомпетентных клеток, то можно достигать значительного усиления антинеопластической терапии (см. обзор Dasgupta, McCarty и др., 2011).
Для усиления противораковой терапии и избирательного приживления аллогенных иммунных клеток указанным аллогенным иммунным клеткам придают устойчивость к лекарственным средствам для их защиты от токсических побочных действий химиотерапевтических агентов. Устойчивость иммунных клеток к лекарственным средствам также приводит к увеличению их количества in vivo или ex vivo, поскольку Т-клетки, которые экспрессируют ген, обусловливающий устойчивость к лекарственному средству, должны выживать и размножаться в отличие от чувствительных к лекарственному средству клеток.
Методы создания Т-клеток, устойчивых к химиотерапевтическим агентам, описаны в РСТ/ЕР2014/075317, документ полностью включен в настоящем описание в качестве ссылки.
В частности, настоящее изобретение относится к способу создания аллогенных клеток, пригодных для иммунотерапии, в которых по меньшей мере один ген, кодирующий компонент Т-клеточного рецептора (TCR), инактивируют, и один ген модифицируют с целью придания устойчивости к лекарственному средству, включающему:
• получение Т-клетки, экспрессирующей анти-CLL1 scCAR;
• модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки путем инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR);
• модификацию указанной экспрессирующей анти-CLL1 scCAR, предпочтительно гуманизированный CLL1 scCAR, Т-клетки для придания устойчивости к лекарственному средству указанной экспрессирующей анти-CLL1 scCAR Т-клетке;
• размножение указанной сконструированной экспрессирующей анти-CLL1 scCAR Т-клетки в присутствии указанного лекарственного средства.
Альтернативно этому, настоящее изобретение относится к способу, включающему:
- получение Т-клетки, экспрессирующей анти-CLL1 scCAR;
предпочтительно гуманизированный CLL1 scCAR;
- модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки для придания устойчивости к лекарственному средству указанной экспрессирующей анти-CLL1 scCAR Т-клетке;
- модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки путем инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR);
- размножение указанной сконструированной экспрессирующей анти-CLL1 scCAR Т-клетки в присутствии указанного лекарственного средства.
В частности, настоящее изобретение относится также к способу создания аллогенных клеток, пригодных для иммунотерапии, в которых по меньшей мере один ген, кодирующий компонент Т-клеточного рецептора (TCR), инактивируют и один ген модифицируют с целью придания устойчивости к лекарственному средству, включающему:
- получение Т-клетки, экспрессирующей анти-CLL1 scCAR;
предпочтительно гуманизированный CLL1 scCAR;
- модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки путем инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR);
- модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки для придания устойчивости к лекарственному средству указанной экспрессирующей анти-CLL1 scCAR Т-клетке;
- размножение указанной сконструированной экспрессирующей анти-CLL1 scCAR Т-клетки в присутствии указанного лекарственного средства.
Альтернативно этому, настоящее изобретение относится к способу, включающему:
- получение Т-клетки, экспрессирующей анти-CLL1 scCAR;
предпочтительно гуманизированный CLL1 scCAR;
- модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки для придания устойчивости к лекарственному средству указанной экспрессирующей анти-CLL1 scCAR Т-клетке;
- модификацию указанной экспрессирующей анти-CLL1 scCAR Т-клетки путем инактивации по меньшей мере одного гена, кодирующего компонент Т-клеточного рецептора (TCR);
- размножение указанной сконструированной экспрессирующей анти-CLL1 scCAR Т-клетки в присутствии указанного лекарственного средства.
Экспрессия генов, обусловливающих устойчивость к лекарственным средствам, в экспрессирующих анти-CLL1 scCAR иммунных клетках
В конкретном варианте осуществления изобретения указанную устойчивость к лекарственным средствам можно придавать Т-клетке путем экспрессии по меньшей мере одного гена, обусловливающего устойчивость к лекарственному средству. Указанный ген, обусловливающий устойчивость к лекарственному средству, представляет собой нуклеотидную последовательность, которая кодирует «устойчивость» к агенту, такому как химиотерапевтический агент (например, метотрексат). Другими словами, экспрессия гена, обусловливающего устойчивость к лекарственному средству, в клетке обеспечивает пролиферацию клетки в присутствии агента в большей степени, чем пролиферация соответствующей клетки, в которой отсутствует ген, обусловливающий устойчивость к лекарственному средству. Экспрессия гена, обусловливающего устойчивость к лекарственному средству, в клетке обеспечивает пролиферацию клеток в присутствии агента и не влияет на их эффективность. Ген, обусловливающий устойчивость к лекарственному средству, может кодировать устойчивость к антиметаболиту, метотрексату, винбластину, цисплатину, алкилирующим агентам, антрациклинам, цитотоксическим антибиотикам, анти-иммунофилинам, их аналогам или производным и т.п.
В одном из вариантов осуществления изобретения ген, обусловливающий устойчивость к лекарственному средству, предлагаемый в изобретении, может придавать устойчивость к лекарственному средству (или агенту), в частности, к противораковому лекарственному средству, выбранному из арацитина, цитозин-арабинозида, амсакрина, даунорубицина, идарубицина, новантрона, митоксантрона, вепезида, этопозида (VP16), триоксида мышьяка, транс-ретиноевой кислоты, комбинации триоксида мышьяка и транс-ретиноевой кислоты, мехлорэтамина, прокарбазина, хлорамбуцила, цитарабина, антрациклинов, 6-тиогуанина, гидроксимочевины, преднизона и их комбинации.
Идентифицировано несколько генов, обусловливающих устойчивость к лекарственному средству, которые потенциально пригодны для придания устойчивости к лекарственному средству требуемым клеткам (Takebe, Zhao и др., 2001; Sugimoto, Tsukahara и др., 2003; Zielske, Reese и др., 2003; Nivens, Felder и др., 2004; Bardenheuer, Lehmberg и др., 2005; Kushman, Kabler и др., 2007).
Одним из примеров гена, обусловливающего устойчивость к лекарственному средству, может являться также мутантная или модифицированная форма дигидрофолат-редуктазы (DHFR). DHFR представляет собой фермент, участвующий в регулировании количества тетрагидрофолата в клетке, и является важным для синтеза ДНК. Аналоги фолата, такие как метотрексат (МТХ), ингибируют DHFR и поэтому применяются в качестве антинеопластических агентов в клинических условиях. Описаны различные мутантные формы DHFR, которые обладают повышенной устойчивостью к ингибированию антифолатными агентами, которые применяют в терапии. В конкретном варианте осуществления изобретения ген, обусловливающий устойчивость к лекарственному средству, предлагаемый в настоящем изобретении, может представлять собой нуклеотидную последовательность, кодирующую мутантную форму человеческой DHFR дикого типа (GenBank: ААН71996.1), которая содержит по меньшей мере одну мутацию, обусловливающую устойчивость к лечению антифолатным средством, таким как метотрексат. В конкретном варианте осуществления изобретения мутантная форма DHFR содержит по меньшей мере одну измененную в результате мутации аминокислоту в положении G15, L22, F31 или F34, предпочтительно в положениях L22 или F31 (Schweitzer, Dicker и др., 1990); международная заявка на патент WO 94/24277; патент США US №6642043). В конкретном варианте осуществления изобретения указанная мутантная форма DHFR содержит две измененные в результате мутации аминокислоты в положении L22 и F31. Соответствие аминокислотным положениям, указанным в настоящем описании, часто выражают в виде положений аминокислот в полипептиде DHFR дикого типа, зарегистрированного в GenBank: AAH71996.1. В конкретном варианте осуществления изобретения остаток серина в положении 15 предпочтительно заменяют на остаток триптофана. В другом конкретном варианте осуществления изобретения остаток лейцина в положении 22 предпочтительно заменяют на аминокислоту, которая может нарушать связывание мутантной DHFR с антифолатными агентами, предпочтительно на незаряженные аминокислотные остатки, такие как фенилаланин или тирозин. В другом конкретном варианте осуществления изобретения остаток фенилаланина в положениях 31 или 34 предпочтительно заменяют на остаток низкомолекулярной гидрофильной аминокислоты, такой как аланин, серин или глицин.
В контексте настоящего описания понятие «антифолатный агент» или «аналоги фолатов» относится к молекуле, которая должна оказывать некоторое воздействие на путь метаболизма фолатов. Примеры антифолатных агентов включают, например, метотрексат (МТХ); аминоптерин; триметрексат (Neutrexin™); эдатрексат; N10-пропаргил-5,8-дидеазафолиевую кислоту (СВ3717); ZD1694 (тумодекс), 5,8-дидеазафолиевую кислоту (IAHQ); 5,10-дидеазатетрагидрофолиевую кислоту (DDATHF); 5-деазафолиевую кислоту; РТ523 (N-альфа-(4-амино-4-дезоксиптероил)-N-дельта-гемифталоил-L-орнитин); 10-этил-10-дезаминоптерин (DDATHF, ломатрексол); пиритрексим; 10-EDAM; ZD1694; GW1843; пеметрексат и PDX (10-пропаргил-Ю-дезаминоптерин).
Другим примером гена, обусловливающего устойчивость к лекарственному средству, может являться также мутантная или модифицированная форма инозин-5'-монофосфатдегидрогеназы II (IMPDH2), фермента, ограничивающего скорость синтеза de novo гуанозиновых нуклеотидов. Мутантная или модифицированная форма IMPDH2 представляет собой ген, обусловливающий устойчивость к ингибитору IMPDH. Ингибиторы IMPDH могут представлять собой микофеноловую кислоту (МРА) или ее пролекарство микофенолата мофетил (MMF). Мутант IMPDH2 может содержать по меньшей мере одну, предпочтительно две мутации в сайте связывания MAP человеческой IMPDH2 дикого типа (NP_000875.2), которые приводят к значительному повышению устойчивости к ингибитору IMPDH. Мутации предпочтительно затрагивают положения Т333 и/или S351 (Yam, Jensen и др., 2006; Sangiolo, Lesnikova и др., 2007; Jonnalagadda, Brown и др., 2013). В конкретном варианте осуществления изобретения остаток треонина в положении 333 заменяют на остаток изолейцина и остаток серина в положении 351 заменяют на остаток тирозина. Соответствие аминокислотным положениям, указанным в настоящем описании, часто выражают в виде положений аминокислот в человеческом полипептиде IMPDH2 дикого типа, зарегистрированном под номером NP_000875.2.
Другим геном, обусловливающим устойчивость к лекарственному средству, является мутантная форма кальциневрина. Кальциневрин (РР2В) представляет собой повсеместно экспрессируемую серин/треониновую протеинфосфатазу, которая участвует во многих биологических процессах и которая играет центральную роль в активации Т-клеток. Кальциневрин представляет собой гетеродимер, состоящий из каталитической субъединицы (CnA; три изоформы) и регуляторной субъединицы (CnB; две изоформы). После контакта с Т-клеточным рецептором кальциневрин дефосфоририлирует фактор транскрипции NFAT, что приводит к его транслокации в ядро, где он активирует имеющий решающее значение ген, такой как IL2. FK506 в комплексе с FKBP12 или циклоспорин А (CsA) в комплексе с СуРА блокируют доступ NFAT к активному сайту кальциневрина, препятствуя его дефосфорилированию и ингибируя тем самым активацию Т-клеток (Brewin, Mancao и др., 2009). Ген, обусловливающий устойчивость к лекарственному средству, предлагаемый в настоящем изобретении, может представлять собой нуклеотидную последовательность, которая кодирует мутантную форму кальциневрина, устойчивую к ингибитору кальциневрина, такому как FK506 и/или CsA. В конкретном варианте осуществления изобретения указанная мутантная форма может содержать по меньшей мере одну мутантную аминокислоту в гетеродимере кальциневрина дикого типа в положениях: V314, Y341, М347, Т351, W352, L354, K360, предпочтительно двойные мутации в положениях Т351 и L354 или V314 и Y341. В конкретном варианте осуществления изобретения остаток валина в положении 341 можно заменять на остаток лизина или аргинина, остаток тирозина в положении 341 можно заменять на остаток фенилаланина; метионин в положении 347 можно заменять на остаток глутаминовой кислоты, аргинина или триптофана; треонин в положении 351 можно заменять на остаток глутаминовой кислоты; остаток триптофана в положении 352 можно заменять на остаток цистеин, глутаминовой кислоты или аланина, серин в положении 353 можно заменять на остаток гистидина или аспарагина, лейцин в положении 354 можно заменять на остаток аланина; лизин в положении 360 можно заменять на остаток аланина или фенилаланина в последовательности, соответствующей последовательности, зарегистрированной в GenBank: ACX34092.1. Соответствие аминокислотным положениям, указанным в настоящем описании, часто выражают в виде положений аминокислот в человеческом полипептиде гетеродимера кальциневрина дикого типа, зарегистрированном в GenBank: ACX34092.1.
В другом конкретном варианте осуществления изобретения указанная мутантная форма может содержать по меньшей мере одну мутантную аминокислоту в гетеродимере кальциневрина дикого типа в положениях: V120, N123, L124 или K125, предпочтительно двойные мутации в положениях L124 и K125. В конкретном варианте осуществления изобретения валин в положении 120 можно заменять на остаток серина, аспарагиновой кислоты, фенилаланина или лейцина; аспарагин в положении 123 можно заменять на триптофан, лизин, фенилаланин, аргинин, гистидин или серин; лейцин в положении 124 можно заменять на остаток треонина; лизин в положении 125 можно заменять на аланин, глутаминовую кислоту, триптофан, или два остатка, такие как лейцин-аргинин или изолейцин-глутаминовая кислота, можно добавлять после лизина в положении 125 в аминокислотную последовательность, соответствующую последовательности, зарегистрированной в GenBank: ACX34095.1. Соответствие аминокислотным положениям, указанным в настоящем описании, часто выражают в виде положений аминокислот в человеческом полипептиде гетеродимера кальциневрина дикого типа, зарегистрированном в GenBank: АСХ34095.1.
Другим геном, обусловливающим устойчивость к лекарственному средству, является ген O(6)-метилгуанинметилтрансферазы (MGMT), кодирующий человеческую алкилгуанинтрансферазу (hAGT). AGT представляет собой репарирующий ДНК белок, который придает устойчивость к цитотоксическим действиям алкилирующих агентов, таких как нитрозомочевины и темозоломид (TMZ). 6-бензилгуанин (6-BG) является ингибитором AGT, который усиливает токсичность нитрозомочевины и его вводят совместно с TMZ для усиления цитотоксических действий этого агента. Несколько мутантных форм MGMT, которые кодируют варианты AGT, обладают высокой устойчивостью к инактивации с помощью 6-BG, но сохраняют их способность к репарации повреждения ДНК (Maze, Kurpad и др., 1999). В конкретном варианте осуществления изобретения мутантная форма AGT может содержать мутантную аминокислоту в положении Р140 AGT дикого типа в аминокислотной последовательности, представленной в базе данных UniProtKB под регистрационным номеров Р16455. В предпочтительном варианте осуществления изобретения указанный пролин в положении 140 заменяют на остаток лизина.
Другим геном, обусловливающим устойчивость к лекарственному средству, может являться ген обусловливающего устойчивость ко многим лекарственным средствам белка 1 (MDR1). Этот ген кодирует мембранный гликопротеин, известный как Р-гликопротеин (P-GP), который участвует в транспорте побочных продуктов метаболизма через клеточную мембрану. Белок P-GP обладает широким спектром специфичности в отношении нескольких структурно неродственных химиотерапевтических агентов.
Установлено, что сверхэкспрессия обусловливающего устойчивость ко многим лекарственным средствам белка 1, приводит к устойчивости к таким лекарственным средства как митоксантрон (Charles S. Morrow, Christina Peklak-Scott, Bimjhana Bishwokarma, Timothy E. Kute, Pamela K. Smitherman и Alan J. Townsend. Multidrug Resistance Protein 1 (MRP1, ABCC1) Mediates Resistance to Mitoxantrone via Glutathione-Dependent Drug Efflux Mol Pharmacol, 69, апрель 2006 г., сс. 1499-1505).
Таким образом, устойчивость к лекарственному средству можно придавать клеткам в результате экспрессии нуклеотидной последовательности, которая кодирует MDR-1 (NP_000918).
Еще один путь получения клеток, устойчивых к лекарственному средству, состоит в получении клеток со специфической(ими) мутацией(ями), такими как мутации Arg486 и Glu571 в гене человеческой топоизомеразы II, для придания устойчивости к амсакрину (S. Patel, В.A. Keller и L.M. Fisher. Molecular Pharmacology, т. 57, 2000, сс. 784-791).
Еще один путь получения клеток, устойчивых к лекарственному средству, состоит в получении клеток, сверхэкспрессирующих микроРНК-21, для придания устойчивости к даунорубицину (Haitao и др.. Involvement of miR-21 in resistance to daunorubicin by regulating PTEN expression in the leukaemia K562 cell line Bai, FEBS Letters, т. 585, вып. 2, сс. 402-408).
В предпочтительном варианте осуществления изобретения клетки, которые несут указанную обусловливающую устойчивость к лекарственному средству мРНК или белок, содержат также ингибирующую мРНК или ген, обусловливающий ее экспрессию, что позволяет избирательно разрушать указанные устойчивые к лекарственному средству клетки в присутствии указанного лекарственного средства или после введения указанного лекарственного средства.
Ген, обусловливающий устойчивость к лекарственному средству, может придавать также устойчивость к цитотоксическим антибиотикам и может представлять собой ген ble или ген mcrA. Эктопическая экспрессия гена ble или гена mcrA в иммунной клетке обеспечивает избирательное преимущество при воздействии химиотерапевтического средства, т.е. блеомицина или митомицина С соответственно.
Наиболее широко распространенным практическим подходом к генной терапии является добавление гена для создания Т-клетки посредством эффективной доставки гена с помощью векторов, предпочтительно вирусного вектора. Таким образом, в конкретном варианте осуществления изобретения указанный ген, обусловливающий устойчивость к лекарственному средству, можно экспрессировать в клетке в результате интродукции в клетку трансгена, предпочтительно кодируемого по меньшей мере одним вектором.
В одном из вариантов осуществления изобретения клетки, которые несут ген, обусловливающий устойчивость к лекарственному средству, или модифицированный ген, обусловливающий устойчивость к лекарственному средству, содержат также индуцибельный ген суицида, индукция которого провоцирует клеточную гибель, что обеспечивает их избирательное разрушение.
Случайная инсерция генов в геном может приводить к несоответствующей экспрессии встроенного гена или гена, расположенного вблизи сайта инсерции. Специфическая генная терапия с использованием гомологичной рекомбинации экзогенной нуклеиновой кислоты, содержащей эндогенные последовательности для направления генов к специфическим сайтам в геноме, может позволять конструировать безопасные Т-клетки. Как описано выше, стадия генетической модификации в способе может включать стадию интродукции в клетки экзогенной нуклеиновой кислоты, которая содержит по меньшей мере последовательность, кодирующую ген, обусловливающий устойчивость к лекарственному средству, и часть эндогенного гена, в результате чего имеет место гомологичная рекомбинация между эндогенным геном и экзогенной нуклеиновой кислотой. В конкретном варианте осуществления изобретения указанный эндогенный ген может представлять собой ген дикого типа, «обусловливающий устойчивость к лекарственному средству», в результате чего после гомологичной рекомбинации ген дикого типа заменяется мутантной формой гена, которая обусловливает устойчивость к лекарственному средству.
Известно, что эндонуклеолитические разрывы стимулируют скорость гомологичной рекомбинации. Таким образом, в конкретном варианте осуществления изобретения способ, предлагаемый в изобретении, дополнительно включает стадию экспрессии в клетке редко расщепляющей эндонуклеазы, которая может расщеплять последовательность-мишень в эндогенном гене. Указанный эндогенный ген может кодировать, например, DHFR, IMPDH2, кальциневрин или AGT. Указанная редко расщепляющая эндонуклеаза может представлять собой TALE-нуклеазу, нуклеазу с цинковыми пальцами, нуклеазу CRISPR/Cas9, MBBBD-нуклеазу или мегануклеазу.
Инактивация генов, повышающих чувствительность к лекарственным средствам в иммунных клетках, экспрессирующих анти-CLL1 CAR
В другом конкретном варианте осуществления изобретения указанную устойчивость к лекарственному средству можно придавать клетке, предлагаемой в изобретении (экспрессирующей анти-CLL1 CAR иммунной клетке), путем инактивации гена, повышающего чувствительность к лекарственному средству.
При создании изобретения предпринимали попытку инактивировать потенциально повышающий чувствительность к лекарственному средству ген для создания предназначенной для иммунотерапии Т-клетки, в частности, для создания экспрессирующей анти-CLL1 CAR иммунной клетки, которую можно применять в комбинации с терапевтическим агентом (противораковым лекарственным средством).
Под инактивацией гена подразумевается, что представляющий интерес ген не экспрессируется в форме функционально белка. В конкретном варианте осуществления изобретения метод генетической модификации, применяемый при конструировании клеток, основывается на экспрессии одной редко расщепляющей эндонуклеазы, которая специфически катализирует расщепление в одном гене-мишени, инактивируя тем самым указанный ген-мишень. В конкретном варианте осуществления изобретения стадия инактивации по меньшей мере одного повышающего чувствительность к лекарственному средству гена включает интродукцию в клетку редко расщепляющей эндонуклеазы, которая может разрушать по меньшей мере один повышающий чувствительность к лекарственному средству ген. В более конкретном варианте осуществления изобретения указанные клетки трансформируют нуклеиновой кислотой, которая кодирует редко расщепляющую эндонуклеазу, которая может разрушать повышающий чувствительность к лекарственному средству ген, и экспрессируют указанную редко расщепляющую эндонуклеазу в указанных клетках. Указанная редко расщепляющая эндонуклеаза может представлять собой мегануклеазу, нуклеазу с «цинковыми пальцами», нуклеазу CRISPR/Cas9, MBBBD-нуклеазу или TALE-нуклеазу. В предпочтительном варианте осуществления изобретения указанная редко расщепляющая эндонуклеаза представляет собой TALE-нуклеазу.
В предпочтительном варианте осуществления изобретения повышающий чувствительность к лекарственному средству ген, который можно инактивировать для придания устойчивости к лекарственному средству Т-клетке, представляет собой ген человеческой дезоксицитидинкиназы (dCK). Этот фермент требуется для фосфорилирования дезоксирибонуклеозидов дезоксицитидина (dC), дезоксигуанозина (dG) и дезоксиаденозина (dA). Пуриновые нуклеотидные аналоги (PNA) метаболизируются dCK с образованием моно-, ди- и трифосфата PNA. Указанные трифосфатные формы, и прежде всего клофарабина трифосфат, конкурируют с АТФ при синтезе ДНК, действуют в качестве проапоптозного агента и являются сильными ингибиторами рибонуклеотидредуктазы (RNR), которая участвует в производстве тринуклеотидов.
Предпочтительно инактивация dCK в Т-клетках опосредуется TALE-нуклеазой. Для достижения указанной цели разработано несколько пар TALE-нуклеаз для dCK, которые собраны на полинуклеотидном уровне и подтверждены секвенированием. Примеры пар TALE-нуклеаз, которые можно применять согласно изобретению, описаны в РСТ/ЕР2014/075317.
Указанная инактивация dCK в Т-клетках обусловаливает устойчивость к аналогам пуриновых нуклеозидов (PNA), таким как клофарабин, флударабин или децитабин (дакоген).
В другом предпочтительном варианте осуществления изобретения инактивацию dCK в Т-клетках объединяют с инактивацией генов TRAC, получая указанные Т-клетки с двойным «выключением» (KO), которые обладают устойчивостью к лекарственному средству, такому как клофарабин, и являются менеее аллогенными. Указанные двойные свойства являются наиболее ценными для терапевтичесих целей, позволяя применять «имеющиеся в наличии» аллогенные клетки для иммунотерапии в сочетании с химиотерапией для лечения срадающих раком пациентов. Указанную двойную инактивацию посредством KO dCK/TRAC можно осуществлять одновременно или последовательно. Один из примеров пары TALE-нуклеаза dCK/TRAC, которая обеспечивает успех согласно изобретению, описан в РСТ/ЕР2014/075317, при этом, в частности, последовательности-мишени находятся в 2 локусах (dCK и TRAC).
Другим примером фермента, который можно инактивировать, является ген человеческой гипоксантин-гуанин-фосфорибозилтрансферазы (HPRT) (Genbank: М26434.1). В частности, HPRT можно инактивировать в сконструированных Т-клетках для придания устойчивости к цитостатическому метаболиту 6-тиогуанину (6TG), который под действием HPRT превращается в цитотоксический тиогуаниновый нуклеотид и который в настоящем время применяют для лечения страдающих раком, прежде всего лейкозами, пациентов (Hacke, Treger и др., 2013). Аналоги гуанинов метаболизируются трансферазой HPRT, которая катализирует добавление фосфорибозильного фрагмента и позволяет получать гуаниновые аналоги TGMP, включая 6-меркаптопурин (6МР) и 6-тиогуанин (6TG), которые можно применять в качестве очищающих лимфу лекарственных средств, предназначенных для лечения ALL. Они метаболизируются HPRT (гипоксантин-фосфорибозилтрансфераза, которая катализирует добавление фосфорибозильного фрагмента и позволяет получать TGMP). Их последующее фосфорилирование приводит к образованию их трифосфорилированных форм, которые в конце концов интнгрируются в ДНК. После включения в ДНК тио-ГТФ нарушает правильность репликации ДНК посредством их тиолатной группировки и создают точки случайной мутации, которые являются очень опасными для целостности клеток.
Таким образом, в настоящем изобретении предложена экспрессирующая анти-CLL1 scCAR клетка, прежде всего экспрессирующая анти-CLL1 scCAR Т-клетка, в которой scCAR имеет полипептидную последовательность SEQ ID NO: 35-112 (необязательно гуманизированную) и в которой ген dCK инактивирован.
В другом варианте осуществления изобретения инактивация CD3, который в норме экспрессируется на поверхности Т-клетки, может обусловливать устойчивость к антителам к CD3, таким как теплизумаб.
Устойчивые ко многим лекарственным средствам экспрессирующие анти-CLL1 scCAR иммунные клетки
Согласно другому конкретному варианту осуществления изобретения при создании изобретения предусматривалась разработка стратегии иммунотерапии на основе «имеющихся в наличии» аллогенных Т-клеток, в частности, аллогенной экспрессирующей анти-CLL1 scCAR Т-клетки, устойчивой ко многим лекарственным средствам, для отбора сконструированных Т-клеток, когда пациента лечат различными лекарственными средствами. Терапевтическую эффективность можно значительно повышать путем создания методами генетической инженерии устойчивых ко многим лекарственным средствам аллогенных Т-клеток. Указанная стратегия может быть особенно эффективной при лечении опухолей, которые дают ответ на комбинации лекарственных средств, обладающих синергетическими действиями. Более того, сконструированные Т-клетки с устойчивостью ко многим лекарственным средствам можно размножать и отбирать с использованием минимальной дозы лекарственных средств.
Так, способ, предлагаемый в настоящем изобретении, может включать модификацию Т-клетки для придания устойчивости ко многим лекарственным средствам указанной Т-клетке. Указанную устойчивость ко многим лекарственным средствам можно получать либо путем экспрессии более одного гена, обусловливающего устойчивость к лекарственному средству, либо путем инактивации более одного гена, повышающего чувствительность к лекарственному средству. В другом конкретном варианте осуществления изобретения устойчивость ко многим лекарственным средствам можно придавать указанной Т-клетке путем экспрессии по меньше мере одного гена, обусловливающего устойчивость к лекарственному средству, и инактивации по меньшей мере одного гена, повышающего чувствительность к лекарственному средству. В частности, устойчивость ко многим лекарственным средствам можно придавать указанной Т-клетке путем экспрессии по меньшей мере одного гена, обусловливающего устойчивость к лекарственному средству, такого как мутантная форма DHFR, мутантная форма IMPDH2, мутантная форма кальциневрина, мутантная форма MGMT, ген ble и ген vcrA, и инактивации по меньшей мере одного гена, повышающего чувствительность к лекарственному средству, такого как ген HPRT. В предпочтительном варианте осуществления изобретения устойчивость ко многим лекарственным средствам можно придавать путем инактивации гена HPRT и экспрессии мутантной формы DHFR; или путем инактивации гена HPRT и экспрессии мутантной формы IMPDH2; или путем инактивации гена HPRT и экспрессии мутантной формы кальциневрина; путем инактивации гена HPRT и экспрессии мутантной формы MGMT; путем инактивации гена HPRT и экспрессии гена ble; путем инактивации гена HPRT и экспрессии гена mcrA.
Одним из вариантов осуществления настоящего изобретения является аллогенная экспрессирующая анти-CLL 1 scCAR Т-клетка, которая экспрессирует ген, обусловливающий устойчивость более чем к одному лекарственному средству, или в которой инактивирован ген, повышающий чувствительность более чем к одному лекарственному средству.
Гены суицида в экспрессирующих анти-CLL1 scCAR иммунных клетках
В некоторых случаях, в связи с тем, что сконструированные Т-клетки могут размножаться и сохраняться в течение нескольких лет после введения, может оказаться желательным включать безопасный механизм для избирательной делеции введенных Т-клеток. Так, согласно некоторым вариантам осуществления изобретения способ, предлагаемый в изобретении, может включать трансформацию указанных Т-клеток рекомбинантным геном суицида. Указанный рекомбинантный ген суицида применяют для снижения риска непосредственной токсичности и/или неконтролируемой пролиферации указанных Т-клеток после введения индивидууму (Quintarelli С., Vera F., Blood, 2007; Теу S.K, Dotti G., Rooney C.M, Boil blood marrow transplant, 2007). Гены суицида позволяют осуществлять избирательную делецию трансформированных клеток in vivo. В частности, ген суицида обладает способностью превращать нетоксичное пролекарство в цитотоксическое лекарственное средство или экспрессировать токсичный продукт экспрессии гена. Другими словами, «ген суицида» представляет собой нуклеиновую кислоту, кодирующую продукт, где продукт вызывает гибель клеток сам или в присутствии других соединений.
Репрезентативным примером указанного гена суицида является ген, который кодирует тимидинкиназу вируса герпеса простого. Другими примерами являются тимидинкиназа вируса ветряной оспы и бактериальный ген цитозиндеаминазы, которая может превращать 5-фторцитозин в высокотоксичное соединение 5-фторурацил. Гены суицида также включают, например (но не ограничиваясь только ими) ген каспазы-9 или каспазы-8 или цитозиндеаминазы. Каспазу-9 можно активировать с помощью специфического химического индуктора димеризации (CID).Гены суицида могут также представлять собой гены полипептидов, которые экспрессируются на поверхности клетки и могут придавать клетке чувствительность к терапевтическим моноклональным антителам. В контексте настоящего описания «пролекарство» обозначает любое соединение, которое можно применять в способах, предлагаемых в настоящем изобретении, которое может превращаться в токсичный продукт. В способе, предлагаемом в настоящем изобретении, пролекарство превращается в токсичный продукт с помощью генного продукта гена суицида. Репрезентативным примером такого пролекарства является ганцикловир, который превращается in vivo в токсичное соединение с помощью тимидинкиназы HSV. После этого производное ганцикловира проявляет токсичность в отношении опухолевых клеток. Другие репрезентативные примеры пролекарств включают ацикловир, FIAU [1-(2-дезокси-2-фтор-β-D-арабинофуранозил)-5-йодурпцил], 6-метоксиарабинозид для VZV-TK и 5-фторцитозин для цитозиндеаминазы.
В одной из предпочтительных систем гена суицида применяется рекомбинантный антигенный полипептид, который содержит антигенный мотив, распознаваемый МАт к CD20 ритуксимабом, прежде всего QBen10, такой как так называемый полипептид RQR8, описанный в WO 2013/153391, который экспрессируется независимо от анти-CLL1 CAR. Ритуксимаб, запатентованное лекарственное средство, представляющее собой антитело, затем можно применять при необходимости для клеточного истощения.
Одним из вариантов осуществления настоящего изобретения является аллогенная экспрессирующая анти-CLL1 scCAR Т-клетка, которая экспрессирует ген, обусловливающий устойчивость более чем к одному лекарственному средству, или в которой инактивирован ген, повышающий чувствительность более чем к одному лекарственному средству, и в которой присутствует ген суицида, позволяющий разрушать указанные клетки.
В частности, настоящее изобретение относится к аллогенной Т-клетке, в частности, аллогенной экспрессирующей анти-CLL1 scCAR Т-клетке, и предпочтительно аллогенной экспрессирующей анти-CLL1 scCAR Т-клетке, которая содержит пептид, идентичный на 80%-100% scFv из антител SC02-357, SC02-378, SC02-161, М26, М31, G4, М22, М29, М2, М5, G12, 21.26 или 1075.7, указанная аллогенная экспрессирующая анти-CLL1 scCAR Т-клетка, которая содержит пептид, идентичный на 80%-100% scFv из антител SC02-357, SC02-378, SC02-161, М26, М31, G4, М22, М29, М2, М5, G12, 21.26 и 1075.7, обладает более предпочтительной устойчивостью к лекарственным средствам и особенно пригодна для иммунотерапии.
Устойчивость к лекарственному средству можно придавать путем инактивации генов, повышающих чувствительность к лекарственным средствам, или путем экспрессии генов, обусловливающих устойчивость к лекарственным средствам. Некоторыми примерами лекарственных средств, которые можно применять согласно изобретению, являются аналоги пуриновых нуклеозидов (PNA), такие как клофарабин и флударабин, или другие лекарственные средства, такие как 6-меркаптопурин (6МР) и 6-тиогуанин (6TG).
Одним из объектов настоящего изобретения являются способы конструирования иммунных клеток таким образом, чтобы сделать их устойчивыми к аналогам пуриновых нуклеотидов (PNA), таким как клофарабин или флударабин, в результате чего их можно применять для противораковой иммунотерпии пациентов, которых предварительно лечили указанными общепринятыми химиотерапевтическими средствами.
Устойчивость к лекарственным средствам можно придавать Т-клеткам путем инактивации одного или нескольких гена(ов), ответственного(ых) за чувствительность клеток к лекарственному средству (повышающий(ие) чувствительность к лекарственному средству ген(ы)), такие как гены dcK и/или HPRT.
Согласно другому объекту изобретения устойчивость к лекарственным средствам можно придавать Т-клетке путем экспрессии гена, обусловливающего устойчивость к лекарственному средству. Установлено, что аллельные варианты некоторых генов, таких как ген дигидрофолат-редуктазы (DHFR), инозин-монофосфат-дегидрогеназы 2 (IMPDH2), кальциневрина или метилгуанинтрансферазы (MGMT), придают устойчивость к лекарственному средству клетке, предлагаемой в изобретении.
Например, CD52 и глюкортикоидные рецепторы (GR), которые являются мишенями лекарственных средств при лечении Campath® (алемтузумаб) или ритуксимабом и глюкокортикоидами, можно инактивировать, придавая клеткам устойчивость к указанным обработкам и давая конкурентное преимущество по сравнению с собственными Т-клетками пациента, не наделенными анти-CLL1 scCAR. Можно также подавлять или снижать экспрессию гена CD3, придавая устойчивость к теплизумабу, который представляет собой другое иммуносупрессорное средство. Согласно изобретению можно также подавлять или снижать экспрессию HPRT, придавая устойчивость к 6-тиогуанину, цитостатическому агенту, обычно применяемому для химиотерапии, прежде всего для лечения острого лимфобластного лейкоза.
Клетки со сконструированными иммунными контрольными точками
Согласно другому объекту изобретения на иммунные клетки можно дополнительно оказывать воздействие, делая их более активными или ограничивая их истощение, путем инактивации генов, кодирующих белки, которые действуют в качестве «иммунных контрольных точек», выступая в качестве регуляторов активации Т-клеток, таких как гены, выбранные из CTLA4, РРР2СА, РРР2СВ, PTPN6, PTPN22, PDCD1, LAG3, HAVCR2, BTLA, CD160, TIGIT, CD96, CRTAM, LAIR1, SIGLEC7, SIGLEC9, CD244, TNFRSF10B, TNFRSF10A, CASP8, CASP10, CASP3, CASP6, CASP7, FADD, FAS, TGFBRII, TGFBRI, SMAD2, SMAD3, SMAD4, SMAD10, SKI, SKIL, TGIF1, IL10RA, IL10RB, НМОХ2, IL6R, IL6ST, CSK, PAG1, SIT1, FOXP3, PRDM1 (orblimp1), BATF, GUCY1A2, GUCY1A3, GUCY1B2, GUCY1B3, предпочтительно указанный ген представляет собой PDCD1 или CTLA-4. Примеры генов, экспрессию которых можно снижать или подавлять, представлены в таблице 9.
В настоящем изобретении предложены также аллогенные Т-клетки, экспрессирующие анти-CLL1 scCAR, в частности, анти-CLL1, в которых по меньшей мере один ген, экспрессирующий один или несколько компонентов Т-клеточного рецептора (TCR), инактивирован и/или инактивирован один ген, выбранный из следующих генов: CTLA4, РРР2СА, РРР2СВ, PTPN6, PTPN22, PDCD1, LAG3, HAVCR2, BTLA, CD160, TIGIT, CD96, CRTAM, LAIR1, SIGLEC7, SIGLEC9, CD244, TNFRSF10B, TNFRSF10A, CASP8, CASP10, CASP3, CASP6, CASP7, FADD, FAS, TGFBRII, TGFBRI, SMAD2, SMAD3, SMAD4, SMAD10, SKI, SKIL, TGIF1, IL10RA, IL10RB, НМОХ2, IL6R, IL6ST, CSK, PAG1, SIT1, FOXP3, PRDM1 (orblimp1), BATF, GUCY1A2, GUCY1A3, GUCY1B2, GUCY1B3, что описано в WO 2014/184741.
В одном из вариантов осуществления изобретения указанный ген представляет собой ген, который действует в качестве регулятора активации Т-клеток, кодирующий белок бета-2-микроглобулина.
Согласно другому объекту изобретения несущие анти-CLL1 scCAR иммунные клетки, предлагаемые в изобретении, можно подвергать дополнительным манипуляциям, придавая им устойчивость к лекарственному средству, в частности к лекарственному средству, которое применяют при химиотерапии против рака, в частности, рака, опосредуемого экспрессирующими CLL1 клетками, такого как AML. Для достижения этого можно интродуцировать ген, придающий устойчивость к указанному лекарственному средству. Указанный ген можно «включать» и «выключать», используя описанный ранее ген, индуцирующий ингибирование/экспрессию системы (Garcia E.L., Mills A.A. Getting around lethality with inducible Cremediated excision. Semin Cell Dev Biol 13, 2002, сс. 151-158; Lewandoski M., Conditional control of gene expression in the mouse. Nat Rev Genet 2, 2001, сс. 743-755; Scharfenberger L., Hennerici Т., Kirly G. и др., Transgenic mouse technology in skin biology: Generation of complete or tissue-specific knockout mice. J Invest Dermatol 134:e16, 2014; Schwenk F., Kuhn R., Angrand P.O. и др., Temporally and spatially regulated somatic mutagenesis in mice. Nucleic Acids Res 26, 1998, сс. 1427-1432).
Таким образом, объектом настоящего изобретения является экспрессирующая анти-CLL1 scCAR устойчивая к лекарственному средству иммунная клетка, в которой (I) по меньшей мере один ген, экспрессирующий один или несколько компонентов Т-клеточного рецептора (TCR), инактивирован, (II) по меньшей мере один ген, обусловливающий устойчивость к лекарственному средству, встроен или ген, обусловливающий чувствительность к указанному лекарственному средству, удален или изменен в результате мутации для его инактивации, (III) необязательно другой ген, выбранный из генов, указанных в таблице 9, инактивирован.
В настоящем изобретении предложены выделенные несущие анти-CLL1 scCAR иммунные клетки или клеточные линии, которые можно получать способом, предлагаемым в изобретении, более конкретно выделенные клетки, содержащие любые белки, полипептиды, аллельные варианты, измененные или удаленные гены или векторы, представленные в настоящем описании.
Иммунные клетки, предлагаемые в настоящем изобретении, или клеточные линии могут содержать дополнительно экзогенные рекомбинантные полинуклеотиды, в частности, scCAR, или гены суицида, и они могут содержать измененные гены или из них могут быть удалены гены, которые кодируют белки контрольных точек или их лиганды, которые влияют на их эффективность в качестве терапевтического продукта, в идеальном варианте «имеющегося в наличии» продукта. Другим объектом настоящего изобретения является способ лечения или предупреждения рака у пациента, включающий введение по меньшей мере одной сконструированной иммунной клетки, которую можно получать с помощью описанных выше способов.
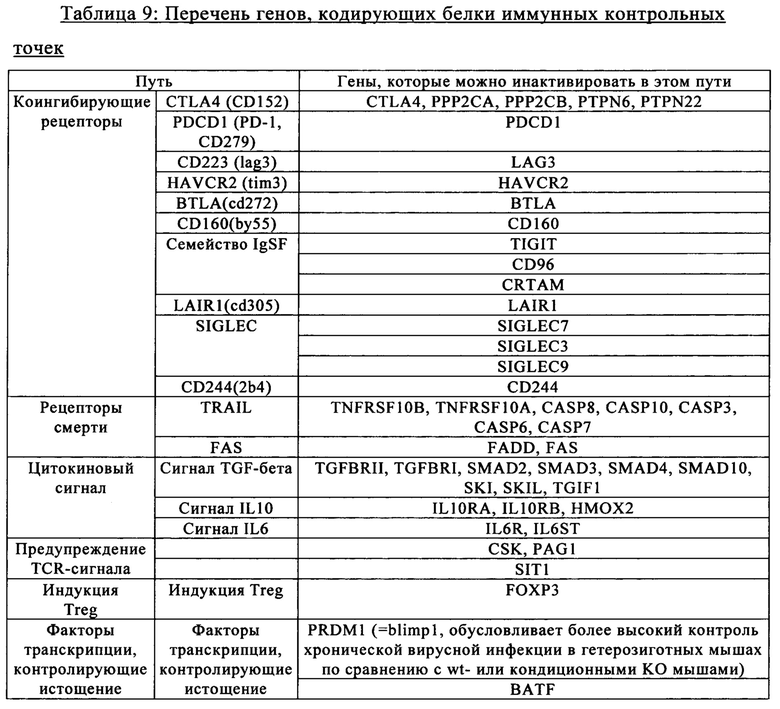

Устойчивые к лекарственному средству CLL1+/luc+-клетки Дауди для тетирования цитотоксичности устойчивых к лекарственному средству аллогенных scCAR-T-клеток
Настоящее изобретения относится также к способу получения клеток-мишеней, которые экспрессируют как рецептор клеточной поверхности, специфический в отношении scCAR Т-клеток, так и ген устойчивости. Указанные клетки-мишени являются особенно ценными для тестирования цитотоксичности scCAR-T-клеток. Эти клетки уже являются устойчивыми к применяемой в клинических условиях дозе клофарабина и обладают люциферазной активностью. Указанная комбинация признаков позволяет отслеживать их на мышиной модели in vivo или при необходимости разрушать их.
Более конкретно, их можно применять для оценки цитотоксических свойств устойчивых к лекарственному средству Т-клеток у мышей в присутствии клофарабина или других PNA. Устойчивые к клофарабину клетки Дауди имитируют физиологическое состояние страдающих острым миелолейкозом (AML) пациентов с рецидивом после индукционной терапии, которые имеют устойчивые к лекарственному средству В-клеточные злокачественные состояния. Таким образом, указанные клетки представляют большой интерес для оценки достоверности и цитотоксичности устойчивых к лекарственному средству scCAR-T-клеток. Предпочтительно эти клетки-мишени представляют собой CLL1+-люцифераза+-клетки Дауди.
Инсерция по меньшей мере одного эпитопа во внеклеточный домен анти-CLL1-одноцепочечного CAR
В случае анти-CLL1 CAR, предлагаемого в изобретении, во внеклеточный домен указанного CAR можно включать посредством инсерции по меньшей мере один эпитоп.Это должно приводить к предварительному истощению иммунных клеток, наделенных CAR, в случае вредных воздействий in vivo, таких как цитокиновый шторм. Кроме того, указанная инсерция эпитопа или «эпитопное мечение» можно применять для сортинга или очистки сконструированных иммунных клеток in vitro в процессе их получения. Указанный по меньшей мере один эпитоп может представлять собой любой антигенный пептид, который является в достаточной степени иммуногенным для того, чтобы связываться специфическим антителом, которое распознает такой пептид. Например, для достижения этого можно встраивать по меньшей мере одну и предпочтительно две копии мимеотопа CD20, предпочтительно имеющего последовательность PYSNPSLCS (SEQ ID NO: 113), в полипептидную последовательность CAR. Далее для простоты принимается следующий порядок расположения scFv, начиная с N-конца к С-концу: VH-цепь и затем VL-цепь. Однако под объем настоящего изобретения подпадает случай с обратным порядком расположения: VL-цепь и затем VH-цепь.
Различные положения по меньшей мере одного мимеотопа CD20 в анти-CLL1 CAR, предлагаемом в изобретении, схематически изображены на фиг. 3. Указанные две копии мимеотопа CD20 можно сшивать друг с другом, а также с VL с помощью линкера. Их можно также встраивать между анти-CLL1 scFv и шарниром (таким как шарнир CD8альфа) с помощью необязательного линкера. Мимеотопы CD20 могут связываться с антителами к CD20, такими как ритуксимаб (McLaughlin P. и др., 1998).
Таким образом, анти-CLL1 CAR, предлагаемый в настоящем изобретении, может содержать VH- и VL-цепи, которые могут связываться с антигеном клеточной поверхности CLL1, необязательно гуманизированным, линкер L, домен суицида, шарнир или его часть, трансмембранный домен, костимуляторный домен и стимуляторный домен.
Согласно другому варианту осуществления изобретения эпитоп представляет собой мимеотоп.В качестве макромалекулы, часто пептида, которая имитирует структуру эпитопа, преимуществом мимеотопа является меньший по сравнению с обычным эпитопом размер, и это является ценным для неконформационной последовательности и повышает простоту воспроизводства в длинном полипептиде, таком как CAR. Мимеотопы известны для некоторых МАт, разрешенных для применения в качестве фармацевтических средств, например, два состоящих из 10 аминокислот пептида для цетуксимаба (Riemer и др., 2005), или 24 ак для паливизумаба (Arbiza и др., 1992). Поскольку эти мимеотопы можно идентифицировать с помощью фагового дисплея, то можно использовать некоторые из них для получения последовательности, которая не нарушают scFv этого же МАт.Кроме того, их применение может повышать комплементзависимую цитотоксичность (CDC).
Некоторые примеры таких эпитопов и мимеотопов в сочетании со связывающими их МАт приведены ниже в таблице 10.
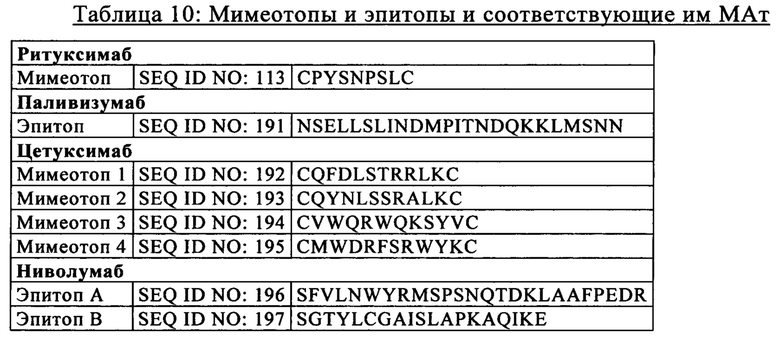
В предпочтительном варианте осуществления изобретения эпитоп, интродуцированный в химерный scFv, представляет собой мимеотоп CD20 (SEQ ID NO: 113), и неслитый МАт, обладающий аффинностью к этому мимеотопу, который применяют для цели(ей) сортировки и/или истощения, представляет собой ритуксимаб.
В одном из вариантов осуществления изобретения указанный по меньшей мере один эпитоп встраивают между VH-и VL-цепями анти-CLL1.1 CAR, который необязательно связывают с указанными VH- и VL-цепями с помощью одного линкера.
В некоторых вариантах осуществления изобретения понятие «линкер» в контексте scFv относится к пептидному линкеру, который состоит из аминокислот, таких как остатки глицина и/или серина, которые используют индивидуально или в комбинации, предназначенному для соединения вариабельной области тяжелой цепи и вариабельной области легкой цепи. В одном из вариантов осуществления изобретения гибкий полипептидный линкер представляет собой глицин/сериновый линкер, и он содержит аминокислотную последовательность (Gly-Gly-Gly-Ser)n или (Gly-Gly-Gly-Gly-Ser)n, в которой n обозначает положительное целое число, равное или превышающее 1. Например, n=1, n=2, n=3, n=4, n=5, n=6, n=7, n=8, n=9 и n=10. В одном из вариантов осуществления изобретения гибкие полипептидные линкеры представляют собой (но не ограничиваясь только ими) (Gly4Ser)4 или (Gly4Ser)3. В другом варианте осуществления изобретения линкеры включают несколько повторов (GlyxSer)n, где х=1, 2, 3, 4 или 5 и n обозначает 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10, например, несколько повторов (GlySer), (Gly2Ser) или (Gly5Ser). Под объем изобретения подпадают также линкеры, описанные в WO 2012/138475, которая включена в настоящее описание в качестве ссылки.
В предпочтительном варианте осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, в которой один мимеотоп CD20 встроен между VH- и VL-цепями анти-CLL1.1 CAR, который необязательно связан с указанными VH- и VL-цепями с помощью одного линкера.
В более предпочтительном варианте осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг.2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета, и в которой встроен один мимеотоп CD20 между VH- и VL-цепями анти-CLL1.1 CAR, который необязательно связан с указанными VH- и VL-цепями с помощью одного линкера.
В другом варианте осуществления изобретения указанный по меньшей мере один эпитоп встроен на N-конец CAR - т.е. перед scFv-, который необязательно связан с VH-цепью и с N-концом CAR с помощью одного линкера.
В предпочтительном варианте осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный домен из 4-1ВВ, сигнальный домен CD3дзeта, и в которой один эпитоп встроен на N-конец CAR -т.е. перед scFv-, который необязательно связан с VH-цепью и с N-концом CAR с помощью одного линкера.
В более предпочтительном варианте осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг.2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8a, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета, и в которой один эпитоп встроен на N-конец CAR, т.е. перед scFv-, который необязательно связан с VH-цепью и с N-концом CAR с помощью одного линкера.
В другом варианте осуществления изобретения указанный по меньшей мере один эпитоп встроен между scFv-фрагментами и шарниром CAR, который необязательно связан с VL-цепью и с шарниром с помощью одного линкера.
В предпочтительном варианте осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8a, костимуляторный домен из 4-1ВВ, сигнальный домен CD3дзета, и в которой один эпитоп встроен между scFv-фрагментами и шарниром CAR, который необязательно связан с VL-цепью и с шарниром с помощью одного линкера.
В более предпочтительном варианте осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета, и в которой один эпитоп встроен между scFv-фрагментами и шарниром CAR, который необязательно связан с VL-цепью и с шарниром с помощью одного линкера.
В предпочтительном варианте осуществления изобретения по меньшей мере два эпитопа встроены во внеклеточный домен анти-CLL1 CAR, предлагаемого в настоящем изобретении.
В одном из вариантов осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета и два мимеотопа CD20, где
указанный внеклеточный связывающий домен содержит VH- и VL-цепи, направленные против CLL1, и шарнир FcγRIIIα или CD8α, или IgG1;
указанные два эпитопа встроены в виде тандема между scFv и указанным шарниром,
и необязательно линкер (SEQ ID NO: 10) помещен между двумя эпитопами и/или между VH и двумя эпитопами.
В одном из вариантов осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен CD3дзeта и два мимеотопа CD20, где
указанный внеклеточный связывающий домен содержит VH- и VL-цепи, направленные против CLL1, и шарнир FcyRIIIa или CD8α, или IgG1;
указанные два эпитопа встроены в виде тандема перед scFv, т.е. на N-конце CAR,
и необязательно линкер (SEQ ID NO: 10) помещен между двумя эпитопами и/или на N-конце CAR.
Согласно одному из вариантов осуществления изобретения по меньшей мере два эпитопа встроены во внеклеточный домен таким образом, чтобы VH был локализован между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В более предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный домен из 4-1ВВ, сигнальный домен CD3 дзета, и в которой два эпитопа встроены во внеклеточный домен таким образом, чтобы VH был локализован между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В более предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен СВ3дзета, и в которой два эпитопа встроены во внеклеточный домен таким образом, чтобы VL был локализован между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
Согласно другому варианту осуществления изобретения два эпитопа встроены во внеклеточный домен таким образом, чтобы VL был локализован между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный домен из 4-1ВВ, сигнальный домен CD3 дзета, и в которой два эпитопа встроены во внеклеточный домен таким образом, чтобы VL был локализован между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В более предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета, и в которой два эпитопа встроены во внеклеточный домен таким образом, чтобы VL был локализован между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
Согласно другому варианту осуществления изобретения указанный CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) содержит внеклеточный связывающий домен, где по меньшей мере два эпитопа встроены во внеклеточный домен таким образом, чтобы VH- и VL-цепи были локализованы между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный домен из 4-1ВВ, сигнальный домен CD3дзета, и в которой два эпитопа встроены во внеклеточный домен таким образом, чтобы VH- и VL-цепи были локализованы между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В более предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен СD3дзета, и в которой два эпитопа встроены во внеклеточный домен таким образом, чтобы VH- и VL-цепи были локализованы между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В другом варианте осуществления изобретения три эпитопа встраивают во внеклеточный домен анти-CLL1 CAR, предлагаемого в настоящем изобретении.
Согласно конкретному варианту осуществления изобретения указанный CLL1-специфический CAR, предлагаемый в изобретении, содержит внеклеточный связывающий домен, где три эпитопа встроены во внеклеточный домен таким образом, чтобы VH- и VL-цепи были локализованы между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный домен из 4-1ВВ, сигнальный домен СD3дзета, и в которой три эпитопа встроены во внеклеточный домен таким образом, чтобы VH- и VL-цепи были локализованы между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В более предпочтительном варианте осуществления изобретения CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур версии V3, проиллюстрированных на фиг.2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8a, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета, и в которой три эпитопа встроены во внеклеточный домен таким образом, чтобы VH- и VL-цепи были локализованы между ними, где все указанные компоненты необязательно разделены с помощью по меньшей мере одного линкера.
В другом варианте осуществления изобретения, CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8α, костимуляторный из домен 4-1ВВ, сигнальный домен CD3 дзета и три эпитопа CD20, где
указанный внеклеточный связывающий домен содержит VH- и VL-цепи, направленные против CLL1, и шарнир FcγRIIIα или CD8α, или IgG1;
указанные три эпитопа встроены в виде тандема между scFv и указанным шарниром,
и необязательно линкер (SEQ ID NO: 10) помещен между тремя эпитопами и/или между VH и тремя эпитопами.
В другом варианте осуществления изобретения, CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR) имеет одну из полипептидных структур, выбранную из V1, V3 или V5, проиллюстрированных на фиг. 2, где указанная структура содержит по меньшей мере анти-CLL1 внеклеточный лигандсвязывающий домен, трансмембранный домен CD8a, костимуляторный домен из 4-1ВВ, сигнальный домен CD3дзета, два эпитопа CD20 и один эпитоп CD34;где
указанный внеклеточный связывающий домен содержит VH- и VL-цепи, направленные против CLL1, и шарнир FcγRIIIα или CD8α, или IgG1;
указанные два эпитопа встроены в виде тандема между scFv и указанным шарниром и
указанный эпитоп CD34 встроен между двумя указанными эпитопами CD20, где все компоненты разделены между собой с помощью линкера (SEQ ID NO: 10) и линкера между эпитопом и между VH и тремя эпитопами.
Все вышеизложенные варианты осуществления изобретения относятся к эпитопсодержащим анти-CLL1 CAR, где VH- и VL-цепи, которые используются в качестве внеклеточного связывающего домена, связываются предпочтительно с человеческим расположенным на мембране CLL1-1.
В предпочтительном варианте осуществления изобретения указанные выше анти-CLL1 CAR содержат по меньшей мере внеклеточный лигандсвязывающий домен, который включает VH- и VL-цепи, полученные из моноклональных антител к CLL1.
Более конкретно в CAR, предлагаемый в настоящем изобретении можно включать следующие варианты эпитопов:
В некоторых вариантах осуществления изобретения внеклеточный связывающий домен содержит по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 МАт-специфических эпитопов.
В некоторых вариантах осуществления изобретения внеклеточный связывающий домен содержит по меньшей мере 1, 2 или 3 МАт-специфических эпитопа.
В некоторых вариантах осуществления изобретения, когда внеклеточный связывающий домен содержит несколько МАт-специфических эпитопов, все МАт-специфические эпитопы являются идентичными.
В некоторых вариантах осуществления изобретения, когда внеклеточный связывающий домен содержит несколько МАт-специфических эпитопов, МАт-специфические эпитопы не являются идентичными. Например, внеклеточный связывающий домен может содержать три МАт-специфических эпитопа, два из которых являются идентичными, а третий отличается от них.
В некоторых вариантах осуществления изобретения внеклеточный связывающий домен содержит VH, VL, один или несколько МАт-специфических эпитопов, предпочтительно 1, 2 или 3, более предпочтительно 2 или 3 МАт-специфических эпитопа.
В некоторых вариантах осуществления изобретения внеклеточный связывающий домен содержит следующую последовательность (N-конец расположен слева):
V1-L1-V2-(L)х-эпитоп1-(L)x;
V1-L1-V2-(L)х-эпитоп1-(L)x-эпитоп2-(L)x;
V1-L1-V2-(L)х-эпитоп1-(L)x-эпитоп2-(L)х-эпитоп3-(L)x;
(L)х-эпитоп1-(L)x-V1-L1-V2;
(L)х-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2;
эпитоп1-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-V1-L1-V2;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x;
(L)x-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2-(L)x-эпитоп3-(L)x;
(L)x-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x;
V1-(L)x-эпитоп1-(L)x-V2;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x;
(L)x-эпитоп1-(L)x-V1-(L)x-эпитоп2-(L)x-V2;
(L)х-эпитоп1-(L)x-V1-(L)х-эпитоп2-(L)х-V2-(L)х-эпитоп3-(L)x;
V1-L1-V2-L-эпитоп1;
V1-L1-V2-L-эпитоп1-L;
V1-L1-V2-L-эпитоп1-L-эпитоп2;
V1-L1-V2-L-эпитоп1-L-эпитоп2-L;
V1-L1-V2-L-эпитоп1-L-эпитоп2-L-эпитоп3;
V1-L1-V2-L-эпитоп1-L-эпитоп2-L-эпитоп3-L;
V1-L1-V2-эпитоп1;
V1-L1-V2-эпитоп1-L;
V1-L1-V2-эпитоп1-эпитоп1-L-эпитоп2;
V1-L1-V2-эпитоп1-эпитоп1-L-эпитоп2-L;
V1-L1-V2-эпитоп1-эпитоп1-L-эпитоп2-L-эпитоп3;
V1-L1-V2-эпитоп1-L-эпитоп2-L-эпитоп3-L;
эпитоп1-V1-L1-V2;
эпитоп1-L-V1-L1-V2;
L-эпитоп1-V1-L1-V2;
L-эпитоп1-L-V1-L1-V2;
эпитоп1-L-эпитоп2-V1-L1-V2;
эпитоп1-L-эпитоп2-L-V1-L1-V2;
L-эпитоп1-L-эпитоп2-V1-L1-V2;
L-эпитоп1-L-эпитоп2-L-V1-L1-V2;
эпитоп1-L-эпитоп2-L-эпитоп3-V1-L1-V2;
эпитоп1-L-эпитоп2-L-эпитоп3-L-V1-L1-V2;
L-эпитоп1-L-эпитоп2-L-эпитоп3-V1-L1-V2;
L-эпитоп1-L-эпитоп2-L- эпитоп3-L-V1-L1-V2;
V1-L-эпитоп1-L-V2;
L-эпитоп1-L-V1-L-эпитоп2-L-V2;
V1-L-эпитоп1-L-V2-L- эпитоп2-L;
V1-L-эпитоп1-L-V2-L-эпитоп2-L-эпитоп3;
V1-L-эпитоп1-L-V2-L-эпитоп2-эпитоп3;
V1-L-эпитоп1-L-V2-L-эпитоп2-L-эпитоп3-эпитоп4;
L-эпитоп1-L-V1-L-эпитоп2-L-V2-L-эпитоп3-L;
эпитоп1-L-V1-L-эпитоп2-L-V2-L-эпитоп3-L;
L-эпитоп1-L-V1-L-эпитоп2-L-V2-L-эпитоп3;
L-эпитоп1-L- V1-L1-V2-L-эпитоп2-L;
L-эпитоп1-L-V1-L1-V2-L-эпитоп2-L-эпитоп 3;
L-эпитоп1-L-V1-L1-V2-L-эпитоп2-эпитоп3 или
эпитоп1-L-V1-L1-V2-L-эпитоп2-L-эпитоп3-эпитоп4,
где
V1 и V2 обозначают VH и VL scFv (т.е. V1 обозначает VL и V2 обозначает VH или V1 обозначает VH и V2 обозначает VL);
L1 обозначает любой линкер, пригодный для соединения VH-цепи с VL-цепью в scFv;
L обозначает линкер, предпочтительно содержащий глициновые и сериновые остатки, и каждый из присутствующих во внеклеточном связывающем домене L может быть идентичен или отличаться от другого L, присутствующего в том же самом внеклеточном связывающем домене, и
х обозначает 0 или 1 и каждый из присутствующих х выбирают независимо от других; и
эпитоп 1, эпитоп 2 и эпитоп 3 представляют собой МАт-специфические эпитопы, и они могут быть идентичными или различными.
В некоторых вариантах осуществления изобретения внеклеточный связывающий домен содержит следующую последовательность (N-конец расположен слева):
VH-L1-VL-L-эпитоп1-L-эпитоп2-L;
L-эпитоп1-L-VH-L- эпитоп2-L-VL-L-эпитоп3-L;
VL-L1-VH-L- эпитоп1-L- эпитоп2-L; или
L-эпитоп1-L-VL-L-эпитоп2-L-VH-L-эпитоп3-L.
где L, L1, эпитоп 1, эпитоп 2 и эпитоп 3 имеют указанные выше значения.
В некоторых вариантах осуществления изобретения L1 обозначает линкер, содержащий глицин и/или серин. В некоторых вариантах осуществления изобретения L1 обозначает линкер, содержащий аминокислотную последовательность (Gly-Gly-Gly-Ser)n или (Gly-Gly-Gly-Gly-Ser)n, где n обозначает 1, 2, 3, 4 или 5. В некоторых вариантах осуществления изобретения L1 обозначает (Gly4Ser)4 или (C1y4Ser)3.
В некоторых вариантах осуществления изобретения L обозначает гибкий линкер, предпочтительно содержащий глицин и/или серин. В некоторых вариантах осуществления изобретения L имеет аминокислотную последовательность, выбранную из SGG, GGS, SGGS, SSGGS, GGGG, SGGGG, GGGGS, SGGGGS, GGGGGS, SGGGGGS, SGGGGG, GSGGGGS, GGGGGGGS, SGGGGGGG, SGGGGGGGS или SGGGGSGGGGS, предпочтительно SGG, SGGS, SSGGS, GGGG, SGGGGS, SGGGGGS, SGGGGG, GSGGGGS или SGGGGSGGGGS. В некоторых вариантах осуществления изобретения, в которых внеклеточный связывающий домен содержит несколько указанных L, все L являются идентичными. В некоторых вариантах осуществления изобретения, в которых внеклеточный связывающий домен содержит несколько указанных L, не все L являются идентичными. В некоторых вариантах осуществления изобретения L обозначает SGGGGS. В некоторых вариантах осуществления изобретения внеклеточный связывающий домен содержит несколько указанных L, и все L представляют собой SGGGGS.
В некоторых вариантах осуществления изобретения эпитоп 1, эпитоп 2 и эпитоп 3 являются идентичными или различными и их выбирают из МАт-специфических эпитопов, которые имеют любую из аминокислотных последовательностей SEQ ID NO: 33-SEQ ID NO: 42.
В некоторых вариантах осуществления изобретения эпитоп 1, эпитоп 2 и эпитоп 3 являются идентичными или различными и их выбирают из МАт-специфических эпитопов, которые специфическим распознаются ибритумомабом, тиоксетаном, муромонабом-CD3, тозитумомабом, абциксимабом, базиликсимабом, брентуксимаб ведотином, цетуксимабом, инфликсимабом, ритуксимабом, алемтузумабом, бевацизумабом, цертолизумаба пеголом, даклизумабом, экулизумабом, эфализумабом, гемтузумабом, натализумабом, омализумабом, паливизумабом, ранибизумабом, тоцилизумабом, трастузумабом, ведолизумабом, адалимумабом, белимумабом, канакинумабом, деносумабом, голимумабом, ипилимумабом, офатумумабом, панитумумабом, QBEND-10, алемтузумабом или устекинумабом.
В некоторых вариантах осуществления изобретения эпитоп 1 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID NO: 33.
В некоторых вариантах осуществления изобретения эпитоп 2 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID NO: 35.
В некоторых вариантах осуществления изобретения эпитоп 3 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID NO: 36.
В некоторых вариантах осуществления изобретения эпитоп 4 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID NO: 37.
В некоторых вариантах осуществления изобретения эпитоп 4 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID NO: 38.
В некоторых вариантах осуществления изобретения эпитоп 2 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID NO: 33, и эпитоп 3 представляет собой МАт-специфический эпитоп, который имеет аминокислотную последовательность SEQ ID N0:41 или 42.
В некоторых вариантах осуществления изобретения один из эпиопов, таких как эпитоп 1, эпитоп 2, эпитоп 3 и эпитоп 4, представляет собой эпитоп CD34, предпочтительно эпитоп, имеющий SEQ ID NO: 41 или 42. В некоторых вариантах осуществления изобретения один из эпитопов, таких как эпитоп 1, эпитоп 2, эпитоп 3 и эпитоп 4, представляет собой эпитоп CD34, предпочтительно эпитоп, имеющий SEQ ID NO: 41 или 42, а другие МАт-специфические эпитопы представляют собой мимеотопы CD20, предпочтительно мимеотоп, имеющий SEQ ID NO: 33.
Способ истощения экспрессирующих CAR иммунных клеток
Иммунные клетки, экспрессирующие CLL1-специфический CAR, предлагаемый в настоящем изобретении, могут содержать эпитоп(ы) во внеклеточном домене, например, описанный(ые) выше, поэтому их можно истощать у пациента в случае неблагоприятного или слишком острого иммунного ответа (например, цитокинового шторма) путем введения указанному пациенту антитела, предпочтительно моноклонального, специфического в отношении указанного(ых) эпитопа(ов).
Под «истощением in vivo» в настоящем изобретении подразумевается введение млекопитающему лекарственного средства, способствующего прекращению пролиферации экспрессирующих CAR иммунных клеток путем ингибирования или элиминации.
Одним из объектов изобретения является способ истощения in vivo сконструированной иммунной клетки, экспрессирующей CAR, который содержит ранее описанный МАт-специфический эпитоп, включающий приведение в контакт указанной сконструированной иммунной клетки или указанной экспрессирующей CAR иммунной клетки по меньшей мере с одним из эпитопспецифических МАт. Другим объектом изобретения является способ истощения in vivo экспрессирующей CAR иммунной клетки, который содержит указанный выше химерный scFv (созданный путем инсерции МАт-специфического эпитопа), включающий приведение в контакт указанной сконструированной иммунной клетки с эпитопспецифическими антителами.
Предпочтительно указанные иммунные клетки представляют собой Т-клетки и/или антитела являются моноклональными.
Согласно одному из вариантов осуществления изобретения истощение in vivo иммунной сконструированной клетки осуществляют, используя сконструированную иммунную клетку, ранее отсортированную с помощью предлагаемого в настоящем изобретении способа in vitro. В этом случае следует с помощью такой же инфузии применять МАт.
Согласно предпочтительному варианту осуществления изобретения специфическим антигеном для МАт является антиген CD20 и эпитопспецифическое МАт представляет собой ритуксимаб.
Некоторыми вариантами осуществления изобретения является способ истощения in vivo описанной выше сконструированной иммунной клетки, экспрессирующей CAR, который содержит МАт-специфический эпитоп (экспрессирующая CAR иммунная клетка), у пациента, включающий приведение в контакт указанной экспрессирующей CAR иммунной клетки по меньшей мере с одним из эпитопспецифических МАт.
В некоторых вариантах осуществления изобретения указанный МАт-специфический эпитоп представляет собой эпитоп или мимеотоп CD20, предпочтительно имеющий SEQ ID NO: 35, и эпитопспецифическое МАт представляет собой ритуксимаб.
В некоторых вариантах осуществления изобретения стадия приведения в контакт указанной сконструированной иммунной клетки или указанной экспрессирующей CAR иммунной клетки по меньшей мере с одним эпитопспецифическим МАт включает введение пациенту путем инфузии эпитопспецифического МАт, предпочтительно ритуксимаба.
В одном из вариантов осуществления изобретения, в котором истощают иммунные клетки, экспрессирующие CAR, который содержит МАт-специфический эпитоп (экспрессирующие CAR иммунные клетки), по данным анализа CDC с использованием специфического в отношении эпитопа МАт количество жизнеспособных экспрессирующих CAR иммунных клеток снижают предпочтительно по меньшей мере на 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80% или 90%. Предпочтительно анализ CDC представляет собой анализ, описанный в примере 3, примере 4 или примере 7.4. В некоторых вариантах осуществления изобретения указанный МАт-специфический эпитоп представляет собой эпитоп или мимеотоп CD20, предпочтительно имеющий SEQ ID NO: 35, и эпитопспецифический МАт представляет собой ритуксимаб.
Помимо обеспечения возможности истощения in vivo иммунных клеток, предлагаемых в изобретении, эпитопы, встроенные во внеклеточный домен CAR могут быть ценными для стадий сортинга или очистки иммунных клеток, экспрессирующих указанные CAR, в качестве стадии способа их получения.
Выделенные клетки
Образовавшиеся клетки представляют собой сконструированные иммунные клетки, экспрессирующие на поверхности клеточной мембраны CLL1-специфический химерный антигенный рецептор, описанный выше, в частности, сконструированные иммунные клетки, полученные из первичных Т-лимфоцитов, необязательно устойчивые к противораковому лекарственному средству и несущие делецию в гене, который кодирует альфа-цепь TCR или бета-цепь TCR.
В настоящем изобретении описана сконструированная иммунная клетка, указанная выше, в которой подавлена экспрессия TCR.
В настоящем изобретении описана сконструированная иммунная клетка, указанная выше, где экспрессия по меньшей мере одного белка ГКГС, предпочтительно β2m или HLA, понижена или подавлена в указанной сконструированной иммунной клетке. β2m обозначает бета 2 микроглобулин, а HLA обозначает человеческий лейкоцитарный антиген. Белок ГКГС представляет собой белок ГКГС класса I или класса II.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, где указанная сконструированная иммунная клетка подвергнута мутации для придания устойчивости по меньшей мере к одному иммуносупрессорному лекарственному средству, химиотерапевтическому лекарственному средству или противораковому лекарственному средству.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, предназначенная для применения в терапии.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где пациент представляет собой человека.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где состояние представляет собой предзлокачественное или злокачественное связанное с раком состояние, отличающееся наличием экспрессирующих CLL1 клеток.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где состояние представляет собой состояние, отличающееся избыточным количеством экспрессирующих CLL1 клеток.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где состояние представляет собой состояние, связанное с гематологическим раком.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где состояние, связанное с гематологическим раком, представляет собой лейкоз или злокачественные лимфопролиферативные нарушения.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где указанный лейкоз выбран из группы, состоящей из острого миелогенного лейкоза, хронического миелогенного лейкоза, мелодиспластического синдрома, острого лимфоидного лейкоза, хронического лимфоидного лейкоза и миелодиспластического синдрома.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где указанный лейкоз представляет собой острый миелогенный лейкоз (AML).
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где указанный гематологический рак представляет собой злокачественное лимфопролиферативное нарушение.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где указанное лимфопролиферативное нарушение представляет собой лимфому.
В настоящем изобретении предложена сконструированная иммунная клетка, описанная выше, которая предназначена для применения в терапии, где указанная лимфома выбрана из группы, состоящей из множественной миеломы, неходжкинской лимфомы, лимфомы Беркитта и фолликулярной лимфомы (мелкоклеточной и крупноклеточной).
В настоящем изобретении предложен способ разрушения клетки гематологического рака, включающий приведение в контакт указанной клетки гематологического рака со сконструированной клеткой, которая экспрессирует по меньшей мере описанный выше анти-CLL1, в количестве, эффективном для разрушения указанной раковой клетки.
В настоящем изобретении предложен способ создания иммунной клетки, включающий:
(а) получение иммунной клетки,
(б) экспрессию на поверхности указанной клетки по меньшей мере одного CLL1-специфического одноцепочечного химерного антигенного рецептора, описанного выше.
В настоящем изобретении предложен способ создания описанной выше иммунной клетки, включающий:
(а) получение иммунной клетки,
(б) интродукцию в указанную клетку по меньшей мере одного полинуклеотида, который кодирует указанный CLL1-специфический одноцепочечный химерный антигенный рецептор,
(в) экспрессию указанного полинуклеотида в указанной клетке.
В настоящем изобретении предложен способ создания описанной выше иммунной клетки, включающий:
(а) получение иммунной клетки,
(б) интродукцию в указанную клетку по меньшей мере одного полинуклеотида, который кодирует указанный CLL1-специфический одноцепочечный химерный антигенный рецептор,
(в) интродукцию по меньшей мере одного другого химерного антигенного рецептора, не специфического в отношении CLL1.
В настоящем изобретении предложен также способ лечения индивидуума, который нуждается в этом, включающий:
(а) получение иммунной клетки, экспрессирующей на поверхности CLL1-специфический одноцепочечный химерный антигенный рецептор, описанный выше,
(в) введение указанных иммунных клеток указанному пациенту.
В настоящем изобретении предложен способ лечения указанного выше индивидуума, нуждающегося в этом, в котором указанную иммунную клетку получают из донора.
В настоящем изобретении предложен способ лечения указанного выше индивидуума, нуждающегося в этом, в котором указанную иммунную клетку получают из организма самого пациента.
Терапевтические применения
В другом варианте осуществления изобретения выделенные клетки, полученные различными способами, или клеточную линию, полученную из указанной выделенной клетки, согласно описанному ранее методу, можно применять в качестве лекарственного средства.
В другом варианте осуществления изобретения указанное лекарственное средство можно применять для лечения рака, прежде всего для лечения лейкоза у пациента, нуждающегося в этом.
В другом варианте осуществления изобретения указанную выделенную клетку, предлагаемую в изобретении, или клеточную линию, полученную из указанной выделенной клетки, можно применять для приготовления лекарственного средства, предназначенного для лечения рака у пациента, нуждающегося в этом.
В конкретном варианте осуществления изобретения предложена экспрессирующая анти-CLL1 CAR Т-клетка в качестве лекарственного средства, предназначенного для лечения AML, подтипа AML, связанного с AML осложнения или связанного с AML состояния.
Согласно другому варианту осуществления изобретения указанное лекарственное средство можно применять для лечения опосредуемого экспрессирующей CLL1 клеткой патологического состояния или состояния, отличающегося прямой или косвенной активностью экспрессирующей CLL1 клетки.
Другой объект настоящего изобретения относится к способам лечения пациентов, нуждающихся в этом, где указанный способ включает по меньшей мере одну из следующих стадий, на которых:
(а) получают иммунную клетку, которую можно создавать с помощью одного из ранее описанных способов;
(б) вводят указанные трансформированные иммунные клетки указанному пациенту.
В одном из вариантов осуществления изобретения указанные Т-клетки можно подвергать интенсивному Т-клеточному размножению in vivo и их можно сохранять в течение продолжительного периода времени.
Указанное лечение может быть облегчающим, терапевтическим или профилактическим. Оно может представлять собой либо составную часть аутологичного иммунотерапевтического лечения, либо составную часть аллогенного иммунотерапевтического лечения. Понятие «аутологичный» означает, что клетки, клеточная линия или популяция клеток, применяемые для лечения пациентов, получают из организма указанного пациента или из организма совместимого по человеческому лейкоцитарному антигену (HLA) донора. Понятие «аллогенный» означает, что клетки или популяция клеток, применяемые для лечения пациентов, получают не из организма указанного пациента, а получают из организма донора.
В предыдущих разделах описаны клетки, которые можно применять в описанных выше способах. Указанное лечение можно применять для лечения пациентов, у которых диагностировано предзлокачественное или злокачественное связанное с раком состояние, отличающееся наличием экспрессирующих CLL1 клеток, прежде всего избыточным количеством экспрессирующих CLL1 клеток. Такие состояния имеют место при гематологических видах рака, таких как лейкоз.
Одним из вариантов осуществления настоящего изобретения является композиция, предназначенная для применения для лечения заболевания, опосредуемого экспрессирующими CLL1 клетками, в частности, опосредуемого экспресирующими CLL1 клетками гематологического рака, указанная композиция содержит указанную экспресирующую анти-CLL1 scCAR Т-клетку, предлагаемую в изобретении.
Любые другие опосредуемые CLL1 или включающие CLL1 злокачественные лимфопролиферативные нарушения, указанные в настоящем описании, можно облегчать с помощью экспрессирующих анти-CLL1 CAR клеток, предлагаемых в настоящем изобретении.
В предпочтительном варианте осуществления изобретения рак, который можно лечить с помощью экспрессирующих анти-CLL1 CAR клеток, предлагаемых в настоящем изобретении, представляет собой лейкоз, заболевание, ассоциированное с лейкозом или с его осложнением. Лейкозы, которые можно лечить с помощью экспрессирующих анти-CLL1 CAR клеток, предлагаемых в настоящем изобретении, могут представлять собой острый миелогенный лейкоз (AML). AML или подтипы AML, которые можно лечить с помощью экспрессирующих анти-CLL1 scCAR клеток, предлагаемых в настоящем изобретении, могут представлять собой, в частности, острый миелобластный лейкоз, минимально-дифференцированный острый миелобластный лейкоз, острый миелобластный лейкоз без созревания (клеток), острый миелобластный лейкоз с созреванием гранулоцитов, промиелоцитарный или острый промиелоцитарный лейкоз (APL), острый миеломоноцитарный лейкоз, миеломоноцитарный лейкоз в сочетании с эозинофилией костного мозга, острый монобластный лейкоз (М5а) или острый моноцитарный лейкоз (M5b), острый эритроидный лейкоз, включая эритролейкоз (М6а) и очень редкий чистый эритроидный лейкоз (M6b), острый мегакариобластный лейкоз, острый базофильный лейкоз, острый панмиелоз с миелофиброзом, в которых участвуют CLL1-позитивные клетки.
Подтипы AML включают также волосатоклеточный лейкоз, лейкоз, позитивный по филадельфийской хромосоме. AML можно классифицировать как AML со специфическим генетическими аномалиями. Классификация основывается на возможности прогнозировать на основе кариотипа ответ на индукционную терапию, риск рецидива, продолжительность жизни.
Таким образом, AML, который можно лечить с помощью экспрессирующих анти-CLL1 CAR клеток, предлагаемых в настоящем изобретении, может представлять собой AML с транслокацией между хромосомами 8 и 21, AML с транслокацией или инверсией в хромосоме 16, AML с транслокацией между хромосомами 9 и 11, APL (M3) с транслокацией между хромосомами 15 и 17, AML с транслокацией между хромосомами 6 и 9, AML с транслокацией или инверсией в хромосоме 3, AML (мегакариобластный) с транслокацией между хромосомами 1 и 22.
Настоящее изобретение является наиболее ценным для лечения AML, ассоциированного с указанными конкретными цитогенетическими маркерами.
В настоящем изобретении предложена также экспрессирующая анти-CLL1 CAR Т-клетка для лечения пациентов со специфическими цитогенетическими субпопуляциями AML, например, пациентов с t(15;17)(q22;q21), идентифицированных с помощью полностью транс-ретиноевой кислоты (ATRA) 16-19, и для лечения пациентов с t(8;21)(q22;q22) или inv(16)(p13q22)/t(16;16)(pl3;q22), идентифицированных с помощью повторяющихся высоких доз цитарабина.
Предпочтительно в настоящем изобретении предложена экспрессирующая анти-CLL1 CAR Т-клетка для лечения пациентов с аберрациям, такими как -5/del(5q), -7, аномалиями 3q или комплексным кариотипом, у которых обнаружены плохие уровни полной ремиссии и выживаемости.
В контексте настоящего описания понятия «терапевтический агент», «химиотерапевтический агент» или «лекарственное средство», или «противораковое лекарственное средство» относятся к медикаментозному препарату, предпочтительно соединению или его производному, которое может оказывать воздействие на раковую клетку, тем самым снижая пролиферативный статус клетки и/или уничтожая клетку. Примерами химиотерапевтических лекарственных средств или «противораковых лекарственных средств» являются (но не ограничиваясь только ими) алкилирующие агенты (например, бусульфан, карбоплатин, хлорамбуцил, цисплатин, циклофосфамид, ифосфамид, мелфалан, мехлорэтамин, оксалиплатин, урамустин, темозоломид, фотемустин), антагонисты метаболизма (например, антиметаболит пуриновых нуклеозидов, такой как клофарабин, флударабин или 2'-дезоксиаденозин, метотрексат (МТХ), 5-фторурацил или его производные, азатиоприн, капецитабин, цитарабин, флоксуридин, фторурацил, гемцитабин, метотрексат, пеметрексед), противоопухолевые антибиотики (например, митомицин, адриамицин, блеомицин, даунорубицин, доксорубицин, эпирубицин, гидроксимочевина, ударубицин, митомицин С, митоксантрон), противоопухолевые агенты растительного происхождения (например, винкристин, виндезин, таксол, винбластин, винорелбин, доцетаксел, паклитаксел), ингибитор топоизомеразы (иринотекан, топотекан, этопозид).
В предпочтительном варианте осуществления изобретения понятия «терапевтический агент», «химиотерапевтическое лекарственное средство» в контексте настоящего описания относятся к соединению или его производному, которое можно применять для лечения рака, в частности, гематопоэтической раковой клетки и более конкретно AML, снижая тем самым пролиферативный статус раковой клетки и/или уничтожая раковую клетку. Примеры химиотерапевтических агентов включают (но не ограничиваясь только ими) арацитин, цитозин-арабинозид, амсакрин, даунорубицин, идарубицин, новантрон, митоксантрон, вепезид, этопозид (VP16), триоксид мышьяка, транс-ретиноевую кислоту, мехлорэтамин, прокарбазин, хлорамбуцил и их комбинации.
В других вариантах осуществления настоящего изобретения клетки, предлагаемые в изобретении, вводят пациенту в сочетании с лекарственным средством (или агентом), выбранным из арацитина, цитозин-арабинозида, амсакрина, даунорубицина, идарубицина, новантрона, митоксантрона, вепезида, этопозида (VP16), триоксида мышьяка, транс-ретиноевой кислоты, цитарабина, антрациклинов, 6-тиогуанина, гидроксимочевины, преднизона и их комбинации.
Указанные агенты могут включать также (но не ограничиваясь только ими) противораковые агенты TRIMETHOTRIXATE™ (TMTX), TEMOZOLOMIDE™, RALTRITREXED™, S-(4-нитробензил)-6-тиоинозин (NBMPR), 6-бензилгуанидин (6-BG), бис-хлорнитрозомочевину (BCNU) и CAMPTOTHECIN™ или терапевтическое производное любого из указанных средств.
В более предпочтительном варианте осуществления изобретения Т-клетку, экспрессирующую анти-CLL1 scCAR, вводят пациенту в комбинации по меньшей мере с одним терапевтическим агентом, выбранным из арацитина, цитозин-арабинозида, амсакрина, даунорубицина, идарубицина, новантрона, митоксантрона, вепезида, этопозида (VP16), триоксида мышьяка, транс-ретиноевой кислоты и их комбинации.
В контексте настоящего описания понятие «клетка, устойчивая или толерантная» к агенту относится к клетке, которую генетически модифицировали так, чтобы клетка размножалась в присутствии агента в количестве, которое ингибирует или предупреждает пролиферацию клетки без модификации.
Группа пациентов
Предпочтительным вариантом осуществления изобретения является лечение лейкоза, в частности, у страдающих AML пациентов, возрастом старше 60 лет, пациентов возрастом моложе 20 лет.
Более предпочтительным вариантом осуществления настоящего изобретения является педиатрическое лечение, в частности педиатрическое лечение AML или ассоциированных с AML заболеваний или осложнений.
Еще более предпочтительным вариантом осуществления изобретения является применение для лечения страдающих AML пациентов с низким, плохим или неблагоприятным статусом, основой для определения которого является прогнозируемая выживаемость, составляющая менее 5 лет. В этой группе пациенты, страдающие AML со следующими цитогенетическими характеристиками: -5; 5q; -7; 7q- ; 11q23; non t(9; 11); inv(3); t(3; 3); t(6; 9); t(9; 22), имеют статус, ассоциированный с риском неблагоприятного исхода (Byrd J.C. и др., Blood, 100 (13), 15 декабря 2002 г.; и для них прежде всего пригодно лечение, предлагаемое в настоящем изобретении, или лечение на основе объекта настоящего изобретения.
В одном из вариантов осуществления изобретения экспрессирующую анти-CLL1 CAR Т-клетку, предлагаемую в настоящем изобретении, можно применять в качестве индукционной терапии, в качестве терапии после ремиссии AML или в качестве консолидирующей терапии для пациентов с AML.
В одном из вариантов осуществления изобретения экспрессирующую анти-CLL1 CAR Т-клетку, предлагаемую в настоящем изобретении, можно применять в случае рецидива AML или в случае рефракторного или устойчивого AML и более предпочтительно в комбинации по меньшей мере с одним другим противораковым лекарственным средством.
В другом предпочтительном варианте осуществления изобретения по меньшей мере одну клетку, экспрессирующую анти-CLL1 CAR, применяют для предупреждения развития раковых клеток, что имеет место, в частности, после противоракового лечения, в процессе деплеции (очистки) костного мозга или перед трансплантацией костного мозга, после разрушения костного мозга.
Связанные с AML осложнения
Одним из предпочтительных вариантов осуществления изобретения является лекарственное средство, которое улучшает состояние здоровья пациента, в частности, пациента с осложнениями, связанными с AML. Более предпочтительно указанная сконструированная экспрессирующая анти-CLL1 CAR Т-клетка, предлагаемая в изобретении, экспрессирует по меньшей мере один анти-CLL1 CAR и ее применяют в качестве лекарственного средства для лечения осложнения, связанного с AML.
Осложнение или заболевание, связанное с AML, может включать предшествующую миелодисплазии фазу, вторичный лейкоз, в частности, вторичный AML, высокое содержание лейкоцитов и отсутствие палочек Ауэра. Среди прочего, в качестве осложнения или заболевания, связанного с AML, рассматриваются лейкостаз и затрагивающий центральную нервную систему (ЦНС) гиперлейкоцитоз, остаточные явления болезни также рассматриваются в качестве осложнения или заболевания, связанного с AML.
Ассоциированные с AML заболевания
Одним из вариантов осуществления настоящего изобретения является также экспрессирующая анти-CLL1 CAR Т-клетка, предназначенная для лечения патологического состояния, связанного с AML.
В настоящем изобретении предложена терапия связанных с AML миелоидных неоплазм, острого миелоидного лейкоза и миелодиспластического синдрома, лечение рецидивирующего или рефракторного острого миелоидного лейкоза, лечение рецидивирующего или рефракторного острого промиелоцитарного лейкоза у взрослых, лечение острого промиелоидного лейкоза, лечение острого миелоидного лейкоза у взрослых возрастом старше 60 лет.
Другим объектом настоящего изобретения является композиция, предназначенная для лечения ассоциированных с AML заболеваний, в частности, гематологических злокачественных связанных с AML заболеваний.
Гематологические злокачественные связанные с AML состояния включают миелодиспластический синдром (MDS, ранее известный как «предлейкоз»), который представляет собой широкий спектр гематологических состояний, объединенных неэффективным производством (или дисплазией) миелоидных клеток крови и риск трансформации в AML.
Другие патологические состояния или генетические синдромы, ассоциированные с риском AML, можно улучшать посредством соответствующего применения настоящего изобретения, указанные генетические синдромы включают синдром Дауна, трисомию, анемию Фанкони, синдром Блума, атаксию-телеангиэктазию, анемию Даймонда-Блекфена, синдром Швахмана-Даймонда, синдром Ли-Фраумени, нейтрофиброматоз типа 1, серьезную врожденную нейтропению (которую называют также синдромом Костмана).
Другие опосредуемые CLL1 патологические состояния
Другим объектом настоящего изобретения является композиция, предназначенная для лечения опосредуемых CLL1+-клеткой заболеваний. Указанные опосредуемые CLL1+-клеткой заболевания включают воспалительные заболевания, такие как ревматоидный артрит.
В частности, настоящее изобретение можно применять для лечения опосредуемых CLL1+-клеткой заболеваний, таких как воспаление и более предпочтительно ревматоидный артрит.
Композиции
Настоящее изобретение относится также к композиции, содержащей сконструированные Т-клетки, предлагаемые в изобретении, для применения в способе лечения заболевания.
Согласно одному из объектов изобретения заболевание представляет собой гематологический рак, в частности рак из стволовых клеток, включая (но не ограничиваясь только ими) лейкоз (такой как острый миелогенный лейкоз (AML)) или связанные с ним осложнения.
В настоящем изобретении предложена также композиция, предназначенная для применения, или способ ингибирования пролиферации или снижения численности популяции экспрессирующих CLL1 клеток или их активности у пациента. Приведенный в качестве примера способ включает приведение в контакт популяции клеток, содержащих экспрессирующую CLL1 клетку, с экспрессирующей анти-CLL1 CAR Т-клеткой, и в частности с scCAR-T-клеткой (scCART), предлагаемой в изобретении, которая связывается с экспрессирующей CLL1 клеткой.
Более конкретным объектом настоящего изобретения является предназначенная для применения композиция или способ ингибирования пролиферации или снижения численности популяции раковых клеток, которые экспрессируют CLL1, у пациента, где способы включают приведение в контакт популяции клеток, содержащей экспрессирующую CLL1 клетку, с экспрессирующей CLL1 CAR Т-клеткой, и в частности с scCART, предлагаемой в изобретении, которая связывается с экспрессирующей CLL1 клеткой, связывание экспрессирующей анти-CLL1 CAR клетки, и в частности scCART, предлагаемой в изобретении, с экспрессирующей CLL1 клеткой приводит к разрушению экспрессирующих CLL1 раковых клеток.
В некоторых объектах изобретения анти-CLL1 CAR Т-клетка, и в частности scCART, предлагаемая в изобретении, снижает уровень, число, количество или процент клеток и/или раковых клеток по меньшей мере на 25%, по меньшей мере на 30%, по меньшей мере на 40%, по меньшей мере на 50%, по меньшей мере на 65%, по меньшей мере на 75%, по меньшей мере на 85%, по меньшей мере на 95% или по меньшей мере на 99% (до неподдающегося обнаружению уровня) у индивидуума или на животной модели миелоидного лейкоза или другого вида рака, ассоциированного с экспрессирующими CLL1 клетками, относительно отрицательного контроля.
В настоящем изобретении предложены также предназначенная для применения композиция или способ предупреждения, лечения и/или контроля нарушения или состояния, ассоциированного с экспрессирующими CLL1 клетками (например, ассоциированного с гематологическим раком), где способы включают введение индивидууму, который нуждается в этом, экспрессирующей анти-CLL1 CAR Т-клетки, и в частности scCART, предлагаемой в изобретении, которая связывается с экспрессирующей CLL1 клеткой. В одном из объектов изобретения индивидуум представляет собой человека. Примерами нарушений, ассоциированных с экспрессирующими CLL1 клетками, являются (но не ограничиваясь только ими) воспалительные нарушения (такие как ревматоидный артрит) и разные виды рака (например, виды гематологического рака, в частности, AML или связанные с AML осложнения).
Настоящее изобретение относится также к предназначенной для применения композиции или способу предупреждения, лечения и/или контроля заболевания, ассоциированного с экспрессирующими CLL1 клетками, где способ включает введение индивидууму, который нуждается в этом, анти-CLL1 CAR Т-клетки, и в частности scCART, предлагаемой в изобретении, которая связывается с экспрессирующей CLL1 клеткой. В одном из объектов изобретения индивидуум представляет собой человека. Примерами заболеваний, ассоциированных с экспрессирующими CLL1 клетками, являются, в частности (но не ограничиваясь только ими), острый миелоидный лейкоз (AML),
Настоящее изобретение относится также к предназначенной для применения композиции или способу лечения или предупреждения рецидива рака, ассоциированного с экспрессирующими CLL1 клетками, где способ включает введение индивидууму, который нуждается в этом, анти-CLL1 CAR Т-клетки, и в частности scCART, предлагаемой в изобретении, которая связывается с экспрессирующей CLL1 клеткой. Согласно другому объекту изобретения способы включают введение индивиууму, который нуждается в этом, в эффективном количестве анти-CLL1 CAR Т-клетки, и в частности scCART, предлагаемой в изобретении, которая связывается с экспрессирующей CLL1 клеткой, в сочетании с применяемой в эффективном количестве другой терапией.
В одном из объектов изобретения CLL1 рассматривается в качестве маркера «раковой стволовой клетки» при AML. Таким образом, анти-CLL1 CAR Т-клетка, и в частности scCART, предлагаемая в изобретении, может предупреждать рецидив AML или даже лечить AML, который главным образом является CLL1-негативным, но имеет популяцию «стволовых» CLL1+-клеток (экспрессирующих CLL1 клеток).
Одним из объектов изобретения являются композиции и способы, предназначенные для лечения индивидуумов, которых подвергают лечению по поводу заболевания или нарушения, ассоциированного с повышенными уровнями экспрессии CD 19, и которые имеют заболевание или нарушение, ассоциированное с повышенными уровнями CLL1.
Лечение с помощью сконструированных иммунных клеток, предлагаемых в изобретении, можно осуществлять в сочетании с применением одного или нескольких типов противораковой терапии, выбранных из группы, включающей терапию на основе антител, химиотерапию, цитокиновую терапию, терапию с использованием дендритных клеток, генную терапию, гормональную терапию, свето-лазерную терапию и лучевую терапию.
Предпочтительно лечение сконструированными иммунными клетками, предлагаемыми в изобретении, можно осуществлять в комбинации (например, до, одновременно или после) с одним или несколькими противораковыми терапевтическими средствами, выбранным из арацитина, цитозин-арабинозида, амсакрина, даунорубицина, идарубицина, новантрона, митоксантрона, вепезида, этопозида (VP16), триоксида мышьяка, транс-ретиноевои кислоты, комбинации триоксида мышьяка, транс-ретиноевой кислоты, мехлорэтамина, прокарбазина, хлорамбуцила и их комбинации.
Согласно предпочтительному варианту осуществления изобретения указанное лечение можно применять для пациентов, подвергающихся иммуносупрессорной терапии. Фактически настоящее изобретение предпочтительно относится к клеткам или популяциям клеток, которым придана устойчивость по меньшей мере к одному иммуноспрессорному агенту в результате инактивации гена, который кодирует рецептор для указанного иммуносупрессорного агента. В этом аспекте иммуноспрессорное лечение должно способствовать отбору и размножению Т-клеток, предлагаемых в изобретении, в организме пациента.
Введение клеток или популяции клеток, предлагаемых в настоящем изобретении, можно осуществлять любым пригодным методом, включая ингаляцию аэрозоля, инъекцию, прием внутрь, трансфузию, имплантацию или трансплантацию. Композиции, представленные в настоящем описании, можно вводить пациенту подкожно, внутрикожно, внутрь опухоли, интранодально, интрамедуллярно, внутримышечно, путем внутривенной или внутрилимфатической инъекции, или внутрибрюшинно. В одном из вариантов осуществления изобретения клеточные композиции, предлагаемые в настоящем изобретении, предпочтительно вводят путем внутривенной инъекции.
Введение клеток или популяции клеток может заключаться во введении 104-109 клеток/кг веса тела, предпочтительно от 105 до 106 клеток/кг веса тела, включая все целочисленные количества клеток в указанных диапазонах. Клетки или популяцию клеток можно вводить в виде одной или нескольких доз. В другом варианте осуществления изобретения указанное эффективное количество клеток вводят в виде однократной дозы. В другом варианте осуществления изобретения указанное эффективное количество клеток вводят в виде более чем одной дозы в течение некоторого периода времени. График введения находится в компетенции лечащего врача и зависит от клинического состояния пациента. Клетки или популяцию клеток можно получать из любого источника, такого как банк крови или донор. Поскольку индивидуальные потребности варьируются, определение оптимальных диапазонов эффективных количеств рассматриваемого типа клеток для конкретного заболевания или состояний, находится в компетенции специалиста в данной области. Эффективное количество представляет собой количество, которое обеспечивает терапевтическое или профилактическое полезное действие. Вводимая доза должна зависеть от возраста, состояния здоровья и веса реципиента, вида одновременно применяемого лечения, если это имеет место, частоты обработки и природы требуемого действия.
В другом варианте осуществления изобретения клетки или композицию, содержащую эти клетки, вводят в указанном эффективном количестве парентерально. Указанное введение может представлять собой внутривенное введение. Указанное введение можно осуществлять непосредственно путем инъекции в опухоль.
В некоторых вариантах осуществления настоящего изобретения клетки вводят пациенту в сочетании с осуществлением (например, до, одновременно или после) любого количества соответствующих форм лечения, включая (но не ограничиваясь только ими) лечение с помощью средств противовирусной терапии, таких как цидофовир и интерлейкин-2, цитарабин (известный также под названием ARA-C), или лечение натализумабом пациентов с MS (рассеянный склероз), или лечение эфализумабом пациентов с псориазом, или другие виды лечения пациентов с PML (прогрессирующая многоочаговая лейкоэнцефалопатия). В других вариантах осуществления изобретения Т-клетки, предлагаемые в изобретении, можно применять в сочетании с химиотерапией, лучевой терапией, иммунодепрессантами, такими как циклоспорин, азатиоприн, метотрексат, микофенолат и FK506, антителами или другими иммунодеструктивными агентами, такими как САМРАТН, антитела к CD3, или с другими видами терапии на основе антител, цитоксином, флударибином, циклоспорином, FK506, рапамицином, микофеноловой кислотой, стероидами, FR901228, цитокинами и облучением. Эти лекарственные средства или ингибируют кальций-зависимую фосфатазу кальцинеурин (циклоспорин и FK506), или ингибируют киназу p70S6, которая важна для индуцируемой фактором роста передачи сигнала (рапамицин) (Henderson, Naya и др., 1991; Liu, Albers и др., 1992; Bierer, Hollander и др., 1993).
В другом варианте осуществления изобретения клеточные композиции, предлагаемые в настоящем изобретении, вводят пациенту в сочетании с (например, до, одновременно или после) трансплантацией костного мозга, Т-клеточной абляционной терапией с использованием либо химиотерапевтических средств, таких как флударабин, либо внешней лучевой терапии (XRT), циклофосфамида, либо антител, таких как OKT3 или САМРАТН.
В другом варианте осуществления изобретения клеточные композиции, предлагаемые в настоящем изобретении, вводят после осуществления В-клеточной абляционной терапии с использованием агентов, которые взаимодействуют с CD20, таких, например, как ритуксан. Например, в одном из вариантов осуществления изобретения индивидуумов можно подвергать стандартной обработке химиотерапевтическими средствами в высокой дозе с последующей трансплантацией стволовых клеток периферической крови. В некоторых вариантах осуществления изобретения после трансплантации индивидуумам вводят путем инфузии размноженные иммунные клетки, предлагаемые в настоящем изобретении. В еще одном варианте осуществления изобретения размноженные клетки вводят до или после хирургического вмешательства.
В некоторых вариантах осуществления настоящего изобретения экспрессирующие анти-CLL1 scCAR клетки водят пациенту в сочетании с (например, до, одновременно или после) лекарственным средством, выбранным из арацитина, цитозин-арабинозида, амсакрина, даунорубицина, идарубицина, новантрона, митоксантрона, вепезида, этопозида (VP16), триоксида мышьяка, транс-ретиноевои кислоты, мехлорэтамина, прокарбазина, хлорамбуцила и их комбинации. В указанных вариантах осуществления изобретения экспрессирующие анти-CLL1 scCAR клетки могут обладать устойчивостью к конкретному лекарственному средству или комбинации лекарственных средств, которое/которые вводят в сочетании с экспрессирующими анти-CLL1 scCAR клетками.
В других вариантах осуществления настоящего изобретения экспрессирующие анти-CLL1 scCAR клетки вводят пациенту в сочетании с лекарственным средством, выбранным из цитарабина, антрациклинов, 6-тиогуанина, гидроксимочевины, преднизона и их комбинации.
Другие определения
- Если специально не указано иное, то множественное число и единственное число и понятие «по меньшей мере один» используются взаимозаменяемо и означают «один» или «более чем один».
- Аминокислотные остатки в полипептидной последовательности обозначают в настоящем описании с помощью однобуквенного кода, согласно которому, например, Q обозначает Gln или остаток глутамина, R обозначает Arg или остаток аргинина и D обозначает Asp или остаток аспарагиновой кислоты.
- Аминокислотная замена обозначает замену одного аминокислотного остатка на другой, например, замена остатка аргинина на остаток глутамина в пептидной последовательности представляет собой аминокислотную замену.
- Нуклеотиды обозначают следующим образом: однобуквенный код используют для обозначения основания нуклеозида: а обозначает аденин, t обозначает тимин, с обозначает цитозин и g обозначает гуанин. Для вырожденных нуклеотидов r обозначает g или а (пуриновые нуклеотиды), k обозначает g или t, s обозначает g или с, w обозначает а или t, m обозначает а или с, у обозначает t или с (пиримидиновые нуклеотиды), d обозначает g, а или t, v обозначает g, а или с, b обозначает g, t или с, h обозначает а, t или с и n обозначает g, a, t или с.
- В контексте настоящего описания понятия «нуклеиновая кислота» или «полинуклеотиды» относятся к нуклеотидам и/или полинуклеотидам, таким как дезоксирибонуклеиновая кислота (ДНК) или рибонуклеиновая кислота (РНК), олигонуклеотидам, фрагментам, созданным с помощью полимеразной цепной реакции (ПЦР), и фрагментам, созданным путем лигирования, расщепления, обработки эндонуклеазами и обработки экзонуклеазами. Молекулы нуклеиновой кислоты могут состоять из мономеров, которые являются встречающимися в естественных условиях нуклеотидами (такими как ДНК или РНК) или аналогами встречающихся в естественных условиях нуклеотидов (например, энантиомерными формами встречающихся в естественных условиях нуклеотидов), или представляют собой их комбинацию. Модифицированные нуклеотиды могут иметь изменения в сахарных фрагментах и/или в фрагментах, содержащих пиримидиновые или пуриновые основания. Модификации сахаров включают, например, замену одной или нескольких гидроксильных групп на галогены, алкильные группы, амины и азидогруппы, или сахара могут быть функционализированы до простых или сложных эфиров. Кроме того, весь сахарный фрагмент может быть заменен стерически и электронно сходными структурами, такими как аза-сахара и карбоциклические аналоги сахаров. Примерами модификаций во фрагменте, представляющем собой основание, могут служить алкилированные пурины и пиримидины, ацилированные пурины или пиримидины или другие хорошо известные гетероциклические заместители. Мономеры нуклеиновой кислоты могут быть сшиты фосфодиэфирными связями или аналогами таких связей. Нуклеиновые кислоты могут быть одноцепочечными или двухцепочечными.
- Под «вектором для доставки» или «векторами для доставки» понимают любой доставляющий вектор, который можно применять согласно настоящему изобретению для приведения в контакт с клеткой (т.е. для «контактирования») или доставки внутрь клетки или в субклеточные компартменты (т.е. для «интродукции») агентов/химических веществ и молекул (белков или нуклеиновых кислот), требующихся для осуществления настоящего изобретения. К ним относятся (но не ограничиваясь только ими) липосомальные векторы для доставки, вирусные векторы для доставки, векторы для доставки лекарственного средства, химические носители, полимерные носители, липоплексы, полиплексы, дендримеры, микропузырьки (ультразвуковые контрастные агенты), наночастицы, эмульсии или другие пригодные векторы для переноса. Такие векторы для доставки позволяют доставлять молекулы, химические вещества, макромолекулы (гены, белки) или другие векторы, такие как плазмиды, пептиды, разработанные фирмой Diatos. В этих случаях векторы для доставки представляют собой молекулы-носители. Под «вектором для доставки» или «векторами для доставки» понимают также методы доставки, предназначенные для осуществления трансфекции.
- Понятия «вектор» или «векторы» относятся к молекуле нуклеиновой кислоты, обладающей способностью транспортировать другую нуклеиновую кислоту, с которой она сцеплена. К «векторам», применяемым согласно настоящему изобретению, относятся (но не ограничиваясь только ими) вирусный вектор, плазмида, РНКовый вектор или молекула линейной или кольцевой ДНК или РНК, которые могут состоять из хромосомальных, нехромосомальных, полусинтетических или синтетических нуклеиновых кислот.Предпочтительными векторами являются векторы, которые способны автономно реплицировать (эписомальный вектор) и/или экспрессировать нуклеиновые кислоты, с которыми они сцеплены (экспрессионные векторы). Специалистам в данной области известно большое количество пригодных векторов, которые поступают в продажу.
К вирусным векторам относятся ретровирус, аденовирус, парвовирус (например, аденоассоциированный вирусы), коронавирус, РНК-вирусы с негативной цепью, такие как ортомиксовирус (например, вирус гриппа), рабдовирус (например, вирус бешенства и вирус везикулярного стоматита), парамиксовирус (например, вирус кори и вирус Сендай), РНК-вирусы с позитивной цепью, такие как пикорнавирус и альфавирус, и вирусы с двухцепочечной ДНК, включая аденовирус, вирус герпеса (например, вирус герпеса простого типов 1 и 2, вирус Эпштейна-Барра, цитомегаловирус) и вирус оспы (например, вирус коровьей оспы, вирус оспы птиц и вирус оспы канарейки). Другие вирусы включают, например, вирус Норвалк, тогавирус, флавивирус, реовирусы, паповирус, гепаднавирус и вирус гепатита. Примерами ретровирусов являются: вирус птичьего лейкоза-саркомы, вирусы млекопитающих С-типа, В-типа, вирусы D-типа, группа HTLV-BLV, лентивирус, спумавирус (Coffin J.M., Retroviridae: The viruses and their replication, в: Fundamental Virology, 3-е изд., под ред. В.N. Fields и др., изд-во Lippincott-Raven Publishers, Philadelphia, 1996).
- Под «лентивирусным вектором» подразумевают лентивирусные векторы на основе ВИЧ, которые являются очень перспективными с точки зрения доставки гена благодаря их относительно большой способности к упаковке, пониженной иммуногенности и их способности стабильно трансдуцировать с высокой эффективностью большой спектр различных типов клеток. Лентивирусные векторы, как правило, создают посредством кратковременной трансфекции клеток-продуцентов тремя (упаковывающая плазмида, оболочечная плазмида и плазмида переноса) или большим количеством плазмид. Подобно ВИЧ, лентивирусные векторы проникают в клетку-мишень посредством взаимодействия гликопротеинов вирусной поверхности с рецепторами на клеточной поверхности. После проникновения вирусная РНК подвергается обратной транскрипции, опосредуемой комплексом вирусной обратной транскриптазы. Продуктом обратной транскрипции является двухцепочечная линейная вирусная ДНК, которая служит субстратом для вирусной интеграции в ДНК инфицированных клеток. Понятие «интегрирующие лентивирусные векторы (или LV)» обозначает, например (но не ограничиваясь только ими), такие векторы, которые способны интегрироваться в геном клетки-мишени. В противоположность этому, понятие «неинтегрирующие лентивирусные векторы (или NILV)» обозначает эффективные векторы для доставки генов, которые не интегрируются в геном клетки-мишени под действием вирусной интегразы.
- Векторы для доставки и векторы можно объединять или комбинировать с любыми методами клеточной пермеабилизации, такими как сонопорация или электропорация, или вариантами этих методов.
- Под клеткой или клетками подразумевают любые эукариотические живые клетки, первичные клетки и клеточные линии, полученные из указанных организмов для получения культур in vitro.
- Под «первичной клеткой» или «первичными клетками» подразумевают клетки, взятые непосредственно из живой ткани (т.е. материал, полученный путем биопсии) и подготовленные для выращивания in vitro, в популяции которых имело место лишь небольшое количество удвоений, и следовательно они являются более репрезентативными с точки зрения основных функциональных компонентов и характеристик тканей, из которых они происходят, по сравнению с непрерывно поддерживаемыми или искусственно иммортализованными онкогенными клеточными линиями.
Примерами таких клеточных линий могут являться (но не ограничиваясь только ими) клеточные линии, выбранные из группы, состоящей из клеток линии СНО-K1; клеток линии HEK293; клеток линии Сасо2; клеток линии U2-OS; клеток линии NIH 3Т3; клеток линии NSO; клеток линии SP2; клеток линии CHO-S; клеток линии DG44; клеток линии K-562, клеток линии U-937; клеток линии MRC5; клеток линии IMR90; клеток линии Jurkat; клеток линии HepG2; клеток линии HeLa; клеток линии НТ-1080; клеток линии НСТ-116; клеток линии Hu-h7; клеток линии Huvec; клеток линии Molt 4.
Все эти клеточные линии можно модифицировать с помощью способа, предлагаемого в настоящем изобретении, создавая модели клеточных линий для получения, экспрессии, количественной оценки, обнаружения, изучения представляющего интерес гена или белка; эти модели можно применять также для скрининга представляющих интерес биологически активных молекул для исследовательских и промышленных целей в различных областях, например, таких как (но не ограничиваясь только ими) химия, биотопливо, терапевтические средства и агрономия.
- Под «мутацией» подразумевают замену, делецию или инсерцию одного/одной, двух, трех, четырех, пяти, шести, семи, восьми, девяти, десяти, одиннадцати, двенадцати, тринадцати, четырнадцати, пятнадцати, двадцати, двадцати пяти, тридцати, сорока, пятидесяти или большего количества нуклеотидов/аминокислот в полинуклеотидной (кДНК, ген) или полипептидной последовательности. Мутация может влиять на кодирующую последовательность гена или его регуляторную последовательность. Она может влиять также на структуру геномной последовательности или структуру/стабильность кодируемой мРНК.
- Под «вариантом(ами)» подразумевают вариант повтора, вариант, ДНК-связывающий вариант, вариант TALE-нуклеазы, вариант полипептида, полученный в результате мутации или замены по меньшей мере одного остатка в аминокислотной последовательности родительской молекулы.
- Под «функциональным вариантом» подразумевают обладающий каталитической активностью мутант белка или домена белка; такой мутант может обладать такой же активностью, что и родительский белок или домен белка, или дополнительными свойствами, или более высокой или более низкой активностью.
- Понятие «идентичность» относится к идентичности последовательностей двух молекул нуклеиновых кислот или полипептидов. Идентичность можно оценивать путем сравнения положений в каждой из последовательностей, которые можно выравнивать для целей сравнения. Если положение в сравниваемой последовательности занято тем же самым основанием, то молекулы являются идентичными в этом положении. Степень сходства или идентичности между двумя нуклеотидными или аминокислотными последовательностями является функцией количества идентичных или совпадающих нуклеотидов в положениях нуклеотидных последовательностей. Для расчета идентичности двух последовательностей можно использовать различные алгоритмы и/или программы сравнительного анализа первичной структуры последовательностей, включая FASTA или BLAST, которые доступны в виде компонента пакета программ анализа последовательностей GCG (Университет Висконсина, Мэдисон, шт. Висконсин), и их можно применять, например, с использованием задаваемых по умолчанию параметров. Например, можно рассматривать полипептиды, обладающие идентичностью, составляющей по меньшей мере 70%, 85%, 90%, 95%, 98% или 99%, с конкретными полипептидами, представленными в настоящем описании, и предпочтительно обладающие практически такими же функциями, а также полинуклеотиды, кодирующие такие полипептиды.
- Понятие «сходство» характеризует степень родства между аминокислотными последовательностями двух или большего количества полипептидов. BLASTP можно применять также для идентификации аминокислотной последовательности, идентичной по меньшей мере на 70%, 75%, 80%, 85%, 87,5%, 90%, 92,5%, 95%, 97,5%, 98%, 99% аминокислотной референс-последовательности, с использованием матриц сходства, таких как BLOSUM45, BLOSUM62 или BLOSUM80. Если специально не указано иное, то указанные в описании баллы сходства получены с использованием BLOSUM62. При использовании BLASTP процент сходства основан на рассчитанном программой BLASTP балле положительных оценок, а процент идентичности последовательностей основан на рассчитанном BLASTP балле идентичности. Рассчитанные с помощью BLASTP «идентичности» представляют собой количество и пропорцию идентичных остатков относительно общего количества остатков в парах последовательностей с высокими баллами; а рассчитанные с помощью BLASTP «положительные оценки» представляют собой количество и пропорцию остатков, для которых полученные при сравнительном анализе первичной структуры последовательностей баллы, являются положительными, и которые сходны друг с другом. В настоящей заявке предложены и подпадают под объем изобретения аминокислотные последовательности, имеющие указанные степени идентичности или сходства, или любую промежуточную степень идентичности или сходства с представленными в настоящем описании аминокислотными последовательностями. Полинуклеотидные последовательности сходных полипептидов выводят с использованием генетического кода и их можно получать с помощью общепринятых методов. Например, функциональный вариант рТальфа может иметь последовательность, сходную на 70%, 75%, 80%, 85%, 87,5%, 90%, 92,5%, 95%, 97.5%, 98%, 99% аминокислотной последовательности SEQ ID NO: 107, описанной в WO 2013/176916. Полинуклеотид, кодирующий такой функциональный вариант, можно получать путем обратной трансляции его аминокислотной последовательности с использованием генетического кода.
В контексте настоящего описания понятие «индивидуум» или «пациент» включает всех представителей царства животных, в том числе приматов кроме человека и человека.
Понятие «наличие рецидива» относится к ситуации, когда у индивидуума или млекопитающего, у которого наблюдалась ремиссия рака после терапии, вновь появляются раковые клетки.
Понятие «рефракторая или устойчивая» относится к ситуации, когда у индивидуума или млекопитающего даже после интенсивного лечения в организме присутствуют оставшиеся раковые клетки.
Понятие «устойчивость к лекарственному средству» относится к состоянию, когда заболевание не реагирует на лечение лекарственным средством или лекарственными средствами. Устойчивость к лекарственному средству может быть либо врожденная (или первичная устойчивость), это означает, что заболевание никогда не обладало чувствительностью к лекарственному средству или лекарственным средствам, либо может быть приобретенной, это означает, что заболевание перестало реагировать на лекарственное средство или лекарственные средства, к которому(ым) оно ранее обладало чувствительностью (вторичная устойчивость). В некоторых вариантах осуществления изобретения устойчивость к лекарственному средству является врожденной. В некоторых вариантах осуществления изобретения устойчивость к лекарственному средству является приобретенной.
Понятие «гематологическое злокачественное заболевание» или «гематологический рак» относится к раку крови - костного мозга и/или лимфатической ткани организма. Примеры гематологических злокачественных заболеваний включают, например, острый миелоидный лейкоз (AML), AML с трехлинейной миелодисплазией (AML/TMDS), смешанную линию лейкоза (миелоидная/лимфоидная) (MLL) и другие связанные с AM патологии.
Понятие «лейкоз» относится к злокачественным неоплазмам кроветворных тканей, включая, в частности, острый миелоидный лейкоз или острый миелогенный лейкоз (AML).
В представленном выше описании изобретения изложены подход и способ его осуществления и применения, так что любой специалист в данной области способен осуществлять и применять его, эта возможность относится, прежде всего, к сущности изобретения, изложенной в прилагаемой формуле изобретения, которая представляет собой составную часть исходного описания.
Если в настоящем описании указаны численные пределы или численный диапазон, то они включают их границы. Кроме того, считается, что включены все значения и поддиапазоны, находящиеся в указанных пределах или указанном численном диапазоне, как если бы они были специально указаны.
Приведенное выше описание представлено для того, чтобы дать возможность специалисту в данной области осуществлять и применять изобретение, и оно изложено в контексте конкретного применения и требуемых для этого условий. Специалистам в данной области должны быть очевидны различные модификации предпочтительных вариантов осуществления изобретения, и общие принципы, указанные в нем могут быть применены для других вариантов осуществления изобретения и применений без отклонения от сущности и объема изобретения. Таким образом, не следует считать, что настоящее изобретение ограничено представленными вариантами осуществления изобретения, напротив, оно соответствует наиболее широкому объему, согласующемуся с принципами и отличительными признаками, указанными в настоящем описании.
Имея общее описание настоящего изобретения, можно достичь более полного понимания после ознакомления с некоторыми конкретными примерами, которые представлены в настоящем описании только для целей иллюстрации и не направлены на ограничение объема, если специально не указано иное.
Примеры
Общие методы
Культуры первичных Т-клеток
Т-клетки очищали из образцов лейкоцитарных пленок, полученных на фирме EFS (Etablissement Francais du Sang, Париж, Франция), используя среду с градиентом плотности фиколла. Выделяли слой РВМС и Т-клетки очищали, используя поступающий в продажу набор для обогащения Т-клеток. Очищенные Т-клетки активировали в среде X-Vivo™-15 (фирма Lonza), дополненной человеческим IL-2 в концентрации 20 нг/мл, человеческой сывороткой в концентрации 5%, и используя гранулы Dynabeads® с активатором человеческих Т-клеток CD3/CD28 при соотношении гранулы:клетки 1:1 (фирма Life Technologies).
Трансфекция мРНК scCAR
Трансфекции осуществляли в день 4 или день 11 после очистки и активации Т-клеток. 5 миллионов клеток трансфектировали 15 мкг мРНК, кодирующих различные конструкции scCAR. мРНК scCAR получали, применяя трансфекции с использованием мРНК-полимеразы Т7 с помощью технологии Cytopulse путем применения двух импульсов по 0,1 мс при 3000 В/см с последующими 4 импульсами по 0,2 мс при 325 В/см в кюветах с расстоянием между электродами 0,4 см в конечном объеме 200 мкл буфера Т для цитопорации («Cytoporation buffer Т») (устройство ВТХ Harvard). Клетки немедленно разводили в среде X-Vivo™-15 (фирма Lonza) и инкубировали при 37°С в атмосфере с 5% СО2. IL-2 добавляли через 2 ч после электропорации в концентрации 20 нг/мл.
Анализ дегрануляции (мобилизация CD107a)
Т-клетки инкубировали в 96-луночных планшетах (40000 клеток/лунку) вместе с равным количеством клеток, экспрессирующих различные уровни белка CLL1. Совместные культуры поддерживали в конечном объеме 100 мкл среды Х-Vivo™-15 (фирма Lonza) в течение 6 ч при 37°С в атмосфере с 5% СО2. Окрашивание CD107a осуществляли в процессе стимуляции клеток, добавляя флуоресцентное антитело к CD107a в начале совместного культивирования, вместе с антителом к CD49d в концентрации 1 мкг/мл, антителом к CD28 в концентрации 1 мкг/мл, и 1-кратным раствором монензина. После периода инкубации, составляющего 6 ч, клетки окрашивали фиксирующим витальным красителем и конъюгированным с флуорохромом антителом к CD8 и анализировали с помощью проточной цитометрии. Дегранулирующую активность определяли в виде % CD8+/CD107a+-клеток и на основе определения средней интенсивности сигнала флуоресцении (MFI) для окрашивания CD107a в популяции CD8+-клеток. Анализы дегрануляции осуществляли через 24 ч после трансфекции мРНК.
Анализ высвобождения IFN гамма
Т-клетки инкубировали в 96-луночных планшетах (40000 клеток/лунку) вместе с клеточными линиями, экспрессирующими различные уровни белка CLL1. Совместные культуры поддерживали в конечном объеме 100 мкл среды X-Vivo™-15 (фирма Lonza) в течение 24 ч при 37°С в атмосфере с 5% СО2. После указанного периода инкубации планшеты центрифугировали при 1500 об/мин в течение 5 мин и супернатанты переносили в новый планшет.Обнаружение IFN гамма в супернатантах клеточных культур осуществляли с помощью ELISA-анализа. Анализы высвобождения IFN гамма осуществляли в начале совместного культивирования клеток через 24 ч после трансфекции мРНК.
Анализ цитотоксичности
Т-клетки инкубировали в 96-луночных планшетах (100000 клеток/лунку) вместе с 10000 клеток-мишеней (экспрессирующих CLL1) и 10000 контрольных (CLL1neg) клеток в одной и той же лунке. Клетки-мишени и контрольные клетки метили флуоресцентными внутриклеточными красителями (CFSE или Cell Trace Violet) перед их совместным культивированием с scCAR+-Т-клетками. Совместные культуры инкубировали в течение 4 ч при 37°С в атмосфере с 5% СО2. После указанного периода инкубации клетки метили фиксирующим витальным красителем и анализировали с помощью проточной цитометрии. Определяли жизнеспособность каждой клеточной популяции (клетки-мишени или контрольные CLL1neg-клетки) и рассчитывали % удельного клеточного лизиса. Анализы цитотоксичности осуществляли через 48 ч после трансфекции мРНК.
Трансдукция Т-клеток
Трансдукцию Т-клеток рекомбинантными лентивирусными векторами, экспрссирующими scCAR, осуществляли через 3 дня после очистки/активации Т-клеток. Обнаружение scCAR на поверхности Т-клеток осуществляли, используя рекомбинантный белок, который состоит из белка внеклеточного домена человеческого белка CLL1, слитого с Fc-фрагментом мышиного IgG1. Связывание указанного белка с молекулой scCAR определяли с помощью конъюгированного с флуорохромом вторичного антитела, мишенью которого является белок мышиного Fc-фрагмента, и анализировали с помощью проточной цитомерии.
Мышиная модель для оценки противоопухолевого действия
Мышам с иммунодефицитом линии NOG инъецировали внутривенно (iv) клетки, экспрессирующие CLL1_MOLM13-люциферазу, в качестве мышиной модели ксенотрансплантата AML. Необязательно мышей подвергали противоопухолевой обработке. Затем мышам инъецировали iv (например, через 2 или 7 дней после инъекции линии опухолевых клеток) scCAR+-Т-клетки в тестируемых различных дозах или Т-клетки, не трансдуированные несущим scCAR лентивирусным вектором. Биолюминисцентые сигналы определяли в день инъекции Т-клеток (D0), в D7, 14, 21, 28 и 40 после инъекции Т-клеток, для отслеживания развития опухолей у различных животных.
Пример 1: Пролиферация клеток с инактивированным ТСRальфа, экспрессирующих CLL1-scCAR
Конструировали и получали гетеродимерную TALE-нуклеазу, нацеленную на две последовательности длиной 17 пар оснований (которые обозначали как полумишени), разделенные состоящим из 15 пар оснований спейсером в гене константной области альфа-цепи Т-клеточного рецептора (TRAC). Каждая из полумишеней распознавалась повторами половин TALE-нуклеаз, представленными в таблице 10.

Каждую конструкцию TALE-нуклеазы субклонировали с использованием расщепления рестриктазами в экспрессионном векторе для клеток млекопитающих под контролем промотора Т7. мРНК, кодирующую TALE-нуклеазу, которая расщепляла геномную последовательность TRAC, синтезировали с использованием плазмиды, несущей кодирующую последовательность, расположенную в прямом направлении относительно промотора Т7.
Очищенные Т-клетки, предварительно активированные в течение 72 ч покрытыми антителом к CD3/CD28 гранулами, трансфектировали каждой из 2 мРНК, кодирующих обе половины TRAC_T01 TALE-нуклеаз. Через 48 ч после трансфекции различные группы Т-клеток из одного и того же донора соответственно трансдуцировали лентивирусным вектором, кодирующим один из ранее описанных анти-CLL1 scCAR (SEQ ID NO: 35-112). Через 2 для после трансдукции СD3NEG-клетки очищали с использованием покрытых антителом к CD3 магнитных гранул и через 5 дней после трансдукции клетки реактивировали растворимым антителом к CD28 (5 мкг/мл).
Мониторинг клеточной пролиферации осуществляли в течение периода времени вплоть до 30 дней после реактивации, подсчитывая количество клеток 2 раза в неделю. Обнаружено увеличение пролиферации клеток с инактивированным ТСRальфа, экспрессирующих CLL1 scCAR, прежде всего в том случае, когда их реактивировали антителом к CD28, по сравнению с нетрансдуцированными клетками.
Для изучения вопроса о том, находятся ли человеческие Т-клетки, экспрессирующие CLL1 scCAR, в активированном состоянии, анализировали экспрессию маркера активации CD25 с помощью FACS через 7 дней после трансдукции. Очищенные клетки, трансдуцированные лентивирусным вектором, кодирующим CLL1 scCAR, анализировали в отношении экспрессии CD25 на их поверхности для оценки их активации по сравнению с нетрансдуцированными клетками. Повышенный уровень экспрессии CD25 обнаружен как в условиях после реактивации антителом к CD28, так и в условиях без реактивации.
Пример 2: Конструирование CLL1 scCAR с использованием фрагментов различных антител к CLL1
Культуры первичных Т-клеток
Т-клетки очищали из образцов лейкоцитарных пленок, полученных на фирме EFS (Etablissement Francais du Sang, Париж, Франция), используя среду с градиентом плотности фиколла (среда Ficoll Paque PLUS/фирма GE Healthcare Life Sciences). Выделяли слой РВМС и Т-клетки очищали, используя поступающий в продажу набор для обогащения Т-клеток (фирма Stem Cell Technologies). Очищенные Т-клетки активировали в среде X-Vivo™-15 (фирма Lonza), дополненной человеческим IL-2 в концентрации 20 нг/мл (фирма Miltenyi Biotech), человеческой сывороткой в концентрации 5% (фирма Sera Laboratories) и гранулами Dynabeads® с активатором человеческих Т-клеток CD3/CD28 при соотношении гранулы: клетки 1:1 (фирма Life Technologies). После активации клетки выращивали и поддерживали в среде X-Vivo™-15 (фирма Lonza), дополненной человеческим IL-2 в концентрации 20 нг/мл (фирма Miltenyi Biotech) и человеческой сывороткой в концентрации 5% (фирма Sera Laboratories).
Трансфекция мРНК scCAR
Трансфекции осуществляли в день 4 или день 11 после очистки и активации Т-клеток. 5 миллионов клеток трансфектировали 15 мкг мРНК, кодирующих различные конструкции scCAR. мРНК scCAR получали, применяя набор mMESSAGE mMACHINE T7 (фирма Life Technologies) и очищали, используя вращающиеся колонки RNeasy Mini (фирма Qiagen). Трансфекции осуществляли, используя технологию Cytopulse, применяя два импульса по 0,1 мс при 3000 В/см с последующими 4 импульсами по 0,2 мс при 325 В/см в кюветах с расстоянием между электродами 0,4 см в конечном объеме 200 мкл буфера Т для цитопорации («Cytoporation buffer Т») (фирма ВТХ Harvard). Клетки немедленно разводили в среде X-Vivo™-15 (фирма Lonza) и инкубировали при 37°С в атмосфере с 5% СО2. IL-2 (фирма Miltenyi Biotec) добавляли через 2 ч после электропорации в концентрации 20 нг/мл.
Анализ дегрануляции (мобилизация СР107а)
Т-клетки инкубировали в 96-луночных планшетах (40000 клеток/лунку) вместе с равным количеством клеток, экспрессирующих различные уровни белка CLL1. Совместные культуры поддерживали в конечном объеме 100 мкл среды X-Vivo™-15 (фирма Lonza) в течение 6 ч при 37°С в атмосфере с 5% СО2. Окрашивание CD107a осуществляли в процессе стимуляции клеток, добавляя флуоресцентное антитело к CD107a (конъюгированное с АРС, фирма Miltenyi Biotec) в начале совместного культивирования, вместе с антителом к CD49d (фирма BD Pharmingen) в концентрации 1 мкг/мл, антителом к CD28 (фирма Miltenyi Biotec) в концентрации 1 мкг/мл, и 1-кратным раствором монензина (фирма eBioscience). После периода инкубации, составляющего 6 ч, клетки окрашивали фиксирующим витальным красителем(eFluor 780, фирма eBioscience) и конъюгированным с флуорохромом антителом к CD8 (конъюгированное с РЕ, фирма Miltenyi Biotec) и анализировали с помощью проточной цитометрии. Дегранулирующую активность определяли в виде % CD8+/CD107a+-клеток и на основе определения средней интенсивности сигнала флуоресценции (MFI) для окрашивания CD107a в популяции CD8+-клеток. Анализы дегрануляции осуществляли через 24 ч после трансфекции мРНК.
Анализ высвобождения IFN гамма
Т-клетки инкубировали в 96-луночных планшетах (40000 клеток/лунку) вместе с клеточными линиями, экспрессирующими различные уровни белка CLL1. Совместные культуры поддерживали в конечном объеме 100 мкл среды X-Vivo™-15 (фирма Lonza) в течение 24 ч при 37°С в атмосфере с 5% СО2. После указанного периода инкубации планшеты центрифугировали при 1500 об/мин в течение 5 мин и супернатанты переносили в новый планшет.Обнаружение IFN гамма в супернатантах клеточных культур осуществляли с помощью ELISA-анализа (набор Human IFN-gamma Quantikine ELISA, фирма R&D Systems). Анализы высвобождения IFN гамма осуществляли в начале совместного культивирования клеток через 24 ч после трансфекции мРНК.
Анализ цитотоксичности
Т-клетки инкубировали в 96-луночных планшетах (100000 клеток/лунку) вместе с 10000 клеток-мишеней (экспрессирующих CLL1) и 10000 контрольных (CLL1neg) клеток в одной и той же лунке. Клетки-мишени и контрольные клетки метили флуоресцентными внутриклеточными красителями (CFSE или Cell Trace Violet, фирма Life Technologies) перед их совместным культивированием с scCAR+-Т-клетками. Совместные культуры инкубировали в течение 4 ч при 37°С в атмосфере с 5% СО2. После указанного периода инкубации клетки метили фиксирующим витальным красителем (eFluor 780, фирма eBioscience) и анализировали с помощью проточной цитометрии. Определяли жизнеспособность каждой клеточной популяции (клетки-мишени или контрольные CLL1neg-клетки) и рассчитывали % удельного клеточного лизиса. Анализы цитотоксичности осуществляли через 48 ч после трансфекции мРНК.
Примеры анти-CLL1 одноцепочечных химерных антигенных рецепторов
sc SC02-357-CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 35)

sc SC02-357-CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 36)

sc SC02-357-CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 37)


sc SC02-357-CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 38)

sc SC02-357-CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 39)

sc SC02-357-CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 40)

sc SC02-378 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 41)

sc SC02-378 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 42)


sc SC02-378 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 43)

sc SC02-378 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 44)

sc SC02-378 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 45)


sc SC02-378 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 46)

sc SC02-161 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 47)

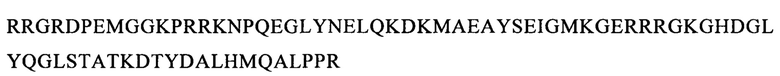
sc SC02-161 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 48)

sc SC02-161 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 49)

sc SC02-161 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 50)


sc SC02-161 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 51)

sc SC02-161 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 52)

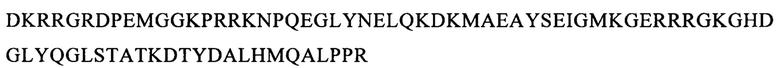
sc M26 CAR-vl (SEQ ID NO: 1+SEQ ID NO: 53)

sc M26 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 54)

sc M26 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 55)


sc M26 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 56)

sc M26 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 57)

sc M26 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 58)

sc M31 CAR-vl (SEQ ID NO: 1+SEQ ID NO: 59)

sc M31 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 60)


sc M31 CAR-v3 (SEQ ID NO: 1+SEQ ID NO. 61)

sc M31 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 62)

sc M31 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 63)


sc M31 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 64)

sc G4 CAR-vl (SEQ ID NO: 1+SEQ ID NO: 65)


sc G4 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 66)

sc G4 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 67)

sc G4 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 68)
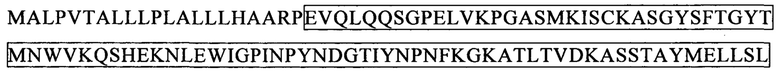

sc G4 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 69)

sc G4 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 70)


sc M22 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 71)

sc M22 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 72)

sc M22 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 73)


sc M22 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 74)

sc M22 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 75)


sc M22 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 76)

sc M29 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 77)

sc M29 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 78)

sc M29 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 79)

sc M29 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 80)

sc M29 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 81)

sc M29 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 82)

sc M2 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 83)

sc M2 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 84)

sc M2 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 85)

sc M2 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 86)

sc M2 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 87)

sc M2 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 88)


sc M5 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 89)

sc M5 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 90)

sc M5 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 91)

sc M5 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 92)

sc M5 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 93)


sc MS CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 94)

sc G12 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 95)


sc G12 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 96)

sc G12 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 97)

sc G12 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 98)


sc G12 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 99)

sc G12 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 100)


sc 21.26 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 101)

sc 21.26 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 102)

sc 21.26 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 103)


sc 21.26 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 104)

sc 21.26 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 105)

sc 21.26 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 106)

sc 1075.7 CAR-v1 (SEQ ID NO: 1+SEQ ID NO: 107)

sc 1075.7 CAR-v2 (SEQ ID NO: 1+SEQ ID NO: 108)


sc 1075.7 CAR-v3 (SEQ ID NO: 1+SEQ ID NO: 109)

sc 1075.7 CAR-v4 (SEQ ID NO: 1+SEQ ID NO: 110)

sc 1075.7 CAR-v5 (SEQ ID NO: 1+SEQ ID NO: 111)

sc 1075.7 CAR-v6 (SEQ ID NO: 1+SEQ ID NO: 112)

Список процитированной литературы
Arbiza J., Taylor G.,  , Furze J., Wyld S., Whyte P., Stott E.J., Wertz G., Sullender W., Trudel M. и др., Characterization of two антигенный sites recognized by neutralizing моноклональн antibodies directed against the fusion glycoprotein of human respiratory syncytial virus, J Gen Virol.;73 (9), 1992, cc. 2225-2234.
, Furze J., Wyld S., Whyte P., Stott E.J., Wertz G., Sullender W., Trudel M. и др., Characterization of two антигенный sites recognized by neutralizing моноклональн antibodies directed against the fusion glycoprotein of human respiratory syncytial virus, J Gen Virol.;73 (9), 1992, cc. 2225-2234.
Arimondo P.B., C.J. Thomas и др., "Exploring the cellular activity of camptothecin-triple-helix-forming oligonucleotide conjugates", Mol Cell Biol 26(1), 2006, cc. 324-333.
Atkins J.F., N.M. Wills и др., "A case for "StopGo": reprogramming translation to augment codon meaning of GGN by promoting unconventional termination (Stop) after addition of glycine and then allowing continued translation (Go)." Rna 13(6), 2007, cc. 803-810.
Bakker A.B., Van den Oudenrijn S, Bakker A.Q., Feller N., van Meijer M., Bia J.A., Jongeneelen M.A., Visser T.J., Bijl N., Geuijen C.A., Marissen W.E., Radosevic K., Throsby M., Schuurhuis G.J., Ossenkoppele G.J., de KruifJ., Goudsmit J., Kruisbeek A.M., "C-type lectin-like molecule-1: a novel myeloid cell surface marker associated with acute myeloid leukemia", Cancer Res. 64(22), 2004, cc. 8443-8450).
Bardenheuer W., K. Lehmberg и др., "Resistance to cytarabine and gemcitabine and in vitro selection of transduced cells after retroviral expression of cytidine deaminase in human hematopoietic progenitor cells", Leukemia 19(12), 2005, cc. 2281-8.
Betts M.R., J.M. Brenchley и др., "Sensitive and viable identification of antigen-specific CD8+ T cells by a flow cytometric assay for degranulation." J Immunol Methods 281(1-2), 2003, cc. 65-78.
Bierer B.E., G. Hollander и др., "Cyclosporin A and FK506: molecular mechanisms of immunosuppression and probes for transplantation biology", Curr Opin Immunol 5(5), 1993, cc. 763-773.
Boch J., H. Scholze и др., "Breaking the code of DNA binding specificity of TAL-type III effectors." Science 326(5959), 2009, cc. 1509-1512.
Brewin J., C. Mancao и др., "Generation of EBV-specific cytotoxic T cells that are resistant to calcineurin inhibitors for the treatment of posttransplantation lymphoproliferative disease." Blood 114(23), 2009, cc. 4792-4803.
Choulika A., A. Perrin и др., "Induction of homologous recombination in mammalian chromosomes by using the I-SceI system of Saccharomyces cerevisiae." Mol Cell Biol 15(4), 1995, cc. 1968-1973.
Christian M., T. Cermak и др., "Targeting DNA double-strand breaks with TAL effector nucleases." Genetics 186(2), 2010, cc. 757-761.
Cong L., F.A. Ran и др., "Multiplex genome engineering using CRISPR/Cas systems." Science 339(6121), 2013, cc. 819-823.
Critchlow S.E. и S.P. Jackson, "DNA end-joining: from yeast to man." Trends Biochem Sci 23(10), 1998, cc. 394-398.
Dasgupta A., D. McCarty и др., "Engineered drug-resistant immunocompetent cells enhance tumor cell killing during a chemotherapy challenge." Biochem Biophys Res Commun 391(1), 2011, cc. 170-175.
Deltcheva E., K. Chylinski и др., "CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III." Nature 471(7340), 2011, cc. 602-607.
Deng, D.C. Yan и др., "Structural basis for sequence-specific recognition of DNA by TAL effectors". Science 335(6069), 2012, cc. 720-723.
Donnelly M. и G. Elliott, "Nuclear localization and shuttling of herpes simplex virus tegument protein VP13/14", J Virol 75(6), 2001, cc. 2566-2574.
Doronina V.A., C. Wu и др., "Site-specific release of nascent chains from ribosomes at a sense codon." Mol Cell Biol 28(13), 2008, cc. 4227-4239.
Eisenschmidt K., T. Lanio и др., "Developing a programmed restriction endonuclease for highly specific DNA cleavage". Nucleic Acids Res 33(22), 2005, cc. 7039-7047.
Garneau J.E., M.E. Dupuis и др., "The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA", Nature 468(7320) 2010), cc. 67-71.
Gasiunas G., R. Barrangou и др., "Cas9-crRNA ribonucleoprotein complex mediates специфическ DNA cleavage for adaptive immunity in bacteria", Proc Natl Acad Sci USA 109(39), 2012, cc. E2579-2586.
Geissler R., H. Scholze и др., "Transcriptional activators of human genes with programmable DNA-specificity", PLoS One 6(5), 2011, cc. e19509.
Hacke K., J.A. Treger и др., "Genetic modification of mouse bone marrow by lentiviral vector-mediated delivery of hypoxanthine-Guanine phosphoribosyltransferase short hairpin RNA confers chemoprotection against 6-thioguanine cytotoxicity". Transplant Proc 45(5), 2013, cc. 2040-2044.
Henderson D.J., I. Naya и др., "Comparison of the effects of FK-506, cyclosporin A and rapamycin on IL-2 production". Immunology 73(3), 1991, cc. 316-321.
Huang P., A. Xiao и др., "Heritable gene targeting in zebrafish using customized TALENs", Nat Biotechnol 29(8), 2011, cc. 699-700.
Jena B., G. Dotti и др., "Redirecting T-cell specificity by introducing a tumor-specific chimeric antigen receptor". Blood 116(7), 2010, cc. 1035-1044.
Jinek M., K. Chylinski и др., "A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity". Science 337(6096), 2012, cc. 816-821.
Jonnalagadda M., C.E. Brown и др., "Engineering human T cells for resistance to methotrexate and mycophenolate mofetil as an in vivo cell selection strategy", PLoS One 8(6), 2013, c. e65519.
Kalish J.M. и P.M. Glazer, "Targeted genome modification via triple helix formation", Ann N Y Acad Sci 1058, 2005, cc. 151-161.
Kushman M.E., S.L. Kabler и др., "Expression of human glutathione S-transferase PI confers resistance to benzo[a]pyrene or benzo[a]pyrene-7,8-dihydrodiol mutagenesis, macromolecular alkylation and formation of stable N2-Gua-BPDE adducts in stably transfected V79MZ cells co-expressing hCYP1A1." Carcinogenesis 28(1), 2007, cc. 207-214.
Larsen H.∅., Roug A.S., Just T., Brown G.D., Hokland P., "Expression of the hMICL in acute myeloid leukemia-a highly reliable disease marker at diagnosis and during follow-up", Cytometry B Clin Cytom. 82(1), 2012, cc. 3-8.
Li L., M.J. Piatek и др., "Rapid and highly efficient construction of TALE-based transcriptional regulators and nucleases for genome modification". Plant Mol Biol 78(4-5), 2012, cc. 407-416.
Li T., S. Huang и др., "TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain". Nucleic Acids Res 39(1), 2011, cc. 359-372.
Li T., S. Huang и др., "Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes". Nucleic Acids Res 39(14), 2011,cc. 6315-6325.
Liu J., M.W. Albers и др., "Inhibition of T cell signaling by immunophilin-лиганд complexes correlates with loss of calcineurin phosphatase activity", Biochemistry 31(16), 1992, cc. 3896-3901.
Ma J.L., E.M. Kim и др., "Yeast Mre11 and Rad1 proteins define a Kuindependent mechanism to repair double-strand breaks lacking overlapping end sequences", Mol Cell Biol 23(23), 2003, cc. 8820-8828.
Mahfouz M.M., L. Li и др., "Targeted transcriptional repression using a chimeric TALE-SRDX repressor protein". Plant Mol Biol 78(3), 2012, cc. 311-321.
Mahfouz M.M., L. Li и др., "De novo-engineered transcription activator-like effector (TALE) hybrid nuclease with novel DNA binding specificity creates double-strand breaks", Proc Natl Acad Sci USA 108(6), 2011, cc. 2623-2628.
Mak A.N., P. Bradley и др., "The crystal structure of TAL effector PthXo1 bound to its DNA target". Science 335(6069), 2012, cc. 716-719.
Mali P., L. Yang и др., "RNA-guided human genome engineering via Cas9", Science 339(6121), 2013, cc. 823-826.
McLaughlin P и др., Rituximab chimeric anti-CD20 monoclonal antibody therapy for relapsed indolent lymphoma: half of patients respond to a four-dose treatment program, J Clin Oncol. 16(8), 1998, cc. 2825-8233.
Miller J.C., S. Tan и др., "A TALE nuclease architecture for efficient genome editing", Nat Biotechnol 29(2), 2011, cc. 143-148.
Morbitzer R., P. Romer и др., "Regulation of selected genome loci using de novo-engineered transcription activator-like effector (TALE)-type transcription factors", Proc Natl Acad Sci USA 107(50), 2011, cc. 21617-2122.
Moscou M.J. и A.J. Bogdanove, "A simple cipher governs DNA recognition by TAL effectors". Science 326(5959), 2009, c. 1501.
Mussolino C., R. Morbitzer и др., "A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity". Nucleic Acids Res 39(21), 2011, cc. 9283-9293.
Nivens M.C., T. Felder и др., "Engineered resistance to camptothecin and antifolates by retroviral coexpression of tyrosyl DNA phosphodiesterase-I and thymidylate synthase". Cancer Chemother Pharmacol 53(2), 2004, cc. 107-115.
Paques F. и P. Duchateau, "Meganucleases and DNA double-strand break-induced recombination: perspectives for gene therapy", Curr Gene Ther 7(1), 2007, cc. 49-66.
Park T.S., S.A. Rosenberg h и др., "Treating cancer with genetically engineered T cells". Trends Biotechnol 29(11), 2011, cc. 550-557.
Peipp M., D. Saul и др., "Efficient eukaryotic expression of fluorescent scFv fusion proteins directed against CD antigens for FACS applications", J Immunol Methods 285(2), 2004, cc. 265-280.
Perrin A., M. Buckle и др., "Asymmetrical recognition and activity of the I-SceI endonuclease on its site and on intron-exon junctions", Embo J 12(7), 1993, cc. 2939-2347.
Pingoud A. и G.H. Silva, "Precision genome surgery", Nat Biotechnol 25(7), 2007,cc. 743-744.
Porteus M.H. и D. Carroll, "Gene targeting using zinc finger nucleases", Nat Biotechnol 23(8), 2005, cc. 967-973.
Ravetch J.V., Perussia B., "Alternative membrane forms of Fc gamma RIII(CD16) on human natural killer cells and neutrophils. Cell type-specific expression of two genes that differ in single nucleotide substitutions", J. Exp. Med. 170, 1989.cc. 481-497.
Riemer A.B., Kurz H., Klinger M., Scheiner O., Zielinski C. и Jensen-Jarolim E., Vaccination with cetuximab mimotopes and biological properties of induced antiepidermal growth factor receptor antibodies, J Natl Cancer Inst.; 97(22), 2005, cc. 1663-1670.
Rouet P., F. Smih и др., "Introduction of double-strand breaks into the genome of mouse cells by expression of a rare-cutting endonuclease", Mol Cell Biol 14(12), 1994, cc. 8096-8106.
Sander J.D., L. Cade и др., "Targeted gene disruption in somatic zebrafish cells using engineered TALENs", Nat Biotechnol 29(8), 2011, cc. 697-698.
Sangiolo D., M. Lesnikova и др., "Lentiviral vector conferring resistance to mycophenolate mofetil and sensitivity to ganciclovir for in vivo T-cell selection", Gene Ther 14(21), 2007, cc. 1549-1554.
Schweitzer B.I., A.P. Dicker и др., "Dihydrofolate reductase as a therapeutic target", Faseb J 4(8), 1990, cc. 2441-2452.
Sorek R., C.M. Lawrence и др., "CRISPR-mediated Adaptive Immune Systems in Bacteria and Archaea", Annu Rev Biochem., 2013).
Stoddard B.L., "Homing endonuclease structure and function." Q Rev Biophys 38(1), 2005, cc. 49-95.
Sugimoto Y., S. Tsukahara и др., "Drug-selected co-expression of P-glycoprotein and gp91 in vivo from an MDR1-bicistronic retrovirus vector Ha-MDR-IRES-gp91", J Gene Med 5(5), 2003, cc. cc. 366-376.
Takebe N., S.C. Zhao и др., "Generation of dual resistance to 4-hydroperoxycyclophosphamide and methotrexate by retroviral transfer of the human aldehyde dehydrogenase class 1 gene and a mutated dihydrofolate reductase gene", Mol Ther 3(1), 2001, cc. 88-96.
Tesson L., C. Usal и др., "Knockout rats generated by embryo microinjection of TALENs", Nat Biotechnol 29(8), 2011, cc. 695-696.
Van Rhenen A., van Dongen G.A., Kelder A., Rombouts E.J., Feller N., Moshaver B., Stigter-van Walsum M., Zweegman S., Ossenkoppele G.J., Jan Schuurhuis G., Blood. "The novel AML stem cell associated антиген CLL-1 aids in discrimination between normal and leukemic stem cells. Blood, 110(7), 2007, cc. 2659-2666.
Weber E., R. Gruetzner и др., "Assembly of designer TAL effectors by Golden Gate cloning", PLoS One 6(5), 2011, c. e19722.
Yam P., M. Jensen и др., "Ex vivo selection and expansion of cells based on expression of a mutated inosine monophosphate dehydrogenase 2 after HIV vector transduction: effects on lymphocytes, monocytes, and CD34+ stem cells", Mol Ther 14(2), 2006, cc. 236-244.
Zhang Hongyong, Luo Juntao, Li Yuanpei, Henderson Paul T., Wachsmann-Hogiu Sebastian, Lam Kit S. и Pan Chong-xian, «Characterization of high-affinity peptides and their feasibility for use in nanotherapeutics targeting leukemia stem cells", Nanomedicine; 8(7), 2011, cc. 1116-1124.
Zhang F., L. Cong и др., "Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription", Nat Biotechnol 29(2), 2011, cc. 149-153.
Zielske S.P., J.S. Reese и др., "In vivo selection of MGMT(P140K) lentivirus-transduced human NOD/SCID repopulating cells without pretransplant irradiation conditioning", J Clin Invest 112(10), 2003, cc. 1561-1570.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> СЕЛЛЕКТИС
<120> АНТИ-CLL1-СПЕЦИФИЧЕСКИЕ ОДНОЦЕПОЧЕЧНЫЕ ХИМЕРНЫЕ АНТИГЕННЫЕ
РЕЦЕПТОРЫ (scCAR) ДЛЯ ИММУНОТЕРАПИИ РАКА
<130> P81504846PCT00
<150> PA201570044
<151> 2015-01-26
<160> 199
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> PRT
<213> artificial sequence
<220>
<223> signal peptide
<400> 1
Met Glu Thr Asp Thr Leu Leu Leu Trp Val Leu Leu Leu Trp Val Pro
1 5 10 15
Gly Ser Thr Gly
20
<210> 2
<211> 21
<212> PRT
<213> artificial sequence
<220>
<223> signal peptide
<400> 2
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro
20
<210> 3
<211> 16
<212> PRT
<213> homo sapiens
<220>
<223> FcgRIIIa hinge
<400> 3
Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln
1 5 10 15
<210> 4
<211> 45
<212> PRT
<213> homo sapiens
<220>
<223> CD8alpha hinge
<400> 4
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 5
<211> 231
<212> PRT
<213> homo sapiens
<220>
<223> IgG1 hinge
<400> 5
Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
20 25 30
Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val Thr Cys Val Val Val
35 40 45
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
50 55 60
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
65 70 75 80
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
85 90 95
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
100 105 110
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
115 120 125
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
130 135 140
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
145 150 155 160
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
165 170 175
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
180 185 190
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
195 200 205
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
210 215 220
Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 6
<211> 24
<212> PRT
<213> homo sapiens
<220>
<223> CD8alpha transmembrane domain
<400> 6
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr Cys
20
<210> 7
<211> 27
<212> PRT
<213> homo sapiens
<220>
<223> 41BB transmembrane domain
<400> 7
Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu
1 5 10 15
Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
20 25
<210> 8
<211> 42
<212> PRT
<213> homo sapiens
<220>
<223> Fragment of 4-1BB (residues 214-255)
<400> 8
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 9
<211> 112
<212> PRT
<213> homo sapiens
<220>
<223> fragment of T-cell surface glycoprotein CD3 zeta chain
<400> 9
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 10
<211> 15
<212> PRT
<213> artificial sequence
<220>
<223> linker
<400> 10
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 11
<211> 115
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 heavy chain variable region
<400> 11
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Ser
85 90 95
Ser Ser Gly Gly Phe Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 12
<211> 118
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 heavy chain variable region
<400> 12
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 13
<211> 118
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 heavy chain variable region
<400> 13
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 14
<211> 107
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 & SC02-378 & SC02-161 light chain variable region
<400> 14
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 15
<211> 120
<212> PRT
<213> artificial sequence
<220>
<223> M26 heavy chain variable region
<400> 15
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 16
<211> 108
<212> PRT
<213> artificial sequence
<220>
<223> M26 light chain variable region
<400> 16
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Ser Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly
50 55 60
Asn Arg Ser Gly Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 17
<211> 120
<212> PRT
<213> artificial sequence
<220>
<223> M31 heavy chain variable region
<400> 17
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Val Met His Trp Val Lys Gln Lys Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Pro Ile Tyr Phe Asp Asn Asp Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Lys Val Ser Ser
115 120
<210> 18
<211> 111
<212> PRT
<213> artificial sequence
<220>
<223> M31 light chain variable region
<400> 18
Thr Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr
20 25 30
Gly Asn Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp
65 70 75 80
Pro Val Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn
85 90 95
Tyr Asp Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 19
<211> 120
<212> PRT
<213> artificial sequence
<220>
<223> G4 heavy chain variable region
<400> 19
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Gly Tyr
20 25 30
Thr Met Asn Trp Val Lys Gln Ser His Glu Lys Asn Leu Glu Trp Ile
35 40 45
Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile Tyr Asn Pro Asn Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Thr Asp Asp Tyr Asp Asp Tyr Thr Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 20
<211> 107
<212> PRT
<213> artificial sequence
<220>
<223> G4 light chain variable region
<400> 20
Glu Ile Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 21
<211> 121
<212> PRT
<213> artificial sequence
<220>
<223> M22 heavy chain variable region
<400> 21
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Arg Tyr
20 25 30
Trp Met His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His Tyr Asn Gln Gln Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 22
<211> 113
<212> PRT
<213> artificial sequence
<220>
<223> M22 light chain variable region
<400> 22
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser
20 25 30
Gly Asn Gln Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn
85 90 95
Asp Tyr Ser Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
100 105 110
Lys
<210> 23
<211> 119
<212> PRT
<213> artificial sequence
<220>
<223> M29 heavy chain variable region
<400> 23
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Ile Phe Thr Ser Tyr
20 25 30
Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Leu Thr Val Ser Ser
115
<210> 24
<211> 106
<212> PRT
<213> artificial sequence
<220>
<223> M29 light chain variable region
<400> 24
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Gly Lys Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr
20 25 30
Ile Ala Trp Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile
35 40 45
His Tyr Thr Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 25
<211> 120
<212> PRT
<213> artificial sequence
<220>
<223> M2 heavy chain variable region
<400> 25
Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Phe Met His Trp Val Lys Gln Lys Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 26
<211> 108
<212> PRT
<213> artificial sequence
<220>
<223> M2 light chain variable region
<400> 26
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Ser Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 27
<211> 115
<212> PRT
<213> artificial sequence
<220>
<223> M5 heavy chain variable region
<400> 27
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala Tyr Ala Ser Lys Phe
50 55 60
Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr
100 105 110
Val Ser Ser
115
<210> 28
<211> 112
<212> PRT
<213> artificial sequence
<220>
<223> M5 light chain variable region
<400> 28
Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Val Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Ser Asn Gln Lys Asn Asn Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 29
<211> 117
<212> PRT
<213> artificial sequence
<220>
<223> G12 heavy chain variable region
<400> 29
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Pro Ser Ser
20 25 30
Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Ile Tyr Phe Cys
85 90 95
Ala Arg Val Tyr Asn Trp His Phe Asp Val Trp Gly Ala Gly Thr Thr
100 105 110
Val Thr Val Ser Ser
115
<210> 30
<211> 112
<212> PRT
<213> artificial sequence
<220>
<223> G12 light chain variable region
<400> 30
Asn Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr
20 25 30
Gly Asp Ile Phe Met Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp
65 70 75 80
Pro Val Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn
85 90 95
Glu Asp Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
<210> 31
<211> 120
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 heavy chain variable region
<400> 31
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Thr
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Arg Tyr
20 25 30
Trp Met His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser Tyr Asn Glu Lys Val
50 55 60
Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser Ser Thr Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser
115 120
<210> 32
<211> 106
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 light chain variable region
<400> 32
Gln Ile Val Leu Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
100 105
<210> 33
<211> 123
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 heavy chain variable region
<400> 33
Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr Ser Ile Thr Ser Ala
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly Asn Lys Leu Glu Trp
35 40 45
Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser Lys Asn Gln Phe Phe
65 70 75 80
Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr Tyr Ala Met Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 34
<211> 107
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 light chain variable region
<400> 34
Glu Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser
20 25 30
Tyr Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp
35 40 45
Ile Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu
65 70 75 80
Ala Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro
85 90 95
Leu Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
100 105
<210> 35
<211> 455
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 scCAR-v1 polypeptide
<400> 35
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe
260 265 270
Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
275 280 285
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
290 295 300
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
305 310 315 320
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
325 330 335
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg
450 455
<210> 36
<211> 458
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 scCAR-v2 polypeptide
<400> 36
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe
260 265 270
Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr
275 280 285
Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
290 295 300
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
305 310 315 320
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
325 330 335
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
340 345 350
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
355 360 365
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
370 375 380
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
385 390 395 400
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
405 410 415
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
420 425 430
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
435 440 445
Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 37
<211> 484
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 scCAR-v3 polypeptide
<400> 37
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
260 265 270
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
275 280 285
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
290 295 300
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
305 310 315 320
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
325 330 335
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
340 345 350
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
355 360 365
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
370 375 380
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
385 390 395 400
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
405 410 415
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
420 425 430
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
435 440 445
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
450 455 460
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
465 470 475 480
Leu Pro Pro Arg
<210> 38
<211> 487
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 scCAR-v4 polypeptide
<400> 38
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
260 265 270
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
275 280 285
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
290 295 300
Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu
305 310 315 320
Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly
325 330 335
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
340 345 350
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
355 360 365
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
370 375 380
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
385 390 395 400
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
405 410 415
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg
485
<210> 39
<211> 670
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 scCAR-v5 polypeptide
<400> 39
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys
260 265 270
Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe
275 280 285
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val
290 295 300
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
305 310 315 320
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
325 330 335
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
340 345 350
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
355 360 365
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
370 375 380
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
385 390 395 400
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
405 410 415
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
420 425 430
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
435 440 445
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
450 455 460
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
465 470 475 480
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Tyr Ile Trp
485 490 495
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
500 505 510
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
515 520 525
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
530 535 540
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
545 550 555 560
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
565 570 575
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
580 585 590
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
595 600 605
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
610 615 620
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
625 630 635 640
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
645 650 655
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
660 665 670
<210> 40
<211> 673
<212> PRT
<213> artificial sequence
<220>
<223> SC02-357 scCAR-v6 polypeptide
<400> 40
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys
260 265 270
Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe
275 280 285
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val
290 295 300
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
305 310 315 320
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
325 330 335
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
340 345 350
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
355 360 365
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
370 375 380
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
385 390 395 400
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
405 410 415
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
420 425 430
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
435 440 445
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
450 455 460
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
465 470 475 480
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Ile Ser Phe
485 490 495
Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu
500 505 510
Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr
515 520 525
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
530 535 540
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
545 550 555 560
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
565 570 575
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
580 585 590
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
595 600 605
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
610 615 620
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
625 630 635 640
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
645 650 655
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
660 665 670
Arg
<210> 41
<211> 455
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 scCAR-v1 polypeptide
<400> 41
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe
260 265 270
Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
275 280 285
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
290 295 300
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
305 310 315 320
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
325 330 335
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg
450 455
<210> 42
<211> 458
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 scCAR-v2 polypeptide
<400> 42
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe
260 265 270
Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr
275 280 285
Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
290 295 300
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
305 310 315 320
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
325 330 335
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
340 345 350
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
355 360 365
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
370 375 380
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
385 390 395 400
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
405 410 415
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
420 425 430
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
435 440 445
Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 43
<211> 484
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 scCAR-v3 polypeptide
<400> 43
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
260 265 270
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
275 280 285
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
290 295 300
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
305 310 315 320
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
325 330 335
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
340 345 350
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
355 360 365
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
370 375 380
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
385 390 395 400
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
405 410 415
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
420 425 430
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
435 440 445
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
450 455 460
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
465 470 475 480
Leu Pro Pro Arg
<210> 44
<211> 487
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 scCAR-v4 polypeptide
<400> 44
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
260 265 270
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
275 280 285
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
290 295 300
Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu
305 310 315 320
Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly
325 330 335
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
340 345 350
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
355 360 365
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
370 375 380
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
385 390 395 400
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
405 410 415
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg
485
<210> 45
<211> 670
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 scCAR-v5 polypeptide
<400> 45
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys
260 265 270
Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe
275 280 285
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val
290 295 300
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
305 310 315 320
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
325 330 335
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
340 345 350
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
355 360 365
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
370 375 380
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
385 390 395 400
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
405 410 415
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
420 425 430
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
435 440 445
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
450 455 460
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
465 470 475 480
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Tyr Ile Trp
485 490 495
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
500 505 510
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
515 520 525
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
530 535 540
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
545 550 555 560
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
565 570 575
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
580 585 590
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
595 600 605
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
610 615 620
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
625 630 635 640
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
645 650 655
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
660 665 670
<210> 46
<211> 673
<212> PRT
<213> artificial sequence
<220>
<223> SC02-378 scCAR-v6 polypeptide
<400> 46
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys
260 265 270
Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe
275 280 285
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val
290 295 300
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
305 310 315 320
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
325 330 335
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
340 345 350
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
355 360 365
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
370 375 380
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
385 390 395 400
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
405 410 415
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
420 425 430
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
435 440 445
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
450 455 460
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
465 470 475 480
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Ile Ser Phe
485 490 495
Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu
500 505 510
Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr
515 520 525
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
530 535 540
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
545 550 555 560
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
565 570 575
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
580 585 590
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
595 600 605
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
610 615 620
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
625 630 635 640
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
645 650 655
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
660 665 670
Arg
<210> 47
<211> 455
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 scCAR-v1 polypeptide
<400> 47
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe
260 265 270
Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
275 280 285
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
290 295 300
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
305 310 315 320
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
325 330 335
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg
450 455
<210> 48
<211> 458
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 scCAR-v2 polypeptide
<400> 48
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe
260 265 270
Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr
275 280 285
Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
290 295 300
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
305 310 315 320
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
325 330 335
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
340 345 350
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
355 360 365
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
370 375 380
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
385 390 395 400
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
405 410 415
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
420 425 430
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
435 440 445
Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 49
<211> 484
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 scCAR-v3 polypeptide
<400> 49
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
260 265 270
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
275 280 285
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
290 295 300
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
305 310 315 320
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
325 330 335
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
340 345 350
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
355 360 365
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
370 375 380
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
385 390 395 400
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
405 410 415
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
420 425 430
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
435 440 445
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
450 455 460
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
465 470 475 480
Leu Pro Pro Arg
<210> 50
<211> 487
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 scCAR-v4 polypeptide
<400> 50
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
260 265 270
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
275 280 285
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
290 295 300
Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu
305 310 315 320
Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly
325 330 335
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
340 345 350
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
355 360 365
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
370 375 380
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
385 390 395 400
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
405 410 415
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg
485
<210> 51
<211> 670
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 scCAR-v5 polypeptide
<400> 51
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys
260 265 270
Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe
275 280 285
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val
290 295 300
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
305 310 315 320
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
325 330 335
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
340 345 350
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
355 360 365
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
370 375 380
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
385 390 395 400
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
405 410 415
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
420 425 430
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
435 440 445
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
450 455 460
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
465 470 475 480
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Tyr Ile Trp
485 490 495
Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile
500 505 510
Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
515 520 525
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
530 535 540
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
545 550 555 560
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
565 570 575
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
580 585 590
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
595 600 605
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
610 615 620
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
625 630 635 640
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
645 650 655
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
660 665 670
<210> 52
<211> 673
<212> PRT
<213> artificial sequence
<220>
<223> SC02-161 scCAR-v6 polypeptide
<400> 52
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Glu Thr Leu Ser Leu Thr Cys Val Val Ser Gly Gly
35 40 45
Ser Ile Ser Ser Ser Asn Trp Trp Ser Trp Val Arg Gln Pro Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Ser Arg Val Thr Ile Ser Val Asp Lys Ser
85 90 95
Lys Asn Gln Phe Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln
145 150 155 160
Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr
165 170 175
Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln
180 185 190
Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu
195 200 205
Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
210 215 220
Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr
225 230 235 240
Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro Thr Phe Gly Gln Gly Thr
245 250 255
Lys Val Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys
260 265 270
Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe
275 280 285
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val
290 295 300
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
305 310 315 320
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
325 330 335
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
340 345 350
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
355 360 365
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
370 375 380
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
385 390 395 400
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
405 410 415
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
420 425 430
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
435 440 445
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
450 455 460
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
465 470 475 480
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Ile Ser Phe
485 490 495
Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu
500 505 510
Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr
515 520 525
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
530 535 540
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
545 550 555 560
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
565 570 575
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
580 585 590
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
595 600 605
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
610 615 620
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
625 630 635 640
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
645 650 655
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
660 665 670
Arg
<210> 53
<211> 458
<212> PRT
<213> artificial sequence
<220>
<223> M26 scCAR-v1 polypeptide
<400> 53
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly Asn Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Gly Leu Ala Val Ser Thr Ile Ser
260 265 270
Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala
275 280 285
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
290 295 300
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
305 310 315 320
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
325 330 335
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
340 345 350
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
355 360 365
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
370 375 380
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
385 390 395 400
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
405 410 415
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
420 425 430
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
435 440 445
Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 54
<211> 461
<212> PRT
<213> artificial sequence
<220>
<223> M26 scCAR-v2 polypeptide
<400> 54
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly Asn Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Gly Leu Ala Val Ser Thr Ile Ser
260 265 270
Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu
275 280 285
Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe
290 295 300
Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
305 310 315 320
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
325 330 335
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
340 345 350
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
355 360 365
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
370 375 380
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
385 390 395 400
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
405 410 415
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
420 425 430
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
435 440 445
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 55
<211> 487
<212> PRT
<213> Artificial sequence
<220>
<223> M26 scCAR-v3 polypeptide
<400> 55
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly Asn Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro
260 265 270
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
275 280 285
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
290 295 300
Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
305 310 315 320
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
325 330 335
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
340 345 350
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
355 360 365
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
370 375 380
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
385 390 395 400
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
405 410 415
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg
485
<210> 56
<211> 490
<212> PRT
<213> Artificial sequence
<220>
<223> M26 scCAR-v4 polypeptide
<400> 56
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly Asn Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro
260 265 270
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
275 280 285
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
290 295 300
Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr
305 310 315 320
Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
325 330 335
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
340 345 350
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
355 360 365
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
370 375 380
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
385 390 395 400
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
405 410 415
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
420 425 430
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
435 440 445
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
450 455 460
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
465 470 475 480
Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 57
<211> 673
<212> PRT
<213> Artificial sequence
<220>
<223> M26 scCAR-v5 polypeptide
<400> 57
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly Asn Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Glu Pro Lys Ser Pro Asp Lys Thr
260 265 270
His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
305 310 315 320
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile
485 490 495
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
500 505 510
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
515 520 525
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
530 535 540
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
545 550 555 560
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
565 570 575
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
580 585 590
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
595 600 605
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
610 615 620
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
625 630 635 640
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
645 650 655
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
660 665 670
Arg
<210> 58
<211> 676
<212> PRT
<213> Artificial sequence
<220>
<223> M26 scCAR-v6 polypeptide
<400> 58
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Ile His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Thr Gln Glu Leu Ser Gly Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly Asn Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ile Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Glu Pro Lys Ser Pro Asp Lys Thr
260 265 270
His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
305 310 315 320
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile
485 490 495
Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu
500 505 510
Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys
515 520 525
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
530 535 540
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
545 550 555 560
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
565 570 575
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
580 585 590
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
595 600 605
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
610 615 620
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
625 630 635 640
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
645 650 655
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
660 665 670
Leu Pro Pro Arg
675
<210> 59
<211> 463
<212> PRT
<213> Artificial sequence
<220>
<223> M31 scCAR-v1 polypeptide
<400> 59
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Val Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Phe Ser Cys Cys Ala Arg Pro Ile Tyr Phe Asp Asn Asp
115 120 125
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Lys Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
145 150 155 160
Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg
165 170 175
Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr Gly Asn
180 185 190
Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu
195 200 205
Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val
225 230 235 240
Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Tyr Asp
245 250 255
Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala
260 265 270
Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile
275 280 285
Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val
290 295 300
Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
305 310 315 320
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
325 330 335
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
340 345 350
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
355 360 365
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
370 375 380
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
385 390 395 400
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
405 410 415
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
420 425 430
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
435 440 445
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 60
<211> 466
<212> PRT
<213> Artificial sequence
<220>
<223> M31 scCAR-v2 polypeptide
<400> 60
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Val Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Phe Ser Cys Cys Ala Arg Pro Ile Tyr Phe Asp Asn Asp
115 120 125
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Lys Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
145 150 155 160
Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg
165 170 175
Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr Gly Asn
180 185 190
Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu
195 200 205
Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val
225 230 235 240
Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Tyr Asp
245 250 255
Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala
260 265 270
Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser
275 280 285
Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe
290 295 300
Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu
305 310 315 320
Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu
325 330 335
Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys
340 345 350
Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
355 360 365
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
370 375 380
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
385 390 395 400
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
405 410 415
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
420 425 430
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
435 440 445
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
450 455 460
Pro Arg
465
<210> 61
<211> 492
<212> PRT
<213> Artificial sequence
<220>
<223> M31 scCAR-v3 polypeptide
<400> 61
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Val Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Phe Ser Cys Cys Ala Arg Pro Ile Tyr Phe Asp Asn Asp
115 120 125
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Lys Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
145 150 155 160
Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg
165 170 175
Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr Gly Asn
180 185 190
Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu
195 200 205
Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val
225 230 235 240
Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Tyr Asp
245 250 255
Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr
260 265 270
Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro
275 280 285
Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val
290 295 300
His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro
305 310 315 320
Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu
325 330 335
Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
340 345 350
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
355 360 365
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
370 375 380
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
385 390 395 400
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
405 410 415
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
420 425 430
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
435 440 445
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
450 455 460
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
465 470 475 480
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 62
<211> 495
<212> PRT
<213> Artificial sequence
<220>
<223> M31 scCAR-v4 polypeptide
<400> 62
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Val Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Phe Ser Cys Cys Ala Arg Pro Ile Tyr Phe Asp Asn Asp
115 120 125
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Lys Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
145 150 155 160
Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg
165 170 175
Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr Gly Asn
180 185 190
Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu
195 200 205
Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val
225 230 235 240
Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Tyr Asp
245 250 255
Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr
260 265 270
Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro
275 280 285
Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val
290 295 300
His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu
305 310 315 320
Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu
325 330 335
Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
340 345 350
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
355 360 365
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
370 375 380
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
385 390 395 400
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
405 410 415
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
420 425 430
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
435 440 445
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
450 455 460
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
465 470 475 480
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490 495
<210> 63
<211> 678
<212> PRT
<213> Artificial sequence
<220>
<223> M31 scCAR-v5 polypeptide
<400> 63
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Val Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Phe Ser Cys Cys Ala Arg Pro Ile Tyr Phe Asp Asn Asp
115 120 125
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Lys Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
145 150 155 160
Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg
165 170 175
Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr Gly Asn
180 185 190
Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu
195 200 205
Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val
225 230 235 240
Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Tyr Asp
245 250 255
Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys
260 265 270
Ser Pro Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val
275 280 285
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ala Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Lys Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
500 505 510
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
515 520 525
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
530 535 540
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
545 550 555 560
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
565 570 575
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
580 585 590
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
595 600 605
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
610 615 620
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
625 630 635 640
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
645 650 655
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
660 665 670
Gln Ala Leu Pro Pro Arg
675
<210> 64
<211> 681
<212> PRT
<213> Artificial sequence
<220>
<223> M31 scCAR-v6 polypeptide
<400> 64
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Val Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Phe Ser Cys Cys Ala Arg Pro Ile Tyr Phe Asp Asn Asp
115 120 125
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Lys Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
145 150 155 160
Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg
165 170 175
Ala Thr Ile Ser Cys Arg Ala Ser Glu Ser Val Asp Ser Tyr Gly Asn
180 185 190
Ser Phe Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu
195 200 205
Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val
225 230 235 240
Glu Ala Asp Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Tyr Asp
245 250 255
Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys
260 265 270
Ser Pro Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val
275 280 285
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ala Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Lys Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala
500 505 510
Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys
515 520 525
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
530 535 540
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
545 550 555 560
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
565 570 575
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
580 585 590
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
595 600 605
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
610 615 620
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
625 630 635 640
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
645 650 655
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
660 665 670
Leu His Met Gln Ala Leu Pro Pro Arg
675 680
<210> 65
<211> 459
<212> PRT
<213> Artificial sequence
<220>
<223> G4 scCAR-v1 polypeptide
<400> 65
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ser Phe Thr Gly Tyr Thr Met Asn Trp Val Lys Gln Ser His Glu Lys
50 55 60
Asn Leu Glu Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile
65 70 75 80
Tyr Asn Pro Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Thr Asp Asp Tyr Asp Asp Tyr
115 120 125
Thr Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
145 150 155 160
Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly Asp Arg
165 170 175
Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile Tyr Tyr
195 200 205
Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp Thr Phe
245 250 255
Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala Val Ser Thr Ile
260 265 270
Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu
275 280 285
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
290 295 300
Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
305 310 315 320
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
325 330 335
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser
340 345 350
Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr
355 360 365
Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys
370 375 380
Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn
385 390 395 400
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
405 410 415
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
420 425 430
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
435 440 445
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 66
<211> 462
<212> PRT
<213> Artificial sequence
<220>
<223> G4 scCAR-v2 polypeptide
<400> 66
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ser Phe Thr Gly Tyr Thr Met Asn Trp Val Lys Gln Ser His Glu Lys
50 55 60
Asn Leu Glu Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile
65 70 75 80
Tyr Asn Pro Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Thr Asp Asp Tyr Asp Asp Tyr
115 120 125
Thr Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
145 150 155 160
Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly Asp Arg
165 170 175
Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile Tyr Tyr
195 200 205
Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp Thr Phe
245 250 255
Gly Gly Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala Val Ser Thr Ile
260 265 270
Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala
275 280 285
Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg
290 295 300
Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
305 310 315 320
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
325 330 335
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
340 345 350
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
355 360 365
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
370 375 380
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
385 390 395 400
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
405 410 415
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
420 425 430
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
435 440 445
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 67
<211> 488
<212> PRT
<213> Artificial sequence
<220>
<223> G4 scCAR-v3 polypeptide
<400> 67
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ser Phe Thr Gly Tyr Thr Met Asn Trp Val Lys Gln Ser His Glu Lys
50 55 60
Asn Leu Glu Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile
65 70 75 80
Tyr Asn Pro Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Thr Asp Asp Tyr Asp Asp Tyr
115 120 125
Thr Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
145 150 155 160
Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly Asp Arg
165 170 175
Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile Tyr Tyr
195 200 205
Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp Thr Phe
245 250 255
Gly Gly Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg
260 265 270
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
275 280 285
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
290 295 300
Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
305 310 315 320
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
325 330 335
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
340 345 350
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
355 360 365
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
370 375 380
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
385 390 395 400
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
405 410 415
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
420 425 430
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
435 440 445
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
450 455 460
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
465 470 475 480
His Met Gln Ala Leu Pro Pro Arg
485
<210> 68
<211> 491
<212> PRT
<213> Artificial sequence
<220>
<223> G4 scCAR-v4 polypeptide
<400> 68
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ser Phe Thr Gly Tyr Thr Met Asn Trp Val Lys Gln Ser His Glu Lys
50 55 60
Asn Leu Glu Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile
65 70 75 80
Tyr Asn Pro Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Thr Asp Asp Tyr Asp Asp Tyr
115 120 125
Thr Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
145 150 155 160
Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly Asp Arg
165 170 175
Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile Tyr Tyr
195 200 205
Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp Thr Phe
245 250 255
Gly Gly Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg
260 265 270
Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg
275 280 285
Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly
290 295 300
Leu Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser
305 310 315 320
Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val
325 330 335
Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
340 345 350
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
355 360 365
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser
370 375 380
Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr
385 390 395 400
Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys
405 410 415
Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn
420 425 430
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
435 440 445
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
450 455 460
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
465 470 475 480
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 69
<211> 674
<212> PRT
<213> Artificial sequence
<220>
<223> G4 scCAR-v5 polypeptide
<400> 69
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ser Phe Thr Gly Tyr Thr Met Asn Trp Val Lys Gln Ser His Glu Lys
50 55 60
Asn Leu Glu Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile
65 70 75 80
Tyr Asn Pro Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Thr Asp Asp Tyr Asp Asp Tyr
115 120 125
Thr Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
145 150 155 160
Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly Asp Arg
165 170 175
Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile Tyr Tyr
195 200 205
Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp Thr Phe
245 250 255
Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys
260 265 270
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser
275 280 285
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg
290 295 300
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
305 310 315 320
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
325 330 335
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
340 345 350
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
355 360 365
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
370 375 380
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
385 390 395 400
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
405 410 415
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
420 425 430
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
435 440 445
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
450 455 460
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
465 470 475 480
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
485 490 495
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
500 505 510
Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu
515 520 525
Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu
530 535 540
Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys
545 550 555 560
Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
565 570 575
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
580 585 590
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
595 600 605
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
610 615 620
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
625 630 635 640
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
645 650 655
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
660 665 670
Pro Arg
<210> 70
<211> 677
<212> PRT
<213> Artificial sequence
<220>
<223> G4 scCAR-v6 polypeptide
<400> 70
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ser Phe Thr Gly Tyr Thr Met Asn Trp Val Lys Gln Ser His Glu Lys
50 55 60
Asn Leu Glu Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile
65 70 75 80
Tyr Asn Pro Asn Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ala
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Leu Ser Leu Thr Ser Asp Asp Pro
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Thr Asp Asp Tyr Asp Asp Tyr
115 120 125
Thr Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Ile
145 150 155 160
Gln Met Thr Gln Thr Pro Ser Ser Leu Ser Ala Ser Leu Gly Asp Arg
165 170 175
Val Thr Ile Ser Cys Arg Ala Ser His Asp Ile Ser Asn Tyr Leu Asn
180 185 190
Trp Tyr Gln Gln Lys Pro Asp Gly Thr Leu Lys Leu Leu Ile Tyr Tyr
195 200 205
Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
210 215 220
Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln Glu Asp
225 230 235 240
Ile Ala Thr Tyr Phe Cys Gln Gln Gly Lys Thr Leu Leu Trp Thr Phe
245 250 255
Gly Gly Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys
260 265 270
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser
275 280 285
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg
290 295 300
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
305 310 315 320
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
325 330 335
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
340 345 350
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
355 360 365
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
370 375 380
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
385 390 395 400
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
405 410 415
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
420 425 430
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
435 440 445
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
450 455 460
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
465 470 475 480
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
485 490 495
Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu
500 505 510
Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys
515 520 525
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
530 535 540
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
545 550 555 560
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
565 570 575
Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
580 585 590
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
595 600 605
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
610 615 620
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
625 630 635 640
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
645 650 655
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
660 665 670
Ala Leu Pro Pro Arg
675
<210> 71
<211> 464
<212> PRT
<213> Artificial sequence
<220>
<223> M22 scCAR-v1 polypeptide
<400> 71
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His
65 70 75 80
Tyr Asn Gln Gln Phe Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala
115 120 125
Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly Glu Lys Val
165 170 175
Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser Gly Asn Gln
180 185 190
Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys
195 200 205
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg
210 215 220
Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
225 230 235 240
Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn Asp Tyr Ser
245 250 255
Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Gly Leu
260 265 270
Ala Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr
275 280 285
Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu
290 295 300
Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile
305 310 315 320
Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp
325 330 335
Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
340 345 350
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
355 360 365
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
370 375 380
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
385 390 395 400
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
405 410 415
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
420 425 430
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
435 440 445
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 72
<211> 467
<212> PRT
<213> Artificial sequence
<220>
<223> M22 scCAR-v2 polypeptide
<400> 72
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His
65 70 75 80
Tyr Asn Gln Gln Phe Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala
115 120 125
Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly Glu Lys Val
165 170 175
Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser Gly Asn Gln
180 185 190
Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys
195 200 205
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg
210 215 220
Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
225 230 235 240
Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn Asp Tyr Ser
245 250 255
Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Gly Leu
260 265 270
Ala Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile
275 280 285
Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe
290 295 300
Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu
305 310 315 320
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
325 330 335
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
340 345 350
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
355 360 365
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
370 375 380
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
385 390 395 400
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
405 410 415
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
420 425 430
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
435 440 445
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
450 455 460
Pro Pro Arg
465
<210> 73
<211> 493
<212> PRT
<213> Artificial sequence
<220>
<223> M22 scCAR-v3 polypeptide
<400> 73
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His
65 70 75 80
Tyr Asn Gln Gln Phe Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala
115 120 125
Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly Glu Lys Val
165 170 175
Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser Gly Asn Gln
180 185 190
Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys
195 200 205
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg
210 215 220
Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
225 230 235 240
Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn Asp Tyr Ser
245 250 255
Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Thr Thr
260 265 270
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
275 280 285
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
290 295 300
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala
305 310 315 320
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
325 330 335
Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
340 345 350
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
355 360 365
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
370 375 380
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
385 390 395 400
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
405 410 415
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
420 425 430
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
435 440 445
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
450 455 460
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
465 470 475 480
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 74
<211> 496
<212> PRT
<213> Artificial sequence
<220>
<223> M22 scCAR-v4 polypeptide
<400> 74
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His
65 70 75 80
Tyr Asn Gln Gln Phe Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala
115 120 125
Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly Glu Lys Val
165 170 175
Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser Gly Asn Gln
180 185 190
Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys
195 200 205
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg
210 215 220
Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
225 230 235 240
Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn Asp Tyr Ser
245 250 255
Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Thr Thr
260 265 270
Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln
275 280 285
Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala
290 295 300
Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe
305 310 315 320
Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr
325 330 335
Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile
340 345 350
Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp
355 360 365
Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
370 375 380
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
385 390 395 400
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
405 410 415
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
420 425 430
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
435 440 445
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
450 455 460
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
465 470 475 480
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490 495
<210> 75
<211> 679
<212> PRT
<213> Artificial sequence
<220>
<223> M22 scCAR-v5 polypeptide
<400> 75
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His
65 70 75 80
Tyr Asn Gln Gln Phe Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala
115 120 125
Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly Glu Lys Val
165 170 175
Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser Gly Asn Gln
180 185 190
Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys
195 200 205
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg
210 215 220
Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
225 230 235 240
Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn Asp Tyr Ser
245 250 255
Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Glu Pro
260 265 270
Lys Ser Pro Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro
275 280 285
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
290 295 300
Leu Met Ile Ala Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
305 310 315 320
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
325 330 335
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
340 345 350
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
355 360 365
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
370 375 380
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
385 390 395 400
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
405 410 415
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
420 425 430
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
435 440 445
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
450 455 460
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
465 470 475 480
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
485 490 495
Leu Ser Pro Gly Lys Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
500 505 510
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
515 520 525
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
530 535 540
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
545 550 555 560
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
565 570 575
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
580 585 590
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
595 600 605
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
610 615 620
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
625 630 635 640
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
645 650 655
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
660 665 670
Met Gln Ala Leu Pro Pro Arg
675
<210> 76
<211> 682
<212> PRT
<213> Artificial sequence
<220>
<223> M22 scCAR-v6 polypeptide
<400> 76
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His
65 70 75 80
Tyr Asn Gln Gln Phe Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala
115 120 125
Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Val
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Thr Val Thr Ala Gly Glu Lys Val
165 170 175
Thr Met Ser Cys Lys Ser Ser Gln Asn Leu Leu Asn Ser Gly Asn Gln
180 185 190
Lys Lys Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys
195 200 205
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val Pro Asp Arg
210 215 220
Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser
225 230 235 240
Val Gln Ala Glu Asp Leu Ala Val Tyr Phe Cys Gln Asn Asp Tyr Ser
245 250 255
Tyr Pro Phe Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Glu Pro
260 265 270
Lys Ser Pro Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro
275 280 285
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
290 295 300
Leu Met Ile Ala Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
305 310 315 320
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
325 330 335
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
340 345 350
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
355 360 365
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
370 375 380
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
385 390 395 400
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
405 410 415
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
420 425 430
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
435 440 445
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
450 455 460
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
465 470 475 480
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
485 490 495
Leu Ser Pro Gly Lys Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr
500 505 510
Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
515 520 525
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
530 535 540
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
545 550 555 560
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
565 570 575
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
580 585 590
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
595 600 605
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
610 615 620
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
625 630 635 640
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
645 650 655
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
660 665 670
Ala Leu His Met Gln Ala Leu Pro Pro Arg
675 680
<210> 77
<211> 457
<212> PRT
<213> Artificial sequence
<220>
<223> M29 scCAR-v1 polypeptide
<400> 77
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ile Phe Thr Ser Tyr Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Gly Lys Val
165 170 175
Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr Ile Ala Trp
180 185 190
Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile His Tyr Thr
195 200 205
Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
210 215 220
Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro Glu Asp Ile
225 230 235 240
Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser
260 265 270
Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala Gly
275 280 285
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
290 295 300
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
305 310 315 320
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
325 330 335
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
340 345 350
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
355 360 365
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
370 375 380
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
385 390 395 400
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
405 410 415
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
420 425 430
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
435 440 445
Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 78
<211> 460
<212> PRT
<213> Artificial sequence
<220>
<223> M29 scCAR-v2 polypeptide
<400> 78
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ile Phe Thr Ser Tyr Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Gly Lys Val
165 170 175
Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr Ile Ala Trp
180 185 190
Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile His Tyr Thr
195 200 205
Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
210 215 220
Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro Glu Asp Ile
225 230 235 240
Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser
260 265 270
Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr
275 280 285
Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser
290 295 300
Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
305 310 315 320
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
325 330 335
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 79
<211> 486
<212> PRT
<213> Artificial sequence
<220>
<223> M29 scCAR-v3 polypeptide
<400> 79
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ile Phe Thr Ser Tyr Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Gly Lys Val
165 170 175
Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr Ile Ala Trp
180 185 190
Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile His Tyr Thr
195 200 205
Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
210 215 220
Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro Glu Asp Ile
225 230 235 240
Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg
485
<210> 80
<211> 489
<212> PRT
<213> Artificial sequence
<220>
<223> M29 scCAR-v4 polypeptide
<400> 80
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ile Phe Thr Ser Tyr Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Gly Lys Val
165 170 175
Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr Ile Ala Trp
180 185 190
Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile His Tyr Thr
195 200 205
Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
210 215 220
Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro Glu Asp Ile
225 230 235 240
Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala
305 310 315 320
Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys
325 330 335
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
340 345 350
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
355 360 365
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
370 375 380
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
385 390 395 400
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
405 410 415
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
420 425 430
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
435 440 445
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
450 455 460
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
465 470 475 480
Leu His Met Gln Ala Leu Pro Pro Arg
485
<210> 81
<211> 672
<212> PRT
<213> artificial sequence
<220>
<223> M29 scCAR-v5 polypeptide
<400> 81
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ile Phe Thr Ser Tyr Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Gly Lys Val
165 170 175
Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr Ile Ala Trp
180 185 190
Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile His Tyr Thr
195 200 205
Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
210 215 220
Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro Glu Asp Ile
225 230 235 240
Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His
260 265 270
Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
275 280 285
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro
290 295 300
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
305 310 315 320
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
325 330 335
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
340 345 350
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
355 360 365
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
370 375 380
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
385 390 395 400
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
405 410 415
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
420 425 430
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
435 440 445
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
450 455 460
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
465 470 475 480
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Tyr
485 490 495
Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu
500 505 510
Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile
515 520 525
Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp
530 535 540
Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
545 550 555 560
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
565 570 575
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
580 585 590
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
595 600 605
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
610 615 620
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
625 630 635 640
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
645 650 655
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
660 665 670
<210> 82
<211> 675
<212> PRT
<213> artificial sequence
<220>
<223> M29 scCAR-v6 polypeptide
<400> 82
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Ile Phe Thr Ser Tyr Val Met Tyr Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Ser Cys Cys Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
145 150 155 160
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Gly Lys Val
165 170 175
Thr Ile Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr Ile Ala Trp
180 185 190
Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile His Tyr Thr
195 200 205
Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly Ser Gly Ser
210 215 220
Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro Glu Asp Ile
225 230 235 240
Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Tyr Leu Trp Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His
260 265 270
Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
275 280 285
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro
290 295 300
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
305 310 315 320
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
325 330 335
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
340 345 350
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
355 360 365
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
370 375 380
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
385 390 395 400
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
405 410 415
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
420 425 430
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
435 440 445
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
450 455 460
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
465 470 475 480
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Ile
485 490 495
Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe
500 505 510
Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu
515 520 525
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
530 535 540
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
545 550 555 560
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
565 570 575
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
580 585 590
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
595 600 605
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
610 615 620
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
625 630 635 640
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
645 650 655
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
660 665 670
Pro Pro Arg
675
<210> 83
<211> 458
<212> PRT
<213> artificial sequence
<220>
<223> M2 scCAR-v1 polypeptide
<400> 83
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly Ser Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Gly Leu Ala Val Ser Thr Ile Ser
260 265 270
Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala
275 280 285
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
290 295 300
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
305 310 315 320
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
325 330 335
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
340 345 350
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
355 360 365
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
370 375 380
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
385 390 395 400
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
405 410 415
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
420 425 430
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
435 440 445
Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 84
<211> 461
<212> PRT
<213> artificial sequence
<220>
<223> M2 scCAR-v2 polypeptide
<400> 84
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly Ser Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Gly Leu Ala Val Ser Thr Ile Ser
260 265 270
Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu
275 280 285
Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe
290 295 300
Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
305 310 315 320
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
325 330 335
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
340 345 350
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
355 360 365
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
370 375 380
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
385 390 395 400
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
405 410 415
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
420 425 430
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
435 440 445
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 85
<211> 487
<212> PRT
<213> artificial sequence
<220>
<223> M2 scCAR-v3 polypeptide
<400> 85
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly Ser Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro
260 265 270
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
275 280 285
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
290 295 300
Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys
305 310 315 320
Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly
325 330 335
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
340 345 350
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
355 360 365
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
370 375 380
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
385 390 395 400
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
405 410 415
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
420 425 430
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
435 440 445
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
450 455 460
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
465 470 475 480
Met Gln Ala Leu Pro Pro Arg
485
<210> 86
<211> 490
<212> PRT
<213> artificial sequence
<220>
<223> M2 scCAR-v4 polypeptide
<400> 86
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly Ser Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro
260 265 270
Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro
275 280 285
Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu
290 295 300
Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr
305 310 315 320
Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
325 330 335
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
340 345 350
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
355 360 365
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
370 375 380
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
385 390 395 400
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
405 410 415
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
420 425 430
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
435 440 445
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
450 455 460
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
465 470 475 480
Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 87
<211> 673
<212> PRT
<213> artificial sequence
<220>
<223> M2 scCAR-v5 polypeptide
<400> 87
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly Ser Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Glu Pro Lys Ser Pro Asp Lys Thr
260 265 270
His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
305 310 315 320
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile
485 490 495
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
500 505 510
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
515 520 525
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
530 535 540
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
545 550 555 560
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
565 570 575
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
580 585 590
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
595 600 605
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
610 615 620
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
625 630 635 640
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
645 650 655
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
660 665 670
Arg
<210> 88
<211> 676
<212> PRT
<213> artificial sequence
<220>
<223> M2 scCAR-v6 polypeptide
<400> 88
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Arg Gln Ser Gly Pro Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Ser Tyr Phe Met His Trp Val Lys Gln Lys Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys
65 70 75 80
Tyr Asn Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Glu Leu Asn Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met
145 150 155 160
Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly Glu Arg Val Ser
165 170 175
Leu Thr Cys Arg Ala Ser Gln Glu Ile Ser Val Tyr Leu Ser Trp Leu
180 185 190
Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser
195 200 205
Thr Leu Asp Ser Gly Val Pro Glu Arg Phe Ser Gly Ser Arg Ser Gly
210 215 220
Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala
225 230 235 240
Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Tyr Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Arg Glu Pro Lys Ser Pro Asp Lys Thr
260 265 270
His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
305 310 315 320
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile
485 490 495
Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu
500 505 510
Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys
515 520 525
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
530 535 540
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
545 550 555 560
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
565 570 575
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
580 585 590
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
595 600 605
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
610 615 620
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
625 630 635 640
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
645 650 655
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
660 665 670
Leu Pro Pro Arg
675
<210> 89
<211> 457
<212> PRT
<213> artificial sequence
<220>
<223> M5scCAR-v1 polypeptide
<400> 89
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu
20 25 30
Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe
35 40 45
Asn Ile Lys Asp Asp Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln
50 55 60
Gly Leu Glu Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala
65 70 75 80
Tyr Ala Ser Lys Phe Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser
85 90 95
Ser Asn Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Ser Gln Ser Pro Ser
145 150 155 160
Ser Leu Ala Val Ser Val Gly Glu Lys Val Thr Met Ser Cys Lys Ser
165 170 175
Ser Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala Trp
180 185 190
Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala
195 200 205
Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser
210 215 220
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu
225 230 235 240
Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser
260 265 270
Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala Gly
275 280 285
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
290 295 300
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
305 310 315 320
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
325 330 335
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
340 345 350
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
355 360 365
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
370 375 380
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
385 390 395 400
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
405 410 415
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
420 425 430
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
435 440 445
Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 90
<211> 460
<212> PRT
<213> artificial sequence
<220>
<223> M5 scCAR-v2 polypeptide
<400> 90
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu
20 25 30
Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe
35 40 45
Asn Ile Lys Asp Asp Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln
50 55 60
Gly Leu Glu Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala
65 70 75 80
Tyr Ala Ser Lys Phe Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser
85 90 95
Ser Asn Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Ser Gln Ser Pro Ser
145 150 155 160
Ser Leu Ala Val Ser Val Gly Glu Lys Val Thr Met Ser Cys Lys Ser
165 170 175
Ser Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala Trp
180 185 190
Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala
195 200 205
Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser
210 215 220
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu
225 230 235 240
Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Gly Leu Ala Val Ser Thr Ile Ser Ser
260 265 270
Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr
275 280 285
Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser
290 295 300
Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
305 310 315 320
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
325 330 335
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 91
<211> 486
<212> PRT
<213> artificial sequence
<220>
<223> M5 scCAR-v3 polypeptide
<400> 91
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu
20 25 30
Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe
35 40 45
Asn Ile Lys Asp Asp Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln
50 55 60
Gly Leu Glu Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala
65 70 75 80
Tyr Ala Ser Lys Phe Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser
85 90 95
Ser Asn Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Ser Gln Ser Pro Ser
145 150 155 160
Ser Leu Ala Val Ser Val Gly Glu Lys Val Thr Met Ser Cys Lys Ser
165 170 175
Ser Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala Trp
180 185 190
Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala
195 200 205
Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser
210 215 220
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu
225 230 235 240
Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
305 310 315 320
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg
325 330 335
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
340 345 350
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
355 360 365
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
370 375 380
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
385 390 395 400
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
405 410 415
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
420 425 430
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
435 440 445
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
450 455 460
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
465 470 475 480
Gln Ala Leu Pro Pro Arg
485
<210> 92
<211> 489
<212> PRT
<213> artificial sequence
<220>
<223> M5 scCAR-v4 polypeptide
<400> 92
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu
20 25 30
Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe
35 40 45
Asn Ile Lys Asp Asp Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln
50 55 60
Gly Leu Glu Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala
65 70 75 80
Tyr Ala Ser Lys Phe Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser
85 90 95
Ser Asn Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Ser Gln Ser Pro Ser
145 150 155 160
Ser Leu Ala Val Ser Val Gly Glu Lys Val Thr Met Ser Cys Lys Ser
165 170 175
Ser Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala Trp
180 185 190
Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala
195 200 205
Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser
210 215 220
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu
225 230 235 240
Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Thr Thr Thr Pro Ala Pro Arg Pro Pro
260 265 270
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
275 280 285
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
290 295 300
Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala
305 310 315 320
Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys
325 330 335
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
340 345 350
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
355 360 365
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
370 375 380
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
385 390 395 400
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
405 410 415
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
420 425 430
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
435 440 445
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
450 455 460
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
465 470 475 480
Leu His Met Gln Ala Leu Pro Pro Arg
485
<210> 93
<211> 672
<212> PRT
<213> artificial sequence
<220>
<223> M5 scCAR-v5 polypeptide
<400> 93
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu
20 25 30
Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe
35 40 45
Asn Ile Lys Asp Asp Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln
50 55 60
Gly Leu Glu Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala
65 70 75 80
Tyr Ala Ser Lys Phe Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser
85 90 95
Ser Asn Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Ser Gln Ser Pro Ser
145 150 155 160
Ser Leu Ala Val Ser Val Gly Glu Lys Val Thr Met Ser Cys Lys Ser
165 170 175
Ser Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala Trp
180 185 190
Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala
195 200 205
Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser
210 215 220
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu
225 230 235 240
Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His
260 265 270
Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
275 280 285
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro
290 295 300
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
305 310 315 320
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
325 330 335
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
340 345 350
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
355 360 365
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
370 375 380
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
385 390 395 400
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
405 410 415
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
420 425 430
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
435 440 445
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
450 455 460
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
465 470 475 480
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Tyr
485 490 495
Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu
500 505 510
Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile
515 520 525
Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp
530 535 540
Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
545 550 555 560
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
565 570 575
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
580 585 590
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
595 600 605
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
610 615 620
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
625 630 635 640
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
645 650 655
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
660 665 670
<210> 94
<211> 675
<212> PRT
<213> artificial sequence
<220>
<223> M5 scCAR-v6 polypeptide
<400> 94
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu
20 25 30
Val Arg Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe
35 40 45
Asn Ile Lys Asp Asp Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln
50 55 60
Gly Leu Glu Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala
65 70 75 80
Tyr Ala Ser Lys Phe Gln Asp Lys Ala Thr Ile Thr Ser Asp Thr Ser
85 90 95
Ser Asn Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Thr Leu Thr Gly Arg Phe Asp Tyr Trp Gly Gln
115 120 125
Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Asp Ile Val Met Ser Gln Ser Pro Ser
145 150 155 160
Ser Leu Ala Val Ser Val Gly Glu Lys Val Thr Met Ser Cys Lys Ser
165 170 175
Ser Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala Trp
180 185 190
Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala
195 200 205
Ser Thr Arg Glu Ser Gly Val Pro Asp Arg Phe Thr Gly Ser Gly Ser
210 215 220
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Leu
225 230 235 240
Ala Val Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly
245 250 255
Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser Pro Asp Lys Thr His
260 265 270
Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe
275 280 285
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro
290 295 300
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
305 310 315 320
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
325 330 335
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
340 345 350
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
355 360 365
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
370 375 380
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
385 390 395 400
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
405 410 415
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
420 425 430
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
435 440 445
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
450 455 460
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
465 470 475 480
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Ile
485 490 495
Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe
500 505 510
Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu
515 520 525
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
530 535 540
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
545 550 555 560
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
565 570 575
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
580 585 590
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
595 600 605
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
610 615 620
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
625 630 635 640
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
645 650 655
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
660 665 670
Pro Pro Arg
675
<210> 95
<211> 461
<212> PRT
<213> artificial sequence
<220>
<223> G12 scCAR-v1 polypeptide
<400> 95
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Pro Ser Ser Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser
65 70 75 80
Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Ile Tyr Phe Ser Cys Cys Ala Arg Val Tyr Asn Trp His Phe Asp
115 120 125
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Ile Val Leu Thr
145 150 155 160
Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile
165 170 175
Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met
180 185 190
Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr
195 200 205
Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
210 215 220
Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val Glu Ala Asp
225 230 235 240
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Glu Asp Pro Tyr Thr
245 250 255
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Gly Leu Ala Val Ser
260 265 270
Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala
275 280 285
Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr
290 295 300
Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
305 310 315 320
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
325 330 335
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
340 345 350
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
355 360 365
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
370 375 380
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
385 390 395 400
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
405 410 415
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
420 425 430
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
435 440 445
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 96
<211> 464
<212> PRT
<213> artificial sequence
<220>
<223> G12 scCAR-v2 polypeptide
<400> 96
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Pro Ser Ser Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser
65 70 75 80
Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Ile Tyr Phe Ser Cys Cys Ala Arg Val Tyr Asn Trp His Phe Asp
115 120 125
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Ile Val Leu Thr
145 150 155 160
Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile
165 170 175
Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met
180 185 190
Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr
195 200 205
Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
210 215 220
Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val Glu Ala Asp
225 230 235 240
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Glu Asp Pro Tyr Thr
245 250 255
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Gly Leu Ala Val Ser
260 265 270
Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe
275 280 285
Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr
290 295 300
Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile
305 310 315 320
Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp
325 330 335
Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
340 345 350
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
355 360 365
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
370 375 380
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
385 390 395 400
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
405 410 415
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
420 425 430
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
435 440 445
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 97
<211> 490
<212> PRT
<213> artificial sequence
<220>
<223> G12 scCAR-v3 polypeptide
<400> 97
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Pro Ser Ser Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser
65 70 75 80
Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Ile Tyr Phe Ser Cys Cys Ala Arg Val Tyr Asn Trp His Phe Asp
115 120 125
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Ile Val Leu Thr
145 150 155 160
Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile
165 170 175
Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met
180 185 190
Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr
195 200 205
Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
210 215 220
Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val Glu Ala Asp
225 230 235 240
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Glu Asp Pro Tyr Thr
245 250 255
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Thr Thr Pro Ala
260 265 270
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
275 280 285
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
290 295 300
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
305 310 315 320
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
325 330 335
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
340 345 350
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
355 360 365
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg
370 375 380
Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn
385 390 395 400
Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg
405 410 415
Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro
420 425 430
Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala
435 440 445
Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His
450 455 460
Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp
465 470 475 480
Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 98
<211> 493
<212> PRT
<213> artificial sequence
<220>
<223> G12 scCAR-v4 polypeptide
<400> 98
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Pro Ser Ser Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser
65 70 75 80
Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Ile Tyr Phe Ser Cys Cys Ala Arg Val Tyr Asn Trp His Phe Asp
115 120 125
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Ile Val Leu Thr
145 150 155 160
Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile
165 170 175
Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met
180 185 190
Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr
195 200 205
Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
210 215 220
Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val Glu Ala Asp
225 230 235 240
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Glu Asp Pro Tyr Thr
245 250 255
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Thr Thr Pro Ala
260 265 270
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
275 280 285
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
290 295 300
Arg Gly Leu Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu
305 310 315 320
Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe
325 330 335
Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
340 345 350
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
355 360 365
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
370 375 380
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
385 390 395 400
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
405 410 415
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
420 425 430
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
435 440 445
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
450 455 460
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
465 470 475 480
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 99
<211> 676
<212> PRT
<213> artificial sequence
<220>
<223> G12 scCAR-v5 polypeptide
<400> 99
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Pro Ser Ser Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser
65 70 75 80
Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Ile Tyr Phe Ser Cys Cys Ala Arg Val Tyr Asn Trp His Phe Asp
115 120 125
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Ile Val Leu Thr
145 150 155 160
Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile
165 170 175
Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met
180 185 190
Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr
195 200 205
Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
210 215 220
Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val Glu Ala Asp
225 230 235 240
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Glu Asp Pro Tyr Thr
245 250 255
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Glu Pro Lys Ser Pro
260 265 270
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
275 280 285
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
290 295 300
Ala Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
305 310 315 320
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
325 330 335
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
340 345 350
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
355 360 365
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
370 375 380
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
385 390 395 400
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
405 410 415
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
420 425 430
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
435 440 445
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
450 455 460
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
465 470 475 480
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
485 490 495
Gly Lys Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
500 505 510
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
515 520 525
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
530 535 540
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
545 550 555 560
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
565 570 575
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
580 585 590
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
595 600 605
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
610 615 620
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
625 630 635 640
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
645 650 655
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
660 665 670
Leu Pro Pro Arg
675
<210> 100
<211> 679
<212> PRT
<213> artificial sequence
<220>
<223> G12 scCAR-v6 polypeptide
<400> 100
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Ala Ser Met Lys Met Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Pro Ser Ser Asn Ile His Trp Leu Lys Gln Thr Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser
65 70 75 80
Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr Asp Lys Ser
85 90 95
Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Ile Tyr Phe Ser Cys Cys Ala Arg Val Tyr Asn Trp His Phe Asp
115 120 125
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Ile Val Leu Thr
145 150 155 160
Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly Gln Arg Ala Thr Ile
165 170 175
Ser Cys Arg Ala Ser Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met
180 185 190
Leu Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Leu Leu Ile Tyr
195 200 205
Phe Ala Ser Asn Leu Glu Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
210 215 220
Gly Ser Arg Thr Asp Phe Thr Leu Thr Ile Asp Pro Val Glu Ala Asp
225 230 235 240
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Asn Asn Glu Asp Pro Tyr Thr
245 250 255
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Glu Pro Lys Ser Pro
260 265 270
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly
275 280 285
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
290 295 300
Ala Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
305 310 315 320
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
325 330 335
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
340 345 350
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
355 360 365
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
370 375 380
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
385 390 395 400
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
405 410 415
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
420 425 430
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
435 440 445
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
450 455 460
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
465 470 475 480
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
485 490 495
Gly Lys Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu
500 505 510
Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly
515 520 525
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
530 535 540
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
545 550 555 560
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
565 570 575
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
580 585 590
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
595 600 605
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
610 615 620
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
625 630 635 640
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
645 650 655
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
660 665 670
Met Gln Ala Leu Pro Pro Arg
675
<210> 101
<211> 456
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 scCAR-v1 polypeptide
<400> 101
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Thr Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser
65 70 75 80
Tyr Asn Glu Lys Val Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser
85 90 95
Ser Thr Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Val Leu
145 150 155 160
Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly Glu Lys Val Thr
165 170 175
Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met His Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe Ala Thr Ser Asn
195 200 205
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
210 215 220
Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu Asp Ala Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr Phe Gly Ala
245 250 255
Gly Thr Lys Leu Glu Leu Gly Leu Ala Val Ser Thr Ile Ser Ser Phe
260 265 270
Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr
275 280 285
Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg
290 295 300
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
305 310 315 320
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
325 330 335
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
340 345 350
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
355 360 365
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
370 375 380
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
385 390 395 400
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
405 410 415
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
420 425 430
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
435 440 445
His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 102
<211> 459
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 scCAR-v2 polypeptide
<400> 102
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Thr Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser
65 70 75 80
Tyr Asn Glu Lys Val Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser
85 90 95
Ser Thr Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Val Leu
145 150 155 160
Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly Glu Lys Val Thr
165 170 175
Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met His Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe Ala Thr Ser Asn
195 200 205
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
210 215 220
Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu Asp Ala Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr Phe Gly Ala
245 250 255
Gly Thr Lys Leu Glu Leu Gly Leu Ala Val Ser Thr Ile Ser Ser Phe
260 265 270
Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser
275 280 285
Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val
290 295 300
Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
305 310 315 320
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
325 330 335
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser
340 345 350
Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr
355 360 365
Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys
370 375 380
Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn
385 390 395 400
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
405 410 415
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
420 425 430
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
435 440 445
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455
<210> 103
<211> 485
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 scCAR-v3 polypeptide
<400> 103
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Thr Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser
65 70 75 80
Tyr Asn Glu Lys Val Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser
85 90 95
Ser Thr Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Val Leu
145 150 155 160
Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly Glu Lys Val Thr
165 170 175
Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met His Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe Ala Thr Ser Asn
195 200 205
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
210 215 220
Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu Asp Ala Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr Phe Gly Ala
245 250 255
Gly Thr Lys Leu Glu Leu Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr
260 265 270
Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala
275 280 285
Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe
290 295 300
Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val
305 310 315 320
Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys
325 330 335
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
340 345 350
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
355 360 365
Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro
370 375 380
Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly
385 390 395 400
Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro
405 410 415
Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
420 425 430
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
435 440 445
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
450 455 460
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
465 470 475 480
Ala Leu Pro Pro Arg
485
<210> 104
<211> 488
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 scCAR-v4 polypeptide
<400> 104
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Thr Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser
65 70 75 80
Tyr Asn Glu Lys Val Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser
85 90 95
Ser Thr Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Val Leu
145 150 155 160
Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly Glu Lys Val Thr
165 170 175
Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met His Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe Ala Thr Ser Asn
195 200 205
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
210 215 220
Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu Asp Ala Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr Phe Gly Ala
245 250 255
Gly Thr Lys Leu Glu Leu Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr
260 265 270
Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala
275 280 285
Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe
290 295 300
Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu
305 310 315 320
Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg
325 330 335
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
340 345 350
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
355 360 365
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
370 375 380
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
385 390 395 400
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
405 410 415
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
420 425 430
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
435 440 445
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
450 455 460
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
465 470 475 480
His Met Gln Ala Leu Pro Pro Arg
485
<210> 105
<211> 671
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 scCAR-v5 polypeptide
<400> 105
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Thr Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser
65 70 75 80
Tyr Asn Glu Lys Val Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser
85 90 95
Ser Thr Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Val Leu
145 150 155 160
Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly Glu Lys Val Thr
165 170 175
Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met His Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe Ala Thr Ser Asn
195 200 205
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
210 215 220
Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu Asp Ala Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr Phe Gly Ala
245 250 255
Gly Thr Lys Leu Glu Leu Glu Pro Lys Ser Pro Asp Lys Thr His Thr
260 265 270
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
275 280 285
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu
290 295 300
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
305 310 315 320
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
325 330 335
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
340 345 350
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
355 360 365
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
370 375 380
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
385 390 395 400
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
405 410 415
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
420 425 430
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
435 440 445
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
450 455 460
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
465 470 475 480
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Tyr Ile
485 490 495
Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val
500 505 510
Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
515 520 525
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
530 535 540
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
545 550 555 560
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
565 570 575
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
580 585 590
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
595 600 605
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
610 615 620
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
625 630 635 640
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
645 650 655
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
660 665 670
<210> 106
<211> 674
<212> PRT
<213> artificial sequence
<220>
<223> 21.26 scCAR-v6 polypeptide
<400> 106
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu
20 25 30
Val Lys Pro Gly Thr Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr
35 40 45
Thr Phe Thr Arg Tyr Trp Met His Trp Val Lys Gln Arg Pro Gly Gln
50 55 60
Gly Leu Glu Trp Ile Gly Met Ile His Pro Ser Ser Gly Ser Thr Ser
65 70 75 80
Tyr Asn Glu Lys Val Lys Asn Lys Ala Thr Leu Thr Val Asp Arg Ser
85 90 95
Ser Thr Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser
100 105 110
Ala Val Tyr Tyr Cys Ala Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ile Val Leu
145 150 155 160
Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly Glu Lys Val Thr
165 170 175
Met Thr Cys Arg Ala Ser Ser Ser Ile Asn Tyr Met His Trp Tyr Gln
180 185 190
Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Phe Ala Thr Ser Asn
195 200 205
Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr
210 215 220
Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu Asp Ala Ala Thr
225 230 235 240
Tyr Tyr Cys Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr Phe Gly Ala
245 250 255
Gly Thr Lys Leu Glu Leu Glu Pro Lys Ser Pro Asp Lys Thr His Thr
260 265 270
Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu
275 280 285
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala Arg Thr Pro Glu
290 295 300
Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys
305 310 315 320
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
325 330 335
Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu
340 345 350
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
355 360 365
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
370 375 380
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
385 390 395 400
Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
405 410 415
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
420 425 430
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
435 440 445
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
450 455 460
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
465 470 475 480
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Ile Ser
485 490 495
Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe
500 505 510
Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu
515 520 525
Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu
530 535 540
Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys
545 550 555 560
Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
565 570 575
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
580 585 590
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
595 600 605
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
610 615 620
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
625 630 635 640
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
645 650 655
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
660 665 670
Pro Arg
<210> 107
<211> 460
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 scCAR-v1 polypeptide
<400> 107
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr
35 40 45
Ser Ile Thr Ser Ala Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly
50 55 60
Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser
85 90 95
Lys Asn Gln Phe Phe Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr
100 105 110
Ala Thr Tyr Tyr Cys Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr
115 120 125
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
145 150 155 160
Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly Glu
165 170 175
Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr
180 185 190
Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp Ile
195 200 205
Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala
225 230 235 240
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Leu
245 250 255
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Gly Leu Ala Val Ser Thr
260 265 270
Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Tyr Ile Trp Ala Pro
275 280 285
Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu
290 295 300
Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
305 310 315 320
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
325 330 335
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 108
<211> 463
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 scCAR-v2 polypeptide
<400> 108
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr
35 40 45
Ser Ile Thr Ser Ala Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly
50 55 60
Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser
85 90 95
Lys Asn Gln Phe Phe Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr
100 105 110
Ala Thr Tyr Tyr Cys Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr
115 120 125
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
145 150 155 160
Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly Glu
165 170 175
Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr
180 185 190
Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp Ile
195 200 205
Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala
225 230 235 240
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Leu
245 250 255
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Gly Leu Ala Val Ser Thr
260 265 270
Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln Ile Ile Ser Phe Phe Leu
275 280 285
Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu
290 295 300
Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
305 310 315 320
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
325 330 335
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg
340 345 350
Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln
355 360 365
Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp
370 375 380
Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro
385 390 395 400
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
405 410 415
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
420 425 430
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
435 440 445
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 109
<211> 489
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 scCAR-v3 polypeptide
<400> 109
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr
35 40 45
Ser Ile Thr Ser Ala Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly
50 55 60
Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser
85 90 95
Lys Asn Gln Phe Phe Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr
100 105 110
Ala Thr Tyr Tyr Cys Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr
115 120 125
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
145 150 155 160
Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly Glu
165 170 175
Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr
180 185 190
Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp Ile
195 200 205
Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala
225 230 235 240
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Leu
245 250 255
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Thr Thr Thr Pro Ala Pro
260 265 270
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
275 280 285
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
290 295 300
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
305 310 315 320
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys
325 330 335
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
340 345 350
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
355 360 365
Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser
370 375 380
Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu
385 390 395 400
Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg
405 410 415
Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln
420 425 430
Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr
435 440 445
Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp
450 455 460
Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala
465 470 475 480
Leu His Met Gln Ala Leu Pro Pro Arg
485
<210> 110
<211> 492
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 scCAR-v4 polypeptide
<400> 110
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr
35 40 45
Ser Ile Thr Ser Ala Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly
50 55 60
Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser
85 90 95
Lys Asn Gln Phe Phe Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr
100 105 110
Ala Thr Tyr Tyr Cys Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr
115 120 125
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
145 150 155 160
Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly Glu
165 170 175
Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr
180 185 190
Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp Ile
195 200 205
Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala
225 230 235 240
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Leu
245 250 255
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Thr Thr Thr Pro Ala Pro
260 265 270
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
275 280 285
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
290 295 300
Gly Leu Asp Phe Ala Cys Asp Ile Ile Ser Phe Phe Leu Ala Leu Thr
305 310 315 320
Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser
325 330 335
Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
340 345 350
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
355 360 365
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
370 375 380
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
385 390 395 400
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
405 410 415
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
420 425 430
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
435 440 445
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
450 455 460
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
465 470 475 480
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 111
<211> 675
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 scCAR-v5 polypeptide
<400> 111
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr
35 40 45
Ser Ile Thr Ser Ala Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly
50 55 60
Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser
85 90 95
Lys Asn Gln Phe Phe Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr
100 105 110
Ala Thr Tyr Tyr Cys Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr
115 120 125
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
145 150 155 160
Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly Glu
165 170 175
Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr
180 185 190
Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp Ile
195 200 205
Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala
225 230 235 240
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Leu
245 250 255
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Glu Pro Lys Ser Pro Asp
260 265 270
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro
275 280 285
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala
290 295 300
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
305 310 315 320
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
325 330 335
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
340 345 350
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
355 360 365
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
370 375 380
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
385 390 395 400
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
405 410 415
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
420 425 430
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
435 440 445
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
450 455 460
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
465 470 475 480
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
485 490 495
Lys Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
500 505 510
Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu
515 520 525
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
530 535 540
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
545 550 555 560
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
565 570 575
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
580 585 590
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
595 600 605
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
610 615 620
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
625 630 635 640
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
645 650 655
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
660 665 670
Pro Pro Arg
675
<210> 112
<211> 678
<212> PRT
<213> artificial sequence
<220>
<223> 1075.7 scCAR-v6 polypeptide
<400> 112
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Ile Gln Leu Gln Glu Ser Gly Pro Gly Leu
20 25 30
Val Lys Pro Ser Gln Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr
35 40 45
Ser Ile Thr Ser Ala Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly
50 55 60
Asn Lys Leu Glu Trp Met Gly Tyr Ile Ser Tyr Asp Gly Arg Asn Asn
65 70 75 80
Tyr Asn Pro Ser Leu Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser
85 90 95
Lys Asn Gln Phe Phe Leu Lys Leu Asn Ser Val Thr Thr Glu Asp Thr
100 105 110
Ala Thr Tyr Tyr Cys Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr
115 120 125
Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
145 150 155 160
Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly Glu
165 170 175
Lys Val Thr Met Thr Cys Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr
180 185 190
Val His Trp Tyr Gln Gln Arg Ser Gly Ala Ser Pro Lys Leu Trp Ile
195 200 205
Tyr Ser Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
210 215 220
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala
225 230 235 240
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Leu
245 250 255
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Glu Pro Lys Ser Pro Asp
260 265 270
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro
275 280 285
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ala
290 295 300
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
305 310 315 320
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
325 330 335
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
340 345 350
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
355 360 365
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
370 375 380
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
385 390 395 400
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
405 410 415
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
420 425 430
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
435 440 445
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
450 455 460
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
465 470 475 480
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
485 490 495
Lys Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe
500 505 510
Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val Lys Arg Gly Arg
515 520 525
Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln
530 535 540
Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu
545 550 555 560
Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
565 570 575
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
580 585 590
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
595 600 605
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
610 615 620
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
625 630 635 640
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
645 650 655
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
660 665 670
Gln Ala Leu Pro Pro Arg
675
<210> 113
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CD20 mimotope
<400> 113
Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser
1 5 10
<210> 114
<211> 49
<212> DNA
<213> artificial sequence
<220>
<223> Target TALEN TRAC_T01
<400> 114
ttgtcccaca gatatccaga accctgaccc tgccgtgtac cagctgaga 49
<210> 115
<211> 530
<212> PRT
<213> artificial sequence
<220>
<223> TAL binding domain TRAC_T01-L
<400> 115
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
65 70 75 80
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
100 105 110
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
130 135 140
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala
245 250 255
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
260 265 270
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys
275 280 285
Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala
290 295 300
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
305 310 315 320
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
325 330 335
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
340 345 350
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
355 360 365
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
370 375 380
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
385 390 395 400
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
405 410 415
Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala
420 425 430
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
435 440 445
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
450 455 460
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
465 470 475 480
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
485 490 495
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
500 505 510
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
515 520 525
Leu Glu
530
<210> 116
<211> 530
<212> PRT
<213> artificial sequence
<220>
<223> TAL binding domain TRAC_T01-R
<400> 116
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
65 70 75 80
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
130 135 140
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
245 250 255
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
260 265 270
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
275 280 285
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
290 295 300
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
305 310 315 320
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
325 330 335
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
340 345 350
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
355 360 365
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
370 375 380
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
385 390 395 400
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
405 410 415
Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala
420 425 430
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
435 440 445
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
450 455 460
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
465 470 475 480
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
485 490 495
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
500 505 510
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
515 520 525
Leu Glu
530
<210> 117
<211> 2814
<212> DNA
<213> artificial sequence
<220>
<223> polynucleotide encoding TRAC_T01-L TALEN
<400> 117
atgggcgatc ctaaaaagaa acgtaaggtc atcgattacc catacgatgt tccagattac 60
gctatcgata tcgccgatct acgcacgctc ggctacagcc agcagcaaca ggagaagatc 120
aaaccgaagg ttcgttcgac agtggcgcag caccacgagg cactggtcgg ccacgggttt 180
acacacgcgc acatcgttgc gttaagccaa cacccggcag cgttagggac cgtcgctgtc 240
aagtatcagg acatgatcgc agcgttgcca gaggcgacac acgaagcgat cgttggcgtc 300
ggcaaacagt ggtccggcgc acgcgctctg gaggccttgc tcacggtggc gggagagttg 360
agaggtccac cgttacagtt ggacacaggc caacttctca agattgcaaa acgtggcggc 420
gtgaccgcag tggaggcagt gcatgcatgg cgcaatgcac tgacgggtgc cccgctcaac 480
ttgacccccc agcaggtggt ggccatcgcc agcaatggcg gtggcaagca ggcgctggag 540
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ccagcaggtg 600
gtggccatcg ccagcaataa tggtggcaag caggcgctgg agacggtcca gcggctgttg 660
ccggtgctgt gccaggccca cggcttgacc ccccagcagg tggtggccat cgccagcaat 720
ggcggtggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 780
cacggcttga ccccggagca ggtggtggcc atcgccagcc acgatggcgg caagcaggcg 840
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 900
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 960
ctgttgccgg tgctgtgcca ggcccacggc ttgaccccgg agcaggtggt ggccatcgcc 1020
agccacgatg gcggcaagca ggcgctggag acggtccagc ggctgttgcc ggtgctgtgc 1080
caggcccacg gcttgacccc ggagcaggtg gtggccatcg ccagcaatat tggtggcaag 1140
caggcgctgg agacggtgca ggcgctgttg ccggtgctgt gccaggccca cggcttgacc 1200
ccggagcagg tggtggccat cgccagccac gatggcggca agcaggcgct ggagacggtc 1260
cagcggctgt tgccggtgct gtgccaggcc cacggcttga ccccggagca ggtggtggcc 1320
atcgccagca atattggtgg caagcaggcg ctggagacgg tgcaggcgct gttgccggtg 1380
ctgtgccagg cccacggctt gaccccccag caggtggtgg ccatcgccag caataatggt 1440
ggcaagcagg cgctggagac ggtccagcgg ctgttgccgg tgctgtgcca ggcccacggc 1500
ttgaccccgg agcaggtggt ggccatcgcc agcaatattg gtggcaagca ggcgctggag 1560
acggtgcagg cgctgttgcc ggtgctgtgc caggcccacg gcttgacccc ccagcaggtg 1620
gtggccatcg ccagcaatgg cggtggcaag caggcgctgg agacggtcca gcggctgttg 1680
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagcaat 1740
attggtggca agcaggcgct ggagacggtg caggcgctgt tgccggtgct gtgccaggcc 1800
cacggcttga ccccccagca ggtggtggcc atcgccagca atggcggtgg caagcaggcg 1860
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 1920
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 1980
ctgttgccgg tgctgtgcca ggcccacggc ttgacccctc agcaggtggt ggccatcgcc 2040
agcaatggcg gcggcaggcc ggcgctggag agcattgttg cccagttatc tcgccctgat 2100
ccggcgttgg ccgcgttgac caacgaccac ctcgtcgcct tggcctgcct cggcgggcgt 2160
cctgcgctgg atgcagtgaa aaagggattg ggggatccta tcagccgttc ccagctggtg 2220
aagtccgagc tggaggagaa gaaatccgag ttgaggcaca agctgaagta cgtgccccac 2280
gagtacatcg agctgatcga gatcgcccgg aacagcaccc aggaccgtat cctggagatg 2340
aaggtgatgg agttcttcat gaaggtgtac ggctacaggg gcaagcacct gggcggctcc 2400
aggaagcccg acggcgccat ctacaccgtg ggctccccca tcgactacgg cgtgatcgtg 2460
gacaccaagg cctactccgg cggctacaac ctgcccatcg gccaggccga cgaaatgcag 2520
aggtacgtgg aggagaacca gaccaggaac aagcacatca accccaacga gtggtggaag 2580
gtgtacccct ccagcgtgac cgagttcaag ttcctgttcg tgtccggcca cttcaagggc 2640
aactacaagg cccagctgac caggctgaac cacatcacca actgcaacgg cgccgtgctg 2700
tccgtggagg agctcctgat cggcggcgag atgatcaagg ccggcaccct gaccctggag 2760
gaggtgagga ggaagttcaa caacggcgag atcaacttcg cggccgactg ataa 2814
<210> 118
<211> 2832
<212> DNA
<213> artificial sequence
<220>
<223> polynucleotide encoding TRAC_T01-R TALEN
<400> 118
atgggcgatc ctaaaaagaa acgtaaggtc atcgataagg agaccgccgc tgccaagttc 60
gagagacagc acatggacag catcgatatc gccgatctac gcacgctcgg ctacagccag 120
cagcaacagg agaagatcaa accgaaggtt cgttcgacag tggcgcagca ccacgaggca 180
ctggtcggcc acgggtttac acacgcgcac atcgttgcgt taagccaaca cccggcagcg 240
ttagggaccg tcgctgtcaa gtatcaggac atgatcgcag cgttgccaga ggcgacacac 300
gaagcgatcg ttggcgtcgg caaacagtgg tccggcgcac gcgctctgga ggccttgctc 360
acggtggcgg gagagttgag aggtccaccg ttacagttgg acacaggcca acttctcaag 420
attgcaaaac gtggcggcgt gaccgcagtg gaggcagtgc atgcatggcg caatgcactg 480
acgggtgccc cgctcaactt gaccccggag caggtggtgg ccatcgccag ccacgatggc 540
ggcaagcagg cgctggagac ggtccagcgg ctgttgccgg tgctgtgcca ggcccacggc 600
ttgacccccc agcaggtggt ggccatcgcc agcaatggcg gtggcaagca ggcgctggag 660
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ggagcaggtg 720
gtggccatcg ccagccacga tggcggcaag caggcgctgg agacggtcca gcggctgttg 780
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagcaat 840
attggtggca agcaggcgct ggagacggtg caggcgctgt tgccggtgct gtgccaggcc 900
cacggcttga ccccccagca ggtggtggcc atcgccagca ataatggtgg caagcaggcg 960
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 1020
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 1080
ctgttgccgg tgctgtgcca ggcccacggc ttgacccccc agcaggtggt ggccatcgcc 1140
agcaatggcg gtggcaagca ggcgctggag acggtccagc ggctgttgcc ggtgctgtgc 1200
caggcccacg gcttgacccc ccagcaggtg gtggccatcg ccagcaataa tggtggcaag 1260
caggcgctgg agacggtcca gcggctgttg ccggtgctgt gccaggccca cggcttgacc 1320
ccccagcagg tggtggccat cgccagcaat aatggtggca agcaggcgct ggagacggtc 1380
cagcggctgt tgccggtgct gtgccaggcc cacggcttga ccccccagca ggtggtggcc 1440
atcgccagca atggcggtgg caagcaggcg ctggagacgg tccagcggct gttgccggtg 1500
ctgtgccagg cccacggctt gaccccggag caggtggtgg ccatcgccag caatattggt 1560
ggcaagcagg cgctggagac ggtgcaggcg ctgttgccgg tgctgtgcca ggcccacggc 1620
ttgaccccgg agcaggtggt ggccatcgcc agccacgatg gcggcaagca ggcgctggag 1680
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ggagcaggtg 1740
gtggccatcg ccagcaatat tggtggcaag caggcgctgg agacggtgca ggcgctgttg 1800
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagccac 1860
gatggcggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 1920
cacggcttga ccccccagca ggtggtggcc atcgccagca ataatggtgg caagcaggcg 1980
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gacccctcag 2040
caggtggtgg ccatcgccag caatggcggc ggcaggccgg cgctggagag cattgttgcc 2100
cagttatctc gccctgatcc ggcgttggcc gcgttgacca acgaccacct cgtcgccttg 2160
gcctgcctcg gcgggcgtcc tgcgctggat gcagtgaaaa agggattggg ggatcctatc 2220
agccgttccc agctggtgaa gtccgagctg gaggagaaga aatccgagtt gaggcacaag 2280
ctgaagtacg tgccccacga gtacatcgag ctgatcgaga tcgcccggaa cagcacccag 2340
gaccgtatcc tggagatgaa ggtgatggag ttcttcatga aggtgtacgg ctacaggggc 2400
aagcacctgg gcggctccag gaagcccgac ggcgccatct acaccgtggg ctcccccatc 2460
gactacggcg tgatcgtgga caccaaggcc tactccggcg gctacaacct gcccatcggc 2520
caggccgacg aaatgcagag gtacgtggag gagaaccaga ccaggaacaa gcacatcaac 2580
cccaacgagt ggtggaaggt gtacccctcc agcgtgaccg agttcaagtt cctgttcgtg 2640
tccggccact tcaagggcaa ctacaaggcc cagctgacca ggctgaacca catcaccaac 2700
tgcaacggcg ccgtgctgtc cgtggaggag ctcctgatcg gcggcgagat gatcaaggcc 2760
ggcaccctga ccctggagga ggtgaggagg aagttcaaca acggcgagat caacttcgcg 2820
gccgactgat aa 2832
<210> 119
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of SC02-357 heavy chain variable region
<400> 119
Gly Ser Ile Ser Ser Ser Asn Trp Trp Ser
1 5 10
<210> 120
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of SC02-357 heavy chain variable region
<400> 120
Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asp Tyr
1 5 10
<210> 121
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of SC02-357 heavy chain variable region
<400> 121
Lys Val Ser Thr Gly Gly Phe Phe Asp Tyr
1 5 10
<210> 122
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of SC02-378 heavy chain variable region
<400> 122
Gly Ser Ile Ser Ser Ser Asn Trp Trp Ser
1 5 10
<210> 123
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of SC02-378 heavy chain variable region
<400> 123
Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn Tyr
1 5 10
<210> 124
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of SC02-378 heavy chain variable region
<400> 124
Arg Ser Ser Ser Gly Gly Phe Phe Asp Tyr
1 5 10
<210> 125
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of SC02-161 heavy chain variable region
<400> 125
Gly Ser Ile Ser Ser Ser Asn Trp Trp Ser
1 5 10
<210> 126
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of SC02-161 heavy chain variable region
<400> 126
Trp Ile Gly Glu Ile Tyr His Ser Gly Ser Pro Asn Tyr
1 5 10
<210> 127
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of SC02-161 heavy chain variable region
<400> 127
Arg Gln Thr Thr Ala Gly Ser Phe Asp Tyr
1 5 10
<210> 128
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of SC02-357 & SC02-378 & SC02-161 light chain variable region
<400> 128
Gln Ser Ile Ser Ser Tyr Leu Asn
1 5
<210> 129
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of SC02-357 & SC02-378 & SC02-161 light chain variable region
<400> 129
Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
1 5 10
<210> 130
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of SC02-357 & SC02-378 & SC02-161 light chain variable region
<400> 130
Gln Gln Ser Tyr Ser Thr Pro Pro
1 5
<210> 131
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M26 heavy chain variable region
<400> 131
Gly Tyr Thr Phe Thr Ser Tyr Phe Ile His
1 5 10
<210> 132
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M26 heavy chain variable region
<400> 132
Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Ser Lys Tyr
1 5 10
<210> 133
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M26 heavy chain variable region
<400> 133
Thr Arg Asp Asp Gly Tyr Tyr Gly Tyr Ala Met Asp Tyr
1 5 10
<210> 134
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M26 light chain variable region
<400> 134
Gln Glu Leu Ser Gly Tyr Leu Ser
1 5
<210> 135
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M26 light chain variable region
<400> 135
Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser
1 5 10
<210> 136
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M26 light chain variable region
<400> 136
Leu Gln Tyr Ala Ile Tyr Pro Tyr
1 5
<210> 137
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M31 heavy chain variable region
<400> 137
Gly Tyr Thr Phe Thr Ser Tyr Val Met His
1 5 10
<210> 138
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M31 heavy chain variable region
<400> 138
Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Lys Tyr
1 5 10
<210> 139
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M31 heavy chain variable region
<400> 139
Ala Arg Pro Ile Tyr Phe Asp Asn Asp Tyr
1 5 10
<210> 140
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M31 light chain variable region
<400> 140
Glu Ser Val Asp Ser Tyr Gly Asn Ser Phe Met His
1 5 10
<210> 141
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M31 light chain variable region
<400> 141
Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser
1 5 10
<210> 142
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M31 light chain variable region
<400> 142
Gln Gln Asn Asn Tyr Asp Pro Trp
1 5
<210> 143
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of G4 heavy chain variable region
<400> 143
Gln Gln Asn Asn Tyr Asp Pro Trp
1 5
<210> 144
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of G4 heavy chain variable region
<400> 144
Trp Ile Gly Pro Ile Asn Pro Tyr Asn Asp Gly Thr Ile Tyr
1 5 10
<210> 145
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of G4 heavy chain variable region
<400> 145
Ala Arg Thr Asp Asp Tyr Asp Asp Tyr Thr Met Asp Tyr
1 5 10
<210> 146
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of G4 light chain variable region
<400> 146
His Asp Ile Ser Asn Tyr Leu Asn
1 5
<210> 147
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of G4 light chain variable region
<400> 147
Leu Leu Ile Tyr Tyr Thr Ser Arg Leu His Ser
1 5 10
<210> 148
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of G4 light chain variable region
<400> 148
Gln Gln Gly Lys Thr Leu Leu Trp
1 5
<210> 149
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M22 heavy chain variable region
<400> 149
Gly Tyr Thr Phe Thr Arg Tyr Trp Met His
1 5 10
<210> 150
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M22 heavy chain variable region
<400> 150
Trp Ile Gly Asn Ile Asp Pro Ser Asp Thr Glu Thr His Tyr
1 5 10
<210> 151
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M22 heavy chain variable region
<400> 151
Ala Ile Tyr Tyr Gly Asn Pro Ser Tyr Tyr Ala Met Asp Tyr
1 5 10
<210> 152
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M22 light chain variable region
<400> 152
Gln Asn Leu Leu Asn Ser Gly Asn Gln Lys Lys Tyr Leu Asn
1 5 10
<210> 153
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M22 light chain variable region
<400> 153
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser
1 5 10
<210> 154
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M22 light chain variable region
<400> 154
Gln Asn Asp Tyr Ser Tyr Pro Phe
1 5
<210> 155
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M29 heavy chain variable region
<400> 155
Gly Tyr Ile Phe Thr Ser Tyr Val Met Tyr
1 5 10
<210> 156
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M29 heavy chain variable region
<400> 156
Trp Ile Gly Tyr Ile Asn Pro Tyr
1 5
<210> 157
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M29 heavy chain variable region
<400> 157
Ala Arg Tyr Tyr Asp Tyr Asp Tyr Tyr Phe Asp Tyr
1 5 10
<210> 158
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M29 light chain variable region
<400> 158
Gln Asp Ile Asn Lys Tyr Ile Ala
1 5
<210> 159
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M29 light chain variable region
<400> 159
Leu Leu Ile His Tyr Thr Ser Thr Leu Gln Pro
1 5 10
<210> 160
<211> 7
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M29 light chain variable region
<400> 160
Leu Gln Tyr Asp Tyr Leu Trp
1 5
<210> 161
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M2 heavy chain variable region
<400> 161
Gly Tyr Thr Phe Thr Ser Tyr Phe Met His
1 5 10
<210> 162
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M2 heavy chain variable region
<400> 162
Trp Ile Gly Phe Ile Asn Pro Tyr Asn Asp Gly Thr Lys Tyr
1 5 10
<210> 163
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M2 heavy chain variable region
<400> 163
Thr Arg Asp Asp Gly Tyr Tyr Asp Tyr Ala Met Asp Tyr
1 5 10
<210> 164
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M2 light chain variable region
<400> 164
Gln Glu Ile Ser Val Tyr Leu Ser
1 5
<210> 165
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M2 light chain variable region
<400> 165
Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser
1 5 10
<210> 166
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M2 light chain variable region
<400> 166
Leu Gln Tyr Ala Ser Tyr Pro Tyr
1 5
<210> 167
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M5 heavy chain variable region
<400> 167
Gly Phe Asn Ile Lys Asp Asp Tyr Ile His
1 5 10
<210> 168
<211> 16
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M5 heavy chain variable region
<400> 168
Trp Ile Gly Trp Ile Asp Pro Glu Lys Gly Asp Thr Ala Tyr Ala Ser
1 5 10 15
<210> 169
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M5 heavy chain variable region
<400> 169
Thr Leu Thr Gly Arg Phe Asp Tyr
1 5
<210> 170
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of M5 light chain variable region
<400> 170
Gln Ser Leu Leu Tyr Ser Ser Asn Gln Lys Asn Asn Leu Ala
1 5 10
<210> 171
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of M5 light chain variable region
<400> 171
Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser
1 5 10
<210> 172
<211> 7
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of M5 light chain variable region
<400> 172
Gln Gln Tyr Tyr Ser Tyr Arg
1 5
<210> 173
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of G12 heavy chain variable region
<400> 173
Gly Tyr Thr Phe Pro Ser Ser Asn Ile His
1 5 10
<210> 174
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of G12 heavy chain variable region
<400> 174
Trp Ile Gly Val Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr
1 5 10
<210> 175
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of G12 heavy chain variable region
<400> 175
Ala Ile Tyr Phe Val Tyr Asn Trp His Phe Asp Val
1 5 10
<210> 176
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of G12 light chain variable region
<400> 176
Glu Ser Val Asp Gly Tyr Gly Asp Ile Phe Met Leu
1 5 10
<210> 177
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of G12 light chain variable region
<400> 177
Leu Leu Ile Tyr Phe Ala Ser Asn Leu Glu Ser
1 5 10
<210> 178
<211> 8
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of G12 light chain variable region
<400> 178
Gln Gln Asn Asn Glu Asp Pro Tyr
1 5
<210> 179
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of 21.26 heavy chain variable region
<400> 179
Gly Tyr Thr Phe Thr Arg Tyr Trp Met His
1 5 10
<210> 180
<211> 16
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of 21.26 heavy chain variable region
<400> 180
Met Ile His Pro Ser Ser Gly Ser Thr Ser Tyr Asn Glu Lys Val Lys
1 5 10 15
<210> 181
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of 21.26 heavy chain variable region
<400> 181
Arg Asp Gly Asp Tyr Tyr Tyr Gly Thr Gly Asp Tyr
1 5 10
<210> 182
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of 21.26 light chain variable region
<400> 182
Arg Ala Ser Ser Ser Ile Asn Tyr Met His
1 5 10
<210> 183
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of 21.26 light chain variable region
<400> 183
Pro Trp Ile Phe Ala Thr Ser Asn Leu Ala Ser
1 5 10
<210> 184
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of 21.26 light chain variable region
<400> 184
Gln Gln Trp Arg Ser Asp Arg Ala Leu Thr
1 5 10
<210> 185
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of 1075.7 heavy chain variable region
<400> 185
Gly Tyr Ser Ile Thr Ser Ala Tyr Tyr Trp Asn
1 5 10
<210> 186
<211> 16
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of 1075.7 heavy chain variable region
<400> 186
Tyr Ile Ser Tyr Asp Gly Arg Asn Asn Tyr Asn Pro Ser Leu Lys Asn
1 5 10 15
<210> 187
<211> 16
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of 1075.7 heavy chain variable region
<400> 187
Ala Lys Glu Gly Asp Tyr Asp Val Gly Asn Tyr Tyr Ala Met Asp Tyr
1 5 10 15
<210> 188
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> CDR1 of 1075.7 light chain variable region
<400> 188
Arg Ala Ser Ser Asn Val Ile Ser Ser Tyr Val His
1 5 10
<210> 189
<211> 11
<212> PRT
<213> artificial sequence
<220>
<223> CDR2 of 1075.7 light chain variable region
<400> 189
Leu Trp Ile Tyr Ser Thr Ser Asn Leu Ala Ser
1 5 10
<210> 190
<211> 9
<212> PRT
<213> artificial sequence
<220>
<223> CDR3 of 1075.7 light chain variable region
<400> 190
Gln Gln Tyr Ser Gly Tyr Pro Leu Thr
1 5
<210> 191
<211> 24
<212> PRT
<213> artificial sequence
<220>
<223> Epitope for palivizumab
<400> 191
Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp
1 5 10 15
Gln Lys Lys Leu Met Ser Asn Asn
20
<210> 192
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> Mimotope 1 for cetuximab
<400> 192
Cys Gln Phe Asp Leu Ser Thr Arg Arg Leu Lys Cys
1 5 10
<210> 193
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> Mimotope 2 for cetuximab
<400> 193
Cys Gln Tyr Asn Leu Ser Ser Arg Ala Leu Lys Cys
1 5 10
<210> 194
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> Mimotope 3 for cetuximab
<400> 194
Cys Val Trp Gln Arg Trp Gln Lys Ser Tyr Val Cys
1 5 10
<210> 195
<211> 12
<212> PRT
<213> artificial sequence
<220>
<223> Mimotope 4 for cetuximab
<400> 195
Cys Met Trp Asp Arg Phe Ser Arg Trp Tyr Lys Cys
1 5 10
<210> 196
<211> 25
<212> PRT
<213> artificial sequence
<220>
<223> Epitope 1 for nivolumab
<400> 196
Ser Phe Val Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp
1 5 10 15
Lys Leu Ala Ala Phe Pro Glu Asp Arg
20 25
<210> 197
<211> 19
<212> PRT
<213> artificial sequence
<220>
<223> Epitope 2 for nivolumab
<400> 197
Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu Ala Pro Lys Ala Gln
1 5 10 15
Ile Lys Glu
<210> 198
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> Epitope 1 for CD34
<400> 198
Thr Phe Ser Asn Val Ser Thr Asn Val Ser Tyr Gln Glu Thr
1 5 10
<210> 199
<211> 14
<212> PRT
<213> artificial sequence
<220>
<223> Epitope 2 for CD34
<400> 199
Asn Thr Asn Ser Ser Val Gln Ser Gln Thr Ser Val Ile Ser
1 5 10
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Химерные антигенные рецепторы, нацеливающиеся на CD70 | 2019 |
|
RU2801824C2 |
| Т-КЛЕТКИ С ДВУМЯ ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ CAR | 2021 |
|
RU2839662C1 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА АНТИГЕН СОЗРЕВАНИЯ В-КЛЕТОК, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2829097C2 |
| Т-КЛЕТКИ С КОСТИМУЛИРУЮЩИМ ХИМЕРНЫМ АНТИГЕННЫМ РЕЦЕПТОРОМ, НАЦЕЛЕННЫЕ НА IL13Rα2 | 2015 |
|
RU2749922C2 |
| Т-КЛЕТКИ, МОДИФИЦИРОВАННЫЕ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА, НАЦЕЛЕННЫМ НА CS1, ДЛЯ ЛЕЧЕНИЯ АМИЛОИДОЗА AL | 2018 |
|
RU2774895C2 |
| CD123-СПЕЦИФИЧЕСКИЕ ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ ДЛЯ ИММУНОТЕРАПИИ РАКА | 2015 |
|
RU2727290C2 |
| ВИДЫ КОМБИНИРОВАННОЙ ТЕРАПИИ С ИСПОЛЬЗОВАНИЕМ ХИМЕРНЫХ АНТИГЕННЫХ РЕЦЕПТОРОВ И ИНГИБИТОРОВ PD-1 | 2017 |
|
RU2809160C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА ПОДОБНЫЙ FC-РЕЦЕПТОРУ БЕЛОК 5, И ИХ ПРИМЕНЕНИЕ | 2015 |
|
RU2779747C2 |
| Т-КЛЕТКИ, МОДИФИЦИРОВАННЫЕ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА, НАЦЕЛЕННЫМ НА CS1 | 2015 |
|
RU2727451C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА FLT3 | 2018 |
|
RU2820859C2 |

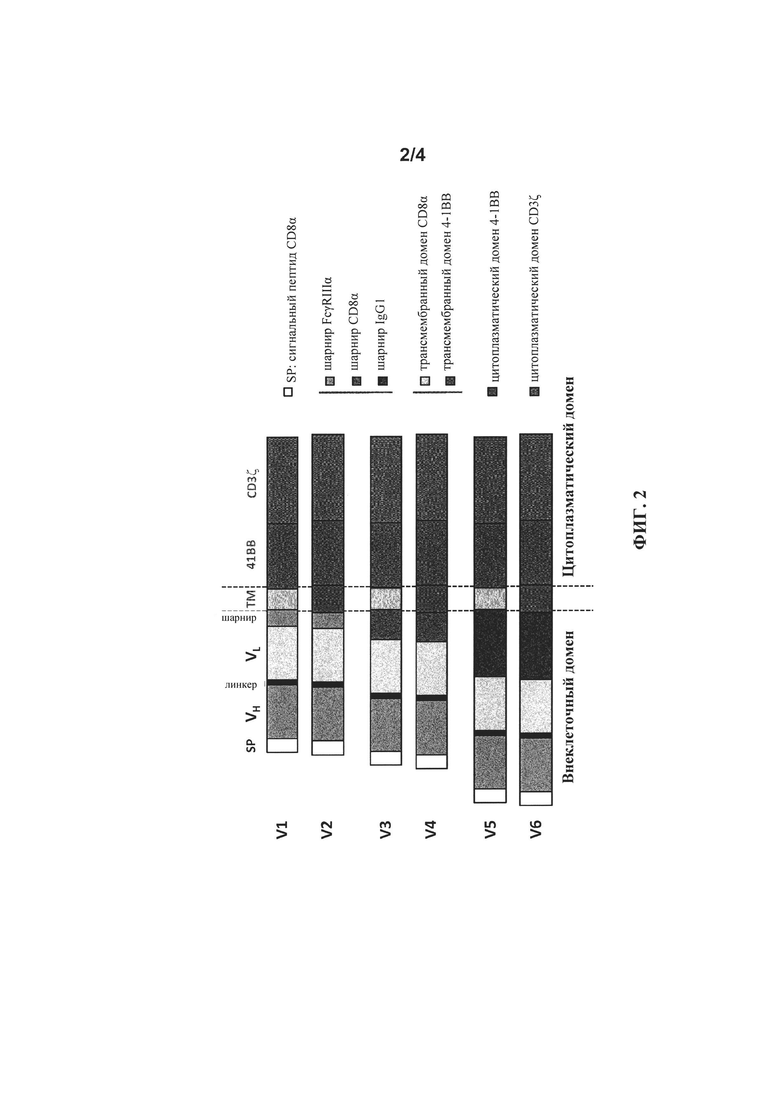
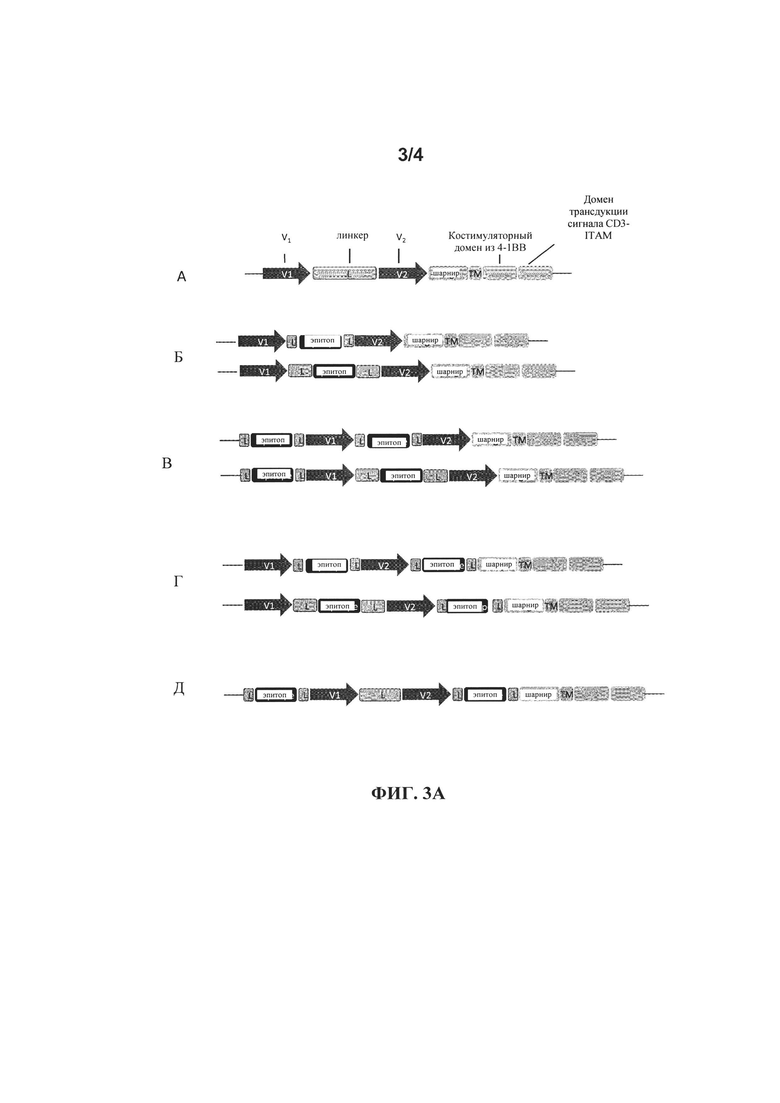

Изобретение относится к области биотехнологии. Предложен CLL1-специфический химерный антигенный рецептор (CAR), обладающий способностью перенаправлять специфичность и реактивность иммунных клеток на CLL1-позитивные клетки. Также изобретение относится к кодирующим CAR полинуклеотидным молекулам, к получению и использованию сконструированной Т-клетки, экспрессирующей на своей клеточной поверхности CAR по изобретению. Сконструированные иммунные клетки, наделенные указанными CAR, наиболее пригодны для иммунотерапии при лечении рака, в частности лейкозов. 6 н. и 39 з.п. ф-лы, 4 ил., 11 табл., 2 пр.
1. CLL1-специфический химерный антигенный рецептор (анти-CLL1 CAR), который содержит по меньшей мере:
- внеклеточный лигандсвязывающий домен анти-CLL1, который содержит фрагмент одноцепочечного антитела (scFv), содержащий вариабельные фрагменты тяжелой (VH) и легкой (VL) цепей анти-CLL1 моноклонального антитела, связанные гибким линкером, в котором указанный VH содержит CDRs с SEQ ID NO: 131, SEQ ID NO: 132 и SEQ ID NO: 133 и указанный VL содержит CDRs с SEQ ID NO: 134, SEQ ID NO: 135 и SEQ ID NO: 136, или в котором указанный VH содержит CDRs с SEQ ID NO: 161, SEQ ID NO: 162 и SEQ ID NO: 163 и указанный VL содержит CDRs с SEQ ID NO: 164, SEQ ID NO: 165 и SEQ ID NO: 166 соответственно,
- шарнир, выбранный из шарниров FcγRIIIα, CD8α или IgG1,
- CD8α трансмембранный домен,
- цитоплазматический домен, включающий сигнальный домен CD3-дзета и костимуляторный домен 4-1 ВВ или CD28.
2. CLL1-специфический CAR по п.1, в котором указанный CAR содержит шарнир FcγRIIIα.
3. CLL1-специфический CAR по п.1, в котором указанный CAR содержит шарнир CD8α.
4. CLL1-специфический CAR по п.1, в котором указанный CAR содержит шарнир IgG1.
5. CLL1-специфический CAR по одному из пп.1-4, в котором указанный VH имеет полипептидную последовательность SEQ ID NO: 15 и указанный VL - полипептидную последовательность SEQ ID NO: 16; или в котором указанная VH имеет полипептидную последовательность SEQ ID NO: 25 и указанный VL - полипептидную последовательность SEQ ID NO: 26 соответственно.
6. CLL1-специфический CAR по одному из пп.1-5, в котором костимуляторный домен из 4-1 ВВ имеет полипептидную последовательность SEQ ID NO: 8.
7. CLL1-специфический CAR по одному из пп.1-6, в котором указанный сигнальный домен CD3-дзета имеет полипептидную последовательность SEQ ID NO: 9.
8. CLL1-специфический CAR по п.1 или 2, в котором указанный шарнир FcγRIIIα имеет полипептидную последовательность SEQ ID NO: 3.
9. CLL1-специфический CAR по одному из пп.1 или 3, в котором указанный шарнир CD8α имеет полипептидную последовательность SEQ ID NO: 4.
10. CLL1-специфический CAR по п.1 или 4, в котором указанный шарнир IgG1 имеет полипептидную последовательность SEQ ID NO: 5.
11. CLL1-специфический CAR по одному из пп.1-10, в котором указанный трансмембранный домен CD8α имеет полипептидную последовательность SEQ ID NO: 6.
12. CLL1-специфический CAR по п.1, который содержит полипептидную последовательность SEQ ID NO: 53 или 83.
13. CLL1-специфический CAR по п.1, который содержит полипептидную последовательность SEQ ID NO: 55 или 85.
14. CLL1-специфический CAR по п.1, который содержит полипептидную последовательность SEQ ID NO: 57 или 87.
15. CLL1-специфический CAR по одному из пп.1-11, дополнительно содержащий сигнальный пептид.
16. CLL1-специфический CAR по п.15, в котором указанный сигнальный пептид имеет последовательность SEQ ID NO: 1 или SEQ ID NO: 2.
17. CLL1-специфический CAR по одному из пп.1-11, в котором внеклеточный связывающий домен включает по меньшей мере один мимеотоп CD20.
18. CLL1-специфический CAR по п.17, в котором мимеотоп CD20 специфически распознается ритуксимабом.
19. CLL1-специфический CAR по п.17 или 18, в котором мимеотоп CD20 имеет аминокислотную последовательность SEQ ID NO: 113.
20. CLL1-специфический CAR по одному из пп.1-11, в котором внеклеточный связывающий домен содержит следующую последовательность
V1-L1-V2-(L)x-эпитоп 1 -(L)x-;
V1-L1-V2-(L)x-эпитоп1-(L)x-эпитоп2-(L)x-;
V1-L1-V2-(L)x-эпитоп1-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-;
(L)x-эпитоп1-(L)x-V1-L1-V2;
(L)x-эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2;
эпитоп1-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-V1-L1-V2;
(L)х-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x;
(L)х-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-;
(L)x-эпитоп1-(L)x-V1-L1-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x-;
(L)x- эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2-(L)x-эпитоп3-(L)x-;
(L)x- эпитоп1-(L)x-эпитоп2-(L)x-V1-L1-V2-(L)x-эпитоп3-(L)x-3эпитоп4-(L)x-;
V1-(L)x-эпитоп1-(L)x-V2;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x;
V1-(L)x-эпитоп1-(L)x-V2-(L)x-эпитоп2-(L)x-эпитоп3-(L)x-эпитоп4-(L)x;
(L)x-эпитоп1-(L)x-V1-(L)x-эпитоп2-(L)x-V2; или
(L)x-эпитоп1-(L)x-V1-(L)x-эпитоп2-(L)x-V2-(L)x-эпитоп3-(L)x;
где
V1 обозначает VL и V2 обозначает VH или V1 обозначает VH и V2 обозначает VL; L1 обозначает линкер, который можно применять для связи VH-цепи с VL-цепью; L обозначает линкер, содержащий глициновые и сериновые остатки, и каждый из присутствующих во внеклеточном связывающем домене L может быть идентичен или отличаться от другого L, присутствующего в том же самом внеклеточном связывающем домене, и
x обозначает 0 или 1 и каждый из присутствующих x выбирают независимо от других; и
эпитоп 1, эпитоп 2 и эпитоп 4 представляют собой МАт-специфические эпитопы, имеющие аминокислотную последовательность SEQ ID NO: 113, и эпитоп 3, выбранный из МАт-специфических эпитопов, имеющих аминокислотную последовательность SEQ ID NO: 113, SEQ ID NO: 198 или SEQ ID NO: 199.
21. CLL1-специфический CAR по п.20, в котором эпитоп 1, эпитоп 2 и эпитоп 4 представляют собой МАт-специфические эпитопы, имеющие аминокислотную последовательность SEQ ID NO: 113, и эпитоп 3 представляет собой МАт-специфический эпитоп, имеющий аминокислотную последовательность SEQ ID NO: 198 или SEQ ID NO: 199.
22. CLL1-специфический CAR по п.20 или 21, в котором L1 обозначает линкер, содержащий глицин и/или серин.
23. CLL1-специфический CAR по п.22, в котором L1 обозначает линкер, содержащий аминокислотную последовательность (Gly-Gly-Gly-Ser)n или (Gly-Gly-Gly-Gly-Ser)n, где n обозначает 1, 2, 3, 4 или 5.
24. CLL1-специфический CAR по п.23, в котором L1 обозначает линкер, содержащий аминокислотную последовательность (Gly4Ser)4 или (Gly4Ser)3.
25. CLL1-специфический CAR по любому из пп.20-24, в котором L обозначает линкер, содержащий глицин и/или серин.
26. CLL1-специфический CAR по п.25, в котором L обозначает линкер, имеющий аминокислотную последовательность, выбранную из SGG, GGS, SGGS, SSGGS, GGGG, SGGGG, GGGGS, SGGGGS, GGGGGS, SGGGGGS, SGGGGG, GSGGGGS, GGGGGGGS, SGGGGGGG, SGGGGGGGS или SGGGGSGGGGS.
27. CLL1-специфический CAR по п.26, в котором L обозначает SGGGG, GGGGS или SGGGGS.
28. Полинуклеотид, кодирующий химерный антигенный рецептор по любому из пп.1-27.
29. Экспрессионный вектор, содержащий полинуклеотид по п.28.
30. Сконструированная Т-клетка, трансдуцированная экспрессионным вектором по п.29 и экспрессирующая на поверхности клеточной мембраны анти-CLL1 CAR по любому из пп.1-27.
31. Сконструированная Т-клетка по п.30, происходящая из воспалительных Т-лимфоцитов, цитотоксических Т-лимфоцитов, регуляторных Т-лимфоцитов или хелперных Т-лимфоцитов.
32. Сконструированная Т-клетка по любому из пп.30 или 31, где экспрессия TCR подавляется в указанной Т-клетке.
33. Сконструированная Т-клетка по любому из пп.30-32, где экспрессия по меньшей мере одного белка ГКГС, предпочтительно β2m или HLA, ингибируется или подавляется в указанной Т-клетке.
34. Сконструированная Т-клетка по любому из пп.30-33, где указанная Т-клетка изменена в результате мутации для придания устойчивости по меньшей мере к одному иммуносупрессорному или химиотерапевтическому лекарственному средству.
35. Сконструированная Т-клетка по любому из пп.30-34 для применения в терапии пациента, которым является человек и у которого диагностировали предзлокачественное или состояние, связанное со злокачественным раком, отличающееся присутствием экспрессирующих CLL1 клеток.
36. Сконструированная Т-клетка для применения по п.35, где состояние отличается избыточным количеством экспрессирующих CLL1 клеток.
37. Сконструированная Т-клетка для применения по п.35 или 36, где состояние представляет собой связанное с гематологическим раком состояние.
38. Сконструированная Т-клетка для применения по п.37, где связанное с гематологическим раком состояние представляет собой лейкоз.
39. Сконструированная Т-клетка для применения по п.38, где лейкоз представляет собой острый миогенный лейкоз (AML).
40. Способ конструирования Т-клетки, включающий:
(а) получение Т-клетки,
(б) интродукцию в указанную клетку по меньшей мере одного полинуклеотида, который кодирует по меньшей мере один CLL1-специфический химерный антигенный рецептор по одному из пп.1-27,
(в) экспрессию указанного полинуклеотида в указанной клетке.
41. Способ конструирования Т-клетки по п.40, в котором указанная сконструированная Т-клетка экспрессирует на поверхности по меньшей мере один CLL1-специфический химерный антигенный рецептор по одному из пп.1-27.
42. Способ конструирования Т-клетки по п.40, включающий:
(а) получение Т-клетки,
(б) интродукцию в указанную клетку по меньшей мере одного полинуклеотида, который кодирует по меньшей мере один CLL1-специфический химерный антигенный рецептор по одному из пп.1-27,
(в) интродукцию по меньшей мере одного другого полинуклеотида, кодирующего химерный антигенный рецептор, не специфический в отношении CLL1.
43. Способ лечения пациента, у которого диагностировали предзлокачественное или состояние, связанное со злокачественным раком, отличающееся присутствием экспрессирующих CLL1 клеток, включающий:
(а) получение Т-клетки, экспрессирующей на поверхности анти-CLL1 специфический химерный антигенный рецептор по одному из пп.1-27;
(б) введение указанных Т-клеток указанному пациенту.
44. Способ по п.43, в котором Т-клетку получают из организма донора.
45. Способ по п.43, в котором указанную Т-клетку получают из организма самого пациента.
| WO 2014191128 A1, 04.12.2014 | |||
| WO 2014145252 А2, 18.09.2014 | |||
| WO 2014130635 A1, 28.08.2014 | |||
| US 2013295118 A1, 07.11.2013 | |||
| US 8536310 B2, 17.09.2013 | |||
| SADELAIN M | |||
| et al., The basic principles of chimeric antigen receptor design, Cancer discovery, 2013, Volume 3, Issue 4, pp.388-398 | |||
| ПРОСКУРЯКОВ С.Я | |||
| и др., Современное состояние и перспективы |
Авторы
Даты
2020-09-04—Публикация
2016-01-25—Подача